用于鉴定具有活化的孕酮受体的癌症的系统和方法与流程
本申请要求2014年3月12日提交的美国临时专利申请系列号61/951,650的优先权。以上提及的申请通过引用如同完整重申地被并入本文。本文引用的所有参考文献(包括但不限于专利和专利申请)通过引用以其整体并入。
背景
几十年来,已经运用了相当大的努力来理解靶组织诸如乳腺和子宫内膜中孕酮(progesterone)信号传导的分子机理。孕酮通过其细胞核受体(PR)调节转录,所述细胞核受体(PR)与染色质上特异性靶位点缔合。PR结合的共有脱氧核糖核酸(DNA)序列(孕酮应答元件(PRE))由反向重复序列:RGNACAnnnTGTNCY中的6个核酸碱基对组成[1,2,3]。DNA结合的PR招募转录辅激活物(co-activator)和相关的辅因子(co-factor),其修饰局部的染色质结构并且有利于转录活化,导致PR靶基因的活化或阻遏[4,5,6,7]。除了通过蛋白-蛋白相互作用与PR调节复合物缔合的辅调节物(co-regulator)和辅因子之外,PR招募染色质重塑因子,其修饰局部DNA架构以增强PR相互作用和转录活化[8]。已知参与在孕激素(progestin)调节位点的染色质重塑的因子包括SWI/SNF染色质重塑复合物[8,9]和转录因子NF1,所述转录因子NF1与PR合作用于MMTV的结合和活化[10,11]。对于其他细胞核受体包括雌激素受体(ER)和雄激素受体(AR),先锋因子(pioneer factor)诸如FOXA1(其与凝聚染色质相互作用)是转录靶的细胞核受体活化必需的[12,13,14,15,16,17]。除了在PRE与DNA直接相互作用之外,已报道PR经由与其他转录因子,包括AP-1、SP1和Stat3的拴系而与靶基因缔合[18,19,20,21,22]。
尽管已经描述了体外管理PR的转录活性的决定因素,对于体内孕酮的多形作用(pleomorphic role)的分子基础知之甚少。孕酮对于正常的生殖组织的功能是必需的[23],并且在子宫中支持分化以及抑制子宫内膜的增殖[24]。与之相比,在乳腺中,孕酮与增加的增殖、导管侧分支和小叶肺泡发育相关[25]。与孕酮在这两个组织中的不同作用一致,在乳腺和子宫内膜中存在对孕酮的不同的转录应答[23,26,27,28,29,30]。
暴露于激素替代疗法中的外源孕激素与增加的乳腺癌风险相关[31,32,33,34,35]。有趣的是,与正常的乳腺相比,在乳腺癌细胞中孕激素调节不同的转录组[36],所以似乎合理的是,孕激素对乳腺癌风险的影响可以通过癌前和/或癌性乳腺组织中孕激素作用的改变的特异性来介导。如果PR的改变的细胞特异性构成孕激素对乳腺癌风险的不利影响的基础,那么孕激素作用的细胞特异性的决定因素则需要阐明。
概述
在一方面,提供了用于鉴定多种组织类型中活化的孕酮受体的系统和方法。在另一方面,肿瘤组织中活化的孕酮受体通过检测孕酮受体与基因组(DNA)结合位点的结合来鉴定。这些示例性系统和方法可被用于鉴定和治疗怀疑患有对通过抗孕激素(例如,奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮)的生长抑制和癌细胞凋亡敏感的恶性肿瘤的患者。在一方面,怀疑患有对通过抗孕激素的生长抑制敏感的恶性肿瘤(癌症)的患者可以用抗孕激素治疗。在另一方面,对用抗孕激素的治疗敏感的癌症,包括但不限于乳腺、脑、脑膜瘤、前列腺、卵巢、子宫内膜、子宫肉瘤、子宫平滑肌瘤和肺。在另一方面,抗孕激素可以以每天从约10mg到约200mg的量向患者施用。任选地,抗肿瘤化合物(例如,依维莫司、曲妥珠单抗、TM1-D、抗HER2药物、贝伐单抗、紫杉醇、多西他赛、紫杉烷、阿霉素、脂质体阿霉素、聚乙二醇化脂质体阿霉素、蒽环类、蒽二酮、卡铂、顺铂、5-FU、吉西他滨、环磷酰胺、抗雌激素、选择性雌激素受体调节剂、芳香化酶抑制剂和抗雄激素)还可以与用抗孕激素的治疗同时地、在其之前或在其之后向患者施用。
本文描述的方面提供了用于鉴定对用抗孕激素治疗敏感的癌症和肿瘤的方法和系统。在一方面,如本文描述的与特定基因组DNA区域结合的活化的孕酮受体或PR,可被鉴定和定量以鉴定对用抗孕激素治疗敏感的恶性肿瘤。
在另一方面,测试样品中特定基因组DNA区域的定量为例如比测定中的比较阴性对照参考样品中的相同的特定区域的检测大至少约四倍的水平,指示活化的PR的存在。用于此类定量的示例性比较阴性对照样品(在一方面,本文统称为“对照DNA序列”)包括但不限于(1)来自例如在ChIP-seq测定(如本文描述的)中的预清洁的样品(a pre-cleared sample)的分离的输入基因组DNA;和(2)在例如在非特异性免疫球蛋白或无一抗的存在下进行的并且在其他方面以与测试样品的特异性PR染色质免疫沉淀相同的方式处理的ChIP-PCR测定中的测试样品的染色质免疫沉淀物。
在又另一方面,适合本文使用的抗孕激素包括但不限于:
奥那司酮(例如,(8S,11R,13R,14S,17S)-11-[4-(二甲氨基)苯基]-17-羟基-17-(3-羟丙基)-13-甲基-1,2,6,7,8,11,12,14,15,16-十氢环戊并[a]菲-3-酮)具有以下的化学结构:
其他抗孕激素包括:促孕的3-(6,6-亚乙基-17B-羟基-3-氧代-17A-孕甾-4-烯-17A-基)丙酸G-内酯、3-(6,6-亚乙基-17.β.-羟基-3-氧代-17.α.-孕甾-4-烯-17.α.-基)丙酸.γ.-内酯和以下:
米非司酮
(10S,11S,14S,15S,17R)-17-[4-(二甲氨基)苯基]-14-羟基-15-甲基-14-(丙-1-炔-1-基)四环[8.7.0.0^{2,7}.0^{11,15}]十七碳-1,6-二烯-5-酮
利洛司酮
(11-β,17-β,17(z))-丙烯基);雌甾-4,9-二烯-3-酮、11-(4-(二甲氨基)苯基)-17-羟基-17-(3-羟基-1-丙烯基;11β-[4-(二甲氨基)苯基]-17β-羟基-17-[(Z)-3-羟基-1-丙烯基]雌甾-4,9-二烯-3-酮
ORG2058
(8R,9S,10R,13S,14S,16R,17S)-16-乙基-17-(2-羟基乙酰基)-13-甲基-2,6,7,8,9,10,11,12,14,15,16,17-十二氢-1H-环戊并[a]菲-3-酮
洛那立生
(8S,11R,13S,14S,17S)-11-(4-乙酰基苯基)-17-羟基-13-甲基-17-(1,1,2,2,2-五氟乙基)-1,2,6,7,8,11,12,14,15,16-十氢环戊并[a]菲-3-酮
Asoprisnil
(8S,11R,13S,14S,17S)-11-[4-[(E)-羟基亚氨基甲基]苯基]-17-甲氧基-17-(甲氧基甲基)-13-甲基-1,2,6,7,8,11,12,14,15,16-十氢环戊并[a]菲-3-酮
乌利司他
(8S,11R,13S,14S,17R)-17-乙酰基-11-[4-(二甲氨基)苯基]-17-羟基-13-甲基-1,2,6,7,8,11,12,14,15,16-十氢环戊并[a]菲-3-酮
PF-2413873
4-[3-环丙基-1-(甲磺酰基甲基)-5-甲基-1H-吡唑-4-基]氧基,-2,6-二甲基苄腈
阿来司酮(8S,11R,13S,14S,17R)-11-(4-二甲基氨基苯基)-17-羟基-13-甲基-17-[(Z)-丙-1-烯基]-1,2,6,7,8,11,12,14,15,16-十氢环戊并[a]菲-3-酮:
另外的抗孕激素包括以下:
ZM172406–(R)-N-(3-氯-4-氰基苯基-3,3,3-三氟-2-羟基-2-甲基丙酰胺:
ZM172405–(S)-N-(3-氯-4-氰基苯基)-3,3,3-三氟-2-羟基-2-甲基丙酰胺:
ZM150271–N-(3-氯-4-氰基苯基)-3,3,3,-三氟-2-羟基-2-甲基丙酰胺:
卵巢激素孕酮的转录作用是多效的,并且与细胞核孕酮受体(PR)(一种配体活化的转录因子)的DNA结合,导致多种靶组织中的多种结果。为了确定促成PR转录组的细胞特异性的PR的基因组相互作用的不同模式,我们比较了乳腺癌细胞和永生化正常乳腺细胞中PR的基因组结合位点。在两个细胞系中,PR结合与转录结果相关,其中60%的孕激素调节基因与一个或更多个PR结合区域相关。
在两个细胞系的PR顺反组(cistrome)之间存在出乎意料地低的重叠并且在转录靶中存在类似地低的重叠。在来自乳腺癌和正常的乳腺细胞系二者的PR DNA结合区域中鉴定了保守的PR结合元件(DNA的区域),但存在不同模式的已知的辅因子结合基序的富集,其中FOXA1位点在乳腺癌细胞结合区域中过度代表(over-represented),且NF1和AP-1基序在永生化正常细胞系中独特地富集。下游分析表明,不同的辅因子可用性可以产生这些不同的PR顺反组(例如,基因组DNA上特定的孕酮受体结合位点),暗示辅因子水平可以调节PR对DNA结合区域的特异性。PR结合的细胞特异性可以由关键的结合辅因子的协调作用决定。该信息还可被用于鉴定和治疗怀疑患有对通过抗孕激素(例如,奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮)的生长抑制敏感的肿瘤的患者。
附图
图1A和1B图解了T-47D和AB32细胞中各个PR结合区域距最近的基因转录起始位点(TSS)的距离的分布,如图1A和1B中分别示出的;
图1C和1D图解了T-47D(图1C)和AB32(图1D)中结合区域对每染色体基因数目的线性回归分析;
图1E图解了结合到上调基因的PR的中值距离;
图1F图解了相对于AB32细胞中上调和下调基因的结合区域分布;
图2A-F图解了在用10nM ORG2058处理后的2小时、6小时和24小时确定的T-47D和AB32细胞中的基因表达谱;
图3A图解了在T-47D和AB32细胞之间共有的PR结合区域;
图3B图解了在用10nM ORG2058(ORG)或媒介物处理持续2小时、6小时和24小时的T-47D和AB32细胞中通过全基因组微阵列测量的转录本表达;
图3C图解了在2小时、6小时或24小时在T-47D和AB32细胞中受到孕激素调节的转录本之间的重叠;
图3D图解了在单独的处理时间在T-47D和AB32中孕激素调节的转录本的数目;
图4A图解了在T-47D和AB32细胞中的结合区域中鉴定的全长和核心PRE;
图4B-C图解了使用MEME-ChIP和Homer分析PR结合区域序列的保守的序列基序的统计富集;
图4D-E图解了PRE分类为强的、中等的、或弱的/不存在的;
图5A图解了在FOXA1的存在和不存在下,在AB32细胞中响应孕激素的转录谱的无监督聚簇分析;
图5B图解了与T-47D细胞中的内源表达相比,在病毒转导之前和之后,在AB32细胞和亲本MCF-10A细胞中的FOXA1蛋白表达;
图5C图解了在FOXA1的存在和不存在下,AB32细胞中孕激素调节的转录本的数目;
图5D图解了在PR、ER和FOXA1结合位点的FOXA1结合强度的比较;
图5E图解了针对随着FOXA1表达丢失、获得或保持孕激素调节的基因,PR结合区域与AB32细胞中被调节的基因的比率;
图5F图解了从AB32中PR结合区域到随着FOXA1表达丢失、获得或保持孕激素调节的基因的距离;
图6图解了孕酮受体(PR)结合与T-47D和AB32细胞中染色体分布相关;
图7图解了PR结合和孕激素调节的时间之间的关系;
图8图解了PR结合区域的位置;
图9图解了T-47D细胞中转录调节的模式;
图10图解了AB32细胞中转录调节的模式;
图11图解了在T-47D和AB32细胞中的PR结合区域;
图12图解了验证在ChIP-seq中鉴定的细胞类型特异性的PR结合区域;
图13图解了ORG2058处理的T-47D和AB32细胞中PR结合区域与孕酮(P4)处理之后T-47D细胞中的结合的重叠;
图14图解了在另外的乳腺细胞系中基因表达的孕激素调节;
图15图解了在T-47D和AB32细胞中在调节相关的结合位点中PRE和辅因子基序的富集;
图16图解了在T-47D和AB32细胞中的PR结合区域中PRE位置的分布;
图17是图解PRE强度不预测PR结合的示例性实验;
图18图解了细胞系中FOXA1转录本表达;
图19图解了在具有或不具有预测的FOXA1基序的PR结合区域处的FOXA1结合;和
图20图解了在T-47D、AB32和AB9细胞中的PR表达;
图21示出了HCC1428乳腺癌细胞中的T-47D中活化的PR的ChIP验证;
图22提供了T-47D和HCC1428乳腺癌细胞中ChIP验证实验的总结;
图23提供了证实T-47D和HCC1428乳腺癌细胞中PR活化特征(signature)的PR结合靶的示例性列表;和
图24示出了奥那司酮对T-47D和HCC1428乳腺癌细胞中通过ORG2058配合的孕酮受体的结合的影响。
描述
在一方面,PR结合的应答元件的DNA序列、转录辅因子的可用性和靶细胞的染色质架构对PR转录组的特异性具有组合的作用。在另一方面,正常乳腺细胞和乳腺癌细胞中孕酮受体(PR)应答元件、转录辅因子和染色质架构的贡献可使用与高通量测序联合的全基因组PR染色质免疫沉淀比较示例性乳腺癌细胞系(T-47D)和永生化正常乳腺细胞中基因组DNA上的PR相互作用来确定,所述永生化正常乳腺细胞稳定地表达PR-A和PR-B亚型二者(AB32,MCF-10A的稳定的PR表达克隆)。在又另一方面,鉴定并且表征了示例性PR顺反组。这些PR顺反组可以被用于例如鉴定患有对用如本文描述的抗孕激素治疗敏感的恶性肿瘤(例如,乳腺癌、子宫内膜癌、卵巢癌、前列腺癌、肺癌和子宫肉瘤)的患者。鉴定和治疗对用抗孕激素治疗敏感的患者可以影响此类患者中癌症的发展、增殖、生长和转移。
多种正常的和肿瘤发生的组织中的孕酮受体的活性和状态可被预测。参见例如美国专利申请号13/644,872,其通过引用以其整体并入本文。该信息可被用于鉴定和治疗怀疑患有对通过抗孕激素(例如,奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮)的生长抑制敏感的肿瘤的患者。在本文的一方面,我们描述了用于通过以下确定孕酮受体的活性和状态的方法和系统:使取自患者的怀疑是肿瘤发生或癌性的组织样品中的孕酮受体与基因组DNA靶结合;检测在组织样品中和在阴性对照样品中一个或更多个活化的孕酮受体缔合的DNA靶的水平或丰度;和如果一个或更多个活化的孕酮受体缔合的DNA靶的水平或丰度比阴性对照样品中的水平大至少约4倍或统计上显著地大于阴性对照样品中的,则向患者施用抗孕激素。
在一方面,术语“顺反组(cistrome)”指基因组内的DNA区域的集合,所述DNA区域被特定的顺式作用转录调节物(例如,PR)结合。
如本文使用的,术语“活化的孕酮受体缔合的DNA靶”指基因组DNA的区域,孕酮受体能够与所述基因组DNA的区域结合,并且与基因组DNA的对照区域相比,所述基因组DNA的区域与孕酮受体顺反组的转录的增加相关。
术语“施用”指提供一种药物或更多种药物,开一种或更多种药物的处方,或将一种或更多种药物放置在处方集(formulary)上。术语“提供”指通过任何合适的施用途径(例如,口服、注射、静脉内、肌肉内和经皮等)向患者直接分配药物,或向患者提供进行所述分配的说明。
术语“孕激素(progestin)”指模拟孕酮(progesterone)的一些或全部作用的天然的或合成的促孕物质,还被称作孕酮受体调节剂(PRM)或选择性孕酮受体调节剂(SPRM)。
术语“抗孕激素”指抑制促孕剂的形成、转运或作用或使促孕剂失活的物质,包括但不限于奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮。PRM或SPRM可以具有一些抗孕激素性质,并且取决于使用的环境可以被认为是抗孕激素或孕激素。
术语“结合(bind)”或“结合(binding)”指分子或化学化合物上的一个或更多个部分通过相互作用或化学键(例如,氢键、疏水键、离子键和共价键)的缔合。“交联(cross-link)”或“交联(cross-linking)”可以是“结合(bind)”或“结合(binding)”的一种形式。
本文提供了用于确定患者是否对用抗孕激素治疗敏感的方法和系统。在一方面,用抗孕激素治疗患者的方法包括:从患者获得怀疑是肿瘤发生或癌性的组织样品;使组织样品中的孕酮受体与基因组DNA结合;检测组织样品中与孕酮受体缔合的基因组DNA;检测组织样品中和阴性对照样品中一个或更多个活化的孕酮受体缔合的DNA靶的转录或丰度水平;和如果一个或更多个活化的孕酮受体缔合的DNA靶的水平比阴性对照样品中的水平大至少约4倍或统计上显著地大于阴性对照样品中的水平,则向患者施用抗孕激素。
组织样品或活组织检查物可以从患者获得,例如由外科医生、内科医生、护士或医务人员从怀疑患有肿瘤或呈现癌症或其他异常细胞生长的症状的患者获得。
组织样品可以通过例如,机械破坏和通过暴露于磷酸盐缓冲盐水(PBS)中的1%的甲醛交联样品被处理或制备用于分析。样品可以重悬在裂解缓冲液中,并且可以通过声处理使基因组DNA片段化。
通过与抗PR一抗一起孵育,孕酮受体(PR)结合的基因组DNA可被免疫沉淀,所述抗PR一抗与二抗-磁珠缀合物络合。在一方面,可以使用至少一式两份测定(determinations)和含有样品而不使用一抗的匹配的阴性对照孵育物进行测定。磁珠复合物可以用缓冲液洗涤以减少非特异性信号。
基因组DNA可以通过以下从磁珠洗脱:例如,将珠与洗脱缓冲液(1%SDS、0.1M NaHCO3)在室温孵育两次,持续15min,以及收集含有分离的基因组DNA的洗脱液。交联可以通过在高盐存在下加热到例如65℃持续至少四小时被逆转。基因组DNA片段可以使用PCR纯化试剂,例如由Qiagen提供的QIAquick PCR纯化试剂盒纯化。
活化的PR缔合的DNA靶可以使用与活化的PR缔合的DNA靶互补的寡核苷酸引物,通过定量实时PCR来检测。在一方面,样品以一式三份被扩增。在另一方面,至少四个活化的PR缔合的DNA靶被选择。
测试样品中活化的PR缔合的DNA靶的丰度可相对于在特异性一抗不存在下孵育的阴性对照/参考样品被确定。在一方面,倍数差异=2^-(平均Ct测试-平均Ct参考)。倍数差异的统计显著性可例如,通过不成对的重复计数测定(unpaired replicate count determination)的学生t检验被确定。在一方面,约四倍或更大的倍数差异和小于0.05的p值指示活化的PR的存在。
在另一方面,如果检测到活化的PR,则可以向患者施用抗孕激素。
在又另一方面,组织样品选自由以下组成的组:乳腺、脑、脑膜瘤、前列腺、卵巢、子宫内膜、子宫肉瘤、子宫平滑肌瘤和肺部组织。
在一方面,抗孕酮选自由以下组成的组:奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮。
在另一方面,抗孕激素以每天从约10mg到约200mg的量向患者施用。
在又另一方面,除了抗孕激素之外,抗肿瘤化合物(例如,依维莫司、曲妥珠单抗、TM1-D、抗HER2药物、贝伐单抗、紫杉醇、多西他赛、紫杉烷、阿霉素、脂质体阿霉素、聚乙二醇化脂质体阿霉素、蒽环类、蒽二酮、卡铂、顺铂、5-FU、吉西他滨、环磷酰胺、抗雌激素、选择性雌激素受体调节剂、芳香化酶抑制剂和抗雄激素)可以被施用或共施用至患者。
在一方面,活化的孕酮受体缔合的DNA靶包括靠近ACSL1和PACSIN1的PR结合区域。
在另一方面,可以使用单克隆抗PR抗体,例如hPRa6和hPRa7进行免疫沉淀。
其他方面提供了用于确定组织样品中孕酮受体的活化状态的系统。系统可以包括例如,抗PR一抗和针对一个或更多个活化的孕酮受体缔合的DNA靶的一个或更多个寡核苷酸引物对。
在另一方面,寡核苷酸引物可以选自由以下组成的引物组:用于检测靠近ACSL1和PACSIN1的结合区域的寡核苷酸引物。
其他方面提供了用于通过以下抑制对通过抗孕激素的生长抑制敏感的肿瘤的生长的方法:从患者的肿瘤获得怀疑是肿瘤发生或癌性的组织样品;使组织样品中的孕酮受体与基因组DNA结合(例如,交联);检测组织样品中与孕酮受体缔合的基因组DNA;检测组织样品中和阴性对照样品中一个或更多个活化的孕酮受体缔合的DNA靶的水平;和如果一个或更多个活化的孕酮受体缔合的DNA靶的水平比阴性对照样品中的水平大至少约4倍或统计上显著地大于阴性对照样品中的水平,则向患者施用抗孕激素。
另外的方面提供了通过以下用抗孕激素治疗患者的方法:从患者获得怀疑是肿瘤发生或癌性的组织样品;检测组织样品中与孕酮受体缔合的基因组DNA;检测一个或更多个活化的孕酮受体缔合的DNA靶的丰度的水平,所述一个或更多个活化的孕酮受体缔合的DNA靶选自由以下组成的组:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516(下表);以及检测组织样品中的阴性对照测定中的相同DNA区域。如果一个或更多个活化的孕酮受体缔合的DNA靶的转录水平比阴性对照中的相同DNA区域的水平大至少约4倍和/或统计上显著地大于阴性对照中的,则可以向患者施用抗孕激素,所述一个或更多个活化的孕酮受体缔合的DNA靶选自由以下组成的组:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516。
另外的方面提供了用抗孕激素治疗患者的方法,所述方法包括:从患者获得怀疑是肿瘤发生或癌性的组织样品;检测组织样品中与孕酮受体缔合的基因组DNA;检测组织样品中的一个或更多个活化的孕酮受体缔合的DNA靶的第一转录水平,其中DNA靶选自由以下组成的组:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516;检测阴性对照组织样品中一个或更多个活化的孕酮受体缔合的DNA靶的第二转录水平,其中DNA靶选自由以下组成的组:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516;比较第一转录水平和第二转录水平;和如果一个或更多个活化的孕酮受体缔合的DNA靶的转录水平比阴性对照样品中的水平大至少约4倍,则向患者施用抗孕激素。
在一方面,组织样品选自由以下组成的组:乳腺、脑、脑膜瘤、前列腺、卵巢、子宫内膜、子宫肉瘤、子宫平滑肌瘤和肺组织。
在另一方面,抗孕激素选自由以下组成的组:奥那司酮、洛那立生、米非司酮、PF-02413873、特拉司酮、利洛司酮、ORG2058、asoprisnil、乌利司他、ZM172406、ZM150271、ZM172405和阿来司酮。
在又另一方面,抗孕激素以每天从约10mg到约200mg的量向患者施用。
另外的方面提供了施用抗肿瘤化合物(例如,依维莫司、曲妥珠单抗、TM1-D、抗HER2药物、贝伐单抗、紫杉醇、多西他赛、紫杉烷、阿霉素、脂质体阿霉素、聚乙二醇化脂质体阿霉素、蒽环类、蒽二酮、卡铂、顺铂、5-FU、吉西他滨、环磷酰胺、抗雌激素、选择性雌激素受体调节剂、芳香化酶抑制剂和抗雄激素)。
在一方面,检测了辅因子结合基序(例如FOXA1)的存在。在另一方面,基因组DNA通过免疫沉淀检测。免疫沉淀可以使用单克隆抗体(例如,hPRa6和hPRa7)进行。在又另一方面,与基因组DNA缔合的孕酮受体通过使用抗PR一抗的免疫沉淀检测。
另外的方面提供了基本上分离的核酸,所述基本上分离的核酸包含选自由以下组成的组的核酸:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516。核酸可以是例如DNA或RNA。核酸可以与选自由以下组成的组的核酸互补:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516。
另外的方面提供了用于检测核酸的转录水平的试剂盒,所述核酸选自由以下组成的组:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516。在一方面,试剂盒包含选自由以下组成的组的核酸:T47D2822、T47D299、T47D3514、T47D4414、T47D4818、T47D5045和T47D5516;和用于检测基因组DNA靶的存在的单克隆抗体(例如,hPRa6和hPRa7)。
在描述本文描述的若干示例性方面之前,应当理解,本发明不限于以下描述中陈述的结构或方法步骤的细节。本文描述的方面能够以多种方式被实践或实施。
实施例
实施例1-全基因组PR相互作用谱的产生
在一方面,绘制T-47D乳腺癌细胞和AB32细胞系中的PR基因组相互作用的图谱,所述AB32细胞系是MCF-10A永生化正常乳腺细胞系的稳定的PR表达克隆。用孕激素ORG2058(10nM,45分钟)处理细胞,随后为PR染色质免疫沉淀(ChIP)和Illumina测序。将序列与人类基因组比对,并且使用Bowtie[37]和ERANGE[38]软件工具鉴定比对中富集的基因组区域(错误发现率0.27%)。
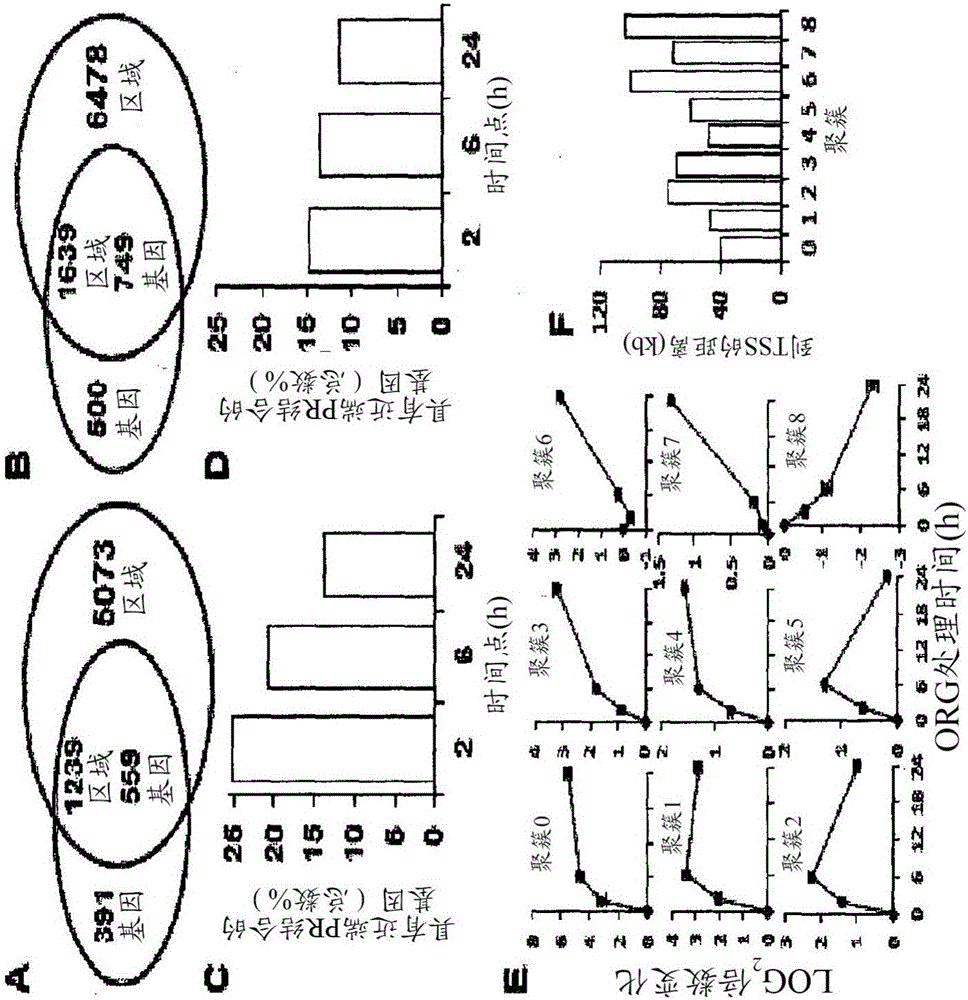
在T-47D细胞中,鉴定出6312个PR结合峰,且在AB32细胞中检测到8117个结合区域(表1)。大多数PR结合区域(T-47D中的88%和AB32中的73%)在最靠近的基因的100kb以内,其中T-47D中57%的结合区域和AB32中54%的结合区域在50kb以内。然而,少许结合区域(T-47D中的21%和AB32中的20%)落在基因TSS的10kb以内(表1)。T-47D和AB32细胞中各个PR结合区域距最靠近的基因转录起始位点(TSS)的距离的分布分别显示在图1A和1B中。
检测了全部染色体上的PR结合区域,并且尽管观察到一些可变性,每条染色体的结合位点的数目反映了染色体尺寸和该特定染色体上的基因的数目。T-47D(图1C)和AB32(图1D)细胞中每条染色体的结合区域对基因数目的线性回归分析揭示了AB32细胞中与基因数目的较强的相关性(AB32中R2=0.76,相比于T-47D中R2=0.58)。总体说来,在两个细胞系之间的每条染色体的结合区域的数目之间存在相关性(R2=0.66,补充图1),然而注意到一些例外。相比于AB32,T-47D数据集中染色体2和8上的结合区域代表不足(under-represented),而染色体11上的区域则过度代表。相比于AB32细胞,T-47D细胞的核型[39]显示显著的重排和重复,并且这可以部分地解释观察到的结合差异,因为T-47D细胞含有染色体11的4个拷贝而不是2个拷贝。然而,T-47D细胞中染色体2是正常的,然而与该染色体上的区域的结合是在AB32细胞中检测到的频率的一半。AB32中被PR结合的染色体2上的被调节的基因的功能注释揭示了参与代谢的基因的富集(补充表1),表明癌细胞系中对孕激素的改变的或减弱的代谢应答。
实施例2-PR基因组相互作用和转录应答之间的关系
在另一方面,与ChIP-seq平行进行的基因表达谱分析揭示了PR结合区域集中在被调节的基因周围。每个基因的PR结合区域的密度,例如,对于被调节的基因而言(每个被调节的基因的结合区域的密度:T-47D细胞中为2.23,AB32细胞中为2.19;表1)高于整体PR结合区域的密度(T-47D细胞中对于所有基因为每基因0.73,AB32细胞中为0.74;表1)。另外,相比于所有基因的50kb以内的PR结合区域的比例(T-47D细胞中57%的PR结合区域在TSS的50kb以内,AB32细胞中为54%,表1),PR结合峰更可能在被调节的基因中的基因转录起始位点的50kb以内(T-47D和AB32细胞中被调节的基因的74%和69%)。
与靠近下调基因的区域相比,T-47D细胞中的PR结合区域平均更接近上调基因TSS。在T-47D细胞中,PR结合与上调基因的中值距离是44kb,而与下调基因的中值距离是75kb(图1E)。这在该细胞系中距上调和下调基因的结合区域距离的累积频率分布中的统计显著整体差异中得到反映(图1E,p=0.001,柯尔莫哥洛夫-斯米尔诺夫(Kolmogorov-Smirnov)双样本检验)。相比之下,关于AB32细胞中的上调和下调基因,未观察到结合区域分布中的显著的差异(图1F,p=0.305)。
除了更接近上调基因的PR结合区域之外,存在靠近上调基因的更多的PR结合区域,例如,相比于1.5个结合区域每下调基因,T-47D细胞中为平均2.3个结合区域每上调基因;且AB32细胞中分别为2.4和1.9个平均区域每上调基因和每下调基因(表1)。此外,与下调基因相比(T-47D中50/98-51%的下调基因和AB32中325/621-52%的下调基因,表1),更高比例的上调基因(T-47D中509/786-65%的上调基因和AB32中439/546-80%的上调基因)与PR结合区域缔合。
实施例3-PR结合与转录调节相关
在另一方面,两个细胞系中的大多数被调节的基因(T-47D中559/950(59%)和AB32中749/1249(60%),图2A和B,表1)在100kb以内具有一个或更多个PR结合区域。在ORG2058处理之后的较早时间点,在PR结合和转录调节之间存在较强的相关性,表明受PR直接地调节的基因比为间接靶的那些基因更可能在转录水平上被及早检测到(补充图2)。这一关系在T-47D细胞中最强,且在AB32细胞中,这一关系仅对于相对靠近被调节的基因的TSS的结合区域是如此,如由两个细胞系中在较早的时间的启动子近端PR结合区域(5’UTR并多达上游10kb)的较高代表性显示的(图2C和D)。
在另一个方面,关于基因内和基因间区域的PR结合区域的整体分布在两个细胞系中是类似的(补充图3):在被调节的基因的上游和5’UTR中观察到最大比例的PR结合区域,代表与被调节的基因相关的区域的43%-45%。
T-47D细胞中与PR结合区域相关的孕激素调节的转录本的自组装映射(SOM)聚簇分析(图2E)和相应的结合区域的分析显示,与下调基因或在随后的时间点被调节的基因的TSS相比,PR结合区域显著地更接近快速上调的基因的TSS(克鲁斯卡尔-沃利斯单因素方差分析,p值<0.001,图2F-比较聚簇0-5和6-8)。所有孕激素调节的转录本的自组装映射聚簇分析揭示了整体类似于通过与PR结合相关的基因的子集观察到的调节模式的调节模式(图9)。然而,在较大的数据集中检测到26个转录本的聚簇,所述较大的数据集代表通过ORG2058在所有时间点减少,但在24h时开始回到基线水平的转录本。尽管该聚簇中的12个转录本还出现在与PR结合相关的转录本的集中,14个仅在完全的孕激素调节的转录组中发现,且代表被早期调节但到24h时大部分恢复的转录本,表明由直接的PR结合介导的转录沉默可以比间接调节更持续。
在AB32细胞中,与通过ORG2058增加的转录本相比,更多的转录本通过ORG2058减少(图10),并且在调节聚簇之间未观察到平均的结合区域到TSS的距离中的显著差异(克鲁斯卡尔-沃利斯p值=0.209,未显示)。
实施例4-细胞系之间PR结合的不同模式反映了相异的转录应答
在T-47D中被PR结合的6312个区域和AB32中被PR结合的8117个区域中,仅1824个结合区域(组合总数的14%)是两个细胞系共有的,代表了T-47D中结合区域的29%和AB32结合区域的22%(图3)。两个细胞系共有的结合区域更不可能与被调节的基因相关:在AB32和T-47D二者中发现的1824个结合区域中,AB32中的431个(24%)与孕激素调节相关且T-47D中为345个(19%)-与表1中示出的所有结合区域与被调节的基因的相关性类似。157个(9%)结合区域与两个细胞系中被调节的基因相关(数据未显示)。图11中示出了在一个细胞系中专有地检测到的或两个细胞系共有的结合峰的实例。定向的ChIP证实与AB32、T-47D或两个细胞系中被调节的基因结合的PR的不同模式(图12)。此外,T-47D和AB32细胞中PR结合与T-47D细胞中的另一个PR顺反组[40]之间的重叠的直接检查揭示,与对于AB32PR数据集的相比,在两个T-47D数据集之间的结合区域中的较高的重叠(图13)。
在两个细胞系之间的结合位点中的重叠的缺乏在孕激素处理的2h、6h和24h的转录谱中的类似地低的重叠中得到反映(图3C-E)。两个细胞系中孕激素靶中小的重叠在检查的所有时间点是类似的(图3E)。该重叠的缺乏在两个另外的细胞系,ZR-75-1乳腺癌细胞和另外的PR表达细胞系MCF-10A克隆AB9中被证实,这揭示了当彼此直接比较时(图14)或与T-47D或AB32细胞比较时,孕激素应答的类似地低的重叠。
实施例5-在T-47D和AB32细胞中的PR结合区域中鉴定的保守PRE
与被调节的基因相关的PR结合区域的从头基序富集分析鉴定了与之前预测的孕酮应答元件(PRE)一致的保守PR结合基序(图4A)的高度显著富集。MEME-ChIP(软件分析程序)和Homer基序分析(软件分析程序)工具二者鉴定了由被三个非特定的碱基分开的六个碱基对的反向重复序列RGNACA组成的共有PR结合基序,与PR结合元件的经典生化研究一致[2,3],并且与近期简要描述的元件类似[40]。另外,Homer中的从头分析鉴定了高度富集的较短的元件,代表反向重复序列的中心核心(图4A),表明PRE的该更为强烈的保守的部分是对于PR结合最关键的。在T-47D细胞中,与被调节的基因相关的782/1239PR结合区域(63%)含有一个或更多个全长或核心PRE基序(图15A),并且在33%的结合区域中,这包括至少一个高度保守的全长PRE。在AB32细胞中,62%的调节相关的PR结合区域含有PRE(图15B)。相当比例的PR结合可能由与这些基序的直接的基因组相互作用介导,因为在T-47D和AB32细胞二者中存在PRE关于结合区域的中心的正态分布(图16,和单样本柯尔莫哥洛夫-斯米尔诺夫检验)。两个细胞系中的若干结合区域含有多于一个PRE,尽管PRE的数目与峰高不相关(皮尔逊(Pearson)回归R平方=0.0048),表明PRE数目与结合强度不相关。
针对通过在两个细胞系中的从头绘制基序图谱(de novo motif mapping)定义的全长PRE的位置特异性概率矩阵被用于将所有PR结合区域中的PRE强度分类。PRE强度不预测转录结果,因为相同比例的调节相关的和不相关的PR结合区域含有强的PRE(数据未显示)。基于PRE p值,将调节相关的PR结合区域分组(图4B-C,强(+++)-p<1x10-5,中等(++)-p=1x10-5-1x10-3,弱/不存在(+)-p>1x10-3)。通过这些标准,T-47D细胞中77%的调节相关的结合区域和AB32中76%的调节相关的结合区域含有一个或更多个中等或强PRE(p<0.001)。大多数的PR结合区域不含有强PRE,表明PR结合位点选择方面的广泛的灵活性,并且还意味着PR结合强度不仅由DNA核酸序列决定,并且可能受二级DNA结构和其他DNA结合PR辅因子影响。PR结合峰高与转录结果正相关,表明其是结合强度的量度。在T-47D和AB32细胞二者中,在上调基因的50kb以内的结合区域的平均峰高显著地大于远离任何被调节的基因的那些结合区域的平均峰高(图4D-E,非配对t检验,T-47D p=2.89x10-8,AB32 p=3.8x10-6)。当将PRE强度与峰高直接地比较时,未观察到相关性,说明单独的PRE品质不决定PR结合强度(图17)。该发现结果得到T-47D和AB32细胞中依据峰高的上部(top)PR结合区域的定向的ChIP验证的支持。这些区域的分析揭示了T-47D细胞中的仅两个区域含有强PRE且AB32细胞为三个区域。前十位中的大多数区域含有中等强度PRE,并且在每个细胞系中在除了一个结合区域以外的所有结合区域中证实了PR结合(表S2)。
实施例6-T-47D和AB32中的结合区域具有不同的基序富集
我们分析了距每个结合峰多达400bp的序列的其他富集的基序的存在。T-47D细胞中的结合区域显著地富集有先锋因子FOXA1的基序(图4F),而来自AB32细胞的PR结合区域中不存在该因子的显著富集。在T-47D细胞中的548/1239(44%)的调节相关的PR结合区域中鉴定了FOXA1结合基序(图15A)。在AB32细胞中的结合区域中,存在用于AP-1复合物和用于DNA结合PR辅因子NF1的结合位点的强的富集。AP-1结合基序存在于AB32细胞中的454/1639(28%)的调节相关的PR结合区域中,且在AB32细胞中的380/1639(23%)的调节相关的PR结合区域中鉴定出NF1位点。这些细胞中相对少的结合区域(74/1639,4.5%)含有用于两种因子的结合基序。任何转录因子基序在靠近被ORG2058上调或下调的基因的结合区域中的发生率中不存在差异(t检验,p>0.05)。此外,上调相关的结合区域的单独的基序分析和与下调相关的那些结合区域的单独的基序分析未揭示不同的转录因子基序的富集(未显示)。
实施例7-先锋因子FOXA1改变PR转录应答
FOXA1转录本相对于AB32和AB9细胞在T-47D和ZR-75-1细胞中丰富地表达(图18),表明FOXA1的内源水平在调节PR转录应答中可以发挥作用。因此,用慢病毒递送的FOXA1感染缺乏内源水平的FOXA1的AB32细胞(图5)。这导致在6h和24h的孕激素调节的转录组中的极大的改变(图5A)。FOXA1转导导致在仅用对照pCDH病毒转导的细胞中未被调节的303个转录本的孕激素调节(图5C)。这些靶中的几乎一半(146/303,48%)被检测为聚簇在一起的基因的不同的盒(图5A,红色条)。功能分析揭示,FOXA1表达后获得孕激素调节的基因参与了血管形态发生和细胞运动性的调节(表S3且数据未显示)。这些类别包括诸如转化生长因子β3、CD44和碱性成纤维细胞生长因子的基因,表明较广泛的发育功能。出人意外地,当FOXA1不存在时被调节的大比例的转录本(不存在FOXA1时,在6h、24h或二者被调节的1333个转录本,图5C)在先锋因子的表达后失去了调节,并且在多个聚簇中是明显的(图5A,蓝色条)。功能分析揭示了FOXA1表达对参与细胞凋亡的负调节的基因的主要影响:不存在FOXA1时这些基因被孕激素增加,但当FOXA1表达时失去了孕激素应答(表S3且数据未显示)。被孕激素减少的该类别中的基因不被FOXA1表达改变,表明FOXA1的净作用是促进响应孕激素的细胞凋亡。FOXA1表达对孕激素调节的削弱性作用表明,先锋因子在PR作用中可以发挥双重作用,与其在雄激素受体信号传导中的作用类似,其中先锋因子作为对一个子集的雄激素靶的活化物并且对其他雄激素靶的辅阻遏物起作用[41]。仅168个转录本的孕激素调节不受改变的FOXA1水平的影响(图5C)。这些基因的功能分析揭示了孕激素介导的参与细胞周期进程的基因的增强,表明孕激素的增殖作用可能不需要FOXA1。
由于FOXA1表现出具有对PR转录的与对ER观察到的不同的作用,我们将T-47D细胞中PR结合区域周围的FOXA1 ChIP-seq相互作用的密度[42]与在FOXA1或ER结合区域观察到的那些密度进行比较(图5D)。不受限于理论,ER结合区域周围的FOXA1的结合非常高,指示雌激素信号传导中对该因子的需求。相比之下,尽管在PR结合区域附近观察到FOXA1相互作用的峰,序列富集显著地较低(图5D),表明虽然FOXA1可以在一些区域参与PR结合,其代表了结合事件的一个子集。
该结论得到以下发现结果支持:FOXA1结合在其中已预测了FOXA1基序的PR结合区域比在未发现基序的区域中强得多,并且与在ER结合区域中整体观察到的结合密度类似(图19)。为了测试靠近在FOXA1表达后获得孕激素调节的基因的PR结合位点的数目是否不同,我们比较了FOXA1阴性AB32细胞中靠近当FOXA1表达时丢失、获得或保持孕激素调节的基因的PR结合峰的数目。尽管靠近获得调节的基因存在稍微较少的PR结合区域(图5E),差异不显著。这表明FOXA1影响PR结合和靶基因的转录调节的能力可能与PR结合位点密度不相关;PR可以形成靠近“获得”基因子集的弱的缔合,但对于相互作用变的富有成效,则需要FOXA1。我们还检查了与当FOXA1表达时丢失、获得或保留孕激素调节的基因相关的PR结合区域中用于NF1和AP-1的基序的富集水平,并且发现在组之间无差异(未显示)。
FOXA1经由其DNA弯曲活性(bending activity)影响转录因子活性[43,44,45]。我们推测,需要FOXA1来影响转录的PR结合区域可能比不需要FOXA1来影响转录的那些PR结合区域更加远离靶基因,并且靠近那些区域的FOXA1的结合导致DNA弯曲,这使得PR转录复合物更接近靶基因。从PR结合区域到通过FOXA1获得调节的基因的距离的检查揭示了情况就是这样,并且该区域的子集比靠近在FOXA1不存在时被调节的基因的结合区域显著地远离被调节的基因(图5F,p=0.003,非配对t检验)。
总之,两个不同的示例性细胞系(例如,T-47D和AB32)中的ChIP-seq谱分析已揭示PR结合的预料不到地不同的模式。这些不同的顺反组在对孕激素的转录应答的显著差异中得到反映。在两个细胞系中的PR结合由高度类似的PRE介导,说明DNA相互作用的类似的模式,但辅因子特别是FOXA1结合位点富集的关键差异表明,这些辅因子的表达水平影响PR的细胞特异性结合和配体应答。
PR基因组相互作用的全基因组调查的这一首次详细描述已在永生化的正常和恶性乳腺细胞中鉴定了非重叠的PR结合位点;显示,PR相互作用发生在近端启动子的远端,支持以下观点:PR的作用跨越比之前预期的用于直接顺式作用转录因子更长的距离被介导;并且说明了转录辅因子是对细胞特异性PR活性的重要的贡献者。
实施例8-PR结合区域远离TSS
大多数PR结合区域位于距离被调节的基因的TSS超过10kb,其中在两个细胞系中少于35%的被调节的基因具有在TSS的10kb以内的PR结合区域,并且少于4%的被调节的基因具有在TSS的1kb以内的结合区域。在两个乳腺细胞类型中,结合与基因调节相关,其中大多数孕激素调节的基因具有在50kb以内的一个或更多个PR结合区域,并且通过孕激素增加的基因比被减少的基因更可能与PR结合位点相关。这些发现结果与在可比较的实验系统中通过其他细胞核受体观察到的结果一致。Reddy等在地塞米松处理的A549细胞中通过ChIP-seq鉴定了4392个GR结合位点(2%FDR)[46]。Welboren等在MCF-7细胞中鉴定了7713和10205个之间的雌激素依赖的ER结合位点,取决于使用的峰调用算法[47]。ER和GR二者表明在结合和基因调节之间的相关性,并且与该研究的发现结果一致,相对低比例的启动子近端结合被报道[14,46,47]。针对ER[14]和GR[46],还已描述了结合与转录上调之间比结合与转录下调之间的更强的相关性。
在一方面,发现的PR结合位点的数目超过孕激素调节的转录靶的数目,并且许多PR结合位点与活性转录不相关(例如,20%的PR结合区域与每个细胞系中的转录调节相关)。在一方面,这被认为代表PR结合位点与基因的相关的“接近”。该发现结果与其他细胞核受体的结果一致[14,17,46,47]。提出了若干可能的解释[48]。一些结合事件可以以低于全基因组表达谱分析的检测阈值的水平调节转录。此外,一个子集的结合位点可以代表仅在一个子集的细胞中发生的较弱的缔合或结合,以使得转录调节不以显著的水平发生。我们的数据支持该可能性,因为相比于非调节相关的结合区域,靠近被调节的基因的PR结合峰信号强度显著地更高。还表明与转录调节不相关的结合事件可以是在细胞类型特异性位点,其需要仅在一个子集的环境可得的结合辅因子的合作[48,49,50]。还必须假定,一定比例的结合区域代表非特异性相互作用,尽管PRE在调节相关的和非相关的结合区域中是类似地普遍的发现结果将争辩非特异性相互作用解释整体结合的小部分。
实施例9-PR结合区域中的PRE
针对具有与之前描述的孕酮应答元件一致的序列的结合元件,PR结合区域被显著地富集[2,3]。在15个碱基对的回文结构的应答元件中在特定碱基位置的相对保守性是可变的并且与对于GR[46,50]和AR[51]观察到的保守性的模式一致。还检测了代表核心的高度保守的元件的较短的基序(CA/t nnn TGTnC,图4A),表明PR结合元件中的这些位置的特别的重要性。存在PRE序列的高度的可变性,如通过共有序列指示的,允许在若干位置的显著的变化,并且许多结合区域含有与理想的分歧相当大的弱序列。此外,一定比例的PR结合区域完全缺少共有PRE,提出了这些PR结合区域是否直接结合PR的问题。为了解决该问题,我们寻找仅在那些区域中的基序富集,并且确实以比较低的显著性水平鉴定了PRE样基序(未显示)。这表明缺乏共有PRE的许多结合区域确实含有与PRE一致的序列。报道了关于GR的类似的发现结果[46]。
尽管在PR结合区域中鉴定的PRE的存在和强度中存在可变性,这不是特定的区域是否与转录活性相关的决定因素,因为PRE强度既不与PR结合峰强度相关也不与转录结果相关。这表明PRE强度不是PR是否将与特定的结合区域相互作用并且调节基因表达的唯一的决定因素,并且其他序列特征以及DNA结合辅因子的影响可能是重要的决定因素。这得到了FOXA1、AP-1和NF1在检查的两个细胞系中被鉴定作为PR的潜在的细胞类型特异性结合辅因子的支持。
实施例10-T-47D和AB32细胞中的PR顺反组是非重叠的
T-47D和AB32细胞中的PR结合区域中存在相对地小的重叠。这与以下观察结果一致:对孕激素的转录应答在两个细胞系之间也是非重叠的。此外,两个细胞系共有的结合区域更不可能与转录结果相关。另外两个细胞系(ZR-75-1乳腺癌细胞和独立的PR阳性MCF-10A克隆(AB9))中的表达谱分析揭示了通过孕激素的转录调节中的类似地低的重叠。MCF-7乳腺癌细胞与表达ER的U2OS骨肉瘤细胞中的ER结合模式的比较揭示了结合位点和转录组中的类似地低的重叠[49]。在那个研究中,提出了不同的启动子甲基化作为该差异的基础。然而,AB32细胞中DNA甲基化的整体抑制,尽管增强存在的转录靶,未显著地改变孕激素应答的转录组(数据未显示)。
在我们的发现结果的支持方面,Yin和同事近期报道了当暴露于拮抗剂RU486时T-47D细胞和子宫平滑肌瘤细胞中的PR基因组相互作用中的类似地低的重叠[52]。将本文描述的示例性T-47D和AB32PR顺反组与公开可得的数据集比较揭示了两个T-47D数据集之间(51%报道的T-47D PR结合位点在我们的研究中同样在T-47D中被发现)比与AB32细胞中的结合(28%)的更大的重叠,支持了不同结合模式的观察结果的正确性。
在发表的研究中,用孕酮处理T-47D细胞,所述孕酮具有与ORG2058类似的药物代谢动力学谱,但比合成的类似物更快地从PR解离。此外,已经示出了PR在基因组DNA的移动性是配体特异性的[53],并且与孕酮相比,当结合ORG2058时可以不同。第二,通过不同的方法检测了PR结合:在该研究中使用了ChIP-seq,而发表的数据源自ChIP-chip。ChIP-seq以无偏性全基因组方式调查结合。ChIP-chip依赖于存在于使用的阵列上的序列,并且可受到杂交偏差影响。在MCF-7细胞中通过ChIP-seq和ChIP-chip检测的ER结合位点中观察到类似的重叠[47]。最后,用于产生发表的数据的分析方法不同于我们的研究中使用的。
实施例11-染色质结构和先锋因子FOXA1的作用
先锋因子诸如FOXA1能够与紧密包装的异染色质结合,打开DNA结构以允许被细胞核受体包括ER、GR和AR结合和调节[12,14,15,17,41,54]。对FOXA1需要的水平和FOXA1在受体信号传导中发挥的作用在受体间不同。FOXA1的表达对于通过ER的转录活化是关键的,尽管特定的基因靶在细胞系之间可以不同。在最近的研究中,Hurtado和同事在三种乳腺癌细胞系MCF-7、T-47D和ZR-75-1中绘制了ER和FOXA1结合的图谱,并且确定了沉默因子CTCF的定位在三个细胞系之间不同,并且定义了哪些ER靶通过FOXA1结合在转录上被增强。在这些细胞系中,FOXA1对于ER的作用是关键的[15]。
相比之下,FOXA1表现为在雄激素信号传导中发挥双重作用,其中FOXA1促进一些AR靶的雄激素应答而充当其他的AR靶的阻遏物。这得到在LNCaP前列腺癌细胞中的近期研究的支持,其中FOXA1的耗竭引起AR结合模式的显著重塑以及雄激素调节的转录本的显著的增加[41]。在该环境中,FOXA1是结合位点选择的决定因素,并且取决于靶位点而充当AR结合的促进物和阻遏物二者。
我们的数据表明,FOXA1可能在PR信号传导中发挥与AR类似的作用,因为FOXA1不是孕激素应答绝对需要的,并且在缺乏内源的FOXA1的AB32细胞中,FOXA1的过量表达引起那些细胞中的孕激素调节的基因的数目的显著的减少。在FOXA1丰富表达的T-47D细胞中,用于先锋因子的结合基序在PR结合区域中统计地富集。FOXA1在PR信号传导中通过含有FOXA1基序的区域的作用得到以下发现结果的支持:T-47D细胞中在这些位点的FOXA1结合水平大于在不含有预测的FOXA1基序的PR结合区域的相互作用。然而,T-47D细胞中所有PR结合区域周围的平均FOXA1结合与在ER相互作用位点的平均FOXA1结合的比较,揭示了与ER相比,靠近PR的FOXA1结合的显著较低的整体富集,表明了FOXA1不是所有PR相互作用必需的。总之,我们的数据表明,FOXA1可以充当T-47D中鉴定的许多靶的PR转录活化的增强剂,而在AB32细胞中,FOXA1表达的缺乏允许通常可以被FOXA1阻遏的PR靶的结合。
在T-47D和FOXA1转导的AB32细胞中的孕激素调节之间的重叠是低的,表明FOXA1表达未引起AB32细胞在其孕激素应答方面变得更像T-47D细胞。这与以下我们的观察结果一致:FOXA1可以不是T-47D细胞中的所有PR结合事件绝对需要的。还表明,尽管FOXA1可以影响PR结合,其他细胞特异性因子或特征在决定PR结合中是重要的,所述其他细胞特异性因子或特征可能是通过ChIP不可鉴定的。已经显示ER和AR二者以细胞类型和启动子特异性的方式与组蛋白修饰因子缔合[55,56],其被招募到增强子作为大的辅调节复合物的一部分,并且通过基序分析将是不可鉴定的。已经显示GR顺反组的性质高度地取决于染色质的可及性[57],这也是细胞类型特异性的。可能的是,相同的因子以细胞类型特异性的方式影响PR结合。
实施例12-AP-1和NF-1
已经显示,细胞核受体包括PR通过与AP-1的拴系,独立于激素应答元件地与DNA缔合[18,19,20]。在ER的情况中,该机制被报道为通过雌激素介导靶转录本的转录阻遏[14]。这些发现结果表明,AP-1结合位点在缺乏PRE的结合区域中可能更常见,并且可与下调的基因相关。在AB32结合区域中,AP-1位点存在于27%的含有PRE的区域中和29%的缺乏PRE的区域中。该比例整体上比在未观察到AP-1位点富集的T-47D细胞中的高(在T-47D中,12%的具有PRE的区域和10.7%的缺乏PRE的区域含有AP-1位点),然而,其在两个结合区域子集之间并无差异。不存在AP1位点在下调的基因中比在上调的基因中更普遍的证据(数据未显示)。这些数据表明,尽管在AB32细胞中的一个子集的结合位点上AP-1可以与PR合作,AP-1在孕酮信号传导中的作用可能比用于雌激素的更次要。
转录辅因子NF1与DNA的结合需要通过PR的共缔合,并且NF1和PR对基因表达具有协同作用[11],表明这些转录因子的共表达的潜能导致增强的转录结果。在乳腺中,NF1同种型的协调的表达参与了控制泌乳和退化(involution)[58]。NF1在乳腺中的作用是环境特异性的,并且当乳腺上皮细胞保持与富含层粘连蛋白的细胞外基质接触时被诱导[59]。NF1在乳腺中的发育特异性和环境特异性的作用表明,NF1与PR的相互影响可能受到NF1和PR二者的水平调节,并且这些可能对在包括癌发生的生理环境下的调节敏感。NF1结合基序在AB32细胞中但不在乳腺癌细胞中的PR结合区域中的富集,支持该观点,并且表明NF1是细胞类型限制性的PR辅因子。
染色质重塑辅因子的组合对于乳腺中的孕酮应答是重要的,并且这些辅因子的相对表达和协调的作用决定PR顺反组。孕酮在正常和恶性靶组织中具有不同范围的作用,并且该研究的结果表明,针对孕酮调节的顺反组的在关键辅因子和PR之间的相互影响有助于孕酮作用的环境特异性,并且在乳腺癌中的异常的孕激素作用中发挥核心作用。
实施例13-细胞培养
T-47D和ZR-75-1乳腺癌细胞系从E.G.and G.Mason Research Institute(Worcester,MA)获得。MCF-10A永生化正常乳腺细胞和HEK293T肾细胞从美国模式培养物保藏所(American Type Culture Collection)(atcc.org,Manassas,VA)获得。将T-47D和ZR-75-1细胞维持在含有10%胎牛血清和0.25单位/ml胰岛素的RPMI1640培养基中。将HEK293T维持在补充有10%胎牛血清的达尔伯克改良伊格尔培养基(Dulbecco's Modified Eagle's Medium)中。通过将PRA和PRB从病毒载体共引入MCF-10A细胞系和使用嘌呤霉素的克隆筛选,产生AB32和AB9细胞系。通过双重免疫荧光分析并且通过蛋白质印迹法表征了克隆的PRA和PRB的表达。图20中示出了将AB32和AB9中的PR表达与T-47D细胞中的PR水平进行比较的蛋白质印迹。将细胞维持在补充有霍乱毒素(0.1μg/ml)、胰岛素(0.28iu/ml)、氢化可的松(0.5μg/ml)、表皮生长因子(0.02μg/μl)和5%马血清的1∶1DMEM:Hams-F12培养基中。合成的孕激素ORG2058从Amersham Biosciences(GE Healthcare,Rydalmere,Australia)获得。TSA和5AdC从Sigma-Aldrich(Castle Hill,Australia)获得。
实施例14-染色质免疫沉淀
将细胞在15cm组织培养皿中培养到80%汇合,然后用10nM ORG2058或媒介物处理持续45分钟。随后,通过向培养基中添加甲醛到1%的终浓度并且在37℃孵育持续10分钟,使染色质交联。立即去除培养基,并且用冷的磷酸盐缓冲盐水洗涤细胞,并且通过刮擦收获细胞。通过离心收集细胞,并且将沉淀物在4℃的SDS缓冲液(1%SDS;10mM EDTA;50mM Tris-HCl,pH 8)中裂解10分钟。使用7次以40%振幅和60%负荷(duty)的一分钟脉冲(burst)(每次被至少一分钟的静止隔开),使用Branson 450超声波破碎器在4℃将裂解物进行声处理。将裂解物在4℃以13,000x g离心15分钟以去除碎片。从裂解物的等分部分分离基因组DNA,并且通过凝胶电泳检验以证实声处理已产生具有200bp到400bp平均尺寸的片段化的DNA。用IP缓冲液(0.5%NP-40;50mM Tris,pH 8;120mM NaCl;0.5mM PMSF;全蛋白酶抑制剂混合物,Roche,Ryde,Australia)以1:10稀释上清液,并且通过与Dynabeads M-280绵羊抗小鼠IgG磁珠(Invitrogen,Mulgrave,Australia)一起孵育,伴随在4℃温和旋转持续至少2h以减少与二抗珠的非特异性结合,进行预清洁。通过在4℃与内部的hPRa6和hPRa7小鼠单克隆抗PR抗体[60]和新配制的绵羊抗小鼠IgG磁珠(40ul每1.4ml稀释的裂解物)旋转过夜,分离结合PR的基因组DNA片段。将珠用IP缓冲液、高盐洗涤缓冲液(0.5%NP-40,50mM Tris,pH 8,500mM NaCl,0.5mM PMSF)、锂洗涤缓冲液(250mM LiCl,0.5%NP-40,1%脱氧胆酸钠,1mM EDTA,pH 8,10mM Tris-HCl,pH 8)和TE(10mM Tris,pH 8,1mM EDTA)顺序地洗涤。使用洗脱缓冲液(1%SDS,0.1M NaHCO3)在室温将分离的DNA片段洗脱两次,持续15分钟。通过添加0.25M NaCl并且加热到65℃持续至少6h使交联逆转。使用Qiagen PCR纯化柱(Qiagen,Doncaster,Australia)纯化DNA片段。将通过PR染色质免疫沉淀从ORG2058处理的细胞分离的DNA片段在基因功能分析Ramaciotti中心(the Ramaciotti Centre for Gene Function Analysis)(University of New South Wales,Australia)和GeneWorks(Hindmarsh,Australia)的Illumina GA-IIx测序仪上测序。将从预清洁的ORG2058处理的样品中分离的输入DNA测序作为基线对照。从ORG2058处理的、预清洁的样品中分离的DNA的等分部分充当新一代测序中的输入DNA对照样品。
实施例15-分析
使用Bowtie 0.12.0.1[37],将序列与重复序列屏蔽的人类基因组hg18(NCBI版本(build)36)比对。在比对的序列中,允许多达2个错配。多重比对允许多达10的多重性,但仅报告最佳排序的比对。该策略导致42%到48%的读段的比对。T-47D中的结果代表三个独立的生物重复和两个重复输入对照的组合的结果。AB32的结果来自两次独立的ChIP和每次的输入对照样品。除了来自T-47D细胞的一个ChIP和一个匹配的输入对照样品之外,所有序列是36bp读段长度。这些样品被处理成具有64个碱基对的读段长度,但在数据处理期间被修剪到36个碱基对,以避免在下游分析中的偏差。使用ERANGE开源软件工具[38]确定了富集的PR结合的区域。使用ERANGE findall.py(分析软件程序)中的位移学习函数确定了峰位移为70bp。将峰阈值设为如从对照输入DNA序列确定的背景的四倍。将区域以内的读段的最小数目(RPM)设为10。多个读段以1/多重性的值加权。以错误发现率0.27%调用峰。使用CisGenome v1.1[61]和Homer[62],相对于邻近的基因注释PR结合的区域。使用Homer和基序分析工具的MEME程序组版本4[63,64],鉴定了已知的和从头富集的结合基序。使用Fisher Exact Test确定了MEME中发现的结合基序的富集的显著性。富集的E值代表p值乘以测试的序列的数目。使用将结合区域序列与匹配的基因组背景比较的积累的超几何分布分析[62],在Homer中对基序富集评分。使用通过MEME发现的两个细胞系中的位置特异性概率矩阵,将MEME中的FIMO程序用于分类AB32和T-47D细胞中PR结合区域中的全长PRE的发生。报道了对于与共有PRE的相似性具有p值<0.01的序列,并且p值范围从0.01到1x10-10。较低的p值表示相比于共有PRE的较大的序列保守,并且为了比较的目的,p值<1x10-5被认为代表强的PRE。为了将T-47D细胞中的PR基因组相互作用与发表的ER和FOXA1相互作用物组(interactome)[42]比较,在Homer中从所有三个数据集产生了序列标签文库,并且对每个数据集使用相同的参数鉴定了结合峰。然后,使用Homer中的峰注释功能,确定了在PR、ER和FOXA1峰的平均FOXA1标签密度。通过ChIP-seq和基因表达谱分析产生的所有原始数据已被放置在Gene Expression Omnibus(www.ncbi.nlm.nih.gov/geo/)上,并且通过GEO登录号GSE31130可访问。基因表达数据符合MIAME指导方针。
实施例16-实时PCR
使用与关于ChIP-seq描述的相同的方案进行定向的ChIP。在RotorGene 6000实时PCR仪上使用Platinum Sybr Green试剂(Invitrogen)将靶模板定量。如以上描述的进行定向的ChIP,并且在通过实时qPCR定量之前,将纯化的gDNA片段稀释2到5倍。用于特定的靶验证的引物序列是:ACSL1-fwd 5'-TGC AAA GAG CAA GAC AGA AAA G-3'、rev-5'-GCG GTC ATA GAG ACA CAA TTC C-3',DHRS9-fwd 5'-GGC TGT CTG AGT GAA TCT GTA GTG-3'、rev-5'-AGT TAC ATT TGC CCT TGA TTC C-3',FLRT3-fwd 5'-GGA GAA ACA GAC TTT ACC TGA CC-3'、rev-5'-TGT TGC AGT CAA GGA GAC AGA G-3',NOTCH 2-fwd 5'-GCC TGT TCC TAT TAA GTG TCC TG-3'、rev-5'-GGC TGT AAA GTT ATT TGC TAG ATT G-3',PACSIN1-fwd 5'-AAC GTC CTC TTC CTG CTC TTG-3'、rev-5'-GAG CTT TGA TGT AGA CGG AAT-3'G,PDK4-fwd 5'-CG AGC AGC AAT AAC TTT CC-3'、rev-5'-ACG CAA GAA CAC AGT GAG TAG C-3'。
实施例17-慢病毒转导
从PlasmID Dana Faber/Harvard Cancer Center DNA Resource Core(Boston,MA)获得FOXA1cDNA。通过高保真PCR扩增开放阅读框,并且转移到pCDH-CMV-MCS-EF1-copGFP的多克隆位点。通过测序证实插入物的完整性。通过将pCDH-FOXA1-GFP载体和慢病毒包装构建体共转染进入HEK293T细胞,并且允许病毒在培养基中积累持续48h,产生慢病毒颗粒。使用Facs Calibur流式细胞仪来评价GFP阳性,AB32细胞中经过稀释系列的病毒滴度被评价。以预测产生70%感染率的水平感染AB32细胞,并且在37℃孵育持续24h以允许FOXA1的表达,随后用10nM ORG2058处理持续0h、6h和24h。用亲本pCDH-CMV-MCS-EF1-copGFP病毒感染的匹配的对照样品被包括在每个时间点。
实施例18-基因表达微阵列
使用RNAqueous纯化柱(Invitrogen)分离总RNA。使用Illumina TotalPrep试剂(Invitrogen)扩增总RNA(500ng),并且用生物素标记。使用推荐的Illumina试剂并且遵循制造商的方案,将扩增的样品(750ng)与人类全基因组HT-12基因表达珠阵列杂交。使用Genome Studio软件(Illumina)分析原始数据。在背景减除数据和三次样条归一化之后,使用Genome Studio的Illumina定制模型确定差异表达的p值。使用Microsoft Excel和SPSS统计软件进一步分析数据。使用GenePattern[65]进行分层聚簇分析(hierarchical clustering)和自组装映射聚簇分析。
实施例19-蛋白提取物制备和免疫印迹法
通过胰蛋白酶消化收集待通过蛋白免疫印迹法分析的细胞,用冷的磷酸盐缓冲盐水溶液洗涤,并且通过离心收集为细胞沉淀物。全细胞提取物通过在含有10mM NaMoO4、1%抑肽酶、全蛋白酶抑制剂(Roche,Castle Hill,Australia)和0.5mM苯甲基磺酰氟的RIPA缓冲液(10mM NaPO4(pH 7.0)、150mM NaCl、2mM EDTA、1%脱氧胆酸钠、1%NP-40、0.1%β-巯基乙醇)中裂解细胞,并且在4℃旋转15min来制备。通过在4℃以14,000x g离心15min,去除不溶的碎片。使用Bradford染料试剂(Bio-Rad,Regents Park,Australia)评价蛋白浓度。蛋白通过变性的7.5%聚丙烯酰胺-SDS凝胶通过电泳被分级,并且转移到硝酸纤维素膜,如之前描述的[66]。为了检测FOXA1的表达,以100μg每泳道加载T-47D全细胞提取物和以10μg每泳道加载转导的细胞的提取物。使用以1∶800稀释的山羊抗人类FOXA1多克隆抗体(Abcam ab5089,Sapphire Biosciences,Waterloo,Australia),以及兔抗山羊辣根过氧化物酶缀合的二抗(Dako Cytomation,Kingsgrove,Australia)检测FOXA1。为了检测PR蛋白的表达,如所示的加载全细胞提取物。使用hPRa6和hPRa7内部鼠单克隆抗体(各自1:100)和山羊抗小鼠辣根过氧化物酶缀合的二抗(Dako)检测PR。通过使用ECL试剂(Quantum Scientific,Murrarie,Australia)的化学发光反应,并且曝光于胶片或使用Kodak Image Station(Carestream Health,Richmond,Australia)成像使蛋白条带可视化。
实施例20-T-47D和AB32细胞中的PR结合位点的基因组分布。
如图1中示出的,使用Bowtie将通过ChIP-seq鉴定的孕激素依赖的PR结合DNA片段与基因组比对,并且使用ERANGE鉴定结合的峰,具有0.27%的FDR。(A和B)使用CisGenome确定(A)T-47D和(B)AB32细胞中PR结合区域相对于RefSeq基因的转录起始位点的位置。(C和D)在(C)T-47D和(D)AB32细胞中按基因数目排序的根据染色体的PR结合区域分布。(E和F)在(E)T-47D和(F)AB32细胞中在距TSS的给定距离以内具有PR结合区域的孕激素调节的基因的百分比。实线-与上调基因相关的结合区域,虚线-与下调基因相关的结合区域。指示了上调和下调基因到最靠近的TSS的中值距离。
实施例21-与孕激素应答基因相关的PR结合。
如图2中示出的,确定了在用10nM ORG2058处理后2h、6h和24h的T-47D和AB32细胞中的基因表达谱。相比于未处理的对照,在2h、6h和/或24h显著地差异地表达的转录本被认为被孕激素调节,并且与相同的细胞系中的PR结合区域的列表比较。(A和B)在(A)T-47D和(B)AB32细胞中PR结合区域与孕激素调节的基因之间的重叠。(C和D)在(C)T-47D和(D)AB32细胞中在2h、6h和24h在被调节的基因的10kb以内或在5’-UTR中的PR结合区域。(E)使用Gene Pattern通过SOM聚簇分析鉴定了T-47D细胞中分组在一起的具有孕激素调节模式的基因。调节模式被绘制为相对于未处理的对照的平均log倍数变化。误差条代表平均值的标准误差。(F)与(E)中示出的每个SOM聚簇相关的PR结合区域到TSS的平均距离。
实施例22-在T-47D和AB32中的差异PR结合和转录调节。
参考图3A,使用Bed Tools中的IntersectBed功能鉴定了T-47D和AB32细胞之间共有的PR结合区域。示出了T-47D或AB32细胞独特的或是两个细胞系共有的区域的数目和百分比。
参考图3B,用10nM ORG2058(ORG)或媒介物处理T-47D和AB32细胞持续2h、6h和24h。通过全基因组微阵列测量转录表达。在T-47D或AB32细胞中在一个或更多个时间点在ORG2058处理的样品中相对于未处理的对照差异表达的基因,通过无监督分层聚簇分析被比较。红色-相对于媒介物处理的对照增加的表达,绿色-相对于媒介物处理的对照减少的表达。
参考图3C,示出了在2h、6h或24h在T-47D和AB32细胞中被孕激素调节的转录本之间的重叠。示出了在T-47D或AB32中被孕激素独特地调节的转录本和是两个细胞系共有的调节的转录本的数目和百分比。
参考图3D,示出了在单独的处理时间在T-47D和AB32中孕激素调节的转录本的数目。示出了在特定的时间点在T-47D或AB32细胞中被独特地调节的或在二者中均被调节的转录本的数目。
实施例23-PR结合区域中的基序富集。
参考图4A,示出了在T-47D和AB32细胞中的结合区域中鉴定的全长和核心PRE。使用MEME-ChIP和Homer分析调节相关的PR结合区域序列的保守的序列基序的统计富集。在两个细胞系中发现了全长和核心PR结合元件。示出了通过MEME-ChIP鉴定的全长PRE和Homer中鉴定的核心元件。
参考图4B和C,示出了(B)T-47D和(C)AB32细胞中依据p值的PRE强度。PRE被分类为强(+++,p<1x10-5)、中等(++,p=1x10-5到1x10-3)或弱/不存在(+,p>1x10-3)。
参考图4D和4E,示出了(D)T-47D和(E)AB32细胞中调节和非调节相关的PR结合区域中的PR峰高。(F)T-47D和AB32细胞中上部转录因子结合基序的富集。
实施例24-将FOXA1引入AB32细胞内改变孕激素应答。
参考图5A,示出了在FOXA1的存在和不存在下,在AB32细胞中响应孕激素的转录谱的无监督聚簇分析。用包含pCDH-FOXA1构建体或空pCDH对照的病毒颗粒转导AB32细胞持续24h。用10nM ORG2058(ORG)或媒介物处理细胞6h和24h。通过全基因组微阵列测量基因的表达。通过无监督分层聚簇分析,分析与对照相比在ORG2058处理的细胞中在任何时间点差异表达的基因。红色-增加的log倍数表达,绿色-减少的log倍数表达。
参考图5B,示出了与T-47D细胞中的内源表达相比,在病毒转导之前和之后,在AB32细胞和亲本MCF-10A细胞中的FOXA1蛋白表达。
参考图5C,示出了在FOXA1的存在和不存在下,AB32细胞中孕激素调节的转录本的数目。
参考图5D,示出了在PR、ER和FOXA1结合位点的FOXA1结合强度的比较。
参考图5E,示出了针对随着FOXA1表达丢失、获得或保持孕激素调节的基因,PR结合区域与AB32细胞中被调节的基因的比率。
参考图5F,示出了AB32中从PR结合区域到随着FOXA1表达丢失、获得或保持孕激素调节的基因的距离。误差条代表平均值的标准误差。
实施例26-T-47D和AB32细胞中PR结合区域相对染色体的分布。
参考图6,通过T-47D和AB32数据集之间的线性回归比较每条染色体的PR结合区域的总数目。示出了拟合的线和95%置信区间。标签代表染色体号。
实施例27-PR结合和孕激素调节的时间之间的关系。
参考图7,确定了(A)T-47D和(B)AB32细胞中在处理后2h、6h或24h,与一个或更多个PR结合区域相关的孕激素调节的基因的比例。
实施例28-PR结合区域的位置。
参考图8,使用CisGenome v1.1确定了(A)T-47D和(B)AB32细胞中所有PR结合区域相对于最靠近的基因的分布。
实施例29-PR结合区域的位置。
参考图9,使用CisGenome v1.1确定了(A)T-47D和(B)AB32细胞中所有PR结合区域相对于最靠近的基因的分布。
实施例30-AB32细胞中转录调节的模式。
参考图10,通过基因表达谱分析鉴定了在用10nM ORG2058处理2h、6h和24h的AB32细胞中孕激素调节的转录本。使用Gene Pattern,通过自组装映射聚簇分析,鉴定了经过24h时间进程的转录调节的模式。
实施例31-T-47D和AB32细胞中的PR结合区域。
参考图11,T-47D或AB32细胞独特的或是两个细胞系共有的PR结合区域的实例被显示为在UCSC基因组浏览器中展示的定制的轨迹(custom track)。
实施例32-验证在ChIP-seq中鉴定的细胞类型特异性的PR结合区域。
参考图12,使用结合区域特异性的引物和定量实时PCR,通过定向的PR-ChIP验证在(A)T-47D、(B)AB32或(C)两个细胞系中通过ChIP-seq鉴定的PR结合区域。靠近在T-47D中但未在AB32中被调节的ACSL1和PACSIN1结合的区域,产生在T-47D细胞中且未在AB32中的结合的片段的显著的富集。相反的情况对于在AB32中但未在T-47D中鉴定的PR靶区域是真实的。FLRT3和DHRS9(二者都是仅在AB32中的转录靶)在AB32中被PR强烈地结合,但在T-47D细胞中显示弱的缔合。PDK4和Notch 2(其在两个细胞系中都被孕激素调节)在两个细胞系中被PR结合,尽管在T-47D中的缔合更强(在T-47D与AB32中,PDK-4的85倍与42倍结合富集和Notch 2结合的300倍与37倍富集)。
实施例33-ORG2058处理的T-47D和AB32中PR结合区域与孕酮(P4)处理之后的T-47D中的结合的重叠(图13)。
将我们的数据与在Tang等[1]中概述的并且在http://cistrome.dfci.harvard.edu/NR_Cistrome/可得的T-47D中孕酮配合的PR结合比较。
实施例34-另外的乳腺细胞系中基因表达的孕激素调节。
参考图14,用10nM ORG2058或媒介物处理ZR-75-1乳腺癌细胞和AB9PR阳性转化的正常乳腺细胞2h、6h或24h,然后收集细胞并分离总RNA。通过Illumina HT-12微阵列评价基因表达水平。使用Genome Studio软件分析数据。相比于媒介物处理的细胞,具有在ORG2058中显著地不同的(diff p值<0.01)并且具有1.5或更多的倍数变化的水平的转录本被认为被孕激素调节。(A)ZR-75-1或AB9细胞或二者中孕激素调节的转录本的数目。(B)使用Gene Pattern,对在一个或两个细胞系中被孕激素调节的转录本的子集进行阵列(皮尔逊相关)和基因表达倍数变化(非中心的相关)的无监督平均连锁分层聚簇分析。红色-相对于媒介物通过ORG2058增加的表达,绿色-相对于媒介物通过ORG2058减少的表达。
实施例35-T-47D和AB32细胞中在调节相关的结合位点中PRE和辅因子基序的富集。
参考图15,示出了含有PRE具有或不具有一个或更多个上部富集的转录辅因子结合基序的调节相关的PR结合区域的相对比例。示出了(A)T-47D和(B)AB32的基序分布。
实施例36-T-47D和AB32细胞中PR结合区域中PRE位置的分布。
参考图16,(A)T-47D和(B)AB32细胞中的PR结合区域中PRE基序相对于峰中心的位置被绘制成频率分布。
实施例37-PRE强度不预测PR结合。
参考图17,使用MEME中的FIMO程序[2],将PR结合区域中的PRE基序分类。将每个结合区域中最强的候选PRE的强度(如由p值确定的)相对于峰高绘图,作为PR结合强度的指示。评价了估计的拟合线和皮尔逊相关R2值。示出了(A)T-47D和(B)AB32细胞的数据。
实施例38-细胞系中FOXA1的转录表达。
参考图18,将在Illumina HT-12阵列上测量的在乳腺癌(T-47D、ZR-75-1)和转化的正常乳腺细胞(AB9、AB32)中的FOXA1转录表达比较。FOXA1水平相对于AB32细胞中的水平表示。
实施例39-在具有或不具有预测的FOXA1基序的PR结合区域的FOXA1结合。
参考图19,使用Homer软件预测PR结合区域中FOXA1基序的存在。预测为结合FOXA1的PR结合区域和缺乏FOXA1结合基序的区域被单独地分析实际的FOXA1结合富集。示出了T-47D中来自ChIP-seq的在含有FOXA1基序(蓝色线)的PR结合区域和缺乏任何预测的FOXA1基序(红色线)的PR结合区域的平均FOXA1结合强度。
实施例40-在T-47D、AB32和AB9细胞中的PR表达。
参考图20,以所示的加载的来自全细胞提取物的蛋白,通过变性的7.5%聚丙烯酰胺-SDS凝胶电泳被分级并且转移到硝酸纤维素膜。PR蛋白条带被可视化,如本文中描述的。
实施例41-表S1:AB32中根据染色体的基因功能注释的小结
确定了AB32细胞中染色体2、8和11上PR结合区域相关的基因的子集的基因本体(gene ontology)方面的富集。
实施例42-表S2:上部PR结合区域的验证
通过定向的ChIP-PCR证实PR与T-47D和AB32细胞中选择的位点结合。选择T-47D中的10个结合区域和AB32细胞中的9个用于验证。
T-47D
AB32
实施例43-表S3:随着FOXA1的表达丢失、获得和保留孕激素调节的基因簇的功能分析。
对在表达FOXA1后AB32细胞中丢失、获得和保留孕激素调节的基因的组进行功能注释聚簇分析。
实施例44-通过奥那司酮减弱PR结合(图21-24)
我们选择了若干基因组区域,所述基因组区域我们之前在T-47D细胞中通过ChIP-seq显示被PR结合,并且靠近这些细胞中被孕激素ORG2058(使用10nM的药物浓度在6h)调节的基因。
我们在T-47D细胞中和第二乳腺癌细胞系HCC1428中用ORG2058、奥那司酮、其组合或媒介物进行ChIP-PCR。使用的浓度是1nM ORG2058和100nM奥那司酮。这是基于揭示在该浓度的ORG作用最大并且被奥那司酮消除的初步实验。
在两个细胞系中测试了十个PR结合区域,并且证实了10个中的9个被ORG2058配合的PR结合并被奥那司酮减弱。在一方面,我们观察到在单独的奥那司酮的存在下,PR与一些靶结合。这主要在T-47D细胞中,并且考虑到奥那司酮被报道阻断DNA被PR结合,是出人意料的。
实施例45-表达谱分析(图21-24)
表达谱分析揭示了309个差异地表达的探针,代表在一个或两个细胞系中被1nM ORG2058调节的267个基因。除了T-47D中的5个基因和HCC1428中的6个基因之外,奥那司酮消除了所有基因的孕激素调节。通常孕激素应答在T-47D细胞中更显著,所述T-47D细胞以显著高于HCC1428的水平表达PR。
总体说来,在两个不相关的细胞系中的数据证实,被孕激素活化的PR可再现地与一系列的定义的DNA靶结合,并且该结合被奥那司酮减弱。
尽管以上的描述涉及特定的方面,应该理解这些方面仅仅是阐述性的。对本领域技术人员将明显的是,可以对本文描述的多态形式和方法做出多种修改和变化。因此,本描述意图包括在描述的范围内的修改和变化形式及其等价物。
参考文献
本文中引用的参考文献通过引用以其整体并入。
1.Hubler TR,Scammell JG(2004)Intronic hormone response elements mediate regulation of FKBP5by progestins and glucocorticoids.Cell Stress Chaperones 9:243-252.
2.Lieberman BA,Bona BJ,Edwards DP,Nordeen SK(1993)The constitution of a progesterone response element.Mol Endocrinol 7:515-527.
3.Nelson CC,Hendy SC,Shukin RJ,Cheng H,Bruchovsky N,等(1999)Determinants of DNA sequence specificity of the androgen,progesterone,and glucocorticoid receptors:evidence for differential steroid receptor response elements.Mol Endocrinol 13:2090-2107.
4.Onate SA,Tsai SY,Tsai MJ,O'Malley BW(1995)Sequence and characterization of a coactivator for the steroid hormone receptor superfamily.Science 270:1354-1357.
5.Liu Z,Wong J,Tsai SY,Tsai MJ,O'Malley BW(2001)Sequential recruitment of steroid receptor coactivator-1(SRC-1)and p300 enhances progesterone receptor-dependent initiation and reinitiation of transcription from chromatin.Proc Natl Acad Sci U S A 98:12426-12431.
6.McKenna NJ,O'Malley BW(2002)Combinatorial control of gene expression by nuclear receptors and coregulators.Cell 108:465-474.
7.Scarpin KM,Graham JD,Mote PA,Clarke CL(2009)Progesterone action in human tissues:regulation by progesterone receptor(PR)isoform expression,nuclear positioning and coregulator expression.Nucl Recept Signal7:e009.
8.Vicent GP,Zaurin R,Nacht AS,Li A,Font-Mateu J,等(2009)Two chromatin remodeling activities cooperate during activation of hormone responsive promoters.PLoS Genet 5:e1000567.
9.Vicent GP,Ballare C,Zaurin R,Saragueta P,Beato M(2006)Chromatin remodeling and control of cell proliferation by progestins via cross talk of progesterone receptor with the estrogen receptors and kinase signaling pathways.Ann N Y Acad Sci 1089:59-72.
10.Vicent GP,Nacht AS,Zaurin R,Ballare C,Clausell J,等(2010)Minireview:role of kinases and chromatin remodeling in progesterone signaling to chromatin.Mol Endocrinol 24:2088-2098.
11.Vicent GP,Zaurin R,Nacht AS,Font-Mateu J,Le Dily F,等(2010)Nuclear factor 1 synergizes with progesterone receptor on the mouse mammary tumor virus promoter wrapped around a histone H3/H4tetramer by facilitating access to the central hormone-responsive elements.J Biol Chem 285:2622-2631.
12.Friedman JR,Kaestner KH(2006)The Foxa family of transcription factors in development and metabolism.Cell Mol Life Sci 63:2317-2328.
13.Bernardo GM,Lozada KL,Miedler JD,Harburg G,Hewitt SC,等(2010)FOXA1is an essential determinant of ERalpha expression and mammary ductal morphogenesis.Development 137:2045-2054.
14.Carroll JS,Meyer CA,Song J,Li W,Geistlinger TR,等(2006)Genome-wide analysis of estrogen receptor binding sites.Nat Genet 38:1289-1297.
15.Hurtado A,Holmes KA,Ross-Innes CS,Schmidt D,Carroll JS(2011)FOXA1is a key determinant of estrogen receptor function and endocrine response.Nat Genet 43:27-33.
16.Lupien M,Eeckhoute J,Meyer CA,Wang Q,Zhang Y,等(2008)FoxA1translates epigenetic signatures into enhancer-driven lineage-specific transcription.Cell 132:958-970.
17.Wang Q,Li W,Liu XS,Carroll JS,Janne OA,等(2007)A hierarchical network of transcription factors governs androgen receptor-dependent prostate cancer growth.Mol Cell 27:380-392.
18.Cicatiello L,Addeo R,Sasso A,Altucci L,Petrizzi VB,等(2004)Estrogens and progesterone promote persistent CCND1 gene activation during G1by inducing transcriptional derepression via c-Jun/c-Fos/estrogen receptor(progesterone receptor)complex assembly to a distal regulatory element and recruitment of cyclin D1 to its own gene promoter.Mol Cell Biol 24:7260-7274.
19.Kushner PJ,Agard DA,Greene GL,Scanlan TS,Shiau AK,等(2000)Estrogen receptor pathways to AP-1.J Steroid Biochem Mol Biol 74:311-317.
20.Tseng L,Tang M,Wang Z,Mazella J(2003)Progesterone receptor(hPR)upregulates the fibronectin promoter activity in human decidual fibroblasts.DNA Cell Biol 22:633-640.
21.Beguelin W,Diaz Flaque MC,Proietti CJ,Cayrol F,Rivas MA,等(2010)Progesterone receptor induces ErbB-2nuclear translocation to promote breast cancer growth via a novel transcriptional effect:ErbB-2 function as a coactivator of Stat3.Mol Cell Biol 30:5456-5472.
22.Faivre EJ,Daniel AR,Hillard CJ,Lange CA(2008)Progesterone receptor rapid signaling mediates serine 345phosphorylation and tethering to specificity protein 1transcription factors.Mol Endocrinol 22:823-837.
23.Graham JD,Clarke CL(1997)Physiological action of progesterone in target tissues.Endocr Rev 18:502-519.
24.Clarke CL,Sutherland RL(1990)Progestin regulation of cellular proliferation.Endocr Rev 11:266-301.
25.Longacre TA,Bartow SA(1986)A correlative morphologic study of human breast and endometrium in the menstrual cycle.Am J Surg Pathol 10:382-393.
26.Ace CI,Okulicz WC(2004)Microarray profiling of progesterone-regulated endometrial genes during the rhesus monkey secretory phase.Reprod Biol Endocrinol 2:54.
27.Borthwick JM,Charnock-Jones DS,Tom BD,Hull ML,Teirney R,等(2003)Determination of the transcript profile of human endometrium.Mol Hum Reprod 9:19-33.
28.Graham JD,Yager ML,Hill HD,Byth K,O’Neill GM,等(2005)Altered progesterone receptor isoform expression remodels progestin responsiveness of breast cancer cells.Mol Endocrinol 19:2713-2735.
29.Kao LC,Tulac S,Lobo S,Imani B,Yang JP,等(2002)Global gene profiling in human endometrium during the window of implantation.Endocrinology 143:2119-2138.
30.Richer JK,Jacobsen BM,Manning NG,Abel MG,Wolf DM,等(2002)Differential gene regulation by the two progesterone receptor isoforms in human breast cancer cells.J Biol Chem 277:5209-5218.
31.Beral V,Million Women Study C(2003)Breast cancer and hormone-replacement therapy in the Million Women Study.Lancet 362:419-427.
32.Beral V,Reeves G,Bull D,Green J Breast Cancer Risk in Relation to the Interval Between Menopause and Starting Hormone Therapy.J Natl Cancer Inst 103:296-305.
33.Rossouw JE,Anderson GL,Prentice RL,LaCroix AZ,Kooperberg C,等(2002)Risks and benefits of estrogen plus progestin in healthy postmenopausal women:principal results From the Women's Health Initiative randomized controlled trial.JAMA 288:321-333.
34.Beral V,Reeves G,Bull D,Green J(2011)Breast cancer risk in relation to the interval between menopause and starting hormone therapy.J Natl Cancer Inst 103:296-305.
35.Chlebowski RT,Anderson GL,Gass M,Lane DS,Aragaki AK,等(2010)Estrogen plus progestin and breast cancer incidence and mortality in postmenopausal women.JAMA 304:1684-1692.
36.Graham JD,Mote PA,Salagame U,van Dijk JH,Balleine RL,等(2009)DNA replication licensing and progenitor numbers are increased by progesterone in normal human breast.Endocrinology 150:3318-3326.
37.Langmead B,Trapnell C,Pop M,Salzberg SL(2009)Ultrafast and memory-efficient alignment of short DNA sequences to the human genome.Genome Biol 10:R25.
38.Mortazavi A,Williams BA,McCue K,Schaeffer L,Wold B(2008)Mapping and quantifying mammalian transcriptomes by RNA-Seq.Nat Methods 5:621-628.
39.Roschke AV,Tonon G,Gehlhaus KS,McTyre N,Bussey KJ,等(2003)Karyotypic complexity of the NCI-60drug-screening panel.Cancer Res 63:8634-8647.
40.Tang Q,Chen Y,Meyer C,Geistlinger T,Lupien M,等(2011)A comprehensive view of nuclear receptor cancer cistromes.Cancer Res71:6940-6947.
41.Wang D,Garcia-Bassets I,Benner C,Li W,Su X,等(2011)Reprogramming transcription by distinct classes of enhancers functionally defined by eRNA.Nature 474:390-394.
42.Joseph R,Orlov YL,Huss M,Sun W,Kong SL,等(2010)Integrative model of genomic factors for determining binding site selection by estrogen receptor-alpha.Mol Syst Biol 6:456.
43.Bravieri R,Shiyanova T,Chen TH,Overdier D,Liao X(1997)Different DNA contact schemes are used by two winged helix proteins to recognize a DNA binding sequence.Nucleic Acids Res 25:2888-2896.
44.Cirillo LA,Zaret KS(2007)Specific interactions of the wing domains of FOXA1 transcription factor with DNA.J Mol Biol 366:720-724.
45.Pierrou S,Hellqvist M,Samuelsson L,Enerback S,Carlsson P(1994)Cloning and characterization of seven human forkhead proteins:binding site specificity and DNA bending.EMBO J 13:5002-5012.
46.Reddy TE,Pauli F,Sprouse RO,Neff NF,Newberry KM,等(2009)Genomic determination of the glucocorticoid response reveals unexpected mechanisms of gene regulation.Genome Res 19:2163-2171.
47.Welboren WJ,van Driel MA,Janssen-Megens EM,van Heeringen SJ,Sweep FC,等(2009)ChIP-Seq of ERalpha and RNA polymerase II defines genes differentially responding to ligands.EMBO J 28:1418-1428.
48.Macquarrie KL,Fong AP,Morse RH,Tapscott SJ(2011)Genome-wide transcription factor binding:beyond direct target regulation.Trends Genetics 27:141-148.
49.Krum SA,Miranda-Carboni GA,Lupien M,Eeckhoute J,Carroll JS,等(2008)Unique ERalpha cistromes control cell type-specific gene regulation.Molecular Endocrinology 22:2393-2406.
50.So AY,Chaivorapol C,Bolton EC,Li H,Yamamoto KR(2007)Determinants of cell-and gene-specific transcriptional regulation by the glucocorticoid receptor.PLoS Genet 3:e94.
51.Bolton EC,So AY,Chaivorapol C,Haqq CM,Li H,等(2007)Cell-and gene-specific regulation of primary target genes by the androgen receptor.Genes Dev 21:2005-2017.
52.Yin P,Roqueiro D,Huang L,Owen JK,Xie A,等(2012)Genome-wide progesterone receptor binding:cell type-specific and shared mechanisms in T47D breast cancer cells and primary leiomyoma cells.PLoS One 7:e29021.
53.Rayasam GV,Elbi C,Walker DA,Wolford R,Fletcher TM,等(2005)Ligand-specific dynamics of the progesterone receptor in living cells and during chromatin remodeling in vitro.Mol Cell Biol 25:2406-2418.
54.Belikov S,Astrand C,Wrange O(2009)FoxA1 binding directs chromatin structure and the functional response of a glucocorticoid receptor-regulated promoter.Mol Cell Biol 29:5413-5425.
55.Garcia-Bassets I,Kwon Y-S,Telese F,Prefontaine GG,Hutt KR,等(2007)Histone methylation-dpendent mechanisms impose ligand dependency for gene activation by nuclear receptors.Cell 128:505-518.
56.Metzger E,Wissmann M,Yin N,Muller JM,Schneider R,等(2005)LSD1 demethylates repressive histone marks to promote androgen-receptor-dependent transcription.Nature 437:436-439.
57.John S,Sabo PJ,Thurman RE,Sung MH,Biddie SC,等(2011)Chromatin accessibility pre-determines glucocorticoid receptor binding patterns.Nat Genet 43:264-268.
58.Murtagh J,Martin F,Gronostajski RM(2003)The Nuclear Factor I(NFI)gene family in mammary gland development and function.J Mammary Gland Biol Neoplasia 8:241-254.
59.Streuli CH,Edwards GM,Delcommenne M,Whitelaw CB,Burdon TG,等(1995)Stat5as a target for regulation by extracellular matrix.J Biol Chem 270:21639-21644.
60.Clarke CL,Zaino RJ,Feil PD,Miller JV,Steck ME,等(1987)Monoclonal antibodies to human progesterone receptor:characterization by biochemical and immunohistochemical techniques.Endocrinology 121:1123-1132.
61.Ji H,Jiang H,Ma W,Johnson DS,Myers RM,等(2008)An integrated software system for analyzing ChIP-chip and ChIP-seq data.Nat Biotechnol 26:1293-1300.
62.Heinz S,Benner C,Spann N,Bertolino E,Lin YC,等(2010)Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities.Mol Cell 38:576-589.
63.Bailey TL,Williams N,Misleh C,Li WW(2006)MEME:discovering and analyzing DNA and protein sequence motifs.Nucleic Acids Res 34:W369-373.
64.Bailey TL,Boden M,Whitington T,Machanick P(2010)The value of position-specific priors in motif discovery using MEME.BMC Bioinformatics 11:179.
65.Reich M,Liefeld T,Gould J,Lerner J,Tamayo P,等(2006)GenePattern 2.0.Nat Genet 38:500-501.
66.Graham JD,Yeates C,Balleine RL,Harvey SS,Milliken JS,等(1995)Characterization of progesterone receptor A and B expression in human breast cancer.Cancer Res 55:5063-5068.
67.Tang Q,Chen Y,Meyer C,Geistlinger T,Lupien M,等(2011)A comprehensive view of nuclear receptor cancer cistromes.Cancer Res 71:6940-6947.
68.Bailey TL,Williams N,Misleh C,Li WW(2006)MEME:discovering and analyzing DNA and protein sequence motifs.Nucleic Acids Res 34:W369-373.
缩略词(通过引用以其整体并入本文)
AP-1 活化物蛋白1
AR 雄激素受体
Bowtie 序列比对软件(http://bowtie-bio.sourceforge.net)
CD44 细胞表面糖蛋白CD44
ChIP 染色质免疫沉淀
Ct 定量实时PCR反应中的循环阈值
CTCF CCCTC-结合因子
ER 雌激素受体
ERANGE findall.py ChIP 峰鉴定软件工具
(http://woldlab.caltech.edu/rnaseq)
FDR 错误发现率
FOXA1 叉头框A1
GR 糖皮质激素受体
Homer 用于ChIP峰鉴定和DNA结合基序鉴定的序列分
析软件(http://biowhat.ucsd.edu/homer/)
MEME-ChIP 用于特别地在ChIP-seq数据中的DNA结合基序鉴
定的在线序列分析工具(http://meme.nbcr.net/)
NF1 细胞核因子1
ORG Organon 2058
PRE 孕酮应答元件
SOM 自组装映射
SP1 特化蛋白1
Stat3 转录的信号转导物和活化物
TSS 转录起始位点
- 还没有人留言评论。精彩留言会获得点赞!
- 作为5-HT<sub>2A</sub>血清素受体调节剂适用于治疗与其相关的病症的3-苯基-吡唑衍生物的制作方法
- 作为5-HT<sub>2A</sub> 血清素受体调节剂适用于治疗与其相关的病症的3-苯基-吡唑衍生物的制作方法
- 用于诊断表达her2受体或其截短形式的癌症的方法
- 人T2R受体hT2R50的新单体型及其在鉴定人苦味调节剂的测定法中的用途的制作方法
- 一种新的多肽——嗅觉受体27.61 和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——t1/st2受体结合蛋白10.23和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人嘌呤受体9.46和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人嘌呤受体13.75和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人sigma受体15.07和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——补体受体12和编码这种多肽的多核苷酸的制作方法
- 5‑HT2B拮抗剂的制造方法与工艺
- 1‑(噻唑‑2‑基)哌啶‑4‑醇的合成方法与制造工艺
- 一种大麻酚类化合物的合成方法与制造工艺
- 一类对GPR84具有激动作用的化合物及其制备方法和用途与制造工艺
- eGFP标记GPR120阳性细胞的小鼠模型构建方法
- 靶向生长抑素受体的Al<sup>18</sup>F?NOTA?PEG<sub>6</sub>?TATE及其制备方法和应用
- 吡咯烷衍生物的口服制剂的制作方法
- 胰高血糖素样肽-1受体激动剂制备治疗肺动脉高压药物的应用
- 一种armc5基因检测试剂盒及突变位点的制作方法
- 作为mglu5受体的负性别构调节剂的6-氟-2-[4-(吡啶-2-基)丁-3-炔-1-基]咪唑并[1,2a ...的制作方法