非生物胁迫条件下改良农艺性状的植物和方法与流程
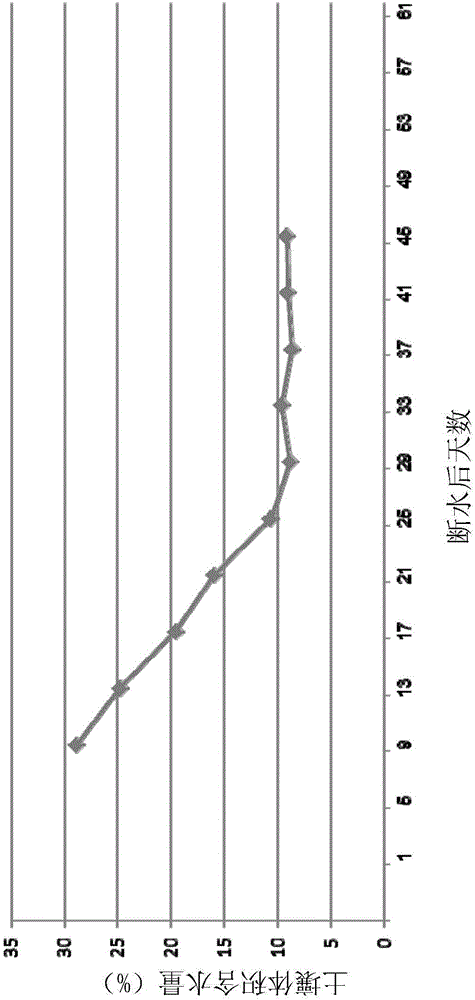
本发明涉及植物育种和遗传学,特别是,涉及用于提高植物诸如干旱的非生物胁迫抗性的重组DNA载体。
背景技术:
:生物和非生物原因可以对植物产生胁迫,例如造成生物胁迫的原因包括病原菌感染、昆虫取食、一种植物对另一种植物的寄生,例如槲寄生;非生物胁迫包括诸如有效水分的过量或者不足、极限温度和诸如除草剂的合成化学药品。非生物胁迫是造成世界范围内农作物减产的主要原因,造成主要农作物平均减产50%以上(Boyer,J.S.(1982)Science218:443-448;Bray,E.A.等(2000)InBiochemistryandMolecularBiologyofPlants,editedbyBuchannan,B.B.等,Amer.Soc.PlantBiol.,pp.1158-1249)。植物固着于地面,必须调整以适应周围的环境条件,这就导致了植物发育中基因调控、形态建成和新陈代谢的巨大可塑性。植物的适应和防御策略包括激活编码重要蛋白的基因,这些蛋白可以使植物适应或防御不同胁迫条件。干旱(有效水分不足)是一种主要的非生物胁迫,限制了世界范围内农作物的生产。在植物生长发育阶段,将植物暴露于水分限制环境下,将会激活植物各种不同生理和发育变化。近年来,尽管对非生物胁迫响应的分子机制和植物耐旱性的遗传调控网络进行了广泛的研究(Valliyodan,B.和Nguyen,H.T.(2006)Curr.Opin.PlantBiol.9:189-195;Wang,W.等(2003)Planta218:1-14;Vinocur,B.和Altman,A.(2005)Curr.Opin.Biotechnol.16:123-132;Chaves,M.M.和Oliveira,M.M.(2004)J.Exp.Bot.55:2365-2384;Shinozaki,K.等(2003)Curr.Opin.PlantBiol.6:410-417;Yamaguchi-Shinozaki,K.和Shinozaki,K.(2005)TrendsPlantSci.10:88-94),但是植物如何感受、传导干旱胁迫信号和其耐旱性的生物化学和分子机制仍然是生物学研究中面临的一个主要挑战。遗传学研究表明植物的耐旱性是一个由多基因调控的数量性状,分子标记辅助育种能够提高农作物的耐旱性,但是标记的准确性和育种效率仍然是有疑问的(AshrafM.(2010)Biotechnol.Adv.28:169-183)。转基因途径提高农作物的耐旱性已经取得了很大进展(VinocurB.andAltmanA.(2005)Curr.Opin.Biotechnol.16:123-132;LawlorDW.(2013)J.Exp.Bot.64:83-108)。早期非生物胁迫响应分子方面的研究主要借助差异和/或加减法分析(Bray,E.A.(1993)PlantPhysiol.103:1035-1040;Shinozaki,K.和Yamaguchi-Shinozaki,K.(1997)PlantPhysiol.115:327-334;Zhu,J.-K.等(1997)Crit.Rev.PlantSci.16:253-277;Thomashow,M.F.(1999)Annu.Rev.PlantPhysiol.PlantMol.Biol.50:571-599);和其它的方法,如分离候选基因,分析胁迫条件下该基因的表达或活性产物,或进行特定胁迫条件下功能互补分析(Xiong,L.和Zhu,J.-K.(2001)PhysiologiaPlantarum112:152-166)。另外,用于鉴定和分离调控基因突变体的正向和反向遗传学研究为胁迫条件下基因表达的变化提供了证据(Xiong,L.和Zhu,J.-K.(2001)PhysiologiaPlantarum112:152-166)。激活标签可以用于鉴定影响植物性状的基因,这一方法已经用于模式植物拟南芥的研究中(Weigel,D.等(2000)PlantPhysiol.122:1003-1013)。转录增强子序列的插入主要激活和/或提高邻近内源基因的表达,因此该方法可以用于分离具有重要农艺性状表型的基因,包括提高非生物胁迫如耐旱性的基因。发明概述本发明包括以下具体实施方案:在一个实施方案中,本发明包括一种分离的多核苷酸,所述多核苷酸包括(a)一种多核苷酸,其核苷酸序列与SEQIDNO:3、6、9、12、15或18的序列一致性至少为85%;(b)一种多核苷酸,其核苷酸序列与SEQIDNO:4、7、10、13、16或19的序列一致性至少为85%;(c)一种多核苷酸,其编码多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20的序列一致性至少为90%;或(d)核苷酸序列(a)、(b)或(c)的全长互补序列,其中过量表达所述多核苷酸能够提高植物的耐旱性。所述分离的多核苷酸包括SEQIDNO:3、4、6、7、9、10、12、13、15、16、18或19所示的核苷酸序列;所述多肽包括SEQIDNO:5、8、11、14、17或20所示的氨基酸序列。在另一个实施方案中,本发明包括一个重组DNA构建体,所述重组DNA构建体包括一个分离的多核苷酸和与其可操作的连接至少一个异源调控序列,其中所述多核苷酸包括(a)一种多核苷酸,其核苷酸序列与SEQIDNO:3、4、6、7、9、10、12、13、15、16、18或19的序列一致性至少为85%;(b)一种多核苷酸,其编码多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20的序列一致性至少为90%;或者(c)核苷酸序列(a)或(b)的全长互补序列。在另一个实施例中,本发明包括一种转基因的植物或种子,所述植物或种子包括一个重组DNA构建体,所述重组DNA构建体包括一个多核苷酸和与其可操作的连接至少一个调控序列,其中所述多核苷酸包括(a)一个多核苷酸,其核苷酸序列与SEQIDNO:3、4、6、7、9、10、12、13、15、16、18或19的序列一致性至少为85%;(b)一种多核苷酸,其编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20的序列一致性至少为90%;或(c)核苷酸序列(a)或(b)的全长互补序列。在另一个实施例中,本发明包括一个转基因植物,所述转基因植物在其基因组中包括一个重组DNA构建体,所述重组DNA构建体包括一个多核苷酸和与其可操作的连接至少一个调控序列,其中所述多核苷酸序列包括(a)一个多核苷酸,其核苷酸序列与SEQIDNO:3、4、6、7、9、10、12、13、15、16、18或19的序列一致性至少为85%;(b)一个多核苷酸,其编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20的序列一致性至少为90%;或(c)核苷酸序列(a)或(b)的全长互补序列,其中,与对照植物相比,所述转基因植物显示提高的耐旱性能。在另一个实施方案中,本发明包括任一公开的植物,所述植物选自水稻、玉米、大豆、向日葵、高粱、油菜、小麦、苜蓿、棉花、大麦、粟、甘蔗或柳枝稷。在另一实施方案中,公开了提高植物耐旱性的方法,所述方法包括的步骤为:(a)将重组DNA构建体转入可再生植物细胞,所述重组DNA构建体包括一个多核苷酸和与其可操作连接的至少一个调控序列,其中所述多核苷酸编码的多肽的氨基酸能序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%的序列一致性;(b)步骤(a)后由可再生植物细胞再生转基因植物,其中所述转基因在其基因组中含有重组DNA构建体;和(c)获得步骤(b)转基因植物的子代植物,其中所述子代植物在其基因组中含有重组DNA构建体;与不含有重组DNA构建体的对照植物相比,所述子代植物显示出提高的耐旱性。在另一实施方案中,公开了评估植物耐旱性的方法,所述方法包括的步骤为:(a)将重组DNA构建体转入可再生植物细胞,所述重组DNA构建体包括一个多核苷酸和与其可操作连接的至少一个调控序列,其中所述多核苷酸编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%的序列一致性;(b)步骤(a)后由可再生植物细胞再生出转基因植物,其中所述转基因植物在其基因组中含有重组DNA构建体;(c)获得转基因植物的子代植物,其中所述子代植物在其基因组中含有重组DNA构建体;和(d)与不含有重组DNA构建体的对照植物相比,评估所述子代植物的耐旱性。在另一个实施方案中,本发明涉及重组DNA构建体,所述重组DNA构建体包含本发明任一分离的多核苷酸和与其可操作连接至少一个调控序列;以及含有所述重组DNA构建体的细胞、植物或种子。所述细胞可以是真核细胞,如酵母、昆虫或植物细胞;也可以是原核细胞,诸如细菌细胞。附图说明和序列表根据以下发明详述和附图及序列表,可以更全面的理解本发明,以下发明详述和附图及序列表形成本发明的一部分。图1.为OsGSTU41转基因水稻第一次田间干旱试验中不同发育阶段的土壤体积含水量。OsGSTU41转基因水稻在断水后26天开始抽穗,断水后63天成熟。图2.为OsGSTU41转基因水稻第二次田间干旱试验中不同发育阶段的土壤体积含水量。OsGSTU41转基因水稻在断水后43天开始抽穗,断水后73天成熟。图3.为实时PCR分析测定的不同转基因水稻株系叶片中OsGSTU41基因的相对表达量。DP0043.15中的表达量设置为1.00,其它转基因株系表达量柱上面的数字表示与DP0043.15相比的变化倍数,ZH11为野生型水稻。图4.为实时PCR分析测试的不同转基因水稻株系叶片中OsPPCK4基因的相对表达量。ZH11-TC中的OsPPCK4基因表达水平设置为1.00,OsPPCK4转基因株系中相对表达量为与ZH11-TC相比的倍数变化,ZH11-TC为组织培养的ZH11水稻植株,DP0158为空载体转化的ZH11水稻植株。图5.为实时PCR分析测试的不同转基因水稻株系叶片中OsDN-DTP4基因的相对表达量。ZH11-TC中的表达量设置为1.00,表达量柱上的数字为与ZH11-TC水稻相比的倍数变化,ZH11-TC为组织培养的ZH11水稻植株,DP0158为空载体转化的ZH11水稻植株。图6.为实时PCR分析测试的不同转基因水稻株系叶片中OsLecRK4.1基因的相对表达量。ZH11-TC中的表达量设置为1.00,表达量柱上的数字为与ZH11-TC水稻相比的倍数变化,ZH11-TC为组织培养的ZH11水稻植株。图7.为实时PCR分析测试的不同转基因水稻株系叶片中OsLecRK4.2基因的相对表达量。ZH11-TC中的表达量设置为1.00,表达量柱上的数字为与ZH11-TC水稻相比的倍数变化,ZH11-TC为组织培养的ZH11水稻植株。图8.为OsPPCK4转基因水稻干旱试验中不同发育阶段土壤体积含水量的变化。图9.为OsCAM2转基因水稻第一次田间干旱测试中不同发育阶段土壤体积含水量。OsCAM2转基因水稻在断水后31天开始抽穗。图10.为OsCAM2转基因水稻第二次田间干旱测试中不同发育阶段土壤体积含水量。OsCAM2转基因水稻在断水后24天开始抽穗。图11.为OsLecRK4.1转基因水稻田间干旱测试中不同发育阶段土壤体积含水量。OsLecRK4.1转基因水稻在断水后22天开始抽穗。图12.为OsLecRK4.2转基因水稻田间干旱试验中不同发育阶段体积含水量。OsLecRK4.2转基因水稻在断水后25天开始抽穗。表1.序列表中核苷酸和氨基酸序列的编号表2.水稻基因名称、基因ID(TIGR)和构建体的ID表3.克隆水稻耐旱性基因的引物表4.克隆耐旱性基因的PCR反应混合液表5.PCR循环状况表6.温室条件下OsGSTU41转基因水稻增强耐旱性(第一次试验)表7.温室条件下OsGSTU41转基因水稻增强耐旱性(第二次试验,构建体水平)表8.温室条件下OsGSTU41转基因水稻增强耐旱性(第二次试验,转基因株系水平)表9.温室条件下OsPPCK4转基因水稻增强耐旱性(第一次试验)表10.温室条件下OsPPCK4转基因水稻增强耐旱性(第二次试验,构建体水平)表11.温室条件下OsPPCK4转基因水稻增强耐旱性(第二次试验,转基因株系水平)表12.温室条件下OsCAM2转基因水稻增强耐旱性(第一次试验)表13.温室条件下OsCAM2转基因水稻增强耐旱性(第二次试验,构建体水平)表14.温室条件下OsCAM2转基因水稻增强耐旱性(第二次试验,转基因株系水平)表15.温室条件下OsCAM2转基因水稻增强耐旱性(第三次试验,构建体水平)表16.温室条件下OsCAM2转基因水稻增强耐旱性(第三次试验,转基因株系水平)表17.温室条件下OsDN-DTP4转基因水稻的耐旱性试验(第一次试验)表18.温室条件下OsDN-DTP4转基因水稻的耐旱性试验(第二次试验,构建体水平)表19.温室条件下OsDN-DTP4转基因水稻的耐旱性试验(第二次试验,转基因株系水平)表20.温室条件下OsDN-DTP4转基因水稻的耐旱性试验(第三次试验,构建体水平)表21.温室条件下OsDN-DTP4转基因水稻的耐旱性试验(第三次试验,转基因株系水平)表22.OsGSTU41转基因水稻在田间干旱条件下的籽粒产量分析(第一次试验)表23.OsGSTU41转基因水稻在田间干旱条件下籽粒产量分析(第二次试验)表24.OsPPCK4转基因水稻在田间干旱条件下的籽粒产量分析表25.OsCAM2转基因水稻在田间干旱条件下籽粒产量分析(第一次试验)表26.OsCAM2转基因水稻在田间干旱条件下籽粒产量分析(第二次试验)表27.OsLecRK4.1转基因水稻在田间干旱条件下籽粒产量分析表28.OsLecRK4.2转基因水稻在田间干旱条件下籽粒产量分析表29.OsGSTU41转基因水稻百草枯耐性试验(第一次试验,转基因株系水平)表30.OsGSTU41转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表31.OsPPCK4转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)表32.OsPPCK4转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表33.OsPPCK4转基因水稻的百草枯耐性试验(第三次试验,转基因株系水平)表34.OsCAM2转基因水稻的百草枯耐性试验(第一次试验,转基因株系)表35.OsCAM2转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表36.OsDN-DTP4转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)表37.OsDN-DTP4转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表38.OsLecRK4.1转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)表39.OsLecRK4.1转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表40.OsLecRK4.2转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)表41.OsLecRK4.2转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)表1.序列表中核苷酸和氨基酸序列的编号序列表包含核苷酸序列字符的单字母码以及氨基酸的三字母码,如遵照IUPAC-IUBMB标准所定义的,该标准在NucleicAcidsRes.13:3021-3030(1985)以及在BiochemicalJ.219(No.2):345-373(1984)中有所描述,这两篇文献以引用的方式并入本发明。用于核苷酸和氨基酸序列数据的符号和格式遵循37C.F.R.§1.822中列出的规则。SEQIDNO:1为DP0005载体的核苷酸序列。SEQIDNO:2为DsRed表达盒的核苷酸序列。SEQIDNO:3为OsGSTU41基因cDNA的核苷酸序列。SEQIDNO:4为OsGSTU41基因CDS的核苷酸序列。SEQIDNO:5为OsGSTU41的氨基酸序列。SEQIDNO:6为OsPPCK4基因cDNA的核苷酸序列。SEQIDNO:7为OsPPCK4基因CDS的核苷酸序列。SEQIDNO:8为OsPPCK4的氨基酸序列。SEQIDNO:9为OsCAM2基因cDNA的核苷酸序列。SEQIDNO:10为OsCAM2基因CDS的核苷酸序列。SEQIDNO:11为OsCAM2的氨基酸序列。SEQIDNO:12为OsDN-DTP4基因gDNA的核苷酸序列。SEQIDNO:13为OsDN-DTP4基因CDS的核苷酸序列。SEQIDNO:14为OsDN-DTP4的氨基酸序列。SEQIDNO:15为OsLecRK4.1基因gDNA的核苷酸序列。SEQIDNO:16为OsLecRK4.1基因CDS的核苷酸序列。SEQIDNO:17为OsLecRK4.1的氨基酸序列。SEQIDNO:18为OsLecRK4.2基因gDNA的核苷酸序列。SEQIDNO:19为OsLecRK4.2基因CDS的核苷酸序列。SEQIDNO:20为OsLecRK4.2的氨基酸序列。SEQIDNO:21为克隆OsGSTU41基因cDNA的正向引物。SEQIDNO:22为克隆OsGSTU41基因cDNA的反向引物。SEQIDNO:23为克隆OsPPCK4基因cDNA的正向引物。SEQIDNO:24为克隆OsPPCK4基因cDNA的反向引物。SEQIDNO:25为克隆OsCAM2基因cDNA的正向引物。SEQIDNO:26为克隆OsCAM2基因cDNA的反向引物。SEQIDNO:27为克隆OsDN-DTP4基因gDNA的正向引物。SEQIDNO:28为克隆OsDN-DTP4基因gDNA的反向引物。SEQIDNO:29为克隆OsLecRK4.1基因gDNA的正向引物。SEQIDNO:30为克隆OsLecRK4.1基因gDNA的反向引物。SEQIDNO:31为克隆OsLecRK4.2基因gDNA的正向引物。SEQIDNO:32为克隆OsLecRK4.2基因gDNA的反向引物。SEQIDNO:33为OsGSTU41基因实时PCR分析的正向引物。SEQIDNO:34为OsGSTU41基因实时PCR分析的反向引物。SEQIDNO:35为OsPPCK4基因实时PCR分析的正向引物。SEQIDNO:36为OsPPCK4基因实时PCR分析的反向引物。SEQIDNO:37为OsDN-DTP4基因实时PCR分析的正向引物。SEQIDNO:38为OsDN-DTP4基因实时PCR分析的反向引物。SEQIDNO:39为OsLecRK4.1基因实时PCR分析的正向引物。SEQIDNO:40为OsLecRK4.1基因实时PCR分析的反向引物。SEQIDNO:41为OsLecRK4.2基因实时PCR分析的正向引物。SEQIDNO:42为OsLecRK4.2基因实时PCR分析的反向引物。详细描述本发明中所列出的每篇参考文献的公开内容的全文均以引用的方式并入本发明。如本发明所用的并在所附权利要求书中的单数形式“一个”和“所述”包括复数涵义,除非上下文中清楚地另有指明。因此,例如,“一株植物”的涵义包括多株该类植物。“一个细胞”的涵义包括一个或多个细胞及其本领域的技术人员已知的等同物,等等。如本发明所述:术语“OsGSTU41”是谷胱甘肽S-转移酶TAU41(glutathioneS-transferaseTAU41),指水稻基因位点LOC_Os01g72160.1编码的,赋予植物耐旱性和百草枯耐性表型的水稻多肽。“GSTU41多肽”此处指OsGSTU41多肽和其它植物中的同源物。OsGSTU41多肽(SEQIDNO:5)是水稻基因位点LOC_Os01g72160.1的编码序列(CDS)(SEQIDNO:4)或核苷酸序列(SEQIDNO:3)编码的氨基酸序列,该多肽在TIGR中注释为“谷胱甘肽S-转移酶,推测,表达”。术语“OsPPCK4”是磷酸烯醇式丙酮酸羧化酶激酶4(phosphoenolpyruvatecarboxylasekinase4),指水稻基因位点LOC_Os02g56310.1编码的,赋予植物耐旱性和百草枯耐性表型的水稻多肽。“PPCK4多肽”此处指OsPPCK4多肽和其它植物中的同源物。OsPPCK4多肽(SEQIDNO:8)是水稻基因位点LOC_Os02g56310.1的编码序列(CDS)(SEQIDNO:7)或核苷酸序列(SEQIDNO:6)的氨基酸序列。该多肽在TIGR中注释为“钙依赖蛋白激酶异构体AK1,推测,表达”,在NCBI(www.ncbi.nlm.nih.gov)中注释为“假定磷酸烯醇式丙酮酸羧化酶激酶”。术语“OsCAM2”是钙调素2(Calmodulin2),指水稻基因位点LOC_Os05g41210.1编码的,赋予植物耐旱性和百草枯耐性表型的水稻多肽。“CAM2多肽”此处指OsCAM2多肽和其它植物中的同源物。OsCAM2多肽(SEQIDNO:11)是水稻基因位点LOC_Os05g41210.1的编码序列(CDS)(SEQIDNO:10)或核苷酸序列(SEQIDNO:9)编码的氨基酸序列。该多肽在TIGR中注释为“Oscam2,钙调素,表达”。术语“OsDN-DTP4”是耐旱性蛋白4(droughttoleranceprotein4),指水稻基因位点LOC_Os07g04720.1编码的,赋予植物耐旱性和百草枯耐性表型的水稻多肽。“DN-DTP4多肽”此处指OsDN-DTP4多肽和其它植物中的同源物。OsDN-DTP4多肽(SEQIDNO:14)是水稻基因位点LOC_Os07g04720.1的编码序列(CDS)(SEQIDNO:13)或核苷酸序列(SEQIDNO:12)编码的氨基酸序列。该多肽在TIGR中注释“表达的蛋白”。术语“OsLecRK4.1”是植物血凝素-类似受体激酶4.1(lectin-likereceptorkinase4.1),指水稻基因位点LOC_Os04g44900.1编码的,赋予植物耐旱性和百草枯耐性表型的水稻多肽。“LecRK4.1多肽”此处指OsLecRK4.1多肽和其它植物中的同源物。OsLecRK4.1多肽(SEQIDNO:17)是水稻基因位点LOC_Os04g44900.1的编码序列(CDS)(SEQIDNO:16)或核苷酸序列(SEQIDNO:15)编码的氨基酸序列。该多肽在TIGR中注释为“植物血凝素-类似受体激酶,推定,表达”。术语“OsLecRK4.2”是植物血凝素-类似受体激酶4.2(lectin-likereceptorkinase4.2),指水稻基因位点LOC_Os04g44910.1编码的,赋予植物耐旱性和百草枯耐性的水稻多肽。“LecRK4.2多肽”指OsLecRK4.2多肽和其它植物中的同源物。OsLecRK4.2多肽(SEQIDNO:20)是水稻基因位点LOC_Os04g44910.1的编码序列(CDS)(SEQIDNO:19)或核苷酸序列(SEQIDNO:18)编码的氨基酸序列。该多肽在TIGR中注释为“受体类似蛋白激酶,推测,表达”。本发明中的单子叶植物包括禾本科的植物;双子叶植物包括十字花科、豆科和茄科的植物。“全长互补序列”是指给定核苷酸序列的互补序列,互补序列和核苷酸序列含有相同的核苷酸数,并且100%的互补。“表达序列标签”(EST)是从cDNA文库中获得的DNA序列,代表已经转录的序列。EST通常通过cDNA插入序列单程测序获取。将完整的cDNA插入序列称为“全长插入序列”(“FIS”)。“重叠群”序列是由选自但不限于EST、FIS和PCR序列的两个或更多个序列装配成的序列。将编码完整或功能性蛋白的序列称为“完全基因序列”(“CGS”),该序列可得自FIS或重叠群。“性状”指植物或特定植物材料或细胞的生理的、形态的、生化的或物理的特征。在一些实施例中,这些特征可以是肉眼可见的,比如种子、植株的大小等;可用生物化学技术测定的指标,如种子或叶片中蛋白、淀粉或油份的含量等;可观察的代谢或生理过程,如测定对水分胁迫、特定盐、糖或氮浓度的耐性;可检测的基因表达水平;或可观察渗透胁迫的耐性或产量等农艺性状。“农艺性状”是可测量的指标参数,包括但不限于:叶片绿色、籽粒产量、生长速率、总生物量或积累速率、成熟时的鲜重、成熟时的干重、果实产量、种子产量、植物总氮含量、果实氮含量、种子氮含量、植物营养组织氮含量、植物总游离氨基酸含量、果实游离氨基酸含量、种子游离氨基酸含量、植物营养组织游离氨基酸含量、植物总蛋白含量、果实蛋白含量、种子蛋白含量、植物营养组织蛋白质含量、耐旱性、氮的吸收、根的倒伏、收获指数、茎的倒伏、株高、穗高、穗长、耐盐性、分蘖数、圆锥花序的大小、早期苗的活力和低温胁迫下的出苗状况。可以测量增加的生物量,例如与对照植物相比,增加的植物高度、植物总叶面积、植物鲜重、植物干重或植物种子产量等。增加植株生物量或大小的能力具有多种重要商业应用,作物栽培品种可用于产生较高植物营养组织部分,用于食品、饲料、纤维和/或生物燃料。人们对叶片大小的增加特别有兴趣。增加的叶片的生物量可用于增加植物源药或工业产品的生产,增加的分蘖数可以增加产量,增加植物叶片面积可提高植物总的光合作用,增加的光合合成能力可以增加特定植物组织的产量,并使植物在低光强或高光强下生长,所述特定植物组织包括叶片、根、果实或种子。其它组织如根的生物量的改变有利于提高植物在严酷条件下生长的能力,所述严酷条件包括干旱、营养贫瘠,因为大量的根系可以更好地吸收水分和营养。对于观赏植物,人们所期望获得较大的品种,许多植物包括果树,用于木材生产的树,作为视图或风帘的乔木或灌木,可以通过增加其大小以获得较大的产量或增加屏障。“转基因的”指其基因组因异源核酸(如重组DNA构建体)的存在而发生改变的任何细胞、细胞系、愈伤组织、组织、植物部分或植物,包括那些最初的转基因事件以及从最初的转基因事件通过有性杂交或无性生殖而产生的那些。如本发明所用的术语“转基因”不涵盖通过常规植物育种方法或通过诸如随机异花受精、非重组病毒感染、非重组细菌转化、非重组转座或自发突变之类的自然发生事件导致的基因组(染色体基因组或染色体外基因组)改变。“对照”、“对照植物”或“对照植物细胞”为测定受测试植物或植物细胞的表型变化提供参考,由于转化,受测植物或植物细胞的基因组改变影响到目的基因,测试的植物或植物细胞可以是上述改变并含有上述变化的植物或植物细胞的子代。对照植物或对照植物细胞包括,例如:(a)用于基因改变产生受测植物或细胞的相同基因型的作为起始材料的野生型植物或细胞;(b)相同基因组作为起始材料但是转入空载体(如带有标记基因的并对目标的性状没有影响的载体)的植物或植物细胞;(c)转基因植物或植物细胞性状分离,获得的非转基因的子代植物或植物细胞;(d)没有暴露于可以诱导基因表达的条件或刺激下的,与转基因植物或植物细胞基因组相同的植物或植物细胞;(e)特定的目标基因不表达情况下的,转基因植物或者植物细胞本身。“基因组”在用于植物细胞时不仅涵盖存在于细胞核中的染色体DNA,而且还包括存在于细胞的亚细胞组分(如线粒体、质粒)中的细胞器DNA。“植物”包括整个植株、植物器官、植物组织、种子和植物细胞以及同一植株的子代。植物细胞包括但不限于得自下列物质的细胞:种子、悬浮培养物、胚、分生区域、愈伤组织、叶、根、芽、配子体、孢子体、花粉和小孢子。“子代”包括植物的任何后续世代。“转基因的植物”包括在其基因组内包含异源多核苷酸的植物。例如异源多核苷酸能够稳定地整合进基因组中,且多聚核苷酸能够遗传到后续世代。异源多核苷酸可单独地或作为重组DNA构建体的部分整合进基因组中。T0植物直接源于转化和再生过程,T0植物的子代为T1代(第一个子代),T2代(第二个子代)等。针对序列而言的“异源”意指来自外来物种的序列,或者如果来自相同物种,则指通过蓄意的人为干预而从其天然形式发生了组成和/或基因座的显著改变的序列。“多核苷酸”、“核酸序列”、“核苷酸序列”或“核酸片段”可互换使用并且是任选含有合成的、非天然的或改变的核苷酸碱基的单链或双链RNA或DNA聚合物。核苷酸(通常以它们的5’-单磷酸形式存在)通过如下它们的单个字母名称来指代:“A”为腺苷酸或脱氧腺苷酸(分别对应RNA或DNA),“C”表示胞苷酸或脱氧胞苷酸,“G”表示鸟苷酸或脱氧鸟苷酸,“U”表示尿苷酸,“T”表示脱氧胸苷酸,“R”表示嘌呤(A或G),“Y”表示嘧啶(C或T),“K”表示G或T,“H”表示A或C或T,“I”表示肌苷,并且“N”表示任何核苷酸。“多肽”、“肽”、“氨基酸序列”和“蛋白质”在本发明中可互换使用,指氨基酸残基的聚合物。该术语适用于其中一个或多个氨基酸残基是相应的天然存在的氨基酸的人工化学类似物的氨基酸聚合物,以及适用于天然存在的氨基酸聚合物。术语“多肽”、“肽”、“氨基酸序列”和“蛋白质”还可包括修饰形式,包括但不限于糖基化、脂质连接、硫酸盐化、谷氨酸残基的γ羧化、羟化和ADP-核糖基化。“信使RNA(mRNA)”指无内含子并且可通过细胞翻译成蛋白质的RNA。“cDNA”指与mRNA模板互补并且利用逆转录酶从mRNA模板合成的DNA。cDNA可为单链的或者可用DNA聚合成酶I的Klenow片段合成双链形式。“成熟”蛋白质指经翻译后加工的多肽;即已经去除了存在于初级翻译产物中的任何前肽或原肽的多肽。“前体”蛋白质指mRNA的翻译初级产物;即带有前肽和原肽的蛋白质。前肽和原肽可为并且不限于细胞内定位信号。“分离的”指物质,例如核酸和/或蛋白质,该物质基本上不含在天然存在的环境中通常伴随该物质或与其反应的组分,或者说是该物质被从所述组分移出。分离的多核苷酸可从它们天然存在的宿主细胞中纯化。技术人员已知的常规核酸纯化方法可用于获得分离的多核苷酸。该术语也涵盖重组多核苷酸和化学合成的多核苷酸。“重组体”指(例如)通过化学合成或者通过用基因工程技术操纵分离的核酸片段来实现的两个原本分离的序列片段的人工组合。“重组体”也包括指已经通过引入异源核酸而进行了修饰的细胞或载体,或源于经这样修饰的细胞的细胞,但不涵盖由天然发生的事件(如自发突变、自然转化/转导/转座)对细胞或载体的改变,例如没有蓄意人为干扰而发生的那些。“重组DNA构建体”指在自然界中通常不会一起存在的核酸片段的组合。因此,重组DNA构建体可包含源于不同来源的调控序列和编码序列,或源于相同来源但以不同于通常天然存在的方式排列的调控序列和编码序列。术语“入门克隆”和“入门载体”本发明可互换使用。“调控序列”和“调控元件”可互换使用,指位于编码序列的上游(5'非编码序列)、中间或下游(3'非编码序列),并且影响相关编码序列的转录、RNA加工或稳定性或者翻译的核苷酸序列。调控序列可包括但不限于启动子、翻译前导序列、内含子和多腺苷酸化识别序列。“启动子”指能够控制另一核酸片段转录的核酸片段。“植物中有功能启动子”是能够控制植物细胞中基因转录的启动子,无论其是否来源于植物细胞。“组织特异性启动子”和“组织优选启动子”可互换使用,并且指主要但非必须专一地在一种组织或器官中表达,而且也可在一种特定细胞或细胞型中表达的启动子。“发育调控启动子”指其活性由发育事件决定的启动子。术语“可操作地连接”指核酸片段连接成单一片段,使得其中一个核酸片段的功能受到另一个核酸片段的调控。例如,在启动子能够调节核酸片段的转录时,该启动子与该核酸片段进行了可操作地连接。“表达”指功能产物的产生。例如,核酸片段的表达可指核酸片段的转录(如转录生成mRNA或功能RNA)和/或RNA翻译成前体或成熟蛋白质。“表型”意指细胞或生物体的可检测的特征。有关将核酸片段(例如重组DNA构建体)插入细胞内的“导入”是指“转染”或“转化”或“转导”,并且包括指将核酸片段整合进真核或原核细胞中,在该细胞中核酸片段可整合进细胞的基因组(如染色体、质粒、质体或线粒体DNA)内,转变成自主的复制子或瞬时表达(如转染的mRNA)。“转化细胞”是将核酸片段(如重组DNA构建体)导入其中的任何细胞。本发明所用的“转化”指稳定转化和瞬时转化两者。“稳定转化”指将核酸片段导入宿主生物体的基因组中,导致基因稳定遗传。一旦稳定转化,核酸片段稳定地整合进宿主生物体和任何连续世代的基因组中。“瞬时转化”指将核酸片段导入宿主生物体的核中或包含DNA的细胞器中,引起基因表达而没有基因稳定遗传。“等位基因”是占据染色体上给定位点的基因的几种供选择形式的其中一种。当二倍体植物中一对同源染色体上给定基因座上存在的等位基因相同时,该植物在该基因座处是纯合的。如果二倍体植物中一对同源染色体上给定基因座上存在的等位基因不同,则该植物在该基因座处是杂合的。如果转基因存在于二倍体植物中一对同源染色体中的其中之一上,则该植物在该基因座处是半合子的。“叶绿体转运肽”是与蛋白协同翻译并将蛋白导向叶绿体或翻译蛋白的细胞中存在的其它质体类型的氨基酸序列。“叶绿体转运序列”指编码叶绿体转运肽的核苷酸序列。“信号肽”是一种与蛋白协同翻译并将蛋白导向分泌器官的氨基酸序列(Chrispeels,M.(1991)Ann.Rev.PlantPhys.PlantMol.Biol.42:21-53)。如果将所述蛋白导向液泡,可另外加上液泡靶向信号,或者如果将所述蛋白导向内质网,可加上内质网驻留信号。如果将蛋白导向细胞核,将移除任何存在的信号肽并用以核定位信号替代(Raikhel(1992)PlantPhys.100:1627-1632)。“线粒体信号肽”是引导前体蛋白进入线粒体的氨基酸序列(Zhang和Glaser(2002)TrendsPlantSci7:14-21)。目前已知有多种可以用来研究各种多聚核苷酸和多肽序列之间关系的方法。本发明中,“参考序列”是一个界定序列,用作序列比对的基础。参考序列是一个特定序列的子集或全部,例如全长cDNA/基因序列的片段,抑或完整的cDNA/基因序列。此处用到的“比对窗口”,涉及到一个多聚核苷酸或多肽序列的共有或特定片段,其中两个序列的最佳比对时,与参考序列(没有添加或者删除)相比,比较窗口中序列可能会包含添加或删除(换言之既间隙)。通常比对窗口中至少包含20个连续的碱基或氨基酸,还可以选择30、40、50、100甚至更长。本领域的技术人员为了避免因引入序列间隙而导致的与参考序列的高相似性,引入空位罚分,并减去相应的匹配数目。使用数学算法可以计算任意两条序列的序列一致性(百分比)。序列比对算法的例子包括Myers和Miller运算法则(CABIOS4:11-17,1988);局部联配算法(smith等,Adv.Appl.Math.2:482,1981);全局联配算法(Needleman和Wunsch,J.Mol.Biol.48:443-453,1970);局部搜索联配算法(Pearson和Lipman,Proc.Natl.Acad.Sci.85:2444-2448,1988);和Karlin和Altschul运算法则(Proc.Natl.Acad.Sci.USA872264(1990),Karlin和Altschul于1993年修订Proc.Natl.Acad.Sci.USA90:5873-5877)。计算机可以利用这些算法来确定序列的一致性,这些应用包括但不限于CLUSTALinthePC/GeneProgram(Intelligenetics,MountainView,美国加州);ALIGN(Ver2.0)和GCGWisconsin遗传学软件包(Ver10)(AccelrysInc.,9685美国加州圣地亚哥斯克兰顿路)中的GAP、BESTFIT、BLAST、FASTA和TFASTA;和bioinformaticscomputingsuite(Inc.,Madison,WI)的使用缺省参数就可以通过上述程序开展序列联配工作。Higgins等(Gene73:237-244,1988),Higgins等(CABIOS5:151-153,1989),Corpet等(NucleicAcidsRes.16:10881-10890,1988),Huang等(CABIOS8:155-165,1992)和Pearson等(Meth.Mol.Biol.24:307-331,1994)对CLUSTAL进行了详细的描述;ALIGN程序则以Myers和Miller提出的supra算法(1988)为基础。进行氨基酸序列比对时,当使用PAM120残基重量表,且空位罚分为12和空位罚分为4时,可以与ALIGN程序相结合对氨基酸序列进行比较。BLAST程序(Altschul等,J.Mol.Biol.215:403,1990)以Karlin和Altschul的supra算法(1990)为基础,可以使用BLASTN对核苷酸序列进行检索,score=100,wordlength=12,以获得本发明所涉及蛋白质对应编码核苷酸序列的同源序列;可以使用BLASTX,score=50,wordlength=3,以获得本发明所涉及蛋白质或多肽的同源氨基酸序列。GappedBLAST(BLAST2.0)(Altschul等,(1997)NucleicAcidsRes.25:3389)可以用于Gap比对。作为一种选择,PSI-BLAST(BLAST2.0)可以开展交互式搜索,从而检测到亲缘关系较远的两个分子(Altschul等(1997)supra)。当使用BLAST时,GappedBLAST、PSI-BLAST和相应程序的缺省参数(例如用于核甘酸序列的BLASTN,用于蛋白质的BLASTX)也可以一并使用(美国政府国立卫生研究院国立医学图书馆国家生物技术信息中心)。序列联配也可以用于人工检索。通过使用GAP(Ver10)和以下参数可以得到配对序列一致性/相似性值:使用GAPWeight50、LengthWeight3以及nwsgapdna.cmp得分矩阵来计算核酸序列的一致性(%)和相似性(%);使用GAPWeight8、LengthWeight2以及BLOSUM62得分矩阵可以计算氨基酸序列的一致性(%)和相似性(%);或者任何等同程序。任何序列比较程序都可以被看作“等同程序”,因为任意两条序列进行比较,都会产生一个联配结果,包含一致的核苷酸或氨基酸碱基配对。当与GAPVer10所产生序列联配结果进行比较时,还会产生序列一致性百分比。GAP利用Needleman和Wunsch算法((1970)J.Mol.Biol.48:443-453),以发现两个全长序列联配的最大匹配数和最小间隙数。GAP考虑所有可能的联配和gap位点,并创造联配位点数目最大且gap数目结果,它允许匹配碱基单元中存在间隙创造罚分和间隙延展罚分,GAP必须利用每个插入间隙的间隙创造罚分匹配数,另外,如果选择间隙延展罚分大于0,GAP必须利用每个插入间隙的长度间隙次数,间隙延展罚分。GCGWisconsin遗传学软件包Ver10中,缺省间隙创造罚分值和间隙延展罚分值分别为8和2,核苷酸序列中,缺省间隙创造罚分是50,缺省间隙延展罚分为3,间隙创造和间隙延展罚分可以是0-200中的整数,因此间隙创造和间隙延展罚分可以是0、1、2、3、4、5、6、7、8、9、10、15、20、25、30、35、40、45、50、55、60、65或更大。GAP作为最好的序列比对工具之一,而且在序列比对质量上表现最为优异。GAP主要表现四个比对的性能指数:质量、比率、一致性和相似性,质量指度量化为比对序列,比率指质量除以较短的片段中碱基数,一致性百分数指匹配符号的百分数,相似性百分数指类似符号的百分数。符号对面的差距被忽略,当配对符号得分矩阵值大于或等于0.50(相似性阈值),需要计算相似度。GCGWisconsin遗传学软件程序包(Ver10)使用的得分矩阵是BLOSUM62(Henikoff和Henikoff.Proc.Natl.Acad.Sci.USA89:10915,1989)。除非特别说明,本发明中的多序列比对采用ClustalV比对方法(Higgins和Sharp.(1989)CABIOS.5:151-153)和缺省参数(间隙罚分=10,间隙长度罚分=10),两个比对的缺省参数和计算氨基酸序列一致性百分数使用ClustalV方法:KTUPLE=1,间隙罚分=3,窗口=5和对角线拯救=5;用于核酸序列的参数KTUPLE=2,间隙罚分=5,窗口=4和对角线拯救=4。序列比对后应用ClustalV程序,通过观察“序列距离”表,可以得到“一致性百分比”和“散度”值,除非特别说明,一致性百分比和散度值以相同的方式获得。本发明中,当在特定比对窗口中最大一致性比对时,两个多聚核苷酸或多肽序列的“序列一致性”或“一致性”指参考两序列中残基相同,当序列一致性百分数用于提及的蛋白质,被认为不同残基位置与保守氨基酸替代不同,氨基酸残基被具有相似化学性质的其它氨基酸残基(如电荷或疏水性)替代,而不改变分子的功能特性,当序列在保守区替代不同,序列一致性百分数可以上调以更正替代的保守性,保守替代不同的序列被认为具有“序列相似性”或“相似性”。本领域的技术人员熟悉进行调整的方法,包括计算作为部分的而不是全长错配的保守替代,因而增加序列一致性百分数,因此,相同氨基酸给与1分,不保守的替代给与0分,保守替代给与0-1分,计算保守替代的得分,如在PC/GENE执行(Intelligenetics,MountainView,加州)。本发明中“序列一致性百分数”的计算包括确定两个序列中相同核苷酸碱基或氨基酸残基的位置数目,获得匹配位置数,然后将匹配的位置数除以比对窗口中位置总数再乘以100。本发明中使用的标准重组DNA和分子克隆技术为本领域技术人员熟知,并且在如下文献中有更全面的描述:Sambrook,J.,Fritsch,E.F.和Maniatis,T.,MolecularCloning:ALaboratoryManual;ColdSpringHarborLaboratoryPress:ColdSpringHarbor,1989(下文称为“Sambrook”)。实施方案包括分离的多核苷酸和多肽、赋予耐旱性的重组DNA构建体、包含重组DNA构建体的诸如植物或种子的组合物、以及利用这些重组DNA构建体的方法。分离的多核苷酸和多肽:本发明包括如下分离的多核苷酸和多肽:一种分离的多核苷酸,包括(i)一种核酸序列,其编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;或(ii)核酸序列(i)的全长互补序列,其中核酸序列(i)与全长互补序列具有相同数目的核苷酸,并且100%的互补。前述任一分离的多核苷酸可用于构建本发明的任一重组DNA构建体,过量表达所述编码的多肽可提高植物的耐旱性和/或百草枯耐性。一种分离的多肽,所述多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性。所述多肽优选为耐旱性多肽,过量表达所述多肽增加植物的耐旱性和/或百草枯耐性。一种分离的多核苷酸,包括(i)一种核酸序列,其核苷酸序列与SEQIDNO:4、7、10、13、16或19相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;(ii)一种核酸序列,其核苷酸序列与SEQIDNO:3、6、9、12、15或18相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;或(iii)核酸序列(i)或(ii)的全长互补序列。前述任一分离的多核苷酸可用于构建本发明任一重组DNA构建体。所述分离的多核苷酸优选为编码耐旱性多肽,过量表达所述多肽提高植物的耐旱性和/或百草枯耐性。重组DNA构建体:一方面,本发明包括重组DNA构建体。在一个实施方案中,一种重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列(如植物中有功能的启动子),其中所述多核苷酸包括(i)一种核酸序列,其编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;或(ii)核酸序列(i)的全长互补序列。在另一个实施方案中,一种重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列(如植物中有功能的启动子),其中,所述多核苷酸包括(i)一种核酸序列,其核苷酸序列与SEQIDNO:4、7、10、13、16或19相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;(ii)一种核酸序列,其核苷酸序列与SEQIDNO:3、6、9、12、15或18相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;或(iii)核酸序列(i)或(ii)的全长互补序列。在另一个实施方案中,一种重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列(如植物中有功能的启动子),其中所述多核苷酸编码一个耐旱性多肽,所述多肽优选为具有耐旱性和/或百草枯耐性,并可来自,例如水稻(Oryzasativa)、野生稻(Oryzaaustraliensis)、短舌野生稻(Oryzabarthii)、非洲型稻(Oryzaglaberrima)、阔叶稻(Oryzalatifolia)、长雄野生稻(Oryzalongistaminata)、南方野生稻(Oryzameridionalis)、药用野生稻(Oryzaofficinalis)、斑点野生稻(Oryzapunctata)、普通野生稻(Oryzarufipogon)(红稻)、印度野生稻(Oryzanivara)、拟南芥(Arabidopsisthaliana)、玉米(Zeamays)、大豆(Glycinemax)、烟豆(Glycinetabacina)、野大豆(Glycinesoja)和短绒野大豆(Glycinetomentella)。应当理解(正如本领域技术人员将会理解的),本发明不仅仅涵盖这些具体的示例性序列。导致给定位点处产生化学上等价的氨基酸但不影响所编码多肽的功能特性的核酸片段中的改变是本领域众所周知的。例如,丙氨酸(一种疏水性氨基酸)的密码子可被编码另一个疏水性较弱的残基(例如甘氨酸)或疏水性较强的残基(例如缬氨酸、亮氨酸或异亮氨酸)的密码子取代。类似地,导致一个带负电荷的残基替换为另一个带负电荷的残基(例如,天冬氨酸替代谷氨酸)或者一个带正电荷的残基替换为另一个带正电荷的残基(例如,赖氨酸替换精氨酸)的改变也可预期产生功能上等价的产物。导致多肽分子的N-末端和C-末端部分改变的核苷酸变化也将预计不会改变多肽的活性。所提出的修饰中的每一种均完全在本领域常规技术内,如测定所编码的产物的生物活性的保留。“抑制DNA构建体”是在转化或稳定整合进植物基因组时,导致该植物中的靶基因“沉默”的重组DNA构建体。对该植物来说,该靶基因可为内源性的基因或是转入的基因。如本文针对靶基因所使用的,“沉默”通常指在由靶基因表达的mRNA或蛋白质/酶的水平上的抑制,和/或在酶活性或蛋白质功能性的水平上的抑制。本文中可交换使用的术语“抑制”、“抑制性”以及“沉默”包括降低、减少、减退、减小、抑制、消除或防止。“沉默”或“基因沉默”不特指机理并且包括但不限于反义、病毒-抑制、发夹抑制、茎-环抑制、基于RNAi的方法以及基于小RNAi的方法。抑制DNA构建体可包含源自所关注的靶基因的区域并且可包含所关注的靶基因的有义链(或反义链)的核酸序列的全部或部分。取决于所要利用的方法,所述区域可与所关注基因的有义链(或反义链)的全部或部分100%相同或者具有少于100%序列的一致性(如,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%的一致性)。抑制DNA构建体是本领域所熟知的,一旦选定所关注的靶基因就很容易构建,并且包括但不限于共抑制构建体、反义构建体、病毒-抑制构建体、发夹抑制构建体、茎-环抑制构建体、产生双链RNA的构建体,以及更通常的是,RNAi(RNA干扰)构建体和小RNA构建体,例如siRNA(短干扰RNA)构建体和miRNA(微RNA)构建体。“反义抑制”指产生能够抑制靶基因或基因产物表达的反义RNA转录物。“反义RNA”指与靶初级转录物或mRNA的全部或部分互补,并阻断分离的靶核酸片段表达的RNA转录物(美国专利号5,107,065)。反义RNA可与特定基因转录物的任何部分,即5′非编码序列、3′非编码序列、内含子或编码序列互补。“共抑制”指产生正义RNA转录物可以抑制目的基因或基因产物的表达;“正义”RNA涉及RNA转录物,包括在细胞内或者生物体外翻译成蛋白质的mRNA,过去,植物中的共抑制载体用于在正义方向过表达与天然mRNA同源的核酸序列,因此减少所有与过表达序列同源的所有RNA(Vaucheret等(1998)PlantJ.16:651-659;和Gura.(2000)Nature404:804-808)。RNA干扰(RNAi)是指由短干扰性RNA(siRNA)介导的动物中序列特异性转录后基因沉默的过程(Fire等人,(1998)Nature391:806)。在植物中的对应过程通常称为转录后基因沉默(PTGS)或RNA沉默,并且在真菌中也称为阻抑作用(quelling)。据信PTGS转录后基因沉默过程是用于防止外来基因表达的进化保守性细胞防御机制,并且通常由不同植物区系和门所共有(Fire等人,(1999)TrendsGenet.15:358)。小RNA在控制基因表达中起重要作用。很多发育过程(包括开花)的调节是由小RNA控制的。现在有可能通过使用在植物中产生小RNA的转基因构建体来以基因工程手段改变植物基因的表达。小RNA似乎是通过与互补RNA或DNA靶序列碱基配对来行使功能的。当与RNA结合时,小RNA或者引发靶序列的RNA裂解或者引发翻译抑制。当与DNA靶序列结合时,小RNA可介导靶序列的DNA甲基化。无论具体机制是什么,这些事件的后果是基因表达受到抑制。微RNA(miRNA)是长度为约19至约24个核苷酸(nt)的已经在动物和植物中鉴定出的非编码RNA(Lagos-Quintana等人,(2001)Science294:853-858;Lagos-Quintana等人,(2002)Curr.Biol.12:735-739;Lau等人,(2001)Science294:858-862;Lee和Ambros,Science294:(2001)862-864;Llave等人,(2002)PlantCell14:1605-1619;Mourelatos等人,(2002)Genes.Dev.16:720-728;Park等人,(2002)Curr.Biol.12:1484-1495;Reinhart等人,(2002)Genes.Dev.16:1616-1626)。它们是由大小为大约70至200nt较长的前体转录物加工生成的,并且这些前体转录物能够形成稳定的发夹结构。微RNA(miRNA)通过与位于由这些基因产生的转录物中的互补序列结合来调节靶基因。miRNA可进入至少两条靶基因调控途径:(1)翻译抑制;和(2)RNA裂解。进入RNA裂解途径的微RNA类似于在动物中的RNA干涉作用(RNAi)和植物中的转录后基因沉默(PTGS)期间生成的21-25nt的短干涉RNA(siRNA),并且可能掺入了RNA诱导沉默复合物(RISC),该复合物与RNAi相似或相同。调控序列:本发明的重组DNA构建体包含至少一个调控序列。调控序列可以是启动子。多种启动子可用于本发明的重组DNA构建体,启动子根据期望的结果选择,可包括宿主生物体中表达的组成型、组织特异型、诱导型、或其它启动子。一般将引起基因在多数细胞类型中在多数情况下表达的启动子称为“组成型启动子”。当组成型启动子驱动候选基因表达时能够评估候选基因的效应,但候选基因在35S或UBI启动子控制下的高水平、组成型表达可具有多重效应。使用组织特异和/或胁迫特异启动子可消除不需要的效应但保留提高植物耐旱性的能力。在拟南芥属中已经观察到了该效应(Kasuga等人(1999)NatureBiotechnol.17:287-91)。适用于植物宿主细胞的组成型启动子包括(例如)Rsyn7启动子的核心启动子和在WO99/43838和美国专利6,072,050中公开的其它组成型启动子;CaMV35S核心启动子(Odell等人,(1985)Nature313:810-812);稻肌动蛋白(McElroy等人,(1990)PlantCell2:163-171);泛素启动子(Christensen)等人,(1989)PlantMol.Biol.12:619-632和Christensen等人,(1992)PlantMol.Biol.18:675-689);pEMU(Last等人,(1991)Theor.Appl.Genet.81:581-588);MAS(Velten等人,(1984)EMBOJ.3:2723-2730);ALS启动子(美国专利5,659,026)等。其它组成型启动子包括例如在美国专利5,608,149、5,608,144、5,604,121、5,569,597、5,466,785、5,399,680、5,268,463、5,608,142和6,177,611中公开的那些启动子。在选择启动子用于本发明方法时,期望使用组织特异性启动子或发育调节启动子。组织特异性启动子或发育调节启动子是这样的DNA序列,其调节DNA序列选择性地在对雄穗发育、结籽或两者重要的植物细胞/组织中表达,并限制这种DNA序列只在植物的雄穗发育或种子成熟期间表达。任何引起所需时空表达的可鉴定启动子均可用于本发明的方法中。本领域的技术人员知道多种叶片优选的启动子(Yamamoto等(1997)PlantJ.12(2):255-265;Kwon等(1994)PlantPhysiol.105:357-367;Yamamoto等(1994)PlantCellPhysiol.35(5):773-778;Gotor等(1993)PlantJ.3:509-518;Orozco等(1993)PlantMol.Biol.23(6):1129-1138;和Matsuoka等(1993)Proc.Natl.Acad.Sci.USA90(20):9586-9590)。可用于本发明的种子或胚特异性启动子包括大豆Kunitz胰蛋白酶抑制剂(Kti3,Jofuku和Goldberg,(1989)PlantCell1:1079-1093),伴豌豆球蛋白、豌豆球蛋白和豆球蛋白(豌豆子叶)(Rerie,W.G.等,(1991)Mol.Gen.Genet.259:149-157;Newbigin,E.J.等,(1990)Planta180:461-470;Higgins,T.J.V.等,(1988)Plant.Mol.Biol.11:683-695),玉米蛋白(玉米胚乳)(Schemthaner,J.P.等,(1988)EMBOJ.7:1249-1255),菜豆蛋白(菜豆子叶)(Segupta-Gopalan,C.等,(1985)Proc.Natl.Acad.Sci.U.S.A.82:3320-3324),植物血球凝集素(菜豆子叶)(Voelker,T.等,(1987)EMBOJ.6:3571-3577),B-伴球蛋白和大豆球蛋白(大豆子叶)(Chen,Z-L等,(1988)EMBOJ.7:297-302),谷蛋白(稻胚乳),大麦醇溶蛋白(大麦胚乳)(Marris,C.等,(1988)PlantMol.Biol.10:359-366),麦谷蛋白和麦醇溶蛋白(小麦胚乳)(Colot,V.等,(1987)EMBOJ.6:3559-3564)。可操作地连接至嵌合基因构建体异源编码区的种子特异性基因的启动子在转基因植物中保持它们的时空表达模式。这样的实施例包括在拟南芥和甘蓝型油菜(Brassicanapus)种子中表达脑啡肽的拟南芥属2S种子储藏蛋白基因启动子(Vanderkerckhove等,(1989)Bio/Technology7:L929-932),表达荧光素酶的菜豆凝集素和β-菜豆蛋白启动子(Riggs等,(1989)PlantSci.63:47-57),以及表达氯霉素乙酰转移酶的小麦谷蛋白启动子(Colot等,(1987)EMBOJ6:3559-3564)。诱导启动子响应内源性或外源性刺激的存在,例如,通过化合物(化学诱导剂),或响应环境、激素、化学信号和/或发育信号而选择性表达可操纵连接的DNA序列。诱导启动子或受调控的启动子包括(例如)受光、热、胁迫、水涝或干旱、植物激素、创伤或诸如乙醇、茉莉酮酸酯、水杨酸或安全剂之类的化学品调控的启动子。用于本发明的启动子包括以下启动子:1)胁迫诱导型RD29A启动子(Kasuga等,(1999)NatureBiotechnol.17:287-91);2)胁迫诱导启动子Rab17(Vilardell等,(1991)PlantMol.Bio.17:985-993;KampBusk等(1997)PlantJ11(6):1285-1295);3)大麦启动子B22E;B22E是发育中玉米籽粒中的柄所特异表达的启动子(“PrimaryStructureofaNovelBarleyGeneDifferentiallyExpressedinImmatureAleuroneLayers”(在未成熟糊粉层中差异表达的新大麦基因的一级结构)Klemsdal,S.S.等,(1991)Mol.Gen.Genet.228(1/2):9-16);和4)玉米启动子Zag2(“IdentificationandmolecularcharacterizationofZAG1,themaizehomologoftheArabidopsisfloralhomeoticgeneAGAMOUS”,Schmidt,R.J.等,(1993)PlantCell5(7):729-737;“Structuralcharacterization,chromosomallocalizationandphylogeneticevaluationoftwopairsofAGAMOUS-likeMADS-boxgenesfrommaize”,Theissen等,(1995)Gene156(2):155-166;NCBIGenBank登录号X80206))。Zag2转录物可在授粉前5天至授粉后(DAP)7至8天被检测到,并且引导Ciml在发育中的雌花序的心皮中表达,Ciml是发育玉米籽粒的籽仁特异的启动子。Ciml转录物在授粉前4至5天至授粉后6至8天被检测到。其它可用的启动子包括可源自其表达与发育中的雌小花母系相关的基因的任何启动子。对于在发育种子组织中表达的多聚核苷酸,特定的启动子包括种子优选的启动子,尤其是早期籽粒/胚启动子和晚期籽粒/胚启动子,授粉后籽粒的发育大致可以分为三个基本阶段,籽粒生长的停滞期起始于授粉后0天到10-12天,在此期间,籽粒不再明显的生长,但是决定籽粒活力的重要事件将在此期间发生(如细胞建成数目)。线性籽粒灌浆期起始于授粉后的10-12天并延续到授粉后的40天左右,在此籽粒发育期间,籽粒达到最终的质量,并产生多种储藏物质如淀粉、蛋白质和油等;成熟期起始于授粉后大约40天到收获,在这一过程中,籽粒开始休眠,变干并为萌芽前的种子休眠做准备。本发明中的“早期籽粒/胚启动子”指主要在种子发育的停滞期(也就是授粉第0天到授粉后第12天期间)驱动基因表达的启动子;“后期籽粒/胚启动子”主要驱动基因在授粉后12天到成熟过程中的种子中基因表达;表达窗口可能会有一些重叠,将根据使用的ABA偶联的序列和期望的表型选择启动子。早期籽粒/胚启动子包括,Cim1,其在授粉后第5天活跃在特定组织中(WO00/11177);其它的早期籽粒/胚胎启动子包括种子偏爱启动子end1,其在授粉后7-10天表达,和end2,其在授粉后9-14天在整个籽粒中表达,在授粉后10天在胚乳和果皮中表达(WO00/12733),此处以全文引用方式并入本文。本发明中的特定方法使用的其它早期籽粒/胚乳启动子包括种子偏爱启动子ltp2(美国专利号5,525,716);玉米Zm40启动子(美国专利号6,403,862);玉米nuc1c(美国专利号6,407,315);玉米ckx1-2启动子(美国专利号6,921,815和美国专利申请公开号2006/0037103);玉米lec1启动子(美国专利号7,122,658);玉米ESR启动子(美国专利号7,276,596);玉米ZAP启动子(美国专利申请公开号20040025206和20070136891);玉米启动子eep1(美国专利申请公开号20070169226);和玉米启动子ADF4(美国专利申请号60/963,878,2007年8月7日申请)。本发明中调控核酸序列在植物中表达的其它启动子是茎特异启动子,包括苜蓿S2A启动子(GenBank登录号EF030816;Abrahams等(1995)PlantMol.Biol.27:513-528)和S2B启动子(GenBank登录号EF030817)和类似的启动子。启动子可以整个源于天然基因,或者由源于不同的天然存在的启动子的不同元件组成,或者甚至包括合成的DNA片段。用于本发明特定实施例的启动子包括:RIP2、mLIP15、ZmCOR1、Rab17、CaMV35S、RD29A、B22E、Zag2、SAM合成酶、泛素、CaMV19S、nos、Adh、蔗糖合成酶、R-等位基因、维管组织偏爱启动子S2A(Genbank登录号EF030816)和S2B(Genbank登录号EF030817)及来自玉米的组成型启动子GOS2。其它启动子还包括根偏爱启动子,例如玉米NAS2启动子、玉米Cyclo启动子(US2006/0156439,公开于2006年7月13日)、玉米ROOTMET2启动子(WO05063998,公开于2005年7月14日)、CRlBIO启动子(WO06055487,公开于2006年5月26日)、CRWAQ81(WO05035770,公开于2005年4月21日)和玉米ZRP2.47启动子(NCBI登录号:U38790;GINo.1063664)。本发明的重组DNA构建体也可包括其它调控序列,包括但不限于翻译前导序列、内含子和多腺苷酸化识别序列。在特定实施方案中,重组DNA构建体还包括增强子或沉默子。内含子序列可加至5’非翻译区、蛋白编码区或3’非翻译区以增加积聚在胞浆中的成熟信息的量。已经显示,在植物和动物两者的表达构建体的转录单位中包含可剪接内含子可使基因表达在mRNA和蛋白质水平上均增强高达1000倍。参见Buchman和Berg,(1988)Mol.CellBiol.8:4395-4405;Callis等,(1987)GenesDev.1:1183-1200。任何植物都可以选择用来鉴定将用于本发明重组DNA构建体的调控序列和基因。适用于分离基因和调控序列的靶植物的实例应该包括但不限于苜蓿、苹果、杏、拟南芥、洋蓟、芝麻菜、芦笋、鳄梨、香蕉、大麦、豆类、甜菜、黑莓、蓝莓、西兰花、抱子甘蓝、卷心菜、加拿大油菜、香瓜、胡萝卜、木薯、蓖麻子、菜花、芹菜、樱桃、菊苣、芫荽、柑桔、克莱门氏小柑橘、三叶草、椰子、咖啡、玉米、棉、蔓越莓、黄瓜、花旗松、茄子、菊苣、茅菜、桉树、茴香、无花果、大蒜、葫芦、葡萄、柚子树、白兰瓜、豆薯、猕猴桃、生菜、韭葱、柠檬、酸橙、火炬松、亚麻子、芒果、甜瓜、蘑菇、油桃、坚果、燕麦、油棕、油菜、秋葵、橄榄树、洋葱、橙、观赏植物、棕榈、木瓜树、欧芹、欧洲防风草、豌豆、桃树、花生、梨树、胡椒、柿树、松树、菠萝、车前草、李树、石榴树、白杨、马铃薯、南瓜、温柏、辐射松、红菊苣、萝卜、油菜、树莓、稻、黑麦、高粱、南方松、大豆、菠菜、南瓜、草莓、甜菜、甘蔗、向日葵、甘薯、枫香树、柑橘、茶、烟草、蕃茄、黑小麦、草皮草、芜菁、葡萄树、西瓜、小麦、薯蓣和西葫芦。组合物:本发明的组合物是其基因组中包含本发明的任何重组DNA构建体(例如上面所讨论的任何一种构建体)的植物。组合物还包括所述植物的任何子代,以及获取自所述植物或其子代的任何种子,其中所述子代或种子在其基因组中包含重组DNA构建体。所述子代包括植物通过自花授粉或异型杂交而获得的连续世代。子代也包括杂交种和自交系。在杂交种子繁殖的农作物中,成熟的转基因植物可自花授粉而产生纯合的自交系植物。该自交系植物产生的种子含有新导入的重组DNA构建体。这些种子可生长成植物,所述植物表现出改变的农学特性(如水分限制条件下增加农业特性),或者可用于育种程序以产生杂交种子,这些杂交种子可生长成植物,所述植物将会表现出如改变的农学特性。所述种子可为玉米种子或水稻种子。植物可为单子叶植物或双子叶植物,例如水稻、玉米或大豆植物,如玉米杂种植物或玉米自交系植物。植物还可为向日葵、高梁、油菜、小麦、苜蓿、棉花、大麦、粟、甘蔗或柳枝稷。重组DNA构建体可稳定地整合进植物的基因组中。特定的实施方案包括但不限于以下实施方案:1.在其基因组中包含重组DNA构建体的转基因植物,如水稻、玉米或大豆,所述重组DNA构建体包含一个多核苷酸和与其可操作连接至少一个调控序列,其中所述多核苷酸编码一个多肽,所述多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;与对照植物相比,所述植物表现出增加的耐旱性和/或百草枯耐性,且所述植物进一步表现出至少一个改变的农艺性状。2.在其基因组中包含重组DNA构建体的转基因植物,如水稻、玉米或大豆,所述重组DNA构建体包含一个多核苷酸和与其可操作连接至少一个调控序列,其中所述多核苷酸编码一个耐旱性多肽,与对照植物相比,所述植物表现出增加的耐旱性和/或百草枯耐性,且所述植物进一步表现出至少一个改变的农艺性状。3.在其基因组中包含重组DNA构建体的转基因植物,例如水稻、玉米或大豆,所述重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列,其中所述多核苷酸编码一个耐旱性多肽,与对照植物相比,所述植物表现出至少一个农艺性状的变化。4.实施方案1-3所述的植物的任何子代植物,实施方案1-3所述的植物的任何种子,实施方案1-3所述的植物的任何子代植物的种子,和源于实施方案1-3的植物和其子代植物的细胞。前述1-4任一实施方案或其它实施方案中,所述耐旱性多肽可来自水稻(Oryzasativa)、野生稻(Oryzaaustraliensis)、短舌野生稻(Oryzabarthii)、非洲型稻(Oryzaglaberrima)、阔叶稻(Oryzalatifolia)、长雄野生稻(Oryzalongistaminata)、南方野生稻(Oryzameridionalis)、药用野生稻(Oryzaofficinalis)、斑点野生稻(Oryzapunctata)、普通野生稻(Oryzarufipogon)(红稻)、印度野生稻(Oryzanivara)、拟南芥(Arabidopsisthaliana)、玉米(Zeamays)、大豆(Glycinemax)、烟豆(Glycinetabacina)、野大豆(Glycinesoja)和短绒野大豆(Glycinetomentella)。前述1-4任一实施方案或其它实施方案中,重组DNA构建体包含至少一个植物中有功能的启动子作为调控序列。前述1-4任一实施方案或其它实施方案中,至少一种农艺性状的变化可以是增加也可以是减少。前述1-4任一实施方案或其它实施方案中,至少一种农艺性状选自叶片绿色、籽粒产量、生长速率、生物量、成熟时的鲜重、成熟时的干重、果实产量、种子产量、植物总氮含量、果实氮含量、种子氮含量、植物营养组织氮含量、植物总游离氨基酸含量、果实游离氨基酸含量、种子游离氨基酸含量、植物营养组织游离氨基酸含量、植物总蛋白含量、果实蛋白含量、种子蛋白含量、植物营养组织蛋白含量、耐旱性、氮吸收能力、根倒伏、收获指数、茎倒伏、株高、穗高、穗长、耐盐性、分蘖数、圆锥花序大小、幼苗活力和低温胁迫下的出苗情况。例如,至少一个农艺性状的改变可以是籽粒产量、叶绿素含量或生物量的增加。前述1-4任一实施方案或其它实施方案中,与对照植物相比,所述植物在水分限制条件下表现出至少一个农艺性状的变化。前述1-4任一实施方案或其它实施方案中,与对照植物相比,所述植物在氧化胁迫即百草枯胁迫下表现出至少一个农艺性状的变化。“干旱”指植物可用水分的减少,特别是较长时间的缺水或者在植物重要的生长阶段发生,会造成植物损伤,或阻止植物的生长(限制植物的生长,降低籽粒的产量)。“耐旱性”指干旱胁迫下能够存活并且没有实质性的生理或物理改变的能力,和/或一段时间干旱后复水能够恢复的能力。多肽的“耐旱能力”表示与参照或对照植物相比,过表达该多肽可以提高转基因植物的耐旱性能。植物“增强的耐旱性”是与参照或对照植物相比测定的,反映了植物在干旱胁迫下存活的能力,并且与参照或对照相比,具有较小的生理或物理损伤,或者干旱胁迫后复水时,恢复较快的能力。“环境条件”指植物生长的环境,诸如可用的水分、可用的营养物质或存在的昆虫或病害。“百草枯”(1,1-二甲基-4,4-联吡啶二氯化物)是一类叶面喷施的非选择性的吡啶除草剂,能够引起光氧化胁迫,并进一步造成植物的损害或者阻止植物的正常生长。“百草枯耐性”是植物的一种性状,反映了百草枯溶液处理后,与参照或对照植物相比,植物的存活或生长良好的能力。植物“增加的百草枯耐性”是相对于参照或对照植物测定的,反映了植物在百草枯溶液处理后,与参照或对照植物相比,能够存活并具有较小的生理或物理伤害的能力。通常,针对较低浓度的百草枯溶液的耐性用作诸如干旱胁迫的非生物胁迫耐性的一种指标。“氧化胁迫”反映了活性氧分子的生成和生物系统清除活性氧中间体或修复损伤的能力之间的不平衡。扰乱细胞正常的氧化还原状态会导致产生过氧化氢和自由基的毒性效应,过氧化氢和自由基能够损害包括蛋白、脂质和DNA在内的细胞组成部件。下面的例子描述一些代表性的方法或技术用于模拟干旱条件和/或评估耐旱性;模拟氧化条件。本领域的技术人员也可以测试模拟或者自然发生的干旱条件下的植物保持足够产量(至少75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或者100%的产量)的能力评估耐旱性,例如,干旱条件下产生与非干旱条件下相比实质相当的产量;与对照或参照植物相比,干旱条件下产量减少较少。本发明中涉及的参数包括与对照细胞或对照植物相比的恢复度、存活率、百草枯耐性率、基因表达水平、水分利用效率、编码蛋白的水平或活性和其它参数。“对照”、“对照植物”或“对照植物细胞”为测定受测试植物或植物细胞的表型变化提供参考,由于转化,受测植物或植物细胞的基因组改变影响到目的基因,测试的植物或植物细胞可以是转基因植物或转基因植物细胞的子代,并含有上述的基因的改变。本领域技术人员很容易找到适当的对照或参照植物,当采用本发明描述的组合物或方法评估或测定转基因植物一个农艺性状或表型时。例如,但不限于下述举例:1.转化植物的子代,所述转化植物对于重组DNA构建体来说是半合子的,所述子代分离成包含或不包含该重组DNA构建体的植株:包含该重组DNA构建体的子代将通常相对于不包含该重组DNA构建体的子代来进行测量,即不包含该重组DNA构建体的子代是对照或参照植物。2.重组DNA构建体基因渗入至近交系中,例如在水稻和玉米中,或基因渗入进变种中,例如在大豆中:基因渗入品系将通常相对于亲本近交系或变种品系进行测量(即,亲本近交系或品种品系是对照或参照植物)。3.双杂交系,其中第一杂交系由两个亲本近交系产生,而第二杂交系由相同的两个亲本近交系产生,不同的是其中一个亲本近交系含有重组DNA构建体:第二杂交系通常将相对于第一杂交系进行测量(即第一杂交系为对照植物或参照植物)。4.包含重组DNA构建体的植物:该植株可以相对于这样的对照植物进行评价或测量,该对照植物不包含重组DNA构建体,但具有与该植株相当的遗传背景(例如,与包含重组DNA构建体的植株相比较,核遗传物质具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列同一性)。存在许多可用于分析、比较和表征植物遗传背景的实验室的技术;其中这些技术是同工酶电泳、限制性片段长度多态性(RFLPs)、随机扩增多态性DNA(RAPDs)、任何引物聚合成酶链式反应(AP-PCR)、DNA扩增指纹(DAF)、序列特异扩增区域(SCARs)、扩增片段长度多态性()和也称为微卫星的简单序列重复(SSRs)。对照植物或对照植物细胞包括,例如:(a)用于基因改变产生受测植物或细胞的野生型植物或细胞;(b)相同基因组作为起始材料但是转入空载体(如带有标记基因的并对目标的性状没有影响的载体)的植物或植物细胞;(c)转基因植物或植物细胞性状分离,获得的非转基因的子代植物或植物细胞;(d)没有暴露于可以诱导基因表达的条件或刺激下的,与转基因植物或植物细胞基因组相同的植物或植物细胞;(e)特定的目标基因不表达情况下的,转基因植物或者植物细胞本身。对照可以包括多种代表上述一种或多种类型的个体,例如,(c)中性状分离后非转基因的材料的混合通常认为是空白对照。本发明中,株系空白(Linenull)、空白对照(Bulknull)、ZH11-TC、和VC指对照植物。株系空白(Linenull)表示半合子转基因水稻株系分离的相应空白,空白对照(Bulknull)表示半合子转基因株系分离的非转基因空白对照的混合,ZH11-TC表示通过组织培养中花11获得的水稻植株,VC表示转化空载体DP0005或DP0158获得水稻植株。方法:方法包括但不限于:提高植物耐旱性的方法,评估植物耐旱性的方法,提高植物百草枯耐受性的方法,改变植物农艺性状的方法,确定植物农艺性状变化的方法,和生产种子的方法。植物可以是单子叶植物,也可以是双子叶植物,例如水稻、玉米或大豆,植物也可以是向日葵、油菜、小麦、苜蓿、棉花、大麦、粟、甘蔗或高粱,种子是玉米或大豆种子,例如玉米杂交种子或玉米自交种子。方法包括但不限于:转化细胞方法包括将本发明公开的任意一个或多个分离的多核苷酸转化细胞,其中,在特定的实施方案中,所述细胞是真核细胞,如酵母、昆虫或植物细胞;或原核细胞如细菌细胞。产生转基因植物的方法包括将本发明公开的任一分离的核苷酸或重组DNA构建体转化植物细胞和由转化的植物细胞再生转基因植物,其中本方法获得转基因植物和转基因种子可用于本发明的其它方法。从细胞或细胞培养物中分离本发明的多肽的方法,其中,所述细胞包含重组DNA构建体,所述重组DNA构建体包含本发明的一个多核苷酸和与其可操作连接的至少一个调控序列,所述宿主细胞在适于重组DNA构建体表达的条件下生长。改变本发明多肽在宿主细胞中表达水平的方法包括:(a)将本发明重组DNA构建体转化宿主细胞;和(b)使转化宿主细胞在适于重组DNA构建体表达的条件下生长,其中重组DNA构建体的表达导致转化宿主细胞中本发明多肽含量的变化。一种提高植物耐旱性和/或百草枯耐受性的方法,包括(a)将重组DNA构建体导入到可再生的植物细胞中,所述重组DNA构建体包含一个多核苷酸和与其可操作地连接至少一种调控序列(例如在植物中有功能的启动子),其中所述多核苷酸编码多肽,所述所述多肽的氨基酸序列在与SEQIDNO:5、8、11、14、17或20进行比较时,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;(b)在步骤(a)之后,由所述可再生的植物细胞再生转基因植物,其中所述转基因植物在其基因组中包含所述重组DNA构建体并且在与对照植物进行比较时表现出提高的耐旱性和/或百草枯耐性;和进一步的(c)获得来源于所述转基因植物的子代植物,其中所述子代植物在其基因组中包含重组DNA构建体,与对照植物相比,表现出提高的耐旱性和/或百草枯耐性。一种评估植物耐旱性和/或百草枯耐性的方法,包括(a)获得转基因植物,所述转基因植物在其基因组中包含重组DNA构建体,所述重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列(如,植物中有功能的启动子),其中,所述多核苷酸编码一个多肽,所述多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;(b)获得源于所述转基因植物的子代植物,其中所述子代植物在其基因组中包含重组DNA构建体;和(c)与对照植物相比,评估子代植物的耐旱性和/或百草枯耐性。确定植物一种农艺性状变化的方法,包括(a)获得转基因植物,所述转基因植物在其基因组中包含重组DNA构建体,所述重组DNA构建体包含一个多核苷酸和与其可操作连接至少一个调控序列(如,植物中有功能的启动子),其中所述多核苷酸编码的多肽的氨基酸序列与SEQIDNO:5、8、11、14、17或20相比,具有至少50%、51%、52%、53%、54%、55%、56%、57%、58%、59%、60%、61%、62%、63%、64%、65%、66%、67%、68%、69%、70%、71%、72%、73%、74%、75%、76%、77%、78%、79%、80%、81%、82%、83%、84%、85%、86%、87%、88%、89%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%的序列一致性;(b)获得源于所述转基因植物的子代植物,其中所述子代植物的基因组中包含重组DNA构建体;和(c)水分限制条件下,确定与对照植物相比,所述子代植物是否表现出至少一个农艺性状的变化。一种生产种子的方法包括前述的任一方法,进一步还包括从所述子代植物获得种子,其中所述种子在其基因组中包含重组DNA构建体。在前述任一的方法或本文其它实施方案披露的任一方法中,所述引入步骤中所述再生植物细胞可包括愈伤细胞、胚胎愈伤细胞、配子细胞、分生细胞、或不成熟的胚胎细胞。可再生植物细胞可源于自交玉米植株。在前述任一方法或本文披露的其它实施方案中的任一方法中,所述再生步骤可包括:(i)在含有胚促激素培养基上培养所述转化的植物细胞直至长出愈伤组织;(ii)将步骤(i)所述的转化植物细胞转移至含有组织促激素的第一培养基上;和(iii)将步骤(ii)所述转化植物细胞接种到第二培养基上,使其茎伸长、根发育或两者均发育。在前述任一方法或本发明的其它实施方案的方法中,确定转基因植物农艺性状的变化的步骤,如果可行,可包括在可变的环境条件下,确定与不含有重组DNA构建体的对照植物相比,所述转基因植物是否表现出至少一个农艺性状的变化。在前述任一方法或者本发明其它实施方案的任一方法中,如果可能,确定子代植物农艺性状变化的步骤包括可变的环境条件下,确定与不含有重组DNA构建体的对照植物相比,所述子代植物是否表现出至少一个农艺性状的变化。在前述任一方法或者本发明其它实施方案的任一方法中,与对照植物相比,所述植物在水分限制条件下表现出至少一个农艺性状的变化。在前述任一方法或者本发明其它实施方案的任一方法中,存在将重组DNA构建体导入可再生植物的供选方案,所述重组DNA构建体包含一个多核苷酸和与其可操作连接的至少一个调控序列,例如可以将一个调控序列(如一个或多个增强子、任选的转座子元件的一部分)导入可再生植物细胞,接着筛选带有调控序列和与其可操作连接的编码本发明多肽的内源基因的转基因事件。可以通过任何适当的技术将本发明的重组DNA构建体导入植物中,所述技术包括但不限于直接DNA吸收、化学药品处理、电穿孔、显微注射、细胞融合、感染、载体介导的DNA转移、基因枪轰击或农杆菌的转化。植物转化和再生的技术在国际专利公开号WO2009/006276中描述,其全部内容并入作为参考。另外,也有修饰或改变宿主内源基因组DNA的方法,包括改变宿主天然DNA序列或包括调控元件、编码或非编码序列等前体转基因序列。这些方法也可以用于将核酸序列靶向到基因组中改造目标识别序列。例如本文中转基因修饰的细胞或植物使用传统的基因工程核酸酶如产生修饰植物基因组的归位核酸内切酶产生(例如WO2009/114321;Gao等(2010)PlantJournal1:176-187)。其它的位点定向工程是通过使用锌指结构域识别偶联的限制性内切酶的限制性特点修饰内源基因(例如Urnov等(2010)NatRevGenet.11(9):636-46;Shukla等(2009)Nature459(7245):437-41)。转录激活样(TAL)效应因子-DNA修饰酶(transcriptionactivator-likeeffector-DNAmodifyingenzyme,TALE或TALEN)可用于基因工程修饰植物基因组,参见例如US20110145940,Cermak等(2011)NucleicAcidsRes.39(12)和Boch等(2009),Science326(5959):1509-12。植物基因组定点修饰也可以使用细菌II型CRISPR(成簇的规律的间隔的短回文的重复序列,clusteredregularlyinterspacedshortpalindromicrepeats)/Cas(CRISPR关联蛋白,CRISPR-associated)系统,参考例如Belhaj等(2013),PlantMethods9:39.CRISPR/Cas系统可以允许可定制的小的非编码RNA引导的基因组DNA靶向切割。本领域的技术人员熟悉将编码期望多肽的外源基因转化植物和培育再生植物的方法。再生植物可以自花授粉产生纯合子转基因植物,或者将再生植物的花粉与种子长出的农艺重要的植物杂交,或者农艺重要的植物的花粉与再生的转基因植物杂交。本领域的技术人员熟知培育本文披露的含有期望多肽的转基因植物的方法。实施例本文的具体实施进一步在下列实例中示明。在这些实例中,除非特别说明,采用摄氏/公制。在这些实例中,仅用举例说明具体的实施过程。通过上述讨论和具体事例,本领域的专业人员可以查明本发明的基本特征,经过各种改变和修改将本发明应用于各种用途和条件,并不偏离本发明主体和范围。因此,除了本专利表述和讨论的各种修改外,本领域内的专业人员所做的不偏离本发明主题的修改也将落入本专利的权力要求范围内。实施例1.耐旱性基因的克隆和构建体的构建根据水稻激活标签突变体库初步的筛选结果和表2的基因ID序列信息,设计引物,克隆了水稻非生物胁迫耐性基因OsGSTU41、OsPPCK4、OsCAM2、OsDN-DTP4、OsLecRK4.1和OsLecRK4.2。引物序列和扩增基因片段的长度如表3所示。以中花11号水稻叶、茎和根混合的cDNA库为模板克隆OsGSTU41、OsPPCK4和OsCAM2的cDNA;以ZH11基因组DNA为模板克隆OsDN-DTP4、OsLecRK4.1和OsLecRK4.2的gDNA。PCR反应混合液和PCR程序如表4和5所示。表2.水稻基因名称、基因ID(TIGR)和构建体ID基因名称LOCID构建体IDOsGSTU41LOC_Os01g72160DP0043OsPPCK4LOC_Os02g56310DP0058OsCAM2LOC_Os05g41210DP0059OsDN-DTP4LOC_Os07g04720DP0167OsLecRK4.1LOC_Os04g44900DP0173OsLecRK4.2LOC_Os04g44910DP0209表3.克隆水稻耐旱性基因的引物表4.克隆耐旱性基因的PCR反应混合液表5.PCR循环状况PCR扩增产物在琼脂糖凝胶电泳分离后采用柱式试剂盒回收,并与TA克隆载体连接。通过测序确定PCR产物的核酸序列和在构建体中的方向,然后将基因克隆到植物双元载体DP0005(pCAMBIA1300-AsRed,SEQIDNO:1)或DP0158中,DP0158为将DsRed基因表达盒(SEQIDNO:2)连接入DP0005载体后获得的。OsGSTU41、OsPPCK4和OsCAM2基因克隆入DP0005构建体中,产生的过表达构建体列在表2中。DP0043构建体中克隆的核苷酸序列和OsGSTU41的编码序列如SEQIDNO:3和4所示,OsGSTU41的氨基酸序列如SEQIDNO:5所示;DP0058构建体中克隆的核苷酸序列和OsPPCK4的编码序列如SEQIDNO:6和7所示,OsPPCK4的氨基酸序列如SEQIDNO:8所示;DP0059构建体中克隆的核苷酸序列和OsCAM2的编码序列如SEQIDNO:9和10所示,OsCAM2的氨基酸序列如SEQIDNO:11所示。OsDN-DTP4、OsLecRK4.1和OsLecRK4.2克隆入DP0158构建体中,DP0167构建体中克隆的核苷酸序列和OsDN-DTP4的编码序列如SEQIDNO:12和13所示,OsDN-DTP4的氨基酸序列如SEQIDNO:14所示;DP0173构建体中克隆的核苷酸序列和OsLecRK4.1的编码序列如SEQIDNO:15和16所示,OsLecRK4.1的氨基酸序列如SEQIDNO:17所示;和DP0209构建体中克隆的核苷酸序列和OsLecRK4.2的编码序列如SEQIDNO:18和19所示,OsLecRK4.2的氨基酸序列如SEQIDNO:20所示。实施例2.转化获得转基因水稻本研究中,所有过表达构建体和空载体(DP0005和DP0158)采用林拥军和张启发((2005)PlantCellRep.23:540-547)描述的农杆菌介导的方法转化入中花11号水稻。中花11号水稻为中国农业科学院作物研究所培育的品种,第一批种子由北京未名凯拓农业生物公司提供。将含有构建体的农杆菌侵染胚诱导产生的愈伤组织,转化实验室获得的T0代转基因幼苗移栽至田间水田中获得T1种子,T1和T2代种子储藏在4℃冷库中。OsGSTU41、OsPPCK4和OsCAM2转基因种子在绿色荧光灯下不能显示红色,使用潮霉素筛选T1代转基因植株,将生长到1-2cm高的水稻植株在50mg/L的潮霉素溶液中培养,存活的植株即潮霉素抗性植株种植到田间收获T2代种子,只有潮霉素抗性的T2代转基因种子用于性状筛选。OsDN-DTP4、OsLecRK4.1和OsLecRK4.2转基因种子在绿色荧光灯下显示红色即为转基因种子,用于下述试验。实施例3.基因表达分析采用标准的实时RT-PCR程序诸如源于的反转录试剂盒和实时RT-PCR(SYBRRPremixExTaqTM,宝生物)分析转基因水稻植株中基因的表达水平。EF1α基因用作内参显示转基因水稻和对照植物的扩增和上样量类似。以EF1αmRNA水平为参照确定基因表达量。如图3所示,OsGSTU41基因在DP0043.15水稻叶片中的表达量设置为1.00,OsGSTU41在所有10个测试的转基因水稻株系中过量表达,而在ZH11-TC植株中没有检测到OsGSTU41基因表达,实时PCR的引物为:DP0043-3:5'-GGCTGTCGTTCTGGATGG-3'(SEQIDNO:33)DP0043-4:5'-GCAGTGAACAAGGCGACG-3'(SEQIDNO:34)如图4所示,OsPPCK4基因在ZH11-TC水稻叶片中的表达量设置为1.00,OsPPCK4在所有转基因株系中过量表达,而OsPPCK4基因在ZH11-TC和DP0158对照中的表达量较低。DP0058-F1:5'-GCTCTACATGATGCTCTCCG-3'(SEQIDNO:35)DP0058-R1:5'-GAGACGTCCTTGCAGAGC-3'(SEQIDNO:36)OsDN-DTP4基因在ZH11-TC水稻中的表达量设置为1.00,OsDN-DTP4基因在所有9个转基因株系中过量表达(图5.)。DP0167-F1:5'-CCAGTTCAGAGTACGGTGCCG-3'(SEQIDNO:37)DP0167-R1:5'-GTGTCCACGTCAGCCTCCTTTC-3'(SEQIDNO:38)OsLecRK4.1基因在ZH11-TC水稻中的表达量设置为1.00,OsLecRK4.1在所有9个转基因水稻株系中过量表达(图6.)。DP0173-F1:5'-CGCTCAACATCTCATCCC-3'(SEQIDNO:39)DP0173-R1:5'-CCGCATGAACACGAACAC-3'(SEQIDNO:40)如图7所示,OsLecRK4.2基因在ZH11-TC水稻中的表达量设置为1.00,OsLecRK4.2在所有测试的转基因株系中过量表达,而OsLecRK4.2基因在ZH11-TC植株中表达量较低。DP0209-F1:5'-CCGACGATGGTGAGCTAC-3'(SEQIDNO:41)DP0209-R1:5'-GTGACGGTGGAAGGGAAG-3'(SEQIDNO:42)实施例4.转基因水稻植株的干旱验证水稻幼苗的耐旱性筛选试验在温室中进行。温室中设置比例为1:1的钠灯和金属卤化物灯作为光源,光照/黑暗时间为16h/8h,光源设置在苗床上部大约1.5m处,晴天时,高于苗床30cm处的光强度为10,000-20,000lx,阴天时为6,000-10,000lx;温室相对湿度为30%-90%,温度为20-35℃。干旱验证方法:T2转基因种子和对照种子首先采用800ppm的多菌灵在32℃消毒8h,然后使用蒸馏水冲洗3~5次,然后32℃浸种16h,在35-37℃培养箱中萌发18h。发芽的种子种植在装有有机质、蛭石和沙子(V:V:V=3:3:2)的托盘或小盆中。幼苗在正常的温室条件下生长,并浇灌改良的IRRI营养液,当幼苗生长到3-叶期时,停止浇水,并将水稻幼苗放置在干燥的位置,直至水稻叶片变干和卷曲(大约9~15天,根据季节不同),然后再将托盘放在水槽中使水稻苗复水5-7天,并计算恢复程度。应用以下的打分系统:大于半个绿茎=1,大于2/3的绿叶=1,大于1/3绿叶并低于2/3绿叶=0.5,低于1/3绿叶=0.2,零绿叶或绿茎=0。恢复度是绿色组织得分的总和,数据利用混合模型(MixedModel)进行统计分析,表现显著优于对照的水稻株系被认为是阳性株系(P<0.05)。存活率指存活株数除以总株数的百分数,是干旱验证的一个指标。本试验中采用两种实验设计:1)采用拉丁方设计,每个株系的16株水稻种植在同一托盘的不同位置,种在同一托盘中的组织培养后的野生型中花11(ZH11-TC)和/或空载体转基因转化对照(DP0005或DP0158)用作对照,耐旱型品种绵恢501作为阳性对照、敏旱性品种东北引2号作为阴性对照种植在同一个托盘中;2)随机区组设计用于从构建体水平验证水稻的功能,同一构建体的9-12个转基因株系种植在一个试验单元中,采用混合模型(MixedModel)考虑构建体、株系和环境效应来评估基因的功能。如果转基因水稻的存活率或恢复度显著高于对照(P<0.05),则认为测试的基因具有耐旱性的功能。GH干旱试验结果:1)OsGSTU41转基因水稻(DP0043)的GHDRT验证结果第一次试验中采用拉丁方设计对10个OsGSTU41转基因株系进行测试,不同的株系种植在不同的托盘上,同一托盘上的ZH11-TC和DP0005幼苗用作相应的对照。表6表明10个转基因株系的存活率和恢复度高于ZH11-TC对照,6个株系的恢复度显著高于ZH11-TC对照。8个转基因株系的存活率和恢复度高于DP0005对照,4个株系的恢复度显著高于DP0005对照。这些结果表明OsGSTU41转基因水稻增强了苗期耐旱性。表6.温室条件下OsGSTU41转基因水稻增强耐旱性(第一次试验)第二次试验中采用构建体水平的实验设计对9个转基因株系进行测试,当生长到三叶期,水稻植株第一次干旱胁迫17天后,在水中恢复6天,随后进行第二次干旱胁迫22天,恢复7天。如表7所示,108株OsGSTU41转基因幼苗中,69株存活;而24株ZH11-TC植株中,9株存活;12株DP0158幼苗中,5株存活。构建体水平,OsGSTU41转基因水稻的存活率和平均恢复度均高于ZH11-TC和DP0158对照(表7)。转基因株系水平的分析表明8个株系的存活率和平均恢复度高于ZH11-TC和DP0158对照(表8)。这些结果表明OsGSTU41转基因水稻增强了苗期耐旱性,OsGSTU41基因在提高转基因植物耐旱性中发挥作用。表7.温室条件下OsGSTU41转基因水稻增强耐旱性(第二次试验,构建体水平)表8.温室条件下OsGSTU41转基因水稻增强耐旱性(第二次试验,转基因株系水平)2)OsPPCK4转基因水稻(DP0058)的GHDRT验证结果第一次试验中对12个OsPPCK4转基因株系进行干旱胁迫测试,不同的株系种植在不同的托盘上,同一托盘上的ZH11-TC幼苗用作相应的对照。表9表明10个转基因株系的存活率和恢复度高于ZH11-TC对照,5个株系的恢复度显著高于ZH11-TC对照,表明OsPPCK4转基因水稻提高了苗期耐旱性。表9.温室条件下OsPPCK4转基因水稻增强耐旱性(第一次试验)第二次试验中采用构建体水平的实验设计对9个转基因株系进行测试,当生长到三叶期,水稻植株干旱胁迫17天后,在水中恢复7天。108株OsPPCK4转基因水稻植株中,49株存活;而24株ZH11-TC植株中,6株存活;12株DP0158幼苗中,只有1株存活。构建体水平,OsPPCK4转基因水稻的存活率高于ZH11-TC和DP0158幼苗,平均恢复度显著高于ZH11-TC和DP0158幼苗(表10)。转基因株系水平的分析表明6个株系的存活率高于ZH11-TC和DP0158对照,9个株系的恢复度高于两个对照(表11)。这些结果表明OsPPCK4转基因水稻增强了苗期耐旱性,OsPPCK4基因在提高转基因植物耐旱性中发挥作用。表10.温室条件下OsPPCK4转基因水稻增强耐旱性(第二次试验,构建体水平)表11.温室条件下OsPPCK4转基因水稻增强耐旱性(第二次试验,转基因株系水平)3)OsCAM2转基因水稻(DP0059)的GHDRT验证结果第一次试验中采用拉丁方设计对8个OsCAM2转基因株系进行测试,不同的株系种植在不同的托盘上,同一托盘上的ZH11-TC幼苗用作相应的对照。表12表明6个转基因株系的存活率和恢复度高于ZH11-TC对照,4个株系的恢复度显著高于ZH11-TC对照,表明OsCAM2转基因水稻提高了苗期耐旱性。第二次试验中采用构建体水平的实验设计对8个OsCAM2转基因株系进行测试。如表13所示,干旱胁迫后,96株OsCAM2转基因幼苗中,83株存活。构建体水平,OsCAM2转基因水稻的存活率和恢复度高于DP0158对照(P值=0.0679),并显著高于ZH11-TC对照(P值=0.0090)。转基因株系水平的分析表明8个株系的存活率和平均恢复度高于ZH11-TC和DP0158对照,5个株系的恢复度显著高于ZH11-TC对照,3个株系的恢复度显著高于DP0158对照(表14)。这些结果进一步表明OsCAM2基因增强了植物耐旱性。表12.温室条件下OsCAM2转基因水稻增强耐旱性(第一次试验)表13.温室条件下OsCAM2转基因水稻增强耐旱性(第二次试验,构建体水平)表14.温室条件下OsCAM2转基因水稻增强耐旱性(第二次试验,转基因株系水平)第三次试验中采用构建体水平的实验设计对8个OsCAM2转基因株系进行测试。当生长到三叶期,水稻植株干旱胁迫15天后,在水中恢复7天。96株OsCAM2转基因水稻植株中,55株存活;而24株ZH11-TC幼苗中,4株存活;12株DP0158幼苗中,4株存活。构建体水平,OsCAM2转基因水稻的存活率高于ZH11-TC和DP0158幼苗,平均恢复度显著高于ZH11-TC对照(表15)。转基因株系水平的分析表明8个株系的存活率和平均恢复度高于ZH11-TC和DP0158对照(表16)。三次试验结果中,OsCAM2转基因水稻显示出增强的苗期耐旱性。这些结果表明OsCAM2基因在提高转基因植物耐旱性中发挥作用。表15.温室条件下OsCAM2转基因水稻增强耐旱性(第三次试验,构建体水平)表16.温室条件下OsCAM2转基因水稻增强耐旱性(第三次试验,转基因株系水平)4)OsDN-DTP4转基因水稻(DP0167)的GHDRT验证结果第一次试验中采用拉丁方设计对12个OsDN-DTP4转基因株系进行测试,不同的株系种植在不同的托盘上,同一托盘上的ZH11-TC幼苗用作相应的对照。表17表明8个转基因株系的存活率和恢复度高于ZH11-TC对照,4个株系的恢复度显著高于ZH11-TC对照。这些结果表明OsDN-DTP4转基因水稻提高了苗期耐旱性。第二次和第三次试验中采用构建体水平的实验设计对9个OsDN-DTP4转基因株系进行测试。如表18所示,第二次试验中,构建体水平,所测试的OsDN-DTP4转基因水稻的存活率和恢复度高于ZH11-TC和DP0158对照;转基因株系水平的分析表明9个株系的存活率和恢复度高于ZH11-TC和DP0158对照(表19)。第三次试验的结果表现出相同的趋势(表20和21),所测试的OsDN-DTP4转基因水稻的恢复度高于ZH11-TC和DP0158对照。这些结果进一步表明OsDN-DTP4基因增强了植物耐旱性。表17.温室条件下OsDN-DTP4转基因水稻耐旱性试验(第一次试验)表18.温室条件下OsDN-DTP4转基因水稻耐旱性试验(第二次试验,构建体水平)表19.温室条件下OsDN-DTP4转基因水稻耐旱性试验(第二次试验,转基因株系水平)表20.温室条件下OsDN-DTP4转基因水稻耐旱性试验(第三次试验,构建体水平)表21.温室条件下OsDN-DTP4转基因水稻耐旱性试验(第三次试验,转基因株系水平)实施例5.成熟转基因水稻的田间耐旱性试验开花期干旱胁迫是农业生产中严重的问题。转基因水稻进一步在田间干旱条件下进行验证。田间干旱试验中,每个基因构建体选择9-12个转基因株系。T2代种子首先参照实施例4描述的方法消毒,萌发的种子种植在田间苗床上,三叶期时,将水稻幼苗移栽到田间试验地,设置三到四个重复,每个重复每转基因株系8-10株苗,并将三到四个重复种植在同一地块。根据统计分析,同一地块中,邻近种植转基因株系种植的ZH11-TC、DP0158或空白对照(BulkNull或LineNull)用作统计分析中的对照。水稻植株正常管理,并使用相应的杀虫剂和化肥,幼穗期分化II期停止浇水,因此开花期时产生干旱胁迫,干旱时间长短取决于温度和湿度等天气条件。干旱期间,使用TDR30(SpectrumTechnologies,Inc.)在每块地的不同位点每四天测定土壤相对含水量。试验过程中,观察并记录植株表型,植株的表型主要包括抽穗期、卷叶程度、敏旱性和抗旱性,尤其关注中午时植株的卷叶程度。收获时,每个株系在每行中间挑选约6株具有代表性的植株,并称量每株水稻籽粒的重量,利用混合线性模型(mixedlinearmodel)对籽粒重量进行统计分析。根据分析结果选择阳性转基因株系。田间干旱试验结果:1)OsGSTU41转基因水稻(DP0043)的田间DRT验证结果第一次试验中,22个OsGSTU41转基因株系在海南省进行验证,邻近种植的株系空白(linenull)用作相应的对照。每个株系种植10株,并设置4个重复,种在同一个地块。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从30%降到10%(图1)。收获时,每个株系在每行中间挑选约6株具有代表性的植株,并称量每株水稻籽粒的重量。如表22所示,13个株系的单株籽粒产量高于相应的linenull,7个株系的单株籽粒产量显著高于相应的linenull(P<0.05)。这些结果表明,干旱胁迫后,OsGSTU41水稻植株的单株籽粒产量高于对照。第二次试验在海南省进行,对12个OsGSTU41转基因株系进行验证,邻近种植的空白对照(bulknull)水稻植株用作对照,每个株系种植8株,设置三个重复。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从27%降到6%(图2)。3个转基因株系DP0043.10、DP0043.16和DP0043.22显示出耐旱性表型,如卷叶程度较轻和叶色较绿;4个株系DP0043.10、DP0043.16、DP0043.22、DP0043.26在成熟期显示出较好的结实率。收获时,每个株系在每行中间挑选约6株具有代表性的植株,并称量每株水稻籽粒的重量。如表23所示,11个OsGSTU41转基因水稻的单株籽粒产量高于bulknull对照,2个株系的单株籽粒产量显著高于bulknull对照(P<0.1)。这些结果进一步表明干旱胁迫后,OsGSTU41转基因水稻的单株籽粒产量高于对照。如实施例4所述,OsGSTU41转基因水稻增强了苗期耐旱性,这些结果表明OsGSTU41基因在增强植物苗期和成熟期耐旱性中发挥作用。表22.OsGSTU41转基因水稻在田间干旱条件下的籽粒产量分析(第一次试验)表23.OsGSTU41转基因水稻在田间干旱条件下的籽粒产量分析(第二次试验)2)OsPPCK4转基因水稻(DP0058)的田间DRT验证结果第一次试验中,9个OsPPCK4转基因株系在北京进行验证,ZH11-TC和DP0158用作对照。每个转基因株系种植8株,并设置3个重复,种植在同一地块中。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从50%降到10%(图8)。转基因株系DP0058.14表现出耐旱性表型,如保持叶片伸展和叶色浓绿。收获时,每个株系在每行中间挑选约6株具有代表性的植株,并称量每株水稻籽粒的重量。如表24所示,9个株系的单株籽粒产量高于DP0158对照,4个株系的单株籽粒产量高于ZH11-TC对照。这些结果表明,干旱胁迫后,OsPPCK4水稻植株的单株籽粒产量高于对照。表24.OsPPCK4转基因水稻在田间干旱条件下的籽粒产量分析3)OsCAM2转基因水稻(DP0059)的田间DRT验证结果第一次试验中,8个OsCAM2转基因株系在北京进行验证,ZH11-TC和DP0158用作对照。每个转基因株系种植8株,并设置3个重复,种植在同一地块中。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从50%降到10%(图9)。转基因株系DP0059.05在干旱胁迫过程中表现出较好的耐旱性表型,并表现出较好的结实率。收获时,每个株系在每行中间挑选约6株具有代表性的植株,并称量每株水稻籽粒的重量。如表25所示,构建体水平上,OsCAM2转基因水稻的单株籽粒产量显著高于DP0158对照;8个株系的单株籽粒产量高于DP0158对照,3个株系的单株籽粒产量高于ZH11-TC对照。这些结果表明,干旱胁迫后,OsCAM2水稻植株的单株籽粒产量高于对照。表25.OsCAM2转基因水稻在田间干旱条件下的籽粒产量分析(第一次试验)第二次试验中,同样的8个OsCAM2转基因株系在海南省进行验证,ZH11-TC和DP0158用作对照。每个转基因株系种植10株,并设置4个重复。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从35%降到5%(图10)。5个转基因株系DP0059.04、DP0059.05、DP0059.09、DP0059.11和DP0059.14表现出耐旱性表型,卷叶程度比对照轻和叶色比对照更绿。收获时,OsCAM2转基因水稻没有表现出较高的单株籽粒产量,只有2个株系的单株籽粒产量较高(表26)。这些结果表明,OsCAM2过表达转基因水稻植株获得了苗期耐旱性,过量表达OsCAM2基因提高了植物耐旱性。表26.OsCAM2转基因水稻在田间干旱条件下的籽粒产量分析(第二次试验)4)OsLecRK4.1转基因水稻(DP0173)的田间DRT验证结果第一次试验中,对12个OsLecRK4.1转基因株系进行验证,ZH11-TC和DP0158水稻植株用作对照。每个转基因株系种植10株,并设置4个重复,种植在同一地块中。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从40%降到5%(图11)。4个转基因株系DP0173.01、DP0173.04、DP0173.08和DP0173.14比邻近种植的ZH11-TC和DP0158对照叶色更绿,卷叶程度较轻。DP0173.04在成熟期还表现出较好的结实率。构建体水平上,OsLecRK4.1转基因水稻的单株籽粒产量低于ZH11-TC对照但高于DP0158对照;表27为转基因株系水平的分析。这些结果表明,OsLecRK4.1转基因水稻植株提高了苗期耐旱性,并提高了的干旱胁迫后单株籽粒产量。表27.OsLecRK4.1转基因水稻在田间干旱条件下的籽粒产量分析5)OsLecRK4.2转基因水稻(DP0209)的田间DRT验证结果第一次试验中,对12个OsLecRK4.2转基因株系进行验证,ZH11-TC和DP0158水稻植株用作对照。每个转基因株系种植10株,并设置4个重复,种植在同一地块中。幼穗分化II期断水直至种子成熟,产生较严重的干旱胁迫,抽穗和成熟过程中土壤体积含水量从35%降到5%(图12)。4个转基因株系DP0209.13、DP0209.26、DP0209.28和DP0209.34比邻近种植的ZH11-TC和DP0158对照叶色更绿,卷叶程度较轻。DP0209.34在成熟期还表现出较好的结实率。构建体水平上,OsLecRK4.2转基因水稻的单株籽粒产量低于ZH11-TC和DP0158对照;表28为转基因株系水平的分析。这些结果表明,OsLecRK4.2转基因水稻植株在干旱胁迫后提高了苗期耐旱性。表28.OsLecRK4.2转基因水稻在田间干旱条件下的籽粒产量分析实施例6.转基因水稻植株的实验室百草枯试验百草枯(1,1-二甲基-4,4-联吡啶二氯化物)是一类叶面喷施的非选择性的吡啶除草剂,是世界范围内广泛应用一种除草剂,能够控制大量作物如玉米、水稻、大豆等中生长的杂草。在植物细胞中,百草枯主要靶向叶绿体,百草枯通过接受光系统I的电子,然后与氧气发生化学反应生成过氧化物和过氧化氢,而过氧化物和过氧化氢可以导致产生光氧化胁迫。干旱胁迫通常导致植物中产生活性氧(ROS),有时,植物的耐旱性与增强的抗活性氧能力相关。百草枯是强有力的氧化胁迫诱导剂,能够大大的增加活性氧(ROS)的产生,同时抑制抗氧化系统活性所需的的还原物和化合物的再生。非生物胁迫增加了ROS的产生,而植物响应耐性到死亡的范围取决于胁迫力度和与其相关的ROS水平。相对较低水平的百草枯能够模拟胁迫相关的ROS产生,并用作植物胁迫生物学中胁迫耐性的标记(HasaneenM.N.A.(2012)Herbicide-Properties,SynthesisandControlofWeedsbook)。因此,进一步采用百草枯验证耐旱性的转基因水稻。百草枯试验方法:每个构建体的水稻选择8-10个转基因株系用于百草枯试验,组培中花11(ZH11-TC)和空载体转化植株DP0158用作对照。T2代种子参照实施例4描述的方法消毒和萌发。百草枯试验在温度28-30℃,湿度30%的生长室中进行。萌发的种子放置在底部有孔的离心管中,采用水稻水培方法,培养5天,至一叶一心期;然后选择高度大约3.5~4cm的一致的幼苗用于百草枯试验。本实验采用随机区组设计,在同一个筛选水槽内设置5个区组;区组内包含所有测试的8-10个转基因株系、ZH11-TC和DP0158;区组的行列为16*12,每一行为一份测试材料,故每个转基因株系在区组内各12株,对照ZH11-TC和DP0158在区组内各3行;区组内的所有转基因株系和对照均随机排布。幼苗用终浓度为0.8μM的百草枯溶液进行处理7天,光周期为10h黑暗/14h光照,每两天更换一次溶液,在处理和更换溶液后,保证幼苗首先进入光周期的黑暗时期。处理7天后,计算绿色的幼苗。绿色没有损伤的幼苗为百草枯耐性幼苗;叶片、茎部变白褪色的幼苗为非百草枯耐性的幼苗。耐性率是百草枯试验的一个指标,指保持绿色并显示百草枯耐性表型的幼苗数除以总幼苗数的百分数。试验数据在构建体水平(所有的转基因幼苗与对照幼苗相比)和转基因株系水平(不同的转基因株系与对照相比)进行分析,采用的统计模型为“Y~seg+line(seg)+rep+error”,随机效应为“rep”,统计方法是“PROCGLIMMIX”。百草枯试验结果:1)OsGSTU41转基因水稻(DP0043)的百草枯验证结果第一次试验中,0.8μM百草枯溶液处理7天后,540株OsGSTU41转基因幼苗中,231株保持绿色并显示出百草枯耐性表型;而240株ZH11-TC幼苗中,48株具有百草枯耐性表型;180株DP0158幼苗中,55株显示百草枯耐性表型。OsGSTU41转基因幼苗的百草枯耐性率为43%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为20%和31%。OsGSTU41转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0087)对照。0.8μM百草枯溶液处理后,与ZH11-TC和DP0158幼苗相比,OsGSTU41转基因幼苗生长的更好,这些结果表明OsGSTU41转基因幼苗在构建体水平显示出高于ZH11-TC和DP0158幼苗的百草枯耐性。表29为进一步在转基因株系水平的分析,表明8个OsGSTU41转基因株系的百草枯耐性率高于ZH11-TC对照,且其中5个株系的百草枯耐性率显著高于ZH11-TC对照;5个株系的百草枯耐性率高于DP0158对照,其中4个株系的百草枯耐性率显著高于DP0158对照。这些结果表明,过量表达OsGSTU41基因提高了转基因植物百草枯耐性或抗氧化能力。表29.OsGSTU41转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,0.8μM百草枯溶液处理7天后,600株OsGSTU41转基因幼苗中,372株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,86株具有百草枯耐性表型;180株DP0158幼苗中,73株显示百草枯耐性表型。OsGSTU41转基因幼苗的百草枯耐性率为62%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为48%和41%。OsGSTU41转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0008)和DP0158(P值=0.0000)对照。转基因株系水平的分析,表明9个OsGSTU41转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,且其中5个株系的百草枯耐性率显著高于ZH11-TC对照,8个株系的百草枯耐性率显著高于DP0158对照(表30)。这些结果进一步表明,OsGSTU41转基因水稻增强了百草枯耐性,过量表达OsGSTU41基因提高了转基因植物百草枯耐性或抗氧化能力。如实施例4和5所述,过量表达OsGSTU41基因提高了水稻植株在苗期和成熟期的耐旱性,这些交叉验证进一步证明OsGSTU41基因在提高植物抗氧化能力并进一步提高耐旱性中发挥作用。表30.OsGSTU41转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)2)OsPPCK4转基因水稻(DP0058)的百草枯验证结果第一次试验中,0.8μM百草枯溶液处理7天后,600株OsPPCK4转基因幼苗中,380株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,76株具有百草枯耐性表型;180株DP0158幼苗中,65株显示百草枯耐性表型。OsPPCK4转基因幼苗的百草枯耐性率为63%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为42%和36%。OsPPCK4转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。与ZH11-TC和DP0158幼苗相比,OsPPCK4转基因幼苗生长的更好,这些结果表明OsPPCK4转基因幼苗在构建体水平显示出高于ZH11-TC和DP0158幼苗的百草枯耐性。表31为进一步在转基因株系水平的分析,9个OsPPCK4转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中6个株系的百草枯耐性率显著高于ZH11-TC和DP0158对照,这些结果表明,与ZH11-TC和DP0158两个对照相比,OsPPCK4转基因水稻在构建体水平和转基因株系水平均提高了幼苗的百草枯耐性。表31.OsPPCK4转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,0.8μM百草枯溶液处理7天后,540株OsPPCK4转基因幼苗中,265株保持绿色并显示出百草枯耐性表型;而240株ZH11-TC幼苗中,88株具有百草枯耐性表型;180株DP0158幼苗中,75株显示百草枯耐性表型。OsPPCK4转基因幼苗的百草枯耐性率为49%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为37%和42%。构建体水平上,OsPPCK4转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0030)和并高于DP0158(P值=0.1075)对照。这些结果表明OsPPCK4转基因幼苗在构建体水平显示增强的百草枯耐性。转基因株系水平的分析表明5个OsPPCK4转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照(表32),其中5个株系的百草枯耐性率显著高于ZH11-TC,和4个株系的百草枯耐性率显著高于DP0158对照。这些结果表明,与ZH11-TC和DP0158两个对照相比,OsPPCK4转基因水稻在构建体水平和转基因株系水平均提高了幼苗的百草枯耐性。表32.OsPPCK4转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)第三次试验中,0.8μM百草枯溶液处理7天后,540株OsPPCK4转基因幼苗中,290株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,77株具有百草枯耐性表型;180株DP0158幼苗中,89株显示百草枯耐性表型。OsPPCK4转基因幼苗的百草枯耐性率为54%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为43%和49%。构建体水平上,OsPPCK4转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0086)和并高于DP0158(P值=0.2236)对照。转基因株系水平的分析表明6个OsPPCK4转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照(表33),其中4个株系的百草枯耐性率显著高于ZH11-TC,和2个株系的百草枯耐性率显著高于DP0158对照。这些结果表明,与ZH11-TC和DP0158两个对照相比,OsPPCK4转基因水稻在构建体水平和转基因株系水平均提高了幼苗的百草枯耐性,过量表达OsPPCK4基因增强了转基因植物的百草枯耐性或抗氧化能力。表33.OsPPCK4转基因水稻的百草枯耐性试验(第三次试验,转基因株系水平)如实施例4或5所示,过量表达OsPPCK4基因提高了转基因水稻的耐旱性,三个不同试验的交叉验证证明OsPPCK4基因提高了植物的耐旱性。3)OsCAM2转基因水稻(DP0059)的百草枯验证结果第一次试验中,百草枯溶液处理后,480株OsCAM2转基因幼苗中,341株保持绿色并显示出百草枯耐性表型;而300株ZH11-TC幼苗中,165株具有百草枯耐性表型;180株DP0158幼苗中,71株显示百草枯耐性表型。OsCAM2转基因幼苗的百草枯耐性率为71%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为55%和39%。构建体水平上,OsCAM2转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。百草枯溶液处理后,与ZH11-TC和DP0158幼苗相比,OsCAM2转基因幼苗生长的更好,这些结果表明OsCAM2转基因幼苗在构建体水平显示出高于ZH11-TC和DP0158幼苗的百草枯耐性。表34为转基因株系水平的分析,8个OsCAM2转基因株系的百草枯耐性率高于ZH11-TC和DP0158幼苗,其中8个株系的百草枯耐性率显著高于DP0158对照,这些结果进一步表明,过量表达OsCAM2基因增强转基因植物在构建体水平和转基因株系水平的苗期百草枯耐性。表34.OsCAM2转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,百草枯溶液处理后,480株OsCAM2转基因幼苗中,287株保持绿色并显示出百草枯耐性表型;而300株ZH11-TC幼苗中,134株具有百草枯耐性表型;180株DP0158幼苗中,75株显示百草枯耐性表型。OsCAM2转基因幼苗的百草枯耐性率为60%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为45%和42%。构建体水平上,OsCAM2转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。表35为转基因株系水平的分析,5个OsCAM2转基因株系的百草枯耐性率高于ZH11-TC和DP0158幼苗,其中4个株系的百草枯耐性率显著高于ZH11-TC和DP0158对照,这些结果进一步表明,OsCAM2转基因水稻显示出较好的百草枯耐性和/或抗氧化能力,过量表达OsCAM2基因增强转基因植物在构建体水平和转基因株系水平的苗期百草枯耐性。如实施例4和5所述,过量表达OsCAM2基因提高了转基因植物的耐旱性,两个不同试验的交叉验证表明OsCAM2基因提高了植物的耐旱性。表35.OsCAM2转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)4)OsDN-DTP4转基因水稻(DP0167)的百草枯验证结果第一次试验中,所有测试的OsDN-DTP4转基因幼苗在构建体水平的分析表明,600株OsDN-DTP4转基因幼苗中,486株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,101株具有百草枯耐性表型;180株DP0158幼苗中,71株显示百草枯耐性表型。OsDN-DTP4转基因幼苗的百草枯耐性率为81%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为56%和39%。构建体水平上,所测试的OsDN-DTP4转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。这些结果表明0.8μM百草枯溶液处理后,与ZH11-TC或DP0158幼苗相比,OsDN-DTP4转基因幼苗生长的更好,并显示出增强的百草枯耐性。进一步在转基因株系水平的分析表明所有10个OsDN-DTP4转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中9个株系的百草枯耐性率显著高于ZH11-TC对照,10个株系的百草枯耐性率显著高于DP0158对照(表36)。这些结果表明,与ZH11-TC和DP0158两个对照相比,OsDN-DTP4转基因水稻在构建体水平和转基因株系水平均提高了幼苗的百草枯耐性。表36.OsDN-DTP4转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,百草枯溶液处理后,600株OsDN-DTP4转基因幼苗中,366株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,63株具有百草枯耐性表型;180株DP0158幼苗中,98株显示百草枯耐性表型。OsDN-DTP4转基因幼苗的百草枯耐性率为61%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为35%和54%。所测试的OsDN-DTP4转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0491)对照。这些结果表明,与ZH11-TC或DP0158幼苗相比,OsDN-DTP4转基因幼苗生长的更好,并显示出增强的百草枯耐性。在构建体水平显示出高于ZH11-TC和DP0158幼苗的百草枯耐性。转基因株系水平的分析表明8个OsDN-DTP4转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中8个株系的百草枯耐性率显著高于ZH11-TC对照,2个株系的百草枯耐性率显著高于DP0158对照(表37)。这些结果进一步表明,OsDN-DTP4转基因水稻提高了苗期百草枯耐性和/或抗氧化能力,过量表达OsDN-DTP4基因提高了转基因植物百草枯耐性或抗氧化能力。如实施例4所述,过量表达OsDN-DTP4基因提高了转基因植物的耐旱性,两个不同试验的交叉验证表明,过量表达OsDN-DTP4基因提高植物的耐旱性。表37.OsDN-DTP4转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)5)OsLecRK4.1转基因水稻(DP0173)的百草枯验证结果第一次试验中,百草枯溶液处理后,600株OsLecRK4.1转基因幼苗中,462株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,99株具有百草枯耐性表型;180株DP0158幼苗中,95株显示百草枯耐性表型。OsLecRK4.1转基因幼苗的百草枯耐性率为77%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为55%和53%。OsLecRK4.1转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。百草枯溶液处理后,与ZH11-TC或DP0158幼苗相比,OsLecRK4.1转基因水稻生长的较好,这些结果表明OsLecRK4.1转基因幼苗在构建体水平显示出高于ZH11-TC和DP0158幼苗的百草枯耐性。如表38所示,转基因株系水平,9个OsLecRK4.1转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中7个株系的百草枯耐性率显著高于ZH11-TC对照,8个株系的百草枯耐性率显著高于DP0158对照。这些结果进一步表明,过量表达OsLecRK4.1基因提高了转基因水稻的百草枯耐性或抗氧化能力。表38.OsLecRK4.1转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,百草枯溶液处理后,600株OsLecRK4.1转基因幼苗中,425株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,103株具有百草枯耐性表型;180株DP0158幼苗中,108株显示百草枯耐性表型。OsLecRK4.1转基因幼苗的百草枯耐性率为71%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为57%和60%。OsLecRK4.1转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0002)和DP0158(P值=0.0020)对照。转基因株系水平的分析表明9个OsLecRK4.1转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中,4个株系的百草枯耐性率显著高于ZH11-TC对照,3个株系的百草枯耐性率显著高于DP0158对照(表39)。这些结果进一步表明,过量表达OsLecRK4.1基因提高了转基因水稻的百草枯耐性或抗氧化能力。如实施例5所述,过量表达OsLecRK4.1基因提高了转基因水稻的耐旱性,两个不同试验的交叉验证表明OsLecRK4.1基因在提高植物耐旱性中发挥作用。表39.OsLecRK4.1转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)6)OsLecRK4.2转基因水稻(DP0209)的百草枯验证结果第一次试验中,将600株OsLecRK4.2转基因幼苗作为整体在构建体水平分析,600株OsLecRK4.2转基因幼苗中,221株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,30株具有百草枯耐性表型;180株DP0158幼苗中,29株显示百草枯耐性表型。OsLecRK4.2转基因幼苗的百草枯耐性率为37%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为17%和16%。构建体水平上,OsLecRK4.2转基因幼苗的百草枯耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0000)对照。这些结果表明OsLecRK4.2转基因幼苗在构建体水平显示出增强的百草枯耐性,0.8μM百草枯溶液处理后,与ZH11-TC和DP0158幼苗相比,OsLecRK4.2转基因幼苗生长的更好。转基因株系水平的分析表明9个OsLecRK4.2转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中8个株系的百草枯耐性率显著高于ZH11-TC和DP0158对照(表40)。这些结果表明,与ZH11-TC和DP0158两个对照相比,OsLecRK4.2转基因水稻在构建体水平和转基因株系水平均提高了幼苗的百草枯耐性,过量表达OsLecRK4.2基因提高了转基因植物百草枯耐性或抗氧化能力。表40.OsLecRK4.2转基因水稻的百草枯耐性试验(第一次试验,转基因株系水平)第二次试验中,600株OsLecRK4.2转基因幼苗中,351株保持绿色并显示出百草枯耐性表型;而180株ZH11-TC幼苗中,66株具有百草枯耐性表型;180株DP0158幼苗中,85株显示百草枯耐性表型。OsLecRK4.2转基因幼苗的百草枯耐性率为59%,ZH11-TC和DP0158幼苗的百草枯耐性率分别为37%和47%。构建体水平上,OsLecRK4.2转基因幼苗的耐性率显著高于ZH11-TC(P值=0.0000)和DP0158(P值=0.0080)对照。这些结果表明OsLecRK4.2转基因幼苗在构建体水平显示增强的百草枯耐性。转基因株系水平的分析表明8个OsLecRK4.2转基因株系的百草枯耐性率高于ZH11-TC和DP0158对照,其中8个株系的百草枯耐性率显著高于ZH11-TC对照,5个株系的百草枯耐性率显著高于DP0158对照(表41)。这些结果进一步表明,OsLecRK4.2转基因水稻提高了幼苗的百草枯耐性,过量表达OsLecRK4.2基因提高了转基因植物百草枯耐性或抗氧化能力。如实施例5所示,过量表达OsLecRK4.2基因提高了转基因水稻的耐旱性,两个不同试验的交叉验证证实OsLecRK4.2基因能够提高植物的耐旱性。表41.OsLecRK4.2转基因水稻的百草枯耐性试验(第二次试验,转基因株系水平)实施例7.水稻耐旱基因转化玉米并评估在玉米中的抗旱性将水稻耐旱性基因或者玉米、拟南芥或其它物种中的相应的同源基因转化玉米植株,并使基因过量表达,以组成型的启动子如玉米泛素启动子(Christensen等(1989)PlantMol.Biol.12:619-632和Christensen等(1992)PlantMol.Biol.18:675-689)或诸如胁迫响应启动子或组织偏爱启动子的其它启动子驱动玉米转化载体中基因的表达,采用微粒轰击法(国际专利公布WO2009/006276)将构建的重组DNA载体转化玉米细胞,也可以以采用农杆菌介导法(Zhao等,Meth.Mol.Biol.318:315-323(2006)和Zhao等,Mol.Breed.8:323-333(2001)和美国专利号5,981,840,1999年11月9日公布)转化玉米植株。农杆菌介导转化的过程包括细菌的接种、共培养、静止、筛选和植株的再生。再生植株的子代如T1代植株可以种在土壤中进行干旱胁迫,采用图像分析,测量干旱胁迫前和干旱胁迫过程中多个时期点植株面积、体积、生长速率和叶片颜色。与对照相比,明显延迟胁迫期间植株的萎蔫,叶面积的减小,黄色素积累的减少,和/或促进生长速率的增加被认为GLR基因提高玉米的耐旱性能。实施例8.转化玉米株系衍生的GaspeFlint并评价其在玉米中的功能如实施例7所述,可以转化玉米使其过表达水稻的GLR基因或者其它物种的同源基因。在某些情况下,具有短生活周期(快速循环)、小株型和高转化潜力的玉米(Tomes等,美国专利7,928,287)也可以成为植物受体细胞。以玉米胚作为受体,转化获得的转基因玉米(T0)群体,然后将转基因玉米按照优化的随机分组设计种植在条件可控的温室中减小或消除环境误差,比如每个重复30株包括24株转化获得植株和6株对照植株种植在盆中排成一排摆放在温室中的苗床上,每个对照或试验植株随机摆在不同设计的位置,一个实验的多个重复组种植在同一温室中,根据最小化空间需求量和环境效应来确定温室中重复组的排列,这样的排列可以被认为是压缩的温室排列。在研究过程中,转基因库中每一个植株都会被识别和追踪调查,植物上采集的数据自动与植株相关连,因此采集的数据可以与转化植株中基因相关联,比如每一个植物容器有可机读的标签(如通用产品码(UPC)条形码)包括植物身份信息,并与温室的位置相互关联,因此采集的数据自动与植物相关联。本研究中可以利用任何有效,机器可读的植物识别系统如二维矩阵码或射频识别标签(RFID)收到的数据,并用无线电频率接收器/数据处理器(美国专利7,403,855和7,702,462)翻译。对照和T0代温室植株用于目标农艺性状的分析,每个植株农艺数据会被记录或以一定的方式保存,并因此与植物的识别信息相关联。T1代植株采用前述类似的实验设计确认植株的表型(基因效应)。实施例9.水稻耐旱基因转化的拟南芥的实验室干旱验证为了验证水稻耐旱性基因是否提高双子叶植物的耐旱性或其它性状,采用农杆菌介导的花浸转化法将水稻耐旱性基因过表达载体转化拟南芥(Cloumbia),并鉴定转基因拟南芥(Clough,S.T.和Bent,A.F.(1998)ThePlantJournal16,735–743;Zhang,X.等(2006)NatureProtocols1:641-646)。一个称为pBC-yellow的16.8-kbT-DNA双元载体用于本试验。该载体包含一个RD29a启动子驱动ZS-Yellow基因表达,ZS-Yellow基因赋予转化种子黄色荧光。参照实施例1描述的方法克隆水稻耐旱基因,并构建gateway载体;随后采用INVITROGENTM的技术,一个LR重组反应在含有定向克隆的PCR产物的进入克隆和pBC-yellow载体之间进行,获得过表达载体。T2代种子用于实验室干旱试验,拟南芥的干旱试验是一种基于土壤的水分限制试验,在生长室中进行,光照强度为145μMol,温度22℃白天/20℃夜晚,湿度为60%。转基因种子采用COPASTM(复杂对象的参数分析和分选机、种子分选机,UnionBiometrica)分离,然后层积放置在0.1%的琼脂溶液中,4℃放置3天。野生型拟南芥用作对照,按照上述方法打破休眠。36株过表达拟南芥和36株野生型等距种植,并成“Z”字型排布。土壤的成分为3份泥煤苔、2份蛭石和1份珍珠岩,除此之外,肥料和杀真菌剂以下述的浓度添加在土壤中:NPK(氮磷钾)-1m/kg土壤、微量营养物-0.5g/kg土壤、杀真菌剂-0.5g/kg土壤。间苗后,每盆中有9株拟南芥,一个平盘中有72株拟南芥,前12天正常浇水,最后以1L去离子水使拟南芥的土壤饱和30min,多余的水分完全流出。种子萌发后28天到36天,采用成像仪使植株成像,并分析成像数据。种子种植后的第二天开始每天转动平盘直至成像的最后一天。成像系统产生的文件转变成XLS文件,并转换成Stan’s格式,发送给ESL产生每个试验株系的Stan’s分数。干旱条件下损害或萎蔫的速率用作测试参数,截止点分数=1.5。序列表<110>先锋海外公司<120>非生物胁迫条件下改良农艺性状的植物和方法<130>RTS14370S<150>PCT/CN2014/081601<151>2014-07-03<160>42<170>PatentInversion3.5<210>1<211>10952<212>DNA<213>合成序列<220><223>DP0005载体的核苷酸序列<400>1gaattctctagtcccgatctagtaacatagatgacaccgcgcgcgataatttatcctagt60ttgcgcgctatattttgttttctatcgcgtattaaatgtataattgcgggactctaatca120taaaaacccatctcataaataacgtcatgcattacatgttaattattacatgcttaacgt180aattcaacagaaattatatgataatcatcgcaagaccggcaacaggattcaatcttaaga240aacgcggccgcttcagttgtggcccagcttggaggtcgactcgcgaggatcctctagtcc300cgatctagtaacatagatgacaccgcgcgcgataatttatcctagtttgcgcgctatatt360ttgttttctatcgcgtattaaatgtataattgcgggactctaatcataaaaacccatctc420ataaataacgtcatgcattacatgttaattattacatgcttaacgtaattcaacagaaat480tatatgataatcatcgcaagaccggcaacaggattcaatcttaagaaacgcggccgcttc540agttgtggcccagcttggagggggcggcgtcgcagtagcggcccacggcggcctcgtact600gcttgtagcacttgcccttctccacctcctccaggatctcgatgcggtggtcctcgaagt660ggaagccgggcatcttcagggcggaggcgggcttcttggagcggtaggtggtgtgcaggt720ggcaggtcaggtggcgaccgccggggcactccagggccatcagggactggccgcgcagca780cgccgtccacctcgtacacgatctcggtggagggctcccagcggccggccttgttctgca840tcacggggccgtcggcggggaagttgttgcccaggatcttcaccttgtacaccaggcagt900cgccgtccagggaggtgtcctggtgggcggtcaggaagccgccgtcctcgtaggtggtgg960tgcgctcccaggtgaagccctcggggagggactgcttgaagtagtcggggatgccggaca1020cgtacttgatgaaggccttggagccgtacatgcaggaggtggacaggatgtggaaggcga1080agggcagggggccgccctcgatcacctcgatcttcatctcctgggtgccctccagggggt1140tgccctcgcccttgccggtgcacttgaagtagtggccgttcacggtgccctcgatggtgg1200tcctgaagggcatggtcttcttcagcaaagaggccatggtggcgaccggtaccagatctc1260tgcagagagatagatttgtagagagagactggtgatttcagcgtgtcctctccaaatgaa1320atgaacttccttatatagaggaagggtcttgcgaaggatagtgggattgtgcgtcatccc1380ttacgtcagtggagatatcacatcaatccacttgctttgaagacgtggttggaacgtctt1440ctttttccacgatgctcctcgtgggtgggggtccatctttgggaccactgtcggcagagg1500catcttgaacgatagcctttcctttatcgcaatgatggcatttgtaggtgccaccttcct1560tttctactgtccttttgatgaagtgacagatagctgggcaatggaatccgaggaggtttc1620ccgatattaccctttgttgaaaagtctcaatagccctttggtcttctgagactgtatctt1680tgatattcttggagtagacgagagtgtcgtgctccaccatgttcacatcaatccacttgc1740tttgaagacgtggttggaacgtcttctttttccacgatgctcctcgtgggtgggggtcca1800tctttgggaccactgtcggcagaggcatcttgaacgatagcctttcctttatcgcaatga1860tggcatttgtaggtgccaccttccttttctactgtccttttgatgaagtgacagatagct1920gggcaatggaatccgaggaggtttcccgatattaccctttgttgaaaagtctcaatagcc1980ctttggtcttctgagactgtatctttgatattcttggagtagacgagagtgtcgtgctcc2040accatgttgccaagctgctctaagcttggcactggccgtcgttttacaacgtcgtgactg2100ggaaaaccctggcgttacccaacttaatcgccttgcagcacatccccctttcgccagctg2160gcgtaatagcgaagaggcccgcaccgatcgcccttcccaacagttgcgcagcctgaatgg2220cgaatgctagagcagcttgagcttggatcagattgtcgttactatcagtgtttgacagga2280tatattggcgggtaaacctaagagaaaagagcgtttattagaataacggatatttaaaag2340ggcgtgaaaaggtttatccgttcgtccatttgtatgtgcatgccaaccacagggttcccc2400tcgggatcaaagtactttgatccaacccctccgctgctatagtgcagtcggcttctgacg2460ttcagtgcagccgtcttctgaaaacgacatgtcgcacaagtcctaagttacgcgacaggc2520tgccgccctgcccttttcctggcgttttcttgtcgcgtgttttagtcgcataaagtagaa2580tacttgcgactagaaccggagacattacgccatgaacaagagcgccgccgctggcctgct2640gggctatgcccgcgtcagcaccgacgaccaggacttgaccaaccaacgggccgaactgca2700cgcggccggctgcaccaagctgttttccgagaagatcaccggcaccaggcgcgaccgccc2760ggagctggccaggatgcttgaccacctacgccctggcgacgttgtgacagtgaccaggct2820agaccgcctggcccgcagcacccgcgacctactggacattgccgagcgcatccaggaggc2880cggcgcgggcctgcgtagcctggcagagccgtgggccgacaccaccacgccggccggccg2940catggtgttgaccgtgttcgccggcattgccgagttcgagcgttccctaatcatcgaccg3000cacccggagcgggcgcgaggccgccaaggcccgaggcgtgaagtttggcccccgccctac3060cctcaccccggcacagatcgcgcacgcccgcgagctgatcgaccaggaaggccgcaccgt3120gaaagaggcggctgcactgcttggcgtgcatcgctcgaccctgtaccgcgcacttgagcg3180cagcgaggaagtgacgcccaccgaggccaggcggcgcggtgccttccgtgaggacgcatt3240gaccgaggccgacgccctggcggccgccgagaatgaacgccaagaggaacaagcatgaaa3300ccgcaccaggacggccaggacgaaccgtttttcattaccgaagagatcgaggcggagatg3360atcgcggccgggtacgtgttcgagccgcccgcgcacgtctcaaccgtgcggctgcatgaa3420atcctggccggtttgtctgatgccaagctggcggcctggccggccagcttggccgctgaa3480gaaaccgagcgccgccgtctaaaaaggtgatgtgtatttgagtaaaacagcttgcgtcat3540gcggtcgctgcgtatatgatgcgatgagtaaataaacaaatacgcaaggggaacgcatga3600aggttatcgctgtacttaaccagaaaggcgggtcaggcaagacgaccatcgcaacccatc3660tagcccgcgccctgcaactcgccggggccgatgttctgttagtcgattccgatccccagg3720gcagtgcccgcgattgggcggccgtgcgggaagatcaaccgctaaccgttgtcggcatcg3780accgcccgacgattgaccgcgacgtgaaggccatcggccggcgcgacttcgtagtgatcg3840acggagcgccccaggcggcggacttggctgtgtccgcgatcaaggcagccgacttcgtgc3900tgattccggtgcagccaagcccttacgacatatgggccaccgccgacctggtggagctgg3960ttaagcagcgcattgaggtcacggatggaaggctacaagcggcctttgtcgtgtcgcggg4020cgatcaaaggcacgcgcatcggcggtgaggttgccgaggcgctggccgggtacgagctgc4080ccattcttgagtcccgtatcacgcagcgcgtgagctacccaggcactgccgccgccggca4140caaccgttcttgaatcagaacccgagggcgacgctgcccgcgaggtccaggcgctggccg4200ctgaaattaaatcaaaactcatttgagttaatgaggtaaagagaaaatgagcaaaagcac4260aaacacgctaagtgccggccgtccgagcgcacgcagcagcaaggctgcaacgttggccag4320cctggcagacacgccagccatgaagcgggtcaactttcagttgccggcggaggatcacac4380caagctgaagatgtacgcggtacgccaaggcaagaccattaccgagctgctatctgaata4440catcgcgcagctaccagagtaaatgagcaaatgaataaatgagtagatgaattttagcgg4500ctaaaggaggcggcatggaaaatcaagaacaaccaggcaccgacgccgtggaatgcccca4560tgtgtggaggaacgggcggttggccaggcgtaagcggctgggttgtctgccggccctgca4620atggcactggaacccccaagcccgaggaatcggcgtgacggtcgcaaaccatccggcccg4680gtacaaatcggcgcggcgctgggtgatgacctggtggagaagttgaaggccgcgcaggcc4740gcccagcggcaacgcatcgaggcagaagcacgccccggtgaatcgtggcaagcggccgct4800gatcgaatccgcaaagaatcccggcaaccgccggcagccggtgcgccgtcgattaggaag4860ccgcccaagggcgacgagcaaccagattttttcgttccgatgctctatgacgtgggcacc4920cgcgatagtcgcagcatcatggacgtggccgttttccgtctgtcgaagcgtgaccgacga4980gctggcgaggtgatccgctacgagcttccagacgggcacgtagaggtttccgcagggccg5040gccggcatggccagtgtgtgggattacgacctggtactgatggcggtttcccatctaacc5100gaatccatgaaccgataccgggaagggaagggagacaagcccggccgcgtgttccgtcca5160cacgttgcggacgtactcaagttctgccggcgagccgatggcggaaagcagaaagacgac5220ctggtagaaacctgcattcggttaaacaccacgcacgttgccatgcagcgtacgaagaag5280gccaagaacggccgcctggtgacggtatccgagggtgaagccttgattagccgctacaag5340atcgtaaagagcgaaaccgggcggccggagtacatcgagatcgagctagctgattggatg5400taccgcgagatcacagaaggcaagaacccggacgtgctgacggttcaccccgattacttt5460ttgatcgatcccggcatcggccgttttctctaccgcctggcacgccgcgccgcaggcaag5520gcagaagccagatggttgttcaagacgatctacgaacgcagtggcagcgccggagagttc5580aagaagttctgtttcaccgtgcgcaagctgatcgggtcaaatgacctgccggagtacgat5640ttgaaggaggaggcggggcaggctggcccgatcctagtcatgcgctaccgcaacctgatc5700gagggcgaagcatccgccggttcctaatgtacggagcagatgctagggcaaattgcccta5760gcaggggaaaaaggtcgaaaaggtctctttcctgtggatagcacgtacattgggaaccca5820aagccgtacattgggaaccggaacccgtacattgggaacccaaagccgtacattgggaac5880cggtcacacatgtaagtgactgatataaaagagaaaaaaggcgatttttccgcctaaaac5940tctttaaaacttattaaaactcttaaaacccgcctggcctgtgcataactgtctggccag6000cgcacagccgaagagctgcaaaaagcgcctacccttcggtcgctgcgctccctacgcccc6060gccgcttcgcgtcggcctatcgcggccgctggccgctcaaaaatggctggcctacggcca6120ggcaatctaccagggcgcggacaagccgcgccgtcgccactcgaccgccggcgcccacat6180caaggcaccctgcctcgcgcgtttcggtgatgacggtgaaaacctctgacacatgcagct6240cccggagacggtcacagcttgtctgtaagcggatgccgggagcagacaagcccgtcaggg6300cgcgtcagcgggtgttggcgggtgtcggggcgcagccatgacccagtcacgtagcgatag6360cggagtgtatactggcttaactatgcggcatcagagcagattgtactgagagtgcaccat6420atgcggtgtgaaataccgcacagatgcgtaaggagaaaataccgcatcaggcgctcttcc6480gcttcctcgctcactgactcgctgcgctcggtcgttcggctgcggcgagcggtatcagct6540cactcaaaggcggtaatacggttatccacagaatcaggggataacgcaggaaagaacatg6600tgagcaaaaggccagcaaaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttc6660cataggctccgcccccctgacgagcatcacaaaaatcgacgctcaagtcagaggtggcga6720aacccgacaggactataaagataccaggcgtttccccctggaagctccctcgtgcgctct6780cctgttccgaccctgccgcttaccggatacctgtccgcctttctcccttcgggaagcgtg6840gcgctttctcatagctcacgctgtaggtatctcagttcggtgtaggtcgttcgctccaag6900ctgggctgtgtgcacgaaccccccgttcagcccgaccgctgcgccttatccggtaactat6960cgtcttgagtccaacccggtaagacacgacttatcgccactggcagcagccactggtaac7020aggattagcagagcgaggtatgtaggcggtgctacagagttcttgaagtggtggcctaac7080tacggctacactagaaggacagtatttggtatctgcgctctgctgaagccagttaccttc7140ggaaaaagagttggtagctcttgatccggcaaacaaaccaccgctggtagcggtggtttt7200tttgtttgcaagcagcagattacgcgcagaaaaaaaggatctcaagaagatcctttgatc7260ttttctacggggtctgacgctcagtggaacgaaaactcacgttaagggattttggtcatg7320cattctaggtactaaaacaattcatccagtaaaatataatattttattttctcccaatca7380ggcttgatccccagtaagtcaaaaaatagctcgacatactgttcttccccgatatcctcc7440ctgatcgaccggacgcagaaggcaatgtcataccacttgtccgccctgccgcttctccca7500agatcaataaagccacttactttgccatctttcacaaagatgttgctgtctcccaggtcg7560ccgtgggaaaagacaagttcctcttcgggcttttccgtctttaaaaaatcatacagctcg7620cgcggatctttaaatggagtgtcttcttcccagttttcgcaatccacatcggccagatcg7680ttattcagtaagtaatccaattcggctaagcggctgtctaagctattcgtatagggacaa7740tccgatatgtcgatggagtgaaagagcctgatgcactccgcatacagctcgataatcttt7800tcagggctttgttcatcttcatactcttccgagcaaaggacgccatcggcctcactcatg7860agcagattgctccagccatcatgccgttcaaagtgcaggacctttggaacaggcagcttt7920ccttccagccatagcatcatgtccttttcccgttccacatcataggtggtccctttatac7980cggctgtccgtcatttttaaatataggttttcattttctcccaccagcttatatacctta8040gcaggagacattccttccgtatcttttacgcagcggtatttttcgatcagttttttcaat8100tccggtgatattctcattttagccatttattatttccttcctcttttctacagtatttaa8160agataccccaagaagctaattataacaagacgaactccaattcactgttccttgcattct8220aaaaccttaaataccagaaaacagctttttcaaagttgttttcaaagttggcgtataaca8280tagtatcgacggagccgattttgaaaccgcggtgatcacaggcagcaacgctctgtcatc8340gttacaatcaacatgctaccctccgcgagatcatccgtgtttcaaacccggcagcttagt8400tgccgttcttccgaatagcatcggtaacatgagcaaagtctgccgccttacaacggctct8460cccgctgacgccgtcccggactgatgggctgcctgtatcgagtggtgattttgtgccgag8520ctgccggtcggggagctgttggctggctggtggcaggatatattgtggtgtaaacaaatt8580gacgcttagacaacttaataacacattgcggacgtttttaatgtactgaattaacgccga8640attaattcgggggatctggattttagtactggattttggttttaggaattagaaatttta8700ttgatagaagtattttacaaatacaaatacatactaagggtttcttatatgctcaacaca8760tgagcgaaaccctataggaaccctaattcccttatctgggaactactcacacattattat8820ggagaaactcgagcttgtcgatcgacagatccggtcggcatctactctatttctttgccc8880tcggacgagtgctggggcgtcggtttccactatcggcgagtacttctacacagccatcgg8940tccagacggccgcgcttctgcgggcgatttgtgtacgcccgacagtcccggctccggatc9000ggacgattgcgtcgcatcgaccctgcgcccaagctgcatcatcgaaattgccgtcaacca9060agctctgatagagttggtcaagaccaatgcggagcatatacgcccggagtcgtggcgatc9120ctgcaagctccggatgcctccgctcgaagtagcgcgtctgctgctccatacaagccaacc9180acggcctccagaagaagatgttggcgacctcgtattgggaatccccgaacatcgcctcgc9240tccagtcaatgaccgctgttatgcggccattgtccgtcaggacattgttggagccgaaat9300ccgcgtgcacgaggtgccggacttcggggcagtcctcggcccaaagcatcagctcatcga9360gagcctgcgcgacggacgcactgacggtgtcgtccatcacagtttgccagtgatacacat9420ggggatcagcaatcgcgcatatgaaatcacgccatgtagtgtattgaccgattccttgcg9480gtccgaatgggccgaacccgctcgtctggctaagatcggccgcagcgatcgcatccatag9540cctccgcgaccggttgtagaacagcgggcagttcggtttcaggcaggtcttgcaacgtga9600caccctgtgcacggcgggagatgcaataggtcaggctctcgctaaactccccaatgtcaa9660gcacttccggaatcgggagcgcggccgatgcaaagtgccgataaacataacgatctttgt9720agaaaccatcggcgcagctatttacccgcaggacatatccacgccctcctacatcgaagc9780tgaaagcacgagattcttcgccctccgagagctgcatcaggtcggagacgctgtcgaact9840tttcgatcagaaacttctcgacagacgtcgcggtgagttcaggctttttcatatctcatt9900gccccccgggatctgcgaaagctcgagagagatagatttgtagagagagactggtgattt9960cagcgtgtcctctccaaatgaaatgaacttccttatatagaggaaggtcttgcgaaggat10020agtgggattgtgcgtcatcccttacgtcagtggagatatcacatcaatccacttgctttg10080aagacgtggttggaacgtcttctttttccacgatgctcctcgtgggtgggggtccatctt10140tgggaccactgtcggcagaggcatcttgaacgatagcctttcctttatcgcaatgatggc10200atttgtaggtgccaccttccttttctactgtccttttgatgaagtgacagatagctgggc10260aatggaatccgaggaggtttcccgatattaccctttgttgaaaagtctcaatagcccttt10320ggtcttctgagactgtatctttgatattcttggagtagacgagagtgtcgtgctccacca10380tgttatcacatcaatccacttgctttgaagacgtggttggaacgtcttctttttccacga10440tgctcctcgtgggtgggggtccatctttgggaccactgtcggcagaggcatcttgaacga10500tagcctttcctttatcgcaatgatggcatttgtaggtgccaccttccttttctactgtcc10560ttttgatgaagtgacagatagctgggcaatggaatccgaggaggtttcccgatattaccc10620tttgttgaaaagtctcaatagccctttggtcttctgagactgtatctttgatattcttgg10680agtagacgagagtgtcgtgctccaccatgttggcaagctgctctagccaatacgcaaacc10740gcctctccccgcgcgttggccgattcattaatgcagctggcacgacaggtttcccgactg10800gaaagcgggcagtgagcgcaacgcaattaatgtgagttagctcactcattaggcacccca10860ggctttacactttatgcttccggctcgtatgttgtgtggaattgtgagcggataacaatt10920tcacacaggaaacagctatgaccatgattacg10952<210>2<211>1921<212>DNA<213>合成序列<220><223>DsRed表达盒的核苷酸序列<400>2cgaagctggccgctctagaactagtggatctcgatgtgtagtctacgagaagggttaacc60gtctcttcgtgagaataaccgtggcctaaaaataagccgatgaggataaataaaatgtgg120tggtacagtacttcaagaggtttactcatcaagaggatgcttttccgatgagctctagta180gtacatcggacctcacatacctccattgtggtgaaatattttgtgctcatttagtgatgg240gtaaattttgtttatgtcactctaggttttgacatttcagttttgccactcttaggtttt300gacaaataatttccattccgcggcaaaagcaaaacaattttattttacttttaccactct360tagctttcacaatgtatcacaaatgccactctagaaattctgtttatgccacagaatgtg420aaaaaaaacactcacttatttgaagccaaggtgttcatggcatggaaatgtgacataaag480taacgttcgtgtataagaaaaaattgtactcctcgtaacaagagacggaaacatcatgag540acaatcgcgtttggaaggctttgcatcacctttggatgatgcgcatgaatggagtcgtct600gcttgctagccttcgcctaccgcccactgagtccgggcggcaactaccatcggcgaacga660cccagctgacctctaccgaccggacttgaatgcgctaccttcgtcagcgacgatggccgc720gtacgctggcgacgtgcccccgcatgcatggcggcacatggcgagctcagaccgtgcgtg780gctggctacaaatacgtaccccgtgagtgccctagctagaaacttacacctgcaactgcg840agagcgagcgtgtgagtgtagccgagtagatcctcgccaccatggcctcctccgagaacg900tcatcaccgagttcatgcgcttcaaggtgcgcatggagggcaccgtgaacggccacgagt960tcgagatcgagggcgagggcgagggccgcccctacgagggccacaacaccgtgaagctga1020aggtgacgaagggcggccccctgcccttcgcctgggacatcctgtccccccagttccagt1080acggctccaaggtgtacgtgaagcaccccgccgacatccccgactacaagaagctgtcct1140tccccgagggcttcaagtgggagcgcgtgatgaacttcgaggacggcggcgtggcgaccg1200tgacccaggactcctccctgcaggacggctgcttcatctacaaggtgaagttcatcggcg1260tgaacttcccctccgacggccccgtgatgcagaagaagaccatgggctgggaggcctcca1320ccgagcgcctgtacccccgcgacggcgtgctgaagggcgagacccacaaggccctgaagc1380tgaaggacggcggccactacctggtggagttcaagtccatctacatggccaagaagcccg1440tgcagctgcccggctactactacgtggacgccaagctggacatcacctcccacaacgagg1500actacaccatcgtggagcagtacgagcgcaccgagggccgccaccacctgttcctgtagc1560ggcccatggatattcgaacgcgtaggtaccacatggttaacctagacttgtccatcttct1620ggattggccaacttaattaatgtatgaaataaaaggatgcacacatagtgacatgctaat1680cactataatgtgggcatcaaagttgtgtgttatgtgtaattactagttatctgaataaaa1740gagaaagagatcatccatatttcttatcctaaatgaatgtcacgtgtctttataattctt1800tgatgaaccagatgcatttcattaaccaaatccatatacatataaatattaatcatatat1860aattaatatcaattgggttagcaaaacaaatctagtctaggtgtgttttgcgaatgcggc1920c1921<210>3<211>727<212>DNA<213>水稻<400>3aaatggttaagctaatcagcgccttcgggagcccattcgggcaccgcgccgaggcggcgc60tccggctgaaaggcgtgcagtacgagctcctcctggaagacctccgcagcaagagcgacc120tgctgctcgcccacaaccccgtccacaagctcgtccccgtcctcctccactccgacggcc180gctccgtcgccgagtccctcgtcgtcgtccagtacgtcgacgacgccttccatggcccgc240ccctcctccccgccgacccatacgctcgtgcccaggcccgtttctgggcccaattcatcg300atgataagttctcgaggccgttctggctgtcgttctggatggaggacggggagaagaagg360aggcgttcgtgagggaagcgaaggagaatctgcggccgctggaggcgcagctcgacggcg420gcaacaagaggttcttcggcggcgacgccattggcctcgtggacatcgccgccagtgggc480tggctcactgggtcggggtgttcgaggaggtcaccggcgtgagcttggtgagcgagcggg540agttccccgcgctgtgccggtggtcgcagcgctacgtcaacgatggagccgtgaggcagt600gcttgccgagcagagatgaactcgtcgccttgttcactgcaaacaaagaggcgtatacac660tgctggccaaggcaaagttgcagaaataattttattactcgtgaatttcgatgctggaat720atgcgta727<210>4<211>687<212>DNA<213>水稻<400>4atggttaagctaatcagcgccttcgggagcccattcgggcaccgcgccgaggcggcgctc60cggctgaaaggcgtgcagtacgagctcctcctggaagacctccgcagcaagagcgacctg120ctgctcgcccacaaccccgtccacaagctcgtccccgtcctcctccactccgacggccgc180tccgtcgccgagtccctcgtcgtcgtccagtacgtcgacgacgccttccatggcccgccc240ctcctccccgccgacccatacgctcgtgcccaggcccgtttctgggcccaattcatcgat300gataagttctcgaggccgttctggctgtcgttctggatggaggacggggagaagaaggag360gcgttcgtgagggaagcgaaggagaatctgcggccgctggaggcgcagctcgacggcggc420aacaagaggttcttcggcggcgacgccattggcctcgtggacatcgccgccagtgggctg480gctcactgggtcggggtgttcgaggaggtcaccggcgtgagcttggtgagcgagcgggag540ttccccgcgctgtgccggtggtcgcagcgctacgtcaacgatggagccgtgaggcagtgc600ttgccgagcagagatgaactcgtcgccttgttcactgcaaacaaagaggcgtatacactg660ctggccaaggcaaagttgcagaaataa687<210>5<211>228<212>PRT<213>水稻<400>5MetValLysLeuIleSerAlaPheGlySerProPheGlyHisArgAla151015GluAlaAlaLeuArgLeuLysGlyValGlnTyrGluLeuLeuLeuGlu202530AspLeuArgSerLysSerAspLeuLeuLeuAlaHisAsnProValHis354045LysLeuValProValLeuLeuHisSerAspGlyArgSerValAlaGlu505560SerLeuValValValGlnTyrValAspAspAlaPheHisGlyProPro65707580LeuLeuProAlaAspProTyrAlaArgAlaGlnAlaArgPheTrpAla859095GlnPheIleAspAspLysPheSerArgProPheTrpLeuSerPheTrp100105110MetGluAspGlyGluLysLysGluAlaPheValArgGluAlaLysGlu115120125AsnLeuArgProLeuGluAlaGlnLeuAspGlyGlyAsnLysArgPhe130135140PheGlyGlyAspAlaIleGlyLeuValAspIleAlaAlaSerGlyLeu145150155160AlaHisTrpValGlyValPheGluGluValThrGlyValSerLeuVal165170175SerGluArgGluPheProAlaLeuCysArgTrpSerGlnArgTyrVal180185190AsnAspGlyAlaValArgGlnCysLeuProSerArgAspGluLeuVal195200205AlaLeuPheThrAlaAsnLysGluAlaTyrThrLeuLeuAlaLysAla210215220LysLeuGlnLys225<210>6<211>760<212>DNA<213>水稻<400>6ggagaagagaatcgcggagatagcatgagcagcggcggcgagctcgcggagcaggagccc60aaggtgcacctcgtcgcgtcggcgccggggcccggggccagcccgcacgtcgtcgcgctc120cacgccgccttcgaggacgacgccgcggtgcacctcgtggtcgacctctgcgcgggcggg180gacctcctctccctcgtctcctcccgcggccgcctcccggagcacgaggccgcggacctc240gtggcgcaactggcctccgcgctcgcgtcctgccaccgccgcggggtagcgcaccgcgac300gtgaagcccgacaacctcctgttcgacggcggcggcgtgctcaagctcggcgacttcggg360tcggcggggtggttcggggacgggaggccgatgacggggctggtcgggacgccctactac420gtggcgccggaggtggtggccgggagggagtacggcgagaaggtggacgtgtggagcgcc480ggggtggtgctctacatgatgctctccgggaccctgcccttctacggcgccaccgccgcg540gaggtcttccagtgcgtgctccgcggcaacctccgcttcccgccgcgcgcgttcgcctcc600gtctcgccggaggccaaggacctgatgcgccgcatgctctgcaaggacgtctccagaagg660ttctccgccgaccaagtcctgaggcatccttggatcgtgagccgtgggggagccgcggtg720atgggctagttcgttcgatcgaggatgatgatgaactcca760<210>7<211>705<212>DNA<213>水稻<400>7atgagcagcggcggcgagctcgcggagcaggagcccaaggtgcacctcgtcgcgtcggcg60ccggggcccggggccagcccgcacgtcgtcgcgctccacgccgccttcgaggacgacgcc120gcggtgcacctcgtggtcgacctctgcgcgggcggggacctcctctccctcgtctcctcc180cgcggccgcctcccggagcacgaggccgcggacctcgtggcgcaactggcctccgcgctc240gcgtcctgccaccgccgcggggtagcgcaccgcgacgtgaagcccgacaacctcctgttc300gacggcggcggcgtgctcaagctcggcgacttcgggtcggcggggtggttcggggacggg360aggccgatgacggggctggtcgggacgccctactacgtggcgccggaggtggtggccggg420agggagtacggcgagaaggtggacgtgtggagcgccggggtggtgctctacatgatgctc480tccgggaccctgcccttctacggcgccaccgccgcggaggtcttccagtgcgtgctccgc540ggcaacctccgcttcccgccgcgcgcgttcgcctccgtctcgccggaggccaaggacctg600atgcgccgcatgctctgcaaggacgtctccagaaggttctccgccgaccaagtcctgagg660catccttggatcgtgagccgtgggggagccgcggtgatgggctag705<210>8<211>234<212>PRT<213>水稻<400>8MetSerSerGlyGlyGluLeuAlaGluGlnGluProLysValHisLeu151015ValAlaSerAlaProGlyProGlyAlaSerProHisValValAlaLeu202530HisAlaAlaPheGluAspAspAlaAlaValHisLeuValValAspLeu354045CysAlaGlyGlyAspLeuLeuSerLeuValSerSerArgGlyArgLeu505560ProGluHisGluAlaAlaAspLeuValAlaGlnLeuAlaSerAlaLeu65707580AlaSerCysHisArgArgGlyValAlaHisArgAspValLysProAsp859095AsnLeuLeuPheAspGlyGlyGlyValLeuLysLeuGlyAspPheGly100105110SerAlaGlyTrpPheGlyAspGlyArgProMetThrGlyLeuValGly115120125ThrProTyrTyrValAlaProGluValValAlaGlyArgGluTyrGly130135140GluLysValAspValTrpSerAlaGlyValValLeuTyrMetMetLeu145150155160SerGlyThrLeuProPheTyrGlyAlaThrAlaAlaGluValPheGln165170175CysValLeuArgGlyAsnLeuArgPheProProArgAlaPheAlaSer180185190ValSerProGluAlaLysAspLeuMetArgArgMetLeuCysLysAsp195200205ValSerArgArgPheSerAlaAspGlnValLeuArgHisProTrpIle210215220ValSerArgGlyGlyAlaAlaValMetGly225230<210>9<211>576<212>DNA<213>水稻<400>9ctcgcggaaccttctcgaagcttccacacccccaacctcgcctccaccaccaacccccca60tggcggaccagctcaccgacgagcagatcgccgagttcaaggaggcgttcagcctcttcg120acaaggacggcgacggttgcatcactactaaggagcttggaaccgtgatgcggtcccttg180gtcagaacccaactgaggcggagctgcaggacatgatcaacgaggttgatgctgatggca240atgggaccattgacttcccagagttcctgaacctgatggcgaagaagatgaaggataccg300actctgaggaggagctcaaggaggccttccgtgtgtttgacaaggaccagaacggtttca360tctcggctgctgagctccgccacgtcatgaccaaccttggtgagaagctgaccgacgagg420aagtcgacgagatgatccgtgaggctgacgtcgatggcgatggccagatcaactacgagg480agttcgttaaggtcatgatggccaagtgaggagggttcccattaaataagttctgtctga540agtgaactaaaactgtcagggcctacaacaaagctg576<210>10<211>450<212>DNA<213>水稻<400>10atggcggaccagctcaccgacgagcagatcgccgagttcaaggaggcgttcagcctcttc60gacaaggacggcgacggttgcatcactactaaggagcttggaaccgtgatgcggtccctt120ggtcagaacccaactgaggcggagctgcaggacatgatcaacgaggttgatgctgatggc180aatgggaccattgacttcccagagttcctgaacctgatggcgaagaagatgaaggatacc240gactctgaggaggagctcaaggaggccttccgtgtgtttgacaaggaccagaacggtttc300atctcggctgctgagctccgccacgtcatgaccaaccttggtgagaagctgaccgacgag360gaagtcgacgagatgatccgtgaggctgacgtcgatggcgatggccagatcaactacgag420gagttcgttaaggtcatgatggccaagtga450<210>11<211>149<212>PRT<213>水稻<400>11MetAlaAspGlnLeuThrAspGluGlnIleAlaGluPheLysGluAla151015PheSerLeuPheAspLysAspGlyAspGlyCysIleThrThrLysGlu202530LeuGlyThrValMetArgSerLeuGlyGlnAsnProThrGluAlaGlu354045LeuGlnAspMetIleAsnGluValAspAlaAspGlyAsnGlyThrIle505560AspPheProGluPheLeuAsnLeuMetAlaLysLysMetLysAspThr65707580AspSerGluGluGluLeuLysGluAlaPheArgValPheAspLysAsp859095GlnAsnGlyPheIleSerAlaAlaGluLeuArgHisValMetThrAsn100105110LeuGlyGluLysLeuThrAspGluGluValAspGluMetIleArgGlu115120125AlaAspValAspGlyAspGlyGlnIleAsnTyrGluGluPheValLys130135140ValMetMetAlaLys145<210>12<211>1184<212>DNA<213>水稻<400>12gcggcaaaaacgatgtcagtggctagacagcggccagcggggaggcagctggcggcgagg60ctaagtggtggcggcggccgctgacctcgccgatggcggagacactacggcgaatccgag120atggcggcgccatgctcctcctcgtcctcctcaccatcgcagcgcaggtgcagcaggagt180acgaggtcacctcctcctcgtcctccttgccatcgcggcgtggcgcgggcgagggccatc240agcagctgccttgggcgcagcaggagtacgaagtcacctctgccgccactgcggggccct300gcgatgcctacctcgtgttccgctcctccccaccgctctacgcctccgccgtctctatct360tcaacctcctcaacgtcaccgccacccccggcgacgaggtcgtccgcggaggagatggag420gagcaccaaaactggaagaagaacgcgctggtgctctatgacctcgtcatctcccagccg480ctcgagtggccatcgctcaccgtccagtggctcccctcccactcccggtcaccggactcc540accctctcctaccgcctcatcctcggaacccacacctccgacgagacgtgccaaccacct600cctgctcgccgacgccaccctcccgcttccaccctgcctggcggcggcagcctcggcggc660gagcggcgccgtcccgaccccgcttgtgtccatctcccgctcggtgccgcacaatggcga720ggtcaaccacgcccgctgcatgccgcagaggccgtacacggtggctaccaagacctgtgt780ggatgaggtgcatgtgtaccatcttggtgacggcggcgagaagagcgacgtcgatgtggt840gctcagggggcatgaagctgaggggtatgggctggcgccagttcagagtacggtgccgtt900ccaggtgagcatctccaccagccgatccaccatcgcagtcgacgtgccgacctgtcggag960cacggcctcgccgagccgcgggttcgccgcgacattgacgaagatgacgaagggagggag1020agagagagagatgaggaagggagagaagaggggaaagagggaaaggaggctgacgtggac1080acctgacatgtgggtcctatgctgactcagccgtcacgtagaataaaaccggagttaaaa1140ccaccgaaggatctcgggagaccggttttatatagttaagggac1184<210>13<211>813<212>DNA<213>水稻<400>13atggcggagacactacggcgaatccgagatggcggcgccatgctcctcctcgtcctcctc60accatcgcagcgcaggtgcagcaggagtacgaggtcacctcctcctcgtcctccttgcca120tcgcggcgtggcgcgggcgagggccatcagcagctgccttgggcgcagcaggagtacgaa180gtcacctctgccgccactgcggggccctgcgatgcctacctcgtgttccgctcctcccca240ccgctctacgcctccgccgtctctatcttcaacctcctcaacgtcaccgccacccccggc300gacgaggtcgtccgcggaggagatggaggagcaccaaaactggaagaagaacgcgctgcc360tcggcggcgagcggcgccgtcccgaccccgcttgtgtccatctcccgctcggtgccgcac420aatggcgaggtcaaccacgcccgctgcatgccgcagaggccgtacacggtggctaccaag480acctgtgtggatgaggtgcatgtgtaccatcttggtgacggcggcgagaagagcgacgtc540gatgtggtgctcagggggcatgaagctgaggggtatgggctggcgccagttcagagtacg600gtgccgttccaggtgagcatctccaccagccgatccaccatcgcagtcgacgtgccgacc660tgtcggagcacggcctcgccgagccgcgggttcgccgcgacattgacgaagatgacgaag720ggagggagagagagagagatgaggaagggagagaagaggggaaagagggaaaggaggctg780acgtggacacctgacatgtgggtcctatgctga813<210>14<211>270<212>PRT<213>水稻<400>14MetAlaGluThrLeuArgArgIleArgAspGlyGlyAlaMetLeuLeu151015LeuValLeuLeuThrIleAlaAlaGlnValGlnGlnGluTyrGluVal202530ThrSerSerSerSerSerLeuProSerArgArgGlyAlaGlyGluGly354045HisGlnGlnLeuProTrpAlaGlnGlnGluTyrGluValThrSerAla505560AlaThrAlaGlyProCysAspAlaTyrLeuValPheArgSerSerPro65707580ProLeuTyrAlaSerAlaValSerIlePheAsnLeuLeuAsnValThr859095AlaThrProGlyAspGluValValArgGlyGlyAspGlyGlyAlaPro100105110LysLeuGluGluGluArgAlaAlaSerAlaAlaSerGlyAlaValPro115120125ThrProLeuValSerIleSerArgSerValProHisAsnGlyGluVal130135140AsnHisAlaArgCysMetProGlnArgProTyrThrValAlaThrLys145150155160ThrCysValAspGluValHisValTyrHisLeuGlyAspGlyGlyGlu165170175LysSerAspValAspValValLeuArgGlyHisGluAlaGluGlyTyr180185190GlyLeuAlaProValGlnSerThrValProPheGlnValSerIleSer195200205ThrSerArgSerThrIleAlaValAspValProThrCysArgSerThr210215220AlaSerProSerArgGlyPheAlaAlaThrLeuThrLysMetThrLys225230235240GlyGlyArgGluArgGluMetArgLysGlyGluLysArgGlyLysArg245250255GluArgArgLeuThrTrpThrProAspMetTrpValLeuCys260265270<210>15<211>2346<212>DNA<213>水稻<400>15accggggccgtgacttgactgacacaatagaagcagcaaagcttgggcatttggtgaggc60actgattgaagctcagcactaggcgaagtgcagggcatggtgcttcccaaaccagaaatg120ccgttctttgtcctgctcctcttcctcggcctcggctgcctccgcccggcggcggcgacc180gacgagcggttcgtcttcaacgggttcaccggcgccaacctcagcttcgacggcatggca240acggtgacctcgaacgggctgctcatgctgaccaacggcacgaaccagctcaagggccac300gccttcttcccgtcgccgctccagttccagagggggcccaacagcacggcgatgcagtcc360ttctccacggccttcgtcatcggcatcatcggcgcgttcgaggacctgagcagccacggc420atggcgttcatcatcgccaagagcaagaacctcacctcggcgctgccggggcagttcatg480gggctcgtcaactccgccaacaacggcaacgcgaccaaccacctcttcgccgtggagttc540gacaccatcctcaactcggagttcaacgacatgagcggcaaccatgtcgggatcgacgtc600aacggcctcaactccgtcgacgccgacaacgccgggtactacgacgacggcaccggcgac660ttcaagaacatgagcctggtgagccgcaggccgatgcaggtgtgggtggacttcgacggc720cagaccatgcaggtcaatgtcaccatggcgccgctggaggtggcgcggccaaagaagccc780ctgctgtccaaaatcgtcaacatctcctccgtcattgatgacaccgcctacgtcggcttc840tcctcggcgaccggcatcctcttctgccgccactacgtgctcggctggagcttcaagatg900aacggcgccgcgccggcgctcaacatctcatccctgccctccctgccggtcacgttcccc960aagccgcggtccaagacgcttgagatcgtgctgccgatagcctcggcggtgctcgtcttc1020gcggtggccgccgccgtgttcgtgttcatgcggcggcggcgcatgttctcggagctcaag1080gaggagtgggaggtgacgttcgggcctcacaggttctcgtacaaggacctgttccacgcc1140accgacgggttcagcgacaagcggctcctcggcatcggcgggttcgggcgcgtgtaccgt1200ggcgtgctcccgtcgtccaaggcggaggtcgccgtgaagaaggtggcgcacgggtcgagg1260caggggatgagggagttcgtggcggaggtggtcagcatcggccggctccggcaccggaac1320ctcgtgcagctgctcggctactgccggcgcaagggcgagctcctgctggtgtacgactac1380atgcccaacggcagcctggacaagcagctgtacgaccagggcaagatcaccctgagatgg1440gcgcagaggttccgcatcatcaggggcgtcgcgtccgggctgctctacctccacgaggac1500tgggagcaggtggtggtgcacagggacatcaaggccagcaacgtgctgctcgacgccgac1560atgaacggccggctcggcgacttcggcctggcgcggctgtacgaccacggcaccgacccg1620cacacgacgcacgtggtgggcaccatgggctacctggcgccggagctggggcacaccggc1680aaggcgtccaaggcgtcggacgtgttcgcgttcggggccttcatgctggaggtggcgtgc1740gggcggaagcccgtggcgcaggacgcgcgcgacaaccgcgtcgtgctcgtcgactgggtg1800ctcgaccggtggcgcgccggggcgatcacggacacggtggacccgcgcctgcacggcgac1860ttcgtcgagagcgaggcgagcctcgtgctgcggctgggcctgctgtgctcgcacccgctg1920cccggcgcacggccggggacgaggcagcttgtgcagtacctggaaggcgacgtgccgctg1980ccggagctgtcgccgacgtaccagagcttcaacatgctggcgctcatgcaggaccagggg2040ttcgacccctacgtcatgtcctacccgatgacatccaccagcgcaggcaccttctctgac2100ctctctggaggtagatgatgacccgttcccatcagccggatgtagttaatcagtacctaa2160ctagtatcatcctgttttgtttaagatcagaactttggtgttgttatgtttatatctatg2220atgtaaagtggtgttacgagaatagctcagtgttcagataagagctgagaaataacagat2280ctgaaattaaacatttcgggctttacctcccactataaattttctcctctgatctgattg2340tcgacg2346<210>16<211>2022<212>DNA<213>水稻<400>16atggtgcttcccaaaccagaaatgccgttctttgtcctgctcctcttcctcggcctcggc60tgcctccgcccggcggcggcgaccgacgagcggttcgtcttcaacgggttcaccggcgcc120aacctcagcttcgacggcatggcaacggtgacctcgaacgggctgctcatgctgaccaac180ggcacgaaccagctcaagggccacgccttcttcccgtcgccgctccagttccagaggggg240cccaacagcacggcgatgcagtccttctccacggccttcgtcatcggcatcatcggcgcg300ttcgaggacctgagcagccacggcatggcgttcatcatcgccaagagcaagaacctcacc360tcggcgctgccggggcagttcatggggctcgtcaactccgccaacaacggcaacgcgacc420aaccacctcttcgccgtggagttcgacaccatcctcaactcggagttcaacgacatgagc480ggcaaccatgtcgggatcgacgtcaacggcctcaactccgtcgacgccgacaacgccggg540tactacgacgacggcaccggcgacttcaagaacatgagcctggtgagccgcaggccgatg600caggtgtgggtggacttcgacggccagaccatgcaggtcaatgtcaccatggcgccgctg660gaggtggcgcggccaaagaagcccctgctgtccaaaatcgtcaacatctcctccgtcatt720gatgacaccgcctacgtcggcttctcctcggcgaccggcatcctcttctgccgccactac780gtgctcggctggagcttcaagatgaacggcgccgcgccggcgctcaacatctcatccctg840ccctccctgccggtcacgttccccaagccgcggtccaagacgcttgagatcgtgctgccg900atagcctcggcggtgctcgtcttcgcggtggccgccgccgtgttcgtgttcatgcggcgg960cggcgcatgttctcggagctcaaggaggagtgggaggtgacgttcgggcctcacaggttc1020tcgtacaaggacctgttccacgccaccgacgggttcagcgacaagcggctcctcggcatc1080ggcgggttcgggcgcgtgtaccgtggcgtgctcccgtcgtccaaggcggaggtcgccgtg1140aagaaggtggcgcacgggtcgaggcaggggatgagggagttcgtggcggaggtggtcagc1200atcggccggctccggcaccggaacctcgtgcagctgctcggctactgccggcgcaagggc1260gagctcctgctggtgtacgactacatgcccaacggcagcctggacaagcagctgtacgac1320cagggcaagatcaccctgagatgggcgcagaggttccgcatcatcaggggcgtcgcgtcc1380gggctgctctacctccacgaggactgggagcaggtggtggtgcacagggacatcaaggcc1440agcaacgtgctgctcgacgccgacatgaacggccggctcggcgacttcggcctggcgcgg1500ctgtacgaccacggcaccgacccgcacacgacgcacgtggtgggcaccatgggctacctg1560gcgccggagctggggcacaccggcaaggcgtccaaggcgtcggacgtgttcgcgttcggg1620gccttcatgctggaggtggcgtgcgggcggaagcccgtggcgcaggacgcgcgcgacaac1680cgcgtcgtgctcgtcgactgggtgctcgaccggtggcgcgccggggcgatcacggacacg1740gtggacccgcgcctgcacggcgacttcgtcgagagcgaggcgagcctcgtgctgcggctg1800ggcctgctgtgctcgcacccgctgcccggcgcacggccggggacgaggcagcttgtgcag1860tacctggaaggcgacgtgccgctgccggagctgtcgccgacgtaccagagcttcaacatg1920ctggcgctcatgcaggaccaggggttcgacccctacgtcatgtcctacccgatgacatcc1980accagcgcaggcaccttctctgacctctctggaggtagatga2022<210>17<211>673<212>PRT<213>水稻<400>17MetValLeuProLysProGluMetProPhePheValLeuLeuLeuPhe151015LeuGlyLeuGlyCysLeuArgProAlaAlaAlaThrAspGluArgPhe202530ValPheAsnGlyPheThrGlyAlaAsnLeuSerPheAspGlyMetAla354045ThrValThrSerAsnGlyLeuLeuMetLeuThrAsnGlyThrAsnGln505560LeuLysGlyHisAlaPhePheProSerProLeuGlnPheGlnArgGly65707580ProAsnSerThrAlaMetGlnSerPheSerThrAlaPheValIleGly859095IleIleGlyAlaPheGluAspLeuSerSerHisGlyMetAlaPheIle100105110IleAlaLysSerLysAsnLeuThrSerAlaLeuProGlyGlnPheMet115120125GlyLeuValAsnSerAlaAsnAsnGlyAsnAlaThrAsnHisLeuPhe130135140AlaValGluPheAspThrIleLeuAsnSerGluPheAsnAspMetSer145150155160GlyAsnHisValGlyIleAspValAsnGlyLeuAsnSerValAspAla165170175AspAsnAlaGlyTyrTyrAspAspGlyThrGlyAspPheLysAsnMet180185190SerLeuValSerArgArgProMetGlnValTrpValAspPheAspGly195200205GlnThrMetGlnValAsnValThrMetAlaProLeuGluValAlaArg210215220ProLysLysProLeuLeuSerLysIleValAsnIleSerSerValIle225230235240AspAspThrAlaTyrValGlyPheSerSerAlaThrGlyIleLeuPhe245250255CysArgHisTyrValLeuGlyTrpSerPheLysMetAsnGlyAlaAla260265270ProAlaLeuAsnIleSerSerLeuProSerLeuProValThrPhePro275280285LysProArgSerLysThrLeuGluIleValLeuProIleAlaSerAla290295300ValLeuValPheAlaValAlaAlaAlaValPheValPheMetArgArg305310315320ArgArgMetPheSerGluLeuLysGluGluTrpGluValThrPheGly325330335ProHisArgPheSerTyrLysAspLeuPheHisAlaThrAspGlyPhe340345350SerAspLysArgLeuLeuGlyIleGlyGlyPheGlyArgValTyrArg355360365GlyValLeuProSerSerLysAlaGluValAlaValLysLysValAla370375380HisGlySerArgGlnGlyMetArgGluPheValAlaGluValValSer385390395400IleGlyArgLeuArgHisArgAsnLeuValGlnLeuLeuGlyTyrCys405410415ArgArgLysGlyGluLeuLeuLeuValTyrAspTyrMetProAsnGly420425430SerLeuAspLysGlnLeuTyrAspGlnGlyLysIleThrLeuArgTrp435440445AlaGlnArgPheArgIleIleArgGlyValAlaSerGlyLeuLeuTyr450455460LeuHisGluAspTrpGluGlnValValValHisArgAspIleLysAla465470475480SerAsnValLeuLeuAspAlaAspMetAsnGlyArgLeuGlyAspPhe485490495GlyLeuAlaArgLeuTyrAspHisGlyThrAspProHisThrThrHis500505510ValValGlyThrMetGlyTyrLeuAlaProGluLeuGlyHisThrGly515520525LysAlaSerLysAlaSerAspValPheAlaPheGlyAlaPheMetLeu530535540GluValAlaCysGlyArgLysProValAlaGlnAspAlaArgAspAsn545550555560ArgValValLeuValAspTrpValLeuAspArgTrpArgAlaGlyAla565570575IleThrAspThrValAspProArgLeuHisGlyAspPheValGluSer580585590GluAlaSerLeuValLeuArgLeuGlyLeuLeuCysSerHisProLeu595600605ProGlyAlaArgProGlyThrArgGlnLeuValGlnTyrLeuGluGly610615620AspValProLeuProGluLeuSerProThrTyrGlnSerPheAsnMet625630635640LeuAlaLeuMetGlnAspGlnGlyPheAspProTyrValMetSerTyr645650655ProMetThrSerThrSerAlaGlyThrPheSerAspLeuSerGlyGly660665670Arg<210>18<211>2464<212>DNA<213>水稻<400>18gtagcgaggagtgtgaacgatgtgatgccactgccagtatgccacacacaaccagtaaac60ctgggataatggttggtgtccggtgttcatgcattgactcgaatagcgagtcttctgttc120agacatttgatgctatgactcgaatagcggtgactacacatcatatgccctgtgctaatt180gcaactcccaaacggaagcaaatctcccgatgggtaatcaccgcttgctgctgctcctcc240tcctcctgctcgcggtcgttggctctgatcatgggggcgtgctcgccgccgacgagttca300cctacaacggcttcggcggtgccaacctcacgctcgacggcatggccgccgtggcgccca360acggcctcctcgtgctcagcaacggcacgaaccagatggccgggcacgcgttccacccga420cgccgatccgcctgcggggcggcgcggcgggcggcgccgtccagtccttctcggccgcgt480tcgtcttcgccatcgtgtccaacttcaccgtgctgagcgacaacggcatggcgttcgtgg540tcgcgcccagcacgcggctctccaccttcaacgccggccagtacctcggcatcctcaacg600tcaccgacaacggcaacgccgataacaacatcttcgccgtcgagctcgacaccatgctca660acccggagttccaggacatgaacagcaaccacatcggcgtcgacatcaacagcatgaagt720ccgtgcagaaccacagcgccggctactacgacgaagccacgggggccttcaacaatctga780gcttgatcagccgccagccgatgcaggtgtgggtggactacgacggcgccaccacggtgc840tcaacgtgacgatggcgccgctcgacgttcccaagcccagtaagcccctcatctccgcgc900ccgtcaacctctcgtccgtcgtgaccgacacggcgtacgtcgggttctcggcggccacgg960gcgtcatctacacgcggcactacgttctcggctggagcttctcccagaacggcgccgctc1020cttctctccacacctcaagcctcccggcgctgccgcggttcgggccgaagccccgttcca1080aggtgctggagatcgtgctcccgatcgccaccgcggcgttcgtcctcgcgctggtcatcg1140ccgccttcctgttcgtccggaggcgggtgaggtacgccgaggtgcgggaggactgggagg1200tggagttcggaccgcaccgcttctcctacaaggagctctaccaggcgacaaaggggttca1260agaacaagcagctgctcggcaccggcggattcggcagggtgtacaagggcgtgctcgcga1320aatccaacctcgagatcgccgtgaagagggtgtcgcacgactcgaagcaagggatgaagg1380agttcatcgcggaggtcgtcagcatcggccacctccggcaccgcaacctcgtgcagctgc1440tcggctactgccggcgcaagggcgagctcctgctggtgtacgactacatgtccaacggca1500gcctcgacaagtacttgtacgacaagaccaagcctgttcttgattgggggcagaggtttc1560agatcatcaagggcgtcgcctccggcctgctctacctccacgaggactgggagcaggtcg1620tcatccaccgggacatcaaggcgagcaacgtgctcctcgacggcgagatgaacggcaggc1680tgggcgacttcggcctcgcgaggctgtacgaccacggcgttgacccgcagacgacgcacg1740tcgtcggcaccatgggttacctcgccccggagctggtgcgcacgggcaaggcgacgccgg1800tcaccgacgtgttcgcgttcggcgtgttcgtgctggaggtcacctgcgggcggcggccgc1860tcggctgcatcgcgcccgacgaccagaacgtgctgctggactgggtgcaggagcacgagc1920gccggcacgcggccctcgacacggtggacgcgaggctgtgcggcaagtacgacgccgacg1980aggcgaggctggcgctcaagctggggctcatgtgcgcgcacccgttgcccgacgcgcgcc2040ccaccatgcgccaggtcacgcagtacctggacggcgacgcacccatgccggaggtggcgc2100cgacgatggtgagctacaccatgctggcgctgatgcagaacgacgggttcgactcgttcg2160ccatgtccttcccttccaccgtcacgtcgaccgccagccccatgtccgccgacgtctcgg2220ccgtgtccggcctctccggtggaaggtgaacaaacaaacaaaaaggccgccgcaaaataa2280tggtcacaacaacccgttctcctggtgacttgtcatctatcgtgtaacatagtgtagagc2340actagtgactacgtagtagtgaggtcactgttcactgctgattaccacagtatatcccca2400tattagtttaccaatcaagcagtaatacgtatattacagtgagtgtgcaaagcttcgaga2460aggc2464<210>19<211>2211<212>DNA<213>水稻<400>19atgccacacacaaccagtaaacctgggataatggttggtgtccggtgttcatgcattgac60tcgaatagcgagtcttctgttcagacatttgatgctatgactcgaatagcggtgactaca120catcatatgccctgtgctaattgcaactcccaaacggaagcaaatctcccgatgggtaat180caccgcttgctgctgctcctcctcctcctgctcgcggtcgttggctctgatcatgggggc240gtgctcgccgccgacgagttcacctacaacggcttcggcggtgccaacctcacgctcgac300ggcatggccgccgtggcgcccaacggcctcctcgtgctcagcaacggcacgaaccagatg360gccgggcacgcgttccacccgacgccgatccgcctgcggggcggcgcggcgggcggcgcc420gtccagtccttctcggccgcgttcgtcttcgccatcgtgtccaacttcaccgtgctgagc480gacaacggcatggcgttcgtggtcgcgcccagcacgcggctctccaccttcaacgccggc540cagtacctcggcatcctcaacgtcaccgacaacggcaacgccgataacaacatcttcgcc600gtcgagctcgacaccatgctcaacccggagttccaggacatgaacagcaaccacatcggc660gtcgacatcaacagcatgaagtccgtgcagaaccacagcgccggctactacgacgaagcc720acgggggccttcaacaatctgagcttgatcagccgccagccgatgcaggtgtgggtggac780tacgacggcgccaccacggtgctcaacgtgacgatggcgccgctcgacgttcccaagccc840agtaagcccctcatctccgcgcccgtcaacctctcgtccgtcgtgaccgacacggcgtac900gtcgggttctcggcggccacgggcgtcatctacacgcggcactacgttctcggctggagc960ttctcccagaacggcgccgctccttctctccacacctcaagcctcccggcgctgccgcgg1020ttcgggccgaagccccgttccaaggtgctggagatcgtgctcccgatcgccaccgcggcg1080ttcgtcctcgcgctggtcatcgccgccttcctgttcgtccggaggcgggtgaggtacgcc1140gaggtgcgggaggactgggaggtggagttcggaccgcaccgcttctcctacaaggagctc1200taccaggcgacaaaggggttcaagaacaagcagctgctcggcaccggcggattcggcagg1260gtgtacaagggcgtgctcgcgaaatccaacctcgagatcgccgtgaagagggtgtcgcac1320gactcgaagcaagggatgaaggagttcatcgcggaggtcgtcagcatcggccacctccgg1380caccgcaacctcgtgcagctgctcggctactgccggcgcaagggcgagctcctgctggtg1440tacgactacatgtccaacggcagcctcgacaagtacttgtacgacaagaccaagcctgtt1500cttgattgggggcagaggtttcagatcatcaagggcgtcgcctccggcctgctctacctc1560cacgaggactgggagcaggtcgtcatccaccgggacatcaaggcgagcaacgtgctcctc1620gacggcgagatgaacggcaggctgggcgacttcggcctcgcgaggctgtacgaccacggc1680gttgacccgcagacgacgcacgtcgtcggcaccatgggttacctcgccccggagctggtg1740cgcacgggcaaggcgacgccggtcaccgacgtgttcgcgttcggcgtgttcgtgctggag1800gtcacctgcgggcggcggccgctcggctgcatcgcgcccgacgaccagaacgtgctgctg1860gactgggtgcaggagcacgagcgccggcacgcggccctcgacacggtggacgcgaggctg1920tgcggcaagtacgacgccgacgaggcgaggctggcgctcaagctggggctcatgtgcgcg1980cacccgttgcccgacgcgcgccccaccatgcgccaggtcacgcagtacctggacggcgac2040gcacccatgccggaggtggcgccgacgatggtgagctacaccatgctggcgctgatgcag2100aacgacgggttcgactcgttcgccatgtccttcccttccaccgtcacgtcgaccgccagc2160cccatgtccgccgacgtctcggccgtgtccggcctctccggtggaaggtga2211<210>20<211>736<212>PRT<213>水稻<400>20MetProHisThrThrSerLysProGlyIleMetValGlyValArgCys151015SerCysIleAspSerAsnSerGluSerSerValGlnThrPheAspAla202530MetThrArgIleAlaValThrThrHisHisMetProCysAlaAsnCys354045AsnSerGlnThrGluAlaAsnLeuProMetGlyAsnHisArgLeuLeu505560LeuLeuLeuLeuLeuLeuLeuAlaValValGlySerAspHisGlyGly65707580ValLeuAlaAlaAspGluPheThrTyrAsnGlyPheGlyGlyAlaAsn859095LeuThrLeuAspGlyMetAlaAlaValAlaProAsnGlyLeuLeuVal100105110LeuSerAsnGlyThrAsnGlnMetAlaGlyHisAlaPheHisProThr115120125ProIleArgLeuArgGlyGlyAlaAlaGlyGlyAlaValGlnSerPhe130135140SerAlaAlaPheValPheAlaIleValSerAsnPheThrValLeuSer145150155160AspAsnGlyMetAlaPheValValAlaProSerThrArgLeuSerThr165170175PheAsnAlaGlyGlnTyrLeuGlyIleLeuAsnValThrAspAsnGly180185190AsnAlaAspAsnAsnIlePheAlaValGluLeuAspThrMetLeuAsn195200205ProGluPheGlnAspMetAsnSerAsnHisIleGlyValAspIleAsn210215220SerMetLysSerValGlnAsnHisSerAlaGlyTyrTyrAspGluAla225230235240ThrGlyAlaPheAsnAsnLeuSerLeuIleSerArgGlnProMetGln245250255ValTrpValAspTyrAspGlyAlaThrThrValLeuAsnValThrMet260265270AlaProLeuAspValProLysProSerLysProLeuIleSerAlaPro275280285ValAsnLeuSerSerValValThrAspThrAlaTyrValGlyPheSer290295300AlaAlaThrGlyValIleTyrThrArgHisTyrValLeuGlyTrpSer305310315320PheSerGlnAsnGlyAlaAlaProSerLeuHisThrSerSerLeuPro325330335AlaLeuProArgPheGlyProLysProArgSerLysValLeuGluIle340345350ValLeuProIleAlaThrAlaAlaPheValLeuAlaLeuValIleAla355360365AlaPheLeuPheValArgArgArgValArgTyrAlaGluValArgGlu370375380AspTrpGluValGluPheGlyProHisArgPheSerTyrLysGluLeu385390395400TyrGlnAlaThrLysGlyPheLysAsnLysGlnLeuLeuGlyThrGly405410415GlyPheGlyArgValTyrLysGlyValLeuAlaLysSerAsnLeuGlu420425430IleAlaValLysArgValSerHisAspSerLysGlnGlyMetLysGlu435440445PheIleAlaGluValValSerIleGlyHisLeuArgHisArgAsnLeu450455460ValGlnLeuLeuGlyTyrCysArgArgLysGlyGluLeuLeuLeuVal465470475480TyrAspTyrMetSerAsnGlySerLeuAspLysTyrLeuTyrAspLys485490495ThrLysProValLeuAspTrpGlyGlnArgPheGlnIleIleLysGly500505510ValAlaSerGlyLeuLeuTyrLeuHisGluAspTrpGluGlnValVal515520525IleHisArgAspIleLysAlaSerAsnValLeuLeuAspGlyGluMet530535540AsnGlyArgLeuGlyAspPheGlyLeuAlaArgLeuTyrAspHisGly545550555560ValAspProGlnThrThrHisValValGlyThrMetGlyTyrLeuAla565570575ProGluLeuValArgThrGlyLysAlaThrProValThrAspValPhe580585590AlaPheGlyValPheValLeuGluValThrCysGlyArgArgProLeu595600605GlyCysIleAlaProAspAspGlnAsnValLeuLeuAspTrpValGln610615620GluHisGluArgArgHisAlaAlaLeuAspThrValAspAlaArgLeu625630635640CysGlyLysTyrAspAlaAspGluAlaArgLeuAlaLeuLysLeuGly645650655LeuMetCysAlaHisProLeuProAspAlaArgProThrMetArgGln660665670ValThrGlnTyrLeuAspGlyAspAlaProMetProGluValAlaPro675680685ThrMetValSerTyrThrMetLeuAlaLeuMetGlnAsnAspGlyPhe690695700AspSerPheAlaMetSerPheProSerThrValThrSerThrAlaSer705710715720ProMetSerAlaAspValSerAlaValSerGlyLeuSerGlyGlyArg725730735<210>21<211>26<212>DNA<213>合成序列<220><223>克隆OsGSTU41基因cDNA的正向引物<400>21aaatggttaagctaatcagcgccttc26<210>22<211>27<212>DNA<213>合成序列<220><223>克隆OsGSTU41基因cDNA的反向引物<400>22tacgcatattccagcatcgaaattcac27<210>23<211>24<212>DNA<213>合成序列<220><223>克隆OsPPCK4基因cDNA的正向引物<400>23ggagaagagaatcgcggagatagc24<210>24<211>24<212>DNA<213>合成序列<220><223>克隆OsPPCK4基因cDNA的反向引物<400>24tggagttcatcatcatcctcgatc24<210>25<211>24<212>DNA<213>合成序列<220><223>克隆OsCAM2基因cDNA的正向引物<400>25ctcgcggaaccttctcgaagcttc24<210>26<211>22<212>DNA<213>合成序列<220><223>克隆OsCAM2基因cDNA的反向引物<400>26cagctttgttgtaggccctgac22<210>27<211>26<212>DNA<213>合成序列<220><223>克隆OsDN-DTP4基因gDNA的正向引物<400>27gcggcaaaaacgatgtcagtggctag26<210>28<211>29<212>DNA<213>合成序列<220><223>克隆OsDN-DTP4基因gDNA的反向引物<400>28gtcccttaactatataaaaccggtctccc29<210>29<211>23<212>DNA<213>合成序列<220><223>克隆OsLecRK4.1基因gDNA的正向引物<400>29accggggccgtgacttgactgac23<210>30<211>25<212>DNA<213>合成序列<220><223>克隆OsLecRK4.1基因gDNA的反向引物<400>30cgtcgacaatcagatcagaggagaa25<210>31<211>28<212>DNA<213>合成序列<220><223>克隆OsLecRK4.2基因gDNA的正向引物<400>31gtagcgaggagtgtgaacgatgtgatgc28<210>32<211>28<212>DNA<213>合成序列<220><223>克隆OsLecRK4.2基因gDNA的反向引物<400>32gccttctcgaagctttgcacactcactg28<210>33<211>18<212>DNA<213>合成序列<220><223>OsGSTU41基因实时PCR分析的正向引物<400>33ggctgtcgttctggatgg18<210>34<211>18<212>DNA<213>合成序列<220><223>OsGSTU41基因实时PCR分析的反向引物<400>34gcagtgaacaaggcgacg18<210>35<211>20<212>DNA<213>合成序列<220><223>OsPPCK4基因实时PCR分析的正向引物<400>35gctctacatgatgctctccg20<210>36<211>18<212>DNA<213>合成序列<220><223>OsPPCK4基因实时PCR分析的反向引物<400>36gagacgtccttgcagagc18<210>37<211>21<212>DNA<213>合成序列<220><223>OsDN-DTP4基因实时PCR分析的正向引物<400>37ccagttcagagtacggtgccg21<210>38<211>22<212>DNA<213>合成序列<220><223>OsDN-DTP4基因实时PCR分析的反向引物<400>38gtgtccacgtcagcctcctttc22<210>39<211>18<212>DNA<213>合成序列<220><223>OsLecRK4.1基因实时PCR分析的正向引物<400>39cgctcaacatctcatccc18<210>40<211>18<212>DNA<213>合成序列<220><223>OsLecRK4.1基因实时PCR分析的反向引物<400>40ccgcatgaacacgaacac18<210>41<211>18<212>DNA<213>合成序列<220><223>OsLecRK4.2基因实时PCR分析的正向引物<400>41ccgacgatggtgagctac18<210>42<211>18<212>DNA<213>合成序列<220><223>OsLecRK4.2基因实时PCR分析的反向引物<400>42gtgacggtggaagggaag18当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1