MIT生物标志物和使用它们的方法与流程
本申请要求临时的2014年5月23日提交的美国申请No.62/002,612;2014年10月3日提交的美国申请No.62/059,362;和2015年1月30日提交的美国申请No.62/109,775的优先权,据此通过援引将其完整收录。
序列表
本申请含有序列表,已经经EFS-Web提交且据此通过援引将其完整收录。2015年5月21日创建的所述ASCII拷贝命名为P05829-WO_SL.txt且大小为85,027个字节。
发明领域
提供的是涉及病理性状况(诸如癌症)的治疗的疗法。
发明背景
肾癌在美国每年占~60,000例新病例和~13,000例死亡(Siegel et al.,2013)。约85%的肾癌是肾细胞癌(RCC),其源自肾上皮。占75%RCC的透明细胞RCC(ccRCC)是表征最好的肾癌亚型(Pena-Llopis et al.,2012;Sato et al.,2013;TCGA,2013)。广泛分类为非透明细胞RCC(nccRCC)的剩余25%RCC代表独特的肿瘤亚型,包括乳头状(pRCC;10-15%)和嫌色(chRCC;4-5%)(Osunkoya,2010;Picken,2010;Young,2010;Yusenko,2010a,b)。在针芯活检中,chRCC有时难以与肾瘤细胞瘤(oncocytoma)(RO)区分,后者是发病率~5%的一种良性肾上皮肿瘤(Osunkoya,2010;Picken,2010;Young,2010;Yusenko,2010a,b)。它们的诊断仍然具有挑战性且因显示RO和chRCC二者特征的混合肿瘤的存在而变得复杂(Osunkoya,2010;Picken,2010;Young,2010;Yusenko,2010a,b)。其它nccRCC类型包括收集管(<1%),易位(tRCC;罕见)和髓质(罕见)。约4-5%的肿瘤仍然未分类(Bellmunt and Dutcher,2013)。虽然不频繁,但是tRCC趋于影响青少年和年轻成人且是特别破坏性的。一旦nccRCC转移,该疾病一般仍然是无法治愈的。虽然数种药物最近已经批准用于转移性RCC,但是登记试验几乎不牵涉具有ccRCC的患者,而且没有治疗证明nccRCC亚型中的功效(Bellmunt and Dutcher,2013)。
仍然需要更好的了解癌症(特别是人肾细胞癌)的发病机理,还有鉴定新的治疗靶。
发明概述
提供了用于测定MiT生物标志物表达(测定MiT生物标志物的存在)的方法,其包括测定来自个体的样品是否表达MiT生物标志物的步骤。在一些实施方案中,MiT是MITF。在一些实施方案中,MiT是TFEB。在一些实施方案中,MiT是TFEC。在一些实施方案中,MiT是TFE3。在一些实施方案中,MiT是SBNO2。
在本发明的任何方法的一些实施方案中,生物标志物的存在通过存在升高的生物标志物表达水平(例如与参照表达水平相比)来指示。在一些实施方案中,一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位或倒位(例如重排和/或融合)。在一些实施方案中,易位是MITF易位。在一些实施方案中,该MITF易位包含ACTG1和MITF。在一些实施方案中,该MITF易位包含ACTG1外显子3。在一些实施方案中,该MITF易位包含ACTG1外显子3和MITF外显子3。在一些实施方案中,MITF易位包含SEQ ID NO:13和/或30。在一些实施方案中,MITF易位包含SEQ ID NO:30。在一些实施方案中,该MITF易位是通过由SEQ ID NO:11和/或12组成或包含SEQ ID NO:11和/或12的引物可检测的。在一些实施方案中,该MITF易位是通过由SEQ ID NO:9,10,11和/或12组成或包含SEQ ID NO:9,10,11和/或12的引物可检测的。在一些实施方案中,该MITF易位是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位包含AP3S1和MITF。在一些实施方案中,该MITF易位包含AP3S1外显子3。在一些实施方案中,该MITF易位包含ACTG1外显子3和MITF外显子3。在一些实施方案中,其中该MITF易位是由AP3S1启动子驱动的。在一些实施方案中,该易位是TFEB易位。在一些实施方案中,该TFEB易位包含CLTC和TFEB。在一些实施方案中,该TFEB易位包含CLTC外显子17。在一些实施方案中,该TFEB易位包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位包含SEQ ID NO:19。在一些实施方案中,该TFEB易位是通过由SEQ ID NO:17和/或18组成或包含SEQ ID NO:17和/或18的引物可检测的。在一些实施方案中,该TFEB易位是通过由SEQ ID NO:15,16,17和/或18组成或包含SEQ ID NO:15,16,17和/或18的引物可检测的。在一些实施方案中,TFEB易位是由CLTC启动子驱动的。在一些实施方案中,该易位是SBNO2倒位。在一些实施方案中,SBNO2倒位包含MIDN和SBNO2。在一些实施方案中,SBNO2倒位包含MIDN启动子。在一些实施方案中,该SBNO2倒位包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2倒位包含SEQ ID NO:25。在一些实施方案中,该SBNO2倒位是通过由SEQ ID NO:23和/或24组成或包含SEQ ID NO:23和/或24的引物可检测的。在一些实施方案中,该SBNO2倒位是通过由SEQ ID NO:21,22,23,和/或25组成或包含SEQ ID NO:21,22,23,和/或25的引物可检测的。在一些实施方案中,SBNO2倒位是由CLTC启动子驱动的。
在本发明的任何方法的一些实施方案中,该MiT易位导致升高的MET表达水平(例如与没有该MiT易位的参照相比)。
在本发明的任何方法的一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的MET活性和/或活化(例如与没有该MiT易位的参照相比)。
在本发明的任何方法的一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的BIRC7表达水平(例如与没有该MiT易位的参照相比)。
在本发明的任何方法的一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的BIRC7活性和/或活化(例如与没有该MiT易位的参照相比)。
在本发明的任何方法的一些实施方案中,该易位是体细胞易位。
在本发明的任何方法的一些实施方案中,该易位是染色体内易位。
在本发明的任何方法的一些实施方案中,该易位是染色体间易位。
在本发明的任何方法的一些实施方案中,该易位是倒位
在本发明的任何方法的一些实施方案中,该易位是删除。
在本发明的任何方法的一些实施方案中,该易位是易位融合多核苷酸(例如功能性MiT易位融合多核苷酸)和/或功能性易位融合多肽(例如功能性MiT易位融合多肽)。
在本发明的任何方法的一些实施方案中,该样品是癌症样品。
在本发明的任何方法的一些实施方案中,该癌症是鳞状细胞癌(例如上皮鳞状细胞癌),肺癌包括小细胞肺癌(SCLC),非小细胞肺癌(NSCLC),肺的腺癌和肺的鳞癌,腹膜癌,肝细胞癌,胃的癌或胃癌包括胃肠癌,胰腺癌,成胶质细胞瘤,宫颈癌,卵巢癌,肝癌,膀胱癌,肝瘤,乳腺癌(包括转移性乳腺癌),结肠癌,直肠癌,结直肠癌,子宫内膜或子宫癌,唾液腺癌,肾癌或肾的癌,前列腺癌,阴门癌,甲状腺癌,肝的癌,肛门癌,阴茎癌,睾丸癌,食道癌,胆管肿瘤,以及头和颈癌。
在本发明的任何方法的一些实施方案中,该癌症是肾细胞癌(RCC)。在一些实施方案中,该RCC是非透明细胞肾细胞癌(nccRCC)或易位RCC(tRCC)。在一些实施方案中,该RCC是nccRCC。
提供的是治疗个体中的癌症的方法,其包括对该个体施用有效量的MiT拮抗剂,其中治疗是基于该个体具有包含MiT过表达的癌症。在一些实施方案中,该癌症包含MiT易位,该方法包括提供有效量的MiT拮抗剂。
本文中提供的是治疗个体中的癌症的方法,前提是该个体已经发现具有包含MiT易位的癌症,该方法包括对该个体施用有效量的MiT拮抗剂。
本文中提供的是治疗个体中的癌症的方法,该方法包括:测定自该个体获得的样品包含MiT易位,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该癌症得到治疗。
本文中提供的是治疗癌症的方法,其包括:(a)选择具有癌症的个体,其中该癌症包含MiT易位;并(b)对如此选择的个体施用有效量的MiT拮抗剂,由此该癌症得到治疗。
本文中提供的是鉴定更有可能或不太可能展现受益于用包含MiT拮抗剂的抗癌疗法治疗的具有癌症的个体的方法,该方法包括:测定自该个体获得的样品中MiT易位的存在或缺失,其中该样品中存在该MiT易位指示该个体更有可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗或缺失该MiT易位指示该个体不太可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗。
本文中提供的是预测具有癌症的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定MiT易位,由此存在该MiT易位指示该个体更有可能有效响应用该MiT拮抗剂治疗且缺失该MiT易位指示该个体不太可能有效响应用该MiT拮抗剂治疗。
本文中提供的是预测具有癌症的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,该方法包括检测自该个体获得的样品中MiT易位的存在或缺失,其中存在该MiT易位预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且缺失该MiT易位预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。
在本发明的任何方法的一些实施方案中,该方法进一步包括对该个体施用有效量的MiT拮抗剂。
本文中提供的是抑制nccRCC癌细胞增殖的方法,其包括使该癌细胞接触有效量的MiT易位拮抗剂。
本文中提供的是治疗个体中的nccRCC的方法,其包括对该个体施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。
在本发明的任何方法的一些实施方案中,该癌症或癌细胞包含MiT易位。
在本发明的任何方法的一些实施方案中,该癌症或癌细胞包含MiT过表达。
在本发明的任何方法的一些实施方案中,该癌症或癌细胞包含BIRC7过表达。
在本发明的任何方法的一些实施方案中,MiT是MITF。
在本发明的任何方法的一些实施方案中,MiT是TFEB。
在本发明的任何方法的一些实施方案中,MiT是TFEC。
在本发明的任何方法的一些实施方案中,MiT是TFE3。
在本发明的任何方法的一些实施方案中,MiT是SBNO2。
在本发明的任何方法的一些实施方案中,MiT易位是MITF易位。
在本发明的任何方法的一些实施方案中,MiT易位是TFEB易位。
在本发明的任何方法的一些实施方案中,MiT易位是TFEC易位。
在本发明的任何方法的一些实施方案中,MiT易位是TFE3易位。
在一些实施方案中,该MiT易位是使用本文中公开的测定MiT易位(检测MiT易位的存在)的任何方法检测的。
在本发明的任何方法的一些实施方案中,该癌症或癌症是鳞状细胞癌(例如上皮鳞状细胞癌),肺癌包括小细胞肺癌(SCLC),非小细胞肺癌(NSCLC),肺的腺癌和肺的鳞癌,腹膜癌,肝细胞癌,胃的癌或胃癌包括胃肠癌,胰腺癌,成胶质细胞瘤,宫颈癌,卵巢癌,肝癌,膀胱癌,肝瘤,乳腺癌(包括转移性乳腺癌),结肠癌,直肠癌,结直肠癌,子宫内膜或子宫癌,唾液腺癌,肾癌或肾的癌,前列腺癌,阴门癌,甲状腺癌,肝的癌,肛门癌,阴茎癌,睾丸癌,食道癌,胆管肿瘤,以及头和颈癌。
在本发明的任何方法的一些实施方案中,该癌症或癌症是肾细胞癌(RCC)。在一些实施方案中,该RCC是非透明细胞肾细胞癌(nccRCC)或易位RCC(tRCC)。在一些实施方案中,该RCC是nccRCC。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是抗体,结合多肽,小分子,或多核苷酸。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是MET拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是BIRC7拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是MITF拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是TFEB拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是TFEC拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂是TFE3拮抗剂。
在本发明的任何方法的一些实施方案中,该MiT拮抗剂结合MITF易位。在一些实施方案中,该MITF易位包含ACTG1和MITF。在一些实施方案中,MITF易位包含ACTG1外显子3。在一些实施方案中,MITF易位包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位包含SEQ ID NO:13和/或30。在一些实施方案中,该MITF易位包含SEQ ID NO:30。在一些实施方案中,该MITF易位是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位包含AP3S1和MITF。在一些实施方案中,MITF易位包含AP3S1外显子3。在一些实施方案中,该MITF易位包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位是由AP3S1启动子驱动的。在一些实施方案中,MiT拮抗剂结合TFEB易位。在一些实施方案中,该TFEB易位包含CLTC和TFEB。在一些实施方案中,该TFEB易位包含CLTC外显子17。在一些实施方案中,该TFEB易位包含CLTC外显子17和TFEB外显子6。在一些实施方案中,TFEB易位包含SEQ ID NO:19。在一些实施方案中,TFEB易位是由CLTC启动子驱动的。在一些实施方案中,该MiT拮抗剂结合SBNO2易位。在一些实施方案中,该SBNO2易位是倒位。在一些实施方案中,该SBNO2倒位包含MIDN和SBNO2。在一些实施方案中,SBNO2倒位包含MIDN启动子。在一些实施方案中,该SBNO2倒位包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2倒位包含SEQ ID NO:25。在一些实施方案中,该SBNO2倒位是由CLTC启动子驱动的。
在本发明的任何方法的一些实施方案中,该方法进一步包括施用别的治疗剂。
附图简述
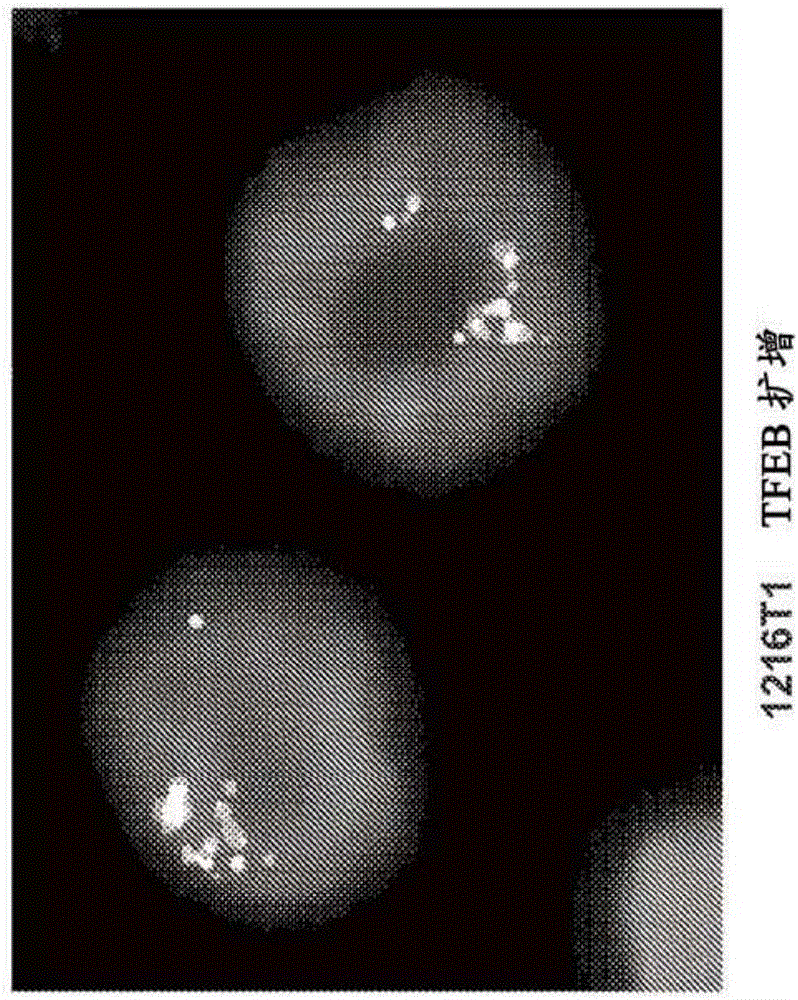
图1A,B,C,D。FISH(荧光原位杂交)图像,显示(A)肿瘤细胞中的TFEB扩增,(B)正常细胞中的TFE3,(C-D)肿瘤细胞中的TFE3(C)和TFEB(D)基因重排。使用杂交至染色体条带Xp11.2中的TFE3基因或染色体条带6p21中的TFEB基因的5’或3’侧的DNA探针集(Agilent Technologies,CA)。使用的TFE3探针:TFE3 5'Xp11标记的红色和TFE3 3'Xp11标记的绿色。使用的TFEB探针:TFEB 5'6p21.1标记的红色和TFEB 3'6p21.1标记的绿色。基因的完整拷贝产生黄色(Y)信号,其来自红色(R)和绿色(G)标记物的合并(小图B)。在小图C中,与TFE3易位(融合)一致,我们在男性患者中观察到涉及分开的红色和绿色信号的样式(在超过80%的细胞中看到)。类似地,在小图D中,与TFEB易位一致,我们观察到涉及清楚分开的红色和绿色信号的样式。在小图A中,90%的细胞显示TFEB基因的多体性且~25%显示3’TFEB信号的额外拷贝,与在SNP阵列上检测到的扩增一致。
图2。所示nccRCC亚型中的TFE3表达的框线图。
图3A,B。MIDN-SBNO2基因融合。(A)描绘MIDN-SBNO2融合在基因组上的位置,取向和外显子-内含子架构的卡通图。显示了使用RNA-seq数据鉴定的MIDN(e1)-SBNO2(e2)融合的读出证据。RT-PCR衍生产物的代表性Sanger测序层析图,确认了MIDN(e1)-SBNO2(e2)融合接合处。(B)所得MIDN-SBNO2融合蛋白的示意图。
图4A-D。肿瘤中的SBNO2表达的柱形图,如通过RNA-seq测量的。
图5A,B。CLTC-TFEB基因融合。(A)描绘CLTC-TFEB融合在基因组上的位置,取向和外显子-内含子架构的卡通图。显示了使用RNA-seq数据鉴定的CLTC(e17)-TFEB(e6)融合的读出证据。RT-PCR衍生产物的代表性Sanger测序层析图,确认了CLTC(e17)-TFEB(e6)融合接合处。(B)所得CLTC-TFEB融合蛋白的示意图。CL-P-网格蛋白_推进;CL-网格蛋白-连接;CH-L-网格蛋白_H_连接;富Gln-富甘氨酸;AD-活化域;B-碱性;HLH-螺旋-环-螺旋;及LZ-亮氨酸拉链。
图6A-G。nccRCC的基于RNA-seq的分类。(A)描绘样品1216T中的局灶性TFEB扩增的拷贝数比图。(B)肿瘤中的TFEB表达的框线图显示样品1216T中高水平的TFEB表达。
图7A,B,C。MITF基因融合。(A)描绘ACTG1-MITF融合在基因组上的位置,取向和外显子-内含子架构的卡通图。显示了使用RNA-seq数据鉴定的ACTG1(e3)-MITF(e3)融合的读出证据和RT-PCR衍生产物的代表性Sanger测序层析图,确认了ACTG1(e3)-MITF(e3)融合接合处。(B)ACTG1-MITF融合蛋白的示意图。(C)包含MITF融合的肿瘤中的MITF表达。AD-活化域;B-碱性;HLH-螺旋-环-螺旋;及LZ-亮氨酸拉链。
图8A,B,C,D,E。ACTG1-MITF基因融合促进锚定不依赖性生长。(A)经MITF WT或融合构建体转染的HEK293T细胞中MITF靶基因的表达。所示值来自三份复制品。(误差棒代表SEM;**p<0.01;***p<0.001)。(B)使用Western印迹评估的环己酰亚胺处理后经所示构建体转染的HEK293T细胞中MITF融合蛋白随时间的稳定性。(C)显示表达所示构建体的NIH3T3细胞中带Flag标签的ACTG1,MITF和ACTG1-MITF融合蛋白的表达的Western印迹。使用Hsp90作为加载对照。(D)描绘稳定表达所示构建体的NIH3T3细胞的集落形成的代表性图像(EV=空载体)。(E)小图(D)中所示集落数目(>300uM直径)的量化。所示数据是均值±SEM(n=3,***p<0.001)。
图9。瞬时转染的293T细胞中WT MITF和ACTG1-MITF的表达水平。使用这来标准化图8A中所示靶基因的表达。
图10A-B。与没有易位的样品相比,具有MITF/TFE易位或融合的肿瘤中BIRC7的表达。BIRC7的表达在具有MITF/TFE事件的样品中是显著的(t检验p值0.002308467)。
发明详述
I.定义
术语“MITF”在本文中指来自任何脊椎动物来源,包括哺乳动物诸如灵长类(例如人)和啮齿类(例如小鼠和大鼠)的天然MITF,除非另有说明。该术语涵盖“全长”,未加工的MITF以及因细胞中的加工所致的任何形式的MITF。该术语还涵盖MITF的天然存在变体,例如剪接变体或等位变体。一种例示性人MITF的核酸序列的序列为SEQ ID NO:2。
“MITF变体”或其变型意指与如本文中公开的任何MITF具有至少约80%氨基酸序列同一性的MITF多肽或多核苷酸,一般为或编码活性MITF多肽,如本文中定义的。例如,此类MITF变体包括如下的MITF,其中添加或删除一个或多个核酸或氨基酸残基。通常,MITF变体与如本文中公开的MITF会具有至少约80%序列同一性,或者至少约81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%序列同一性。通常,MITF变体的长度是至少约10个残基,或者长度为至少约20,30,40,50,60,70,80,90,100,110,120,130,140,150,160,170,180,190,200,210,220,230,240,250,260,270,280,290,300,310,320,330,340,350,360,370,380,390,400,410,420,430,440,450,460,470,480,490,500,510,520,530,540,550,560,570,580,590,600个,或更多个。任选地,MITF变体会具有或编码具有与MITF相比不超过1处保守氨基酸替代,或者与MITF相比不超过2,3,4,5,6,7,8,9,或10处保守氨基酸替代的序列。
术语“TFEB”在本文中指来自任何脊椎动物来源,包括哺乳动物诸如灵长类(例如人)和啮齿类(例如小鼠和大鼠)的天然TFEB,除非另有说明。该术语涵盖“全长”,未加工的TFEB以及因细胞中的加工所致的任何形式的TFEB。该术语还涵盖TFEB的天然存在变体,例如剪接变体或等位变体。一种例示性人TFEB的核酸序列的序列为SEQ ID NO:4。
“TFEB变体”或其变型意指与如本文中公开的任何TFEB具有至少约80%氨基酸序列同一性的TFEB多肽或多核苷酸,一般为或编码活性TFEB多肽,如本文中定义的。例如,此类TFEB变体包括如下的TFEB,其中添加或删除一个或多个核酸或氨基酸残基。通常,TFEB变体与如本文中公开的TFEB会具有至少约80%序列同一性,或者至少约81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%序列同一性。通常,TFEB变体的长度是至少约10个残基,或者长度为至少约20,30,40,50,60,70,80,90,100,110,120,130,140,150,160,170,180,190,200,210,220,230,240,250,260,270,280,290,300,310,320,330,340,350,360,370,380,390,400,410,420,430,440,450,460,470,480,490,500,510,520,530,540,550,560,570,580,590,600个,或更多个。任选地,TFEB变体会具有或编码具有与TFEB相比不超过1处保守氨基酸替代,或者与TFEB相比不超过2,3,4,5,6,7,8,9,或10处保守氨基酸替代的序列。
术语“TFE3”在本文中指来自任何脊椎动物来源,包括哺乳动物诸如灵长类(例如人)和啮齿类(例如小鼠和大鼠)的天然TFE3,除非另有说明。该术语涵盖“全长”,未加工的TFE3以及因细胞中的加工所致的任何形式的TFE3。该术语还涵盖TFE3的天然存在变体,例如剪接变体或等位变体。一种例示性人TFE3的核酸序列的序列为SEQ ID NO:6。
“TFE3变体”或其变型意指与如本文中公开的任何TFE3具有至少约80%氨基酸序列同一性的TFE3多肽或多核苷酸,一般为或编码活性TFE3多肽,如本文中定义的。例如,此类TFE3变体包括如下的TFE3,其中添加或删除一个或多个核酸或氨基酸残基。通常,TFE3变体与如本文中公开的TFE3会具有至少约80%序列同一性,或者至少约81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%序列同一性。通常,TFE3变体的长度是至少约10个残基,或者长度为至少约20,30,40,50,60,70,80,90,100,110,120,130,140,150,160,170,180,190,200,210,220,230,240,250,260,270,280,290,300,310,320,330,340,350,360,370,380,390,400,410,420,430,440,450,460,470,480,490,500,510,520,530,540,550,560,570,580,590,600个,或更多个。任选地,TFE3变体会具有或编码具有与TFE3相比不超过1处保守氨基酸替代,或者与TFE3相比不超过2,3,4,5,6,7,8,9,或10处保守氨基酸替代的序列。
术语“TFEC”在本文中指来自任何脊椎动物来源,包括哺乳动物诸如灵长类(例如人)和啮齿类(例如小鼠和大鼠)的天然TFEC,除非另有说明。该术语涵盖“全长”,未加工的TFEC以及因细胞中的加工所致的任何形式的TFEC。该术语还涵盖TFEC的天然存在变体,例如剪接变体或等位变体。一种例示性人TFEC的核酸序列的序列为SEQ ID NO:8。
“TFEC变体”或其变型意指与如本文中公开的任何TFEC具有至少约80%氨基酸序列同一性的TFEC多肽或多核苷酸,一般为或编码活性TFEC多肽,如本文中定义的。例如,此类TFEC变体包括如下的TFEC,其中添加或删除一个或多个核酸或氨基酸残基。通常,TFEC变体与如本文中公开的TFEC会具有至少约80%序列同一性,或者至少约81%,82%,83%,84%,85%,86%,87%,88%,89%,90%,91%,92%,93%,94%,95%,96%,97%,98%,或99%序列同一性。通常,TFEC变体的长度是至少约10个残基,或者长度为至少约20,30,40,50,60,70,80,90,100,110,120,130,140,150,160,170,180,190,200,210,220,230,240,250,260,270,280,290,300,310,320,330,340,350,360,370,380,390,400,410,420,430,440,450,460,470,480,490,500,510,520,530,540,550,560,570,580,590,600个,或更多个。任选地,TFEC变体会具有或编码具有与TFEC相比不超过1处保守氨基酸替代,或者与TFEC相比不超过2,3,4,5,6,7,8,9,或10处保守氨基酸替代的序列。
术语“MiT”指蛋白质MITF,TFEB,TFE3,TFEC,和SBNO2。
术语“MiT易位”在本文中指如下的MiT,其中断裂染色体包括例如编码MiT,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与MiT天然位置不同的染色体位置或MiT天然位置之中和/或周围与第二基因天然位置不同的染色体位置。MiT易位可以是MITF易位,TFEB易位,TFE3易位,TFEC易位和/或SBNO2易位。
术语“MITF易位”在本文中指如下的MITF,其中断裂染色体包括例如编码MITF,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与MITF天然位置不同的染色体位置或MITF天然位置之中和/或周围与第二基因天然位置不同的染色体位置。
术语“TFEB易位”在本文中指如下的TFEB,其中断裂染色体包括例如编码TFEB,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与TFEB天然位置不同的染色体位置或TFEB天然位置之中和/或周围与第二基因天然位置不同的染色体位置。
术语“TFE3易位”在本文中指如下的TFE3,其中断裂染色体包括例如编码TFE3,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与TFE3天然位置不同的染色体位置或TFE3天然位置之中和/或周围与第二基因天然位置不同的染色体位置。
术语“TFEC易位”在本文中指如下的TFEC,其中断裂染色体包括例如编码TFEC,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与TFEC天然位置不同的染色体位置或TFEC天然位置之中和/或周围与第二基因天然位置不同的染色体位置。
术语“SBNO2易位”在本文中指如下的SBNO2,其中断裂染色体包括例如编码SBNO2,其变体,或片段或第二基因,其变体,或片段的多核苷酸的一部分重附着于不同的染色体位置,例如与SBNO2天然位置不同的染色体位置或SBNO2天然位置之中和/或周围与第二基因天然位置不同的染色体位置。
术语“MiT易位融合多核苷酸”在本文中指MiT易位基因产物或融合多核苷酸的核酸序列。MiT易位融合多核苷酸可以是MITF易位融合多核苷酸,TFEB易位融合多核苷酸,TFE3易位融合多核苷酸,TFEC易位融合多核苷酸和/或SBNO2易位融合多核苷酸。术语“MiT易位融合多肽”在本文中指MiT易位基因产物或融合多核苷酸的氨基酸序列。MiT易位融合多肽可以是MITF易位融合多肽,TFEB易位融合多肽,TFE3易位融合多肽,TFEC易位融合多肽和/或SBNO2易位融合多肽。
如本文中定义的,术语“MiT易位拮抗剂”指部分或完全阻断,抑制,或中和由MiT易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合MiT易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。MiT易位拮抗剂可以是MITF易位拮抗剂,TFEB易位拮抗剂,TFE3易位拮抗剂,TFEC易位拮抗剂和/或SBNO2易位拮抗剂。
如本文中定义的,术语“MITF易位拮抗剂”指部分或完全阻断,抑制,或中和由MITF易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合MITF易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“TFEB易位拮抗剂”指部分或完全阻断,抑制,或中和由TFEB易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合TFEB易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“TFE3易位拮抗剂”指部分或完全阻断,抑制,或中和由TFE3易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合TFE3易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“TFEC易位拮抗剂”指部分或完全阻断,抑制,或中和由TFEC易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合TFEC易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“SBNO2易位拮抗剂”指部分或完全阻断,抑制,或中和由SBNO2易位融合多肽介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合SBNO2易位融合多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“MET途径拮抗剂”指部分或完全阻断,抑制,或中和由MET途径介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合MET途径多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是抗体拮抗剂。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
如本文中定义的,术语“BIRC7途径拮抗剂”指部分或完全阻断,抑制,或中和由BIRC7途径介导的生物学活性的任何分子。在一些实施方案中,此类拮抗剂结合BIRC7途径多肽。依照一个实施方案,拮抗剂是多肽。依照另一个实施方案,拮抗剂是抗体拮抗剂。依照另一个实施方案,拮抗剂是小分子拮抗剂。依照另一个实施方案,拮抗剂是多核苷酸拮抗剂。
“多核苷酸”或“核酸”在本文中可互换使用,指任何长度的核苷酸聚合物,包括DNA和RNA。核苷酸可以是脱氧核糖核苷酸,核糖核苷酸,经过修饰的核苷酸或碱基,和/或其类似物,或者是可通过DNA或RNA聚合酶,或者通过合成反应掺入聚合物中的任何底物。多核苷酸可包含经过修饰的核苷酸,诸如甲基化核苷酸及其类似物。核苷酸序列可以由非核苷酸组分中断。多核苷酸可包含合成后进行的修饰,诸如缀合至标记物。其它类型的修饰包括例如“帽”,将一个或多个天然存在的核苷酸用类似物替代,核苷酸间修饰诸如例如具有不带电荷连接(例如甲基膦酸酯,磷酸三酯,磷酰胺酯(phosphoamidate),氨基甲酸酯等)和具有带电荷连接(例如硫代磷酸酯,二硫代磷酸酯等)的修饰,含有悬垂模块(pendant moiety)诸如例如蛋白质(例如核酸酶,毒素,抗体,信号肽,聚L-赖氨酸等)的修饰,具有嵌入剂(例如吖啶,补骨脂素等)的修饰,含有螯合剂(例如金属,放射性金属,硼,氧化性金属等)的修饰,含有烷化剂的修饰,具有经修饰连接(例如α端基异构核酸(anomeric nucleic acid)等)的修饰,以及未修饰形式的多核苷酸。另外,通常存在于糖类中的任何羟基基团可以用例如膦酸(phosphonate)基团,磷酸(phosphate)基团替换,用标准保护基团保护,或活化以制备与别的核苷酸的别的连接,或者可缀合至固体或半固体支持物。可磷酸化或者用胺或1-20个碳原子的有机加帽基团模块取代5’和3’末端OH。其它羟基也可衍生成标准保护基团。多核苷酸也可含有本领域普遍知道的核糖或脱氧核糖糖类的类似物形式,包括例如2’-氧-甲基-,2’-氧-烯丙基-,2’-氟-或2’-叠氮-核糖,碳环糖类似物,α-端基异构糖,差向异构糖诸如阿拉伯糖,木糖或来苏糖,吡喃糖,呋喃糖,景天庚酮糖,无环类似物及脱碱基核苷类似物诸如甲基核糖核苷。可用备选连接基团替换一个或多个磷酸二酯连接。这些备选连接基团包括但不限于以下实施方案,其中磷酸酯用P(O)S(“硫代酸酯”(thioate)),P(S)S(“二硫代酸酯”(dithioate)),(O)NR2(“酰胺酯”(amidate)),P(O)R,P(O)OR’,CO或CH2(“甲缩醛”(formacetal))替代,其中R或R’各自独立为H或者取代或未取代的烃基(1-20个C),任选含有醚(-O-)连接,芳基,烯基,环烃基,环烯基或芳烃基(araldyl)。并非多核苷酸中的所有连接都必需是相同的。前述描述适用于本文中提及的所有多核苷酸,包括RNA和DNA。
如本文中所使用的,“寡核苷酸”一般指单链的合成的多核苷酸,其在长度上一般小于约200个核苷酸,但这不是必须的。术语“寡核苷酸”和“多核苷酸”并不互相排斥。上文关于多核苷酸的描述同样且完全可适用于寡核苷酸。
术语“引物”指能够与核酸杂交并允许互补核酸的聚合作用(一般通过提供游离的3’-OH基团)的单链多核苷酸。
术语“小分子”指具有约2000道尔顿或更小,优选约500道尔顿或更小的分子量的任何分子。
术语“宿主细胞”,“宿主细胞系”,和“宿主细胞培养物”可互换使用,并且指已经导入有外源核酸的细胞,包括此类细胞的后代。宿主细胞包括“转化体”和“经转化的细胞”,其包括原代的经转化的细胞及自其衍生的后代而不考虑传代的次数。后代在核酸内容物上可以与亲本细胞不完全相同,而是可以含有突变。本文中包括具有与在初始转化细胞中筛选或选择的相同功能或生物学活性的突变体后代。
如本文中使用的,术语“载体”指能够扩增与其连接的另一种核酸的核酸分子。该术语包括作为自身复制型核酸结构的载体及整合入接受其导入的宿主细胞的基因组中的载体。某些载体能够指导与其可操作连接的核酸的表达。此类载体在本文中称为“表达载体”。
“分离的”抗体指已经与其天然环境的组分分开的抗体。在一些实施方案中,抗体纯化至大于95%或99%的纯度,如通过例如电泳(例如SDS-PAGE,等电聚焦(IEF),毛细管电泳)或层析(例如离子交换或反相HPLC)测定的。关于用于评估抗体纯度的方法的综述,见例如Flatman et al.,J.Chromatogr.B 848:79-87(2007)。
“分离的”核酸指已经与其天然环境的组分分开的核酸分子。分离的核酸包括通常含有核酸分子的细胞中含有的核酸分子,但是核酸分子在染色体外或在与其天然染色体位置不同的染色体位置处存在。
本文中的术语“抗体”以最广义使用,并且涵盖各种抗体结构,包括但不限于单克隆抗体,多克隆抗体,多特异性抗体(例如双特异性抗体),和抗体片段,只要它们展现出期望的抗原结合活性。
“抗体片段”指与完整抗体不同的分子,其包含完整抗体的如下部分,该部分结合完整抗体所结合的抗原。抗体片段的例子包括但不限于Fv,Fab,Fab’,Fab’-SH,F(ab’)2;双抗体;线性抗体;单链抗体分子(例如scFv);和由抗体片段形成的多特异性抗体。
与参照抗体“结合相同表位的抗体”指在竞争测定法中将参照抗体对其抗原的结合阻断50%或更多的抗体,且相反,参照抗体在竞争测定法中将该抗体对其抗原的结合阻断50%或更多。本文中提供了例示性的竞争测定法。
术语“全长抗体”,“完整抗体”,和“全抗体”在本文中可互换使用,指与天然抗体结构具有基本上类似的结构或者具有含有如本文中所限定的Fc区的重链的抗体。
如本文中使用的,术语“单克隆抗体”指从一群基本上同质的抗体获得的抗体,即构成群体的各个抗体是相同的和/或结合相同表位,除了例如含有天然存在的突变或在单克隆抗体制备物的生成期间发生的可能的变体抗体外,此类变体一般以极小量存在。与通常包含针对不同决定簇(表位)的不同抗体的多克隆抗体制备物不同,单克隆抗体制备物的每个单克隆抗体针对抗原上的单一决定簇。如此,修饰语“单克隆”指示抗体自一群基本上同质的抗体获得的特性,而不应解释为要求通过任何特定方法来生成抗体。例如,可以通过多种技术来生成要依照本发明使用的单克隆抗体,包括但不限于杂交瘤方法,重组DNA方法,噬菌体展示方法,和利用含有所有或部分人免疫球蛋白基因座的转基因动物的方法,本文中描述了用于生成单克隆抗体的此类方法和其它例示性方法。
“天然抗体”指具有不同结构的天然存在的免疫球蛋白分子。例如,天然IgG抗体是约150,000道尔顿的异四聚糖蛋白,由二硫化物键合的两条相同轻链和两条相同重链构成。从N至C端,每条重链具有一个可变区(VH),也称作可变重域或重链可变域,接着是三个恒定域(CH1,CH2,和CH3)。类似地,从N至C端,每条轻链具有一个可变区(VL),也称作可变轻域或轻链可变域,接着是一个恒定轻(CL)域。根据其恒定域氨基酸序列,抗体轻链可归入两种型中的一种,称作卡帕(κ)和拉姆达(λ)。
术语“嵌合”抗体指其中的重和/或轻链的一部分自特定的来源或物种衍生,而重和/或轻链的剩余部分自不同来源或物种衍生的抗体。
“人抗体”指拥有与由人或人细胞生成的或利用人抗体全集或其它人抗体编码序列自非人来源衍生的抗体的氨基酸序列对应的氨基酸序列的抗体。人抗体的此定义明确排除包含非人抗原结合残基的人源化抗体。
“人源化”抗体指包含来自非人HVR的氨基酸残基和来自人FR的氨基酸残基的嵌合抗体。在某些实施方案中,人源化抗体会包含至少一个,通常两个基本上整个可变域,其中所有或基本上所有HVR(例如CDR)对应于非人抗体的那些,且所有或基本上所有FR对应于人抗体的那些。任选地,人源化抗体可以至少包含自人抗体衍生的抗体恒定区的一部分。抗体,例如非人抗体的“人源化形式”指已经经历人源化的抗体。
抗体的“类”指其重链拥有的恒定域或恒定区的类型。抗体有5大类:IgA,IgD,IgE,IgG,和IgM,并且这些中的几种可以进一步分成亚类(同种型),例如,IgG1,IgG2,IgG3,IgG4,IgA1,和IgA2。与不同类免疫球蛋白对应的重链恒定域分别称作α,δ,ε,γ,和μ。
“效应器功能”指那些可归于抗体Fc区且随抗体同种型而变化的生物学活性。抗体效应器功能的例子包括:C1q结合和补体依赖性细胞毒性(CDC);Fc受体结合;抗体依赖性细胞介导的细胞毒性(ADCC);吞噬作用;细胞表面受体(例如B细胞受体)下调;和B细胞活化。
本文中的术语“Fc区”用于定义免疫球蛋白重链中至少含有恒定区一部分的C端区。该术语包括天然序列Fc区和变体Fc区。在一个实施方案中,人IgG重链Fc区自Cys226,或自Pro230延伸至重链的羧基端。然而,Fc区的C端赖氨酸(Lys447)可以存在或不存在。除非本文中另有规定,Fc区或恒定区中的氨基酸残基的编号方式依照EU编号系统,也称作EU索引,如记载于Kabat等,Sequences of Proteins of Immunological Interest,第5版,Public Health Service,National Institutes of Health,Bethesda,MD,1991。
“框架”或“FR”指除高变区(HVR)残基以外的可变域残基。可变域的FR一般由4个FR域组成:FR1,FR2,FR3,和FR4。因而,HVR和FR序列在VH(或VL)中一般以如下的顺序出现:FR1-H1(L1)-FR2-H2(L2)-FR3-H3(L3)-FR4。
“人共有框架”指代表人免疫球蛋白VL或VH框架序列选集中最常存在的氨基酸残基的框架。通常,人免疫球蛋白VL或VH序列选集来自可变域序列亚组。通常,序列亚组是如Kabat等,Sequences of Proteins of Immunological Interest,第五版,NIH Publication 91-3242,Bethesda MD(1991),第1-3卷中的亚组。在一个实施方案中,对于VL,亚组是如Kabat等,见上文中的亚组κI。在一个实施方案中,对于VH,亚组是如Kabat等,见上文中的亚组III。
出于本文中的目的,“受体人框架”指包含自人免疫球蛋白框架或如下文定义的人共有框架衍生的轻链可变域(VL)框架或重链可变域(VH)框架的氨基酸序列的框架。自人免疫球蛋白框架或人共有框架“衍生”的受体人框架可以包含其相同的氨基酸序列,或者它可以含有氨基酸序列变化。在一些实施方案中,氨基酸变化的数目是10或更少,9或更少,8或更少,7或更少,6或更少,5或更少,4或更少,3或更少,或2或更少。在一些实施方案中,VL受体人框架与VL人免疫球蛋白框架序列或人共有框架序列在序列上相同。
术语“可变区”或“可变域”指抗体重或轻链中牵涉抗体结合抗原的域。天然抗体的重链和轻链可变域(分别为VH和VL)一般具有类似的结构,其中每个域包含4个保守的框架区(FR)和3个高变区(HVR)(见例如Kindt等,Kuby Immunology,第6版,W.H.Freeman and Co.,第91页(2007))。单个VH或VL域可以足以赋予抗原结合特异性。此外,可以分别使用来自结合抗原的抗体的VH或VL域筛选互补VL或VH域的文库来分离结合特定抗原的抗体。见例如,Portolano等,J.Immunol.150:880-887(1993);Clarkson等,Nature352:624-628(1991)。
如本文中使用的,术语“高变区”或“HVR”指抗体可变域中在序列上高变的和/或形成结构上限定的环(“高变环”)的每个区域。一般地,天然的四链抗体包含6个HVR;三个在VH中(H1,H2,H3),且三个在VL中(L1,L2,L3)。HVR一般包含来自高变环和/或来自互补决定区(CDR)的氨基酸残基,后一种是最高序列变异性的和/或牵涉抗原识别。例示性的高变环存在于氨基酸残基26-32(L1),50-52(L2),91-96(L3),26-32(H1),53-55(H2),和96-101(H3)(Chothia and Lesk,J.Mol.Biol.196:901-917(1987))。例示性的CDR(CDR-L1,CDR-L2,CDR-L3,CDR-H1,CDR-H2,和CDR-H3)存在于L1的氨基酸残基24-34,L2的50-56,L3的89-97,H1的31-35B,H2的50-65,和H3的95-102(Kabat et al.,Sequences of Proteins of Immunological Interest,5th Ed.Public Health Service,National Institutes of Health,Bethesda,MD(1991))。除了VH中的CDR1外,CDR一般包含形成高变环的氨基酸残基。CDR还包含“特异性决定残基”,或“SDR”,其是接触抗原的残基。SDR包含在称作缩短的-CDR,或a-CDR的CDR区域内。例示性的a-CDR(a-CDR-L1,a-CDR-L2,a-CDR-L3,a-CDR-H1,a-CDR-H2,和a-CDR-H3)存在于L1的氨基酸残基31-34,L2的50-55,L3的89-96,H1的31-35B,H2的50-58,和H3的95-102(参见Almagro and Fransson,Front.Biosci.13:1619-1633(2008))。除非另有指示,可变域中的HVR残基和其它残基(例如FR残基)在本文中依照Kabat等,见上文编号。
“亲和力”指分子(例如抗体)的单一结合位点与其结合配偶(例如抗原)之间全部非共价相互作用总和的强度。除非另有指示,如本文中使用的,“结合亲和力”指反映结合对的成员(例如抗体和抗原)之间1:1相互作用的内在结合亲和力。分子X对其配偶Y的亲和力通常可以用解离常数(Kd)来表述。亲和力可以通过本领域知道的常用方法来测量,包括本文中所描述的方法。下文描述了用于测量结合亲和力的具体的说明性和例示性的实施方案。
“亲和力成熟的”抗体指在一个或多个高变区(HVR)中具有一处或多处改变的抗体,与不拥有此类改变的亲本抗体相比,此类改变导致该抗体对抗原的亲和力改善。
术语“抗MiT抗体”和“结合MiT的抗体”指能够以足够亲和力结合MiT多肽,使得该抗体可作为诊断剂和/或治疗剂用于靶向MiT的抗体。在一个实施方案中,根据例如通过放射免疫测定法(RIA)的测量,抗MiT抗体结合无关的非MiT多肽的程度小于该抗体对MiT易位融合多肽的结合的约10%。在某些实施方案中,结合MiT的抗体具有≤1μM,≤100nM,≤10nM,≤1nM,≤0.1nM,≤0.01nM,或≤0.001nM(例如10-8M或更少,例如10-8M至10-13M,例如10-9M至10-13M)的解离常数(Kd)。MiT可以是MITF易位,TFEB易位,TFE3易位,TFEC易位和/或SBNO2易位。
术语“抗MiT易位抗体”和“结合MiT易位融合多肽的抗体”指能够以足够亲和力结合MiT易位融合多肽,使得该抗体可作为诊断剂和/或治疗剂用于靶向MiT易位的抗体。在一个实施方案中,根据例如通过放射免疫测定法(RIA)的测量,抗MiT易位抗体结合无关的非MiT易位融合多肽和/或非易位MiT多肽的程度小于该抗体对MiT易位融合多肽的结合的约10%。在某些实施方案中,结合MiT易位融合多肽的抗体具有≤1μM,≤100nM,≤10nM,≤1nM,≤0.1nM,≤0.01nM,或≤0.001nM(例如10-8M或更少,例如10-8M至10-13M,例如10-9M至10-13M)的解离常数(Kd)。在某些实施方案中,抗MiT易位抗体结合在MiT易位间独特的MiT易位表位。MiT易位可以是MITF易位,TFEB易位,TFE3易位,TFEC易位和/或SBNO2易位。
“阻断性”抗体或“拮抗性”抗体是抑制或降低其结合的抗原的生物学活性的抗体。优选的阻断性抗体或拮抗性抗体实质性或完全抑制抗原的生物学活性。
“裸抗体”指未与异源模块(例如细胞毒性模块)或放射性标记物缀合的抗体。裸抗体可以存在于药物配制剂中。
“免疫缀合物”指与一种或多种异源分子,包括但不限于细胞毒剂缀合的抗体。
关于参考多肽序列的“百分比(%)氨基酸序列同一性”定义为比对序列并在必要时引入缺口以实现最大百分比序列同一性后,且不将任何保守替代视为序列同一性的一部分时,候选序列中与参考多肽序列中的氨基酸残基相同的氨基酸残基的百分率。为测定百分比氨基酸序列同一性目的的比对可以本领域技术范围内的多种方式进行,例如使用公众可得到的计算机软件,诸如BLAST,BLAST-2,ALIGN或Megalign(DNASTAR)软件。本领域技术人员可决定用于比对序列的适宜参数,包括对所比较序列全长实现最大比对所需的任何算法。然而,为了本文中的目的,%氨基酸序列同一性值是使用序列比较计算机程序ALIGN-2生成的。ALIGN-2序列比较计算机程序由Genentech公司编写,源代码已经连同用户文档一起提交给美国版权局(US Copyright Office,Washington D.C.,20559),并以美国版权注册号TXU510087注册。公众可自Genentech公司(South San Francisco,California)得到ALIGN-2程序,或者可以自源代码编译。ALIGN-2程序应当编译成在UNIX操作系统,包括数码UNIX V4.0D上使用。所有序列比较参数由ALIGN-2程序设定且不变。
在采用ALIGN-2来比较氨基酸序列的情况中,给定氨基酸序列A相对于(to),与(with),或针对(against)给定氨基酸序列B的%氨基酸序列同一性(或者可表述为具有或包含相对于,与,或针对给定氨基酸序列B的某一%氨基酸序列同一性的给定氨基酸序列A)如下计算:
分数X/Y乘100
其中X是由序列比对程序ALIGN-2在该程序的A和B比对中评分为相同匹配的氨基酸残基数,且其中Y是B中的氨基酸残基总数。可以领会,若氨基酸序列A的长度与氨基酸序列B的长度不相等,则A相对于B的%氨基酸序列同一性将不等于B相对于A的%氨基酸序列同一性。除非另有具体说明,本文中所使用的所有%氨基酸序列同一性值都是依照上一段所述,使用ALIGN-2计算机程序获得的。
术语“检测”包括任何检测手段,包括直接和间接检测。
如本文中使用的,术语“生物标志物”指可在样品中检测的指示物,例如预测性,诊断性和/或预后性的指示物。生物标志物可以充当由特定的分子,病理学,组织学和/或临床特征表征的特定疾病或病症(例如癌症)亚型的指示物。在一些实施方案中,生物标志物是一种基因。在一些实施方案中,生物标志物是基因的变异(例如突变和/或多态性)。在一些实施方案中,生物标志物是易位。生物标志物包括但不限于,多核苷酸(例如DNA和/或RNA),多肽,多肽和多核苷酸修饰(例如翻译后修饰),碳水化合物和/或基于糖脂的分子标志物。
生物标志物与个体增加的临床益处有关的“存在”,“量”,或“水平”是生物学样品中的可检测水平。这些可通过本领域技术人员已知的及本文中公开的方法来测量。所评估的生物标志物的表达水平或量可用于确定对治疗的响应。
术语“表达的水平”或“表达水平”一般可互换使用,一般指生物学样品中生物标志物的量。“表达”一般指信息(例如基因编码和/或表观遗传)转化成细胞中存在并运行的结构的过程。因此,如本文中使用的,“表达”可以指转录成多核苷酸,翻译成多肽,或甚至多核苷酸和/或多肽修饰(例如多肽的翻译后修饰)。转录的多核苷酸的,翻译的多肽的,或多核苷酸和/或多肽修饰(例如多肽的翻译后修饰)的片段也应视为表达的,无论它们是源自通过可变剪接生成的转录物或经过降解的转录物,或者是源自多肽的翻译后加工(例如通过蛋白水解)。“表达的基因”包括转录成多核苷酸(如mRNA),然后翻译成多肽的基因,还有转录成RNA但不翻译成多肽的基因(例如转运和核糖体RNA)。
“升高的表达”,“升高的表达水平”或“升高的水平”指相对于对照如不具有疾病或病症(例如癌症)的个体或内部对照(例如持家型生物标志物),个体中生物标志物的增加的表达或增加的水平。
“降低的表达”,“降低的表达水平”或“降低的水平”指相对于对照诸如没有罹患疾病或病症(例如癌症)的个体或内部对照(例如持家型生物标志物),个体中生物标志物的降低的表达或降低的水平。
术语“持家型生物标志物”指通常相似地存在于所有细胞类型中的一种生物标志物或一组生物标志物(例如多核苷酸和/或多肽)。在一些实施方案中,持家型生物标志物是“持家基因”。“持家基因”在本文中指一种基因或一组基因,其编码活性对于细胞功能维持而言必要的蛋白质,且通常相似地存在于所有细胞类型中。
如本文中使用的,“扩增”一般指生成多拷贝的期望序列的过程。“多拷贝”指至少2个拷贝。“拷贝”不必然意味着与模板序列有完全的序列互补性或同一性。例如,拷贝可以包含核苷酸类似物诸如脱氧肌苷,有意的序列变化(诸如经由包含与模板可杂交但不互补的序列的引物引入的序列变化)和/或扩增期间发生的序列错误。
术语“多重PCR”指出于在单个反应中扩增两种或更多种DNA序列的目的,使用超过一套引物在自单一来源(例如个体)获得的核酸上进行的单个PCR反应。
杂交反应的“严格性”可以由本领域中的普通技术人员容易地确定,而且通常根据探针长度,洗涤温度和盐浓度凭经验计算。一般而言,较长的探针要求较高的温度以正确退火,而较短的探针需要较低的温度。杂交通常依赖于当互补链存在于低于其解链温度的环境中时变性DNA重新退火的能力。探针与可杂交序列之间的期望同源性程度越高,可使用的相对温度也越高。结果,可推出较高相对温度将趋向于使反应条件更为严格,而较低温度也就较不严格。关于杂交反应严格性的其它细节和解释,参见Ausubel et al.,Current Protocols in Molecular Biology,Wiley Interscience Publishers,(1995)。
如本文中所定义的,“严格条件”或“高严格条件”可由以下鉴定:(1)对于洗涤采用低离子强度和高温,例如0.015M氯化钠/0.0015M柠檬酸钠/0.1%十二烷基硫酸钠,50℃;(2)在杂交期间使用变性剂,诸如甲酰胺,例如50%(v/v)甲酰胺及0.1%牛血清清蛋白/0.1%Ficoll/0.1%聚乙烯吡咯烷酮/50mM磷酸钠缓冲液pH 6.5及750mM氯化钠,75mM柠檬酸钠,42℃;或(3)在使用50%甲酰胺,5x SSC(0.75M NaCl,0.075M柠檬酸钠),50mM磷酸钠(pH6.8),0.1%焦磷酸钠,5x登哈特(Denhardt)氏溶液,超声处理的鲑精DNA(50μg/ml),0.1%SDS和10%硫酸右旋糖苷的溶液中在42℃过夜杂交,在42℃于0.2x SSC(氯化钠/柠檬酸钠)洗涤10分钟,然后在55℃进行由含有EDTA的0.1x SSC组成的10分钟高严格洗涤。
“中等严格条件”可以如Sambrook et al.,Molecular Cloning:A Laboratory Manual,New York:Cold Spring Harbor Press,1989所描述的来定义,而且包括使用与上文所述那些相比较不太严格的洗涤溶液和杂交条件(例如温度,离子强度和%SDS)。中等严格条件的一个例子是在含:20%甲酰胺,5x SSC(150mM NaCl,15mM柠檬酸三钠),50mM磷酸钠(pH7.6),5x登哈特氏溶液,10%硫酸右旋糖苷和20mg/ml变性的剪切的鲑精DNA的溶液中于37℃温育过夜,然后于约37-50℃在1x SSC中洗涤滤膜。熟练技术人员会认识到如何根据适应诸如探针长度等因素的需要来调节温度,离子强度等。
术语“诊断”在本文中用于指对分子或病理学状态,疾病或疾患(例如癌症)的鉴定或分类。例如,“诊断”可以指特定癌症类型的鉴定。“诊断”还可以指特定癌症亚型的分类,例如通过组织病理学标准或分子特征(例如由一种或一组生物标志物(例如特定基因或由所述基因编码的蛋白质)的表达表征的亚型)。
术语“帮助做出诊断”在本文中用于指帮助进行关于特定类型的疾病或病症(例如癌症)的症状或状况的存在或性质的临床确定的方法。例如,帮助做出疾病或状况(例如癌症)诊断的方法可以包括在来自个体的生物学样品中检测特定的生物标志物。
术语“样品”在用于本文时指获得自或衍生自感兴趣的受试者和/或个体的组合物,其包含有待根据例如物理,生化,化学和/或生理特点来表征和/或鉴定的细胞和/或其它分子实体。例如,短语“疾病样品”或其变体指得自感兴趣的受试者的任何样品,预计或已知其包含待表征的细胞和/或分子实体。样品包括但不限于,原代或培养的细胞或细胞系,细胞上清,细胞裂解物,血小板,血清,血浆,玻璃体液,淋巴液,滑液,滤泡液(follicular fluid),精液,羊水,乳,全血,血液衍生的细胞,尿液,脑脊髓液,唾液,痰,泪,汗液,粘液,肿瘤裂解物,和组织培养液(tissue culture medium),组织提取物如匀浆化的组织,肿瘤组织,细胞提取物,及其组合。
“组织样品”或“细胞样品”意指从受试者或个体的组织获得的相似细胞的集合。组织或细胞样品的来源可以是实体组织如来自新鲜,冷冻和/或保存的器官,组织样品,活检和/或抽吸物;血液或任何血液成分如血浆;体液如脑脊髓液,羊水,腹膜液或间隙液(interstitial fluid);来自受试者的妊娠或发育中任意时间的细胞。组织样品还可以是原代或培养的细胞或细胞系。任选地,组织或细胞样品自患病的组织/器官获得。组织样品可以含有在自然界中不天然与组织混杂的化合物,如防腐剂,抗凝血剂,缓冲剂,固定剂,营养物,抗生素等。
如本文中使用的,“参照样品”,“参照细胞”,“参照组织”,“对照样品”,“对照细胞”或“对照组织”指用于比较目的的样品,细胞,组织,标准,或水平。在一个实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织自同一受试者或个体的健康和/或无疾病身体部位(例如组织或细胞)获得。例如,临近于患病细胞或组织的健康和/或无疾病细胞或组织(例如临近肿瘤的细胞或组织)。在另一个实施方案中,参照样品自同一受试者或个体的身体的未治疗组织和/或细胞获得。在又一个实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织自不是该受试者或个体的个体的健康和/或无疾病身体部位(例如组织或细胞)获得。在再一个实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织自不是该受试者或个体的个体身体的未治疗组织和/或细胞获得。
为本发明目的,组织样品的“切片”意指一块或一片组织样品,例如从组织样品上切下来的一薄片组织或细胞。应当了解,可以制作多片组织样品切片并进行分析,只要理解可以将组织样品的同一切片用于形态学和分子两个水平的分析或者针对多肽和多核苷酸二者进行分析。
“关联”或“联系”意指以任何方式将第一分析或方案的性能和/或结果与第二分析或方案的性能和/或结果进行比较。例如,可以将第一分析或方案的结果用于实施第二方案,和/或,可以使用第一分析或方案的结果来决定是否应当实施第二分析或方案。就多核苷酸分析或方案的实施方案而言,可以使用多核苷酸表达分析或方案的结果来决定是否应当实施特定治疗方案。
“个体响应”或“响应”可使用指示对个体益处的任何终点评估,包括但不限于:(1)一定程度地抑制疾病进展(例如癌症进展),包括减缓和完全阻滞;(2)缩小肿瘤尺寸;(3)抑制(即减轻,减缓或完全终止)癌细胞浸润入临近周围器官和/或组织;(4)抑制(即减轻,减缓或完全终止)转移;(5)一定程度地减轻与疾病或病症(例如癌症)有关的一种或多种症状;(6)无进展存活的长度延长;和/或(9)治疗后给定时间点的死亡率降低。
如本文中使用的,短语“基本上相似”表示两个数值之间足够高的相似程度(通常一个与分子有关,另一个与参照/比较分子有关),以致本领域技术人员会认为在用所述数值(例如Kd值)测量的生物学特征背景内两个数值之间的差异没有统计学显著性。作为参照/比较值的函数,所述两个数值之间的差异可以例如小于约20%,小于约10%,和/或小于约5%。短语“基本上正常”表示与参照(例如正常参照)基本上相似。
短语“实质性不同”表示两个数值之间足够高的差异程度(通常一个与分子有关,另一个与参照/比较分子有关),以致本领域技术人员会认为在用所述数值(例如Kd值)测量的生物学特征背景内两个数值之间的差异具有统计学显著性。作为参照/比较分子值的函数,所述两个数值之间的差异可以例如大于约10%,大于约20%,大于约30%,大于约40%,和/或大于约50%。
词语“标记物”在用于本文时指可检测的化合物或组合物。标记物通常与试剂如多核苷酸探针或抗体直接或间接缀合或融合,而且协助对其所缀合或融合的试剂的检测。标记物可以是通过自身就可检测的(例如放射性同位素标记物或荧光标记物),或者在酶标记物的情况中,可催化产生可检测产物的底物化合物或组合物的化学改变。
“有效量”指在必需的剂量和时间段上有效实现期望的治疗或预防结果的量。
本发明的物质/分子,激动剂或拮抗剂的“治疗有效量”可根据诸如个体的疾病状态,年龄,性别和重量及该物质/分子,激动剂或拮抗剂在个体中引发期望应答的能力等因素而变化。治疗有效量还指物质/分子,激动剂或拮抗剂的治疗有益效果胜过任何有毒或有害效果的量。“预防有效量”指在必需的剂量和时间段上有效实现期望的预防结果的量。通常而非必然,由于预防剂量是在疾病之前或在疾病的早期用于受试者的,因此预防有效量将低于治疗有效量。
术语“药物配制剂”指处于如下的形式,使得允许其中含有的活性组分的生物学活性是有效的,且不含对会接受配制剂施用的受试者具有不可接受的毒性的其它成分的制剂。
“药学可接受载剂”指药物配制剂中除活性组分以外,且对受试者无毒的组分。药学可接受载剂包括但不限于缓冲剂,赋形剂,稳定剂,或防腐剂。
如本文中使用的,“治疗/处理”(及其语法变型)指试图改变所治疗个体的天然过程的临床干预,并且可以为了预防或者在临床病理学的过程期间实施。治疗的期望效果包括但不限于预防疾病的发生或复发,减轻症状,减轻/减少疾病的任何直接或间接病理后果,预防转移,降低疾病进展速率,改善或减轻疾病状态,和消退或改善的预后。在一些实施方案中,使用本发明的抗体来延迟疾病的形成或减缓疾病的进展。
术语“癌症”和“癌性”指向或描述哺乳动物中典型特征为细胞生长/增殖不受调控的生理疾患。癌症的例子包括但不限于癌瘤,淋巴瘤(例如何杰金(Hodgkin)氏和非何杰金氏淋巴瘤),母细胞瘤,肉瘤,和白血病。此类癌症的更具体例子包括鳞状细胞癌,小细胞肺癌,非小细胞肺癌,肺的腺癌,肺的鳞癌,腹膜癌,肝细胞癌,胃肠癌,胰腺癌,胶质瘤,宫颈癌,卵巢癌,肝癌(liver cancer),膀胱癌,肝瘤(hepatoma),乳腺癌,结肠癌,结直肠癌,子宫内膜癌或子宫癌,唾液腺癌,肾癌,肝癌(liver cancer),前列腺癌,外阴癌,甲状腺癌,肝癌(hepatic carcinoma),白血病和其它淋巴增殖性病症,及各种类型的头和颈癌。在一些实施方案中,该癌症是肾细胞癌(例如非透明细胞肾细胞癌(nccRCC),易位RCC(tRCC)),黑素瘤,血管周围上皮样肿瘤,泡状软组织肉瘤,透明细胞肉瘤,血管肌脂瘤,淋巴管平滑肌瘤,透明细胞糖肺肿瘤,胰腺,子宫,许旺细胞瘤,细胞蓝色痣,神经纤维瘤,生殖细胞瘤,淋巴瘤,脂肪肉瘤。
术语“抗癌疗法”指在治疗癌症中有用的疗法。抗癌治疗剂的例子包括但不限于例如化疗剂,生长抑制剂,细胞毒剂,放射疗法中所使用的药剂,抗血管发生剂,凋亡剂,抗微管蛋白剂,和其它治疗癌症的药剂,抗CD20抗体,血小板衍生生长因子抑制剂(例如GleevecTM(Imatinib Mesylate)),COX-2抑制剂(例如celecoxib),干扰素,细胞因子,结合一种或多种以下靶物的拮抗剂(例如中和性抗体)(PDGFR-β,BlyS,APRIL,BCMA受体,TRAIL/Apo2),和其它生物活性和有机化学剂,等。本发明还包括它们的组合。
术语“细胞毒剂”在用于本文时指抑制或防止细胞功能和/或引起细胞死亡或破坏的物质。细胞毒剂包括但不限于放射性同位素(例如At211,I131,I125,Y90,Re186,Re188,Sm153,Bi212,P32,Pb212和Lu的放射性同位素);化疗剂或化疗药物(例如甲氨蝶呤(methotrexate),阿霉素(adriamicin),长春花生物碱类(vinca alkaloids)(长春新碱(vincristine),长春碱(vinblastine),依托泊苷(etoposide)),多柔比星(doxorubicin),美法仑(melphalan),丝裂霉素(mitomycin)C,苯丁酸氮芥(chlorambucil),柔红霉素(daunorubicin)或其它嵌入剂);生长抑制剂;酶及其片段,诸如溶核酶;抗生素;毒素,诸如小分子毒素或者细菌,真菌,植物或动物起源的酶活毒素,包括其片段和/或变体;及下文披露的各种抗肿瘤药或抗癌药。
“化疗剂”指可用于治疗癌症的化学化合物。化疗剂的实例包括烷化剂类(alkylating agents),诸如塞替派(thiotepa)和环磷酰胺(cyclophosphamide)磺酸烷基酯类(alkyl sulfonates),诸如白消安(busulfan),英丙舒凡(improsulfan)和哌泊舒凡(piposulfan);氮丙啶类(aziridines),诸如苯佐替派(benzodopa),卡波醌(carboquone),美妥替派(meturedopa)和乌瑞替派(uredopa);乙撑亚胺类(ethylenimines)和甲基蜜胺类(methylamelamines),包括六甲蜜胺(altretamine),三乙撑蜜胺(triethylenemelamine),三乙撑磷酰胺(triethylenephosphoramide),三乙撑硫代磷酰胺(triethylenethiophosphoramide)和三羟甲蜜胺(trimethylomelamine);番荔枝内酯类(acetogenin)(尤其是布拉他辛(bullatacin)和布拉他辛酮(bullatacinone));δ-9-四氢大麻酚(tetrahydrocannabinol)(屈大麻酚(dronabinol),β-拉帕醌(lapachone);拉帕醇(lapachol);秋水仙素类(colchicines);白桦脂酸(betulinicacid);喜树碱(camptothecin)(包括合成类似物托泊替康(topotecan)CPT-11(伊立替康(irinotecan),乙酰喜树碱,东莨菪亭(scopolectin)和9-氨基喜树碱);苔藓抑素(bryostatin);callystatin;CC-1065(包括其阿多来新(adozelesin),卡折来新(carzelesin)和比折来新(bizelesin)合成类似物);鬼臼毒素(podophyllotoxin);鬼臼酸(podophyllinic acid);替尼泊苷(teniposide);隐藻素类(cryptophycins)(特别是隐藻素1和隐藻素8);多拉司他汀(dolastatin);duocarmycin(包括合成类似物,KW-2189和CB1-TM1);艾榴塞洛素(eleutherobin);pancratistatin;sarcodictyin;海绵抑素(spongistatin);氮芥类(nitrogen mustards),诸如苯丁酸氮芥(chlorambucil),萘氮芥(chlornaphazine),胆磷酰胺(chlorophosphamide),雌莫司汀(estramustine),异环磷酰胺(ifosfamide),双氯乙基甲胺(mechlorethamine),盐酸氧氮芥(mechlorethamine oxide hydrochloride),美法仑(melphalan),新氮芥(novembichin),苯芥胆甾醇(phenesterine),泼尼莫司汀(prednimustine),曲磷胺(trofosfamide),尿嘧啶氮芥(uracil mustard);亚硝脲类(nitrosoureas),诸如卡莫司汀(carmustine),氯脲菌素(chlorozotocin),福莫司汀(fotemustine),洛莫司汀(lomustine),尼莫司汀(nimustine)和雷莫司汀(ranimnustine);抗生素类,诸如烯二炔类抗生素(enediyne)(如加利车霉素(calicheamicin),尤其是加利车霉素γ1I和加利车霉素ωI1(参见例如Nicolaou et al.,Angew.Chem Intl.Ed.Engl.,33:183-186(1994));CDP323,一种口服α-4整联蛋白抑制剂;蒽环类抗生素(dynemicin),包括dynemicin A;埃斯波霉素(esperamicin);以及新制癌素(neocarzinostatin)发色团和相关色蛋白烯二炔类抗生素发色团),阿克拉霉素(aclacinomysin),放线菌素(actinomycin),氨茴霉素(authramycin),偶氮丝氨酸(azaserine),博来霉素(bleomycin),放线菌素C(cactinomycin),carabicin,洋红霉素(carminomycin),嗜癌霉素(carzinophilin),色霉素(chromomycin),放线菌素D(dactinomycin),柔红霉素(daunorubicin),地托比星(detorubicin),6-二氮-5-氧-L-正亮氨酸,多柔比星(doxorubicin)(包括吗啉代多柔比星,氰基吗啉代多柔比星,2-吡咯代多柔比星,盐酸多柔比星脂质体注射剂脂质体多柔比星TLC D-99PEG化脂质体多柔比星和脱氧多柔比星),表柔比星(epirubicin),依索比星(esorubicin),伊达比星(idarubicin),麻西罗霉素(marcellomycin),丝裂霉素类(mitomycins)诸如丝裂霉素C,霉酚酸(mycophenolic acid),诺拉霉素(nogalamycin),橄榄霉素(olivomycin),培洛霉素(peplomycin),泊非霉素(porfiromycin),嘌呤霉素(puromycin),三铁阿霉素(quelamycin),罗多比星(rodorubicin),链黑菌素(streptonigrin),链佐星(streptozocin),杀结核菌素(tubercidin),乌苯美司(ubenimex),净司他丁(zinostatin),佐柔比星(zorubicin);抗代谢物类,诸如甲氨蝶呤,吉西他滨(gemcitabine)替加氟(tegafur)卡培他滨(capecitabine)埃坡霉素(epothilone)和5-氟尿嘧啶(5-FU);叶酸类似物,诸如二甲叶酸(denopterin),甲氨蝶呤,蝶酰三谷氨酸(pteropterin),三甲曲沙(trimetrexate);嘌呤类似物,诸如氟达拉滨(fludarabine),6-巯基嘌呤(mercaptopurine),硫咪嘌呤(thiamiprine),硫鸟嘌呤(thioguanine);嘧啶类似物,诸如安西他滨(ancitabine),阿扎胞苷(azacitidine),6-氮尿苷,卡莫氟(carmofur),阿糖胞苷(cytarabine),双脱氧尿苷(dideoxyuridine),去氧氟尿苷(doxifluridine),依诺他滨(enocitabine),氟尿苷(floxuridine);雄激素类,诸如卡鲁睾酮(calusterone),丙酸屈他雄酮(dromostanolone propionate),表硫雄醇(epitiostanol),美雄烷(mepitiostane),睾内酯(testolactone);抗肾上腺类,诸如氨鲁米特(aminoglutethimide),米托坦(mitotane),曲洛司坦(trilostane);叶酸补充剂,诸如亚叶酸(frolinic acid);醋葡醛内酯(aceglatone);醛磷酰胺糖苷(aldophosphamide glycoside);氨基乙酰丙酸(aminolevulinic acid);恩尿嘧啶(eniluracil);安吖啶(amsacrine);bestrabucil;比生群(bisantrene);依达曲沙(edatraxate);地磷酰胺(defofamine);地美可辛(demecolcine);地吖醌(diaziquone);elfornithine;依利醋铵(elliptinium acetate);epothilone;依托格鲁(etoglucid);硝酸镓;羟脲(hydroxyurea);香菇多糖(lentinan);氯尼达明(lonidainine);美登木素生物碱类(maytansinoids),诸如美登素(maytansine)和安丝菌素(ansamitocin);米托胍腙(mitoguazone);米托蒽醌(mitoxantrone);莫哌达醇(mopidanmol);二胺硝吖啶(nitraerine);喷司他丁(pentostatin);蛋氨氮芥(phenamet);吡柔比星(pirarubicin);洛索蒽醌(losoxantrone);2-乙基酰肼(ethylhydrazide);丙卡巴肼(procarbazine);多糖复合物(JHS Natural Products,Eugene,OR);雷佐生(razoxane);根霉素(rhizoxin);西索菲兰(sizofiran);螺旋锗(spirogermanium);细交链孢菌酮酸(tenuazonic acid);三亚胺醌(triaziquone);2,2’,2”-三氯三乙胺;单端孢菌素类(trichothecenes)(尤其是T-2毒素,疣孢菌素(verracurin)A,杆孢菌素(roridin)A和蛇行菌素(anguidine));乌拉坦(urethan);长春地辛(vindesine)达卡巴嗪(dacarbazine);甘露醇氮芥(mannomustine);二溴甘露醇(mitobronitol);二溴卫矛醇(mitolactol);哌泊溴烷(pipobroman);gacytosine;阿糖胞苷(arabinoside)(“Ara-C”);塞替派(thiotepa);类紫杉醇(taxoid),例如帕利他塞(paclitaxel)清蛋白改造的纳米颗粒剂型帕利他塞(ABRAXANETM)和多西他塞(docetaxel)苯丁酸氮芥(chloranbucil);6-硫鸟嘌呤(thioguanine);巯基嘌呤(mercaptopurine);甲氨蝶呤(methotrexate);铂剂,诸如顺铂(cisplatin),奥沙利铂(oxaliplatin)(例如和卡铂(carboplatin);长春药类(vincas),其阻止微管蛋白聚合形成微管,包括长春碱(vinblastine)长春新碱(vincristine)长春地辛(vindesine)和长春瑞滨(vinorelbine)依托泊苷(etoposide)(VP-16);异环磷酰胺(ifosfamide);米托蒽醌(mitoxantrone);亚叶酸(leucovorin);能灭瘤(novantrone);依达曲沙(edatrexate);道诺霉素(daunomycin);氨基蝶呤(aminopterin);伊本膦酸盐(ibandronate);拓扑异构酶抑制剂RFS 2000;二氟甲基鸟氨酸(DMFO);类维A酸(retinoids),诸如维A酸(retinoic acid),包括贝沙罗汀(bexarotene)二膦酸盐类(bisphosphonates),诸如氯膦酸盐(clodronate)(例如或依替膦酸钠(etidronate)NE-58095,唑来膦酸/唑来膦酸盐(zoledronic acid/zoledronate)阿伦膦酸盐(alendronate)帕米膦酸盐(pamidronate)替鲁膦酸盐(tiludronate)或利塞膦酸盐(risedronate)以及曲沙他滨(troxacitabine)(1,3-二氧戊环核苷胞嘧啶类似物);反义寡核苷酸,特别是抑制牵涉异常细胞增殖的信号途经中的基因表达的反义寡核苷酸,诸如例如PKC-α,Raf,H-Ras和表皮生长因子受体(EGF-R);疫苗,诸如疫苗和基因疗法疫苗,例如疫苗,疫苗和疫苗;拓扑异构酶1抑制剂(例如rmRH(例如BAY439006(sorafenib;Bayer);SU-11248(sunitinib,Pfizer);哌立福辛(perifosine),COX-2抑制剂(如塞来考昔(celecoxib)或艾托考昔(etoricoxib)),蛋白体抑制剂(例如PS341);bortezomibCCI-779;tipifarnib(R11577);orafenib,ABT510;Bcl-2抑制剂,诸如oblimersen sodiumpixantrone;EGFR抑制剂(见下文定义);酪氨酸激酶抑制剂(见下文定义);丝氨酸-苏氨酸激酶抑制剂,诸如雷帕霉素(rapamycin)(sirolimus,法尼基转移酶抑制剂,诸如lonafarnib(SCH 6636,SARASARTM);及任何上述各项的药学可接受盐,酸或衍生物;以及两种或更多种上述各项的组合,诸如CHOP(环磷酰胺,多柔比星,长春新碱和泼尼松龙联合疗法的缩写)和FOLFOX(奥沙利铂(ELOXATINTM)联合5-FU和亚叶酸的治疗方案的缩写)。
如本文中定义的化疗剂包括起调节,降低,阻断或抑制可促进癌生长的激素效果作用的“抗激素剂”或“内分泌治疗剂”类。它们自身可以是激素,包括但不限于:具有混合的激动剂/拮抗剂特性的抗雌激素类,包括他莫昔芬(tamoxifen)4-羟基他莫昔芬,托瑞米芬(toremifene)艾多昔芬(idoxifene),屈洛昔芬(droloxifene),雷洛昔芬(raloxifene)曲沃昔芬(trioxifene),那洛昔芬(keoxifene),和选择性雌激素受体调节剂类(SERM),诸如SERM3;没有激动剂特性的纯的抗雌激素类,诸如氟维司群(fulvestrant)和EM800(此类药剂可阻断雌激素受体(ER)二聚化,抑制DNA结合,提高ER周转,和/或遏制ER水平);芳香酶抑制剂类,包括类固醇芳香酶抑制剂类,诸如福美坦(formestane)和依西美坦(exemestane)和非类固醇芳香酶抑制剂类,诸如阿那曲唑(anastrazole)来曲唑(letrozole)和氨鲁米特(aminoglutethimide),和其它芳香酶抑制剂类,包括伏氯唑(vorozole)醋酸甲地孕酮(megestrol acetate)法倔唑(fadrozole)和4(5)-咪唑;促黄体生成激素释放激素激动剂类,包括亮丙瑞林(leuprolide)和戈舍瑞林(goserelin),布舍瑞林(buserelin)和曲普瑞林(tripterelin);性类固醇类(sex steroids),包括妊娠素类(progestine),诸如醋酸甲地孕酮(megestrol acetate)和醋酸甲羟孕酮(medroxyprogesterone acetate),雌激素类,诸如二乙基己烯雌酚(diethylstilbestrol)和普雷马林(premarin),和雄激素类/类视黄酸类,诸如氟甲睾酮(fluoxymesterone),所有反式视黄酸(transretionic acid)和芬维A胺(fenretinide);奥那司酮(onapristone);抗孕酮类;雌激素受体下调剂类(ERD);抗雄激素类,诸如氟他米特(flutamide),尼鲁米特(nilutamide)和比卡米特(bicalutamide);及任何上述物质的药剂学可接受的盐,酸或衍生物;以及两种或多种上述物质的组合。
术语“前药”在用于本申请时指与母药(parent drug)相比对肿瘤细胞的细胞毒性较小并能够酶促活化或转变为更具活性母药形式的药用活性物质的前体或衍生物形式。参见例如Wilman,“Prodrugs in Cancer Chemotherapy”Biochemical Society Transactions,14,pp.375-382,615th Meeting Belfast(1986)和Stella et al.,“Prodrugs:A Chemical Approach to Targeted Drug Delivery,”Directed Drug Delivery,Borchardt et al.,(ed.),pp.247-267,Humana Press(1985)。本发明的前药包括但不限于含磷酸盐/酯前药,含硫代磷酸盐/酯前药,含硫酸盐/酯前药,含肽前药,D-氨基酸修饰前药,糖基化前药,含β-内酰胺前药,含任选取代苯氧基乙酰胺的前药或含任选取代苯乙酰胺的前药,可转化为更具活性而无细胞毒性的药物的5-氟胞嘧啶和其它5-氟尿苷前药。可衍生为本发明使用的前药形式的细胞毒性药物的例子包括但不限于上文描述的那些化疗剂。
“生长抑制剂”在用于本文时指抑制细胞(例如在体外或在体内其生长依赖于MiT基因和/或MITF易位表达的细胞)生长的化合物或组合物。生长抑制剂的例子包括阻断细胞周期行进(处于S期以外的位置)的药剂,诸如诱导G1停滞和M期停滞的药剂。经典的M期阻断剂包括长春药类(vincas)(长春新碱(vincristine)和长春碱(vinblastine)),紫杉烷类(taxanes),和拓扑异构酶II抑制剂诸如多柔比星(doxorubicin),表柔比星(epirubicin),柔红霉素(daunorubicin),依托泊苷(etoposide)和博来霉素(bleomycin)。那些阻滞G1的药剂也溢出进入S期停滞,例如DNA烷化剂类诸如他莫昔芬(tamoxifen),泼尼松(prednisone),达卡巴嗪(dacarbazine),双氯乙基甲胺(mechlorethamine),顺铂(cisplatin),甲氨蝶呤(methotrexate),5-氟尿嘧啶(5-fluorouracil)和ara-C。更多信息可参见The Molecular Basis of Cancer,Mendelsohn and Israel,eds.,Chapter 1,entitled"Cell cycle regulation,oncogenes,and antineoplastic drugs"by Murakami et al.(WB Saunders:Philadelphia,1995),尤其是第13页。紫杉烷类(帕利他赛(paclitaxel)和多西他赛(docetaxel))是衍生自紫杉树的抗癌药。衍生自欧洲紫杉的多西他赛Rhone-Poulenc Rorer)是帕利他赛Bristol-Myers Squibb)的半合成类似物。帕利他赛和多西他赛促进由微管蛋白二聚体装配成微管并通过防止解聚使微管稳定,导致对细胞中有丝分裂的抑制。
“放射疗法”指使用定向伽马射线或贝塔射线来诱发对细胞的足够损伤,以限制细胞正常发挥功能的能力或全然破坏细胞。应当领会,本领域知道许多方式来确定治疗的剂量和持续时间。典型的治疗作为一次施用来给予,而典型的剂量范围为每天10-200个单位(戈瑞(Gray))。
“个体”或“受试者”指哺乳动物。哺乳动物包括但不限于驯养动物(例如牛,绵羊,猫,犬,和马),灵长类(例如人和非人灵长类诸如猴),兔,和啮齿类(例如小鼠和大鼠)。在某些实施方案中,个体或受试者是人。
术语“并行”在本文中用于指施用两种或更多种治疗剂,其中至少部分施用在时间上交叠。因而,并行施用包括如下的剂量给药方案,一种或多种药剂的施用中断后继续施用一种或多种其它药剂。
“降低或抑制”指引起20%,30%,40%,50%,60%,70%,75%,80%,85%,90%,95%,或更大的总体降低的能力。降低或抑制可以指所治疗的病症的症状,转移的存在或大小,或原发性肿瘤的大小。
术语“包装插页”用于指通常包括在治疗用产品的商业包装中的说明书,它们包含有关涉及此类治疗用产品应用的适应征,用法,剂量,施用,组合疗法,禁忌症和/或警告的信息。
“制品”是包含至少一种试剂,例如用于治疗疾病或病症(例如癌症)的药物或用于特异性检测本文所述生物标志物的探针的任何制造物(例如包装或容器)或试剂盒。在某些实施方案中,制造物或试剂盒以用于实施本文所述方法的单位推销,分销或销售。
“目标受众”指接受如通过推销或做广告(尤其为了特定用途,治疗,或适应症)进行的特定药物宣传或意图进行的特定药物宣传的人群或机构,诸如个体,群体,报纸,医学文献,和杂志读者,电视或因特网观众,无线电或因特网听众,内科医生,药品公司等。
如本领域技术人员理解的,本文中提述“约”某值或参数包括(且描述)针对该值或参数本身的实施方案。例如,提述“约X”的描述包括对“X”的描述。
应当理解,本文中描述的发明的方面和实施方案包括由和/或基本上由各方面和实施方案“组成”。如本文中使用的,单数形式“一个”,“一种”和“所述/该”包括复数提及物,除非另外指示。
II.方法和用途
本文中提供的是利用MiT拮抗剂的方法。特别是,本文中提供的是利用MiT易位拮抗剂的方法。例如,本文中提供的是抑制癌细胞的细胞增殖的方法,其包括使该癌细胞与有效量的MiT易位拮抗剂接触。本文中还提供的是在个体中治疗癌症的方法,其包括对该个体施用有效量的MiT易位拮抗剂。在一些实施方案中,该癌症或癌症包含MiT易位。
本文中还提供的是在个体中治疗癌症的方法,其包括对该个体施用有效量的抗癌疗法,其中治疗是基于该个体具有包含一种或多种生物标志物的癌症。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,提供的是在个体中治疗癌症的方法,其包括对该个体施用有效量的MiT拮抗剂,其中治疗是基于该个体具有包含MiT过表达和/或MiT易位的癌症。例如,提供的是在个体中治疗癌症的方法,其包括对该个体施用有效量的MiT拮抗剂,其中治疗是基于该个体具有包含MET过表达和/或BIRC7过表达的癌症。在一些实施方案中,该MiT拮抗剂是MITF,TFEB,TFE3,TFEC和/或SBNO2拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂(例如MITF,TFEB,TFE3,TFEC和/或SBNO2易位拮抗剂)。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中还提供的是在个体中治疗nccRCC的方法,其包括对该个体施用有效量的抗癌疗法,其中治疗是基于该个体具有包含一种或多种生物标志物的nccRCC癌症。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,提供的是在个体中治疗nccRCC的方法,其包括对该个体施用有效量的MiT拮抗剂,其中治疗是基于该个体具有包含MiT过表达和/或MiT易位的癌症。例如,提供的是在个体中治疗nccRCC的方法,其包括对该个体施用有效量的MiT拮抗剂,其中治疗是基于该个体具有包含MET过表达和/或BIRC7过表达的癌症。在一些实施方案中,该MiT拮抗剂是MITF,TFEB,TFE3,TFEC和/或SBNO2拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂(例如MITF,TFEB,TFE3,TFEC和/或SBNO2易位拮抗剂)。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中进一步提供的是在个体中治疗癌症的方法,前提是该个体已经发现具有包含一种或多种生物标志物的癌症,该治疗包括对该个体施用有效量的抗癌疗法。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,本文中提供的是在个体中治疗癌症的方法,前提是该个体已经发现具有包含MiT易位的癌症,该治疗包括对该个体施用有效量的MiT拮抗剂。例如,本文中提供的是在个体中治疗癌症的方法,前提是该个体已经发现具有包含MET过表达和/或BIRC7过表达的癌症,该治疗包括对该个体施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中进一步提供的是在个体中治疗nccRCC的方法,前提是该个体已经发现具有包含一种或多种生物标志物的nccRCC,该治疗包括对该个体施用有效量的抗癌疗法。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,本文中提供的是在个体中治疗nccRCC的方法,前提是该个体已经发现具有包含MiT易位的nccRCC,该治疗包括对该个体施用有效量的MiT拮抗剂。例如,本文中提供的是在个体中治疗nccRCC的方法,前提是该个体已经发现具有包含MET过表达和/或BIRC7过表达的nccRCC,该治疗包括对该个体施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是处理癌细胞的方法,其中该癌细胞包含一种或多种生物标志物,该方法包括提供有效量的MiT拮抗剂。例如,本文中提供的是处理癌细胞的方法,其中该癌细胞包含MiT易位,该方法包括提供有效量的MiT拮抗剂。例如,本文中提供的是处理癌细胞的方法,其中该癌细胞包含MET过表达和/或BIRC7过表达,该方法包括提供有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是处理nccRCC细胞的方法,其中该癌细胞包含一种或多种生物标志物,该方法包括提供有效量的MiT拮抗剂。例如,本文中提供的是处理nccRCC癌细胞的方法,其中该癌细胞包含MiT易位,该方法包括提供有效量的MiT拮抗剂。例如,本文中提供的是处理nccRCC癌细胞的方法,其中该癌细胞包含MET过表达和/或BIRC7过表达,该方法包括提供有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是用于在个体中治疗癌症的方法,该方法包括:测定自该个体获得的样品包含一种或多种生物标志物,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该癌症得到治疗。例如,本文中提供的是用于在个体中治疗癌症的方法,该方法包括:测定自该个体获得的样品包含MiT过表达和/或MiT易位,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该癌症得到治疗。
在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是用于在个体中治疗nccRCC的方法,该方法包括:测定自该个体获得的样品包含一种或多种生物标志物,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该nccRCC得到治疗。例如,本文中提供的是用于在个体中治疗nccRCC的方法,该方法包括:测定自该个体获得的样品包含MiT过表达和/或MiT易位,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该nccRCC得到治疗。例如,本文中提供的是用于在个体中治疗nccRCC的方法,该方法包括:测定自该个体获得的样品包含MET过表达和/或BIRC7过表达,并对该个体施用有效量的包含MiT拮抗剂的抗癌疗法,由此该nccRCC得到治疗。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中还提供的是治疗癌症的方法,其包括:(a)选择具有癌症的个体,其中该癌症包含一种或多种生物标志物;并(b)对如此选择的个体施用有效量的MiT拮抗剂,由此该癌症得到治疗。例如,本文中还提供的是治疗癌症的方法,其包括:(a)选择具有癌症的个体,其中该癌症包含MiT易位;并(b)对如此选择的个体施用效量的MiT拮抗剂,由此该癌症得到治疗。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中还提供的是治疗nccRCC的方法,其包括:(a)选择具有nccRCC的个体,其中该nccRCC包含一种或多种生物标志物;并(b)对如此选择的个体施用有效量的MiT拮抗剂,由此该nccRCC得到治疗。例如,本文中还提供的是治疗nccRCC的方法,其包括:(a)选择具有nccRCC的个体,其中该nccRCC包含MiT易位;并(b)对如此选择的个体施用有效量的MiT拮抗剂,由此该nccRCC得到治疗。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。例如,本文中还提供的是治疗nccRCC的方法,其包括:(a)选择具有nccRCC的个体,其中该nccRCC包含MET过表达和/或BIRC7过表达;并(b)对如此选择的个体施用有效量的MiT拮抗剂,由此该nccRCC得到治疗。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中进一步提供的是鉴定更有可能或不太可能展现受益于用抗癌疗法治疗的具有癌症的个体的方法,该方法包括:测定自该个体获得的样品中一种或多种生物标志物的存在或缺失,其中该样品中该一种或多种生物标志物的存在指示该个体更有可能展现受益于用该抗癌疗法治疗或该一种或多种生物标志物的缺失指示该个体不太可能展现受益于用该抗癌疗法治疗。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,本文中提供的是鉴定更有可能或不太可能展现受益于用包含MiT拮抗剂的抗癌疗法治疗的具有癌症的个体的方法,该方法包括:测定自该个体获得的样品中MiT过表达和/或MiT易位的存在或缺失,其中该样品中该MiT过表达和/或MiT易位的存在指示该个体更有可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗或该MiT过表达和/或MiT易位的缺失指示该个体不太可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中进一步提供的是鉴定更有可能或不太可能展现受益于用抗癌疗法治疗的具有nccRCC的个体的方法,该方法包括:测定自该个体获得的样品中一种或多种生物标志物的存在或缺失,其中该样品中该一种或多种生物标志物的存在指示该个体更有可能展现受益于用该抗癌疗法治疗或该一种或多种生物标志物的缺失指示该个体不太可能展现受益于用该抗癌疗法治疗。在一些实施方案中,该抗癌疗法包含MiT拮抗剂。例如,本文中提供的是鉴定更有可能或不太可能展现受益于用包含MiT拮抗剂的抗癌疗法治疗的具有nccRCC的个体的方法,该方法包括:测定自该个体获得的样品中MiT过表达和/或MiT易位的存在或缺失,其中该样品中该MiT过表达和/或MiT易位的存在指示该个体更有可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗或该MiT过表达和/或MiT易位的缺失指示该个体不太可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗。例如,本文中提供的是鉴定更有可能或不太可能展现受益于用包含MiT拮抗剂的抗癌疗法治疗的具有nccRCC的个体的方法,该方法包括:测定自该个体获得的样品中MET过表达和/或BIRC7过表达的存在或缺失,其中该样品中该MiT过表达和/或MiT易位的存在指示该个体更有可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗或该MiT过表达和/或MiT易位的缺失指示该个体不太可能展现受益于用包含该MiT拮抗剂的该抗癌疗法治疗。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是用于预测具有癌症的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定一种或多种生物标志物,由此该一种或多种生物标志物的存在指示该个体更有可能有效响应用该MiT拮抗剂治疗且该一种或多种生物标志物的缺失指示该个体不太可能有效响应用该MiT拮抗剂治疗。例如,本文中提供的是用于预测具有癌症的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定MiT过表达和/或MiT易位,由此该MiT过表达和/或MiT易位的存在指示该个体更有可能有效响应用该MiT拮抗剂治疗且该MiT过表达和/或MiT易位的缺失指示该个体不太可能有效响应用该MiT拮抗剂治疗。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是用于预测具有nccRCC的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定一种或多种生物标志物,由此该一种或多种生物标志物的存在指示该个体更有可能有效响应用该MiT拮抗剂治疗且该一种或多种生物标志物的缺失指示该个体不太可能有效响应用该MiT拮抗剂治疗。例如,本文中提供的是用于预测具有nccRCC的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定MiT过表达和/或MiT易位,由此该MiT过表达和/或MiT易位的存在指示该个体更有可能有效响应用该MiT拮抗剂治疗且该MiT过表达和/或MiT易位的缺失指示该个体不太可能有效响应用该MiT拮抗剂治疗。例如,本文中提供的是用于预测具有nccRCC的个体是否更有可能或不太可能有效响应用包含MiT拮抗剂的抗癌疗法治疗的方法,该方法包括测定MET过表达和/或BIRC7过表达,由此该MET过表达和/或BIRC7过表达的存在指示该个体更有可能有效响应用该MiT拮抗剂治疗且该MiT过表达和/或MiT易位的缺失指示该个体不太可能有效响应用该MiT拮抗剂治疗。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是预测具有癌症的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,其包括检测自该个体获得的样品中一种或多种生物标志物的存在或缺失,其中该一种或多种生物标志物的存在预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且该一种或多种生物标志物的缺失预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。例如,本文中提供的是预测具有癌症的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,其包括检测自该个体获得的样品中MiT过表达和/或MiT易位的存在或缺失,其中该MiT过表达和/或MiT易位的存在预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且该MiT过表达和/或MiT易位的缺失预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
本文中提供的是预测具有nccRCC的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,其包括检测自该个体获得的样品一种或多种生物标志物的存在或缺失,其中该一种或多种生物标志物的存在预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且该一种或多种生物标志物的缺失预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。例如,本文中提供的是预测具有nccRCC的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,其包括检测自该个体获得的样品中MiT过表达和/或MiT易位的存在或缺失,其中该MiT过表达和/或MiT易位的存在预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且该MiT过表达和/或MiT易位的缺失预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。例如,本文中提供的是预测具有nccRCC的个体对包含MiT拮抗剂的抗癌疗法有响应或无响应的方法,其包括检测自该个体获得的样品中MET表达和/或BIRC7表达的存在或缺失,其中该MET表达和/或BIRC7表达的存在预示该个体对包含该MiT拮抗剂的该抗癌疗法有响应且该MET表达和/或BIRC7表达的缺失预示该个体对包含该MiT拮抗剂的该抗癌疗法无响应。在一些实施方案中,该方法进一步包括施用有效量的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MET,HIF1A,APEX1,和BIRC7的基因。在一些实施方案中,一种或多种生物标志物的存在包含一种或多种选自MET,HIF1A,APEX1,和BIRC7的基因的变异(例如多态性或突变)的存在。在一些实施方案中,该变异(例如多态性或突变)是体细胞变异(例如多态性或突变)。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)。在一些实施方案中,该一种或多种生物标志物的存在通过一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MITF。在一些实施方案中,该一种或多种生物标志物包含TFEB。在一些实施方案中,该一种或多种生物标志物包含TFE3。在一些实施方案中,该一种或多种生物标志物包含TFEC。在一些实施方案中,该一种或多种生物标志物包含SBNO2。
在任何方法的一些实施方案中,该一种或多种生物标志物包含MET和/或BIRC7。在一些实施方案中,该一种或多种生物标志物的存在通过MET和/或BIRC7的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MET。在一些实施方案中,该一种或多种生物标志物包含BIRC7。
在任何方法的一些实施方案中,该一种或多种生物标志物包含MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在通过MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7中一种或多种的升高的表达水平(例如与参照相比)的存在来指示。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如倒位,重排和/或融合)。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)的存在。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种的过表达的存在。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是MITF易位(例如重排和/或融合)。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:13和/或30。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:30。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ IDNO:9,10,11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。在一些实施方案中,该ASP3S1-MITF易位存在于透明细胞RCC中。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是TFEB易位(例如重排和/或融合)。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含SEQ ID NO:19。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:15,16,17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是由CLTC启动子驱动的。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是SBNO2易位(例如重排和/或融合)。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN和SBNO2。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含SEQ ID NO:25。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:23和/或24的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:21,22,23,和/或25的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是由CLTC启动子驱动的。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的MiT表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的MiT活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的MET表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的MET活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的BIRC7表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的BIRC7活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MiT表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位突变导致升高的MiT活性和/或活化(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MITF突变是下述一种或多种:G259A,A260G,A260G,T403G,G426C,A/T(-3),G712A,G1120A。在一些实施方案中,该MITF突变是下述一种或多种:E318K,I212M和E213D4TΔ2B,L135V,L142F,G244R,D380N。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MET表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MET活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的BIRC7表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的BIRC7活性和/或活化(例如与没有该MiT易位的参照相比)。
在任何易位(例如重排和/或融合)的一些实施方案中,该易位(例如重排和/或融合)是体细胞易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是染色体内易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是染色体间易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是倒位。在一些实施方案中,该易位(例如重排和/或融合)是删除。在一些实施方案中,该易位(例如重排和/或融合)是功能性易位融合多核苷酸(例如功能性MiT易位融合多核苷酸)和/或功能性易位融合多肽(例如功能性MiT易位融合多肽)。在一些实施方案中,该功能性易位融合多肽(例如功能性MiT易位融合多肽)活化已知受到该易位基因之一调控的途径(例如BIRC7途径)。在一些实施方案中,测定途径活化的方法是本领域已知的且包括如本文中描述的萤光素酶报告物测定法。
癌症和癌细胞的例子包括但不限于癌,淋巴瘤,母细胞瘤(包括髓母细胞瘤和视网膜母细胞瘤),肉瘤(包括脂肪肉瘤和滑膜细胞肉瘤),神经内分泌肿瘤(包括类癌瘤,胃泌素瘤,和胰岛细胞癌),间皮瘤,许旺细胞瘤(包括听神经瘤),脑脊膜瘤,腺癌,黑素瘤,和白血病或淋巴样恶性。此类癌症的更具体例子包括权利要求81的方法,其中该MiT拮抗剂结合。
可以基于本领域中已知的任何适宜标准定性和/或定量地测定生物标志物(例如MiT易位或MiT突变)的存在和/或表达水平/量,所述标准包括但不限于DNA,mRNA,cDNA,蛋白质,蛋白质片段和/或基因拷贝数。在某些实施方案中,第一样品中生物标志物的存在和/或表达水平/量相比于第二样品中的存在/缺失和/或表达水平/量升高。在某些实施方案中,第一样品中生物标志物的存在/缺失和/或表达水平/量相比于第二样品中的存在和/或表达水平/量降低。在某些实施方案中,第二样品是参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织。本文中描述了用于测定基因的存在/缺失和/或表达水平/量的其他公开内容。
在任何方法的一些实施方案中,升高的表达指生物标志物(例如蛋白质或核酸(例如基因或mRNA))水平中相比于参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织的任意的约10%,20%,30%,40%,50%,60%,70%,80%,90%,95%,96%,97%,98%,99%或更高的总体增加,其通过标准的本领域已知方法诸如本文中描述的那些检测。在某些实施方案中,升高的表达指样品中生物标志物的表达水平/量的增加,其中所述增加是参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织中相应生物标志物表达水平/量的任意的至少约1.5倍,1.75倍,2倍,3倍,4倍,5倍,6倍,7倍,8倍,9倍,10倍,25倍,50倍,75倍或100倍。在一些实施方案中,升高的表达指相比参照样品,参照细胞,参照组织,对照样品,对照细胞,对照组织或内部对照(例如持家基因)高约1.5倍,约1.75倍,约2倍,约2.25倍,约2.5倍,约2.75倍,约3.0倍或约3.25倍的总体增加。
在任何方法的一些实施方案中,降低的表达指生物标志物(例如蛋白质或核酸(例如基因或mRNA))水平中相比于参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织的任意的约10%,20%,30%,40%,50%,60%,70%,80%,90%,95%,96%,97%,98%,99%或更高的总体降低,其通过标准的本领域已知方法诸如本文中描述的那些检测。在某些实施方案中,降低的表达指样品中生物标志物的表达水平/量的降低,其中所述降低是参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织中相应生物标志物表达水平/量的任意的至少约0.9倍,0.8倍,0.7倍,0.6倍,0.5倍,0.4倍,0.3倍,0.2倍,0.1倍,0.05倍或0.01倍。
在一个方面,提供的是用于测定MiT易位表达(测定MiT易位的存在)的方法,其包括测定来自患者的(例如来自患者的癌症的)样品是否具有MiT易位的步骤。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)。在一些实施方案中,该一种或多种生物标志物的存在通过一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MITF。在一些实施方案中,该一种或多种生物标志物包含TFEB。在一些实施方案中,该一种或多种生物标志物包含TFE3。在一些实施方案中,该一种或多种生物标志物包含TFEC。在一些实施方案中,该一种或多种生物标志物包含SBNO2。
在任何方法的一些实施方案中,该一种或多种生物标志物包含MET和/或BIRC7。在一些实施方案中,该一种或多种生物标志物的存在通过MET和/或BIRC7的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MET。在一些实施方案中,该一种或多种生物标志物包含BIRC7。
在任何方法的一些实施方案中,该一种或多种生物标志物包含MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7)中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在通过MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7中的一种或多种的升高的表达水平(例如与参照相比)的存在来指示。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)的存在。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种的过表达的存在。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是MITF易位(例如重排和/或融合)。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:13和/或30。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:30。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:9,10,11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。在一些实施方案中,该ASP3S1-MITF易位存在于透明细胞RCC中。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是TFEB易位(例如重排和/或融合)。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含SEQ ID NO:19。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:15,16,17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是由CLTC启动子驱动的。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是SBNO2易位(例如重排和/或融合)。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN和SBNO2。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含SEQ ID NO:25。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:23和/或24的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:21,22,23,和/或25的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是由CLTC启动子驱动的。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的MiT表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的MiT活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的MET表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的MET活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)易位(例如重排和/或融合)导致升高的BIRC7表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位(例如重排和/或融合)导致升高的BIRC7活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MiT表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT易位突变导致升高的MiT活性和/或活化(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MITF突变是下述一种或多种:G259A,A260G,A260G,T403G,G426C,A/T(-3),G712A,G1120A。在一些实施方案中,该MITF突变是下述一种或多种:E318K,I212M和E213D4TΔ2B,L135V,L142F,G244R,D380N。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MET表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的MET活性和/或活化(例如与没有该MiT易位的参照相比)。
在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的BIRC7表达水平(例如与没有该MiT易位的参照相比)。在一些实施方案中,该MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)突变导致升高的BIRC7活性和/或活化(例如与没有该MiT易位的参照相比)。
在任何易位(例如重排和/或融合)的一些实施方案中,该易位(例如重排和/或融合)是体细胞易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是染色体内易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是染色体间易位(例如重排和/或融合)。在一些实施方案中,该易位(例如重排和/或融合)是倒位。在一些实施方案中,该易位(例如重排和/或融合)是删除。在一些实施方案中,该易位(例如重排和/或融合)是功能性易位融合多核苷酸(例如功能性MiT易位融合多核苷酸)和/或功能性易位融合多肽(例如功能性MiT易位融合多肽)。在一些实施方案中,该功能性易位融合多肽(例如功能性MiT易位融合多肽)活化已知受到该易位基因之一调控的途径(例如BIRC7途径)。在一些实施方案中,测定途径活化的方法是本领域已知的且包括如本文中描述的萤光素酶报告物测定法。
可通过许多方法学来分析样品中各种生物标志物的存在和/或表达水平/量,其中许多是本领域中已知且熟练技术人员理解的,包括但不限于免疫组织化学(“IHC”),Western印迹分析,免疫沉淀,分子结合测定法,ELISA,ELIFA,荧光激活细胞分选(“FACS”),MassARRAY,蛋白组学,基于血液的定量测定法(如例如血清ELISA),生化酶活性测定法,原位杂交,Southern分析,Northern分析,全基因组测序,聚合酶链式反应(“PCR”)(包括定量实时PCR(“qRT-PCR”)和其他扩增类型检测方法,如例如分支的DNA,SISBA,TMA等),RNA-Seq,FISH,微阵列分析,基因表达概况分析,和/或基因表达系列分析(“SAGE”),以及可通过蛋白质,基因和/或组织阵列分析实施的许多种测定法中的任一种。用于评估基因和基因产物状态的典型方案见于例如Ausubel et al.,eds.,1995,Current Protocols In Molecular Biology,Units 2(Northern Blotting),4(Southern Blotting),15(Immunoblotting)和18(PCR Analysis)。还可使用多重免疫测定法如那些可从Rules Based Medicine或Meso Scale Discovery(“MSD”)获得的。
在一些实施方案中,使用包括以下的方法测定生物标志物的存在和/或表达水平/量:(a)在样品(如受试者癌症样品)上实施基因表达概况分析,PCR(诸如rtPCR),RNA-seq,微阵列分析,SAGE,MassARRAY技术,或FISH;并(b)测定生物标志物在样品中的存在和/或表达水平/量。在一些实施方案中,微阵列方法包括使用微阵列芯片,其具有一种或多种能在严格条件下与编码上文所述基因的核酸分子杂交的核酸分子或具有一种或多种能与一种或多种由上文所述基因编码的蛋白质结合的多肽(诸如肽或抗体)。在一个实施方案中,PCR方法是qRT-PCR。在一个实施方案中,PCR方法是多重PCR。在一些实施方案中,通过微阵列测量基因表达。在一些实施方案中,通过qRT-PCR测量基因表达。在一些实施方案中,通过多重PCR来测量表达。
用于评估细胞中mRNA的方法是众所周知的,包括例如使用互补DNA探针的杂交测定法(诸如使用经标记的特异于一种或多种基因的核糖核酸探针的原位杂交,Northern印迹和相关技术)和各种核酸扩增测定法(诸如使用特异于一种或多种基因的互补引物的RT-PCR,及其它扩增型检测方法,诸如例如分支DNA,SISBA,TMA等)。
可以使用Northern,点印迹或PCR分析方便地对来自哺乳动物的样品测定mRNA。另外,此类方法可以包括一个或多个如下步骤,其容许测定生物学样品中靶mRNA的水平(例如通过同时检查“持家”基因诸如肌动蛋白家族成员的比较性对照mRNA序列的水平)。任选的是,可以测定所扩增靶cDNA的序列。
本发明的任选方法包括通过微阵列技术在组织或细胞样品中检查或检测mRNA如靶mRNA的方案。使用核酸微阵列,将来自测试和对照组织样品的测试和对照mRNA样品逆转录并标记以生成cDNA探针。然后,将探针与固定化于固体支持物上的核酸阵列杂交。阵列配置为使得阵列每个成员的序列和位置是已知的。例如,可将其表达与抗血管生成疗法的增加或降低的临床益处相关的基因选择集在固体支持物上呈阵列。经标记探针与特定阵列成员的杂交指示得到该探针的样品表达该基因。
依照一些实施方案,通过观察前述基因的蛋白质表达水平来测量存在和/或表达水平/量。在某些实施方案中,所述方法包括使生物学样品与针对本文所述生物标志物的抗体(例如抗MiT抗体,抗MiT易位抗体)在允许生物标志物结合的条件下接触,并检测抗体与生物标志物之间是否形成复合物。这类方法可以是体外或体内方法。在一个实施方案中,使用抗体来选择符合使用MiT拮抗剂(特别是MiT易位拮抗剂)疗法的资格的受试者,例如用于选择个体的生物标志物。
在某些实施方案中,使用IHC和染色方案来检查样品中生物标志物蛋白质的存在和/或表达水平/量。已显示对组织切片的IHC染色是一种测定或检测样品中蛋白质的存在的可靠方法。在一个方面,使用包括下述的方法来测定生物标志物的表达水平:(a)用抗体对样品(诸如受试者癌症样品)实施IHC分析;和(b)测定样品中生物标志物的表达水平。在一些实施方案中,IHC染色强度是相对于参照值确定的。
IHC可与另外的技术如形态学染色和/或荧光原位杂交组合实施。IHC有两种一般性方法;直接和间接测定法。依照第一种测定法,直接测定抗体对靶抗原的结合。该直接测定法使用经标记的试剂,如荧光标签或酶标记的一抗,其可以在没有别的抗体相互作用的情况下可视化。在典型的间接测定法中,未缀合的一抗结合抗原,然后经标记的二抗结合该一抗。在二抗缀合于酶标记物的情况下,添加生色或产荧光底物以提供抗原的可视化。发生信号扩增,因为几个二抗可以与一抗上的不同表位反应。
用于IHC的一抗和/或二抗通常用可检测模块标记。存在大量标记,其一般可分组成以下类别:(a)放射性同位素,如35S,14C,125I,3H和131I;(b)胶体金颗粒;(c)荧光标记物,包括但不限于稀土螯合物(铕螯合物),Texas Red,罗丹明,荧光素,丹酰,丽丝胺(Lissamine),伞形酮(umbelliferone),藻红蛋白(phycocrytherin),藻蓝蛋白,或商品化的荧光团如SPECTRUM ORANGE7和SPECTRUM GREEN7和/或上述任意一种或多种的衍生物;(d)存在各种酶-底物标记物,且美国专利No.4,275,149提供了对其中一些的综述。酶标记物的例子包括萤光素酶(例如萤火虫萤光素酶和细菌萤光素酶;美国专利No.4,737,456),萤光素,2,3-二氢酞嗪二酮(dihydrophthalazinedione),苹果酸脱氢酶,脲酶,过氧化物酶如辣根过氧化物酶(HRPO),碱性磷酸酶,β-半乳糖苷酶,葡糖淀粉酶,溶菌酶,糖氧化酶(例如葡萄糖氧化酶,半乳糖氧化酶,和葡萄糖-6-磷酸脱氢酶),杂环氧化酶(如尿酸酶和黄嘌呤氧化酶),乳过氧化物酶(lactoperoxidase),微过氧化物酶(microperoxidase)等。
酶-底物组合的例子包括,例如辣根过氧化物酶(HRPO)和作为底物的过氧化氢酶;碱性磷酸酶(AP)和作为生色底物的对硝基苯基磷酸;和β-D-半乳糖苷酶(β-D-Gal)与生色底物(例如对硝基苯基-β-D-半乳糖苷酶)或产荧光底物(例如4-甲基伞形基(methylumbelliferyl)-β-D-半乳糖苷酶)。对于这些的一般性综述,参见美国专利No.4,275,149和4,318,980。
可将如此制备的标本封固并盖上盖玻片。然后确定载玻片评估,例如使用显微镜,并可采用本领域中常规使用的染色强度标准。在一些实施方案中,约1+或更高的染色模式得分是诊断性和/或预后性的。在某些实施方案中,IHC测定法中约2+或更高的染色模式得分是诊断性和/或预后性的。在其它实施方案中,约3或更高的染色模式得分是诊断性和/或预后性的。在一个实施方案中,理解当使用IHC检查来自肿瘤或结肠腺瘤的细胞和/或组织时,通常在肿瘤细胞和/或组织中测定或评估染色(相对于可能存在于样品中的基质或周围组织)。
在备选的方法中,可使样品与特异于所述生物标志物的抗体(例如抗MiT抗体,抗MiT易位抗体)在足以形成抗体-生物标志物复合物的条件下接触,然后检测该复合物。可以许多途径来检测生物标志物的存在,如通过Western印迹和ELISA规程,其用于测定很多种组织和样品,包括血浆或血清。有大量使用这类测定形式的免疫测定技术,参见例如美国专利No.4,016,043,4,424,279和4,018,653。这些包括非竞争型的单位点和双位点二者或“三明治式”测定法,以及传统的竞争性结合测定法。这些测定法还包括经标记抗体对靶生物标志物的直接结合。
例示性的MITF抗体包括c5(abcam),D5(abcam),HPA003259(Sigma),和本文中描述和例示的别的抗MITF抗体。例示性的TFEB抗体包括ab113372(abcam),ab113372(abcam),LS-B5907(LSBio),和本文中描述和例示的别的抗TFEB抗体。例示性的TFEC抗体包括13547-1-AP(proteinTech),和本文中描述和例示的别的抗TFEC抗体。例示性的TFE3抗体包括MRQ-37(Ventana),HPA023881(Sigma),和本文中描述和例示的别的抗TFE3抗体。例示性的MET抗体包括SP44(Ventana),Met4。例示性的BIRC7抗体包括1D12(OriGene)。
选定的生物标志物在组织或细胞样品中的存在和/或表达水平/量还可经由功能性或基于活性的测定法来检查。例如,如果生物标志物是酶,可以进行本领域中已知的测定法来测定或检测给定的酶活性在组织或细胞样品中的存在。
在某些实施方案中,针对测定的生物标志物的量中的差异和使用的样品质量中的可变性,以及测定轮数之间的变异性两者将样品标准化。这类标准化可通过检测并纳入特定标准化生物标志物(包括公知的看家基因如ACTB)表达来实现。或者,标准化可基于所有测定基因或其较大子集的均值或中值信号(全局标准化办法)。在一个基因接一个基因的基础上,将受试者肿瘤mRNA或蛋白质的测量的经标准化的量与在参照集中发现的量比较。每种mRNA或蛋白质每份测试肿瘤每位受试者的经标准化表达水平可表示为在参照集中测量的表达水平的百分数。在要分析的特定受试者样品中测量的存在和/或表达水平/量将落在该范围内的某个百分数处,这可通过本领域中公知的方法来测定。
在某些实施方案中,如下测定基因的相对表达水平:
相对表达基因1样品1=2exp(Ct看家基因–Ct基因1),Ct在样品中测定。
相对表达基因1参照RNA=2exp(Ct看家基因–Ct基因1),Ct在参照样品中测定。
标准化的相对表达基因1样品1=(相对表达基因1样品1/相对表达基因1参照RNA)x 100
Ct是阈值循环。Ct是反应中生成的荧光与阈值线相交的循环数。
将所有实验均对参照RNA标准化,参照RNA是来自各种组织来源的RNA的广泛混合物(例如参照RNA#636538,来自Clontech,Mountain View,CA)。将等同的参照RNA包含在每次qRT-PCR运行中,从而允许在不同的实验运行之间比较结果。
在一个实施方案中,所述样品是临床样品。在另一个实施方案中,所述样品用在诊断性测定法中。在一些实施方案中,所述样品从原发性或转移性肿瘤获得。经常使用组织活检来获得肿瘤组织的代表性的片/块。或者,可以以已知或认为含有感兴趣肿瘤细胞的组织或流体的形式间接获得肿瘤细胞。例如,可通过切除,支气管镜检,细针抽吸,支气管刷检,或从痰,胸膜液或血液获得肺癌损伤的样品。可从癌症或肿瘤组织或从其他身体样品如尿液,痰,血清或血浆检测基因或基因产物。上文论述的用于检测癌性样品中靶基因或基因产物的相同技术可应用于其他身体样品。癌细胞可能从癌损伤脱落并出现在这类身体样品中。通过筛选这类身体样品,可实现对这些癌症的简单的早期诊断。另外,通过测试这类身体样品中的靶基因或基因产物能更容易地监测治疗的进展。
在某些实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织是来自同一受试者或个体的单一样品或组合的多重样品,其在不同于获得测试样品时的一个或多个时间点获得。例如,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织在早于获得测试样品时的时间点从同一受试者或个体获得。如果参照样品在癌症的初始诊断期间获得而测试样品在癌症变成转移性时的更晚时候获得,那么这类参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织可以是有用的。
在某些实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织是来自一个或多个并非该受试者或个体的健康个体的组合的多重样品。在某些实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织是来自患有疾病或病症(例如癌症)的,并非该受试者或个体的一个或多个个体的组合的多重样品。在某些实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织是来自并非该受试者或个体的一个或多个个体的正常组织的合并RNA样品或合并的血浆或血清样品。在某些实施方案中,参照样品,参照细胞,参照组织,对照样品,对照细胞或对照组织是来自患有疾病或病症(例如癌症)的,并非该受试者或个体的一个或多个个体的肿瘤组织的合并RNA样品或合并的血浆或血清样品。
在任何方法的一些实施方案中,该MiT拮抗剂是MiT易位拮抗剂。在任何方法的一些实施方案中,该MiT拮抗剂,特别是MiT易位拮抗剂是抗体,结合多肽,结合小分子,或多核苷酸。在一些实施方案中,该MiT拮抗剂,特别是MiT易位拮抗剂是抗体。在一些实施方案中,该抗体是单克隆抗体。在一些实施方案中,该抗体是人,人源化,或嵌合抗体。在一些实施方案中,该抗体是抗体片段且该抗体片段结合MiT多肽,特别是MiT拮抗剂和/或MiT易位融合多肽。
在任何方法的一些实施方案中,依照任何上述实施方案的个体可以是人。
在任何方法的一些实施方案中,该方法包括对具有此类癌症的个体施用有效量的MiT拮抗剂,特别是MiT易位拮抗剂。在一个此类实施方案中,该方法进一步包括对该个体施用有效量的至少一种下文所述别的治疗剂。在一些实施方案中,个体可以是人。
可以在疗法中单独或与其它药剂组合使用本文中描述的MiT拮抗剂,特别是MiT易位拮抗剂。例如,可以与至少一种别的治疗剂(包括另一种MiT拮抗剂)共施用本文中描述的MiT拮抗剂,特别是MiT易位拮抗剂。在某些实施方案中,别的治疗剂是化疗剂。
上文记载的此类组合疗法涵盖组合施用(其中两种或更多种治疗剂包含在同一配制剂或分开的配制剂中),和分开施用(在该情况中MiT拮抗剂,特别是MiT易位拮抗剂可以在别的治疗剂和/或佐剂施用之前,同时,和/或之后施用)。MiT拮抗剂,特别是MiT易位拮抗剂还可以与放射疗法组合使用。
可以通过任何合适的手段,包括胃肠外,肺内,和鼻内,及若期望用于局部治疗的话,损伤内施用来施用本文中描述的MiT拮抗剂,特别是MiT易位拮抗剂(例如抗体,结合多肽,和/或小分子)(及任何别的治疗剂)。胃肠外输注包括肌肉内,静脉内,动脉内,腹膜内,或皮下施用。部分根据施用是短暂的还是长期的,给药可以通过任何合适的路径,例如通过注射,诸如静脉内或皮下注射进行。本文中涵盖各种给药日程表,包括但不限于单次施用或在多个时间点里的多次施用,推注施用,和脉冲输注。
本文中描述的MiT拮抗剂,特别是MiT易位拮抗剂(例如抗体,结合多肽,和/或小分子)可以一种符合良好的医学实践的方式配制,确定剂量及施用。在此背景中考虑的因素包括在治疗的特定病症,在治疗的特定哺乳动物,个体的临床状态,病症原因,药剂递送部位,施用方法,施用日程以及其它为从业医生所知的因素。MiT拮抗剂,特别是MiT易位拮抗剂无需但可任选地与一种或多种目前用于预防或治疗所述病症的药剂一起配制。这类其它药剂的有效量取决于配方中所存在的MiT拮抗剂(特别是MiT易位拮抗剂)的量,病症或治疗的类型,以及其它上述讨论的因素。这些药剂通常以相同的剂量使用并具有本文中所描述的施用途径,或以约1-99%的本文所描述的剂量使用,或以任何剂量并通过任何途径使用,所述剂量和途径是凭经验确定的/经临床测定合适的。
为了预防或治疗疾病,本文中描述的MiT拮抗剂(特别是MiT易位拮抗剂)(当单独或与一种或多种其它别的治疗剂联合使用时)的合适剂量会取决于所要治疗的疾病的类型,疾病的严重性和病程,施用MiT拮抗剂(特别是MiT易位拮抗剂)是出于预防还是治疗目的,之前的疗法,受试者的临床史和对MiT拮抗剂的响应,以及主治医师的斟酌决定。MiT拮抗剂(特别是MiT易位拮抗剂)适合于在一次或一系列的治疗中给予个体。一个典型的日剂量可在约1μg/kg-100mg/kg或更多的范围内,取决于上文所述因素。对于在数天或更长时间内的重复施用,根据状况,治疗一般将持续直至发生期望的对疾病症状的抑制。这类剂量可间歇施用,如每周或每三周施用,例如使得个体接受约2-约20剂,或例如约6剂的MiT拮抗剂。可施用初始较高的负荷剂量,接着施用一个或多个较低的剂量。例示性的给药方案包括施用。然而,可使用其它给药方案。通过常规技术和测定法易于监测该治疗的进展。
理解的是,任何上述配制剂或治疗性方法可使用本发明的免疫缀合物代替或补充MiT拮抗剂,特别是MiT易位拮抗剂来进行。
III.治疗性组合物
本文中提供的是在本文所述方法中有用的MiT拮抗剂。在一些实施方案中,该MiT拮抗剂是抗体,结合多肽,结合小分子,和/或多核苷酸。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7拮抗剂。
抗体拮抗剂
在一些实施方案中,该MET途径拮抗剂是抗体。在一个实施方案中,该药物是抗体,包括但不限于结合人c-met的抗体。在一些实施方案中,该抗体干扰(例如阻断)c-met结合肝细胞生长因子(HGF)。在一些实施方案中,该抗体结合c-met。在一些实施方案中,该抗体结合HGF。在一个实施方案中,该抗c-met抗体是奥奴珠单抗(onartuzumab)。本文中描述了且本领域知道适合用于本发明方法的其它抗c-met抗体。例如,WO05/016382中公开的抗c-met抗体(包括但不限于抗体13.3.2,9.1.2,8.70.2,8.90.3);由以ICLC编号PD 03001保藏于CBA(Genoa)的杂交瘤细胞系生成或识别HGF受体β链胞外域上表位(所述表位与该单克隆抗体识别的表位相同)的抗c-met抗体;WO2007/126799中公开的抗c-met抗体(包括但不限于04536,05087,05088,05091,05092,04687,05097,05098,05100,05101,04541,05093,05094,04537,05102,05105,04696,04682);WO2009/007427中公开的抗c-met抗体(包括但不限于于2007年3月14日以编号I-3731,于2007年3月14日以编号I-3732,于2007年7月6日以编号I-3786,于2007年3月14日以编号I-3724保藏于CNCM(Institut Pasteur,Paris,France)的抗体);20110129481中公开的抗c-met抗体;US20110104176中公开的抗c-met抗体;WO2009/134776中公开的抗c-met抗体;WO2010/059654中公开的抗c-met抗体;WO2011020925中公开的抗c-met抗体(包括但不限于自于2008年3月12日以编号I-3949保藏于CNCM(Institut Pasteur,Paris,France)的杂交瘤和于2010年1月14日以编号I-4273保藏的杂交瘤分泌的抗体)。
在一些实施方案中,MET途径拮抗剂是抗肝细胞生长因子(HGF)抗体,包括但不限于人源化抗HGF抗体TAK701,rilotumumab,Ficlatuzumab,和/或WO2007/143090中描述的人源化抗体2B8。在一些实施方案中,抗HGF抗体是US7718174B2中描述的抗HGF抗体。
在一些实施方案中,BIRC7拮抗剂是抗BIRC7抗体。例示性抗体是本领域已知的。
在又一个方面,抗体实施方案,特别是依照任何上述实施方案的,可单一地或组合地掺入下文各节中描述的任何特征:
抗体亲和力
在某些实施方案中,本文中提供的抗体具有≤1μM的解离常数(Kd)。在一个实施方案中,Kd是通过如下述测定法所述用Fab型式的感兴趣抗体及其抗原实施的放射性标记抗原结合测定法(RIA)来测量的。通过在存在未标记抗原的滴定系列的情况中用最小浓度的(125I)标记抗原平衡Fab,然后用抗Fab抗体包被板捕捉结合的抗原来测量Fab对抗原的溶液结合亲和力(见例如Chen等,J.Mol.Biol.293:865-881(1999))。为了建立测定法的条件,将多孔板(Thermo Scientific)用50mM碳酸钠(pH 9.6)中的5μg/ml捕捉用抗Fab抗体(Cappel Labs)包被过夜,随后用PBS中的2%(w/v)牛血清清蛋白于室温(约23℃)封闭2-5小时。在非吸附板(Nunc#269620)中,将100pM或26pM[125I]-抗原与连续稀释的感兴趣Fab(例如与Presta等,Cancer Res.57:4593-4599(1997)中抗VEGF抗体,Fab-12的评估一致)混合。然后将感兴趣的Fab温育过夜;然而,温育可持续更长时间(例如约65小时)以确保达到平衡。此后,将混合物转移至捕捉板,于室温温育(例如1小时)。然后除去溶液,并用PBS中的0.1%聚山梨酯20洗板8次。平板干燥后,加入150μl/孔闪烁液(MICROSCINT-20TM;Packard),然后在TOPCOUNTTM伽马计数器(Packard)上对平板计数10分钟。选择各Fab给出小于或等于最大结合之20%的浓度用于竞争性结合测定法。
依照另一个实施方案,Kd是使用表面等离振子共振测定法使用或(BIAcore,Inc.,Piscataway,NJ)于25℃使用固定化抗原CM5芯片在约10个响应单位(RU)测量的。简言之,依照供应商的用法说明书用盐酸N-乙基-N’-(3-二甲氨基丙基)-碳二亚胺(EDC)和N-羟基琥珀酰亚胺(NHS)活化羧甲基化右旋糖苷生物传感器芯片(CM5,BIACORE,Inc.)。将抗原用10mM乙酸钠pH 4.8稀释至5μg/ml(约0.2μM),然后以5μl/分钟的流速注射以获得约10个响应单位(RU)的偶联蛋白质。注入抗原后,注入1M乙醇胺以封闭未反应基团。为了进行动力学测量,于25℃以约25μl/分钟的流速注入在含0.05%聚山梨酯20(TWEEN-20TM)表面活性剂的PBS(PBST)中两倍连续稀释的Fab(0.78nM至500nM)。使用简单一对一朗格缪尔(Langmuir)结合模型Evaluation Software version 3.2)通过同时拟合结合和解离传感图计算结合速率(kon)和解离速率(koff)。平衡解离常数(Kd)以比率koff/kon计算。见例如Chen等,J.Mol.Biol.293:865-881(1999)。如果根据上文表面等离振子共振测定法,结合速率超过106M-1S-1,那么结合速率可使用荧光淬灭技术来测定,即根据分光计诸如配备了断流装置的分光光度计(Aviv Instruments)或8000系列SLM-AMINCOTM分光光度计(ThermoSpectronic)中用搅拌比色杯的测量,在存在浓度渐增的抗原的情况中,测量PBS pH 7.2中20nM抗抗原抗体(Fab形式)于25℃的荧光发射强度(激发=295nm;发射=340nm,16nm带通)的升高或降低。
抗体片段
在某些实施方案中,本文中提供的抗体是抗体片段。抗体片段包括但不限于Fab,Fab’,Fab’-SH,F(ab’)2,Fv,和scFv片段,及下文所描述的其它片段。关于某些抗体片段的综述,见Hudson等Nat.Med.9:129-134(2003)。关于scFv片段的综述,见例如Pluckthün,于The Pharmacology of MonoclonalAntibodies,第113卷,Rosenburg和Moore编,(Springer-Verlag,New York),第269-315页(1994);还可见WO 93/16185;及美国专利No.5,571,894和5,587,458。关于包含补救受体结合表位残基,并且具有延长的体内半衰期的Fab和F(ab’)2片段的讨论,见美国专利No.5,869,046。
双抗体是具有两个抗原结合位点的抗体片段,其可以是二价的或双特异性的。见例如EP 404,097;WO 1993/01161;Hudson等,Nat.Med.9:129-134(2003);及Hollinger等,Proc.Natl.Acad.Sci.USA 90:6444-6448(1993)。三抗体和四抗体也记载于Hudson等,Nat.Med.9:129-134(2003)。
单域抗体是包含抗体的整个或部分重链可变域或整个或部分轻链可变域的抗体片段。在某些实施方案中,单域抗体是人单域抗体(Domantis,Inc.,Waltham,MA;见例如美国专利No.6,248,516B1)。
可以通过多种技术,包括但不限于对完整抗体的蛋白水解消化及重组宿主细胞(例如大肠杆菌或噬菌体)的生成来生成抗体片段,如本文中所描述的。
嵌合的和人源化的抗体
在某些实施方案中,本文中提供的抗体是嵌合抗体。某些嵌合抗体记载于例如美国专利No.4,816,567;及Morrison等,Proc.Natl.Acad.Sci.USA,81:6851-6855(1984))。在一个例子中,嵌合抗体包含非人可变区(例如,自小鼠,大鼠,仓鼠,家兔,或非人灵长类,诸如猴衍生的可变区)和人恒定区。在又一个例子中,嵌合抗体是“类转换的”抗体,其中类或亚类已经自亲本抗体的类或亚类改变。嵌合抗体包括其抗原结合片段。
在某些实施方案中,嵌合抗体是人源化抗体。通常,将非人抗体人源化以降低对人的免疫原性,同时保留亲本非人抗体的特异性和亲和力。一般地,人源化抗体包含一个或多个可变域,其中HVR,例如CDR(或其部分)自非人抗体衍生,而FR(或其部分)自人抗体序列衍生。任选地,人源化抗体还会至少包含人恒定区的一部分。在一些实施方案中,将人源化抗体中的一些FR残基用来自非人抗体(例如衍生HVR残基的抗体)的相应残基替代,例如以恢复或改善抗体特异性或亲和力。
人源化抗体及其生成方法综述于例如Almagro和Fransson,Front.Biosci.13:1619-1633(2008),并且进一步记载于例如Riechmann等,Nature332:323-329(1988);Queen等,Proc.Nat’l Acad.Sci.USA 86:10029-10033(1989);美国专利No.5,821,337,7,527,791,6,982,321和7,087,409;Kashmiri等,Methods 36:25-34(2005)(描述了SDR(a-CDR)嫁接);Padlan,Mol.Immunol.28:489-498(1991)(描述了“重修表面”);Dall’Acqua等,Methods 36:43-60(2005)(描述了“FR改组”);及Osbourn等,Methods 36:61-68(2005)和Klimka等,Br.J.Cancer,83:252-260(2000)(描述了FR改组的“引导选择”方法)。
可以用于人源化的人框架区包括但不限于:使用“最佳拟合(best-fit)”方法选择的框架区(见例如Sims等J.Immunol.151:2296(1993));自轻或重链可变区的特定亚组的人抗体的共有序列衍生的框架区(见例如Carter等Proc.Natl.Acad.Sci.USA,89:4285(1992);及Presta等J.Immunol.,151:2623(1993));人成熟的(体细胞突变的)框架区或人种系框架区(见例如Almagro和Fransson,Front.Biosci.13:1619-1633(2008));和通过筛选FR文库衍生的框架区(见例如Baca等,J.Biol.Chem.272:10678-10684(1997)及Rosok等,J.Biol.Chem.271:22611-22618(1996))。
人抗体
在某些实施方案中,本文中提供的抗体是人抗体。可以使用本领域中已知的多种技术来生成人抗体。一般地,人抗体记载于van Dijk和van de Winkel,Curr.Opin.Pharmacol.5:368-74(2001)及Lonberg,Curr.Opin.Immunol.20:450-459(2008)。
可以通过对转基因动物施用免疫原来制备人抗体,所述转基因动物已经修饰为响应抗原性攻击而生成完整人抗体或具有人可变区的完整抗体。此类动物通常含有所有或部分人免疫球蛋白基因座,其替换内源免疫球蛋白基因座,或者其在染色体外存在或随机整合入动物的染色体中。在此类转基因小鼠中,一般已经将内源免疫球蛋白基因座灭活。关于自转基因动物获得人抗体的方法的综述,见Lonberg,Nat.Biotech.23:1117-1125(2005)。还可见例如美国专利No.6,075,181和6,150,584,其描述了XENOMOUSETM技术;美国专利No.5,770,429,其描述了技术;美国专利No.7,041,870,其描述了技术,和美国专利申请公开文本No.US 2007/0061900,其描述了技术)。可以例如通过与不同人恒定区组合进一步修饰来自由此类动物生成的完整抗体的人可变区。
也可以通过基于杂交瘤的方法生成人抗体。已经描述了用于生成人单克隆抗体的人骨髓瘤和小鼠-人异骨髓瘤细胞系(见例如Kozbor J.Immunol.,133:3001(1984);及Boerner等,J.Immunol.,147:86(1991))。经由人B细胞杂交瘤技术生成的人抗体也记载于Li等,Proc.Natl.Acad.Sci.USA,103:3557-3562(2006)。其它方法包括那些例如记载于美国专利No.7,189,826(其描述了自杂交瘤细胞系生成单克隆人IgM抗体)和Ni,Xiandai Mianyixue,26(4):265-268(2006)(其描述了人-人杂交瘤)的。人杂交瘤技术(Trioma技术)也记载于Vollmers和Brandlein,Histology and Histopathology,20(3):927-937(2005)及Vollmers和Brandlein,Methods and Findings inExperimental and Clin.Pharma.,27(3):185-91(2005)。
也可以通过分离自人衍生的噬菌体展示文库选择的Fv克隆可变域序列生成人抗体。然后,可以将此类可变域序列与期望的人恒定域组合。下文描述了自抗体文库选择人抗体的技术。
文库衍生的抗体
可以通过对组合文库筛选具有期望的一种或多种活性的抗体来分离本发明的抗体。例如,用于生成噬菌体展示文库并对此类文库筛选拥有期望结合特征的抗体的多种方法是本领域中已知的。此类方法综述于例如Hoogenboom等于METHODS IN MOL.BIOL.178:1-37(O’Brien等编,Human Press,Totowa,NJ,2001),并且进一步记载于例如McCafferty等,Nature348:552-554;Clackson等,Nature 352:624-628(1991);Marks等,J.Mol.Biol.222:581-597(1992);Marks和Bradbury,于METHODS IN MOL.BIOL.248:161-175(Lo编,Human Press,Totowa,NJ,2003);Sidhu等,J.Mol.Biol.338(2):299-310(2004);Lee等,J.Mol.Biol.340(5):1073-1093(2004);Fellouse,Proc.Natl.Acad.Sci.USA 101(34):12467-12472(2004);及Lee等,J.Immunol.Methods 284(1-2):119-132(2004)。
在某些噬菌体展示方法中,将VH和VL基因的全集分别通过聚合酶链式反应(PCR)克隆,并在噬菌体文库中随机重组,然后可以对所述噬菌体文库筛选抗原结合噬菌体,如记载于Winter等,Ann.Rev.Immunol.,12:433-455(1994)的。噬菌体通常以单链Fv(scFv)片段或以Fab片段展示抗体片段。来自经免疫的来源的文库提供针对免疫原的高亲和力抗体,而不需要构建杂交瘤。或者,可以(例如自人)克隆未免疫全集以在没有任何免疫的情况中提供针对一大批非自身和还有自身抗原的抗体的单一来源,如由Griffiths等,EMBO J,12:725-734(1993)描述的。最后,也可以通过自干细胞克隆未重排的V基因区段,并使用含有随机序列的PCR引物编码高度可变的CDR3区并在体外实现重排来合成生成未免疫文库,如由Hoogenboom和Winter,J.Mol.Biol.,227:381-388(1992)所描述的。描述人抗体噬菌体文库的专利公开文本包括例如:美国专利No.5,750,373,和美国专利公开文本No.2005/0079574,2005/0119455,2005/0266000,2007/0117126,2007/0160598,2007/0237764,2007/0292936和2009/0002360。
认为自人抗体文库分离的抗体或抗体片段是本文中的人抗体或人抗体片段。
多特异性抗体
在某些实施方案中,本文中提供的抗体是多特异性抗体,例如双特异性抗体。多特异性抗体是对至少两个不同位点具有结合特异性的单克隆抗体。在某些实施方案中,结合特异性之一针对MiT多肽诸如MiT易位融合多肽,而另一种针对任何其它抗原。在某些实施方案中,双特异性抗体可以结合MiT多肽诸如MiT易位融合多肽的两个不同表位。也可以使用双特异性抗体来将细胞毒剂定位于表达MiT多肽诸如MiT易位融合多肽的细胞。双特异性抗体可以以全长抗体或抗体片段制备。
用于生成多特异性抗体的技术包括但不限于具有不同特异性的两对免疫球蛋白重链-轻链对的重组共表达(见Milstein和Cuello,Nature 305:537(1983)),WO 93/08829,和Traunecker等,EMBO J.10:3655(1991)),和“突起-入-空穴”工程化(见例如美国专利No.5,731,168)。也可以通过用于生成抗体Fc-异二聚体分子的工程化静电操纵效应(WO 2009/089004A1);交联两个或更多个抗体或片段(见例如美国专利No.4,676,980,及Brennan等,Science,229:81(1985));使用亮氨酸拉链来生成双特异性抗体(见例如Kostelny等,J.Immunol.,148(5):1547-1553(1992));使用用于生成双特异性抗体片段的“双抗体”技术(见例如Hollinger等,Proc.Natl.Acad.Sci.USA,90:6444-6448(1993));及使用单链Fv(sFv)二聚体(见例如Gruber等,J.Immunol.,152:5368(1994));及如例如Tutt等J.Immunol.147:60(1991)中所描述的,制备三特异性抗体来生成多特异性抗体。
本文中还包括具有三个或更多个功能性抗原结合位点的工程化改造抗体,包括“章鱼抗体”(见例如US 2006/0025576)。
本文中的抗体或片段还包括包含结合MiT多肽诸如MiT易位融合多肽及另一种不同抗原的抗原结合位点的“双重作用FAb”或“DAF”(见例如US2008/0069820)。
抗体变体
糖基化变体
在某些实施方案中,改变本文中提供的抗体以提高或降低抗体糖基化的程度。可以通过改变氨基酸序列,使得创建或消除一个或多个糖基化位点来方便地实现对抗体的糖基化位点的添加或删除。
在抗体包含Fc区的情况中,可以改变其附着的碳水化合物。由哺乳动物细胞生成的天然抗体通常包含分支的,双触角寡糖,其一般通过N连接附着于Fc区的CH2域的Asn297。见例如Wright等TIBTECH 15:26-32(1997)。寡糖可以包括各种碳水化合物,例如,甘露糖,N-乙酰葡糖胺(GlcNAc),半乳糖,和唾液酸,以及附着于双触角寡糖结构“主干”中的GlcNAc的岩藻糖。在一些实施方案中,可以对本发明的抗体中的寡糖进行修饰以创建具有某些改善的特性的抗体变体。
在一个实施方案中,提供了抗体变体,其具有缺乏附着(直接或间接)于Fc区的岩藻糖的碳水化合物结构。例如,此类抗体中的岩藻糖量可以是1%至80%,1%至65%,5%至65%或20%至40%。通过相对于附着于Asn297的所有糖结构(例如,复合的,杂合的和高甘露糖的结构)的总和,计算Asn297处糖链内岩藻糖的平均量来测定岩藻糖量,如通过MALDI-TOF质谱术测量的,例如如记载于WO 2008/077546的。Asn297指位于Fc区中的约第297位(Fc区残基的Eu编号方式)的天冬酰胺残基;然而,Asn297也可以由于抗体中的微小序列变异而位于第297位上游或下游约±3个氨基酸,即在第294位和第300位之间。此类岩藻糖基化变体可以具有改善的ADCC功能。见例如美国专利公开文本No.US 2003/0157108(Presta,L.);US 2004/0093621(Kyowa Hakko Kogyo Co.,Ltd)。涉及“脱岩藻糖基化的”或“岩藻糖缺乏的”抗体变体的出版物的例子包括:US 2003/0157108;WO 2000/61739;WO 2001/29246;US 2003/0115614;US 2002/0164328;US 2004/0093621;US 2004/0132140;US 2004/0110704;US 2004/0110282;US 2004/0109865;WO 2003/085119;WO 2003/084570;WO 2005/035586;WO 2005/035778;WO2005/053742;WO2002/031140;Okazaki等J.Mol.Biol.336:1239-1249(2004);Yamane-Ohnuki等Biotech.Bioeng.87:614(2004)。能够生成脱岩藻糖基化抗体的细胞系的例子包括蛋白质岩藻糖基化缺陷的Lec13CHO细胞(Ripka等Arch.Biochem.Biophys.249:533-545(1986);US 2003/0157108,Presta,L;及WO 2004/056312,Adams等,尤其在实施例11),和敲除细胞系,诸如α-1,6-岩藻糖基转移酶基因FUT8敲除CHO细胞(见例如Yamane-Ohnuki等Biotech.Bioeng.87:614(2004);Kanda,Y.等,Biotechnol.Bioeng.,94(4):680-688(2006);及WO2003/085107)。
进一步提供了具有两分型寡糖的抗体变体,例如其中附着于抗体Fc区的双触角寡糖是通过GlcNAc两分的。此类抗体变体可以具有降低的岩藻糖基化和/或改善的ADCC功能。此类抗体变体的例子记载于例如WO 2003/011878(Jean-Mairet等);美国专利No.6,602,684(Umana等);及US 2005/0123546(Umana等)。还提供了在附着于Fc区的寡糖中具有至少一个半乳糖残基的抗体变体。此类抗体变体可以具有改善的CDC功能。此类抗体变体记载于例如WO 1997/30087(Patel等);WO 1998/58964(Raju,S.);及WO 1999/22764(Raju,S.)。
Fc区变体
在某些实施方案中,可以将一处或多处氨基酸修饰引入本文中提供的抗体的Fc区中,由此生成Fc区变体。Fc区变体可以包含在一个或多个氨基酸位置包含氨基酸修饰(例如替代)的人Fc区序列(例如,人IgG1,IgG2,IgG3或IgG4Fc区)。
在某些实施方案中,本发明涵盖拥有一些但不是所有效应器功能的抗体变体,所述效应器功能使其成为如下应用的期望候选物,其中抗体的体内半衰期是重要的,而某些效应器功能(诸如补体和ADCC)是不必要的或有害的。可以进行体外和/或体内细胞毒性测定法以确认CDC和/或ADCC活性的降低/消减。例如,可以进行Fc受体(FcR)结合测定法以确保抗体缺乏FcγR结合(因此有可能缺乏ADCC活性),但是保留FcRn结合能力。介导ADCC的主要细胞NK细胞仅表达FcγRIII,而单核细胞表达FcγRI,FcγRII和FcγRIII。在Ravetch和Kinet,Annu.Rev.Immunol.9:457-492(1991)的第464页上的表3中汇总了造血细胞上的FcR表达。评估感兴趣分子的ADCC活性的体外测定法的非限制性例子记载于美国专利No.5,500,362(见例如Hellstrom,I.等Proc.Nat’l Acad.Sci.USA 83:7059-7063(1986))和Hellstrom,I等,Proc.Nat’l Acad.Sci.USA 82:1499-1502(1985);5,821,337(见Bruggemann,M.等,J.Exp.Med.166:1351-1361(1987))。或者,可以采用非放射性测定方法(见例如用于流式细胞术的ACTITM非放射性细胞毒性测定法(CellTechnology,Inc.Mountain View,CA;和CytoTox 非放射性细胞毒性测定法(Promega,Madison,WI))。对于此类测定法有用的效应细胞包括外周血单个核细胞(PBMC)和天然杀伤(NK)细胞。或者/另外,可以在体内评估感兴趣分子的ADCC活性,例如在动物模型中,诸如披露于Clynes等Proc.Nat’l Acad.Sci.USA95:652-656(1998)的。也可以实施C1q结合测定法以确认抗体不能结合C1q,并且因此缺乏CDC活性。见例如WO 2006/029879和WO 2005/100402中的C1q和C3c结合ELISA。为了评估补体激活,可以实施CDC测定法(见例如Gazzano-Santoro等,J.Immunol.Methods 202:163(1996);Cragg,M.S.等,Blood 101:1045-1052(2003);及Cragg,M.S.和M.J.Glennie,Blood103:2738-2743(2004))。也可以使用本领域中已知的方法来实施FcRn结合和体内清除/半衰期测定(见例如Petkova,S.B.等,Int’l.Immunol.18(12):1759-1769(2006))。
具有降低的效应器功能的抗体包括那些具有Fc区残基238,265,269,270,297,327和329中的一个或多个的替代的(美国专利No.6,737,056)。此类Fc突变体包括在氨基酸位置265,269,270,297和327中的两处或更多处具有替代的Fc突变体,包括残基265和297替代成丙氨酸的所谓的“DANA”Fc突变体(美国专利No.7,332,581)。
描述了具有改善的或降低的对FcR的结合的某些抗体变体(见例如美国专利No.6,737,056;WO 2004/056312,及Shields等,J.Biol.Chem.9(2):6591-6604(2001))。在某些实施方案中,抗体变体包含具有改善ADCC的一处或多处氨基酸替代,例如Fc区的位置298,333,和/或334(残基的EU编号方式)的替代的Fc区。在一些实施方案中,对Fc区做出改变,其导致改变的(即,改善的或降低的)C1q结合和/或补体依赖性细胞毒性(CDC),例如,如记载于美国专利No.6,194,551,WO 99/51642,及Idusogie等J.Immunol.164:4178-4184(2000)的。
具有延长的半衰期和改善的对新生儿Fc受体(FcRn)的结合的抗体记载于US2005/0014934A1(Hinton等),新生儿Fc受体(FcRn)负责将母体IgG转移至胎儿(Guyer等,J.Immunol.117:587(1976)及Kim等,J.Immunol.24:249(1994))。那些抗体包含其中具有改善Fc区对FcRn结合的一处或多处替代的Fc区。此类Fc变体包括那些在Fc区残基238,256,265,272,286,303,305,307,311,312,317,340,356,360,362,376,378,380,382,413,424或434中的一处或多处具有替代,例如,Fc区残基434的替代的(美国专利No.7,371,826)。还可见Duncan和Winter,Nature 322:738-40(1988);美国专利No.5,648,260;美国专利No.5,624,821;及WO 94/29351,其关注Fc区变体的其它例子。
经半胱氨酸工程化改造的抗体变体
在某些实施方案中,可以期望创建经半胱氨酸工程化改造的抗体,例如,“thioMAb”,其中抗体的一个或多个残基用半胱氨酸残基替代。在具体的实施方案中,替代的残基存在于抗体的可接近位点。通过用半胱氨酸替代那些残基,反应性硫醇基团由此定位于抗体的可接近位点,并且可以用于将抗体与其它模块,诸如药物模块或接头-药物模块缀合,以创建免疫缀合物,如本文中进一步描述的。在某些实施方案中,可以用半胱氨酸替代下列残基之任一个或多个:轻链的V205(Kabat编号方式);重链的A118(EU编号方式);和重链Fc区的S400(EU编号方式)。可以如例如美国专利No.7,521,541所述生成经半胱氨酸工程化改造的抗体。
免疫缀合物
本发明还提供包含本文中抗MiT(诸如MiT易位融合多肽)抗体的免疫缀合物,该抗体与一种或多种细胞毒剂诸如化疗剂或药物,生长抑制剂,毒素(例如蛋白质毒素,细菌,真菌,植物,或动物起源的酶活性毒素,或其片段),或放射性同位素缀合。
在一个实施方案中,免疫缀合物是抗体-药物缀合物(ADC),其中抗体与一种或多种药物缀合,包括但不限于美登木生物碱(maytansinoid)(参见美国专利No.5,208,020,No.5,416,064和欧洲专利EP 0 425 235B1);auristatin诸如单甲基auristatin药物模块DE和DF(MMAE和MMAF)(参见美国专利No.5,635,483和No.5,780,588,及No.7,498,298);多拉司他汀(dolastatin);加利车霉素(calicheamicin)或其衍生物(参见美国专利No.5,712,374,No.5,714,586,No.5,739,116,No.5,767,285,No.5,770,701,No.5,770,710,No.5,773,001,和No.5,877,296;Hinman等人,Cancer Res.53:3336-3342(1993);及Lode等人,Cancer Res.58:2925-2928(1998));蒽环类抗生素诸如道诺霉素(daunomycin)或多柔比星(doxorubicin)(参见Kratz等人,Current Med.Chem.13:477-523(2006);Jeffrey等人,Bioorganic&Med.Chem.Letters 16:358-362(2006);Torgov等人,Bioconj.Chem.16:717-721(2005);Nagy等人,Proc.Natl.Acad.Sci.USA 97:829-834(2000);Dubowchik等人,Bioorg.&Med.Chem.Letters 12:1529-1532(2002);King等人,J.Med.Chem.45:4336-4343(2002);及美国专利No.6,630,579);甲氨蝶呤;长春地辛(vindesine);紫杉烷诸如多西他赛(docetaxel),帕利他赛(paclitaxel),larotaxel,tesetaxel,和ortataxel;单端孢菌素(trichothecene);和CC1065。
在另一个实施方案中,免疫缀合物包含本文所述抗体,该抗体与酶活性毒素或其片段缀合,包括但不限于白喉毒素A链,白喉毒素的非结合活性片段,外毒素A链(来自铜绿假单胞菌Pseudomonas aeruginosa),蓖麻毒蛋白(ricin)A链,相思豆毒蛋白(abrin)A链,蒴莲根毒蛋白(modeccin)A链,α-帚曲霉素(sarcin),油桐(Aleurites fordii)毒蛋白,香石竹(dianthin)毒蛋白,美洲商陆(Phytolaca americana)毒蛋白(PAPI,PAPII和PAP-S),苦瓜(Momordica charantia)抑制物,麻疯树毒蛋白(curcin),巴豆毒蛋白(crotin),肥皂草(sapaonaria officinalis)抑制物,白树毒蛋白(gelonin),丝林霉素(mitogellin),局限曲菌素(restrictocin),酚霉素(phenomycin),依诺霉素(enomycin)和单端孢菌素(tricothecenes)。
在另一个实施方案中,免疫缀合物包含本文所述抗体,该抗体与放射性原子缀合以形成放射缀合物。多种放射性同位素可用于生成放射缀合物。实例包括At211,I131,I125,Y90,Re186,Re188,Sm153,Bi212,P32,Pb212和Lu的放射性同位素。在将放射缀合物用于检测时,它可包含放射性原子用于闪烁照相研究,例如Tc99或I123,或自旋标记物用于核磁共振(NMR)成像(也称为磁共振成像,MRI),诸如碘-123,碘-131,铟-111,氟-19,碳-13,氮-15,氧-17,钆,锰或铁。
可使用多种双功能蛋白质偶联剂来制备抗体和细胞毒剂的缀合物,诸如N-琥珀酰亚氨基-3-(2-吡啶基二硫代)丙酸酯(SPDP),琥珀酰亚氨基-4-(N-马来酰亚氨基甲基)环己烷-1-羧酸酯(SMCC),亚氨基硫烷(IT),亚氨酸酯(诸如盐酸己二酰亚氨酸二甲酯),活性酯类(诸如辛二酸二琥珀酰亚氨基酯),醛类(诸如戊二醛),双叠氮化合物(诸如双(对-叠氮苯甲酰基)己二胺),双重氮衍生物(诸如双(对-重氮苯甲酰基)乙二胺),二异氰酸酯(诸如甲苯2,6-二异氰酸酯),和双活性氟化合物(诸如1,5-二氟-2,4-二硝基苯)的双功能衍生物。例如,可如Vitetta等,Science 238:1098(1987)中所述制备蓖麻毒蛋白免疫毒素。碳-14标记的1-异硫氰酸苯甲基-3-甲基二亚乙基三胺五乙酸(MX-DTPA)是用于将放射性核苷酸与抗体缀合的例示性螯合剂。参见WO 94/11026。接头可以是便于在细胞中释放细胞毒药物的“可切割接头”。例如,可使用酸不稳定接头,肽酶敏感接头,光不稳定接头,二甲基接头,或含二硫化物接头(Chari等,Cancer Res.52:127-131(1992);美国专利No.5,208,020)。
本文中的免疫缀合物或ADC明确涵盖但不限于用下列交联剂制备的此类缀合物,包括但不限于:商品化(如购自Pierce Biotechnology Inc.,Rockford,IL,U.S.A)的BMPS,EMCS,GMBS,HBVS,LC-SMCC,MBS,MPBH,SBAP,SIA,SIAB,SMCC,SMPB,SMPH,sulfo-EMCS,sulfo-GMBS,sulfo-KMUS,sulfo-MBS,sulfo-SIAB,sulfo-SMCC,和sulfo-SMPB,和SVSB(琥珀酰亚胺基-(4-乙烯基砜)苯甲酸酯)。
C.结合多肽
本文中提供了作为本文所述任何方法中的MiT拮抗剂使用的MiT结合多肽拮抗剂。MiT结合多肽拮抗剂是如下的多肽,其结合,优选特异性结合MiT多肽。在任何MiT结合多肽拮抗剂的一些实施方案中,MiT结合多肽拮抗剂为嵌合多肽。
在任何结合多肽的一些实施方案中,该MiT结合多肽拮抗剂是MITF结合多肽拮抗剂。在一些实施方案中,该MiT结合多肽拮抗剂是MITF易位结合多肽拮抗剂。在任何结合多肽的一些实施方案中,该MiT结合多肽拮抗剂是TFEB结合多肽拮抗剂。在一些实施方案中,该MiT结合多肽拮抗剂是TFEB易位结合多肽拮抗剂。在任何结合多肽的一些实施方案中,该MiT结合多肽拮抗剂是TFEC结合多肽拮抗剂。在一些实施方案中,该MiT小分子拮抗剂是TFEC易位结合多肽拮抗剂。在任何小分子的一些实施方案中,该MiT结合多肽拮抗剂是TFE3结合多肽拮抗剂。在一些实施方案中,该MiT结合多肽拮抗剂是TFE3易位结合多肽拮抗剂。
在任何结合多肽的一些实施方案中,该结合多肽结合MITF易位融合多肽。在一些实施方案中,结合多肽特异性结合MITF易位融合多肽,但是并非实质性结合野生型MITF和/或第二易位基因。在一些实施方案中,该MITF易位融合多肽包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位融合多肽包含SEQ ID NO:30和/或14。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。
在任何结合多肽的一些实施方案中,该结合多肽结合TFEB易位融合多肽。在一些实施方案中,结合多肽特异性结合TFEB易位融合多肽,但是并非实质性结合野生型TFEC和/或第二易位基因。在一些实施方案中,该TFEB易位融合多肽包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该MITF易位融合多肽包含SEQ ID NO:20和/或31。
在任何结合多肽的一些实施方案中,该结合多肽结合TFEC易位融合多肽。在一些实施方案中,结合多肽特异性结合TFEC易位融合多肽,但是并非实质性结合野生型TFEC和/或第二易位基因。
在任何结合多肽的一些实施方案中,该结合多肽结合TFE3易位融合多肽。在一些实施方案中,结合多肽特异性结合TFE3易位融合多肽,但是并非实质性结合野生型TFE3和/或第二易位基因。
在一些实施方案中,该MiT结合多肽拮抗剂是MET途径结合多肽拮抗剂。例如,特异性结合HGF的c-met受体分子或其片段能在本发明的方法中使用,例如用于结合和隔绝HGF蛋白质,由此阻止其信号传导。优选地,c-met受体分子或其HGF结合片段是可溶性形式。在一些实施方案中,所述受体的可溶性形式通过结合HGF,由此阻止它结合其在靶细胞表面上存在的天然受体而对c-met蛋白质的生物学活性发挥抑制效果。还包括c-met受体融合蛋白,下文描述了它的例子。在一些实施方案中,该MET途径结合多肽拮抗剂包含可溶性形式的c-met受体,包括嵌合受体蛋白。特异性结合c-met并阻断或降低c-met活化,由此阻止它发信号的HGF分子或其片段能在本发明的方法中使用。
在一些实施方案中,该MiT结合多肽拮抗剂是BIRC7结合多肽拮抗剂。在一个实施方案中,该BIRC7结合多肽拮抗剂包含EERTCKVCLDRAVSIVFVPCGHLVCAECAPGLQLCPICRAPVRSRVRTFL(SEQ ID NO:)。在一个实施方案中,该BIRC7结合多肽拮抗剂包含CRAPVRSRVRTFLS(SEQ ID NO:)。
结合多肽可以使用已知的多肽合成方法学化学合成,或者可使用重组技术制备和纯化。结合多肽的长度通常是至少约5个氨基酸,或者长度为至少约6,7,8,9,10,11,12,13,14,15,16,17,18,19,20,21,22,23,24,25,26,27,28,29,30,31,32,33,34,35,36,37,38,39,40,41,42,43,44,45,46,47,48,49,50,51,52,53,54,55,56,57,58,59,60,61,62,63,64,65,66,67,68,69,70,71,72,73,74,75,76,77,78,79,80,81,82,83,84,85,86,87,88,89,90,91,92,93,94,95,96,97,98,99或100个氨基酸或更长,其中此类结合多肽能够结合,优选特异性结合本文所述的靶物,即MiT多肽。结合多肽无需过多试验就可使用公知技术来鉴定。在这点上,注意到用于对多肽文库筛选能够特异性结合多肽靶物的结合多肽的技术是本领域公知的(参见例如美国专利5,556,762,5,750,373,4,708,871,4,833,092,5,223,409,5,403,484,5,571,689,5,663,143;PCT公开号WO 84/03506和WO 84/03564;Geysen等,Proc.Natl.Acad.Sci.U.S.A.81:3998-4002(1984);Geysen等,Proc.Natl.Acad.Sci.U.S.A.82:178-182(1985);Geysen等,in Synthetic Peptides as Antigens,130-149(1986);Geysen等,J.Immunol.Meth.102:259-274(1987);Schoofs等,J.Immunol.140:611-616(1988);Cwirla,S.E.等,(1990)Proc.Natl.Acad.Sci.USA 87:6378;Lowman,H.B.等,(1991)Biochemistry 30:10832;Clackson,T.等,(1991)Nature 352:624;Marks,J.D.等,(1991),J.Mol.Biol.222:581;Kang,A.S.等,(1991)Proc.Natl.Acad.Sci.USA 88:8363;Smith,G.P.,(1991)Current Opin.Biotechnol.2:668)。
在这点上,噬菌体展示是一种可以筛选大型多肽文库来鉴定那些文库中能够特异性结合靶多肽,即MiT多肽的成员的公知技术。噬菌体展示是将变体多肽作为与外壳蛋白的融合蛋白展示在噬菌体颗粒表面的一种技术(Scott,J.K.and Smith,G.P.(1990)Science 249:386)。噬菌体展示的效用在于,可以对选择性随机化蛋白质变体(或随机克隆cDNA)的大型文库迅速且有效地分选那些以高亲和力结合靶分子的序列的事实。在噬菌体上展示肽(Cwirla,S.E.等,(1990)Proc.Natl.Acad.Sci.USA 87:6378)或蛋白质(Lowman,H.B.等,(1991)Biochemistry 30:10832;Clackson,T.等,(1991)Nature 352:624;Marks,J.D.等,(1991)J.MoI.Biol.222:581;Kang,A.S.等,(1991)Proc.Natl.Acad.Sci.USA 88:8363)文库已经用于对数百万多肽或寡肽筛选具有特异结合特性的那些多肽或寡肽(Smith,G.P.(1991)Current Opin.Biotechnol.2:668)。分选随机突变体的噬菌体文库需要构建和扩充大量变体的策略,使用靶受体进行亲和纯化的流程,及评估结合富集的结果的手段。参见美国专利5,223,409,5,403,484,5,571,689和5,663,143。
尽管大多数噬菌体展示方法已经使用丝状噬菌体,λ类(lambdoid)噬菌体展示系统(WO 95/34683;US 5,627,024),T4噬菌体展示系统(Ren等,Gene 215:439(1998);Zhu等,Cancer Research 58(15):3209-3214(1998);Jiang等,Infection&Immunity 65(11):4770-4777(1997);Ren等,Gene 195(2):303-311(1997);Ren,Protein Sci.5:1833(1996);Efimov等,Virus Genes 10:173(1995))和T7噬菌体展示系统(Smith and Scott,Methods in Enzymology 217:228-257(1993);US 5,766,905)也是已知的。
别的改进增强了展示系统对肽文库筛选与选定靶分子的结合及展示功能性蛋白质的能力,所述功能性蛋白质具有对这些蛋白质筛选期望特性的潜力。已经开发了用于噬菌体展示反应的组合反应装置(WO 98/14277),而且噬菌体展示文库已经用于分析和控制生物分子相互作用(WO 98/20169;WO 98/20159)和受约束(constrained)螺旋肽的特性(WO 98/20036)。WO 97/35196描述了分离亲和配体的方法,其中使噬菌体展示文库接触第一种溶液和第二种溶液以选择性分离结合的配体,在第一种溶液中配体将结合靶分子,而在第二种溶液中亲和配体将不会结合靶分子。WO 97/46251描述了这样一种方法,即用亲和纯化的抗体生物淘选随机噬菌体展示库,然后分离结合的噬菌体,随后使用微量板的孔进行淘选过程以分离高亲和力结合的噬菌体。已经报道了金黄色葡萄球菌(Staphylococcus aureus)蛋白A作为亲和标签的使用(Li等,(1998)Mol.Biotech.9:187)。WO 97/47314描述了底物扣除文库用于区别酶特异性的用途,其中使用可以是噬菌体展示库的组合文库。WO 97/09446描述了使用噬菌体展示选择适用于洗涤剂的酶的方法。美国专利5,498,538,5,432,018和WO 98/15833中描述了选择特异性结合的蛋白质的其它方法。
产生肽文库和筛选这些文库的方法还公开于美国专利5,723,286,5,432,018,5,580,717,5,427,908,5,498,530,5,770,434,5,734,018,5,698,426,5,763,192,和5,723,323。
D.结合小分子
本文中提供了作为本文所述任何方法中的MiT拮抗剂使用的MiT小分子拮抗剂。
在任何小分子的一些实施方案中,该MiT小分子拮抗剂是MITF小分子拮抗剂。在一些实施方案中,该MiT小分子拮抗剂是MITF易位小分子拮抗剂。在任何小分子的一些实施方案中,该MiT小分子拮抗剂是TFEB小分子拮抗剂。在一些实施方案中,该MiT小分子拮抗剂是TFEB易位小分子拮抗剂。在任何小分子的一些实施方案中,该MiT小分子拮抗剂是TFEC小分子拮抗剂。在一些实施方案中,该MiT小分子拮抗剂是TFEC易位小分子拮抗剂。在任何小分子的一些实施方案中,该MiT小分子拮抗剂是TFE3小分子拮抗剂。在一些实施方案中,该MiT小分子拮抗剂是TFE3易位小分子拮抗剂。
在任何小分子的一些实施方案中,该小分子结合MITF易位融合多肽。在一些实施方案中,小分子特异性结合MITF易位融合多肽,但是并非实质性结合野生型MITF和/或第二易位基因。在一些实施方案中,该MITF易位融合多肽包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位融合多肽包含SEQ ID NO:30和/或14。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。
在任何小分子的一些实施方案中,该小分子结合TFEB易位融合多肽。在一些实施方案中,小分子特异性结合TFEB易位融合多肽,但是并非实质性结合野生型TFEC和/或第二易位基因。在一些实施方案中,该TFEB易位融合多肽包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该MITF易位融合多肽包含SEQ ID NO:20和/或31。
在任何小分子的一些实施方案中,该小分子结合TFEC易位融合多肽。在一些实施方案中,小分子特异性结合TFEC易位融合多肽,但是并非实质性结合野生型TFEC和/或第二易位基因。
在任何小分子的一些实施方案中,该小分子结合TFE3易位融合多肽。在一些实施方案中,小分子特异性结合TFE3易位融合多肽,但是并非实质性结合野生型TFE3和/或第二易位基因。
优选地,小分子指本文所定义的结合多肽或抗体以外,结合,优选特异性结合本文所述MiT多肽的有机分子。有机小分子可以使用已知方法学来鉴定和化学合成(参见例如PCT公开号WO 00/00823和WO 00/39585)。有机小分子的大小通常小于约2000道尔顿,或者其大小小于约1500,750,500,250或200道尔顿,其中此类能够结合,优选特异性结合本文所述多肽的有机小分子无需过多实验就可使用公知技术来鉴定。在这点上,注意到用于对有机小分子文库筛选能够结合多肽靶物的分子的技术是本领域公知的(参见例如PCT公开号WO 00/00823和WO 00/39585)。有机小分子可以是例如醛,酮,肟,腙,缩氨基脲(semicarbazone),卡巴肼(carbazide),伯胺,仲胺,叔胺,N-取代的肼,酰肼,醇,醚,硫醇,硫醚,二硫化物,羧酸,酯,酰胺,脲,氨基甲酸酯(carbamate),碳酸酯(carbonate),缩酮,硫缩酮(thioketal),缩醛,硫缩醛,芳基卤,芳基磺酸酯(aryl sulfonate),烃基卤,烃基磺酸酯(alkylsulfonate),芳香族化合物,杂环化合物,苯胺,烯烃,炔烃,二醇,氨基醇,口恶唑烷,口恶唑啉,噻唑烷,噻唑啉,烯胺,磺酰胺(sulfonamide),环氧化物,吖丙啶(aziridine),异氰酸酯(isocyanate),磺酰氯,重氮化合物,酰基氯(acid chloride)等。
在任何小分子的一些实施方案中,该MiT小分子拮抗剂是c-met小分子拮抗剂。在一个实施方案中,该c-met拮抗剂结合c-met胞外域。在一些实施方案中,该c-met小分子拮抗剂结合c-met激酶域。在一些实施方案中,该c-met小分子拮抗剂与HGF竞争c-met结合。在一些实施方案中,该c-met小分子拮抗剂与c-met竞争HGF结合。在一些实施方案中,该c-met小分子拮抗剂结合HGF。
在某些实施方案中,c-met小分子拮抗剂是下述任一种:SGX-523,Crizotinib;JNJ-38877605(CAS no.943540-75-8),BMS-698769,PHA-665752(Pfizer),SU5416,INC-280(Incyte;SU11274(Sugen;[(3Z)-N-(3-氯苯基)-3-({3,5-二甲基-4-[(4-甲基哌嗪-1-基)羰基]-1H-吡咯-2-基}亚甲基)-N-甲基-2-氧二氢吲哚-5-磺酰胺;CAS no.658084-23-2]),Foretinib,MGCD-265(MethylGene;MGCD-265靶向c-MET,VEGFR1,VEGFR2,VEGFR3,Ron和Tie-2受体;CAS no.875337-44-3),Tivantinib(ARQ 197),LY-2801653(Lilly),LY2875358(Lilly),MP-470,EMD 1214063(Merck Sorono),EMD1204831(Merck Serono),NK4,Cabozantinib(carbozantinib是met和VEGFR2的一种双重抑制剂),MP-470(SuperGen;是c-KIT,MET,PDGFR,Flt3,和AXL的一种抑制剂),Comp-1,E7050(Cas no.1196681-49-8;E7050是一种双重c-met和VEGFR2抑制剂(Esai);MK-2461(Merck;N-((2R)-1,4-二恶烷-2-基甲基)-N-甲基-N'-[3-(1-甲基-1H-吡唑-4-基)-5-氧-5H-苯并[4,5]环庚[1,2-b]吡啶-7-基]磺酰胺;CAS no.917879-39-1);MK8066(Merck),PF4217903(Pfizer),AMG208(Amgen),SGX-126,RP1040,LY2801653,AMG458,EMD637830,BAY-853474,DP-3590。在某些实施方案中,c-met小分子拮抗剂是crizotinib,tivantinib,carbozantinib,MGCD-265,h224G11,DN-30,MK-2461,E7050,MK-8033,PF-4217903,AMG208,JNJ-38877605,EMD1204831,INC-280,LY-2801653,SGX-126,RP1040,LY2801653,BAY-853474,和/或LA480中任一种或多种。在某些实施方案中,c-met小分子拮抗剂是crizotinib,tivantinib,carbozantinib,MGCD-265,和/或foretinib中任一种或多种。在某些实施方案中,该c-met小分子拮抗剂是crizotinib,foretinib或carbozantinib中任一种或多种。
在任何小分子的一些实施方案中,该MiT小分子拮抗剂是BIRC7途径小分子拮抗剂。在一些实施方案中,该BIRC7途径小分子拮抗剂是下述任一项或多项:MV1,BV6,GDC-0152,LBW242,SM-164,HGS1029,TL32711。
在一些实施方案中,该BIRC7途径拮抗剂是单价拮抗剂。在一些实施方案中,该BIRC7途径拮抗剂是二价拮抗剂。
E.拮抗性多核苷酸
本文中提供了作为本文所述任何方法中的MiT拮抗剂使用的MiT多核苷酸拮抗剂。多核苷酸可以是反义核酸和/或核酶。反义核酸包含至少与MiT基因的RNA转录物的一部分互补的序列。然而,尽管优选绝对互补性,但是其不是必要的。在一些实施方案中,MiT多核苷酸是MITF多核苷酸。在一些实施方案中,MiT多核苷酸是TFEB多核苷酸。在一些实施方案中,MiT多核苷酸是TFE3多核苷酸。在一些实施方案中,MiT多核苷酸是TFEC多核苷酸。在一些实施方案中,MiT多核苷酸是SBNO2多核苷酸。
在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合MITF多核苷酸。在一些实施方案中,该多核苷酸结合TFEB易位多核苷酸。在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFEB多核苷酸。在一些实施方案中,该多核苷酸结合TFEB易位多核苷酸。在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFEC多核苷酸。在一些实施方案中,该多核苷酸结合TFEC易位多核苷酸。在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFE3多核苷酸。在一些实施方案中,该多核苷酸结合TFE3易位多核苷酸。
在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合MITF易位融合多肽。在一些实施方案中,该多核苷酸特异性结合MITF易位融合多核苷酸,但是并非实质性结合野生型MITF多核苷酸和/或第二易位基因。在一些实施方案中,该MITF易位融合多核苷酸包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位融合多核苷酸包含SEQ ID NO:13。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。
在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFEB易位融合多核苷酸。在一些实施方案中,该多核苷酸特异性结合TFEB易位融合多核苷酸,但是并非实质性结合野生型TFEC多核苷酸和/或第二易位基因。在一些实施方案中,该TFEB易位融合多核苷酸包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位融合多核苷酸包含SEQ ID NO:19。
在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFEC易位融合多核苷酸。在一些实施方案中,该多核苷酸特异性结合TFEC易位融合多核苷酸,但是并非实质性结合野生型TFEC和/或第二易位基因。
在任何多核苷酸拮抗剂的一些实施方案中,该多核苷酸结合TFE3易位融合多核苷酸。在一些实施方案中,该多核苷酸特异性结合TFE3易位融合多核苷酸,但是并非实质性结合野生型TFE3多核苷酸和/或第二易位基因。
本文中提及的“至少与RNA的一部分互补的”序列意指具有足够的互补性以能够与RNA杂交,形成稳定的双链体的序列;在双链MiT反义核酸的情况中,如此可以测试双链体DNA的单链,或者可以测定三链体形成。杂交能力会取决于互补性程度和反义核酸的长度两者。一般地,杂交的核酸越大,与MiT RNA的碱基错配越多,它可以含有且仍形成稳定的双链体(或三链体,情况也可以如此)。本领域技术人员可以通过使用标准规程测定杂交复合物的熔点来确认可容许的错配程度。
与信息5’端(例如5’非翻译序列直至AUG起始密码子且包括AUG起始密码子)互补的多核苷酸在抑制翻译方面应当最有效起作用。然而,已经显示了与mRNA的3’非翻译序列互补的序列也有效抑制mRNA的翻译。一般见Wagner,R.,1994,Nature 372:333-335。如此,与MiT基因的5’-或3’-非翻译,非编码区互补的寡核苷酸可以在反义方法中使用以抑制内源MiT mRNA的翻译。与mRNA的5’非翻译区互补的多核苷酸应当包括AUG起始密码子的互补物。与mRNA编码区互补的反义多核苷酸是不太有效的翻译抑制剂,但是可以依照本发明使用。不论设计为与MiT mRNA的5’,3’或编码区杂交,反义核酸的长度应当是至少6个核苷酸,并且优选是长度范围为6至约50个核苷酸的寡核苷酸。在具体的方面,寡核苷酸是至少10个核苷酸,至少17个核苷酸,至少25个核苷酸或至少50个核苷酸。
在一个实施方案中,本发明的MiT反义核酸通过自外源序列转录在细胞内生成。例如,转录载体或其部分,生成MiT基因的反义核酸(RNA)。此类载体会含有编码MiT反义核酸的序列。此类载体可以仍然是附加体的或者变为染色体整合的,只要它可以被转录以生成期望的反义RNA。可以通过本领域中标准的重组DNA技术方法构建此类载体。载体可以是用于在脊椎动物细胞中复制和表达的质粒,病毒,或本领域中已知的其它载体。可以通过本领域中已知在脊椎动物(优选人细胞)中起作用的任何启动子表达编码MiT或其片段的序列。此类启动子可以是诱导型或组成性的。此类启动子包括但不限于SV40早期启动子区(Bernoist and Chambon,Nature 29:304-310(1981),劳氏肉瘤病毒的3’长末端重复中含有的启动子(Yamamoto等,Cell 22:787-797(1980),疱疹胸苷启动子(Wagner等,Proc.Natl.Acad.Sci.U.S.A.78:1441-1445(1981),金属硫蛋白基因的调节序列(Brinster等,Nature 296:39-42(1982)),等等。
F.抗体和结合多肽变体
在某些实施方案中,涵盖本文中提供的抗体和/或结合多肽的氨基酸序列变体。例如,可以期望改善抗体和/或结合多肽的结合亲和力和/或其它生物学特性。可以通过将合适的修饰引入编码抗体和/或结合多肽的核苷酸序列中,或者通过肽合成来制备抗体和/或结合多肽的氨基酸序列变体。此类修饰包括例如对抗体和/或结合多肽的氨基酸序列内的残基的删除,和/或插入和/或替代。可以进行删除,插入,和替代的任何组合以得到最终的构建体,只要最终的构建体拥有期望的特征,例如,靶物结合。
在某些实施方案中,提供了具有一处或多处氨基酸替代的抗体变体和/或结合多肽变体。替代诱变感兴趣的位点包括HVR和FR。保守替代在表1中在“保守替代”的标题下显示。更实质的变化在表1中在“例示性替代”的标题下提供,并且如下文参照氨基酸侧链类别进一步描述。可以将氨基酸替代引入感兴趣的抗体和/或结合多肽中,并且对产物筛选期望的活性,例如保留/改善的抗原结合,降低的免疫原性,或改善的ADCC或CDC。
表1
依照共同的侧链特性,氨基酸可以如下分组:
(1)疏水性的:正亮氨酸,Met,Ala,Val,Leu,Ile;
(2)中性,亲水性的:Cys,Ser,Thr,Asn,Gln;
(3)酸性的:Asp,Glu;
(4)碱性的:His,Lys,Arg;
(5)影响链取向的残基:Gly,Pro;
(6)芳香族的:Trp,Tyr,Phe。
非保守替代会需要用这些类别之一的成员替换另一个类别的。
一类替代变体牵涉替代亲本抗体(例如人源化或人抗体)的一个或多个高变区残基。一般地,为进一步研究选择的所得变体相对于亲本抗体会具有某些生物学特性的改变(例如改善)(例如升高的亲和力,降低的免疫原性)和/或会基本上保留亲本抗体的某些生物学特性。例示性的替代变体是亲和力成熟的抗体,其可以例如使用基于噬菌体展示的亲和力成熟技术诸如本文中所描述的那些技术来方便地生成。简言之,将一个或多个HVR残基突变,并将变体抗体在噬菌体上展示,并对其筛选特定的生物学活性(例如结合亲和力)。
可以对HVR做出变化(例如,替代),例如以改善抗体亲和力。可以对HVR“热点”,即由在体细胞成熟过程期间以高频率经历突变的密码子编码的残基(见例如Chowdhury,Methods Mol.Biol.207:179-196(2008)),和/或SDR(a-CDR)做出此类变化,其中对所得的变体VH或VL测试结合亲和力。通过次级文库的构建和再选择进行的亲和力成熟已经记载于例如Hoogenboom等于METHODS IN MOL.BIOL.178:1-37(O’Brien等编,Human Press,Totowa,NJ,(2001))。在亲和力成熟的一些实施方案中,通过多种方法(例如,易错PCR,链改组,或寡核苷酸指导的诱变)中的任何方法将多样性引入为成熟选择的可变基因。然后,创建次级文库。然后,筛选文库以鉴定具有期望的亲和力的任何抗体变体。另一种引入多样性的方法牵涉HVR指导的方法,其中将几个HVR残基(例如,一次4-6个残基)随机化。可以例如使用丙氨酸扫描诱变或建模来特异性鉴定牵涉抗原结合的HVR残基。特别地,经常靶向CDR-H3和CDR-L3。
在某些实施方案中,可以在一个或多个HVR内发生替代,插入,或删除,只要此类变化不实质性降低抗体结合抗原的能力。例如,可以对HVR做出保守变化(例如,保守替代,如本文中提供的),其不实质性降低结合亲和力。此类变化可以在HVR“热点”或SDR外部。在上文提供的变体VH和VL序列的某些实施方案中,每个HVR或是未改变的,或是含有不超过1,2或3处氨基酸替代。
一种可用于鉴定抗体和/或结合多肽中可以作为诱变靶位的残基或区域的方法称作“丙氨酸扫描诱变”,如由Cunningham和Wells(1989)Science,244:1081-1085所描述的。在此方法中,将残基或靶残基的组(例如,带电荷的残基诸如arg,asp,his,lys,和glu)鉴定,并用中性或带负电荷的氨基酸(例如,丙氨酸或多丙氨酸)替换以测定抗体与抗原的相互作用是否受到影响。可以在对初始替代表明功能敏感性的氨基酸位置引入进一步的替代。或者/另外,利用抗原-抗体复合物的晶体结构来鉴定抗体与抗原间的接触点。作为替代的候选,可以靶向或消除此类接触残基和邻近残基。可以筛选变体以确定它们是否含有期望的特性。
氨基酸序列插入包括长度范围为1个残基至含有100或更多个残基的多肽的氨基和/或羧基端融合,及单个或多个氨基酸残基的序列内插入。末端插入的例子包括具有N端甲硫氨酰基残基的抗体。抗体分子的其它插入变体包括抗体的N或C端与酶(例如对于ADEPT)或延长抗体的血清半衰期的多肽的融合物。
G.抗体和结合多肽衍生物
在某些实施方案中,可进一步修饰本文中提供的抗体和/或结合多肽以包含本领域中已知的且容易获得的别的非蛋白质性质模块。适合于抗体和/或结合多肽衍生化的模块包括但不限于水溶性聚合物。水溶性聚合物的非限制性例子包括但不限于聚乙二醇(PEG),乙二醇/丙二醇的共聚物,羧甲基纤维素,右旋糖苷(dextran),聚乙烯醇,聚乙烯吡咯烷酮,聚-1,3-二氧杂环戊烷,聚-1,3,6-三氧杂环戊烷,乙烯/马来酸酐共聚物,聚氨基酸(或是同聚物或是随机共聚物),右旋糖苷或聚(n-乙烯吡咯烷酮)聚乙二醇,聚丙二醇(propropylene glycol)同聚物,聚环氧丙烷(prolypropylene oxide)/环氧乙烷共聚物,聚氧乙烯化多元醇(例如甘油),聚乙烯醇,及其混合物。聚乙二醇丙醛由于其在水中的稳定性而有利于制备。聚合物可以是任何分子量的,且可以是分支的或不分支的。附着于抗体和/或结合多肽的聚合物数目是变化的,且若附着超过一个聚合物,则它们可以是相同的或不同的分子。通常,可基于以下考虑确定用于衍生化的聚合物的数目和/或类型,所述考虑包括但不限于,待改进之抗体和/或结合多肽的具体特性或功能,抗体衍生物和/或结合多肽衍生物是否会在限定条件下用于治疗,等等。
在另一个实施方案中,提供了抗体和/或结合多肽与可通过暴露于辐射而选择性加热的非蛋白质性质模块的缀合物。在一个实施方案中,非蛋白质性质模块是碳纳米管(Kam等,Proc.Natl.Acad.Sci.USA 102:11600-11605(2005))。辐射可以是任何波长的,且包括但不限于不会伤害普通细胞但将非蛋白质性质模块加热至邻近抗体和/或结合多肽-非蛋白质性质模块的细胞被杀死的温度的波长。
H.重组方法和组合物
可以使用重组方法和组合物来生成抗体和/或结合多肽,例如,如记载于美国专利No.4,816,567的。在一个实施方案中,分离的核酸编码抗MiT抗体。此类核酸可以编码包含抗体VL的氨基酸序列和/或包含抗体VH的氨基酸序列(例如,抗体的轻和/或重链)。在又一个实施方案中,提供了包含此类核酸的一种或多种载体(例如,表达载体),所述核酸编码抗体和/或结合多肽。在又一个实施方案中,提供了包含此类核酸的宿主细胞。在一个此类实施方案中,宿主细胞包含(例如,已经用下列载体转化):(1)包含核酸的载体,所述核酸编码包含抗体的VL的氨基酸序列和包含抗体的VH的氨基酸序列,或(2)第一载体和第二载体,所述第一载体包含编码包含抗体的VL的氨基酸序列的核酸,所述第二载体包含编码包含抗体的VH的氨基酸序列的核酸。在一个实施方案中,宿主细胞是真核的,例如中国仓鼠卵巢(CHO)细胞或淋巴样细胞(例如,Y0,NS0,Sp20细胞)。在一个实施方案中,提供了生成抗体诸如抗MiT抗体和/或结合多肽的方法,其中该方法包括在适合于表达抗体和/或结合多肽的条件下培养包含编码抗体和/或结合多肽的核酸的宿主细胞,如上文提供的,并且任选地,自宿主细胞(或宿主细胞培养液)回收抗体和/或多肽。
对于抗体诸如抗MiT抗体和/或结合多肽的重组生成,将编码抗体和/或结合多肽的核酸(例如如上文所描述的)分离,并插入一种或多种载体中,以在宿主细胞中进一步克隆和/或表达。可以使用常规规程将此类核酸容易地分离并测序(例如,通过使用寡核苷酸探针来进行,所述寡核苷酸探针能够特异性结合编码抗体的重和轻链的基因)。
适合于克隆或表达载体的宿主细胞包括本文中所描述的原核或真核细胞。例如,可以在细菌中生成抗体,特别是在不需要糖基化和Fc效应器功能时。对于抗体片段和多肽在细菌中的表达,见例如美国专利No.5,648,237,5,789,199和5,840,523(还可见Charlton,METHODS IN MOL.BIOL.,第248卷(B.K.C.Lo编,Humana Press,Totowa,NJ,2003),第245-254页,其描述了抗体片段在大肠杆菌(E.coli.)中的表达)。表达后,可以将抗体在可溶性级分中自细菌细胞团糊分离,并可以进一步纯化。
在原核生物外,真核微生物诸如丝状真菌或酵母是适合于载体的克隆或表达宿主,包括其糖基化途径已经“人源化”,导致生成具有部分或完全人的糖基化样式的抗体的真菌和酵母菌株。见Gerngross,Nat.Biotech.22:1409-1414(2004),及Li等,Nat.Biotech.24:210-215(2006)。
适合于表达糖基化抗体和/或糖基化结合多肽的宿主细胞也自多细胞生物体(无脊椎动物和脊椎动物)衍生。无脊椎动物细胞的例子包括植物和昆虫细胞。已经鉴定出许多杆状病毒株,其可以与昆虫细胞一起使用,特别是用于转染草地夜蛾(Spodoptera frugiperda)细胞。
也可以利用植物细胞培养物作为宿主。见例如美国专利No.5,959,177,6,040,498,6,420,548,7,125,978和6,417,429(其描述了用于在转基因植物中生成抗体的PLANTIBODIESTM技术)。
也可以使用脊椎动物细胞作为宿主。例如,适合于在悬浮液中生长的哺乳动物细胞系可以是有用的。有用的哺乳动物宿主细胞系的其它例子是经SV40转化的猴肾CV1系(COS-7);人胚肾系(293或293细胞,如记载于例如Graham等,J.Gen Virol.36:59(1977)的);幼年仓鼠肾细胞(BHK);小鼠塞托利(sertoli)细胞(TM4细胞,如记载于例如Mather,Biol.Reprod.23:243-251(1980)的);猴肾细胞(CV1);非洲绿猴肾细胞(VERO-76);人宫颈癌细胞(HELA);犬肾细胞(MDCK;牛鼠(buffalo rat)肝细胞(BRL 3A);人肺细胞(W138);人肝细胞(Hep G2);小鼠乳房肿瘤(MMT 060562);TRI细胞,如记载于例如Mather等,Annals N.Y.Acad.Sci.383:44-68(1982)的;MRC 5细胞;和FS4细胞。其它有用的哺乳动物宿主细胞系包括中国仓鼠卵巢(CHO)细胞,包括DHFR-CHO细胞(Urlaub等,Proc.Natl.Acad.Sci.USA 77:4216(1980));和骨髓瘤细胞系诸如Y0,NS0和Sp2/0。关于适合于抗体生成和/或结合多肽生成的某些哺乳动物宿主细胞系的综述,见例如Yazaki和Wu,METHODS IN MOL.BIOL.,第248卷(B.K.C.Lo编,Humana Press,Totowa,NJ),第255-268页(2003)。
虽然描述主要涉及通过培养用含有抗体和结合多肽编码核酸的载体转化或转染的细胞来生成抗体和/或结合多肽。当然涵盖了可采用本领域公知的备选方法来制备抗体和/或结合多肽。例如,可使用固相技术通过直接肽合成来生成适宜的氨基酸序列或其部分[见例如Stewart等,Solid-Phase Peptide Synthesis,W.H.Freeman Co.,San Francisco,CA(1969);Merrifield,J.Am.Chem.Soc.,85:2149-2154(1963)]。体外蛋白质合成可使用手工技术或通过自动化来进行。自动化合成可例如使用Applied Biosystems肽合成仪(Foster City,CA)使用制造商的说明书来完成。抗体和/或结合多肽的多个部分可分开化学合成,并使用化学或酶促方法组合以生成期望的抗体和/或结合多肽。
IV.筛选和/或鉴定具有期望功能的MiT拮抗剂的方法
上文已经描述了用于生成MiT拮抗剂诸如抗体,结合多肽,和/或小分子的技术。对于别的MiT拮抗剂,诸如本文中提供的抗MiT抗体,结合多肽,小分子,和/或多核苷酸,可以通过本领域中已知的多种测定法对其鉴定,筛选,或表征其物理/化学特性和/或生物学活性。
本文中提供筛选和/或鉴定抑制MiT信号传导,诱导癌细胞周期停滞,抑制癌细胞增殖和/或促进癌细胞死亡的MiT拮抗剂的方法,所述方法包括:(a)使(i)包含一种或多种生物标志物的癌细胞,癌症组织和/或癌症样品和(ii)参照细胞,参照组织和/或参照样品与MiT候选拮抗剂接触,(b)测定MiT结合的存在或缺失,MiT信号传导的水平,细胞周期阶段的分布,细胞增殖的水平和/或癌细胞死亡的水平,其中包含一种或多种生物标志物的癌细胞,癌症组织和/或癌症样品与参照癌细胞,参照癌症组织和/或参照癌症样品之间降低的MiT信号传导水平,细胞周期阶段分布的差异,降低的细胞增殖水平和/或升高的癌细胞死亡水平鉴定出作为MiT拮抗剂的MiT候选拮抗剂,其结合MiT,抑制MiT信号传导,诱导癌细胞周期停滞,抑制癌细胞增殖和/或促进癌细胞癌症死亡。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
另外,本文中提供筛选和/或鉴定结合MiT,抑制MiT信号传导,诱导癌细胞周期停滞,抑制癌细胞增殖和/或促进癌细胞死亡的MiT拮抗剂的方法,所述方法包括:(a)使癌细胞,癌症组织和/或癌症样品与MiT候选拮抗剂接触,其中该癌细胞,癌症组织和/或癌症样品包含一种或多种生物标志物,(b)相对于在MiT候选拮抗剂缺失下的癌细胞,癌症组织和/或癌症样品,测定MiT结合的存在或缺失,MiT信号传导的水平,细胞周期阶段的分布,细胞增殖的水平和/或癌细胞死亡的水平,其中在MiT候选拮抗剂存在下的癌细胞,癌症组织和/或癌症样品与在MiT候选拮抗剂缺失下的癌细胞,癌症组织和/或癌症样品与之间降低的MiT结合,MiT信号传导水平,细胞周期阶段分布的差异,降低的细胞增殖水平和/或升高的癌细胞死亡水平鉴定出作为MiT拮抗剂的MiT候选拮抗剂,其不结合MiT,抑制MiT信号传导,诱导癌细胞周期停滞,抑制癌细胞增殖和/或促进癌细胞癌症死亡。在一些实施方案中,该MiT拮抗剂是MET途径拮抗剂。在一些实施方案中,该MiT拮抗剂是BIRC7途径拮抗剂。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)。在一些实施方案中,该一种或多种生物标志物的存在通过一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MITF。在一些实施方案中,该一种或多种生物标志物包含TFEB。在一些实施方案中,该一种或多种生物标志物包含TFE3。在一些实施方案中,该一种或多种生物标志物包含TFEC。在一些实施方案中,该一种或多种生物标志物包含SBNO2。
在任何方法的一些实施方案中,该一种或多种生物标志物包含MET和/或BIRC7。在一些实施方案中,该一种或多种生物标志物的存在通过MET和/或BIRC7的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MET。在一些实施方案中,该一种或多种生物标志物包含BIRC7。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种MiT突变(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2中的一种或多种的突变)。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的扩增。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位或倒位(例如重排和/或融合)。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)的存在。
在任何方法的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位或倒位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种的过表达的存在。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是MITF易位(例如重排和/或融合)。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:13和/或30。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:30。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:9,10,11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。在一些实施方案中,该ASP3S1-MITF易位存在于透明细胞RCC中。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是TFEB易位(例如重排和/或融合)。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含SEQ ID NO:19。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:15,16,17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是由CLTC启动子驱动的。
在任何方法的一些实施方案中,该易位(例如重排和/或融合)是SBNO2易位(例如重排和/或融合)。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN和SBNO2。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2易位(例如重排和/或融合)包含SEQ ID NO:25。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:23和/或24的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是通过包括SEQ ID NO:21,22,23,和/或25的引物可检测的。在一些实施方案中,该SBNO2易位(例如重排和/或融合)是由CLTC启动子驱动的。
测定MiT信号传导水平的方法是本领域已知的且在本文实施例中有描述。在一些实施方案中,使用实施例中所述萤光素酶报告物测定法测定MiT信号传导水平。在一些实施方案中,该MiT拮抗剂通过将MiT信号传导水平降低约10,20,30,40,50,60,70,80,90,或100%任一而抑制MiT信号传导。
可通过本领域中已知的方法来评估本文描述的MiT拮抗剂的生长抑制作用,例如使用内源性表达或在用相应基因转染后表达MiT的细胞。例如,可将适宜的肿瘤细胞系和经MiT多肽转染的细胞用各种浓度的本文中描述的MiT拮抗剂处理几天(例如2-7天)并用结晶紫或MTT染色,或通过一些其他比色测定法分析。另一种测量增殖的方法将通过比较在存在或不存在本发明的抗体,结合多肽,小分子和/或多核苷酸的情况下处理的细胞的3H-胸苷摄取。在处理后,收获细胞并在闪烁计数器中对掺入DNA的放射性量定量。适宜的阳性对照包括将选定的细胞系用已知抑制该细胞系生长的生长抑制性抗体处理。可以以本领域已知的多种方式来测定肿瘤细胞的体内生长抑制。
测定细胞周期阶段的分布,细胞增殖水平和/或细胞死亡水平的方法是本领域中已知的。在一些实施方案中,癌细胞周期停滞是G1中的停滞。
在一些实施方案中,所述MiT拮抗剂会将癌细胞,癌症组织或癌症样品的体外或体内癌细胞增殖相比于未处理的癌细胞,癌症组织或癌症样品抑制约25-100%,更优选地约30-100%,甚至更优选地约50-100%或约70-100%。例如,生长抑制可在细胞培养物中MiT拮抗剂浓度为约0.5至约30μg/ml或约0.5nM至约200nM时测量,其中在肿瘤细胞暴露于MiT候选拮抗剂后1-10天测定生长抑制。如果以约1μg/kg至约100mg/kg体重的MiT候选拮抗剂施用导致从MiT候选拮抗剂的首次施用起约5天至3个月内(优选约5至30天内)肿瘤大小降低或肿瘤细胞增殖降低,则所述MiT拮抗剂是体内生长抑制性的。
为了选择诱导癌细胞死亡的MiT拮抗剂,可评估由例如碘化丙啶(PI),锥虫蓝(trypan blue)或7AAD摄取指示的相对于参照的膜完整性的丧失。可在缺乏补体和免疫效应器细胞的情况下实施PI摄取测定法。将表达MiT的肿瘤细胞与单独的培养基或含有适宜的MiT拮抗剂的培养基温育。将细胞温育达3天时间段。在每份处理后,清洗细胞并等分试样到35mm过滤器加盖的12x75管(1ml每管,3个管每处理组)中以除去细胞块。然后管接收PI(10μg/ml)。可以使用流式细胞器和CellQuest软件(Becton Dickinson)来分析样品。可将诱导统计学显著的细胞死亡水平(如通过PI摄取测定)的那些MiT拮抗剂选为细胞死亡诱导性抗体,结合多肽,小分子和/或多核苷酸。
为了筛选结合表位或与目标抗体结合的多肽相互作用的MiT拮抗剂,可以实施常规的交联阻断测定法,如记载于Antibodies,A Laboratory Manual,Cold Spring Harbor Laboratory,Ed Harlow和David Lane(1988)的。该测定法可用于确定候选MiT拮抗剂是否与已知抗体结合相同的位点或表位。或者/另外地,可通过本领域中已知的方法实施表位作图。例如,抗体和/或结合多肽序列可如通过丙氨酸扫描诱变以鉴定接触残基。首先对突变抗体测试与多克隆抗体和/或结合多肽的结合以确保正确折叠。在一种不同的方法中,可在竞争测定法中使用对应于多肽不同区域的肽,使用几种候选抗体和/或结合多肽,或者一种候选抗体和/或结合多肽和具有已表征或已知表位的抗体。
在任何筛选和/或鉴定方法的一些实施方案中,所述MiT候选拮抗剂是抗体,结合多肽,小分子或多核苷酸。在一些实施方案中,所述MiT候选拮抗剂是抗体。在一些实施方案中,所述MiT拮抗剂是小分子。
在一个方面,例如通过已知方法诸如ELISA,Western印迹,等对MiT拮抗剂测试它的抗原结合活性。
V.药物配制剂
通过将具有期望纯度的此类抗体与一种或多种任选的药学可接受载体(REMINGTON’S PHARMA.SCI.,第16版,Osol,A.编(1980))混合以冻干配制剂或水性溶液形式制备如本文中所描述的MiT拮抗剂的药物配制剂。在一些实施方案中,MiT拮抗剂是小分子,抗体,结合多肽,和/或多核苷酸。一般地,药学可接受载体在所采用的剂量和浓度对接受者是无毒的,而且包括但不限于缓冲剂,诸如磷酸盐,柠檬酸盐,和其它有机酸;抗氧化剂,包括抗坏血酸和甲硫氨酸;防腐剂(诸如氯化十八烷基二甲基苄基铵;氯化己烷双胺;苯扎氯铵,苄索氯铵;酚,丁醇或苯甲醇;对羟基苯甲酸烃基酯,诸如对羟基苯甲酸甲酯或丙酯;邻苯二酚;间苯二酚;环己醇;3-戊醇;和间甲酚);低分子量(少于约10个残基)多肽;蛋白质,诸如血清清蛋白,明胶或免疫球蛋白;亲水性聚合物,诸如聚乙烯吡咯烷酮;氨基酸,诸如甘氨酸,谷氨酰胺,天冬酰胺,组氨酸,精氨酸或赖氨酸;单糖,二糖和其它碳水化合物,包括葡萄糖,甘露糖或糊精;螯合剂,诸如EDTA;糖类,诸如蔗糖,甘露醇,海藻糖或山梨醇;成盐相反离子,诸如钠;金属复合物(例如Zn-蛋白质复合物);和/或非离子表面活性剂,诸如聚乙二醇(PEG)。本文中的例示性的药学可接受载体进一步包含间质药物分散剂诸如可溶性中性活性透明质酸酶糖蛋白(sHASEGP),例如人可溶性PH-20透明质酸酶糖蛋白,诸如rHuPH20Baxter International,Inc.)。某些例示性的sHASEGP和使用方法,包括rHuPH20记载于美国专利公开文本No.2005/0260186和2006/0104968。在一个方面,将sHASEGP与一种或多种别的糖胺聚糖酶诸如软骨素酶组合。
例示性的冻干配制剂记载于美国专利No.6,267,958。水性抗体配制剂包括那些记载于美国专利No.6,171,586和WO2006/044908的,后一种配制剂包含组氨酸-乙酸盐缓冲液。
本文中的配制剂还可含有超过一种所治疗具体适应症所必需的活性组分,优选那些活性互补且彼此没有不利影响的组分。此类活性组分适于以有效用于所需目的的量而组合存在。
活性成分可包载于例如通过凝聚技术或通过界面聚合制备的微胶囊中(例如分别是羟甲基纤维素或明胶微胶囊和聚(甲基丙烯酸甲酯)微胶囊),在胶状药物投递系统中(例如脂质体,清蛋白微球体,微乳剂,纳米颗粒和纳米胶囊),或在粗滴乳状液中。此类技术披露于REMINGTON’S PHARMA.SCI.,第16版,Osol,A.编(1980)。
可以制备持续释放制剂。持续释放制剂的合适的例子包括含有MiT拮抗剂的固体疏水性聚合物的半透性基质,该基质为成形商品形式,例如膜,或微胶囊。
用于体内施用的配制剂一般是无菌的。无菌性可容易地实现,例如通过穿过无菌滤膜过滤。
VI.制品
在本发明的另一个方面,提供了含有上文所述可用于治疗,预防和/或诊断病症的材料的制品。所述制品包括容器和所述容器上或与所述容器相连的标签或包装插页。合适的容器包括例如药瓶,药管,注射器,IV溶液袋,等。所述容器可以用多种材料制成,诸如玻璃或塑料。所述容器装有独自或与另一组合物组合有效治疗,预防和/或诊断所述疾患的组合物,而且可以具有无菌存取口(例如所述容器可以是静脉内溶液袋或带有皮下注射针可刺穿的塞子的管形瓶)。所述组合物中的至少一种活性药剂是本文所述MiT拮抗剂(例如MiT拮抗剂,例如MiT易位拮抗剂)。所述标签或包装插页指明该组合物用于治疗选择的疾患。此外,制品可包括(a)其中装有组合物的第一容器,其中所述组合物包含MiT拮抗剂(例如MiT拮抗剂,例如MiT易位拮抗剂);和(b)其中装有组合物的第二容器,其中所述组合物包含其它的细胞毒性或其它方面治疗性的药剂。
在一些实施方案中,所述制品包含容器,所述容器上的标签和所述容器内含有的组合物;其中所述组合物包含一种或多种试剂(例如结合一种或多种生物标志物的一抗,或针对本文所述一种或多种生物标志物的探针和/或引物),指示该组合物可用于评估样品中一种或多种生物标志物的存在的容器上的标签,和用于使用所述试剂来评估样品中一种或多种生物标志物的存在的说明书。所述制品可进一步包括一套用于制备样品和利用试剂的说明书和材料。在一些实施方案中,所述制品可以包含试剂如一抗和二抗两者,其中二抗缀合于一种标记物,例如酶标记物。在一些实施方案中,所述制品包含针对本文所述一种或多种生物标志物的一种或多种探针和/或引物。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)。在一些实施方案中,该一种或多种生物标志物的存在通过一种或多种MiT(例如MITF,TFEB,TFE3,TFEC,和/或SBNO2)的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MITF。在一些实施方案中,该一种或多种生物标志物包含TFEB。在一些实施方案中,该一种或多种生物标志物包含TFE3。在一些实施方案中,该一种或多种生物标志物包含TFEC。在一些实施方案中,该一种或多种生物标志物包含SBNO2。
在任何制品的一些实施方案中,该一种或多种生物标志物包含MET和/或BIRC7。在一些实施方案中,该一种或多种生物标志物的存在通过MET和/或BIRC7的升高的表达水平(例如与参照相比)的存在来指示。在一些实施方案中,该表达是多肽表达。在一些实施方案中,该表达是核酸表达。在一些实施方案中,该一种或多种生物标志物包含MET。在一些实施方案中,该一种或多种生物标志物包含BIRC7。
在任何制品的一些实施方案中,该一种或多种生物标志物包含MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在通过MITF,TFEB,TFE3,TFEC,SBNO2,MET和/或BIRC7中的一种或多种升高的表达水平(例如与参照相比)的存在来指示。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)的存在。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的突变。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的扩增。
在任何制品的一些实施方案中,该一种或多种生物标志物包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合),和MET和/或BIRC7中的一种或多种。在一些实施方案中,该一种或多种生物标志物的存在包含一种或多种选自MITF,TFEB,TFE3,TFEC,和/或SBNO2的基因的易位(例如重排和/或融合)的存在,和MET和/或BIRC7中的一种或多种的过表达。
在任何制品的一些实施方案中,该易位(例如重排和/或融合)是MITF易位(例如重排和/或融合)。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含SEQ ID NO:13和/或30。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ ID NO:11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是通过包括SEQ IDNO:9,10,11和/或12的引物可检测的。在一些实施方案中,该MITF易位(例如重排和/或融合)是由ACTG1启动子驱动的。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1和MITF。在一些实施方案中,该MITF易位(例如重排和/或融合)包含AP3S1外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)包含ACTG1外显子3和MITF外显子3。在一些实施方案中,该MITF易位(例如重排和/或融合)是由AP3S1启动子驱动的。
在任何制品的一些实施方案中,该易位(例如重排和/或融合)是TFEB易位(例如重排和/或融合)。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC和TFEB。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含CLTC外显子17和TFEB外显子6。在一些实施方案中,该TFEB易位(例如重排和/或融合)包含SEQ ID NO:19。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是通过包括SEQ ID NO:15,16,17和/或18的引物可检测的。在一些实施方案中,该TFEB易位(例如重排和/或融合)是由CLTC启动子驱动的。
在任何制品的一些实施方案中,该易位(例如重排和/或融合)是SBNO2倒位(例如重排和/或融合)。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)包含MIDN和SBNO2。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)包含MIDN启动子和SBNO2外显子1。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)包含SEQ ID NO:25。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)是通过包括SEQ ID NO:23和/或24的引物可检测的。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)是通过包括SEQ ID NO:21,22,23,和/或25的引物可检测的。在一些实施方案中,该SBNO2倒位(例如重排和/或融合)是由CLTC启动子驱动的。
在任何制品的一些实施方案中,该制品包含引物。在一些实施方案中,该引物是SEQ ID NO:9,10,11,12,15,16,17,18,21,22,23,和/或24任一。在一些实施方案中,该引物是SEQ ID NO:9,10,11,和/或12中的任一种或多种。在一些实施方案中,该引物是SEQ ID NO:15,16,17,和/或18中的任一种或多种。在一些实施方案中,该引物是SEQ ID NO:21,22,23,和/或24中的任一种或多种。
在任何制品的一些实施方案中,该MiT拮抗剂是抗体,结合多肽,小分子,或多核苷酸。在一些实施方案中,该MiT拮抗剂是小分子。在一些实施方案中,该MiT拮抗剂是抗体。在一些实施方案中,该抗体是单克隆抗体。在一些实施方案中,该抗体是人,人源化或嵌合抗体。在一些实施方案中,该抗体是抗体片段且该抗体片段结合MiT多肽。
本发明的此实施方案中的制品还可包括指示所述组合物可用于治疗特定疾患的包装插页。或者/另外,所述制品可进一步包括第二(或第三)容器,其中装有药学可接受的缓冲液,诸如注射用抑菌水(BWFI),磷酸盐缓冲盐水,林格(Ringer)氏溶液和右旋糖溶液。它可进一步包括从商业和用户立场出发需要的其它材料,包括其它缓冲液,稀释剂,滤器,针头,和注射器。
制品中的其它任选成分包括一种或多种缓冲液(例如封闭缓冲液,清洗缓冲液,底物缓冲液等),其它试剂诸如被酶标记物化学改变的底物(例如色原),表位修复液,对照样品(阳性和/或阴性对照),对照载玻片等。
理解的是,任何上述制品可包括本文所述免疫缀合物代替或补充MiT拮抗剂。
序列
实施例
以下是本发明的方法和组合物的实施例。应当理解,鉴于上文提供的一般描述,可以实施各种其它实施方案。
用于实施例的材料和方法
样品,DNA和RNA制备:在这项研究中分析了167份人原代nccRCC样品及其邻近的正常组织。nccRCC样品包括67份pRCC,49份chRCC,35份RO,8份未分类类型的癌症,6份tRCC和2份具有肉瘤样去分化的样品(表2和3)。
表2:样品汇总表
表3:样品信息
该研究中使用的样品是自为了RCC的肾块或转移在Saint Paul University医院,Parkland Memorial医院和Zale Lipshy University医院经历手术的患者获得的。这些医院代表三级护理转诊中心(Saint Paul和Zale Lipshy)以及县级医院(Parkland Memorial)且服务极其广泛的多个种族的患者,包括高加索人,西班牙裔,非洲裔美国人,亚裔和南亚裔。如果患者已知具有HIV,HBV,HCV或TB感染,那么将他们排除在外。
这项研究是在有适当的IRB批准和患者知情同意书的情况下进行的。人组织样品在使用它们之前去除标识且依照美国人体健康服务部规章和相关指南(45CFR Part 46)不认为是人受试者研究。肿瘤和邻近的正常肾样品在液氮至新鲜冷冻并保存于-80℃。紧邻所有冷冻的肿瘤和正常组织的1-3mm厚碎片侧翼的垂直切片由病理学家查看以确认诊断和肿瘤含量1。在可得的情况下,该研究中的患者样品的基本人口统计学信息包括在表3中。组织加工以及自相同样品同时提取高质量的基因组DNA和总RNA如先前描述的那样实施1。
RNA-seq:我们获得了159份肿瘤样品的RNA-seq数据(119份有肿瘤/正常对的数据)。使用TruSeq RNA样品制备试剂盒(Illumina,CA)制备了RNA-seq文库。该文库重复每个泳道三次并在HiSeq 2000上测序以获得平均而言每份样品~68x106个成对末端(2x 75bp)读出。
序列数据加工:使用Bioconductor ShortRead package2对所有测序读出评估了质量。为了确认所有样品得到正确鉴定,比较了与Illumina 2.5M阵列数据交叠的所有外显子组和RNA-seq数据变量并检查了一致性。在别的数据分析之前对种系变体进行了一项全部对全部样品比较以确认患者匹配的肿瘤-正常配对。
变量调用:使用BWA软件3(设置为缺省参数)将测序读出定位至UCSC人基因组(GRCh37/hg19)。如先前所述4实施局部重新比对,重复标记和原始变体调用。使用Strelka5对肿瘤和它的匹配正常BAM文件实施体细胞变体调用。我们使用最小Strelka变体质量得分1来过滤变体。对所有样品过滤出在dbSNP Build 1316或6515个先前发表的正常外显子组7中有呈现但在COSMIC v628中未呈现的已知种系变体。另外,去除在肿瘤和正常样品二者中都存在的种系变体。为了评估这种算法的性能,我们随机选择了178种蛋白质改变性变体并如先前所述9使用Sequenom核酸技术验证它们。其中,92%(164/178)验证为体细胞的。自最终的集合去除所有未得到Sequenom验证的变体。标记为经过验证的:RNA-Seq的变体在RNA-seq数据中显示该变体经过确认的表达。在上文所述dbSNP变体过滤以外,未配对的样品针对来自这个数据集的正常变体以及来自先前发表的结肠数据集10的正常过滤它们的初始调用变体。使用PolyPhen11,SIFT12,和Condel13预测所有非同义体细胞突变对基因功能的影响。使用Ensembl(release 63,www.ensembl.org)注释所有变体。
RNA-seq数据分析:使用GSNAP20将RNA-seq读出与人基因组版本NCBI GRCh37比对。通过对由NCBI和Ensembl基因注释和RefSeq mRNA序列定义的在一对内协调排列且对每个基因座唯一的读出的数目计数,获得表达计数/基因。使用edgeR21和DESeq222实施的差异基因表达分析来计算方差稳定化的表达值,用于表达热图绘图。使用GATK4确定RNA-seq数据中的变量。
基因融合检测和验证:使用我们开发的称作GSTRUCT-融合的计算管道18鉴定推定的融合。只包括有至少3个读出定位至融合接合处且在任何正常样品中没有找到的融合事件用于进一步考虑。而且,我们去除了包括未注释的外显子或具有密切相关序列的融合配偶的事件,因为这些很可能是假阳性。如先前描述的那样18用nccRCC肿瘤和匹配的正常样品使用RT-PCR进行基因融合的验证。
细胞和质粒:在补充有10%FBS的DMEM中维持自Genentech细胞库获得的NIH3T3和HEK293T细胞。自pCMV6表达载体表达C端带Myc/DDK标签的MITF,ACTG1和MET的克隆购自Origene,MD。通过交叠PCR使用剪接生成了具有3’C端Myc/DDK标签序列的ACTG1-MITF融合物并克隆入pCMV6。CA)。
FISH分析:在带正电荷的载玻片上封固FFPE(福尔马林固定,石蜡包埋)组织的3微米切片。由委员会认证的病理学家(PK)实施苏木精和曙红(H&E)染色的载片上靶区和组织的选择。使用H&E载片作为参照,在要检定的未染色的载片的背面上用金刚石尖端蚀刻机蚀刻靶区。依照DAKO组织学FISH附属试剂盒手册(SSK5799CE_001/EFG/LMA/2012)中的微波方法实施预处理,杂交和后洗。针对TFE3(Xp11.2)和TFEB(6p21.1)的DNA探针集是自AgilentTechnologies,CA获得的。
MITF稳定性分析:依照制造商的说明书(Roche,CA)使用Fugene 6用pCMV6-MITF(0.5μg)和pCMV6-ACTG1-MITF(0.3μg)转染HEK293T(1X105个细胞/孔)。我们在转染中使用较小量的pCMV6-ACTG1-MITF,因为我们发现它的表达与野生型MITF相比要高。在转染后24小时时,用50μg/ml环乙酰亚胺(Sigma,MO)处理细胞以阻断翻译。如先前描述的那样28,加工样品并提交Western印迹。使用小鼠抗c-myc抗体(9E10,Genentech Inc.,CA)通过Western印迹评估MITF蛋白质。使用家兔抗hsp90抗体(Santa Cruz Biotechnology,CA)评估hsp90表达并用作加载对照。在LI-COR Odyssey成像仪(LI-COR Biotechnology,NE)上使用适宜的二抗分析表达。
Western印迹分析:如先前描述的那样28,制备来自稳定表达野生型或突变型任一的NIH3T3(5x 106个细胞)的细胞裂解物并用于Western印迹。使用抗phosho-Met(Y1234/35)抗体(Cell signaling,CA)评估MET的磷酸化状态。如图中指出的那样,使用抗Flag抗体(Sigma,MO)或抗hsp90抗体(Santa Cruz,CA)通过Western印迹评估NIH3T3稳定系中MET,ACTG1,MITF,ACTG1-MITF,hsp90的表达。如先前描述的那样28,使用适宜的二抗实施免疫印迹。
锚定不依赖性生长:如先前描述的那样实施该测定法28。简言之,将20,000个稳定表达带Flag标签的MET-WT,MET突变体,ACTG1,MITF或ACTG1-MITF的NIH3T3细胞与DMEM(高葡萄糖)中的0.35%琼脂混合并一式三份在6孔板中在0.5%基础琼脂上涂板。然后用完全生长培养基(1ml)覆盖涂板的细胞并于37℃温育。3周后使用GelCount(Oxford Optronix Ltd,UK)评估每块板中形成的集落的数目。使用GraphPad Prism 5.00(GraphPad Software,San Diego,CA)用Student氏t检验(双尾)进行统计学分析以比较各处理组。p值<0.05认为是统计学显著的(*p<0.05和**p<0.01)。
细胞生长测定法:在完全培养基中分配表达野生型或D153Y突变型MET的NIH3T3稳定细胞。24小时后用无血清培养基替换完全培养基并用指定浓度的重组HGF(R&D system,MN)处理。3天后如先前描述的那样28用Cell Titer-Glo发光细胞存活力试剂盒(Promega Corp.,WI)测量细胞生长。使用GraphPad Prism 5.00(GraphPad Software,San Diego,CA)用Student氏t检验(双尾)进行统计学分析以比较各处理组。p值<0.05认为是统计学显著的(*p<0.05和**p<0.01)。
定量PCR分析:使用VILOTM大师级混合物(Life Technologies,CA)逆转录转染后24小时时使用RNeasy迷你试剂盒(Qiagen)自用MITF或MITF融合构建体转染的细胞分离的RNA(1ug)以生成cDNA。然后稀释cDNA并用于使用Taqman基因表达大师级混合物在ViiA 7实时PCR系统(Life Technologies,CA)上进行的定量PCR。用于Taqman基因表达测定法的引物和探针套组(20x)是自Life Technologies获得的。所使用的引物和探针套组包括GAPDH(Hs02758991_g1),MITF(Hs01117294_m1),HIF1A(Hs00153153_m1),MET(Hs01565584_m1),和APEX1(Hs00959050_g1)。将相对基因表达值针对GAPDH表达标准化,然后针对经转染HEK293T细胞中的MITF mRNA水平进一步标准化。
结果
在突变,拷贝数变化和表达改变以外,越来越多地认识到基因融合在实体瘤中发挥重大驱动者作用64。MiTF碱性螺旋-环-螺旋(bHLH)转录因子中的一些,TFE3,TFEB,TFEC,和MITF65在癌症中失调。已知tRCC中牵涉TFE3和TFEB的易位。通过IHC检测TFE3表达和/或用FISH检测它的易位已经用于TFE3融合阳性tRCC的分类。TFEB易位是低频率事件且常常在临床上遗漏。我们分析了我们的RNA-seq数据以评估它用于在tRCC中发现新颖融合和检测已知融合的效用。在通过TFE3IHC分类为tRCC的样品中,我们为先前报告的ASPSCR1-TFE3融合18,66,67和PRCC-TFE3融合67找到了证据。这通过FISH得到进一步确认(图1B,C)。我们在两份tRCC样品(14336T和PtS1T)中没有检测到牵涉TFE3(或其它MiTF成员)的融合事件,尽管它们显示升高的TFE3表达(图2)。而且,我们在这些样品中没有为TFE3扩增找到证据,提示牵涉另一种机制导致它的上调。然而,在缺乏TFE3融合的tRCC样品之一(14336T)中,我们鉴定出牵涉中脑核仁蛋白(midnolin,MIDN)(一种核仁蛋白质)和草莓刻缺蛋白同系物2(SBNO2)(DExD/H螺旋酶家族辅阻抑物)的融合。MIDN和SBNO2位于染色体19p13.3且由相对的链编码(图3)。观察到的融合很可能是由于19p13.3中的基因组倒位,它在转录和剪接时将MIDN的非编码外显子放置于SBNO2第二外显子的5’端。这产生在MIDN启动子控制下的编码全长SBNO2的转录物(图3)。有趣的是,具有MIDN-SBNO2的样品具有第二位最高水平的SBNO2表达(图4)。而且,第二份缺乏TFE3融合的tRCC样品(PtS1T)具有最高水平的SBNO2表达,尽管导致SBNO2上调的确切机制仍然有待确定(图4)。最近,已经显示SBNO2经由MITF68活化在骨稳态中发挥至关重要作用。然而,SBNO2是否能调控包括TFE3在内的其它MiTF成员的水平和转录活性需要进一步调查。
在TFE3融合以外,我们在指派为未分类的一份nccRCC样品(8432T;图5)中找到了牵涉CLTC和TFEB的一种未报告的基因融合(CLTC-TFEB)。我们使用FISH确认了此融合(图1D)。CLTC-TFEB编码一种同框融合蛋白,其含有TFEB的活化和HLH域,正如在其它已知TFEB融合中观察到的66,指示它很可能是功能性的。
类似地,我们在另一份未分类样品(20825T1)中找到了PRCC-TFE3融合。基于融合的存在,8432T和20825T1均遵循别的病理学审查重分类为tRCC。
一份pRCC样品(1216T)显示染色体6中包括TFEB的一个490Kb区域的扩增(图6A)。我们使用FISH确认了扩增事件(图1A)。与扩增一致,此样品具有与所有其它样品相比最高水平的TFEB表达(图6E),指示在先前已知的tRCC中的TFEB易位以外,扩增可能是pRCC亚型中的另一种癌症相关TFEB改变。鉴于TFEB扩增,我们重评估了此样品的组织学并发现它还具有与tRCC一致的特征。
虽然已经发现了MITF中的ccRCC倾向性种系突变,但是就我们所知,尚无nccRCC中牵涉MITF的基因融合的报告66。我们在这项研究中鉴定出牵涉ACTG1和MITF的ACTG1-MITF基因融合(图7)。我们验证和确认了ACTG1-MITF是体细胞的(图7)。表达ACTG1-MITF融合的样品(159T)在组织学上分类为pRCC。由ACTG1-MITF编码的融合蛋白的大小与野生型MITF大致相同,只是MITF的头118个氨基酸被ACTG1的N端121个氨基酸替换(图7B)。我们发现了表达该融合的肿瘤具有与匹配的正常相比更高水平的MITF(图7C)。为了进一步了解ACTG1-MITF融合蛋白,我们将表达该融合蛋白的cDNA转染入HEK293T细胞并测试已知MITF靶基因的表达69,70。我们发现了ACTG1-MITF融合显示与野生型MITF相比HIF1A,MET和APEX1基因诱导的显著升高(图8和图9)。鉴于MITF中的ccRCC倾向性突变认为是通过提高它的稳定性来发挥功能的71,我们通过跟踪它在细胞中的周转对ACTG1-MITF融合评估它的稳定性。我们发现了ACTG1-MITF蛋白与野生型MITF相比更加稳定(图8B)。而且,通过在NIH3T3细胞中稳定表达融合和野生型MITF蛋白并对它们评估锚定不依赖性生长,我们测试了ACTG1-MITF的转化能力(图8C-E)。我们发现了与野生型MITF相比,表达ACTG1-MITF的细胞具有显著数目的锚定不依赖性集落(图8D-E)。总之,这些数据提示像TFE3/TFEB融合一样,MITF融合能促进nccRCC中的肿瘤发生。
MiTF蛋白同二聚化或以各种组合与其它家族成员异二聚化且结合相似DNA元件以调控基因表达72。鉴于此,我们对具有MiTF融合/扩增的样品评估了在它们之间共同上调的基因,它们能充当药物靶。我们发现了MiTF融合/扩增样品中的大多数(4/5)显示升高的BIRC7表达(图10)。BIRC7(一种抗凋亡蛋白)是已知在数种癌症中上调的MITF靶基因69,70。我们的数据提示BIRC7表达可能有助于诊断易位癌症和过表达MiTF成员的癌症。敏化癌细胞发生凋亡的小分子BIRC7抑制剂处于临床开发中69且可能证明在治疗当前仍然难处理的MiTF融合阳性或过表达肿瘤中是有效的。
已知tRCC中牵涉MiTF成员TFEB和TFE3的易位。然而,当前组织学诊断不依赖于MiTF融合的全面评估。我们显示了RNA-seq在检测已知TFEB和TFE3融合中是有效且全面的。它在发现未报告的融合(像ACTG1-MITF和CLTC-TFEB)中也是有用的。还有,RNA-seq显示了仅仅基于TFE3表达而分类为tRCC的样品中融合的缺失。然而,融合阴性样品过表达TFE3,尽管这种上调的精确机制需要进一步调查。我们的分析说明了单独基于组织学对tRCC样品分类中所涉及的复杂性并提倡使用整合组织基因组办法进行诊断。
我们使用RNA-seq的全面易位分析鉴定出牵涉MITF的ACTG1-MITF融合和先前未报告的TFEB融合,其牵涉一种新的融合配偶CLTC,扩充了在nccRCC中观察到的融合。我们显示了MITF融合能够诱导下游靶基因表达且与野生型MITF相比更加稳定。在nccRCC的一个子集中,MiTF成员的上调看来是一种共同的根本机制,其经由基因融合,扩增或其它有待发现的机制来实现。这提示具有MiTF融合,涵盖tRCC,和/或MiTF(MITF/TFE3/TFEB)过表达的肿瘤很可能形成一种独特的nccRCC MiTF高亚型。
我们在MiTF高亚型中鉴定药物靶的努力显示这些肿瘤中的大多数表达抗凋亡蛋白BIRC7。这提示MitF高肿瘤可能是牵涉BIRC7抑制剂的疗法的候选,它能敏化这些肿瘤以诱导凋亡。
部分参考文献列表
1.Argani,P.,Olgac,S.,Tickoo,S.K.,Goldfischer,M.,Moch,H.,Chan,D.Y.,Eble,J.N.,Bonsib,S.M.,Jimeno,M.,Lloreta,J.,et al.(2007).Xp11 translocation renal cell carcinoma in adults:expanded clinical,pathologic,and genetic spectrum.The American journal of surgical pathology 31,1149-1160.
2.Beleut,M.,Zimmermann,P.,Baudis,M.,Bruni,N.,Buhlmann,P.,Laule,O.,Luu,V.D.,Gruissem,W.,Schraml,P.,and Moch,H.(2012).Integrative genome-wide expression profiling identifies three distinct molecular subgroups of renal cell carcinoma with different patient outcome.BMC Cancer 12,310.
3.Bellmunt,J.,and Dutcher,J.(2013).Targeted therapies and the treatment of non-clear cell renal cell carcinoma.Annals of oncology:official journal of the European Society for Medical Oncology/ESMO 24,1730-1740.
4.Burwinkel,B.,Scott,J.W.,Buhrer,C.,van Landeghem,F.K.,Cox,G.F.,Wilson,C.J.,Grahame Hardie,D.,and Kilimann,M.W.(2005).Fatal congenital heart glycogenosis caused by a recurrent activating R531Q mutation in the gamma 2-subunit of AMP-activated protein kinase(PRKAG2),not by phosphorylase kinase deficiency.Am J Hum Genet 76,1034-1049.
5.Calderaro,J.,Labrune,P.,Morcrette,G.,Rebouissou,S.,Franco,D.,Prevot,S.,Quaglia,A.,Bedossa,P.,Libbrecht,L.,Terracciano,L.,et al.(2013).Molecular characterization of hepatocellular adenomas developed in patients with glycogen storage disease type I.Journal of hepatology 58,350-357.
6.Carling,D.,Mayer,F.V.,Sanders,M.J.,and Gamblin,S.J.(2011).AMP-activated protein kinase:nature's energy sensor.Nat Chem Biol 7,512-518.
7.Cheng,H.,Fukushima,T.,Takahashi,N.,Tanaka,H.,and Kataoka,H.(2009).Hepatocyte growth factor activator inhibitor type 1regulates epithelial to mesenchymal transition through membrane-bound serine proteinases.Cancer Res 69,1828-1835.
8.Choueiri,T.K.,Vaishampayan,U.,Rosenberg,J.E.,Logan,T.F.,Harzstark,A.L.,Bukowski,R.M.,Rini,B.I.,Srinivas,S.,Stein,M.N.,Adams,L.M.,et al.(2013).Phase II and biomarker study of the dual MET/VEGFR2 inhibitor foretinib in patients with papillary renal cell carcinoma.Journal of clinical oncology:official journal of the American Society of Clinical Oncology 31,181-186.
9.Contractor,H.,Zariwala,M.,Bugert,P.,Zeisler,J.,and Kovacs,G.(1997).Mutation of the p53 tumour suppressor gene occurs preferentially in the chromophobe type of renal cell tumour.The Journal of pathology 181,136-139.
10.Davies,J.K.,Wells,D.J.,Liu,K.,Whitrow,H.R.,Daniel,T.D.,Grignani,R.,Lygate,C.A.,Schneider,J.E.,Noel,G.,Watkins,H.,et al.(2006).Characterization of the role of gamma2 R531G mutation in AMP-activated protein kinase in cardiac hypertrophy and Wolff-Parkinson-White syndrome.American journal of physiology Heart and circulatory physiology 290,H1942-1951.
11.de Moor,R.A.,Schweizer,J.J.,van Hoek,B.,Wasser,M.,Vink,R.,and Maaswinkel-Mooy,P.D.(2000).Hepatocellular carcinoma in glycogen storage disease type IV.Arch Dis Child 82,479-480.
12.Dynek,J.N.,Chan,S.M.,Liu,J.,Zha,J.,Fairbrother,W.J.,and Vucic,D.(2008).Microphthalmia-associated transcription factor is a critical transcriptional regulator of melanoma inhibitor of apoptosis in melanomas.Cancer Res 68,3124-3132.
13.Farley,M.N.,Schmidt,L.S.,Mester,J.L.,Pena-Llopis,S.,Pavia-Jimenez,A.,Christie,A.,Vocke,C.D.,Ricketts,C.J.,Peterson,J.,Middelton,L.,et al.(2013).A novel germline mutation in BAP1 predisposes to familial clear-cell renal cell carcinoma.Molecular cancer research:MCR 11,1061-1071.
14.Forbes,S.A.,Tang,G.,Bindal,N.,Bamford,S.,Dawson,E.,Cole,C.,Kok,C.Y.,Jia,M.,Ewing,R.,Menzies,A.,et al.(2010).COSMIC(the Catalogue of Somatic Mutations in Cancer):a resource to investigate acquired mutations in human cancer.Nucleic Acids Res 38,D652-657.
15.Fu,L.,Wang,G.,Shevchuk,M.M.,Nanus,D.M.,and Gudas,L.J.(2011).Generation of a Mouse Model of Von Hippel-Lindau Kidney Disease Leading to Renal Cancers by Expression of a Constitutively Active Mutant of HIF1 Cancer Res 71,6848-6856.
16.Gad,S.,Lefevre,S.H.,Khoo,S.K.,Giraud,S.,Vieillefond,A.,Vasiliu,V.,Ferlicot,S.,Molinie,V.,Denoux,Y.,Thiounn,N.,et al.(2007).Mutations in BHD and TP53 genes,but not in HNF1beta gene,in a large series of sporadic chromophobe renal cell carcinoma.Br J Cancer 96,336-340.
17.Gandino,L.,Longati,P.,Medico,E.,Prat,M.,and Comoglio,P.M.(1994).Phosphorylation of serine 985 negatively regulates the hepatocyte growth factor receptor kinase.The Journal of biological chemistry 269,1815-1820.
18.Gonzalez-Perez,A.,and Lopez-Bigas,N.(2011).Improving the assessment of the outcome of nonsynonymous SNVs with a consensus deleteriousness score,Condel.Am J Hum Genet 88,440-449.
19.Hagenkord,J.M.,Gatalica,Z.,Jonasch,E.,and Monzon,F.A.(2011).Clinical genomics of renal epithelial tumors.Cancer genetics 204,285-297.
20.Hakimi,A.A.,Pham,C.G.,and Hsieh,J.J.(2013).A clear picture of renal cell carcinoma.Nat Genet 45,849-850.
21.Haq,R.,and Fisher,D.E.(2011).Biology and clinical relevance of the micropthalmia family of transcription factors in human cancer.Journal of clinical oncology:official journal of the American Society of Clinical Oncology 29,3474-3482.
22.Hoek,K.S.,Schlegel,N.C.,Eichhoff,O.M.,Widmer,D.S.,Praetorius,C.,Einarsson,S.O.,Valgeirsdottir,S.,Bergsteinsdottir,K.,Schepsky,A.,Dummer,R.,et al.(2008).Novel MITF targets identified using a two-step DNA microarray strategy.Pigment cell&melanoma research 21,665-676.
23.Jeon,S.M.,Chandel,N.S.,and Hay,N.(2012).AMPK regulates NADPH homeostasis to promote tumour cell survival during energy stress.Nature 485,661-665.
24.Kan,Z.,Jaiswal,B.S.,Stinson,J.,Janakiraman,V.,Bhatt,D.,Stern,H.M.,Yue,P.,Haverty,P.M.,Bourgon,R.,Zheng,J.,et al.(2010).Diverse somatic mutation patterns and pathway alterations in human cancers.Nature 466,869-873.
25.Lager,D.J.,Huston,B.J.,Timmerman,T.G.,and Bonsib,S.M.(1995).Papillary renal tumors.Morphologic,cytochemical,and genotypic features.Cancer 76,669-673.
26.Lawrence,M.S.,Stojanov,P.,Polak,P.,Kryukov,G.V.,Cibulskis,K.,Sivachenko,A.,Carter,S.L.,Stewart,C.,Mermel,C.H.,Roberts,S.A.,et al.(2013).Mutational heterogeneity in cancer and the search for new cancer-associated genes.Nature 499,214-218.
27.Linehan,W.M.,and Ricketts,C.J.(2013).The metabolic basis of kidney cancer.Semin Cancer Biol 23,46-55.
28.Manzia,T.M.,Angelico,R.,Toti,L.,Cillis,A.,Ciano,P.,Orlando,G.,Anselmo,A.,Angelico,M.,and Tisone,G.(2011).Glycogen Storage Disease Type Ia and VI Associated With Hepatocellular Carcinoma:Two Case Reports.Transplantation Proceedings 43,1181-1183.
29.Maruyama,K.,Uematsu,S.,Kondo,T.,Takeuchi,O.,Martino,M.M.,Kawasaki,T.,and Akira,S.(2013).Strawberry notch homologue 2 regulates osteoclast fusion by enhancing the expression of DC-STAMP.The Journal of experimental medicine 210,1947-1960.
30.Miller,M.,Ginalski,K.,Lesyng,B.,Nakaigawa,N.,Schmidt,L.,and Zbar,B.(2001).Structural basis of oncogenic activation caused by point mutations in the kinase domain of the MET proto-oncogene:modeling studies.Proteins 44,32-43.
31.Monzon,F.A.,Hagenkord,J.M.,Lyons-Weiler,M.A.,Balani,J.P.,Parwani,A.V.,Sciulli,C.M.,Li,J.,Chandran,U.R.,Bastacky,S.I.,and Dhir,R.(2008).Whole genome SNP arrays as a potential diagnostic tool for the detection of characteristic chromosomal aberrations in renal epithelial tumors.Modern pathology:an official journal of the United States and Canadian Academy ofPathology,Inc 21,599-608.
32.Nagashima,Y.,Kuroda,N.,and Yao,M.(2013).Transition of organizational category on renal cancer.Jap J Clin Oncol 43,233-242.
33.Ng,P.C.,and Henikoff,S.(2002).Accounting for human polymorphisms predicted to affect protein function.Genome Res 12,436-446.
34.Osunkoya,A.O.(2010).Editorial comment to molecular pathology of chromophobe renal cell carcinoma:a review.International journal of urology:official journal of the Japanese Urological Association 17,600-601.
35.Palmer,R.E.,Lee,S.B.,Wong,J.C.,Reynolds,P.A.,Zhang,H.,Truong,V.,Oliner,J.D.,Gerald,W.L.,and Haber,D.A.(2002).Induction of BAIAP3 by the EWS-WT1 chimeric fusion implicates regulated exocytosis in tumorigenesis.Cancer Cell 2,497-505.
36.Palmieri,F.(2013).The mitochondrial transporter family SLC25:identification,properties and physiopathology.Molecular aspects of medicine 34,465-484.
37.Pena-Llopis,S.,and Brugarolas,J.(2013).Simultaneous isolation of high-quality DNA,RNA,miRNA and proteins from tissues for genomic applications.Nature protocols 8,2240-2255.
38.Pena-Llopis,S.,Vega-Rubin-de-Celis,S.,Liao,A.,Leng,N.,Pavia-Jimenez,A.,Wang,S.,Yamasaki,T.,Zhrebker,L.,Sivanand,S.,Spence,P.,et al.(2012).BAP1 loss defines a new class of renal cell carcinoma.Nat Genet 44,751-759.
39.Picken,M.M.(2010).Editorial comment from Dr Picken to Molecularpathology of renal oncocytoma:a review.International journal of urology:official journal of the Japanese Urological Association 17,613-614.
40.Pleasance,E.D.,Stephens,P.J.,O’Meara,S.,McBride,D.J.,Meynert,A.,Jones,D.,Lin,M.-L.,Beare,D.,Lau,K.W.,Greenman,C.,et al.(2009).A small-cell lung cancer genome with complex signatures of tobacco exposure.Nature 463,184-190.
41.Popova,T.,Hebert,L.,Jacquemin,V.,Gad,S.,Caux-Moncoutier,V.,Dubois-d'Enghien,C.,Richaudeau,B.,Renaudin,X.,Sellers,J.,Nicolas,A.,et al.(2013).Germline BAP1 mutations predispose to renal cell carcinomas.Am J Hum Genet 92,974-980.
42.Ramensky,V.,Bork,P.,and Sunyaev,S.(2002).Human non-synonymous SNPs:server and survey.Nucleic Acids Res 30,3894-3900.
43.Righi,L.,Rapa,I.,Votta,A.,Papotti,M.,and Sapino,A.(2012).Human achaete-scute homolog-1 expression in neuroendocrine breast carcinoma.Virchows Archiv:an international journal of pathology 460,415-421.
44.Rohan,S.,Tu,J.J.,Kao,J.,Mukherjee,P.,Campagne,F.,Zhou,X.K.,Hyjek,E.,Alonso,M.A.,and Chen,Y.T.(2006).Gene expression profiling separates chromophobe renal cell carcinoma from oncocytoma and identifies vesicular transport and cell junction proteins as differentially expressed genes.Clinical cancer research:an official journal of the American Association for Cancer Research 12,6937-6945.
45.Sato,Y.,Yoshizato,T.,Shiraishi,Y.,Maekawa,S.,Okuno,Y.,Kamura,T.,Shimamura,T.,Sato-Otsubo,A.,Nagae,G.,Suzuki,H.,et al.(2013).Integrated molecular analysis of clear-cell renal cell carcinoma.Nat Genet 45,860-867.
46.Schmidt,L.,Duh,F.M.,Chen,F.,Kishida,T.,Glenn,G.,Choyke,P.,Scherer,S.W.,Zhuang,Z.,Lubensky,I.,Dean,M.,et al.(1997).Germline and somatic mutations in the tyrosine kinase domain of the MET proto-oncogene in papillary renal carcinomas.Nat Genet 16,68-73.
47.Schmidt,L.,Junker,K.,Nakaigawa,N.,Kinjerski,T.,Weirich,G.,Miller,M.,Lubensky,I.,Neumann,H.P.,Brauch,H.,Decker,J.,et al.(1999).Novel mutations of the MET proto-oncogene in papillary renal carcinomas.Oncogene 18,2343-2350.
48.Scott,J.W.,Ross,F.A.,Liu,J.K.,and Hardie,D.G.(2007).Regulation of AMP-activated protein kinase by a pseudosubstrate sequence on the gamma subunit.The EMBO journal 26,806-815.
49.Siegel,R.,Naishadham,D.,and Jemal,A.(2013).Cancer statistics,2013.CA:a cancer journal for clinicians 63,11-30.
50.Srigley,J.R.,Delahunt,B.,Eble,J.N.,Egevad,L.,Epstein,J.I.,Grignon,D.,Hes,O.,Moch,H.,Montironi,R.,Tickoo,S.K.,et al.(2013).The International Society of Urological Pathology(ISUP)Vancouver Classification of Renal Neoplasia.The American journal of surgical pathology 37,1469-1489.
51.Steinberg,G.R.,and Kemp,B.E.(2009).AMPK in Health and Disease.Physiol Rev 89,1025-1078.
52.Steingrimsson,E.,Copeland,N.G.,and Jenkins,N.A.(2004).Melanocytes and the microphthalmia transcription factor network.Annu Rev Genet 38,365-411.
53.TCGA(2013).Comprehensive molecular characterization of clear cell renal cell carcinoma.Nature 499,43-49.
54.Toker,L.,Bersudsky,Y.,Plaschkes,I.,Chalifa-Caspi,V.,Berry,G.T.,Buccafusca,R.,Moechars,D.,Belmaker,R.H.,and Agam,G.(2014).Inositol-related gene knockouts mimic lithium's effect on mitochondrial function.Neuropsychopharmacology:official publication of the American College of Neuropsychopharmacology 39,319-328.
55.Venkateswarlu,K.,and Cullen,P.J.(1999).Molecular cloning and functional characterization of a human homologue of centaurin-alpha.Biochem Biophys Res Commun 262,237-244.
56.Wang,W.,Marimuthu,A.,Tsai,J.,Kumar,A.,Krupka,H.I.,Zhang,C.,Powell,B.,Suzuki,Y.,Nguyen,H.,Tabrizizad,M.,et al.(2006).Structural characterization of autoinhibited c-Met kinase produced by coexpression in bacteria with phosphatase.Proceedings of the National Academy of Sciences of the United States of America 103,3563-3568.
57.Weimer,J.M.,Chattopadhyay,S.,Custer,A.W.,and Pearce,D.A.(2005).Elevation of Hook1 in a disease model of Batten disease does not affect a novel interaction between Ankyrin G and Hook1.Biochem Biophys Res Commun 330,1176-1181.
58.Yokoyama,S.,Woods,S.L.,Boyle,G.M.,Aoude,L.G.,MacGregor,S.,Zismann,V.,Gartside,M.,Cust,A.E.,Haq,R.,Harland,M.,et al.(2011).A novel recurrent mutation in MITF predisposes to familial and sporadic melanoma.Nature 480,99-103.
59.Young,A.N.(2010).Editorial comment from Dr Young to Molecular pathology of renal oncocytoma:A review.International journal of urology:official journal of the Japanese Urological Association 17,612-613.
60.Yusenko,M.V.(2010a).Molecular pathology of chromophobe renal cell carcinoma:a review.International journal of urology:official journal of the Japanese Urological Association 17,592-600.
61.Yusenko,M.V.(2010b).Molecular pathology of renal oncocytoma:a review.International journal of urology:official journal of the Japanese Urological Association 17,602-612.
62.Yusenko,M.V.,Kuiper,R.P.,Boethe,T.,Ljungberg,B.,van Kessel,A.G.,and Kovacs,G.(2009).High-resolution DNA copy number and gene expression analyses distinguish chromophobe renal cell carcinomas and renal oncocytomas.BMC Cancer 9,152.
63.Zimmermann,P.(2006).The prevalence and significance of PDZ domain-phosphoinositide interactions.Biochimica et biophysica acta 1761,947-956.
虽然为了理解清楚的目的,上述发明已经通过例示和实施例较为详细地描述,但是描述和实施例不应解释为限制本发明的范围。通过援引明确完整收录本文中引用的所有专利和科学文献的公开内容。
序列表
<110> 基因泰克公司(GENENTECH, INC.)等
<120> MIT生物标志物和使用它们的方法
<130> P5829R1-WO
<140>
<141>
<150> 62/109,775
<151> 2015-01-30
<150> 62/059,362
<151> 2014-10-03
<150> 62/002,612
<151> 2014-05-23
<160> 57
<170> PatentIn version 3.5
<210> 1
<211> 419
<212> PRT
<213> 人(Homo sapiens)
<400> 1
Met Leu Glu Met Leu Glu Tyr Asn His Tyr Gln Val Gln Thr His Leu
1 5 10 15
Glu Asn Pro Thr Lys Tyr His Ile Gln Gln Ala Gln Arg Gln Gln Val
20 25 30
Lys Gln Tyr Leu Ser Thr Thr Leu Ala Asn Lys His Ala Asn Gln Val
35 40 45
Leu Ser Leu Pro Cys Pro Asn Gln Pro Gly Asp His Val Met Pro Pro
50 55 60
Val Pro Gly Ser Ser Ala Pro Asn Ser Pro Met Ala Met Leu Thr Leu
65 70 75 80
Asn Ser Asn Cys Glu Lys Glu Gly Phe Tyr Lys Phe Glu Glu Gln Asn
85 90 95
Arg Ala Glu Ser Glu Cys Pro Gly Met Asn Thr His Ser Arg Ala Ser
100 105 110
Cys Met Gln Met Asp Asp Val Ile Asp Asp Ile Ile Ser Leu Glu Ser
115 120 125
Ser Tyr Asn Glu Glu Ile Leu Gly Leu Met Asp Pro Ala Leu Gln Met
130 135 140
Ala Asn Thr Leu Pro Val Ser Gly Asn Leu Ile Asp Leu Tyr Gly Asn
145 150 155 160
Gln Gly Leu Pro Pro Pro Gly Leu Thr Ile Ser Asn Ser Cys Pro Ala
165 170 175
Asn Leu Pro Asn Ile Lys Arg Glu Leu Thr Ala Cys Ile Phe Pro Thr
180 185 190
Glu Ser Glu Ala Arg Ala Leu Ala Lys Glu Arg Gln Lys Lys Asp Asn
195 200 205
His Asn Leu Ile Glu Arg Arg Arg Arg Phe Asn Ile Asn Asp Arg Ile
210 215 220
Lys Glu Leu Gly Thr Leu Ile Pro Lys Ser Asn Asp Pro Asp Met Arg
225 230 235 240
Trp Asn Lys Gly Thr Ile Leu Lys Ala Ser Val Asp Tyr Ile Arg Lys
245 250 255
Leu Gln Arg Glu Gln Gln Arg Ala Lys Glu Leu Glu Asn Arg Gln Lys
260 265 270
Lys Leu Glu His Ala Asn Arg His Leu Leu Leu Arg Ile Gln Glu Leu
275 280 285
Glu Met Gln Ala Arg Ala His Gly Leu Ser Leu Ile Pro Ser Thr Gly
290 295 300
Leu Cys Ser Pro Asp Leu Val Asn Arg Ile Ile Lys Gln Glu Pro Val
305 310 315 320
Leu Glu Asn Cys Ser Gln Asp Leu Leu Gln His His Ala Asp Leu Thr
325 330 335
Cys Thr Thr Thr Leu Asp Leu Thr Asp Gly Thr Ile Thr Phe Asn Asn
340 345 350
Asn Leu Gly Thr Gly Thr Glu Ala Asn Gln Ala Tyr Ser Val Pro Thr
355 360 365
Lys Met Gly Ser Lys Leu Glu Asp Ile Leu Met Asp Asp Thr Leu Ser
370 375 380
Pro Val Gly Val Thr Asp Pro Leu Leu Ser Ser Val Ser Pro Gly Ala
385 390 395 400
Ser Lys Thr Ser Ser Arg Arg Ser Ser Met Ser Met Glu Glu Thr Glu
405 410 415
His Thr Cys
<210> 2
<211> 4472
<212> DNA
<213> 人(Homo sapiens)
<400> 2
ctcgggatac cttgtttata gtaccttctc tttgccagtc catcttcaaa ttggaattat 60
agaaagtaga gggagggata gtctaccgtc tctcactgga ttggtgccac ctaaaacatt 120
gttatgctgg aaatgctaga atataatcac tatcaggtgc agacccacct cgaaaacccc 180
accaagtacc acatacagca agcccaacgg cagcaggtaa agcagtacct ttctaccact 240
ttagcaaata aacatgccaa ccaagtcctg agcttgccat gtccaaacca gcctggcgat 300
catgtcatgc caccggtgcc ggggagcagc gcacccaaca gccccatggc tatgcttacg 360
cttaactcca actgtgaaaa agagggattt tataagtttg aagagcaaaa cagggcagag 420
agcgagtgcc caggcatgaa cacacattca cgagcgtcct gtatgcagat ggatgatgta 480
atcgatgaca tcattagcct agaatcaagt tataatgagg aaatcttggg cttgatggat 540
cctgctttgc aaatggcaaa tacgttgcct gtctcgggaa acttgattga tctttatgga 600
aaccaaggtc tgcccccacc aggcctcacc atcagcaact cctgtccagc caaccttccc 660
aacataaaaa gggagctcac agcgtgtatt tttcccacag agtctgaagc aagagcactg 720
gccaaagaga ggcagaaaaa ggacaatcac aacctgattg aacgaagaag aagatttaac 780
ataaatgacc gcattaaaga actaggtact ttgattccca agtcaaatga tccagacatg 840
cgctggaaca agggaaccat cttaaaagca tccgtggact atatccgaaa gttgcaacga 900
gaacagcaac gcgcaaaaga acttgaaaac cgacagaaga aactggagca cgccaaccgg 960
catttgttgc tcagaataca ggaacttgaa atgcaggctc gagctcatgg actttccctt 1020
attccatcca cgggtctctg ctctccagat ttggtgaatc ggatcatcaa gcaagaaccc 1080
gttcttgaga actgcagcca agacctcctt cagcatcatg cagacctaac ctgtacaaca 1140
actctcgatc tcacggatgg caccatcacc ttcaacaaca acctcggaac tgggactgag 1200
gccaaccaag cctatagtgt ccccacaaaa atgggatcca aactggaaga catcctgatg 1260
gacgacaccc tttctcccgt cggtgtcact gatccactcc tttcctcagt gtcccccgga 1320
gcttccaaaa caagcagccg gaggagcagt atgagcatgg aagagacgga gcacacttgt 1380
tagcgaatcc tccctgcact gcattcgcac aaactgcttc ctttcttgat tcgtagattt 1440
aataacttac ctgaaggggt tttcttgata attttccttt aatatgaaat tttttttcat 1500
gctttatcaa tagcccagga tatattttat ttttagaatt ttgtgaaaca gacttgtata 1560
ttctatttta caactacaaa tgcctccaaa gtattgtaca aataagtgtg cagtatctgt 1620
gaactgaatt caccacagac tttagctttc tgagcaagag gattttgcgt cagagaaatg 1680
tctgtccatt tttattcagg ggaaacttga tttgagattt ttatgcctgt gacttccttg 1740
gaaatcaaat gtaaagttta attgaaagaa tgtaaagcaa ccaaaaagaa aaaaaaaaag 1800
aaagaaagag gaaaagaaat ccatactaac ccttttccat tttataaatg tattgattca 1860
ttggtactgc cttaaagata cagtacccct ctagctttgt ttagtcttta tactgcaaac 1920
tatttaaaga aatatgtatt ctgtaaaaga aaaaaaaaat gcggcctttt catgaggatc 1980
gtctggttag aaaacataac tgataccaac cgaaactgaa gggagttaga ccaaggctct 2040
gaaatataaa gtctaatctt gctctctttt attctgtgct gttacagttt tcttcatcaa 2100
tgagtgtgat ccagtttttc ataagatatt ttattttgaa atggaaatta atgtcctctc 2160
aaagtaaaat attgaggagc actgaaagta tgttttactt tttttttatt ttatttttgc 2220
ttttgataag aaaaccgaac tgggcatatt tctaattggc tttactattt ttatttttaa 2280
attatgtttt actgttcatt tgatttgtac agattcttta ttatcattgt tcttttcaat 2340
atatttgtat taatttgtaa gaatatgcat cttaaaatgg caagttttcc atatttttac 2400
aactcactgg tggttttccg cattctttgt acacccatga aagaaaactt ttatgcaagg 2460
tcttgcattt aaaagacagc tttgcgaata ttttgtaaat tacagtctca ctcagaactg 2520
tttttggaca catttaaggt gtagtattaa taggttaaaa ccaggctttc tagaaagaat 2580
aaacttacat atttattttt aggacatgaa aatagcaata ttcttggaga ttgataacca 2640
tagcattaat acgcccatta tggtcattta aattggggtt tatttcagca aacttgttga 2700
atttattttt aagaaagaaa tactgtattg ggaagttact gttacttgat aacaatgttt 2760
taacaagaag caatgttata aagttagttt cagtgcatta tctacttgtg tagtcctatg 2820
caataacagt agtgttacat gtatcaagcc tagatgtttt atacagatgc catatagtgt 2880
tatgagccag gctgttgaat ggaatttctc agtagcagcc tacaactgaa tagcaagtgg 2940
cataaagcat atccattcag aatgaagtgc cttaaatata gcagtagtct tttttggact 3000
agcactgact gaactgtaat gtaggggaaa gtttcatgat ggtatctata gtcaagacga 3060
acatgtagca tggtgcctat gtagacaata taagagcttc caattttcct tcagatattt 3120
ttaatattaa atatatttta gtgacagagt gccaacttct ttcatcagga aaccttattc 3180
aggagggttt ttaaaaagtg tttaaatgtc aaatgtgaat tggtgatggg tgatggaggg 3240
ttcagagagg agtgatcgtc agatgtgtga atggacggtt taggtgaaaa taatcaactg 3300
catagttccc atgcacgctg ggcaatgaga atccttggaa acattggtga tgctatcagt 3360
tttatagctt tatttcttaa gggggtaggg aaaattagtt cccattcttt caaccccctt 3420
aactgtatag ctcttttcct agaatagtga cgcaaatctg catgaacagc taattgtacc 3480
atagtgttca ttgatacaat catagcattg tctatttttc tcttcatatt tatatggggg 3540
ggagggcgct ggatgcaaaa gttgaagatc gtgatgctat gatgttagtt ttccttagct 3600
gattttgagg gtttttaaaa ataaagcaag gttgactaac ctacggccac gggaacagga 3660
ccatggttaa gcaaccatat agaaagcttt gttgaaagaa agtatggcat cttgtaccac 3720
tgccctgact gtcacaactc ctaaccttgc cattgcctgc ctccccctcc ccttctcctt 3780
aagagacaat ttctgcaggt ggcaggtgag caagcccagg agaatgctgc aatcttgggg 3840
gtggttttat ttatttcttt tttgccaaat agagtgtgga ttcatttcag gggctagcta 3900
agccaagagg cagtggtttg ggcttgttgt ttgtaacaag aaaatgatcc acaccactcc 3960
cccgattccc gggtgcagaa ttgtaactcg gggttgggcc tctatatgga gtgaccaaaa 4020
tgccaaaatt gtccatctgc ctctgagtag ggcaatggaa ataccaaacc ttctgacttt 4080
gccaaaaagc atacaagcaa cctggtcata cataggatga caaaattctt tctggttgtt 4140
tttaaacaat aaagcaataa gaacaaatac aatacatagg aagttaaaag cacaaaggaa 4200
tgaacttatt aatatttttg aaaaatgcac tgggaaaaag ttgatgtcaa taacagtata 4260
aaacagccct atttcttgat aaaaaatgac aaatgactgt ctcttgcgga tgcttggtac 4320
tgtaatgtta ataatagtca cctgctgttg gatgcagcaa taatttctgt atggtccata 4380
gcactgtata ttatggatcg atattaatgt atccaatgaa ataatcgact tgttcttgat 4440
agcctcatta aagcatttgg tttttcacat ag 4472
<210> 3
<211> 476
<212> PRT
<213> 人(Homo sapiens)
<400> 3
Met Ala Ser Arg Ile Gly Leu Arg Met Gln Leu Met Arg Glu Gln Ala
1 5 10 15
Gln Gln Glu Glu Gln Arg Glu Arg Met Gln Gln Gln Ala Val Met His
20 25 30
Tyr Met Gln Gln Gln Gln Gln Gln Gln Gln Gln Gln Leu Gly Gly Pro
35 40 45
Pro Thr Pro Ala Ile Asn Thr Pro Val His Phe Gln Ser Pro Pro Pro
50 55 60
Val Pro Gly Glu Val Leu Lys Val Gln Ser Tyr Leu Glu Asn Pro Thr
65 70 75 80
Ser Tyr His Leu Gln Gln Ser Gln His Gln Lys Val Arg Glu Tyr Leu
85 90 95
Ser Glu Thr Tyr Gly Asn Lys Phe Ala Ala His Ile Ser Pro Ala Gln
100 105 110
Gly Ser Pro Lys Pro Pro Pro Ala Ala Ser Pro Gly Val Arg Ala Gly
115 120 125
His Val Leu Ser Ser Ser Ala Gly Asn Ser Ala Pro Asn Ser Pro Met
130 135 140
Ala Met Leu His Ile Gly Ser Asn Pro Glu Arg Glu Leu Asp Asp Val
145 150 155 160
Ile Asp Asn Ile Met Arg Leu Asp Asp Val Leu Gly Tyr Ile Asn Pro
165 170 175
Glu Met Gln Met Pro Asn Thr Leu Pro Leu Ser Ser Ser His Leu Asn
180 185 190
Val Tyr Ser Ser Asp Pro Gln Val Thr Ala Ser Leu Val Gly Val Thr
195 200 205
Ser Ser Ser Cys Pro Ala Asp Leu Thr Gln Lys Arg Glu Leu Thr Asp
210 215 220
Ala Glu Ser Arg Ala Leu Ala Lys Glu Arg Gln Lys Lys Asp Asn His
225 230 235 240
Asn Leu Ile Glu Arg Arg Arg Arg Phe Asn Ile Asn Asp Arg Ile Lys
245 250 255
Glu Leu Gly Met Leu Ile Pro Lys Ala Asn Asp Leu Asp Val Arg Trp
260 265 270
Asn Lys Gly Thr Ile Leu Lys Ala Ser Val Asp Tyr Ile Arg Arg Met
275 280 285
Gln Lys Asp Leu Gln Lys Ser Arg Glu Leu Glu Asn His Ser Arg Arg
290 295 300
Leu Glu Met Thr Asn Lys Gln Leu Trp Leu Arg Ile Gln Glu Leu Glu
305 310 315 320
Met Gln Ala Arg Val His Gly Leu Pro Thr Thr Ser Pro Ser Gly Met
325 330 335
Asn Met Ala Glu Leu Ala Gln Gln Val Val Lys Gln Glu Leu Pro Ser
340 345 350
Glu Glu Gly Pro Gly Glu Ala Leu Met Leu Gly Ala Glu Val Pro Asp
355 360 365
Pro Glu Pro Leu Pro Ala Leu Pro Pro Gln Ala Pro Leu Pro Leu Pro
370 375 380
Thr Gln Pro Pro Ser Pro Phe His His Leu Asp Phe Ser His Ser Leu
385 390 395 400
Ser Phe Gly Gly Arg Glu Asp Glu Gly Pro Pro Gly Tyr Pro Glu Pro
405 410 415
Leu Ala Pro Gly His Gly Ser Pro Phe Pro Ser Leu Ser Lys Lys Asp
420 425 430
Leu Asp Leu Met Leu Leu Asp Asp Ser Leu Leu Pro Leu Ala Ser Asp
435 440 445
Pro Leu Leu Ser Thr Met Ser Pro Glu Ala Ser Lys Ala Ser Ser Arg
450 455 460
Arg Ser Ser Phe Ser Met Glu Glu Gly Asp Val Leu
465 470 475
<210> 4
<211> 4728
<212> DNA
<213> 人(Homo sapiens)
<400> 4
ggccgggcgg gcatgggcct tcccggcccg gagctgggag tcgaaggggc gggaggcgtg 60
atggtgaact cgcaagaagt ttgagggacg cgcgggcccc gcgcccactc cccctccacc 120
ggacacggct ggggccggcg atgcctgaga gggggtcgga ggacgcagtg aacatatatg 180
catgtacagt gtggatcctc atctgagagg agggagatga aaacacaccc acctcacagg 240
ctgttgtgag gactaagggt gcggcagtgc ctggtacatg ggagccagcg ccggcagcca 300
ccatggcgtc acgcataggg ttgcgcatgc agctcatgcg ggagcaggcg cagcaggagg 360
agcagcggga gcgcatgcag caacaggctg tcatgcatta catgcagcag cagcagcagc 420
agcaacagca gcagctcgga gggccgccca ccccggccat caataccccc gtccacttcc 480
agtcgccacc acctgtgcct ggggaggtgt tgaaggtgca gtcctacctg gagaatccca 540
catcctacca tctgcagcag tcgcagcatc agaaggtgcg ggagtacctg tccgagacct 600
atgggaacaa gtttgctgcc cacatcagcc cagcccaggg ctctccgaaa cccccaccag 660
ccgcctcccc aggggtgcga gctggacacg tgctgtcctc ctccgctggc aacagtgctc 720
ccaatagccc catggccatg ctgcacattg gctccaaccc tgagagggag ttggatgatg 780
tcattgacaa cattatgcgt ctggacgatg tccttggcta catcaatcct gaaatgcaga 840
tgcccaacac gctacccctg tccagcagcc acctgaatgt gtacagcagc gacccccagg 900
tcacagcctc cctggtgggc gtcaccagca gctcctgccc tgcggacctg acccagaagc 960
gagagctcac agatgctgag agcagggccc tggccaagga gcggcagaag aaagacaatc 1020
acaacttaat tgaaaggaga cgaaggttca acatcaatga ccgcatcaag gagttgggaa 1080
tgctgatccc caaggccaat gacctggacg tgcgctggaa caagggcacc atcctcaagg 1140
cctctgtgga ttacatccgg aggatgcaga aggacctgca aaagtccagg gagctggaga 1200
accactctcg ccgcctggag atgaccaaca agcagctctg gctccgtatc caggagctgg 1260
agatgcaggc tcgagtgcac ggcctcccta ccacctcccc gtccggcatg aacatggctg 1320
agctggccca gcaggtggtg aagcaggagc tgcctagcga agagggccca ggggaggccc 1380
tgatgctggg ggctgaggtc cctgaccctg agccactgcc agctctgccc ccgcaagccc 1440
cgctgcccct gcccacccag ccaccatccc cattccatca cctggacttc agccacagcc 1500
tgagctttgg gggcagggag gacgagggtc ccccgggcta ccccgaaccc ctggcgccgg 1560
ggcatggctc cccattcccc agcctgtcca agaaggatct ggacctcatg ctcctggacg 1620
actcactgct accgctggcc tctgatccac ttctgtccac catgtccccc gaggcctcca 1680
aggccagcag ccgccggagc agcttcagca tggaggaggg cgatgtgctg tgaccctggc 1740
tgcccctgtg ccagggaaca ggggccggcc tgggggctgg gagggccagg ggcacctccc 1800
tcccaccctt caggctgcac tgtgtgtgaa gtagccacct gccctgcctc cctcctcccc 1860
gttggcccct gtttggactt agtgcctgtc tggcagcctg tggggtcagg agaagcaccc 1920
ccagggcagc cctcttgact ggcgcagtgg gaagaggcct tcagcccctc tcccggagat 1980
ggaatcgcgg ggcagggagg ggcagggtgt tctagaggtg agaagagggc ctggtggaga 2040
ttccctgtct tctgagcccg agcccctcat taccagtgaa ggacatgctt gaggggttcg 2100
ggaagctcct catctgaggc aactggtcct gggggtgctc aggcctgcct ttttgggact 2160
cagatggcag gaggtccacc ccgcagcctg gtcctcggct ctcccacagg tgggcacccc 2220
ccactttggt gctaatagct ctccaccagg tggtgtgagc gcgggggctg ccagaagcgg 2280
gaggggtcac tgccggaaga gcagctgccc tccgacccct cactttgtgc ctttagtaaa 2340
cactgtgctt tgtaaaaaaa aaaaggccgg gcgggcatgg gccttcccgg cccggagctg 2400
ggagtcgaag gggcgggagg cgtgatggtg aactcgcaag aagtttgagg gacgcgcggg 2460
ccccgcgccc actccccctc caccggacac ggctggggcc ggcgatgcct gagagggggt 2520
cggaggacgc agtgaacata tatgcatgta cagtgtggat cctcatctga gaggagggag 2580
atgaaaacac acccacctca caggctgttg tgaggactaa gggtgcggca gtgcctggta 2640
catgggagcc agcgccggca gccaccatgg cgtcacgcat agggttgcgc atgcagctca 2700
tgcgggagca ggcgcagcag gaggagcagc gggagcgcat gcagcaacag gctgtcatgc 2760
attacatgca gcagcagcag cagcagcaac agcagcagct cggagggccg cccaccccgg 2820
ccatcaatac ccccgtccac ttccagtcgc caccacctgt gcctggggag gtgttgaagg 2880
tgcagtccta cctggagaat cccacatcct accatctgca gcagtcgcag catcagaagg 2940
tgcgggagta cctgtccgag acctatggga acaagtttgc tgcccacatc agcccagccc 3000
agggctctcc gaaaccccca ccagccgcct ccccaggggt gcgagctgga cacgtgctgt 3060
cctcctccgc tggcaacagt gctcccaata gccccatggc catgctgcac attggctcca 3120
accctgagag ggagttggat gatgtcattg acaacattat gcgtctggac gatgtccttg 3180
gctacatcaa tcctgaaatg cagatgccca acacgctacc cctgtccagc agccacctga 3240
atgtgtacag cagcgacccc caggtcacag cctccctggt gggcgtcacc agcagctcct 3300
gccctgcgga cctgacccag aagcgagagc tcacagatgc tgagagcagg gccctggcca 3360
aggagcggca gaagaaagac aatcacaact taattgaaag gagacgaagg ttcaacatca 3420
atgaccgcat caaggagttg ggaatgctga tccccaaggc caatgacctg gacgtgcgct 3480
ggaacaaggg caccatcctc aaggcctctg tggattacat ccggaggatg cagaaggacc 3540
tgcaaaagtc cagggagctg gagaaccact ctcgccgcct ggagatgacc aacaagcagc 3600
tctggctccg tatccaggag ctggagatgc aggctcgagt gcacggcctc cctaccacct 3660
ccccgtccgg catgaacatg gctgagctgg cccagcaggt ggtgaagcag gagctgccta 3720
gcgaagaggg cccaggggag gccctgatgc tgggggctga ggtccctgac cctgagccac 3780
tgccagctct gcccccgcaa gccccgctgc ccctgcccac ccagccacca tccccattcc 3840
atcacctgga cttcagccac agcctgagct ttgggggcag ggaggacgag ggtcccccgg 3900
gctaccccga acccctggcg ccggggcatg gctccccatt ccccagcctg tccaagaagg 3960
atctggacct catgctcctg gacgactcac tgctaccgct ggcctctgat ccacttctgt 4020
ccaccatgtc ccccgaggcc tccaaggcca gcagccgccg gagcagcttc agcatggagg 4080
agggcgatgt gctgtgaccc tggctgcccc tgtgccaggg aacaggggcc ggcctggggg 4140
ctgggagggc caggggcacc tccctcccac ccttcaggct gcactgtgtg tgaagtagcc 4200
acctgccctg cctccctcct ccccgttggc ccctgtttgg acttagtgcc tgtctggcag 4260
cctgtggggt caggagaagc acccccaggg cagccctctt gactggcgca gtgggaagag 4320
gccttcagcc cctctcccgg agatggaatc gcggggcagg gaggggcagg gtgttctaga 4380
ggtgagaaga gggcctggtg gagattccct gtcttctgag cccgagcccc tcattaccag 4440
tgaaggacat gcttgagggg ttcgggaagc tcctcatctg aggcaactgg tcctgggggt 4500
gctcaggcct gcctttttgg gactcagatg gcaggaggtc caccccgcag cctggtcctc 4560
ggctctccca caggtgggca ccccccactt tggtgctaat agctctccac caggtggtgt 4620
gagcgcgggg gctgccagaa gcgggagggg tcactgccgg aagagcagct gccctccgac 4680
ccctcacttt gtgcctttag taaacactgt gctttgtaaa aaaaaaaa 4728
<210> 5
<211> 575
<212> PRT
<213> 人(Homo sapiens)
<400> 5
Met Ser His Ala Ala Glu Pro Ala Arg Asp Gly Val Glu Ala Ser Ala
1 5 10 15
Glu Gly Pro Arg Ala Val Phe Val Leu Leu Glu Glu Arg Arg Pro Ala
20 25 30
Asp Ser Ala Gln Leu Leu Ser Leu Asn Ser Leu Leu Pro Glu Ser Gly
35 40 45
Ile Val Ala Asp Ile Glu Leu Glu Asn Val Leu Asp Pro Asp Ser Phe
50 55 60
Tyr Glu Leu Lys Ser Gln Pro Leu Pro Leu Arg Ser Ser Leu Pro Ile
65 70 75 80
Ser Leu Gln Ala Thr Pro Ala Thr Pro Ala Thr Leu Ser Ala Ser Ser
85 90 95
Ser Ala Gly Gly Ser Arg Thr Pro Ala Met Ser Ser Ser Ser Ser Ser
100 105 110
Arg Val Leu Leu Arg Gln Gln Leu Met Arg Ala Gln Ala Gln Glu Gln
115 120 125
Glu Arg Arg Glu Arg Arg Glu Gln Ala Ala Ala Ala Pro Phe Pro Ser
130 135 140
Pro Ala Pro Ala Ser Pro Ala Ile Ser Val Val Gly Val Ser Ala Gly
145 150 155 160
Gly His Thr Leu Ser Arg Pro Pro Pro Ala Gln Val Pro Arg Glu Val
165 170 175
Leu Lys Val Gln Thr His Leu Glu Asn Pro Thr Arg Tyr His Leu Gln
180 185 190
Gln Ala Arg Arg Gln Gln Val Lys Gln Tyr Leu Ser Thr Thr Leu Gly
195 200 205
Pro Lys Leu Ala Ser Gln Ala Leu Thr Pro Pro Pro Gly Pro Ala Ser
210 215 220
Ala Gln Pro Leu Pro Ala Pro Glu Ala Ala His Thr Thr Gly Pro Thr
225 230 235 240
Gly Ser Ala Pro Asn Ser Pro Met Ala Leu Leu Thr Ile Gly Ser Ser
245 250 255
Ser Glu Lys Glu Ile Asp Asp Val Ile Asp Glu Ile Ile Ser Leu Glu
260 265 270
Ser Ser Tyr Asn Asp Glu Met Leu Ser Tyr Leu Pro Gly Gly Thr Thr
275 280 285
Gly Leu Gln Leu Pro Ser Thr Leu Pro Val Ser Gly Asn Leu Leu Asp
290 295 300
Val Tyr Ser Ser Gln Gly Val Ala Thr Pro Ala Ile Thr Val Ser Asn
305 310 315 320
Ser Cys Pro Ala Glu Leu Pro Asn Ile Lys Arg Glu Ile Ser Glu Thr
325 330 335
Glu Ala Lys Ala Leu Leu Lys Glu Arg Gln Lys Lys Asp Asn His Asn
340 345 350
Leu Ile Glu Arg Arg Arg Arg Phe Asn Ile Asn Asp Arg Ile Lys Glu
355 360 365
Leu Gly Thr Leu Ile Pro Lys Ser Ser Asp Pro Glu Met Arg Trp Asn
370 375 380
Lys Gly Thr Ile Leu Lys Ala Ser Val Asp Tyr Ile Arg Lys Leu Gln
385 390 395 400
Lys Glu Gln Gln Arg Ser Lys Asp Leu Glu Ser Arg Gln Arg Ser Leu
405 410 415
Glu Gln Ala Asn Arg Ser Leu Gln Leu Arg Ile Gln Glu Leu Glu Leu
420 425 430
Gln Ala Gln Ile His Gly Leu Pro Val Pro Pro Thr Pro Gly Leu Leu
435 440 445
Ser Leu Ala Thr Thr Ser Ala Ser Asp Ser Leu Lys Pro Glu Gln Leu
450 455 460
Asp Ile Glu Glu Glu Gly Arg Pro Gly Ala Ala Thr Phe His Val Gly
465 470 475 480
Gly Gly Pro Ala Gln Asn Ala Pro His Gln Gln Pro Pro Ala Pro Pro
485 490 495
Ser Asp Ala Leu Leu Asp Leu His Phe Pro Ser Asp His Leu Gly Asp
500 505 510
Leu Gly Asp Pro Phe His Leu Gly Leu Glu Asp Ile Leu Met Glu Glu
515 520 525
Glu Glu Gly Val Val Gly Gly Leu Ser Gly Gly Ala Leu Ser Pro Leu
530 535 540
Arg Ala Ala Ser Asp Pro Leu Leu Ser Ser Val Ser Pro Ala Val Ser
545 550 555 560
Lys Ala Ser Ser Arg Arg Ser Ser Phe Ser Met Glu Glu Glu Ser
565 570 575
<210> 6
<211> 3467
<212> DNA
<213> 人(Homo sapiens)
<400> 6
ttcctgtgga gtttccccat ccctgggagg agggaggagg gggaagaaga cgagggggag 60
gagggcggtc gtccggggtt aggttgaggg ggggcgtcgg tccgttctgg gcgggggatg 120
actcacagcc catcccatct ccccgacgcc gcccgcccgc gcagtgctag ctccatggct 180
tagcggagga ggcggcggtg gcgagctggg gggagggggg actcttattt tgttaggggg 240
accgggccga ggcccgaccg gcctggcagg gctcgcccgg ggccgggcgt catgtctcat 300
gcggccgaac cagctcggga tggcgtagag gccagcgcgg agggccctcg agccgtgttc 360
gtgctgttgg aggagcgcag gccggccgac tcggctcagc tgctcagcct gaactctttg 420
cttccggaat ccgggattgt tgctgacata gaattagaaa acgtccttga tcctgacagc 480
ttctacgagc tcaaaagcca acccttaccc cttcgctcaa gcctcccaat atcactgcag 540
gccacaccag ccaccccagc tacactctct gcatcgtctt ctgcaggggg ctccaggacc 600
cctgccatgt cgtcatcttc ttcatcgagg gtcttgctgc ggcagcagct aatgcgggcc 660
caggcgcagg agcaggagag gcgtgagcgt cgggaacagg ccgccgcggc tcccttcccc 720
agtcctgcac ctgcctctcc tgccatctct gtggttggcg tctctgctgg gggccacaca 780
ttgagccgtc caccccctgc tcaggtgccc agggaggtgc tcaaggtgca gacccatctg 840
gagaacccaa cgcgctacca cctgcagcag gcgcgccggc agcaggtgaa acagtacctg 900
tccaccacac tcgggcccaa gctggcttcc caggccctca ccccaccgcc ggggcccgca 960
agtgcccagc cactgcctgc ccctgaggct gcccacacta ccggccccac aggcagtgcg 1020
cccaacagcc ccatggcgct gctcaccatc gggtccagct cagagaagga gattgatgat 1080
gtcattgatg agatcatcag cctggagtcc agttacaatg atgaaatgct cagctatctg 1140
cccggaggca ccacaggact gcagctcccc agcacgctgc ctgtgtcagg gaatctgctt 1200
gatgtgtaca gtagtcaagg cgtggccaca ccagccatca ctgtcagcaa ctcctgccca 1260
gctgagctgc ccaacatcaa acgggagatc tctgagaccg aggcaaaggc ccttttgaag 1320
gaacggcaga agaaagacaa tcacaaccta attgagcgtc gcaggcgatt caacattaac 1380
gacaggatca aggaactggg cactctcatc cctaagtcca gtgacccgga gatgcgctgg 1440
aacaagggca ccatcctgaa ggcctctgtg gattatatcc gcaagctgca gaaggagcag 1500
cagcgctcca aagacctgga gagccggcag cgatccctgg agcaggccaa ccgcagcctg 1560
cagctccgaa ttcaggaact agaactgcag gcccagatcc atggcctgcc agtacctccc 1620
actccagggc tgctttcctt ggccacgact tcggcttctg acagcctcaa gccagagcag 1680
ctggacattg aggaggaggg caggccaggc gcagcaacgt tccatgtagg ggggggacct 1740
gcccagaatg ctccccatca gcagccccct gcaccgccct cagatgccct tctggacctg 1800
cactttccca gcgaccacct gggggacctg ggagacccct tccacctggg gctggaggac 1860
attctgatgg aggaggagga gggggtggtg ggaggactgt cggggggtgc cctgtcccca 1920
ctgcgggctg cctccgatcc cctgctctct tcagtgtccc ctgctgtctc caaggccagc 1980
agccgccgca gcagcttcag catggaagag gagtcctgat caggcctcac ccctcccctg 2040
ggactttccc acccaggaaa ggaggaccag tcaggatgag gccccgcctt ttcccccacc 2100
ctcccatgag actgccctgc ccaggtatcc tgggggaaga ggagatgtga tcaggcccca 2160
cccctgtaat caggcaagga ggaggagtca gatgaggccc tgcaccttcc ccaaaggaac 2220
cgcccagtgc aggtatttca gaaggagaag gctggagaag gacatgagat cagggcctgc 2280
cccctgggga tcacagcctc acccctgccc ctgtgggact catccttgcc caggtgaggg 2340
aaggagacag gatgaggtct cgaccctgtc ccctagggac tgtcctagcc aggtctcctg 2400
ggaaagggag atgtcaggat gttgctccat cctttgtctt ggaaccacca gtctagtccg 2460
tcctggcaca gaagaggagt caagtaatgg aggtcccagc cctgggggtt taagctctgc 2520
cccttcccca tgaaccctgc cctgctctgc ccaggcaagg aacagaagtg aggatgagac 2580
ccagcccctt cccctgggaa ctctcctggc cttctaggaa tggaggagcc aggccccacc 2640
ccttccctat aggaacagcc cagcacaggt atttcaggtg tgaaagaatc agtaggacca 2700
ggccaccgct agtgcttgtg gagatcacag ccccaccctt gtccctcagc aacatcccat 2760
ctaagcattc cacactgcag ggaggagtgg tacttaagct cccctgcctt aacctgggac 2820
caacctgacc taacctagga gggctctgag ccaaccttgc tcttggggaa ggggacagat 2880
tatgaaattt catggatgaa ttttccagac ctatatctgg agtgagaggc ccccaccctt 2940
gggcagagtc ctgccttctt ccttgagggg cagtttggga aggtgatggg tattagtggg 3000
ggactgagtt caggttacca gaaccagtac ctcagtattc tttttcaaca tgtagggcaa 3060
gaggatgaag gaaggggcta tcctgggacc tccccagccc aggaaaaact ggaagccttc 3120
ccccagcaag gcagaagctt ggaggagggt tgtaaaagca tattgtaccc cctcatttgt 3180
ttatctgatt tttttattgc tccgcatact gagaatctag gccaccccaa cctctgttcc 3240
ccacccagtt cttcatttgg aggaatcacc ccatttcaga gttatcaaga gacactcccc 3300
cctccattcc cacccctcat acctacaccc aaggttgtca gctttggatt gctggggcca 3360
ggccccatgg agggtatact gaggggtcta taggtttgtg attaaaataa taaaagctag 3420
gcgtgtttga tgcgctttta actttggcaa aaaaaaaaaa aaaaaaa 3467
<210> 7
<211> 347
<212> PRT
<213> 人(Homo sapiens)
<400> 7
Met Thr Leu Asp His Gln Ile Ile Asn Pro Thr Leu Lys Trp Ser Gln
1 5 10 15
Pro Ala Val Pro Ser Gly Gly Pro Leu Val Gln His Ala His Thr Thr
20 25 30
Leu Asp Ser Asp Ala Gly Leu Thr Glu Asn Pro Leu Thr Lys Leu Leu
35 40 45
Ala Ile Gly Lys Glu Asp Asp Asn Ala Gln Trp His Met Glu Asp Val
50 55 60
Ile Glu Asp Ile Ile Gly Met Glu Ser Ser Phe Lys Glu Glu Gly Ala
65 70 75 80
Asp Ser Pro Leu Leu Met Gln Arg Thr Leu Ser Gly Ser Ile Leu Asp
85 90 95
Val Tyr Ser Gly Glu Gln Gly Ile Ser Pro Ile Asn Met Gly Leu Thr
100 105 110
Ser Ala Ser Cys Pro Ser Ser Leu Pro Met Lys Arg Glu Ile Thr Glu
115 120 125
Thr Asp Thr Arg Ala Leu Ala Lys Glu Arg Gln Lys Lys Asp Asn His
130 135 140
Asn Leu Ile Glu Arg Arg Arg Arg Tyr Asn Ile Asn Tyr Arg Ile Lys
145 150 155 160
Glu Leu Gly Thr Leu Ile Pro Lys Ser Asn Asp Pro Asp Met Arg Trp
165 170 175
Asn Lys Gly Thr Ile Leu Lys Ala Ser Val Glu Tyr Ile Lys Trp Leu
180 185 190
Gln Lys Glu Gln Gln Arg Ala Arg Glu Leu Glu His Arg Gln Lys Lys
195 200 205
Leu Glu Gln Ala Asn Arg Arg Leu Leu Leu Arg Ile Gln Glu Leu Glu
210 215 220
Ile Gln Ala Arg Thr His Gly Leu Pro Thr Leu Ala Ser Leu Gly Thr
225 230 235 240
Val Asp Leu Gly Ala His Val Thr Lys Gln Gln Ser His Pro Glu Gln
245 250 255
Asn Ser Val Asp Tyr Cys Gln Gln Leu Thr Val Ser Gln Gly Pro Ser
260 265 270
Pro Glu Leu Cys Asp Gln Ala Ile Ala Phe Ser Asp Pro Leu Ser Tyr
275 280 285
Phe Thr Asp Leu Ser Phe Ser Ala Ala Leu Lys Glu Glu Gln Arg Leu
290 295 300
Asp Gly Met Leu Leu Asp Asp Thr Ile Ser Pro Phe Gly Thr Asp Pro
305 310 315 320
Leu Leu Ser Ala Thr Ser Pro Ala Val Ser Lys Glu Ser Ser Arg Arg
325 330 335
Ser Ser Phe Ser Ser Asp Asp Gly Asp Glu Leu
340 345
<210> 8
<211> 6700
<212> DNA
<213> 人(Homo sapiens)
<400> 8
acttctcttt tctctttcct caactaactg gattccaacc agcccaaagt attcattact 60
cacggctaga tcgtttactt tggttgtccc ttctggcatg gtgcatatgt tatgggaaga 120
gggattataa tttggtgctg tttgtagaga tgacaacact gataaaatcc actcattgct 180
ggtcccagca cacctggaaa gttctgcaag gcctcagcta cagaaagccc agagacagaa 240
agtaaactct ttcatgaccc ttgatcatca gatcatcaat ccaactctta aatggtcaca 300
acctgcagtg ccaagtggtg ggcctcttgt gcagcatgca cacacaactc tggacagtga 360
tgctggcctc acagaaaacc cactcaccaa gttactagct attgggaaag aagatgacaa 420
tgcacaatgg catatggagg acgttattga ggatataatc ggtatggaat caagttttaa 480
agaggaagga gcagactctc ctctgctaat gcaaagaaca ttatctggaa gtattttgga 540
tgtgtatagc ggtgaacaag gaatttcacc aattaacatg gggcttacaa gtgcttcttg 600
tccaagtagt ctaccaatga aaagagaaat tacagaaact gacactagag ctttagcaaa 660
agagagacaa aaaaaggaca accacaacct cattgaaaga agaagaaggt ataatattaa 720
ttaccgaatc aaggagcttg gcactcttat tccaaagtct aatgatcctg atatgcgctg 780
gaacaaagga accattctaa aagcatcagt ggagtacatc aagtggctac aaaaagaaca 840
acagagagcc cgagaattgg aacacagaca gaagaaatta gagcaggcta acaggcgact 900
tctacttcgg attcaggaac tagaaattca ggctcgtact catggtctgc caaccctggc 960
ttcacttggc acggttgatt taggtgctca tgtcaccaaa cagcagagcc atcctgagca 1020
gaattcagta gactattgcc aacaactgac tgtgtctcag gggccaagcc ctgagctctg 1080
tgatcaagct atagcctttt ctgatccttt gtcatacttc acagatttat catttagtgc 1140
tgcattgaaa gaggaacaaa gattggatgg catgctattg gatgacacaa tctctccatt 1200
tggaacagat cctctgctat ctgccacttc ccctgcagtt tccaaagaaa gcagtaggag 1260
aagtagcttt agctcagatg atggtgatga attataagaa ataaacagac ccaattcatc 1320
aactggaaag caattctatg ctggtgctat gcaattatgc tctgtgtttc atatgttgct 1380
ttggcttatt ttttttctta aaggaatgtg ttgttcatga aaaactgata gaagcaacag 1440
aagaattcgc aggaagaaaa atcatagtgt taatgaatta ttgagggcga aaaaaaggtg 1500
ttttcttctt tgactacgga gtccaaatcc acttaaattc tgttttcctg aaaagaggta 1560
cagcataaga aatagctctt tattgatgtt ttaaaagcag caacttggtg gtgtactact 1620
ggaactaatg actgcaaagt gttaaacgac tgaaatatac aaacagtctc ttagttactc 1680
atttccatct tctcttcaac tttcacatca gtcttccgga atcaagatca acatatcagg 1740
tggtcattgc ctttctccat tgtctagtag acatgtctaa agttcaaact ttataggata 1800
aataaatgta taatagatta tctgtcactt gtggttgaaa ggcaaatcta caataaatgt 1860
gagaattttc cacaataaaa tatggataac ttataaaaac attggttact aaaattagat 1920
cctcatttta ttgtagttgg ttcaattaca ctaattctaa aagcatccat gcatatttat 1980
atctccagtc tctgttcagg aaaaggaaca tattgaatat tttcctcagg aatatggacc 2040
agaattgtat cccttcacac aaacatacac atacacatat gcacatcatt caggtagtat 2100
atgttctttt gttttcttca tgcttctgac tgcatcagaa tcacattcca aattctcttt 2160
tcttatgaag aagagatgtc agatcatcaa ttttagtaaa taaaatataa aatgtccccc 2220
tgcaaggaca gttttcaggt acttaaaact tttcatcagt attggacaga aatcaattag 2280
ttgttgattt ggtttttctc caaatggata aaatattgaa aattgaattg ccaattgaca 2340
aaataattat tacaacaaac tatttcttat tattttcagt tctgagagga acgtaaggtt 2400
ctatttctat aaacacttag agtgtcctat gatctttggt tgcactgtta gcatttatta 2460
taagcactta taactatgat gcttcattta gatttttatc tcttgcgctt gttttaggtt 2520
aggaaattaa gttaccaagc acgttgctct gtgctggacc tccaagagtg atcatccgat 2580
caatgagtat tcatggaggc atactcagaa ctgcagtgag tcctgagaaa atgtagaaga 2640
gttgaaaaag atatggtctt actcctaagg tagttaatga gaatgcatga aaaacaaaaa 2700
caaaagatag aagacatata gaataaaaaa tctaagcata aactgtaaat acagaaggtc 2760
agagaaaatg aaaatgactg acaaacttta tgggaacagc agaatttgac ttctctttga 2820
aaggcattaa ttgagtgaaa gtgagggcat ataaaggaat aataccaacc aaatgataaa 2880
agtgagaata ataggggaag tatctttata ggactttaat tagaaagatc tggatgccct 2940
agaagatgtc tgttggggag ttctgaagca taagactgac aacaggttgt gaaggccctt 3000
agaaaactat ggtgaaataa tatagtatta atataataga aacaaatctt tttagtagca 3060
gggaggatgc taaaaactaa aatatatatt actaatgagt gagatgtaat ctttcatgaa 3120
tattaagctt tgggataaat tttgaaagga tcttttagtt gcttctcaca tggaaaatat 3180
gtcactatga aatgtgagac cctaatgctg ataaaagtag aaaaatagta aatcattatg 3240
gtataataat tatctcaggc ttagcttgta tcatttgctc agaatttgga gaattaaaat 3300
aattttattg tttgacatgg aaaaataatt actaacttct gggtacagtc attaaagcct 3360
gatagataaa ttcatctgaa atatgccatg tgaaacagct attagataca tcttctcaat 3420
aattttcaag acgatattac atacttttaa ttttataaca aaattccact tcattatctg 3480
agttgataat ttgattaaat gttaaaaatt atagataagg aatatatttt ggagttcata 3540
gaaaacacac cactttatta gatagagact ggcttcagtt ctcattttat ctgctcttct 3600
catttcttca tattttgagt agacttgcac tgatgatgtc attactcagt cattattttc 3660
tgttctgtta aggtacaact actgggcctt gaaatctatc cttcacaatc ttggctgaga 3720
taaggacttg gcacaggatg atagagcctg gacacaagag atctggagag ggaactgcta 3780
tttgctgcta cattcagatt atgagatgtc agggaactgt aaaaagggta atgaattttt 3840
aaagagagta aagagttatg ctgtgtcccg gtcaactgaa atacattaaa aattaattag 3900
atggtagttc ttaacttttt aaggcagata ccttgagaat ctagtgagtt atagaccctc 3960
tccctagaaa agtatagaac tgtttatatg gacaataatt cacatgcatt cagagatctt 4020
agggatctcc tgaatgtttt cttggatccc aagtgaaaaa ttcctgaatc aggtctctta 4080
ggtgtgcggt taatgtcatg tataacaatg gggggaccct tacttatcta aatataatta 4140
tcccaatcct aataatgatg aggttgcata ggaaagtaat agtgtaccaa actttagatt 4200
attcactaaa aaagttgttc atttatgaag tagtcgttta tcaaacatgt cctccccact 4260
cagcaaacta tgccttctat ttattatata tgggtcagtt tcactataat tactgagtgt 4320
tataaacatc tgaccataac attttgaaat gatgcaaata aatttccaaa caaaaatagt 4380
gtgaatttaa aagcaaatta tttgagtatc taagaaacaa gatagacttc tagaaaaatt 4440
tgactctcta gaatatttct tgcagaaatg agatttttca taatagtaaa agaggcatat 4500
gtttatcaaa caatgctgtc acaaaaagca tcaactgtaa tgggactatt aatgcataat 4560
tattgattta ttcattcaat taatatacaa ttatcccttt tcatttaaag atttaattca 4620
taattacaat tataataaaa cttcctttaa agtaagatac aataatttta ttgtttttca 4680
ttcttttttc aacaaaatat cccatccaat cattttttat attattaaat attggctgct 4740
ttttcttgga ttcacattaa acagcccttt ccaacttcca attgtcttaa aataatgatg 4800
acctcctgtg agtagataca gctctttaca atttttttct ttagtgcctt ttcttcttga 4860
atttttccta tatcaaatgg agaatatatg tacagatggt attttctcag tttataggca 4920
tatcagtgac catggctttc tttatatagg tttttaaaaa agccctaaat aataaatagc 4980
cagatgagct ggggacattg agaaatagcc ttcctcttcc tttttcaact catttttttc 5040
ccacctacat gactgtaaat caaatattta atagctctta cttaaaaaaa cagatacaaa 5100
gaatgtcttg atttggtgtg ctcatttacc ataatgtcat gaggggaatt agatttcaca 5160
actttaaaag gaatatattt ttattttatt ttgaaaaact gagtcatata ggaattttct 5220
tatacttcaa ggcatcatgg aaacactttt ttcctgtttg gatattgtgg aatttaaacg 5280
ttcaaataaa taaatggcat aactaagtgt tccaaatttt tttacaatgt ctttgaccct 5340
attcaaacac tttaggtatt tactgaccgt ctgatgtgta agatgtggaa taaaactgga 5400
atcaattaat tatttcactg tgttatcagc gcaagatcaa ccatctgggt ttcttaaaga 5460
cacccgaagg attgaatttt gtttcagtat tgataatggc atagtctcta tgtgctacat 5520
ggaattacat catttatccc tccagtgccc tatatgttga taagtatgtc agtttgactt 5580
agtatacata tatacagaga tttcattaca tttacctaat aaatacaaaa tacatttcga 5640
gtgttactga tctcctatgt tacatgagcc tcctgtaatc attgtgcatt gatgttccaa 5700
tgttttattg tttgtatgaa ttttaatttg aaaacaagga aacaatccaa aagcagaaaa 5760
aatagctttt cttaaaattt tcagtgctac attttccctc tgaggtccat agagatttga 5820
atgtatagga gattatccga aaacagctat tttgattaaa aaatatatct cccaggattc 5880
aaccaactta atgatgaagt actattgtct actgctttat acataaaagg gaacttttta 5940
tctgcttgta aagggatttt tatgtgtatt tctgcataat cagatgactt ctattgtgtt 6000
ttctactgat gaaattctct gtaaaatgtc tttttcttac attatccaac aggcataaag 6060
aataacagta aagacttttg tgtttgtaat actacctctt ttatccctgc actactgttt 6120
tattgcaaaa attctatatt gtcactgtat tttttccata gaatataaat tttgttcttg 6180
tgctaaagct ggtagtttat gtagcagaca aaatatacaa ataaaagaag agactgattt 6240
tgctgaaaga attatatata atcaagaagt tacatattgt tctttaaata tgatactgaa 6300
ttttaaaagc aaacgaaatt caagaatctt atctaacagc atagcagttg cttatggcat 6360
acaaggctaa aattaattca gctatttaat cttaataatt attatgtagt taaaaatctt 6420
tgactttaat agtgttttac atatacaaat agctgaagta acattcctat aattttaatc 6480
tgacattggt tagatcaaga aaacattgtt aataagactg tagaatttgt aattattgct 6540
atttttcatt tttaataaca aagtaatgtg tcttattttc taagaaaatg gagaactttg 6600
gtgtacttta atacatacaa aaatctttgt aaaaatacct taaaatgtac caatattttc 6660
tttgcatata ttaaatgaaa gactataatt atgaaatgtt 6700
<210> 9
<211> 62
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 9
cccagcagga gcaggagcag gagcgggagc gggatcccca gcaggagcag gagcgggagc 60
gg 62
<210> 10
<211> 65
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 10
ctgcctgtgt cagggaatct gcttgatgtg tacagtagtc aaggcgtggc cacaccagcc 60
atcac 65
<210> 11
<211> 21
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 11
cattgagcat ggcatcgtca c 21
<210> 12
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 12
ggtttggaca tggcaagctc 20
<210> 13
<211> 287
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 13
cattgagcat ggcatcgtca ccaactggga cgacatggag aagatctggc accacacctt 60
ctacaacgag ctgcgcgtgg ccccggagga gcacccagtg ctgctgaccg aggcccccct 120
gaaccccaag gccaacagag agaagatgac tcaggtgcag acccacctcg aaaaccccac 180
caagtaccac atacagcaag cccaacggca gcaggtaaag cagtaccttt ctaccacttt 240
agcaaataaa catgccaacc aagtcctgag cttgccatgt ccaaacc 287
<210> 14
<211> 95
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多肽"
<400> 14
Ile Glu His Gly Ile Val Thr Asn Trp Asp Asp Met Glu Lys Ile Trp
1 5 10 15
His His Thr Phe Tyr Asn Glu Leu Arg Val Ala Pro Glu Glu His Pro
20 25 30
Val Leu Leu Thr Glu Ala Pro Leu Asn Pro Lys Ala Asn Arg Glu Lys
35 40 45
Met Thr Gln Val Gln Thr His Leu Glu Asn Pro Thr Lys Tyr His Ile
50 55 60
Gln Gln Ala Gln Arg Gln Gln Val Lys Gln Tyr Leu Ser Thr Thr Leu
65 70 75 80
Ala Asn Lys His Ala Asn Gln Val Leu Ser Leu Pro Cys Pro Asn
85 90 95
<210> 15
<211> 70
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 15
tgagaagaga gatccacatc tggcctgtgt tgcttatgaa cgtggccaat gtgatctgga 60
acttattaat 70
<210> 16
<211> 66
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 16
ctacccctgt ccagcagcca cctgaatgtg tacagcagcg acccccaggt cacagcctcc 60
ctggtg 66
<210> 17
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 17
gcttctgcct tggctagagg 20
<210> 18
<211> 21
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 18
gtcgctgctg tacacattca g 21
<210> 19
<211> 268
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 19
gcttctgcct tggctagagg ccagaattca tgagggctgt gaggagcctg ctactcacaa 60
tgccttagcc aaaatctaca tagacagtaa taacaacccg gagagatttc ttcgtgaaaa 120
tccctactat gacagtcgcg ttgttggaaa gtattgtgag aagagagatc cacatctggc 180
ctgtgttgct tatgaacgtg gccaatgtga tctggaactt attaatctac ccctgtccag 240
cagccacctg aatgtgtaca gcagcgac 268
<210> 20
<211> 89
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多肽"
<400> 20
Leu Leu Pro Trp Leu Glu Ala Arg Ile His Glu Gly Cys Glu Glu Pro
1 5 10 15
Ala Thr His Asn Ala Leu Ala Lys Ile Tyr Ile Asp Ser Asn Asn Asn
20 25 30
Pro Glu Arg Phe Leu Arg Glu Asn Pro Tyr Tyr Asp Ser Arg Val Val
35 40 45
Gly Lys Tyr Cys Glu Lys Arg Asp Pro His Leu Ala Cys Val Ala Tyr
50 55 60
Glu Arg Gly Gln Cys Asp Leu Glu Leu Ile Asn Leu Pro Leu Ser Ser
65 70 75 80
Ser His Leu Asn Val Tyr Ser Ser Asp
85
<210> 21
<211> 69
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 21
cggccagcgc gcattcggcc ccggacgaag gtactcgcag cacttggagc gcagaaccgg 60
ccgcgcccg 69
<210> 22
<211> 70
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 22
atcatgatgc tgccgccacc gccgccacca cggagcgaga agcccagata gacgccccgg 60
cggccccggg 70
<210> 23
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 23
gtactcgcag cacttggagc 20
<210> 24
<211> 21
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的引物"
<400> 24
atctgggctt ctcgctccgt g 21
<210> 25
<211> 88
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 25
gtactcgcag cacttggagc gcagaaccgg ccgcgcccga tcatgatgct gccgccaccg 60
ccgccaccac ggagcgagaa gcccagat 88
<210> 26
<211> 4659
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 26
atggaagaag agatcgccgc gctggtcatt gacaatggct ccggcatgtg caaagctggt 60
tttgctgggg acgacgctcc ccgagccgtg tttccttcca tcgtcgggcg ccccagacac 120
cagggcgtca tggtgggcat gggccagaag gactcctacg tgggcgacga ggcccagagc 180
aagcgtggca tcctgaccct gaagtacccc attgagcatg gcatcgtcac caactgggac 240
gacatggaga agatctggca ccacaccttc tacaacgagc tgcgcgtggc cccggaggag 300
cacccagtgc tgctgaccga ggcccccctg aaccccaagg ccaacagaga gaagatgact 360
caggtgcaga cccacctcga aaaccccacc aagtaccaca tacagcaagc ccaacggcag 420
caggtaaagc agtacctttc taccacttta gcaaataaac atgccaacca agtcctgagc 480
ttgccatgtc caaaccagcc tggcgatcat gtcatgccac cggtgccggg gagcagcgca 540
cccaacagcc ccatggctat gcttacgctt aactccaact gtgaaaaaga gggattttat 600
aagtttgaag agcaaaacag ggcagagagc gagtgcccag gcatgaacac acattcacga 660
gcgtcctgta tgcagatgga tgatgtaatc gatgacatca ttagcctaga atcaagttat 720
aatgaggaaa tcttgggctt gatggatcct gctttgcaaa tggcaaatac gttgcctgtc 780
tcgggaaact tgattgatct ttatggaaac caaggtctgc ccccaccagg cctcaccatc 840
agcaactcct gtccagccaa ccttcccaac ataaaaaggg agctcacaga gtctgaagca 900
agagcactgg ccaaagagag gcagaaaaag gacaatcaca acctgattga acgaagaaga 960
agatttaaca taaatgaccg cattaaagaa ctaggtactt tgattcccaa gtcaaatgat 1020
ccagacatgc gctggaacaa gggaaccatc ttaaaagcat ccgtggacta tatccgaaag 1080
ttgcaacgag aacagcaacg cgcaaaagaa cttgaaaacc gacagaagaa actggagcac 1140
gccaaccggc atttgttgct cagaatacag gaacttgaaa tgcaggctcg agctcatgga 1200
ctttccctta ttccatccac gggtctctgc tctccagatt tggtgaatcg gatcatcaag 1260
caagaacccg ttcttgagaa ctgcagccaa gacctccttc agcatcatgc agacctaacc 1320
tgtacaacaa ctctcgatct cacggatggc accatcacct tcaacaacaa cctcggaact 1380
gggactgagg ccaaccaagc ctatagtgtc cccacaaaaa tgggatccaa actggaagac 1440
atcctgatgg acgacaccct ttctcccgtc ggtgtcactg atccactcct ttcctcagtg 1500
tcccccggag cttccaaaac aagcagccgg aggagcagta tgagcatgga agagacggag 1560
cacacttgtt agcgaatcct ccctgcactg cattcgcaca aactgcttcc tttcttgatt 1620
cgtagattta ataacttacc tgaaggggtt ttcttgataa ttttccttta atatgaaatt 1680
ttttttcatg ctttatcaat agcccaggat atattttatt tttagaattt tgtgaaacag 1740
acttgtatat tctattttac aactacaaat gcctccaaag tattgtacaa ataagtgtgc 1800
agtatctgtg aactgaattc accacagact ttagctttct gagcaagagg attttgcgtc 1860
agagaaatgt ctgtccattt ttattcaggg gaaacttgat ttgagatttt tatgcctgtg 1920
acttccttgg aaatcaaatg taaagtttaa ttgaaagaat gtaaagcaac caaaaagaaa 1980
aaaaaaaaga aagaaagagg aaaagaaatc catactaacc cttttccatt ttataaatgt 2040
attgattcat tggtactgcc ttaaagatac agtacccctc tagctttgtt tagtctttat 2100
actgcaaact atttaaagaa atatgtattc tgtaaaagaa aaaaaaaatg cggccttttc 2160
atgaggatcg tctggttaga aaacataact gataccaacc gaaactgaag ggagttagac 2220
caaggctctg aaatataaag tctaatcttg ctctctttta ttctgtgctg ttacagtttt 2280
cttcatcaat gagtgtgatc cagtttttca taagatattt tattttgaaa tggaaattaa 2340
tgtcctctca aagtaaaata ttgaggagca ctgaaagtat gttttacttt ttttttattt 2400
tatttttgct tttgataaga aaaccgaact gggcatattt ctaattggct ttactatttt 2460
tatttttaaa ttatgtttta ctgttcattt gatttgtaca gattctttat tatcattgtt 2520
cttttcaata tatttgtatt aatttgtaag aatatgcatc ttaaaatggc aagttttcca 2580
tatttttaca actcactggt ggttttccgc attctttgta cacccatgaa agaaaacttt 2640
tatgcaaggt cttgcattta aaagacagct ttgcgaatat tttgtaaatt acagtctcac 2700
tcagaactgt ttttggacac atttaaggtg tagtattaat aggttaaaac caggctttct 2760
agaaagaata aacttacata tttattttta ggacatgaaa atagcaatat tcttggagat 2820
tgataaccat agcattaata cgcccattat ggtcatttaa attggggttt atttcagcaa 2880
acttgttgaa tttattttta agaaagaaat actgtattgg gaagttactg ttacttgata 2940
acaatgtttt aacaagaagc aatgttataa agttagtttc agtgcattat ctacttgtgt 3000
agtcctatgc aataacagta gtgttacatg tatcaagcct agatgtttta tacagatgcc 3060
atatagtgtt atgagccagg ctgttgaatg gaatttctca gtagcagcct acaactgaat 3120
agcaagtggc ataaagcata tccattcaga atgaagtgcc ttaaatatag cagtagtctt 3180
ttttggacta gcactgactg aactgtaatg taggggaaag tttcatgatg gtatctatag 3240
tcaagacgaa catgtagcat ggtgcctatg tagacaatat aagagcttcc aattttcctt 3300
cagatatttt taatattaaa tatattttag tgacagagtg ccaacttctt tcatcaggaa 3360
accttattca ggagggtttt taaaaagtgt ttaaatgtca aatgtgaatt ggtgatgggt 3420
gatggagggt tcagagagga gtgatcgtca gatgtgtgaa tggacggttt aggtgaaaat 3480
aatcaactgc atagttccca tgcacgctgg gcaatgagaa tccttggaaa cattggtgat 3540
gctatcagtt ttatagcttt atttcttaag ggggtaggga aaattagttc ccattctttc 3600
aaccccctta actgtatagc tcttttccta gaatagtgac gcaaatctgc atgaacagct 3660
aattgtacca tagtgttcat tgatacaatc atagcattgt ctatttttct cttcatattt 3720
atatgggggg gagggcgctg gatgcaaaag ttgaagatcg tgatgctatg atgttagttt 3780
tccttagctg attttgaggg tttttaaaaa taaagcaagg ttgactaacc tacggccacg 3840
ggaacaggac catggttaag caaccatata gaaagctttg ttgaaagaaa gtatggcatc 3900
ttgtaccact gccctgactg tcacaactcc taaccttgcc attgcctgcc tccccctccc 3960
cttctcctta agagacaatt tctgcaggtg gcaggtgagc aagcccagga gaatgctgca 4020
atcttggggg tggttttatt tatttctttt ttgccaaata gagtgtggat tcatttcagg 4080
ggctagctaa gccaagaggc agtggtttgg gcttgttgtt tgtaacaaga aaatgatcca 4140
caccactccc ccgattcccg ggtgcagaat tgtaactcgg ggttgggcct ctatatggag 4200
tgaccaaaat gccaaaattg tccatctgcc tctgagtagg gcaatggaaa taccaaacct 4260
tctgactttg ccaaaaagca tacaagcaac ctggtcatac ataggatgac aaaattcttt 4320
ctggttgttt ttaaacaata aagcaataag aacaaataca atacatagga agttaaaagc 4380
acaaaggaat gaacttatta atatttttga aaaatgcact gggaaaaagt tgatgtcaat 4440
aacagtataa aacagcccta tttcttgata aaaaatgaca aatgactgtc tcttgcggat 4500
gcttggtact gtaatgttaa taatagtcac ctgctgttgg atgcagcaat aatttctgta 4560
tggtccatag cactgtatat tatggatcga tattaatgta tccaatgaaa taatcgactt 4620
gttcttgata gcctcattaa agcatttggt ttttcacat 4659
<210> 27
<211> 523
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多肽"
<400> 27
Met Glu Glu Glu Ile Ala Ala Leu Val Ile Asp Asn Gly Ser Gly Met
1 5 10 15
Cys Lys Ala Gly Phe Ala Gly Asp Asp Ala Pro Arg Ala Val Phe Pro
20 25 30
Ser Ile Val Gly Arg Pro Arg His Gln Gly Val Met Val Gly Met Gly
35 40 45
Gln Lys Asp Ser Tyr Val Gly Asp Glu Ala Gln Ser Lys Arg Gly Ile
50 55 60
Leu Thr Leu Lys Tyr Pro Ile Glu His Gly Ile Val Thr Asn Trp Asp
65 70 75 80
Asp Met Glu Lys Ile Trp His His Thr Phe Tyr Asn Glu Leu Arg Val
85 90 95
Ala Pro Glu Glu His Pro Val Leu Leu Thr Glu Ala Pro Leu Asn Pro
100 105 110
Lys Ala Asn Arg Glu Lys Met Thr Gln Val Gln Thr His Leu Glu Asn
115 120 125
Pro Thr Lys Tyr His Ile Gln Gln Ala Gln Arg Gln Gln Val Lys Gln
130 135 140
Tyr Leu Ser Thr Thr Leu Ala Asn Lys His Ala Asn Gln Val Leu Ser
145 150 155 160
Leu Pro Cys Pro Asn Gln Pro Gly Asp His Val Met Pro Pro Val Pro
165 170 175
Gly Ser Ser Ala Pro Asn Ser Pro Met Ala Met Leu Thr Leu Asn Ser
180 185 190
Asn Cys Glu Lys Glu Gly Phe Tyr Lys Phe Glu Glu Gln Asn Arg Ala
195 200 205
Glu Ser Glu Cys Pro Gly Met Asn Thr His Ser Arg Ala Ser Cys Met
210 215 220
Gln Met Asp Asp Val Ile Asp Asp Ile Ile Ser Leu Glu Ser Ser Tyr
225 230 235 240
Asn Glu Glu Ile Leu Gly Leu Met Asp Pro Ala Leu Gln Met Ala Asn
245 250 255
Thr Leu Pro Val Ser Gly Asn Leu Ile Asp Leu Tyr Gly Asn Gln Gly
260 265 270
Leu Pro Pro Pro Gly Leu Thr Ile Ser Asn Ser Cys Pro Ala Asn Leu
275 280 285
Pro Asn Ile Lys Arg Glu Leu Thr Glu Ser Glu Ala Arg Ala Leu Ala
290 295 300
Lys Glu Arg Gln Lys Lys Asp Asn His Asn Leu Ile Glu Arg Arg Arg
305 310 315 320
Arg Phe Asn Ile Asn Asp Arg Ile Lys Glu Leu Gly Thr Leu Ile Pro
325 330 335
Lys Ser Asn Asp Pro Asp Met Arg Trp Asn Lys Gly Thr Ile Leu Lys
340 345 350
Ala Ser Val Asp Tyr Ile Arg Lys Leu Gln Arg Glu Gln Gln Arg Ala
355 360 365
Lys Glu Leu Glu Asn Arg Gln Lys Lys Leu Glu His Ala Asn Arg His
370 375 380
Leu Leu Leu Arg Ile Gln Glu Leu Glu Met Gln Ala Arg Ala His Gly
385 390 395 400
Leu Ser Leu Ile Pro Ser Thr Gly Leu Cys Ser Pro Asp Leu Val Asn
405 410 415
Arg Ile Ile Lys Gln Glu Pro Val Leu Glu Asn Cys Ser Gln Asp Leu
420 425 430
Leu Gln His His Ala Asp Leu Thr Cys Thr Thr Thr Leu Asp Leu Thr
435 440 445
Asp Gly Thr Ile Thr Phe Asn Asn Asn Leu Gly Thr Gly Thr Glu Ala
450 455 460
Asn Gln Ala Tyr Ser Val Pro Thr Lys Met Gly Ser Lys Leu Glu Asp
465 470 475 480
Ile Leu Met Asp Asp Thr Leu Ser Pro Val Gly Val Thr Asp Pro Leu
485 490 495
Leu Ser Ser Val Ser Pro Gly Ala Ser Lys Thr Ser Ser Arg Arg Ser
500 505 510
Ser Met Ser Met Glu Glu Thr Glu His Thr Cys
515 520
<210> 28
<211> 4298
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 28
atggcccaga ttctgccaat tcgttttcag gagcatctcc agctccagaa cctgggtatc 60
aacccagcaa acattggctt cagtaccctg actatggagt ctgacaaatt catctgcatt 120
agagaaaaag taggagagca ggcccaggtg gtaatcattg atatgaatga cccaagtaat 180
ccaattcgaa gaccaatttc agcagacagc gccatcatga atccagctag caaagtaatt 240
gcactgaaag ctgggaaaac tcttcagatt tttaacattg aaatgaaaag taaaatgaag 300
gctcatacca tgactgatga tgtcaccttt tggaaatgga tctctttgaa tacggttgct 360
cttgttacgg ataatgcagt ttatcactgg agtatggaag gagagtctca gccagtgaaa 420
atgtttgatc gccattctag ccttgcaggg tgccagatta tcaattaccg tacagatgca 480
aaacaaaagt ggttacttct gactggtata tctgcacagc aaaatcgtgt ggtgggagct 540
atgcagctat attctgtaga taggaaagtg tctcagccca ttgaaggaca tgcagctagc 600
tttgcacagt ttaagatgga aggaaatgca gaagaatcaa cgttattttg ttttgcagtt 660
cggggccaag ctggagggaa gttacatatt attgaagttg gcacaccacc tacagggaac 720
cagccctttc caaagaaggc agtggatgtc ttctttcctc cagaagcaca aaatgatttt 780
cctgttgcaa tgcagatcag tgaaaagcat gatgtggtgt tcttgataac caagtatggt 840
tatatccacc tctatgatct tgagactggt acctgcatct acatgaatag aatcagtgga 900
gaaacaattt ttgttactgc acctcatgaa gccacagctg gaataattgg agtaaacaga 960
aagggacaag ttctgtcagt gtgtgtggaa gaagaaaaca taattcctta catcaccaat 1020
gttctacaaa atcctgattt ggctctgaga atggctgtac gtaataactt agccggtgct 1080
gaagaactct ttgcccggaa atttaatgct ctttttgccc agggaaatta ctcggaggca 1140
gcaaaggtgg ctgctaatgc accaaaggga attcttcgta ctccagacac tatccgtcgg 1200
ttccagagtg tcccagccca gccaggtcaa acttctcctc tacttcagta ctttggtatc 1260
cttttggacc agggacagct caacaaatac gaatccttag agctttgtag gcctgtactt 1320
cagcaagggc gaaaacagct tttggagaaa tggttaaaag aagataagct ggaatgttct 1380
gaagaactgg gtgatcttgt gaaatctgtg gaccctacat tggcacttag tgtgtaccta 1440
agggctaacg tcccaaataa agtcattcag tgctttgcag aaacaggtca agtccaaaag 1500
attgttttat atgctaaaaa agttggatac actccagatt ggatatttct gctgagaaat 1560
gtaatgcgaa tcagtccaga tcagggacag cagtttgccc aaatgttagt tcaagatgaa 1620
gagcctcttg ctgacatcac acagattgta gatgtcttta tggaatacaa tctaattcag 1680
cagtgtactg cattcttgct tgatgctctg aagaataatc gcccatctga aggtccttta 1740
cagacgcggt tacttgagat gaaccttatg catgcgcctc aagttgcaga tgctattcta 1800
ggcaatcaga tgttcacaca ttatgaccgg gctcatattg ctcaactgtg tgaaaaggct 1860
ggcctactgc agcgtgcatt agaacatttc actgatttat atgatataaa acgtgcagtg 1920
gttcacaccc atcttcttaa ccctgagtgg ttagtcaact actttggttc cttatcagta 1980
gaagactccc tagaatgtct cagagccatg ctgtctgcca acatccgtca gaatctgcag 2040
atttgtgttc aggtggcttc taaatatcat gaacaactgt caactcagtc tctgattgaa 2100
ctttttgaat ctttcaagag ttttgaaggt ctcttttatt ttctgggatc cattgttaac 2160
tttagccagg acccagatgt gcactttaaa tatattcagg cagcttgcaa gactgggcaa 2220
atcaaagaag tagaaagaat ctgtagagaa agcaactgct acgatcctga gcgagtcaag 2280
aattttctta aggaagcaaa actaacagat cagctaccac ttatcattgt gtgtgatcga 2340
tttgactttg tccatgattt ggtgctctat ttatatagaa ataatcttca aaagtatata 2400
gagatatatg tacagaaggt gaatccaagt cgacttcctg tagttattgg aggattactt 2460
gatgttgact gttctgaaga tgtcataaaa aacttgattc ttgttgtaag aggtcaattc 2520
tctactgatg agcttgttgc tgaggttgaa aaaagaaaca gattgaaact gcttctgcct 2580
tggctagagg ccagaattca tgagggctgt gaggagcctg ctactcacaa tgccttagcc 2640
aaaatctaca tagacagtaa taacaacccg gagagatttc ttcgtgaaaa tccctactat 2700
gacagtcgcg ttgttggaaa gtattgtgag aagagagatc cacatctggc ctgtgttgct 2760
tatgaacgtg gccaatgtga tctggaactt attaatctac ccctgtccag cagccacctg 2820
aatgtgtaca gcagcgaccc ccaggtcaca gcctccctgg tgggcgtcac cagcagctcc 2880
tgccctgcgg acctgaccca gaagcgagag ctcacagatg ctgagagcag ggccctggcc 2940
aaggagcggc agaagaaaga caatcacaac ttaattgaaa ggagacgaag gttcaacatc 3000
aatgaccgca tcaaggagtt gggaatgctg atccccaagg ccaatgacct ggacgtgcgc 3060
tggaacaagg gcaccatcct caaggcctct gtggattaca tccggaggat gcagaaggac 3120
ctgcaaaagt ccagggagct ggagaaccac tctcgccgcc tggagatgac caacaagcag 3180
ctctggctcc gtatccagga gctggagatg caggctcgag tgcacggcct ccctaccacc 3240
tccccgtccg gcatgaacat ggctgagctg gcccagcagg tggtgaagca ggagctgcct 3300
agcgaagagg gcccagggga ggccctgatg ctgggggctg aggtccctga ccctgagcca 3360
ctgccagctc tgcccccgca agccccgctg cccctgccca cccagccacc atccccattc 3420
catcacctgg acttcagcca cagcctgagc tttgggggca gggaggacga gggtcccccg 3480
ggctaccccg aacccctggc gccggggcat ggctccccat tccccagcct gtccaagaag 3540
gatctggacc tcatgctcct ggacgactca ctgctaccgc tggcctctga tccacttctg 3600
tccaccatgt cccccgaggc ctccaaggcc agcagccgcc ggagcagctt cagcatggag 3660
gagggcgatg tgctgtgacc ctggctgccc ctgtgccagg gaacaggggc cggcctgggg 3720
gctgggaggg ccaggggcac ctccctccca cccttcaggc tgcactgtgt gtgaagtagc 3780
cacctgccct gcctccctcc tccccgttgg cccctgtttg gacttagtgc ctgtctggca 3840
gcctgtgggg tcaggagaag cacccccagg gcagccctct tgactggcgc agtgggaaga 3900
ggccttcagc ccctctcccg gagatggaat cgcggggcag ggaggggcag ggtgttctag 3960
aggtgagaag agggcctggt ggagattccc tgtcttctga gcccgagccc ctcattacca 4020
gtgaaggaca tgcttgaggg gttcgggaag ctcctcatct gaggcaactg gtcctggggg 4080
tgctcaggcc tgcctttttg ggactcagat ggcaggaggt ccaccccgca gcctggtcct 4140
cggctctccc acaggtgggc accccccact ttggtgctaa tagctctcca ccaggtggtg 4200
tgagcgcggg ggctgccaga agcgggaggg gtcactgccg gaagagcagc tgccctccga 4260
cccctcactt tgtgccttta gtaaacactg tgctttgt 4298
<210> 29
<211> 1225
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多肽"
<400> 29
Met Ala Gln Ile Leu Pro Ile Arg Phe Gln Glu His Leu Gln Leu Gln
1 5 10 15
Asn Leu Gly Ile Asn Pro Ala Asn Ile Gly Phe Ser Thr Leu Thr Met
20 25 30
Glu Ser Asp Lys Phe Ile Cys Ile Arg Glu Lys Val Gly Glu Gln Ala
35 40 45
Gln Val Val Ile Ile Asp Met Asn Asp Pro Ser Asn Pro Ile Arg Arg
50 55 60
Pro Ile Ser Ala Asp Ser Ala Ile Met Asn Pro Ala Ser Lys Val Ile
65 70 75 80
Ala Leu Lys Ala Gly Lys Thr Leu Gln Ile Phe Asn Ile Glu Met Lys
85 90 95
Ser Lys Met Lys Ala His Thr Met Thr Asp Asp Val Thr Phe Trp Lys
100 105 110
Trp Ile Ser Leu Asn Thr Val Ala Leu Val Thr Asp Asn Ala Val Tyr
115 120 125
His Trp Ser Met Glu Gly Glu Ser Gln Pro Val Lys Met Phe Asp Arg
130 135 140
His Ser Ser Leu Ala Gly Cys Gln Ile Ile Asn Tyr Arg Thr Asp Ala
145 150 155 160
Lys Gln Lys Trp Leu Leu Leu Thr Gly Ile Ser Ala Gln Gln Asn Arg
165 170 175
Val Val Gly Ala Met Gln Leu Tyr Ser Val Asp Arg Lys Val Ser Gln
180 185 190
Pro Ile Glu Gly His Ala Ala Ser Phe Ala Gln Phe Lys Met Glu Gly
195 200 205
Asn Ala Glu Glu Ser Thr Leu Phe Cys Phe Ala Val Arg Gly Gln Ala
210 215 220
Gly Gly Lys Leu His Ile Ile Glu Val Gly Thr Pro Pro Thr Gly Asn
225 230 235 240
Gln Pro Phe Pro Lys Lys Ala Val Asp Val Phe Phe Pro Pro Glu Ala
245 250 255
Gln Asn Asp Phe Pro Val Ala Met Gln Ile Ser Glu Lys His Asp Val
260 265 270
Val Phe Leu Ile Thr Lys Tyr Gly Tyr Ile His Leu Tyr Asp Leu Glu
275 280 285
Thr Gly Thr Cys Ile Tyr Met Asn Arg Ile Ser Gly Glu Thr Ile Phe
290 295 300
Val Thr Ala Pro His Glu Ala Thr Ala Gly Ile Ile Gly Val Asn Arg
305 310 315 320
Lys Gly Gln Val Leu Ser Val Cys Val Glu Glu Glu Asn Ile Ile Pro
325 330 335
Tyr Ile Thr Asn Val Leu Gln Asn Pro Asp Leu Ala Leu Arg Met Ala
340 345 350
Val Arg Asn Asn Leu Ala Gly Ala Glu Glu Leu Phe Ala Arg Lys Phe
355 360 365
Asn Ala Leu Phe Ala Gln Gly Asn Tyr Ser Glu Ala Ala Lys Val Ala
370 375 380
Ala Asn Ala Pro Lys Gly Ile Leu Arg Thr Pro Asp Thr Ile Arg Arg
385 390 395 400
Phe Gln Ser Val Pro Ala Gln Pro Gly Gln Thr Ser Pro Leu Leu Gln
405 410 415
Tyr Phe Gly Ile Leu Leu Asp Gln Gly Gln Leu Asn Lys Tyr Glu Ser
420 425 430
Leu Glu Leu Cys Arg Pro Val Leu Gln Gln Gly Arg Lys Gln Leu Leu
435 440 445
Glu Lys Trp Leu Lys Glu Asp Lys Leu Glu Cys Ser Glu Glu Leu Gly
450 455 460
Asp Leu Val Lys Ser Val Asp Pro Thr Leu Ala Leu Ser Val Tyr Leu
465 470 475 480
Arg Ala Asn Val Pro Asn Lys Val Ile Gln Cys Phe Ala Glu Thr Gly
485 490 495
Gln Val Gln Lys Ile Val Leu Tyr Ala Lys Lys Val Gly Tyr Thr Pro
500 505 510
Asp Trp Ile Phe Leu Leu Arg Asn Val Met Arg Ile Ser Pro Asp Gln
515 520 525
Gly Gln Gln Phe Ala Gln Met Leu Val Gln Asp Glu Glu Pro Leu Ala
530 535 540
Asp Ile Thr Gln Ile Val Asp Val Phe Met Glu Tyr Asn Leu Ile Gln
545 550 555 560
Gln Cys Thr Ala Phe Leu Leu Asp Ala Leu Lys Asn Asn Arg Pro Ser
565 570 575
Glu Gly Pro Leu Gln Thr Arg Leu Leu Glu Met Asn Leu Met His Ala
580 585 590
Pro Gln Val Ala Asp Ala Ile Leu Gly Asn Gln Met Phe Thr His Tyr
595 600 605
Asp Arg Ala His Ile Ala Gln Leu Cys Glu Lys Ala Gly Leu Leu Gln
610 615 620
Arg Ala Leu Glu His Phe Thr Asp Leu Tyr Asp Ile Lys Arg Ala Val
625 630 635 640
Val His Thr His Leu Leu Asn Pro Glu Trp Leu Val Asn Tyr Phe Gly
645 650 655
Ser Leu Ser Val Glu Asp Ser Leu Glu Cys Leu Arg Ala Met Leu Ser
660 665 670
Ala Asn Ile Arg Gln Asn Leu Gln Ile Cys Val Gln Val Ala Ser Lys
675 680 685
Tyr His Glu Gln Leu Ser Thr Gln Ser Leu Ile Glu Leu Phe Glu Ser
690 695 700
Phe Lys Ser Phe Glu Gly Leu Phe Tyr Phe Leu Gly Ser Ile Val Asn
705 710 715 720
Phe Ser Gln Asp Pro Asp Val His Phe Lys Tyr Ile Gln Ala Ala Cys
725 730 735
Lys Thr Gly Gln Ile Lys Glu Val Glu Arg Ile Cys Arg Glu Ser Asn
740 745 750
Cys Tyr Asp Pro Glu Arg Val Lys Asn Phe Leu Lys Glu Ala Lys Leu
755 760 765
Thr Asp Gln Leu Pro Leu Ile Ile Val Cys Asp Arg Phe Asp Phe Val
770 775 780
His Asp Leu Val Leu Tyr Leu Tyr Arg Asn Asn Leu Gln Lys Tyr Ile
785 790 795 800
Glu Ile Tyr Val Gln Lys Val Asn Pro Ser Arg Leu Pro Val Val Ile
805 810 815
Gly Gly Leu Leu Asp Val Asp Cys Ser Glu Asp Val Ile Lys Asn Leu
820 825 830
Ile Leu Val Val Arg Gly Gln Phe Ser Thr Asp Glu Leu Val Ala Glu
835 840 845
Val Glu Lys Arg Asn Arg Leu Lys Leu Leu Leu Pro Trp Leu Glu Ala
850 855 860
Arg Ile His Glu Gly Cys Glu Glu Pro Ala Thr His Asn Ala Leu Ala
865 870 875 880
Lys Ile Tyr Ile Asp Ser Asn Asn Asn Pro Glu Arg Phe Leu Arg Glu
885 890 895
Asn Pro Tyr Tyr Asp Ser Arg Val Val Gly Lys Tyr Cys Glu Lys Arg
900 905 910
Asp Pro His Leu Ala Cys Val Ala Tyr Glu Arg Gly Gln Cys Asp Leu
915 920 925
Glu Leu Ile Asn Leu Pro Leu Ser Ser Ser His Leu Asn Val Tyr Ser
930 935 940
Ser Asp Pro Gln Val Thr Ala Ser Leu Val Gly Val Thr Ser Ser Ser
945 950 955 960
Cys Pro Ala Asp Leu Thr Gln Lys Arg Glu Leu Thr Asp Ala Glu Ser
965 970 975
Arg Ala Leu Ala Lys Glu Arg Gln Lys Lys Asp Asn His Asn Leu Ile
980 985 990
Glu Arg Arg Arg Arg Phe Asn Ile Asn Asp Arg Ile Lys Glu Leu Gly
995 1000 1005
Met Leu Ile Pro Lys Ala Asn Asp Leu Asp Val Arg Trp Asn Lys
1010 1015 1020
Gly Thr Ile Leu Lys Ala Ser Val Asp Tyr Ile Arg Arg Met Gln
1025 1030 1035
Lys Asp Leu Gln Lys Ser Arg Glu Leu Glu Asn His Ser Arg Arg
1040 1045 1050
Leu Glu Met Thr Asn Lys Gln Leu Trp Leu Arg Ile Gln Glu Leu
1055 1060 1065
Glu Met Gln Ala Arg Val His Gly Leu Pro Thr Thr Ser Pro Ser
1070 1075 1080
Gly Met Asn Met Ala Glu Leu Ala Gln Gln Val Val Lys Gln Glu
1085 1090 1095
Leu Pro Ser Glu Glu Gly Pro Gly Glu Ala Leu Met Leu Gly Ala
1100 1105 1110
Glu Val Pro Asp Pro Glu Pro Leu Pro Ala Leu Pro Pro Gln Ala
1115 1120 1125
Pro Leu Pro Leu Pro Thr Gln Pro Pro Ser Pro Phe His His Leu
1130 1135 1140
Asp Phe Ser His Ser Leu Ser Phe Gly Gly Arg Glu Asp Glu Gly
1145 1150 1155
Pro Pro Gly Tyr Pro Glu Pro Leu Ala Pro Gly His Gly Ser Pro
1160 1165 1170
Phe Pro Ser Leu Ser Lys Lys Asp Leu Asp Leu Met Leu Leu Asp
1175 1180 1185
Asp Ser Leu Leu Pro Leu Ala Ser Asp Pro Leu Leu Ser Thr Met
1190 1195 1200
Ser Pro Glu Ala Ser Lys Ala Ser Ser Arg Arg Ser Ser Phe Ser
1205 1210 1215
Met Glu Glu Gly Asp Val Leu
1220 1225
<210> 30
<211> 8
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的肽"
<400> 30
Lys Met Thr Gln Val Gln Thr His
1 5
<210> 31
<211> 11
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的肽"
<400> 31
Asp Leu Glu Leu Ile Asn Leu Pro Leu Ser Ser
1 5 10
<210> 32
<211> 50
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多肽"
<400> 32
Glu Glu Arg Thr Cys Lys Val Cys Leu Asp Arg Ala Val Ser Ile Val
1 5 10 15
Phe Val Pro Cys Gly His Leu Val Cys Ala Glu Cys Ala Pro Gly Leu
20 25 30
Gln Leu Cys Pro Ile Cys Arg Ala Pro Val Arg Ser Arg Val Arg Thr
35 40 45
Phe Leu
50
<210> 33
<211> 14
<212> PRT
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的肽"
<400> 33
Cys Arg Ala Pro Val Arg Ser Arg Val Arg Thr Phe Leu Ser
1 5 10
<210> 34
<211> 21
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 34
ggccgcgccc gatcatgatg c 21
<210> 35
<211> 120
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 35
agcgcgcatt cggccccgga cgaaggtact cgcagcactt ggagcgcaga accggccgcg 60
cccgatcatg atgctgccgc caccgccgcc accacggagc gagaagccca gatagacgcc 120
<210> 36
<211> 75
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 36
gcgcgcattc ggccccggac gaaggtactc gcagcacttg gagcgcagaa ccggccgcgc 60
ccgatcatga tgctg 75
<210> 37
<211> 75
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 37
cgcattcggc cccggacgaa ggtactcgca gcacttggag cgcagaaccg gccgcgcccg 60
atcatgatgc tgccg 75
<210> 38
<211> 75
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 38
aggtactcgc agcacttgga gcgcagaacc ggccgcgccc gatcatgatg ctgccgccac 60
cgccgccacc acgga 75
<210> 39
<211> 75
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 39
acttggagcg cagaaccggc cgcgcccgat catgatgctg ccgccaccgc cgccaccacg 60
gagcgagaag cccag 75
<210> 40
<211> 75
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 40
agcgcagaac cggccgcgcc cgatcatgat gctgccgcca ccgccgccac cacggagcga 60
gaagcccaga tagac 75
<210> 41
<211> 69
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 41
ccggccgcgc ccgatcatga tgctgccgcc accgccgcca ccacggagcg agaagcccag 60
atagacgcc 69
<210> 42
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 42
acttattaat ctacccctgt 20
<210> 43
<211> 116
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 43
gatccacatc tggcctgtgt tgcttatgaa cgtggccaat gtgatctgga acttattaat 60
ctacccctgt ccagcagcca cctgaatgtg tacagcagcg acccccaggt cacagc 116
<210> 44
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 44
ccacatctgg cctgtgttgc ttatgaacgt ggccaatgtg atctggaact tattaatcta 60
cccctgtcca gcagcc 76
<210> 45
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 45
tggcctgtgt tgcttatgaa cgtggccaat gtgatctgga acttattaat ctacccctgt 60
ccagcagcca cctgaa 76
<210> 46
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 46
ttatgaacgt ggccaatgtg atctggaact tattaatcta cccctgtcca gcagccacct 60
gaatgtgtac agcagc 76
<210> 47
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 47
gtggccaatg tgatctggaa cttattaatc tacccctgtc cagcagccac ctgaatgtgt 60
acagcagcga ccccca 76
<210> 48
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 48
aatgtgatct ggaacttatt aatctacccc tgtccagcag ccacctgaat gtgtacagca 60
gcgaccccca ggtcac 76
<210> 49
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 49
atgtgatctg gaacttatta atctacccct gtccagcagc cacctgaatg tgtacagcag 60
cgacccccag gtcaca 76
<210> 50
<211> 20
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 50
gatgactcag gtgcagaccc 20
<210> 51
<211> 114
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的多核苷酸"
<400> 51
ctgctgaccg aggcccccct gaaccccaag gccaacagag agaagatgac tcaggtgcag 60
acccacctcg aaaaccccac caagtaccac atacagcaag cccaacggca gcag 114
<210> 52
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 52
tgctgaccga ggcccccctg aaccccaagg ccaacagaga gaagatgact caggtgcaga 60
cccacctcga aaaccc 76
<210> 53
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 53
cgaggccccc ctgaacccca aggccaacag agagaagatg actcaggtgc agacccacct 60
cgaaaacccc accaag 76
<210> 54
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 54
ctgaacccca aggccaacag agagaagatg actcaggtgc agacccacct cgaaaacccc 60
accaagtacc acatac 76
<210> 55
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 55
cccaaggcca acagagagaa gatgactcag gtgcagaccc acctcgaaaa ccccaccaag 60
taccacatac agcaag 76
<210> 56
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 56
aggccaacag agagaagatg actcaggtgc agacccacct cgaaaacccc accaagtacc 60
acatacagca agccca 76
<210> 57
<211> 76
<212> DNA
<213> 人工序列
<220>
<221> 来源
<223> /注解="人工序列的说明: 合成的寡核苷酸"
<400> 57
agagagaaga tgactcaggt gcagacccac ctcgaaaacc ccaccaagta ccacatacag 60
caagcccaac ggcagc 76
- 还没有人留言评论。精彩留言会获得点赞!