木糖甙木糖醇寡聚物的生产方法与流程
本申请要求2014年6月24日提交的美国临时申请第62/016,555号的权益,其全部内容通过引用并入本文。
提交ASCII文本文件的序列表
下列以ASCII文本文件提交的内容通过引用全部并入本文:序列表的计算机可读形式(CRF)(文件名称:416272008040SEQLIST.TXT,记录日期:2015年6月18日,大小:25KB)。
发明领域
本公开总体来说涉及木糖甙-木糖醇寡聚物(xylosyl-xylitol oligomer)的生产,更具体地涉及在宿主细胞中利用生物法生产木糖甙-木糖醇寡聚物。
背景技术:
源自植物细胞壁的木质纤维生物质是用于生产生物能源和可再生化合物最丰富的原材料。半纤维素约占木质纤维生物质总重量的30%。半纤维素的主要形式是杂木聚糖(heteroxylan),其含有1,4-连接的β-D-木糖聚合物的主链和各种装饰的阿拉伯糖、阿魏酸、半乳糖、葡糖醛酸和乙酰酯。与纤维素相比,在预处理过程中,半纤维素组分容易解聚成短的寡聚物释放到液相中。将释放出来的半纤维素组分转化成燃料或其它高附加值的化合物,对构建经济的生物质转化过程是非常有意义的。
据估计,在健康成人中微生物的数量比人类细胞多十倍(Savage et al.,1977)。人们日益认识到,微生物菌群和人体器官之间的共生对健康十分重要。源自半纤维素的低聚木糖(Xylooligosaccharides)和木糖醇,分别对肠道菌群和口腔菌群大有卑益。
低聚木糖(XOS,也称为木寡糖、木糖糊精)是在竹笋、水果、蔬菜、牛奶和蜂蜜中发现的天然存在的低聚糖(Vazquez et al.,2000)。工业上,可以通过水热法或酶水解法处理价格低廉的木质纤维素原料进行大规模生产XOS(Aachary et al.,2011)。XOS的广泛应用已被证实,包括作为功能性食品、预防和治疗胃肠道感染、鱼类和家禽的动物饲料、农业增产和催熟剂、治疗骨质疏松、皮肤瘙痒症症、耳炎、皮肤和头发失调等药物的活性剂。在当前市场上,XOS最重要的应用是作为益生元或调配的合生元等功能性食品的配料(Schrezenmeir et al.,2001)。益生元(Prebiotics)是“一种不可被消化的食品成分,它通过选择性的刺激一种或少数几种结肠中的细菌生长和/或活性,对宿主产生有益的影响,从而改善宿主健康”(Gibson et al.,1995)。XOS已证实能够在体外(Okazaki et al.,1990)和体内(Hsu et al.,2004)促进有益菌青春双歧杆菌(Bifidobacterium adolescentis)的生长。据估计,到2018年益生元的市场将达到48亿美元。目前XOS最大的生产商(山东龙力生物科技股份有限公司)年产量达1万吨。
木糖醇是另一种源自半纤维素、对人体健康有益的化合物。对于许多细菌和酵母来说,摄入无法利用的木糖醇将会妨碍己糖的利用,这有助于人体重建一个健康的微生物菌群。木糖醇已经用于预防中耳炎(Azarpazhooh et al.,2011)和蛀牙(Maguire et al.,2003; et al.,1992)。此外,木糖醇与蔗糖具有相似的甜度,但其卡路里含量却比蔗糖少33%(Hyvonen et al.,1982),已被广泛用于甜味剂的替代品。虽然工业上主要是通过木糖的化学氢化来生产木糖醇,但是由于转化成本低,微生物发酵法在新建的工厂中变得更加普遍。根据2007年的一份报告,木糖醇的年产量在2万至4万吨( et al.,2007)。因此,改进低聚木糖及其相关化合物如含有木糖醇组分的低聚木糖的生产方法非常有必要。
发明简述
一种生产木糖甙-木糖醇寡聚物(xylosyl-xylitol oligomer)的方法,所述方法包括:a)提供宿主细胞,其包含一种重组的木寡糖转运蛋白多肽和一种包含木寡糖还原酶活性的重组木糖还原酶多肽,其中所述木寡糖转运蛋白多肽可将木寡糖转运到宿主细胞中;和b)在含有木寡糖的培养基中培养所述宿主细胞,所述的宿主细胞能利用木寡糖生产木糖甙-木糖醇寡聚物。在一些实施方案中,宿主细胞是一种真菌宿主细胞。在一些实施方案中,宿主细胞是酿酒酵母(Saccharomyces cerevisiae)。在一些可能与上述任一个实施方案相结合的实施方案中,重组木寡糖转运蛋白多肽包含与来自粗糙脉孢霉(Neurospora crassa)的CDT-2具有至少80%氨基酸一致性的氨基酸序列。在一些可能与上述任一个实施方案相结合的实施方案中,重组木糖还原酶多肽包含与来自树干毕赤酵母(Scheffersomyces stipitis)的XYL1具有至少80%氨基酸一致性的氨基酸序列。在一些可能与上述任一个实施方案相结合的实施方案中,一种或多种的木寡糖是指木二糖或木三糖。在一些可能与上述任一个实施方案相结合的实施方案中,至少一种木糖甙-木糖醇寡聚物是木糖甙-木糖醇或木二糖甙-木糖醇。在一些可能与上述任一个实施方案相结合的实施方案中,与相应的对照细胞相比,在宿主细胞中一种或多种β-木糖苷酶多肽的木糖甙-木糖醇水解酶活性降低。在一些实施方案中,与相应的对照细胞相比,一种或多种β-木糖苷酶多肽的活性降低是由于一种或多种多肽的丰度降低。在一些实施方案中,一种或多种β-木糖苷酶多肽的丰度由于一种或多种多肽的表达降低而降低。在一些实施方案中,一种或多种β-木糖苷酶多肽的表达由于一种突变而降低。在一些可能与上述任一个实施方案相结合的实施方案中,与相应的对照细胞相比,宿主细胞中一种或多种木糖醇脱氢酶多肽的活性降低。在一些实施方案中,一种或多种木糖醇脱氢酶多肽的丰度由于一种或多种多肽的表达降低而降低。在一些实施方案中,一种或多种木糖醇脱氢酶多肽的表达由于一种突变而降低。在一些可能与上述任一个实施方案相结合的实施方案中,所述的方法还包括在b)之后从培养基中初步纯化木糖甙-木糖醇寡聚物的方法。
另一方面,本公开涉及一种组合物,包括用前述方法纯化得到的木糖甙-木糖醇寡聚物。在一些实施方案中,组合物至少包括一种从木糖甙-木糖醇和木二糖甙-木糖醇中初步纯化的木糖甙-木糖醇寡聚物。
另一方面,本公开涉及一种生产木糖甙-木糖醇寡聚物的方法,所述方法包括:a)提供宿主细胞,其包含一种木寡糖转运蛋白多肽,其中所述木寡糖转运蛋白多肽将木寡糖转运到宿主细胞中和一种包含木寡糖还原酶活性的木糖还原酶多肽,其中与相应的对照细胞相比,在宿主细胞中一种或多种β-木糖苷酶多肽的木糖甙-木糖醇水解酶活性降低;和b)在含有木寡糖的培养基中培养所述宿主细胞,其中宿主细胞能利用木寡糖生产木糖甙-木糖醇寡聚物。在一些实施方案中,宿主细胞是真菌宿主细胞。在一些实施方案中,宿主细胞是粗糙脉孢霉(Neurospora crassa)。在一些可能与上述任一个实施方案相结合的实施方案中,与相应的对照细胞相比,所述一种或多种β-木糖苷酶多肽的活性由于所述一种或多种多肽的丰度降低而降低。在一些实施方案中,一种或多种β-木糖苷酶多肽的丰度由于所述一种或多种多肽的表达降低而降低。在一些实施方案中,一种或多种β-木糖苷酶多肽的表达由于一种突变而降低。在一些可能与上述任一个实施方案相结合的实施方案中,与相应的对照细胞相比,所述宿主细胞中一种或多种木糖醇脱氢酶多肽的活性降低。在一些实施方案中,一种或多种木糖醇脱氢酶多肽的丰度由于所述一种或多种多肽的表达降低而降低。在一些实施方案中,一种或多种木糖醇脱氢酶多肽的表达由于一种突变而降低。在一些可能与上述任一个实施方案相结合的实施方案中,一种或多种的木寡糖是木二糖或木三糖。在一些可能与上述任一个实施方案相结合的实施方案中,至少一种木糖甙-木糖醇寡聚物是木糖甙-木糖醇或木二糖甙-木糖醇。在一些可能与上述任一个实施方案相结合的实施方案中,所述的方法还包括在b)之后从培养基中初步纯化木糖甙-木糖醇寡聚物的方法。
另一方面,本公开涉及一种组合物,包括用前述方法纯化得到的木糖甙-木糖醇寡聚物。在一些实施方案中,组合物至少包括一种从木糖甙-木糖醇和木二糖甙-木糖醇中初步纯化的木糖甙-木糖醇寡聚物。
附图说明
本专利或申请文件包含至少一张彩色附图。具有彩色附图的本专利或申请公开的专利副本由专利局根据请求并支付必要费用后提供。
图1说明了表达粗糙脉孢霉转运蛋白的酿酒酵母将木寡糖转运到细胞质中。每种纯化组分的木寡糖起始浓度为100μM。培养基中残余的木糖(X1)和木寡糖是用带有空质粒的酿酒酵母(vector)或单独表达CDT-1或CDT-2转运蛋白的酿酒酵母来表示。所用的木寡糖所括木二糖(X2)、木三糖(X3)、木四糖(X4)和木五糖(X5)。
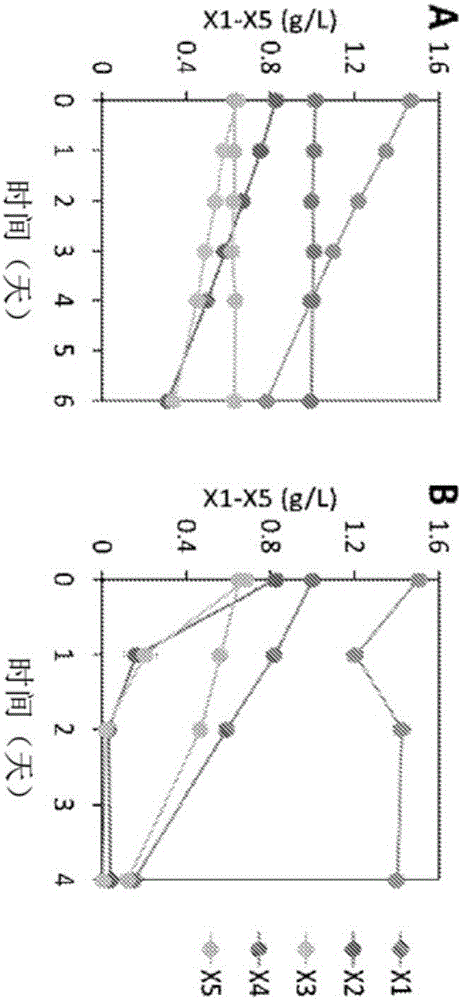
图2A-2B说明了通过改造的酿酒酵母消耗木寡糖。图2A显示了以木糖和木寡糖为碳源,在好氧条件下,以OD600=1的起始细胞密度接种表达了XR/XDH木糖消耗途径(CDT-2和GH43-2)的酿酒酵母培养物。图2B显示了图2A的培养条件,只是起始细胞密度OD600=20。两张图中,显示了在不同时间段后培养液的木糖(X1)和具有较高聚合度(DP)的木寡糖(X2-X5)的浓度。
图3A-3D说明了酿酒酵母中木寡糖消耗途径的组分。图3A显示了菌株WT D452-2在起始细胞密度OD600=1时,在不同时间段后培养液中残糖浓度。图3B显示了具有树干毕赤酵母木糖利用途径(质粒pLNL78)的菌株D452-2在起始细胞密度OD600=1时,在不同时间段后培养液中残糖浓度。图3C显示了菌株WT D452-2在起始细胞密度OD600=20时,在不同时间段后培养液中残糖浓度。图3D显示了具有树干毕赤酵母木糖利用途径(质粒pLNL78)的菌株D452-2在起始细胞密度OD600=20时,在不同时间段后培养液中残糖浓度。在所有图中,显示了木糖(X1)和高级DP的木糖糊精(X2-X5)。
图4A-4B说明了木糖甙-木糖醇的液相色谱-质谱(LC-MS)和液相色谱-质谱/质谱(LC-MS/MS)谱图。图4A显示了木糖甙-木糖醇的LC-MS和LC-MS/MS谱图(结构已示)。高分辨率的MS谱图说明了负离子模式的m/z(质荷比)。分别以0.32和0.33ppm的精确度测定去质子化和甲酸盐加合物离子。下图中的MS/MS谱图显示与预测片段匹配的产物离子。母离子[xylosyl-xylitol-H]-用黑色的菱形标记。图4B显示了木二糖甙-木糖醇的LC-MS和LC-MS/MS谱图(结构已示)。分别以0.51和0.37ppm的精确度测定去质子化和甲酸盐加合物离子。下图中的MS/MS谱图显示与预测片段匹配的产物离子。母离子[xylosyl-xylosyl-xylitol-H]-用黑色的菱形标记。
图5说明了用共培养酵母菌株的木寡糖代谢来鉴定生产木糖甙-木糖醇所需要的酶的来源。将起始细胞密度OD600=1.0的木糖利用菌株(SR8)和起始细胞密度OD600=20木寡糖水解菌株(含有表达CDT-2和FH43-2基因的pXD8.4质粒的D452-2)在含有2%木寡糖的培养基上共培养。
图6A-6B说明了在体外用来自粗糙脉孢霉的木糖还原酶XRY-1生产木糖甙-木糖醇寡聚物。图6A显示了在加入XYR-1之前反应液中木寡糖X1-X4的含量。图6B显示了反应之后木寡糖和木糖甙-木糖醇寡聚物的浓度。木糖、聚合度2-4的木寡糖以及它们还原的产物木糖甙-木糖寡聚物分别标记为X1-X4(木寡糖)和xlt1-xlt4(木糖甙-木糖寡聚物)。
图7说明了粗糙脉孢霉Δcdt-2菌株在木聚糖培养上清液中的木寡糖含量。在CarboPac PA200柱上通过HPAEC分析25μL 1:200稀释的粗糙脉孢霉木聚糖培养上清液。在野生型菌株(品红色线)培养上清液中没有检测到可溶性的糖,而在Δcdt-2菌株(蓝色线)培养上清液中存在高浓度的未修饰和修饰的木寡糖。此外,还发现少量的木糖,意味着木糖是通过不同转运蛋白转运的。
图8A-8B说明了GH43-2和GH43-7β-木糖苷酶对各种木寡糖或木糖甙-木糖醇底物的活性。图8A显示了GH43-7对木糖甙-木糖醇的水解。将纯化的GH43-2或GH43-7与木糖甙-木糖醇和木二糖孵育通过离子排阻HPLC测量所得产物的浓度(mM)。图8B显示了如图8A所示的β-木糖苷酶对木糖甙-木糖醇的水解产物色谱图。酶促反应的产物通过离子排阻HPLC进行解析。使用峰面积对反应结束后的底物和产物浓度进行定量。
图9A-9H说明了各种工程化酵母生长和代谢情况。图9A显示了好氧条件下酵母细胞浓度OD600=1,以木寡糖为唯一碳源的生长曲线。图中是显示了无质粒的SR8或转化了表达CDT-2和GH43-2的质粒(pXD8.4)的酵母的生长。图9B显示了微好氧条件下带有pXD8.4的酵母在起始细胞浓度OD600=20,以木寡糖为唯一碳源的情况下木二糖的消耗。图9C显示了微好氧条件下带有pXD8.6的酵母在起始细胞浓度OD600=20,以木寡糖为唯一碳源的情况下,木二糖消耗。图9D显示了在微好氧条件下带有pXD8.7的酵母在起始细胞浓度为OD600=20,以木寡糖为唯一碳源的情况下木二糖的消耗。图9E显示了微好氧条件下带有pXD8.4的酵母在起始细胞浓度OD600=80,以木寡糖为唯一碳源的情况下木二糖的消耗情况。图9F显示了微好氧条件下带有pXD8.6的酵母在起始细胞浓度OD600=80,以木寡糖为唯一碳源的情况下木二糖的消耗情况。图9G显示了微好氧条件下带有pXD8.7的酵母在起始细胞浓度OD600=80,以木寡糖为唯一碳源的情况下木二糖的消耗情况。图9H显示了微好氧条件下带有pXD8.4、pXD8.6或pXD8.7的酵母在起始细胞浓度OD600=80,以木寡糖为唯一碳源的情况下乙醇的生产。所有的生长实验都进行三次生物学重复实验,误差线表示实验间的标准差。
图10说明了酵母生长过程中碳水化合物及其衍生物的分布。酵母以木寡糖为唯一碳源,在好氧条件下起始细胞密度OD600=20或在微好氧条件下起始细胞密度OD600=80的生长。图上显示了转化了表达CDT-2和GH43-2的pXD8.4质粒、表达CDT-2和GH43-7的pXD8.6质粒或者表达CDT-2、GH43-2和GH43-7的pXD8.7质粒的酵母SR8菌株。所有的生长实验都进行三次生物学重复实验,误差线表示实验间的标准差。
图11说明了GH43-2(NCU01900)和GH43-7(NCU09652)家族中β-木糖苷酶的系统发生分布。用于构建系统发生树的序列的NCBI GI编号在物种名称旁边指示。分支上显示的是进行1000次拔靴法重复取样计算获得的支持率。比例尺显示了每个氨基酸残基有0.2个取代。右边显示了使用Phyre2服务器对GH43-2和GH43-7进行的同源建模的模型。GH43-7与GH43-2相比,虽然都具有十分相似的催化结构域(绿色),但GH43-7具有一个额外的C-末端辅助结构域(品红色),这可能导致它对木糖甙-木糖醇底物的选择性。
图12说明了在酿酒酵母中重建粗糙脉孢霉的两条寡糖消耗途径。细胞内纤维二糖的利用需要CDT-1或CDT-2与β-葡萄糖苷酶一起(Galazka et al.,2010),经过己糖激酶(HXK)的磷酸化后进入糖酵解途径形成葡萄糖-6-磷酸(Glc-6-P)。细胞内木寡糖的利用同样也需要CDT-2与细胞内的GH43-2和GH43-7β-葡萄糖苷酶。所得的木糖可以通过含有木糖/木寡糖还原酶(XR)、木糖醇脱氢酶(XDH)和木酮糖激酶(XK)的磷酸戊糖途径同化。
图13A-13B说明了酵母转化木寡糖生成木糖甙-木糖醇(xlt2)和木二糖甙-木糖醇(xlt3)。图13A显示了工程改造的(木糖甙)n-木糖醇生产途径。木寡糖通过木寡糖转运蛋白进入细胞,木糖还原酶将木寡糖转化为木糖甙-木糖醇寡聚物。图13B显示了工程改造的酿酒酵母其(木糖甙)n-木糖醇发酵曲线。
图14说明了通过工程改造的酿酒酵母发酵生产高级的木糖甙-木糖醇寡聚物。
图15说明了芽孢杆菌(BsGH43-7)和大肠杆菌(EcGH43-7)两种细菌GH43-7在木二糖底物(X2)或木糖甙-木糖醇底物(xlt2)的酶活(μM/min)。
图16说明了木糖、XOS(低聚木糖)、木糖醇和(木糖甙)n-木糖醇的结构。R1和R2代表一个羟基基团或β-1,4-连接的木糖单体。
图17说明了木二糖与假丝酵母木糖还原酶(CtXR)的计算机对接模型。木二糖是黄色,NADH辅因子是品红色,蛋白的二级结构是深绿色,活性位点的残基是亮绿色并显示侧链。部分CtXR表面显示为描绘活性位点口袋的形状。黑色的虚线显示的是CtXR和木二糖非还原末端残之间预测的氢键。
图18A显示了来源于植物细胞壁的两个寡糖组分。来自纤维素的纤维糊精是葡萄糖的主要来源。来自半纤维素的木寡糖是木糖的主要来源。6-甲氧基(蓝色)是区分葡萄糖衍生物和木糖的基团。R1、R2=H、纤维二糖或木二糖;R1=高度聚合的纤维糊精中β-1,4-连接的葡萄糖单体;R2=高度聚合的木寡糖中β-1,4-连接的木糖单体。图18B显示了在不同碳源生长的粗糙脉孢霉中表达的转运蛋白的转录水平。转录水平用发表在(Coradetti et al.,2012;Sun et al.,2012)上的FPKM表示,即每1百万个reads中map到外显子上的每1K个碱基上的片段数来表示。*CBT-1转运纤维二糖,它是溶解性多糖单加氧酶(LPMOs或CaZy家族AA9和AA10)的反应产物(Xiong et al.,2014)。
图19说明了不同碳源培养基中粗糙脉孢霉的生长情况。野生型(WT)粗糙脉孢霉,或敲除了转运蛋白cdt-1(Δcdt-1)或cdt-2(Δcdt-2)的粗糙脉孢霉在含有奇岗(Miscanthus giganteus)植物细胞壁,或纯化的植物细胞壁的培养基上生长。Avicel是一种源自植物细胞壁的纤维素。黑框表示Δcdt-2菌株在木聚糖培养基上生长迟缓的表型。
图20A-20B说明了所预测的β-木糖苷酶GH43-2的木二糖酶活性。图20A显示了GH43-2对聚合度至少为2-8(X2-X8)的木寡糖的水解。为了更清楚地对比说明,将30min后的色谱图进行了偏移。图20B显示了通过测定木二糖到木糖的水解程度优化GH43-2的最适pH值。图上显示木糖的HPAEC色谱峰面积变化。
图21说明了预测的丝状真菌胞内β-木糖苷酶GH43-2的系统发生分布。通过与NCBI蛋白数据库的Blast查询(Altschul et al.,1997),确认GH43-2(NCU01900)的同源蛋白。选择来自多样化分类的、具有代表性的序列用MUSCLE(Edgar 2004)算法进行比对。使用MEGA v6.05(Tamura et al.,2013)软件,根据Jones-Taylor-Thornton比对模型构建了最大可能性进化树。木聚糖诱导的胞外GH43-3(NCU05965)为外类群。用于构建系统发生树的序列的NCBI GI编号在物种名称旁边指示。分支上显示的是进行1000次拔靴法重复取样计算获得的支持率。比例尺显示了每个氨基酸残基有0.2个取代。
图22说明了表达CDT-2和GH43-7(质粒pXD8.4)的SR8U菌株培养基上清液的碳水化合物情况,这些化合物通过HPAEC分离,其缩写如下:X1,木糖;X2,木二糖;X3,木三糖;X4,木四糖;xlt,木糖醇;xlt2,木糖甙-木糖醇;xlt3,木二糖甙-木糖醇。
图23说明了GH43-7的系统进化。脉孢霉GH43-2作为外类群。分支上显示的是进行1000次拔靴法重复取样计算获得的支持率。比例尺显示了每个氨基酸残基有0.2个取代。用于构建系统发生树的序列的NCBI GI编号在物种名称旁边指示。
图24A-24B说明了通过一系列微生物生产的木糖甙-木糖醇和木二糖甙-木糖醇。图24A显示色谱图上粗糙脉孢霉、里氏木霉、构巢曲霉和芽孢杆菌胞内代谢物中木寡糖衍生的碳水化合物水平。化合物的缩写如下:X1,木糖;X2,木二糖;X3,木三糖;X4,木四糖;xlt,木糖醇;xlt2,木糖甙-木糖醇;xlt3,木二糖甙-木糖醇。图24B显示了在本文所描述的实验中,在木寡糖培养上能生产木糖甙-木糖醇的微生物系统进化树。年代数据来自(Wellman et al.,Osterloff,and Mohiuddin 2003;Galagan et al.,2005;Hedges,Dudley,and Kumar 2006)。
图25说明了用液体色谱-质谱/质谱(LC-MS/MS)检测生长在木寡糖培养基上的微生物的培养基上清液中木糖甙-木糖醇含量的色谱图。图上所示的是,对生长在木寡糖培养基上的脉孢霉、里氏木霉、构巢曲霉和芽孢杆菌胞内代谢物用液相色谱分离后,用MS/MS对木糖甙-木糖醇的转变(红色,m/z 283.1035→151.0612转变)和木二糖甙-木糖醇的转变(绿色,m/z 415.1457→151.0612转变)进行分析的结果。
图26说明了酵母生长在木寡糖培养基上碳水化合物及其衍生物分布。酵母在好氧条件下细胞密度OD600=20,以木寡糖为唯一碳源(g/L)。酵母SR8菌株分别转化了表达CDT-2和GH43-2的质粒(pXD8.4)、表达CDT-2和GH43-7的质粒(pXD8.6)或者表达CDT-2、GH43-2和GH43-7的质粒(pXD8.7)。所有的生长实验都进行三次生物学重复实验,误差线表示实验间的标准差。
图27A-27B说明了木寡糖与木糖或葡萄糖的共厌氧发酵。图27A显示了在分批补料反应器中进行的木寡糖与木糖的厌氧发酵。表达CDT-2、GH43-2和GH43-7(质粒pXD8.7)的SR8U菌株起始OD600为20。实线代表培养基中化合物的浓度。蓝色虚线显示了随时间推移添加到培养物中的木糖的总量。误差线代表生物学重复实验的标准差。图27B显示了在分批补料反应器中进行的木寡糖与葡萄糖的厌氧发酵。在发酵液中未能检测到葡萄糖。误差线代表生物学重复实验的标准差。
图28说明了在木糖存在时,厌氧条件下木寡糖的利用。带有完整木寡糖途径(CDT-2,GH43-2,GH43-7,XR/XDH)的菌株厌氧条件下在含有4%木糖和3%木寡糖的oMM培养基上(Lin et al.,2014)生长。木糖耗尽后木二糖(X2)和木三糖(X3)的消耗停止,在48小时补充木糖后又继续消耗。
图29A-29B说明了厌氧控制条件下,表达完整木寡糖途径的酿酒酵母发酵情况。使用的是带有表达CDT-2、GH43-2和GH43-7质粒pXD8.7的菌株SR8,起始OD600为20。实线代表培养基中化合物的浓度。蓝色虚线显示了随时间推移添加到培养物中的木糖的总量。图29A显示了反应器中,菌株在含有4%木寡糖的oMM培养基中,不补加木糖的发酵曲线。图29B显示了反应器中,菌株在含有4%木寡糖的oMM培养基中,持续补加木糖的发酵曲线。
图30说明了XI和XR/XDH途径发酵性能的比较。图上显示了使用菌株SR8(XR/XDH途径)和SXA-R2P-E(XI途径),在不同细胞起始接种浓度条件下木糖的发酵情况。所用的发酵培养基为oMM。纵坐标,g/L;横坐标,时间(小时)。
图31说明了使用对数生长期的培养物为种子,菌株SR8(XR/XDH)和SXA-R2P-E(XI)木糖发酵性能。菌株和起始OD600都标记在每张图上方。所用的发酵培养基为oMM。纵坐标,g/L;横坐标,时间(小时)。
图32说明了使用oMM或YSC培养基,菌株SR8(XR/XDH)和SXA-R2P-E(XI)木糖发酵性能。培养基、菌株和起始OD600都标记在每张图上方。纵坐标,g/L;横坐标,时间(小时)。
图33A-33B木寡糖和蔗糖共发酵情况。图33A是蔗糖的发酵。纵坐标,g/L;横坐标,时间(小时)。图33B是使用带有表达CDT-2、GH43-2和GH43-7(质粒pXD8.7)的SR8U菌株进行木寡糖和蔗糖的分批共发酵。纵坐标,g/L;横坐标,时间(小时)。以10g/L提供包含木二糖(4.2g/L)和木三糖(2.3g/L)的木寡糖。在该实验时间内未能发酵的,除半纤维素改性剂如乙酸酯之外,木寡糖样品还包括木四糖和木五糖。
发明详述
以下提供的描述能够让本领域普通技术人员制作和使用各种实施方案。所描述的具体设备、技术和应用仅作为示例提供。对本文描述的示例的各种修改对于本领域普通技术人员将是显而易见的,并且在不脱离各种实施例的精神和范围的情况下,本文定义的一般原理可以应用于其他示例和应用。因此,各种实施例并不旨在限于本文所描述和示出的示例,而是符合与权利要求一致的范围。
本公开总体来说涉及木糖甙-木糖醇寡聚物(xylosyl-xylitol oligomer)的生产,更具体地涉及在宿主细胞中利用生物法生产木糖甙-木糖醇寡聚物。
本公开是基于,或者至少部分基于申请人发现含有木寡糖转运蛋白和木糖还原酶的重组酿酒酵母能够生产和积累木糖甙-木糖醇寡聚物。申请人的发现是特别令人惊讶的。因为最初认为,该重组菌株可以通过代谢低聚木糖为相应的单体,如木糖和木糖醇,从而利用木寡糖为能源。相反,申请人发现在这些酵母细胞内的木糖还原酶可将木寡糖转化为木糖甙-木糖醇寡聚物,而不会进一步将这些化合物代谢为相应的单体。由于木糖甙-木糖醇寡聚物具有许多潜在的有益用途,比如作为甜味剂,申请人已经证明建立了利用活细胞生物法生产木糖甙-木糖醇寡聚物的生物平台。
因此,申请人在此提供在宿主细胞中生产木糖甙-木糖醇寡聚物的方法。同时还提供适合生产木糖甙-木糖醇寡聚物的宿主细胞。将本公开的宿主细胞培养在含有木寡糖或木寡糖来源的生长培养基上,可以生产木糖甙-木糖醇寡聚物。
本公开的多肽
本公开涉及使用宿主细胞生产木糖甙-木糖醇寡聚物。本公开的宿主细胞具有生产和/或积累木糖甙-木糖醇寡聚物的特征。在一些实施方案中,本公开的宿主细胞含有促进木糖甙-木糖醇寡聚物产生的多肽。在一些实施方案中,本公开的宿主细胞具有降低的蛋白质活性,否则其将起作用阻碍木糖甙-木糖醇寡聚物生产和/或积累。
如本文所用,“多肽”是包括多个连续聚合的氨基酸残基(例如,至少约15个连续的聚合氨基酸残基)的氨基酸序列。如本文所用,“多肽”是指氨基酸序列,寡肽,肽,蛋白质或其部分,术语“多肽”和“蛋白质”可互换使用。
木寡糖转运蛋白多肽
本公开的宿主细胞含有一种木寡糖转运蛋白多肽。木寡糖转运蛋白多肽通常是指能够将木寡糖转运穿过细胞膜的任何一种糖转运蛋白。如本文所用,“木寡糖”,是指各种长度的木糖聚合物,例如,包括木二糖(木糖二聚体)、木三糖(木糖三聚体)、木四糖(木糖四聚体)、木五糖(木糖五聚体)、木六糖(木糖六聚体)等。在一些实施方案中,本公开的木寡糖转运蛋白多肽,可以具有一种或多种的木二糖转运活性、木三糖转运活性、木四糖转运活性、木五糖转运活性、木六糖转运活性等。“木寡糖”和“低聚木糖”在本文中可互换使用。
各种木寡糖转运蛋白在本领域是已知,并且可以用于本公开的方法中。此外,还可以用本领域熟知的任何合适的方法,例如本文公开的那些,筛选假定的木寡糖转运蛋白多肽的活性。
例如,适宜的木寡糖转运蛋白可以包括主要协同转运蛋白超家族的糖转运蛋白家族成员,例如NCU08114。主要协同转运蛋白超家族(MFS,转运蛋白分类#2.A.1)的转运蛋白成员,大多包含12个跨膜的α-螺旋、在胞内的N-末端和C-末端(S.S.Pao,I.T.Paulsen,M.H.Saier,Jr.,Microbiol Mol Biol Rev 62,1.Mar.1998)。尽管MFS转运蛋白的一级结构差异很大,但都认为它们与大肠杆菌乳糖通透酶(LacY)(J.Abramson et al.,Science 301,610,Aug.2003)和Pi/3-磷酸甘油转运蛋白(GlpT)(Y.Huang,M.J.Lemieux,J.Song,M.Auer,D.N.Wang,Science 301,616,Aug.2003)具有共同的三级结构。在这些实施例中,6个N-和C-末端螺旋形成通过螺旋6和7之间的长胞质环连接的两个不同的结构域。这种对称结构与被认为产生MFS的复制事件相对应。底物与N-末端结构域的第1、2、4和5螺旋以及C-末端结构域的第7、8、10和11螺旋形成的亲水空腔结合。该空腔通过第3、6、9和12螺旋稳定。在某些实施方案中,本公开的木寡糖转运蛋白可以包含12个跨膜的α-螺旋,1个位于第6和第7α-螺旋的环,并且具有位于胞内的N-和C-末端。
具有木寡糖转运活性的MFS糖转运蛋白被一种或多种、两种或多种、三种或多种、四种或多种、五种或多种或六种下列的PROSITE模体所鉴定:R-x-L-Y-T-G-Y-D-G(SEQ ID NO:7);G-F-G-N-S-x(2)-Q(SEQ ID NO:8);L-L-x(1,30)-H-P-Q-H-R(SEQ ID NO:9);E-[FY]-x-E-I-[RK]-[DE]-T(SEQ ID NO:10);W-S-G-L(SEQ ID NO:11);和G-P-T-L-E-E(SEQ ID NO:12)。因此,在某些实施方案中,本公开的木寡糖转运蛋白可以包括以下模体:R-x-L-Y-T-G-Y-D-G(SEQ ID NO:7),G-F-G-N-S-x(2)-Q(SEQ ID NO:8),L-L-x(1,30)-H-P-Q-H-R(SEQ ID NO:9),E-[FY]-x-E-I-[RK]-[DE]-T(SEQ ID NO:10),W-S-G-L(SEQ ID NO:11),和/或G-P-T-L-E-E(SEQ ID NO:12)。
在某些实施方案中,连接具有木寡糖转运活性的MFS糖转运蛋白的跨膜螺旋1和跨膜螺旋2的环可以包含R-x-L-Y-T-G-Y-D-G(SEQ ID NO:7)模体。在其它的实施方案中,具有木寡糖转运活性的MFS糖转运蛋白的跨膜螺旋4可以包含G-F-G-N-S-x(2)-Q(SEQ ID NO:8)模体。在进一步的实施方案中,连接具有木寡糖转运活性的MFS糖转运蛋白的跨膜螺旋4和跨膜螺旋5的环可以包含L-L-x(1,30)-H-P-Q-H-R(SEQ ID NO:9)模体。在一些实施方案中,具有木寡糖转运活性的MFS糖转运蛋白的跨膜螺旋6和跨膜螺旋7之间的序列可以包含E-[FY]-x-E-I-[RK]-[DE]-T(SEQ ID NO:10)模体。在其它的实施方案中,具有木寡糖转运活性的MFS糖转运蛋白的跨膜螺旋10可以包含W-S-G-L(SEQ ID NO:11)模体。在进一步的实施方案中,具有木寡糖转运活性的MFS糖转运蛋白的胞内区域可以包含G-P-T-L-E-E(SEQ ID NO:12)模体。
因此,在某些实施方案中,本公开的木寡糖转运蛋白可以是具有木寡糖转运活性的MFS糖转运蛋白。MFS糖转运蛋白含有12个跨膜的α-螺旋(如α-螺旋1、α-螺旋2、α-螺旋3、α-螺旋4、α-螺旋5、α-螺旋6、α-螺旋7、α-螺旋8、α-螺旋9、α-螺旋10、α-螺旋11、α-螺旋12),并且至少含有一种、二种、三种、四种、五种或六种下列的模体:R-x-L-Y-T-G-Y-D-G(SEQ ID NO:7),G-F-G-N-S-x(2)-Q(SEQ ID NO:8),L-L-x(1,30)-H-P-Q-H-R(SEQ ID NO:9),E-[FY]-x-E-I-[RK]-[DE]-T(SEQ ID NO:10),W-S-G-L(SEQ ID NO:11)和G-P-T-L-E-E(SEQ ID NO:12)。此外,至少一种、二种、三种、四种、五种或六种的模体序列可以与其它任何数目的模体组合。例如,在某些实施方案中,本公开的重组的木寡糖转运蛋白可以含有α-螺旋1、α-螺旋2、α-螺旋3、α-螺旋4、α-螺旋5、α-螺旋6、α-螺旋7、α-螺旋8、α-螺旋9、α-螺旋10、α-螺旋11、α-螺旋12,可以含有一种或多种下列的模体:R-x-L-Y-T-G-Y-D-G(SEQ ID NO:7),G-F-G-N-S-x(2)-Q(SEQ ID NO:8),L-L-x(1,30)-H-P-Q-H-R(SEQ ID NO:9),E-[FY]-x-E-I-[RK]-[DE]-T(SEQ ID NO:10),W-S-G-L(SEQ ID NO:11)和G-P-T-L-E-E(SEQ ID NO:12)。
示例性的木寡糖转运蛋白可以包括,如:GI336463171,GI85111305,GI336265400,GI317144741,GI115492463,GI367019682,GI169613751,GI116180882,GI396476342,GI238485660,GI330924560,GI83768202,GI189198948,GI389624607,GI340924204,GI378727300,GI310790351,GI310800345,GI156057085,GI171687197,GI380474010,GI169851388,GI429858279,GI472245111,GI317451470,GI477537114,GI159123739,GI347840412,GI70983456,GI380480795,GI451851885,GI345565206,GI451995308,GI409045374,GI389739875,GI255954533,GI393212245,GI392561917,GI389747805,GI449542817,GI317431879,GI392589230,GI393212288,GI392594130,GI402080963,GI392589229,GI477517948,GI477532537,GI471566562,GI392570497,GI336375423,GI342887383,GI302675837,GI403412898,GI346979623,GI393243817,GI50980922,GI429856489,GI302885243,GI50980920,GI402220822,GI119484188,GI336384853,GI50980814,GI395327133,GI407921652,GI403417250,GI395333241,GI50980916,GI157674373,GI336372105,GI389743154,GI390598972,GI170099628,GI46126507,GI50980918,GI409076933,GI475675010,GI429850133,GI426195279,GI408391526,GI380481283,GI50980914,GI449544581,GI346970758,GI451847000,GI361124441,GI409048565,GI395329450,GI452002698,GI477527980,GI115386538,GI449542872,GI390598040,GI302403881,GI398395223,GI449295152,GI361129867,GI389628216,GI46121949,GI408391130,GI409045849,GI342872438,GI406859506,GI402079797,GI452980716,GI393212779,GI403420153,GI310795966,GI299748721,GI367039407,GI390598898,GI189192086,GI408392461,GI46139961,GI353237496,GI406867787,GI121700448,GI330920776,GI396501117,GI393212304,GI471897438,GI159130218,GI70992963,GI477517573,GI330944461,GI119473893,GI477593444,GI451856188,GI471572208,GI169600747,GI119473383,GI342877339,GI302887372,GI389643896,GI302890289,GI471879149,GI429848762,GI169784774,GI406701640,GI115397337,GI121712210,GI367023388,GI401881295,GI238508000,GI171682982,GI367040823,GI402079610,GI336372104,GI67515463,GI402219043,GI378733150,GI477532665,GI353237498,GI302881468,GI477521642,GI408398452,GI380482112,GI380491182,GI393241491,GI46114604,GI83768524,GI340946132,GI392594043,GI46139085,GI391864453,GI398388786,GI408398213,GI238490288,GI353234628,GI475673276,GI46126477,GI408391512,GI477523847,GI342889863,GI302679092,GI429848658,GI475666583,GI477531560,GI449540330,GI358386965,GI342876695,GI255954603,GI302880307,GI342879157,GI477511570,GI402221884,GI477509075,GI452003196,GI310797035,GI393244761,GI477517292,GI170098821,GI392594044,GI154297060,GI70985968,GI242772225,GI426199525,GI409078516,GI429856851,GI402224495,GI440471626,GI477528319,GI391865969,GI336370919,GI310794698,GI302410075,GI212529706,GI409048830,GI408398456,GI302684805,GI393213085,GI346980084,GI346974165,GI358398653,GI392568136,GI302416575,GI367039713,GI242820800,GI336383675,GI402224782,GI320589783,GI302900235,GI317431855,GI353237497,GI395332594,GI408393532,GI443921814,GI189209525,GI46126625,GI212541650,GI342873777,GI477520190,GI342882753,GI395329777,GI302886316,GI46115532,GI475674542,GI353234629,GI169615919,GI477513409,GI408395147,GI449549369,GI475673984,GI409050432,GI336373956,GI255941274,GI342873441,GI402222263,GI443917241,GI302418580,GI393241388,GI242787981,GI353242496,GI320584092,GI409079438,GI169865238,GI426192620,GI389749836,GI475675183,GI86196491,GI392586908,GI46139335,GI50426457,GI440472215,GI449302102,GI212534032,GI440639149,GI393233992,GI380477434,GI392594946,GI452987659,GI402222514,GI146422137,GI238494754,GI302917301,GI336262574,GI475675239,GI429853891,GI169777579,GI347829750,GI429855681,GI407919583,GI302888323,GI477511581,GI398411145,GI344233577,GI402221284,GI429859607,GI389646653,GI302887434,GI440482625,GI190348766,GI146412796,GI353239284,GI389647635,GI86196013,GI67902250,GI169859598,GI380489737,GI429849376,GI477532141,GI134119066,GI408392325,GI380492851,GI380485843,GI302881155,GI336465735,GI350287441,GI429858924,GI115491233,GI164424491,GI471564797,GI378725745,GI346978042,GI345566254,GI429856885,GI302415056,GI402219282,GI145248423,GI350639100,GI452002162,GI342882290,GI344304410,GI358370507,GI317034943,GI169594826,GI302901784,GI350632927,GI452001921,GI67524313,GI407916978,GI477533791,GI294659451,GI477523640,GI429847594,GI389624925,GI350630030,GI46103327,GI425767943,GI429863729,GI405123292,GI358374365,GI429852368,GI459366747,GI340519452,GI350639301,GI407923780,GI317451460,GI380488960,GI260951447,GI477535671,GI358393072,GI389637768,GI58260536,GI134117103,GI358385446,GI255730163,GI145235529,GI402220490,GI342890350,GI440470256,GI477511506,GI308198145,GI310791878,GI126273966,GI391873356,GI46114596,GI171691520,GI380486599,GI260951445,GI402076217,GI429862052,GI310795192,GI146421205,GI169785253,GI190346119,GI302883799,GI475674623,GI477533270,GI302882471,GI402080455,GI317031224,GI406861400,GI322697407,GI85094247,GI336467806,GI406607550,GI238506229,GI126132458,GI367033975,GI126273939,GI302884874,GI396464906,GI475674549,GI322711180,GI134077570,GI477512227,GI477515015,GI429851103,GI336471923,GI358383783,GI342878508,GI342876702,GI119494457,GI321265766,GI380095555,GI477511576,GI396475175,GI408396504,GI302898747,GI475670215,GI402076475,GI150865436,GI344229190,GI342883283,GI58262550,GI346976696,GI255940738,GI475675360,GI477513860,GI119473356,GI477523296,GI302883821,GI336270802,GI396493052,GI310800862,GI317451430,GI477516994,GI380493642,GI85085012,GI367039241,GI116200915,GI336266077,GI46138341,GI302913423,GI471562806,GI477526206,GI358376032,GI212538607,GI403418185,GI119473409,GI46125945,GI367026193,GI406701892,GI402216469,GI342873809,GI475672478,GI134081460,GI169774135,GI475677330,GI121700434,GI452979822,GI347828193,GI406859968,GI340939620,GI342889050,GI477509642,GI255931303,GI451854957,GI238497377,GI452001121,GI477514839,GI126138322,GI121700751,GI83771606,GI171677314,GI406607553,GI475674291,GI402219269,GI70996500,GI408388106,GI477527586,GI159130224,GI342883062,GI70992951,GI115391487,GI46102882,GI46114928,GI408389965,GI342875112,GI459367863,GI385301207,GI302415575,GI119494209,GI119482834,GI344229911,GI346977093,GI429860850,GI406607552,GI342876303,GI358394391,GI242792392,GI342872176,GI242799333,GI330907139,GI302886675,GI429857088,GI346321540,GI449299447,它们的同源蛋白,和直系同源蛋白。
在一些实施方案中,木寡糖转运蛋白多肽所包含的氨基酸序列与脉孢霉CDT-2的氨基酸序列(NCU08114)(SEQ ID NO:1)具有至少有10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
本公开的木寡糖转运蛋白多肽可以是内源存在细胞中,或者可以是重组表达于本公开的细胞中。在某些实施例中,本公开的重组木寡糖转运蛋白多肽包括,例如,含有突变的木寡糖转运蛋白多肽。这些突变的木寡糖转运蛋白多肽含有一种或多种增加特异性、功能和/或木寡糖转运活性的突变。在一些实施方案中,本公开的重组木寡糖转运蛋白多肽可以具有一种或多种额外的转运蛋白活性,如葡萄糖、纤维素或纤维糊精转运活性。因此,在某些实施方案中,本公开的突变木寡糖转运蛋白多肽优先转运木寡糖。也就是说,本公开的突变木寡糖转运蛋白多肽,与缺少一个或多个突变的相应的木寡糖转运蛋白相比,对木寡糖有更高的特异性。含有一个或多个突变的重组木寡糖转运蛋白可通过突变编码本公开的木寡糖转运蛋白的多核苷酸来产生。在一些实施方案中,本公开的一种突变的木寡糖转运蛋白可以含有一个或多个的突变,其包括,例如,点突变、错义突变、取代突变、移码突变、插入突变、重复突变、扩增突变、易位突变或倒置突变。与相应的不含一个或多个突变的木寡糖转运蛋白相比这些突变导致木寡糖转运蛋白对木寡糖的特异性增强和/或木寡糖转运活性增加。
在一些实施方案中,木寡糖转运蛋白是保持将一种或多种木寡糖分子到细胞内和/或从细胞内转运到细胞外的能力的功能片段。木寡糖转运蛋白多肽的功能片段,可以包括,例如具有至少10个、12个、14个、16个、18个或20个连续的氨基酸的木寡糖转运蛋白多肽。
木糖还原酶多肽
本公开的宿主细胞含有一种木糖还原酶多肽。木糖还原酶多肽通常理解为催化以下反应的酶:木糖+NAD(P)H+H+=木糖醇+NAD(P)+(E.C.1.1.1.21)。然而,申请人已表明,木糖还原酶多肽可以同样也作用于木寡糖底物上,催化下列的一般反应:木寡糖+NAD(P)H+H+=木糖甙-木糖醇寡聚物+NAD(P)+。例如,当底物是木二糖时,本公开的一种木糖还原酶多肽可以催化下列反应:木二糖+NAD(P)H+H+=木糖甙-木糖醇+NAD(P)+。可以作用于木寡糖底物,产生木糖甙-木糖醇寡聚物的木糖还原酶多肽,具有木寡糖还原酶活性。同样地,本公开方法所使用的木糖还原酶多肽具有木寡糖还原酶活性。
本公开的木糖还原酶多肽,所具有的木糖还原酶活性和木寡糖还原酶活性,并不一定是相互排斥的。例如本公开的木糖还原酶多肽可以同时具有木糖还原酶活性和木寡糖还原酶活性。
各种木糖还原酶多肽是本领域已知的,并且可以用于本公开的方法中。此外,还可以用本领域熟知的任何合适的方法,例如,在此公开的那些方法,筛选具有木糖还原酶活性和/或木寡糖还原酶活性的假定的木寡糖转运蛋白多肽。
适宜的木糖还原酶可以从任何能够代谢木糖和/或木寡糖的微生物中获得。这些微生物包括,例如,毕赤酵母、假丝酵母、曲霉和粗糙脉孢霉。这些适宜的木糖还原酶可以包括,例如,来自树干毕赤酵母的木糖还原酶XYL1(GenBank:ADQ89193.1)、来自粗糙脉孢霉的木糖还原酶XYR-1(NCU08384)。其它的木糖还原酶可以包括,例如,AAF86345.1、AAO91803.1、AEY80024.1、AAC25601.1和AAW34373.1。与上述登录号相关的每个序列通过引用并入本文。其它的木糖还原酶为本领域的技术人员公知的,并且可以用于本公开的宿主细胞。
在一些实施方案中,木糖还原酶多肽所包含的氨基酸序列与树干酵母XYL1的氨基酸序列(GenBank:ADQ89193.1)(SEQ ID NO:2)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。在一些实施方案中,木糖还原酶多肽所包含的氨基酸序列与粗糙脉孢霉XYR-1的氨基酸序列(NCU08384)(SEQ ID NO:3)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
本公开的木糖还原酶多肽可以是内源存在细胞中,或者可以是重组表达于本公开的细胞中。在某些实施例中,本公开的重组木糖还原酶多肽包括,例如,含有突变的木糖还原酶多肽。这些突变的木糖还原酶多肽含有一个或多个增加特异性、功能和/或木糖还原酶活性的突变。例如,在一些实施方案中,本公开的突变或内源性的木糖还原酶多肽优先作用于低聚木糖底物。也就是说,本公开的突变木糖还原酶多肽,与缺少一个或多个突变的相应木糖还原酶多肽相比,对木寡糖有更高的特异性。重组的木糖还原酶多肽含有一个或多个突变,可以通过突变编码本公开的木糖还原酶的多核苷酸来产生。在一些实施方案中,本公开的一种突变的木糖还原酶多肽可以含有一个或多个的突变,这些突变包括,例如,点突变、错义突变、取代突变、移码突变、插入突变、重复突变、扩增突变、易位突变或倒置突变。。与相应的不含一个或多个突变的木糖还原酶多肽相比这些突变导致一种木糖还原酶多肽对木寡糖(而不是木糖)的特异性增强。
在一些实施方案中,木糖还原酶多肽是维持催化活性(例如,转化木寡糖底物到木糖甙-木糖醇寡聚物的能力)的功能片段。木糖还原酶多肽的功能片段,可以包括,例如具有至少10个、12个、14个、16个、18个或20个连续的氨基酸的木糖还原酶多肽。
β-木糖苷酶多肽
在一些方面,本公开的宿主细胞与相应的野生型细胞相比,一种或多种β-木糖苷酶多肽的活性降低。β-木糖苷酶多肽通常被理解为β-1,4-D-木聚糖木糖苷水解酶(E.C.3.2.1.37),它可以催化水解木寡糖中D-木糖残基的非还原末端,释放出D-木糖。然而,申请人还已显示β-木糖苷酶可以具有作用于木糖甙-木糖醇寡聚物底物的活性。可以催化水解木糖甙-木糖醇寡聚物的β-木糖苷酶多肽具有木糖甙-木糖醇水解酶活性。因此,本公开方法使用的β-木糖苷酶多肽具有木糖甙-木糖醇水解酶活性。
本公开的β-木糖苷酶多肽,所具有的β-木糖苷酶活性和木糖甙-木糖醇水解酶活性,并不一定是相互排斥的。例如本公开的β-木糖苷酶多肽可以同时具有β-木糖苷酶活性和木糖甙-木糖醇水解酶活性。
各种β-木糖苷酶多肽是本领域已知的。用本公开的方法可以把这些β-木糖苷酶多肽作为降低活性的靶标。本公开所用的具有活性降低的β-木糖苷酶,可以包括,例如,糖苷水解酶GH3家族的成员、糖苷水解酶GH39家族的成员、糖苷水解酶GH43家族的成员、糖苷水解酶GH52家族的成员、糖苷水解酶GH54家族的成员。
可以通过本领域已知的任何合适的方法(包括例如本文公开的那些)筛选蛋白质,例如GH3、GH39GH43、GH52或GH54糖苷水解酶的木糖甙-木糖醇水解酶活性。在一些实施方案中,具有木糖甙-木糖醇水解酶活性的β-木糖苷酶多肽作为降低活性的靶标。
在一些实施方案中,降低活性的β-木糖苷酶所包含的氨基酸序列与粗糙脉孢霉GH43-7的氨基酸序列(NCU09652)(SEQ ID NO:4)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
在一些实施方案中,降低活性的β-木糖苷酶所包含的氨基酸序列与枯草芽孢杆菌β-木糖苷酶的氨基酸序列(AAB41091.1)(SEQ ID NO:5)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
在一些实施方案中,降低活性的β-木糖苷酶所包含的氨基酸序列与大肠杆菌β-木糖苷酶的氨基酸序列(WP_000406872.1)(SEQ ID NO:6)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
木糖醇脱氢酶多肽
在一些方面,本公开的宿主细胞与野生细胞相比,一种或多种木糖醇脱氢酶(XDH)多肽的活性降低。木糖醇脱氢酶通常是指催化以下反应的酶(E.C.1.1.1.9):木糖醇+NAD+=D-木酮糖+NADH+H+。木糖醇脱氢酶其它名称包括,例如,“D-木酮糖还原酶”、“NAD-依赖的木糖醇脱氢酶”、“赤藻糖醇脱氢酶”、“2,3-顺式多元醇(DPN)脱氢酶(C3-5)”、“戊五醇-DPN脱氢酶”、“木糖醇-2-脱氢酶”和“木糖醇:NAD+2-氧化还原酶(D-木酮糖形成)”。
各种木糖醇脱氢酶(XDH)多肽是本领域已知,并且用本公开的方法可以把这些木糖醇脱氢酶多肽作为降低活性的靶标。本公开所用的具有活性降低的木糖醇脱氢酶,可以包括,例如,来自毕赤酵母、假丝酵母、曲霉和粗糙脉孢霉的木糖醇脱氢酶。在一些实施方案中,木糖醇脱氢酶是来自树干毕赤酵母的木糖醇脱氢酶XYL2(登录号P22144)。在一些实施方案中,木糖醇脱氢酶包括,例如,NCBI登录号为ACI01079.1、XP_456938.2和ABA39795.1的多肽。与上述登录号相关的每个序列通过引用并入本文。其它的木糖醇脱氢酶是本领域的技术人员众所周知的。
在一些实施方案中,降低活性的木糖醇脱氢酶所包含的氨基酸序列与木糖醇脱氢酶P22144的氨基酸序列(WP_000406872.1)(SEQ ID NO:6)具有至少10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%的一致性。
编码多肽的多核苷酸
本公开进一步涉及到编码本公开多肽的多核苷酸。编码多肽的多核苷酸在本文中也称为“基因”。例如,提供了编码如本文所述的任何已知或推定的木寡糖转运蛋白、木糖还原酶、β-木糖苷酶或木糖醇脱氢酶的多核苷酸。用于判断多肽与编码多肽的多核苷酸之间的关系的方法是本领域技术人员众所周知的。类似地,确定由多核苷酸序列编码的多肽序列的方法是本领域技术人员众所周知的。。
如本文所用,术语“多核苷酸”、“核酸序列”、“核酸”及其变体应属于多脱氧核糖核苷酸(含有2-脱氧-D-核糖)、多核糖核苷酸(含有D-核糖)、N-糖苷的嘌呤或嘧啶碱基的任何其它类型的多核苷酸,以及含有非核苷酸主链的其它聚合物。只要是所述聚合物含有允许碱基配对和碱基堆积的构型,如在DNA和RNA所发现的。因此,这些术语包括了已知类型的核酸序列修饰,例如,用类似物取代一个或多个天然存在的核苷酸,以及核苷酸间的修饰。如本文所用,核苷酸和多核苷酸的符号是由IUPAC-IUB生物化学命名委员会推荐的那些符号。
本公开的多核苷酸序列可以用本领域已知的各种方法制备,包括例如直接化学合成或克隆。对于直接化学合成,核酸聚合物的形成通常涉及将3'-封闭的和5'-封闭的核苷酸单体依次加入到延伸的核苷酸链的末端5'-羟基,其中每次加入通过亲核作用在所添加的单体的3'-位上的延伸链的末端5'-羟基,其通常是磷衍生物,例如磷酸三酯,亚磷酰胺等。这种方法是本领域普通技术人员已知的,并且描述在相关的文本和文献中(如Matteucci et al.,(1980)Tetrahedron Lett 21:719-722;U.S.Pat.Nos.4,500,707;5,436,327;和5,700,637)。此外,可通过使用适当的内切酶剪切DNA,用凝胶电泳分离,之后用本领域普通技术人员已知的方法,从凝胶里回收所需的多核苷酸序列,从天然来源分离所需的序列,例如利用聚合酶链反应(PCR,如U.S.Pat.No.4,683,195)。
序列相似性的鉴定方法
本领域的技术人员已知用于鉴定多肽和/或多核苷酸序列相似性(如同源物,直系同源物,旁系同源物)的各种方法,包括系统发生方法,序列相似性分析和杂交方法。
可以通过使用诸如CLUSTAL(Thompson et al.,Nucleic Acids Res.22:4673-4680(1994);Higgins et al.,Methods Enzymol 266:383-402(1996))或MEGA(Tamura et al.,Mol.Biol.&Evo.24:1596-1599(2007))程序构建基因家族的系统发生树。来自物种的基因的初始树一旦建立构建,可以将潜在的直系同源序列置于系统发生树中,并且可以确定它们与来自目的物种的基因的关系。进化关系也可以通过邻位-相接法(Saitou and Nei,Mol.Biol.&Evo.4:406-425(1987))来推断。同源的系列也可以通过交互式的BLAST策略来鉴定。进化距离可以使用泊松校正方法(Zuckerkandl and Pauling,pp.97-166in Evolving Genes and Proteins,edited by V.Bryson and H.J.Vogel.Academic Press,New York(1965))来计算。
此外,进化信息可以用来预测基因的功能。通过关注基因如何在序列上变得相似(即通过进化过程)而不是序列本身相似性(Eisen,Genome Res.8:163-167(1998)),可以极大地改善基因的功能预测。存在许多具体实例,其中已经显示基因功能与基因系统发生良好相关(Eisen,Genome Res.8:163-167(1998))。通过使用系统发生分析,本领域技术人员将认识到推断密切相关的多肽赋予的相似功能的能力是可预测的。
当使用系统发育程序如CLUSTAL分析一组相关序列时,紧密相关的序列通常聚集在一起或在相同的进化枝(一组相似的基因)中。多组相似的基因也可以通过成对的BLAST分析来鉴定(Feng and Doolittle,J.Mol.Evol.25:351-360(1987))。分析落在同一个分枝上的多组具有相似功能的相似基因,可以得到这个分枝特有的子序列。这些子序列,被称为保守序列,不仅可以用来定义每个分枝内的序列,还可以定义这些基因的功能。位于同一个分枝内的基因可能含有旁系同源序列或具有相同功能的直系同源序列(见Mount,Bioinformatics:Sequence and Genome Analysis Cold Spring Harbor Laboratory Press,Cold Spring Harbor,N.Y.,page 543(2001))。
为了找到与参考序列同源的序列,可以使用BLASTN程序(得分设为100,字长为12),以获得编码本公开的蛋白的核苷酸序列同源的核苷酸序列。可以用BLASTX程序(得分设为50,字长为3)进行BLAST蛋白质搜索,以获得与本公开的蛋白质或多肽同源的氨基酸序列。可以用Altschul et al.,(1997)Nucleic Acids Res.25:3389所描述的Gapped BLAST(在BLAST 2.0中)获得序列比较所需要的带空位的比对。或者可以用PSI-Blast对距离较远的分子进行迭代搜索(见上面的Altschul et al.,(1997))。使用BLAST、Gapped BLAST或PSI-BLAST时,可以使用各自程序(如核酸序列用的BLASTN,蛋白序列用的BLASTX)的默认参数。
用于序列比对和用于分析多肽和多核苷酸序列的相似性和一致性的方法是本领域众所周知的。
本文所用的“序列一致性”是指所分析的序列在相同位置相同碱基的百分比。如本文所用的“序列相似性”是指所分析的序列在相同位置具有相似的生理/生化特性(如电荷、大小、疏水性)的残基的百分比。
用于比较的序列比对方法是本领域公知的,包括手动比对和计算机辅助的序列比对和分析。由于计算机辅助比对方法能够大量地处理数据,后一种方法是本公开中的优选方法。如下所述,有许多可以用来进行序列比对的计算机程序,也可以由技术人员自己研发。
两条任意序列间的一致性和/或相似性百分比的计算,可以使用数学的算法完成。例如,这些数学的算法有:Myers and Miller算法(CABIOS 4:11-17(1988))、Smith局部相似性算法(Smith et al.,Adv.Appl.Math.2:482(1981))、Needleman和Wunsch的同源比对算法(Needleman and Wunsch,J.Mol.Biol.48:443-453(1970)1970)、Pearson和Lipman的搜索相似性算法(Pearson and Lipman,Proc.Natl.Acad.Sci.85:2444-2448(1988)、Karlin和Altschul算法(Karlin and Altschul,Proc.Natl.Acad.Sci.USA 87:2264-2268(1990))、修改的Karlin和Altschul算法(Karlin and Altschul,Proc.Natl.Acad.Sci.USA 90:5873-5877(1993)。
由计算机执行的这些数学算法可以用来比较序列,确定序列的一致性和/或相似性。这些执行的程序可以包括:PC/Gene程序中的CLUSTAL(可从Intelligenetics,Mountain View,Calif.获得)、AlignX程序,版本10.3.0(Invitrogen,Carlsbad,CA)和Wisconsin Genetics软件包中的GAP、BESTFIT、BLAST、FASTA和TFASTA,版本 8(可从Genetics Computer Group(GCG),575Science Drive,Madison,Wis.,USA获得)。使用这些程序进行比对时,可以使用默认的参数。CLUSTAL程序在以下的文献中充分描述(Higgins et al.,Gene 73:237-244(1988);Higgins et al.,CABIOS 5:151-153(1989);Corpet et al.,Nucleic Acids Res.16:10881-90(1988);Huang et al.,CABIOS 8:155-65(1992);and Pearson et al.,Meth.Mol.Biol.24:307-331(1994))。BLAST程序(Altschul et al.,J.Mol.Biol.215:403-410(1990))是基于上文的Karlin和Altschul算法(Karlin and Altschul(1990))。
与参考序列同源的多核苷酸可以在严紧条件或高度严紧条件下的杂交来鉴定。在各种良好表征的物理-化学力,如氢键、溶剂排除力、碱基堆积力和相类似的力的作用下,单链多核苷酸发生杂交。杂交的严紧度反应了反涉及的核酸序列的一致性的程度。严紧度越高,两条多核苷酸链越相似。严紧度受杂交和洗涤溶液和温育(及其次数)中存在的多种因素的影响,包括温度、盐浓度和组成、有机和非有机添加剂、溶剂等,如在下文引用的参考文献中详细描述(如,Sambrook et al.,Molecular Cloning:A Laboratory Manual,2nd Ed.,Vol.1-3,Cold Spring Harbor Laboratory,Cold Spring Harbor,N.Y.("Sambrook")(1989);Berger and Kimmel,Guide to Molecular Cloning Techniques,Methods in Enzymology,vol.152Academic Press,Inc.,San Diego,Calif.("Berger and Kimmel")(1987);and Anderson and Young,"Quantitative Filter Hybridisation."In:Hames and Higgins,ed.,Nucleic Acid Hybridisation,A Practical Approach.Oxford,TRL Press,73-111(1985))。
本公开涵盖能够在各种严格条件下与所公开的多核苷酸序列及其片段杂交的多核苷酸序列(见Wahl and Berger,Methods Enzymol.152:399-407(1987);and Kimmel,Methods Enzymo.152:507-511,(1987))。可以使用公知的多核苷酸杂交方法鉴定和分离本公开的多核苷酸的全长cDNA、同源物、直系同源物和旁系同源物。
此外,本公开可以使用的基因和蛋白包括这里所描述基因和蛋白的保守性修饰的变体。这本文所用的“保守性修饰的变体”包括对多肽序列单个的替换、缺失或添加,结果导致氨基酸被化学性质相似的氨基酸替换。提供功能相似的氨基酸的保守性替换表是本领域熟知的。此类保守性修饰的变体是本公开的多态变体、种间同源物、等位基因和各种替代,且不具有排它性。以下8组包含彼此保守取代的氨基酸:1)丙氨酸(A),甘氨酸(G);2)天冬氨酸(D),谷氨酸(E);3)天冬酰胺(N),谷氨酰胺(Q);4)精氨酸(R),赖氨酸(K);5)异亮氨酸(I),亮氨酸(L),甲硫氨酸(M),缬氨酸(V);6)苯丙氨酸(F),酪氨酸(Y),色氨酸(W);7)丝氨酸(S),苏氨酸(T);和8)半胱氨酸(C),甲硫氨酸(M)(见Creighton,Proteins(1984)).
用于表达多核苷酸的载体
本公开的每条多核苷酸可以克隆到表达质粒。“表达载体”或“载体”是指一种混合物或组合物可以转导、转化或感染宿主细胞,除该细胞天然的多核苷酸和/或蛋白质以外的多核苷酸和/或蛋白质,或以非原有的方式表达。“表达载体”含有可在宿主中表达的多核苷酸序列(通常是RNA或DNA)。可选的,表达载体还包括一些有助于多核苷酸成功进入宿主的材料,如病毒、脂质体、蛋白外衣等。考虑用于本公开内容的表达载体包括可以插入多核苷酸序列的表达载体,以及任何优选的或需要的操作元件。此外,表达载体必须是可以被转移到宿主细胞内,并在其中复制的载体。优先的表达载体是质粒,特别是那些已经已经被充分证明的限制性位点和具有转录多核苷酸序列的优选或必需的操作元件的质粒。这些质粒,以及其他的表达载体,是本领域熟知的。
单个多核苷酸的克隆可以通过已知的方法完成,包括可以使用限制酶性内切酶(例如BamHI,EcoRI,HhaI,XhoI,XmaI等)切割表达载体,如质粒,的特定位点。限制性内切酶产生的单链末端,可以与表达载体的粘性末端退火,这种末端可以是自带的或合成的。使用适当的酶,例如DNA连接酶进行退火。如本领域的普通技术人员将理解的,表达载体和所需的多核苷酸通常用相同的限制内切酶切割,从而确保表达载体的末端和多核苷酸的末端彼此互补。此外,DNA接头可用于辅助多核苷酸序列连接到表达载体中。
一系列单个多核苷酸也可以通过利用本领域已知的方法(例如,U.S.Pat.No.4,683,195)进行组合。例如,每个所需的多核苷酸可以在单独的PCR中产生。此后,设计特异性引物,使得PCR产物的末端含有互补序列。当PCR产物混合、变性和重新退火时,在3'端具有匹配序列的链会重叠,并且可以充当彼此的引物。通过DNA聚合酶对该重叠进行延伸,产生把原始序列“剪接”在一起的分子。以这种方式,一系列单个多核苷酸可以“剪接”在一起,并随后同时转导到宿主细胞中。因此,多个多核苷酸中的每一个都能表达。
单个多核苷酸或“剪接的”多核苷酸接着克隆到表达载体中。本公开不限于将多核苷酸克隆到表达载体的过程。本领域普通技术人员熟悉将多核苷酸克隆到表达载体的必要步骤。一个典型的表达载体包含:在所需的多核苷酸前有一个或多个调节区,以及一个核糖体结合位点,例如长度为3-9个核苷酸并位于大肠杆菌起始密码子上游3-11个核苷酸的核苷酸序列。见Shine和Dalgarno(1975)Nature 254(5495):34-38和Steitz(1979)Biological Regulation and Development(Goldberger,R.F.),1:349-399(Plenum,New York)。
本文所用的术语“可操作地连接”是指这样的构型:将控制序列置于相对于DNA序列或多核苷酸的编码序列的适当位置,使得控制序列能够指导多肽的表达。
调节区域包括,例如含有启动子和操纵子的那些区域。启动子与所需的多核苷酸可操作地连接,从而通过RNA聚合酶启动多核苷酸的转录。操纵子是与启动子相邻的多核苷酸序列,它包含可以与阻遏蛋白结合的蛋白结构域。在不存在阻遏蛋白的情况下,转录通过启动子启动。当存在阻遏蛋白时,阻遏蛋白与蛋白结合结构域特异性地结合,从而抑制转录。以这种方式,使用特定的调控区域和相应的阻遏蛋白的存在或不存在来完成转录的控制。实例包括乳糖启动子(当与乳糖接触时,Lad阻遏蛋白构象发生变化,从而阻止Lad阻遏蛋白与操纵子结合)和色氨酸启动子(当与色氨酸结合时,TrpR阻遏蛋白具有与操纵基因结合的构象;在不存在色氨酸时,TrpR阻抑蛋白不具有结合操纵基因的构象)。另一个实例是tac启动子(见de Boer et al.,(1983)Proc Natl Acad Sci USA 80(1):21-25)。
构建本公开的宿主细胞的方法可以包括将含有本公开的重组核酸的表达载体导入或转移到宿主细胞中。这些将表达载体转移到宿主细胞中的方法是本领域普通技术人员熟知的。例如,用表达载体转化细胞的一种方法包括氯化钙处理,通过钙沉淀物转化入表达载体。也可以按类似的方法使用其它盐,如磷酸钙。此外,电穿孔(即,使用电流以增加细胞对核酸序列的渗透性)可用于转化宿主细胞。细胞也可以通过使用原生质球转化(Schweizer,M,Proc.Natl.Acad.Sci.,78:5086-5090(1981))。此外,显微注射的核酸序列也具有转染宿主细胞的能力。也可以使用脂质复合物,脂质体和树状聚合物。本领域普通技术人员可以使用这些或其它方法将所需的序列转染宿主细胞。
在一些情况下,在转化前将细胞制备为原生质体或原生质球。可以通过用酶处理具有细胞壁的细胞以降解细胞壁来制备原生质体或原生质球。真菌细胞可以用几丁质酶处理。
载体可以是自主复制的载体,即作为染色体外存在的载体,其复制独立于染色体的复制,例如质粒、染色体外元件、微染色体或人工染色体。载体可以包含用于确保自我复制的任何元件。或者,载体可以是导入宿主时整合到基因组中并与染色体一起复制的载体。此外,可以使用单个载体或质粒或两个或多个载体或质粒,一起含有待导入宿主基因组的总DNA。也可以使用转座子。
载体优选含有一个或多个选择标记,这些标记能够可方便地选择转化的宿主细胞。选择标记是一种基因,其产物能够提供抗生素或病毒抗性、对重金属的抗性、营养缺陷型的原养性等。细菌细胞可以通过由诸如amp、gpt、neo和hyg基因赋予的抗生素抗性进行选择。
用于真菌宿主细胞的选择标记可以包括amdS(乙酰胺酶)、argB(鸟氨酸氨甲酰基转移酶)、bar(草胺膦乙酰转移酶)、hph(潮霉素磷酸转移酶)、niaD(硝酸还原酶)、pyrG(乳清酸核苷-5′-磷酸脱羧酶)、sC(硫酸腺苷酰转移酶)和trpC(邻氨基苯甲酸合成酶),及其等同物。
载体可以含有允许载体整合到宿主基因组中的元件,允许载体在细胞中独立于基因组的自主复制的元件。
为了整合到宿主基因组中,载体可以通过基因的序列或载体的任何其它元件进行同源或非同源重组,将载体整合到基因组中。或者,载体可含有额外的核苷酸序列,用于指导同源重组整合到宿主的基因组中。额外的核苷酸序列使得载体能够精确定位到染色体中的位置,整合到宿主基因组中。为了增加精确定位整合的可能性,整合元件应当包含足够数量的核酸,例如100至10,000个碱基对、400至10,000个碱基对或800至10,000个碱基对,以增强与相应的靶序列同源重组的概率。整合元件可以是与宿主基因组中的靶序列同源的任何序列。此外,整合元件可以是非编码或编码核苷酸序列。另一方面,载体可以通过非同源重组整合到宿主的基因组中。
对于自主复制,载体可以进一步包含一个复制起点位点,使得载体能够在所讨论的宿主中自主复制。复制起点可以是在细胞中倡导自主复制的任何质粒复制子。术语“复制起点”或“质粒复制子”在本文中定义为使质粒或载体能够在体内复制的序列。
用于调节载体中本公开的重组核酸的表达的各种启动子是本领域熟知的,可以有组成型启动子和诱导型启动子。启动子在Sambrook,et al.,Molecular Cloning:A Laboratory Manual,第3版,Cold Spring Harbor Laboratory Press,(2001)等中有描述。启动子可以是病毒,细菌,真菌,哺乳动物或植物启动子。另外,启动子可以是组成型启动子、诱导型启动子、环境调节型启动子或发育调节型启动子。用于调节本公开的重组核酸的合适启动子的实例可以是:粗糙脉孢霉ccg-1组成型启动子,其对粗糙脉孢霉昼夜节律和营养条件有响应;粗糙脉孢霉gpd-1(甘油醛-3-磷酸脱氢酶-1)强组成型启动子;粗糙脉孢霉vvd(光)诱导型启动子;粗糙脉孢霉qa-2(奎尼酸)诱导型启动子;构巢曲霉gpdA启动子;构巢曲霉trpC组成型启动子;粗糙脉孢霉tef-1(转录延长因子)高度组成型启动子;和粗糙脉孢霉xlr-1(XlnR同源物)启动子(经常用于曲霉菌属)。在一些实施方案中,本公开的重组多肽的表达处于异源启动子的控制下。
可以将不止一个拷贝的基因插入宿主中以增加基因产物的生产。可以通过将基因的至少一个额外拷贝整合到宿主基因组中,或通过包含具有核苷酸序列的可扩增选择性标记基因来获得基因拷贝数的增加。含有扩增拷贝选择标记基因的细胞,可通过在适当的选择剂存在的条件,筛选获得基因的额外拷贝。
用于连接上述元件以构建本公开的重组表达载体的步骤是本领域技术人员熟知的(可见Sambrook et al.,1989,同上)。当仅使用单个表达载体(不添加中间体)时,载体将包含所有必需的核酸序列。
本公开的宿主细胞
本公开的宿主细胞能够从木寡糖底物产生木糖甙-木糖醇寡聚物。“宿主细胞”和“宿主微生物”在本文中可互换使用,并且是指可以用于本公开方法中的活的生物细胞。在一些实施方案中,可以通过插入一个或多个重组核酸(例如DNA或RNA)转化宿主细胞。这样的重组核酸可以在一个或多个表达载体中。因此,本文所述的宿主微生物或细胞可以是原核生物(例如,真细菌界的生物)或真核细胞。如本领域的普通技术人员将理解的,原核细胞缺乏被膜包围的细胞核,而真核细胞具有被膜包围的细胞核。
各种原核或真核宿主细胞可以用于本公开,只要其在转化核酸序列后保持存活即可。优选地,宿主细胞不受必要的核酸序列的转导、随之的蛋白表达或所得中间体的不利影响。合适的真核细胞包括例如真菌、植物、昆虫和哺乳动物细胞。
在一些实施方案中,宿主是真菌菌株。本文所用的“真菌”包括例如子囊菌门,担子菌门(Ascomycota),壶菌门(Basidiomycota)和接合菌门(Zygomycota)(如Hawksworth,et al.,In,Ainsworth and Bisby's Dictionary of The Fungi,第8版,1995,CAB International,University Press,剑桥,英国)以及卵菌门(Oomycota)(如Hawksworth,et al.,1995,同上,第171页中所述)和所有有丝分裂孢子真菌(Hawksworth,et al.,1995,同上)。
在某些实施方案中,真菌细胞是酵母细胞。本文所用的“酵母”包括产子囊酵母(内孢霉目)、产担孢子酵母(basidiosporogenous yeast)和属于半知菌的酵母(Blastomycetes)。由于酵母的分类在将来可能改变,为了本公开的目的,酵母应按照Biology and Activities of Yeast定义(Skinner,F.A.,Passmore,S.M.和Davenport,R.R.,eds,Soc.App.Bacteriol.Symposium Series No.9,1980)。
在一些实施方案中,酵母宿主是假丝酵母属(Candida)、汉逊酵母属(Hansenula)、伊萨酵母属(Issatchenkia)、克鲁维酵母属(Kluyveromyces)、毕赤酵母属(Pichia)、酵母属(Saccharomyces)、裂殖酵母属(Schizosaccharomyces)或耶氏酵母属(Yarrowia)菌株。在其他实施方案中,酵母宿主是卡氏酵母(Saccharomyces carlsbergensis)(Todka,2010)、酿酒酵母(Saccharomyces cerevisiae)(Duarte et al.,2009)、糖化酵母(Saccharomyces diastaticus)、道格拉斯酵母(Saccharomyces douglasii)、克鲁弗酵母(Saccharomyces kluyveri)、诺地酵母(Saccharomyces norbensis)、摩纳酵母(Saccharomyces monacensis)(GB-Analysts Reports,2008)、贝酵母(Saccharomyces bayanus)(Kristen Publicover,2010)、巴氏酵母(Saccharomyces pastorianus)(Nakao et al.,2007)、粟酒酵母(Saccharomyces pombe)(Mousdale,2008)或卵形酵母(Saccharomyces oviformis)菌株。在其他实施方案中,酵母宿主是乳酸克鲁维酵母(Kluyveromyces lactis)(O.W.Merten,2001)、脆壁克鲁维酵母(Kluyveromyces fragilis)(Pestal et al.,2006;Siso,1996)、马克斯克鲁维酵母(Kluyveromyces marxiamus)(K.Kourkoutas et al.,2008)、树干毕赤酵母(Pichia stipitis)(Almeida et al.,2008)、休哈塔假丝酵母(Candida shehatae)(Ayhan Demirbas,2003)或热带假丝酵母(Candida tropicalis)(Jamai et al.,2006)。在其他实施方案中,酵母宿主可以是解脂耶氏酵母(Yarrowia lipolytica)(Biryukova E.N.,2009)、卡斯酒香酵母(Brettanomyces custersii)(Spindler D.D.et al.,1992)或鲁氏接合酵母(Zygosaccharomyces roux)(Chabane et al.,2006)。在一些实施方案中,酵母细胞是酿酒酵母(Saccharomyces cerevisiae)。
在其它实施方案中,真菌宿主是丝状真菌菌株。“丝状真菌”包括真菌门(Eumycota)和卵菌门(Oomycota)的所有丝状形式(如Hawksworth et al.,1995,同上所定义)。丝状真菌通常特征在于由几丁质、纤维素、葡聚糖、壳聚糖、甘露聚糖和其它复合多糖组成的菌丝壁。营养生长是通过菌丝伸长,碳分解代谢是专性好氧的。相比之下,酵母如酿酒酵母的营养生长是通过单细胞菌体的出芽,并且碳分解代谢可以是发酵的。
在一些实施方案中,丝状真菌宿主可以是支顶孢属(Acremonium)、曲霉属(Aspergillus)、镰孢属(Fusarium)、腐质霉属(Humicola)、毛霉属(Mucor)、毁丝霉属(Myceliophthora)、脉孢霉属(Neurospora)、青霉属(Penicillium)、节格孢属(Scytalidium)、梭孢壳属(Thielavia)、弯颈霉属(Tolypocladium)或木霉属(Trichoderma)的菌株。在一些实施方案中,宿主细胞是粗糙脉孢霉(Neurospora crassa)。
在一些实施方案中,丝状真菌宿主是泡盛曲霉(Aspergillus awamori)、臭曲霉(Aspergillus foetidus)、日本曲霉(Aspergillus japonicus),构巢曲霉(Aspergillus nidulans)、黑曲霉(Aspergillus niger)或米曲霉(Aspergillus oryzae)菌株。在另一些实施方案中,丝状真菌宿主是杆抱状镰孢菌(Fusarium bactridioides)、禾谷谷类镰孢菌(Fusarium cerealis)、克鲁克威尔镰孢菌(Fusarium crookwellense)、黄色镰孢菌(Fusarium culmorum)、正禾谷镰孢菌(Fusarium graminearum)、禾赤镰孢菌(Fusarium graminum)、异孢镰孢菌(Fusarium heterosporum)、合欢镰孢菌(Fusarium negundi)、尖镰孢菌(Fusarium oxysporum)、多枝镰孢菌(Fusarium reticulatum)、粉红镰孢菌(Fusarium roseum)、接骨木镰孢菌(Fusarium sambucinum)、肤色镰孢菌(Fusarium sarcochroum)、拟枝孢镰孢菌(Fusarium sporotrichioides)、硫色镰孢菌(Fusarium sulphureum)、簇镰孢菌(Fusarium torulosum)、拟丝孢镰刀菌(Fusarium trichothecioides)或镶片镰孢菌(Fusarium venenatum)菌株。在一些实施方案中,丝状真菌宿主是特异腐质霉(Humicola insolens)、高温毛壳霉(Humicola lanuginosa)、(Mucor miehei)、嗜热毁丝霉(Myceliophthora thermophila)、产紫青霉(Penicillium purpurogenum)、嗜热柱霉(Scytalidium thermophilum)、嗜热侧孢霉(Sporotrichum thermophile)(Topakas et al.,2003)、或太瑞斯梭孢壳霉(Thielavia terrestris)菌株。在一些实施方案中,丝状真菌宿主是哈茨木霉(Trichoderma harzianum)、康宁木霉(Trichoderma koningii)、长梗木霉(Trichoderma longibrachiatum)、里氏木霉(Trichoderma reesei)或绿色木霉菌株(Trichoderma viride)。
在一些实施方案中,宿主细胞是原核的,并且在某些实施方案中,原核生物是大肠杆菌(E.coli)(Dien,B.S.et al.,2003;Yomano,L.P.et al.,1998;Moniruzzaman et al.,1996)、枯草芽孢杆菌(Bacillus subtilis)(Susana Romero et al.,2007)、运动发酵单胞菌(Zymomonas mobilis)(B.S.Dien et al.,2003;Weuster Botz,1993;Alterthum and Ingram,1989)、解热厌氧杆菌(Thermoanaerobacterium saccharolyticum)(Marietta Smith,2009)或产酸克雷伯式菌(Klebsiella oxytoca)(Dien B.S.et al.,2003;Zhou et al.,2001;Brooks and Ingram,1995)。在其他实施方案中,原核宿主细胞是互营单孢菌(Carboxydocella sp.)、谷氨酸棒杆菌(Corynebacterium glutamicum)(Masayuki Inui et al.,2004)、肠杆菌(Enterobacteriaceae)(Ingram et al.,1995)、菊欧文菌(Erwinia chrysanthemi)(Zhou和Ingram,2000;Zhou et al.,2001)、乳杆菌(Lactobacillus sp.)(McCaskey,T.A.et al.,1994)、嗜酸小球菌(Pediococcus acidilactici)(Zhou,S.et al.,2003)、荚膜红假单胞菌(Rhodopseudomonas capsulata)(X.Y.Shi et al.,2004)、乳酸链球菌(Streptococcus lactis)(J.C.Tang et al.,1988)、弗尼斯弧菌(Vibrio furnissii)(L.P.Wackett,2010)、弗尼斯弧菌M1(Vibrio furnissii M1)(Park et al.,2001)、解糖热纤维素菌(Caldicellulosiruptor saccharolyticus)(Z.Kadar et al.,2004)或野油菜黄单胞菌(Xanthomonas campestris)(S.T.Yang et al.,1987)。在其他实施方案中,宿主细胞是蓝细菌(cyanobacteria)。其它细菌宿主细胞的实例包括例如分类到大肠杆菌属(Escherichia)、肠杆菌属(Enterobacter)、固氮菌属(Azotobacter)、欧文氏菌属(Erwinia)、杆菌属(Bacillus)、假单胞菌属(Pseudomonas)、克雷伯菌属(Klebsiella)、变形杆菌属(Proteus)、沙门氏菌属(Salmonella)、沙雷氏菌属(Serratia)、志贺氏菌属(Shigella)、根瘤菌属(Rhizobia)、透明颤菌属(Vitreoscilla)、集胞藻属(Synechococcus)、集胞藻属(Synechocystis)和副球菌属(Paracoccus)的细菌。
本公开的宿主细胞可以是遗传修饰的,因为重组核酸已经被导入宿主细胞,因此自然界没有这种经遗传修饰的宿主细胞。本公开的适宜的宿主细胞是能够表达编码一种或多种不同功能的蛋白的一种或多种核酸组件的宿主细胞。
如本文所用的“重组核酸”或“异源核酸”或“重组多核苷酸”是指核酸的聚合物,其中至少下列之一是真的:(a)对于给定的宿主细胞,核酸序列是外来的(即,不是天然存在于宿主细胞内);(b)该序列可以在给定的宿主细胞中天然存在,但是它的量是非天然的(例如大于预期);或(c)核酸序列含有两个或多个在自然界中彼此不具有相同关系的子序列。例如,关于实例(c),重组核酸序列将来自无关基因的两条或更多条序列,重排在一起,开成具有新功能的核酸。具体地,本公开描述了将表达载体引入宿主细胞中,其中表达载体含有编码通常不在宿主细胞中存在的蛋白的核酸序列,或虽含有编码通常存在宿主细胞中的蛋白的核酸序列,但处于不同调控序列的控制之下。然后,与参考宿主细胞的基因组相比,编码蛋白的核酸序列是重组的。被称为重组的蛋白通常意味着其由宿主细胞中的重组核酸序列编码。
本公开的“重组”多肽、蛋白或酶是由“重组核酸”或“异源核酸”或“重组多核苷酸”编码的多肽,蛋白或酶。
在一些实施方案中,宿主细胞中编码所需蛋白的基因对于宿主细胞来说可以是异源的,或者这些基因对于宿主细胞来说可以是内源性的,但可操作地连接到异源启动子和/或控制区,导致其在宿主细胞中基因表达更高。在某些实施方案中,宿主细胞不会自然地产生期望的蛋白,并且含有能够表达一种或多种产生这些分子所必需的基因的异源核酸组件。
如本文所用的关于核酸分子或多肽和特定细胞或微生物“内源的”,是指存在细胞中并且不使用重组工程技术引入细胞中的核酸序列或多肽;例如当细胞最初从自然界分离时就存在于细胞中的基因。在一些实施方案中,本公开的某些多肽内源地存在于产生木糖甙-木糖醇寡聚物方法的宿主细胞中。
“经遗传工程改造的”或“遗传修饰的”是指用于构建能够表达蛋白的原核或真核宿主细胞的任何重组DNA或RNA方法,该蛋白表达可以是上升的形式、下降的形式或非内源性(例如突变的)形式。换句话说,已经用重组多核苷酸分子转染、转化或转导宿主细胞,从而改变宿主细胞以使细胞改变所需蛋白的表达。用于遗传工程改造宿主细胞的方法和载体是本领域公知的;例如各种技术在Ausubel等人编辑的Current Protocols in Molecular Biology上有阐述(Wiley&Sons,New York,1988,每季度更新)。遗传工程技术包括例如表达载体,和定点同源重组和基因激活(例如见U.S.Pat.No.5,272,071)。
改变多肽活性的方法
与相应的对照细胞,例如相应的野生型细胞相比,本公开的宿主细胞可具有修饰的多肽活性,以促进和/或增加木糖甙-木糖醇寡聚物的生产。可以修饰多肽活性,使得本公开的一种或多种多肽具有增加的活性或降低的活性。在一些实施方案中,宿主细胞可以具有一种或多种具有增加的活性的多肽以及一种或多种具有降低的活性的多肽。修饰(例如增加和/或降低)本公开的一种或多种多肽的活性的方法是本领域熟知的并且在本文中描述。
降低多肽的活性
本公开的宿主细胞可以含有与相应的对照细胞例如野生型细胞相比具有降低的/降低的活性的一种或多种多肽。例如,与相应的对照细胞相比,一种或多种β-木糖苷酶蛋白(例如具有木糖甙-木糖醇水解酶活性的那些)或一种或多种木糖醇脱氢酶可以在宿主细胞中具有降低的活性。降低多肽的表达,丰度和/或活性的方法是本领域熟知的并且在本文中描述。
在一些实施方案中,降低多肽的活性涉及过表达作为多肽抑制蛋白的多肽。宿主细胞可以过表达抑制一种或多种蛋白的表达和/或活性的抑制蛋白,所述蛋白用于防止木糖甙-木糖醇寡聚物的产生和/或积累,例如β-木糖苷酶具有木糖甙-木糖醇水解酶的活性。
在一些实施方案中,降低多肽(例如一种或多种β-木糖苷酶和/或一种或多种木糖醇脱氢酶)的活性涉及降低编码多肽的核酸的表达。降低核酸的表达可以通过将遗传突变引入靶核酸中来实现。突变方法可以用于通过产生突变来破坏或“敲除”靶基因的表达。在一些实施方案中,突变导致靶基因的部分缺失。在其他实施方案中,突变导致靶基因的完全缺失。微生物突变的方法是本领域熟知的,包括例如随机突变和定点突变以引入突变。随机突变的方法的实例包括例如化学诱变(例如使用乙磺酸甲酯),插入突变和辐射。在一些实施方案中,本公开的宿主细胞具有一种或多种β-木糖苷酶多肽和/或一种或多种木糖醇脱氢酶多肽的突变。
用于减少或抑制靶基因表达的一种方法是通过遗传修饰靶基因并通过同源重组引入宿主细胞的基因组中以替换野生型版本的基因(例如,描述在U.S.Pat.No.6,924,146中)。
用于减少或抑制靶基因表达的另一种方法是通过使用根癌农杆菌(Agrobacterium tumefaciens)的T-DNA或转座子的插入突变(参见Winkler et al.,Methods Mol.Biol.82:129-136,1989,和Martienssen,Proc.Natl.Acad.Sci.95:2021-2026,1998)。在产生插入突变体后,突变体可以经过筛选以鉴定在靶基因中含有插入的突变体。通过插入突变来破坏靶基因的方法描述于例如U.S.Pat.No.5,792,633。通过转座子突变来破坏靶基因的方法描述于例如U.S.Pat.No.6,207,384。
破坏靶基因的另一种方法是通过使用cre-lox系统(例如,如U.S.Pat.No.4,959,317中所述)。
破坏靶基因的另一种方法是通过使用PCR突变(例如,如U.S.Pat.No.7,501,275中所述)。
内源基因表达还可以通过RNA干扰(RNAi)来减少或抑制,其使用具有与靶基因序列相同或相似的序列的双链RNA。RNAi可以包括使用microRNA(例如人工miRNA)抑制基因的表达。
RNAi是当将具有与靶基因的序列相同或相似的序列的双链RNA导入细胞中时,插入的外源基因和靶内源基因两者的表达被抑制的现象。双链RNA可以由两条分开的互补RNA形成,或者可以是具有形成双链RNA的内部互补序列的单条RNA。
因此,在一些实施方案中,使用RNAi技术实现基因表达的降低或抑制。例如,为了使用RNAi实现编码蛋白的DNA的表达的降低或抑制,使用具有编码蛋白的DNA序列或与其基本上相似的序列的双链RNA(包括那些工程改造的不翻译蛋白的序列)或其片段,引入到感兴趣的宿主细胞中。如本文所用,RNAi和dsRNA均指通过引入双链RNA分子诱导的基因特异性沉默,参见例如U.S.Pat.Nos.6,506,559和6,573,099,并且包括具有双链区域的分子,例如短发夹RNA分子。然后可以对所得细胞筛选与靶基因表达降低相关的表型,例如降低的纤维素酶表达,和/或通过监测靶基因转录的RNA稳定水平。尽管用于RNAi的序列不需要与靶基因完全相同,但它们可以是至少70%、80%、90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或更多与与靶基因相同。参见例如美国专利申请公开号2004/0029283。构建编码具有与靶基因无关的茎-环结构的RNA分子的组件,并且其位于所感兴趣的基因特异性的序列的远端,也可以用于抑制靶基因表达。参见例如美国专利申请公开号2003/0221211。
RNAi核酸可以包括全长靶RNA或对应于靶RNA的片段。在一些情况下,所述片段将比对应的靶序列少100、200、300、400或500个核苷酸。另外,在一些方面,这些片段的长度至少50、100、150、200或更多个核苷酸。干扰RNA可基于短双链体(即,双链序列的短区)设计。通常,短双链体的长度为至少约15、20或25-50个核苷酸(例如,双链RNA的每个互补序列的长度为15-50个核苷酸),通常约20-30个核苷酸,例如20、21、22、23、24、25、26、27、28、29或30个核苷酸。在一些情况下,用于RNAi的片段所对应的靶蛋白的区域,不存在于生物体中的其它蛋白中或与生物体中的其他转录物几乎没有相似性的。例如通过分析与比较公开可用的序列数据库进行选择。类似地,可以通过与感兴趣的基因家族(例如本文所述的那些)的保守序列的相似性或一致性来选择RNAi片段,使得RNAi可以针对含有保守序列的多种不同基因转录物。
RNAi可以作为较大DNA组件的一部分引入宿主细胞。通常,这样的组件允许RNAi在引入后在细胞中稳定表达,例如通过将组件整合到宿主基因组中。因此,转染细胞后能连续表达RNAi的表达载体可用于本公开。例如,可以使用表达小发夹或茎-环结构的RNA或microRNA前体的载体。microRNA在体内可加工成能够进行基因特异性沉默的小RNAi分子(Brummelkamp et al.,Science 296:550-553,(2002);和Paddison,et al.,Genes&Dev.16:948-958,(2002))。用双链RNA进行转录后基因沉默的更详细的讨论见Hammond et al.,Nature Rev Gen 2:110-119,(2001);Fire et al.,Nature 391:806-811,(1998);和Timmons and Fire,Nature 395:854,(1998)。
用于选择和设计产生RNAi的序列的方法是本领域众所周知的(例如U.S.Pat.Nos.6,506,559;6,511,824;和 6,489,127)。
降低或抑制靶基因在宿主细胞中的表达,还可以通过将基于靶基因核酸序列的反义元件引入宿主细胞中来实现。对于反义抑制,靶序列相对于表达载体中的启动子序列反向方向排列。引入的序列不需要是全长cDNA或基因,并且不需要与靶cDNA或待转化细胞中的基因相同。然而,一般地,当引入的序列具有较短的长度时,使用与天然靶序列同源性更高的序列可实现有效的反义抑制。在一些方面,载体中引入的反义序列的长度为至少30个核苷酸,并且通常在反义序列的长度增加时,反义抑制效果提高。在一些方面,载体中反义序列的长度将大于100个核苷酸。所述的反义元件的转录导致RNA分子的产生,该RNA分子与内源靶基因转录的mRNA分子反向互补。靶基因表达的抑制也可以使用核酶实现。核酶的制备和使用公开于U.S.Pat.Nos.4,987,071和5,543,508。
可以使用本领域熟知的方法构建含有编码靶基因表达的抑制剂,例如反义或siRNA的核酸表达框。元件包括调节元件,包括启动子和用于表达和筛选表达元件的细胞的其它序列。通常,真菌和/或细菌转化载体包括一个或多个克隆的编码序列(基因组或cDNA)和显性选择标记,编码序列序列受5'和3'调节序列。这样的转化载体通常还包含启动子(例如,控制诱导型或组成型、环境或发育调节的表达的调节区)、转录起始起始位点、RNA加工信号(例如内含子剪接位点)、转录终止位点,和/或polyA信号。
在某些实施方案中,可以修饰靶核酸的一部分,例如编码催化结构域的区域、编码区或编码区表达所需的控制序列。基因的这种控制序列可以是启动子序列或其部分功能,即足以影响基因表达的部分。例如,启动子序列可以是失活的,导致没有表达,或者可以用较弱的启动子替代天然启动子序列以降低编码序列的表达。可供改造的其它控制序列可包括例如前导序列、前肽序列、信号序列、转录终止子和转录激活子。
提高多肽的活性
本公开的宿主细胞可以含有与相应的对照细胞(例如相应的野生型细胞)相比具有增加的表达水平和/或活性的一种或多种多肽。在一些实施方案中,本公开的宿主细胞含有编码重组多肽的重组核酸。在某些实施方案中,重组核酸在宿主细胞中异常地表达(例如,组成型表达,可诱导表达等),使得与相应的对照细胞相比,异常表达导致多肽活性的增加。在一些实施方案中,含有编码重组多肽的重组核酸的宿主细胞,其多肽的量比不含相应重组核酸的对照细胞更多。当宿主细胞中蛋白或核酸以大于正常量的量产生或维持时,蛋白或核酸被“过表达”。在一些实施方案中,本公开的宿主细胞过表达多肽,例如,木寡糖转运蛋白或木糖还原酶多肽。宿主细胞可以过表达一种或多种多肽,使得与相应的对照细胞相比,宿主细胞中一种或多种这些蛋白的活性增加。相应的对照细胞可以是,例如没有过表达宿主细胞中过表达的一种或多种多肽的细胞,例如野生型细胞。各种对照细胞对于本领域技术人员是显而易见的。
增加多肽表达的各种方法是本领域已知的。例如,可以改造涉及控制编码多肽的核酸表达的其他遗传区域,例如增强子序列,从而增加核酸的表达。核酸的表达水平可以通过检测该基因编码的mRNA的水平和/或通过检测该核酸编码的多肽的水平或活性来评估。
在一些实施方案中,宿主细胞过表达是一种或多种多肽(例如木糖寡糖转运蛋白和/或木糖还原酶)的活化蛋白的多肽。激活蛋白多肽的过表达可以导致多肽的丰度和活性增加。激活蛋白可以增加靶多肽的表达。活化蛋白可以增加靶多肽的活性。
增加本公开多肽(例如木寡糖转运蛋白和/或木糖还原酶)的丰度以增加多肽活性可以通过过表达多肽来实现。增加多肽丰度的其他方法是本领域已知的。例如,通过细胞降解机制(例如蛋白酶体)降低多肽的降解可增加多肽的稳定性和丰度。多肽可以被遗传改造,增强它们对细胞蛋白水解的抗性,但它们在分子活性上没有变化。可以将作为参与一种或多种多肽降解的细胞因子的抑制蛋白多肽引入宿主细胞中以增加一种或多种多肽的丰度,所述多肽是定向增加活性的。此外,宿主细胞可以用蛋白酶体的化学抑制剂如放线菌酮处理,以增加本公开的一种或多种多肽的丰度。
生产木糖甙-木糖醇寡聚物的方法
本公开涉及生产木糖甙-木糖醇寡聚物的方法。本公开用于生产木糖甙-木糖醇寡聚物的宿主细胞具有木糖葡聚糖转运蛋白和木糖还原酶。可以使用各种木寡糖转运蛋白,只要它们能够将一种或多种木寡糖运输到宿主细胞中即可。可以使用各种木糖还原酶多肽,只要它们能够作用于木寡糖底物以将木寡糖转化成木糖甙-木糖醇寡聚物。另外,细胞中,一种或多种β-木糖苷酶蛋白的活性可以降低,只要特定的β-木糖苷酶具有木糖甙-木糖醇水解酶活性。具有木糖甙-木糖醇水解酶活性的蛋白将水解木糖甙-木糖醇寡聚物并阻止它们在宿主细胞中积累,因此细胞中的木糖甙-木糖醇水解酶活性应该降低或去除。此外,可以在细胞中降低一种或多种木糖醇脱氢酶的活性。
为了产生木糖甙-木糖醇寡聚物,本公开的宿主细胞应在含有木寡糖或木寡糖源的生长培养基中培养。可以使用各种木寡糖,包括例如木二糖(2个木糖单体),木三糖(3个木糖单体),木四糖(4个木糖单体),木五糖(5个木糖单体)和木六糖(6个木糖单体)。如本领域技术人员将理解的,可以针对宿主细胞中特定的木寡糖转运蛋白选择特定的木寡糖,因为不同的木寡糖转运蛋白可以具有对不同类型和长度的木寡糖的不同亲和力和/或不同的转运能力。木寡糖可以商业购买,或者它们可以衍生自其它木寡糖。
木寡糖的来源是本领域公知的并且在本文中描述。例如,木寡糖的来源可以是含有木寡糖或其分解释放木寡糖的材料。如本文所用,“含木寡糖的材料”是含有或能够产生木寡糖的任何材料,包括生物质,例如含有植物材料的生物质。适合在目前公开的方法中使用的生物质包括各种含木寡糖的材料,例如芒草、柳枝稷、禾草、黑麦草、芦苇草、甜象草、普通芦苇、麦秆、大麦秸秆、油菜秸秆、燕麦秸秆、玉米秸秆、大豆秸秆、燕麦壳、高粱、稻壳、黑麦壳、小麦壳、甘蔗渣、椰肉粉、椰肉颗粒、棕榈仁粉、玉米纤维、具有可溶物的干酒糟(DDGS)、蓝茎、玉米芯、松木、桦木、柳木、白杨木、杨木、能源甘蔗、废纸、锯屑、林业废物、城市固体废物、作物残渣、其他草和其他木材。木寡糖的来源材料可能需要预处理以产生和/或释放木寡糖,例如用高温或高压处理。这样的处理是本领域技术人员公知的。在宿主细胞在某些木寡糖源例如木质纤维素生物质上生长的实施方案中,应当使用能够从生物质中释放木寡糖的宿主细胞。示例性细胞对于本领域技术人员是显而易见的。
本公开的宿主细胞应在合适的条件下培养,使得宿主细胞能从木寡糖底物产生木糖甙-木糖醇寡聚物。在本公开的方法中用于培养宿主细胞的生长培养基应当包含合适的碳源。“碳源”通常是指适合用作细胞生长的碳源的底物或化合物。碳源可以是各种形式,例如聚合物、碳水化合物、酸、醇、醛、酮、氨基酸、肽等。这些包括例如各种单糖、寡糖、多糖、生物质聚合物(如纤维素或半纤维素)、木糖、阿拉伯糖、二糖(如蔗糖)、饱和或不饱和脂肪酸、琥珀酸盐、乳酸盐、乙酸盐、乙醇等,或其混合物。除了合适的碳源之外,培养基可以包含本领域技术人员已知的、适合培养物生长和促进参与木糖甙-木糖醇寡聚物生产途径的合适的矿物质、盐、辅因子、缓冲液和其他组分。用于宿主细胞例如微生物的标准培养环境是本领域公知的并且在本文中描述。在一些实施方案中,本发明的宿主细胞好氧培养或微好氧培养。
本公开的方法可以导致木糖甙-木糖醇寡聚物(木糖甙n-木糖醇)在宿主细胞中的产生和积累。宿主细胞可以生产各种木糖甙-木糖醇寡聚物,包括例如木糖甙-木糖醇、木二糖甙-木糖醇(木糖甙2-木糖醇)、木三糖甙-木糖醇(木糖甙3-木糖醇)、木四糖甙-木糖醇(木糖甙4-木糖醇)、木五糖甙-木糖醇(木糖甙5-木糖醇)和木六糖甙-木糖醇(木糖甙6-木糖醇)。本领域技术人员很容易理解,所生产的特定木糖甙-木糖醇寡聚物取决于多种因素,例如当作用于木寡糖底物时,特定的木寡糖底物和特定的木糖还原酶的活性。
在一些实施方案中,本公开的方法涉及的宿主细胞与相应的对照细胞相比,具有增加的木糖甙-木糖醇寡聚物生产速率。具有增加的木糖甙-木糖醇寡聚物生产速率的宿主细胞可以是具有降低的一种或多种β-木糖苷酶和/或一种或多种木糖醇脱氢酶的活性和/或一种或多种木糖醇转运蛋白和/或木糖还原酶的表达或活性增加。本公开的宿主细胞,木糖甙-木糖醇寡聚物产生的速率比野生型细胞或不具有改造的多肽活性的细胞可以高至少0.1倍、至少0.2倍、至少0.3倍、至少0.4倍、至少0.5倍、至少0.6倍至少1.0倍、至少0.7倍、至少0.8倍、至少0.9倍、至少1倍、至少1.25倍、至少1.5倍、至少1.75倍、至少2倍、至少2.25倍、至少2.5倍、至少2.75倍、至少3倍、至少3.25倍、至少3.5倍、至少3.75倍、至少4倍、至少4.25倍、至少4.5倍、至少4.75倍、至少5倍、至少5.25倍、至少5.5倍、至少5.75倍、至少6倍、至少7倍、至少8倍、至少9倍、至少10倍、至少20倍或至少30倍或更多。在一些实施方案中,相应的对照细胞不积累任何木糖甙-木糖醇寡聚物。对照细胞可能不能产生木糖甙-木糖醇寡聚物,因为例如对照细胞不含有产生木糖甙-木糖醇寡聚物所需的分子机制。不能产生木糖甙-木糖醇寡聚物的示例性细胞可以包括例如不具有木寡糖转运蛋白和/或木糖还原酶多肽的细胞。对照细胞可能不能积累木糖甙-木糖醇寡聚物,因为例如对照细胞具有水解木糖甙-木糖醇寡聚物的分子机制,例如具有木糖甙-木糖醇水解酶活性的β-木糖苷酶。
在一些实施方案中,本公开的方法涉及的宿主细胞,与相应的对照细胞相比木糖甙-木糖醇寡聚物消耗速率降低。木糖甙-木糖醇寡聚物消耗速率降低的宿主细胞可以是降低木糖甙-木糖醇水解酶活性的一种或多种β-木糖苷酶和/或一种或多种木糖醇脱氢酶的活性。本公开的宿主细胞,与对应的对照细胞例如野生型细胞或未改造的多肽活性的细胞相双,对木糖甙-木糖醇寡聚物的水解可以降低约5%、约10%、约15%、约20%、约25%、约30%、约35%、约40%、50%、约45%、约50%、约55%、约60%、约65%、约70%、约75%、约80%、约85%、约90%、约95%或约100%。在一些实施方案中,与相应的对照细胞相比,本公开的宿主细胞消耗木糖甙-木糖醇寡聚物减少约100%。
测量宿主细胞或细胞群木糖甙-木糖醇寡聚物的积累、产生和/或消耗/降解/水解速率的方法是本领域熟知的并且在本文中描述。例如,测定培养物中细胞或细胞群的木糖甙-木糖醇寡聚物消耗/降解速率可以通过允许细胞在含有木寡糖的培养基中生长,并且在指定的时间点测量生长培养基中的木寡糖随时间的消耗,测定所绘曲线的斜率以确定木寡糖消耗/降解速率(例如木二糖/L/h)。类似地,测定木糖甙-木糖醇寡聚物积聚或产生可以通过在含有木寡糖的培养基中培养细胞或细胞群,并且在指定的时间点测量从木寡糖中产生的木糖甙-木糖醇寡聚物。
在一些实施方案中,在本公开的宿主细胞产生木糖甙-木糖醇寡聚物之后,木糖甙-木糖醇寡聚物可以从培养基中沉淀、移出或初步纯化。从培养基中初步纯化木糖甙-木糖醇寡聚物的方法对于本领域技术人员是显而易见的并且在本文中描述。例如,各种柱层析技术可用于从培养基中纯化木糖甙-木糖醇寡聚物。
含有木糖甙-木糖醇寡聚物的组合物
本发明还提供了含有木糖甙-木糖醇寡聚物的组合物。根据本公开的方法产生的包括木糖甙-木糖醇寡聚物在内的各种组合物在本文中描述并且对于本领域技术人员是显而易见的。例如,在从培养基中纯化木糖甙-木糖醇寡聚物的一些实施方案中,木糖甙-木糖醇寡聚物可以包含在组合物中,产生含有木糖甙-木糖醇的组合物。在其它实施方案中,含有木糖甙-木糖醇寡聚物的组合物可以含有未从培养基中初步纯化出来的寡聚物。例如,可以制备含有由本公开的宿主细胞产生的含有木糖甙-木糖醇寡聚物的培养基的组合物。可以从培养基中除去宿主细胞,使得组合物含有由宿主细胞产生的无细胞生长的培养基和木糖甙-木糖醇寡聚物。
实施例
提供以下实施例用于说明并帮助本领域技术人员更好地理解本公开的各种实施例。以下实施例不以任何方式限制本公开的范围。
实施例1:木糖甙-木糖醇寡聚物及其微生物和酶制备
本实施例展示木糖甙-木糖醇寡聚物是由含有木寡糖转运和利用途径的酿酒酵母工程菌株产生。异源表达木寡糖转运蛋白和木糖还原酶的工程菌株,能将木寡糖运到体内并转化成主要产物木糖甙-木糖醇和木二糖甙-木糖醇。因此,该工作展示了建立木糖甙-木糖醇寡聚物生物法生产的生物平台。从木寡糖底物生产木糖甙-木糖醇寡聚物的生物途径的概述见图13A。
材料与方法
粗糙脉孢霉菌株
粗糙脉孢霉菌株来自真菌遗传学库存中心(FGSC)(McCluskey et al.,2003),包括WT(FGSC 2489)和两种寡糖转运蛋白的缺失菌株:NCU00801(FGSC 16575)和NCU08114(FGSC 17868)。
异源表达的质粒和酵母菌株
按(http://www.fgsc.net/fgn35/lee35.pdf)和Harju等人所述提取粗糙脉孢霉WT菌株(FGSC 2489)和酿酒酵母S288C菌株的模板基因组DNA(gDNA)。从粗糙脉孢霉gDNA模板扩增β-木糖苷酶基因NCU01900和NCU09652(分别为GH43-2和GH43-7)的开放阅读框(ORF)。NCBI上粗糙脉孢霉CDT-2的登录号为XP_963873.1。GenBank上的树干毕赤酵母木糖还原酶的登录号为ADQ89193.1。为方便生化分析,每个ORF的C末端与6个组氨酸标签融合,并与2μ酵母质粒pRS423骨架中的酿酒酵母PTEF1启动子和CYC1转录终止子侧接。质粒pRS426_NCU08114先前已有描述(Galazka et al.,2010)。含有树干毕赤酵母木糖利用途径(木糖还原酶、木糖醇脱氢酶和木酮糖激酶)的质粒pLNL78来自John Dueber的实验室(Latimer et al.,提交)。通过使用In-Fusion克隆试剂盒(Clontech)将NCU08114和NCU01900表达框整合到pLNL78中构建质粒pXD2,即单一质粒形式的木寡糖途径。质粒pXD8.4,衍生自质粒pRS316(Sikorski et al.,1989)用于表达带有PCCW12启动子的CDT-2和GH43-2。质粒pXD8.6,衍生自pXD8.4,用GH43-7的ORF替代GH43-2的ORF。pXD8.7包含使用各自的PCCW12启动子的所有三种表达框(CDT-2,GH43-2和GH43-7)。酿酒酵母菌株D452-2(MATa leu2 his3 ura3 can1)(Kurtzman et al.,1994)和SR8U(进化的木糖快速利用菌株SR8的尿嘧啶自养型)(Kim et al.,2013)用于酵母实验的受体菌株。从粗糙脉孢霉gDNA扩增粗糙脉孢霉木糖还原酶(XYR-1,NcXR)的ORF,通过重叠PCR除去内含子。从pLNL78扩增树干毕赤酵母木糖还原酶(SsXR)的ORF。将每个木糖还原酶ORF的C-末端与6个组氨酸标签融合,并与酿酒酵母PCCW12启动子和CYC1转录终止子侧接,并插入质粒pRS316中。
基于酵母细胞的木寡糖摄取测定
酿酒酵母在缺乏尿嘧啶的优化基础培养基(oMM)中生长至对数期后期。oMM含有1.7g/L YNB(Sigma,Y1251),2倍适当的CSM无氮源混合物,10g/L(NH4)2SO4,1g/L MgSO4.7H2O,6g/L KH2PO4,100mg/L,100mg/L腺嘌呤半硫酸盐,10mg/L肌醇,100mg/L谷氨酸,20mg/L赖氨酸,375mg/L丝氨酸和100mM 4-吗啉乙磺酸(MES),pH 6.0(Lin et al.,提交)。然后收获细胞并用测定缓冲液(5mM MES,100mM NaCl,pH 6.0)洗涤三次,并重悬至最终OD600为40。用相同测定缓冲液制备浓度为200μM的底物母液。通过混合等体积的细胞悬浮液和底物母液开始转运的测定。反应在30℃下连续振荡孵育30分钟。将样品在4℃以14,000rpm离心5分钟除去酵母细胞。将各样品上清液400μL转移到含有100μL 0.5M NaOH的HPLC小瓶中,如下所述通过HPAEC测定剩余底物的浓度。
酶的纯化
将转化酶表达质粒的酿酒酵母菌株在缺少组氨酸的oMM中生长到对数后期,离心收集菌体。将酵母细胞沉淀重悬于含有50mM Tris-HCl、100mM NaCl、0.5mM DTT(pH 7.4)和蛋白酶抑制剂混合物(Pierce)的缓冲液中。将细胞用Avestin匀浆机裂解,将澄清的上清液加载到HisTrap柱(GE Healthcare)上。组氨酸标记的酶用咪唑梯度纯化,将缓冲液置换为20mM Tris-HCl、100mM NaCl(pH 7.4),并浓缩至5mg/mL。
酶的测定
对于木寡糖底物的GH43-2β-木糖苷酶的测定:将0.5μM纯化的酶与0.1%自制的木寡糖或1mM木二糖(Megazyme)在30℃,1×PBS中孵育。在30分钟时取样,并加入5倍体积的0.1mM NaOH灭活。如下所述通过HPAEC分析产物。对于pH分析,加入0.1M的浓度pH 4.0、4.5、5.0、5.5、6.0的醋酸盐缓冲液和6.5、7.0、7.5、8的磷酸盐缓冲液。对于木糖甙-木糖醇底物的GH43-2和GH43β-木糖苷酶的测定:将10μM纯化的酶与4.5mM木糖甙-木糖醇和0.5mM木二糖在20mM MES缓冲液(pH 7.0)和1mM CaCl2在30℃孵育。在3小时取样,并通过在99℃加热10分钟灭活。如下所述通过离子排阻HPLC分析产物。
对于树干毕赤酵母木糖还原酶XR和粗糙脉孢霉木糖还原酶XR的测定:在30℃下将1μM纯化的酶与0.06%木寡糖和2mM NADPH(在1×PBS中)一起孵育。在30分钟时取样,并通过在99℃下加热10分钟灭活。如下所述通过LC-QToF分析产物。
寡糖制备
木寡糖购自Cascade Analytical Reagents and Biochemicals公司,或根据公开的流程(Akpinar et al.,2009),稍作修改制备。简言之,将20g榉木木聚糖(Sigma-Aldrich)完全悬浮在1000mL水中,向其中加入13.6mL 18.4M H2SO4。将混合物在150℃油浴中连续搅拌孵育。30分钟后,将反应物倒入冰上的2L塑料容器中,同时搅拌以使其冷却。此后,缓慢加入0.25mol CaCO3以中和pH并沉淀硫酸盐。将上清液过滤并在旋转蒸发器上50℃浓缩至干。自制的木寡糖含有约30%的木糖单体和70%的低聚物。为了获得较大部分的短链木寡糖,将商业木寡糖按20%质量体积比(重量/体积)溶解,并与2mg/mL木聚糖酶在37℃下孵育48小时。使用前进行热失活和过滤。
(木糖甙)n-木糖醇纯化
从含有质粒pXD8.4的菌株SR8的木寡糖培养液中纯化木糖甙-木糖醇和寡聚体。将50mL培养物上清液在50℃的旋转蒸发器上浓缩至约5mL。将过滤的样品加载到填充有SupelcleanTM ENVI-CarbTM填料(Sigma-Aldrich)的XK 16/70柱子上(GE Healthcare),用Purifier(GE Healthcare)进行纯化。在室温下用乙腈梯度以3.0mL/min的流速洗脱柱。将通过LC-MS验证的纯化组分合并并浓缩。最终的产品,含有90%木糖甙-木糖醇和10%木二糖用于酶活测定的底物和HPLC标准品。
酵母在木寡糖上培养
好氧条件下,将酵母菌株在含有2%葡萄糖的oMM培养基中预培养过夜,用水洗涤3次,并重悬于oMM培养基中。对于好氧生长,菌株按起始OD600为1.0或20接种到50mL含4%w/v(重量/体积)木寡糖的oMM培养基的250mL锥形瓶中,并用4层miracl布覆盖,220rpm振荡培养。对于微好氧生长,菌株按起始OD600 80接种到50mL含4%w/v(重量/体积)木寡糖的oMM培养基的125mL锥形瓶中,并用4层miracl布覆盖,100rpm振荡培养。在指定的时间点,取出0.8mL样品并离心。取20μL上清液通过离子排阻HPLC分析测定木糖、木糖醇、甘油和乙醇浓度。25μL 1:200稀释或2μL 1:100稀释的上清液分别通过HPAEC或LC-QToF分析以测定木寡糖浓度。
图13B中,具体地,将工程化的酵母菌株在含有2%葡萄糖的oMM培养基中好氧预培养过夜,用水洗涤3次,并重悬于oMM培养基中。不含碳源的oMM培养基每升含有1.7g YNB(不含硫酸铵,Sigma,Y1251)、2倍适当的CSM无氮混合物(MP Biomedicals)、10g(NH4)2SO4,1g MgSO4.7H2O、6g KH2PO4、100mg腺嘌呤半硫酸盐、10mg肌醇,以及另外的100mg谷氨酸、20mg赖氨酸和375mg丝氨酸。然后将细胞按起始OD600为80接种到50mL含有4%w/v(重量/体积)木寡糖的oMM培养基的125mL锥形瓶中,4层miracl布覆盖,100rpm振荡培养。在指定的时间点,取出0.8mL样品并离心。通过LC-QToF质谱鉴定和测量XOS消耗和(木糖甙)n-木糖醇生产。在第5天结束时,消耗了93%的木二糖和71%的木三糖。木糖甙-木糖醇和木二糖甙-木糖醇的产率分别为90%和54%。
离子排阻HPLC分析
在装有示差检测器的Shimadzu Prominence HPLC仪上进行离子排阻HPLC。样品在离子排阻柱(Aminex HPX-87H柱,300×7.8mm,Bio-Rad)上使用流动相为0.01N H2SO4,在50℃条件下,以0.6mL/min的流速分离。
HPACE分析
在30℃下使用CarboPac PA200分析柱(150×3mm)和CarboPac PA200保护柱(3×30mm)在ICS-3000HPLC仪(Thermo Fisher)上进行HPAEC分析。在注射25μL稀释的样品后,用0.1M NaOH在乙酸钠梯度的流动相中以0.4mL/min进行洗脱。对于木寡糖和木糖甙-木糖醇分离,乙酸盐梯度为:0mM 1分钟,在8分钟内增加至80mM,在1分钟内增加至300mM,保持30mM 2分钟,然后在0mM下重新平衡3分钟。使用脉冲电流检测器(PAD)检测碳水化合物,并使用Chromeleon软件包分析和定量峰。
质谱分析
所有质谱分析在Agilent 1200 LC-Agilent 6520 Accurate-Mass Q-TOF上进行,使用的柱子是100×7.8mm Rezex RFQ-Fast Fruit H+8%柱(Phenomenex),以0.5%甲酸为流动相,在55℃条件下,以0.3mL/min的流速分离样品。
为了确定未知代谢物的精确质量,取2μL 1:100稀释的酵母培养物上清液进行LC-QTOF分析。使用氮气为载气。在负离子模式下,源电压(Vcap)为3,000V,碰撞电压为100V。干燥气温度为300℃;干燥气流量为7L/min;雾化器压力为45psi。ESI离子源使用单独的雾化器持续、低量地引入参考质量化合物(112.985587,1033.988109)以维持质量轴的校准。以1Hz采集速率收集数据,扫描范围m/z 50至1100,以棒状图形式存储。
以LC-MS/MS确认木糖甙-木糖醇和木二糖甙-木糖醇。保留时间(RT)为5.8分钟且m/z比为283.103的化合物和RT为4.7分钟且m/z比为415.15的化合物用10、20和40eV的碰撞量碎片化。获得MS/MS谱图,产物离子与ChemBioDraw Ultra v13中的碎裂工具产生的计算碎片离子进行比较和匹配。
为了定量培养物中的碳水化合物和碳水化合物衍生物,将培养物上清液用水中稀释100倍,取2μL通过LC-QToF分析。将谱图导入到Agilent MassHunter Workstation软件的定性分析模块中,使用从校准样品获得的m/z和保留时间值来搜索数据中的目标离子。这些搜索基于目标化合物列表生成提取离子色谱图(EIC)。将峰进行积分并与标准曲线比较以计算浓度。从oMM培养基中制备的校准样品(与所有样品一样)计算出校准曲线,并且对于每个化合物的曲线拟合R2为0.999。以4-吗啉乙磺酸(MES)为内标(IS)进行浓度校准,MES化合物在oMM培养基中具有恒定浓度且不会被酵母利用。
系统发育分析
使用BLAST(Altschul et al.,1997)将各自的序列与NCBI蛋白数据进行查询,找到GH43-2和GH43-7的同源物。选择来自多样化分类的、具有代表性的序列用MUSCLE(Edgar et al.,2004)算法进行比对。使用MEGA v6.05(Tamura et al.,2013)软件,根据Jones-Taylor-Thornton比对模型构建了最大可能性进化树。使用Phyre2服务器对GH43-2和GH43-7进行同源建模。
结果
申请人对探索木寡糖代谢的生物学方法感兴趣。利用生物法从植物生物质生产生物燃料和可再生化学品,需要经济的方式将来自植物细胞壁的复杂碳水化合物聚合物转化为可被微生物发酵的单糖(Carroll et al.,2009;Chundawat et al.,2011)。在目前的工业方法中,植物细胞壁中的两种主要多糖,纤维素和半纤维素(Somerville et al.,2004)通常分别加工成葡萄糖和木糖的单体(Chundawat et al.,2011)。然而,除了对生物质的苛刻的预处理之外,还需要大量的纤维素酶和半纤维素酶混合物以从植物细胞壁中释放单糖,导致许多未解决的经济和后勤的挑战(Chundawat et al.,2011;Himmel et al.,2007;Jarboe et al.,2010;Lynd et al.,2002)。生物乙醇工业目前使用酵母酿酒酵母将来自玉米淀粉或蔗糖发酵成乙醇(Hong et al.,2012),但是酿酒酵母需要大量的改造才能发酵来源于植物细胞壁的糖,例如纤维二糖和木糖(Hong et al.,2012;Ha et al.,2011;Kuyper et al.,2005;Young et al.,2014;Jeffries et al.,2006)。
与酿酒酵母相反,许多纤维素分解真菌包括粗糙脉孢霉(Tian et al.,2009)在植物细胞壁的纤维素和半纤维素组分上生长良好。如图12所示,多种分子组分参与纤维素和半纤维素作为碳源的转运和利用,包括用于将纤维糊精和/或木寡糖运输到细胞中的转运蛋白、细胞内β-葡糖苷酶和β-木糖苷酶分别水解纤维糊精和木寡糖、以及促进这些水解产物作为碳源的利用的各种组分。对于木糖,它是木寡糖水解的产物,木糖还原酶(XR),木糖醇脱氢酶(XDH)和木酮糖激酶(XK)可以利用木糖。
如上所述,来自植物细胞壁的纤维糊精和木寡糖不能被野生型酿酒酵母分解代谢(Galazka et al.,2010;Young et al.,2010;Matsushika et al.,2009)。之前发现,在酿酒酵母中重构来自粗糙脉孢霉的纤维糊精转运和消耗途径,使得该酵母能够发酵纤维二糖(Galazka et al.,2010)。此外,酿酒酵母之前被工程化改造,通过引入木糖异构酶(XI)或木糖还原酶(XR)和木糖醇脱氢酶(XDH)消耗木糖(Jeffries et al.,2006;Matsushika et al.,2009)。申请人推断表达来自粗糙脉孢霉的、具有功能的木寡糖转运和消耗系统,可以进一步扩大酿酒酵母利用植物来源的木寡糖作为碳源的能力。
木寡糖运输和利用菌株的构建
申请人感兴趣于在酿酒酵母中构建功能性木寡糖转运和消耗系统,并且开始研究粗糙脉孢霉中木寡糖转运的可能机制。申请人分析了粗糙脉孢霉的转录分析数据(Tian et al.,2009)和敲除菌株生长表型,鉴定了粗糙脉孢霉消耗由其分泌酶释放的纤维糊精和木寡糖的单独途径(Galazka et al.,2010)。缺失了先前鉴定的纤维糊精转运蛋白(CDT-2,NCU08114)的菌株(Galazka et al.,2010)不能在木聚糖上生长,并且木寡糖留在培养物上清液中,表明CDT-2可能是木糖转运蛋白。作为CDT-2的转运功能的直接测试,表达cdt-2的酿酒酵母菌株能够导入木二糖、木三糖和木四糖(图1)。与CDT-1一样,CDT-2的直系同源物广泛分布在真菌界(Galazka et al.,2010),这表明许多真菌消耗来自植物细胞壁的木寡糖。
在运输进入细胞后,木寡糖的水解将允许它们进一步用作碳源。值得注意的是,当在木聚糖上生长时,粗糙脉孢霉能表达假定的细胞内β-木糖苷酶GH43-2(NCU01900)(Sun et al.,2012)。申请人发现纯化的GH43-2对于具有2至8的聚合度(DP)和最适pH 7附近的木寡糖显示出很强的水解酶活性。此外,如同细胞内β-glucosidases(Galazka et al.,2010)一样,细胞内β-木糖苷酶也广泛存在于真菌中(Sun et al.,2012)。
申请人因此鉴定了可将木寡糖转运到细胞中的蛋白(CDT-2)和可水解木寡糖的蛋白(GH43-2)。为了测试酿酒酵母是否可以利用木寡糖,酿酒酵母菌株工程改造了源自树干毕赤酵母的XR/XDH途径进行工程改造,该途径类似于粗糙脉孢霉中使用的途径(Sun et al.,2012),还对酿酒酵母菌株工程改造了源自粗糙脉孢霉的木寡糖转运和消耗途径。表达来自粗糙脉孢霉的CDT-2、来自粗糙脉孢霉的胞内β-木糖苷酶GH43-2和来自树干毕赤酵母的XR和XDH的木糖利用酵母,能够直接利用具有2或3个DP(聚合度)的木寡糖(图2A)。对照野生型酿酒酵母菌株不能直接利用木寡糖(图3A和3C)。类似地,工程改造了木糖利用途径而不是木寡糖转运途径的酿酒酵母菌株不能代谢木寡糖(图3B和3D)。
工程菌株生产木糖甙-木糖醇寡聚物
进行与图2A相似的实验,除了使用更高的细胞密度培养物(OD600=20)(见图2B)。值得注意的是,工程化酵母的高细胞密度培养物能够消耗高达DP 5的木寡糖,但是木糖水平保持很高(图2B),这表明在工程化酵母中存在未知的瓶颈。这些结果与之前尝试工程化酿酒酵母用于木寡糖消耗的那些结果类似,那些都报道了木糖在培养基中积累(Fujii et al.,2011)。申请人关注为什么在该培养物中木寡糖浓度降低,但木糖浓度随时间保持相对稳定(不降低)。不希望受理论束缚,认为来自树干毕赤酵母的木糖还原酶会将木糖(由来自粗糙脉孢霉的β-木糖苷酶GH43-2水解木寡糖产生的)转化为木糖醇,然后木糖醇将进一步代谢。在这点上,认为木糖将被消耗,而不是如所观察到的在该培养物中积累。申请人试图探索这个未知的瓶颈并分析培养物上清液的内容物。对来自具有木寡糖的培养物的上清液的分析意外地揭示,通过工程化酵母将木寡糖转化成木糖甙-木糖醇寡聚物,一组以前未知的代谢中间体,而不是水解成木糖并消耗(图4A和4B)。所得木糖甙-木糖醇寡聚物是有效的末端产物,不能被该工程酵母菌株进一步代谢。
申请人发现XR/XDH/CDT-2/GH43-2酿酒酵母菌株能产生木糖甙-木糖醇寡聚物是令人惊讶的,因为之前认为该菌株将有效地将木寡糖代谢成木糖用于消耗。此外,这个令人惊讶的发现可能是非常有益的。不希望受理论束缚,认为木糖甙-木糖醇寡聚物可用于各种目的,例如用作甜味剂,用作益生元或作为抗感染药物。因此,申请人希望更好地理解工程化的酵母菌株中木糖甙-木糖醇寡聚物的产生。
木糖醇寡聚体生产的研究机制
为了测试木糖甙-木糖醇寡聚物产生是否由木寡糖与酿酒酵母内源酶的副反应导致,将两种单独的酵母菌株在组合培养物中孵育:一种菌株含有由CDT-2和GH43-2组成的木寡糖水解途径,另一种菌株用XR/XDH木糖消耗途径工程改造。表达CDT-2和GH43-2的菌株能够将木寡糖切割成木糖,然后被分泌并用作表达木糖消耗途径(XR和XDH)的菌株的碳源。相比之下,表达XR和XDH但不是木寡糖转运和水解途径(CD-2和GH43-2)的工程酵母将仅能够消耗木糖。当这些菌株共培养时,发现木寡糖被消耗而不产生木糖甙-木糖醇副产物(图5)。X2和X3减少,并且产生木糖醇单体,但是值得注意的是,未检测到木糖甙-木糖醇寡聚物。这些结果表明酵母内源性的酶和GH43-2的转糖酵解活性不负责产生木糖甙-木糖醇副产物,表明它们必须由木糖还原酶(XR)产生。在这种情况下该酶来自树干毕赤酵母。
纯化的木糖还原酶催化由木寡糖底物制备木糖甙-木糖醇寡聚物
不希望受理论束缚,认为如果木糖甙-木糖醇副产物由来自树干毕赤酵母的XR产生,则类似的副产物可能由粗糙脉孢霉中的XR产生。为了直接测试木糖还原酶从木寡糖底物产生木糖甙-木糖醇寡聚物的能力,用来自粗糙脉孢霉的木糖还原酶XYR-1进行体外分析。如图6A和6B所示,当与聚合度2至4的木寡糖底物一起孵育时,XYR-1能够直接产生具有聚合度达3(xlt3)的木糖甙-木糖醇寡聚物。
β-木糖苷酶GH43-7水解木糖甙-木糖醇寡聚物
从上面在图6中描述的实验,发现来自粗糙脉孢霉的木糖还原酶XYR-1(NCU08384)可以在体外由木寡糖产生木糖甙-木糖醇产物。然而,当在培养物中生长时,在粗糙脉孢霉培养基中没有木糖甙-木糖醇副产物积聚(图7),表明在粗糙脉孢霉中存在能够代谢木糖甙-木糖醇寡聚物的内源途径。发现在木聚糖上生长的粗糙脉孢霉,其假定的第二个β-木糖苷酶表达上调。第二个β-木糖苷酶GH43-7(NCU09625)(Sun et al.,2012)具有很弱的β-木糖苷酶活性,但却能快速水解木糖甙-木糖醇转化成木糖和木糖醇(图8A和8B)。尽管该酶的活性从木糖消耗的观点来看是有益的,但是该蛋白的活性不允许木糖甙-木糖醇寡聚物的积累。然而,不希望受理论束缚,因为该蛋白(GH43-7)已显示具有直接木糖甙-木糖醇水解酶活性,申请人相信,降低该蛋白或其它具有相似功能的蛋白的活性或敲除该蛋白或其它具有相似功能的蛋白将导致木糖甙-木糖醇寡聚物的重新的积累。
进一步的实验
为了进一步研究由各种工程化酵母菌株产生的各种产物,进行了一系列发酵实验。图9A显示野生型酿酒酵母SR8酵母、转化了CDT-2和GH43-2(pXD8.4)的SR8酵母、转化了CDT-2和GH43-7(pXD8.6)的SR8酵母或转化CDT-2、GH43-2和GH43-7(pXD8.7)的SR8酵母在以木寡糖作为唯一碳源生长时的生长速度。发现工程化的酵母菌株与野生型相比生长具有一些改善。具有pXD8.4的菌株最类似于野生型,推测是因为该菌株积累木糖甙-木糖醇寡聚物而不是利用来自木寡糖水解的木糖。图9B、9C和9D显示了在微好氧条件下,指定的酵母菌株在木寡糖上生长,起始培养物OD600=20时,木寡糖和木糖甙-木糖醇寡聚物水平。仅在具有质粒pXD8.4的SR8菌株中观察到木糖甙-木糖醇积累。图9E、9F和9G显示了在微好氧条件下,指定的酵母菌株在木寡糖上生长,起始培养物OD600=80时,木寡糖和木糖甙-木糖醇寡聚物水平。同样,只有具有pXD8.4的菌株可以积累木糖甙-木糖醇寡聚物。类似的结果在图10中给出,但分析了不同的木寡糖及其衍生物。如图9H所示,具有pXD8.7的菌株是最有效的乙醇生产菌株,该菌株不能积累木糖甙-木糖醇寡聚物。结果进一步证实,木糖还原酶能够从木寡糖底物中产生木糖甙-木糖醇寡聚物,并且粗糙脉孢霉酶GH43-7具有木糖甙-木糖醇水解酶活性并且能够代谢木糖甙-木糖醇寡聚物。
为了进一步研究工程化的酵母产生木糖甙-木糖醇寡聚物的能力,进行了另外的实验。其中一个实验,类似于图9和图10,稍有不同,例如更长的孵育时间和对不同的木糖甙-木糖醇产物分析。实验中,使用具有XR/CDT-2/GH43-2的酿酒酵母菌株的,起始培养物OD600为80。如图13B所示,在第5天结束时,消耗了93%的木二糖和71%的木三糖。木糖甙-木糖醇和木二糖甙-木糖醇的产率分别为90%和54%。图13A显示了由木寡糖底物产生木糖甙-木糖醇寡聚物的可能机制。
还进行具有XR/CDT-2/GH43-2的酿酒酵母菌株的发酵延长到第10天的实验。这与先前的发酵相反,之前第6天或更早终止。如图14所示,在培养物中生长10天后,具有木寡糖转运蛋白和木糖还原酶的工程化酵母能够产生聚合度高达6的木糖甙-木糖醇寡聚物,虽然速率较低,并且最终产率低于观察到的较短的寡聚体,例如木二糖甙-木糖醇。
还进行实验以进一步研究其它β-木糖苷酶的潜在木糖甙-木糖醇水解酶活性。如图11所示,发现枯草芽孢杆菌和大肠杆菌都具有与GH43-7同源的蛋白。为了观察这些蛋白是否实际上具有木糖甙-木糖醇水解酶活性,将纯化的枯草芽孢杆菌GH43-7和纯化的大肠杆菌GH43-7与木二糖(X2)或木糖甙-木糖醇(xlt2)一起孵育,并分析它们对这些底物的活性。如图15所示,枯草芽孢杆菌GH43-7和大肠杆菌GH43-7都对木糖甙-木糖醇底物表现出木糖甙-木糖醇水解酶活性。这两种蛋白还表现出对木二糖底物的β-木糖苷酶活性。因此,这些蛋白是表现出β-木糖苷酶活性(在木寡糖底物上)和木糖甙-木糖醇水解酶活性(在木糖甙-木糖醇寡聚物底物上)的β-木糖苷酶的实例。这与粗糙脉孢霉GH43-7的β-木糖苷酶相反,其表现出弱的β-木糖苷酶活性(在木寡糖底物上),但具有强的木糖甙-木糖醇活性(在木糖甙-木糖醇寡聚物底物上)。总之,测试了来自细菌枯草芽孢杆菌(AAB41091.1)和大肠杆菌(WP_000406872.1)的两种GH43酶并确认它们能切割木糖甙-木糖醇。
讨论
申请人发现广泛用于工业微生物木糖醇生产的真菌木糖还原酶同样会减少低聚木糖(XOS)。产物是木糖甙-木糖醇寡聚物((木糖甙)n-木糖醇),这是一组以前不知道是木寡糖代谢中间体的化合物。木糖、XOS,木糖醇和(木糖甙)n-木糖醇的结构描述于图16中。结果表明能够作用于木寡糖(XOS)的木糖还原酶可以用于木糖甙-木糖醇寡聚物的生产。
此外,新鉴定的木糖甙-木糖醇-特异性β-木糖苷酶GH43-7广泛分布在真菌和细菌中(图11)。这些结果揭示了广泛使用的XR/XDH途径天然具有对木寡糖的宽底物特异性,并且真菌和细菌可以使用平行的细胞内β-木糖苷酶水解木寡糖和木糖甙-木糖醇以产生单体木糖和木糖醇。在该实施例中描述的木寡糖转运和代谢途径的总结提供在图12中。为了改善从具有木糖还原酶活性的细胞中用木寡糖底物生产木糖甙-木糖醇寡聚物,应当降低具有木糖甙-木糖醇水解酶活性的蛋白的活性,或完全从细胞中除去,以允许木糖甙-木糖醇寡聚物的积累。
实施例2:具有木寡糖还原酶活性的木糖还原酶的建模
本实施例描述了模拟作用于木寡糖底物的木糖还原酶的建模实验。
结果
从实施例1,申请人发现并提供了显示具有木寡糖还原酶活性的木糖还原酶的实例。传统上认为木糖还原酶催化木糖转化为木糖醇,但申请人已经表明,这些酶还可以具有木寡糖还原酶活性,可以催化低聚木糖(木寡糖)转化成木糖甙-木糖醇寡聚物。
为了进一步探索木糖还原酶的木糖还原酶催化活性,申请人进行了建模实验。在该研究中,分析了假丝酵母木糖还原酶(CtXR,AAC25601.1)(Kavanagh et al.,2002)的结构,CtXR是木干毕赤酵母蛋白XYL1(SsXR)的高度同源物。回想实施例1,SsXR是发现具有木寡糖还原酶活性的木糖还原酶。
CtXR含有开放的活性位点空腔,可与木糖结合,位于NADH辅因子的结合位点附近(Kavanagh et al.,2002;Kratzer et al.,2006)。申请人发现,特别地,活性位点的开放形状可以很容易地适应较长的木寡糖底物的结合(图17)。使用计算对接算法(Trott et al.,2010),发现木二糖在口袋中很好地适应。此外,在蛋白中似乎没有阻止更长的木寡糖寡聚物结合的任何阻碍物。
总之,申请人已经使用计算对接分析来显示木糖还原酶(XR)通常具有开放的活性位点,可以容易地适应作为底物的木寡糖。不希望受理论束缚,认为具有可容纳作为底物木寡糖的活性位点的木糖还原酶将表现出木寡糖还原酶活性。此外,并且不希望受理论束缚,认为这有助于解释实施例1中木糖甙-木糖醇寡聚物积累的令人惊奇的发现,因为所分析的木糖还原酶可能能够适应木寡糖作为底物,通过木寡糖还原酶活性产生木糖甙-木糖醇寡聚物。
实施例3:在酵母中扩展木糖代谢用于植物细胞壁转化为生物燃料
本实施例详述了实施例1和2中提供的实验、数据和信息。用可再生生物质生产可持续生物燃料,将需要有效和完全利用植物细胞壁中的所有丰富的糖。使用纤维素分解真菌粗糙脉孢霉作为模型,申请人已经鉴定了其在半纤维素上生长所需的木寡糖运输和消耗途径。在酿酒酵母中重建这种木寡糖利用途径揭示了真菌木糖还原酶作为木寡糖还原酶,能产生木糖甙-木糖醇寡聚物作为代谢中间体。然后通过两种水解酶将木寡糖和木糖甙-木糖醇寡聚物水解以产生细胞内木糖和木糖醇。在蔗糖和葡萄糖的共发酵中使用木寡糖转运蛋白、木寡糖还原酶和串联的胞内水解酶的木寡糖消耗极大地扩展了酵母使用植物细胞壁衍生的糖的能力,并且具有提高第一代和第二代生物燃料生产效率的潜力。
材料和方法
粗糙脉孢霉菌株
粗糙脉孢霉菌株来自真菌遗传学库存中心(FGSC)(McCluskey,Wiest,and Plamann 2010),包括WT(FGSC 2489)和两种寡糖转运蛋白的缺失菌株:NCU00801(FGSC 16575)和NCU08114(FGSC 17868)(Colot et al.,2006)。
粗糙脉孢霉生长测定
将分生孢子以106个/mL浓度接种到24深孔板中,每个孔有3mL含有2%w/v(重量/体积)奇岗草粉(Energy Bioscience Institute,UC-Berkeley)、Avicel PH 101(Sigma)、榉木木聚糖(Sigma)或果胶(Sigma)的Vogel培养基(Vogel,1956)。将板用Corning TM透气密封带密封,并在恒定光照、25℃条件下振动(200rpm)培养。在48小时时拍摄图像。如下所述,在Dionex高效阴离子交换色谱(HPAEC)分析之前,将培养物上清液用0.1M NaOH稀释200倍。
质粒和酵母菌株
按(http://www.fgsc.net/fgn35/lee35.pdf)和Harju等人所述提取粗糙脉孢霉WT菌株(FGSC 2489)和酿酒酵母S288C菌株的模板基因组DNA(gDNA)(McCluskey and Plamann,2010)。从粗糙脉孢霉gDNA模板扩增β-木糖苷酶基因NCU01900和NCU09652(分别为GH43-2和GH43-7)的开放阅读框(ORF)。为方便生化分析,每个ORF的C末端与6个组氨酸标签融合,并与2μ酵母质粒pRS423骨架中的酿酒酵母PTEF1启动子和CYC1转录终止子侧接。质粒pRS426_NCU08114先前已有描述(Galazka et al.,2010)。含有树干毕赤酵母木糖利用途径(木糖还原酶、木糖醇脱氢酶和木酮糖激酶)的质粒pLNL78来自John Dueber的实验室(Latimer et al.,2014)。通过使用In-Fusion克隆试剂盒(Clontech)将NCU08114(CDT-2)和NCU01900(GH43-2)表达框整合到pLNL78中构建质粒pXD2,即单一质粒形式的木寡糖途径。质粒pXD8.4,衍生自质粒pRS316(Sikorski and Hieter,1989)用于表达带有PCCW12启动子的CDT-2和GH43-2。质粒pXD8.6,衍生自pXD8.4,用GH43-7的ORF替代GH43-2的ORF。pXD8.7包含使用各自的PCCW12启动子的所有三种表达框(CDT-2,GH43-2和GH43-7)。酿酒酵母菌株D452-2(MATa leu2his3ura3can1)(Kurtzman et al.,1994)和SR8U(进化的木糖快速利用菌株SR8的尿嘧啶自养型)(Kim et al.,2013)用于酵母实验的受体菌株。从粗糙脉孢霉gDNA扩增粗糙脉孢霉木糖还原酶(xyr-1,NcXR)的ORF,通过重叠PCR除去内含子。XR ORF C-末端与6个组氨酸标签融合,并与酿酒酵母PCCW12启动子和CYC1转录终止子侧接,插入质粒pRS313中。表1列出了本研究中使用的质粒。
表1:质粒列表
基于酵母细胞的木寡糖摄取测定
酿酒酵母在缺乏尿嘧啶的优化基础培养基(oMM)中生长至对数期后期。oMM含有1.7g/L YNB(Sigma,Y1251),2倍适当的CSM无氮源混合物,10g/L(NH4)2SO4,1g/L MgSO4.7H2O,6g/L KH2PO4,100mg/L,100mg/L腺嘌呤半硫酸盐,10mg/L肌醇,100mg/L谷氨酸,20mg/L赖氨酸,375mg/L丝氨酸和100mM 4-吗啉乙磺酸(MES),pH 6.0(Lin et al.,提交)。然后收获细胞并用测定缓冲液(5mM MES,100mM NaCl,pH 6.0)洗涤三次,并重悬至最终OD600为40。用相同测定缓冲液制备浓度为200μM的底物母液。通过混合等体积的细胞悬浮液和底物母液开始转运的测定。反应在30℃下连续振荡孵育30分钟。将样品在4℃以14,000rpm离心5分钟除去酵母细胞。将各样品上清液400μL转移到含有100μL 0.5M NaOH的HPLC小瓶中,如下所述通过HPAEC测定剩余底物的浓度。
酶的纯化
将转化酶表达质粒的酿酒酵母菌株在缺少组氨酸的oMM中生长到对数后期,离心收集菌体。将酵母细胞沉淀重悬于含有50mM Tris-HCl、100mM NaCl、0.5mM DTT(pH 7.4)和蛋白酶抑制剂混合物(Pierce)的缓冲液中。将细胞用Avestin匀浆机裂解,将澄清的上清液加载到HisTrap柱(GE Healthcare)上。组氨酸标记的酶用咪唑梯度纯化,将缓冲液置换为20mM Tris-HCl、100mM NaCl(pH 7.4),并浓缩至5mg/mL。
酶的测定
对于木寡糖底物的GH43-2β-木糖苷酶的测定:将0.5μM纯化的酶与0.1%自制的木寡糖或1mM木二糖(Megazyme)在30℃,1×PBS中孵育。在30分钟时取样,并加入5倍体积的0.1mM NaOH灭活。如下所述通过HPAEC分析产物。对于pH分析,加入0.1M的浓度pH 4.0、4.5、5.0、5.5、6.0的醋酸盐缓冲液和6.5、7.0、7.5、8的磷酸盐缓冲液。对于木糖甙-木糖醇底物的GH43-2和GH43β-木糖苷酶的测定:将10μM纯化的酶与4.5mM木糖甙-木糖醇和0.5mM木二糖在20mM MES缓冲液(pH 7.0)和1mM CaCl2在30℃孵育。在3小时取样,并通过在99℃加热10分钟灭活。如下所述通过离子排阻HPLC分析产物。
对于树干毕赤酵母木糖还原酶XR和粗糙脉孢霉木糖还原酶XR的测定:在30℃下将1μM纯化的酶与0.06%木寡糖和2mM NADPH(在1×PBS中)一起孵育。在30分钟时取样,并通过在99℃下加热10分钟灭活。如下所述通过LC-QToF分析产物。
寡糖制备
木寡糖购自Cascade Analytical Reagents and Biochemicals公司,或根据公开的流程(Akpinar et al.,2009),稍作修改制备。简言之,将20g榉木木聚糖(Sigma-Aldrich)完全悬浮在1000mL水中,向其中加入13.6mL 18.4M H2SO4。将混合物在150℃油浴中连续搅拌孵育。30分钟后,将反应物倒入冰上的2L塑料容器中,同时搅拌以使其冷却。此后,缓慢加入0.25mol CaCO3以中和pH并沉淀硫酸盐。将上清液过滤并在旋转蒸发器上50℃浓缩至干。自制的木寡糖含有约30%的木糖单体和70%的低聚物。为了获得较大部分的短链木寡糖,将商业木寡糖按20%质量体积比(重量/体积)溶解,并与2mg/mL木聚糖酶在37℃下孵育48小时。使用前进行热失活和过滤。
从含有质粒pXD8.4的菌株SR8的木寡糖培养液中纯化木糖甙-木糖醇和寡聚体。将50mL培养物上清液在50℃的旋转蒸发器上浓缩至约5mL。将过滤的样品加载到填充有SupelcleanTM ENVI-CarbTM填料(Sigma-Aldrich)的XK 16/70柱子上(GE Healthcare),用Purifier(GE Healthcare)进行纯化。在室温下用乙腈梯度以3.0mL/min的流速洗脱柱。将通过LC-MS验证的纯化组分合并并浓缩。最终的产品,含有90%木糖甙-木糖醇和10%木二糖用于酶活测定的底物和HPLC标准品。
真菌和枯草芽孢杆菌木糖甙-木糖醇生产的测量
粗糙脉孢霉菌株(FGSC 2489)和构巢曲霉储存并分生在含2%葡萄糖的Volgel培养基(Vogel,1956)的琼脂斜面上。里氏木霉(菌株QM6a)在马铃薯葡萄糖琼脂(PDA)平板上分生孢子。通过重悬浮于水中收集每种真菌的分生孢子,并以10 6个/mL细胞的浓度接种。粗糙脉孢霉和构巢曲霉接种到含有2%木寡糖的Volgel培养基中。里氏木霉接种到含有2%木寡糖的木霉基本培养基(et al.,1987)中。粗糙脉孢霉、构巢曲霉和里氏木霉分别在25℃、37℃和30℃下在摇瓶中生长。40小时后,收集2mL培养物的菌丝体,并在玻璃纤维过滤器上用水洗涤,并转移至预冷的具有螺旋盖的2mL试管中。试管中含有0.5mL Zirconia氧化锆珠(0.5mm)和1.2mL酸性乙腈提取溶液(80%乙腈,20%H2O和0.1M甲酸)(Rabinowitz和Kimball 2007),然后将试管插入液氮中。收集过程控制在30秒内,将样品保持在-80℃直至如下所述提取。
枯草芽孢杆菌储存在0.5×LB(1%胰蛋白胨,0.5%酵母提取物和0.5%NaCl)琼脂平板上。将单个菌落接种到含有1%葡萄糖的0.5×LB液体培养基中,37℃过夜振荡培养。将过夜培养物接种到具有1%木聚糖的新鲜0.5×LB液体培养基中,初始OD600为0.2。40小时后,取2mL培养物离心并用冷PBS溶液洗涤。向细胞沉淀中加入Zirconia氧化锆珠和酸性乙腈提取溶液。试管立即快速冷冻管并保持在-80℃直至提取。
为了提取,使所有样品在4℃解冻10分钟,珠磨(bead beat)2分钟,并在4℃涡旋20分钟。取每个样品的50μL上清液通过LC-MS/MS(参见质谱分析部分)分析。
酵母在木寡糖上好氧培养
酵母菌株在含有2%葡萄糖的oMM培养基中好氧预培养过夜,用水洗涤3次,并重悬于oMM培养基中。对于好氧生长,将细胞按起始OD600为1.0接种到50mL含有3%w/v(重量/体积)木寡糖的oMM培养基的250mL锥形瓶中,4层miracl布覆盖,220rpm振荡培养。在指定的时间点,取出0.8mL样品并离心。取20μL上清液通过离子排阻HPLC分析测定木糖、木糖醇、甘油和乙醇浓度。25μL 1:200稀释或2μL 1:100稀释的上清液分别通过HPAEC或LC-QToF分析以测定木寡糖浓度。
补料分批厌氧发酵
在1L搅拌罐生物反应器(DASGIP生物反应器系统,DGCS4型,Eppendorf AG)中进行厌氧发酵实验,培养基为含有3%w/v(重量/体积)木寡糖的oMM培养基,接种的初始细胞浓度OD600=20。在30℃温度,发酵时间107小时,培养物搅拌转速200rpm下,并用6L/h的氮气恒定地吹扫。对于木糖和木寡糖共发酵,以0.8mL/h连续流加25%木糖母液。在发酵期间,每4小时用自动进样器通过陶瓷取样针(Seg-Flow Sampling System,Flownamics)取3mL无细胞样品。取20μL上清液通过离子排阻HPLC分析测定木糖、木糖醇、甘油和乙醇浓度。2μL 1:100稀释的上清液通过LC-QToF分析以测定木寡糖浓度。对于葡萄糖和木寡糖共发酵,以2mL/h连续流加10%葡萄糖母液。如木糖和木寡糖共发酵所述检测分析物,增加培养基中葡萄糖浓度的测定。
木糖发酵比较
为了对菌株SR8(XR/XDH)和SXA-R2P-E(XI)之间的木糖发酵进行比较,首先建立YPD上生长的每个菌株每OD600的克细胞干重,发现两株菌相当,对于菌株SR8为0.433±0.01g/OD,对于菌株SXA-R2P-E为0.423±0.01g/OD。将酵母菌株SR8或SXA-R2P-E在含有2%葡萄糖的oMM或YSC培养基中好氧预培养至对数中期或后期,用水洗涤3次,根据分批发酵情况,重悬于oMM或YSC培养基中。用如图所示的一系列起始OD值接种到含有4%w/v(重量/体积)木糖的培养基中,然后用N2吹扫。在装有50mL的oMM或YSC培养基的125mL血清瓶中进行发酵,在30℃摇床中以220rpm振荡培养。在指定的时间点,用注射器取出1mL样品并离心。取5μL上清液通过离子排阻HPLC分析测定木糖、木糖醇、甘油和乙醇浓度。
蔗糖与木寡糖的共发酵
将含有质粒pXD8.7的酵母菌株SR8U在缺乏尿嘧啶并含有2%葡萄糖的oMM培养基中好氧预培养至对数后期,用水洗涤,并重悬于oMM培养基中。用20OD洗涤的酵母种子培养物接种到含有75g/L蔗糖加或减15g/L木寡糖的培养基中,并用N2吹扫。在装有50mL的oMM或YSC培养基的125mL血清瓶中进行发酵,在30℃摇床中以220rpm振荡培养。在指定的时间点,用注射器取出1mL样品并离心。取5μL上清液通过离子排阻HPLC分析测定木糖、木糖醇、甘油和乙醇浓度。如下所述,取2μL 1:100稀释的上清液通过LC-QToF分析以测定木寡糖浓度。
离子排阻HPLC分析
在装有示差检测器的Shimadzu Prominence HPLC仪上进行离子排阻
HPLC。样品通过Rezex RFQ-Fast Fruit H+8%柱(100×7.8mm Phenomenex),在50℃条件下,以1mL/min的流速分离。木寡糖发酵样品在Aminex HPX-87H柱(300×7.8mm,Bio-Rad)上使用流动相为0.01N H2SO4,在50℃条件下,以0.6mL/min的流速分离。
HPACE分析
在30℃下使用CarboPac PA200分析柱(150×3mm)和CarboPac PA200保护柱(3×30mm)在ICS-3000HPLC仪(Thermo Fisher)上进行HPAEC分析。在注射25μL稀释的样品后,用0.1M NaOH在乙酸钠梯度的流动相中以0.4mL/min进行洗脱。对于木寡糖和木糖甙-木糖醇分离,乙酸盐梯度为:0mM 1分钟,在8分钟内增加至80mM,在1分钟内增加至300mM,保持30mM 2分钟,然后在0mM下重新平衡3分钟。使用脉冲电流检测器(PAD)检测碳水化合物,并使用Chromeleon软件包分析和定量峰。
质谱分析
所有质谱分析在Agilent 1200 LC-Agilent 6520 Accurate-Mass Q-TOF上进行,使用的柱子是100×7.8mm Rezex RFQ-Fast Fruit H+8%柱(Phenomenex),以0.5%甲酸为流动相,在55℃条件下,以0.3mL/min的流速分离样品。
为了确定未知代谢物的精确质量,取2μL 1:100稀释的酵母培养物上清液进行LC-QTOF分析。使用氮气为载气。在负离子模式下,源电压(Vcap)为3,000V,碰撞电压为100V。干燥气温度为300℃;干燥气流量为7L/min;雾化器压力为45psi。ESI离子源使用单独的雾化器持续、低量地引入参考质量化合物(112.985587,1033.988109)以维持质量轴的校准。以1Hz采集速率收集数据,扫描范围m/z 50至1100,以棒状图形式存储。
以LC-MS/MS确认木糖甙-木糖醇和木二糖甙-木糖醇。保留时间(RT)为5.8分钟且m/z比为283.103的化合物和RT为4.7分钟且m/z比为415.15的化合物用10、20和40eV的碰撞量碎片化。获得MS/MS谱图,产物离子与ChemBioDraw Ultra v13中的碎裂工具产生的计算碎片离子进行比较和匹配。
为了定量培养物中的碳水化合物和碳水化合物衍生物,将培养物上清液用水中稀释100倍,取2μL通过LC-QToF分析。将谱图导入到Agilent MassHunter Workstation软件的定性分析模块中,使用从校准样品获得的m/z和保留时间值来搜索数据中的目标离子。这些搜索基于目标化合物列表生成提取离子色谱图(EIC)。将峰进行积分并与标准曲线比较以计算浓度。从oMM培养基中制备的校准样品(与所有样品一样)计算出校准曲线,并且对于每个化合物的曲线拟合R2为0.999。以4-吗啉乙磺酸(MES)为内标(IS)进行浓度校准,MES化合物在oMM培养基中具有恒定浓度且不会被酵母利用。
结果
利用生物法从植物生物质生产生物燃料和可再生化学品,需要经济的方式将来自植物细胞壁的复杂碳水化合物聚合物转化为可被微生物发酵的单糖(Carroll et al.,2009;Chundawat et al.,2011)。在目前的工业方法中,植物细胞壁中的两种主要多糖,纤维素和半纤维素(Somerville et al.,2004)通常分别加工成葡萄糖和木糖的单体(Chundawat et al.,2011)。然而,除了对生物质的苛刻的预处理之外,还需要大量的纤维素酶和半纤维素酶混合物以从植物细胞壁中释放单糖,导致许多未解决的经济和后勤的挑战(Chundawat et al.,2011;Himmel et al.,2007;Jarboe et al.,2010;Lynd et al.,2002)。生物乙醇工业目前使用酵母酿酒酵母将来自玉米淀粉或蔗糖发酵成乙醇(Hong et al.,2012),但是酿酒酵母需要大量的改造才能发酵来源于植物细胞壁的糖,例如纤维二糖和木糖(Kuyper et al.,2005;Jeffries,2006;van Maris et al.,2007;Ha et al.,2011;Hong and Nielsen,2012;Young et al.,2014)。
与酿酒酵母相反,许多纤维素分解真菌包括粗糙脉孢霉(Tian et al.,2009)在植物细胞壁的纤维素和半纤维素组分上自然生长良好。通过分析粗糙脉孢霉的转录分析数据(Tian et al.,2009)和敲除菌株生长表型,申请人鉴定了粗糙脉孢霉消耗由其分泌酶释放的纤维糊精(Galazka et al.,2010)和木寡糖的单独途径(图18A和18B)。缺失了先前鉴定的纤维糊精转运蛋白(CDT-2,NCU08114)的菌株(Galazka et al.,2010)不能在木聚糖上生长(图19),并且木寡糖留在培养物上清液中(见图17)。作为CDT-2的转运功能的直接测试,表达cdt-2的酿酒酵母菌株能够导入木二糖、木三糖和木四糖(图1)。值得注意的是,当在木聚糖上生长时,粗糙脉孢霉能表达假定的细胞内β-木糖苷酶GH43-2(NCU01900)(Sun et al.,2012)。纯化的GH43-2对于具有2至8的聚合度(DP)和最适pH 7附近的木寡糖显示出很强的水解酶活性(图20A、20B)。在(Cai et al.,2014)中独立获得CDT-2和GH43-2的结果。与cdt-1一样,cdt-2的直系同源物广泛分布在真菌界(Galazka et al.,2010),这表明许多真菌消耗来自植物细胞壁的木寡糖。此外,如同细胞内β-glucosidases(Galazka et al.,2010)一样,细胞内β-木糖苷酶也广泛存在于真菌中(Sun et al.,2012)(图21)。
来自植物细胞壁的纤维糊精和木寡糖不能被野生型酿酒酵母分解代谢(Matsushika et al.,2009;Galazka et al.,2010;Young and Alper,2010)。在酿酒酵母中重建来自粗糙脉孢霉的纤维糊精运输和消耗途径使得该酵母能够发酵纤维二糖(Galazka et al.,2010)。因此,申请人推断,表达来自粗糙脉孢霉的、具有功能的木寡糖转运和消耗系统,可以进一步扩大酿酒酵母利用植物来源的木寡糖作为碳源的能力。之前,酿酒酵母通过工程改造引入木糖异构酶(XI)或木糖还原酶(XR)和木糖醇脱氢酶(XDH)消耗木糖(Jeffries,2006;van Maris et al.,2007;Matsushika et al.,2009)。为了测试酿酒酵母是否可以利用木寡糖,酿酒酵母菌株工程改造了源自树干毕赤酵母的XR/XDH途径进行工程改造,该途径类似于粗糙脉孢霉中使用的途径(Sun et al.,2012),还对酿酒酵母菌株工程改造了源自粗糙脉孢霉的木寡糖转运(CDT-2)和消耗途径(GH43-2)。表达来自粗糙脉孢霉的CDT-2、来自粗糙脉孢霉的胞内β-木糖苷酶GH43-2和来自树干毕赤酵母的XR和XDH的木糖利用酵母,能够直接利用具有2或3个DP(聚合度)的木寡糖(见图2A、图3A-3D)。
值得注意的是,尽管工程化酵母的高细胞密度培养物能够消耗高达DP 5的木寡糖,但是木糖水平保持很高(见图2B),这表明在工程化酵母中存在许多未知的瓶颈。这些结果与之前尝试工程化酿酒酵母用于木寡糖消耗的那些结果类似,那些都报道了木糖在培养基中积累(Fujii et al.,2011)。对表达CDT-2,GH43-2和树干毕赤酵母XR/XDH途径的酵母菌株的培养物的上清液的分析令人惊讶地揭示,木寡糖被转化成木糖甙-木糖醇寡聚物,一组以前未知的代谢中间体,而不是水解成木糖并消耗(见图16、图4A-4B、图22)。所得木糖甙-木糖醇寡聚物是有效的末端产物,不能被进一步代谢。
由于之前没有报道作为中间代谢物木糖甙-木糖醇寡聚物产生,需要检测木糖甙-木糖醇寡聚物生成中涉及的分子成分。为了测试木糖甙-木糖醇寡聚物产生是否由木寡糖与酿酒酵母内源酶的副反应导致,申请人将两种单独的酵母菌株在组合培养物中孵育:一种菌株含有由CDT-2和GH43-2组成的木寡糖水解途径,另一种菌株用XR/XDH木糖消耗途径工程改造。表达CDT-2和GH43-2的菌株能够将木寡糖切割成木糖,然后木糖可以通过内源性转运蛋白分泌(Hamacher et al.,2002),并作为表达木糖消耗途径(XR和XDH)的菌株的碳源。表达XR和XDH的工程化酵母能够消耗木糖(见图2A)。当共培养时,这些菌株消耗木寡糖而不产生木糖甙-木糖醇副产物(见图5)。这些结果表明内源性酵母酶和GH43-2的转糖酵解活性不负责产生木糖甙-木糖醇副产物,意味着它们一定由来自树干毕赤酵母(SsXR)的XR产生的。
真菌木糖还原酶如SsXR已经在工业上广泛用于木糖发酵。然而,尚未建立XR活性位点与底物结合的结构细节。为了探索低聚木寡糖的XR还原的分子基础,分析了与SsXR高度同源的假丝酵母木糖还原酶(CtXR)(Kavanagh et al.,2002)的结构。CtXR含有开放的活性位点空腔,位于NADH辅因子的结合位点附近,可与木糖结合,(Kavanagh et al.,2002;Kratzer et al.,2006)。值得注意的是,活性位点的开放形状可以容易地适应较长的木寡糖底物的结合(图17)。使用计算对接算法(Trott and Olson,2010),发现木二糖在口袋中很好地适应。此外,在蛋白中没有阻止更长的木寡糖低聚物结合的阻碍(图17)。
申请人推断,如果木糖甙-木糖醇副产物是由类似于来自树干毕赤酵母的真菌XR产生的,则应在粗糙脉孢霉中产生类似的副产物,从而需要额外的途径来消耗它们。与该假设一致,来自粗糙脉孢霉的木糖还原酶XYR-1(NCU08384)也能从木寡糖产生木糖甙-木糖醇产物(见图6A-6B)。然而,当粗糙脉孢霉在木聚糖上生长时,培养基中并没有木糖甙-木糖醇副产物积聚(图7)。因此,粗糙脉孢霉中想必存在能消耗木糖甙-木糖醇寡聚物的额外酶活性。与该假说一致,发现在木聚糖上生长的粗糙脉孢霉,其假定的第二个β-木糖苷酶表达上调。第二个β-木糖苷酶GH43-7(NCU09625)(Sun et al.,2012)具有很弱的β-木糖苷酶活性,但却能快速水解木糖甙-木糖醇转化成木糖和木糖醇(图8A和8B)。新鉴定的β-木糖苷酶GH43-7,能够代谢木糖甙-木糖醇,广泛分布在真菌和细菌中(见图11、图23),表明其被各种微生物用于消耗木寡糖。实际上,来自细菌枯草芽孢杆菌和大肠杆菌的GH43-7能切割木寡糖和木糖甙-木糖醇(见图15)。
为了测试木糖甙-木糖醇是否通常是微生物在半纤维素上生长产生的中间代谢物,申请人提取并分析了许多在木寡糖上生长的子囊菌物和枯草芽孢杆菌的代谢物。值得注意的是,当在木寡糖上生长时,这些广泛分歧的真菌和枯草芽孢杆菌都能产生木糖甙-木糖醇(图24A和图25)。这些生物跨越了10亿年的进化(图24B),表明使用木寡糖还原酶消耗植物半纤维素是广泛的。
接下来测试完整的木寡糖消耗途径的整合是否将克服酿酒酵母很差的木寡糖利用能力(见图2A-2B)(Fujii et al.,2011)。当与原始的木寡糖途径(CDT-2加GH43-2)组合时,GH43-7使得酿酒酵母在木寡糖上更快地生长(见图9A),并消除木糖甙-木糖醇中间体的积累(见图9B-9D、图10和图26)。木糖和葡萄糖的存在极大地改善了木寡糖的厌氧发酵(图27A-27B、图28和图29A-29B),表明具有完全木寡糖途径的酿酒酵母中的代谢途径可能还需要进行附加的调节(Youk and van Oudenaarden,2009)才能用于最优的木寡糖发酵。总之,这些结果揭示了广泛用于工程化酿酒酵母中的XR/XDH途径天然具有对木寡糖的宽底物特异性,并且完全重构天然存在的木寡糖途径使得酿酒酵母能有效地消耗木寡糖。
已经有人提出将木糖发酵为生物燃料的最佳方法是使用XI而不是XR/XDH途径(van Maris et al.,2007;Zhou et al.,2012;Lee and Alper,2014)。尽管XI可以原则上替代用于产生细胞内木酮糖的木寡糖消耗途径中的XR/XDH部分,但是未见发表工程改造使用XI或XR/XDH的最佳酵母菌株的明确比较(Karhumaa et al.,2007)。因此,申请人在厌氧木糖发酵实验中,比较了在公共领域中表现最佳的两种工程酵母菌株:使用XR/XDH的菌株SR8(Kim et al.,2013)和使用XI的菌株SXA-R2P-E(Lee and Alper,2014)。与表达XI的菌株SXA-R2P-E相比(图30、表2),在一系列细胞起始接种浓度(OD600 2.5-20)下,表达XR/XDH的菌株SR8显示出更高的乙醇生产速率。无论对于从对数中期或后期培养物制备的种子以及在两种不同的已知成分的培养基中结果都是一样的(Lin et al.,2014;Lee and Alper,2014)(图31)。相比之下,菌株SXA-R2P-E具有略微更高的乙醇产率,并且产生更少的木糖醇副产物(图30、表2和图31)。值得注意的是,当木寡糖用于发酵时,XR/XDH途径比木糖产生少得多的木糖醇(图27A-27B和图29B)。因此,通过XR/XDH途径的木寡糖消耗可以导致酵母菌株乙醇生产率和产率都很高,而没有木糖醇形成。
表2:两种利用木糖的酿酒酵母菌株的发酵性能比较
1在分批条件下使用三角瓶和40g/L木糖进行厌氧发酵。
2木糖或乙醇的克数/g细胞干重/小时的最大值。
3g乙醇产量/g木糖。
葡萄糖能促进木葡聚糖发酵,这个观察结果(图27B)表明,木寡糖途径可以更普遍地用于共发酵以增强生物燃料生产。因此,测试木寡糖发酵是否可以与蔗糖发酵同时进行,作为增加甘蔗乙醇产量的手段。在这种情况下,通过热水处理释放的木寡糖(Hendriks and Zeeman,2009;Agbor et al.,2011;Evangelina et al.,2012)可以加入到经过木寡糖消耗途径工程改造的酵母的蔗糖发酵中。为了测试这一点,申请人蔗糖和木寡糖的组合发酵中使用了含有木寡糖途径(CDT-2、GH43-2和GH43-7)工程改造的菌株SR8。申请人观察到蔗糖和木寡糖的同时发酵,且乙醇产量增加(图33)。值得注意的是,如同在葡萄糖的共发酵中观察到的现象(图27B),蔗糖共发酵也同样发现木糖醇产生水平低(图33)。
使用酵母作为示例性平台,申请人鉴定了粗糙脉孢霉(图12)中的木寡糖消耗途径,令人惊讶地,涉及到一种由许多真菌和细菌广泛产生的新的代谢中间体。在细菌如枯草芽孢杆菌中,木糖甙-木糖醇可以通过已知具有宽底物特异性的醛-酮还原酶产生(Barski et al.,2008)。在纤维素分解真菌中发现的木寡糖消耗途径和纤维消耗途径(Galazka et al.,2010),可以降解植物细胞壁的两种主要的糖组分,为工程改造酵母发酵植物生物质来源的糖,提供了许多模式(图12)。XR/XDH途径还可为来自半纤维素的糖的发酵提供显著的优点。乙酰化的半纤维素的分解(Sun et al.,2012)释放高毒性乙酸盐,降低酿酒酵母发酵的性能(Bellissimi et al.,2009;Sun et al.,2012)。由XR/XDH途径产生的额外还原力(最初被认为是一个问题)可用于驱动乙酸盐的还原,从而使发酵培养基解毒并增加乙醇产量(Wei et al.,2013)。
通过优化,新鉴定的木寡糖消耗途径提供了将玉米淀粉或甘蔗为原料的第一代生物乙醇生产扩展为包括来自植物细胞壁的半纤维素的新机会。例如,如本文所述的实验,申请人提出通过使用压缩热水处理从甘蔗渣中的半纤维素释放的木寡糖(Hendriks and Zeeman,2009;Agbor et al.,2011;Evangelina et al.,2012),可以被经工程改造、消耗木寡糖的酵母直接发酵(图33)。结合葡萄糖或纤维糊精消耗的木寡糖消耗(图12)还可以改进木质纤维素原料生产二代生物燃料遇到的需要许多预处理的问题(Hendriks and Zeeman,2009;Evangelina et al.,2012)。这些途径可以广泛用于克服使用可持续和经济来源的木质纤维素原料发酵生产生物燃料和可再生化学品的所剩余的瓶颈。
参考文献
D.C.Savage,Microbial ecology of the gastrointestinal tract.Annu Rev Microbiol 31,107(1977).
M.J.Vazquez,J.L.Alonso,H.Dominguez,J.C.Parajo,Xylooligosaccharides:manufacture and applications.Trends Food Sci Tech 11,387(Nov,2000).
A.A.Aachary,S.G.Prapulla,Xylooligosaccharides(XOS)as an Emerging Prebiotic:
Microbial Synthesis,Utilization,Structural Characterization,Bioactive Properties,and Applications.Compr Rev Food Sci F 10,2(Jan,2011).
J.Schrezenmeir,M.de Vrese,Probiotics,prebiotics,and synbiotics‐approaching a definition.American Journal of Clinical Nutrition 73,361s(Feb,2001).
G.R.Gibson,M.B.Roberfroid,Dietary modulation of the human colonic microbiota:introducing the concept of prebiotics.J Nutr 125,1401(Jun,1995).
M.Okazaki,S.Fujikawa,N.Matsumoto,Effects of xylooligosaccharide on growth of bifidobacteria.Nippon Eiyo Shokuryo Gakkaishi=Journal of the Japanese Society of Nutrition and Food Science 43,395(1990).
C.K.Hsu,J.W.Liao,Y.C.Chung,C.P.Hsieh,Y.C.Chan,Xylooligosaccharides and fructooligosaccharides affect the intestinal microbiota and precancerous colonic lesion development in rats.J Nutr 134,1523(Jun,2004).
A.Azarpazhooh,H.Limeback,H.P.Lawrence,P.S.Shah,Xylitol for preventing acute otitis media in children up to 12 years of age.Cochrane Database Syst Rev,CD007095(2011).
A.Maguire,A.J.Rugg‐Gunn,Xylitol and caries prevention‐‐is it a magic bullet?Br Dent J 194,429(Apr 26,2003).
K.Dietary prevention of dental caries by xylitol‐clinical effectiveness and safety.J Appl Nutr 44,16(1992).
L.Hyvonen,P.Koivistoinen,F.Voirol,Food Technological Evaluation of Xylitol.Adv Food Res 28,373(1982).
T.B.K.Izumori,M.Leisola,A rare sugar xylitol.Part II:biotechnological production and future applications of xylitol.Applied microbiology and biotechnology 74,273(2007).
A.Carroll,C.Somerville,Cellulosic biofuels.Annual Review of Plant Biology 60,165(2009).
S.P.Chundawat,G.T.Beckham,M.E.Himmel,B.E.Dale,Deconstruction of lignocellulosic biomass to fuels and chemicals.Annual Review of Chemical and Biomolecular Engineering 2,121(2011).
C.Somerville et al.,Toward a systems approach to understanding plant cell walls.Science306,2206(2004).
M.E.Himmel et al.,Biomass recalcitrance:engineering plants and enzymes for biofuels production.Science 315,804(2007).
L.R.Jarboe et al.,Metabolic engineering for production of biorenewable fuels and chemicals:contributions of synthetic biology.Journal of Biomedicine&Biotechnology 2010,761042(2010).
L.R.Lynd,P.J.Weimer,W.H.van Zyl,I.S.Pretorius,Microbial cellulose utilization:fundamentals and biotechnology.Microbiology and Molecular Biology Reviews 66,506(2002).
K.K.Hong,J.Nielsen,Metabolic engineering of Saccharomyces cerevisiae:a key cell factory platform for future biorefineries.Cellular and Molecular Life Sciences 69,2671(2012).
S.J.Ha et al.,Engineered Saccharomyces cerevisiae capable of simultaneous cellobiose and xylose fermentation.Proceedings of the National Academy of Sciences of the United States of America 108,504(2011).
M.Kuyper et al.,Metabolic engineering of a xylose-isomerase-expressing Saccharomyces cerevisiae strain for rapid anaerobic xylose fermentation.FEMS Yeast Research 5,399(2005).
E.M.Young,A.Tong,H.Bui,C.Spofford,H.S.Alper,Rewiring yeast sugar transporter preference through modifying a conserved protein motif.Proceedings of the National Academy of Sciences of the United States of America 111,131(2014).
T.W.Jeffries,Engineering yeasts for xylose metabolism.Current Opinion in Biotechnology 17,320(2006).
C.Tian et al.,Systems analysis of plant cell wall degradation by the model filamentous fungus Neurospora crassa.Proceedings of the National Academy of Sciences of the United States of America 106,22157(2009).
J.M.Galazka et al.,Cellodextrin transport in yeast for improved biofuel production.Science330,84(2010).
J.Sun,C.Tian,S.Diamond,N.L.Glass,Deciphering transcriptional regulatory mechanisms associated with hemicellulose degradation in Neurospora crassa.Eukaryotic Cell 11,482(2012).
E.Young,S.M.Lee,H.Alper,Optimizing pentose utilization in yeast:the need for novel tools and approaches.Biotechnology for Biofuels 3,24(2010).
A.Matsushika,H.Inoue,T.Kodaki,S.Sawayama,Ethanol production from xylose in engineered Saccharomyces cerevisiae strains:current state and perspectives.Applied Microbiology and Biotechnology 84,37(2009).
T.Fujii et al.,Ethanol production from xylo-oligosaccharides by xylose-fermenting Saccharomyces cerevisiae expressing beta-xylosidase.Bioscience,Biotechnology,and Biochemistry 75,1140(2011).
K.McCluskey,The Fungal Genetics Stock Center:from molds to molecules.Advances in Applied Microbiology 52,245(2003).
S.Harju,H.Fedosyuk,K.R.Peterson,Rapid isolation of yeast genomic DNA:Bust n'Grab.BMC Biotechnol 4,8(2004).
Latimer,Luke N,Michael E Lee,Daniel Medina-Cleghorn,Rebecca A Kohnz,Daniel K Nomura,and John E Dueber.2014.“Employing a Combinatorial Expression Approach to Characterize Xylose Utilization in Saccharomyces cerevisiae.”Metabolic Engineering 25(September):20–29.doi:10.1016/j.ymben.2014.06.002.
R.S.Sikorski,P.Hieter,A system of shuttle vectors and yeast host strains designed for efficient manipulation of DNA in Saccharomyces cerevisiae.Genetics 122,19(1989).
C.P.Kurtzman,Molecular taxonomy of the yeasts.Yeast 10,1727(1994).
Kim et al.,Rational and evolutionary engineering approaches uncover a small set of genetic changes efficient for rapid xylose fermentation in Saccharomyces cerevisiae.PloS One 8,e57048(2013).
Lin,Yuping,Kulika Chomvong,Ligia Acosta-Sampson,Raíssa Estrela,Jonathan M Galazka,Soo Rin Kim,Yong-Su Jin,and Jamie HD Cate.2014.“Leveraging Transcription Factors to Speed Cellobiose Fermentation by Saccharomyces cerevisiae.”Biotechnol Biofuels 7(1):126.doi:10.1186/s13068-014-0126-6.
O.Akpinar,K.Erdogan,S.Bostanci,Production of xylooligosaccharides by controlled acid hydrolysis of lignocellulosic materials.Carbohydr Res 344,660(2009).
S.F.Altschul et al.,Gapped BLAST and PSI-BLAST:a new generation of protein database search programs.Nucleic Acids Research 25,3389(1997).
R.C.Edgar,MUSCLE:multiple sequence alignment with high accuracy and high throughput.Nucleic Acids Research 32,1792(2004).
K.Tamura,G.Stecher,D.Peterson,A.Filipski,S.Kumar,MEGA6:Molecular Evolutionary Genetics Analysis version 6.0.Molecular Biology and Evolution 30,2725(2013).
L.A.Kelley,M.J.Sternberg,Protein structure prediction on the Web:a case study using the Phyre server.Nature Protocols 4,363(2009).
K.L.Kavanagh,M.Klimacek,B.Nidetzky,D.K.Wilson,The structure of apo and holo forms of xylose reductase,a dimeric aldo-keto reductase from Candida tenuis.Biochemistry 41,8785-8795(2002).
R.Kratzer,S.Leitgeb,D.K.Wilson,B.Nidetzky,Probing the substrate binding site of Candida tenuis xylose reductase(AKR2B5)with site-directed mutagenesis.The Biochemical journal 393,51-58(2006).
O.Trott,A.J.Olson,AutoDock Vina:improving the speed and accuracy of docking with a new scoring function,efficient optimization,and multithreading.Journal of computational chemistry 31,455-461(2010).
Agbor,V B,N Cicek,R Sparling,A Berlin,and D B Levin.2011.“Biomass Pretreatment:Fundamentals Toward Application.”Biotechnol Adv 29(6):675–85.doi:10.1016/j.biotechadv.2011.05.005.
Barski,Oleg A,Srinivas M Tipparaju,and Aruni Bhatnagar.2008.“The Aldo-Keto Reductase Superfamily and Its Role in Drug Metabolism and Detoxification.”Drug Metabolism Reviews 40(4):553–624.doi:10.1080/03602530802431439.
Bellissimi,Eleonora,Johannes P van Dijken,Jack T Pronk,and Antonius J A van Maris.2009.“Effects of Acetic Acid on the Kinetics of Xylose Fermentation by an Engineered,Xylose-Isomerase-Based Saccharomyces cerevisiae Strain.”FEMS Yeast Research 9(3):358–64.doi:10.1111/j.1567-1364.2009.00487.x.
Cai,Pengli,Ruimeng Gu,Bang Wang,Jingen Li,Li Wan,Chaoguang Tian,and Yanhe Ma.2014.“Evidence of a Critical Role for Cellodextrin Transporte 2(CDT-2)in Both Cellulose and Hemicellulose Degradation and Utilization in Neurospora crassa.”Edited by Michael Freitag.PloS One 9(2):e89330.doi:10.1371/journal.pone.0089330.
Colot,Hildur V,Gyungsoon Park,Gloria E Turner,Carol Ringelberg,Christopher M Crew,Liubov Litvinkova,Richard L Weiss,Katherine A Borkovich,and Jay C Dunlap.2006.“A High-Throughput Gene Knockout Procedure for Neurospora Reveals Functions for Multiple Transcription Factors.”Proceedings of the National Academy of Sciences of the United States of America 103(27):10352–57.doi:10.1073/pnas.0601456103.
Coradetti,Samuel T,James P Craig,Yi Xiong,Teresa Shock,Chaoguang Tian,and N Louise Glass.2012.“Conserved and Essential Transcription Factors for Cellulase Gene Expression in Ascomycete Fungi.”Proceedings of the National Academy of Sciences of the United States of America 109(19):7397–7402.doi:10.1073/pnas.1200785109.
Evangelina Vallejos,Maria,Marcia Dib Zambon,Maria Cristina Area,and Antonio Aprigio da Silva Curvelo.2012.“Low Liquid-Solid Ratio(LSR)Hot Water Pretreatment of Sugarcane Bagasse.”Green Chemistry 14(7):1982–89.doi:10.1039/c2gc35397k.
Galagan,James E,Matthew R Henn,Li-Jun Ma,Christina A Cuomo,and Bruce Birren.2005.“Genomics of the Fungal Kingdom:Insights Into Eukaryotic Biology..”Genome Research 15(12):1620–31.doi:10.1101/gr.3767105.
Hamacher,Tanja,Jessica Becker,Márk Gárdonyi,and Eckhard Boles.2002.“Characterization of the Xylose-Transporting Properties of Yeast Hexose Transporters and Their Influence on Xylose Utilization.”Microbiology(Reading,England)148(Pt 9):2783–88.
Hedges,S Blair,Joel Dudley,and Sudhir Kumar.2006.“TimeTree:a Public Knowledge-Base of Divergence Times Among Organisms.”Bioinformatics(Oxford,England)22(23):2971–72.doi:10.1093/bioinformatics/btl505.
Hendriks,A T,and G Zeeman.2009.“Pretreatments to Enhance the Digestibility of Lignocellulosic Biomass.”Bioresour Technol 100(1):10–18.doi:10.1016/j.biortech.2008.05.027.
Karhumaa,Kaisa,Rosa Garcia Sanchez,and Marie-F Gorwa-Grauslund.2007.“Comparison of the Xylose Reductase-Xylitol Dehydrogenase and the Xylose Isomerase Pathways for Xylose Fermentation by Recombinant Saccharomyces cerevisiae.”Microbial Cell Factories 6(1):5.doi:10.1186/1475-2859-6-5.
Lee,Sun-Mi,Taylor Jellison,and Hal S Alper.2014.“Systematic and Evolutionary Engineering of a Xylose Isomerase-Based Pathway in Saccharomyces cerevisiae for Efficient Conversion Yields.”Biotechnol Biofuels 7(1):122.doi:10.1186/s13068-014-0122-x.
McCluskey,K,A Wiest,and M Plamann.2010.“The Fungal Genetics Stock Center:a Repository for 50 Years of Fungal Genetics Research.”Journal of Biosciences 35(1):119–26.
M,H Nevalainen,ME Salminen,and J Knowles.1987.“A Versatile Transformation System for the Cellulolytic Filamentous Fungus Trichoderma reesei.”Gene 61(2):155–64.
Rabinowitz,Joshua D,and Elizabeth Kimball.2007.“Acidic Acetonitrile for Cellular Metabolome Extraction From Escherichia coli.”Analytical Chemistry 79(16):6167–73.doi:10.1021/ac070470c.
van Maris,Antonius J A,Aaron A Winkler,Marko Kuyper,Wim T A M de Laat,Johannes P van Dijken,and Jack T Pronk.2007.“Development of Efficient Xylose Fermentation in Saccharomyces cerevisiae:Xylose Isomerase as a Key Component.”Advances in Biochemical Engineering/Biotechnology 108(Chapter 57).Berlin,Heidelberg:Springer Berlin Heidelberg:179–204.doi:10.1007/10_2007_057.
Vogel,Henry J.1956.“A Convenient Growth Medium for Neurospora(Medium N).”Microbial Genetics Bulletin 13:42–43.
Wei,Na,Josh Quarterman,Soo Rin Kim,Jamie H D Cate,and Yong-Su Jin.2013.“Enhanced Biofuel Production Through Coupled Acetic Acid and Xylose Consumption by Engineered Yeast.”Nature Communications 4(October).doi:10.1038/ncomms3580.
Wellman,Charles H,Peter L Osterloff,and Uzma Mohiuddin.2003.“Fragments of the Earliest Land Plants.”Nature 425(6955):282–85.doi:10.1038/nature01884.
Xiong,Yi,Samuel T Coradetti,Xin Li,Marina A Gritsenko,Therese Clauss,Vlad Petyuk,David Camp,et al.2014.“The Proteome and Phosphoproteome of Neurospora crassa in Response to Cellulose,Sucrose and Carbon Starvation.”Fungal Genetics and Biology:FG&B 72(November):21–33.doi:10.1016/j.fgb.2014.05.005.
Youk,Hyun,and Alexander van Oudenaarden.2009.“Growth Landscape Formed by Perception and Import of Glucose in Yeast.”Nature 462(7275):875–79.doi:10.1038/nature08653.
Zhou,Hang,Jing-Sheng Cheng,Benjamin L Wang,Gerald R Fink,and Gregory Stephanopoulos.2012.“Xylose Isomerase Overexpression Along with Engineering of the Pentose Phosphate Pathway and Evolutionary Engineering Enable Rapid Xylose Utilization and Ethanol Production by Saccharomyces cerevisiae.”Metabolic Engineering 14(6):611–22.doi:10.1016/j.ymben.2012.07.011.
序列表
<110> 詹姆斯·H·杜德纳·凯特
李欣
<120> 木糖甙木糖醇寡聚物的生产方法
<130> PN1651687PZC
<140> PCT/US2015/037115
<141> 2015-06-23
<150> 62/016,555
<151> 2014-06-24
<160> 12
<170> FastSEQ for Windows Version 4.0
<210> 1
<211> 525
<212> PRT
<213> 粗糙脉孢霉(N. crassa)
<400> 1
Met Gly Ile Phe Asn Lys Lys Pro Val Ala Gln Ala Val Asp Leu Asn
1 5 10 15
Gln Ile Gln Glu Glu Ala Pro Gln Phe Glu Arg Val Asp Trp Lys Lys
20 25 30
Asp Pro Gly Leu Arg Lys Leu Tyr Phe Tyr Ala Phe Ile Leu Cys Ile
35 40 45
Ala Ser Ala Thr Thr Gly Tyr Asp Gly Met Phe Phe Asn Ser Val Gln
50 55 60
Asn Phe Glu Thr Trp Ile Lys Tyr Phe Gly Asp Pro Arg Gly Ser Glu
65 70 75 80
Leu Gly Leu Leu Gly Ala Leu Tyr Gln Ile Gly Ser Ile Gly Ser Ile
85 90 95
Pro Phe Val Pro Leu Leu Thr Asp Asn Phe Gly Arg Lys Thr Pro Ile
100 105 110
Ile Ile Gly Cys Val Ile Met Ile Val Gly Ala Val Leu Gln Ala Thr
115 120 125
Ala Lys Asn Leu Asp Thr Phe Met Gly Gly Arg Thr Met Leu Gly Phe
130 135 140
Gly Asn Ser Leu Ala Gln Ile Ala Ser Pro Met Leu Leu Thr Glu Leu
145 150 155 160
Ala His Pro Gln His Arg Ala Arg Leu Thr Thr Ile Tyr Asn Cys Leu
165 170 175
Trp Asn Val Gly Ala Leu Val Val Ser Trp Leu Ala Phe Gly Thr Asn
180 185 190
Tyr Ile Asn Asn Asp Trp Ser Trp Arg Ile Pro Ala Leu Leu Gln Ala
195 200 205
Phe Pro Ser Ile Ile Gln Leu Leu Gly Ile Trp Trp Val Pro Glu Ser
210 215 220
Pro Arg Phe Leu Ile Ala Lys Asp Lys His Asp Glu Ala Leu His Ile
225 230 235 240
Leu Ala Lys Tyr His Ala Asn Gly Asp Pro Asn His Pro Thr Val Gln
245 250 255
Phe Glu Phe Arg Glu Ile Lys Glu Thr Ile Arg Leu Glu Met Glu Ser
260 265 270
Thr Lys Asn Ser Ser Tyr Leu Asp Phe Phe Lys Ser Arg Gly Asn Arg
275 280 285
Tyr Arg Leu Ala Ile Leu Leu Ser Leu Gly Phe Phe Ser Gln Trp Ser
290 295 300
Gly Asn Ala Ile Ile Ser Asn Tyr Ser Ser Lys Leu Tyr Glu Thr Ala
305 310 315 320
Gly Val Thr Asp Ser Thr Ala Lys Leu Gly Leu Ser Ala Gly Gln Thr
325 330 335
Gly Leu Ala Leu Ile Val Ser Val Thr Met Ala Leu Leu Val Asp Lys
340 345 350
Leu Gly Arg Arg Leu Ala Phe Leu Ala Ser Thr Gly Gly Met Cys Gly
355 360 365
Thr Phe Val Ile Trp Thr Leu Thr Ala Gly Leu Tyr Gly Glu His Arg
370 375 380
Leu Lys Gly Ala Asp Lys Ala Met Ile Phe Phe Ile Trp Val Phe Gly
385 390 395 400
Ile Phe Tyr Ser Leu Ala Trp Ser Gly Leu Leu Val Gly Tyr Ala Ile
405 410 415
Glu Ile Leu Pro Tyr Arg Leu Arg Gly Lys Gly Leu Met Val Met Asn
420 425 430
Met Ser Val Gln Cys Ala Leu Thr Leu Asn Thr Tyr Ala Asn Pro Val
435 440 445
Ala Phe Asp Tyr Phe Gly Pro Asp His Ser Trp Lys Leu Tyr Leu Ile
450 455 460
Tyr Thr Cys Trp Ile Ala Ala Glu Phe Val Phe Val Phe Phe Met Tyr
465 470 475 480
Val Glu Thr Lys Gly Pro Thr Leu Glu Glu Leu Ala Lys Val Ile Asp
485 490 495
Gly Asp Glu Ala Asp Val Ala His Ile Asp Ile His Gln Val Glu Lys
500 505 510
Glu Val Glu Ile His Glu His Glu Gly Lys Ser Val Ala
515 520 525
<210> 2
<211> 318
<212> PRT
<213> 树干毕赤酵母(Scheffersomyces stipitis)
<400> 2
Met Pro Ser Ile Lys Leu Asn Ser Gly Tyr Asp Met Pro Ala Val Gly
1 5 10 15
Phe Gly Cys Trp Lys Val Asp Val Asp Thr Cys Ser Glu Gln Ile Tyr
20 25 30
Arg Ala Ile Lys Thr Gly Tyr Arg Leu Phe Asp Gly Ala Glu Asp Tyr
35 40 45
Ala Asn Glu Lys Leu Val Gly Ala Gly Val Lys Lys Ala Ile Asp Glu
50 55 60
Gly Ile Val Lys Arg Glu Asp Leu Phe Leu Thr Ser Lys Leu Trp Asn
65 70 75 80
Asn Tyr His His Pro Asp Asn Val Glu Lys Ala Leu Asn Arg Thr Leu
85 90 95
Ser Asp Leu Gln Val Asp Tyr Val Asp Leu Phe Leu Ile His Phe Pro
100 105 110
Val Thr Phe Lys Phe Val Pro Leu Glu Glu Lys Tyr Pro Pro Gly Phe
115 120 125
Tyr Cys Gly Lys Gly Asp Asn Phe Asp Tyr Glu Asp Val Pro Ile Leu
130 135 140
Glu Thr Trp Lys Ala Leu Glu Lys Leu Val Lys Ala Gly Lys Ile Arg
145 150 155 160
Ser Ile Gly Val Ser Asn Phe Pro Gly Ala Leu Leu Leu Asp Leu Leu
165 170 175
Arg Gly Ala Thr Ile Lys Pro Ser Val Leu Gln Val Glu His His Pro
180 185 190
Tyr Leu Gln Gln Pro Arg Leu Ile Glu Phe Ala Gln Ser Arg Gly Ile
195 200 205
Ala Val Thr Ala Tyr Ser Ser Phe Gly Pro Gln Ser Phe Val Glu Leu
210 215 220
Asn Gln Gly Arg Ala Leu Asn Thr Ser Pro Leu Phe Glu Asn Glu Thr
225 230 235 240
Ile Lys Ala Ile Ala Ala Lys His Gly Lys Ser Pro Ala Gln Val Leu
245 250 255
Leu Arg Trp Ser Ser Gln Arg Gly Ile Ala Ile Ile Pro Lys Ser Asn
260 265 270
Thr Val Pro Arg Leu Leu Glu Asn Lys Asp Val Asn Ser Phe Asp Leu
275 280 285
Asp Glu Gln Asp Phe Ala Asp Ile Ala Lys Leu Asp Ile Asn Leu Arg
290 295 300
Phe Asn Asp Pro Trp Asp Trp Asp Lys Ile Pro Ile Phe Val
305 310 315
<210> 3
<211> 322
<212> PRT
<213> 粗糙脉孢霉(N. crassa)
<400> 3
Met Val Pro Ala Ile Lys Leu Asn Ser Gly Phe Asp Met Pro Gln Val
1 5 10 15
Gly Phe Gly Leu Trp Lys Val Asp Gly Ser Ile Ala Ser Asp Val Val
20 25 30
Tyr Asn Ala Ile Lys Ala Gly Tyr Arg Leu Phe Asp Gly Ala Cys Asp
35 40 45
Tyr Gly Asn Glu Val Glu Cys Gly Gln Gly Val Ala Arg Ala Ile Lys
50 55 60
Glu Gly Ile Val Lys Arg Glu Glu Leu Phe Ile Val Ser Lys Leu Trp
65 70 75 80
Asn Thr Phe His Asp Gly Asp Arg Val Glu Pro Ile Val Arg Lys Gln
85 90 95
Leu Ala Asp Trp Gly Leu Glu Tyr Phe Asp Leu Tyr Leu Ile His Phe
100 105 110
Pro Val Ala Leu Glu Tyr Val Asp Pro Ser Val Arg Tyr Pro Pro Gly
115 120 125
Trp His Phe Asp Gly Lys Ser Glu Ile Arg Pro Ser Lys Ala Thr Ile
130 135 140
Gln Glu Thr Trp Thr Ala Met Glu Ser Leu Val Glu Lys Gly Leu Ser
145 150 155 160
Lys Ser Ile Gly Val Ser Asn Phe Gln Ala Gln Leu Leu Tyr Asp Leu
165 170 175
Leu Arg Tyr Ala Lys Val Arg Pro Ala Thr Leu Gln Ile Glu His His
180 185 190
Pro Tyr Leu Val Gln Gln Asn Leu Leu Asn Leu Ala Lys Ala Glu Gly
195 200 205
Ile Ala Val Thr Ala Tyr Ser Ser Phe Gly Pro Ala Ser Phe Arg Glu
210 215 220
Phe Asn Met Glu His Ala Gln Lys Leu Gln Pro Leu Leu Glu Asp Pro
225 230 235 240
Thr Ile Lys Ala Ile Gly Asp Lys Tyr Asn Lys Asp Pro Ala Gln Val
245 250 255
Leu Leu Arg Trp Ala Thr Gln Arg Gly Leu Ala Ile Ile Pro Lys Ser
260 265 270
Ser Arg Glu Ala Thr Met Lys Ser Asn Leu Asn Ser Leu Asp Phe Asp
275 280 285
Leu Ser Glu Glu Asp Ile Lys Thr Ile Ser Gly Phe Asp Arg Gly Ile
290 295 300
Arg Phe Asn Gln Pro Thr Asn Tyr Phe Ser Ala Glu Asn Leu Trp Ile
305 310 315 320
Phe Gly
<210> 4
<211> 543
<212> PRT
<213> 粗糙脉孢霉(N. crassa)
<400> 4
Met Pro Leu Val Lys Asn Pro Ile Leu Pro Gly Phe Asn Pro Asp Pro
1 5 10 15
Ser Val Leu Arg Val Gly Gln Asp Tyr Tyr Ile Ala Thr Ser Thr Phe
20 25 30
Glu Trp Tyr Pro Gly Val Gln Ile His His Ser Thr Asp Leu Ala Asn
35 40 45
Trp Thr Leu Leu Thr Arg Pro Leu Asn Arg Lys Ser Gln Leu Asp Met
50 55 60
Arg Gly Asp Pro Asp Ser Cys Gly Val Trp Ala Pro Cys Leu Ser His
65 70 75 80
Asp Gly Asp Lys Phe Trp Leu Val Tyr Thr Asp Val Lys Arg Lys Asp
85 90 95
Gly Ser Phe Lys Asp Ala His Asn Phe Ile Val Trp Ala Asp Lys Ile
100 105 110
Asp Gly Glu Trp Ser Asp Pro Val Tyr Val Asn Ser Ser Gly Phe Asp
115 120 125
Pro Ser Leu Phe His Asp Pro Asp Ser Gly Lys Lys Tyr Phe Val Asn
130 135 140
Met Leu Trp Asp His Arg Arg Arg Pro Leu Leu Phe Ala Gly Ile Ala
145 150 155 160
Val Gln Glu Trp Asp Ala Lys Thr Lys Lys Leu Val Gly Glu Arg Lys
165 170 175
Asn Val Tyr Gln Gly Thr Glu Leu Ala Leu Ala Glu Ala Pro His Val
180 185 190
Tyr Lys Arg Asn Gly Trp Tyr Tyr Leu Leu Ile Ala Glu Gly Gly Thr
195 200 205
Gly Tyr Glu His Ala Cys Thr Leu Ala Arg Ser Lys Asp Ile Trp Gly
210 215 220
Pro Tyr Lys Thr His Pro Glu Lys His Val Leu Thr Ser Lys Asp His
225 230 235 240
Pro Arg Ala Ala Leu Gln Arg Ala Gly His Gly Asp Ile Val Glu Ile
245 250 255
Glu Asp Gly Arg Thr Tyr Leu Val His Leu Thr Gly Arg Pro Thr Thr
260 265 270
Gln Leu Arg Arg Cys Val Leu Gly Arg Glu Thr Ala Ile Gln Glu Cys
275 280 285
Tyr Trp Lys Asp Asp Trp Leu Tyr Val Lys Asn Gly Pro Val Pro Ser
290 295 300
Leu Trp Val Asp Leu Leu Gly Glu Arg Asp Glu Ser Lys Tyr Trp Glu
305 310 315 320
Glu Lys Arg Tyr Thr Phe Lys Asp Gly Leu His Lys Asp Phe Gln Trp
325 330 335
Leu Arg Thr Pro Glu Thr Glu Arg Ile Phe Asn Val Lys Asp Gly Lys
340 345 350
Leu Thr Leu Ile Gly Arg Glu Ala Ile Gly Ser Trp Phe Glu Gln Ala
355 360 365
Leu Val Ala Arg Arg Gln Glu His Phe Ser Tyr Asp Ala Glu Thr Val
370 375 380
Ile Asp Phe Ser Pro Thr Asp Glu Arg Gln Phe Ala Gly Leu Thr Ala
385 390 395 400
Tyr Tyr Cys Arg Tyr Asn Phe Phe Tyr Leu Thr Val Thr Ala His Ser
405 410 415
Asp Gly Gln Arg Glu Leu Leu Ile Met Ser Ser Glu Ala Ser Trp Pro
420 425 430
Ile Gly Asn Leu Asn Thr Pro Tyr Val Pro Tyr Val Gln Ile Pro Asn
435 440 445
Glu Gly Lys Val Lys Leu Ala Leu Thr Ile Arg Gly Asn Gln Leu Gln
450 455 460
Phe Tyr Tyr Ala Val Leu Asp Ser Gly Asp Glu Glu Leu Lys Lys Ile
465 470 475 480
Gly Pro Val Phe Asp Ala Ser Ile Val Ser Asp Glu Cys Gly Gly His
485 490 495
Gln Gln His Gly Ser Phe Thr Gly Ala Phe Val Gly Val Ala Ala Ser
500 505 510
Asp Leu Asn Gly Leu Ala Ala Glu Ala Lys Phe Asp Tyr Leu Leu Tyr
515 520 525
Lys Pro Val Lys Asn Gln Gln Asp Ala Tyr Val Arg Asp Asn Glu
530 535 540
<210> 5
<211> 533
<212> PRT
<213> 枯草芽孢杆菌(Bacillus subtilis)
<400> 5
Met Lys Ile Thr Asn Pro Val Leu Lys Gly Phe Asn Pro Asp Pro Ser
1 5 10 15
Ile Cys Arg Ala Gly Glu Asp Tyr Tyr Ile Ala Val Ser Thr Phe Glu
20 25 30
Trp Phe Pro Gly Val Gln Ile His His Ser Lys Asp Leu Val Asn Trp
35 40 45
His Leu Val Ala His Pro Leu Gln Arg Val Ser Gln Leu Asp Met Lys
50 55 60
Gly Asn Pro Asn Ser Gly Gly Val Trp Ala Pro Cys Leu Ser Tyr Ser
65 70 75 80
Asp Gly Lys Phe Trp Leu Ile Tyr Thr Asp Val Lys Val Val Asp Gly
85 90 95
Ala Trp Lys Asp Cys His Asn Tyr Leu Val Thr Cys Glu Thr Ile Asn
100 105 110
Gly Asp Trp Ser Glu Pro Ile Lys Leu Asn Ser Ser Gly Phe Asp Ala
115 120 125
Ser Leu Phe His Asp Thr Asp Gly Lys Lys Tyr Leu Leu Asn Met Leu
130 135 140
Trp Asp His Arg Ile Asp Arg His Ser Phe Gly Gly Ile Val Ile Gln
145 150 155 160
Glu Tyr Ser Asp Lys Glu Gln Lys Leu Ile Gly Lys Pro Lys Val Ile
165 170 175
Phe Glu Gly Thr Asp Arg Lys Leu Thr Glu Ala Pro His Leu Tyr His
180 185 190
Ile Gly Asn Tyr Tyr Tyr Leu Leu Thr Ala Glu Gly Gly Thr Arg Tyr
195 200 205
Glu His Ala Ala Thr Ile Ala Arg Ser Ala Asn Ile Glu Gly Pro Tyr
210 215 220
Glu Val His Pro Asp Asn Pro Ile Leu Thr Ser Trp His Asp Pro Gly
225 230 235 240
Asn Pro Leu Gln Lys Cys Gly His Ala Ser Ile Val Gln Thr His Thr
245 250 255
Asp Glu Trp Tyr Leu Ala His Leu Thr Gly Arg Pro Ile His Pro Asp
260 265 270
Asp Asp Ser Ile Phe Gln Gln Arg Gly Tyr Cys Pro Leu Gly Arg Glu
275 280 285
Thr Ala Ile Gln Lys Leu Tyr Trp Lys Asp Glu Trp Pro Tyr Val Val
290 295 300
Gly Gly Lys Glu Gly Ser Leu Glu Val Asp Ala Pro Ser Ile Pro Glu
305 310 315 320
Thr Ile Phe Glu Ala Thr Tyr Pro Glu Val Asp Glu Phe Glu Asp Ser
325 330 335
Thr Leu Asn Ile Asn Phe Gln Thr Leu Arg Ile Pro Phe Thr Asn Glu
340 345 350
Leu Gly Ser Leu Thr Gln Ala Pro Asn His Leu Arg Leu Phe Gly His
355 360 365
Glu Ser Leu Thr Ser Thr Phe Thr Gln Ala Phe Val Ala Arg Arg Trp
370 375 380
Gln Ser Leu His Phe Glu Ala Glu Thr Ala Val Glu Phe Tyr Pro Glu
385 390 395 400
Asn Phe Gln Gln Ala Ala Gly Leu Val Asn Tyr Tyr Asn Thr Glu Asn
405 410 415
Trp Thr Ala Leu Gln Val Thr His Asp Glu Glu Leu Gly Arg Ile Leu
420 425 430
Glu Leu Thr Ile Cys Asp Asn Phe Ser Phe Ser Gln Pro Leu Asn Asn
435 440 445
Lys Ile Val Ile Pro Arg Glu Val Lys Tyr Val Tyr Leu Arg Val Asn
450 455 460
Ile Glu Lys Asp Lys Tyr Tyr Tyr Phe Tyr Ser Phe Asn Lys Glu Asp
465 470 475 480
Trp His Lys Ile Asp Ile Ala Leu Glu Ser Lys Lys Leu Ser Asp Asp
485 490 495
Tyr Ile Arg Gly Gly Gly Phe Phe Thr Gly Ala Phe Val Gly Met Gln
500 505 510
Cys Gln Asp Thr Gly Gly Asn His Ile Pro Ala Asp Phe Arg Tyr Phe
515 520 525
Arg Tyr Lys Glu Lys
530
<210> 6
<211> 536
<212> PRT
<213> 大肠杆菌(Escherichia coli)
<400> 6
Met Glu Ile Thr Asn Pro Ile Leu Thr Gly Phe Asn Pro Asp Pro Ser
1 5 10 15
Leu Cys Arg Gln Gly Glu Asp Tyr Tyr Ile Ala Thr Ser Thr Phe Glu
20 25 30
Trp Phe Pro Gly Val Arg Ile Tyr His Ser Arg Asp Leu Lys Asn Trp
35 40 45
Ser Leu Val Ser Thr Pro Leu Asp Arg Val Ser Met Leu Asp Met Lys
50 55 60
Gly Asn Pro Asp Ser Gly Gly Ile Trp Ala Pro Cys Leu Ser Tyr Ala
65 70 75 80
Asp Gly Lys Phe Trp Leu Leu Tyr Thr Asp Val Lys Ile Val Asp Ser
85 90 95
Pro Trp Lys Asn Gly Arg Asn Phe Leu Val Thr Ala Pro Ser Ile Glu
100 105 110
Gly Pro Trp Ser Glu Pro Ile Pro Met Gly Asn Gly Gly Phe Asp Pro
115 120 125
Ser Leu Phe His Asp Asp Asp Gly Arg Lys Tyr Tyr Ile Tyr Arg Pro
130 135 140
Trp Gly Pro Arg His His Ser Asn Pro His Asn Thr Ile Val Leu Gln
145 150 155 160
Ala Phe Asp Pro Gln Thr Gly Thr Leu Ser Pro Glu Arg Lys Thr Leu
165 170 175
Phe Thr Gly Thr Pro Leu Cys Tyr Thr Glu Gly Ala His Leu Tyr Arg
180 185 190
His Ala Gly Trp Tyr Tyr Leu Met Val Ala Glu Gly Gly Thr Ser Tyr
195 200 205
Glu His Ala Val Val Val Leu Arg Ser Lys Asn Ile Asp Gly Pro Tyr
210 215 220
Glu Leu His Pro Asp Val Thr Met Met Thr Ser Trp His Leu Pro Glu
225 230 235 240
Asn Pro Leu Gln Lys Ser Gly His Gly Ser Leu Leu Gln Thr His Thr
245 250 255
Gly Glu Trp Tyr Met Ala Tyr Leu Thr Ser Arg Pro Leu Arg Leu Pro
260 265 270
Gly Val Pro Leu Leu Ala Ser Gly Gly Arg Gly Tyr Cys Pro Leu Gly
275 280 285
Arg Glu Thr Gly Ile Ala Arg Ile Glu Trp Arg Asp Gly Trp Pro Tyr
290 295 300
Val Glu Gly Gly Lys His Ala Gln Leu Thr Val Lys Gly Pro Gln Val
305 310 315 320
Ala Glu Gln Pro Ala Ala Val Pro Gly Asn Trp Arg Asp Asp Phe Asp
325 330 335
Ala Ser Ser Leu Asp Pro Glu Leu Gln Thr Leu Arg Ile Pro Phe Asp
340 345 350
Asp Thr Leu Gly Ser Leu Thr Ala Arg Pro Gly Phe Leu Arg Leu Tyr
355 360 365
Gly Asn Asp Ser Leu Asn Ser Thr Phe Thr Gln Ser Thr Val Ala Arg
370 375 380
Arg Trp Gln His Phe Ala Phe Arg Ala Glu Thr Arg Met Glu Phe Ser
385 390 395 400
Pro Val His Phe Gln Gln Ser Ala Gly Leu Thr Cys Tyr Tyr Asn Ser
405 410 415
Lys Asn Trp Ser Tyr Cys Phe Val Asp Tyr Glu Glu Gly Gln Gly Arg
420 425 430
Thr Ile Lys Val Ile Gln Leu Asp His Asn Val Pro Ser Trp Pro Leu
435 440 445
His Glu Gln Pro Ile Pro Val Pro Glu His Ala Glu Ser Val Trp Leu
450 455 460
Arg Val Asp Val Asp Thr Leu Val Tyr Arg Tyr Ser Tyr Ser Phe Asp
465 470 475 480
Gly Glu Thr Trp His Thr Val Pro Val Thr Tyr Glu Ala Trp Lys Leu
485 490 495
Ser Asp Asp Tyr Ile Gly Gly Arg Gly Phe Phe Thr Gly Ala Phe Val
500 505 510
Gly Leu His Cys Glu Asp Ile Ser Gly Asp Gly Cys Tyr Ala Asp Phe
515 520 525
Asp Tyr Phe Thr Tyr Glu Pro Val
530 535
<210> 7
<211> 9
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<220>
<221> 变体
<222> 2
<223> Xaa = 任何氨基酸
<400> 7
Arg Xaa Leu Tyr Thr Gly Tyr Asp Gly
1 5
<210> 8
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<220>
<221> 变体
<222> 6, 7
<223> Xaa = 任何氨基酸
<400> 8
Gly Phe Gly Asn Ser Xaa Xaa Gln
1 5
<210> 9
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<220>
<221> 变体
<222> 3
<223> Xaa = 任何氨基酸
<400> 9
Leu Leu Xaa His Pro Gln His Arg
1 5
<210> 10
<211> 8
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<220>
<221> 变体
<222> 2
<223> Xaa = Phe或Tyr
<220>
<221> 变体
<222> 3
<223> Xaa = 任何氨基酸
<220>
<221> 变体
<222> 6
<223> Xaa = Arg或Lys
<220>
<221> 变体
<222> 7
<223> Xaa = Asp或Glu
<400> 10
Glu Xaa Xaa Glu Ile Xaa Xaa Thr
1 5
<210> 11
<211> 4
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 11
Trp Ser Gly Leu
1
<210> 12
<211> 6
<212> PRT
<213> 人工序列
<220>
<223> 合成构建体
<400> 12
Gly Pro Thr Leu Glu Glu
1 5
- 还没有人留言评论。精彩留言会获得点赞!