抗人Gas6单克隆抗体的制作方法
[关联申请]本说明书包含作为本申请的优先权基础的美国临时申请us62/066,687(2014年10月21日申请)的说明书记载的内容。本发明涉及:一种单克隆抗体或该抗体片段,其为与人生长停滞特异性蛋白6(hgas6)特异性结合的抗体,与人gas6的第314、315及316位的氨基酸残基中的至少一个氨基酸残基结合;含有编码上述抗体或抗体片段的碱基序列的核酸;含有上述核酸的转化细胞;上述抗体或抗体片段的制造方法;使用上述抗体或抗体片段的gas6的检测或测定用试剂;含有上述抗体或抗体片段作为有效成分的gas6相关疾病的治疗药或诊断药;或者,上述抗体或抗体片段在制造gas6相关疾病的治疗药或诊断药中的应用。
背景技术:
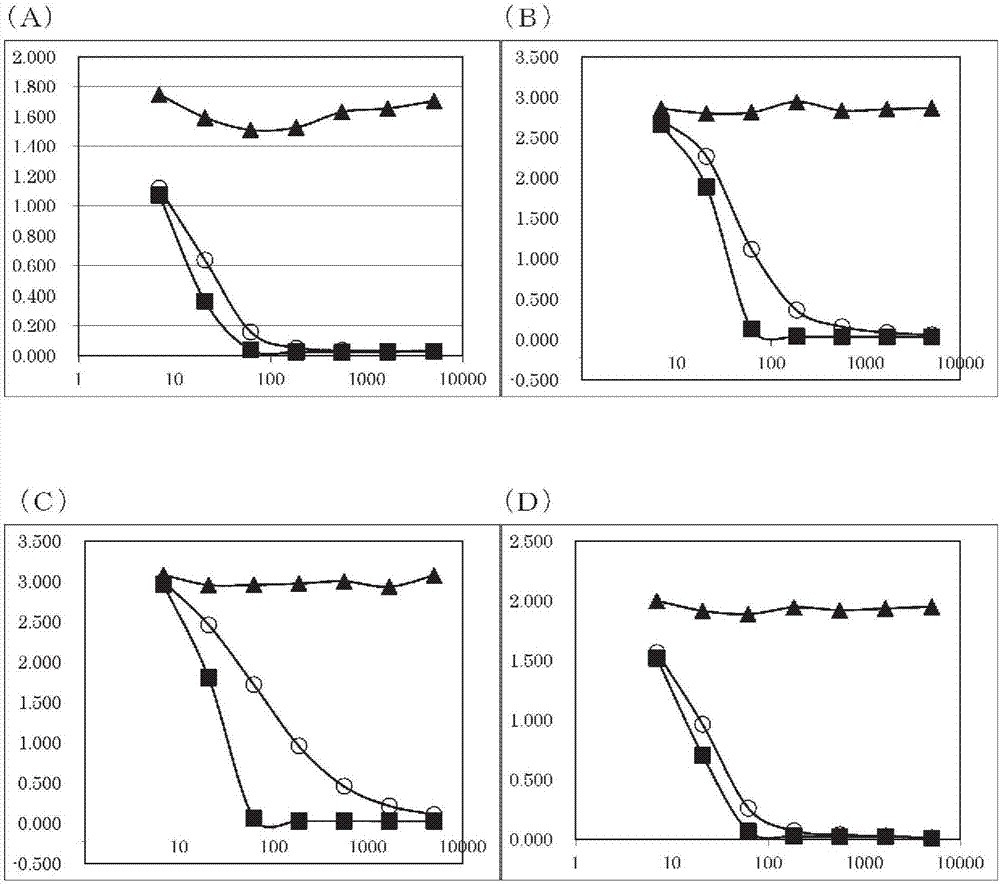
:生长停滞特异性蛋白6(growtharrestspecific6,gas6)基因(也称为axl酪氨酸激酶受体配体(axltyrosinekinasereceptorligand),axllg)作为一种在血清饥饿状态的细胞中表达亢进的基因,于1998年被克隆(非专利文献1)。gas6由678个氨基酸构成,为由三个结构域构成的蛋白,所述三个结构域从n末端侧起为γ-羧基谷氨酸(γ-carboxyglutamicacid,gla)结构域、表皮生长因子(epidermalgrowthfactor,egf)样结构域及含有两个层粘连蛋白g(lamining-type,lg)结构域的性激素结合球蛋白(sexhormonebindingglobulin,shbg)样结构域(非专利文献2)。gas6与蛋白s的氨基酸序列同源性高(44%),与蛋白s同样分类为维生素k依赖性的gla蛋白家族。gla是指谷氨酸残基的γ位的碳被γ谷氨酰羧化酶(ggcx)羧基化的产物(非专利文献3)。未gla化的gas6与gla化的gas6相比,与受体的结合活性下降到十分之一左右(非专利文献4),生理活性也下降。另外已知,c末端的shbg结构域为与受体axl结合的结构域(非专利文献5)。其它的作为维生素k依赖性蛋白质的凝血酶原、第x因子等主要在肝脏中合成,gas6的mrna则在肝脏中几乎检测不到(非专利文献2)。在人体中,在肺、肠、骨髓、内皮中观察到mrna表达,此外,在小鼠中,在心脏、胃、肾脏中确认到mrna的表达。另外,在蛋白质水平,已经确认在人的血浆中以13~23ng/ml存在,且明确了不存在年龄、性别所致的偏差。此外报道了gas6敲除(ko)小鼠小鼠的妊娠、分娩正常,新生仔的体重、大小、繁殖能力没有问题(非专利文献6)。作为gas6的受体,已知有axl(axlreceptortyrosinekinase,axl受体酪氨酸激酶)、sky(rse、tyro3(tyro3proteintyrosinekinase,tyro3蛋白酪氨酸激酶))、mertk(mertyrosinekinaseprotooncogene,mer酪氨酸激酶原癌基因)这3种。这3种受体均为1次跨膜型的酪氨酸激酶,细胞外结构域由两个免疫球蛋白样结构域及随后的两个纤连蛋白-iii样结构域构成(非专利文献7)。gas6与这3种受体的亲和性非常高。虽然各文献所报道的解离常数(kd值)不同,但据报道亲和性高的情况下,axl时为5×10-11m、sky时为3×10-11m、mer时为3×10-10m(非专利文献8)。已知在肾病中,gas6、axl的表达量亢进(非专利文献9、10)。另外,在使用作为进行性肾小球肾炎模型的ntn模型的试验中,确认gas6ko小鼠与野生型小鼠相比病状得到显著抑制。另外确认到,在gas6ko小鼠中得到抑制的病状通过给药gas6而再次恶化(非专利文献11)。进而,在显示出系膜增生性肾小球肾炎样病状的thy1肾炎大鼠的肾脏中,确认到由病状诱发引起的gas6和axl的表达亢进,在对thy1肾炎大鼠给药axl的fc体的实验中,确认到病状大幅改善(非专利文献12)。在作为慢性疾病的i型糖尿病性肾病(stz)模型中,也确认到gas6ko小鼠中病状得到抑制(非专利文献13)。作为通过gas6的中和来抑制肾病的恶化的作用机制,报道了pdgf的表达及肾小球系膜细胞增殖的抑制(非专利文献14)、抗炎症作用(非专利文献15)或抗血小板作用(非专利文献16、17)等。另外,近年,关于gas6及gas6受体与癌的病状的关系也有多篇报道(非专利文献18、19、20、21)。目前为止,作为抗人gas6单克隆抗体,已知有wg1(专利文献1)及cnto300(非专利文献22)。据报道,wg1及cnto300具有在体外抑制gas6与作为其受体的axl结合的活性。另外,其它的gas6中和抗体尚属未知。现有技术文献专利文献专利文献1:us7547767非专利文献非专利文献1:cell54,787-793(1988)非专利文献2:molcellbiol13,4976-4985(1993)非专利文献3:journalofthrombosisandhaemostasis3,1873-1878(2005)非专利文献4:febslett408,306-310(1997)非专利文献5:emboj25,80-87,(2006)非专利文献6:natmed7,215-221,(2001)非专利文献7:jthrombhaemost3,733-741,(2005)非专利文献8:biochemj387,727-735,(2005)非专利文献9:amjkidneydis43,286-295(2004)非专利文献10:transplant27:4166-4172(2012)非专利文献11:jclininvest110,239-246,doi:10.1172/jci14861(2002)非专利文献12:amjpathol158,1423-1432,(2001)非专利文献13:thejournalofbiologicalchemistry278,20,18229-18234(2003)非专利文献14:thejournalofbiologicalchemistry276,45,42364-42369(2001)非专利文献15:blood111,8,4096-4105(2008)非专利文献16:thejournalofclinicalinvestigation115,237-246(2005)非专利文献17:naturemedicine7,2,215-221(2001)非专利文献18:thejournalofclinicalinvestigation123,8,3231-3242(2013)非专利文献19:expertopinthertargets.14,19,1073-1090(2010)非专利文献20:naturegenetics44,8,852-860(2012)非专利文献21:journalofcellularphysiology204,36-44(2005)非专利文献22:biochem.j.,727-735387(2005)技术实现要素:发明所要解决的问题本发明的课题在于,提供一种与人gas6的特定位点特异性结合、具有高中和活性的新的抗人gas6单克隆抗体、以及利用了上述抗体的gas6相关疾病的治疗药及诊断药。用于解决问题的方法作为用于解决上述课题的方法,本发明提供一种与人gas6的第314、315及316位中的至少一个氨基酸残基结合的人gas6单克隆抗体。即,本发明涉及以下的(1)~(19)。(1)一种单克隆抗体或该抗体片段,其与人gas6的第314、315及316位的氨基酸残基中的至少一个氨基酸残基结合。(2)根据(1)所述的单克隆抗体或该抗体片段,其中,单克隆抗体为与人gas6的第314、315及316位的氨基酸残基结合的单克隆抗体。(3)根据(1)或(2)所述的单克隆抗体或该抗体片段,其中,单克隆抗体为选自下述(a)~(e)中的任意一种抗体。(a)抗体的vh的cdr1~3的氨基酸序列分别为序列号79、80及81所述的氨基酸序列、并且vl的cdr1~3的氨基酸序列分别为序列号82、83及84所述的氨基酸序列的抗体。(b)抗体的vh的cdr1~3的氨基酸序列分别为序列号85、86及87所述的氨基酸序列、并且vl的cdr1~3的氨基酸序列分别为序列号88、89及90所述的氨基酸序列的抗体。(c)与上述(a)或(b)所述的抗体竞争性地与人gas6结合的抗体。(d)与含有上述(a)或(b)所述的抗体所结合的表位的表位结合的抗体,即,竞争性地与含有上述(a)或(b)所述的抗体所结合的表位的表位结合的抗体。(e)与和上述(a)或(b)所述的抗体所结合的表位相同的表位结合的抗体,即,竞争性地与上述(a)或(b)所述的抗体所结合的表位结合的抗体。(4)根据(1)~(3)中任一项所述的单克隆抗体或该抗体片段,其中,单克隆抗体为选自下述(a)~(e)中的任意一种抗体。(a)抗体的vh的氨基酸序列为序列号69所述的氨基酸序列、并且vl的氨基酸序列为序列号72所述的氨基酸序列的抗体。(b)抗体的vh的氨基酸序列为序列号75所述的氨基酸序列、并且vl的氨基酸序列为序列号78所述的氨基酸序列的抗体。(c)抗体的vh的氨基酸序列为序列号135所述的氨基酸序列、并且vl的氨基酸序列为序列号123所述的氨基酸序列的抗体。(d)抗体的vh的氨基酸序列为序列号195所述的氨基酸序列、并且vl的氨基酸序列为序列号174所述的氨基酸序列的抗体。(e)抗体的vh的氨基酸序列为序列号186所述的氨基酸序列、并且vl的氨基酸序列为序列号180所述的氨基酸序列的抗体。(5)根据(1)~(4)中任一项所述的单克隆抗体或该抗体片段,其中,单克隆抗体为基因重组抗体。(6)根据(5)所述的单克隆抗体或该抗体片段,其中,基因重组抗体为选自人嵌合抗体、人源化抗体及人抗体中的基因重组抗体。(7)根据(1)~(6)中任一项所述的抗体片段,其中,抗体片段选自fab、fab’、(fab’)2、单链抗体(scfv)、二聚体化v区(双链抗体,diabody)、二硫键稳定的v区(dsfv)及含有cdr的肽。(8)一种核酸,其具有编码(1)~(7)中任一项所述的抗体或该抗体片段的碱基序列。(9)一种转化细胞,其含有(8)所述的核酸。(10)(1)~(7)所述的抗体或该抗体片段的制造方法,其含有如下步骤:在培养基中培养(9)所述的细胞,从培养液收集该抗体或该抗体片段。(11)一种gas6的检测或测定用试剂,其含有(1)~(7)所述的抗体或该抗体片段(根据期望,同时含有药理学上可接受的载体)。(12)一种gas6相关疾病的治疗药,其含有(1)~(7)所述的抗体或该抗体片段作为有效成分(根据期望,同时含有药理学上可接受的载体)。(13)根据(12)所述的治疗药,其中,gas6相关疾病为肾病或癌症。(14)根据(13)所述的治疗药,其中,肾病为进行性肾小球肾炎、系膜增生性肾小球肾炎、糖尿病性肾病或iga肾病。(15)根据(13)所述的治疗药,其中,癌症为肺癌、乳腺癌、卵巣癌、前列腺癌、胰腺癌、肾癌或成胶质细胞瘤。(16)一种gas6相关疾病的诊断药,其含有(1)~(7)所述的抗体或该抗体片段作为有效成分(根据期望,同时含有药理学上可接受的载体)。(17)一种gas6相关疾病的诊断方法,其含有如下步骤:使用(1)~(7)所述的抗体或该抗体片段进行gas6的检测或测定。(18)(1)~(7)中任一项所述的抗体或该抗体片段在制造gas6相关疾病的治疗药中的应用。(19)(1)~(7)中任一项所述的抗体或该抗体片段在制造gas6相关疾病的诊断药中的应用。发明效果本发明的单克隆抗体与人gas6的特定位点特异性结合,抑制gas6与gas6受体的结合,抑制gas6受体表达细胞内的信号转导的活化,或者抑制gas6受体表达细胞的细胞增殖亢进。因此,本发明的单克隆抗体可以作为gas6相关疾病的治疗药及诊断药来利用。附图说明图1为通过elisa测定所得到的抗gas6单克隆抗体与各种抗原的结合性的结果。elisa以n=2来实施,图中示出其平均值。任一图中,纵轴表示吸光度,横轴表示抗体浓度(μg/ml)。作为抗原,(a)使用人gas6-f,(b)使用食蟹猴gas6-f,(c)使用大鼠gas6-f,(d)使用小鼠gas6-f,(e)使用bap-f,(f)使用蛋白s。○为km5320-mkg1抗体的结果,□为km5321-mkg1抗体的结果,▲为cnto抗体的结果。图2是对于抗gas6单克隆抗体评价抑制各种gas6与axl的结合的活性((a)人、(b)大鼠、(c)小鼠、(d)食蟹猴)的结果。实验以n=2来实施,图中示出其平均值。任一图中,纵轴表示吸光度,横轴表示抗体浓度(ng/ml)。○为km5320-mkg1抗体的结果,■为km5321-mkg1抗体的结果,▲为cnto抗体的结果。图3为通过报告基因测定评价抗gas6单克隆抗体对细胞内信号转导的作用的结果。实验以n=3来实施,图中示出其平均值。另外,使用标准偏差(sd)的值作为误差棒。图的纵轴表示发光强度,横轴表示使细胞强制表达的gas6受体。白色柱为km5320-mkg1抗体的结果,带有横线的柱为km5321-mkg1抗体的结果,黑色柱为同种型对照的结果,带有斜线的柱为仅培养基的结果。图4为通过蛋白质印迹评价抗gas6单克隆抗体对于由人gas6引起的akt磷酸化水平亢进的作用的结果。关于图中的记作hgas6的项目,添加0.1μg/ml的hgas6时以+表示,未添加hgas6时以-表示。图中的记作ab的项目表示添加于孔中的受试物质。km5320表示km5320-mkg1抗体,km5321表示km5321-mkg1抗体,同种型表示igg1同种型对照(阴性对照),axl-hfc表示axl受体的细胞外结构域和人igg1抗体的fc区的融合蛋白(阳性对照)。记载于各受试物质名的下部的数值表示浓度(μg/ml)。图中的axl、akt及p-akt分别表示axl受体、akt蛋白及磷酸化akt蛋白。图5为评价本发明的抗gas6单克隆抗体km5320抗体、km5321抗体与cnto抗体是否竞争性地与hgas6结合的结果。实验以n=2来实施,图中示出其平均值。图的纵轴表示吸光度,横轴表示抗体浓度(μg/ml)。◇为仅1%bsa-pbs(缓冲液)的结果,■为cnto抗体的结果,▲为km5320-mkg1抗体的结果,×为km5321-mkg1抗体的结果。图6为对于抗gas6单克隆抗体评价癌细胞的细胞增殖抑制活性的结果。图的纵轴表示荧光量,横轴表示添加于孔中的样品名。图中的gas6表示hgas6-f,axl-fc表示axl受体的细胞外结构域与igg1抗体的人fc的融合蛋白(阳性对照),km5320表示km5320-rkg1抗体,km5321表示km5321-rkg1抗体,dnp表示抗dnp抗体(阴性对照)。图7示出不含信号序列的km5320抗体的轻链可变区及km5320人源化抗体(以下记作hzkm5320抗体)的轻链可变区(lv0、lv1a、lv1b、lv2a、lv2b、lv3、lv5、lv6)的氨基酸序列。各序列中的被框包围的区域表示cdr的氨基酸序列。图8示出不含信号序列的km5320抗体的重链可变区及hzkm5320抗体的重链可变区(hv0、hv1、hv2、hv3a、hv3b、hv3c、hv4、hv6、hv8)的氨基酸序列。各序列中的被框包围的区域表示cdr的氨基酸序列。图9示出不含信号序列的km5321抗体的轻链可变区及km5321人源化抗体(以下记作hzkm5321抗体)的轻链可变区(lv0、lv1a、lv1b、lv1c、lv3、lv4、lv6、lv7a、lv7b、lv9)的氨基酸序列。各序列中的被框包围的区域表示cdr的氨基酸序列。图10示出不含信号序列的km5321抗体的重链可变区及hzkm5321抗体的重链可变区(hv0、hv1、hv2a、hv2b、hv3a、hv3b、hv4a、hv4b、hv5、hv7)的氨基酸序列。各序列中的被框包围的区域表示cdr的氨基酸序列。图11示出(a)km5320嵌合抗体及hzkm5320抗体、(b)km5321嵌合抗体及hzkm5321抗体的与人gas6蛋白的结合活性。实验以n=2来实施,图中示出其平均值。任一图中,纵轴表示吸光度,横轴表示抗体浓度(ng/ml)。(a)中,■表示km5320嵌合抗体,◇表示hzkm5320lv5hv2,●表示hzkm5320lv1bhv0,○表示阴性对照抗dnp抗体。(b)中,■表示km5321嵌合抗体,◇表示hzkm5321lv6hv2b,●表示hzkm5321lv7bhv0,○表示阴性对照抗dnp抗体。图12示出(a)km5320嵌合抗体及hzkm5320抗体、(b)km5321嵌合抗体及hzkm5321抗体的抑制人gas6蛋白与axl的结合的活性。实验以n=2来实施,图中示出其平均值。任一图中,纵轴表示吸光度,横轴表示抗体浓度(ng/ml)。(a)中,■表示km5320嵌合抗体,◇表示hzkm5320lv5hv2,●表示hzkm5320lv1bhv0,○表示阴性对照抗dnp抗体。(b)中,■表示km5321嵌合抗体,◇表示hzkm5321lv6hv2b,●表示hzkm5321lv7bhv0,○表示阴性对照抗dnp抗体。具体实施方式本发明涉及与人gas6的氨基酸序列的第314、315及316位的氨基酸残基中的至少1个氨基酸残基结合的单克隆抗体或该抗体片段。具体而言,涉及与存在于人gas6的氨基酸序列的shbg结构域的第314、315及316位的氨基酸残基中的至少1个氨基酸残基结合的单克隆抗体或该抗体片段、与含有序列号4所示的氨基酸序列的人gas6的第314、315及316位的氨基酸残基中的至少1个氨基酸残基结合的单克隆抗体或该抗体片段。作为本发明的抗体,可以列举:与含有序列号4所示的氨基酸序列的人gas6的第314位的氨基酸残基结合的抗体、与第315位的氨基酸残基结合的抗体、与第316位的氨基酸残基结合的抗体、与第314及315位的氨基酸残基结合的抗体、与第314及316位的氨基酸残基结合的抗体、与第315及316位的氨基酸残基结合的抗体或者与第314、315及316位的氨基酸残基结合的抗体。另外,作为本发明的抗体,具体可以列举选自下述(a)~(e)中的任意一种抗体。(a)抗体的重链可变区(heavychainvariableregion)(以下称为vh)的互补决定区(complementaritydeterminingregion)(以下称为cdr)1~3的氨基酸序列分别为序列号79、80及81所述的氨基酸序列、并且轻链可变区(lightchainvariableregion)(以下称为vl)的cdr1~3的氨基酸序列分别为序列号82、83及84所述的氨基酸序列的抗体。(b)抗体的vh的cdr1~3的氨基酸序列分别为序列号85、86及87所述的氨基酸序列、并且vl的cdr1~3的氨基酸序列分别为序列号88、89及90所述的氨基酸序列的抗体。(c)与上述(a)或(b)所述的抗体竞争性地与人gas6结合的抗体。(d)与含有上述(a)或(b)所述的抗体所结合的表位的表位结合的抗体。(e)与和上述(a)或(b)所述的抗体所结合的表位相同的表位结合的抗体。本发明中,作为上述(a)所述的抗体中、抗体的vh的cdr1~3的氨基酸序列分别为序列号79、80及81所述的氨基酸序列、并且vl的cdr1~3的氨基酸序列分别为序列号82、83及84所述的氨基酸序列的抗体的一个方式,可以列举:小鼠抗人gas6单克隆抗体km5320-mkg1、抗人gas6小鼠-大鼠嵌合抗体km5320-rkg1及抗人gas6人源化抗体hzkm5320等。本发明中,作为上述(a)所述的抗体中、抗体的vh的cdr1~3的氨基酸序列分别为序列号85、86及87所述的氨基酸序列、并且vl的cdr1~3的氨基酸序列分别为序列号88、89及90所述的氨基酸序列的抗体的一个方式,可以列举:小鼠抗人gas6单克隆抗体km5321-mkg1、抗人gas6小鼠-大鼠嵌合抗体km5321-rkg1、抗人gas6人源化抗体hzkm5321等。另外,本发明的上述(c)的抗体是指:在将上述(a)所述的抗体设为第1抗体、且将第1抗体所结合的表位设为第1表位时,与含有该第1表位的第2表位结合的第2抗体。另外,作为本发明的抗体,具体而言,还可以列举选自下述(a)~(e)中的任意一种抗体。(a)抗体的vh的氨基酸序列为序列号69所述的氨基酸序列、并且vl的氨基酸序列为序列号72所述的氨基酸序列的抗体。(b)抗体的vh的氨基酸序列为序列号75所述的氨基酸序列、并且vl的氨基酸序列为序列号78所述的氨基酸序列的抗体。(c)抗体的vh的氨基酸序列为序列号135所述的氨基酸序列、并且vl的氨基酸序列为序列号123所述的氨基酸序列的抗体。(d)抗体的vh的氨基酸序列为序列号195所述的氨基酸序列、并且vl的氨基酸序列为序列号174所述的氨基酸序列的抗体。(e)抗体的vh的氨基酸序列为序列号186所述的氨基酸序列、并且vl的氨基酸序列为序列号180所述的氨基酸序列的抗体。作为上述(a)的抗体的一个方式,可以列举:km5320-mkg1、km5320-rkg1等。作为上述(b)的抗体的一个方式,可以列举:km5321-mkg1、km5321-rkg1等。作为上述(c)的抗体的一个方式,可以列举:抗人gas6人源化抗体hzkm5320lv5hv2等。作为上述(d)的抗体的一个方式,可以列举:抗人gas6人源化抗体hzkm5321lv6hv2b等。作为上述(e)的抗体的一个方式,可以列举:抗人gas6人源化抗体hzkm5321lv7bhv0等。本发明中,人生长停滞特异性蛋白6(growtharrest-specific6,gas6)也称为axl受体激酶配体(axlreceptorkinaseligand,axllg)或axl刺激因子(axlstimulatoryfactor,axsf)。本发明中,作为人gas6,可以列举:含有序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列的多肽;由序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列中缺失、置换或添加1个以上氨基酸的氨基酸序列构成、且具有人gas6的功能的多肽;或者,由与序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列具有60%以上、优选80%以上、进一步优选90%以上、最优选95%以上的同源性的氨基酸序列构成、且具有人gas6的功能的多肽等。具有在序列号4所述的氨基酸序列或ncbi登录号np_000811所示的氨基酸序列中缺失、置换或添加1个以上氨基酸的氨基酸序列的多肽可以如下得到:使用定点诱变法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)、currentprotocolsinmolecularbiology,johnwiley&sons(1987-1997)、nucleicacidsresearch,10,6487(1982)、proc.natl.acad.sci.usa,79,6409(1982)、gene,34,315(1985)、nucleicacidsresearch,13,4431(1985)、proc.natl.acad.sci.usa,82,488(1985)]等,在例如编码含有序列号4的氨基酸序列的多肽的dna中导入位点特异性突变,从而得到。对缺失、置换或添加的氨基酸个数没有特别限定,优选为1个~数十个、例如1~20个、更优选为1个~数个、例如1~5个氨基酸。作为编码人gas6的基因,可以列举:序列号3所述的碱基序列、ncbi登录号nm_000820的碱基序列。本发明的编码人gas6的基因中,还包含:含有由在序列号3所述的碱基序列或nm_000820的碱基序列中缺失、置换或添加1个以上碱基的碱基序列构成、且编码具有人gas6的功能的多肽的dna的基因;含有下述dna的基因,所述dna由与序列号3所述的碱基序列或nm_000820的碱基序列具有至少60%以上的同源性的碱基序列、优选具有80%以上的同源性的碱基序列、进一步优选具有95%以上的同源性的碱基序列构成,且编码具有人gas6的功能的多肽;或者,由与含有序列号3所述的碱基序列、或nm_000820的碱基序列的dna在严格条件下杂交的dna构成、且编码具有人gas6的功能的多肽的基因;等。作为在严格条件下杂交的dna,是指使用含有序列号3所述的碱基序列或nm_000820的碱基序列的dna作为探针,通过菌落杂交法、噬菌斑杂交法、dna印迹杂交法或dna微阵列法等得到的能够杂交的dna。具体可以列举可以通过如下方法鉴定的dna:使用固定有来自所杂交的菌落或噬菌斑的dna、具有该序列的pcr产物或寡dna的滤膜或载玻片,在0.7~1.0mol/l的氯化钠存在下、在65℃下进行杂交法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)、currentprotocolsinmolecularbiology,johnwiley&sons(1987-1997)、dnacloning1:coretechniques,apracticalapproach,secondedition,oxforduniversity,(1995)]后,使用0.1~2倍浓度的ssc溶液(1倍浓度的ssc溶液的组成由150mmol/l氯化钠、15mmol/l柠檬酸钠构成),在65℃条件下洗涤滤膜或载玻片。作为能够杂交的dna,可以列举:与序列号3所述的碱基序列或nm_000820的碱基序列具有至少60%以上的同源性的dna、优选具有80%以上的同源性的dna、进一步优选具有95%以上的同源性的dna。编码真核生物的蛋白质的基因的碱基序列中,常常可以观察到基因的多态性。本发明的编码人gas6的基因中还包含:本发明所使用的基因中,由于这类多态性而使碱基序列产生了小规模的突变的基因。除特别说明的情况以外,本发明中的同源性的数值可以是使用本领域技术人员公知的同源性检索程序计算的数值,对于碱基序列而言,可以列举使用blast[j.mol.biol.,215,403(1990)]中默认的参数计算的数值等,对于氨基酸序列而言,可以列举使用blast2[nucleicacidsres.,25,3389(1997)、genomeres.,7,649(1997)、http://www.ncbi.nlm.nih.gov/education/blastinfo/information3.html]中默认的参数计算的数值等。作为默认的参数,g(起始空位罚分,costtoopengap)在碱基序列的情况下为5,在氨基酸序列的情况下为11;-e(空位延伸罚分,costtoextendgap)在碱基序列的情况下为2,在氨基酸序列的情况下为1;-q(核苷酸错配罚分,penaltyfornucleotidemismatch)为-3;-r(核苷酸匹配得分,rewardfornucleotidematch)为1;-e(期望值,expectvalue)为10;-w(字长大小,wordsize)在碱基序列的情况下为11个残基,在氨基酸序列的情况下为3个残基;-y[非空位延伸下降的阈值,dropoff(x)forblastextensionsinbits]在blastn的情况下为20,在blastn以外的程序中为7;-x(空位比对的下降阈值,xdropoffvalueforgappedalignmentinbits)为15;以及,-z(最终空位比对的下降值,finalxdropoffvalueforgappedalignmentinbits)在blastn的情况下为50,在blastn以外的程序中为25(http://www.ncbi.nlm.nih.gov/blast/html/blastcgihelp.html)。含有序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列的部分序列的多肽可以通过本领域技术人员公知的方法来制作。具体而言,可以通过使编码序列号4的氨基酸序列的dna的一部分缺失并培养导入有含有该dna的表达载体的转化体来制作。另外,通过与上述同样的方法,可以得到具有在序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列中缺失、置换或添加1个以上的氨基酸的氨基酸序列的多肽。进而,由序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列构成的多肽、或具有在序列号4所述的氨基酸序列或ncbi登录号np_000811的氨基酸序列中缺失、置换或添加1个以上的氨基酸的氨基酸序列的多肽还可以通过芴甲氧羰基(fmoc)法、叔丁氧羰基(tboc)法等化学合成法来制造。作为本发明中的单克隆抗体,可以列举:由杂交瘤产生的抗体;或者,由用含有抗体基因的表达载体转化后的转化体产生的基因重组抗体。单克隆抗体为单一克隆的抗体产生细胞所分泌的抗体,仅识别一个表位(也称为抗原决定基),构成单克隆抗体的氨基酸序列(一级序列)是一致的。表位可以列举:单克隆抗体所识别、结合的单一氨基酸序列;由氨基酸序列构成的立体结构;通过翻译后修饰而经修饰的氨基酸序列;以及,由该氨基酸序列构成的立体结构等。作为通过翻译后修饰而经修饰的氨基酸序列,可以列举结合有糖链结合于具有oh取代基的tyr及ser而成的o结合型糖链、结合于具有nh2取代基的gln及asn而成的n结合型糖链以及硫酸分子结合于具有oh取代基的tyr而成的硫酸基等的氨基酸序列。作为本发明的抗体所结合的存在于人gas6上的氨基酸残基或表位,可以列举:与gas6受体的结合部分的表位;存在于人gas6的shbg结构域的表位;存在于人gas6的氨基酸序列的shbg结构域的含有选自第314、315及316位中的至少1个氨基酸残基的表位;存在于人gas6的氨基酸序列的shbg结构域的由第314、315及316位构成的表位。本发明的抗体与人gas6结合这一点可以通过下述方法来确认:使用固相夹心法等放射免疫测定;或使用酶免疫测定法(elisa)等的针对人gas6的公知的免疫学检测法;或使用bicacore系统(通用电气医疗公司制)等的表面等离子激元共振法等。另外,还可以组合公知的免疫学检测法[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、antibodies-alaboratorymanual,coldspringharborlaboratory(1988)、单克隆抗体实验手册,講談社サイエンテイフイツク(1987)]等来进行确认。本发明的抗体所结合的人gas6的氨基酸残基或表位可以使用使人gas6的部分结构域缺失的缺失体、与来自其它蛋白质的结构域置换后的突变体以及人gas6的部分肽片段等进行抗体结合实验来确定。或者,本发明的抗体所结合的人gas6的氨基酸残基或表位还可以如下确定:在用蛋白水解酶消化后的人gas6的肽片段中添加本发明的抗体,使用已知的质谱法进行表位作图。另外,作为本发明的抗体,具体可以列举与人gas6的氨基酸序列的第314、315及316位的氨基酸残基中的至少1个氨基酸残基结合、从而对人gas6具有结合活性及中和活性的抗体。作为人gas6的功能,已知gas6结合于gas6受体而活化该受体、从而引起细胞内信号转导的活化、细胞增殖亢进的功能。本发明中,作为gas6受体,具体可以列举:axl、sky、mertk等。本发明中,中和活性是指抑制人gas6的功能、抑制上述gas6受体活化及与活化相伴随的各种反应的活性。具体可以列举:抑制gas6和gas6受体的结合、从而抑制gas6受体的活性的活性;抑制gas6添加所引起的gas6受体表达细胞内的信号转导的活化的活性;或抑制gas6添加所引起的gas6受体表达细胞的细胞增殖亢进的活性等。本发明的抗体与gas6特异性结合及具有抑制gas6与gas6受体结合的活性这一点可以通过下述方法来确认:elisa等公知的免疫学检测法;使用biacore(注册商标)系统(通用电气医疗公司制)等的表面等离子激元共振法;或这些的组合。本发明的抗体具有抑制由gas6结合引起的gas6受体表达细胞内的信号转导的活化的活性这一点可以如下确认:使用公知的报告基因测定来检测特定基因产物的表达量,或者使用蛋白质印迹法或流式细胞术等检测特定信号转导物质的磷酸化水平。或者,还可以通过使用微阵列网罗性地检测基因的活化状态或表达量等来确认。本发明的抗体具有抑制由gas6引起的gas6受体表达细胞的细胞增殖亢进的活性这一点可以使用公知的细胞增殖测定法来确认。公知的细胞增殖测定法具体可以列举:使用mtt、wst-1等四唑盐测定细胞生存活性的方法;或者,使用[3h]-胸苷等放射性同位素测定细胞内的dna合成的方法等。本发明中,本发明的单克隆抗体所具有的高结合活性或高中和活性是指:与公知的抗人gas6抗体或市售的抗人gas6抗体所具有的hgas6结合活性或中和活性相比,结合活性或中和活性更强。具体是指:本发明的抗hgas6单克隆抗体与抗hgas6单克隆抗体wg1(us7,547,767)相比,具有更高的与hgas6的结合活性及中和活性。抗体分子也称为免疫球蛋白(以下记作ig),人抗体根据分子结构的不同而分为iga1、iga2、igd、ige、igg1、igg2、igg3、igg4及igm这些同种型。也将氨基酸序列的同源性较高的igg1、igg2、igg3及igg4统称为igg。抗体分子由称为重链(heavychain,以下记作h链)及称为轻链(lightchain,以下记作l链)的多肽构成。另外,h链从n末端侧起由h链可变区(也记作vh)、h链恒定区(也记作ch)的各区域构成,l链从n末端侧起由l链可变区(也记作vl)、l链恒定区(也记作cl)的各区域构成。ch在各亚类中分别已知α、δ、ε、γ及μ链。进而,ch从n末端侧起由ch1结构域、铰链结构域、ch2结构域、ch3结构域各结构域构成。结构域是指构成抗体分子的各多肽的功能性结构单元。另外,将ch2结构域和ch3结构域合称为fc区,或者简称为fc。cl已知有cλ链及cκ链。本发明中的ch1结构域、铰链结构域、ch2结构域、ch3结构域及fc区可以按照eu索引[kabatetal.,sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]用从n末端起的氨基酸残基的编号来特别指定。具体而言,ch1特别指定为eu索引第118~215位的氨基酸序列,铰链特别指定为eu索引第216~230位的氨基酸序列,ch2特别指定为eu索引第231~340位的氨基酸序列,ch3特别指定为eu索引第341~447位的氨基酸序列。作为本发明的抗体,还特别地包含:通过基因工程方法制作的小鼠抗体、大鼠抗体、人嵌合抗体(以下也简记为嵌合抗体)、人源化抗体[也称为人互补决定区(complementaritydeterminingregion;cdr)移植抗体]及人抗体等基因重组抗体。嵌合抗体是指由人以外的动物(非人动物)的抗体的vh及vl与人抗体的ch及cl构成的抗体。作为非人动物,只有是小鼠、大鼠、仓鼠、兔等能够制作杂交瘤的非人动物,则任意非人动物均可以使用。杂交瘤是指将用抗原免疫非人动物而得到的b细胞、与来自小鼠等的骨髓瘤细胞进行细胞融合而得到的、产生具有期望的抗原特异性的单克隆抗体的细胞。因此,构成杂交瘤所产生的抗体的可变区由非人动物抗体的氨基酸序列构成。人嵌合抗体可以如下制造:由生产单克隆抗体的来自非人动物细胞的杂交瘤得到编码该单克隆抗体的vh及vl的cdna,分别插入具有编码人抗体的ch及cl的dna的动物细胞用表达载体中,构建人嵌合抗体表达载体,将其导入动物细胞中从而使其表达。人源化抗体是指将非人动物抗体的vh及vl的cdr的氨基酸序列移植到人抗体的vh及vl的对应cdr处而成的抗体。vh及vl的cdr以外的区域称为框架区(以下记作fr)。人源化抗体可以如下制造:构建编码如下vh的氨基酸序列的cdna和编码如下vl的氨基酸序列的cdna,分别插入具有编码人抗体的ch及cl的dna的动物细胞用表达载体中,构建人源化抗体表达载体,将其导入动物细胞中从而使其表达;所述vh由非人动物抗体的vh的cdr的氨基酸序列和任意的人抗体的vh的fr的氨基酸序列构成,所述vl由非人动物抗体的vl的cdr的氨基酸序列和任意的人抗体的vl的fr的氨基酸序列构成。作为本发明的人源化抗体,具体而言,关于km5320,可以列举包含cdr1~3分别含有序列号79~81所示的氨基酸序列的抗体的vh、且包含cdr1~3分别含有序列号82~84所示的氨基酸序列的抗体的vl的人源化抗体。关于km5321,可以列举包含cdr1~3分别含有序列号85~87所示的氨基酸序列的抗体的vh、且包含cdr1~3分别含有序列号88~90所示的氨基酸序列的抗体的vl的人源化抗体。作为本发明的人源化抗体,关于km5320,具体可以列举含有以下的(a)vl及(b)vh的至少一个的人源化抗体;关于km5321,具体可以列举含有以下的(c)vl及(d)vh的至少一个的人源化抗体。(a)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列,或序列号105所示的氨基酸序列中的选自第2位的val、第15位的leu、第46位的leu、第73位的leu、第78位的leu及第87位的tyr中的至少1个氨基酸残基被置换为其它氨基酸残基的氨基酸序列(b)含有如下氨基酸序列的抗体的vh:序列号129所示的氨基酸序列,或序列号129所示的氨基酸序列的选自第2位的val、第9位的ser、第20位的val、第38位的arg、第46位的glu、第77位的ser、第93位的val及第95位的tyr中的至少1个氨基酸残基被置换为其它氨基酸残基的氨基酸序列(c)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列,或序列号156所示的氨基酸序列中的选自第4位的leu、第13位的ala、第15位的val、第43位的ala、第64位的gly、第73位的leu、第78位的leu、第85位的thr及第104位的val中的至少1个氨基酸残基被置换为其它氨基酸残基的氨基酸序列(d)含有如下氨基酸序列的抗体的vh:序列号186所示的氨基酸序列,或序列号186所示的氨基酸序列的选自第2位的val、第9位的ser、第38位的arg、第46位的glu、第79位的ser、第93位的val及第112位的val中的至少1个氨基酸残基被置换为其它氨基酸残基的氨基酸序列进而,作为本发明的km5320人源化抗体所含的vl,优选以下的(1)~(7)的vl。(1)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第2位的val、第15位的leu、第46位的leu、第73位的leu、第78位的leu及第87位的tyr被置换为其它氨基酸残基的氨基酸序列(2)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第2位的val、第46位的leu、第73位的leu、第78位的leu及第87位的tyr被置换为其它氨基酸残基的氨基酸序列(3)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第46位的leu、第73位的leu及第87位的tyr被置换为其它氨基酸残基的氨基酸序列(4)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第15位的leu及第73位的leu被置换为其它氨基酸残基的氨基酸序列(5)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第78位的leu及第87位的tyr被置换为其它氨基酸残基的氨基酸序列(6)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第78位的leu被置换为其它氨基酸残基的氨基酸序列(7)含有如下氨基酸序列的抗体的vl:序列号105所示的氨基酸序列中的第87位的tyr被置换为其它氨基酸残基的氨基酸序列作为上述vl的氨基酸序列,可以列举例如导入了选自如下改变中的至少1种改变的氨基酸序列:将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第15位的leu置换为ala、将第46位的leu置换为val、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe。作为导入了6个改变的vl的氨基酸序列,具体可以列举例如:将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第15位的leu置换为ala、将第46位的leu置换为val、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列。作为导入了5个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第46位的leu置换为val、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(2)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第15位的leu置换为ala、将第46位的leu置换为val、将第73位的leu置换为phe及将第87位的tyr置换为phe的氨基酸序列(3)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第15位的leu置换为ala、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(4)将序列号105所示的氨基酸序列中的第15位的leu置换为ala、将第46位的leu置换为val、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列作为导入了4个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号105所示的氨基酸序列中的第46位的leu置换为val、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(2)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第73位的leu置换为phe、将第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(3)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第46位的leu置换为val、将第73位的leu置换为phe及将第78位的leu置换为val的氨基酸序列(4)将序列号105所示的氨基酸序列中的第2位的val置换为ile、将第15位的leu置换为ala、将第73位的leu置换为phe及将第87位的tyr置换为phe的氨基酸序列作为导入了3个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号105所示的氨基酸序列中的第46位的leu置换为val、将第73位的leu置换为phe及将第87位的tyr置换为phe的氨基酸序列(2)将序列号105所示的氨基酸序列中的第15位的leu置换为ala、将第46位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(3)将序列号105所示的氨基酸序列中的第15位的leu置换为ala、将第46位的leu置换为val及将第78位的leu置换为val的氨基酸序列(4)将序列号105所示的氨基酸序列中的第46位的leu置换为val、将第73位的leu置换为phe及将第78位的leu置换为val的氨基酸序列作为导入了2个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号105所示的氨基酸序列中的第78位的leu置换为val及将第87位的tyr置换为phe的氨基酸序列(2)将序列号105所示的氨基酸序列中的第15位的leu置换为ala及将第73位的leu置换为phe的氨基酸序列(3)将序列号105所示的氨基酸序列中的第46位的leu置换为val及将第78位的leu置换为val的氨基酸序列(4)将序列号105所示的氨基酸序列中的第2位的val置换为ile及将第15位的leu置换为ala的氨基酸序列作为导入了1个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号105所示的氨基酸序列中的第2位的val置换为ile的氨基酸序列(2)将序列号105所示的氨基酸序列中的第46位的leu置换为val的氨基酸序列(3)将序列号105所示的氨基酸序列中的第78位的leu置换为val的氨基酸序列(4)将序列号105所示的氨基酸序列中的第87位的tyr置换为phe的氨基酸序列另外,作为本发明的km5320人源化抗体所含的vh,优选以下的(1)~(8)的vh。(1)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第2位的val、第9位的ser、第20位的val、第38位的arg、第46位的glu、第77位的ser、第93位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(2)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第9位的ser、第20位的val、第38位的arg、第46位的glu、第93位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(3)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第9位的ser、第46位的glu、第93位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(4)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第46位的glu、第93位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(5)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第2位的val、第20位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(6)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第9位的ser、第38位的arg及第46位的glu被置换为其它氨基酸残基的氨基酸序列(7)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第93位的val及第95位的tyr被置换为其它氨基酸残基的氨基酸序列(8)含有如下氨基酸序列的vh:序列号129所示的氨基酸序列中的第46位的glu被置换为其它氨基酸残基的氨基酸序列作为上述vh的氨基酸序列,可以列举例如导入了选自如下改变中的至少1种改变的氨基酸序列:将序列号129所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第20位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第77位的ser置换为thr、将第93位的val置换为thr及将第95位的tyr置换为phe。作为导入了8个改变的vh的氨基酸序列,具体可以列举例如:将序列号129所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第20位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第77位的ser置换为thr、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列。作为导入了6个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号129所示的氨基酸序列中的第20位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第77位的ser置换为thr、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列(2)将序列号129所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第20位的val置换为ile、将第38位的arg置换为lys、将第77位的ser置换为thr及将第93位的val置换为thr的氨基酸序列(3)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第20位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列(4)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys、将第77位的ser置换为thr、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列作为导入了4个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第20位的val置换为ile、将第46位的glu置换为lys及将第93位的val置换为thr的氨基酸序列(2)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第46位的glu置换为lys、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列(3)将序列号129所示的氨基酸序列中的第20位的val置换为ile、将第46位的glu置换为lys、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列(4)将序列号129所示的氨基酸序列中的第38位的arg置换为lys、将第46位的glu置换为lys、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列作为导入了3个改变的vh的氨基酸序列,具体可以列举例如:选自以下的(1)~(4)中的1个氨基酸序列。(1)将序列号129所示的氨基酸序列中的第2位的val置换为ile、将第20位的val置换为ile及将第95位的tyr置换为phe的氨基酸序列(2)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第38位的arg置换为lys及将第46位的glu置换为lys的氨基酸序列(3)将序列号129所示的氨基酸序列中的第46位的glu置换为lys、将第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列(4)将序列号129所示的氨基酸序列中的第9位的ser置换为pro、将第38位的arg置换为lys及将第95位的tyr置换为phe的氨基酸序列作为导入了2个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号129所示的氨基酸序列中的第9位的ser置换为pro及将第38位的arg置换为lys的氨基酸序列(2)将序列号129所示的氨基酸序列中的第20位的val置换为ile及将第77位的ser置换为thr的氨基酸序列(3)将序列号129所示的氨基酸序列中的第46位的glu置换为lys及第95位的tyr置换为phe的氨基酸序列(4)将序列号129所示的氨基酸序列中的第93位的val置换为thr及将第95位的tyr置换为phe的氨基酸序列作为导入了1个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)序列号129所示的氨基酸序列中的第20位的val置换为ile的氨基酸序列(2)序列号129所示的氨基酸序列中的第38位的arg置换为lys的氨基酸序列(3)序列号129所示的氨基酸序列中的第46位的glu置换为lys的氨基酸序列(4)序列号129所示的氨基酸序列中的第95位的tyr置换为phe的氨基酸序列另外,作为本发明的km5320人源化抗体的具体例子,可以列举以下的(1)~(3)的人源化抗体等。(1)抗体的vh含有序列号135所示的氨基酸序列和/或抗体的vl含有序列号123所示的氨基酸序列的人源化抗体(2)抗体的vh含有图8所示的任一氨基酸序列和/或抗体的vl含有序列号123所示的氨基酸序列的人源化抗体(3)抗体的vh含有序列号135所示的氨基酸序列和/或抗体的vl含有图7所示的任一氨基酸序列的人源化抗体进而,作为本发明的km5321人源化抗体所含的vl,优选以下的(1)~(7)的vl。(1)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第4位的leu、第13位的ala、第15位的val、第43位的ala、第64位的gly、第73位的leu、第78位的leu、第85位的thr及第104位的val被置换为其它氨基酸残基的氨基酸序列(2)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第13位的ala、第15位的val、第64位的gly、第73位的leu、第78位的leu、第85位的thr及第104位的val被置换为其它氨基酸残基的氨基酸序列(3)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第13位的ala、第15位的val、第43位的ala、第73位的leu、第78位的leu及第85位的thr被置换为其它氨基酸残基的氨基酸序列(4)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第13位的ala、第15位的val、第73位的leu及第78位的leu被置换为其它氨基酸残基的氨基酸序列(5)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第15位的val、第78位的leu及第85位的thr被置换为其它氨基酸残基的氨基酸序列(6)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第13位的ala及第43位的ala被置换为其它氨基酸残基的氨基酸序列(7)含有如下氨基酸序列的抗体的vl:序列号156所示的氨基酸序列中的第43位的ala被置换为其它氨基酸残基的氨基酸序列作为上述vl的氨基酸序列,可以列举例如导入了选自如下改变中的至少1种改变的氨基酸序列:将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr、将第85位的thr置换为asp及将第104位的val置换为leu。作为导入了9个改变的vl的氨基酸序列,具体可以列举例如:将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr、将第85位的thr置换为asp及将第104位的val置换为leu的氨基酸序列。作为导入了7个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第13位的ala置换为val、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr、将第85位的thr置换为asp及将第104位的val置换为leu的氨基酸序列(2)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列(3)将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第78位的leu置换为thr、将第85位的thr置换为asp及将第104位的val置换为leu的氨基酸序列(4)将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列作为导入了6个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第85位的thr置换为asp及将第104位的val置换为leu的氨基酸序列(2)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列(3)将序列号156所示的氨基酸序列中的第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列(4)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列作为导入了4个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第4位的leu置换为val、将第13位的ala置换为val、将第15位的val置换为thr及将第43位的ala置换为pro的氨基酸序列(2)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro及将第85位的thr置换为asp的氨基酸序列(3)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr、将第73位的leu置换为phe及将第78位的leu置换为thr的氨基酸序列(4)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr、将第73位的leu置换为phe及将第85位的thr置换为asp的氨基酸序列作为导入了3个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第15位的val置换为thr及将第73位的leu置换为phe的氨基酸序列(2)将序列号156所示的氨基酸序列中的第13位的ala置换为val、将第73位的leu置换为phe及将第85位的thr置换为asp的氨基酸序列(3)将序列号156所示的氨基酸序列中的第15位的val置换为thr、将第78位的leu置换为thr及将第85位的thr置换为asp的氨基酸序列(4)将序列号156所示的氨基酸序列中的第43位的ala置换为pro、将第73位的leu置换为phe及将第78位的leu置换为thr的氨基酸序列作为导入了2个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第13位的ala置换为val及将第15位的val置换为thr的氨基酸序列(2)将序列号156所示的氨基酸序列中的第13位的ala置换为val及将第43位的ala置换为pro的氨基酸序列(3)将序列号156所示的氨基酸序列中的第15位的val置换为thr及将第85位的thr置换为asp的氨基酸序列(4)将序列号156所示的氨基酸序列中的第43位的ala置换为pro及将第64位的gly置换为ser的氨基酸序列作为导入了1个改变的vl的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号156所示的氨基酸序列中的第13位的ala置换为val的氨基酸序列(2)将序列号156所示的氨基酸序列中的第43位的ala置换为pro的氨基酸序列(3)将序列号156所示的氨基酸序列中的第73位的leu置换为phe的氨基酸序列(4)将序列号156所示的氨基酸序列中的第85位的thr置换为asp的氨基酸序列另外,作为本发明的km5321人源化抗体所含的vh,优选以下的(1)~(8)的vh。(1)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第2位的val、9位的ser、38位的arg、46位的glu、79位的ser、93位的val及第112位的val被置换为其它氨基酸残基的氨基酸序列(2)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第2位的val、38位的arg、46位的glu、79位的ser及第112位的val被置换为其它氨基酸残基的氨基酸序列(3)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第2位的val、9位的ser、79位的ser及第112位的val被置换为其它氨基酸残基的氨基酸序列(4)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第9位的ser、46位的glu及第93位的val被置换为其它氨基酸残基的氨基酸序列(5)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第38位的arg、46位的glu及第93位的val被置换为其它氨基酸残基的氨基酸序列(6)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第38位的arg及第46位的glu被置换为其它氨基酸残基的氨基酸序列(7)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第9位的ser及第93位的val被置换为其它氨基酸残基的氨基酸序列(8)含有如下氨基酸序列的vh:序列号186所示的氨基酸序列中的第46位的glu被置换为其它氨基酸残基的氨基酸序列作为上述vh的氨基酸序列,可以列举例如导入了选自如下改变中的至少1种改变的氨基酸序列:将序列号186所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys、将第79位的ser置换为ala、将第93位的val置换为thr及将第112位的val置换为ile。作为导入了7个改变的vh的氨基酸序列,具体可以列举例如:将序列号186所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys、将第79位的ser置换为ala、将第93位的val置换为thr及将第112位的val置换为ile的氨基酸序列。作为导入了5个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号186所示的氨基酸序列中的第2位的val置换为ile、将第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys及将第112位的val置换为ile的氨基酸序列(2)将序列号186所示的氨基酸序列中的第2位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第79位的ser置换为ala及将第112位的val置换为ile的氨基酸序列(3)将序列号186所示的氨基酸序列中的第2位的val置换为ile、将第38位的arg置换为lys、将第46位的glu置换为lys、将第79位的ser置换为ala及将第93位的val置换为thr的氨基酸序列(4)将序列号186所示的氨基酸序列中的第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys、将第93位的val置换为thr及将第112位的val置换为ile的氨基酸序列作为导入了4个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号186所示的氨基酸序列中的、第2位的val置换为ile、将第9位的ser置换为pro、将第79位的ser置换为ala及将第112位的val置换为ile的氨基酸序列(2)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys及将第79位的ser置换为ala的氨基酸序列(3)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys及将第93位的val置换为thr的氨基酸序列(4)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro、将第46位的glu置换为lys、将第79位的ser置换为ala及将第112位的val置换为ile的氨基酸序列作为导入了3个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro、将第46位的glu置换为lys及将第93位的val置换为thr的氨基酸序列(2)将序列号186所示的氨基酸序列中的、第2位的val置换为ile、将第38位的arg置换为lys及将第46位的glu置换为lys的氨基酸序列(3)将序列号186所示的氨基酸序列中的、第38位的arg置换为lys、将第46位的glu置换为lys及将第79位的ser置换为ala的氨基酸序列(4)将序列号186所示的氨基酸序列中的、第38位的arg置换为lys、将第46位的glu置换为lys及将第93位的val置换为thr的氨基酸序列作为导入了2个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号186所示的氨基酸序列中的、第38位的arg置换为lys及将第46位的glu置换为lys的氨基酸序列(2)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro及将第93位的val置换为thr的氨基酸序列(3)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro及将第38位的arg置换为lys的氨基酸序列(4)将序列号186所示的氨基酸序列中的、第79位的ser置换为ala及将第93位的val置换为thr的氨基酸序列作为导入了1个改变的vh的氨基酸序列,具体可以列举例如:以下的(1)~(4)的氨基酸序列。(1)将序列号186所示的氨基酸序列中的、第9位的ser置换为pro的氨基酸序列(2)将序列号186所示的氨基酸序列中的、第38位的arg置换为lys的氨基酸序列(3)将序列号186所示的氨基酸序列中的、第46位的glu置换为lys的氨基酸序列(4)将序列号186所示的氨基酸序列中的、第93位的val置换为thr的氨基酸序列另外,作为本发明的km5321人源化抗体的具体例子,可以列举以下的(1)~(6)的人源化抗体等。(1)抗体的vh含有序列号195所示的氨基酸序列和/或抗体的vl含有序列号174所示的氨基酸序列的人源化抗体(2)抗体的vh含有图10所示的任一氨基酸序列和/或抗体的vl含有序列号174所示的氨基酸序列的人源化抗体(3)抗体的vh含有序列号195所示的氨基酸序列和/或抗体的vl含有图9所示的任一氨基酸序列的人源化抗体(4)抗体的vh含有序列号186所示的氨基酸序列和/或抗体的vl含有序列号180所示的氨基酸序列的人源化抗体(5)抗体的vh含有图10所示的任一氨基酸序列和/或抗体的vl含有序列号180所示的氨基酸序列的人源化抗体(6)抗体的vh含有序列号186所示的氨基酸序列和/或抗体的vl含有图9所示的任一氨基酸序列的人源化抗体对于人抗体而言,原本是指天然地存在于人体内的抗体,但也包括从随着最近的基因工程、细胞工程、发育工程的技术进步而制作的人抗体噬菌体文库和产生人抗体的转基因动物得到的抗体等。人抗体可以通过用期望的抗原免疫保有人免疫球蛋白基因的小鼠(tomizukak.et.al.,procnatlacadsciusa.97,722-7,2000.)来得到。另外,使用从来自人的b细胞扩增了抗体基因的噬菌体展示文库来选择具有期望的结合活性的人抗体,从而不进行免疫也可以得到人抗体(winterg.et.al.,annurevimmunol.12:433-55.1994)。进而,使用eb病毒使人b细胞永生化,从而制作生产具有期望的结合活性的人抗体的细胞,可以得到人抗体(rosena.et.al.,nature267,52-54.1977)。存在于人体内的抗体例如可以如下得到:使eb病毒等感染由人外周血分离出的淋巴细胞而使其永生化后进行克隆,从而可以得到产生该抗体的淋巴细胞,可以从培养该淋巴细胞而得的培养物中纯化该抗体。人抗体噬菌体文库是通过将由人b细胞制备的抗体基因插入噬菌体基因中而使fab、scfv等抗体片段在表面表达的噬菌体文库。可以以对固定有抗原的底物的结合活性为指标,从该文库回收表达具有期望的抗原结合活性的抗体片段的噬菌体。该抗体片段还可以进一步通过基因工程方法变换为由2条完整的h链及2条完整的l链构成的人抗体分子。产生人抗体的转基因动物是指宿主动物的染色体内整合有人抗体基因的动物。具体而言,向小鼠es细胞中导入人抗体基因,将该es细胞移植到其它小鼠的早期胚胎中,然后使其发育,从而可以制作产生人抗体的转基因动物。来自产生人抗体的转基因动物的人抗体的制作方法为:通过用通常的人以外的动物进行的杂交瘤制作方法得到产生人抗体的杂交瘤并进行培养,从而在培养物中产生并蓄积人抗体。作为本发明的抗体的vh及vl的氨基酸序列,可以为将人抗体的vh及vl的氨基酸序列、非人动物抗体的vh及vl的氨基酸序列或非人动物抗体的cdr移植到任意的人抗体的框架而成的人源化抗体的vh及vl的氨基酸序列中的任一种。具体可以列举:杂交瘤所产生的非人动物抗体的vh及vl的氨基酸序列、人源化抗体的vh及vl的氨基酸序列、人抗体的vh及vl的氨基酸序列等。作为本发明的抗体中的cl的氨基酸序列,可以为人抗体的氨基酸序列或非人动物抗体的氨基酸序列中的任一种,优选人抗体的氨基酸序列的cκ或cλ。作为本发明的抗体的ch,只要属于免疫球蛋白则可以为任意的ch,可以优选使用属于igg类的亚类、γ1(igg1)、γ2(igg2)、γ3(igg3)、γ4(igg4)中的任一种。作为本发明的抗体,fc与抗体片段结合而成的fc融合蛋白、fc与天然存在的配体或受体结合而成的fc融合蛋白(也称为免疫粘附素)、使多个fc区融合而成的fc融合蛋白等也包含在本发明中。另外,为了使抗体稳定化及为了控制血中半衰期,在本发明的抗体中也可以使用改变了氨基酸残基的fc区等。本发明的抗体或该抗体片段还包含含有进行了翻译后修饰的任意氨基酸残基的抗体。作为翻译后修饰,可以列举例如:h链c末端的赖氨酸残基的缺失[赖氨酸剪切(lysineclipping)]及多肽的n末端的谷氨酰胺残基向焦谷氨酰胺(pyroglu)的变换等[becketal,analyticalchemistry,85,715-736(2013)]。本发明中,作为抗体片段,可以列举:fab、fab′、f(ab′)2、scfv、双链抗体(diabody)、dsfv、含有多个cdr的肽等。fab是通过用蛋白酶木瓜蛋白酶处理igg抗体而得到的片段中(在h链的第224位的氨基酸残基处被切断)的、h链n末端侧的约一半和整个l链通过二硫键(s-s键)结合而成的分子量约5万的具有抗原结合活性的抗体片段。f(ab′)2是用蛋白水解酶胃蛋白酶处理igg而得到的片段中(在h链的第234位的氨基酸残基处被切断)的、与fab介由铰链区的s-s键结合而成的片段相比稍大的、分子量约10万的具有抗原结合活性的抗体片段。fab’是将上述f(ab’)2的铰链区的s-s键切断而成的分子量约5万的具有抗原结合活性的抗体片段。scfv是使用将任意个由4个gly及1个ser残基构成的接头(g4s)连接而成的接头肽等适当的肽接头(p)、将一条vh与一条vl连接而成的vh-p-vl或vl-p-vh多肽,是具有抗原结合活性的抗体片段。双链抗体是抗原结合特异性相同或不同的scfv形成了二聚体的抗体片段,是对相同的抗原具有二价抗原结合活性或者对不同的抗原具有特异性抗原结合活性的抗体片段。dsfv是指:使将vh和vl中的各一个氨基酸残基置换成半胱氨酸残基的多肽介由该半胱氨酸残基之间的s-s键结合而成的抗体片段。含有cdr的肽含有vh或vl的cdr中的至少1个以上区域而构成。含有多个cdr的肽可以使cdr彼此直接结合或者介由适当的肽接头来结合。可以如下制造:构建编码本发明的改变抗体的vh及vl的cdr的dna,将该dna插入原核生物用表达载体或真核生物用表达载体中,并将该表达载体导入原核生物或真核生物中而使其表达,从而制造。另外,含有cdr的肽也可以通过fmoc法或tboc法等化学合成法来制造。本发明的单克隆抗体中,包含在本发明的与人gas6结合的单克隆抗体或该抗体片段上通过化学方法或基因工程方法结合有放射性同位素、低分子药剂、高分子药剂、蛋白质或抗体药物等的抗体衍生物。抗体衍生物可以如下制造:通过化学方法[抗体工学入门,地人书馆(1994)]使放射性同位素、低分子药剂、高分子药剂、免疫刺激剂、蛋白质、抗体药物或核酸药物等结合于本发明的与人gas6结合的单克隆抗体或该抗体片段的h链或l链的n末端侧、c末端侧、抗体分子中的适当取代基或侧链、以及糖链等。另外,可以通过如下基因工程方法来制造:将编码本发明的与人gas6结合的单克隆抗体或该抗体片段的dna、和编码想要结合的蛋白质或抗体药物的dna连接并插入表达载体,将该表达载体导入适当的宿主细胞而使其表达。作为放射性同位素,可以列举例如:111in、131i、125i、90y、64cu、99tc、77lu或211at等。放射性同位素可以通过氯胺t法等直接与抗体进行结合。另外,也可以在抗体上结合用于螯合放射性同位素的物质。作为螯合剂,可以列举例如1-异硫氰酸苄酯基-3-甲基二亚乙基三胺五乙酸(mx-dtpa)等。作为低分子药剂,可以列举:烷化剂、亚硝基脲剂、代谢拮抗剂、抗生素、植物生物碱、拓扑异构酶抑制剂、激素疗法制剂、激素拮抗剂、芳香化酶抑制剂、p糖蛋白抑制剂、铂络合物衍生物、m期抑制剂或激酶抑制剂等抗癌剂[临床肿瘤学,癌症与化学疗法出版社(1996)],或氢化可的松、泼尼松龙等甾体制剂,阿司匹林、吲哚美辛等非甾体制剂,硫代苹果酸金、青霉胺等免疫调节剂,环磷酰胺、硫唑嘌呤等免疫抑制剂、马来酸氯苯那敏、氯马斯汀之类的抗组胺剂等抗炎剂[炎症与抗炎疗法,医齿药出版株式会社(1982)]等。作为抗癌剂,可以列举:阿米福汀(氨磷汀)、顺铂、达卡巴嗪(dtic)、更生霉素、二氯甲基二乙胺(氮芥)、链脲霉素、环磷酰胺、异环磷酰胺、卡莫司汀(bcnu)、洛莫司汀(ccnu)、多柔比星(阿霉素)、表柔比星、吉西他滨(健择)、柔红霉素、丙卡巴肼、丝裂霉素、阿糖胞苷、依托泊苷、甲氨蝶呤、5-氟尿嘧啶、氟尿嘧啶、长春碱、长春新碱、博来霉素、道诺霉素、培洛霉素、雌莫司汀、紫杉醇(泰素)、多西紫杉醇(多西他奇)、阿地白介素、天冬酰胺酶、白消安、卡铂、奥沙利铂、奈达铂、克拉屈滨、喜树碱、10-羟基-7-乙基-喜树碱(sn38)、氟尿苷、氟达拉滨、羟基脲、伊达比星、美司那、依立替康(cpt-11)、拓扑替康、米托蒽醌、托泊替康、亮丙瑞林、甲地孕酮、美法仑、巯基嘌呤、羟基脲、普卡霉素、密妥坦、培门冬酶、喷司他丁、哌泊溴烷、他莫昔芬、戈舍瑞林、亮丙瑞林、氟他胺、替尼泊苷、睾内酯、硫鸟嘌呤、塞替派、乌拉莫司汀、长春瑞滨、苯丁酸氮芥、氢化可的松、泼尼松龙、甲基泼尼松龙、长春地辛、尼莫司汀、司莫司汀、卡培他滨、雷替曲塞、氮胞苷、uft、奥沙利铂、吉非替尼(易瑞沙)、伊马替尼(sti571)、厄洛替尼、fms样酪氨酸激酶3(flt3)抑制剂、血管内皮生长因子受体(vegfr)抑制剂、成纤维细胞生长因子受体(fgfr)抑制剂、易瑞沙、特罗凯等表皮生长因子受体egfr)抑制剂、根赤壳菌素、17-烯丙基氨基-17-去甲氧基格尔德霉素、雷帕霉素、安吖啶、全反式维甲酸、沙利度胺、来那度胺、阿那曲唑、法曲唑、来曲唑、依西美坦、硫代苹果酸金、d-青霉胺、布西拉明、硫唑嘌呤、咪唑立宾、环孢素、雷帕霉素、氢化可的松、蓓萨罗丁(塔革雷汀)、他莫昔芬、地寒米松、孕激素类、雌激素类、阿那曲唑(瑞宁德)、利普安、阿司匹林、吲哚美辛、塞来昔布、青霉胺、硫代苹果酸金、马来酸氯苯那敏、氯苯那敏、氯马斯汀、维甲酸、蓓萨罗丁、砷、硼替佐米、别嘌呤醇、卡奇霉素、替伊莫单抗、塔革雷汀、奥唑米星、克拉霉素、瘤可维、酮康唑、氨鲁米特、苏拉明、类美登醇或其衍生物等。作为使低分子药剂与抗体结合的方法,可以列举:介由戊二醛使药剂和抗体的氨基间结合的方法、或者介由水溶性碳二亚胺使药剂的氨基与抗体的羧基结合的方法等。作为高分子药剂,可以列举:聚乙二醇(以下记作peg)、白蛋白、葡聚糖、聚环氧乙烷、苯乙烯马来酸共聚物、聚乙烯基吡咯烷酮、吡喃共聚物或羟丙基甲基丙烯酰胺等。通过使这些高分子化合物与抗体或该抗体片段结合,可以期待如下效果:(1)对化学性、物理性或生物性的各种因素的稳定性提高;(2)血中半衰期显著延长;(3)抑制免疫原性消失或抑制抗体产生;等[バイオコンジュゲ一ト医薬品,广川书店(1993)]。例如,作为使peg与抗体结合的方法,可以列举使其与peg化修饰试剂反应的方法等[バイオコンジュゲ一ト医薬品,广川书店(1993)]。作为peg化修饰试剂,可以列举:赖氨酸的ε-氨基的修饰剂(日本特开昭61-178926号)、天冬氨酸及谷氨酸的羧基的修饰剂(日本特开昭56-23587号)或精氨酸的胍基的修饰剂(日本特开平2-117920号)等。作为免疫刺激剂,可以是作为免疫佐剂已知的天然物,作为具体例子,使免疫亢进的药剂可以列举:β(1→3)葡聚糖(香菇多糖、西佐糖)或α-半乳糖神经酰胺(krn7000)等。作为免疫刺激剂,可以是作为免疫佐剂已知的天然物,作为具体例子,使免疫亢进的药剂可以列举:β(1→3)葡聚糖(香菇多糖、西佐糖)或α-半乳糖神经酰胺(krn7000)等。作为蛋白质,可以列举:活化nk细胞、巨噬细胞或中性粒细胞等免疫活性细胞的细胞因子或增殖因子、或者毒素蛋白等。作为细胞因子或增殖因子,可以列举例如:干扰素(以下记作ifn)-α、inf-β、inf-γ、白细胞介素(以下记作il)-2、il-12、il-15、il-18、il-21、il-23、粒细胞集落刺激因子(g-csf)、粒细胞巨噬细胞集落刺激因子(gm-csf)或巨噬细胞集落刺激因子(m-csf)等。作为毒素蛋白,可以列举:蓖麻毒素、白喉毒素或ontak等,还包括为调节毒性而向蛋白质中导入突变而得到的蛋白毒素。作为抗体药物,可以列举针对下述抗原的抗体:通过抗体的结合诱导凋亡的抗原、与肿瘤的病状形成相关的抗原或调节免疫功能的抗原、与病变部位的血管新生相关的抗原。作为通过抗体的结合来诱导凋亡的抗原,可以列举:分化抗原(以下记作cd)19、cd20、cd21、cd22、cd23、cd24、cd37、cd53、cd72、cd73、cd74、cdw75、cdw76、cd77、cdw78、cd79a、cd79b、cd80(b7.1)、cd81、cd82、cd83、cdw84、cd85、cd86(b7.2)、人类白细胞抗原(hla)ii类或表皮生长因子受体(egfr)等。作为与肿瘤的病状形成相关的抗原或调节免疫功能的抗体的抗原,可以列举:cd4、cd40、cd40配体、b7家族分子(cd80、cd86、cd274、b7-dc、b7-h2、b7-h3或b7-h4)、b7家族分子的配体(cd28、ctla-4、icos、pd-1或btla)、ox-40、ox-40配体、cd137、肿瘤坏死因子肿瘤坏死因子(tnf)受体家族分子(dr4、dr5、tnfr1或tnfr2)、tnf相关的凋亡诱导配体受体(trail)家族分子、trail家族分子的受体家族(trail-r1、trail-r2、trail-r3或trail-r4)、核因子κb受体活化因子配体(rank)、rank配体、cd25、叶酸受体4、细胞因子[il-1α、il-1β、il-4、il-5、il-6、il-10、il-13、转化生长因子(tgf)β或tnfα等]、上述细胞因子的受体、趋化因子(slc、elc、i-309、tarc、mdc或ctack等)或上述趋化因子的受体。作为抑制病变部位的血管新生的抗体的抗原,可以列举:血管内皮生长因子(vegf)、血管生成素、成纤维细胞生长因子(fgf)、egf、肝细胞生长因子(hgf)、血小板源性生长因子(pdgf)、胰岛素样生长因子(igf)、促红细胞生成素(epo)、tgfβ、il-8、ephrin、sdf-1或这些的受体等。与蛋白质或抗体药物的融合抗体可以如下制造:使编码蛋白质或编码抗体药物中所含的抗体的cdna与编码单克隆抗体或抗体片段的cdna连接,构建编码融合抗体的dna,将该dna插入原核生物或真核生物用表达载体中,并将该表达载体导入原核生物或真核生物中而使其表达,来制造融合抗体。作为核酸药物,可以列举含有通过控制基因功能而作用于生物的小干扰rna(sirna)、microrna等核酸的药品。例如,可以考虑与抑制th17细胞的关键转录因子rorγt的核酸药物的偶联物。在将本发明的抗体衍生物用于检测方法、测定方法或诊断方法时或者将本发明的抗体衍生物作为检测用试剂、测定用试剂或诊断药使用时,作为该抗体所结合的药剂,可以列举通常的免疫学检测或测定法中使用的标记物。作为标记物,可以列举:碱性磷酸酶、过氧化物酶或荧光素酶等酶;吖啶酯或洛酚碱等发光物质;或者,荧光素异硫氰酸酯(fitc)或四甲基罗丹明异硫氰酸酯(ritc)等荧光物质等。另外,本发明涉及人gas6相关疾病的治疗药,其含有与人gas6结合的单克隆抗体或该抗体片段作为有效成分。另外,本发明涉及人gas6相关疾病的治疗方法,其含有给药与人gas6结合的单克隆抗体或该抗体片段的步骤。作为人gas6相关疾病,只要是与人gas6或人gas6受体相关的疾病则可以为任意疾病,可以列举例如:肾病、癌症。作为肾病,可以列举:肾小球肾炎、iga肾病、糖尿病性肾病等。作为肾小球肾炎,可以列举:进行性肾小球肾炎、系膜增生性肾小球肾炎等。另外,作为癌症,具体可以列举:肺癌、乳腺癌、卵巣癌、前列腺癌、胰腺癌、肾癌、成胶质细胞瘤等。作为其它疾病,可以列举:血栓栓塞、缺血性疾病、静脉血栓栓塞、动脉血栓、静脉血栓、肺栓塞、再狭窄、糖尿病性血管病变或同种异体移植动脉粥样硬化等。含有本发明的抗体或该抗体片段的治疗剂可以仅含有作为有效成分的该抗体或该抗体片段,但通常期望以与药理学上可接受的1种以上载体一起混合、通过制剂学
技术领域:
中公知的任意方法制造的药物制剂形式来提供。就给药途径而言,期望使用在治疗时最为有效的给药途径,可以列举:经口给药或口腔内、气道内、直肠内、皮下、肌肉内或静脉内等非经口给药,优选列举静脉内给药。作为给药形态,可以列举:喷雾剂、胶囊剂、片剂、散剂、颗粒剂、糖浆剂、乳剂、栓剂、注射剂、软膏或胶带剂等。给药量或给药次数根据目标治疗效果、给药方法、治疗期、年龄及体重等而不同,通常,成人每天为10μg/kg~10mg/kg。本发明涉及gas6的检测或测定方法,其使用含有与人gas6结合的单克隆抗体或该抗体片段的gas6的检测或测定用试剂、或与人gas6结合的单克隆抗体或该抗体片段。本发明中,作为检测或测定人gas6的方法,可以列举任意的公知方法。可以列举例如:免疫学检测或测定方法等。免疫学检测或测定方法是指使用带有标记的抗原或抗体来检测或测定抗体量或抗原量的方法。作为免疫学检测或测定方法,可以列举:放射免疫测定法(ria)、酶免疫测定法(eia或elisa)、荧光免疫测定法(fia)、发光免疫测定法(luminescentimmunoassay)、蛋白质印迹法或物理化学方法等。本发明涉及gas6相关疾病的诊断方法,其含有如下步骤:使用含有与人gas6结合的单克隆抗体或该抗体片段作为有效成分的gas6相关疾病的诊断药、或与人gas6结合的单克隆抗体或该抗体片段,来进行gas6的检测或测定。通过使用本发明的单克隆抗体或该抗体片段按照上述方法来检测或测定表达人gas6的细胞,可以诊断与人gas6相关的疾病。本发明中,作为成为检测或测定人gas6的对象的生物试样,只要是组织、细胞、血液、血浆、血清、胰液、尿、粪便、组织液或培养液等可能含有人gas6或表达人gas6的细胞的生物试样,则没有特别限定。含有本发明的单克隆抗体或该抗体片段的诊断药可以根据目标诊断法而含有用于进行抗原抗体反应的试剂、该反应的检测用试剂。作为用于进行抗原抗体反应的试剂,可以列举:缓冲剂、盐等。作为检测用试剂,可以列举:识别该单克隆抗体或该抗体片段的标记后的二抗、或与标记对应的底物等通常用于免疫学检测或测定法的试剂。另外,本发明涉及抗人gas6单克隆抗体或该抗体片段在制造gas6相关疾病的治疗药或诊断药中的应用。以下对本发明的抗体的制造方法、疾病的治疗方法及疾病的诊断方法进行具体说明。1.抗体的制造方法(1)抗原的制备成为抗原的人gas6可如下得到:从在大肠杆菌、酵母、昆虫细胞或动物细胞等中导入了含有编码人gas6全长或其部分长度的cdna的表达载体的人gas6表达细胞进行纯化。另外,还可以从大量表达人gas6的各种人细胞株、人细胞、人组织等纯化人gas6,从而得到。进而,还可以通过fmoc法或tboc法等化学合成法制备含有人gas6的部分序列的合成肽,并作为抗原使用。人gas6或具有人gas6的部分序列的合成肽还可以在c末端或n末端附加有flag或his等公知标签。本发明中使用的人gas6可以使用molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)、currentprotocolsinmolecularbiology,johnwiley&sons(1987-1997)等中记载的方法等、例如通过以下方法使宿主细胞中表达编码该人gas6的dna,从而制造。首先,将含有编码人gas6的部分的全长cdna插入适当的表达载体的启动子的下游,从而制作重组载体。也可以代替上述全长cdna而使用基于全长cdna制备的、含有编码多肽的部分的适当长度的dna片段。然后,将得到的该重组载体导入适合于该表达载体的宿主细胞中,从而可以得到生产多肽的转化体。作为表达载体,只要是能够在使用的宿主细胞中进行自主复制或者能够整合到染色体中、并且在能够转录编码多肽的dna的位置含有适当的启动子的表达载体,则均可以使用。作为宿主细胞,只要是大肠杆菌等属于埃希氏菌属等的微生物、酵母、昆虫细胞或动物细胞等可以表达目标基因的宿主细胞,则均可以使用。在使用大肠杆菌等原核生物作为宿主细胞的情况下,重组载体优选为能够在原核生物中自主复制并且含有启动子、核糖体结合序列、含有编码人gas6的部分的dna及转录终止序列的载体。另外,该重组载体不一定需要转录终止序列,但优选在紧接结构基因的下游配置转录终止序列。进而,该重组载体还可以含有调控启动子的基因。作为该重组载体,优选使用将作为核糖体结合序列的夏因-达尔加诺序列(也称为sd序列)与起始密码子的间隔调节为适当距离(例如6~18个碱基)而得到的质粒。另外,作为该编码人gas6的dna的碱基序列,可以对碱基进行置换以形成最适于在宿主内表达的密码子,从而可以提高目标人gas6的生产率。作为表达载体,只要是可以在使用的宿主细胞中发挥功能的表达载体,则均可以使用,可以列举例如:、pbtrp2、pbtac1、pbtac2(以上为罗氏诊断公司制)、pkk233-2(法玛西亚公司制)、pse280(invitrogen公司制)、pgemex-1(promega公司制)、pqe-8(凯杰公司制)、pkyp10(日本特开昭58-110600号)、pkyp200[agriculturalbiologicalchemistry,48,669(1984)]、plsa1[agric.biol.chem.,53,277(1989)]、pgel1[proc.natl.acad.sci.usa,82,4306(1985)]、pbluescriptiisk(-)(stratagene公司制)、ptrs30[由大肠杆菌jm109/ptrs30(fermbp-5407)制备]、ptrs32[由大肠杆菌jm109/ptrs32(fermbp-5408)制备]、pgha2[由大肠杆菌igha2(fermbp-400)制备,日本特开昭60-221091号]、pgka2[由大肠杆菌igka2(fermbp-6798)制备,日本特开昭60-221091号]、pterm2(us4686191、us4939094、us5160735)、psupex、pub110、ptp5、pc194、peg400[j.bacteriol.,172,2392(1990)]、pgex(法玛西亚公司制)、pet系统(novagen公司制)或pme18sfl3等。作为启动子,只要是可以在所使用的宿主细胞中发挥功能的启动子,则可以为任意启动子。可以列举例如:trp启动子(ptrp)、lac启动子、pl启动子、pr启动子或t7启动子等来自大肠杆菌或噬菌体等的启动子。另外,也可以使用两个ptrp串联而成的串联启动子、tac启动子、lact7启动子或leti启动子等人为设计改变后的启动子等。作为宿主细胞,可以列举例如:大肠杆菌xl1-blue、大肠杆菌xl2-blue、大肠杆菌dh1、大肠杆菌mc1000、大肠杆菌ky3276、大肠杆菌w1485、大肠杆菌jm109、大肠杆菌hb101、大肠杆菌no.49、大肠杆菌w3110、大肠杆菌ny49或大肠杆菌dh5α等。作为向宿主细胞中导入重组载体的方法,只要是向使用的宿主细胞中导入dna的方法,则均可以使用,可以列举例如:使用钙离子的方法[proc.natl.acad.sci.usa,69,2110(1972)、gene,17,107(1982)、molecular&generalgenetics,168,111(1979)]。在使用动物细胞作为宿主时,作为表达载体,只要是可以在动物细胞中发挥功能的表达载体,则均可以使用,可以列举例如:pcdnai、pcdm8(funakoshi公司制)、page107[日本特开平3-22979;cytotechnology,3,133(1990)]、pas3-3(日本特开平2-227075)、pcdm8[nature,329,840(1987)]、pcdnai/amp(invitrogen公司制)、pcdna3.1(invitrogen公司制)、prep4(invitrogen公司制)、page103[j.biochemistry,101,1307(1987)]、page210、pme18sfl3、pkantex93(wo97/10354)、n5kg1val(us6,001,358)、inpep4(biogen-idec公司制)及转座子载体(wo2010/143698)等。作为启动子,只要是可以在动物细胞中发挥功能的启动子,则均可以使用,可以列举例如:巨细胞病毒(cmv)的即刻早期(ie)基因的启动子、sv40的早期启动子、逆转录病毒的启动子、金属硫蛋白启动子、热休克启动子、srα启动子或莫洛尼小鼠白血病病毒的启动子或增强子。另外,也可以将人cmv的ie基因的增强子与启动子一同使用。作为宿主细胞,可以列举:人白血病细胞namalwa细胞、猴细胞cos细胞、中华仓鼠卵巣细胞cho细胞(journalofexperimentalmedicine,108,945(1958);proc.natl.acad.sci.usa,60,1275(1968);genetics,55,513(1968);chromosoma,41,129(1973);methodsincellscience,18,115(1996);radiationresearch,148,260(1997);proc.natl.acad.sci.usa,77,4216(1980);proc.natl.acad.sci.,60,1275(1968);cell,6,121(1975);molecularcellgenetics,appendixi,ii(pp.883-900);)、二氢叶酸还原酶基因(以下记作dhfr)缺失的cho细胞(proc.natl.acad.sci.usa,77,4216(1980))、cho-k1(atccccl-61)、dukxb11(atccccl-9096)、pro-5(atccccl-1781)、cho-s(lifetechnologies,cat#11619)、pro-3、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(或者,也称为yb2/0)、小鼠骨髓瘤细胞nso、小鼠骨髓瘤细胞sp2/0-ag14、叙利亚仓鼠细胞bhk或hbt5637(日本特开昭63-000299)等。作为向宿主细胞中导入重组载体的方法,只要是向动物细胞中导入dna的方法,则均可以使用,可以列举例如:电穿孔法[cytotechnology,3,133(1990)]、磷酸钙法(日本特开平2-227075)或脂质体转染法[proc.natl.acad.sci.usa,84,7413(1987)]等。在培养基中培养按照上述方式得到的保有整合有编码人gas6的dna的重组载体的、来自微生物或动物细胞等的转化体,在培养液中生成并蓄积该人gas6,并从该培养液中进行收集,从而能够制造人gas6。在培养基中培养该转化体的方法可以按照宿主的培养中使用的常规方法来进行。在来自真核生物的细胞中表达时,可以得到附加有糖或糖链的人gas6。另外,为了制作在gas6的gla结构域结合有γ-羧基谷氨酸残基(gla)的gas6蛋白,还可以使用导入有作为参与谷氨酸残基的γ-羧基化的酶的维生素k环氧化物还原酶(vkor)、γ-谷氨酰羧化酶(ggcx)的细胞。优选的是,为了促进还原型维生素k的衍生,优选使用导入有维生素k环氧化物还原酶复合体亚单位1(vkorc1)及ggcx两者的细胞。另外,作为vkor、vkorc1及ggcx,只要可以有效地在gas6中导入gla残基则可以为任意酶,可以使用人、大鼠、小鼠等任意物种的酶,可以根据所使用的宿主细胞来选择、使用。优选使用导入有人或大鼠的vkorc1基因及ggcx基因的细胞来制作gla化gas6。在培养通过使用诱导性启动子的重组载体转化的微生物时,可以根据需要在培养基中添加诱导剂。例如,在培养通过使用lac启动子的重组载体转化的微生物时,可以在培养基中添加异丙基-β-d-硫代半乳糖吡喃糖苷等,在培养通过使用trp启动子的重组载体转化的微生物时,可以在培养基中添加吲哚丙烯酸等。作为用于培养以动物细胞为宿主而得到的转化体的培养基,可以使用通常使用的rpmi1640培养基[thejournaloftheamericanmedicalassociation,199,519(1967)]、伊戈尔(eagle)的mem培养基[science,122,501(1952)]、杜尔贝科(dulbecco)改良mem培养基[virology,8,396(1959)]、199培养基[proc.soc.exp.biol.med.,73,1(1950)]、伊思考夫(iscove)改良的杜尔贝科培养基(imdm)培养基或向这些培养基中添加胎牛血清(fbs)等而得到的培养基等。培养通常在ph6~8、30~40℃、5%co2存在下等条件下进行1~7天。另外,培养中可以根据需要向培养基中添加卡那霉素、青霉素等抗生素。作为编码人gas6的基因的表达方法,除了直接表达以外,还可以使用分泌生产或融合蛋白表达等方法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)]。作为人gas6的生产方法,有在宿主细胞内生产的方法、分泌到宿主细胞外的方法或在宿主细胞外膜上生产的方法,可以通过变更所使用的宿主细胞或生产的人gas6的结构来选择适当的方法。在宿主细胞内或宿主细胞外膜上生产人gas6时,可以通过使用鲍尔森等人的方法[j.biol.chem.,264,17619(1989)]、罗等人的方法[proc.natl.acad.sci.,usa,86,8227(1989)、genesdevelop.,4,1288(1990)]、日本特开平05-336963或wo94/23021等中记载的方法来使人gas6主动地分泌到宿主细胞外。另外,还可以利用使用二氢叶酸还原酶基因等的基因扩增体系(日本特开平2-227075)来提高人gas6的生产量。得到的人gas6例如可以按照以下方式来分离、纯化。人gas6在细胞内以溶解状态表达的情况下,培养结束后通过离心分离回收细胞,悬浮于水系缓冲液中后,利用超声波破碎机、法式压滤壶、manton-gulin匀浆器或卧式砂磨机等将细胞破碎,得到无细胞提取液。从将该无细胞提取液离心分离而得到的上清中利用通常的蛋白质分离纯化方法,即单独使用或者组合使用溶剂提取法、利用硫酸铵等的盐析法、脱盐法、利用有机溶剂的沉淀法、使用二乙氨基乙基(deae)-琼脂糖、diaionhpa-75(三菱化学公司制)等树脂的阴离子交换色谱法、使用s-sepharoseff(法玛西亚公司制)等树脂的阳离子交换色谱法、使用丁基琼脂糖、苯基琼脂糖等树脂的疏水色谱法、使用分子筛的凝胶过滤法、亲和色谱法、聚焦色谱法或等电点电泳等电泳法等方法,可以得到纯化标准品。人gas6在细胞内形成不溶物而表达的情况下,与上述同样地回收细胞后将其破碎,并进行离心分离,从而以沉淀组分的形式回收该人gas6的不溶物。利用蛋白变性剂使回收的该人gas6的不溶物增溶。对该增溶液进行稀释或透析,从而使该人gas6恢复至正常的立体结构,然后,通过与上述同样的分离纯化方法,可以得到多肽的纯化标准品。人gas6或其糖基化物等衍生物被分泌到细胞外的情况下,可以在培养上清中回收该人gas6或其糖基化物等衍生物。将该培养物与上述同样地利用离心分离等方法进行处理,从而得到可溶性组分,使用与上述同样的分离纯化方法,从而能够从该可溶性组分中得到纯化标准品。另外,本发明中使用的人gas6也可以通过fmoc法或tboc法等化学合成法来制造。此外,也可以利用advancedchemtech公司、珀金埃尔默公司、法玛西亚公司、proteintechnologyinstrument公司、synthecell-vega公司、パ一セプチブ公司或岛津制作所公司制造等肽合成仪进行化学合成。(2)动物的免疫和融合用抗体产生细胞的制备用(1)所得到的抗原免疫3~20周龄的小鼠、大鼠或仓鼠等动物,采集该动物的脾、淋巴结、外周血中的抗体产生细胞。另外,也可以使用小鼠gas6敲除小鼠作为被免疫动物。免疫通过将抗原连同例如弗氏完全佐剂或者氢氧化铝凝胶和百日咳菌疫苗等适当的佐剂一起给药到动物的皮下或静脉内或腹腔内来进行。在抗原为部分肽的情况下,与bsa(牛血清白蛋白)或klh(keyholelimpethemocyanin,匙孔血蓝蛋白)等载体蛋白制成偶联物,将其作为免疫原使用。关于抗原的给药,在第1次给药之后,每隔1~2周进行5~10次。每次给药后第3~7天从眼底静脉丛采血,使用酶免疫测定法[antibodies-alaboratorymanual,coldspringharborlaboratory(1988)]等测定其血清的抗体效价。将其血清对免疫所用的抗原显示出充分的抗体效价的动物作为融合用抗体产生细胞的供给源。在抗原的末次给药后第3~7天,从免疫后的动物摘除脾脏等含有抗体产生细胞的组织,提取抗体产生细胞。在使用脾脏细胞的情况下,将脾脏细细切碎并搅散后,进行离心分离,进而除去红细胞,得到融合用抗体产生细胞。(3)骨髓瘤细胞的制备作为骨髓瘤细胞,使用由小鼠得到的细胞系,例如,使用8-氮杂鸟嘌呤抗性小鼠(来自balb/c)骨髓瘤细胞株p3-x63ag8-u1(p3-u1)[currenttopicsinmicrobiologyandimmunology,18,1(1978)]、p3-ns1/1-ag41(ns-1)[europeanj.immunology,6,511(1976)]、sp2/0-ag14(sp-2)[nature,276,269(1978)]、p3-x63-ag8653(653)[j.immunology,123,1548(1979)]或p3-x63-ag8(x63)[nature,256,495(1975)]等。该骨髓瘤细胞用正常培养基[加入了谷氨酰胺、2-巯基乙醇、庆大霉素、fbs及8-氮杂鸟嘌呤的rpmi1640培养基]进行传代,在细胞融合前的3~4天传代到正常培养基中,在融合当天确保2×107个以上的细胞数。(4)细胞融合和产生单克隆抗体的杂交瘤的制备将(2)所得到的融合用抗体产生细胞和(3)中得到的骨髓瘤细胞用最低必需培养基(mem)或pbs(磷酸氢二钠1.83g、磷酸二氢钾0.21g、氯化钠7.65g、蒸馏水1升、ph7.2)充分洗涤,按照使细胞数达到融合用抗体产生细胞∶骨髓瘤细胞=5~10∶1的方式进行混合,离心分离后,除去上清。将沉淀的细胞群充分搅散后,在37℃下一边搅拌一边加入聚乙二醇-1000(peg-1000)、mem培养基及二甲亚砜的混合液。进而,每1~2分钟加入1~2ml的mem培养基,重复数次后,加入mem培养基使总量达到50ml。离心分离后,除去上清。将沉淀的细胞群轻轻搅散后,将融合用抗体产生细胞轻轻地悬浮于hat培养基[添加有次黄嘌呤、胸腺嘧啶核苷及氨基蝶呤的正常培养基]中。将该悬浮液在5%co2的培养箱中在37℃下培养7~14天。培养后,抽取一部分培养上清,通过后述的结合测定等杂交瘤选择方法选择与包含人gas6的抗原反应且与不含人gas6的抗原不反应的细胞群。另外,通过后述的竞争测定等选择产生抑制人gas6与axl等gas6受体的结合的抗人gas6抗体的杂交瘤细胞群。然后,通过有限稀释法进行克隆,选择稳定地确认到强抗体效价的细胞作为产生单克隆抗体的杂交瘤。(5)纯化单克隆抗体的制备对姥鲛烷处理[腹腔内给药0.5ml的2,6,10,14-四甲基十五碳烷(pristane)并饲养2周]后的8~10周龄的小鼠或裸小鼠腹腔内注射(4)中得到的产生单克隆抗体的杂交瘤。在10~21天杂交瘤产生癌性腹水。从该小鼠中收集腹水,离心分离除去固态成分后,用40~50%的硫酸铵盐析,利用辛酸沉淀法、deae-琼脂糖柱、蛋白a柱或凝胶过滤柱进行纯化,收集igg或igm组分,得到纯化单克隆抗体。另外,将(4)所得到的产生单克隆抗体的杂交瘤在添加有10%fbs的rpmi1640培养基等中培养,然后,通过离心分离除去上清,悬浮于杂交瘤sfm培养基中,培养3~7天。将得到的细胞悬浮液离心分离,利用蛋白a柱或蛋白g柱进行纯化,而由所得到的上清收集igg组分,从而也可以得到纯化单克隆抗体。需要说明的是,杂交瘤-sfm培养基中也可以添加5%ダイゴgf21。抗体的亚类的决定使用亚类分型试剂盒通过酶免疫测定法来进行。蛋白质量的定量通过劳里法或280nm下的吸光度来计算。(6)单克隆抗体的选择单克隆抗体的选择通过以下所示的利用酶免疫测定法的结合测定或竞争测定等来进行。除了这些方法以外,还可以通过利用biacore(注册商标)的动力学分析等来选择单克隆抗体。另外,还可以利用公知的方法[theprostate,67,1163(2007)]鉴定抗体的靶抗原,从而选择单克隆抗体。(6-a)结合测定作为抗原,使用将(1)所得到的、含有编码人gas6的cdna的表达载体导入大肠杆菌、酵母、昆虫细胞或动物细胞等中而得到的重组蛋白,或者由人组织得到的纯化多肽或部分肽等。在抗原为重组蛋白时,可以附加有flag或his等标签。在抗原为部分肽时,可以制作与bsa或klh等载体蛋白偶联物并使用该偶联物。将抗原分注到96孔板等板中并固定,然后分注作为第1抗体的血清、杂交瘤的培养上清或纯化单克隆抗体等受试物质并使其反应。用pbs或pbs-吐温等充分洗涤后,分注作为第2抗体的用生物素、酶、化学发光物质或放射性化合物等标记后的抗免疫球蛋白抗体并使其反应。用pbs-吐温充分洗涤后,进行与第2抗体的标记物质相应的反应,来选择与免疫原特异性反应的单克隆抗体。另外,与含有本发明的与人gas6结合的单克隆抗体所结合的表位的表位结合的抗体可以如下得到:用公知的方法鉴定通过上述结合测定体系得到的抗体的表位,制作含有鉴定出的表位的合成肽等并进行免疫。进而,与和本发明的与人gas6结合的单克隆抗体所结合的表位相同的表位结合的抗体可以如下得到:对通过上述结合测定体系得到的抗体的表位进行鉴定,制作鉴定出的表位的部分合成肽或模拟表位的立体结构的合成肽等并进行免疫。(6-b)竞争测定按照(1)所述的方法制备人axl细胞外结构域和人igg1重链恒定区融合蛋白(haxl-hfc)。haxl-hfc中,在人axl细胞外结构域和igg1重链恒定区之间可以具有适当的限制酶识别序列。将得到的haxl-hfc分注到96孔板中并固定。然后,将杂交瘤培养上清或纯化单克隆抗体等受试物质、及(1)所得到的带标签的hgas6的混合溶液分注到孔中而使其反应。用pbs或pbs-吐温等充分洗涤后,分注作为检测用抗体的用生物素、酶、化学发光物质或放射性化合物等标记后的针对上述标签的抗体而使其反应。用pbs-吐温充分洗涤后,进行与检测用抗体的标记物质相应的反应,选择抑制hgas6与haxl-hfc的结合的单克隆抗体。(6-c)利用biacore(注册商标)的动力学分析使用biacore(注册商标)t100测定抗原与受试物质之间的结合的动力学,将其结果用设备附带的分析软件进行分析。将抗小鼠igg抗体通过胺偶联法固定在传感器芯片cm5上后,使杂交瘤培养上清或纯化单克隆抗体等受试物质流过,从而使其以适当量结合,进而使浓度已知的多个浓度的抗原流过,对结合、解离进行测定。将得到的数据用设备附带的软件利用1∶1结合模型来进行动力学分析,得到各种参数。或者,将人gas6、其部分肽、其部分肽与载体蛋白的偶联物通过例如胺偶联法固定在传感器芯片上后,使浓度已知的多个浓度的纯化单克隆抗体流过,对结合及解离进行测定。将得到的数据用机器附带的软件利用二价结合模型进行动力学分析,得到各种参数。2.基因重组抗体的制作作为基因重组抗体的制作例子,以下示出人嵌合抗体及人源化抗体的制作方法。(1)基因重组抗体表达用载体的构建基因重组抗体表达用载体是整合有编码人抗体的ch及cl的dna的动物细胞用表达载体,可以通过将编码人抗体的ch及cl的dna分别克隆到动物细胞用表达载体中来构建。人抗体的c区可以使用任意的人抗体的ch及cl。例如,使用人抗体的γ1亚类的ch及κ类的cl等。作为编码人抗体的ch及cl的dna,使用edna,也可以使用含有外显子和内含子的染色体dna。作为动物细胞用表达载体,只要是可以整合并表达编码人抗体的c区的基因的载体,则可以使用的载体。可以使用例如page107[cytotechnol.,3,133(1990)]、page103[j.biochem.,101,1307(1987)〕、phsg274[gene,27,223(1984)]、pkcr[proc.natl.acad.sci.usa,78,1527(1981)]、psg1bd2-4[cytotechnol.,4,173(1990)]或pse1uk1sed1-3[cytotechnol.,13,79(1993)]等。作为动物细胞用表达载体中的启动子和增强子,使用sv40的早期启动子[j.biochem.,101,1307(1987)]、莫洛尼小鼠白血病病毒ltr[biochem.biophys.res.commun.,149,960(1987)]或免疫球蛋白h链的启动子[cell,41,479(1985)]和增强子[cell,33,717(1983)]等。从基因重组抗体表达载体的构建的容易度、向动物细胞中导入的容易度、抗体h链及l链在动物细胞内的表达量的平衡均衡等观点出发,基因重组抗体表达用载体使用抗体h链及l链存在于同一载体上的类型(串联型)的基因重组抗体表达用载体[j.immunol.methods,167,271(1994)],但也可以使用抗体h链及l链存在于不同载体上的类型。作为串联型基因重组抗体表达用载体,使用pkantex93(wo97/10354)、pee18[hybridoma,17,559(1998)]等。(2)编码来自人类以外的动物的抗体的v区的cdna的得到及氨基酸序列的分析编码非人抗体的vh及vl的cdna的得到及氨基酸序列的分析可以如下进行。从产生非人抗体的杂交瘤细胞中提取mrna,并合成cdna。将合成的cdna克隆到噬菌体或质粒等载体中,制作cdna文库。使用编码小鼠抗体的c区部分或v区部分的dna作为探针,从该文库中分别分离出具有编码vh或vl的cdna的重组噬菌体或重组质粒。分别确定重组噬菌体或重组质粒上的目标小鼠抗体的vh或vl的全部碱基序列,由碱基序列分别推测出vh或vl的全部氨基酸序列。作为制作产生非人抗体的杂交瘤细胞的人类以外的动物,使用小鼠、大鼠、仓鼠或兔等,只要能够制作杂交瘤细胞,则任何动物均可以使用。由杂交瘤细胞制备总rna时,使用异硫氰酸胍三氟乙酸铯法[methodsinenzymol.,154,3(1987)]或rnaeasykit(凯杰公司制)等试剂盒等。由总rna制备mrna时,使用固定有寡聚(dt)的纤维素柱法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)]或oligo-dt30<super>mrnapurification(注册商标)试剂盒(宝生物公司制)等试剂盒等。另外,也可以使用fasttrackmrnaisolation(注册商标)kit(invitrogen公司制)或quickprepmrnapurification(注册商标)kit(法玛西亚公司制)等试剂盒从杂交瘤细胞中制备mrna。cdna的合成及cdna文库的制作时,使用公知的方法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)、currentprotocolsinmolecularbiology,supplement1,johnwiley&sons(1987-1997)]或superscriptplasmidsystemforcdnasynthesisandplasmidcloning(invitrogen公司制)或zap-cdnasynthesis(注册商标)kit(stratagene公司制)等试剂盒等。制作cdna文库时,整合以由杂交瘤细胞提取的mrna为模板而合成的cdna的载体只要是可以整合该cdna的载体,则均可以使用。可以使用例如zapexpress[strategies,5,58(1992)]、pbluescriptiisk(+)[nucleicacidsresearch,17,9494(1989)]、λzapii(stratagene公司制)、λgt10、λgt11[dnacloning:apracticalapproach,i,49(1985)]、lambdabluemid(クロ一ンテツク公司制)、λexcell、pt7t3-18u(法玛西亚公司制)、pcd2[mol.cell.biol.,3,280(1983)]或puc18[gene,33,103(1985)]等。作为用于导入由噬菌体或质粒载体构建的cdna文库的大肠杆菌,只要是可以导入、表达及维持该cdna文库的大肠杆菌,则均可以使用。可以使用例如xl1-bluemrf’[strategies,5,81(1992)]、c600[genetics,39,440(1954)]、y1088、y1090[science,222,778(1983)]、nm522[j.mol.biol.,166,1(1983)]、k802[j.mol.biol.,16,118(1966)]或jm105[gene,38,275(1985)]等。从cdna文库中选择编码非人抗体的vh或vl的cdna克隆时,使用利用同位素或荧光标记的探针的集落杂交法或噬菌斑杂交法[molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)]等。另外,制备引物,以由mrna合成的cdna或cdna文库为模板来进行聚合酶链式反应法[以下记作pcr法;molecularcloning,alaboratorymanual,secondedition,coldspringharborlaboratorypress(1989)、currentprotocolsinmolecularbiology,supplement1,johnwiley&sons(1987-1997)],从而也可以制备编码vh或vl的cdna。将选择出的cdna用适当的限制酶等切断后,克隆到pbluescriptsk(-)(stratagene公司制)等质粒中,利用通常使用的碱基序列分析方法等确定该cdna的碱基序列。作为碱基序列分析方法,进行例如双脱氧法[proc.natl.acad.sci.usa,74,5463(1977)]等反应后,使用abiprism3700(pebiosystems公司制)或a.l.f.dna测序仪(法玛西亚公司制)等碱基序列自动分析装置等。由确定的碱基序列分别推测出vh及vl的全部氨基酸序列,并与已知的抗体的vh及vl的全部氨基酸序列[sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]进行比较,从而分别确认所得到的cdna是否编码含有分泌信号序列的抗体的vh及vl的完整氨基酸序列。关于含有分泌信号序列的抗体的vh及vl的完整氨基酸序列,通过与已知的抗体的vh及vl的全部氨基酸序列[sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]进行比较,能够推测出分泌信号序列的长度及n末端氨基酸序列,进而可以获知它们所属的亚类。另外,关于vh及vl的各cdr的氨基酸序列,也可以通过与已知的抗体的vh及vl的氨基酸序列[sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]进行比较来找出。另外,使用所得到的vh及vl的完整氨基酸序列,对例如swiss-prot或pir-protein等任意的数据库利用blast法[j.mol.biol.,215,403(1990)]等进行同源性检索,可以确认vh及vl的完整氨基酸序列是否是新的。(3)人嵌合抗体表达载体的构建将分别编码非人抗体的vh或vl的cdna分别克隆到(1)所得到的基因重组抗体表达用载体的编码人抗体的ch或cl的各基因的上游,从而可以构建人嵌合抗体表达载体。为了使编码非人抗体的vh或vl的cdna的3’末端侧与人抗体的ch或cl的5’末端侧连接,制作按照如下方式设计的vh及vl的cdna:连接部分的碱基序列编码适当的氨基酸,并且成为适当的限制酶识别序列。将所制作的vh及vl的cdna分别克隆到(1)所得到的基因重组抗体表达用载体的编码人抗体的ch或cl的各基因的上游,并且使它们以适当的形式表达,从而构建人嵌合抗体表达载体。另外,也可以使用在两端具有适当的限制酶识别序列的合成dna,通过pcr法分别扩增编码非人抗体vh或vl的cdna,并克隆到(1)所得到的基因重组抗体表达用载体中。(4)编码人源化抗体的v区的cdna的构建编码人源化抗体的vh或vl的cdna可以如下构建。分别选择用于移植非人抗体的vh或vl的cdr的氨基酸序列的人抗体的vh或vl的fr的氨基酸序列。选择的fr的氨基酸序列只要是人抗体来源的氨基酸序列,则均可以使用。例如,使用蛋白数据库(proteindatabank)等数据库中登录的人抗体的fr的氨基酸序列或人抗体的fr的各亚类的共有氨基酸序列[sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]等。为了抑制抗体的结合活性的降低,选择与原来的抗体的vh或vl的fr的氨基酸序列具有尽量高的同源性(至少60%以上)的fr的氨基酸序列。然后,向选择出的人抗体的vh或vl的fr的氨基酸序列中分别移植原抗体的cdr的氨基酸序列,从而分别设计出人源化抗体的vh或vl的氨基酸序列。通过考虑在抗体基因的碱基序列中出现的密码子的使用频率[sequencesofproteinsofimmunologicalinterest,usdept.healthandhumanservices(1991)]而将设计出的氨基酸序列变换成dna序列,从而分别设计出编码人源化抗体的vh或vl的氨基酸序列的dna序列。基于设计出的dna序列,合成多条含有100碱基左右的长度的合成dna,使用这些合成dna进行pcr反应。这种情况下,从pcr反应中的反应效率及能够合成的dna的长度考虑,优选对于vh、vl分别设计各6条合成dna。进而,通过在位于两端的合成dna的5’或3’末端导入适当的限制酶识别序列,从而可以将编码人源化抗体的vh或vl的cdna容易地克隆到(1)所得到的基因重组抗体表达用载体中。pcr反应后,将扩增产物分别克隆岛pbluescriptsk(-)(stratagene公司制)等质粒中,通过与(2)所述的方法同样的方法确定碱基序列,得到具有编码期望的人源化抗体的vh或vl的氨基酸序列的dna序列的质粒。或者,也可以基于设计出的dna序列以1条长链dna形式分别合成出vh全长及vl全长,使用其来代替上述pcr扩增产物。进而,通过在合成长链dna的两端导入适当的限制酶识别序列,从而可以将编码人源化抗体的vh或vl的cdna容易地克隆岛(1)所得到的基因重组抗体表达用载体中。(5)人源化抗体的v区的氨基酸序列的改变仅将非人抗体的vh及vl的cdr移植到人抗体的vh及vl的fr中时,人源化抗体的抗原结合活性与原来的非人抗体相比降低[bio/technology,9,266(1991)]。在人源化抗体中,鉴定出人抗体的vh及vl的fr的氨基酸序列中直接与抗原结合相关的氨基酸残基、与cdr的氨基酸残基相互作用的氨基酸残基以及维持抗体的立体结构并间接与抗原结合相关的氨基酸残基,将这些氨基酸残基置换为原来的非人抗体的氨基酸残基,从而可以使降低的抗原结合活性提高。为了鉴定与抗原结合活性相关的fr的氨基酸残基,可以通过使用x射线晶体分析[j.mol.biol.,112,535(1977)]或计算机模拟分析[proteinengineering,7,1501(1994)]等来进行抗体的立体结构的构建及分析。另外,通过针对各个抗体制作多种改变体并反复研究它们与各抗原结合活性的相关性,通过进行各种尝试可以得到具有所需要的抗原结合活性的人源化抗体。人抗体的vh及vl的fr的氨基酸残基可以通过使用改变用合成dna进行(4)所述的pcr反应来进行改变。关于pcr反应后的扩增产物,通过(2)所述的方法确定碱基序列,来确认已实施了目标改变。(6)人源化抗体表达载体的构建将构建的编码基因重组抗体的vh或vl的cdna分别克隆到(1)所得到的基因重组抗体表达用载体的编码人抗体的ch或cl的各基因的上游,从而可以构建人源化抗体表达载体。例如,通过在(4)及(5)所得到的构建人源化抗体的vh或vl时使用的合成dna中、位于两端的合成dna的5’或3’末端导入适当的限制酶识别序列,并分别克隆到(1)所得到的人源化抗体表达用载体的编码人抗体的ch或cl的各基因的上游,并使它们以适当的形式表达。(7)基因重组抗体的瞬时表达使用(3)及(6)所得到的基因重组抗体表达载体或对它们进行了改变的表达载体进行基因重组抗体的瞬时表达,从而可以高效地评价所制作的多种人嵌合抗体、人源化抗体的抗原结合活性。作为导入表达载体的宿主细胞,只要是可以表达基因重组抗体的宿主细胞则任意细胞均可以使用,例如,使用cos-7细胞[americantypeculturecollection(atcc)编号:crl1651][methodsinnucleicacidsres.,crcpress,283(1991)]。为了向cos-7细胞中导入表达载体,使用deae-葡聚糖法[methodsinnucleicacidsres.,crcpress(1991)]或脂质体转染法[proc.natl.acad.sci.usa,84,7413(1987)]等。导入表达载体后,使用酶免疫抗体法[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、antibodies-alaboratorymanual,coldspringharborlaboratory(1988)、单克隆抗体实验手册,講談社サイエンテイフイック(1987)]等测定培养上清中的基因重组抗体的表达量及抗原结合活性。(8)稳定表达基因重组抗体的转化株的得到和基因重组抗体的制备通过将(3)及(6)所得到的基因重组抗体表达载体导入适当的宿主细胞,从而可以得到稳定表达基因重组抗体的转化株。为了向宿主细胞中导入表达载体,使用电穿孔法[日本特开平2-257891、cytotechnology,3,133(1990)]等。作为导入基因重组抗体表达载体的宿主细胞,只要是可以表达基因重组抗体的宿主细胞,则任意细胞均可以使用。例如,使用cho-k1(atccccl-61)、dukxb11(atccccl-9096)、pro-5(atccccl-1781)、cho-s(lifetechnologies,cat#11619)、大鼠骨髓瘤细胞yb2/3hl.p2.g11.16ag.20(atcc编号:crl1662,或者也称为yb2/0)、小鼠骨髓瘤细胞ns0、小鼠骨髓瘤细胞sp2/0-ag14(atcc编号:crl1581)、小鼠p3x63-ag8.653细胞(atcc编号:crl1580)、二氢叶酸还原酶基因(dihydroforatereductase,以下记作dhfr)缺失的cho细胞[proc.natl.acad.sci.usa,77,4216(1980)]等。另外,还可以使用与细胞内糖核苷酸gdp-岩藻糖的合成相关的酶等蛋白、或与使岩藻糖的1位与n-糖苷键复合型糖链的还原末端的n-乙酰葡糖胺的6位形成α键的糖链修饰相关的酶等蛋白、或与细胞内糖核苷酸gdp-岩藻糖向高尔基体的转运相关的蛋白等的活性下降或缺失的宿主细胞、例如α1,6-岩藻糖转移酶基因缺失的cho细胞(wo2005/035586、wo02/31140)、得到了凝集素抗性的lec13[somaticcellandmoleculargenetics,12,55(1986)]等。导入表达载体后,通过含有g418硫酸盐(以下记作g418)等药剂的动物细胞培养用培养基进行培养,从而选择稳定表达基因重组抗体的转化株(日本特开平2-257891)。作为动物细胞培养用培养基,使用rpmi1640培养基(invitrogen公司制)、git培养基(日本制药公司制)、ex-cell301培养基(jrh公司制)、imdm培养基(invitrogen公司制)、hybridoma-sfm培养基(invitrogen公司制)或在这些培养基添加有fbs等各种添加物的培养基等。通过在培养基中培养所得到的转化株,从而表达基因重组抗体并蓄积在培养上清中。培养上清中的基因重组抗体的表达量及抗原结合活性可以通过elisa法等来测定。另外,可以利用dhfr基因扩增体系(日本特开平2-257891)等来提高转化株所产生的基因重组抗体的表达量。基因重组抗体使用蛋白a柱从转化株的培养上清中纯化[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、antibodies-alaboratorymanual,coldspringharborlaboratory(1988)]。另外,还可以将凝胶过滤、离子交换色谱及超滤等用于蛋白质纯化的方法组合。纯化后的基因重组抗体的h链、l链或完整抗体分子的分子量可以使用聚丙烯酰胺凝胶电泳法[nature,227,680(1970)]或蛋白质印迹法[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、antibodies-alaboratorymanual,coldspringharborlaboratory(1988)]等来测定。3.纯化单克隆抗体或该抗体片段的活性评价纯化后的本发明的单克隆抗体或该抗体片段的活性评价可以按照以下方式来进行。与人gas6的结合活性使用上述1-(6a)所述的结合测定来测定、以及使用利用(6c)所述的biacore(注册商标)系统等的表面等离子激元共振法来测定。另外,可以使用荧光抗体法[cancerimmunol.immunother.,36,373(1993)]等来测定。人gas6与gas6受体的结合抑制活性例如可以使用上述1-(6b)所述的竞争测定来测定。4.使用本发明的抗人gas6单克隆抗体或该抗体片段的疾病的治疗方法只要是人gas6依赖性的细胞增殖、病变等与gas6相关的疾病,则任意的人gas6相关疾病的治疗中均可以使用本发明的单克隆抗体或该抗体片段。含有本发明的单克隆抗体或该抗体片段的治疗剂可以仅含有作为有效成分的该抗体或该抗体片段,但通常以与药理学上可接受的1种以上载体一起混合、通过制剂学
技术领域:
中公知的方法制造的药物制剂形式来提供。作为给药途径,可以列举:经口给药或口腔内、气管内、直肠内、皮下、肌肉内或静脉内等非经口给药。作为给药形式,可以列举例如:喷雾剂、胶囊剂、片剂、散剂、颗粒剂、糖浆剂、乳剂、栓剂、注射剂、软膏或胶带剂等。适于经口给药的制剂为乳剂、糖浆剂、胶囊剂、片剂、散剂或颗粒剂等。乳剂或糖浆剂这样的液体制备物使用如下成分作为添加剂来制造:水、蔗糖、山梨糖醇或果糖等糖类;聚乙二醇或丙二醇等二醇类;芝麻油、橄榄油或大豆油等油类;对羟基苯甲酸酯类等防腐剂;或者,草莓香料或薄荷等香料类等。胶囊剂、片剂、散剂或颗粒剂等使用如下成分作为添加剂来制造:乳糖、葡萄糖、蔗糖或甘露醇等赋形剂;淀粉或藻酸钠等崩解剂;硬脂酸镁或滑石等润滑剂;聚乙烯醇、羟丙基纤维素或明胶等粘结剂;脂肪酸酯等表面活性剂;或甘油等增塑剂等。作为适于非经口给药的制剂,为注射剂、栓剂或喷雾剂等。注射剂使用包含盐溶液、葡萄糖溶液或者这两者的混合物的载体等来制造。栓剂使用可可脂、氢化脂肪或羧酸等载体来制造。喷雾剂使用不刺激接受者的口腔及气道粘膜、并且使本发明的单克隆抗体或该抗体片段以微细粒子的形式分散从而易于吸收的载体等来制造。作为载体,使用例如乳糖或甘油等。另外,也可以制成气溶胶或干粉。进而,上述非经口剂中,也可以添加在适于经口给药的制剂中作为添加剂所例示的成分。5.使用本发明的抗人gas6单克隆抗体或该抗体片段的疾病的诊断方法通过使用本发明的单克隆抗体或该抗体片段来检测或测定人gas6或表达人gas6的细胞,从而可以诊断人gas6相关疾病。作为人gas6相关疾病的肾病或癌症的诊断例如可以通过免疫学方法来检测或测定存在于患者体内的人gas6而进行。另外,可以使用流式细胞仪等免疫学方法来检测在患者体内的细胞中表达的人gas6,从而进行诊断。免疫学方法是指使用带有标记的抗原或抗体来检测或测定抗体量或抗原量的方法。例如,使用放射免疫测定法、酶免疫测定法、荧光免疫测定法、发光免疫测定法、蛋白质印迹法或物理化学方法等。放射免疫测定法中,例如,使本发明的抗体或该抗体片段与抗原或表达抗原的细胞等反应,进而与带有放射线标记的抗免疫球蛋白抗体或结合片段反应后,用闪烁记数器等测定。酶免疫测定法中,例如,使本发明的抗体或该抗体片段与抗原或表达抗原的细胞等反应,进而与带有标记的抗免疫球蛋白抗体或结合片段反应后,用吸光光度计测定显色色素。例如,使用夹心elisa法等。作为酶免疫测定法中使用的标记物,可以使用公知[酶免疫测定法,医学书院(1987)]的酶标记。例如,使用碱性磷酸酶标记、过氧化物酶标记、荧光素酶标记或生物素标记等。夹心elisa法是使抗体结合于固相后使其捕获作为检测或测定对象的抗原、并使第2抗体与所捕获的抗原反应的方法。该elisa法中,准备2种识别所要检测或测定的抗原、且抗原识别位点不同的抗体或抗体片段,将其中的第1抗体或抗体片段预先吸附在板(例如,96孔板)上,然后将第2抗体或抗体片段用fitc等荧光物质、过氧化物酶等酶或生物素等标记。使由生物体内分离出的细胞或其破碎液、组织或其破碎液、细胞培养上清、血清、胸水、腹水或眼液等与上述吸附有抗体的板反应后,与标记后的单克隆抗体或抗体片段反应,进行与标记物质相应的检测反应。由将浓度已知的抗原阶梯稀释而制作的标准曲线计算受试样品中的抗原浓度。作为夹心elisa法所使用的抗体,可以使用多克隆抗体或单克隆抗体中的任一种,也可以使用fab、fab’或f(ab)2等抗体片段。作为夹心elisa法中使用的2种抗体的组合,可以是识别不同表位的单克隆抗体或抗体片段的组合,也可以是多克隆抗体和单克隆抗体或抗体片段的组合。荧光免疫测定法通过文献[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、单克隆抗体实验手册、kodan-shascientific(1987)]等所述的方法来进行测定。作为荧光免疫测定法中使用的标记物,可以使用公知[荧光抗体法,ソフトサイエンス社(1983)]的荧光标记。例如,使用fitc或ritc等。发光免疫测定法通过文献[生物发光和化学发光临床检查42,广川书店(1998)]等所述的方法来进行测定。作为发光免疫测定法中使用的标记物,可以列举公知的发光体标记,使用吖啶酯、洛酚碱等。蛋白质印迹法中,将抗原或表达抗原的细胞等用sds(十二烷基硫酸钠)-page(聚丙烯酰胺凝胶)[antibodies-alaboratorymanualcoldspringharborlaboratory(1988)]分级后,将该凝胶印迹于聚偏氟乙烯(pvdf)膜或硝酸纤维素膜,使识别抗原的抗体或抗体片段与该膜反应,进而与带有fitc等荧光物质、过氧化物酶等酶标记或生物素标记等的抗小鼠igg抗体或结合片段反应后,对该标记可视化而进行测定。以下示出一例。将表达具有序列号4的氨基酸序列的多肽的细胞、组织溶解,在还原条件下,使每个泳道的蛋白量为0.1~30μg,通过sds-page法进行电泳。将电泳后的蛋白质转印到pvdf膜上,在含有1~10%bsa的pbs(以下记作bsa-pbs)中在室温下反应30分钟,从而进行封闭操作。这里,使本发明的单克隆抗体反应,用含有0.05~0.1%的吐温20的pbs(以下记作吐温-pbs)洗涤,与过氧化物酶标记后的山羊抗小鼠igg在室温下反应2小时。用吐温-pbs洗涤,使用ecl蛋白质印迹检测试剂(安玛西亚公司制)等检测结合有单克隆抗体的条带,从而检测具有序列号4的氨基酸序列的多肽。作为蛋白质印迹中的检测时使用的抗体,使用可以与未保持天然型立体结构的多肽结合的抗体。物理化学方法如下进行:例如,通过使作为抗原的人gas6与本发明的单克隆抗体或该抗体片段结合而形成凝集体,对该凝集体进行检测。此外,作为物理化学方法,还可以使用毛细管法、一维免疫扩散法、免疫比浊法或胶乳免疫比浊法[临床检查法提要,金原出版(1998)]等。胶乳免疫比浊法中,使用以抗体或抗原致敏的粒径0.1~1μm程度的聚苯乙烯胶乳等载体,利用相应的抗原或抗体引起抗原抗体反应则反应液中的散射光增加、透射光减少。通过以吸光度或积分球浊度形式检测该变化,从而测定受试样品中的抗原浓度等。表达人gas6的细胞的检测或测定可以使用公知的免疫学检测法,其中优选使用免疫沉降法、免疫细胞染色法、免疫组织染色法或荧光抗体染色法等。免疫沉淀法中,使表达人gas6的细胞等与本发明的单克隆抗体或该抗体片段反应后,加入对蛋白g-琼脂糖等免疫球蛋白具有特异性结合能力的载体,从而使抗原抗体复合物沉降。或者,也可以通过如下方法来进行。使上述本发明的单克隆抗体或该抗体片段固定在elisa用96孔板上后,通过bsa-pbs进行封闭。在抗体为例如杂交瘤培养上清等未纯化的状态的情况下,使抗小鼠免疫球蛋白、抗大鼠免疫球蛋白、蛋白a或蛋白g等预先固定在elisa用96孔板上,用bsa-pbs封闭。分注杂交瘤培养上清而使其进行结合。接着,弃去bsa-pbs并用pbs充分洗涤,然后,使表达人gas6的细胞或组织的溶解液进行反应。用sds-page用样品缓冲液从充分洗涤后的板上提取免疫沉降物,通过上述蛋白免疫印迹进行检测。免疫细胞染色法或免疫组织染色法为如下方法:将表达抗原的细胞或组织等根据情况用表面活性剂或甲醇等进行处理以增加抗体的透过性,然后,与本发明的单克隆抗体反应,进而,与带有fitc等荧光标记、过氧化物酶等酶标记或生物素标记等的抗免疫球蛋白抗体或其结合片段反应,然后,使该标记可视化并利用显微镜进行镜检。另外,可以通过使荧光标记的抗体与细胞反应、使用流式细胞仪进行分析的荧光抗体染色法[monoclonalantibodies-principlesandpractice,thirdedition,academicpress(1996)、单克隆抗体实验手册,講談社サイエンテイフイック(1987)]进行检测。特别地,与人gas6结合的、本发明的单克隆抗体或该抗体片段可以实现保持天然型立体结构的细胞的检测。另外,荧光抗体染色法中,在使用fmat8100hts系统(アプライドバイオシステム公司制)等的情况下,可以在不将所形成的抗体-抗原复合体和未参与抗体-抗原复合体形成的游离的抗体或抗原分离的情况下来测定抗原量或抗体量。以下,通过实施例更具体地说明本发明,但本发明不受下述实施例限定。实施例[实施例1]gas6敲除(以下记作ko)小鼠的获得由taconic公司购入gas6杂合体ko小鼠的精子。所购入的gas6杂合体ko小鼠的背景为129/svev-c57bl/6。通过cleajapan,inc.进行该gas6杂合体ko小鼠的精子与c57bl/6njcl小鼠的卵子的体外受精后,将受精卵移植到受体小鼠中,得到仔鼠。对于得到的仔鼠,通过公知的方法进行基因分型,确认已敲除了gas6基因,得到gas6纯合体ko小鼠。[实施例2]各种gas6重组体的制作为了用于免疫及筛选,通过以下所述的方法制作在c末端附加了flag标签的人、食蟹猴、大鼠及小鼠的gas6重组蛋白。以下分别记作hgas6-f、cgas6-f、rgas6-f及mgas6-f。(1)hgas6-f表达载体的构建由整合有人gas6的基因序列(序列号3,genbank登录号:nm_000820)的质粒(invitrogen公司制),按照以下方式制作整合有hgas6-f基因序列的动物细胞表达载体。以上述质粒为模板,使用引物1、2(序列号1、2)通过聚合酶链反应(pcr)来扩增含有hgas6-f基因的dna片段。pcr中,制备含有模板质粒、各10pmol的上述2种引物及kodfx(东洋纺公司制)的20μl的反应液,在94℃下保温2分钟后,将在94℃下15秒、58℃下30秒及68℃下2.5分钟作为1个循环,进行30次循环反应。将得到的pcr产物供于琼脂糖凝胶电泳,使用qiaquickgelextractionkit(qiagen公司制)回收约2kbp的扩增dna片段(含有hgas6-f基因的dna片段)。用zeroblunttopopcrcloningkit(invitrogen公司制)将所得到的扩增dna片段插入pcr4-blunt-topo载体中,得到含有质粒pcr4-hgas6-f的反应液。使用该反应液按照常规方法转化大肠杆菌dh5α株(toyobo公司制),由得到的转化体提取质粒pcr4-hgas6-f。对于得到的pcr4-hgas6-f,选择具有不存在起因于pcr的突变的插入基因序列的克隆,用于此后的实验。然后,将pcr4-hgas6-f用限制酶ecori及bamhi进行酶处理。将反应液供于琼脂糖凝胶电泳后,用qiaquickgelextractionkit回收约2kbp的dna片段(以下记作hgas6-f-ecori-bamhi)。同样地,将动物细胞表达用载体pkantex93(wo97/10354)用ecori及bamhi进行酶处理。将反应液供于琼脂糖凝胶电泳后,用qiaquickgelextractionkit回收约9.3kbp的dna片段(以下记作pkantex93-ecori-bamhi)。将得到的2种dna片段用ligationhighver.2(东洋纺公司制)连接,用该反应液转化大肠杆菌dh5α株(toyobo)。由得到的转化体得到作为hgas6-f动物细胞表达载体的pkantex-hgas6-f。(2)cgas6-f表达载体的构建通过以下的方法构建整合有cgas6-f基因的动物细胞表达载体。首先,先进行食蟹猴gas6基因的克隆。通过pcr扩增含有食蟹猴gas6基因的dna片段。pcr中,制备含有作为模板的来自食蟹猴肺的cdna(cytomol公司制)、各10pmol的引物3、4(序列号5、6)、kod-plus-(东洋纺公司制)及2%dmso的20μl的反应液,在94℃下保温2分钟后,将在94℃下15秒、65℃下30秒及68℃下2.5分钟作为1个循环,进行35个循环。此后,通过与(1)同样的方法得到在pcr4-blunt-topo载体中插入有扩增dna片段(含有cgas6基因的dna片段)的质粒pcr4-cgas6。对于得到的质粒,通过常规方法进行序列分析,将得到的食蟹猴gas6的碱基序列示于序列号7,将由该碱基序列推测出的食蟹猴gas6的氨基酸序列示于序列号8。然后,通过pcr法扩增含有cgas6-f基因的dna片段,并整合到动物细胞表达用载体pkantex93中。pcr中,制备含有作为模板的pcr4-gas6、各10pmol的引物5、6(序列号9、10)及primestarhsdnapolymerase(宝生物公司制)的20μl的反应液,在94℃下保温5分钟后,将在98℃下10秒、68℃下2分20秒作为1个循环,进行35个循环。得由到的pcr产物及pkantex93中,回收通过与(1)同样的方法进行限制酶消化后的dna片段(cgas6-f-ecori-bamhi及pkantex93-ecori-bamhi)。将得到的2种dna片段用in-fusionhd克隆试剂盒(clontechlaboratories,inc.制)连接,通过与(1)同样的方法得到cgas6-f动物细胞表达用载体pkantex-cgas6-f。对于得到的载体,选自具有不存在起因于pcr的突变的插入基因的克隆,用于此后的实验。(3)rgas6-f表达载体的构建通过以下的方法制作整合有rgas6-f基因的动物细胞表达载体。通过pcr法扩增含有rgas6-f基因的dna片段。pcr中,制备含有作为模板的来自大鼠心脏或肝脏的cdna(宝生物公司制)、各10pmol的引物7、8(序列号11、12)及kodfx(东洋纺公司制)的20μl的反应液,在94℃下保温2分钟后,将在94℃下15秒、58℃下30秒及68℃下2.5分钟作为1个循环,进行30个循环。通过与(1)同样的方法在pcr4-blunt-topo载体中插入用pcr扩增出的dna片段(含有rgas6-f基因的dna片段),得到质粒pcr4-rgas6-f。得到的质粒所具有的大鼠gas6的碱基序列与genbank登录号:nm_057100所示的大鼠gas6的碱基序列(序列号13)一致,从而确认未产生起因于pcr的基因突变。以得到的pcr4-rgas6-f为基础,通过与(1)同样的方法在pkantex93中插入rgas6-f基因,得到作为rgas6-f动物细胞表达载体的pkantex-rgas6-f。(4)mgas6-f表达载体的构建通过以下所述的方法制作整合有mgas6-f基因的动物细胞表达载体。通过pcr扩增含有mgas6-f基因的dna片段。pcr中,制备含有作为模板的来自小鼠肾脏或肺的cdna(ambion公司制)、各10pmol的引物7、9(序列号11及15)的引物及kodfx(东洋纺公司制)的20μl的反应液,在94℃下保温2分钟后,将在94℃下15秒、58℃下30秒及68℃下2.5分钟作为1个循环,进行30个循环。通过与(1)同样的方法在pcr4-blunt-topo载体中插入用pcr扩增出的dna片段(含有mgas6-f基因的dna片段),得到质粒pcr4-mgas6-f。得到的质粒所具有的小鼠gas6的碱基序列与genbank登录号:nm_019521所示的小鼠gas6的碱基序列(序列号16)一致,确认未产生起因于pcr的基因突变。以得到的pcr4-mgas6-f为基础,通过与(1)同样的方法在pkantex93中插入mgas6-f基因,得到作为mgas6-f动物细胞表达载体的pkantex-mgas6-f。(5)hgas6-f稳定表达细胞株的建立为了建立hgas6-f的稳定表达细胞株,用电穿孔法[サイトテクノロジ一、3,133(199)]按照以下方式将(1)所制作的hgas6-f表达载体pkantex-hgas6-f导入dhfr缺失的cho细胞[proc.natl.acad.sci.usa,77,4216(1980)]中。就细胞的培养而言,在常规传代中,使用基础培养基[含有10%透析fbs(gibco公司制)、1×ht溶液(invitrogen公司制)、50μg/ml庆大霉素(nacalaitesque公司制)的imdm(invitrogen公司制)]。在基因导入后的细胞选拔时,使用含有50nm、200nm或500nm的氨甲喋呤水合物(sigma-aldrich公司制)(以下记作mtx)的基础培养基(mtx培养基)。任一细胞均在37℃、5%co2条件下静置培养。将含有10μg的pkantex-hgas6-f的质粒溶液[将(1)所得到的pkantex-hgas6-f溶解于灭菌水而得的溶液]添加到电穿孔用样品池(bio-rad公司制)中。在该样品池中加入用k-pbs[将137mmol/l的kcl、2.7mmol/l的nacl、8.1mmol/l的na2hpo4、1.5mmol/l的kh2po4、4.0mmol/l的mgcl2混合而成的溶剂]制备的8×106个/ml的细胞悬液,混和后,使用genepulser(bio-rad)在脉冲电压350v、电容量250μf的条件下进行基因导入。将基因导入后的细胞悬液悬浮在50ml的不含ht溶液的基础培养基中,以100μl/孔播种在5块96孔板中。在培养开始14天后将培养基交换成50nmmtx培养基,在第22天交换成200nmmtx培养基,进行mtx抗性细胞株的选拔。在培养第35天,对于可以确认到菌落的细胞株,使用humangas6elisa试剂盒(r&d公司制)测定培养上清中的hgas6-f的表达量。将hgas6-f的表达量高的细胞株放大到24孔板中,在培养开始42天后,将培养基交换成500nmmtx培养基。对于500nmmtx抗性细胞株,通过与上述同样的方法测定培养上清的hgas6-f的表达量,选择hgas6-f的表达量最高的细胞株作为hgas6-f稳定表达细胞株。(6)cgas6-f、rgas6-f及mgas6-f的稳定表达细胞株的制作将(2)~(4)所制作的pkantex-cgas6-f、pkantex-rgas6-f及pkantex-mgas6,通过与(5)同样的方法导入宿主细胞中,来建立cgas6-f、rgas6-f及mgas6-f的稳定表达细胞株。上述载体均通过使用限制酶muli进行酶处理而线形化。通过苯酚/氯仿提取及乙醇沉淀来纯化该线形化载体,溶解于灭菌水而供于实验。培养上清中的cgas6-f的表达量使用humangas6elisa试剂盒(r&d公司制)来测定。另外,rgas6-f及mgas6-f的表达量使用mousegas6elisa试剂盒(r&d公司制)来测定。(7)大鼠vkor及人ggcx表达串联载体的构建为了得到活性型gas6蛋白,需要用作为gla化修饰相关酶的ggcx将gas6的gla结构域所含的谷氨酸残基的γ位的碳羧基化。另外,对于ggcx的活化而言,还原维生素k是必须的,还原维生素k通过vkor(vitaminkepoxidereductasecomplexsubunit1)还原维生素k环氧化物而形成[journalofthrombosisandhaemostasis3,1873-1878(2005)]。因此,为了得到活性型gas6,制作人ggcx(以下记作hggcx)及大鼠vkor(以下记作rvkor)表达载体。首先,通过与(1)同样的方法将rvkor基因整合到pcr4-blunt-topo载体中,得到质粒pcr4-rvkor。pcr中,制备含有作为模板的来自大鼠肝脏的cdna(宝生物公司制)、各10pmol的引物10、11(序列号18、19)的引物及kod-plus-(东洋纺公司制)的反应液,供于实验。得到的质粒所具有的rvkor基因序列与genbank登录号nm_203335所示的大鼠vkor的碱基序列(序列号20)一致,确认未产生起因于pcr的基因突变。通过与(1)同样的方法,将用限制酶hindiii及smai进行酶处理后的pcr4-rvkor插入到用相同限制酶处理后的page249表达载体(j.biol.chem.,278,3466-3473,2003)中,得到page-rvkor。将page-rvkor用限制酶clai进行酶处理,使其与5’末端磷酸化的二种合成寡dna(引物18、19)(序列号38、39)退火,通过与(1)同样的方法得到在page-rvkor中插入有xhoi酶位点的载体page-rvkor(xhoi)。通过与(1)同样的方法得到将hggcx基因整合到pcr4-blunt-topo载体中的质粒pcr4-hggcx。pcr中,制备含有作为模板的来自人肝脏的cdna(ambion公司制)、10pmol的引物12、13(序列号22、23)的引物及kod-plus-(东洋纺公司制)的反应液,供于实验。得到的质粒所具有的hggcx基因序列为genbank登录号nm_000821所示的hggcx基因序列(序列号24)的第145位的胞嘧啶被置换为丙氨酸的序列。但是,这些碱基序列所编码的hggcx的氨基酸序列相同,因此将得到的质粒用于此后的实验。通过与(1)同样的方法,将用限制酶sali及smai进行酶消化后的pcr4-hggcx插入用相同限制酶处理后的page249表达载体中,得到page-hggcx。将page-rvkor(xhoi)和page-hggcx分别用xhoi进行酶处理,通过与(1)同样的方法将含有来自page249的启动子区域的hggcx片段插入page-rvkor(xhoi)中,得到page-vkor-hggcx。(8)page-vkor-hggcx向各种gas6-f稳定表达细胞株中的导入为了制作活性型的各种gas6-f的稳定表达株,通过与(5)同样的方法在(5)及(6)所制作的各种gas6-f稳定表达细胞株中导入(7)所制作的gla化修饰相关酶表达载体page-vkor-hggcx。将基因导入后的细胞悬液悬浮在10ml的500nmmtx培养基中,播种到125cm2烧瓶中。第二天,将培养基交换成mtx·潮霉素培养基[含有500μg/ml的潮霉素(wako公司制)的500mmmtx培养基],在培养开始约1个月后,在175cm2烧瓶中进行放大培养。将得到的细胞株记作活性型的各种gas6-f稳定表达细胞株。(9)各种gas6-f的纯化将(8)所建立的活性型的各种gas6-f稳定表达细胞株悬浮于mtx·潮霉素培养基,接着在细胞用烧瓶中培养3天。然后,将培养基交换成无血清培养基[在ex-cell302(sigma-aldrich公司制)中添加有6mml-谷氨酰胺(invitrogen公司制)、100ng/ml维生素k3(nacalaitesque公司制)、500nmmtx、500μg/ml潮霉素、100nm3,3′,5-三碘代-l-甲状腺原氨酸钠盐(sigma-aldrich公司制)及50μg/ml庆大霉素的培养基],培养5天后,回收培养基。将回收的培养基离心分离,将得到的培养上清用0.22μm过滤器(thermoscientific公司制)进行过滤灭菌。使用回收的培养上清来纯化各种gas6-f。纯化中,使用填充有anti-flagm2affinitygel(sigma-aldrich公司制)的开放柱。在柱中添加培养上清后,用平衡化缓冲液[50mmtris(nacalaitesque公司制)、150mmnacl(nacalaitesque公司制)、0.5%聚氧乙烯失水山梨醇单月桂酸酯(nacalaitesque公司制)(ph8.2)]洗涤柱。然后,用不含聚氧乙烯失水山梨醇单月桂酸酯的平衡化缓冲液洗涤柱后,用洗脱缓冲液[0.1m甘氨酸(nacalaitesque公司制)(ph3.5)或3m氯化镁(nacalaitesque公司制)]洗脱各种gas6-f。得到的各种gas6-f溶液在用nap(gehealthcare公司制)将缓冲液置换为gas6用缓冲液(20mmtris,150mmnacl、ph8.2)后,用0.22μm过滤器进行过滤灭菌,然后用于试验。蛋白质的吸光系数通过将摩尔吸光系数除以分子量而计算(proteinscience,4,2411-2423(1995))。hgas6-f及cgas6-f的吸光系数为0.95。另外,rgas6-f及mgas6-f的吸光系数为0.89。蛋白质溶液中的蛋白浓度使用nanodrop(thermoscientific公司制)来测定。[实施例3]axl细胞外结构域和igg1重链恒定区的复合体的制作(1)人axl细胞外结构域和人igg1重链恒定区的复合体(以下记作haxl-hfc)表达载体的构建通过以下方法制作整合有haxl-hfc基因序列的动物细胞表达用载体。进行含有haxl-hfc的基因序列(序列号28)的全合成,得到pmd19-haxl-hfc(宝生物公司)。序列号28所示的碱基序列中,从5’末端起包含bglii及mlui限制酶识别序列、编码人axl的细胞外结构域的碱基序列(编码序列号26所示的人axl全长的碱基序列中的第1~1314位的碱基序列)、bamhi、sali及ecori限制酶识别序列以及编码人igg1重链恒定区的碱基序列。将pmd19-haxl-hfc及动物细胞表达载体pktabex-tc26.2(wo2013/005649)用限制酶bglii及bamhi进行酶处理,将反应液供于琼脂糖凝胶电泳后,用qiaquickgelextractionkit分别得到约2kbp的dna片段(以下记作haxl-hfc-bglii-bamhi)及约9.6kbp的dna片段(以下记作pktabex-bglii-bamhi)。用ligationhighver.2(东洋纺公司制)使上述2种dna片段结合,通过与实施例2(1)同样的方法得到haxl-hfc重组体表达载体pktabex-haxl-hfc。(2)猴axl细胞外结构域和人igg1重链恒定区的复合体(以下记作caxl-hfc)表达载体的构建构建制备caxl-hfc所必需的表达载体。先通过与实施例2(1)同样的方法得到在pcr4-blunt-topo载体中整合有食蟹猴axl基因的质粒。pcr中,制备含有作为模板的来自食蟹猴肾脏的cdna(cytomol公司制)、各10pmol的引物14、15(序列号30、31)、kod-fx(东洋纺公司制)及2%dmso的20μl的反应液,在94℃下保温2分钟后,将在94℃下15秒、60℃下30秒及68℃下3.5分钟作为1个循环,进行30个循环。将得到的质粒所具有的食蟹猴axl的碱基序列示于序列号32。另外,将由该碱基序列推测出的食蟹猴axl的氨基酸序列示于序列号33。然后,以得到的质粒为模板进行pcr,从容扩增含有编码食蟹猴axl细胞外结构域的碱基序列的dna片段。pcr中,制备含有模板质粒、引物16、17(序列号34、35)各10pmol及primestarhsdnapolymerase(宝生物公司制)的20μl的反应液,在94℃下保温5分钟后,将在94℃下15秒、55℃下10秒、68℃下1分40秒作为1个循环,进行30个循环。将pcr产物供于琼脂糖凝胶电泳,使用qiaquickgelextractionkit(qiagen公司制)得到约1.3kbp的dna片段(以下记作caxl-bglii-ecori)。另外,将(1)所制作的pktabex-haxl-hfc用限制酶bglii及ecori进行酶处理。将反应液供于琼脂糖凝胶电泳后,用qiaquickgelextractionkit得到约9.6kbp的dna片段(以下记作pktabex-hfc-bglii-ecori)。最后,用in-fusionhd克隆试剂盒(clontechlaboratories,inc.制)在pktabex-hfc-bglii-ecori中插入caxl-bglii-ecori,得到caxl-hfc重组体表达载体pktabex-caxl-hfc。将pktabex-caxl-hfc所具有的caxl-hfc的碱基序列示于序列号36,将由该碱基序列推测出的caxl-hfc的氨基酸序列示于序列号37。(3)大鼠axl细胞外结构域和人igg1重链恒定区的复合体(以下记作raxl-hfc)表达载体的构建通过以下所述的方法构建制备raxl-hfc所必需的表达载体。通过genscript公司进行大鼠axl的细胞外结构域的碱基序列的全合成,得到整合于puc57质粒的puc57-raxl。大鼠axl的细胞外结构域的碱基序列使用序列号40所示的大鼠axl的碱基序列(genbank登录号nm_0317941)的1~1329位的碱基。然后,以puc57-raxl为基础,通过与(2)同样的方法得到raxl-hfc重组体表达载体pktabex-raxl-hfc。pcr中,制备含有作为模板的puc57-raxl、各10pmol的引物20、21(序列号42、43)及primestarhsdnapolymerase(宝生物公司制)的20μl的反应液,在94℃下保温5分钟后,将在94℃下15秒、55℃下10秒、68℃下1分40秒作为1个循环,进行30个循环。将raxl-hfc的碱基序列示于序列号40,将由该碱基序列推测出的raxl-hfc的氨基酸序列示于序列号41。(4)小鼠axl细胞外结构域和小鼠igg1重链恒定区的复合体(以下记作maxl-mfc)表达载体的构建通过以下所述的方法构建制备maxl-mfc所必需的表达载体。通过宝生物公司进行含有序列号48所示的maxl-mfc的碱基序列的全合成,得到整合于pmd19质粒的pmd19-maxl-mfc。maxl-mfc从5’末端起包含bglii及mlui识别序列、编码小鼠axl的细胞外结构域的碱基序列(序列号46所示的小鼠axl的碱基序列中的第1~1329位的碱基序列)、bamhi、sali及ecori识别序列以及编码小鼠igg1重链恒定区的碱基序列。将pmd19-maxl-mfc用限制酶bglii及bamhi进行酶处理,通过与(1)同样的方法插入pktabex-tc26.2中,得到maxl-mfc重组体表达载体pktabex-maxl-mfc。(5)haxl-hfc及maxl-mfc稳定表达细胞株的制作为了制作haxl-hfc及maxl-mfc的稳定表达株,在宿主细胞中导入表达载体。使用cho-k1(理研)作为haxl-hfc及maxl-mfc表达用宿主细胞。对于细胞培养,常规传代中使用含有4mml-谷氨酰胺(invitrogen公司制)及50μg/ml庆大霉素(nacalaitesque公司制)的ex-cell325pf(nichireicorporation制)(基础培养基)。另外,在基因导入株的选拔时,使用含有3μg/ml放线菌酮预制备液(cycloheximidereadymadesolution)(sigma公司制)的基础培养基(chx培养基)。任一细胞均在37℃、5%co2条件下下振荡培养。基因向细胞中的导入通过与实施例2(5)同样的方法来进行。将含有(1)所得到的10μg的pktabex-haxl-hfc、20μg的转移酶表达载体(wo2010/143698)(以下记作tpex_pmug)的溶液添加到电穿孔用样品池(genepulsercuvette,bio-rad公司制)。将400μl用pbs制备的4×106个/ml的cho-k1细胞悬液加入到该样品池中。将样品池中的细胞悬液混和后,用genepulser(bio-rad)在脉冲电压300v、电容量500μf的条件下进行基因导入。基因导入后,将样品池中的细胞悬浮在20ml的基础培养基中,以200μl/孔播种在1块96孔板中。培养开始4天后将培养基交换成chx培养基,进行基因导入株的选拔。培养第29天,将培养基变成黄色的细胞株放大至24孔,使用进一步培养3天的上清,通过以下所述的方法测定haxl-hfc的表达量。为了测定haxl-hfc的表达量,进行以下的elisa。在96孔板(nunc公司制)中,以50μl/孔分注作为一抗的用pbs稀释750倍的goatanti-humanigg(h&l)(americanqualex公司制),在4℃下静置一晚而固定。将板用含有0.05~0.1%的吐温-20的pbs(以下记作吐温20-pbs)(wako公司制)洗涤5次,以100μl/孔将含有1%bsa的pbs(以下记作1%bsa-pbs)(nacalaitesque公司制)分注到elisa板中,在室温下静置2小时,从而进行封闭。将板用吐温20-pbs(wako公司制)洗涤5次,以50μl/孔分注作为受试体的用1%bsa-pbs稀释的培养上清,静置1小时。作为标准品,使用公知的人igg1抗体。用吐温20-pbs(wako公司制)洗涤5次后,以50μl/孔分注作为二抗的用1%bsa-pbs稀释2000倍的goatanti-humanigg(h&l)-hrp(americanqualex公司制),静置1小时。在用吐温20-pbs洗涤后,以50μl/孔分注abts(2,2-azino-bis(3-ethylbenzothiazoline)-6-sulfonicacid,2,2-连氮-双(3-乙基苯并噻唑啉-6-磺酸)(thermofisher公司制)进行显色。以50μl/孔分注5%sds溶液从而终止显色。用酶标仪测定样品波长415nm、参比波长490nm下的吸光度(415nm-490nm)。将haxl-hfc表达量高的细胞株从6孔板依次放大到125ml三角烧瓶中,再次测定haxl-hfc表达量。选择结果中表达量最高的细胞株作为haxl-hfc稳定表达细胞株。同样地,使用(4)所得到的pktabex-maxl-mfc得到maxl-mfc稳定表达细胞株。maxl-mfc的表达量与上述同样地通过elisa法进行测定。使用用pbs稀释100倍的polyclonalrabbitanti-mouseimmunoglobulins(dako公司制)作为一抗,使用用1%bsa-pbs稀释400倍的polyclonalrabbitanti-mouseimmunoglobulinshrp(dako公司制)作为二抗。作为标准品,使用公知的小鼠igg1抗体。(6)caxl-hfc及raxl-hfc瞬时表达细胞株的制作为了制作caxl-hfc及raxl-hfc的瞬时表达株,将表达载体导入宿主细胞中。作为caxl-hfc及raxl-hfc表达用宿主细胞,使用cho-s(lifetecnologies公司制)。细胞的传代中,使用含有4mml-谷氨酰胺(invitrogen公司制)的freestylecho(invitrogen公司制),在37℃、5%co2条件下振荡培养。将(2)所制作的1.25mg的pktabex-caxl-hfc溶解于20ml的opti-prosfm(invitrogen公司制)中,另外,将1.25ml的freestylemaxreagent(invitrogen公司制)溶解于20ml的optiprosfm中,在室温下放置5分钟。将上述两液体混合,在室温下放置15分钟。将该混合溶液滴加到cho-s培养液中,得到caxl-hfc瞬时表达细胞株。同样地,使用(3)所制作的pktabex-raxl-hfc得到raxl-hfc瞬时表达细胞株。(7)各种axl-hfc及maxl-mfc的纯化将(5)所得到的haxl-hfc及maxl-mfc稳定表达细胞株悬浮于公知的蛋白质表达用培养基,在三角烧瓶中培养7天后,回收培养上清。对回收的培养上清进行离心分离,将得到的培养上清用0.22μm过滤器进行过滤,从而制备含有haxl-hfc的培养上清。通过同样的方法制备含有maxl-mfc的培养上清。另外,将(6)所得到的caxl-hfc及raxl-hfc瞬时表达细胞株悬浮于添加有4mm的l-谷氨酰胺(invitrogen公司制)的freestylecho(invitrogen公司制)中,用三角烧瓶培养5天后,回收培养上清。对回收的培养上清进行离心分离,将得到的培养上清用0.22μm过滤器进行过滤,从而制备含有caxl-hfc的培养上清。通过同样的方法制备含有raxl-hfc的培养上清。由制备的培养上清中,通过常规方法纯化各种axl-fc。树脂使用hitrapmabselectsure(gehealthcare公司制)。得到的纯化蛋白质溶液在用0.22μm过滤器进行过滤灭菌后,用于试验。用实施例2(9)所述的方法计算各蛋白质的吸光系数。haxl-hfc、maxl-mfc、caxl-hfc及raxl-hfc的吸光系数分别为1.38、1.54、1.42及1.8。[实施例4]现有的抗人gas6单克隆抗体的制作(1)cnto抗体表达载体的制作以us7547767专利说明书所述的抗gas6单克隆抗体wg1的vh及vl的碱基序列(us7547767专利说明书的seqidno:25、27)为基础,通过以下所述方法制作该抗体(以下记作cnto抗体)的表达载体。通过idt公司进行cnto抗体的vh及vl的碱基序列的全合成,并且整合于适当的质粒中。将cnto抗体的vh及vl的碱基序列分别示于序列号61、63。另外,将cnto抗体的vh及vl的氨基酸序列分别示于序列号62、64。us7547767中,序列号25所示的wg1的vh的碱基序列的第305位记载为n,因此根据kabat的人抗体序列信息(sequencesofproteinsofimmunologicalinterest,usdepthealthandhumanservices(1991))以及与第305位的碱基形成密码子的第304、306位的碱基序列,使用胸苷作为305位的碱基。在表达用载体pkantex93(wo97/10354)的适当位置通过公知的方法插入cnto抗体的基因序列,构建作为cnto抗体表达载体的pkantex-cnto。(2)cnto抗体稳定表达细胞株的制作通过与实施例2(5)同样的方法在dhfr缺失的cho细胞中导入(1)所制备的pkantex-cnto,制作cnto抗体稳定表达细胞株。(3)cnto抗体的纯化将(2)所得到的cnto抗体稳定表达细胞株悬浮于500nmmtx培养基,然后在细胞用烧瓶中培养3天。然后,将培养基交换成ex-cell302(含有6mml-谷氨酰胺、100nm3,3′,5-三碘代-l-甲状腺原氨酸钠盐、50μg/ml庆大霉素)并培养5天后,回收培养上清。对回收的培养上清进行离心分离,将上清用0.22μm过滤器进行过滤,从而制备含有cnto抗体的培养上清。从制备的培养上清中,通过常规方法纯化cnto抗体。树脂使用mabselectsure(gehealthcare公司制)。得到的cnto抗体在用0.22μm过滤器进行过滤灭菌后,用于试验。cnto抗体的吸光系数为1.43。[实施例5]抗人gas6单克隆抗体的制作(1)对动物的免疫和抗体产生细胞的制备将实施例2(9)所制作的hgas6-f或rgas6-f对实施例1所得到的ko小鼠进行免疫。小鼠的免疫中,使用氢氧化铝(antibodiesalaboratorymanual,coldspringharborlaboratory,p99,1988)及百日咳疫苗(nacalaitesque公司制)作为佐剂。具体而言,相对于1只,使用80μl的氢氧化铝及5μl的百日咳疫苗制备与hgas6-f或rgas6-f的悬浮液。按照每1只给药30μg的hgas6-f或rgas6-f的量,将该悬浮液给药到ko小鼠的腹腔内。仅首次免疫中使用佐剂,包括末次强化在内,共进行4次免疫。分成仅使用hgas6-f进行免疫的群、交替给药rgas6-f和hgas6-f的群,每组分别对4只进行免疫。末次免疫的4天后摘出脾脏。将摘出的脾脏在mem培养基(invitrogen公司制)中弄碎后,通过离心分离(1200rpm、5分钟)回收脾细胞组分。得到的脾细胞组分含有红细胞,因此添加redbloodcelllysingbuffer(sigma公司制)在37℃下进行反应,从而除去红细胞。得到的脾细胞用mem培养基洗涤2次后,供于细胞融合。(2)小鼠骨髓瘤细胞的制备将8-氮杂鸟嘌呤抗性小鼠骨髓瘤细胞株p3x63ag8u.1(由p3-u1:atcc购入)用含有10%fcs(moregatebiotech公司制)的rpmi1640(wako公司制)培养,作为细胞融合的亲株。(3)杂交瘤的制作将(1)所得到的小鼠脾细胞与(2)所得到的骨髓瘤细胞按照8:1的比例混合并离心分离(1200rpm、5分钟)。对于得到的沉淀组分(细胞群),一边平稳地晃动一边缓慢地加入0.5ml的聚乙二醇-1000(罗氏诊断公司制)。然后,在37℃的水浴中,每隔1分钟在该细胞液中加入1ml的mem,添加5次,最后加入45ml的mem。然后进行离心分离(900rpm、5分钟)。将得到的沉淀组分(细胞群)悬浮于hat培养基(10%胎牛血清的rpmi1640培养基中添加有hatmediasupplement(invitrogen公司制)的培养基),制备成脾细胞数1.5×107个/平板。将该细胞悬液在96孔板中各播种200μl,在37℃、5%co2的条件下培养。在孔内的细胞达到适于筛选的细胞数的前一天,用hat培养基进行培养基交换。(4)杂交瘤的筛选对于(3)所制作的杂交瘤,通过以下所述的竞争elisa法来选择产生抑制人及大鼠的gas6与axl的结合的抗体的杂交瘤。首先,在96孔的elisa用板(nunc公司制)中以50μl/孔分注2μg/ml的haxl-hfc溶液(将实施例3(7)所得到的haxl-hfc溶液用pbs(nacalaitesque公司制)稀释后的溶液),在4℃下静置一晚进行吸附。将该板用吐温20-pbs洗涤5次后,以300μl/孔加入1%bsa-pbs(nacalaitesque公司制),在室温下静置1小时进行封闭,用吐温20-pbs(wako公司制)洗涤5次。然后,在该板中以50μl/孔分注通过以下方法制备的反应液,在室温下静置1小时后,用吐温20-pbs洗涤5次。反应液如下制备:将100ng/ml的hgas6-f溶液(将实施例2(9)所得到的hgas6-f溶液用1%bsa-pbs稀释后的溶液)与杂交瘤的培养上清或杂交瘤用培养基(阴性对照)等量混合,在4℃下静置30分钟。然后,以50μl/孔分注作为检测用抗体的、用1%bsa-pbs稀释2000倍的monoclonalanti-flagm2-peroxidase(hrp)antibodyproducedinmouse(sigma-aldrich),在室温下静置1小时。将该板用吐温20-pbs洗涤5次,以50μl/孔添加tmb(sigma-aldrich公司制)并使其反应。在适当的时刻,以50μl/孔添加1n盐酸(wako公司制)而终止反应,对于各孔的溶液,用酶标仪测定样品波长450nm、参比波长570nm下的吸光度(450nm-570nm)。本测定体系中,当杂交瘤培养上清中含有抑制hgas6与haxl的结合的抗体时,添加了该培养上清的孔的吸光度与添加了阴性对照的孔的吸光度相比是下降的。因此,选择与添加了阴性对照的孔相比吸光度低的孔,选择与添加于该孔的培养上清对应的杂交瘤。通过同样的方法,来选择产生抑制rgas6与raxl的结合的抗体的杂交瘤。作为样品,使用实施例2(9)、实施例3(7)所得到的rgas6-f、raxl-hfc。(5)使用固定抗原的gas6结合活性测定elisa通过以下所述的抗原结合elisa法,来确认(4)所选择的杂交瘤的培养上清中的抗体与hgas6-f及rgas6-f结合、不与和hgas6同源性高的人蛋白s及flag标签(bap-f)结合。作为吸附于elisa用板的抗原,使用与实施例2(9)所纯化的hgas6-f、rgas6-f、hgas6同源性高的人蛋白s(来自人血清,enzymeresearchlaboratories公司制)或carboxy-terminalflag-bapfusionprotein(以下记作bap-f)(sigma-aldrich公司制)。首先,在96孔的elisa用板(nunc公司制)中,以50μl/孔分注2μg/ml的上述抗原溶液(将各抗原用pbs(nacalaitesque公司制)制备而成的溶液),在4℃下静置一晚进行吸附。用吐温20-pbs洗涤5次后,以300μl/孔加入1%bsa-pbs(nacalaitesque公司制),在室温下静置1小时仅封闭,用吐温20-pbs(wako公司制)洗涤5次。然后,以50μl/孔分注作为受试物质的杂交瘤的培养上清,在室温下静置1小时后,用吐温20-pbs洗涤5次。然后,以50μl/孔分注用1%bsa-pbs稀释为2000倍的polyclonalgoatanti-mouseimmunoglobulins/hrp(dako,p0447),在室温下静置1小时。将板用吐温20-pbs洗涤5次,以50μl/孔添加tmb(sigma-aldrich公司制)并使其反应,在适当的时候,以50μl/孔添加1n盐酸(wako公司制)来终止反应,用酶标仪(spectramax,moleculardevices公司制)测定样品波长450nm、参比波长570nm下的吸光度(450nm-570nm)。(6)杂交瘤的克隆化将(4)及(5)所选择的杂交瘤用克隆用培养基(添加有10%胎牛血清、1%htsupllement(invitrogen公司制)、0.2%硫酸庆大霉素溶液(nacalaitesque公司制)的s-clone克隆介质(eidiaco.,ltd制))进行有限稀释,播种在96孔板中进行克隆化。克隆化仅进行1次。通过以上操作,分离出两株与人及大鼠gas6结合、进而产生具有抑制人及大鼠gas6与axl的结合的活性的抗体的杂交瘤。(7)由杂交瘤得到抗体将(6)所分离出的杂交瘤按照细胞密度达到1×107个/100ml的量播种到悬浮烧瓶中。作为培养基,使用含有5%的fetalbovineserum-ultralowigg(invitrogen公司制)的hybridomasfm培养基(invitrogen公司制)。将细胞在37℃下静置培养7天后,回收含有细胞的培养基。对回收的培养基进行离心分离,将得到的培养上清用0.22μm过滤器进行过滤。由用过滤器过滤出的培养上清中,使用常规方法纯化来自杂交瘤的抗人gas6小鼠单克隆抗体km5320抗体(以下也记作km5320-mkg1抗体)、km5321抗体(以下也记作km5321-mkg1抗体)。树脂使用proteingsepharose4fastflow(gehealthcare公司制)。得到的抗体溶液在用0.22μm过滤器进行灭菌后,用于实验。通过实施例2(9)所述的方法计算吸光系数,km5320-mkg1抗体、km5321-mkg1抗体的吸光系数分别为1.54和1.45。[实施例6]使用悬浮抗原的gas6结合活性评价使用以下所述的竞争elisa法,来确认获得抗体对更接近天然状态的各种gas6的抗体结合性。首先,在96孔的elisa用板(nunc公司制)中,以50μl/孔分注2μg/ml的抗flag抗体[将monoclonalanti-flagm2antibodyproducedinmouse(sigma-aldrich公司制)用pbs(nacalaitesque公司制)稀释后的抗体],在4℃下静置一晚进行吸附。除去固定化液后,以300μl/孔加入1%bsa-pbs(nacalaitesque公司制),在室温下静置1小时进行封闭,用吐温20-pbs(wako公司制)洗涤5次。然后,以50μl/孔分注1μg/ml的各种gas6-f或bap-f溶液[将实施例2(9)所得到的各种gas6-f溶液或bap-f(sigma-aldrich公司制)用1%bsa-pbs稀释后的溶液],在室温下静置1小时后,用吐温20-pbs洗涤5次。然后,将km5320-mkg、km5321-mkg及cnto抗体用biotinlabelingkit-nh2(dojindo公司制)生物素化,用1%bsa-pbs制备成适当的浓度,将其以50μl/孔分注,在室温下静置1小时。将该板用吐温20-pbs洗涤5次后,以50μl/孔分注用1%bsa-pbs稀释200倍的streptavidinhrpconjugate(r&d公司制),在室温下静置1小时。将该板用吐温20-pbs洗涤5次,以50μl/孔添加tmb(sigma-aldrich公司制)而进行反应,在适当时候,以50μl/孔添加1n盐酸(wako公司制)而终止反应,用酶标仪测定样品波长450nm、参比波长570nm下的吸光度(450nm-570nm)。另外,通过与实施例5(5)同样的方法测定获得抗体与人蛋白s的结合活性。就样品而言,将作为受试物质的上述制备的生物素化km5320-mkg、km5321-mkg及cnto抗体用1%bsa-pbs稀释成适当的浓度后使用。作为二次检测试剂,使用用1%bsa-pbs稀释200倍的streptavidinhrpconjugate(r&d公司制)。将结果示于图1。km5320-mkg1抗体及km5321-mkg1抗体与人gas6-f、猴gas6-f、大鼠gas6-f及小鼠gas6-f结合,不与人proteins及bap-f结合。该结果表明:km5320-mkg1抗体及km5321-mkg1抗体为与人gas6、猴gas6、大鼠gas6及小鼠gas6特异性结合的抗体。另外,就km5320-mkg1抗体及km5321-mkg1抗体与人gas6-f的结合活性而言,在抗体浓度0.0003μg/ml下可检测,在0.04μg/ml下达到最大活性。另一方面,就cnto抗体与人gas6-f的结合活性而言,在抗体浓度0.04μg/ml下可检测,在5μg/ml下也仅显示出km5320-mkg1抗体及km5321-mkg1的最大活性的一半以下的活性(图1-a)。该结果表明:与cnto抗体相比,km5320-mkg1抗体及km5321-mkg1抗体与各种gas6的结合更强。[实施例7]抗gas6单克隆抗体的抑制gas6与axl结合的活性的评价对于km5320-mkg1抗体、km5321-mkg1抗体及cnto抗体,通过与实施例5(4)同样的方法测定抑制各种gas6与axl结合的活性。作为反应液,使用将100ng/ml的各种gas6-f溶液(将实施例2(9)所得到的各种gas6-f溶液用1%bsa-pbs稀释后的溶液)和最终浓度的2倍浓度的各抗体溶液(将实施例4(3)及实施例5(7)所得到的抗体溶液用1%bsa-pbs稀释后的溶液)等量混合的溶液。将结果示于图2。km5320-mkg1抗体、km5321-mkg1抗体抑制人、猴、大鼠及小鼠gas6与来自各个物种的axl的结合。km5320-mkg1抗体的情况下,在对50ng/ml的人gas6添加约200ng/ml的抗体时,几乎完全抑制了人gas6与人axl的结合。由于人gas6的分子量为约70kda、抗体的分子量为约140~150kda,因此该结果表明:抗体与gas6为2:1的摩尔浓度比时,km5320-mkg1抗体完全抑制人gas6与人axl的结合。同样地,km5321-mkg1抗体的情况下,对于50ng/ml的人gas6添加约60ng/ml的抗体时,几乎完全抑制了人gas6与人axl的结合。该结果表明:抗体与gas6为1∶2的摩尔浓度比时,km5321-mkg1抗体完全抑制人gas6与人axl的结合。由于1个抗体分子最多结合2个抗原分子,因此表明km5321-mkg1抗体具有非常强的结合活性。另一方面,cnto抗体在所研究的抗体浓度下未完全抑制各种gas6与axl的结合。以上表明:km5320-mkg1抗体、km5321-mkg1抗体与cnto抗体相比,具有更强的中和活性。[实施例8]获得抗体对gas6依赖性的细胞内信号的作用作为gas6的受体,除了axl以外,还已知sky及mer。当gas6与细胞上表达的该受体结合时,在细胞内,该受体介导的信号转导通路被活化。为了确认获得抗体对该细胞内信号转导的作用,使用gas6受体的强制表达细胞株实施了报告基因测定。在该gas6受体的强制表达细胞株中,导入有egr1(earlygrowthresponese1)的识别序列的载体,该egr1为gas6受体的与下游信号转导相关的转录因子之一。另外,载体中的egr1识别序列的下游整合有荧光素酶基因。本测定体系中,当gas6与上述细胞株上的gas6受体结合时,在细胞内,信号转导通路被活化,egr-1的表达上调。egr1结合于egr1识别序列则使荧光素酶基因的表达上调。因此,通过检测生物合成出的荧光素酶的发光强度,可以确认由gas6添加导致的细胞内信号转导通路的活化状态。(1)gas6受体表达载体的构建首先,构建人axl的表达载体。该载体是以整合有序列号26所示的人axl的基因序列(genecopoeia公司制)(genbank登录号nm_021913)的质粒为模板、通过以下所述的方法来制作的。制备含有模板质粒、各10pmol的引物22、23(序列号50、51)及primestarhsdnapolymerase(宝生物公司制)的20μl的反应液,在94℃下保温5分钟后,将在98℃下10秒、55℃下5秒、72℃下3分30秒作为1个循环,进行30个循环的pcr。将得到的pcr产物供于琼脂糖凝胶电泳,使用qiaquickgelextractionkit(qiagen公司制)回收约2.7kbp的扩增dna片段(含有haxl的碱基序列的dna片段)。另外,将公知的适当的动物细胞表达载体用ecori及bamhi进行酶消化,供于琼脂糖凝胶电泳后,用qiaquickgelextractionkit回收目标dna片段。用in-fusionhd克隆试剂盒(clontechlaboratories,inc.制)将得到的2种dna片段结合,得到haxl重组体表达载体。对于得到的表达载体中所含的人axl基因序列进行分析,结果序列号26所示的人axl基因的第1546位的胞嘧啶被置换成了胸苷。但是,对于任一碱基序列,推测出的氨基酸序列是相同(genbank登录号np_068713)的,存在同一碱基序列的基因多态性,因此将本序列用于此后的实验。通过同样的方法将人sky基因重组到适当的公知的动物细胞表达载体中,制备人sky重组体表达载体。作为pcr的模板,使用整合有序列号52所示的人sky基因序列(genbank登录号nm_006293))的质粒(invitrogen公司制)。pcr中,制备含有上述模板质粒、各10pmol的引物24、25(序列号54、55)及primestarmaxdnapolymerase(宝生物公司制)的20μl的反应液,在98℃下保温10分钟后,将98℃下10秒、55℃下5秒、72℃下1分30秒作为1个循环,进行30个循环的pcr。对于得到的表达载体所含的人sky的碱基序列进行分析,结果序列号52所示的人sky的碱基序列的2168位的胸苷被置换成了胞嘧啶。但是,对于任一碱基序列,推测出的氨基酸序列是相同(genbank登录号np_06284)的,存在同一碱基序列的基因多态性,因此决定使用本序列。通过同样的方法将人mer基因整合到适当的公知的动物细胞表达载体中,制备人mer重组体表达载体。作为pcr的模板质粒,使用整合有序列号56所示的人mer基因序列、(genbank登录号nm_006343))的质粒(genecopoeia公司制)。pcr中,制备含有上述模板质粒、各10pmol的引物26、27(序列号58、59)及primestarmaxdnapolymerase(宝生物公司制)的20μl的反应液,在98℃下保温10分钟后,将98℃下10秒、55℃下5秒、72℃下5秒作为1个循环,进行30个循环的pcr。(2)向宿主细胞中导入gas6受体表达载体及荧光素酶报告基因载体通过以下所述的方法将上述(1)所制作的3种gas6受体表达载体及报告基因载体导入宿主细胞hek293f细胞(invitrogen公司)中。细胞培养的培养基使用了freestyle293expressionmedium(invitrogen公司制),在37℃、5%co2条件下振荡培养。报告基因载体使用了在作为荧光素酶报告基因载体的pgl3载体(promega公司制)中插入有小鼠的egr1识别序列(序列号60)的pgl3-megr1(nature444,770-774,2006、pnas85(21),7857-61,1988)。将各gas6受体表达载体及pgl3-megr1溶解于opti-prosfm(invitrogen公司制),另外,将293fectintransfectionreagent(invitrogen公司制)溶解于optiprosfm,在室温下放置5分钟。将上述两液体混合,在室温下放置20分钟后,滴加于hek293f细胞培养液中,振荡培养5小时。将这样得到的细胞株分别记作人axl-megr1转录因子启动子的报告基因细胞株、人sky-megr1转录因子启动子的报告基因细胞株、人mer-megr1转录因子启动子的报告基因细胞株。(3)报告基因测定将(2)所制作的3种gas6受体(axl、sky或mer)-megr1转录因子启动子的报告基因细胞株按照达到1.6×104个/孔的量在96孔黑色板中各播种80μl。然后,以10μl/孔添加含有80μg/ml(最终浓度的10倍浓度)的km5320-mkg1或km5321-mkg1的培养基[将实施例5(7)所得到的抗体溶液用freestyle293expressionmedium(invitrogen公司制)稀释后的溶液],静置培养1小时。作为同种型对照,以10μl/孔添加通过与上述同样的方法制备的含有80μg/ml的igg1同种型对照(purifiedmousemonoclonaligg1、r&d公司制)的培养基。然后,以10μl/孔添加含有20μg/ml(最终浓度的10倍浓度)的hgas6-f的培养基[将实施例2(9)所制备的hgas6-f溶液用freestyle293expressionmedium稀释后的溶液],静置培养。另外,还准备了仅添加上述细胞株和培养基的孔作为阴性对照。在12~14小时后,以100μl/孔添加化学发光试剂(steadygloluciferaseassaysystem、promega公司制),用照度计(veritas公司制)测定各孔的发光强度。将结果示于图3。添加hgas6-f和同种型对照时,任一的gas6受体强制表达细胞中,与仅添加培养基时相比,发光强度均增加了3倍左右。另一方面,添加hgas6-f和km5320-mkg1或km5321-mkg1时,显示出与仅添加培养基时同等程度的发光强度。如上所述,本测定体系中,随着细胞内信号转导通路的活化,检测出的荧光素酶的发光强度也增加。因此表明:将人gas6添加于各种gas6受体强制表达细胞株时,细部内信号转导通路被活化,并且km5320-mkg1、km5321-mkg1均抑制该活化。另外,通过与实施例7同样的方法计算时,km5320-mkg1抗体、km5321-mkg1抗体均在抗体与gas6为2∶1的摩尔浓度下完全抑制由gas6导致的细胞内信号转导通路的活化。该结果表明:km5320-mkg1抗体、km5321-mkg1抗体具有非常强的中和活性。[实施例9]本发明的抗人gas6单克隆抗体对人肾肾小球系膜细胞中的磷酸化信号的作用在与实施例8相比更接近生物体的条件下,确认获得抗体对由于gas6和gas6受体的结合而产生的细胞内信号的作用。已知gas6受体由于gas6的结合而活化时,在该受体表达细胞内,akt被磷酸化。因此,使用天然表达各gas6受体的人肾肾小球系膜细胞(sciencell公司制,以下简记为人肾小球系膜细胞),通过以下所述的方法来检测由gas6添加所引起的akt的磷酸化水平。人肾小球系膜细胞中表达axl、sky、mer的各gas6受体这一点通过用公知的方法进行facs分析来确认。关于facs分析,为了检测各gas6受体,分别使用anti-axlantibody(abcam公司制、mm0098-2n33)、humandtkmab(r&d公司制、clone96201)、humanmermab(r&d公司制、clone125518)。作为二抗,使用alexafluor488goatanti-mouseigg(h+l)antibody(invitrogen公司制)。将人肾小球系膜细胞悬浮于在肾小球系膜细胞培养基(sciencell公司制,以下记作mcm)中添加有2%fbs及1%mesangialcellgrowthsupplement(mcm所附带的)的培养基中,按照达到0.5×104个/孔的量播种在12孔板中,在37℃、5%co2条件下静置培养。2天后,将培养基交换成不含添加物的mcm,静置培养。1天后,将孔用mcm洗涤一次,重新添加400μl/孔的mcm。然后,以50μl/孔添加用mcm稀释成最终浓度的10倍浓度的实施例4(3)所制备的抗体样品、haxl-hfc及同种型对照抗体,静置培养1小时。作为同种型对照抗体(阴性对照),使用igg1同种型对照(purifiedmousemonoclonaligg1、r&d公司制)。然后,以50μl/孔添加用mcm稀释成最终浓度(0.1μg/ml)的10倍浓度的hgas6-f或仅mcm,静置培养10分钟。在冰上除去培养基,用含有proteaseinhibitorcocktail(シゲマアルドリッチ公司制)的pbs洗涤后,以120μl/孔添加用含有proteaseinhibitorcocktail和2-巯基乙醇(nacalaitesque公司制)的pbs稀释5倍的lanemarkernon-reducingsamplebuffer(thermofisher公司制)。回收各孔中的样品,在95℃下加热10分钟。将加热后的样品供于e·pagel(5~20%、atto公司制)后,通过sds丙烯酰胺凝胶电泳法分离蛋白质。分离后,将蛋白质通过半干印迹法转录到pvdf膜上,进行蛋白质印迹。作为一抗,使用将抗akt抗体(cellsignaling公司制、#4691)稀释1000倍、或将抗phospho-akt(ser273)抗体(cellsignaling公司制、#4060)稀释2000倍的抗体。作为二抗,使用抗兔igg抗体-hrp(dako公司制、p0448)。上述抗体的稀释中,使用含有5%的eclblockingagent(gehealthcare公司制)的1×tbst(santacruz公司制)。作为显色底物,使用eclselectwesternblottingdetectionreagent(gehealthcare公司制),使用imagequantlas500(gehealthcare公司制)来检测化学发光。将结果示于图4。在肾小球系膜细胞中添加hgas6时,akt的磷酸化水平亢进。另一方面,haxl-hfc、km5320-mkg1抗体及km5321-mkg1抗体均浓度依赖性地抑制由hgas6引起的akt磷酸化水平亢进。另外,同种型对照未抑制由hgas6引起的akt磷酸化水平亢进。该结果表明:km5320-mkg1抗体、km5321-mkg1抗体在原本就表达gas6受体的人肾小球系膜细胞中也抑制由于添加hgas6而发生的细胞内信号转导通路的活化。[实施例10]本发明的抗人gas6单克隆抗体和抗人gas6单克隆抗体cnto抗体的竞争抑制实验使用竞争elisa来确认获得抗体与cnto抗体是否竞争性地与人gas6结合。通过以下所述的方法进行竞争elisa。在96孔的elisa用板(nunc公司制)中,以50μl/孔分注将monoclonalanti-flagm2antibodyproducedinmouse(sigma-aldrich公司制)用pbs(nacalaitesque公司制)制备成2μg/ml的液体,在4℃下静置一晚进行吸附。除去固定化液后,以300μl/孔加入1%bsa-pbs(nacalaitesque公司制),在室温下静置1小时进行封闭,用吐温20-pbs(wako公司制)洗涤5次。然后,以50μl/孔分注将hgas6-f用1%bsa-pbs制备成1μg/ml的浓度的液体,在室温下静置1小时。然后,将实施例4(3)所制备的cnto抗体用biotinlabelingkit-nh2(dojindo公司制)生物素化,按照达到最终浓度(2μg/ml)的二倍浓度的方式用1%bsa-pbs稀释,制备生物素化cnto抗体溶液。另外,作为受试物质,将实施例5(7)所制备的未标记的km5320-mkg1抗体、km5321-mkg1抗体及实施例4(3)所制备的未标记的cnto300按照达到最终浓度的二倍浓度的方式用1%bsa-pbs稀释并分别与生物素化cnto抗体溶液等量混合,在室温下静置1小时。将板用吐温20-pbs洗涤5次后,以50μl/孔分注上述混合样品,在室温下静置1小时。将上述板用吐温20-pbs洗涤5次后,以50μl/孔分注用1%bsa-pbs稀释200倍的streptavidinhrpconjugate(r&d公司制),在室温下静置1小时。将板用吐温20-pbs洗涤5次,以50μl/孔添加tmb(sigma-aldrich公司制)进行显色,在得到适当的显色的时候,以50μl/孔添加1n盐酸(wako公司制),用酶标仪测定样品波长450nm、参比波长570nm下的吸光度(450nm-570nm)。将结果示于图5。以cnto抗体为受试物质时,与阴性对照相比吸光度下降。另一方面,以km5320-mkg1抗体、km5321-mkg1抗体为受试物质时,为与阴性对照同等程度的吸光度。作为受试物质的上述未标记的gas6单克隆抗体与生物素化cnto抗体竞争性地与人gas6结合时,检测的吸光度与阴性对照相比也下降。因此表明:km5320-mkg1抗体及km5321-mkg1抗体与cnto抗体并非竞争性地与hgas6结合。[实施例11]编码抗人gas6单克隆抗体的vh及vl的基因序列的分离(1)由产生抗人gas6单克隆抗体的杂交瘤细胞制备总rna由5×106个分别产生km5320-mkg1抗体、km5321-mkg1抗体的杂交瘤中,使用rnaeasyminikit(qiagen公司制)及qiashredder(qiagen公司制)制备总rna。(2)抗人gas6单克隆抗体的vh及vl的基因克隆由(1)所得到的1μg的总rna,使用smarterracecdnaamplificationkit(clontech公司制)制作cdna。制备含有作为模板的所得到的cdna、试剂盒所附的通用引物amix(含有正向引物)和编码小鼠igg1重链恒定区的反向引物(引物28(序列号65))、以及primestarmaxdnapolymerase(宝生物公司制)的25μl的反应液,进行pcr。pcr中,在98℃下保温10秒后,以98℃下10秒、55℃下5秒及72℃下5秒作为1个循环,进行30个循环,来扩增含有各抗体的vh的基因的dna片段。另外,使用通用引物a和小鼠ig(κ)特异性的引物(引物29(序列号66))同样地进行pcr,来扩增含有各抗体的vl的基因的dna片段。将pcr产物供于琼脂糖凝胶电泳,使用qiaquickgelextractionkit(qiagen公司制)回收扩增dna片段。将得到的扩增dna片段用zeroblunttopopcrcloningkit(invitrogen公司制)插入pcr4-blunt-topo载体中,通过与实施例2(1)同样的方法得到质粒。对得到的质粒的碱基序列进行分析,确认得到了在cdna的5’末端存在推测为起始密码子的atg序列的全长的vhcdna及vlcdna。(3)抗人gas6单克隆抗体v区的基因序列的分析将(2)所得到的km5320-mkg1抗体的vh的全部碱基序列示于序列号67,将由该序列推测出的含有信号序列的vh的全部氨基酸序列示于序列号68,将序列号68所示的氨基酸序列中除去信号序列后的氨基酸序列示于序列号69。另外,将km5320-mkg1抗体的vl的全部碱基序列示于序列号70,将由该序列推测出的含有信号序列的vl的全部氨基酸序列示于序列号71,将序列号71所示的氨基酸序列中除去信号序列后的氨基酸序列示于序列号72。将km5321-mkg1抗体的vh的全部碱基序列示于序列号73,将由该序列推测出的含有信号序列的vh的全部氨基酸序列示于序列号74,将序列号74所示的氨基酸序列中除去信号序列后的氨基酸序列示于序列号75。另外,将km5321-mkg1抗体的vl的全部碱基序列示于序列号76,将由该序列推测出的含有信号序列的vl的全部氨基酸序列示于序列号77,将序列号77所示的氨基酸序列中除去信号序列后的序列示于序列号78。通过与已知的小鼠抗体的序列数据[sequencesofproteinsofimmunologicalinterest、usdept.healthandhumanservices(1991)]比较,确认了分离出的各cdna为编码包含分泌信号序列的km5320-mkg1抗体、km5321-mkg1抗体的全长cdna。通过与已知抗体的氨基酸序列进行比较,来鉴定各单克隆抗体的vh及vl的cdr。将km5320-mkg1抗体的vh的cdr1、cdr2及cdr3的氨基酸序列示于序列号79、80及81,将vl的cdr1、cdr2及cdr3的氨基酸序列示于序列号82、83及84。将km5321-mkg1抗体的vh的cdr1、cdr2及cdr3的氨基酸序列示于序列号85、86及87,将vl的cdr1、cdr2及cdr3的氨基酸序列示于序列号88、89及90。[实施例12]抗人gas6小鼠-大鼠嵌合抗体的制备通过以下所述的方法,由抗gas6小鼠单克隆抗体km5320-mkg1抗体及km5321-mkg1抗体来制作小鼠-大鼠igg1嵌合抗体(以下简记为大鼠嵌合抗体)。以下将各大鼠嵌合抗体记作km5320-rkg1抗体及km5321-rkg1抗体。(1)大鼠嵌合抗体表达载体的构建将km5320-rkg1抗体的重链全长的碱基序列(序列号91)及轻链全长的碱基序列(序列号93)用常规方法串联整合到公知的适当的动物细胞表达载体的适当的位置。序列号91所示的km5320-rkg1抗体的重链全长的碱基序列由km5320抗体的vh的全部碱基序列(序列号67)及含有大鼠igg1重链恒定区的基因序列构成。另外,序列号93所示的km5320-rkg1抗体的轻链全长的碱基序列由km5320抗体的vl的全部碱基序列(序列号70)和含有大鼠ig(κ)恒定区的基因序列构成。同样地,将km5321-rkg1抗体的重链全长的碱基序列(序列号95)及轻链全长的碱基序列(序列号97)用常规方法串联整合到公知的适当的动物细胞表达载体的适当的位置。序列号95所示的km5321-rkg1抗体的重链全长的碱基序列由km5321抗体的vh的全部碱基序列(序列号73)及含有大鼠igg1重链恒定区的基因序列构成。另外,序列号97所示的km5321-rkg1抗体的轻链全长的碱基序列由km5321抗体的vl的全部碱基序列(序列号76)及含有大鼠ig(κ)恒定区的基因序列构成。(2)大鼠嵌合抗体稳定表达细胞株的制作基于实施例2(5)及常规方法将(1)所制作的表达载体导入cho-k1细胞(europeancollectionofcellcultures;ecacc)中,制作大鼠嵌合抗体稳定表达细胞株。(3)大鼠嵌合抗体的纯化将(2)所制作的大鼠嵌合抗体稳定表达细胞用公知的蛋白质表达用的培养基培养数天,回收培养上清。基于实施例3(7)及公知的方法从所回收的培养上清中纯化km5320-rkg1及km5321-rkg1。基于实施例2(9)所述的方法测定抗体的吸光度,km5320-rkg1抗体的吸光系数为1.54、km5321-rkg1抗体的吸光系数为1.45。对于得到的大鼠嵌合抗体,通过与实施例6及实施例7同样的方法确认了与hgas6的结合活性及抑制hgas6与haxl-fc结合的活性与各自的亲抗体同等。[实施例13]抗人gas6单克隆抗体的表位分析为了对获得抗体的表位进行分析,制备人gas6结构域缺失体及将该蛋白质的氨基酸的一部分置换为丙氨酸的突变蛋白质,来确认获得抗体的结合活性的变化。(1)人gas6突变体(结构域缺失体)表达载体的制备为了得到从hgas6中除去gla结构域、且c末端附加有flag标签及his标签的gas6突变体(以下记作hgas6-fh),通过以下所述的方法制作该蛋白质的表达载体。通过genscrip公司进行序列号99所示的碱基序列(hgas6-delta)的全合成,并整合于适当的质粒中。序列号99所示的碱基序列从5’末端起由ecori识别序列、hgas6-fh的碱基序列及bamhi识别序列构成。hgas6-fh的碱基序列为在序列号3所示的hgas6的碱基序列中除去第91位~第273位的碱基序列、且3’末端结合有编码公知的flag标签及his标签的碱基序列的碱基序列。将得到的质粒及动物细胞表达用载体inpep4(biogen-idec公司制)用ecori及bamhi进行酶处理,通过与实施例2(1)同样的方法得到作为hgas6-fh表达载体的inpep-hgas6-fh。(2)人gas6突变体(丙氨酸置换体)表达载体的制备制作将(1)所述的hgas6-fh的氨基酸序列中的、与序列号4所示的人gas6的全长氨基酸序列的第314位的亮氨酸、315位的谷氨酰胺、316位的脯氨酸对应的氨基酸全部置换成丙氨酸的突变体(以下记作hgas6-fh-l314a、q315a,p316a或简单记作丙氨酸置换体)的表达载体。通过以下所述的方法在(1)所制作的inpep-hgas6-fh中进行位点特异性突变导入,从而制作表达上述丙氨酸突变体的载体。制备含有作为模板的inpep-hgas6-fh、各10pmol的引物30、31(序列号101、102)及primestarmaxdnapolymerase(宝生物公司制)的25μl的反应液,进行pcr。pcr中,在98℃下保温10秒后,将在98℃下10秒、55℃下5秒及72℃下5秒作为1个循环,进行30个循环,来扩增含有各突变体的碱基序列的dna片段。在pcr产物中添加dpni,在37℃下进行1小时的限制酶处理,从而进行未插入突变的模板载体的消化。将dpni消化后的pcr产物导入大肠杆菌dh5α中,由所得到的转化体得到具有插入了期望突变的基因的质粒。(3)人gas6突变体瞬时表达细胞株的制备在人gas6突变体(hgas6-f及丙氨酸置换体)的表达细胞株的制备中,使用expi293细胞(invitrogen公司制)。作为细胞培养的培养基,使用expi293tmexpressionmedium(invitrogen公司制),在37℃、5%co2条件下振荡培养。在expi293细胞中导入(1)、(2)所制作的各种人gas6突变体表达载体,得到各种人gas6突变体的瞬时表达细胞株。在细胞中导入表达载体时,按照所附说明书来使用expifectamine293transfectionkit(invitrogen公司制)。(4)由含有各种人gas6突变体的培养上清纯化人gas6突变体将(3)所得到的各种人gas6突变体瞬时表达细胞株按照expifectamine293transfectionkit(invitrogen公司制)的所附说明书培养4天,回收培养基。对回收的培养基进行离心分离,将得到的培养上清用0.22μm过滤器进行过滤,从而制备含有各种人gas6突变体的培养上清。通过与实施例2(9)同样的方法,用anti-flagm2affinitygel(sigma-aldrich公司制)来纯化培养上清。作为洗脱缓冲液,使用3m氯化镁(nacalaitesque公司制),得到的人gas6突变体溶液的缓冲液在置换成pbs(nacalaitesque公司制)并用0.22μm过滤器过滤灭菌后,用于试验。(5)获得抗体与各种人gas6突变体的结合活性的评价为了确定获得抗体所结合的gas6的表位,基于实施例6所述的方法来评价与(4)所纯化出的各种人gas6突变体的结合活性。实验以n=2来进行,将得到的吸光度的平均值为0.1以下时判定为未结合,将为2以上时判定为结合。作为抗原样品,使用实施例2(9)所制备的hgas6-f及(3)所制备的各种人gas6突变体。作为抗体样品,将实施例12(3)所制备的km5320-rkg1及km5321-rkg1抗体用biotinlabelingkit-nh2(dojindo公司制)生物素化后,用1%bsa-pbs按照达到1μg/ml的方式稀释后使用。另外,作为阳性对照,将anti-humangas6(r&d公司制,dy885)用1%bsa-pbs按照成为0.2ng/ml的方式稀释后使用。将结果示于表1。在抗体与各抗原结合时以○来表示,未结合时以×来表示。[表1]抗人gas6抗体与人gas6突变体的结合km5320-rkg1及km5321-rkg1抗体以同等程度与hgas6-f及hgas6-fh结合。另一方面,这些抗体不与hgas6-fh-l314a、q315a、p316a结合。以上表明:km5320-rkg1及km5321-rkg1抗体与序列号4所示的人gas6的全长氨基酸序列中的第314位的亮氨酸、第315位的谷氨酰胺、第316位的脯氨酸中的至少任意一个结合。[实施例14]抗人gas6单克隆抗体的癌细胞株的增殖抑制活性评价为了确认抗人gas6单克隆抗体对gas6依赖性的癌细胞的增殖的作用,使用3种人癌细胞株进行细胞增殖测定。使用胰腺癌细胞株panc-1细胞(americantypeculturecollection)、恶性黑素瘤细胞株a375细胞(大日本制药)、胃癌细胞株mkn7细胞(理研细胞银行)进行增殖测定。用流式细胞术通过与实施例9同样的方法测定各细胞中有无gas6受体的表达,分别确认到表达axl和mer、axl和sky、axl和sky。将panc-1细胞、a375细胞悬浮于在dmem(lifetechnologies公司制)中添加有10%fbs(lifetechnologies公司制)的培养基中,将mkn7细胞悬浮于在rpmi1640(lifetechnologies公司制)中添加有10%fbs的培养基中。将各细胞悬液分别按照成为2.0×104、1.0×104、0.6×104个/孔的方式播种于96孔板中,在37℃、5%co2条件下静置培养。1天后将培养基交换为fbs无添加培养基并静置培养。1天后弃掉培养基,以200μl/孔添加用fbs无添加培养基调整为下述最终浓度的受试体并静置培养。受试体使用了最终浓度1μg/ml的hgas6-f以及最终浓度20μg/ml的haxl-hfc、km5320-rkg1抗体、km5321-rkg1抗体及通过公知的方法制作的抗二硝基苯肼(dnp)抗体(motokiket.al.,clin.cancerres.11,3126-3135,2005)。另外,作为阴性对照,使用用上述培养基稀释20倍的pbs。3天后弃掉培养基,以50μl/孔添加溶解于celltiter-glosubstrate(promega公司制)所附的celltiter-globuffer中的celltiter-gloreagent,用振荡器搅拌1分钟,在室温下静置10分钟后检测发光。对于panc-1细胞,将得到的结果示于图6。在panc-1细胞中添加hgas6时,与添加pbs时相比,细胞数增加至约2倍。与此相对,km5320-rkg1和km5321-rkg1将由hgas6导致的细胞增殖亢进抑制至与pbs同等程度。抗dnp抗体对由hgas6导致的细胞增殖亢进没有影响。另外,panc-1细胞以外的另外2个细胞株也为同样的结果。这些结果表明:km5320-rkg1和km5321-rkg1在表达gas6受体的癌细胞株中也抑制hgas6依赖性的细胞增殖。[实施例15]km5320、km5321人源化抗体的轻链及重链可变区的设计(1)km5320人源化抗体的vl及vh的氨基酸序列的设计通过以下所述的方法设计km5320人源化抗体的各种vl及vh的氨基酸序列。此后的说明中,作为具有各种vl、vh的氨基酸序列的km5320人源化抗体的总称而记作hzkm5320抗体。首先,为了选择适于km5320抗体的cdr的氨基酸序列的移植的、已知的人抗体的fr的氨基酸,利用thenationalcenterforbiotechnologyinformation提供的blastp数据库,对于vl及vh分别检索与km5320抗体的框架(以下记作fr)的氨基酸序列同源性高的已知的人抗体的fr的氨基酸序列。其结果是,genbank登录号aaw69164.1(anti-tetanustoxoidimmunoglobulinlightchainvariableregion)及ddbj登录号bac01510.1(immunoglobulinheavychainvhdjregion)所示的氨基酸序列(以下记作分别aaw69164.1、bac01510.1)的fr的氨基酸序列分别与km5320抗体的vl及vh的fr的氨基酸序列的同源性最高。因此,将序列号82、83、84所示的km5320的vl的cdr1~3的氨基酸序列分别移植到aaw69164.1的fr的氨基酸序列的适当的位置,从而设计hzkm5320lv0(序列号105)。另外,将序列号79、80、81所示的km5320vh的cdr1~3的氨基酸序列分别移植到bac01510.1的fr的氨基酸序列的适当的位置,从而设计hzkm5320hv0(序列号129)。按照上述那样设计的hzkm5320lv0及hzkm5320hv0为在所选择的人抗体的fr的氨基酸序列中仅移植了来自作为小鼠单克隆抗体的km5320的cdr的氨基酸序列的氨基酸序列。但是,通常在制作人源化抗体时,仅将来自啮齿类的抗体的cdr的氨基酸序列移植到人抗体的fr的氨基酸序列中的情况下,人源化抗体的结合活性多数情况下会下降。为了避免这种结合活性的下降,在移植cdr的氨基酸序列的同时,对于人抗体与啮齿类抗体中不同的fr的氨基酸残基中、认为会对抗体的结合活性产生影响的氨基酸残基进行改变。因此,本实施例中,还按照以下方式对认为会对抗体的结合活性产生影响的fr的氨基酸残基进行了鉴定、改变。首先,分别将vl、vh中具有上述设计出的hzkm5320lv0及hzkm5320hv0的抗体记作hzkm5320lv0hv0抗体或简记为hzkm5320lv0hv0。对于其它hzkm5320抗体,也用同样的方法来记载。用计算机模拟的方法构建hzkm5320lv0hv0抗体的可变区的三维结构。三维结构座标制作及三维结构的表示中使用discoverystudio(accelrys公司)。另外,也同样地构建了km5320抗体的可变区的三维结构的计算机模型。进而,制作将hzkm5320lv0hv0抗体的vl及vh的fr的氨基酸序列中的、与km5320抗体不同的氨基酸残基置换成存在于km5320抗体的相同位点的氨基酸残基的氨基酸序列,同样地构建三维结构模型。对上述制作的km5320抗体、hzkm5320lv0hv0抗体及改变体的可变区的三维结构进行比较,来鉴定推测会对抗体的结合活性产生影响的氨基酸残基。其结果是,在hzkm5320lv0hv0抗体的可变区的fr的氨基酸残基中,作为认为会改变抗原结合位点的三维结构而对抗体的结合活性产生影响的氨基酸残基,在vl中选择了序列号105所示的氨基酸序列的第2位的val、第15位的leu、第46位的leu、第73位的leu、第78位的leu及第87位的tyr,vh中选择了序列号129所示的氨基酸序列的第2位的val、第9位的ser、第20位的val、第38位的arg、第46位的glu、第77位的ser、第93位的val及第95位的tyr。将上述所选择的氨基酸残基中的至少1个以上氨基酸残基置换为存在于km5320抗体的相同位点的氨基酸残基,从而设计了具有各种改变的人源化抗体的vl及vh。具体而言,对于vl,导入下述氨基酸改变中的至少1个改变,所述改变为:将序列号105所示的氨基酸序列的第2位的val置换成ile、将第15位的leu置换成ala、将第46位的leu置换成val、将第73位的leu置换成phe、将第78位的leu置换成val及将第87位的tyr置换成phe。从而作为hzkm5320抗体的vl,设计出hzkm5320lv0(序列号105)、lv1a(序列号108)、lv1b(序列号111)、lv2a(序列号114)、lv2b(序列号117)、lv3(序列号120)、lv5(序列号123)及lv6(序列号126),将各氨基酸序列示于图7。另外,对于vh,导入下述氨基酸改变中的至少1个改变,所述改变为:将序列号129所示的氨基酸序列的第2位的val置换成ile、将第9位的ser置换成pro、将第20位的val置换成ile、将第38位的arg置换成lys、将第46位的glu置换成lys、将第77位的ser置换成thr、将第93位的val置换成thr及将第95位的tyr置换成phe。从而作为hzkm5320抗体的vh,设计出hzkm5320hv0(序列号129)、hv1(序列号132)、hv2(序列号135)、hv3a(序列号138)、hv3b(序列号141)、hv3c(序列号144)、hv4(序列号147)、hv6(序列号150)及hv8(序列号153),将各氨基酸序列示于图8。(2)km5321人源化抗体的vl及vh的氨基酸序列的设计还通过与实施例15(1)同样的方法设计了km5321人源化抗体的各种vl及vh的氨基酸序列。此后的说明中,作为具有各种vl、vh的氨基酸序列的km5321人源化抗体的总称而记作hzkm5321抗体。通过在genbank登录号aaw67414.1(rotavirus-specificintestinal-homingantibodylightchainvariableregion)所示的人抗体的vl的fr的氨基酸序列的适当的位置移植km5321抗体的vl的cdr1~3的氨基酸序列(各序列号88、89、90),从而设计出hzkm5321lv0(序列号156)。另外,通过在embl登录号caj13496.1(immunoglobulinheavychainvariableregion)所示的人抗体的fr的氨基酸序列的适当的位置移植km5321抗体vh的cdr1~3的氨基酸序列(各序列号85、86、87),从而设计出hzkm5321hv0(序列号186)。对于认为会对hzkm5321抗体的结合活性产生影响的fr的氨基酸残基,也通过与hzkm5320抗体情况下同样的方法分别对vl、vh进行选择。将选择的氨基酸残基中的至少1个以上氨基酸序列置换为存在于km5321抗体的相同位点的氨基酸残基,从而设计出具有各种改变的人源化抗体的vl及vh。具体而言,对于vl,导入下述氨基酸改变中的至少1个改变,所述改变为:将序列号156所示的氨基酸序列的第4位的leu置换为val、将第13位的ala置换为val、将第15位的val置换为thr、将第43位的ala置换为pro、将第64位的gly置换为ser、将第73位的leu置换为phe、将第78位的leu置换为thr、将第85位的thr置换为asp及将第104位的val置换为leu。从而作为hzkm5321抗体的vl,设计出hzkm5321lv0(序列号156)、lv1a(序列号159)、lv1b(序列号162)、lv1c(序列号165)、lv3(序列号168)、lv4(序列号171)、lv6(序列号174)、lv7a(序列号177)、lv7b(序列号180)及lv9(序列号183),将各氨基酸序列示于图9。另外,对于vh,导入下述氨基酸改变中的至少1个改变,所述改变为:将序列号186所示的氨基酸序列的第2位的val置换为ile、将第9位的ser置换为pro、将第38位的arg置换为lys、将第46位的glu置换为lys、将第79位的ser置换为ala、将第93位的val置换为thr及将第112位的val置换为ile。从而作为hzkm5321抗体的vh,设计出hzkm5321hv0(序列号186)、hv1(序列号189)、hv2a(序列号192)、hv2b(序列号195)、hv3a(序列号198)、hv3b(序列号201)、hv4a(序列号204)、hv4b(序列号207)、hv5(序列号210)及hv7(序列号213),将各氨基酸序列示于图10。此后的说明中,将vl、vh中分别具有hzkm5321lv0及hzkm5321hv0的抗体记作hzkm5321lv0hv0抗体或hzkm5321lv0hv0。对于其它的hzkm5321抗体,也通过同样的方法来记载。(3)人源化抗体的可变区基因的设计使用在动物细胞高频使用的密码子来设计编码人源化抗体(hzkm5320,hzkm5321抗体)的可变区的氨基酸序列的碱基序列。使用这些碱基序列构建人源化抗体的表达载体并表达人源化抗体。[实施例16]hzkm5320、hzkm5321抗体的表达载体的构建通过以下所述的方法构建了表2所示的hzkm5320、hzkm5321抗体的表达载体。[表2]制作的hzkm5320抗体和hzkm5321抗体hzkm5320抗体hzkm5321抗体hzkm5320lv0hv0hzkm5321lv0hv0hzkm5320lv1ahv0hzkm5321lv1ahv0hzkm5320lv1bhv0hzkm5321lv1bhv0hzkm5320lv2ahv0hzkm5321lv1chv0hzkm5320lv2bhv0hzkm5321lv3hv0hzkm5320lv3hv0hzkm5321lv4hv0hzkm5320lv5hv0hzkm5321lv6hv0hzkm5320lv6hv0hzkm5321lv7ahv0hzkm5320lv0hv8hzkm5321lv7bhv0hzkm5320lv1ahv8hzkm5321lv9hv0hzkm5320lv1bhv8hzkm5321lv0hv7hzkm5320lv2ahv8hzkm5321lv1ahv7hzkm5320lv2bhv8hzkm5321lv1bhv7hzkm5320lv3hv8hzkm5321lv1chv7hzkm5320lv5hv8hzkm5321lv3hv7hzkm5320lv6hv8hzkm5321lv4hv7hzkm5320lv5hv1hzkm5321lv6hv7hzkm5320lv5hv2hzkm5321lv7ahv7hzkm5320lv5hv3ahzkm5321lv7bhv7hzkm5320lv5hv3bhzkm5321lv9hv7hzkm5320lv5hv3chzkm5321lv6hv1hzkm5320lv5hv4hzkm5321lv6hv2ahzkm5320lv5hv6hzkm5321lv6hv2bhzkm5321lv6hv3ahzkm5321lv6hv3bhzkm5321lv6hv4ahzkm5321lv6hv4bhzkm5321lv6hv5首先,对于表3所述的编码各人源化抗体的含有信号序列的可变区氨基酸序列的碱基序列,通过fasmac公司合成必要的基因片段。[表3]使用上述合成基因片段、导入有适当的抗体分泌信号的人κ链恒定区的表达载体(econi/bsiwi处理)及人重链恒定区的表达载体(fspai/nhei处理),通过in-fuisionhd克隆试剂盒(clontech公司)进行向载体中的亚克隆。转化大肠杆菌dh5α感受态细胞(宝生物公司),确认所得到的质粒的序列。选拔产生插入有正确碱基序列的质粒的大肠杆菌菌落,使用nucleobondxtramidief试剂盒(宝生物公司)制备质粒。为了表达包含含有euindexs228p、l235e及r409k的氨基酸残基置换的突变人igg4恒定区的抗人gas6人源化抗体,使用如下载体作为人重链恒定区的表达载体:将n5kg1载体(美国专利第6,001,358号)中的编码轻链恒定区及重链恒定区的碱基序列用限制酶bgl2及bamhi除去,将该部分置换成编码上述突变人igg4的恒定区的碱基序列。[实施例17]hzkm5320、hzkm5321抗体的瞬时表达及纯化使用expi293fexpressionsystemkit(lifetechnologies公司制)使所制作的人源化抗体进行瞬时表达。质粒的导入的方法按照所附文件来进行。轻链的表达载体和重链的表达载体以1∶2的比率混合后进行导入。将导入质粒后的细胞在120ml的培养液中在37℃、5%co2、125rpm的条件下培养3天。然后,进行细胞培养液的离心分离,使其通过0.2μm过滤器(thermoscientific公司)并回收培养上清。从培养上清中,通过使用mabselectsure(gehealthcare公司制)的亲和纯化来得到纯化抗体。具体而言,将填充在柱中的树脂用pbs平衡化后,在该柱中添加培养上清,用pbs洗涤2次,用wash缓冲液1(pbs,含1mnacl)、wash缓冲液2(20mm柠檬酸、50mmnacl,ph5.0)各洗涤1次后,用洗脱缓冲液(20mm柠檬酸、50mmnacl,ph3.4)洗脱抗体。在得到的抗体溶液中加入1/10量的中和缓冲液(1m磷酸-naoh,ph7.0)进行中和,用nap25(gehealthcare公司制)将抗体溶液的溶剂置换为保存缓冲液(10mm柠檬酸、150mmnacl、ph6.0)。对于缓冲液置换后的抗体溶液,用amiconultra-4centrifugalfilterunits(millipore公司制)通过超滤而进行浓缩,使用nanodrop(thermoscientific公司)测定吸光度a280,进行抗体溶液的浓度的测定和调整。[实施例18]利用biacore(注册商标)进行的hzkm5320、hzkm5321抗体与人gas6蛋白的结合活性评价基于实施例16所述的方法,制作使km5320及km5321的可变区氨基酸序列结合于包含euindexs228p、l235e、r409k的氨基酸残基置换的突变人igg4恒定区的人嵌合抗体(以下分别记作km5320嵌合抗体、km5321嵌合抗体)。为了比较这些嵌合抗体、和实施例17所得到的hzkm5320、hzkm5321抗体与人gas6的结合活性,使用实施例2所制作的人gas6,通过表面等离子激元共振法(spr法)实施结合性试验。作为测定设备,使用biacore(注册商标)t100(gehealthcare公司制)。将抗人igg抗体用humanantibodycapturekit(gehealthcare公司制)按照所附说明书固定在cm5传感器芯片(gehealthcare公司制)上。将制备成1μg/ml的受试抗体以10μl/min的流速向流动池中添加10秒。然后,以30μl/min分的流速添加作为分析物的从10μg/ml以每次3倍的倍率进行5次阶梯稀释的人gas6蛋白溶液(用含有0.1%bsa的hbs-ep+稀释),对各抗体和分析物的结合反应进行2分钟测定,对解离反应进行10分钟测定。测定通过单周期动力学法进行。将得到的传感器图用biaevaluationsoftware(gehealthcare公司制)进行分析,计算各抗体的动力学常数。在本测定的预备试验中已经明确:在长时间地测定多个抗体的结合活性期间,即使是同一抗体,与测定的前半期相比,后半期所测定的ka值下降。可以认为,该现象是由于作为分析物使用的人gas6蛋白在测定中逐渐吸附于小瓶(バイアル)、实际浓度下降而产生的。为了排除分析物吸附于小瓶对测定结果产生的影响,本测定中,在小瓶中添加人gas6蛋白溶液后静置足够长的时间,在人gas6蛋白向小瓶的吸附达到稳定的状态下开始测定对各抗体的结合活性。将计算的km5320嵌合抗体及hzkm5320抗体与人gas6的结合速率常数(ka)的最小值、解离速率常数(kd)及解离常数[kd/ka=kd]的最大值示于表4。对于km5321嵌合抗体及hzkm5321抗体,也通过同样的方法测定结合活性,将得到的结果示于表6。另外,仅对于km5320嵌合抗体、hzkm5320lv5hv2抗体及hzkm5320lv1bhv0抗体,再次通过spr法进行结合性试验,将计算ka、kd及kd的结果示于表5。再次试验中,不像上述试验那样将添加于小瓶中的分析物静置足够长的时间,而是减少所测定的抗体的个数和分析物的浓度的变化,在短时间(数小时以内)进行测定。分析物使用了从10μg/ml以每次3倍的倍率进行3次阶梯稀释的gas6蛋白溶液(用含有0.1%bsa的hbs-ep+稀释)。从而可以使分析物对小瓶的吸附达到最低限度,与上述试验相比,可以计算更正确的ka、kd的值。对于km5321嵌合抗体及hzkm5321lv6hv2b抗体及hzkm5321lv7bhv0抗体,也通过同样的方法再次测定结合活性,将得到的结果示于表7。根据表4,km5320嵌合抗体(km5320chimera)和各种hzkm5320抗体的ka值均为同等程度。hzkm5320lv0hv0抗体与km5320嵌合抗体相比,kd值增加至约6倍,伴随该增加,kd值也增加了10倍以上,但其它hzkm5320抗体中,半数以上的抗体中,相对于km5320嵌合抗体的kd值的增加被抑制在2倍左右。另外,根据表6,km5321嵌合抗体(km5321chimera)和各种hzkm5321抗体的ka值均为同等程度。hzkm5321lv0hv0抗体与km5321嵌合抗体相比,kd值、kd值增加至约3倍,其它hzkm5321抗体中,三分之一以上的抗体中,相对于km5321嵌合抗体的kd值的增加被抑制在2倍程度。根据以上可以明确:仅将km5320、km5321抗体的cdr移植到人抗体的fr的hzkm5320lv0hv0抗体、hzkm5321lv0hv0抗体与km5320、km5321嵌合抗体相比,与gas6蛋白的结合活性大幅下降。但是,通过将hzkm5320lv0hv0、hzkm5321lv0hv0抗体的fr的氨基酸残基的一部分置换成存在于km5320抗体、km5321抗体的fr的相同位置的氨基酸残基,上述抗体结合活性的下降得到抑制,可以制作保持了km5320、km5321嵌合抗体的50%左右的结合活性的多个人源化抗体。另外,根据表5,km5320嵌合抗体的kd值为0.73nm,与此相对地,hzkm5320lv5hv2抗体的kd值为1.29nm,与表4的结果同样地,可以确认hzkm5320lv5hv2抗体保持了km5320嵌合抗体的50%以上的结合活性。进而,根据表7,km5321嵌合抗体的kd值为0.2nm,与此相对地,hzkm5321lv6hv2b抗体的kd值为0.48nm、hzkm5321lv7bhv0抗体的kd值为0.40nm,可以再次确认这些人源化抗体保持了km5321嵌合抗体的50%左右的结合活性。[表4]km5320嵌合抗体和hzkm5320抗体与人gas6的结合性(1)mabka(1/ms)kd(1/s)se(kd)kd(m)a-01.km5320chimera>8.37e+41.56e-41.18e-6<1.87e-9a-02.hzkm5320lv0hv0>6.68e+49.24e-42.59e-6<13.84e-9a-03.hzkm5320lv1ahv0>6.57e+48.29e-42.22e-6<12.63e-9a-04.hzkm5320lv1bhv0>7.20e+411.53e-43.49e-6<16.01e-9a-05.hzkm5320lv2ahv0>6.91e+410.50e-43.33e-6<15.18e-9a-06.hzkm5320lv2bhv0>7.26e+410.45e-43.03e-6<14.40e-9a-07.hzkm5320lv3hv0>7.19e+42.09e-41.48e-6<2.91e-9a-08.hzkm5320lv5hv0>6.67e+41.90e-41.58e-6<2.85e-9a-09.hzkm5320lv6hv0>6.72e+42.16e-41.80e-6<3.22e-9a-10.hzkm5320lv0hv8>7.29e+47.14e-43.00e-6<9.79e-9a-11.hzkm5320lv1ahv8>7.20e+46.94e-42.65e-6<9.64e-9a-12.hzkm5320lv1bhv8>7.79e+48.56e-43.19e-6<10.98e-9a-13.hzkm5320lv2ahv8>7.86e+48.00e-43.03e-6<10.17e-9a-14.hzkm5320lv2bhv8>8.10e+47.91e-43.70e-6<9.77e-9a-15.hzkm5320lv3hv8>7.56e+41.74e-41.66e-6<2.30e-9a-16.hzkm5320lv5hv8>7.36e+41.55e-42.08e-6<2.11e-9a-17.hzkm5320lv6hv8>7.25e+41.58e-42.21e-6<2.18e-9mabka(1/ms)kd(1/s)se(kd)kd(m)a-01.km5320chimera>8.37e+41.56e-41.18e-6<1.87e-9a-08.hzkm5320lv5hv0>6.67e+41.90e-41.58e-6<2.85e-9a-19.hzkm5320lv5hv1>7.41e+41.84e-42.17e-6<2.48e-9a-20.hzkm5320lv5hv2>7.43e+41.58e-42.39e-6<2.13e-9a-21.hzkm5320lv5hv3a>7.71e+41.77e-42.06e-6<2.30e-9a-22.hzkm5320lv5hv3b>7.63e+41.53e-42.18e-6<2.01e-9a-23.hzkm5320lv5hv3c>7.79e+41.40e-42.20e-6<1.79e-9a-24.hzkm5320lv5hv4>7.92e+41.73e-42.26e-6<2.19e-9a-25.hzkm5320lv5hv6>8.17e+41.47e-42.57e-6<1.80e-9a-16.hzkm5320lv5hv8>7.36e+41.55e-42.08e-6<2.11e-9[表5]km5320嵌合抗体和hzkm5320抗体与人gas6的结合性(2)mabka(1/ms)se(ka)kd(1/s)se(kd)kd(m)a-01.km5320chimera1.86e+51681.35e-46.95e-70.73e-9a-20.hzkm5320lv5hv21.52e+51971.97e-49.63e-71.29e-9a-04.hzkm5320lv1bhv00.90e+550714.89e-441.57e-716.51e-9[表6]km5321嵌合抗体和hzkm5321抗体与人gas6的结合性(1)mabka(1/ms)kd(1/s)se(kd)kd(m)b-01.km5321chimera>1.21e+51.41e-41.29e-6<1.16e-9b-02.lv0hv0>1.31e+54.68e-42.08e-6<3.57e-9b-03.lv1ahv0>1.26e+54.92e-42.22e-6<3.92e-9b-04.lv1bhv0>1.27e+55.43e-42.66e-6<4.26e-9b-05.lv1chv0>1.26e+55.11e-42.96e-6<4.05e-9b-06.lv3hv0>1.53e+52.85e-44.89e-6<1.86e-9b-07.lv4hv0>1.26e+55.51e-43.34e-6<4.38e-9b-08.lv6hv0>1.34e+51.59e-43.02e-6<1.19e-9b-09.lv7ahv0>1.33e+51.88e-43.13e-6<1.41e-9b-10.lv7bhv0>1.29e+52.42e-41.93e-6<1.88e-9b-11.lv9hv0>1.26e+52.47e-42.27e-6<1.96e-9b-12.lv0hv7>1.00e+56.72e-41.65e-6<6.74e-9b-13.lv1ahv7>1.00e+56.84e-41.94e-6<6.83e-9b-14.lv1bhv7>1.00e+57.51e-41.92e-6<7.49e-9b-15.lv1chv7>1.01e+57.50e-42.09e-6<7.45e-9b-16.lv3hv7>1.01e+57.12e-41.95e-6<7.03e-9b-17.lv4hv7>1.00e+58.59e-42.45e-6<8.59e-9b-18.lv6hv7>1.00e+52.81e-41.57e-6<2.81e-9b-19.lv7ahv7>1.03e+52.99e-41.64e-6<2.91e-9b-20.lv7bhv7>1.07e+53.29e-41.61e-6<3.07e-9b-21.lv9hv7>1.06e+53.52e-41.76e-6<3.33e-9mabka(1/ms)kd(1/s)se(kd)kd(m)b-01.km5321chimera>1.10e+51.37e-41.77e-6<1.25e-9b-08.lv6hv0>1.03e+52.38e-42.32e-6<2.32e-9b-22.lv6hv1>1.02e+52.39e-42.06e-6<2.35e-9b-23.lv6hv2a>1.02e+52.12e-41.99e-6<2.07e-9b-24.lv6hv2b>1.03e+52.39e-41.97e-6<2.33e-9b-25.lv6hv3a>1.05e+52.26e-41.94e-6<2.15e-9b-26.lv6hv3b>1.06e+52.00e-41.93e-6<1.88e-9b-27.lv6hv4a>1.06e+53.41e-42.41e-6<3.20e-9b-28.lv6hv4b>1.06e+52.04e-42.03e-6<1.92e-9b-29.lv6hv5>1.06e+52.77e-42.47e-6<2.61e-9b-18.lv6hv7>1.07e+52.78e-42.44e-6<2.59e-9[表7]km5321嵌合抗体和hzkm5321抗体与人gas6的结合性(2)mabka(1/ms)se(ka)kd(1/s)se(kd)kd(m)b-01.km5321chimera3.80e+55157.66e-59.92e-72.02e-10b-24.lv6hv2b3.19e+539715.43e-59.37e-74.84e-10b-10.lv7bhv03.53e+560714.23e-512.80e-74.03e-10[实施例19]利用elisa来评价hzkm5320、hzkm5321抗体与人gas6蛋白的结合活性基于实施例6所述的方法,通过elisa测定了hzkm5320抗体和hzkm5321抗体与人gas6蛋白的结合活性。由于嵌合抗体及人源化抗体是恒定区来自于人的抗体,因此作为二抗,使用含有用1%bsa-pbs稀释3000倍的山羊抗人igg(h&l)adstoms、rb、bv、hohorseradishperoxidase(americanqualex公司制、a110pd)的溶液。为了降低测定值的背景,二抗稀释液使用与抗dnp小鼠igg1抗体50μg/ml混合后在室温下孵育1小时的液体。将得到的结果示于图11。对于图11的图,利用每种抗体在各浓度下的吸光值,进行logistic曲线的曲线拟合,用统计分析语言r(ver.3.02)计算km5320、km5321嵌合抗体及hzkm5320、hzkm5321抗体的结合的ec50值及其se值。将结果示于表8。hzkm5320lv5hv2抗体显示出与km5320嵌合抗体同等程度的结合活性。另外,hzkm5321lv6hv2b、lv7bhv0抗体也显示出与km5321嵌合抗体同等程度的结合活性。[表8]km5320、km5321嵌合抗体、hzkm5320抗体和hzkm5321抗体与人gas6蛋白的结合性(elisa)抗体ec50[ng/ml]s.e.(ec50)km5320chimera11.321.235hzkm5320lv5hv212.191.546hzkm5320lv1bhv019.052.407km5321chimera6.781.221hzkm5321lv6hv2b5.951.342hzkm5321lv7bhv06.691.386dnp--[实施例20]hzkm5320、hzkm5321抗体的抑制人gas6蛋白与axl的结合的活性的评价通过与实施例7同样的方法,来测定hzkm5320、hzkm5321抗体的抑制人gas6蛋白与axl的结合的活性。将得到的结果示于图12。对于图12的图,利用每种抗体在各浓度下的elisa的吸光值,进行logistic曲线的拟合,使用统计分析语言r计算km5320、km5321嵌合抗体及hzkm5320、hzkm5321抗体的结合抑制的ic50值及其se值。将结果示于表9。明确了hzkm5320lv5hv2抗体维持了km5320嵌合抗体的7成左右的结合抑制活性。另外明确了:hzkm5321lv6hv2b抗体及hzkm5321lv7bhv0抗体维持了与km5321嵌合抗体同等程度的结合抑制活性。[表9]km5320、km5321嵌合抗体和hzkm5320、hzkm5321抗体的抑制人gas6蛋白与alx的结合的活性(elisa)抗体ic50[ng/ml]s.e.(ic50)km5320chimera21.804.485kzkm5320lv5hv233.626.328hzkm5320lv1bhv0742.2796.190km5321chimera12.323.354hzkm5321lv6hv2b12.652.651hzkm5321lv7bhv011.782.351dnp--产业上的可利用性本发明的单克隆抗体在肾病或癌症等gas6相关疾病的治疗及诊断中是有用的。本说明书中引用的全部出版物、专利及专利申请直接以参考形式纳入本说明书中。序列表自由文本序列号1-人工序列的说明:引物1序列号2-人工序列的说明:引物2序列号5-人工序列的说明:引物3序列号6-人工序列的说明:引物4序列号9-人工序列的说明:引物5序列号10-人工序列的说明:引物6序列号11-人工序列的说明:引物7序列号12-人工序列的说明:引物8序列号15-人工序列的说明:引物9序列号18-人工序列的说明:引物10序列号19-人工序列的说明:引物11序列号22-人工序列的说明:引物12序列号23-人工序列的说明:引物13序列号28-人工序列的说明:haxl-hfc序列号29-合成结构序列号30-人工序列的说明:引物14序列号31-人工序列的说明:引物15序列号34-人工序列的说明:引物16序列号35-人工序列的说明:引物17序列号36-人工序列的说明:caxl-hfc序列号37-合成结构序列号38-人工序列的说明:引物18序列号39-人工序列的说明:引物19序列号42-人工序列的说明:引物20序列号43-人工序列的说明:引物21序列号44-人工序列的说明:raxl-hfc序列号45-合成结构序列号48-人工序列的说明:maxl-mfc序列号49-合成结构序列号50-人工序列的说明:引物22序列号51-人工序列的说明:引物23序列号54-人工序列的说明:引物24序列号55-人工序列的说明:引物25序列号58-人工序列的说明:引物26序列号59-人工序列的说明:引物27序列号61-人工序列的说明:cntovh序列号62-合成结构序列号63-人工序列的说明:cntovl序列号64-合成结构序列号65-人工序列的说明:引物28序列号66-人工序列的说明:引物29序列号67-人工序列的说明:km5320vh序列号68-合成结构序列号69-人工序列的说明:除信号序列以外的km5320vh的氨基酸序列序列号70-人工序列的说明:km5320vl序列号71-合成结构序列号72-人工序列的说明:除信号序列以外的km5320vl的氨基酸序列序列号73-人工序列的说明:km5321vh序列号74-合成结构序列号75-人工序列的说明:除信号序列以外的km5321vh的氨基酸序列序列号76-人工序列的说明:km5321vl序列号77-合成结构序列号78-人工序列的说明:除信号序列以外的km5321vl的氨基酸序列序列号79-人工序列的说明:km5320vhcdr1的氨基酸序列序列号80-人工序列的说明:km5320vhcdr2的氨基酸序列序列号81-人工序列的说明:km5320vhcdr3的氨基酸序列序列号82-人工序列的说明:km5320vlcdr1的氨基酸序列序列号83-人工序列的说明:km5320vlcdr2的氨基酸序列序列号84-人工序列的说明:km5320vlcdr3的氨基酸序列序列号85-人工序列的说明:km5321vhcdr1的氨基酸序列序列号86-人工序列的说明:km5321vhcdr2的氨基酸序列序列号87-人工序列的说明:km5321vhcdr3的氨基酸序列序列号88-人工序列的说明:km5321vlcdr1的氨基酸序列序列号89-人工序列的说明:km5321vlcdr2的氨基酸序列序列号90-人工序列的说明:km5321vlcdr3的氨基酸序列序列号91-人工序列的说明:km5320-rigg1序列号92-合成结构序列号93-人工序列的说明:km5320-r_kappa序列号94-合成结构序列号95-人工序列的说明:km5321-rigg1序列号96-合成结构序列号97-人工序列的说明:km5321-r_kappa序列号98-合成结构序列号99-人工序列的说明:hgas6-delta序列号100-合成结构序列号101-人工序列的说明:引物30序列号102-人工序列的说明:引物31序列号103-人工序列的说明:hzkm5320lv0序列序列号104-合成结构序列号105-人工序列的说明:除信号序列以外的hzkm5320lv0的氨基酸序列序列号106-人工序列的说明:hzkm5320lv1a序列序列号107-合成结构序列号108-人工序列的说明:除信号序列以外的hzkm5320lv1a的氨基酸序列序列号109-人工序列的说明:hzkm5320lv1b序列序列号110-合成结构序列号111-人工序列的说明:除信号序列以外的hzkm5320lv1b的氨基酸序列序列号112-人工序列的说明:hzkm5320lv2a序列序列号113-合成结构序列号114-人工序列的说明:除信号序列以外的hzkm5320lv2a的氨基酸序列序列号115-人工序列的说明:hzkm5320lv2b序列序列号116-合成结构序列号117-人工序列的说明:除信号序列以外的hzkm5320lv2b的氨基酸序列序列号118-人工序列的说明:hzkm5320lv3序列序列号119-合成结构序列号120-人工序列的说明:除信号序列以外的hzkm5320lv3的氨基酸序列序列号121-人工序列的说明:hzkm5320lv5序列序列号122-合成结构序列号123-人工序列的说明:除信号序列以外的hzkm5320lv5的氨基酸序列序列号124-人工序列的说明:hzkm5320lv6序列序列号125-合成结构序列号126-人工序列的说明:除信号序列以外的hzkm5320lv6的氨基酸序列序列号127-人工序列的说明:hzkm5320hv0序列序列号128-合成结构序列号129-人工序列的说明:除信号序列以外的hzkm5320hv0的氨基酸序列序列号130-人工序列的说明:hzkm5320hv1序列序列号131-合成结构序列号132-人工序列的说明:除信号序列以外的hzkm5320hv1的氨基酸序列序列号133-人工序列的说明:hzkm5320hv2序列序列号134-合成结构序列号135-人工序列的说明:除信号序列以外的hzkm5320hv2的氨基酸序列序列号136-人工序列的说明:hzkm5320hv3a序列序列号137-合成结构序列号138-人工序列的说明:除信号序列以外的hzkm5320hv3a的氨基酸序列序列号139-人工序列的说明:hzkm5320hv3b序列序列号140-合成结构序列号141-人工序列的说明:除信号序列以外的hzkm5320hv3b的氨基酸序列序列号142-人工序列的说明:hzkm5320hv3c序列序列号143-合成结构序列号144-人工序列的说明:除信号序列以外的hzkm5320hv3c的氨基酸序列序列号145-人工序列的说明:hzkm5320hv4序列序列号146-合成结构序列号147-人工序列的说明:除信号序列以外的hzkm5320hv4的氨基酸序列序列号148-人工序列的说明:hzkm5320hv6序列序列号149-合成结构序列号150-人工序列的说明:除信号序列以外的hzkm5320hv6的氨基酸序列序列号151-人工序列的说明:hzkm5320hv8序列序列号152-合成结构序列号153-人工序列的说明:除信号序列以外的hzkm5320hv8的氨基酸序列序列号154-人工序列的说明:hzkm5321lv0序列序列号155-合成结构序列号156-人工序列的说明:除信号序列以外的hzkm5321lv0的氨基酸序列序列号157-人工序列的说明:hzkm5321lv1a序列序列号158-合成结构序列号159-人工序列的说明:除信号序列以外的hzkm5321lv1a的氨基酸序列序列号160-人工序列的说明:hzkm5321lv1b序列序列号161-合成结构序列号162-人工序列的说明:除信号序列以外的hzkm5321lv1b的氨基酸序列序列号163-人工序列的说明:hzkm5321lv1c序列序列号164-合成结构序列号165-人工序列的说明:除信号序列以外的hzkm5321lv1c的氨基酸序列序列号166-人工序列的说明:hzkm5321lv3序列序列号167-合成结构序列号168-人工序列的说明:除信号序列以外的hzkm5321lv3的氨基酸序列序列号169-人工序列的说明:hzkm5321lv4序列序列号170-合成结构序列号171-人工序列的说明:除信号序列以外的hzkm5321lv4的氨基酸序列序列号172-人工序列的说明:hzkm5321lv6序列序列号173-合成结构序列号174-人工序列的说明:除信号序列以外的hzkm5321lv6的氨基酸序列序列号175-人工序列的说明:hzkm5321lv7a序列序列号176-合成结构序列号177-人工序列的说明:除信号序列以外的hzkm5321lv7a的氨基酸序列序列号178-人工序列的说明:hzkm5321lv7b序列序列号179-合成结构序列号180-人工序列的说明:除信号序列以外的hzkm5321lv7b的氨基酸序列序列号181-人工序列的说明:hzkm5321lv9序列序列号182-合成结构序列号183-人工序列的说明:除信号序列以外的hzkm5321lv9的氨基酸序列序列号184-人工序列的说明:hzkm5321hv0序列序列号185-合成结构序列号186-人工序列的说明:除信号序列以外的hzkm5321hv0的氨基酸序列序列号187-人工序列的说明:hzkm5321hv1序列序列号188-合成结构序列号189-人工序列的说明:除信号序列以外的hzkm5321hv1的氨基酸序列序列号190-人工序列的说明:hzkm5321hv2a序列序列号191-合成结构序列号192-人工序列的说明:除信号序列以外的hzkm5321hv2a的氨基酸序列序列号193-人工序列的说明:hzkm5321hv2b序列序列号194-合成结构序列号195-人工序列的说明:除信号序列以外的hzkm5321hv2b的氨基酸序列序列号196-人工序列的说明:hzkm5321hv3a序列序列号197-合成结构序列号198-人工序列的说明:除信号序列以外的hzkm5321hv3a的氨基酸序列序列号199-人工序列的说明:hzkm5321hv3b序列序列号200-合成结构序列号201-人工序列的说明:除信号序列以外的hzkm5321hv3b的氨基酸序列序列号202-人工序列的说明:hzkm5321hv4a序列序列号203-合成结构序列号204-人工序列的说明:除信号序列以外的hzkm5321hv4a的氨基酸序列序列号205-人工序列的说明:hzkm5321hv4b序列序列号206-合成结构序列号207-人工序列的说明:除信号序列以外的hzkm5321hv4b的氨基酸序列序列号208-人工序列的说明:hzkm5321hv5序列序列号209-合成结构序列号210-人工序列的说明:除信号序列以外的hzkm5321hv5的氨基酸序列序列号211-人工序列的说明:hzkm5321hv7序列序列号212-合成结构序列号213-人工序列的说明:除信号序列以外的hzkm5321hv7的氨基酸序列序列表<110>协和发酵麒麟株式会社(kyowahakkokirinco.,ltd.)<120>抗人gas6单克隆抗体(anti-humangas6monoclonalantibody)<130>pkh-9002wo<150>us62/066687<151>2014-10-21<160>213<170>patentinversion3.3<210>1<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer1<400>1gccgaattcgccaccatggccccttcgctctcgc34<210>2<211>52<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer2<400>2gccggatccctacttgtcgtcgtcgtccttgtagtcggctgcggcgggctcc52<210>3<211>2037<212>dna<213>homosapiens<220><221>cds<222>(1)..(2037)<400>3atggccccttcgctctcgcccgggcccgccgccctgcgccgcgcgccg48metalaproserleuserproglyproalaalaleuargargalapro151015cagctgctgctgctgctgctggccgcggagtgcgcgcttgccgcgctg96glnleuleuleuleuleuleualaalaglucysalaleualaalaleu202530ttgccggcgcgcgaggccacgcagttcctgcggcccaggcagcgccgc144leuproalaargglualathrglnpheleuargproargglnargarg354045gcctttcaggtcttcgaggaggccaagcagggccacctggagagggag192alapheglnvalphegluglualalysglnglyhisleugluargglu505560tgcgtggaggagctgtgcagccgcgaggaggcgcgggaggtgttcgag240cysvalglugluleucysserarggluglualaarggluvalpheglu65707580aacgaccccgagacggattatttttacccaagatacttagactgcatc288asnaspprogluthrasptyrphetyrproargtyrleuaspcysile859095aacaagtatgggtctccgtacaccaaaaactcaggcttcgccacctgc336asnlystyrglyserprotyrthrlysasnserglyphealathrcys100105110gtgcaaaacctgcctgaccagtgcacgcccaacccctgcgataggaag384valglnasnleuproaspglncysthrproasnprocysasparglys115120125gggacccaagcctgccaggacctcatgggcaacttcttctgcctgtgt432glythrglnalacysglnaspleumetglyasnphephecysleucys130135140aaagctggctgggggggccggctctgcgacaaagatgtcaacgaatgc480lysalaglytrpglyglyargleucysasplysaspvalasnglucys145150155160agccaggagaacgggggctgcctccagatctgccacaacaagccgggt528serglngluasnglyglycysleuglnilecyshisasnlysprogly165170175agcttccactgttcctgccacagcggcttcgagctctcctctgatggc576serphehiscyssercyshisserglyphegluleuserseraspgly180185190aggacctgccaagacatagacgagtgcgcagactcggaggcctgcggg624argthrcysglnaspileaspglucysalaaspserglualacysgly195200205gaggcgcgctgcaagaacctgcccggctcctactcctgcctctgtgac672glualaargcyslysasnleuproglysertyrsercysleucysasp210215220gagggctttgcgtacagctcccaggagaaggcttgccgagatgtggac720gluglyphealatyrserserglnglulysalacysargaspvalasp225230235240gagtgtctgcagggccgctgtgagcaggtctgcgtgaactccccaggg768glucysleuglnglyargcysgluglnvalcysvalasnserprogly245250255agctacacctgccactgtgacgggcgtgggggcctcaagctgtcccag816sertyrthrcyshiscysaspglyargglyglyleulysleusergln260265270gacatggacacctgtgaggacatcttgccgtgcgtgcccttcagcgtg864aspmetaspthrcysgluaspileleuprocysvalpropheserval275280285gccaagagtgtgaagtccttgtacctgggccggatgttcagtgggacc912alalysservallysserleutyrleuglyargmetpheserglythr290295300cccgtgatccgactgcgcttcaagaggctgcagcccaccaggctggta960provalileargleuargphelysargleuglnprothrargleuval305310315320gctgagtttgacttccggacctttgaccccgagggcatcctcctcttt1008alaglupheasppheargthrpheaspprogluglyileleuleuphe325330335gccggaggccaccaggacagcacctggatcgtgctggccctgagagcc1056alaglyglyhisglnaspserthrtrpilevalleualaleuargala340345350ggccggctggagctgcagctgcgctacaacggtgtcggccgtgtcacc1104glyargleugluleuglnleuargtyrasnglyvalglyargvalthr355360365agcagcggcccggtcatcaaccatggcatgtggcagacaatctctgtt1152serserglyprovalileasnhisglymettrpglnthrileserval370375380gaggagctggcgcggaatctggtcatcaaggtcaacagggatgctgtc1200glugluleualaargasnleuvalilelysvalasnargaspalaval385390395400atgaaaatcgcggtggccggggacttgttccaaccggagcgaggactg1248metlysilealavalalaglyaspleupheglnprogluargglyleu405410415tatcatctgaacctgaccgtgggaggtattcccttccatgagaaggac1296tyrhisleuasnleuthrvalglyglyileprophehisglulysasp420425430ctcgtgcagcctataaaccctcgtctggatggctgcatgaggagctgg1344leuvalglnproileasnproargleuaspglycysmetargsertrp435440445aactggctgaacggagaagacaccaccatccaggaaacggtgaaagtg1392asntrpleuasnglygluaspthrthrileglngluthrvallysval450455460aacacgaggatgcagtgcttctcggtgacggagagaggctctttctac1440asnthrargmetglncyspheservalthrgluargglyserphetyr465470475480cccgggagcggcttcgccttctacagcctggactacatgcggacccct1488proglyserglyphealaphetyrserleuasptyrmetargthrpro485490495ctggacgtcgggactgaatcaacctgggaagtagaagtcgtggctcac1536leuaspvalglythrgluserthrtrpgluvalgluvalvalalahis500505510atccgcccagccgcagacacaggcgtgctgtttgcgctctgggccccc1584ileargproalaalaaspthrglyvalleuphealaleutrpalapro515520525gacctccgtgccgtgcctctctctgtggcactggtagactatcactcc1632aspleuargalavalproleuservalalaleuvalasptyrhisser530535540acgaagaaactcaagaagcagctggtggtcctggccgtggagcatacg1680thrlyslysleulyslysglnleuvalvalleualavalgluhisthr545550555560gccttggccctaatggagatcaaggtctgcgacggccaagagcacgtg1728alaleualaleumetgluilelysvalcysaspglyglngluhisval565570575gtcaccgtctcgctgagggacggtgaggccaccctggaggtggacggc1776valthrvalserleuargaspglyglualathrleugluvalaspgly580585590accaggggccagagcgaggtgagcgccgcgcagctgcaggagaggctg1824thrargglyglnsergluvalseralaalaglnleuglngluargleu595600605gccgtgctcgagaggcacctgcggagccccgtgctcacctttgctggc1872alavalleugluarghisleuargserprovalleuthrphealagly610615620ggcctgccagatgtgccggtgacttcagcgccagtcaccgcgttctac1920glyleuproaspvalprovalthrseralaprovalthralaphetyr625630635640cgcggctgcatgacactggaggtcaaccggaggctgctggacctggac1968argglycysmetthrleugluvalasnargargleuleuaspleuasp645650655gaggcggcgtacaagcacagcgacatcacggcccactcctgccccccc2016glualaalatyrlyshisseraspilethralahissercyspropro660665670gtggagcccgccgcagcctag2037valgluproalaalaala675<210>4<211>678<212>prt<213>homosapiens<400>4metalaproserleuserproglyproalaalaleuargargalapro151015glnleuleuleuleuleuleualaalaglucysalaleualaalaleu202530leuproalaargglualathrglnpheleuargproargglnargarg354045alapheglnvalphegluglualalysglnglyhisleugluargglu505560cysvalglugluleucysserarggluglualaarggluvalpheglu65707580asnaspprogluthrasptyrphetyrproargtyrleuaspcysile859095asnlystyrglyserprotyrthrlysasnserglyphealathrcys100105110valglnasnleuproaspglncysthrproasnprocysasparglys115120125glythrglnalacysglnaspleumetglyasnphephecysleucys130135140lysalaglytrpglyglyargleucysasplysaspvalasnglucys145150155160serglngluasnglyglycysleuglnilecyshisasnlysprogly165170175serphehiscyssercyshisserglyphegluleuserseraspgly180185190argthrcysglnaspileaspglucysalaaspserglualacysgly195200205glualaargcyslysasnleuproglysertyrsercysleucysasp210215220gluglyphealatyrserserglnglulysalacysargaspvalasp225230235240glucysleuglnglyargcysgluglnvalcysvalasnserprogly245250255sertyrthrcyshiscysaspglyargglyglyleulysleusergln260265270aspmetaspthrcysgluaspileleuprocysvalpropheserval275280285alalysservallysserleutyrleuglyargmetpheserglythr290295300provalileargleuargphelysargleuglnprothrargleuval305310315320alaglupheasppheargthrpheaspprogluglyileleuleuphe325330335alaglyglyhisglnaspserthrtrpilevalleualaleuargala340345350glyargleugluleuglnleuargtyrasnglyvalglyargvalthr355360365serserglyprovalileasnhisglymettrpglnthrileserval370375380glugluleualaargasnleuvalilelysvalasnargaspalaval385390395400metlysilealavalalaglyaspleupheglnprogluargglyleu405410415tyrhisleuasnleuthrvalglyglyileprophehisglulysasp420425430leuvalglnproileasnproargleuaspglycysmetargsertrp435440445asntrpleuasnglygluaspthrthrileglngluthrvallysval450455460asnthrargmetglncyspheservalthrgluargglyserphetyr465470475480proglyserglyphealaphetyrserleuasptyrmetargthrpro485490495leuaspvalglythrgluserthrtrpgluvalgluvalvalalahis500505510ileargproalaalaaspthrglyvalleuphealaleutrpalapro515520525aspleuargalavalproleuservalalaleuvalasptyrhisser530535540thrlyslysleulyslysglnleuvalvalleualavalgluhisthr545550555560alaleualaleumetgluilelysvalcysaspglyglngluhisval565570575valthrvalserleuargaspglyglualathrleugluvalaspgly580585590thrargglyglnsergluvalseralaalaglnleuglngluargleu595600605alavalleugluarghisleuargserprovalleuthrphealagly610615620glyleuproaspvalprovalthrseralaprovalthralaphetyr625630635640argglycysmetthrleugluvalasnargargleuleuaspleuasp645650655glualaalatyrlyshisseraspilethralahissercyspropro660665670valgluproalaalaala675<210>5<211>19<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer3<400>5cggcctcccgggcgctctg19<210>6<211>21<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer4<400>6cctccgctgtttcggacagag21<210>7<211>2040<212>dna<213>macacafascicularis<220><221>cds<222>(1)..(2040)<400>7atggccccttcgccctcgcccgggcccgccgccccgcgcgccgcgccg48metalaproserproserproglyproalaalaproargalaalapro151015ctgcggctgctgctgctgctgctggcctcggagtgcgcgctcgccgcg96leuargleuleuleuleuleuleualaserglucysalaleualaala202530ttgttgccggcgcgcgaggccacgcagttcctgcggcccaggcagcgc144leuleuproalaargglualathrglnpheleuargproargglnarg354045cgcgccttccaggtcttcgaggaggccaagcagggccacctggagagg192argalapheglnvalphegluglualalysglnglyhisleugluarg505560gagtgcgtggaggagctgtgcagccgcgaggaggcgcgggaggtgttc240glucysvalglugluleucysserarggluglualaarggluvalphe65707580gagaacgaccccgagacggactatttttacccgagatacttaggctgc288gluasnaspprogluthrasptyrphetyrproargtyrleuglycys859095atcgaaaagtatgggtctccctacgccaaagacccaggcttcgccacc336ileglulystyrglyserprotyralalysaspproglyphealathr100105110tgcgtgcagaacctgcctgaccagtgcacgcccaacccctgtgataag384cysvalglnasnleuproaspglncysthrproasnprocysasplys115120125aaggggacccaagcctgccaagacctcatgggcaacttcttctgcctg432lysglythrglnalacysglnaspleumetglyasnphephecysleu130135140tgtaaagctggctgggggggccggctctgtgacagagatgtcaatgaa480cyslysalaglytrpglyglyargleucysaspargaspvalasnglu145150155160tgcagccaggagaatgggggctgcctccagatctgccacaacaagccg528cysserglngluasnglyglycysleuglnilecyshisasnlyspro165170175ggcagcttccactgtgcctgccacagcggcttccagctctcctctgac576glyserphehiscysalacyshisserglypheglnleuserserasp180185190ggcaggacctgccaagacatagacgagtgtgcagactcggaggcctgc624glyargthrcysglnaspileaspglucysalaaspserglualacys195200205ggggaggcacgctgcaagaacctgcccggctcctactcctgcctctgt672glyglualaargcyslysasnleuproglysertyrsercysleucys210215220gacaagggctttgcatacagctcccaggagaaggcctgccgagatgtg720asplysglyphealatyrserserglnglulysalacysargaspval225230235240gatgagtgtctgcagggccgctgtgagcaggtctgtgtgaactccccg768aspglucysleuglnglyargcysgluglnvalcysvalasnserpro245250255ggcagctacacctgccactgtgatgggcgcgggggcctcaagctgtcc816glysertyrthrcyshiscysaspglyargglyglyleulysleuser260265270caggacatggacacctgtgaggacatcttgccgtgcgtgcccttcagc864glnaspmetaspthrcysgluaspileleuprocysvalpropheser275280285atggccaagagcgtgaagtccttgtacctgggccggatgttcagtggg912metalalysservallysserleutyrleuglyargmetphesergly290295300acccccgtgatccgactgcgcttcaagaggctgcagcccaccaggctc960thrprovalileargleuargphelysargleuglnprothrargleu305310315320gtagctgagtttgacttccggacctttgaccccgagggcatcctcctc1008valalaglupheasppheargthrpheaspprogluglyileleuleu325330335ttcgctggaggccaccaggacagcacctggatcgtgctggccctgcga1056phealaglyglyhisglnaspserthrtrpilevalleualaleuarg340345350gccggccggctggagctgcagctgcgctacaatggcgtcggccgcgtc1104alaglyargleugluleuglnleuargtyrasnglyvalglyargval355360365accagcagcggcccggtcatcaaccacggcatgtggcagacgatctct1152thrserserglyprovalileasnhisglymettrpglnthrileser370375380gttgaggagctggcgcggaatctggtcatcaaggtcaacagggacgct1200valglugluleualaargasnleuvalilelysvalasnargaspala385390395400gtcatgaaaatcgcggtggccggggacttgttccaaccggagcgagga1248valmetlysilealavalalaglyaspleupheglnprogluarggly405410415ctgtatcatctgaacctcaccgtaggaggcattcccttccatgagaag1296leutyrhisleuasnleuthrvalglyglyileprophehisglulys420425430gacctcgtgcagcctataaaccctcgtctggatggctgtatgaggagc1344aspleuvalglnproileasnproargleuaspglycysmetargser435440445tggaactggctgaacggagaagacaccaccatccaagaaacggtgaaa1392trpasntrpleuasnglygluaspthrthrileglngluthrvallys450455460gcgaacacgaaaatgcagtgcttctcggtgacagagagaggctccttt1440alaasnthrlysmetglncyspheservalthrgluargglyserphe465470475480tacccggggagtggctttgccttctacagcctggactacatgcggacc1488tyrproglyserglyphealaphetyrserleuasptyrmetargthr485490495cctctggacatcgggacagaatcgacctgggaaatagaagtcgtggct1536proleuaspileglythrgluserthrtrpgluilegluvalvalala500505510cacatccgcccggccgcagacacaggggtgctgttcgcactctgggtc1584hisileargproalaalaaspthrglyvalleuphealaleutrpval515520525cccgacctccgtgccgtgcctctctctgtggccctggtagactatcac1632proaspleuargalavalproleuservalalaleuvalasptyrhis530535540tccacaaagaagctcaagaagcagctggtggtcctggccgtggagcac1680serthrlyslysleulyslysglnleuvalvalleualavalgluhis545550555560gtggccttggccctgatggagatcaaggtctgcgatggtcaagagcac1728valalaleualaleumetgluilelysvalcysaspglyglngluhis565570575atggtcaccatctcgctgagggagggtgaggccaccctggaggtggac1776metvalthrileserleuarggluglyglualathrleugluvalasp580585590ggcaccaggggccagagcgaggtgagcgccgcgcagctgcaggagagg1824glythrargglyglnsergluvalseralaalaglnleuglngluarg595600605ctggccgtgctcgagaagcacctgcggagccccgtgctcacctttgcc1872leualavalleuglulyshisleuargserprovalleuthrpheala610615620ggtggcctgccagatgtgccggtgacttcagcgccagtcaccgcattc1920glyglyleuproaspvalprovalthrseralaprovalthralaphe625630635640taccgtggctgcatgacactggaggtcaaccggaggctgctggacctg1968tyrargglycysmetthrleugluvalasnargargleuleuaspleu645650655gacgaggcggcatacaagcacagtgacatcaccgcccactcctgccca2016aspglualaalatyrlyshisseraspilethralahissercyspro660665670cccgtggagcccgccgcagcctag2040provalgluproalaalaala675<210>8<211>679<212>prt<213>macacafascicularis<400>8metalaproserproserproglyproalaalaproargalaalapro151015leuargleuleuleuleuleuleualaserglucysalaleualaala202530leuleuproalaargglualathrglnpheleuargproargglnarg354045argalapheglnvalphegluglualalysglnglyhisleugluarg505560glucysvalglugluleucysserarggluglualaarggluvalphe65707580gluasnaspprogluthrasptyrphetyrproargtyrleuglycys859095ileglulystyrglyserprotyralalysaspproglyphealathr100105110cysvalglnasnleuproaspglncysthrproasnprocysasplys115120125lysglythrglnalacysglnaspleumetglyasnphephecysleu130135140cyslysalaglytrpglyglyargleucysaspargaspvalasnglu145150155160cysserglngluasnglyglycysleuglnilecyshisasnlyspro165170175glyserphehiscysalacyshisserglypheglnleuserserasp180185190glyargthrcysglnaspileaspglucysalaaspserglualacys195200205glyglualaargcyslysasnleuproglysertyrsercysleucys210215220asplysglyphealatyrserserglnglulysalacysargaspval225230235240aspglucysleuglnglyargcysgluglnvalcysvalasnserpro245250255glysertyrthrcyshiscysaspglyargglyglyleulysleuser260265270glnaspmetaspthrcysgluaspileleuprocysvalpropheser275280285metalalysservallysserleutyrleuglyargmetphesergly290295300thrprovalileargleuargphelysargleuglnprothrargleu305310315320valalaglupheasppheargthrpheaspprogluglyileleuleu325330335phealaglyglyhisglnaspserthrtrpilevalleualaleuarg340345350alaglyargleugluleuglnleuargtyrasnglyvalglyargval355360365thrserserglyprovalileasnhisglymettrpglnthrileser370375380valglugluleualaargasnleuvalilelysvalasnargaspala385390395400valmetlysilealavalalaglyaspleupheglnprogluarggly405410415leutyrhisleuasnleuthrvalglyglyileprophehisglulys420425430aspleuvalglnproileasnproargleuaspglycysmetargser435440445trpasntrpleuasnglygluaspthrthrileglngluthrvallys450455460alaasnthrlysmetglncyspheservalthrgluargglyserphe465470475480tyrproglyserglyphealaphetyrserleuasptyrmetargthr485490495proleuaspileglythrgluserthrtrpgluilegluvalvalala500505510hisileargproalaalaaspthrglyvalleuphealaleutrpval515520525proaspleuargalavalproleuservalalaleuvalasptyrhis530535540serthrlyslysleulyslysglnleuvalvalleualavalgluhis545550555560valalaleualaleumetgluilelysvalcysaspglyglngluhis565570575metvalthrileserleuarggluglyglualathrleugluvalasp580585590glythrargglyglnsergluvalseralaalaglnleuglngluarg595600605leualavalleuglulyshisleuargserprovalleuthrpheala610615620glyglyleuproaspvalprovalthrseralaprovalthralaphe625630635640tyrargglycysmetthrleugluvalasnargargleuleuaspleu645650655aspglualaalatyrlyshisseraspilethralahissercyspro660665670provalgluproalaalaala675<210>9<211>35<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer5<400>9cccacaagctgaattcgccaccatggccccttcgc35<210>10<211>63<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer6<400>10cgcgactagtggatccctacttgtcgtcgtcgtccttgtagtcggctgcggcgggctcca60cgg63<210>11<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer7<400>11ccggaattcgccaccatgccgccaccgcccgggc34<210>12<211>54<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer8<400>12gccggatccctacttgtcgtcgtcgtccttgtagtcggctgtgacgtgctccac54<210>13<211>2025<212>dna<213>rattusnorvegicus<220><221>cds<222>(1)..(2025)<400>13atgccgccaccgcccgggcccaccgccgccctgggcactgcgcttctg48metproproproproglyprothralaalaleuglythralaleuleu151015ctgctcctgctggcctccgagtcttcgcacactgtgctgttgcgggcg96leuleuleuleualasergluserserhisthrvalleuleuargala202530cgtgaggcggcgcagttcctgcggcccaggcagcgccgcgcctaccaa144argglualaalaglnpheleuargproargglnargargalatyrgln354045gtcttcgaggaggccaagcagggccacctggaacgggagtgcgtggag192valphegluglualalysglnglyhisleugluargglucysvalglu505560gaggtgtgcagcaaggaggaggctagagaggtgttcgagaacgacccc240gluvalcysserlysgluglualaarggluvalphegluasnasppro65707580gagacggactatttctatccaagatatcaagagtgcatgaggaaatat288gluthrasptyrphetyrproargtyrglnglucysmetarglystyr859095ggccggcccgaagataaaaacccaaatttcgccacctgtgttaagaac336glyargprogluasplysasnproasnphealathrcysvallysasn100105110ttacctgaccaatgcaccccaaacccctgtgataagaagggcactcaa384leuproaspglncysthrproasnprocysasplyslysglythrgln115120125ctctgccaagacctcatgggcaacttcttctgcttgtgcaaagatggc432leucysglnaspleumetglyasnphephecysleucyslysaspgly130135140tggggaggccggctctgtgacaaagatgtcaacgagtgtagtcagaag480trpglyglyargleucysasplysaspvalasnglucysserglnlys145150155160aatgggggctgcagccaggtctgccataacaaaccaggaagcttccaa528asnglyglycysserglnvalcyshisasnlysproglyserphegln165170175tgtgcctgccacagtggcttctcacttcaatcagacaacaagagctgc576cysalacyshisserglypheserleuglnseraspasnlyssercys180185190caagatatagatgaatgcacagactcagacacctgtggggatgcgcgt624glnaspileaspglucysthraspseraspthrcysglyaspalaarg195200205tgcaagaaccttccgggctcctactcctgcctctgcgacaaggggtac672cyslysasnleuproglysertyrsercysleucysasplysglytyr210215220acttacagctccaaggagaagacctgccaagatgtggatgagtgccag720thrtyrserserlysglulysthrcysglnaspvalaspglucysgln225230235240caggaccgttgtgagcagacctgtgtcaactccccaggcagctatacc768glnaspargcysgluglnthrcysvalasnserproglysertyrthr245250255tgccactgtaatgggcgcgggggcctaaaactgtccccagacatggat816cyshiscysasnglyargglyglyleulysleuserproaspmetasp260265270acctgtgaggacatcttaccgtgtgtgcccttcagcatggccaagagc864thrcysgluaspileleuprocysvalprophesermetalalysser275280285gtcaagtccttgtacctgggccgcatgttcagcgggacccccgtgatt912vallysserleutyrleuglyargmetpheserglythrprovalile290295300agactacgcttcaagaggctccagcctaccaggctgctggccgaattt960argleuargphelysargleuglnprothrargleuleualagluphe305310315320gacttccgtacttttgaccctgagggagtcctcttcttcgccggaggt1008asppheargthrpheaspprogluglyvalleuphephealaglygly325330335cgctcggatagcacctggatcgtcctgggcttcagggctgggcgactt1056argseraspserthrtrpilevalleuglypheargalaglyargleu340345350gagttgcagctacggtacaatggcgttggacgcatcaccagcagtggg1104gluleuglnleuargtyrasnglyvalglyargilethrsersergly355360365ccaaccatcaaccacggcatgtggcaaacgatctctgtggaagaactg1152prothrileasnhisglymettrpglnthrileservalglugluleu370375380gaccgcaaccttgtcatcaaggtcaacaaagatgccgtgatgaagatt1200aspargasnleuvalilelysvalasnlysaspalavalmetlysile385390395400gcggtggctggggggctgttccagctagagagaggcctgtaccacctg1248alavalalaglyglyleupheglnleugluargglyleutyrhisleu405410415aatctcactgtggggggcattcccttcaaggagagtgacctcgtccag1296asnleuthrvalglyglyileprophelysgluseraspleuvalgln420425430ccgattaaccctcgcctggacgggtgcatgaggagctggaactggctg1344proileasnproargleuaspglycysmetargsertrpasntrpleu435440445aatggggaagacagtgccattcaggaaacggtcaaggccaatacaaaa1392asnglygluaspseralaileglngluthrvallysalaasnthrlys450455460atgcagtgcttctctgtgacagagaggggctccttcttcccggggaat1440metglncyspheservalthrgluargglyserphepheproglyasn465470475480ggatttgccttctatagcctcaactacacccggacatcgctggatgtc1488glyphealaphetyrserleuasntyrthrargthrserleuaspval485490495ggcacggaaaccacctgggaagtagaagtcgtggctcgcattcgccct1536glythrgluthrthrtrpgluvalgluvalvalalaargileargpro500505510gccactgacacgggggtgctgatggcactggtgggggacaaagacgtc1584alathraspthrglyvalleumetalaleuvalglyasplysaspval515520525gtcctcctctctgtggccctggtcgactaccactccacaaagaagctc1632valleuleuservalalaleuvalasptyrhisserthrlyslysleu530535540aagaagcagctggtggtcctggcagttgagaatgttgccctggccctg1680lyslysglnleuvalvalleualavalgluasnvalalaleualaleu545550555560atggaaatcaaggtgtgcgacagccaggaacacactgtcactgtctcc1728metgluilelysvalcysaspserglngluhisthrvalthrvalser565570575ctgcgggatggcgaggccaccctggaagtggatggtaccaagggccag1776leuargaspglyglualathrleugluvalaspglythrlysglygln580585590agcgaagtgagcaccgcacagctgcaggagcgactggacctgcttaag1824sergluvalserthralaglnleuglngluargleuaspleuleulys595600605acacgtctgcaaggctccgtgctcacctttgtggggggcctgccagat1872thrargleuglnglyservalleuthrphevalglyglyleuproasp610615620gtacaagtgacttccacacccgtcacggcgttctaccgtggatgcatg1920valglnvalthrserthrprovalthralaphetyrargglycysmet625630635640actctggaggtaaacgggaagaccctggacctggatacggcctcctac1968thrleugluvalasnglylysthrleuaspleuaspthralasertyr645650655aagcacagtgacatcacctcccactcctgcccgcctgtggagcacgtc2016lyshisseraspilethrserhissercysproprovalgluhisval660665670acagcctag2025thrala<210>14<211>674<212>prt<213>rattusnorvegicus<400>14metproproproproglyprothralaalaleuglythralaleuleu151015leuleuleuleualasergluserserhisthrvalleuleuargala202530argglualaalaglnpheleuargproargglnargargalatyrgln354045valphegluglualalysglnglyhisleugluargglucysvalglu505560gluvalcysserlysgluglualaarggluvalphegluasnasppro65707580gluthrasptyrphetyrproargtyrglnglucysmetarglystyr859095glyargprogluasplysasnproasnphealathrcysvallysasn100105110leuproaspglncysthrproasnprocysasplyslysglythrgln115120125leucysglnaspleumetglyasnphephecysleucyslysaspgly130135140trpglyglyargleucysasplysaspvalasnglucysserglnlys145150155160asnglyglycysserglnvalcyshisasnlysproglyserphegln165170175cysalacyshisserglypheserleuglnseraspasnlyssercys180185190glnaspileaspglucysthraspseraspthrcysglyaspalaarg195200205cyslysasnleuproglysertyrsercysleucysasplysglytyr210215220thrtyrserserlysglulysthrcysglnaspvalaspglucysgln225230235240glnaspargcysgluglnthrcysvalasnserproglysertyrthr245250255cyshiscysasnglyargglyglyleulysleuserproaspmetasp260265270thrcysgluaspileleuprocysvalprophesermetalalysser275280285vallysserleutyrleuglyargmetpheserglythrprovalile290295300argleuargphelysargleuglnprothrargleuleualagluphe305310315320asppheargthrpheaspprogluglyvalleuphephealaglygly325330335argseraspserthrtrpilevalleuglypheargalaglyargleu340345350gluleuglnleuargtyrasnglyvalglyargilethrsersergly355360365prothrileasnhisglymettrpglnthrileservalglugluleu370375380aspargasnleuvalilelysvalasnlysaspalavalmetlysile385390395400alavalalaglyglyleupheglnleugluargglyleutyrhisleu405410415asnleuthrvalglyglyileprophelysgluseraspleuvalgln420425430proileasnproargleuaspglycysmetargsertrpasntrpleu435440445asnglygluaspseralaileglngluthrvallysalaasnthrlys450455460metglncyspheservalthrgluargglyserphepheproglyasn465470475480glyphealaphetyrserleuasntyrthrargthrserleuaspval485490495glythrgluthrthrtrpgluvalgluvalvalalaargileargpro500505510alathraspthrglyvalleumetalaleuvalglyasplysaspval515520525valleuleuservalalaleuvalasptyrhisserthrlyslysleu530535540lyslysglnleuvalvalleualavalgluasnvalalaleualaleu545550555560metgluilelysvalcysaspserglngluhisthrvalthrvalser565570575leuargaspglyglualathrleugluvalaspglythrlysglygln580585590sergluvalserthralaglnleuglngluargleuaspleuleulys595600605thrargleuglnglyservalleuthrphevalglyglyleuproasp610615620valglnvalthrserthrprovalthralaphetyrargglycysmet625630635640thrleugluvalasnglylysthrleuaspleuaspthralasertyr645650655lyshisseraspilethrserhissercysproprovalgluhisval660665670thrala<210>15<211>51<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer9<400>15ggatccctacttgtcgtcgtcgtccttgtagtcggctgtgacgtgctccac51<210>16<211>2025<212>dna<213>musmusculus<220><221>cds<222>(1)..(2025)<400>16atgccgccaccgcccgggcccgccgccgccctgggcactgcgcttctg48metproproproproglyproalaalaalaleuglythralaleuleu151015ctgctcctgctggcttccgagtcttctcacactgtgctgttgcgggcg96leuleuleuleualasergluserserhisthrvalleuleuargala202530cgtgaggcggcgcagtttctgcggcccaggcagcgccgcgcctaccaa144argglualaalaglnpheleuargproargglnargargalatyrgln354045gtcttcgaggaggccaagcagggccacctggaacgggagtgcgtggag192valphegluglualalysglnglyhisleugluargglucysvalglu505560gaggtgtgcagcaaagaggaggccagagaggtgttcgagaacgacccc240gluvalcysserlysgluglualaarggluvalphegluasnasppro65707580gagacggagtatttctatccacgatatcaagagtgcatgagaaaatat288gluthrglutyrphetyrproargtyrglnglucysmetarglystyr859095ggcaggcctgaagaaaaaaacccagatttcgccaaatgtgttcagaac336glyargprogluglulysasnproaspphealalyscysvalglnasn100105110ttgcctgaccagtgcaccccaaacccttgtgataagaagggtactcat384leuproaspglncysthrproasnprocysasplyslysglythrhis115120125atctgccaagacctcatgggcaacttcttctgcgtgtgcacagatggc432ilecysglnaspleumetglyasnphephecysvalcysthraspgly130135140tggggaggccggctctgtgacaaagatgtcaatgagtgtgtccagaag480trpglyglyargleucysasplysaspvalasnglucysvalglnlys145150155160aatgggggctgcagccaggtctgccacaacaaaccaggaagcttccaa528asnglyglycysserglnvalcyshisasnlysproglyserphegln165170175tgtgcctgccatagtggcttctcgcttgcatcagacggccagacctgc576cysalacyshisserglypheserleualaseraspglyglnthrcys180185190caagatatcgatgaatgcacagactcagacacctgtggggacgcgcga624glnaspileaspglucysthraspseraspthrcysglyaspalaarg195200205tgcaagaacttgccaggctcctactcttgcctctgcgatgagggatat672cyslysasnleuproglysertyrsercysleucysaspgluglytyr210215220acatacagctccaaggagaagacctgccaagatgtggacgagtgccag720thrtyrserserlysglulysthrcysglnaspvalaspglucysgln225230235240caggatcgctgtgagcagacctgtgtcaactccccaggcagctatacc768glnaspargcysgluglnthrcysvalasnserproglysertyrthr245250255tgccactgtgatgggcgagggggcctaaaactatccccagacatggat816cyshiscysaspglyargglyglyleulysleuserproaspmetasp260265270acttgtgaggacatcttaccatgtgtgcccttcagcatggccaagagc864thrcysgluaspileleuprocysvalprophesermetalalysser275280285gtgaagtccttgtacctgggccgcatgttcagcgggacccccgtgatt912vallysserleutyrleuglyargmetpheserglythrprovalile290295300agactacgcttcaagaggcttcagcctaccaggctgctggctgaattt960argleuargphelysargleuglnprothrargleuleualagluphe305310315320gacttccgcacttttgaccctgaaggagtcctcttcttcgctggaggc1008asppheargthrpheaspprogluglyvalleuphephealaglygly325330335cgttcagacagcacctggattgtcctgggcctaagagctgggcggctt1056argseraspserthrtrpilevalleuglyleuargalaglyargleu340345350gagctgcagcttcggtacaatggcgttgggcgcatcaccagcagcggg1104gluleuglnleuargtyrasnglyvalglyargilethrsersergly355360365ccaaccatcaaccacggcatgtggcaaactatctccgtggaagagctg1152prothrileasnhisglymettrpglnthrileservalglugluleu370375380gaacgtaaccttgtcatcaaggtcaacaaagatgctgtaatgaagatc1200gluargasnleuvalilelysvalasnlysaspalavalmetlysile385390395400gcggtagctggggagctgtttcagctggagaggggcctctatcacctg1248alavalalaglygluleupheglnleugluargglyleutyrhisleu405410415aatctcaccgtgggcggcattcccttcaaggagagtgagctcgtccag1296asnleuthrvalglyglyileprophelysglusergluleuvalgln420425430ccgattaaccctcgcctggatgggtgcatgaggagttggaactggctg1344proileasnproargleuaspglycysmetargsertrpasntrpleu435440445aacggggaagacagcgccatccaggagacagtcaaggcaaacacaaaa1392asnglygluaspseralaileglngluthrvallysalaasnthrlys450455460atgcagtgcttctctgtgacagaaaggggctccttcttcccggggaat1440metglncyspheservalthrgluargglyserphepheproglyasn465470475480ggatttgctacctacaggctcaactacacccgaacatcgctggatgtc1488glyphealathrtyrargleuasntyrthrargthrserleuaspval485490495ggcacggaaaccacctgggaagttaaagttgtggctcggatccgccct1536glythrgluthrthrtrpgluvallysvalvalalaargileargpro500505510gccacggacacgggggtgctgctggcgctggtgggggacgacgatgtc1584alathraspthrglyvalleuleualaleuvalglyaspaspaspval515520525gtccccatctctgtggccctagtcgactaccactctacaaagaagctc1632valproileservalalaleuvalasptyrhisserthrlyslysleu530535540aagaagcagttggtggtcctggcagttgaggatgttgccctggcactg1680lyslysglnleuvalvalleualavalgluaspvalalaleualaleu545550555560atggaaatcaaggtgtgcgacagccaggaacacacggtcactgtctcc1728metgluilelysvalcysaspserglngluhisthrvalthrvalser565570575ctgcgggagggtgaggccaccctagaagtggatggcacaaagggccag1776leuarggluglyglualathrleugluvalaspglythrlysglygln580585590agtgaagtgagcactgcccagctgcaggagcgactggacacacttaag1824sergluvalserthralaglnleuglngluargleuaspthrleulys595600605acacatctgcaaggctctgtgcacacctatgttggaggcctgccagaa1872thrhisleuglnglyservalhisthrtyrvalglyglyleuproglu610615620gtatcggtgatttctgcacccgtcactgcgttctaccgcggatgcatg1920valservalileseralaprovalthralaphetyrargglycysmet625630635640actctggaggtaaacgggaaaatcctggacctggatacggcctcgtac1968thrleugluvalasnglylysileleuaspleuaspthralasertyr645650655aagcacagtgacatcacctcccactcctgcccgcctgtggagcatgcc2016lyshisseraspilethrserhissercysproprovalgluhisala660665670accccctag2025thrpro<210>17<211>674<212>prt<213>musmusculus<400>17metproproproproglyproalaalaalaleuglythralaleuleu151015leuleuleuleualasergluserserhisthrvalleuleuargala202530argglualaalaglnpheleuargproargglnargargalatyrgln354045valphegluglualalysglnglyhisleugluargglucysvalglu505560gluvalcysserlysgluglualaarggluvalphegluasnasppro65707580gluthrglutyrphetyrproargtyrglnglucysmetarglystyr859095glyargprogluglulysasnproaspphealalyscysvalglnasn100105110leuproaspglncysthrproasnprocysasplyslysglythrhis115120125ilecysglnaspleumetglyasnphephecysvalcysthraspgly130135140trpglyglyargleucysasplysaspvalasnglucysvalglnlys145150155160asnglyglycysserglnvalcyshisasnlysproglyserphegln165170175cysalacyshisserglypheserleualaseraspglyglnthrcys180185190glnaspileaspglucysthraspseraspthrcysglyaspalaarg195200205cyslysasnleuproglysertyrsercysleucysaspgluglytyr210215220thrtyrserserlysglulysthrcysglnaspvalaspglucysgln225230235240glnaspargcysgluglnthrcysvalasnserproglysertyrthr245250255cyshiscysaspglyargglyglyleulysleuserproaspmetasp260265270thrcysgluaspileleuprocysvalprophesermetalalysser275280285vallysserleutyrleuglyargmetpheserglythrprovalile290295300argleuargphelysargleuglnprothrargleuleualagluphe305310315320asppheargthrpheaspprogluglyvalleuphephealaglygly325330335argseraspserthrtrpilevalleuglyleuargalaglyargleu340345350gluleuglnleuargtyrasnglyvalglyargilethrsersergly355360365prothrileasnhisglymettrpglnthrileservalglugluleu370375380gluargasnleuvalilelysvalasnlysaspalavalmetlysile385390395400alavalalaglygluleupheglnleugluargglyleutyrhisleu405410415asnleuthrvalglyglyileprophelysglusergluleuvalgln420425430proileasnproargleuaspglycysmetargsertrpasntrpleu435440445asnglygluaspseralaileglngluthrvallysalaasnthrlys450455460metglncyspheservalthrgluargglyserphepheproglyasn465470475480glyphealathrtyrargleuasntyrthrargthrserleuaspval485490495glythrgluthrthrtrpgluvallysvalvalalaargileargpro500505510alathraspthrglyvalleuleualaleuvalglyaspaspaspval515520525valproileservalalaleuvalasptyrhisserthrlyslysleu530535540lyslysglnleuvalvalleualavalgluaspvalalaleualaleu545550555560metgluilelysvalcysaspserglngluhisthrvalthrvalser565570575leuarggluglyglualathrleugluvalaspglythrlysglygln580585590sergluvalserthralaglnleuglngluargleuaspthrleulys595600605thrhisleuglnglyservalhisthrtyrvalglyglyleuproglu610615620valservalileseralaprovalthralaphetyrargglycysmet625630635640thrleugluvalasnglylysileleuaspleuaspthralasertyr645650655lyshisseraspilethrserhissercysproprovalgluhisala660665670thrpro<210>18<211>38<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer10<400>18ccgaagcttgccaccatgggcaccacctggaggagccc38<210>19<211>36<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer11<400>19ggcccgggtcagggctttttgaccttgtgttctggc36<210>20<211>486<212>dna<213>rattusnorvegicus<220><221>cds<222>(1)..(486)<400>20atgggcaccacctggaggagccctggacgtttgcggcttgcactatgc48metglythrthrtrpargserproglyargleuargleualaleucys151015ctcgctggcctagccctctcactgtacgcactgcacgtgaaggcggcg96leualaglyleualaleuserleutyralaleuhisvallysalaala202530cgcgcccgcaatgaggattaccgcgcgctctgcgacgtgggcacggcc144argalaargasngluasptyrargalaleucysaspvalglythrala354045atcagctgttcccgcgtcttctcctctcggtggggccggggctttggg192ilesercysserargvalpheserserargtrpglyargglyphegly505560ctggtggagcatgtgttaggagctgacagcatcctcaaccaatccaac240leuvalgluhisvalleuglyalaaspserileleuasnglnserasn65707580agcatatttggttgcatgttctacaccatacagctgttgttaggttgc288serilepheglycysmetphetyrthrileglnleuleuleuglycys859095ttgaggggacgttgggcctctatcctactgatcctgagttccctggtg336leuargglyargtrpalaserileleuleuileleuserserleuval100105110tctgtcgctggttctctgtacctggcctggatcctgttctttgtcctg384servalalaglyserleutyrleualatrpileleuphephevalleu115120125tatgatttctgcattgtttgcatcaccacctatgccatcaatgcgggc432tyraspphecysilevalcysilethrthrtyralaileasnalagly130135140ctgatgttgcttagcttccagaaggtgccagaacacaaggtcaaaaag480leumetleuleuserpheglnlysvalprogluhislysvallyslys145150155160ccctga486pro<210>21<211>161<212>prt<213>rattusnorvegicus<400>21metglythrthrtrpargserproglyargleuargleualaleucys151015leualaglyleualaleuserleutyralaleuhisvallysalaala202530argalaargasngluasptyrargalaleucysaspvalglythrala354045ilesercysserargvalpheserserargtrpglyargglyphegly505560leuvalgluhisvalleuglyalaaspserileleuasnglnserasn65707580serilepheglycysmetphetyrthrileglnleuleuleuglycys859095leuargglyargtrpalaserileleuleuileleuserserleuval100105110servalalaglyserleutyrleualatrpileleuphephevalleu115120125tyraspphecysilevalcysilethrthrtyralaileasnalagly130135140leumetleuleuserpheglnlysvalprogluhislysvallyslys145150155160pro<210>22<211>35<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer12<400>22cggtcgacgccaccatggcggtgtctgccgggtcc35<210>23<211>32<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer13<400>23ggcccgggtcagaactctgagtggacaggatc32<210>24<211>2277<212>dna<213>homosapiens<220><221>cds<222>(1)..(2277)<400>24atggcggtgtctgccgggtccgcgcggacctcgcccagctcagataaa48metalavalseralaglyseralaargthrserproserserasplys151015gtacagaaagacaaggctgaactgatctcagggcccaggcaggacagc96valglnlysasplysalagluleuileserglyproargglnaspser202530cgaatagggaaactcttgggttttgagtggacagatttgtccagttgg144argileglylysleuleuglypheglutrpthraspleusersertrp354045cggaggctggtgaccctgctgaatcgaccaacggaccctgcaagctta192argargleuvalthrleuleuasnargprothraspproalaserleu505560gctgtctttcgttttctttttgggttcttgatggtgctagacattccc240alavalpheargpheleupheglypheleumetvalleuaspilepro65707580caggagcgggggctcagctctctggaccggaaataccttgatgggctg288glngluargglyleuserserleuasparglystyrleuaspglyleu859095gatgtgtgccgcttccccttgctggatgccctacgcccactgccactt336aspvalcysargpheproleuleuaspalaleuargproleuproleu100105110gactggatgtatcttgtctacaccatcatgtttctgggggcactgggc384asptrpmettyrleuvaltyrthrilemetpheleuglyalaleugly115120125atgatgctgggcctgtgctaccggataagctgtgtgttattcctgctg432metmetleuglyleucystyrargilesercysvalleupheleuleu130135140ccatactggtatgtgtttctcctggacaagacatcatggaacaaccac480protyrtrptyrvalpheleuleuasplysthrsertrpasnasnhis145150155160tcctatctgtatgggttgttggcctttcagctaacattcatggatgca528sertyrleutyrglyleuleualapheglnleuthrphemetaspala165170175aaccactactggtctgtggacggtctgctgaatgcccataggaggaat576asnhistyrtrpservalaspglyleuleuasnalahisargargasn180185190gcccacgtgcccctttggaactatgcagtgctccgtggccagatcttc624alahisvalproleutrpasntyralavalleuargglyglnilephe195200205attgtgtacttcattgcgggtgtgaaaaagctggatgcagactgggtt672ilevaltyrpheilealaglyvallyslysleuaspalaasptrpval210215220gaaggctattccatggaatatttgtcccggcactggctcttcagtccc720gluglytyrsermetglutyrleuserarghistrpleupheserpro225230235240ttcaaactgctgttgtctgaggagctgactagcctgctggtcgtgcac768phelysleuleuleuserglugluleuthrserleuleuvalvalhis245250255tggggtgggctgctgcttgacctctcagctggtttcctgctctttttt816trpglyglyleuleuleuaspleuseralaglypheleuleuphephe260265270gatgtctcaagatccattggcctgttctttgtgtcctacttccactgc864aspvalserargserileglyleuphephevalsertyrphehiscys275280285atgaattcccagcttttcagcattggtatgttctcctacgtcatgctg912metasnserglnleupheserileglymetphesertyrvalmetleu290295300gccagcagccctctcttctgctcccctgagtggcctcggaagctggtg960alaserserproleuphecysserproglutrpproarglysleuval305310315320tcctactgcccccgaaggttgcaacaactgttgcccctcaaggcagcc1008sertyrcysproargargleuglnglnleuleuproleulysalaala325330335cctcagcccagtgtttcctgtgtgtataagaggagccggggcaaaagt1056proglnproservalsercysvaltyrlysargserargglylysser340345350ggccagaagccagggctgcgccatcagctgggagctgccttcaccctg1104glyglnlysproglyleuarghisglnleuglyalaalaphethrleu355360365ctctacctcctggagcagctattcctgccctattctcattttctcacc1152leutyrleuleugluglnleupheleuprotyrserhispheleuthr370375380cagggctataacaactggacaaatgggctgtatggctattcctgggac1200glnglytyrasnasntrpthrasnglyleutyrglytyrsertrpasp385390395400atgatggtgcactcccgctcccaccagcacgtgaagatcacctaccgt1248metmetvalhisserargserhisglnhisvallysilethrtyrarg405410415gatggccgcactggcgaactgggctaccttaaccctggggtatttaca1296aspglyargthrglygluleuglytyrleuasnproglyvalphethr420425430cagagtcggcgatggaaggatcatgcagacatgctgaagcaatatgcc1344glnserargargtrplysasphisalaaspmetleulysglntyrala435440445acttgcctgagccgcctgcttcccaagtataatgtcactgagccccag1392thrcysleuserargleuleuprolystyrasnvalthrgluprogln450455460atctactttgatatttgggtctccatcaatgaccgcttccagcagagg1440iletyrpheaspiletrpvalserileasnaspargpheglnglnarg465470475480atttttgaccctcgtgtggacatcgtgcaggccgcttggtcacccttt1488ilepheaspproargvalaspilevalglnalaalatrpserprophe485490495cagcgcacatcctgggtgcaaccactcttgatggacctgtctccctgg1536glnargthrsertrpvalglnproleuleumetaspleuserprotrp500505510agggccaagttacaggaaatcaagagcagcctagacaaccacactgag1584argalalysleuglngluilelysserserleuaspasnhisthrglu515520525gtggtcttcattgcagatttccctggactgcacttggagaattttgtg1632valvalpheilealaasppheproglyleuhisleugluasnpheval530535540agtgaagacctgggcaacactagcatccagctgctgcagggggaagtg1680sergluaspleuglyasnthrserileglnleuleuglnglygluval545550555560actgtggagcttgtggcagaacagaagaaccagactcttcgagaggga1728thrvalgluleuvalalagluglnlysasnglnthrleuargglugly565570575gaaaaaatgcagttgcctgctggtgagtaccataaggtgtatacgaca1776glulysmetglnleuproalaglyglutyrhislysvaltyrthrthr580585590tcacctagcccttcttgctacatgtacgtctatgtcaacactacagag1824serproserprosercystyrmettyrvaltyrvalasnthrthrglu595600605cttgcactggagcaagacctggcatatctgcaagaattaaaggaaaag1872leualaleugluglnaspleualatyrleuglngluleulysglulys610615620gtggagaatggaagtgaaacagggcctctacccccagagctgcagcct1920valgluasnglysergluthrglyproleuproprogluleuglnpro625630635640ctgttggaaggggaagtaaaagggggccctgagccaacacctctggtt1968leuleugluglygluvallysglyglyprogluprothrproleuval645650655cagacctttcttagacgccaacaaaggctccaggagattgaacgccgg2016glnthrpheleuargargglnglnargleuglngluilegluargarg660665670cgaaatactcctttccatgagcgattcttccgcttcttgttgcgaaag2064argasnthrprophehisgluargphepheargpheleuleuarglys675680685ctctatgtctttcgccgcagcttcctgatgacttgtatctcacttcga2112leutyrvalpheargargserpheleumetthrcysileserleuarg690695700aatctgatattaggccgtccttccctggagcagctggcccaggaggtg2160asnleuileleuglyargproserleugluglnleualaglngluval705710715720acttatgcaaacttgagaccctttgaggcagttggagaactgaatccc2208thrtyralaasnleuargpropheglualavalglygluleuasnpro725730735tcaaacacggattcttcacattctaatcctcctgagtcaaatcctgat2256serasnthraspserserhisserasnproprogluserasnproasp740745750cctgtccactcagagttctga2277provalhissergluphe755<210>25<211>758<212>prt<213>homosapiens<400>25metalavalseralaglyseralaargthrserproserserasplys151015valglnlysasplysalagluleuileserglyproargglnaspser202530argileglylysleuleuglypheglutrpthraspleusersertrp354045argargleuvalthrleuleuasnargprothraspproalaserleu505560alavalpheargpheleupheglypheleumetvalleuaspilepro65707580glngluargglyleuserserleuasparglystyrleuaspglyleu859095aspvalcysargpheproleuleuaspalaleuargproleuproleu100105110asptrpmettyrleuvaltyrthrilemetpheleuglyalaleugly115120125metmetleuglyleucystyrargilesercysvalleupheleuleu130135140protyrtrptyrvalpheleuleuasplysthrsertrpasnasnhis145150155160sertyrleutyrglyleuleualapheglnleuthrphemetaspala165170175asnhistyrtrpservalaspglyleuleuasnalahisargargasn180185190alahisvalproleutrpasntyralavalleuargglyglnilephe195200205ilevaltyrpheilealaglyvallyslysleuaspalaasptrpval210215220gluglytyrsermetglutyrleuserarghistrpleupheserpro225230235240phelysleuleuleuserglugluleuthrserleuleuvalvalhis245250255trpglyglyleuleuleuaspleuseralaglypheleuleuphephe260265270aspvalserargserileglyleuphephevalsertyrphehiscys275280285metasnserglnleupheserileglymetphesertyrvalmetleu290295300alaserserproleuphecysserproglutrpproarglysleuval305310315320sertyrcysproargargleuglnglnleuleuproleulysalaala325330335proglnproservalsercysvaltyrlysargserargglylysser340345350glyglnlysproglyleuarghisglnleuglyalaalaphethrleu355360365leutyrleuleugluglnleupheleuprotyrserhispheleuthr370375380glnglytyrasnasntrpthrasnglyleutyrglytyrsertrpasp385390395400metmetvalhisserargserhisglnhisvallysilethrtyrarg405410415aspglyargthrglygluleuglytyrleuasnproglyvalphethr420425430glnserargargtrplysasphisalaaspmetleulysglntyrala435440445thrcysleuserargleuleuprolystyrasnvalthrgluprogln450455460iletyrpheaspiletrpvalserileasnaspargpheglnglnarg465470475480ilepheaspproargvalaspilevalglnalaalatrpserprophe485490495glnargthrsertrpvalglnproleuleumetaspleuserprotrp500505510argalalysleuglngluilelysserserleuaspasnhisthrglu515520525valvalpheilealaasppheproglyleuhisleugluasnpheval530535540sergluaspleuglyasnthrserileglnleuleuglnglygluval545550555560thrvalgluleuvalalagluglnlysasnglnthrleuargglugly565570575glulysmetglnleuproalaglyglutyrhislysvaltyrthrthr580585590serproserprosercystyrmettyrvaltyrvalasnthrthrglu595600605leualaleugluglnaspleualatyrleuglngluleulysglulys610615620valgluasnglysergluthrglyproleuproprogluleuglnpro625630635640leuleugluglygluvallysglyglyprogluprothrproleuval645650655glnthrpheleuargargglnglnargleuglngluilegluargarg660665670argasnthrprophehisgluargphepheargpheleuleuarglys675680685leutyrvalpheargargserpheleumetthrcysileserleuarg690695700asnleuileleuglyargproserleugluglnleualaglngluval705710715720thrtyralaasnleuargpropheglualavalglygluleuasnpro725730735serasnthraspserserhisserasnproprogluserasnproasp740745750provalhissergluphe755<210>26<211>2685<212>dna<213>homosapiens<220><221>cds<222>(1)..(2685)<400>26atggcgtggcggtgccccaggatgggcagggtcccgctggcctggtgc48metalatrpargcysproargmetglyargvalproleualatrpcys151015ttggcgctgtgcggctgggcgtgcatggcccccaggggcacgcaggct96leualaleucysglytrpalacysmetalaproargglythrglnala202530gaagaaagtcccttcgtgggcaacccagggaatatcacaggtgcccgg144glugluserprophevalglyasnproglyasnilethrglyalaarg354045ggactcacgggcacccttcggtgtcagctccaggttcagggagagccc192glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560cccgaggtacattggcttcgggatggacagatcctggagctcgcggac240progluvalhistrpleuargaspglyglnileleugluleualaasp65707580agcacccagacccaggtgcccctgggtgaggatgaacaggatgactgg288serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095atagtggtcagccagctcagaatcacctccctgcagctttccgacacg336ilevalvalserglnleuargilethrserleuglnleuseraspthr100105110ggacagtaccagtgtttggtgtttctgggacatcagaccttcgtgtcc384glyglntyrglncysleuvalpheleuglyhisglnthrphevalser115120125cagcctggctatgttgggctggagggcttgccttacttcctggaggag432glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140cccgaagacaggactgtggccgccaacacccccttcaacctgagctgc480progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160caagctcagggacccccagagcccgtggacctactctggctccaggat528glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175gctgtccccctggccacggctccaggtcacggcccccagcgcagcctg576alavalproleualathralaproglyhisglyproglnargserleu180185190catgttccagggctgaacaagacatcctctttctcctgcgaagcccat624hisvalproglyleuasnlysthrserserphesercysglualahis195200205aacgccaagggggtcaccacatcccgcacagccaccatcacagtgctc672asnalalysglyvalthrthrserargthralathrilethrvalleu210215220ccccagcagccccgtaacctccacctggtctcccgccaacccacggag720proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240ctggaggtggcttggactccaggcctgagcggcatctaccccctgacc768leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255cactgcaccctgcaggctgtgctgtcagacgatgggatgggcatccag816hiscysthrleuglnalavalleuseraspaspglymetglyilegln260265270gcgggagaaccagaccccccagaggagcccctcacctcgcaagcatcc864alaglygluproaspproproglugluproleuthrserglnalaser275280285gtgcccccccatcagcttcggctaggcagcctccatcctcacacccct912valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tatcacatccgcgtggcatgcaccagcagccagggcccctcatcctgg960tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320acccactggcttcctgtggagacgccggagggagtgcccctgggcccc1008thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335cctgagaacattagtgctacgcggaatgggagccaggccttcgtgcat1056progluasnileseralathrargasnglyserglnalaphevalhis340345350tggcaagagccccgggcgcccctgcagggtaccctgttagggtaccgg1104trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365ctggcgtatcaaggccaggacaccccagaggtgctaatggacataggg1152leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380ctaaggcaagaggtgaccctggagctgcagggggacgggtctgtgtcc1200leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400aatctgacagtgtgtgtggcagcctacactgctgctggggatggaccc1248asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415tggagcctcccagtacccctggaggcctggcgcccagggcaagcacag1296trpserleuprovalproleuglualatrpargproglyglnalagln420425430ccagtccaccagctggtgaaggaaccttcaactcctgccttctcgtgg1344provalhisglnleuvallysgluproserthrproalaphesertrp435440445ccctggtggtatgtactgctaggagcagtcgtggccgctgcctgtgtc1392protrptrptyrvalleuleuglyalavalvalalaalaalacysval450455460ctcatcttggctctcttccttgtccaccggcgaaagaaggagacccgt1440leuileleualaleupheleuvalhisargarglyslysgluthrarg465470475480tatggagaagtgtttgaaccaacagtggaaagaggtgaactggtagtc1488tyrglygluvalphegluprothrvalgluargglygluleuvalval485490495aggtaccgcgtgcgcaagtcctacagtcgtcggaccactgaagctacc1536argtyrargvalarglyssertyrserargargthrthrglualathr500505510ttgaacagcctgggcatcagtgaagagctgaaggagaagctgcgggat1584leuasnserleuglyileserglugluleulysglulysleuargasp515520525gtgatggtggaccggcacaaggtggccctggggaagactctgggagag1632valmetvalasparghislysvalalaleuglylysthrleuglyglu530535540ggagagtttggagctgtgatggaaggccagctcaaccaggacgactcc1680glyglupheglyalavalmetgluglyglnleuasnglnaspaspser545550555560atcctcaaggtggctgtgaagacgatgaagattgccatctgcacgagg1728ileleulysvalalavallysthrmetlysilealailecysthrarg565570575tcagagctggaggatttcctgagtgaagcggtctgcatgaaggaattt1776sergluleugluasppheleuserglualavalcysmetlysgluphe580585590gaccatcccaacgtcatgaggctcatcggtgtctgtttccagggttct1824asphisproasnvalmetargleuileglyvalcyspheglnglyser595600605gaacgagagagcttcccagcacctgtggtcatcttacctttcatgaaa1872gluarggluserpheproalaprovalvalileleuprophemetlys610615620catggagacctacacagcttcctcctctattcccggctcggggaccag1920hisglyaspleuhisserpheleuleutyrserargleuglyaspgln625630635640ccagtgtacctgcccactcagatgctagtgaagttcatggcagacatc1968provaltyrleuprothrglnmetleuvallysphemetalaaspile645650655gccagtggcatggagtatctgagtaccaagagattcatacaccgggac2016alaserglymetglutyrleuserthrlysargpheilehisargasp660665670ctggcggccaggaactgcatgctgaatgagaacatgtccgtgtgtgtg2064leualaalaargasncysmetleuasngluasnmetservalcysval675680685gcggacttcgggctctccaagaagatctacaatggggactactaccgc2112alaasppheglyleuserlyslysiletyrasnglyasptyrtyrarg690695700cagggacgtatcgccaagatgccagtcaagtggattgccattgagagt2160glnglyargilealalysmetprovallystrpilealailegluser705710715720ctagctgaccgtgtctacaccagcaagagcgatgtgtggtccttcggg2208leualaaspargvaltyrthrserlysseraspvaltrpserphegly725730735gtgacaatgtgggagattgccacaagaggccaaaccccatatccgggc2256valthrmettrpgluilealathrargglyglnthrprotyrprogly740745750gtggagaacagcgagatttatgactatctgcgccagggaaatcgcctg2304valgluasnsergluiletyrasptyrleuargglnglyasnargleu755760765aagcagcctgcggactgtctggatggactgtatgccttgatgtcgcgg2352lysglnproalaaspcysleuaspglyleutyralaleumetserarg770775780tgctgggagctaaatccccaggaccggccaagttttacagagctgcgg2400cystrpgluleuasnproglnaspargproserphethrgluleuarg785790795800gaagatttggagaacacactgaaggccttgcctcctgcccaggagcct2448gluaspleugluasnthrleulysalaleuproproalaglnglupro805810815gacgaaatcctctatgtcaacatggatgagggtggaggttatcctgaa2496aspgluileleutyrvalasnmetaspgluglyglyglytyrproglu820825830ccccctggagctgcaggaggagctgaccccccaacccagccagaccct2544proproglyalaalaglyglyalaaspproprothrglnproasppro835840845aaggattcctgtagctgcctcactgcggctgaggtccatcctgctgga2592lysaspsercyssercysleuthralaalagluvalhisproalagly850855860cgctatgtcctctgcccttccacaacccctagccccgctcagcctgct2640argtyrvalleucysproserthrthrproserproalaglnproala865870875880gataggggctccccagcagccccagggcaggaggatggtgcctga2685aspargglyserproalaalaproglyglngluaspglyala885890<210>27<211>894<212>prt<213>homosapiens<400>27metalatrpargcysproargmetglyargvalproleualatrpcys151015leualaleucysglytrpalacysmetalaproargglythrglnala202530glugluserprophevalglyasnproglyasnilethrglyalaarg354045glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560progluvalhistrpleuargaspglyglnileleugluleualaasp65707580serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095ilevalvalserglnleuargilethrserleuglnleuseraspthr100105110glyglntyrglncysleuvalpheleuglyhisglnthrphevalser115120125glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175alavalproleualathralaproglyhisglyproglnargserleu180185190hisvalproglyleuasnlysthrserserphesercysglualahis195200205asnalalysglyvalthrthrserargthralathrilethrvalleu210215220proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255hiscysthrleuglnalavalleuseraspaspglymetglyilegln260265270alaglygluproaspproproglugluproleuthrserglnalaser275280285valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335progluasnileseralathrargasnglyserglnalaphevalhis340345350trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415trpserleuprovalproleuglualatrpargproglyglnalagln420425430provalhisglnleuvallysgluproserthrproalaphesertrp435440445protrptrptyrvalleuleuglyalavalvalalaalaalacysval450455460leuileleualaleupheleuvalhisargarglyslysgluthrarg465470475480tyrglygluvalphegluprothrvalgluargglygluleuvalval485490495argtyrargvalarglyssertyrserargargthrthrglualathr500505510leuasnserleuglyileserglugluleulysglulysleuargasp515520525valmetvalasparghislysvalalaleuglylysthrleuglyglu530535540glyglupheglyalavalmetgluglyglnleuasnglnaspaspser545550555560ileleulysvalalavallysthrmetlysilealailecysthrarg565570575sergluleugluasppheleuserglualavalcysmetlysgluphe580585590asphisproasnvalmetargleuileglyvalcyspheglnglyser595600605gluarggluserpheproalaprovalvalileleuprophemetlys610615620hisglyaspleuhisserpheleuleutyrserargleuglyaspgln625630635640provaltyrleuprothrglnmetleuvallysphemetalaaspile645650655alaserglymetglutyrleuserthrlysargpheilehisargasp660665670leualaalaargasncysmetleuasngluasnmetservalcysval675680685alaasppheglyleuserlyslysiletyrasnglyasptyrtyrarg690695700glnglyargilealalysmetprovallystrpilealailegluser705710715720leualaaspargvaltyrthrserlysseraspvaltrpserphegly725730735valthrmettrpgluilealathrargglyglnthrprotyrprogly740745750valgluasnsergluiletyrasptyrleuargglnglyasnargleu755760765lysglnproalaaspcysleuaspglyleutyralaleumetserarg770775780cystrpgluleuasnproglnaspargproserphethrgluleuarg785790795800gluaspleugluasnthrleulysalaleuproproalaglnglupro805810815aspgluileleutyrvalasnmetaspgluglyglyglytyrproglu820825830proproglyalaalaglyglyalaaspproprothrglnproasppro835840845lysaspsercyssercysleuthralaalagluvalhisproalagly850855860argtyrvalleucysproserthrthrproserproalaglnproala865870875880aspargglyserproalaalaproglyglngluaspglyala885890<210>28<211>2034<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:haxl-hfc<220><221>cds<222>(1)..(2034)<400>28agatctacgcgtgccaccatggcgtggcggtgccccaggatgggcagg48argserthrargalathrmetalatrpargcysproargmetglyarg151015gtcccgctggcctggtgcttggcgctgtgcggctgggcgtgcatggcc96valproleualatrpcysleualaleucysglytrpalacysmetala202530cccaggggcacgcaggctgaagaaagtcccttcgtgggcaacccaggg144proargglythrglnalaglugluserprophevalglyasnprogly354045aatatcacaggtgcccggggactcacgggcacccttcggtgtcagctc192asnilethrglyalaargglyleuthrglythrleuargcysglnleu505560caggttcagggagagccccccgaggtacattggcttcgggatggacag240glnvalglnglygluproprogluvalhistrpleuargaspglygln65707580atcctggagctcgcggacagcacccagacccaggtgcccctgggtgag288ileleugluleualaaspserthrglnthrglnvalproleuglyglu859095gatgaacaggatgactggatagtggtcagccagctcagaatcacctcc336aspgluglnaspasptrpilevalvalserglnleuargilethrser100105110ctgcagctttccgacacgggacagtaccagtgtttggtgtttctggga384leuglnleuseraspthrglyglntyrglncysleuvalpheleugly115120125catcagaccttcgtgtcccagcctggctatgttgggctggagggcttg432hisglnthrphevalserglnproglytyrvalglyleugluglyleu130135140ccttacttcctggaggagcccgaagacaggactgtggccgccaacacc480protyrpheleuglugluprogluaspargthrvalalaalaasnthr145150155160cccttcaacctgagctgccaagctcagggacccccagagcccgtggac528propheasnleusercysglnalaglnglyproprogluprovalasp165170175ctactctggctccaggatgctgtccccctggccacggctccaggtcac576leuleutrpleuglnaspalavalproleualathralaproglyhis180185190ggcccccagcgcagcctgcatgttccagggctgaacaagacatcctct624glyproglnargserleuhisvalproglyleuasnlysthrserser195200205ttctcctgcgaagcccataacgccaagggggtcaccacatcccgcaca672phesercysglualahisasnalalysglyvalthrthrserargthr210215220gccaccatcacagtgctcccccagcagccccgtaacctccacctggtc720alathrilethrvalleuproglnglnproargasnleuhisleuval225230235240tcccgccaacccacggagctggaggtggcttggactccaggcctgagc768serargglnprothrgluleugluvalalatrpthrproglyleuser245250255ggcatctaccccctgacccactgcaccctgcaggctgtgctgtcagac816glyiletyrproleuthrhiscysthrleuglnalavalleuserasp260265270gatgggatgggcatccaggcgggagaaccagaccccccagaggagccc864aspglymetglyileglnalaglygluproaspproprogluglupro275280285ctcacctcgcaagcatccgtgcccccccatcagcttcggctaggcagc912leuthrserglnalaservalproprohisglnleuargleuglyser290295300ctccatcctcacaccccttatcacatccgcgtggcatgcaccagcagc960leuhisprohisthrprotyrhisileargvalalacysthrserser305310315320cagggcccctcatcctggacccactggcttcctgtggagacgccggag1008glnglyprosersertrpthrhistrpleuprovalgluthrproglu325330335ggagtgcccctgggcccccctgagaacattagtgctacgcggaatggg1056glyvalproleuglyproprogluasnileseralathrargasngly340345350agccaggccttcgtgcattggcaagagccccgggcgcccctgcagggt1104serglnalaphevalhistrpglngluproargalaproleuglngly355360365accctgttagggtaccggctggcgtatcaaggccaggacaccccagag1152thrleuleuglytyrargleualatyrglnglyglnaspthrproglu370375380gtgctaatggacatagggctaaggcaagaggtgaccctggagctgcag1200valleumetaspileglyleuargglngluvalthrleugluleugln385390395400ggggacgggtctgtgtccaatctgacagtgtgtgtggcagcctacact1248glyaspglyservalserasnleuthrvalcysvalalaalatyrthr405410415gctgctggggatggaccctggagcctcccagtacccctggaggcctgg1296alaalaglyaspglyprotrpserleuprovalproleuglualatrp420425430cgcccagggcaagcacagccagtccaccagctggtggtcgacgaattc1344argproglyglnalaglnprovalhisglnleuvalvalaspgluphe435440445gacaaaactcacacatgcccaccgtgcccagcacctgaactcctgggg1392asplysthrhisthrcysproprocysproalaprogluleuleugly450455460ggaccgtcagtcttcctcttccccccaaaacccaaggacaccctcatg1440glyproservalpheleupheproprolysprolysaspthrleumet465470475480atctcccggacccctgaggtcacatgcgtggtggtggacgtgagccac1488ileserargthrprogluvalthrcysvalvalvalaspvalserhis485490495gaagaccctgaggtcaagttcaactggtacgtggacggcgtggaggtg1536gluaspprogluvallyspheasntrptyrvalaspglyvalgluval500505510cataatgccaagacaaagccgcgggaggagcagtacaacagcacgtac1584hisasnalalysthrlysproargglugluglntyrasnserthrtyr515520525cgtgtggtcagcgtcctcaccgtcctgcaccaggactggctgaatggc1632argvalvalservalleuthrvalleuhisglnasptrpleuasngly530535540aaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatc1680lysglutyrlyscyslysvalserasnlysalaleuproalaproile545550555560gagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtg1728glulysthrileserlysalalysglyglnproarggluproglnval565570575tacaccctgcccccatcccgggatgagctgaccaagaaccaggtcagc1776tyrthrleuproproserargaspgluleuthrlysasnglnvalser580585590ctgacctgcctggtcaaaggcttctatcccagcgacatcgccgtggag1824leuthrcysleuvallysglyphetyrproseraspilealavalglu595600605tgggagagcaatgggcagccggagaacaactacaagaccacgcctccc1872trpgluserasnglyglnprogluasnasntyrlysthrthrpropro610615620gtgctggactccgacggctccttcttcctctacagcaagctcaccgtg1920valleuaspseraspglyserphepheleutyrserlysleuthrval625630635640gacaagagcaggtggcagcaggggaacgtcttctcatgctccgtgatg1968asplysserargtrpglnglnglyasnvalphesercysservalmet645650655catgaggctctgcacaaccactacacgcagaagagcctctccctgtct2016hisglualaleuhisasnhistyrthrglnlysserleuserleuser660665670ccgggcaaatgaggatcc2034proglylysglyser675<210>29<211>675<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>29argserthrargalathrmetalatrpargcysproargmetglyarg151015valproleualatrpcysleualaleucysglytrpalacysmetala202530proargglythrglnalaglugluserprophevalglyasnprogly354045asnilethrglyalaargglyleuthrglythrleuargcysglnleu505560glnvalglnglygluproprogluvalhistrpleuargaspglygln65707580ileleugluleualaaspserthrglnthrglnvalproleuglyglu859095aspgluglnaspasptrpilevalvalserglnleuargilethrser100105110leuglnleuseraspthrglyglntyrglncysleuvalpheleugly115120125hisglnthrphevalserglnproglytyrvalglyleugluglyleu130135140protyrpheleuglugluprogluaspargthrvalalaalaasnthr145150155160propheasnleusercysglnalaglnglyproprogluprovalasp165170175leuleutrpleuglnaspalavalproleualathralaproglyhis180185190glyproglnargserleuhisvalproglyleuasnlysthrserser195200205phesercysglualahisasnalalysglyvalthrthrserargthr210215220alathrilethrvalleuproglnglnproargasnleuhisleuval225230235240serargglnprothrgluleugluvalalatrpthrproglyleuser245250255glyiletyrproleuthrhiscysthrleuglnalavalleuserasp260265270aspglymetglyileglnalaglygluproaspproprogluglupro275280285leuthrserglnalaservalproprohisglnleuargleuglyser290295300leuhisprohisthrprotyrhisileargvalalacysthrserser305310315320glnglyprosersertrpthrhistrpleuprovalgluthrproglu325330335glyvalproleuglyproprogluasnileseralathrargasngly340345350serglnalaphevalhistrpglngluproargalaproleuglngly355360365thrleuleuglytyrargleualatyrglnglyglnaspthrproglu370375380valleumetaspileglyleuargglngluvalthrleugluleugln385390395400glyaspglyservalserasnleuthrvalcysvalalaalatyrthr405410415alaalaglyaspglyprotrpserleuprovalproleuglualatrp420425430argproglyglnalaglnprovalhisglnleuvalvalaspgluphe435440445asplysthrhisthrcysproprocysproalaprogluleuleugly450455460glyproservalpheleupheproprolysprolysaspthrleumet465470475480ileserargthrprogluvalthrcysvalvalvalaspvalserhis485490495gluaspprogluvallyspheasntrptyrvalaspglyvalgluval500505510hisasnalalysthrlysproargglugluglntyrasnserthrtyr515520525argvalvalservalleuthrvalleuhisglnasptrpleuasngly530535540lysglutyrlyscyslysvalserasnlysalaleuproalaproile545550555560glulysthrileserlysalalysglyglnproarggluproglnval565570575tyrthrleuproproserargaspgluleuthrlysasnglnvalser580585590leuthrcysleuvallysglyphetyrproseraspilealavalglu595600605trpgluserasnglyglnprogluasnasntyrlysthrthrpropro610615620valleuaspseraspglyserphepheleutyrserlysleuthrval625630635640asplysserargtrpglnglnglyasnvalphesercysservalmet645650655hisglualaleuhisasnhistyrthrglnlysserleuserleuser660665670proglylys675<210>30<211>19<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer14<400>30gctgggcagagccggtggc19<210>31<211>23<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer15<400>31gtgcttagcttggatcctgagag23<210>32<211>2685<212>dna<213>macacafascicularis<220><221>cds<222>(1)..(2685)<400>32atggcgtggcggtgccccaggatgggcagggtcccgctggcctggtgc48metalatrpargcysproargmetglyargvalproleualatrpcys151015ttggcgctgtgcggctgggtgtgcatggcccccaggggcacacaggct96leualaleucysglytrpvalcysmetalaproargglythrglnala202530gaagaaagtcctttcgtgggtaacccagggaatatcacaggtgcccgg144glugluserprophevalglyasnproglyasnilethrglyalaarg354045ggactcacgggcacccttcggtgtcagctccaggttcagggagagccc192glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560cccgaggtacactggcttcgggacggacagatcctggagctcgcggac240progluvalhistrpleuargaspglyglnileleugluleualaasp65707580agcacccagacccaggtgcccctgggtgaagatgagcaggatgactgg288serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095atagtggtcagccagctcagaatcgcctccctacagctttccgacgcg336ilevalvalserglnleuargilealaserleuglnleuseraspala100105110ggacagtaccagtgtttggtgtttctgggacatcagaacttcgtgtcc384glyglntyrglncysleuvalpheleuglyhisglnasnphevalser115120125cagcctggctacgtagggctggagggcttgccttacttcctggaggag432glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140cctgaggacaggactgtggccgccaacacccccttcaacctgagctgc480progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160caagcccagggacccccagagcccgtggacctactctggctccaggat528glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175gctgtccccctggccacagctccaggtcatggtccccagcgcaacctg576alavalproleualathralaproglyhisglyproglnargasnleu180185190catgttccagggctgaacaagacatcctctttctcctgcgaagcccat624hisvalproglyleuasnlysthrserserphesercysglualahis195200205aacgccaagggagtcaccacatcccgcacggccaccatcacagtgctc672asnalalysglyvalthrthrserargthralathrilethrvalleu210215220ccccagcagccccgtaacctccatctggtctcccgccaacccacggag720proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240ctggaggtggcttggactccaggcctgagcggcatctaccccctgacc768leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255cactgcaccctgcaggctatgctgtcagacaatgaggtgggcatccag816hiscysthrleuglnalametleuseraspasngluvalglyilegln260265270gcgggagaaccagaccccccagaggagcccctcaccttgcaagcatct864alaglygluproaspproproglugluproleuthrleuglnalaser275280285gtgcccccccaccagcttcggctgggcagcctccatcctcacacccct912valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tatcacatccgtgtggcatgcaccagcagccagggcccctcatcctgg960tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320acacactggcttcctgtggagacgccggagggagtgcccctgggcccc1008thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335cctgagaacattagtgccacgcggaatgggagccaggccttcgtgcat1056progluasnileseralathrargasnglyserglnalaphevalhis340345350tggcaggagccccgggcgcccctgcagggtaccctgttagggtaccgg1104trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365ctggcgtatcaaggccaggacaccccagaggtgctaatggacataggg1152leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380ctaaggcaagaggtgaccctggagctgcagggggacgggtctgtgtcc1200leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400aatctgacagtgtgtgtggcagcctacactgctgctggggatggaccc1248asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415tggagcctcccagtacccctggaggcctggcgcccagggcaagcacag1296trpserleuprovalproleuglualatrpargproglyglnalagln420425430ccagtccaccagctggtgaaggaaacttcagctcctgccttctcgtgg1344provalhisglnleuvallysgluthrseralaproalaphesertrp435440445ccctggtggtatatactgctaggagcagtcgtggccgctgcctgtgtc1392protrptrptyrileleuleuglyalavalvalalaalaalacysval450455460ctcatcttggctctcttccttgtccaccggcgaaagaaggagacccgt1440leuileleualaleupheleuvalhisargarglyslysgluthrarg465470475480tatggagaagtgttcgagccaacagtggaaagaggtgaactggtagtc1488tyrglygluvalphegluprothrvalgluargglygluleuvalval485490495aggtaccgcgtgcgcaagtcctacagtcgccggaccactgaagctacc1536argtyrargvalarglyssertyrserargargthrthrglualathr500505510ttgaacagcctgggcatcagtgaagagctgaaggagaagctgcgggat1584leuasnserleuglyileserglugluleulysglulysleuargasp515520525gtgatggtggaccggcacaaggtggccctggggaagactctgggagaa1632valmetvalasparghislysvalalaleuglylysthrleuglyglu530535540ggagagtttggagccgtgatggaaggccagctcaaccaggacgactcc1680glyglupheglyalavalmetgluglyglnleuasnglnaspaspser545550555560atcctcaaggtggctgtgaagacaatgaagattgccatctgcacaagg1728ileleulysvalalavallysthrmetlysilealailecysthrarg565570575tcagagctggaggatttcctgagtgaagcagtctgcatgaaggaattc1776sergluleugluasppheleuserglualavalcysmetlysgluphe580585590gaccatcccaatgtcatgaggctcatcggtgtctgtttccagggttct1824asphisproasnvalmetargleuileglyvalcyspheglnglyser595600605gaacgagagagctttccagcacctgtggtcatcttacctttcatgaag1872gluarggluserpheproalaprovalvalileleuprophemetlys610615620catggagacctacacagcttcctcctctattcccggcttggggaccag1920hisglyaspleuhisserpheleuleutyrserargleuglyaspgln625630635640ccagtgtacctgcccactcagatgctagtgaagttcatggcggacatc1968provaltyrleuprothrglnmetleuvallysphemetalaaspile645650655gccagtggcatggaatatctgagtaccaagagattcatacaccgggac2016alaserglymetglutyrleuserthrlysargpheilehisargasp660665670ctggcggccaggaactgcatgctgaatgagaacatgtccgtgtgtgtg2064leualaalaargasncysmetleuasngluasnmetservalcysval675680685gcggacttcgggctctccaagaagatctacaacggggactactaccgc2112alaasppheglyleuserlyslysiletyrasnglyasptyrtyrarg690695700cagggacgtatcgccaagatgccagtcaagtggattgccattgagagt2160glnglyargilealalysmetprovallystrpilealailegluser705710715720ctagctgaccgtgtctacacgagcaagagtgatgtgtggtccttcggg2208leualaaspargvaltyrthrserlysseraspvaltrpserphegly725730735gtgacaatgtgggagattgccacaagaggccaaaccccatatccaggc2256valthrmettrpgluilealathrargglyglnthrprotyrprogly740745750gtggagaacagcgagatttatgactatctgcgccagggaaatcgcctg2304valgluasnsergluiletyrasptyrleuargglnglyasnargleu755760765aagcagcctgcggactgtctggatggactgtatgccttgatgtcgcgg2352lysglnproalaaspcysleuaspglyleutyralaleumetserarg770775780tgctgggagctaaatccccaggaccggccaagttttacagagctgcgg2400cystrpgluleuasnproglnaspargproserphethrgluleuarg785790795800gaagatttggagaacacactgaaggccttgcctcctgcccaggagcct2448gluaspleugluasnthrleulysalaleuproproalaglnglupro805810815gacgaaatcctctatgtcaacatggatgaaggtggaggttatcctgaa2496aspgluileleutyrvalasnmetaspgluglyglyglytyrproglu820825830cctcccggcgctgctggaggagctgaccccccaacccagctagaccct2544proproglyalaalaglyglyalaaspproprothrglnleuasppro835840845aaggattcctgtagctgcctcacttcggctgaggtccatcctgctgga2592lysaspsercyssercysleuthrseralagluvalhisproalagly850855860cgctatgtcctctgcccttccacagcccctagccccgctcagcctgct2640argtyrvalleucysproserthralaproserproalaglnproala865870875880gataggggctccccagcagccccagggcaggaggatggcgcctga2685aspargglyserproalaalaproglyglngluaspglyala885890<210>33<211>894<212>prt<213>macacafascicularis<400>33metalatrpargcysproargmetglyargvalproleualatrpcys151015leualaleucysglytrpvalcysmetalaproargglythrglnala202530glugluserprophevalglyasnproglyasnilethrglyalaarg354045glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560progluvalhistrpleuargaspglyglnileleugluleualaasp65707580serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095ilevalvalserglnleuargilealaserleuglnleuseraspala100105110glyglntyrglncysleuvalpheleuglyhisglnasnphevalser115120125glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175alavalproleualathralaproglyhisglyproglnargasnleu180185190hisvalproglyleuasnlysthrserserphesercysglualahis195200205asnalalysglyvalthrthrserargthralathrilethrvalleu210215220proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255hiscysthrleuglnalametleuseraspasngluvalglyilegln260265270alaglygluproaspproproglugluproleuthrleuglnalaser275280285valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335progluasnileseralathrargasnglyserglnalaphevalhis340345350trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415trpserleuprovalproleuglualatrpargproglyglnalagln420425430provalhisglnleuvallysgluthrseralaproalaphesertrp435440445protrptrptyrileleuleuglyalavalvalalaalaalacysval450455460leuileleualaleupheleuvalhisargarglyslysgluthrarg465470475480tyrglygluvalphegluprothrvalgluargglygluleuvalval485490495argtyrargvalarglyssertyrserargargthrthrglualathr500505510leuasnserleuglyileserglugluleulysglulysleuargasp515520525valmetvalasparghislysvalalaleuglylysthrleuglyglu530535540glyglupheglyalavalmetgluglyglnleuasnglnaspaspser545550555560ileleulysvalalavallysthrmetlysilealailecysthrarg565570575sergluleugluasppheleuserglualavalcysmetlysgluphe580585590asphisproasnvalmetargleuileglyvalcyspheglnglyser595600605gluarggluserpheproalaprovalvalileleuprophemetlys610615620hisglyaspleuhisserpheleuleutyrserargleuglyaspgln625630635640provaltyrleuprothrglnmetleuvallysphemetalaaspile645650655alaserglymetglutyrleuserthrlysargpheilehisargasp660665670leualaalaargasncysmetleuasngluasnmetservalcysval675680685alaasppheglyleuserlyslysiletyrasnglyasptyrtyrarg690695700glnglyargilealalysmetprovallystrpilealailegluser705710715720leualaaspargvaltyrthrserlysseraspvaltrpserphegly725730735valthrmettrpgluilealathrargglyglnthrprotyrprogly740745750valgluasnsergluiletyrasptyrleuargglnglyasnargleu755760765lysglnproalaaspcysleuaspglyleutyralaleumetserarg770775780cystrpgluleuasnproglnaspargproserphethrgluleuarg785790795800gluaspleugluasnthrleulysalaleuproproalaglnglupro805810815aspgluileleutyrvalasnmetaspgluglyglyglytyrproglu820825830proproglyalaalaglyglyalaaspproprothrglnleuasppro835840845lysaspsercyssercysleuthrseralagluvalhisproalagly850855860argtyrvalleucysproserthralaproserproalaglnproala865870875880aspargglyserproalaalaproglyglngluaspglyala885890<210>34<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer16<400>34tcgcgtcgaaagatctgccaccatggcgtggcgg34<210>35<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer17<400>35gagttttgtcgaattccaccagctggtggactgg34<210>36<211>2004<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:caxl-hfc<220><221>cds<222>(1)..(2004)<400>36atggcgtggcggtgccccaggatgggcagggtcccgctggcctggtgc48metalatrpargcysproargmetglyargvalproleualatrpcys151015ttggcgctgtgcggctgggtgtgcatggcccccaggggcacacaggct96leualaleucysglytrpvalcysmetalaproargglythrglnala202530gaagaaagtcctttcgtgggtaacccagggaatatcacaggtgcccgg144glugluserprophevalglyasnproglyasnilethrglyalaarg354045ggactcacgggcacccttcggtgtcagctccaggttcagggagagccc192glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560cccgaggtacactggcttcgggacggacagatcctggagctcgcggac240progluvalhistrpleuargaspglyglnileleugluleualaasp65707580agcacccagacccaggtgcccctgggtgaagatgagcaggatgactgg288serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095atagtggtcagccagctcagaatcgcctccctacagctttccgacgcg336ilevalvalserglnleuargilealaserleuglnleuseraspala100105110ggacagtaccagtgtttggtgtttctgggacatcagaacttcgtgtcc384glyglntyrglncysleuvalpheleuglyhisglnasnphevalser115120125cagcctggctacgtagggctggagggcttgccttacttcctggaggag432glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140cctgaggacaggactgtggccgccaacacccccttcaacctgagctgc480progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160caagcccagggacccccagagcccgtggacctactctggctccaggat528glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175gctgtccccctggccacagctccaggtcatggtccccagcgcaacctg576alavalproleualathralaproglyhisglyproglnargasnleu180185190catgttccagggctgaacaagacatcctctttctcctgcgaagcccat624hisvalproglyleuasnlysthrserserphesercysglualahis195200205aacgccaagggagtcaccacatcccgcacggccaccatcacagtgctc672asnalalysglyvalthrthrserargthralathrilethrvalleu210215220ccccagcagccccgtaacctccatctggtctcccgccaacccacggag720proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240ctggaggtggcttggactccaggcctgagcggcatctaccccctgacc768leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255cactgcaccctgcaggctatgctgtcagacaatgaggtgggcatccag816hiscysthrleuglnalametleuseraspasngluvalglyilegln260265270gcgggagaaccagaccccccagaggagcccctcaccttgcaagcatct864alaglygluproaspproproglugluproleuthrleuglnalaser275280285gtgcccccccaccagcttcggctgggcagcctccatcctcacacccct912valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tatcacatccgtgtggcatgcaccagcagccagggcccctcatcctgg960tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320acacactggcttcctgtggagacgccggagggagtgcccctgggcccc1008thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335cctgagaacattagtgccacgcggaatgggagccaggccttcgtgcat1056progluasnileseralathrargasnglyserglnalaphevalhis340345350tggcaggagccccgggcgcccctgcagggtaccctgttagggtaccgg1104trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365ctggcgtatcaaggccaggacaccccagaggtgctaatggacataggg1152leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380ctaaggcaagaggtgaccctggagctgcagggggacgggtctgtgtcc1200leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400aatctgacagtgtgtgtggcagcctacactgctgctggggatggaccc1248asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415tggagcctcccagtacccctggaggcctggcgcccagggcaagcacag1296trpserleuprovalproleuglualatrpargproglyglnalagln420425430ccagtccaccagctggtggaattcgacaaaactcacacatgcccaccg1344provalhisglnleuvalglupheasplysthrhisthrcyspropro435440445tgcccagcacctgaactcctggggggaccgtcagtcttcctcttcccc1392cysproalaprogluleuleuglyglyproservalpheleuphepro450455460ccaaaacccaaggacaccctcatgatctcccggacccctgaggtcaca1440prolysprolysaspthrleumetileserargthrprogluvalthr465470475480tgcgtggtggtggacgtgagccacgaagaccctgaggtcaagttcaac1488cysvalvalvalaspvalserhisgluaspprogluvallyspheasn485490495tggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgg1536trptyrvalaspglyvalgluvalhisasnalalysthrlysproarg500505510gaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtc1584glugluglntyrasnserthrtyrargvalvalservalleuthrval515520525ctgcaccaggactggctgaatggcaaggagtacaagtgcaaggtctcc1632leuhisglnasptrpleuasnglylysglutyrlyscyslysvalser530535540aacaaagccctcccagcccccatcgagaaaaccatctccaaagccaaa1680asnlysalaleuproalaproileglulysthrileserlysalalys545550555560gggcagccccgagaaccacaggtgtacaccctgcccccatcccgggat1728glyglnproarggluproglnvaltyrthrleuproproserargasp565570575gagctgaccaagaaccaggtcagcctgacctgcctggtcaaaggcttc1776gluleuthrlysasnglnvalserleuthrcysleuvallysglyphe580585590tatcccagcgacatcgccgtggagtgggagagcaatgggcagccggag1824tyrproseraspilealavalglutrpgluserasnglyglnproglu595600605aacaactacaagaccacgcctcccgtgctggactccgacggctccttc1872asnasntyrlysthrthrproprovalleuaspseraspglyserphe610615620ttcctctacagcaagctcaccgtggacaagagcaggtggcagcagggg1920pheleutyrserlysleuthrvalasplysserargtrpglnglngly625630635640aacgtcttctcatgctccgtgatgcatgaggctctgcacaaccactac1968asnvalphesercysservalmethisglualaleuhisasnhistyr645650655acgcagaagagcctctccctgtctccgggcaaatga2004thrglnlysserleuserleuserproglylys660665<210>37<211>667<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>37metalatrpargcysproargmetglyargvalproleualatrpcys151015leualaleucysglytrpvalcysmetalaproargglythrglnala202530glugluserprophevalglyasnproglyasnilethrglyalaarg354045glyleuthrglythrleuargcysglnleuglnvalglnglyglupro505560progluvalhistrpleuargaspglyglnileleugluleualaasp65707580serthrglnthrglnvalproleuglygluaspgluglnaspasptrp859095ilevalvalserglnleuargilealaserleuglnleuseraspala100105110glyglntyrglncysleuvalpheleuglyhisglnasnphevalser115120125glnproglytyrvalglyleugluglyleuprotyrpheleugluglu130135140progluaspargthrvalalaalaasnthrpropheasnleusercys145150155160glnalaglnglyproprogluprovalaspleuleutrpleuglnasp165170175alavalproleualathralaproglyhisglyproglnargasnleu180185190hisvalproglyleuasnlysthrserserphesercysglualahis195200205asnalalysglyvalthrthrserargthralathrilethrvalleu210215220proglnglnproargasnleuhisleuvalserargglnprothrglu225230235240leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255hiscysthrleuglnalametleuseraspasngluvalglyilegln260265270alaglygluproaspproproglugluproleuthrleuglnalaser275280285valproprohisglnleuargleuglyserleuhisprohisthrpro290295300tyrhisileargvalalacysthrserserglnglyprosersertrp305310315320thrhistrpleuprovalgluthrprogluglyvalproleuglypro325330335progluasnileseralathrargasnglyserglnalaphevalhis340345350trpglngluproargalaproleuglnglythrleuleuglytyrarg355360365leualatyrglnglyglnaspthrprogluvalleumetaspilegly370375380leuargglngluvalthrleugluleuglnglyaspglyservalser385390395400asnleuthrvalcysvalalaalatyrthralaalaglyaspglypro405410415trpserleuprovalproleuglualatrpargproglyglnalagln420425430provalhisglnleuvalglupheasplysthrhisthrcyspropro435440445cysproalaprogluleuleuglyglyproservalpheleuphepro450455460prolysprolysaspthrleumetileserargthrprogluvalthr465470475480cysvalvalvalaspvalserhisgluaspprogluvallyspheasn485490495trptyrvalaspglyvalgluvalhisasnalalysthrlysproarg500505510glugluglntyrasnserthrtyrargvalvalservalleuthrval515520525leuhisglnasptrpleuasnglylysglutyrlyscyslysvalser530535540asnlysalaleuproalaproileglulysthrileserlysalalys545550555560glyglnproarggluproglnvaltyrthrleuproproserargasp565570575gluleuthrlysasnglnvalserleuthrcysleuvallysglyphe580585590tyrproseraspilealavalglutrpgluserasnglyglnproglu595600605asnasntyrlysthrthrproprovalleuaspseraspglyserphe610615620pheleutyrserlysleuthrvalasplysserargtrpglnglngly625630635640asnvalphesercysservalmethisglualaleuhisasnhistyr645650655thrglnlysserleuserleuserproglylys660665<210>38<211>8<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer18<400>38cgctcgag8<210>39<211>8<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer19<400>39gagctcgc8<210>40<211>2667<212>dna<213>rattusnorvegicus<220><221>cds<222>(1)..(2667)<400>40atgggcagggtcccgctggcctggtgcgtggcgctgtgctgctggggg48metglyargvalproleualatrpcysvalalaleucyscystrpgly151015tgtgcagcccctaaggacacacagaccgaggctgacagcccattcgtg96cysalaalaprolysaspthrglnthrglualaaspserpropheval202530gggaacccagggaatatcacgggtgccagaggactcacggggaccctt144glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045cggtgtgagctccaggttcagggggagccccctgaggtgatgtggctt192argcysgluleuglnvalglnglygluproprogluvalmettrpleu505560cgagatggacagatcctagaactggctgataacacccagacccaggtg240argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580cctctgggcgaagactggcaagatgaatggaaagtcgtcagtcagctc288proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095agaatctcagccctgcaactttcagatgcaggagagtaccagtgtatg336argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110gtgcacctggaaggacggacctttgtgtctcagccgggctttgtagga384valhisleugluglyargthrphevalserglnproglyphevalgly115120125ctggaaggtctcccgtacttcctggaggaacctgaagacaaagctgtg432leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140cctgccaacacccccttcaacctaagctgccaggcccagggacccccg480proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gaacccgtgaccctgctctggcttcaagatgctgtccctctggcccca528gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175gtcgcaggatacagctttcagcacagtttgcaagctccaggcctgaac576valalaglytyrserpheglnhisserleuglnalaproglyleuasn180185190aagacatcttctttctcatgtgaagcccacaatgccaagggagtcacc624lysthrserserphesercysglualahisasnalalysglyvalthr195200205acctcccgcacagctaccatcacagtgctcccacagagacctcacaat672thrserargthralathrilethrvalleuproglnargprohisasn210215220ctccacgtggtttccagacatcccacggagctagaggtagcttggatc720leuhisvalvalserarghisprothrgluleugluvalalatrpile225230235240cctaccctgagtggcatctacccgctcacccactgcaccctgcaggct768prothrleuserglyiletyrproleuthrhiscysthrleuglnala245250255gtgctgtcaaacgatggggtgggcgtctggctgggaaagtcagatcct816valleuserasnaspglyvalglyvaltrpleuglylysserasppro260265270cctgaagaacccctcaccgtgcaagtatcagtgcccccccaccagctt864proglugluproleuthrvalglnvalservalproprohisglnleu275280285cggctggaaaagctccttcctcacaccccatatcacatccgggtatcc912argleuglulysleuleuprohisthrprotyrhisileargvalser290295300tgcactagcagccagggcccctcaccttggacccactggcttcctgtg960cysthrserserglnglyproserprotrpthrhistrpleuproval305310315320gagaccacggagggagtgcccttgggtccccctgagaacgttagcgcc1008gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335atgcggaatgggagccaggccctcgtgcgttggcaggagccaagggag1056metargasnglyserglnalaleuvalargtrpglngluproargglu340345350cccttgcagggcaccctgttagggtaccggctggcatatcgaggccag1104proleuglnglythrleuleuglytyrargleualatyrargglygln355360365gacacccccgaggtacttatggatatagggctaactcgagaggtgacc1152aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380ttggaacttcggggggacaggcctgtggctaacctgactgtgtctgtg1200leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400gcagcctatacctcagctggggatgggccctggagccttcctgtgccc1248alaalatyrthrseralaglyaspglyprotrpserleuprovalpro405410415ctagagccctggcgcccagggcaaggacagccactccaccatctggtg1296leugluprotrpargproglyglnglyglnproleuhishisleuval420425430agtgaacccccacctcccgccttctcgtggccttggtggtatgtactg1344sergluproproproproalaphesertrpprotrptrptyrvalleu435440445ctgggagcacttgtggccgccgcctgtgtccttatcttgaccctgttc1392leuglyalaleuvalalaalaalacysvalleuileleuthrleuphe450455460cttgtccatcggaggaagaaggagacgagatatggggaggtgttcgag1440leuvalhisargarglyslysgluthrargtyrglygluvalpheglu465470475480ccaactgtggaaaggggtgaactggtagtcaggtaccgtgcccgaaag1488prothrvalgluargglygluleuvalvalargtyrargalaarglys485490495tcctacagtcgccggaccacggaagccaccttgaacagtctgggcatc1536sertyrserargargthrthrglualathrleuasnserleuglyile500505510agcgaagagctgaaggagaaactacgagacgtcatggtagatcggcat1584serglugluleulysglulysleuargaspvalmetvalasparghis515520525aaggtggccttggggaagaccctgggagagggagaatttggtgctgtg1632lysvalalaleuglylysthrleuglygluglyglupheglyalaval530535540atggagggccagctcaaccaggatgactccatcctcaaggtcgctgtg1680metgluglyglnleuasnglnaspaspserileleulysvalalaval545550555560aagaccatgaaaattgccatctgcacaagatcagagctggaggatttc1728lysthrmetlysilealailecysthrargsergluleugluaspphe565570575ctgagtgaagctgtctgcatgaaggaatttgaccaccccaacgtcatg1776leuserglualavalcysmetlysglupheasphisproasnvalmet580585590aggctcattggcgtctgtttccagggttctgaccgagagggtttccca1824argleuileglyvalcyspheglnglyserasparggluglyphepro595600605gaaccggtggtcatcttgcctttcatgaaacatggagacctccacagt1872gluprovalvalileleuprophemetlyshisglyaspleuhisser610615620ttcctcctgtactcgcggctcggggaccagccagtgttcctgcccact1920pheleuleutyrserargleuglyaspglnprovalpheleuprothr625630635640cagatgctagtgaagtttatggccgacattgccagtggcatggagtac1968glnmetleuvallysphemetalaaspilealaserglymetglutyr645650655ctcagtaccaagagattcatacaccgggacctagctgccaggaactgc2016leuserthrlysargpheilehisargaspleualaalaargasncys660665670atgctgaatgagaacatgtccgtgtgcgtggcagacttcgggctctcc2064metleuasngluasnmetservalcysvalalaasppheglyleuser675680685aagaagatctacaatggggattactaccgccaagggcgcattgccaag2112lyslysiletyrasnglyasptyrtyrargglnglyargilealalys690695700atgccagtcaagtggattgctatcgagagtctggcagatcgagtctac2160metprovallystrpilealailegluserleualaaspargvaltyr705710715720accagcaagagtgacgtgtggtccttcggtgtgacaatgtgggagatc2208thrserlysseraspvaltrpserpheglyvalthrmettrpgluile725730735gccacccgaggccaaactccctatccaggggtggagaacagtgagatt2256alathrargglyglnthrprotyrproglyvalgluasnsergluile740745750tacgactacctacgtcaaggaaatcgcctgaaacagcctctggactgt2304tyrasptyrleuargglnglyasnargleulysglnproleuaspcys755760765ctggatggcctctatgccctgatgtcccggtgctgggagctgaaccct2352leuaspglyleutyralaleumetserargcystrpgluleuasnpro770775780cgagaccggccaagttttgcagagctccgggaagacttggagaacaca2400argaspargproserphealagluleuarggluaspleugluasnthr785790795800ttgaaggctctaccccctgctcaggagcctgatgaaatcctctatgtc2448leulysalaleuproproalaglngluproaspgluileleutyrval805810815aacatggatgagggcggaagtcaccttgaaccccgtggggctgctgga2496asnmetaspgluglyglyserhisleugluproargglyalaalagly820825830ggagctgaccccccaacccaacctgatcctaaggattactgtagctgt2544glyalaaspproprothrglnproaspprolysasptyrcyssercys835840845ctcactgcagctgacgtccactcagctggacgctatgtcctttgtcct2592leuthralaalaaspvalhisseralaglyargtyrvalleucyspro850855860tctacagccccaggacccactctgtctgctgacagaggctgcccagca2640serthralaproglyprothrleuseralaaspargglycysproala865870875880cctccagggcaggaggacggagcctga2667proproglyglngluaspglyala885<210>41<211>888<212>prt<213>rattusnorvegicus<400>41metglyargvalproleualatrpcysvalalaleucyscystrpgly151015cysalaalaprolysaspthrglnthrglualaaspserpropheval202530glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045argcysgluleuglnvalglnglygluproprogluvalmettrpleu505560argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110valhisleugluglyargthrphevalserglnproglyphevalgly115120125leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175valalaglytyrserpheglnhisserleuglnalaproglyleuasn180185190lysthrserserphesercysglualahisasnalalysglyvalthr195200205thrserargthralathrilethrvalleuproglnargprohisasn210215220leuhisvalvalserarghisprothrgluleugluvalalatrpile225230235240prothrleuserglyiletyrproleuthrhiscysthrleuglnala245250255valleuserasnaspglyvalglyvaltrpleuglylysserasppro260265270proglugluproleuthrvalglnvalservalproprohisglnleu275280285argleuglulysleuleuprohisthrprotyrhisileargvalser290295300cysthrserserglnglyproserprotrpthrhistrpleuproval305310315320gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335metargasnglyserglnalaleuvalargtrpglngluproargglu340345350proleuglnglythrleuleuglytyrargleualatyrargglygln355360365aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400alaalatyrthrseralaglyaspglyprotrpserleuprovalpro405410415leugluprotrpargproglyglnglyglnproleuhishisleuval420425430sergluproproproproalaphesertrpprotrptrptyrvalleu435440445leuglyalaleuvalalaalaalacysvalleuileleuthrleuphe450455460leuvalhisargarglyslysgluthrargtyrglygluvalpheglu465470475480prothrvalgluargglygluleuvalvalargtyrargalaarglys485490495sertyrserargargthrthrglualathrleuasnserleuglyile500505510serglugluleulysglulysleuargaspvalmetvalasparghis515520525lysvalalaleuglylysthrleuglygluglyglupheglyalaval530535540metgluglyglnleuasnglnaspaspserileleulysvalalaval545550555560lysthrmetlysilealailecysthrargsergluleugluaspphe565570575leuserglualavalcysmetlysglupheasphisproasnvalmet580585590argleuileglyvalcyspheglnglyserasparggluglyphepro595600605gluprovalvalileleuprophemetlyshisglyaspleuhisser610615620pheleuleutyrserargleuglyaspglnprovalpheleuprothr625630635640glnmetleuvallysphemetalaaspilealaserglymetglutyr645650655leuserthrlysargpheilehisargaspleualaalaargasncys660665670metleuasngluasnmetservalcysvalalaasppheglyleuser675680685lyslysiletyrasnglyasptyrtyrargglnglyargilealalys690695700metprovallystrpilealailegluserleualaaspargvaltyr705710715720thrserlysseraspvaltrpserpheglyvalthrmettrpgluile725730735alathrargglyglnthrprotyrproglyvalgluasnsergluile740745750tyrasptyrleuargglnglyasnargleulysglnproleuaspcys755760765leuaspglyleutyralaleumetserargcystrpgluleuasnpro770775780argaspargproserphealagluleuarggluaspleugluasnthr785790795800leulysalaleuproproalaglngluproaspgluileleutyrval805810815asnmetaspgluglyglyserhisleugluproargglyalaalagly820825830glyalaaspproprothrglnproaspprolysasptyrcyssercys835840845leuthralaalaaspvalhisseralaglyargtyrvalleucyspro850855860serthralaproglyprothrleuseralaaspargglycysproala865870875880proproglyglngluaspglyala885<210>42<211>35<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer20<400>42tcgcgtcgaaagatctgccaccatgggcagggtcc35<210>43<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer21<400>43gagttttgtcgaattcaggccacgagaaggcggg34<210>44<211>2019<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:raxl-hfc<220><221>cds<222>(1)..(2019)<400>44atgggcagggtcccgctggcctggtgcgtggcgctgtgctgctggggg48metglyargvalproleualatrpcysvalalaleucyscystrpgly151015tgtgcagcccctaaggacacacagaccgaggctgacagcccattcgtg96cysalaalaprolysaspthrglnthrglualaaspserpropheval202530gggaacccagggaatatcacgggtgccagaggactcacggggaccctt144glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045cggtgtgagctccaggttcagggggagccccctgaggtgatgtggctt192argcysgluleuglnvalglnglygluproprogluvalmettrpleu505560cgagatggacagatcctagaactggctgataacacccagacccaggtg240argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580cctctgggcgaagactggcaagatgaatggaaagtcgtcagtcagctc288proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095agaatctcagccctgcaactttcagatgcaggagagtaccagtgtatg336argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110gtgcacctggaaggacggacctttgtgtctcagccgggctttgtagga384valhisleugluglyargthrphevalserglnproglyphevalgly115120125ctggaaggtctcccgtacttcctggaggaacctgaagacaaagctgtg432leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140cctgccaacacccccttcaacctaagctgccaggcccagggacccccg480proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gaacccgtgaccctgctctggcttcaagatgctgtccctctggcccca528gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175gtcgcaggatacagctttcagcacagtttgcaagctccaggcctgaac576valalaglytyrserpheglnhisserleuglnalaproglyleuasn180185190aagacatcttctttctcatgtgaagcccacaatgccaagggagtcacc624lysthrserserphesercysglualahisasnalalysglyvalthr195200205acctcccgcacagctaccatcacagtgctcccacagagacctcacaat672thrserargthralathrilethrvalleuproglnargprohisasn210215220ctccacgtggtttccagacatcccacggagctagaggtagcttggatc720leuhisvalvalserarghisprothrgluleugluvalalatrpile225230235240cctaccctgagtggcatctacccgctcacccactgcaccctgcaggct768prothrleuserglyiletyrproleuthrhiscysthrleuglnala245250255gtgctgtcaaacgatggggtgggcgtctggctgggaaagtcagatcct816valleuserasnaspglyvalglyvaltrpleuglylysserasppro260265270cctgaagaacccctcaccgtgcaagtatcagtgcccccccaccagctt864proglugluproleuthrvalglnvalservalproprohisglnleu275280285cggctggaaaagctccttcctcacaccccatatcacatccgggtatcc912argleuglulysleuleuprohisthrprotyrhisileargvalser290295300tgcactagcagccagggcccctcaccttggacccactggcttcctgtg960cysthrserserglnglyproserprotrpthrhistrpleuproval305310315320gagaccacggagggagtgcccttgggtccccctgagaacgttagcgcc1008gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335atgcggaatgggagccaggccctcgtgcgttggcaggagccaagggag1056metargasnglyserglnalaleuvalargtrpglngluproargglu340345350cccttgcagggcaccctgttagggtaccggctggcatatcgaggccag1104proleuglnglythrleuleuglytyrargleualatyrargglygln355360365gacacccccgaggtacttatggatatagggctaactcgagaggtgacc1152aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380ttggaacttcggggggacaggcctgtggctaacctgactgtgtctgtg1200leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400gcagcctatacctcagctggggatgggccctggagccttcctgtgccc1248alaalatyrthrseralaglyaspglyprotrpserleuprovalpro405410415ctagagccctggcgcccagggcaaggacagccactccaccatctggtg1296leugluprotrpargproglyglnglyglnproleuhishisleuval420425430agtgaacccccacctcccgccttctcgtggcctgaattcgacaaaact1344sergluproproproproalaphesertrpproglupheasplysthr435440445cacacatgcccaccgtgcccagcacctgaactcctggggggaccgtca1392histhrcysproprocysproalaprogluleuleuglyglyproser450455460gtcttcctcttccccccaaaacccaaggacaccctcatgatctcccgg1440valpheleupheproprolysprolysaspthrleumetileserarg465470475480acccctgaggtcacatgcgtggtggtggacgtgagccacgaagaccct1488thrprogluvalthrcysvalvalvalaspvalserhisgluasppro485490495gaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgcc1536gluvallyspheasntrptyrvalaspglyvalgluvalhisasnala500505510aagacaaagccgcgggaggagcagtacaacagcacgtaccgtgtggtc1584lysthrlysproargglugluglntyrasnserthrtyrargvalval515520525agcgtcctcaccgtcctgcaccaggactggctgaatggcaaggagtac1632servalleuthrvalleuhisglnasptrpleuasnglylysglutyr530535540aagtgcaaggtctccaacaaagccctcccagcccccatcgagaaaacc1680lyscyslysvalserasnlysalaleuproalaproileglulysthr545550555560atctccaaagccaaagggcagccccgagaaccacaggtgtacaccctg1728ileserlysalalysglyglnproarggluproglnvaltyrthrleu565570575cccccatcccgggatgagctgaccaagaaccaggtcagcctgacctgc1776proproserargaspgluleuthrlysasnglnvalserleuthrcys580585590ctggtcaaaggcttctatcccagcgacatcgccgtggagtgggagagc1824leuvallysglyphetyrproseraspilealavalglutrpgluser595600605aatgggcagccggagaacaactacaagaccacgcctcccgtgctggac1872asnglyglnprogluasnasntyrlysthrthrproprovalleuasp610615620tccgacggctccttcttcctctacagcaagctcaccgtggacaagagc1920seraspglyserphepheleutyrserlysleuthrvalasplysser625630635640aggtggcagcaggggaacgtcttctcatgctccgtgatgcatgaggct1968argtrpglnglnglyasnvalphesercysservalmethisgluala645650655ctgcacaaccactacacgcagaagagcctctccctgtctccgggcaaa2016leuhisasnhistyrthrglnlysserleuserleuserproglylys660665670tga2019<210>45<211>672<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>45metglyargvalproleualatrpcysvalalaleucyscystrpgly151015cysalaalaprolysaspthrglnthrglualaaspserpropheval202530glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045argcysgluleuglnvalglnglygluproprogluvalmettrpleu505560argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110valhisleugluglyargthrphevalserglnproglyphevalgly115120125leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175valalaglytyrserpheglnhisserleuglnalaproglyleuasn180185190lysthrserserphesercysglualahisasnalalysglyvalthr195200205thrserargthralathrilethrvalleuproglnargprohisasn210215220leuhisvalvalserarghisprothrgluleugluvalalatrpile225230235240prothrleuserglyiletyrproleuthrhiscysthrleuglnala245250255valleuserasnaspglyvalglyvaltrpleuglylysserasppro260265270proglugluproleuthrvalglnvalservalproprohisglnleu275280285argleuglulysleuleuprohisthrprotyrhisileargvalser290295300cysthrserserglnglyproserprotrpthrhistrpleuproval305310315320gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335metargasnglyserglnalaleuvalargtrpglngluproargglu340345350proleuglnglythrleuleuglytyrargleualatyrargglygln355360365aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400alaalatyrthrseralaglyaspglyprotrpserleuprovalpro405410415leugluprotrpargproglyglnglyglnproleuhishisleuval420425430sergluproproproproalaphesertrpproglupheasplysthr435440445histhrcysproprocysproalaprogluleuleuglyglyproser450455460valpheleupheproprolysprolysaspthrleumetileserarg465470475480thrprogluvalthrcysvalvalvalaspvalserhisgluasppro485490495gluvallyspheasntrptyrvalaspglyvalgluvalhisasnala500505510lysthrlysproargglugluglntyrasnserthrtyrargvalval515520525servalleuthrvalleuhisglnasptrpleuasnglylysglutyr530535540lyscyslysvalserasnlysalaleuproalaproileglulysthr545550555560ileserlysalalysglyglnproarggluproglnvaltyrthrleu565570575proproserargaspgluleuthrlysasnglnvalserleuthrcys580585590leuvallysglyphetyrproseraspilealavalglutrpgluser595600605asnglyglnprogluasnasntyrlysthrthrproprovalleuasp610615620seraspglyserphepheleutyrserlysleuthrvalasplysser625630635640argtrpglnglnglyasnvalphesercysservalmethisgluala645650655leuhisasnhistyrthrglnlysserleuserleuserproglylys660665670<210>46<211>2667<212>dna<213>musmusculus<220><221>cds<222>(1)..(2667)<400>46atgggcagggtcccgctggcctggtggttggcgctgtgctgctggggg48metglyargvalproleualatrptrpleualaleucyscystrpgly151015tgtgcagcccataaggacacacagaccgaggctggcagcccgtttgtg96cysalaalahislysaspthrglnthrglualaglyserpropheval202530gggaacccagggaatatcacaggtgccagaggactcacggggacactt144glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045cggtgtgagctccaggttcagggggaaccccctgaggtggtgtggctt192argcysgluleuglnvalglnglygluproprogluvalvaltrpleu505560cgagatggacagatcctagaactggctgataacacccagacccaggtg240argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580cctctgggcgaagactggcaagatgaatggaaagttgtcagtcagctc288proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095agaatctcagccctgcaactttcagatgcaggggagtaccagtgtatg336argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110gtgcatctagaaggacggacctttgtgtctcagccgggctttgtaggg384valhisleugluglyargthrphevalserglnproglyphevalgly115120125ctggaaggtctcccgtacttcctggaggagcctgaggacaaagctgtg432leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140cctgccaacacccctttcaacctaagctgccaggcccagggacccccg480proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gaacccgtgaccctactctggcttcaagatgctgtccccctggcccca528gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175gtcacaggacacagctcccagcacagtctgcaaactccaggcctgaac576valthrglyhisserserglnhisserleuglnthrproglyleuasn180185190aagacatcttctttctcatgtgaagcccacaatgccaagggagtcacc624lysthrserserphesercysglualahisasnalalysglyvalthr195200205acctcccgcacagccaccatcacagtgctcccccagaggcctcaccat672thrserargthralathrilethrvalleuproglnargprohishis210215220ctccacgtggtttccagacaacctacggagctagaggtagcttggacc720leuhisvalvalserargglnprothrgluleugluvalalatrpthr225230235240cctggcctgagtggcatctacccgctcacccactgcaacctgcaggcc768proglyleuserglyiletyrproleuthrhiscysasnleuglnala245250255gtgctgtcagacgatggggtgggtatctggctgggaaagtcagatcct816valleuseraspaspglyvalglyiletrpleuglylysserasppro260265270cctgaagaccccctcaccttgcaagtatcagtgcccccccaccagctt864progluaspproleuthrleuglnvalservalproprohisglnleu275280285cggctggaaaagctccttcctcacaccccgtatcacatccggatatcc912argleuglulysleuleuprohisthrprotyrhisileargileser290295300tgcagcagcagccagggcccctcaccttggacccactggcttcctgtg960cysserserserglnglyproserprotrpthrhistrpleuproval305310315320gagaccacagagggagtgcccttgggtccccctgagaacgttagcgcc1008gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335atgcggaatgggagccaggtcctcgtgcgttggcaggagccaagggtg1056metargasnglyserglnvalleuvalargtrpglngluproargval340345350cccctgcaaggcaccctgttagggtaccggctggcatatcgaggccag1104proleuglnglythrleuleuglytyrargleualatyrargglygln355360365gacacccccgaggtacttatggatatagggctaactcgagaggtgacc1152aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380ttggaactgcggggggacaggcctgtggctaacctgactgtgtctgtg1200leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400acagcctatacctcggctggggatgggccctggagccttcctgtgccc1248thralatyrthrseralaglyaspglyprotrpserleuprovalpro405410415ctagagccctggcgcccagggcaaggacagccactccaccatctggtg1296leugluprotrpargproglyglnglyglnproleuhishisleuval420425430agtgaacccccacctcgcgccttctcgtggccttggtggtatgtactg1344sergluproproproargalaphesertrpprotrptrptyrvalleu435440445ctgggagcacttgtggctgccgcctgcgtcctcatcttggccctgttc1392leuglyalaleuvalalaalaalacysvalleuileleualaleuphe450455460cttgtccatcggaggaagaaggagactcgatatggggaggtgtttgag1440leuvalhisargarglyslysgluthrargtyrglygluvalpheglu465470475480ccaaccgtggaaagaggtgaactggtagtcaggtaccgtgtccgaaag1488prothrvalgluargglygluleuvalvalargtyrargvalarglys485490495tcctacagccggcggaccactgaagccaccttgaacagtctgggcatc1536sertyrserargargthrthrglualathrleuasnserleuglyile500505510agtgaagagctgaaggagaaactacgagacgtcatggtagatcggcat1584serglugluleulysglulysleuargaspvalmetvalasparghis515520525aaggtggccttggggaagaccctgggagaaggagaatttggcgctgtg1632lysvalalaleuglylysthrleuglygluglyglupheglyalaval530535540atggaaggtcagctcaatcaggatgactccatcctcaaggtcgctgtg1680metgluglyglnleuasnglnaspaspserileleulysvalalaval545550555560aagaccatgaaaattgccatctgcacaagatcagagctggaggatttc1728lysthrmetlysilealailecysthrargsergluleugluaspphe565570575ctgagtgaagctgtctgcatgaaggaatttgaccaccccaacgtcatg1776leuserglualavalcysmetlysglupheasphisproasnvalmet580585590aggctcattggcgtctgttttcagggctctgacagagagggtttccca1824argleuileglyvalcyspheglnglyserasparggluglyphepro595600605gaacctgtggtcatcttgcctttcatgaaacacggagacctacacagt1872gluprovalvalileleuprophemetlyshisglyaspleuhisser610615620ttcctcctgtactcccggctcggggaccagccagtgttcctgcccact1920pheleuleutyrserargleuglyaspglnprovalpheleuprothr625630635640cagatgctagtgaagttcatggccgacattgccagtggtatggagtac1968glnmetleuvallysphemetalaaspilealaserglymetglutyr645650655ctgagtaccaagagattcatacatcgggacctggctgccaggaactgc2016leuserthrlysargpheilehisargaspleualaalaargasncys660665670atgctgaatgagaacatgtccgtgtgtgtggcagacttcgggctctcc2064metleuasngluasnmetservalcysvalalaasppheglyleuser675680685aagaagatctacaacggggattactaccgccaagggcgcattgccaag2112lyslysiletyrasnglyasptyrtyrargglnglyargilealalys690695700atgccagtcaagtggattgctattgagagtctggcagatcgggtctac2160metprovallystrpilealailegluserleualaaspargvaltyr705710715720accagcaagagcgatgtgtggtccttcggtgtgacaatgtgggagatc2208thrserlysseraspvaltrpserpheglyvalthrmettrpgluile725730735gccacccgaggccaaactccctatccaggggtggagaacagtgagatt2256alathrargglyglnthrprotyrproglyvalgluasnsergluile740745750tacgactacctgcgtcaaggaaatcggctgaaacagcctgtggactgt2304tyrasptyrleuargglnglyasnargleulysglnprovalaspcys755760765ctggacggcctgtatgccctgatgtctcggtgctgggaactgaaccct2352leuaspglyleutyralaleumetserargcystrpgluleuasnpro770775780cgagaccggccaagttttgcggagctccgggaagacttggagaacaca2400argaspargproserphealagluleuarggluaspleugluasnthr785790795800ctgaaggctctgccccctgctcaggagccagatgaaatcctctatgtc2448leulysalaleuproproalaglngluproaspgluileleutyrval805810815aacatggatgagggcggaagccaccttgaaccccgtggggctgctgga2496asnmetaspgluglyglyserhisleugluproargglyalaalagly820825830ggagctgaccccccaacccaacctgatcctaaggattcctgtagctgt2544glyalaaspproprothrglnproaspprolysaspsercyssercys835840845ctcactgcagctgacgtccactcagctggacgctatgtcctttgtcct2592leuthralaalaaspvalhisseralaglyargtyrvalleucyspro850855860tctacagccccaggacccactctgtctgctgacagaggctgcccagca2640serthralaproglyprothrleuseralaaspargglycysproala865870875880cctccagggcaggaggacggagcctga2667proproglyglngluaspglyala885<210>47<211>888<212>prt<213>musmusculus<400>47metglyargvalproleualatrptrpleualaleucyscystrpgly151015cysalaalahislysaspthrglnthrglualaglyserpropheval202530glyasnproglyasnilethrglyalaargglyleuthrglythrleu354045argcysgluleuglnvalglnglygluproprogluvalvaltrpleu505560argaspglyglnileleugluleualaaspasnthrglnthrglnval65707580proleuglygluasptrpglnaspglutrplysvalvalserglnleu859095argileseralaleuglnleuseraspalaglyglutyrglncysmet100105110valhisleugluglyargthrphevalserglnproglyphevalgly115120125leugluglyleuprotyrpheleuglugluprogluasplysalaval130135140proalaasnthrpropheasnleusercysglnalaglnglypropro145150155160gluprovalthrleuleutrpleuglnaspalavalproleualapro165170175valthrglyhisserserglnhisserleuglnthrproglyleuasn180185190lysthrserserphesercysglualahisasnalalysglyvalthr195200205thrserargthralathrilethrvalleuproglnargprohishis210215220leuhisvalvalserargglnprothrgluleugluvalalatrpthr225230235240proglyleuserglyiletyrproleuthrhiscysasnleuglnala245250255valleuseraspaspglyvalglyiletrpleuglylysserasppro260265270progluaspproleuthrleuglnvalservalproprohisglnleu275280285argleuglulysleuleuprohisthrprotyrhisileargileser290295300cysserserserglnglyproserprotrpthrhistrpleuproval305310315320gluthrthrgluglyvalproleuglyproprogluasnvalserala325330335metargasnglyserglnvalleuvalargtrpglngluproargval340345350proleuglnglythrleuleuglytyrargleualatyrargglygln355360365aspthrprogluvalleumetaspileglyleuthrarggluvalthr370375380leugluleuargglyaspargprovalalaasnleuthrvalserval385390395400thralatyrthrseralaglyaspglyprotrpserleuprovalpro405410415leugluprotrpargproglyglnglyglnproleuhishisleuval420425430sergluproproproargalaphesertrpprotrptrptyrvalleu435440445leuglyalaleuvalalaalaalacysvalleuileleualaleuphe450455460leuvalhisargarglyslysgluthrargtyrglygluvalpheglu465470475480prothrvalgluargglygluleuvalvalargtyrargvalarglys485490495sertyrserargargthrthrglualathrleuasnserleuglyile500505510serglugluleulysglulysleuargaspvalmetvalasparghis515520525lysvalalaleuglylysthrleuglygluglyglupheglyalaval530535540metgluglyglnleuasnglnaspaspserileleulysvalalaval545550555560lysthrmetlysilealailecysthrargsergluleugluaspphe565570575leuserglualavalcysmetlysglupheasphisproasnvalmet580585590argleuileglyvalcyspheglnglyserasparggluglyphepro595600605gluprovalvalileleuprophemetlyshisglyaspleuhisser610615620pheleuleutyrserargleuglyaspglnprovalpheleuprothr625630635640glnmetleuvallysphemetalaaspilealaserglymetglutyr645650655leuserthrlysargpheilehisargaspleualaalaargasncys660665670metleuasngluasnmetservalcysvalalaasppheglyleuser675680685lyslysiletyrasnglyasptyrtyrargglnglyargilealalys690695700metprovallystrpilealailegluserleualaaspargvaltyr705710715720thrserlysseraspvaltrpserpheglyvalthrmettrpgluile725730735alathrargglyglnthrprotyrproglyvalgluasnsergluile740745750tyrasptyrleuargglnglyasnargleulysglnprovalaspcys755760765leuaspglyleutyralaleumetserargcystrpgluleuasnpro770775780argaspargproserphealagluleuarggluaspleugluasnthr785790795800leulysalaleuproproalaglngluproaspgluileleutyrval805810815asnmetaspgluglyglyserhisleugluproargglyalaalagly820825830glyalaaspproprothrglnproaspprolysaspsercyssercys835840845leuthralaalaaspvalhisseralaglyargtyrvalleucyspro850855860serthralaproglyprothrleuseralaaspargglycysproala865870875880proproglyglngluaspglyala885<210>48<211>2034<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:maxl-mfc<220><221>cds<222>(1)..(2034)<400>48agatctacgcgtgccaccatgggcagggtcccgctggcctggtggttg48argserthrargalathrmetglyargvalproleualatrptrpleu151015gcgctgtgctgctgggggtgtgcagcccataaggacacacagaccgag96alaleucyscystrpglycysalaalahislysaspthrglnthrglu202530gctggcagcccgtttgtggggaacccagggaatatcacaggtgccaga144alaglyserprophevalglyasnproglyasnilethrglyalaarg354045ggactcacggggacacttcggtgtgagctccaggttcagggggaaccc192glyleuthrglythrleuargcysgluleuglnvalglnglyglupro505560cctgaggtggtgtggcttcgagatggacagatcctagaactggctgat240progluvalvaltrpleuargaspglyglnileleugluleualaasp65707580aacacccagacccaggtgcctctgggcgaagactggcaagatgaatgg288asnthrglnthrglnvalproleuglygluasptrpglnaspglutrp859095aaagttgtcagtcagctcagaatctcagccctgcaactttcagatgca336lysvalvalserglnleuargileseralaleuglnleuseraspala100105110ggggagtaccagtgtatggtgcatctagaaggacggacctttgtgtct384glyglutyrglncysmetvalhisleugluglyargthrphevalser115120125cagccgggctttgtagggctggaaggtctcccgtacttcctggaggag432glnproglyphevalglyleugluglyleuprotyrpheleugluglu130135140cctgaggacaaagctgtgcctgccaacacccctttcaacctaagctgc480progluasplysalavalproalaasnthrpropheasnleusercys145150155160caggcccagggacccccggaacccgtgaccctactctggcttcaagat528glnalaglnglyproprogluprovalthrleuleutrpleuglnasp165170175gctgtccccctggccccagtcacaggacacagctcccagcacagtctg576alavalproleualaprovalthrglyhisserserglnhisserleu180185190caaactccaggcctgaacaagacatcttctttctcatgtgaagcccac624glnthrproglyleuasnlysthrserserphesercysglualahis195200205aatgccaagggagtcaccacctcccgcacagccaccatcacagtgctc672asnalalysglyvalthrthrserargthralathrilethrvalleu210215220ccccagaggcctcaccatctccacgtggtttccagacaacctacggag720proglnargprohishisleuhisvalvalserargglnprothrglu225230235240ctagaggtagcttggacccctggcctgagtggcatctacccgctcacc768leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255cactgcaacctgcaggccgtgctgtcagacgatggggtgggtatctgg816hiscysasnleuglnalavalleuseraspaspglyvalglyiletrp260265270ctgggaaagtcagatcctcctgaagaccccctcaccttgcaagtatca864leuglylysseraspproprogluaspproleuthrleuglnvalser275280285gtgcccccccaccagcttcggctggaaaagctccttcctcacaccccg912valproprohisglnleuargleuglulysleuleuprohisthrpro290295300tatcacatccggatatcctgcagcagcagccagggcccctcaccttgg960tyrhisileargilesercysserserserglnglyproserprotrp305310315320acccactggcttcctgtggagaccacagagggagtgcccttgggtccc1008thrhistrpleuprovalgluthrthrgluglyvalproleuglypro325330335cctgagaacgttagcgccatgcggaatgggagccaggtcctcgtgcgt1056progluasnvalseralametargasnglyserglnvalleuvalarg340345350tggcaggagccaagggtgcccctgcaaggcaccctgttagggtaccgg1104trpglngluproargvalproleuglnglythrleuleuglytyrarg355360365ctggcatatcgaggccaggacacccccgaggtacttatggatataggg1152leualatyrargglyglnaspthrprogluvalleumetaspilegly370375380ctaactcgagaggtgaccttggaactgcggggggacaggcctgtggct1200leuthrarggluvalthrleugluleuargglyaspargprovalala385390395400aacctgactgtgtctgtgacagcctatacctcggctggggatgggccc1248asnleuthrvalservalthralatyrthrseralaglyaspglypro405410415tggagccttcctgtgcccctagagccctggcgcccagggcaaggacag1296trpserleuprovalproleugluprotrpargproglyglnglygln420425430ccactccaccatctggtgagtgaacccccacctcgcgccttctcgtgg1344proleuhishisleuvalsergluproproproargalaphesertrp435440445cctgtcgacgaattcggttgtaagccttgcatatgtacagtcccagaa1392provalaspglupheglycyslysprocysilecysthrvalproglu450455460gtatcatctgtcttcatcttccccccaaagcccaaggatgtgctcacc1440valserservalpheilepheproprolysprolysaspvalleuthr465470475480attactctgactcctaaggtcacgtgtgttgtggtagacatcagcaag1488ilethrleuthrprolysvalthrcysvalvalvalaspileserlys485490495gatgatcccgaggtccagttcagctggtttgtagatgatgtggaggtg1536aspaspprogluvalglnphesertrpphevalaspaspvalgluval500505510cacacagctcagacgcaaccccgggaggagcagttcaacagcactttc1584histhralaglnthrglnproargglugluglnpheasnserthrphe515520525cgctcagtcagtgaacttcccatcatgcaccaggactggctcaatggc1632argservalsergluleuproilemethisglnasptrpleuasngly530535540aaggagttcaaatgcagggtcaacagtgcagctttccctgcccccatc1680lysgluphelyscysargvalasnseralaalapheproalaproile545550555560gagaaaaccatctccaaaaccaaaggcagaccgaaggctccacaggtg1728glulysthrileserlysthrlysglyargprolysalaproglnval565570575tacaccattccacctcccaaggagcagatggccaaggataaagtcagt1776tyrthrileproproprolysgluglnmetalalysasplysvalser580585590ctgacctgcatgataacagacttcttccctgaagacattactgtggag1824leuthrcysmetilethraspphepheprogluaspilethrvalglu595600605tggcagtggaatgggcagccagcggagaactacaagaacactcagccc1872trpglntrpasnglyglnproalagluasntyrlysasnthrglnpro610615620atcatggacacagatggctcttacttcgtctacagcaagctcaatgtg1920ilemetaspthraspglysertyrphevaltyrserlysleuasnval625630635640cagaagagcaactgggaggcaggaaatactttcacctgctctgtgtta1968glnlysserasntrpglualaglyasnthrphethrcysservalleu645650655catgagggcctgcacaaccaccatactgagaagagcctctcccactct2016hisgluglyleuhisasnhishisthrglulysserleuserhisser660665670cctggtaaatgaggatcc2034proglylysglyser675<210>49<211>675<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>49argserthrargalathrmetglyargvalproleualatrptrpleu151015alaleucyscystrpglycysalaalahislysaspthrglnthrglu202530alaglyserprophevalglyasnproglyasnilethrglyalaarg354045glyleuthrglythrleuargcysgluleuglnvalglnglyglupro505560progluvalvaltrpleuargaspglyglnileleugluleualaasp65707580asnthrglnthrglnvalproleuglygluasptrpglnaspglutrp859095lysvalvalserglnleuargileseralaleuglnleuseraspala100105110glyglutyrglncysmetvalhisleugluglyargthrphevalser115120125glnproglyphevalglyleugluglyleuprotyrpheleugluglu130135140progluasplysalavalproalaasnthrpropheasnleusercys145150155160glnalaglnglyproprogluprovalthrleuleutrpleuglnasp165170175alavalproleualaprovalthrglyhisserserglnhisserleu180185190glnthrproglyleuasnlysthrserserphesercysglualahis195200205asnalalysglyvalthrthrserargthralathrilethrvalleu210215220proglnargprohishisleuhisvalvalserargglnprothrglu225230235240leugluvalalatrpthrproglyleuserglyiletyrproleuthr245250255hiscysasnleuglnalavalleuseraspaspglyvalglyiletrp260265270leuglylysseraspproprogluaspproleuthrleuglnvalser275280285valproprohisglnleuargleuglulysleuleuprohisthrpro290295300tyrhisileargilesercysserserserglnglyproserprotrp305310315320thrhistrpleuprovalgluthrthrgluglyvalproleuglypro325330335progluasnvalseralametargasnglyserglnvalleuvalarg340345350trpglngluproargvalproleuglnglythrleuleuglytyrarg355360365leualatyrargglyglnaspthrprogluvalleumetaspilegly370375380leuthrarggluvalthrleugluleuargglyaspargprovalala385390395400asnleuthrvalservalthralatyrthrseralaglyaspglypro405410415trpserleuprovalproleugluprotrpargproglyglnglygln420425430proleuhishisleuvalsergluproproproargalaphesertrp435440445provalaspglupheglycyslysprocysilecysthrvalproglu450455460valserservalpheilepheproprolysprolysaspvalleuthr465470475480ilethrleuthrprolysvalthrcysvalvalvalaspileserlys485490495aspaspprogluvalglnphesertrpphevalaspaspvalgluval500505510histhralaglnthrglnproargglugluglnpheasnserthrphe515520525argservalsergluleuproilemethisglnasptrpleuasngly530535540lysgluphelyscysargvalasnseralaalapheproalaproile545550555560glulysthrileserlysthrlysglyargprolysalaproglnval565570575tyrthrileproproprolysgluglnmetalalysasplysvalser580585590leuthrcysmetilethraspphepheprogluaspilethrvalglu595600605trpglntrpasnglyglnproalagluasntyrlysasnthrglnpro610615620ilemetaspthraspglysertyrphevaltyrserlysleuasnval625630635640glnlysserasntrpglualaglyasnthrphethrcysservalleu645650655hisgluglyleuhisasnhishisthrglulysserleuserhisser660665670proglylys675<210>50<211>33<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer22<400>50ctcactatagaattcgccaccatggcgtggcgg33<210>51<211>38<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence;primer23<400>51caataaacaagttggatcctcaggcaccatcctcctgc38<210>52<211>2673<212>dna<213>homosapiens<220><221>cds<222>(1)..(2673)<400>52atggcgctgaggcggagcatggggcggccggggctcccgccgctgccg48metalaleuargargsermetglyargproglyleuproproleupro151015ctgccgccgccaccgcggctcgggctgctgctggcggctctggcttct96leuproproproproargleuglyleuleuleualaalaleualaser202530ctgctgctcccggagtccgccgccgcaggtctgaagctcatgggagcc144leuleuleuprogluseralaalaalaglyleulysleumetglyala354045ccggtgaagctgacagtgtctcaggggcagccggtgaagctcaactgc192provallysleuthrvalserglnglyglnprovallysleuasncys505560agtgtggaggggatggaggagcctgacatccagtgggtgaaggatggg240servalgluglymetglugluproaspileglntrpvallysaspgly65707580gctgtggtccagaacttggaccagttgtacatcccagtcagcgagcag288alavalvalglnasnleuaspglnleutyrileprovalserglugln859095cactggatcggcttcctcagcctgaagtcagtggagcgctctgacgcc336histrpileglypheleuserleulysservalgluargseraspala100105110ggccggtactggtgccaggtggaggatgggggtgaaaccgagatctcc384glyargtyrtrpcysglnvalgluaspglyglygluthrgluileser115120125cagccagtgtggctcacggtagaaggtgtgccatttttcacagtggag432glnprovaltrpleuthrvalgluglyvalprophephethrvalglu130135140ccaaaagatctggcagtgccacccaatgcccctttccaactgtcttgt480prolysaspleualavalproproasnalapropheglnleusercys145150155160gaggctgtgggtccccctgaacctgttaccattgtctggtggagagga528glualavalglyproprogluprovalthrilevaltrptrparggly165170175actacgaagatcgggggacccgctccctctccatctgttttaaatgta576thrthrlysileglyglyproalaproserproservalleuasnval180185190acaggggtgacccagagcaccatgttttcctgtgaagctcacaaccta624thrglyvalthrglnserthrmetphesercysglualahisasnleu195200205aaaggcctggcctcttctcgcacagccactgttcaccttcaagcactg672lysglyleualaserserargthralathrvalhisleuglnalaleu210215220cctgcagcccccttcaacatcaccgtgacaaagctttccagcagcaac720proalaalapropheasnilethrvalthrlysleuserserserasn225230235240gctagtgtggcctggatgccaggtgctgatggccgagctctgctacag768alaservalalatrpmetproglyalaaspglyargalaleuleugln245250255tcctgtacagttcaggtgacacaggccccaggaggctgggaagtcctg816sercysthrvalglnvalthrglnalaproglyglytrpgluvalleu260265270gctgttgtggtccctgtgcccccctttacctgcctgctccgggacctg864alavalvalvalprovalproprophethrcysleuleuargaspleu275280285gtgcctgccaccaactacagcctcagggtgcgctgtgccaatgccttg912valproalathrasntyrserleuargvalargcysalaasnalaleu290295300gggccctctccctatgctgactgggtgccctttcagaccaagggtcta960glyproserprotyralaasptrpvalpropheglnthrlysglyleu305310315320gccccagccagcgctccccaaaacctccatgccatccgcacagattca1008alaproalaseralaproglnasnleuhisalaileargthraspser325330335ggcctcatcttggagtgggaagaagtgatccccgaggcccctttggaa1056glyleuileleuglutrpglugluvalileproglualaproleuglu340345350ggccccctgggaccctacaaactgtcctgggttcaagacaatggaacc1104glyproleuglyprotyrlysleusertrpvalglnaspasnglythr355360365caggatgagctgacagtggaggggaccagggccaatttgacaggctgg1152glnaspgluleuthrvalgluglythrargalaasnleuthrglytrp370375380gatccccaaaaggacctgatcgtacgtgtgtgcgtctccaatgcagtt1200aspproglnlysaspleuilevalargvalcysvalserasnalaval385390395400ggctgtggaccctggagtcagccactggtggtctcttctcatgaccgt1248glycysglyprotrpserglnproleuvalvalserserhisasparg405410415gcaggccagcagggccctcctcacagccgcacatcctgggtacctgtg1296alaglyglnglnglyproprohisserargthrsertrpvalproval420425430gtccttggtgtgctaacggccctggtgacggctgctgccctggccctc1344valleuglyvalleuthralaleuvalthralaalaalaleualaleu435440445atcctgcttcgaaagagacggaaagagacgcggtttgggcaagccttt1392ileleuleuarglysargarglysgluthrargpheglyglnalaphe450455460gacagtgtcatggcccggggagagccagccgttcacttccgggcagcc1440aspservalmetalaargglygluproalavalhispheargalaala465470475480cggtccttcaatcgagaaaggcccgagcgcatcgaggccacattggac1488argserpheasnarggluargprogluargileglualathrleuasp485490495agcttgggcatcagcgatgaactaaaggaaaaactggaggatgtgctc1536serleuglyileseraspgluleulysglulysleugluaspvalleu500505510atcccagagcagcagttcaccctgggccggatgttgggcaaaggagag1584ileprogluglnglnphethrleuglyargmetleuglylysglyglu515520525tttggttcagtgcgggaggcccagctgaagcaagaggatggctccttt1632pheglyservalargglualaglnleulysglngluaspglyserphe530535540gtgaaagtggctgtgaagatgctgaaagctgacatcattgcctcaagc1680vallysvalalavallysmetleulysalaaspileilealaserser545550555560gacattgaagagttcctcagggaagcagcttgcatgaaggagtttgac1728aspilegluglupheleuargglualaalacysmetlysglupheasp565570575catccacacgtggccaaacttgttggggtaagcctccggagcagggct1776hisprohisvalalalysleuvalglyvalserleuargserargala580585590aaaggccgtctccccatccccatggtcatcttgcccttcatgaagcat1824lysglyargleuproileprometvalileleuprophemetlyshis595600605ggggacctgcatgccttcctgctcgcctcccggattggggagaacccc1872glyaspleuhisalapheleuleualaserargileglygluasnpro610615620tttaacctacccctccagaccctgatccggttcatggtggacattgcc1920pheasnleuproleuglnthrleuileargphemetvalaspileala625630635640tgcggcatggagtacctgagctctcggaacttcatccaccgagacctg1968cysglymetglutyrleuserserargasnpheilehisargaspleu645650655gctgctcggaattgcatgctggcagaggacatgacagtgtgtgtggct2016alaalaargasncysmetleualagluaspmetthrvalcysvalala660665670gacttcggactctcccggaagatctacagtggggactactatcgtcaa2064asppheglyleuserarglysiletyrserglyasptyrtyrarggln675680685ggctgtgcctccaaactgcctgtcaagtggctggccctggagagcctg2112glycysalaserlysleuprovallystrpleualaleugluserleu690695700gccgacaacctgtatactgtgcagagtgacgtgtgggcgttcggggtg2160alaaspasnleutyrthrvalglnseraspvaltrpalapheglyval705710715720accatgtgggagatcatgacacgtgggcagacgccatatgctggcatc2208thrmettrpgluilemetthrargglyglnthrprotyralaglyile725730735gaaaacgctgagatttacaactacctcattggcgggaaccgcctgaaa2256gluasnalagluiletyrasntyrleuileglyglyasnargleulys740745750cagcctccggagtgtatggaggacgtgtatgatctcatgtaccagtgc2304glnproproglucysmetgluaspvaltyraspleumettyrglncys755760765tggagtgctgaccccaagcagcgcccgagctttacttgtctgcgaatg2352trpseralaaspprolysglnargproserphethrcysleuargmet770775780gaactggagaacatcttgggccagctgtctgtgctatctgccagccag2400gluleugluasnileleuglyglnleuservalleuseralasergln785790795800gaccccttatacatcaacatcgagagagctgaggagcccactgcggga2448aspproleutyrileasnilegluargalaglugluprothralagly805810815ggcagcctggagctacctggcagggatcagccctacagtggggctggg2496glyserleugluleuproglyargaspglnprotyrserglyalagly820825830gatggcagtggcatgggggcagtgggtggcactcccagtgactgtcgg2544aspglyserglymetglyalavalglyglythrproseraspcysarg835840845tacatactcacccccggagggctggctgagcagccagggcaggcagag2592tyrileleuthrproglyglyleualagluglnproglyglnalaglu850855860caccagccagagagtcccctcaatgagacacagaggcttttgctgctg2640hisglnprogluserproleuasngluthrglnargleuleuleuleu865870875880cagcaagggctactgccacacagtagctgttag2673glnglnglyleuleuprohissersercys885890<210>53<211>890<212>prt<213>homosapiens<400>53metalaleuargargsermetglyargproglyleuproproleupro151015leuproproproproargleuglyleuleuleualaalaleualaser202530leuleuleuprogluseralaalaalaglyleulysleumetglyala354045provallysleuthrvalserglnglyglnprovallysleuasncys505560servalgluglymetglugluproaspileglntrpvallysaspgly65707580alavalvalglnasnleuaspglnleutyrileprovalserglugln859095histrpileglypheleuserleulysservalgluargseraspala100105110glyargtyrtrpcysglnvalgluaspglyglygluthrgluileser115120125glnprovaltrpleuthrvalgluglyvalprophephethrvalglu130135140prolysaspleualavalproproasnalapropheglnleusercys145150155160glualavalglyproprogluprovalthrilevaltrptrparggly165170175thrthrlysileglyglyproalaproserproservalleuasnval180185190thrglyvalthrglnserthrmetphesercysglualahisasnleu195200205lysglyleualaserserargthralathrvalhisleuglnalaleu210215220proalaalapropheasnilethrvalthrlysleuserserserasn225230235240alaservalalatrpmetproglyalaaspglyargalaleuleugln245250255sercysthrvalglnvalthrglnalaproglyglytrpgluvalleu260265270alavalvalvalprovalproprophethrcysleuleuargaspleu275280285valproalathrasntyrserleuargvalargcysalaasnalaleu290295300glyproserprotyralaasptrpvalpropheglnthrlysglyleu305310315320alaproalaseralaproglnasnleuhisalaileargthraspser325330335glyleuileleuglutrpglugluvalileproglualaproleuglu340345350glyproleuglyprotyrlysleusertrpvalglnaspasnglythr355360365glnaspgluleuthrvalgluglythrargalaasnleuthrglytrp370375380aspproglnlysaspleuilevalargvalcysvalserasnalaval385390395400glycysglyprotrpserglnproleuvalvalserserhisasparg405410415alaglyglnglnglyproprohisserargthrsertrpvalproval420425430valleuglyvalleuthralaleuvalthralaalaalaleualaleu435440445ileleuleuarglysargarglysgluthrargpheglyglnalaphe450455460aspservalmetalaargglygluproalavalhispheargalaala465470475480argserpheasnarggluargprogluargileglualathrleuasp485490495serleuglyileseraspgluleulysglulysleugluaspvalleu500505510ileprogluglnglnphethrleuglyargmetleuglylysglyglu515520525pheglyservalargglualaglnleulysglngluaspglyserphe530535540vallysvalalavallysmetleulysalaaspileilealaserser545550555560aspilegluglupheleuargglualaalacysmetlysglupheasp565570575hisprohisvalalalysleuvalglyvalserleuargserargala580585590lysglyargleuproileprometvalileleuprophemetlyshis595600605glyaspleuhisalapheleuleualaserargileglygluasnpro610615620pheasnleuproleuglnthrleuileargphemetvalaspileala625630635640cysglymetglutyrleuserserargasnpheilehisargaspleu645650655alaalaargasncysmetleualagluaspmetthrvalcysvalala660665670asppheglyleuserarglysiletyrserglyasptyrtyrarggln675680685glycysalaserlysleuprovallystrpleualaleugluserleu690695700alaaspasnleutyrthrvalglnseraspvaltrpalapheglyval705710715720thrmettrpgluilemetthrargglyglnthrprotyralaglyile725730735gluasnalagluiletyrasntyrleuileglyglyasnargleulys740745750glnproproglucysmetgluaspvaltyraspleumettyrglncys755760765trpseralaaspprolysglnargproserphethrcysleuargmet770775780gluleugluasnileleuglyglnleuservalleuseralasergln785790795800aspproleutyrileasnilegluargalaglugluprothralagly805810815glyserleugluleuproglyargaspglnprotyrserglyalagly820825830aspglyserglymetglyalavalglyglythrproseraspcysarg835840845tyrileleuthrproglyglyleualagluglnproglyglnalaglu850855860hisglnprogluserproleuasngluthrglnargleuleuleuleu865870875880glnglnglyleuleuprohissersercys885890<210>54<211>35<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer24<400>54ctcactatagaattcgccaccatggcgctgaggcg35<210>55<211>34<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer25<400>55caataaacaagttggatccctaacagctactgtg34<210>56<211>3000<212>dna<213>homosapiens<220><221>cds<222>(1)..(3000)<400>56atggggccggccccgctgccgctgctgctgggcctcttcctccccgcg48metglyproalaproleuproleuleuleuglyleupheleuproala151015ctctggcgtagagctatcactgaggcaagggaagaagccaagccttac96leutrpargargalailethrglualaarggluglualalysprotyr202530ccgctattcccgggaccttttccagggagcctgcaaactgaccacaca144proleupheproglypropheproglyserleuglnthrasphisthr354045ccgctgttatcccttcctcacgccagtgggtaccagcctgccttgatg192proleuleuserleuprohisalaserglytyrglnproalaleumet505560ttttcaccaacccagcctggaagaccacatacaggaaacgtagccatt240pheserprothrglnproglyargprohisthrglyasnvalalaile65707580ccccaggtgacctctgtcgaatcaaagcccctaccgcctcttgccttc288proglnvalthrservalgluserlysproleuproproleualaphe859095aaacacacagttggacacataatactttctgaacataaaggtgtcaaa336lyshisthrvalglyhisileileleusergluhislysglyvallys100105110tttaattgctcaatcagtgtacctaatatataccaggacaccacaatt384pheasncysserileservalproasniletyrglnaspthrthrile115120125tcttggtggaaagatgggaaggaattgcttggggcacatcatgcaatt432sertrptrplysaspglylysgluleuleuglyalahishisalaile130135140acacagttttatccagatgatgaagttacagcaataatcgcttccttc480thrglnphetyrproaspaspgluvalthralaileilealaserphe145150155160agcataaccagtgtgcagcgttcagacaatgggtcgtatatctgtaag528serilethrservalglnargseraspasnglysertyrilecyslys165170175atgaaaataaacaatgaagagatcgtgtctgatcccatctacatcgaa576metlysileasnasnglugluilevalseraspproiletyrileglu180185190gtacaaggacttcctcactttactaagcagcctgagagcatgaatgtc624valglnglyleuprohisphethrlysglnproglusermetasnval195200205accagaaacacagccttcaacctcacctgtcaggctgtgggcccgcct672thrargasnthralapheasnleuthrcysglnalavalglypropro210215220gagcccgtcaacattttctgggttcaaaacagtagccgtgttaacgaa720gluprovalasnilephetrpvalglnasnserserargvalasnglu225230235240cagcctgaaaaatccccctccgtgctaactgttccaggcctgacggag768glnproglulysserproservalleuthrvalproglyleuthrglu245250255atggcggtcttcagttgtgaggcccacaatgacaaagggctgaccgtg816metalavalphesercysglualahisasnasplysglyleuthrval260265270tccaagggagtgcagatcaacatcaaagcaattccctccccaccaact864serlysglyvalglnileasnilelysalaileproserproprothr275280285gaagtcagcatccgtaacagcactgcacacagcattctgatctcctgg912gluvalserileargasnserthralahisserileleuilesertrp290295300gttcctggttttgatggatactccccgttcaggaattgcagcattcag960valproglypheaspglytyrserpropheargasncysserilegln305310315320gtcaaggaagctgatccgctgagtaatggctcagtcatgatttttaac1008vallysglualaaspproleuserasnglyservalmetilepheasn325330335acctctgccttaccacatctgtaccaaatcaagcagctgcaagccctg1056thrseralaleuprohisleutyrglnilelysglnleuglnalaleu340345350gctaattacagcattggtgtttcctgcatgaatgaaataggctggtct1104alaasntyrserileglyvalsercysmetasngluileglytrpser355360365gcagtgagcccttggattctagccagcacgactgaaggagccccatca1152alavalserprotrpileleualaserthrthrgluglyalaproser370375380gtagcacctttaaatgtcactgtgtttctgaatgaatctagtgataat1200valalaproleuasnvalthrvalpheleuasngluserseraspasn385390395400gtggacatcagatggatgaagcctccgactaagcagcaggatggagaa1248valaspileargtrpmetlysproprothrlysglnglnaspglyglu405410415ctggtgggctaccggatatcccacgtgtggcagagtgcagggatttcc1296leuvalglytyrargileserhisvaltrpglnseralaglyileser420425430aaagagctcttggaggaagttggccagaatggcagccgagctcggatc1344lysgluleuleuglugluvalglyglnasnglyserargalaargile435440445tctgttcaagtccacaatgctacgtgcacagtgaggattgcagccgtc1392servalglnvalhisasnalathrcysthrvalargilealaalaval450455460accagagggggagttgggcccttcagtgatccagtgaaaatatttatc1440thrargglyglyvalglypropheseraspprovallysilepheile465470475480cctgcacacggttgggtagattatgccccctcttcaactccggcgcct1488proalahisglytrpvalasptyralaproserserthrproalapro485490495ggcaacgcagatcctgtgctcatcatctttggctgcttttgtggattt1536glyasnalaaspprovalleuileilepheglycysphecysglyphe500505510attttgattgggttgattttatacatctccttggccatcagaaaaaga1584ileleuileglyleuileleutyrileserleualailearglysarg515520525gtccaggagacaaagtttgggaatgcattcacagaggaggattctgaa1632valglngluthrlyspheglyasnalaphethrglugluaspserglu530535540ttagtggtgaattatatagcaaagaaatccttctgtcggcgagccatt1680leuvalvalasntyrilealalyslysserphecysargargalaile545550555560gaacttaccttacatagcttgggagtcagtgaggaactacaaaataaa1728gluleuthrleuhisserleuglyvalserglugluleuglnasnlys565570575ctagaagatgttgtgattgacaggaatcttctaattcttggaaaaatt1776leugluaspvalvalileaspargasnleuleuileleuglylysile580585590ctgggtgaaggagagtttgggtctgtaatggaaggaaatcttaagcag1824leuglygluglyglupheglyservalmetgluglyasnleulysgln595600605gaagatgggacctctctgaaagtggcagtgaagaccatgaagttggac1872gluaspglythrserleulysvalalavallysthrmetlysleuasp610615620aactcttcacagcgggagatcgaggagtttctcagtgaggcagcgtgc1920asnserserglnarggluilegluglupheleuserglualaalacys625630635640atgaaagacttcagccacccaaatgtcattcgacttctaggtgtgtgt1968metlysasppheserhisproasnvalileargleuleuglyvalcys645650655atagaaatgagctctcaaggcatcccaaagcccatggtaattttaccc2016ileglumetserserglnglyileprolysprometvalileleupro660665670ttcatgaaatacggggacctgcatacttacttactttattcccgattg2064phemetlystyrglyaspleuhisthrtyrleuleutyrserargleu675680685gagacaggaccaaagcatattcctctgcagacactattgaagttcatg2112gluthrglyprolyshisileproleuglnthrleuleulysphemet690695700gtggatattgccctgggaatggagtatctgagcaacaggaattttctt2160valaspilealaleuglymetglutyrleuserasnargasnpheleu705710715720catcgagatttagctgctcgaaactgcatgttgcgagatgacatgact2208hisargaspleualaalaargasncysmetleuargaspaspmetthr725730735gtctgtgttgcggacttcggcctctctaagaagatttacagtggcgat2256valcysvalalaasppheglyleuserlyslysiletyrserglyasp740745750tattaccgccaaggccgcattgctaagatgcctgttaaatggatcgcc2304tyrtyrargglnglyargilealalysmetprovallystrpileala755760765atagaaagtcttgcagaccgagtctacacaagtaaaagtgatgtgtgg2352ilegluserleualaaspargvaltyrthrserlysseraspvaltrp770775780gcatttggcgtgaccatgtgggaaatagctacgcggggaatgactccc2400alapheglyvalthrmettrpgluilealathrargglymetthrpro785790795800tatcctggggtccagaaccatgagatgtatgactatcttctccatggc2448tyrproglyvalglnasnhisglumettyrasptyrleuleuhisgly805810815cacaggttgaagcagcccgaagactgcctggatgaactgtatgaaata2496hisargleulysglnprogluaspcysleuaspgluleutyrgluile820825830atgtactcttgctggagaaccgatcccttagaccgccccaccttttca2544mettyrsercystrpargthraspproleuaspargprothrpheser835840845gtattgaggctgcagctagaaaaactcttagaaagtttgcctgacgtt2592valleuargleuglnleuglulysleuleugluserleuproaspval850855860cggaaccaagcagacgttatttacgtcaatacacagttgctggagagc2640argasnglnalaaspvaliletyrvalasnthrglnleuleugluser865870875880tctgagggcctggcccagggctccacccttgctccactggacttgaac2688sergluglyleualaglnglyserthrleualaproleuaspleuasn885890895atcgaccctgactctataattgcctcctgcactccccgcgctgccatc2736ileaspproaspserileilealasercysthrproargalaalaile900905910agtgtggtcacagcagaagttcatgacagcaaacctcatgaaggacgg2784servalvalthralagluvalhisaspserlysprohisgluglyarg915920925tacatcctgaatgggggcagtgaggaatgggaagatctgacttctgcc2832tyrileleuasnglyglysergluglutrpgluaspleuthrserala930935940ccctctgctgcagtcacagctgaaaagaacagtgttttaccgggggag2880proseralaalavalthralaglulysasnservalleuproglyglu945950955960agacttgttaggaatggggtctcctggtcccattcgagcatgctgccc2928argleuvalargasnglyvalsertrpserhissersermetleupro965970975ttgggaagctcattgcccgatgaacttttgtttgctgacgactcctca2976leuglyserserleuproaspgluleuleuphealaaspaspserser980985990gaaggctcagaagtcctgatgtga3000gluglysergluvalleumet995<210>57<211>999<212>prt<213>homosapiens<400>57metglyproalaproleuproleuleuleuglyleupheleuproala151015leutrpargargalailethrglualaarggluglualalysprotyr202530proleupheproglypropheproglyserleuglnthrasphisthr354045proleuleuserleuprohisalaserglytyrglnproalaleumet505560pheserprothrglnproglyargprohisthrglyasnvalalaile65707580proglnvalthrservalgluserlysproleuproproleualaphe859095lyshisthrvalglyhisileileleusergluhislysglyvallys100105110pheasncysserileservalproasniletyrglnaspthrthrile115120125sertrptrplysaspglylysgluleuleuglyalahishisalaile130135140thrglnphetyrproaspaspgluvalthralaileilealaserphe145150155160serilethrservalglnargseraspasnglysertyrilecyslys165170175metlysileasnasnglugluilevalseraspproiletyrileglu180185190valglnglyleuprohisphethrlysglnproglusermetasnval195200205thrargasnthralapheasnleuthrcysglnalavalglypropro210215220gluprovalasnilephetrpvalglnasnserserargvalasnglu225230235240glnproglulysserproservalleuthrvalproglyleuthrglu245250255metalavalphesercysglualahisasnasplysglyleuthrval260265270serlysglyvalglnileasnilelysalaileproserproprothr275280285gluvalserileargasnserthralahisserileleuilesertrp290295300valproglypheaspglytyrserpropheargasncysserilegln305310315320vallysglualaaspproleuserasnglyservalmetilepheasn325330335thrseralaleuprohisleutyrglnilelysglnleuglnalaleu340345350alaasntyrserileglyvalsercysmetasngluileglytrpser355360365alavalserprotrpileleualaserthrthrgluglyalaproser370375380valalaproleuasnvalthrvalpheleuasngluserseraspasn385390395400valaspileargtrpmetlysproprothrlysglnglnaspglyglu405410415leuvalglytyrargileserhisvaltrpglnseralaglyileser420425430lysgluleuleuglugluvalglyglnasnglyserargalaargile435440445servalglnvalhisasnalathrcysthrvalargilealaalaval450455460thrargglyglyvalglypropheseraspprovallysilepheile465470475480proalahisglytrpvalasptyralaproserserthrproalapro485490495glyasnalaaspprovalleuileilepheglycysphecysglyphe500505510ileleuileglyleuileleutyrileserleualailearglysarg515520525valglngluthrlyspheglyasnalaphethrglugluaspserglu530535540leuvalvalasntyrilealalyslysserphecysargargalaile545550555560gluleuthrleuhisserleuglyvalserglugluleuglnasnlys565570575leugluaspvalvalileaspargasnleuleuileleuglylysile580585590leuglygluglyglupheglyservalmetgluglyasnleulysgln595600605gluaspglythrserleulysvalalavallysthrmetlysleuasp610615620asnserserglnarggluilegluglupheleuserglualaalacys625630635640metlysasppheserhisproasnvalileargleuleuglyvalcys645650655ileglumetserserglnglyileprolysprometvalileleupro660665670phemetlystyrglyaspleuhisthrtyrleuleutyrserargleu675680685gluthrglyprolyshisileproleuglnthrleuleulysphemet690695700valaspilealaleuglymetglutyrleuserasnargasnpheleu705710715720hisargaspleualaalaargasncysmetleuargaspaspmetthr725730735valcysvalalaasppheglyleuserlyslysiletyrserglyasp740745750tyrtyrargglnglyargilealalysmetprovallystrpileala755760765ilegluserleualaaspargvaltyrthrserlysseraspvaltrp770775780alapheglyvalthrmettrpgluilealathrargglymetthrpro785790795800tyrproglyvalglnasnhisglumettyrasptyrleuleuhisgly805810815hisargleulysglnprogluaspcysleuaspgluleutyrgluile820825830mettyrsercystrpargthraspproleuaspargprothrpheser835840845valleuargleuglnleuglulysleuleugluserleuproaspval850855860argasnglnalaaspvaliletyrvalasnthrglnleuleugluser865870875880sergluglyleualaglnglyserthrleualaproleuaspleuasn885890895ileaspproaspserileilealasercysthrproargalaalaile900905910servalvalthralagluvalhisaspserlysprohisgluglyarg915920925tyrileleuasnglyglysergluglutrpgluaspleuthrserala930935940proseralaalavalthralaglulysasnservalleuproglyglu945950955960argleuvalargasnglyvalsertrpserhissersermetleupro965970975leuglyserserleuproaspgluleuleuphealaaspaspserser980985990gluglysergluvalleumet995<210>58<211>40<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer26<400>58ctcactatagaattcgccaccatggggccggccccgctgc40<210>59<211>43<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer27<400>59caataaacaagttggatcctcacatcaggacttctgagccttc43<210>60<211>1027<212>dna<213>musmusculus<400>60aacagatcctggcggggacttaggactgacctagaacaatcagggttccgcaatccaggt60ccccaaaggtgggatcctcaaccgcaggacggagggaatagcctttcgattctgggtggt120gcattggaagccccaggctctaaaacccccaacctactgactggtggccgagtatgcacc180cgactgctagctaggcagtgtcccaagaaccagtagccaaatgtcttggcctcagttttc240ccggtgacacctggaaagtgaccctgccattagtagaggctcaggtcagggccccgcctc300tcctgggcggcctctgccctagcccgccctgccgctcctcctctccgcaggctcgctccc360acggtccccgaggtgggcgggtgagcccaggatgacggctgtagaaccccggcctgactc420gccctcgcccccgcgccgggcctgggcttccctagcccagctcgcacccgggggccgtcg480gagccgccgcgcgcccagctctacgcgcctggcgccctccccacgcgggcgtccccgact540cccgcgcgcgctcaggctcccagttgggaaccaaggagggggaggatgggggggggggtg600tgcgccgacccggaaacgccatataaggagcaggaaggatcccccgccggaacagacctt660atttgggcagcgccttatatggagtggcccaatatggccctgccgcttccggctctggga720ggaggggcgagcgggggttggggcgggggcaagctgggaactccaggcgcctggcccggg780aggccactgctgctgttccaatactaggctttccaggagcctgagcgctcgcgatgccgg840agcgggtcgcagggtggaggtgcccaccactcttggatgggagggcttcacgtcactccg900ggtcctcccggccggtccttccatattagggcttcctgcttcccatatatggccatgtac960gtcacggcggaggcgggcccgtgctgttccagacccttgaaatagaggccgattcgggga1020gtcgcga1027<210>61<211>438<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:cntovh<220><221>cds<222>(1)..(438)<400>61atggagtttgggctgacctgggttttcctcgttgctcttttaagaggt48metglupheglyleuthrtrpvalpheleuvalalaleuleuarggly151015gtccagtgtcaggtgcagctggtggagtctgggggaggcgtggtccag96valglncysglnvalglnleuvalgluserglyglyglyvalvalgln202530cctgggaggtccctgagactctcctgtgcagcgtctggattcaccttc144proglyargserleuargleusercysalaalaserglyphethrphe354045agtagctatggcatgcactgggtccgccaggctccaggcaaggggctg192sersertyrglymethistrpvalargglnalaproglylysglyleu505560gagtgggtggcagttatatggtatgatggaagtaataaatactatgca240glutrpvalalavaliletrptyraspglyserasnlystyrtyrala65707580gactccgtgaaggggcgattcaccatctccagagacaattccaagaac288aspservallysglyargphethrileserargaspasnserlysasn859095acgctgtatctgcaaatgaacagcctgagagccgaggacacggctatg336thrleutyrleuglnmetasnserleuargalagluaspthralamet100105110tatttctgtgcgagagaagggttttattacgatattttgactgcttat384tyrphecysalaarggluglyphetyrtyraspileleuthralatyr115120125tcccttgaatacttccagcactggggccagggcaccctggtcaccgtc432serleuglutyrpheglnhistrpglyglnglythrleuvalthrval130135140tcctca438serser145<210>62<211>146<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>62metglupheglyleuthrtrpvalpheleuvalalaleuleuarggly151015valglncysglnvalglnleuvalgluserglyglyglyvalvalgln202530proglyargserleuargleusercysalaalaserglyphethrphe354045sersertyrglymethistrpvalargglnalaproglylysglyleu505560glutrpvalalavaliletrptyraspglyserasnlystyrtyrala65707580aspservallysglyargphethrileserargaspasnserlysasn859095thrleutyrleuglnmetasnserleuargalagluaspthralamet100105110tyrphecysalaarggluglyphetyrtyraspileleuthralatyr115120125serleuglutyrpheglnhistrpglyglnglythrleuvalthrval130135140serser145<210>63<211>378<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:cntovl<220><221>cds<222>(1)..(378)<400>63atgtcgccatcacaactcattgggtttctgctgctctgggttccagcc48metserproserglnleuileglypheleuleuleutrpvalproala151015tccaggggtgaaattgtgctgactcagtctccagactttcagtctgtg96serargglygluilevalleuthrglnserproasppheglnserval202530actccaaaggagaaggtcaccatcacctgccgggccagtcagagcatt144thrprolysglulysvalthrilethrcysargalaserglnserile354045ggtagtagcttacactggtaccagcagaaaccagatcagtctccaaag192glyserserleuhistrptyrglnglnlysproaspglnserprolys505560ctcctcatcaagtatgcttcccagtccttctcaggggtcccctcgagg240leuleuilelystyralaserglnserpheserglyvalproserarg65707580ttcagtggcagtggatctgggacagatttcaccctcaccatcaatagc288pheserglyserglyserglythraspphethrleuthrileasnser859095ctggaagctgaagatgctgcagcgtattactgtcatcagagtagtagt336leuglualagluaspalaalaalatyrtyrcyshisglnserserser100105110ttaccgtacacttttggccaggggaccaagctggagatcaaa378leuprotyrthrpheglyglnglythrlysleugluilelys115120125<210>64<211>126<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>64metserproserglnleuileglypheleuleuleutrpvalproala151015serargglygluilevalleuthrglnserproasppheglnserval202530thrprolysglulysvalthrilethrcysargalaserglnserile354045glyserserleuhistrptyrglnglnlysproaspglnserprolys505560leuleuilelystyralaserglnserpheserglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileasnser859095leuglualagluaspalaalaalatyrtyrcyshisglnserserser100105110leuprotyrthrpheglyglnglythrlysleugluilelys115120125<210>65<211>30<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer28<400>65ccagggtcaccatggagttagtttgggcag30<210>66<211>32<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer29<400>66ctaacactcattcctgttgaagctcttgacaa32<210>67<211>414<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5320vh<220><221>cds<222>(1)..(414)<400>67atggattggctgtggaacttgctattcctgatggcagctgcccaaagt48metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015gcccaagcacagatccagttggtgcagtctggacctgagctgaagaag96alaglnalaglnileglnleuvalglnserglyprogluleulyslys202530cctggagagacagtcaagatctcctgcaaggcttctgggtataccttc144proglygluthrvallysilesercyslysalaserglytyrthrphe354045acaaactatggaatgaactgggtgaagcaggctccaggaaagggttta192thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560aagtggatgggctggataaacactgagactggagagccaacatattct240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gatgacttcaagggacggtttgccttctctttggaaacctctgccacc288aspaspphelysglyargphealapheserleugluthrseralathr859095actgcctatttgcagatcaacaacctcagaaatgaggacatggctaca336thralatyrleuglnileasnasnleuargasngluaspmetalathr100105110tatttctgtgcaagagaggatggttactacggtactttggactactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggtcaaggaacctcagtcaccgtctcctca414glyglnglythrservalthrvalserser130135<210>68<211>138<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>68metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015alaglnalaglnileglnleuvalglnserglyprogluleulyslys202530proglygluthrvallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphealapheserleugluthrseralathr859095thralatyrleuglnileasnasnleuargasngluaspmetalathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrservalthrvalserser130135<210>69<211>119<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vhexcludingsignalsequence<400>69glnileglnleuvalglnserglyprogluleulyslysproglyglu151015thrvallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglylysglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphealapheserleugluthrseralathrthralatyr65707580leuglnileasnasnleuargasngluaspmetalathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrservalthrvalserser115<210>70<211>381<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5320vl<220><221>cds<222>(1)..(381)<400>70atgaagtcacagacccaggtcttcgtatttctactgctctgtgtgtct48metlysserglnthrglnvalphevalpheleuleuleucysvalser151015ggtgctcatgggagtattgtgatgacccagactcccaaattcctgctt96glyalahisglyserilevalmetthrglnthrprolyspheleuleu202530gtatcagcaggagacagggttaccataacctgcaaggccagtcagagt144valseralaglyaspargvalthrilethrcyslysalaserglnser354045gtgagtaatgatgtagcttggtaccaacagaagccagggcagtctcct192valserasnaspvalalatrptyrglnglnlysproglyglnserpro505560aaagtgctgatatactatgcatccaatcgctacactggagtccctgat240lysvalleuiletyrtyralaserasnargtyrthrglyvalproasp65707580cgcttcactggcagtggatatgggacggatttcactttcaccatcagc288argphethrglyserglytyrglythraspphethrphethrileser859095actgtgcaggctgaagacctggcagtttatttctgtcagcaggattat336thrvalglnalagluaspleualavaltyrphecysglnglnasptyr100105110agctctccgtggacgttcggtggaggcaccaagctggaaatcaaa381serserprotrpthrpheglyglyglythrlysleugluilelys115120125<210>71<211>127<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>71metlysserglnthrglnvalphevalpheleuleuleucysvalser151015glyalahisglyserilevalmetthrglnthrprolyspheleuleu202530valseralaglyaspargvalthrilethrcyslysalaserglnser354045valserasnaspvalalatrptyrglnglnlysproglyglnserpro505560lysvalleuiletyrtyralaserasnargtyrthrglyvalproasp65707580argphethrglyserglytyrglythraspphethrphethrileser859095thrvalglnalagluaspleualavaltyrphecysglnglnasptyr100105110serserprotrpthrpheglyglyglythrlysleugluilelys115120125<210>72<211>107<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vlexcludingsignalsequence<400>72serilevalmetthrglnthrprolyspheleuleuvalseralagly151015aspargvalthrilethrcyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysvalleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphethrgly505560serglytyrglythraspphethrphethrileserthrvalglnala65707580gluaspleualavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglyglythrlysleugluilelys100105<210>73<211>405<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5321vh<220><221>cds<222>(1)..(405)<400>73atggattggctgtggaacttgctattcctgatggcagctgcccaaagt48metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015atccaagcacagatccagttggtgcagtctggacctgagctgaagaag96ileglnalaglnileglnleuvalglnserglyprogluleulyslys202530cctggagagacagtcaagatctcctgcagggcttctgggtataccttc144proglygluthrvallysilesercysargalaserglytyrthrphe354045acaaactatggaatgaactgggtgaagcaggctccaggaaagggttta192thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560aagtggatgggctggataaacaccaacactggagagccaacttatact240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaagagttcaagggacggtttgccttctctttggagacctctgccact288glugluphelysglyargphealapheserleugluthrseralathr859095actgcctatttgcagatcaacgacctcaaaaatgaggacacggctaca336thralatyrleuglnileasnaspleulysasngluaspthralathr100105110tatttctgtgcaagagatgagggatggtttgtttactggggccaaggg384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctgatcactgtctctgca405thrleuilethrvalserala130135<210>74<211>135<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>74metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015ileglnalaglnileglnleuvalglnserglyprogluleulyslys202530proglygluthrvallysilesercysargalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphealapheserleugluthrseralathr859095thralatyrleuglnileasnaspleulysasngluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuilethrvalserala130135<210>75<211>116<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vhexcludingsignalsequence<400>75glnileglnleuvalglnserglyprogluleulyslysproglyglu151015thrvallysilesercysargalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglylysglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphealapheserleugluthrseralathrthralatyr65707580leuglnileasnaspleulysasngluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuile100105110thrvalserala115<210>76<211>381<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5321vl<220><221>cds<222>(1)..(381)<400>76atgctctcactagctcctctcctcagccttcttctcctctgtgtctct48metleuserleualaproleuleuserleuleuleuleucysvalser151015gattctagggcagaaacaactgtgacccagtctccagcatccctgtcc96aspserargalagluthrthrvalthrglnserproalaserleuser202530gtggctacaggagaaaaagtcactatcagatgcataaccagtactgat144valalathrglyglulysvalthrileargcysilethrserthrasp354045attgatgatgatatgaactggtaccaacagaagccaggggaacctcct192ileaspaspaspmetasntrptyrglnglnlysproglyglupropro505560aagctccttatttcagaaggcaatactcttcgtcctggagtcccatcc240lysleuleuilesergluglyasnthrleuargproglyvalproser65707580cgattctccagcagtggctatggcacagattttgtttttacaattgaa288argpheserserserglytyrglythraspphevalphethrileglu859095aacacgctctcagaagatgttgcagattactactgtttgcaaactgat336asnthrleusergluaspvalalaasptyrtyrcysleuglnthrasp100105110agcgtgcctctcacgttcggtgctgggaccaagctggagctgaaa381servalproleuthrpheglyalaglythrlysleugluleulys115120125<210>77<211>127<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>77metleuserleualaproleuleuserleuleuleuleucysvalser151015aspserargalagluthrthrvalthrglnserproalaserleuser202530valalathrglyglulysvalthrileargcysilethrserthrasp354045ileaspaspaspmetasntrptyrglnglnlysproglyglupropro505560lysleuleuilesergluglyasnthrleuargproglyvalproser65707580argpheserserserglytyrglythraspphevalphethrileglu859095asnthrleusergluaspvalalaasptyrtyrcysleuglnthrasp100105110servalproleuthrpheglyalaglythrlysleugluleulys115120125<210>78<211>107<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vlexcludingsignalsequence<400>78gluthrthrvalthrglnserproalaserleuservalalathrgly151015glulysvalthrileargcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglygluproprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargpheserser505560serglytyrglythraspphevalphethrilegluasnthrleuser65707580gluaspvalalaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyalaglythrlysleugluleulys100105<210>79<211>5<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vhcdr1<400>79asntyrglymetasn15<210>80<211>17<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vhcdr2<400>80trpileasnthrgluthrglygluprothrtyrseraspaspphelys151015gly<210>81<211>10<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vhcdr3<400>81gluaspglytyrtyrglythrleuasptyr1510<210>82<211>11<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vlcdr1<400>82lysalaserglnservalserasnaspvalala1510<210>83<211>7<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vlcdr2<400>83tyralaserasnargtyrthr15<210>84<211>9<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5320vlcdr3<400>84glnglnasptyrserserprotrpthr15<210>85<211>5<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vhcdr1<400>85asntyrglymetasn15<210>86<211>17<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vhcdr2<400>86trpileasnthrasnthrglygluprothrtyrthrglugluphelys151015gly<210>87<211>7<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vhcdr3<400>87aspgluglytrpphevaltyr15<210>88<211>11<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vlcdr1<400>88ilethrserthraspileaspaspaspmetasn1510<210>89<211>7<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vlcdr2<400>89gluglyasnthrleuargpro15<210>90<211>9<212>prt<213>artificialsequence<220><223>descriptionoftheartificialsequence:aminoacidsequenceofkm5321vlcdr3<400>90leuglnthraspservalproleuthr15<210>91<211>1395<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5320-rigg1<220><221>cds<222>(1)..(1395)<400>91atggattggctgtggaacttgctattcctgatggcagctgcccaaagt48metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015gcccaagcacagatccagttggtgcagtctggacctgagctgaagaag96alaglnalaglnileglnleuvalglnserglyprogluleulyslys202530cctggagagacagtcaagatctcctgcaaggcttctgggtataccttc144proglygluthrvallysilesercyslysalaserglytyrthrphe354045acaaactatggaatgaactgggtgaagcaggctccaggaaagggttta192thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560aagtggatgggctggataaacactgagactggagagccaacatattct240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gatgacttcaagggacggtttgccttctctttggaaacctctgccacc288aspaspphelysglyargphealapheserleugluthrseralathr859095actgcctatttgcagatcaacaacctcagaaatgaggacatggctaca336thralatyrleuglnileasnasnleuargasngluaspmetalathr100105110tatttctgtgcaagagaggatggttactacggtactttggactactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggtcaaggaacctcagtcaccgtctcctcagctgaaacaacagccccg432glyglnglythrservalthrvalserseralagluthrthralapro130135140tctgtctatccactggctcctggaactgctctcaaaagtaactccatg480servaltyrproleualaproglythralaleulysserasnsermet145150155160gtgaccctgggatgcctggtcaagggctatttccctgagccagtcacc528valthrleuglycysleuvallysglytyrpheprogluprovalthr165170175gtgacctggaactctggagccctgtccagcggtgtgcacaccttccca576valthrtrpasnserglyalaleuserserglyvalhisthrphepro180185190gctgtcctgcagtctgggctctacactctcaccagctcagtgactgta624alavalleuglnserglyleutyrthrleuthrserservalthrval195200205ccctccagcacctggcccagccagaccgtcacctgcaacgtagcccac672proserserthrtrpproserglnthrvalthrcysasnvalalahis210215220ccggccagcagcaccaaggtggacaagaaaattgtgcccagaaactgt720proalaserserthrlysvalasplyslysilevalproargasncys225230235240ggaggtgattgcaagccttgtatatgtacaggctcagaagtatcatct768glyglyaspcyslysprocysilecysthrglysergluvalserser245250255gtcttcatcttccccccaaagcccaaagatgtgctcaccatcactctg816valpheilepheproprolysprolysaspvalleuthrilethrleu260265270actcctaaggtcacgtgtgttgtggtagacattagccaggacgatccc864thrprolysvalthrcysvalvalvalaspileserglnaspasppro275280285gaggtccatttcagctggtttgtagatgacgtggaagtccacacagct912gluvalhisphesertrpphevalaspaspvalgluvalhisthrala290295300cagactcgaccaccagaggagcagttcaacagcactttccgctcagtc960glnthrargproproglugluglnpheasnserthrpheargserval305310315320agtgaactccccatcctgcaccaggactggctcaatggcaggacgttc1008sergluleuproileleuhisglnasptrpleuasnglyargthrphe325330335agatgcaaggtcaccagtgcagctttcccatcccccatcgagaaaacc1056argcyslysvalthrseralaalapheproserproileglulysthr340345350atctccaaacccgaaggcagaacacaagttccgcatgtatacaccatg1104ileserlysprogluglyargthrglnvalprohisvaltyrthrmet355360365tcacctaccaaggaagagatgacccagaatgaagtcagtatcacctgc1152serprothrlysgluglumetthrglnasngluvalserilethrcys370375380atggtaaaaggcttctatcccccagacatttatgtggagtggcagatg1200metvallysglyphetyrproproaspiletyrvalglutrpglnmet385390395400aacgggcagccacaggaaaactacaagaacactccacctacgatggac1248asnglyglnproglngluasntyrlysasnthrproprothrmetasp405410415acagatgggagttacttcctctacagcaagctcaatgtgaagaaggaa1296thraspglysertyrpheleutyrserlysleuasnvallyslysglu420425430aaatggcagcagggaaacacgttcacgtgttctgtgctgcatgaaggc1344lystrpglnglnglyasnthrphethrcysservalleuhisglugly435440445ctgcacaaccaccatactgagaagagtctctcccactctccgggtaaa1392leuhisasnhishisthrglulysserleuserhisserproglylys450455460tga1395<210>92<211>464<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>92metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015alaglnalaglnileglnleuvalglnserglyprogluleulyslys202530proglygluthrvallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphealapheserleugluthrseralathr859095thralatyrleuglnileasnasnleuargasngluaspmetalathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrservalthrvalserseralagluthrthralapro130135140servaltyrproleualaproglythralaleulysserasnsermet145150155160valthrleuglycysleuvallysglytyrpheprogluprovalthr165170175valthrtrpasnserglyalaleuserserglyvalhisthrphepro180185190alavalleuglnserglyleutyrthrleuthrserservalthrval195200205proserserthrtrpproserglnthrvalthrcysasnvalalahis210215220proalaserserthrlysvalasplyslysilevalproargasncys225230235240glyglyaspcyslysprocysilecysthrglysergluvalserser245250255valpheilepheproprolysprolysaspvalleuthrilethrleu260265270thrprolysvalthrcysvalvalvalaspileserglnaspasppro275280285gluvalhisphesertrpphevalaspaspvalgluvalhisthrala290295300glnthrargproproglugluglnpheasnserthrpheargserval305310315320sergluleuproileleuhisglnasptrpleuasnglyargthrphe325330335argcyslysvalthrseralaalapheproserproileglulysthr340345350ileserlysprogluglyargthrglnvalprohisvaltyrthrmet355360365serprothrlysgluglumetthrglnasngluvalserilethrcys370375380metvallysglyphetyrproproaspiletyrvalglutrpglnmet385390395400asnglyglnproglngluasntyrlysasnthrproprothrmetasp405410415thraspglysertyrpheleutyrserlysleuasnvallyslysglu420425430lystrpglnglnglyasnthrphethrcysservalleuhisglugly435440445leuhisasnhishisthrglulysserleuserhisserproglylys450455460<210>93<211>705<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5320-r_kappa<220><221>cds<222>(1)..(705)<400>93atgaagtcacagacccaggtcttcgtatttctactgctctgtgtgtct48metlysserglnthrglnvalphevalpheleuleuleucysvalser151015ggtgctcatgggagtattgtgatgacccagactcccaaattcctgctt96glyalahisglyserilevalmetthrglnthrprolyspheleuleu202530gtatcagcaggagacagggttaccataacctgcaaggccagtcagagt144valseralaglyaspargvalthrilethrcyslysalaserglnser354045gtgagtaatgatgtagcttggtaccaacagaagccagggcagtctcct192valserasnaspvalalatrptyrglnglnlysproglyglnserpro505560aaagtgctgatatactatgcatccaatcgctacactggagtccctgat240lysvalleuiletyrtyralaserasnargtyrthrglyvalproasp65707580cgcttcactggcagtggatatgggacggatttcactttcaccatcagc288argphethrglyserglytyrglythraspphethrphethrileser859095actgtgcaggctgaagacctggcagtttatttctgtcagcaggattat336thrvalglnalagluaspleualavaltyrphecysglnglnasptyr100105110agctctccgtggacgttcggtggaggcaccaagctggaaatcaaacgg384serserprotrpthrpheglyglyglythrlysleugluilelysarg115120125gctgatgcggcaccaactgtatctatcttcccaccatccacggaacag432alaaspalaalaprothrvalserilepheproproserthrglugln130135140ttagcaactggaggtgcctcagtcgtgtgcctcatgaacaacttctat480leualathrglyglyalaservalvalcysleumetasnasnphetyr145150155160cccagagacatcagtgtcaagtggaagattgatggcactgaacgacga528proargaspileservallystrplysileaspglythrgluargarg165170175gatggtgtcctggacagtgttactgatcaggacagcaaagacagcacg576aspglyvalleuaspservalthraspglnaspserlysaspserthr180185190tacagcatgagcagcaccctctcgttgaccaaggctgactatgaaagt624tyrsermetserserthrleuserleuthrlysalaasptyrgluser195200205cataacctctatacctgtgaggttgttcataagacatcatcctcaccc672hisasnleutyrthrcysgluvalvalhislysthrserserserpro210215220gtcgtcaagagcttcaacaggaatgagtgttag705valvallysserpheasnargasnglucys225230<210>94<211>234<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>94metlysserglnthrglnvalphevalpheleuleuleucysvalser151015glyalahisglyserilevalmetthrglnthrprolyspheleuleu202530valseralaglyaspargvalthrilethrcyslysalaserglnser354045valserasnaspvalalatrptyrglnglnlysproglyglnserpro505560lysvalleuiletyrtyralaserasnargtyrthrglyvalproasp65707580argphethrglyserglytyrglythraspphethrphethrileser859095thrvalglnalagluaspleualavaltyrphecysglnglnasptyr100105110serserprotrpthrpheglyglyglythrlysleugluilelysarg115120125alaaspalaalaprothrvalserilepheproproserthrglugln130135140leualathrglyglyalaservalvalcysleumetasnasnphetyr145150155160proargaspileservallystrplysileaspglythrgluargarg165170175aspglyvalleuaspservalthraspglnaspserlysaspserthr180185190tyrsermetserserthrleuserleuthrlysalaasptyrgluser195200205hisasnleutyrthrcysgluvalvalhislysthrserserserpro210215220valvallysserpheasnargasnglucys225230<210>95<211>1386<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5321-rigg1<220><221>cds<222>(1)..(1386)<400>95atggattggctgtggaacttgctattcctgatggcagctgcccaaagt48metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015atccaagcacagatccagttggtgcagtctggacctgagctgaagaag96ileglnalaglnileglnleuvalglnserglyprogluleulyslys202530cctggagagacagtcaagatctcctgcagggcttctgggtataccttc144proglygluthrvallysilesercysargalaserglytyrthrphe354045acaaactatggaatgaactgggtgaagcaggctccaggaaagggttta192thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560aagtggatgggctggataaacaccaacactggagagccaacttatact240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaagagttcaagggacggtttgccttctctttggagacctctgccact288glugluphelysglyargphealapheserleugluthrseralathr859095actgcctatttgcagatcaacgacctcaaaaatgaggacacggctaca336thralatyrleuglnileasnaspleulysasngluaspthralathr100105110tatttctgtgcaagagatgagggatggtttgtttactggggccaaggg384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctgatcactgtctctgcagctgaaacaacagccccgtctgtctat432thrleuilethrvalseralaalagluthrthralaproservaltyr130135140ccactggctcctggaactgctctcaaaagtaactccatggtgaccctg480proleualaproglythralaleulysserasnsermetvalthrleu145150155160ggatgcctggtcaagggctatttccctgagccagtcaccgtgacctgg528glycysleuvallysglytyrpheprogluprovalthrvalthrtrp165170175aactctggagccctgtccagcggtgtgcacaccttcccagctgtcctg576asnserglyalaleuserserglyvalhisthrpheproalavalleu180185190cagtctgggctctacactctcaccagctcagtgactgtaccctccagc624glnserglyleutyrthrleuthrserservalthrvalproserser195200205acctggcccagccagaccgtcacctgcaacgtagcccacccggccagc672thrtrpproserglnthrvalthrcysasnvalalahisproalaser210215220agcaccaaggtggacaagaaaattgtgcccagaaactgtggaggtgat720serthrlysvalasplyslysilevalproargasncysglyglyasp225230235240tgcaagccttgtatatgtacaggctcagaagtatcatctgtcttcatc768cyslysprocysilecysthrglysergluvalserservalpheile245250255ttccccccaaagcccaaagatgtgctcaccatcactctgactcctaag816pheproprolysprolysaspvalleuthrilethrleuthrprolys260265270gtcacgtgtgttgtggtagacattagccaggacgatcccgaggtccat864valthrcysvalvalvalaspileserglnaspaspprogluvalhis275280285ttcagctggtttgtagatgacgtggaagtccacacagctcagactcga912phesertrpphevalaspaspvalgluvalhisthralaglnthrarg290295300ccaccagaggagcagttcaacagcactttccgctcagtcagtgaactc960proproglugluglnpheasnserthrpheargservalsergluleu305310315320cccatcctgcaccaggactggctcaatggcaggacgttcagatgcaag1008proileleuhisglnasptrpleuasnglyargthrpheargcyslys325330335gtcaccagtgcagctttcccatcccccatcgagaaaaccatctccaaa1056valthrseralaalapheproserproileglulysthrileserlys340345350cccgaaggcagaacacaagttccgcatgtatacaccatgtcacctacc1104progluglyargthrglnvalprohisvaltyrthrmetserprothr355360365aaggaagagatgacccagaatgaagtcagtatcacctgcatggtaaaa1152lysgluglumetthrglnasngluvalserilethrcysmetvallys370375380ggcttctatcccccagacatttatgtggagtggcagatgaacgggcag1200glyphetyrproproaspiletyrvalglutrpglnmetasnglygln385390395400ccacaggaaaactacaagaacactccacctacgatggacacagatggg1248proglngluasntyrlysasnthrproprothrmetaspthraspgly405410415agttacttcctctacagcaagctcaatgtgaagaaggaaaaatggcag1296sertyrpheleutyrserlysleuasnvallyslysglulystrpgln420425430cagggaaacacgttcacgtgttctgtgctgcatgaaggcctgcacaac1344glnglyasnthrphethrcysservalleuhisgluglyleuhisasn435440445caccatactgagaagagtctctcccactctccgggtaaatga1386hishisthrglulysserleuserhisserproglylys450455460<210>96<211>461<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>96metasptrpleutrpasnleuleupheleumetalaalaalaglnser151015ileglnalaglnileglnleuvalglnserglyprogluleulyslys202530proglygluthrvallysilesercysargalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglylysglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphealapheserleugluthrseralathr859095thralatyrleuglnileasnaspleulysasngluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuilethrvalseralaalagluthrthralaproservaltyr130135140proleualaproglythralaleulysserasnsermetvalthrleu145150155160glycysleuvallysglytyrpheprogluprovalthrvalthrtrp165170175asnserglyalaleuserserglyvalhisthrpheproalavalleu180185190glnserglyleutyrthrleuthrserservalthrvalproserser195200205thrtrpproserglnthrvalthrcysasnvalalahisproalaser210215220serthrlysvalasplyslysilevalproargasncysglyglyasp225230235240cyslysprocysilecysthrglysergluvalserservalpheile245250255pheproprolysprolysaspvalleuthrilethrleuthrprolys260265270valthrcysvalvalvalaspileserglnaspaspprogluvalhis275280285phesertrpphevalaspaspvalgluvalhisthralaglnthrarg290295300proproglugluglnpheasnserthrpheargservalsergluleu305310315320proileleuhisglnasptrpleuasnglyargthrpheargcyslys325330335valthrseralaalapheproserproileglulysthrileserlys340345350progluglyargthrglnvalprohisvaltyrthrmetserprothr355360365lysgluglumetthrglnasngluvalserilethrcysmetvallys370375380glyphetyrproproaspiletyrvalglutrpglnmetasnglygln385390395400proglngluasntyrlysasnthrproprothrmetaspthraspgly405410415sertyrpheleutyrserlysleuasnvallyslysglulystrpgln420425430glnglyasnthrphethrcysservalleuhisgluglyleuhisasn435440445hishisthrglulysserleuserhisserproglylys450455460<210>97<211>705<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:km5321-r_kappa<220><221>cds<222>(1)..(705)<400>97atgctctcactagctcctctcctcagccttcttctcctctgtgtctct48metleuserleualaproleuleuserleuleuleuleucysvalser151015gattctagggcagaaacaactgtgacccagtctccagcatccctgtcc96aspserargalagluthrthrvalthrglnserproalaserleuser202530gtggctacaggagaaaaagtcactatcagatgcataaccagtactgat144valalathrglyglulysvalthrileargcysilethrserthrasp354045attgatgatgatatgaactggtaccaacagaagccaggggaacctcct192ileaspaspaspmetasntrptyrglnglnlysproglyglupropro505560aagctccttatttcagaaggcaatactcttcgtcctggagtcccatcc240lysleuleuilesergluglyasnthrleuargproglyvalproser65707580cgattctccagcagtggctatggcacagattttgtttttacaattgaa288argpheserserserglytyrglythraspphevalphethrileglu859095aacacgctctcagaagatgttgcagattactactgtttgcaaactgat336asnthrleusergluaspvalalaasptyrtyrcysleuglnthrasp100105110agcgtgcctctcacgttcggtgctgggaccaagctggagctgaaacgg384servalproleuthrpheglyalaglythrlysleugluleulysarg115120125gctgatgcggcaccaactgtatctatcttcccaccatccacggaacag432alaaspalaalaprothrvalserilepheproproserthrglugln130135140ttagcaactggaggtgcctcagtcgtgtgcctcatgaacaacttctat480leualathrglyglyalaservalvalcysleumetasnasnphetyr145150155160cccagagacatcagtgtcaagtggaagattgatggcactgaacgacga528proargaspileservallystrplysileaspglythrgluargarg165170175gatggtgtcctggacagtgttactgatcaggacagcaaagacagcacg576aspglyvalleuaspservalthraspglnaspserlysaspserthr180185190tacagcatgagcagcaccctctcgttgaccaaggctgactatgaaagt624tyrsermetserserthrleuserleuthrlysalaasptyrgluser195200205cataacctctatacctgtgaggttgttcataagacatcatcctcaccc672hisasnleutyrthrcysgluvalvalhislysthrserserserpro210215220gtcgtcaagagcttcaacaggaatgagtgttag705valvallysserpheasnargasnglucys225230<210>98<211>234<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>98metleuserleualaproleuleuserleuleuleuleucysvalser151015aspserargalagluthrthrvalthrglnserproalaserleuser202530valalathrglyglulysvalthrileargcysilethrserthrasp354045ileaspaspaspmetasntrptyrglnglnlysproglyglupropro505560lysleuleuilesergluglyasnthrleuargproglyvalproser65707580argpheserserserglytyrglythraspphevalphethrileglu859095asnthrleusergluaspvalalaasptyrtyrcysleuglnthrasp100105110servalproleuthrpheglyalaglythrlysleugluleulysarg115120125alaaspalaalaprothrvalserilepheproproserthrglugln130135140leualathrglyglyalaservalvalcysleumetasnasnphetyr145150155160proargaspileservallystrplysileaspglythrgluargarg165170175aspglyvalleuaspservalthraspglnaspserlysaspserthr180185190tyrsermetserserthrleuserleuthrlysalaasptyrgluser195200205hisasnleutyrthrcysgluvalvalhislysthrserserserpro210215220valvallysserpheasnargasnglucys225230<210>99<211>1896<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:hgas6-delta<220><221>cds<222>(1)..(1896)<400>99atggccccttcgctctcgcccgggcccgccgccctgcgccgcgcgccg48metalaproserleuserproglyproalaalaleuargargalapro151015cagctgctgctgctgctgctggccgcggagtgcgcgcttgcctactta96glnleuleuleuleuleuleualaalaglucysalaleualatyrleu202530gactgcatcaacaagtatgggtctccgtacaccaaaaactcaggcttc144aspcysileasnlystyrglyserprotyrthrlysasnserglyphe354045gccacctgcgtgcaaaacctgcctgaccagtgcacgcccaacccctgc192alathrcysvalglnasnleuproaspglncysthrproasnprocys505560gataggaaggggacccaagcctgccaggacctcatgggcaacttcttc240asparglysglythrglnalacysglnaspleumetglyasnphephe65707580tgcctgtgtaaagctggctgggggggccggctctgcgacaaagatgtc288cysleucyslysalaglytrpglyglyargleucysasplysaspval859095aacgaatgcagccaggagaacgggggctgcctccagatctgccacaac336asnglucysserglngluasnglyglycysleuglnilecyshisasn100105110aagccgggtagcttccactgttcctgccacagcggcttcgagctctcc384lysproglyserphehiscyssercyshisserglyphegluleuser115120125tctgatggcaggacctgccaagacatagacgagtgcgcagactcggag432seraspglyargthrcysglnaspileaspglucysalaaspserglu130135140gcctgcggggaggcgcgctgcaagaacctgcccggctcctactcctgc480alacysglyglualaargcyslysasnleuproglysertyrsercys145150155160ctctgtgacgagggctttgcgtacagctcccaggagaaggcttgccga528leucysaspgluglyphealatyrserserglnglulysalacysarg165170175gatgtggacgagtgtctgcagggccgctgtgagcaggtctgcgtgaac576aspvalaspglucysleuglnglyargcysgluglnvalcysvalasn180185190tccccagggagctacacctgccactgtgacgggcgtgggggcctcaag624serproglysertyrthrcyshiscysaspglyargglyglyleulys195200205ctgtcccaggacatggacacctgtgaggacatcttgccgtgcgtgccc672leuserglnaspmetaspthrcysgluaspileleuprocysvalpro210215220ttcagcgtggccaagagtgtgaagtccttgtacctgggccggatgttc720pheservalalalysservallysserleutyrleuglyargmetphe225230235240agtgggacccccgtgatccgactgcgcttcaagaggctgcagcccacc768serglythrprovalileargleuargphelysargleuglnprothr245250255aggctggtagctgagtttgacttccggacctttgaccccgagggcatc816argleuvalalaglupheasppheargthrpheaspprogluglyile260265270ctcctctttgccggaggccaccaggacagcacctggatcgtgctggcc864leuleuphealaglyglyhisglnaspserthrtrpilevalleuala275280285ctgagagccggccggctggagctgcagctgcgctacaacggtgtcggc912leuargalaglyargleugluleuglnleuargtyrasnglyvalgly290295300cgtgtcaccagcagcggcccggtcatcaaccatggcatgtggcagaca960argvalthrserserglyprovalileasnhisglymettrpglnthr305310315320atctctgttgaggagctggcgcggaatctggtcatcaaggtcaacagg1008ileservalglugluleualaargasnleuvalilelysvalasnarg325330335gatgctgtcatgaaaatcgcggtggccggggacttgttccaaccggag1056aspalavalmetlysilealavalalaglyaspleupheglnproglu340345350cgaggactgtatcatctgaacctgaccgtgggaggtattcccttccat1104argglyleutyrhisleuasnleuthrvalglyglyileprophehis355360365gagaaggacctcgtgcagcctataaaccctcgtctggatggctgcatg1152glulysaspleuvalglnproileasnproargleuaspglycysmet370375380aggagctggaactggctgaacggagaagacaccaccatccaggaaacg1200argsertrpasntrpleuasnglygluaspthrthrileglngluthr385390395400gtgaaagtgaacacgaggatgcagtgcttctcggtgacggagagaggc1248vallysvalasnthrargmetglncyspheservalthrgluarggly405410415tctttctaccccgggagcggcttcgccttctacagcctggactacatg1296serphetyrproglyserglyphealaphetyrserleuasptyrmet420425430cggacccctctggacgtcgggactgaatcaacctgggaagtagaagtc1344argthrproleuaspvalglythrgluserthrtrpgluvalgluval435440445gtggctcacatccgcccagccgcagacacaggcgtgctgtttgcgctc1392valalahisileargproalaalaaspthrglyvalleuphealaleu450455460tgggcccccgacctccgtgccgtgcctctctctgtggcactggtagac1440trpalaproaspleuargalavalproleuservalalaleuvalasp465470475480tatcactccacgaagaaactcaagaagcagctggtggtcctggccgtg1488tyrhisserthrlyslysleulyslysglnleuvalvalleualaval485490495gagcatacggccttggccctaatggagatcaaggtctgcgacggccaa1536gluhisthralaleualaleumetgluilelysvalcysaspglygln500505510gagcacgtggtcaccgtctcgctgagggacggtgaggccaccctggag1584gluhisvalvalthrvalserleuargaspglyglualathrleuglu515520525gtggacggcaccaggggccagagcgaggtgagcgccgcgcagctgcag1632valaspglythrargglyglnsergluvalseralaalaglnleugln530535540gagaggctggccgtgctcgagaggcacctgcggagccccgtgctcacc1680gluargleualavalleugluarghisleuargserprovalleuthr545550555560tttgctggcggcctgccagatgtgccggtgacttcagcgccagtcacc1728phealaglyglyleuproaspvalprovalthrseralaprovalthr565570575gcgttctaccgcggctgcatgacactggaggtcaaccggaggctgctg1776alaphetyrargglycysmetthrleugluvalasnargargleuleu580585590gacctggacgaggcggcgtacaagcacagcgacatcacggcccactcc1824aspleuaspglualaalatyrlyshisseraspilethralahisser595600605tgcccccccgtggagcccgccgcagccgactacaaggacgacgacgac1872cysproprovalgluproalaalaalaasptyrlysaspaspaspasp610615620aagcaccaccaccaccaccactag1896lyshishishishishishis625630<210>100<211>631<212>prt<213>artificialsequence<220><223>syntheticconstruct<400>100metalaproserleuserproglyproalaalaleuargargalapro151015glnleuleuleuleuleuleualaalaglucysalaleualatyrleu202530aspcysileasnlystyrglyserprotyrthrlysasnserglyphe354045alathrcysvalglnasnleuproaspglncysthrproasnprocys505560asparglysglythrglnalacysglnaspleumetglyasnphephe65707580cysleucyslysalaglytrpglyglyargleucysasplysaspval859095asnglucysserglngluasnglyglycysleuglnilecyshisasn100105110lysproglyserphehiscyssercyshisserglyphegluleuser115120125seraspglyargthrcysglnaspileaspglucysalaaspserglu130135140alacysglyglualaargcyslysasnleuproglysertyrsercys145150155160leucysaspgluglyphealatyrserserglnglulysalacysarg165170175aspvalaspglucysleuglnglyargcysgluglnvalcysvalasn180185190serproglysertyrthrcyshiscysaspglyargglyglyleulys195200205leuserglnaspmetaspthrcysgluaspileleuprocysvalpro210215220pheservalalalysservallysserleutyrleuglyargmetphe225230235240serglythrprovalileargleuargphelysargleuglnprothr245250255argleuvalalaglupheasppheargthrpheaspprogluglyile260265270leuleuphealaglyglyhisglnaspserthrtrpilevalleuala275280285leuargalaglyargleugluleuglnleuargtyrasnglyvalgly290295300argvalthrserserglyprovalileasnhisglymettrpglnthr305310315320ileservalglugluleualaargasnleuvalilelysvalasnarg325330335aspalavalmetlysilealavalalaglyaspleupheglnproglu340345350argglyleutyrhisleuasnleuthrvalglyglyileprophehis355360365glulysaspleuvalglnproileasnproargleuaspglycysmet370375380argsertrpasntrpleuasnglygluaspthrthrileglngluthr385390395400vallysvalasnthrargmetglncyspheservalthrgluarggly405410415serphetyrproglyserglyphealaphetyrserleuasptyrmet420425430argthrproleuaspvalglythrgluserthrtrpgluvalgluval435440445valalahisileargproalaalaaspthrglyvalleuphealaleu450455460trpalaproaspleuargalavalproleuservalalaleuvalasp465470475480tyrhisserthrlyslysleulyslysglnleuvalvalleualaval485490495gluhisthralaleualaleumetgluilelysvalcysaspglygln500505510gluhisvalvalthrvalserleuargaspglyglualathrleuglu515520525valaspglythrargglyglnsergluvalseralaalaglnleugln530535540gluargleualavalleugluarghisleuargserprovalleuthr545550555560phealaglyglyleuproaspvalprovalthrseralaprovalthr565570575alaphetyrargglycysmetthrleugluvalasnargargleuleu580585590aspleuaspglualaalatyrlyshisseraspilethralahisser595600605cysproprovalgluproalaalaalaasptyrlysaspaspaspasp610615620lyshishishishishishis625630<210>101<211>27<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer30<400>101gagggcggcggccaccaggctggtagc27<210>102<211>29<212>dna<213>artificialsequence<220><223>descriptionoftheartificialsequence:primer31<400>102gtggccgccgccctcttgaagcgcagtcg29<210>103<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv0sequence<220><221>cds<222>(1)..(378)<400>103atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggatgttgtcatgactcagtcacccgactccttggcagtt96serserseraspvalvalmetthrglnserproaspserleualaval202530tcattgggggaacgtgtgactattaattgtaaagcctctcagtctgta144serleuglygluargvalthrileasncyslysalaserglnserval354045agcaacgatgtggcttggtatcagcagaagccaggccaatcccccaag192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560ctcttgatctactacgcctctaaccggtatactggagtgcctgacagg240leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttttctggttccggaagcgggactgattttacacttaccattagcagc288pheserglyserglyserglythraspphethrleuthrileserser859095ttgcaggctgaggatgtggccgtctattattgtcagcaggactactct336leuglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110tcaccttggacatttggccagggaaccaagttggaaattaag378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>104<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>104metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>105<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv0excludingsignalsequence<400>105aspvalvalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysleuleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrleuthrileserserleuglnala65707580gluaspvalalavaltyrtyrcysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>106<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv1asequence<220><221>cds<222>(1)..(378)<400>106atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggatgtggtgatgacacaatctccagactcactcgccgtg96serserseraspvalvalmetthrglnserproaspserleualaval202530agtctgggtgagcgagtcactatcaactgcaaggctagccaatccgtg144serleuglygluargvalthrileasncyslysalaserglnserval354045agtaatgatgtagcttggtaccagcaaaagcccggacagtcccccaaa192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560cttctgatttactacgcttctaaccgttatacaggcgtgcccgatcgc240leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttctctggatctgggagcgggactgactttaccctcacaatctctagc288pheserglyserglyserglythraspphethrleuthrileserser859095ctgcaagccgaggacgttgccgtctacttctgtcagcaggattacagc336leuglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110agcccatggacatttggtcaggggacaaagttggagattaaa378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>107<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>107metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>108<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv1aexcludingsignalsequence<400>108aspvalvalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysleuleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrleuthrileserserleuglnala65707580gluaspvalalavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>109<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv1bsequence<220><221>cds<222>(1)..(378)<400>109atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggacgtcgtgatgacacaatctccagactctcttgccgta96serserseraspvalvalmetthrglnserproaspserleualaval202530tcactgggtgagcgcgtgacaatcaactgcaaagccagtcagtccgtg144serleuglygluargvalthrileasncyslysalaserglnserval354045tccaatgacgttgcctggtatcaacagaaaccagggcagtcaccaaaa192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560ctgctcatctattatgccagtaataggtacacaggtgtgccagaccga240leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttcagcggctccggctctggtaccgacttcaccctcacaatatcatcc288pheserglyserglyserglythraspphethrleuthrileserser859095gttcaggccgaggacgttgccgtgtattattgccagcaggattacagc336valglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110tccccctggaccttcgggcaggggacaaagctcgaaatcaaa378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>110<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>110metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrleuthrileserser859095valglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>111<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv1bexcludingsignalsequence<400>111aspvalvalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysleuleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrleuthrileserservalglnala65707580gluaspvalalavaltyrtyrcysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>112<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv2asequence<220><221>cds<222>(1)..(378)<400>112atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggacgtggtgatgactcagtctcccgattctctcgccgtc96serserseraspvalvalmetthrglnserproaspserleualaval202530tccctgggggagcgggtaactatcaattgcaaggctagccagtcagtc144serleuglygluargvalthrileasncyslysalaserglnserval354045agtaacgatgtagcctggtaccagcaaaagccaggtcagtcccctaag192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560ctcttgatatactacgcatctaaccgttacaccggagtacccgaccgc240leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttctccgggagcggaagtggcaccgacttcaccctgaccatcagctct288pheserglyserglyserglythraspphethrleuthrileserser859095gtgcaggcagaggacgttgctgtctacttttgccagcaggactacagc336valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110tctccatggacattcggacaaggcactaaactcgagattaaa378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>113<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>113metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrleuthrileserser859095valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>114<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv2aexcludingsignalsequence<400>114aspvalvalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysleuleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrleuthrileserservalglnala65707580gluaspvalalavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>115<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv2bsequence<220><221>cds<222>(1)..(378)<400>115atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggatgtggtcatgacacagagcccagacagcctcgctgtg96serserseraspvalvalmetthrglnserproaspserleualaval202530agcgccggagaaagggtcaccattaactgcaaggcctctcaaagcgtg144seralaglygluargvalthrileasncyslysalaserglnserval354045agtaatgacgtcgcctggtatcaacagaagcctggtcaatccccaaag192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560ctgctcatatactatgctagtaatcggtacaccggcgtgccagaccgc240leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttctctggctcagggagtgggacagacttcacctttactatcagcagt288pheserglyserglyserglythraspphethrphethrileserser859095ctgcaggctgaagatgttgccgtgtattactgccagcaggactatagt336leuglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110agcccttggaccttcggacagggtactaagcttgaaatcaag378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>116<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>116metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530seralaglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560leuleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrphethrileserser859095leuglnalagluaspvalalavaltyrtyrcysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>117<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv2bexcludingsignalsequence<400>117aspvalvalmetthrglnserproaspserleualavalseralagly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysleuleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrphethrileserserleuglnala65707580gluaspvalalavaltyrtyrcysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>118<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv3sequence<220><221>cds<222>(1)..(378)<400>118atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggacgtggtgatgacacagtctcccgactctttggcagtc96serserseraspvalvalmetthrglnserproaspserleualaval202530agcctgggcgagcgggtcactatcaattgcaaagcatctcagagcgta144serleuglygluargvalthrileasncyslysalaserglnserval354045tcaaacgacgtagcttggtaccagcagaaacctggccagagccctaaa192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560gtattgatctactacgcaagcaaccgctatacaggcgtgcccgatcgc240valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttctccgggtctggctctggtacagattttacatttaccattagtagc288pheserglyserglyserglythraspphethrphethrileserser859095ctccaggccgaagacgtcgccgtctacttctgccagcaggactatagt336leuglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110agcccttggacattcgggcagggaacaaaactcgagatcaaa378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>119<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>119metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspvalvalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrphethrileserser859095leuglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>120<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv3excludingsignalsequence<400>120aspvalvalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysvalleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrphethrileserserleuglnala65707580gluaspvalalavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>121<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv5sequence<220><221>cds<222>(1)..(378)<400>121atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggacatcgtgatgacacagtcccccgactccctggccgtc96serserseraspilevalmetthrglnserproaspserleualaval202530tctctgggtgagagggtaaccatcaactgcaaggccagccaaagcgtt144serleuglygluargvalthrileasncyslysalaserglnserval354045agcaacgatgttgcttggtatcaacagaagcctggccaatcacccaag192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560gttctcatttattatgcctctaatagatacactggagtccctgataga240valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttcagtggctcaggcagcggcaccgacttcacattcactatctctagt288pheserglyserglyserglythraspphethrphethrileserser859095gtgcaggcagaggacgtggccgtgtatttctgccagcaggattacagc336valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110tccccatggaccttcggtcagggaactaaacttgagatcaag378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>122<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>122metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspilevalmetthrglnserproaspserleualaval202530serleuglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrphethrileserser859095valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>123<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv5excludingsignalsequence<400>123aspilevalmetthrglnserproaspserleualavalserleugly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysvalleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrphethrileserservalglnala65707580gluaspvalalavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>124<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320lv6sequence<220><221>cds<222>(1)..(378)<400>124atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggatattgttatgacacagtcccctgatagtctggctgtc96serserseraspilevalmetthrglnserproaspserleualaval202530agcgcaggcgagcgcgtaaccataaattgtaaggcctcccagagcgtc144seralaglygluargvalthrileasncyslysalaserglnserval354045tctaacgacgtcgcctggtaccaacagaagcccggacagtcacctaaa192serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560gtactcatttactatgcttcaaaccgttacacaggagtgcccgacagg240valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580ttctctggctccggatcaggcaccgattttacctttactatcagcagc288pheserglyserglyserglythraspphethrphethrileserser859095gtccaggctgaggacgtcgccgtatacttctgccagcaagactactca336valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110agcccatggacattcgggcaaggaacaaagctggagataaaa378serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>125<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>125metlysleuprovalargleuleuvalleumetphetrpileproala151015serserseraspilevalmetthrglnserproaspserleualaval202530seralaglygluargvalthrileasncyslysalaserglnserval354045serasnaspvalalatrptyrglnglnlysproglyglnserprolys505560valleuiletyrtyralaserasnargtyrthrglyvalproasparg65707580pheserglyserglyserglythraspphethrphethrileserser859095valglnalagluaspvalalavaltyrphecysglnglnasptyrser100105110serprotrpthrpheglyglnglythrlysleugluilelys115120125<210>126<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320lv6excludingsignalsequence<400>126aspilevalmetthrglnserproaspserleualavalseralagly151015gluargvalthrileasncyslysalaserglnservalserasnasp202530valalatrptyrglnglnlysproglyglnserprolysvalleuile354045tyrtyralaserasnargtyrthrglyvalproaspargphesergly505560serglyserglythraspphethrphethrileserservalglnala65707580gluaspvalalavaltyrphecysglnglnasptyrserserprotrp859095thrpheglyglnglythrlysleugluilelys100105<210>127<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv0sequence<220><221>cds<222>(1)..(414)<400>127atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcaacttgtacagtccggtagtgaactgaaaaaa96valglncysglnvalglnleuvalglnserglysergluleulyslys202530cctggcgctagcgtcaaggtctcctgcaaggcatctggctacaccttc144proglyalaservallysvalsercyslysalaserglytyrthrphe354045accaactatggaatgaactgggttaggcaggcaccaggacaaggtctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gagtggatgggatggattaatacagaaacaggggagcctacatattca240glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgacttcaagggccggttcgtgttttccctggataccagcgtctcc288aspaspphelysglyargphevalpheserleuaspthrservalser859095acagcctatttgcagatcagtagcctgaaggctgaggacacagccgtt336thralatyrleuglnileserserleulysalagluaspthralaval100105110tactactgtgctcgggaagatggctattacgggacccttgactactgg384tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125gggcagggcaccacagtgacagtatcctcc414glyglnglythrthrvalthrvalserser130135<210>128<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>128metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglysergluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralaval100105110tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>129<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv0excludingsignalsequence<400>129glnvalglnleuvalglnserglysergluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralavaltyrtyrcys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>130<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv1sequence<220><221>cds<222>(1)..(414)<400>130atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaagtgcagctggtgcaaagcggcagtgaactgaagaag96valglncysglnvalglnleuvalglnserglysergluleulyslys202530cctggcgctagtgtgaaggtttcttgcaaggcttctggctacaccttt144proglyalaservallysvalsercyslysalaserglytyrthrphe354045actaattacggcatgaactgggtgcgtcaggcacctggccagggcctc192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560aaatggatggggtggattaacactgagaccggtgagcccacatactca240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gatgattttaaaggccggtttgtcttttccctggacacatctgttagc288aspaspphelysglyargphevalpheserleuaspthrservalser859095accgcttatctgcagatatcaagtctgaaagccgaagatactgccgtg336thralatyrleuglnileserserleulysalagluaspthralaval100105110tattattgtgcccgcgaagatggttactatggaaccctggactactgg384tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggccagggcactaccgttactgtgtccagt414glyglnglythrthrvalthrvalserser130135<210>131<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>131metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglysergluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralaval100105110tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>132<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv1excludingsignalsequence<400>132glnvalglnleuvalglnserglysergluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralavaltyrtyrcys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>133<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv2sequence<220><221>cds<222>(1)..(414)<400>133atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaagttcagcttgtgcagagtggaagcgaactcaagaaa96valglncysglnvalglnleuvalglnserglysergluleulyslys202530cccggtgcctctgtaaaagtctcatgtaaagcttcaggctacacattt144proglyalaservallysvalsercyslysalaserglytyrthrphe354045acaaattatggtatgaattgggtccgacaggctcccggtcaaggactc192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gaatggatgggctggataaatactgagacaggggagcccacttactct240glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgatttcaagggcagattcgtgttcagcttggacacatcagtgtca288aspaspphelysglyargphevalpheserleuaspthrservalser859095accgcttatttgcagatttccagcctgaaagctgaggatactgctact336thralatyrleuglnileserserleulysalagluaspthralathr100105110tatttctgtgcccgcgaggatgggtactatggaactctggactactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggacagggcacaactgtcactgttagcagt414glyglnglythrthrvalthrvalserser130135<210>134<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>134metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglysergluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>135<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv2excludingsignalsequence<400>135glnvalglnleuvalglnserglysergluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>136<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv3asequence<220><221>cds<222>(1)..(414)<400>136atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcagcttgtgcagtctggatctgaacttaaaaag96valglncysglnvalglnleuvalglnserglysergluleulyslys202530cctggcgcttcagtcaaggtttcttgtaaggcaagcggatacaccttt144proglyalaservallysvalsercyslysalaserglytyrthrphe354045actaattatggtatgaattgggtacgccaagccccaggccaaggtctc192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560aagtggatgggttggattaacaccgagaccggagagcctacctattcc240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgatttcaagggccgcttcgtgttctcattggacactagcgtgagt288aspaspphelysglyargphevalpheserleuaspthrservalser859095accgcctacctccagatctccagtttgaaagcagaggacacagccacc336thralatyrleuglnileserserleulysalagluaspthralathr100105110tacttctgtgccagggaggacggatactacggaaccctggactactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125gggcagggcacaaccgtgacagtgtcctct414glyglnglythrthrvalthrvalserser130135<210>137<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>137metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglysergluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>138<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv3aexcludingsignalsequence<400>138glnvalglnleuvalglnserglysergluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>139<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv3bsequence<220><221>cds<222>(1)..(414)<400>139atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccagatacagctggtgcagtccggcagcgaactgaaaaag96valglncysglnileglnleuvalglnserglysergluleulyslys202530cctggggcctctgtcaaaatctcttgcaaggctagtgggtacacattt144proglyalaservallysilesercyslysalaserglytyrthrphe354045acaaactacggtatgaattgggtgcggcaagccccaggtcagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gaatggatgggttggattaatacagagactggggagccaacttacagc240glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gatgatttcaaaggtcgcttcgtcttcagcctcgacacttctgtctct288aspaspphelysglyargphevalpheserleuaspthrservalser859095accgcttacttgcagatcagcagcctgaaggcagaagacaccgcagtc336thralatyrleuglnileserserleulysalagluaspthralaval100105110tatttctgtgccagggaagatggctactacggcactctggattattgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggtcagggtactaccgtgactgtcagcagt414glyglnglythrthrvalthrvalserser130135<210>140<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>140metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnileglnleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralaval100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>141<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv3bexcludingsignalsequence<400>141glnileglnleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralavaltyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>142<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv3csequence<220><221>cds<222>(1)..(414)<400>142atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggttcagctggtgcagagcgggcccgagctgaaaaag96valglncysglnvalglnleuvalglnserglyprogluleulyslys202530cctggcgctagtgtaaaggtttcatgtaaggctagcggctataccttt144proglyalaservallysvalsercyslysalaserglytyrthrphe354045actaactatggtatgaactgggtcaaacaagcccctggacaaggtttg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aaatggatggggtggatcaatacagaaaccggtgagccaacatattct240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgacttcaaaggccgcttcgtcttctctctggatacatccgtgagc288aspaspphelysglyargphevalpheserleuaspthrservalser859095actgcctacctgcagatcagcagcctcaaggctgaagacacagcagtg336thralatyrleuglnileserserleulysalagluaspthralaval100105110tattattgcgctagggaggacggatactatggcactctggattactgg384tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggtcagggcaccaccgtgactgtttctagc414glyglnglythrthrvalthrvalserser130135<210>143<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>143metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglyprogluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralaval100105110tyrtyrcysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>144<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv3cexcludingsignalsequence<400>144glnvalglnleuvalglnserglyprogluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralavaltyrtyrcys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>145<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv4sequence<220><221>cds<222>(1)..(414)<400>145atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggttcagttggttcagtctggccccgaactgaaaaag96valglncysglnvalglnleuvalglnserglyprogluleulyslys202530cccggggcaagcgtgaaggtctcttgtaaggcaagcggctatacattt144proglyalaservallysvalsercyslysalaserglytyrthrphe354045acaaactacggcatgaactgggtgcgacaggctcctggccaggggctt192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560aagtggatgggatggatcaacacagagacaggtgaaccaacatatagc240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgacttcaagggcagatttgtcttttccttggacacttctgtctcc288aspaspphelysglyargphevalpheserleuaspthrservalser859095accgcctacctgcagatttctagcctgaaggcagaggacacagccaca336thralatyrleuglnileserserleulysalagluaspthralathr100105110tacttttgcgcccgcgaagacggctactacgggacactggactactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggacaaggtaccaccgtgacagtttctagc414glyglnglythrthrvalthrvalserser130135<210>146<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>146metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglyprogluleulyslys202530proglyalaservallysvalsercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>147<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv4excludingsignalsequence<400>147glnvalglnleuvalglnserglyprogluleulyslysproglyala151015servallysvalsercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>148<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv6sequence<220><221>cds<222>(1)..(414)<400>148atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcagctggtgcagtcaggacccgagctgaagaag96valglncysglnvalglnleuvalglnserglyprogluleulyslys202530ccaggagcatcagtaaagatttcttgtaaagcttcaggctacacattt144proglyalaservallysilesercyslysalaserglytyrthrphe354045actaattatgggatgaattgggttaaacaggctcccggtcagggtctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggatggattaataccgaaactggagagccaacctactct240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gatgacttcaaagggcgcttcgtgttcagcctggatacttctgtttct288aspaspphelysglyargphevalpheserleuaspthrservalser859095actgcctatctgcagatctcctccctcaaggccgaggacaccgctact336thralatyrleuglnileserserleulysalagluaspthralathr100105110tacttctgcgcccgagaagacggatattacggcaccctggattactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggccaaggcacaacagtcaccgtatcatct414glyglnglythrthrvalthrvalserser130135<210>149<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>149metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalglnleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalser859095thralatyrleuglnileserserleulysalagluaspthralathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>150<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv6excludingsignalsequence<400>150glnvalglnleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalserthralatyr65707580leuglnileserserleulysalagluaspthralathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>151<211>414<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5320hv8sequence<220><221>cds<222>(1)..(414)<400>151atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccagattcagcttgtgcagtccggtccagaactgaagaag96valglncysglnileglnleuvalglnserglyprogluleulyslys202530ccaggagcaagcgtaaagatctcttgtaaggcatctggttacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045acaaactacggcatgaattgggtaaagcaagcacccggtcaggggctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggttggattaatactgaaaccggagagcctacatattct240lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580gacgacttcaaaggacggttcgtcttctcactggatacaagtgtaaca288aspaspphelysglyargphevalpheserleuaspthrservalthr859095accgcctacctgcagatttcctctttgaaggcagaagacacagccacc336thralatyrleuglnileserserleulysalagluaspthralathr100105110tacttctgcgctcgagaagatggttactatggaaccctggattactgg384tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125ggacagggtaccacagtcactgtgtcttcc414glyglnglythrthrvalthrvalserser130135<210>152<211>138<212>prt<213>artificial<220><223>syntheticconstruct<400>152metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnileglnleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrgluthrglygluprothrtyrser65707580aspaspphelysglyargphevalpheserleuaspthrservalthr859095thralatyrleuglnileserserleulysalagluaspthralathr100105110tyrphecysalaarggluaspglytyrtyrglythrleuasptyrtrp115120125glyglnglythrthrvalthrvalserser130135<210>153<211>119<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5320hv8excludingsignalsequence<400>153glnileglnleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrgluthrglygluprothrtyrseraspaspphe505560lysglyargphevalpheserleuaspthrservalthrthralatyr65707580leuglnileserserleulysalagluaspthralathrtyrphecys859095alaarggluaspglytyrtyrglythrleuasptyrtrpglyglngly100105110thrthrvalthrvalserser115<210>154<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv0sequence<220><221>cds<222>(1)..(378)<400>154atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgct96sersersergluthrthrleuthrglnserproserserleuserala202530tccgtgggggatcgcgtgaccatcacctgtatcacttccactgacatc144servalglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcactctgactatctcctcc288pheserglyserglyserglythraspphethrleuthrileserser859095ctgcagcccgaggacttcgccacctactactgcctgcagactgattcc336leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>155<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>155metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserala202530servalglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>156<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv0excludingsignalsequence<400>156gluthrthrleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>157<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv1asequence<220><221>cds<222>(1)..(378)<400>157atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgct96sersersergluthrthrleuthrglnserproserserleuserala202530tccgtgggggatcgcgtgactatcacctgtatcacttccactgacatc144servalglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaagcctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcactctgactatctcctcc288pheserglyserglyserglythraspphethrleuthrileserser859095ctgcagcccgaggacttcgccacctactactgcctgcagactgattcc336leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>158<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>158metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserala202530servalglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>159<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv1aexcludingsignalsequence<400>159gluthrthrleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysproprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>160<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv1bsequence<220><221>cds<222>(1)..(378)<400>160atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgct96sersersergluthrthrleuthrglnserproserserleuserala202530tccgtgggggatcgcgtgaccatcacctgtatcacttccactgacatc144servalglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcactctgactatctcctcc288pheserglyserglyserglythraspphethrleuthrileserser859095ctgcagcccgaggacttcgccgactactactgcctgcagactgattcc336leuglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>161<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>161metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserala202530servalglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>162<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv1bexcludingsignalsequence<400>162gluthrthrleuthrglnserproserserleuseralaservalgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>163<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv1csequence<220><221>cds<222>(1)..(378)<400>163atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgtg96sersersergluthrthrleuthrglnserproserserleuserval202530tccgtgggggatcgcgtgaccatcacctgtatcacttccactgacatc144servalglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcactctgactatctcctcc288pheserglyserglyserglythraspphethrleuthrileserser859095ctgcagcccgaggacttcgccacctactactgcctgcagactgattcc336leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>164<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>164metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserval202530servalglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileserser859095leuglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>165<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv1cexcludingsignalsequence<400>165gluthrthrleuthrglnserproserserleuservalservalgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserleuglnpro65707580gluaspphealathrtyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>166<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv3sequence<220><221>cds<222>(1)..(378)<400>166atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgcc96sersersergluthrthrleuthrglnserproserserleuserala202530tccactggggatcgcgtgaccatcacctgtatcacttccactgacatc144serthrglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcactctgactatctcctcc288pheserglyserglyserglythraspphethrleuthrileserser859095actcagcccgaggacttcgccgactactactgcctgcagactgattcc336thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>167<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>167metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserala202530serthrglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrleuthrileserser859095thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>168<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv3excludingsignalsequence<400>168gluthrthrleuthrglnserproserserleuseralaserthrgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrleuthrileserserthrglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>169<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv4sequence<220><221>cds<222>(1)..(378)<400>169atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgtg96sersersergluthrthrleuthrglnserproserserleuserval202530tccactggggatcgcgtgactatcacctgtatcacttccactgacatc144serthrglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctccggctctggctctgggactgacttcaccttcactatctcctcc288pheserglyserglyserglythraspphethrphethrileserser859095actcagcccgaggacttcgccacctactactgcctgcagactgattcc336thrglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>170<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>170metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserval202530serthrglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserglyserglyserglythraspphethrphethrileserser859095thrglnprogluaspphealathrtyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>171<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv4excludingsignalsequence<400>171gluthrthrleuthrglnserproserserleuservalserthrgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargphesergly505560serglyserglythraspphethrphethrileserserthrglnpro65707580gluaspphealathrtyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>172<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv6sequence<220><221>cds<222>(1)..(378)<400>172atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgtg96sersersergluthrthrleuthrglnserproserserleuserval202530tccactggggatcgcgtgaccatcacctgtatcacttccactgacatc144serthrglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaagcctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctcctcttccggctccgggactgacttcactctgactatctcctcc288pheserserserglyserglythraspphethrleuthrileserser859095actcagcccgaggacttcgccgactactactgcctgcagactgattcc336thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>173<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>173metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserval202530serthrglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserserserglyserglythraspphethrleuthrileserser859095thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>174<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv6excludingsignalsequence<400>174gluthrthrleuthrglnserproserserleuservalserthrgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysproprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargpheserser505560serglyserglythraspphethrleuthrileserserthrglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>175<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv7asequence<220><221>cds<222>(1)..(378)<400>175atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactctgactcagtccccctcctccctgtccgtg96sersersergluthrthrleuthrglnserproserserleuserval202530tccactggggatcgcgtgactatcacctgtatcacttccactgacatc144serthrglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaagcctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctcctcttccggctccgggactgacttcaccttcactatctcctcc288pheserserserglyserglythraspphethrphethrileserser859095actcagcccgaggacttcgccgactactactgcctgcagactgattcc336thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaaagtggagatcaag378valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>176<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>176metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrleuthrglnserproserserleuserval202530serthrglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserserserglyserglythraspphethrphethrileserser859095thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysvalgluilelys115120125<210>177<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv7aexcludingsignalsequence<400>177gluthrthrleuthrglnserproserserleuservalserthrgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysproprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargpheserser505560serglyserglythraspphethrphethrileserserthrglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysvalgluilelys100105<210>178<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv7bsequence<220><221>cds<222>(1)..(378)<400>178atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactgtgactcagtccccctcctccctgtccgtg96sersersergluthrthrvalthrglnserproserserleuserval202530tccgtgggggatcgcgtgactatcacctgtatcacttccactgacatc144servalglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaaggctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctcctcttccggctccgggactgacttcaccttcactatctcctcc288pheserserserglyserglythraspphethrphethrileserser859095actcagcccgaggacttcgccgactactactgcctgcagactgattcc336thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaagctggagatcaag378valproleuthrpheglyglnglythrlysleugluilelys115120125<210>179<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>179metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrvalthrglnserproserserleuserval202530servalglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysalaprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserserserglyserglythraspphethrphethrileserser859095thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysleugluilelys115120125<210>180<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv7bexcludingsignalsequence<400>180gluthrthrvalthrglnserproserserleuservalservalgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysalaprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargpheserser505560serglyserglythraspphethrphethrileserserthrglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysleugluilelys100105<210>181<211>378<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321lv9sequence<220><221>cds<222>(1)..(378)<400>181atgaagctgcccgttcgcctgcttgtgctgatgttctggatccccgcg48metlysleuprovalargleuleuvalleumetphetrpileproala151015tcctcctcggagactactgtgactcagtccccctcctccctgtccgtg96sersersergluthrthrvalthrglnserproserserleuserval202530tccactggggatcgcgtgactatcacctgtatcacttccactgacatc144serthrglyaspargvalthrilethrcysilethrserthraspile354045gacgatgacatgaactggtaccagcagaagcctgggaagcctcccaag192aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560ctgctgatctccgaggggaacactctgcgccctggcgttccttcccgc240leuleuilesergluglyasnthrleuargproglyvalproserarg65707580ttctcctcttccggctccgggactgacttcaccttcactatctcctcc288pheserserserglyserglythraspphethrphethrileserser859095actcagcccgaggacttcgccgactactactgcctgcagactgattcc336thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110gtgcccctgactttcgggcaggggactaagctggagatcaag378valproleuthrpheglyglnglythrlysleugluilelys115120125<210>182<211>126<212>prt<213>artificial<220><223>syntheticconstruct<400>182metlysleuprovalargleuleuvalleumetphetrpileproala151015sersersergluthrthrvalthrglnserproserserleuserval202530serthrglyaspargvalthrilethrcysilethrserthraspile354045aspaspaspmetasntrptyrglnglnlysproglylysproprolys505560leuleuilesergluglyasnthrleuargproglyvalproserarg65707580pheserserserglyserglythraspphethrphethrileserser859095thrglnprogluaspphealaasptyrtyrcysleuglnthraspser100105110valproleuthrpheglyglnglythrlysleugluilelys115120125<210>183<211>107<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321lv9excludingsignalsequence<400>183gluthrthrvalthrglnserproserserleuservalserthrgly151015aspargvalthrilethrcysilethrserthraspileaspaspasp202530metasntrptyrglnglnlysproglylysproprolysleuleuile354045sergluglyasnthrleuargproglyvalproserargpheserser505560serglyserglythraspphethrphethrileserserthrglnpro65707580gluaspphealaasptyrtyrcysleuglnthraspservalproleu859095thrpheglyglnglythrlysleugluilelys100105<210>184<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321hv0sequence<220><221>cds<222>(1)..(405)<400>184atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggctccgagctgaagaag96valglncysglnvalhisleuvalglnserglysergluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgagacaggctcctggccagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gaatggatgggctggatcaacaccaacactggcgagcctacctacacc240glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctgtg336thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125accctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>185<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>185metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>186<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv0excludingsignalsequence<400>186glnvalhisleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralavaltyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>187<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321hv1sequence<220><221>cds<222>(1)..(405)<400>187atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggctccgagctgaagaag96valglncysglnvalhisleuvalglnserglysergluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgagacaggctcctggccagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacactggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctgtg336thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>188<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>188metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>189<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv1excludingsignalsequence<400>189glnvalhisleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralavaltyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>190<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321hv2asequence<220><221>cds<222>(1)..(405)<400>190atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggctccgagctgaagaag96valglncysglnvalhisleuvalglnserglysergluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgaagcaggctcctggccagggcctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacactggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctgtg336thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>191<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>191metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralaval100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>192<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv2aexcludingsignalsequence<400>192glnvalhisleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralavaltyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>193<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv2bsequence<220><221>cds<222>(1)..(405)<400>193atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggccctgagctgaagaag96valglncysglnvalhisleuvalglnserglyprogluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgagacaggctcctggccagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gaatggatgggctggatcaacaccaacactggcgagcctacctacacc240glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctacc336thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125accctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>194<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>194metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>195<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv2bexcludingsignalsequence<400>195glnvalhisleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>196<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv3asequence<220><221>cds<222>(1)..(405)<400>196atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggccctgagctgaagaag96valglncysglnvalhisleuvalglnserglyprogluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgagacaggctcctggccagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacactggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctacc336thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>197<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>197metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>198<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv3aexcludingsignalsequence<400>198glnvalhisleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>199<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:hzkm5321hv3bsequence<220><221>cds<222>(1)..(405)<400>199atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggctccgagctgaagaag96valglncysglnvalhisleuvalglnserglysergluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgaagcaggctcctggccagggcctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacactggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctacc336thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>200<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>200metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>201<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv3bexcludingsignalsequence<400>201glnvalhisleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>202<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv4asequence<220><221>cds<222>(1)..(405)<400>202atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccagatccacctcgtgcagtctggccctgagctgaagaag96valglncysglnilehisleuvalglnserglyprogluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgagacaggctcctggccagggcctg192thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560gaatggatgggctggatcaacaccaacaccggcgagcctacctacacc240glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctccgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095accgcttacctgcagatctccaccctgaaggctgaggataccgctgtg336thralatyrleuglnileserthrleulysalagluaspthralaval100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125accctgatcaccgtgtcctcc405thrleuilethrvalserser130135<210>203<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>203metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnilehisleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvalargglnalaproglyglnglyleu505560glutrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thralatyrleuglnileserthrleulysalagluaspthralaval100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuilethrvalserser130135<210>204<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv4aexcludingsignalsequence<400>204glnilehisleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvalargglnalaproglyglnglyleuglutrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthralatyr65707580leuglnileserthrleulysalagluaspthralavaltyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuile100105110thrvalserser115<210>205<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv4bsequence<220><221>cds<222>(1)..(405)<400>205atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccaggtgcatctcgtgcagtctggccctgagctgaagaag96valglncysglnvalhisleuvalglnserglyprogluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgaagcaggctcctggccagggcctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacactggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctccctggacacctctgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095acctcctacctgcagatctccaccctgaaggctgaggataccgctacc336thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125actctcgtgaccgtgtcctcc405thrleuvalthrvalserser130135<210>206<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>206metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnvalhisleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thrsertyrleuglnileserthrleulysalagluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuvalthrvalserser130135<210>207<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv4bexcludingsignalsequence<400>207glnvalhisleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthrsertyr65707580leuglnileserthrleulysalagluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuval100105110thrvalserser115<210>208<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv5sequence<220><221>cds<222>(1)..(405)<400>208atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccagatccacctcgtgcagtctggctccgagctgaagaag96valglncysglnilehisleuvalglnserglysergluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgaagcaggctcctggccagggcctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacaccggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctctctggacacctccgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095accgcttacctgcagatctccaccctgaaggctgaggataccgctgtg336thralatyrleuglnileserthrleulysalagluaspthralaval100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125accctgatcaccgtgtcctcc405thrleuilethrvalserser130135<210>209<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>209metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnilehisleuvalglnserglysergluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thralatyrleuglnileserthrleulysalagluaspthralaval100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuilethrvalserser130135<210>210<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv5excludingsignalsequence<400>210glnilehisleuvalglnserglysergluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthralatyr65707580leuglnileserthrleulysalagluaspthralavaltyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuile100105110thrvalserser115<210>211<211>405<212>dna<213>artificial<220><223>descriptionofartificialsequence:丂hzkm5321hv7sequence<220><221>cds<222>(1)..(405)<400>211atgaatctggggctgtcgctgatcttcctggcgctgatcctgaagggc48metasnleuglyleuserleuilepheleualaleuileleulysgly151015gtgcagtgccagatccacctcgtgcagtctggccctgagctgaagaag96valglncysglnilehisleuvalglnserglyprogluleulyslys202530cctggcgcttctgtgaagatctcctgcaaggcttctggctacaccttc144proglyalaservallysilesercyslysalaserglytyrthrphe354045accaactacggcatgaactgggtgaagcaggctcctggccagggcctg192thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560aagtggatgggctggatcaacaccaacaccggcgagcctacctacacc240lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580gaggagttcaagggcagattcgtgttctctctggacacctccgtgacc288glugluphelysglyargphevalpheserleuaspthrservalthr859095accgcttacctgcagatctccaccctgaaggctgaggataccgctacc336thralatyrleuglnileserthrleulysalagluaspthralathr100105110tacttctgcgctcgagatgagggctggtttgtgtactggggccagggc384tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125accctgatcaccgtgtcctcc405thrleuilethrvalserser130135<210>212<211>135<212>prt<213>artificial<220><223>syntheticconstruct<400>212metasnleuglyleuserleuilepheleualaleuileleulysgly151015valglncysglnilehisleuvalglnserglyprogluleulyslys202530proglyalaservallysilesercyslysalaserglytyrthrphe354045thrasntyrglymetasntrpvallysglnalaproglyglnglyleu505560lystrpmetglytrpileasnthrasnthrglygluprothrtyrthr65707580glugluphelysglyargphevalpheserleuaspthrservalthr859095thralatyrleuglnileserthrleulysalagluaspthralathr100105110tyrphecysalaargaspgluglytrpphevaltyrtrpglyglngly115120125thrleuilethrvalserser130135<210>213<211>116<212>prt<213>artificial<220><223>descriptionofartificialsequence:aminoacidsequenceofhzkm5321hv7excludingsignalsequence<400>213glnilehisleuvalglnserglyprogluleulyslysproglyala151015servallysilesercyslysalaserglytyrthrphethrasntyr202530glymetasntrpvallysglnalaproglyglnglyleulystrpmet354045glytrpileasnthrasnthrglygluprothrtyrthrglugluphe505560lysglyargphevalpheserleuaspthrservalthrthralatyr65707580leuglnileserthrleulysalagluaspthralathrtyrphecys859095alaargaspgluglytrpphevaltyrtrpglyglnglythrleuile100105110thrvalserser115当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1