强直性脊柱炎微生物标志物的制作方法
本发明涉及生物标志物领域,具体的,本发明涉及强直性脊柱炎微生物标志物,更具体的,本发明涉及一种强直性脊柱炎标志物、强直性脊柱炎标志物的用途、一种利用强直性脊柱炎标志物确定个体的状态的方法、一种利用强直性脊柱炎标志物确定个体的状态的装置、一种利用强直性脊柱炎标志物对多个个体进行分类的方法、一种治疗强直性脊柱炎的药物以及一种制备治疗强直性脊柱炎药物的方法。
背景技术:
强直性脊柱炎(ankylosingspondylitis,as)是一种主要侵犯脊柱,并累及骶髂关节和周围关节的慢性进行性炎性疾病。起病多为15~30岁的男性,现在研究显示,该病男女之比为2:1~3:1,强直性脊柱炎的病因前尚未完全明确,近年来研究结果表明该病与遗传素质、感染及免疫等因素有关。
强直性脊柱炎一般起病比较隐匿,早期可无任何临床症状,有些病人在早期可表现出轻度的全身症状,如乏力、消瘦、长期或间断低热、厌食、轻度贫血等。由于病情较轻,病人大多不能早期发现,致使病情延误,失去最佳治疗时机。
强直性脊柱炎与人体白细胞抗原hla-b27密切相关,据流行病学调查,强直性脊柱炎病人hla-b27阳性率高达90%~96%,而普通人群hla-27阳性率仅4%~9%;hla-b27阳性者强直性脊柱炎发病率约为10%~20%,而普通人群发病为1‰~2‰,相差约100倍。虽然hla-b27检测对强直性脊柱炎的诊断有一定的帮助,但绝大部分的患者还要通过病史、体征和x线检查作出诊断。目前,强直性脊柱炎的早期诊断主要是用ct、放射性核扫描、磁共振等影像学进行检查,检查过程较复杂。
随着人体基因组测序完成及高通量测序技术的高速发展,基因筛查成为强直性脊柱炎诊断的方向,对于发现强直性脊柱炎潜在人群很有优势。研究表明超过70%的强直性脊柱炎患者患有肠道炎症,这些患者中5~10%肠道炎症很严重,可能会发展为临床上的炎症性肠病(ibd)或克罗恩氏病(cd)(mielantsetal.,1985)。克罗恩氏病(cd)的一些标记基因与强直性脊柱炎相关联(parkesetal.,2013),说明两种疾病可能有类似发病机制,可能与肠道失调相关。有研究表明与强直性脊柱炎相关的多基因在肠道免疫中起重要作用,如涉及 到白介素il-23途径的基因对肠道健康的调节起重要作用(wellcomeetal.,2007)。与健康对照相比,强直性脊柱炎患者及其直系亲属肠道通透性增加,再次说明肠道微生物在强直性脊柱炎中的重要作用(mielantsetal.,1991)。到目前为止,还没有强直性脊柱炎患者肠道微生物标记物的相关报道。
技术实现要素:
本发明旨在至少在一定程度上解决上述技术问题之一或至少提供一种商业选择。
依据本发明的第一方面,提供一种强直性脊柱炎微生物标志物,所述标志物由以下微生物组成:放线菌(actinomycessp.icm47)、短双歧杆菌(bifidobacteriumbreve)、双歧杆菌(bifidobacteriumsp.12_1_47bfaa)、长双歧杆菌(bifidobacteriumlongum)、产气柯林斯菌(collinsellaaerofaciens)、两岐双岐杆菌(bifidobacteriumbifidum)和假小链双歧杆菌(bifidobacteriumpseudocatenulatum)。
上述本发明一方面提供的强直性脊柱炎微生物标志物是发明人通过对大量患强直性脊柱炎个体和大量健康对照个体的粪便样本中的微生物的丰度的差异比较分析、以及验证,而确定下来的,明确了肠道微生物中强直性脊柱炎相关的微生物标志物。利用所称的强直性脊柱微生物炎标志物,能够确定个体处于患有强直性脊柱炎状态的概率高低或者处于健康状态的概率高低,能够用于非侵入性的早期发现或辅助检测强直性脊柱炎。
依据本发明的第二方面,本发明提供上述强直性脊柱炎微生物标志物在检测强直性脊柱炎、监测治疗强直性脊柱炎、制备治疗强直性脊柱炎的药物和/或制备功能性食品中的用途。
上述强直性脊柱炎微生物标志物是发明人通过差异比较分析各种肠道微生物在强直性脊柱炎疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。相较于在健康群体组,所称强直性脊柱炎标志物在强直性脊柱炎患者中显著富集,所称显著富集是指与在健康组中的丰度相比,上述强直性脊柱炎标志物所包含的各种微生物在疾病组中的丰度均具有统计意义地高于或者明显地、实质性地高于在健康组中的丰度。能够使其丰度减小的物质能够用于治疗强直性脊柱炎或者益于强直性脊柱炎患者服用,能够使其丰度减少的物质不限于治疗强直性脊柱炎的药物和有益肠道菌群平衡的功能性食品,上述本发明一方面提供的强直性脊柱炎标志物能够用于制备治疗强直性脊柱炎的药物和/或用于制备益于平衡肠道菌群的功能性食品、保健药等。
依据本发明的第三方面,提供一种利用上述强直性脊柱炎微生物标志物确定个体的状态的方法,该方法包括:(1)确定所述个体的粪便样本中的所述强直性脊柱炎标志物中的 各种微生物的丰度;(2)分别将(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度与其在对照组中的丰度进行比较,依据获得的比较结果确定所述个体的状态,所述对照组由一组或多组相同状态的个体的粪便样本组成,所述状态包括患有强直性脊柱炎和不患有强直性脊柱炎。所称强直性脊柱炎标志物是发明人通过差异比较分析各种肠道微生物在强直性脊柱炎疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。
上述本发明这一方面的方法基于检测个体的粪便样本中的强直性脊柱炎微生物标志物中的各种微生物的丰度,分别将检测确定的各种微生物的丰度与其在对照组中丰度进行比较,依据获得的比较结果能够确定个体为强直性脊柱炎个体或者为健康个体的概率。为早期发现强直性脊柱炎提供一种非侵入性的辅助检测方法。
上述本发明的这一方面的利用强直性脊柱炎微生物标志物确定个体的状态的方法的全部或部分步骤,可以利用包含可拆分的相应单元功能模块的装置/系统来施行,或者将方法程序化、存储于机器可读介质,利用机器运行该可读介质来实现。
依据本发明的第四方面,提供一种利用上述强直性脊柱炎微生物标志物确定个体的状态的装置,该装置用以实施本发明一方面的利用强直性脊柱炎微生物标志物确定个体的状态的方法的全部或部分步骤,该装置包括:数据输入单元,用于输入数据;数据输出单元,用于输出数据;处理器,用于执行可执行程序,执行所述可执行程序包括完成上述利用强直性脊柱炎确定个体的状态的方法的所有或部分步骤;存储单元,与所述数据输入单元、所述数据输出单元和所述处理器相连,用于存储数据,其中包括所述可执行程序。上述对本发明一方面的利用强直性脊柱炎微生物标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的装置,在此不再赘述。
依据本发明的第五方面,提供一种利用上述强直性脊柱炎微生物标志物确定个体状态的系统,该系统用以实施本发明一方面的利用强直性脊柱炎微生物标志物确定个体的状态的方法的全部或部分步骤,该系统包括:微生物丰度确定单元,用于确定所述个体的粪便样本中的所述强直性脊柱炎标志物中的各种微生物的丰度;个体状态确定单元,用于分别将来自所述微生物丰度确定单元的所述强直性脊柱炎标志物中的各种微生物的丰度与其在对照组中的丰度进行比较,依据获得的比较结果确定所述个体的状态,所述对照组由一组或多组相同状态的个体的粪便样本组成,所述状态包括患有强直性脊柱炎和不患有强直性脊柱炎。上述对本发明一方面的利用强直性脊柱炎标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的系统,在此不再赘述。
依据本发明的第六方面,提供一种利用上述强直性脊柱炎微生物标志物对多个个体进 行分类的方法,该方法包括:分别利用上述本发明一方面的确定个体状态的方法确定各个个体的状态;依据获得的各个个体的状态对所述各个个体进行分类。该方法能够依据个体的状态的不同区分开多个个体或者区分开多个未知的粪便样本,便于归类、标记管理。另外,上述对本发明一方面的利用强直性脊柱炎微生物标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的方法,在此不再赘述。
依据本发明的第七方面,提供一种治疗强直性脊柱炎的药物,所述药物促使上述强直性脊柱炎微生物标志物中的各种微生物的丰度减少。所称强直性脊柱炎微生物标志物是发明人通过差异分析各种肠道微生物在强直性脊柱炎疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。相较于在健康群体组,所称强直性脊柱炎标志物在强直性脊柱炎疾病组中显著富集,所称显著富集是指与在健康组中的丰度相比,上述强直性脊柱炎标志物所包含的各种微生物在疾病组中的丰度均具有统计意义地高于或者明显地、实质性地高于在健康组中的丰度。能够使其丰度减少的物质能够用于治疗强直性脊柱炎或者益于强直性脊柱炎患者服用,能够使其丰度减少的物质可以作为治疗强直性脊柱炎的药物。
利用本发明这一方面的药物或者功能性食品,合理有效地应用确定的强直性脊柱炎微生物标志物,扶持肠道有益菌的生长和/或抑制肠道潜在致病菌,对于辅助降低血内毒素水平和/或减轻强直性脊柱炎的临床症状具有重要意义。
依据本发明的第八方面,本发明提供一种制备或筛选上述本发明一方面的治疗强直性脊柱炎的药物的方法,该方法包括制备或者筛选能够促使上述本发明一方面的强直性脊柱炎标志物中的各个微生物的丰度减少的物质作为所述药物的步骤。
利用本发明这一方面的生产或筛选治疗强直性脊柱炎的药物的方法,通过合理有效地应用确定的强直性脊柱炎生物标志物进行筛选,能够获得能扶持肠道有益菌的生长和/或抑制肠道潜在致病菌的药物,对于辅助降低血内毒素水平和/或减轻强直性脊柱炎的临床症状具有重要意义。
附图说明
本发明的上述和/或附加的方面和优点从结合下面附图对实施方式的描述中将变得明显和容易理解,其中:
图1是本发明的实施例中的筛选鉴定强直性脊柱炎微生物标志物的试验分析流程示意图。
图2是本发明的实施例中的微生物物种标志物作为诊断指标的auc评价结果示意图。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中,自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,仅用于解释本发明,而不能理解为对本发明的限制。需要说明的,本文中所使用的术语“第一”或者“第二”等仅为方便描述,不能理解为指示或暗示相对重要性,也不能理解为之间有先后顺序关系。
在本发明的描述中,除非另有说明,“多个”的含义是两个或两个以上。在本文中,除非另有明确的规定和限定,术语“相连”、“连接”等术语应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或一体地连接;可以是机械连接,也可以是电连接;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通。
生物学标志物是从生物学介质中可以检测到的细胞、生物化学或分子改变。生物学介质包括各种体液、组织、细胞、粪便、头发、呼气等。
所称的某种微生物的丰度指在某一微生物群体中该种微生物的丰富程度,例如在肠道微生物群体中该种微生物的丰富程度,可表示为该种微生物在该群体中的含量。
所称序列的一致性(identity)和序列的相似度(similarity)分别指序列间相同或相似的程度。
根据本发明的一个实施方式提供的一种强直性脊柱炎生物标志物,检测该生物标志物的变化可以确定个体患有强直性脊柱炎的相对概率高低。所述强直性脊柱炎微生物标志物由以下微生物组成:放线菌(actinomycessp.icm47)、短双歧杆菌(bifidobacteriumbreve)、双歧杆菌(bifidobacteriumsp.12_1_47bfaa)、长双歧杆菌(bifidobacteriumlongum)、产气柯林斯菌(collinsellaaerofaciens)、两岐双岐杆菌(bifidobacteriumbifidum)和假小链双歧杆菌(bifidobacteriumpseudocatenulatum)。
所称的强直性脊柱炎微生物标志物是发明人通过对大量强直性脊柱炎个体和大量健康对照个体的粪便样本中的微生物的丰度进行差异比较分析、以及验证,而确定下来的,明确了肠道微生物中强直性脊柱炎相关的微生物标志物。利用所称的强直性脊柱炎标志物,能够确定个体处于患有强直性脊柱炎状态的概率或者处于健康状态的概率,能够用于非侵入性的早期发现、辅助检测强直性脊柱炎等。
根据本发明一个实施方式提供的上述强直性脊柱炎微生物标志物在检测强直性脊柱炎、监测治疗强直性脊柱炎、制备治疗强直性脊柱炎的药物和/或在制备功能性食品中的用途。所强直性脊柱炎微生物标志物是发明人通过差异比较分析各种肠道微生物在强直性脊柱炎疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而 确定下来的。相较于在健康群体组,所称强直性脊柱炎微生物标志物在强直性脊柱炎疾病组中显著富集,所称显著富集是指与在健康组中的丰度相比,上述强直性脊柱炎标志物所包含的各种微生物在疾病组中的丰度均具有统计意义地高于或者明显地、实质性地高于在健康组中的丰度。
能够使所称强直性脊柱炎标志物丰度减小的物质能够用于治疗强直性脊柱炎或者益于强直性脊柱炎患者服用,能够使所称强直性脊柱炎标志物丰度减小的物质不限于治疗强直性脊柱炎的药物和有益肠道菌群平衡的功能性食品,该实施方式提供的强直性脊柱炎标志物能够用于制备治疗强直性脊柱炎的药物和/或用于制备益于平衡肠道菌群的功能性食品、保健药等。
根据本发明的另一个实施方式提供的一种利用上述实施例中的强直性脊柱炎标志物确定个体的状态的方法,该方法包括以下步骤(1)和(2):
(1)确定待测个体中的强直性脊柱炎微生物标志物的丰度。
确定待测个体的粪便样本中的强直性脊柱炎微生物标志物中的各种微生物的丰度。
根据本发明的实施例,进行以下以完成该步骤:获得所述个体的粪便样本中的核酸序列的测序数据,所述测序数据包括多个读段;将所述读段比对至所述标志物中的各种微生物的基因组,获得比对结果;依据所述比对结果,确定所述标志物中的各种微生物的丰度。
所称的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平台的不同,可选择但不限于半导体测序技术平台比如pgm、ionproton、bgiseq-100平台,合成边测序的技术平台比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所称的获得的比对结果包含各条读段与各物种的基因组的比对情况,包括读段是否能够比对上某个或某些物种的基因组、只唯一比对到一种物种还是比对到多种物种的基因组、比对到物种的基因组的位置、比对到物种基因组的唯一位置还是多个位置等信息。根据本发明的一个实施例,利用soapalign2.21进行比对,设置参数为–r2–m100–x1000。
所称微生物的基因组指预先确定的该微生物物种的序列,可以是预先获得的待测样本所属或者所包含的生物类别的任意参考模板,例如,目标是待测样本中的微生物,参考序 列可选择ncbi数据库中的各种微生物的参考基因组和/或hmp、metahit项目公开的dacc肠道基因组,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源的个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。根据本发明的一个实施例,各种微生物的参考基因组从公开的数据库中获得,通常的,一种微生物的参考基因组有多个版本,即一种微生物有多个公开的基因组。
reads与物种的参考基因组比对,比对上的可以被分为两部分:a)uniquereads(u):唯一比对上一个物种的基因组;称这些reads为uniquereads。即,如果reads比对上的基因组均来自同一物种,定义这些reads为uniquereads。b)multiplereads(m):比对上一个以上物种的基因组,定义为multiplereads。即,如果reads比对上的基因组来自至少两种物种,定义这些reads为multiplereads。
根据本发明的一个实施例,所称依据测序数据,所述依据比对结果,确定所述标志物中的各种微生物的丰度,包括利用以下公式确定微生物物种s的丰度:微生物物种s的丰度ab(s)=ab(us)+ab(ms),其中,ab(us)=us/ls,us为唯一比对上该种微生物s的读段数目,ls为比对上的该种微生物s的基因组的长度,
对于给定的微生物物种s,其丰度为ab(s),与uniquereads和multiplereads相关,上述公式中的ab(u)和ab(m)分别为该物种s的uniquereads和multiplereads贡献的丰度。每个multiplereads,有特异的物种丰度系数co,假设一条multiplereads比对上n个物种,可以利用下列公式计算该条multiplereads的co:
(2)丰度比较,以确定个体状态。
分别将(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度与其在对照组中的丰度进行比较,依据获得的比较结果确定所述个体的状态,所述对照组由一组或多组相同状态的个体的粪便样本组成,所述状态包括患有强直性脊柱炎状态和健康状态。所称强直性 脊柱炎标志物是发明人通过差异比较分析各种肠道微生物在强直性脊柱炎疾病组和健康组的粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。
所称标志物中的各种微生物在对照组中的丰度可以与待测个体的同时测定或者先后测定。根据本发明的一个实施例,将标志物中的各种微生物在对照组中的丰度预先测定保存,用以作为设定阈值的依据。将标志物中的微生物在待测个体样本中的丰度与所称的阈值比较,基于二者的差异程度,进行待测个体的状态的确定。所称阈值可以为一数值或者数值范围,例如,基于某种微生物在对照组中的丰度均值,该微生物对应的阈值可以设为该丰度均值的95%的置信区间。
根据本发明的实施例,对照组由多个患强直性脊柱炎的个体的粪便样本组成,该步骤(2)包括:当步骤(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度都与其在对照组中的丰度无差异,确定所述个体的状态为患有强直性脊柱炎。根据本发明的实施例,所述无差异为来自所述微生物丰度确定单元的强直性脊柱炎标志物中的各种微生物的丰度落入其在对照组中的丰度的第一预定置信区间,任选的所述第一预定置信区间为95%置信区间。
所称无差异为步骤(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度都落入其在对照组中的丰度的第一预定置信区间。所称的置信区间是指由样本统计量所构造的总体参数的估计区间。在统计学中,一个概率样本的置信区间(confidenceinterval)是对这个样本的某个总体参数的区间估计。置信区间展现的是这个参数的真实值有一定概率落在测量结果的周围的程度。置信区间给出的是被测量参数的测量值的可信程度,即前面所要求的“一定概率”。这个概率被称为置信水平。
根据本发明的实施例,对照组由多个健康个体的粪便样本组成,该步骤(2)包括:当步骤(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度都与其在对照组中的丰度无差异,确定所述个体的状态为没有患强直性脊柱炎。所称无差异为(1)中确定的强直性脊柱炎标志物中的各种微生物的丰度都落入其在对照组中的丰度的第二预定置信区间,任选的所述第二预定置信区间为95%置信区间。
需要说明的是,根据目的或要求不同,可能对确定个体状态结果的可信程度有不同的要求,本领域技术人员可以选择不同的显著性水平(α),即选择不同的可能犯错误的概率,如此,确定的个体的状态的可信程度为1-α。例如,利用该实施例确定个体处于所确定出的状态95%是可靠的。
该方法基于检测个体的粪便样本中的强直性脊柱炎标志物中的各种微生物的丰度,分别将检测确定的各种微生物的丰度与其在对照组中丰度进行比较,依据获得的比较结果能 够确定个体为强直性脊柱炎个体或者为健康个体的概率。为早期发现强直性脊柱炎提供一种非侵入性的辅助检测或者辅助干预治疗的方法。
以上任一实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的全部或部分步骤,可以利用包含可拆分的相应单元功能模块的装置/系统来施行,或者将方法程序化、存储于机器可读介质,利用机器运行该可读介质来实现。
根据本发明的一个实施方式提供的一种利用上述本发明实施例中的强直性脊柱炎标志物确定个体的状态的装置,该装置用以实施上述本发明实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的全部或部分步骤,该装置包括:数据输入单元,用于输入数据;数据输出单元,用于输出数据;处理器,用于执行可执行程序,执行所述可执行程序包括完成上述本发明人一实施例中的确定个体的状态的方法;存储单元,与所述数据输入单元、所述数据输出单元和所述处理器相连,用于存储数据,其中包括所述可执行程序。上述对本发明任一实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的装置,在此不再赘述。
根据本发明的一个实施方式提供的一种利用上述本发明一实施例中的强直性脊柱炎标志物确定个体的状态的系统,该装置用以实施上述本发明任一实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的全部或部分步骤,该系统包括:微生物丰度确定单元,用于确定所述个体的粪便样本中的所述强直性脊柱炎标志物中的各种微生物的丰度;个体状态确定单元,用于分别将来自所述微生物丰度确定单元的所述强直性脊柱炎标志物中的各种微生物的丰度与其在对照组中的丰度进行比较,依据获得的比较结果确定所述个体的状态,所述对照组由一组或多组相同状态的个体的粪便样本组成,所述状态包括患有强直性脊柱炎和不患有强直性脊柱炎。上述对本发明任一实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的系统。
根据本发明的实施例,所述微生物丰度确定单元用于进行以下:获得所述个体的粪便样本中的核酸序列的测序数据,所述测序数据包括多个读段;将所述读段比对至所述标志物中的各种微生物的基因组,获得比对结果;依据所述比对结果,确定所述标志物中的各种微生物的丰度。
所称的测序数据通过对样本中的核酸序列进行测序得来,测序依据所选的测序平台的不同,可选择但不限于半导体测序技术平台比如pgm、ionproton、bgiseq-100平台,合成边测序的技术平台比如illumina公司的hiseq、miseq序列平台以及单分子实时测序平台比如pacbio序列平台。测序方式可以选择单端测序,也可以选择双末端测序,获得的下机数据是测读出来的片段,称为读段(reads)。
比对可以利用已知比对软件进行,例如soap、bwa和teramap等,在比对过程中,一般对比对参数进行设置,设置一个或者一对读段(reads)最多允许有s个碱基错配(mismatch),例如设置s≤2,若reads中有超过s个碱基发生错配,则视为该reads无法比对到(比对上)该组装片段上。所称的获得的比对结果包含各条读段与各物种的基因组的比对情况,包括读段是否能够比对上某个或某些物种的基因组、只唯一比对到一种物种还是比对到多种物种的基因组、比对到物种的基因组的位置、比对到物种基因组的唯一位置还是多个位置等信息。根据本发明的一个实施例,利用soapalign2.21进行比对,设置参数为–r2–m100–x1000。
所称微生物的基因组指预先确定的该微生物物种的序列,可以是预先获得的待测样本所属或者所包含的生物类别的任意参考模板,例如,目标是待测样本中的微生物,参考序列可选择ncbi数据库中的各种微生物的参考基因组和/或hmp、metahit项目公开的dacc肠道基因组,进一步地,也可以预先配置包含更多参考序列的资源库,例如依据待测样本来源的个体的状态、地域等因素选择或是测定组装出更接近的序列作为参考序列。根据本发明的一个实施例,各种微生物的参考基因组从公开的数据库中获得,通常的,一种微生物的参考基因组有多个版本,即一种微生物有多个公开的基因组。
reads与物种的参考基因组比对,比对上的可以被分为两部分:a)uniquereads(u):唯一比对上一个物种的基因组;称这些reads为uniquereads。即,如果reads比对上的基因组均来自同一物种,定义这些reads为uniquereads。b)multiplereads(m):比对上一个以上物种的基因组,定义为multiplereads。即,如果reads比对上的基因组来自至少两种物种,定义这些reads为multiplereads。
根据本发明的一个实施例,所称依据测序数据,所述依据比对结果,确定所述标志物中的各种微生物的丰度,包括利用以下公式确定微生物物种s的丰度:微生物物种s的丰度ab(s)=ab(us)+ab(ms),其中,ab(us)=us/ls,us为唯一比对上该种微生物s的读段数目,ls为比对上的该种微生物s的基因组的长度,
对于给定的微生物物种s,其丰度为ab(s),与uniquereads和multiplereads相关,上述公式中的ab(u)和ab(m)分别为该物种s的uniquereads和multiplereads贡献的丰度。每个multiplereads,有特异的物种丰度系数co,假设一条multiplereads比对上n个物种,可以利用下列公式计算该条multiplereads的co:
根据本发明的实施例,所述个体状态确定单元中的对照组包括多个患强直性脊柱炎的个体的粪便样本,所述个体状态确定单元用于进行以下:当来自所述微生物丰度确定单元的所述强直性脊柱炎标志物中的各种微生物的丰度都与其在对照组中的丰度无差异,确定所述个体的状态为患有强直性脊柱炎。
根据本发明的实施例,所述无差异为来自所述微生物丰度确定单元的强直性脊柱炎标志物中的各种微生物的丰度落入其在对照组中的丰度的第一预定置信区间,任选的所述第一预定置信区间为95%置信区间。
根据本发明的实施例,所述个体状态确定单元中的对照组包括多个健康个体的粪便样本,所述个体状态确定单元用于进行以下:当来自所述微生物丰度确定单元的所述强直性脊柱炎标志物中的各种微生物的丰度都与其在对照组中的丰度无差异,确定所述个体的状态为没有患强直性脊柱炎。
根据本发明的实施例,所述无差异为来自所述微生物丰度确定单元的强直性脊柱炎标志物中的各种微生物的丰度落入其在对照组中的丰度的第二预定置信区间,任选的所述第二预定置信区间为95%置信区间。
根据本发明的另一个实施方式提供的一种利用上述本发明任一实施例的强直性脊柱炎标志物对多个个体进行分类的方法,该方法包括:分别利用上述本发明任一实施例中的确定个体的状态的方法确定各个个体的状态;依据获得的各个个体的状态对各个个体进行分类。该方法能够依据个体的状态的不同区分开多个个体或者区分开多个未知的粪便样本,便于归类、标记管理。另外,上述对本发明任一实施例中的利用强直性脊柱炎标志物确定个体的状态的方法的技术特征和优点的描述,同样适用本发明这一方面的方法,在此不再赘述。
根据本发明的又一个实施方式提供的一种治疗强直性脊柱炎的药物,所述药物能够促使上述本发明任一实施例中的强直性脊柱炎标志物中的各个微生物的丰度减少。所称强直性脊柱炎标志物是发明人通过差异分析各种肠道微生物在强直性脊柱炎疾病组和健康组的 粪便样本中的丰度,以及经过大量已知状态的粪便样本的验证,而确定下来的。相较于在健康群体组,所称强直性脊柱炎标志物在强直性脊柱炎疾病组中显著富集,所称显著富集是指与在健康组中的丰度相比,上述强直性脊柱炎标志物所包含的各种微生物在疾病组中的丰度均具有统计意义地高于或者明显地、实质性地高于在健康组中的丰度。能够使其丰度减少的物质能够用于治疗强直性脊柱炎或者益于强直性脊柱炎患者服用,能够使其丰度减少的物质可以作为治疗强直性脊柱炎的药物。
利用这一实施方式的药物或者功能性食品,合理有效地应用确定的强直性脊柱炎微生物标志物,扶持肠道有益菌的生长和/或抑制肠道潜在致病菌,可以阻止肠道屏障的缺损,改善并恢复肠道微生态结构,对于辅助降低血内毒素水平和/或减轻强直性脊柱炎的临床症状具有重要意义。
根据本发明的又一个实施方式,提供一种制备或筛选上述实施方式中的治疗强直性脊柱炎的药物的方法,该方法包括制备或者筛选能够促使上述本发明任一实施例中的强直性脊柱炎标志物中的各个微生物的丰度减少的物质作为所述药物的步骤。
利用本发明该实施方式中的生产或筛选治疗强直性脊柱炎的药物的方法,通过合理有效地应用确定的强直性脊柱炎生物标志物进行筛选,能够获得能扶持肠道有益菌的生长和/或抑制肠道潜在致病菌的药物,可以阻止肠道屏障的缺损,改善并恢复肠道微生态结构,对于辅助降低血内毒素水平和/或减轻强直性脊柱炎的临床症状具有重要意义。
以下结合具体实施例对本发明的方法和/或装置进行详细的描述。除另有交待,以下实施例中涉及的未特别交待的试剂、序列(接头、标签和引物)、软件及仪器,都是常规市售产品或者开源的,例如购买illumina的转录组文库构建试剂盒。
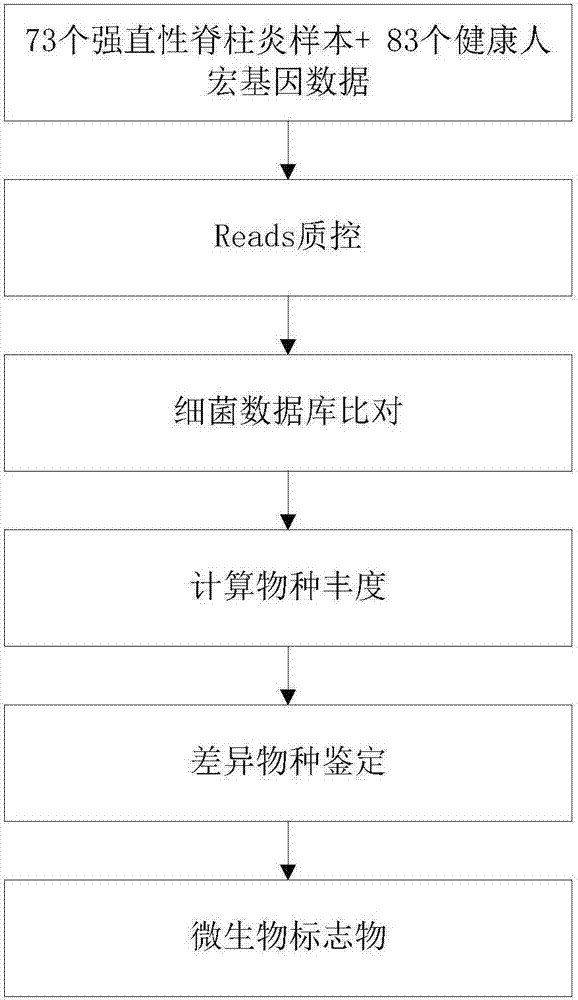
以下实施例包括第一阶段和第二阶段,即对应发现阶段和验证阶段。发现阶段包括:基于分析比较73个强直性脊柱炎患者及83个健康对照组的肠道微生物成分及功能改变,以确定物种标志物;验证阶段包括:利用24个as患者及31个健康对照组验证第一阶段结果的准确性。
实施例1
该示例中,发明人从73个强直性脊柱炎患者、83个健康对照的粪便样品开展整个肠道菌群微生物的关联分析研究描述粪便微生物群落及功能成分特征。总的来说,发明人下载约428.09gb高质量的测序数据(lc健康人)以及实验测序得到的294gb高质量测序数据构建了强直性脊柱炎参照基因集,并且和下载的lc基因集、igc基因集构建一个更加完整的基因集。宏基因组分析显示,17个微生物物种与强直性脊柱炎疾病密切相关,其中7个菌在健康人的肠道微生物中富集,10个菌在强直性脊柱炎病人的肠道微生物中富集。
1、测序数据的获取
1.1样本收集和dna提取
强直性脊柱炎患者来自杭州浙江中医药大学第一附属医院,实验共采集了73个中国强直性脊柱炎患者的粪便样品,其中每个个体的新鲜粪便样品分成200mg/份,共5份,立即-80℃冰箱冷冻保存。
73个中国强直性脊柱炎患者的粪便样品中提取总dna。苯酚三氯甲烷处理提取dna方法提取dna。
1.2构建文库及测序,以及参照数据下载
dna建库按仪器制造商(illumina)的操作指南进行。对文库进行pe100bp测序。illuminahiseq2000(illumina,sandiego,ca)平台对73个样品的文库进行测序。每个样本平均产生4.03gb(sd.±0.64gb)高质量测序结果,总计294gb测序数据量。
从ebi下载83个健康中国人的测序数据,accession号:erp005860。
参照图1的实验流程,鉴定强直性脊柱炎的相关生物标志物,其中省略的步骤或者细节为本领域技术人员所熟知,几个重要步骤介绍如下面步骤所述。
2、生物标志物的鉴定
2.1测序数据的基本处理
1)测序数据经过质控:获得第一阶段的73个as样品的测序数据以后,对其进行过滤,质控按以下标准进行:a)去除低质量碱基(q20)大于50%的reads;b)移除大于5个n碱基的reads;c)移除尾部低质量(q20)和n碱基。丢失成对reads的序列被认为是单条reads用于组装。
2)同样处理下载的健康人数据。
3)lc基因集获自qin,n.etal.alterationsofthehumangutmicrobiomeinlivercirrhosis.nature513,59-64(2014).,从ftp://climb.genomics.cn/pub/10.5524/100001_101000/100064/1.genecatalogs/igc.fa.gz下载得到igc基因集。
2.2物种丰度分析
利用soapalign2.21将经过上步骤处理的paired-endcleanreads比对(匹配)到冗余基因组,这里,所称的冗余基因组来自各数据库中公开的的细菌的参考基因组。比对参数为–r2–m100–x1000。reads与冗余基因组比对,可能被分为两部分:a)uniquereads(u):reads只比对上一个物种的基因组;这些reads被定义为uniquereads。即,如果这些基因组来自同一物种,reads与一个以上的基因组比对,发明人将这些reads定义为uniquereads。b)multiplereads(m):如果reads比对上一个物种以上的基因组,定义为multiplereads。即, 如果比对上的基因组来自不同物种,发明人定义这些reads为multiplereads。
对于物种s,其丰度为ab(s),与特有的ureads和共享的mreads相关,丰度的计算方式如下:
ab(s)=ab(u)+ab(m)
ab(u)=u/l
ab(u)和ab(m)分别为来自该物种s的uniquereads和multiplereads的丰度,l表示基因组的长度。每个multiplereads,有特异物种系数co;假设某一multiplereads比对上n个物种,按以下方法计算该条multiplereads的co:
对于这些reads,发明人以加和的n个物种的uniquereads的丰度作为标准。即对于multiplereads,发明人把其所比对上的n个物种的uniquereads丰度之和作为分母。
2.3关联分析/筛选物种标记物
为了获得与强直性脊柱炎疾病密切相关的肠道微生物物种标记物,发明人利用强直性脊柱炎患者组(73例)与健康人组(83例)两组肠道微生物物种丰度数据,在物种级别做了一个与疾病相关性的研究。
基于上步骤得到的物种丰度表,发明人设置标准如下:(1)强直性脊柱炎患者组或健康人组物种丰度的中位数必须大于0.00001;(2)通过结合benjaminihochberg的多重检验的wilcoxon秩和检验进行检验,得到每个物种和强直性脊柱炎疾病的相关性p值和q值;(3)使用一个相对非常严格的阈值(q_values<5.0e-05)。然后,发明人得到28个与强直性脊柱炎疾病密切相关的肠道微生物物种。其中在强直性脊柱炎(as)患者中富集的有14个物种,在健康人(hd)中富集的有14个物种。这28个微生物物种标记物如下表1所示。
表1
实施例2
为了证实实施例1中的分析结果可以用以作为强直性脊柱炎物种标志物,进一步比较验证群体中的31个健康人及24个强直性脊柱炎患者的物种丰度,并对最终微生物物种标记物根据验证情况做出筛选。验证群体的测序数据的获得与处理,参照实施例1进行。
验证结果如下:上述富集在健康人群中的14个物种中,7个在验证集中得到高质量的验证(p_values<0.05),健康人富集的微生物物种标志物验证的p值和q值情况如下表2所示。微生物物种分别为毛螺科菌(lachnospiraceaebacterium6_1_63faa)、死亡梭杆菌(fusobacteriummortiferum)、拟杆菌([bacteroides]pectinophilus)、阴沟肠杆菌(enterobactercloacae)、挑剔真杆菌(eubacteriumeligens)、毛螺科菌(lachnospiraceaebacterium8_1_57faa)、梭菌(clostridiumsp.l2-50)。拟杆菌存在于人体肠道中,主要是帮助分解食物以提供人体所需的营养和能量。
表2
对于上述富集在强直性脊柱炎患者中的14个物种,其中10个在验证集中得到高质量的验证(p_values<0.05),强直性脊柱炎患者富集的微生物物种标志物验证的p值和q值情况如下表3所示,分别为放线菌(actinomycessp.icm47)、短双歧杆菌(bifidobacteriumbreve)、双歧杆菌(bifidobacteriumsp.12_1_47bfaa)、长双歧杆菌(bifidobacteriumlongum)、产气柯林斯菌(collinsellaaerofaciens)、两岐双岐杆菌(bifidobacteriumbifidum)、假小链双歧杆菌(bifidobacteriumpseudocatenulatum)、迟缓埃格特菌(eggerthellalenta)、链状双歧杆菌(bifidobacteriumcatenulatum)、青春双歧杆菌(bifidobacteriumadolescentis)。
双歧杆菌可以预防腹泻,减少便秘,即双向调节。有研究表明超过70%的强直性脊柱炎患者患有肠道炎症,这些患者中5~10%肠道炎症很严重,可能会发展为临床上的炎症性肠病(ibd)或克罗恩氏病(cd)(mielantsetal.,1985),研究发现ibd病人肠道中拟杆菌、梭状芽胞杆菌、厚壁菌下降,放线菌、变形菌比例升高。
表3
发明人认为,可以将从健康人富集的7个微生物物种标记物,作为强直性脊柱炎疾病患病的反向指标,或作为治疗强直性脊柱炎进行研发的微生物制剂药物菌群成分,或作为检测强直性脊柱炎、监测强直性脊柱炎治疗进程的恢复指标;将强直性脊柱炎患者富集的10个微生物物种标记物,作为强直性脊柱炎疾病患病的正向指标,特别用于强直性脊柱炎疾病非创伤式的检测和诊断。
发明人利用这17个微生物物种标记物,构建一个综合指标,估计roc(receiver-operatingcharacteristic)曲线下面积auc,auc越大,表示诊断能力越高,评价综合得分对应其对强直性脊柱炎的诊断能力。通过对一期(第一阶段)的156个样品和二期(第二阶段)的55个样品进行评测,具体情况如图2所示,都表现出了很好的诊断能力,在一期得到auc=96.62%,如图2左边的图所示,置信区间为93.93%-99.31%;二期得到auc=89.92%,如图2右边的图所示,置信区间为81.28%-98.56%。
实施例3
利用45个粪便样本进行样本来源的个体状态的检测。
参照实施例2的方法确定各粪便样本中的表3的放线菌(actinomycessp.icm47)、短双歧杆菌(bifidobacteriumbreve)、双歧杆菌(bifidobacteriumsp.12_1_47bfaa)、长双歧杆菌(bifidobacteriumlongum)、产气柯林斯菌(collinsellaaerofaciens)、两岐双岐杆菌(bifidobacteriumbifidum)和假小链双歧杆菌(bifidobacteriumpseudocatenulatum)的丰度,判断各样本中的这七种菌株丰度是否落入各自在疾病对照组或者健康对照组的丰度的95%的置信区间,判定这七种菌种的丰度均落入疾病组的对应区间的样本所对应的个体的状态为强直性脊柱炎患者,判定七种菌种的丰度均落入健康组的对应区间的样本所对应的个体的状态为非强直性脊柱炎患者。
结果显示,利用上述方法能够对其中的41个样本进行个体状态判断,而且对这41个样本中的38个样本对应个体的状态的判断,与记录的该样本来源个体的状态一致。
另外,发明人发现对表2和表3中的物种联合检测,例如检测表3中的物种标志物被富集,同时表2中物种标记物不被富集,能够更准确的判断发现强直性脊柱炎患者或易感人群。
在利用标志物治疗强直性脊柱炎的方案中,发明人发现使3中的物种标志物生长得到抑制或者清除,同时使表2中物种标记物被富集,治疗效果极佳。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不一 定指的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任何的一个或多个实施例或示例中以合适的方式结合。
尽管已经示出和描述了本发明的实施例,本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同物限定。
- 还没有人留言评论。精彩留言会获得点赞!