一种抗肿瘤侵袭转移的多靶点融合蛋白的制备及用途的制作方法
本发明属于生物工程药用蛋白技术领域,涉及一类TRAIL蛋白相关的多靶点抗肿瘤融合蛋白制备及其用途。
背景技术:
:
肿瘤坏死因子相关凋亡诱导配体(tumor necrosis factor-related apoptosis-inducing ligand,TRAIL),又称为凋亡素2配体(Apo2 ligand,Apo2L),是机体自身合成的一种细胞因子,表达于少数淋巴细胞表面,也以可溶的游离形式存在,其生理功能主要是调节机体的免疫反应,参与免疫监视以及抑制肿瘤发生、转移。研究表明,人可溶性TRAIL(sTRAIL),即TRAIL蛋白的114至281位氨基酸序列,具有完整TRAIL蛋白的活性(Pitti RM,J.Biol.Chem,1996),可以诱导多种肿瘤细胞发生凋亡,而靶向特异性使其对正常细胞几乎没有毒性作用,然而临床试验效果不佳,耐药性的产生局限了其在临床上的应用。基于TRAIL相关的融合蛋白,可以提高肿瘤细胞对TRAIL的敏感性,一定程度上逆转其耐药性。重组人可溶性TRAIL(rhTRAIL)及靶向TRAIL受体的激动剂型抗体具有很好的抗肿瘤活性,并在临床试验中取得很大的进展。但是,耐药性的产生以及个体差异等,是TRAIL及靶向TRAIL受体的激动剂型抗体在临床应用的最大难题。研究发现。与sTRAIL重组后的多靶点药物抗肿瘤活性可得到明显的提升,例如EGFR单链抗体与sTRAIL偶联后的重组蛋白,对EGFR表达水平高的肿瘤细胞具有更强的凋亡诱导作用。
RGD肽是一类含有精氨酸-甘氨酸-天冬氨酸(Arg-Gly-Asp)序列的短肽,是整合素与配体蛋白相互作用识别的位点。整合素ανβ3和ανβ5在肿瘤的侵袭转移中发挥着重要的作用,通常高表达于肿瘤新生血管和一部分肿瘤细胞如乳腺癌、肺癌、前列腺癌、胶质母细胞瘤等,因此可以作为肿瘤治疗的理想靶点。RGD肽已广泛应用于肿瘤的诊断与肿瘤治疗研究。利用RGD肽与整合素ανβ3和ανβ5的特异结合,可以将抗肿瘤药物靶向运输至肿瘤部位,有效降低对正常组织或器官的损伤。研究报道,RGD与sTRAIL重组后的融合蛋白,其活性明显优于sTRAIL。本发明中采用的环形RGD肽的氨基酸序列为ACDCRGDCFC(Koivunen E,Bio-Technology,1995)也称为RGD4C。
NGR肽是一种抗菌肽、靶向肽(Asn-Gly-Arg),通过氨肽酶N(aminopeptidase N,CD13)与新生血管发生特异性结合,通过NGR修饰可以将细胞毒性药物、细胞因子、抗血管生成药物以及造影剂等靶向新生血管,提高药效降低对正常组织的损伤,目前已大量被用于治疗性药物靶向运输的研究,其中NGR-hTNF已经进入临床ΙΙ、ΙΙΙ期研究。研究报道sTRAIL对血管生成具有一定的抑制作用,若将NGR与sTRAIL重组,得到的融合蛋白不仅可以诱导肿瘤细胞发生凋亡,并可将融合蛋白靶向运输至肿瘤新生血管部位,抑制肿瘤新生血管的生成。但是, 迄今为止,尚未见有NGR肽与sTRAIL偶联及其抗肿瘤作用的相关报道。本发明应用的NGR肽的氨基酸序列是CNGRCVSGCAGRC(Arap W,Science,1998)。
基于以上现状,本发明利用TRAIL对肿瘤细胞的选择性凋亡诱导作用,RGD肽和NGR肽的靶向作用,分别于sTRAIL的两端引入上述RGD肽和NGR肽,研发了TRAIL相关的抗肿瘤细胞及抑制肿瘤细胞转移的多靶点融合蛋白,其具有更强的肿瘤细胞凋亡诱导作用并能有效抑制肿瘤细胞体内外转移。
技术实现要素:
:
本发明提供了一类多靶点抗肿瘤融合蛋白RGD-TRAIL-NGR和TRAIL-NGR。
本发明提供了TRAIL相关融合蛋白RGD-TRAIL-NGR和TRAIL-NGR的编码基因及氨基酸序列。
本发明提供了TRAIL相关融合蛋白的制备方法。
本发明提供了TRAIL相关的融合蛋白在抗肿瘤中的应用。
本发明还提供了以所述融合蛋白为有效成分与药学上可接受的载体组成的组合物及其在抗肿瘤中的应用。
有关TRAIL相关融合蛋白制备的已有报道几乎均采用原核表达系统。本发明利用基因工程技术,于sTRAIL的两端分别引入了RGD肽和NGR肽,构建了TRAIL相关融合蛋白RGD-TRAIL-NGR的重组载体,通过毕赤酵母分泌表达系统表达目的蛋白,经纯化获得一种多功能药用蛋白。
本发明所提供的一种抗肿瘤侵袭转移多靶点融合蛋白,其结构为:RGD-连接肽(G4S)-sTRAIL-连接肽(G4S)-NGR,所述蛋白具有SEQ ID NO:1氨基酸序列,共208个氨基酸。
本发明提供所述融合蛋白的编码基因,其具有SEQ ID NO:2所示核苷酸序列,共624bp。
本发明提供的另一种抗肿瘤侵袭转移多靶点融合蛋白TRAIL-NGR的氨基酸序列为SEQ ID NO:3,共192个氨基酸,具有SEQ ID NO:4所示的核苷酸序列,共576bp。
本发明采用基因工程技术,提供了TRAIL相关新型多靶点融合蛋白的制备方法(实施例2)。本发明的优点在于,利用毕赤酵母表达系统分泌表达了RGD-TRAIL-NGR、RGD-TRAIL和TRAIL-NGR三种融合蛋白,并比较了它们的抗肿瘤作用。所获得的三种融合蛋白,可高效结合相关细胞表面因子表达的肿瘤细胞,对肿瘤细胞的增殖抑制效果显著,融合蛋白还能明显抑制肿瘤细胞的体内外转移作用。RGD肽和NGR肽的加入,能够增强TRAIL的抗肿瘤效果,并部分逆转TRAIL的耐药性,表现出更好的应用前景。其中RGD-TRAIL已有相关报道,通过比较表明,本发明构建的RGD-TRAIL-NGR融合蛋白具有更佳的抗肿瘤活性。
附图说明:
图1—TRAIL相关融合蛋白表达载体的构建及鉴定(M1:DNA marker DL 2000,1:PCR产物,
2:重组质粒,3:SnaB I和Not I双酶切,4:Sal I酶切,M2:DNA marker DL 15000)
其中:图A为RGD-TRAIL表达载体的构建及鉴定结果;
图B为TRAIL-NGR表达载体的构建及鉴定结果;
图C为RGD-TRAIL-NGR表达载体的构建及鉴定结果。
图2—TRAIL相关融合蛋白的分离纯化及鉴定(M:蛋白Marker,1:Washing buffer I洗脱液,
2:Washing buffer II洗脱液,3:Elution buffer洗脱液,4:抗His-tag免疫印迹结果,5:
抗TRAIL免疫印迹结果)
其中:图A为RGD-TRAIL分离纯化及鉴定结果;
图B为TRAIL-NGR分离纯化及鉴定结果;
图C为NGR-TRAIL-NGR分离纯化及鉴定结果。
图3—TRAIL相关融合蛋白与肿瘤细胞的亲和活性
其中:图A为多种肿瘤细胞表面受体αν、CD13、DR4和DR5的表达情况;
图B为ELISA法检测TRAIL相关融合蛋白与肿瘤细胞H460、A549、HT1080、PANC-1的结合情况。
图4—MTT法检测TRAIL相关融合蛋白对肿瘤细胞H460、A549、HT1080、PANC-1的增殖抑制作用
图5—划痕实验检测TRAIL相关融合蛋白对A549和HT1080迁移抑制作用
其中,图A为A549细胞;
图B为HT1080细胞。
图6—Transwell实验检测TRAIL相关融合蛋白对A549和HT1080迁移抑制作用
其中,图A为A549细胞;
图B为HT1080细胞。
图7—流式细胞术和Western blot法分析TRAIL相关融合蛋白对肿瘤细胞A549和HT1080的凋亡诱导作用
其中,图A为A549细胞;
图B为HT1080细胞。
图8—TRAIL相关融合蛋白对裸鼠肺转移模型的转移抑制作用
其中:图A为各组别荧光强度;
图B为各组肺转移瘤结节的数量;
图C为各组裸鼠体重变化曲线。
具体实施方式:
以下实施例仅为帮助本领域技术人员更好地理解本发明,但不以任何方式限制本发明。《实施例1》重组表达载体pHBM-rgd-trail-ngr、pHBM-rgd-trail和pHBM-trail-ngr的构建
利用酶切、PCR、连接分子生物学技术,采用pHBM表达载体构建重组质粒pHBM‐rgd‐trail‐ngr和pHBM‐trail‐ngr。
融合蛋白RGD-连接肽(G4S)-TRAIL-连接肽(G4S)-NGR全基因序列由金斯瑞公司根 据毕赤酵母偏爱密码子优化合成,融合蛋白RGD-TRAIL和TRAIL-NGR的基因序列通过设计引物利用分子生物学技术进行构建,引物由InvitrogenTM公司合成,表达载体为pHBM(CHKD博硕士论文全文数据库,2014)。
P1:5’-TCTGTACGTAGCTTGTGATTGTAGAGGAG-3’
(下划线为SnaB I酶切位点,其后为RGD肽编码基因起始序列)
P2:5’-ATAAGAATGCGGCCGCTTAGTGGTGGTGGTGGTGGTGACCAACCAAAAAGGC-3’
(下划线为Not I酶切位点,其后为His-tag编码基因及TRAIL蛋白编码基因结尾互补序列)
P3:5’-TCTGTACGTAGTTAGAGAAAGAGGACCTCAG-3’
(下划线为SnaB I酶切位点,其后为TRAIL编码基因起始序列)
P4:5’-ATAAGAATGCGGCCGCTTAGTGGTGGTG-3’
(下划线为Not I酶切位点,其后为His-tag编码基因结尾互补序列)
1)提取金斯瑞公司提供的pUC57-rgd-trail-ngr质粒,用SnaB I和Not I双酶切后,与经过双酶切的pHBM质粒载体进行连接,得到重组质粒pHBM-rgd-trail-ngr;
2)以质粒pHBM-rgd-trail-ngr为模板,引物P1和P2进行PCR扩增反应,胶回收PCR产物,用SnaB I和Not I双酶切后,与经过双酶切的pHBM质粒载体进行连接,得到重组质粒pHBM-rgd-trail;
3)以质粒pHBM-rgd-trail-ngr为模板,引物P3和P4进行PCR扩增反应,胶回收PCR产物,用SnaB I和Not I双酶切后,与经过双酶切的pHBM质粒载体进行连接,得到重组质粒pHBM-trail-ngr。
毕赤酵母表达的融合蛋白RGD-TRAIL-NGR基因序列如SEQ ID NO:2,全长624bp,编码208个氨基酸,RGD肽基因与TRAIL蛋白基因之间以及TRAIL蛋白基因和NGR肽基因之间为GGGGS连接肽基因(编码5个氨基酸),TRAIL-NGR蛋白基因序列如SEQ ID NO:4,全长576bp,编码192个氨基酸。
《实施例2》TRAIL相关融合蛋白在毕赤酵母中的表达及纯化
将构建的表达质粒pHBM-rgd-trail、pHBM-trail-ngr、pHBM-rgd-trail-ngr转化DH5α感受态细胞,双酶切及测序鉴定后,提取质粒,用Sal I酶切线性化(图1),胶回收分子量大的片段,电转化毕赤酵母GS115感受态细胞,涂布MD平板,分别挑取单菌落进行菌落PCR鉴定,挑取His+菌落进行诱导表达,选取表达量相对较高的菌株作为表达菌进行较大规模发酵。所述的菌株被命名为GS115-RTN,于2016年09月19日送交中国微生物菌种保藏管理委员会普通微生物中心保藏,其编号为CGMCC No.13016。首先接种融合蛋白的表达菌株于BMGY培养基(1%酵母提取物,2%蛋白胨,1.34%YNB,4×10-5%生物素,100mM pH 6.0磷酸钾缓冲液,1%甘油)中,30℃培养36h,室温静置或离心收集菌体,将其转移至BMMY培养基(1%酵母提取物,2%蛋白胨,1.34%YNB,4×10-5%生物素,100mM pH 6.0磷酸钾缓冲 液,1%甲醇),20℃继续培养48-96h(每24h补加100%甲醇至终浓度为1%,诱导融合蛋白表达)。融合蛋白在毕赤酵母中以可溶性的形式表达于培养基中,上清经0.45μm滤膜过滤后,用GE公司的Ni2+亲和柱进行纯化,具体操作按照说明书进行。通过12%SDS-PAGE检测融合蛋白的表达及纯化情况,经Western blot鉴定,纯化后的蛋白含有TRAIL蛋白结构和His-tag结构(图2)。
《实施例3》融合蛋白与肿瘤细胞亲和活性分析
1)Western blot检测不同肿瘤细胞表面αν、CD13、DR4和DR5的表达水平
肿瘤细胞HEK293、3T3、A549、H460、PANC-1、BxPC-3、MIA、HCT-15和HT1080均由本室传代保存。将处于对数生长期的肿瘤细胞用预冷的PBS润洗3次,加入适量的细胞裂解液(50mmol/L Tris-Hcl,150mmol/L Nacl,0.2%SDS,2%NP-40,0.5%脱氧胆酸钠,pH 8.0。临用前加入1%PMSF),冰上裂解30min。于4℃,12000rpm离心20min,上清液转移至新的1.5ml EP管。用BCA试剂盒对各种细胞裂解样品进行蛋白定量,每种细胞裂解样品按照相同总蛋白量制备,与适量5×上样缓冲液混合,沸水浴中变性10min,冷却后上样进行SDS-PAGE电泳分析。电泳完毕后进行转膜,将蛋白转移至PVDF膜上,经5%脱脂奶粉封闭液封闭后,根据待检测目的蛋白分子量大小裁剪PVDF膜,用相应的一抗4℃孵育过夜,用TBST溶液洗膜三次,再孵育二抗,TBST溶液洗膜三次后,用显影液进行显影并拍照。所用的一抗为αν。所检测的细胞中,H460细胞不表达CD13,但αν和DR4/DR5的表达水平较高;A549、PANC-1和HT1080均表达上述细胞表面受体,因此主要应用H460、A549、PANC-1和HT1080四种肿瘤细胞来比较构建的TRAIL相关融合蛋白的生物学活性。
2)ELISA法分析融合蛋白对不同肿瘤细胞的亲和活性
肿瘤细胞H460,A549,HT1080和PANC-1以24h长满96孔板为原则接种于96孔板(根据细胞体积的大小及生长速度的快慢调整每种肿瘤细胞的接种量,约1×104个细胞/孔),37℃培养24h后用PBS洗2次(3min/次),加入4℃预冷的0.05%戊二醛50μl/孔,放至4℃固定细胞20min;固定后的细胞用PBS洗3次(3min/次),甩干残留液体后,用5%脱脂牛奶溶液以200μl/孔在室温摇床上轻摇固定封闭2h;用PBST缓冲液(PBS中含有0.05%的Tween-20)洗3次(3min/次);将融合蛋白用PBS倍比稀释后加入到96孔板中,每个浓度设3个平行孔,50μl/孔,37℃孵育2h;用PBST洗3次(3min/次)后,加入抗His-tag单克隆抗体(Abmart公司,1:2000稀释),50μl/孔,37℃孵育2h;用PBST洗3次(3min/次)后,加入HRP标记的羊抗鼠IgG抗体(1:2500稀释),50μl/孔,37℃孵育2h;用PBST洗5次(5min/次)后,每孔加入辣根过氧化物酶的底物可溶型单组分TMB溶液(北京天根生化科技有限公司)100μl,室温避光反应10—20min,当液体由无色变为蓝色后,每孔加入2mol/L的硫酸100μl终止反应,立即在酶标仪上测定450nm处的吸光值。结果如图3所示,三种蛋白对H460、A549、PANC-1和HT1080细胞均呈现出较强的亲和活性,从图中可以看出四种融合蛋白与上述肿瘤细胞的亲和活性差异不大,表明靶向肽的引入对TRAIL蛋白的结合活 性影响不大,同样也没有显著提高融合蛋白与肿瘤细胞的亲和力。
《实施例4》MTT法检测融合蛋白对各种肿瘤细胞增殖抑制作用
处于对数生长期的肿瘤细胞H460、A549、HT1080和PANC-1经胰酶消化后,进行细胞计数,根据细胞的生长速度,每孔铺2500-5000个细胞,37℃培养24h使细胞贴壁;将融合蛋白用PBS进行倍比稀释后,每孔加入100μl,每个融合蛋白浓度设三个复孔,同时设立对照组和空白组,37℃继续培养48h;每孔加入20μl浓度为5mg/ml MTT,37℃继续培养4h;小心将孔内液体吸尽,每孔加入150μl DMSO,室温下低速振荡10min,酶标仪测定570nm处的吸光值;将平行孔吸光值取平均值,按照公式计算细胞的存活率:存活率=(AT-AB)/(AC-AB)×100%,其中AB,AC,AT分别代表空白组、对照组、加重组蛋白组平均吸光值。以细胞存活率为纵坐标,融合蛋白浓度为横坐标作浓度反应曲线,利用SPSS软件计算IC50值。
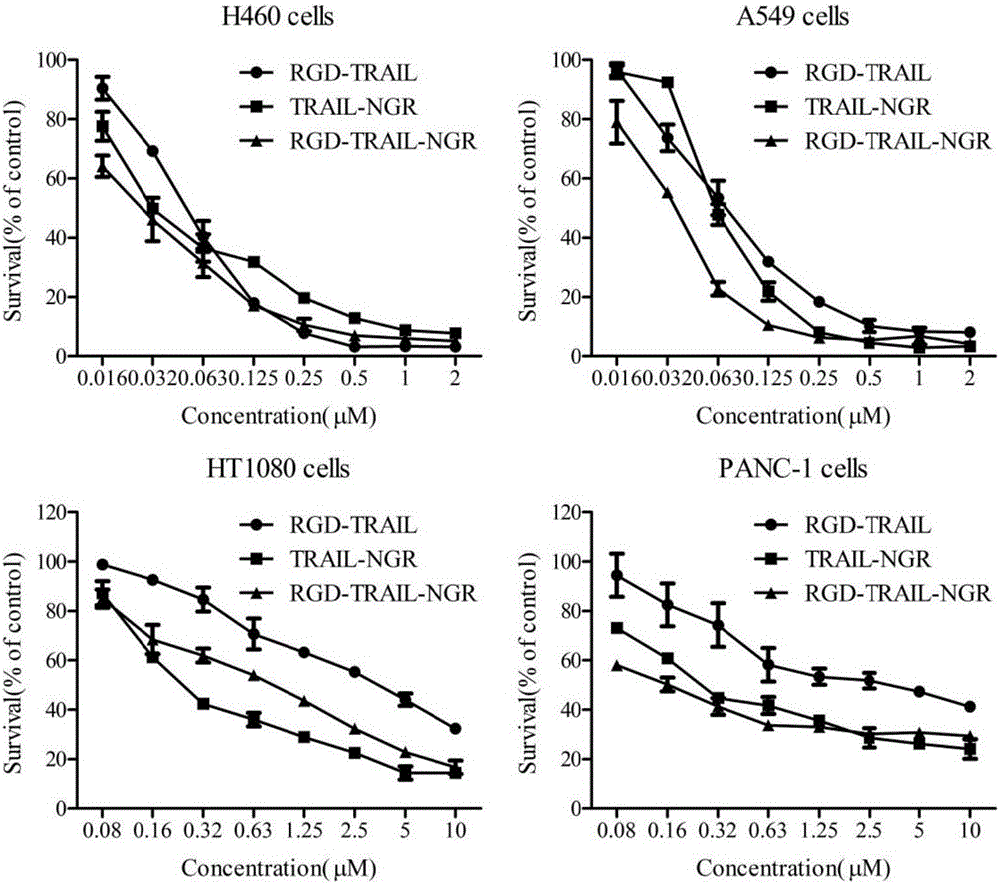
从MTT结果(图4)可以看出,这3种蛋白对H460和A549细胞的增殖抑制作用没有特别显著的差别。事实上,H460和A549细胞对TRAIL相对敏感,而HT1080和PANC-1细胞对TRAIL相对不敏感。因此在H460和A549细胞中经过靶向肽修饰后的融合蛋白活性差异并不大,RGD-TRAIL-NGR的活性稍强,而单个靶向肽修饰后的融合蛋白活性稍弱;在PANC-1细胞中,RGD-TRAIL与另两种融合蛋白活性差异较为显著,表明NGR肽可以增强融合蛋白的活性;在HT1080细胞中,融合蛋白TRAIL-NGR的增殖抑制效果最强,其次是RGD-TRAIL-NGR,这可能与HT1080细胞表面CD13表达水平较高有关,并且其作用对于HT1080细胞尤为重要,而RGD-TRAIL-NGR融合蛋白可能由于RGD肽的存在,空间上对NGR肽作用的充分发挥具有一定的限制作用,因此其活性较TRAIL-NGR稍差(表1)。
表1.融合蛋白RGD-TRAIL、TRAIL-NGR和RGD-TRAIL-NGR对各种细胞的IC50值
《实施例5》融合蛋白对肿瘤细胞迁移抑制作用
1)划痕法检测融合蛋白对细胞迁移的影响
将传代培养的肿瘤细胞接种于6孔板,37℃培养24h使细胞贴壁;用无菌的200μl黄枪头沿孔的直径紧贴板底划线,用PBS润洗3次,洗掉漂浮的细胞,加入新的细胞培养基及相应浓度的融合蛋白;分别于划线后0h、24h在倒置显微镜下观察拍照。实验结果如图5所示,由于HT1080和A549细胞对融合蛋白敏感性不同因此,融合蛋白的浓度分别设为0.12μmol·L-1和0.016μmol·L-1,与对照组相比,融合蛋白能够明显抑制肿瘤细胞HT1080和A549的迁移作 用,其中抑制作用由弱至强分别为RGD-TRAIL、TRAIL-NGR和RGD-TRAIL-NGR,表明双靶向肽的引入均能够发挥抑制肿瘤细胞迁移的作用,其中NGR肽在迁移抑制方面作用更为显著。
2)Transwell法检测融合蛋白对细胞迁移的影响
向Transwell小室内及外室分别加入100μl和600μl的无血清培养基,静置于37℃培养箱平衡1-2h,提高细胞的贴附效率;将传代培养的肿瘤细胞消化后,用无血清培养基洗2次,进行细胞计数,根据不同细胞的迁移能力,调整细胞密度,A549细胞浓度为8×105个/ml,HT1080细胞密度为5×105个/ml;弃掉小室和外室的液体,分别加入细胞悬液和相应浓度的融合蛋白,总体积100μl,向外室加入600μl含有血清的培养基,其中A549细胞用20%血清,而HT1080细胞用10%血清;于37℃培养24h后,取出小室,弃掉小室内和外室的培养液,把嵌套膜用PBS洗一次,用预冷的甲醇固定细胞10min;用PBS润洗3次,放入0.1%结晶紫染色液,室温下染色30min;用PBS润洗3次,用棉签拭去嵌套膜小室内侧的液体和没有转移的细胞,保留嵌套膜外侧的细胞,擦干后于显微镜下观察并拍照;为了更客观的评价肿瘤细胞的迁移情况,用刀片将嵌套膜沿边缘切下,放入96孔板内,加入150μl 33%的醋酸溶解结晶紫,低速振荡溶解10min,小心取出膜,在酶标仪上测定OD 570处吸光值。
Transwell实验中融合蛋白的浓度与划痕实验一致,结果如图6所示,经融合蛋白作用后细胞的迁移明显得到了抑制,且实验结果与划痕实验相一致,进一步表明融合蛋白能够发挥靶向肽RGD和NGR的作用。
《实施例6》融合蛋白对肿瘤细胞A549和HT1080凋亡诱导作用
1)流式细胞术检测融合蛋白诱导A549和HT1080细胞凋亡
将A549和HT1080细胞接种于6孔板,37℃培养过夜使其贴壁;加入相应浓度的融合蛋白,继续培养24h;收集细胞,经PBS润洗2次后,加入100μl 1×Annexin V Binding Solution将细胞重悬;向细胞悬液中加入5μl Annexin V,FITC结合物,再加入5μl PI,室温下避光培养15min;加入400μl 1×Annexin V Binding Solution,流式细胞仪检测。
根据MTT实验结果,分别选择浓度为0.05和1μmol·L-1的融合蛋白作用于A549和HT1080细胞。如图7所示,在对TRAIL相对敏感的A549细胞中,融合蛋白RGD-TRAIL-NGR对细胞凋亡诱导作用最强,其次是TRAIL-NGR,结果与MTT相符;但是在HT1080细胞中,虽然细胞凋亡率与MTT结果趋势相同,但凋亡率较低且差异较小,表明对TRAIL相对不敏感的HT1080细胞,融合蛋白虽能诱导其发生凋亡,但效果并不显著,同时表明RGD肽和NGR肽能够发挥作用。
2)Western blot检测融合蛋白对A549和HT1080凋亡相关蛋白诱导作用
分别将A549、HT1080细胞接种于6孔板中,37℃培养24h使细胞贴壁;分别加入适当浓度的融合蛋白,继续培养24h;然后收集细胞,冰上裂解收集蛋白,经BCA试剂盒定量后,电泳并将蛋白转移至PVDF膜上,用5%脱脂牛奶室温下封闭2h,用封闭液稀释相关一抗, 孵育过夜;TBST润洗3次,加入用TBST稀释的二抗,继续孵育2h,再用TBST润洗5次;将Millipore公司发光检测液A和B按1:1混合后,加在PVDF膜上进行显色,凝胶成像系统拍照。
根据MTT实验结果,分别选择浓度为0.025、0.05μmol·L-1的RGD-TRAIL、TRAIL-NGR和RGD-TRAIL-NGR蛋白作用于A549细胞,选择浓度为0.5、1μmol·L-1的上述3种蛋白作用于HT1080细胞,24h后提取总蛋白进行Western blot检测凋亡相关蛋白PARP和caspase-3的表达情况(如图7),在A549细胞中,低浓度的四种蛋白均能诱导肿瘤细胞发生凋亡,说明融合蛋白能够发挥TRAIL蛋白部分的作用,相比之下RGD-TRAIL-NGR的效果更加明显,这与MTT实验结果相符,表明RGD肽与NGR肽均能发挥相应的作用。而在HT1080细胞中也能检测到凋亡相关蛋白的表达,但是caspase-3剪接体并不明显,这与HT1080细胞对TRAIL相对不敏感有关,并且三种蛋白的效果相似,MTT结果显示带有NGR肽的融合蛋白更能够抑制HT1080细胞的增殖,说明可能是NGR肽发挥了较强的抑制效果。
《实施例7》融合蛋白对裸鼠肺转移模型的转移抑制作用
实施例5体外转移抑制作用结果显示,TRAIL-NGR和RGD-TRAIL-NGR对细胞迁移抑制较为明显,而RGD-TRAIL作用较弱,因此本实验检测TRAIL-NGR和RGD-TRAIL-NGR两种融合蛋白的体内转移抑制效果,并以TRAIL组作为对照。HT1080LUC细胞由本室构建,BALB/C裸鼠购于斯贝福(北京)生物技术有限公司。收集HT1080LUC细胞,将其稀释为7.5×106的细胞悬液。每只裸鼠从尾静脉注射200μl细胞悬液。一周后腹腔注射荧光素酶底物D-luciferin(150mg·kg-1),将动物置于Xenogen活体动物成像系统检测仪密闭的暗箱中成像观察肺部肿瘤的生长状况。根据荧光强度,将裸鼠随机分成4组:对照组、TRAIL组、TRAIL-NGR组和RGD-TRAIL-NGR组,每组5只,其中融合蛋白组以TRAIL-NGR组剂量10mg·kg-1为基准,TRAIL和RGD-TRAIL-NGR组分别给予相同摩尔质量的剂量,接种后的第8天开始通过尾静脉注射给药,每3天注射一次,共给药4次。29天活体成像后处死动物,取各只裸鼠完整的肺组织,用Bouin’s液中固定48h,正常的肺组织呈棕黄色,而癌灶为白色隆起,统计肺表面的转移瘤结节数。
结果如图8所示,TRAIL组与TRAIL-NGR组抑制作用近似,与对照组有显著性差异(P<0.05),而RGD-TRAIL-NGR组更加明显地抑制了肿瘤细胞的转移,与TRAIL组和TRAIL-NGR组相比差异显著(P<0.05)。从而证明,RGD-TRAIL-NGR该多靶点融合蛋白具有较好的体内治疗效果。
序列表
<110> 中国医学科学院医药生物技术研究所
<120> 一种抗肿瘤侵袭转移的多靶点融合蛋白的制备及用途
<160> 4
<210> 1
<211> 208
<212> PRT
<213> 人工序列
<400> 1
Ala Cys Asp Cys Arg Gly Asp Cys Phe Cys Gly Gly Gly Gly Gly Ser
5 10 15
Val Arg Glu Arg Gly Pro Gln Arg Val Ala Ala His Ile Thr Gly Thr
20 25 30
Arg Gly Arg Ser Asn Thr Leu Ser Ser Pro Asn Ser Lys Asn Glu Lys
35 40 45
Ala Leu Gly Arg Lys Ile Asn Ser Trp Glu Ser Ser Arg Ser Gly His
50 55 60
Ser Phe Leu Ser Asn Leu His Leu Arg Asn Gly Glu Leu Val Ile His
65 70 75 80
Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser Gln Thr Tyr Phe Arg Phe Gln
85 90 95
Glu Glu Ile Lys Glu Asn Thr Lys Asn Asp Lys Gln Met Val Gln Tyr
100 105 110
Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp Pro Ile Leu Leu Met Lys Ser
115 120 125
Ala Arg Asn Ser Cys Trp Ser Lys Asp Ala Glu Tyr Gly Leu Tyr Ser
130 135 140
Ile Tyr Gln Gly Gly Ile Phe Glu Leu Lys Glu Asn Asp Arg Ile Phe
145 150 155 160
Val Ser Val Thr Asn Glu His Leu Ile Asp Met Asp His Glu Ala Ser
165 170 175
Phe Phe Gly Ala Phe Leu Val Gly Gly Gly Gly Gly Ser Cys Asn Gly
180 185 190
Arg Cys Val Ser Gly Cys Ala Gly Arg Cys His His His His His His
195 200 205
<210> 2
<211> 624
<212> DNA
<213> 人工序列
<400> 2
gcttgtgatt gtagaggaga ctgtttttgt ggtggtggtg gtggtagtgt tagagaaaga 60
ggacctcaga gagttgctgc ccatatcact ggaacaagag gtagatccaa cactttgtct 120
tccccaaact caaagaatga aaaagccctt ggaagaaaga ttaattcttg ggagtcaagt 180
agatctggtc attccttttt gtcaaacttg caccttagaa atggagaact tgttattcat 240
gagaagggtt tctactacat ctactctcaa acctacttca gattccagga agagattaag 300
gaaaacacta agaatgataa gcaaatggtc cagtacatct acaaatacac aagttaccca 360
gaccctatct tgcttatgaa gtctgcaaga aacagttgtt ggtctaaaga tgctgagtat 420
ggattgtaca gtatctacca aggtggaatc tttgaactta aggagaacga cagaattttc 480
gtttccgtca ctaatgaaca tttgatcgat atggaccacg aggcttcctt tttcggtgcc 540
tttttggttg gtggaggtgg aggttcatgt aacggaagat gcgtctctgg atgtgctgga 600
agatgccacc accaccacca ccac 624
<210> 3
<211> 192
<212> PRT
<213> 人工序列
<400> 3
Val Arg Glu Arg Gly Pro Gln Arg Val Ala Ala His Ile Thr Gly Thr
5 10 15
Arg Gly Arg Ser Asn Thr Leu Ser Ser Pro Asn Ser Lys Asn Glu Lys
20 25 30
Ala Leu Gly Arg Lys Ile Asn Ser Trp Glu Ser Ser Arg Ser Gly His
35 40 45
Ser Phe Leu Ser Asn Leu His Leu Arg Asn Gly Glu Leu Val Ile His
50 55 60
Glu Lys Gly Phe Tyr Tyr Ile Tyr Ser Gln Thr Tyr Phe Arg Phe Gln
65 70 75 80
Glu Glu Ile Lys Glu Asn Thr Lys Asn Asp Lys Gln Met Val Gln Tyr
85 90 95
Ile Tyr Lys Tyr Thr Ser Tyr Pro Asp Pro Ile Leu Leu MET Lys Ser
100 105 110
Ala Arg Asn Ser Cys Trp Ser Lys Asp Ala Glu Tyr Gly Leu Tyr Ser
115 120 125
Ile Tyr Gln Gly Gly Ile Phe Glu Leu Lys Glu Asn Asp Arg Ile Phe
130 135 140
Val Ser Val Thr Asn Glu His Leu Ile Asp Met Asp His Glu Ala Ser
145 150 155 160
Phe Phe Gly Ala Phe Leu Val Gly Gly Gly Gly Gly Ser Cys Asn Gly
165 170 175
Arg Cys Val Ser Gly Cys Ala Gly Arg Cys His His His His His His
180 185 190
<210> 4
<211> 576
<212> DNA
<213> 人工序列
<400> 4
gttagagaaa gaggacctca gagagttgct gcccatatca ctggaacaag aggtagatcc 60
aacactttgt cttccccaaa ctcaaagaat gaaaaagccc ttggaagaaa gattaattct 120
tgggagtcaa gtagatctgg tcattccttt ttgtcaaact tgcaccttag aaatggagaa 180
cttgttattc atgagaaggg tttctactac atctactctc aaacctactt cagattccag 240
gaagagatta aggaaaacac taagaatgat aagcaaatgg tccagtacat ctacaaatac 300
acaagttacc cagaccctat cttgcttatg aagtctgcaa gaaacagttg ttggtctaaa 360
gatgctgagt atggattgta cagtatctac caaggtggaa tctttgaact taaggagaac 420
gacagaattt tcgtttccgt cactaatgaa catttgatcg atatggacca cgaggcttcc 480
tttttcggtg cctttttggt tggtggaggt ggaggttcat gtaacggaag atgcgtctct 540
ggatgtgctg gaagatgcca ccaccaccac caccac 576
- 还没有人留言评论。精彩留言会获得点赞!