GNAS基因突变体及其应用的制作方法
本发明属于医药生物领域,涉及GNAS基因突变体、一种用于显性遗传疾病的家系筛查和产前基因筛查的GNAS基因突变体及其应用。
背景技术:
GNAS基因位于人类20号染色体的长臂,是一个复合的印记基因,能转录产生多种基因产物。其中最重要的产物是其1-13号外显子编码的刺激性G蛋白α亚单位(Gsα)。G蛋白是一种无所不在的信号传导蛋白,能通过产生第二信使环磷酸腺苷(cAMP)介导多种激素发挥作用。编码Gsα的GNAS基因突变可以导致Gsα表达减少和/或功能下降导致Albright遗传性骨营养不良(AHO)。同时GNAS还是一个组织特异的印记基因,在近端肾小管、甲状腺、垂体和卵巢组织中主要表达母系等位基因。母系来源的突变导致AHO表型和多种激素如甲状旁腺素(PTH),促甲状腺激素(TSH)和促性腺激素抵抗,即假性甲状旁腺功能减退Ia型。而父系来源的突变仅导致AHO表型,即假-假性甲状旁腺功能减退[1]。
GNAS基因突变分布在整个Gsα编码和剪接区域,将近50%为导致移码突变的缺失或插入突变,其他突变还包括错义突变、无义突变、剪接位点突变、框内缺失或插入以及全部或部分基因缺失。其中位于外显子7的密码子189-190位的4bp缺失是个热点突变。在亚洲人群中的研究并不多,突变类型以移码突变和错义突变为主,也发现了少量无义突变、剪接位点突变和全部基因缺失[1]。
PCR即聚合酶链反应,该技术是在模板DNA,引物和四种脱氧核糖核苷酸存在下,依赖于DNA聚合酶的酶促合成反应。DNA聚合酶以单链DNA为模板,通过人工合成的寡核苷酸引物与单链DNA模板中的一段互补序列结合,形成部分双链。在适宜的温度和环境下,DNA聚合酶将脱氧单核苷酸加到引物3’-OH末端,并以此为起始点,沿模板5’→3’方向延伸,合成一条新的DNA互补链。降落PCR(touchdown PCR)为一种PCR技术,主要用于PCR的条件的优化。在许多情况下引物的设计使得PCR难以进行,例如特异性不够易错配等。降落PCR提供了一个较为简易的优化方法,其原理大致为:首先在较高的温度下扩增,此时虽然扩增效率低,但非特异扩增基本没有。随着退火温度的降低,非特异扩增会逐步增多。但由于此时特异的扩增产物已经达到一定的数量优势,因此会对非特异扩增产生强烈的竞争抑制,从而大幅提高PCR的特异性和效率。退火温度从高于Tm值5℃开始,每个循环递减1℃,直到低于Tm值5℃,一般用10个循环,之后用低于Tm值5℃作为退火温度,再进行20-25个循环。
基因测序原理简介:DNA双脱氧测序法是Sanger等于1977年首创,该技术利用双脱氧核苷三磷酸(ddNTP)作为测序反应链终止剂。ddNTP与普通脱氧核糖核苷三磷酸(dNTP)不同之处在于它们在脱氧核糖的3’端缺少一个羟基。它们可以在DNA聚合酶作用下通过5’端三磷酸集团掺入正常延伸的DNA链中。但由于没有3’羟基,它们不能同后续的dNTP形成磷酸二酯键,因此,正在延伸的DNA链不能继续延伸而终止。美国ABI PRISM 3100测序仪所用试剂BigDye中包含4种荧光标志物,4种标志物分别与4种ddNTP相连。因此,测序反应中形成随机终止的核酸片段混合物。该混合物经测序仪上的毛细管电泳,通过特殊波长的激光扫描和计算机处理后,把核苷酸链上的各碱基信号以图谱形式表现出来,这就是我们所见到的测序图谱。
技术实现要素:
本研究需要通过目的基因DNA提取、引物设计、降落PCR以及基因测序技术找到患病个体中的GNAS基因突变位点和类型,从而指导临床医生对患者家系成员进行同一基因位点的筛查,以及指导产前基因筛查。
本发明提供了一种分离的GNAS基因突变体的核酸及其多肽。
一种分离的GNAS基因突变体的核酸,所述GNAS基因(SEQ ID NO:1)c.310delG缺失(即第4外显子第53位G碱基缺失,序列为SEQ ID NO:2)。
一种分离的多肽,所述多肽是GNAS基因(SEQ ID NO:1)c.310delG缺失的核酸编码的(序列为SEQ ID NO:4)。
另外本发明还提供了一种检测GNAS基因突变体的方法。
一种非诊断目的检测所述GNAS基因突变体的方法,包括使用如下引物进行扩增的步骤:上游引物5’-TACTCCTAACTGACATGGTG-3’(序列为SEQ ID NO:5);下游引物5’-ATATGGACACTGTGCTCAGG-3’(序列为SEQ ID NO:6)。
具体包括如下步骤:
(1)基因组DNA的提取
采用血液基因组DNA提取系统(0.1-20ml)RelaxGene Blood DNA System提取全血基因组DNA;
(2)GNAS基因1-13号外显子的PCR扩增
1)PCR反应体系
GNAS基因1-13号外显子引物序列
25μl体系:
2)PCR反应程序(1-13号外显子)
(3)PCR产物的检测、回收和测序
PCR产物采用1%琼脂糖凝胶电泳进行检测,严格按照天根普通琼脂糖凝胶DNA回收试剂盒的操作步骤进行PCR产物纯化,并送杭州擎科梓熙生物技术有限公司测序。
更进一步的,本发明还提供了非诊断目的的筛选假性甲状旁腺功能减退症的生物样品的方法,包括如下步骤:
从生物样品中提取核酸样本;
通过上述方法确定所述核酸样本的核酸序列,并进行检测;
若所述核酸样品的核酸序列的GNAS基因c.310delG缺失是所述生物样品或生物样品来源及其家族成员易患假性甲状旁腺功能减退症的指示。
另外,本发明提供了检测试剂盒。
一种用于筛选假性甲状旁腺功能减退症的生物样品的试剂盒,包含检测GNAS基因突变体的试剂,所述GNAS基因c.310delG缺失。
一种用于胎儿产前筛查的试剂盒,包含检测GNAS基因突变体的试剂,所述GNAS基因c.310delG缺失。
其中,所述试剂为核酸探针。
所述试剂盒(或核酸探针)包括一组引物对,上游引物5’-TACTCCTAACTGACATGGTG-3’;下游引物5’-ATATGGACACTGTGCTCAGG-3’。
另外本发明提供试剂盒在假性甲状旁腺功能减退症家系筛查和产前筛查中的应用。
本发明通过全血标本采集、引物设计、基因组DNA的提取、GNAS基因1-13号外显子的PCR扩增、PCR产物的检测、回收和测序获得GNAS基因突变结果:
GNAS基因1-13号外显子(包含紧邻的内含子片段)均扩增出目标条带(附图1)。通过Chromas及DNAMAN软件对测序结果进行比对分析,发现患者GNAS第4外显子第53位G碱基缺失导致移码突变,根据国际上默认的表示方法,表示为c.310delG(附图2)。
通过对突变序列编码的氨基酸和蛋白结构进行模拟预测(DNAMAN软件和Swiss model在线同源建模(http://swissmodel.expasy.org//SWISS-MODEL.html)[3],发现患者GNAS基因c.310delG的缺失突变可能导致蛋白质翻译移码,并可能导致GNAS基因编码的第104位谷氨酸变为赖氨酸,并于第110位氨基酸处发生提前终止(附图3),可能产生截短蛋白或被降解(附图4)。
本发明的优点和效果:该方法简便,只需要对患者家系成员针对性地检测已发现的突变,即可从分子生物学角度诊断假性甲状旁腺功能减退症。对患者的胎儿进行产前筛查,可指导优生。
附图说明
图1为GNAS基因1-13号外显子扩增电泳图。条带从左到右依次为外显子1,2,3,4/5,6,7/8,9/10,11/12,13,阴性对照,分子标记物。
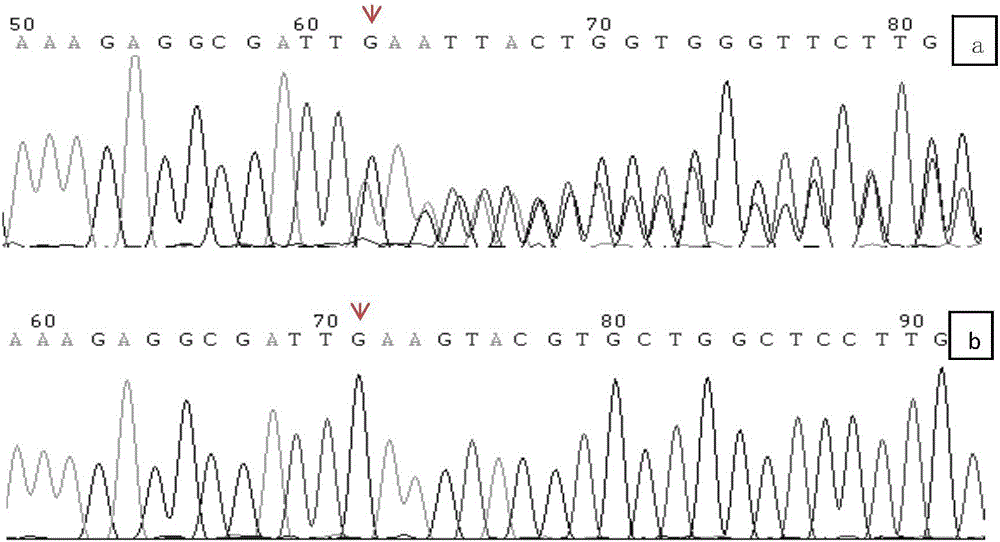
图2 患者(a)和正常人(b)的GNAS外显子4/5基因序列。a图中出现了两种序列,一种为正常序列,另一种为突变序列。由于箭头所指位置G(黑色信号)缺失,下一位点的A(绿色信号)提前出现,即突变位点之后的每一个核苷酸都提前了一个位点。
图3 正常GNAS和突变GNAS编码序列对应的氨基酸序列。
图4 同源建模后的正常Gsα蛋白三维结构(左图)与突变后Gsα蛋白三维结构(右图)。
具体实施方式
下面结合具体实施例进一步阐述本发明,应理解,以下实施例仅用于说明本发明而不用于限制本发明的保护范围。
本发明实施例中所需要的材料、试剂除特殊说明均可市场购得。所使用的方法非特殊说明均为本领域常规方法。
本发明提供了一种分离的GNAS基因突变体的核酸及其多肽。
分离的GNAS基因突变体的核酸,所述GNAS基因(SEQ ID NO:1)c.310delG缺失(即第4外显子第53位G碱基缺失,序列为SEQ ID NO:2)。
GNAS基因1-13号外显子(包含紧邻的内含子片段)均扩增出目标条带(附图1)。通过Chromas及DNAMAN软件对测序结果进行比对分析,发现患者GNAS第4外显子第53位G碱基缺失导致移码突变,根据国际上默认的表示方法,表示为c.310delG(附图2)。
正常GNAS基因核苷酸序列:(NCBI参考序列NM_000516.4)
突变GNAS基因核苷酸序列:
一种分离的多肽,所述多肽是GNAS基因(SEQ ID NO:1)c.310delG缺失的核酸编码的(序列为SEQ ID NO:4)。
通过对突变序列编码的氨基酸和蛋白结构进行模拟预测(DNAMAN软件和Swiss model在线同源建模(http://swissmodel.expasy.org//SWISS-MODEL.html)[3],发现患者GNAS基因c.310delG的缺失突变可能导致蛋白质翻译移码,并可能导致GNAS基因编码的第104位谷氨酸变为赖氨酸,并于第110位氨基酸处发生提前终止(附图3),可能产生截短蛋白或被降解(附图4)。进而判断是否易患假性甲状旁腺功能减退症。
正常GNAS基因编码的氨基酸序列:
突变GNAS基因编码的氨基酸序列:
本发明提供了检测GNAS基因突变体的方法,包括如下步骤:
(1)全血标本采集
全血(由浙江大学第一附属医院提供)装入含EDTANa4的抗凝管,放入一80℃冰箱保存,以备提取DNA时应用。
(2)引物设计
引物参考已有文献。根据GNAS基因序列(Genebank ID:NT 011362)、应用Primmer5软件对GNAS基因1-13号外显子(包含紧邻的内含子片段)分别设计特异性引物,应用MethPrimer软件每个甲基化区域分别设计两对引物,甲基化特异性引物MF/MR及非甲基化特异性引物UF/UR,所有引物由杭州擎科梓熙生物技术有限公司合成。引物参见表1.1[2]。
表1.1 GNAS基因1-13号外显子引物序列
(3)基因组DNA的提取
采用血液基因组DNA提取系统(0.1-20ml)RelaxGene Blood DNA System提取全血基因组DNA,按照说明书进行。
(4)GNAS基因1-13号外显子的PCR扩增
1)PCR反应体系
25μl体系:
2)PCR反应程序(1-13号外显子)
(5)PCR产物的检测、回收和测序
PCR产物采用1%琼脂糖凝胶电泳进行检测,严格按照天根普通琼脂糖凝胶DNA回收试剂盒的操作步骤进行PCR产物纯化,并送杭州擎科梓熙生物技术有限公司测序。
本发明还提供根据检测GNAS基因突变体,进而筛选假性甲状旁腺功能减退症的生物样品的方法。
(1)全血标本采集
假性甲状旁腺功能减退症患者及正常人的全血(由浙江大学第一附属医院提供)装入含EDTANa4的抗凝管,放入一80℃冰箱保存,以备提取DNA时应用。
(2)引物设计
引物参考已有文献。根据GNAS基因序列(Genebank ID:NT 011362)、应用Primmer5软件对GNAS基因1-13号外显子(包含紧邻的内含子片段)分别设计特异性引物,应用MethPrimer软件每个甲基化区域分别设计两对引物,甲基化特异性引物MF/MR及非甲基化特异性引物UF/UR,所有引物由杭州擎科梓熙生物技术有限公司合成。引物参见表1.1[2]。
表1.1 GNAS基因1-13号外显子引物序列
(3)基因组DNA的提取
采用血液基因组DNA提取系统(0.1-20ml)RelaxGene Blood DNA System提取全血基因组DNA,按照说明书进行。
(4)GNAS基因1-13号外显子的PCR扩增
1)PCR反应体系
25μl体系:
2)PCR反应程序(1-13号外显子)
(5)PCR产物的检测、回收和测序
PCR产物采用1%琼脂糖凝胶电泳进行检测,严格按照天根普通琼脂糖凝胶DNA回收试剂盒的操作步骤进行PCR产物纯化,并送杭州擎科梓熙生物技术有限公司测序。
GNAS基因1-13号外显子(包含紧邻的内含子片段)均扩增出目标条带(附图1)。通过Chromas及DNAMAN软件对测序结果进行比对分析,发现患者GNAS第4外显子第53位G碱基缺失导致移码突变,根据国际上默认的表示方法,表示为c.310delG(附图2)。
参考文献
[1]Turan S,Bastepe M.The GNAS complex locus and human diseases associated with loss-of-function mutations or epimutations within this imprinted gene.Hormone research in paediatrics 2013;80:229-41.
[2]Zhang XL.Analysis of Pseudohypoparathyroidism and GNAS1Gene[D].Zhengzhou:Zhengzhou University,2011:21
[3]Arnold K.,Bordoli L.,Kopp J.,and Schwede T.(2006).The SWISS-MODEL Workspace:A web-based environment for protein structure homology modeling.Bioinformatics,22,195-201.
SEQUENCE LISTING
<110> 浙江大学
<120> GNAS基因突变体及其应用
<130> 21-2016
<160> 6
<170> PatentIn version 3.3
<210> 1
<211> 1926
<212> DNA
<213> 人类Homo sapiens (human)
<400> 1
ggcgggggcc cggccgaggc aataagagcg gcggcggcgg cagcggcggc agcagctccc 60
gcagctcctg ctctggtccg cctcggcccg gcggcggcca tcagccccct cggcctcggc 120
tcgaggggcg gggagctgcg cgcgcccctc ggtccgaccg acaccctccc cttcccgccc 180
gtccgcgcgc cccgcggccc gcggcccgca gtccgccccg cgcgctcctt gccgaggagc 240
cgagcccgcg cccggcccgc ccgcccggcg ctgccccggc cctcccggcc cgcgtgaggc 300
cgcccgcgcc cgccgccgcc gcagcccggc cgcgccccgc cgccgccgcc gccgccatgg 360
gctgcctcgg gaacagtaag accgaggacc agcgcaacga ggagaaggcg cagcgtgagg 420
ccaacaaaaa gatcgagaag cagctgcaga aggacaagca ggtctaccgg gccacgcacc 480
gcctgctgct gctgggtgct ggagaatctg gtaaaagcac cattgtgaag cagatgagga 540
tcctgcatgt taatgggttt aatggagagg gcggcgaaga ggacccgcag gctgcaagga 600
gcaacagcga tggtgagaag gcaaccaaag tgcaggacat caaaaacaac ctgaaagagg 660
cgattgaaac cattgtggcc gccatgagca acctggtgcc ccccgtggag ctggccaacc 720
ccgagaacca gttcagagtg gactacatcc tgagtgtgat gaacgtgcct gactttgact 780
tccctcccga attctatgag catgccaagg ctctgtggga ggatgaagga gtgcgtgcct 840
gctacgaacg ctccaacgag taccagctga ttgactgtgc ccagtacttc ctggacaaga 900
tcgacgtgat caagcaggct gactatgtgc cgagcgatca ggacctgctt cgctgccgtg 960
tcctgacttc tggaatcttt gagaccaagt tccaggtgga caaagtcaac ttccacatgt 1020
ttgacgtggg tggccagcgc gatgaacgcc gcaagtggat ccagtgcttc aacgatgtga 1080
ctgccatcat cttcgtggtg gccagcagca gctacaacat ggtcatccgg gaggacaacc 1140
agaccaaccg cctgcaggag gctctgaacc tcttcaagag catctggaac aacagatggc 1200
tgcgcaccat ctctgtgatc ctgttcctca acaagcaaga tctgctcgct gagaaagtcc 1260
ttgctgggaa atcgaagatt gaggactact ttccagaatt tgctcgctac actactcctg 1320
aggatgctac tcccgagccc ggagaggacc cacgcgtgac ccgggccaag tacttcattc 1380
gagatgagtt tctgaggatc agcactgcca gtggagatgg gcgtcactac tgctaccctc 1440
atttcacctg cgctgtggac actgagaaca tccgccgtgt gttcaacgac tgccgtgaca 1500
tcattcagcg catgcacctt cgtcagtacg agctgctcta agaagggaac ccccaaattt 1560
aattaaagcc ttaagcacaa ttaattaaaa gtgaaacgta attgtacaag cagttaatca 1620
cccaccatag ggcatgatta acaaagcaac ctttcccttc ccccgagtga ttttgcgaaa 1680
cccccttttc ccttcagctt gcttagatgt tccaaattta gaaagcttaa ggcggcctac 1740
agaaaaagga aaaaaggcca caaaagttcc ctctcacttt cagtaaaaat aaataaaaca 1800
gcagcagcaa acaaataaaa tgaaataaaa gaaacaaatg aaataaatat tgtgttgtgc 1860
agcattaaaa aaaatcaaaa taaaaattaa atgtgagcaa agaatgaaaa aaaaaaaaaa 1920
aaaaaa 1926
<210> 2
<211> 1925
<212> DNA
<213> 人类Homo sapiens (human)
<400> 2
ggcgggggcc cggccgaggc aataagagcg gcggcggcgg cagcggcggc agcagctccc 60
gcagctcctg ctctggtccg cctcggcccg gcggcggcca tcagccccct cggcctcggc 120
tcgaggggcg gggagctgcg cgcgcccctc ggtccgaccg acaccctccc cttcccgccc 180
gtccgcgcgc cccgcggccc gcggcccgca gtccgccccg cgcgctcctt gccgaggagc 240
cgagcccgcg cccggcccgc ccgcccggcg ctgccccggc cctcccggcc cgcgtgaggc 300
cgcccgcgcc cgccgccgcc gcagcccggc cgcgccccgc cgccgccgcc gccgccatgg 360
gctgcctcgg gaacagtaag accgaggacc agcgcaacga ggagaaggcg cagcgtgagg 420
ccaacaaaaa gatcgagaag cagctgcaga aggacaagca ggtctaccgg gccacgcacc 480
gcctgctgct gctgggtgct ggagaatctg gtaaaagcac cattgtgaag cagatgagga 540
tcctgcatgt taatgggttt aatggagagg gcggcgaaga ggacccgcag gctgcaagga 600
gcaacagcga tggtgagaag gcaaccaaag tgcaggacat caaaaacaac ctgaaagagg 660
cgattaaacc attgtggccg ccatgagcaa cctggtgccc cccgtggagc tggccaaccc 720
cgagaaccag ttcagagtgg actacatcct gagtgtgatg aacgtgcctg actttgactt 780
ccctcccgaa ttctatgagc atgccaaggc tctgtgggag gatgaaggag tgcgtgcctg 840
ctacgaacgc tccaacgagt accagctgat tgactgtgcc cagtacttcc tggacaagat 900
cgacgtgatc aagcaggctg actatgtgcc gagcgatcag gacctgcttc gctgccgtgt 960
cctgacttct ggaatctttg agaccaagtt ccaggtggac aaagtcaact tccacatgtt 1020
tgacgtgggt ggccagcgcg atgaacgccg caagtggatc cagtgcttca acgatgtgac 1080
tgccatcatc ttcgtggtgg ccagcagcag ctacaacatg gtcatccggg aggacaacca 1140
gaccaaccgc ctgcaggagg ctctgaacct cttcaagagc atctggaaca acagatggct 1200
gcgcaccatc tctgtgatcc tgttcctcaa caagcaagat ctgctcgctg agaaagtcct 1260
tgctgggaaa tcgaagattg aggactactt tccagaattt gctcgctaca ctactcctga 1320
ggatgctact cccgagcccg gagaggaccc acgcgtgacc cgggccaagt acttcattcg 1380
agatgagttt ctgaggatca gcactgccag tggagatggg cgtcactact gctaccctca 1440
tttcacctgc gctgtggaca ctgagaacat ccgccgtgtg ttcaacgact gccgtgacat 1500
cattcagcgc atgcaccttc gtcagtacga gctgctctaa gaagggaacc cccaaattta 1560
attaaagcct taagcacaat taattaaaag tgaaacgtaa ttgtacaagc agttaatcac 1620
ccaccatagg gcatgattaa caaagcaacc tttcccttcc cccgagtgat tttgcgaaac 1680
ccccttttcc cttcagcttg cttagatgtt ccaaatttag aaagcttaag gcggcctaca 1740
gaaaaaggaa aaaaggccac aaaagttccc tctcactttc agtaaaaata aataaaacag 1800
cagcagcaaa caaataaaat gaaataaaag aaacaaatga aataaatatt gtgttgtgca 1860
gcattaaaaa aaatcaaaat aaaaattaaa tgtgagcaaa gaatgaaaaa aaaaaaaaaa 1920
aaaaa 1925
<210> 3
<211> 394
<212> PRT
<213> 人类Homo sapiens (human)
<400> 3
Met Gly Cys Leu Gly Asn Ser Lys Thr Glu Asp Gln Arg Asn Glu Glu
1 5 10 15
Lys Ala Gln Arg Glu Ala Asn Lys Lys Ile Glu Lys Gln Leu Gln Lys
20 25 30
Asp Lys Gln Val Tyr Arg Ala Thr His Arg Leu Leu Leu Leu Gly Ala
35 40 45
Gly Glu Ser Gly Lys Ser Thr Ile Val Lys Gln Met Arg Ile Leu His
50 55 60
Val Asn Gly Phe Asn Gly Glu Gly Gly Glu Glu Asp Pro Gln Ala Ala
65 70 75 80
Arg Ser Asn Ser Asp Gly Glu Lys Ala Thr Lys Val Gln Asp Ile Lys
85 90 95
Asn Asn Leu Lys Glu Ala Ile Glu Thr Ile Val Ala Ala Met Ser Asn
100 105 110
Leu Val Pro Pro Val Glu Leu Ala Asn Pro Glu Asn Gln Phe Arg Val
115 120 125
Asp Tyr Ile Leu Ser Val Met Asn Val Pro Asp Phe Asp Phe Pro Pro
130 135 140
Glu Phe Tyr Glu His Ala Lys Ala Leu Trp Glu Asp Glu Gly Val Arg
145 150 155 160
Ala Cys Tyr Glu Arg Ser Asn Glu Tyr Gln Leu Ile Asp Cys Ala Gln
165 170 175
Tyr Phe Leu Asp Lys Ile Asp Val Ile Lys Gln Ala Asp Tyr Val Pro
180 185 190
Ser Asp Gln Asp Leu Leu Arg Cys Arg Val Leu Thr Ser Gly Ile Phe
195 200 205
Glu Thr Lys Phe Gln Val Asp Lys Val Asn Phe His Met Phe Asp Val
210 215 220
Gly Gly Gln Arg Asp Glu Arg Arg Lys Trp Ile Gln Cys Phe Asn Asp
225 230 235 240
Val Thr Ala Ile Ile Phe Val Val Ala Ser Ser Ser Tyr Asn Met Val
245 250 255
Ile Arg Glu Asp Asn Gln Thr Asn Arg Leu Gln Glu Ala Leu Asn Leu
260 265 270
Phe Lys Ser Ile Trp Asn Asn Arg Trp Leu Arg Thr Ile Ser Val Ile
275 280 285
Leu Phe Leu Asn Lys Gln Asp Leu Leu Ala Glu Lys Val Leu Ala Gly
290 295 300
Lys Ser Lys Ile Glu Asp Tyr Phe Pro Glu Phe Ala Arg Tyr Thr Thr
305 310 315 320
Pro Glu Asp Ala Thr Pro Glu Pro Gly Glu Asp Pro Arg Val Thr Arg
325 330 335
Ala Lys Tyr Phe Ile Arg Asp Glu Phe Leu Arg Ile Ser Thr Ala Ser
340 345 350
Gly Asp Gly Arg His Tyr Cys Tyr Pro His Phe Thr Cys Ala Val Asp
355 360 365
Thr Glu Asn Ile Arg Arg Val Phe Asn Asp Cys Arg Asp Ile Ile Gln
370 375 380
Arg Met His Leu Arg Gln Tyr Glu Leu Leu
385 390
<210> 4
<211> 109
<212> PRT
<213> 人类Homo sapiens (human)
<400> 4
Met Gly Cys Leu Gly Asn Ser Lys Thr Glu Asp Gln Arg Asn Glu Glu
1 5 10 15
Lys Ala Gln Arg Glu Ala Asn Lys Lys Ile Glu Lys Gln Leu Gln Lys
20 25 30
Asp Lys Gln Val Tyr Arg Ala Thr His Arg Leu Leu Leu Leu Gly Ala
35 40 45
Gly Glu Ser Gly Lys Ser Thr Ile Val Lys Gln Met Arg Ile Leu His
50 55 60
Val Asn Gly Phe Asn Gly Glu Gly Gly Glu Glu Asp Pro Gln Ala Ala
65 70 75 80
Arg Ser Asn Ser Asp Gly Glu Lys Ala Thr Lys Val Gln Asp Ile Lys
85 90 95
Asn Asn Leu Lys Glu Ala Ile Lys Pro Leu Trp Pro Pro
100 105
<210> 5
<211> 20
<212> DNA
<213> 人类Homo sapiens (human)
<400> 5
tactcctaac tgacatggtg 20
<210> 6
<211> 20
<212> DNA
<213> 人类Homo sapiens (human)
<400> 6
atatggacac tgtgctcagg 20
- 还没有人留言评论。精彩留言会获得点赞!