与猪瘟病毒E2蛋白特定区域结合多肽配基设计及应用的制作方法
本发明涉及与猪瘟病毒E2蛋白特定区域结合多肽配基设计及应用,属于多肽设计及病毒抗原检测领域。
背景技术:
基于分子对接的虚拟筛选技术是一种新兴的研究多肽与蛋白质之间相互作用的技术手段。这一技术主要是运用计算机快速运算实现多肽与相应靶标蛋白在空间构象上的对接,将虚拟多肽数据库中的分子逐一与靶标蛋白晶体结构的特定活性位点进行对接,通过计算机快速运算并不断调整多肽与靶标蛋白结合的位置、构象、分子内部可旋转键的二面角和靶标蛋白的氨基酸残基侧链和骨架,寻找多肽分子与靶标蛋白在空间结构上的最佳构象,并预测两者之间的结合模式与亲和力,通过评分值挑选出接近天然构象的与靶标蛋白亲和力最佳的多肽配体的一种理论模拟分子间相互作用的方法。
猪瘟病毒(Classical Swine Fever Virus,CSFV)是造成猪群爆发猪霍乱,我国也称之为烂肠瘟的主要病原。它是以发病猪群出现高热、出血灶和白细胞减少症为主要特征的一种高发病率和死亡率的烈性传染病。该病毒在全世界范围内广泛分布,给全球的养殖业带来了极大的影响与损失。CSFV是黄病毒科,瘟病毒属的成员之一,与牛病毒性腹泻病毒(Bovine Viral Diarrhea Virus,BVDV)在结构上具有很高的相似性。CSFV属于有囊膜的单股正链RNA病毒,其基因组长约12.3kb,形成一个大的开放阅读框(open reading frame,ORF),编码一个由3989个氨基酸组成的多聚蛋白。E2蛋白是一种典型的位于跨膜区的囊膜糖蛋白,在病毒吸附及侵入宿主细胞过程中发挥着至关重要的作用。同时,它作为CSFV的免疫优势蛋白,可以诱导机体产生高滴度的中和抗体水平,从而使机体具备可抵抗强毒株感染的能力,是研制针对CSFV新型疫苗的首选靶蛋白,因此E2蛋白也成为许多科研工作者的主要研究对象。
技术实现要素:
本发明借助于分子对接及虚拟筛选技术,在黄病毒科病毒E2蛋白晶体结构的基础上,通过分子对接技术,搜寻虚拟多肽库中与靶标蛋白结合模式与亲和力最佳的多肽配基,其多肽配基序列为FYKRTSSS,即Pep1。人工合成Pep1,利用表达纯化的CSFV E2蛋白进行表面等离子体共振(Surface Plasmon Resonance,SPR)与ELISA结合试验,结果表明,人工合成的Pep1与CSFV E2蛋白有良好的结合能力,由此证明,借助本发明设计而成的多肽配基,可用于对猪瘟病毒抗原进行定量和定性的快速检测。
为了实现上述目的,本发明所采用的技术方案是:
与猪瘟病毒E2蛋白特定区域结合的多肽配基,所述的多肽配基序列为FYKRTSSS。
所述的与猪瘟病毒E2蛋白特定区域结合的多肽配基,包括以所述的多肽配基序列为核心,任何对该多肽配基序列所进行的相应调整或修饰;修饰材料包括但不限制在纳米材料、荧光材料、酶类、生物素及特定的蛋白质。
将所述的多肽配基序列用于猪瘟病毒或者牛病毒性腹泻病毒的E2蛋白的鉴定,其中包括但不局限于酶联免疫吸附试验(ELISA)检测。
一种所述的多肽配基在猪瘟病毒或者牛病毒性腹泻病毒E2蛋白的快速检测中的应用。
一种所述的多肽配基在对猪瘟病毒抗原进行定量和定性检测中的应用。
本发明的有益效果:
1、本发明借助于分子对接及虚拟筛选技术,在黄病毒科病毒E2蛋白晶体结构的基础上,通过分子对接技术,搜寻虚拟多肽库中与靶标蛋白结合模式与亲和力最佳的多肽配体,最终得到可与猪瘟病毒E2蛋白结合的多肽配基,其多肽配基序列为FYKRTSSS,即Pep1。人工合成Pep1,并与猪瘟病毒E2蛋白进行亲和力鉴定,Pep1序列与人工表达的CSFV E2蛋白之间相互作用的平衡解离常数KD为4.72×10-6M,即4.72μM,说明亲和力较好。
2、由于猪瘟病毒纯化技术的局限性,针对猪瘟病毒的抗体获得很难,本发明设计得到的Pep1序列很好地避免了这一难题,实现快速人工合成与低廉检测成本。
3、本发明设计而成的Pep1序列与人工接种CSFV、人工表达CSFV E2蛋白均可以结合,除与BVDV E2蛋白反应性较高外,与其它病毒蛋白均不发生交叉性反应,特异性较好。
4、本发明与传统噬菌体多肽库筛选相比,具有简单,快速及成本低的特点;通过计算机辅助实施分子对接,可为实现猪瘟病毒E2蛋白结构功能解析提供较好的理论指导。
5、本发明与人工表达纯化的蛋白进行免疫来获得针对蛋白抗体的过程相比,具有操作简单,省时省力,费用低等优点;通过对筛选Pep1序列进行标记,可实现对猪瘟病毒E2蛋白进行定性和定量的快速检测。
附图说明
图1为Pep1序列与CSFV E2蛋白的对接结果展示。
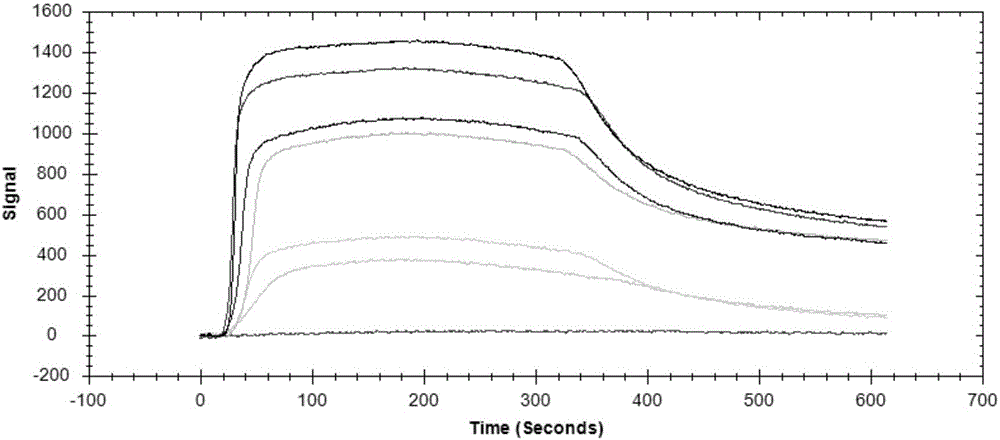
图2为Pep1序列与人工表达CSFV E2蛋白的SPR亲和力鉴定结果。其中,纵坐标代表传感器所检测到的信号值;横坐标代表样品在传感器中相互作用的时间。图中,从上到下各曲线对应的Pep1溶液浓度逐渐降低。
图3为Pep1序列与人工表达CSFV E2蛋白的ELISA鉴定结果。
图4为Pep1序列与人工接种CSFV的ELISA鉴定结果。
具体实施方式
以下结合实施例对本发明的具体实施方式作进一步详细说明。
实施例1分子对接及虚拟肽库的筛选
1、E2蛋白的准备
从PDB数据库中搜寻黄病毒科病毒E2蛋白的晶体结构(4JNT),借助计算机程序对晶体结构进行分析,选定其第800到900氨基酸残基作为对接设定区域,以进行分子对接。
2、虚拟多肽库的设计
建立不同氨基酸残基的空间结构,借助计算机程序,实现对输入目标多肽进行批量生成,以满足分子对接计算机程序的自动调用和处理。虚拟多肽库均以直链形式生成,不对任何侧链和首尾氨基和羧基进行修饰。单一虚拟多肽库的生成以不超过四位氨基酸残基为宜。
3、对接结果的评断
分别计算多肽与蛋白结合的自由能,氢链,范得华力等力学参数进行综合评定,以此来判定筛选结果,并筛选得到Pep1,其多肽序列为苯丙氨酸-酪氨酸-赖氨酸-精氨酸-苏氨酸-丝氨酸-丝氨酸-丝氨酸(Phe-Tyr-Lys-Arg-Thr-Ser-Ser-Ser,简写FYKRTSSS)(SEQ ID NO.1),其与CSFV E2蛋白对接的相互作用位置结果见图1。
实施例2 Pep1序列与人工表达CSFV E2蛋白的亲和力鉴定
1、将人工表达纯化的CSFV E2蛋白采用PBS缓冲液(pH 7.4)稀释为1μg/ml(蛋白量计),采用活泼酯法,分别将1-(3-二甲氨基丙基)-3-乙基碳二亚胺/N-羟基琥珀酰亚胺(EDC/NHS,1:1)、CSFV E2蛋白注入安装有氨基芯片的SPR检测仪中,保证EDC/NHS和CSFV E2蛋白分别与氨基芯片相互作用5min,实施CSFV E2蛋白与氨基芯片的偶联。偶联完成后,该传感器就可以用于测量CSFV E2蛋白与Pep1序列之间的相互作用。
2、向传感器中注入250μl PBS缓冲液(pH 7.4),以最大流速(150μl/min)运行缓冲液,达到信号基线,将缓冲液的流速降至20μl/min,以获得较为稳定的基线。
3、将人工合成并在氨基端生物素化修饰的Pep1干粉使用PBS缓冲液(pH 7.4)分别稀成浓度为187.185μM、93.5925μM、46.7962μM、23.3981μM、11.699μM、5.8495μM、0.7311μM的Pep1溶液,从低浓度开始依次向传感器中注入250μl的Pep1溶液,每次注入样品均采用20μl/min的流速并且与传感器相互作用5min,最后用PBS缓冲液(pH 7.4)冲洗5min。以得到的不同浓度Pep1溶液与CSFV E2蛋白相互作用的结合与解离曲线为依据,进行Pep1序列与CSFV E2蛋白相结合的亲和力分析(见图2)。
结果表明,Pep1序列与人工表达的CSFV E2蛋白具有较好的亲和性结合,两者之间相互作用的平衡解离常数KD为4.72×10-6M,即4.72μM。
实施例3 Pep1序列与人工表达CSFV E2蛋白的ELISA鉴定
1、将人工表达纯化的CSFV E2蛋白以1μg/ml(蛋白量计)进行酶标板包被;以同样方式将不同病毒表达纯化的蛋白,即牛病毒性腹泻病毒E2蛋白(BVDV-E2)、乙脑病毒E2蛋白(JEV-E2)、圆环病毒Cap蛋白(PCV-Cap)、伪狂犬病毒gE蛋白(PRV-gE),和质量分数2%牛血清白蛋白(BSA)、PBS缓冲液进行酶标板包被,作为对照。其中,包被抗原均采用碳酸盐(CBS)缓冲液进行稀释,每孔50μl加入96孔酶标板中,置于4℃条件下过夜,用PBST缓冲液洗涤5次后再用质量分数2%的BSA液进行封闭。
2、将人工合成并在氨基端生物素化修饰的Pep1干粉使用PBS缓冲液(pH 7.4)稀释成500ng/ml的浓度,以每孔50μl的体积加入到上述酶标板中,混匀后,置于37℃条件下,避光温育30min。
3、用PBST缓冲液洗涤5次,并甩干酶标板孔中的液体;使用PBST缓冲液稀释偶联辣根过氧化物酶的链霉亲和素(1:1000),以每孔50μl的体积加入甩干的酶标板中,混匀后,置于37℃条件下,避光温育30min。
4、根据试验所需量,将TMB显色液以每孔100μl的体积加入到上述酶标板中,充分混匀30s之后,室温条件下,显色10min。
5、将2M的硫酸终止液以每孔50μl的体积加入上述酶标板中,充分混匀30s之后,在酶标仪器上,读取各孔在450nm处的吸光值,判定结果。
结果表明,Pep1序列与人工表达CSFV E2蛋白具有较好的亲和性与特异性结合,除与牛病毒性腹泻病毒E2蛋白的交叉反应较高外,与其它病毒蛋白均不发生反应(见图3)。
实施例4 Pep1序列与人工接种CSFV的ELISA鉴定
(1)将接种CSFV的PK-15细胞培养液进行超声破碎,然后以200TCID50(病毒含量计)进行酶标板包被;以同样方式将不同病毒培养液,即牛病毒性腹泻病毒(BVDV)、乙脑病毒(JEV)、圆环病毒(PCV)、伪狂犬病毒(PRV)的培养液,和同等体积的未接种病毒的PK-15细胞培养液进行酶标板包被,作为对照。其中,包被抗原均采用CBS缓冲液进行稀释,以每孔50μl的体积加入96孔酶标板中,置于4℃条件下过夜,用PBST缓冲液洗涤5次后再用质量分数2%的BSA液封闭。
(2)将人工合成并在氨基端生物素化修饰的Pep1干粉使用PBS缓冲液(pH 7.4)稀释成500ng/ml的浓度,以每孔50μl的体积加入到上述酶标板中,混匀后,置于37℃条件下,避光温育30min。
(3)用PBST缓冲液洗涤5次,并甩干酶标板孔中的液体;使用PBST缓冲液稀释偶联辣根过氧化物酶的链霉亲和素(1:1000),每孔50μl加入甩干的酶标板中,混匀后,置于37℃条件下,避光温育30min。
(4)根据试验所需量,将TMB显色液以每孔100μl的体积加入到上述酶标板中,充分混匀30s之后,室温条件下,显色10min。
(5)将2M的硫酸终止液以每孔50μl的体积加入上述酶标板中,充分混匀30s之后,在酶标仪器上,读取各孔在450nm处的吸光值,判定结果。
结果表明,Pep1序列与人工接种的CSFV细胞培养液具有较好的亲和性与特异性结合,除与牛病毒性腹泻病毒细胞培养液有交叉反应较高外,与其它病毒培养液均不发生反应(见图4)。
SEQUENCE LISTING
<110> 河南省农业科学院
<120> 与猪瘟病毒E2蛋白特定区域结合多肽配基设计及应用
<160> 1
<170> PatentIn version 3.5
<210> 1
<211> 8
<212> PRT
<213> 人工序列
<400> 1
Phe Tyr Lys Arg Thr Ser Ser Ser
1 5
- 还没有人留言评论。精彩留言会获得点赞!