一种催化丙氨酸脱羧的蛋白质及其基因和应用的制作方法
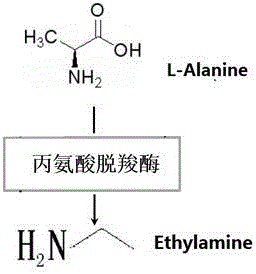
本发明属于生物基因工程技术领域,具体涉及一种催化丙氨酸脱羧的蛋白质及其基因和应用。
背景技术:
茶氨酸(L-Theanine)是茶叶中特有的游离氨基酸,茶氨酸是谷氨酸γ-乙基酰胺,有甜味。茶叶中含有大量的茶氨酸,是形成茶叶风味的主要成分。茶氨酸含量通常占茶叶中其他氨基酸总量的50%以上。
研究发现茶氨酸合成受到前提物质乙胺的调控。在植物组织培养合成茶氨酸的研究中,有学者已经证实向培养基中添加前体物质-盐酸乙胺时,可以大幅度提高愈伤组织中的茶氨酸合成量,而直接添加丙氨酸,则对茶氨酸累积影响不大。茶氨酸合成中的乙胺系由丙氨酸脱羧(CsAlaDC)而来,催化该反应即为丙氨酸脱羧酶。该酶在子叶和根中活性高,在绿色幼苗根中活性高于黄化幼苗,预示着它积极参与茶氨酸合成中所需乙胺的形成。
目前,有关CsAlaDC基因的克隆鉴定未见任何报道。鉴于乙胺的添加可以极大提高茶树愈伤组织和悬浮细胞合成茶氨酸,因此CsAlaDC基因的发现对于研究茶树氮吸收与转运机制具有重要的意义。
技术实现要素:
针对现有技术存在的问题,本发明的目的在于设计提供一种茶树特有丙氨酸脱羧蛋白CsAlaDC及其编码基因和应用的技术方案。
所述的一种催化丙氨酸脱羧的蛋白质,其特征在于该蛋白质的氨基酸序列为:
1)SEQ ID No.2 所示的氨基酸序列 ;或
2)SEQ ID No.2 所示的氨基酸序列经替换、缺失和/或添加一个或几个氨基酸残基形成的具有同等功能的氨基酸序列。
所述编码所述蛋白质的基因,其特征在于该基因的核苷酸序列为:
1)SEQ ID No.1 所示的核苷酸;或
2)SEQ ID No.1 所示的核苷酸序列经取代一个或几个核苷酸,得到编码 CsAlaDC的核苷酸序列。
所述编码基因的重组载体。
所述的编码基因在催化丙氨酸脱羧形成乙胺中的应用。
所述的编码基因在调控茶树茶氨酸合成中的应用。
所述的一种催化丙氨酸脱羧形成乙胺的方法,其特征在于包括如下步骤:将编码基因导入大肠杆菌中,诱导表达得到重组蛋白,将重组蛋白纯化后与丙氨酸溶液混合进行催化得到乙胺,所述的编码基因的核苷酸序列为:
1)SEQ ID No.1 所示的核苷酸;或
2)SEQ ID No.1 所示的核苷酸序列经取代一个或几个核苷酸,得到编码 CsAlaDC的核苷酸序列。
所述的一种催化丙氨酸脱羧形成乙胺的方法,其特征在于丙氨酸脱羧酶的催化反应的条件为10mM 丙氨酸, 100mM的磷酸钾缓冲液,0.08-0.12 mM 磷酸吡哆醛,5mM二硫苏糖醇,1mM K2EDTA,10%的甘油,反应溶液pH 为:pH 7.2-pH 7.8反应温度为35℃-45℃。
本发明的实验证明,本发明在茶树中发现丙氨酸脱羧酶蛋白CsAlaDC,将该蛋白对应的基因在大肠杆菌中过表达,纯化的重组蛋白同时具有催化丙氨酸脱羧的活性,验证了该基因具有催化丙氨酸脱羧的功能。
附图说明
图1 丙氨酸脱酸酶催化反应图示;
图2 茶树CsAlaDC的基因序列;
图3 不同组织CsAlaDC基因qPCR相对定量结果;
图4 CsAlaDC蛋白原核纯化的SDS-PAGE;
图5 重组CsAlaDC蛋白催化丙氨酸脱羧形成乙胺的高效液相(HPLC)检测图谱;
图6 催化反应2h与24h后,反应液中生成乙胺的浓度。
具体实施方式
以下结合实施例来进一步说明本发明。
实施例
(1) 基因克隆:以龙井43的幼根为材料,用试剂盒法提取总RNA;根据CsAlaDC的mRNA序列设计引物,用反转录PCR法获得该基因的全长,并测序验证。反转录基因克隆所采用的引物为SDC-RT-F1: 5′- CTCCTCGGATTCTCAAACCTC AC-3′;SDC-RT-R: 5′- CACACAAACGATAGTAGAACCAACA-3′。
(2)实时荧光定量分析: 荧光定量PCR采用ABI 7500实时PCR系统(Applied Biosystems),以SYBR Green染料进行标记。内参基因选择茶树GAPDH基因(GE651107)。采用的引物为SDC-QRT-F: 5′-GGGAAGAACGTGCACACAAC-3′; SDC-QRT-R: 5′-CTAACCAAGACACCGGCCAT-3′; GAPDH-F:5′-TTGGCATCGTTGAGGG TCT-3′及 GAPDH-R:5′-CAGTGGGAACACGGAAAGC-3′。反应体系为25ml,包含0.5mL LATaq, 5mL PCR缓冲液, 2 mL dNTP(2.5 mM), 0.5mL引物(10 M), 1mL cDNA(40 ng)和15.5mL ddH2O。 反应条件为: 94℃,3分钟; 95℃ ,30秒;59℃,30秒;72℃,1分钟;30个循环。72℃,10分钟;4℃保存。每组样品3个重复。如图3所示,三个茶树品种中根、成熟叶片、一芽二叶等不同组织部位CsAlaDC基因qPCR相对定量结果,说明茶树CsAlaDC基因在茶树根中特异表达。茶树CsAlaDC的基因序列如图2所示。茶树CsAlaDC的基因序列如SEQ ID No.1 所示,其编码的蛋白质氨基酸序列如SEQ ID No.2 所示。
(3)转基因蛋白重组表达纯化:将茶树CsAlaDC全长cDNA的编码框克隆到含有T7启动子的原核表达载体pET28b上。具体步骤包括:设计特异引物:正向引物5’-CGGGATCC(BamH I)ATGGAAGGGACTGTGTCAGTGCTATC and reverse primer 5’-CCCAAGCTT(Hind III)GTTTATGAAGATCACAATCACAATT CTCAC;表达载体构建:PCR 扩增的CsAlaDC基因经酚、酚/氯仿(1:1)、氯仿/异戊醇(24:1)抽提纯化后,用BamH I 和Hind III 双酶切,低熔点胶电泳分离回收1 500 bp左右的片断。再将回收片段与经相同双酶切的表达载体pET-28b 连接,从而将CsAlaDC基因定向克隆到表达载体pET-28b 中,并将含有插入片断的重组质粒命名为pET-AlaDC,将重组质粒pET-AlaDC 转化到E.coli BL21 中,构建工程菌;IPTG诱导大肠表达及蛋白纯化:挑取经DNA 测序验证的工程菌的单菌落,接入5 ml 含氨苄青霉素(50μg/ml)的LB 培养基,37℃振荡培养过夜,取50μl 接入5 ml 含氨苄青霉素(50μg/ml)的LB 培养基,37℃振荡培养2 h 左右,加入IPTG 至终浓度0.05 mmol/L,于16-22℃诱导表达12-18 h,室温5 000 r/min 离心5 min收集菌体。菌体破碎后,用镍柱亲和层析纯化重组蛋白,洗脱液为160mM 咪唑,透析液为25mM Tris-HCl(pH 7.4),150mM NaCl,10%甘油。如图4所示,CsAlaDC蛋白原核纯化的聚丙烯酰胺凝胶电泳(SDS-PAGE)图。
(4)丙氨酸脱羧活性验证
将10μL纯酶液加到90μL反应缓冲液A中(10mM 丙氨酸, 100mM pH 7.2-pH 7.8的磷酸钾缓冲液,0.08-0.12 mM 磷酸吡哆醛,5mM二硫苏糖醇,1mM K2EDTA,10%的甘油),混匀后,于35℃-45℃水浴2h和24h,分别取45μL反应液于新的离心管中,加5μL 10%的三氯乙酸停止反应,静置5min后,加入400μL超纯水稀释,按照Waters AccQ Tag氨基酸分析包中的方法进行衍生,高效液相色谱仪检测产物含量。其中丙氨酸脱酸酶催化反应如图1所示。HPLC检测发现,催化反应2h时检测到乙胺,催化反应24h时,乙胺大量累积(增加了6倍),说明该重组蛋白具有催化丙氨酸脱羧形成乙胺的能力(图5;图6)。
SEQUENCE LISTING
<110> 中国农业科学院茶叶研究所
<120> 一种催化丙氨酸脱羧的蛋白质及其基因和应用
<130> 11
<160> 2
<170> PatentIn version 3.3
<210> 1
<211> 1437
<212> DNA
<213> 茶树
<400> 1
atggaaggga ctgtgtcagt gctatcgaat gtgagcaagg tggagctgtt gtcgaagtgc 60
tttgatctca ttaccatccc tgtggaaccc ttgcctccgg ttgtggcttc caacggagtt 120
gctggcggag agacgaagaa gatgaaggag aaggatattg ttctggggaa gaacgtgcac 180
acaacaagcc tcaccatcac ggagcctgat gtggacgatg actccaccag cgatatggag 240
gccttcatgg ccggtgtctt ggttaggtat cgcaaaactc tcattgagaa gaccaagtat 300
catttaggct atccatttaa tctggacttg gattatggtc ctctagcgga attgcagcat 360
ttcgccataa acaaccttgg cgatccattt attgaaagca actatggtgt tcattcaaga 420
caatttgaag tgggtgtttt ggattggttt gcccgtctat gggaaataga gcagaaagaa 480
tactggggat acattacaaa tggtggcaca gaaggcaatc ttcatggaat cctggttgga 540
agagaagtgt ttccagatgg aattttttat acgtcgcaag aatcacatta ctctatcttc 600
aaagcagcac ggatgtacag aatggaatgc gttaaggtcg gcactttaat caatggggag 660
attgattgtg cagatttcaa agcaaagcta ctttctaaca aggacaaacc agccataatt 720
aatttgaaca taggtactac tgtcaaagga gcggttgatg atattgatct tgttatacaa 780
acccttgaag aatgtggatt ctcgcatgat cgattctaca tccactgtga tggggctctg 840
tttggattca tgatgccatt tctcaaccgt ggaccgaaaa taaccttcaa gaagcccatt 900
ggaagtgtga gtgtttctgg ccacaagttt atgggatgtc caacgccgtg cggtgtccag 960
ataacaaggc ttgagcacat taatgcctta tcaaggaatg tcgaatacct tgcttcaagg 1020
gatgccacaa tcacaggaag ccggaacggc cactctccaa tcattctgtg gtacgcgctg 1080
aacagaaaag gtttcaaggg gttccagaaa gaagtccaaa agtgcctcag aaatgctcac 1140
tatttgaaag accgccttag ggaagcaggt attagtgcca tgctaaatga gcttagtagc 1200
acggttgtgt ttgagcgacc tctagatgag gagtttgttc ggcgttggca acttgcatgc 1260
gagggaaata tggcacatgt tattgtgatg cctaatgtca ccattgagaa gctggatgaa 1320
ttcttgaatg aattagttca aaagcgcgcg aattggtaca acgatgggaa agctggacct 1380
ccttgtcttg caccagatat aggaagtgag aattgtgatt gtgatcttca taaatga 1437
<210> 2
<211> 478
<212> PRT
<213> 茶树
<400> 2
Met Glu Gly Thr Val Ser Val Leu Ser Asn Val Ser Lys Val Glu Leu Leu Ser Lys Cys
1 5 10 15 20
Phe Asp Leu Ile Thr Ile Pro Val Glu Pro Leu Pro Pro Val Val Ala Ser Asn Gly Val
25 30 35 40
Ala Gly Gly Glu Thr Lys Lys Met Lys Glu Lys Asp Ile Val Leu Gly Lys Asn Val His
45 50 55 60
Thr Thr Ser Leu Thr Ile Thr Glu Pro Asp Val Asp Asp Asp Ser Thr Ser Asp Met Glu
65 70 75 80
Ala Phe Met Ala Gly Val Leu Val Arg Tyr Arg Lys Thr Leu Ile Glu Lys Thr Lys Tyr
85 90 95 100
His Leu Gly Tyr Pro Phe Asn Leu Asp Leu Asp Tyr Gly Pro Leu Ala Glu Leu Gln His
105 110 115 120
Phe Ala Ile Asn Asn Leu Gly Asp Pro Phe Ile Glu Ser Asn Tyr Gly Val His Ser Arg
125 130 135 140
Gln Phe Glu Val Gly Val Leu Asp Trp Phe Ala Arg Leu Trp Glu Ile Glu Gln Lys Glu
145 150 155 160
Tyr Trp Gly Tyr Ile Thr Asn Gly Gly Thr Glu Gly Asn Leu His Gly Ile Leu Val Gly
165 170 175 180
Arg Glu Val Phe Pro Asp Gly Ile Phe Tyr Thr Ser Gln Glu Ser His Tyr Ser Ile Phe
185 190 195 200
Lys Ala Ala Arg Met Tyr Arg Met Glu Cys Val Lys Val Gly Thr Leu Ile Asn Gly Glu
205 210 215 220
Ile Asp Cys Ala Asp Phe Lys Ala Lys Leu Leu Ser Asn Lys Asp Lys Pro Ala Ile Ile
225 230 235 240
Asn Leu Asn Ile Gly Thr Thr Val Lys Gly Ala Val Asp Asp Ile Asp Leu Val Ile Gln
245 250 255 260
Thr Leu Glu Glu Cys Gly Phe Ser His Asp Arg Phe Tyr Ile His Cys Asp Gly Ala Leu
265 270 275 280
Phe Gly Phe Met Met Pro Phe Leu Asn Arg Gly Pro Lys Ile Thr Phe Lys Lys Pro Ile
285 290 295 300
Gly Ser Val Ser Val Ser Gly His Lys Phe Met Gly Cys Pro Thr Pro Cys Gly Val Gln
305 310 315 320
Ile Thr Arg Leu Glu His Ile Asn Ala Leu Ser Arg Asn Val Glu Tyr Leu Ala Ser Arg
325 330 335 340
Asp Ala Thr Ile Thr Gly Ser Arg Asn Gly His Ser Pro Ile Ile Leu Trp Tyr Ala Leu
345 350 355 360
Asn Arg Lys Gly Phe Lys Gly Phe Gln Lys Glu Val Gln Lys Cys Leu Arg Asn Ala His
365 370 375 380
Tyr Leu Lys Asp Arg Leu Arg Glu Ala Gly Ile Ser Ala Met Leu Asn Glu Leu Ser Ser
385 390 395 400
Thr Val Val Phe Glu Arg Pro Leu Asp Glu Glu Phe Val Arg Arg Trp Gln Leu Ala Cys
405 410 415 420
Glu Gly Asn Met Ala His Val Ile Val Met Pro Asn Val Thr Ile Glu Lys Leu Asp Glu
425 430 435 440
Phe Leu Asn Glu Leu Val Gln Lys Arg Ala Asn Trp Tyr Asn Asp Gly Lys Ala Gly Pro
445 450 455 460
Pro Cys Leu Ala Pro Asp Ile Gly Ser Glu Asn Cys Asp Cys Asp Leu His Lys
465 470 475
- 还没有人留言评论。精彩留言会获得点赞!