一个具ABC1‑likekinase结构域的基因TaABC1‑2及其应用的制作方法
本发明属于基因工程领域,公开了一个具ABC1-like kinase结构域的基因TaABC1-2及其应用。
背景技术:
小麦是我国乃至世界最重要的粮食作物之一,其生长发育却受多种非生物和生物胁迫。由小麦白粉菌(Blumeria graminis f.sp tritici)引起的小麦白粉病是小麦主要的病害之一。它属于专化寄生,可以在整个生育期侵染小麦的叶部、茎秆、穗部等,其中主要危害小麦叶片,严重时影响小麦正常的光合作用,从而影响其正常的生长,降低产量。一般情况下,小麦白粉病能够引起小麦减产5-19%,严重情况下能够使小麦减产30%以上。从九十年代以后,全国每年小麦白粉病的发病面积在600万公顷以上,严重危害小麦生产安全,因此小麦白粉病的防治越来越受到人们的关注。
选育抗白粉病小麦品种是最有效的方法。小麦和白粉菌在长期的进化过程中形成了复杂的互作系统,一方面小麦进化出了一系列抗病基因和复杂的抗病机制去抵抗白粉菌的侵染达到抗病目的,所以在小麦中导入抗病基因可以提高小麦对白粉病的抗性;另一方面白粉菌也进化出了一系列致病效应因子和复杂的致病机制去侵染植物达到致病目的,病原菌致病性的发挥不仅需要病原菌自身致病效应因子的参与,还需要激活或利用小麦的感病基因去达到成功侵染的目的,所以在小麦中降低感病基因的表达可以干扰白粉菌的侵染,同样可以达到提高小麦对白粉病抗性的目的,但是目前可供基因工程利用的小麦感白粉病基因缺乏。
南农9918是一个广谱抗白粉病的小麦品种,与其遗传背景类似的亲本扬麦158是一个感白粉病小麦品种。利用转录谱技术获得抗病南农9918和感病扬麦158受白粉菌诱导前后的基因转录谱,通过比较两个材料之间转录谱的差异,获得了一个在感病扬麦158中受白粉菌诱导表达的基因TaABC1-2,该基因可能在白粉菌侵染过程中起到协助病原菌入侵和发育的作用,从而导致扬麦158感病。所以在感病材料中降低TaABC1-2的表达,可以干扰病原菌的生产,起到提高感病材料抗白粉病的能力。
技术实现要素:
本发明的目的是针对现有技术的上述缺陷,提供一种具ABC1-like kinase结构域的基因TaABC1-2的重组干扰载体和应用。
本发明的目的可通过如下技术方案实现:
具ABC1-like kinase结构域的基因TaABC1-2来自普通小麦(Triticum asetivum L.)扬麦158,其核苷酸序列为SEQ ID NO.1。
该具ABC1-like kinase结构域的基因的蛋白质为TaABC1-2,其氨基酸序列为SEQ ID NO.2。
含有所述的具ABC1-like kinase结构域的基因TaABC1-2的重组干扰载体。
所述的重组干扰载体pWMB006:TaABC RNAi优选以pWMB006为出发载体,将TaABC1-2基因的正向序列插入到pWMB006的BamHI和KpnI酶切位点间、同时反向序列插入到pWMB006的SpeI和SacI之间所得。
所述的基因TaABC1-2在构建抗白粉病小麦品种中的应用;优选抑制易感白粉病小麦品种中所述的基因TaABC1-2的表达,能够提高易感白粉病小麦品种的对白粉病的抗性。
所述的含具ABC1-like kinase结构域的基因TaABC1-2的表达载体在构建抗白粉病小麦品种中的应用;优选将所述的重组干扰表达载体导入易感白粉病小麦品种中,可以提高易感白粉病小麦品种对白粉病的抗性。
有益效果
本发明从小麦中克隆得到了一个具ABC1-like kinase结构域的基因TaABC1-2,该基因在含有感白粉病的小麦扬麦158中受白粉菌诱导表达,而抗白粉病的小麦南农9918中不受白粉菌诱导表达。将其插入表达载体pWMB006,得到的该基因的重组干扰表达载体pWMB006:TaABC RNAi导入感病小麦品种中,可以提高感白粉病小麦品种对白粉病的抗性。TaABC1-2用于基因工程育种,将其导入易感白粉病小麦品种中,能够提高小麦的白粉病抗性。
附图说明
图1利用Q-PCR分析TaABC1-2在抗白粉病南农9918和感白粉病扬麦158中的表达
0小时:TaABC1-2在未诱导样品中的相对表达量;24小时:TaABC1-2在白粉菌诱导样品中的相对表达量
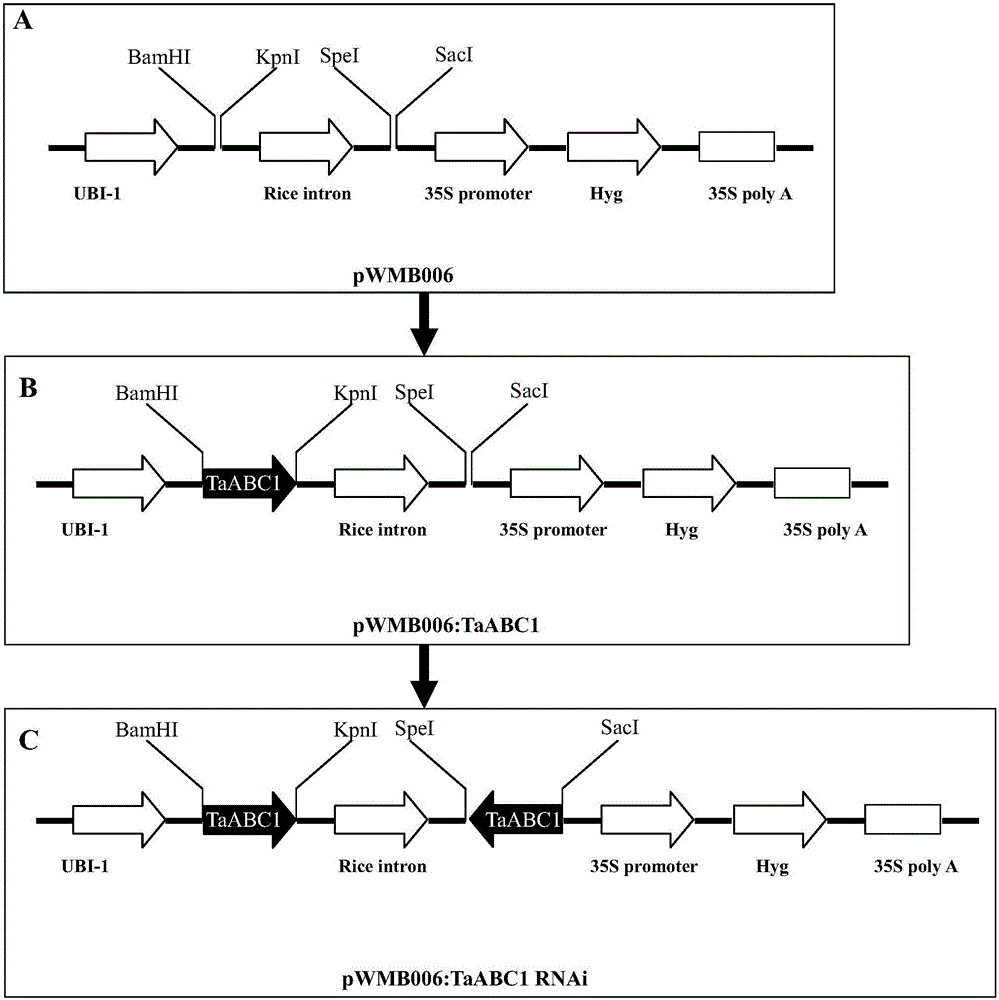
图2 TaABC1-2 RNAi载体pWMB006:TaABC1-2 RNAi的构建
A:pWMB006;B:pWMB006:TaABC1-2中间载体;C:pWMB006:TaABC1-2 RNAi干扰载体;
图3 pBi220:TaABC1-2转基因植株T0代PCR分子鉴定
Marker:DL2000 DNA标准分子量;质粒:pWMB006:TaABC1-2 RNAi阳性对照;扬麦158:阴性对照;T0-8,T0-12,T0-15,T0-16:阳性转基因植株。
图4 TaABC1-2转化扬麦158的T1代阳性植株的白粉病抗性鉴定
A:T1-8株系抗性鉴定;B:T1-12株系抗性鉴定;C:T1-15株系抗性鉴定;D:T1-16株系抗性鉴定
CK:感病扬麦158;R:阳性转基因株系中分离出的抗病单株;S:阳性转基因株系中分离出的感病单株。
具体实施方式
实施例1扬麦158中受白粉菌诱导表达的具ABC1-like kinase结构域基因TaABC1-2的克隆
南农9918是南京农业大学细胞遗传所运用现代生物技术与常规育种技术相结合,以扬麦158/92R137//扬麦158杂交选育而成的抗病、高产、优质小麦新品种(陈佩度,张守忠,王秀娥,王苏玲,周波,冯祎高,刘大钧.抗白粉病高产小麦新品种南农9918.南京农业大学学报,2002,25(4):1438-1444)。扬麦158是江苏里下河地区农科所培育的大面积推广的小麦品种,对白粉病表现感病。
为了筛选扬麦158在感白粉病过程中哪些基因受白粉菌诱导表达,本实验室在前期研究中,采用高通量测序的方法获得抗白粉病南农9918与感白粉病小麦扬麦158中在白粉菌诱导前后的转录谱,并通过比较获得感白粉病扬麦158中受白粉菌诱导上调表达的基因。具体流程如下:将抗白粉病小麦南农9918的种子和感白粉病小麦扬麦158的种子播于培养皿中发芽,露白后移栽到盆钵,一叶期把从感白粉病小麦材料上搜集的白粉菌孢子接种到苗上进行诱导,并于接种前和接种24小时取样,分别提取RNA(用Invitrogen公司的Trizol试剂提取),形成四个样品R0(南农9918诱导0小时的样品)、R24(南农9918诱导24小时的样品)和Y0(扬麦158诱导0小时的样品)、Y24(扬麦158诱导24小时的样品),并送交华大公司进行转录谱测序。将R24与R0的转录谱进行比较,筛选在南农9918中受白粉菌诱导上调表达的基因形成数据库R-data,同时将Y24与Y0的转录谱进行比较,筛选在扬麦158中受白粉菌诱导上调表达的基因形成数据库Y-data。进一步将Y-data与R-data进行比较,筛选只出现在Y-data中的基因,这类基因即为只在感病扬麦158中受白粉菌诱导上调表达,而在南农9918中受白粉菌诱导下调表达的基因。在扬麦158受白粉菌诱导表达的基因中,有一个基因Ta#S58896846具有ABC1-like kinase结构域,初步判断该基因与扬麦158的感病性具有相关性。
以扬麦158经白粉菌诱导24小时的叶片提取出的RNA反转录成cDNA为模板、以根据芯片探针Ta#S58896846设计的引物P1(ATGTCCGTCGCGGCCGCGGC,SEQ ID NO.3)和P2(GGCTATGCTACACCACCTCT,SEQ ID NO.4)为引物进行RT-PCR,获得了TaABC1-2的全长基因;其ORF序列如SEQ ID NO.1所示,编码的蛋白序列如SEQ ID NO.2所示。。
实施例2 TaABC1-2在抗病南农9918和感病扬麦158中的表达分析
为了研究TaABC1-2在抗感白粉病材料中的表达模式,利用抗病材料南农9918和感病材料扬麦158经白粉菌诱导0、24小时的RNA反转录cDNA为模板,利用P3(TCCTCTCCGATAGCTGCAAG,SEQ ID NO.5)和P4(GTTAGGCCGGTATGCTGTTG,SEQ ID NO.6)为引物进行实时荧光定量PCR(Q-PCR)分析。PCR程序为:PCR反应在实时荧光定量PCR仪(MyIQ,Bio-Rad公司,美国)上扩增并检测荧光。20uL PCR反应体系中含2×SYBR Green PCR Master Mix 10uL,0.5μM引物P3和P4,反转录cDNA模板2uL。扩增参数为:95℃10分钟,然后95℃15秒、60℃30秒,72℃1分钟,共40个循环。反应结束后,进行熔解曲线的测定。检测基因表达水平用MyiQ系统软件进行分析。结果表明,在92R137中,TaABC1-2受条锈菌诱导上调表达,12小时上调显著,24h后表达水平最高,48和72小时表达已下降;在扬麦158中,TaABC1-2没有受条锈菌诱导表达的特征,各个时间段的表达量均低于在92R137中的表达量(图1)。
实施例3 TaABC1-2干扰载体pWMB006:TaABC RNAi的构建
干扰TaABC1-2基因表达pWMB006:TaABC RNAi出发载体是pWMB006(Tingting Chen,Jin Xiao,Jun Xu,Wentao Wan,Bi Qin,Aizhong Cao,Wei Chen,Liping Xing,Chen Du,Xiquan Gao,Shouzhong Zhang,Ruiqi Zhang,Wenbiao Shen,Haiyan Wang and Xiue Wang.Two members of TaRLK family confer powdery mildew resistance in common wheat.BMC Plant Biology 201616:27 DOI:10.1186/s12870-016-0713-8)。构建流程如下:1、以已经克隆的TaABC1-2的基因序列为模板,设计引物P5(TTGGATCCAGATCGACTGCACCC,SEQ ID NO.7)和P6(TTGGTACCCACGAGTCACGACCT,SEQ ID NO.8),且P5带有BamHI的酶切位点,P6带有KpnI的酶切位点。2、以含有TaABC1-2基因的质粒为模板,使用引物对P5和P6进行PCR扩增,回收扩增片段。3、用BamHI和KpnI对扩增产物进行双酶切,将酶切产物插入到BamHI和KpnI双酶切后的载体pWMB006中,将TaABC1-2正向置于35S启动子后面的多克隆位点处。由此将目标基因TaABC1-2克隆到强启动子35S的下游,获得表达载体pWMB006:TaABC1-2。4、以已经克隆的TaABC1-2的基因序列为模板,设计引物P7(TTGAGCTCAGATCGACTGCACCC,SEQ ID NO.9)和P8(TTACTAGTCACGAGTCACGACCT,SEQ ID NO.10),且P7带有SacI的酶切位点,P8带有SpeI的酶切位点。5、以含有TaABC1-2基因的质粒pWMB006:TaABC1-2为模板,使用引物对P7和P8进行PCR扩增,回收扩增片段。6、用SacI和SpeI对扩增产物进行双酶切,将酶切产物插入到SacI和SpeI双酶切后的载体pWMB006:TaABC1-2中,将TaABC1-2反向置于35S启动子后面的多克隆位点处。由此将目标基因TaABC1-2的发卡结构克隆到强启动子35S的下游,获得如图2所示的干扰表达载体pWMB006:TaABC RNAi。
实施例4 pWMB006:TaABC RNAi转化普通小麦扬麦158及阳性植株的分子鉴定
利用基因枪转化方法将pWMB006:TaABC RNAi转入感白粉病受体扬麦158的转化方法如下:1、挑取预培养7天约2000块扬麦158幼胚愈伤组织,在高渗培养基(MS+ABA0.5mg/L+水解酪蛋白500mg/L+2,4-D2mg/L+葡萄糖30g/L+0.4mol/L甘露醇,pH5.8)上预处理4–5小时;2、将携有目的基因TaABC1-2的干扰表达载体pWMB006:TaABC RNAi通过基因枪轰击法转化到扬麦158愈伤组织,轰击后在高渗培养基上继续培养16小时。3、将愈伤组织转移至恢复培养基(1/2MS+水解酪蛋白500mg/L+2,4-D2mg/L+蔗糖30g/L,pH5.8)上暗培养2周;4、将愈伤组织转移至含有除草剂的筛选培养基上(1/2MS+ABA0.5mg/L+水解酪蛋白500mg/L+2,4-D1mg/L+蔗糖30g/L+4mg/L Bialaphos,pH5.8)筛选培养2周;5、将具有除草剂抗性的愈伤组织转移到分化培养基中(1/2MS+L-谷氨酞胺l mmol/L+水解酪蛋白200mg/L+KT 1mg/L+IAA 0.5mg/L+蔗糖30g/L+琼脂0.8%,pH5.8)进行分化,待分化芽长至2–4cm时将其转移至生根培养基(1/2MS+KT 1mg/L+蔗糖30g/L+琼脂0.8%,pH5.8)中。6、至再生苗长约8cm、根系较健壮时,即可开管炼苗1–2天,最后洗去根系携带的培养基残渣便可移栽入盆钵。获得再生植株共135棵。
提取所有再生植株基因组DNA,对转化植株利用启动子内部引物P9(GCCCCATATCTCCAGGTACC,SEQ ID NO.11)和基因内部引物P10(ACTGCCGTGGAACTGTCATA,SEQ ID NO.12)进行PCR扩增,以鉴定阳性植株。PCR程序:10-50ng基因组DNA模板,10μM的P9和P10各0.5μl;2.5μl 10×buffer;2.5μl 2.5mM的dNTP;1.5μl 25mM的Mg2+;0.25μl(5U/μl)Taq polymerase(TaKaRa),加水至25μl。PCR反应条件为:94℃预变性3分钟;94℃45秒,55℃45秒,72℃2分钟,33个循环;72℃延伸10分钟。PCR产物经1%的琼脂糖凝胶电泳检测。其中4株可以扩增出目的条带,鉴定为阳性植株。图3所示为部分阳性植株的筛选,包括T0-8、T0-12、T0-15、T0-16。
实施例5 pWMB006:TaABC RNAi转扬麦158阳性植株的白粉病抗性鉴定
TaABC1-2转基因T0代阳性植株分单株收获后,将阳性单株的T1代种子种于盆钵,以感病受体扬麦158为对照,进行离体叶片的白粉病抗性鉴定。结果表明:T1-8,T1-12,T1-15,和T1-16四个株系表现抗病,且每个株系间均出现抗感分离现象。感病对照植株扬麦158对白粉菌表现高感,无过敏性坏死斑,叶片上布满白粉菌孢子堆;转基因T1代阳性植株与未转化扬麦158相比,白粉病抗性水平得到显著提高,叶片上只有少量孢子,而且叶片上产生了抗病的过敏性坏死斑,这是叶片对病原菌产生抗性的标志(图4)。以上鉴定结果表明:TaABC1-2在感病材料中的降低表达量后,可提高感病材料的白粉病抗性,该基因可用于利用基因工程手段培育抗白粉病小麦。
<110> 南京农业大学
<120> 一个具ABC1-like kinase结构域的基因TaABC1-2及其应用
<160> 12
<210> 1
<211> 2265
<212> DNA
<213> 普通小麦(Triticum asetivum L.)扬麦158
<220>
<223> 具ABC1-like kinase结构域的基因TaABC1-2
<400> 1
atgtccgtcg cggccgcggc cgcctccctc gtcgcctcct cttcgctctc cgttcccgac 60
cacctccgcc tccgccgccc cccgccaccg ctccgcttcc gccgccgatc aaaggaccgc 120
ctcgttcttg ctgttctcga ggagaaatcc tcctctgcgc ttactgagga ggaagcaagg 180
aagttcgggc tcaacgggag cgcgagcagg ctcgggtacg acgatgccgc ggtggaggcg 240
tacctcgggt ccaacggcaa cggaaacagc aacagcaaca ggagtgcgag taggagtggg 300
aacggcgcga gcgtgaagcc cgtgccatcc gggtctggca cttctgtggt atccggccgt 360
gggccgggtg aggatgagag gaggaggaag gagagagtgg aggagatagg cagggaggac 420
gcatggttta agcgaagcga cggagaggcc atgcccaagg tgtctgttgt tcctggtggt 480
cgttggaatc ggtttaaaac ctattcaacg attcaaagaa ccttggagat atggggctct 540
gtcttcgcat ttatattcaa ggtttggctc aacaaccaga agttcactta tcgagggggg 600
atgacggaag agaaaagggt aatgaggagg aaagttcttg ccaagtggct caaggagagt 660
attttgagat taggccccac atttattaaa ataggacagc aattctccac cagggtggat 720
attcttccaa aagaatatgt ggatcaatta tctgaattgc aggatcaggt ccctccattc 780
ccttcagaga cagctgtgtc aactattgaa gaagagctgg gagcatctgt aaatgagata 840
tttgaacgat ttgacgttga accattagct gctgctagcc tcggccaggt tcatcgggca 900
tgcctaaatg gccaagaagt tgtactcaag gtgcaaaggc ctggtctgaa agagctgttt 960
gatattgatc tgaagaattt aagggtaata gcagaatacc ttcagaaagt ggatcccaag 1020
tcagatggtg ccaagagaga ctgggttgct atttatgatg aatgtgcaaa tgtattatat 1080
caggaaatag actataccaa agaagcattt aatgctgaaa agtttgctga aaacttcaaa 1140
aatatggatt atgtgaaggt tccagaaatt tactgggagt atactacacc tcaggttcta 1200
acaatggagt atgtcccagg aatcaaaata aataggatac agcagataga taagttaggg 1260
cttgatcgga aaaggttagg ccggtatgct gttgagtcct accttgagca gattttatcg 1320
catggatttt tccacgccga cccgcatcca gggaacattg ctgttgatga tgccaatggt 1380
gggaggctta tcttctacga ctttgggatg atgggaagta ttagcccaaa tatccgcgaa 1440
ggactgcttg aagtatttta tggagtttat gaaaaagatc ctgataaagt gcttcaagca 1500
atggttcaaa tgggtgtcct tgttcctact ggagatatga cagccgtcag aagaacagct 1560
caatttttcc tggatagctt tgaagagcgt ctagctgcac aaaggaaaga gagagagatg 1620
gcaaccgtgg aacctggatt taaaaaacaa ttatctaagg aggaaaagtt tgaaaagaag 1680
aagcagagac ttgcagctat cggagaggat cttttggcaa ttgctgctga tcaaccattt 1740
cggtttcctg ccacctttac atttgtggtc agagcattct cagtactgga tggtattggg 1800
aaaggtttag atcctagatt tgatattaca gagattgcta agccttatgc catggagttg 1860
ctcagattta atgaagctgg tgttgaagta cttgttaagg atgcaaggaa gcgatgggac 1920
aggcagtctc gtgcattcta caatgtgttc cgtcaacctg acagagttga aaaacttgca 1980
cagatcattg agcgtttgga gcaaggtgat ctcaagcttc gtgtcagagc attagaatcg 2040
gaaagatcgt ttcaaagagt tgccgctgta cagaaaacaa ttggatatgg agtggctgcg 2100
ggcagtttgg taaacctcgc taccattctg cacctcaatt cgatccggat gccagcaacc 2160
atcggatact gtctttgtgc gttctttggg ctacaaatcc tgcttggcgt tctcaaggtg 2220
aagaagctgg accaacaaga gagattgata accggcactg cctga 2265
<210> 2
<211> 754
<212> PRT
<213> 普通小麦(Triticum asetivum L.)扬麦158
<220>
<223> 蛋白TaABC1-2
<400> 2
Met Ser Val Ala Ala Ala Ala Ala Ser Leu Val Ala Ser Ser Ser Leu
1 5 10 15
Ser Val Pro Asp His Leu Arg Leu Arg Arg Pro Pro Pro Pro Leu Arg
20 25 30
Phe Arg Arg Arg Ser Lys Asp Arg Leu Val Leu Ala Val Leu Glu Glu
35 40 45
Lys Ser Ser Ser Ala Leu Thr Glu Glu Glu Ala Arg Lys Phe Gly Leu
50 55 60
Asn Gly Ser Ala Ser Arg Leu Gly Tyr Asp Asp Ala Ala Val Glu Ala
65 70 75 80
Tyr Leu Gly Ser Asn Gly Asn Gly Asn Ser Asn Ser Asn Arg Ser Ala
85 90 95
Ser Arg Ser Gly Asn Gly Ala Ser Val Lys Pro Val Pro Ser Gly Ser
100 105 110
Gly Thr Ser Val Val Ser Gly Arg Gly Pro Gly Glu Asp Glu Arg Arg
115 120 125
Arg Lys Glu Arg Val Glu Glu Ile Gly Arg Glu Asp Ala Trp Phe Lys
130 135 140
Arg Ser Asp Gly Glu Ala Met Pro Lys Val Ser Val Val Pro Gly Gly
145 150 155 160
Arg Trp Asn Arg Phe Lys Thr Tyr Ser Thr Ile Gln Arg Thr Leu Glu
165 170 175
Ile Trp Gly Ser Val Phe Ala Phe Ile Phe Lys Val Trp Leu Asn Asn
180 185 190
Gln Lys Phe Thr Tyr Arg Gly Gly Met Thr Glu Glu Lys Arg Val Met
195 200 205
Arg Arg Lys Val Leu Ala Lys Trp Leu Lys Glu Ser Ile Leu Arg Leu
210 215 220
Gly Pro Thr Phe Ile Lys Ile Gly Gln Gln Phe Ser Thr Arg Val Asp
225 230 235 240
Ile Leu Pro Lys Glu Tyr Val Asp Gln Leu Ser Glu Leu Gln Asp Gln
245 250 255
Val Pro Pro Phe Pro Ser Glu Thr Ala Val Ser Thr Ile Glu Glu Glu
260 265 270
Leu Gly Ala Ser Val Asn Glu Ile Phe Glu Arg Phe Asp Val Glu Pro
275 280 285
Leu Ala Ala Ala Ser Leu Gly Gln Val His Arg Ala Cys Leu Asn Gly
290 295 300
Gln Glu Val Val Leu Lys Val Gln Arg Pro Gly Leu Lys Glu Leu Phe
305 310 315 320
Asp Ile Asp Leu Lys Asn Leu Arg Val Ile Ala Glu Tyr Leu Gln Lys
325 330 335
Val Asp Pro Lys Ser Asp Gly Ala Lys Arg Asp Trp Val Ala Ile Tyr
340 345 350
Asp Glu Cys Ala Asn Val Leu Tyr Gln Glu Ile Asp Tyr Thr Lys Glu
355 360 365
Ala Phe Asn Ala Glu Lys Phe Ala Glu Asn Phe Lys Asn Met Asp Tyr
370 375 380
Val Lys Val Pro Glu Ile Tyr Trp Glu Tyr Thr Thr Pro Gln Val Leu
385 390 395 400
Thr Met Glu Tyr Val Pro Gly Ile Lys Ile Asn Arg Ile Gln Gln Ile
405 410 415
Asp Lys Leu Gly Leu Asp Arg Lys Arg Leu Gly Arg Tyr Ala Val Glu
420 425 430
Ser Tyr Leu Glu Gln Ile Leu Ser His Gly Phe Phe His Ala Asp Pro
435 440 445
His Pro Gly Asn Ile Ala Val Asp Asp Ala Asn Gly Gly Arg Leu Ile
450 455 460
Phe Tyr Asp Phe Gly Met Met Gly Ser Ile Ser Pro Asn Ile Arg Glu
465 470 475 480
Gly Leu Leu Glu Val Phe Tyr Gly Val Tyr Glu Lys Asp Pro Asp Lys
485 490 495
Val Leu Gln Ala Met Val Gln Met Gly Val Leu Val Pro Thr Gly Asp
500 505 510
Met Thr Ala Val Arg Arg Thr Ala Gln Phe Phe Leu Asp Ser Phe Glu
515 520 525
Glu Arg Leu Ala Ala Gln Arg Lys Glu Arg Glu Met Ala Thr Val Glu
530 535 540
Pro Gly Phe Lys Lys Gln Leu Ser Lys Glu Glu Lys Phe Glu Lys Lys
545 550 555 560
Lys Gln Arg Leu Ala Ala Ile Gly Glu Asp Leu Leu Ala Ile Ala Ala
565 570 575
Asp Gln Pro Phe Arg Phe Pro Ala Thr Phe Thr Phe Val Val Arg Ala
580 585 590
Phe Ser Val Leu Asp Gly Ile Gly Lys Gly Leu Asp Pro Arg Phe Asp
595 600 605
Ile Thr Glu Ile Ala Lys Pro Tyr Ala Met Glu Leu Leu Arg Phe Asn
610 615 620
Glu Ala Gly Val Glu Val Leu Val Lys Asp Ala Arg Lys Arg Trp Asp
625 630 635 640
Arg Gln Ser Arg Ala Phe Tyr Asn Val Phe Arg Gln Pro Asp Arg Val
645 650 655
Glu Lys Leu Ala Gln Ile Ile Glu Arg Leu Glu Gln Gly Asp Leu Lys
660 665 670
Leu Arg Val Arg Ala Leu Glu Ser Glu Arg Ser Phe Gln Arg Val Ala
675 680 685
Ala Val Gln Lys Thr Ile Gly Tyr Gly Val Ala Ala Gly Ser Leu Val
690 695 700
Asn Leu Ala Thr Ile Leu His Leu Asn Ser Ile Arg Met Pro Ala Thr
705 710 715 720
Ile Gly Tyr Cys Leu Cys Ala Phe Phe Gly Leu Gln Ile Leu Leu Gly
725 730 735
Val Leu Lys Val Lys Lys Leu Asp Gln Gln Glu Arg Leu Ile Thr Gly
740 745 750
Thr Ala
<210> 3
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P1
<400> 3
atgtccgtcg cggccgcggc 20
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P2
<400> 4
ggctatgcta caccacctct 20
<210> 5
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P3
<400> 5
tcctctccga tagctgcaag 20
<210> 6
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P4
<400> 6
gttaggccgg tatgctgttg 20
<210> 7
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物P5
<400> 7
ttggatccag atcgactgca ccc 23
<210> 8
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物P6
<400> 8
ttggtaccca cgagtcacga cct 23
<210> 9
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物P7
<400> 9
ttgagctcag atcgactgca ccc 23
<210> 10
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物P8
<400> 10
ttactagtca cgagtcacga cct 23
<210> 11
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P9
<400> 11
gccccatatc tccaggtacc 20
<210> 12
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物P10
<400> 12
actgccgtgg aactgtcata 20
- 还没有人留言评论。精彩留言会获得点赞!
- IL-15Rα sushi结构域作为通过IL-15Rβ/γ的IL-15作用的选择性和有效的增强剂以及超 ...的制作方法
- 含有hiv转导结构域的生存素突变体及制备方法和应用的制作方法
- 突变的无激活作用的IgG2结构域和插入该结构域的抗CD3抗体的制作方法
- 基于IL-15和IL-15Rα SUSHI结构域的免疫细胞因子的制作方法
- 用于gnn的锌指结合结构域的制作方法
- 截短的丙肝病毒ns5结构域以及含有该截短结构域的融合蛋白的制作方法
- 用于gnn的锌指结合结构域的制作方法
- 癌胚抗原的n-结构域及其组合物、方法和用途的制作方法
- Adamalysin的解离素结构域在抗血管生成、抗侵入和抗转移组合物中的应用的制作方法
- 一种透皮结构域及其衍生的具有透皮作用的融合蛋白和其制备方法