用于基因组编辑的CRISPR/CAS9复合物的制作方法
本申请要求2015年6月17日提交的美国临时申请no.62/181,138和2015年12月11日提交的美国临时申请no.62/266,316的权益,这两件申请据此以引用的方式全文并入本文中。
背景
成簇规律间隔的短回文重复序列(crispr)-相关联的(cas)体系(crispr-cas9体系)用于哺乳动物细胞中所需基因组位点处的基因编辑。在crispr-cas9体系中,cas9核酸酶通过与指导rna复合而靶向至基因组位点,所述指导rna与基因组中的靶位点杂交。这导致经由双链或单链dna修复模板而引发基因组dna的非同源性末端接合(nhej)或同源引导的修复(hdr)的双链断裂。然而,经由hdr修复基因组位点效率不高。
概述
本文提供了一种用于校正细胞或细胞群的基因组中的突变的复合物。所述复合物包含指导rna(grna),该指导rna包含:第一核苷酸序列,其与细胞基因组中的靶dna杂交,其中所述靶dna包含突变;以及第二核苷酸序列,其与定点核酸酶相互作用。所述复合物还包含可操作地连接至超荷电蛋白的重组定点核酸酶,其中所述定点核酸酶包含与指导rna的第二核苷酸序列相互作用的rna结合部分,并且其中所述定点核酸酶特异性结合并且切割靶dna以产生双链断裂。所述复合物还包含单链供体寡核苷酸(ssodn),其与靶dna中双链断裂侧翼的基因组序列杂交并且整合到靶dna中以校正靶dna中的突变。
还提供了在细胞或细胞群中对靶dna进行位点特异性修饰的方法。所述方法包括引入用于校正细胞基因组中的突变的复合物,其中在允许同源引导的修复(hdr)和ssodn向靶dna中整合的条件下将所述复合物引入细胞中。所述方法还提供了经校正的细胞中的高细胞存活率。
还提供了治疗受试者的与编码血红蛋白的基因组序列中的突变相关联的疾病的方法。所述方法包括在允许同源引导的修复(hdr)的条件下将用于校正编码血红蛋白的基因组序列中的突变的复合物引入从受试者获得的细胞群中,以校正编码血红蛋白的基因组序列中的突变并将经校正的细胞移植到受试者中。
附图说明
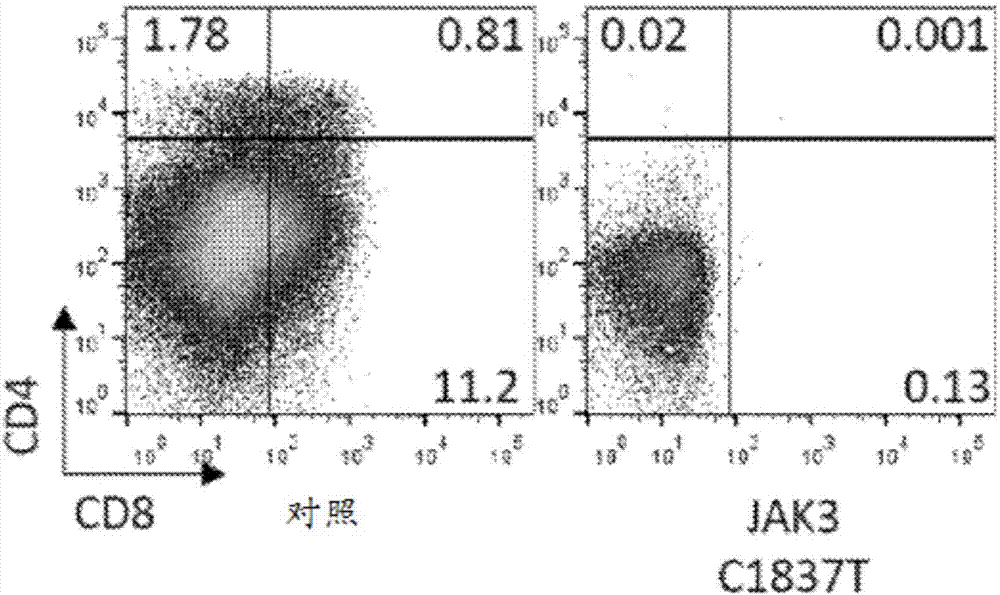
图1a-1c示出jak3c1837t患者的诱导性多能干细胞(ipsc)的体外分化再现了scid表型。图1a和1b示出ipsc来源的t细胞的流式细胞术。jak3wtipsc(对照)和jak3缺陷型ipsc(jak3c1837t)在op9基质细胞上分化成cd34+细胞,随后在op9-dl4单层上分化成t细胞。与对照相比,缺乏来自jak3缺陷型ipsc的t细胞分化;未观察到cd3+t细胞或cd3-cd16+cd56+nk细胞(图1a),并且未检测到cd4+cd8+双阳性(dp)、cd4+单阳性(sp)或cd8+单阳性(sp)t细胞(图1b)。图1c示出针对调节t细胞谱系的规范期间的早期事件的关键基因转录物的rt-qpcr测定结果。示出相对于gapdh表达的rna水平。
图2a-2c示出bcl2部分挽救了jak3缺陷型体外来源细胞中的t细胞发育缺陷。图2a示出与jak3wt对照相比,jak3缺陷型ipsc来源的t细胞的凋亡。在t细胞诱导第10天(td10)和第17天(td17)分析膜联蛋白v阳性细胞。用对照jak3wt细胞(对照)进行四次独立实验,并且用jak3缺陷型细胞(jak3c1837t)进行5次独立实验。*p<0.005。图2b示出来自jak3wt(对照)和jak3缺陷型细胞(jak3c1837t)的两列(1和2)中抗凋亡bcl2和促凋亡bax表达的rt-qpcr测定结果。nd,未确定(由于jak3qpcr信号不显著)。示出相对于gapdh表达的rna水平。图2c示出用bcl2-2a-gfp慢病毒转导的jak3缺陷型ipsc来源的t细胞的流式细胞术,以评估对nk(cd16+56+)和t细胞(cd3+)发育以及dp(cd4+cd8+)至sp(cd4+或cd8+)t细胞成熟的影响。
图3a-3d示出crispr/cas9增强了患者特异性ipsc中jak3c1837t突变的校正。图3a描绘了使用crispr/cas9诱导jak3基因座中的双链断裂以及用于同源引导的修复的模板进行基因组修饰的策略。顶线,jak3基因的结构。空心框,外显子。星号,c1837t突变。箭头,指导rna。图3b的顶部示出证明同源重组的pcr分析;指示用于5'和3'分析的引物。(左下)证明jak3wt(对照)、jak3缺陷型(jak3c1837t)和经校正的(jak3校正的)t细胞中的jak3mrna表达的rt-pcr分析。(右下)证明jak3wt(对照)、jak3缺陷型(jak3c1837t)和经校正的(jak3校正的)t细胞中的jak3蛋白表达的蛋白印迹分析。图3c提供了指导rna的靶向效率的汇总。(图3d)来自亲本jak3ipsc(左)、杂合校正的ipsc(中)和纯合校正的ipsc(右)的pcr扩增子的sanger测序。用grna2+野生型cas9来校正两个杂合克隆,并用grna1+grna2+切口酶cas9(d10a)来校正纯合子克隆。
图4a-4c示出jak3校正的患者ipsc的体外分化产生具有成熟t细胞的表型和功能特征的t细胞。图4a示出t细胞发育标志物在jak3wt(对照,n=3)、jak3缺陷型(jak3c1837t,n=5)和jak3-校正的(jak3校正的,n=6)t细胞中的表达。将细胞用指定的抗体染色并在t细胞诱导第14天、21天、28天和35天(td14、21、28和35)通过流式细胞术进行分析。图4b示出jak3校正的t细胞的t细胞受体(tcr)vβ分析。高度多样的tcrvβ库展示于源自校正的scid患者ipsc的t细胞中。图4c示出证明jak3校正的t细胞中的t细胞活化的流式细胞术。在分析活化标志物cd25和cd69之前,将源自jak3wt(对照)和jak3校正的ipsc的t细胞用抗cd3/28珠粒刺激3天。针对cd3+群对数据进行设门。
图5a-5c示出通过与人骨髓基质细胞(hmsc)共培养而从hipsc体外产生cd34+hsc。在分析造血标志物cd34和cd43之前,将人ipsc在hmsc上培养18天(图a)。cd34+细胞在珠粒上纯化并分化成t细胞(图b)、红系细胞和骨髓细胞(图c)。为了产生t细胞,将纯化的cd34+细胞铺在op9-dl4细胞上并保持3至4周。对于产生骨髓和红系细胞的cfc测定,根据制造商的方案将纯化的cd34+细胞铺在methoculth4434经典培养基中。这些数据证明,在hmsc上共培养之后,hipsc可以有效地分化成多能hsc。
图6a-6c示出通过用hmsc-dl4培养hipsc来源的cd34+细胞来体外产生t细胞。为了产生cd7+t祖细胞,将hipsc来源的cd34+细胞在hmsc-dl4上共培养3至4周(图6a)。当将图6a中的cd7+细胞在磁珠上纯化并在op9-dl4上共培养时,在10天或更短时间内产生完全成熟的cd4+/cd8+/cd3+/tcr‐αβ+细胞(图b和c)。这些数据证明hipsc可以在于hmsc-dl4上共培养之后有效地分化成cd7+淋巴样祖细胞。
图7示出来自hipsc的γδt细胞的体外产生。将人ipsc用携带与2a-gfpcdna片段连接的预先重排的人vγδ1cdna的慢病毒载体转导。与op9共培养18天后,在磁珠上纯化hipsc来源的cd34+细胞。随后将这些细胞铺在op9-dl4细胞上以进行t细胞分化。在第32天收获细胞,通过facs分析t细胞表面标志物。gfp+群代表vδ1-2a-gfp慢病毒转导的细胞。高百分比的这些gfp阳性细胞表达vδ1(66%)。低百分比的gfp阴性细胞表达vδ1(1%)。这些结果证明表达重组t细胞受体(tcr)的vδt细胞可有效地从基因修饰的ipsc产生。表达对肿瘤抗原具有特异性的重组t细胞受体(tcr)的vδt细胞的产生提供了用于许多类型的癌症的强效细胞疗法。
图8示出包含指导rna、修饰的cas9和单链寡核苷酸供体序列(ssodn)的校正复合物可校正镰状细胞突变。通过核穿孔(nucleoporation)将该复合物引入镰状ipsc中,并在2天后通过数字pcr(ddpcr)分析基因组dna并测序。超过65%的细胞含有至少一个经校正的基因。结果确认如下。在引入校正复合物两天后,将细胞铺在培养皿中,并分离出43个单独的ipsc集落。从这些集落中分离基因组dna,并对β-珠蛋白基因进行测序。百分之六十五的集落含有至少一个经校正的β-珠蛋白基因(s校正为a)。
图9示出将镰状细胞校正复合物(grna修饰的重组cas9-ssodn)引入患者原代骨髓cd34+细胞中可校正镰状细胞突变。在体外分化十二天之后,通过数字pcr(ddpcr)分析dna并测序。观察到大约等量的βa和βsmrna。
图10是来自图9的经校正的镰状患者cd34+细胞的体外分化的红细胞的等电聚焦(ief)凝胶,示出hba(正常血红蛋白)与hbs(具有镰状细胞突变的血红蛋白)的比率为约1:3,其足以抑制镰状形成和治疗镰状细胞性贫血。
图11示出工程改造的带正电荷的cas9rnp/ssodn(epccas9rnp/ssodn)有效地校正了人患者ipsc中的镰状突变。将野生型cas9(cas9wt)rnp和八个工程改造的带正电荷的(epccas9)rnp与校正ssodn共同核核穿孔到人镰状ipsc中。在核转染后两天通过sanger测序来确定汇合的细胞中的镰状校正效率。箭头指示镰状校正的位置(t->a),并且剪刀指示镰状hbbdna上的cas9wt-36gfprnp切割位点。
图12示出人镰状ipsc群中在靶(on-target)修饰的深度测序的结果。示出用cas9wtrnp/ssodn、cas9wt-egfp或四种epccas9rnp/ssodn进行核穿孔的人镰状ipsc的在靶深度测序分析。黑色条指示经校正的基准,黑色条下面的空间表示镰状细胞突变。阴性对照和单独的ssodn两者均显示出仅镰状细胞突变。所有ipsc样品还包含镰状突变附近(右侧的列)的snp。
图13示出tat-cas9wt-egfprnp抑制在靶插入缺失(indel)。用cas9wt和tat-cas9wt-egfprnp(具有(+ssodn)或没有校正ssodn(-ssodn))对人镰状ipsc进行核穿孔。通过在核穿孔后两天进行sanger测序来分析插入缺失和校正效率。箭头指示镰状校正的位置(t->a),并且剪刀表示镰状hbbdna上的cas9wt-36gfprnp切割位点。
图14示出epccas9rnp抑制人镰状ipsc中的在靶插入缺失。用cas9wt和5种epccas9rnp(含有或不含校正ssodn)对人镰状ipsc进行核穿孔。通过在核穿孔后两天进行sanger测序来分析插入缺失和校正效率。箭头指示镰状校正的位置(t->a),并且剪刀表示镰状hbbdna上的cas9wt-36gfprnp切割位点。
图15示出epccas9rnp增强了在人镰状ipsc中进行核穿孔后的细胞存活。用cas9wtrnp和7种epccas9rnp(含有或不含校正ssodn)对人镰状ipsc进行核穿孔。核转染后两天通过光学显微镜评估细胞存活。
图16a和16b示出人ipsc中镰状校正的ssodn:cas9rnp比率。校正ssodn和cas9wt-36gfp/t2rnp以0、0.2、0.5、1.0、1.15、1.35、1.5和2.0的摩尔比核穿孔到镰状患者ipsc中。(对于这些实验,固定1:1.35的cas9wt-36gfp:t2grna摩尔比。例如,下图中r=0.5的值是0.5ssodn:1.0cas9wt-36gfp:1.35t2grna。)在ssodn:cas9wt-36gfprnp核穿孔后48小时,通过微滴式数字pcr(ddpcr)(图16a)和sanger测序(图16b)来对镰状校正进行定量。将校正百分比(βa/βs等位基因×100)相对于r(ssodn:cas9wt-36gfprnp)进行绘图。用数据点对s形虚曲线进行拟合。(b)箭头指示镰状校正的位置(t->a),并且剪刀指示镰状hbbdna上的cas9wt-36gfprnp切割位点。
图17示出人ipsc中镰状校正的cas9:sgrna比率。对1:1.15、1:1.35和1:1.50的cas9-36gfp:sgrna摩尔比以及1.15或1.35的ssodn摩尔比进行测试以确定患者ipsc中镰状突变的最佳校正效率。将混合物核穿孔到人镰状ips细胞中,并在核转染后两天分析汇合细胞的sanger测序结果。箭头指示镰状校正的位置(t->a),并且剪刀指示镰状hbbdna上的cas9wt-36gfprnp切割位点。
图18示出epccas9rnp/ssodn对人镰状ipsc的校正。对用tat-cas9wt-36gfp-inf7rnp/ssodn核转染的汇合的人镰状ips细胞进行sanger测序分析。箭头指示镰状校正的位置(t->a),并且剪刀指示epccas9rnp诱导的dsb在镰状hbbdna上的位置。
图19a和19b示出用epccas9rnp和摆动ssodn对人ipsc的校正。用tat-cas9-36gfp-inf7rnp和在grna切割位点附近含有摆动碱基的ssodn对人镰状ipsc进行核穿孔。(a)对用t1grna和t1-wbssodn核穿孔的ipsc群进行sanger测序。图19a左侧的箭头指示镰状突变的位置,位于镰状突变下游的3个箭头指示摆动碱基的位置。剪刀指向t1切割位点。(b)对用t2grna和t2-wbssodn核穿孔的ipsc群进行sanger测序。左侧的箭头指示镰状突变的位置,并且镰状突变下游的2个箭头指示摆动碱基的位置。剪刀指向t2切割位点。
图20a和20b示出用tat-cas9wt-36gfp-inf7rnp/ssodn校正的4个ipsc克隆的全基因组测序(wgs)分析的结果。(a)在靶序列分析证明镰状校正和摆动碱基取代。(b)示出对与t1和t2sgrna具有同源性的基因组基因座的wgs脱靶分析。
图21a-d示出镰状患者骨髓cd34+hspc的基因校正。(a)通过cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp/ssodn对人镰状骨髓cd34+细胞进行核穿孔。核转染后6天分析汇合的细胞群的基因校正效率。箭头表示镰状校正的位置(t->a),并且剪刀表示镰状hbbdna上的cas9wt-36gfprnp切割位点。(b)通过在于红系细胞分化培养基中培养10天后收获的cas9wt-36gfp核穿孔的镰状cd34+细胞中进行rt-pcr和sanger测序来进行mrna校正。(c)来自cas9wt-36gfprnp/ssodn核转染的镰状cd34+细胞的体外分化rbc的ief凝胶分析。还上样代表hbf、hbs和hba蛋白的人镰状儿童患者血液裂解液(ss)和人正常成人血液裂解液(aa)作为对照。(d)源自用cas9wt-36gfprnp/ssodn核转染的镰状cd34+细胞的体外分化rbc的质谱分析。峰证明来自未经校正的hbs蛋白和经校正的hba蛋白的信号。
图22a-c示出源自单cd34+祖细胞的集落的校正。(a)源自核穿孔的人镰状cd34+细胞的bfu-e和cfu-gemm集落。(b)在用tat-cas9wt-36gfp-inf7rnp/ssodn进行核穿孔后从人镰状cd34+细胞获得的集落的代表性sanger测序结果。(c)用cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp外加ssodn进行核穿孔后的集落存活。
图23是来自表6的深度测序数据的图形化汇总。
图24示出cas9wtrnp靶向位点附近的非特异性修饰。来自cas9wtrnp/ssodn核穿孔的镰状cd34+细胞的bfu-e集落包含似乎未在切割位点引发的插入缺失。标记为“上游”的顶部序列代表预期切割位点上游的非特异性修饰。标记为“下游”的底部序列代表在预期切割位点下游观察到的非特异性修饰。箭头指示镰状突变的位置,并且剪刀指示cas9wtrnp的预期切割位点。
图25示出在将用cas9rnp/ssodn核穿孔的镰状小鼠胎肝c-kit+细胞初次移植到经辐照的c57bi/6小鼠中以校正镰状细胞突变后6周对血液进行的等电聚焦(ief)凝胶分析。小鼠胎肝c-kit+细胞相当于人脐带血cd34+细胞。
图26示出在移植到经辐照的c57bi6小鼠中后12周对facs纯化的骨髓细胞的ddpcr分析。核穿孔和移植后12周,校正了大约50%的红系细胞(ter119+)和骨髓细胞(cd11b+和cd11b+/gr1+)。红系细胞和骨髓细胞存活相对较短;因此,这些细胞源自移植的hsc。b细胞和t细胞中的校正水平在12周(总共24周)的二次移植后可升高至大约50%。24周后,大多数(即使不是全部)的造血细胞将源自长期的hsc。
图27示出在将用cas9rnp/ssodn核穿孔的细胞初次移植以校正镰状细胞突变后12周以及二次移植以校正镰状细胞突变后6周对来自小鼠的血液进行的ief凝胶分析。在将用cas9rnp/ssodn核穿孔的hsc进行移植以校正镰状细胞突变后,在小鼠体内中产生人hba。
详述
本文提供了用于细胞的基因组修饰的crispr/cas9复合物。使用本文提供的复合物的方法导致修饰效率提高,细胞存活率提高和/或细胞中hdr与nhej的比率增加。这些复合物和方法可用于治疗目的,例如校正细胞中的突变,其中所述突变与疾病或病症相关。
本发明提供一种用于校正细胞基因组中的突变的复合物,其包含(a)指导rna(grna),其包含:与所述细胞基因组中的靶dna杂交的第一核苷酸序列,其中所述靶dna包含突变;和与定点核酸酶相互作用的第二核苷酸序列;(b)重组定点核酸酶,其可操作地连接至超荷电蛋白,其中所述定点核酸酶包含与所述指导rna的所述第二核苷酸序列相互作用的rna结合部分,并且其中所述定点核酸酶特异性结合并切割所述靶dna以产生双链断裂;和(c)单链供体寡核苷酸(ssodn),其与所述靶dna中双链断裂侧翼的基因组序列杂交并整合到所述靶dna中以校正所述靶dna中的突变。
应当理解,本文所述的包含指导rna(grna)、重组定点核酸酶和供体核苷酸的复合物在自然界中不存在。然而,该复合物向必需元件提供了所需的构型和化学计量学,以高效且有效地修饰细胞。grna分子结合定点核酸酶并将核酸酶靶向至靶dna内的特定位置。grna包含:第一核苷酸序列,其与细胞基因组中的靶dna杂交,其中所述靶dna包含突变;以及第二核苷酸序列,其与定点核酸酶相互作用。本文所述的复合物可包含一个或两个独立的grna。因此,术语指导rna包括单指导rna和双指导rna两者。可用于校正与镰状细胞性贫血相关联的突变的指导序列的实例在本文中被阐述为taacggcagacttctccac(seqidno:1)。包含用于cas9结合的茎环的指导序列的实例在本文中被提供为gtaacggcagacttctccacgttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcttttttt(seqidno:2)。应注意的是,在体外转录期间,seqidno:2的5'g添加了t7。
在本文所述的复合物中,重组定点核酸酶可为rna指导的定点核酸酶,例如来自任何细菌种类的cas蛋白或其功能片段。例如,cas蛋白可为cas9蛋白或其功能片段。如本文所用,术语“cas9”是指存在于编码ii型crispr/cas9体系的任何细菌种类中的cas9蛋白或其片段。参见例如,makarova等,naturereviews,microbiology,9:467-477(2011),包括补充信息,其全部内容据此以引用方式并入。例如,cas9蛋白或其片段可来自酿脓链球菌(streptococcuspyogenes)。全长cas9是包含识别结构域和两个核酸酶结构域(分别是hnh和ruvc)的核酸内切酶。在氨基酸序列中,hnh是线性连续的,而ruvc被分成三个区,一个在识别结构域的左侧,另外两个在hnh结构域侧翼的识别结构域的右侧。来自酿脓链球菌的cas9通过与指导rna相互作用靶向至细胞中的基因组位点,所述指导rna与紧接在由cas9识别的ngg基序之前的20个核苷酸的dna序列杂交。这导致双链断裂,该双链断裂通过供体核苷酸经由hdr进行修复,所述供体核苷酸为例如ssodn或双链dna构建体,其与靶dna中的双链断裂侧翼的基因组序列杂交并整合到靶dna中以校正靶dna中的突变。
在本文提供的复合物中,grna、可操作地连接至超荷电蛋白的定点核酸酶以及ssodn的摩尔比可为约1:1:0.2至约1.5:1:2.0。例如,grna、可操作地连接至超荷电蛋白的定点核酸酶以及ssodn的摩尔比可为约1:1:1、1.1:1:1、1:1:1.15、1:1:1.25、1:1:1.30;1:1:1.35;1:1:1.40;1:1:1.50、1.2:1:1、1.3:1:1.1.4:1:1、1.5:1:1、1.5:1:1.15、1.5:1:1.25、1.5:1:1.35;1.5:1:1.40、1.5:1:1.45;1.5:1:1.50;1.5:1:1.55;1.5:1:1.60;1.5:1:1.65;1.5:1:1.70;1.5:1:1.75;1.5:1:1.80;1.5:1:1.85;1.5:1:1.90;1.5:1:1.95;1.5:1:2.0或这些比率之间的任何比率。具有这些摩尔比的复合物可用于本文所述的任何方法中。在将复合物引入细胞或细胞群之前制备复合物的方法在实施例中阐述。
如本文所用,超荷电蛋白可为具有比其对应的未修饰蛋白更大的总正电荷的超荷正电蛋白。例如,超荷正电蛋白可为具有约+5至约+40的总正电荷的超荷正电绿色荧光蛋白(gfp)。例如,总正电荷可为约+5、+6、+7、+8、+9、+10、+11、+12、+13、+14、+15、+16、+17、+18、+19、+20、+21、+22、+23、+24、+25、+26、+27、+28、+29、+30、+31、+32、+33、+34、+35、+36、+37、+38、+39或+40。
超荷电蛋白可可操作地连接至核酸酶的氨基末端或羧基末端。还考虑超荷电蛋白可与核酸酶缔合,而不必与核酸酶共价连接。超荷电蛋白的实例是超荷正电gfp,例如+36gfp。+36gfp可可操作地连接至cas9或其功能片段的氨基末端或羧基末端。参见例如mcnaughton等,“mammaliancellpenetration,sirnatransfection,anddnatransfectionbysuperchargedproteins,”pnas106(15):6111-6116。本文以seqidno:3提供了包含可操作地连接至cas9的羧基末端的+36gfp的多肽的实例。
核酸酶还可可操作地连接至超荷电蛋白和一个或多个带正电荷的肽,例如,一个或多个反式作用转录活化因子(tat)肽可可操作地连接至核酸酶的氨基末端或羧基末端。例如但不限于,超荷正电蛋白可可操作地连接至核酸酶的羧基末端并且一个或多个tat肽(例如1xtat、2xtat、3xtat、4xtat等)可可操作地连接至核酸酶的氨基末端。本文以seqidno:4提供了多肽的实例,所述多肽包含可操作地连接至核酸酶的氨基末端的tat肽和可操作地连接至核酸酶的羧基末端的超荷正电gfp。还提供了与seqidno:3或seqidno:4具有至少约75%同一性的多肽序列。例如,还提供了至少约75%、80%、85%、90%、95%、99%或其间任何百分比的多肽序列。
核酸酶还可可操作地连接至超荷电蛋白,并且一个或多个带负电荷的肽,例如,长度为约10至约25个氨基酸的带负电荷的肽,例如seqidno:50,可可操作地连接至定点核酸酶的羧基末端。例如但不限于,超荷正电蛋白可可操作地连接至核酸酶的羧基末端,而带负电荷的肽可可操作地连接至超荷正电蛋白的羧基末端。
如通篇所用,重组是两个多核苷酸之间交换遗传信息的过程。同源引导的修复(hdr)是指例如在修复细胞中的双链断裂期间发生的dna修复。此过程需要核苷酸序列同源性,并使用供体分子,例如单链或双链核苷酸序列作为模板来修复靶基因组序列,即具有双链断裂的基因组序列,并导致遗传信息从供体转移到靶基因组序列。同源引导的修复可导致对靶基因组序列的序列修饰。例如,hdr可导致靶基因组序列中的插入、缺失或突变。供体多核苷酸的部分或全部序列可以并入靶dna中。还考虑供体多核苷酸、供体多核苷酸的一部分、供体多核苷酸的拷贝或供体多核苷酸的拷贝的一部分整合到靶dna中。
如通篇所用,所谓非同源性末端接合(nhej)意指通过将断裂端彼此直接连接而不需要同源模板来修复dna中的双链断裂(与同源引导的修复形成对比,所述同源引导的修复需要同源序列来指导修复)。
本文提供的复合物和方法可用于通过hdr来校正靶dna中的任何突变。例如但不限于,所述复合物可用于在基因组中的特定位点用正确的核苷酸序列来替换不正确的核苷酸序列(例如,以恢复由于功能突变缺失而引起的受损的靶多核苷酸序列的功能,即,snp)。举例来说,这些突变可以与自身免疫性疾病、遗传疾病、血液病症、t细胞病症、单基因病症、癌症、神经退行性疾病、心血管疾病或传染病相关联。例如但不限于,本文提供的复合物和方法可用于校正与镰状细胞病相关联的突变(即,血红蛋白基因中的突变,例如在β-珠蛋白基因的第6位密码子处导致谷氨酸至缬氨酸取代的gag至gtg突变)、与重度联合免疫缺陷症(scid)相关联的突变(例如,jak3中的突变)、与β地中海贫血或维-奥二氏综合征(wiskott-aldrichsyndrome)相关联的突变。
单个突变或多个突变的校正可用一种或多种复合物进行。本文提供的复合物和方法还可用于将序列插入基因组中的特定位点以校正缺失,而非是进行校正或取代。本文提供的复合物和方法还可用于将编码功能性多肽的核苷酸序列插入细胞基因组中的特定位点,以便在细胞中表达功能性多肽。功能性多肽可为细胞内源性(即,通常由细胞表达)或外源性(即,通常不由细胞表达)的多肽。例如,可将嵌合抗原受体(car)序列插入t细胞前体的基因组中以产生用于治疗癌症的癌症特异性t细胞。在另一个实例中,本文提供的复合物和方法可用于抑制细胞基因组中特定位点的基因活性。例如,本文提供的复合物和方法可用于将序列插入cxcr4或ccr5受体中以治疗或预防hiv感染。
与常规的crispr/cas体系相比,本文提供的复合物可以意外高的效率修饰或改变靶dna。细胞群中的改变效率可为至少约5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%或80%或更高或这些百分比之间的任何百分比。改变效率还可大于或等于约80%。因此,本文还提供了细胞群,其中至少5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%或80%或更高或其间的任何百分比发生改变。例如,与镰状细胞病或其他病症相关联的突变已被校正。如果将包含与镰状细胞病相关联的突变的细胞群与本文所述的crispr/cas复合物接触,并且突变在约5%的细胞中被校正,则修饰或改变的效率为约5%。任选地,其中与镰状细胞病相关联的突变在约30%的细胞(包括例如27%、28%和29%)中得到校正的细胞群在移植到患有镰状细胞病的受试者时足以治疗镰状细胞病。任选地,与镰状细胞病相关联的突变在群中约40%、50%、60%、70%、80%、90%或更高或其间任何百分比的细胞中被校正。
除了高效地改变靶dna之外,本文提供的复合物还可增加与复合物接触的细胞群中的hdr与nhej的比率。hdr/nhej比率可为约10至约0.5。例如,hdr/nhej比率可为约10、9、8、7、6、5、4、3、2、1、0.5或更小或者这些比率之间的任何比率。除了高校正效率和高hdr与nhej比率之外,经校正的细胞的细胞存活率可为至少约50%、60%、70%、80%、90%或更高以及其间的任何百分比。
可使用本文所述的复合物来修饰或获得任何细胞。将复合物引入细胞中可为细胞周期依赖性的或为非细胞周期依赖性的。使细胞同步以增加特定阶段(例如s阶段)中的细胞比例的方法是本领域已知的。参见例如takahashi等“efficientintroductionofageneintohematopoieticcellsins-phasebyelectroporation,”exp.hematol.19(5):343-346(1991)。根据待修饰的细胞的类型,本领域技术人员可容易地确定细胞周期同步是否是必要的。
细胞可为真核细胞,例如哺乳动物细胞。细胞还可为原核细胞或植物细胞。细胞可为人细胞。细胞可为生殖细胞、体细胞、干细胞、前体细胞或祖细胞。前体细胞可为例如多能干细胞(pluripotentstemcell)或多能干细胞(multipotentstemcell),如造血干细胞。如通篇所用,多能细胞包括诱导性多能干细胞。制备诱导性多能干细胞的方法是本领域已知的并且描述于实施例中。细胞还可为cd34+细胞,其任选地源自诱导性多能干细胞。cd34+细胞可选自原代cd34+造血祖细胞、cd34+外周血细胞、cd34+脐带血细胞和cd34+骨髓细胞。细胞还可为原代细胞,例如原代cd34+造血祖细胞。细胞是非癌细胞的细胞、非肿瘤细胞的细胞或非转化细胞的细胞。可以在校正之前或之后对细胞进行筛选,以获得不希望的遗传特征的证据。还提供了包含本文所述的任何复合物的细胞。细胞可为体外的、离体的或体内的。
还提供了对细胞群中的靶dna进行位点特异性修饰的方法,其包括向细胞中引入本文所述的任何复合物,其中在允许同源引导的修复(hdr)以及将供体核苷酸(例如,ssodn或双链核苷酸序列)整合到靶dna中的条件下将复合物引入细胞中。复合物经由核穿孔而引入细胞中。核穿孔的方法是本领域已知的。参见,例如,maasho等,“efficientgenetransferintothehumannaturalkillercellline,nkl,usingtheamaxanucleofectionsystem,”journalofimmunologicalmethods284(1-2):133-140(2004);和aluigi等,“nucleofectionisanefficientnon-viraltransductiontechniqueforhumanbonemarrowderivedmesenchymalstemcells,”stemcells24(2):454-461(2006)),这两篇文献均以引用的方式全文并入本文中。
在本文提供的一些方法中,供体核苷酸(例如ssodn或双链核苷酸序列)整合到靶dna中并校正靶dna中的突变。在本文提供的方法中,细胞群中hdr与nhej的比率相对于其他crispr-cas9递送方法增加。hdr/nhej比率可为约10至约0.5。例如,hdr/nhej比率可为约10、9、8、7、6、5、4、3、2、1、0.5或更小或者这些比率之间的任何比率。在本文提供的方法中,hdr的改变效率可为至少约5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%或更高或者这些百分比之间的任何百分比。hdr的改变效率还可大于或等于约80%。例如,如果将包含与镰状细胞性贫血相关联的突变的细胞群与本文所述的crispr/cas复合物接触,并且突变在约5%的细胞中被校正,则hdr的改变效率为约5%。细胞群可从患有病症的受试者获得,使得至少约5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%或80%或更高或这些百分比之间的任何百分比的细胞历经hdr以校正与所述病症相关联的突变。在一些情况下,高于80%的来自受试者的细胞将经历hdr以校正与所述病症相关联的突变。在本文所述的方法中,介于约50%和99%之间的细胞在引入复合物后存活。例如,在引入复合物之后,高于约50%、60%、70%、80%、90%、95%、99%或这些百分比之间的任何百分比的经校正的细胞存活。
还提供了一种治疗受试者的与编码血红蛋白的基因组序列中的突变相关联的疾病的方法,其包括:(a)将复合物引入从所述受试者获得的细胞群中,所述复合物包含(1)指导rna(grna),其包含:与所述细胞基因组中的靶dna杂交的第一核苷酸序列,其中所述靶dna是包含突变的血红蛋白基因;和与定点核酸酶相互作用的第二核苷酸序列;(2)重组定点核酸酶,其可操作地连接至超荷电蛋白,其中所述定点核酸酶包含与所述指导rna的所述第二核苷酸序列相互作用的rna结合部分,并且其中所述定点核酸酶特异性结合并切割所述靶dna以产生双链断裂;和(3)单链供体寡核苷酸(ssodn),其与所述靶dna中双链断裂侧翼的基因组序列杂交并整合到所述靶dna中以校正血红蛋白基因中的突变;以及(b)将经校正的细胞移植到所述受试者中。
在用于治疗受试者的与编码血红蛋白的基因组序列中的突变相关联的疾病例如镰状细胞性贫血的方法中,患有镰状细胞性贫血的受试者可任选地是输血依赖性受试者或患有至少一种沉默性梗塞的受试者。受试者的年龄还可小于约12个月、11个月、10个月、9个月、8个月、7个月、6个月、5个月、4个月、3个月、2个月或1个月。由于婴儿接受常规的镰状细胞病筛查,因此可在疾病症状显示之前对婴儿进行治疗。本文提供的方法还可包括诊断患有病症(例如镰状细胞病)的受试者。
如上所述,可从患有所述疾病的受试者或从相关供体获得细胞。例如,可从受试者获得或收获骨髓细胞。骨髓收获包括用放入骨的柔软中央部分(骨髓)的针来采集干细胞。骨髓可例如从受试者的髋骨或胸骨中收获。可从受试者获得约500ml至约1升的骨髓。
在本文提供的任何方法中,细胞可为真核细胞,例如人细胞。细胞可为生殖细胞、干细胞、前体细胞。前体细胞可为例如多能干细胞或造血干细胞。如通篇所用,多能细胞包括诱导性多能干细胞。制备诱导性多能干细胞的方法是本领域已知的并且描述于实施例中。细胞还可为cd34+细胞。cd34+细胞可选自原代cd34+造血祖细胞、cd34+外周血细胞、cd34+脐带血细胞和cd34+骨髓细胞。细胞还可为原代细胞,例如原代cd34+造血祖细胞。细胞是非癌细胞的细胞、非肿瘤细胞的细胞或非转化细胞的细胞。细胞可为在体外的或离体的。细胞还可为药学上可接受的组合物。
本文提供的方法还可包括培养用hdr校正的细胞。例如,可在扩增条件下或在促进经校正的细胞分化成t细胞的条件下培养细胞。例如但不限于,使用本文提供的方法,在于诱导性多能干细胞中经由hdr校正突变之后,可以将经校正的细胞与人骨髓基质细胞共培养以产生cd34+细胞。然后可在使cd34+细胞分化成t细胞的条件下培养cd34+细胞。
本文提供的方法还可包括在移植之前针对适当校正、其他突变或nej筛选经校正的细胞。任选地,可筛选细胞以检测具有一种或多种校正的细胞。
在本文提供的方法中,可将细胞在修饰后(例如在通过hdr校正突变之后)移植到受试者中。可将细胞在分化或未分化的情况下移植到受试者中。例如,修饰的造血干细胞(hsc)可以骨髓移植物的形式施用,其中使hsc在受试者体内分化和成熟。或者,可在移植之前使修饰的细胞分化成所需的细胞群。
如本文所使用,将向受试者移植、引入或施用细胞是指将细胞置于受试者中。例如,包含根据本文所述的方法校正或修饰的靶dna序列的细胞可通过适当的途径移植到受试者中,所述途径使得移植的细胞至少部分定位在所需部位。细胞可直接植入所需部位,或替代地,可通过任何适当的途径施用,所述途径使得可递送至受试者的所需位置,在所述所需位置至少一部分植入细胞保持活力。例如,细胞可经由静脉内输注进行全身施用。施用给受试者后的细胞活力期可短至数小时例如二十四小时、数天,甚至几年。
对于离体方法,细胞可为自体细胞,即,取自需要修饰一个或多个细胞中的靶dna的受试者的一个或多个细胞(即,供体和受体是同一个体)。如本文所述,修饰可为例如校正突变、插入抑制蛋白活性的序列或插入增加蛋白表达的序列,例如插入编码可用于靶向癌细胞的嵌合抗原受体的序列。自体细胞可用于避免可导致细胞排斥的免疫反应。换句话说,当使用自体细胞时,供体和受体是同一受试者。或者,细胞可为异源的,例如取自供体,优选相关供体。第二受试者可为相同或不同的物种。通常,当细胞来自供体时,它们将来自与受体具有足够免疫相容性的供体,以降低移植排斥的可能性,和/或减少对免疫抑制疗法的需求。细胞还可从异种来源获得,即已被遗传工程改造成与受体或受体物种具有足够免疫相容性的非人哺乳动物。治疗本文所述的病症的任何方法还可包括向受试者施用一种或多种免疫抑制剂。
在涉及移植的方法中,在移植本文所述的任何细胞之前,受试者任选地历经清髓性疗法(myeloablativetherapy)。清髓性疗法可包括施用一次或多次剂量的化学疗法、放射疗法或两者,其导致健康骨髓细胞的严重或完全耗竭。在另一个实例中,受试者可历经亚清髓性核疗法,其包括施用使健康骨髓细胞的一部分耗竭的一次或多次剂量的化学疗法、放射疗法或两者。细胞还可移植到已历经非消融性化疗的受试者中。例如,细胞可移植到已用白消安(busulfan)、氟达拉滨(fludarabine)和/或苏消安(treosulfan)治疗的受试者中。
在涉及移植的方法中,向受试者施用有效剂量或量的校正细胞。术语有效量和有效剂量可互换使用。术语有效量被定义为产生所需生理反应所需要的任何量。在一些方法中,可以施用约1×106至约7×106个校正的细胞/千克,但该量可根据相关联的病症而变化。经校正的细胞的百分比、施用这些细胞的有效量和时间安排可以凭经验确定,并且做出此类确定在本领域技术人员的能力范围内。施用的剂量范围是大到足以产生所需效果(例如治疗疾病,例如镰状细胞性贫血)的剂量范围。剂量不应过大以致引起显著的不良副作用,例如不希望的交叉反应、过敏反应等。通常,剂量将随着年龄、状况、性别、疾病类型、疾病或病症的程度,施用途径或其他药物是否包括在方案中而变化,并且可由本领域的技术人员确定。如有任何禁忌症,则可由个体医师对剂量进行调整。剂量可变化,并且可根据需要将药剂以每天一次或多次剂量进行施用,持续一天或多天。
如通篇所用,受试者可为脊椎动物、更具体而言是哺乳动物(例如、人、马、猫、狗、牛、猪、绵羊、山羊、小鼠、兔、大鼠和豚鼠)。该术语不表示具体的年龄或性别。因此,意在涵盖成年人和新生儿,无论是男性还是女性。如本文所用,患者或受试者可以互换使用,并且可指患有病症或处于发展成病症危险中的受试者。术语患者或受试者包括人和兽医受试者。
如本文所用,术语治疗是指通过在受试者中引发免疫应答来减轻病症的一种或多种效应或减轻病症的一种或多种症状的方法,所述病症例如为镰状细胞病。因此,在所公开的方法中,治疗可指将镰状细胞病或其他病症的严重程度减轻10%、20%、30%、40%、50%、60%、70%、80%、90%或100%。例如,治疗镰状细胞病的方法被认为是与对照相比时使受试者的感染的一种或多种症状减轻10%的治疗。因此,与天然或对照水平相比,减轻可为10%、20%、30%、40%、50%、60%、70%、80%、90%、100%,或介于10%与100%之间的任何百分比的减轻。应当理解,治疗不一定是指治愈或完全消除病症或病症的症状。
还提供了一种校正与t细胞病症相关联的突变的方法,其包括:将复合物引入从患有t细胞病症的受试者获得的细胞群中,所述复合物包含:(a))指导rna(grna),其包含:与所述细胞基因组中的靶dna杂交的第一核苷酸序列,其中所述靶dna包含与所述t细胞病症相关联的突变;和与定点核酸酶相互作用的第二核苷酸序列;(b)重组定点核酸酶,其可操作地连接至超荷电蛋白,其中所述定点核酸酶包含与所述grna的所述第二核苷酸序列相互作用的rna结合部分,并且其中所述定点核酸酶特异性结合并切割所述靶dna以在所述靶dna中产生双链断裂,所述靶dna包含与t细胞病症相关联的突变;和(c)单链供体寡核苷酸(ssodn),其包含第三核苷酸序列,所述第三核苷酸序列与所述靶dna中双链断裂侧翼的基因组序列杂交并整合到所述靶dna中以校正与所述t细胞病症相关联的突变,其中在允许同源引导的修复(hdr)的条件下将所述复合物引入所述细胞中以校正与t细胞病症相关联的突变。
在本文提供的方法中,包含与t细胞病症相关联的突变的靶dna可为编码与t淋巴细胞发育相关联的蛋白的靶dna。例如,靶dna可编码jak3。这种经校正的细胞可用于例如治疗scid。
除了校正细胞基因组中的突变以外,本文提供的复合物和方法还可用于将功能性多肽插入细胞基因组的特定位点,使得多肽由细胞表达。多肽可在细胞中或细胞表面上表达。
还提供了一种制备肿瘤特异性t细胞前体细胞的方法,其包括:将复合物引入t细胞前体细胞群中,所述复合物包含:(a)指导(grna),其包含与所述t细胞前体细胞的基因组中的靶dna杂交的第一核苷酸序列,和与定点核酸酶相互作用的第二核苷酸序列;(b)重组定点核酸酶,其可操作地连接至超荷电蛋白,其中所述定点核酸酶包含与所述grna的所述第二核苷酸序列相互作用的rna结合部分,并且其中所述定点核酸酶特异性结合并切割所述靶dna以产生双链断裂;(c)供体核苷酸序列,其包含编码嵌合抗原受体(car)的第三核苷酸序列和与所述靶dna中双链断裂侧翼的基因组序列杂交的第四核苷酸序列,其中在允许同源引导的修复(hdr)以及将第三核苷酸序列整合到靶dna中的条件下将所述复合物引入所述t细胞前体细胞中,以形成表达car的经修饰的t细胞前体细胞。
t细胞前体细胞可从患有癌症的受试者获得。如上所述,hdr/nhej比率可为约10至约0.5。例如,hdr/nhej比率可为约10、9、8、7、6、5、4、3、2、1、0.5或这些比率之间的任何比率。在本文提供的方法中,hdr的改变效率可为至少约5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%、80%或这些百分比之间的任何百分比。hdr的改变效率还可大于或等于约80%。例如,当使用本文所述的方法时,如果编码功能性多肽的核苷酸序列(例如编码car的核苷酸序列)插入约5%的细胞中,则hdr的改变效率为约5%。细胞群可从患有癌症的受试者获得,使得至少约5%、10%、15%、20%、25%、30%、35%、40%、45%、50%、55%、60%、65%、70%、75%或80%或这些百分比之间的任何百分比的细胞历经hdr以插入编码嵌合抗原受体(car)的核苷酸序列并形成表达car的细胞。在一些情况下,高于80%的来自受试者的细胞将经历hdr以校正与所述病症相关联的突变。
表达car的经修饰的t细胞前体细胞可移植到患有癌症的受试者中。如本文所用,癌症是由异常细胞的快速和不受控的生长所表征的疾病。癌细胞可以局部扩散或通过血流和淋巴系统扩散到身体的其他部位。癌症的实例包括但不限于乳腺癌、前列腺癌、卵巢癌、宫颈癌、皮肤癌、胰腺癌、结直肠癌、肾癌、肝癌、脑癌、淋巴瘤、白血病、肺癌等。当抗原结合结构域结合其对应的抗原时,表达car的经修饰的t细胞前体细胞表现出抗肿瘤免疫性。
公开了可用于所公开的方法和组合物、可与所公开的方法和组合物联合使用、可用于制备所公开的方法和组合物、或者是所公开的方法和组合物的产品的材料、组合物和组分。这些和其他材料在本文中公开,并且应当理解的是,当这些材料的组合、子集、相互作用、群组等被公开时,虽然可能没有明确地公开每种不同的单独和集体组合和排列的具体的引用,但每一种均被具体地涵盖并且在本文描述。例如,如果一种方法被公开并被讨论,并且讨论了可对该方法步骤进行的许多修改,则方法步骤的每个和每一个组合和排列以及可能的修改均被具体地涵盖,除非有具体相反的说明。同样地,这些的任何子集或组合也被具体地涵盖和公开。这个概念适用于本公开的所有方面,包括但不限于使用所公开的组合物的方法中的步骤。因此,如果存在可执行的多个附加步骤,应当理解的是,这些附加步骤中的每一者可与所公开的方法的任何特定的方法步骤或方法步骤的组合一起执行,并且每个此类组合或组合的子集均被具体地涵盖并且应被视为公开。
本文引用的出版物及其引用的材料据此以引用的方式明确地全文并入。
实施例
实施例1
通过crispr/cas9增强的基因替代来校正scid
janus家族激酶jak3基因的突变引起重度联合免疫缺陷症(scid)。人体jak3缺陷的特征是缺少循环t细胞和自然杀伤(nk)细胞,并且具有正常数目的功能不良的b细胞(t–b+nk–)。如本文所示,使用scid患者特异性诱导性多能干细胞(ipsc)和t细胞体外分化系统,证明了jak3缺陷型细胞的早期t细胞发育中的完整阻断。通过crispr/cas9增强的基因替代来校正新型jak3突变使得恢复了正常t细胞发育,包括产生具有宽泛t细胞受体(tcr)库的成熟t细胞群。经校正细胞的全基因组测序证明没有crispr/cas9脱靶修饰。因此,本文提供了用于研究人淋巴细胞生成的新型方法和用于具有免疫缺陷的人中的基因替代疗法的方法。
同种异体基因造血干细胞(hsc)移植是目前唯一建立的针对scid的疗法;然而,延迟的免疫恢复和移植物抗宿主疾病的风险存在显著的风险。对于x连锁scid,已经成功地证明了通过基于逆转录病毒的基因疗法所进行的治疗。然而,已经观察到逆转录病毒基因疗法具有插入诱变的严重副作用。自失活慢病毒载体已被有效地用于最近的临床试验,但需要长期随访以彻底解决安全问题。
本文提供了一种替代治疗策略,其中获得患者特异性诱导性多能干细胞(ipsc),并且通过使用crispr-cas9复合物进行基因替代来校正致病性突变。这些经校正的ipsc可以任选地分化成造血祖细胞以移植到患者中来治疗疾病(hanna等,“treatmentofsicklecellanemiamousemodelwithipscellsgeneratedfromautologousskin,”science318:1920-1923(2007))。如本文所示,jak3缺陷型人t细胞的分化在早期发育阶段被阻断。还证明了通过crispr/cas9增强的基因替代校正人jak3突变恢复了早期t细胞祖细胞的分化潜能。这些经校正的祖细胞能够产生nk细胞和表达宽泛的t细胞抗原受体(tcr)库的成熟t细胞群。这些研究建立了一种强效体系,该体系用于确定人scid患者免疫缺陷的机制以及测试该病症的药理学和遗传学疗法。
患者信息
根据赫尔辛基宣言(declarationofhelsinki),男性患者被纳入伦理审查委员会(institutionalreviewboard)批准的研究。家族史对免疫缺陷为阴性。在前8个月龄的时候,他的体重增加不良,具有腹泻以及需要频繁住院的复发性毛细支气管炎。他在8个月龄时被送入医院,伴有严重的呼吸窘迫和鹅口疮。采用支气管肺泡灌洗的支气管镜检查证明有细菌(假单胞菌,h型流感,肺炎链球菌)和病毒生物体(呼吸道合胞病毒)。免疫学评价证明具有重度低丙种球蛋白血症,ige<3、iga<4、igg=29、igm=26。外周血的免疫表型分型证明完全缺乏cd3+t细胞和nk细胞,但存在b细胞(绝对b细胞计数=875)。有丝分裂原研究证明对伴刀豆球蛋白a、美洲商陆有丝分裂原(pokeweedmitogen)和植物血球凝集素a完全没有应答。scid的诊断通过基因检验得到确认,jak3基因外显子14的纯合c>t核苷酸置换导致精氨酸密码子(cga)在第613位氨基酸处被终止密码子(tga)替代。这是将此jak3变体(rs149316157)与scid临床病例联系起来的第一份报告。患者接历经强度降低的经调整匹配的无关骨髓移植,并且在脱离完全免疫重建治疗两年后现在表现良好。
人ipsc重编程和表征
对于ipsc诱导,将5×104个原代角质形成细胞接种到6孔板的一个孔中。在第二天,用1ml病毒上清液和1ml含聚凝胺的人角质形成细胞培养基以4μg/ml的终浓度转导角质形成细胞。将角质形成细胞以800×g离心转染45分钟(第1天)。第二天再次重复转导过程。在第3天,将细胞改换至新鲜的人角质形成细胞培养基并再培养两天。在第5天,将角质形成细胞用胰蛋白酶消化并转移到预先接种了经丝裂霉素c处理的鼠胚胎成纤维细胞(mef)的10cm培养皿中并在人角质形成细胞培养基中培养。在第7天,将细胞改换至人es培养基并在同一培养皿中连续培养3-4周。每天更换es培养基。2-3周后可看到潜在的ipsc集落。将这些集落单独挑选并在mef上扩增以进行分析。为了去除整合的慢病毒和多顺反子序列,用表达cre的腺病毒(rad-cre-ie)感染ipsc。挑选单个集落并使用引物gctaattcactcccaaagaagacaag(seqidno:5)和cttcagcaagccgagtcctg(seqidno:6)通过pcr来验证cre介导的对侧接loxp位点的(floxed)序列的去除。
用op9共培养产生cd34+细胞和t细胞
先前描述了该程序(chang等,“broadt-cellreceptorrepertoireint-lymphocytesderivedfromhumaninducedpluripotentstemcells,”plosone9,e97335(2014))。使用该方法进行了以下修改。如ohnuki等(ohnukim,“generationandcharacterizationofhumaninducedpluripotentstemcells.currprotocstemcellbiolchapter4:unit4a2(2009))所述,将6孔板的一个孔中的hipsc培养物用ctk溶液处理以制备小细胞团块。然后将细胞团块转移至10cm板,所述板在含有10%fbs,1x青霉素/链霉素和100μm单硫代甘油的基于α-mem的培养基中预先接种2天龄的op9细胞。每隔一天更换培养基,细胞培养18天而不分裂。共培养18天后,通过用解离溶液(α-mem培养基中的0.15%胶原酶iv和0.015%透明质酸酶)处理约30分钟来收获细胞,然后再用0.25%胰蛋白酶处理30分钟。然后在抗cd34+磁珠(microbeadkit;miltenyibiotec,bergischgladbach,germany)上纯化cd34+细胞。对于t细胞分化,将这些cd34+细胞铺在op9-dl4细胞上,并用含有20%fbs,5ng/mlhflt3-l,5ng/mlhil-7和10ng/mlhscf的α-mem培养基进行培养。培养基每隔一天更换一次,将细胞每4天转移到新的op9-dl4平板上。
t细胞刺激
在通过流式细胞术分析之前,根据制造商的方案,通过与cd3/28珠粒(invitrogen,carlsbad,ca)温育3天来刺激来自hipsc的体外来源t细胞,如先前所描述(chang等,2014)。
流式细胞术
收获细胞并洗涤,然后用lsrfortessa细胞分析仪(bdbioscience,sanjose,ca)进行分析。对于细胞表面染色,使用碘化丙啶(pi,sigma-aldrich,st.louis,mo)来排除死细胞。对于凋亡测定,将收获的细胞首先用细胞表面抗体染色30分钟。用1xpbs洗涤一次后,将细胞重悬于含有膜联蛋白v-647(invitrogen,carlsbad,ca)和pi的100μl膜联蛋白结合缓冲液(invitrogen,carlsbad,ca)中,并温育15分钟,然后添加含有pi的400μl膜联蛋白结合缓冲液。除非另外指明,从bdbiosciences获得抗体:cd3(percp-cy5-5,克隆ucht1)、cd4(pe-cy7,克隆sk3)、cd7(apc,bv510,克隆m-t701)、cd8(apc-cy7,克隆sk1)、cd16(pe,克隆b73.1)、cd25(fitc,克隆2a3)、cd34(pe-cy7,克隆wm59)、cd43(pe,克隆1g10)、cd56-pe(克隆my31)、cd69(fitc,克隆l78)、nkg2d-pe(克隆1d11)、tcr-αβ(fitc,pe,克隆t10b9.1a-31)、tcr-vδ1-fitc(fisherscientific,pittsburgh,pa,克隆ts8.2)、tcr-vδ2-pe(克隆b6)、tcrvγ9-fitc(克隆b3)、tnf-α-pe-cy7(克隆mab11)、betamarktcr库试剂盒(beckmancoulter,atlanta,ga)。
载体构建
先前描述了多顺反子oskm载体(chang等,polycistroniclentiviralvectorfor“hitandrun”reprogrammingofadultskinfibroblaststoinducedpluripotentstemcells,”stemcells27:1042-1049(2009))。通过将pcr扩增的人dl4cdna(openbiosystems,lafayette,co)、ires片段(openbiosystems)和mcherrycdna克隆到含有ef1α启动子的慢病毒载体(pdl171)中来构建lenti-hdl4-mcherry质粒。使用primestar聚合酶(takara,mountainview)进行pcr反应。
为了构建crispr质粒,设计grna寡核苷酸并按照zhang实验室方案(addgene,cambridge,ma)将其引入px330和px335质粒中。为了构建jak3修复质粒,使用jak3引物组(5'臂:gtcgacgtcgacgctcagtgaagctgaagtattccttctgcttcacagggcgaccactac(seqidno:7)和atttaaatcctcccctcgaacccttaccaaactcctatgcatactacag(seqidno:8);3'臂:ttaattaattaattagcattttaggttcaggttgtgagaacactagaagagaacaagtca(seqidno:9)和gtatacgtatacgcatacctggagaggggacaaggtcttgagatgcgagggt(seqidno:10)对野生型人基因组dna进行pcr扩增。用酶(5'臂:sali和swai;3'臂:paci和bstz17i)消化后,将pcr产物克隆到含有loxp-pgk-neo-loxp片段的质粒中。本研究中使用的所有寡核苷酸均由integrateddnatechnologies(idt,coralville,ia)合成。为了构建bcl2慢病毒质粒,使用引物组(正向:agccaccttaattaagccaccatggcgcacgctgggagaacggggtacgata(seqidno:11)和反向:taacagagagaagttcgtggctccggatcccttgtggcccagataggcacccagggtgat(seqidno:12))来扩增人bcl2cdna(openbiosystems)片段。产物通过pcr经由2a序列与gfp连接,并克隆到pdl171载体中。grna-f1caccgtgagatacagatacagaca(seqidno:13)grna-r1aaactgtctgtatctgtatctcac(seqidno:14)grna-f2caccgaatgatttgcctggaatgcc(seqidno:14)grna-r2aaacggcattccaggcaaatcattc(seqidno:15)grna-f3caccgcagcctaggcaaaggcctgc(seqidno:16)grna-r3aaacgcaggcctttgcctaggctgc(seqidno:17)grna-f4caccgtgccaacagaactgcctgat(seqidno:18)grna-r4aaacatcaggcagttctgttggcac(seqidno:19)grna-f5caccgaccagggtgcaagtgtgga(seqidno:20)grna-r5aaactccacacttgcaccctggtc(seqidno:21)grna-f6caccgctcctcagcctggcattca(seqidno:22)grna-r6aaactgaatgccaggctgaggagc(seqidno:23)
细胞培养
在由补充有1x非必需氨基酸、1x青霉素-链霉素、1xl-谷氨酰胺(全部来自mediatech)、20%knockout血清替代物(invitrogen)、2-βme(sigma)和5-10ng/mlbfgf(invitrogen)的dmemf-12组成的es细胞培养基中,在源自e14.5cf-1胚胎的经丝裂霉素c处理的mef上培养ipsc。在dermalifekmediumcompletekit(lifelinecelltechnology,frederick,md)中培养人原代角质形成细胞。op9细胞购自atcc,并在含有20%fbs和青霉素-链霉素的α-mem培养基中生长。通过用含有hdl4和mcherry的慢病毒转导op9细胞来建立op9-dl4细胞。
病毒生产
为了制备慢病毒,通过fugene6(roche,nutley,njorpromega,madison,wi)将10μg慢病毒载体、2.5μg包膜质粒(pmdg)和7.5μg包装质粒(pcmbvdr8.9.1)共转染到5×106个293t细胞中。转染后2天收集含病毒的上清液,并通过0.45μm过滤器。
基因靶向
将ipsc用0.25%胰蛋白酶处理5分钟以产生单细胞悬液。用1xpbs洗涤两次后,将1至2百万个细胞与5μgjak3修复质粒和5μg用于核转染的px330-jak3或px335-jak3质粒(humanstemcellnucleofectorkit,programa-023,lonza,alpharetta,ga)进行混合并铺在mef上。二至四天后,将含有30μg/ml的g418的hes培养基加入平板以选择耐药性集落。3至4周后挑选集落并进行扩增以用于基因组dna提取。对于pcr基因分型,使用5'引物组(tgctaaagcgcatgctccagact(seqidno:24)和gtcttcatctcagggtcggct(seqidno:25))以及3'引物组(cctctctgtgcattatggcag(seqidno:26)和gccttctatcgccttcttg(seqidno:27))。为了去除neo选择标志物,用表达cre的腺病毒(rad-cre-ie)感染hipsc。
rt-pcr
用trizol试剂(invitrogen,carlsbad,ca)从体外来源细胞中分离总rna。使用superscript第一链合成系统(first-strandsynthesissystem)(invitrogen)根据制造商的说明用0.5至2μg的总rna合成cdna。根据制造商的说明将sybrgreenpcrmastermix(lifetechnologies,carlsbad,ca)用于qpcr。用于qpcr的引物组是gapdh(f:actcctccacctttgacgct(seqidno:28),r:tcccctcttcaagggtctacatg(seqidno:29));pu.1(f:gtgcaaaatggaagggtttc(seqidno:30),r:ggagctccgtgaagttgttc(seqidno:31));gata3(f:tgtttcctttcactggccaca(seqidno:32),r:aacggcaactggtgaacggta(seqidno:33));bcl11b(f:ggcgatgccagaatagatgccg(seqidno:34),r:ccaggccacttggctcctctatctccaga(seqidno:35));rag1(f:ccttactgttgagactgcaatatcc(seqidno:36),r:ctgaagtcccagtatatacttcacac(seqidno:37));rag2(f:cccagaagcagtaataatcatcgag(seqidno:38),r:atgtgggatgtagtagatcttgc(seqidno:39));pta(f:gggtcttacctcagcagttac(seqidno:40),r:cctcacacagtgtgacgcag(seqidno:41));bcl2(f:gactgagtacctgaaccggc(seqidno:42),r:gggccaaactgagcagagtc(seqidno:43));bax(f:aagaccagggtggttgggac(seqidno:44),r:gtaagaaaaatgcccacgtc(seqidno:45));和jak3(f:agtcagacgtctggagcttc(seqidno:46),r:gtgagcagtgaaggcatgagtc(seqidno:47))。所有值均相对于gapdh表达归一化。
全基因组测序和分析
使用covariss2聚焦超声发生器剪切来自ipsc的dna:在扫频模式下,使微型管中的130μl样品接受两个40秒循环,其中占空比为10%,强度为4,每次破碎200个循环。使用agilent2100生物分析仪的dna芯片(dna1000kit;agilenttechnologies,santaclara,ca)分析表明平均片段大小为400bp。使用用于illumina的nebnextultradna库进行库制备(neb#e7370)进行库制备,并且使用kapaillumina库量化试剂盒(kk4835;kapabiosystems,wilmington,ma)和appliedbiosystemsviia7实时pcr系统(lifetechnologies)通过qpcr来测定最终库浓度。使用illuminatruseqpeclusterkitv3–cbot–hs(pe-401-3001)和illuminacbot在流动细胞上产生测序簇。使用illuminatruseqsbs试剂盒v3–hs–200循环(fc-401-3001)和illuminahiseq2500升级进行wgs,以生成用于生物信息学分析的2×100单索引配对末端读段。通过如下方式来鉴定可能的脱靶位点:使用embossfuzznuc软件(v6.6.0.0)将crispr/cas9指导序列与hg19参照基因组比对(rice等,“emboss:theeuropeanmolecularbiologyopensoftwaresuite,“trendsingenetics:tig16:276-277(2000)),并允许最多三个不匹配;对于第一指导序列(gtgagatacagatacagaca)(seqidno:48)预测了1193个位点,对于第二指导序列(aatgatttgcctggaatgcc)(seqidno:49)预测了257个位点。使用bwa(v0.7.5a)mem算法(li和durbin,“fastandaccuratelong-readalignmentwithburrows-wheelertransform,”bioinformatics26:589-595(2010))将针对每个样品的所有来自wgs的读段映射到hg19参考基因组,并且使用picard-工具(v1.100)(http://picard.sourceforge.net)去除重复的读段。使用gatk(v2.7-2)进行局部重新比对以及碱基质量值重校正(mckenna等,“thegenomeanalysistoolkit:amapreduceframeworkforanalyzingnext-generationdnasequencingdata,”genomeresearch20:1297-1303(2010))。使用gatkhaplotypecaller来调用snv和插入缺失。此外,按照gatk最佳实践(gatkbestpractices)中的描述分别对snv和插入缺失进行了重新校正,并且应用了质量过滤器。将来自参考基因组的所有四个ipsc样品共有的变体从crispr/cas9脱靶分析中排除。使用bedtools(v2.17.0)(quinlan和hall,“bedtools:aflexiblesuiteofutilitiesforcomparinggenomicfeatures,”bioinformatics26:841-842(2010))来筛选未排除的变体以确定它们是否落入可能的脱靶位点。分析显示,这些变体均不存在于脱靶位点中,并表明这些突变是随机累积的。然后使用annovar软件包对具有低等位基因频率(<1%,dbsnp138)的所有功能变体(排除的和未排除的)进行注释,并筛选已知与hgmd和clinvar(v20140902)中疾病的关联;此外,还使用gwascatalog和cosmic(v70)对具有高cadd得分(cadd>=20)的所有命中的与复杂疾病的关联进行了筛选。在所查询的数据库中未鉴定出经验证的疾病相关变体。特别要关注的是,jak3c1837t(p.r613x)突变也未被验证与疾病相关联,但snp(rs149316157)被预测显著有害,其中gerp得分为3.85,cadd得分(caddphred样得分)38。因此,jak3c1837t变体首次与scid的临床病例相关联。
登录号
可在ncbisra数据库中以登录号srp056149访问wgs数据。
jak3缺陷型人t细胞表达低水平的bcl2并在早期发育阶段死亡
从scid患者的皮肤角质形成细胞产生ipsc(chang等,2009),该患者对于jak3基因的外显子14中的c>t核苷酸取代而言是纯合的。该突变用tga终止密码子(p.r613x)替代cga密码子(613处的精氨酸)。如上所述,4个月龄的患者呈现出t-b+nk-临床表型。为了确定这种scid表型是否可以在体外再现,尝试使用两步op9和op9-dl4体系(chang等,2014)将患者特异性ipsc分化成t淋巴细胞。jak3缺陷型ipsc的生长速率与源自健康供体的对照ipsc相当,并且这些ipsc在op9基质细胞单层上有效地分化成cd34+造血祖细胞(hp)。然而,当jak3缺陷型ipsc来源的cd34+hp铺在op9-dl4基质单层上时,与对照相比,缺乏t-细胞分化(图1)。未观察到cd3+t细胞或cd3-cd16+cd56+nk细胞(图1a),并且未检测到cd4+cd8+双阳性(dp),cd4+单阳性(sp)或cd8+单阳性(sp)t细胞(图1b)。由于在生产性tcr重排之前cd4-cd8-双阴性(dn)阶段的胸腺细胞分化中的阻断,jak3敲除(ko)小鼠具有小的胸腺。为了进一步理解由人的jak3突变所导致的发育缺陷,测定了与正常jak3野生型对照相比,jak3缺陷型细胞的t谱系定型和成熟。将ipsc来源的cd34+细胞铺在op9-dl4单层上,收获细胞并在t细胞诱导日(td)14、21、28和35(图4a)分析淋巴细胞标志物。在正常对照中,在td14时从1-2x106个cd34+细胞产生1.2x107个cd7+细胞(在淋巴门中计数的细胞的84%)。在t细胞成熟后依次检测t细胞标志物cd4、cd8、cd3和tcrαβ。在td35,超过50%的群是cd8sp细胞。在jak3缺陷型细胞中,在td14从1-2x106个cd34+细胞仅产生4.5x104个cd7+细胞(在淋巴门中计数的细胞的38.9%)。延长培养期间cd7+细胞数量减少,并且t细胞标志物cd3、cd4、cd8和tcrαβ未被显著表达。在通过早期t细胞祖细胞(etp)的过渡期间,cd4-cd8-(dn)至cd4+cd8+(dp)阶段通过精确活化和抑制特定转录因子来引导。在对照细胞中,pu.1的沉默以及gata3和bcl11b的诱导(图1c)表明,这些细胞进行t谱系定型(dn2至dn3),然后是tcr重排。相比之下,在jak3缺陷型细胞中pu.1积累,并且gata3和bcl11b水平降低(图1c)。这些数据表明人jak3缺陷型细胞在dn2阶段之前或在dn2阶段阻滞,该阶段与t细胞在jak3ko小鼠中死亡的阶段相似。有趣的是,人jak3缺陷型细胞可以表达足够的rag1、rag2和ptcra(图1c)来进行tcr重排,但细胞存活的时间长度不足以进行至此重要的发育阶段。这些在jak3缺陷型细胞的淋巴细胞发育中的严重缺陷可通过在淋巴样祖细胞存活和分化中起重要作用的il-7信号传导的缺乏来解释。il-7/jak3信号传导通过调节凋亡调节因子的bcl2家族来维持胸腺细胞稳态。来自jak3ko小鼠的胸腺细胞和外周t细胞部分通过选择性提高bax(促凋亡因子)以及通过降低抗凋亡因子bcl2的表达而具有高凋亡指数。类似地,在这些研究中,与td10(9%至2.2%)和td17(7%至1.9%)(图2a)中的对照相比,体外来源的人jak3缺陷型细胞的凋亡增加。与此表型一致,与对照相比,jak3缺陷型细胞中的bax水平提高并且bcl2水平降低(图2b)。bcl2的强制表达挽救了γc–缺陷小鼠中的t而非b或nk细胞的发育(kondo等,immunity7:155-162(1997))。对jak3ko小鼠移植表达bcl2的jak3ko骨髓细胞也增加了外周t细胞数(wen等,molecularandcellularbiology21:678-689(2001))。为了确定bcl2的过表达是否会挽救人jak3缺陷型细胞的t细胞发育缺陷,将体外来源的jak3缺陷型cd34+细胞用含有被ef1a启动子驱动的bcl2-2a-gfp多顺反子的慢病毒转导。转导后,将cd34+细胞铺在op9-dl4单层上,并在td28时测定nk和t细胞标志物。在gfp-(jak3-;bcl2低)或gfp+细胞(jak3-;bcl2+)中未发现cd3-cd16+cd56+nk细胞(图2c)。这些发现表明bcl2在jak3缺陷型细胞的dn阶段释放了阻断。有趣的是,在dp阶段具有明显的第二次发育阻滞;在gfp+细胞中未观察到cd8+cd4+dp阳性细胞的进一步分化(图2c)。概括地说,上述研究证明人scid表型可以在体外用患者来源的ipsc再现。jak3缺陷导致dn胸腺细胞的增殖性缺陷。bcl2的强制表达增强了dn细胞的存活,该细胞进一步分化成dp胸腺细胞。然而,dp胸腺细胞不能成熟为spt细胞,并且这种缺陷可能是由于缺乏il7/jak3信号传导所致。
通过crispr/cas9增强的基因替代来校正scidhipsc中的jak3缺陷
为了确定在jak3缺陷型scid患者细胞中可否恢复正常的t细胞发育,通过crispr/cas9增强的基因替代在ipsc中对jak3突变进行校正。外显子14上游和下游内含子中的6个指导rna被设计成靶向靠近c1837t突变的wtcas9或ncas9,并且使用校正模板进行基因替代(图3a)。用两种表达d10acas9切口酶的质粒和配对的指导rna或表达野生型cas9的单个质粒和单指导rna对ipsc进行核转染。在核转染后,使细胞在含有g418的培养基中生长2周。挑选单个集落、扩增并通过pcr进行基因分型(图3b顶部)。图3c中示出crispr/cas9介导的jak3基因校正的效率。通过sanger测序来进一步验证来自wtcas9+grna#1的三个克隆、来自wtcas9+grna#2的3个克隆和来自cas9切口酶+配对的grna#1和#2的6个克隆。在12个测序克隆中,鉴定了2个纯合子校正的克隆(来自cas9切口酶+配对的grna#1和#2的1个克隆,以及来自wtcas9+grna#1的1个克隆)和10个杂合子校正的克隆(图3d)。通过rt-pcr(jak3mrna)(图3b;左下图)和蛋白印迹(jak3蛋白)(图3b;右下)证明jak3基因表达的恢复。
crispr/cas9引导的jak3校正的特异性
脱靶crispr/cas9引导的基因组修饰的潜在性引起对在人类中使用这种疗法途径的担忧。在癌细胞系中,已经描述了由cas9-grna引起的相对高水平的脱靶诱变。为了确定crispr/cas9引导的jak3校正在人scidipsc中的特异性,在基因替代之前和之后进行全基因组测序。对两个杂合子校正的的克隆和一个纯合子校正的克隆的基因组进行测序。用grna#2+野生型cas9校正两个杂合子克隆,并用grna#1+grna#2+切口酶cas9(d10a)校正纯合子克隆。将20个碱基的crispr指导序列映射到人参考基因组,从而允许多达3个错配以鉴定可能的脱靶位点。然后分析这些位点在crispr/cas9引导的基因替代后的ipsc样品中的变化。对一个纯合子和两个杂合子校正的ipsc系的wgs分析证明没有突变(snv或插入缺失)被引入预测的脱靶位点,表明crispr/cas9引导的基因替代的强特异性。
在crispr/cas9引导的jak3校正后的t细胞发育的恢复
为了确定jak3基因校正后t细胞发育是否恢复,测定了t细胞谱系定型和成熟。t细胞分化依次通过体内观察到的中间体:cd34+cd7+t/nk定型阶段;cd7+cd4+cd8-未成熟、sp阶段;cd4+cd8+dp阶段;最后是cd3+cd8+tcrαβ成熟阶段。成熟t细胞是多克隆的,增殖并且响应于有丝分裂原分泌细胞因子。因此,jak3校正的hipsc在op9单层上分化成造血祖细胞,并且cd34+细胞在抗cd34磁珠上被正向选择。将这些细胞铺在op9-dl4单层上,并且在td14、21、28和35时分析非粘附细胞的淋巴细胞标志物(图4)。与对照细胞类似,在td14时,1-2×106个cd34+jak3校正的细胞分化成4.7x106个cd7+细胞(在淋巴门中计数的细胞的91%)。进一步分化至td21、td28和td35后,观察到大量的t细胞成熟标志物cd3、cd4、cd8和tcrαβ(图4a)。为了确定是否在jak3校正的t细胞中重建tcr重排,通过流式细胞术进行tcrvβ分型并在图4b中汇总。jak3校正的t细胞表达我们测试的所有vβ区段(25中的19个);因此,恢复了宽泛的tcr库。最后,通过测量抗cd3/cd28刺激后细胞表面活化标志物来检查tcr信号传导路径(jak3校正的t细胞中的t细胞功能的替代物)的完整性。在刺激后第3天,jak3校正的t细胞中cd3+cd25+cd69+t细胞的百分比从0.68%增加到59.7%,与在对照细胞中观察到的增加相似(0.01%到37.6%)(图4c)。上述这些数据和结果证明,在体外ipsc模型体系中通过crispr/cas9增强的基因替代校正jak3c1837t(p.r613x)突变,恢复了正常t细胞发育,使得能够产生具有宽泛tcr库的功能性成熟t细胞群。
在人中,scid患者外周血中的淋巴细胞的表型已被充分描述,但一直难以进行关于淋巴定型和胸腺细胞发育的关键步骤。从未经治疗的scid患者获取骨髓和胸腺细胞样品是具有挑战性的,因为这些情况是罕见的,并且婴儿通常存在危及生命的感染,需要紧急hsc移植才能存活。本文描述的用于研究人scid的策略避免了这些限制;大量的造血祖细胞可以由患者特异性ipsc在体外产生,并且可以精确地确定造成免疫缺陷的机制。本文证明了人jak3缺陷型scid中的t细胞发育在cd4-cd8-(dn2)阶段之前或在该阶段被完全阻断。有趣的是,bcl2的强制表达增强了dn细胞的存活,该细胞进一步分化成dp胸腺细胞。然而,dp胸腺细胞不能成熟为spt细胞,并且这种缺陷可能是由于缺乏il7/jak3信号传导所致。还证明了通过crispr/cas9增强的基因替代校正人jak3突变恢复了早期t细胞祖细胞的分化潜能。经校正的祖细胞能够产生表达宽泛tcr库的nk细胞和成熟t细胞群。对一个纯合子和两个杂合子校正的ipsc系的全基因组测序分析证明没有突变(snv或插入缺失)被引入预测的脱靶位点,表明crispr/cas9引导的基因替代的强特异性。
在本文所述的方法中,可以通过与人骨髓基质干(hmsc)细胞共培养,从hipsc产生cd34+hsc(参见图5)。通过此方法从基因校正/修饰后的患者特异性ipsc产生的hsc可以移植回患者,以治疗诸如镰状细胞病(scd)、scid或癌症的疾病。在本文所述的方法中,可以通过将hipsc来源的cd34+细胞与hmsc-dl4共培养来培养hipsc来源的cd34+细胞,从而产生t细胞(参见图6)。通过此方法从校正/修饰后的患者特异性ipsc产生的hsc可以移植回患者以治疗疾病。t细胞可包括γδt细胞。如图7所示,表达重组t细胞受体(tcr)的γδt细胞可有效地从基因修饰的ipsc产生。表达对肿瘤抗原具有特异性的tcr的γδt细胞的产生提供了用于癌症的细胞疗法。
实施例2
通过crispr/cas9增强的基因替代来校正与镰状细胞性贫血相关联的突变
载体构建
将具有n末端和c末端核定位序列(nls-cas9-nls)的人密码子优化的酿脓链球菌(s.pyogenes)cas9从px330载体(addgeneid:42230)pcr克隆到在n末端具有his6-sumo标签的经修饰的pet-28b(emdbiosciences)载体中。将含有短接头肽,然后是带有净电荷+36的超荷电gfp以及23氨基酸的流感病毒血凝素ha-2变体肽inf7(glfeaiegfiengwegmidgwyg)(seqidno:50)的基因块盒针对大肠杆菌进行密码子优化,然后合成(idtdna)并克隆以与nls-cas9-nls的c末端融合。还合成hiv-tat肽(ygrkkrrqrrrppq))(seqidno:51)编码序列(idtdna)并将其进行克隆以与nls-cas9-nls的n末端融合。
蛋白质过表达和纯化
将pet-sumo-sccas9质粒转化到lb培养基中的大肠杆菌菌株rosettatm2(de3)细胞(emdmillipore,billerica,ma)中。使细胞在37℃下生长,直至600nm时的光密度达到0.6。通过加入0.5mm异丙基-1-硫代-β-d-吡喃半乳糖苷(iptg)并在18℃下在振荡器中培养过夜来实现蛋白过表达的诱导。将收获的细胞重悬于ni-结合缓冲液(20mmtris-hclph8.0,1.5mnacl,25mm咪唑和0.2mmtcep)中并通过emulsiflexc3高压均化器(avestin)裂解。将终浓度为0.4%的聚乙烯亚胺(pei)加入澄清的裂解液中以沉淀核酸。然后通过硫酸铵使离心后上清液中的蛋白沉淀以去除pei,并且重新溶解于ni结合缓冲液中。将蛋白质首先通过histrap镍亲和柱(gehealthcare)纯化,然后在4℃下用sumo蛋白酶ulp1过夜消化。然后经由第二个histrap柱去除切割的his-sumo标签。将含有sccas9蛋白的流过物稀释以达到0.5m的最终nacl浓度,并通过用含有20mmtris-hclph8.0、2.0mnacl和0.2mmtcep的缓冲液进行梯度洗脱来在hitrap肝素柱(gehealthcare)上纯化。将洗脱的sccas9蛋白在凝胶过滤缓冲液(20mmtris-hclph8.0、0.5mnacl和0.2mmtcep)中通过尺寸排阻柱superdex20016/600(gehealthcare)来进行进一步纯化,经由通过0.22μm过滤器进行灭菌,并通过具有100kda截留值的amicon离心单元(emdmillipore)浓缩。将浓缩的蛋白质通过紫外分光光度计定量并在液氮中快速冷冻。
指导rna制备
使用引物组,添加t7启动子和polya序列,通过pcr来产生用于sgrna转录的模板dna。根据制造商的手册,使用t7ribomax表达系统(promega,madison,wi)通过t7rna聚合酶来在体外转录sgrna。按如下方式纯化转录的rna:通过酚:氯仿提取,乙醇沉淀,然后用megacleartm转录清除试剂盒(ambion,austin,tx)进行柱纯化。将纯化的grna通过紫外分光光度计定量并储存在-80℃冰箱中。
单链dna供体
单链dna(ssodn)供体由idtdna合成。
细胞培养
人镰状患者ipsc源自皮肤成纤维细胞,并维持在含有青霉素/链霉素的mtesrtm1培养基(stemcelltechnologies,vancouver,ca)中的基质胶(bd)上。
sccas9-sgrna-ssodn复合物的制备和核转染
将1/10体积的10xpbs加入sgrna中以达到1x终浓度。使sgrna在pcr热循环仪上退火,温度从95℃缓慢降低至4℃。退火后,将sccas9蛋白以1:1.5的蛋白与rna的摩尔比加入sgrna中,并通过轻敲管快速混合,直至所有瞬时沉淀消失。将混合物在黑暗中在室温下温育10分钟。然后,将1摩尔比量的ssodn加入混合物中并在黑暗中另外温育10分钟以形成sccas9-sgrna-ssodn复合物。
核转染前一天,将细胞通过accutase(stemcelltechnologies)分离,并将1×106个细胞/孔细胞接种在具有10μmrock抑制剂(y-27632)(emdmillipore)的6孔板上。对于每个实验,将5×105个hsipsc作为单细胞重悬于100μl补充的人干细胞核转染溶液1(lonza)中,然后将sccas9-sgrna-ssodn复合物与细胞溶液混合。使用nucleofectorii设备(lonza,basel,switzerland)通过程序a-023对细胞进行核转染。核转染两天后通过ddpcr来分析hbb基因组校正的效率。
通过ddpcr来检测镰状校正
按照制造商的手册,通过prepgem组织dna提取试剂(zygem,hamilton,nz)来裂解用sccas9-sgrna-ssodn复合物核转染的细胞,并用水进行1:3稀释。在22μlddpcr反应中,将11μl2×ddpcr混合物(bio-rad)与1μl各5μm下述等位基因特异性fam或victaqman探针、0.2μl各100μm正向引物和反向引物和8.6μl稀释的基因组dna混合。根据制造商的手册,通过qx200微滴生成器来(bio-rad,hercules,ca)产生微滴。然后将反应混合物转移到96孔pcr板中,并在标准热循环仪(bio-rad)上进行pcr。用于pcr的程序为:步骤1:95℃10分钟;步骤2:95℃30秒;步骤3:55℃1分钟;将步骤2-3重复39次;步骤4:98℃10分钟;步骤5:8℃,保持。pcr完成后,然后通过qx200微滴读取仪(bio-rad)对板进行分析。
taqman探针:
ddpcr引物:
如上所述,可以将包含指导rna(grna)、经修饰的重组cas9蛋白(mrcas9)和单链寡脱氧核糖核苷酸(ssodn)的复合物引入人干细胞或其衍生物中,以校正引起疾病的单碱基突变。表1和图8示出将镰状细胞校正复合物(grna-mrcas9-ssodn)引入源自镰状细胞患者的皮肤细胞的诱导性多能干细胞(ipsc)中的结果。如实施例1所述获得ipsc。通过核穿孔将校正复合物引入镰状ipsc中,2天后使用上述引物通过数字pcr来分析基因组dna,并进行测序。超过65%的细胞含有至少一个经校正的基因。一个经校正的基因足以治愈所述疾病。结果确认如下。在引入校正复合物两天后,将细胞铺在培养皿中,并分离出43个单独的ipsc集落。从这些集落中分离基因组dna,并对β-珠蛋白基因进行测序。百分之六十五的集落含有至少一个经校正的β-珠蛋白基因(s校正为a)。
表1
用患者原代骨髓cd34+细胞进行类似的研究。方案如下。通过irb批准的方案从镰状患者获得骨髓。在miltenyi抗cd34+珠粒(miltenyi,bergischgladbach,germany)上纯化cd34+细胞。用如上所述制备的复合物对细胞进行核穿孔。在核穿孔之后,在2周后挑选接种于methycult中的细胞以及bfu-e、cfu-e和cfu-gemm集落,并分析经校正的等位基因。表2和图9示出将镰状细胞校正复合物(grna-mrcas9-ssodn)引入患者原代骨髓cd34+细胞中的结果。在体外分化十二天之后,通过数字pcr(ddpcr)分析dna并测序。观察到大约等量的βa和βsmrna(见图9)。在核穿孔之后,立即将一些细胞在红系细胞分化培养基中培养最多十八天,并分析无核的红细胞的hba。来自校正的镰状患者cd34+细胞的体外分化的红细胞的等电聚焦(ief)凝胶示出hba(正常血红蛋白)与hbs(具有镰状细胞突变的血红蛋白)的比率为约1:3,其足以抑制镰状形成和治疗所述疾病(见图10)。
表2
实施例3
通过crispr/cas9增强的基因替代来校正与镰状细胞性贫血相关联的突变
ipsc具有产生所有细胞类型,包括hspc(人干/祖细胞)的潜能;因此,基于ipsc的基因疗法可为镰状细胞病提供治愈性疗法。校正镰状ipsc可提供无限数量的细胞以由这些细胞产生经校正的hspc,并且这些经校正的hspc可用于自体移植。重要的是,可在移植前对经校正的ipsc和源自它们的hspc进行充分表征并进行安全性评价。以下描述的是对来自镰状患者成纤维细胞的ipsc的crispr/cas9增强的基因校正。
细胞培养
人镰状ipsc
人镰状ipsc源自从uabkirklinclinic的同意的镰状患者获得的皮肤活组织检查的成纤维细胞。将细胞维持在含有青霉素/链霉素的mtesrtm1培养基(stemcelltechnologies)中的基质胶(bd)上。通过将集落与accutase(stemcelltechnologies)温育,每3-4天对人镰状ipsc进行传代,并将单细胞接种在含有10μmrock抑制剂(y-27632)(emdmillipore)的基质胶涂布的板上。一天之后,更换不含rock抑制剂的培养基。
人镰状骨髓cd34+细胞
在uab的成人镰状诊所抽取来自同意的镰状患者的骨髓。将cd34+细胞在抗cd34+珠粒上纯化,等分并储存在液氮中。
用于大肠杆菌过表达的cas9表达质粒
cas9wt
将具有n末端和c末端融合核定位序列(nls-cas9wt-nls)的酿脓链球菌)cas9wt编码序列从px330载体(addgeneid:42230)pcr克隆到在n末端具有his6-sumo标签的经修饰的pet-28b(emdbiosciences)载体中,从而产生psumo-cas9wt质粒。
tat-cas9wt-egfp
将包含短接头肽的合成基因块(idtdna)和egfp的编码区连接至nls-cas9wt-nls的c末端并克隆。还合成了用于hiv-tat肽(ygrkkrrqrrrppq)(seqidno:51)的编码序列,将其连接至nls-cas9wt-nls的n末端并克隆,从而产生psumo-tat-cas9wt-egfp质粒。
cas9wt-36gfp
包含带有+36的净正电荷的超荷电gfp的大肠杆菌密码子优化的编码序列(idtdna)(lawrence等,“superchargingproteinscanimpartunusualresilience,”j.am.chem.soc.129(33):10110(2007)))和短接头肽的合成基因块与nls-cas9wt-nls的c末端连接并克隆,从而产生psumo-cas9wt-36gfp质粒。
tat-cas9wt-36gfp
合成hiv-tat肽(ygrkkrrqrrrppq)(seqidno:51)的编码序列,将其与cas9wt-36gfp的c末端连接并克隆,从而产生psumo-tat-cas9wt-36gfp载体。
tat-cas9wt-36gfp-inf7
将含有短接头肽,然后是带有净电荷+36的超荷电gfp(lawrence,2007)以及23个氨基酸的流感病毒血凝素ha-2变体肽inf7(glfeaiegfiengwegmidgwyg)(seqidno:50)(plank,1994)的合成基因块(idtdna)针对大肠杆菌进行密码子优化,然后与nls-cas9wt-nls的c末端连接并克隆。还合成了hiv-tat肽(ygrkkrrqrrrppq)(seqidno:51)编码序列,将其与nls-cas9-nls的n末端连接并克隆,从而产生psumo-tat-cas9wt-36gfp-inf7质粒。
cas9wt-3xtat
将用短接头(ygrkkrrqrrrppqagggsggsygrkkrrqrrrppqagggsggsygrkkrrqrrrppqag)(seqidno:61)分离的hiv-tat肽的编码区的3个串联重复的编码序列针对大肠杆菌进行密码子优化,合成,与nls-cas9wt的c末端连接并克隆,从而产生psumo-cas9wt-3xtat质粒。
tat-cas9wt-3xtat
合成hiv-tat肽的编码序列(ygrkkrrqrrrppq)(seqidno:51),与nls-cas9wt-3xtat的n末端连接并克隆,从而产生psumo-tat-cas9wt-3xtat质粒。
蛋白质过表达和纯化
将cas9wt或工程改造的带正电荷的cas9(epccas9)表达质粒转化到lb培养基中的大肠杆菌菌株rosettatm2(de3)细胞(emdmillipore)中。使细胞在37℃下生长,直至600nm时的光密度达到0.6。通过加入0.5mm异丙基-1-硫代-β-d-吡喃半乳糖苷(iptg)并在18℃下在振荡培养箱中培养过夜来实现蛋白质过表达的诱导。将收获的细胞重悬于ni-结合缓冲液(20mmtris-hclph8.0,1.5mnacl,25mm咪唑和0.2mmtcep)中并用emulsiflexc3高压均化器(avestin)裂解。将聚乙烯亚胺(pei)添加至澄清的裂解液上清液中至终浓度为0.4%以沉淀核酸。然后通过硫酸铵使离心后的上清液沉淀以去除pei,并且将蛋白沉淀重新溶解于ni结合缓冲液中。将蛋白溶液首先通过histrap镍亲和柱(gehealthcare,atlanta,ga)纯化,然后在4℃下用sumo蛋白酶ulp1过夜消化。然后经由通过第二个histrap柱去除切割的his-sumo标签。将含有cas9蛋白的流过物稀释以达到0.5m的最终nacl浓度,并通过用含有20mmtris-hclph8.0、2.0mnacl和0.2mmtcep的缓冲液进行梯度洗脱来在hitrap肝素柱(gehealthcare)上纯化。将洗脱的cas9蛋白在凝胶过滤缓冲液(20mmtris-hclph8.0、0.5mnacl和0.2mmtcep)中通过尺寸排阻柱superdex20016/600(gehealthcare)来进行进一步纯化,经由通过0.22μm过滤器进行灭菌,并通过具有100kda截留值的amicon离心单元(emdmillipore)浓缩。将浓缩的蛋白通过紫外分光光度计定量,在液氮中快速冷冻并在-80℃储存。
单指导rna制备
使用引物,在5'末端添加t7启动子并且在3'末端添加polya序列,通过pcr来产生用于sgrna体外转录的dna模板。根据制造商的手册,使用t7ribomax表达试剂盒(promega),通过t7rna聚合酶在体外转录sgrna。然后按如下方式分离转录的rna:通过酚:氯仿提取,乙醇沉淀和用megacleartm转录清除试剂盒(ambion)进行柱纯化。将sgrna在无核酸酶的水中洗脱,并通过紫外分光光度计测量浓度。然后将储备sgrna等分并储存在-80℃冰箱中。
cas9rnp/ssodn组装
在与cas9蛋白复合之前,将10xpbs加入储备sgrna溶液中以达到1xpbs最终盐浓度。通过将温度从95℃缓慢降低至4℃,使sgrna在热循环仪上退火。为了形成cas9rnp,将储备cas9蛋白以1:1.5蛋白:rna的摩尔比加入退火的sgrna中,并通过快速轻敲管充分混合,直至所有瞬时沉淀消失。将混合物在黑暗中在室温下温育10分钟。随后,将ssodn以与cas9rnp1:1的摩尔比加入以用于核穿孔。
使用cas9rnp/ssodn对人镰状ipsc进行核穿孔
在核穿孔前一天,用accutase(stemcelltechnologies)分离人镰状ipsc,并在补充有10μmrock抑制剂(stemcelltechnologies)的mtesr1培养基中温育以获得单细胞悬液。将这种单细胞悬液以5×105个细胞/孔的密度接种到6孔板中。在核穿孔当天,如上所述用accutase制备5×105个人镰状ipsc细胞,并将其重悬于100μl人干细胞核转染溶液1(lonza)中,并将7.5μmcas9rnp/ssodn与细胞悬液在核穿孔杯池(cuvette)中混合。使用nucleofectorii(lonza)通过程序a-023对细胞进行核穿孔,并立即转移到预热的培养基中。在核穿孔后2天测定对细胞群的校正效率。
通过ddpcr来检测镰状校正
在核穿孔2至5天后,按照制造商的手册,通过prepgem组织dna提取试剂(zygem)裂解cas9rnp/ssodn核穿孔的细胞,并且将细胞裂解液用水进行1:3稀释。在22μlddpcr反应中,将11μl2×ddpcr混合物(bio-rad)与1μl各5μm等位基因特异性fam或victaqman探针、0.2μl各100μm正向引物和反向引物和8.6μl稀释的细胞裂解液混合。根据制造商的说明,通过qx200微滴生成器(bio-rad)产生微滴。然后将反应混合物转移到96孔pcr板中,并在标准热循环仪(bio-rad)上进行pcr。用于pcr的程序为:步骤1:95℃10分钟;步骤2:95℃30秒;步骤3:55℃1分钟;将步骤2-3重复39次;步骤4:98℃10分钟;步骤5:8℃,保持。pcr完成后,通过qx200微滴读取仪(bio-rad)对板进行分析。
在cas9rnp/ssodn核穿孔后产生单ipsc克隆
为了产生单ipsc克隆,在系列稀释至20、10和5个细胞/孔的密度后,将cas9rnp/ssodn核穿孔的镰状ipsc接种在bd基质凝胶涂布的96孔板中。在培养的第一个6天期间,每2天更换含10μm抑制剂的新鲜mtesr1培养基。在第6天后每天更换不含抑制剂的mtesr1培养基。接种后十至十二天,挑选单ipsc集落,通过sanger测序对细胞裂解液的基因组修饰进行分析。
人患者骨髓镰状cd34+细胞的活化和核穿孔
为了活化细胞周期,将冷冻的人镰状骨髓cd34+细胞解冻并重悬于补充有cc110细胞因子混合物(stemcelltechnology)的预热的stemspan培养基中。将细胞在含有5%co2的37℃培养箱中培养,每天部分地更换新鲜培养基,持续2天,然后进行核穿孔。在核穿孔当天,用1xpbs冲洗5×105个活cd34+细胞,并通过150g离心15分钟收获。将细胞沉淀重悬于100μlp4原代细胞核转染溶液(lonza)中,并将15μmcas9rnp/ssodn复合物与细胞悬液在核穿孔杯池中混合。使用4d-nucleofector(lonza)通过程序dn-100对细胞进行核穿孔,并立即转移到预热的培养基中。核穿孔后6天分析基因校正的效率。
对cas9rnp核穿孔的cd34+细胞的红系细胞集落形成单位(cfu)测定
在用cas9rnp/ssodn复合物进行核穿孔后,在35mm组织培养板中以500-1000个细胞/ml的密度将cd34+细胞接种到methocult培养基(stemcelltechnologies)中。使细胞在含有5%co2的37℃培养箱中生长12-15天,直至集落大到足以单独挑选用于分析。
cd34+hspc体外红系细胞分化成rbc
在用cas9rnp/ssodn复合物对cd34+细胞进行核穿孔后一天,将培养基更换成补充有1u/ml促红细胞生成素(epo)、2nm地塞米松(dex),1nmβ-雌二醇、20ng/ml人scf和5ng/ml人il-3的红系细胞扩增培养基(stemspansfem(stemcelltechnologies)。每2天更换一次培养基。在扩增和分化的前7天后,向培养基补充更高浓度的epo(2u/ml),直至在15-18天收获分化的rbc。
rbc中经校正的血红蛋白β蛋白的质谱分析
通过page分离从人镰状骨髓cd34+hspc分化的rbc的溶血产物。将珠蛋白带从凝胶中切下并用胰蛋白酶消化。通过lc-ms/ms分离并分析肽。
序列
体外转录的sgrna序列:
t1sgrna:
gggucugccguuacugcccugguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu(seqidno:62)
t2sgrna:
ggguaacggcagacuucuccacguuuuagagcuagaaauagcaaguuaaaauaaggcuaguccguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu(seqidno:63)
91-nt校正ssodn:
atccacgttcaccttgccccacagggcagtaacggcagacttctcctcaggagtcaggtgcaccatggtgtctgtttgaggttgctagtga(seqidno:52)
95-ntt2摆动ssodn:
cttcatccacgttcaccttgccccacagggcagtaacggcagatttttcctcaggagtcaggtgcaccatggtgtctgtttgaggttgctagtga(seqidno:53)
90-ntt1摆动ssodn:
acctcaaacagacaccatggtgcacctgactcctgaggagaagtctgccgtcacagctctgtgggcaaggtgaacgtggatgaagttgg(seqidno:64)
用于t2sgrna体外转录模板的pcr引物
t7-t2f:taatacgactcactatagggtaacggcagacttctccac(seqidno:54)t7-r:aaaaagcaccgactcggtgcc(seqidno:55)
用于t1sgrna体外转录模板的pcr引物
t7-t1f:taatacgactcactatagggtctgccgttactgccctg(seqidno:65)t7-r:aaaaagcaccgactcggtgcc(seqidno:55)
用于在靶sanger测序的pcr引物
r157:tccacatgcccagtttctat(seqidno:66)
r158:agtagcaatttgtactgatggtatg(seqidno:67)
工程改造的带正电荷的cas9rnp/ssodn(epccas9rnp/ssodn)有效地校正了人患者ipsc(诱导性多能干细胞)中的镰状突变
为了校正镰状hbb基因,使用cas9wt/t2rnp、cas9wt-egfp/t2或8种不同的epccas9/t2rnp(工程改造的带正电荷的cas9/t2rnp)以及91-nt的ssodn校正模板(seqidno:51)对人镰状患者来源的ipsc进行核穿孔。cas9/t2rnp诱导镰状突变附近(下游2bp)的双链断裂。切割位点与突变的接近利用91-ntssodn校正模板增强了镰状突变(t->a)的hdr。针对ipsc群的在靶sanger测序数据证明在cas9wtrnp/ssodn核穿孔细胞中以高效率校正了镰状突变(图11)。在cas9wt的c末端添加egfp(增强的绿色荧光蛋白)结构域不影响校正水平。
用8种不同的epccas9srnp进行核穿孔的细胞中的校正效率不同。在cas9wt-egfp(tat-cas9wt-egfp)的n末端添加带正电荷的hivtat肽导致与cas9wt和cas9wt-egfp相比校正效率的小幅降低以及插入缺失的少量减少。在cas9wt-egfp(3xtat-cas9wt-egfp)的n末端添加3x串联重复的tat几乎完全消除了校正和插入缺失水平,指示出由此修饰引起的cas9酶活性的丧失。此结果表明,连接至cas9的n末端的相对高数量的正电荷严重抑制酶活性。有趣的是,在cas9的c末端添加正电荷(cas9wt-3xtat或cas9wt-36gfp)导致高的校正水平和较低的插入缺失水平。这些结果表明,连接至cas9的c末端的正电荷显著抑制切割末端的外切核酸酶消化并刺激末端的再连接而不形成插入缺失。从epccas9观察到类似水平的校正和插入缺失,其中c末端添加3x串联重复的tat肽或带正电荷的+36gfp。
具有n末端和c末端带正电荷的修饰(tat-cas9wt-3xtat和tat-cas9wt-36gfp)的epccas9产生显著更少的插入缺失。有趣的是,与tat-cas9wt-36gfp相比,向tat-cas9wt-36gfp(tat-cas9wt-36gfp–inf7)的c末端进一步添加带负电荷的inf7肽显著提高了校正效率。在使用cas9wt和选择的epccas9rnp进行核核穿孔后,通过对ipsc群的在靶校正和插入缺失进行深度测序分析来验证sanger测序结果(图12)。
epccas9rnp抑制人镰状ipsc中的在靶插入缺失
为了进一步研究带正电荷的修饰对基于hdr的基因校正和基于nhej的插入效率的影响,使用cas9rnp+/-91-ntssodn校正模板对人镰状ipsc进行核穿孔。在靶sanger测序分析证明,向cas9wtrnp和tat-cas9wt-egfp中添加ssodn(+ssodn)以类似的高效率校正了镰状突变(图13)。然而,在不存在ssodn(-ssodn)的情况下,与cas9wt相比,tat-cas9wt-egfp的插入缺失形成显著较低。由于hdr需要dsb(双链断裂),cas9的酶活性显然不因加入1xtat而降低。因此,插入缺失形成的大的差异不是由于tat-cas9wt-egfp的低转导效率或低酶活性所致。
为了确认这些观察结果,评价了其他5种含有(+)或不含(-)ssodn的epccas9rnp的校正和插入缺失效率(图14)。sanger测序分析确认,在不存在ssodn(-ssodn)的情况下,所有epccas9rnp导致显著更少的在靶插入缺失。尽管epccas9rnps/+ssodn的校正效率随着不同的带正电荷的修饰而改变(图14),但所有带正电荷的cas9修饰均抑制插入缺失形成。
epccas9rnp增强人镰状ipsc中核穿孔后的细胞存活
为了确定带正电荷的修饰是否影响细胞存活,使用cas9wtrnp或7种含有(+)或不含(-)校正ssodn的不同epccas9对镰状ipsc进行核穿孔。在核穿孔后立即将细胞铺在培养皿中,48小时后检查生长情况。采用cas9wt的细胞存活率较差,并且随着带正电荷的修饰的增加,细胞存活率急剧增加(图15)。用含有n末端和c末端带正电荷的修饰的cas9wt-36gfp和epccas9(图15)获得优异的细胞存活。考虑到细胞存活和校正/插入缺失效率,用cas9wt-36gfp和tat-cas9-36gfp-inf7rnp在人镰状ipsc中实现了高校正、低插入缺失形成和优异细胞存活的最佳平衡。
针对人ipsc中镰状校正的ssodn:cas9rnp比率
ssodn校正模板与cas9rnp(ssodn:cas9rnp)的比率对于hdr和细胞存活至关重要。单链odn对细胞具有毒性;因此,高ssodn:cas9rnp比率可导致核穿孔后细胞存活较差。然而,低ssodn:cas9rnp比率低可导致低效hdr。为了以高细胞存活率获得高校正效率,对ssodn:cas9rnp比率进行了优化。确定了镰状患者ipsc中随ssodn剂量增加的镰状突变校正效率。对于这些实验,固定1:1.35的cas9wt-36gfp:t2sgrna摩尔比,并且ssodn:cas9wt-36gfprnp的摩尔比的范围为0至2.0(r=0、0.2、0.5、1.0、1.15、1.35、1.5和2.0)。例如,图6中的r=0.5值是0.5ssodn:1.0cas9wt-36gfp:1.35t2sgrna。在ssodn:cas9wt-36gfprnp核穿孔后48小时,通过微滴式数字pcr(ddpcr)(图16a)和sanger测序(图16b)来对镰状校正进行定量。将校正百分比(βa/βs等位基因×100)相对于r(ssodn:cas9wt-36gfprnp)进行绘图。校正效率随着r=0.2至r=1.0而提高,并在1.15(65.7%)达到平稳段。r增加到1.15以上不会显著提高校正效率以及显著抑制细胞存活。
针对人ipsc中镰状校正的cas9:sgrna比率
理论上,最佳的cas9:sgrna摩尔比为1:1。用sgrna对cas9蛋白进行饱和确保了最大的cas9酶活性并降低了游离cas9与其他小rna相互作用的可能性,该相互作用可能产生不可预测的脱靶基因组修饰。小rna对核酸酶敏感;因此,大于1:1的cas9:sgrna摩尔比对于饱和cas9而言可能是必需的。以1.15或1.35的ssodn摩尔比测试1:1.15、1:1.35和1:1.5的cas9-36gfp:sgrna摩尔比,以确定患者ipsc中镰状突变的最佳校正效率。sanger测序结果和细胞存活分析证明,以1:1:35:1.15的cas9-36gfp:sgrna:ssodn摩尔比实现了最佳的校正效率和细胞存活(图17)。
针对人ipsc中镰状校正的集落分析
使用摩尔比为1.0:1.35:1.0的tat-cas9wt-36gfp-inf7:t2sgrna:ssodn对人镰状ipsc进行核穿孔,以研究细胞群中的校正效率(图18)以及随后在单细胞水平下的校正效率(表3)。对于单细胞分析,在系列稀释后,将核穿孔的ipsc铺在96孔板中。两周后,挑选单ipsc集落,分离基因组dna,并进行sanger测序。对43个单ipsc集落进行了在靶修饰的分析。表3汇总了这些ipsc克隆的sanger测序结果。这43个集落中的28个包含至少一个经校正的等位基因(a/a、a/s或a/插入缺失);因此,65.1%的克隆包含至少一个经校正的等位基因。包含至少一个经校正的等位基因的ipsc将产生不具有镰状的红细胞。
表3.通过epccas9rnp/ssodn校正的ipsc集落的sanger测序结果汇总
还在这些ipsc克隆的等位基因水平下评估了基因组编辑事件。86个等位基因中的42个(48.8%)被校正,86个等位基因中的28个(32.6%)包含插入缺失,并且86个等位基因中的16个(18.6%)未被修饰。这种高基因组修饰率(81.4%的等位基因和93%的细胞)证明采用生化复合物的高效基因靶向是可能的。
用epccas9rnp和摆动ssodn校正人ipsc
经校正的dna的重新靶向对于基于hdr的基因校正中的crispr/cas体系而言是潜在的缺陷。与质粒或病毒递送相比,由于rnp的半衰期短,因此cas9rnp的重新靶向的风险低;然而,重新靶向难以完全避免。在此实例中,镰状突变位于t2sgrna靶向序列内,并且仅为2个来自pam的碱基对。用ssodn校正后,经校正的dna含有1个与sgrna靶序列错配的碱基。这种差异减少了但并没有消除重新靶向。防止重新靶向的一个策略是将摆动碱基变化引入校正模板中。这些碱基变化不会改变翻译的蛋白序列,但会改变pam序列处或附近的dna序列,使得经校正的dna不再是cas9rnp的靶标。基于此策略,用tat-cas9wt-36gfp-inf7/t1sgrna/t1wb-ssodn和tat-cas9wt-36gfp-inf7/t2sgrna/t2wb-ssodn对镰状ipsc进行核穿孔,以确定epccas9rnp是否能通过摆动ssodn以高效率校正镰状菌突变。
核穿孔的细胞群的sanger测序结果验证了两个核穿孔的细胞群中的镰状突变的校正(图19)。采用t2wb-ssodn的镰状校正效率(图19b)与不含摆动碱基的ssodn的校正效率(图18)类似。然而,采用t1sgrna和t1wbssodn的镰状校正效率低于t2wb-ssodn,这可能是由于sgrna靶向效率、从镰状突变到sgrna切割位点的距离以及摆动碱基数目的差异所致。因此,t2wb-ssodn是优选的ssodn。
epccas9校正的ipsc集落的全基因组测序分析
为了确定人镰状患者ipsc的epccas9rnp引导的校正的特异性,对未经校正的镰状ipsc进行全基因组测序(wgs),并用tat-cas9wt-36gfp-inf7rnp产生4个纯合子校正的克隆。在4个经校正的ipsc克隆中,用t2sgrna和不含摆动碱基的91-ntssodn校正了2个克隆(t2-cl1和t2-cl2);用t2sgrna和95-ntt2wbssodn校正了1个克隆(t1w);以及用t1sgrna和90-ntt1wbssodn校正了1个克隆(t1w)(表4)。这些wgs数据确认了4个纯合子校正的ipsc克隆中的镰状突变的纯合子校正和在靶插入缺失的不存在(图20a)。对与t1sgrna具有同源性的4720个潜在脱靶位点以及与t2sgrna具有同源性的1476个潜在脱靶位点(1-5个错配)的分析证明没有脱靶修饰(图20b)。此外,如chang等所述的全基因组序列数据的分析(cellreports12(10):1668-77(2015)证明在与sgrna具有或不具有同源性的序列中没有致病变体。用tat-cas9wt-36gfp-inf7rnp产生四个纯合子校正的克隆。
表4.epccas9校正的ipsc集落的全基因组测序分析
镰状患者骨髓cd34+hspc的基因校正
对来自镰状患者的原代cd34+hspc进行校正,然后进行自体移植是一种强效且简单的scd基因疗法途径。为了确定epccas9rnp是否还可校正骨髓祖细胞中的镰状突变,获得的cd34+hspc是从同意的镰状细胞患者的骨髓中获得。将镰状cd34+细胞在抗cd34珠粒上纯化,并且通过在具有特定细胞因子(scf、tpo和flt-3)的培养基中培养2天来使细胞周期活化。随后,用cas9wt、cas9-36gfp或tat-cas9-3xtat+t2sgrna和ssodn对细胞进行核穿孔。在核穿孔后6天通过sanger测序来确定镰状校正的效率(图21a)。用cas9wt获得最高的校正效率;然而,插入缺失频率高。尽管采用2种epccas9rnp的校正效率低于采用cas9wt的校正效率,但插入缺失的频率显著降低。
在mrna和蛋白质水平上验证了采用一种epccas9(cas9-36gfp)对镰状突变的校正(图21b-d)。将核穿孔的细胞在人红系细胞扩增培养基中扩增10天后,进行rt-pcr和sanger测序(图21b)。观察到大约等量的βa和βsmrna(峰基本上重叠)。还将细胞在含有促红细胞生成素(epo)的人红系细胞分化培养基中培养15-18天。裂解来自此培养物的红细胞(rbc),并在ief凝胶上溶解血红蛋白(图21c)。总血红蛋白中大约35%是hba(图21c),并且此结果通过质谱法来确认(图21d)。在体内,含有hba的rbc的存活时间是仅含有hbs的rbc的存活时间的5-10倍。因此,如果移植后骨髓中约30%的细胞被校正,则外周血中将实现60-70%的hba水平。
epccas9rnp提高镰状患者骨髓cd34+hspc中的校正/插入缺失比率
除了检查患者骨髓cd34+细胞群中镰状突变的校正之外,我们还分析了源自单cd34+祖细胞的集落。在用tat-cas9wt-36gfp-inf7进行核穿孔后,将cd34+细胞与半固体methocult培养基混合并铺在培养皿中。铺板两周后,分离源自单细胞的集落,提取dna并进行sanger测序。我们检查的集落是bfu-e(爆发集落形成单位-红系细胞)、cfu-e(集落形成单位-红系细胞)和cfu-gemm(集落形成单位-粒细胞、红细胞、单核细胞、巨核细胞)。图11示出典型的bfu-e和cfu-gemm集落(a)和获得的六种基因型的代表性sanger测序结果(b)。表5汇总了分别在cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp和ssodn的核穿孔后获得的95、96和96个集落(bfu-e、cfu-e和cfu-gemm)的sanger测序结果。采用cas9wt获得最高的校正效率(51.6%);然而,用cas9wt处理的细胞中的插入缺失缺失/插入缺失频率也非常高(40.0%)。这种插入缺失/插入缺失的水平可能导致β-地中海贫血,因为这些hsc将有效地竞争有限数量的骨髓微环境(niche),而来自这些hsc的红细胞不能合成hba。尽管用cas9wt-36gfprnp获得的校正效率较低(28.1%),但这种校正水平足以治愈如上所述的疾病,并且插入缺失的频率(8.3%)更加安全。对于tat-cas9wt-3xtatrnp,校正效率(32.3%)和插入缺失频率(14.6%)是中等的。cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp+ssodn的细胞核穿孔后的校正/插入缺失比率分别为1.29(51.6/40.0)、3.39(28.1/8.3)和2.21(32.3/14.6)。因此,校正/插入缺失比率为3.39的cas9wt-36gfp是我们优选的epccas9。
表5.在分别用cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp以及ssodn对人镰状患者骨髓cd34+hspc进行核穿孔后获得的95、96和96个集落(bfu-e、cfu-e和cfu-gemm)的sanger测序结果的汇总。
如上所述,cas9wt-36gfprnp/ssodn复合物(总cfu的28.1%;cfu-gemm的25%)的镰状校正效率足以治愈所述疾病。移植后骨髓中的这种校正水平将导致外周血中60-70%的rbc得到校正。另外,仅8.3%的集落是纯合的插入缺失(插入缺失/插入缺失);因此移植后不太可能导致地中海贫血。
epccas9rnp增强在镰状患者骨髓cd34+hspc中核穿孔后的细胞存活
图22c中的数据证明epccas9rnp增强了在镰状患者骨髓cd34+hspc中核穿孔后的细胞存活。将在镰状患者骨髓cd34+hspc的核穿孔后获得的红系细胞集落(bfu-e和cfu-e)的数量与cas9wt、cas9wt-36gfp和tat-cas9wt-3xtatrnp+ssodn进行比较。将采用cas9wtrpn/ssodn获得的集落数量归一化为1。采用cas9wt-36gfprnp/ssodn获得的集落数量是cas9wt对照的2.5倍,并且tat-cas9wt-3xtatrnp/ssodn是该对照的1.6倍。结论是,epc(工程改造正电荷)保护人骨髓祖细胞/干细胞免受单链寡聚脱氧核苷酸(ssodn)的毒性作用。
这些结果是显著的,因为cd34+hpsc的剂量对于移植后的骨髓重建是关键的。一般来说,将两百万个cd34+细胞/千克移植给人受体。低于此水平的细胞剂量导致长期重建不良。体重75kg的患者需要大约1.5亿个细胞的剂量。可从麻醉状态下的75kg的患者收获1升骨髓,并且可分离出大约2亿个cd34+细胞进行移植。如上所述,与cas9-36gfprnp/ssodn相比,用cas9wtrnp/ssodn对cd34+细胞进行核穿孔后获得少于2.5倍的细胞。因此,我们优选的校正复合物是cas9wt-36gfprnp/ssodn。
epccas9导致较高的基因组编辑特异性
为了评价epccas9rnp在核穿孔的cd34+细胞中进行基因组编辑的特异性,在5个潜在的脱靶基因组基因座处进行了深度测序分析。基于与sgrna的序列同源性,五个潜在的脱靶位点是由zhangmit服务器(http://crispr.mit.edu)预测的前5个位点。在cas9rnp/ssodn核穿孔的镰状患者cd34+细胞中,深度测序测出在ot5位点处有大约0.1%的脱靶插入缺失(表6)。相比之下,在cas9wt-36gfp或tat-cas9wt-3xtatrnp/ssodn核穿孔细胞中,未观察到脱靶修饰(图23)。
表6对5个潜在的脱靶基因组基因座进行深度测序分析以评价epccas9rnp在核穿孔的cd34+细胞中的编辑特异性
另外,在源自cas9wtrnp/ssodn核穿孔的镰状cd34+细胞的红系细胞集落中,观察到95个集落中有5个集落在靶向位点附近(上游或下游)含有非特异性修饰(图24)。这些非特异性修饰是似乎在预期的cas9rnp切割位点不被引发的随机基因替换或插入缺失。相比之下,源自epccas9rnp核穿孔细胞的96个集落中没有集落包含非特异性修饰。
实施例4
小鼠中镰状细胞突变的校正
图25示出在将用cas9rnp/ssodn核穿孔的镰状小鼠胎肝c-kit+细胞的初次移植以校正镰状细胞突变后6周对血液进行的等电聚焦(ief)凝胶分析。小鼠胎肝c-kit+细胞相当于人脐带血cd34+细胞。图26示出在移植到经辐照的c57bl6小鼠中后的12周对facs纯化的骨髓细胞的ddpcr分析。核穿孔和移植后12周,校正了大约50%的红系细胞(ter119+)和骨髓细胞(cd11b+和cd11b+/gr1+)。红系细胞和骨髓细胞存活相对较短;因此,这些细胞源自移植的hsc。b细胞和t细胞中的校正水平在12周(总共24周)的二次移植后应升高至大约50%。24周后,大多数(即使不是全部)的造血细胞将源自长期的hsc。图27示出在将用cas9rnp/ssodn核穿孔的细胞初次移植以校正镰状细胞突变后12周以及二次移植以治疗镰状细胞突变后6周对小鼠血液进行的ief凝胶分析。在将用cas9rnp/ssodn核穿孔的hsc进行移植以校正镰状细胞突变后,在小鼠体内中产生人hba。小鼠血红蛋白带将再在六个星期内消失。
序列
seqidno:1
taacggcagacttctccac
seqidno:2
gtaacggcagacttctccacgttttagagctagaaatagcaagttaaaataaggctagtccgttatcaacttgaaaaagtggcaccgagtcggtgcttttttt
seqidno:3
cas9超荷电gfp构建体
mdykdhdgdykdhdidykddddkmapkkkrkvgihgvpaadkkysigldigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaeatrlkrtarrrytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmrkpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirrdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdhivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesilpkrnsdkliarkkdwdpkkyggfdsptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasagelqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaehiihlftltnlgapaafkyfdttidrkrytstkevldatlihqsitglyetridlsqlggdkrpaatkkagqakkkkgsgsngssgsaskgerlfrgkvpilvelkgdvnghkfsvrgkgkgdatrgkltlkficttgklpvpwptlvttltygvqcfsrypkhmkrhdffksampkgyvqertisfkkdgkyktraevkfegrtlvnriklkgrdfkekgnilghklrynrnshkvyitadkrkngikakfkirhnvkdgsvqladhyqqntpigrgpvllprnhylstrsklskdpkekrdhmvllefvtaagikhgrderyk
seqidno:4
tat-cas9超荷电gfp构建体
ygrkkrrqrrrppqaggsmdykdhdgdykdhdidykddddkmapkkkrkvgihgvpaadkkysigldigtnsvgwavitdeykvpskkfkvlgntdrhsikknligallfdsgetaeatrlkrtarrrytrrknricylqeifsnemakvddsffhrleesflveedkkherhpifgnivdevayhekyptiyhlrkklvdstdkadlrliylalahmikfrghfliegdlnpdnsdvdklfiqlvqtynqlfeenpinasgvdakailsarrlsksrrlenliaqlpgekknglfgnlialslgltpnfksnfdlaedaklqlskdtydddldnllaqigdqyadlflaaknlsdaillsdilrvnteitkaplsasmikrydehhqdltllkalvrqqlpekykeiffdqskngyagyidggasqeefykfikpilekmdgteellvklnredllrkqrtfdngsiphqihlgelhailrrqedfypflkdnrekiekiltfripyyvgplargnsrfawmtrkseetitpwnfeevvdkgasaqsfiermtnfdknlpnekvlpkhsllyeyftvyneltkvkyvtegmrkpaflsgeqkkaivdllfktnrkvtvkqlkedyfkkiecfdsveisgvedrfnaslgtyhdllkiikdkdfldneenediledivltltlfedremieerlktyahlfddkvmkqlkrrrytgwgrlsrklingirdkqsgktildflksdgfanrnfmqlihddsltfkediqkaqvsgqgdslhehianlagspaikkgilqtvkvvdelvkvmgrhkpeniviemarenqttqkgqknsrermkrieegikelgsqilkehpventqlqneklylyylqngrdmyvdqeldinrlsdydvdhivpqsflkddsidnkvltrsdknrgksdnvpseevvkkmknywrqllnaklitqrkfdnltkaergglseldkagfikrqlvetrqitkhvaqildsrmntkydendklirevkvitlksklvsdfrkdfqfykvreinnyhhahdaylnavvgtalikkypklesefvygdykvydvrkmiakseqeigkatakyffysnimnffkteitlangeirkrplietngetgeivwdkgrdfatvrkvlsmpqvnivkktevqtggfskesilpkrnsdkliarkkdwdpkkyggfdsptvaysvlvvakvekgkskklksvkellgitimerssfeknpidfleakgykevkkdliiklpkyslfelengrkrmlasagelqkgnelalpskyvnflylashyeklkgspedneqkqlfveqhkhyldeiieqisefskrviladanldkvlsaynkhrdkpireqaeniihlftltnlgapaafkyfdttidrkrytstkevldatlihqsitglyetridlsqlggdkrpaatkkagqakkkkgsgsngssgsaskgerlfrgkvpilvelkgdvnghkfsvrgkgkgdatrgkltlkficttgklpvpwptlvttltygvqcfsrypkhmkrhdffksampkgyvqertisfkdgkyktraevkfegrtlvnriklkgrdfkekgnilghklrynfnshkvyitadkrkngikakfkirhnvkdgsvqladhyqqntpigrgpvllpmhylstrsklskdpkekrdhmvllefvtaagikhgrderykggsggsvdglfeaiegfiengwegmidgwyg
序列表
<110>uab研究基金会(uabresearchfoundation)
蒂姆•汤斯(townes,tim)
丁磊(ding,lei)
詹家伟(chang,chia-wei)
<120>用于基因组编辑的crispr/cas9复合物
<130>035979-1014678(177wo1)
<150>us62/181,138
<151>2015-06-17
<150>us62/266,316
<151>2015-12-11
<160>69
<170>siposequencelisting1.0
<210>1
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>1
taacggcagacttctccac19
<210>2
<211>103
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>2
gtaacggcagacttctccacgttttagagctagaaatagcaagttaaaataaggctagtc60
cgttatcaacttgaaaaagtggcaccgagtcggtgcttttttt103
<210>3
<211>1671
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>peptide
<222>()..()
<223>seqidno:4
<400>3
metasptyrlysasphisaspglyasptyrlysasphisaspileasp
151015
tyrlysaspaspaspasplysmetalaprolyslyslysarglysval
202530
glyilehisglyvalproalaalaasplyslystyrserileglyleu
354045
aspileglythrasnservalglytrpalavalilethraspglutyr
505560
lysvalproserlyslysphelysvalleuglyasnthrasparghis
65707580
serilelyslysasnleuileglyalaleuleupheaspserglyglu
859095
thralaglualathrargleulysargthralaargargargtyrthr
100105110
argarglysasnargilecystyrleuglngluilepheserasnglu
115120125
metalalysvalaspaspserphephehisargleuglugluserphe
130135140
leuvalglugluasplyslyshisgluarghisproilepheglyasn
145150155160
ilevalaspgluvalalatyrhisglulystyrprothriletyrhis
165170175
leuarglyslysleuvalaspserthrasplysalaaspleuargleu
180185190
iletyrleualaleualahismetilelyspheargglyhispheleu
195200205
ilegluglyaspleuasnproaspasnseraspvalasplysleuphe
210215220
ileglnleuvalglnthrtyrasnglnleupheglugluasnproile
225230235240
asnalaserglyvalaspalalysalaileleuseralaargleuser
245250255
lysserargargleugluasnleuilealaglnleuproglyglulys
260265270
lysasnglyleupheglyasnleuilealaleuserleuglyleuthr
275280285
proasnphelysserasnpheaspleualagluaspalalysleugln
290295300
leuserlysaspthrtyraspaspaspleuaspasnleuleualagln
305310315320
ileglyaspglntyralaaspleupheleualaalalysasnleuser
325330335
aspalaileleuleuseraspileleuargvalasnthrgluilethr
340345350
lysalaproleuseralasermetilelysargtyraspgluhishis
355360365
glnaspleuthrleuleulysalaleuvalargglnglnleuproglu
370375380
lystyrlysgluilephepheaspglnserlysasnglytyralagly
385390395400
tyrileaspglyglyalaserglnglugluphetyrlyspheilelys
405410415
proileleuglulysmetaspglythrglugluleuleuvallysleu
420425430
asnarggluaspleuleuarglysglnargthrpheaspasnglyser
435440445
ileprohisglnilehisleuglygluleuhisalaileleuargarg
450455460
glngluaspphetyrpropheleulysaspasnargglulysileglu
465470475480
lysileleuthrpheargileprotyrtyrvalglyproleualaarg
485490495
glyasnserargphealatrpmetthrarglysserglugluthrile
500505510
thrprotrpasnpheglugluvalvalasplysglyalaseralagln
515520525
serpheilegluargmetthrasnpheasplysasnleuproasnglu
530535540
lysvalleuprolyshisserleuleutyrglutyrphethrvaltyr
545550555560
asngluleuthrlysvallystyrvalthrgluglymetarglyspro
565570575
alapheleuserglygluglnlyslysalailevalaspleuleuphe
580585590
lysthrasnarglysvalthrvallysglnleulysgluasptyrphe
595600605
lyslysileglucyspheaspservalgluileserglyvalgluasp
610615620
argpheasnalaserleuglythrtyrhisaspleuleulysileile
625630635640
lysasplysasppheleuaspasnglugluasngluaspileleuglu
645650655
aspilevalleuthrleuthrleuphegluaspargglumetileglu
660665670
gluargleulysthrtyralahisleupheaspasplysvalmetlys
675680685
glnleulysargargargtyrthrglytrpglyargleuserarglys
690695700
leuileasnglyileargasplysglnserglylysthrileleuasp
705710715720
pheleulysseraspglyphealaasnargasnphemetglnleuile
725730735
hisaspaspserleuthrphelysgluaspileglnlysalaglnval
740745750
serglyglnglyaspserleuhisgluhisilealaasnleualagly
755760765
serproalailelyslysglyileleuglnthrvallysvalvalasp
770775780
gluleuvallysvalmetglyarghislysprogluasnilevalile
785790795800
glumetalaarggluasnglnthrthrglnlysglyglnlysasnser
805810815
arggluargmetlysargileglugluglyilelysgluleuglyser
820825830
glnileleulysgluhisprovalgluasnthrglnleuglnasnglu
835840845
lysleutyrleutyrtyrleuglnasnglyargaspmettyrvalasp
850855860
glngluleuaspileasnargleuserasptyraspvalasphisile
865870875880
valproglnserpheleulysaspaspserileaspasnlysvalleu
885890895
thrargserasplysasnargglylysseraspasnvalproserglu
900905910
gluvalvallyslysmetlysasntyrtrpargglnleuleuasnala
915920925
lysleuilethrglnarglyspheaspasnleuthrlysalagluarg
930935940
glyglyleusergluleuasplysalaglypheilelysargglnleu
945950955960
valgluthrargglnilethrlyshisvalalaglnileleuaspser
965970975
argmetasnthrlystyraspgluasnasplysleuilearggluval
980985990
lysvalilethrleulysserlysleuvalseraspphearglysasp
99510001005
pheglnphetyrlysvalarggluileasnasntyrhishisalahis
101010151020
aspalatyrleuasnalavalvalglythralaleuilelyslystyr
1025103010351040
prolysleuglusergluphevaltyrglyasptyrlysvaltyrasp
104510501055
valarglysmetilealalyssergluglngluileglylysalathr
106010651070
alalystyrphephetyrserasnilemetasnphephelysthrglu
107510801085
ilethrleualaasnglygluilearglysargproleuilegluthr
109010951100
asnglygluthrglygluilevaltrpasplysglyargasppheala
1105111011151120
thrvalarglysvalleusermetproglnvalasnilevallyslys
112511301135
thrgluvalglnthrglyglypheserlysgluserileleuprolys
114011451150
argasnserasplysleuilealaarglyslysasptrpaspprolys
115511601165
lystyrglyglypheaspserprothrvalalatyrservalleuval
117011751180
valalalysvalglulysglylysserlyslysleulysservallys
1185119011951200
gluleuleuglyilethrilemetgluargserserpheglulysasn
120512101215
proileasppheleuglualalysglytyrlysgluvallyslysasp
122012251230
leuileilelysleuprolystyrserleuphegluleugluasngly
123512401245
arglysargmetleualaseralaglygluleuglnlysglyasnglu
125012551260
leualaleuproserlystyrvalasnpheleutyrleualaserhis
1265127012751280
tyrglulysleulysglyserprogluaspasngluglnlysglnleu
128512901295
phevalgluglnhislyshistyrleuaspgluileilegluglnile
130013051310
serglupheserlysargvalileleualaaspalaasnleuasplys
131513201325
valleuseralatyrasnlyshisargasplysproileargglugln
133013351340
alagluasnileilehisleuphethrleuthrasnleuglyalapro
1345135013551360
alaalaphelystyrpheaspthrthrileasparglysargtyrthr
136513701375
serthrlysgluvalleuaspalathrleuilehisglnserilethr
138013851390
glyleutyrgluthrargileaspleuserglnleuglyglyasplys
139514001405
argproalaalathrlyslysalaglyglnalalyslyslyslysgly
141014151420
serglyserasnglyserserglyseralaserlysglygluargleu
1425143014351440
pheargglylysvalproileleuvalgluleulysglyaspvalasn
144514501455
glyhislyspheservalargglylysglylysglyaspalathrarg
146014651470
glylysleuthrleulyspheilecysthrthrglylysleuproval
147514801485
protrpprothrleuvalthrthrleuthrtyrglyvalglncysphe
149014951500
serargtyrprolyshismetlysarghisaspphephelysserala
1505151015151520
metprolysglytyrvalglngluargthrileserphelyslysasp
152515301535
glylystyrlysthrargalagluvallysphegluglyargthrleu
154015451550
valasnargilelysleulysglyargaspphelysglulysglyasn
155515601565
ileleuglyhislysleuargtyrasnpheasnserhislysvaltyr
157015751580
ilethralaasplysarglysasnglyilelysalalysphelysile
1585159015951600
arghisasnvallysaspglyservalglnleualaasphistyrgln
160516101615
glnasnthrproileglyargglyprovalleuleuproargasnhis
162016251630
tyrleuserthrargserlysleuserlysaspprolysglulysarg
163516401645
asphismetvalleuleugluphevalthralaalaglyilelyshis
165016551660
glyargaspgluargtyrlys
16651670
<210>4
<211>1720
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>peptide
<222>()..()
<223>合成构建体
<400>4
tyrglyarglyslysargargglnargargargproproglnalagly
151015
glysermetasptyrlysasphisaspglyasptyrlysasphisasp
202530
ileasptyrlysaspaspaspasplysmetalaprolyslyslysarg
354045
lysvalglyilehisglyvalproalaalaasplyslystyrserile
505560
glyleuaspileglythrasnservalglytrpalavalilethrasp
65707580
glutyrlysvalproserlyslysphelysvalleuglyasnthrasp
859095
arghisserilelyslysasnleuileglyalaleuleupheaspser
100105110
glygluthralaglualathrargleulysargthralaargargarg
115120125
tyrthrargarglysasnargilecystyrleuglngluilepheser
130135140
asnglumetalalysvalaspaspserphephehisargleugluglu
145150155160
serpheleuvalglugluasplyslyshisgluarghisproilephe
165170175
glyasnilevalaspgluvalalatyrhisglulystyrprothrile
180185190
tyrhisleuarglyslysleuvalaspserthrasplysalaaspleu
195200205
argleuiletyrleualaleualahismetilelyspheargglyhis
210215220
pheleuilegluglyaspleuasnproaspasnseraspvalasplys
225230235240
leupheileglnleuvalglnthrtyrasnglnleupheglugluasn
245250255
proileasnalaserglyvalaspalalysalaileleuseralaarg
260265270
leuserlysserargargleugluasnleuilealaglnleuprogly
275280285
glulyslysasnglyleupheglyasnleuilealaleuserleugly
290295300
leuthrproasnphelysserasnpheaspleualagluaspalalys
305310315320
leuglnleuserlysaspthrtyraspaspaspleuaspasnleuleu
325330335
alaglnileglyaspglntyralaaspleupheleualaalalysasn
340345350
leuseraspalaileleuleuseraspileleuargvalasnthrglu
355360365
ilethrlysalaproleuseralasermetilelysargtyraspglu
370375380
hishisglnaspleuthrleuleulysalaleuvalargglnglnleu
385390395400
proglulystyrlysgluilephepheaspglnserlysasnglytyr
405410415
alaglytyrileaspglyglyalaserglnglugluphetyrlysphe
420425430
ilelysproileleuglulysmetaspglythrglugluleuleuval
435440445
lysleuasnarggluaspleuleuarglysglnargthrpheaspasn
450455460
glyserileprohisglnilehisleuglygluleuhisalaileleu
465470475480
argargglngluaspphetyrpropheleulysaspasnargglulys
485490495
ileglulysileleuthrpheargileprotyrtyrvalglyproleu
500505510
alaargglyasnserargphealatrpmetthrarglyssergluglu
515520525
thrilethrprotrpasnpheglugluvalvalasplysglyalaser
530535540
alaglnserpheilegluargmetthrasnpheasplysasnleupro
545550555560
asnglulysvalleuprolyshisserleuleutyrglutyrphethr
565570575
valtyrasngluleuthrlysvallystyrvalthrgluglymetarg
580585590
lysproalapheleuserglygluglnlyslysalailevalaspleu
595600605
leuphelysthrasnarglysvalthrvallysglnleulysgluasp
610615620
tyrphelyslysileglucyspheaspservalgluileserglyval
625630635640
gluaspargpheasnalaserleuglythrtyrhisaspleuleulys
645650655
ileilelysasplysasppheleuaspasnglugluasngluaspile
660665670
leugluaspilevalleuthrleuthrleuphegluaspargglumet
675680685
ileglugluargleulysthrtyralahisleupheaspasplysval
690695700
metlysglnleulysargargargtyrthrglytrpglyargleuser
705710715720
arglysleuileasnglyileargasplysglnserglylysthrile
725730735
leuasppheleulysseraspglyphealaasnargasnphemetgln
740745750
leuilehisaspaspserleuthrphelysgluaspileglnlysala
755760765
glnvalserglyglnglyaspserleuhisgluhisilealaasnleu
770775780
alaglyserproalailelyslysglyileleuglnthrvallysval
785790795800
valaspgluleuvallysvalmetglyarghislysprogluasnile
805810815
valileglumetalaarggluasnglnthrthrglnlysglyglnlys
820825830
asnserarggluargmetlysargileglugluglyilelysgluleu
835840845
glyserglnileleulysgluhisprovalgluasnthrglnleugln
850855860
asnglulysleutyrleutyrtyrleuglnasnglyargaspmettyr
865870875880
valaspglngluleuaspileasnargleuserasptyraspvalasp
885890895
hisilevalproglnserpheleulysaspaspserileaspasnlys
900905910
valleuthrargserasplysasnargglylysseraspasnvalpro
915920925
serglugluvalvallyslysmetlysasntyrtrpargglnleuleu
930935940
asnalalysleuilethrglnarglyspheaspasnleuthrlysala
945950955960
gluargglyglyleusergluleuasplysalaglypheilelysarg
965970975
glnleuvalgluthrargglnilethrlyshisvalalaglnileleu
980985990
aspserargmetasnthrlystyraspgluasnasplysleuilearg
99510001005
gluvallysvalilethrleulysserlysleuvalseraspphearg
101010151020
lysasppheglnphetyrlysvalarggluileasnasntyrhishis
1025103010351040
alahisaspalatyrleuasnalavalvalglythralaleuilelys
104510501055
lystyrprolysleuglusergluphevaltyrglyasptyrlysval
106010651070
tyraspvalarglysmetilealalyssergluglngluileglylys
107510801085
alathralalystyrphephetyrserasnilemetasnphephelys
109010951100
thrgluilethrleualaasnglygluilearglysargproleuile
1105111011151120
gluthrasnglygluthrglygluilevaltrpasplysglyargasp
112511301135
phealathrvalarglysvalleusermetproglnvalasnileval
114011451150
lyslysthrgluvalglnthrglyglypheserlysgluserileleu
115511601165
prolysargasnserasplysleuilealaarglyslysasptrpasp
117011751180
prolyslystyrglyglypheaspserprothrvalalatyrserval
1185119011951200
leuvalvalalalysvalglulysglylysserlyslysleulysser
120512101215
vallysgluleuleuglyilethrilemetgluargserserpheglu
122012251230
lysasnproileasppheleuglualalysglytyrlysgluvallys
123512401245
lysaspleuileilelysleuprolystyrserleuphegluleuglu
125012551260
asnglyarglysargmetleualaseralaglygluleuglnlysgly
1265127012751280
asngluleualaleuproserlystyrvalasnpheleutyrleuala
128512901295
serhistyrglulysleulysglyserprogluaspasngluglnlys
130013051310
glnleuphevalgluglnhislyshistyrleuaspgluileileglu
131513201325
glnileserglupheserlysargvalileleualaaspalaasnleu
133013351340
asplysvalleuseralatyrasnlyshisargasplysproilearg
1345135013551360
gluglnalagluasnileilehisleuphethrleuthrasnleugly
136513701375
alaproalaalaphelystyrpheaspthrthrileasparglysarg
138013851390
tyrthrserthrlysgluvalleuaspalathrleuilehisglnser
139514001405
ilethrglyleutyrgluthrargileaspleuserglnleuglygly
141014151420
asplysargproalaalathrlyslysalaglyglnalalyslyslys
1425143014351440
lysglyserglyserasnglyserserglyseralaserlysglyglu
144514501455
argleupheargglylysvalproileleuvalgluleulysglyasp
146014651470
valasnglyhislyspheservalargglylysglylysglyaspala
147514801485
thrargglylysleuthrleulyspheilecysthrthrglylysleu
149014951500
provalprotrpprothrleuvalthrthrleuthrtyrglyvalgln
1505151015151520
cyspheserargtyrprolyshismetlysarghisaspphephelys
152515301535
seralametprolysglytyrvalglngluargthrileserphelys
154015451550
lysaspglylystyrlysthrargalagluvallysphegluglyarg
155515601565
thrleuvalasnargilelysleulysglyargaspphelysglulys
157015751580
glyasnileleuglyhislysleuargtyrasnpheasnserhislys
1585159015951600
valtyrilethralaasplysarglysasnglyilelysalalysphe
160516101615
lysilearghisasnvallysaspglyservalglnleualaasphis
162016251630
tyrglnglnasnthrproileglyargglyprovalleuleuproarg
163516401645
asnhistyrleuserthrargserlysleuserlysaspprolysglu
165016551660
lysargasphismetvalleuleugluphevalthralaalaglyile
1665167016751680
lyshisglyargaspgluargtyrlysglyglyserglyglyserval
168516901695
aspglyleupheglualailegluglypheilegluasnglytrpglu
170017051710
glymetileaspglytrptyrgly
17151720
<210>5
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>5
gctaattcactcccaaagaagacaag26
<210>6
<211>20
<212>dna
<213>合成构建体(syntheticconstruct)
<400>6
cttcagcaagccgagtcctg20
<210>7
<211>60
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>7
gtcgacgtcgacgctcagtgaagctgaagtattccttctgcttcacagggcgaccactac60
<210>8
<211>49
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>8
atttaaatcctcccctcgaacccttaccaaactcctatgcatactacag49
<210>9
<211>60
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>9
ttaattaattaattagcattttaggttcaggttgtgagaacactagaagagaacaagtca60
<210>10
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>10
gtatacgtatacgcatacctggagaggggacaaggtcttgagatgcgagggt52
<210>11
<211>52
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>11
agccaccttaattaagccaccatggcgcacgctgggagaacggggtacgata52
<210>12
<211>60
<212>dna
<213>合成构建体(syntheticconstruct)
<400>12
taacagagagaagttcgtggctccggatcccttgtggcccagataggcacccagggtgat60
<210>13
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>13
caccgtgagatacagatacagaca24
<210>14
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>14
aaactgtctgtatctgtatctcac24
<210>15
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>15
aaacggcattccaggcaaatcattc25
<210>16
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>16
caccgcagcctaggcaaaggcctgc25
<210>17
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>17
aaacgcaggcctttgcctaggctgc25
<210>18
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>18
caccgtgccaacagaactgcctgat25
<210>19
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>19
aaacatcaggcagttctgttggcac25
<210>20
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>20
caccgaccagggtgcaagtgtgga24
<210>21
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>21
aaactccacacttgcaccctggtc24
<210>22
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>22
caccgctcctcagcctggcattca24
<210>23
<211>24
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>23
aaactgaatgccaggctgaggagc24
<210>24
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>24
tgctaaagcgcatgctccagact23
<210>25
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>25
gtcttcatctcagggtcggct21
<210>26
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>26
cctctctgtgcattatggcag21
<210>27
<211>19
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>27
gccttctatcgccttcttg19
<210>28
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>28
actcctccacctttgacgct20
<210>29
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>29
tcccctcttcaagggtctacatg23
<210>30
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>30
gtgcaaaatggaagggtttc20
<210>31
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>31
ggagctccgtgaagttgttc20
<210>32
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>32
tgtttcctttcactggccaca21
<210>33
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>33
aacggcaactggtgaacggta21
<210>34
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>34
ggcgatgccagaatagatgccg22
<210>35
<211>29
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>35
ccaggccacttggctcctctatctccaga29
<210>36
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>36
ccttactgttgagactgcaatatcc25
<210>37
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>37
ctgaagtcccagtatatacttcacac26
<210>38
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>38
cccagaagcagtaataatcatcgag25
<210>39
<211>23
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>39
atgtgggatgtagtagatcttgc23
<210>40
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>40
gggtcttacctcagcagttac21
<210>41
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>41
cctcacacagtgtgacgcag20
<210>42
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>42
gactgagtacctgaaccggc20
<210>43
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>43
gggccaaactgagcagagtc20
<210>44
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>44
aagaccagggtggttgggac20
<210>45
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>45
gtaagaaaaatgcccacgtc20
<210>46
<211>20
<212>dna
<213>人工序列(artificialsequence)
<400>46
agtcagacgtctggagcttc20
<210>47
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>47
gtgagcagtgaaggcatgagtc22
<210>48
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>48
gtgagatacagatacagaca20
<210>49
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>49
aatgatttgcctggaatgcc20
<210>50
<211>23
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>peptide
<222>()..()
<223>合成构建体
<400>50
glyleupheglualailegluglypheilegluasnglytrpglugly
151015
metileaspglytrptyrgly
20
<210>51
<211>14
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>peptide
<222>()..()
<223>合成构建体
<400>51
tyrglyarglyslysargargglnargargargproprogln
1510
<210>52
<211>91
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>52
atccacgttcaccttgccccacagggcagtaacggcagacttctcctcaggagtcaggtg60
caccatggtgtctgtttgaggttgctagtga91
<210>53
<211>95
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>53
cttcatccacgttcaccttgccccacagggcagtaacggcagatttttcctcaggagtca60
ggtgcaccatggtgtctgtttgaggttgctagtga95
<210>54
<211>39
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>54
taatacgactcactatagggtaacggcagacttctccac39
<210>55
<211>21
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>55
aaaaagcaccgactcggtgcc21
<210>56
<211>14
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>56
tcctgaggaaaaat14
<210>57
<211>16
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>57
tgactcctgaggagaa16
<210>58
<211>15
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>58
actcctgtggagaag15
<210>59
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>59
cagagccatctattgcttacatttg25
<210>60
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>60
ggcctcaccaccaacttcat20
<210>61
<211>60
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>peptide
<222>()..()
<223>合成构建体
<400>61
tyrglyarglyslysargargglnargargargproproglnalagly
151015
glyglyserglyglysertyrglyarglyslysargargglnargarg
202530
argproproglnalaglyglyglyserglyglysertyrglyarglys
354045
lysargargglnargargargproproglnalagly
505560
<210>62
<211>104
<212>rna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>62
gggucugccguuacugcccugguuuuagagcuagaaauagcaaguuaaaauaaggcuagu60
ccguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu104
<210>63
<211>105
<212>rna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>63
ggguaacggcagacuucuccacguuuuagagcuagaaauagcaaguuaaaauaaggcuag60
uccguuaucaacuugaaaaaguggcaccgagucggugcuuuuuuu105
<210>64
<211>90
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>64
acctcaaacagacaccatggtgcacctgactcctgaggagaagtctgccgtcacagctct60
gtggggcaaggtgaacgtggatgaagttgg90
<210>65
<211>38
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>65
taatacgactcactatagggtctgccgttactgccctg38
<210>66
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>66
tccacatgcccagtttctat20
<210>67
<211>25
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>67
agtagcaatttgtactgatggtatg25
<210>68
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>68
ggtctgccgttactgccctg20
<210>69
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>gene
<222>()..()
<223>合成构建体
<400>69
gtaacggcagacttctccac20
- 还没有人留言评论。精彩留言会获得点赞!