一种鉴定菊花耐涝性的dCAPS标记开发及应用的制作方法
本发明属于生物技术领域,涉及一种鉴定菊花耐涝性的dcaps标记开发方法及应用,可用于菊花耐涝性分子标记辅助育种。
背景技术:
涝渍是危害世界菊花生产的严重自然灾害,培育耐涝性菊花新品种是解决这一问题的直接有效方法。因此,开发与菊花耐涝性相关的特异分子标记具有重要意义。目前,rapd、aflp、srap、issr、ssr等传统的分子标记已经成功应用在菊花种质资源鉴定、遗传多样性分析、连锁图谱构建和关联分析中[1-5]。随着现代分子生物学的发展及基因组测序计划的进行,snp标记应运而生。snp(singienucieotidepoiymorphism),即单核苷酸多态性,是指基因组水平上由单核苷酸的变异所引起的dna序列多态性,主要包括单个碱基的插入、缺失、转换和颠倒等。snp在基因组中分布广泛、数量多,遗传稳定性高、遗传分析重现性好且准确性高,易于快速且高通量地进行基因型分型[6]。基于以上优点,snp已经被广泛地应用于多个物种上的全基因组关联分析(genomewideassociationstudy,gwas)中[7-9]。在菊花上,chong等人[10]对199份菊花资源利用简化基因组测序slaf(specific-locusamplifiedfragmentsequencing)技术,检测到>46万个snp,并据此分析了菊花的进化关系。
酶切扩增多态性序列(cleavedamplifiedpolymorphicsequence,caps)标记是利用特异引物pcr与限制性酶切相结合而产生的一种检测snp位点的dna标记[11]。其原理是snp突变位点位于某种内切酶的识别序列上,经过特异引物pcr扩增后,用相应内切酶对pcr产物进行酶切,从而产生酶切片段的多态性,这样通过琼脂糖或聚丙烯酰胺凝胶电泳、染色即可显示。但是snp恰好位于某种限制性酶切位点的这种情况还比较少,于是michaels和amasino[12]及neff[13]在caps标记基础上,开发了人工引入错配碱基的衍生酶切扩增多态性标记(derivedcleavedamplifiedpolymorphicsequence,dcaps),从而几乎能够将所有snp位点转换成可以电泳检测的标记。无论是caps还是dcaps,均具有共显性、位点特异性、操作简单、检测快速、成本低廉且不依赖于精密仪器设备等特点,可用于用于植物基因分型、定位、遗传多样性分析、品种鉴定等方面。
菊花属于高度杂合的异源多倍体,复杂的遗传背景限制了其基因组研究的发展。目前应用在菊花上的分子标记大多为显性标记,且较难直接应用在菊花育种工作中。此前,本发明人已经公开了一种采用盆栽模拟淹水法模拟菊花涝害处理以及利用隶属函数法评价菊花耐涝性的专利(106446596a)[14];此外本发明人在2016年公开了199份菊花资源的snp标记[11],本专利在以上基础上,首先利用gwas检测到与菊花耐涝性显著关联的snp,据此成功开发了一个共显性dcaps标记,并在具有极端耐涝性差异的两个群体进行验证,结果表明该标记可以用于指导菊花耐涝性分子标记辅助育种。
[1]zhangf,chens,chenf,fangw,lif.apreliminarygeneticlinkagemapofchrysanthemum(chrysanthemummorifolium)cultivarsusingrapd,issrandaflpmarkers.scientiahorticulturae,2010,125(3):422-428
[2]zhangf,chens,chenf,fangw,cheny,lif.srap-basedmappingandqtldetectionforinflorescence-relatedtraitsinchrysanthemum(dendranthemamorifolium).molecularbreeding,2011,27(1):11-23
[3]wangc,zhangf,guanz,chens,jiangj,fangw,chenf.inheritanceandmolecularmarkersforaphid(macrosiphoniellasanbourni)resistanceinchrysanthemum(chrysanthemummorifoliumramat.).scientiahorticulturae,2014,180:220-226
[4]pengh,zhangf,jiangj,chens,fangw,guanz,chenf.identificationofquantitativetraitlociforbranchingtraitsofspraycutchrysanthemum.euphytica,2015,202(3):385-392
[5]lip,zhangf,chens,jiangj,wangh,suj,fangw,guanz,chenf.geneticdiversity,populationstructureandassociationanalysisincutchrysanthemum(chrysanthemummorifoliumramat.).molgenetgenomics,2016,291(3):1117–1125
[6]唐立群,肖层林,王伟平.snp分子标记的研究及其应用进展[j].中国农学通报,2012,28(12):154-158
[7]陈广凤,陈建省,田纪春.小麦株高相关性状与snp标记全基因组关联分析[j].作物学报,2015,41(10):1500-1509
[8]nimmakayalap,abburivl,saminathant,almeidaa,davenportb,davidsonj,c.v.chandramr,geraldh,andrease,doilc,stommelj,umeshkr.genome-widedivergenceandlinkagedisequilibriumanalysesforcapsicumbaccatumrevealedbygenome-anchoredsinglenucleotidepolymorphisms.
frontiersinplantscience,2016,7.doi:10.3389/fpls.2016.01646
[9]zhouq,zhouc,zhengw,masonas,fans,wuc,fud,huangy.genome-widesnpmarkersbasedonslaf-sequncoverbreedingtracesinrapeseed(brassicanapusl.).frontiersinplantscience,2017,8.doi:10.3389/fpls.2017.00648
[10]chongx,zhangf,wuy,yangx,zhaon,wangh,guanz,fangw,chenf.asnp-enabledassessmentofgeneticdiversity,evolutionaryrelationshipsandtheidentificationofcandidategenesinchrysanthemum.genomebiologyandevolution,2016,8(12):3661-3671
[11]leega,kohhj,chunghk,dixita,chungjw,makh,leesy,leejr,leegs,gwagjg.developmentofsnp-basedcapsanddcapsmarkersineightdifferentgenesinvolvedinstarchbiosynthesisinrice.molecularbreeding,2009,24(1):93–101.
[12]michaelssd,amasinorm.arobustmethodfordetectingsingle-nucleotidechangesaspolymorphicmarkersbypcr.plantjournal,1998,14:381–385
[13]neffmm,neffjd,choryj,pepperae.dcaps,asimpletechniqueforthegeneticanalysisofsinglenucleotidepolymorphisms:experimentalapplicationsinarabidopsisthalianagenetics.plantjournal,1998,14:387–392
[14]陈发棣,苏江硕,张飞,王海滨,管志勇,房伟民,廖园.一种与菊花耐涝性显著相关的分子标记及其鉴定方法和应用[p].中国专利:106446596a,2017-02-22(审中—实审)
技术实现要素:
为了克服现有技术的不足,本发明通过全基因组关联分析(gwas)方法筛选出与菊花耐涝性显著关联的snp位点,据此开发了一个快速、简单易行的共显性dcaps标记,命名为wt-dcaps1。我们在两个群体的极端耐涝和不耐涝个体中对该标记进行验证,平均准确率为78.9%,初步认为可用于菊花耐涝性分子标记辅助选择育种,大大缩短育种周期,从而提高育种效率。
本发明的目的通过以下技术手段获得:
一种鉴定菊花耐涝性的dcaps标记的筛选方法,包括以下步骤:
a、通过gwas方法挖掘与菊花耐涝性显著关联的snp位点
选取多份来源不同且无直接亲缘关系的菊花品种,采用盆栽模拟淹水法(106446596a)对该群体进行了三次耐涝性鉴定,获得表型数据;
基于已有的snp标记数据分别采用admixture软件和spagedi软件对该群体进行分析,获得群体结构q矩阵和亲缘关系系数k矩阵;在tassel5.0软件中,根据三次耐涝鉴定试验表型数据的平均值,利用一般线性模型(generallinearmodel,glm)和(mixedlinearmodel,mlm)两种分析模型进行全基因关联分析(gwas),以两种模型均能检测到的显著snp位点(p<1e-3)为与菊花耐涝性紧密关联的位点,同时得出该标记的贡献率r2;
b、snp突变位点特异性酶切位点分析及dcaps引物设计
(1)从步骤a所得的与菊花耐涝性紧密关联的位点中,选择p值最小,且贡献率较大的snp位点进行后续dcaps标记开发;
(2)利用在线酶切识别软件dcapsfinder2.0(http://helix.wustl.edu/dcaps/dcaps.html)查找步骤(1)中所选择的snp位点突变所引起的限制性内切酶信息,选用合适的内切酶,且人工引入错配碱基;
(3)利用primerprimer5.0软件设计pcr引物,引物设计标准为18-25bp;gc含量40-60%;退火温度为55℃-65℃,且上下游引物的gc含量和退火温度尽量保持接近;无引物二聚体及发卡结构;
c、结合基因型与表型数据预期酶切扩增多态性
对步骤b中选择的snp位点在该群体进行基因型分析,对选择的snp位点的基因型分别计算平均表型值,并用student’st检验方法分析耐涝差异显著性,结合基因型与表型值预期酶切扩增多态性;
d、dcaps标记在自然耐涝极端群体的验证
(1)提取多份耐涝菊花品种材料和多份不耐涝菊花品种材料的基因组dna,用上述步骤b中设计的特异引物对耐涝菊花品种材料和不耐涝菊花品种材料进行pcr扩增,获得目的片段全长;
(2)将扩增产物用步骤b中选用的内切酶进行酶切,并对酶切产物进行电泳检测、银染法染色;酶切扩增多态性分析,如果检测结果与步骤c中的预期相符,则将该snp位点作为鉴定菊花耐涝性的dcaps标记,将该标记命名为wt-dcaps1标记;
e、在f1后代耐涝极端群体中对wt-dcaps1标记进行进一步验证
选取耐涝性菊花品种和不耐涝性菊花品种进行杂交,在f1杂交群体中选择极端耐涝株系和极端不耐涝株系进一步验证所筛选的wt-dcaps1标记。
步骤b的(1)中选择的p值最小且贡献率较大的snp位点为标签marker6619的75位置处的snp位点(marker6619-75)。
步骤b的(2)中选用识别序列为gctagc的内切酶nhei,在marker6619-74处引入错配碱基(t→c),步骤b的(3)中设计的引物序列分别为:f5’-atgcacaaggcattggtcttgc-3’;r5’-cctcaaagttttggctattctcc-3’。
步骤c中对marker6619-75位点进行基因型分析,共有c和y两种基因型,y代表ct兼并碱基,根据内切酶nhei的识别序列可知,若该snp位点为c,不能被酶切,若为y,则可以部分酶切;对marker6619-75位点的两种基因型分别计算平均表型值,并用student’st检验方法分析耐涝差异显著性,显示c基因型的材料耐涝性显著高于y基因型(p<0.01),即不耐涝的菊花品种的pcr产物更有可能被酶切。
步骤d的(2)中将扩增产物用内切酶nhei进行酶切,耐涝菊花品种材料的扩增产物不能酶切;不耐涝菊花品种材料的扩增产物酶切产物电泳检测结果包含两条主带170bp和149bp(部分酶切的产物包括170bp,149bp和21bp三个片段,其中21bp的条带太小,电泳检测时不能被检测到)。
上述方法筛选的鉴定菊花耐涝性wt-dcaps标记。
上述的鉴定菊花耐涝性wt-dcaps标记在耐涝性菊花育种中的应用。
一种菊花耐涝性dcaps分子标记辅助选择育种方法,该dcaps分子标记为marker6619-75,选用识别序列为gctagc的内切酶nhei,设计上下游引物序列:f5’-atgcacaaggcattggtcttgc-3’和r5’-cctcaaagttttggctattctcc-3’,以菊花的基因组dna为模板,以所设计的上下游引物进行pcr扩增,获得目的片段全长,共170bp;将扩增产物用内切酶nhei进行酶切,如果酶切产物只包含一条170bp的主带,则待测菊花为耐涝菊花品种;若酶切产物的电泳检测结果包含两条主带170bp和149bp,则待测菊花为不耐涝菊花品种。
本发明的有益效果:
本发明建立了一种鉴定菊花耐涝性的dcaps标记开发体系,首先使用全基因组关联分析方法挖掘和菊花耐涝性显著关联的snp位点,然后将获得的snp进一步转化成1个dcaps标记,并在自然群体和f1后代群体中的极端耐涝和不耐涝个体中进行验证。与目前技术相比,其优点是:
(1)使用在基因组中分布广泛、数量多,遗传稳定性高且遗传分析重现性好、准确性高的snp标记进行gwas分析。此外,我们进行了三次表型鉴定试验,并以glm和mlm两种模型下均能检测到的显著snp位点(p<1e-3)为与菊花耐涝性紧密关联的标记,在一定程度上较少了外界因素对关联分析结果的影响。
(2)选择了最有可能和耐涝性显著关联的一个snp进行dcaps标记开发,结合该位点的基因型和表型数据首先分析得到预期的酶切情况,再和实际获得的酶切扩增多态性进行比较,提高了标记开发的准确性。
(3)对开发的dcaps标记在两个群体中进行验证,增加了标记开发的准确性。此外,本发明开发的是和菊花耐涝性共分离的功能性dcaps共显性标记,初步认为可以应用于菊花耐涝性分子标记辅助育种,这样不用进行田间统计即可快速筛选菊花耐涝性,既避免了环境因子对表型鉴定的影响又大大缩短了育种周期,对于培育耐涝性菊花新品种具有重要的理论和实践意义。
附图说明
图1为三次耐涝性表型鉴定数据的箱图。
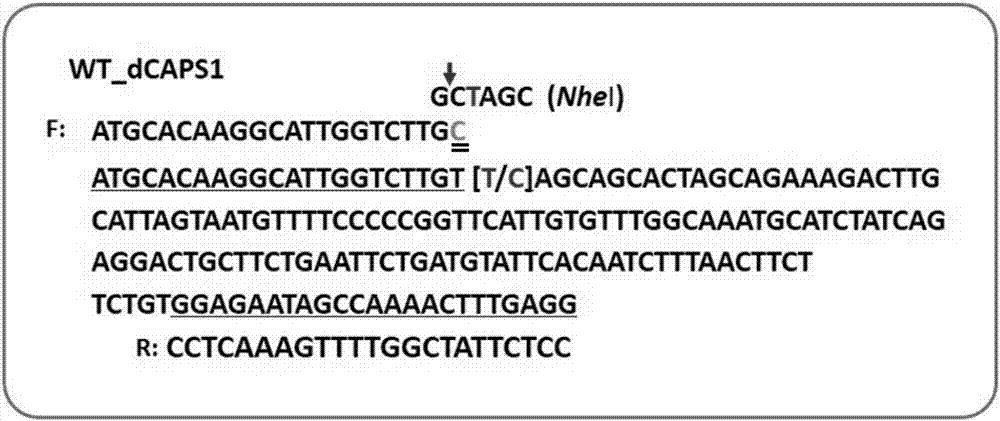
图2为wt-dcaps1的pcr扩增片段序列及引物设计图解。
其中,双下划线处的“c”碱基为人工引入的错配碱基,大括号内的碱基[t/c]表示snp突变位点,箭头表示酶切位点。
图3为marker6619-75在关联分析群体中两种基因型的耐涝性隶属函数值(mfvw)箱图。
其中,标号为1的箱子表示c基因型;标号为2箱子表示y基因型;箱子上方的不同大、小写字母表示两者的表型值在p<0.01和p<0.05水平上均具有显著性差异。
图4为开发的wt-dcaps1标记在自然群体(a)和f1后代群体(b)中的验证。
其中,1-13为13个耐涝品种,2-26为13个不耐涝品种;1为菊花品种‘南农雪峰’,14为菊花品种‘蒙白’,f1群体为‘南农雪峰’和‘蒙白’杂交所得;a-m为13个耐涝株系,n-z为13个不耐涝株系。
具体实施方式
下面结合实施例对本发明做进一步说明,下列实施例中未注明具体条件的实验方法,通常按照本领域的公知手段。
(一)挖掘与菊花耐涝性显著关联的snp
(1)试验材料和耐涝表型数据的获得:选取不同来源且无直接关系的88份菊花资源作为试验材料,所有材料保存于“中国菊花种质资源保存中心”,本领域技术人员可以从“中国菊花种质资源保存中心”中获得上述种质。采用盆栽模拟淹水法对该自然群体在菊花苗期进行三次耐涝性鉴定试验,淹水处理保持超过水面3cm,淹水3d后统计萎蔫指数,8d后根据形态变化进行打分并统计黄叶率,采用隶属函数法对以上三个指标进行每个品种的菊花耐涝性隶属函数值(mfvw)的计算,以此作为各品种最终的耐涝性鉴定数据,其中mfvw值越大,说明耐涝性越好。
利用spss20.0软件对三次耐涝性鉴定数据进行方差分析和表型箱图绘制,发现三次试验之间无显著性差异(图1),故将三次试验的mfvw平均值用于后续的gwas分析(表1)。
表1供试的88个菊花品种及其mfvw
(2)分子数据的获得:本发明所采用的snp标记为现有技术已公开的数据。首先对468,521个snp原始数据在88份资源中进行筛选,筛选标准为:snp完整度大于50%且最小等位基因频率(minorallelefrequency,maf)大于5%。过滤后获得92,811个有效snp。基于此,通过admixture软件分析样品的群体结构,假设样品的分群数(k值)为1-10,进行聚类,对聚类结果进行交叉验证,根据交叉验证错误率的谷值确定该群体的最优分群数为3,因此在k=3时生成的q-矩阵用于后续gwas分析;使用spagedi软件对该自然群体两两个体间的亲缘关系(relativekinship)进行估计。亲缘关系本身是定义两特定材料之间的遗传相似度与任意材料之间的遗传相似度的相对值,因此当结果出现两材料之间的亲缘关系值小于0时,则直接定义为0,据此建立亲缘关系k-矩阵。
(3)与菊花耐涝性显著关联的snp的确定:根据不考虑或者考虑(2)中群体结构和亲缘关系对于关联分析结果的影响,采用glm和mlm两种分析模型,结合(1)中所获得表型数据通过tassel5.0进行gwas分析。以两种模型均能检测到的显著snp位点(p<1e-3)为与菊花耐涝性紧密关联的标记,同时得出该标记的贡献率r2。共检测到10个符合该要求的snp(表2)。进一步分析发现标签marker6619的75位置处的snp位点(即marker6619-75)在两个模型下的p值均最小,且在glm模型下的贡献率也最大,为20.67%,在mlm模型下的贡献率为16.90%,因此选择该snp位点进行后续dcaps标记开发。
表2与菊花耐涝性显著相关的snp(p<0.001)
(二)snp突变位点特异性酶切位点分析及dcaps引物设计
(1)特异性酶切位点分析:根据(一)中获得的与耐涝性最为显著关联的marker6619-75位点,提取slaf标签marker6619的序列(双末端测序各100bp):caccatttcttcttcagtggacatttccgaactatctatttctaacgtctcaatgcacaaggcattggtcttgt[y/c]agcagcactagcagaaagacttgcannnnnnnnnntgaattctgatgtattcacaatctttaacttcttctgtggagaatagccaaaactttgaggtaaattaaatttgtctttatcatattcataaccatttcg。利用在线酶切识别软件dcapsfinder2.0(http://helix.wustl.edu/dcaps/dcaps.html)查找marker6619-75突变所引起的限制性内切酶信息,选用内切酶nhei,该酶的酶切识别序列为gctagc,需要在marker6619-74位置处人工引入错配碱基(t→c)。
(2)基因型与表型分析:对(1)中的marker6619-75位点在gwas分析群体中进行基因型分析,共有c和y两种基因型,其中y代表ct兼并碱基,若某材料的基因型为y表示该材料在该位点为杂合状态;根据上述(1)中nhei的识别序列可知,若该snp位点为c,不能被酶切,若为y,则可以部分酶切;对marker6619-75位点的两种基因型分别计算平均表型值,发现c基因型的平均mfvw为0.62±0.15,y基因型的平均mfvw为0.29±0.11。用student’st检验方法分析耐涝差异显著性,发现c基因型的材料耐涝性显著高于y基因型(p<0.01),综合上述(1)和(2)得出不耐涝的菊花品种的pcr产物更有可能被酶切。
(3)引物设计:利用primerprimer5.0软件设计pcr引物,引物设计标准为长度18-25bp;gc含量40-60%;退火温度为55℃-65℃,且上下游引物的gc含量和退火温度尽量保持接近;无引物二聚体及发卡结构;上游引物必需包含错配碱基而决不能包含突变碱基,据此设计的上游引物序列为5’-atgcacaaggcattggtcttgc-3’,下游引物序列为5’-cctcaaagttttggctattctcc-3’。引物设计详情见下表3。
表3wt_dcaps1标记引物信息
(三)pcr扩增及产物测序
(1)采用ctab法提取13份耐涝菊花品种材料和13份不耐涝菊花品种材料的基因组dna(表4),用核酸仪检测dna的浓度和纯度后,用ddh2o稀释至300ng/μl备用。
表4用于wt-dcaps1验证的26份菊花品种
(2)用上述步骤(二)中设计的特异引物对(1)中的26份材料进行pcr扩增。扩增体系为25μl,其中包括:10×buffer(含mg2+)2.5μl,dntp(2.5mmeach)2.0μl,前后引物各1μl,taqdna聚合酶1u,模板dna1μl,ddh2o17.3μl。pcr扩增程序为:94℃预变性3min;94℃变性30s,57℃退火30s,72℃延伸30s,共35个循环;72℃延伸7min。对pcr产物采用1.5%琼脂糖凝胶电泳检测,获得单一条带,说明引物特异性好。
(3)由于slaf技术是针对双末端各100bp进行测序,中间的一段序列为未知,因此对(2)中获得的pcr产物进行测序,获得目的片段全长,共170bp:atgcacaaggcattggtcttg|c[y/c]agcagcactagcagaaagacttgcattagtaatgttttcccccggttcattgtgtttggcaaatgcatctatcagaggactgcttctgaattctgatgtattcacaatctttaacttcttctgtggagaatagccaaaactttgagg。nhei的识别序列为gctagc,竖线位置为nhei酶切位点,再根据(二)中基因型与表型的分析,推断耐涝菊花品种不能被酶切,而不耐涝菊花品种能够被部分酶切,且酶切产物分别为分子量大小为149bp和21bp的两个条带。值得注意的是,该步骤的pcr产物测序并非必要步骤,但是通过对产物测序既可以验证引物设计的准确性,也能因此推断酶切条带的大小。
(四)dcaps标记在自然耐涝极端群体的验证
对(三)中获得的pcr产物进行37℃酶切3h。酶切反应体系为50μl,其中包括:10×nebuffer5μl,pcr产物10μl,ddh2o34μl,nhei内切酶0.1μl。所用试剂购自newenglandbiolabs公司,酶切结束后在80℃下热失活20mim。用10%非变性聚丙烯酰胺胶对酶切产物进行电泳检测、银染法染色。酶切扩增多态性分析发现:13份耐涝菊花品种材料中11份不能酶切(84.6%),而13份不耐涝菊花品种材料中9份能够部分酶切(69.2%),酶切条带大小符合(三)中的预期(由于另一条21bp的酶切条带太小,电泳时沉到电泳液中不能显示),所开发的和菊花耐涝性状共分离的wt-dcaps1标记平均准确率为76.9%(图4a)。
(五)dcaps标记在f1后代耐涝极端群体的进一步验证
由(四)可知耐涝菊花品种‘南农雪峰’不可以被酶切,而不耐涝菊花品种‘蒙白’可以被部分酶切(图4a)。因此本发明在‘南农雪峰’ב蒙白’的f1杂交群体中(162个株系)选择13个极端耐涝株系和13个极端不耐涝株系进一步验证开发的wt-dcaps1标记和菊花耐涝性状共分离(表5)。dna提取、pcr扩增、酶切反应、电泳检测等均与上述26个自然品种的验证方法相同。酶切扩增多态性分析发现:13个耐涝株系中10份不能酶切(76.9%),而13份不耐涝菊花品种材料中11份能够部分酶切(84.6%),平均准确率为80.8%(图4b),从而进一步验证了本发明开发的鉴定菊花耐涝性的dcaps标记的可行性。
表5用于wt-dcaps1验证的26个‘南农雪峰’ב蒙白’f1后代株系
以上是为了阐述本发明的发明原理而叙述的本发明的示例性实施方式和具体的操作过程,然而本发明的保护范围不仅限于此,本领域技术人员在不脱离本发明实质情况下,可以做出各种改进和变更,这些改进和变更也属于本发明的保护。
序列表
<110>南京农业大学
<120>一种鉴定菊花耐涝性的dcaps标记开发及应
<160>3
<170>siposequencelisting1.0
<210>1
<211>22
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgcacaaggcattggtcttgc22
<210>2
<211>23
<212>dna
<213>人工序列(artificialsequence)
<400>2
cctcaaagttttggctattctcc23
<210>3
<211>210
<212>dna
<213>人工序列(artificialsequence)
<400>3
caccatttcttcttcagtggacatttccgaactatctatttctaacgtctcaatgcacaa60
ggcattggtcttgtcagcagcactagcagaaagacttgcannnnnnnnnntgaattctga120
tgtattcacaatctttaacttcttctgtggagaatagccaaaactttgaggtaaattaaa180
tttgtctttatcatattcataaccatttcg210
- 还没有人留言评论。精彩留言会获得点赞!