多菌灵降解酶CbmA及其编码基因和应用的制作方法
本发明属于应用环境微生物和农业领域,涉及多菌灵降解酶cbma及其编码基因和应用。
背景技术:
杀菌剂是防治作物病害最重要的武器。多菌灵(carbendazim),化学名称为苯并咪唑-2-氨基甲酸酯,别名有棉萎灵、苯并咪唑44号、棉萎丹、mbc等,分子式为c9h9n3o2,分子量为191.19g·mol-1,为浅灰色粉末,水溶性为8mg·l-1。于1973年开发,是农业生产过程中使用广泛,频率较高的一种杀菌剂,市场上有多种复配剂。多菌灵是全世界广泛用于农业、林业中真菌病害防治的广谱、内吸性苯并咪唑类杀菌剂。多用于防治谷物和水果,如棉花、花生、小麦、甜菜、甘蔗、番茄、柑橘、香蕉、草莓、菠萝和梨果等植物的病害。多菌灵也是很多苯并咪唑类杀菌剂的水解产物和活性成分。由于多菌灵多用于作物生长后期的病害防治及采摘后的贮存,所以,在农产品中较容易检测到残留。这些残留经常出现在日常生活中常见的食物,如水果、蔬菜、粮食、油料和中药材等。2014年,国家发布实施的《食品中最大农药残留量限量标准》(gb2763-2014)中,多菌灵在绝大多数粮食及果蔬中含量不得超过0.5mg·kg-1。在欧盟被视为危险物质,在美国已被列入禁用列表,但仍然在许多发展中国家,如中国、印度等广泛使用。
要利用微生物(酶)降解技术解决多菌灵残留问题首先要获得优良的菌株和基因资源。目前已经报道了一些降解多菌灵的菌株,但多菌灵水解酶的报道只有一个酰胺酶mhei,表达效果也并不理想,这就限制了人们利用微生物(酶)降解技术消除土壤和农产品上的多菌灵残留。
技术实现要素:
本发明的目的是针对现有技术的不足,提供新的多菌灵降解酶cbma与其编码基因cbma以及二者的应用。基因cbma可用于构建降解多菌灵的降解菌液,亦可用于生产降解多菌灵的酶制剂,用于消除土壤、水体和农产品中多菌灵残留。
本发明的目的可通过如下技术方案实现:
一株多菌灵降解菌djl-6-2,保藏于中国典型培养物保藏中心,保藏编号为cctccno:m2017441。
基因cbma的出发菌株rhodococcusjialingiaedjl-6-2保存于中国典型培养物保藏中心,保藏编号为cctccno:m2017441。质谱分析结果表明菌株djl-6-2的粗酶液可以通过水解多菌灵的c-n键来降解多菌灵。
一种多菌灵降解酶基因cbma,其核苷酸序列为seqidno.1。
克隆多菌灵降解酶基因的策略为蛋白纯化结合基因组学的方法。通过硫酸铵分级沉淀、deae-sepharoseff离子柱层析、q-sepharoseff离子柱层析和superdex-200凝胶层析4步纯化流程获得了满足maldi-tof-ms技术分析条件的多菌灵水解酶样品。肽指纹图谱鉴定结合菌株基因组完成图测序结果分析,确定编码该蛋白的orf为orf04383。orf分析显示,该基因全长为1434bp,编码477aa。通过氨基酸序列比对(swiss-prot)发现,cbma与来自rhodococcuserythropolis的acylamidase具有最高的相似性(96%),其余蛋白的相似性都小于34%,与之前的多菌灵水解酶mhei相似性很低,表明cbma是一个新的多菌灵水解酶。
所述cbma基因所编码的酶cbma,其氨基酸序列为:seqidno.2。
含有所述cbma基因的重组表达载体。
所述的重组表达载体优选将所述cbma基因插入pet-29a(+)的ndei和xhoi位点之间所得。
含有所述cbma的基因工程菌。
所述的基因工程菌的出发菌株为大肠杆菌bl21(de3)。
所述cbma基因在构建降解多菌灵的基因工程菌中的应用。
所述cbma基因在去除农产品、土壤和水体中多菌灵残留中的应用。
所述降解酶cbma在降解多菌灵中的应用。
所述降解酶cbma在去除农产品、土壤和水体中多菌灵残留中的应用。
所述重组表达载体在构建降解多菌灵的的降解菌剂中的应用。
所述含重组表达载体的基因工程菌在去除水体中多菌灵残留中的应用。
有益效果
1.本发明筛选获得一株rhodococcusjialingiaedjl-6-2,质谱分析结果表明菌株djl-6-2的粗酶液可以水解多菌灵生成2-氨基苯并咪唑。在此基础上,本发明用蛋白纯化的方法成功从菌株djl-6-2(cctccno.m2017441)中克隆出基因cbma。在genbank比对结果表明该基因为一个酰胺酶基因,全长(从起始密码子到终止密码子)为1434bp,编码477个氨基酸。
2.本发明中的降解酶cbma或携带cbma基因的工程菌能高效降解多菌灵。基因cbma可用于构建降解多菌灵的转基因微生物或植物,亦可用于生产降解多菌灵的酶制剂,用于消除土壤、水体和农产品中多菌灵残留。
附图说明
图1水解酶基因cbma肽指纹图谱鉴定结果。
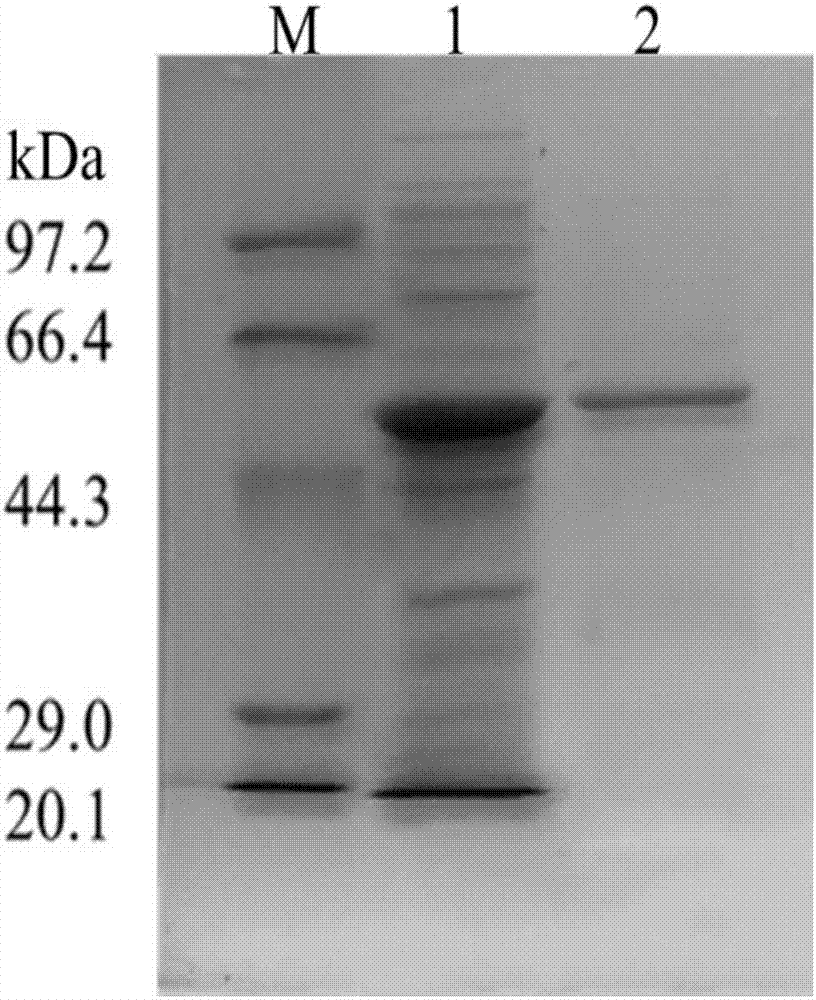
图2水解酶cbma蛋白电泳图谱;
其中泳道1为蛋白质marker,泳道2为破碎粗酶液,泳道3为纯化的水解酶cbma蛋白。
图3水解酶cbma催化的降解多菌灵的hplc/ms图谱;
a:保留时间为5.15min多菌灵的一级质谱图;b:保留时间为3.72min2-氨基苯并咪唑的一级质谱图。
生物材料保藏信息
降解菌djl-6-2,分类命名为rhodococcusjialingiaedjl-6-2,保存在中国典型培养物保藏中心(cctcc),地址为中国武汉武汉大学,保藏编号为cctccno:m2017441。
具体实施方式
实施例1
1.1多菌灵降解菌djl-6-2(cctccno:m2017441)的分离
取5g处理多菌灵废水的活性污泥加入100ml的富集培养基中,于温度为30℃,转速为160rpm的摇床中富集培养,以5%的接种量每5d转接到新鲜的msmc培养基中。转接到第3次时,设计空白对照,即不接种的msmc培养基同富集培养基在相同条件中培养5d。通过紫外分光光度计和高效液相色谱测定多菌灵的含量,与空白对照相比,多菌灵的含量明显下降,即为有降解活性的富集液。将富集液以10-3~10-8进行梯度稀释,各取0.2ml涂布于加有100mg·l-1多菌灵的msm固体培养基上,30℃恒温培养箱中倒置培养至有菌落长出,挑取具有不同形态特征的单菌落进行分离纯化,并接种于装有20mlmsmc培养基的50ml三角瓶中,经30℃,160rpm摇床培养5d后,对单菌进行降解活性检测,对有降解活性的菌株按1∶1加入30%已灭菌甘油,保存至-70℃超低温冰箱。
基础盐培养基(msm)配方为(1l):1.0gnh4no3,1.0gnacl,1.5gk2hpo4,0.5gkh2po4,0.2gmgso4·7h2o.ph7.0.固体培养基加15.0g琼脂。
富集分离培养基(msmc):加入多菌灵的基础盐培养基。如无特别说明,添加多菌灵浓度为50mg·l-1作为唯一碳源。
降解效果的验证方法:
(1)紫外扫描法:加入等体积二氯甲烷于待测样品中,在涡旋振荡器上震荡5~10min,保证多菌灵能被充分萃取。剧烈振荡结束后静置分层,吸取下层有机相或去弃上层水相,加入过量的无水硫酸钠去除残余水分。取处理好的有机相使用岛津uv-2401pc紫外扫描仪于200~350nm连续扫描,根据280nm处多菌灵的特征吸收峰的高低值来确定降解活性。
(2)高效液相色谱法(hplc):将待测样品同二氯甲烷按等体积混合后进行全量提取,在涡旋振荡器上剧烈振荡后,静置分层,弃上层水相,与有机相中加入过量无水硫酸钠进行脱水处理,取1ml有机相至1.5ml离心管中,放置于通风厨中至溶剂完全挥发,然后用1ml甲醇溶解,并用0.22μm尼龙滤膜过滤后进行hplc分析。液相色谱条件:液相色谱仪型号为岛津rid-10a;液相色谱柱为c18反相柱,规格为250mm×4.6mm×5μm;流动相为甲醇∶乙腈∶水(30∶20∶50)加0.5%乙酸;柱温为40℃;流速为0.8ml·min-1;检测波长为280nm和240nm。
从富集液中,分离到1株多菌灵降解菌,命名为djl-6-2,该菌在2d内几乎完全降解100mg·l-1的多菌灵。
1.2多菌灵降解菌djl-6-2的鉴定和生物学特性
多菌灵降解菌djl-6-2革兰氏阳性菌,好氧,不运动,无孢子形态,抗酸染色阴性,有球杆状变化。在lb固体培养基上菌落呈不透明,浅粉红色,潮湿,边缘光滑凸起。温度生长范围为4-35℃,ph生长范围为5.0-9.0。nacl耐受浓度为7%。氧化酶,h2s产生,吲哚,明胶液化,淀粉水解,硝酸盐还原,甲基红等反应为阴性;接触酶,v-p试验,尿酶反应为阳性。菌株djl-6-2能够利用d-阿拉伯糖,d-果糖,d-甘露醇,myo-肌醇,七叶苷柠檬酸铁,苯菌灵,多菌灵,儿茶酚等为唯一碳源;而d-鼠李唐,d-山梨醇糖,d-蔗糖,d-海藻糖,d-木糖,菊淀粉等不能被利用。菌株djl-6-2细胞壁组分中具有分类学意义的氨基酸为meso-dap,丙氨酸,谷氨酸,甘氨酸,天门冬氨酸。糖类型为核糖,阿拉伯糖和半乳糖等。磷脂为:dpg,pel,pg,pe,pim,pi,pme和cl。菌株djl-6-2中主要的醌为mk-8(h2),含有枝菌酸。主要脂肪酸组成为c14:0(9.95%),c15:0(1.25%),c16:0(36.77%),c17:1ω8c(1.44%),c18:1ω9c(27.29%),c18:0(4.89%)和10-methylc18:0(tuberculostearicacid,tbsa)(3.70%)。dnag+cmol%含量为60.1%.
16srrna基因序列系统发育分析:菌株总dna的提取采用高盐法,并以总dna作为模板,进行16srrna基因序列的扩增。用于扩增反应的引物为一对通用引物,上游引物:5’-agagtttgatcctggctcag-3’(seqidno.5),下游引物:5’-taccttgttacgactt-3’(seqidno.6)。25μlpcr反应体系为:模板1μl,dntp(25mmol/l)2μl,引物(1mmol/l)各1μl,10×taq缓冲液2.5μl,mg2+(25mmol/l)1.5μl,taq酶(5u/μl)0.5μl,超纯水15.5μl。聚合酶链式反应条件:95℃预变性5min;94℃变性0.5min,52℃退火1min,72℃延伸1.5min,循环30次;72℃延伸10min。采用pcr回收试剂盒(axygen公司)回收16srdna的基因片段,琼脂糖凝胶电泳检测扩增产物的大小(1.5kb左右),ta克隆后进行测序(由金斯瑞公司完成)。测序结果通过在线分析,与ezbiocloud数据库(http://www.ezbiocloud.net/)中的模式菌株16srrna基因序列进行相似性比较。结果显示菌株djl-6-2与rhodococcusqingshengiidjl-6t,rhodococcusbaikonurensisdsm44587t,rhodococcuserythropolisdsm43066t和rhodococcusgloberulusdsm43954t的亲缘关系较近,16srdna同源性分别为99.80%、99.78%、99.12%和98.75%。结合生理生化特征将该菌鉴定为rhodococcussp.,保存在中国典型培养物保藏中心(cctcc),地址为中国武汉大学,保藏编号为cctccno:m2017441。
实施例2基因cbma的克隆
克隆该基因采取的策略为蛋白纯化结合基因组学的方法。对菌株djl-6-2的粗酶液通过硫酸铵分级沉淀、deae-sepharoseff离子柱层析、q-sepharoseff离子柱层析和superdex-200凝胶层析四步进行纯化得到可能的目的蛋白,再通过肽指纹图谱鉴定结合菌株基因组测序结果对目的蛋白序列进行确定。
(1)菌株djl-6-2的培养和粗酶液的制备
将保藏的菌株djl-6-2从-70℃冰箱中取出,在含有100mg·l-1多菌灵的lb固体平板上进行划线活化。30℃培养3d后,挑取单菌落接种于含有30mg·l-1多菌灵的100mllb液体培养基中,置于30℃摇床,160rpm振荡培养至对数生长期;于4℃,12000×g离心10min弃上清,收集菌体;用5ml预冷的20mmtris-hcl(ph7.4)重悬菌体,4℃,12000×g离心10min收集菌体,重复此步骤直至lb被完全去除;用10ml预冷的20mmtris-hcl重悬菌体。在冰浴下,使用超声波破碎仪破碎菌体,程序设置为破碎2s,停3s,破碎至悬浮液澄清,破碎结束后,4℃,12000×g离心30min,所得上清即为粗酶液。
(2)硫酸铵分级沉淀
按0~40%、40~50%、50~60%、60~70%、70~80%和80~100%的硫酸铵饱和梯度对粗酶液进行沉淀。将所需体积的粗酶液加入内有磁性转子的三角烧瓶中,三角烧瓶置于磁力搅拌器上的烧杯中冰浴,打开磁力搅拌器至匀速。按粗酶液的体积计算硫酸铵的质量,向粗酶液中缓慢均匀加入硫酸铵,使饱和度达到20%,此过程约为30min,4℃,12000×g离心20min,沉淀用适量tris-hcl缓冲液(20mm,ph7.4)重悬,用于后续测定蛋白含量和酶活力;向上清中继续添加硫酸铵,使饱和度达到40%,按上述步骤,直至硫酸铵的饱和度达到100%。将重悬后的各级组分于4℃,20mmtris-hcl(ph7.4)缓冲液中透析过夜,透析袋截留分子量为10kda。透析好的各级酶液即可测定蛋白量和相对酶活力,选择比酶活较高的组分继续纯化。(3)deae-sepharoseff离子柱层析
将透析后有酶活力的酶液加入到用20mmtris-hcl(ph7.4)平衡好的deae-sepharoseff阴离子柱上,用含有0、0.2、0.3、0.4、0.5和1.0mnacl的20mmtris-hcl(ph7.4)进行梯度洗脱,每个梯度缓冲液的体积为20ml,流速为1ml·min-1,每2ml收集一管。将不同梯度收集的酶液,于4℃,20mmtris-hcl(ph7.4)缓冲液中分别透析过夜。透析好的各梯度酶液即可测定蛋白量和相对酶活力。将透析后酶活较高的酶液用超滤离心管(10kda)4℃,3000×g进行超滤浓缩,进行下一步纯化。
(4)q-sepharoseff离子柱层析
将上一步有酶活力的酶液加入到用20mmtris-hcl(ph7.4)平衡好的q-sepharoseff阴离子柱上,用含有0、0.4、0.5、0.6、0.7和1.0mnacl的20mmtris-hcl(ph7.4)进行梯度洗脱,每个梯度缓冲液的体积为20ml,流速为1ml·min-1,每2ml收集一管。将不同梯度收集的酶液,于4℃,20mmtris-hcl(ph7.4)缓冲液中分别透析过夜。透析好的各梯度酶液即可测定蛋白和酶活力。将透析后酶活较高的酶液用超滤离心管(10kda)4℃,3000×g进行超滤浓缩,进行下一步纯化。
(5)superdex-200凝胶层析
将上一步超滤浓缩后的酶液加入到用20mmtris-hcl(ph7.4)平衡好的superdex-200凝胶层析柱上,用含有0.1mnacl的20mmtris-hcl(ph7.4)进行洗脱,aktapurifier全自动层析仪(aktapurifier10upc)自动收集,流速为0.4ml·min-1,每500μl收集一管。每管取适量酶液测活性,将有活性的收集管中的酶液进行超滤浓缩至100μl。
(6)肽指纹图谱分析
将目的蛋白委托上海博苑生物科技有限公司进行肽指纹图谱分析(maldi-tofmassspectrometryanalysis),通过肽段序列与菌株djl-6-2基因组完成图中的氨基酸序列进行同源性比对分析(图1),多菌灵降解酶基因cbma的氨基酸序列为seqidno.2,根据cbma氨基酸序列在基因组完成图中对应的核苷酸序列为seqidno.1。
实施例3多菌灵水解酶cbma在bl21(pet-29a(+))中的高效表达、纯化及其功能测定
(1)基因cbma的pcr扩增
以正向引物:5’-taagaaggagatatacatatgaccgaacagaatctgcattg-3’(ndei)(seqidno.3)和反向引物:5’-gtggtggtggtggtgctcgagtgcaacaaccaggctacctgc-3’(xhoi)(seqidno.4)为引物,用pcr从rhodococcusjialingiaedjl-6-2(cctccno.m2017441)基因组dna中扩增出cbma基因片段
扩增体系:
pcr反应条件:
(2)pcr产物用ndei和xhoi双酶切。
酶切体系:
在37℃水浴中,反应30min。酶切产物进行0.75%的琼脂糖凝胶电泳切胶回收。
(3)pet-29a(+)用ndei和xhoi双酶切(参考(2))。
(4)表达载体pet-29a-cbma的构建
将双酶切后切胶回收的pet-29a(+)片段(5.23kb)和目的基因片段(1434bp)按照同源重组一步克隆试剂盒(
同源重组体系如下(20μl):
(5)cbma的表达、纯化和功能验证
重组菌株bl-29-cbma经iptg诱导表达多菌灵水解酶cbma,经多次试验和参考文献后发现iptg浓度为0.5mm,16℃诱导8h时表达效果最好。诱导后收集菌体进行破碎得粗酶液,利用ni-nta柱纯化粗酶液,纯化后蛋白的sds-page如图2所示。在350mm的咪唑洗脱液中,目的条带单一,大小约为50kda,经过透析后可以满足后续酶学特性研究的要求。
(6)cbma降解多菌灵产物的测定
酶活反应体系(3ml):20mmtris-hcl(ph7.5),0.3mm(50mg·l-1)靶标底物(多菌灵)、反应酶量(实施例3步骤(5)中纯化所得)50μl,35℃反应10min。每个反应以加入酶开始计时,用3ml二氯甲烷终止反应并萃取,分层后有机相经无水硫酸钠脱水,多菌灵的含量用hplc测定(具体方法见实施例1步骤(2))。hplc结果表明,当50mg·l-1的多菌灵作为唯一碳源时,培养24h后发现有中间代谢产物的积累。hplc色谱图中出现2个物质峰,其保留时间分别为3.720min和5.147min,与标准物质多菌灵和2-氨基苯并咪唑的保留时间对照进行比较发现:保留时间3.720min的吸收峰情况与标准品2-氨基苯并咪唑的保留时间基本一致;保留时间5.147min的吸收峰情况与标准品多菌灵的保留时间基本一致。对此两个响应峰进行ms分析,分别得到相对分子质量([m+h]+)为192和135的碎片峰,该结果进一步证明hplc中两个响应峰对应的物质分别为多菌灵和2-氨基苯并咪唑(图3)。可以推断,cbma将多菌灵降解为2-氨基苯并咪唑,降解路径如下:
本发明使用的微生物来源如下:
大肠杆菌dh5α购自南京诺维赞生物科技限公司,
大肠杆菌高表达载体pet-29a(+)购自novegen公司,
表达宿主菌大肠杆菌bl21(de3)购自南京诺维赞生物科技限公司。
序列表
<110>南京农业大学
<120>多菌灵降解酶cbma及其编码基因和应用
<160>2
<170>siposequencelisting1.0
<210>1
<211>1434
<212>dna
<213>djl-6-2(rhodococcusjialingiaedjl-6-2)
<400>1
atgaccgaacagaatctgcattggctctccgctaccgagatggcggcgtcggtcgcgtcg60
aacagtctctcgcccaacgagattgccgaagcgatgatccagcgcgtcgacgccgtcaat120
ccgtcgatcaacgcgatagtccagttcgatcgcgagcaggtgacgcgcgatgcggccgaa180
ctctcacggcaacaggaagcgggcgagaaactcggcccgctgcacggcgttccgttcacg240
atcaaagatgtgacggcagtcgacgggctgccgaccacgttcgggatgaagccgatggcc300
gacaacatcgcgacgggaaatgccgtcgtcgtggacaggctgcgaggcgccggcggactg360
ttcctgggaaagacgaacactcccgaaagtgcttactacggtggcacggacaaccacctg420
tacgggccgacgcacaacccgtggaagctcggcaacagcgcgggcgggtccagtggcggt480
gcgtcggctgccgtggctgcaggcctcgggccactcgccgagggcagtgacggcgcggga540
tcggtgcgtatcccgtcggcgctctgcggggtcgtcgggttgaagccgaccaccggcgtc600
atcccgcagaccatcctggccgggcgcttctacaactgggggtatcacggtccgatcacc660
cggactgttgccgacaacgcgctgatgctcgacatcatggccgggccggacaatgcggac720
ccgctcacgatcgagcgtgtcgagacctcgtacgtcgaagcctcgaagggcgacgtgaag780
ggccttcgcgtggcgtggtcgacgaatctcggtctcggccatgttgatccggaggtgctg840
gcggtgtgtctcgacgcgttgtcggcattcgaggaattgggtgcccggatcaccgaggcg900
accccgcagtggggaaatccgtcggagtcgatgtggaacggcatctgggttcccggttac960
gcttccgaatacgacttgctcgactgggagaaccagcgcggcgaggtcgacgacaacctg1020
atcgagatcatgcacgaggccgagcggctcaccggtgtcgacgtcgggcgggccgacgca1080
ttccgcggcgtcatgtgggacacgtggaccgcgttcatgaacgactacgacgtgttggtc1140
tcgccgaccttggcttcggccacgttcccgctcagtcagttcgcgccgtcgtggctcgaa1200
ggtgcgtcgttgcgtgagcagttgctcgattggctcttcacctacccgtacaacatgctc1260
aacaaccccgcgatcaccgtgccggccggattcaccgccgacggtcgaccggtggggctg1320
cagatcgccgcacgccaccgccaggacgcactggtcctgcggactgccgcgaacttcgag1380
gcggtgcgtccgtgggcggacaggaagccggccggttcactggtggtggcctag1434
<210>2
<211>477
<212>prt
<213>djl-6-2(rhodococcusjialingiaedjl-6-2)
<400>2
metthrgluglnasnleuhistrpleuseralathrglumetalaala
151015
servalalaserasnserleuserproasngluilealaglualamet
202530
ileglnargvalaspalavalasnproserileasnalailevalgln
354045
pheasparggluglnvalthrargaspalaalagluleuserarggln
505560
glnglualaglyglulysleuglyproleuhisglyvalprophethr
65707580
ilelysaspvalthralavalaspglyleuprothrthrpheglymet
859095
lysprometalaaspasnilealathrglyasnalavalvalvalasp
100105110
argleuargglyalaglyglyleupheleuglylysthrasnthrpro
115120125
gluseralatyrtyrglyglythraspasnhisleutyrglyprothr
130135140
hisasnprotrplysleuglyasnseralaglyglyserserglygly
145150155160
alaseralaalavalalaalaglyleuglyproleualagluglyser
165170175
aspglyalaglyservalargileproseralaleucysglyvalval
180185190
glyleulysprothrthrglyvalileproglnthrileleualagly
195200205
argphetyrasntrpglytyrhisglyproilethrargthrvalala
210215220
aspasnalaleumetleuaspilemetalaglyproaspasnalaasp
225230235240
proleuthrilegluargvalgluthrsertyrvalglualaserlys
245250255
glyaspvallysglyleuargvalalatrpserthrasnleuglyleu
260265270
glyhisvalaspprogluvalleualavalcysleuaspalaleuser
275280285
alapheglugluleuglyalaargilethrglualathrproglntrp
290295300
glyasnproserglusermettrpasnglyiletrpvalproglytyr
305310315320
alaserglutyraspleuleuasptrpgluasnglnargglygluval
325330335
aspaspasnleuilegluilemethisglualagluargleuthrgly
340345350
valaspvalglyargalaaspalapheargglyvalmettrpaspthr
355360365
trpthralaphemetasnasptyraspvalleuvalserprothrleu
370375380
alaseralathrpheproleuserglnphealaprosertrpleuglu
385390395400
glyalaserleuarggluglnleuleuasptrpleuphethrtyrpro
405410415
tyrasnmetleuasnasnproalailethrvalproalaglyphethr
420425430
alaaspglyargprovalglyleuglnilealaalaarghisarggln
435440445
aspalaleuvalleuargthralaalaasnpheglualavalargpro
450455460
trpalaasparglysproalaglyserleuvalvalala
465470475
- 还没有人留言评论。精彩留言会获得点赞!