HLA分型A位点常见的模棱两可型别的检测方法与流程
本发明涉及一种hla分型a位点常见的模棱两可型别的检测方法,及用于区分hla分型a位点常见模棱两可型别的特异性引物,属于基因检测技术领域。
背景技术:
人类白细胞抗原(hla)是迄今为止人类最复杂遗传系统,具有高度的多态性。准确的hla分型技术和分型数据已被广泛应用于器官和造血干细胞移植中寻找hla相匹配的供者、hla与疾病相关性的研究、亲子关系鉴定、个体识别以及以肽为基础的病毒和癌症疫苗的研发等应用领域。供受者间hla等位基因的配合程度与移植效果密切相关,精确的hla分型有助于提高移植后的存活率。
目前以dna为基础的hla基因分型方法已得到广泛应用,其中直接测序分析基础上的基因分型技术(pcr-sbt)是who推荐的高分辨水平hla基因分型的实验方法,被认为是hla基因分型的金标准,可直接获得dna序列,鉴定所有存在的多态性,为最直接、最直观的方法。随着基因测序仪的普及和临床移植配型的要求,pcr-sbt方法在大规模的hla分型中逐渐被广泛采用。但实际操作过程中,由于目的基因序列的特殊性、引物设计等多种因素,使得该方法存在较大的缺陷,即存在较高比例的模棱两可分型结果(两组或两组以上的两个杂合等位基因的组合序列相同)。而且目前国内使用非血缘关系供受者进行器官移植及造血干细胞移植已普遍展开,更多的hla新等位基因的不断被发现,模棱两可结果的组合种类和比例也在不断上升。
pcr-sbt主要分析hla-ⅰ类和ⅱ类基因的可变区以及一些保守区,并与可能的等位基因组合的数据库相比较而判读。其基因分型模棱两可组合结果出现的其中一个很主要的原因,是由于sbt方法同时对2个等位基因扩增和序列分析,当出现因杂合碱基对组合而产生相同的序列情况时,无法分别鉴别2个等位基因对应的序列结果,即检测区杂合序列一致的模棱两可结果。大多数模棱两可结果可用高分辨水平pcr-ssp试剂盒进一步确认,但是pcr-ssp的成本相对较高,且随着等位基因的继续增多,模棱两可的比例也不断提高,pcr-ssp已经不能完全解决高分辨确认精确分型的问题。
据发明人所在实验室统计资料显示,pcr-sbt法进行hla分型,hla-a、hla-b、hla-c、hla-drb1、hla-dqb1出现模棱两可型的比例分别为>50%、>35%、>15%、>8%、>7%。因此,找到快速高效区分检测区内杂合序列一致导致的模棱两可结果的解决方案,对于大规模供者hla分型具有重要的实践意义。
技术实现要素:
针对上述现有技术,本发明提供了一种hla分型a位点常见的模棱两可型别的检测方法,以及用于鉴别hla分型a位点常见的模棱两可型别的特异性引物。针对a位点检测区杂合序列一致的常见的模棱两可型别,本发明根据组间碱基的差异性设计了具有普遍适用性的特性引物,来确认a位点的模棱两可型别,该方法耗能低,流程短,易实现高通量,较省时、经济。
本发明是通过以下技术方案实现的:
hla分型a位点常见模棱两可型别的检测方法,包括以下步骤:
(1)通过hla分型结果得到a位点的模棱两可型(通过hlasbtutype7.0分型软件进行hla分型,在常规a位点分型过程中,发现部分样本0错配存在模棱两可型别),选择区分该模棱两可型别所需的特异性引物;所述特异性引物有9条,核苷酸序列如表1所示,每条特异性引物可同时确认一个或者多个模棱两可型别,每条特异性引物能够区分的模棱两可型别如表1所示;
(2)利用上述选择的特异性引物对纯化后的hla-a基因扩增产物进行测序pcr反应;
(3)将测序后的产物纯化,通过测序仪得到产物序列,将得到的序列与模棱两可型别进行比对,从而区分模棱两可型别,最终确认hla-a型别信息。
用于区分hla分型a位点常见模棱两可型别的特异性引物,包括9条特异性引物,其核苷酸序列如表1所示,如seqidno.1~9所示。
yf-a-amb-1:5’-gctctggttgtagtagcgga-3’;如seqidno.1所示;
yf-a-amb-2:5’-caggtatctgcggagcccg-3’;如seqidno.2所示;
yf-a-amb-3:5’-cactccatgaggtatttctacacc-3’;如seqidno.3所示;
yf-a-amb-4:5’-gtccccaggtccactcggtg-3’;如seqidno.4所示;
yf-a-amb-5:5’-gagccactccacgcacgt-3’;如seqidno.5所示;
yf-a-amb-6:5’-gctctcagctgctccgccac-3’;如seqidno.6所示;
yf-a-amb-7:5’-gaggccggttctcacacca-3’;如seqidno.7所示;
yf-a-amb-8:5’-gcagggtccccaggtcca-3’;如seqidno.8所示;
yf-a-amb-9:5’-ccagatgatgtatggctgcc-3’;如seqidno.9所示。
表1:hla-a位点常见的模棱两可型别及可确认型别的引物信息
与传统的pcr-ssp方法确认基因的模棱两可型相比,本发明对型别的判定足够准确及精确,且成本大大降低,操作简单,易实现高通量,对于大规模供者hla分型不失为一个省时经济的选择。
本发明的引物的设计过程为:本发明是基于hlasbtutype7.0分型软件进行hla分型发现需要进行区分的模棱两可型别设计的,这些待区分的模棱两可型别属于wcd表(中国常见及确认的hla等位基因)亚洲人群中可能出现的型别组合,必须进行区分,才能准确出具报告。引物在设计之初是对比了多个模棱两可型别,并归类总结出哪些模棱两可型可以用同一个引物区分。比如引物yf-a-amb-1,如表1所示,当运用hlasbtutype7.0进行hla分型过程中,a位点出现如表中所示的模棱两可型别,均可运用引物yf-a-amb-1进行区分。比如a*03:01/24:02,03:08/24:07这组模棱两可型可以用yf-a-amb-1引物区分,其具体区分原理如图1。本发明在序列比对以及引物设计所使用的工具均为vectornti10.0。
本发明使用的各种术语和短语具有本领域技术人员公知的一般含义。
附图说明
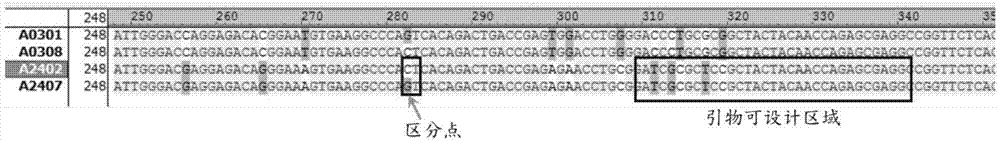
图1:引物yf-a-amb-1区分模棱两可型a*03:01/24:02,03:08/24:07的原理图。理论上,该引物可以和a*24:02以及a*24:07结合,向左延伸,区分点在282bp处,若结合在a*24:07上,那么型别即为a*03:08/24:07;若结合在a*24:02上,那么型别即为a*03:01/24:02。
图2:0错配存在模棱两可型别。
图3:分析结果示意图。
具体实施方式
下面结合实施例对本发明作进一步的说明。然而,本发明的范围并不限于下述实施例。本领域的专业人员能够理解,在不背离本发明的精神和范围的前提下,可以对本发明进行各种变化和修饰。
下述实施例中所涉及的仪器、试剂、材料等,若无特别说明,均为现有技术中已有的常规仪器、试剂、材料等,可通过正规商业途径获得。下述实施例中所涉及的实验方法,检测方法等,若无特别说明,均为现有技术中已有的常规实验方法,检测方法等。
实施例1hla分型a位点常见模棱两可型别的检测方法
步骤如下:
(1)通过hla分型结果得到a位点的模棱两可型(通过hlasbtutype7.0分型软件进行hla分型,在常规a位点分型过程中,发现部分样本0错配存在模棱两可型别),选择区分该模棱两可型别所需的特异性引物;所述特异性引物有9条,核苷酸序列如表1所示,每条特异性引物可同时确认一个或者多个模棱两可型别,每条特异性引物能够区分的模棱两可型别如表1所示;
(2)利用上述选择的特异性引物对纯化后的hla-a基因扩增产物进行测序pcr反应;
(3)将测序后的产物纯化,通过测序仪得到产物序列,将得到的序列与模棱两可型别进行比对,从而区分模棱两可型别,最终确认hla-a型别信息。
应用实例hla分型a位点常见模棱两可型别的检测
具体实施步骤如下:
(1)常规a位点分型过程中,发现0错配存在模棱两可型别,如图2,那么需要采用引物yf-a-amb-1来区分模棱两可型a*03:01/24:02,03:08/24:07。
(2)采用引物yf-a-amb-1进行增补实验,测序结果继续导入原数据进行分析,结果如图3所示。
结果显示,增补序列即为引物yf-a-amb-1所测的序列,模棱两可型a*03:01/24:02,03:08/24:07在282bp处存在一个区分点,增补序列显示的碱基是c,可结合到a*24:02型上,因此该位点的型别为a*03:01/24:02。
所以引物yf-a-amb-1即是通过以上方式将模棱两可型别区分开来。其他引物亦是如此。
给本领域技术人员提供上述实施例,以完全公开和描述如何实施和使用所主张的实施方案,而不是用于限制本文公开的范围。对于本领域技术人员而言显而易见的修饰将在所附权利要求的范围内。
序列表
<110>银丰基因科技有限公司
银丰生物工程集团有限公司
<120>hla分型a位点常见的模棱两可型别的检测方法
<141>2018-12-26
<160>9
<170>siposequencelisting1.0
<210>1
<211>20
<212>dna
<213>artificialsequence
<400>1
gctctggttgtagtagcgga20
<210>2
<211>19
<212>dna
<213>artificialsequence
<400>2
caggtatctgcggagcccg19
<210>3
<211>24
<212>dna
<213>artificialsequence
<400>3
cactccatgaggtatttctacacc24
<210>4
<211>20
<212>dna
<213>artificialsequence
<400>4
gtccccaggtccactcggtg20
<210>5
<211>18
<212>dna
<213>artificialsequence
<400>5
gagccactccacgcacgt18
<210>6
<211>20
<212>dna
<213>artificialsequence
<400>6
gctctcagctgctccgccac20
<210>7
<211>19
<212>dna
<213>artificialsequence
<400>7
gaggccggttctcacacca19
<210>8
<211>18
<212>dna
<213>artificialsequence
<400>8
gcagggtccccaggtcca18
<210>9
<211>20
<212>dna
<213>artificialsequence
<400>9
ccagatgatgtatggctgcc20
- 还没有人留言评论。精彩留言会获得点赞!