一种快速鉴别非洲猪瘟病毒的检测试剂盒及其检测方法与流程
本发明属于分子生物检测技术领域,尤其涉及一种检测试剂盒及检测方法,具体涉及是一种快速鉴别非洲猪瘟病毒核酸的检测试剂盒及其检测方法。
背景技术:
非洲猪瘟是由非洲猪瘟病毒(africanswinefevervirus,asfv)引起的一种高烈性、高度接触性动物传染病,猪一旦感染之后死亡率可达到100%,世界动物卫生组织(oie)将其列为必须报告的动物疫病,我国也将非洲猪瘟列为一类动物疫病。近年来,中国周边国家格鲁吉亚、亚美尼亚、阿塞拜疆、俄罗斯、乌克兰等国先后发生多起非洲猪瘟疫情,自2018年8月辽宁省沈阳市首例非洲猪瘟疫情确诊以来,全国多个省市都相继报告非洲猪瘟疫病例。截至2018年10月29日,全球有37个国家和地区向oie报告发生非洲猪瘟疫情。特别是2018年以来,波兰、俄罗斯、拉脱维亚、捷克、罗马尼亚、摩尔多瓦、乌克兰、匈牙利、保加利亚、比利时、科特迪瓦、南非、乍得、赞比亚和中国等15个国家共计发生了4183起非洲猪瘟疫情。由于目前世界上还没有研发出有效的针对非洲猪瘟的疫苗,为防止疾病传播疫点内的生猪只能被全部扑杀和无害化处理,给养殖业带来巨大经济损失。因此,在当前严峻的防控形势下研发简便、快速、精确的非洲猪瘟现场检测技术就显得非常迫切。
非洲猪瘟病毒属于非洲猪瘟病毒科(asfarviridae)非洲猪瘟病毒属(asfivirus),是一种有囊膜的双链dna病毒,能够耐受低温环境,4℃可以存活1年,在冷冻猪肉中可以存活数年之久;但是非洲猪瘟病毒对高温比较敏感,25-37℃可以存活数周,56℃大约可以存活70分钟,而60℃则仅可存活20分钟。乙醚、氯仿、2%氢氧化钠、3%次氯酸钠和0.3%福尔马林等多种消毒剂均可有效杀灭非洲猪瘟病毒。非洲猪瘟病毒粒子主要由病毒基因组(genome)、核心壳(coreshell)、内膜(innerenvelope)、衣壳(capsid)和囊膜(externalenvelope)组成,外形似正六边形,直径约200nm。非洲猪瘟病毒基因组不具有感染性,复制主要发生在胞浆,但有一个短暂的入核过程,对病毒的复制起始有着重要作用,因此非洲猪瘟病复制机制与痘病毒比较相似。
非洲猪瘟病毒基因组为双链闭合线性dna分子,大小约为170–194kb,含有150–160个开放阅读框(orf),总共编码54种结构蛋白和100多种非结构蛋白。非洲猪瘟病毒基因组主要由末端的发卡环结构、中间部分是比较稳定的基因区和由串联重复序列和多基因家族构成的可变区三部分组成,其中可变区基因拷贝数的变化是造成基因组大小不等的主要因素。根据编码主要衣壳蛋白的p72(b646l)基因末端约500bp核苷酸的差异已鉴定出24种基因型,但是基因型不能反映出病毒血清学特性,也不能反映毒株的免疫学特。利用pcr技术对不同病毒基因型进行遗传演化分析发现,西非地区流行的主要是基因ⅰ型,东非和南非则有20多种基因型。基因ii型流行于非洲东南部,2007年传至格鲁吉亚、俄罗斯和东欧地区;2017年该基因型病毒传播至俄罗斯伊尔库茨克地区。目前我国流行的毒株为基因ii型,与俄罗斯及东欧流行的毒株属同一分支。
原核生物(如细菌和古细菌等)通过crispr和crispr相关(cas)蛋白介导适应性免疫,crispr-cas系统的发现引起基因编辑技术领域发生了革命性突破。特别是crispr-cas9迅速风靡全球,与之类似的crispr-cas12a(最初称为cpf1)被发现也可以用于切割目标dna,而且研究发现cas12a能够在cas9不能作用的位置切割双链dna,留下的不规则边缘使得插入新基因更加容易。最特别的是,cas12a在切割目标双链dna后会对所有的单链dna进行任意切割,根据cas12a对单链dna可以进行“任意切割”的这种特性可以开发出对细胞、血液、唾液、尿液和粪便等样本中病毒dna进行快速、准确检测的检测系统。
由于当前非洲猪瘟防控形势非常严峻,开发新型的猪瘟病毒检测工具必须确保具有较高的灵敏度和较强的特异性,并且还要能够在条件简陋的现场检测环境下实现快速准确报告结果。
技术实现要素:
针对现有技术中的上述技术问题,本发明提供了一款快速准确鉴别非洲猪瘟病毒的检测试剂盒及其检测方法,通过本发明制备的检测试剂盒能够在常温条件下对样本进行rpa等温扩增迅速增加病毒的拷贝数,通过crispr-cas12a和crrna结合后靶向切割病毒核酸并且进一步切割荧光淬灭分子探针的单链dna产生荧光信号,因此在仪器设备简单、常温下的现场检测中即可快速准确的鉴别非洲猪瘟病毒。
本发明通过下述技术方案实现上述技术效果:
一种用于快速鉴别非洲猪瘟病毒核酸的基因序列,其核苷酸序列如seq.no.1所示。该基因序列根据美国国立生物技术信息中心(nationalcenterforbiotechnologyinformation,ncbi)数据库检索到asfv-sy18毒株的p72(b646l)基因的核苷酸序列(genbank:mh713612.1)合成长度为1941bp的基因片段。该序列能强烈激活crispr-cas12a切割酶活性。
本发明还提供了一种快速鉴别非洲猪瘟病毒核酸的方法,其包括如下步骤:
1)根据编码非洲猪瘟病毒主要衣壳蛋白的p72(b646l)基因的核苷酸序列设计一对特异性引物,利用重组酶聚合酶扩增(rpa)体系对样本非洲猪瘟病毒核酸进行等温扩增以增加非洲猪瘟病毒核酸的拷贝数;
2)通过体外重组表达的方法获得crispr-cas12a酶,根据非洲猪瘟病毒p72(b646l)基因序列设计切割靶点,构建能够特异性切割非洲猪瘟病毒核酸的向导rna,通过crispr-cas12a酶、向导rna与非洲猪瘟病毒共同形成的反应体系激活crispr-cas12a酶的切割活性;
3)构建一种包含荧光基团和淬灭基团的单链dna分子探针,被激活的cas12a酶通过切割分子探针的单链dna产生荧光信号,从而即时、准确的鉴别检测非洲猪瘟病毒核酸。
优选地,制备非洲猪瘟病毒核酸标准品的步骤中,所述的核苷酸序列是根据美国国立生物技术信息中心(nationalcenterforbiotechnologyinformation,ncbi)数据库检索到asfv-sy18毒株的p72(b646l)基因的核苷酸序列(genbank:mh713612.1)合成长度为1941bp的基因片段,将其克隆到pgem-t载体上,经测序验证后转化大肠杆菌扩增,采用质粒提取试剂盒提取重组质粒后,测定浓度为265.8ng/μl,作为重组酶聚合酶扩增(rpa)模板的标准品(换算成拷贝数为8.9×1010copy/μl)。
在建立重组酶聚合酶扩增(rpa)反应的体系时,首先使用primer5软件设计特异性的扩增引物和相应的荧光分子探针。优选地,所述的特异性引物中正向引物为p1,其序列如seq.no.6所示,反向引物为p2:其序列如seq.no.7所示。所述的荧光分子探针的序列为5'-seq.no.8[fam]c[thf]t[bhq]seq.no.9-3'。
优选地,50μl重组酶聚合酶扩增(rpa)反应体系包括如下体积的组分:正向引物2.1μl,反向引物2.1μl,荧光分子探针0.6μl,无引物水合缓冲液29.5μl,模板标准品10μl,分子级双蒸水3.2μl和醋酸镁2.5μl,轻微震荡使所有反应试剂充分混合,然后短暂离心。将反应管放入荧光检测设备,在37-40℃条件下扩增20分钟,通过实时监测相对荧光强度确定重组酶聚合酶扩增效果。
所述的crispr-cas12a蛋白酶的体外重组方法包括下述步骤:将crispr-cas12a蛋白酶基因连接到相应载体后转化高效表达bl-21大肠杆菌,通过氯霉素抗性培养基筛选出包含重组质粒的单克隆菌落,添加终浓度为0.5-1mmol/l的iptg后16℃诱导培养12-16小时,通过聚丙烯酰胺凝胶电泳(sds-page)检测crispr-cas12a蛋白的表达量;收集crispr-cas12a蛋白样品并采用ni-nta亲和层析方法纯化即可得到携带多组氨酸标签(his-tag)的重组crispr-cas12a蛋白酶。
优选地,在建立20μlcrispr-cas12a蛋白酶:crrna体外切割体系时具体包括如下体积的组分:2.88μlcrispr-cas12a蛋白酶,1.6μlcrrna,2μlnebbuffer3,1μl荧光淬灭探针,12.52μl分子级使用的双蒸水。将上述混合物与18μl重组酶聚合酶扩增(rpa)反应产物充分混合,以pcv,prv,ppv,csfv和jev等常见的猪病毒核酸样本为对照,在37℃条件下通过酶标仪监测相对荧光强度30分钟,确定该试剂盒的特异性和灵敏性。
本发明还提供了3条与crispr-cas12a酶结合后能够特异性切割非洲猪瘟病毒核酸的向导rna,分别为crrna1、crrna2和crrna3,其序列分别如seq.no.3,seq.no.4和seq.no.5所示。所述步骤3)中单链dna分子探针序列为5'-[fam]seq.no.2[tamra]-3'。
本发明还提供了rpa等温体系与crispr-cas12a-crrna切割系统连用后鉴别检测非洲猪瘟病毒的检测试技术,具有较高的实用价值。
本发明还请求保护一种用于快速鉴别非洲猪瘟病毒的检测试剂盒,该试剂盒中包括:
(1)重组酶聚合酶扩增反应体系:50μl重组酶聚合酶扩增反应体系包括如下体积的组分:正向引物2.1μl,反向引物2.1μl,荧光分子探针0.6μl,无引物水合缓冲液29.5μl,模板标准品10μl,分子级双蒸水3.2μl和醋酸镁2.5μl;
(2)crispr-cas12a蛋白酶:crrna体外切割体系:20μlcrispr-cas12a蛋白酶:crrna体外切割体系包括如下体积的组分:2.88μlcrispr-cas12a蛋白酶,1.6μlcrrna,2μlnebbuffer3,1μl荧光淬灭探针,12.52μl分子级使用的双蒸水。
所述的检测试剂盒的使用方法为:获得相关样本的质粒,将一定浓度的质粒与重组酶聚合酶扩增反应体系混合,轻微震荡使反应试剂充分混合,将反应管放入荧光检测设备,在37-40℃条件下扩增20分钟;取20μlcrispr-cas12a蛋白酶:crrna体外切割体系和18μl重组酶聚合酶扩增反应产物,在37℃条件下通过酶标仪监测相对荧光强度30分钟,通过荧光强度的变化即可鉴别非洲猪瘟病毒。
本发明还请求保护所述的检测试剂盒在快速鉴定非洲猪瘟病毒核酸中的应用。本发明所述的rpa等温扩增技术与crispr-cas12a切割系统连用快速、准确鉴别检测非洲猪瘟病毒的检测技术,与现有技术相比具有如下技术优势:
1、本发明对非洲猪瘟病毒采用了等温扩增技术,其与传统的扩增技术相比无需变性退火过程中的温度变化,其在条件简单的现场检测环境下迅速获得样本的非洲猪瘟病毒核酸扩增产物。
2、本发明采用crispr-cas12a酶和特异性切割非洲猪瘟病毒p72(b646l)基因序列的向导rna(crrna),能够在30分钟内检测到100拷贝以下的非洲猪瘟病毒核酸样本,其检测限度相对于以往技术大大降低,同时检测时间短,可以简单快捷的非洲猪瘟病毒的早期感染样本。
3、本发明建立的crispr-cas12a酶和向导rna(crrna)切割体系通过切割荧光淬灭分子探针的单链dna产生荧光信号,其肉眼识别性强,因而可以即时、准确鉴别检测非洲猪瘟病毒核酸。
附图说明
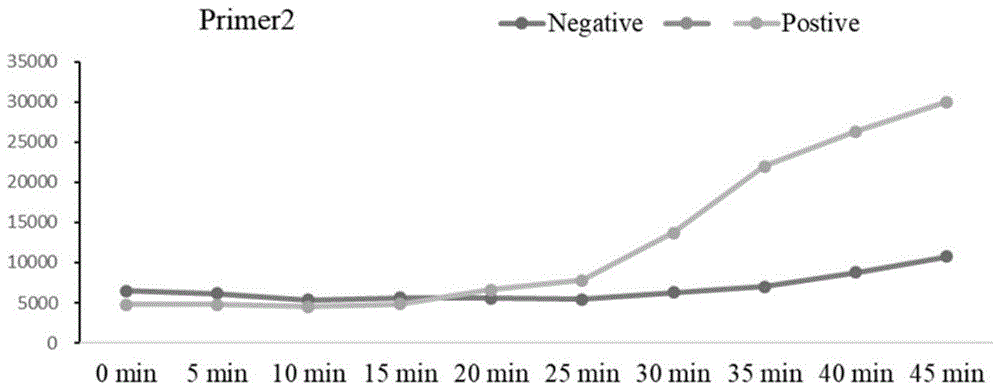
图1为非洲猪瘟病毒p72和阴性样本的rpa扩增反应的荧光强度。
图2为本发明非洲猪瘟病毒核酸检测方法的灵敏度测定。
图3为本发明非洲猪瘟病毒核酸检测方法的特异性测定。
具体实施方式
以下通过具体实施例进一步描述本发明,但本领域技术人员应该知晓,所述实施例并不以任何方式限定本发明的保护范围。
实施例1一种非洲猪瘟病毒p72(b646l)基因的重组酶聚合酶扩增的方法
按照美国国立生物技术信息中心(ncbi)数据库检索到非洲猪瘟病毒asfv-sy18毒株的p72(b646l)基因的核苷酸序列。本发明中委托上海华津生物科技有限公司合成长度为1941bp的基因片段,其序列如seq.no1所示。选择bamhⅰ和ecorⅰ酶切位点,将目的基因克隆于puc19载体上,经测序验证质粒准确无误后转化大肠杆菌,提取重组质粒后测定浓度为265.8ng/μl,作为重组酶聚合酶扩增(rpa)模板的标准品(换算成拷贝数为8.9×1010copy/μl)。
使用primer5软件设计特异性的扩增引物和相应的荧光分子探针。所述的特异性引物中正向引物为p1,其序列如seq.no.6所示,反向引物为p2:其序列如seq.no.7所示。所述的荧光分子探针的序列为5'-seq.no.8[fam]c[thf]t[bhq]seq.no.9-3'。
重组酶聚合酶扩增(rpa)反应的体系分别添加以下试剂:浓度为10μmol/l的正向引物2.1μl,浓度为10μmol/l的反向引物2.1μl,浓度为10μmol/l的荧光分子探针0.6μl,无引物再水合缓冲液29.5μl,模板标准品10μl,分子级双蒸水3.2μl和浓度为280mmol/l的醋酸镁2.5μl,总体积50μl。轻微震荡使所有反应试剂充分混合后,短暂离心。将反应管放入荧光检测设备,在37-40℃条件下扩增20分钟,通过实时监测相对荧光强度确定扩增效果。
rpa扩增反应结果如图1所示,可以看出,所述的特异性引物在20min后可以特异性的扩增非洲猪瘟病毒asfv-sy18毒株上的基因片段,并可以通过荧光强度的指标监控其扩增效率。
实施例2原核重组表达crispr-cas12a蛋白并分离纯化制备酶的方法
一种原核重组表达crispr-cas12a蛋白的方法,其具体包括下述步骤:
首先将crispr-cas12a基因连接到相应载体后转化高效表达bl-21大肠杆菌,通过含100μg/l氯霉素的选择培养基筛选出包含重组质粒的单克隆菌落,测定验证序列构建正确后进行诱导表达。将含有crispr-cas12a重组质粒的bl-21大肠杆菌在37℃条件下培养达到对数生长期后,添加终浓度为0.5-1mmol/l的iptg后在16℃条件下继续诱导培养12-16小时。采用裂解平衡缓冲液(lebuffer的具体组成为:50mmna2hpo4,0.3mnacl,ph8.0)收集培养液中的菌体,加入0.1-1mmol/l的pmsf蛋白酶抑制剂,冰浴中进行超声破碎30分钟(工作时间30s,暂停时间10s,功率200j),通过聚丙烯酰胺凝胶电泳(sds-page)检测crispr-cas12a蛋白的表达量,收集crispr-cas12a蛋白样品,采用ni-nta亲和层析方法纯化携带多组氨酸标签(his-tag)的重组蛋白。
所述的重组蛋白的纯化方法包括:首先以0.5ml/min流速的裂解平衡缓冲液(lebuffer)上样使蛋白结合到分离柱上,再以流速为1ml/min的洗涤缓冲液(washbuffer的具体组成为:50mmna2hpo4,0.3mnacl,10mmimidazole,ph8.0)洗涤分离柱以去除杂蛋白,然后用流速为0.5-1ml/min洗脱缓冲液(elutionbuffer的具体组成为:50mmna2hpo4,0.3mnacl,250mmimidazole,ph8.0)将蛋白系脱下来,分别收集流出液并通过紫外分光光度计检测a280吸收值,检测到a280值突然上升时开始收集分离峰即可获得分离纯化后的crispr-cas12a酶。
实施例3一种利用crispr-cas12a蛋白酶:crrna体系鉴别检测非洲猪瘟病毒的方法及其灵敏度、特异性测定
利用crispr-cas12a蛋白酶:crrna体系鉴别检测非洲猪瘟病毒时,首先根据asfv-sy18毒株的p72(b646l)基因设计3条特异性的crrna,分别为crrna1、crrna2和crrna3,其序列分别如seq.no.3,seq.no.4和seq.no.5所示。所述的crispr-cas12a蛋白酶:crrna体外切割反应体系每20μl具体包括如下含量的组分:浓度为1mg/ml的crispr-cas12a蛋白2.88μl,10μmol/lcrrna1.6μl,非洲猪瘟病毒2μl,nebbuffer3缓冲液2μl,分子级的双蒸水11.52μl,总体积为20μl,短暂振荡并快速离心,在37℃条件孵育30分钟后添加浓度为1μl荧光淬灭探针,然后在37℃条件下通过酶标仪监测相对荧光强度30分钟以确定非洲猪瘟病毒核酸的含量。将拷贝数为8.9×1010copy/μl非洲猪瘟病毒p72(b646l)基因质粒标准品按照1:10的比例倍比稀释,逐渐稀释到8.9×100copy/μl,分别检测不同浓度质粒标准品以确定该检测方法的灵敏性;同时分别以pcv,prv,ppv,csfv和jev等常见的猪病毒核酸样本为对照,以确定该检测方法的特异性。
所得试验结果如图2和图3所示,其中图2为本发明检测方法的灵敏度检测,可以看出,本发明所述的方法的检测限为asfv在101,其检测限相对于现有技术大大降低。图3为本发明检测方法的检测特异性检测。可以看出,随着时间的延长,样本为asfv-sy18的检测组的荧光强度逐渐降低,而其他的常见猪病毒核酸样本(pcv,prv,ppv,csfv和jev)则基本无变化。
实施例4rpa联合crispr-cas12a单反应体系检测非洲猪瘟病毒的方法
在现场试验设备简单、条件简陋的条件下,利用重组酶聚合酶扩增(rpa)技术可以实现非洲猪瘟病毒核酸的等温扩增而迅速获得较多拷贝数的检测样本,同时联合使用crispr-cas12a蛋白酶:crrna反应体系切割荧光淬灭分子探针释放荧光信号,既可以快速、准确的鉴别检测非洲猪瘟病毒核酸的样本。具体步骤如下:总体积为50μl重组酶聚合酶扩增(rpa)反应的体系分别添加以下试剂:浓度为10μmol/l的正向引物2.1μl,浓度为10μmol/l的反向引物2.1μl,无引物再水合缓冲液29.5μl,待测样本10μl,双蒸水3.8μl和浓度为280mmol/l的醋酸镁2.5μl,充分混合后短暂离心,37℃反应10分钟。吸取18μlrpa反应的产物与20ul的crispr-cas12a:crrna:荧光淬灭分子探针反应体系混合。crispr-cas12a:crrna反应体系分别添加以下试剂:浓度为1mg/ml的crispr-cas12a蛋白2.88μl,10μmol/l的crrna1.6μl,1μmol/l荧光淬灭分子探针1μl,nebbuffer3缓冲液2μl和分子级的双蒸水12.52μl。短暂振荡并快速离心后在37℃条件孵育1-2h,同时通过酶标仪监测相对荧光强度(激发波长为485nm,发射波长为535nm),根据相对荧光强度值的大小确定非洲猪瘟病毒核酸的含量。
序列表
<110>中国农业科学院上海兽医研究所
<120>一种快速鉴别非洲猪瘟病毒的检测试剂盒及其检测方法
<160>9
<170>siposequencelisting1.0
<210>1
<211>1941
<212>dna
<213>africanswinefevervirussy18
<400>1
ttaggtactgtaacgcagcacagctgaaccgttctgaagaagaagaaagttaatagcaga60
tgccgataccacaagatcagccgtagtgatagaccccacgtaatccgtgtcccaactaat120
ataaaattctcttgctctggatacgttaatatgaccactgggttggtattcctcccgtgg180
cttcaaagcaaaggtaatcatcatcgcacccggatcatcgggggttttaatcgcattgcc240
tccgtagtggaagggtatgtaagagctgcagaactttgatggaaatttatcgataagatt300
gataccatgagcagttacggaaatgtttttaataataggtaatgtgatcggatacgtaac360
ggggctaatatcagatatagatgaacatgcgtctggaagagctgtatctctatcctgaaa420
gcttatctctgcgtggtgagtgggctgcataatggcgttaacaacatgtccgaacttgtg480
ccaatctcggtgttgatgaggattttgatcggagatgttccaggtaggttttaatcctat540
aaacatatattcaatgggccatttaagagcagacattagtttttcatcgtggtggttatt600
gttggtgtgggtcacctgcgttttatggacacgtatcagcgaaaagcgaacgcgttttac660
aaaaaggttgtgtatttcaggggttacaaacaggttattgatgtaaagttcattattcgt720
gagcgagatttcattaatgactcctgggataaaccatggtttaaagcgtatattgcgtct780
actggggcgtccagctataaaacgtgactggcgtacaaaaagtccaggaaattcattcac840
caaatccttttgcgatgcaagctttatggtgataaagcgctcgccgaagggaatggatac900
tgagggaatagcaaggttcacgttctcattaaaccaaaagcgcaacttaatccagagcgc960
aagagggggctgatagtatttaggggtttgaggtccattacagctgtaatgaacattacg1020
tcttatgtccagatacgttgcgtccgtgataggagtaatatcttgtttacctgctgtttg1080
gatattgtgagagttctcgggaaaatgctgtgaaagaaatttcgggttggtatggctaca1140
cgttcgctgcgtatcattttcatcggtaagaataggtttgctttggtgcggcttgtgcaa1200
atcatgaatgttgcataggagagggccactggttccctccaccgatacctcctggccaac1260
caagtgcttatatccagtcattttatcccctgggatgcaaaatttgcgcacaagcgttgt1320
gacatccgaactatattcgtctagggaatttccatttacatcgaatcttacgttttcata1380
aagtcgttctccggggtattcgcagtagtaaaccaagtttcggtacgcattctttgtgcc1440
gggtacaatgggtcttccaaaaggatctacaagcgtgtaaacggcgccctctaagggtgt1500
ttggttgtcccagtcatatccgttgcgaggaaacgtttgaagctgcccatgggcccccat1560
ctgggacgtgccctgaatcggagcatcctgccaggatgaatgacatgcacccaatatatg1620
atggcccaccatatcatggaaaaagtctccgtactggggaataccaaaggtaagcttgtt1680
tcccaaggtgggggtacccgtatgcgggcgtactttattgtattcaaaccctactggaac1740
ataaggcttaaaatgcgcattaaaatgcaccaaatgtgtttcttcgatttgactcaaagt1800
gggttcgggatcgggtttcccataacttttgttcacatttttaatgttagagatcctgct1860
attcagcaagtcttgggccaatataatcttgtcggccttcccatcgttagcaataagaca1920
aaaagctcctcctgatgccat1941
<210>2
<211>5
<212>dna
<213>africanswinefevervirussy18
<400>2
ttatt5
<210>3
<211>40
<212>rna
<213>africanswinefevervirussy18
<400>3
aauuucuacuguuguagauucgauaagauugauaccauga40
<210>4
<211>40
<212>rna
<213>africanswinefevervirussy18
<400>4
aauuucuacuguuguagauccuccaccgauaccuccuggc40
<210>5
<211>40
<212>rna
<213>africanswinefevervirussy18
<400>5
aauuucuacuguuguagaucgcacaagcguugugacaucc40
<210>6
<211>22
<212>dna
<213>africanswinefevervirussy18
<400>6
ggtatgtaagagctgcagaact22
<210>7
<211>25
<212>dna
<213>africanswinefevervirussy18
<400>7
taagctttcaggatagagatacagc25
<210>8
<211>28
<212>dna
<213>africanswinefevervirussy18
<400>8
agagctgcagaactttgatggaaattta28
<210>9
<211>13
<212>dna
<213>africanswinefevervirussy18
<400>9
aagattgatacca13
- 还没有人留言评论。精彩留言会获得点赞!