修饰型菊酸酯酶的制作方法
本发明涉及对菊酸乙酯显示水解活性的酶(菊酸酯酶)的修饰。详细而言,涉及立体选择性发生变化的修饰型菊酸酯酶和其用途。本申请基于2018年12月6日申请的日本专利申请第2018-229479号主张优先权,并将该专利申请通过参照援引于此。
背景技术:
菊酸作为合成拟除虫菊酯系杀虫剂的合成原料被广泛利用。菊酸存在4种异构体((1r,3r)-菊酸、(1r,3s)-菊酸、(1s,3s)-菊酸和(1s,3r)-菊酸),但(1r,3r)-菊酸的杀虫活性最高。工业上利用化学合成法制造菊酸。另一方面,还利用菊酸酯酶,从而通过酶反应制造菊酸。将关于菊酸酯酶的过去的报告示于以下(专利文献1、非专利文献1~3)。专利文献1中记载了来自球形节杆菌(arthrobacterglobiformis)的酯酶将1r,3r((+)-trans)菊酸乙酯特异性地分解(表1)。另外,非专利文献1中记载了利用来自球形节杆菌的酯酶、其它来源的酯酶将1r,3r((+)-trans)菊酸乙酯特异性地分解或者将1s,3r((-)-trans)菊酸乙酯特异性地分解。非专利文献2中记载了作为菊酸的各异构体的杀虫效果,1r,3r((+)-trans)和1r,3s((+)-cis)显示不同的杀虫效果。非专利文献3为关于利用来自球形节杆菌的酯酶分解菊酸乙酯的研究报告,通过特定的变异(k62r突变)而显示立体选择性发生变化(对(1s,3r)-菊酸乙酯显示反应性)。
现有技术文献
专利文献
专利文献1:日本特开平5-56787号公报
非专利文献
非专利文献1:agric.biol.chem.,55(11),2865-2870,1991
非专利文献2:最近的合成拟除虫菊酯和其立体异构体的化学和作用,吉冈宏辅、有机合成化学第38卷第12号(1980)、1151-1162
非专利文献3:关于使用微生物酶的光学活性拟除虫菊酯中间体的制造的研究,奈良尖端科学技术大学院大学物质创成科学研究科、西泽雅子、2011年3月
技术实现要素:
作为菊酸的异构体之一的(1r,3s)-菊酸也具有杀虫活性,其自身是有用的。高效地生成(1r,3s)-菊酸的酶(即,对(1r,3s)-菊酸乙酯的选择性高的酯酶)是未知的,目前通过菊酸乙酯异构体混合物的化学水解和之后的光学拆分(利用手性柱的纯化)得到(1r,3s)-菊酸。从收率、成本的方面或排水限制的观点考虑,希望利用酶制造(1r,3s)-菊酸,但并没有报告选择性或特异性地作用于(1r,3s)-菊酸乙酯的酶。因此,本发明的课题在于提供对(1r,3s)-菊酸的制造有用的酯酶。
为了解决上述课题,本发明人等尝试了来自节杆菌属微生物(球形节杆菌)的酯酶的通过蛋白质工程学方法进行的立体选择性的修饰。该酯酶对(1r,3r)-菊酸乙酯是特异性的,事实上无法用于(1r,3s)-菊酸的制造。重复多次试验最终成功地确定了对针对(1r,3s)-菊酸乙酯的反应性或选择性的提高有效的多个变异点(氨基酸残基),得到有用性高的变异体(修饰型酯酶)。特别是2个位置的氨基酸被取代的变异体(a328g/a221f)是特异性地作用于(1r,3s)-菊酸乙酯的酶,其有用性和利用价值极高。应予说明,该变异体中也包含的变异a328g也可以说是规定底物的立体选择性的重要变异。
以下的发明基于以上的成果和考察。
[1]一种修饰型酯酶,具有在序列号1的氨基酸序列中包含以下的(1)~(23)中的任一个氨基酸取代的氨基酸序列或与该氨基酸序列显示90%以上同源性的氨基酸序列(其中,在所述氨基酸取代的位置以外的部分产生氨基酸序列的差异),与由序列号1的氨基酸序列构成的酯酶相比,对(1r,3s)-菊酸乙酯的反应性提高,
(1)变异点为f58,取代后的氨基酸为e、i、c、a、m或y,
(2)变异点为d227,取代后的氨基酸为i、s、a、w、h、g、f或v,
(3)变异点为i228,取代后的氨基酸为y、s、t或w,
(4)变异点为l229,取代后的氨基酸为y、f或v,
(5)变异点为l231,取代后的氨基酸为m、a或g,
(6)变异点为s244,取代后的氨基酸为f,
(7)变异点为a245,取代后的氨基酸为n或g,
(8)变异点为v297,取代后的氨基酸为r、w、q、a或i,
(9)变异点为f298,取代后的氨基酸为s,
(10)变异点为a328,取代后的氨基酸为n,
(11)变异点为a328,取代后的氨基酸为g,
(12)变异点为a328和f58,变异点a328的取代后的氨基酸为g、变异点f58的取代后的氨基酸为l或e,
(13)变异点为a328和l151,变异点a328的取代后的氨基酸为g、变异点l151的取代后的氨基酸为q、r、h、w、a、v、m、n、c、t或i,
(14)变异点为a328和f217,变异点a328的取代后的氨基酸为g、变异点f217的取代后的氨基酸为w、g、a、d、t、r、e、v或y,
(15)变异点为a328和s220,变异点a328的取代后的氨基酸为g、变异点s220的取代后的氨基酸为q、l、n或g,
(16)变异点为a328和a221,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、m、l、v、e、r、g或s,
(17)变异点为a328和d227,变异点a328的取代后的氨基酸为g、变异点d227的取代后的氨基酸为e或c,
(18)变异点为a328和i228,变异点a328的取代后的氨基酸为g、变异点i228的取代后的氨基酸为l或g,
(19)变异点为a328和l229,变异点a328的取代后的氨基酸为g、变异点l229的取代后的氨基酸为r、v、i或h,
(20)变异点为a328和v297,变异点a328的取代后的氨基酸为g、变异点v297的取代后的氨基酸为c或l,
(21)变异点为a328、a221和d326,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v,
(22)变异点为a328、a221、d326和f298,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v、变异点f298的取代后的氨基酸为l,
(23)变异点为a328、a221、d326、f298和n222,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v、变异点f298的取代后的氨基酸为l、变异点n222的取代后的氨基酸为d。
[2]根据[1]所述的修饰型酯酶,其中,(1)的取代后的氨基酸为e、i、c、a或m,
(2)的取代后的氨基酸为i、s、a、w、h或g,
(3)的取代后的氨基酸为y或s,
(4)的取代后的氨基酸为y或f,
(5)的取代后的氨基酸为m或a,
(8)的取代后的氨基酸为r、w或q,
(12)的变异点f58的取代后的氨基酸为l,
(13)的变异点l151的取代后的氨基酸为q或r,
(14)的变异点f217的取代后的氨基酸为w、g、a、d、t、r或e,
(15)的变异点s220的取代后的氨基酸为q或l,
(16)的变异点a221的取代后的氨基酸为f、m、l、v、e、r或g,
(19)的变异点l229的取代后的氨基酸为r、v或i。
[3]根据[1]所述的修饰型酯酶,其中,(4)的取代后的氨基酸为y,
(5)的取代后的氨基酸为m,
(8)的取代后的氨基酸为r或w,
(13)的变异点l151的取代后的氨基酸为q,
(14)的变异点f217的取代后的氨基酸为w、g、a、d或t,
(16)的变异点a221的取代后的氨基酸为f、m、l、v、e或r。
[4]根据[1]所述的修饰型酯酶,其中,氨基酸取代为(11)~(23)中的任一个,对(1r,3s)-菊酸乙酯的选择性提高。
[5]根据[4]所述的修饰型酯酶,其中,氨基酸取代为a328g、a328g/f217w、a328g/a221m、a328g/a221l、a328g/a221v或a328g/i228l。
[6]根据[4]所述的修饰型酯酶,其中,氨基酸取代为a328g/a221f,特异性地作用于(1r,3s)-菊酸乙酯。
[7]根据[4]所述的修饰型酯酶,其中,氨基酸取代为a328g/a221f/d326v。
[8]根据[4]所述的修饰型酯酶,其中,氨基酸取代为a328g/a221f/d326v/f298l。
[9]根据[4]所述的修饰型酯酶,其中,氨基酸取代为a328g/a221f/d326v/f298l/n222d。
[10]根据[1]~[9]中任一项所述的修饰型酯酶,其中,所述同源性为95%以上。
[11]根据[1]所述的修饰型酯酶,由序列号2~11中的任一氨基酸序列构成。
[12]一种基因,编码[1]~[11]中任一项所述的修饰型酯酶。
[13]根据[12]所述的基因,包含序列号13~22中的任一碱基序列。
[14]一种重组dna,包含[12]或[13]所述的基因。
[15]一种微生物,保有[14]所述的重组dna。
[16]一种酶剂,包含[1]~[11]中任一项所述的修饰型酯酶。
[17]一种修饰型酯酶的制备方法,包括以下的步骤(i)~(iii):
(i)准备编码[1]~[11]中任一项的修饰型酯酶的氨基酸序列的核酸的步骤,
(ii)使所述核酸表达的步骤,以及
(iii)回收表达产物的步骤。
[18]根据[17]所述的制备方法,其中,所述氨基酸序列为序列号2~11中的任一氨基酸序列。
[19]根据[17]所述的制备方法,其中,所述核酸包含序列号13~22中的任一碱基序列。
[20]一种(1r,3s)-菊酸的制造方法,其特征在于,使[1]~[11]中任一项的修饰型酯酶作用于(1r,3s)-菊酸乙酯。
[21]根据[20]所述的制造方法,其中,(1r,3s)-菊酸乙酯为与(1r,3r)-菊酸乙酯、(1s,3s)-菊酸乙酯和(1s,3r)-菊酸乙酯共存的状态。
附图说明
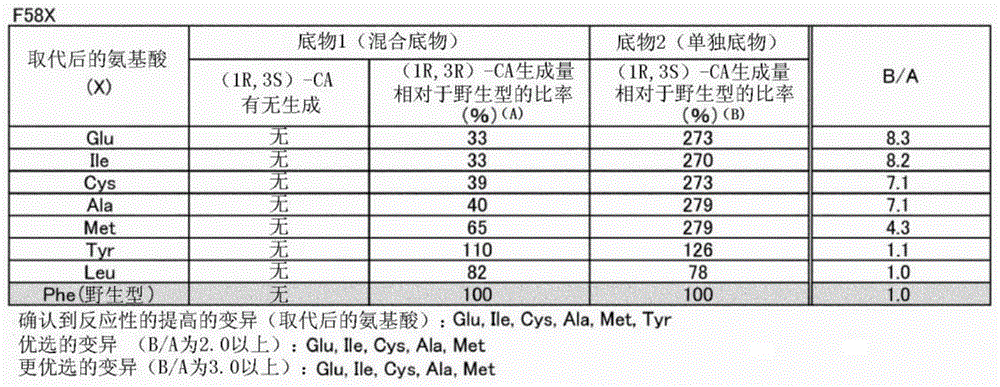
图1是变异f58x的评价结果。利用“使用(1r,3s)-菊酸乙酯(单独底物)的反应中的(1r,3s)-菊酸的生成量相对于野生型酶的比率(%)”(b)除以“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于野生型酶的比率(%)”(a)而得到的值(b/a)来评价“对(1r,3s)-菊酸乙酯的反应性”(对于以下的突变也同样)。
图2是变异f217x、s220x、d227x的评价结果。
图3是变异i228x、l229x、l231x的评价结果。
图4是变异s244x、a245x、v297x的评价结果。
图5是变异f298x、a328x的评价结果。
图6是组合变异a328g/f58x、a328g/l151x、a328g/f217x的评价结果。利用“使用混合底物的反应中的(1r,3s)-菊酸的生成量相对于a328g变异体的比率(%)”(c)除以“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于a328g变异体的比率(%)”(d)而得到的值(c/d)来评价“对(1r,3s)-菊酸乙酯的选择性”(对于以下的组合变异也同样)。还示出了(1r,3s)-菊酸的生成量/(1r,3r)-菊酸的生成量的值。
图7是组合变异a328g/s220x、a328g/a221x、a328g/d227x的评价结果。
图8是组合变异a328g/i228x、a328g/l229x、a328g/v297x的评价结果。
图9是组合变异a328g/a221f、a328g/a221f/d326v、a328g/a221f/d326v/f298l、a328g/a221f/d326v/f298l/n222d的评价结果。
具体实施方式
为了方便说明,以下对与本发明相关而使用的术语的一部分进行定义。
(术语)
术语“修饰型酯酶”是将作为基准的酯酶(以下,称为“基准酯酶”)修饰或变异而得到的酶。基准酯酶典型而言是具有序列号1的氨基酸序列的来自节杆菌属微生物(球形节杆菌)的酯酶。
在本发明中,作为修饰或变异,进行“氨基酸的取代”。因此,将修饰型酯酶和基准酯酶进行比较时,确认到一部分氨基酸残基有差异。应予说明,在本说明书中,将修饰型酯酶也称为修饰型酶或变异体。
在本说明书中,按照惯例,如下所示,将各氨基酸用1个字母表示。
甲硫氨酸:m、丝氨酸:s、丙氨酸:a、苏氨酸:t、缬氨酸:v、酪氨酸:y、亮氨酸:l、天冬酰胺:n、异亮氨酸:i、谷氨酰胺:q、脯氨酸:p、天冬氨酸:d、苯丙氨酸:f、谷氨酸:e、色氨酸:w、赖氨酸:k、半胱氨酸:c、精氨酸:r、甘氨酸:g、组氨酸:h
在本说明书中,将n末端的氨基酸残基作为第1位,通过从n末端向c末端标出的编号来确定变异点的位置。
在本说明书中,用表示取代前的氨基酸残基的1个字母、表示变异点(发生氨基酸取代的氨基酸残基的位置)的数字和表示取代后的氨基酸残基的1个字母的组合来表达基于氨基酸取代的变异。例如,如果是328位的丙氨酸被取代为甘氨酸的变异,则表达为“a328g”。另外,在表示两个变异的组合(并用)的情况下,使用符号“/”。例如,如果是328位的丙氨酸被取代为甘氨酸的变异和221位的丙氨酸被取代为苯丙氨酸的变异的组合,则表达为“a328g/a221f”。
1.修饰型酯酶
本发明的第1方面涉及一种修饰型酯酶(修饰型酶)。本发明的修饰型酶典型而言具有在序列号1的氨基酸序列中包含一或两个以上特定的氨基酸取代(变异)的氨基酸序列。根据该特征,与由序列号1的氨基酸序列构成的酯酶相比,对(1r,3s)-菊酸乙酯的反应性或选择性或者这两者提高。应予说明,序列号1的氨基酸序列为来自球形节杆菌的酯酶的序列。
“对(1r,3s)-菊酸乙酯的反应性”是也包含与对(1r,3s)-菊酸乙酯的光学异构体((1r,3r)-菊酸乙酯、(1s,3s)-菊酸乙酯、(1s,3r)-菊酸乙酯)的反应性进行比较时的反应性(相对反应性)的表达。因此,“对(1r,3s)-菊酸乙酯的反应性的提高”也可通过对(1r,3s)-菊酸乙酯的选择性的提高、对(1r,3s)-菊酸乙酯的活性的上升、对(1r,3s)-菊酸乙酯的光学异构体((1r,3r)-菊酸乙酯、(1s,3s)-菊酸乙酯、(1s,3r)-菊酸乙酯)的活性的降低等而带来。
在此,来自球形节杆菌的酯酶(野生型酶)对(1r,3r)-菊酸乙酯具有高选择性,4种光学异构体((1r,3r)-菊酸乙酯、(1r,3s)-菊酸乙酯、(1s,3s)-菊酸乙酯、(1s,3r)-菊酸乙酯)共存时,优先将(1r,3r)-菊酸乙酯水解。因此,通常,作为产物,仅检测到(1r,3r)-菊酸。另外,野生型酶也具有对(1r,3s)-菊酸乙酯的水解活性,但其反应速度与对(1r,3r)-菊酸的水解活性的反应速度相比,压倒性地慢,因此,在(1r,3r)-菊酸乙酯共存下,表观上不与(1r,3s)-菊酸乙酯反应。另一方面,如果使(1r,3s)-菊酸乙酯单独作为底物反应时,则检测到其产物(1r,3s)-菊酸。
在本申请中,考虑到野生型酶的上述特性,“对(1r,3s)-菊酸乙酯的反应性”基于仅使用(1r,3s)-菊酸乙酯作为底物(单独底物)的反应中的(1r,3s)-菊酸的生成量和使用(1r,3r)-菊酸乙酯、(1r,3s)-菊酸乙酯、(1s,3s)-菊酸乙酯和(1s,3r)-菊酸乙酯的混合物即混合底物的反应中的(1r,3r)-菊酸乙酯的生成量进行评价。具体而言,利用“使用(1r,3s)-菊酸乙酯(单独底物)的反应中的(1r,3s)-菊酸的生成量相对于野生型酶的比率(%)”除以“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于野生型酶的比率(%)”而得到的值评价“对(1r,3s)-菊酸乙酯的反应性”,该值超过1时,判定为“对(1r,3s)-菊酸乙酯的反应性”提高。在优选的修饰型酶中,该值为2.0以上,反应性提高的程度高。在进一步优选的修饰型酶中,该值为3.0以上,反应性进一步提高。应予说明,野生型酶为修饰前的酶、即基准酯酶。
如上所述,野生型酶对(1r,3r)-菊酸乙酯具有高选择性,与混合底物反应时,未检测到(1r,3s)-菊酸(实质上未生成)。另一方面,如后述的实施例所示,将野生型酶的328位氨基酸丙氨酸取代为甘氨酸的变异体(a328g变异体)对(1r,3s)-菊酸乙酯的选择性提高,即使在与混合底物反应的情况下,也显示对(1r,3s)-菊酸乙酯的水解活性,确认到(1r,3s)-菊酸的生成。因此,在本申请中,“对(1r,3s)-菊酸乙酯的选择性”的评价利用a328g变异体。具体而言,利用“使用混合底物的反应中的(1r,3s)-菊酸的生成量相对于a328g变异体的比率(%)”除以“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于a328g变异体的比率(%)”而得到的值评价“对(1r,3s)-菊酸乙酯的选择性”。该值为1以上时,为与a328g变异体同等或其以上的选择性,判定为“对(1r,3s)-菊酸乙酯的选择性”提高。在优选的修饰型酶中,该值为1.5以上,选择性提高的程度高。在进一步优选的修饰型酶中,该值为2.0以上,选择性进一步提高。应予说明,(1r,3r)-菊酸的生成量为检测限以下时,即,实质未生成(1r,3r)-菊酸时,“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于a328g变异体变异体的比率”为零,上述值无法计算。带来这样的结果的修饰型酶(a328g/a221f变异体符合)可以说是(1r,3s)-菊酸乙酯特异性。
在本说明书中,“包含氨基酸取代”是指变异点(即,产生特定的氨基酸取代的氨基酸残基的位置)为取代后的氨基酸。因此,如果将包含氨基酸取代的氨基酸序列(变异氨基酸序列)与不含氨基酸取代的序列号1的氨基酸序列(基准氨基酸序列)进行比较,则在该氨基酸取代的位置确认到氨基酸残基的差异。
以下,列举本发明的修饰型酶所含有且带来对(1r,3s)-菊酸乙酯的反应性的提高的氨基酸取代(变异点和取代后的氨基酸)。应予说明,(11)~(23)除提高对(1r,3s)-菊酸乙酯的反应性以外,也提高对(1r,3s)-菊酸乙酯的选择性,故更优选。
(1)变异点为f58,取代后的氨基酸为e、i、c、a、m或y。优选取代后的氨基酸为e、i、c、a或m。
(2)变异点为d227,取代后的氨基酸为i、s、a、w、h、g、f或v。优选取代后的氨基酸为i、s、a、w、h或g。进一步优选取代后的氨基酸为i、s、a、w或h。
(3)变异点为i228,取代后的氨基酸为y、s、t或w。优选取代后的氨基酸为y或s。
(4)变异点为l229,取代后的氨基酸为y、f或v。优选取代后的氨基酸为y或f。进一步优选取代后的氨基酸为y。
(5)变异点为l231,取代后的氨基酸为m、a或g。优选取代后的氨基酸为m或a。进一步优选取代后的氨基酸为m。
(6)变异点为s244,取代后的氨基酸为f。
(7)变异点为a245,取代后的氨基酸为n或g
(8)变异点为v297,取代后的氨基酸为r、w、q、a或i。优选取代后的氨基酸为r、w或q。进一步优选取代后的氨基酸为r或w。
(9)变异点为f298,取代后的氨基酸为s。
(10)变异点为a328,取代后的氨基酸为n。
(11)变异点为a328,取代后的氨基酸为g。
(12)变异点为a328和f58,变异点a328的取代后的氨基酸为g、变异点f58的取代后的氨基酸为l或e。优选变异点f58的取代后的氨基酸为l。
(13)变异点为a328和l151,变异点a328的取代后的氨基酸为g、变异点l151的取代后的氨基酸为q、r、h、w、a、v、m、n、c、t或i。优选变异点l151的取代后的氨基酸为q或r。进一步优选变异点l151的取代后的氨基酸为q。
(14)变异点为a328和f217,变异点a328的取代后的氨基酸为g、变异点f217的取代后的氨基酸为w、g、a、d、t、r、e、v或y。优选变异点f217的取代后的氨基酸为w、g、a、d、t、r或e。进一步优选变异点f217的取代后的氨基酸为w、g、a、d或t。
(15)变异点为a328和s220,变异点a328的取代后的氨基酸为g、变异点s220的取代后的氨基酸为q、l、n或g。优选变异点s220的取代后的氨基酸为q或l。
(16)变异点为a328和a221,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、m、l、v、e、r、g或s。优选变异点a221的取代后的氨基酸为f、m、l、v、e、r或g。进一步优选变异点a221的取代后的氨基酸为f、m、l、v、e或r。
(17)变异点为a328和d227,变异点a328的取代后的氨基酸为g、变异点d227的取代后的氨基酸为e或c。
(18)变异点为a328和i228,变异点a328的取代后的氨基酸为g、变异点i228的取代后的氨基酸为l或g。
(19)变异点为a328和l229,变异点a328的取代后的氨基酸为g、变异点l229的取代后的氨基酸为r、v、i或h。优选变异点l229的取代后的氨基酸为r、v或i。
(20)变异点为a328和v297,变异点a328的取代后的氨基酸为g、变异点v297的取代后的氨基酸为c或l。
(21)变异点为a328、a221和d326,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v。
(22)变异点为a328、a221、d326和f298,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v、变异点f298的取代后的氨基酸为l。
(23)变异点为a328、a221、d326、f298和n222,变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f、变异点d326的取代后的氨基酸为v、变异点f298的取代后的氨基酸为l、变异点n222的取代后的氨基酸为d。
以上的氨基酸取代中,对于(14)和(15),尽管变异点f217和变异点s220单独时未带来反应性的提高,但如果成为与a328g变异的组合,则对(1r,3s)-菊酸乙酯的选择性提高,值得注意。另外,对于变异点为a328和a221、变异点a328的取代后的氨基酸为g、变异点a221的取代后的氨基酸为f,带来对(1r,3s)-菊酸乙酯的选择性的飞跃性的提高,能够制备与(1r,3s)-菊酸乙酯特异性地作用的修饰型酶,是极其优选的氨基酸取代。
作为本发明的修饰型酶的具体例,可举出由序列号2~11中的任一氨基酸序列构成的酯酶(依次对应于包含(11)的氨基酸取代a328g的变异体、包含(14)的氨基酸取代之一的a328g/f217w的变异体、包含(16)的氨基酸取代之一的a328g/a221f的变异体、包含(16)的氨基酸取代之一的a328g/a221m的变异体、包含(16)的氨基酸取代之一的a328g/a221l的变异体、包含(16)的氨基酸取代之一的a328g/a221v的变异体、包含(18)的氨基酸取代之一的a328g/i228l的变异体、包含(21)的氨基酸取代之一的a328g/a221f/d326v的变异体、包含(22)的氨基酸取代之一的a328g/a221f/d326v/f298l的变异体、包含(23)的氨基酸取代之一的a328g/a221f/d326v/f298l/n222d的变异体)。如后述的实施例所示,这些变异体的(1r,3s)-菊酸乙酯选择性特别提高。其中,a328g/a221f变异体特异性地作用于(1r,3s)-菊酸乙酯,有用性特别高。另外,a328g/a221f/d326v变异体、a328g/a221f/d326v/f298l变异体、和a328g/a221f/d326v/f298l/n222d变异体除了对(1r,3s)-菊酸乙酯的选择性非常高以外,反应性也非常高,极其有用。
通常,使某一蛋白质的氨基酸序列的一部分变异时,有时变异后的蛋白质与变异前的蛋白质具有同等的功能。即,有时氨基酸序列的变异对蛋白质的功能不产生实质的影响,蛋白质的功能在变异前后被维持。另一方面,两个蛋白质的氨基酸序列的同源性高时,两者显示同等的特性的可能性高。考虑到这些技术常识,虽然不与上述修饰型酶的氨基酸序列、即“在序列号1的氨基酸序列中包含上述(1)~(23)中的任一氨基酸取代的氨基酸序列(该氨基酸序列的具体例为序列号2~11的氨基酸序列)”完全相同(即100%的同源性),但只要是具有显示高同源性的氨基酸序列且确认到期望的特性变化的修饰型酶,则可以看作与上述修饰型酶实质上相同的酶(实质相同的酯酶)。在此的同源性为60%以上、70%以上、80%以上、82%以上、85%以上、90%以上、93%以上、95%以上、98%或99%以上。同源性越高越优选。因此,在最优选的方案中,同源性为99%以上。
如果将上述修饰型酶和实质相同的酯酶进行比较,则确认到氨基酸序列的细微差异。但是,氨基酸序列的差异在实施了上述氨基酸取代的位置以外的位置产生。因此,例如,同源性的基准为序列号2的氨基酸序列时,在328位g以外的位置产生氨基酸序列的差异,同源性的基准为序列号3的氨基酸序列时,在328位g和217位w以外的位置产生氨基酸序列的差异,同源性的基准为序列号4的氨基酸序列时,在328位g和221位f以外的位置产生氨基酸序列的差异,同源性的基准为序列号5的氨基酸序列时,在328位g和221位m以外的位置产生氨基酸序列的差异,同源性的基准为序列号6的氨基酸序列时,在328位g和221位l以外的位置产生氨基酸序列的差异,同源性的基准为序列号7的氨基酸序列时,在328位g和221位v以外的位置产生氨基酸序列的差异,同源性的基准为序列号8的氨基酸序列时,在328位g和228位l以外的位置产生氨基酸序列的差异,同源性的基准为序列号9的氨基酸序列时,在328位g、221位f和326位v以外的位置产生氨基酸序列的差异,同源性的基准为序列号10的氨基酸序列时,在328位g、221位f、326位v和298位l以外的位置产生氨基酸序列的差异,同源性的基准为序列号11的氨基酸序列时,在328位g、221位f、326位v、298位l和222位d以外的位置产生氨基酸序列的差异。换言之,在与序列号2的氨基酸序列显示上述同源性(60%以上、70%以上、80%以上、82%以上、85%以上、90%以上、93%以上、95%以上、98%或99%以上)的氨基酸序列中,328位氨基酸为g。同样地,在与序列号3的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、217位氨基酸为w,在与序列号4的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为f,在与序列号5的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为m,在与序列号6的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为l,在与序列号7的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为v,在与序列号8的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、228位氨基酸为l,在与序列号9的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为f、326位氨基酸为v,在与序列号10的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为f、326位氨基酸为v、298位氨基酸为l,在与序列号11的氨基酸序列显示上述同源性的氨基酸序列中,328位氨基酸为g、221位氨基酸为f、326位氨基酸为v、298位氨基酸为l、222位氨基酸为d。
在此的“氨基酸序列的细微差异”是通过氨基酸的缺失、取代、附加、插入或它们的组合而产生的。典型而言,是指通过构成氨基酸序列的1~多个(上限例如为3个、5个、7个、10个)的氨基酸的缺失、取代、或1~多个(上限例如为3个、5个、7个、10个)的氨基酸的附加、插入、或它们的组合而在氨基酸序列产生变异(变化)。“氨基酸序列的细微差异”优选通过保守性氨基酸取代产生。在此的“保存性氨基酸取代”是指将某一氨基酸残基取代成具有同样性质的侧链的氨基酸残基。氨基酸残基根据其侧链而分类为碱性侧链(例如赖氨酸、精氨酸、组氨酸)、酸性侧链(例如天冬氨酸、谷氨酸)、非带电极性侧链(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸)、非极性侧链(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸、色氨酸)、β分支侧链(例如苏氨酸、缬氨酸、异亮氨酸)、芳香族侧链(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)之类的若干族。保守性氨基酸取代优选为同一族内的氨基酸残基间的取代。应予说明,已知来自球形节杆菌的酯酶(序列号1)的活性残基为59位氨基酸丝氨酸、325位氨基酸组氨酸、326位氨基酸天冬氨酸,因此,在实施变异时对该酸残基没有影响即可。
可是,两个氨基酸序列或两个碱基序列(以下,使用“两个序列”作为包含它们的术语)的同源性(%)例如可以通过以下的步骤来确定。首先,以能够进行最佳比较的方式排列两个序列(例如,可以向第一序列导入空位而将与第二序列的比对优化)。第一序列的特定位置的分子(氨基酸残基或核苷酸)与第二序列中的对应的位置的分子相同时,可以说该位置的分子是相同的。两个序列的同源性是在该两个序列中共同的同一位置的数量的函数(即,同源性(%)=同一位置的数量/位置的总数×100),优选也将比对的优化所需的空位的数量和尺寸考虑在内。
两个序列的比较和同源性的确定可以使用数学算法来实现。作为可用于序列的比较的数学算法的具体例,有在karlin和altschul(1990)proc.natl.acad.sci.usa87:2264-68中记载、在karlin和altschul(1993)proc.natl.acad.sci.usa90:5873-77中改变的算法,但并不限定于此。这样的算法被整合到altschul等人(1990)j.mol.biol.215:403-10中记载的nblast程序和xblast程序(版本2.0)中。为了得到等价的核苷酸序列,只要用例如nblast程序,设为score=100、wordlength=12进行blast核苷酸检索即可。为了得到等价的氨基酸序列,只要用例如xblast程序,设为score=50、wordlength=3进行blast多肽检索即可。为了得到用于比较的空位比对,可以利用altschul等人(1997)aminoacidsresearch25(17):3389-3402中记载的gappedblast。利用blast和gappedblast时,可以使用对应的程序(例如xblast和nblast)的默认参数。详细而言,请参照http://www.ncbi.nlm.nih.gov。作为可用于序列的比较的其它数学算法的例子,有myers和miller(1988)computapplbiosci.4:11-17中记载的算法。这样的算法被整合到例如可以在genestream网站服务器(ighmontpellier、法国)或isrec服务器中利用的align程序中。氨基酸序列的比较利用align程序时,例如使用pam120残基质量表,设为空位长度罚分=12、空位罚分=4。
可以使用gcg软件包的gap程序,使用blossom62matrix或pam250matrix,设为空位权重=12、10、8、6或4、空位长度权重=2、3或4来确定两个氨基酸序列的同源性。另外,可以使用gcg软件包(可以在http://www.gcg.com中利用)的gap程序,设为空位权重=50、空位长度权重=3来确定两个碱基序列的同源性。
典型而言,通过由序列号1的氨基酸序列构成的酯酶、即来自球形节杆菌的酯酶发生变异(上述(1)~(23)中的任一氨基酸取代),成为本发明的修饰型酶。对于上述的实质相同的酯酶,可以通过对由序列号1的氨基酸序列构成的酯酶发生变异(上述(1)~(23)中的任一氨基酸取代)而成的酯酶进一步加入变异、对来自与产生由序列号1的氨基酸序列构成的酯酶的球形节杆菌株同种同属的酯酶等由与序列号1的氨基酸序列同源性高的氨基酸序列构成的酯酶加入同等的变异、或对通过该变异而得到的酯酶进一步加入变异而得到。对于此处的“同等的变异”,是在与序列号1的氨基酸序列同源性高的氨基酸序列中,相当于本发明中的变异点(上述(1)~(23)中的任一氨基酸取代的变异点)的氨基酸残基的氨基酸残基被取代。应于说明,作为由与序列号1的氨基酸序列同源性高的氨基酸序列构成的酯酶的例子,可举出来自节杆菌属(arthrobactersp.)rue61a且具有序列号23的氨基酸序列的酯酶(氨基酸列的同源性81%)。
在本说明书中关于氨基酸残基使用时的术语“相当”是指在被比较的蛋白质(酶)之间,对其功能的发挥带来同等的贡献。例如,以考虑一级结构(氨基酸序列)的部分相同性并且能够进行最佳比较的方式将比较对象的氨基酸序列相对于基准酯酶的氨基酸序列(序列号1的氨基酸序列)进行排列时(此时也可以根据需要导入空位,将比对优化),可以将与基准的氨基酸序列中的特定的氨基酸对应的位置的氨基酸确定为“相当的氨基酸”。也可以代替一级结构彼此的比较或在此基础上通过立体结构(三维结构)彼此的比较来确定“相当的氨基酸”。通过利用立体结构信息,得到可靠性高的比较结果。此时,可以采用一边比较多个酶的立体结构的原子坐标一边进行比对的方法。变异对象酶的立体结构信息例如能够从proteindatabank(http://www.pdbj.org/index_j.html)获得。
以下示出利用x射线结晶结构解析确定蛋白质立体结构的方法的一个例子。
(1)将蛋白质结晶化。结晶化是为了进行立体结构确定所必不可少的,除此以外,作为蛋白质的高纯度的纯化法、高密度下稳定的保存法也具有产业上的有用性。此时,可以将结合有底物或其类似化合物作为配体的蛋白质结晶化。
(2)对制作的结晶照射x射线来收集衍射数据。应予说明,蛋白质结晶通过x射线照射而受到破坏,衍射能力劣化的情形较多。此时,将结晶急剧地冷却到-173℃左右,在该状态下收集衍射数据的低温测定技术最近已经普及。应予说明,最终,为了收集用于结构确定的高分辨率数据,利用亮度高的同步辐射光。
(3)在进行结晶结构解析中,除衍射数据以外,还需要相位信息。对于目标蛋白质,近缘蛋白质的结晶结构未知时,利用分子取代法进行结构确定是不可能的,必须通过重原子同晶取代法解决相位问题。重原子同晶取代法是将汞、铂等原子序号大的金属原子导入结晶,利用金属原子大的x射线散射能力对于x射线衍射数据的贡献而得到相位信息的方法。被确定的相位可以通过将结晶中的溶剂区域的电子密度平滑化而改善。由于溶剂区域的水分子波动大而几乎观察不到电子密度,因此,通过将该区域的电子密度近似为0,能够接近真正的电子密度,进而能够改善相位。另外,非对称单元含有多个分子时,通过将这些分子的电子密度平均化,能够进一步大幅地改善相位。在使用如此改善的相位进行计算而得到的电子密度图中将蛋白质的模型拟合。该工艺在计算机绘图上,使用msi公司(美国)的quanta等程序进行。然后,使用msi公司的x-plor等程序进行结构精密化,完成结构解析。对于目标蛋白质,近缘蛋白质的结晶结构已知时,可以使用已知蛋白质的原子坐标并通过分子取代法来确定。分子取代和结构精密化可以使用程序cns_solvever.11等进行。
2.编码修饰型酯酶的核酸等
本发明的第2方面提供与本发明的修饰型酶相关的核酸。即,提供编码修饰型酶的基因、能够作为用于鉴定编码修饰型酶的核酸的探针使用的核酸、能够作为用于使编码修饰型酶的核酸扩增或突变等的引物使用的核酸。
编码修饰型酶的基因典型而言被用于修饰型酶的制备。根据使用编码修饰型酶的基因的基因工程制备方法,能够得到更均质状态的修饰型酶。另外,该方法在制备大量的修饰型酶时也可以说是优选的方法。应予说明,编码修饰型酶的基因的用途并不限于修饰型酶的制备。例如,作为以阐明修饰型酶的作用机制等为目的的实验用工具,或作为用于设计或制作酶的进一步修饰体的工具,也可以利用该核酸。
本说明书中“编码修饰型酶的基因”是指使其表达时可得到该修饰型酶的核酸,当然包括具有与该修饰型酶的氨基酸序列对应的碱基序列的核酸,也包含对这样的核酸附加不编码氨基酸序列的序列而成的核酸。另外,还考虑了密码子的简并。
将编码修饰型酶的基因的序列(碱基序列)的例子示于序列号13~22。这些序列如下所述编码后述的实施例所示的变异体。
序列号13:a328g变异体
序列号14:a328g/a217w变异体
序列号15:a328g/a221f变异体
序列号16:a328g/a221m变异体
序列号17:a328g/a221l变异体
序列号18:a328g/a221v变异体
序列号19:a328g/i228l变异体
序列号20:a328g/a221f/d326v变异体
序列号21:a328g/a221f/d326v/f298l变异体
序列号22:a328g/a221f/d326v/f298l/n222d变异体
使本发明的基因在宿主内表达时,可以在上述的序列(序列号13~22中的任一者)的5'末端侧附加编码信号肽的序列(信号序列)。信号序列只要根据宿主选择即可。只要是能够表达目标变异体的信号序列,则可以使用于本发明。作为可利用的信号序列,可例示编码α-因子的信号肽的序列(proteinengineering,1996,vol9,p.1055-1061)、编码α-因子受体的信号肽的序列、编码suc2蛋白质的信号肽的序列、编码pho5蛋白质的信号肽的序列、编码bgl2蛋白质的信号肽的序列、编码aga2蛋白质的信号肽的序列、编码tora(三甲胺n-氧化还原酶)的信号肽的序列、编码来自枯草芽孢杆菌的phod(磷酸酯酶)的信号肽的序列、编码来自枯草芽孢杆菌的lipa(脂肪酶)的信号肽的序列、编码来自米曲霉的高峰淀粉酶的信号肽的序列(日本特开2009-60804号公报)、编码来自解淀粉芽孢杆菌的α-淀粉酶的信号肽的序列(eur.j.biochem.155,577-581(1986))、编码来自枯草芽孢杆菌的中性蛋白酶的信号肽的序列(appliedandenvironmentalmicrobiology,apr.1995,p.1610-1613vol.61,no.4)、编码来自bacillus属细菌的纤维素酶的信号肽的序列(日本特开2007-130012号公报)、编码用于pet系的pelb的序列。
本发明的核酸可以通过将本说明书或添加的序列表所公开的序列信息作为参考,使用标准的基因工程方法、分子生物学方法、生化学方法、化学合成等而制备成分离的状态。
在本发明的其它方案中,提供与编码本发明修饰型酶的基因的碱基序列进行比较时其所编码的蛋白质的功能同等但一部分碱基序列有差异的核酸(以下,也称为“相同核酸”。另外,将规定相同核酸的碱基序列称为“相同碱基序列”)。作为相同核酸的例子,可举出将编码本发明修饰型酶的核酸的碱基序列作为基准由含有1个或多个碱基的取代、缺失、插入、附加或倒位的碱基序列构成、编码具有修饰型酶特征性的酶活性(即酯酶活性)的蛋白质的dna。碱基的取代、缺失等可以在多个部位产生。在此的“多个”根据该核酸所编码的蛋白质的立体结构中的氨基酸残基的位置、种类而不同,例如为2~40碱基,优选为2~20碱基,更优选为2~10碱基。
相同核酸相对于作为基准的碱基序列,具有例如60%以上、优选70%以上、更优选80%以上、更进一步优选85%以上、进一步优选约90%以上、更进一步优选95%以上、最优选99%以上的同源性。
如上所述的相同核酸例如通过限制性内切酶处理、利用核酸外切酶、dna连接酶等的处理、利用位置指定突变导入法(molecularcloning,thirdedition,chapter13,coldspringharborlaboratorypress,newyork)、随机突变导入法(molecularcloning,thirdedition,chapter13,coldspringharborlaboratorypress,newyork)的变异导入等而得到。另外,也可以通过紫外线照射等其它方法而得到相同核酸。
本发明的其它方案涉及具有与编码本发明修饰型酶的基因的碱基序列互补的碱基序列的核酸。本发明的再一其它方案提供相对于编码本发明修饰型酶的基因的碱基序列或与其互补的碱基序列具有至少约60%、70%、80%、90%、95%、99%、99.9%相同的碱基序列的核酸。
本发明的再一其它方案涉及具有对编码本发明修饰型酶的基因的碱基序列或与该相同碱基序列互补的碱基序列在严格的条件下进行杂交的碱基序列的核酸。在此的“严格的条件”是指形成所谓的特异性杂交、不形成非特异性杂交的条件。这样的严格的条件是本领域技术人员是公知的,例如可以参照molecularcloning(thirdedition,coldspringharborlaboratorypress,newyork)、currentprotocolsinmolecularbiology(editedbyfrederickm.ausubeletal.,1987)进行设定。作为严格的条件,例如可举出使用杂交液(50%甲酰胺、10×ssc(0.15mnacl,15mm柠檬酸钠,ph7.0)、5×denhardt溶液、1%sds、10%硫酸葡聚糖、10μg/ml的变性鲑鱼精子dna、50mm磷酸缓冲液(ph7.5)),在约42℃~约50℃孵育,然后,使用0.1×ssc、0.1%sds在约65℃~约70℃进行清洗的条件。作为进一步优选的严格的条件,例如可举出作为杂交液使用50%甲酰胺、5×ssc(0.15mnacl,15mm柠檬酸钠,ph7.0)、1×denhardt溶液、1%sds、10%硫酸葡聚糖、10μg/ml的变性鲑鱼精子dna、50mm磷酸缓冲液(ph7.5))的条件。
本发明的再一其它方案提供具有编码本发明修饰型酶的基因的碱基序列或与其互补的碱基序列的一部分的核酸(核酸片段)。这样的核酸片段能够用于对具有编码本发明修饰型酶的基因的碱基序列的核酸等进行检测、鉴定和/或扩增等。核酸片段例如以至少含有与编码本发明修饰型酶的基因的碱基序列中连续的核苷酸部分(例如约10~约100碱基长、优选为约20~约100碱基长、进一步优选为约30~约100碱基长)进行杂交的部分的方式进行设计。作为探针利用时,可以将核酸片段标记化。标记化例如可以使用荧光物质、酶、放射性同位素。
本发明的再一其它方面涉及含有本发明的基因(编码修饰型酶的基因)的重组dna。本发明的重组dna例如以载体的形态提供。本说明书中,术语“载体”是指能够将插入其中的核酸运输至细胞等靶内的核酸性分子。
根据使用目的(克隆、蛋白质的表达),还考虑宿主细胞的种类而选择适当的载体。作为以大肠杆菌作为宿主的载体,可例示pet21或其修饰体、m13噬菌体或其修饰体、λ噬菌体或其修饰体、pbr322或其修饰体(pb325、pat153、puc8等)等,作为以酵母作为宿主的载体,可例示pyepsec1、pmfa、pyes2、ppic3.5k等,作为以昆虫细胞作为宿主的载体,可例示pac、pvl等,作为以哺乳类细胞作为宿主的载体,可例示pcdm8、pmt2pc等。
本发明的载体优选为表达载体。“表达载体”是指能够将插入其中的核酸导入目标细胞(宿主细胞)内且在该细胞内能够表达的载体。表达载体通常包含插入的核酸的表达所需要的启动子序列、促进表达的增强子序列等。也可以使用含有选择标记物的表达载体。使用该表达载体时,可以利用选择标记物来确认表达载体有无导入(和其程度)。
本发明的核酸向载体的插入、选择标记物基因的插入(需要时)、启动子的插入(需要时)等可以使用标准的重组dna技术(例如,可以参照molecularcloning,thirdedition,1.84,coldspringharborlaboratorypress,newyork的使用限制性内切酶和dna连接酶的众所周知的方法)进行。
作为宿主细胞,从处理容易性的方面考虑,可以使用曲霉(例如米曲霉)、芽孢杆菌属细菌(例如枯草芽孢杆菌)、大肠杆菌(大肠杆菌)、出芽酵母(酿酒酵母)等微生物,只要是重组dna能够复制且修饰型酶的基因能够表达的宿主细胞就可以利用。可以优选使用大肠杆菌(大肠杆菌)、出芽酵母(酿酒酵母)。作为大肠杆菌的例子,利用t7系启动子时,可举出大肠杆菌bl21(de3),不是这种情况时,可举出大肠杆菌jm109。另外,作为出芽酵母的例子,可举出出芽酵母shy2、出芽酵母ah22或出芽酵母invsc1(invitrogen公司)。
本发明的其它方面涉及保有本发明的重组dna的微生物(即转化体)。本发明的微生物可以通过使用了上述本发明载体的转染或转化而得到。例如,可通过氯化钙法(journalofmolecularbiology(j.mol.biol.)、第53卷、第159页(1970))、hanahan法(journalofmolecularbiology、第166卷、第557页(1983))、sem法(gene、第96卷、第23页(1990))、chung等的方法(proceedingsofthenationalacademyofsciencesoftheusa、第86卷、第2172页(1989))、磷酸钙共沉降法、电穿孔(potter,h.etal.,proc.natl.acad.sci.u.s.a.81,7161-7165(1984))、脂转染(felgner,p.l.etal.,proc.natl.acad.sci.u.s.a.84,7413-7417(1984))等来实施。应予说明,本发明的微生物能够用于生产本发明的修饰型酶。
3.含有修饰型酯酶的酶剂
本发明的修饰型酶例如可以以酶剂的形态提供。酶剂除有效成分(本发明的修饰型酶)以外,还可以含有赋形剂、缓冲剂、悬浮剂、稳定剂、保存剂、防腐剂、生理盐水等。作为赋形剂,可以使用淀粉、糊精、麦芽糖、海藻糖、乳糖、d-葡萄糖、山梨糖醇、d-甘露醇、白糖、甘油等。作为缓冲剂,可以使用磷酸盐、柠檬酸盐、乙酸盐等。作为稳定剂,可以使用丙二醇、抗坏血酸等。作为保存剂,可以使用苯酚、苯扎氯铵、苄醇、氯丁醇、对羟基苯甲酸甲酯等。作为防腐剂,可以使用乙醇、苯扎氯铵、对羟基苯甲酸、氯丁醇等。
4.修饰型酯酶的制备方法
本发明的又一方面涉及修饰型酶的制备方法。在本发明的制备方法的一个方案中,通过基因工程方法制备本发明人等成功取得的修饰型酶。在该方案的情况下,准备编码序列号2~11中任一氨基酸序列的核酸(步骤(i))。在此,“编码特定的氨基酸序列的核酸”是使其表达时得到具有该氨基酸序列的多肽的核酸,由与该氨基酸序列对应的碱基序列构成的核酸当然可以,也可以对这样的核酸附加多余的序列(可以为编码氨基酸序列的序列,也可以为不编码氨基酸序列的序列)。另外,也可以考虑密码子的简并。“编码序列号2~11中任一氨基酸序列的核酸”可以通过参照本说明书或附加的序列表所公开的序列信息,使用标准的基因工程方法、分子生物学方法、生物化学手法等制备成分离的状态。在此,序列号2~11的氨基酸序列均是对来自球形节杆菌的酯酶的氨基酸序列实施变异而得到的氨基酸序列。因此,通过对编码来自球形节杆菌的酯酶的基因施加必要的变异,也能够得到编码序列号2~11中的任一氨基酸序列的核酸(基因)。用于位置特异性碱基序列取代的方法在该技术领域中已知有很多(例如,参照molecularcloning,thirdedition,coldspringharborlaboratorypress,newyork),可以从其中选择适当的方法而使用。作为位置特异性变异导入法,可以采用位置特异性氨基酸饱和变异法。位置特异性氨基酸饱和变异法是以蛋白质的立体结构为基础,推定与所要求的功能相关的位置,导入氨基酸饱和变异的“semi-rational,semi-random”方法(j.mol.biol.331,585-592(2003))。例如可以使用quickchange(stratagene公司)等试剂盒、overlapextentionpcr(nucleicacidres.16,7351-7367(1988))导入位置特异性氨基酸饱和变异。pcr中使用的dna聚合酶可以使用taq聚合酶等。其中,优选使用kod-plus-(东洋纺公司)、pfuturbo(stratagene社)等精度高的dna聚合酶。
步骤(i)之后,使准备的核酸表达(步骤(ii))。例如,首先准备插入有上述核酸的表达载体,使用其将宿主细胞进行转化。
接着,在产生表达产物即修饰型酶的条件下培养转化体。转化体的培养只要依照常规方法进行即可。作为培养基中使用的碳源,只要是可同化的碳化合物即可,例如可以使用葡萄糖、蔗糖、乳糖、麦芽糖、糖蜜、丙酮酸等。另外,作为氮源,只要是可利用的氮化合物即可,例如可以使用蛋白胨、肉提取物、酵母提取物、酪蛋白水解物、豆粕碱性提取物等。此外,可以根据需要使用磷酸盐、碳酸盐、硫酸盐、镁、钙、钾、铁、锰、锌等盐类、特定的氨基酸、特定的维生素等。
另一方面,培养温度可以在30℃~40℃的范围内(优选为37℃附近)进行设定。培养时间可以考虑培养对象的转化体的生长特性、修饰型酶的产生特性等而进行设定。培养基的ph在转化体生长且产生酶的范围内进行制备。优选使培养基的ph为6.0~9.0左右(优选为ph7.0附近)。
接下来,回收表达产物(修饰型酶)(步骤(iii))。也将含有培养后的菌体的培养液直接或者在经过浓缩、杂质除去等后作为酶溶液利用,但通常从培养液或菌体暂时回收表达产物。表达产物如果为分泌型蛋白质,则可以从培养液回收,如果是其以外的蛋白质,则可以从菌体内回收。从培养液回收时,例如过滤培养上清液,进行离心处理而除去不溶物后,通过将减压浓缩、膜浓缩、利用硫酸铵或硫酸钠的盐析、利用甲醇、乙醇或丙酮等的分别沉淀法、透析、加热处理、等电点处理、凝胶过滤、吸附色谱法、离子交换色谱法、亲和色谱法等各种色谱法(例如,利用葡聚糖(sephadex)凝胶(gehealthcarescience)等的凝胶过滤、deae琼脂糖cl-6b(gehealthcarescience)、辛基琼脂糖cl-6b(gehealthcarescience)、cm琼脂糖cl-6b(gehealthcarescience))等组合而进行分离、纯化,由此能够得到修饰型酶的纯化品。另一方面,从菌体内回收时,通过将培养液进行过滤、离心处理等而采取菌体,接着,通过加压处理、超声波处理、物理破碎碎处理等机械方法或利用溶菌酶等的酶方法将菌体破坏后,与上述同样地进行分离、纯化,由此能够得到修饰型酶的纯化品。
也可以将如上得到的纯化酶通过例如冷冻干燥、真空干燥或喷雾干燥等进行粉末化而提供。此时,可以将纯化酶预先溶解于磷酸缓冲液、三乙醇胺缓冲液、tris盐酸缓冲液、good的缓冲液。可以优选使用磷酸缓冲液、三乙醇胺缓冲液。应予说明,在此作为good的缓冲液,可举出pipes、mes或mops。
通常如上利用适当的宿主-载体系统进行基因的表达~表达产物(修饰型酶)的回收,但也可以利用无细胞合成系统。在此,无细胞合成系统(无细胞转录系统、无细胞转录/翻译系统)”是指不使用活细胞,使用来自活细胞的(或通过基因工程方法得到的)核糖体、转录·翻译因子等,根据作为模板的核酸(dna、mrna)在体外合成其所编码的mrna、蛋白质。在无细胞合成系统中,通常使用根据需要将细胞破碎液纯化而得到的细胞提取液。在细胞提取液中,通常含有蛋白质合成所需要的核糖体、起始因子等各种因子、trna等各种酶。进行蛋白质的合成时,在该细胞提取液中添加各种氨基酸、atp、gtp等能量源、磷酸肌酸等蛋白质的合成所需要的其它物质。当然,在蛋白质合成时,可以根据需要补充另外准备的核糖体、各种因子和/或各种酶等。
也报告了将蛋白质合成所需要的各分子(因子)重建的转录/翻译系统的开发(shimizu,y.etal.:naturebiotech.,19,751-755,2001)。在该合成系统中,从大肠杆菌基因组将由构成细菌的蛋白质合成系统的3种起始因子、3种延伸因子、与终止相关的4种因子、使各氨基酸与trna结合的20种氨基酰基trna合成酶以及甲硫氨酰trna甲酰转移酶组成的31种因子的基因进行扩增,使用它们在体外重建蛋白质合成系统。在本发明中可以利用这样重建的合成系统。
术语“无细胞转录/翻译系统”可以与无细胞蛋白质合成系统、体外翻译系统或体外转录/翻译系统交换使用。在体外翻译系统中,使用rna作为模板来合成蛋白质。作为模板rna,可以使用总rna、mrna、体外转录产物等。在其他的体外转录/翻译系统中,使用dna作为模板。模板dna应含有核糖体结合区域,另外,优选含有适当的终止子序列。应予说明,在体外转录/翻译系统中,以转录反应和翻译反应连续进行的方式设定添加各反应所需要的因子的条件。
5.修饰型酯酶的用途
本发明的再一方面涉及修饰型酶的用途,提供使用本发明的修饰型酶的(1r,3s)-菊酸的制造方法。在本发明的制造方法中,使本发明的修饰型酶作用于(1r,3s)-菊酸乙酯而进行水解,生成(1r,3s)-菊酸。只要可实现期望的水解反应,则反应条件没有特别限定。例如,可以利用甘氨酸-naoh、pipes等缓冲液,在ph6~11、温度40℃~60℃的条件下进行反应。作为底物,可以使用纯度高的(1r,3s)-菊酸乙酯(例如纯度为98%以上),或者也可以使用(1r,3r)-菊酸乙酯、(1s,3s)-菊酸乙酯和(1s,3r)-菊酸乙酯共存的状态的底物(例如、(1r,3r):(1s,3s):(1r,3s):(1s,3r)=3:3:2:2的混合菊酸乙酯等)。反应产物根据需要进行光学拆分后,被用于各种用途。光学拆分可以使用柱色谱法。修饰型酶可以使用经固定化的修饰型酶。
实施例
来自球形节杆菌的野生型酯酶(将氨基酸序列示于序列号1,将基因序列示于序列号12)对(1r,3r)-菊酸乙酯具有高选择性。尝试了该酯酶的通过蛋白质工程方法的立体选择性的改变。
1.变异体文库的制作
通过以下的方法制作变异体文库(castinglibrary和2点组合变异体)。
(1)变异的导入·转化
设计变异导入用pcr引物,在以下的条件下进行pcr,导入变异。
<模板质粒>
pet21b(导入野性型酶基因)约30ng/μl
<pcr反应组成>(总量20μl)
5×primestargxlbuffer(takarabio):4μl
dntpmixture(2.5mmeach):1.6μl
模板(约30ng/μl):0.25μl
f-引物:0.2μl
r-引物:0.2μl
primestargxldnapolymerase(takarabio):0.8μl
将以上成分混合,用超纯水(milliq水)调整为20μl
<pcr条件>
在98℃反应1分钟
将在98℃反应10秒、在60℃反应15秒和在68℃反应2分钟进行20个循环
在68℃反应5分钟
在4℃放置
在pcr反应液15μl中添加dpni1.5μl进行处理(37℃、3小时以上)。接着,使用dpni处理液2μl,通过以下的连接反应组成进行连接处理(16℃、o/n)。
<连接反应组成>(总量5.5μl)
dpni处理pcr产物:2μl
t4多核苷酸激酶:1μl
ligationhigh(东洋纺):2.5μl
使用连接反应液5.5μl,转化(在42℃处理45秒后,在冰上骤冷)于e.colibl21(de3)25μl。将转化溶液约30μl涂布于添加有氨苄青霉素(最终浓度0.1mg/ml)的lb琼脂培养基板上,以37℃、o/n进行培养(前培养)。
(2)酶提取液的取得
将前培养后的各变异株接种于添加有氨苄青霉素(最终浓度0.1mg/ml)的tb培养基(invitrogen)1ml。在33℃培养48小时(在培养开始24小时的时刻添加iptg(最终浓度0.1mm)),进行离心分离(3000g×10分钟)。除去上清液并回收菌体后,利用溶菌剂将菌体溶菌(25℃、2小时以上)而提取酶。将溶菌液进行离心分离(3000g×10分钟)后,回收上清液而作为酶提取液。
(3)组合变异体的制作
以a328g变异体作为模板,通过与上述(1)同样的操作进行pcr。将pcr产物回收,通过与上述(2)同样的操作得到酶提取液。应予说明,基于castinglibrary的评价结果和利用moe(molsis公司)的对接模拟的结果选定与a328g组合的变异点的候选。
(4)多重变异体的制作
以a328g/a221f变异体作为模板,通过error-pronepcr进行随机变异导入。使用反式-pnp菊酸,以游离的pnp量为指标进行筛选。以得到的变异体为模板重复同样的变异导入和筛选,实施多次,由此,制作对(1r,3s)-菊酸乙酯的反应性增加了的多重变异体。
2.变异体的评价
2-1.castinglibrary的评价
通过以下的方法评价castinglibrary。为了评价对(1r,3s)-菊酸乙酯的反应性的变化,使用以下的2种底物。
底物1(混合底物):菊酸乙酯(纯度95%以上、(1r,3r)体、(1r,3s)体、(1s,3r)体、(1s,3s)体的混合物((1r,3r):(1s,3s):(1r,3s):(1s,3r)=3:3:2:2)、东京化学工业)
底物2(单独底物):(1r,3s)-菊酸乙酯
将1μl的底物(底物1或底物2)分注于反应容器,加入测定样品(上述1.(2)中制备的酶提取液)99μl,使其反应(40℃、24小时)。反应后,加入6nhcl10μl和己烷150μl。充分搅拌后,进行离心分离,采取有机层100μl。将提取物干固,再溶解于分析用溶剂后,用超临界流体色谱法(sfc)进行分析。将分析条件示于以下。
<超临界流体色谱仪(sfc)的分析条件>
装置:acquityupc2pda系统(waters公司)
柱:acquityupc2trefoilamy1(waters公司)2.5μm,3.0×150mm
流动相:co2/10mm含有乙酸铵的2-丙醇(0.5%乙酸)=92/8
流量:2.0ml/分钟
注入量:1μl
分析时间:1.5分钟
pda检测机:205-400nm
分辨率:1.2nm
以野生型酶的反应性作为基准评价对(1r,3s)-菊酸乙酯的反应性的变化。具体而言,由sfc的区域面积算出“使用(1r,3s)-菊酸乙酯(单独底物)的反应中的(1r,3s)-菊酸的生成量相对于野生型酶的比率(%)”/“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于野生型酶的比率(%)”的值,如果该值超过1,则判断为对(1r,3s)-菊酸乙酯的反应性增加。
将评价结果示于图1~5。作为对(1r,3s)-菊酸乙酯的反应性增加的变异,鉴定有以下的变异。
f58e、f58i、f58c、f58a、f58m、f58y
d227i、d227s、d227a、d227w、d227h、d227g、d227f、d227v
i228y、i228s、i228t、i228w
l229y、l229f、l229v
l231m、l231a、l231g
s244f
a245n、a245g
v297r、v297w、v297q、v297a、v297i
f298s
a328n、a328g
以下的变异显示2.0以上的值,对于对(1r,3s)-菊酸乙酯的反应性的提高更优选。
f58e、f58i、f58c、f58a、f58m
d227i、d227s、d227a、d227w、d227h、d227g
i228y、i228s
l229y、l229f
l231m、l231a
v297r、v297w、v297q
a328g
以下的变异的值为3.0以上,可以说更进一步优选。应予说明,a328g在使用底物1(混合底物)的反应中也确认到(1r,3s)-菊酸的生成,是特别有效的变异。
f58e、f58i、f58c、f58a、f58m
d227i、d227s、d227a、d227w、d227h
i228y、i228s
l229y
l231m
v297r、v297w
a328g
2-2.组合变异体的评价
将1μl的底物1分注于反应容器,加入测定样品(上述1.(3)中制备的酶提取液)99μl,使其反应(40℃、24小时)。反应后,加入6nhcl10μl和己烷150μl。充分搅拌后,进行离心分离,采取有机层100μl。将提取物干固,再溶解于分析用溶剂后,通过超临界流体色谱法(sfc)进行分析。分析条件与castinglibrary的评价的情况同样。
以具有带来对(1r,3s)-菊酸乙酯的选择性的提高的变异(a328g)的变异酶(a328g变异体)的反应性/选择性作为基准来评价对(1r,3s)-菊酸乙酯的选择性的变化。具体而言,由sfc的区域面积算出“使用混合底物的反应中的(1r,3s)-菊酸的生成量相对于a328g变异体的比率(%)”/“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于a328g变异体的比率(%)”的值,如果该值为1以上,则判断是对(1r,3s)-菊酸乙酯的选择性的提高有效的变异。
将评价结果示于图6~8。作为对(1r,3s)-菊酸乙酯的选择性的提高有效的变异,鉴定有以下的变异的组合。应予说明,令人吃惊的是,a328g/f217e、a328g/f217v、a328g/f217y、a328g/s220q、a328g/s220l、a328g/s220n和a328g/s220g尽管变异点f217和变异点s220单独时未带来反应性的提高,但如果是与a328g变异的组合,则对(1r,3s)-菊酸乙酯的选择性提高。
a328g/f58l、a328g/f58e
a328g/l151q、a328g/l151r、a328g/l151h、a328g/l151w、a328g/l151a、a328g/l151v、a328g/l151m、a328g/l151n、a328g/l151c、a328g/l151t、a328g/l151i
a328g/f217w、a328g/f217g、a328g/f217a、a328g/f217d、a328g/f217t、a328g/f217r、a328g/f217e、a328g/f217v、a328g/f217y
a328g/s220q、a328g/s220l、a328g/s220n、a328g/s220g
a328g/a221f、a328g/a221m、a328g/a221l、a328g/a221v、a328g/a221e、a328g/a221r、a328g/a221g、a328g/a221s
a328g/d227e、a328g/d227c
a328g/i228l、a328g/i228g
a328g/l229r、a328g/l229v、a328g/l229i、a328g/l229h
a328g/v297c、a328g/v297l
以下的变异的组合显示1.5以上的值,对于对(1r,3s)-菊酸乙酯的选择性的提高更优选。
a328g/f58l
a328g/l151q、a328g/l151r
a328g/f217w、a328g/f217g、a328g/f217a、a328g/f217d、a328g/f217t、a328g/f217r、a328g/f217e
a328g/s220q、a328g/s220l
a328g/a221f、a328g/a221m、a328g/a221l、a328g/a221v、a328g/a221e、a328g/a221r、a328g/a221g
a328g/i228l、a328g/i228g
a328g/l229r、a328g/l229v、a328g/l229i
以下的变异的组合的值为2.0以上,可以说更进一步优选。应予说明,特别应注意的是变异a328g/a221f,对(1r,3s)-菊酸乙酯显示特异性的反应性。
a328g/l151q
a328g/f217w、a328g/f217g、a328g/f217a、a328g/f217d、a328g/f217t
a328g/a221f、a328g/a221m、a328g/a221l、a328g/a221v、a328g/a221e、a328g/a221r
a328g/i228l、a328g/i228g
2-3.多重变异体的评价
将2μl的底物1分注于反应容器,加入测定样品(上述1.(3)中制备的酶提取液)98μl,使其反应(40℃、24小时)。反应后,加入6nhcl10μl和己烷150μl。充分搅拌后,进行离心分离,采取有机层100μl。将提取物干固,再溶解于分析用溶剂后,通过超临界流体色谱法(sfc)进行分析。分析条件与castinglibrary的评价的情况同样。
以具有带来对(1r,3s)-菊酸乙酯的选择性的提高的变异(a328g)的变异酶(a328g变异体)的反应性/选择性作为基准来评价对(1r,3s)-菊酸乙酯的选择性的变化。具体而言,由sfc的区域面积算出“使用混合底物的反应中的(1r,3s)-菊酸的生成量相对于a328g变异体的比率(%)”/“使用混合底物的反应中的(1r,3r)-菊酸的生成量相对于a328g变异体的比率(%)”的值,如果该值为1以上,则判断是对(1r,3s)-菊酸乙酯的选择性的提高有效的变异,(1r,3r)-菊酸乙酯的生成量相对于a328g变异体的比率越高,越是对(1r,3s)-菊酸乙酯的生产率提高有用的变异。
将评价结果示于图9。作为对(1r,3s)-菊酸乙酯的选择性和反应性非常高的变异,鉴定有以下的变异的组合。
a328g/a221f/d326v
a328g/a221f/d326v/f298l
a328g/a221f/d326v/f298l/n222d
3.总结
成功鉴定了对于对(1r,3s)-菊酸乙酯的反应性/选择性的提高有效的多个变异。a328g/a221f变异体显示(1r,3s)-菊酸乙酯特异性的反应性,是极具特征的。a328g/a221f/d326v/f298l/n222d变异体除了对(1r,3s)-菊酸乙酯的选择性大幅提高以外,反应性也大幅提高,极其有用。将包含这些变异体在内含有特别有效的变异的变异体的氨基酸序列和编码其的基因(一个例子)示于以下。
a328g变异体:序列号2(氨基酸序列)、序列号13(基因序列)
a328g/f217w变异体:序列号3(氨基酸序列)、序列号14(基因序列)
a328g/a221f变异体:序列号4(氨基酸序列)、序列号15(基因序列)
a328g/a221m变异体:序列号5(氨基酸序列)、序列号16(基因序列)
a328g/a221l变异体:序列号6(氨基酸序列)、序列号17(基因序列)
a328g/a221v变异体:序列号7(氨基酸序列)、序列号18(基因序列)
a328g/i228l变异体:序列号8(氨基酸序列)、序列号19(基因序列)
a328g/a221f/d326v变异体:序列号9(氨基酸序列)、序列号20(基因序列)
a328g/a221f/d326v/f298l变异体:序列号10(氨基酸序列)、序列号21(基因序列)
a328g/a221f/d326v/f298l/n222d变异体:序列号11(氨基酸序列)、序列号22(基因序列)
产业上的可利用性
本发明的修饰型酯酶对(1r,3s)-菊酸乙酯的反应性高,对于(1r,3s)-菊酸的制造有用。通过使用本发明的修饰型酯酶,能够通过酶法有效地制造(1r,3s)-菊酸,可解决以往的制造法(化学水解)所具有的问题。
本发明并不受上述发明的实施方式和实施例的说明任何限定。在不脱离要求保护范围的记载且本领域技术人员能够容易地想到的范围的各种变形方案也包含在本发明中。本说明书中所示的论文、公开专利公报和专利公报等内容通过援引其全部内容而进行引用。
序列表自由文本
序列号2:人工序列的说明:a328g变异体
序列号3:人工序列的说明:a328g/f217w变异体
序列号4:人工序列的说明:a328g/a221f变异体
序列号5:人工序列的说明:a328g/a221m变异体
序列号6:人工序列的说明:a328g/a221l变异体
序列号7:人工序列的说明:a328g/a221v变异体
序列号8:人工序列的说明:a328g/i228l变异体
序列号9:人工序列的说明:a328g/a221f/d326v变异体
序列号10:人工序列的说明:a328g/a221f/d326v/f298l变异体
序列号11:人工序列的说明:a328g/a221f/d326v/f298l/n222d变异体
序列号13:人工序列的说明:a328g变异体
序列号14:人工序列的说明:a328g/f217w变异体
序列号15:人工序列的说明:a328g/a221f变异体
序列号16:人工序列的说明:a328g/a221m变异体
序列号17:人工序列的说明:a328g/a221l变异体
序列号18:人工序列的说明:a328g/a221v变异体
序列号19:人工序列的说明:a328g/i228l变异体
序列号20:人工序列的说明:a328g/a221f/d326v变异体
序列号21:人工序列的说明:a328g/a221f/d326v/f298l变异体
序列号22:人工序列的说明:a328g/a221f/d326v/f298l/n222d变异体
序列表
<110>天野酶制品株式会社
<120>修饰型菊酸酯酶
<130>ae17007p
<150>jpp2018-229479
<151>2018-12-06
<160>23
<170>patentinversion3.5
<210>1
<211>375
<212>prt
<213>球形节杆菌
<400>1
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuseralaasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyalaseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>2
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g
<400>2
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuseralaasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>3
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/f217w
<400>3
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhistrpglyleuseralaasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>4
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221f
<400>4
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuserpheasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>5
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221m
<400>5
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleusermetasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>6
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221l
<400>6
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuserleuasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>7
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221v
<400>7
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuservalasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>8
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/i228l
<400>8
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuseralaasnserala
210215220
valglyaspleuleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisaspglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>9
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v
<400>9
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuserpheasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalpheglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisvalglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>10
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v/f298l
<400>10
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuserpheasnserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalleuglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisvalglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>11
<211>375
<212>prt
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v/f298l/n222d
<400>11
valaspalaglnthrilealaproglyphegluservalalagluleu
151015
pheglyargpheleusergluaspargglutyrseralaglnleuala
202530
alatyrhisargglyvallysvalleuaspileserglyglyprohis
354045
argargproaspservalthrglyvalphesercysserlysglyval
505560
serglyleuvalilealaleuleuvalglnaspglypheleuaspleu
65707580
aspalagluvalvallystyrtrpproglupheglyalagluglyser
859095
alathrilethrvalalaglnleuleuserhisglnalaglyleuleu
100105110
glyvalgluglyglyleuthrleualaglutyrasnasnsergluleu
115120125
alaalaalalysleualaglnmetargproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyvalphemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluiletyrgluglnarg
165170175
ileargservalthrglyalahisphepheleuglyleuprogluser
180185190
glugluproargtyralathrleuargtrpalaalaaspprosergln
195200205
protrpileaspproalaserhispheglyleuserpheaspserala
210215220
valglyaspileleuaspleuproasnleuarggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
valtyralaalaalaleuthrglyleualaalaasnglyaspargala
260265270
alavalalaproleuleuserglugluthrileglnthrvalthrala
275280285
gluglnvalpheglyileaspargvalleuglygluthrsercysphe
290295300
glythrvalphemetlysserhisalaargserprotyrglysertyr
305310315320
argalapheglyhisvalglyglyseralaserleuglyphealaasp
325330335
provaltyrgluleualapheglytyrvalproglnglnalaglupro
340345350
glyglyalaglycysargasnleugluleuseralaalavalarglys
355360365
alavalthrgluleualagln
370375
<210>12
<211>1128
<212>dna
<213>球形节杆菌
<400>12
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
gcaaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgccagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>13
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g
<400>13
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
gcaaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>14
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/f217w
<400>14
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccattggggcctttcc660
gtgaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>15
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221f
<400>15
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
tttaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>16
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221m
<400>16
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
atgaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>17
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221l
<400>17
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
ctgaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>18
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221v
<400>18
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
gtgaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>19
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/i228l
<400>19
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
gtgaactcggccgtgggggacctgcttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgacggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>20
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v
<400>20
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
tttaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgttcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgtcggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>21
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v/f298l
<400>21
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
tttaactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgctcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgtcggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>22
<211>1128
<212>dna
<213>人工序列
<220>
<223>变异体a328g/a221f/d326v/f298l/n222d
<400>22
gtggatgcacagacgattgcccctggattcgaatcagtcgccgaactctttggccgtttc60
ctgagcgaagaccgggaatattcagcccagctcgcggcctaccaccgcggagtcaaggta120
ttggacatcagcggtgggccgcaccgccgcccggattccgtgaccggtgttttctcctgc180
tccaagggagtatccgggctggtcatcgcacttttggtccaggacggcttcctcgacctc240
gacgccgaagtggtcaagtactggccggaattcggcgccgaaggaaaggccacgattacc300
gtggcccagctgctctcccaccaggccgggcttctgggagtcgaaggcggactcaccctc360
gcggaatacaacaactccgaactggccgccgccaagctcgcgcagatgcggccgctgtgg420
aagcccgggaccgccttcgggtaccacgccctgaccatcggcgtcttcatggaggagctt480
tgccgccggatcaccgggtccacgctccaggaaatctacgaacagcggatccgctcggtc540
acgggcgcccacttcttcctgggactgcctgagtccgaggaaccccgctatgccaccctc600
cgttgggctgcagacccctcccagccgtggattgatcccgccagccatttcggcctttcc660
tttgactcggccgtgggggacatccttgacctgcccaacctccgcgaggtccgcgcagcc720
ggcctgagttcagccgccggagtcgccagcgcggaaggcatggcccgcgtctacgctgcg780
gcactcaccggacttgccgccaacggcgaccgagccgccgtcgcgcccctcctcagcgaa840
gagaccatccaaaccgtcacggccgagcaggtcttcggcatcgaccgggtgctcggcgag900
acgagctgctttgggacagtgttcatgaaatcgcatgcacgctcgccttatggcagctac960
cgggcgttcgggcacgtcggcgggagcgcatctttggggttcgctgaccctgtgtatgaa1020
ctcgccttcgggtacgtgccgcaacaggccgagccgggcggagcgggatgccgcaacctt1080
gagctgagcgccgccgtgcggaaggcagtcaccgaactggctcagtag1128
<210>23
<211>372
<212>prt
<213>节杆菌属rue61a
<400>23
methisserglnvalilealaproglyphegluprovalalagluleu
151015
pheglyvalpheleugluglnaspproasptyrseralaglnvalala
202530
alatyrhisargglyvallysvalleuaspleuserglyglyprohis
354045
ileargproaspservalthrglyvalphesercysserlysglymet
505560
alaglyleuvalmetalaleuleuvalglnaspglygluleuaspleu
65707580
glualagluvalvallystyrtrpproglupheglyvalgluglylys
859095
serserilethrvalalaglnleuleuserhisargalaglyleuleu
100105110
glyvalgluglyglyleuthrleuhisgluvalasnasnsergluleu
115120125
alaalaalalysleualagluleuproproleutrplysproglythr
130135140
alapheglytyrhisalaleuthrileglyilephemetglugluleu
145150155160
cysargargilethrglyserthrleuglngluvalphegluglnarg
165170175
ileargalavalthrglyalaasnphetyrleuglyleuprogluser
180185190
glugluserargphealaglnpheargtrpalaalaaspprosertrp
195200205
protrpvalaspproalaserhispheglyleualaalaasnalaala
210215220
valglyaspileleuaspleuproasnilearggluvalargalaala
225230235240
glyleuserseralaalaglyvalalaseralagluglymetalaarg
245250255
iletyralaalaalaleuthrglyleugluglylysseralametpro
260265270
proleuleuthrglugluthrileargthrvalseralagluglnval
275280285
pheglyileaspargvalpheglygluthrglycyspheglythrval
290295300
phemetlysserhisthrargmetpropheglysertyrargalaphe
305310315320
glyhisaspglyalaseralaserleuglyphealaaspprovaltyr
325330335
gluleuglypheglytyrvalproglnthralagluproglyglyval
340345350
glycysargasnpheglnleuserseralavalarggluvalileala
355360365
glyphealaala
370
- 还没有人留言评论。精彩留言会获得点赞!