具有植酸酶活性的多肽的制作方法
本发明涉及具有植酸酶活性的多肽,其生产方法,以及包含这种多肽的组合物。
背景技术:
植酸(phyticacid)(植酸化物(phytate)、肌醇六磷酸化物(inositolhexakisphosphate)、ip6)在许多植物中被发现,它们在这些植物中起着储存磷酸盐的作用。储存在ip6分子中的磷酸盐可以作为无机磷酸盐的形式释放。当从植酸分子中释放出无机磷酸盐时,ip6转化为低级肌醇磷酸化物即五磷酸化物(ip5)、四磷酸化物(ip4)、三磷酸化物(ip3)、双磷酸化物(ip2)和单磷酸化物(ip)。
植酸酶是一组催化植酸水解的磷酸酶。市售的植酸酶均属于组氨酸酸性磷酸酶(hap)蛋白家族。属于hap蛋白家族的植酸酶共有保守的n末端活性位点七肽基序rhgxrxp和c末端的催化活性hd-二肽。组氨酸酸性磷酸酶是较大的组氨酸磷酸酶超家族的一部分。所述组氨酸磷酸酶超家族是一大类功能多样的蛋白质。它们共有以组氨酸为中心的保守催化核心,该组氨酸在反应过程中被磷酸化。pfam基序his_phos_2(pf00328)代表所述组氨酸磷酸酶超家族的分支2,该分支主要含有酸性磷酸酶和植酸酶。
植酸酶被用于饲料中,以通过植酸化物降解来提高植物来源(例如小麦、大麦、玉米、大豆)的饲料成分的磷酸盐利用率。对于单胃动物,如家禽、鱼和猪,这尤其令人感兴趣,因为这些的上肠道内的肠植酸化物降解是有限的。这种局限性不仅限制了磷的利用,而且由于肌醇磷酸化物的螯合作用,还限制了其他营养素的利用率,并且这是为什么至少ip6至ip4应该尽可能地去磷酸化的原因。目前在动物饲料应用中使用的几种现有的植酸酶的效率相当有效,但是ip6降解为ip1的效率仍应提高,以进一步增加动物对磷酸盐和其他营养素的利用率。
植酸酶分解植酸化物伴随着ip6逐步降解为低级肌醇磷酸酯(ip5、ip4、ip3、ip2和ip1)。已表明,使用工业标准水平的植酸酶可如预期显著降低体外和体内的ip6水平。然而,随着ip6降解,回肠消化物中检测到ip4和ip3增加,这表明第一个磷酸基团的水解裂解不是植酸化物降解的唯一限制步骤(例如zeller等,2015;menezes-blackburn等,2015)。因为即使这些低级肌醇磷酸化物也由于结合不同营养素如矿物质而具有抗营养特性(xu等,1992),所以动物饲养的目标是降解ip酯直至回肠末端(bedford和walk,2016)。达到使肌醇完全释放的目的的另一个原因是,增加游离肌醇的含量已显示出可以通过不同的机制改善动物的生长表现,但仍在研究中(lee和bedford,2016)。
动物肠道中用植酸酶补充剂使肌醇磷酸酯发生最佳降解的部分是胃,由于胃的ph值低,从而导致最佳的底物(植酸化物)溶解性。在胃中的保留时间很短,并且部分内容物可能会迅速冲到ph值为中性的肠道。因此,快速且更有效地作用于ip6以及作用于低级肌醇磷酸化物如ip4和ip3异构体的植酸酶的进一步开发对动物饲养业具有重要意义。这些类型的植酸酶将进一步提高磷酸盐和肌醇的肠道利用率。
技术实现要素:
本发明的一个目的是提供具有植酸酶活性并且适用于饲料和食品应用的新型多肽。本发明的多肽通过从存在于植物来源的饲料或食品中的植酸中释放出无机磷酸盐,可用于增加饲料或食品的营养价值。本发明的另一个目的是提供新型的饲料和添加剂。
本发明的多肽对植物材料中发现的植酸和/或低级肌醇磷酸化物或从植酸和/或低级肌醇磷酸化物降解的低级肌醇磷酸化物具有改进的植酸化物降解活性和/或特异性。本发明的多肽优选地为组氨酸酸性磷酸酶,并且它们具有改进的从植物材料中释放无机磷酸盐的能力,从而使所述多肽适合用于动物饲料中以提高饲料的营养价值。与现有的植酸酶相比,本发明的多肽在将ip6连续降解为低级肌醇磷酸化物方面具有改进的活性,并且在降解低级ip如ip4方面具有优越性,所述先前的植酸酶例如在实施例中描述的测试中用作基准商业产品的quantumblue(qb)中的大肠杆菌(e.coli)突变型植酸酶。
本发明人已经发现了一组有限的组氨酸酸性磷酸酶,其可用于工业规模的生产并且具有使其适于饲料应用的特性。本发明人还发现,不可能仅基于序列相似性或筛选一种或几种特性(例如最佳ph、生产收率或温度稳定性)来预测候选植酸酶的性能。因此,基于序列分析被注释为植酸酶的数据库多肽在本发明植酸酶的预期应用中不一定有用。此外,不能基于序列分析预测酶对特定肌醇磷酸化物的特异性。
本发明人发现,对于例如用于饲料应用的候选植酸酶的测试,在模拟动物如家禽的胃肠道状况(胃肠道模拟测定法,git)的环境中分析的测试具有更多信息,因为它结合了与植酸酶用作饲料添加剂的有效性相关的几种情况。实际上,大多数被注释为推定具有植酸酶活性的候选植酸酶均未显示任何显著的植酸酶活性,或者它们在重组宿主中的产生不成功。因此,本发明的多肽一起构成了统一的植酸酶组,并且所述多肽在git测定法中显示出有效性,并且适合于饲料应用。
根据第一方面,本发明提供了一种具有植酸酶活性的多肽,其中所述多肽选自:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列;
其中所述多肽是分离的、重组的或合成的多肽。
第一方面的所有多肽体现了本发明的共同发明构思。它们在植酸酶类别中在结构上相似,并且它们共有相同的功能特征。它们还对低级肌醇磷酸化物如ip4具有高活性,并且可以单独地或与其他植酸酶组合用于饲料应用。因此,本发明的一个目的是提供植酸酶和含有它们的组合物,所述植酸酶对ip4具有优先性,以增强高级肌醇磷酸酯降解为低级肌醇磷酸酯轻质组分。
根据第二方面,本发明提供了一种组合物,其包含第一方面的多肽和选自以下物质中的至少一者的至少一种饲料(饲料成分)或添加剂:稳定剂、防腐剂、矿物质和营养素。在一个实施方式中,营养素包括油、维生素、微量矿物质和氨基酸中的至少一者。
所述组合物可用于饲养动物,因为所述组合物可给予动物并且所述植酸酶可有效地降解植酸化物并改善营养素从所述组合物的组分中或从饲料中的释放。
根据第三方面,本发明提供了一种组合物,其包含至少两种具有植酸酶活性的多肽和至少一种饲料或添加剂,其中:
第一多肽具有植酸酶活性,并且选自与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;并且
第二多肽具有植酸酶活性,不具有与所述第一多肽相同的氨基酸序列,并且选自大肠杆菌突变型植酸酶qb,与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列。
所述组合物可以通过将其组分混合在一起来制造。
根据第四方面,本发明提供了动物饲料,其包含第一方面的多肽或者第二或第三方面的组合物;至少一种植物来源的蛋白质源;以及
a.任选地至少一种另外的酶,其选自蛋白酶、淀粉酶、植酸酶、木聚糖酶、内切葡聚糖酶、β-葡聚糖酶或其组合;和
b.任选地至少一种填充剂,其选自麦芽糊精、面粉、盐、氯化钠、硫酸盐、硫酸钠或其组合。
根据第四方面,本发明提供了一种饲料补充剂,其包含第一方面的多肽或第二方面的组合物;以及
a.任选地至少一种另外的酶,其选自蛋白酶、淀粉酶、植酸酶、木聚糖酶、内切葡聚糖酶、β-葡聚糖酶或其组合;和
b.任选地至少一种填充剂,其选自麦芽糊精、面粉、盐、氯化钠、硫酸盐、硫酸钠、矿物质、氨基酸、益生元、益生菌或其组合。
根据第五方面,本发明提供了第一方面的多肽或包含所述多肽的组合物用于降解植酸的用途。在一个实施方式中,所述用途涉及降解ip4或ip6,或者降解ip4和ip6两者,和/或任选地降解ip3和ip2。在另一个实施方式中,所述用途涉及降解ip4、ip5和ip6,以及任选地降解ip3和ip2。
根据第六方面,本发明提供了一种在重组宿主细胞中产生植酸酶的方法,所述方法包括
a.提供包含用于产生植酸酶的遗传元件的多核苷酸,所述植酸酶包含的氨基酸序列与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性;以及
b.在重组宿主细胞中表达所述多核苷酸;
其中所述植酸酶能够从植酸中释放磷酸盐。
根据第七方面,本发明提供了一种重组宿主细胞,其包含用于产生至少一种第一方面的多肽的遗传元件。
根据另一方面,本发明提供了第一方面的多肽在动物饲料中、在动物饲料添加剂中、在制备用于动物饲料的组合物中和/或用于改善饲料消化率的用途和使用方法。
根据另一方面,本发明提供了第一方面的多肽在食品中、在食品添加剂中、在制备用于食品的组合物中和/或用于改善食品的消化率的用途和使用方法。
根据另一方面,本发明提供了一种用于生产动物饲料的方法,其包括将包含碳水化合物、脂肪和蛋白质中的一者或多者的营养素组分与第一方面的多肽组合,从而生产所述动物饲料。
在另一方面,本发明提供了一种用于增加动物饲料的营养价值的方法,其包括提供包含碳水化合物、脂肪和蛋白质中的一者或多者以及第一方面的多肽的动物饲料。
如实施例所证明,根据本发明的多肽和组合物具有使其能够在重组宿主细胞中产生并使其可用于工业应用的酶组合物中的结构、组成和特性。所述酶组合物由于良好的稳定性、性能和特定活性而特别适合于饲料制剂。
附图说明
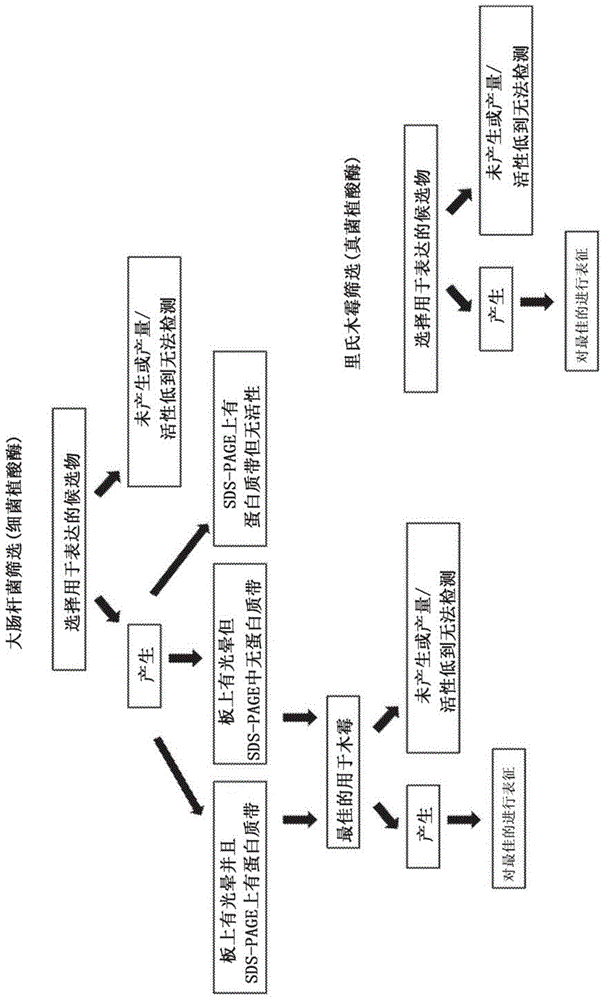
图1示出了在大肠杆菌和里氏木霉(t.reesei)中候选植酸酶筛选程序的概述。
图2示出了候选多肽的系统发育树。
图3示出了分裂的系统发育树。
图4示出了在ph3和ph5下,所选植酸酶对ip6和ip4降解的效率(反应中吸收的变化)。图4a和图4b,突变型大肠杆菌植酸酶(quantumblue产品,qb);图4c和图4d,ba59;图4e和图4f,ye76;图4g和图4h,psd34;图4i和图4j,psd35;图4k和图4l,psd65;图4m和图4n,psd67;图4o和图4p,aspaw;图4q和图4r,psf203;图4s和图4t,psk256;图4u和图4v,psk260;图4w和图4x,psk267。
图5示出了利用玉米-豆粕为底物和每千克玉米-豆粕混合物250ftu植酸酶进行git测定后,使用选自筛选的植酸酶获得的ip6至ip3肌醇磷酸化物的量。quantumblue(qb)产品和以与新型植酸酶类似的发酵方式产生的qb中的大肠杆菌突变型植酸酶(参考)用作参考。图5a,psf203(两次平行试验的结果)、ba59、psd35、psd67;图5b,qb、psd65(两次平行试验的结果)、psk260、psk256、ye76。-,未添加植酸酶。
图6示出了相对于未添加植酸酶的空白,来自使用不同剂量(125、250、500和1000ftu/kg底物)的ba59、psd65和以与新型植酸酶类似的发酵方式产生的qb中的大肠杆菌突变型植酸酶(参考)的git测试的结果。图6a示出了以相比于空白的%计,使用玉米豆粕混合物作为底物进行git测定后,包括ip5和ip4异构体的ip6-ip3肌醇磷酸化物的量。图6b示出了图6a中给出的测试结果的ip6-4的总和(以相比于空白的%计的ip6、ip5、ip4的总和)。
图7示出了相对于未添加植酸酶的空白,来自使用不同剂量(250、500和1000ftu/kg饲料)的ba59、psd65和以与新型植酸酶类似的发酵方式产生的qb中的大肠杆菌突变型植酸酶(参考)的git测试的结果。图7a示出了使用p和ca水平低的基于玉米-大豆-油菜籽粕的复合饲料(阴性对照)作为底物进行git测定后,包括异构体的ip6-3肌醇磷酸化物。图7b示出了图7a中给出的测试结果的ip6-4的总和(ip6、ip5、ip4的总和)。
图8示出了相对于未添加植酸酶的空白,来自使用每千克饲料250ftu的ba59、psd65和以与新型植酸酶类似的发酵方式产生的qb中的大肠杆菌突变型植酸酶(参考)的git测试结果。图8a示出了使用玉米-豆粕混合物(cs)、基于玉米-大豆-油菜籽粕的复合饲料的普通饲料(pc,阳性对照)或降低的p和ca水平(nc,阴性对照)作为底物进行git测定后,包括异构体的ip6-3肌醇磷酸化物的量。图8b示出了图8a中给出的测试结果的ip6-4的总和(以相比于空白的%计的ip6、ip5、ip4的总和)。
图9示出了相对于未添加植酸酶的空白,来自将ba59或psd65与以与新型植酸酶类似的发酵方式产生的qb中的大肠杆菌突变型植酸酶(参考)组合使用时的git测试的结果。使用250ftu/kg的参考并且添加62.5、125和250ftu/kg的ba59、psd65或参考。图9a。使用玉米-豆粕混合物作为底物并利用单独的250ftu/kg的参考(-)或利用添加了递增剂量的ba59、psd65或参考进行git测定后,包括异构体的ip6-3肌醇磷酸化物的量。图9b示出了图9a中给出的测试结果的ip6-pi4的总和(以相比于空白的%计的ipip6、ip5和ip4的总和)。
图10:一组新型植酸酶之间的成对同一性%。推导的氨基酸序列在没有其信号序列的情况下用于比对。ecoliwt(uniprotppa_ecoli)和aspaw(uniprotppa_aspaw)是参考序列。表1中列出了所述新型植酸酶的seqid编号。
序列表
seqidno:1:全长植酸酶的ba59氨基酸序列。
seqidno:2:表达盒中使用的ba59核苷酸序列。
seqidno:3:全长植酸酶的ye76氨基酸序列。
seqidno:4:表达盒中使用的ye76核苷酸序列。
seqidno:5:全长植酸酶的psd32氨基酸序列。
seqidno:6:表达盒中使用的psd32核苷酸序列。
seqidno:7:全长植酸酶的psd34氨基酸序列。
seqidno:8:表达盒中使用的psd34核苷酸序列。
seqidno:9:全长植酸酶的psd35氨基酸序列。
seqidno:10:表达盒中使用的psd35核苷酸序列。
seqidno:11:全长植酸酶的psd65氨基酸序列。
seqidno:12:表达盒中使用的psd65核苷酸序列。
seqidno:13:全长植酸酶的psd67氨基酸序列。
seqidno:14:表达盒中使用的psd67核苷酸序列。
seqidno:15:全长植酸酶的psf203氨基酸序列。
seqidno:16:表达盒中使用的psf203核苷酸序列。
seqidno:17:全长植酸酶的psf207氨基酸序列。
seqidno:18:表达盒中使用的psf207核苷酸序列。
seqidno:19:全长植酸酶的psk235氨基酸序列。
seqidno:20:表达盒中使用的psk235核苷酸序列。
seqidno:21:全长植酸酶的psk252氨基酸序列。
seqidno:22:表达盒中使用的psk252核苷酸序列。
seqidno:23:全长植酸酶的psk256氨基酸序列。
seqidno:24:表达盒中使用的psk256核苷酸序列。
seqidno:25:全长植酸酶的psk260氨基酸序列。
seqidno:26:表达盒中使用的psk260核苷酸序列。
seqidno:27:全长植酸酶的psk267_ii氨基酸序列。与数据库条目相比,从第二个蛋氨酸开始。
seqidno:28:表达盒中使用的psk267_ii核苷酸序列。
seqidno:29:全长植酸酶的psk268_3mc氨基酸序列。与数据库条目相比,从第三个蛋氨酸开始。
seqidno:30:表达盒中使用的psk268_3mc核苷酸序列。
具体实施方式
在一个实施方式中,所述多肽是优选地来自细菌或真菌的组氨酸酸性磷酸酶。
在一个实施方式中,所述多肽包含以下其他氨基酸序列种的至少一个:信号序列、分泌序列、载体多肽、标签、酶活性或其任何组合。
本发明的多肽具有ip4降解活性、ip6降解活性、或者ip4和ip6降解活性两者。本发明的多肽还可以具有ip3和/或ip2降解活性。
在一个实施方式中,所述多肽在ph3下具有比ip6降解活性更高的ip4降解活性。这种特性使得本发明的植酸酶可用于例如饲料应用中,因为所述植酸酶能够有效降解饲料中的植酸。与饲料应用中先前使用的ip6植酸酶相比,本发明的植酸酶可以更进一步进行肌醇磷酸化物的降解,并且更有效地释放饲料中的营养素,因为低级肌醇磷酸化物也被有效降解。
在一个实施方式中,所述多肽在约ph4下具有最佳植酸化物水解活性,以在以下条件下释放无机磷酸盐的能力来测量:反应温度37℃,反应时间15分钟,作为底物的植酸钠初始浓度为10g/l。
在一个实施方式中,与大肠杆菌突变型植酸酶qb相比,所述多肽具有改进或增加的ip降解活性,所述ip降解活性表示为ip6、ip5和ip4降解活性的总和。在一个实施方式中,使用植物来源的肌醇磷酸化物底物测定所述降解活性。
在一个实施方式中,所述组合物是食品或饲料,并且它还包含含有植酸的植物材料。
在一个实施方式中,所述组合物是食品添加剂或饲料添加剂,所述组合物还包含以下物质中的至少一者:至少一种微量矿物质,至少一种氨基酸,特别是赖氨酸,水溶性维生素,脂溶性维生素,益生元,益生菌。
在一个实施方式中,所述组合物是食品添加剂或饲料添加剂,所述组合物符合欧洲议会和理事会于2003年9月22日关于动物营养用添加剂的第1831/2003号法规(ec)的要求。
在一个实施方式中,所述组合物是液体组合物或固体组合物例如溶液、分散体、糊剂、粉末、颗粒、包衣颗粒、片剂、饼状物、晶体、晶体浆液、凝胶、挤出物、沉淀物、任选地与其他添加剂的预混物或丸粒的形式。
在一个实施方式中,通过在重组宿主细胞中产生而获得所述组合物中的至少第一多肽或第二多肽。
在一个实施方式中,所述重组宿主细胞选自植物细胞、真菌细胞、丝状真菌细胞、酵母和细菌细胞。
“成熟多肽”是指采取在翻译和任何翻译后修饰后的形式的多肽,所述翻译后修饰例如n末端加工、c末端截短、糖基化和磷酸化。
在一个实施方式中,ba59的成熟氨基酸序列包含seqidno:1的氨基酸残基26-439。
在一个实施方式中,psd65的成熟氨基酸序列包含seqidno:11的氨基酸残基24-424。
在一个实施方式中,psd67的成熟氨基酸序列包含seqidno:13的氨基酸残基25-403。
在一个实施方式中,psf203的成熟氨基酸序列包含seqidno:15的氨基酸残基16-469。
在一个实施方式中,所述多肽与seqidno:1的成熟氨基酸序列具有至少85%,例如,至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的氨基酸序列同一性。
在一个实施方式中,所述多肽与seqidno:11的成熟氨基酸序列具有至少85%,例如,至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的氨基酸序列同一性。
在一个实施方式中,所述多肽与seqidno:13的成熟氨基酸序列具有至少85%,例如,至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的氨基酸序列同一性。
在一个实施方式中,所述多肽与seqidno:15的成熟氨基酸序列具有至少85%,例如,至少86%、至少87%、至少88%、至少89%、至少90%、至少91%、至少92%、至少93%、至少94%、至少95%、至少96%、至少97%、至少98%、至少99%或100%的氨基酸序列同一性。
在一个实施方式中,所述多肽或所述植酸酶是组氨酸酸性磷酸酶。在另一个实施方式中,其含有pfam基序his_phos_2(pf00328)。在另一个实施方式中,所述多肽或所述植酸酶包含活性位点序列基序rhgxrxp和c末端的催化活性hd-二肽。这些结构特征中的每一者是根据本发明的多肽共有的共同特征。
在一个实施方式中,通过在异源宿主细胞,优选地真菌宿主细胞,例如木霉(trichoderma)中重组生产来获得多肽。
在另一个实施方式中,所述多肽是通过在植物细胞中,即在转基因植物中重组生产而获得的。
术语“功能片段”或“有效片段”是指保留大致相同的酶功能或作用的所述多肽的片段或部分。
如本文所用,“分离的”是指自然界中不存在的形式或环境中的物质。分离的物质的非限制性实例包括(1)任何非天然存在的物质;(2)至少部分地从一种或多种或所有的与物质自然相关的天然存在成分中取出的任何物质,包括任何酶、变体、核酸、蛋白质、肽或辅因子;(3)相对于自然界中发现的物质进行人为修饰的任何物质,例如变体;或(4)通过增加或减少物质相对于与其自然相关的其他组分的量而修饰的任何物质(例如,宿主细胞中的重组生产;编码所述物质的基因的一个或多个拷贝;以及使用与编码所述物质的基因天然相关的启动子的替代启动子)。在一个实施方式中,本发明的多肽、酶、变体、多核苷酸、宿主细胞或组合物是分离的。
如本文所用,术语“包含”包括“包括”、“含有”和“涵盖”的更广泛含义,以及较窄范围的表述“由……组成”和“仅由……组成”。
如本文所用,“肽”和“多肽”是包含多个连续聚合的氨基酸残基的氨基酸序列。为了本发明的目的,肽是包含至多20个氨基酸残基的分子,而多肽包含多于20个氨基酸残基。肽或多肽可以包含修饰的氨基酸残基、没有密码子编码的天然存在的氨基酸残基和非天然存在的氨基酸残基。如本文所用,“蛋白质”可以指任何大小的肽或多肽。蛋白质可以是酶、蛋白质、抗体、膜蛋白、肽激素、调节剂或任何其他蛋白质。
术语“多核苷酸”表示从5'端至3'端读取的脱氧核糖核苷酸或核糖核苷酸碱基的单链或双链聚合物。多核苷酸包括rna和dna,并且可以从天然来源分离,体外合成,或由天然和合成分子的组合制备。
如本文所用,“同一性”是指相对于在两个比对序列中均存在残基的位置数量,两个序列之间的氨基酸残基精确匹配的百分比。当一个序列的一个残基在另一序列中没有相应的残基时,比对程序允许在比对中有空位,并且所述位置不计入同一性计算的分母中。同一性是利用embl-ebi网站(www.ebi.ac.uk/tools/psa/emboss_needle/)上的逐对序列比对工具embossneedle确定的值。与具有酶活性的多肽的较高序列同一性可能意味着所述多肽具有更多相似的功能特性和相似的结构。然而,尽管一级结构有所不同,但具有较低序列同一性的多肽也可能具有相似的特性:具有低序列同一性的多肽可能能够采用例如在底物结合位点中、在与相互作用或构象变化有关的位点中以及在活性位点中的关键氨基酸的相似折叠和构象。在一个实施方式中,所列举的序列同一性在序列的整个长度上延伸。
如本文所用,“宿主细胞”是指易于用包含多核苷酸的核酸构建体或表达载体转化、转染、转导、交配、杂交等的任何细胞类型。术语“宿主细胞”涵盖由于复制过程中发生的突变而导致不相同的任何后代。宿主细胞的非限制性实例是植物细胞,真菌细胞,来自子囊菌门(ascomycota)、盘菌亚门(pezizomycotina)的丝状真菌细胞;优选地选自粪壳菌纲(sordariomycetes)、肉座菌亚纲(hypocreomycetidae)、肉座菌目(hypocreales)和小囊菌目(microascales),以及曲霉属(aspergillus)、金孢子菌属(chrysosporium)、毁丝霉属(myceliophthora)和腐质霉属(humicola)的成员;更优选地选自肉座菌科(hypocreacea)、丛赤壳科(nectriaceae)、麦角菌科(clavicipitaceae)、小囊菌科(microascaceae),以及木霉(trichoderma)(肉座菌属(hypocrea)的无性型)、镰刀菌属(fusarium)、赤霉菌属(gibberella)、丛赤壳属(nectria)、葡萄穗霉属(stachybotrys)、麦角菌属(claviceps)、绿僵菌属(metarhizium)、绿核菌属(villosiclava)、线虫草属(ophiocordyceps)、头孢霉属(cephalosporium)和丝孢菌属(scedosporium);更优选地选自里氏木霉(红褐肉座菌(hypocreajecorina))、柠檬绿木霉(t.citrinoviridae)、长枝木霉(t.longibrachiatum)、绿木霉(t.virens)、哈茨木霉(t.harzianum)、棘孢木霉(t.asperellum)、深绿木霉(t.atroviridae)、近里氏木霉(t.parareesei)、尖孢镰刀菌(fusariumoxysporum)、禾谷镰刀菌(f.gramineanum)、假禾谷镰刀菌(f.pseudograminearum)、镶片镰孢霉(f.venenatum)、藤仓赤霉(gibberellafujikuroi)、串珠赤霉(g.moniliformis)、玉蜀黍赤霉(g.zeaea)、血红丛赤壳菌(血赤壳菌)(nectria(haematonectria)haematococca)、纸葡萄穗霉(stachybotryschartarum)、氯卤葡萄穗霉(s.chlorohalonata)、黑麦麦角菌(clavicepspurpurea)、蝗绿僵菌(metarhiziumacridum)、金龟子绿僵菌(m.anisopliae)、稻绿核菌(villosiclavavirens)、冬虫夏草菌(ophiocordycepssinensis)、顶头孢霉(头孢霉菌)(acremonium(cephalosporium)chrysogenum)和尖端赛多孢子菌(scedosporiumapiospermum),以及黑曲霉(aspergillusniger)、泡盛曲霉(aspergillusawamori)、米曲霉(aspergillusoryzae)、鲁克文金孢子菌(chrysosporiumlucknowense)、嗜热毁丝霉(myceliophthorathermophila)、特异腐质霉(humicolainsolens)和灰腐质霉(humicolagrisea),最优选地为里氏木霉。宿主细胞的非限制性实例是细菌细胞,优选地革兰氏阳性杆菌(例如枯草芽孢杆菌(bacillussubtilis)、地衣芽孢杆菌(b.licheniformis)、巨大芽孢杆菌(b.megaterium)、解淀粉芽孢杆菌(b.amyloliquefaciens)、短小芽孢杆菌(b.pumilus)),革兰氏阴性细菌(例如大肠杆菌(escherichiacoli)),放线菌目(例如链霉菌属种(streptomyces),以及酵母(例如酿酒酵母(saccharomycescerevisiae)、巴斯德毕赤酵母(pichiapastoris)、解脂耶氏酵母(yarrowialipolytica))。
在一个实施方式中,所述宿主细胞是真菌细胞,优选地丝状真菌细胞,例如木霉或里氏木霉。在一个实施方式中,所述宿主细胞是细菌细胞,优选地革兰氏阳性杆菌细胞,例如枯草芽孢杆菌、地衣芽孢杆菌、巨大芽孢杆菌、解淀粉芽孢杆菌、短小芽孢杆菌。
如本文所用,“表达”包括在宿主细胞中产生多肽所涉及的任何步骤,包括但不限于转录、翻译、翻译后修饰和分泌。表达之后可以是收获,即回收宿主细胞或所表达的产物。
术语“表达载体”表示线性或环状的dna分子,其包含编码目的多肽的区段,所述目的多肽与提供其转录的其他区段可操作地连接。所述其他区段可以包括启动子和终止子序列,并且可以任选地包括一个或多个复制起点、一个或多个选择性标记、增强子、聚腺苷酸化信号、载体等。表达载体通常源自质粒或病毒dna,或者可以包含两者的元件。表达载体可以是方便地进行重组dna程序的任何表达载体,并且载体的选择通常取决于要引入载体的宿主细胞。因此,载体可以是自主复制的载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒。或者,载体可以是当引入宿主细胞中时被整合到宿主细胞基因组中并与已经整合了载体的一个或多个染色体一起复制。在一个实施方式中,本发明载体是表达载体。
本文中与多肽或蛋白质的产生结合使用的术语“重组的”或“重组产生的”是根据本领域的标准定义来定义的。
如本文与特定微生物来源结合使用的术语“获自”和“可获得的”是指多核苷酸由特定来源表达(同源表达),或由已插入来源中的基因的细胞表达(异源表达)。
术语“酶组合物”是指可能从单个微生物物种中分离和纯化的常规酶发酵产物,这种制剂通常包含多种不同的酶活性;或单组分酶的混合物,优选地为通过使用常规重组技术源自细菌或真菌物种的酶,这些酶已分别发酵并可能分离和纯化,并且其可能源自不同物种,优选地为真菌或细菌物种或用作产生重组多肽的宿主细胞的微生物的发酵产物,但所述微生物同时产生其他酶。酶组合物可以是干燥产品,例如粉末、丸粒、挤出物或颗粒。
在另一个实施方式中,所述多肽以如下形式配制在组合物中:至少部分溶解形式,稀释或压缩固体或液体形式,包括但不限于具有某些目标特性如例如受控的流变性、粘度或酶释放的溶液、悬浮液、乳液、半固体、固体、糊剂、丸粒、饼状物、凝胶、片剂,薄膜或包衣。
在本发明的一个实施方式中,提供了包含本发明的多肽或组合物的颗粒。所述颗粒任选地被包衣层包衣,该包衣层包封核心以形成基本上连续的层并控制核心的释放。包衣层优选地是适用于食品和/或饲料的食品级或饲料级材料。
术语“可操作地连接”当涉及dna区段时表示所述区段被排列成使它们能够按预期目的协同作用,例如转录在启动子中起始并通过编码区段进行到终止子。
术语“启动子”表示包含提供rna聚合酶的结合和转录起始的dna序列的基因的一部分。启动子序列通常但并非总是存在于基因的5'非编码区中。
术语“分泌信号序列”或“信号序列”表示编码多肽(“分泌肽”)的dna序列,所述分泌肽作为较大多肽的组分,通过产生所述较大多肽的宿主细胞的分泌途径引导所述较大多肽。分泌信号序列可以是天然的,或者其可以用来自另一个来源的分泌信号序列或载体序列代替。取决于宿主细胞,较大的肽可以在通过分泌途径转运的过程中被裂解以除去分泌肽。
术语“核心区域”或“催化结构域”表示酶的一个结构域,其可能会或可能不会被修饰或改变,但保留了其原始活性的至少一部分。
有效量是指足以在所选应用中降解植酸或低级ip形式的量。
术语“稳定性”包括储存稳定性和在使用过程中的稳定性,例如在饲料生产条件下或在动物消化道中的稳定性,并反映了根据本发明的多肽随时间变化的稳定性,例如将多肽保存在溶液中、饲料中或用于应用时保留多少活性。稳定性受许多因素例如ph、温度、蛋白酶等影响。可以使用如实施例中所述的“活性测定法”来测量植酸酶稳定性。
在一个实施方式中,如实施例2中所述,利用ppu测定法、ftu测定法、平板测定法或4-mup测定法测定植酸酶活性。
在本申请中,“动物”是指任何动物。动物的实例是单胃动物(非反刍动物),包括但不限于猪或豕(包括但不限于小猪,生长中的猪和母猪);家禽,例如火鸡、鸭子、鹌鹑、珍珠鸡、鹅、鸽子(包括乳鸽)和鸡(包括但不限于肉用仔鸡(在本文中称为肉鸡)、小鸡、生蛋母鸡(在本文中称为蛋鸡));鱼;马(包括但不限于热血马、冷血马和温血马)。在一个实施方式中,所述动物是任何非人类动物。
“饲料”,例如动物饲料,是指适合或打算被动物摄入的任何化合物、制剂或混合物。单胃动物的动物饲料通常包括浓缩物,其包含任何饲料成分,如但不限于玉米、小麦、豆粕以及维生素、矿物质、酶、直接饲喂的微生物、氨基酸和/或其他饲料成分(例如以预混物形式),而反刍动物的动物饲料通常包含草料(包括粗饲料和青贮饲料),并且还可以包含浓缩物以及维生素、矿物质、酶、直接饲喂的微生物、氨基酸和/或其他饲料成分(例如以预混物形式)。
在不限制专利权利要求书的范围和解释的情况下,以下列出了本文公开的一个或多个方面或实施方式的某些技术效果:技术效果是植酸、特别是低级肌醇磷酸化物的降解或修饰。
前述描述已经通过本发明的特定实现方式和实施方式的非限制性实例的方式,提供了发明人目前构想的用于实施本发明的最佳模式的完整且信息丰富的描述。然而,对于本领域技术人员而言清楚的是,本发明不限于以上呈现的实施方式的细节,而是可以在不背离本发明的特征的情况下使用等效手段在其他实施方式中实现本发明。
此外,可以在不相应使用其他特征的情况下有利地使用本发明的上述方面和实施方式的一些特征。因而,前面的描述应被认为仅是本发明原理的示例,而不是对其的限制。因此,本发明的范围仅由所附的专利权利要求书来限制。
在一个实施方式中,所述多肽或核酸是非天然存在的多肽或核酸,其中其序列与用于鉴定它的相应seqidno不是100%相同。在一个实施方式中,对所述核酸进行用于在所选择的宿主细胞中产生的密码子优化,以进行重组生产。用这种密码子优化的核酸产生的多肽提供了增加的多肽生产收率。
在一个实施方式中,与本发明组合物的至少一种组分来源的相应天然组分相比,所述至少一种组分具有不同的化学、结构或物理特征。在一个实施方式中,所述特征是以下中的至少一者:均一大小、均匀分散、不同同工型、不同密码子简并性、不同翻译后修饰、不同甲基化、不同三级或四级结构、不同酶活性、不同亲和力、不同结合活性和不同免疫原性。
描述了以下条款:
条款1.一种组合物,所述组合物包含多肽和至少一种选自稳定剂、防腐剂、矿物质和营养素的饲料或添加剂,其中所述多肽选自:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列;并且
其中所述多肽是分离的、重组的或合成的多肽。
条款2.根据条款1所述的组合物,其中所述多肽在ph3下具有比ip6降解活性更高的ip4降解活性。
条款3.一种组合物,所述组合物包含至少两种具有植酸酶活性的多肽和至少一种添加剂,其中:
第一多肽具有植酸酶活性,并且选自与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;并且
第二多肽具有植酸酶活性,不具有与所述第一多肽相同的氨基酸序列,并且选自大肠杆菌突变型植酸酶qb,与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列。
条款4.根据条款1-3所述的组合物,其中所述组合物是食品添加剂或饲料添加剂,所述组合物还包含以下物质中的至少一者:矿物质、氨基酸、益生元、益生菌。
条款5.根据条款1-4所述的组合物,所述组合物为液体组合物或固体组合物例如溶液、分散体、糊剂、粉末、颗粒、包衣颗粒、片剂、饼状物、晶体、晶体浆液、凝胶、挤出物或丸粒的形式。
条款6.根据条款1-5所述的组合物,其中所述第一多肽和所述第二多肽中的至少一者是通过在重组宿主细胞中生产而获得的。
条款7.一种动物饲料,所述动物饲料包含如条款1-6所述的组合物和至少一种植物来源的蛋白质源,以及
a.任选地至少一种另外的酶,其选自蛋白酶、淀粉酶、植酸酶、木聚糖酶、内切葡聚糖酶、β-葡聚糖酶或其组合;和
b.任选地至少一种填充剂,其选自麦芽糊精、面粉、盐、氯化钠、硫酸盐、硫酸钠或其组合。
条款8.一种饲料补充剂,所述饲料补充剂包含如条款1-6所述的组合物;以及
a.任选地至少一种另外的酶,其选自蛋白酶、淀粉酶、植酸酶、木聚糖酶、内切葡聚糖酶、β-葡聚糖酶或其组合;和
b.任选地至少一种填充剂,其选自麦芽糊精、面粉、盐、氯化钠、硫酸盐、硫酸钠、矿物质、氨基酸、益生元、益生菌或其组合。
条款9.一种多肽用于降解植酸的用途,所述多肽选自:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列。
条款10.一种在重组宿主细胞中产生植酸酶的方法,所述方法包括
a.提供包含用于产生植酸酶的遗传元件的多核苷酸,所述植酸酶包含与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;以及
b.在重组宿主细胞中表达所述多核苷酸;
其中所述植酸酶能够从植酸中释放磷酸盐。
条款11.一种重组宿主细胞,所述重组宿主细胞包含用于产生至少一种选自以下的多肽的遗传元件:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列。
条款12.根据条款11所述的重组宿主细胞,其中所述重组宿主细胞选自植物细胞、真菌细胞、丝状真菌细胞、酵母和细菌细胞。
条款13.一种多肽在动物饲料中、在动物饲料添加剂中、在制备用于动物饲料的组合物中和/或用于改善饲料的消化率的用途,所述多肽选自:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列。
条款14.一种多肽在食品中、在食品添加剂中、在制备用于食品的组合物中和/或用于改善食品的消化率的用途,其中所述多肽选自:
a.与ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469具有至少85%序列同一性的氨基酸序列;和
b.包含ba59(seqidno:1)的氨基酸26-439、psd65(seqidno:11)的氨基酸24-424、psd67(seqidno:13)的氨基酸25-403或psf203(seqidno:15)的氨基酸16-469的功能片段的氨基酸序列。
实施例
提供以下实施例以说明本发明的各个方面。它们无意限制由所附权利要求书限定的本发明。
实施例1.序列搜索以鉴定新型候选植酸酶
为了鉴定新型植酸酶候选物,进行蛋白质家族分析。选择组氨酸酸性磷酸酶组(hap蛋白质家族)用于搜索。
为了开始组氨酸酸性磷酸酶的蛋白质家族分析,收集了属于该蛋白质家族的所有蛋白质。通过基于同源性的blast搜索以及通过蛋白质基序搜索来检索蛋白质。在blast搜索中,使用了泡盛曲霉(uniprotphya_aspaw)和大肠杆菌(uniprotppa_ecoli)的已知植酸酶序列作为查询。
针对几种序列数据库进行了blast搜索,以最大程度地覆盖可用序列。使用命令行blast查询以下数据库:ncbi蛋白质数据库(nr,pataa,tsa_nr,env_nr),ncbi核苷酸数据库(nt,patnt,env_nt,tsa_nt,nt,other_genomic),uniprot蛋白质数据库(包括swissprot和trembl),从jgi(https://genome.jgi.doe.gov/)下载的真菌基因组,真菌和细菌物种的专有基因组,经由sequencebase获得的专利序列数据,即来自thomsonreutersgeneseqtm、
所述blast搜索是在unix环境中使用shell脚本进行的。该脚本会遍历数据库,并针对每个数据库同时使用两个查询蛋白质序列进行批量blast搜索。使用1e-30的e值阈值。所述blast被请求为每个查询返回10000个最佳命中。否则,将使用默认参数。然后,脚本检索匹配命中的dna/aa序列。对于来自核苷酸数据库的blast命中,使用专门构建的脚本来处理xml格式的blast输出,并提取匹配部分作为氨基酸序列。使用基于同源性的blast搜索检索到总共14998个序列。
通过uniprot(2017-02版)检索基于蛋白质家族基序的序列。uniprot数据库已对uniprot中的所有序列注释了所有蛋白质基序。从uniprot中检索与组氨酸酸性磷酸酶蛋白质家族pfam-基序pf00328匹配的所有序列。使用这种方法检索到11924个序列。
使用上述过程可能会多次检索到相同的同一性序列。为了避免重复,使用聚类方法来鉴定和去除相同的氨基酸序列。cd-hit(http://weizhongli-lab.org/cd-hit/)是用于聚类和比较蛋白质或核苷酸序列的程序,其被用于进行聚类。在聚类程序之后,保留了8130个唯一的序列。
在检索了组氨酸酸性磷酸酶蛋白质家族的所有成员后,构建系统发育树以了解组氨酸酸性磷酸酶家族的多样性和亚家族。系统发育树的构建包括序列聚类以去除冗余,接着进行多序列比对和系统发育树的构建。
在进行多序列比对和系统发育树构建之前将蛋白质序列聚类。这样做是为了去除冗余。为了获得序列空间的均匀覆盖,使用cd-hit聚类程序将序列聚类为95%的同一性。最后剩下2309个序列。这2309个序列中的每个序列都是其自身或者是一组与其具有95%以上同一性的序列的代表。
使用多序列比对程序mafft(https://mafft.cbrc.ip/alignment/software/)对2309个所选候选物进行比对。使用fasttree(http://www.microbesonline.org/fasttree/)算法基于多序列比对来构建系统发育树(图2)。fasttree从核苷酸或蛋白质序列的比对中推断出近似最大似然的系统发育树。
根据系统发育树中包含的序列的描述和注释对所述树进行注释。除植酸酶外,所述树还含有葡萄糖-1-磷酸酶、肌醇六磷酸和二磷酸肌醇-五磷酸激酶、酸性磷酸酶(包括溶血磷脂酸磷酸酶和前列腺酸磷酸酶)、多种肌醇-多磷酸化物磷酸酶和一大批未表征的蛋白质,其中一些是暂时注释为磷酸甘油酸变位酶样蛋白。
所述树的大约三分之一(759个序列)在一个主要分支内,该分支含有已知的细菌植酸酶和已知的细菌葡萄糖-1-磷酸酶。该分支含有许多未表征的序列,其推定的注释范围介于植酸酶、葡萄糖-1-磷酸酶和磷酸酐磷酸水解酶之间。
所述树的大约三分之二(1373个序列)在一个主要分支内,该分支含有已知的真菌植酸酶和真菌酸性磷酸酶,以及暂时注释为磷酸甘油酸变位酶样的真菌未表征序列的大分支。在该较大的分支内,含有真菌植酸酶a型蛋白质的分支具有553个序列,而含有酵母磷酸酶和真菌植酸酶b型蛋白质的分支具有205个序列。后者含有主要通过pfam结构域搜索检索的序列,因为与用作blast查询的真菌植酸酶(曲霉属phya)的同源性很低。
从细菌和真菌分支中选择用于表达的候选序列。在系统发育树的不同部分之间的选择策略有所不同。从含有具有经过验证功能的序列的分支中挑取更多的候选物,而从仅含有具有未知功能的序列的分支中挑取极少的序列。为了帮助手动选择过程,通过在不同深度分裂系统发育树,将所述树分为几类。为每个主要分支选择合适的分割深度,得到不相交的小的子树的列表(图3)。从每个不相交的子树中选择单个代表性序列。
在最接近于已知植酸酶的子树中选择16个新型序列。从附近的分支中,总共选择160个候选物,以使得它们具有彼此至多80%的同一性。最后,从含有葡萄糖-1-磷酸酶的分支中选择彼此至多80%同一性的16个候选物。
在真菌分支中,从含有来自真菌的植酸酶a序列的子树中选择56个序列(彼此至多80%同一性)。所述真菌子树的不太有前景的部分在较高水平上被分割,产生22个不相交的子树以及因此22个候选物。这22个候选物的成熟氨基酸序列具有彼此至多45%同一性。
实施例2.使用的活性测定方法
平板测定法
在半定量植酸酶的平板测定法中,混浊的植酸化物在被水解时变得澄清。因此,通过所施加样品周围变澄清使植酸酶活性可视化。对于大肠杆菌细胞裂解物,情况就是这样。在使用平板测定法来选择木霉或芽孢杆菌转化体的情况下,所用样品是培养物上清液。
用于测定法的平板通过以下方法制备:将1.3%的植酸钠、4%的麦芽提取物、6.5%的酵母提取物、1%的酶促水解酪蛋白、0.01m的乙酸钠(使用1m溶液,ph4.5)和0.05m的cacl2(使用1m溶液)添加至50%的矿物盐溶液(4gkh2po4、6gna2hpo4、0.2gfeso4×7h2o、1mgcacl2、1g(nh4)2so4,加1l自来水)。使用去离子水并添加4%琼脂。将准备好的培养基高压灭菌(121℃下15分钟)并倒在培养皿上。为了施加酶样品,从硬化的琼脂平板上缝出小孔,并且将所述样品吸移到所述孔中。对于大肠杆菌细胞裂解物和芽孢杆菌培养物上清液,使用20μl的裂解物,并将平板在37℃下温育18小时。通过拍摄平板的照片来记录结果。
ppu测定法
植酸酶作用于植酸化物(肌醇六磷酸化物)以释放无机磷酸盐。通过分析从底物释放的无机磷酸盐的量来确定植酸酶活性。ppu分析方法用于筛选和表征在大肠杆菌、短小芽孢杆菌和里氏木霉中产生的新型植酸酶。在这种方法中,一个活性单位(称为ppu)是在以下条件下在一分钟内释放出1μmol的无机磷酸盐的酶量:ph5.0,反应温度37℃,反应时间15分钟,作为底物的植酸钠(c6h6o24p6na12,sigmap0109)浓度为10g/l。所述分析是基于通过磷钼酸盐复合物的还原而形成的颜色。
在所述测定法中,首先将酶样品在反应缓冲液(0.2m柠檬酸盐缓冲液,ph5.0)中稀释。将1ml的酶溶液用于分析。将1ml底物添加到酶样品中,并将混合物在37℃下精确地温育15分钟。通过添加2.0ml的15%(w/v)tca溶液来终止反应,之后将反应管混合并使其冷却至室温。水解反应混合物冷却后,通过吸移0.4ml样品并添加3.6ml水,将其以1:10稀释到试管中。添加由3份体积的1m硫酸、1份体积的钼酸铵(25g的钼酸铵(nh4)6mo7o24·4h2o,merck1182,于100ml水中)和1份体积的含10%抗坏血酸的水组成的4.0ml新鲜配制的显色试剂,并混合试管的内容物。将试管在50℃温育20分钟,然后在820nm处测量吸收。对于酶的空白值,用三氯乙酸淬灭后加入酶样品。
经由与已知浓度的磷酸盐溶液的显色反应的校准曲线来确定释放的磷酸盐的量。
为了分析ph3下的植酸酶活性,对ppu测定法进行了如下修改:使用ph3.0的0.2m甘氨酸-hcl来替代使用ph5.0的0.2m柠檬酸盐反应缓冲液。
4-mup测定法
人工底物4-甲基伞形酮-磷酸化物(4-mup)用于在筛选大肠杆菌中产生的植酸酶时直接动力学测量植酸酶活性。在测定中生成荧光信号。信号强度在酶促反应过程中发生变化。确定并给出信号相对于标准值变化的斜率。
在测定法中使用溶解于50mm柠檬酸钠缓冲液(ph5.00)中的0.25mm4-mup作为底物。从样品的不同稀释度分析斜率。将50μl的样品、空白和标准溶液吸移到96孔微量滴定板的孔中,然后将板转移到微量滴定板读取器,例如bmgpolarstaromega或bmgclariostar。在板读取器上通过将200μl的底物溶液吸移(使用板读取器的注射器)至每个孔来开始反应。随后监测酶反应(激发360nm,发射450nm,间隔时间1.00s)。样品一式三份进行分析。在孔中包含一式三份的标准物和空白。
ftu测定法
在ftu测定法中,检测通过植酸酶的水解酶作用从植酸钠底物释放的无机磷酸盐。通过分光光度法测量的颜色形成是钼酸盐和钒酸盐离子与无机磷酸盐络合的结果。一个植酸酶单位(ftu)是使用60分钟的温育时间,每分钟从37℃、ph5.50的植酸钠中释放出1μmol无机磷酸盐的酶量。当在ph3下进行活性分析时,将缓冲液和底物的ph调节至ph3.0,而不是ph5.5(参见下文)。
在所述测定法中,将2.0ml的1%植酸钠底物(labchemee05501,在ph5.5的250mm乙酸钠缓冲液中,并包括1mmcacl2·2h2o和0.01%tween20)吸移到塑料离心管中。将底物管在37℃下预温育5-10分钟,然后加入1.0ml稀释的酶样品。精确地温育60分钟后,添加2.0ml显色终止溶液,并通过涡旋混合试管内容物。与所述样品类似地制备酶空白,但是在添加稀释的酶样品之前将显色终止溶液添加到底物管中。对于显色反应,将试管在室温下温育20分钟,然后将其以4000rpm离心10分钟。在415nm处针对酶空白测量样品吸光度。对于活性单位,制备磷酸钾标准曲线(ph5.50)(干燥的kh2po4,merck1.04873.1用于标准;在称重之前在105℃下干燥2小时)。
终止溶液的制备如下(在即将使用前制备):对于100ml的显色终止溶液,将25ml的七钼酸铵储备溶液(20g的(nh4)6mo7o24·4h2o,merck1182于180ml水中,添加2ml氢氧化铵(nh4oh,sigma-aldrich22122828-30%),最终体积200ml)与25ml钒酸铵储备溶液(0.47g钒酸铵(nh4vo3,riedeldehaen31153)于160ml水中;一旦完全溶解,就加入4ml的22.75%硝酸溶液,最终体积200ml)混合。然后,加入16.5ml的22.75%的硝酸溶液(hno3,merck1.00456),然后加入蒸馏水以使容量瓶中的体积达到100ml。
使用肌醇-6-磷酸化物(insp6,ip6)和肌醇-4-磷酸化物(insp4,ip4)作为底物的植酸酶的动力学分析
使用来自里氏木霉转化体的发酵培养物上清液分析了ip6和ip4底物的磷酸盐形成动力学。所述测定法在ph3.0和ph5.0下进行。首先,如下使用pd10柱将酶制剂脱盐。将2.5ml的植酸酶材料装载到pd10柱上,并使用ph5.0的0.2m柠檬酸盐缓冲液洗脱。收集3.5ml的洗脱液。用ph5.0的0.2m柠檬酸钠/0.15mnacl或用ph3.0的0.2m甘氨酸/hcl/0.15mnacl稀释洗脱液,分别用于ph5和ph3分析。
分析中使用的植酸十二钠盐(ip6)购自sigma-aldrich(德国的陶夫基兴)。主要生成ip4特异性异构体级分的方法以4个步骤进行。在第一步骤中,如greiner&konietzky,(1996)中所述,将quantumblue植酸酶固定在5mlhitrapnhs活化的琼脂糖凝胶柱(来自美国波士顿的generalelectric)上。在第二步骤中,固定化的植酸酶在ph5.0的0.1m乙酸钠缓冲液中逐步降解ip6成低级肌醇磷酸化物。5ml/min的流量实现了ip4的最高部分。下一步骤用于从溶液中去除磷酸盐,并将ip4与其他不需要的肌醇磷酸化物分离。因此,用来自biorad(美国的赫拉克勒斯)的ag1-x4树脂手动填充的阴离子交换柱装载所产生的肌醇磷酸化物混合物,并用0.5mhcl洗脱ip4。在最后的步骤中,用旋转蒸发仪去除hcl,并将ip4重新溶解在水中。
以ip6和ip4为底物的动力学反应在37℃下进行10分钟,并追踪在820nm处的吸光度。
实施例3.一组细菌源潜在植酸酶基因在大肠杆菌中的表达作为预筛选
在大肠杆菌中对从基因库搜索获得的细菌源序列(实施例1)进行预筛选,然后在里氏木霉(实施例5)和短小芽孢杆菌(实施例6)生产平台中表达所选分子。选择这种方法是因为它可以更快地表达大量分子。在木霉中直接筛选来自数据库搜索的真菌源植酸酶基因(实施例4)。
从公共数据库的序列研究(实施例1)中选择总共192个细菌hap基因在大肠杆菌中表达。将从genewiz订购的序列克隆到质粒pet28-(a)中以进行细胞内大肠杆菌表达。通过热休克法将获得的质粒转化到大肠杆菌shufflet7express(neb)中。将转化体在摇瓶中培养。因此,从具有单个菌落的新鲜平板接种10mllb+30mg/ml卡那霉素的预培养物,并在200rpm摇动下于30℃温育过夜。用0.5ml所述预培养物接种在50mllb+30mg/ml卡那霉素中进行主培养,并在30℃和250rpm下温育直至达到od600≈1.0。通过添加1mm(最终浓度)iptg引发诱导。然后,将所述培养物在16℃和250rpm下进一步温育18小时。完成培养后,通过离心收集细胞,使用10%甘油以1/4的培养体积洗涤,并将细胞团块冷冻在-20℃。使用dna酶裂解缓冲液(50mmtris,2.5mmmgcl2,0.5mmcacl2,1mg/ml溶菌酶,1u/mldna酶i)裂解细胞。将团块重悬于dna酶裂解缓冲液中,并将悬浮液在37℃下温育1小时。通过离心去除细胞碎片。用代表整个细胞内蛋白质提取物的上清液进行进一步分析。
对每种蛋白质提取物进行半定量板活性测定(实施例2)、sds-page和基于定量高通量荧光的磷酸酶活性测定(4-mup;实施例2)。在这些测试中显示出活性的那些分子另外被定向到更特异性的植酸酶活性测定(以ip6为底物的ppu测定,实施例2)。选择在大肠杆菌中产生的显示良好植酸酶活性的分子以在木霉中表达(图1),并且还选择一组植酸酶在芽孢杆菌中表达。对于里氏木霉表达,重新订购使用了里氏木霉的优化密码子的的编码这些分子的基因(实施例5)。对于短小芽孢杆菌表达,密码子使用是大肠杆菌表达中使用的密码子,或者订购了对于短小芽孢杆菌优化的基因。
在192种新型潜在植酸酶基因中,在大肠杆菌中产生了89种(约46%),而103种(约54%)未产生或作为蛋白质或植酸酶活性的生产水平低到无法检测。在上述69种植酸酶中,有35种(约39%)在植酸化物选择板上显示活性(形成光晕),并且在sds-page分析中检测到蛋白质带,有13种(约15%)在板上具有活性但没有在sds-page中检测到质量与预期植酸酶质量相对应的蛋白质带,并且即使在sds-page中检测到了预期分子量的蛋白质带,有21种(约24%)在板上也没有活性。在89个候选物中,有20个的蛋白质带非常微弱或者在植酸化物板上只形成很小的光晕,表明生产水平非常低。
选择总共27种在所进行的分析中显示出最佳活性的新型细菌植酸酶用于在里氏木霉中表达(实施例5)。
实施例4.一组真菌源植酸酶基因在里氏木霉中的表达
选择来自数据库搜索(实施例1)的源自真菌(酵母或丝状真菌)的总共78种新型植酸酶以在里氏木霉中表达。
从geneart(thermofisherscientific)订购具有天然信号序列的相应基因作为具有里氏木霉密码子使用的合成基因。将所述基因克隆到表达盒中,在其中将它们与里氏木霉cbh1(纤维二糖水解酶1)启动子融合(直接融合)。通过里氏木霉cbh2(纤维二糖水解酶2)终止子终止转录。编码乙酰胺酶的合成基因(amds)用作质粒中的转化标记。psk268_3mc是一个例外,因为它是使用类似于里氏木霉中表达的细菌源植酸酶的载体多肽生产的(实施例5)。使用原生质体转化将包括表达盒的质粒转化到里氏木霉中。从每组中,在摇瓶中的纤维素酶诱导培养基中培养3-10个转化体,并在sds-page凝胶中分析培养物上清液以确认是否产生了植酸酶并分泌到真菌培养物上清液中。从培养物上清液分析植酸酶活性,其中检测到预期分子量的可见蛋白质带。在分析中使用ppu测定法(实施例2)。在发酵罐培养中产生在里氏木霉培养物上清液中显示最佳植酸酶活性的候选物(实施例7),并对其进行更详细的表征(实施例8-9)。分析中使用了来自所选转化体发酵的培养物上清液。如实施例10-13中所述,在体外动物模拟试验中进行所选候选物在植酸化物降解中的效率。
从所表达的78种真菌植酸酶,仅16种(约21%)在里氏木霉中良好产生。其余没有产生或生产收率非常低,这表明植酸酶蛋白质对里氏木霉蛋白酶的低的表达或生产水平或敏感性。
实施例5.所选细菌源植酸酶基因在里氏木霉中的表达
从geneart(thermofisherscientific)订购从大肠杆菌筛选(实施例3)中选择的27种新型细菌植酸酶作为合成基因。将基因订购为成熟的植酸酶编码序列(无天然信号序列),并与表达质粒中的载体多肽编码序列融合。所使用的载体是里氏木霉cbhiicbd(a)和天然cbhii的铰链(b)区(paloheimo等,2003)。在所述载体与植酸酶编码序列之间包括编码kex2切割位点(rdkr)的序列。使用里氏木霉cbh1启动子表达遗传构造。使用里氏木霉cbh2终止子终止转录。包括编码乙酰胺酶的合成基因(amds)作为质粒中的转化标记。使用原生质体转化将包括表达盒的质粒转化到里氏木霉中。培养从每个转化中选择的转化体,并以与关于真菌源植酸酶所述类似的方式(实施例4)表征所产生的植酸酶。
在所表达的细菌植酸酶中,在里氏木霉中产生了13种(48%)。其余没有产生或生产收率非常低,这表明植酸酶蛋白质的低表达或生产水平或对里氏木霉蛋白酶的敏感性。
实施例6.所选细菌源植酸酶基因在短小芽孢杆菌中的表达
选择了总共15种植酸酶基因在短小芽孢杆菌中表达,短小芽孢杆菌可替代地用作新型植酸酶的生产宿主。在这些候选物中,有13种未在木霉中产生,或者它们的收率非常低。编码植酸酶psf203和ba59的两个基因在木霉中充分产生。对于候选物中的10个,对大肠杆菌和芽孢杆菌优化的dna序列均进行了表达测试,以分析密码子使用对表达水平的差异影响。所述芽孢杆菌优化序列购自genewiz,并且大肠杆菌优化序列如同用于大肠杆菌预筛选的那些。
将植酸酶基因克隆到芽孢杆菌高拷贝表达质粒中的信号肽下游,以进行细胞外分泌。所述质粒带有卡那霉素抗性盒以供选择。所述质粒的构建是使用带有用于诱导感受态的质粒的高感受态枯草芽孢杆菌构建宿主进行的。通过使用电穿孔将所述质粒转化到短小芽孢杆菌生产宿主中。在摇瓶中通过在具有20mg/ml卡那霉素的10mllb培养基中接种预培养物并在37℃在摇动(180rpm)下温育过夜以进行表达。用1ml的预培养物接种主培养物,并在180rpm和37℃下温育31小时。在无抗生素的主培养物中使用20ml培养基(2%葡萄糖、6%玉米浆粉、1.32%(nh4)2hpo4、0.05%mgso4×7h2o、0.5%caco3,ph调节至7.3并在121℃高压灭菌30分钟)。将培养物离心以去除细胞,并将上清液在-20℃储存以供进一步分析。所述样品的分析包括植酸酶平板测定、sds-page和在ph5下的ppu分析以及在ph3下的ftu分析(实施例2)。
从15个候选物中的11个检测到sds-page中的蛋白质信号,并且8个候选物在植酸酶平板测定中显示出活性。在某些情况下,芽孢杆菌优化序列的蛋白质收率略高。在ph3下,只有3个测试候选物(包括也在里氏木霉中表达的psd65和psd67)具有活性。然而,与在木霉中表达相应基因时获得的活性相比,所述活性水平非常低。
实施例7.选择用于表征的新型植酸酶
选择了在里氏木霉培养物上清液中具有最佳测量活性的总共15种新型植酸酶用于表征(表1)。在所选候选物中,有7种是细菌来源的并且有8种是真菌来源的。用于表征的植酸酶在里氏木霉中产生。
表1.选择用于表征的新型植酸酶。示出了植酸酶的名称、基因来源、全长氨基酸序列的seqidno、表达盒中使用的核苷酸序列的seqidno和原始数据库条目号。
新型植酸酶的氨基酸序列的同一性比较在图10中示出。使用needleman-wunsch全局比对(成本矩阵:blosum62,空位开放罚分12,空位延伸罚分3)进行比较。在分析之前将信号序列去除。使用signalp4.1(http://www.cbs.dtu.dk/services/siqnalp/)对信号序列进行分析。
所述植酸酶之间的氨基酸序列同一性较低(图10)。都来自狄克氏菌属物种的psd34和psd35之间的同一性最高,为75.9%。其他植酸酶彼此之间的同一性为12%至60.2%(图10)。
实施例8.分析所选植酸酶的ph和温度曲线
确定ph和温度曲线
从相应的里氏木霉转化体的培养物上清液测量植酸酶的ph和温度曲线。均在里氏木霉中产生的曲霉植酸酶(aspaw)、大肠杆菌野生型植酸酶和来自产品quantumblue(qb)的大肠杆菌突变型植酸酶用作参考。
在50℃下在ph3.0、4.0、5.0和6.0中以植酸酶活性(ppu,实施例2)测量ph曲线。使用柠檬酸将反应缓冲液(0.2m柠檬酸盐缓冲液)的ph调节至反应ph。酶在每个ph下的活性显示为相对于在所讨论酶的最佳ph下的活性值归一化的相对活性(%)(表2)。
表2.选择的新型植酸酶的ph曲线。示出了与植酸酶在其最佳ph下的活性相比的相对植酸酶活性(%)。参考是曲霉植酸酶(aspaw)以及大肠杆菌野生型和突变型植酸酶。
大多数的新型细菌植酸酶以及细菌参考在ph4下具有其最佳ph,而大多数的真菌植酸酶和真菌参考在ph5下具有其最佳ph。ba59、ye76、psd35、psd65、psd67、psf203和psk267_ii以及参考植酸酶在ph3下仍具有相对较好的活性(40%或更高)。
确定温度曲线
以在ph5.0下在40-80℃的温度下的植酸酶活性(ppu,实施例2)测定温度曲线(表3)。在每个温度下的活性显示为相对于每种植酸酶在最佳温度下的值归一化的相对活性(%)。
表3.选择的新型植酸酶的温度曲线。示出了与最佳温度下的活性相比的相对植酸酶活性(%)。参考是曲霉植酸酶(aspaw)以及大肠杆菌野生型和突变型植酸酶。
大肠杆菌野生型参考、ba59、psd65、psd67、psk252和psk267_ii的最佳温度为60℃,而ye76、psd32、psd34、psd35、psf207、psk256、psk260和psk268_3mc的最佳温度为50℃。psd65在70℃下仍具有高活性(62%)。大肠杆菌突变型植酸酶的最佳温度为70℃,并且在80℃下仍具有高活性(83%)。psf203和真菌参考植酸酶(aspaw)的最佳温度在70℃。psk235的最佳温度在40℃。
在高温中的稳定性是有利的,例如当所述多肽用于在制造期间在高温下加工的饲料制剂中时,例如在颗粒状制剂的生产或饲料调理和粒化中。
实施例9.使用ip6和ip4异构体的纯化植酸酶的动力学分析
如实施例2所述,将在里氏木霉中产生的所选新型植酸酶用于动力学分析,其中以ip6和ip4异构体作为底物。商业quantumblue植酸酶产品(qb)和曲霉源aspaw用作分析中的参考。
在ph3和5下,参考植酸酶qb和aspaw比起降解ip4更有效地降解ip6(分别为图4a-b和图4o-p)。几种新型植酸酶在降解ip4方面比参考更有效(图4)。在ph3和5下,psd65、psd67和psk256降解ip4的效率均高于降解ip6的效率。psf203、ba59和ye76在ph3下可有效降解ip4,而psd35在ph5下有效降解ip4。在两个ph下,其他测试的植酸酶(psd34、psk260和psk267_ii)降解ip6的效率与ip4相比更高。
实施例10.胃肠模拟(git)测试
使用胃肠模拟(git)测试系统对所选的新型植酸酶候选物进行比较,它们在降解饲料材料中的植酸化物的能力方面显示良好的ip4降解(并以psk260作为阴性对照)。所述git(sommerfeld等,2017)是一项三步骤连续体外模拟测试,以分析在动物消化条件下植酸酶降解饲料材料中植酸化物的能力。反应在40℃下以及在相应的ph和ph变化下进行,如在肉鸡的嗉囊、砂囊和小肠中。也添加相应的消化酶。为了成功进行git测定,所述植酸酶需要具有有利的生化特性的组合。它需要抵抗不同的ph和消化道的温度并起作用。它还需要对消化道的蛋白酶具有抗性。
在git筛选测试中,每千克底物材料使用250ftu的植酸酶。将植酸酶样品添加到ph5.2的1g的细磨玉米-豆粕材料中,并且首先在40℃温育30分钟,然后通过添加hcl将ph降至2.9。加入胃蛋白酶(3000u/g)。温育45分钟后,通过添加naoh使ph增加至6.1。此外,加入胰酶(18.5mg/g)。将混合物温育60分钟。在冰上用总共10ml、ph8.00的0.1mnaf、0.2medta终止反应。在终止溶液中将肌醇磷酸化物提取2×30分钟。在使用高效离子色谱法(hpic)分析肌醇磷酸化物(ip6-ip3)之前,用30kda离心过滤器从上清液中去除植酸酶。ip6-ip3分析是根据blaabjerg等,2010(k.blaabjerg等,/j.chromatogr.b878(2010)347-354)进行的。
来自植酸酶产生里氏木霉转化体的发酵培养的培养物上清液用作git测定中的样品。新型植酸酶ba59、psd35、psd65、psd67和psf203与参考相比更有效地将底物降解为低级ip。ye76、psk256和psk260在ip6降解方面不比参考更好。获得的结果如图5所示,并汇总在表4中。
表4.git结果概述。qb,quantumblue植酸酶产品;参考,大肠杆菌突变型植酸酶,以与新型植酸酶类似的发酵中在里氏木霉中产生的quantumblue产品的植酸酶。
进行了剂量效应试验研究,其中添加了不同量的活性单位(125、250、500和1000ftu/kg底物材料)。用两种新型植酸酶ba59和psd65进行了剂量效应研究。来自quantumblue产品的突变型大肠杆菌植酸酶用作参考。在这些测试中,与参考相比,随着剂量增加,ba59和psd65明显地降解更多的ip4并产生远远更多的低级肌醇磷酸化物(图6)。
实施例11.使用添加矿物质的饲料作为底物的胃肠模拟(git)测定
在该实验中,使用添加有矿物质和其他饲料成分的饲料材料代替纯玉米-豆粕混合物,利用ba59、psd65和参考植酸酶进行git测定。所述饲料材料用于鉴定植酸酶候选物之间在降解植酸化物时对矿物质和其他饲料成分的脆弱性差异。给与250、500和1000ftu/kg的植酸酶。
当使用含矿物质的饲料材料作为底物时,与参考相比,随着剂量增加,ba59和psd65也产生远远更多的低级肌醇磷酸化物(图7)。
实施例12.使用三种底物的胃肠模拟(git)测试
在该实验中,使用以下三种底物利用ba59、psd65和参考植酸酶进行git测定:玉米-豆粕和两种添加有其他饲料成分的混合饲料,一种含5.4g/kg的磷和9g/kg的钙(“阴性对照饲料”,nc)而另一种含8g/kg的磷和9g/kg的钙的饲料(“阳性对照饲料”,pc)。所述实验中使用剂量为250ftu/kg的植酸酶。
与参考相比,ba59和psd65从所有底物都产生更多的低级肌醇磷酸化物(图8)。
也确认了ba59和psd65在动物饲养试验中的效率。
实施例13.新型植酸酶与商业quantumblue(qb)产品中包含的突变型大肠杆菌植酸酶组合的胃肠模拟(git)测试
由于一些新型植酸酶在ip4降解方面优于qb,因此使用git测定法测试了新型植酸酶与来自qb的大肠杆菌突变型植酸酶组合的效率。这些植酸酶根据植酸酶活性(ftu/kg,ph5.5)组合在一起,其中设定活性的大肠杆菌突变型植酸酶(250ftu/kg)和增加剂量的ba59或psd65(62.5、125和250ftu/kg)组合。作为参考,添加另外的相同剂量的大肠杆菌突变型植酸酶。
当添加62.5ftu/kgba59和psd65时,与参考测试相比,ip6、ip5和ip4的降解效率更高。与来自qb的大肠杆菌突变型植酸酶相比,ba59和psd65都降解更多的ip4,psd65效率更高。
当添加相同活性的大肠杆菌突变型植酸酶时,随着相对于250ftu/kg大肠杆菌突变型植酸酶ba59和psd65的剂量增加,更多的ip4被降解。对于两种剂量(125和250ftu/kg),psd65最有效。在250ftu/kg的剂量下,与大肠杆菌突变型植酸酶形式的相同活性相比,psd65降解ip4多一半以上。与大肠杆菌突变型植酸酶相比,ba59的所有组合剂量均导致更好的降解,而ip4降解中最有效的是添加250ftu/kg。
结果(两次重复测试的平均值)在图9中示出。
参考文献:
bedford,m.r.和c.l.walk.2016.植酸化物还原为四磷酸化物(ip4)至三磷酸化物(ip3)或者甚至更低级不会消除其抗营养特性(reductionofphytatetotetrakisphosphate(ip4)totrisphosphate(ip3),orperhapsevenlower,doesnotremoveitsantinutritiveproperties).在:《植酸化物破坏-精细动物营养后果》(phytatedestruction-consequencesforprecisionanimalnutrition).walk,c.l.,kühn,i.,stein,h.h.,kidd,m.t.和rodehutscord编,m.wageningen学术出版社:45-52中。
lee,s.a.和m.r.bedford.2016.肌醇-有效的生长促进剂?(inositol-aneffectivegrowthpromotor?)world'spoultrysciencejournal.72:743-760。
menezes-blackburn,d.,s.gabler和r.greiner.2015.七种商业植酸酶在家禽消化道体外模拟中的性能(performanceofsevencommercialphytasesinaninvitrosimulationofpoultrydigestivetract).jagric.foodchem.63:6142-6149。
xu,p.,j.price,a.wise和p.j.aggett.1992.肌醇磷酸化物与钙、锌和组氨酸的相互作用(interactionofinositolphosphateswithcalcium,zinc,andhistidine).journalofinorganicbiochemistry47:119-130。
zeller,e.,m.schollenberger,i.kühn和m.rodehutscord.2015.肉鸡消化道的不同区段中在未补充或补充植酸酶的情况下植酸化物的水解和肌醇磷酸化物异构体的形成(hydrolysisofphytateandformationofinositolphosphateisomerswithoutorwithsupplementedphytasesindifferentsegmentsofthedigestivetractofbroilers).journalnutritionalscience4,e1:1-12。
pct/fi2019/05087509.12.2019序列表
<110>生化酶股份有限公司(abenzymes)
<120>具有植酸酶活性的多肽
<130>31747
<160>30
<170>patentin3.5版
<210>1
<211>439
<212>prt
<213>水生布戴约维采菌(budviciaaquatica)
<400>1
metgluileileaspargleuserlysleuseralaleumetleuleu
151015
glycyssercysleuproalaleualavalserthrprovalasngly
202530
tyrvalleugluargvalvalileleuserarghisglyvalargser
354045
prothrlysglnthraspleumetasnasnvalthrproasplystrp
505560
proglntrpprovalglnalaglytyrleuthrproglnglygluhis
65707580
leumetthrleumetglyglyphetyrargasptyrpheargserhis
859095
asnleuleuproserglnglycysprothraspglyserleutyrval
100105110
trpalaaspileaspglnargthrargleuthrglyglnalapheleu
115120125
alaglyvalalaproglucysaspleulysilehishisglnalaasp
130135140
leulysthrthraspalaleuphehisprovalglualaglyilecys
145150155160
lysleuasplysglnglnthrglnglnalavalglulysglnleugly
165170175
alaproleualathrleuserglnargtyralaleuproleualagln
180185190
metglygluileleuasnphealaalaserprotyrcyslysglumet
195200205
glnthrlysglyglnsercysaspphealaserphethrproasngln
210215220
ilehisleuserproasnglyglnlysvalserleuserglyproleu
225230235240
alaleuserserthrleusergluilepheleuleuglntyralagln
245250255
glymetprogluvalalatrpglnargleuserglygluaspasntrp
260265270
argserleumetserleuhisasngluglnpheasnleumetalalys
275280285
thrprotyrilealaserhislysglythrproleuleulysgluile
290295300
seralathrleualaglyglnglnglythrleulysargproalaasn
305310315320
asnargileleupheilealaglyhisaspthrasnilealaasnile
325330335
alaglymetleuglyleuasntrpgluleuprohisglnproaspasn
340345350
thrproproglyglyglyleuvalphegluleutrpasnasnprogln
355360365
asphisglnglntyrvalservallysmetphetyrglnthrmetglu
370375380
glnleuargasnglyglulysleuaspmethishisproalaglymet
385390395400
valglnvalalailealaglycysgluasnserasnserservalleu
405410415
cysserleulysaspleuglnlyslysvalserglnalaileglnpro
420425430
alacysglnleusermetgln
435
<210>2
<211>1245
<212>dna
<213>水生布戴约维采菌(budviciaaquatica)
<400>2
gtcagcacgcccgttaacggctatgtcctcgagcgcgtcgtcatcctcagccgccacggc60
gtccgaagccctaccaaacagacggacctcatgaacaacgtcacccccgataaatggcct120
cagtggcccgttcaagctggctacctcacccctcaaggtgagcatttaatgaccctcatg180
ggcggcttttaccgcgactactttcgcagccacaatttactgccctcgcaaggttgccct240
accgacggcagcctctacgtgtgggccgacatcgaccagcgcacccgactcaccggtcaa300
gcttttctcgctggcgtcgcccccgaatgcgacctcaagatccaccatcaagctgattta360
aagacgaccgacgctttatttcaccccgtcgaggctggcatctgcaaactggacaagcag420
cagacccagcaagctgtcgaaaagcagctcggcgcccctctcgccacgctgagccagcga480
tacgctctgcccctcgcccaaatgggcgagatcctcaattttgccgccagcccctactgc540
aaggaaatgcagaccaagggccaatcgtgcgacttcgcttcgttcacccccaaccagatc600
catctcagccccaacggccagaaagtgtcgctgagcggtcctctcgccctcagctcgacc660
ctcagcgagatctttttactccaatacgcccaaggtatgcccgaagtcgcttggcaacgc720
ctctcgggcgaggacaactggcgctctttaatgtctttacacaacgagcagttcaattta780
atggccaagaccccctacatcgccagccacaagggcacccctttactcaaggagatcagc840
gccaccctcgctggccagcaaggtaccctcaagcgccccgctaacaaccgcatcctcttc900
atcgccggtcacgacaccaacatcgccaacatcgctggcatgctgggcctcaactgggaa960
ctcccccaccagcccgataatacgcctcccggcggtggcctcgtctttgagctgtggaac1020
aacccccaagatcaccagcagtacgtcagcgtgaagatgttttaccagacgatggagcag1080
ctgcgcaacggcgagaagctggatatgcaccatcccgctggcatggtgcaagttgccatc1140
gctggctgcgagaacagcaatagcagcgtgctgtgtagcctcaaggatttacagaagaag1200
gtcagccaagccatccaacccgcttgtcagctctcgatgcagtaa1245
<210>3
<211>454
<212>prt
<213>嗜虫耶尔森菌(yersiniaentomophaga)
<400>3
metilethrilelysarglysleuserleuseralametileleuthr
151015
pheileglyseralailethrileprolysvalvalalaglugluval
202530
glytyrthrleuglulysvalvalileleuserarghisglyvalarg
354045
serprothrlysglnthrgluleumetasngluvalthrproasplys
505560
trpproglutrpprovallysalaglytyrleuthrproargglyglu
65707580
glnleuvallysilemetglyhisphetyrglyglntyrphearghis
859095
thrglyleuleuprolysgluargcysproalagluglyglnvaltyr
100105110
valtyrseraspileaspglnargthrleuleuthrglyglnalaleu
115120125
ileaspglyilealaproglncysglyphelysilephehisglnlys
130135140
asnleulysglnileaspproleuphehisprovalglualaglyile
145150155160
cysalaleuasnalalyslysthrglnglualaileglulyslysleu
165170175
glyalaproilethrthrleuserglnargtyralalysserleuala
180185190
leumetglylysvalleuasnpheseralaserprotyrcysglnlys
195200205
metglulysglnglymetaspcysasnphealaaspleuserproasn
210215220
gluvalglnvalasnglngluglyserlysalaserleuserglypro
225230235240
valalaleuserserthrleualagluilepheleuleuglnasnser
245250255
glnglymetproilevalalatrphisargleuleuthrarggluasp
260265270
trpglnthrleumetleuleuhisasnserglnpheaspleumetala
275280285
lysthrprotyrilealaglnhislysglythrproleuleuglngln
290295300
ileasnvalalaleuleuglnprogluglnglylysalaglyleuasp
305310315320
lysglnproalathrlyslysthrleuserasnthrglnleuproasp
325330335
asnasnalavalpheileleuglyglyhisaspthrasnilealaasn
340345350
valalaglymetleuglyleuasntrpthrleuproleuglnproasp
355360365
asnthrproproglyglyglyleuvalphegluargtrpargaspgln
370375380
hisglyglnserphevalalailelysmetphetyrglnthrleuasn
385390395400
glnleuargasnmetglulysleuaspleuserileasnproalagly
405410415
metvalserileaspileproglycysgluasngluglygluasnlys
420425430
leucysargleualathrpheglnlyslysvalvalglualaileglu
435440445
proalacysasnleupro
450
<210>4
<211>1296
<212>dna
<213>嗜虫耶尔森菌(yersiniaentomophaga)
<400>4
atccccaaggtggtcgccgaggaggtcggttacaccctcgagaaggtggtcattttatct60
cgtcacggtgtccgcagccccacgaagcagaccgagctcatgaacgaggtcacccccgac120
aagtggcccgaatggcccgtcaaagccggttacctcaccccccgaggcgagcagctcgtc180
aagatcatgggccacttctacggccagtacttccgccacaccggcctcctccccaaggag240
cgatgtcccgctgagggccaagtttacgtctacagcgacattgatcagcgaacgctgctg300
accggccaagctctgatcgacggcatcgccccccagtgcggcttcaagatcttccaccag360
aagaacctcaaacagatcgaccctttattccaccccgttgaggctggcatttgcgccctc420
aacgccaaaaagacccaagaagctatcgaaaagaagctgggcgcccccatcaccacttta480
agccagcgctacgccaagtcgctcgccctcatgggtaaagtcctcaacttcagcgccagc540
ccctactgtcagaagatggagaagcaaggtatggactgcaacttcgccgacctctcgccc600
aacgaggtccaagttaatcaagaaggctcgaaggcctctttatcgggtcccgtcgctctc660
agcagcaccctcgccgagatttttttattacagaattcgcaaggtatgcccatcgtcgct720
tggcatcgtttactcacgcgcgaggattggcagactttaatgctgctccataactcgcaa780
ttcgatctcatggccaagaccccctacatcgcccagcacaagggtacccctctgctgcag840
cagatcaacgtcgctttactgcaacccgaacaaggtaaggccggcctcgacaagcagccc900
gctaccaagaagaccctctcgaacacccagctccccgataacaacgccgtgttcatcctc960
ggcggccacgacacgaacatcgccaacgtggccggcatgctgggcctcaactggacttta1020
cctctccagcccgataacacccctcccggtggtggcctcgtctttgagcgctggcgagac1080
cagcacggccagtcgtttgtcgccatcaagatgttctaccagaccctcaaccagctccgc1140
aacatggagaagctcgacctcagcatcaaccccgccggcatggtgagcatcgacatcccc1200
ggttgtgagaacgagggcgagaacaagctgtgccgcctcgctaccttccagaagaaggtg1260
gtcgaagccatcgaacccgcttgtaacctcccctaa1296
<210>5
<211>432
<212>prt
<213>假单胞菌属种(pseudomonassp.)
<400>5
metlysthrleuleuproleutyrthrglyleuleuleualasermet
151015
leuserproalaalaleualaglnthrprothrseraspglytyrala
202530
leuasplysvalvalglnvalserarghisglyvalargproprothr
354045
lysserasnglulysleuleuthralavalthrglnargglntrppro
505560
thrtrpleuvalpropheglyasnleuthrglyhisglytyrthrgly
65707580
alavalgluleuglyargtyrargglygluvalleuargthralagly
859095
leuileprolysglycysproaspalaserglnleuleuvalhisala
100105110
serproleuglnargthrlysalathralaglnalaleuleuaspgly
115120125
metpheproglycysglyleuglnprothrtyrvalserglyaspgln
130135140
aspalaleupheglnalaasnglumetprophealaalaleuasppro
145150155160
vallysalalysalaaspileleulysalaleuglyglyservalglu
165170175
alaalaglnalaargtyrlysprophegluglulysleuarggluile
180185190
alacysleuproalaalaaspcysprotyrglylysglnalatrpthr
195200205
leugluglnasnalalysglyargphealailelysglyleuserala
210215220
glyalaasnmetglygluvalpheargleuglutyrserglnglyleu
225230235240
provalasplysvalalapheglyasnglyargthralaalagluval
245250255
serserleumetvalleuthrlysalalystyrasptyrleuasnasp
260265270
ileprotyrilealaserargglyalasergluleumetasnglnile
275280285
serleuglnleulysglnglythrproleualaprolysaspalagln
290295300
serileproproaspvalproleumetleuleuvalalahisaspthr
305310315320
asnilesertyrleuargthrmetleuglypheglytrplysglngly
325330335
asptyrilegluasnasnileproprovalglythrleuglnpheglu
340345350
argtyrlysgluvallysthrglyglutyrpheleuargilealaphe
355360365
glualaglnsermetaspglnileargasnleuthrglnleuthrala
370375380
glyglnlysproleulysthrasppheasnseralalysaspcysarg
385390395400
asnthrservalglyleuleucysproleulysglyalametalathr
405410415
valaspglnasnileaspprothralaleuthrprotyralapheglu
420425430
<210>6
<211>1230
<212>dna
<213>假单胞菌属种(pseudomonassp.)
<400>6
cagacccccacgagcgacggctacgctttagataaggtcgtccaagtcagccgccacggc60
gtccgccctcccaccaagagcaacgagaagctcctcacggctgtcacccaacgccagtgg120
cccacttggctggtccccttcggcaacctcacgggccacggttatacgggcgccgtcgag180
ctcggccgctatcgcggtgaggtcctccgaacggccggcctcatccctaagggctgcccc240
gatgctagccagctgctggtccacgctagccctctgcagcgcaccaaggctacggcccaa300
gctctcctcgatggcatgttccccggttgcggcctccaacccacctacgtcagcggcgac360
caagatgccctctttcaagctaacgaaatgccttttgccgccctcgaccccgttaaggct420
aaggccgacattttaaaggctctcggcggcagcgtcgaggccgcccaagctcgctacaag480
cccttcgaggagaaactccgcgagattgcttgtctgcccgccgctgactgcccctatggc540
aagcaagcttggaccctcgagcagaacgccaagggccgcttcgccattaagggtctcagc600
gccggcgccaacatgggcgaagtgttccgtttagaatacagccaaggtctccccgtcgac660
aaggtcgcttttggcaacggccgaaccgccgccgaggtgagcagcctcatggttttaacc720
aaggccaagtacgactatttaaacgacatcccttacattgcctctcgtggtgccagcgag780
ctcatgaaccagatcagcctccagctgaaacaaggtacgcccctcgcccctaaggacgcc840
caatcgatcccccccgatgtccctttaatgctcctcgtcgcccacgacacgaacatctcg900
tatttacgcaccatgctcggtttcggctggaagcaaggtgactacatcgagaacaacatt960
ccccccgtgggcactttacaatttgagcgctacaaggaggtcaagaccggcgagtacttc1020
ctccgcatcgcctttgaggctcaaagcatggaccagatccgcaacctcacgcaactcacg1080
gccggccagaagcccctcaagacggactttaacagcgccaaggactgccgcaacacgagc1140
gtcggcctcctctgccctctgaagggcgctatggctaccgtcgaccagaacatcgacccc1200
accgccctcaccccttacgccttcgagtaa1230
<210>7
<211>424
<212>prt
<213>狄克氏菌属(dickeya)
<400>7
metilethrargthrthralaalaglyleuleuleuseralaleupro
151015
leupheasnthrglnalaglnthrthrthrasptrpglnleuglulys
202530
valvalgluileserarghisglyvalargproprothrgluglyasn
354045
metlysthrileglnalaglythrglyargalatrpproglutrpleu
505560
thrargtyrglygluleuthrglyhisglytyralaalaalavallys
65707580
lysglyglntyrgluglyasntyrleuargglnhisglyleuleuthr
859095
glnglycysproglnproglygluleuphevaltrpalaserproleu
100105110
glnargthrarggluthralametalaleumetaspglyvalphepro
115120125
glycysglyvalalaileglnglyproalasergluasphisaspthr
130135140
leuphehisalagluaspalaglyvalthrleuaspglnalaglnval
145150155160
lysalaalaleuasphisalametglnglyglnseralaglyglnile
165170175
glnthrargleuglnprovalileasplysleulysglnalavalcys
180185190
leuproasnthralacysproalapheaspthrglutrpalailelys
195200205
gluserlyslysglyasnleuserleuhisglyprogluthrleuala
210215220
asnmetalagluthrileargleualatyrserasnasnasnproleu
225230235240
serglnvalalapheglyhisalaargseralaalagluvalglythr
245250255
leuleuthrleualathralaglntyraspphethrasnaspleupro
260265270
tyrvalalaargargglyalaserthrleumethisglnilealaleu
275280285
alaleuthrprogluglnargproaspalaproproglualalystrp
290295300
leuleutyrvalalahisaspthrasnilealalysleuargthrleu
305310315320
leuglyphethrtrpglnglnglyasptyrproargglyasnilepro
325330335
proalaglyserleuilephegluargtrpargasnproglnsergly
340345350
gluargpheleuargiletyrpheglnalaglnserleuaspglnile
355360365
argasnleuaspproleuaspasnglnhisproproleuargserglu
370375380
pheservalalaglycysglulysthrgluvalglythrvalcyspro
385390395400
leuaspglyalaleulysargleuasnaspalavalaspasnserala
405410415
leuleuprovalglntyrsergln
420
<210>8
<211>1209
<212>dna
<213>狄克氏菌属(dickeya)
<400>8
cagaccaccacggactggcagctggaaaaggtcgtcgagatcagccgccatggcgtccgc60
cctcctaccgaaggcaacatgaagaccatccaagctggcaccggccgcgcttggcccgaa120
tggctgacccgatacggcgagctgacgggccacggttatgccgctgccgtgaagaagggc180
cagtacgagggcaactatttacgacagcacggcctcctcacccaaggttgtccccagccc240
ggtgagctgttcgtctgggccagccctctccagcgaacccgagagaccgctatggccctc300
atggacggcgtcttccccggttgtggcgtggccattcaaggtcccgctagcgaggatcac360
gacactttattccacgccgaggacgctggtgtcacgctggaccaagctcaagtcaaagcc420
gctctcgaccatgctatgcaaggtcagagcgccggccagattcagacccgactccagccc480
gtcatcgacaagctgaagcaagctgtctgtctccccaacaccgcttgtcccgcttttgac540
acggagtgggccatcaaggaaagcaagaaaggcaacctctctttacatggccccgaaacc600
ctcgccaacatggccgagaccatccgcctcgcctacagcaacaacaaccctttaagccaa660
gttgcctttggccatgctcgcagcgccgctgaagtcggtaccctcctcaccctcgccacc720
gcccagtatgacttcacgaacgatttaccttatgtcgcccgacgcggtgccagcaccctc780
atgcaccagatcgccctcgccctcacgcccgaacaacgccccgatgcccctcccgaagcc840
aagtggctgctctacgtcgcccacgacaccaacatcgccaagctccgcaccttattaggc900
ttcacttggcagcaaggtgactaccctcgaggcaacatccctcccgctggctcgctcatc960
ttcgagcgatggcgcaatccccagtcgggcgagcgctttttacgcatctatttccaagct1020
cagagcctcgaccaaatccgcaatttagaccctctggacaatcagcacccccccctccgc1080
agcgagtttagcgtcgccggctgcgagaagaccgaggtcggtaccgtctgccctttagac1140
ggtgctctgaagcgcctcaatgacgccgtcgacaactcggccctcctccccgttcagtac1200
agccagtaa1209
<210>9
<211>426
<212>prt
<213>达旦提狄克氏菌(dickeyadadantii)
<400>9
metvalphethrarglysileilealaglyleuleuleuseralaleu
151015
protrpmetglyalaglnalaaspthrproserserglytrpthrleu
202530
glulysvalvalgluvalserarghisglyvalargproprothrlys
354045
glyasnvallysthrileglngluglythraspargglntrpprothr
505560
trpleuthrglntyrglygluleuthrglyhisglytyralaalaala
65707580
valleulysglyhistyrgluglyglntyrleuargglnhisglyleu
859095
leuserglnalacysproalaproglyaspvalphevaltrpalaser
100105110
proleuglnargthrarggluthralametalaleumetaspglyval
115120125
pheproglycysglyvalalavalglnglyprogluaspgluaspasn
130135140
aspproleuphehisalaglualaalaglyvallysleuaspglngln
145150155160
glnvallysalaaspleuglnasnalametglnasplysseralaala
165170175
glnileglnalaglntrpglnproalaileaspargleulysglnala
180185190
valcysleuproasplysprocysproalapheserthrprotrpglu
195200205
vallysgluserlyslysglyasnileserleuhisglyprogluala
210215220
leualaasnilealagluthrileargleusertyrserasnasnasn
225230235240
proleusergluvalalapheglyhisalalysthralaalagluval
245250255
alaalaleumetproleuleuthralaasntyraspphethrasnasp
260265270
leuprotyrmetalaargargglyalaservalleumetasnglnile
275280285
alaleualaleuasnthrgluhisglnalaaspvalproproaspval
290295300
lystrpleuleutyrvalalahisaspthrasnilealalysleuarg
305310315320
thrmetleuglyphethrtrpglnmetglyasptyrproargglyasn
325330335
ileproproalaglyserleuilephegluargtrpargasnglngln
340345350
serglyglnargleuvalargiletyrpheglnalaglnserleuasp
355360365
glnileargglyleualaalaleuaspaspserhisproproleuarg
370375380
sergluphesermetalaaspcysglnlysthraspvalglythrleu
385390395400
cysprotyraspalavalmetlysargleuasnaspalavalasparg
405410415
thralaleuleuprovalargtyrthrpro
420425
<210>10
<211>1212
<212>dna
<213>达旦提狄克氏菌(dickeyadadantii)
<400>10
gacaccccctcgagcggctggacgctcgagaaggtcgtcgaagtcagccgccatggcgtc60
cgccctcctaccaagggcaacgtcaagacgatccaagaaggcaccgaccgccaatggcct120
acttggctgacccagtatggcgagctgacgggccacggctacgctgccgctgttttaaaa180
ggccattacgagggccagtacctccgacagcacggcctcctcagccaagcttgtcccgct240
cccggtgacgtctttgtctgggctagccctttacaacgcacgcgagagaccgccatggcc300
ctcatggacggtgtcttccccggttgtggcgtcgccgtccaaggtcccgaagacgaggac360
aacgaccctctcttccacgccgaggccgctggtgtcaagctcgaccagcagcaagttaag420
gccgatttacagaacgccatgcaagataagagcgccgcccagatccaagcccagtggcag480
cccgctatcgaccgtttaaagcaagctgtctgcctccccgataagccttgtcccgctttc540
agcaccccttgggaggtcaaagagtcgaagaagggcaacatcagcctccatggccccgaa600
gccctcgccaacatcgccgagacgatccgtttaagctatagcaacaacaaccctttaagc660
gaggtcgctttcggtcacgccaagaccgccgctgaagtggccgctttaatgcctctgctc720
accgccaactacgactttaccaacgatttaccctatatggcccgccgaggcgctagcgtg780
ctgatgaaccagattgccctcgctttaaacaccgaacaccaagctgacgtccctcccgac840
gtcaagtggctgctctacgtggcccacgatacgaacatcgccaagctccgcaccatgctc900
ggtttcacttggcagatgggcgactaccctcgcggcaatattccccccgccggttcgctg960
atcttcgagcgctggcgaaatcaacagtcgggccagcgtttagtccgcatctacttccaa1020
gctcagtcgctcgaccagatccgaggtttagccgctctcgacgattcgcaccccccttta1080
cgaagcgagttcagcatggccgattgccagaagacggacgtgggcacgctctgcccttac1140
gacgccgtcatgaagcgactcaacgacgctgtcgatcgaaccgccttattacccgtccgc1200
tacacgccctaa1212
<210>11
<211>424
<212>prt
<213>沃氏嗜胆菌(bilophilawadsworthia)
<400>11
metlystrpglyleuserileleualaleucysalaleuleualaala
151015
thralaprogluglyglyalaalagluglnglyglyglyaspalalys
202530
leuleulysmetvalvalleuserarghisglyvalargserprothr
354045
glnsersergluthrleuglusertrpserarglysasptrpproglu
505560
trpprovallysargglygluleuthrproargglyalalysleuval
65707580
thralamettrpgluglnglualaalapheleuargglualaglyleu
859095
leuproserlysglycysproglualaglythrilealavalargala
100105110
aspargaspglnargthrargvalthrglyglualavalleuglugly
115120125
leualaproglycysglyphelysproilevalasngluthrasphis
130135140
proaspproleuphehisproleuglualaglytyrcysalaleuasp
145150155160
proalavalvalarglysgluileprovalglyalailegluglyleu
165170175
gluglnserleuserglyproileglygluleualaalaileleugly
180185190
proalaserprogluphecysarglyshisglnleuprogluglycys
195200205
thrvalalaaspvalprothrargleuthrleualalysaspasnarg
210215220
thrvalhisleuaspglylysleuglythralaserseralaalaglu
225230235240
ilemetleuleuglutyrglyglntrpasphisproalaglytrpgly
245250255
alavalasplysglyalaleuglnargleuleuprovalhisserthr
260265270
valpheaspalavalasnargalaproservalalaalaglyarggly
275280285
sergluleuleuleuaspmetalaasnalaleuthrglyargtyrala
290295300
aspproalavalasnlysalalysvalvalvalphevalglyhisasp
305310315320
thrasnilealaasnileglyglymetleuglyleuhistrpglnleu
325330335
proglytyralaproaspgluileproproalaseralaleuvalleu
340345350
thrleutrpleuglnasnaspvaltyrglnleuargalaargmetile
355360365
glyglnserleuaspthrleuhisaspproalametlysglygluval
370375380
leuargglnaspilegluvalprotrpcysglyprotyrgluaspgly
385390395400
lysasncysthrleuthraspphegluleuargvalargaspvalleu
405410415
argproglucysvalarggluarg
420
<210>12
<211>1206
<212>dna
<213>沃氏嗜胆菌(bilophilawadsworthia)
<400>12
gccgagcaaggtggcggtgacgccaagctgctcaagatggtcgttttaagccgccatggt60
gtccgcagccctacgcagagcagcgagacgctggagagctggagccgcaaggactggccc120
gaatggcccgttaagcgcggtgagctcacccctcgaggcgccaagctggtgacggctatg180
tgggagcaagaagccgctttcctccgagaggccggtctcctccctagcaagggttgcccc240
gaggctggcaccatcgccgtgcgagctgaccgagatcagcgcacccgcgtcacgggtgaa300
gccgtcctcgagggtctggcccccggttgcggctttaagcccatcgtcaacgagaccgac360
caccccgaccctctcttccaccctttagaagctggctattgcgccctcgaccccgctgtc420
gtgcgcaaggagatccccgtgggcgccatcgagggcctcgaacagagcctcagcggtccc480
attggtgagctcgctgccattctcggccccgcttcgcccgaattctgccgaaagcaccag540
ctccccgagggctgtaccgtcgctgatgtccctacccgcctcactttagccaaggacaac600
cgcaccgtccacctcgatggcaagctgggtaccgcctcgagcgccgccgagatcatgctc660
ctcgaatacggccagtgggatcaccccgccggttggggtgctgtcgacaagggtgcttta720
cagcgactgctgcccgttcacagcacggtgttcgacgccgtcaaccgcgctccctcggtg780
gccgccggtcgcggcagcgagctcttattagatatggccaacgccctcaccggccgctac840
gccgatcccgccgtcaacaaggccaaggtcgtcgtcttcgtcggccacgacaccaatatc900
gccaacattggcggcatgctcggcctccactggcagctccccggttacgctcccgatgag960
atccctcccgcttcggccctcgttttaaccctctggctgcagaacgacgtctaccagctc1020
cgcgctcgcatgatcggccagtcgctggacacgctccatgaccccgctatgaagggcgaa1080
gtgctgcgccaagatatcgaggtcccttggtgcggcccttacgaggacggcaagaactgc1140
actttaaccgacttcgagctgcgcgtccgcgatgtcctccgccccgagtgtgtccgcgag1200
cgctaa1206
<210>13
<211>403
<212>prt
<213>希贝琥珀酸单胞菌(succinatimonashippei)
<400>13
metlysleuleuasnlysmetseralaalacysalavalilepheile
151015
serserphepheglyvalmetalaalagluasnserphetyralaphe
202530
alaprocysalaaspalalysleuglulysilevalvalleuserarg
354045
hisglyvalargserprothrglnasptyrglnthrleusersertrp
505560
serlysasnasptrpproglntrpasnvalproserglytyrleuthr
65707580
aspargglytyrpheleuilelysserglntrpgluhisleulyslys
859095
ileleuilelysaspservaltyrlysasnasnleuphelysserval
100105110
lysileilealaaspseraspglnargthrvallysthralagluala
115120125
ilearggluglyilesersergluasnthriletyrserasplyslys
130135140
aspvaltyrproleuphehisprovallysalaglyilevallysleu
145150155160
asnalaglnaspvalglngluserileileasnargvalasnserthr
165170175
lysglnglnaspalaileasnleuileglnthrilethrasncyscys
180185190
serglnserilecysglyargasnalaprocysasnleuserasnile
195200205
glnaspservalthrvallysaspasnaspvallysleuleuglyglu
210215220
lysalailealaserserilealagluilepheleuleuglutyrgly
225230235240
glntrpthraspargasnalaglytrpglyglnvalaspglulysthr
245250255
leuthrlysleuleuserleuhisasngluvalpheaspalaleuasn
260265270
argglnasnalavalalaleuserargglyglythrleuleuasnala
275280285
leulysasngluileasnaspargalaserhisilealaphepheval
290295300
glyhisaspthrasnileserasnileglyglyleualaaspleuthr
305310315320
trpserileproaspglnglyvalasnserileproproglycysala
325330335
ilevalpheleuargtrpasnsergluthrgluasnasnglntyrval
340345350
thralaalailealaalaproserilelyspheilehissergluasn
355360365
proasplysgluargleupheleuileseralaasnleuasncysglu
370375380
glylysilecysserleuthrlyspheasnlyslysileaspargala
385390395400
leuasnarg
<210>14
<211>1140
<212>dna
<213>希贝琥珀酸单胞菌(succinatimonashippei)
<400>14
gccgagaacagcttctacgccttcgctccttgtgctgacgccaagctggagaagatcgtc60
gtcctcagccgacatggcgtgcgctcgcctacgcaagattaccagacgctcagcagctgg120
agcaagaacgactggcctcagtggaacgtccctagcggctacctcaccgaccgaggctac180
tttctgatcaagagccagtgggagcacctcaaaaagattttaattaaggacagcgtctac240
aaaaacaatttattcaagtcggtgaagatcatcgccgacagcgaccagcgcaccgtcaag300
accgctgaggccatccgcgagggcatctcgagcgagaacacgatctatagcgacaagaag360
gacgtgtaccctctcttccaccccgtgaaagccggcattgtcaagctgaatgcccaagat420
gtccaagaaagcatcatcaaccgcgtcaactcgaccaagcaacaagatgctatcaatctg480
attcagacgattaccaactgttgctcgcagagcatctgtggccgcaacgccccttgtaat540
ttaagcaatatccaagattcggtcacggtgaaggacaacgatgtcaagctgctgggcgag600
aaggctatcgcttcgtcgatcgccgagatctttttactcgagtacggccagtggaccgat660
cgaaacgccggctggggtcaagtcgacgagaagaccctcacgaagttattaagcctccac720
aatgaggtcttcgacgccctcaaccgacagaacgccgtcgccctctctcgtggtggcact780
ttactgaacgctctcaagaacgagatcaacgaccgcgccagccacatcgcctttttcgtc840
ggccacgacacgaacatcagcaacattggcggcctcgccgacctcacttggtcgatcccc900
gatcaaggtgtgaactcgatcccccccggttgcgccatcgtctttctccgctggaactcg960
gaaaccgagaacaaccagtacgtcaccgccgccatcgctgcccccagcatcaagttcatc1020
cacagcgaaaaccccgacaaggagcgcctcttcctcatcagcgccaatttaaactgcgag1080
ggcaagatctgttctttaaccaagttcaacaagaagatcgaccgcgctttaaaccgctaa1140
<210>15
<211>469
<212>prt
<213>食腺嘌呤芽生葡萄孢酵母(blastobotrysadeninivorans)
<400>15
metleuservalleumettrpalaleuproleuvalvalserglyhis
151015
leuglnvalleuthrproproalaaspaspileleuargleuthrgly
202530
thrlysglyprotyrvalaspargleuserglnglyleugluleuasp
354045
proproglnglycysservalgluglnvalilemetvalmetarghis
505560
glyalaargtyrprothralaasnalatyrproaspleuglnglyala
65707580
leualalysleugluasnlysthrpheserserglyproleuglnphe
859095
pheasnasptrpthrprotyrvalaspasnleuglytrpleuglugln
100105110
gluthrpheserglyprotyralaglyleuleuseralatyrglnarg
115120125
glythrglutyrargalaargtyrglyhisleutrpaspglygluser
130135140
ilevalprophetrpserseraspsergluargvalilemetthrala
145150155160
arghispheglygluglyphepheglytyrasntyrserthrasnala
165170175
alaleuasnilevalserglualagluserglnglyalaasnserleu
180185190
thrprovalcyshisalavalaspprolysserglyaspvalcysser
195200205
asnleuasnvallysglnsertyrproleualaservalalaalaasp
210215220
argleuasnlysargtyrglyleuasnleuthrglyaspaspvaltyr
225230235240
asnleuglnglnleualavalphegluleuasnvalargglyaspser
245250255
protrpleuaspalaphethrargaspglutrpvalalapheasntyr
260265270
tyrcysglyleuaspphephetyrcyslysglyproglyseralagly
275280285
servalalaalaglyservalvalalaasnalaserleuserleuleu
290295300
glnglnglyprosergluhisalaglnalaglyglylysmettyrtrp
305310315320
asnpheserhisaspseraspilevalproileleualaalaleugly
325330335
leualatyrproaspglulysaspvalprotyrasnglyserileala
340345350
phethralalystyrasnalavalaspilemetprometalaglyhis
355360365
valvalphegluargleuhiscysthrgluaspthrproalatrpser
370375380
alaglythrtyrvalargvalileleuasngluglnvalleuproval
385390395400
glnaspcysglnasnglyproglyphesercyslysleugluasptyr
405410415
ilealaargalalyslysargleuvalasptyrasnserphecyslys
420425430
thrproaspasptrpglnlyshisleuserphetrptrpasnproasn
435440445
thrthrglnglntyrasntyrglnlysglyproileglytyrglngln
450455460
lysthrthrleuglu
465
<210>16
<211>1410
<212>dna
<213>食腺嘌呤芽生葡萄孢酵母(blastobotrysadeninivorans)
<400>16
atgctcagcgtcctcatgtgggccctgcctctcgtcgtcagcggccacctccaggtcctc60
acgcctcctgccgacgacatcctccgcctcaccggcacgaagggcccctacgtcgaccgc120
ctcagccagggcctcgagctggaccctcctcagggctgcagcgtcgagcaggtcatcatg180
gtcatgcgccacggcgctcgctaccccaccgccaacgcttaccccgacctccagggcgct240
ctcgccaagctcgagaacaagaccttcagcagcggccctctccagttcttcaacgactgg300
acgccttacgtcgacaacctcggctggctcgagcaggagactttcagcggcccgtacgcc360
ggcctcctcagcgcctaccagcgcggcaccgagtacagggcccgctacggccacctgtgg420
gacggcgagagcatcgtccccttttggtcctccgacagcgagcgcgtcatcatgactgcc480
cgccacttcggcgagggctttttcggctacaactacagcaccaacgccgctctcaacatc540
gtcagcgaggccgagagccagggcgccaactcgctcacgcccgtctgccacgccgtcgat600
cccaagagcggcgacgtctgcagcaacctcaacgtcaagcagagctaccctctcgccagc660
gtcgccgccgaccgcctgaacaagcgatacggcctcaacctgaccggcgacgacgtctac720
aacctgcagcagctcgccgtctttgagctgaacgtccgaggcgacagcccctggctcgac780
gccttcacgcgcgacgagtgggtcgccttcaactactactgcggcctcgacttcttctac840
tgcaagggccctggcagcgccggctccgtcgctgccggctcggtcgtcgccaacgccagc900
ctcagcctcctccagcagggccccagcgagcacgcccaggctggcggcaagatgtactgg960
aacttcagccacgacagcgacattgtccccatcctcgctgccctcggcctggcgtacccc1020
gacgagaaggacgtcccctacaacggcagcattgccttcaccgccaagtacaacgccgtg1080
gacatcatgcccatggctggccacgtcgtgttcgagcgcctccactgcaccgaggacacg1140
cccgcctggtccgccggcacctacgtccgcgtcatcctcaacgagcaggttctccccgtc1200
caggactgccagaacggccctggcttcagctgcaagcttgaggactacattgcccgagcc1260
aagaagcgcctcgtcgactacaacagcttctgcaagacgcccgacgactggcagaagcac1320
ctctcgttctggtggaaccccaacaccacgcagcagtacaactaccagaagggccctatc1380
ggctaccagcagaagaccacgctcgagtaa1410
<210>17
<211>449
<212>prt
<213>威克汉姆西弗酵母(wickerhamomycesciferrii)
<400>17
metglnleuserthrthrvalleuseralaleualaleuserilethr
151015
alavalaspalaleuproileglyserlyssergluserileargpro
202530
tyrserglnglualaleuaspglnpheservalleuargpheleugly
354045
thralaserprotyrvalglnasnthrglytyrglyilegluargasp
505560
alaprotyrglncyslysvalthrglnalaasnleuileserarghis
65707580
glygluargtyrprothrserasnglnglylyslysmetalalyshis
859095
phegluglnileglnasnglythrgluthrilegluglyproleuser
100105110
pheleulysasptyrgluphelysalaleuaspglugluglyleuasp
115120125
glngluthrglylysglyprotyrserglyleuleuaspleutyrlys
130135140
hisglyalaleupheargaspargtyraspaspleutyrasnglugly
145150155160
aspgluilelysphetyrseralaserglnlysargvalvalgluser
165170175
alalyslysphealaglnglypheleuglyglusertyrasngluser
180185190
tyrileglngluileaspgluaspthrasnaspleuglyalaasnser
195200205
leuthrprovalasnalacysasnasntyrasplyshisileasngln
210215220
asplysileaspgluleuserthrserpheleuasnlysthralaasp
225230235240
argleuasnasnglnserproglyileasnleuileprogluglnval
245250255
glyserleuiletyrtyrcysglyphegluleuasnvalgluglyser
260265270
asnglnilecysgluilephethrthraspgluleuleualatyrser
275280285
tyrthrlysaspvalsertyrtyrtyrglulysglyproglyasnasn
290295300
leuseralathrileglyservaltyrileaspalailethrargleu
305310315320
ilelysaspaspserlysasnleuthrleuserphealahisaspthr
325330335
aspilephetyrilevalserleuleuglyleupheaspglygluleu
340345350
prothrasphisglnserpheasnhisleutrplysileserasnile
355360365
alaprometglyalaargleuvalilegluargleuglucysgluasp
370375380
glnglugluprophevalargileilehisasnaspalavalleupro
385390395400
ileproglyhissergluglyproglytyrserileserileserasp
405410415
pheglulystyrilegluasplysleuaspglylysthrtyrglulys
420425430
aspcysglythrlysaspglyvalprothrgluleuthrphephetrp
435440445
asp
<210>18
<211>1350
<212>dna
<213>威克汉姆西弗酵母(wickerhamomycesciferrii)
<400>18
atgcagctcagcaccaccgtcctcagcgccctcgctctcagcatcaccgccgtcgacgcc60
ctgcctatcggcagcaagagcgagagcattcgcccctacagccaggaggccctcgaccag120
ttcagcgtcctccgcttcctcggcaccgcctcgccttacgtccagaacaccggctacggc180
atcgagcgcgacgccccttaccagtgcaaggtcacccaggccaacctcatcagccgccac240
ggcgagcgctaccccaccagcaaccagggcaagaagatggccaagcacttcgagcagatc300
cagaacggcaccgagactatcgagggcccgctcagcttcctcaaggactacgagttcaag360
gccctggacgaggagggcctcgatcaggagactggcaagggcccctactccggcctcctc420
gacctctacaagcacggcgccctcttccgcgaccgctacgacgacctgtacaacgagggc480
gacgagatcaagttctacagcgccagccagaagcgcgtcgtcgagagcgccaagaagttc540
gcccagggcttcctgggcgagagctacaacgagtcgtacatccaggagatcgacgaggac600
accaacgacctcggcgccaactcgctcacgcccgtcaacgcctgcaacaactacgacaag660
cacatcaaccaggataagattgacgagctgagcacctcgttcctcaacaagaccgccgac720
cgcctcaacaaccagtcgcctggcatcaacctcattccggagcaggtcggcagcctcatc780
tactactgcggcttcgagcttaacgtcgagggctccaaccagatctgcgagatcttcacc840
accgacgagctgctcgcctacagctacaccaaggacgtcagctactactacgagaagggc900
cctggcaacaacctgagcgccaccatcggcagcgtctacatcgacgccatcacgcgcctc960
atcaaggacgacagcaagaacctcacgctgagcttcgcccacgacaccgacatcttctac1020
atcgtcagcctgctcggcctcttcgacggcgagctgcccaccgaccaccagagcttcaac1080
cacctctggaagatcagcaacattgcccctatgggcgctcgcctcgtcattgagcgcctc1140
gagtgcgaggaccaggaggagcccttcgtccgcatcatccacaacgacgccgtcctgcct1200
attcctggccacagcgagggccctggctacagcatcagcatcagcgacttcgagaagtac1260
atcgaggacaagctcgacggcaagacctatgagaaggactgcggcaccaaggatggcgtc1320
cctaccgagctgacgttcttctgggactaa1350
<210>19
<211>452
<212>prt
<213>栎迷孔菌(daedaleaquercina)
<400>19
metthrthrleuleucysilealaleuthrphetyralaphecysgly
151015
tyrilealaleuglyargproproaspaspilevalvalaspileglu
202530
serglygluthrglyphealaalaaspproserglypheglyalaasp
354045
serthrasntrpserglntyrserprotyrtyrprovalgluprotyr
505560
seralaproprogluglycysvalvalaspglnvalhisileilegln
65707580
arghisglyalaargtyrprothrserglyalaalalysargilegln
859095
alaalaleualalysleuglnthralathrileglnserasnserser
100105110
leualaphealaleuasntyrsertyrthrleuglyglnaspserleu
115120125
valserleuglyalaargglusertyrglyalaglyglnglualatyr
130135140
alaargtyralaserilevalaspalaglulysmetprophevalarg
145150155160
alaserglysergluargvalvalglnseralathrasntrpthrala
165170175
glyphealaalaalaserasnaspvaltyrthrprovalleuserval
180185190
ileilesergluglnglyasnaspthrleuaspaspsersercyspro
195200205
leuhisthrglyseraspileproglythrtyrileaspvaltyrala
210215220
alaasnmetthraspvalleuasnthrglyalatyrglyalaasnleu
225230235240
serasnalaaspthrhisalaleuvalthrleucysmetphegluser
245250255
valalalysglugluargseralatrpcysaspleuphealagluleu
260265270
glyalaleuaspglyphealahistrpalaalaleuasplystyrtyr
275280285
glythrglytyrglyasngluleuglyprovalglnglyvalglytyr
290295300
ileasngluleuleualaargleuthrasnthrprovalasnaspser
305310315320
thrserthrasnserthrleuaspserasnprogluthrpheproleu
325330335
asnargthrvaltyralaasppheserhisaspasnleuileilepro
340345350
valphealaalametglyleupheprothraspproleuaspprothr
355360365
glyalaalaglyglnalathrtrpasnvalserthrmetvalprophe
370375380
alaglyargmetvalvalgluargleualacysalaglyglyglugly
385390395400
glylystyrvalargvalleuvalasnglnalavalglnproleuval
405410415
phecysglyalaglualaaspglyvalcysserleuaspalapheval
420425430
gluserglnalatyralaargserargglyasnglythrtrpalaala
435440445
cysphealaser
450
<210>20
<211>1359
<212>dna
<213>栎迷孔菌(daedaleaquercina)
<400>20
atgaccacgctcctctgcattgccctgaccttctacgccttctgcggctacattgccctc60
ggccgacctccggacgacatcgtcgtcgacatcgagagcggcgagactggcttcgccgcc120
gatcctagcggctttggcgccgacagcaccaactggtcccagtacagcccttactacccc180
gtcgagccctacagcgcccctcctgagggctgcgtcgtcgatcaggtccacatcatccag240
cgccacggcgctcgctaccccaccagcggcgctgccaagcgcatccaggctgccctcgcc300
aagctccagaccgccaccatccagagcaacagcagcctcgccttcgctctcaactacagc360
tacaccctcggccaggacagcctcgtcagcctgggcgctcgcgagagctacggcgctggc420
caggaggcttacgcccgctacgccagcatcgtcgacgccgagaagatgcccttcgtccgc480
gccagcggcagcgagcgcgtcgtccagagcgccacgaactggaccgccggctttgccgct540
gccagcaacgacgtctacacgcccgtcctcagcgtcatcatcagcgagcagggcaacgac600
accctggacgacagcagctgccctctccacaccggcagcgacatccccggcacctacatt660
gacgtctacgccgccaacatgaccgacgtcctcaacaccggcgcctacggcgccaacctc720
agcaacgccgacacgcacgccctcgtcaccctctgcatgttcgagagcgtcgccaaggag780
gagcgcagcgcctggtgcgacctcttcgccgagctgggcgccctcgacggcttcgcccac840
tgggccgctctggataagtactacggcaccggctacggcaacgagcttggccccgtccag900
ggcgtgggctacatcaacgagctgctcgcccgcctcaccaacactcccgtcaacgactcc960
acctccaccaactcgaccctcgacagcaaccccgagacttttcccctcaaccgcaccgtg1020
tacgccgacttcagccacgacaacctcatcatccccgtctttgccgccatgggcctgttt1080
ccgacggaccctctcgaccccacgggcgctgccggccaggccacctggaacgtcagcacg1140
atggtccctttcgctggccgcatggtcgtcgagcgcctggcctgcgctggcggcgagggc1200
ggcaagtacgtccgcgtcctcgtcaaccaggccgtccagcctctcgtcttttgcggcgct1260
gaggccgacggcgtctgcagcctcgacgcctttgtcgagagccaggcctacgctcgctcg1320
cgcggcaacggcacctgggccgcctgcttcgccagctaa1359
<210>21
<211>453
<212>prt
<213>嗜热子囊菌(thermoascusaurantiacus)
<400>21
mettyrleuleuleuglyvalleualaglyvalpheileleuserlys
151015
glusercysserthrvalaspargglytyrglncysasnproasnile
202530
thrhisphetrpglyglnasnserprotyrpheserleuseraspval
354045
sergluileserprogluileproalaglycysservalthrpheala
505560
glnvalleuserarghisglyalaargtyrprothralalyslysthr
65707580
glnleutyrseraspileilethrargileglnglnseralaglnser
859095
pheasnglyaspphealapheleugluargtyrasntyrthrleugly
100105110
alagluaspleuthralapheglygluaspglumetthrlyssergly
115120125
ileglnphetyrasnargtyrlysthrleualaserserilevalpro
130135140
phevalargalaserglyseraspargvalilealaserglygluarg
145150155160
pheleugluglypheglnseralalysmetmetaspproserserasn
165170175
lysasnaspserleuprolysileservalileilealagluserasp
180185190
serserasnasnthrleuasphisserglycysalaalaphegluasp
195200205
aspasnthrlysaspilevalglnserglupheleugluvalpheala
210215220
proalavalleuaspargphelysalaglyleuproglyvalasnleu
225230235240
servalseraspvalproaspilemetaspilecysalaphegluthr
245250255
valalaasnthrproaspalaserglnleuserprophecysserleu
260265270
phethrglugluglutrpleuglntyrasptyrtyrglnserleugly
275280285
lystyrtyrglytyrglyaspglyasnproleuglyalathrglngly
290295300
valglyphevalasngluleuilealaargleuthrasnthrproval
305310315320
gluasphisthrthrvalasnhisthrleuaspserasnprogluthr
325330335
pheproleuasnalathrleutyralaasppheserhisaspasnthr
340345350
ilealaserilehisalaalaleuglyleutyrasnglythrlyslys
355360365
leuserthrthrargvalglnservalalagluthrasnglytyrser
370375380
alaalatrpthrvalpropheseralaargthrtyrvalglumetmet
385390395400
glncyssersergluasngluprophevalargileleuvalasnasp
405410415
argvalvalproleuhisglycysgluvalaspserleuglyargcys
420425430
lysargaspaspphevalargglyleuserphealaargserglygly
435440445
asntrpalasercys
450
<210>22
<211>1362
<212>dna
<213>嗜热子囊菌(thermoascusaurantiacus)
<400>22
atgtacctgctcctcggcgtcctcgccggcgtctttatcctcagcaaggagagctgcagc60
accgtcgaccgaggctaccagtgcaaccccaacatcacccacttttggggccagaacagc120
ccctacttcagcctcagcgacgtcagcgagatcagccccgagattcctgccggctgcagc180
gtcacgttcgcccaggtcctcagccgccacggcgctcgctaccccaccgccaagaagacc240
cagctctacagcgacatcatcacccgcatccagcagagcgcccagagcttcaacggcgac300
ttcgccttcctcgagcgctacaactacaccctgggcgccgaggacctcaccgccttcggc360
gaggatgagatgaccaagagcggcatccagttctacaaccgctacaagaccctcgccagc420
agcatcgtccccttcgtccgcgccagcggcagcgaccgcgtcattgcctccggcgagcgc480
tttctcgagggcttccagagcgccaagatgatggaccccagcagcaacaagaacgactcg540
ctccccaagatcagcgtcatcattgccgagagcgacagctccaacaacaccctcgaccac600
agcggctgcgccgccttcgaggacgacaacaccaaggacatcgtccagagcgagttcctc660
gaggtctttgcccctgccgtcctcgaccgcttcaaggctggcctgcctggcgtcaacctc720
agcgtgagcgacgtccccgacatcatggacatctgcgcctttgagactgtcgccaacacg780
cccgacgccagccagctgagccccttctgcagcctgttcaccgaggaggagtggctccag840
tacgactactaccagagcctcggcaagtactacggctacggcgacggcaaccctctcggc900
gccacgcagggcgtcggcttcgtcaacgagctgatcgcccgcctcaccaacactcccgtc960
gaggaccacaccaccgtcaaccacacgctcgacagcaaccccgagacttttcccctcaac1020
gccacgctctacgccgacttcagccacgataacacgatcgccagcatccacgctgccctc1080
ggcctctacaacggcaccaagaagctcagcaccacgcgcgtccagtccgtcgccgagact1140
aacggctacagcgccgcctggacggtccccttcagcgcccgcacctacgtcgagatgatg1200
cagtgcagcagcgagaacgagccctttgtccgcatcctggtcaacgaccgagtcgtcccg1260
ctccacggctgcgaggtcgacagcctcggccgctgcaagcgcgacgacttcgtccgaggc1320
ctcagcttcgcccgcagcggcggcaactgggccagctgctaa1362
<210>23
<211>466
<212>prt
<213>米曲霉(aspergillusoryzae)
<400>23
metalavalleuservalleuleuproilethrpheleuleuserser
151015
valalaglythrprovalthrserproargglnglnsercysasnthr
202530
valaspgluglytyrglncyspheserglyvalserhisleutrpgly
354045
glntyrserprotyrpheservalaspaspgluserleuleuserglu
505560
aspileproasphiscysglnvalthrphealaglnvalleuserarg
65707580
hisglyalaargtyrprothrgluserlysserglulystyralalys
859095
leuilelysalavalglnhisasnalathrserpheserglylystyr
100105110
alapheleulyssertyrasntyrserleuglyalaaspaspleuthr
115120125
propheglygluasnglnleuvalaspserglyilelysphetyrgln
130135140
argtyrglugluleualalysasnvalvalpropheileargalaser
145150155160
glyseraspargvalilealaserglyglulyspheilegluglyphe
165170175
glnlysalalysleuglyaspserlysserlysargglyglnproala
180185190
proilevalasnvalvalilethrgluthrgluglypheasnasnthr
195200205
leuasphisserleucysthralaphegluasnserlysthrglyasp
210215220
aspalagluasplysphethralavalphethrproserilealaglu
225230235240
argleuglulysaspleuproglythrthrleuserserlysgluval
245250255
valtyrleumetaspmetcyssertyraspthrvalalaleuthrarg
260265270
aspglyserargileserprophecysalaleuphethrglngluglu
275280285
trpvalglntyrasptyrleuglnserileserlystyrtyrglytyr
290295300
glyalaglyasnproleuglyproalaglnglyileglyphealaasn
305310315320
gluleuilealaargleuthrlysserprovallysasphisthrthr
325330335
thrasnthrthrleuaspserasnproalathrpheproleuasnala
340345350
thrleutyralaasppheserhisaspasnthrmetthrservalphe
355360365
phealaleuglyleutyrasnthrthrgluproleuserglnthrser
370375380
valglnserthrglugluthrasnglytyrserseralatrpthrval
385390395400
propheglyalaargalatyrvalglumetmetglncysthrglyglu
405410415
lysgluproleuvalargvalleuvalasnaspargvalileproleu
420425430
glnglycysaspalaaspglutyrglyargcyslysargaspaspphe
435440445
ilegluglyleuserphevalthrserglyglyasntrpglyglucys
450455460
pheala
465
<210>24
<211>1401
<212>dna
<213>米曲霉(aspergillusoryzae)
<400>24
atggccgtcctcagcgtcctcctgcctatcaccttcctgctcagcagcgtcgccggcacg60
cccgtcacctcgcctcgccagcagagctgcaacaccgtcgacgagggctaccagtgcttc120
agcggcgtcagccacctctggggccagtacagcccctacttcagcgtcgacgacgagagc180
ctcctcagcgaggacatccccgaccactgccaggtcacgttcgcccaggtcctcagccgc240
cacggcgctcgctaccccaccgagagcaagagcgagaagtacgccaagctcattaaggcc300
gtccagcacaacgccaccagcttctccggcaagtacgccttcctcaagagctacaactac360
agcctgggcgccgacgacctcacgcctttcggcgagaaccagctcgtcgacagcggcatc420
aagttctaccagcgctacgaggagctggccaagaacgtcgtccccttcatccgcgccagc480
ggcagcgaccgcgtcattgccagcggcgagaagttcatcgagggcttccagaaggccaag540
ctcggcgactccaagagcaagcgcggccagcctgctcctatcgtcaacgtcgtcatcacc600
gagactgagggcttcaacaacaccctcgaccacagcctctgcaccgccttcgagaacagc660
aagaccggcgacgacgccgaggacaagttcaccgccgtctttacgccctcgatcgccgag720
cgcctcgagaaggacctgcctggcacgaccctcagcagcaaggaggtcgtctacctcatg780
gacatgtgcagctacgacacggtcgccctcacgcgcgacggcagccgcatcagccccttt840
tgcgccctcttcacccaggaggagtgggtccagtacgactacctccagagcatcagcaag900
tactacggctacggcgctggcaaccctctcggccctgctcagggcatcggcttcgccaac960
gagctgatcgcccgcctcaccaagtcgcccgtcaaggaccacaccaccaccaacaccacg1020
ctcgacagcaaccccgccacctttcctctcaacgccacgctctacgccgacttcagccac1080
gacaacaccatgaccagcgtctttttcgccctcggcctctacaacaccaccgagcctctc1140
agccagacctccgtccagagcaccgaggagactaacggctacagcagcgcctggacggtc1200
ccctttggcgctcgcgcctacgtcgagatgatgcagtgcacgggcgagaaggagcccctc1260
gtccgcgtcctcgtcaacgaccgagtcattcccctccagggctgcgacgccgacgagtac1320
ggccgctgcaagcgcgacgactttatcgagggcctcagcttcgtcaccagcggcggcaac1380
tggggcgagtgcttcgcctaa1401
<210>25
<211>460
<212>prt
<213>蜜蜂球囊菌(ascosphaeraapis)
<400>25
metleuileleuvalpropheileleuglyalaasnalaleuserile
151015
proserserhisvalserthrlysaspglyalacysserserprogly
202530
aspglytyrhiscysphegluglutyrserargargtrpglyglutyr
354045
serprotyrpheservallysaspvalsersertyrprovalaspile
505560
proserglucysgluvalthrphealaglnvalleuserarghisgly
65707580
alaargtyrprothralahislysglylysthrtyralaaspleuile
859095
gluargileglnasnglnthralaargtyrglyseraspphehisphe
100105110
ilegluserpheglutyrserleuasnthraspserleuvalgluphe
115120125
glylysgluglnleuphegluserglyvalserphetyrgluargtyr
130135140
alaasnleuserargthrasnvalprophevalargsersertrpthr
145150155160
proargvalilegluseralagluhisphemetargglypheserglu
165170175
serarglysglnalaasnglygluprovalasnlysthralavalasp
180185190
valvalmettyraspglythrglypheasnserserleuasnleugly
195200205
alacysproalapheglulysgluserlysileglnprotrphisgln
210215220
glyphemetglulyshispheserlysthrleuglnargvallyslys
225230235240
asnleuproglyalaglnleuaspvallysaspileiletyrleumet
245250255
aspleucysserphehisthrvalseralathrproaspalathrphe
260265270
leuserprophecysproleuphethrglugluglutrpglualatyr
275280285
asptyrpheserthrleuarglystyrtyrlyspheglylysglyasn
290295300
gluleualaproproasnglyilealaphevalasngluleuileala
305310315320
argleuthrasnglnprovalhisasphisserserileasnargthr
325330335
leuaspseraspprolysthrpheproleuglyleuproleutyrala
340345350
asppheserhisaspasnglumetalaproiletyralaalametgly
355360365
ilealaglyproileleuasnlyslysserilepheglnprogluasp
370375380
valsertyrphethrserserargleuileprophealaalaargmet
385390395400
tyrvalglulysleuglucysglyserglulysgluglutyrvalarg
405410415
valleuileasnaspargvalmetprovalgluglycysalaalaasp
420425430
hisleuglyargcyslysleuseraspphevalasnglymetthrtyr
435440445
alaserserglytyralatrplysleucystyrglu
450455460
<210>26
<211>1383
<212>dna
<213>蜜蜂球囊菌(ascosphaeraapis)
<400>26
atgctcattctcgtccctttcatcctcggcgccaacgctctcagcatccccagcagccac60
gtcagcaccaaggacggcgcctgcagcagccctggcgacggctaccactgcttcgaggag120
tacagccgccgctggggcgagtactctccctacttcagcgtcaaggacgtcagcagctac180
cccgtcgacatcccctccgagtgcgaggtcacgttcgcccaggtcctcagccgccacggc240
gctcgctaccccaccgctcacaagggcaagacctacgccgacctcatcgagcgcatccag300
aaccagacggcccgctacggcagcgacttccacttcatcgagagcttcgagtactccctc360
aacaccgacagcctcgtcgagttcggcaaggagcagctgttcgagagcggcgtcagcttc420
tacgagcgctacgccaacctgagccgcaccaacgtgcccttcgtccgcagcagctggacc480
cctcgcgtcatcgagtccgccgagcacttcatgcgcggcttcagcgagagccgcaagcag540
gccaacggcgagcccgtcaacaagaccgccgtcgacgtcgtgatgtacgacggcaccggc600
ttcaacagcagcctcaacctcggcgcttgccccgccttcgagaaggagagcaagatccag660
ccttggcaccagggcttcatggagaagcacttcagcaagaccctccagcgcgtcaagaag720
aacctgcctggcgctcagctcgacgtgaaggacatcatctacctcatggacctctgcagc780
ttccacaccgtcagcgcgacgcccgacgctaccttcctctcgccgttctgccctctgttc840
accgaggaggagtgggaggcctacgactacttctcgaccctccgcaagtactacaagttt900
ggcaagggcaacgagctggcccctcctaacggcattgccttcgtgaacgagctgatcgcc960
cgcctcaccaaccagcctgtccacgaccacagctccatcaaccgcacgctcgacagcgac1020
cccaagacgtttcccctcggcctgcctctgtacgccgacttcagccacgacaacgagatg1080
gctcccatctacgccgccatgggcattgctggccccattctcaacaagaagtccatcttt1140
cagcccgaggacgtgagctactttaccagcagccgactcatccccttcgccgctcgcatg1200
tacgtcgagaagctcgagtgcggctcggagaaggaggagtacgtccgcgtcctcatcaac1260
gaccgcgtcatgcccgtcgagggctgcgccgctgaccacctcggccgctgcaagctctcc1320
gacttcgtcaacggcatgacgtacgccagcagcggctacgcctggaagctctgctacgag1380
taa1383
<210>27
<211>491
<212>prt
<213>根串珠霉孔菌(thielaviopsispunctulata)
<400>27
metalaprolysasnargarglysleuglnphethrleuvalsermet
151015
alaserleuleuvalphealavalleuileglymetargtyrleuphe
202530
serserproserleuleuserthrserilelyspropheilevalasn
354045
argserprohishisprohisglyserserseralacystyraspcys
505560
mettyrmetalathrproserleutrpglyglntyrserprometphe
65707580
servalprosergluleucysalaaspvalproprolyscysthrleu
859095
thrpheglyglnvalleuserarghisglyalaargaspprothrser
100105110
serlysthrvalglutyrasnargthrileglnargilehisaspser
115120125
valthrglutyrglyglualavallyspheleuargasntyrthrtyr
130135140
serleuglyalaasphisleuthrlyspheglyglnaspglumetmet
145150155160
valserglyvalasnphepheglnargtyrlysserleualaalaser
165170175
servalpropheileargalaserglyglnglnargvalvalgluser
180185190
alaleuasntrpthrglnglyphetyralaalaargvalserhisgly
195200205
ileaspprovalglygluserlysglylysleuleuileileproglu
210215220
glyaspthrserasnasnthrleuasnasnglyleucysthralaleu
225230235240
gluserglylystyrserglnvalglyaspaspalalysaspalaphe
245250255
leualathrpheilegluproilethralaargleuasnleuasnleu
260265270
proglyalaasnleuthrasnlysglualavaltyrmetmetaspleu
275280285
cyspropheasnthrvalalathralaaspalathrvalsergluphe
290295300
cysleuleuphethrilegluasptrpargasntyrasptyrtyrgln
305310315320
thrmetasplystyrtyrglyhisglyasnglyasnprometglyala
325330335
thrserglyvalglytrpalaasngluleuilealaargleuthrasp
340345350
thrprovalvalasphisthrservalasnhisthrleuaspserser
355360365
asnalathrpheproleuglyargalaleutyralaasppheserhis
370375380
aspasnaspmetthrglyilepheseralaleuargleutyraspala
385390395400
thrproleuleuprothraspargargvalalaprogluglualagly
405410415
glyphealasersertrpvalvalprophealaalaargmettyrval
420425430
glulysmetargcysgluglygluserglugluphevalargpheleu
435440445
valasnaspargvalileproleuvalaspcysglyvalasplysphe
450455460
glyargcyslysleuserlystrpvalaspthrmetglyphealagln
465470475480
serglyglyasntrpserglucyspheserser
485490
<210>28
<211>1476
<212>dna
<213>根串珠霉孔菌(thielaviopsispunctulata)
<400>28
atggcccctaagaaccgccgcaagctccagttcaccctcgtcagcatggccagcctgctc60
gtctttgccgtcctcatcggcatgcgctacctcttcagcagccccagcctcctcagcacc120
agcatcaagcccttcatcgtcaaccgctcgcctcaccatcctcacggcagcagcagcgcc180
tgctacgactgcatgtatatggccacgcctagcctctggggccagtacagccccatgttc240
agcgtccccagcgagctgtgcgccgacgtgcctcctaagtgcaccctcaccttcggccag300
gtcctcagccgccacggcgctcgcgaccccaccagcagcaagaccgtcgagtacaaccgc360
accatccagcgcatccacgacagcgtcaccgagtacggcgaggccgtcaagttcctccgc420
aactacacctacagcctgggcgccgaccacctcaccaagtttggccaggacgagatgatg480
gtcagcggcgtcaacttcttccagcgctacaagagcctcgccgccagctcggtccccttc540
atccgcgccagcggccagcagcgcgtcgtcgagagcgccctcaactggacccagggcttc600
tacgccgctcgcgtcagccacggcatcgaccccgtcggcgagagcaagggcaagctcctc660
atcatccccgagggcgacaccagcaacaacaccctcaacaacggcctctgcacggccctc720
gagagcggcaagtacagccaggtcggcgacgacgccaaggacgccttcctcgccaccttc780
atcgagcccatcactgcccgcctcaacctcaacctgcctggcgccaacctgaccaacaag840
gaggccgtctacatgatggatctctgccccttcaacaccgtcgccaccgccgacgccacc900
gtcagcgagttctgcctcctgttcaccatcgaggactggcgcaactacgactactaccag960
accatggacaagtactacggccatggcaacggcaaccccatgggcgccacctccggcgtc1020
ggctgggccaacgagctgatcgcccgcctgaccgacacgcccgtcgtcgaccacaccagc1080
gtcaaccacacgctcgacagcagcaacgccacctttcctcttggccgcgctctctacgcc1140
gacttcagccacgacaacgacatgaccggcatcttcagcgccctccgcctctacgacgcc1200
acgcctctgctccccaccgaccgccgcgtcgctcccgaggaggctggcggcttcgccagc1260
tcctgggtcgtccccttcgctgcccgcatgtacgtcgagaagatgcgctgcgagggcgag1320
agcgaggagttcgtccgcttcctcgtcaacgaccgcgtcattcccctcgtcgactgcggc1380
gtcgacaagttcggccgctgcaagctcagcaagtgggtcgacaccatgggcttcgcccag1440
agcggcggcaactggtccgagtgcttcagctcctaa1476
<210>29
<211>465
<212>prt
<213>稻瘟菌(magnaportheoryzae)
<400>29
metphethrleuvalleuleupheglnleutrpseralaleuilearg
151015
alathrhisalavalgluproglyserglnglyaspcysaspthrpro
202530
aspglyglypheglncysasnserserileserhisasntrpglygln
354045
tyrserprophepheleualaprosergluileaspalaservalpro
505560
serglycysgluvalthrphealaglnileleuserarghisglyala
65707580
argaspprothralaseralathralaalatyrglygluthrileglu
859095
glnileglnserservalthrasntyrglygluglypheglupheile
100105110
lysasptyrglntyrthrleuglyalaaspglnleuseraspphegly
115120125
argglnglumetpheasnserglyvalhisphetyrasnargtyrgln
130135140
ileleualaargasnasnthrprophepheargseraspglyglngln
145150155160
argvalvalgluserglyglnlystrpthrgluglyphehisglnala
165170175
leuleuglyaspserglyargalaglyglyproalaaspgluphepro
180185190
tyrlysmetvalileileproasngluaspglythrasnasnthrleu
195200205
asnhisasnleucysthralaphegluasnthrlysleuglylysglu
210215220
alaglnlysgluphemetgluseralametglyglyilethrgluarg
225230235240
leuasnasnglyleugluglyalaasnleuthrthrlysglnalaval
245250255
glnilemetgluleucysprophegluthrvalalaaspproglnala
260265270
thrleuserglnphecysthrleuphethrglnargasptrpgluala
275280285
tyrasptyrleuglnthrleuglylystrptyrglytyrglyasngly
290295300
asnproleuglyserthrglnglyvalglyphevalasngluleuile
305310315320
alaargleuleuglnlysprovalgluasphisthrasnthrasnser
325330335
thrleuaspseraspproserthrpheproleuasplyslysleutyr
340345350
alaasppheserhisaspasnaspmetleuglyiletyralaalaleu
355360365
glyiletyrasnalathralaproaspservalprolyslysgluarg
370375380
argseralaglngluleuserglyphesersersertrpalavalpro
385390395400
phealaalaargmetphevalglulysmetthrcysalaglyglnasn
405410415
glugluleuvalargileleuvalasnaspargvalthrproleugln
420425430
asncysaspalaaspsermetglyargcysthrleuserlyspheval
435440445
gluserleuserphealaargserglyglyargtrpaspglncysphe
450455460
val
465
<210>30
<211>1338
<212>dna
<213>稻瘟菌(magnaportheoryzae)
<400>30
gtcgagcccggtagccaaggtgactgcgacacgcccgacggcggctttcagtgcaatagc60
agcatctcgcataactggggccagtactcgcccttctttttagccccttcggagatcgac120
gcttcggtccccagcggttgcgaggtcaccttcgcccagattctgtctcgtcacggtgcc180
cgcgatcctacggcttcggctaccgccgcctacggcgagaccatcgagcaaatccagagc240
tcggtcaccaactacggcgagggcttcgagtttatcaaagactaccaatacaccctcggc300
gctgatcagctgagcgacttcggccgacaagaaatgttcaacagcggcgtccacttctac360
aaccgctaccagattttagcccgcaacaacacgccctttttccgctcggacggtcaacag420
cgcgtcgtcgagagcggtcagaagtggacggagggtttccaccaagctctgctcggcgac480
tcgggtcgcgccggcggccccgctgacgagttcccctacaagatggtcatcatccccaac540
gaggacggtacgaacaataccctcaaccataatttatgcacggccttcgaaaacaccaag600
ctcggcaaggaggcccagaaagagttcatggagtcggccatgggtggcattaccgagcga660
ctcaacaacggtctcgagggcgctaatttaacgacgaagcaagctgtccagatcatggag720
ctgtgcccctttgagaccgtcgccgatccccaagctactttatcgcagttctgcaccctc780
tttacgcagcgcgactgggaggcctacgactacctccagaccctcggcaagtggtacggc840
tacggcaacggcaaccctctcggctcgacccaaggtgtcggctttgtcaacgagctcatc900
gcccgcctcctccagaagcccgttgaggaccacacgaacaccaactcgactttagatagc960
gaccccagcaccttccctttagacaagaagctgtacgccgacttctcgcacgacaacgac1020
atgctgggcatctacgccgctctgggcatctataatgccacggcccccgacagcgtgccc1080
aagaaagagcgccgcagcgcccaagaactgagcggcttcagcagcagctgggctgtcccc1140
ttcgctgcccgcatgtttgtcgagaagatgacgtgcgccggtcagaacgaggagctcgtc1200
cgcatcctcgtcaacgaccgcgtcacccccctccagaactgcgacgccgacagcatgggc1260
cgctgcacgctcagcaagtttgtcgagtcgctcagcttcgcccgaagcggtggtcgctgg1320
gaccagtgctttgtctaa1338
- 还没有人留言评论。精彩留言会获得点赞!