磷酸乙醇胺的定量方法、定量用的氧化还原酶、定量用组合物、定量用试剂盒和定量用传感器与流程
本发明涉及磷酸乙醇胺的定量方法、定量用的氧化还原酶、定量用组合物、定量用试剂盒或定量用传感器。
背景技术:
人的血液中含有磷酸乙醇胺(eap)。在专利文献1中记载了eap是用于诊断抑郁症的生物标志物。在非专利文献1中记载了能够以eap浓度1.5μm以下作为基准值而诊断抑郁症的情况。
另外,在专利文献2中记载了利用eap磷酸化酶(phosphorylase)和乙醛脱氢酶(acetaldehydedehydrogenase)的eap浓度的测定方法。
(现有技术文献)
(专利文献)
专利文献1:国际公开2011/019072号
专利文献2:国际公开2013/069645号
(非专利文献)
非专利文献1:kawamuran,"plasmametabolomeanalysisofpatientswithmajordepressivedisorder."psychiatryclinneurosci.2018may;72(5):349-361
技术实现要素:
(发明所要解决的问题)
到目前为止,尚未报道除专利文献2中公开的方法之外的eap浓度的测定方法,期望开发新的测定方法。
本发明的目的之一在于提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组合物、定量用试剂盒或定量用传感器。
(解决问题所采用的措施)
根据本发明的实施方式之一,提供向含有磷酸乙醇胺的样品中添加氧化还原酶的磷酸乙醇胺的定量方法。
可以通过添加氧化还原酶来还原介体,使还原后的介体与试剂反应,确定磷酸乙醇胺的浓度。
根据本发明的实施方式之一,提供在磷酸乙醇胺的定量方法中使用的氧化还原酶。
氧化还原酶可以是属于ec号1.4或ec号1.5的氧化还原酶。
属于ec号1.4或ec号1.5的氧化还原酶可以是从伯胺脱氢酶、单胺脱氢酶、二胺脱氢酶、多胺脱氢酶、乙醇胺脱氢酶、酪胺脱氢酶、苯基乙胺脱氢酶、苄胺脱氢酶、组胺脱氢酶、5-羟色胺脱氢酶、精胺脱氢酶、亚精胺脱氢酶、β-丙氨酸脱氢酶、γ-氨基丁酸(gaba)脱氢酶、牛磺酸脱氢酶、尸胺酸脱氢酶以及胍丁胺脱氢酶中选择的氧化还原酶。
牛磺酸脱氢酶也可以包含大亚基(largesubunit)。
属于ec号1.4或ec号1.5的氧化还原酶也可以是ec号1.4.3或ec号1.5.3的氧化酶。
氧化酶可以是从伯胺氧化酶、单胺氧化酶、二胺氧化酶、多胺氧化酶、乙醇胺氧化酶、酪胺氧化酶、苯基乙胺氧化酶、苄胺氧化酶、组胺氧化酶、5-羟色胺氧化酶、精胺氧化酶、亚精胺氧化酶、β-丙氨酸氧化酶、γ-氨基丁酸(gaba)氧化酶、牛磺酸氧化酶、尸胺酸氧化酶以及胍丁胺氧化酶中选择的氧化酶。
根据本发明的实施方式之一,提供含有所述任一种氧化还原酶的磷酸乙醇胺的定量用组合物。
磷酸乙醇胺的定量用组合物也可以含有通过添加氧化还原酶而被还原的介质和与被还原的所述介质反应的试剂。
根据本发明的实施方式之一,提供一种磷酸乙醇胺的定量用试剂盒,其中含有所述任一种氧化还原酶、通过添加所述氧化还原酶而被还原的介质、和与被还原的所述介质反应的试剂。
在所述磷酸乙醇胺的定量方法中,氧化还原酶是氧化酶,可以使通过添加氧化酶而产生的过氧化氢与试剂反应,确定磷酸乙醇胺的浓度。
根据本发明的实施方式之一,提供在磷酸乙醇胺的定量方法中作为氧化还原酶而使用的氧化酶。
氧化酶也可以是属于ec号1.4.3或ec号1.5.3的氧化酶。
氧化酶可以是从伯胺氧化酶、单胺氧化酶、二胺氧化酶、多胺氧化酶、乙醇胺氧化酶、酪胺氧化酶、苯基乙胺氧化酶、苄胺氧化酶、组胺氧化酶、5-羟色胺氧化酶、精胺氧化酶、亚精胺氧化酶、β-丙氨酸氧化酶、γ-氨基丁酸(gaba)氧化酶、牛磺酸氧化酶、尸胺酸氧化酶以及胍丁胺氧化酶中选择的氧化酶。
根据本发明的实施方式之一,提供含有所述任一种氧化酶的磷酸乙醇胺的定量用组合物。
磷酸乙醇胺的定量用组合物也可以含有与通过添加氧化酶而产生的过氧化氢反应的试剂。
根据本发明的实施方式之一,提供一种磷酸乙醇胺的定量用试剂盒,其中含有所述任一种氧化酶和与通过添加氧化酶而生成的过氧化氢反应的试剂。
根据本发明的实施方式之一,提供含有所述任一项所述的氧化还原酶或所述任一项所述的氧化酶的电极。
根据本发明的实施方式之一,提供一种具备所述电极作为工作电极的传感器芯片。
根据本发明的实施方式之一,提供一种具备所述传感器芯片的传感器。
(发明的效果)
根据本发明,提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组合物、定量用试剂盒或定量用传感器。
附图说明
图1是表示本发明的实施例的eap浓度与酶活性(u/ml)的相关性的图。
图2是表示本发明的实施例的eap浓度与吸光度(a438、mabs)的关系的图。
图3是表示本发明的实施例的酶促反应的ph依赖性的图。
图4是表示本发明的实施例的eap浓度与吸光度(a555、mabs)的关系的图。
图5是表示本发明的实施例的eap浓度与吸光度(a555、mabs)的关系的图。
图6是表示本发明的实施例的eap浓度与电流值(μa)的关系的图。
图7是表示本发明的实施例的eap浓度与电流值(μa)的关系的图。
图8的(a)是本发明的实施方式之一的传感器芯片10的示意图,图8的(b)~(d)是表示构成传感器芯片10的部件的示意图。
图9的(a)是本发明的实施方式之一的传感器100的示意图,图9的(b)是本发明的实施方式之一的传感器100的框结构图。
具体实施方式
以下,说明本发明的对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组合物、定量用试剂盒以及定量用传感器。但是,本发明的对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组合物、定量用试剂盒以及定量用传感器并不限定于以下所示的实施方式以及实施例的记载内容来解释。
在实施方式之一中,本发明中使用的氧化还原酶是作为底物作用于eap的脱氢酶。本发明中使用的氧化还原酶被认为最适合使用eap脱氢酶。然而,直到提交本申请时,eap脱氢酶尚未被鉴别。
因此,可以使用有效地作用于具有与eap类似结构的底物的脱氢酶作为本发明中使用的氧化还原酶的替代物。类似结构是指从结构、电子、立体化学的观点等来看被认为是类似的物理化学结构。
例如,作为在结构上类似于eap的底物,存在具有ch-nh2键或ch-nh键的底物,作为该底物的酶,存在属于ec号1.4或ec号1.5的氧化还原酶。
例如,作为属于ec号1.4或ec号1.5的氧化还原酶,有伯胺脱氢酶、单胺脱氢酶、二胺脱氢酶、多胺脱氢酶、乙醇胺脱氢酶、酪胺脱氢酶、苯基乙胺脱氢酶、苄胺脱氢酶、组胺脱氢酶、5-羟色胺脱氢酶、精胺脱氢酶、亚精胺脱氢酶、β-丙氨酸脱氢酶、γ-氨基丁酸(gaba)脱氢酶、牛磺酸脱氢酶、尸胺酸脱氢酶以及胍丁胺脱氢酶。特别地,作为在结构上与eap类似的底物,有牛磺酸,作为该底物的脱氢酶,可以优选使用牛磺酸脱氢酶(tdh)。
本发明中使用的氧化还原酶可以是多聚体也可以是单体。例如,在可以仅由构成作为多聚体的氧化还原酶的几个亚基中的某一亚基(单体)催化从底物中提取氢并将其传递给氢受体的脱氢反应的情况下,用于本发明的氧化还原酶可以是多聚体,也可以是该亚基(单体)。
更具体地说,本发明使用的tdh包含具有序列号2的碱基序列的大亚基(latdh)和具有序列号4的碱基序列的小亚基(smtdh),不仅可以由具有latdh和smtdh的多聚体、还可以仅由latdh来催化从底物中提取氢并将其传递给氢受体的脱氢反应。因此,用于本发明的tdh可以具有latdh和smtdh,也可以仅具有latdh。
此外,属于ec号1.4或ec号1.5的氧化还原酶也可以是属于ec号1.4.3或ec号1.5.3的氧化酶。
例如,作为属于ec号1.4.3或ec号1.5.3的氧化酶,有伯胺氧化酶、单胺氧化酶、二胺氧化酶、多胺氧化酶、乙醇胺氧化酶、酪胺氧化酶、苯基乙胺氧化酶、苄胺氧化酶、组胺氧化酶、5-羟色胺氧化酶、精胺氧化酶、亚精胺氧化酶、β-丙氨酸氧化酶、γ-氨基丁酸(gaba)氧化酶、牛磺酸氧化酶、尸胺酸氧化酶以及胍丁胺氧化酶。特别地,可以优选使用苯基乙胺氧化酶(peaox)。
本发明使用的氧化还原酶的反应条件可以是作用于eap并有效催化氧化反应的任何条件。不过,酶一般具有表现出最高活性的最适温度、最适ph。因此,反应条件优选在最适温度、最适ph附近。例如,tdh的反应条件可以优选使用后述的温度30℃、ph8.5,但并不局限于此。另外,例如peaox的反应条件可以优选使用后述的温度37℃、ph8.5,但并不局限于此。
本发明的氧化还原酶可以是自然界中存在的微生物所产生的氧化还原酶,也可以是转化后的微生物所产生的氧化还原酶。从高效地大量表达酶的观点出发,通过使用转化后的微生物可以高效地大量表达酶。
产生本发明的氧化还原酶的微生物没有特别限定,但可以优选使用副球菌(paracoccus)属、甲基小盒菌(methylarcula)属、马特尔菌(martelella)属、红细菌(rhodobacter)属、玫瑰杆菌(roseobacter)属、芽殖杆菌(gemmobacter)属、节杆菌(arthrobacter)属、类节杆菌(paenarthrobacter)属、污物假节杆菌(pseudarthrobacter)属、耐冷冷杆菌(cryobacterium)属、芽孢杆菌(bacillus)属、中华单胞菌(sinomonas)属、tersicoccus属、考克氏菌(kocuria)属、微球菌(micrococus)属、短杆菌(brevibacterium)属、刘志恒氏菌(zhihengliuella)属、柠檬球菌(citricoccus)属、geodematophilus属、红球菌(rhodococcus)属、拟无枝酸菌(amycolatopsis)属、诺卡氏菌(nocardia)属、多栏纯洁杆菌(modestobacter)属、谷氨酸杆菌(glutamicibacter)属、伪诺卡氏菌(psudonocardia)属、戈登氏菌(gordonia)属、链霉菌(streptomyces)属、地嗜皮菌(geodermatophilus)属、纤维菌(cellulomonas)属、分枝杆菌(mycobacterium)属、分枝杆菌(mycolicibacterium)属、伪谷氨酸杆菌(psudoglutamicibacter)属、棒状杆菌(corynebacterium)属、类诺卡氏菌(nocardiopsis)属、野野村菌(nonomuraea)属、糖多孢菌(saccharomonospora)属、普劳瑟尔氏(prauserella)属、胺杆菌(amnibacterium)属、放线菌(actinobacteria)属、雷公藤糖多孢菌(saccharopolyspora)属、雷弗松氏菌(leifsonia)属、壤霉菌(agromyces)属、米色链霉菌(streptacidiphilus)属、木聚糖单胞菌(xylanimonas)属、冢村菌(tsukamurella)属、干草威廉姆氏菌(williamsia)属、浅野氏菌(asanoa)属、植物内生菌(plantactinospora)属、盐孢菌(salinispora)属、阿格雷氏菌(agreia)属、克拉菌(cryocola)属、短小杆菌(curtobacterium)属、murinocardiopsis属、北方栖低境菌(subtercola)属、小双包菌(microbispora)属、姜成林氏菌(jiangella)属、芽殖球菌(blastococcus)属、马杜拉放线菌(actinomadura)属、游动放线菌(actinoplanes)属、细小链孢菌(catenulispora)属、横梗霉(lichtheimia)属、并头状菌(syncephalastrum)属的微生物。
例如,本发明的氧化还原酶可以是由脱氮副球菌(paracoccusdenitrificans)产生的tdh,也可以是由在含有来自双歧杆菌的tdh基因的质粒的作用下转化的大肠杆菌产生的tdh,但是通过使用在含有来自双歧杆菌的tdh基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
另外,例如,本发明的氧化还原酶可以是由球形节杆菌(arthrobacterglobiformis)产生的peaox,也可以是由在含有来自球形节杆菌的peaox基因的质粒的作用下转化的大肠杆菌产生的peaox,但是通过使用在含有来自球形节杆菌的peaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
另外,例如,本发明的氧化还原酶可以是由伞状毛霉菌(lichtheimiacorymbifera)产生的胺氧化酶(lcaox),也可以是由在含有来自伞状毛霉菌的具有序列号16的碱基序列的lcaox基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但是通过使用在含有来自伞状毛霉菌的lcaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
另外,例如,本发明的氧化还原酶可以是由总状横梗霉(lichtheimiaramosa)产生的假设蛋白(hypotheticalprotein)(lrhp),也可以是由在含有来自总状横梗霉的具有序列号21的碱基序列的lrhp基因的质粒作用下转化的大肠杆菌产生的假设蛋白,但是通过使用在含有来自总状横梗霉的lrhp基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
此外,例如,本发明的氧化还原酶可以是由总状共头霉(syncephalastrumracemosum)产生的胺氧化酶(sraox3925),也可以是由在含有来自总状共头霉的具有序列号26的碱基序列的sraox3925基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但是通过使用在含有来自总状共头霉的sraox3925基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
此外,例如,本发明的氧化还原酶可以是由总状共头霉产生的胺氧化酶(sraox3926),也可以是由在含有来自总状共头霉的具有序列号31的碱基序列的sraox3926基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但是通过使用在含有来自总状共头霉的sraox3926基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
此外,例如,本发明的氧化还原酶可以是由总状共头霉产生的乙醇胺氧化酶(sreaox),也可以是由在含有来自总状共头霉的具有序列号36的碱基序列的sreaox基因的质粒的作用下转化的大肠杆菌产生的乙醇胺氧化酶,但是通过使用在含有来自总状共头霉的sreaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由脱氮副球菌产生的latdh的氨基酸序列(序列号1)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号1的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由球形节杆菌产生的peaox的氨基酸序列(序列号9)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号9的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由伞状毛霉菌产生的lcaox的氨基酸序列(序列号15)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号15的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由总状横梗霉产生的lrhp的氨基酸序列(序列号20)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号20的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由总状共头霉产生的sraox3925的氨基酸序列(序列号25)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号25的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由总状共头霉产生的sraox3926的氨基酸序列(序列号30)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号30的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
在实施方式之一中,本发明的氧化还原酶可以列举对于由总状共头霉产生的sreaox的氨基酸序列(序列号35)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化还原酶以及具有在序列号35的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化还原酶。
(氨基酸序列的一致性)
氨基酸序列的一致性可以通过genetyx(注册商标)(泽尼特公司(genetyxcorporation))的最大匹配、搜索同源(homology)等程序,或dnasis(注册商标)pro(日立解决方案公司)的最大匹配、多重比对(multiplealignment),或clustalw的多重比对等程序来计算。为了计算氨基酸序列的一致性而对两个以上的氧化还原酶的氨基酸序列进行对比时,可以检查在所述两个以上的氧化还原酶中一致的氨基酸的位置。基于这些信息,可以确定氨基酸序列中的一致区域。在此,对于两个以上的氨基酸序列,一致性%是指,在利用blosum62等算法进行两个以上的氨基酸序列的比对时,将能够比对的区域的总氨基酸数作为分母,将其中由相同的氨基酸占据的位置的数量作为分子时的百分比。因此,通常,当在两个以上的氨基酸序列中存在完全不具有一致性的区域时,例如,当在一方的氨基酸序列中存在在氨基端(n-端)或碳端(c-端)中完全不具有一致性的附加序列时,不可能比对所述不具有一致性的区域,因此不被用于计算一致性%。
(酶的制备方法)
以下,对本发明的氧化还原酶的制备方法进行说明。
(表达用质粒的构建)
通过通常使用的方法获得本发明的氧化还原酶表达用的质粒。例如,从产生本发明的氧化还原酶的微生物中提取dna,制备dna文库。从制作完成的dna文库中鉴别并分离编码本发明的氧化还原酶的dna片段。使用将分离后的dna片段作为模板的互补引物,通过聚合酶链反应(pcr)扩增该dna片段,并克隆编码本发明的氧化还原酶的基因。将扩增后的dna片段连接到载体(vector),获得具有对本发明的氧化还原酶进行编码的dna片段的质粒。
或者,化学合成编码本发明的氧化还原酶的dna片段,并将该dna片段连接到载体,获得具有对本发明的氧化还原酶进行编码的dna的质粒。
用获得的质粒转化大肠杆菌等菌株,得到具有对本发明的氧化还原酶进行编码的dna的大肠杆菌等菌株。
另外,例如,可以用获得的质粒转化酵母等菌株,获得具有对本发明的氧化还原酶进行编码的dna的酵母等菌株。作为转化为酵母的方法,可以优选使用公知的方法,例如使用乙酸锂的方法(methodsmol.cell.biol.,5,255-269(1995))、电穿孔(jmicrobiolmethods55(2003)481-484)等,但不限于此,可以使用包括原生质球法、玻璃珠法等的各种任意方法进行转化。作为分类为酵母的微生物,例如可以举出属于接合酵母(zygosaccharomyces)属、酵母(saccharomyces)属、毕赤酵母(pichia)属、假丝酵母(candida)属等酵母。具有编码本发明的氧化还原酶的dna的质粒也可以含有用于实现对于转化后的细胞的选择的标记基因。作为标记基因,可以举出与宿主的营养缺陷型互补的基因,例如ura3和trp1。另外,具有对本发明的氧化还原酶进行编码的dna的质粒期望含有能够在宿主细胞中表达本发明的氧化还原酶基因的启动子或其它调控序列(regulatorysequence)(例如分泌信号序列、增强子序列、终止子序列或多腺苷酸化(polyadenylation)序列等)。作为启动子,具体地,可以举出例如gal1启动子、adh1启动子等。
另外,作为宿主细胞的其他例子,可以举出例如曲霉(aspergillus)属、木霉(trichoderma)属等丝状真菌。丝状真菌的转化体的制备方法没有特别限制,例如,根据常规方法,举出编码本发明的氧化还原酶的dna以表达的方式插入宿主丝状真菌中的方法。具体地说,制备在表达诱导启动子以及终止子之间插入了编码本发明的氧化还原酶的基因的dna构建体(construct),然后用含有编码本发明的氧化还原酶的基因的dna构建体转化宿主丝状真菌,由此获得过表达编码氧化还原酶的基因的转化体。在本说明书中,为了转化宿主丝状真菌而制备的、由表达诱导启动子-编码氧化还原酶的基因-终止子组成的dna片段和包含该dna片段的重组载体统称为dna构建体。
编码氧化还原酶的基因以表达的方式插入宿主丝状真菌内的方法没有特别限制,例如可以举出通过使用同源重组而直接插入宿主生物体的染色体上的方法,或者通过连接到质粒载体上而导入宿主丝状真菌内的方法等。
在利用同源重组的方法中,dna构建体可以连接在与染色体上的重组位点的上游区域以及下游区域相同的序列之间,并插入宿主丝状真菌的基因组中。在宿主丝状真菌自身的高表达启动子控制下,在宿主丝状真菌内过表达,由此能够获得由自我克隆而产生的转化体。高表达启动子没有特别限制,例如,可以举出作为翻译延伸因子的tef1基因(tef1)的启动子区、α-淀粉酶基因(amy)的启动子区、碱性蛋白酶基因(alp)的启动子区等。
在利用载体的方法中,可以通过常规方法将dna构建体整合到用于转化丝状真菌的质粒载体中,并可以通过常规方法转化相应的宿主丝状真菌。
像这种优选的载体-宿主系统只要是可以在宿主丝状真菌中产生本发明的氧化还原酶的系统即可,不做特别限定,例如可以举出puc19以及丝状真菌的系统、psta14(mol.gen.genet.218,99-104,1989)以及丝状真菌的系统等。
优选将dna构建体导入宿主丝状真菌的染色体而使用,但作为其他方法,通过将dna构建体整合到自主复制型的载体(ozekietal.biosci.biotechnol.biochem.59,1133(1995)),也可以以不导入染色体的形式使用。
dna构建体也可以含有用于实现对于转化后的细胞的选择的标记基因。标记基因没有特别限定,例如可以举出如pyrg、niad、adea般与宿主的营养缺陷型互补的基因;对于吡啶硫胺(pyrithiamin)、潮霉素b、寡霉素等药物的耐药基因等。此外,dna构建体优选含有可以实现在宿主细胞中过表达对本发明的氧化还原酶进行编码的基因的启动子、终止子、其它的调控序列(例如增强子,多腺苷酸化序列等)。启动子没有特别限定,可以列举适当的表达诱导型启动子、组成型启动子,例如可以列举tef1启动子、alp启动子、amy启动子等。终止子也没有特别限定,例如可以举出alp终止子、amy终止子、tef1终止子等。
在dna构建体中,关于编码本发明的氧化还原酶的基因的表达调控序列,插入的含有对本发明的氧化还原酶进行编码的基因的dna片段并非必须具有表达调控功能的序列。另外,在通过共转化法来进行转化的情况下,dna构建体存在不具有标记基因也可的情况。
dna构建体的实施方式之一是例如将tef1基因启动子、编码氧化还原酶的基因、alp基因终止子以及pyrg标记基因连接到位于puc19的多克隆位点的in-fusion克隆位点(in-fusioncloningsite)而获得的dna构建体。
作为转化为丝状真菌的方法,可以适当选择本领域技术人员已知的方法,例如,在制备宿主丝状真菌的原生质体后,可以采用使用聚乙二醇和氯化钙的原生质体peg法(例如,参照mo1.gen.genet.218,99-104,1989、日本特开2007-222055号公报等)。根据所使用的宿主丝状真菌和转化标记基因,使用合适的用于使转化丝状真菌再生的培养基。例如,在使用酱油曲霉(aspergillussojae)作为宿主丝状真菌、使用pyrg基因作为转化标记基因的情况下,转化丝状真菌的再生例如可以用含有0.5%琼脂以及1.2m山梨醇的czapek-dox最低限度培养基(minimalmedium)(difco公司制造)进行。
(酶的重组表达)
在培养基中培养具有对本发明的氧化还原酶进行编码的dna的大肠杆菌等菌株。在培养微生物宿主细胞时,可以通过通风搅拌深层培养、振荡培养、静置培养等在10℃~42℃的培养温度下,优选在约25℃的培养温度下进行数小时~数天,更优选在约25℃的培养温度下优选进行1天~7天。作为培养上述微生物宿主细胞的培养基,例如,使用在酵母提取物、胰胨(胰蛋白胨)、胨(蛋白胨)、肉类提取物、玉米浆或大豆或麦麸的浸出液等一种以上的氮源中,添加氯化钠、磷酸一钾、磷酸二钾、硫酸镁、氯化镁、氯化铁、硫酸铁或硫酸锰等无机盐类中的一种以上,并根据需要适当添加糖质原料、维生素等的培养基。通过离心分离而从培养得到的培养液中分离菌体。将分离得到的菌体进行超声波粉碎、磨碎等,或者利用溶菌酶、yatalase等溶菌酶进行处理,制成悬浊液,将该悬浊液离心分离,由得到的级分得到粗酶液。
(酶的纯化)
酶的纯化方法可以是任何方法,只要是可以从粗酶液中纯化酶的方法即可。例如,可以通过离子交换色谱法、凝胶过滤层析法等通常使用的方法,从粗酶液中纯化酶。
(酶的活性测定)
酶的活性测定的方法可以是任何方法,只要是直接或间接地测定酶所催化的氧化还原反应所产生的产物的方法即可。例如,酶催化氧化还原反应而产生还原产物,该还原产物将电子传递到电极而产生电流值,可以通过测量该电流值来测定酶活性。优选地,可以通过使通过酶催化的氧化还原反应而产生的还原产物和含有与该还原产物反应的吸光物质的试剂(以下称为“吸光试剂”)反应并进行吸光度测定来测定酶活性。
(eap的定量)
使作用于eap的氧化还原酶作用于含有eap的样品。样品中的eap浓度并不特别限制,例如可以是0.1μm~1000μm。作用时间可以为例如5秒~120分钟,优选为0.5分钟~60分钟,更优选为1分钟~30分钟,更加优选为1分钟~10分钟。作用温度也取决于所使用的酶的最适温度,例如可以适当地选择用于通常的酶反应的、20℃~45℃的温度。
本发明所用的作用于eap的氧化还原酶的适当用量,例如,添加使得终浓度为0.001u/ml~50u/ml,优选为0.01u/ml~10u/ml即可。通常,样品溶液中所含的底物的浓度越低,则添加的氧化还原酶的终浓度越高为好。考虑氧化还原酶的最适ph,优选使用缓冲剂(bufferingagent)来调节作用时的ph值,以使该ph值成为适合于反应的ph值,但是该ph值不限于此,只要是可进行作用的ph值即可。例如为ph3~ph11,优选为ph5~ph9。作为可以使用的缓冲剂,例如可以举出n-[三(羟甲基)甲基]甘氨酸、磷酸盐、乙酸盐、碳酸盐、三(羟甲基)-氨基甲烷、硼酸盐、柠檬酸盐、二甲基谷氨酸盐、曲辛、hepes、mes、bis-tris、ada、pipes、aces、mopso、bes、mops、tes、dipso、tapso、popso、heppso、epps、曲辛(tricine)、n-二(羟乙基)甘氨酸(bicine)、taps、邻苯二甲酸、酒石酸等。
本发明提供一种通过作用于eap的氧化还原酶来还原介体(mediator),使被还原的介体与显色或褪色试剂反应而测定eap的方法。作为本发明所用的显色或褪色底物,除dcip(2,6-dichlorophenolindophenol,二氯酚靛酚)以外,还可举出例如四唑类化合物(tetrazoliumblue、nitro-tetrazoliumblue、水溶性四氮唑-1(watersolubletetrazolium(wst)-1)、wst-3、wst-4、wst-5、wst-8、wst-9)等。
在本发明的eap测定方法中使用的样品可以是来自可能含有eap的任意生物学样品,例如血液、血浆等的样品。样品可以适当地被加工处理。例如,也可以通过离心浓缩装置进行浓缩。
(含有氧化还原酶的组合物以及磷酸乙醇胺的定量用试剂盒)
本发明的使用氧化还原酶的eap的定量方法可以通过提供含有氧化还原酶和产物反应试剂的组合物来进行,也可以通过组合氧化还原酶与市售的产物反应试剂来进行。例如,可以作为含有氧化还原酶的磷酸乙醇胺的定量用组合物、进一步含有通过添加氧化还原酶而被还原的介体和与被还原的介体反应的试剂的磷酸乙醇胺的定量用组合物而被提供。此外,可以作为含有氧化还原酶、通过添加氧化还原酶而被还原的介体和与被还原的介体反应的试剂的磷酸乙醇胺的定量用试剂盒而被提供。
用于本发明的测定方法或定量用试剂盒的介体(也称为人造电子介体、人造电子受体或电子介体)没有特别限定,只要是能够从氧化还原酶接受电子的介体即可。作为介体,可以举例醌类、酚嗪类、紫精类、细胞色素类、吩噁嗪类、吩噻嗪类、铁氰化物、例如铁氰化钾、铁氧还蛋白类、二茂铁、锇络合物及其衍生物等,作为酚嗪化合物,可以举出例如5-甲基吩嗪硫酸甲酯(5-methylphenaziniummethosulfate)(pms)、甲氧基pms,但不限于此。
(传感器芯片和电极)
图8的(a)是本发明的实施方式之一的传感器芯片10的示意图,图8的(b)~图8的(d)是表示构成传感器芯片10的部件的示意图。传感器芯片10具备配置在基材11上的两个以上的电极。基材11由绝缘材料构成。在图8的(a)以及图8的(b)中,作为一个例子,工作电极1、对电极(相对电极)3以及参考电极5配置在基材11上。各电极分别与布线部7电连接,布线部7与位于各电极的布线方向的相反侧的端子9电连接。工作电极1、对电极3以及参考电极5彼此分开配置。另外,工作电极1、对电极3以及参考电极5优选与布线部7以及端子9一体地构成。另外,也可以使对电极3和参考电极5集成在一起。
如图8的(a)以及图8的(c)所示,在与布线部7平行的基材11的端部配置有间隔件(spacer)13,配置有覆盖工作电极1、对电极3、参考电极5以及间隔件13的盖15。间隔件13和盖15由绝缘材料构成。间隔件13具有与工作电极1、对电极3以及参考电极5大致相同的厚度,并优选为与工作电极1、对电极3和参考电极5紧密接触。此外,间隔件13和盖15可以一体地构成。盖15是防止布线部7暴露在外部空气中而劣化、防止由测定样品的渗入而引起的短路的保护层。
在实施方式之一中,本发明的氧化还原酶可以被涂布、吸附或固定于电极。优选地,本发明的氧化还原酶被涂布、吸附或固定于工作电极。在另一种实施方式中,还可以将介体与氧化还原酶一起涂布、吸附或固定于电极。氧化还原酶,或氧化还原酶和介体可包含在配置在工作电极1、对电极3以及参考电极5上的反应层19。作为电极,可以使用碳电极、铂、金、银、镍以及钯等金属电极等。在为碳电极的情况下,作为材料,可以举出热解石墨(pg)、玻璃碳(gc)、碳糊以及泡沫塑料碳(pfc)等。测定系统可以是二电极系统,也可以是三电极系统,例如,酶可以固定在工作电极上。作为参照电极,可以举出标准氢电极、可逆氢电极、银-氯化银电极(ag/agcl)、钯氢电极以及饱和甘汞电极等,从稳定性、再现性的观点出发,优选使用ag/agcl。
酶可以通过交联、透析膜覆盖、向聚合物基体(polymermatrix)包封、使用光交联聚合物、使用导电聚合物、使用氧化/还原聚合物等来固定于电极。另外,也可以将酶与介体一起固定在聚合物中或吸附固定于电极,也可以组合这些方法。
用于组合物、试剂盒、电极或传感器芯片的介体(也称为人造电子介体、人造电子受体或电子介体)没有特别限定,只要是能够从氧化还原酶接受电子即可。作为介体,可以举例醌类、酚嗪类、紫精类、细胞色素类、吩噁嗪类、吩噻嗪类、铁氰化物、例如铁氰化钾、铁氧还蛋白类、二茂铁、锇络合物以及其衍生物等,作为酚嗪化合物,可以举例pms、甲氧基pms,但不限于此。
本发明的氧化还原酶通过使用恒电位仪、恒电流仪等,可应用于各种电化学测定方法。作为电化学测定方法,可以举例安培法(amperometry)、电位法(potentiometry)以及库仑法(coulometry)等各种方法。例如,通过使用安培法,通过测定由过氧化氢电极对氧化还原酶与eap反应时产生的过氧化氢施加+600mv~+1000mv(vs.ag/agcl)而产生的电流值,可以计算样品中的eap的浓度。例如,针对已知的eap浓度(0、50、100、150、200μm)而测定电流值,并绘制相对于eap浓度的电流值,由此可以创建校正曲线。通过测定未知的eap的电流值,可以从校正曲线中获得eap浓度。作为过氧化氢电极,例如,可以使用碳电极、铂电极。此外,使用固定了过氧化物酶、过氧化氢酶等还原酶的电极代替过氧化氢电极,通过测定施加-400mv~+100mv(vs.ag/agcl)而产生的还原电流值,可以定量过氧化氢的量,测定eap的值。
此外,将介体混入反应溶液中,通过例如安培法将氧化还原酶与eap反应时产生的电子传递给氧化型介体,产生还原型介体,进而测定通过施加-1000mv~+500mv(vs.ag/agcl)而产生的电流值,由此可以计算样品中的eap的浓度。作为对电极,优选碳电极、铂电极。例如,针对已知的eap浓度(0、50、100、150、200μm)而测定电流值,并绘制相对于eap浓度的电流值,由此可以创建校正曲线。通过测定未知的eap的电流值,可以从校正曲线中获得eap浓度。
此外,可以使用印刷电极(传感器芯片)来减少测定所需的溶液量。在这种情况下,电极优选形成在由绝缘基板构成的基材上。具体地说,期望通过光刻技术、诸如丝网印刷、凹版印刷以及柔性版印刷等印刷技术来在基板上形成电极。另外,作为绝缘基板的材料,可以举出硅、玻璃、陶瓷、聚氯乙烯、聚乙烯、聚丙烯以及聚酯等,但优选使用对各种溶剂、化学品的耐性强的材料。
[eap测定传感器]
在实施方式之一中,提供使用本发明的氧化还原酶的eap测定传感器。图9的(a)是本发明的实施方式之一的传感器100的示意图。传感器是使用本发明的氧化还原酶的eap测定装置,具备含有氧化还原酶的传感器芯片和测定部。测定部30例如也可以具备作为输入部的开关31和作为显示部的显示器33。开关31例如可以用于控制测定部30的电源的接通(on)/断开(off),也可以用于控制传感器100中的eap测定的开始、中断。显示器33例如可以显示eap的测定值,也可以具备作为控制测定部30的输入部的触摸面板。
图9的(b)是本发明的实施方式之一的传感器100的框结构图。传感器100例如可以在测定部30具备控制部110、显示部120、输入部130、存储部140、通信部150以及电源160,它们也可以通过导线190而相互电连接。另外,稍后描述的传感器芯片10的端子与测定部30的端子电连接,传感器芯片10所产生的电流由控制部110检测。控制部110是控制传感器100的控制装置,例如由公知的中央处理器(cpu)和控制传感器100的操作程序构成。或者,控制部110具备中央处理器和操作系统(os),也可以具备用于进行eap测定的应用程序或模块。
显示部120例如具备公知的显示器33,可以显示eap的测定值、测定部30的状态、对测定者的操作请求。输入部130是测定者操作传感器100的输入装置,例如,也可以是开关31、配置在显示器33的触摸面板。多个开关31可以配置在测定部30。
存储部140由主存储装置(存储器)构成,也可以在外部配置辅助存储装置(硬盘)。主存储装置(存储器)也可以由只读存储器(rom)和/或随机存取存储器(ram)构成。操作程序、操作系统、应用程序或模块被存储在存储部140中,由中央处理器执行而构成控制部110。另外,测量值、电流值可以存储在存储部140中。
通信部150是连接传感器100或测定部30与外部的装置(计算机、打印机或网络)的公知的通信装置。通信部150和外部的装置通过有线或无线通信连接。另外,电源160是向传感器100或测定部30供给电源的公知的电源装置。
如上所述,本发明的eap的定量方法、定量用的氧化还原酶、定量用组合物以及定量用试剂盒通过含有氧化还原酶,可以提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组合物、定量用试剂盒以及定量用传感器。
(利用氧化酶活性的eap的定量方法)
本发明使用的氧化酶是作为底物而作用于eap的氧化酶。关于本发明所使用的氧化酶,被认为最合适使用eap氧化酶。然而,直到提交本申请时,eap氧化酶尚未被鉴别。作为在结构上与eap类似的底物,存在具有ch-nh2键或ch-nh键的底物,作为该底物的酶,存在胺氧化酶。
更具体而言,作为在结构上与eap类似的底物,有苯基乙胺、乙醇胺、酪胺、苄胺、组胺、5-羟色胺、精胺、亚精胺、β-丙氨酸、γ-氨基丁酸(gaba)、牛磺酸、尸胺、胍丁胺等,作为该底物的氧化酶,可以优选使用苯基乙胺氧化酶(peaox)、乙醇胺氧化酶、酪胺氧化酶、苄胺氧化酶、5-羟色胺氧化酶、精胺氧化酶、亚精胺氧化酶、β-丙氨酸氧化酶、γ-氨基丁酸(gaba)氧化酶、牛磺酸氧化酶、尸胺氧化酶、胍丁胺氧化酶等。
本发明使用的氧化酶的反应条件可以是作用于eap并有效催化氧化反应的任何条件。不过,酶一般具有表现出最高活性的最适温度、最适ph。因此,反应条件优选在最适温度、最适ph附近。例如,作为peaox的反应条件,可以优选使用后述的温度37℃、ph8.5,但并不局限于此。
在本发明中使用的氧化酶的反应过程中,当本发明的氧化酶作用于eap时,各种化学物质可以参与其中。例如,当本发明的氧化酶作用于eap时,氧可以作为氧化还原反应的电子受体而参与其中。
氧化酶可以是自然界中存在的微生物所产生的氧化酶,也可以是转化后的微生物所产生的氧化酶。从高效地大量表达酶的观点出发,通过使用转化后的微生物可以高效地大量表达酶。
例如,本发明的氧化酶可以是由球形节杆菌产生的氧化酶(agpeaox),也可以是由在含有来自球形节杆菌的peaox基因的质粒的作用下转化的大肠杆菌产生的氧化酶,但是通过使用在含有来自球形节杆菌的peaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
另外,例如,本发明的氧化酶可以是由伞状毛霉菌产生的lcaox,也可以是由在含有来自伞状毛霉菌的具有序列号16的碱基序列的lcaox基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但是通过使用在含有来自伞状毛霉菌的lcaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
此外,例如,本发明的氧化酶可以是由总状横梗霉产生的lrhp,也可以是由在含有来自总状横梗霉的具有序列号21的碱基序列的lrhp基因的质粒的作用下转化的大肠杆菌产生的假设蛋白,但是通过使用在含有来自总状横梗霉的lrhp基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
另外,例如,本发明的氧化酶可以是由总状共头霉产生的sraox3925,也可以是由在含有来自总状共头霉的具有序列号26的碱基序列的sraox3925基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但通过使用在含有来自总状共头霉的sraox3925基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
此外,例如,氧化酶可以是由总状共头霉产生的sraox3926,也可以是由在含有来自总状共头霉的具有序列号31的碱基序列的sraox3926基因的质粒的作用下转化的大肠杆菌产生的胺氧化酶,但是通过使用在含有来自总状共头霉的sraox3926基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
此外,例如,氧化酶可以是由总状共头霉产生的sreaox,也可以是由在含有来自于总状共头霉的具有序列号36的碱基序列的sreaox基因的质粒的作用下转化的大肠杆菌产生的乙醇胺氧化酶,但是通过使用在含有自总状共头霉的sreaox基因的质粒的作用下转化的大肠杆菌,可以高效地大量表达氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由球形节杆菌产生的peaox的氨基酸序列(序列号9)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号9的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由伞状毛霉菌产生的lcaox的氨基酸序列(序列号15)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号15的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由总状横梗霉产生的lrhp的氨基酸序列(序列号20)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号20的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由总状共头霉产生的sraox3925的氨基酸序列(序列号25)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号25的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由总状共头霉产生的sraox3926的氨基酸序列(序列号30)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号30的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
在实施方式之一中,本发明的氧化酶可以列举对于由总状共头霉产生的sreaox的氨基酸序列(序列号35)具有高的序列一致性(如70%以上、71%以上、72%以上、73%以上、74%以上、75%以上、76%以上、77%以上、78%以上、79%以上、80%以上、81%以上、82%以上、83%以上、84%以上、85%以上、86%以上、87%以上、88%以上、89%以上、90%以上、91%以上、92%以上、93%以上、94%以上、95%以上、96%以上、97%以上、98%以上、例如99%以上)的氧化酶以及具有在序列号35的氨基酸序列中,一个以上的氨基酸被改变或变异,或者,被删减、被置换、增加和/或被插入的氨基酸序列的氧化酶。
此外,由于这些氧化酶的氨基酸序列的一致性是通过与氧化还原酶的氨基酸序列的一致性相同的方法计算的,因此省略详细描述。
(酶的制备方法)
以下,对本发明的氧化酶的制备方法进行说明。
(表达质粒的构建)
通过通常使用的方法获得本发明的氧化酶表达质粒。例如,从产生本发明的氧化酶的微生物中提取dna,制备dna文库。从制作完成的dna文库中鉴别并分离编码本发明的氧化酶的dna片段。使用将分离后的dna片段作为模板的互补引物,通过聚合酶链反应(pcr)扩增该dna片段,并克隆编码本发明的氧化酶的基因。将扩增后的dna片段连接到载体,获得具有对本发明的氧化酶进行编码的dna片段的质粒。
或者,化学合成编码本发明的氧化酶的dna片段,并将该dna片段连接到载体,获得具有对本发明的氧化酶进行编码的dna片段的质粒。
用得到的质粒转化大肠杆菌等菌株,得到具有对本发明的氧化酶进行编码的dna的大肠杆菌等菌株。
酵母或丝状真菌可用作用于本发明的氧化酶的表达的宿主细胞。该方法可以遵循与上述氧化还原酶的表达相同的方法,省略详细描述。
本发明的氧化酶可以具有增加对eap的反应性的氨基酸置换。例如,可以在与来自具有序列号9的氨基酸序列的球形节杆菌的peaox的105位的苯丙氨酸相对应的位置和/或与在358位的亮氨酸相对应的位置具有氨基酸置换。
(酶的重组表达、纯化)
氧化酶的表达以及纯化可以通过与上述的氧化还原酶的表达和纯化相同的方法进行,省略详细描述。
(酶的活性测定)
酶活性测定的方法可以是任何方法,只要是直接或间接地测定酶所催化的反应所产生的产物的方法即可。例如,若使酶所催化的反应所产生的产物和与该产物反应的试剂(以下称为“产物反应试剂”)反应,测定由该反应产生的吸光物质,则可以通过进行吸光度测定来测定酶的活性。
(eap的定量)
使作用于eap的氧化酶作用于含有eap的样品。样品中的eap浓度并不特别限定,例如可以是0.1μm~1000μm。作用时间可以为例如5秒~120分钟,优选为0.5分钟~60分钟,更优选为1分钟~30分钟,更加优选为1分钟~10分钟。作用温度也取决于所使用的酶的最适温度,例如可以适当地选择用于通常的酶反应的、20℃~45℃的温度。
本发明所用的作用于eap的氧化酶的适当用量,例如,添加使得终浓度为0.001u/ml~50u/ml,优选为0.01u/ml~10u/ml即可。通常,样品溶液中所含的底物的浓度越低,则添加的氧化酶的终浓度越高为好。考虑氧化酶的最适ph,优选使用缓冲剂来调节作用时的ph值,以使该ph值成为适合于反应的ph值,然而,该ph值不限于此,只要是可进行作用的ph值即可。例如,为ph3~ph11,优选为ph5~ph9。可用的缓冲剂与通过氧化还原酶来定量eap的情况相同。
本发明提供一种通过测定作用于eap的氧化酶的作用的产物或消耗物来测定eap的方法,举出过氧化氢作为身为易于测定的产物的、优选作为测定对象的物质。通过氧化酶的作用而产生的过氧化氢可以通过显色底物等检测,作为在本发明中使用的显色底物,除了4-氨基安替比林以外,还可以列举例如ados(n-乙基-n-(2-羟基-3-磺丙基)-间氨基苯甲醚)、alos(n-乙基-n-(2-羟基-3-磺丙基)苯胺)、toos(n-乙基-n-(2-羟基-3-磺丙基)-间甲苯胺钠)、da-67(10-(羧甲基氨基羰基)-3,7-双(二甲氨基)-吩噻嗪)、da-64(n-(羧甲基氨基羰基)-4,4'-双(二甲氨基)-二苯胺)等。ados、alos、toos在与4-氨基安替比林缩合时显色。da-64、da-67不需要4-氨基安替比林,单独使用即可显色。在任意一种情况下,显色反应都是由过氧化物酶催化的。另外,作为要测量的消耗物,举出溶解氧,可以使用溶氧计等来测定反应液中的溶氧量。例如,可以通过分光光度计或生物化学自动分析装置等来测定上述测定试剂的显色的程度(吸光度变化量),并将其与标准样品的吸光度进行比较,从而测定样品中所含的eap。
在本发明的eap测定方法中使用的样品可以是来自可能含有eap的任意生物学样品,例如血液、血浆等的样品。样品可以适当地被加工处理。例如,也可以通过离心浓缩装置进行浓缩。
(含有氧化酶的组合物以及磷酸乙醇胺的定量用试剂盒)
本发明的使用氧化酶的eap的定量方法可以通过提供含有氧化酶和产物反应试剂的组合物来进行,也可以通过组合氧化酶与市售的产物反应试剂来进行。例如,可以作为含有氧化酶的磷酸乙醇胺的定量用组合物、进一步含有与通过添加氧化酶而产生的过氧化氢反应的试剂的磷酸乙醇胺的定量用组合物而被提供。此外,磷酸可以作为含有氧化酶和与通过添加氧化酶而产生的过氧化氢反应的试剂的磷酸乙醇胺的定量用试剂盒而被提供。
(传感器芯片和电极)
在实施方式之一中,本发明的氧化酶可以被涂布、吸附或固定于电极。优选地,本发明的氧化酶被涂布、吸附或固定于工作电极。电极的结构可以应用与针对使用氧化还原酶的电极而说明的结构相同的结构,因此省略详细描述。此外,氧化酶可以通过交联、由透析膜覆盖、向聚合物基体包封、使用光交联聚合物、使用导电聚合物、使用氧化/还原聚合物等来固定于电极。
本发明的氧化酶通过使用恒电位仪、恒电流仪等,可应用于各种电化学测定方法。作为电化学测定方法,可以举例安培法、电位法以及库仑法等各种方法。例如,通过使用安培法,通过测定由过氧化氢电极对氧化酶与eap反应时产生的过氧化氢施加+600mv~+1000mv(vs.ag/agcl)而产生的电流值,可以计算样品中的eap的浓度。例如,针对已知的eap浓度(0、50、100、150、200μm)而测定电流值,并绘制相对于eap浓度的电流值,由此可以创建校正曲线。通过测定未知的eap的电流值,可以从校正曲线中获得eap浓度。作为过氧化氢电极,例如,可以使用碳电极、铂电极。此外,使用固定了过氧化物酶、过氧化氢酶等还原酶的电极代替过氧化氢电极,通过测定施加-400mv~+100mv(vs.ag/agcl)而产生的还原电流值,可以定量过氧化氢的量,测定eap的值。
此外,可以使用印刷电极(传感器芯片)来减少测定所需的溶液量。在这种情况下,电极优选形成在由绝缘基板构成的基材上。使用氧化酶的传感器芯片的结构可以是与使用氧化还原酶的传感器芯片的结构相同的结构,省略详细说明。
[eap测量传感器]
在实施方式之一中,提供使用本发明的氧化酶的eap测定传感器。传感器是使用本发明的氧化酶的eap测定装置,具备:含有氧化酶的传感器芯片;和测定部。使用氧化酶的eap测定传感器的结构可以是与使用氧化还原酶的eap测定传感器的结构相同的结构,省略详细说明。
如上所述,本发明的eap的定量方法、定量用氧化酶、定量用组合物以及定量用试剂盒通过含有氧化酶,可以提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用酶、定量用组合物、定量用试剂盒和定量用传感器。
实施例
示出并更详细地说明上述的本发明的定量方法、定量用的氧化还原酶、定量用组合物以及定量用试剂盒的具体实施例以及试验结果。
(重组质粒pet22b(+)-latdhdna的制备)
全合成具有在两端含有限制性内切酶位点ndei和bamhi的序列号2的碱基序列的latdh基因以及具有序列号4的碱基序列的smtdh基因,首先,将latdh基因插入pet22b(+)的限制性内切酶位点ndei和bamhi之间,并使用它来转化大肠杆菌jm109。
将具有重组质粒的大肠杆菌jm109(pet22b(+)-latdh)菌株接种于2.5mllb-amp培养基[1%(w/v)细菌用胰胨(bactotryptone)、0.5%(w/v)酵母提取物、0.5%(w/v)nacl、50μg/ml氨苄西林(ampicillin)]中,在37℃下振荡培养20小时,得到培养物。
将培养物在7000rpm下离心分离5分钟,由此集菌而得到菌体。然后,使用qiagen(注册商标)tip-100(qiagen公司制造)从该菌体提取重组质粒pet22b(+)-latdh并纯化,得到2.5μg重组质粒pet22b(+)-latdh的dna。
(重组质粒pet22b(+)-latdh-smtdhdna的制备)
以具有序列号4的碱基序列的smtdh基因为模板,使用序列号5、6的合成寡核苷酸、primestar(注册商标)maxdnapolymerase(takarabio公司制造),在以下条件下进行pcr反应。即,加入primestarmaxpremix(2×)25μl、成为模板的smtdh基因100pg、上述的合成寡核苷酸各15pmol,用无菌水将总量设定为50μl。使用热循环仪(thermalcycler)(艾本德(eppendorfag)公司制造)在98℃下将制备完毕的反应液温育(incubate)2分钟,然后重复30次“98℃,10秒”-“55℃,5秒”-“72℃,35秒”的循环。同样地,以获得的重组质粒pet22b(+)-latdh为模板,使用序列号7、8的合成寡核苷酸进行pcr反应。将反应液用1.0%琼脂糖凝胶电泳,切割并纯化扩增后的目的dna。
然后,通过使用in-fusion(注册商标)hdcloningkit(克隆科技公司(clontech)制造)连接所获得的dna片段,制备出在pet22b(+)-latdh中的latdh的3’末端侧插入了smtdh基因的共表达载体。
与以上记述相同地,转化大肠杆菌jm109而得到了2.5μg重组质粒pet22b(+)-latdh-smtdh的dna。
(latdh、smtdh的生产)
使用通过上述步骤获得的上述重组质粒转化大肠杆菌bl21(de3)菌株。将各个大肠杆菌bl21(de3)菌株在2.5mlzyp-5052培养液(0.5%丙三醇(glycerol)0、0.05%葡萄糖(glucose)、0.2%乳糖(lactose)、50mm(nh4)2so4、50mmkh2po4、50mmna2hpo4、1mmmgso4)中,在25℃下培养27小时。
此后,将各菌体用ph8.5的0.05mches-naoh酸缓冲液清洗、超声波破碎、在15000rpm下离心分离10分钟,制备出含有具有序列号1的氨基酸序列的latdh的粗酶液和含有latdh和具有序列号3的氨基酸序列的smtdh的粗酶液各1.5ml。
同样地,也培养仅用pet22b(+)载体转化的大肠杆菌bl21(de3)菌株,进行超声波破碎处理,制备出1.5ml粗酶液。
(各种粗酶液的底物特异性的评价)
在96孔板中,进行了使用二氯酚靛酚(2,6-dichlorophenolindophenol,(dcip))的活性测定。如表1所示,将145μl的由磷酸钾缓冲液ph8.0、5-甲基吩嗪硫酸甲酯(5-methylphenaziniummethosulfate)(pms、富士胶片和光纯药株式会社制造)、二氯酚靛酚(2,6-dichlorophenolindophenol(dcip、sigma-aldrich公司制造))以及底物制成的试剂混合到5μl粗酶液中,观察到脱氢酶活性所引起的来自dcip的蓝色的消失(颜色变化)。使用了牛磺酸(富士胶片和光纯药株式会社制造)、磷酸乙醇胺(eap,富士胶片和光纯药株式会社制造)、乙醇胺(东京化成工业株式会社制造)和苄胺(东京化成工业株式会社制造)作为底物。
[表1]
(latdh活性测定)
将表1的化合物中的缓冲液从磷酸钾缓冲液ph8.0更改为ches-naoh酸缓冲液ph8.5、底物定为eap,并将eap浓度调整为5mm、10mm、30mm、100mm的试剂与latdh-smtdh或latdh的粗酶液混合,测定600nm的吸光度变化量,以此计算出latdh-smtdh或latdh的酶活性。
吸光度的变化量如下求出:将1400μl的含有ches-naoh酸缓冲液、dcip、eap的试剂在30℃下温育5分钟后,添加50μlpms和50μl粗酶液,使用分光光度计(u-3900、日立高新技术公司(hitachihigh-techsciencecorporation)制造)测定出在30℃下的每1分钟的a600变化量(δas)。
其次,代替底物溶液地,添加50μl0.05mches-naoh酸缓冲液并进行混合,测定出在30℃下每1分钟的a600变化量(δa0)。
根据以下公式计算出脱氢酶活性。
脱氢酶活性(u/ml)
=(δas-δa0)×1.5×df/(21.4×0.05)
=1.4×(δas-δa0)×df
21.4:dcip色素对于波长600nm的光的毫摩尔消光系数(mm-1cm-1)
df:酶溶液的稀释率
(测试结果:各种粗酶液的底物特异性的评价)
通过观察来自dcip的蓝色的消失程度,获得了共表达latdh-smtdh的粗酶液以及表达latdh的粗酶液都与牛磺酸的反应最大,也与磷酸乙醇胺反应的结果。另一方面,获得了共表达latdh-smtdh的粗酶液和表达latdh的粗酶液都不与乙醇胺、苄胺反应的结果。也就是说,获得了latdh-smtdh以及latdh可以将eap识别为底物的结果。
(试验结果:基于latdh的eap测定结果)
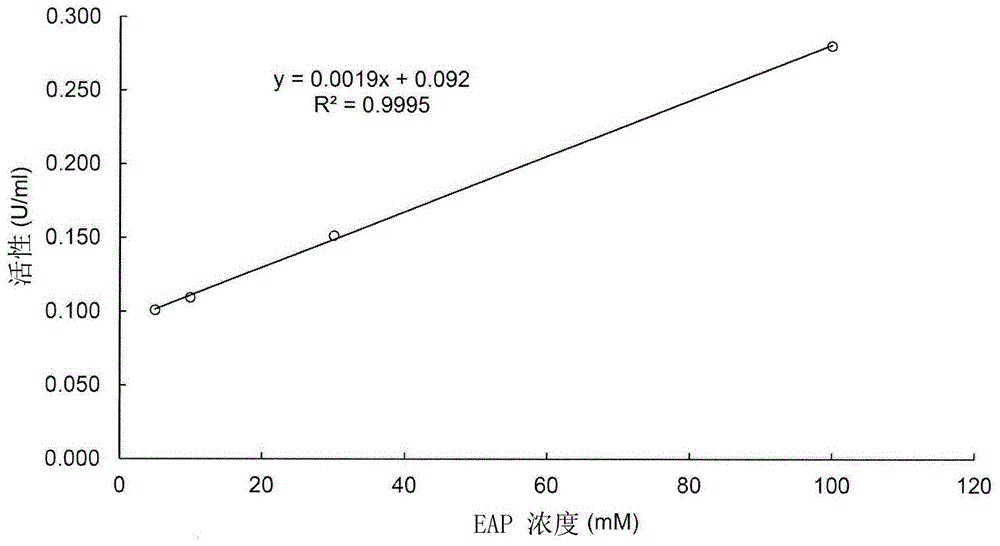
图1是表示本发明的实施例的eap浓度与酶活性(u/ml)的相关性的图。图1显示,在5mm~100mm的范围内,作为eap浓度和酶活性(u/ml)的相关指标的决定系数(r2)为0.9995,因此得到了eap浓度(mm)与酶活性(u/ml)之间具有相关性的结果。即,获得了通过利用latdh,可以在5mm-100mm的范围内测定eap浓度(mm)的结果。
(agpeaox的表达质粒的构建)
使用in-fusion(注册商标)hdcloningkit(克隆科技公司制造)制备来自具有序列号9的氨基酸序列的球形节杆菌的苯基乙胺氧化酶(agapeaox,uniprotidp46881)的表达质粒(pkk223-3-agpeaox)。
关于载体(pkk223-3)的片段,使用pkk223-3-cfp-t7(参照wo2007/125779公报)作为模板,使用pkk223-3hindiii3fw(5′-aagcttggctgttttggcggatgagagaag-3′)以及pkk223-3ecori5rv(5′-gaattctgtttcctgtgtgaaattgttatc-3′)作为引物,通过pcr制备。
在pcr后的溶液中加入1.0μldpni(newenglandbiolabs制造),在37℃下处理1小时后,进行琼脂糖凝胶电泳,切割含有目的片段(约4.6kbp)的凝胶,用illustra(注册商标)gfxpcrdnaandgelbandpurificationkit(ge医疗制造)从凝胶中提取。
将具有序列号10的碱基序列的agpeaox基因分割为从5'末端向3'末端的方向序列号11和序列号12的dna序列依次结合的前半部分(agpeaox_1-325)和从5'末端向3'末端的方向序列号13和序列号14的dna序列依次结合的后半部分(agpeaox_321-638)而委托给integrateddnatechnologies合成。agpeaox_1-325的5'末端侧的15个碱基(序列号11:caggaaacagaattc)和agpeaox_321-638的3’末端侧的15个碱基(序列号14:aagcttggctgtttt)表示来自pkk223-3载体的序列。agpeaox_1-325的3'末端侧的15个碱基(atcacgtacctgtcc)和agpeaox_321-638的5’末端侧的15个碱基(atcacgtacctgtcc)表示在agpeaox基因的前半与后半重叠的序列。
用pkk223-3的载体片段以及两个agpeaox基因片段而由表2的组成进行in-fusion反应(50℃,15分钟),得到agpeaox的表达质粒(pkk223-3-agpeaox)。用得到的质粒转化e.colijm109菌株。
[表2]
(agpeaox的重组表达)
将agpeaox生产株接种在装入试管的2.5mllb-amp培养基(氨苄西林浓度50μg/ml)中,并在37℃、160rpm下种子培养一晚。将1ml种子培养液接种在装入坂口烧瓶的150ml的含有0.1mmcuso4以及0.1mmiptg的lb-amp培养基(氨苄西林浓度50μg/ml)中,并在25℃下培养16小时。
将六个坂口烧瓶的培养液以6500×g离心10分钟而得到的颗粒物再悬浮于含有2mmcuso4的20mmtris-hclph8.0。
使用amicon(注册商标)ultraultracel-30k(密理博(millipore)公司制造)而在20mmtris-hclph8.0对在超声波粉碎菌体悬浊液后,以20400×g的速度离心15分钟而得到的上清液进行缓冲液置换,得到agpeaox的粗酶液。
(agpeaox的纯化)
使agpeaox的粗酶液涂覆于用20mmtris-hclph8.0平衡化的hiscreen(注册商标)captoq(ge医疗制造,树脂含量4.7ml)而与阴离子交换树脂结合。
然后,用含有47ml(10cv)的150mmnacl的20mmtris-hcl(ph8.0)清洗树脂,一边使20mmtris-hcl(ph8.0)中含有的nacl浓度从150mm直线上升至500mm,一边输液164.5ml(35cv),使与树脂结合的agpeaox溶出。
将溶出级分用amiconultracel-30k浓缩,用hiload(注册商标)26/60superdex200柱纯化。在树脂的平衡化、溶出中采用了20mmtris-hcl(ph8.0)。
通过sds-page评价溶出的各级分的纯度,回收不含杂质蛋白质的级分作为agpeaox的纯化标准品。
(agpeaox的脱氢酶活性测定)
将609μl的由表3的组成所构成的试剂在37℃下温育5分钟后,添加21μl底物溶液(1500mmeap)并混合,使用分光光度计(u-3900、日立高新技术公司制造)测定出37℃下的每1分钟的a438变化量(δas)。
接着,代替底物溶液地,添加21μl离子交换水并进行混合,测定出37℃下的每1分钟的a438变化量(δa0)。使用了由同仁化学研究所生产的1-methoxypes(1-mpes)以及2-(4-碘苯)-3-(4-硝基苯)-5-(2,4-二磺基苯)-2h-四氮唑钠盐(wst-1)。
【表3】
根据以下公式计算出脱氢酶活性。
脱氢酶活性(u/ml)
=(δas-δa0)×630×df/(37.0×x)
=17.0×(δas-δa0)×df/x
37.0:wst-1甲
df:酶溶液的稀释率
(基于agpeaox的eap的定量)
将609μl的由表4的组成所构成的试剂在37℃下温育5分钟后,添加eap溶液(30mm~300mm)21μl或离子交换水并混合,使用分光光度计(u-3900)测定出37℃下的10分钟的吸光度(a438)的变化。以测定开始10分钟后的a438为纵轴,eap浓度为横轴,评价a438与eap浓度的相关性。
[表4]
*关于活性(u),使用终浓度50mmeap,在ph8.5下进行了测定。
(试验结果:基于agpeaox的eap测定结果)
图2是表示eap浓度与10分钟后的吸光度(a438、mabs)的相关性的图。根据图2,在1mm~10mm的范围内,作为eap浓度与10分钟后的吸光度(a438、mabs)的相关指标的决定系数(r2)为0.975,因此得到了eap浓度(mm)与吸光度(a438(mabs))之间存在相关性的结果。即,获得了通过利用agpeaox所具有的脱氢酶活性,可以在1mm~10mm范围内测定eap浓度(mm)的结果。可以设想,增加使用的agpeaox量的话,更容易定量低浓度的eap,减少使用的agpeaox量的话,更容易定量高浓度的eap。
如上所述,通过本发明的在含有eap的样品中添加氧化还原酶的eap的定量方法、在含有eap的样品中添加的eap的定量用的氧化还原酶、含有在含有eap的样品中添加的氧化还原酶的eap的定量用组成物、以及含有在含有eap的样品中添加的氧化还原酶的eap的定量用试剂盒以及含有在含有eap的样品中添加的氧化还原酶的eap的定量用传感器,可以提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组成物、定量用试剂盒以及定量用传感器。
(agpeaox的氧化酶活性测定)
测定了用上述方法表达、纯化的agpeaox的氧化酶活性。将609μl的由表5的组成所构成的试剂在37℃下温育5分钟后,添加21μl底物溶液(1500mmeap)并混合,使用分光光度计(u-3900、日立高新科技公司制造)测定出在37℃下的每1分钟的a555变化量(δas)。
接着,代替底物溶液地,添加21μl离子交换水并进行混合,测定出在37℃下的每1分钟的a555变化量(δa0)。使用了由富士胶片和光纯药制造的4-氨基安替比林(4-aa)以及eap,由同仁化学研究所制造的n-乙基-n-(2-羟基-3-磺丙基)-3-甲基苯胺(toos),由东洋纺制造的辣根过氧化物酶(pod)。
[表5]
根据以下公式计算出氧化酶活性。
氧化酶活性(u/ml)
=(δas-δa0)×630×df/(39.2×0.5×x)
=32.1×(δas-δa0)×df/x
39.2:4-aa-toos缩合色素对于波长555nm的光的毫摩尔消光系数(mm-1cm-1)
df:酶溶液的稀释率
(agpeaox的催化反应的ph依赖性测定)
在测定agpeaox的催化反应的ph依赖性时,将表5的活性测定用试剂所含有的bicine-naoh缓冲液ph8.5置换为调整到规定的ph的磷酸钾缓冲液、mops-naoh缓冲液或bicine-naoh而进行活性测定。
(基于agpeaox的eap的定量)
将609μl的由表6的组成所构成的试剂在37℃下温育5分钟后,添加eap溶液(6、12、18、24或30mm)21μl或离子交换水并进行混合,使用分光光度计(u-3900)测定出在37℃下的5分钟的a555变化。以测量开始5分钟后的a555为纵轴,eap浓度为横轴,评价a555与eap浓度的相关性。
【表6】
*关于活性(u),采用终浓度50mm的eap,在ph8.5下进行了测定。
(试验结果:agpeaox的催化反应的ph依赖性)
图3是表示本发明的实施例的酶反应的ph依赖性的图。纵轴表示agpeaox的相对活性(relativeactivity(%)),横轴表示ph值。相对活性是指将bicine-naoh缓冲液ph8.5的氧化酶活性作为100%时的相对氧化酶活性。从图3获得了agpeaox的催化反应的最适ph为8.5的结果。因此,制备ph8.5的试剂,并用该试剂测定eap浓度。
(试验结果:基于agpeaox的eap测定结果)
图4是表示eap浓度与5分钟后的吸光度(a555、mabs)的相关性的图。根据图4,在200μm~1000μm的范围内,作为eap浓度与5分钟后的吸光度(a555、mabs)的相关指标的决定系数(r2)为0.993,因此得到了eap浓度(μm)与吸光度(a555(mabs))之间存在相关性的结果。即,获得了通过利用agpeaox,可以在200μm~1000μm的范围内测定eap浓度的结果。可以设想,增加使用的agpeaox量的话,更容易定量低浓度的eap,减少使用的agpeaox量的话,更容易定量高浓度的eap。
如上所述,通过本发明的在含有eap的样品中添加氧化酶的eap的定量方法、在含有eap的样品中添加的eap的定量用的氧化酶、有含在含有eap的样品中添加的氧化酶的eap的定量用组成物、含有在含有eap的样品中添加的氧化酶的eap的定量用试剂盒以及含有在含有eap的样品中添加的氧化酶的eap的定量用传感器,可以提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组成物、定量用试剂盒以及定量用传感器。
(lcaox的表达质粒的构建)
来自具有序列号15的氨基酸序列的伞状毛霉菌的胺氧化酶(lcaox、genbankidcdh56199.1)的表达质粒(pkk223-3-lcaox)是使用in-fusion(注册商标)hdcloningkit(clontech克隆科技公司制造)制成的。
根据(agpeaox的表达质粒的构建)中描述的方法制备了载体(pkk223-3)的片段。
将具有序列号16的碱基序列的lcaox基因分割为从5'末端向3'末端的方向序列号11和序列号17的dna序列依次结合的前半部分(lcaox_frag1)、序列号18记载的中间部分(lcaox_frag2)、以及从5'末端向3'末端的方向序列号19和序列号14的dna序列依次结合的后半部分(lcaox_frag3)而委托给integrateddnatechnologies合成。为了利用in-fusion反应而分别重复lcaox_frag1的3′末端侧和lcaox_frag2的5′末端侧的15个碱基(cccgaacaccttggt),以及lcaox_frag2的3′末端侧和lcaox_frag3的5′末端侧的15个碱基(cagcatcatcaacat)。
用pkk223-3的载体片段以及3个lcaox基因片段而由表7的组成进行in-fusion反应(50℃,15分钟),得到了lcaox的表达质粒(pkk223-3-lcaox)。用得到的质粒转化了e.colijm109菌株。
[表7]
(lrhp的表达质粒的构建)
来自具有序列号20的氨基酸序列的总状横梗霉的假设蛋白(lrhp、genbankidcds02610.1)的表达质粒(pkk223-3-lrhp)是使用in-fusion(注册商标)hdcloningkit(clontech克隆科技公司制造)制成的。
根据(agpeaox的表达质粒的构建)中描述的方法制备了载体(pkk223-3)的片段。
将具有序列号21的碱基序列的lrhp基因分割为从5'末端向3'末端的方向序列号11和序列号22的dna序列依次结合的前半部分(lrhp_frag1)、序列号23记载的中间部分(lrhp_frag2)、以及从5'末端向3'末端的方向序列号24和序列号14的dna序列依次结合的后半部分(lrhp_frag3)而委托给integrateddnatechnologies合成。为了利用in-fusion反应而分别重复lrhp_frag1的3′末端侧和lrhp_frag2的5′末端侧的15个碱基(catttagggcaagat)、以及lrhp_frag2的3′末端侧和lrhp_frag3的5′末端侧的15个碱基(caccatcaacatttg)。
用pkk223-3的载体片段以及3个lrhp基因片段而由表8的组成进行in-fusion反应(50℃,15分钟),得到了lrhp的表达质粒(pkk223-3―lrhp)。用得到的质粒转化了e.colijm109菌株。
[表8]
(sraox3925的表达质粒的构建)
来自具有序列号25的氨基酸序列的总状共头霉的胺氧化酶(sraox3925、genbankidorz03925.1)的表达质粒(pkk223-3-sraox3925)是使用in-fusion(注册商标)hdcloningkit(克隆科技公司制造)制成的。
根据(agpeaox的表达质粒的构建)中描述的方法制备了载体(pkk223-3)的片段。
将具有序列号26的碱基序列的sraox3925基因分割为从5'末端向3'末端的方向序列号11和序列号27的dna序列依次结合的前半部分(sraox3925_frag1)、序列号28记载的中间部分(sraox3925_frag2)、以及从5'末端向3'末端的方向序列号29和序列号14的dna序列依次结合的后半部分(sraox3925_frag3)而委托给integrateddnatechnologies合成。为了利用in-fusion反应而分别重复sraox3925_frag1的3′末端侧和sraox3925_frag2的5′末端侧的15个碱基(cagtttttaccagag)、以及sraox3925_frag2的3′末端侧和sraox3925_frag3的5′末端侧的15个碱基(gtcgtaggccagcat)分别重叠。
用pkk223-3的载体片段以及3个sraox3925基因片段而由表9的组成进行in-fusion反应(50℃,15分钟),得到了sraox3925的表达质粒(pkk223-3-sraox3925)。用得到的质粒转化了e.colijm109菌株。
[表9]
(sraox3926的表达质粒的构建)
来自具有序列号30的氨基酸序列的总状共头霉的胺氧化酶(sraox3926、genbankidorz03926.1)的表达质粒(pkk223-3-sraox3926)是使用in-fusion(注册商标)hdcloningkit(克隆科技公司制造)制成的。
根据(agpeaox的表达质粒的构建)中描述的方法制备了载体(pkk223-3)的片段。
将具有序列号31的碱基序列的sraox3926基因分割为从5'末端向3'末端的方向序列号11和序列号32的dna序列依次结合的前半部分(sraox3926_frag1)、序列号33记载的中间部分(sraox3926_frag2)、以及从5'末端向3'末端的方向序列号34和序列号14的dna序列依次结合的后半部分(sraox3926_frag3)而委托给integrateddnatechnologies合成。为了利用in-fusion反应而分别重复sraox3926_frag1的3′末端侧和sraox3926_frag2的5′末端侧的15个碱基(ttgcgcaaagatatt)、以及sraox3926_frag2的3′末端侧和sraox3926_frag3的5′末端侧的15个碱基(gatccgatggtagac)。
用pkk223-3的载体片段以及3个sraox3926基因片段而由表10的组成进行了in-fusion反应(50℃,15分钟),得到了sraox3926的表达质粒(pkk223-3-sraox3926)。用得到的质粒转化了e.colijm109菌株。
[表10]
(sreaox的表达质粒的构建)
来自具有序列号35的氨基酸序列的总状共头霉的乙醇胺氧化酶(sreaox、genbankidbau20376.1)的表达质粒(pkk223-3-sreaox)是使用in-fusion(注册商标)hdcloningkit(克隆科技公司制造)制成的。
根据(agpeaox的表达质粒的构建)中描述的方法制备了载体(pkk223-3)的片段。
将具有序列号36的碱基序列的sreaox基因分割为从5'末端向3'末端的方向序列号11和序列号37的dna序列依次结合的前半部分(sreaox_frag1)、序列号38记载的中间部分(sreaox_frag2)、以及从5'末端向3'末端的方向序列号39和序列号14的dna序列依次结合的后半部分(sreaox_frag3)而委托给integrateddnatechnologies合成。为了利用in-fusion反应而分别重复sreaox_frag1的3′末端侧和sreaox_frag2的5′末端侧的15个碱基(ctccgcaaagatata)、以及sreaox_frag2的3′末端侧和sreaox_frag3的5′末端侧的15个碱基(ccaatggtagatgga)。
用pkk223-3的载体片段以及3个sreaox基因片段而由表11的组成进行in-fusion反应(50℃,15分钟),得到了sreaox的表达质粒(pkk223-3-sreaox)。用得到的质粒转化了e.colijm109菌株。
[表11]
(酶的重组表达)
将lcaox、lrhp、sraox3925、sraox3926以及sreaox生产株接种于装入试管的2.5mllb-amp培养基(氨苄西林浓度50μg/ml),在37℃,160rpm下种子培养一晚。将1.5ml种子培养液接种于装入坂口烧瓶的150ml的含有0.02mmcuso4以及0.1mmiptg的lb-amp培养基(氨苄西林浓度50μg/ml),在25℃下培养16小时。
将150ml的培养液以6500×g离心10分钟而得到的颗粒物再悬浮于20mmtris-hclph7.5。将菌体悬浊液超声波破碎后,以20400×g离心15分钟而回收上清液作为粗酶液。
(lcaox、lrhp、sraox3925的纯化)
使lcaox、lrhp或sraox3925的粗酶液涂覆于以20mmtris-hclph7.5平衡化的hiscreen(注册商标)captoq(ge医疗制造,树脂含量4.7ml)而与阴离子交换树脂结合。
然后,用47ml(10cv)的20mmtris-hcl(ph7.5)清洗树脂,一边使20mmtris-hcl(ph7.5)中含有的nacl浓度从0mm直线上升到500mm,一边输液117.5ml(25cv),溶出与树脂结合的lcaox、lrhp或sraox3925。
将溶出级分用离子交换水稀释3倍而降低盐分浓度后,使其涂覆于以20mmtris-hclph7.5平衡化的hiscreen(注册商标)captoqinpres(ge医疗制造,树脂含量4.7ml)而与阴离子交换树脂结合。
然后,用23.5ml(5cv)的20mmtris-hcl(ph7.5)清洗树脂,一边使20mmtris-hcl(ph7.5)中含有的nacl浓度从0mm直线上升至300mm,一边输液141ml(30cv),溶出与树脂结合的lcaox、lrhp或sraox3925。
将溶出级分用amiconultracel-30k浓缩,用hiload(注册商标)26/60superdex200柱纯化。在树脂的平衡化、溶出中使用了含有150mmnacl的10mmbis-tris-hcl(ph7.0)。
通过sds-page评价溶出的各级分的纯度,回收不含杂质蛋白质的级分作为lcaox、lrhp或sraox3925的纯化标准品。
(lcaox、lrhp、sraox3925、sraox3926、sreaox的氧化酶活性测定)
测定了用上述方法表达的lcaox、lrhp、sraox3925、sraox3926、sreaox的氧化酶活性。将580μl的由表12的组成所构成的试剂在37℃下温育5分钟后,添加20μl底物溶液(1500mmeap)并混合,使用分光光度计(u-3900,日立高新技术公司制造)测定出在37℃下的每1分钟的a555变化量(δas)。接着,代替底物溶液地,添加20μl离子交换水并进行混合,测定出在37℃下的每1分钟的a555变化量(δa0)。
[表12]
根据以下公式计算出氧化酶活性。
氧化酶活性(u/ml)
=(δas-δa0)×600.0×df/(39.2×0.5×x)
=30.6×(δas-δa0)×df/x
39.2:4-aa-toos缩合色素对于波长555nm的光的毫摩尔消光系数(mm-1cm-1)
df:酶溶液的稀释率
(基于lrhp的eap的定量)
将580.0μl的由表13的组成所构成的试剂在37℃下温育5分钟后,添加eap溶液(3、6、18或30mm)20.0μl或离子交换水并混合,使用分光光度计(u-3900)测定出在37℃下的20分钟的a555变化。以测量开始20分钟后的a555为纵轴,eap浓度为横轴,评价a555与eap浓度的相关性。
[表13]
*关于活性(u),采用终浓度50mmeap,在ph7.5下进行了测定。
(基于电化学法的lrhp的eap的定量)
在丝网印刷电极(screen-printedelectrodes)(dropsens公司制造,产品号drp-c110)上涂布、混合20μl的150mmbicine-naoh缓冲液(ph7.5)、5μl的1.5m氯化钾液15μl,lrhp液(0.14u/ml)。然后,使用专用连接器(drp-cac)连接到als电化学分析仪814d。在+600mv(ag/agcl)下进行计时电流法(chronoamperometry)测定。接着,加入2μl各浓度的eap溶液,记录测量开始100秒后的电流值。作为对照实验,使用5μl超纯水(离子交换水)代替lrhp溶液而进行同样的实验。
(试验结果:lcaox、lrhp、sraox3925、sraox3926、sreaox的氧化酶活性测定)
lcaox、lrhp、sraox3925、sraox3926、sreaox的粗酶液的对于eap的氧化酶活性分别为1.8、5.8、2.6、0.2和16u/l。对于lcaox、lrhp、sraox3925,纯化后还分别表现出0.11、0.31和0.16u/ml的活性。因此,表示lcaox、lrhp、sraox3925各自单独地对氧化eap而生成过氧化氢的反应进行催化。sraox3926、sreaox被认为在纯化到不含杂质蛋白质的水平之后也表现出对于eap的氧化酶活性。
(试验结果:基于lrhp的eap测定结果)
图5是表示eap浓度与20分钟后的吸光度(a555、mabs)的相关性的图。根据图5,在100μm~1000μm的范围内,作为eap浓度与20分钟后的吸光度(a555、mabs)的相关指标的决定系数(r2)为0.908,因此得到了eap浓度(μm)与吸光度(a555(mabs))之间存在相关性的结果。即,获得了通过利用lrhp,可以在100μm~1000μm的范围内测定eap浓度(μm)的结果。可以设想,如果增加使用的lrhp量,则更容易定量低浓度的eap,如果减少使用的lrhp量,则更容易定量高浓度的eap。认为即使在使用lcaox、sraox3925、sraox3926或sreaox代替lrhp的情况下,也可以测定eap浓度(μm)。
(试验结果:基于电化学方法的lrhp的eap的定量)
图6是绘制了添加0μm~1400μm的eap时的、从测定开始100秒后的电流值的图。图7是绘制了添加0μm~170μm的eap时的、从测定开始100秒后的电流值的图。两个结果都表明,随着eap浓度的升高,电流值也随之升高。作为对照,在使用5μl超纯水(离子交换水)代替eapox溶液时进行同样的实验,但添加eap时没有观察到响应电流的增加。因此,也可以通过电化学测定来对eap进行定量。可以设想,如果增加使用的lrhp的量,则更容易定量低浓度的eap,如果减少使用的lrhp的量,则更容易定量高浓度的eap。认为即使在使用agpeaox、lcaox、sraox3925、sraox3926或sreaox代替lrhp的情况下,也可以测定eap浓度(μm)。
如上所述,通过本发明的在含有eap的样品中添加氧化酶的eap的定量方法、在含有eap的样品中添加的eap的定量用的氧化酶、含有在含有eap的样品中添加的氧化酶的eap的定量用组成物、含有在含有eap的样品中添加的氧化酶的eap的定量用试剂盒以及含有在含有eap的样品中添加的氧化酶的eap的定量用传感器,可以提供对作为抑郁症的生物标志物的eap浓度进行定量的新的定量方法、定量用的酶、定量用组成物、定量用试剂盒以及定量用传感器。
(附图标记的说明)
1:工作电极;3:对电极;5:参考电极;7:布线部;9:端子;
10:传感器芯片;11:基材;13:间隔件;15:盖;
19:反应层;30:测定部;31:开关;33:显示器;100:传感器;
110:控制部;120:显示部;130:输入部;140:存储部;
150:通信部;160:电源;190:导线。
序列表
<110>龟甲万株式会社(kikkomancorporation)
<120>磷酸乙醇胺的定量方法、定量用的氧化还原酶、定量用组合物、定量用试剂盒和定量用传感器
<130>km18i001
<160>39
<170>patentin3.5版
<210>1
<211>463
<212>prt
<213>脱氮副球菌(paracoccusdenitrificans)
<400>1
metglnalaileglulysargalaglyalaleuprotyraspproleu
151015
tyraspprovalthralaargglyleuglyproargserasptyrala
202530
prothrtyrtrpileglythralaglyalaproproproaspaspgly
354045
provalthrglyaspmetaspalaaspvalvalvalileglysergly
505560
tyrthrglyleusercysalaleuhisleualalysmethisglyile
65707580
lysalavalvalleuglualaasnglyvalalatyrglycysserthr
859095
argasnglyglyglnalaglnvalserserglyargleulysargser
100105110
glntrpilegluargtrpglyleuaspvalalaargargleuhisala
115120125
gluvalcysgluglypheaspleupheargglyleuileargasphis
130135140
alaileaspcysaspproglnaspglyglyhistyrtyrilealahis
145150155160
lysalaseralametproalaleuglulysgluthralaleuleuarg
165170175
aspthrpheglytyraspalaargmetileserargaspgluleuhis
180185190
gluthrvalalaargaspglnglualahisglyalamettrpgluala
195200205
aspglyvalglyilehisalaalalysleualapheglytyrleuarg
210215220
alaalaarggluleuglyalaargvalhisvalaspserprovalgln
225230235240
glytrpglutyrargasnglyvalhishisleuargthrproglygly
245250255
thrvalargalaargargvalalavalalathralaalatyralapro
260265270
argserleuhisproargleuargaspargleumetproilemetser
275280285
asnserilevalthrargvalleuthrproalagluleuglualaval
290295300
glyilearglysleuserproleuthraspthrargthrleuarghis
305310315320
tyrtyrargleuleuproaspasnargleuglnileglyserargala
325330335
alailethrglyargaspalaalaasnproalahisleuaspalaleu
340345350
arggluglymetalaarglyspheproalaleuargglyilealaleu
355360365
asptyrsertrptrpglytrpvalaspvalserhisaspmetmetpro
370375380
argilethrglyleuproaspleuproglyalaphetyralaleugly
385390395400
tyrglyglyasnglyvalmettyrseralametalaglyargargmet
405410415
alaglnleuvalalaglyglualavalproaspleuproilepheasn
420425430
asngluleuprohisgluglytrpargthrpropheargargleugly
435440445
glntrpglyleutyrlysphetyrhistyrargaspgluargarg
450455460
<210>2
<211>1392
<212>dna
<213>脱氮副球菌(paracoccusdenitrificans)
<400>2
atgcaagctattgaaaaacgtgcaggcgccttgccttacgaccctctttatgatcctgtt60
accgcacgtggactgggtccgcgctccgattatgctccgacctactggatcggcaccgca120
ggagcacctcccccggatgatggcccggttaccggtgatatggacgcagatgtagttgtt180
attgggagcggttacaccgggctgtcatgcgctttacatctggcaaaaatgcatgggatc240
aaggcagtggtcttggaggccaatggtgttgcctatggctgctctactcggaatggcggc300
caggcgcaggtttcatctggccgccttaagagatctcaatggattgaacgctggggtctt360
gatgtcgcccgtcgtcttcacgccgaagtttgtgagggtttcgatctgttccgtggcctt420
attcgtgatcacgctatcgattgtgatccgcaagacggtgggcattattatatcgcacat480
aaagcaagcgctatgccggccttagagaaggagacagccctgctgcgtgatacttttgga540
tatgacgcccgtatgattagccgtgacgagttgcatgaaaccgttgcgcgtgaccaggaa600
gcacatggtgccatgtgggaagccgatggtgtgggtatacacgcggcaaaattagccttc660
ggttatttacgtgccgcaagagagctgggggcccgtgttcacgtagacagcccggtgcaa720
ggctgggaataccgtaatggtgtgcatcacctgcgcaccccgggcggaactgttcgcgcg780
cgtcgcgtggcagtggccacagcggcctacgcgccgcgttcattacatcctcgccttcgt840
gaccgtctgatgcctatcatgagtaacagcatcgtgacacgcgtgcttactccggcggaa900
cttgaagcagtcggtattagaaaactgagtccgctgacggatacacggacactgcgccat960
tattatcggttgttaccagacaatcgcctgcagattggtagtagagcagcgataacgggt1020
cgggacgcggctaatcctgcgcatctggacgcgctgcgcgagggtatggcgcgcaaattt1080
cccgctctgagaggcattgctctggattactcctggtggggttgggtagatgtctcgcac1140
gatatgatgccacgcataacggggttacccgatctgcccggagctttttacgctttaggg1200
tatggcggcaacggagtaatgtattcggctatggctggccgccggatggcgcagttggta1260
gcgggggaagctgtcccagatttaccaatttttaacaacgaactgccacacgaaggatgg1320
cggacgccatttcgccggctgggacagtggggattgtataaattttatcattatagagat1380
gaaagacggtaa1392
<210>3
<211>139
<212>prt
<213>脱氮副球菌(paracoccusdenitrificans)
<400>3
metproargthrleuthralaalaaspleuglnalathrpheaspala
151015
pheasnarghisaspileaspglyvalmetthrhisphealaaspasp
202530
cysvalphetyrthrvalserglygluhisglutyrglyasnargile
354045
gluglylysalaalailealaargalapheglualavaltrpthrthr
505560
metproaspvalglntrpalagluhisthrhispheleusergluasp
65707580
glythrargglyvalserglntrpthrpheargalathrasnproasp
859095
glyserargthrgluvalglnglyvalaspleupheargilealaasp
100105110
glyargilevalglulysglnalailearglysglnargproalaile
115120125
proalaalathralaproalaleualaglylys
130135
<210>4
<211>420
<212>dna
<213>脱氮副球菌(paracoccusdenitrificans)
<400>4
atgccacgtactttgaccgctgccgacttacaggccacattcgacgcatttaacagacac60
gatattgacggtgtcatgacgcactttgccgatgattgtgtattctataccgtttccggc120
gaacatgagtacggcaatcgcattgaaggcaaagcggcgatagcgcgtgcattcgaagcc180
gtttggaccaccatgcccgatgtgcaatgggcagaacatacacattttctgagcgaagac240
ggaactcgcggtgttagtcagtggacgtttcgcgcgactaatccggatggatcacgtacg300
gaggtgcagggggtggatctgtttcgtatcgcggatgggcggattgtcgagaaacaagca360
attcggaagcagagacctgcaatcccggctgctacagcaccggctcttgccggtaaataa420
<210>5
<211>30
<212>dna
<213>人工序列
<220>
<223>人工合成的引物序列
<400>5
agacggtaaggatccatgccacgtactttg30
<210>6
<211>30
<212>dna
<213>人工序列
<220>
<223>人工合成的引物序列
<400>6
acggagctcgaattcttatttaccggcaag30
<210>7
<211>30
<212>dna
<213>人工序列
<220>
<223>人工合成的引物序列
<400>7
ggatccttaccgtctttcatctctataatg30
<210>8
<211>30
<212>dna
<213>人工序列
<220>
<223>人工合成的引物序列
<400>8
gaattcgagctccgtcgacaagcttgcggc30
<210>9
<211>638
<212>prt
<213>球形节杆菌(arthrobacterglobiformis)
<400>9
metthrproserthrileglnthralaserpropheargleualaser
151015
alaglygluilesergluvalglnglyileleuargthralaglyleu
202530
leuglyproglulysargilealatyrleuglyvalleuaspproala
354045
argglyalaglyserglualagluaspargargpheargvalpheile
505560
hisaspvalserglyalaargproglngluvalthrvalservalthr
65707580
asnglythrvalileseralavalgluleuaspthralaalathrgly
859095
gluleuprovalleugluglugluphegluvalvalgluglnleuleu
100105110
alathraspgluargtrpleulysalaleualaalaargasnleuasp
115120125
valserlysvalargvalalaproleuseralaglyvalpheglutyr
130135140
alaglugluargglyargargileleuargglyleualaphevalgln
145150155160
asppheprogluaspseralatrpalahisprovalaspglyleuval
165170175
alatyrvalaspvalvalserlysgluvalthrargvalileaspthr
180185190
glyvalpheprovalproalagluhisglyasntyrthraspproglu
195200205
leuthrglyproleuargthrthrglnlysproileserilethrgln
210215220
progluglyproserphethrvalthrglyglyasnhisileglutrp
225230235240
glulystrpserleuaspvalglypheaspvalarggluglyvalval
245250255
leuhisasnilealapheargaspglyaspargleuargproileile
260265270
asnargalaserilealaglumetvalvalprotyrglyaspproser
275280285
proileargsertrpglnasntyrpheaspthrglyglutyrleuval
290295300
glyglntyralaasnserleugluleuglycysaspcysleuglyasp
305310315320
ilethrtyrleuserprovalileseraspalapheglyasnproarg
325330335
gluileargasnglyilecysmethisglugluasptrpglyileleu
340345350
alalyshisseraspleutrpserglyileasntyrthrargargasn
355360365
argargmetvalileserphephethrthrileglyasntyrasptyr
370375380
glyphetyrtrptyrleutyrleuaspglythrilegluphegluala
385390395400
lysalathrglyvalvalphethrseralapheprogluglyglyser
405410415
aspasnileserglnleualaproglyleuglyalaprophehisgln
420425430
hisilepheseralaargleuaspmetalaileaspglyphethrasn
435440445
argvalgluglugluaspvalvalargglnthrmetglyproglyasn
450455460
gluargglyasnalapheserarglysargthrvalleuthrargglu
465470475480
serglualavalargglualaaspalaargthrglyargthrtrpile
485490495
ileserasnprogluserlysasnargleuasngluprovalglytyr
500505510
lysleuhisalahisasnglnprothrleuleualaaspproglyser
515520525
serilealaargargalaalaphealathrlysaspleutrpvalthr
530535540
argtyralaaspaspgluargtyrprothrglyaspphevalasngln
545550555560
hisserglyglyalaglyleuprosertyrilealaglnaspargasp
565570575
ileaspglyglnaspilevalvaltrphisthrpheglyleuthrhis
580585590
pheproargvalgluasptrpproilemetprovalaspthrvalgly
595600605
phelysleuargprogluglyphepheaspargserprovalleuasp
610615620
valproalaasnproserglnserglyserhiscyshisgly
625630635
<210>10
<211>1914
<212>dna
<213>球形节杆菌(arthrobacterglobiformis)
<400>10
atgacaccgtcaacgattcaaactgccagtccatttcgtttagcatcggctggcgagatc60
agcgaagtacaaggaatcctgcgcactgcaggcttattgggtcccgagaaacgcattgct120
tacttgggggtccttgatcctgctcgtggggccggcagcgaagcggaagatcgccgtttc180
cgcgtttttatccacgatgtctcgggagcgcgccctcaagaagtgactgtgtcggtaacg240
aacggtacggtcatttccgccgtcgaattggatacagccgccactggtgaattaccggta300
ttggaagaagagttcgaggttgtagaacaactgctggcaacggatgagcgttggttaaag360
gcacttgcggctcgtaaccttgatgttagtaaagtacgcgtagctcctctttctgctggt420
gtctttgagtacgccgaagaacgcggccgtcgcatccttcgtggcttggcttttgtacag480
gacttcccagaagattcggcttgggcacatcccgtggacgggcttgtggcctacgttgac540
gtggtatcaaaggaagtcacccgtgtaattgatactggggttttccccgtaccggcggaa600
catgggaactacacagatcccgagttgacaggtcctcttcgcacgacacagaagcctatc660
agcattacacagcccgaaggtccttcatttacggtgacgggcggcaaccatatcgagtgg720
gagaaatggtccctggacgtcggattcgacgttcgtgagggtgtggtgcttcataacatt780
gcttttcgtgacggagatcgccttcgcccgattatcaaccgcgcgtccattgctgaaatg840
gtcgtcccttacggggatccttctcccattcgctcgtggcagaactatttcgatacgggg900
gagtacttagtcggccagtacgccaactcattggaacttggctgtgattgtcttggagat960
atcacgtacctgtcccctgttatctcagatgcgttcggaaaccctcgtgaaatccgcaac1020
gggatctgtatgcatgaagaagattgggggattcttgcaaaacattcggatttatggtcc1080
ggcatcaactatacgcgtcgcaatcgtcgtatggtgatttcatttttcacaaccatcggg1140
aattatgattatgggttttattggtatctgtacctggacggaacgatcgagtttgaagca1200
aaggcaactggagtggtattcacgtctgcgtttcccgagggcgggtccgataatattagc1260
cagcttgccccaggccttggcgctccgtttcatcaacacatcttctcggcccgtttagac1320
atggctatcgatggattcacgaatcgtgttgaggaagaggatgttgtgcgccaaacgatg1380
ggacccggcaacgaacgtgggaatgcgttcagtcgtaaacgtacggttctgacgcgcgaa1440
tcagaagcggtacgtgaggcagacgcacgtacaggtcgcacctggatcatctcaaatccc1500
gagtctaagaatcgtcttaatgaacccgtagggtataagttacacgcccacaaccaaccg1560
accttactggcggaccctggaagctcaatcgctcgtcgcgcggccttcgccaccaaagat1620
ctgtgggtaacacgttacgccgacgatgagcgctatcccacaggagacttcgtaaaccag1680
cattccgggggcgcgggtttgccatcctatattgcccaggaccgcgacatcgatggccaa1740
gacatcgtagtatggcatacatttggacttacccactttccacgtgtggaggactggcca1800
attatgccggtggacactgtaggtttcaagcttcgcccagaaggattctttgaccgcagt1860
cctgtgctggatgtaccagccaatccctcccaatctggttcacactgccatgga1914
<210>11
<211>15
<212>dna
<213>人工序列
<220>
<223>pkk223-3
<400>11
caggaaacagaattc15
<210>12
<211>975
<212>dna
<213>球形节杆菌(arthrobacterglobiformis)
<400>12
atgacaccgtcaacgattcaaactgccagtccatttcgtttagcatcggctggcgagatc60
agcgaagtacaaggaatcctgcgcactgcaggcttattgggtcccgagaaacgcattgct120
tacttgggggtccttgatcctgctcgtggggccggcagcgaagcggaagatcgccgtttc180
cgcgtttttatccacgatgtctcgggagcgcgccctcaagaagtgactgtgtcggtaacg240
aacggtacggtcatttccgccgtcgaattggatacagccgccactggtgaattaccggta300
ttggaagaagagttcgaggttgtagaacaactgctggcaacggatgagcgttggttaaag360
gcacttgcggctcgtaaccttgatgttagtaaagtacgcgtagctcctctttctgctggt420
gtctttgagtacgccgaagaacgcggccgtcgcatccttcgtggcttggcttttgtacag480
gacttcccagaagattcggcttgggcacatcccgtggacgggcttgtggcctacgttgac540
gtggtatcaaaggaagtcacccgtgtaattgatactggggttttccccgtaccggcggaa600
catgggaactacacagatcccgagttgacaggtcctcttcgcacgacacagaagcctatc660
agcattacacagcccgaaggtccttcatttacggtgacgggcggcaaccatatcgagtgg720
gagaaatggtccctggacgtcggattcgacgttcgtgagggtgtggtgcttcataacatt780
gcttttcgtgacggagatcgccttcgcccgattatcaaccgcgcgtccattgctgaaatg840
gtcgtcccttacggggatccttctcccattcgctcgtggcagaactatttcgatacgggg900
gagtacttagtcggccagtacgccaactcattggaacttggctgtgattgtcttggagat960
atcacgtacctgtcc975
<210>13
<211>957
<212>dna
<213>球形节杆菌(arthrobacterglobiformis)
<400>13
atcacgtacctgtcccctgttatctcagatgcgttcggaaaccctcgtgaaatccgcaac60
gggatctgtatgcatgaagaagattgggggattcttgcaaaacattcggatttatggtcc120
ggcatcaactatacgcgtcgcaatcgtcgtatggtgatttcatttttcacaaccatcggg180
aattatgattatgggttttattggtatctgtacctggacggaacgatcgagtttgaagca240
aaggcaactggagtggtattcacgtctgcgtttcccgagggcgggtccgataatattagc300
cagcttgccccaggccttggcgctccgtttcatcaacacatcttctcggcccgtttagac360
atggctatcgatggattcacgaatcgtgttgaggaagaggatgttgtgcgccaaacgatg420
ggacccggcaacgaacgtgggaatgcgttcagtcgtaaacgtacggttctgacgcgcgaa480
tcagaagcggtacgtgaggcagacgcacgtacaggtcgcacctggatcatctcaaatccc540
gagtctaagaatcgtcttaatgaacccgtagggtataagttacacgcccacaaccaaccg600
accttactggcggaccctggaagctcaatcgctcgtcgcgcggccttcgccaccaaagat660
ctgtgggtaacacgttacgccgacgatgagcgctatcccacaggagacttcgtaaaccag720
cattccgggggcgcgggtttgccatcctatattgcccaggaccgcgacatcgatggccaa780
gacatcgtagtatggcatacatttggacttacccactttccacgtgtggaggactggcca840
attatgccggtggacactgtaggtttcaagcttcgcccagaaggattctttgaccgcagt900
cctgtgctggatgtaccagccaatccctcccaatctggttcacactgccatggataa957
<210>14
<211>15
<212>dna
<213>人工序列
<220>
<223>pkk223-3
<400>14
aagcttggctgtttt15
<210>15
<211>676
<212>prt
<213>伞状毛霉菌(lichtheimiacorymbifera)
<400>15
metserserserserseralathrprolysprohisproleuasppro
151015
leuseralaaspgluileargargalaalaalaileileargglulys
202530
argglyglnaspthrsertyrvalpheasnserleuthrleulysglu
354045
proalalysglnglnmetmetleutyrleuglytrpvalasnserser
505560
serglnprolysprovalthrileasparggluvalphealavalleu
65707580
ileaspargproserglyleuvalhisglumetthrvalasnleuasp
859095
lysseralavalthrsertrpasnlysvalgluglyargglnprothr
100105110
ileasnilepheglumetleuglualagluarggluileleulysasp
115120125
gluargvallysaspglncysargglnleuglyilethraspmetser
130135140
metvaltyralaaspprotrpglyvalglytyrhisgluilelysgly
145150155160
lysargleuvalglnalaleuleutyralaargthrserproaspasp
165170175
asnglntyralahisproleuasppheasnproleutyraspleuasn
180185190
alalyslysvalleuaspilevalvalserlysargargasnserser
195200205
phegluargprovalileprometalaasnhishispheleuproglu
210215220
hisleuglygluaspargleuarglysaspilelysproilegluile
225230235240
thrglnproglnglyvalserpheserileargaspglyhisglnleu
245250255
histrpglnlystrpaspmethisleuserpheasntyrargglugly
260265270
leuvalileasnasnleusertyrargaspmetaspserthrvalarg
275280285
proileiletyrargmetserleualaglumetvalvalprotyrala
290295300
asnprotyrlysprotyrasnhislysmetalapheaspvalglyglu
305310315320
tyrglyleuglyasnleuthrasnserleugluleuglycysaspcys
325330335
valglylysileglytyrleuaspalavalleuseraspleuasngly
340345350
aspprotrphisileproasnalailecysilehisglugluaspthr
355360365
glyleuleuphelyshisserasptyrargthrglylysalahisser
370375380
alaargserargargleuvalileserhisilevalthralaalaasn
385390395400
tyrasptyrglyleutyrtyrtyrphetyrglnaspglythrilegln
405410415
tyrgluvallysalathrglygluleuasnthrglnvalleualaglu
420425430
aspgluaspalaalaprotyrglythrilevalalaproglnvalasp
435440445
alaglnhishisglnhisleuphesermetargileaspprometleu
450455460
aspglyproasnasnservalalagluvalaspvalvalalaserasp
465470475480
leuprovalglyhisprohisasnservalglyasnserphetyrpro
485490495
valthrlysvalpheasnthrthraspglualalysthrmetalaser
500505510
valgluarghisargthrtrplysileileasngluserlysilehis
515520525
protyralalysglnprovalglyphelysmetmetalahisprothr
530535540
proproleuleuprolysproglyserilevaltyrgluargalamet
545550555560
phealaserlysthrleutrpvalthrprohisasnasplysglnleu
565570575
tyrproglyglyphetyrcystyrglnsergluprosergluasnleu
580585590
glyleuproglntrpthrlysgluthrglnasnvalargaspthrasp
595600605
ilevalcystrpleuasnpheglyilethrhisileproargvalglu
610615620
asppheproilemetproilegluthrcysglyilemetleulyspro
625630635640
alaasnphepheleucysasnproglyileaspileproproserthr
645650655
argglnserthrlysseralatyralaasnaspalathrcyscysarg
660665670
lysasnasnleu
675
<210>16
<211>2031
<212>dna
<213>伞状毛霉菌(lichtheimiacorymbifera)
<400>16
atgtcctcttcctcaagcgccacgccaaagcctcatccattagaccctttgtccgcagat60
gagatccgccgcgcagcagccattatccgcgaaaaacgtgggcaggatacttcttatgtg120
ttcaacagtttaactttgaaggaacctgcgaaacagcaaatgatgttgtacttgggttgg180
gtcaacagttcatcgcagcctaagcccgttacaattgaccgcgaagttttcgcagttctt240
attgatcgtccaagcggtctggttcacgagatgacagttaacttagataagtcggcggtt300
acctcctggaataaagtggaaggacgccagcccaccattaacatctttgaaatgttagag360
gcggaacgcgaaatcctgaaggatgagcgtgtcaaggaccaatgtcgccagctgggcatc420
accgacatgtcaatggtctatgctgacccgtggggggtaggctatcatgagatcaaaggt480
aaacgtcttgttcaagcgttgttatacgcacgcacgtcccctgacgacaatcaatacgcg540
cacccccttgacttcaatcccttatacgatctgaacgctaagaaggtgcttgacatcgtc600
gtgtccaaacgtcgtaattcctcatttgagcgccctgtgatccccatggcgaatcatcac660
tttctgcccgaacaccttggtgaagaccgtcttcgtaaggatattaagccaatcgaaatt720
acacaaccgcagggcgtatcgttttccattcgtgacggtcatcagcttcattggcagaag780
tgggacatgcatttgtcattcaactaccgcgaaggccttgttatcaataatcttagctac840
cgtgatatggactccaccgtccgtccaattatttaccgtatgagccttgctgagatggtc900
gtgccgtatgctaacccctacaaaccgtacaatcataagatggcgttcgacgtaggagag960
tacggcttagggaatttaacgaatagccttgagttaggttgtgactgtgttggtaagatt1020
ggttatttagatgcggtgttgtcggatttaaacggggatccctggcatatccctaatgca1080
atttgcattcatgaggaggatacggggttgttatttaaacactcggactaccgcacaggg1140
aaggcgcactctgctcgtagccgtcgtttggtaatttcacatatcgtgacggcggccaac1200
tatgattatggactgtactactacttctaccaggatggtaccatccagtacgaagttaag1260
gcaacgggtgaattgaacacccaggtactggccgaggatgaagacgccgcaccctacggc1320
accattgtggctcctcaagtcgatgcccagcatcatcaacatttgttctccatgcgtatc1380
gacccaatgttagacggaccgaacaactcggtcgcagaggttgacgtggttgcctccgat1440
cttcccgtcggacaccctcacaattctgtgggaaacagcttttatccagtaactaaagtc1500
ttcaacactaccgacgaggccaaaacaatggcgtcagttgagcgtcaccgtacatggaaa1560
atcattaacgaaagtaaaatccatccctatgccaagcagccggttggttttaagatgatg1620
gctcatcccaccccgccgcttctgcctaagccagggagcatcgtatatgaacgcgcaatg1680
tttgctagtaagacgttatgggtgacgcctcataacgacaagcagttatatcctggcggt1740
ttttattgctatcagtcagagccgtcagaaaaccttgggcttccacaatggacgaaggag1800
acacaaaatgtgcgcgataccgatatcgtctgttggctgaactttggcatcactcatatc1860
ccacgcgtagaagactttcctatcatgcctattgaaacttgtggaattatgcttaagccg1920
gcgaacttcttcttatgtaatcccgggatcgatattccgccctccacacgtcaatcgacc1980
aagagcgcctatgcgaatgatgcaacttgttgtcgtaaaaacaatttataa2031
<210>17
<211>681
<212>dna
<213>伞状毛霉菌(lichtheimiacorymbifera)
<400>17
atgtcctcttcctcaagcgccacgccaaagcctcatccattagaccctttgtccgcagat60
gagatccgccgcgcagcagccattatccgcgaaaaacgtgggcaggatacttcttatgtg120
ttcaacagtttaactttgaaggaacctgcgaaacagcaaatgatgttgtacttgggttgg180
gtcaacagttcatcgcagcctaagcccgttacaattgaccgcgaagttttcgcagttctt240
attgatcgtccaagcggtctggttcacgagatgacagttaacttagataagtcggcggtt300
acctcctggaataaagtggaaggacgccagcccaccattaacatctttgaaatgttagag360
gcggaacgcgaaatcctgaaggatgagcgtgtcaaggaccaatgtcgccagctgggcatc420
accgacatgtcaatggtctatgctgacccgtggggggtaggctatcatgagatcaaaggt480
aaacgtcttgttcaagcgttgttatacgcacgcacgtcccctgacgacaatcaatacgcg540
cacccccttgacttcaatcccttatacgatctgaacgctaagaaggtgcttgacatcgtc600
gtgtccaaacgtcgtaattcctcatttgagcgccctgtgatccccatggcgaatcatcac660
tttctgcccgaacaccttggt681
<210>18
<211>696
<212>dna
<213>伞状毛霉菌(lichtheimiacorymbifera)
<400>18
cccgaacaccttggtgaagaccgtcttcgtaaggatattaagccaatcgaaattacacaa60
ccgcagggcgtatcgttttccattcgtgacggtcatcagcttcattggcagaagtgggac120
atgcatttgtcattcaactaccgcgaaggccttgttatcaataatcttagctaccgtgat180
atggactccaccgtccgtccaattatttaccgtatgagccttgctgagatggtcgtgccg240
tatgctaacccctacaaaccgtacaatcataagatggcgttcgacgtaggagagtacggc300
ttagggaatttaacgaatagccttgagttaggttgtgactgtgttggtaagattggttat360
ttagatgcggtgttgtcggatttaaacggggatccctggcatatccctaatgcaatttgc420
attcatgaggaggatacggggttgttatttaaacactcggactaccgcacagggaaggcg480
cactctgctcgtagccgtcgtttggtaatttcacatatcgtgacggcggccaactatgat540
tatggactgtactactacttctaccaggatggtaccatccagtacgaagttaaggcaacg600
ggtgaattgaacacccaggtactggccgaggatgaagacgccgcaccctacggcaccatt660
gtggctcctcaagtcgatgcccagcatcatcaacat696
<210>19
<211>684
<212>dna
<213>伞状毛霉菌(lichtheimiacorymbifera)
<400>19
cagcatcatcaacatttgttctccatgcgtatcgacccaatgttagacggaccgaacaac60
tcggtcgcagaggttgacgtggttgcctccgatcttcccgtcggacaccctcacaattct120
gtgggaaacagcttttatccagtaactaaagtcttcaacactaccgacgaggccaaaaca180
atggcgtcagttgagcgtcaccgtacatggaaaatcattaacgaaagtaaaatccatccc240
tatgccaagcagccggttggttttaagatgatggctcatcccaccccgccgcttctgcct300
aagccagggagcatcgtatatgaacgcgcaatgtttgctagtaagacgttatgggtgacg360
cctcataacgacaagcagttatatcctggcggtttttattgctatcagtcagagccgtca420
gaaaaccttgggcttccacaatggacgaaggagacacaaaatgtgcgcgataccgatatc480
gtctgttggctgaactttggcatcactcatatcccacgcgtagaagactttcctatcatg540
cctattgaaacttgtggaattatgcttaagccggcgaacttcttcttatgtaatcccggg600
atcgatattccgccctccacacgtcaatcgaccaagagcgcctatgcgaatgatgcaact660
tgttgtcgtaaaaacaatttataa684
<210>20
<211>673
<212>prt
<213>总状横梗霉(lichtheimiaramosa)
<400>20
metserserserproilelysprotyrproleuaspproleuthrala
151015
aspgluileargargalaalaalaleuilearggluthrargglygln
202530
aspthrthrtyrvalpheasnserleuthrleulysgluproserlys
354045
glnglnmetmetsertyrleuglytrpthrasnserproserhispro
505560
lysprovalthrileasparggluvalphealavalleuileasparg
65707580
proserglyleuvalhisglumetthrvalasnleuasplysasngln
859095
valthrargtrpasnlysvalgluglyargglnprothrvalasnval
100105110
pheglumetleuglualagluarggluileleulysaspgluargval
115120125
lysgluglncysargglnleuglyileasnaspmetsermetvalphe
130135140
alaaspprotrpglyvalglytyrhisgluilelysglylysargleu
145150155160
valglnalaleumettyralaargthrserproaspaspasnglntyr
165170175
alahisproleuasppheasnproleutyraspvalasnalalyslys
180185190
valileaspileilevalserlysargargasnserserpheasparg
195200205
provalileprometalaasnhishispheleuprogluhisleugly
210215220
glnaspargleuarglysaspilelysproilegluilethrglnpro
225230235240
glnglyvalserpheserileargaspglyhisglnleuhistrpgln
245250255
lystrpaspmethisleuserpheasntyrarggluglyleuvalile
260265270
asnasnleusertyrargaspmetaspglythrvalargproileile
275280285
tyrargmetserleuserglumetvalvalprotyralaasnprotyr
290295300
lysprotyrasnhislysmetalapheaspvalglyglutyrglyleu
305310315320
glyasnleuthrasnserleugluleuglycysaspcysvalglylys
325330335
ilecystyrleuaspalathrleuseraspleuasnglyaspprotrp
340345350
glnileproasnalailecysilehisglugluaspthrglyleuleu
355360365
phelyshisthrasptyrargthrglylysalahisseralaargser
370375380
argargleuvalileserhisilevalthralaalaasntyrasptyr
385390395400
glyleutyrtyrtyrphetyrglnaspglythrileglntyrgluval
405410415
lysalathrglygluleuasnthrglnvalleualaalaaspgluasp
420425430
alaalaprotyrglythrilevalalaproglnvalaspalaglnhis
435440445
hisglnhisleuphesermetargileaspprometvalaspglypro
450455460
asnasnservalalagluvalaspvalvalalaseraspleuproval
465470475480
glyhisprohisasnserileglyasnserphetyrprovalthrlys
485490495
valpheasnthrthraspglualalysthrmetalaserilegluarg
500505510
hisargsertrplysileileasngluasnlysilehisprotyrala
515520525
lysglnprovalglyphelysmetmetalahisprothrproproleu
530535540
leuprolysproglyserilevaltyrgluargalametphealaser
545550555560
lysthrleutrpvalthrprotyrasnglugluglnargtyrprogly
565570575
glyphetyrcystyrglnsergluprosergluasnleuglyleupro
580585590
glntrpthrlysgluthrglnasnvalargaspthraspilevalcys
595600605
trpleuasnpheglyilethrhisileproargvalgluaspphepro
610615620
ilemetproilegluthrcysglyvalmetleulysprovalasnphe
625630635640
pheleuglyasnproglyileaspileproproserthrargglnser
645650655
thrlysseralatyralathrglualathrcyscysarglysasnasn
660665670
leu
<210>21
<211>2022
<212>dna
<213>总状横梗霉(lichtheimiaramosa)
<400>21
atgtcgtcctcaccgatcaagccgtatcctttagatccattaacagccgatgagattcgc60
cgcgcagcagcccttatccgtgaaacgcgcgggcaggacaccacctatgtgttcaactct120
ctgacactgaaggaaccttcgaagcagcaaatgatgtcttaccttgggtggactaactcg180
cctagtcaccccaagccggtcactattgaccgcgaagtctttgcggtgttaatcgaccgt240
ccatccgggttggtccatgaaatgaccgtgaacttggataaaaaccaggtgacccgctgg300
aataaggttgaaggtcgccaacctacggtaaatgtattcgagatgttggaggcggaacgc360
gagattttgaaagacgagcgtgttaaagagcaatgccgccaattggggattaatgatatg420
agtatggtctttgccgatccctggggtgtaggatatcacgaaattaaaggcaaacgtttg480
gttcaggccttgatgtatgcacgtacctctccggatgacaaccaatacgctcatccactg540
gactttaaccctctgtacgatgtgaacgccaagaaagtaatcgacattatcgtgtcaaaa600
cgtcgcaacagtagtttcgatcgtccagtcattcccatggcaaatcatcatttcttgccg660
gagcatttagggcaagatcgccttcgcaaagacatcaagcctattgagatcactcaacct720
cagggggtttctttcagcatccgcgatggacaccaattacattggcagaagtgggacatg780
catttgtcatttaattaccgtgaaggacttgtgatcaacaacttgagctatcgcgacatg840
gatggcactgtccgccccattatttatcgtatgtcgctttccgagatggttgtgccctat900
gcgaatccatataagccgtataaccacaaaatggccttcgacgttggtgagtacgggctt960
ggtaaccttacaaactctcttgaattagggtgtgactgcgtcggaaagatttgctattta1020
gacgcgacattgtcggatttgaatggagacccgtggcagattcccaatgcaatctgcatc1080
catgaagaagacacaggacttctgtttaagcacacggattaccgcacgggaaaagcacac1140
tcggcgcgttctcgtcgtttggttatctctcatatcgttacggcggcaaattacgactac1200
ggtttatattattacttctatcaagacggtaccattcagtatgaagttaaagccaccggc1260
gagttgaacactcaggttcttgcggcagacgaagacgctgctccttacggaactattgtg1320
gccccgcaggttgacgctcaacaccatcaacatttgttcagtatgcgtatcgacccaatg1380
gtcgatggcccgaataactcagtggcggaagtcgacgttgtggcatctgatcttccggtc1440
gggcacccgcacaattctattggtaacagtttttaccccgtaacgaaagtatttaacacg1500
actgatgaggcaaaaacgatggcctccattgaacgccaccgctcatggaaaattattaac1560
gagaacaaaattcatccctatgcaaagcagcccgtaggtttcaaaatgatggcccaccct1620
acgccgcctcttctgcccaaaccaggttctatcgtctacgaacgcgccatgtttgcgagc1680
aaaacattgtgggttactccatacaacgaggagcaacgttatcccggggggttttattgt1740
taccagtcggaaccttcggagaacttaggactgccacagtggacaaaggaaactcagaat1800
gttcgcgatacggatattgtatgttggttgaattttggcatcacccacatccctcgcgtt1860
gaggatttcccaatcatgcctatcgaaacgtgcggcgtaatgctgaagccggtaaatttt1920
tttttagggaatcccggcattgacatcccgccctccactcgccaaagcacgaagagtgcg1980
tacgcgacggaagcgacttgctgccgcaaaaataatttataa2022
<210>22
<211>678
<212>dna
<213>总状横梗霉(lichtheimiaramosa)
<400>22
atgtcgtcctcaccgatcaagccgtatcctttagatccattaacagccgatgagattcgc60
cgcgcagcagcccttatccgtgaaacgcgcgggcaggacaccacctatgtgttcaactct120
ctgacactgaaggaaccttcgaagcagcaaatgatgtcttaccttgggtggactaactcg180
cctagtcaccccaagccggtcactattgaccgcgaagtctttgcggtgttaatcgaccgt240
ccatccgggttggtccatgaaatgaccgtgaacttggataaaaaccaggtgacccgctgg300
aataaggttgaaggtcgccaacctacggtaaatgtattcgagatgttggaggcggaacgc360
gagattttgaaagacgagcgtgttaaagagcaatgccgccaattggggattaatgatatg420
agtatggtctttgccgatccctggggtgtaggatatcacgaaattaaaggcaaacgtttg480
gttcaggccttgatgtatgcacgtacctctccggatgacaaccaatacgctcatccactg540
gactttaaccctctgtacgatgtgaacgccaagaaagtaatcgacattatcgtgtcaaaa600
cgtcgcaacagtagtttcgatcgtccagtcattcccatggcaaatcatcatttcttgccg660
gagcatttagggcaagat678
<210>23
<211>693
<212>dna
<213>总状横梗霉(lichtheimiaramosa)
<400>23
catttagggcaagatcgccttcgcaaagacatcaagcctattgagatcactcaacctcag60
ggggtttctttcagcatccgcgatggacaccaattacattggcagaagtgggacatgcat120
ttgtcatttaattaccgtgaaggacttgtgatcaacaacttgagctatcgcgacatggat180
ggcactgtccgccccattatttatcgtatgtcgctttccgagatggttgtgccctatgcg240
aatccatataagccgtataaccacaaaatggccttcgacgttggtgagtacgggcttggt300
aaccttacaaactctcttgaattagggtgtgactgcgtcggaaagatttgctatttagac360
gcgacattgtcggatttgaatggagacccgtggcagattcccaatgcaatctgcatccat420
gaagaagacacaggacttctgtttaagcacacggattaccgcacgggaaaagcacactcg480
gcgcgttctcgtcgtttggttatctctcatatcgttacggcggcaaattacgactacggt540
ttatattattacttctatcaagacggtaccattcagtatgaagttaaagccaccggcgag600
ttgaacactcaggttcttgcggcagacgaagacgctgctccttacggaactattgtggcc660
ccgcaggttgacgctcaacaccatcaacatttg693
<210>24
<211>681
<212>dna
<213>总状横梗霉(lichtheimiaramosa)
<400>24
caccatcaacatttgttcagtatgcgtatcgacccaatggtcgatggcccgaataactca60
gtggcggaagtcgacgttgtggcatctgatcttccggtcgggcacccgcacaattctatt120
ggtaacagtttttaccccgtaacgaaagtatttaacacgactgatgaggcaaaaacgatg180
gcctccattgaacgccaccgctcatggaaaattattaacgagaacaaaattcatccctat240
gcaaagcagcccgtaggtttcaaaatgatggcccaccctacgccgcctcttctgcccaaa300
ccaggttctatcgtctacgaacgcgccatgtttgcgagcaaaacattgtgggttactcca360
tacaacgaggagcaacgttatcccggggggttttattgttaccagtcggaaccttcggag420
aacttaggactgccacagtggacaaaggaaactcagaatgttcgcgatacggatattgta480
tgttggttgaattttggcatcacccacatccctcgcgttgaggatttcccaatcatgcct540
atcgaaacgtgcggcgtaatgctgaagccggtaaatttttttttagggaatcccggcatt600
gacatcccgccctccactcgccaaagcacgaagagtgcgtacgcgacggaagcgacttgc660
tgccgcaaaaataatttataa681
<210>25
<211>668
<212>prt
<213>总状共头霉(syncephalastrumracemosum)
<400>25
metthrilevalserproserhisproleuaspproleuthrproser
151015
gluilearghisvalalagluilevalargalathrargserproasp
202530
asplysthrproargasptyrilepheserserilecysleulysglu
354045
prohislysasplysthrleualatyrleuglnservalsergluglu
505560
alaalaalaleumetprogluargglualaleuvalileleuileasp
65707580
argproserglyleuvalhisgluileleuvalserilethraspglu
859095
lysvallysseralalysthrleulysasnvalglnprothrglnhis
100105110
valleuglumetileglualaglulysileilethrlysaspproser
115120125
valilegluglucysarglysleuglyilethraspmetlysasnval
130135140
tyralaaspprotrpthrvalglytyrhisalaglnphelysserser
145150155160
lysargleumetglnalaleumettyrmetargthrserproaspasp
165170175
asnglntyralahisproleuaspphevalproiletyraspvalasn
180185190
alaglnlysvalvalgluileleuargglngluthrserglualaser
195200205
lystyraspargprothrvalproleugluasnhisglnpheleupro
210215220
gluhisileglyvalgluasnleuarglysaspilelysproileglu
225230235240
ilethrglnprogluglyvalserphethrvalargglyarggluile
245250255
glutrpglnasntrpsermethisvalglypheasntyrargglugly
260265270
valileileasnasnvalsertyrlysasplysglyasnvalargpro
275280285
leuphetyrargvalservalserglumetvalvalprotyralahis
290295300
prolysglupropheasnhislysmetalapheaspvalglyglutyr
305310315320
glyleuglyasnleuthrasnserleugluleuglycysaspcysleu
325330335
glyseriletyrtyrmetaspglyvalcysasnasnleuaspglyglu
340345350
protrpvalileproasnalailecysilehisglugluaspthrgly
355360365
leuleuphelyshisthrasptyrargthrasplysalahisserala
370375380
argserargargleuvalileserglnilevalthralaalaasntyr
385390395400
asptyrglyleutyrphetyrphetyrglnaspglythrpheglntyr
405410415
gluvallysalathrglygluleuasnthrglnvalphealagluasp
420425430
gluasnproalaprotyrglythralavalalaproglnvalvalgly
435440445
glnhishisglnhisleuphemetmetargileaspprometleuasp
450455460
glyargleuasnservalalaglnvalaspvalleuproserglutyr
465470475480
provalglyhisvalgluasnproileglyasnalapheserproile
485490495
thrthriletyrseraspthrthrglualaglnalahisglyasnleu
500505510
gluserserargthrtrplysileileasngluserlysleuhispro
515520525
tyrthrlysgluprovalglytyrlysleuvalserproasnthrpro
530535540
prometleuprolysproglyserleuvaltyrgluargalalysphe
545550555560
alathrlysthriletrpvalthrprotyraspproaspglniletyr
565570575
proalaglyphetyrcysserglnserproglyaspaspsermetgly
580585590
leuproalatrpthrlysgluproglnservalargglyargaspval
595600605
valvaltrpleuthrpheglyleuthrhisileproargvalgluasp
610615620
pheprovalmetprovalgluthrcysglytrpalaleulysalacys
625630635640
asnphepheleuglyasnproglyileaspileproalaalaglnlys
645650655
glythrserlysargvalasnglyalacyscysala
660665
<210>26
<211>2007
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>26
atgaccattgtctctccttcacaccccttagacccgcttacccctagtgaaattcgccac60
gtagccgaaatcgtacgcgccactcgctctcctgatgataagaccccgcgcgattatatt120
ttttccagcatttgcttaaaagaacctcataaggacaagacgttggcatacttgcaatcg180
gtgtccgaagaagctgccgctctgatgcctgaacgtgaagctctggtaattctgatcgat240
cgcccgtcgggccttgtacacgagattttagtttctatcacagatgagaaggtgaaatct300
gcaaagacccttaagaatgttcaacctactcagcatgtcttagagatgatcgaagcggaa360
aagatcatcacgaaggatccttcagttatcgaagaatgtcgcaagttaggcattacggat420
atgaagaatgtctatgctgatccgtggacggtgggttaccatgctcaatttaagtccagt480
aaacgtttaatgcaggcccttatgtacatgcgcacctctcccgacgacaaccagtatgcg540
catcctttagattttgtaccgatttatgatgttaacgcacaaaaggtcgtcgagattttg600
cgccaggaaacttcggaagcaagcaagtatgatcgtccgacagttccgttagaaaaccat660
cagtttttaccagagcacattggcgtggaaaacttacgcaaggacatcaagcccatcgaa720
attacccaaccggagggggtgtcttttacagtgcgcgggcgtgaaatcgaatggcaaaat780
tggagtatgcatgtcgggttcaactatcgtgaaggagttatcattaacaacgtgtcctat840
aaggacaagggcaatgtgcgtccgttattctaccgcgtgtcggtaagtgaaatggtcgta900
ccgtacgcacaccccaaggagccgttcaaccataagatggcctttgacgtcggggagtac960
ggtttggggaaccttactaacagccttgagctgggctgtgattgtttgggctcgatttat1020
tatatggatggtgtctgtaataatctggatggcgaaccctgggtcatccccaatgcaatt1080
tgcatccatgaagaggatacgggtttgttattcaaacacaccgattaccgcactgataaa1140
gctcacagcgcacgtagccgccgtttggttatctcgcaaattgtgaccgccgccaactat1200
gattatggcttgtatttctacttctaccaagacggtacgtttcagtacgaggtcaaagct1260
accggggagctgaacactcaagtttttgcggaagacgaaaacccagcaccttatggtacc1320
gcagtcgctccccaagtcgtaggccagcatcatcaacacttatttatgatgcgcattgac1380
ccaatgttagatggtcgtctgaactctgttgcccaggttgatgttttaccctccgagtac1440
ccagtcggtcacgtcgagaatccaatcggcaacgcctttagtcctattacgactatttac1500
agtgacacgactgaggcgcaggcacatgggaatttagagtcgtcgcgtacatggaagatc1560
attaacgaatctaaactgcatccatatacaaaagaaccagtaggttacaaacttgtgagt1620
cctaacactcccccgatgcttcccaaacctggatcccttgtctatgaacgcgcaaaattc1680
gctactaaaaccatctgggtaactccctatgaccccgatcagatttatcccgccggattc1740
tattgctcacaatcaccgggtgacgattcgatgggattgcctgcatggactaaagaacca1800
caaagcgtacgtggacgtgacgtagttgtgtggcttaccttcggacttacgcacatccca1860
cgcgtggaagacttccccgttatgcctgtagagacgtgcgggtgggcgctgaaagcgtgt1920
aattttttcttagggaatccagggatcgacattccggcagcacagaaggggacctccaag1980
cgcgtaaatggtgcatgttgtgcgtaa2007
<210>27
<211>675
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>27
atgaccattgtctctccttcacaccccttagacccgcttacccctagtgaaattcgccac60
gtagccgaaatcgtacgcgccactcgctctcctgatgataagaccccgcgcgattatatt120
ttttccagcatttgcttaaaagaacctcataaggacaagacgttggcatacttgcaatcg180
gtgtccgaagaagctgccgctctgatgcctgaacgtgaagctctggtaattctgatcgat240
cgcccgtcgggccttgtacacgagattttagtttctatcacagatgagaaggtgaaatct300
gcaaagacccttaagaatgttcaacctactcagcatgtcttagagatgatcgaagcggaa360
aagatcatcacgaaggatccttcagttatcgaagaatgtcgcaagttaggcattacggat420
atgaagaatgtctatgctgatccgtggacggtgggttaccatgctcaatttaagtccagt480
aaacgtttaatgcaggcccttatgtacatgcgcacctctcccgacgacaaccagtatgcg540
catcctttagattttgtaccgatttatgatgttaacgcacaaaaggtcgtcgagattttg600
cgccaggaaacttcggaagcaagcaagtatgatcgtccgacagttccgttagaaaaccat660
cagtttttaccagag675
<210>28
<211>690
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>28
cagtttttaccagagcacattggcgtggaaaacttacgcaaggacatcaagcccatcgaa60
attacccaaccggagggggtgtcttttacagtgcgcgggcgtgaaatcgaatggcaaaat120
tggagtatgcatgtcgggttcaactatcgtgaaggagttatcattaacaacgtgtcctat180
aaggacaagggcaatgtgcgtccgttattctaccgcgtgtcggtaagtgaaatggtcgta240
ccgtacgcacaccccaaggagccgttcaaccataagatggcctttgacgtcggggagtac300
ggtttggggaaccttactaacagccttgagctgggctgtgattgtttgggctcgatttat360
tatatggatggtgtctgtaataatctggatggcgaaccctgggtcatccccaatgcaatt420
tgcatccatgaagaggatacgggtttgttattcaaacacaccgattaccgcactgataaa480
gctcacagcgcacgtagccgccgtttggttatctcgcaaattgtgaccgccgccaactat540
gattatggcttgtatttctacttctaccaagacggtacgtttcagtacgaggtcaaagct600
accggggagctgaacactcaagtttttgcggaagacgaaaacccagcaccttatggtacc660
gcagtcgctccccaagtcgtaggccagcat690
<210>29
<211>672
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>29
gtcgtaggccagcatcatcaacacttatttatgatgcgcattgacccaatgttagatggt60
cgtctgaactctgttgcccaggttgatgttttaccctccgagtacccagtcggtcacgtc120
gagaatccaatcggcaacgcctttagtcctattacgactatttacagtgacacgactgag180
gcgcaggcacatgggaatttagagtcgtcgcgtacatggaagatcattaacgaatctaaa240
ctgcatccatatacaaaagaaccagtaggttacaaacttgtgagtcctaacactcccccg300
atgcttcccaaacctggatcccttgtctatgaacgcgcaaaattcgctactaaaaccatc360
tgggtaactccctatgaccccgatcagatttatcccgccggattctattgctcacaatca420
ccgggtgacgattcgatgggattgcctgcatggactaaagaaccacaaagcgtacgtgga480
cgtgacgtagttgtgtggcttaccttcggacttacgcacatcccacgcgtggaagacttc540
cccgttatgcctgtagagacgtgcgggtgggcgctgaaagcgtgtaattttttcttaggg600
aatccagggatcgacattccggcagcacagaaggggacctccaagcgcgtaaatggtgca660
tgttgtgcgtaa672
<210>30
<211>683
<212>prt
<213>总状共头霉(syncephalastrumracemosum)
<400>30
metthrvalseralavalilehisproleuaspproleuserproglu
151015
gluilearghisalaseralaileileargarggluargseralaasp
202530
lysthrilepheilepheasnserileserleuarggluproprolys
354045
aspglnvalleualahispheglytrpalaalaglyprolysproala
505560
glnileaspargglnalaphevalvalleuileaspargprosergly
65707580
leuvalhisgluileilevalserleuthrthrserservalvalser
859095
trpasnarglysglnglyvalglnprothrleuhisvalglnglumet
100105110
leuglualaglugluvalmetleulysaspgluargvalilegluglu
115120125
cysarglysleuglyilegluaspmetserleuvalphealaaspthr
130135140
trpglyvalglytrphislysthrlysglylysargleumetglnala
145150155160
leumettyrmetargthrserproaspaspasnglntyralahispro
165170175
leuaspphethrproleutyraspvalasngluglnlysvalileasp
180185190
valleuvalalalysargargasnserlysphegluargprovalile
195200205
proargalaasphisglnpheleuprogluhisleuglygluglyasn
210215220
leuarglysaspilelysproilegluileileglnprogluglyval
225230235240
serpheglnileargglyhisgluileasptrpglnlystrpasnleu
245250255
hisileglypheasntyrarggluglyleuvalileserasnvalser
260265270
tyrlysaspmetaspglythrvalargprovalphetyrargvalser
275280285
leualaglumetvalvalprotyralaasnprotyrgluprotyrasn
290295300
hislysmetalapheaspvalglyglutyrglyleuglyasnleuthr
305310315320
asnserleugluleuglycysaspcysvalglyserilephetyrmet
325330335
aspglyvalcysseraspleulysglyaspalatrpvalileproasn
340345350
alailecysilehisglugluaspthrglyleuleuphelyshisthr
355360365
asppheargasnasnlysalahisseralaargserargargleuval
370375380
ileserhisilevalthralaalaasntyrasptyrglyleutyrtyr
385390395400
tyrphetyrglnaspglythrpheglntyrgluvallysalathrgly
405410415
gluleuasnthrhisvalleualagluaspgluasnproalaprotyr
420425430
glythrilevalalaproglnileaspalaglnhishisglnhisleu
435440445
phesermetargileaspprometvalaspglyprothrasnserval
450455460
alaglnvalaspvalvalalaseraspleuprovalglyhisproasp
465470475480
asnalavalglyasnalapheserproilethrthriletyralaasn
485490495
thraspglualaglnalaargalaasnglygluthrserargtyrtrp
500505510
lysileileasngluasplysilehisprotyrthrarggluproval
515520525
glyphelysleumetcysproasnthrproprometleuprolyspro
530535540
glyserilealatyrgluargalavalphealaserlysthrvaltrp
545550555560
valthrprotyraspalagluglnleupheproglyglyphetyrcys
565570575
tyrglnseraspproalaaspargleuglyleuproglutrpthrlys
580585590
glylyslysaspvallysasnlysaspilevalleutrpleuthrphe
595600605
glyleuthrhisileproargvalgluasppheproilemetproval
610615620
gluthrcysglyphemetleulysprocysasnphepheleualaasn
625630635640
proglyileaspileproalaserasplyshisserthrlysserala
645650655
tyralahisalaleuthrasnglyalaasnglythrglyasnglyser
660665670
thrasnglysersercyscysasnlysasntyr
675680
<210>31
<211>2052
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>31
atgacagtttctgccgtgattcatcctttggaccccttgtctcccgaggagatccgtcat60
gcatcagcgattatccgccgcgagcgtagtgccgacaagacaatctttatcttcaacagc120
atctcgctgcgtgaaccccccaaagaccaggtgcttgcacactttggctgggctgctggc180
ccaaaacctgctcagattgatcgtcaggctttcgtcgtccttatcgatcgcccgtccggc240
ttagtgcatgaaattatcgtgtccttaacgacgtccagcgtggtttcatggaaccgtaaa300
caaggcgtacaaccaacgctgcacgttcaggagatgcttgaggcagaagaggtgatgttg360
aaagatgaacgtgtcatcgaggaatgccgcaaattaggaatcgaagatatgagtcttgtg420
tttgccgacacgtggggagtgggctggcataaaacaaagggcaaacgtcttatgcaggct480
ttgatgtatatgcgtacgagccctgatgataaccagtatgcgcatcccttggactttaca540
ccattatacgatgtcaatgagcaaaaagtcatcgatgtgcttgtcgccaagcgccgtaac600
tcaaagttcgaacgtcccgtcatcccccgtgcggaccaccagtttctgccggaacacctg660
ggggagggcaatttgcgcaaagatattaagcccattgaaattattcaaccagagggtgtc720
agctttcaaatccgcgggcatgagatcgactggcaaaagtggaaccttcacatcggattc780
aattatcgcgagggacttgttatcagcaacgttagttacaaagacatggacggcacggta840
cgtcctgtattctatcgcgtatctctggcagaaatggtagtcccctacgcgaatccttac900
gaaccctacaaccataagatggccttcgatgtaggagagtacggcctgggcaacttgacg960
aacagtttagaactgggatgtgattgcgtagggagcatcttctacatggacggggtctgt1020
tcggatctgaagggagacgcgtgggtcatccccaatgcgatctgcatccatgaagaggac1080
accgggttattgttcaagcacacagattttcgcaacaacaaagcgcactcagctcgtagt1140
cgtcgtttggttatttcccatatcgttaccgccgccaattatgattatgggttgtattac1200
tatttttaccaggatggtactttccaatacgaagtcaaggccacaggagaacttaacact1260
cacgtcttagcggaggatgagaaccccgctccctacggaacaattgtggcacctcagatc1320
gatgcgcaacaccaccaacacttattttccatgcgcattgatccgatggtagacggaccg1380
actaactcggtggctcaggtagacgtggtagctagcgacttgcctgtcggacaccctgac1440
aacgccgtggggaatgcattctcgccaattaccacaatctatgcgaacacagacgaggca1500
caagcgcgcgccaatggagagacttcccgttactggaagatcatcaacgaagataaaatt1560
cacccatatacacgtgaacctgttggttttaagcttatgtgtccgaacacaccacccatg1620
ctgccgaaaccgggatccattgcgtatgagcgtgcagtcttcgcttcgaaaacggtctgg1680
gttaccccgtacgacgcagaacaattgttcccaggtggtttctactgttatcagtcggac1740
cccgccgatcgtttaggcctgccggagtggacaaaagggaaaaaggacgtaaagaacaag1800
gacattgtcctgtggttaacgttcgggctgacccacattccgcgtgtggaagactttccc1860
attatgcccgtagaaacctgcggctttatgcttaaaccctgtaacttcttccttgcaaac1920
ccaggaattgacatcccggcctccgataagcactcgactaaatcggcgtacgctcatgcg1980
cttacgaatggagccaatgggactgggaacggtagcacaaacggctccagctgctgcaat2040
aagaattactaa2052
<210>32
<211>687
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>32
atgacagtttctgccgtgattcatcctttggaccccttgtctcccgaggagatccgtcat60
gcatcagcgattatccgccgcgagcgtagtgccgacaagacaatctttatcttcaacagc120
atctcgctgcgtgaaccccccaaagaccaggtgcttgcacactttggctgggctgctggc180
ccaaaacctgctcagattgatcgtcaggctttcgtcgtccttatcgatcgcccgtccggc240
ttagtgcatgaaattatcgtgtccttaacgacgtccagcgtggtttcatggaaccgtaaa300
caaggcgtacaaccaacgctgcacgttcaggagatgcttgaggcagaagaggtgatgttg360
aaagatgaacgtgtcatcgaggaatgccgcaaattaggaatcgaagatatgagtcttgtg420
tttgccgacacgtggggagtgggctggcataaaacaaagggcaaacgtcttatgcaggct480
ttgatgtatatgcgtacgagccctgatgataaccagtatgcgcatcccttggactttaca540
ccattatacgatgtcaatgagcaaaaagtcatcgatgtgcttgtcgccaagcgccgtaac600
tcaaagttcgaacgtcccgtcatcccccgtgcggaccaccagtttctgccggaacacctg660
ggggagggcaatttgcgcaaagatatt687
<210>33
<211>702
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>33
ttgcgcaaagatattaagcccattgaaattattcaaccagagggtgtcagctttcaaatc60
cgcgggcatgagatcgactggcaaaagtggaaccttcacatcggattcaattatcgcgag120
ggacttgttatcagcaacgttagttacaaagacatggacggcacggtacgtcctgtattc180
tatcgcgtatctctggcagaaatggtagtcccctacgcgaatccttacgaaccctacaac240
cataagatggccttcgatgtaggagagtacggcctgggcaacttgacgaacagtttagaa300
ctgggatgtgattgcgtagggagcatcttctacatggacggggtctgttcggatctgaag360
ggagacgcgtgggtcatccccaatgcgatctgcatccatgaagaggacaccgggttattg420
ttcaagcacacagattttcgcaacaacaaagcgcactcagctcgtagtcgtcgtttggtt480
atttcccatatcgttaccgccgccaattatgattatgggttgtattactatttttaccag540
gatggtactttccaatacgaagtcaaggccacaggagaacttaacactcacgtcttagcg600
gaggatgagaaccccgctccctacggaacaattgtggcacctcagatcgatgcgcaacac660
caccaacacttattttccatgcgcattgatccgatggtagac702
<210>34
<211>693
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>34
gatccgatggtagacggaccgactaactcggtggctcaggtagacgtggtagctagcgac60
ttgcctgtcggacaccctgacaacgccgtggggaatgcattctcgccaattaccacaatc120
tatgcgaacacagacgaggcacaagcgcgcgccaatggagagacttcccgttactggaag180
atcatcaacgaagataaaattcacccatatacacgtgaacctgttggttttaagcttatg240
tgtccgaacacaccacccatgctgccgaaaccgggatccattgcgtatgagcgtgcagtc300
ttcgcttcgaaaacggtctgggttaccccgtacgacgcagaacaattgttcccaggtggt360
ttctactgttatcagtcggaccccgccgatcgtttaggcctgccggagtggacaaaaggg420
aaaaaggacgtaaagaacaaggacattgtcctgtggttaacgttcgggctgacccacatt480
ccgcgtgtggaagactttcccattatgcccgtagaaacctgcggctttatgcttaaaccc540
tgtaacttcttccttgcaaacccaggaattgacatcccggcctccgataagcactcgact600
aaatcggcgtacgctcatgcgcttacgaatggagccaatgggactgggaacggtagcaca660
aacggctccagctgctgcaataagaattactaa693
<210>35
<211>683
<212>prt
<213>总状共头霉(syncephalastrumracemosum)
<400>35
metthrleuprothrthrilehisproleuaspproleuserproglu
151015
gluilearghisvalsergluileilearglysglnargalaalaasp
202530
gluthrthrtyrilepheasnserilealaleuarggluproprolys
354045
gluglnileleualahispheglytrpthraspglyprolysproval
505560
glnileaspargglnalaphealavalleuileaspargprosergly
65707580
leuvalhisgluileilevalserilethrthralaserilevalser
859095
trpgluthrlysgluglyvalglnprothrleuhisvalglnglumet
100105110
leuglualagluglnvalmetleulysaspgluargvalilegluglu
115120125
cysarglysleuglyilegluaspmetsermetvalphealaaspthr
130135140
trpglyvalglytrphislysthrlysglylysargleumetglnala
145150155160
leumettyrmetargthrserproaspaspasnglntyralahispro
165170175
leuaspphethrproleutyraspvalasngluglnlysvalileasp
180185190
valleuvalalalysargargasnserlysphegluargprovalile
195200205
proargalaaspargglnpheleuprogluhisleuglyglugluasn
210215220
leuarglysaspilelysproilegluilethrglnproglnglyval
225230235240
serpheglnileargglyhisgluileasptrpglnlystrpasnleu
245250255
hisvalglypheasntyrarggluglyleuvalileasnasnvalser
260265270
tyrlysaspmetaspglythrvalargprometphetyrargvalser
275280285
leualaglumetvalvalprotyralaasnprotyrgluprotyrasn
290295300
hislysmetalapheaspvalglyglutyrglyleuglyasnleuthr
305310315320
asnserleugluleuglycysaspcysvalglyserilephetyrmet
325330335
aspglyvalcysseraspilelysglyaspalatrpvalileproasn
340345350
alailecysilehisglugluaspthrglyleuleuphelyshisthr
355360365
asppheargasnasnlysalahisseralaargserargargleuval
370375380
ileserhisilevalthralaalaasntyrasptyrglyleutyrtyr
385390395400
tyrphetyrglnaspglythrpheglntyrgluvallysalathrgly
405410415
gluleuasnthrhisvalleualagluaspgluaspproalaprotyr
420425430
glythrilevalalaproglnvalaspalaglnhishisglnhisleu
435440445
phesermetargileaspprometvalaspglyprothrasnserval
450455460
alaglnvalaspvalvalalaseraspleuprovalglyhisproasp
465470475480
asnalavalglyasnalapheserprovalthrthriletyralaasp
485490495
thraspglualaargalaargalaasnglygluthrserargtyrtrp
500505510
lysileileasngluthrargilehisprotyrthrlysgluproval
515520525
glyphelysleumetcysproasnthrproprometleuprolyspro
530535540
glyserilealatyrgluargalavalphealaserasnthrvaltrp
545550555560
valthrprotyraspalagluglnleupheproglyglyphetyrcys
565570575
tyrglnseraspproalaaspargleuglyleuproglutrpthrarg
580585590
glulyslysaspvallysasnlysaspilevalleutrpleuthrphe
595600605
glyleuthrhisileproargvalgluasppheproilemetproval
610615620
gluthrcysglyphemetleulysprocysasnphepheleualaasn
625630635640
proglyileaspileproalaserasparghisserserlysserala
645650655
tyralaproalavalalaasnglyglutyrglyilethrasnglythr
660665670
thrasnglysersercyscysserlysglyhis
675680
<210>36
<211>2052
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>36
atgacactccccactactatccacccattggatccgctgagtcccgaggaaattcgtcat60
gttagtgagatcatccgaaaacaacgggccgccgacgaaacaacctatatattcaactcc120
atagcactccgtgaaccgcctaaggaacagatcctcgcgcatttcggttggacagatgga180
cctaaaccagttcagattgatcggcaagcgttcgctgtcctcatagaccgcccttcggga240
ttggtccatgagataattgtgagcatcacgaccgcctccatagtttcctgggagactaag300
gaaggcgttcagcccactctgcatgtacaagaaatgctggaggcagagcaagtgatgctc360
aaggatgaacgtgtcatcgaggaatgccgaaaactcggtattgaagatatgtcgatggta420
ttcgctgatacatggggagtgggttggcacaaaaccaagggcaaacgattgatgcaagcc480
ctcatgtacatgcgcactagtccagacgacaatcagtatgcccatcccctggatttcaca540
cccctgtatgatgtaaacgagcaaaaagtgatagatgttctggtggcgaaaaggcgaaat600
tcgaaattcgagcgaccagtcattccacgagccgaccgccagttcttgccagagcacctc660
ggagaggaaaatctccgcaaagatataaaaccgattgaaattacccagccccaaggtgtc720
agtttccagattaggggccacgagatcgactggcaaaagtggaacctgcatgtcggattc780
aattatcgagaaggcctcgtaattaacaatgtcagttacaaggacatggatggcactgtt840
aggcccatgttctatcgagtgagtctggcagaaatggtcgtcccgtatgcgaacccgtac900
gagccttacaatcacaagatggcttttgacgtgggtgaatatggactcggtaacctcaca960
aatagcttggagctcggatgtgattgtgtaggtagtatattttatatggacggagtgtgt1020
agcgacatcaagggtgacgcttgggtaattcccaatgcgatttgcatccacgaagaagac1080
acgggcctcctgttcaagcacactgattttaggaacaataaggcgcatagcgctcgaagc1140
cgcaggctggtgatttcccacatcgtcacagcggcaaattacgattatggcctgtactat1200
tatttttatcaggacggtacatttcagtacgaagtgaaggcgactggagagctcaacact1260
cacgtattggcggaagacgaagaccctgcaccttacggcactattgttgctcctcaggta1320
gacgcgcagcatcatcaacacctctttagtatgcgtattgatccaatggtagatggacct1380
accaatagtgtggctcaagtagacgtcgtggccagcgatttgcccgtcggtcatcccgat1440
aacgccgtgggtaacgcgttcagtccggttacaacgatttacgcagacactgacgaagcg1500
cgggcacgagctaatggagaaacctcccgctactggaagataattaacgaaactcgcata1560
catccctacactaaagaaccagtaggtttcaaactcatgtgccctaatacacctccgatg1620
ctcccgaaaccgggaagcattgcgtacgaacgcgcagtatttgcctcgaatacagtttgg1680
gttactccgtacgatgccgagcaactgtttccgggtggattctactgctatcaaagcgat1740
cccgccgaccgcctgggtctgccggagtggactagggagaagaaggacgtgaaaaacaag1800
gacatagttctgtggctgacgtttggtctcactcatattcccagggttgaggattttcca1860
atcatgccggtagaaacatgtggattcatgctgaagccttgtaatttcttcttggcaaac1920
ccgggaattgatatccctgctagcgatcgtcatagtagtaagtccgcatatgcaccggcg1980
gttgcaaacggagaatatggcataacaaatggaacgacgaatggctcgtcctgttgcagc2040
aagggacactaa2052
<210>37
<211>687
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>37
atgacactccccactactatccacccattggatccgctgagtcccgaggaaattcgtcat60
gttagtgagatcatccgaaaacaacgggccgccgacgaaacaacctatatattcaactcc120
atagcactccgtgaaccgcctaaggaacagatcctcgcgcatttcggttggacagatgga180
cctaaaccagttcagattgatcggcaagcgttcgctgtcctcatagaccgcccttcggga240
ttggtccatgagataattgtgagcatcacgaccgcctccatagtttcctgggagactaag300
gaaggcgttcagcccactctgcatgtacaagaaatgctggaggcagagcaagtgatgctc360
aaggatgaacgtgtcatcgaggaatgccgaaaactcggtattgaagatatgtcgatggta420
ttcgctgatacatggggagtgggttggcacaaaaccaagggcaaacgattgatgcaagcc480
ctcatgtacatgcgcactagtccagacgacaatcagtatgcccatcccctggatttcaca540
cccctgtatgatgtaaacgagcaaaaagtgatagatgttctggtggcgaaaaggcgaaat600
tcgaaattcgagcgaccagtcattccacgagccgaccgccagttcttgccagagcacctc660
ggagaggaaaatctccgcaaagatata687
<210>38
<211>705
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>38
ctccgcaaagatataaaaccgattgaaattacccagccccaaggtgtcagtttccagatt60
aggggccacgagatcgactggcaaaagtggaacctgcatgtcggattcaattatcgagaa120
ggcctcgtaattaacaatgtcagttacaaggacatggatggcactgttaggcccatgttc180
tatcgagtgagtctggcagaaatggtcgtcccgtatgcgaacccgtacgagccttacaat240
cacaagatggcttttgacgtgggtgaatatggactcggtaacctcacaaatagcttggag300
ctcggatgtgattgtgtaggtagtatattttatatggacggagtgtgtagcgacatcaag360
ggtgacgcttgggtaattcccaatgcgatttgcatccacgaagaagacacgggcctcctg420
ttcaagcacactgattttaggaacaataaggcgcatagcgctcgaagccgcaggctggtg480
atttcccacatcgtcacagcggcaaattacgattatggcctgtactattatttttatcag540
gacggtacatttcagtacgaagtgaaggcgactggagagctcaacactcacgtattggcg600
gaagacgaagaccctgcaccttacggcactattgttgctcctcaggtagacgcgcagcat660
catcaacacctctttagtatgcgtattgatccaatggtagatgga705
<210>39
<211>690
<212>dna
<213>总状共头霉(syncephalastrumracemosum)
<400>39
ccaatggtagatggacctaccaatagtgtggctcaagtagacgtcgtggccagcgatttg60
cccgtcggtcatcccgataacgccgtgggtaacgcgttcagtccggttacaacgatttac120
gcagacactgacgaagcgcgggcacgagctaatggagaaacctcccgctactggaagata180
attaacgaaactcgcatacatccctacactaaagaaccagtaggtttcaaactcatgtgc240
cctaatacacctccgatgctcccgaaaccgggaagcattgcgtacgaacgcgcagtattt300
gcctcgaatacagtttgggttactccgtacgatgccgagcaactgtttccgggtggattc360
tactgctatcaaagcgatcccgccgaccgcctgggtctgccggagtggactagggagaag420
aaggacgtgaaaaacaaggacatagttctgtggctgacgtttggtctcactcatattccc480
agggttgaggattttccaatcatgccggtagaaacatgtggattcatgctgaagccttgt540
aatttcttcttggcaaacccgggaattgatatccctgctagcgatcgtcatagtagtaag600
tccgcatatgcaccggcggttgcaaacggagaatatggcataacaaatggaacgacgaat660
ggctcgtcctgttgcagcaagggacactaa690
- 还没有人留言评论。精彩留言会获得点赞!