一种生物鲁棒性提高的大肠杆菌底盘细胞及其构建方法与应用与流程
本发明属于基因工程技术领域,具体涉及一种生物鲁棒性提高的大肠杆菌底盘细胞及其构建方法与应用。
背景技术:
合成生物学技术的发展极大地提高了构建微生物细胞工厂生产大宗化学品和天然产物的能力。合成生物学在构建细胞工厂的关键技术包括最优合成途径的设计、合成途径的创建与优化、细胞性能的优化。细胞性能优化是基于底盘细胞自身的能量或物质代谢方面进行优化,从而提高底盘细胞中功能性生物元件的活力,以高效合成目标产物。然而微生物在诸如环境中ph值、温度、渗透压、有毒代谢产物等压力条件会对细胞生长产生不利影响并限制代谢产物的产生。工业微生物的鲁棒性状体现在能够应对复杂多样的恶劣工业环境,并且具有与正常环境下可比拟的生产性能。因此,在外界压力条件下维持细胞正常生长和提高目标产物产量是微生物工业生产中最重要的目标。
大肠杆菌是一种常用的异源合成底盘细胞。它具有清晰的遗传背景、成熟的基因操作技术与工具、丰富的基因表达元件、可靠经济的大规模发酵技术。然而大肠杆菌在遇到代谢压力时会生长缓慢,产生诸多次级代谢产物影响其目标产物的生成。例如:高密度发酵时乙酸的积累影响抑制大肠杆菌的生长速率。因此提高大肠杆菌底盘细胞的鲁棒性使其在环境胁迫或代谢压力时仍保持细胞系统功能的稳定是工业应用的关键问题。
大肠杆菌作为表达系统在表达外源蛋白时会产生物质和能量代谢负担从而影响蛋白的表达效果。截至目前,大多数大肠杆菌合成生物学研究主要集中在天然产物或小分子化合物的生物合成方面,对于通过代谢调控或是代谢工程手段提高单一外源蛋白表达量的研究仍很薄弱。然而底盘细胞增殖的速度、基因表达水平以及细胞过表达异源基因的能力是相互依赖的。因此在重组蛋白的生产中,调节细菌资源分配,即将细菌资源从生物量的合成转移到用于重组蛋白的生产仍然是生物技术领域的巨大挑战。
技术实现要素:
本发明的目的是提供生物鲁棒性提高、细胞性能优良的大肠杆菌底盘细胞及其在外源蛋白表达中的应用。
为了实现上述目的,本发明首先提供了prpd蛋白和/或malk蛋白的新用途。
本发明提供了prpd蛋白和/或malk蛋白在如下a1)-a5)任一种中的应用:
a1)调控大肠杆菌鲁棒性;
a2)调控大肠杆菌生长;
a3)调控大肠杆菌中乙酸积累量;
a4)调控大肠杆菌中atp产量;
a5)调控大肠杆菌中外源蛋白的表达量和/或活性。
为了实现上述目的,本发明又提供了与prpd蛋白相关的生物材料和/或与malk蛋白相关的生物材料的新用途。
本发明提供了与prpd蛋白相关的生物材料和/或与malk蛋白相关的生物材料在如下b1)-b6)任一种中的应用:
b1)调控大肠杆菌鲁棒性;
b2)调控大肠杆菌生长;
b3)调控大肠杆菌中乙酸积累量;
b4)调控大肠杆菌中atp产量;
b5)调控大肠杆菌中外源蛋白的表达量和/或活性;
b6)制备鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp含量提高的大肠杆菌底盘细胞;
所述与prpd蛋白相关的生物材料为下述c1)-c8)中的任一种:
c1)编码prpd蛋白的核酸分子;
c2)含有c1)所述核酸分子的表达盒;
c3)含有c1)所述核酸分子的重组载体;
c4)含有c2)所述表达盒的重组载体;
c5)含有c1)所述核酸分子的重组微生物;
c6)含有c2)所述表达盒的重组微生物;
c7)含有c3)所述重组载体的重组微生物;
c8)含有c4)所述重组载体的重组微生物;
所述与malk蛋白相关的生物材料为下述d1)-d8)中的任一种:
d1)编码malk蛋白的核酸分子;
d2)含有d1)所述核酸分子的表达盒;
d3)含有d1)所述核酸分子的重组载体;
d4)含有d2)所述表达盒的重组载体;
d5)含有d1)所述核酸分子的重组微生物;
d6)含有d2)所述表达盒的重组微生物;
d7)含有d3)所述重组载体的重组微生物;
d8)含有d4)所述重组载体的重组微生物。
上述应用中,c1)所述核酸分子为如下1)或2)或3)所示的基因:
1)其编码序列是seqidno.1所示的cdna分子或基因组dna分子;
2)与1)限定的核苷酸序列具有75%或75%以上同一性,且编码prpd蛋白的cdna分子或基因组dna分子;
3)在严格条件下与1)或2)限定的核苷酸序列杂交,且编码prpd蛋白的cdna分子或基因组dna分子;
d1)所述核酸分子为如下4)或5)或6)所示的基因:
4)其编码序列是seqidno.4所示的cdna分子或基因组dna分子;
5)与4)限定的核苷酸序列具有75%或75%以上同一性,且编码malk蛋白的cdna分子或基因组dna分子;
6)在严格条件下与4)或5)限定的核苷酸序列杂交,且编码malk蛋白的cdna分子或基因组dna分子。
上述应用中,所述调控大肠杆菌鲁棒性为提高大肠杆菌鲁棒性;
所述调控大肠杆菌生长为促进大肠杆菌生长;
所述调控大肠杆菌中乙酸积累量为降低大肠杆菌中乙酸积累量;
所述调控大肠杆菌中atp产量为提高大肠杆菌中atp产量;
所述调控大肠杆菌中外源蛋白的表达量和/或活性为提高大肠杆菌中外源蛋白的表达量和/或活性。
为了实现上述目的,本发明还提供了如下e1)-e7)任一种方法:
e1)一种提高大肠杆菌鲁棒性的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌;所述重组大肠杆菌的鲁棒性高于所述目的大肠杆菌;
e2)一种促进大肠杆菌生长的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌;所述重组大肠杆菌的生长速率高于所述目的大肠杆菌;
e3)一种降低大肠杆菌中乙酸积累量的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌;所述重组大肠杆菌中的乙酸积累量低于所述目的大肠杆菌;
e4)一种提高大肠杆菌中atp产量的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌;所述重组大肠杆菌中的atp产量高于所述目的大肠杆菌;
e5)一种提高大肠杆菌中外源蛋白的表达量和/或活性的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌;所述重组大肠杆菌中的外源蛋白表达量和/或活性高于所述目的大肠杆菌;
e6)一种制备鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞的方法,包括如下步骤:提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性,得到重组大肠杆菌,即为所述鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞;
e7)一种外源蛋白的表达方法,包括利用按照e6)所述方法制备得到的鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞表达外源蛋白的步骤。
上述方法中,所述e1)-e6)中,所述提高目的大肠杆菌中prpd蛋白和/或malk蛋白的含量和/或活性的方法为在目的大肠杆菌中过表达prpd蛋白和/或malk蛋白。
进一步的,所述在目的大肠杆菌中过表达prpd蛋白和/或malk蛋白的方法为将prpd蛋白的编码基因序列和/或malk蛋白的编码基因序列导入目的大肠杆菌中。
再进一步的,所述prpd蛋白的编码基因序列和/或所述malk蛋白的编码基因序列通过重组载体甲导入目的大肠杆菌中;所述重组载体甲为将所述prpd蛋白的编码基因序列和/或所述malk蛋白的编码基因序列插入表达载体甲的多克隆位点后得到的载体。所述表达载体甲可为现有技术中常见的表达载体,如pacyc-duet-1、pacyc-184、prsfduet1、pet-duet1等载体。
所述重组载体甲中用于启动prpd蛋白的编码基因序列表达的启动子或用于启动malk蛋白的编码基因序列表达的启动子可为组成型启动子或诱导型启动子。由于不同的外源蛋白在折叠表达的速率不一致会给大肠杆菌自身代谢带来的影响也不尽相同,所以在调控malk或prpd的表达强度上也需要根据具体表达的外源蛋白来决定。
当外源蛋白为udp-糖基转移酶76g1时,所述重组载体甲中,用于启动prpd蛋白的编码基因序列表达的启动子优选为组成型启动子(如tetpromoter),用于启动malk蛋白的编码基因序列表达的启动子优选为诱导型启动子(如t7promoter&lacoperator)。
当外源蛋白为过氧化氢酶时,所述重组载体甲中,用于启动prpd蛋白的编码基因序列表达的启动子优选为组成型启动子(如tetpromoter),用于启动malk蛋白的编码基因序列表达的启动子优选为诱导型启动子(如t7promoter&lacoperator)。
当外源蛋白为udp-糖基转移酶hv1时,所述重组载体甲中,用于启动prpd蛋白的编码基因序列表达的启动子优选为诱导型启动子(如t7promoter&lacoperator),用于启动malk蛋白的编码基因序列表达的启动子优选为诱导型启动子(如t7promoter&lacoperator)。
当外源蛋白为淀粉酶时,所述重组载体甲中,用于启动prpd蛋白的编码基因序列表达的启动子优选为诱导型启动子(如t7promoter&lacoperator),用于启动malk蛋白的编码基因序列表达的启动子优选为组成型启动子(如tetpromoter)。
在实际应用中,在更换要表达的外源蛋白时需要根据表达量调整malk和/或prpd的表达强度即通过更换其相应的启动子的方式完成,以此方式来找出最适合该外源蛋白表达的对应的malk和/或prpd的表达强度。
更进一步的,所述重组载体甲为pacyc-184/prpd、pacyc-184/malk、pacyc-duet-1/prpdi-malki、pacyc-duet-g1/prpdc-malki、pacyc-duet-g2/prpdi-malkc或pacyc-duet-g3/prpdc-malkc。
所述pacyc-184/prpd为将prpd基因插入到载体pacyc-184多克隆位点后得到载体。
所述pacyc-184/malk为将malk基因插入到载体pacyc-184多克隆位点后得到载体。
所述pacyc-duet-1/prpdi-malki为将prpd基因和malk基因插入到载体pacyc-duet-1的两个不同的多克隆位点后得到的载体。
所述pacyc-duet-g1/prpdc-malki为将prpd基因和malk基因插入到载体pacyc-duet1-g1的两个不同的多克隆位点后得到的载体。
所述pacyc-duet-g2/prpdi-malkc为将prpd基因和malk基因插入到载体pacyc-duet1-g2的两个不同的多克隆位点后得到的载体。
所述pacyc-duet-g3/prpdc-malkc为将prpd基因和malk基因插入到载体pacyc-duet1-g3的两个不同的多克隆位点后得到的载体。
所述pacyc-duet-g1是在载体pacyc-duet-1的基础上,将其多克隆位点mcs-1前的t7promoter&lacoperator序列更换为tetpromoter序列后得到的载体;所述pacyc-duet-g2是在载体pacyc-duet-1的基础上,将其多克隆位点mcs-2前的t7promoter&lacoperator序列更换为tetpromoter序列后得到的载体;所述载体pacyc-duet-g3是在所述载体pacyc-duet-g2的基础上,将其多克隆位点mcs-1前的t7promoter&lacoperator序列更换为tetpromoter序列后得到的载体。
所述e7)中,所述利用按照e6)所述方法制备得到的鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞表达外源蛋白的方法为在鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞中过表达外源蛋白。
进一步的,所述在鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞中过表达外源蛋白的方法为将外源蛋白的编码基因序列导入鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞中。
再进一步的,所述外源蛋白的编码基因序列通过重组载体乙导入鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞中;所述重组载体乙为将所述外源蛋白的编码基因序列插入表达载体乙的多克隆位点后得到的载体。所述表达载体乙可为现有技术中常见的表达载体,如pet26b、pet32a等载体。
更进一步的,所述重组载体乙为pet32a-76g1或pet32a-hv1或pet26b-kata或pet26b-amy。
所述pet32a-76g1为将seqidno.9所示的植物来源的udp-糖基转移酶76g1的编码基因序列插入到载体pet32a的ecori与noti酶切位点之间后得到的载体。
所述pet32a-hv1为将seqidno.10所示的植物来源的udp-糖基转移酶hv1的编码基因序列插入到载体pet32a的ecori与noti酶切位点之间后得到的载体。
所述pet26b-kata为将seqidno.11所示的来源于枯草芽孢杆菌(b.subtilius168)的过氧化氢酶kata的编码基因序列插入载体pet26b的noci和xhoi酶切位点之间后得到的载体。
所述pet26b-amy为将seqidno.12所示的淀粉酶amy的编码基因序列插入到载体pet-26b的noci和xhoi酶切位点之间后得到的载体。
为了实现上述目的,本发明还提供了如下任一产品:
f1)按照上述e6)所述方法制备得到的鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞;
f2)由组成型启动子和prpd基因组成的产品;
f3)由诱导型启动子和prpd基因组成的产品;
f4)由组成型启动子和malk基因组成的产品;
f5)由诱导型启动子和malk基因组成的产品;
f6)由f2)所示的产品和f4)所示的产品组成的成套产品;
f7)由f2)所示的产品和f5)所示的产品组成的成套产品;
f8)由f3)所示的产品和f4)所示的产品组成的成套产品;
f9)由f3)所示的产品和f5)所示的产品组成的成套产品;
f10)prpd基因和pacyc-184载体组成的成套产品;
f11)malk基因和pacyc-184载体组成的成套产品;
f12)prpd基因、malk基因和pacyc-duet-1载体组成的成套产品;
f13)prpd基因、malk基因和pacyc-duet-g1载体组成的成套产品;
f14)prpd基因、malk基因和pacyc-duet-g2载体组成的成套产品;
f15)prpd基因、malk基因和pacyc-duet-g3载体组成的成套产品。
上述产品中,所述组成型启动子可为tetpromoter、t7promoter、sp6promoter;所述tetpromoter的核苷酸序列具体如seqidno.7第42-70位所示。
所述诱导型启动子可为t7promoter&lacoperator、lacpromoter、ptacpromoter、arabadpromoter;所述t7promoter&lacoperator的核苷酸序列具体如seqidno.8第21-64位所示。
上述产品中,所述f1)中,所述鲁棒性提高和/或生长速率提高和/或乙酸积累量下降和/或atp产量提高的大肠杆菌底盘细胞具体为含有上述pacyc-184/prpd或上述pacyc-184/malk或上述pacyc-duet-1/prpdi-malki或上述pacyc-duet-g1/prpdc-malki或上述pacyc-duet-g2/prpdi-malkc或上述pacyc-duet-g3/prpdc-malkc的大肠杆菌。
上述产品在如下e1)-e12)任一种中的应用也属于本发明的保护范围:
e1)提高大肠杆菌鲁棒性;
e2)制备提高大肠杆菌鲁棒性的产品;
e3)促进大肠杆菌生长;
e4)制备促进大肠杆菌生长的产品;
e5)降低大肠杆菌中乙酸积累量;
e6)制备降低大肠杆菌中乙酸积累量的产品;
e7)提高大肠杆菌中atp产量;
e8)制备提高大肠杆菌中atp产量的产品;
e9)表达外源蛋白;
e10)制备表达外源蛋白的产品;
e11)提高外源蛋白的表达量和/或活性;
e12)制备提高外源蛋白的表达量和/或活性的产品。
上述任一所述应用或方法或产品中,所述大肠杆菌或目的大肠杆菌可为大肠杆菌bl21(de3)系列菌株、大肠杆菌bl21(de3)plyss、大肠杆菌rosetta(de3)。所述大肠杆菌bl21(de3)系列菌株包括大肠杆菌bl21-trxb(de3)和大肠杆菌bl21(de3)。
上述任一所述应用或方法或产品中,所述外源蛋白可为来源于动物、植物、微生物中的任一种外源蛋白。在本发明的具体实施例中,所述外源蛋白为udp-糖基转移酶76g1或udp-糖基转移酶hv1或过氧化氢酶kata或淀粉酶amy。
上述任一所述应用或方法或产品中,所述prpd蛋白的氨基酸序列如seqidno.13所示。所述prpd基因的核苷酸序列如seqidno.1所示。
所述malk蛋白的氨基酸序列如seqidno.14所示。所述malk基因的核苷酸序列如seqidno.4所示。
所述udp-糖基转移酶76g1为seqidno.9所示的基因编码的蛋白。所述udp-糖基转移酶76g1基因为seqidno.9所示的dna分子。
所述udp-糖基转移酶hv1为seqidno.10所示的基因编码的蛋白。所述udp-糖基转移酶hv1基因为seqidno.10所示的dna分子。
所述过氧化氢酶kata为seqidno.11所示的基因编码的蛋白。所述过氧化氢酶kata基因为seqidno.11所示的dna分子。
所述淀粉酶amy为seqidno.12所示的基因编码的蛋白。所述淀粉酶amy基因为seqidno.12所示的dna分子。
本发明提供了一种生物鲁棒性能提高的大肠杆菌底盘细胞及其构建方法和应用。本发明通过单独或同时过表达大肠杆菌本源的基因prpd和malk的方法,构建一生长速率加快、atp能量供应稳定充足、乙酸积累受抑制的鲁棒性能提高的大肠杆菌底盘细胞,并命名为:escherichiacolibl21(de3)mp,此优化后的底盘细胞在外界压力条件下仍能保持大肠杆菌的系统功能的稳定性。鲁棒性提高的大肠杆菌可以应用到表达外源蛋白中,针对不同的外源蛋白,分别或同时增强内源prpd和malk的表达均可显著提高不同外源蛋白在大肠杆菌中的表达量和/或活性,在本发明中,外源蛋白的表达量可提高80%-200%。本发明的大肠杆菌底盘细胞适用于动物、植物、微生物等不同来源的外源蛋白在大肠杆菌中的表达,解决了因更换不同重组目标蛋白而需对宿主菌重新改造的问题。除此之外,应用本发明的大肠杆菌底盘细胞提高重组蛋白表达量的方法工艺简化、通用性高、便于实验研究及工业化放大应用。
附图说明
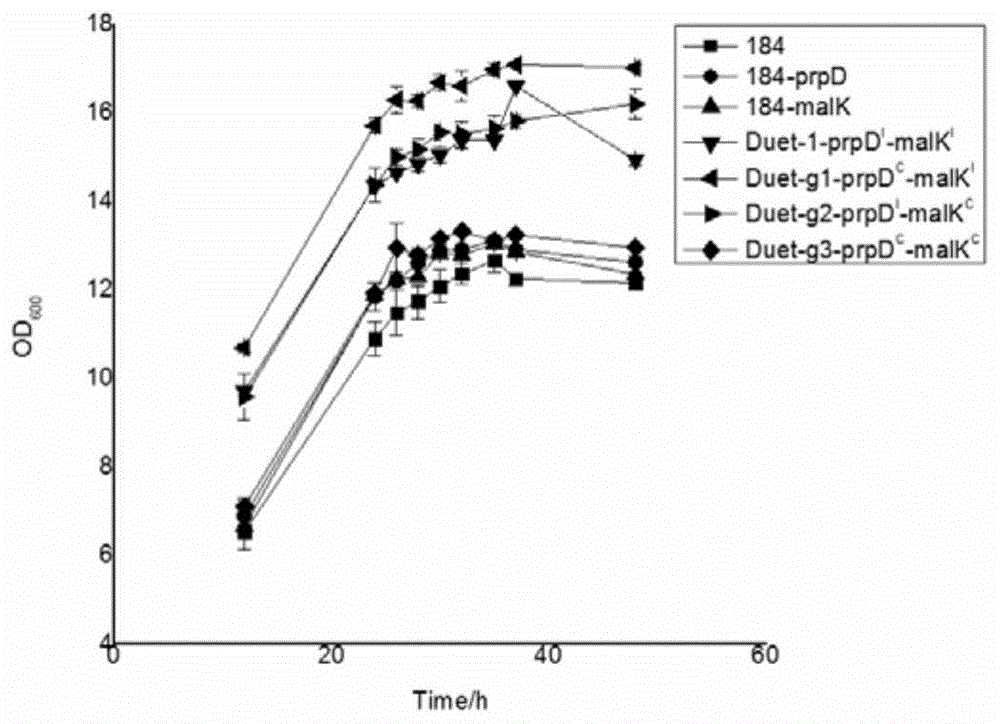
图1为实施例2中的各重组大肠杆菌的生长曲线图。
图2为实施例2中的各重组大肠杆菌的相对光度值(rlu)。图中所示rlu为放大5*106倍的数值。
图3为实施例3中udp-糖基转移酶76g1表达量提高的sds-page图。
图4为实施例4中过氧化氢酶酶活提高的对比图。
图5为实施例5中淀粉酶酶活提高的对比图。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。以下实施例中的定量试验,均设置三次重复实验,结果取平均值。
实施例1、重组质粒的构建
通过诱导型启动子、组成型启动子与prpd、malk之间不同的组合方式构建不同表达强度的prpd、malk重组质粒,不同表达强度的prpd与malk均可使大肠杆菌底盘细胞的鲁棒性能改善,但不同的启动子的替换间接影响基因的表达强度,进而影响其细胞的性能。
1、根据seqidno.1所示的prpd基因的核苷酸序列设计两对引物f1/r1、f2/r2,以bl21(de3)基因组dna作为模板采用引物f1/r1进行pcr,得到seqidno.2所示的核酸序列,以bl21(de3)基因组dna作为模板采用引物f2/r2进行pcr,seqidno.3所示的核苷酸序列。
f1:gcagtcaggcaccgtgtatgatgtcagctcaaatcaacaacatc;
r1:gtgaatccgttagcgagtcattaaatgacgtacaggtcgag;
f2:aggagatataccatgggcagcatgtcagctcaaatcaacaacatc;
r2:atcctggctgtggtgatgatgttaaatgacgtacaggtcgag。
2、在质粒pacyc-184(北京全式金生物技术有限公司)的tcr序列前后设计引物f3/r3,以质粒pacyc-184为模板采用引物f3/r3进行pcr,得到线性化的pacyc-184质粒;在质粒pacyc-duet-1(北京全式金生物技术有限公司)的多克隆位点mcs-1中设计引物f4/r4,以质粒pacyc-duet-1为模板采用引物f4/r4进行pcr,得到线性化的pacyc-duet-1质粒。
f3:tgactcgctaacggattcaccac;
r3:catacacggtgcctgactg;
f4:catcatcaccacagccag;
r4:gctgcccatggtatatctcct。
3、用无缝克隆连接酶将步骤1中的seqidno.2所示的核苷酸序列和步骤2中的线性化的pacyc-184质粒连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-184/prpd;
用无缝克隆连接酶将步骤1中的seqidno.3所示的核苷酸序列和步骤2中的线性化的pacyc-duet-1质粒连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-duet-1/prpd。
4、根据seqidno.4所示核苷酸序列设计两对引物分别为f5/r5和f6/r6,以bl21(de3)基因组dna作为模板采用引物f5/r5进行pcr,得到seqidno.5所示的核苷酸序列,以bl21(de3)基因组dna作为模板采用引物f6/r6进行pcr,得到seqidno.6所示的核苷酸序列。
f5:cagtcaggcaccgtgtatgatggcgagcgtacagctg;
r5:gtggtgaatccgttagcgagttaaacgcccggctccttatgc;
f6:ggcagatctcaattggatatcgatggcgagcgtacagctg;
r6:gctagtagacgagtccatttaaacgcccggctccttatgc。
5、在质粒pacyc-duet-1/prpd的多克隆位点mcs-2中设计引物f7/r7,以pacyc-duet-1/prpd质粒为模板采用引物f7/r7进行pcr,得到线性化的pacyc-duet-1/prpd质粒。
f7:atggactcgtctactagcg;
r7:cgatatccaattgagatctgcc。
6、用无缝克隆连接酶将步骤2中的线性化的pacyc-184质粒和步骤4中的seqidno.5所示的核苷酸序列连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-184/malk。
用无缝克隆连接酶将步骤4中的seqidno.6所示的核苷酸序列和步骤5中的线性化的pacyc-duet-1/prpd质粒连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-duet-1/prpdi-malki。
7、在质粒pacyc-184上的tetpromoter区域前后设计引物f8/r8,以质粒pacyc-184为模板采用引物f8/r8进行pcr,得到seqidno.7所示的核苷酸序列。
f8:ctgaagtcagccccatacga;
r8:catacacggtgcctgactg。
8、在质粒pacyc-duet-1/prpdi-malki上prpd序列、malk序列前的t7promoter&lacoperator区域前后设计引物f9/r9和f10/r10,以质粒pacyc-duet-1/prpdi-malki为模板采用引物f9/r9进行pcr,得到线性化的质粒pacyc-duet-1/g1;以质粒pacyc-duet-1/prpdi-malki为模板采用引物f10/r10进行pcr,得到线性化的质粒pacyc-duet-1/g2。
f9:tatcgtatggggctgacttcagcataagggagagcgtcgaga;
r9:gcagtcaggcaccgtgtatgatgtcagctcaaatcaacaacatc;
f10:tcgtatggggctgacttcagcgacttaagcattatgcggc;
r10:gcagtcaggcaccgtgtatgatggcgagcgtacagctg。
9、用无缝克隆连接酶将步骤7中的seqidno.7所示的核苷酸序列和步骤8中的pacyc-duet-1/g1连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-duet-g1/prpdc-malki。
用无缝克隆连接酶将步骤7中的seqidno.7所示的核苷酸序列和步骤8中的pacyc-duet/g2连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-duet-g2/prpdi-malkc。
10、以质粒pacyc-duet-g2/prpdi-malkc为模板采用引物f9/r9进行pcr,得到线性化的质粒pacyc-duet/g3。
11、用无缝克隆连接酶将步骤7中的seqidno.7所示的核苷酸序列和步骤10中的线性化的质粒pacyc-duet/g3连接,得到连接产物,并将连接产物热转化入bl21(de3)大肠杆菌感受态细胞。转化液涂布含氯霉素(25ug/ml)的lb平板,提取质粒测序验证构建的重组质粒pacyc-duet-g3/prpdc-malkc。
上述测序工作由华大基因完成。最终构建得到如下重组质粒:pacyc-184/prpd、pacyc-184/malk、pacyc-duet-1/prpdi-malki、pacyc-duet-g1/prpdc-malki、pacyc-duet-g2/prpdi-malkc、pacyc-duet-g3/prpdc-malkc。其中,c代表组成型启动子tetpromoter;i代表诱导型启动子t7promoter&lacoperator。
实施例2、重组大肠杆菌生长曲线、乙酸积累量及atp含量的测定
一、重组大肠杆菌的构建
将pacyc-184热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌184bl21(de3)。
将实施例1中构建的pacyc-184/prpd热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌184-prpdbl21(de3)。
将实施例1中构建的pacyc-184/malk热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌184-malkbl21(de3)。
将实施例1中构建的pacyc-duet-1/prpdi-malki热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌duet-1/prpdi-malkibl21(de3)。其中prpd、malk的启动子均为诱导型启动子。
将实施例1中构建的pacyc-duet-g1/prpdc-malki热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌duet-g1-prpdc-malkibl21(de3)。其中prpd的启动子是组成型启动子,malk是诱导型启动子。
将实施例1中构建的pacyc-duet-g2/prpdi-malkc热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌duet-g2-prpdi-malkcbl21(de3)。其中prpd的启动子是诱导型启动子,malk是组成型启动子。
将实施例1中构建的pacyc-duet-g3/prpdc-malkc热转化至bl21(de3)大肠杆菌中,得到重组大肠杆菌duet-g3-prpdc-malkcbl21(de3)。其中prpd、malk的启动子均为组成型启动子。
二、重组大肠杆菌的培养及生长曲线测定
将步骤一中构建的各个重组大肠杆菌甘油管按照1%接种量接种至tb培养基(12g/l胰蛋白胨、24g/l酵母粉、9.4g/lk2hpo4、2.2g/lkh2po4、4ml/l甘油)中,在37℃、220rpm条件下进行培养,分别于培养12h、22h、24h、28h、30h、32h、34h、36h、48h后进行取样测od600。同时以转pacyc-184质粒的bl21(de3)大肠杆菌作为对照。
结果如图1所示,结果表明:相比对照组,共表达prpd或malk基因中的一个或两个对大肠杆菌均有促进生长的效果,其中duet-1-prpdi-malki、duet-g1-prpdc-malki、duet-g2-prpdi-malkc生长速率提高幅度较大且在培养30h后仍保持平稳的生长,生长速率的增长及24h后的稳定,说明大肠杆菌能量代谢在30h后仍可处于稳定的平台期。
三、hplc法测定重组大肠杆菌的乙酸积累量
大肠杆菌在有氧发酵过程中能产生胞外副产物乙酸,乙酸在中性条件下能以乙酸盐的形式存在于发酵液中。并且,大肠杆菌大量积累乙酸,乙酸作为代谢的副产物能够改变代谢流,抑制目的产物的合成。其次乙酸的积累将消耗本可用于菌体生长和蛋白产物合成的碳源。
采用hplc法测定各重组大肠杆菌发酵液中的乙酸积累量。具体步骤如下:
1、将以下6个甘油菌184-prpdbl21(de3)、184-malkbl21(de3)、duet--prpdi-malkibl21(de3)、duet-g1-prpdc-malkibl21(de3)、duet-g2-prpdi-malkcbl21(de3)、duet-g3-prpdc-malkcbl21(de3)分别接种至5ml含lb培养基的试管中,37℃、220rpm培养12h后转接入含有2.5mg/ml的麦芽糖的tb培养基中,每个样品设置3个平行。同时以大肠杆菌bl21(de3)作为对照(control)。
2、37℃、220rpm进行摇瓶发酵,于发酵培养24h后取2ml发酵液8000rpm离心10min取上清,用0.22μm滤膜过滤备用,得到的滤液加入液相瓶中。采用hplc测定滤液中的乙酸含量。hplc条件如下:流动相为10mmol/lnah2po4水溶液(ph2.98):甲醇=95:5,流速1.0ml/min;检测波长210nm;柱温30℃;进样量10μl。
表1
乙酸含量检测结果如表1所示,从表中可以看出:在同时共表达malk和prpd比单独过表达malk或prpd时乙酸的积累量明显降低,特别是共表达诱导型启动子调控下的malk和组成型启动子调控下的prpd时乙酸积累量降低最显著。
四、重组大肠杆菌atp含量的测定
取步骤三的2中发酵培养24h后的发酵液进行胞内atp含量的测定,具体步骤如下:
1、平板计数:取发酵液100μl,用无菌培养液稀释至1ml,混匀后取10μl再次稀释至1ml,再次混匀后重复取10μl稀释一次,取100μl稀释10倍,取2μl用于涂板。
2、相对光度值(rlu):取发酵液100μl于黑色96孔板中,加入平衡至室温的bac-titer-glo试剂(promega)(bac-titer-glo试剂中含有荧光素酶,此酶只有在atp条件下分解产生荧光信号,细胞中atp含量越高分解产生荧光信号越高,相对吸光度越大)100μl,混匀后反应5min,此时细胞裂解,荧光素酶在atp作用下分解荧光素发光,用多功能酶标仪测560nm下各样品相对光度值(rlu)。
3、atp相对含量的计算:将所测相对光度值除以计数的菌体个数得到单个细胞内atp的相对含量。各样品相对光度值(rlu)的计算结果如图2所示,在同时共表达malk和prpd比单独过表达malk或prpd时atp含量明显升高,特别是共表达诱导型启动子调控下的malk和组成型启动子调控下的prpd时atp含量升高约是单独过表达此两个基因的50倍;共表达诱导型启动子调控下的malk和prpd时atp含量升高约是分别单独过表达此两个基因的30倍。
综上所述,与野生型大肠杆菌相比:分别单独过表达prpd和malk的大肠杆菌的生长速率、atp含量均有提高且乙酸积累量也受到抑制;与分别单独过表达prpd和malk的大肠杆菌相比,同时过表达prpd和malk的大肠杆菌的生长速率、atp含量提高显著,且乙酸积累明显受抑制。
实施例3、pacyc-duet-g1/prpdc-malki重组质粒在提高重组蛋白udp-糖基转移酶76g1表达量中的应用
一、重组质粒pet32a-76g1的构建
将seqidno.9所示的植物来源的udp-糖基转移酶76g1基因插入到表达载体pet32a(北京全式金生物技术有限公司)的ecori与noti酶切位点之间,得到重组质粒pet32a-76g1。
二、重组大肠杆菌pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)的构建
1、将步骤一构建的含目标蛋白的质粒(pet32a-76g1)热转化入bl21-trxb(de3)大肠杆菌(北京全式金生物技术有限公司)感受态细胞,转化液涂布含氨苄青霉素(100ug/ml)的lb平板,挑取单菌落经pcr验证正确后将该重组大肠杆菌制作成感受态细胞。
2、将实施例1中构建的重组质粒pacyc-duet-g1/prpdc-malki热转化入步骤1中获得的重组大肠杆菌感受态细胞,转化液涂布含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml)的lb平板,挑取单菌落经pcr验证,得到可以提高重组udp-糖基转移酶76g1蛋白表达量的重组菌,并将其记作pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)。
三、重组大肠杆菌pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)的发酵培养
1、摇瓶发酵培养
1)挑取重组大肠杆菌pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)单菌落于15mllb液体培养基(含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml))过夜培养,得到种子液。
2)将步骤1)获得的种子液接种到含25mltb液体培养基(含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml))的100ml摇瓶中,37℃、220r/min培养至od600=0.7左右,向摇瓶中添加终浓度0.5mmiptg诱导,27℃、220r/min培养30h。
同时设置不含有pacyc-duet-g1/prpdc-malki重组质粒且含有pet32a-76g1的重组大肠杆菌pet32a-76g1bl21-trxb(de3)作为对照组。
2、udp-糖基转移酶76g1大肠杆菌粗酶液的制备及酶活测定
1)将培养30h后的菌体离心去上清,取菌体沉淀,用磷酸钾缓冲液(100mmol/l,ph7.2)洗涤两次,洗涤后的沉淀悬浮于磷酸钾缓冲液中,超声处理细胞(功率300w,超声5s,间歇5s,共5min),破碎后的液体于8000rpm,4℃离心15min,取上清即为粗酶液。
2)udp-糖基转移酶76g1酶活测定方法如下:甜菊糖为底物酶法合成莱胞迪苷a(reba),根据甜菊糖的转化率判断udp-糖基转移酶76g1酶活。即甜菊糖的转化率越高,udp-糖基转移酶76g1酶活越高。
76g1酶活测定对照组:在反应体系中依次加入1.0g甜菊糖和4.0g蔗糖,取对照组pet32a-76g1bl21-trxb(de3)超声破碎后粗酶液(2mg/ml总蛋白量)及0.1mol/l磷酸缓冲液(ph7.2)至终体积20ml,混合均匀后置于40℃水浴,200rpm搅拌反应24h后加入70%甲醇终止反应。取500μl反应液,置于95℃水浴15min,再12,000rpm离心1min,取上清液过滤膜后用高效液相色谱检测。甜菊糖的转化率为54%。
76g1酶活测定实验组:在反应体系中依次加入1.0g甜菊糖和4.0g蔗糖,取实验组pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)超声破碎后的粗酶液(2mg/ml总蛋白量)及0.1mol/l磷酸缓冲液(ph7.2)至终体积20ml,混合均匀后置于40℃水浴,200rpm搅拌反应24h后加入70%甲醇终止反应。取500μl反应液,置于95℃水浴15min,再12,000rpm离心1min,取上清液过滤膜后用高效液相色谱检测。甜菊糖的转化率为98%。
udp-糖基转移酶76g1酶活测定结果表明:含有pacyc-duet-g1/prpdc-malki重组质粒的菌株发酵酶活(实验组)比不含有pacyc-duet-g1/prpdc-malki重组质粒菌株(对照组)udp-糖基转移76g1酶活提高约80%。从图3的sds-page图中可明显看出蛋白表达量的差异。此结果说明本发明过表达prpd和malk对重组蛋白udp-糖基转移76g1表达量的提高具有显著作用。
实施例4、pacyc-duet-1/prpdi-malki重组质粒在提高重组蛋白udp-糖基转移酶hv1表达量中的应用
一、重组质粒pet32a-hv1的构建
将seqidno.10所示的植物来源的udp-糖基转移酶hv1基因插入到表达载体pet32a(北京全式金生物技术有限公司)的ecori与noti酶切位点之间,得到重组质粒pet32a-hv1。
二、重组大肠杆菌pet32a-hv1pacyc-duet-1/prpdi-malkibl21-trxb(de3)的构建
1、将步骤一构建的含目标蛋白的质粒(pet32a-hv1)热转化入bl21-trxb(de3)大肠杆菌感受态细胞,转化液涂布含氨苄青霉素(100ug/ml)lb平板,挑取单菌落经pcr验证正确后将该重组大肠杆菌制作成感受态细胞。
2、将实施例1中构建的重组质粒pacyc-duet-1/prpdi-malki热转化入步骤1中获得的重组大肠杆菌感受态细胞,转化液涂布含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml)的lb平板,挑取单菌落经pcr验证,得到可以提高重组udp-糖基转移酶hv1蛋白表达量的重组大肠杆菌pet32a-hv1pacyc-duet-1/prpdi-malkibl21-trxb(de3)。
三、重组大肠杆菌pet32a-hv1pacyc-duet-1/prpdi-malkibl21-trxb(de3)的发酵培养
1、摇瓶发酵培养
1)摇瓶发酵
挑取重组大肠杆菌pet32a-hv1pacyc-duet-1/prpdi-malkibl21-trxb(de3)单菌落于15mllb液体培养基(含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml))过夜培养,得到种子液。
2)将步骤1)获得的种子液接种到含25mltb液体培养基(含氨苄青霉素(100ug/ml)和氯霉素(25ug/ml))的100ml摇瓶中,37℃、220r/min培养至od600=0.7左右,向摇瓶中添加终浓度0.5mmiptg诱导,27℃、220r/min培养30h。
同时设置不含有pacyc-duet-1/prpdi-malki重组质粒且含有pet32a-hv1的重组大肠杆菌pet32a-hv1bl21-trxb(de3)作为对照组。
2、udp-糖基转移酶hv1大肠杆菌粗酶液的制备及酶活测定
1)将培养30h后的菌体离心去上清,取菌体沉淀,用磷酸钾缓冲液(100mmol/l,ph7.2)洗涤两次,洗涤后的沉淀悬浮于磷酸钾缓冲液中,超声处理细胞(功率300w,超声5s,间歇5s,共5min),破碎后的液体于8000rpm,4℃离心15min,取上清即为粗酶液。
2)udp-糖基转移酶hv1酶活测定方法如下:莱胞迪苷d(rd)为底物酶法合成莱胞迪苷m(rm),根据甜菊糖的转化率判断udp-糖基转移酶hv1酶活。即莱胞迪苷d的转化率越高,udp-糖基转移酶hv1酶活越高。
hv1酶活测定对照组:在反应体系中依次加入20mm莱胞迪苷d和4.0g蔗糖,取对照组pet32a-hv1bl21-trxb(de3)超声破碎后粗酶液(2mg/ml总蛋白量)及0.1mol/l磷酸缓冲液(ph7.2)至终体积20ml,混合均匀后置于40℃水浴,200rpm搅拌反应24h后加入70%甲醇终止反应。取500μl反应液,置于95℃水浴15min,再12,000rpm离心1min,取上清液过滤膜后用高效液相色谱检测。rd的转化率为28%。
hv1酶活测定实验组:在反应体系中依次加入20mm莱胞迪苷d和4.0g蔗糖,取实验组pet32a-hv1pacyc-duet-1/prpdi-malkibl21-trxb(de3)超声破碎后的粗酶液(2mg/ml总蛋白量)及0.1mol/l磷酸缓冲液(ph7.2)至终体积20ml,混合均匀后置于40℃水浴,200rpm搅拌反应24h后加入70%甲醇终止反应。取500μl反应液,置于95℃水浴15min,再12,000rpm离心1min取上清液过滤膜后用高效液相色谱检测。rd的转化率为86%。
udp-糖基转移酶hv1酶活测定结果表明:含有pacyc-duet-1/prpdi-malki重组质粒的菌株发酵酶活(实验组)比不含有pacyc-duet-1/prpdi-malki重组质粒菌株(对照组)udp-糖基转移hv1酶活提高约200%。从图3的sds-page图中可明显看出蛋白表达量的差异。此结果说明此发明所述方法对重组蛋白udp-糖基转移hv1表达量的提高具有显著作用。
实施例5、pacyc-duet-g1/prpdc-malki重组质粒在提高过氧化氢酶表达量中的应用
一、重组质粒的pet26b-kata的构建
将seqidno.11所示的来源于枯草芽孢杆菌(b.subtilius168)的过氧化氢酶基因kata插入到表达载体pet26b(北京全式金生物技术有限公司)的noci和xhoi酶切位点之间,得到重组质粒pet26b-kata。
二、重组大肠杆菌pet26b-katapacyc-duet-g1/prpdc-malkibl21(de3)的构建
1、将步骤一构建的含目标蛋白的质粒(pet26b-kata)热转化入bl21(de3)大肠杆菌感受态细胞,转化液涂布含卡那霉素(50ug/ml)的lb平板,挑取单菌落经pcr验证正确后将该重组大肠杆菌制作成感受态细胞。
2、将实施例1中构建的重组质粒pacyc-duet-g1/prpdc-malki热转化入步骤1中获得的重组大肠杆菌感受态细胞,转化液涂布含卡那霉素(50ug/ml)和氯霉素(25ug/ml)的lb平板,挑取单菌落经pcr验证,得到可以提高重组过氧化氢酶蛋白表达量的重组菌,并将其记作pet26b-katapacyc-duet-g1/prpdc-malkibl21(de3)。
三、重组大肠杆菌pet32a-76g1pacyc-duet-g1/prpdc-malkibl21-trxb(de3)的发酵培养
1、摇瓶发酵培养
1)挑取pet26b-katapacyc-duet-g1/prpdc-malkibl21(de3)单菌落于15mllb液体培养基(含卡那霉素(50ug/ml)和氯霉素(25ug/ml))过夜培养,得到种子液。
2)将步骤1)获得的种子液接种到含25mltb液体培养基(含卡那霉素(50ug/ml)和氯霉素(25ug/ml))的100ml摇瓶中,37℃、220r/min培养至od600=0.7左右,向摇瓶中添加终浓度0.5mmiptg诱导,27℃、220r/min培养30h。
同时设置不含有pacyc-duet-g1/prpdc-malki重组质粒且含有pet26b-kata的重组大肠杆菌pet26b-katabl21(de3)作为对照组。
2、过氧化氢酶酶活测定
使用分光光度法测定样品酶活。具体方法如下(反应体系为200μl):在37℃条件下将1μl待测酶液样品快速加入199μl含10mmol/lh2o2的kh2po4-k2hpo4缓冲液(50mmol/l,ph7.0)中,用紫外光度计在240nm波长下测定h2o2的分解速率。酶活定义为:37℃下,每分钟分解1μmolh2o2所需的酶量为一个酶活单位。
过氧化氢酶酶活测定结果表明:含有pacyc-duet-g1/prpdc-malki重组质粒的实验组过氧化氢酶酶活比不含有pacyc-duet-g1/prpdc-malki重组质粒的对照组过氧化氢酶酶活提高28.7%(图4)。
实施例6、pacyc-duet-g2/prpdi-malkc重组质粒在提高淀粉酶表达量中的应用
一、重组质粒pet26b-amy的构建
将seqidno.12所示的淀粉酶基因amy插入到表达载体pet-26b的noci和xhoi酶切位点之间,得到重组质粒pet26b-amy。
二、重组大肠杆菌pet26b-amypacyc-duet-g2/prpdi-malkcbl21(de3)的构建
1、将步骤一构建的含目标蛋白的质粒(pet26b-amy)热转化入bl21(de3)大肠杆菌感受态细胞,转化液涂布含卡那霉素(50ug/ml)的lb平板,挑取单菌落经pcr验证正确后将该重组大肠杆菌制作成感受态细胞。
2、将实施例1中构建的重组质粒pacyc-duet-g2/prpdi-malkc热转化入步骤1中的重组大肠杆菌感受态细胞,转化液涂布含卡那霉素(50ug/ml)和氯霉素(25ug/ml)的lb平板,挑取单菌落经pcr验证,得到可以提高重组淀粉酶蛋白表达量的重组菌,并将其记作pet26b-amypacyc-duet-g2/prpdi-malkcbl21(de3)。
三、重组大肠杆菌pet26b-amypacyc-duet-g2/prpdi-malkcbl21(de3)的发酵培养
1、摇瓶发酵培养
1)挑取pet26b-amypacyc-duet-g2/prpdi-malkcbl21(de3)单菌落于15mllb液体培养基(含卡那霉素那霉素(50ug/ml)和氯霉素(25ug/ml))过夜培养,得到种子液。
2)将步骤1)获得的种子液接种到含25mltb液体培养基(含卡那霉素(50ug/ml)和氯霉素(25ug/ml))的100ml摇瓶中,37℃、220r/min培养至od600=0.7左右,向摇瓶中添加终浓度0.5mmiptg诱导,27℃、220r/min培养30h。
同时设置不含有pacyc-duet-g2/prpdi-malkc重组质粒且含有pet26b-amy的重组大肠杆菌pet26b-amybl21(de3)作为对照组。
2、淀粉酶酶活测定
l)吸取浓度为20g/l的可溶性淀粉溶液20ml和磷酸缓冲液(ph=7.0)5ml于试管中。在60℃恒温水浴中预热3-5min。
2)加入稀释好的待测酶液1.00ml(待测酶浓度控制在3.4-4.5u/ml),立即计时,摇匀,准确反应5min。
3)立即吸取1.00ml反应液移入预先装有浓度为0.1mol/l的hcl0.5ml和稀碘液5ml的试管中摇匀。
4)以浓度为0.1mol/lhc10.5ml和稀碘液5ml的混合液作空白对照,于660nm波长下,用10mm比色皿迅速测定吸光度(a)。根据吸光度查表,求得测试酶液的浓度(c)。
酶活力定义及计算方法如下:1ml液体酶,于60℃、ph=6.0条件下,1h液化1g可溶性淀粉,即为1个酶活力单位,以u/ml表示。计算公式如下:x=c*n。式中,x表示样品的酶活力u/ml;c表示测试的酶液浓度u/ml;n表示样品的稀释倍数。
淀粉酶酶活测定结果表明:含有pacyc-g2/prpdi-malkc重组质粒的实验组pet26b-amypacyc-g2/prpdi-malkcbl21(de3)的酶活比不含有pacyc-g2/prpdi-malkc重组质粒的对照组pet26b-amybl21(de3)的酶活提高20%(图5)。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明技术原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>中国科学院天津工业生物技术研究所
<120>一种生物鲁棒性提高的大肠杆菌底盘细胞及其构建方法与应用
<160>14
<170>patentinversion3.5
<210>1
<211>1452
<212>dna
<213>artificialsequence
<400>1
atgtcagctcaaatcaacaacatccgcccggaatttgatcgtgaaatcgttgatatcgtc60
gattacgtcatgaactacgaaatcagctctaaagtggcctacgacaccgcacattactgc120
ctgctcgacacgctcggctgcggtctggaagctctcgaatacccggcctgtaaaaaactg180
ctggggccaattgttcccggcaccgtcgtacccaacggcgtgcgcgtccccggaactcag240
ttccagctcgaccccgtccaggcggcatttaacatcggcgcgatgatccgctggctcgat300
ttcaacgatacctggctggcggcggagtggggccatccttccgacaacctcggcggcatt360
ctggcaacggcggactggctttcgcgcaacgcggtcgccagcggcaaagcgccgttgacc420
atgaaacaggtgctgaccgcaatgatcaaagcccatgaaattcagggctgcatcgcgctg480
gaaaactcctttaaccgcgtcggtctcgaccacgttctgttagtgaaagtggcttccacc540
gccgtggtcgccgaaatgctcggcctgacccgcgaggaaattctcaacgccgtttcgctg600
gcgtgggtggacggtcagtcgctgcgcacctatcgccatgcgccgaacaccggcacgcgt660
aaatcctgggcggcgggcgatgccacttcccgcgcggtacgtctggcactgatggcgaaa720
acgggcgaaatgggttacccgtcagccctgactgcgccggtgtggggcttctacgacgtc780
tcctttaaaggtgaatcgttccgcttccagcgcccgtacggttcctacgttatggaaaat840
gtgctgttcaaaatctccttcccggcggagttccactcccagacggcagttgaagcagcg900
atgacgctctatgaacagatgcaggcagcaggcaaaacggcggcggatatcgaaaaagtg960
accattcgcacccacgaagcctgtattcgcatcatcgacaaaaaagggccgctcaataac1020
ccggcagaccgcgatcactgcattcagtacatggtggcgatcccgctgctattcgggcgc1080
ttaacggcggcagattacgaggacaacgttgcgcaagataaacgcattgacgccctgcgc1140
gagaagatcaattgctttgaagatccggcacttaccgctgactaccacgacccggaaaaa1200
cgcgccatcgccaatgccattacccttgagttcaccgacggcacacgatttgaagaagtg1260
gtggtggagtaccccattggtcatgctcgccgccgtcaggatggtattccgaaactggtc1320
gataaattcaaaatcaatctcgcgcgccagttcccgactcgccagcagcagcgcattctg1380
gaggtttctctcgacagagctcgcctggaacagatgccggtcaatgagtatctcgacctg1440
tacgtcatttaa1452
<210>2
<211>1493
<212>dna
<213>artificialsequence
<400>2
aggagatataccatgggcagcatgtcagctcaaatcaacaacatccgcccggaatttgat60
cgtgaaatcgttgatatcgtcgattacgtcatgaactacgaaatcagctctaaagtggcc120
tacgacaccgcacattactgcctgctcgacacgctcggctgcggtctggaagctctcgaa180
tacccggcctgtaaaaaactgctggggccaattgttcccggcaccgtcgtacccaacggc240
gtgcgcgtccccggaactcagttccagctcgaccccgtccaggcggcatttaacatcggc300
gcgatgatccgctggctcgatttcaacgatacctggctggcggcggagtggggccatcct360
tccgacaacctcggcggcattctggcaacggcggactggctttcgcgcaacgcggtcgcc420
agcggcaaagcgccgttgaccatgaaacaggtgctgaccgcaatgatcaaagcccatgaa480
attcagggctgcatcgcgctggaaaactcctttaaccgcgtcggtctcgaccacgttctg540
ttagtgaaagtggcttccaccgccgtggtcgccgaaatgctcggcctgacccgcgaggaa600
attctcaacgccgtttcgctggcgtgggtggacggtcagtcgctgcgcacctatcgccat660
gcgccgaacaccggcacgcgtaaatcctgggcggcgggcgatgccacttcccgcgcggta720
cgtctggcactgatggcgaaaacgggcgaaatgggttacccgtcagccctgactgcgccg780
gtgtggggcttctacgacgtctcctttaaaggtgaatcgttccgcttccagcgcccgtac840
ggttcctacgttatggaaaatgtgctgttcaaaatctccttcccggcggagttccactcc900
cagacggcagttgaagcagcgatgacgctctatgaacagatgcaggcagcaggcaaaacg960
gcggcggatatcgaaaaagtgaccattcgcacccacgaagcctgtattcgcatcatcgac1020
aaaaaagggccgctcaataacccggcagaccgcgatcactgcattcagtacatggtggcg1080
atcccgctgctattcgggcgcttaacggcggcagattacgaggacaacgttgcgcaagat1140
aaacgcattgacgccctgcgcgagaagatcaattgctttgaagatccggcacttaccgct1200
gactaccacgacccggaaaaacgcgccatcgccaatgccattacccttgagttcaccgac1260
ggcacacgatttgaagaagtggtggtggagtaccccattggtcatgctcgccgccgtcag1320
gatggtattccgaaactggtcgataaattcaaaatcaatctcgcgcgccagttcccgact1380
cgccagcagcagcgcattctggaggtttctctcgacagagctcgcctggaacagatgccg1440
gtcaatgagtatctcgacctgtacgtcatttaatgactcgctaacggattcac1493
<210>3
<211>1494
<212>dna
<213>artificialsequence
<400>3
aggagatataccatgggcagcatgtcagctcaaatcaacaacatccgcccggaatttgat60
cgtgaaatcgttgatatcgtcgattacgtcatgaactacgaaatcagctctaaagtggcc120
tacgacaccgcacattactgcctgctcgacacgctcggctgcggtctggaagctctcgaa180
tacccggcctgtaaaaaactgctggggccaattgttcccggcaccgtcgtacccaacggc240
gtgcgcgtccccggaactcagttccagctcgaccccgtccaggcggcatttaacatcggc300
gcgatgatccgctggctcgatttcaacgatacctggctggcggcggagtggggccatcct360
tccgacaacctcggcggcattctggcaacggcggactggctttcgcgcaacgcggtcgcc420
agcggcaaagcgccgttgaccatgaaacaggtgctgaccgcaatgatcaaagcccatgaa480
attcagggctgcatcgcgctggaaaactcctttaaccgcgtcggtctcgaccacgttctg540
ttagtgaaagtggcttccaccgccgtggtcgccgaaatgctcggcctgacccgcgaggaa600
attctcaacgccgtttcgctggcgtgggtggacggtcagtcgctgcgcacctatcgccat660
gcgccgaacaccggcacgcgtaaatcctgggcggcgggcgatgccacttcccgcgcggta720
cgtctggcactgatggcgaaaacgggcgaaatgggttacccgtcagccctgactgcgccg780
gtgtggggcttctacgacgtctcctttaaaggtgaatcgttccgcttccagcgcccgtac840
ggttcctacgttatggaaaatgtgctgttcaaaatctccttcccggcggagttccactcc900
cagacggcagttgaagcagcgatgacgctctatgaacagatgcaggcagcaggcaaaacg960
gcggcggatatcgaaaaagtgaccattcgcacccacgaagcctgtattcgcatcatcgac1020
aaaaaagggccgctcaataacccggcagaccgcgatcactgcattcagtacatggtggcg1080
atcccgctgctattcgggcgcttaacggcggcagattacgaggacaacgttgcgcaagat1140
aaacgcattgacgccctgcgcgagaagatcaattgctttgaagatccggcacttaccgct1200
gactaccacgacccggaaaaacgcgccatcgccaatgccattacccttgagttcaccgac1260
ggcacacgatttgaagaagtggtggtggagtaccccattggtcatgctcgccgccgtcag1320
gatggtattccgaaactggtcgataaattcaaaatcaatctcgcgcgccagttcccgact1380
cgccagcagcagcgcattctggaggtttctctcgacagagctcgcctggaacagatgccg1440
gtcaatgagtatctcgacctgtacgtcatttaacatcatcaccacagccaggat1494
<210>4
<211>1116
<212>dna
<213>artificialsequence
<400>4
atggcgagcgtacagctgcaaaatgtaacgaaagcctggggcgaggtcgtggtatcgaaa60
gatatcaatctcgatatccatgaaggtgaattcgtggtgtttgtcggaccgtctggctgc120
ggtaaatcgactttactgcgcatgattgccgggcttgagacgatcaccagcggcgacctg180
ttcatcggtgagaaacggatgaatgacactccgccagcagaacgcggcgttggtatggtg240
tttcagtcttacgcgctctatccccacctgtcagtagcagaaaacatgtcatttggcctg300
aaactggctggcgcaaaaaaagaggtgattaaccaacgcgttaaccaggtggcggaagtg360
ctacaactggcgcatttgctggatcgcaaaccgaaagcgctctccggtggtcagcgtcag420
cgtgtggcgattggccgtacgctggtggccgagccaagcgtatttttgctcgatgaaccg480
ctctccaacctcgatgctgcactgcgtgtgcaaatgcgtatcgaaatctcccgtctgcat540
aaacgcctgggccgcacaatgatttacgtcacccacgatcaggtcgaagcgatgacgctg600
gccgacaaaatcgtggtgctggacgccggtcgcgtggcgcaggttgggaaaccgctggag660
ctgtaccactatccggcagaccgttttgtcgccggatttatcggttcgccaaagatgaac720
ttcctgccggtaaaagtgaccgccaccgcaatcgatcaagtgcaggtggagctgccgatg780
ccaaatcgtcagcaagtctggctgccagttgaaagccgtgatgtccaggttggagccaat840
atgtcgctgggtattcgcccggaacatctactgccgagtgatatcgctgacgtcatcctt900
gagggtgaagttcaggtcgtcgagcaactcggcaacgaaactcaaatccatatccagatc960
ccttccattcgtcaaaacctggtgtaccgccagaacgacgtggtgttggtagaagaaggt1020
gccacattcgctatcggcctgccgccagagcgttgccatctgttccgtgaggatggcact1080
gcatgtcgtcgactgcataaggagccgggcgtttaa1116
<210>5
<211>1152
<212>dna
<213>artificialsequence
<400>5
cagtcaggcaccgtgtatggcgagcgtacagctgcaaaatgtaacgaaagcctggggcga60
ggtcgtggtatcgaaagatatcaatctcgatatccatgaaggtgaattcgtggtgtttgt120
cggaccgtctggctgcggtaaatcgactttactgcgcatgattgccgggcttgagacgat180
caccagcggcgacctgttcatcggtgagaaacggatgaatgacactccgccagcagaacg240
cggcgttggtatggtgtttcagtcttacgcgctctatccccacctgtcagtagcagaaaa300
catgtcatttggcctgaaactggctggcgcaaaaaaagaggtgattaaccaacgcgttaa360
ccaggtggcggaagtgctacaactggcgcatttgctggatcgcaaaccgaaagcgctctc420
cggtggtcagcgtcagcgtgtggcgattggccgtacgctggtggccgagccaagcgtatt480
tttgctcgatgaaccgctctccaacctcgatgctgcactgcgtgtgcaaatgcgtatcga540
aatctcccgtctgcataaacgcctgggccgcacaatgatttacgtcacccacgatcaggt600
cgaagcgatgacgctggccgacaaaatcgtggtgctggacgccggtcgcgtggcgcaggt660
tgggaaaccgctggagctgtaccactatccggcagaccgttttgtcgccggatttatcgg720
ttcgccaaagatgaacttcctgccggtaaaagtgaccgccaccgcaatcgatcaagtgca780
ggtggagctgccgatgccaaatcgtcagcaagtctggctgccagttgaaagccgtgatgt840
ccaggttggagccaatatgtcgctgggtattcgcccggaacatctactgccgagtgatat900
cgctgacgtcatccttgagggtgaagttcaggtcgtcgagcaactcggcaacgaaactca960
aatccatatccagatcccttccattcgtcaaaacctggtgtaccgccagaacgacgtggt1020
gttggtagaagaaggtgccacattcgctatcggcctgccgccagagcgttgccatctgtt1080
ccgtgaggatggcactgcatgtcgtcgactgcataaggagccgggcgtttaagagcgatt1140
gcctaagtggtg1152
<210>6
<211>1156
<212>dna
<213>artificialsequence
<400>6
ggcagatctcaattggatatcgatggcgagcgtacagctgcaaaatgtaacgaaagcctg60
gggcgaggtcgtggtatcgaaagatatcaatctcgatatccatgaaggtgaattcgtggt120
gtttgtcggaccgtctggctgcggtaaatcgactttactgcgcatgattgccgggcttga180
gacgatcaccagcggcgacctgttcatcggtgagaaacggatgaatgacactccgccagc240
agaacgcggcgttggtatggtgtttcagtcttacgcgctctatccccacctgtcagtagc300
agaaaacatgtcatttggcctgaaactggctggcgcaaaaaaagaggtgattaaccaacg360
cgttaaccaggtggcggaagtgctacaactggcgcatttgctggatcgcaaaccgaaagc420
gctctccggtggtcagcgtcagcgtgtggcgattggccgtacgctggtggccgagccaag480
cgtatttttgctcgatgaaccgctctccaacctcgatgctgcactgcgtgtgcaaatgcg540
tatcgaaatctcccgtctgcataaacgcctgggccgcacaatgatttacgtcacccacga600
tcaggtcgaagcgatgacgctggccgacaaaatcgtggtgctggacgccggtcgcgtggc660
gcaggttgggaaaccgctggagctgtaccactatccggcagaccgttttgtcgccggatt720
tatcggttcgccaaagatgaacttcctgccggtaaaagtgaccgccaccgcaatcgatca780
agtgcaggtggagctgccgatgccaaatcgtcagcaagtctggctgccagttgaaagccg840
tgatgtccaggttggagccaatatgtcgctgggtattcgcccggaacatctactgccgag900
tgatatcgctgacgtcatccttgagggtgaagttcaggtcgtcgagcaactcggcaacga960
aactcaaatccatatccagatcccttccattcgtcaaaacctggtgtaccgccagaacga1020
cgtggtgttggtagaagaaggtgccacattcgctatcggcctgccgccagagcgttgcca1080
tctgttccgtgaggatggcactgcatgtcgtcgactgcataaggagccgggcgtttaata1140
cctgagcagatgatcg1156
<210>7
<211>120
<212>dna
<213>artificialsequence
<400>7
ctgaagtcagccccatacgatataagttgtaattctcatgtttgacagcttatcatcgat60
aagctttaatgcggtagtttatcacagttaaattgctaacgcagtcaggcaccgtgtatg120
<210>8
<211>116
<212>dna
<213>artificialsequence
<400>8
cgactcctgcattaggaaattaatacgactcactataggggaattgtgagcggataacaa60
ttcccctgtagaaataattttgtttaactttaataaggagatataccatgggcagc116
<210>9
<211>1377
<212>dna
<213>artificialsequence
<400>9
atggaaaataaaacggagaccaccgttcgccggcgccggagaataatattattcccagta60
ccatttcaaggccacattaacccaattcttcagttggccaatgtgttgtactctaaagga120
ttcagtatcaccatctttcacaccaacttcaacaaacccaaaacatctaattaccctcac180
ttcactttcagattcatccttgacaacgacccacaagacgaacgcatttccaatttgcca240
actcatggtccacttgctggtatgcggattccaattatcaacgaacacggagctgacgaa300
ttacgtcgcgaactggaactgttgatgttagcttctgaagaagatgaagaggtatcgtgt360
ttaatcacggatgctctttggtacttcgctcaatctgttgctgacagtcttaaccttcgt420
cggcttgttttgatgacaagcagcttgtttaattttcatgcattggtttcacttcctcag480
tttgatgagcttggttaccttgatcctgatgacaaaacccgtttggaagaacaagctagt540
ggatttcctatgttgaaagtgaaagacatcaagtctgcttattcgaactggcaaatactt600
aaagagatattaggaaagatgataaaacaaacaaaagcatcttcaggagtcatctggaac660
tcatttaaggaacttgaagagtctgagcttgaaactgttatccgtgagatcccagctcca720
agtttcttgataccacttcccaagcatttgacagcctcttccagcagcttattggaccac780
gatcgtaccgtttttcaatggttagaccaacaaccaccaagttcggtactgtatgttagt840
tttggtagtagaagtgaagtggatgagaaagatttcttggaaatagctcgtggattggtt900
gatagcaagcagtcgtttttatgggtggttcgtcctggatttgtcaagggttcgacgtgg960
gtcgaaccattgccagatggattcttgggtgaaagaggacgtattgtgaaatgggttcca1020
cagcaagaagtgttggctcatggagcaataggcgcattctggactcatagcggatggaac1080
tctacgttggaaagcgtttgtgaaggtgttcctatgattttctcggattttggacttgat1140
caaccattgaatgctagatacatgagtgatgttttgaaggtaggagtgtatttggaaaat1200
ggatgggaaagaggagagatagcaaatgcaataagaagagttatggtggatgaagaagga1260
gaatacattagacagaatgcaagagttttgaaacaaaaggcagatgtttctttgatgaag1320
ggtggttcgtcttacgaatcattagagtctttggtttcttacatttcatcgttgtaa1377
<210>10
<211>1377
<212>dna
<213>artificialsequence
<400>10
atggacggtaactcttcctcttctccattgcacgttgttatctgtccatggttggctttg60
ggtcacttgttgccatgtttggacattgctgagagattggcttccagaggtcacagagtt120
tctttcgtgtccactccaagaaacattgccagattgccaccattgaggccagctgttgct180
ccattggttgatttcgttgctttgccattgccacacgttgacggtttgccagaaggtgct240
gaatccactaacgatgtcccatacgacaagttcgagttgcacagaaaggctttcgatggt300
ttggctgctccattctccgagtttttgagagctgcttgtgctgaaggtgcaggttctaga360
cctgactggttgatcgttgatactttccatcattgggctgctgctgccgctgttgaaaac420
aaggttccatgtgtcatgttgttgttgggtgctgctactgttatcgctggtttcgctaga480
ggtgtttctgaacatgctgcagctgccgtcggtaaagaaagaccagctgctgaagctcca540
tctttcgagactgagagaagaaagctgatgactactcagaacgcttccggtatgactgtt600
gccgagagatacttcttgaccctgatgagatctgacttggtcgccattagatcctgtgct660
gaatgggaaccagaatccgttgctgctttgactactttggctggtaagccagttgtccca720
ttgggtttgttaccaccatcacctgaaggtggtagaggtgtctctaaagaggatgctgct780
gttagatggttggacgctcaaccagctaagtccgttgtttacgttgccttgggttctgag840
gttccattgagagccgaacaagttcacgaattggctcttggtttggagttgtccggtgct900
agatttttgtgggccttgagaaagccaactgatgctccagatgctgcagttttgccacca960
ggtttcgaagagagaactagaggtagaggtttggttgttaccggttgggttccacagatt1020
ggtgttttggctcatggtgctgttgctgcctttttgactcattgtggttggaactccacc1080
atcgagggtttgttgtttggtcacccactgattatgctgccaatttcctctgaccaaggt1140
ccaaacgccagattgatggaaggtagaaaggttggtatgcaggttccaagagatgaatcc1200
gacggttcctttagaagagaggatgttgccgctaccgttagagctgttgcagttgaagaa1260
gatggtcgtagagttttcactgccaacgccaagaagatgcaagagatcgttgctgatggt1320
gcctgtcacgagagatgcattgatggtttcatccagcagctgagatcctacaaggct1377
<210>11
<211>1452
<212>dna
<213>artificialsequence
<400>11
atgagttcaaataaactgacaactagctggggcgctccggttggagataatcaaaactca60
atgactgccggttctcgcggaccaactttaattcaagatgtacatttactcgaaaaattg120
gcccatttcaaccgagaacgtgttcctgaacgtgttgttcacgccaaaggagcaggcgca180
cacggatattttgaagtgacaaacgacgtaacaaaatacacgaaagccgctttcctttct240
gaagtcggcaaacgcacaccgttgttcatccgtttctcaacagttgccggtgaacttggc300
tctgctgacacagttcgcgacccgcgcggatttgctgttaaattttatactgaagaagga360
aactacgacatcgtcggcaacaatacgcctgtattctttatccgcgatgcgattaagttc420
cctgatttcatccatacacaaaaaagagatccaaaaacacacctgaaaaaccctacggct480
gtatgggatttctggtcactttcaccagagtcactgcaccaagtgacaatcctgatgtct540
gaccgcggaattcctgcgacacttcgccacatgcacggcttcggaagccatacattcaaa600
tggacaaatgccgaaggcgaaggcgtatggattaaatatcactttaaaacagaacaaggc660
gtgaaaaaccttgatgtcaatacggcagcaaaaattgccggtgaaaaccctgattaccat720
acagaagaccttttcaacgcaatcgaaaacggtgattatcctgcatggaaactatatgtg780
caaatcatgcctttagaagatgcaaatacgtaccgtttcgatccgtttgatgtcacaaaa840
gtttggtctcaaaaagactacccgttaatcgaggtcggacgcatggttctagacagaaat900
ccggaaaactactttgcagaggtagaacaagcgacattttcacctggaaccctcgtgcct960
ggtattgatgtttcaccggataaaatgcttcaaggtcgactttttgcttatcatgatgca1020
caccgctaccgtgtcggtgcaaaccatcaagcgctgccaatcaaccgcgcacgcaacaaa1080
gtaaacaattatcagcgtgatgggcaaatgcgttttgatgataacggcggcggatctgtg1140
tattacgagcctaacagcttcggcggtccaaaagagtcacctgaggataagcaagcagca1200
tatccggtacaaggtatcgctgacagcgtaagctacgatcactacgatcactacactcaa1260
gccggcgatctgtatcgtttaatgagtgaagatgaacgtacccgccttgttgaaaatatc1320
gttaatgccatgaagccggtagaaaaagaagaaatcaagctgcgccaaatcgagcacttc1380
tacaaagcggatcctgaatacggaaaacgcgtggcagaaggccttggattgccgattaaa1440
aaagattcttaa1452
<210>12
<211>1461
<212>dna
<213>artificialsequence
<400>12
atgcatcataatggaacaaatggtactatgatgcaatatttcgaatggtatttgccaaat60
gacgggaatcattggaacaggttgagggatgacgcagctaacttaaagagtaaagggata120
acagctgtatggatcccacctgcatggaaggggacttcccagaatgatgtaggttatgga180
gcctatgatttatatgatcttggagagtttaaccagaaggggacggttcgtacaaaatat240
ggaacacgcaaccagctacaggctgcggtgacctctttaaaaaataacggcattcaggta300
tatggtgatgtcgtcatgaatcataaaggtggagcagatggtacggaaattgtaaatgcg360
gtagaagtgaatcggagcaaccgaaaccaggaaacctcaggagagtatgcaatagaagcg420
tggacaaagtttgattttcctggaagaggaaataaccattccagctttaagtggcgctgg480
tatcattttgatgggacagattgggatcagtcacgccagcttcaaaacaaaatatataaa540
ttcaggggaacaggcaaggcctgggactgggaagtcgatacagagaatggcaactatgac600
tatcttatgtatgcagacgtggatatggatcacccagaagtaatacatgaacttagaaac660
tggggagtgtggtatacgaatacactgaaccttgatggatttagaatagatgcagtgaaa720
catataaaatatagctttacgagagattggcttacacatgtgcgtaacaccacaggtaaa780
ccaatgtttgcagtggctgagttttggaaaaatgaccttggtgcaattgaaaactatttg840
aataaaacaagttggaatcactcggtgtttgatgttcctctccactataatttgtacaat900
gcatctaatagcggtggttattatgatatgagaaatattttaaatggttctgtggtgcaa960
aaacatccaacacatgccgttacttttgttgataaccatgattctcagcccggggaagca1020
ttggaatcctttgttcaacaatggtttaaaccacttgcatatgcattggttctgacaagg1080
gaacaaggttatccttccgtattttatggggattactacggtatcccaacccatggtgtt1140
ccggctatgaaatctaaaatagaccctcttctgcaggcacgtcaaacttttgcctatggt1200
acgcagcatgattactttgatcatcatgatattatcggttggacaagagagggaaatagc1260
tcccatccaaattcaggccttgccaccattatgtcagatggtccaggtggtaacaaatgg1320
atgtatgtggggaaaaataaagcgggacaagtttggagagatattaccggaaataggaca1380
ggcaccgtcacaattaatgcagacggatggggtaatttctctgttaatggagggtccgtt1440
tcggtttgggtgaagcaataa1461
<210>13
<211>483
<212>prt
<213>artificialsequence
<400>13
metseralaglnileasnasnileargproglupheasparggluile
151015
valaspilevalasptyrvalmetasntyrgluileserserlysval
202530
alatyraspthralahistyrcysleuleuaspthrleuglycysgly
354045
leuglualaleuglutyrproalacyslyslysleuleuglyproile
505560
valproglythrvalvalproasnglyvalargvalproglythrgln
65707580
pheglnleuaspprovalglnalaalapheasnileglyalametile
859095
argtrpleuasppheasnaspthrtrpleualaalaglutrpglyhis
100105110
proseraspasnleuglyglyileleualathralaasptrpleuser
115120125
argasnalavalalaserglylysalaproleuthrmetlysglnval
130135140
leuthralametilelysalahisgluileglnglycysilealaleu
145150155160
gluasnserpheasnargvalglyleuasphisvalleuleuvallys
165170175
valalaserthralavalvalalaglumetleuglyleuthrargglu
180185190
gluileleuasnalavalserleualatrpvalaspglyglnserleu
195200205
argthrtyrarghisalaproasnthrglythrarglyssertrpala
210215220
alaglyaspalathrserargalavalargleualaleumetalalys
225230235240
thrglyglumetglytyrproseralaleuthralaprovaltrpgly
245250255
phetyraspvalserphelysglygluserpheargpheglnargpro
260265270
tyrglysertyrvalmetgluasnvalleuphelysileserphepro
275280285
alagluphehisserglnthralavalglualaalametthrleutyr
290295300
gluglnmetglnalaalaglylysthralaalaaspileglulysval
305310315320
thrileargthrhisglualacysileargileileasplyslysgly
325330335
proleuasnasnproalaaspargasphiscysileglntyrmetval
340345350
alaileproleuleupheglyargleuthralaalaasptyrgluasp
355360365
asnvalalaglnasplysargileaspalaleuargglulysileasn
370375380
cysphegluaspproalaleuthralaasptyrhisaspproglulys
385390395400
argalailealaasnalailethrleugluphethraspglythrarg
405410415
pheglugluvalvalvalglutyrproileglyhisalaargargarg
420425430
glnaspglyileprolysleuvalasplysphelysileasnleuala
435440445
argglnpheprothrargglnglnglnargileleugluvalserleu
450455460
aspargalaargleugluglnmetprovalasnglutyrleuaspleu
465470475480
tyrvalile
<210>14
<211>371
<212>prt
<213>artificialsequence
<400>14
metalaservalglnleuglnasnvalthrlysalatrpglygluval
151015
valvalserlysaspileasnleuaspilehisgluglyglupheval
202530
valphevalglyproserglycysglylysserthrleuleuargmet
354045
ilealaglyleugluthrilethrserglyaspleupheileglyglu
505560
lysargmetasnaspthrproproalagluargglyvalglymetval
65707580
pheglnsertyralaleutyrprohisleuservalalagluasnmet
859095
serpheglyleulysleualaglyalalyslysgluvalileasngln
100105110
argvalasnglnvalalagluvalleuglnleualahisleuleuasp
115120125
arglysprolysalaleuserglyglyglnargglnargvalalaile
130135140
glyargthrleuvalalagluproservalpheleuleuaspglupro
145150155160
leuserasnleuaspalaalaleuargvalglnmetargilegluile
165170175
serargleuhislysargleuglyargthrmetiletyrvalthrhis
180185190
aspglnvalglualametthrleualaasplysilevalvalleuasp
195200205
alaglyargvalalaglnvalglylysproleugluleutyrhistyr
210215220
proalaaspargphevalalaglypheileglyserprolysmetasn
225230235240
pheleuprovallysvalthralathralaileaspglnvalglnval
245250255
gluleuprometproasnargglnglnvaltrpleuprovalgluser
260265270
argaspvalglnvalglyalaasnmetserleuglyileargproglu
275280285
hisleuleuproseraspilealaaspvalileleugluglygluval
290295300
glnvalvalgluglnleuglyasngluthrglnilehisileglnile
305310315320
proserileargglnasnleuvaltyrargglnasnaspvalvalleu
325330335
valglugluglyalathrphealaileglyleuproprogluargcys
340345350
hisleuphearggluaspglythralacysargargleuhislysglu
355360365
proglyval
370
- 还没有人留言评论。精彩留言会获得点赞!