一种调控油菜菌核病抗性的蛋白质及其应用
本发明涉及生物基因工程
技术领域:
,特别是涉及一种调控油菜菌核病抗性的蛋白质及其应用。
背景技术:
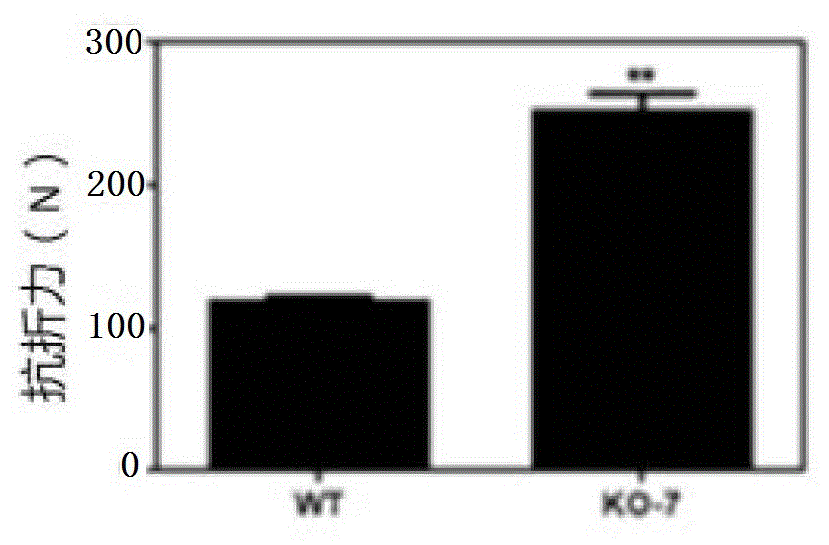
:油菜是世界上食用植物油和植物蛋白的主要来源之一,我国是世界上最大的油菜生产国,自2000年以来年均种植面积已达到1.1亿亩,每年可生产约450万吨菜籽油,面积和总产均占世界四分之一。虽然杂交油菜和双低油菜品种的选育以及大面积推广显著提高了油菜的产量和品质,但油菜菌核病和倒伏等影响增产潜力的因素仍然存在。油菜菌核病是由真菌核盘菌(sclerotiniasclerotiorum(lib)debary)侵染引起的一种世界性病害,由于菌核病菌属广谱性非转化病原物,除了少数品种表现一定程度地抗性,至今尚未找到有效的抗源。现有的农药和栽培措施只能起到抑制作用。倒伏是影响油菜高产优质以及机械化收割的另一重要因素。油菜倒伏后不仅直接导致减产,还会导致含油量降低,并且倒伏后更易于病菌的侵染和扩展。虽然矮化育种已被广泛应用于改良小麦和水稻等作物的抗倒伏性(hedden,2003),但属于无限花序的油菜如果矮化,可能引起结荚数减少从而导致产量下降(muangprometal,2004)。现有技术证明植物木质素含量与植物抗倒伏能力和抗病性有一定相关性,但目前多着重于从栽培管理及植株形态等方面来提高油菜的抗倒伏和抗病性,并没有关于木质素单体含量与植物抗倒伏和抗病性的深入研究报道。技术实现要素:为了解决上述问题,本发明提供了一种调控油菜菌核病抗性的蛋白质及其应用,所述蛋白质能够提高油菜对菌核病的抗病性和抗倒伏性。为了实现上述目的,本发明提供如下技术方案:本发明提供了一种调控油菜菌核病抗性的蛋白质,所述蛋白质的氨基酸序列如:seqidno.1、seqidno.2、seqidno.3或seqidno.4所示。优选的,编码所述蛋白质的基因核苷酸序列如:seqidno.5、seqidno.6、seqidno.7或seqidno.8所示。本发明提供了一种表达上述蛋白质的重组载体,所述重组载体的基础质粒包括pcambia3301。优选的,在所述基础质粒的ecori和hindiii酶切位点处插入编码所述蛋白质的基因。本发明提供了一种包含上述重组表达载体的重组菌。本发明提供了上述蛋白质或上述重组载体或上述重组菌在油菜抗病性育种中的应用。优选的,所述油菜包括甘蓝型油菜。本发明提供了一种改良油菜抗病性的试剂,所述试剂包括上述蛋白质或上述重组载体或上述重组菌。本发明提供了上述试剂在油菜抗病性育种中的应用,所述油菜包括甘蓝型油菜。有益效果:本发明提供了一种调控油菜菌核病抗性的蛋白质,所述蛋白质的氨基酸序列如seqidno.1、seqidno.2、seqidno.3或seqidno.4所示。本发明所述蛋白质,可提高油菜对核盘菌病和抗倒伏的抗性,能够应用于油菜的抗病育种中。实验结果表明:本发明通过基因敲除阿魏酸5-羟化酶(bnf5h)得到获得s型木质素含量明显低于野生型的转基因油菜植株,即s/g含量明显低于野生型;幼苗期离体叶片及成熟期茎秆接种鉴定结果表明,bnf5h基因编辑获得的植株增加了油菜对菌核病的抗性以及抗倒伏能力。本发明为油菜抗性育种提供了一种新选择。附图说明图1为bnf5h家族成员在甘蓝型油菜不同组织器官中的表达,其中root为根,stem为茎,leaf为叶,silique为种子,seed为种子,relativeexpressionlevel为相对表达水平;图2为甘蓝型油菜bnf5h敲除载体的构建,a中m为trans2kdnaladder,ck1为未经酶切的crispr/cas9质粒,1为开环crispr/cas9载体,b中m为trans2kplusdnaladder,1~24依次为单克隆菌液检测(开环cas9载体的胶回收产物与接头磷酸化处理后的grna用quickligase进行快速连接,并转化大肠杆菌dh5α,将得到的菌液富集后涂平板(amp),然后挑取过夜培养的单克隆菌落摇菌,用m13-20+grna-r进行菌液pcr检测),c中m为trans2kdnaladder,ck2为未双酶切的目的片段质粒,1、2为酶切处理过的目的片段质粒,ck3为未双酶切的pcambia3301表达载体,3、4为酶切后的表达载体,d中m为trans2kplusdnaladder,m左侧为引物m13-20+grna-r农杆菌的菌检结果,m右侧为fbar+rbar的菌检结果;图3为再生植株基因组dna提取,其中ck为野生型westar对照,1~23为转基因植株,m为trans2kdnaladder;图4为转基因植株的阳性鉴定,a为检测引物组合m13-20+grna-r,ck为野生型westar对照,m为trans2kplusdnaladder,1~23依次为被检测的株系,即23棵阳性植物,b为检测引物组合fbar-s+rbar-s,ck为野生型westar对照,m为trans2kplusdnaladder,1~23依次为被检测的株系,即23棵阳性植物,c为检测引物组合fbar+rbar,ck为野生型westar对照,m为trans2kplusdnaladder,1~23依次为被检测的株系,即23棵阳性植物;图5为crispr-cas9敲除转基因植株的鉴定及编辑结果的统计图;图6为bnf5h的测序峰图,其中a、d和g分别为野生型westar中bnf5h-4、bnf5h-6、bnf5h-7基因的测序峰图,b和c为转基因植株bnf5h-4的测序峰图;e和f为转基因植株bnf5h-6的测序峰图,h为转基因植株bnf5h-7的测序峰图;其中红色框内为grna序列,红色箭头指向突变位点;图7为转基因植株t2代和野生型对照菌核病抗性鉴定,其中(a)和(b)为叶片菌斑面积及柱形图,(a)和(b)中wt为野生型,ko-7为bnf5h-7敲除转基因阳性植株,(c)和(d)为茎秆菌斑长度及柱形图,(c)中wt为野生型,ko-7为bnf5h-7敲除转基因阳性植株,(d)中wt为野生型,ko-7为bnf5h-7敲除转基因阳性植株;(e)~(j)依次为接种在叶片上的菌斑及菌丝,(e)(f)(g)中wt为野生型,(h)(i)(j)为bnf5h敲除转基因阳性植株;图8为wt和bnf5h基因敲除植株不同部位横切面maule染色结果,wt为野生型,ko-7为bnf5h-7敲除转基因阳性植株;xy是木质部,ve是木质部导管,p是phloem韧皮部,ep是epidermis为表皮,if是interfascicularfibers为维管束间纤维,endocarp为内果皮;r是replum为胚座框;图9为全细胞壁样品及木质素样品中木质素各单体之间的含量图,其中a为木质素单体含量的测定,d为利用gc-ms/ms分析木质素单体含量的化学式,b和c为wt、ko-7全细胞壁样品的2d-hsqc-nmr测定,e和f为wt、ko-7酶木质素样品的2d-hsqc-nmr;全细胞壁光谱为thewholecellwallspectra(wcw),酶木质素核磁共振谱为theenzymelignin(el)nmrspectra;dfrcmonomeryields为木质素单体含量的测定采用优化dfrc协议中所述进行dfrc分析,并表示为细胞壁残留物的百分比(%cwr),crisprf5hline7为bnf5h-7敲除转基因阳性植株ko-7,wtcanolael为用酶木质素核磁共振谱分析野生型油菜,ko-7f5hel为用酶木质素核磁共振谱分析bnf5h-7敲除转基因阳性植株ko-7;图10为bnf5h基因编辑和野生型植株t2代抗折力测定。具体实施方式如无特殊要求,本发明所使用的试剂为本领域技术人员常规购买所得。本发明提供了一种调控油菜菌核病抗性的蛋白质,所述蛋白质的氨基酸序列如seqidno.1、seqidno.2、seqidno.3或seqidno.4所示。在本发明中,所述蛋白质优选通过基因编辑的方法筛选出来。在本发明中,调控s木质素的基因优选包括bnf5h-1、bnf5h-4、bnf5h-6和bnf5h-7;其中bnf5h-1的核苷酸序列优选如seqidno.5所示;所述核苷酸编码的氨基酸序列为seqidno.1。在本发明中,bnf5h-4的核苷酸序列优选如seqidno.6所示;所述核苷酸编码的氨基酸序列为seqidno.2。在本发明中,bnf5h-6的核苷酸序列优选如seqidno.7所示;所述核苷酸编码的氨基酸序列为seqidno.3。在本发明中,bnf5h-7的核苷酸序列优选如seqidno.5所示;所述核苷酸编码的氨基酸序列为seqidno.4。本发明提供了一种表达上述蛋白质的重组载体,所述重组载体的基础质粒包括pcambia3301。本发明优选在所述基础质粒的ecori和hindiii酶切位点处插入编码所述蛋白质的基因。在本发明中,所述ecori和hindiii优选购自于thermoscientific生物公司。本发明提供了一种包含上述重组表达载体的重组菌。在本发明中,所述重组菌的基础菌优选为大肠杆菌dh5α。本发明所述蛋白能够用于油菜抗病性育种,本发明通过敲除bnf5h基因获得s型木质素含量明显低于野生型的转基因油菜植株,即s/g含量明显低于野生型对照,明显增加了油菜植株对菌核病的抗性以及抗倒伏能力。本发明提供了上述蛋白质或上述重组载体或上述重组菌在油菜抗病性育种中的应用。在本发明中,所述油菜优选为甘蓝型油菜。本发明提供了一种改良油菜抗病性的试剂,所述试剂包括上述蛋白质或上述重组载体或上述重组菌。本发明提供了上述试剂在油菜抗病性育种中的应用,所述油菜优选包括甘蓝型油菜。在本发明中,野生型对照植株和转基因植株(敲除bnf5h基因)进行观察时发现,转基因植株的s型木质素的生物合成被阻断,s型木质素含量降低,使得s/g含量明显低于野生型对照,因此能够达到提高植物抗性的效果。为了进一步说明本发明,下面结合附图和实施例对本发明提供的一种调控油菜菌核病抗性的蛋白质及其应用进行详细地描述,但不能将它们理解为对本发明保护范围的限定。实施例1甘蓝型油菜各个组织rna的提取及定量pcr分析利用easyspin植物rna快速提取试剂盒(aidlab)提取甘蓝型油菜中双11各个组织的rna。其中双中11购自于中国农科院油料作物研究所。利用revertaidfirststrandcdnasynthesiskit(mbi)试剂盒反转录,参照revertaidfirststrandcdnasynthesiskit(mbi)说明书进行cdna一链的逆转录,用作定量rt-pcr分析模板。采用iqsybrgreensupermix(bio-rad)试剂分析目的基因的相对表达量。内标基因选择油菜的actin基因(af024716,引物为actin-1(5’-tctcacactgtgccaat-3’,seqidno.25)和actin-2(5’-tccacgtcacacttcat-3’,seqidno.26)。根据brassicanapusgenomeresources网站提供的bnf5h家族各成员基因(nf5h-1、bnf5h-2、bnf5h-3、bnf5h-4、bnf5h-5、bnf5h-6、bnf5h-7和bnf5h-8,bnf5h家族各成员基因的外显子为3~4个,内含子为1~3个,其特征见表1)的参考序列,利用primer5.0软件设计出各个成员基因的qrt-pcr引物,并在线比对检测引物的特异性(http://brassicadb.org/brad/)。bnf5h家族各成员基因的定量pcr引物见序列表2。表1bnf5h家族成员的特征表2引物序列利用sybrgreensupremixssoadvanceduniversalgreensupermix荧光定量试剂盒(bio-rad),使用bio-radcfxmanager荧光定量pcr仪进行。扩增条件为:95℃预变性3min;95℃变性20s,56℃退火20s,72℃延伸30s,40个循环。计算目的基因和内标的比值,目的基因在不同器官组织中的相对表达量,结果见图1:根中bnf5h-1、bnf5h-3、bnf5h-4的表达量高,茎中bnf5h-7的表达量高。叶中bnf5h-1、bnf5h-4、bnf5h-6基因表达量比较高,角果中bnf5h-7和bnf5h-4表达量比较高,种子中bnf5h-7和bnf5h-6的表达量比较高。bnf5h-2和bnf5h-5在各个组织中不表达,bnf5h-8和bnf5h-3的表达量也极低。由此可见,bnf5h家族中的4个优势表达基因为bnf5h-1、bnf5h-4、bnf5h-6和bnf5h-7。实施例2crispr/cas9敲除载体的构建1、基因敲除靶位引物设计利用在线网站(http://crispr.hzau.edu.cn/crispr2/)设计bnf5h-1、bnf5h-4、bnf5h-6和bnf5h-7的共同靶向引物grna序列:tgattgctttgatattgtcac,seqidno.27)。grna序列长20bp,紧邻pam(ngg)区,设计grna引物时优先选择cds序列前2/3区域,同时grna又必须同时位于4个bnf5h成员的保守区域,最后将设计好的grna序列在芸薹属数据库(http://brassicadb.org/brad/)中blast以确保引物的特异性。考虑到实验的重复性,bnf5h-1、bnf5h-4、bnf5h-6、bnf5h-7共设计2对特异性grna引物,分别为grna-1和grna-2,序列如表3所示;所述由上海生工合成。表3引物序列2、cas9载体的酶切处理将crispr/cas9质粒于37℃条件下酶切30min,酶切反应体系如表4所示。其中限制性内切酶购自roche公司。表4酶切反应体系试剂体积cas9载体10μlfastdegestbbii1μlfastap1μl10×fastdigestbuffer2μlddh2o6μltotal(总体积)20μl采用1.2%的琼脂糖凝胶电泳对酶切产物进行检测,并将开环的cas9载体进行胶回收。3、基于甘蓝型油菜bnf5h组织表达特性以及生物信息学分析结果,决定选取优势表达基因bnf5h-1(bnaa01g01400d)、bnf5h-4(bnac01g02400d)、bnf5h-6(bnac07g46040d)、bnf5h-7(bnac07g46050d)进行crispr/cas9植物表达载体的构建和大肠杆菌的转化。紫外灯下,用洁净的刀片切下含目的片段的琼脂糖凝胶块,按照easyspin植物rna快速提取试剂盒(aidlab)的方法回收相应的dna片段。将cas9载体用fastdegestbbii进行单酶切处理(图2中的a),开环cas9载体的胶回收产物与接头磷酸化处理后的grna用quickligase进行快速连接,回收的片段与pcambia3301载体建立如下连接体系:10×t4dna连接缓冲液1μl,载体dna片段1μl,外源连接产物dna片段1μl,t4dna连接酶(thermoscientific生物公司)1μl,用双蒸水补足体积至10μl的连接体系。载体dna片段与外源连接产物dna片段摩尔比为1:3,16℃连接12h。之后将连接产物转化大肠杆菌dh5α(北京博迈德基因技术有限公司)。将得到的菌液富集后涂平板(amp),然后挑取过夜培养的单克隆菌落摇菌,用m13-20+grna-r(表3所述序列)进行菌液(含cas9+grna)pcr检测,如图2中的b所示,扩增片段大小与预期大小一致。用ecori+hindiii(thermoscientific生物公司)将菌液pcr(图2中的b)得到的阳性克隆子的质粒与pcambia3301载体进行双酶切,如图2中的c所示,ck2为未双酶切的目的片段质粒,而1、2为酶切处理过的目的片段质粒,ck3为未双酶切的pcambia3301表达载体,3、4为酶切后的表达载体。酶切产物胶回收后4℃过夜连接并转化大肠杆菌感受态细胞,采用引物m13-20+grna-r(表3所述序列)和fbar+rbar菌检并送样测序,若返回测序结果正确,即可将重组质粒转化根癌农杆菌gv3101感受态细胞,将转化后的菌液再次菌检并测序,如图2中的d所示,marker左侧为引物m13-20+grna-r农杆菌的菌检结果,marker右侧为fbar+rbar的菌检结果,其中a和c中marker为trans2kdnaladder,b和d中marker为trans2kplusdnaladder,扩增条带分别为340bp和528bp左右,与预期大小一致可送样测序。实施例3农杆菌及油菜的遗传转化1、用电激法将构建的植物表达载体质粒导入农杆菌gv3101。参考bio-radmicropulser说明书,将上述植物表达载体通过电激转化法导入农杆菌gv3101(上海唯地生物技术有限公司)。2、特异表达载体整合到甘蓝型油菜基因组采用农杆菌介导的方法对油菜进行遗传转化(刘询,张斌,李浪,等.甘蓝型油菜bnalcr23基因crispr/cas9表达载体的构建及遗传转化[j].分子植物育种,2017(8):3024-3029),将获得的转基因油菜植株进行筛选。3、crispr-cas9敲除bnf5h(bnf5h-1、bnf5h-4、bnf5h-6和bnf5h-7)转基因植株的鉴定及编辑结果的统计采用植物基因组dna提取试剂盒对转基因植株及野生型westar的基因组dna进行提取,然后进行凝胶电泳检测以及浓度测定,如3所示,油菜转化的表达载体质粒的整个t-dna区段约17.8kb,dna条带较亮,且未出现明显降解,说明基因组dna的抽提较为成功,可以用于后续实验。3.1、crispr/cas9再生植株阳性鉴定剪取t0代crispr/cas9再生植株的幼嫩叶片进行dna抽提,以它作为模板,将农杆菌工程菌液作为阳性对照,野生型westar的dna作为阴性对照。以m13-20+grna-r(表3所述序列)、fbar+rbar和fbar-s+rbar-s分别为引物,进行pcr扩增,若3对引物扩增条带大小均与目标条带一致,则crispr/cas9敲除载体转化成功,对应再生植株为阳性,反之为阴性。每个植株重复扩增2次。扩增结果如图4所示,除阴性对照外所有引物均能扩增出与预测大小一致的目的条带,说明检测植株均为阳性,共得到23棵阳性植株。3.2、编辑结果crispr/cas9基因编辑技术对甘蓝型油菜bnf5h家族中的4个优势表达基因bnf5h-1、bnf5h-4、bnf5h-6、bnf5h-7进行编辑,结果显示如图5所示,crispr/cas9系统引起的靶序列或靶序列前后序列处存在碱基的插入(+)或缺失(-)。其中,转基因株系l-7在第二个外显子sgrna处有碱基的插入导致bnf5h-1,bnf5h-4,bnf5h-6和bnf5h-7的移码突变。为了检查后代中可遗传靶向诱变的稳定性,从bnf5h敲除转基因植株t2代中扩增17个单株的片段。发现编辑位点及编辑结果和t1代相同,基因编辑后bnf5h家族各成员(bnf5h-1、bnf5h-4、bnf5h-6、bnf5h-7)的cdna序列依次如序列表seqidno.5~seqidno.8所示。结果表明cas9-sgrna成功地在甘蓝型油菜bnf5h基因中产生了可遗传的突变。在电泳检测后将目的基因片段进行胶回收,吸取4μl胶回收产物加入1μl-t1simplecloningvector并轻轻吹打混合均匀,在pcr仪中设置程序25℃,10min,再将得到的连接产物转化trans1-t1感受态细胞,在含卡那霉素(kan)的lb平板上进行筛选,挑单克隆摇菌,用bnf5h基因编辑结果检测引物进行菌检,将阳性克隆菌液送样,测序引物序列如表5和表6所示,测序返回结果用软件mega5.1进行比对分析,结果如图6所示,其中a、d和g分别为野生型westar中bnf5h-4、bnf5h-6、bnf5h-7基因的测序峰图;b和c为转基因植株bnf5h-4的测序峰图;e和f为转基因植株bnf5h-6的测序峰图,h为转基因植株bnf5h-7的测序峰图;红色框内为grna序列;红色箭头指向突变位点,可见序列发生了突变。表5bnf5h家族各成员针对sgrna-1设计的引物表6bnf5h家族各成员针对sgrna-2设计的引物基因名引物(5'-3')序列号bnf5h-1-fgcgtgctcgggacagcaseqidno.45bnf5h-1-rtgttgatgctggagatgttseqidno.46bnf5h-4-facacgtcgctcatatcaagseqidno.47bnf5h-4-rattgatgctggagatgttgseqidno.48bnf5h-6-fagtccgacaacttctgcgseqidno.49bnf5h-6-raaacgtcggggagcaaaseqidno.50bnf5h-7-ftcaaggtgtggtcccatttseqidno.51bnf5h-7-rgtcctccaagtccaagacagcatseqidno.52实施例4t1代转基因植株菌核病抗性鉴定1、核盘菌纯化与接种体的制备用于菌核病抗性鉴定的核盘菌菌株,由重庆市油菜工程技术中心纯化和保存。具体纯化步骤如下:挑取灰褐色呈鼠粪状的菌核接种到pda培养基中,然后置于22℃培养箱中倒置暗培养,待培养基上长出呈放射状的白色菌丝后,用打孔器在菌丝生长边缘处取一小块菌丝转接到另一个pda培养基上继续培育(带菌丝的一面朝下),待其长出菌丝后再次挑取边缘处的菌丝块进行转接,重复此步骤,待培养基中仅有核盘菌菌丝而不被杂菌污染即可。挑取被活化后的菌丝块,将其接种于准备好的pda培养基上,在温度22℃、湿度85%的环境中培养48h,观察核盘菌的长势待菌丝均匀布满整个培养皿时,用打孔器(直径为6mm)沿着菌丝生长边缘打孔,得到的pda菌丝块即可用于菌核病抗性鉴定实验。2、离体叶片接种鉴定初花期时在t1代bnf5h基因编辑阳性植株(即敲除bnf5h-7基因获得的阳性植株)的同一部位剪取大小基本一致的叶片用于接种实验(倒数第4、5片叶)。在叶柄处用蘸水的棉花将其包裹,以防止水分散失,在准备好的塑料筐中依次铺上湿毛巾和滤纸,并用喷壶均匀洒上等量的水,将叶片整齐摆放在框内,将制备好的菌丝块接种在叶片左右叶腹的中央,带菌丝的一面朝下紧贴叶面,取材以及在后续的操作过程中应动作轻柔不要对叶片表面造成机械损伤从而影响实验结果。在接菌工作完成后,用塑料薄膜将塑料筐密封住,在接种室内放置一个温湿度计,并将温度控制在为22℃,湿度不低于90%,倘若发现湿度不够,这时可适当地补充水分。在接种48h后应及时对菌斑大小进行测量统计,菌斑长径记为a,短径为b,菌斑面积s=(π×a×b)/4。本实验重复3次。本试验中油菜的抗性水平通过比较菌斑面积大小来衡量。2、离体茎杆接种鉴定茎秆接种选取初花期和成熟期的wt和转基因植株的茎秆,每个株系选择3株,取材时保证各个时期植株粗细和wt相同,接菌茎秆两端用保鲜膜缠绕。用打孔器轻微伤害处理后将预先准备好的核盘菌接种体接种于茎秆两端。接菌72h后统计菌斑长度。取wt和各转基因植物幼苗期长势一致的植物叶片及成熟期中部茎秆,分别离体接种核盘菌48h和72h后统计叶片菌斑面积大小和茎秆菌斑长度,实验结果见表7、表8和图7。表7wt和ko-7侵染叶片的病斑面积表8wt和ko-7感染茎秆的病变长度重复1重复2重复3重复4重复5重复6平均wt6.57.57.76.57.67.57.216666667ko-73.94.53.53.53.73.13.7由表7、表8和图7记载的可知,图7中的(a),图7中的(b)所示,bnf5h基因编辑植株叶片菌斑面积小于wt,差异极显著;表7,表8,图7中的(c),图7中的(d)所示茎秆和叶片菌斑长度比wt小,差异显著;图7中的(e)~图7中的(j)所示,菌丝在bnf5h基因编辑植株叶片上的生长速率低于wt,差异显著。由此可见,改变木质素单体比例可以影响甘蓝型油菜对核盘菌的抗性,s/g越小,甘蓝型油菜对核盘菌的抗性越高,可见敲除bnf5h基因能够达到提高甘蓝型油菜的抗病性。3、t1代转基因植株茎秆切片染色观察在盛花期时选取长势一致的t1代crispr/cas9转基因和对照westar植株,每个材料2株,在主茎的第一个分枝上截取长度约15cm的侧枝用染色法进行染色观察。具体过程如下:实验操作前先打开leicacm1800冷冻切片机,待机箱温度降低至-20℃时,截取长度约为0.1~0.3cm的茎秆材料,将其置于样品托头上,然后均匀涂抹包埋剂将其完全裹住,稍等片刻后将样品托头转移到速冻架上,待包埋液完全凝固变白后将其固定到切片机的样品头上调整角度开始切片,切片厚度设置为25~100μm。用预冷的毛刷将组织结构完整的薄片放入盛有冰水的培养皿中,用药匙将切片轻轻放置事先准备好的载玻片上,用滤纸吸去多余的水分,用移液枪在切片上滴加适量的1%高锰酸钾溶液,静置约5min后用蒸馏水清洗3次,然后滴加3%的盐酸溶液浸泡2min,再用蒸馏水清洗3~5次,最后滴加29%的氨水反应2min,盖上盖玻片用滤纸吸取多余水分后方可置于olympusdp80型光学显微镜下进行观察,分别使用软件cellsensstandard、nis-elementsar3.2进行图片采集与分析。结果如图8所示,对照和转基因植株的解剖学观察当bnf5h发生基因编辑时,s木质素的生物合成被阻断,s型木质素含量降低。染色:首先在载玻片上的样品中加入1%高锰酸钾溶液浸染5min,用清水洗涤三次,然后滴加3%hc侵染2min,用清水洗涤三次,最后滴加29%氨水浸染2min,盖上盖玻片,用滤纸吸取多余的染液。用显微镜观察。对染色结果进行调查,maule染色可以使s型木质素被染为红色,而g型木质素被染为棕色,在转基因植物和wt的各种木质化组织中进行了maule组织化学染色。结果如图8所示,wt植株的茎干纤维束间纤维及木质部被染成呈红色,而bnf5h基因编辑植株纤维束间纤维及木质部被染成呈棕褐色。油菜根部在初始开花期的maule试剂染色显示木质部在野生型(wt)中,木质部被染成呈红色,而bnf5h基因编辑植株纤维束间纤维及木质部被染成棕褐色。成熟期角果冰冻切片染色结果显示,bnf5h基因编辑植株内果皮层被染成棕色,而wt植物植株内果皮层被染成红色。实施例5采用2d-hsqc-nmr法测定了t2代wt和bnf5h基因编辑植株全细胞壁样品及木质素样品中木质素各单体之间的含量,结果见表9和图9。表9野生型和转基因系茎细胞壁木质素含量和单信号素注:wcw(thewholecellwallspectra)为全细胞壁光谱,sem为误差值,hoh为h型木质素单体,god为g型木质素单体,soh为s型木质素单体。由表9和图9可知,与wt相比,bnf5h基因编辑植株s型木质素含量明显低于wt植株,s/g含量明显低于wt植株。实施例6利用茎杆强度测定仪分别测量了成熟期wt和各转基因植物(23棵阳性植物)的茎秆强度,结果见表10和图10。表10野生型和转基因系茎的断裂力(n)由表10和图10可知,bnf5h基因编辑植株茎秆抗折力明显高于wt。抗折力提高之后,能够避免植株倒伏之后菌核孢子随着雨水溅到茎干和叶片上的情况的出现,有利于植株抗菌核病。虽然本发明已以较佳的实施例公开如上,但其并非用以限定本发明,任何熟悉此技术的人,在不脱离本发明的精神和范围内,都可以做各种改动和修饰,因此本发明的保护范围应该以权利要求书所界定的为准。序列表<110>西南大学<120>一种调控油菜菌核病抗性的蛋白质及其应用<160>52<170>siposequencelisting1.0<210>1<211>482<212>prt<213>人工序列(artificialsequence)<400>1metgluserserileserglnthrleuserglnvalileaspprothr151015thrglyileleuilevalvalserleupheilepheileglyleuile202530thrargargargargproprotyrproproglyproargglytrppro354045ileileglyasnmetsermetmetaspglnleuthrhisargglyleu505560alaasnleualalyslystyrglyglyleucyshisleuargmetgly65707580pheleuhismettyralavalserserproaspvalalalysglnval859095leuglnvalglnaspservalpheserasnargproalathrileala100105110ilesertyrleuthrtyraspargalaaspmetalaphealahistyr115120125glyprophetrpargglnmetarglysvalcysvalmetlysvalphe130135140serarglysargalaglusertrpalaservalargaspgluvalasp145150155160lysmetileargservalserserasnvalglylysserileasnval165170175glygluglnilephealaleuthrargasnilethrtyrargalaala180185190pheglyseralacysglulysglyglnaspglupheileargileleu195200205glnglupheserlysleupheglyalapheasnvalalaasppheile210215220protyrpheglytrpileaspproglnglyileasnlysargleuval225230235240lysalaargasnaspleuaspglypheileaspaspileileaspglu245250255hismetlyslyslysgluasnglnasnservalaspalaglyaspval260265270valaspthraspmetvalaspaspleuleualaphetyrsergluglu275280285alalysleuvalsergluthralaaspleuglnasnserilelysleu290295300thrargaspasnilelysalaileilemetaspvalmetpheglygly305310315320thrgluthrvalalaseralaileglutrpalaleuthrgluleuleu325330335argserprogluaspleulysargvalglnglngluleualagluval340345350valglyleuaspargargileaspglytyrphevalprolyslysser355360365argvalmetileasnalaphealaileglyargasplysasnsertrp370375380valaspprogluthrpheargproserargpheleugluproglyval385390395400proaspphelysglyserasnpheglupheilepropheglysergly405410415argargsercysproglymetglnleuglyleutyralaleugluleu420425430alavalalahisileleuhiscysphethrtrplysleuproaspgly435440445metlysprosergluleuaspmetglyaspvalpheglyleuthrala450455460prolysalathrargleutyralavalproserthrargleuilecys465470475480serval<210>2<211>476<212>prt<213>人工序列(artificialsequence)<400>2metgluserleuleuserglnthrleuasnglnvalileaspprothr151015proservalleuleuilethrileserleuleuvalvalvaltyrleu202530ileserglntrpphelysproleutyrproproglyprolysglyleu354045provalileglyasnmetleumetmetaspglnleuthrhishisgly505560leualalysleualahislystyrglyglyleuphehisleuargmet65707580glyphehishisvalphealailethrserproaspvalalaarggln859095valleuglnvalglnaspileserpheserasnargprovalthrval100105110alaileasntyrleuthrtyraspleualaaspmetalaphealapro115120125tyrglyprophetrpargglnmetarglysvalcysvalmetlysval130135140pheserarglysargthrglusertrpalaservalargglugluval145150155160asnasnmetvalargserleuserserasnvalglylysprovalasn165170175valglygluleuilephethrleuthrargasnilethrtyrargala180185190alapheglyalaalacysgluthrgluglnaspglupheileargile195200205leuglnglupheserlysleupheglyalapheasnilealaaspphe210215220ilepropheleuglytrppheasppheglnglyileasnlysargleu225230235240vallysalaargasnaspleuaspglypheileaspgluvalileasp245250255gluhismetarglysarggluthrvalasnvalaspgluaspthrasp260265270metvalaspaspleuleualaphetyrsergluaspserserthrasn275280285argasnlysasnthrvallysleuthrargaspasnilelysalaleu290295300valmetaspvalmetpheglyglythrgluthrmetalaserglyile305310315320glutrpalaleuthrgluleuleuargasnproalagluleulysarg325330335leuglnglngluleuthrgluvalvalglyleuaspargargleugln340345350glypheservalprolysglyserargleumetileasnalapheala355360365ilealaargaspprolysleutrpvalaspproglualapheargpro370375380serargphemetgluproglymetproaspphemetglythrasnphe385390395400glupheilepropheglyalaglyargargsercysproglymetgln405410415leuglyleutyralametgluvalalavalalaasnileilehiscys420425430phethrtrplysleuproaspglymetlysprosergluleuaspmet435440445seraspvalmetglyleuthralaproargalathrargleuileala450455460valproaspthrargleuilecysprovalcyspro465470475<210>3<211>480<212>prt<213>人工序列(artificialsequence)<400>3metgluserservalserglnthrleuserglnvalleuaspprothr151015thralaileleuilevalvalserleupheilepheileglyleuile202530thrargargargargsertyrproproglyproargglytrpproile354045ileglyasnmetleumetmetaspglnleuthrhisargglyleuala505560asnleualalyslystyrglyglyleucyshisleuargmetglyphe65707580leuhismettyralavalserserproaspvalalaargglnvalleu859095glnvalglnaspserilepheserasnargproalathrilealaile100105110sertyrleuthrtyraspargalaaspmetalaphealahistyrgly115120125prophetrpargglnmetarglysvalcysvalmetlysvalpheser130135140arglysargalaglusertrpalaservalargaspgluvalasplys145150155160metileargleuvalserserasnvalglylysserileasnvalgly165170175gluglnilephealaleuthrargasnilethrtyrargalaalaphe180185190glyseralacysglulysglyglnaspglupheileargileleugln195200205glupheserlysleupheglyalapheasnvalalaasppheilepro210215220tyrpheglytrpileaspproglnglyileasnlysargleuvallys225230235240alaargasnaspleuaspglypheileaspaspileileaspgluhis245250255metlyslyslysgluasnglnasnthrvalaspaspglytyrvalgly260265270aspthraspmetvalaspaspleuleualaphetyrserglugluala275280285lysleuvalsergluthrthraspleuglnasnserilelysleuthr290295300argaspasnilelysalaileilemetaspvalmetpheglyglythr305310315320gluthrvalalaseralaileglutrpalaleuthrgluleuleuarg325330335serprogluaspleulysargvalglnglngluleualagluvalval340345350glyleuaspargargleuglugluseraspileglulysserargval355360365metileasnalaphealaileglyargaspprolyssertrpproasp370375380alagluthrpheargproserargpheleugluproglyvalalaasp385390395400phelysglyserasnpheglupheilepropheglyserglyargarg405410415sercysproglymetglnleuglyleutyralaleugluleualaval420425430alahisileleuhiscysphethrtrplysleuproaspglymetlys435440445prosergluleuaspmetasnaspvalpheglyleuthralaprolys450455460alathrargleuphealavalproserthrargleuilecysalaval465470475480<210>4<211>482<212>prt<213>人工序列(artificialsequence)<400>4metgluserserileserglnthrleuserglnvalleuaspprothr151015thrglyileleuilevalvalserleupheilepheileglyleuile202530thrargglyargargproprotyrproproglyproargglytrppro354045ileileglyasnmetsermetmetaspglnleuthrhisargglyleu505560alaasnleualalyslystyrglyglyleucyshisleuargmetgly65707580pheleuhismettyralavalserserproaspvalalaargglnval859095leuglnvalglnaspservalpheserasnargproalathrileala100105110ilesertyrleuthrtyraspargalaaspmetalaphealahistyr115120125glyprophetrpargglnmetarglysvalcysvalmetlysvalphe130135140serarglysargalaglusertrpalaservalargaspgluvalasp145150155160argmetileargservalserserasnvalglylysserileasnval165170175glygluglnilephealaleuthrargasnilethrtyrargalaala180185190pheglyseralacysglulysglyglnaspglupheileargileleu195200205glnglupheserlysleupheglyalapheasnvalalaasppheile210215220protyrpheglytrpileaspproglnglyileasnlysargleuval225230235240lysalaargasnaspleuaspglypheileaspaspileileaspglu245250255hisilelyslyslysgluasnglnasnserileaspalaglyaspval260265270valaspthraspmetvalaspaspleuleualaphetyrsergluglu275280285alalysleuvalsergluthralaaspleuglnasnserilelysleu290295300thrargaspasnilelysalaileilemetaspvalmetpheglygly305310315320thrgluthrvalalaseralaileglutrpalaleuthrgluleuleu325330335argserprogluaspleulysargvalglnglngluleualagluval340345350valglyleuaspargargileaspglytyrphevalprolyslysser355360365argvalmetileasnalaphealaileglyargasplysasnsertrp370375380valaspprogluthrpheargproserargpheleugluproglyval385390395400proaspphelysglyserasnpheglupheilepropheglysergly405410415argargsercysproglymetglnleuglyleutyralaleugluleu420425430alavalalahisileleuhiscysphethrtrplysleuproaspgly435440445metlysprosergluleuaspmetseraspvalpheglyleuthrala450455460prolysalathrargleutyralavalproserthrargleuilecys465470475480serval<210>5<211>1449<212>dna<213>人工序列(artificialsequence)<400>5atggagtcttctatatcacaaacactaagccaagtaatagatcccaccacgggtattctc60atcgtcgtctcacttttcatcttcatcggcctcatcacacgacgacgaaggcctccgtat120ccacccggtccacgtggttggcccatcataggcaatatgtcaatgatggaccaactcacc180caccgtggtttagccaacttagctaaaaagtacggtggcttgtgccatctccgcatggga240tttctccacatgtacgccgtttcatcaccagacgtggctaaacaagtccttcaagtccaa300gacagcgtcttctcgaaccgaccagctactatagctataagctatttgacttatgaccga360gccgacatggcgtttgctcactacggaccgttttggagacagatgaggaaagtttgcgtc420atgaaggtgtttagccgtaaacgagccgagtcatgggcttctgttcgtgatgaagtggac480aaaatgatccggtcggtttctagtaacgttggtaagtctatcaacgttggtgagcaaatt540tttgcactgacccgaaacataacttaccgggcagcgttcgggtcagcatgtgaaaaggga600caagacgagttcataagaatcttacaagagttctctaagctttttggagccttcaacgta660gcggatttcataccgtatttcgggtggatcgatcctcaaggaataaacaagcggctcgtg720aaggcccgtaatgacctagacggatttattgacgatatcatcgatgaacacatgaagaag780aaagagaatcaaaacagtgttgatgctggagatgttgttgataccgatatggttgatgat840cttcttgctttttacagtgaagaggcgaaattagtgagcgagacagcggatcttcaaaac900tctatcaaacttacccgtgacaatatcaaagcaatcatcatggacgttatgtttggagga960acggaaacggtagcgtcagcgatagagtgggcattgactgaattattacggagccccgag1020gatctaaaacgagtccaacaagaactcgctgaagttgtcggacttgaccgacgtatcgac1080ggttacttcgttcccaagaaatctcgcgttatgatcaacgcgtttgcgattgggcgcgac1140aagaactcttgggttgaccccgaaacgtttagaccgtctaggtttttggaaccgggcgta1200ccagatttcaaagggagtaacttcgagtttataccattcgggtcgggtcgtaggtcgtgc1260ccgggtatgcaactcgggttatacgcgcttgaactagccgtggcccatatattacattgc1320ttcacgtggaaattacctgatggcatgaaaccaagcgagcttgatatgggcgacgtgttt1380ggtctgacggctcctaaagccacgcgtctctatgctgtcccgagcacgcgccttatttgt1440tctgtttaa1449<210>6<211>1431<212>dna<213>人工序列(artificialsequence)<400>6atggagtctctgttatcacaaacactaaaccaagtaatagatcccacaccgtcagttctt60cttatcaccatctctcttctagttgtagtctacctcatctcacaatggtttaaaccgctc120taccctcccggtcccaaaggcctacccgtaattggaaatatgctaatgatggaccaactc180acacaccatggtctagccaagctagcccataaatacggtggtttgttccatttacgaatg240ggatttcatcacgtgtttgccatcacatcacctgacgtcgcccgacaagtcctccaagtc300caagacatcagcttctcgaaccggcccgtgactgtagccataaactacttaacctacgat360ctagccgacatggccttcgctccttacggacccttttggagacaaatgaggaaagtgtgc420gtcatgaaggtgttcagccggaaacggaccgagtcatgggcctcggtccgtgaagaagta480aacaatatggtccggtctttgtctagcaacgtcggtaagcccgttaacgttggagagctc540attttcacattgacacggaacataacgtaccgagcggcgtttggtgcggcctgcgagaca600gaacaagacgagttcataaggatcttgcaagagttctcaaagttatttggagcattcaac660atcgcggattttataccgttcctagggtggtttgattttcaagggataaacaagaggctt720gttaaggctcgtaacgatcttgacgggttcatcgatgaagttattgatgagcatatgagg780aagagggagactgtaaacgttgatgaagatacagatatggtggatgatctacttgcgttt840tatagcgaggattcatcaactaatcgtaataagaacaccgtaaaactcacacgtgataat900atcaaagccctcgtcatggatgttatgtttggaggaacggagacaatggcgtcagggatc960gaatgggctttgacagagcttctacgtaacccagctgaactcaaacggctccaacaagaa1020ctcaccgaggtcgtgggtcttgaccggcgcctccaaggcttctctgttcccaaaggctca1080cgcttgatgatcaacgccttcgctatcgcacgtgaccccaagttgtgggttgacccggaa1140gcgtttcgaccttctaggtttatggaaccgggtatgcctgatttcatggggactaacttt1200gagtttatacccttcggggcaggtcggagatcgtgtcctggaatgcaacttgggctttat1260gcgatggaggtggctgtggctaacatcattcactgtttcacgtggaagttgcctgacggg1320atgaaacctagtgagctcgacatgagcgacgtcatgggtcttaccgctcccagagccacg1380cgattgatcgcagtgcctgacacgcgcctcatctgtccggtgtgtccttga1431<210>7<211>1443<212>dna<213>人工序列(artificialsequence)<400>7atggagtcttctgtatcacaaacactaagtcaagtattagatcccacaacggctattctc60atcgtcgtctcacttttcatattcatcggcctcatcacacgacggcgaaggtcttaccca120cccggtccacgtggttggcccatcataggaaatatgttaatgatggaccaactcacccac180cgtggtttagccaacttagctaaaaaatatggcggcttgtgccatctccgcatgggcttc240ctccatatgtatgccgtctcatcacctgatgtggctcgacaagtcctccaagtccaagac300agcatcttctcgaaccggccggcaacgatagctataagctatttgacttatgaccgagcc360gacatggcgttcgctcactacggaccgttttggagacagatgaggaaagtgtgtgtcatg420aaggtgtttagccgtaaacgtgcggagtcatgggcttctgttcgagatgaagtggacaaa480atgatccggttggtatctagtaacgttggtaagtctataaacgtcggggagcaaattttc540gccctgacccgaaacataacttaccgggcagcgttcgggtcagcttgcgaaaagggacaa600gacgagttcataagaatcttacaagagttctctaaactttttggagccttcaacgtagca660gatttcataccatatttcgggtggatcgatccacaagggataaacaagcgcctcgtgaag720gcccgtaatgatctagacggatttattgacgatatcatcgacgaacatatgaagaagaaa780gagaatcaaaacactgttgatgatggatatgttggtgataccgatatggttgatgatctt840cttgctttttacagtgaagaggccaaattagtgagcgagacaacggatcttcagaattct900atcaaacttacccgtgacaatatcaaagcaatcatcatggacgtcatgttcggaggaacg960gaaacggtagcgtcggcaatagagtgggccttaacggagttattacggagccccgaggat1020ctaaaacgggtccaacaagaactcgcagaagttgtcggacttgaccgacgtctggaagaa1080tcagacatcgagaaatctcgcgttatgatcaacgcgttcgcgattggacgtgaccctaaa1140tcttggcctgacgccgaaacgtttagaccgtcgaggtttttagaaccgggagtagctgat1200tttaaaggaagtaacttcgagtttataccattcgggtcgggtcgtagatcgtgcccgggt1260atgcaactcgggttatacgcgcttgagttagccgttgctcatatattacactgcttcaca1320tggaaattacctgatgggatgaaacctagcgagcttgacatgaacgacgtgtttggtctc1380acggctcctaaagccactcgtcttttcgccgtgcctagcacacgcctgatttgtgctgtc1440taa1443<210>8<211>1449<212>dna<213>人工序列(artificialsequence)<400>8atggagtcttctatatcacaaacactaagccaagtattagatcccaccacgggtattctc60atcgttgtctcacttttcatcttcatcggcctcatcacacggggacgaaggcctccgtac120ccacccggtccacgtggttggcccatcataggcaatatgtcaatgatggaccaactcact180caccgtggtttagccaacttagctaaaaagtacggtggcttgtgccatctccgcatggga240tttctccacatgtacgccgtttcctcgccagacgtggctaggcaagtccttcaagtccag300gacagcgtcttctcaaaccgaccagcaactatagctataagctatttgacttatgaccga360gccgacatggcgtttgctcactacggaccgttttggagacagatgaggaaagtttgtgtc420atgaaggtgtttagccgtaaacgagccgagtcatgggcttctgttcgtgatgaagtggac480agaatgatccggtcggtttctagtaacgttggtaagtctatcaacgttggtgagcaaatt540tttgcactgacccgaaacataacttaccgggcagcgttcgggtcagcatgtgaaaaggga600caagacgagttcataagaattttacaagagttctctaagctttttggagccttcaacgta660gcggatttcataccgtatttcgggtggatcgatcctcagggaataaacaagcggctcgtg720aaggcccgtaatgacctagacggatttattgacgatatcatcgatgaacacataaagaag780aaagagaatcaaaacagtattgatgctggagatgttgttgataccgatatggttgatgat840cttcttgctttttacagtgaagaggcgaaattagtgagcgagacagcggatcttcagaac900tccatcaaacttacccgtgacaatatcaaagcaatcatcatggacgttatgtttggagga960acggaaacggtagcgtcagcgatagagtgggcattgactgagttattacggagcccagag1020gatctaaaacgagtccaacaagaactcgctgaagttgtcggacttgaccgacgtatcgac1080ggttacttcgttcccaagaaatctcgcgttatgatcaacgcgtttgcgattggacgcgac1140aagaactcttgggttgatcccgaaacgtttagaccgtcgaggtttttggaaccgggcgta1200ccagatttcaaagggagtaacttcgagtttataccattcgggtcgggtcgtcggtcgtgc1260ccgggtatgcagctcgggttatacgcgcttgaactagccgtggcccatatattacattgc1320ttcacgtggaaattacctgatggcatgaaaccaagcgagcttgatatgagcgacgtgttt1380ggtctgacggctcctaaagccacgcgtctctacgctgtcccgagcacgcgccttatttgt1440tctgtttaa1449<210>9<211>20<212>dna<213>人工序列(artificialsequence)<400>9accgatatggttgatgatct20<210>10<211>20<212>dna<213>人工序列(artificialsequence)<400>10ttcttgttggactcgtttta20<210>11<211>18<212>dna<213>人工序列(artificialsequence)<400>11tccttacggacccttttg18<210>12<211>18<212>dna<213>人工序列(artificialsequence)<400>12cgtcttgttctgtctcgc18<210>13<211>18<212>dna<213>人工序列(artificialsequence)<400>13cttacaccttcaaatggg18<210>14<211>18<212>dna<213>人工序列(artificialsequence)<400>14ggttatatcaaacgctcc18<210>15<211>22<212>dna<213>人工序列(artificialsequence)<400>15aagaagaaagagaatcaaaaca22<210>16<211>22<212>dna<213>人工序列(artificialsequence)<400>16taataactcagtcaatgcccac22<210>17<211>21<212>dna<213>人工序列(artificialsequence)<400>17acaagacgagttcataaggat21<210>18<211>21<212>dna<213>人工序列(artificialsequence)<400>18cgctataaaacgcaagtagat21<210>19<211>21<212>dna<213>人工序列(artificialsequence)<400>19tcatcgtcgtctcacttttca21<210>20<211>21<212>dna<213>人工序列(artificialsequence)<400>20cactttcctcatctgtctcca21<210>21<211>20<212>dna<213>人工序列(artificialsequence)<400>21caactcacccaccgtggttt20<210>22<211>20<212>dna<213>人工序列(artificialsequence)<400>22catcttgtcccttttcgcaa20<210>23<211>20<212>dna<213>人工序列(artificialsequence)<400>23aacggcggcaaatctaatga20<210>24<211>20<212>dna<213>人工序列(artificialsequence)<400>24ggagaagaggaatcggaggg20<210>25<211>17<212>dna<213>人工序列(artificialsequence)<400>25tctcacactgtgccaat17<210>26<211>17<212>dna<213>人工序列(artificialsequence)<400>26tccacgtcacacttcat17<210>27<211>21<212>dna<213>人工序列(artificialsequence)<400>27tgattgctttgatattgtcac21<210>28<211>24<212>dna<213>人工序列(artificialsequence)<400>28gattgattgctttgatattgtcac24<210>29<211>24<212>dna<213>人工序列(artificialsequence)<400>29aaacgtgacaatatcaaagcaatc24<210>30<211>24<212>dna<213>人工序列(artificialsequence)<400>30gattggcttgtgccatctccgcat24<210>31<211>24<212>dna<213>人工序列(artificialsequence)<400>31aaacatgcggagatggcacaagcc24<210>32<211>17<212>dna<213>人工序列(artificialsequence)<400>32gtaaaacgacggccagt17<210>33<211>21<212>dna<213>人工序列(artificialsequence)<400>33cgacatccgccgtgccaccga21<210>34<211>21<212>dna<213>人工序列(artificialsequence)<400>34tagatctcggtgacgggcagg21<210>35<211>24<212>dna<213>人工序列(artificialsequence)<400>35gtcaaccactacatcgagacaagc24<210>36<211>20<212>dna<213>人工序列(artificialsequence)<400>36agcaggtgggtgtagagcgt20<210>37<211>17<212>dna<213>人工序列(artificialsequence)<400>37tcggcctcatcacacga17<210>38<211>20<212>dna<213>人工序列(artificialsequence)<400>38aacatctccagcatcaacac20<210>39<211>17<212>dna<213>人工序列(artificialsequence)<400>39catcacacggggacgaa17<210>40<211>18<212>dna<213>人工序列(artificialsequence)<400>40cgcttgtttattccctga18<210>41<211>20<212>dna<213>人工序列(artificialsequence)<400>41ttctcatcgtcgtctcactt20<210>42<211>17<212>dna<213>人工序列(artificialsequence)<400>42ggcgcttgtttatccct17<210>43<211>22<212>dna<213>人工序列(artificialsequence)<400>43gtcaattattagatcccacaac22<210>44<211>17<212>dna<213>人工序列(artificialsequence)<400>44gccgcttgcttatccct17<210>45<211>17<212>dna<213>人工序列(artificialsequence)<400>45gcgtgctcgggacagca17<210>46<211>19<212>dna<213>人工序列(artificialsequence)<400>46tgttgatgctggagatgtt19<210>47<211>19<212>dna<213>人工序列(artificialsequence)<400>47acacgtcgctcatatcaag19<210>48<211>19<212>dna<213>人工序列(artificialsequence)<400>48attgatgctggagatgttg19<210>49<211>18<212>dna<213>人工序列(artificialsequence)<400>49agtccgacaacttctgcg18<210>50<211>17<212>dna<213>人工序列(artificialsequence)<400>50aaacgtcggggagcaaa17<210>51<211>19<212>dna<213>人工序列(artificialsequence)<400>51tcaaggtgtggtcccattt19<210>52<211>23<212>dna<213>人工序列(artificialsequence)<400>52gtcctccaagtccaagacagcat23当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1