一种可用于诊断生长发育迟缓的产品
1.本发明属于生物医药领域,具体涉及一种可用于诊断生长发育迟缓的产品。
背景技术:
2.生长发育迟缓是指生长、发育过程中速度放慢、顺序异常等现象。宫内发育迟缓(intrauterine growth retardation,iugr)一般指胎儿宫内发育迟缓。胎儿宫内发育迟缓是指胎儿的体重低于同龄平均体重的两个标准差,或低于其龄应有体重的第10个百分位数,为妊娠期重要并发症之一。宫内发育迟缓的患儿和同龄儿相比体质量更低,器官发育更不成熟,伴随更严重的全身疾病。2003年,国外首先相对宫内生长发育迟缓而提出宫外生长发育迟缓(extrauterine crowth retardation,eugr)的概念,其在新生儿疾病中发生率较高。在世界范围内,随着对宫外生长发育迟缓研究的深入以及营养强化策略的实施,宫外生长发育迟缓发病率虽然有所下降,但并不明显。近一些年的研究发现,早产儿中仍旧有大约50%的患儿发生了宫外生长发育迟缓。发生宫外生长发育迟缓最主要的原因是早产导致的低出生体重和小胎龄。另外,宫内生长发育迟缓、早期营养摄入不足、胎儿出生后体重下降、恢复与疾病等众多因素也与宫外生长发育迟缓密切相关。近些年来的研究表明,宫外生长发育迟缓带来的后期体重过度追赶增长会增加成年期某些疾病,使其患心血管疾病、糖尿病和高血压等代谢综合征的风险增加。
3.从达尔文的自然选择理论来看,人类携带的共生微生物是微生物(包括细菌、真菌、古细菌及病毒)与人类共同选择、共同进化的结果。人体微生物中不同菌群种类之间,菌群与病毒之间,菌群、宿主与环境之间,始终处于动态平衡状态,形成一个互相依存的复杂生态系。人体微生态系统包括口腔、皮肤、泌尿道、呼吸道、阴道和肠道6个微生态系统,其中以肠道微生态系统最为主要,也最为复杂。人体微生态与感染性疾病以及多种重大慢性疾病的互作关系及机制已成为国际研究热点。但是目前关于肠道菌群与生产发育迟缓的相关研究报道较少,研究肠道菌群与生长发育迟缓之间的关系,有望找到一种诊断、预防或治疗生长发育迟缓的新途径。
技术实现要素:
4.16srrna是原核生物核糖体中30s亚基的组成部分,16srdna是基因组中编码核糖体16srrna分子对应的dna序列。该基因全长约1540bp,存在于所有细菌染色体基因组中,包含10个保守区和9个可变区,其中保守区反映了物种间的亲缘关系,可变区可体现物种间的差异,且变异程度与菌群发育密切相关,是最适于鉴别细菌系统发育和分类的指标。本发明通过16srdna测序技术对受试者样本中的菌群进行动态测序,研究菌群与生长发育迟缓的相关性,为临床诊断、预防或治疗生长发育迟缓提供新的策略。
5.本发明采用了如下技术方案:本发明第一方面提供了检测受试者样本中bacillus niacini含量或丰度的试剂在制备预测或诊断生长发育迟缓的产品中的应用。
6.如本文所用的术语
ꢀ“
诊断”是指确认病理状态的存在或者特征,而本发明的目的不仅在于确认生长发育迟缓的发病与否,还包括判断受试者将来患有生长发育迟缓的风险。
7.进一步,所述的生长发育迟缓包括宫内生长发育迟缓、宫外生长发育迟缓。
8.进一步,所述的生长发育迟缓为宫外生长发育迟缓。
9.如本文所用的术语“受试者”是指任何动物受试者,包括:人、实验室动物、家畜和家养宠物。受试者可以寄居有多种微生物。受试者可在其身体之上和之内的各种生境中具有不同的微生物组。受试者可被诊断出疾病或被怀疑具有患病的高风险。受试者可具有导致疾病的微生物组状态(生态失调)。
10.进一步,所述的受试者为人。
11.进一步,所述的人为早产儿。在本发明的具体实施例中,所述的早产儿为胎龄小于32周的极早产儿。
12.进一步,所述的样本为粪便。
13.进一步,所述的试剂包括引物、探针、反义寡核苷酸、适配体或抗体。
14.在本说明书中,术语“引物”是指可以形成与模板链互补的碱基对,并用作模板链复制起点的七至五十个核酸序列。虽然引物通常是合成的,但也可用于天然存在的核酸中。引物的序列不必一定与模板的序列完全相同,互补性足够也能够与模板进行杂交。可以并入不改变引物本性的其他特征。可以并入的附加特征的例子有甲基化、加帽(capping)、用同源物取代一个或多个核酸、以及核酸之间的修饰等,但不限于此。优选地,本发明的引物可以是能够对bacillus niacini的16s rrna进行扩增的引物。
15.在本说明书中,术语“16s rrna”是指构成原核生物核糖体的30s亚基的、所有物种共有的保守区和能够对特定物种进行分类的高变区的rrna,因此可以通过碱基序列分析来鉴定微生物的存在。特别地,由于同源物种之间几乎没有多样性,而不同物种之间则具有多样性,因此可以通过比较16s rrna的序列来有效地鉴定原核生物。此外,由于16s rdna是编码16s rrna的基因,因此也可以使用16s rdna来鉴定微生物。
16.在一个优选的实施例中,本发明中的引物可用于扩增bacillus niacini的具有特异性的16s rrna(或16s rdna)序列,通过序列扩增的结果是否生成所需产物来检测bacillus niacini的存在。利用引物来扩增序列的方法可以使用本领域已知的各种方法。例如可以使用聚合酶链反应(pcr)、逆转录聚合酶链反应(rt
‑
pcr)、多重pcr(multiplex
‑
pcr)、降落pcr(touchdown pcr)、热启动(hot start) pcr、巢式(nested) pcr、实时(real
‑
time) pcr、差异显示pcr、cdna末端快速扩增技术、反向pcr、小载体pcr、交错式热不对称pcr、连接酶链反应,但不限于此。
17.如本文所述的术语“探针”指能与另一分子的特定序列或亚序列或其它部分结合的分子。除非另有指出,术语“探针”通常指能通过互补碱基配对与另一多核苷酸(往往称为“靶多核苷酸”)结合的多核苷酸探针。根据杂交条件的严谨性,探针能和与该探针缺乏完全序列互补性的靶多核苷酸结合。探针可作直接或间接的标记,其范围包括引物。杂交方式包括,但不限于:溶液相、固相、混合相或原位杂交测定法。
18.此外,在本发明中,微生物检测试剂可以是抗体,并且可以使用基于抗原
‑
抗体反应的免疫学方法来检测所述微生物。为此,分析方法有免疫印迹、酶联免疫吸附测定
(elisa)、放射免疫分析(ria)、放射免疫扩散法、火箭免疫电泳法 、免疫组织化学染色法 、免疫沉淀法、补体结合法或蛋白质芯片等,不限于此。
19.另外,本领域广泛使用的分子和免疫学方法可用于检测本发明的微生物。
20.进一步,所述的产品包括试剂盒、芯片或高通量测序平台。
21.所述的试剂盒不仅包含引物、探针、反义寡核苷酸、适体或抗体等用于检测所述微生物的检测试剂,并且还包括一种或多种适用于分析方法的其他成分组合物、溶液或装置。
22.如本文所用的术语“芯片”可指具有附着有吸附剂的、一般为平面的表面的固体基底。生物芯片的表面可包含多个可寻址的位置,其中每个位置可结合有吸附剂。生物芯片可适合于接合探针接口,并因此用作探针。蛋白质生物芯片适用于捕获多肽,并可包含在可寻址位置处附着有层析或生物特异性吸附剂的表面。微阵列芯片一般用于dna和rna基因表达检测。
23.本发明第二方面提供了一种可用于预测或诊断生长发育迟缓的产品,所述的产品包括检测受试者样本中bacillus niacini含量或丰度的试剂。
24.进一步,所述的产品包括试剂盒、芯片或高通量测序平台。
25.本发明第三方面提供了一种药物组合物,所述的药物组合物包括能够降低bacillus niacini的含量或丰度的物质。
26.本发明第四方面提供了本发明第三方面所述的药物组合物在制备预防或治疗生长发育迟缓的药物中的应用。
27.进一步,所述的生长发育迟缓包括宫内生长发育迟缓、宫外生长发育迟缓。
28.进一步,所述的生长发育迟缓为宫外生长发育迟缓。
29.如本文所用的术语“治疗”可指用于获得有益的或期望的结果的方法,该结果包括但不限于治疗性益处和/或预防性益处。治疗性益处可意指正在被治疗的潜在疾病的根除或改善。另外,治疗性益处也能如下实现:根除或改善与潜在病症相关的一种或多种生理学症状,使得在受试者中观察到改善,尽管该受试者可能仍受到该潜在病症的折磨。预防性效果包括延缓、防止或消除疾病或病状的出现,延缓或消除疾病或病状的症状发作,减慢、终止或逆转疾病或病状的进展,或其任意组合。对于预防性益处,具有发展成特定疾病的风险的受试者或报告疾病的一种或多种生理学症状的受试者可接受治疗,即使可能尚未作出该疾病的诊断。
30.本发明第五方面提供了一种筛选预防或治疗生长发育迟缓的候选药物的方法,所述的方法包括检测待筛选物质干预之前和干预之后的bacillus niacini的含量或丰度,若待筛选的物质可以降低bacillus niacini的含量或丰度,则表明该物质是预防或治疗生长发育迟缓的候选药物。
31.进一步,所述的生长发育迟缓包括宫内生长发育迟缓、宫外生长发育迟缓。
32.进一步,所述的生长发育迟缓为宫外生长发育迟缓。
33.本发明第六方面提供了bacillus niacini在筛选预防或治疗生长发育迟缓的候选药物中的应用。
34.进一步,所述的生长发育迟缓包括宫内生长发育迟缓、宫外生长发育迟缓。
35.进一步,所述的生长发育迟缓为宫外生长发育迟缓。
36.本发明第七方面提供了bacillus niacini在构建预测生长发育迟缓的计算模型
中的应用。
37.进一步,所述的生长发育迟缓包括宫内生长发育迟缓、宫外生长发育迟缓。
38.进一步,所述的生长发育迟缓为宫外生长发育迟缓。
39.本发明还提供了诊断生长发育迟缓的方法,所述的方法包括检测受试者样本中bacillus niacini的含量或丰度。
40.作为一种可选择的实施方式,用于诊断生长发育迟缓的方法包括:获取受试者的样本;处理受试者样本以获取16s rrna基因序列数据;检测样本中微生物的水平,包括用生物信息学软件分析16s rrna基因序列数据;并且当样本中微生物的水平高于对照样本时诊断所述受试者存在发生生长发育迟缓的风险,所述微生物为bacillus niacini。
41.除非另外定义,否则本文所用的技术和科学术语具有与由本领域普通技术人员通常所理解相同的含义。参见例如lackie,dictionary of cell and molecular biology,elsevier(第4版2007);sambrook等,molecular cloning,a laboratory manual,coldsprings harbor press(cold springs harbor,ny 1989)。
42.本技术所用的术语“roc曲线”或“接受者操作特性曲线”是指展示二元分类器系统的性能随着其辨别阈值的变化的图形曲线。通过在各种阈值设置下绘制真阳性率对假阳性率的曲线来创建该曲线。真阳性率也被称为灵敏度。假阳性率计算为1
‑
特异性。因此,roc曲线是一系列截断值范围内的真阳性率对假阳性率(灵敏度vs(1
‑
特异性))的图形显示以及选择最佳截断值进行临床使用的方式。将准确度表示为roc曲线下面积(auc),为比较测试性能提供了有用的参数。接近1的auc表示该测试高度灵敏并且具有高度特异性,而auc接近0.5则表明该测试既不灵敏也不具有特异性。
43.本文中术语“otu”(操作分类单元)是指系统发生树中的末端叶(terminal leaf),由特定的遗传序列以及与该序列在科、属、种或菌株水平上享有序列同一性的全体序列来定义。所述特定遗传序列可以是16s序列或16s序列的一部分,或者可以是在广泛存在于整个真细菌界的功能保守的管家基因。otu之间具有至少70%、75%、80%、85%、90%、95%、96%、97%、98%或99%的序列同一性。otu经常通过比较生物体之间的序列来定义,序列同一性低于95%的序列不视为构成同一otu的一部分,然而在本文所用,一个otu标识可包含序列同一性为0至100%、25%至100%和50%至100%、优选70%至100%、75%至100%、77%至100%、80%至100%、81%至100%、82%至100%、83%至100%、84%、至100%、更优选85%至100%、86%至100%、87%至100%、88%至100%、89%至100%、90%至100%、91%至100%、92%至100%、93%至100%、94%至100%、95%至100%、96%至100%、97%至100%98%至100%和99%至100%的序列。
44.本文中,otu代表此前已经归入或尚未归入属、种和/或株名的细菌,也即otu或otu标识等同于细菌的目、科、属、种或株,在进行生物信息学分析的过程中,通过out聚类,分析其代表的细菌的含量。
45.16s rrna的“v1
‑
v9区”指16s rrna基因的第一至第九高变区,用于细菌样品的基因分型,这是普通技术人员众所周知的。一些实施方式中,用v1、v2、v3、v4、v5、v6、v7、v8和v9区中至少其一来表征微生物标志物。一些实施方式中,用v3和v4区来表征微生物标志物。
46.本发明的优点和有益效果:本发明通过16s rrna测序,首次发现bacillus niacini在生长发育迟缓患者中丰
度显著降低,说明bacillus niacini可作为检测靶标应用于生长发育迟缓的预测和诊断。
47.本发明提供了检测受试者样本中bacillus niacini含量或丰度的试剂在制备预测或诊断生长发育迟缓的产品中的应用。
48.本发明还提供了一种筛选预防或治疗生长发育迟缓的候选药物的方法。
49.附图说明
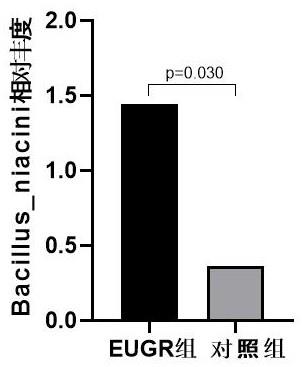
50.图1为bacillus niacini在eugr患儿中的丰度图。
具体实施方式
51.下面结合具体实施例,进一步阐述本发明,仅用于解释本发明,而不能理解为对本发明的限制。本领域的普通技术人员可以理解:在不脱离本发明的原理和宗旨的情况下可以对这些实施例进行多种变化、修改、替换和变型,本发明的范围由权利要求及其等同物限定。下列实施例中未注明具体条件的实验方法,通常按照常规条件或按照厂商所建议的条件实施检测。
52.实施例1 宫外生长发育迟缓患儿肠道菌群分析一、实验方法1、研究设计本研究招募了2018年1月至12月首都医科大学附属北京友谊医院新生儿重症监护室收治的胎龄小于32周的极早产儿,最终纳入22名极早产儿。在出生后4周评估身体发育情况,按体重分为宫外生长发育迟缓组(eugr组)和正常生长组(对照组)。同时,采集粪便样品于
‑
80℃保存,对肠道菌群进行高通量16s rrna测序,分析极早产儿子宫外生长发育迟缓与肠道微生态的关系。本研究经首都医科大学附属北京友谊医院伦理委员会批准。婴儿的父母(或负责任的亲属)给予书面知情同意。所有婴儿给予相同的喂养策略:所有早产儿在入院后24小时内断奶。早期微量喂养(10
‑
15 ml/kg/d),如可耐受,逐渐增加乳量,速度为15
‑
20 ml/kg/d。当不能完全肠内喂养时给予肠外营养支持。
53.2、分组标准根据早产儿fenton曲线图评估身体发育情况:体重低于相应胎龄的第10百分位水平或从出生至给定时间之间体重下降>2sd的婴儿为被认为患有eugr,而那些在第10百分位到第90百分位之间的婴儿视为生长正常。
54.3、纳入和排除标准(1)纳入标准:
①
出生后立即入住新生儿重症监护室;
②
胎龄28~32周,单胎;
③
住院时间> 28天;
④
出生后5天以内抗生素治疗(阿莫西林克拉维酸钾/哌拉西林他唑巴坦)。
55.(2)排除标准:
①
严重先天性畸形和先天性遗传代谢性疾病;
②
自动出院,出院后预后不明确;
③
数据不完整;
④
粪便标本采集过程中接受过益生菌治疗;
⑤
混合喂养。
56.4、样本收集收集早产婴儿的粪便标本。严格按照无菌操作原则,使用一次性无菌粪便容器于婴儿出生后第14、28天采集粪便标本,将粪便标本置于
‑
80℃保存。然后将它们送到allwegene技术公司进行dna提取、测序和生物信息分析。
57.5、16s rrna测序(1)基因dna的提取完成基因组dna抽提后,利用1%琼脂糖凝胶电泳检测抽提的基因组dna。
58.(2)pcr扩增按指定测序区域,合成带有barcode的特异引物,或合成带有错位碱基的融合引物。
59.pcr 采用transgen ap221
‑
02:transstart fastpfu dna polymerase;全部样本按照正式实验条件进行,每个样本3个重复,将同一样本的pcr产物混合后用2%琼脂糖凝胶电泳检测,使用axyprepdna凝胶回收试剂盒(axygen公司)切胶回收pcr产物,tris_hcl洗脱;2%琼脂糖电泳检测。
60.(3)miseq文库构建a.连接“y”字形接头;b.使用磁珠筛选去除接头自连片段;c.利用pcr扩增进行文库模板的富集;d.氢氧化钠变性,产生单链dna片段。
61.(4)miseq上机测序a.dna片段的一端与引物碱基互补,固定在芯片上;b.另一端随机与附近的另外一个引物互补,也被固定住,形成“桥 (bridge)”;c.pcr扩增,产生dna簇;d.dna扩增子线性化成为单链;e.加入改造过的dna聚合酶和带有4种荧光标记的dntp,每次循环只合成一个碱基;f.用激光扫描反应板表面,读取每条模板序列第一轮反应所聚合上去的核苷酸种类;g.将“荧光基团”和“终止基团”化学切割,恢复3'端粘性,继续聚合第二个核苷酸;h.统计每轮收集到的荧光信号结果,获知模板dna片段的序列。
62.6、数据分析(1)数据处理过程和参数miseq测序得到的是pair
‑
end(pe)双端序列数据,对测得的fastq数据进行质控处理,最终得到优质的fasta数据。
63.具体参数如下:a.利用trimmomatic、pear对fastq数据进行质控。对于trimmomatic采用滑动窗口
策略,窗口大小设置50bp,平均质量值为20,最小保留序列长度为120,pear用于去除具有n的序列;b.利用flash、pear根据pe的overlap关系对两端序列进行拼接(merge)处理,最小overlap设置10bp,错配率为0.1,得到fasta序列;c.根据已知数据库用uchime方法比对去除fasta序列的嵌合体,对于未知数据库使用自比对(denovo)方法进行去除,同时去除不合要求的短序列。
64.软件:trimmomatic(v0.36)、pear(v0.9.6)、flash(v1.20)、vsearch(v2.7.1)(2)有效数据统计下机数据(raw pe),在去除barcode和primer并拼接后得到raw_tags,raw_tags经进一步去除嵌合体、短序列后得到优质序列clean_tags。
65.(3)otus聚类分析otus(operational taxonomic units)是在系统发生学或群体遗传学研究中,为了便于进行分析,人为给某一个分类单元(品系,属,种、分组等)设置的同一标志。要了解一个样品测序结果中的菌种、菌属等数目信息,就需要对序列进行归类操作(cluster)。通过归类操作,将序列按照彼此的相似性分归为许多小组,一个小组就是一个otu。可根据不同的相似度水平,对所有序列进行otu划分,通常对97%相似水平下的otu进行生物信息统计分析。
66.平台及软件:qiime(version 1.8.0http://qiime.org/)vsearch(2.7.1 https://github.com/torognes/vsearch)分析方法:将clean tags用聚类(或降噪)方式生成otus。聚类方法可选 uparse[1],uclust[2],cdhit[3],ref参照数据库等方法;降噪法为unoise3[4]方法。默认为uparse聚类法。
[0067]
参考文献[1] edgar, r.c. (2013) uparse: highly accurate otu sequences from microbial amplicon reads, nature methods [pubmed:23955772, dx.doi.org/10.1038/nmeth.2604].[2]edgar, r.c. (2010) search and clustering orders of magnitude faster than blast, bioinformatics 26(19), 2460
‑
2461.doi: 10.1093/bioinformatics/btq461.[3]li et al (2006) cd
‑
hit: a fast program for clustering and comparing large sets of protein or nucleotide sequences.[4]rognes t, flouri t, nichols b, quince c, mah
éꢀ
f. (2016) vsearch: a versatile open source tool for metagenomics. peerj 4:e2584. doi: 10.7717/peerj.2584(4)物种差异分析物种差异分析根据得到的群落丰度数据,运用相关的分析方法进行分析,检测不同组(或样本)微生物群落表现出的丰度差异。物种差异性分析模块的内容包括:组间差异显著性检验、lefse多级物种差异判别分析。
[0068]
组间显著性差异检验根据得到的群落丰度数据,运用严格的统计学方法,对不同组(或样本)微生物群落之间的物种进行假设检验,评估物种丰度差异的显著性水平,获得组(或样本)间显著性差异物种。该分析选择门、纲、目、科、属、种、otu等不同分类水平。
[0069]
组间差异显著性检验的内容包括:a.卡方检验(chi
‑
squaretest)b.费舍尔检验(fisher’exacttest)c.t检验(student’st
‑
test(equalvariance))d.welcht检验(welch’st
‑
test(unknowvariance))e.wilcox秩和检验(mann
‑
whitneyutestorwilcoxonrank
‑
sumtes)f.kruskal_wallis秩和检验(kruskal_wallishtest)g.单因素anova分析(one
‑
wayanova)二、实验结果实验结果显示,如图1所示,与生长正常的婴儿相比,eugr患儿样本中bacillusniacini的丰度显著增加,差异具有统计学意义。
[0070] 实施例2诊断效能分析根据bacillusniacini的相对丰度,使用spss绘制受试者工作特征曲线(roc),计算二项精确置信空间,分析bacillusniacini用于宫外生长发育迟缓诊断的灵敏性和特异性。
[0071]
结果:如表1、表2所示,roc曲线下面积为0.697,cutoff值为1.500,特异性为1.000,敏感性为0.556,该结果证明将bacillusniacini应用于诊断宫外生长发育迟缓具有较高的敏感性、特异性和准确性。
[0072]
表1曲线下方区域表2bacillusniacini诊断效能数据上述实施例的说明只是用于理解本发明的方法及其核心思想。应当指出,对于本领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以对本发明进行若干改进和修饰,这些改进和修饰也将落入本发明权利要求的保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1