基于逐像素点深度卷积网络上皮和基质组织自动分割方法与流程
本发明涉及病理图像信息处理技术领域,特别是基于逐像素点深度卷积网络上皮和基质组织自动分割方法。
背景技术:
上皮组织和基质是乳房组织中的两类基本组织。80%的乳腺肿瘤起源于乳腺中的上皮组织,所以现在有一些学者致力于将计算机辅助诊断系统应用于对病理图像中的上皮和基质组织的异质性分析。自动的鉴别上皮和基质组织是量化这种异质性的前提,这样才使得对上皮细胞核进行单独分析成为可能。然而,由于病理组织图像所具有的复杂性,成功的将两类组织分离是一个具有挑战性的课题。
1)名副其实的大数据:
对于一张完整的病理组织全扫描切片,其尺寸约为100000×700000个像素点,存储在计算机上需要占用1.43个G的硬盘空间,这种高分辨率、大尺度图像对计算机硬件和图像分析算法都是非常具有挑战性的。
2)病理组织结构类型复杂,而且形态差异很大
一张病理切片具有众多的病理结构类型,形态各异。即便是相同的组织,其结构、形态也会千奇百怪。因此难以用一个固定的模型来描述,又大大的提高了对模型鲁棒性的要求。
3)不同病理等级其组织异质性高
随着癌症等级的提高,正常组织的边界不断被癌细胞腐蚀,上皮和基质组织之间的边界信息越来越模糊。而模糊的边界又提高了分割模型的准确性要求。
4)其他挑战
组织图像的背景复杂、噪声大,存在染色不均匀性和成像质量的问题。
由于H&E染色(苏木精-伊红染色)的病理图像能体现病理组织复杂的形态特征,从而在临床中被广泛使用。但是在H&E图像中,不仅背景复杂、图像噪声大,还存在由于切片染色制作过程中产生的染色不均匀,不正确染色等等问题。此外不同的扫描仪成像以及成像质量等问题。这些方面都会对图像处理分析算法带来巨大挑战。
虽然存在着上述的挑战,依然有着不少学者在病理图像的上皮和基质组织自动分割中做出了贡献,推动研究的发展。
与传统的方法不同,深度学习在大量数据的基础上通过组合低层特征来形成更加抽象的高层次的特征。随着深度学习和大数据分析研究的不断深入,使人们的研究目标从简单的图像转变为复杂的大型的图像。而病理组织学图像所具有的复杂性正好符合这一点。
技术实现要素:
本发明所要解决的技术问题是克服现有技术的不足,而提供一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法,与基于块的上皮和基质组织分割方法相比,无论是从定性结果还是定量结果来看,分类的准确率都得到了较大的提升。
本发明为解决上述技术问题采用以下技术方案:
根据本发明提出的一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法,包括以下步骤:
步骤1、对所有病理图像进行预处理操作,去除掉病理图像与病理图像之间的颜色亮度差异;
步骤2、随机选取预处理后的部分病理图像作为训练样本,其余作为测试样本;
步骤3、根据人工标注的组织区域图,从训练样本中的上皮和基质组织内部选取块;
步骤4、根据人工标注的组织区域图,从训练样本中的上皮和基质组织边缘选取块;
步骤5、将步骤3与步骤4得到的块进行整合并随机分为训练集与测试集;
步骤6、构建一个深度卷积神经网络模型DCNN,该模型包含了卷积层、池化层、线性纠正函数激活函数、局部响应归一化层以及分类器;采用步骤5中的训练集和测试集训练该深度卷积神经网络模型;
步骤7、取出步骤2的测试样本中的一张病理图像,以病理图像中每个点为中心,构造一个Q×Q的块;其中,Q为深度卷积神经网络输入尺寸的大小;
步骤8、将步骤7中构造的块输入到步骤6训练好的深度卷积神经网络模型中,得到分类结果。
作为本发明所述的一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法进一步优化方案,根据步骤8得到的分类结果进行伪彩色。
作为本发明所述的一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法进一步优化方案,Q为32。
作为本发明所述的一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法进一步优化方案,所述步骤4具体如下:根据人工标注的组织区域图,找到训练样本中的上皮和基质组织的边界线,对边界线进行膨胀操作得到边界线附近的点的坐标,以这些点为中心构建32×32的块,若中心点落在上皮组织中,则将该块认为是上皮组织小块,反之则是基质组织小块。
作为本发明所述的一种基于逐像素点深度卷积网络上皮和基质组织自动分割的方法进一步优化方案,所述步骤6中构建一个深度卷积神经网络模型DCNN,具体如下:
采用Alex成功区分CIFAR-10数据时所使用的模型中的权重矩阵来初始化深度卷积神经网络;
深度卷积神经网络的具体结构:
1)卷积层
假设滤波器组为每个输入大小为w1-1×w1-1的块通过m1×ml的滤波器滑过整张图像的局部感受域,并与每个局部感受域进行卷积操作,并输出结果;个滤波器一共生成个特征映射图,且每个映射图的大小为(wl-1-ml+1)×(wl-1-ml+1),这个线性滤波表示为其中,是一个l层的一个ml×ml的滤波器,ml代表网络结构第l层中滤波器的大小,是第l层滤波器组Wl中的滤波器的个数;
2)线性纠正函数激活函数的表达式如下:
3)池化层
池化层的操作是在上一层卷积特征映射后进行一个下采样的金字塔操作,在局部的感受域范围内,提取其最大值或平均值作为下一层的特征值,非线性操作后,图像的特征map尺寸大小变为:
其中,s是池化层操作的尺寸;
4)局部响应归一化层
用于局部做减和做除并归一化;
5)输出层
整个网络的最后一层就是输出层,输出层就是一个分类器,分类器的输入是神经网络的最后一层,分类器的输出是类别数,在深度卷积神经网络中,二分类的Softmax分类器的逻辑回归模型为:
其中,x是样本的特征向量,T为转置符号,θ是参数;
Softmax分类器的输入是DCNN网络的最后一层的输出,通过最小化如下的损失函数J(θ)得到Softmax分类器的参数θ;
其中,m为样本数量,y(i)为第i个样本标记,x(i)为第i个样本的特征向量,k为类别数;
θ代表所有的模型参数,如下所示:
其中,是分类为第j类时所采用的参数,同时也是θ这个所有模型参数中的第j行,0<j<k+1且j为整数;
根据得到的Softmax的参数θ,每一个通过滑动窗得到的图像块都会首先进行DCNN的前向传播得到特征向量x(i),再被送到逻辑回归模型中得到一个0~1之间的概率值,最终图像块的类别为:
其中,e为自然底数,k=2,是分类为第l类时所采用的参数,同时也是θ这个所有模型参数中的第l行。
本发明采用以上技术方案与现有技术相比,具有以下技术效果:
(1)在同样的实验条件下,本发明方法的检测准确率比基于像素块的分割方法准确率高;
(2)本发明旨在对每个像素点分类,避免了基于像素块的分割中存在的不同种类的像素点被划分为一个块的问题;
(3)本发明方法针对边缘组织,采取镜像边缘像素的方法来扩充边缘,从而来对它们进行分类;
(4)本发明方法在分割结果的同时在原图上做出展示,便临床医生直接观看,并在此基础上做出后续诊断。
附图说明
图1为深度卷及神经网络的结构图。
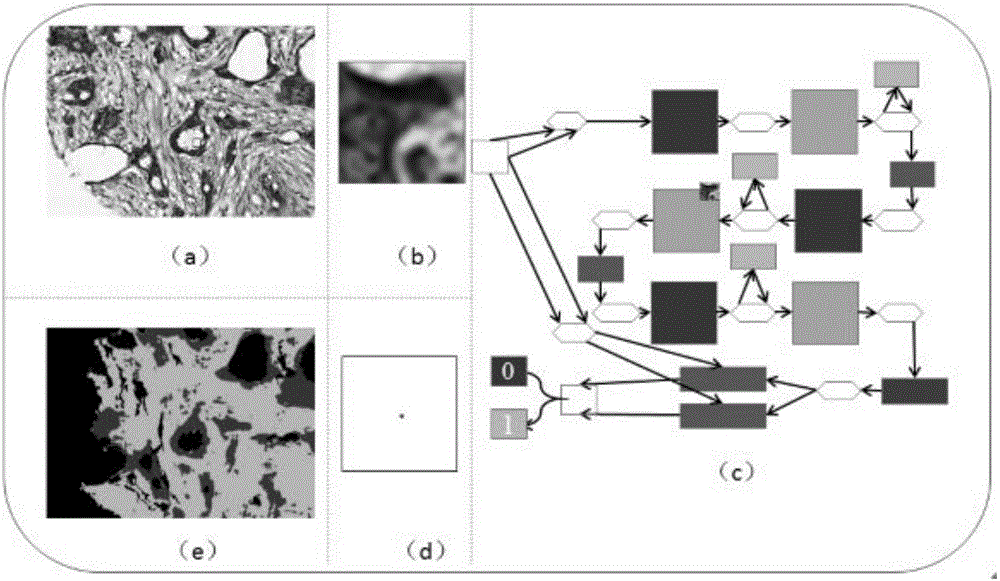
图2为深度卷及神经网络的实验整体流程图;其中,(a)为原始的H&E病理图像;(b)为从(a)中通过滑动窗口取出的32x32的小块;(c)为将小块输入到整个深度卷积神经网络(示意图)中,并得到分类结果;(d)为根据(c)中分类结果对(b)中的小块的中心点像素进行伪彩色染色;(e)为当整张图片所有的滑动小块都被染色之后得到的结果,作为分割结果。
图3是本发明中取出组织边缘的小块方法的示意图;其中,(a)为原始的H&E病理图像;(b)为由病理医生人工标注的结果(深灰色为上皮,浅灰色为基质,黑色为不关心的区域);(c)根据人工标注,得到上皮和基质的分割线并作膨胀处理;(d)在分割线所在区域随机取点,以该点为中心构建小块;(e)为基质小块;(f)为上皮小块。
图4是不同的模型对病理图像中上皮和基质组织分割后的伪彩色结果;其中,(a)是原始的病理图像,(b)是由病理专家精确标注的人工标注,(c)是由本发明提出的基于逐像素点和深度卷积神经网络的方法;(d)-(i)分别是SW-SVM,SW-SMC,Ncut-SVM,Ncut-SMC,SLIC-SVM,DCNN-SLIC-SMC得到的伪彩色分割结果图。
图5a为本发明方法与现有基于像素块的分割方法在NKI数据集上的ROC曲线的对比。
图5b为本发明方法与现有基于像素块的分割方法在VGH数据集上的ROC曲线的对比。
具体实施方式
为了使本发明的目的、技术方案和优点更加清楚,下面将结合附图及具体实施例对本发明进行详细描述。
步骤1、病理图像预处理操作,去除掉图像与图像之间的颜色亮度差异;
该方法预先选取一幅病理图像作为目标图像,其他的病理图像在颜色标准化之后都将与目标图像具有相同的颜色分布。具体方法是将目标图像和待标准化病理图像从RGB颜色空间转换到LAB颜色空间,对三个通道的每一个像素的灰度值进行一个线性变换,随后将线性变换后的LAB颜色空间的待标准化病理图像还原为RGB颜色空间,便可以使待标准化病理图像和目标图像具有一样的颜色分布。
步骤2、取出部分病理图像作为训练样本,其余作为测试样本;
随机的选取数据中的图片,同时确保训练样本与测试样本完全分开。
步骤3、根据专家标注,从上皮和基质组织内部选取图像块;
在病理图像中选取所有像素点都属于上皮组织或基质组织的图像块。关于组织图像块的选取,完全由拥有专业病理知识的临床医生在大幅切片图像中进行组织区域标记,程序会根据这些标记的区域从中选取边长为32个像素的正方形图像块。其中上皮组织中选取的块作为正样本,基质组织中选取的块作为负样本。
步骤4、根据专家标注,从上皮和基质组织边缘选取图像块;
根据专家标注,找到训练样本中的上皮和基质组织的边界,对边界线进行膨胀操作得到边界线附近的点的坐标。以这些点为中心构建32×32的小块,若中心点落在上皮组织中,则将该小块认为是上皮组织小块。反之则是基质组织小块;
步骤5、将步骤3与步骤4得到的小块进行整合并随机分为训练集与测试集;
通过随机筛选整合步骤3、4中得到的数据,其中组织内部小块:组织边缘小块比例大致为1:4。
步骤6、构建一个深度卷积神经网络模型(DCNN),模型包含了卷积层,线性纠正函数激活函数,池化层,局部响应归一化层以及最后的分类器;
深度卷积神经网络(DCNN)是人工神经网络的一种,它的权值共享网络结构使之更类似于生物神经网络,降低了网络模型的复杂度,减少了权值的数量。该优点在网络的输入是多维图像时表现的更为明显,使图像可以直接作为网络的输入,避免了传统识别算法中复杂的特征提取和数据重建过程。深度卷积网络是为识别二维形状而特殊设计的一个多层感知器,这种网络结构对平移、比例缩放、倾斜或者共他形式的变形具有高度不变性。
深度卷积神经网络模型性能的优劣在一定程度上取决于训练样本和初始的神经网络权重。采用随机初始化的方法容易陷于局部最优,所以这里使用采用知名学者Alex成功区分CIFAR-10数据时所使用的模型中的权重矩阵来初始化本发明的深度卷积神经网络。
下面介绍下深度卷积神经网络的具体结构。
1)卷积层
假设滤波器组为每一个(其中)是一个l层的一个ml×ml的滤波器,是第l层滤波器组Wl中的滤波器的个数。每个输入大小为wl-1×wl-1的块通过ml×ml的滤波器滑过整张图像的局部感受域,并与每个局部感受域进行卷积操作,并输出结果。个滤波器一共生成个特征映射图,且每个映射图的大小为(wl-1-ml+1)×(wl-1-ml+1),这个线性滤波可以被简单的表示为
2)ReLu激活函数
为了模仿人脑神经元的工作原理,也为了更好地拟合表示我们的数据信息,对每一层的线性滤波后得到的特征映射图,都要通过一个非线性的激活函数进行激活,在这里使用Relu激活函数,表达式如下:
相比较于传统的sigmod激活函数,Relu激活函数具有不饱和性,在训练梯度下降的时候能够更加快速地收敛,从而加快整个网络的训练速度。
3)Pooling层(S)
Pooling层的操作是在上一层卷积特征map后进行一个下采样的金字塔操作,在局部的感受域范围内,提取其最大值(或平均值)作为下一层的特征值,所以在pooling层是没有参数存在的,只需要做一个非线性操作即可,这样做的原因在于,在一副有意义的图像中,局部区域的信息是有冗余的,而我们要做的就是提取能代表和反映其最大响应的特征。在pooling操作后,图像的特征map尺寸大小变为:
其中s是pooling操作的尺寸。
4)局部响应归一化层
该模块主要进行的是局部做减和做除(local subtractive and divisive normalizations)并归一化,它会迫使在特征map中的相邻特征进行局部竞争,还会迫使在不同特征maps的同一空间位置的特征进行竞争。在一个给定的位置进行减法归一化操作,实际上就是该位置的值减去邻域各像素的加权后的值,权值是为了区分与该位置距离不同影响不同,权值可以由一个高斯加权窗来确定。除法归一化实际上先计算每一个特征maps在同一个空间位置的邻域的加权和的值,然后取所有特征maps这个值的均值,然后每个特征map该位置的值被重新计算为该点的值除以max(那个均值,该点在该map的邻域的加权和的值)。分母表示的是在所有特征maps的同一个空间邻域的加权标准差。实际上如果对于一个图像的话,就是均值和方差归一化,也就是特征归一化。这个实际上是由计算神经科学模型启发得到的。局部响应归一化层层模仿生物神经系统的侧抑制机制,对局部神经元的活动创建竞争机制,使得响应比较大的值相对更大,提高模型泛化能力。实施方式就是在每个给定的位置进行减法归一化操作,实际上就是该位置的值减去邻域各像素的加权后的值,权值是为了区分与该位置距离不同影响不同,权值可以由一个高斯加权窗来确定。
5)输出层
整个网络的最后一层就是输出层,输出层就是一个分类器,分类器的输入是神经网络的最后一层,分类器的输出是类别数,在深度卷积神经网络中,二分类的Softmax分类器的逻辑回归模型为:
其中,训练集由m个已标记的样本构成:{(x(1),y(1)),…,(xm,ym)},x是样本的特征向量,T为转置符号,θ是参数;
Softmax分类器的输入是DCNN网络的最后一层的输出,通过最小化如下的损失函数J(θ)得到Softmax分类器的参数θ;
其中,m为样本数量,y(i)为第i个样本标记,x(i)为第i个样本的特征向量;
为了方便起见,这里使用符号θ代表所有的模型参数,如下所示:
其中θ下标是具体以第几类,上标T是转置符号,k为类别总数;
根据得到的Softmax的参数θ,每一个通过滑动窗得到的图像块都会首先进行DCNN的前向传播得到特征向量x(i),再被送到逻辑回归模型中得到一个0~1之间的概率值,最终图像块的类别为:
其中,e为自然底数,k=2。是分类为第j类时所采用的参数,同时也是θ这个所有模型参数中的第j行。是分类为第l类时所采用的参数,同时也是θ这个所有模型参数中的第l行。
步骤7、取出步骤2中的测试样本中的一张病理图像,以图像中每个点为中心,构造一个32×32的小块;
以每个像素点为中心,往上取15个像素点,往下取16个像素点从而构成一个32×32的小块。针对边缘组织,为了方便取块,采取镜像边缘像素的方法来扩充边缘,从而来对它们进行分类;
步骤8、将步骤7中的小块输入到预先训练好的深度卷积神经网络中,得到分类结果。并根据分类结果进行伪彩色;
将步骤7中取出的小块输入到步骤6中训练好的深度卷积神经网络模型中,并得到最终的输出结果。如果结果为0,则认为该小块的中心像素点为上皮组织像素点,将其染成深灰色。如果结果为1,则认为该小块的中心像素点为基质组织像素点,将其染成浅灰色。同时找到专家标记中的黑色区域位置,将伪彩色结果中的同样位置染成黑色。
为了便于公众理解本发明技术方案,下面给出一个具体实施例。
本实施例将本发明所提供的技术方案应用在苏木精和伊红染色(H&E)的乳腺癌组织图像集上。本发明方法在两个数据库中进行了测试,分别是:荷兰癌症研究所(NKI)和温哥华综合医院(VGH)两个机构分别提供的数据。它包括了由病理专家手动标记出上皮和基质组织的157张病理图像(NKI,106张;VGH,51张)。每张图像都是从20×光学分辨率的H&E染色的乳腺癌组织芯片(TMA)中裁剪出来的,图像尺寸为1128×720。
本实施例中,组织特征提取部分采取深度卷积神经网络,分类部分为softmax分类器,为了验证本发明的基于逐像素点深度卷积网络的上皮和基质组织分割方法的有效性,对比了另外几种常见的使用深度卷积神经网络提取小块特征的上皮和基质组织分割方法,包括SW-SVM(滑动窗口+支持向量机分类),SW-SMC(滑动窗口+softmax分类),Ncut-SVM(规范化图割+支持向量机分类),Ncut-SMC(规范化图割+softmax分类),SLIC-SVM(简单线性迭代聚类+支持向量机分类),SLIC-SMC(简单线性迭代聚类+softmax分类)。
步骤1、病理图像预处理操作,去除掉图像与图像之间的颜色亮度差异;
该方法预先选取一幅病理图像作为目标图像,其他的病理图像在颜色标准化之后都将与目标图像具有相同的颜色分布。具体方法是将目标图像和待标准化病理图像从RGB颜色空间转换到LAB颜色空间,对三个通道的每一个像素的灰度值进行一个线性变换,随后将线性变换后的LAB颜色空间的待标准化病理图像还原为RGB颜色空间,便可以使待标准化病理图像和目标图像具有一样的颜色分布。
像素灰度值线性变化公式:在这里定义分别为LAB各通道所有像素灰度值的均方差和均值。Target为目标图像,original为标准化前的图像,mapped为标准化后的图像。
步骤2、取出部分病理图像作为训练样本,其余作为测试样本;
随机的选取数据中的图片,同时确保训练样本与测试样本完全分开。
步骤3、根据专家标注,从上皮和基质组织内部选取图像块;
在病理图像中选取所有像素点都属于上皮组织或基质组织的图像块。关于组织图像块的选取,完全由拥有专业病理知识的临床医生在大幅切片图像中进行组织区域标记,程序会根据这些标记的区域从中选取边长为32个像素的正方形图像块。其中上皮组织中选取的块作为正样本,基质组织中选取的块作为负样本。
步骤4、根据专家标注,从上皮和基质组织边缘选取图像块;
如图,根据专家标注(图3中的(b)),找到训练样本中的上皮和基质组织的边界,对边界线进行形态学的膨胀操作(图3中的(c))得到膨胀后的边界。从中得到属于该边界线的点的坐标。以这些点为中心构建32×32的小块,若中心点落在上皮组织中,则将该小块认为是上皮组织小块(图3中的(f))。反之则是基质组织小块(图3中的(e));为了更好的展示效果,将原始图像(图3中的(a))与膨胀后的边界(图3中的(c))图像融合得到边界示意图(图3中的(d))。
步骤5、将步骤3与步骤4得到的小块进行整合并随机分为训练集与测试集;
通过随机筛选整合步骤3、4中得到的数据,其中组织内部小块:组织边缘小块比例大致为1:4。样本数量如表1所示。
表1训练样本数量
步骤6、构建一个深度卷积神经网络模型(DCNN),模型包含了卷积层,线性纠正函数激活函数,池化层,局部响应归一化层以及最后的分类器;
对于卷积神经网络,本发明所使用的框架是目前很热门的Caffe框架。网络结构如图1所示:
第一层使用32个卷积核(conv)对图像进行卷积操作(卷积核大小Kernel size=5;步长Stride=1;图像边缘镜像填充像素Pad=2;)。
第二层使用最大值池化(pool)的方式对卷积结果进行下采样(池化核大小Kernel size=3;步长Stride=2;图像边缘镜像填充像素Pad=0;)。
随后使用ReLU激活函数与局部响应归一化(LRN)。
第三层使用32个卷积核对图像进行卷积操作(卷积核大小Kernel size=5;步长Stride=1;图像边缘镜像填充像素Pad=2;)。
随后使用ReLU激活函数。
第四层使用最大值池化的方式对卷积结果进行下采样(池化核大小Kernel size=3;步长Stride=2;图像边缘镜像填充像素Pad=0;)。
随后使用局部响应归一化。
第五层使用64个卷积核对图像进行卷积操作(卷积核大小Kernel size=5;步长Stride=1;图像边缘镜像填充像素Pad=2;)。
随后使用ReLU激活函数。
第六层使用最大值池化的方式对卷积结果进行下采样(池化核大小Kernel size=3;步长Stride=2;图像边缘镜像填充像素Pad=0;)。
第七层使用64个全连接单元(ip)与上一层进行全连接操作。
第八次输出分类结果以及与真实值对比的loss值。
步骤7、取出步骤2中的测试样本中的一张病理图像,以图像中每个点为中心,构造一个32×32的小块;
以每个像素点为中心,往上取15个像素点,往下取16个像素点从而构成一个32×32的小块。针对边缘组织,为了方便取块,采取镜像边缘像素的方法来扩充边缘,从而来对它们进行分类;
步骤8、将步骤7中的小块输入到预先训练好的深度卷积神经网络中,得到分类结果。并根据分类结果进行伪彩色;
将步骤7中取出的小块输入到步骤6中训练好的深度卷积神经网络模型中,并得到最终的输出结果。如果结果为0,则认为该小块的中心像素点为上皮组织像素点,将其染成深灰色。如果结果为1,则认为该小块的中心像素点为基质组织像素点,将其染成浅灰色。同时找到专家标记中的黑色区域位置,将伪彩色结果中的同样位置染成黑色。
图2为深度卷及神经网络的实验整体流程图;其中,(a)为原始的H&E病理图像;(b)为从(a)中通过滑动窗口取出的32x32的小块;(c)为将小块输入到整个深度卷积神经网络(示意图)中,并得到分类结果;(d)为根据(c)中分类结果对(b)中的小块的中心点像素进行伪彩色染色;(e)为当整张图片所有的滑动小块都被染色之后得到的结果,作为分割结果。
为了验证本发明的基于逐像素点深度卷积网络的上皮和基质组织分割的有效性,对比了另外几种常见的使用深度卷积神经网络提取小块特征的基于像素块的上皮和基质组织分割方法,包括SW-SVM(滑动窗口+支持向量机分类),SW-SMC(滑动窗口+softmax分类),Ncut-SVM(规范化图割+支持向量机分类),Ncut-SMC(规范化图割+softmax分类),SLIC-SVM(简单线性迭代聚类+支持向量机分类),SLIC-SMC(简单线性迭代聚类+softmax分类)。
图4展示了不同的模型对病理图像中上皮和基质组织分割后的伪彩色结果。其中,图4中的(a)是原始的病理图像;图4中的(b)是由病理专家精确标注的人工标注,其中深灰色部分代表着上皮组织,浅灰色部分代表着基质组织,黑色部分为背景区域,即不被关注的区域;图4中的(c)是由本章节提出的基于逐像素点和深度卷积神经网络的方法;图4中的(d-i)分别是SW-SVM,SW-SMC,Ncut-SVM,Ncut-SMC,SLIC-SVM,DCNN-SLIC-SMC得到的伪彩色分割结果图,其中深灰色代表分类器分类结果为上皮组织的区域,浅灰色代表分类器分类结果为基质组织的区域,而黑色区域是不被关注的背景区域。
由结果可以看出,撇去背景区域,即专家标注中为黑色的区域,本发明提出的算法和专家标记的结果相似度非常高,具有明显的优势。
为了定量的表示实验结果,使用了混淆矩阵(confused Matrix)中的衍生参数和ROC曲线来比较实验结果。
TP表示真阳性,即专家标记为上皮组织,分类器认为是上皮组织的像素点的个数;
FP表示假阳性,即专家标记为基质组织,分类器认为是上皮组织的像素点的个数;
FN表示假阴性,即专家标记为上皮组织,分类器认为是基质组织的像素点的个数。
TN表示真阴性,即专家标记为基质组织,分类器认为是基质组织的像素点的个数;
混淆矩阵的衍生参数的计算公式如表2所示,真阳性率(TPR),真阴性率(TNR),阳性预测值(PPV)、阴性预测值(NPV),假阳性率(FPR),假阴性率(FNR),伪发现率(FDR),准确性(ACC),F1得分(F1),和马休斯相关系数(MCC)都是通过上述四个混淆矩阵中的参数所衍生出来的评估指标。其中ACC,F1,以及MCC是对模型综合能力评估的指标。
表2混淆矩阵的衍生参数公式
下表3表示不同的模型的分割结果的定量评估(%),其中,粗体字即指标中的最优值。
表3
显示ROC曲线的图被称为“ROC图”。当进行多个学习器的比较时,如果一个学习器的ROC曲线将另一个学习器的曲线完全“包住”,则可以断言前者的性能优于后者;若两个学习器的ROC曲线发生的交叉,则难以断言两者性能孰优孰劣。一个比较合理的判据就比较ROC曲线下面的面积,即AUC(Area Under ROC Curve)。由定义可知,AUC值可以通过对ROC曲线下各部分的面积求和而得。AUC值越大,说明效果越好。图5a展示了几种方法在NKI数据集上分割效果的ROC曲线,图5b展示了几种方法在VGH数据集上分割效果的ROC曲线由AUC值可知,本发明提出的基于逐像素点深度卷积网络的上皮和基质自动分割效果优于基于块的上皮和基质自动分割。
- 还没有人留言评论。精彩留言会获得点赞!