一种基于多重PCR的靶向病原体纳米孔测序快速分析方法与流程
本发明涉及纳米孔测序,具体涉及一种基于多重pcr的靶向病原体纳米孔测序快速分析方法。
背景技术:
1、靶向测序是一种专门用于捕获基因特殊区域并进行高通量测序的方法,pcr是其中一种最为常见的靶向富集方法。用于病原体鉴定的临床样本(如血液、肺泡灌洗液、脑脊液等),往往具有病原体载量低的特性。通过对特异性病原体的目的片段进行扩增,可以达到靶向富集病原体的目的,同时有效减少宿主基因的干扰,实现对低载量病原体的检出。其主要技术及实现方案是提取临床样本中的核酸,利用多对引物进行特异性扩增并构建上机文库,进行测序,待获得测序数据后,使用序列比对软件与病原体序列数据库进行比对,采用一定的比对结果过滤条件进行筛选获得可信结果,进而计算输入的测序数据中病原体来源的序列含量及所占比例等,最终判断测序样本中相应病原体的存在情况。
2、纳米孔测序技术具有长读长的特性,但同时也具有随机错误率高的缺点,如何利用纳米孔测序数据实现对临床样本病原体的快速、准确检测,是当前面临的挑战,也是主要的技术难点。因此,为了提升临床病原检测的时效性和准确性,必须建立一套基于多重pcr的靶向病原体纳米孔测序自动分析方法及系统。
3、目前,对测序数据的处理方式主要包括序列比对和使用k-mer算法进行分析。其中,基于序列相似性比对的软件包括bwa、bowtie 2、blast等,是基于bwt转换算法构建的快速比对算法,在针对二代测序数据的分析中已经广为使用,如solexa序列用bowtie做比对。blast算法是1990年由altschul等人提出的两序列局部比对算法,采用了一种短片段匹配算法和一种有效的统计模型来找出目的序列和数据库之间的最佳局部比对效果,是一种基于局部序列比对的序列比对算法。
4、但是如何分析纳米孔测序产生的长读长、且伴有较高随机错误的序列,还需进一步研究。
5、鉴于上述缺陷,本发明创作者经过长时间的研究和实践终于获得了本发明。
技术实现思路
1、本发明的目的在于解决如何分析纳米孔测序产生的长读长、且伴有较高随机错误的序列的问题,提供了一种基于多重pcr的靶向病原体纳米孔测序自动分析方法。
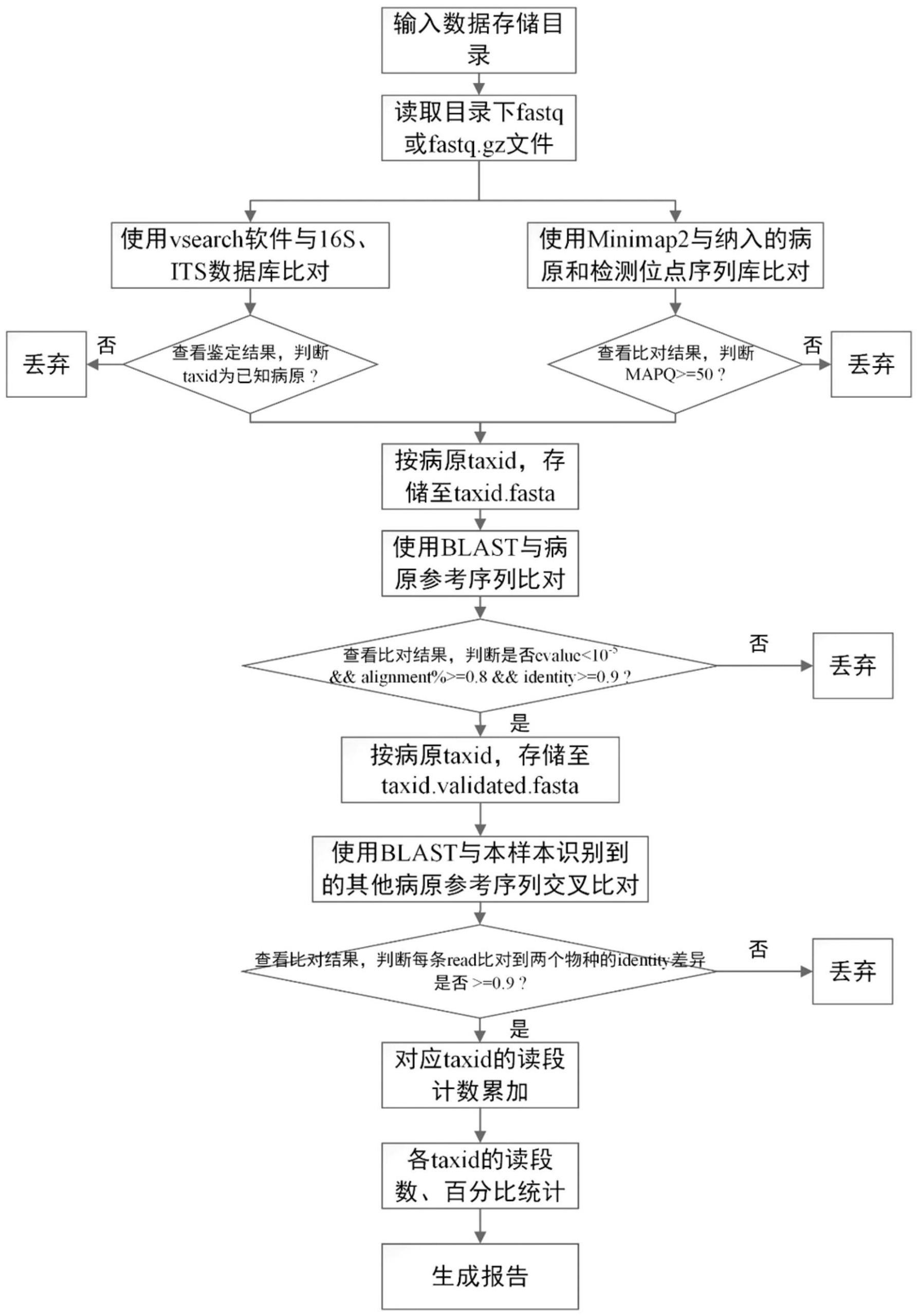
2、为了实现上述目的,本发明公开了一种基于多重pcr的靶向病原体纳米孔测序自动分析方法,包括以下步骤:
3、s1:读取下机数据,采用国际通用的测序数据标准fastq格式,支持gzip方法进行数据压缩以减少存储占用,使用cat或zcat命令合并来自于同一份样本的多个fastq数据文件或压缩的fastq.gz文件;
4、s2:根据数据类型,使用nanofilt软件和fastqc软件和对应参数进行低质量数据过滤;
5、s3:使用samtools软件提取通过数据质控的高质量数据,用于后续分析;
6、s4:第一轮快速比对,通过使用vsearch和minimap2两种快速比对软件和算法进行物种初步判别;
7、s5:基于步骤s3、步骤s4的结果,使用samtools软件提取疑似病原的读段并按初步鉴定的物种进行拆分;
8、s6:将步骤s5拆分的疑似病原读段,与各物种参考序列进行blast比对,提取各读段最优比对,并按照比对长度大于读段长度80%、序列相似度高于90%,比对期望值(evalue)小于1e-5,进行筛选;
9、s7:将步骤s6获得的病原读段,与本样本识别到的其他病原参考序列再次进行blast交叉比对,提取各读段最优比对,并按照比对长度大于读段长度80%、序列相似度高于90%,比对期望值小于1e-5,进行筛选;
10、s8:将步骤s7验证的病原读段按病原类型和基因组类型进行各分类水平的物种进行统计分析,明确该份样本中存在的病原类型、基因组类型、属水平和种水平的读段数量及构成比;
11、s9:根据步骤s8获得的样本中病原体统计信息,补充添加病原体注释信息、样本信息和患者信息等,生成检测报告。
12、所述步骤s2中针对三代测序的fastq测序数据文件进行质控检测和过滤,使用nanofilt软件,筛选平均测序数据质量大于q7的测序读段。
13、所述步骤s2中针对二代数据使用fastqc软件,筛选平均测序数据质量大于q30的测序读段。
14、所述步骤s4中vsearch用于与16s,its数据库比对进行细菌、真菌、病毒和寄生虫识别,minimap2用于针对病毒等基因组变异较大病原体的补充比对,基于minimizer哈希表搜索、chaining算法和动态规划算法。
15、所述步骤s8中病原类型包括细菌、真菌、病毒、寄生虫,基因组类型包括dna和rna。
16、与现有技术比较本发明的有益效果在于:本发明提出的技术方案中建立了针对基于多重pcr的靶向纳米孔长读长测序数据的病原体比对识别方法和系统,能够有效处理纳米孔测序技术产生的长读长数据,很好地解决了长读长、错误率较高的纳米孔测序数据比对分析中准确性和分析速度难以兼顾的问题,将典型数据(1gb纳米孔测序数据)分析耗时降低至10分钟以内,满足了临床中对低丰度病原体进行快速病原检测分析的需求,同时,该方法也能够针对二代测序获得的短读长数据进行分析,具有较好的数据兼容性。
技术特征:
1.一种基于多重pcr的靶向病原体纳米孔测序快速分析方法,其特征在于,包括以下步骤:
2.如权利要求1所述的一种基于多重pcr的靶向病原体纳米孔测序快速分析方法,其特征在于,所述步骤s2中针对三代测序的fastq测序数据文件进行质控检测和过滤,使用nanofilt软件,筛选平均测序数据质量大于q7的测序读段。
3.如权利要求1所述的一种基于多重pcr的靶向病原体纳米孔测序快速分析方法,其特征在于,所述步骤s2中针对二代数据使用fastqc软件,筛选平均测序数据质量大于q30的测序读段。
4.如权利要求1所述的一种基于多重pcr的靶向病原体纳米孔测序快速分析方法,其特征在于,所述步骤s4中vsearch用于与16s,its数据库比对进行细菌、真菌、病毒和寄生虫识别,minimap2用于针对病毒等基因组变异较大病原体的补充比对,基于minimizer哈希表搜索、chaining算法和动态规划算法。
5.如权利要求1所述的一种基于多重pcr的靶向病原体纳米孔测序自动分析方法,其特征在于,所述步骤s8中病原类型包括细菌、真菌、病毒、寄生虫,基因组类型包括dna和rna。
技术总结
本发明涉及纳米孔测序技术领域,具体涉及一种基于多重PCR的靶向病原体纳米孔测序快速分析方法,该方法构建了两轮比对分析的病原识别算法,保证了识别的准确性和时效性。首先在第一轮比对中,通过使用两种快速比对软件——vsearch和minimap2,实现了针对典型数据(1Gb纳米孔测序数据)的物种初步判别;在第二轮验证分析中,使用BLAST精确比对算法将第一步判别的各个物种的读段与该物种参考序列进行比对验证,有效减少了精确比对的计算量,将典型数据耗时降低至10分钟内,通过构建该方法,实现了典型纳米孔测序数据在10分钟内的准确分析和报告生成,并可兼容二代测序数据分析。
技术研发人员:余乐,李寅虎,王志富,武志慧,杨童茜,高军涛
受保护的技术使用者:佛山源生基因生物科技有限公司
技术研发日:
技术公布日:2024/1/15
- 还没有人留言评论。精彩留言会获得点赞!