一种环状RNA亚细胞定位的预测方法
本发明涉及生物信息学领域,具体是一种基于岭回归的用于环状rna亚细胞定位的预测方法。
背景技术:
1、近年来,越来越多的研究揭示了circrna的多方面功能,包括作为rna结合蛋白(rbp)海绵、microrna(mirna)海绵、父基因表达调节因子、可变剪接调节因子和转录调节因子。值得注意的是,少数circrna甚至可以被翻译成肽段或蛋白质。此外,越来越多的证据表明circrna参与了各种疾病的发生,包括癌症、动脉粥样硬化性血管疾病和神经系统疾病,并且在结直肠癌(crc)中表现出异常表达。circrna已被确定为胃癌、衰老甚至是人体唾液中的诊断生物标志物。这些发现表明,circrna在生物发育、疾病发生和进展中具有巨大的潜力发挥独特的调控作用。此外,它们有潜力成为临床诊断、症状和预后的有效标志物,促进对各种复杂疾病及其治疗的深入理解。
2、高通量测序分析表明,circrna存在于不同的亚细胞区域,并通过共定位发挥各种调控功能。与信使rna(mrna)类似,circrna在细胞核中合成,并主要存在于细胞质中。在细胞核内,circrna通过与u1 snrnp结合并招募rna聚合酶ii到父基因的启动子位点来增强mrna的表达。此外,核内circrna阻碍了前核糖体与pes1蛋白之间的相互作用,从而减轻rrna的成熟和加工过程。定位到细胞质的circrna通过与蛋白质和mirna的相互作用发挥各种调控功能。许多circrna已被确定为mirna海绵和蛋白质海绵,它们与相应的mirna或蛋白质结合,减少其对目标的抑制作用。大量circrna定位到外泌体,与其他在相应细胞的细胞核和细胞质中存在的circrna明显不同。外泌体circrna作为不同疾病的靶标,可以实现有效的诊断和治疗。最近的研究揭示了circrna在核糖体中的定位及其翻译成内源性肽段的过程。由于其他ncrna的亚细胞定位已经被阐明,揭示了它们在神经元树突、胚胎发育和基因调控等方面的作用。同时,这加深了我们对于各种生物过程的理解,如基因的转录后调控、表观遗传功能、蛋白质-rna相互作用以及细胞发育和代谢。因此,考虑到它们的多样化生物功能,准确地识别circrna的亚细胞定位模式对于全面了解分子生物学和不同circrna的核心功能至关重要。
3、湿实验室实验仍然是确定不同类型的ncrna亚细胞定位的最常用方法,但它们往往昂贵、资源密集且耗时,这可能不适用于大多数基因组学研究人员进行大规模研究。例如,针对microrna(mirna)的亚细胞定位,使用了免疫荧光共聚焦显微镜、免疫沉淀和亚细胞分离等方法,而针对lncrna,采用了亲和纯化、chip-pcr和双荧光素酶报告基因分析等方法。类似地,通过电子显微镜、rna序列分析和定量聚合酶链反应(qpcr)等方法确定circrna的亚细胞定位。然而,这些湿实验室实验往往需要额外的验证技术来确保实验结果的准确性。这些限制使得实验方法不适合大规模确定circrna的亚细胞定位。
4、各种元数据库的出现,包括rnalocate、ensembl和encode,通过计算手段为大规模确定不同非编码rna(ncrna)的亚细胞定位开辟了新的视角。迄今为止,已经提出了针对多种类型的ncrna的亚细胞定位预测方法,包括长非编码rna(lncrna)、mirna和信使rna(mrna)。这一进展极大地增强了对分子生物学的基本认识,突显了ncrna在关键生物功能中的作用。虽然有基于机器和深度学习的方法可以区分环状rna(circrna)与其他ncrna并预测circrna与蛋白质结合位点,但对circrna的亚细胞定位研究仍然有限。circ-locnet代表了该领域的开创性工作,首次使用计算方法预测circrna的亚细胞定位,并探索了不同序列描述符和机器学习预测器对结果的影响。它评估了使用五种常用的机器学习分类器对七种不同序列描述符的性能,并探讨了使用k阶序列描述符融合不同描述符的影响。研究发现,独立的残基频率基于序列描述符和基于树的分类器最适合预测环状rna的亚细胞定位。此外,k阶序列描述符融合与基于树的分类器相结合提供了最准确的预测。该研究结果可作为开发更准确的计算方法来确定环状rna亚细胞定位的基准。
技术实现思路
1、本发明考虑了各种基于序列的特征,并提出了一种基于岭回归的分类方法来预测circrna的亚细胞定位的方法circlocpred,对不同的circrna亚细胞定位进行计算预测。实验结果表明,circlocpred相比于最先进的方法具有更高的准确度和更均衡的识别能力。
2、以下是实现本发明目的的技术方案,包含如下步骤:
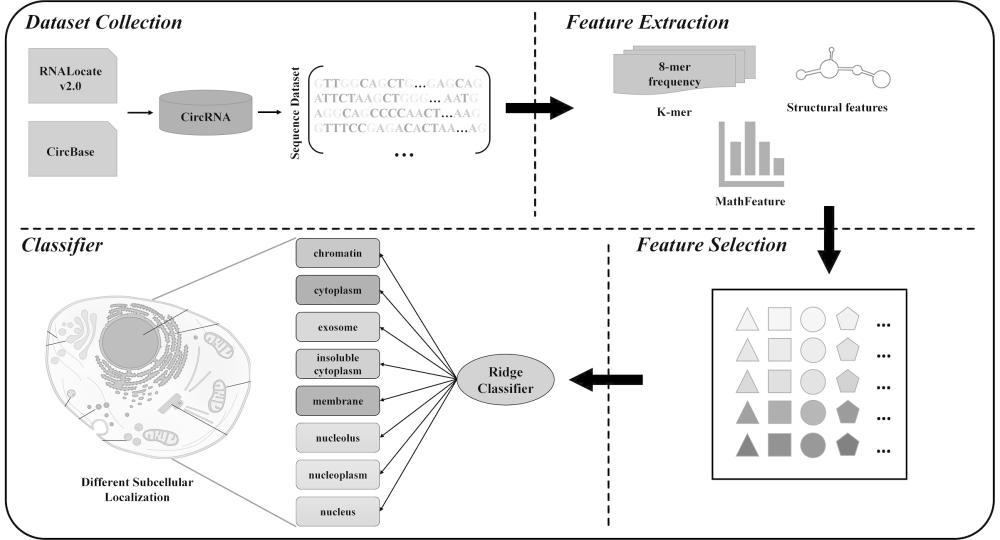
3、1)构建环状rna亚细胞定位数据集:获取含有携带亚细胞定位信息的环状rna序列;
4、2)数据预处理:使用cd-hit软件去除相似度超过80%的同源性序列,获得1230条位于八个不同亚细胞位置的样本;
5、3)生物学特征提取:使用8-mer划分rna序列并计算每个短序列出现的频率特征,根据rna二核苷酸物理化学性质计算自协方差与交叉协方差组成dacc特征;
6、4)数学特征提取:提取rna序列的数学统计特征;
7、5)特征选择与特征融合:使用ifs方法对8-mer特征进行第一次特征选择,经过筛选的特征与dacc特征、数学特征进行融合,然后使用rfe方法进行第二次特征选择;
8、6)获得预测结果:将特征选择后的特征集输入到岭回归模型得到预测结果;
9、7)采用十折交叉验证的评估方式对模型性能进行评估,也即将步骤1)中构建的数据集随机分为10组,9组用来训练,1组用于测试;
10、8)模型评估:根据步骤7)中的测试结果,使用敏感性、特异性、准确率、马修斯相关系数、auc五个评估指标衡量模型。
技术特征:
1.一种环状rna亚细胞定位的预测方法,其特征包含如下步骤:
2.根据权利要求1所述的一种环状rna亚细胞定位的预测方法,其特征在于,步骤4)中,所述的数学特征提取方法,该方法在以往研究中被发现可以有效的提取环状rna序列中蕴含的潜在信息,而且具有通用性,可以仅通过数学手段提取rna序列中的特征,与基于生物学知识提取的序列特征相结合可以显著提高模型预测的能力。
技术总结
本发明公开了一种环状RNA亚细胞定位的预测方法,其特征包含如下步骤:获取含有携带亚细胞定位信息的环状RNA序列;使用CD‑HIT软件去除相似度超过80%的同源性序列;使用8‑mer划分RNA序列并计算每个短序列出现的频率特征,根据RNA二核苷酸物理化学性质计算自协方差与交叉协方差组成DACC特征;使用工具提取RNA序列的数学统计特征;使用IFS方法对8‑mer特征进行第一次特征选择,经过筛选的特征与DACC特征、数学特征进行融合,然后使用RFE方法进行第二次特征选择;将特征选择后的特征集输入到岭回归模型得到预测结果;采用十折交叉验证的评估方式对模型性能进行评估。该方法可以仅通过数学手段提取RNA序列中的特征,与基于生物学知识提取的序列特征相结合可以显著提高模型预测的能力。
技术研发人员:樊永显,潘应捷,孙贵聪,刘梦,卢茜倩,郑梦鑫,李雪萍,郭智,王晨
受保护的技术使用者:桂林电子科技大学
技术研发日:
技术公布日:2024/3/11
- 还没有人留言评论。精彩留言会获得点赞!