一种德谷与门冬胰岛素基于分子模拟的模型构建方法
本发明涉及分子模拟,尤其是涉及一种德谷与门冬胰岛素基于分子模拟的模型构建方法。
背景技术:
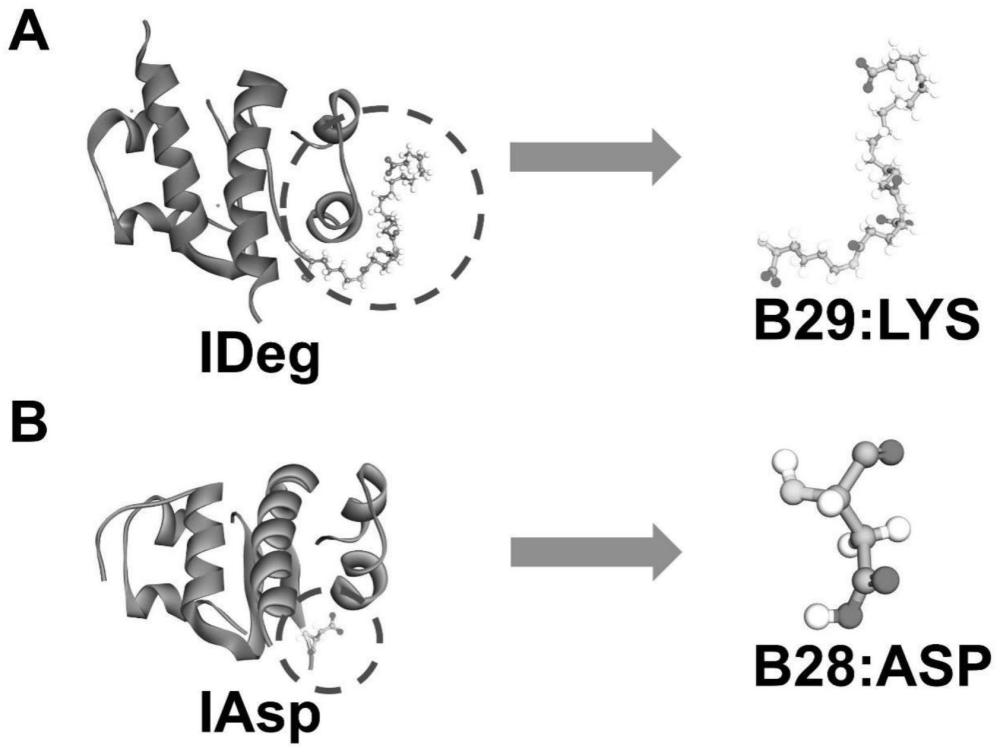
1、目前,随着医疗技术呈多样化的趋势发展,药物产品也可以根据不同个体、不同需求而个性化设计。门冬胰岛素(iasp)是由门冬氨酸替代人胰岛素b链28位的脯氨酸,而形成生物合成的超短效人胰岛素类似物。皮下注射门冬胰岛素以后吸收迅速,起效时间仅为5-15分钟,发挥最大的生物效应在1-1.5小时,持续时间仅仅是4-5个小时;德谷胰岛素(ideg)是一种超长效人胰岛素类似物,与门冬胰岛素作用效果截然相反,它是在人胰岛素b链29位的赖氨酸上接了一条包含22个碳的脂肪二酸链。起效时间非常缓慢,但持续时间超过42小时。门冬胰岛素和德谷胰岛素是根据不同需求而设计产生的蛋白类药物产品,属于新型蛋白。倘若以微观尺度研究新型蛋白作用机理产生差异的原因,通常需要借助分子模拟领域来进行分析。
2、在分子模拟中,传统的蛋白分子模型构建方法有两大关键:找到相应文件和构建模拟力场。传统的蛋白分子可以通过蛋白质数据库(pdb bank)等网站去寻找pdb文件,并且选择gromacs自带的力场(amber,charmm,gromos,opls-aa)。但是,面对新型蛋白分子时,在蛋白质数据库中有时找不到相应的pdb文件,也无法用gromacs现有的力场去识别。此外,药物制备和释放的ph环境不同,而德谷胰岛素和门冬胰岛素的制备环境为强酸性,而释放条件为人体中,故为中性,不同ph值下的力场无法构建。这些问题导致传统的蛋白分子模型构建具有很大的局限性,无法灵活应用于新型的蛋白。
技术实现思路
1、本发明的目的是提供一种德谷与门冬胰岛素基于分子模拟的模型构建方法,打破了传统分子模拟模型构建的局限性,为德谷胰岛素与门冬胰岛素蛋白分子的模型构建提供了方法。
2、为实现上述目的,本发明提供了一种德谷与门冬胰岛素基于分子模拟的模型构建方法,包括以下步骤:
3、s1、通过残基等电点计算,判断德谷胰岛素和门冬胰岛素在不同ph环境下的残基电荷,在gromcas自带的charmm36力场文件中,查找merged.rtp文件中合适的残基力场,修改德谷胰岛素和门冬胰岛素的pdb文件中的残基力场名称,构建德谷胰岛素和门冬胰岛素的残基力场;
4、其中,合适的残基力场需满足:(1)残基力场的电荷与ph条件下的真实残基电荷相吻合;(2)残基力场所对应的结构要与真实的残基结构相吻合;
5、s2、将德谷胰岛素中脂肪二酸链里的原子信息添加到gromacs的力场当中,修改德谷胰岛素的pdb文件和门冬胰岛素的pdb文件;
6、s3、使用gromacs软件将德谷胰岛素和门冬胰岛素的pdb文件转化成gro文件,同时生成拓扑文件,表示分子模拟模型成功构建;
7、利用vmd软件检查分子模拟模型构建是否存在遗漏和错误;
8、s4、将成功构建的分子模拟模型在gromacs中运行md模拟,使胰岛素蛋白模型的能量和密度达到平衡状态,截取该平衡状态下的模型构象,利用vmd查看结构的稳定性;
9、s5、利用伞状抽样分别对两个胰岛素蛋白模型进行分子动力学拖拽,分析验证构建的胰岛素蛋白模型符合胰岛素真实的作用特征。
10、优选的,步骤s2中,修改gromacs自带的charmm36力场文件中的merged.rtp、merged.hdp、residuetype.dat文件,将德谷胰岛素的脂肪二酸链的原子信息编入上述三个文件当中。
11、优选的,脂肪二酸链的原子信息包括:原子的电荷信息、原子之间的成键信息、氢原子与其他原子的匹配信息以及脂肪二酸连的种类信息。
12、优选的,步骤s3中,利用gromacs中的pdb2gmx指令将pdb文件转化成gro文件,指令全文为:
13、gmx pdb2gmx-fxxx.pdb-o xxx.gro-p topol.top-ss-merged all-ignh-ter-water spce;
14、其中,-f表示输入pdb文件名称;-o表示生成gro文件的名称;-p表示生成拓扑文件的名称;-ss表示将胰岛素的二硫键相连;-merged all表示将把胰岛素四条链合并;-ter对胰岛素链的端部进行封端处理;-water代表加入水分子的模型名称。
15、因此,本发明采用上述一种德谷与门冬胰岛素基于分子模拟的模型构建方法,通过gromacs软件构建了德谷与门冬两种胰岛素的模拟模型,将两种胰岛素在不同ph下的模型进行修正并进行md模拟和伞状抽样。该方法为后续德谷与门冬胰岛素在分子模拟领域的研究提供了模型构建以及力场修正的方法。同时,打破了传统分子模拟只能构建蛋白质数据库已有的模型,且无法直接提供不同ph环境下的力场的局限性。
16、下面通过附图和实施例,对本发明的技术方案做进一步的详细描述。
技术特征:
1.一种德谷与门冬胰岛素基于分子模拟的模型构建方法,其特征在于,包括以下步骤:
2.根据权利要求1所述的一种德谷与门冬胰岛素基于分子模拟的模型构建方法,其特征在于,步骤s2中,修改gromacs自带的charmm36力场文件中的merged.rtp、merged.hdp、residuetype.dat文件,将德谷胰岛素的脂肪二酸链的原子信息编入上述三个文件当中。
3.根据权利要求2所述的一种德谷与门冬胰岛素基于分子模拟的模型构建方法,其特征在于,脂肪二酸链的原子信息包括:原子的电荷信息、原子之间的成键信息、氢原子与其他原子的匹配信息以及脂肪二酸连的种类信息。
4.根据权利要求1所述的一种德谷/门冬胰岛素基于分子动力学模拟的模型构建方法,其特征在于,步骤s3中,利用gromacs中的pdb2gmx指令将pdb文件转化成gro文件,指令全文为:
技术总结
本发明公开了一种德谷与门冬胰岛素基于分子模拟的模型构建方法,属于分子模拟技术领域,包括以下步骤:依据德谷胰岛素和门冬胰岛素蛋白分子真实的结构,修改GROMACS中CHARMM36力场中的文件,然后用GROMACS转化成gro结构文件,以此来构建两种胰岛素蛋白的分子模拟模型。在此基础上,通过残基等电点计算和查找merged.rtp文件,修改蛋白分子pdb结构文件的残基力场,构建出了不同pH环境下的分子模拟模型。本发明采用上述的一种德谷与门冬胰岛素基于分子模拟的模型构建方法,打破了传统分子模拟模型构建的局限性,为德谷胰岛素和门冬胰岛素蛋白分子的模型构建提供了方法。
技术研发人员:郭新东,李卓霖,陈博智
受保护的技术使用者:北京化工大学
技术研发日:
技术公布日:2024/2/21
- 还没有人留言评论。精彩留言会获得点赞!