一种用于诊断非吸烟或轻度吸烟人群中肺鳞癌患者的非侵入性标记物及试剂盒的制作方法
本发明属于分子诊断领域,涉及肺鳞癌的非侵入性筛查诊断,具体涉及一种用于诊断非吸烟或轻度吸烟人群中肺鳞癌患者的非侵入性标记物及试剂盒。
背景技术:
肺癌是全球最常见的恶性肿瘤,死亡率居恶性肿瘤之首。我国肺癌发病率呈现逐年上升趋势,年平均增长近2%。肺癌根据病理学特点的不同分为多种组织类型,肺癌组织类型不同,其治疗措施也不同。根据2004版WHO分类,常见肺癌组织病理学分型分为非小细胞癌(NSCLC)和小细胞癌(SCLC)。非小细胞癌又分为鳞状细胞癌(SCC)、腺癌(AC)和大细胞癌(LCC)。不同肺癌临床治疗方案不同,预后效果也不同。因此,为了提高治疗效果,肺癌的治疗策略已经从传统的以分期为基础的治疗模式转变为以组织学类型和基因突变为指导的个体化、精准的多学科治疗模式。个体化治疗提高了肺癌的治疗和预后效果。
通常,环境空气质量下降及吸烟被认为是肺癌最主要的诱因。肺鳞癌和肺腺癌是非小细胞肺癌的两种主要组织学类型。由于它们在病理学基础、临床特征以及与吸烟关系的不同,它们的发病机理也可能不同。例如,不同的抑癌基因可能与不同组织学类型的肺癌发生有关。国外有关人认为吸烟导致不同组织类型肺癌的机理可能与关键性的基因突变有关。这一点已逐渐为人们所重视,特别是原癌基因的激活或抑癌基因失活均与肿瘤的发生相关。
主流观点普遍认为肺腺癌和肺鳞癌在不同性别人群以及吸烟与非吸烟人群之间存在着不同的发病机制,组织病理学存在差异,预后及生存期也大为不同。
申请人于2017年2月20号提交了两件名称分别为“血浆microRNAs用于制备筛查诊断男性群体中肺腺癌患者的诊断试剂的用途”和“血浆microRNAs用于制备筛查诊断女性群体中肺腺癌患者的诊断试剂的用途”的专利申请,提供的两组血浆microRNAs标记物可以分别准确用于筛查诊断男性和女性群体中肺腺癌患者,灵敏度高,特异性强,从而实现男性和女性群体中肺腺癌患者的无介入诊断;而申请人在未将临床样本做性别区分之前一直无法寻找到可以准确用于筛查肺腺癌的microRNAs标记物。
同样,申请人在寻找用于诊断肺鳞癌的microRNAs标记物时,不管单个差异microRNA还是多个差异microRNAs的组合均不具备诊断区分肺鳞癌与健康人的价值。于是,申请人按性别对人群分类,同样未发现具有诊断价值的microRNA或其组合。最终,申请人按是否吸烟以及吸烟指数对人群样本进行了分组,发现了两组用于诊断肺鳞癌的microRNAs标记物,但需要限定临床样本的吸烟指数,且该两组microRNAs标记物无法覆盖所有人群。
技术实现要素:
本发明的目的在于提供用于准确诊断肺鳞癌的血浆microRNAs标记物和诊断试剂盒;具体地,为提高筛查的准确度、敏感度和特异性,根据吸烟严重程度,提供一组用于非侵入性诊断重度吸烟人群中肺鳞癌患者的microRNAs标记物,提供另外一组用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的microRNAs标记物。其中,重度吸烟指吸烟指数≥400;轻度吸烟指吸烟指数<200,吸烟指数=每天吸烟支数×吸烟年数。
上述目的是通过如下技术方案实现的:
重度吸烟人群中肺鳞癌患者的诊断:
一组用于非侵入性诊断重度吸烟人群中肺鳞癌患者的血浆microRNAs标记物,所述重度吸烟人群指吸烟指数≥400的人群,由如下microRNAs组成:miR-432-3p、miR-371a-3p、miR-588、miR-676-5p;miR-432-3p的核苷酸序列如SEQ ID NO.1所示;miR-371a-3p的核苷酸序列如SEQ ID NO.2所示;miR-588的核苷酸序列如SEQ ID NO.3所示;miR-432-3p的核苷酸序列如SEQ ID NO.4所示。
一组用于非侵入性诊断重度吸烟人群中肺鳞癌患者的microRNAs引物、探针的组合,所述重度吸烟人群指吸烟指数≥400的人群,microRNAs引物包括逆转录引物、定量PCR前引物和定量PCR后引物:miR-432-3p的逆转录引物序列如SEQ ID NO.9所示,定量PCR前引物序列如SEQ ID NO.17所示;miR-371a-3p的逆转录引物序列如SEQ ID NO.10所示,定量PCR前引物序列如SEQ ID NO.18所示;miR-588的逆转录引物序列如SEQ ID NO.11所示,定量PCR前引物序列如SEQ ID NO.19所示;miR-676-5p的逆转录引物序列如SEQ ID NO.12所示,定量PCR前引物序列如SEQ ID NO.20所示;各microRNAs定量PCR后引物序列如SEQ ID NO.25所示,各microRNAs探针序列如SEQ ID NO.26所示。
一种用于非侵入性诊断重度吸烟人群中肺鳞癌患者的诊断试剂盒,含有上述microRNAs引物、探针的组合。
进一步地,所述的诊断试剂盒还含有PCR反应常用的酶和试剂,如逆转录酶、缓冲液、dNTPs、MgCl2、DEPC水和Taq酶等,以及标准品和/或对照品。
非吸烟或轻度吸烟人群中肺鳞癌患者的诊断:
一组用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的血浆microRNAs标记物,所述轻度吸烟人群指吸烟指数<200的人群,由如下microRNAs组成:miR-644a、miR-619-5p、miR-222-3p、miR-575;miR-644a的核苷酸序列如SEQ ID NO.5所示;miR-619-5p的核苷酸序列如SEQ ID NO.6所示;miR-222-3p的核苷酸序列如SEQ ID NO.7所示;miR-575的核苷酸序列如SEQ ID NO.8所示。
一组用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的microRNAs引物、探针的组合,所述轻度吸烟人群指吸烟指数<200的人群,microRNAs引物包括逆转录引物、定量PCR前引物和定量PCR后引物:miR-644a的逆转录引物序列如SEQ ID NO.13所示,定量PCR前引物序列如SEQ ID NO.21所示;miR-619-5p的逆转录引物序列如SEQ ID NO.14所示,定量PCR前引物序列如SEQ ID NO.22所示;miR-222-3p的逆转录引物序列如SEQ ID NO.15所示,定量PCR前引物序列如SEQ ID NO.23所示;miR-575的逆转录引物序列如SEQ ID NO.16所示,定量PCR前引物序列如SEQ ID NO.24所示;各microRNAs定量PCR后引物序列如SEQ ID NO.25所示,各microRNAs探针序列如SEQ ID NO.26所示。
一种用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的诊断试剂盒,含有上述microRNAs引物、探针的组合。
进一步地,所述的诊断试剂盒还含有PCR反应常用的酶和试剂,如逆转录酶、缓冲液、dNTPs、MgCl2、DEPC水和Taq酶等,以及标准品和/或对照品。
本发明的有益效果:
为提高筛查的准确度、敏感度和特异性,根据吸烟严重程度,本发明提供一组用于非侵入性诊断重度吸烟人群中肺鳞癌患者的microRNAs标记物,提供另外一组用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的microRNAs标记物,诊断准确度高,敏感度高、特异性强;其中,重度吸烟指吸烟指数≥400;轻度吸烟指吸烟指数<200,吸烟指数=每天吸烟支数×吸烟年数。遗憾的是,该两组microRNAs标记物均不适用于吸烟指数介于200和400之间的吸烟人群中肺鳞癌患者的诊断,准确度低,无临床应用价值。
附图说明
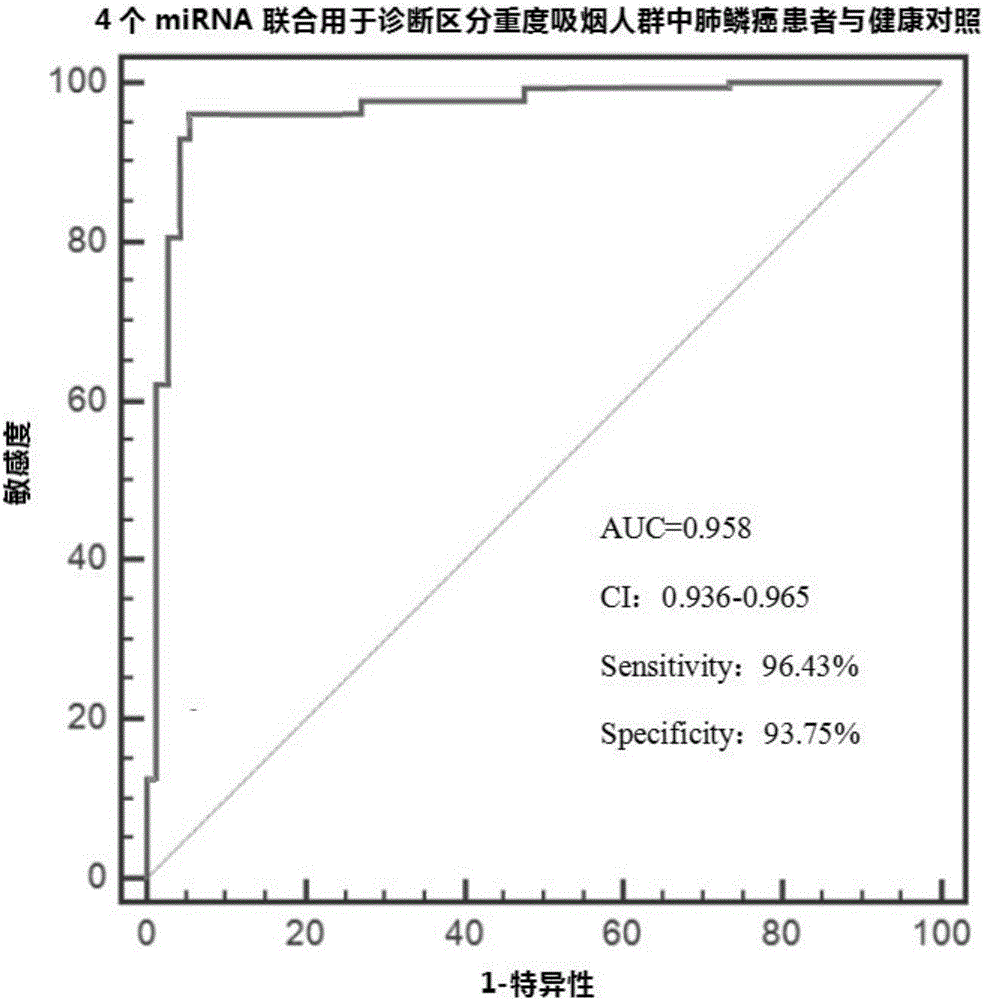
图1为4个microRNAs(miR-432-3p、miR-371a-3p、miR-588、miR-676-5p)联合诊断区分重度吸烟人群中肺鳞癌患者与健康对照的ROC曲线图;
图2为4个microRNAs(miR-644a、miR-619-5p、miR-222-3p、miR-575)联合诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的ROC曲线图;
图3为验证集中用4个microRNAs(miR-432-3p、miR-371a-3p、miR-588、miR-676-5p)联合诊断区分重度吸烟人群中肺鳞癌患者与健康对照的准确性图;
图4为验证集中用4个microRNAs(miR-644a、miR-619-5p、miR-222-3p、miR-575)联合诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的准确性图。
具体实施方式
下面结合具体实施例和附图详细介绍本发明的技术方案和技术效果。未注明具体条件的实验方法,通常按照常规条件,例如教科书和实验指南中所述的条件,或按照制造厂商所建议的条件,为本领域普通技术人员熟知或易于获知。
肺鳞癌患者和健康志愿者自2014年9月至2015年9月期间于南京军区南京总医院和南京鼓楼医院收集,取血经患者同意并通过南京军区南京总医院和南京鼓楼医院伦理委员会审批。重度吸烟人群(吸烟指数≥400)共88例,其中原发性肺鳞癌56例,健康对照32例,样本年龄在35-60岁之间,且原发性肺鳞癌和健康对照人群年龄无统计学差异(P=0.041);非吸烟或轻度吸烟人群(吸烟指数<200)共96例,其中原发性肺鳞癌62例,健康对照34例;样本年龄在25-60岁之间,且原发性肺鳞癌和健康对照人群年龄无统计学差异(P=0.037)。所有肺鳞癌患者和健康对照均经过组织病理确证,无既往肿瘤病史,且血常规正常。
将重度吸烟人群中的肺鳞癌患者和健康对照分别随机对半分成测试集和验证集;将非吸烟或轻度吸烟人群中的肺鳞癌患者和健康对照分别随机对半分成测试集和验证集。测试集用于血浆差异microRNAs的发现,并初步用于评价差异microRNAs作为标记物诊断区分肺鳞癌和健康对照的诊断效能;验证集用于进一步验证microRNAs标记物的诊断准确性。
取血之前均未进行手术、化疗、放疗或内分泌治疗。
实施例1基于microRNA芯片的血浆差异microRNA发现及定量确证
一、实验材料
1、仪器试剂
高速离心机、高速冷冻离心机、紫外分光光度计购自德国Eppendorf公司;NanoDrop 1000分光光度计购自美国赛默飞世尔Thermo Scientific公司;Agilent 2100Bioanalyzer System购自美国Agilent公司;普通PCR反应仪和实时荧光PCR仪购自美国PE公司;DYCP-31B型电泳仪购自北京六一生物科技有限公司;BIO-RAD凝胶成像分析购自美国伯乐;超低温冰箱购自海尔集团。mirvanaTM parisTM kit购自美国Ambion公司;Agilent RNA 6000Pico Kit购自美国Agilent公司;Trizol总RNA提取试剂购自美国Invitrogen公司;TaqMan MicroRNAReverse Transcription Kit、TaqMan Universal PCR Master Mix、cDNA合成试剂盒购自美国赛默飞世尔Thermo Scientific公司;引物、探针委托生工生物工程(上海)股份有限公司合成。未特别强调的仪器和试剂均为本领域技术人员常规使用的仪器和试剂,且易于获得。
2、实验样品
早晨空腹抽取测试集肺鳞癌患者和健康对照外周血2mL,放入EDTA管内,轻轻混匀,使血与管内抗凝物质充分接触,防止血细胞破裂。将EDTA管放入4℃冰箱,在2小时内分离血浆。首先,820g转速离心10分钟,将上层液相小心吸出,避免吸取中间白色细胞层。将上层液相转入事先预冷的1.5mL离心管,16000g转速继续离心10分钟,以便进一步分离血浆和血细胞。将第二步离心以后得到的血浆,以500μL体积分装,转入-80℃冰箱长期保存。
二、实验方法
1、血浆总RNA的提取
mirvanaTM parisTM kit用于抽提血浆总RNA,NanoDrop 1000分光光度计测定总RNA浓度。对RNA质量的评价通过Agilent RNA 6000Pico Kit由Agilent 2100Bioanalyzer System来完成。方法均参照各自说明书。用RNA完整指数(RIN)衡量总RNA的质量,该值从1到10分别反应样本的质量。RIN≥7-10表示质量好,RIN≥5表示质量中等,RIN<5表示质量差。本实验中,以RIN>5作为抽提总RNA合格的标准。
2、全基因组microRNA分析
microRNA芯片检测以及结果分析由上海生物芯片有限公司完成。microRNA芯片包含723个人microRNAs和76个人病毒microRNAs探针,microRNA数据来自Sanger v.10.1数据库。每一张芯片包含八个上样位点。总RNA(100ng)通过Cy3标记,芯片通过XDR Scan(PMT100,PMT5)扫描芯片上的信号。标记和杂交过程按照Agilent microRNA芯片系统说明书操作。芯片图像信息通过软件转换为强度值,消除背景嘈杂信号以后,信号强度值直接输入到软件进行分析。在对样本的检测过程中,发现hsa-miR-1228在血浆中稳定表达,故选用hsa-miR-1228作为内参,将杂交得到的原始信号进行标准化,得到以2为底数的log值。如果一个样本在芯片上重复检测的数值,其变异系数大于15%或者阳性信号值不到5%,认为该样本质量不合格,在下一步试验中将被排除。可以检测到的microRNA定义为在超过50%的样本中,芯片都能检测到阳性信号。
3、实时荧光定量PCR
以总RNA为逆转录模板,使用带有茎环结构的特异性引物将microRNA逆转录为cDNA。将RT的产物cDNA使用TaqMan探针法进行定量PCR检测。PCR扩增釆用特异性前引物及通用后引物,并使用特异性的TaqMan探针检测目的片段。具体参照试剂盒TaqMan MicroRNA Reverse Transcription Kit、TaqMan MicroRNAAssay、TaqMan Universal PCR Master Mix的说明书,每个样品上样量为100ng,反应步骤如下:
100ng RNA标本加入每个RT反应试管中,Nuclease-free water补足体积;加入各反应成分后稍作离心混匀,在PCR反应仪执行如下程序:
16℃30min→42℃30min→85℃5min→4℃保存。
参照TaqMan MicroRNAAssay说明书在实时荧光定量PCR仪上进行,体系和条件如下:
加入各反应成分并充分混匀后稍作离心,每个孔10μL,每个样本做三个复孔。在实时荧光定量PCR仪上执行如下程序:
总共40个循环。60℃时进行荧光数据采集,吸收波长为490nm,释放波长为530nm。Ct值由SDS软件经过二次衍生法计算得到。
4、数据统计分析
以芯片检测结果中最稳定的hsa-miR-1228作为内参对照,利用Agilent Feature Extraction软件进行数据定量分析。芯片的图像信息通过Scanner Control Rev.7.0软件转换为密度值。信号经背景消除后直接导入GeneSpring GX10软件进行标准化。以重复试验获得的平均标准化数据比较研究分析。Benjamini-Hochberg校对的非配对t检验(p≤0.01)。利用hierarchical clustering algorithm软件进行聚类分析,筛选出重度吸烟人群中肺鳞癌与健康对照、非吸烟或轻度吸烟人群中肺鳞癌与健康对照中具有显著性差异的血浆microRNAs。
实时荧光PCR数据采用与芯片相同的内参标准化,非配对t检验确定组间差异。p<0.05设为统计学差异。F检验和T检验分析通过荧光实时定量PCR检测得到的microRNAs表达值之间的显著性差异,通过SPSS软件分析实现,p<0.05认为有统计学意义,均为双侧。
未注明具体条件的实验方法,通常按照常规条件,例如教科书和实验指南中所述的条件,或按照制造厂商所建议的条件,为本领域普通技术人员熟知或易于获知。
三、实验结果
microRNA芯片检测结果发现,重度吸烟人群中,与健康对照相比,肺鳞癌病例出现大量上调或下调表达的microRNAs,部分microRNAs差异表达非常明显;非吸烟或轻度吸烟人群中,与健康对照相比,肺鳞癌病例出现大量上调或下调表达的microRNAs,部分microRNAs差异表达非常明显。由于microRNA芯片对microRNA表达特征的研究是间接的,在分析过程中仍存在特异性和敏感度不高等缺点。因此,所得结果有一定的假阳性,需要辅助其他更为准确的表达研究方法加以验证,本实验利用荧光实时定量RT-PCR来进行鉴定。
部分变化显著的血浆差异microRNAs得到荧光实时定量RT-PCR的确认,表达水平如下:
得到上述变化倍数显著的血浆差异microRNA后,进行下一步实验,采用受试者工作特征曲线(ROC曲线)评价单个血浆差异microRNA及其联合用于诊断区分重度吸烟人群中肺鳞癌患者与健康对照、非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的诊断效能。
实施例2:ROC曲线评价血浆差异microRNA的诊断效能
ROC曲线评价法的原理:
诊断试验的基本评价指标有敏感度、特异性等,综合评价指标有Youden指数、ROC、AUC等。对于诊断试验的评价,首先应该知道受试者的真实类别,即哪些属于健康组,哪些属于疾病组。划分健康组和疾病组的标准就是金标准(如本申请中公认的组织病理诊断法)。对于按金标准确定的疾病组和健康组,采用诊断试验检测的结果可以分为如下情况:
阳性(True Positive,TP);诊断试验检测为阳性(与金标准结果一致);
阴性(True Negative,TN);诊断试验检测为阴性(与金标准结果一致);
假阳性(False Positive,FP):诊断试验检测为阳性(与金标准结果不一致);
假阴性(False Negative,FN):诊断试验检测为阴性(与金标准结果不一致)。
可以用下表表示:
诊断试验的敏感度=A/(A+C);诊断试验的特异性=D/(B+D)。通过敏感度和特异性可以得出诊断试验相对于金标准的诊断灵敏程度和特异程度。敏感度高代表将疾病例诊断为阴性的个数少,漏诊率低;特异性高代表将健康例诊断为阳性的个数少,误诊率低。
ROC曲线正是基于上述敏感度和特异性绘制出的曲线。以诊断试验中可能的诊断界值作为诊断点,根据上述表格计算出相应的敏感度和特异性。然后,以敏感度为纵坐标,1-特异性为横坐标,将各诊断点时各点的敏感度和特异性点在坐标图中标出,连接坐标点得到平滑曲线,该曲线即为ROC曲线。诊断点设置的越多越密,得到的ROC曲线就越平滑。
ROC曲线是以每一个检测结果作为可能的诊断界值,其曲线下面积AUC的大小表明了诊断试验准确度的大小。ROC曲线下面积AUC(ROCAUC)作为诊断试验真实性评价的固有准确度指标已被普遍认可,完全无价值的诊断试验AUC为0.5,理想的诊断试验AUC为1,而一般认为对于一个诊断试验,ROC AUC在0.5~0.7之间时诊断价值较低,在0.7~0.9之间时诊断价值中等,在0.9以上时诊断价值较高。
1、单个血浆差异microRNA用于诊断区分肺鳞癌和健康对照时ROC曲线的绘制方法
分别将测试集重度吸烟人群所有样本中miR-432-3p、miR-371a-3p、miR-588、miR-676-5p的含量经内参标准化后,得到标准化值,以各可能的标准化值作为诊断点,按照上述方法绘制诊断区分重度吸烟人群中肺鳞癌患者与健康对照的ROC曲线;同理,分别将测试集非吸烟或轻度吸烟人群所有样本中miR-644a、miR-619-5p、miR-222-3p、miR-575的含量经内参标准化后,得到标准化值,以各可能的标准化值作为诊断点,按照上述方法绘制诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的ROC曲线。单独microRNA诊断区分肺鳞癌和健康对照的ROC曲线下面积AUC及最佳cut-off值处敏感度和特异性如下表所示:
结果单个差异microRNA诊断区分重度吸烟人群中肺鳞癌患者与健康对照、诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的诊断效能低,AUC在0.5~0.7之间。
2、多个差异microRNAs联合用于诊断区分肺鳞癌和健康对照时ROC曲线的绘制方法
分别将测试集重度吸烟人群所有样本中miR-432-3p、miR-371a-3p、miR-588、miR-676-5p的含量经内参标准化后,得到标准化值,将肺鳞癌样本作为组别1,健康对照样本作为组别2,对miR-432-3p、miR-371a-3p、miR-588、miR-676-5p在两组样本中的标准化值进行二元逻辑回归,得到二元逻辑回归方程;将各样本中miR-432-3p、miR-371a-3p、miR-588、miR-676-5p的标准化值代入该二元逻辑回归方程,即可得到各个样本的回归值,以可能的回归值作为诊断点,按照上述方法绘制诊断区分重度吸烟人群中肺鳞癌患者与健康对照的ROC曲线。
同理,将测试集非吸烟或轻度吸烟人群所有样本中miR-644a、miR-619-5p、miR-222-3p、miR-575的含量经内参标准化后,得到标准化值,将肺鳞癌样本作为组别1,健康对照样本作为组别2,对miR-644a、miR-619-5p、miR-222-3p、miR-575在两组样本中的标准化值进行二元逻辑回归,得二元逻辑回归方程;将各样本miR-644a、miR-619-5p、miR-222-3p、miR-575的标准化值代入该二元逻辑回归方程,即得各个样本的回归值,以可能的回归值作为诊断点,按照上述方法绘制诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照的ROC曲线。
上述ROC曲线即为多个血浆差异microRNAs联合诊断时的诊断曲线,曲线下面积AUC及最佳cut-off值处敏感度和特异性可体现该多个血浆差异microRNAs的联合诊断效能。
结果表明,miR-432-3p、miR-371a-3p、miR-588、miR-676-5p联合用于诊断区分重度吸烟人群中肺鳞癌患者与健康对照具有较高的诊断效能,AUC在0.9以上,敏感度高、特异性强;miR-644a、miR-619-5p、miR-222-3p、miR-575联合用于诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照具有较高的诊断效能,AUC在0.9以上,敏感度高、特异性强。
结果如下表和图1和图2所示。
因此,miR-432-3p、miR-371a-3p、miR-588、miR-676-5p联合用于诊断区分重度吸烟人群中肺鳞癌患者与健康对照时诊断效能高;miR-644a、miR-619-5p、miR-222-3p、miR-575联合用于诊断区分非吸烟或轻度吸烟人群中肺鳞癌患者与健康对照时诊断效能高。
实施例3:在验证集中进一步验证血浆差异microRNAs联合诊断的准确性
一、实验材料
同实施例1。
二、实验方法和结果
1、血浆总RNA的提取和实时荧光定量PCR的方法同实施例1。
2、在验证集中,基于上述二元逻辑回归方程将验证集重度吸烟人群样本中的4个血浆差异microRNAs(miR-432-3p、miR-371a-3p、miR-588、miR-676-5p)含量的标准化值作二元逻辑回归变换,计算出重度吸烟人群样本中的该4个血浆差异microRNAs含量的逻辑回归值。低于最佳cut-off值0.496的预测为健康对照,高于最佳cut-off值0.496的预测为肺鳞癌,最后计算出以该4个代谢标志物水平预测重度吸烟人群中肺鳞癌的准确率。结果如图3,基于上述4个血浆差异microRNAs验证集样本中的预测准确率为95.5%。
3、在验证集中,基于上述二元逻辑回归方程将验证集非吸烟或轻度吸烟人群样本中的4个血浆差异microRNAs(miR-644a、miR-619-5p、miR-222-3p、miR-575)含量的标准化值作二元逻辑回归变换,计算出非吸烟或轻度吸烟人群样本中的该4个血浆差异microRNAs含量的逻辑回归值。低于最佳cut-off值0.443的预测为健康对照,高于最佳cut-off值0.443的预测为肺鳞癌,最后计算出以该4个代谢标志物水平预测非吸烟或轻度吸烟人群中肺鳞癌的准确率。结果如图4,基于上述4个血浆差异microRNAs验证集样本中的预测准确率为95.8%。
实施例4:诊断试剂盒的制备
上述实施例表明,miR-432-3p、miR-371a-3p、miR-588、miR-676-5p联合诊断区分重度吸烟人群中肺鳞癌和健康对照的准确度高,敏感度和特异性强,可基于miR-432-3p、miR-371a-3p、miR-588、miR-676-5p制作诊断重度吸烟人群中肺鳞癌患者的非侵入性诊断试剂盒。该诊断试剂盒中包括miR-432-3p引物、探针;miR-371a-3p引物、探针;miR-588引物、探针;miR-676-5p引物、探针。引物具体包括逆转录引物、定量PCR前引物和定量PCR后引物。当然,诊断试剂盒还含有PCR反应常用的酶和试剂,如逆转录酶、缓冲液、dNTPs、MgCl2、DEPC水和Taq酶等,以及标准品和/或对照品。引物和探针的设计是本领域常规技术手段,下表为引物和探针的一种设计,也可以设计成其他序列。
上述实施例表明,miR-644a、miR-619-5p、miR-222-3p、miR-575联合诊断区分非吸烟或轻度吸烟人群中肺鳞癌和健康对照的准确度高,敏感度和特异性强,可基于miR-644a、miR-619-5p、miR-222-3p、miR-575制作诊断非吸烟或轻度吸烟人群中肺鳞癌患者的非侵入性诊断试剂盒。该诊断试剂盒中包括miR-644a引物、探针;miR-619-5p引物、探针;miR-222-3p引物、探针;miR-575引物、探针。引物具体包括逆转录引物、定量PCR前引物和定量PCR后引物。当然,诊断试剂盒还含有PCR反应常用的酶和试剂,如逆转录酶、缓冲液、dNTPs、MgCl2、DEPC水和Taq酶等,以及标准品和/或对照品。引物和探针的设计是本领域常规技术手段,下表为引物和探针的一种设计,也可以设计成其他序列。
为提高筛查的准确度、敏感度和特异性,根据吸烟严重程度,本发明提供一组用于非侵入性诊断重度吸烟人群中肺鳞癌患者的microRNAs标记物,提供另外一组用于非侵入性诊断非吸烟或轻度吸烟人群中肺鳞癌患者的microRNAs标记物,诊断准确度高,敏感度高、特异性强;其中,重度吸烟指吸烟指数≥400;轻度吸烟指吸烟指数<200,吸烟指数=每天吸烟支数×吸烟年数。遗憾的是,该两组microRNAs标记物均不适用于吸烟指数介于200和400之间的吸烟人群中肺鳞癌患者的诊断,准确度低,无临床应用价值。
SEQUENCE LISTING
<110> 南京九寿堂医药科技有限公司
<120> 一种用于诊断非吸烟或轻度吸烟人群中肺鳞癌患者的非侵入性标记物及试剂盒
<130> 1
<160> 26
<170> PatentIn version 3.3
<210> 1
<211> 21
<212> DNA
<213> 智人
<400> 1
cuggauggcu ccuccauguc u 21
<210> 2
<211> 23
<212> DNA
<213> 智人
<400> 2
aagugccgcc aucuuuugag ugu 23
<210> 3
<211> 21
<212> DNA
<213> 智人
<400> 3
uuggccacaa uggguuagaa c 21
<210> 4
<211> 21
<212> DNA
<213> 智人
<400> 4
ucuucaaccu caggacuugc a 21
<210> 5
<211> 19
<212> DNA
<213> 智人
<400> 5
aguguggcuu ucuuagagc 19
<210> 6
<211> 22
<212> DNA
<213> 智人
<400> 6
gcugggauua caggcaugag cc 22
<210> 7
<211> 21
<212> DNA
<213> 智人
<400> 7
agcuacaucu ggcuacuggg u 21
<210> 8
<211> 19
<212> DNA
<213> 智人
<400> 8
gagccaguug gacaggagc 19
<210> 9
<211> 56
<212> DNA
<213> 人工序列
<400> 9
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacag acatgg 56
<210> 10
<211> 56
<212> DNA
<213> 人工序列
<400> 10
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacac actcaa 56
<210> 11
<211> 56
<212> DNA
<213> 人工序列
<400> 11
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacgt tctaac 56
<210> 12
<211> 56
<212> DNA
<213> 人工序列
<400> 12
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgactg caagtc 56
<210> 13
<211> 56
<212> DNA
<213> 人工序列
<400> 13
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacgc tctaag 56
<210> 14
<211> 56
<212> DNA
<213> 人工序列
<400> 14
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacgg ctcatg 56
<210> 15
<211> 56
<212> DNA
<213> 人工序列
<400> 15
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacac ccagta 56
<210> 16
<211> 56
<212> DNA
<213> 人工序列
<400> 16
gtcgtatcca gtgcgtgtcg tggagtcggc aattgcactg gatacgacgc tcctgt 56
<210> 17
<211> 31
<212> DNA
<213> 人工序列
<400> 17
acactccagc tgggctggat ggctcctcca t 31
<210> 18
<211> 31
<212> DNA
<213> 人工序列
<400> 18
acactccagc tgggaagtgc cgccatcttt t 31
<210> 19
<211> 31
<212> DNA
<213> 人工序列
<400> 19
acactccagc tgggttggcc acaatgggtt a 31
<210> 20
<211> 31
<212> DNA
<213> 人工序列
<400> 20
acactccagc tgggtcttca acctcaggac t 31
<210> 21
<211> 31
<212> DNA
<213> 人工序列
<400> 21
acactccagc tgggagtgtg gctttcttag a 31
<210> 22
<211> 31
<212> DNA
<213> 人工序列
<400> 22
acactccagc tggggctggg attacaggca t 31
<210> 23
<211> 31
<212> DNA
<213> 人工序列
<400> 23
acactccagc tgggagctac atctggctac t 31
<210> 24
<211> 31
<212> DNA
<213> 人工序列
<400> 24
acactccagc tggggagcca gttggacagg a 31
<210> 25
<211> 23
<212> DNA
<213> 人工序列
<400> 25
cgccgcagtg cgtgtcgtgg agt 23
<210> 26
<211> 8
<212> DNA
<213> 人工序列
<400> 26
cgtatcca 8
- 还没有人留言评论。精彩留言会获得点赞!