一种基于深度度量学习的染色体分类方法与流程
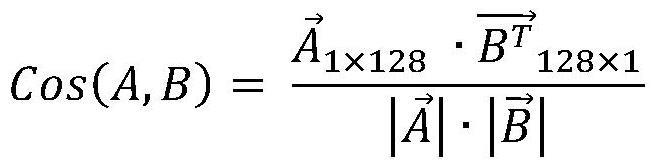
本发明涉及医学影响处理领域,尤其是一种基于深度度量学习的染色体分类方法。
背景技术:
1、针对染色体类别的分类算法是染色体核型分析中的重要环节。传统的核型分析,需要一块具有专业技能和知识的医师,根据染色体的长度、着丝点位置、长短臂比例、随体的有无等特征对染色体的类别进行判断。现有的机器学习算法无法在如下情况下对染色体类别进行准确分类:
2、(1)类别相似性过高:针对不同类别的染色体,人类正常体细胞2n=46,其中常染色体22对,性染色体1对,其类别间相似性非常高。在临床实践中,只有经过专业训练的科研人员能够准确判别其差异并对齐正确分类。在计算机视觉问题上,不同于传统的图像分类任务,染色体类别分类任务存在类别间相似度过高的问题,而容易导致分类不准确。
3、(2)批次差异过大:受限于批次差异,同一类别的染色体图像在不同分裂时间、不同拍摄设备、不同拍摄环境、显带技术、以及空间位置差异。都能够导致拍摄到的同一类别的染色体的形状、显带深度、粗细、长短有较大差异。批次差异由于其受限于可能存在的人为因素,拍摄环境,实验人员的专业素养差异,而不可避免。
技术实现思路
1、为解决上述问题,本发明提出了一种基于深度度量学习的染色体分类方法。
2、本发明的主要内容包括:
3、一种基于深度度量学习的染色体分类方法,包括如下步骤:
4、构建训练集以及三元组损失函数;
5、使用构建的训练集和三元组损失函数对分类识别网络模型以及特征映射网络进行训练;
6、将待识别的染色体图像的融合特征向量以及染色体样本集图像的融合特征向量输入到训练好的分类识别网络模型中,得出待识别的染色体图像的类别;
7、其中,所述三元组损失函数如下:
8、l(x,x+,x-;f)=max(0,‖f-f+‖-‖f-f-‖+m)
9、f为特征映射网络,m是一个大于0的常数;x为锚点样本;x+为与锚点样本x属于同一类的正例样本,x-为与锚点样本x属于不同类的负例样本;‖f-f+‖表示锚点样本与正例样本之间的距离;‖f-f-‖表示锚点样本与负例样本之间的距离;
10、所述特征映射网络包括特征提取层以及特征拼接层,所述特征提取层用于提取输入图像的全局特征以及特定特征,所述特征拼接层用于拼接所述全局特征和所述特定特征,得到融合特征,并对融合特征进行展平编码,得到融合特征向量。
11、优选的,所述特征映射网络将输入图像的原始特征映射至向量空间,包括:
12、将输入网络的染色体图像进行统一尺寸,归一化操作,得到归一化后的染色体图像;
13、将归一化后的染色体图像映射至128维度的向量空间,得到染色体向量空间。
14、优选的,将输入网络的染色体图像进行统一尺寸,归一化操作,包括:将输入网络的染色体图像统一尺寸为256*256。
15、优选的,所述特征映射网络具体包括6层:
16、第一层,用于输入样本集中的重叠染色体图像;输入维度256*256*3;
17、第二层,由一个大小为3的卷积核和大小为128x128的64个特征映射图组成;
18、第三层,由三个大小为3的卷积核和大小为64x64的128个特征映射图组成;
19、第四层,由三个大小为3的卷积核和大小为32x32的256个特征映射图组成;
20、第五层,为最大池化层,将特征图进行最大池化并展平;
21、第六层,为全连接层,将展平特征进行全连接映射并输出128维特征。
22、优选的,还包括对染色体向量空间进行正则化的步骤:
23、对于特征提取网络输出的128维度向量:x=(x1,x2,x3…,x128),对其输入的每一个维度进行归一化:
24、
25、其中,i∈[1,128],μ和σ是该批次向量的均值和标准差,ε是任意小的常数。
26、优选的,根据待识别融合特征向量和样本融合特征向量的相似度,得出待识别的染色体图像的类别,包括:
27、设计距离函数,计算待识别融合特征向量与多个样本融合特征向量之间的距离;
28、选择与待识别融合特征向量的距离最小的样本融合特征向量对应的类别作为待识别融合特征向量的类别。
29、优选的,所述距离函数为:
30、
31、其中是两个128维度的向量,计算量两个向量之间的点积,而为两个向量的模。
32、优选的,构建训练集以及三元组损失函数,包括如下步骤:
33、从带标签的训练样本中随机选取锚点样本,选取同标签样本作为正例样本,选取异标签样本作为负例样本,构成待训练三元组(x,x+,x-);
34、选取满足cos(x,x+)+m<cos(x,x-),得到标准三元组;
35、将标准三元组输入分类识别网络模型中,采用adam优化器最小化三元组损失函数,反向传播,优化分类识别网络模型的参数。
36、优选的,所述特定特征包括染色体的条带特征。
37、本发明的有益效果在于:本发明提出了一种基于深度度量学习的染色体分类方法,能够通过学习不同批次间同一类别染色体的相似特征,在预测过程中克服相同类别染色体之间的批次差异,分类准确性高。
技术特征:
1.一种基于深度度量学习的染色体分类方法,其特征在于,包括如下步骤:
2.根据权利要求1所述的一种基于深度度量学习的染色体分类方法,其特征在于,所述特征映射网络将输入图像的原始特征映射至向量空间,包括:
3.根据权利要求2所述的一种基于深度度量学习的染色体分类方法,其特征在于,将输入网络的染色体图像进行统一尺寸,归一化操作,包括:将输入网络的染色体图像统一尺寸为256*256。
4.根据权利要求2所述的一种基于深度度量学习的染色体分类方法,其特征在于,所述特征映射网络具体包括6层:
5.根据权利要求2所述的一种基于深度度量学习的染色体分类方法,其特征在于,还包括对染色体向量空间进行正则化的步骤:
6.根据权利要求1所述的一种基于深度度量学习的染色体分类方法,其特征在于,根据待识别融合特征向量和样本融合特征向量的相似度,得出待识别的染色体图像的类别,包括:
7.根据权利要求6所述的一种基于深度度量学习的染色体分类方法,其特征在于,所述距离函数为:
8.根据权利要求7所述的一种基于深度度量学习的染色体分类方法,其特征在于,构建训练集以及三元组损失函数,包括如下步骤:
9.根据权利要求1所述的一种基于深度度量学习的染色体分类方法,其特征在于,所述特定特征包括染色体的条带特征。
技术总结
本发明提出了一种基于深度度量学习的染色体分类方法,通过设计的三元损失函数,使用Adam优化器,优化锚点样本与正例的距离,使其小于锚点样本与负例的距离,解决染色体分类问题。本发明的分类方法能够通过学习不同批次间同一类别染色体的相似特征,在预测过程中克服相同类别染色体之间的批次差异,分类准确性高。
技术研发人员:李娜,牛兵,陈思远,朱春阳
受保护的技术使用者:笑纳科技(苏州)有限公司
技术研发日:
技术公布日:2024/1/15
- 还没有人留言评论。精彩留言会获得点赞!