基于spark平台的基因数据分析方法与流程
本发明涉及基因数据测序技术领域,尤其涉及一种基于spark平台的基因数据分析方法。
背景技术:
近年来,基因测序技术得到了迅速发展,尤其是二代测序(ngs,nextgenerationsequence)技术的广泛应用,使得基因测序在疾病监测、生物医疗等领域发挥了重要作用,基因测序相关的医疗产品逐渐成型并展现出巨大的市场潜力。
然而,随着二代测序数据量爆炸性的增长,传统的基因数据分析工具和分析方法已经无法满足海量生物数据的处理需求,基因数据的处理速度逐渐成为整个基因测序流程中的瓶颈。尽管国内外针对基因数据处理进行了大量的优化工作,例如,通过分布式并行处理基因数据或针对基因数据分析工具的优化加速等,但总体而言,相对于原始测序数据的产生,基因数据分析的计算效率较低。
现有技术中,使用的并行优化方法是基于任务调度和共享存储,通常通过数据划分对各个基因数据步骤进行多机并行,这种方法很难进行进程间通信,在编程模式上受到较大局限,很难对数据依赖情况进行处理。而且数据切分会产生大量中间文件,导致磁盘读写速度慢。另外,由于共享文件系统(例如lustre等)对大量小文件的支持较差,也限制了基因数据分析程序的并行性。
此外,基因数据分析流程会根据具体应用场景的不同进行调整,随着基因测序技术的应用场景不断拓展,基因数据分析流程的开发和调整也较为频繁。目前,大多数基因测序产品的核心手段是通过对基因数据进行比对和清理,检测其中的变异位点并出具相应的检测治疗报告。然而,由于不同数据处理流程涉及的基因样本、算法参数等存在较大的差别,使得针对特定测序流程的优化工作难以移植。
因此,需要对现有技术进行改进,以解决基因数据分析过程效率低、可扩展性差等问题。
技术实现要素:
本发明的目的在于克服上述现有技术的缺陷,提供一种基于spark平台的基因数据分析方法。
根据本发明的第一方面,提供了一种基于spark平台的基因数据分析方法,包括以下步骤:
步骤1:获取基因测序数据;
步骤2:利用spark平台将所获取的基因测序数据生成弹性分布式数据集rdd,其中,所述弹性分布式数据集rdd包括多个部分;
步骤3:对所述弹性分布式数据集rdd的每个部分执行与参考基因的比对,以生成包含比对结果的弹性分布式数据集rdd。
在一个实施例中,在步骤2中,对于双端基因测序数据,执行以下步骤:通过hadoopapi接口分别将两个基因测序数据文件加载并生成两个弹性分布式数据集rdd;将所述两个弹性分布式数据集rdd合并为一个弹性分布式数据集rdd;根据测序序列的名称进行groupby操作并通过map操作生成成对的所述弹性分布式数据集rdd。
在一个实施例中,本发明的基因数据分析方法还包括:
步骤4:对所述包含比对结果的弹性分布式数据集rdd进行数据清理,以获得去冗余的弹性分布式数据集rdd;
步骤5:对所述去冗余的弹性分布式数据集rdd依次执行插入缺失重对齐、碱基质量重校验和变异检测,以确定所述基因测序数据中的变异位点。
在一个实施例中,步骤4包括:
对于所述包含比对结果的弹性分布式数据集rdd,去除标志为未比对上、次要的和增补的测序序列;
以测序序列名称为键将同名的测序序列分为一组,不存在两个相同测序序列名称的测序序列作为一个片段;
生成测序对数据结构并生成用于判断是否冗余的签名,其中,所述签名包括测序序列的重叠群、位置和匹配方向;
根据签名将存在冗余的组和片段分为一组;
从所划分的组中选择质量分数最高的组或片段,通过flatmap操作生成去冗余的弹性分布式数据集rdd。
在一个实施例中,所述执行插入缺失重对齐包括:对于所述去冗余的弹性分布式数据集rdd,在每个部分内遍历堆叠,查找需要重新比对的目标区域;对所述目标区域进行重新比对,以获得插入缺失重对齐的弹性分布式数据集rdd。
在一个实施例中,所述执行碱基质量重校验包括:针对完成插入缺失重对齐的弹性分布式数据集rdd,在每个部分内对一组特征的共现次数和错误率进行统计,将分组数据转化为局部的统计结果;根据多个节点上的统计结果,生成全局的bqsr表;对全局bqsr表进行广播操作,使其在各个任务中都有一份拷贝;用全局bqsr表对插入缺失重对齐的弹性分布式数据集rdd进行重新比对,以获得碱基质量重校验的弹性分布式数据集rdd。
在一个实施例中,所述执行变异检测包括:针对碱基质量重校验的弹性分布式数据集rdd,在每一部分内确定有效区域;对所述有效区域进行pair-hmm求似然处理,得到候选基因型并生成gvcf格式的中间结果;对所述中间结果进行过滤和聚合,以得到整个基因测序数据的变异位点。
在一个实施例中,在步骤1中,所述基因测序从spark平台内存中直接读取或从文件系统里读取。
与现有技术相比,本发明的优点在于:利用spark平台对基因测序数据进行比对、数据清理和变异检测等,能够提高基因数据分析的效率以及分析过程的可扩展性。
附图说明
以下附图仅对本发明作示意性的说明和解释,并不用于限定本发明的范围,其中:
图1示出了用于基因数据分析的spark平台的框架示意图;
图2示出了根据本发明一个实施例的基于spark平台进行基因数据分析的开发框架的示意图;
图3示意了根据本发明一个实施例的针对双端测序数据生成数据集的过程;
图4示出了spark平台上各任务获得静态索引的示意图;
图5示出了根据本发明一个实施例的去除冗余数据的过程示意;
图6示出了gatk相关算法的执行模式示意图;
图7示出了一种变异检测算法的执行过程示意图。
具体实施方式
为了使本发明的目的、技术方案、设计方法及优点更加清楚明了,以下结合附图通过具体实施例对本发明进一步详细说明。应当理解,此处所描述的具体实施例仅用以解释本发明,并不用于限定本发明。
本发明结合二代测序基因数据分析和大数据处理技术,提出了一种用于基因数据分析的spark平台开发框架,能够实现主流的基因数据处理算法,并抽象为api(应用程序编程接口)的形式,以便用户使用该开发框架对基因测序流程进行进一步的开发。
spark是一种可扩展的数据分析平台,其使用弹性分布式数据集rdd,提供了分布式内存并行计算引擎,支持快速的迭代计算。spark平台的计算都是通过操作rdd来进行,对rdd的操作分为转化操作(transformation)和行动操作(action),当执行rdd的转化操作时,实际计算并没有被执行,当执行rdd的行动操作时,才会促使计算任务提交以执行相应的计算操作。例如,转化操作包括map、mappartition、flatmap、join、repartition、filter、union等,行动操作包括collect、count、reduce、groupby等。
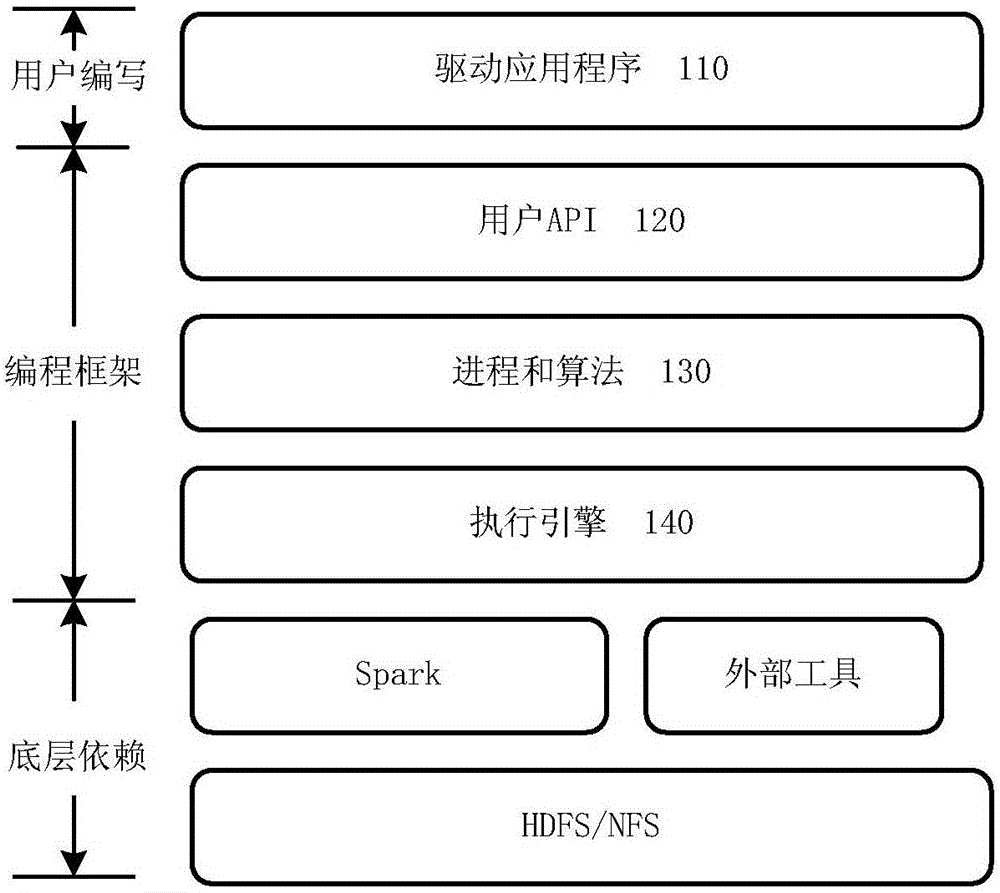
图1示出了根据本发明一个实施例的用于基因数据分析的spark平台开发框架。该开发框架逻辑上可分为三层,最上层是由用户编写的驱动应用程序模块110,中间层的编程框架包括用户api模块120、进程和算法模块130、执行引擎模块140,最下层是编程框架所依赖的spark环境、外部工具和诸如hdfs(hadoop分布式文件系统)和nfs(网络文件系统)的文件系统等。
驱动应用程序模块110由用户编写,包含对基因数据分析特定流程的描述,例如,用户需要运行的基因分析算法以及这些算法执行的顺序。这些特定流程例如包括基因数据比对、去冗余、变异检测等。驱动应用程序模块110可以在rdd上执行转化和行动两种类型的操作,行动操作会在数据集上执行一个计算,并向驱动应用程序模块110返回一个值,而转化操作是从现有的数据集中创建一个新的数据集。
在该编程框架中,基于用户api模块120可以编写驱动应用程序;执行引擎140对驱动应用程序中定义的流程进行解析,分析各步骤之间的数据依赖和运行特点,给出执行顺序,并可采取优化执行的措施。执行引擎140根据执行顺序逐个提交spark操作,具体的作业(job)和阶段(stage)划分等工作由spark自身完成。中间的编程框架会为上层的驱动应用程序提供相应的数据结构和一些相关算法、函数。
底层依赖是指框架运行对环境的依赖,包括spark环境和一些外部的工具,存储方面支持hdfs和nfs(如lustre等)文件系统。这一层根据硬件环境进行资源分配,执行上层程序。
上述三层结构紧密联系,下层为上层提供服务,上层对分析过程进行抽象,协同完成基因分析过程
根据本发明图1所示的开发框架能够实现测序序列与参考基因的比对、基因数据清理和变异检测等算法,此外,本发明的开发框架具备良好的性能、扩展性和容错能力。
图2示出了根据本发明一个实施例的基于spark平台的基因数据分析方法的流程图。简言之,在该实施例中,基因数据分析过程包括:基因测序数据与参考基因进行比对,以生成包含比对结果的数据;从包含比对结果的数据中清除掉冗余数据;对经过冗余清理的数据进行插入缺失重对齐;碱基质量重校验;执行变异检测以识别变异位点。具体包括以下步骤:
步骤s210,将基因测序数据和参考基因进行比对。
具体地,此步骤包括:获取基因测序数据;将基因测序数据生成弹性分布式数据集rdd;对rdd的每个部分通过jni接口执行与参考基因的比对;生成包含比对结果的rdd。
基因测序数据可通过常用的测序技术获得,例如,现有的高通量测序平台产生的基因测序文件类型包括fasta、fastq、gef、bed等,这些文件是存储了生物序列以及相应质量评价的通用格式。本文将以fastq为例阐述本发明的基因数据分析方法。
在利用spark平台进行基因数据分析时,基因测序数据可从内存里直接读取或从文件系统里读取,文件系统包括hdfs、本地文件系统等多种类型。
对fastq文件进行数据划分生成rdd的过程包括:对fastq数据按照记录划分为多个部分(partition),每个partition作为一个区块(chunk),在chunk内通过jni(javanativeinterface)执行基因比对算法(例如,bwamem算法)。
在一个实施例中,对于单端测序数据,通过hadoopapi接口,将fastq文件格式化地读取成fastq记录的rdd,并自动在多个节点上划分为多个partition。
在另一个实施例中,对于双端测序数据,需要读取两个fastq文件,将得到的记录以成对的形式放在一个partition中,具体地,参见图3所示,双端测序数据生成包含比对结果的rdd的过程包括:
步骤11:通过hadoopapi接口分别将两个fastq文件加载生成两个fastq记录的rdd;
步骤12:通过join操作将两个fastq记录的rdd合并成一个fastq记录的rdd;
步骤13:以fastq描述行中的测序序列名称(readname)为键进行groupby操作并通过map操作生成成对的fastq记录;
步骤14:使用mappartition操作,对partition内成对的fastq记录执行基因数据比对算法,例如,bwamem算法;
步骤15:使用flatmap操作得到包含比对结果信息的rdd。
生成的比对结果信息可保存为sam/bam文件格式,比对结果信息包括每条测序序列(read)的起始位置、结束位置、正负链的标志位、对比结果等,例如,比对结果是简要比对信息表达式,使用数字加字母表示,例如,3s6m1p1i4m表示前三个碱基被剪切去除,然后6个碱基比对上了,然后打开了一个缺口,有一个碱基插入,最后是4个碱基比对上了。
需要说明的是,在执行基因数据比对算法bwamem时,需要构建参考序列索引(index),所述参考序列索引用于查找特定的测序序列片段在参考基因组中所有出现的位置,参考序列索引可采用现有的多种方法构建,例如hash等算法。为了降低构建参考序列索的耗时和避免在每个partition调用jni时重复构建索引,在一个实施例中,将参考基因索引设定为进程内的静态变量,这是由于索引是一种只读的资源,多个线程共享不会产生竞争条件。
具体地,图4示出了各任务获得参考索引的模式示意图,如图所示,对于设置为静态变量的参考索引,executor(其是运行在工作节点上的一个进程,负责执行任务)执行任务的过程中,任务3的fastqpartition3和任务4的fastqpartition4可同时获得参考索引实例,而无需等待解锁,相对于对类(class)、方法(method)和静态变量等调用过程中,需要等待解锁才能加载索引的情况,将参考索引设置为静态变量无需每次执行任务时都重复构建索引,此外,各任务也可同时读取参考索引实例,也无需等待另一进程的解锁。
通过上述过程,可生成包含比对结果的rdd,以供后续的基因分析过程使用。
步骤s220,去除冗余数据。
此步骤的目的在于清理掉由于测序原因产生的冗余数据,以避免对后续的分析产生影响,可采用markduplicate算法去除重复数据。
在spark上实现markduplicate算法时,通过使用多个spark原语来保证结果与原有markduplicate算法保持一致。图5示出在spark平台上去冗余的过程,具体包括:
步骤21:在sam记录为元素的rdd上,首先,根据测序数据中的标志过滤掉带有unmapped(即未比对上)、secondary(次要的)和supplementary(增补的)标志的测序序列。
例如,过滤后的数据为p1/1、p1/2、p2/1、f1、f2和p2/2、p3/1、p3/2、f3、p4/1、p4/2。
步骤22:以测序序列名称(readname)为键使用groupby操作,将同名的read分在一组。如果有两个同名的read被分在一组,则构成一个双端分组(pairends),如果没有,则作为一个fragment(片段)。
例如,图5中构成pairends的分组包括p1/1和p1/2、p2/1和p2/2、p3/1和p3/2、p4/1和p4/2,而fragment包括f1、f2、f3。
步骤23:通过map操作,生成测序序列对信息结构(readpairinfo),并生成冗余判定的签名(图5中未示出)。
具体地,readpairinfo中保存了read的引用、质量分数、mapping位置等信息,签名内容包括两条read的contig(堆叠),即基于reads之间的overlap区,拼接获得的序列)、position和匹配方向。
对于一个fragment,那么签名只包括一条read的信息。
步骤24:以签名为键进行groupby操作,使得冗余的pairends和冗余的fragment被分到一个组中。
例如,冗余的pairends有(p1/1、p1/2)和(p2/1、p2/2),以及(p3/1、p3/2)和(p4/1,p4/2),而冗余的fragment是f1和f2,f3没有冗余。
步骤25:在存在冗余的组中选择质量分数最高的pairends和fragment,而其余作为冗余去除,然后通过flatmap操作将rdd重新转化为以sam记录为元素的rdd。
步骤26:由于上述的pairends和fragment的签名一定不相等,可能导致某个pairends和某个fragment存在冗余的情况。为此,再将所有的read单独构建readpairinfo,以签名为键进行groupby操作,使得fragment和pairends中与之重合的一条或多条被分在一个组中。
例如,此时,冗余的分组是p1/2和f1。
步骤27:对于每个分组,通过查看read的标志确定其是否属于一个pairends中的一条。如果一个分组中包含这种read,则去除所有的fragment,例如,假设确定p1/2属于pairends中的一条,则将f1去除。
通过上述过程,生成了一个合并的并去除重复片段的比对数据,以供后续分析使用。
步骤s230,插入缺失重对齐。
此步骤的目的在于,将比对到indel附近的测序序列进行局部重新比对,以将比对的错误率降到最低。通常,绝大部分需要进行重新比对的基因组区域,都是因为插入/缺失的存在,因为在indel附近的比对会出现大量的碱基错配,这些碱基的错配很容易被误认为snp(单核苷酸多态性)。
在一个实施例中,可采用indelrealigner算法来实现插入缺失重对齐。输出的是插入缺失重对齐之后的sam/bam文件。
具体地,对于插入缺失重对齐算法,实际处理的过程可抽象为一个flatmap操作。在flatmap操作中进行如下操作:
步骤31:在partition内部遍历pileup(堆叠),找到需要重新比对的目标区域(targetregion)。
步骤32:根据找到的目标区域进行read重新比对,以获得重新比对的sam记录。
步骤s240,碱基质量重校验。
为了消除测序仪器的误差等原因导致的碱基质量过高或过低,需要对碱基质量值进行重新校正,以提高基因检测的准确性。例如,可采用basequalityscorerecalibratio(bqsr)算法来实现碱基质量重校验。
具体地,对测序序列的碱基质量值进行重新校正,使测序序列中碱基的质量值更加接近真实的与参考基因组之间错配的概率。碱基质量重校验的过程包括:根据一些knownsites(已知的可靠的位点),生成一个校正质量值所需要的数据文件;生成校正后的数据文件;将经过质量值校正的数据输出到新的sam文件中,用于后续的变异检测。
输出的是碱基质量重校验后的sam/bam文件。
对于碱基质量重校验算法,要求收集整个数据集上的特征共现信息,因此,需要分成两个阶段进行,在中间加入一个collect操作收集各个分区的数据。具体执行方法如下:
步骤41:在每个partition内对一组特征的共现次数和错误率进行统计,使用map操作将分组数据转化为局部的统计结果。
步骤42:通过collect操作将多个节点上的统计结果收集到驱动器节点上,生成全局bqsr表。
步骤43:对全局bqsr表进行广播(broadcast)操作,使其在各个任务中都有一份拷贝。
步骤44:使用flatmap操作,用全局bqsr表对sam记录进行重比对。
步骤s250,变异检测
变异检测算法输出的结果是vcf记录(variantcallformat),是用于存储基因序列突变信息的文本格式。vcf文件中包含所有样本的变异位点和基因型信息,例如,可用于描述snp(单个碱基上的变异),indel(插入缺失标记)和sv(结构变异位点)。变异检测算法包括haplotypecaller和mutect等。
变异检测和上述的插入缺失重对齐算法、碱基质量重校验等均为gatk相关算法,这些算法适合于使用粗粒度数据划分的方式并行,其中,有些算法与临近的数据有依赖,可以通过加入一定长度overlap的方式保证正确性。
gatk算法的输入都是reference、sam/bam和已知位点(rod),参见图6所示,gatk相关算法的执行模式包括:
步骤51:读取reference的序列名称、序列长度等信息,生成分区信息。每个分区的两端留出一定长度的overlap。
步骤52:分别将reference(参考基因组)、rod和sam数据按照分区信息得到所属的partitionid,其中,位于overlap中的数据需要被复制并赋予不同的key,然后使用groupby操作划分到多个partition当中。
步骤53:使用join操作将分区内的reference、rod和sam数据组合形成partitionbundle的rdd。
步骤54:对partitionbundle的rdd运行实际算法,合并生成算法结果。
对于变异检测算法,可以通过一个flatmap操作在每个partition内,使用串行的activeregion方法计算activeregion(有效区域)。然而在实际应用中,不同partition中间产生的有效区域个数差别非常大,并且存在个别任务中负载非常重的情况,如果继续在每个partition内对有效区域进行变异检测,会因为任务间负载不均导致严重的长尾效应。这种长尾效应在并行度提高的时候会越发凸显,严重拖慢系统整体性能。为了平衡任务间的负载,图7以两个partition为例,示意了根据本发明一个实施例的负载均衡的方法,包括以下步骤:
步骤61:在各个partition内找到有效区域;
步骤62:通过flatmap操作对有效区域以及其包含的数据进行序列化;
步骤63:通过repartition操作重新划分负载;
步骤64:对有效区域进行拼接、pair-hmm求似然等处理,得到候选基因型,生成gvcf格式的中间结果;
步骤65:使用genotypegvcf算法对中间结果进行进一步过滤和聚合,得到最终整个样本的变异位点。
得到的结果是vcf格式的rdd。
通过变异检测,可以获得检测单核苷酸变异(snp)、基因组在每个位置上发生的小片段序列的插入/缺失(简称indel)或大的结构性变异等。
需要说明的是,虽然上文按照特定顺序描述了各个步骤,但是并不意味着必须按照上述特定顺序来执行各个步骤,实际上,这些步骤中的一些可以并发执行,甚至改变顺序,只要能够实现所需要的功能即可。
本发明可以是系统、方法和/或计算机程序产品。计算机程序产品可以包括计算机可读存储介质,其上载有用于使处理器实现本发明的各个方面的计算机可读程序指令。
计算机可读存储介质可以是保持和存储由指令执行设备使用的指令的有形设备。计算机可读存储介质例如可以包括但不限于电存储设备、磁存储设备、光存储设备、电磁存储设备、半导体存储设备或者上述的任意合适的组合。计算机可读存储介质的更具体的例子(非穷举的列表)包括:便携式计算机盘、硬盘、随机存取存储器(ram)、只读存储器(rom)、可擦式可编程只读存储器(eprom或闪存)、静态随机存取存储器(sram)、便携式压缩盘只读存储器(cd-rom)、数字多功能盘(dvd)、记忆棒、软盘、机械编码设备、例如其上存储有指令的打孔卡或凹槽内凸起结构、以及上述的任意合适的组合。
以上已经描述了本发明的各实施例,上述说明是示例性的,并非穷尽性的,并且也不限于所披露的各实施例。在不偏离所说明的各实施例的范围和精神的情况下,对于本技术领域的普通技术人员来说许多修改和变更都是显而易见的。本文中所用术语的选择,旨在最好地解释各实施例的原理、实际应用或对市场中的技术改进,或者使本技术领域的其它普通技术人员能理解本文披露的各实施例。
- 还没有人留言评论。精彩留言会获得点赞!