一种人体微生物菌群生态网络结构和功能变异的识别方法与流程
本发明属于生物医学信息分析领域,尤其涉及一种人体微生物菌群生态网络结构和功能变异的识别方法。
背景技术:
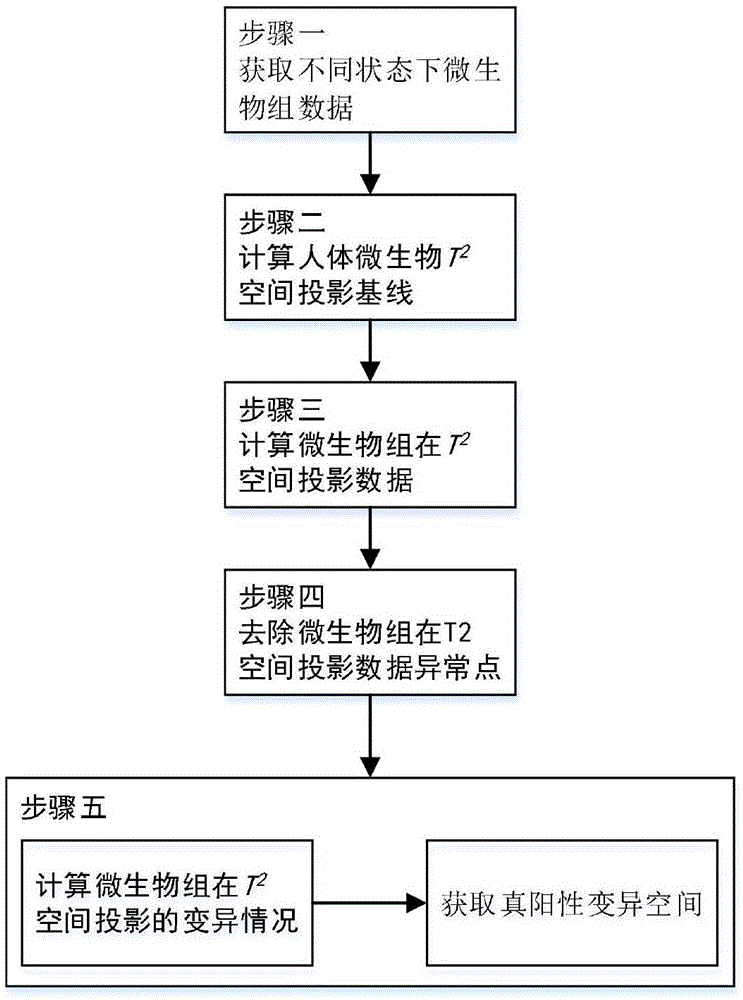
:目前,业内常用的现有技术是这样的:微生物群落是指在一定区域里,或一定生境里,各种微生物种群相互松散结合,或有组织紧凑结合的一种结构单位。微生物以群落的形式存在人体的各个器官中,微生物群落的生态特征可分为结构特征和功能特征,其中结构特征描述微生物群落成员的种类、丰度及其在不同环境条件下的更替。对微生物群落结构特征可以从微生物的各个分类单位如门纲科目属种甚至菌株水平数目来分析,人体不同部位具有不同的生存条件,包括温度、湿度、营养源、酸碱度、富氧程度、生长的基质等,因此,不同身体部位的微生物种类也不相同。与此同时,每个人所携带的微生物种类及其相对数量也是具有个体差异的,“微生物谱”受性别、饮食、气候、年龄、职业、卫生习惯差异的影响。而功能特征则描述群落的行为:底物代谢过程;与宿主或环境以及群落内其他成员相互拮抗和共生的关系。微生物群落的种群结构及种间相互作用是影响其生态功能的决定因素。研究表明,微生物群落之间的相互关系与人类大部分疾病的发生发展有密切的关系。由于传统的微生物培养方法只能分离约1%环境中的微生物,,大量未培养的微生物如同地球上的"暗物质",在相应生境中存在种类到底有多少,如何发挥功能,传统微生物学方法已经不能全面解答。基于高通量测序技术的16srrna测序分析技术和宏基因组测序技术能分别对特定环境中全部微生物的16srrna基因或总dna(也称宏基因组,metagenomic)进行克隆,并通过构建宏基因组文库,测序和生物生息学分析等手段获得各个分类阶层的群落特征,物种功能,属和种间的相互关系。由于不同的人体状态(包括性别、饮食、气候、年龄、职业、卫生习惯、健康状态等)下,例如健康与疾病,偏瘦与肥胖,摄入食物偏碳水化合物与偏肉类蛋白质,男性与女性等,人体所携带的微生物种类及其相对数量也是具有个体差异的,有效且量化的识别并表达该差异,可以为研究微生群落的变异与人类疾病发生发展的关系,奠定基础。综上所述,现有技术存在的问题是:现有技术,例如均值假设检验等典型变异微生物识别方法存在的无法识别离散程度变异情况的缺陷;现有技术仅对单一微生物菌群变异情况识别,无法对微生物菌群之间存在的复杂关系的变化进行描述;对变异微生物的识别停留在定性或半定量阶段,没有提供微生物变异情况的量化的评价方法。解决上述技术问题的难度和意义:微生物变异量化+多维识别的生物学意义,当前微生物生态的网络与疾病的发生发展密切关联,然而常规的差异物种的鉴定方法常常漏掉一些丰度上差异不显著,但在健康状态和疾病状态上功能差异非常明显的物种,该方法在于协助鉴别在疾病发生发展的进程中起关键作用的重要微生物,为下一步通过实验探寻重要的标记物缩小了范围,并奠定了理论基础。在对微生物差异识别时,单一微生物下(一维情况),如何综合考虑微生物丰度以及丰度离散程度,将两者同时纳入到变异评价方法中,是本发明的一个技术难点。在对微生物差异识别时,多种微生物(多维情况),如何综合考虑微生物丰度信息、丰度离散程度信息,以及微生物之间的共生、拮抗关系信息,将三者同时纳入到变异评价方法中,是本发明的一个技术难点。在进行微生物(菌群)变异程度评价时,如何建立起量化的度量方法,使得评价结果横向上(不同的微生物菌群间)、纵向上(不同的样本间)可比较,是本发明的一个技术难点。技术实现要素:针对现有技术存在的问题,本发明提供了一种人体微生物菌群生态网络结构和功能变异的识别方法。具体是通过分析在不同的人体状态下,微生物群落在t2空间投影的差异情况,识别微生物群落的变异,为研究微生菌群的变异与人类疾病发生发展的关系,以及疾病防治和诊断提供支撑。本发明是这样实现的,一种人体微生物菌群生态网络结构和功能变异的识别方法通过计算微生物群落在t2空间投影的差异情况,得到微生物组在不同人体状态下的变异大小,为分析微生群落的变异与人类疾病发生发展的关提供支持。具体包括:步骤一:获取用于开展微生物群落结构和功能变异量化评价与的人体微生物组数据。步骤二:计算人体微生物组t2空间投影基线。步骤三:计算微生物组在t2空间投影数据。步骤四:去除微生物组在t2空间投影数据异常点。步骤五:计算人体微生物组在t2空间投影的变异情况。进一步,步骤一所述获取人体微生物组丰度或相对丰度数据,其具体实现过程如下:(1)获取人体状态数据;(2)建立微生物种群子空间;(3)获取人体微生物组丰度或相对丰度数据。进一步,步骤二所述计算人体微生物组t2空间投影基线,其具体实现过程如下:(1)计算各微生物丰度或相对丰度均值,以及微生物间协方差;(2)微生物组t2空间投影基线。进一步,步骤三所述计算计算微生物组在t2空间投影数据,其具体实现过程如下:(1)计算某状态下(记为sa状态)微生物组数据在其他状态下(记为sb状态)基线下投影数据;(2)计算sb状态微生物组数据在sa状态基线下投影数据;(3)计算sa状态微生物组数据在sa状态基线下投影数据;(4)计算sb状态微生物组数据在sb状态基线下投影数据。进一步,步骤四所述去除微生物组在t2空间投影数据异常点,其具体实现过程如下:(1)去除sa状态下微生物组数据在sb状态基线下投影数据的异常点;(2)去除sb状态微生物组数据在sa状态基线下投影数据异常点;(3)去除sa状态微生物组数据在sa状态基线下投影数据异常点;(4)去除sb状态微生物组数据在sb状态基线下投影数据异常点。进一步,步骤五所述计算人体微生物组在t2空间投影的变异情况,其具体实现过程如下:(1)开展投影数据服从分布的核函数估计;(2)计算子空间内人体微生物组变异程度,获取其量化评价值;(3)对有效子空间进行遍历,获得各个子空间变异程度,识别为变异空间。本发明的另一目的在于提供一种实现所述人体微生物菌群生态网络结构和功能变异的识别方法的计算机程序。本发明的另一目的在于提供一种实现所述人体微生物菌群生态网络结构和功能变异的识别方法的信息数据处理终端。本发明的另一目的在于提供一种计算机可读存储介质,包括指令,当其在计算机上运行时,使得计算机执行所述的人体微生物菌群生态网络结构和功能变异的识别方法。本发明的另一目的在于提供一种实现所述人体微生物菌群生态网络结构和功能变异的识别方法的人体微生物菌群生态网络结构和功能变异的识别设备。综上所述,本发明的优点及积极效果为:本发明利用人体不同状态的各个分类阶层的丰度值,量化各微生物物种间的相互关系,通过分析在不同的人体状态下,微生物群落在t2空间投影的差异情况,识别微生物菌群的变异,为研究微生群落的变异与人类疾病发生发展的关系,提供支持。如图6仿真数据所示,四个个数据集的平均和标准划分是无法区分的,尽管它们明显不同。然而,当比较x轴或y轴pm评分和x-y关系的pm评分时,在大多数成对比较中,可以观察到显著差异。本发明的优点还有:对均值变异与协方差变异共同识别,克服了均值假设检验等典型变异微生物识别方法存在的无法识别离散程度变异情况的缺陷;提出了微生物菌群子空间的概念,实现了对微生物菌群复杂关系的识别,克服了仅对单一微生物菌群变异情况识别的传统方法的缺陷;提供了微生物变异情况的量化的评价方法,量化指标具备对称性。提供了微生物变异情况的量化的评价方法,量化指标线性可比,值域[0,1]之间,值越大,变异情况越大。提供了微生物变异情况的量化的评价方法,该方法适用于单一微生物差异的比较及高维微生物菌落差异的比较,数学形式统一。基于微生物变异情况的量化的评价方法,提出了变异空间识别方法,为研究微生群落的变异与人类疾病发生发展的关提供了量化支持。附图说明图1是本发明实施例提供的人体微生物菌群生态网络结构和功能变异的识别方法流程图。图2是本发明实施例提供的为函数图像。图3是本发明实施例提供的函数图像。图4是本发明实施例提供的函数图像。图5是本发明实施例提供的函数图像。图6是本发明实施例提供的仿真实验中基于均值的统计量与pm的比较图。具体实施方式为了使本发明的目的、技术方案及优点更加清楚明白,以下结合实施例,对本发明进行进一步详细说明。应当理解,此处所描述的具体实施例仅仅用以解释本发明,并不用于限定本发明。下面结合具体分析对本发明的应用作进一步描述。图1本发明实施例提供的人体微生物菌群生态网络结构和功能变异的识别方法,包括:步骤一:获取用于开展微生物群落结构和功能变异量化评价与的人体微生物组数据。1)记人体状态s={s1,s2,…sn};sj下人体微生物样本量为nj;微生物组中包含的微生物种类为otu={otu1,otu2,…otup};2)建立otu的子空间为则otu有效子空间l的数量为个;3)获取人体微生物组丰度或相对丰度数据。处于某状态sj下人体微生物第i个样本丰度或相对丰度数据记为其中i={n1,n2,…nn},有某状态sj下空间内人体微生物第i个样本丰度或相对丰度数据记为不引起歧义的情况下,简记为例:本实施例以zeller,g.etal.potentialoffecalmicrobiotaforearly-stagedetectionofcolorectalcancer.mol.syst.biol.10,766–766(2014)提供的数据展开,部分数据见附录1.该数据集人体状态分别为crc(结肠癌)、h(健康),记为s={s1,s2},s1为crc组,s2为h组。s1样本量为n1=41,s2样本量为n2=74。微生物组中包含的微生物种类为acetanaerobacterium、acidaminococcus、akkermansia、alistipes、anaerosporobacter、ruminococcus等。本实施例数据集中微生物种类有6种,微生物的子空间共有个,本实施例以为例展开,其他子空间的遍历类似。步骤二:计算人体微生物组t2空间投影基线。2.1对sj下内微生物组丰度数据或相对丰度数据计算各微生物均值:计算各微生物间协方差:有2.2建立sj下内微生物组t2空间投影基线为其向量形式记为例:针对计算s1即crc投影基线:有:计算s2即h组投影基线:有:步骤三:计算微生物组在t2空间投影数据。3.1计算内微生物组在状态sa下的丰度数据或相对丰度数据在sb的t2空间投影基线上的投影数据其中:3.2计算内微生物组在状态sb下的丰度数据或相对丰度数据在sa的t2空间投影基线上的投影数据其中:3.3计算内微生物组在状态sa下的丰度数据或相对丰度数据在其t2空间投影基线上的投影数据其中:3.4计算内微生物组在状态sb下的丰度数据或相对丰度数据在其t2空间投影基线上的投影数据其中:例:计算得到微生物组在t2空间投影数据为:见下表:见下表:i1234567投影1.3554542.0808860.7026250.8208380.8631810.5090240.772626i891011121314投影0.2155430.6099960.45480.8813533.33706910.592030.824896i15161718192021投影1.0244711.3318991.601420.3184391.0872140.3593341.195799i22232425262728投影1.1525330.6631641.17026624.076750.4604048.39338129.96924i29303132333435投影66.606231.27648946.475761.6004592.4506861.09735832.26747i36373839404142投影0.5814751.5966186.011975.1475013.1149740.9904451.640455i43444546474849投影0.6907712.7773662.0846789.9676671.0074353.3034883.578533i50515253545556投影0.4350740.817480.7439571.3955730.6760760.2892420.50381i57585960616263投影6.2067370.50104511.338990.5612318.56465210.918621.570757i64656667686970投影0.1458969.9765547.1782270.6021730.417881.011362.004774i71727374---投影0.8751880.3409720.4970940.831668---见下表:i1234567投影0.4015722.8121790.4298541.173770.2283571.0800024.822124i891011121314投影1.9869880.46454316.691880.0310640.9902061.0098160.682768i15161718192021投影0.4867711.0892241.2708241.1307660.3991790.3887380.832443i22232425262728投影0.459372.1230720.5822071.3021590.0645940.0220371.483427i29303132333435投影1.9554940.3755861.6028630.3200970.3043132.9505980.445354i363738394041-投影11.978711.58465810.818351.407410.8188830.997749-见下表:i1234567投影0.9175160.2103430.5513961.3013520.6414990.0791540.542459i891011121314投影0.0943490.5296020.0494590.6018650.5183262.3024840.051108i15161718192021投影0.7778710.8812141.0074920.122510.7495030.2274430.800243i22232425262728投影0.7773810.4566760.0952456.1498270.4688321.8749110.2865i29303132333435投影58.618630.85878512.456821.0069960.2445520.752148.301048i36373839404142投影0.4066291.0050141.2888560.8114210.4521990.7696180.111157i43444546474849投影0.5922190.3733272.9632692.0169681.078410.4779250.571012i50515253545556投影0.1030980.5598490.6023640.9139310.04370.1664450.27173i57585960616263投影1.1557470.2043022.4179620.3543241.7505812.2885360.994435i64656667686970投影0.2371241.9872091.341580.472390.1968060.0662510.155419i71727374---投影0.6257480.3425670.5007140.023636---步骤四:去除微生物组在t2空间投影数据异常点。4.1设定异常点筛选阈值σ,σ>0。4.2计算的1/4,1/2,3/4分位点,分别记为及去除大于的数据,剩余的数据记为4.3计算的1/4,1/2,3/4分位点,分别记为及去除大于的数据,剩余的数据记为4.4计算的1/4,1/2,3/4分位点,分别记为及去除大于的数据,剩余的数据记为4.5计算的1/4,1/2,3/4分位点,分别记为及去除大于的数据,剩余的数据记为例:设定σ=1.5。计算得到的1/4,1/2,3/4分位点,分别为及计算去除异常点后,得到见下表。i1234567投影0.15270.37150.54190.13700.82190.05050.8013i891011121314投影0.31450.31210.29841.22580.11660.53800.2158i15161718192021投影0.98540.84190.10750.34330.12220.56990.2509i22232425262728投影0.00010.34120.20930.96190.20431.00820.1397i2930313233--投影0.50600.45730.99881.31631.3003--计算得到的1/4,1/2,3/4分位点,分别为及计算:去除异常点后,得到见下表。计算得到的1/4,1/2,3/4分位点,分别为及计算:去除异常点后,得到见下表。i1234567投影0.4015720.4298541.173770.2283571.0800021.9869880.464543i891011121314投影0.0310640.9902061.0098160.6827680.4867711.0892241.270824i15161718192021投影1.1307660.3991790.3887380.8324430.45942.12310.5822i22232425262728投影1.30220.06460.02201.4834271.9554940.3755861.602863i29303132333435投影0.3200970.3043130.4453541.5846581.407410.8188830.997749计算得到的1/4,1/2,3/4分位点,分别为及计算:去除异常点后,得到见下表。步骤五:计算人体微生物组在t2空间投影的变异情况。5.1按照scott,d.w.(1992)multivariatedensityestimation.theory,practiceandvisualization.newyork:wiley及sheather,s.j.andjonesm.c.(1991)areliabledata-basedbandwidthselectionmethodforkerneldensityestimation.j.roy.statist.soc.b,683–690提供的方法,分别计算服从分布的核函数估计,分别记估计得出的概率密度分布函数分别为5.2分别计算的最大值、最小值,记:5.3计算:其中5.4计算:其中5.5计算内人体微生物组状态sa与状态sb下变异情况值域为[0,1],值越大,表明状态sa与状态sb下,内人体微生物组变异程度越高。5.6对2p-1个有效子空间进行遍历,获得各个子空间变异程度设定阈值ω,ω>0,识别为变异空间。例:对服从分布进行核函数估计,记估计得出的概率密度分布函数为图像见附图2.对服从分布进行核函数估计,记估计得出的概率密度分布函数为图像见附图3.对服从分布进行核函数估计,记估计得出的概率密度分布函数为图像见附图4.对服从分布进行核函数估计,记估计得出的概率密度分布函数为图像见附图5.计算:有于是于是:有于是于是计算内人体微生物组状态s1(结肠癌crc)与状态sb(健康h)下变异情况即两者变异值为0.1831525.计算有效子空间变异情况如下表。设定ω=0.6,即可以得到变异空间,如下表。附录1:crc_zeller数据集本发明实施例提供一种人体微生物菌群生态网络结构和功能变异的识别设备。下面结合仿真实验对本发明的应用作进一步描述。如图6仿真数据所示,四个个数据集的平均和标准划分是无法区分的,尽管它们明显不同。然而,当比较x轴或y轴pm评分和x-y关系的pm评分时,在大多数成对比较中,可以观察到显著差异。在上述实施例中,可以全部或部分地通过软件、硬件、固件或者其任意组合来实现。当使用全部或部分地以计算机程序产品的形式实现,所述计算机程序产品包括一个或多个计算机指令。在计算机上加载或执行所述计算机程序指令时,全部或部分地产生按照本发明实施例所述的流程或功能。所述计算机可以是通用计算机、专用计算机、计算机网络、或者其他可编程装置。所述计算机指令可以存储在计算机可读存储介质中,或者从一个计算机可读存储介质向另一个计算机可读存储介质传输,例如,所述计算机指令可以从一个网站站点、计算机、服务器或数据中心通过有线(例如同轴电缆、光纤、数字用户线(dsl)或无线(例如红外、无线、微波等)方式向另一个网站站点、计算机、服务器或数据中心进行传输)。所述计算机可读取存储介质可以是计算机能够存取的任何可用介质或者是包含一个或多个可用介质集成的服务器、数据中心等数据存储设备。所述可用介质可以是磁性介质,(例如,软盘、硬盘、磁带)、光介质(例如,dvd)、或者半导体介质(例如固态硬盘solidstatedisk(ssd))等。以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内所作的任何修改、等同替换和改进等,均应包含在本发明的保护范围之内。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1