一种基于es倒排索引的化学分子式检索方法与流程
本发明属于信息检索领域,尤其涉及一种基于es倒排索引的化学分子式检索方法。
背景技术:
化学结构式(分子式)检索主要用在化学品的搜索中,对于拥有海量化学品的企业和研究机构,快速准确地检索到分子式对于生产和研究具有重要的价值。
现有的化学分子式检索方法没有基于大数据平台,没有利用es数据库的倒排索引,化学分子式检索方法通常通过计算分子式的哈希表示来加速检索过程,虽然进行了加速,但这一过程仍然需要与数据库中全部的分子式进行一一比较来实现检索,加速效果有限。
技术实现要素:
本发明在现有的分子式检索方法基础上,结合es(elasticsearch)的倒排索引技术,提出了一种在大数据平台下的检索方法,进一步加快了检索速度。
本发明的目的是通过以下技术方案来实现的:一种基于es倒排索引的化学分子式检索方法,包括以下步骤:
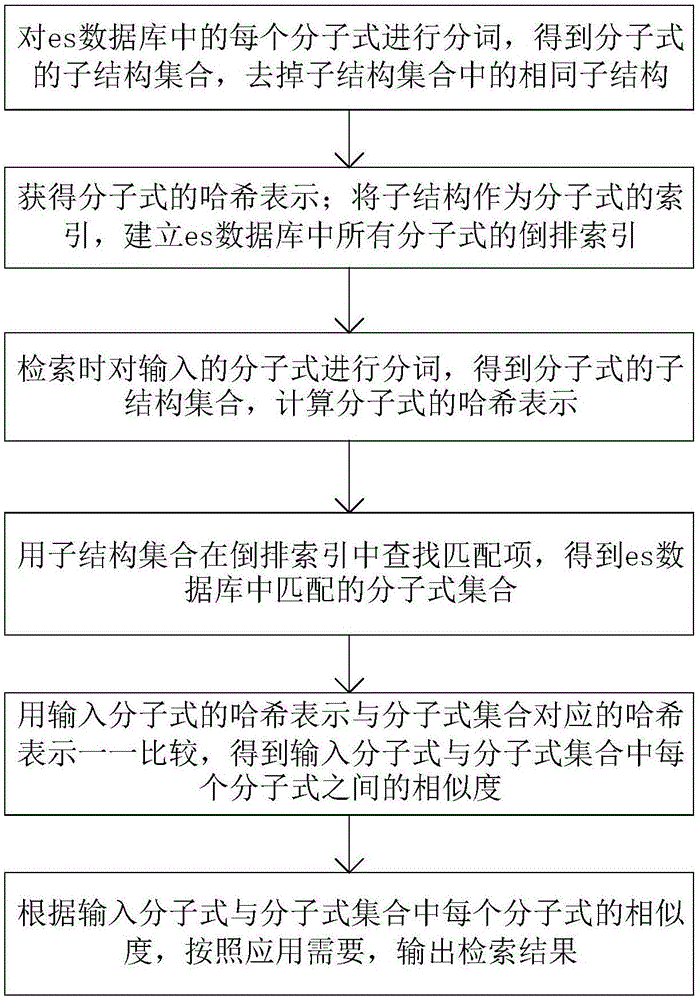
步骤1:对es数据库中的分子式建立倒排索引和哈希表示,具体为:
1.1对es数据库中的每个分子式进行分词,得到分子式的子结构集合,子结构集合包含2个以上用化学键相连的原子,或单个的除常用原子以外的原子;
1.2去掉子结构集合中的相同子结构;
1.3把分子式得到的一系列子结构哈希映射到一个由32位整数组成的数组上,得到分子式的哈希表示;分子式得到的一系列子结构作为分子式的索引,建立es数据库中所有分子式的倒排索引。
步骤2:检索过程,具体为:
2.1检索时对输入的分子式进行分词,得到分子式的子结构集合,计算分子式的哈希表示;
2.2用子结构集合在倒排索引中查找匹配项,即查找包含子结构集合中任一子结构的分子式,得到es数据库中匹配的分子式集合t;
2.3用输入分子式的哈希表示,与得到的分子式集合t对应的哈希表示一一比较,得到输入分子式与分子式集合t中每个分子式之间的相似度;
2.4根据输入分子式与分子式集合t中每个分子式的相似度,按照应用需要,输出t中相似度大于阈值的分子式,或按照相似度从大到小排列,输出前n个最相似的分子式。
进一步地,所述子结构的形成具体为:子结构是通过遍历分子式的原子树形成的,即依次从每一个原子a开始向前、向后遍历,将遍历到的全部原子、化学键和原子a一起形成子结构;遍历停止时即形成一个子结构,子结构记录了形成过程中遍历到的原子、原子之间的化学键、以及多个原子是否形成了环状结构。
进一步地,所述步骤1中,遍历停止条件有两个:①当遍历到的下一个原子与当前的遍历过的几个原子形成环状时,子结构形成过程即停止,得到一个子结构;②当遍历的原子数量达到7个或已经顺序遍历完当前的分子式时即停止,得到一个子结构。
进一步地,所述步骤1中,所述相同子结构有两种情况:某子结构的逆序排列是其相同子结构,环状子结构中任意原子作为起始点,均为相同子结构。
进一步地,所述步骤1中,数组长度设置为32或32的整数倍。
进一步地,所述步骤2中,两个分子式的相似度的计算方式为:每个分子式的哈希表示均看作一个二进制的大整数,具体为:将数组中的每个32位整数依次转化为二进制后,拼接为一个二进制的大整数;将两个分子式对应的两个大整数做按位(bit)与操作得到一个相同长度的二进制大整数a,计算a中1的个数an;做按位或操作得到一个相同长度的二进制大整数b,计算b中1的个数bn;an/bn即得到两个分子式的相似度。
本发明的有益效果是:本发明结合大数据平台对检索过程进行重新设计,首先通过对es数据库中的分子式进行分词得到的子结构建立倒排索引,并对es数据库中的分子式计算哈希表示;检索时对分子式进行分词得到子结构,并计算哈希表示,利用倒排索引得到子结构匹配的分子式集合,再与得到的分子式集合的哈希表示一一比较,减少了比较的范围,加快了分子式的检索。
附图说明
图1为本发明基于es倒排索引的化学分子式检索方法的流程图;
图2为分子式”ccc(=o)cl”的图形。
具体实施方式
下面结合附图和具体实施例对本发明作进一步详细说明。
如图1所示,本发明提供的一种基于es倒排索引的化学分子式检索方法,包括以下步骤:
第一步:对es数据库中的分子式建立倒排索引和哈希表示:
对es数据库中的每个分子式进行分词,得到分子式的子结构集合,子结构集合包含2个以上(7个以下)用化学键相连的原子,或单个的除常用原子以外的原子,所述常用原子包括c、n、o、h等,也可以人为设定;
子结构是通过遍历分子式的原子树(由分子式的原子通过化学键连接构成的树结构)形成的,即依次从每一个原子a开始向前、向后遍历,将遍历到的全部原子、化学键和原子a一起形成子结构;遍历停止时即形成一个子结构,子结构记录了形成过程中遍历到的原子、原子之间的化学键、以及多个原子是否形成了环状结构,遍历的停止条件有两个:①当遍历到的下一个原子与当前的遍历过的几个原子形成环状时,子结构形成过程(遍历过程)即停止,得到一个子结构;②当遍历的原子数量达到7个或已经顺序遍历完当前的分子式时即停止,得到一个子结构。在对所有分子式形成了子结构集合后,去掉集合中的相同子结构,即等同的子结构用同一个原子序列表示;相同子结构有两种情况:某子结构的逆序排列是其相同子结构,环状子结构中任意原子作为起始点,均为相同子结构;
把分子式得到的一系列子结构哈希映射到一个由32位整数组成的数组上,数组长度可以设置为32或32的整数倍,得到分子式的哈希表示;例如:分子式”ccc(=o)cl”的分词,其中1、2表示一价、二价化学键:
c1c
c1c1c
o2c
o2c1c
o2c1c1c
cl
cl1c
cl1c1c
cl1c1c1c
cl1c2o
该分子式的图形如图2所示,其哈希表示如下,其中每个整数都是32位的:
131072000001342177280160768000000003276810737459200000000000131072。
分子式得到的一系列子结构作为分子式的索引,建立es数据库中所有分子式的倒排索引。
第二步:检索过程:
1.检索时对输入的分子式进行上述分词,得到分子式的子结构集合,并用上述过程计算分子式的哈希表示;
2.用子结构集合在倒排索引中查找匹配项,即查找包含子结构集合中任一子结构的分子式,得到es数据库中匹配的分子式集合t;
3.用输入分子式的哈希表示,与得到的分子式集合t对应的哈希表示一一比较,得到输入分子式与分子式集合t中每个分子式之间的相似度;两个分子式的相似度的计算方式为:每个分子式的哈希表示均看作一个二进制的大整数,具体为:将数组中的每个32位整数依次转化为二进制后,拼接为一个二进制的大整数;将两个分子式对应的两个大整数做按位(bit)与操作得到一个相同长度的二进制大整数a,计算a中1的个数an;做按位或操作得到一个相同长度的二进制大整数b,计算b中1的个数bn;an/bn即得到两个分子式的相似度;
4.根据输入分子式与分子式集合t中每个分子式的相似度,按照应用需要,输出t中相似度大于阈值的分子式,或按照相似度从大到小排列,输出前n个最相似的分子式;
由于步骤3中比较的范围为集合t,而不是数据库中全部的分子式,减少了比较的次数,加快了分子式的检索。本发明将一步检索拆成两步,在第一步中通过利用倒排索引的检索减少了第二步比较的次数,进一步加快了检索的过程。
以上所述仅为本发明的较佳实施举例,并不用于限制本发明,凡在本发明精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!