基于药物及靶标信息的药物综合信息库建设方法及系统与流程
本发明涉及药物信息学、生物信息学和化学信息学领域,具体来说,涉及基于药物及靶标信息的药物综合信息库建设方法及系统,系统地整合药物相关的信息资源、提供交互式药物信息检索系统。
背景技术:
随着药物信息学以及信息科学的发展,挖掘已有药物的新适应症(旧药新用或药物重定位)逐渐成为国际上被广泛采用的研发策略,具有更高的投入产出效率,因此也成为众多国际制药企业重视和采用的一种方式;由于已上市药物的药物动力学以及安全性资料较为详尽,新用途的开发能很快能进行ⅱ期临床评估,据评估可节约大概40%的研发费用,并可大幅度缩短研发周期,能够在一定程度上有效规避研发风险、降低整体成本、加快药物上市的步伐,从而迅速满足临床用药需求;挖掘已有药物新的适应症(旧药新用或药物重定位)策略正从依赖临床观察的经验性研究,朝着基于医药大数据分析的系统性研究转变,综上所述,寻找高效的药物研发新策略或新资源成为当务之急。
目前,互联网上涌现了大量丰富的药物信息资源,且日益成为我国药学领域广大教学、科研人员获取学术信息的重要渠道,但网上的各种药学资源分别存储在不同国家、不同地区的服务器,缺乏集中统一的管理机制,整体上处于一种分散的状态,这种高度的自由性和开放性网络资源虽然为网上药学信息资源的发展提供了前所未有的机遇,但因为缺乏必要的过滤和质量控制机制,造成了网上药学资源优劣俱存、良莠不齐等问题,导致用户查找存在检准率低、针对性差、冗余信息太多等问题,使用起来事倍功半。
针对相关技术中的问题,目前尚未提出有效的解决方案。
技术实现要素:
针对相关技术中的上述技术问题,本发明提出一种基于药物及靶标信息的药物综合信息库建设方法及系统,能够为医药工作者提供一个更加高效便捷的获取药物自身及其关联信息的途径,为他们节约大量的宝贵时间,有利于临床、科研和教学任务的顺利展开。
为实现上述技术目的,本发明的技术方案是这样实现的:
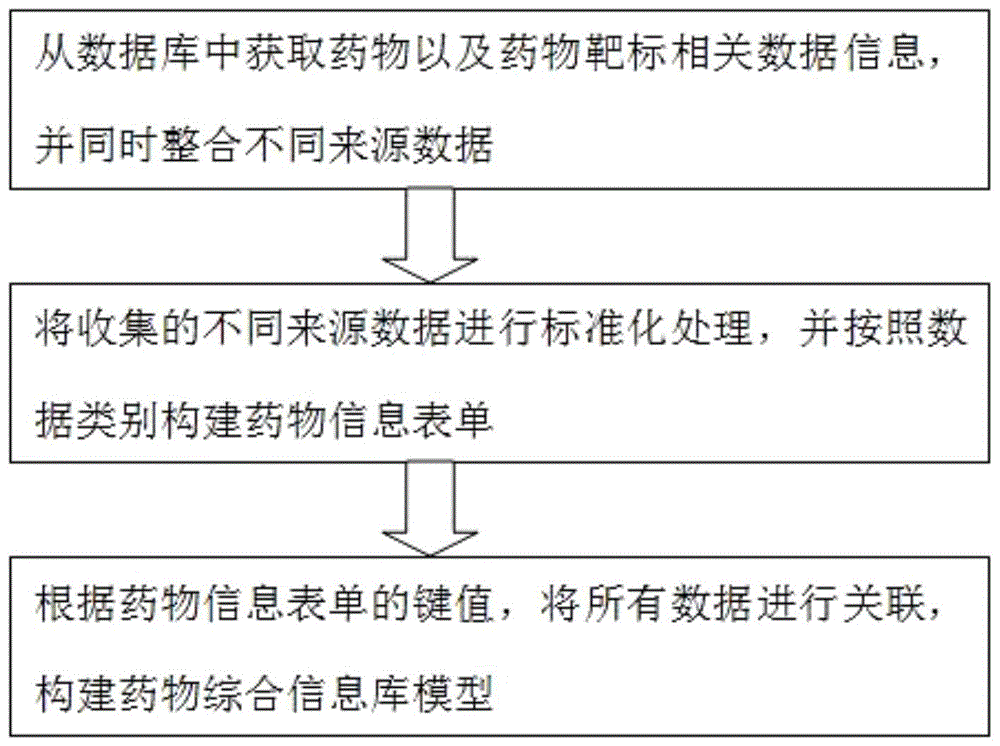
一种基于药物及靶标信息的药物综合信息库建设方法,包括以下步骤:
s1从数据库中获取药物以及药物靶标相关数据信息,并同时整合不同来源数据;
s2将收集的不同来源数据进行标准化处理,并根据数据类别构建药物信息表单;
s3根据药物信息表单的键值,将所有数据进行关联,构建药物综合信息库模型。
进一步地,所述步骤s1中获取药物以及药物靶标相关数据信息的过程需从对应数据库中下载对应的药物关联数据文件,并对获得的文件进行解析和清洗,提取所需的信息。
进一步地,所述步骤s1中整合不同来源数据,去除重复字段和冗余信息内容,对其缺失的信息进行补全。
进一步地,所述步骤s1中数据库包括但不限于drugbank、chembl、tcmid、pubchem、smpdb和keggdrug。
进一步地,所述步骤s2中药物信息表单包括但不限于小分子药物基本信息表、中药信息表、靶标数据信息表、靶标分类数据信息表和药物-靶标相互作用信息表。
进一步地,所述步骤s3中将构建药物综合信息库模型进行展示,其中,展示页面包括但不限于药物识别信息、药物特性、药理信息、毒理信息、药物药物相互作用信息、参考网站外链接信息和药物靶标的网络展示。
本发明的另一方面,提供一种基于药物及靶标信息的药物综合信息库建设系统,包括:
获取模块,用于从数据库中获取药物以及药物靶标相关数据信息,并同时整合不同来源数据;
第一构建模块,用于将收集的不同来源数据进行标准化处理,并根据数据类别构建药物信息表单;
第二构建模块,用于根据药物信息表单的键值,将所有数据进行关联,构建药物综合信息库模型。
进一步地,所述获取模块中获取药物以及药物靶标相关数据信息的过程需从对应数据库中下载对应的药物关联数据文件,并对获得的文件进行解析和清洗,提取所需的信息。
进一步地,所述获取模块中整合不同来源数据,去除重复字段和冗余信息内容,对其缺失的信息进行补全。
进一步地,所述获取模块中数据库包括但不限于drugbank、chembl、tcmid、pubchem、smpdb和keggdrug。
本发明的有益效果:
1、用户能够非常直观便捷的获取关注药物或靶标相关的信息;
2、通过药物和药物靶标实现查询和结果的可视化关联展示。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1是根据本发明实施例所述的基于药物及靶标信息的药物综合信息库建设方法的流程图;
图2是药物综合信息数据库模型;
图3是根据本发明实施例所述的基于药物及靶标信息的药物综合信息库建设系统的结构示意图。
具体实施方式
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员所获得的所有其他实施例,都属于本发明保护的范围。
如图1和2所示,根据本发明实施例所述的基于药物及靶标信息的基于药物及靶标信息的药物综合信息库建设方法,包括以下步骤:
s1从数据库中获取药物以及药物靶标相关数据信息,并同时整合不同来源数据;
具体的,此药物综合信息库构建底层数据依托的主体为drugbank中的数据,drugbank数据库是一个典型的生物信息学和化学信息学资源,包含了详细的药物数据和全面的药物靶点和药物相互作用信息,为每一种药物提供了近200项信息,包括药物作用靶点及其单核苷酸多态性分布等;截至2017年12月,drugbank收录了10000种药物条目,小分子药物为9000余个,其中1600左右个是fda批准上市的小分子化学药,200左右个为通过fda认证的生物药和106个营养补充剂,5030余种为试验药物。
drugbank中最基本的药物信息单元是药物卡片,每个卡片信息分为9块,分别是药物的识别信息:包含药物名称、在各种数据库中的唯一性编号、药物的化学结构、分子式等;药物的分类信息:包含功能基团、亚结构的化学分类信息;药理学信息:包含药物的适应症、作用药理、作用机理、药物动力学参数、毒性等信息;药物商品信息:生产商、专利、剂型和价格信息;药物化学基本特殊性:熔点、溶解性、亲水性等。根据drugbank提供的xml文件通过drugbank中的datasource介绍和xsd文件进行解析。
中药数据来源于tcmid,tcmid中药数据库,详尽的收录了中药相关信息,包括原始植物,具有生物功能的中药组分等,收录了1540余中可以作为中药使用的天然植物。
蛋白相关数据主要来自uniprot,这个数据库整合了swiss-prot、trembl和pir-psd三大数据库,主要是基于基因组测序项目完成后得到的蛋白质序列,包含了大量来自文献的蛋白质的生物功能信息。
s2将收集的不同来源数据进行标准化处理,并根据数据类别构建药物信息表单;
其中,药物信息表单主要包括以下几个部分:
drug表,是小分子药物的基本信息;包含drug_id(pk),drug_name(药物名称),drug_synonyms(药物的替代名称),drug_cas_number(化学文摘服务识别号码),drug_brands(药物所属的品牌名称),drug_type(药物类型—小分子smallmolecule),drug_groups(按药物研发状况所分的组——批准、保健品、非法、试验等中的一个或多个),drug_categories(治疗类别或一般类药物),drug_indication(药物用于治疗的疾病的描述或常见名称),drug_description(药物的一般事实,组成或制备的描述),drug_atccode;drug_structure表,是药物结构的基本信息,其中drug_id是主键,其它均是该结构体在其他公共数据库中的链接:包括2d化学结构以各种形式下载和查看结构的链接;3d结构的图像和链接,以查看结构查看器中的3d结构。
substance表,作为上传数据的表格,信息包括:主键substance_id;常见的理化性质和结构信息;对应的drug_id;上传者资料;上传时间等信息;数据来源信息。
tcm(中药)表,收录中药数据库中的非单体中药信息,主键:tcm_id,中药的多种信息,根据中药数据库中的药物信息制表。
tcm-compound(中药-化合物)表,用来记录中药和化合物成分的关系;主键:关联关系id;tcm_id;drug_id;关系类型;含量信息等。
target表,是靶标信息;其中target_id是主键,target_name是蛋白质或大分子(或其他小分子)的名称,target表通过gene_id和gene表进行关联来获取靶标的基因信息;同时包含靶标对应的基因的相关信息。gene_id(pk),gene_name(基因名称),genebank_gene_id(genbank数据库基因标识符),genebank_protein_id(genbank数据库中蛋白标识符),uniprot_id(uniport数据库中基因标识符),locus(更详细的基因染色体位置信息),orgnism(物种信息),general_function(主要功能3~4字的简单概述),specific_function(具体功能的详细描述,30-40个词汇),pdb_id(pdb数据库中的标识符),go_classification(基因本体分类,包括生物功能,亚细胞定位过程和分子功能),gene_synonyms(基因或蛋白质别名,缩写等),靶标类型(说明靶标是蛋白,小分子,一组类型的分子等中的哪一种)。
drug_target_action表,是药物与靶标作用表。id(pk),interaction_count(相互作用统计),drug_id(药物标识符),target_id(靶点标识符),known_action(已知作用),pathway_id(pathway库中的标识符),pharmgkb_id(药代动力学知识库识别号),relation_type(关系类型),evidence_type(证据类型),source(来源)。
s3根据药物信息表单的键值,将所有数据进行关联,构建药物综合信息库模型,此药物综合信息库模型包括药物-药物数据库、药物-靶标数据库。
具体的,将不同类型数据构建子数据表单,根据不同表单之间的关键词及关联信息,构建药物数据库模型;同时,根据表单中一些关键词,可链接到外部数据库。
本网站的数据库构建采用nodejs+express+mongodb框架;node.js是运行在服务端的javascript,是一个基于chromev8引擎的javascript运行环境,node.js使用了一个事件驱动、非阻塞式i/o的模型,使其轻量又高效,node.js的包管理器npm,是全球最大的开源库生态系统。express是一个基于node.js平台的极简、灵活的web应用开发框架,它提供一系列强大的特性,帮助我们创建各种web和移动设备应用;丰富的http快捷方法和任意排列组合的connect中间件,使得创建健壮、友好的api变得既快速又简单;express不对node.js已有的特性进行二次抽象,只是在它之上扩展了web应用所需的基本功能。
mongodb是由c++语言编写的,是一个基于分布式文件存储的开源数据库系统;在高负载的情况下,添加更多的节点,可以保证服务器性能;mongodb旨在为web应用提供可扩展的高性能数据存储解决方案;mongodb将数据存储为一个文档,数据结构由键值(key=>value)对组成;mongodb文档类似于json对象;字段值可以包含其他文档,数组及文档数组。
首先下载安装node.js,在path环境变量中配置node.js,新建本数据库express项目,上传项目代码到svn版本控制器上,并记录链接,安装mongodb数据库,并导入数据;通过关联数据表、整理数据表与模块展示数据的对应关系,包括药物(drug)模块对应数据表(drug_display、cross_ref_mapping、drug_interaction、drug_targets);靶标(target)模块对应数据表(targets_polypeptide、drugbank_polypeptide、drug_targets、target_classification_tree、drug_target_network);副作用(sider)模块对应数据表(meddra_indications、meddra_se_distinct、meddra_se);通路(pathways)模块对应数据表(drugbank_pathways);中草药(herb)模块对应数据表(tcmid_herb);检索(index)搜索模糊匹配(drug_display、tcmid_herb、drugbank_polypeptide、drugbank_pathways)。
提供对药物及其相关信息的检索和可视化展示,提供药物、靶标、通路、副作用等不同类型的数据检索,不同类型数据检索展示样式不同。
检索功能的search框采用了一种模糊匹配的查找功能。
基于步骤3,首先确定与搜索类型对应的数据库表的名字和字段的名称。包括drug、target、herb、pathway,分别对应的字段名称为:'drug':{'collectionname':'drug_display','queryfield':'name','showfield':'name','keyfield':'drugbankid'},'herb':{'collectionname':'tcmid_herb','queryfield':'herb_pinyin_name','showfield':'herb_pinyin_name','keyfield':'herb_pinyin_name'},'target':{'collectionname':'drugbank_polypeptide','queryfield':'name','showfield':'name','keyfield':'plypeptideid'},'pathway':{'collectionname':'drugbank_pathways','queryfield':'name','showfield':'name','keyfield':'name';
在此基础上将用户选中的搜索类型关联到数据库中的表以及对应的字段,实现相应信息的查找。
检索靶标信息时,提供了靶标信息分类树,该信息树是参考蛋白分类标准对本信息库靶标蛋白进行分类的,靶标信息的详细展示页面提供了靶标名称,基因名称,靶标的分类,go分类,相关的药物信息(以表格的形式展示),部分词条下面提供了药物、疾病和靶标的网络关系的可视化展示,同时提供了uniprot数据库的外链接,可以点击访问参考查看本词条更多的相关信息。
检索通路信息时,展示与不同通路相关的药物信息,同时提供了smpdb数据库的外链接,可点解访问查看本词条更多相关信息。
检索药物副作用时,列举了此副作用和适应症的相对应的药物名称,并提供相关的链接。同时还提供外链接到sider页面查看更多相关信息。
综上所述,根据上述步骤s1、步骤s2和步骤s3实现药物信息的相关检索查询和展示,具体包括药物、靶标、通路和中草药的查询,结构相似小分子药物查询,结构详细靶标查询;药物详细信息页面展示,靶标详细页面展示,通路信息展示和副作用信息展示等。
在本发明的一个具体实施例中,所述步骤s1中获取药物以及药物靶标相关数据信息的过程需从对应数据库中下载对应的药物关联数据文件,并对获得的文件进行解析和清洗,提取所需的信息。
不同数据库所提供的相关数据格式种类包括xml、txt、csv、tsv等多种格式,根据目前流行的药物信息各种资源库,涉及drugbank、chembl、tcmid、pubchem、smpdb、keggdrug等,从官方网站下载数据包,并同时下载相应说明文档,对数据包的数据根据相应的说明文档提供的字段编写python脚本进行解析,并将结果同一保存为csv格式,所涉及的数据来源尽可能的广泛。
以drugbank数据库数据获取为例,注册并登陆drugbank网站,在https://www.drugbank.ca/releases/latest页面下载alldrugs相对应的xml文件,采用python语言,按照网站相关页面https://www.drugbank.ca/documentation#drug-cards提供的datasource说明文档,以及https://www.drugbank.ca/releases/latest网站提供的xml文件结构说明的xsd文档,对该xml文件进行解析,并保存为csv格式文件,获得drugbank数据库中所需要的药物所有信息。
根据解析后的csv文件内容,浏览文件中的相关字段和属性信息,以确定有价值的数据信息,利用python脚本提取相应有价值字段,对文件内容进行重新整合,利用python脚本或数据库操作去除重复字段和冗余信息,从而实现对数据的清洗过程;此处高度考虑到用户使用需求,涵盖目前药物研究者所关心的药物名称、理化性质、药效、药代、药物毒副作用、药物适应症、相关药物信息网站链接、药物靶标信息、靶标蛋白信息、通路信息以及中草药信息。
在本发明的一个具体实施例中,所述步骤s1中整合不同来源数据,去除重复字段和冗余信息内容,对其缺失的信息进行补全。
具体的,将不同来源的数据进行整合,去除重复字段、冗余信息内容,对缺失的信息进行补全;此处包括药物数据整合、主要成分(化合物)整合、蛋白质id整合以及与其他数据库的整合等,根据需要,对药物标签进行分类,主要包括:化学药、生物药、天然产物、分子结构清楚的单体中药。
根据解析后的csv文件内容,浏览文件中的相关字段和属性信息,以确定有价值的数据信息,利用python脚本提取相应有价值字段,对文件内容进行重新整合,利用python脚本或数据库操作去除重复字段和冗余信息,从而实现对数据的清洗过程。本发明此处高度考虑到用户使用需求,涵盖目前药物研究者所关心的药物名称、理化性质、药效、药代、药物毒副作用、药物适应症、相关药物信息网站链接、药物靶标信息、靶标蛋白信息、通路信息以及中草药信息。
在本发明的一个具体实施例中,所述步骤s1中数据库包括但不限于drugbank、chembl、tcmid、pubchem、smpdb和keggdrug。
在本发明的一个具体实施例中,所述步骤s2中药物信息表单包括但不限于小分子药物基本信息表、中药信息表、靶标数据信息表、靶标分类数据信息表和药物-靶标相互作用信息表。
在本发明的一个具体实施例中,所述步骤s3中将构建药物综合信息库模型进行展示,其中,展示页面包括但不限于药物识别信息、药物特性、药理信息、毒理信息、药物药物相互作用信息、参考网站外链接信息和药物靶标的网络展示。
检索药物时,点击药物名称可以进入药物详细信息展示页面,包括药物识别信息、药物特性、药理信息、毒理信息、药物药物相互作用信息、参考网站外链接信息和药物靶标的网络展示。
药物的识别信息:药物描述、同义药名、casnumber、分子质量、分子式、iupac名称等;药物的理化性质包括里宾斯基五规则、氢键受体信息、氢键供体信息、logp值、水溶性等;药理信息:包括药代动力学、药物适应症、药物作用机制、药物吸收性、药物毒性等;药物-药物相互作用包括本词条药物和其他药物之间的相互作用,使药效增强或使药效减弱或产生毒性等;药物参考信息包括chembl、emolecules、pharmgkb、pubchem等中关于本词条药物的其他种类的描述信息,作为对本词条信息的补充;在药物详细信息展示页面提供了此药物相关的靶标的关联信息;为用户提供直观的有关系的药物-靶标关系对的展示图,该图为交互式的,利用悬浮框展示药物靶标属于的分类信息,点击靶标图案可跳转到对应的详细信息页面。
如图2和3所示,本发明的另一方面,提供一种基于药物及靶标信息的药物综合信息库建设系统,包括:
获取模块,用于从数据库中获取药物以及药物靶标相关数据信息,并同时整合不同来源数据;
第一构建模块,用于将收集的不同来源数据进行标准化处理,并根据数据类别构建药物信息表单;
第二构建模块,用于根据药物信息表单的键值,将所有数据进行关联,构建药物综合信息库模型。
在本发明的一个具体实施例中,所述获取模块中获取药物以及药物靶标相关数据信息的过程需从对应数据库中下载对应的药物关联数据文件,并对获得的文件进行解析和清洗,提取所需的信息。
在本发明的一个具体实施例中,所述获取模块中整合不同来源数据,去除重复字段和冗余信息内容,对其缺失的信息进行补全。
在本发明的一个具体实施例中,所述获取模块中数据库包括但不限于drugbank、chembl、tcmid、pubchem、smpdb和keggdrug。
综上所述,借助于本发明的上述技术方案,用户能够非常直观便捷的获取关注药物或靶标相关的信息;通过药物(包括药物名称、pubchemid、drugbankid、atccode等)和药物靶标(包括uniprotid、accessionnumber、keggid等)实现查询和结果的可视化关联展示。
以上所述仅为本发明的较佳实施例而已,并不用以限制本发明,凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
- 还没有人留言评论。精彩留言会获得点赞!