一种面向SSD的基因对比方法及系统与流程
本发明涉及数据处理领域,尤其涉及一种面向ssd的基因对比方法及系统。
背景技术:
在生物学中,基因序列分析是非常关键的一部分。就目前的技术来说,测出某dna片段的序列已非常高效,目前的基因序列分析瓶颈在于readmapping环节,亦就是基因序列对比环节。而在基因序列对比中,一般使用编辑距离来判断两个dna的相似度,但是编辑距离的计算非常耗费时间。
其次,dna片段数据量非常庞大,当某个dna片段需要和百万级别或以上的dna片段进行对比时,频繁的dna数据读写所耗费的时间非常庞大。
在dna基因对比这方面,已经有前人提出了过滤算法,如grim-filters,他们旨在先通过一个简单算法过滤掉一些不需要进行编辑距离计算的dna片段,通过减少编辑距离的计算次数来达到加速dna对比的目的。
因此开发一种新的基因比对方法解决原有方法对基因数据处理效率低时间长的问题具有十分重要的意义。
技术实现要素:
本发明的主要目的在于提供一种面向ssd的基因对比方法,旨在解决现有技术中对基因数据处理效率低、时间长的技术问题。
为实现上述目的,本发明第一方面提供面向ssd的基因对比方法,包括:
获取第一dna片段和第二dna片段,在ssd中对两个dna片段进行对齐处理;
从ssd中读取所述两个dna片段的数据,根据所述两个dna片段的数据及预设的精度参数确定对比参数;
基于对比参数,对所述两个dna片段进行递进式对比,得到对比结果。
为实现上述目的,本发明第二方面提供一种面向ssd的基因对比系统,所述系统包括:
dna数据对齐模块,用于获取第一dna片段和第二dna片段,在ssd中对两个dna片段进行对齐处理;
参数确定模块,用于从ssd中读取所述两个dna片段的数据,根据所述两个dna片段的数据及预设的精度参数确定对比参数;
递进式对比模块,用于基于对比参数,对所述两个dna片段进行递进式对比,得到对比结果。
本发明结合ssd,一方面减少了数据读写次数;另一方面通过对齐策略,递进式对比等手段将数据先进行一个粗略的筛选,大大减少了需要编辑辑距离计算的数据的比例,提升了效率,缩短了计算时间。
附图说明
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
图1为本发明实施例中面向ssd的基因对比方法的流程示意图;
图2为本发明实施例中面向ssd的基因对比系统的结构示意图。
具体实施方式
为使得本发明的发明目的、特征、优点能够更加的明显和易懂,下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而非全部实施例。基于本发明中的实施例,本领域技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
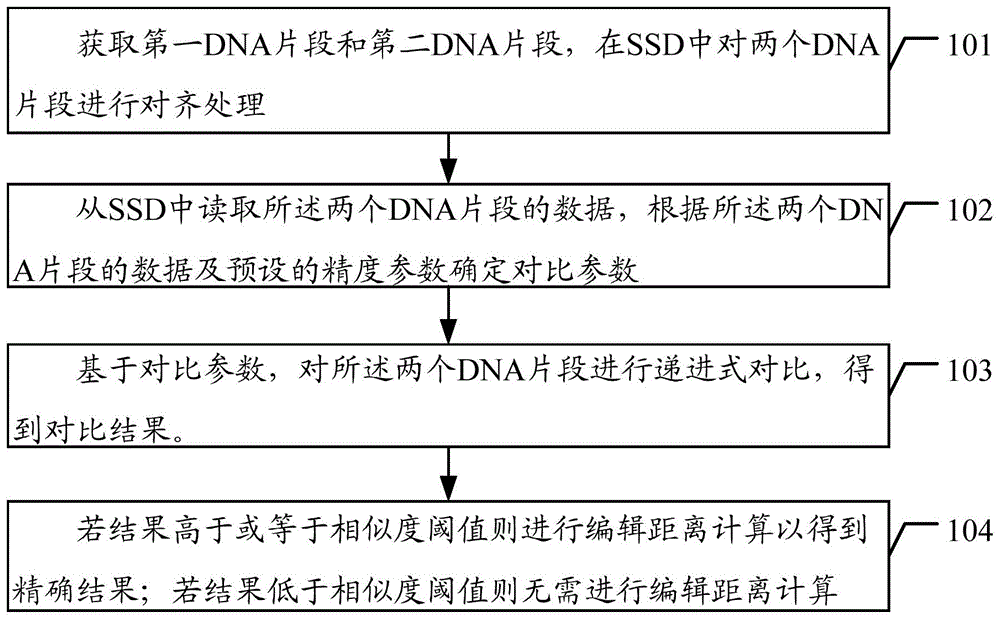
请参阅图1,为本发明实施例面向ssd的基因对比方法的流程示意图,该方法包括:
步骤101、获取第一dna片段和第二dna片段,在ssd中对两个dna片段进行对齐处理;
本发明实施例中,具体的可以获取dna片段的数据,确定n块连续空闲的物理块;所述dna片段包括第一dna片段和第二dna片段;
对于第n个物理块,n的初始值为1,n为小于或等于n的正整数,将dna片段的数据放入第n个物理块,确认第n个物理块是否被放满,若未被放满则代表数据放置完毕,记录下最后一块物理块页数num_final_page;计算总物理页数num_total_page,将dnaid、n、num_total_page这三个数据形成一个节点放在链表dnainfo的尾部;
其中总页数的计算公式为:num_total_page=(n-1)*num_each_block+num_final_page
num_each_block为每个物理块的页数;
若该物理块被放满则将剩余数据放入下一块空闲物理块,直至某一块未放满执行上述操作。
步骤102、从ssd中读取所述两个dna片段的数据,根据所述两个dna片段的数据及预设的精度参数确定对比参数;
本发明实施例中,具体的可以从链表dnainfo中找到第一dna片段的节点及第二dna片段的节点,将第一dna片段的节点及第二dna片段的数据读取到内存,并计算第一dna片段及第二dna片段的大小dna_a_length、dna_b_length;
比较所述两个dna片段大小,得到最小值dna_min_length;
确定精度参数scale_1、scale_2、scale_3得到起始比较次数compare_times,以下用c表示;
其中compare_times=dna_min_length*scale_1;
dna_length=num_total_page*每页的大小。
精度参数scale_1、scale_2、scale_3在0-1之间。
步骤103、基于对比参数,对所述两个dna片段进行递进式对比,得到对比结果;
本发明实施例中,具体的可以从[0,dna_min_length]中等概率选出c个位置,dna_min_length为所述两个dna片段大小的较小值,分别将两个dna片段上的c个位置上的数据逐一对比,得到相同个数count;
若count<c*scale_2则两个dna片段相似度低于阈值;;
若count≥c*scale_2则将compare_times调整为2倍,若2c≤dna_min_length*scale_3则说明这两个dna相似度达到阈值;若2c>dna_min_length*scale_3则回到本步骤的第一步。直到判定出两个dna片段的相似度。
步骤104、若结果高于或等于相似度阈值则进行编辑距离计算以得到精确结果;若结果低于相似度阈值则无需进行编辑距离计算。通过前三个步骤可以筛选出差别较大的dna,最后一步通过准确度更高的距离编辑计算最终确定数据的准确性。
请参阅图2,为本发明实施例中面向ssd的基因对比系统的结构示意图,包括:
dna数据对齐模块,用于获取第一dna片段和第二dna片段,在ssd中对两个dna片段进行对齐处理;
参数确定模块,用于从ssd中读取所述两个dna片段的数据,根据所述两个dna片段的数据及预设的精度参数确定对比参数;
递进式对比模块,用于基于对比参数,对所述两个dna片段进行递进式对比,得到对比结果;
编辑距离计算模块,用于对高于或等于相似度阈值的两个dna片段进行进编辑距离计算。
需要说明的是,图2所示实施例中各个模块的内容与图1所示实施例中各个步骤的内容相似,具体可参阅图1所示实施例中的内容,此处不做赘述。
在本申请所提供的几个实施例中,应该理解到,所揭露的装置和方法,可以通过其它的方式实现。例如,以上所描述的装置实施例仅仅是示意性的,例如,所述模块的划分,仅仅为一种逻辑功能划分,实际实现时可以有另外的划分方式,例如多个模块或组件可以结合或者可以集成到另一个系统,或一些特征可以忽略,或不执行。另一点,所显示或讨论的相互之间的耦合或直接耦合或通信连接可以是通过一些接口,装置或模块的间接耦合或通信连接,可以是电性,机械或其它的形式。
所述作为分离部件说明的模块可以是或者也可以不是物理上分开的,作为模块显示的部件可以是或者也可以不是物理模块,即可以位于一个地方,或者也可以分布到多个网络模块上。可以根据实际的需要选择其中的部分或者全部模块来实现本实施例方案的目的。
另外,在本发明各个实施例中的各功能模块可以集成在一个处理模块中,也可以是各个模块单独物理存在,也可以两个或两个以上模块集成在一个模块中。上述集成的模块既可以采用硬件的形式实现,也可以采用软件功能模块的形式实现。
需要说明的是,对于前述的各方法实施例,为了简便描述,故将其都表述为一系列的动作组合,但是本领域技术人员应该知悉,本发明并不受所描述的动作顺序的限制,因为依据本发明,某些步骤可以采用其它顺序或者同时进行。其次,本领域技术人员也应该知悉,说明书中所描述的实施例均属于优选实施例,所涉及的动作和模块并不一定都是本发明所必须的。
在上述实施例中,对各个实施例的描述都各有侧重,某个实施例中没有详述的部分,可以参见其它实施例的相关描述。
以上为对本发明所提供的一种基于极限学习机的极限ts模糊规则推理方法及系统的描述,对于本领域的技术人员,依据本发明实施例的思想,在具体实施方式及应用范围上均会有改变之处,综上,本说明书内容不应理解为对本发明的限制。
- 还没有人留言评论。精彩留言会获得点赞!