一种提高全基因组预测准确性的方法与流程
本发明属于作物分子育种领域,具体涉及到通过改良全基因组预测(gp)线性模型组分来提高gp预测准确性的方法,具体方向为现代农业技术。
背景技术:
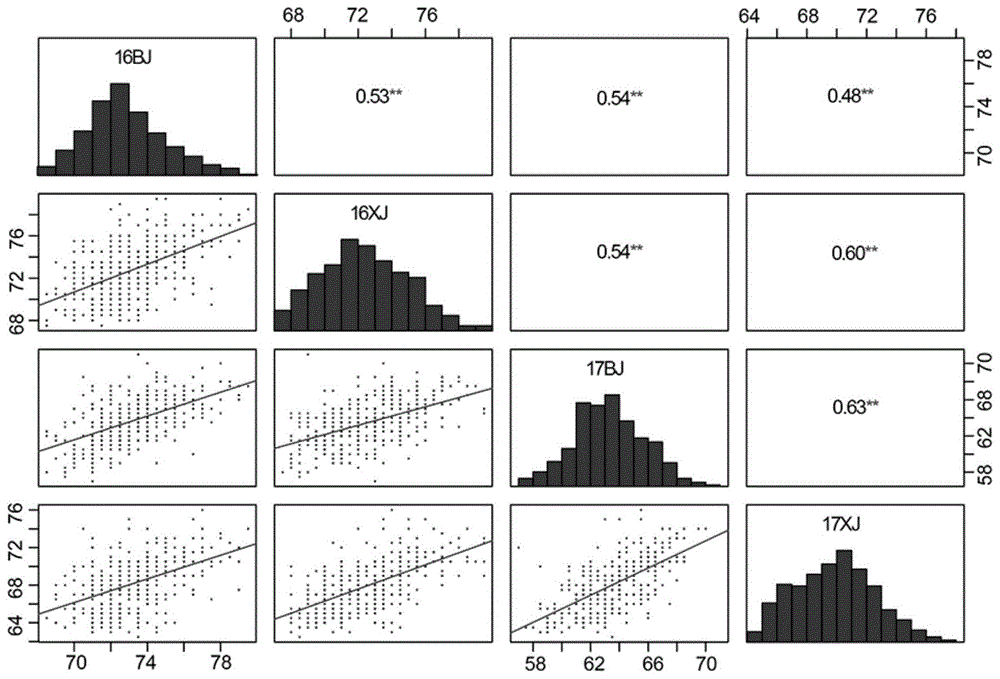
:全基因组预测(gp)是一种新型的分子育种技术,这种技术需要建立两个群体,一个是训练群体,一个是预测群体。其中训练群体需要进行表型和基因型鉴定,利用训练群体估计分子标记的标记效应,从而根据标记效应来估计预测群体的育种值。与常规的分子标记辅助育种(mas)相比,gp具有如下优点,一是gp不需要鉴定显著性的数量性状位点(qtl),二是gp可以照顾到微效qtl的信息,三是gp可以通过加快育种周期和提高遗传增益,从而提高育种效率。提高gp预测的准确性可以提高gp辅助育种的效率,从而更加准确地预测测试材料的表型。前人对gp预测准确性的影响因素的研究主要集中于群体大小、标记密度、遗传基础、群体间的亲缘关系、连锁不平衡程度等。这些影响因素是进行gp育种项目前需要考虑的工作。本发明关注的重点在于,当所有数据已经获得,通过对数据进行处理能不能提高预测的准确性。典型的gp模型是y=xβ+zu+ε,在这个模型中,β是固定效应,u是随机效应。对小麦的抗锈病的研究发现,使用与sr2连锁的标记作为固定效应的gblup模型的预测准确性(pa)比普通gblup更准确。利用一个水稻育种群体进行全基因组关联分析(gwas),把显著的标记作为固定效应的gp模型的预测准确性优于其他六个模型(spindel等,2016)。在玉米中,通常从两个优良自交系杂交产生的f1植株后代中选择育种材料(hallauer等,2010)。因此,有必要研究在玉米双亲群体中验证把效应较大的分子标记作为固定效应的预测效果,目前在玉米中尚未有这方面的研究报道。技术实现要素:为了在不增加成本的基础上提高全基因组选择的效率,本发明提供了一种通过把大效应snp作为固定效应和增加g×e效应来提高gp预测准确性的方法。发明的目的是通过把大效应snps作为固定效应和增加g×e效应,证明将这两种效应合并到一起能否进一步增加gp的预测准确性。本发明首先提供一种提高全基因组预测准确性的方法,其特征在于,包括以下步骤:(1)对目标作物群体进行表型和基因型鉴定,然后基于对整个群体的全基因组关联分析,找到效应最大的n个单碱基变异snps,所述n为正整数,其取值由基于gp预测准确性的显著性检验而定;(2)把效应最大的n个snps作为固定效应,在并全基因组选择模型中加入基因型与环境互作组分,进行全基因组预测。步骤(1)中,对目标作物群体的表型分析,确保目标作物种群在不同环境中有共同的遗传基础。步骤(1)中,进行表型分析时,计算整个群体的最佳线性无偏估计值blue值和h2用于后续关联分析,在计算blues和h2时,利用如下模型:yijm=μ+gi+ej+geij+δ(j)m+εijm,其中yijm是第ith(i=1,2...,481)个基因型在第jth(j=1,2,3,4)环境下表型,mth(m=1,2)表示镶嵌在环境下的重复;μ是总体平均,gi是基因型效应,ej是环境效应,geij是基因型与环境互作效应,δ(j)m是重复效应,εijm是剩余残差。当计算blues时,gi作为固定效应,其他变量作为随机效应。在计算h2时,所有效应为随机效应,从而估计每个效应的方差,h2的计算公式是:在计算h2时,所有效应均为随机效应以便估计每个效应的方差,其中和分别代表遗传方差、遗传与环境互作方差和残差,ne表示环境数,r表示重复数。步骤(1)中,所述全基因组关联分析的模型是:y*=xβ+zg+wτ+ε其中y*是blues,β是固定效应,在这里只包含总体平均值,g是遗传背景效应,τ是标记效应作为随机变量,ε是残差。x、z和w是设计矩阵。步骤(1)中,找到效应最大的4个单碱基变异snp。本发明提供的上述方法的步骤(2)中,在全基因组预测模型中加入基因型与环境互作组分的g×e模型是:y1是第i个环境下的表型,i为从1到n的整数,μi是第i个环境下的表型的总体平均值,x1是基因型矩阵,β是在各环境中保持不变的标记效应,β被分解为两部分,一个是在个环境保持不变的主效应β0,一个是环境特异性的βi,i是指第i个环境,εi是残差。本发明提供的方法,所述的作物包括玉米、水稻、小麦、大豆、花生、高粱、油菜、芝麻、大麦。进一步,本发明提供了一种提高玉米全基因组预测准确性的方法,包括以下步骤:(1)构建包含多个家系的bc1f3:4群体,对群体中所有玉米材料进行表型和基因型鉴定,表型是在多个环境条件下统计玉米开花期,每个环境设置两个重复,基因型是利用包含5.5万个snps的基因芯片进行鉴定;(2)利用bc1f3:4群体进行关联分析,找到效应最大的前50个snps,对这50个snps进行多元线性回归,鉴定出这50个snps的表型贡献率(pve),将这50个snps按照pve从大到小进行排序;利用bc1f3:4群体开花期的blue值进行gp预测,gp预测利用五折交叉验证重复200次,分别把效应最大的前5个snps作为固定效应,检验gp预测准确性增加的情况,发现把效应最大的前5个snps作为固定效应的gp模型预测准确性与把效应最大的4个snps作为固定效应的gp模型预测准确性差异不显著,说明把效应最大的4个snps作为固定效应能最大限度地提高gp预测准确性;(3)证明增加g×e效应的gp模型即g×e模型预测准确性比跨环境的gp模型即a-e模型的预测准确性高:四个环境共计有六种环境组合,利用两种交叉验证模式cv1和cv2对g×e模型和a-e模型进行比较,分别在cv1和cv2模式下,比较当所有标记都是随机变量时和把四个大效应snps作为固定效应时g×e模型和a-e模型的预测准确性;(4)证明g×e模型预测准确性的增加与遗传方差的降低有关,分别利用blue值,单环境表型,a-e模型和g×e模型,在四个大效应snps作为随机变量和固定变量时,估计并比较遗传方差。当把大效应snps作为固定效应时,由于这些大效应的snps作为固定效应时对遗传方差没有贡献,导致遗传方差降低。同时由于这些大效应的snps作为固定效应时可以避免对这些大效应snps效应的估计偏差,从而提高了对全基因组预测的准确性。在本发明的实施例中,主要包括以下内容:构建包含481个家系的bc1f3:4群体,对这481份材料进行表型和基因型鉴定,表型是在两年两个环境条件下统计玉米开花期,每个环境设置两个重复,四个环境分别是2016和2017年北京顺义,2016和2017年新疆昌吉。基因型是利用包含5.5万个snps的基因芯片进行鉴定,利用bc1f3:4群体进行关联分析,找到效应最大的前50(top50)个snps,对这50个snps进行多元线性回归,鉴定出这50个snps的pve,将这50个snps按照pve从大到小进行排序。利用bc1f3:4群体开花期的blue值进行gp预测,gp预测利用五折交叉验证重复200次,分别把pve最大的前1个snp(top1)、top2、top3、top4、top5的snps作为固定效应,检验gp预测准确性增加的情况。证明增加g×e效应的gp模型(g×e模型)预测准确性比跨环境的gp模型(a-e模型)的预测准确性高。四个环境共计有六种环境组合,我们利用两种交叉验证模式(cv1和cv2)对g×e模型和a-e模型进行比较,分别在cv1和cv2模式下,比较当所有标记都是随机变量时和把四个大效应snps作为固定效应时g×e模型和a-e模型的预测准确性。证明g×e模型预测准确性的增加与遗传方差的降低有关,分别利用blue值,单环境表型,a-e模型和g×e模型,在四个大效应snps作为随机变量和固定变量时,估计遗传方差。从而证明遗传方差和把大效应snps作为固定效应时gp预测模型准确性增加的相关性。本发明可以在完成基因型和表型鉴定后,在不增加其他工作量的情况下,仅仅利用现有的数据信息,尽可能地提高预测准确性。该发明应用到分子育种工作中,将会提高预测效率,从而提高预测的可靠性并节约育种成本。附图说明图1为bc1f3:4群体开花期在四个环境下的分布和相关性图。图中**表示p≤0.01,16表示2016年,17表示2017年,bj表示北京,xj表示新疆。图2a为11781个多态性snps在玉米基因组染色体上的分布热图。颜色深浅程度代表每1mb范围内的snp数目差异。chr1,chr2,……chr10代表玉米的十条染色体。标记的物理位置是基于玉米b73参考基因组v3序列版本;图2b为481个bc1f3植株的基因型来源分布,颜色由浅到深分别代表ph4cv背景,杂合背景和郑58背景。图3为大效应snps的鉴定和不同gp模型的比较图,a:gwas分析产生的qq图;b:gwas分析产生的manhattan图;c:利用多元线性回归计算-log10(p)值最大的前50的pve,图中只显示pve大于1%的snps;d:t测验表明把效应最大的前4个snps作为固定效应能最大限度地提高预测准确性,pa代表预测准确性,***表示p<0.001,**表示p<0.01,ns表示不显著。图4为把四个大效应snps作为固定效应的gp模型预测准确性高于随机选择四个snps作为固定效应的gp模型预测准确性图。随机挑选四个snps作为固定效应的gp预测准确性分布图,该过程重复200次,每次用100次五折交叉验证,pa代表预测准确性。右侧三角表示把四个大效应snps作为固定效应的gp模型预测准确性(0.7657),左侧三角表示没有选择snp作为固定效应的gp模型预测准确性(0.7466)。图5a、图5b、图5c为把四个大效应snps作为固定效应可以增加每个环境下gp的预测准确性和减少遗传方差,图5a:把四个大效应snps作为固定效应降低遗传方差;图5b:把四个大效应snps作为固定效应对残差没有影响;图5c:把四个大效应snps作为固定效应提高预测准确性。16bj、16xj、17bj、17xj表示四个环境,fixed表示把四个主效snps作为固定效应的预测模型,random表示没有把任何snp作为固定效应的预测模型,pa表示预测准确性,***表示t测验表明两种模型差异显著性水平小于0.001。具体实施方式以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段,所用原料、试剂、药品均为市售商品。实施例11、表型分析:利用亲本ph4cv和郑58为亲本构建包含481个家系的bc1f3:4群体,其中ph4cv为轮回亲本。对这481份材料进行表型和基因型鉴定,表型是在两年两个环境条件下(共计四个环境)统计玉米开花期,每个环境设置两个重复,四个环境分别是2016和2017年北京顺义,2016和2017年新疆昌吉。表型鉴定显示,四个环境下相关系数都达到极显著水平,相关系数从0.48-0.63,表明种群在不同的环境中具有共同的遗传基础(见图1)。多个环境条件下相对较高的广义遗传力(h2)和较低的变异系数证明了da的稳定性(见表1)。在进行表型分析时,计算了整个群体的blue值用于后续的关联分析,并计算了h2。在计算blues和h2时,利用如下模型:yijm=μ+gi+ej+geij+δ(j)m+εijm,其中yijm是第ith(i=1,2...,481)个基因型在第jth(j=1,2,3,4)环境下表型,mth(m=1,2)表示镶嵌在环境下的重复;μ是总体平均,gi是基因型效应,ej是环境效应,geij是基因型与环境互作效应,δ(j)m是重复效应,εijm是剩余残差。当计算blues时,gi作为固定效应,其他变量作为随机效应。当计算遗传力时,所有的变量都作为随机效应,从而估计出各变量的方差,h2的计算公式是:其中和分别代表遗传方差、遗传与环境互作方差和残差,ne表示环境数,r表示重复数,以上分析使用r语言程序包lme4完成。表1bc1f3:4群体开花期在四个环境下的表型分布特征2、基因型分析:利用481个bc1f3单株叶片提取dna,基因型鉴定利用包含5.5万个snps的基因芯片(xuc,reny,jiany,guoz,zhangy,etal.(2017)developmentofamaize55ksnparraywithimprovedgenomecoverageformolecularbreeding.molbreeding37:20.)进行鉴定。利用如下标准过滤snps:(1)去除任何亲本缺失数据的snps;(2)去掉亲本间非多态性snps;(3)去掉缺失率大于0.05的snps;(4)缺失的标记根据等位基因频率估算的预期值进行填补,填补方法参考文献为(pérezp,deloscamposg(2014)genome-wideregressionandpredictionwiththebglrstatisticalpackage.genetics198:483-495.)。过滤后得到11781个多态性snps,这些标记以足够高的密度分布在整个基因组中(见图2a)。对481株bc1f3的基因型分析表明,大多数植物的背景为纯合ph4cv基因型,平均占基因组的65.44%。纯合郑58基因型和杂合基因型分别占比为16.00%和18.56%(见图2b;表2)。郑58基因型存在于整个基因组中,这表明bc1f3群体在整个基因组中都处于分离状态,有利于进行后续的gwas分析。表2bc1f3群体背景中三种基因型的比例平均值最小值最大值郑58基因型16.0%2.1%38.2%杂合基因型18.6%3.7%38.4%ph4cv基因型65.4%41.4%88.9%3、关联分析和多元线性回归:利用bc1f3:4群体进行gwas分析,进行gwas分析的工具是r语言程序包sommer(参见文献covarrubias-pazarang(2016)genome-assistedpredictionofquantitativetraitsusingtherpackagesommer.plosone11:e0156744.),gwas分析的模型是:y*=xβ+zg+wτ+ε,其中y*是blues,β是固定效应,在这里只包含总体平均值,g是遗传背景效应,τ是标记效应作为随机变量,ε是残差。x、z和w是设计矩阵。gwas分析的qq图显示gwas模型在所研究的群体中是很好的拟合,假阳性信号得到很好的控制。曼哈顿图显示,最高峰值出现在2号染色体上,其次是9号染色体(见图3的a,b)。利用-log10(p)值最大的前50(top50)个snps,对这50个snps进行多元线性回归,得到每一个snp的回归平方和ssreg和总平方和和sstol,每个snp的pve是用ssreg除以sstol,从而鉴定出这50个snps的pve,将这50个snps按照pve进行排序,发现3号染色体上的snp的pve最大,为11.88%,其次是chr2、chr9和chr3上的snps,分别解释了总表型变异的7.52%、4.81%和4.59%(见图3的c)。4、固定不同数目topsnps的gp模型预测准确性比较:利用bc1f3:4群体开花期的blues进行gp预测,gp预测的gblup模型是:y=xβ+zu+ε,其中y是blues,β是固定效应,u是遗传效应作为随机变量,ε是残差,x和z是设计矩阵。上述模型用r语言程序包bglr进行拟合。其中,iterations设置为20000,burn-in设置为5000,进行100次交叉验证(pérez和deloscampos,2014)。当把snps作为固定效应时,β包含截距和作为固定效应的snps,这些snps的基因型加入到矩阵x中。分别把top1、top2、top3、top4、top5的snps作为固定效应,检验gp预测准确性增加的情况。利用双尾t测验检验把n个snps作为固定效应和把n+1个snps(n取值为1到4)作为固定效应时gp模型得到的100个预测准确性值的差异。结果表明,当把top4的snps作为固定效应时,gp预测性已经达到最大,因此我们在后续的研究中把top4的snps作为固定效应(见图3的d)。5、把四个大效应snps作为固定效应能提高gp预测准确性的进一步证明:为了证明把四个大效应snps作为固定效应提高gp预测准确性不是一种偶然现象。本发明在全基因组范围内随机选择四个snps作为固定效应,计算gp预测准确性,重复200次,发现把四个大效应snps作为固定效应时gp的预测准确性总比把四个随机选择的snps作为固定效应时gp的预测准确性高(见图4)。6、证明增加g×e效应的gp模型(g×e模型)预测准确性比跨环境的gp模型(a-e模型)的预测准确性高:四个环境共计有六种环境组合,本实施例利用两种多环境模型,即a-e模型和g×e模型。在a-e模型中,假定每个snp在每个环境中的效应是不变的,如果有n个环境,那么模型是在a-e模型中,y1是第i个环境(1,2,…,n)下的表型,μi是第i个环境下的表型的总体平均值,x1是基因型矩阵,β是在各环境中保持不变的标记效应,εi是残差。在g×e模型中,y1和μi同上,β被分解为两部分,一个是在各环境保持不变的主效应β0,一个是环境特异性的βi(i是指第i个环境),g×e模型是:在a-e模型和g×e模型中加入四个大效应snps作为固定效应的操作过程同步骤4,以上分析用r语言程序包bglr进行(r语言程序包bglr参见pérezp,deloscamposg(2014)genome-wideregressionandpredictionwiththebglrstatisticalpackage.genetics198:483-495.)。表3两种用来比较a-e和g*e模型的交叉验证方案表注:na表示缺失的待预测表型,n代表家系数本实施例利用两种交叉验证模式(cv1和cv2,见表3)对g×e模型和a-e模型进行比较,在cv1模式下,当所有标记都是随机变量时的12中预测模型中,有10中预测模型中g×e模型的预测准确性高于a-e模型;当把四个大效应snps作为固定效应的12中预测模型中,有10中预测模型中g×e模型的预测准确性高于a-e模型。在cv2模式下,当所有标记都是随机变量时的12中预测模型中,有8中预测模型中g×e模型的预测准确性高于a-e模型;当把四个大效应snps作为固定效应的12中预测模型中,有8中预测模型中g×e模型的预测准确性高于a-e模型,见表4。7、证明把四个大效应snps作为固定效应的g×e模型的预测准确性最高:从表4中可以看出,在cv1模式下,在12种预测模型其中10个模型,把四个大效应snps作为固定效应的g×e模型的预测准确性最高。在cv2模式下,在12种预测模型其中8个模型,把四个大效应snps作为固定效应的g×e模型的预测准确性最高。说明结合把四个大效应snps作为固定效应和增加g×e模型可以增加gp模型的预测准确性。表4在gp模型中把四个大效应snps作为固定效应并加入g×e效应提高预测准确性表注:两个环境下的数据作为训练群体,每个环境分别作为验证群体,所以每对环境对应两个预测准确性。a-e模型代表跨环境模型,g×e模型代表g×e互作模型,fixeda-e模型代表在模型中把四个大效应snps作为固定效应的跨环境模型,fixedg×e模型代表在模型中把四个大效应snps作为固定效应的g×e互作模型。√代表同一行中最大的pa值。8、证明把四个大效应snps作为固定效应和增加g×e效应导致的预测准确性增加与遗传方差的降低有关:在单环境模型下,当把四个大效应snps作为固定效应时,方差降低,预测准确性升高,而残差没有改变(见图5a、图5b、图5c)。在a-e模型和g×e模型中,当把四个大效应snps作为固定效应时,遗传方差也降低,但残差没有改变,证明遗传方差的降低与把四个大效应snps作为固定效应有关(见表5)。当比较a-e模型和g×e模型时发现,无论四个大效应snps是否作为固定效应,g×e模型的遗传方差总小于a-e模型的遗传方差,证明遗传方差的降低与加入g×e效应有关(见表5)。表5证明把4个大效应snps作为固定效应和增加g×e效应导致的预测准确性增加与遗传方差的降低有关表注:残差(剩余方差)遗传方差。a-e模型代表跨环境模型,g×e模型代表g×e互作模型,fixeda-e模型代表在模型中把四个大效应snps作为固定效应的跨环境模型,fixedg×e模型代表在模型中把四个大效应snps作为固定效应的g×e互作模型。虽然,上文中已经用一般性说明、具体实施方式及试验,对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1