一种基于深度学习模型的肿瘤新抗原预测方法及新生抗原预测系统与流程
1.本发明属于生物医药领域,涉及一种肿瘤新抗原的预测方法,尤其涉及一种基于深 度学习网络的肿瘤新抗原预测方法、预测系统、装置及其应用。
背景技术:
2.当今,肿瘤免疫已经跻身最火热的赛道。然而,从临床疗效看,肿瘤免疫道阻且长。 以pd-1/pd-l1为例,仅20%-30%的肿瘤患者能够从中受益;而car-t则只对血液肿瘤, 尤其是b淋巴细胞瘤有效,并且副作用严重。大多数肿瘤患者尚无有效的治疗方案,肿 瘤免疫治疗还需要探索更多可能性,个性化肿瘤疫苗便是其中之一。个性化肿瘤疫苗的 研发是一项将基因精准检测和肿瘤免疫治疗相结合的整合型技术。与car-t疗法围绕现 存的少量靶点寻找治疗方案不同,个性化肿瘤疫苗会从肿瘤突变基因中预测出新抗原, 并将合成新抗原回输到患者血液中,激发自体免疫系统对肿瘤细胞的攻击,为解决临床 中对无法进行手术根治、放化疗均无效且无靶向用药的患者提供了一种新的治疗方式。
3.个性化肿瘤疫苗的技术方案包括1)采集肿瘤患者的外周血,癌组织样本,2)外周 血做全外显子测序,肿瘤组织一式两份,一份做全外显子测序,一份做转录组测序,3)根据测序结果,预测潜在的新生抗原多肽,4)新生抗原多肽合成,5)高效体外系统评 测抗原多肽安全性,6)临床患者皮下注射使用。
4.预测新生抗原是个性化疫苗治疗方案中最为关键的一步,如果无法准确预测新生抗 原,后续基于预测结果的治疗效果就会受到影响。肿瘤一般含有几百甚至几千个非同义 突变,但是并不是所有突变都能产生新生抗原。我们需要从这些基因突变中筛选出真正 的新生抗原,即能够在细胞内被加工剪切与hla分子结合,并且被递呈到细胞表面诱导 免疫反应的突变肽链。
5.现在被广泛采用的新生抗原预测的技术方案是将肽链与hla亲和力、rna表达等参 数导入数学模型,给每个肽链进行打分,最终的模型用于预测肽链与hla的亲和力。
6.上述利用数学模型给肽链打分的方式具有以下三个局限性:
7.(1)数据维度少:主要考虑的是肽链的亲和力,没有考虑新生抗原被剪切呈递的过 程。iedb的数据是基于肽链与hla分子在体外的结合,没有考虑肽链是否真的呈递到人 体细胞表面,也没有考虑肽链与hla分子结合前的加工处理和运输环节。用这种方法预 测的肽链只有小于5%是在细胞表面可以被发现的。
8.(2)准确率低:这种预测方式的准确率在30%-40%左右。
9.(3)优化空间小:这种方式只能通过调整参数比重或者增加参数来提高准确率,而且 提高的空间非常有限。
10.因而,目前已经报道的肿瘤新生抗原预测方法准确率低,继而导致基于新生抗原的 抗肿瘤疫苗的疗效不佳。
技术实现要素:
11.本发明要解决的技术问题是提供一种新的获得新生抗原的方法,提高新生抗原预测 准确率。
12.本发明要解决的另一个技术问题是提供新的新生抗原预测系统及其应用。
13.本发明提供了一种新生抗原预测方法,该方法包括以下步骤:
14.(1)采集待预测新生抗原的样本,所述的样本包括肿瘤样本和源自同一个体的正常 样本;
15.(2)分别提取步骤(1)中获得的肿瘤样本和正常样本的dna;
16.(3)对步骤(2)所述的正常样本的dna进行全外显子测序,并且根据正常样本的 外显子测序数据进行人类白细胞抗原(hla)分型分析;
17.(4)对步骤(2)所述的肿瘤样本的dna进行全外显子测序;
18.(5)将步骤(3)和步骤(4)获得的全外显子测序数据与人类参考基因组grch38 版本进行比对拼接,分析肿瘤-正常成对样本的体细胞突变,获得突变肽链序列及其旁侧 序列;
19.(6)提取肿瘤样本的rna,进行转录组测序,对测序数据进行read counts计数,再 进行基因表达水平tpm(transcripts per million)值转换;
20.(7)将步骤(3)获得的hla分型、步骤(5)获得的突变肽链序列及其旁侧序列、 步骤(6)获得的基因表达水平值输入深度学习模型,获得预测的新生抗原。
21.(8)通过神经网络训练的模型进行打分获得预测的新生抗原;所述的神经网络是通 过反复训练的深度学习神经网络。
22.较好的,所述的训练包括:
23.对含有中国人群高频hla亚型细胞系进行转录组测序获得rna表达水平,使用蛋白 免疫沉淀和质谱获取与该hla亚型特异性结合的肽链序列及其旁侧序列;
24.对质谱获得的肽链做阳性标记,未在质谱结果中出现的肽链做阴性标记,作为训练 数据、验证数据、测试数据;
25.先分别计算出肽链呈递到每种hla分型的可能性,再总和获得呈递可能性,把肽链 根据呈递可能性从高到低排列,选出分值靠前的若干条作为潜在抗肿瘤新生抗原。
26.所述的体细胞突变包括但不限于单核苷酸突变、插入/缺失突变、移码突变。
27.步骤(1)中的肿瘤样本和正常样本,可以源自癌组织和癌旁组织,经过组织破碎和 裂解,获得肿瘤样本和源自同一个体的正常样本,例如肿瘤细胞和正常细胞。肿瘤样本 和正常样本也可以从体液、分泌物等离体样本中获取,例如从血液中获得。
28.较好的,步骤(2)和步骤(4)中所述的dna是基因组dna。
29.较好的,步骤(3)中所述的hla分型是指将正常样本的外显子测序数据与人类参考 基因组的序列进行对比,获得hla分型结果。
30.较好的,步骤(5)分析肿瘤样本和正常样本成对样本的体细胞突变,筛选非同义突 变,产生突变肽链以及其旁侧序列。
31.本发明可以使用常规的软件分析样本数据。例如,使用mutect2软件分析肿瘤样本 和正常样本成对样本的体细胞突变。使用fastq软件对测序数据进行质量控制处理。使 用bwa软件将测序数据与人类参考基因组进行比对拼接。用xhla软件对外周血样本外显 子测
序数据进行hla分型分析。通过featurecounts软件对测序数据进行read counts 计数。
32.所述的参考基因组包括但不限于人类参考基因组grch38版本。
33.较好的,分析肿瘤样本和正常样本成对样本的体细胞突变之前,先对测序数据进行 质量控制处理。
34.较好的,所述的肽链序列是样本进行蛋白免疫沉淀和质谱联用,获得与特定hla分 子结合的肽链序列。
35.较好的,所述的旁侧序列的获得方法为:在肽链序列中选取8-11个氨基酸长度的肽 链,并将氨基酸长度小于11的肽链填充到11个,截取其左右各5个氨基酸作为旁侧序 列。
36.较好的,在获得肽链序列过程中,从质谱肽链数据中排除rna表达水平小于等于0 的肽链。
37.在计算tpm时,可以使用以下公式将转录本测序读数(read count)转换为tpm:
38.rpk=read_count/transcript_length*1000
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ(i)39.tpm=rpk/sum(all_rpk)*1000000
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(ii)
40.在构建神经网络时,可以使用训练数据对神经网络进一步完善,提高其准确度。
41.较好的,所述的训练数据的获得方法为:
42.对人源样本进行hla分型分析,通过转录组测序获得rna表达水平,通过蛋白免疫 沉淀和质谱联用分析获得人源样本的肽链序列和旁侧序列;
43.将所述的hla分型、突变肽链序列、旁侧序列和转录组测序获得的rna表达水平, 与从公共数据库中采集的质谱数据输入神经网络;
44.对质谱获得的肽链做阳性标记,把在蛋白质公共数据库里的参考蛋白组中未在质谱 数据中出现的肽链做阴性标记,并将数据分为训练数据和验证数据。
45.所述的人源样本是离体的样本,包括人体的正常组织或者病变组织样本,例如毛发、 赘生物碎片、血液、尿液、脂肪、皮肤、指甲、脏器、粘膜,等等。本发明的一个实施 例中,使用了中国人群高频hla亚型细胞系,该细胞在中国人细胞系基础上使用特定的、 中国人群占比较高的hla亚型代替原有hla。
46.较好的,所述的深度学习模型构建方法为:利用上述训练数据构导入构建的神经网 络,采用深度学习的算法训练模型。
47.较好的,所述的计算肽链递呈到每种hla分型的可能性按照如下模型获得:
48.p(peptide i presented by hla k)=sigmoid{nnk(peptide i
)+nn
flanking
(flankingi) +nn
rna
(log(tpmi))}
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(iii)
49.其中,peptidei:独热编码的肽链i的氨基酸序列;
50.nnk:具有线性最后一层激活的神经网络,模拟肽链序列对呈递概率的贡献;
51.flankingi:独热编码的肽链i的旁侧序列的氨基酸序列;
52.nn
flanking
:具有线性最后一层激活的神经网络,模拟旁侧序列对呈递概率的贡献;
53.tpmi:肽链i来源mrna的tpm表达值;
54.nn
rna
:具有线性最后一层激活的神经网络,模拟rna表达值对呈递概率的贡献。
55.所述的综合肽链递呈到每种hla分型的可能性按照如下模型获得:
56.获取、hla分型分析单元)、新生抗原预测模块。
82.样本收集装置与转录组测序数据分析模块和全外显子测序数据分析模块连接,将样 本细胞运输到转录组测序数据分析模块和/或全外显子测序数据分析模块中,分别检测并 获得样本的转录组测序数据和/或全外显子测序数据;
83.体细胞非同义突变分析单元与全外显子测序数据分析模块连接,接收并比对肿瘤样 本和源自同一个体的正常样本的全外显子测序数据,计算并存储体细胞非同义突变数据;
84.体细胞非同义突变分析与hla分型分析单元连接,将正常样本的全外显子测序数据 呈递给hla分型分析单元,获得正常样本的hla分型数据;
85.突变肽链获取单元与体细胞非同义突变分析单元连接,接收体细胞非同义突变分析 呈递的体细胞非同义突变数据并筛选获得突变肽链序列;
86.旁侧序列获取单元与体细胞非同义突变分析单元连接,接收体细胞非同义突变分析 单元呈递的体细胞非同义突变数据并筛选获得旁侧序列;
87.转录组测序数据分析模块包括rna表达计算单元,将肿瘤样本的转录组测序数据呈 递给rna表达计算单元,获得rna表达水平数据;
88.新生抗原预测模块与转录组测序数据分析模块、突变肽链序列获取单元、旁侧序列 获取单元、hla分型分析单元连接,接收rna表达水平数据、肽链序列、旁侧序列、hla 分型数据并计算获得新生抗原递呈的可能性。
89.较好的,所述的体细胞非同义突变分析单元包括单核苷酸突变子单元、插入突变子 单元、缺失突变子单元和移码突变子单元,分别获取相对应的突变数据。
90.较好的,所述的新生抗原计算装置含有神经网络,包括共享神经网络和各型hla神 经网络;共享神经网络接收转录组测序数据分析模块呈递的rna表达水平结果,以及旁 侧序列模块呈递的旁侧序列结果,并将获得的rna表达水平结果和旁侧序列结果根据hla 分型、分别呈递给各型hla呈递可能性计算模块。
91.突变肽链获取单元将存储的hla信息根据hla分类分别呈递给各类hla神经网络;
92.各型hla呈递可能性计算模块分别接收相应类别的hla神经网络中的肽链序列和共 享神经网络呈递的旁侧序列结果和rna表达水平结果,计算获得递呈的可能性并呈递给 递呈的可能性存储模块。
93.较好的,所述的突变肽链获取单元和旁侧序列模块与质谱装置连接,接受并存储质 谱装置获得的肽链序列和旁侧序列结果。
94.较好的,所述的蛋白免疫沉淀装置和质谱装置由蛋白免疫沉淀和质谱联用装置代替。
95.较好的,所述的深度学习的神经网络由卷积神经网络代替。
96.较好的,所述的样本收集装置包括肿瘤样本收集装置或者正常样本收集装置。
97.较好的,所述的样本收集装置中盛放样本的容器为一次性用品。每次检测一个样品 后更换一次性的盛放容器,以减少反复检测的交叉污染。
98.较好的,所述的hla分型装置为流式细胞仪。
99.较好的,所述的系统还包括训练数据获取部分。训练数据获取部分包括公开数据采集 部分和实验数据采集部分。数据采集部分包括但不限于体细胞非同义突变分析、突变
肽链 和旁侧序列获取、hla分型分析单元、rna水平计算单元。
100.实验数据采集部分包括中国人群高频hla亚型细胞呈递组件、蛋白免疫沉淀装置、 质谱装置、转录组测序数据分析模块、肽链序列筛选单元、旁侧序列限定单元、hla分型 分析单元、rna表达水平计算单元。蛋白免疫沉淀装置和质谱装置对中国人群高频hla亚 型细胞进行检测,获得中国人群高频hla亚型细胞的肽链序列信息和旁侧序列信息,并 将中国人群高频hla亚型细胞的肽链序列信息和旁侧序列信息呈递给神经网络。hla分型 分析单元收集中国人高频hla亚型细胞和人hla保守序列,比对两者从而获得中国hla 分型结果。rna表达水平计算单元接受转录组测序数据、计算并存储中国人高频hla亚型 细胞的rna表达水平结果;公开数据采集部分存储源自公共资源的人类hla信息,包括 肽链序列获取单元、旁侧序列获取单元、hla分型分析单元、rna表达水平计算单元。
101.神经网络接收公开数据采集部分和实验数据采集部分呈递的肽链序列、旁侧序列、 hla分型或者rna表达水平数据。
102.本发明的系统可以用于肿瘤新生抗原的准确预测,提高癌症治疗性疫苗的有效性。
103.较好的,预测或者筛选新生抗原,能够从众多可以作为新生抗原的肽段中排出可能 性低的肽链,提高准确性。
104.本发明的新生抗原预测方案有以下三大优势:
105.(1)多维度模型,训练数据大,数据来源多样化:
106.从新生抗原的加工、亲和、递呈这三个维度考虑,多维度更全面地预测筛选抗肿瘤 新生抗原。ai算法用的训练数据大,且数据来源多样化,除了公开文献中的质谱数据, 我们也对临床患者样本进行了hla i类和hla ii类肽链的质谱数据的采集。不但能准确 预测hla i类和ii类新抗原,更能针对中国人群做出准确预测。本发明还构建了56株 中国人群占比较高的hla亚型细胞系,利用蛋白免疫沉淀和质谱联用的方式,获得对应 特定hla分型的质谱数据,能够更准确地预测出特定hla对应的新抗原。
107.(2)预测准确率高:
108.精度高,继而提高了新抗原疫苗的有效性,也降低了疫苗制备的成本。分别采用传 统预测方式和本发明的预测系统方式,预测小鼠llc肺癌细胞和小鼠ct26结直肠癌细胞 的新生抗原。与传统预测方式相比,本发明的系统的阳性预测率提升了2倍左右(请提 供具体实验步骤和对比结果)。
109.本发明的ai分析系统筛选的新抗原在癌症患者中有显著疗效。其中一例临床案例, 为肺癌晚期,放化疗标准治疗均无效。通过取组织和外周血样本进行高通量测序获得测 序数据后,导入本发明的新抗原ai分析系统,从555个非同义突变中筛选出了13个候 补新抗原。从2019年2月开始接受治疗,7次多肽疫苗注射后,8月达到部分缓解(pr), 总生存期(os)已超过14个月。从cd8指标和nk指标检测,可以表明该患者的自身免 疫反应被激活。
110.(3)算法提升空间大、易优化:
111.算法提升空间大,且优化方式简单。与传统的新抗原预测系统相比,本发明的新抗 原预测方案采用深度学习的算法,利用质谱所得的肽链序列、旁侧序列、rna表达、氨基 酸特性和hla分型等变量训练模型,最终得出的模型用于预测新生抗原。本发明系统的 优势在于,不需要手动调整参数的比重,深度学习将自动调整模型的参数,得出最优化 的模型。
而且,当加入的质谱数据量逐渐增加,预测的准确率也将逐步增加。本发明的 方法和系统预测获得的新生抗原为相关研究和应用提供了准确的数据基础。
附图说明
112.为了更清楚地说明本技术实施例中的技术方案,下面将对实施例中所需要使用的附 图作简单地介绍,显而易见地,下面描述中的附图仅仅是本技术的一些实施例,对于本 领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他 的附图。
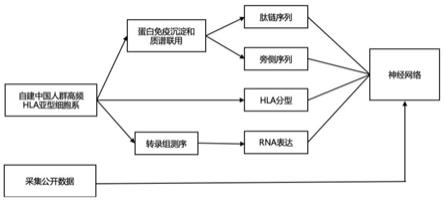
113.图1为训练数据获取途径示意图。
114.图2为深度学习神经网络架构图。
115.图3为新生抗原预测流程图。
具体实施方式
116.下面将对本技术实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施 例仅仅是本技术一部分实施例,而不是全部的实施例。基于本技术中的实施例,本领域 普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本技术保 护的范围。
117.实施例1
118.本发明提供了一种新生抗原预测系统,包括样本收集装置、转录组测序数据分析模 块、全外显子测序数据分析模块(包括体细胞非同义突变分析、突变肽链和旁侧序列获 取、hla分型分析单元)、新生抗原预测装置。
119.样本收集装置包括肿瘤细胞和正常细胞收集装置,将样本运输到转录组测序数据分 析模块和/或全外显子测序数据分析模块中,肿瘤细胞分别进行样本的转录组测序和全外 显子测序,正常细胞进行全外显子测序。全外显子测序数据分析模块接收并比对肿瘤细 胞和正常细胞的全外显子测序数据,计算并将体细胞非同义突变数据呈递给体细胞非同 义突变计算模块。体细胞非同义突变分析包括单核苷酸突变单元、插入突变单元、缺失 突变单元和移码突变单元。全外显子测序数据分析模块与hla分型分析单元连接,后者 将正常细胞的全外显子测序数据与人类参考基因组序列相比较,获得正常样本的hla分 型数据。转录组测序装置与转录组测序数据分析模块连接,将肿瘤细胞的转录组测序数 据呈递给转录组测序数据分析模块,获得rna表达水平数据。
120.蛋白免疫沉淀和质谱联用,获得细胞样本中可以和某一个特定hla分子结合的肽链 序列,选取8-11个氨基酸长度的肽链,并将氨基酸长度小于11的肽链填充到11个,并 截取其左右各5个氨基酸作为旁侧序列。肽链序列模块与体细胞非同义突变分析连接, 接收体细胞非同义突变分析呈递的体细胞非同义突变数据并筛选获得肽链序列。旁侧序 列模块与体细胞非同义突变分析连接,接收体细胞非同义突变分析呈递的体细胞非同义 突变数据并筛选获得旁侧序列。新生抗原预测装置与rna表达单元、肽链序列获取单元、 旁侧序列获取单元、hla分型分析单元连接,接收rna表达水平数据、肽链序列、旁侧 序列、hla分型数据并计算获得新生抗原递呈的可能性。
121.该系统还包括训练数据获取部分,训练数据获取部分包括公开数据采集部分和实
验数 据采集部分。
122.数据采集部分包括但不限于突变肽链和旁侧序列获取单元、hla分型分析单元、rna 表达水平计算单元。
123.实验数据采集部分包括中国人群高频hla亚型细胞呈递组件、蛋白免疫沉淀装置、质 谱装置、转录组测序数据分析模块、中国人群高频hla亚型细胞的肽链序列获取单元、旁 侧序列获取单元、hla分型分析单元、rna表达水平计算单元。蛋白免疫沉淀装置和质谱 装置对中国人群高频hla亚型细胞进行检测,获得中国人群高频hla亚型细胞的肽链序列 信息和旁侧序列信息,并将中国人群高频hla亚型细胞的肽链序列信息和旁侧序列信息呈 递给神经网络。hla分型模块收集中国人高频hla亚型细胞和人hla保守序列,比对两者 从而获得中国hla分型结果。rna表达水平计算单元接受转录组测序数据、计算并存储中 国人高频hla亚型细胞的rna表达水平结果;公开数据采集部分存储源自公共资源的人类 hla信息,包括但不限于突变肽链和旁侧序列获取单元、hla分型分析单元、rna表达水平 计算单元。神经网络接收公开数据采集部分和实验数据采集部分呈递的肽链序列、旁侧序 列、hla分型或者rna表达水平数据。
124.新生抗原预测装置含有神经网络,包括共享神经网络和各型hla神经网络;共享神 经网络接收rna表达水平计算单元呈递的rna表达水平结果,以及旁侧序列获取单元呈 递的旁侧序列结果,并将获得的rna表达水平结果和旁侧序列结果根据hla分型、分别 呈递给各型hla呈递可能性计算模块;肽链序列获取单元将存储的hla信息根据hla分 类分别呈递给各类hla神经网络;各型hla呈递可能性计算单元分别接收相应类别的hla 神经网络中的肽链序列和共享神经网络呈递的旁侧序列结果和rna表达水平结果,计算 获得递呈的可能性并呈递给递呈的可能性存储模块。肽链序列获取单元和旁侧序列获取 单元与质谱装置连接,接受并存储质谱装置获得的肽链序列和旁侧序列结果。
125.使用时,启动系统,将肿瘤细胞和源自同一个体的正常细胞分别放入样品收集装置, 系统按指令启动转录组测序数据分析模块和全外显子测序数据分析模块,获得肽链序列、 旁侧序列、hla分型和rna水平结果并传输给神经网络,计算肽链递呈的可能性,从而 预测效果较好的新生抗原。
126.实施例2
127.新生抗原预测系统神经网络的构建主要分为两步:
128.(1)训练数据获取
129.我们的训练数据获取途径如图1所示。我们构建了中国人群高频hla亚型细胞系。首 先设计针对hla-a、hla-b和hla-c的特定引物,pcr分别扩增b-lcl细胞(crl-2369 tm
)中的hla-a、hla-b和hla-c的基因片段,然后将这些基因片段分别亚克隆到逆转录病 毒载体中,最后用逆转录病毒感染lcl 721.221细胞系(人源hla i类缺失细胞系)( crl-1855
tm
)获得hla亚型细胞系。采用蛋白免疫沉淀和质谱联用,获得可以和某一个特定 hla分子结合的肽链序列,选取8-11个氨基酸长度的肽链,并将氨基酸长度小于11的肽 链填充到11个,并截取其左右各5个氨基酸作为旁侧序列。我们对质谱获得的肽链做阳 性标记,把在蛋白质公共数据库(swissprot)里的参考蛋白组中未在质谱数据中出现的 肽链做阴性标记,并将数据以8:1:1的比列分为训练数据、验证数据和测试数据。hla 亚型样本提取rna,并进行转录组测序,获得rna表达水平(tpm),从质谱肽链数据中排 除tpm小于等于0
熟悉本领域技术的技术人员在本技术公开的技术范围内,可轻易想到的变化或替换,都 应涵盖在本技术的保护范围之内。因此,本技术的保护范围应以所述权利要求的保护范 围为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1