肿瘤微环境的分类的制作方法
肿瘤微环境的分类
1.电子递交的序列表的引用
2.电子递交的序列表(名称:4488_003pc04_seqlisting_st25.txt;大小:17,402字节;以及创建日期:2020年10月30日)的内容通过引用以其整体并入本文。
技术领域
3.本公开涉及用于基于衍生自生物标记物基因表达数据的标志分数或预测模型对肿瘤微环境(tme)进行分类、用于鉴定具有特定tme的癌症患者的亚群以用特定疗法进行治疗以及用于用靶向疗法治疗具有特定tme的患者的方法。
背景技术:
4.癌症临床管理中的一个关键问题是癌症具有高度异质性。选择可以从治疗中获得最大益处的癌症患者的生物标记物通常依赖于免疫组织化学或药物靶(例如,受体)的表达、突变的遗传谱(例如,brca)或循环因子的水平。使用这种方法为少数几种药物开发了成功的诊断方法,并且通常用于癌细胞的靶向疗法,例如(曲妥珠单抗(trastuzumab))作为靶向过表达her2/neu受体的癌症的治疗。对特定疗法的个体癌症应答性的准确预测由于由于调控这种应答性的多种因素,诸如特定受体或其他细胞信号传导开关的存在或不存在而通常不可实现。这往往导致疗法失败或可能导致严重的过度治疗。
5.癌症的临床结果的预测通常通过对原发性肿瘤手术切除期间获得的组织样品进行组织病理学评估来实现。传统的肿瘤分期(ajcc/uicc-tnm分类)总结了关于肿瘤负荷(t)、引流和区域淋巴结中癌细胞的存在(n)以及转移证据(m)的数据。当前的分类提供有限的预后信息,并且不预测对疗法的应答。许多专利申请已描述了例如通过测量免疫学生物标记物,用于对患有实体癌的患者的存活时间进行预后的方法和/或用于评定患有实体癌的患者对抗肿瘤疗法的应答性的方法。参见例如国际申请公布wo2015007625、wo2014023706、wo2014009535、wo2013186374、wo2013107907、wo2013107900、wo2012095448、wo2012072750和wo2007045996,其中的全部通过引用以其整体并入本文。此外,抗癌剂的有效性可能基于患者的独特特征而变化。
6.因此,需要靶向治疗策略来鉴定更有可能对特定抗癌剂产生应答的患者,并且从而改善被诊断患有癌症的患者的临床结果。
技术实现要素:
7.本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme),也称为基质表型或基质亚型的方法,其包括:对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器,其中所述机器学习分类器将所述受试者鉴定为表现出(即,是生物标记物阳性的)或不表现出(即,是生物标记物阴性的)选自由以下组成的组中的tme分类:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合。
8.还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出(即,是生物标记物阳性的)或不表现出(即,是生物标记物阴性的)通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合。
9.本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
10.(i)在施用之前,通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器鉴定表现出(即,是生物标记物阳性的)或不表现出(即,是生物标记物阴性的)tme的受试者,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合;以及
11.(ii)向所述受试者施用tme类别特定疗法。
12.还提供了一种用于鉴定罹患适合用tme类别特定疗法治疗的癌症的人受试者的方法,所述方法包括对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器,其中选自由以下组成的组中的tme的存在(生物标记物阳性,即是生物标记物阳性的)或不存在(生物标记物阴性,即是生物标记物阴性的)指示可以施用tme类别特定疗法来治疗所述癌症:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合。
13.在一些方面,所述机器学习分类器是通过逻辑回归、随机森林、人工神经网络(ann)、支持向量机(svm)、xgboost(xgb)、glmnet、cforest、机器-学习的分类与回归树(cart)、treebag、k最近邻(knn)或其组合获得的模型。在一些方面,所述机器学习分类器是ann。在一些方面,所述ann是前馈型ann。在一些方面,所述ann是多层感知器。
14.在一些方面,所述ann包括输入层、隐藏层和输出层。在一些方面,所述输入层包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、84、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个节点(神经元)。在一些方面,所述输入层中的每个节点(神经元)对应于所述基因套组中的基因。在一些方面,所述基因套组选自表1和表2中(或图28a-g中公开的基因套组(基因集(geneset))中的任一个中)或表5中呈现的基因。
15.在一些方面,所述基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个选自表1的基因和1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60或61个选自表2的基因。在一些方面,所述基因套组是选自表5或图28a-g的基因套组。
16.在一些方面,所述样品包括瘤内组织。在一些方面,所述rna表达水平是转录rna表达水平。在一些方面,所述rna表达水平使用测序或测量rna的任何技术来确定。在一些方
面,所述测序是下一代测序(ngs)。在一些方面,所述ngs选自由以下组成的组:rna-seq、edgeseq、pcr、nanostring、全外显子组测序(wes)或其组合。在一些方面,所述rna表达水平使用荧光来确定。在一些方面,所述rna表达水平使用affymetrix微阵列或agilent微阵列来确定。在一些方面,所述rna表达水平经受分位数归一化。在一些方面,所述分位数归一化包括将输入rna水平值分箱成分位数。在一些方面,所述输入rna水平被分箱成100个分位数、150个分位数、200个分位数或更多。在一些方面,所述分位数归一化包括将所述rna表达水平分位数转换为正态输出分布函数。
17.在一些方面,用训练集训练所述ann,所述训练集包括从多个受试者获得的多个样品中的基因套组中每个基因的rna表达水平,其中每个样品被分配tme分类。在一些方面,分配到所述训练集中的每个样品的所述tme分类通过基于群体的分类器确定。在一些方面,所述基于群体的分类器包括通过测量所述训练集中的每个样品中的基因套组中每个基因的rna表达水平来确定标志1分数和标志2分数;其中用于计算标志1的基因是来自表1或图28a-28g或其组合的基因,并且用于计算标志2的基因是来自表2或图28a-28g或其组合的基因;并且其中
18.(i)如果所述标志1分数是负数并且所述标志2分数是正数,则被分配的所述tme分类是ia(即,所述受试者将被认为是ia生物标记物阳性的);
19.(ii)如果所述标志1分数是正数并且所述标志2分数是正数,则被分配的所述tme分类是is(即,所述受试者将被认为是is生物标记物阳性的);
20.(iii)如果所述标志1分数是负数并且所述标志2分数是负数,则被分配的所述tme分类是id(即,所述受试者将被认为是id生物标记物阳性的);并且(iv)如果所述标志1分数是正数并且所述标志2分数是负数,则被分配的所述tme分类是a(即,所述受试者将被认为是a生物标记物阳性的)。
21.在一些方面,标志1分数的计算包括:
22.(i)测量来自所述受试者的测试样品中的基因套组中的来自表1或图28a-28g或其组合的每个基因的表达水平;
23.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
24.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
25.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根;
26.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
27.在一些方面,标志2分数的计算包括:
28.(i)测量来自所述受试者的测试样品中的基因套组中的来自表2或图28a-28g或其组合的每个基因的表达水平;
29.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
30.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获
得的每个基因的标准偏差;以及
31.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根;
32.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
33.在一些方面,通过反向传播训练所述ann。在一些方面,所述隐藏层包括2个节点(神经元)。在一些方面,将sigmoid激活函数应用于所述隐藏层。在一些方面,所述sigmoid激活函数是双曲正切函数。在一些方面,所述输出层包括4个节点(神经元)。在一些方面,所述输出层中的所述4个输出节点(神经元)中的每一个对应于一种tme输出类别,其中所述4种tme输出类别是ia(免疫活性型)、is(免疫抑制型)、id(免疫沙漠型)和a(血管生成型)。在一些方面,本文公开的ann方法还包括将包括softmax函数的逻辑回归分类器应用于ann的输出,其中所述softmax函数为每种tme输出类别分配概率。在一些方面,所述softmax函数通过额外的神经网络层实现。在一些方面,所述额外的网络层插入在所述隐藏层与所述输出层之间。在一些方面,所述额外的网络层具有与所述输出层相同数量的节点(神经元)。
34.本公开还提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)的ann,其中所述ann使用从来自所述受试者的肿瘤组织样品的基因套组获得的rna表达水平作为输入将所述受试者鉴定为表现出(即,是生物标记物阳性的)或不表现出(即,是生物标记物阴性的)选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,并且其中tme的存在或不存在指示所述受试者可以用tme类别特定疗法进行有效治疗,所述疗法可以是药物、药物组合或具有解决病理学的作用机制的临床疗法。
35.在一些方面,所述ann是前馈型ann。在一些方面,所述ann是多层感知器。在一些方面,所述ann包括输入层、隐藏层和输出层。在一些方面,所述输入层包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、84、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个节点(神经元)。在一些方面,所述输入层中的每个节点(神经元)对应于所述基因套组中的基因。在一些方面,所述基因套组选自表1和表2中(或图28a-g中公开的基因套组(基因集)中的任一个中)或表5中呈现的基因。在一些方面,所述基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个选自表1的基因和1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60或61个选自表2的基因。在一些方面,所述基因套组是选自表5或图28a-g的基因套组。在一些方面,所述样品包括瘤内组织。在一些方面,所述rna表达水平是转录rna表达水平。在一些方面,所述rna表达水平使用测序或测量rna的任何技术来确定。在一些方面,所述测序是下一代测序(ngs)。在一些方面,所述ngs选自由以下组成的组:rna-seq、edgeseq、pcr、nanostring、全外显子组测序(wes)或其组合。
36.在一些方面,所述rna表达水平使用荧光来确定。在一些方面,所述rna表达水平使用affymetrix微阵列或agilent微阵列来确定。在一些方面,rna表达水平经受分位数归一化。在一些方面,所述分位数归一化包括将输入rna水平值分箱成分位数。在一些方面,所述输入rna水平被分箱成100个分位数、150个分位数、200个分位数或更多。在一些方面,所述分位数归一化包括将所述rna表达水平分位数转换为正态输出分布函数。在一些方面,用训练集训练所述ann,所述训练集包括从多个受试者获得的多个样品中的基因套组中每个基因的rna表达水平,其中每个样品被分配tme分类。在一些方面,分配到所述训练集中的每个样品的所述tme分类通过基于群体的分类器确定。
37.在一些方面,所述基于群体的分类器包括通过测量所述训练集中的每个样品中的基因套组中每个基因的rna表达水平来确定标志1分数和标志2分数;其中用于计算标志1的基因是来自表1、图28a-28g或其组合的基因,并且用于计算标志2的基因是来自表2、图28a-28g或其组合的基因;并且其中
38.(i)如果所述标志1分数是负数并且所述标志2分数是正数,则被分配的所述tme分类是ia(即,所述受试者将被认为是ia生物标记物阳性的);
39.(ii)如果所述标志1分数是正数并且所述标志2分数是正数,则被分配的所述tme分类是is(即,所述受试者将被认为是is生物标记物阳性的);
40.(iii)如果所述标志1分数是负数并且所述标志2分数是负数,则被分配的所述tme分类是id(即,所述受试者将被认为是id生物标记物阳性的);并且(iv)如果所述标志1分数是正数并且所述标志2分数是负数,则被分配的所述tme分类是a(即,所述受试者将被认为是a生物标记物阳性的)。
41.在一些方面,标志1分数的计算包括:
42.(i)测量来自所述受试者的测试样品中的基因套组中的来自表1、图28a-28g或其组合的每个基因的表达水平;
43.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
44.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
45.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根;
46.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
47.在一些方面,标志2分数的计算包括:
48.(i)测量来自所述受试者的测试样品中的基因套组中的来自表2、图28a-28g或其组合的每个基因的表达水平;
49.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
50.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
51.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数
的平方根;
52.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。在一些方面,通过反向传播训练所述ann。在一些方面,所述隐藏层包括2、3、4或5个节点(神经元)。在一些方面,将sigmoid激活函数应用于所述隐藏层。在一些方面,所述sigmoid激活函数是双曲正切函数。在一些方面,所述输出层包括4个节点(神经元)。
53.在一些方面,所述输出层中的所述4个输出节点中的每一个对应于一种tme输出类别,其中所述4种tme输出类别是ia(免疫活性型)、is(免疫抑制型)、id(免疫沙漠型)和a(血管生成型)。在一些方面,所述ann还包括将包括softmax函数的逻辑回归分类器应用于ann的输出,其中所述softmax函数为每种tme输出类别分配概率。在一些方面,所述softmax函数通过额外的神经网络层实现。在一些方面,所述额外的网络层插入在所述隐藏层与所述输出层之间。在一些方面,所述额外的网络层具有与所述输出层相同数量的节点。
54.在本公开的方法和ann的一些方面,所述tme类别特定疗法是ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合。在一些方面,tme类别特定疗法的分配是基于特定基质表型的存在,例如,如果受试者呈现ia基质表型(并且因此所述受试者是ia生物标记物阳性的),则将施用ia类别tme疗法。在一些方面,tme类别特定疗法的分配是基于特定基质表型的不存在,例如,如果受试者不呈现ia基质表型(并且因此所述受试者是ia生物标记物阴性的),则将不施用ia类别tme疗法。在一些方面,tme类别特定疗法的分配是基于两种或更多种特定基质表型的存在和/或不存在,例如,如果所述受试者呈现a和is基质表型(并且因此所述受试者是a和is生物标记物阳性的)并且不呈现id和ia基质表型(并且因此所述受试者是id和ia生物标记物阴性的),则将施用特定tme疗法。
55.在一些方面,所述ia类别tme疗法包括检查点调节剂疗法。在一些方面,所述检查点调节剂疗法包括施用刺激性免疫检查点分子的激活剂。在一些方面,所述刺激性免疫检查点分子的激活剂是针对gitr、ox-40、icos、4-1bb或其组合的抗体分子。在一些方面,所述检查点调节剂疗法包括施用rorγ激动剂。在一些方面,所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是针对单独pd-1(例如,信迪利单抗(sintilimab)、替雷利珠单抗(tislelizumab)、派姆单抗(pembrolizumab)或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体,或与以下组合:tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂或cd86激动剂。在一些方面,所述抗pd-1抗体包括纳武单抗(nivolumab)、派姆单抗、西米普利单抗(cemiplimab)、pdr001、cbt-501、cx-188、tsr-042、信迪利单抗、替雷利珠单抗或其抗原结合部分。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042结合相同的表位。在
一些方面,所述抗pd-l1抗体包括阿维单抗(avelumab)、阿替利珠单抗(atezolizumab)、德瓦鲁单抗(durvalumab)、cx-072、ly3300054或其抗原结合部分。在一些方面,所述抗pd-l1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)与阿维单抗、阿替利珠单抗或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。在一些方面,所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;或(iii)其组合。
56.在一些方面,所述is类别tme疗法包括施用(1)检查点调节剂疗法和抗免疫抑制疗法,和/或(2)抗血管生成疗法。在一些方面,所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是针对pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体。在一些方面,所述抗pd-1抗体包括纳武单抗(nivolumab)、派姆单抗、西米普利单抗(cemiplimab)、pdr001、cbt-501、cx-188、tsr-042、信迪利单抗、替雷利珠单抗或其抗原结合部分。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、信迪利单抗、替雷利珠单抗、cx-188或tsr-042交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042结合相同的表位。在一些方面,所述抗pd-l1抗体包括阿维单抗、阿替利珠单抗、cx-072、ly3300054、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。在一些方面,所述抗ctla-4抗体包括伊匹单抗(ipilimumab)或双特异性抗体xmab20717(抗pd-1/抗ctla-4)或其抗原结合部分。在一些方面,所述抗ctla-4与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)结合相同的ctla-4表位。在一些方面,所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗和tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;(iii)抗ctla-4抗体,其是伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4),或(iv)其组合。在一些方面,所述抗血管生成疗法包括施用选自由以下组成的组中的抗vegf抗体:伐利苏单抗(varisacumab)、贝伐珠单抗(bevacizumab)、那赛昔珠单抗(navicixizumab,抗dll4/抗vegf双特异性)及其组合。
57.在一些方面,所述抗血管生成疗法包括施用抗vegf抗体。在一些方面,所述抗vegf抗体是抗vegf双特异性抗体。在一些方面,所述抗vegf双特异性抗体是抗dll4/抗vegf双特异性抗体。在一些方面,所述抗dll4/抗vegf双特异性抗体包括那赛昔珠单抗。在一些方面,所述抗血管生成疗法包括施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2抗体。在一些方面,所述抗vegfr2抗体包括雷莫卢单抗(ramucirumab)。在一些方面,所述抗血
管生成疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。
58.在一些方面,所述抗免疫抑制疗法包括施用抗ps抗体、抗ps靶向抗体、结合β2-糖蛋白1的抗体、pi3kγ的抑制剂、腺苷通路抑制剂、ido的抑制剂、tim的抑制剂、lag3的抑制剂、tgf-β的抑制剂、cd47抑制剂或其组合。在一些方面,所述抗ps靶向抗体是巴维妥昔单抗(bavituximab)或结合β2-糖蛋白1的抗体。在一些方面,所述pi3kγ抑制剂是ly3023414(samotolisib)或ipi-549。在一些方面,所述腺苷通路抑制剂是ab-928。在一些方面,所述tgfβ抑制剂是ly2157299(galunisertib),或者所述tgfβr1抑制剂是ly3200882。在一些方面,所述cd47抑制剂是莫洛利单抗(magrolimab,5f9)。在一些方面,所述cd47抑制剂靶向sirpα。
59.在一些方面,所述免疫抑制疗法包括施用tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂、cd86激动剂或其组合。
60.在一些方面,所述id类别tme疗法包括在施用引发免疫应答的疗法的同时或之后施用检查点调节剂疗法。在一些方面,所述引发免疫应答的疗法是疫苗、car-t或新表位疫苗。在一些方面,所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是针对pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体。在一些方面,所述抗pd-1抗体包括纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042或其抗原结合部分。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042结合相同的表位。在一些方面,所述抗pd-l1抗体包括阿维单抗、阿替利珠单抗、cx-072、ly3300054、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。在一些方面,所述抗ctla-4抗体包括伊匹单抗(ipilimumab)或双特异性抗体xmab20717(抗pd-1/抗ctla-4)或其抗原结合部分。在一些方面,所述抗ctla-4与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)结合相同的ctla-4表位。在一些方面,所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗和tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;(iv)抗ctla-4抗体,其是伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4),或(iii)其组合。
61.在一些方面,所述a类别tme疗法包括vegf靶向疗法和其他抗血管生成剂、血管生成素1(ang1)的抑制剂、血管生成素2(ang2)的抑制剂、dll4的抑制剂、抗vegf和抗dll4的双特异性抑制剂、tki抑制剂、抗fgf抗体、抗fgfr1抗体、抗fgfr2抗体、抑制fgfr1的小分子、抑制fgfr2的小分子、抗plgf抗体、针对plgf受体的小分子、针对plgf受体的抗体、抗vegfb抗体、抗vegfc抗体、抗vegfd抗体、针对vegf/plgf捕获分子的抗体诸如阿柏西普(aflibercept)或ziv-阿柏西普、抗dll4抗体或抗notch疗法,诸如γ-分泌酶的抑制剂。在一些方面,所述tki抑制剂选自由以下组成的组:卡博替尼(cabozantinib)、凡德他尼(vandetanib)、替沃扎尼(tivozanib)、阿西替尼(axitinib)、乐伐替尼(lenvatinib)、索拉非尼(sorafenib)、瑞戈非尼(regorafenib)、舒尼替尼(sunitinib)、呋喹替尼(fruquitinib)、帕唑帕尼(pazopanib)及其任何组合。在一些方面,所述tki抑制剂是呋喹替尼。在一些方面,所述vegf靶向疗法包括施用抗vegf抗体或其抗原结合部分。在一些方面,所述抗vegf抗体包括伐利苏单抗、贝伐珠单抗或其抗原结合部分。在一些方面,所述抗vegf抗体与伐利苏单抗或贝伐珠单抗交叉竞争与人vegf a的结合。在一些方面,所述抗vegf抗体与伐利苏单抗或贝伐珠单抗结合相同的表位。在一些方面,所述vegf靶向疗法包括施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2抗体。在一些方面,所述抗vegfr2抗体包括雷莫卢单抗或其抗原结合部分。
62.在一些方面,所述a类别tme疗法包括施用血管生成素/tie2靶向疗法。在一些方面,所述血管生成素/tie2靶向疗法包括施用内皮糖蛋白和/或血管生成素。在一些方面,所述a类别tme疗法包括施用dll4靶向疗法。在一些方面,所述dll4靶向疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。
63.在一些方面,本文公开的方法还包括:
64.(a)施用化学疗法;
65.(b)进行手术;
66.(c)施用放射疗法;或
67.(d)其任何组合。
68.在一些方面,所述癌症是肿瘤。在一些方面,所述肿瘤是癌。在一些方面,所述肿瘤选自由以下组成的组:胃癌、结直肠癌、肝癌(肝细胞癌,hcc)、卵巢癌、乳腺癌、nsclc、膀胱癌、肺癌、胰腺癌、头颈癌、淋巴瘤、子宫癌、肾脏癌或肾癌、胆管癌、肛门癌、前列腺癌、睾丸癌、尿道癌、阴茎癌、胸癌、直肠癌、脑癌(胶质瘤和胶质母细胞瘤)、子宫颈腮腺癌、食管癌、胃食管癌、喉癌、甲状腺癌、腺癌、神经母细胞瘤、黑色素瘤和默克尔细胞癌。
69.在一些方面,所述癌症是复发性的。在一些方面,所述癌症是难治性的。在一些方面,在至少一种包括施用至少一种抗癌剂的先前疗法后,所述癌症是难治性的。在一些方面,所述癌症是转移性的。在一些方面,所述施用有效地治疗所述癌症。在一些方面,所述施用减少癌症负荷。在一些方面,与所述施用之前的癌症负荷相比,癌症负荷减少至少约10%、至少约20%、至少约30%、至少约40%或约50%。在一些方面,在所述初始施用之后,所述受试者表现出至少约1个月、至少约2个月、至少约3个月、至少约4个月、至少约5个月、至少约6个月、至少约7个月、至少约8个月、至少约9个月、至少约10个月、至少约11个月、至少约一年、至少约18个月、至少约两年、至少约三年、至少约四年或至少约五年的无进展存活。在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4
个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的稳定疾病。
70.在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的部分应答。在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的完全应答。
71.在一些方面,与未表现出tme的受试者的无进展存活概率相比,所述施用使无进展存活概率提高至少约10%、至少约20%、至少约30%、至少约40%、至少约50%、至少约60%、至少约70%、至少约80%、至少约90%、至少约100%、至少约110%、至少约120%、至少约130%、至少约140%或至少约150%。在一些方面,与未表现出tme的受试者的总体存活概率相比,所述施用使总体存活概率提高至少约25%、至少约50%、至少约75%、至少约100%、至少约125%、至少约150%、至少约175%、至少约200%、至少约225%、至少约250%、至少约275%、至少约300%、至少约325%、至少约350%或至少约375%。
72.本公开还提供了一种用于使用包括本文公开的ann的机器学习分类器确定有需要的受试者中的肿瘤的肿瘤微环境的基因套组,其至少包括来自表1的血管生成生物标记物基因和来自表2的免疫生物标记物基因,其中所述肿瘤微环境用于(i)鉴定适合抗癌疗法的受试者;(ii)确定正在经历抗癌疗法的受试者的预后;(iii)启动、暂停或修改抗癌疗法的施用;或(iv)其组合。
73.还提供了一种用于鉴定罹患适合用抗癌疗法治疗的癌症的人受试者的非基于群体的分类器,其包括本文公开的ann,其中所述机器学习分类器将所述受试者鉴定为表现出选自ia、is、id、a类别tme或其组合的tme,其中(i)如果所述tme是ia或主要是ia,则所述疗法是ia类别tme疗法;(ii)如果所述tme是is或主要是is,则所述疗法是is类别tme疗法;(iii)如果所述tme是id或主要是id,则所述疗法是id类别tme疗法;或者(iv)如果所述tme是a或主要是a,则所述疗法是a类别tme疗法。在一些方面,受试者可以表现出多于一种tme,例如,所述受试者对于ia和is、或ia和id、或ia和a等可以是生物标记物阳性的。对于多于一种基质表型是生物标记物阳性和/或生物标记物阴性的受试者可以接受一种或多种tme类别特定疗法。
74.还提供了一种用于治疗有需要的人受试者中的癌症的抗癌疗法,其中根据包括本文公开的ann的机器学习分类器,所述受试者被鉴定为表现出选自ia、is、id或a类别tme或其组合的tme,其中(i)如果所述tme是ia或主要是ia,则所述疗法是ia类别tme疗法;(ii)如果所述tme是is或主要是is,则所述疗法是is类别tme疗法;(iii)如果所述tme是id或主要是id,则所述疗法是id类别tme疗法;或者(iv)如果所述tme是a或主要是a,则所述疗法是a类别tme疗法。在一些方面,受试者可以表现出多于一种tme,例如,所述受试者对于ia和is、或ia和id、或ia和a等可以是生物标记物阳性的。对于多于一种基质表型是生物标记物阳性和/或生物标记物阴性的受试者可以接受一种或多种tme类别特定疗法。
75.还提供了一种为有需要的受试者中的癌症分配tme类别的方法,所述方法包括:(i)通过用训练集训练机器学习方法来生成机器学习模型,所述训练集包括从多个受试者
获得的多个样品中的基因套组中每个基因的rna表达水平,其中每个样品被分配tme分类;和(ii)使用所述机器学习模型分配所述受试者中的所述癌症的tme,其中所述机器学习模型的输入包括从所述受试者获得的测试样品中的所述基因套组中每个基因的rna表达水平。
76.还提供了一种为有需要的受试者中的癌症分配tme类别的方法,所述方法:包括通过用训练集训练机器学习方法来生成机器学习模型,所述训练集包括从多个受试者获得的多个样品中的基因套组中每个基因的rna表达水平,其中每个样品被分配tme分类;其中使用从所述受试者获得的测试样品中的所述基因套组中每个基因的rna表达水平作为输入,所述机器学习模型为所述受试者中的所述癌症分配tme类别。
77.本公开还提供了一种为有需要的受试者中的癌症分配tme类别的方法,所述方法包括:使用机器学习模型来预测所述受试者中的所述癌症的tme,其中通过用训练集训练机器学习方法来生成所述机器学习模型,所述训练集包括从多个受试者获得的多个样品中的基因套组中每个基因的rna表达水平,其中每个样品被分配tme分类。
78.在本文公开的方法的一些方面,机器学习模型通过如本文公开制备的ann生成。在一些方面,分配到所述训练集中的每个样品的所述tme分类通过基于群体的分类器确定。在一些方面,所述基于群体的分类器包括通过测量所述训练集中的每个样品中的基因套组中每个基因的rna表达水平来确定标志1分数和标志2分数;其中用于计算标志1的基因是来自表1、图28a-28g或其组合的基因,并且用于计算标志2的基因是来自表2、图28a-28g或其组合的基因;并且其中
79.(i)如果所述标志1分数是负数并且所述标志2分数是正数,则被分配的所述tme分类是ia(即,所述受试者将被认为是ia生物标记物阳性的);
80.(ii)如果所述标志1分数是正数并且所述标志2分数是正数,则被分配的所述tme分类是is(即,所述受试者将被认为是is生物标记物阳性的);
81.(iii)如果所述标志1分数是负数并且所述标志2分数是负数,则被分配的所述tme分类是id(即,所述受试者将被认为是id生物标记物阳性的);并且(iv)如果所述标志1分数是正数并且所述标志2分数是负数,则被分配的所述tme分类是a(即,所述受试者将被认为是a生物标记物阳性的)。
82.在一些方面,标志1分数的计算包括:
83.(i)测量来自所述受试者的测试样品中的基因套组中的来自表1或其子集的每个基因、或来自图28a-28g的基因的子集的表达水平;
84.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
85.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
86.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根;
87.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
88.在一些方面,标志2分数的计算包括:
89.(i)测量来自所述受试者的测试样品中的基因套组中的来自表2或其子集的每个基因、或来自图28a-28g的基因的子集的表达水平;
90.(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
91.(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
92.(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根;
93.其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
94.在一些方面,所述机器学习模型包括应用于所述的输出的包括softmax函数的逻辑回归分类器,其中所述softmax函数为每种tme输出类别分配概率。
95.在一些方面,所述方法在包括至少一个处理器和至少一个存储器的计算机系统中实现,所述至少一个存储器包括由所述至少一个处理器执行以使所述至少一个处理器实现所述机器学习模型的指令。在一些方面,所述方法还包括(i)将所述机器学习模型输入到所述计算机系统的所述存储器中;(ii)将对应于所述受试者的基因套组输入数据输入到所述计算机系统的所述存储器中,其中所述输入数据包括rna表达水平;(iii)执行所述机器学习模型;或(v)其任何组合。
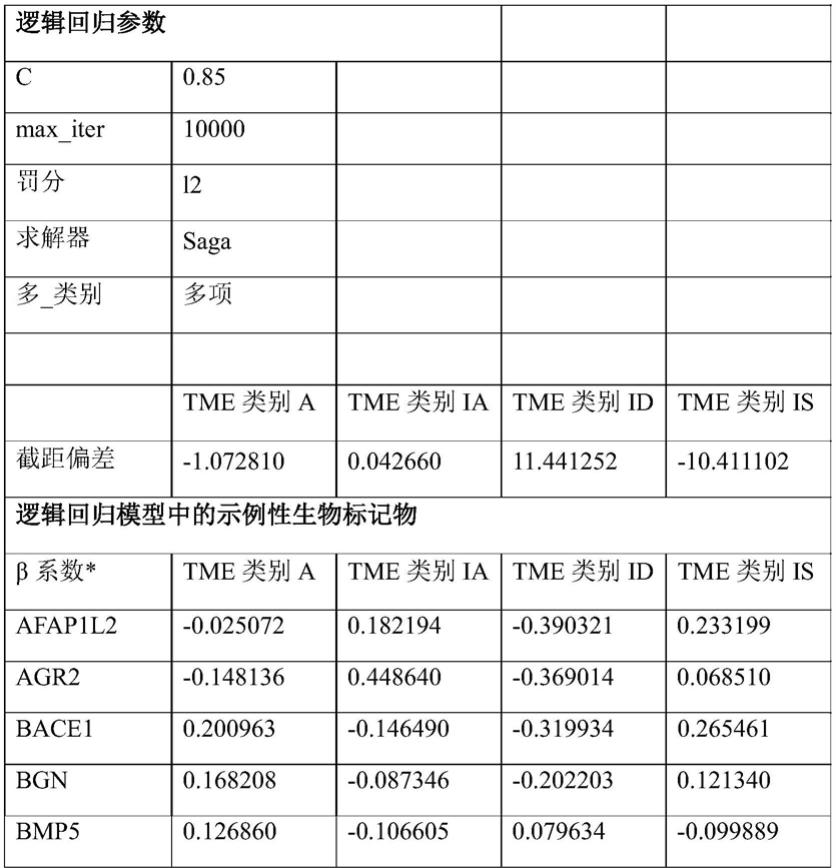
96.在一些方面,所述逻辑回归分类器的概率覆盖在所述ann模型的节点的激活分数的潜在空间图上。在一些方面,所述逻辑回归分类器在所述潜在空间上训练。在一些方面,针对pfs(无进展存活)优化所述逻辑回归分类器。在一些方面,针对以下优化所述逻辑回归分类器:bor(最佳客观应答)、orr(总体应答率)、mss/msi-高(微卫星稳定/微卫星不稳定性-高)状态、pd-1/pd-l1状态、pfs(无进展存活)、nlr(嗜中性粒细胞白细胞比率)、肿瘤突变负荷(tmb)或其任何组合。
附图说明
97.图1示出了分类之前三个数据集的归一化。
98.图2是在将298个患者分类成四种基质亚型(即,基质表型)之后来自acrg数据集的kaplan-meier图的风险曲线比较。
99.图3是在将388个患者分类成四种基质亚型(即,基质表型)之后来自tcga数据集的kaplan-meier图的风险曲线比较。
100.图4是在将192个患者分类成四种基质亚型(即,基质表型)之后来自singapore数据集的kaplan-meier图的风险曲线比较。
101.图5是在分类成四种基质亚型(即,基质表型)之后来自组合的三个数据集(878个患者)的kaplan-meier图的风险曲线比较。
102.图6a和图6b示出了在acrg群组中表示为盒式图的代表性基因本体标志。图6a示出了在acrg数据中作为四种基质亚型(即,基质表型)的函数的来自treg标志的表达水平的值的中值和范围的盒式图。图6b示出了在acrg数据中作为四种基质亚型(即,基质表型)的函数的炎性应答标志的表达水平的值的中值和范围的盒式图。
103.图7a和图7b示出了acrg群组中反应单个图的标题的生物学的代表性基因本体标志。图7a示出了与内皮细胞标志激活相关的标志1激活。图7b示出了与炎性和免疫细胞标志激活相关的标志2激活。
104.图8a和图8b示出了tcga数据集中反应单个图的标题的生物学的代表性基因本体标志。图8a示出了与内皮细胞标志激活相关的标志1激活。图8b示出了与炎性和免疫细胞标志激活相关的标志2激活。
105.图9a和图9b示出了singapore群组中反应单个图的标题的生物学的代表性基因本体标志。图9a示出了与内皮细胞标志激活相关的标志1激活。图9b示出了与炎性和免疫细胞标志激活相关的标志2激活。
106.图10是示出了基于本文公开的分类器的应用的肿瘤微环境(tme)分配以及分配到每种tme类别的治疗类别的图表。
107.图11描绘了逻辑回归模型中使用的逻辑函数。
108.图12a是示例性小决策树。
109.图12b示出了可以通过对来自单个树的预测进行平均来做出对新样品的预测。
110.图13示出了来自随机森林分类器的参数。
111.图14示出了包括多个样品的人工神经网络(ann)训练集的一部分,每一个对应于受试者(a列)、根据本公开的基于群体的分类器分配的受试者癌症的tme类别(b列)以及对应于所选基因套组中的不同基因的rna表达水平(c、d、e列等)。
112.图15示出了在本公开中用作非基于群体的分类器的ann的简化视图。ann包括输入层,其输入对应于基因套组(例如,124基因套组、105基因套组、98基因套组或可替代地87基因套组)中的每个基因;隐藏层,其包括两个神经元(或可替代地3、4或5个神经元);以及输出层,其对应于tme类别分配(即,基质表型分配)。
113.图16是示出了可以用于开发根据本公开的非基于群体的分类器的替代ann架构的示意图。
114.图17示出了对应于基因1至n的mrna水平(x)的ann输入被馈送到隐藏层神经元,并且偏差(b)被应用于隐藏层神经元。神经元的输入通过函数(f)整合,所述函数结合偏差和根据其相应的权重(w1…
wn)归一化的mrna表达水平(x1…
xn)。
115.图18示出了可以应用于隐藏层中的神经元的不同激活函数。
116.图19示出了人工神经元网络(ann)模型架构。“输入层”是来自单个样品的表达式xi、i∈g的向量。“隐藏层”包括两个神经元,每个神经元以基因表达作为输入。“输出层”包括四个神经元,每个神经元以两个隐藏神经元的激活作为输入,将它们用tanh(双曲正切)激活函数转换为加权和以产生(y),接着通过逻辑回归分类器(例如,softmax函数)(zi)产生四种表型类别(ia、id、a、is)的概率。ann的替代方面可以包括例如五个神经元而不是两个神经元。
117.图20示出了用派姆单抗单一疗法治疗的具有已知生物标记物状态和已知结果的胃癌患者群体的kaplan-meier存活曲线。
118.图21a示出了应用机器学习(ann)来优化定义作为应答者相对于非应答者的患者的截止值以及用于患者选择的两个可能选项。
119.图21b示出了除了使用不同于笛卡尔x=0、y=0阈值的线性阈值来定义作为应答
者相对于非应答者的患者,如图21a中例示,可以使用非线性阈值来定义患者群体并且使用此类非线性阈值来进行患者选择。
120.图22示出了具有已知生物标记物状态和已知结果的navi 1b生殖系统癌症患者的kaplan-meier存活曲线。
121.图23示出了以百分比表示的实施例12的派姆单抗患者数据的tme类别的概率轮廓,其覆盖在ann模型的激活分数1和2的潜在空间图(x和y轴)上。左上象限对应于a tme基质表型,左下象限对应于id tme基质表型,右下象限对应于ia tme基质表型,并且右上象限对应于is tme基质表型。患者最佳客观应答结果通过以下表示:进展性疾病(pd)-圆圈;稳定疾病(sd)-三角形;部分应答(pr)-方形;和完全应答(cr)
‑“
x”。实心形状代表pd-l1状态≥1的患者,空心形状代表pd-l1《1。在实施例12的73个患者中,四个患者缺少pd-l1状态,并且因此从图中省略。
122.图24示出了实施例12的派姆单抗患者数据的tme类别的基于大于5个月的无进展存活(pfs)通过逻辑回归分类器通知的生物标记物阳性的概率,其覆盖在ann模型的激活分数1和2的潜在空间图(x和y轴)上。使用pfs》5作为阳性类别,使用小于4的嗜中性粒细胞白细胞比率(nlr《4)基于样品训练分类器。左上象限对应于a tme基质表型,左下象限对应于id tme基质表型,右下象限对应于ia tme基质表型,并且右上象限对应于is tme基质表型。患者最佳客观应答结果通过以下表示:进展性疾病(pd)-圆圈;稳定疾病(sd)-三角形;部分应答(pr)-方形;和完全应答(cr)
‑“
x”。实心形状代表pd-l1状态≥1的患者,空心形状代表pd-l1《1。在实施例12的73个患者中,四个患者缺少pd-l1状态,并且因此从图中省略。
123.图25示出了实施例12的派姆单抗患者数据的tme类别的基于最佳客观应答通过逻辑回归分类器通知的生物标记物阳性的概率,其覆盖在ann模型的激活分数1和2的潜在空间图(x和y轴)上。使用完全应答者和部分应答者(cr+pr)作为阳性类别,使用小于4的嗜中性粒细胞白细胞比率(nlr《4)基于样品训练分类器。左上象限对应于a tme基质表型,左下象限对应于id tme基质表型,右下象限对应于ia tme基质表型,并且右上象限对应于is tme基质表型。患者最佳客观应答结果通过以下表示:进展性疾病(pd)-圆圈;稳定疾病(sd)-三角形;部分应答(pr)-方形;和完全应答(cr)
‑“
x”。实心形状代表pd-l1状态≥1的患者,空心形状代表pd-l1《1。在实施例12的73个患者中,四个患者缺少pd-l1状态,并且因此从图中省略。
124.图26示出了对于所有患者(n=38),实施例7的巴维妥昔单抗和派姆单抗组合疗法临床数据的tme类别的概率,其覆盖在ann模型的激活分数1和2的潜在空间图(x和y轴)上。左上象限对应于a tme基质表型,左下象限对应于id tme基质表型,右下象限对应于ia tme基质表型,并且右上象限对应于is tme基质表型。患者最佳客观应答结果通过以下表示:进展性疾病(pd)-圆圈;稳定疾病(sd)-三角形;部分应答(pr)-方形;和完全应答(cr)
‑“
x”。实心形状代表确认应答的患者,空心形状代表未确认应答。
125.图27示出了每个来自结直肠癌(左,n=370)、胃癌(中心,n=337)和卵巢癌(右,n=392)的组织样品的神经网络激活分数(实心圆,激活分数1(节点1);空心方形,激活分数2(节点2))和预测的tme类别(ann表型调用)。对于不同的疾病组,四种tme类别之间的样品分布是相似的。
126.图28a示出了基因集1至44中124个基因的存在(空心单元)或不存在(实心单元)。
127.图28b示出了基因集45至88中124个基因的存在(空心单元)或不存在(实心单元)。
128.图28c示出了基因集89至132中124个基因的存在(空心单元)或不存在(实心单元)。
129.图28d示出了基因集133至177中124个基因的存在(空心单元)或不存在(实心单元)。
130.图28e示出了基因集178至222中124个基因的存在(空心单元)或不存在(实心单元)。
131.图28f示出了基因集223至267中124个基因的存在(空心单元)或不存在(实心单元)。
132.图28g示出了基因集268至282中124个基因的存在(空心单元)或不存在(实心单元)。
133.图29a是ann模型的第一节点中的基因权重的说明性示意图,其呈现为30个基因权重的样品(x轴)的直方图。开放条,标志1的基因子集,闭合条,标志2基因的子集。权重在y轴上给出。
134.图29b是ann模型的第二节点中的基因权重的说明性示意图,其呈现为30个基因权重的样品(x轴)的直方图。开放条,标志1的基因子集,闭合条,标志2基因的子集。权重在y轴上给出。
具体实施方式
135.本公开提供了根据群体和非群体肿瘤微环境(tme)分类方法对患者和癌症进行分类的方法。本文公开的群体方法(即,基于群体的分类器)不仅可以用作独立式分类器,还可以用作预处理用作训练集以基于机器学习技术的应用生成非群体模型(即,非基于群体的分类器),例如基于人工神经网络(ann)的预测模型的基因表达数据的手段。
136.如本文所用,术语“非基于群体的”方法或分类器可与术语机器学习(ml)方法或ml分类器,例如本公开的ann分类器互换。如本文所用,术语“基于群体的”方法或分类器可与术语z分数方法或z分数分类器互换。
137.在一些方面,可以代表一个或多个生物标志(即,标志1、标志2、标志3、...标志n)的基因集根据本文公开的方法用于计算特征1...n的z分数。这包括群体模型,其可以用于揭示每个标志所代表的主要生物学以及由这些标志的矩阵定义的tme表型。在一些方面,可以例如使用由标志导出的基因集作为特征,并且使用历史患者数据集(例如,acrg(亚洲癌症研究组)患者数据集)作为表达式来训练机器学习模型(例如,ann)。
138.机器学习模型(例如,ann)学习将个体患者分类为特定tme表型的(潜在)基因表达模式。机器学习模型(例如,ann)有效地将高维数据(输入基因集中所有基因的基因表达)压缩到较低维(潜在)空间,例如本文公开的ann中的两个隐藏神经元中。然后机器学习模型(例如,ann)输出表型类别,例如四种tme表型类别,它们本身可以用于以药物特异性方式单独(全部或部分)或彼此组合(再次,全部或部分)定义生物标记物阳性。可替代地,可以在潜在空间上训练二级模型(例如,逻辑回归分类器),以便不学习tme表型,而是基于患者结果标签直接学习生物标记物阳性相对于生物标记物阴性决策边界。
139.在一些方面,可以针对以下优化应用于本公开的方法的ann分类的二级模型(例
如,逻辑回归分类器):bor(最佳客观应答)、orr(总体应答率)、mss/msi-高(微卫星稳定/微卫星不稳定性-高)状态、pd-1/pd-l1状态、pfs(无进展存活)、nlr(嗜中性粒细胞白细胞比率)、肿瘤突变负荷(tmb)或其任何组合。
140.因此,在一些方面,本公开提供了基于多个标志,即与特定基因套组(例如,表3和表4中的那些)中的基因(例如,表1和表2中的那些)的表达相关的总体分数,诸如本文公开的标志1和标志2的整合的群体分类器。这些标志分数允许根据tme对患者和癌症进行分层,并且然后根据特定tme的存在或不存在来指导治疗决策。
141.在其他方面,本公开提供了基于机器学习技术的应用的非群体分类器,例如逻辑回归、随机森林或人工神经网络(ann)。本文公开的ann分类器是基于例如使用根据本文公开的基于群体的分类器预处理的数据集来训练神经网络。
142.本文公开的非基于群体的分类器(ann分类器)相对于本文还公开的基于群体的分类器的优势在于,可以正确地评定来自作为例如临床试验或临床方案的一部分的患者的样品的基质表型或生物标记物阳性而无需参考任何其他当前患者数据。因此,虽然具有每种表型类别的概率的潜在图的可用性是有用的,但不需要正确地评定基质表型或生物标记物阳性。
143.本公开还提供了用于治疗罹患癌症的受试者,例如人受试者的方法,其包括取决于癌症的tme的分类施用特定疗法,所述分类是根据本文公开的基于群体和/或非基于群体的分类器,例如基于一种或多种tme类别分配的存在(生物标记物阳性)和/或不存在(生物标记物阴性)(例如,受试者是否是a和is生物标记物阳性的,和/或id和ia生物标记物阴性的)。
144.还提供了个性化治疗,其可以施用于患有分类为特定tme类别或其组的癌症的受试者(即,受试者对于特定tme类别或其组是生物标记物阳性的),或确定没有分类为特定tme类别或其组的癌症的受试者(即,受试者对于特定tme类别或其组是生物标记物阴性的)。本公开还提供了基因套组(例如,表3和表4中公开的那些),其可以用于鉴定罹患适合用特定治疗剂(例如,tme特定疗法)治疗的癌症的人受试者。
145.本文公开的方法和组合物的应用可以通过将患者与疗法(例如,下文公开的任何tme特定疗法或其组合,取决于受试者的生物标记物阳性和/或生物标记物阴性状态)匹配来改善临床结果,其作用机制靶向一种或多种特定基质亚型(即,基质表型)或肿瘤生物学。
146.主要基质表型可以是定向的,但可以基于一种药物、多种药物或临床方案的作用机制的复杂性针对任何特定药物进行修改。如果例如与对于多于一种基质表型是生物标记物阳性的或主要是一种基质表型的一个患者或患者的组相关,药物或临床方案的组合(即,下文公开的一种或多种tme特定疗法)可以应用于多种基质表型,但是在生物标记物信号中存在其他基质表型的贡献,如在ann模型的概率函数或应用于潜在空间的逻辑回归中所见,如在本公开中。因此,如应用于本文公开的基质表型的术语“主要”指示患者或样品对于特定基质表型(例如,ia)是生物标记物阳性的,但其他基质表型(例如,is、id或a)或其组合对于生物标记物信号也有贡献,如在ml模型,例如本文公开的ann模型的概率函数中所见,或在应用于潜在空间的逻辑回归中所见。
147.在一些方面,患者对于基质表型的特定部分可以是生物标记物阳性的,例如,在高于或低于特定基质表型内的特定阈值或其组合(例如,上阈值和下阈值)时,患者可以被认
为是生物标记物阳性的。换言之,基质表型可以匹配药物(例如,ia基质表型可以匹配药物派姆单抗),但是当药物或药物组合可以改变多种基质表型时,可以将基质表型用作开发药物特异性组合的起点,例如使用巴维妥昔单抗加派姆单抗。因此,确定患者或患者群体对于两种或更多种基质表型是生物标记物阳性的可以用于通过组合两种或更多种tme特定疗法来开发新疗法。例如,巴维妥昔单抗和派姆单抗的临床方案靶向两种基质表型,ia和is,并且因此这种组合的诊断或生物标记物标志将是基于两种基质表型的综合和改进。另一个说明性实例是双特异性抗体那赛昔珠单抗,其是vegf和dll4两者的靶向剂。虽然vegf清楚地靶向a基质表型,但是存在反映dll4生物学的环境的is组的一些特征。因此,如本文所述,利用整合a和is基质表型(或例如其子集,例如由一个或多个阈值定义)的算法的诊断生物标记物标志和额外基因可以用于引出dll4生物学的非血管生成特征。
148.术语
149.为了可以更容易地理解本公开,首先定义某些术语。如本公开中所使用的,除非本文另有明确提供,否则以下术语中的每一个应具有下文所述的含义。另外的定义在整个公开中阐述。
[0150]“施用”是指使用本领域技术人员已知的多种方法和递送系统中的任何一种将包含治疗剂(例如,单克隆抗体)的组合物物理引入受试者。优选施用途径包括静脉内、肌内、皮下、腹膜内、脊柱或其他胃肠外施用途径,例如通过注射或输注。
[0151]
如本文所用的短语“肠胃外施用”意指除肠内和局部施用以外的施用方式,通常是通过注射施用,并且包括但不限于静脉内、肌内、动脉内、鞘内、淋巴管内、病灶内、囊内、眼眶内、心脏内、真皮内、腹膜内、经气管、皮下、表皮下、关节内、囊下、蛛网膜下、脊柱内、眼内、玻璃体内、眼周、硬膜外和胸骨内注射和输注,以及体内电穿孔。其他非肠胃外途径包括口服、局部、表皮或粘膜施用途径,例如鼻内、阴道、直肠、舌下或局部。施用还可以例如一次、多次和/或经一个或多个延长的时间段进行。
[0152]“抗体”(ab)应包括但不限于与抗原特异性结合并包含通过二硫键互连的至少两条重(h)链和两条轻(l)链的糖蛋白免疫球蛋白或其抗原结合部分。每条h链包含重链可变区(本文缩写为vh)和重链恒定区。重链恒定区包含三个恒定结构域,c
h1
、c
h2
和c
h3
。每条轻链包含轻链可变区(本文缩写为v
l
)和轻链恒定区。轻链恒定区包含一个恒定结构域c
l
。vh和v
l
区可以进一步细分为具有高变性的区域,称为互补决定区(cdr),散布有更保守的区域,称为框架区(fr)。每个vh和v
l
包含从氨基末端至羧基末端按以下顺序排列的三个cdr和四个fr:fr1、cdr1、fr2、cdr2、fr3、cdr3和fr4。重链和轻链的可变区含有与抗原相互作用的结合结构域。抗体的恒定区可以介导免疫球蛋白与宿主组织或因子的结合,所述宿主组织或因子包括免疫系统的各种细胞(例如,效应细胞)和经典补体系统的第一组分(c1q)。
[0153]
免疫球蛋白可以衍生自任何通常已知的同种型,包括但不限于iga、分泌型iga、igg和igm。igg亚类也是本领域技术人员熟知的并且包括但不限于人igg1、igg2、igg3和igg4。“同种型”是指由重链恒定区基因编码的抗体类别或亚类(例如,igm或igg1)。
[0154]
术语“抗体”包括例如单克隆抗体;嵌合和人源化抗体;人或非人抗体;全合成抗体;和单链抗体。可以通过重组方法将非人抗体人源化以降低其在人体内的免疫原性。在没有明确说明的情况下并且除非上下文另外指示,否则术语“抗体”还包括任何上述免疫球蛋白的抗原结合片段或抗原结合部分,并且包括单价和二价片段或部分,以及单链抗体。如本
文所用,术语“抗体”不包括天然存在的抗体或多克隆抗体。如本文所用,术语“天然存在的抗体”和“多克隆抗体”不包括由治疗性干预(例如,疫苗)诱导的免疫反应产生的抗体。
[0155]“分离的抗体”是指基本上不含具有不同抗原特异性的其他抗体的抗体(例如,特异性结合pd-1的分离的抗体基本上不含特异性结合除pd-1之外的抗原的抗体)。然而,特异性结合pd-1的分离的抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)可能与其他抗原(诸如来自不同物种的pd-1分子)具有交叉反应性。此外,分离的抗体可以基本上不含其他细胞材料和/或化学品。
[0156]
术语“单克隆抗体”(mab)是指具有单分子组成的抗体分子的非天然制备物,即,其一级序列基本上相同并且对特定表位表现出单一结合特异性和亲和力的抗体分子。单克隆抗体是分离的抗体的一个实例。单克隆抗体可以通过杂交瘤、重组、转基因或本领域技术人员已知的其他技术产生。
[0157]“人抗体”(humab)是指具有可变区的抗体,其中框架区和cdr区两者均来源于人种系免疫球蛋白序列。此外,如果抗体含有恒定区,则所述恒定区也来源于人种系免疫球蛋白序列。本公开的人抗体可以包括并非由人种系免疫球蛋白序列编码的氨基酸残基(例如,在体外通过随机或位点特异性诱变引入的突变或在体内通过体细胞突变引入的突变)。然而,如本文所用,术语“人抗体”不旨在包括其中来源于另一种哺乳动物物种(诸如小鼠)的种系的cdr序列已经被接枝到人框架序列上的抗体。术语“人抗体”和“完全人抗体”作为同义词使用。
[0158]“人源化抗体”是指其中非人抗体的cdr之外的一些、大部分或全部氨基酸被来源于人免疫球蛋白的对应氨基酸替换的抗体。在抗体的人源化形式的一方面,cdr之外的一些、大部分或全部氨基酸已经被来自人免疫球蛋白的氨基酸替换,而一个或多个cdr内的一些、大部分或全部氨基酸未改变。氨基酸的少量添加、缺失、插入、取代或修改是克允许的,只要它们不消除抗体结合特定抗原的能力即可。“人源化抗体”保留与原始抗体类似的抗原特异性。
[0159]“嵌合抗体”是指可变区来源于一个物种并且恒定区来源于另一物种的抗体,诸如可变区来源于小鼠抗体并且恒定区来源于人抗体的抗体。
[0160]
如本文所用,“双特异性抗体”是指包含两个抗原结合位点的抗体,第一结合位点对于第一抗原或表位具有亲和力,并且第二结合位点对于不同于第一抗原或表位的第二抗原或表位具有结合亲和力。
[0161]“抗抗原抗体”是指特异性结合抗原的抗体。例如,抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)特异性结合pd-1,并且抗pd-l1抗体特异性结合pd-l1。
[0162]
抗体的“抗原结合部分”(也称为“抗原结合片段”)是指抗体的一个或多个片段,其保留特异性结合完整抗体所结合的抗原的能力。已经显示,全长抗体的片段可以执行抗体的抗原结合功能。涵盖在术语抗体(例如,本文所述的抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)或抗pd-l1抗体)的“抗原结合部分”内的结合片段的实例包括(i)由v
l
、vh、lc和ch1结构域组成的fab片段(来自木瓜蛋白酶切割的片段)或类似的单价片段;(ii)包含由铰链区的二硫键连接的两个fab片段的f(ab')2片段(来自胃蛋白酶切割的片段)或类似的二价片段;(iii)由vh和ch1结构域组成的fd片段;(iv)由抗体单
臂的v
l
和vh结构域组成的fv片段;(v)dab片段(ward等人,(1989)nature341:544-546),其由vh结构域组成;(vi)分离的互补决定区(cdr)和(vii)两个或更多个分离的cdr的组合,其可以任选地通过合成接头接合。此外,尽管fv片段的两个结构域(v
l
和vh)由独立基因编码,但它们可以使用重组方法通过合成接头接合,所述合成接头能够使它们制成其中v
l
和vh区配对形成单价分子的单一蛋白质链(称为单链fv(scfv);参见例如bird等人(1988)science 242:423-426;和huston等人(1988)proc.natl.acad.sci.usa 85:5879-5883)。此类单链抗体也旨在包含在抗体的“抗原结合部分”术语内。这些抗体片段是使用本领域可用的技术获得的,并且以与完整抗体相同的方式针对效用来筛选所述片段。抗原结合部分可以通过重组dna技术或通过完整的免疫球蛋白的酶促或化学切割来产生。
[0163]
如本文所用,当应用于特定抗原时,术语“抗体”还涵盖包含具有不同结合特异性的其他结合部分的抗体分子。因此,在一方面,术语抗体还涵盖抗体药物缀合物(adc)。在另一个方面,术语抗体涵盖多特异性抗体,例如双特异性抗体。因此,例如,术语抗pd-1抗体也将涵盖包含抗pd-1抗体或其抗原结合部分的adc。类似地,术语抗pd-1抗体将涵盖包含能够特异性结合pd-1的抗原结合部分的双特异性抗体。
[0164]“癌症”是指一组广泛的以体内异常细胞的不受控制的生长为特征的各种疾病。不受调节的细胞分裂和生长导致恶性肿瘤的形成,所述恶性肿瘤侵入邻近组织并且还可以通过淋巴系统或血流转移至身体的远端部位。术语“肿瘤”是指实体癌。术语“癌”是指上皮起源的癌症。
[0165]
术语“免疫疗法”是指通过包括诱导、增强、抑制或以其他方式改变免疫应答的方法对罹患疾病或有患疾病或患有疾病复发风险的受试者进行治疗。受试者的“治疗”或“疗法”是指对受试者进行的任何类型的干预或处理,或向受试者施用活性剂,目的是逆转、减轻、改善、抑制、减缓或预防与疾病相关联的症状、并发症或病症或生化指标的发作、进展、发展、严重程度或复发。
[0166]
在本公开的上下文中,术语“免疫抑制的”或“免疫抑制”描述对癌症的免疫应答状态。肿瘤微环境中的免疫抑制细胞可以抑制患者对癌症的免疫应答,从而阻断、预防或减弱免疫系统对癌症的攻击。在免疫抑制疗法中,目标是通过给患者某些药物来缓解免疫抑制(与引起免疫抑制相反,例如在器官移植物的情况下),使得免疫系统可以攻击癌症。
[0167]
术语“小分子”是指分子量小于约900道尔顿或小于约500道尔顿的有机化合物。所述术语包括具有所需药理学特性的剂,并且包括可以口服或注射的化合物。所述术语包括调控tgf-β和/或与增强或抑制免疫应答相关联的其他分子的活性的有机化合物。
[0168]“程序性死亡-1”(pd-1)是指属于cd28家族的免疫抑制性受体。pd-1主要在体内先前激活的t细胞上表达,并且结合两种配体pd-l1和pd-l2。如本文所用,术语“pd-1”包括人pd-1(hpd-1)、hpd-1的变体、同种型和物种同源物以及与hpd-1具有至少一个共同表位的类似物。完整的hpd-1序列可以在genbank登录号u64863下找到。
[0169]“程序性死亡配体-1”(pd-l1)是pd-1的两种细胞表面糖蛋白配体之一(另一种是pd-l2),其在结合pd-1后下调t细胞激活和细胞因子分泌。如本文所用,术语“pd-l1”包括人pd-l1(hpd-l1)、hpd-l1的变体、同种型和物种同源物以及与hpd-l1具有至少一个共同表位的类似物。完整的hpd-l1序列可以在genbank登录号q9nzq7下找到。人pd-l1蛋白由人cd274基因(ncbi基因id:29126)编码。
[0170]
如本文所用,术语“受试者”包括任何人或非人动物。术语“受试者”和“患者”在本文可互换使用。术语“非人动物”包括但不限于脊椎动物,诸如狗、猫、马、牛、猪、野猪、绵羊、山羊、水牛、野牛、美洲驼、鹿、麋鹿和其他大型动物,以及他们的幼崽,包括小牛和羔羊,以及小鼠、大鼠、兔、豚鼠、灵长类动物诸如猴和其他实验动物。在动物中,哺乳动物是优选的,最优选地,珍贵的和有价值的动物,诸如家养宠物、赛马和用于直接生产(例如,肉)或间接生产(例如,牛奶)供人类食用的食物的动物,尽管实验动物也包括在内。在特定方面,受试者是人。因此,本公开适用于临床、兽医和研究用途。
[0171]
如本文所用,术语“治疗(treat)”、“治疗(treating)”和“治疗(treatment)”是指对受试者进行的任何类型的干预或处理,或向受试者施用活性剂,目的是逆转、减轻、改善、抑制或减缓或预防与疾病相关联的症状、并发症、病症或生化指标的进展、发展、严重程度或复发或增强总体存活。治疗可以是针对患有疾病的受试者或未患有疾病的受试者(例如,用于预防)。如在此所用,术语“治疗(treat)”、“治疗(treating)”和“治疗(treatment)”是指施用有效量或有效剂量。
[0172]
术语“有效剂量(effective dose)”或“有效剂量(effective dosage)”定义为足以实现或至少部分地实现所需效应的量。
[0173]
药物或治疗剂的“治疗有效量”或“治疗有效剂量”是药物在单独地或与另一种治疗剂组合使用时,保护受试者免于疾病的发作或者促进疾病消退的任何量,所述疾病消退是通过疾病症状的严重程度的降低、无疾病症状时期的频率和持续时间的增加或对由于疾病困扰引起的损伤或残疾的预防所证实。
[0174]
药物的治疗有效量或剂量包括“预防有效量”或“预防有效剂量”,它是药物在单独地或与另一种治疗剂组合施用至具有患疾病或遭受疾病复发的风险的受试者时抑制疾病的发生或复发的任何量。
[0175]
此外,关于本文公开的治疗的术语“有效的”和“有效性”包括药理学有效性和生理学安全性两者。药理学有效性是指药物促进患者的癌症消退的能力。生理学安全性是指由药物的施用引起的在细胞、器官和/或生物体水平上的毒性或其他不良生理学影响的水平(不良效应)。
[0176]
可以使用熟练从业人员已知的各种方法评估治疗剂促进疾病消退(例如,癌症消退)的能力,诸如在临床试验期间在人受试者中、在预测在人中的功效的动物模型系统中或通过在体外测定中测定剂的活性来评估。
[0177]
举例来说,“抗癌剂”或其组合促进受试者的癌症消退。在一些方面,治疗有效量的治疗剂促进癌症消退至消除癌症的程度。
[0178]
在本公开的一些方面,抗癌剂作为疗法的组合施用:包括施用(i)抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)和(ii)抗磷脂酰丝氨酸(ps)靶向抗体,例如巴维妥昔单抗的疗法。
[0179]“促进癌症消退”意味着施用有效量的药物的药物或其组合(作为单一治疗组合物一起施用或作为如上文讨论的单独治疗中的单独组合物施用)导致癌症负荷的减小,例如肿瘤生长或大小的减小、肿瘤坏死、至少一种疾病症状的严重程度的降低、无疾病症状时期的频率和持续时间的增加或对由于疾病困扰引起的损伤或残疾的预防。
[0180]
尽管有这些治疗有效性的最终测量,但是免疫治疗药物的评估也必须考虑到免疫
相关应答模式。可以使用本文所述的测定和本领域已知的其他测定来评估治疗剂抑制癌症生长,例如肿瘤生长的能力。可替代地,可以通过检查化合物抑制细胞生长的能力来评估组合物的这种特性,可以通过熟练从业人员已知的测定在体外测量这种抑制。
[0181]
如本文所用,术语“生物样品”或“样品”是指从受试者分离的生物材料。生物样品可以含有适合例如通过对核酸进行测序确定基因表达的任何生物材料。
[0182]
生物样品可以是任何合适的生物组织,例如癌组织。在一方面,样品是肿瘤组织活检,例如福尔马林固定、石蜡包埋(ffpe)的肿瘤组织或新鲜冷冻的肿瘤组织等。在另一方面,使用瘤内样品。在另一方面,生物流体可以存在于肿瘤组织活检中,但生物样品本身不是生物流体。
[0183]
除非上下文另外清楚地说明,否则单数形式“一个”、“一种”以及“所述”包括复数引用。术语“一个”(或“一种”)以及术语“一种或多种”和“至少一种”可以在本文中互换使用。在某些方面,术语“一个”或“一种”意指“单一”。在其他方面,术语“一个”、“一种”包括“两种或更多种”或“多种”。
[0184]
此外,当在本文中使用时将“和/或”视为对两个指定特征或组分中的每一者具有或不具有另一者的具体公开内容。因此,如在本文中以短语诸如“a和/或b”使用的术语“和/或”旨在包括“a和b”、“a或b”、“a”(单独)和“b”(单独)。同样,术语“和/或”如在短语诸如“a、b和/或c”中使用时旨在涵盖以下方面中的每一者:a、b和c;a、b或c;a或c;a或b;b或c;a和c;a和b;b和c;a(单独);b(单独);以及c(单独)。
[0185]
术语“约”、“基本上包含
……”
或“基本上由
……
组成”是指在本领域普通技术人员确定的特定值或组成的可接受误差范围内的值或组成,其部分取决于如何测量或确定值或组成,即测量系统的局限性。例如,根据本领域的实践,“约”、“基本上包含
……”
或“基本上由
……
组成”可以意指在1个或多于1个标准偏差内。可替代地,“约”、“基本上包含
……”
或“基本上由
……
组成”可以意指高达10%的范围。此外,特别是关于生物系统或过程,所述术语可以意指高达值的一个数量级或高达值的5倍。当在本说明书和权利要求中提供特定值或组成时,除非另外说明,否则应假定“约”、“基本上包含
……”
或“基本上由
……
组成”的含义在此特定值或组成的可接受的误差范围内。
[0186]
如本文所用,当应用于一个或多个感兴趣的值时,术语“大约”是指与所陈述参考值类似的值。在某些方面,除非另外说明或者另外从上下文明显看出,否则术语“大约”是指落在所陈述参考值的任一方向(大于或小于)上的10%、9%、8%、7%、6%、5%、4%、3%、2%、1%或更小内的值的范围(这个数字会超过可能值的100%的情况除外)。
[0187]
如本文所述,除非另外指示,否则任何浓度范围、百分比范围、比率范围或整数范围应理解为包括所叙述范围内的任何整数的值,并且在适当时包括它们的分数(诸如整数的十分之一和百分之一)。
[0188]
除非另外定义,否则本文所用的所有技术和科学术语具有与本公开涉及的领域的普通技术人员通常所理解的相同的含义。例如,concise dictionary of biomedicine and molecular biology,juo,pei-show,第2版,2002,crc出版社;the dictionary of cell and molecular biology,第3版,1999,学术出版社;以及oxford dictionary of biochemistry and molecular biology,修订版,2000,牛津大学出版社为技术人员提供了本公开文本中所用的许多术语的通用词典。
[0189]
应理解,本文中无论用语言“包含”描述任何方面,还提供了以“由
……
组成”和/或“基本上由
……
组成”描述的其他类似方面。
[0190]
单位、前缀和符号以其国际单位制(si)公认的形式来表示。本文提供的标题不是对本公开的各个方面的限制,所述各个方面可以通过参考说明书作为整体而获得。因此,通过从整体上参考说明书,可以更全面地定义所定义的术语。
[0191]
本文所用的缩写在整个本公开中定义。在以下小节中进一步详细描述了本公开的各个方面。
[0192]
i.微环境(tme)分类
[0193]
本公开提供了用于对有需要的受试者中的癌症的肿瘤微环境(tme)进行分类的方法。这些分类器可以是基于群体的分类器、非基于群体的分类器或其组合。
[0194]
如本文所用,术语“基于群体的分类器”是指基于计算对应于生物标记物群体(例如,本文公开的生物标记物基因的群体)的一种或多种特征(例如,核酸或蛋白质表达水平)的一个或多个标志的tme分类的方法。在一些方面,使用针对来自本文公开的基因套组的基因集(例如,表1或表2中公开的基因的子集,或图28a-g中公开的基因套组(基因集)中的任一个)获得的基因表达数据(例如,rna表达数据)计算每个标志。
[0195]
如本文所用,术语“非基于群体的分类器”是指基于应用由机器学习生成的预测模型(例如,ann)的tme分类的方法。在一些方面,使用例如训练集生成非基于群体的分类器,所述训练集包括根据本文公开的基于群体的分类器作为训练集预处理的表达数据(例如,rna表达数据)。
[0196]
在一些方面,与新鲜样品(非存档样品)相比,当使用存档样品时,应用如本文公开的基于群体的方法或非基于群体的方法的结果没有差异。实施例7公开了ann方法对新鲜样品(非存档样品)的应用。实施例12公开了ann方法对存档样品的应用。
[0197]
在一些方面,新鲜样品相对于存档样品是优选的。如本文所用,术语“新鲜样品”、“非存档样品”及其语法变体是指在预定时间段之前(例如,在从受试者提取之后一周)已经处理(例如,以确定rna或蛋白质表达)的样品(例如,肿瘤样品)。在一些方面,新鲜样品未冷冻。在一些方面,新鲜样品未固定。在一些方面,新鲜样品在处理之前已经储存少于约两周、少于约一周或少于六天、五天、四天、三天或两天。如本文所用,术语“存档样品”及其语法变体是指在预定时间段之后(例如,在从受试者提取之后一周)已经处理(例如,以确定rna或蛋白质表达)的样品(例如,肿瘤样品)。在一些方面,存档样品已经冷冻。在一些方面,存档样品已经固定。在一些方面,存档样品具有已知的诊断和/或处理历史。在一些方面,存档样品在处理之前已经储存至少一周、至少一个月、至少六个月或至少一年。
[0198]
在一些方面,本公开的基于群体的分类器包括例如确定组合生物标记物,所述组合生物标记物包括通过测量从受试者获得的样品中基因套组(例如,包含来自表1或表2的至少一个基因的基因套组,或图28a-g中公开的基因套组(基因集)中的任一个或其组合)的表达水平确定的至少一个标志分数;其中所述至少一个标志分数允许将受试者的癌症分配到特定tme类别或其组合。
[0199]
在一些方面,本公开的非基于群体的分类器包括测量从受试者获得的样品中基因套组(例如,包含来自表1或表2的至少一个基因的基因套组,或图28a-g中公开的基因套组(基因集)中的任一个或其组合)的表达水平;并且应用通过机器学习生成的预测模型(例
如,逻辑回归、随机森林、人工神经网络或支持向量机模型),这将受试者的癌症分配到特定tme类别或其组合。在一些方面,使用将机器学习模型输出分配到特定tme类别或其组合的统计函数对机器学习模型输出(例如,来自本文公开的ann的输出)进行后处理。
[0200]
之后,将受试者的癌症分配到特定tme或其组合的分类器输出(例如,来自基于群体的分类器、非基于群体的分类器或其组合)将指导一个或多个特定治疗的选择和施用,所述治疗已确定有效治疗具有相同tme的其他受试者的相同类型癌症,即下文公开的tme类别疗法或其组合。
[0201]
如本文所用,术语“肿瘤微环境”和“tme”是指肿瘤细胞周围的环境,包括例如血管、免疫细胞、内皮细胞、成纤维细胞、其他基质细胞、信号传导分子和细胞外基质。在一些方面,术语“基质亚型”、“基质表型”及其语法变体与术语“tme”可互换使用。
[0202]
肿瘤细胞与周围微环境密切相关并且不断相互作用。通常,肿瘤微环境(也称为例如基质表型)涵盖肿瘤基质和肿瘤环境的任何结构和/或功能特征。许多非肿瘤细胞类型可以存在于tme中,例如癌相关成纤维细胞、髓源性抑制细胞、肿瘤相关巨噬细胞、嗜中性粒细胞或肿瘤浸润淋巴细胞。在一些方面,特定tme的分类可以包括分析基质中存在的细胞类型。tme还可以通过特定的功能特征来表征,例如异常的充氧水平、异常的血管通透性或异常水平的特定蛋白质(诸如胶原蛋白、弹性蛋白、糖胺聚糖、蛋白聚糖或糖蛋白)。
[0203]
本文公开的基于群体和非基于群体的分类器可以用于将患者或癌症样品分配到特定tme类别(例如,id、ia、is或a)或其组合(例如,id和ia、id和is、id和a等)。特定tme类别内的患者的特定亚群可以基于阈值的应用进一步分类(例如,通过使用线性阈值或其组合,如图21a例示,或通过使用如图21b例示的非线性阈值或其组合)。
[0204]
此分类充当组合生物标记物,即它是来源于离散生物标记物的生物标记物(例如,tme类别或例如根据线性或非线性阈值或其组合定义的特定tme内的子集),所述离散生物标记物在基于群体的分类器的情况下整合为单个分数或其组合,或者在非基于群体的分类器的情况下整合为模型。因此,对于单种tme类别,例如id、ia、is或a,患者或癌症样品可以是“生物标记物阳性的”,其中患者或样品将被描述为例如id生物标记物阳性的、ia生物标记物阳性的、is生物标记物阳性的或a生物标记物阳性的。在一些方面,患者或癌症样品对于多于一种tme类别可以是生物标记物阳性的。因此,在一些方面,患者或癌症样品对于2、3、4或更多种tme类别可以是生物标记物阳性的。在一些方面,患者或癌症样品可以是例如id和ia生物标记物阳性的;id和is生物标记物阳性的;id和a生物标记物阳性的;ia和is生物标记物阳性的;ia和a生物标记物阳性的;或is和a生物标记物阳性的。在一些方面,患者或癌症样品可以是例如id、ia和is生物标记物阳性的;id、is和a生物标记物阳性的;或id、is和a生物标记物阳性的。
[0205]
在一些方面,使用生物标记物阳性状态的组合概率(即,来自基质表型分类器的一个或多个概率的组合)。可以使用本领域已知的数学技术计算生物标记物阳性状态的组合概率。
[0206]
对于单种tme类别,例如id、ia、is或a,患者或癌症样品也可以定义为“生物标记物阴性的”。因此,患者或样品将被描述为例如id生物标记物阴性的、ia生物标记物阴性的、is生物标记物阴性的或a生物标记物阴性的。在一些方面,患者或癌症样品对于多于一种tme类别可以是生物标记物阴性的。因此,在一些方面,患者或癌症样品对于2、3、4或更多种tme
类别可以是生物标记物阴性的。在一些方面,患者或癌症样品可以是例如id和ia生物标记物阴性的;id和is生物标记物阴性的;id和a生物标记物阴性的;ia和is生物标记物阴性的;ia和a生物标记物阴性的;或is和a生物标记物阴性的。在一些方面,患者或癌症样品可以是例如id、ia和is生物标记物阴性的;id、is和a生物标记物阴性的;或id、is和a生物标记物阴性的。
[0207]
在一些方面,使用生物标记物阴性状态的组合概率(即,来自基质表型分类器的一个或多个概率的组合)。可以使用本领域已知的数学技术计算生物标记物阴性状态的组合概率。
[0208]
在一些方面,tme类别特定疗法的分配是基于特定基质表型的存在,即,如果受试者呈现ia基质表型(并且因此所述受试者是ia生物标记物阳性的),则将施用ia类别tme疗法。在一些方面,tme类别特定疗法的分配是基于特定基质表型的不存在,即,如果受试者不呈现ia基质表型(并且因此所述受试者是ia生物标记物阴性的),则将不施用ia类别tme疗法。
[0209]
在一些方面,将患者或癌症样品分类为tme类别以及将tme类别疗法分配到患者或癌症不是一对一的。换言之,对于多于一种tme类别,可以将患者或癌症样品分类为生物标记物阳性的和/或生物标记物阴性的,并且可以使用多于一种tme类别疗法或其组合来治疗此患者。例如,将患者或癌症样品分类为对于两种不同的tme类别(即,两种基质表型)是生物标记物阳性的可以用于选择一种治疗,其包括在对应于患者或癌症样品对于其是生物标记物阳性的tme类别的tme类别疗法中的药理学方法的组合。此外,如果患者或癌症样品对于特定tme类别是生物标记物阴性的,则这种知识可以用于排除患者或癌症样品对于其是生物标记物阴性的tme类别的tme类别疗法中的特定药理学方法。因此,可用于治疗被分类为对于特定tme类别是生物标记物阳性的癌症样品的药物或其组合、治疗或其组合和/或临床方案或组合可以组合以治疗具有多于一种生物标记物阳性信号(即,具有被分类为对于多于一种的基质表型是生物标记物阳性的癌症样品)的患者。
[0210]
在一些方面,取决于药物或临床方案的作用机制,可以使用不同的分类参数,例如不同的基因套组子集、不同的阈值、不同的ann架构、不同的激活函数或不同的后处理函数产生不同的tme类别,所述类别进而将用于选择适当的tme类别疗法。因此,每种药物或药物方案可以具有不同的诊断基因套组和不同配置的基于群体或非基于群体的分类器,以通知临床医生(诸如医学医生)例如决定是否应选择患者进行治疗、是否应开始治疗、是否应暂停治疗或是否应修改治疗。
[0211]
在一些方面,临床医生可以考虑患者的生物标记物状态的协变量,并且将基质表型或生物标记物状态的概率与msi/mss(微卫星不稳定性/微卫星稳定性-高)状态、ebv(埃-巴二氏病毒)状态、pd-1/pd-l1状态(诸如cps,即组合阳性分数)、嗜中性粒细胞-白细胞比率(nlr)或混淆变量(诸如既往治疗史)组合。
[0212]
在一些方面,临床医生被给予来自算法的二元结果,并且做出如本文所述的治疗或不治疗的决策。在一方面,临床医生给给予例如叠加在潜在空间上并用概率阈值或线性或多项式逻辑回归解释的患者结果的图。
[0213]
i.a.基因套组
[0214]
本公开的基于群体和非基于群体的分类器依赖于选择特定基因套组作为分类器
使用的输入数据的来源。在一些方面,本公开的基因套组中的每个基因被称为“生物标记物”。术语“基因集”和“基因套组”可互换使用。
[0215]
在一些方面,所述生物标记物是核酸生物标记物。如本文所用,术语“核酸生物标记物”是指可以在受试者或来自其的样品(例如,包括例如来自肿瘤的组织、细胞、基质、细胞裂解物和/或其成分的样品)中检测(例如,定量)的核酸(例如,本文公开的基因套组中的基因)。在一些方面,术语核酸生物标记物是指可以在受试者或来自其的样品(例如,包括例如来自肿瘤的组织、细胞、基质、细胞裂解物和/或其成分的样品)中检测(例如,定量)的核酸(例如,本文公开的基因套组中的基因)中感兴趣的特定序列(例如,核酸变体或单核苷酸多态性)的存在或不存在。
[0216]
在一些方面,核酸生物标记物的“水平”可以是指生物标记物的“表达水平”,例如样品中由核酸生物标记物的核酸序列编码的rna或dna的水平。例如,在一些方面,表1或表2中公开的特定基因或图28a-g中公开的基因套组(基因集)中的任一个的表达水平是指从受试者获得的样品中存在的编码这种基因的mrna的量。
[0217]
在一些方面,核酸生物标记物(例如,rna生物标记物)的“水平”可以通过测量下游输出(例如,靶标分子的活性水平或通过核酸生物标记物或其表达产物(例如,rna或dna)调控,例如激活或抑制的效应分子的表达水平)来确定。
[0218]
在一些方面,所述核酸生物标记物是rna生物标记物。如本文所用,“rna生物标记物”是指包含感兴趣的核酸生物标记物的核酸序列的rna,例如编码表1或表2中公开的特定基因或图28a-g中公开的基因套组(基因集)中的任一个的rna。
[0219]
rna生物标记物的“表达水平”通常是指检测到的包含受试者或来自其的样品中存在的感兴趣的核酸序列的rna分子的量,例如,由包含核酸序列的dna分子(例如,受试者或受试者的癌症的基因组)表达的rna分子的量。
[0220]
在一些方面,rna生物标记物的表达水平是肿瘤基质样品中rna生物标记物的量。在一些方面,使用pcr(例如,实时pcr)、测序(例如,深度测序或下一代测序,例如rna-seq)或微阵列表达谱或利用核糖核酸酶保护结合扩增或扩增和新定量方法,诸如rna-seq或其他方法的其他技术来定量rna生物标记物。
[0221]
在一些方面,本文公开的基于群体的分类器包括使用表1和表2中(或图28a-g中公开的基因套组(基因集)中的任一个中)公开的基因的表达水平计算的标志。例如,包括两个标志的基于群体的分类器可以包括从对应于表1中公开的基因或其子集的表达水平获得的标志1,和从对应于表2中公开的基因或其子集的表达水平获得的标志2。在一些特定方面,基于群体的分类器可以使用表3和表4中公开的子集(基因套组)。例如,包括两个标志的基于群体的分类器可以包括从对应于表3中公开的基因套组中的基因的表达水平获得的标志1,和从对应于表4中公开的基因套组中的基因或其子集的表达水平获得的标志2。
[0222]
在本文公开的基于群体的分类器中,从样品群体(例如,来自临床研究的样品)获取的基因套组中的基因的表达水平可以用于根据所计算的标志水平是高于还是低于某些阈值将群体中的样品组分类为属于一种tme类别(或其组合,即样品不仅可以分类为对于单种tme类别是生物标记物阳性的,而且可以分类为对于两种或更多种tme类别是生物标记物阳性的)。随后,从来自测试受试者的一个或多个样品获得的基因套组中的基因的表达水平可以用于将受试者的tme分类为群体中鉴定的tme类别中的一种。
[0223]
在本文公开的非基于群体的分类器中,从样品群体(例如,来自临床研究的样品)获取的基因套组中的基因的表达水平以及其分配到根据本文公开的群体分类器获得的tme类别(或其组合,即样品不仅可以分类为对于单种tme类别是生物标记物阳性的,而且可以分类为对于两种或更多种tme类别是生物标记物阳性的)可以用作用于机器学习,例如使用ann的训练集。机器学习过程可以产生模型,例如ann模型。随后,从来自测试受试者的一个或多个样品获得的基因套组中的基因的表达水平将用作模型的输入,所述模型将受试者的tme分类为特定tme类别(或其组合,即样品不仅可以分类为对于单种tme类别是生物标记物阳性的,而且可以分类为对于两种或更多种tme类别是生物标记物阳性的)。
[0224]
可以通过例如genecards(www.genecards.org)或uniprot(www.uniprot.org)来识别由在整个本公开中使用的标识符指定的蛋白质和基因的标准名称、别名等。
[0225]
表1.标志1基因和登录号(n=63)
[0226]
[0227]
[0228]
[0229]
[0230]
[0231][0232]
表2.标志2基因和登录号(n=61)
[0233]
[0234]
[0235]
[0236]
[0237][0238]
表3:标志1基因套组
[0239]
[0240][0241]
表4:标志2基因套组
[0242][0243][0244]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部
分的基因套组)不包括abcc9、afap1l2、bgn、col4a2、col8a1、fbln5、hey2、igfbp3、lhfp、naalad2、pcdh17、pdgfrb、plxdc2、rgs5、rras、serpine1、steap4、tek、tmem204或其组合。
[0245]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:abcc9、afap1l2、bgn、col4a2、col8a1、fbln5、hey2、igfbp3、lhfp、naalad2、pcdh17、pdgfrb、plxdc2、rgs5、rras、serpine1、steap4、tek和tmem204。
[0246]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括abcc9、col4a2、mest、olfml2a、pcdh17或其组合。
[0247]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:abcc9、col4a2、mest、olfml2a和pcdh17。
[0248]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括adamts4、cd274、cxcl10、ido1、rac2或其组合。
[0249]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:adamts4、cd274、cxcl10、ido1和rac2。
[0250]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括bgn、ccl2、cd19、cd274、cd3e、cd4、cd79a、col4a2、col8a1、ctla4、cxcl9、gzmb、havcr2、ido1、il1b、lag3、pdcd1、pdgfrb、tigit、tnfrsf18、tnfrsf4或其组合。
[0251]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:bgn、ccl2、cd19、cd274、cd3e、cd4、cd79a、col4a2、col8a1、ctla4、cxcl9、gzmb、havcr2、ido1、il1b、lag3、pdcd1、pdgfrb、tigit、tnfrsf18和tnfrsf4。
[0252]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括bgn、ccl2、col4a2、col8a1、ctla4、cxcl10、cxcl9、gzmb、havcr2、il1b、lag3、tigit、tnfrsf18、tnfrsf4或其组合。
[0253]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:bgn、ccl2、col4a2、col8a1、ctla4、cxcl10、cxcl9、gzmb、havcr2、il1b、lag3、tigit、tnfrsf18和tnfrsf4。
[0254]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括bgn、cd19、cd274、cd3e、cd4、cd79a、col4a2、col8a1、ctla4、cxcl10、cxcl9、gzmb、havcr2、ido1、il1b、lag3、pdcd1、pdgfrb、tigit、tnfrsf18、tnfrsf4或其组合。
[0255]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1
分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:bgn、cd19、cd274、cd3e、cd4、cd79a、col4a2、col8a1、ctla4、cxcl10、cxcl9、gzmb、havcr2、ido1、il1b、lag3、pdcd1、pdgfrb、tigit、tnfrsf18和tnfrsf4。
[0256]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括bgn、pdgfrb或其组合。
[0257]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由bgn和pdgfrb组成。
[0258]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括c10orf54、nfatc1或其组合。
[0259]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由c10orf54和nfatc1组成。
[0260]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括capg、dusp4、lag3、plxdc2、tnfrsf18、tnfrsf4或其组合。
[0261]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:capg、dusp4、lag3、plxdc2、tnfrsf18和tnfrsf4。
[0262]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl2、ccl4、cxcl9、gzmb、mgp、mmp12、rac2、timp1或其组合。
[0263]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl2、ccl4、cxcl9、gzmb、mgp、mmp12、rac2和timp1。
[0264]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl2、cd3e、cxcl10、cxcl11、gzmb或其组合。
[0265]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl2、cd3e、cxcl10、cxcl11和gzmb。
[0266]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl2、cd4、cxcl10、mmp13、timp1或其组合。
[0267]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl2、cd4、cxcl10、mmp13和timp1。
[0268]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl3、ccl4、ctla4、etv5、havcr2、ifng、lag3、mta2或其组合。
[0269]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl3、ccl4、ctla4、etv5、havcr2、ifng、lag3和mta2。
[0270]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl4、cd3e、cxcl10、cxcl11、cxcl9、gzmb、havcr2、ido1、ifng、lag3或其组合。
[0271]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl4、cd3e、cxcl10、cxcl11、cxcl9、gzmb、havcr2、ido1、ifng和lag3。
[0272]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl4、cd3e、cxcl10、cxcl11、cxcl9、gzmb、havcr2、ifng、lag3、pdcd1或其组合。
[0273]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl4、cd3e、cxcl10、cxcl11、cxcl9、gzmb、havcr2、ifng、lag3和pdcd1。
[0274]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl4、cxcl10、cxcl11、cxcl9、ido1、ifng ccl4、cxcl10、cxcl11、cxcl9、ifng或其组合。
[0275]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ccl4、cxcl10、cxcl11、cxcl9、ido1、ifng ccl4、cxcl10、cxcl11、cxcl9和ifng。
[0276]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ccl4、gzmb或其组合。
[0277]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由ccl4和gzmb组成。
[0278]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd274、cd3e、cd4、cxcl9、gzmb、ido1、ifng、lag3、pdcd1lg2、tigit或
其组合。
[0279]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd274、cd3e、cd4、cxcl9、gzmb、ido1、ifng、lag3、pdcd1lg2和tigit。
[0280]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd274、cd3e、cd79a、cxcl10、cxcl9、ido1、iqgap3、rac2或其组合。
[0281]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd274、cd3e、cd79a、cxcl10、cxcl9、ido1、iqgap3和rac2。
[0282]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd274、ctla4、cxcl10、cxcl9、gzmb、havcr2、ifng、igfbp3、lag3、pdcd1、pdgfrb、tek、tgfb1、tgfb2、tigit或其组合。
[0283]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd274、ctla4、cxcl10、cxcl9、gzmb、havcr2、ifng、igfbp3、lag3、pdcd1、pdgfrb、tek、tgfb1、tgfb2和tigit。
[0284]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd3e、ctla4、gzmb、lag3、tgfb2或其组合。
[0285]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd3e、ctla4、gzmb、lag3和tgfb2。
[0286]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd4、cd79a、cxcl9或其组合。
[0287]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cd4、cd79a和cxcl9组成。
[0288]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd79a、ctla4、ebf1、epha3、etv5、gnas、pdcd1、pdcd1lg2、pdgfrb、runx1t1或其组合。
[0289]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd79a、ctla4、ebf1、epha3、etv5、gnas、pdcd1、pdcd1lg2、pdgfrb和runx1t1。
[0290]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cd8b、cxcl10、cxcl11、gzmb、ifng或其组合。
[0291]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cd8b、cxcl10、cxcl11、gzmb和ifng。
[0292]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括col4a2。
[0293]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由col4a2组成。
[0294]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ctla4、cxcl10、cxcl11、cxcl9、gzmb、ido1、ifng、tigit或其组合。
[0295]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ctla4、cxcl10、cxcl11、cxcl9、gzmb、ido1、ifng和tigit。
[0296]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ctla4、cxcl10、cxcl11、cxcl9、gzmb、ifng、tigit或其组合。
[0297]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ctla4、cxcl10、cxcl11、cxcl9、gzmb、ifng和tigit。
[0298]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ctla4、cxcl10、cxcl11、tigit或其组合。
[0299]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:ctla4、cxcl10、cxcl11和tigit。
[0300]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ctsb、dusp4、mt2a、serpine2或其组合。
[0301]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由ctsb、dusp4、mt2a和serpine2组成。
[0302]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl10、cxcl12或其组合。
[0303]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl10和cxcl12组成。
[0304]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl10、cxcl9、gzmb、ifng、igfbp3或其组合。
[0305]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:cxcl10、cxcl9、gzmb、ifng和igfbp3。
[0306]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl10、lag3或其组合。
[0307]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl10和lag3组成。
[0308]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl12、pdgfrb、steap4或其组合。
[0309]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl12、pdgfrb和steap4组成。
[0310]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl9、gzmb、ifng或其组合。
[0311]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl9、gzmb和ifng组成。
[0312]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl9、ifng或其组合。
[0313]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl9和ifng组成。
[0314]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括cxcl9、mgp、rac2、timp1或其组合。
[0315]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由cxcl9、mgp、rac2和timp1组成。
[0316]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括ednra、ifng、pdgfrb、tgfb1或其组合。
[0317]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由ednra、ifng、pdgfrb和tgfb1组成。
[0318]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括eln。
[0319]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由eln组成。
[0320]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括nov。
[0321]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由nov组成。
[0322]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括epha3、gnas或其组合。
[0323]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由epha3和gnas组成。
[0324]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括gnas。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由gnas组成。
[0325]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括havcr2、pdcd1、tigit或其组合。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下组成:havcr2、pdcd1和tigit。
[0326]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括havcr2、tigit或其组合。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分
类器中的训练集或模型输入的一部分的基因套组)不由havcr2和tigit组成。
[0327]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括igfbp3、tgfb1或其组合。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由igfbp3和tgfb1组成。
[0328]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括igfbp3。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由igfbp3组成。
[0329]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括pdcd1。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由pdcd1组成。
[0330]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括pdgfrb。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由pdgfrb组成。
[0331]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括rgs5。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由rgs5组成。
[0332]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括tgfb1。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由tgfb1组成。
[0333]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括tigit。在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由tigit组成。
[0334]
在一些方面,用于确定基于群体的分类器中的标志1分数的基因套组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不包括bmp5、gnas、il1b、mmp12、naalad2和stab2。在一些方面,用于确定基于群体的分类器中的标志1分数的基因套
组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不包括1、2、3、4、5或6个选自由以下组成的组中的基因:bmp5、gnas、il1b、mmp12、naalad2和stab2。在一些方面,用于确定基于群体的分类器中的标志1分数的基因套组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不由以下组成:bmp5、gnas、il1b、mmp12、naalad2和stab2。
[0335]
在一些方面,用于确定基于群体的分类器中的标志2分数的基因套组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不包括agr2、c11orf9、cd79a、eif5a、hfe、hp、mest、mst1、mt2a、pla2g4a、plau、strn3、tnfsf18、trim7、usf1和zic2。在一些方面,用于确定基于群体的分类器中的标志2分数的基因套组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15或16个选自由以下组成的组中的基因:agr2、c11orf9、cd79a、eif5a、hfe、hp、mest、mst1、mt2a、pla2g4a、plau、strn3、tnfsf18、trim7、usf1和zic2。在一些方面,用于确定基于群体的分类器中的标志2分数的基因套组或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组不由以下组成:agr2、c11orf9、cd79a、eif5a、hfe、hp、mest、mst1、mt2a、pla2g4a、plau、strn3、tnfsf18、trim7、usf1和zic2。
[0336]
可以根据本文公开的方法使用的基因和基因集在图28a、图28b、图28c、图28d、图28e、图28f或图28g中呈现。图28a-图28g中呈现的基因集中特定基因的存在由空心单元(白色)指示,而图28a-图28g中呈现的基因集中特定基因的不存在由实心单元(黑色)指示。
[0337]
用于确定基于群体的分类器中的标志1或标志2的基因套组或用作本文公开的非基于群体的分类器中的训练集或模型输入的一部分的基因套组包括abcc9、adamts4、afap1l2、agr2、bace1、bgn、bmp5、c11orf9、capg、cavin2、ccl2、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd79a、cd8b、col4a2、col8a1、col8a2、cpxm2、ctla4、ctsb、cxcl10、cxcl11、cxcl12、cxcl9、dusp4、ebf1、ecm2、ednra、eif5a、eln、epha3、etv5、fbln5、folr2、gad1、gnas、gnb4、gucy1a1、gzmb、havcr2、hey2、hfe、hmox1、hp、hspb2、ido1、ifna2、ifnb1、ifng、igfbp3、igll5、il1b、iqgap3、itga9、itpr1、jam2、jam3、kcnj8、lag3、lamb2、lhfpl6、ltbp4、meox1、mest、mgp、mmp12、mmp13、mst1、mt2a、mta2、naalad2、nfatc1、nov、olfml2a、pcdh17、pdcd1、pdcd1lg2、pde5a、pdgfrb、peg3、pla2g4a、plau、plscr2、plxdc2、rac2、reg4、rgs4、rgs5、rnf144a、rnh1、rras、runx1t1、selp、serpine1、serpine2、sgip1、smarca1、spon1、srsf6、stab2、steap4、strn3、tbx2、tek、tgfb1、tgfb2、tigit、timp1、tlr9、tmem204、tnfrsf18、tnfrsf4、tnfsf18、trim7、ttc28、usf1、utrn、vsir和zic2。用于确定基于群体的分类器中的标志1或标志2的基因套组或用作本文公开的非基于群体的分类器中的训练集或模型输入的一部分的基因套组由以下组成:abcc9、adamts4、afap1l2、agr2、bace1、bgn、bmp5、c11orf9、capg、cavin2、ccl2、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd79a、cd8b、col4a2、col8a1、col8a2、cpxm2、ctla4、ctsb、cxcl10、cxcl11、cxcl12、cxcl9、dusp4、ebf1、ecm2、ednra、eif5a、eln、epha3、etv5、fbln5、folr2、gad1、gnas、gnb4、gucy1a1、gzmb、havcr2、hey2、hfe、hmox1、hp、hspb2、ido1、ifna2、ifnb1、ifng、igfbp3、igll5、il1b、iqgap3、itga9、itpr1、jam2、jam3、kcnj8、lag3、lamb2、lhfpl6、ltbp4、meox1、mest、mgp、mmp12、mmp13、mst1、mt2a、mta2、naalad2、nfatc1、nov、olfml2a、pcdh17、pdcd1、pdcd1lg2、pde5a、pdgfrb、peg3、pla2g4a、plau、plscr2、plxdc2、rac2、reg4、rgs4、rgs5、rnf144a、
rnh1、rras、runx1t1、selp、serpine1、serpine2、sgip1、smarca1、spon1、srsf6、stab2、steap4、strn3、tbx2、tek、tgfb1、tgfb2、tigit、timp1、tlr9、tmem204、tnfrsf18、tnfrsf4、tnfsf18、trim7、ttc28、usf1、utrn、vsir和zic2。
[0338]
用于确定基于群体的分类器中的标志1或标志2的基因套组或用作本文公开的非基于群体的分类器中的训练集或模型输入的一部分的基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123或124个选自由以下组成的组中的基因:abcc9、adamts4、afap1l2、agr2、bace1、bgn、bmp5、c11orf9、capg、cavin2、ccl2、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd79a、cd8b、col4a2、col8a1、col8a2、cpxm2、ctla4、ctsb、cxcl10、cxcl11、cxcl12、cxcl9、dusp4、ebf1、ecm2、ednra、eif5a、eln、epha3、etv5、fbln5、folr2、gad1、gnas、gnb4、gucy1a1、gzmb、havcr2、hey2、hfe、hmox1、hp、hspb2、ido1、ifna2、ifnb1、ifng、igfbp3、igll5、il1b、iqgap3、itga9、itpr1、jam2、jam3、kcnj8、lag3、lamb2、lhfpl6、ltbp4、meox1、mest、mgp、mmp12、mmp13、mst1、mt2a、mta2、naalad2、nfatc1、nov、olfml2a、pcdh17、pdcd1、pdcd1lg2、pde5a、pdgfrb、peg3、pla2g4a、plau、plscr2、plxdc2、rac2、reg4、rgs4、rgs5、rnf144a、rnh1、rras、runx1t1、selp、serpine1、serpine2、sgip1、smarca1、spon1、srsf6、stab2、steap4、strn3、tbx2、tek、tgfb1、tgfb2、tigit、timp1、tlr9、tmem204、tnfrsf18、tnfrsf4、tnfsf18、trim7、ttc28、usf1、utrn、vsir和zic2。
[0339]
用于确定基于群体的分类器中的标志1或标志2的基因套组或用作本文公开的非基于群体的分类器中的训练集或模型输入的一部分的基因套组由以下组成:1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123或124个选自由以下组成的组中的基因:abcc9、adamts4、afap1l2、agr2、bace1、bgn、bmp5、c11orf9、capg、cavin2、ccl2、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd79a、cd8b、col4a2、col8a1、col8a2、cpxm2、ctla4、ctsb、cxcl10、cxcl11、cxcl12、cxcl9、dusp4、ebf1、ecm2、ednra、eif5a、eln、epha3、etv5、fbln5、folr2、gad1、gnas、gnb4、gucy1a1、gzmb、havcr2、hey2、hfe、hmox1、hp、hspb2、ido1、ifna2、ifnb1、ifng、igfbp3、igll5、il1b、iqgap3、itga9、itpr1、jam2、jam3、kcnj8、lag3、lamb2、lhfpl6、ltbp4、meox1、mest、mgp、mmp12、mmp13、mst1、mt2a、mta2、naalad2、nfatc1、nov、olfml2a、pcdh17、pdcd1、pdcd1lg2、pde5a、pdgfrb、peg3、pla2g4a、plau、plscr2、plxdc2、rac2、reg4、rgs4、rgs5、rnf144a、rnh1、rras、runx1t1、selp、serpine1、serpine2、sgip1、smarca1、spon1、srsf6、stab2、steap4、strn3、tbx2、tek、tgfb1、tgfb2、tigit、timp1、tlr9、tmem204、tnfrsf18、tnfrsf4、tnfsf18、trim7、ttc28、usf1、utrn、vsir和zic2。
[0340]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括以下中存在的基因:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由黑色单元指示的基因)。
[0341]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下中存在的基因组成:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由黑色单元指示的基因)。
[0342]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)包括以下中存在的基因:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、
66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由黑色单元指示的基因)。
[0343]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)由以下中存在的基因组成:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由黑色单元指示的基因)。
[0344]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不包括以下中不存在的基因:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、
168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由空心单元指示的基因)。
[0345]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)不由以下中不存在的基因组成:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由空心单元指示的基因)。
[0346]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)包括以下中不存在的基因:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、
263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由空心单元指示的基因)。
[0347]
在一些方面,本文公开的基因套组(例如,用于确定基于群体的分类器中的标志1分数或标志2分数的基因套组,或用作非基于群体的分类器中的训练集或模型输入的一部分的基因套组)由以下中不存在的基因组成:基因集1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149、150、151、152、153、154、155、156、157、158、159、160、161、162、163、164、165、166、167、168、169、170、171、172、173、174、175、176、177、178、179、180、181、182、183、184、185、186、187、188、189、190、191、192、193、194、195、196、197、198、199、200、201、202、203、204、205、206、207、208、209、210、211、212、213、214、215、216、217、218、219、220、221、222、223、224、225、226、227、228、229、230、231、232、233、234、235、236、237、238、239、240、241、242、243、244、245、246、247、248、249、250、251、252、253、254、255、256、257、258、259、260、261、262、263、264、265、266、267、268、269、270、271、272、273、274、275、276、277、278、279、280、281或282(图28a-g中由空心单元指示的基因)。
[0348]
i.b.样品和样品处理
[0349]
本文公开的方法包括测量选自样品(例如,从受试者获得的生物样品)的基因套组的表达水平。在一些方面,例如当确定两个标志分数(例如,如本文公开的标志1分数和标志2分数)时,每个样品可以是相同的或者它可以是不同的。因此,在一些方面,分别用于确定第一分数和第二分数的第一样品和第二样品是相同的样品。在其他方面,分别用于确定第一分数和第二分数的第一样品和第二样品是不同的样品。在一些方面,所述样品包括瘤内组织。在一些方面,第一样品和/或第二样品包括瘤内组织。在一些方面,第一样品和/或第二样品可以偶然地包括瘤周组织和/或已经浸润规则或不规则形状肿瘤的健康组织。可以在含有或怀疑含有一个或多个本文公开的生物标记物(例如,rna生物标记物)的任何生物样品(包括来自动物、受试者或患者的任何组织样品或活检,例如受试者的癌组织、肿瘤和/或基质)中测量生物标记物水平(例如,本公开的基因套组中的基因的表达水平)。在一些方面,生物标记物水平来源于肿瘤组织(例如,新鲜组织、冷冻组织或保存的组织)。组织样品的来源可以是实体组织,例如来自新鲜、冷冻和/或保存的器官、组织样品、活检或抽吸物。在一些方面,样品是无细胞样品,例如,包含无细胞核酸(例如,dna或rna)。在一些方面,样品可以包含自然界中不与组织天然混合的化合物,诸如防腐剂、抗凝血剂、缓冲液、固定剂、营养素、抗生素等。
[0350]
在一些情况下,生物标记物水平可以来自固定的肿瘤组织。在一些方面,样品被保存为冷冻样品或福尔马林、甲醛或多聚甲醛固定的石蜡包埋的(ffpe)组织制剂。例如,样品可以包埋在基质中,例如ffpe块或冷冻样品。在一些方面,样品可以包括骨髓;抽吸物;刮取物;骨髓样本;组织活检样本;手术样本等。在一些方面,样品是或包含从个体获得的细胞,
biosciences的pacbio rs系统、htg molecular diagnostics的edgeseq和nanostring technology的hyb&seq ngs技术。
[0360]
ngs技术可以包括一个或多个步骤,例如模板制备、测序和成像以及数据分析,这些将在下文更详细地公开。
[0361]
应注意,模板扩增方法,诸如本领域已知的pcr方法,也可以用于定量生物标记物水平。示例性的模板富集方法包括例如微液滴pcr技术(tewhey r.等人,nature biotech.2009,27:1025-1031)、定制设计的寡核苷酸微阵列(例如,roche/nimblegen寡核苷酸微阵列)和基于溶液的杂交方法(例如,分子倒置探针(mip)(porreca g.j.等人,nature methods,2007,4:931-936;krishnakumar s.等人,proc.natl.acad.sci.usa,2008,105:9296-9310;turner e.h.等人,nature methods,2009,6:315-316)以及生物素化的rna捕获序列(gnirke a.等人,nat.biotechnol.2009;27(2):182-9)。
[0362]
(a)模板制备。用于模板制备的方法可以包括诸如将核酸(例如,rna)随机分解成更小的大小和生成测序模板(例如,片段模板或配对模板)的步骤。空间分离的模板可以附着或固定到固体表面或支撑物,从而允许同时进行大量测序反应。可用于ngs反应的模板类型包括例如源自单个dna分子的克隆扩增模板和单个dna分子模板。用于制备克隆扩增模板的方法包括例如乳液pcr(empcr)和固相扩增。
[0363]
empcr可以用于制备ngs的模板。通常,生成核酸片段的文库,并且将含有通用引发位点的接头连接到片段的末端。然后将片段变性成单链并通过珠捕获。每个珠捕获单个核酸分子。在empcr珠的扩增和富集之后,大量模板可以附着或固定到标准显微镜载玻片上的聚丙烯酰胺凝胶中(例如,polonator),化学交联到氨基涂覆的玻璃表面(例如,life/apg;polonator)或沉积到个体picotiterplate(ptp)孔中(例如,roche/454),在其中可以进行ngs反应。
[0364]
固相扩增也可以用于生成ngs的模板。通常,正向和反向引物共价附着到固体支撑物。扩增片段的表面密度由支撑物上引物与模板的比率来定义。固相扩增可以产生数亿个空间分离的模板簇(例如,illumina/solexa)。模板簇的末端可以与用于ngs反应的通用测序引物杂交。
[0365]
用于制备克隆扩增模板的其他方法还包括例如多重置换扩增(mda)(lasken r.s.curr opin microbiol.2007;10(5):510-6)。mda是非基于pcr的dna扩增技术。所述反应涉及将随机六聚体引物与模板退火并通过高保真酶(通常是噬菌体ф29dna聚合酶)在恒温下进行dna合成。mda可以生成错误频率较低的大尺寸产物。
[0366]
单分子模板是另一种可以用于ngs反应的模板。空间分离的单分子模板可以通过各种方法固定在固体支撑物上。在一种方法中,将个体引物分子共价附着到固体支撑物。将衔接子添加到模板中,并且然后将模板与固定的引物杂交。在另一种方法中,通过从固定的引物引发和延伸单链单分子模板,将单分子模板共价附着到固体支撑物。然后将通用引物与模板杂交。在又一种方法中,将单个聚合酶分子附着到固体支撑物,引发的模板结合到所述固体支撑物。
[0367]
(b)测序和成像。用于ngs的示例性测序和成像方法包括但不限于循环可逆终止(crt)、连接测序(sbl)、单分子添加(焦磷酸测序)和实时测序。
[0368]
crt在循环方法中使用可逆终止子,所述方法最少包括核苷酸并入、荧光成像和切
割的步骤。通常,dna聚合酶将对应于模板碱基的互补核苷酸的单个荧光修饰的核苷酸并入引物中。在添加单个核苷酸之后终止dna合成,并且洗涤掉未并入的核苷酸。进行成像以确定并入的标记核苷酸的身份。然后在切割步骤中,去除终止/抑制基团和荧光染料。使用crt方法的示例性ngs平台包括但不限于illumina/solexa基因组分析仪(ga),其使用克隆扩增模板方法结合通过全内反射荧光(tirf)检测的四色crt方法;和helicos biosciences/heliscope,其使用单分子模板法结合通过tirf检测的单色crt方法。
[0369]
sbl使用dna连接酶和单碱基编码探针或双碱基编码探针进行测序。通常,荧光标记的探针与其邻近的引物模板的互补序列杂交。dna连接酶用于将染料标记的探针与引物连接。在洗掉未连接的探针之后,进行荧光成像以确定连接探针的身份。荧光染料可以通过使用可切割探针来去除,从而为后续的连接循环再生成5
’‑
po4基团。可替代地,在去除旧引物之后,可以将新引物与模板杂交。示例性sbl平台包括但不限于life/apg/solid(支持寡核苷酸连接检测),其使用双碱基编码探针。
[0370]
焦磷酸测序方法是基于用另一种化学发光酶检测dna聚合酶的活性。通常,所述方法允许通过沿其合成互补链,一次一个碱基对,并检测每个步骤中实际添加的碱基来对dna的单链进行测序。模板dna是固定的,并且依次添加a、c、g和t核苷酸的溶液并从反应中去除。仅当核苷酸溶液与模板的第一个未配对碱基互补时才产生光。产生化学发光信号的溶液序列允许确定模板的序列。示例性焦磷酸测序平台包括但不限于roche/454,其使用通过empcr制备的dna模板,其中1-2百万个珠沉积到ptp孔中。
[0371]
实时测序涉及在dna合成过程中对染料标记的核苷酸的连续并入进行成像。示例性实时测序平台包括但不限于pacific biosciences平台,其使用附着到个体零模式波导(zmw)检测器表面的dna聚合酶分子,以在磷酸化核苷酸被并入生长引物链中时获得序列信息;life/visigen平台,其使用具有附着的荧光染料的工程化dna聚合酶,以在通过荧光共振能量转移(fret)并入核苷酸之后生成增强的信号;和li-cor biosciences平台,其在测序反应中使用染料淬灭剂核苷酸。
[0372]
用于ngs的其他测序方法包括但不限于纳米孔测序、杂交测序、基于纳米晶体管阵列的测序、聚合酶克隆测序(polony sequencing)、基于扫描隧道显微镜(stm)的测序和基于纳米线分子传感器的测序。
[0373]
纳米孔测序涉及通过纳米级孔对溶液中的核酸分子进行电泳,所述纳米级孔提供高度受限的空间,在所述空间内可以分析单核酸聚合物。纳米孔测序的示例性方法描述于例如branton d.等人,nat biotechnol.2008;26(10):1146-53。
[0374]
杂交测序是使用dna微阵列的非酶促方法。通常,对单个dna池进行荧光标记并与含有已知序列的阵列杂交。来自阵列上的给定点的杂交信号可以鉴定dna序列。当杂交区域较短或存在专门错配检测蛋白时,dna双螺旋中一条dna链与其互补链的结合甚至对于单碱基错配也是敏感的。杂交测序的示例性方法描述于例如hanna g.j.等人,j.clin.microbiol.2000;38(7):2715
–
21;和edwards j.r.等人,mut.res.2005;573(1-2):3
–
12.。
[0375]
聚合酶克隆测序是基于通过多个单碱基延伸(fisseq)进行的聚合酶克隆扩增和合成测序。聚合酶克隆扩增是在聚丙烯酰胺膜上原位扩增dna的方法。示例性聚合酶克隆测序方法描述于例如美国专利申请公布号2007/0087362。
51:11.14.1-11.14.19)和表达定量(例如,使用featurecounts;liao等人(2014)bioinformatics 30:923-930)。在一些方面,当前的参考人类基因组是ensembl,92版本,参考常见掺入标准进行扩展,诸如ercc(外部rna控制联盟)、外部rna控制和sirv(掺入rna变体)。在其他方面,使用更新的参考人类基因组。在一些方面,作为额外的质量控制步骤,将一百万个读取的样品(例如,使用seqtk工具处理;arc.vt.edu/userguide/seqtk/)映射到所选物种的rrna和珠蛋白序列以确定样品中这些种类的读取的总体比例。结果可以报告例如在诸如multiqc的报告工具的汇总表中。在一些方面,原始和归一化的(例如,tpm,每千碱基的转录本;或fpkm,每千碱基的片段)表达值由软件提供。
[0384]
在本文公开的方法的一些特定方面,在使用基于z分数的模型对样品进行分层之前,tpm归一化表达式可以被分位数转换为正态输出分布,将输入值分箱为例如100个分位数(参见图1)。
[0385]
在一些方面,不同批次的表达数据可以被独立归一化以便训练机器学习模型。当存在明显的批次效应时,可以使用独立的归一化。在一些方面,如本领域已知的,主成分分析可以揭示批次效应,包括在一个非限制性实例中,当除了从不同来源(例如,rna-seq)获得的测序表达值以外,从一个来源(例如,rna外显子组(wes))获得的测序表达值用于训练机器学习模型时可能出现的那些批次效应。在一些方面,样品采集的异步性不是批次效应的来源。在一些方面,样品采集的异步性是批次效应的来源,这可以通过例如归一化技术来解决。
[0386]
对于本文公开的所有平台技术,分位数归一化可以用于跨平台协调,例如在使用illumina和edgeseq(htg molecular diagnostics,inc.)数据时。另一个实例是使用分位数归一化来协调微阵列和rna-seq数据,例如,可以在微阵列数据(例如,来自acrg患者数据集)上训练模型,并且然后将其应用于总rna平台(例如,rna-seq)。
[0387]
输入值可以分箱为例如10、15、20、25、30、35、40、45、50、55、60、65、70、75、80、85、90、95、100或更多个分位数并应用正态或均匀输出分布函数。在一些方面,分位数归一化可以应用于本文公开的z分数分类器的正态分布。在一些方面,分位数归一化可以应用于本文公开的ann分类器的均匀分布。在一些方面,分位数的数量高于、低于或介于上文提供的任何值之间。
[0388]
i.b.1.b蛋白质表达水平
[0389]
用于检测蛋白质(例如,多肽)的表达水平的示例性方法包括但不限于免疫组织化学方法、elisa、western分析、hplc和蛋白质组学测定。在一些方面,蛋白质表达水平使用免疫组织化学方法来确定。例如,福尔马林固定的石蜡包埋的组织与特异性结合本文所述的生物标记物的抗体接触。使用与可检测标记偶联的二级抗体或可检测标记诸如比色标记(例如,具有hrp或ap的酶底物产物)来检测结合的抗体。通过估计阳性肿瘤细胞的比例和阳性肿瘤细胞的平均染色强度来对抗体阳性信号进行评分。将比率和强度分数合并为比较两个因素的总分。
[0390]
在一些方面,蛋白质表达水平通过数字病理学方法来确定。数字病理学方法包括在固体支撑物(诸如玻璃载玻片)上的组织的扫描图像。使用扫描装置将玻璃载玻片扫描成完整的载玻片图像。扫描的图像通常存储在信息管理系统中,以用于档案记录和检索。图像分析工具可以用于从数字载玻片中获得客观的定量测量结果。例如,可以使用适当的图像
分析工具来分析免疫组织化学染色的面积和强度。数字病理学系统可以包括扫描仪、分析工具(可视化软件、信息管理系统和图像分析平台)、存储和通信(共享服务、软件)。数字病理学系统可从许多商业来源获得,诸如aperio technologies,inc.(leica microsystems gmbh的子公司)和ventana medical systems,inc.(现在是roche的一部分)。表达水平可以由商业服务提供商定量,包括flagship biosciences(colorado)、pathology,inc.(california)、quest diagnostics(new jersey)和premier laboratory llc(colorado)。
[0391]
i.c基于群体的分类器
[0392]
本文公开的基于群体的分类器依赖于与例如tme的结构和功能方面相关的多个基因的表达水平的整合,以得出与对特定抗癌疗法的应答相关的分数。因此,确定癌症的特定tme或组合具有特定分数(或如果使用多个基因套组,则为分数组合),允许选择适当的tme类别治疗或其组合。因此,在一方面,本公开提供了用于确定有需要的受试者中的癌症的肿瘤微环境(tme)的方法,其中所述方法包括确定组合生物标记物,其包括:
[0393]
(a)标志1分数(例如,其中基因激活与内皮细胞标志激活相关的标志);和
[0394]
(b)标志2分数(例如,其中激活与炎性和免疫细胞标志激活相关的标志),其中
[0395]
(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且
[0396]
(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定。
[0397]
在一些方面,标志1分数使用选自表3的基因套组来确定,其中所述基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个选自表1的基因。
[0398]
在一些方面,选自表3的基因套组包括abcc9、afap1l2、bace1、bgn、bmp5、col4a2、col8a1、col8a2、cpxm2、cxcl12、ebf1、ecm2、ednra、eln、epha3、fbln5、gnas、gnb4、gucy1a3、hey2、hspb2、il1b、itga9、itpr1、jam2、jam3、kcnj8、lamb2、lhfp、ltbp4、meox1、mgp、mmp12、mmp13、naalad2、nfatc1、nov、olfml2a、pcdh17、pde5a、pdgfrb、peg3、plscr2、plxdc2、rgs4、rgs5、rnf144a、rras、runx1t1、cav2、selp、serpine2、sgip1、smarca1、spon1、stab2、steap4、tbx2、tek、tgfb2、tmem204、ttc28和utrn;或其任何组合。
[0399]
在一些方面,选自表3的基因套组由以下组成:abcc9、afap1l2、bace1、bgn、bmp5、col4a2、col8a1、col8a2、cpxm2、cxcl12、ebf1、ecm2、ednra、eln、epha3、fbln5、gnas、gnb4、gucy1a3、hey2、hspb2、il1b、itga9、itpr1、jam2、jam3、kcnj8、lamb2、lhfp、ltbp4、meox1、mgp、mmp12、mmp13、naalad2、nfatc1、nov、olfml2a、pcdh17、pde5a、pdgfrb、peg3、plscr2、plxdc2、rgs4、rgs5、rnf144a、rras、runx1t1、cav2、selp、serpine2、sgip1、smarca1、spon1、stab2、steap4、tbx2、tek、tgfb2、tmem204、ttc28和utrn。
[0400]
在一些方面,标志2分数使用选自表4的基因套组来确定,其中所述基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60或61个选自表2的基因。
[0401]
在一些方面,选自表4的基因套组包括例如agr2、c11orf9、dusp4、eif5a、etv5、
gad1、iqgap3、mst1、mt2a、mta2、pla2g4a、reg4、srsf6、strn3、trim7、usf1、zic2、c10orf54、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd8b、ctla4、cxcl10、ifna2、ifnb1、ifng、lag3、pdcd1、pdcd1lg2、tgfb1、tigit、tnfrsf18、tnfrsf4、tnfsf18、tlr9、havcr2、cd79a、cxcl11、cxcl9、gzmb、ido1、igll5、adamts4、capg、ccl2、ctsb、folr2、hfe、hmox1、hp、igfbp3、mest、plau、rac2、rnh1、serpine1和timp1;或其任何组合。
[0402]
在一些方面,选自表4的基因套组由以下组成:agr2、c11orf9、dusp4、eif5a、etv5、gad1、iqgap3、mst1、mt2a、mta2、pla2g4a、reg4、srsf6、strn3、trim7、usf1、zic2、c10orf54、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd8b、ctla4、cxcl10、ifna2、ifnb1、ifng、lag3、pdcd1、pdcd1lg2、tgfb1、tigit、tnfrsf18、tnfrsf4、tnfsf18、tlr9、havcr2、cd79a、cxcl11、cxcl9、gzmb、ido1、igll5、adamts4、capg、ccl2、ctsb、folr2、hfe、hmox1、hp、igfbp3、mest、plau、rac2、rnh1、serpine1和timp1。
[0403]
在一些方面,标志1基因可以是血管生成生物标记物。如本文所用,术语“血管生成生物标记物”是指在肿瘤或其基质中差异表达的生物标记物(例如,核酸生物标记物,例如,rna生物标记物),其包括相对于可比较的非癌组织或参考样品的病理学水平的血管生成。示例性血管生成生物标记物在表1中列出。在一些方面,肿瘤或其基质可以表现出表1中列出的多种生物标记物的表达水平的明显升高或降低。
[0404]
在一些方面,肿瘤或其基质表现出例如相对于患有癌症的患者群体的中值水平,表1中列出的生物标记物的至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98、至少约99%或100%的明显升高或降低。
[0405]
在一些方面,标志2基因可以是免疫生物标记物。如本文所用,术语“免疫生物标记物”是指在肿瘤或其基质中差异表达的生物标记物(例如,核酸生物标记物,例如,rna生物标记物),其包括相对于可比较的一个或多个参考样品增加的免疫浸润,使得如果用免疫疗法治疗肿瘤,则可以诱导免疫应答。示例性免疫生物标记物在表2中列出。在一些方面,肿瘤或其基质可以表现出表2中列出的多种生物标记物的表达水平的明显升高或降低。
[0406]
在一些方面,肿瘤或其基质表现出例如相对于患有癌症的患者群体的中值水平,表2中列出的生物标记物的至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约96%、至少约97%、至少约98、至少约99%或100%的明显升高或降低。
[0407]
在本文公开的特定方面,使用两个分类器:标志1分数(由测量对应于表1的生物标记物基因或其子集的表达水平导出);和标志2分数(由测量对应于表2的生物标记物基因或其子集的表达水平导出)。每个分类器考虑两种不同的状态(即,正分数或负分数取决于整合基因套组中的基因的表达值的分数是高于还是低于某个阈值)。这种方法允许将癌症样品分层为四种不同的time。
[0408]
如果将额外的基因套组并入本公开的基于群体的分类器,则tme分类的粒度增加。例如,使用三个标志分数,每个分数具有可能的正值或负值,允许将样品的群体分层为八种不同的tme。可替代地,如果本文所用的相同标志分数不仅具有正状态或负状态,还有基于
两个阈值落在例如3个范围内的额外状态,则粒度也将增加。除了使用多个阈值以外,可以基于其他标准对标志分数值进行分组,例如,基于观察到的分数值的分布,将分数分配到某个三分位数、四分位数或五分位数。
[0409]
应理解,虽然如ann方法使用的标志1和标志2的基因已被证明具有预测性,但是ann方法具有与其他tme的其他基因标志(每个基因标志由包括表1和/或表2中公开的基因的子集的基因套组定义)一起使用的能力,所述其他tme例如本文公开的四种tme、其组合或通过将不同的阈值应用于ann输出或例如使用不同的ann架构、权重或激活函数产生的其他tme。ann方法还具有与标志1和2,任选地与如上所述的其他tme的基因标志和/或与基因活性(例如,表达活性和/或分子生物标记物的表达水平)的一种或多种简化测量组合使用的能力。
[0410]
增加基于群体的分类器的粒度可以增加所选疗法的精度和功效。例如,使用本文公开的分类器(标志1和标志2)但具有三种状态(例如,由两个不同阈值确定的三个范围)将允许将癌症样品群体分层为九种不同的tme。tme群体分类的粒度的这种增加也与治疗选择的粒度增加相关联;换言之,将癌症样品的tme分类为更大数量的tme将允许更精确地确定最佳治疗。例如,将tme分类为四种tme可以足以确定抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)通常是最佳治疗选择,但将tme分类为更大数量的tme可以足以将某种抗pd1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)或某种抗血管生成剂,例如tki抑制剂精准确定为最佳治疗选择。因此,在一些方面,可以通过增加tme类别的数量来增加分类的粒度。在一些方面,分类的粒度还可以通过包括tme类别的组合来增加,例如,将癌症样品分类为对于2种(例如,id和is生物标记物阳性)、3种(例如,id、ia和is生物标记物阳性)或更多种tme类别是生物标记物阳性的。
[0411]
i.c.1分数计算和分类
[0412]
本公开提供了创建能够将基因表达样品分层(或分类)为若干tme类别或其组合的基于群体的z分数分类器(或分类器集)的方法。术语“z分数”,在本领域中也被称为标准分数、z值或正态分数等术语,是用于指示标准偏差的有符号分数的无量纲量,通过其一个事件是高于正在测量的平均值。高于平均值的值具有正z分数,而低于平均值的值具有负z分数。
[0413]
在特定方面,本公开的基于群体的分类器包括两个分类器(标志1和标志2),每个分类器具有两种可能的状态(正或负),其可以将基因表达样本的群体分层为四种不同的tme类别。本公开的基于群体的z分数分类器还能够将患有癌症的受试者的测试样品分类为一种特定tme类别或其组合。根据将受试者的样品分配到特定tme类别或其组合,可以选择已知具有有效治疗受试者的癌症的高可能性的个性化治疗。如本文所用,tme分类也可以被称为基质类型、基质亚型、基质表型或其变体。在一些方面,将不同的权重和参数应用于z分数的计算和/或应用不同的阈值,可以将受试者的样品分配到两种或更多种tme。因此,在一些方面,根据是否考虑分配到两种或更多种tme类别,可以将基因表达样品的群体分层为多于四种不同的tme类别,例如,分层为四种公开的不同tme类别(a、is、id和ia)和/或其组合。
[0414]
i.c.1.a样品分类。
[0415]
可以使用基于群体的分类器,即基于数据(例如,与特定癌症、生物标记物表达水平、治疗和这些治疗的结果相关的参数)的分类系统,将样品分类或分层为特定tme。在一些
方面,本文公开的基于群体的分类器(或基于群体的方法)假定基因表达水平的以零为中心的正态分布(μ=0)。
[0416]
在本文公开的基于群体的分类器的特定方面,在整个患者群体中如上公开确定从表1或表2获得的基因套组或图28a-g中公开的基因套组(基因集)中的任一个的表达水平。在整个患者群体中,根据每个基因的表达水平计算此基因的平均值和标准偏差。这些值可以存储起来,以备将来用作基因套组中的每个基因的参考值。
[0417]
从个体患者样品(测试样品)中,可以针对基因套组中的每个基因确定患者的标准化表达水平。从基因套组中的每个基因的患者表达水平中减去群体平均值。然后将所得值除以此特定基因标准偏差,以产生套组中的此基因的z分数。在一些方面,没有对于对自由度的校正。在其他方面,具有对于对自由度的校正。
[0418]
将对应于基因套组中的基因所有z分数相加,并且然后除以基因数的平方根。结果是根据等式1的激活分数zs(标志值):
[0419][0420]
其中z是指z分数,s是指样品(患者),g是指基因,并且g是指标志基因集(即,基因套组)。|g|指示基因集g(即,基因套组)的大小。z
s,g
是描述远离群体的平均值的量级和方向的向量,并且是无单位的;激活分数zs也是无单位的。
[0421]
当激活分数(即,标志值)等于或大于零时,即zs》=0,则称此标志为正。当激活分数(即,标志值)低于零时,即zs《0,则称此标志为负。
[0422]
在一些方面,标志分数(例如,标志1或标志2)的计算包括:
[0423]
(i)测量来自所述受试者的测试样品中的基因套组中的每个基因的表达水平(例如,mrna表达水平);
[0424]
(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
[0425]
(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
[0426]
(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以基因套组中的基因数的平方根,
[0427]
其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
[0428]
在一些方面,来自受试者的测试样品中的基因套组中的每个基因的表达水平与群体数据(例如,来自本公开的实施例部分中公开的公共数据集的表达数据)合并。
[0429]
应理解,以上公式的变化是可能的,例如,通过对若干基因的表达水平进行分组(例如,按基因家族,或按共同的功能属性,诸如编码与相同受体结合的配体的若干基因)和/或为表达值或z分数分配权重,和/或应用基因特异性阈值。
[0430]
这种基于群体的分类器的概括是并非将患者z分数与零比较,而是与标志特异性阈值(“阈值”)进行比较,其中zs》=阈值意指标志为正(+),并且zs《阈值意指标志为负(-)。取决于被建模的疾病,阈值是分类器的超参数。阈值影响基于群体的分类器的灵敏度和特异性。
[0431]
因此,在一些方面,激活分数zs(标志值)是根据等式2计算的,其中t是适用于激活分数的阈值。
[0432][0433]
在一些方面,激活分数阈值是约+0.01、约+0.02、约+0.03、约+0.04、约+0.05、约+0.06、约+0.07、约+0.08、约+0.09、约+0.10、约+0.15、约+0.20、约+0.25、约+0.30、约+0.35、约+0.40、约+0.45、约+0.50、约+0.55、约+0.60、约+0.65、约+0.70、约+0.75、约+0.80、约+0.85、约+0.90、约+0.95、约+1、约+2、约+3、约+4、约+5、约+6、约+7、约+8、约+9、约+10或高于+10。
[0434]
在一些方面,激活分数阈值是约-0.01、约-0.02、约-0.03、约-0.04、约-0.05、约-0.06、约-0.07、约-0.08、约-0.09、约-0.10、约-0.15、约-0.20、约-0.25、约-0.30、约-0.35、约-0.40、约-0.45、约-0.50、约-0.55、约-0.60、约-0.65、约-0.70、约-0.75、约-0.80、约-0.85、约-0.90、约-0.95、约-1、约-2、约-3、约-4、约-5、约-6、约-7、约-8、约-9、约-10或低于-10。
[0435]
因此,在一些方面,激活分数zs(标志值)是根据等式3计算的,其中t是适用于套组中的每个基因的独立阈值。
[0436][0437]
在一些方面,基因特异性阈值可以是大于平均值至少约5%、至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、或至少约45%,或为零。
[0438]
在一些方面,基因特异性阈值也可以是小于平均值至少约5%、至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、或至少约45%,或为零。
[0439]
在一些方面,无单位的基因特异性阈值可以是约0.05、约0.10、约0.15、约0.20、约0.25、约0.30、约0.35、约0.40、约0.45、约0.50、约0.55、约0.60、约0.65、约0.70、约0.75、约0.80、约0.85、约0.90、约0.95或约1.00或大于平均值,或为零。
[0440]
在一些方面,无单位的基因特异性阈值可以是约0.05、约0.10、约0.15、约0.20、约0.25、约0.30、约0.35、约0.40、约0.45、约0.50、约0.55、约0.60、约0.65、约0.70、约0.75、约0.80、约0.85、约0.90、约0.95或约1.00或小于平均值,或为零。
[0441]
在又其他方面,激活分数zs(标志值)是根据等式4计算的,其中t1是适用于套组中的每个基因的独立阈值,并且t2是适用于激活分数的第二阈值。
[0442][0443]
在一些方面,相同的阈值可以应用于基于群体的分类器中的每个标志,例如标志1
和标志2。在其他方面,不同的阈值可以应用于基于群体的分类器中的每个标志,例如标志1和标志2。因此,在本公开的特定方面,对于标志1和标志2,阈值可以是不同的。
[0444]
在一些方面,可以根据替代方法计算标志分数,诸如:
[0445]
·
标志分数=sum(测试表达式值-参考表达式值),其可以》0或《0。
[0446]
·
标志分数=相对于阈值(测试表达值-参考表达值)的分布的平均值。如果高于阈值,则是正值。如果低于阈值,则是负值。
[0447]
·
标志分数=相对于阈值(测试表达值-参考表达值)的分布的中值。如果高于阈值,则是正值。如果低于阈值,则是负值。
[0448]
在所有这些替代方法中,需要rna表达水平值的正态分布。
[0449]
可以通过将从患者样品获得的激活分数与图10中的表相关联来进行基于如本文公开的两个标志的基于群体的分类器的预后或预测,其提供四种tme(基质表型)。换言之,基于患者z分数的符号和所使用的阈值(例如,正或负zs),通过应用图10中的规则(基于加和标志1和标志2z分数的符号的患者分类规则),可以将患者分类为四种tme中的一种。这四种tme是:
[0450]
(a)ia(免疫活性型):通过负标志1和正标志2定义。
[0451]
(b)is(免疫抑制型):通过正标志1和正标志2定义。
[0452]
(c)id(免疫沙漠型):通过负标志1和负标志2定义。
[0453]
(d)a(血管生成型):通过正标志1和负标志2定义。
[0454]
is tme(基质表型)通常不包括ebv(埃-巴二氏病毒)阳性患者、msi-h(微卫星不稳定性生物标记物高)患者或pd-l1高患者。这些患者通常存在于iatme(基质表型)中。概括是说明性的而不是确定性的。因此,在一些方面,is患者不是ebv阳性患者。在一些方面,is患者不是msi-h患者。在一些方面,is患者不是pd-l1高患者。在一些方面,ia患者是ebv阳性患者。在一些方面,ia患者是msi-h患者。在一些方面,ia患者是pd-l1高患者。
[0455]
在一些方面,接受is类别tme疗法的患者不是ebv阳性患者。在一些方面,接受is类别tme疗法的患者不是msi-h患者。在一些方面,接受is类别tme疗法的患者不是pd-l1高患者。
[0456]
在一些方面,接受ia类别tme疗法的患者是ebv阳性患者。在一些方面,接受ia类别tme疗法的患者是msi-h患者。在一些方面,接受ia类别tme疗法的患者是pd-l1高患者。
[0457]
在一些方面,取决于将不同的权重和参数应用于z分数的计算和应用不同的阈值,可以将样品分配在两种或更多种tme中。在这些方面,肿瘤样品或患者对于两种或更多种tme是生物标记物阳性的,例如a和is生物标记物阳性的。因此,这种肿瘤或患者可以用本文公开的两种或更多种tme类别疗法治疗,例如作为组合疗法,其中每种tme类别疗法对应于肿瘤样品或患者对于其是生物标记物阳性的tme中的一种。
[0458]
对于以免疫活性为主的tme,诸如ia(免疫活性)表型,具有这种生物学的患者对于抗pd1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、抗pd-l1、抗ctla4(检查点抑制剂或cpi)或rorγ激动剂治疗剂(下文更全面描述的所有基质亚型的所有治疗剂)可以具有应答性。
[0459]
对于以血管生成活性为主的tme,诸如被分类为a(血管生成型)表型的患者,具有这种生物学的患者对于vegf靶向疗法、dll4靶向疗法、血管生成素/tie2靶向疗法、抗vegf/
抗dll4双特异性抗体(诸如那赛昔珠单抗)以及抗vegf抗体(诸如伐利苏单抗或贝伐珠单抗)可以具有应答性。
[0460]
对于以免疫抑制为主的tme,这种被分类为is(免疫抑制型)表型的患者对于检查点抑制剂可以具有耐药性,除非还被给予逆转免疫抑制的药物,诸如抗磷脂酰丝氨酸(抗ps)治疗剂、pi3kγ抑制剂、腺苷通路抑制剂、ido、tim、lag3、tgfβ和cd47抑制剂。巴维妥昔单抗是优选的抗ps治疗剂。具有这种生物学的患者也具有潜在的血管生成,并且还可以受益于抗血管生成剂,诸如用于a基质亚型的那些。
[0461]
对于没有免疫活性的tme,诸如被分类为id(免疫沙漠型)表型的患者,具有这种生物学的患者对于检查点抑制剂、抗血管生成剂或其他tme靶向疗法没有应答,并且因此不应用作为单一疗法的抗pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、抗pd-l1、抗ctla-4或rorγ激动剂来治疗。具有这种生物学的患者可以用诱导免疫活性的疗法治疗,从而允许他们然后从检查点抑制剂中受益。对于这些患者可以诱导免疫活性的疗法包括疫苗、car-t、新表位疫苗(包括个性化疫苗)和基于tlr的疗法。
[0462]
在一方面,标志内的不同基因的子集可以同样具有预测性,因为此类基因代表广泛生物学的许多方面。因此,可以使用表1和表2的整个基因集(或图28a-g中公开的基因集中的任一个)或使用来自表1和表2的基因的子集(或来自图28a-g中公开的基因集中的任一个的基因的子集),例如表3和表4中公开的子集来生成如本文公开的四个tme分类器。
[0463]
在一些方面,本文公开的基于群体的分类器用于预后。在一些方面,本文公开的基于群体的分类器在临床环境中被预测性地使用,即,用作预测性生物标记物。
[0464]
在一些方面,如果分类器确定样品或患者对于本文公开的另外两种tme类别是生物标记物阳性的,则可以将群体分层为多于四种类别。例如,可以将群体分层为ia生物标记物阳性的、id生物标记物阳性的、a生物标记物阳性的、is生物标记物阳性的、ia和id生物标记物阳性的、ia和a生物标记物阳性的等。相反,可以将群体分层为ia生物标记物阴性的、id生物标记物阴性的、a生物标记物阴性的、is生物标记物阴性的、ia和id生物标记物阴性的、ia和a生物标记物阴性的等。
[0465]
i.d非基于群体的分类器
[0466]
在一些方面,本公开提供了创建能够将基因表达样品分层(或分类)为若干tme类别的非基于群体的分类器(或分类器集)的方法。四种tme(即,基质亚型或表型)的潜在肿瘤生物学:上文讨论的ia(免疫活性型)、id(免疫沙漠型)、a(血管生成型)和is(免疫抑制型)可以通过应用人工神经网络(ann)方法和其他机器学习技术来揭示。在一些方面,应用本文公开的方法可以将肿瘤样品或患者分类为多于一种本文公开的tme,例如,患者或样品对于两种或更多种tme可以是生物标记物阳性的。
[0467]
在本公开的上下文中,应理解术语分类器包括一个或多个分类器或分类器的组合,它们可以属于相同或不同的类别(例如,群体和/或非群体分类器,或非群体分类器的组合),其中术语分类器用于描述例如将测试样品分配到特定tme类别的数学模型的输出。
[0468]
虽然本文公开的基于群体的分类器依赖于具有许多患者的rna表达值的数据集,然后对这些患者进行分类,但是机器学习方法(例如,ann、逻辑回归或随机森林)复制、概括、再现和/或紧密估计基于群体的分类器的输出。
[0469]
例如,ann方法将本文公开的基因或其子集(即,特征)的基因表达值作为输入,并
且基于表达模式,鉴定具有主要是血管生成表达、主要是激活的免疫基因表达、这些表达模式中两者的混合或均不是两者的患者样品(即,患者)。这四种表型类型可预测对某些类型的治疗的应答。
[0470]
因此,在本公开的一些方面,将tme分类为is(免疫抑制型),如通过本文公开的机器学习方法(例如,ann)分配到患者样品(即,患者),意指患者具有激活的免疫基因表达和血管生成基因表达两者。
[0471]
a(血管生成型)tme分类,如通过本文公开的非基于群体的分类器(例如,ann)分配到患者样品,意指患者样品主要具有血管生成基因表达。ia(免疫活性型)tme分类,如通过本文公开的非基于群体的分类器(例如,ann)分配到患者样品,意指患者样品主要具有激活的免疫基因表达。id(免疫沙漠型)tme,如通过本文公开的非基于群体的分类器(例如,ann)分配到患者样品,意指患者样品没有、具有减少的、低的或非常低的免疫基因表达和血管生成免疫基因表达。
[0472]
在一些方面,本文公开的非基于群体的分类器是通过应用机器学习技术获得的分类器。在一些方面,机器学习技术选自由以下组成的组:逻辑回归、随机森林、人工神经网络(ann)、支持向量机(svm)、xgboost(xgb;针对速度和性能设计的梯度提升决策树的实现)、glmnet(通过惩罚最大似然拟合广义线性模型的包)、cforest(利用条件推理决策树作为基础学习器的随机森林和装袋集成算法的实现)、机器学习的分类和回归树(cart)、treebag(装袋,即引导聚集,用于提高回归和分类问题中模型准确性的算法,其从分离的训练数据子集建立多个模型并构建最终聚集模型)、k最近邻(knn)或其组合。
[0473]
逻辑回归通常被认为是小型数据集的最佳预测指标之一。然而,基于树的模型(例如,随机森林、extratrees)和ann可以揭示特征之间的潜在相互作用。但是,当相互作用很少时,逻辑回归和更复杂的模型具有类似的性能。
[0474]
本文公开的非基于群体的分类器可以用对应于对其获得对应于基因套组的基因表达数据,例如mrna表达数据的样品集的数据进行训练。例如,训练集包括来自表1和表2(或图28a-g中公开的基因套组(基因集)中的任一个)及其任何组合中呈现的基因的表达数据。在一些方面,基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、84、86、87、88、89、90、91、92、93、94、95、96、97、98、99或100个基因。在一些方面,基因套组包括多于100个基因。在一些方面,基因套组包括约10与约20个、约20与约30个、约30与约40个、约40与约50个、约50与约60个、约60与约70个、约70与约80个、约80与约90个、或约90与约100个之间的选自表1和表2(或图28a-g中公开的基因套组(基因集)中的任一个)的基因。
[0475]
在一些方面,训练数据集包括每个样品的另外的变量,例如根据本文公开的基于群体的分类器的样品分类。在其他方面,训练数据包括关于样品的数据,诸如向受试者施用的治疗类型、剂量、剂量方案、施用途径、协同疗法的存在或不存在、对疗法的应答(例如,完全应答、部分应答或无应答)、年龄、体重、性别、种族、肿瘤大小、肿瘤分期、生物标记物的存在或不存在等。
[0476]
在一些方面,如本领域技术人员将理解的,基于包括p值、倍数变化和变异系数的
因素的组合选择训练数据集的基因是有帮助的。在一些方面,使用一个或多个选择标准和随后的排序允许选择前2.5%、5%、7.5%、10%、12.5%、15%、17.5%、20%、30%、40%、50%或更多的基因套组中的排序基因用于输入模型中。如将理解的,因此可以选择表1和2表中所有个体鉴定的基因或基因子集,并且测试所选基因的所有可能组合以鉴定有用的基因组合以产生预测模型。确定组合测试的所选个体基因的数量以及选择可能的基因组合的数量的选择标准将取决于可用于获得基因数据的资源和/或可用于计算和评估由模型产生的分类器的计算机资源。
[0477]
在一些方面,基于机器学习模型的训练结果,基因可能看起来是驱动基因。如本文所用,术语“驱动基因”是指包括驱动基因突变的基因。在一些方面,驱动基因是其中一种或多种获得性突变,例如驱动基因突变,可以与癌症进展有因果关系的基因。在一些方面,驱动基因可以调控一种或多种细胞过程,包括:细胞命运决定、细胞存活和基因组维护。驱动基因可以与一种或多种信号传导通路相关联(例如,可以调控),例如tgf-β通路、mapk通路、stat通路、pi3k通路、ras通路、细胞周期通路、细胞凋亡途径、notch通路、hedgehog(hh)通路、apc通路、染色质修饰通路、转录调控通路、dna损伤控制通路或其组合。示例性驱动基因包括癌基因和肿瘤抑制因子。在一些方面,驱动基因为其所存在于的细胞提供选择性生长优势。在一些方面,驱动基因为其所存在于的细胞提供增殖能力,例如,允许细胞扩增,例如克隆扩增。在一些方面,驱动基因是癌基因。在一些方面,驱动基因是肿瘤抑制因子基因(tsg)。
[0478]
基因集中存在嘈杂的低表达基因可能降低模型的灵敏度。因此,在一些方面,可以从机器学习模型中降低权重或过滤(消除)低表达基因。在一些方面,低表达基因过滤是基于由基因表达(例如,rna水平)计算的统计数据。在一些方面,低表达基因过滤是基于例如基因集中每个基因的原始读取计数的最小值(min)、最大值(max)、平均值(mean)、方差(sd)或其组合。对于每个基因集,可以确定最佳过滤阈值。在一些方面,优化过滤阈值以最大化基因集中差异表达基因的数量
[0479]
随后可以通过确定分类器正确调用每个测试受试者的能力来评估通过本文公开的机器学习方法(例如,ann)生成的非基于群体的分类器。在一些方面,用于导出模型的训练群体的受试者不同于用于测试模型的测试群体的受试者。如本领域技术人员将理解的,这允许人们预测用于训练分类器的基因集的能力,以使其能够正确地表征其基质表型性状表征(例如,tme类别)未知的受试者。
[0480]
输入数学模型中的数据可以是代表被评估的基因产物(例如,mrna)的表达水平的任何数据。根据本公开可用的数学模型包括使用监督和/或无监督学习技术的那些。在本公开的一些方面,所选择的数学模型使用监督学习结合“训练群体”来评估可能的生物标记物组合中的每一个。在一方面,所使用的数学模型选自以下:回归模型、逻辑回归模型、神经网络、聚类模型、主成分分析、最近邻分类器分析、线性判别分析、二次判别分析、支持向量机、决策树、遗传算法、使用装袋的分类器优化、使用boosting的分类器优化、使用随机子空间方法的分类器优化、投影追踪、遗传编程和加权投票。在一些方面,使用逻辑回归模型。在一些方面,使用决策树模型。在一些方面,使用神经网络模型。
[0481]
将本公开的数学模型(例如,ann模型)应用于数据的结果将生成使用一个或多个基因套组的一个或多个分类器。在一些方面,创建了满足给定目的(例如,对tme,即基质表
型进行正确分层)的多个分类器。在这种情况下,在一些方面,生成利用多于一个分类器的公式。例如,可以生成一个公式,它利用串联的分类器(例如,首先获得分类器a的结果,然后是分类器b;例如,分类器a区分tme;并且然后分类器b确定是否将特定处理分配到这种tme)。在另一方面,可以生成通过对多于一个分类器的结果进行加权而产生的公式。其他可能的分类器组合和加权将被理解并涵盖在本文中。在一些方面,应用于相同分类器的不同截止值或应用于相同样品的不同分类器可能导致将样品分类为不同的基质表型。换言之,取决于阈值和/或分类器的组合,样品可以被分类为两种或更多种基质表型(tme),并且因此样品对于本文公开的ia、id、is或a tme类别或其任何组合可以是生物标记物阳性的(例如,受试者可以是a和is生物标记物阳性的和id和ia生物标记物阴性的)。
[0482]
根据本文公开的方法生成的分类器,例如基于非群体的分类器(例如,ann模型)可以用于测试未知或测试受试者。在一方面,由本文鉴定的机器学习方法(例如,ann)生成的模型可以检测个体是否具有特定tme。在一些方面,所述模型可以预测受试者是否将对特定疗法产生应答。在其他方面,所述模型可以选择或用于选择用于施用特定疗法的受试者。
[0483]
在本公开的一方面,使用本领域技术人员已知的方法评估每个分类器正确表征训练群体的每个受试者的能力。例如,可以使用交叉验证、留一法交叉验证(loocv)、n重交叉验证或使用标准统计方法的刀切法分析(jackknife analysis)来评估分类器。在另一方面,评估每个分类器正确表征不用于生成分类器的训练群体的那些受试者的能力。
[0484]
在一些方面,可以使用一个数据集训练分类器,并且在另一个不同的数据集上评估分类器。因此,由于测试数据集与训练数据集不同,因此不需要交叉验证。
[0485]
在一方面,用于评估分类器正确表征训练群体的每个受试者的能力的方法是评估分类器的灵敏度(tpf,真阳性分数)和1-特异性(fpf,假阳性分数)的方法。在一方面,用于测试分类器的方法是接受者操作特征(“roc”),它提供若干参数来评估所生成模型(例如,由应用ann得到的模型)的结果的灵敏度和特异性两者。
[0486]
在一些方面,用于评估分类器正确表征训练群体的每个受试者的能力的度量包括分类准确度(acc)、接受者操作特征曲线下面积(auc roc)、灵敏度(真阳性分数,tpf)、特异性(真阴性分数,tnf)、阳性预测值(ppv)、阴性预测值(npv)或其任何组合。在一个特定方面,用于评估分类器正确表征训练群体的每个受试者的能力的度量是分类准确度(acc)、接受者操作特征曲线下面积(auc roc)、灵敏度(真阳性分数,tpf)、特异性(真阴性分数,tnf)、阳性预测值(ppv)和阴性预测值(npv)。
[0487]
在一些方面,训练集包括至少约10、至少约20、至少约30、至少约40、至少约50、至少约60、至少约70、至少约80、至少约90、至少约100、至少约110、至少约120、至少约130、至少约140、至少约150、至少约160、至少约170、至少约180、至少约190、至少约200、至少约250、至少约300、至少约350、至少约400、至少约450、至少约500、至少约600、至少约700、至少约800、至少约900或至少约1000个受试者的参考群体。
[0488]
在一些方面,本公开中鉴定的一些或所有基因(例如,表1和表2;或图28a-g中呈现的那些)的表达数据,例如mrna表达数据用于回归模型,诸如但不限于逻辑回归模型或线性回归模型,以便鉴定可用于对tme(即,基质表型)分类的分类器。所述模型用于测试表1和表2(或图28a-g)中鉴定的两种或更多种生物标记物基因的各种组合以生成分类器。在逻辑回归模型的情况下,分类器的结果呈等式的形式,所述等式提供因变量y,它表示给定表型(例
如,tme类别)的存在或不存在,其中表示等式中每个生物标记物基因的表达的数据乘以回归模型生成的加权系数。生成的分类器可以用于分析来自测试受试者的表达数据并提供指示测试受试者具有特定tme的概率的结果。
[0489]
通常,感兴趣的多元回归等式可以写成
[0490]
y=α+β1x1+β2x2+
…
+βkxk+ε
[0491]
其中因变量y指示与第一亚组相关联的生物学特征(例如,一种或多种病理的不存在或存在)的存在(当y为正时)或不存在(当y为负时)。此模型表明,因变量y取决于k个解释变量(来自参考群体中第一亚组和第二亚组的受试者的k个选择基因(例如,生物标记物基因)的测量特征值),加上涵盖各种未指定的省略因素的一个误差项。在以上鉴定的模型中,参数β1衡量第一解释变量x1对因变量y(例如,加权因子)的影响,保持其他解释变量不变。类似地,β2给出了解释变量x2对y的影响,保持其余解释变量不变。
[0492]
逻辑回归模型是线性回归的非线性转换。逻辑回归模型通常被称为“logit”模型并且可以表示为
[0493]
ln[p/(1-p)]=α+β1x1+β2x2+
…
+βkxk+ε
[0494][0495]
其中,
[0496]
α和ε是常数
[0497]
ln是自然对数loge,其中e=2.71828...,
[0498]
p是事件y发生的概率,p(y=1),
[0499]
p/(1-p)是“优势比”,
[0500]
ln[p/(1-p)]是对数优势比或“logit”,并且模型的所有其他成分与以上所述的一般线性回归等式相同。α和ε的项可以折叠成单个常数。在一些方面,使用单个项来表示α和ε。“逻辑”分布是s形分布函数。logit分布将估计的概率(p)限制在0与1之间。
[0501]
在一些方面,逻辑回归模型通过最大似然估计(mle)拟合。换言之,系数(例如,α、β1、β2、...)通过最大似然确定。似然是条件概率(例如,p(y|x),给定x的y的概率)。似然函数(l)测量观察到样品数据集中出现的特定因变量值集(y1、y2、...、yn)的概率。它被写为因变量乘积的概率:
[0502]
l=prob(y1*y2***yn)
[0503]
似然函数越高,在样品中观察到y的概率就越高。mle涉及找到使似然函数的对数(ll《0)尽可能大或似然函数的对数的-2倍(-2ll)尽可能小的系数(α、β1、β2、...)。在mle中,对参数α、β1、β2、...进行了一些初始估计。然后计算给定这些参数估计的数据的似然。参数估计得到改进,并且重新计算数据的似然。重复此过程,直至参数估计变化不大(例如,概率的变化小于.01或.001)。逻辑回归和拟合逻辑回归模型的实例见于hastie,the elements of statistical learning,springer,new york,2001,第95-100页。
[0504]
在另一方面,针对本公开的基因套组中的每个生物标记物基因测量的表达,例如mrna水平,可以用于训练神经网络。神经网络是一个两阶段回归或分类模型。神经网络可以是二进制或非二进制的。神经网络具有分层结构,其包括通过权重层连接到输出单元层的输入单元(和偏置)层。对于回归,输出单元层通常仅包括一个输出单元。然而,神经网络可
以无缝方式处理多个定量应答。因此,可以应用神经网络以允许鉴定区分为在多于两个群体(即,多于两个表型性状)之间的生物标记物,例如本文公开的四种tme类别。
[0505]
在一个特定实例中,可以使用来自从受试者群体获得的样品集的表1和表2(或图28a-g)中公开的生物标记物基因的产物(例如,mrna)的表达数据来训练神经网络,以鉴定特定于特定tme的生物标记物的那些组合。神经网络描述于duda等人,2001,pattern classification,第二版,john wiley&sons,inc.,new york;和hastie等人,2001,the elements of statistical learning,springer-verlag,new york。
[0506]
在一些方面,本文公开的神经网络,例如含有具有例如98或87个来自表1和表2(或来自图28a-g)的基因的单个输入层、2个神经元的单个隐藏层和单个输出层中的4个输出的反向传播神经网络(参见,例如abdi,1994,
″
a neural network primer
″
,j.biol system.2,247-283)可以使用easynn-plus版本4.0g软件包(neural planner software inc.)、scikit-learn(scikit-learn.org)或本领域已知的任何其他机器学习包或程序来实现。
[0507]
以上所述的模式分类和统计技术仅仅是可以用于构建可用于诊断或检测例如一种或多种病理的分类器的模型类型的实例,所述模型例如聚类,如例如描述于duda和hart,pattern classification and scene analysis,1973,john wiley&sons,inc.,new york的第211-256页;主成分分析,如例如描述于jolliffe,1986,principal component analysis,springer,new york;最近邻分类器分析,如例如描述于duda,pattern classification,第二版,2001,john wiley&sons,inc和hastie,2001,the elements of statistical learning,springer,new york);线性判别分析,如例如描述于duda,pattern classification,第二版,2001,john wiley&sons,inc;hastie,2001,the elements of statistical learning,springer,new york;或venables&ripley,1997,modern applied statistics with s-plus,springer,new york);支持向量机,如例如描述于cristianini和shawe-taylor,2000,an introduction to support vector machines,cambridge university press,cambridge,boser等人,1992,
″
a training algorithm for optimal margin classifiers,proceedings of the 5th annual acm workshop on computational learning theory,acm press,pittsburgh,pa,第142-152页;或vapnik,1998,statistical learning theory,wiley,new york。
[0508]
在一些方面,非基于群体的分类器包括由ann导出的模型。在一些方面,所述ann是前馈型神经网络。前馈型神经网络是一种人工网络,其中输入节点与输出节点之间的连接不形成循环。如ann的上下文中在此所用,术语“节点”和“神经元”可互换使用。因此,它不同于循环神经网络。在此网络中,信息仅在一个方向上移动,从输入节点向前通过隐藏节点(如果有的话)并到输出节点。所述网络中没有循环或环路。除输入节点外,每个节点都是使用非线性激活函数的神经元,所述函数被开发用于对生物神经元的动作电位或放电频率进行建模。
[0509]
在一些方面,ann是单层感知器网络,其由单层输出节点组成;输入通过一系列权重直接馈送到输出。在每个节点中计算权重和输入的乘积之和,并且如果所述值高于某个阈值(通常为0),则神经元放电并获取激活值(通常为1)。
[0510]
在一些方面,所述ann是多层感知器(mlp)。这类网络由通常以前馈方式互连的多
层计算单元组成。一个层中的每个神经元具有指向下一层神经元的连接。在许多应用中,这些网络的单元应用激活函数,例如sigmoid函数。mlp包括至少三层节点:输入层、隐藏层和输出层。
[0511]
在一些方面,激活函数是根据公式y(vi)=tanh(vi)描述的sigmoid函数,即范围从-1至+1的双曲正切。在一些方面,激活函数是根据公式y(vi)=(1+e-vi
)-1
描述的sigmoid函数,即形状类似于tanh函数但范围是从0至+1的逻辑函数。在这些公式中,yi是第i个节点(神经元)的输出,并且vi是输入连接的加权和。
[0512]
在一些方面,激活函数是整流线性单元(relu)或其变体,例如,噪声relu、泄漏relu、参数relu或指数lu。在一些方面,relu通过公式f(x)=x
+
=max(0,x)定义,其中x是神经元的输入。与双曲正切或逻辑sigmoid相比,relu激活函数能够更好地训练深度神经网络(dnn)。dnn是在输入层与输出层之间具有多层的ann。dnn通常是前馈型网络,其中数据从输入层流向输出层而不返回。由于增加了抽象层,dnn容易过度拟合,这使它们能够对训练数据中的罕见依赖关系进行建模。在一些方面,激活函数是softplus或smoothrelu函数,relu的平滑近似,其通过公式f(x)=ln(1+e
x
)描述。softplus的导数是逻辑函数。
[0513]
在一些方面,mlp包括三个层或更多层(具有一个或多个隐藏层的输入层和输出层)非线性激活节点。其多层和非线性激活将mlp与线性感知器区分开。它可以区分不是线性可分的数据。由于mlp是全连接的,因此一个层中的每个节点以一定的权重w
ij
连接到下一层中的每个节点。通过在处理每条数据之后基于输出中与预期结果相比的误差量改变连接权重,从而在感知器中进行学习。这是监督学习的实例,并且通过反向传播进行。
[0514]
在一些方面,mlp具有3个层。在其他方面,mlp具有多于3个层。在一些方面,mlp具有单个隐藏层。在其他方面,mlp具有多于一个隐藏层。
[0515]
在一些方面,输入层包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123、124、125、126、127、128、129、130、131、132、133、134、135、136、137、138、139、140、141、142、143、144、145、146、147、148、149或150个神经元。
[0516]
在一些方面,输入层包括70与100个之间的神经元。在一些方面,输入层包括70与80个之间的神经元。在一些方面,输入层包括80与90个之间的神经元。在一些方面,输入层包括90与100个之间的神经元。在一些方面,输入层包括70与75个之间的神经元。在一些方面,输入层包括75与80个之间的神经元。在一些方面,输入层包括80与85个之间的神经元。在一些方面,输入层包括85与90个之间的神经元。在一些方面,输入层包括90与95个之间的神经元。在一些方面,输入层包括95与100个之间的神经元。
[0517]
在一些方面,输入层包括至少约1至至少约5个之间、至少约5与至少约10个之间、至少约10与至少约15个之间、至少约15与至少约20个之间、至少约20与至少约25个之间、至少约25与至少约30个之间、至少约30与至少约35个之间、至少约35与至少约40个之间、至少约40与至少约45个之间、至少约45与至少约50个之间、至少约50与至少约55个之间、至少约55与至少约60个之间、至少约60与至少约65个之间、至少约65与至少约70个之间、至少约70
与至少约75个之间、至少约75与至少约80个之间、至少约80与至少约85个之间、至少约85与至少约90个之间、至少约90与至少约95个之间、至少约95与至少约100个之间、至少约100与至少约105个之间、至少约105与至少约110个之间、至少约110与至少约115个之间、至少约115与至少约120个之间、至少约120与至少约125个之间、至少约125与至少约130个之间、至少约130与至少约135个之间、至少约135与至少约140个之间、至少约140与至少约145个之间、或至少约145与至少约150个之间的神经元。
[0518]
在一些方面,输入层包括至少约1与至少约10个之间、至少约10与至少约20个之间、至少约20与至少约30个之间、至少约30与至少约40个之间、至少约40与至少约50个之间、至少约50与至少约60个之间、至少约60与至少约70个之间、至少约70与至少约80个之间、至少约80与至少约90个之间、至少约90与至少约100个之间、至少约100与至少约110个之间、至少约110与至少约120个之间、至少约120与至少约130个之间、至少约130与至少约140个之间、或至少约140与至少约150个之间的神经元。
[0519]
在一些方面,输入层包括至少约1与至少约20个之间、至少约20与至少约40个之间、至少约40与至少约60个之间、至少约60与至少约80个之间、至少约80与至少约100个之间、至少约100与至少约120个之间、至少约120与至少约140个之间、至少约10与至少约30个之间、至少约30与至少约50个之间、至少约50与至少约70个之间、至少约70与至少约90个之间、至少约90与至少约110个之间、至少约110与至少约130个之间、或至少约130与至少约150个之间的神经元。
[0520]
在一些方面,输入层包括多于约1、多于约5、多于约10、多于约15、多于约20、多于约25、多于约30、多于约35、多于约40、多于约45、多于约50、多于约55、多于约60、多于约65、多于约70、多于约75、多于约80、多于约85、多于约90、多于约95、多于约100、多于约105、多于约110、多于约115、多于约120、多于约125、多于约130、多于约135、多于约140、多于约145、或多于约150个神经元。
[0521]
在一些方面,输入层包括少于约1、少于约5、少于约10、少于约15、少于约20、少于约25、少于约30、少于约35、少于约40、少于约45、少于约50、少于约55、少于约60、少于约65、少于约70、少于约75、少于约80、少于约85、少于约90、少于约95、少于约100、少于约105、少于约110、少于约115、少于约120、少于约125、少于约130、少于约135、少于约140、少于约145、或少于约150个神经元。
[0522]
在一些方面,将权重应用于输入层中的每个神经元的输入。
[0523]
在一些方面,ann包括单个隐藏层。在一些方面,ann包括1、2、3、4、5、6、7、8、9或10个隐藏层。在一些方面,单个隐藏层包括1、2、3、4、5、6、7、8、9或10个神经元。在一些方面,单个隐藏层包括至少1个、至少2个、至少3个、至少4个、至少5个、至少6个、至少7个、至少8个、至少9个或至少10个神经元。在一些方面,单个隐藏层包括少于10、少于9、少于8、少于7、少于6、少于5、少于4或少于3个神经元。在一些方面,单个隐藏层包括2个神经元。在一些方面,单个隐藏层包括3个神经元。在一些方面,单个隐藏层包括4个神经元。在一些方面,单个隐藏层包括5个神经元。在一些方面,将偏差应用于隐藏层中的神经元。
[0524]
在一些方面,ann包括对应于不同tme的输出层中的四个神经元。在一些方面,输出层中的四个神经元对应于以上公开的四种tme,ia(免疫活性型)、is(免疫抑制型)、id(免疫沙漠型)和a(血管生成型)。
[0525]
在一些方面,输出层的分类被归一化为预测输出类别上的概率分布,并且分量将加起来为1,使得它们可以被解释为概率。
[0526]
在一些方面,通过应用逻辑回归函数支持将输出层值多类别分类为四种表型类别(ia、id、a和is)。在一些方面,通过应用逻辑回归分类器,例如softmax函数支持将输出层值多类别分类为四种表型类别(ia、id、a和is)。softmax将十进制概率分配到加起来为1.0的每个类别。在一些方面,使用逻辑回归分类器,诸如softmax函数,有助于更快地训练收敛。在一些方面,包括softmax函数的逻辑回归分类器是通过就输出层之前的神经网络层实现的。在一些方面,就在输出层之前的这种神经网络层具有与输出层相同数量的节点。
[0527]
在一些方面,取决于所使用的特定数据集,对逻辑回归分类器(例如,softmax函数)的结果应用各种截止值(参见,例如用于选择特定受试者群体的截止值,例如,对应于特定疗法的那些)。因此,应用不同的截止值集不仅可以将癌症或患者分类在以上公开的四种tme(ia(免疫活性型)、is(免疫抑制型)、id(免疫沙漠型)或a(血管生成型))中的一种中,也可以将癌症或患者分类在多于一种以上公开的tme中。因此,在一些方面,癌症或患者可以被分类为对于ia、is、id、a及其任何组合是生物标记物阳性的。相反,在一些方面,癌症或患者可以被分类为对于ia、is、id、a及其任何组合是生物标记物阴性的。
[0528]
在一些方面,本文公开的mlp ann的隐藏层中的两个神经元对应于在本公开的基于群体的分类器中鉴定的标志1和标志2,其可以用于生成训练数据集。
[0529]
在一些方面,标志1的所有基因或基因子集以及标志2的所有基因或基因子集在每个隐藏层的ann模型中具有正或负基因权重(图29)。
[0530]
在一些方面,本文公开的机器学习方法,例如本文公开的ann,已经使用下表中提供的基因集进行了训练。
[0531]
表5:用于机器学习(例如,ann)训练的基因集。
[0532]
[0533]
[0534]
[0535]
[0536]
[0537][0538]
本公开的机器学习模型的实际行为是表示呈压缩形式的高维数据。压缩数据可以在所谓的潜在空间中直观地表示。这种情况的常见实例是二维图(x轴和轴),其中每个患者被绘制为具有某个向量x和向量y的值。因此,潜在空间是通过本公开的方法生成的标志的投影,例如,是z分数的投影或是隐藏神经元的值的投影。在一些方面,潜在空间可以绘制为三维。
[0539]
每个患者的疾病分数值可以绘制在潜在空间中(即,ann模型的概率结果)。随着时间的推移,可以积累患者数据,或者可以将具有疾病分数的患者数据的回顾性分析结果用作参考图,在其上绘制受试者患者的ann概率结果。
[0540]
在一些方面,潜在空间是ann模型的隐藏神经元的图,并且可以包括这些神经元的所有2路组合。在一些方面,ann模型基于压缩两个隐藏神经元中的数据预测四种表型类别,并且在潜在空间中绘制这些神经元也用作四种输出表型类别的投影。在一些方面,每个患者的表型类别分配在神经元1相对于神经元2的潜在空间中可视化。
[0541]
潜在空间投影可以通过展示输出(表型)分配的概率轮廓来增强。以这种方式,投影不仅可以显示受试者落在潜在空间中的位置,还可以显示每个表型分类的置信度。在一些方面,临床报告可以使用表型类别作为生物标记物逻辑—即,ia=阳性,或ia+is=阳性—然后向临床医生报告表型分配的概率,这已经是模型的输出。潜在空间图还可以用于可视化此患者与决策边界的距离,以帮助临床决策者评估边缘情况和异常情况。
[0542]
在一些方面,tme表型类别之间的边界不在笛卡尔坐标轴(x=0,y=0)上,而是在图中的其他位置。
[0543]
在一些方面,第二模型可以从ann模型潜在空间中学习生物标记物边界。在一些方面,第二模型可以是逻辑回归模型。在一些方面,它可以是任何其他类型的回归或机器学习算法。在一些方面,可以将逻辑回归函数应用于潜在空间。在一些方面,组合表型以定义生物标记物阳性类别,即ia+is,个体表型分配的置信度不等于组合类别分配的置信度。逻辑
回归函数用于了解生物标记物阳性意味着什么,并且直接报告关于生物标记物阳性的统计数据。逻辑回归函数可以用于基于真实患者结果数据微调生物标记物阳性/阴性决策边界。在一些方面,可以通过根据二级模型对潜在空间进行剪切来提高ann模型的准确性。
[0544]
在一些方面,概率函数可以绘制为二维,一个轴表示信号由标志1的基因支配的概率,并且另一个轴表示信号由标志2的基因支配的概率。在一些方面,在血管生成和免疫功能中起作用的基因对每个概率函数都有贡献。潜在空间图的每个象限代表一种基质表型。在另一方面,通过使用逻辑回归来应用阈值。在一些方面,逻辑回归可以是线性的或多项式的。在设定阈值之后,可以根据本文所述的方法分析个体患者结果。
[0545]
i.e.tme特定治疗方法
[0546]
本公开提供了用于根据通过应用衍生自组合生物标记物(例如,对应于基因套组的基因表达数据集)的分类器产生的肿瘤微环境(tme)确定对患者和/或来自这些患者的癌症样品进行分类/分层的方法。在一些方面,分类器是本文公开的非基于群体的分类器,例如,ann模型。在其他方面,分类器是本文公开的基于群体的分类器,其例如整合若干标志分数(例如,在示例性方面中的标志1和标志2)。可以基于特定tme或其组合的存在的鉴定(即,对于本文公开的一种或多种基质表型,患者是否是生物标记物阳性的和/或生物标记物阴性的),选择优选疗法(例如,本文公开的tme类别疗法或其组合)来治疗患者的癌症。
[0547]
在一方面,本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“ia类别tme疗法”,其中在施用之前,通过基于群体的分类器将受试者鉴定为表现出包括以下的组合生物标记物:(a)负标志1分数;和(b)正标志2分数,其中(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定。
[0548]
在一方面,本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“ia类别tme疗法”,其中在施用之前,通过本文公开的非基于群体的分类器,例如ann分类器将受试者鉴定为表现出ia类别tme,其中ia类别tme的存在通过将ann分类器模型应用于数据集来确定,所述数据集包括从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集))的表达水平。
[0549]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0550]
(a)在施用之前通过基于群体的分类器鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[0551]
(a)负标志1分数;和
[0552]
(b)正标志2分数,
[0553]
其中
[0554]
(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且
[0555]
(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定;
[0556]
以及
[0557]
(b)向所述受试者施用ia类别tme疗法。
[0558]
还提供了一种用于鉴定罹患适合用ia类别tme疗法治疗的癌症的人受试者的方
法,所述方法包括:
[0559]
(i)通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平确定标志1分数;以及
[0560]
(ii)通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平确定标志2分数,
[0561]
其中在施用之前通过基于群体的分类器鉴定的包括:
[0562]
(a)负标志1分数;和
[0563]
(b)正标志2分数的组合生物标记物的存在
[0564]
指示可以施用ia类别tme疗法来治疗所述癌症。
[0565]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0566]
(a)在施用之前通过非基于群体的分类器(例如,ann)鉴定表现出ia类别tme的受试者,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;以及
[0567]
(b)向所述受试者施用ia类别tme疗法。
[0568]
在一些方面,如果受试者对于额外基质表型是生物标记物阳性的,则所述ia类别tme疗法可以与本文公开的额外tme类别疗法组合施用。
[0569]
还提供了一种用于鉴定罹患适合用ia类别tme疗法治疗的癌症的人受试者的方法,所述方法包括通过本文公开的非群体分类器(例如,ann)确定受试者中ia类别的存在,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;其中组合ia类别tme的存在指示可以施用ia类别tme疗法来治疗所述癌症。
[0570]
在一些方面,所述ia类别tme疗法包括检查点调节剂疗法。
[0571]
在一些方面,所述检查点调节剂疗法包括施用刺激性免疫检查点分子的激活剂。在一些方面,所述刺激性免疫检查点分子的激活剂是例如针对以下的抗体:gitr(糖皮质激素诱导的肿瘤坏死因子受体,tnfrsf18)、ox-40(tnfrsf4、act35、cd134、imd16、txgp1l、肿瘤坏死因子受体超家族成员4、tnf受体超家族成员4)、icos(诱导型t细胞共刺激物)、4-1bb(tnfrsf9、cd137、cdw137、ila、肿瘤坏死因子受体超家族成员9、tnf受体超家族成员9)或其组合。在一些方面,所述检查点调节剂疗法包括施用rorγ(rorc、nr1f3、rorg、rzr-gamma、rzrg、tor、rar相关孤儿受体γ、imd42、rar相关孤儿受体c)激动剂。
[0572]
在一些方面,所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是例如(1)针对pd-1(pdcd1、cd279、sleb2、hpd-1、hpd-l、hsle1、程序性细胞死亡1)的抗体,例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分;针对pd-l1(cd274、b7-h、b7h1、pdcd1l1、pdcd1lg1、pdl1、cd274分子、程序性细胞死亡配体1、hpd-l1)的抗体;针对pd-l2(pdcd1lg2、b7dc、btdc、cd273、pdcd1l2、pdl2、ba574f11.2、程序性细胞死亡1配体2)的抗体;针对ctla-4(ctla4、alps5、cd、cd152、celiac3、grd4、gse、iddm12、细胞毒性t淋巴细胞相关蛋白4)的抗体;双特异性抗体,其至少包含对单独pd-l1、pd-l2或ctla-4或其组合的结合特异性;或(2)与以下组合的抗体中的任一种:(1)tim-3(含t细胞免疫球蛋白和粘蛋白结构域-3)的抑制剂、lag-3(淋巴细胞激活基因3)的抑制剂、btla(b和t淋巴细胞衰减因子)的抑制剂、tigit(具有ig
和itim结构域的t细胞免疫受体)的抑制剂、vista(t细胞激活的v结构域ig抑制因子)的抑制剂、tgf-β(转化生长因子β)或其受体的抑制剂、cd86(分化簇86)激动剂、lair1(白细胞相关免疫球蛋白样受体1)的抑制剂、cd160(分化簇160)的抑制剂、2b4(自然杀伤细胞受体2b4;分化簇244)的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2(分化簇2)的抑制剂、cd27(分化簇27)的抑制剂、cds(cdp-二酰甘油合成酶1)的抑制剂、icam-1(细胞间粘附分子1)的抑制剂、lfa-1(淋巴细胞功能相关抗原1;cd11a/cd18)的抑制剂、icos(诱导型t细胞共刺激物;cd278)的抑制剂、cd30(分化簇30)的抑制剂、cd40(分化簇40)的抑制剂、baffr(b细胞激活因子受体)的抑制剂、hvem(疱疹病毒侵入介质)的抑制剂、cd7(分化簇7)的抑制剂、light(肿瘤坏死因子超家族成员14;tnfsf14)的抑制剂、nkg2c(杀伤细胞凝集素样受体c2;klrc2、cd159c)的抑制剂、slamf7(slam家族成员7)的抑制剂、nkp80(激活共受体nkp80;凝集素样受体f1;klrf1;杀伤细胞凝集素样受体f1)的抑制剂或其任何组合。
[0573]
在一些方面,所述检查点调节剂疗法包括施用tim-3的调节剂、lag-3的调节剂、btla的调节剂、tigit的调节剂、vista的调节剂、tgf-β或其受体的调节剂、cd86的调节剂、lair1的调节剂、cd160的调节剂、2b4的调节剂、gitr的调节剂、ox40的调节剂、4-1bb(cd137)的调节剂、cd2的调节剂、cd27的调节剂、cds的调节剂、icam-1的调节剂、lfa-1(cd11a/cd18)的调节剂、icos(cd278)的调节剂、cd30的调节剂、cd40的调节剂、baffr的调节剂、hvem的调节剂、cd7的调节剂、light的调节剂、nkg2c的调节剂、slamf7的调节剂、nkp80的调节剂或其组合。
[0574]
如本文所用,术语“调节剂”是指直接或间接与靶标相互作用并且对生物或化学过程或机制产生影响的分子。例如,调节剂可以使生物或化学过程或机制增加、促进、上调、激活、抑制、减少、阻断、阻止、延迟、脱敏、失活、下调等。因此,调节剂可以是靶标的“激动剂”或“拮抗剂”。术语“激动剂”是指增加蛋白质、受体、酶等的内源配体的至少一些作用的化合物。术语“拮抗剂”是指抑制蛋白质、受体、酶等的内源配体的至少一些作用的化合物。
[0575]
因此,在一些方面,所述检查点调节剂疗法包括施用tim-3的激动剂或拮抗剂、lag-3的激动剂或拮抗剂、btla的激动剂或拮抗剂、tigit的激动剂或拮抗剂、vista的激动剂或拮抗剂、tgf-β或其受体的激动剂或拮抗剂、cd86的激动剂或拮抗剂、lair1的激动剂或拮抗剂、cd160的激动剂或拮抗剂、2b4的激动剂或拮抗剂、gitr的激动剂或拮抗剂、ox40的激动剂或拮抗剂、4-1bb(cd137)的激动剂或拮抗剂、cd2的激动剂或拮抗剂、cd27的激动剂或拮抗剂、cds的激动剂或拮抗剂、icam-1的激动剂或拮抗剂、lfa-1(cd11a/cd18)的激动剂或拮抗剂、icos(cd278)的激动剂或拮抗剂、cd30的激动剂或拮抗剂、cd40的激动剂或拮抗剂、baffr的激动剂或拮抗剂、hvem的激动剂或拮抗剂、cd7的激动剂或拮抗剂、light的激动剂或拮抗剂、nkg2c的激动剂或拮抗剂、slamf7的激动剂或拮抗剂、nkp80的激动剂或拮抗剂或其任何组合。
[0576]
在一些方面,所述抗pd-1抗体包括例如纳武单抗、派姆单抗、西米普利单抗、信迪利单抗、替雷利珠单抗或其抗原结合部分。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、信迪利单抗或替雷利珠单抗交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、信迪利单抗或替雷利珠单抗结合相同的表位。
[0577]
在一些方面,所述抗pd-l1抗体包括例如阿维单抗、阿替利珠单抗、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗结合相同的表位。
[0578]
在一些方面,所述检查点调节剂疗法包括施用(i)抗pd-1抗体,例如选自由纳武单抗、派姆单抗、信迪利单抗、替雷利珠单抗和西米普利单抗组成的组中的抗体;(ii)抗pd-l1抗体,例如选自由阿维单抗、阿替利珠单抗和德瓦鲁单抗组成的组中的抗体;或(iii)其组合。
[0579]
本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“is类别tme疗法”,其中在施用之前,通过基于群体的分类器将受试者鉴定为表现出包括以下的组合生物标记物:(a)正标志1分数;和(b)正标志2分数,其中(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定。
[0580]
在一方面,本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“is类别tme疗法”,其中在施用之前,通过本文公开的非基于群体的分类器,例如ann分类器将受试者鉴定为表现出is类别tme,其中is类别tme的存在通过将ann分类器模型应用于数据集来确定,所述数据集包括从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平。
[0581]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括
[0582]
(a)在施用之前通过基于群体的分类器鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[0583]
(a)正标志1分数;和
[0584]
(b)正标志2分数,
[0585]
其中
[0586]
(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且
[0587]
(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定;
[0588]
以及
[0589]
(b)向所述受试者施用is类别tme疗法。
[0590]
还提供了一种用于鉴定罹患适合用is类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[0591]
(i)通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平确定标志1分数;以及
[0592]
(ii)通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平确定标志2分数,
[0593]
其中在施用之前通过基于群体的分类器鉴定的包括:
[0594]
(a)正标志1分数;和
[0595]
(b)正标志2分数的组合生物标记物的存在
[0596]
指示可以施用is类别tme疗法来治疗所述癌症。
[0597]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0598]
(a)在施用之前通过非基于群体的分类器(例如,ann)鉴定表现出is类别tme的受试者,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;以及
[0599]
(b)向所述受试者施用is类别tme疗法。
[0600]
在一些方面,如果受试者对于额外基质表型是生物标记物阳性的,则所述is类别tme疗法可以与本文公开的额外tme类别疗法组合施用。
[0601]
还提供了一种用于鉴定罹患适合用is类别tme疗法治疗的癌症的人受试者的方法,所述方法包括通过本文公开的非群体分类器(例如,ann)确定受试者中is类别的存在,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;其中组合is类别tme的存在指示可以施用is类别tme疗法来治疗所述癌症。
[0602]
在一些方面,所述is类别tme疗法包括例如施用(1)检查点调节剂疗法和抗免疫抑制疗法(例如,包括施用派姆单抗和巴维妥昔单抗的组合疗法)和/或(2)抗血管生成疗法。在一些方面,所述检查点调节剂疗法包括例如施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是例如针对pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体。
[0603]
在一些方面,所述抗pd-1抗体包括例如纳武单抗、派姆单抗、西米普利单抗、斯巴达珠单抗(pdr001)、信迪利单抗、替雷利珠单抗或杰诺单抗(cbt-501)或其抗原结合部分。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、pdr001、信迪利单抗、替雷利珠单抗或cbt-501交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、信迪利单抗、替雷利珠单抗、pdr001或cbt-501结合相同的表位。
[0604]
在一些方面,所述抗pd-l1抗体包括例如阿维单抗、阿替利珠单抗、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗结合相同的表位。
[0605]
在一些方面,所述抗ctla-4抗体包括伊匹单抗或其抗原结合部分。在一些方面,所述抗ctla-4抗体与伊匹单抗交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗结合相同的表位。
[0606]
在一些方面,所述检查点调节剂疗法包括例如施用(i)抗pd-1抗体,其例如选自由纳武单抗、派姆单抗、信迪利单抗、替雷利珠单抗和西米普利单抗组成的组;(ii)抗pd-l1抗体,其例如选自由阿维单抗、阿替利珠单抗和德瓦鲁单抗组成的组;(iii)抗ctla-4抗体,例如伊匹单抗,或(iii)其组合。
[0607]
在一些方面,所述抗血管生成疗法包括例如施用选自由以下组成的组中的抗vegf(血管内皮生长因子)抗体:伐利苏单抗、贝伐珠单抗、那赛昔珠单抗(抗dll4/抗vegf双特异性抗体)及其组合。在一些方面,所述抗血管生成疗法包括例如施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2血管内皮生长因子受体2)抗体。在一些方面,所述抗
vegfr2抗体包括雷莫卢单抗(ramucirumab)。在一些方面,所述抗血管生成疗法包括例如那赛昔珠单抗、abl101(nov1501)或迪帕西单抗(abt165)。
[0608]
在一些方面,所述抗免疫抑制疗法包括例如施用抗ps(磷脂酰丝氨酸)抗体、抗ps靶向抗体、结合β2-糖蛋白1的抗体、pi3kγ(磷脂酰肌醇-4,5-二磷酸3-激酶催化亚基γ同种型)的抑制剂、腺苷通路抑制剂、ido的抑制剂、tim的抑制剂、lag3的抑制剂、tgf-β的抑制剂、cd47抑制剂或其组合。
[0609]
在一些方面,所述抗ps靶向抗体是例如巴维妥昔单抗或结合β2-糖蛋白1的抗体。在一些方面,所述pi3kγ抑制剂是例如ly3023414(samotolisib)或ipi-549(eganelisib)。在一些方面,所述腺苷通路抑制剂是例如ab-928。在一些方面,所述tgfβ抑制剂是例如ly2157299(galunisertib)或tgfβr1抑制剂ly3200882。在一些方面,所述cd47抑制剂是例如莫洛利单抗(5f9)。在一些方面,所述cd47抑制剂靶向sirpα。
[0610]
在一些方面,所述免疫抑制疗法包括施用tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、cd86的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂或其组合。
[0611]
在一些方面,所述抗免疫抑制疗法包括施用tim-3的调节剂、lag-3的调节剂、btla的调节剂、tigit的调节剂、vista的调节剂、tgf-β或其受体的调节剂、cd86的调节剂、lair1的调节剂、cd160的调节剂、2b4的调节剂、gitr的调节剂、ox40的调节剂、4-1bb(cd137)的调节剂、cd2的调节剂、cd27的调节剂、cds的调节剂、icam-1的调节剂、lfa-1(cd11a/cd18)的调节剂、icos(cd278)的调节剂、cd30的调节剂、cd40的调节剂、baffr的调节剂、hvem的调节剂、cd7的调节剂、light的调节剂、nkg2c的调节剂、slamf7的调节剂、nkp80的调节剂或其组合。
[0612]
因此,在一些方面,所述抗免疫抑制疗法包括施用tim-3的激动剂或拮抗剂、lag-3的激动剂或拮抗剂、btla的激动剂或拮抗剂、tigit的激动剂或拮抗剂、vista的激动剂或拮抗剂、tgf-β或其受体的激动剂或拮抗剂、cd86的激动剂或拮抗剂、lair1的激动剂或拮抗剂、cd160的激动剂或拮抗剂、2b4的激动剂或拮抗剂、gitr的激动剂或拮抗剂、ox40的激动剂或拮抗剂、4-1bb(cd137)的激动剂或拮抗剂、cd2的激动剂或拮抗剂、cd27的激动剂或拮抗剂、cds的激动剂或拮抗剂、icam-1的激动剂或拮抗剂、lfa-1(cd11a/cd18)的激动剂或拮抗剂、icos(cd278)的激动剂或拮抗剂、cd30的激动剂或拮抗剂、cd40的激动剂或拮抗剂、baffr的激动剂或拮抗剂、hvem的激动剂或拮抗剂、cd7的激动剂或拮抗剂、light的激动剂或拮抗剂、nkg2c的激动剂或拮抗剂、slamf7的激动剂或拮抗剂、nkp80的激动剂或拮抗剂或其任何组合。
[0613]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“id类别tme疗法”,其中在施用之前,通过基于群体的分类器将受试者鉴定为表现出包括以下的组合生物标记物:(a)负标志1分数;和(b)负标志2分数,其中(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且(ii)标志2分数
通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定。
[0614]
在一方面,本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“id类别tme疗法”,其中在施用之前,通过本文公开的非基于群体的分类器,例如ann分类器将受试者鉴定为表现出id类别tme,其中id类别tme的存在通过将ann分类器模型应用于数据集来确定,所述数据集包括从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平。
[0615]
还提供了一种用于治疗罹患癌症的人受试者的方法,其包括
[0616]
(a)在施用之前通过基于群体的分类器鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[0617]
(a)负标志1分数;和
[0618]
(b)负标志2分数,
[0619]
其中
[0620]
(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且
[0621]
(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定;
[0622]
以及
[0623]
(b)向所述受试者施用id类别tme疗法。
[0624]
还提供了一种用于鉴定罹患适合用id类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[0625]
(i)通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平确定标志1分数;以及
[0626]
(ii)通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平确定标志2分数,
[0627]
其中在施用之前通过基于群体的分类器鉴定的包括:
[0628]
(a)负标志1分数;和
[0629]
(b)负标志2分数的组合生物标记物的存在
[0630]
指示可以施用id类别tme疗法来治疗所述癌症。
[0631]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0632]
(a)在施用之前通过非基于群体的分类器(例如,ann)鉴定表现出id类别tme的受试者,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;以及
[0633]
(b)向所述受试者施用id类别tme疗法。
[0634]
在一些方面,如果受试者对于额外基质表型是生物标记物阳性的,则所述id类别tme疗法可以与本文公开的额外tme类别疗法组合施用。
[0635]
还提供了一种用于鉴定罹患适合用id类别tme疗法治疗的癌症的人受试者的方法,所述方法包括通过本文公开的非群体分类器(例如,ann)确定受试者中id类别的存在,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;其中组合id类别tme的存在指示可以施用id类
别tme疗法来治疗所述癌症。
[0636]
在一些方面,所述id类别tme疗法包括在施用引发免疫应答的疗法的同时或之后施用检查点调节剂疗法。
[0637]
在一些方面,所述引发免疫应答的疗法是疫苗(例如,癌症疫苗)、car-t或新表位疫苗。
[0638]
在一些方面,所述检查点调节剂疗法在施用引发免疫应答的疗法同时或之后施用,并且包括例如施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是例如针对pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体。
[0639]
在一些方面,所述抗pd-1抗体包括例如纳武单抗、派姆单抗、西米普利单抗、pdr001或cbt-501或其抗原结合部分。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、pdr001、信迪利单抗、替雷利珠单抗或cbt-501交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与例如纳武单抗、派姆单抗、西米普利单抗、pdr001、信迪利单抗、替雷利珠单抗或cbt-501结合相同的表位。
[0640]
在一些方面,所述抗pd-l1抗体包括例如阿维单抗、阿替利珠单抗、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与例如阿维单抗、阿替利珠单抗或德瓦鲁单抗结合相同的表位。
[0641]
在一些方面,所述抗ctla-4抗体包括伊匹单抗或其抗原结合部分。在一些方面,所述抗ctla-4抗体与伊匹单抗交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗结合相同的表位。
[0642]
在一些方面,在施用引发免疫应答的疗法同时或之后施用的检查点调节剂疗法包括例如施用(i)抗pd-1抗体,其例如选自由纳武单抗、派姆单抗、信迪利单抗、替雷利珠单抗和西米普利单抗组成的组;(ii)抗pd-l1抗体,其例如选自由阿维单抗、阿替利珠单抗和德瓦鲁单抗组成的组;(iii)抗ctla-4抗体,例如伊匹单抗,或(iii)其组合。
[0643]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“a类别tme疗法”,其中在施用之前,通过基于群体的分类器将受试者鉴定为表现出包括以下的组合生物标记物:(a)正标志1分数;和(b)负标志2分数,其中(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定。
[0644]
在一方面,本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用“a类别tme疗法”,其中在施用之前,通过本文公开的非基于群体的分类器,例如ann分类器将受试者鉴定为表现出a类别tme,其中a类别tme的存在通过将ann分类器模型应用于数据集来确定,所述数据集包括从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平。
[0645]
还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0646]
(a)在施用之前通过基于群体的分类器鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[0647]
(a)正标志1分数;和
[0648]
(b)负标志2分数,
[0649]
其中
[0650]
(i)标志1分数通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平来确定;并且
[0651]
(ii)标志2分数通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平来确定;
[0652]
以及
[0653]
(b)向所述受试者施用a类别tme疗法。
[0654]
本公开还提供了一种用于鉴定罹患适合用a类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[0655]
(i)通过测量从受试者获得的第一样品中选自表3的基因套组的表达水平确定标志1分数;以及
[0656]
(ii)通过测量从受试者获得的第二样品中选自表4的基因套组的表达水平确定标志2分数,
[0657]
其中在施用之前通过基于群体的分类器鉴定的包括:
[0658]
(a)正标志1分数;和
[0659]
(b)负标志2分数的组合生物标记物的存在
[0660]
指示可以施用a类别tme疗法来治疗所述癌症。
[0661]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括:
[0662]
(a)在施用之前通过非基于群体的分类器(例如,ann)鉴定表现出a类别tme的受试者,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;以及
[0663]
(b)向所述受试者施用a类别tme疗法。
[0664]
在一些方面,如果受试者对于额外基质表型是生物标记物阳性的,则所述a类别tme疗法可以与本文公开的额外tme类别疗法组合施用。
[0665]
还提供了一种用于鉴定罹患适合用a类别tme疗法治疗的癌症的人受试者的方法,所述方法包括通过本文公开的非群体分类器(例如,ann)确定受试者中a类别的存在,如通过测量从受试者获得的样品中选自表1和表2的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)的表达水平来确定;其中组合a类别tme的存在指示可以施用a类别tme疗法来治疗所述癌症。
[0666]
在一些方面,所述a类别tme疗法包括vegf靶向疗法和其他抗血管生成剂、血管生成素1和2(ang1和ang2)、dll4(δ样典型notch配体4)、抗vegf和抗dll4的双特异性抗体、tki(酪氨酸激酶抑制剂)诸如呋喹替尼、抗fgf(成纤维细胞生长因子)抗体和抑制fgf受体家族(fgfr1和fgfr2)的抗体或小分子;抗plgf(胎盘生长因子)抗体和小分子抗体以及针对plgf受体的抗体、抗vegfb(血管内皮生长因子b)抗体、抗vegfc(血管内皮生长因子c)抗体、抗vegfd(血管内皮生长因子d);vegf/plgf捕获分子的抗体诸如阿柏西普或ziv-阿柏西普;抗dll4抗体或抗notch疗法,诸如γ-分泌酶的抑制剂。
[0667]
在一些方面,所述抗血管生成疗法包括施用内皮糖蛋白的拮抗剂,例如卡罗妥昔单抗(carotuximab,trc105)。
[0668]
如本文所用,术语“vegf靶向疗法”是指靶向配体,即vegf a(血管内皮生长因子a)、vegf b(血管内皮生长因子b)、vegf c(血管内皮生长因子c)、vegf d(血管内皮生长因子d)、或plgf(胎盘生长因子);受体,例如vegfr1(血管内皮生长因子受体1)、vegfr2(血管内皮生长因子受体2)或vegfr3(血管内皮生长因子受体3);或其任何组合。
[0669]
在一些方面,所述vegf靶向疗法包括施用抗vegf抗体或其抗原结合部分。在一些方面,所述抗vegf抗体包括例如伐利苏单抗、贝伐珠单抗或其抗原结合部分。在一些方面,所述抗vegf抗体与例如伐利苏单抗或贝伐珠单抗交叉竞争与人vegf a的结合。在一些方面,所述抗vegf抗体与例如伐利苏单抗或贝伐珠单抗结合相同的表位。
[0670]
在一些方面,所述vegf靶向疗法包括施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2抗体。在一些方面,所述抗vegfr2抗体包括雷莫卢单抗或其抗原结合部分。
[0671]
在一些方面,所述a类别tme疗法包括施用血管生成素/tie2(tek受体酪氨酸激酶;cdc202b)靶向疗法。在一些方面,所述血管生成素/tie2靶向疗法包括施用内皮糖蛋白和/或血管生成素。
[0672]
在一些方面,所述a类别tme疗法包括施用dll4靶向疗法。在一些方面,所述dll4靶向疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。
[0673]
在以上公开的所有方法,例如治疗受试者或选择用特定疗法治疗的受试者的方法中,其中特定疗法(例如,本文公开的tme类别疗法或其组合)根据使用本文公开的分类器(例如,本公开的基于群体和/或非基于群体的分类器)对癌症的tme(即,癌症对于本文公开的tme类别(即,基质表型)中的至少一种是生物标记物阳性的和/或生物标记物阴性的)进行分类来选择,施用特定疗法(例如,本文公开的tme类别疗法或其组合)可以有效地治疗癌症。
[0674]
在一些方面,施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时),可以减少癌症负荷。在一些方面,与施用疗法(例如,本文公开的tme类别疗法或其组合)之前的癌症负荷相比,向受试者施用本文公开的特定疗法,例如本文公开的tme类别疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)使癌症负荷减少至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%或约100%。
[0675]
在一些方面,施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)产生在初始施用之后至少约1个月、至少约2个月、至少约3个月、至少约4个月、至少约5个月、至少约6个月、至少约7个月、至少约8个月、至少约9个月、至少约10个月、至少约11个月、至少约一年、至少约18个月、至少约两年、至少约三年、至少约四年或至少约五年的无进展存活。
[0676]
在一些方面,在施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)之后,受试者表现出稳定疾病。术语“稳定疾病”是指对癌症存在的诊断,然而癌症已经被治疗并且保持在稳定的状态,即非进展的癌症,例如通过对数据成像和/或最
佳临床判断确定。术语“进展性疾病”是指对高度活跃状态的癌症的存在的诊断,即尚未治疗且不稳定或已经治疗且对疗法没有应答,或已经治疗且活跃疾病仍然存在的癌症,如通过对数据成像和/或最佳临床判断确定。
[0677]“稳定疾病”可以涵盖与治疗开始时(即,治疗之前)的初始肿瘤体积相比,在治疗过程中肿瘤体积的(暂时)肿瘤缩小/减小。在这种情况下,“肿瘤缩小”可以是指与治疗开始时(即,治疗之前)的初始体积相比,治疗之后肿瘤的体积减小。例如,小于100%(例如,治疗开始时初始体积的约99%至约66%)的肿瘤体积可以表示“稳定疾病”。
[0678]“稳定疾病”可以可替代地涵盖与治疗开始时(即,治疗之前)的初始肿瘤体积相比,在治疗过程中肿瘤体积的(暂时)肿瘤生长/增加。在这种情况下,“肿瘤生长”可以是指与治疗开始时(即,治疗之前)的初始体积相比,治疗抑制剂之后肿瘤的体积增加。例如,多于100%(例如,治疗开始时初始体积的约101%至约135%,优选地初始体积的约101%至约110%)的肿瘤体积可以表示“稳定疾病”。
[0679]
术语“疾病稳定”可以包括以下方面。例如,肿瘤体积例如在治疗之后不缩小(即,肿瘤生长停止),或者肿瘤例如在治疗开始时确实缩小但不继续缩小,直至肿瘤消失(即,肿瘤生长首先恢复,在肿瘤具有例如小于初始体积的65%之前,肿瘤再次生长。
[0680]
在参考患者或肿瘤使用时,术语对于本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)的“应答”可以在患者或肿瘤的“完全应答”或“部分应答”中反映。
[0681]
如本文所用,术语“完全应答”可以是指响应于本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时),癌症的所有现象消失。
[0682]
术语“完全应答”和术语“完全缓解”可以在本文中可互换使用。例如,“完全应答”可以在肿瘤的持续缩小(如所附实施例中所示)中反映,直至肿瘤消失。例如,与治疗开始时(即,之前)的初始肿瘤体积(100%)相比,例如0%的肿瘤体积可以表示“完全应答”。
[0683]
用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)进行的治疗可以产生“部分应答”(或部分缓解;例如,响应于治疗,肿瘤大小或体内癌症程度降低)。“部分应答”可以涵盖与治疗开始时(即,治疗之前)的初始肿瘤体积相比,在治疗过程中肿瘤体积的(暂时)肿瘤缩小/减小。
[0684]
因此,在一些方面,在施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)之后,受试者表现出部分应答。在其他方面,在施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)之后,受试者表现出完全应答。
[0685]
术语“应答”可以是指“肿瘤缩小”。因此,在一些方面,向有需要的受试者施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)可以使肿瘤体积减小或缩小。
[0686]
在一些方面,在施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合之后,相对于治疗之前的肿瘤体积,肿瘤的大小可以减小至少约5%、至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%或约100%。
[0687]
在一些方面,在施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)之后,肿瘤体积是治疗之前的初始肿瘤体积的至少约5%、至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%或至少约90%。
[0688]
在一些方面,相对于治疗之前的肿瘤生长速率,施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)可以使肿瘤生长速率减小至少约5%、至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%或约100%。
[0689]
术语“应答”也可以是指肿瘤数量的减少,例如,当癌症已经转移时。
[0690]
在一些方面,与未表现出组合生物标记物的受试者或未用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)治疗的受试者的无进展存活概率相比,施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)使受试者的无进展存活概率提高至少约10%、至少约15%、至少约20%、至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约100%、至少约105%、至少约110%、至少约115%、至少约120%、至少约12%、至少约130%、至少约135%、至少约140%、至少约145%或至少约150%。
[0691]
在一些方面,与未表现出组合生物标记物的受试者或未用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)治疗的受试者的总体存活概率相比,施用本文公开的特定疗法,例如ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别tme疗法或其组合(例如,当受试者对于多于一种基质表型是生物标记物阳性的时)使总体存活概率提高至少约25%、至少约30%、至少约35%、至少约40%、至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%、至少约95%、至少约100%、至少约110%、至少约120%、至少约125%、至少约130%、至少约140%、至少约150%、至少约160%、至少约170%、至少约175%、至少约180%、至少约190%、至少约200%、至少约210%、至少约220%、至少约225%、至少约230%、至少约240%、至少约250%、至少约260%、至少约270%、至少约275%、至少约
280%、至少约290%、至少约300%、至少约310%、至少约320%、至少约325%、至少约330%、至少约340%、至少约350%、至少约360%、至少约370%、至少约375%、至少约380%、至少约390%或至少约400%。
[0692]
本公开还提供了一种用于通过本文公开的基于群体的方法确定有需要的受试者中的肿瘤的肿瘤微环境(tme),即基质表型的基因套组,其至少包括来自表1的标志1生物标记物基因和来自表2的标志2生物标记物基因,其中所述肿瘤微环境或其组合(即,确定所述受试者对于本文公开的tme或其组合是生物标记物阳性的或生物标记物阴性的)用于(i)鉴定适合抗癌疗法的受试者;(ii)确定正在经历抗癌疗法的受试者的预后;(iii)启动、暂停或修改抗癌疗法的施用;或(iv)其组合。在一些方面,根据在此公开的方法使用所述基因套组,以例如对来自患者的肿瘤进行分类并基于此分类施用特定疗法(例如,本文公开的tme类别疗法或其组合)。
[0693]
本公开还提供了一种用于通过本文公开的非基于群体的方法,例如ann确定有需要的受试者中的肿瘤的肿瘤微环境(tme),即基质表型的基因套组,其至少包括来自表1的生物标记物基因和来自表2的生物标记物基因,其中特定肿瘤微环境或其组合的存在或不存在(即,确定所述受试者对于本文公开的tme或其组合是生物标记物阳性的或生物标记物阴性的)用于(i)鉴定适合抗癌疗法的受试者;(ii)确定正在经历抗癌疗法的受试者的预后;(iii)启动、暂停或修改抗癌疗法的施用;或(iv)其组合。在一些方面,根据在此公开的方法使用所述基因套组,以例如对来自患者的肿瘤进行分类(例如,确定肿瘤对于本文公开的tme或其组合是生物标记物阳性的或生物标记物阴性的)并基于此分类施用特定疗法(例如,本文公开的tme类别疗法或其组合)。
[0694]
本公开还提供了一种用于通过基于群体的分类器鉴定罹患适合用抗癌疗法治疗的癌症的人受试者的组合生物标记物,其中所述组合生物标记物包括在从所述受试者获得的样品中测量的标志1分数和标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中表3的基因套组中的基因的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中表4的基因套组中的基因的表达水平来确定,并且其中(a)如果标志1分数为负并且标志2分数为正,则所述疗法是ia类别tme疗法;(b)如果标志1分数为正并且标志2分数为正,则所述疗法是is类别tme疗法;(c)如果标志1分数为负并且标志2分数为负,则所述疗法是id类别tme疗法;或者(d)如果标志1分数为正并且标志2分数为负,则所述疗法是a类别tme疗法。在一些方面,例如,当受试者通过基于群体的分类器被鉴定为对于本文公开的多于一种基质表型是生物标记物阳性的或生物标记物阴性的,例如,受试者对于ia和is是生物标记物阳性的时,可以向受试者施用对应于受试者对于其是生物标记物阳性的基质表型的组合疗法,例如,包括ia类别tme疗法和is类别tme疗法的组合疗法。
[0695]
本公开还提供了一种用于通过非基于群体的分类器(例如,ann)鉴定罹患适合用抗癌疗法治疗的癌症的人受试者的组合生物标记物,其中所述癌症的tme(即,基质表型)通过测量从所述受试者获得的样品中从表1和表2获得的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)或图28a-g中公开的基因套组(基因集)中的任一个中的基因的表达水平,例如mrna表达水平来确定,并且其中(a)如果tme被分配在ia类别中,则所述疗法是ia类别tme疗法;(b)如果tme被分配在is类别中,则所述疗法是is类别tme疗法;(c)如果tme被分配在id上,则所述疗法是id类别tme疗法;或者(d)如果所分配的tme是a类别,则所
述疗法是a类别tme疗法。在一些方面,例如,当受试者通过基于非群体的分类器(例如,ann)被鉴定为对于本文公开的多于一种基质表型是生物标记物阳性的或生物标记物阴性的,例如,受试者对于ia和is是生物标记物阳性的时,可以向受试者施用对应于受试者对于其是生物标记物阳性的基质表型的组合疗法,例如,包括ia类别tme疗法和is类别tme疗法的组合疗法。
[0696]
本公开还提供了一种用于治疗有需要的人受试者中的癌症的抗癌疗法,其中所述受试者通过基于群体的分类器被鉴定为表现出(即,是生物标记物阳性的)或未表现出(即,是生物标记物阴性的)包括标志1分数和标志2分数的组合生物标记物,其中(i)标志1分数通过测量从所述受试者获得的第一样品中表3的基因套组中的基因的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中表4的基因套组中的基因的表达水平来确定,并且其中(a)如果标志1分数为负并且标志2分数为正,则所述疗法是ia类别tme疗法;(b)如果标志1分数为正并且标志2分数为正,则所述疗法是is类别tme疗法;(c)如果标志1分数为负并且标志2分数为负,则所述疗法是id类别tme疗法;或者(d)如果标志1分数为正并且标志2分数为负,则所述疗法是a类别tme疗法。
[0697]
本公开还提供了一种用于治疗有需要的人受试者中的癌症的抗癌疗法,其中所述受试者通过基于非群体的分类器(例如,ann)被鉴定为表现出或未表现出特定类别tme(即,所述受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的),其通过测量从所述受试者获得的样品中从表1和表2获得的基因套组(或图28a-g中公开的基因套组(基因集)中的任一个)或图28a-g中公开的基因套组(基因集)中的任一个中的基因的表达水平,例如mrna表达水平来确定,并且其中(a)如果所分配的tme是ia类别,则所述疗法是ia类别tme疗法;(b)如果所分配的tme是is类别,则所述疗法是is类别tme疗法;(c)如果所分配的tme是id类别,则所述疗法是id类别tme疗法;或者(d)如果所分配的tme是a类别,则所述疗法是a类别tme疗法。在一些方面,如果患者对于多于一种tme类别是生物标记物阳性的,则患者可以接受组合对应于患者对于其是生物标记物阳性的tme类别中的每一种的tme特定疗法的疗法。
[0698]
在一些方面,术语“施用”还可以包括开始疗法、中止或暂停疗法、暂时暂停疗法或修改疗法(例如,增加剂量或剂量频率,或在组合疗法中添加一种或多种治疗剂)。
[0699]
在一些方面,样品可以是例如医疗保健提供者(例如,医生)或医疗保健福利提供者要求的,通过相同或不同的医疗保健提供者(例如,护士、医院)或临床实验室获得和/或处理,并且在处理之后,结果可以转交给原始医疗保健提供者或另一个医疗保健提供者、医疗保健福利提供者或患者。类似地,本文公开的生物标记物的表达水平的定量;生物标记物分数或蛋白质表达水平之间的比较;生物标记物的不存在或存在的评估;相对于某个阈值的生物标记物水平的确定;治疗决策;或其组合可以通过一个或多个医疗保健提供者、医疗保健福利提供者和/或临床实验室进行。
[0700]
如本文所用,术语“医疗保健提供者”是指直接与活体受试者(例如,人患者)交互并对其施药的个体或机构。医疗保健提供者的非限制性实例包括医生、护士、技术人员、治疗师、药剂师、顾问、替代医学从业者、医疗设施、医生办公室、医院、急诊室、诊所、急救中心、替代医学诊所/设施和提供与患者的健康状况的全部或任何部分相关的一般和/或专业治疗、评定、维持、疗法、药物和/或建议的任何其他实体,包括但不限于一般医疗、专业医
疗、手术和/或任何其他类型的治疗、评定、维持、疗法、药物和/或建议。
[0701]
如本文所用,术语“临床实验室”是指用于检查或处理来源于活体受试者例如人类的材料的设施。处理的非限制性实例包括生物、生物化学、血清学、化学、免疫血液学、血液学、生物物理、细胞学、病理学、遗传或来源于人体的材料的其他检查,其目的是提供信息,例如用于诊断、预防或治疗活体受试者例如人类的任何疾病或损害,或评定受试者例如人类的健康。这些检查还可以包括采集或以其他方式获得样品、制备、确定、测量或以其他方式描述活体受试者例如人类的身体或从活体受试者例如人类的身体获得的样品中各种物质的存在或不存在的程序。
[0702]
如本文所用,术语“医疗保健福利提供者”涵盖提供、呈现、供应、支付全部或部分费用或以其他方式与给予患者获得一项或多项医疗保健福利、福利计划、健康保险和/或医疗保健费用账户计划相关联的个人团体、组织或团队。
[0703]
在一些方面,医疗保健提供者可以管理或指导另一个医疗保健提供者施用本文公开的疗法以治疗癌症。医疗保健提供者可以实施或指导另一医疗保健提供者或患者执行以下操作:获得样品、处理样品、提交样品、接收样品、转移样品、分析或测量样品、定量样品、提供分析/测量/定量样品之后获得的结果、接收分析/测量/定量样品之后获得的结果、比较/评分分析/测量/定量一个或多个样品之后获得的结果、提供来自一个或多个样品的比较/分数、获得来自一个或多个样品的比较/分数、施用疗法、开始施用疗法、停止施用疗法、继续施用疗法、暂时中断施用疗法、增加施用的治疗剂的量、降低施用的治疗剂的量、继续施用一定量的治疗剂、增加施用治疗剂的频率、降低施用治疗剂的频率、维持治疗剂的相同给药频率、用至少另一种疗法或治疗剂替代疗法或治疗剂、组合疗法或治疗剂剂与至少另一种疗法或额外的治疗剂。
[0704]
在一些方面,医疗保健提供者可以批准或拒绝例如以下操作:采集样品、处理样品、提交样品、接收样品、转移样品、分析或测量样品、定量样品、提供分析/测量/定量样品之后获得的结果、转移分析/测量/定量样品之后获得的结果、比较/评分分析/测量/定量一个或多个样品之后获得的结果、转移来自一个或多个样品的比较/分数、获得来自一个或多个样品的比较/分数、施用疗法或治疗剂、开始施用疗法或治疗剂、停止施用疗法或治疗剂、继续施用疗法或治疗剂、暂时中断施用疗法或治疗剂、增加施用的治疗剂的量、降低施用的治疗剂的量、继续施用一定量的治疗剂、增加施用治疗剂的频率、降低施用治疗剂的频率、维持治疗剂的相同给药频率、用至少另一种疗法或治疗剂替代疗法或治疗剂或组合疗法或治疗剂剂与至少另一种疗法或额外的治疗剂。
[0705]
此外,医疗保健福利提供者可以例如批准或拒绝治疗处方、批准或拒绝治疗承保、批准或拒绝报销治疗费用、确定或拒绝治疗资格等。
[0706]
在一些方面,临床实验室可以例如采集或获得样品、处理样品、提交样品、接收样品、转移样品、分析或测量样品、定量样品、提供分析/测量/定量样品之后获得的结果、接收分析/测量/定量样品之后获得的结果、比较/评分分析/测量/定量一个或多个样品之后获得的结果、提供来自一个或多个样品的比较/分数、获得来自一个或多个样品的比较/分数或其他相关活动。
[0707]
除了治疗患者或选择用于治疗的患者以外,将患者分配到本文公开的一个或多个特定tme类别(通过应用本文公开的基于群体的分类器和/或非基于群体的分类器产生)还
可以应用于其他治疗或诊断方法。例如,应用于设计新治疗方法的方法(例如,通过选择患者作为用于某种疗法或参与临床试验的候选者),监测治疗剂的功效的方法,或调整治疗(例如,配制品、剂量方案或施用途径)的方法。
[0708]
本文公开的方法还可以包括额外的步骤,诸如至少部分地基于通过应用本文公开的基于群体的分类器和/或非基于群体的分类器确定受试者的癌症中特定tme的存在或不存在(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的),开处方、开始和/或改变预防和/或治疗。
[0709]
本公开还提供了一种确定是否用本文公开的特定tme类别疗法或其组合治疗具有特定tme的患者的方法,所述特定tme通过应用本文公开的基于群体的分类器和/或非基于群体的分类器来鉴定(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)。还提供了基于特定tme的存在和/或不存在选择被诊断患有癌症的患者作为用本文公开的特定tme类别疗法或其组合治疗的候选者的方法,所述特定tme通过应用本文公开的基于群体的分类器和/或非基于群体的分类器来鉴定(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)。
[0710]
在一方面,本文公开的方法包括至少部分地基于受试者中癌症的tme分类(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)进行诊断,其可以是鉴别诊断,其中tme已经通过应用本文公开的基于群体的分类器和/或非基于群体的分类器进行分类。此诊断可以记录在患者病历中。例如,在各个方面,癌症的tme的分类(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)、将患者诊断为可用本文公开的特定tme类别特定疗法或其组合治疗、或所选的治疗可以记录在病历中。病历可以呈纸质形式和/或可以保存在计算机可读介质中。病历可以由实验室、医师办公室、医院、医疗保健维护组织、保险公司和/或个人病历网站维护。
[0711]
在一些方面,基于应用本文公开的基于群体和/或非基于群体的分类器的诊断可以记录在医疗警报物品,诸如卡片、穿戴物品和/或射频识别(rfid)标签的上或之中。如本文所用,术语“穿戴物品”是指可以穿戴在受试者身体上的任何物品,包括但不限于标签、手链、项链或臂章。
[0712]
在一些方面,样品可以由治疗或诊断患者的医疗保健专业人员获得,用于根据医疗保健专业人员的指导(例如,使用如本文所述的特定测定)测量样品中的生物标记物水平。在一些方面,进行测定的临床实验室可以基于患者的癌症是否被分类为属于特定tme类别(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)告知医疗保健提供者患者是否可以受益于用本文公开的特定tme类别疗法或其组合进行的治疗。在一些方面,通过应用本文公开的基于群体的分类器和/或非基于群体的分类器进行的tme分类的结果(即,本文公开的一种或多种基质表型在受试者中存在或不存在,即受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)可以提交给医疗保健福利提供者,以确定患者的保险是否覆盖用本文公开的特定tme类别疗法或其组合进行的治疗。在一些方面,进行测定的临床实验室可以基于癌症的tme分类(即,受试者对于本文公开的一种或多种基质表型是生物标记物阳性的和/或生物标记物阴性的)告知医疗保健提供者患者是否可以受益于用本文公开的特定tme类别疗法
或其组合进行的治疗。
[0713]
i.f tme类别特定疗法
[0714]
用于鉴定肿瘤微环境(tme)的主要生物学的四种基质表型或类别,即特定类型的基质表型,可以用于预测哪些疗法对于治疗特定类别更有效。参见,例如图10。
[0715]
i.f.1ia类别tme疗法
[0716]
对于以免疫活性为主的tme,诸如ia(免疫活性型)表型,具有这种生物学的患者(即,ia生物标记物阳性患者)可能对免疫检查点抑制剂(cpi),诸如抗pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、抗pd-l1或抗ctla-4,或对于rorγ激动剂治疗剂产生应答。
[0717]
检查点抑制剂:在一些方面,免疫检查点抑制剂是结合pd-1的阻断抗体,例如纳武单抗、西米普利单抗(regn2810)、杰诺单抗(cbt-501)、帕克米利单抗(cx-072)、多塔利单抗(tsr-042)、信迪利单抗、替雷利珠单抗和派姆单抗;结合pd-l1的阻断抗体,例如德瓦鲁单抗(medi4736)、阿维单抗、洛达利单抗(ly-3300054)、cx-188和阿替利珠单抗;或结合ctla-4的阻断抗体,例如伊匹单抗和替西木单抗。在一些方面,可以使用此类抗体中的一种或多种的组合。
[0718]
替西木单抗、纳武单抗、德瓦鲁单抗和阿替利珠单抗分别描述于例如美国专利号6,682,736、美国专利号8,008,449、美国专利号8,779,108和美国专利号8,217,149。在一些方面,阿替利珠单抗可以被另一种免疫检查点抗体替代,诸如结合ctla-4、pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1的另一种阻断抗体,或结合任何检查点抑制剂的双特异性阻断抗体。在选择不同的阻断抗体时,本领域普通技术人员将从文献中知道合适的剂量和施用方案。抗ctla-4抗体的合适实例是美国专利号6,207,156中描述的那些。抗pd-l1抗体的其他合适实例是以下中描述的那些:美国专利号8,168,179,其尤其涉及用人抗pd-l1抗体治疗pd-l1过表达癌症,包括化学疗法组合;美国专利号9,402,899,其尤其涉及用针对pd-l1的抗体治疗肿瘤,包括嵌合抗体、人源化抗体和人抗体;和美国专利号9,439,962,其尤其涉及用抗pd-l1抗体和化学疗法治疗癌症。
[0719]
其他合适的针对pd-l1的抗体是美国专利号7,943,743、美国专利号9,580,505和美国专利号9,580,507中的那些、其试剂盒(美国专利号9,580,507)和编码抗体的核酸(美国专利号8,383,796)。此类抗体结合pd-l1并与参考抗体竞争结合;由vh和vl基因定义;或由定义的序列或其保守修饰的重链和轻链cdr3(美国专利号7,943,743)或重链cdr3(美国专利号8,383,796)定义;或与参考抗体具有90%或95%序列同一性。这些抗pd-l1抗体还包括具有定义的定量(包括结合亲和力)和定性特性的那些抗体、免疫偶联物和双特异性抗体。还包括使用此类抗体的方法,以及具有定义的定量(包括结合亲和力)和定性特性的那些抗体,包括呈单链形式的抗体和呈分离cdr形式的那些抗体,以增强免疫应答(美国专利号9,102,725)。如在美国专利号9,102,725中,增强免疫应答可以用于治疗癌症或传染病,诸如病毒、细菌、真菌或寄生虫导致的的病原性感染。
[0720]
其他合适的针对pd-l1的抗体是美国专利申请号2016/0009805中的那些,其涉及针对pd-l1上特定表位的抗体,包括具有定义的cdr序列的抗体和竞争性抗体;核酸、载体、宿主细胞、免疫偶联物;检测、诊断、预后和生物标记物方法;和治疗方法。
[0721]
包括伊匹单抗的特定治疗公开于例如us7,605,238;us8,318,916;8,784,815;和
us8,017,114中。包括替西木单抗的治疗公开于例如us6,682,736、us7,109,003、us7,132,281、us7,411,057、us7,807,797、us7,824,679、us8,143,379、us8,491,895和8,883,984中。使用纳武单抗的治疗公开于例如us8,008,449、us8,779,105、us9,387,247、us9,492,539、us9,492,540、us8,728,474、us9,067,999、us9,073,994和us7,595,048中。使用派姆单抗的治疗公开于例如us8,354,509、us8,900,587和us8,952,136中。使用西米普利单抗的治疗公开于例如us20150203579a1中。使用德瓦鲁单抗的治疗公开于例如us8,779,108和us 9,493,565中。使用阿替利珠单抗的治疗公开于例如us8,217,149中。使用cx-072的治疗公开于例如15/069,622中。使用ly300054的治疗公开于例如us10214586b2中。使用针对pd-1和ctla-4的抗体的组合的肿瘤治疗公开于例如us9,084,776、us8,728,474、us9,067,999和us9,073,994中。使用针对pd-1和ctla-4(包括亚治疗剂量)和pd-l1阴性肿瘤的抗体治疗肿瘤公开于例如us9,358,289中。使用针对pd-l1和ctla-4的抗体治疗肿瘤公开于例如us9,393,301和us9,402,899中。所有这些专利和公布通过引用以其整体并入本文。
[0722]
也定治疗剂和合适的癌症适应症在下表中鉴定。
[0723]
表6
[0724]
[0725]
[0726][0727]
rorγ激动剂治疗剂:在一些方面,rorγ激动剂治疗剂是属于核激素受体家族的rorγ(类视黄醇相关孤儿受体γ)的小分子激动剂。rorγ在胸腺生成和t细胞稳态过程中控制细胞凋亡中起关键作用。临床开发中的小分子激动剂包括lyc-55716(cintirorgon)。
[0728]
替雷利珠单抗
[0729]
替雷利珠单抗(bgb-a317)是针对pd-1的人源化单克隆抗体。它防止pd-1与配体pd-l1和pd-l2结合(因此它是一种检查点抑制剂)。替雷利珠单抗可以用于治疗实体癌,例如霍奇金淋巴瘤(单独或与辅助疗法诸如含铂化学疗法组合使用)、尿路上皮癌、nsclc或肝细胞癌。在一些方面,例如根据本文所述的方法向受试者施用的替雷利珠单抗分子包括替雷利珠单抗。与替雷利珠单抗相关的序列在下表中提供。在本公开的一些方面,替雷利珠单抗或其抗原结合部分可以与巴维妥昔单抗组合施用。
[0730]
表7.替雷利珠单抗序列
[0731][0732][0733]
信迪利单抗
[0734]
信迪利单抗是针对pd-1的全人igg4单克隆抗体。它防止pd-1与配体pd-l1和pd-l2结合(因此它是一种检查点抑制剂)。信迪利单抗可以单独或与辅助疗法组合用于治疗实体癌,例如霍奇金淋巴瘤。在一些方面,例如根据本文所述的方法向受试者施用的信迪利单抗分子包括信迪利单抗。与信迪利单抗相关的序列在下表中提供。在本公开的一些方面,信迪利单抗或其抗原结合部分可以与巴维妥昔单抗组合施用。
[0735]
表8.信迪利单抗序列
[0736][0737]
i.f.2 is类别tme疗法
[0738]
对于以免疫抑制为主的tme,这种被分类为is(免疫抑制型)表型的患者(即,is生物标记物阳性患者)对于检查点抑制剂可以具有耐药性,除非还被给予逆转免疫抑制的药物,诸如抗磷脂酰丝氨酸(抗ps)和抗磷脂酰丝氨酸靶向治疗剂、pi3kγ抑制剂、腺苷通路抑制剂、ido、tim、lag3、tgfβ和cd47抑制剂。
[0739]
巴维妥昔单抗是优选的抗ps靶向治疗剂。具有这种生物学的患者也具有潜在的血管生成,并且还可以受益于抗血管生成剂,诸如用于a基质亚型的那些。
[0740]
现在讨论is生物标记物阳性患者的特定治疗剂。抗ps和ps靶向抗体包括但不限于巴维妥昔单抗;pi3kγ抑制剂,诸如ly3023414(samotolisib)、ipi-549;腺苷通路抑制剂,诸如ab-928(腺苷2a和2b受体的口服拮抗剂);ido抑制剂;抗tim,包括tim和tim-3;抗lag3;tgfβ抑制剂,诸如ly2157299(galunisertib);cd47抑制剂,诸如forty seven的莫洛利单抗(5f9)。
[0741]
is生物标记物阳性患者的特定治疗剂还包括:抗tigit药物,其通过触发树突细胞上的cd155(分化簇155)(以及其他活动)和在肿瘤中表达treg的子集而具有免疫抑制性。优选的抗tigit抗体是ab-154。抗激活素a治疗剂,因为激活素a促进m2样肿瘤巨噬细胞的分化
并抑制nk细胞的生成。抗bmp治疗剂是有用的,因为骨形态发生蛋白(bmp)也促进m2样肿瘤巨噬细胞的分化并抑制ctl和dc。
[0742]
is生物标记物阳性患者的另外的特定治疗剂还包括:tam(tyro3、axl和mer受体)抑制剂或tam产物抑制剂;抗il-10(白细胞介素)或抗il-10r(白细胞介素10受体),因为il-10具有免疫抑制作用;抗m-csf,作为巨噬细胞集落刺激因子(m-csf)拮抗剂已显示耗尽tam;抗ccl2(c-c基序趋化因子配体2)或抗ccl2r(c-c基序趋化因子配体2受体),由这些药物靶向的特定通路将髓样细胞募集到肿瘤;mertk(酪氨酸蛋白激酶mer)拮抗剂,因为此受体酪氨酸激酶的抑制触发促炎tam表型并增加肿瘤cd8+细胞。
[0743]
is生物标记物阳性患者的其他治疗剂包括:sting激动剂,因为通过干扰素基因的刺激剂(sting)进行的细胞溶质dna感应增强抗肿瘤cd8+t细胞的dc刺激,并且激动剂是的一部分;针对ccl3(c-c基序趋化因子3)、ccl4(c-c基序趋化因子4)、ccl5(c-c基序趋化因子5)或其共同受体ccr5(c-c基序趋化因子受体5型)的抗体,因为这些趋化因子是髓源性抑制细胞(mdsc)的产物并激活调节性t细胞(treg)上的ccr5;精氨酸酶-1的抑制剂,因为精氨酸酶-1通过m2样tam产生,减少肿瘤浸润淋巴细胞(til)的产生并增加treg的产生;针对ccr4(c-c基序趋化因子受体4型)的抗体可以用于耗尽treg;针对ccl17(c-c基序趋化因子17)或ccl22(c-c基序趋化因子22)的抗体可以抑制treg上的ccr4(c-c基序趋化因子受体4型)激活;针对gitr(糖皮质激素诱导的tnfr相关蛋白)的抗体可以用于耗尽treg;dna甲基转移酶(dnmt)或组蛋白去乙酰化酶(hdac)的抑制剂,其导致免疫基因的表观遗传沉默的逆转,诸如恩替诺特。
[0744]
在临床前模型中,磷酸二酯酶5的抑制剂西地那非和他达拉非显著抑制mdsc功能,这可以为is患者提供益处。用于将mdsc分化为成熟树突细胞(dc)和巨噬细胞的全反式维甲酸(atra)可以为is患者提供益处。据报道,vegf和c-kit信号传导涉及mdsc的生成。据报道,舒尼替尼治疗转移性肾细胞癌患者减少循环mdsc的数量,这可以为is患者提供益处。
[0745]
is表型(即,is生物标记物阳性),即在所公开的基于群体的分类器中对标志1和2是高的或根据本文公开的非基于群体的分类器被分类为is类别tme的癌症代表用于与检查点抑制剂组合的巴维妥昔单抗治疗的靶群体,所述检查点抑制剂诸如抗pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、抗pd-l1或抗ctla-4。这是因为本公开指出在血管生成存在的情况下发生的免疫应答显示免疫抑制的现象,并且巴维妥昔单抗可以恢复免疫抑制细胞的免疫活性。为了使单剂巴维妥昔单抗起作用,正在进行的免疫应答必须高度活跃,以至于阻断免疫抑制足以释放患者免疫应答的全部潜力。然而,大多数晚期癌症患者需要保持其免疫应答,并且可能需要与巴维妥昔单抗和检查点抑制剂组合使用。因此,本文公开的is表型可以用于确定可能对巴维妥昔单抗和检查点抑制剂产生应答的癌症患者。
[0746]
巴维妥昔单抗
[0747]
巴维妥昔单抗是ps靶向抗体。巴维妥昔单抗在血清存在的情况下与阴离子磷脂强烈结合。巴维妥昔单抗与ps的结合由血清蛋白β2-糖蛋白1(β2gpi)介导。β2gpi也被称为载脂蛋白h。
[0748]
在一些方面,例如根据本文所述的方法向受试者施用的巴维妥昔单抗分子包括巴维妥昔单抗。与巴维妥昔单抗相关的序列在下表中提供。
[0749]
表9.巴维妥昔单抗序列
[0750][0751]
在一些方面,巴维妥昔单抗分子与抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)组合施用。在一些方面,巴维妥昔单抗分子与派姆单抗组合施用。在一些方面,巴维妥昔单抗分子与信迪利单抗组合施用。在一些方面,巴维妥昔单抗分子与替雷利珠单抗组合施用。在一些方面,将巴维妥昔单抗分子施用于患有肝细胞癌、胃癌、nsclc、卵巢癌、乳腺癌、头颈癌或胰腺癌的受试者。
[0752]
i.f.3 id类别tme疗法
[0753]
对于没有免疫活性的tme,诸如被分类为id(免疫沙漠型)表型的患者(即,id生物标记物阳性患者),具有这种生物学的患者对于检查点抑制剂、抗血管生成剂或其他tme靶向疗法的单一疗法没有应答,并且因此不应用作为单一疗法的抗pd-1、抗pd-l1、抗ctla-4或rorγ激动剂来治疗。具有这种生物学的患者可以用诱导免疫活性的疗法治疗,从而允许他们然后从检查点抑制剂或其他tme靶向疗法中受益。对于这些患者可以诱导免疫活性的疗法包括疫苗、car-t、新表位疫苗(包括个性化疫苗)和基于tlr的疗法。
[0754]
car-t疗法是一种治疗方法,其中患者的t细胞(一种免疫系统细胞类型)在实验室中被改变,使得它们攻击癌细胞。t细胞取自患者的血液。然后在实验室中添加结合患者癌细胞上的某种蛋白质的特殊受体的基因。所述特殊受体被称为嵌合抗原受体(car)。大量的car t细胞在实验室中生长并通过输注给予患者。car t细胞疗法正在被研究用于治疗某些类型的癌症。也被称为嵌合抗原受体t细胞疗法。在一些方面,car-t疗法包括施用imm-3、axicabtagene ciloleucel、auto、immunotox、sparx/arc-t疗法或bcma car-t。
[0755]
toll样受体(tlr)(果蝇toll蛋白的哺乳动物同源物),被认为是先天免疫的关键模式识别受体(prr)。癌细胞上的一些tlr可能以炎症依赖性或非依赖性方式促进癌症进
展。通过tlr信号传导刺激的炎性应答可以通过加强肿瘤炎性微环境来促进癌发生。此外,某些类型的癌细胞tlr的表达水平升高促进肿瘤发生,这是tlr衔接子分子所必需的,但与炎症无关。已发现一些tlr激动剂通过间接激活耐受宿主免疫系统以破坏癌细胞来诱导强烈的抗肿瘤活性。因此,tlr的特定激动剂或拮抗剂可以用于治疗癌症。在一些方面,基于tlr的包括施用poly(i:c)。多种tlr激动剂已被考虑用于临床应用。bcg(卡介苗)可以用于例如浅表性膀胱癌或结直肠癌的疗法。tlr3(toll样受体3)配体iph-3102(iph-31xx)可以用于治疗例如乳腺癌。tlr4(toll样受体4)激动剂单磷酰脂质a(mpl)可以用于例如治疗结直肠癌。在一些方面,mpl可以与作为佐剂的cervarix
tm
疫苗一起施用用于预防hpv(人乳头瘤病毒)相关的宫颈癌。在一些方面,鞭毛蛋白衍生的激动剂cblb502(恩替莫德)可以用于治疗晚期实体肿瘤。
[0756]
在一些方面,基于tlr的疗法包括施用bcg(卡介苗)、单磷酰脂质a(mpl)、恩替莫德(cblb502)、咪喹莫特852a(小分子ssrna)、imoxine(cpg-odn)、来非莫德(mgn1703)、(双茎环免疫调节剂)、cpg寡脱氧核苷酸(cpg-odn)、pf3512676(也被称为cpg7909;单独或与化学疗法组合)、1018iss(单独或与化学疗法或组合)、来非莫德、sd-101、莫托莫德(vtx-2337)、imo-2055(imoxine;emd 1201081)、替索莫德(imo-2125)、dv281、cmp-101或cpg7907。
[0757]
治疗性癌症疫苗是基于使用肿瘤抗原对免疫系统进行特异性刺激以引发抗肿瘤应答。在一些方面,癌症疫苗包括例如igv-001(imvax
tm
)、伊利沙定(ilixadencel)、imm-2、tg4010(表达muc-1和il-2的mva)、(表达胎儿癌基因5t4的mva(mva-5t4))、(或psa-)(表达psa的mva)、recmage-a3(重组黑色素瘤相关抗原3)蛋白加as15免疫刺激剂、rindopepimut与gm-csf加替莫唑胺、ima901(10种不同的合成肿瘤相关肽)、替西莫肽(l-blp25)(muc-1衍生的脂肽)、基于dc的疫苗(表达例如细胞因子诸如il-12)、由酪氨酸酶、gp100和mart-1肽构成的多表位疫苗、肽疫苗(egfrviii、epha2、her2/neu肽)(单独或与贝伐珠单抗组合)、hsppc-96(基于肽的个性化疫苗)(单独或与贝伐珠单抗组合)、(基于同种异体细胞的疗法)(单独或与舒尼替尼组合)、pf-06755990(疫苗)(单独或与舒尼替尼和/或替西木单抗组合)、(新抗原肽)(单独或与派姆单抗和/或放射疗法组合)、临床试验中使用的肽疫苗nct02600949(单独或与派姆单抗组合)、dpx-survivac(包封肽)(单独或与派姆单抗和/或化学疗法,例如与环磷酰胺组合)、ptvg-hp(编码pap抗原的dna疫苗)(单独或与纳武单抗和/或cm-csf组合)、(分泌gm-csf的肿瘤细胞)(单独或与纳武单抗和/或化学疗法,例如与环磷酰胺组合)、(表达psa的痘病毒载体)(单独或与纳武单抗组合)、(表达psa的痘病毒载体)(单独或与伊匹单抗组合)、(分泌gm-csf的肿瘤细胞)(单独或与纳武单抗和伊匹单抗组合并且与crs-207和环磷酰胺组合)、基于树突细胞的p53疫苗(单独或与纳武单抗和伊匹单抗组合)、新抗原dna疫苗(与德瓦鲁单抗组合)或cdx-1401疫苗(dec-205/ny-eso-1融合蛋白)(单独或与阿替利珠单抗和化学疗法,例如呱西他滨(guadecitabine)组合)。
[0758]
i.f.4 a类别tme疗法
[0759]
对于以血管生成活性为主的tme,诸如被分类为a(血管生成型)表型的患者(即,a
生物标记物阳性患者),具有这种生物学的患者对于vegf靶向疗法、dll4靶向疗法、血管生成素/tie2靶向疗法、抗vegf/抗dll4双特异性抗体(诸如那赛昔珠单抗)和抗vegf或抗vegf受体抗体(诸如伐利苏单抗、雷莫卢单抗、贝伐珠单抗等)可以具有应答性。
[0760]
在一些方面,可以选择具有抗血管生成作用的双可变结构域免疫球蛋白分子、药物或疗法,诸如具有抗dll4和/或抗vegf活性的那些来治疗被鉴定为对于血管生成标志是生物标记物阳性的或被鉴定为具有a基质表型的患者。在一些方面,双可变结构域免疫球蛋白分子、药物或疗法是迪帕西单抗(abt165)。在一些方面,可以选择具有抗血管生成作用的双靶向蛋白、药物或疗法,诸如具有抗dll4和/或抗vegf活性的那些来治疗被鉴定为对于血管生成标志是生物标记物阳性的或被鉴定为具有a基质表型的患者。在一些方面,双靶向蛋白、药物或疗法是abl001(nov1501,tr009),如美国公开号2016/0159929中所教导的,其通过引用以其整体并入本文。
[0761]
那赛昔珠单抗
[0762]
抗vegf/抗dll4双特异性抗体那赛昔珠单抗详细描述于例如美国专利号9,376,488、9,574,009和9,879,084,其中的每一个都通过引用以其整体并入本文。
[0763]
表10.那赛昔珠单抗序列
[0764][0765][0766]
伐利苏单抗
[0767]
抗vegfa单克隆抗体伐利苏单抗详细描述于例如美国专利号8,394,943、9,421,
256和8,034,905,其中的每一个都通过引用以其整体并入本文。
[0768]
表11.伐利苏单抗序列
[0769][0770]
在一些方面,伐利苏单抗分子与第二抗体,例如抗pd-1抗体(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)组合施用。在一些方面,伐利苏单抗分子与化疗剂,例如紫杉烷,例如紫杉醇或多西他赛组合施用。
[0771]
在一些方面,酪氨酸激酶抑制剂(tki)用于抗血管生成疗法。示例性tki包括卡博替尼、凡德他尼、替沃扎尼、阿西替尼、乐伐替尼、索拉非尼、瑞戈非尼、舒尼替尼、呋喹替尼和帕唑帕尼。在一些方面,可以使用c-met抑制剂。
[0772]
可以作为本文公开的tme类别特定疗法的一部分施用的特定治疗剂包括在表12中。
[0773]
表12:用于作为tme类别特定疗法的一部分施用的治疗剂
[0774]
[0775]
[0776]
[0777]
[0778]
[0779]
[0780][0781]
cpm:检查点调节剂;cpi:检查点抑制剂;aat:抗血管生成疗法;ait:抗免疫抑制疗法;irit:免疫应答启动疗法;vtt/a:vegf靶向疗法/其他血管生成素;attt:促血管生成素/tie2靶向疗法;chemo:化学疗法
[0782]
i.f.5辅助疗法
[0783]
选择用某种疗法治疗的患者的方法以及本文公开的治疗方法还可以包括(i)施用额外的疗法,例如化学疗法、激素疗法或放射疗法,(ii)手术,或(iii)其组合。在一些方面,可以在施用以上公开的tme特定疗法或其组合的同时或顺序地(之前或之后)施用额外的(辅助)疗法。
[0784]
当一种或多种辅助疗法与如本文所述的tme特定疗法或其组合组合使用时,不需要组合结果是单独进行每种治疗时观察到的效果的加成。尽管至少加成效果通常是希望的,但在单一疗法中的任一种之上的增加治疗效果或益处(例如,减少的副作用)都是有价值的。此外,对组合治疗表现出协同效果没有特别要求,尽管这是可能且有利的。
[0785]“新辅助疗法”可以作为缩小肿瘤的第一步进行,然后进行主要治疗,其通常是手术。新辅助疗法的实例包括化学疗法、放射疗法和激素疗法。这是一种诱导疗法。
[0786]
在特定方面,a类别tme疗法可以与化学治疗剂组合施用,例如紫杉烷,诸如紫杉醇或多西他赛。在一些方面,a类别tme疗法可以包括与vegf靶向疗法和/或dll-4靶向疗法组合的化学疗法(例如紫杉烷,诸如紫杉醇或多西他赛)。
[0787]
化学疗法可以作为ia类别tme疗法、is类别tme疗法、id类别tme疗法或其组合的护理标准施用。因此,如果患者或患者的癌症被分配到特定tme类别或其组合(即,患者对于一种或多种tme类别是生物标记物阳性的和/或对于一种或多种tme类别是生物标记物阴性的),则可以将此tme类别或其组合的特定疗法(即,ia类别tme疗法、is类别tme疗法、id类别tme疗法、a类别疗法或其组合)添加到标准护理化学疗法中。
[0788]
有希望的抗肿瘤作用在以下临床试验中报告:在her2阴性转移性乳腺癌患者中使用巴维妥昔单抗与紫杉醇的组合(chalasani等人,cancer med.2015年7月;4(7):1051-9);在晚期非小细胞肺癌nsclc中使用紫杉醇-卡铂(digumarti等人,lung cancer.2014年11月;86(2):231-6);在肝细胞癌中使用索拉非尼(cheng等人,ann surg oncol.2016年12月;23(增刊5):583-5912016);以及在先前治疗的晚期非鳞状nsclc中使用多西他赛(gerber等人,clin lung cancer.2016年3月;17(3):169-762016),所有这些剂都是化疗剂。
[0789]
i.f.5.a化学疗法
[0790]
如本文所述的tme特定疗法可以与一种或多种辅助化疗剂或药物组合施用。
[0791]
术语“化学疗法”是指影响细胞增殖和/或存活的各种治疗方式。所述治疗可以包括施用烷基化剂、抗代谢物、蒽环类、植物生物碱、拓扑异构酶抑制剂和其他抗肿瘤剂,包括单克隆抗体和激酶抑制剂。术语“新辅助化学疗法”涉及由一组激素剂、化疗剂和/或抗体剂组成的术前疗法方案,其旨在缩小原发性肿瘤,从而使局部疗法(手术或放射疗法)破坏性更小或更有效,能够进行保乳手术和评估肿瘤对体内特定剂的敏感性的应答性。
[0792]
化疗药物可以杀死增殖的肿瘤细胞,增强整体治疗产生的坏死区域。药物因此可以增强本公开的主要治疗剂的作用。
[0793]
用于癌症治疗的化疗剂可以根据其作用机制分为若干组。一些化疗剂直接损伤dna和rna。通过破坏dna的复制,此类化疗剂使复制完全停止,或导致产生无义dna或rna。此类别包括例如顺铂柔红霉素阿霉素和依托泊苷另一组癌症化疗剂会干扰核苷酸或脱氧核
糖核苷酸的形成,使得rna合成和细胞复制被阻断。此类别中的药物的实例包括甲氨蝶呤巯基嘌呤氟尿嘧啶和羟基脲第三类化疗剂影响有丝分裂纺锤体的合成或分解,并且因此中断细胞分裂。此类别中的药物的实例包括长春花碱长春新碱和紫杉烷,诸如紫杉醇和多西他赛
[0794]
在一些方面,本文公开的方法包括用紫杉烷衍生物,例如紫杉醇或多西他赛进行的治疗。在一些方面,本文公开的方法包括用蒽环类衍生物,例如像阿霉素、柔红霉素和阿克那霉素进行的治疗。在一些方面,本文公开的方法包括用拓扑异构酶抑制剂,例如像喜树碱、拓扑替康、伊立替康、20-s喜树碱、9-硝基-喜树碱、9-氨基-喜树碱或水溶性喜树碱类似物g1147211进行的治疗。特别设想用这些和其他化疗药物的任何组合进行的治疗。
[0795]
患者可以在手术移除肿瘤后立即接受化学疗法。这种方法通常被称为辅助化学疗法。然而,还可以在手术之前施用化学疗法,如所谓的新辅助化学疗法。
[0796]
i.f.5.a放射疗法
[0797]
如本文所述的tme特定疗法可以与放射疗法组合施用。
[0798]
术语“放射疗法(radiation therapy)”和“放射疗法(radiotherapy)”是指用电离辐射治疗癌症,其包括具有足够动能以从原子或分子发射电子并由此生成离子的粒子。所述术语包括用直接电离辐射,诸如由α粒子(氦核)、β粒子(电子)和原子粒子诸如质子产生的那些和间接电离辐射,诸如光子(包括伽马射线和x射线)进行治疗。放射疗法中使用的电离辐射的实例包括高能x-射线、γ-辐射、电子束、uv辐射、微波和光子束。还设想将放射性同位素直接递送至肿瘤细胞。
[0799]
大多数患者在手术移除肿瘤后立即接受放射疗法。这种方法通常被称为辅助放射疗法。然而,还可以在手术之前施用放射疗法,如所谓的新辅助放射疗法。
[0800]
ii.癌症适应症
[0801]
本文公开的方法和组合物可以用于治疗癌症。“癌症”是指一组广泛的以体内异常细胞的不受控制的生长为特征的各种增殖性疾病。不受调节的细胞分裂和生长导致恶性肿瘤的形成,所述恶性肿瘤侵入邻近组织并且还可以通过淋巴系统或血流转移至身体的远端部位。如本文所用,术语“增殖性”病症或疾病是指多细胞生物体中一个或多个细胞子集的不希望的细胞增殖,导致对多细胞生物体的伤害(即,不适或期望寿命缩短)。例如,如本文所用,增殖性病症或疾病包括赘生性病症和其他增殖性病症。如本文所用,“赘生性”是指导致不正常组织生长的调控异常或不受调控的细胞的任何形式,无论是恶性还是良性的。因此,“赘生性细胞”包括具有调控异常或不受调控的细胞生长的恶性和良性细胞。在一些方面,癌症是肿瘤。如本文所用,“肿瘤”是指所有赘生性细胞生长和增殖(无论恶性或良性)以及所有癌前和癌性细胞和组织。
[0802]
在一些方面,本文公开的方法和组合物用于减小或降低有需要的受试者的肿瘤大小或抑制肿瘤生长。在一些方面,肿瘤是癌(即,上皮起源的癌症)。在一些方面,所述肿瘤例如选自由以下组成的组:胃癌、胃食管交界处癌症(gej)、食管癌、结直肠癌、肝癌(肝细胞癌,hcc)、卵巢癌、乳腺癌、nsclc(非小细胞肺癌)、膀胱癌、肺癌、胰腺癌、头颈癌、淋巴瘤、子宫癌、肾脏癌或肾癌、胆管癌、前列腺癌、睾丸癌、尿道癌、阴茎癌、胸癌、直肠癌、脑癌(胶质
瘤和胶质母细胞瘤)、宫颈癌、腮腺癌、喉癌、甲状腺癌、腺癌、神经母细胞瘤、黑色素瘤和默克尔细胞癌。
[0803]
在一些方面,所述癌症是复发性的。术语“复发性”是指在疗法之后癌症缓解的受试者具有癌细胞返回的情况。在一些方面,所述癌症是难治性的。术语“难治性”或“耐受性”是指即使在强化治疗之后受试者在体内具有残留癌细胞的情况。在一些方面,在至少一种包括施用至少一种抗癌剂的先前疗法后,所述癌症是难治性的。在一些方面,所述癌症是转移性的。
[0804]“癌症”或“癌组织”可以包括各个时期的肿瘤。在某些方面,癌症或肿瘤处于0期,使得例如癌症或肿瘤处于非常早期的发展中并且尚未转移。在一些方面,癌症或肿瘤处于i期,使得例如癌症或肿瘤的大小相对较小,未扩散到附近组织中并且尚未转移。在其他方面,癌症或肿瘤处于ii期或iii期,使得例如癌症或肿瘤比0期或i期大,并且它已经生长到附近组织中但尚未转移,但是可能转移到淋巴结。在其他方面,癌症或肿瘤处于iv期,使得例如癌症或肿瘤已经转移。iv期还可以被称为晚期或转移癌症。
[0805]
在一些方面,癌症可以包括但不限于肾上腺皮质癌、晚期癌症、肛门癌、再生障碍性贫血、胆管癌、膀胱癌、骨癌、骨转移、脑瘤、脑癌、乳腺癌、儿童期癌症、原发性起源未知的癌症、卡斯尔门病、宫颈癌、结肠/直肠癌、子宫内膜癌、食管癌、尤文氏家族肿瘤、眼癌、胆囊癌、胃肠道类癌、胃肠道间质瘤、妊娠滋养细胞疾病、霍奇金病、卡波西肉瘤、肾细胞癌、喉和下咽癌、急性淋巴细胞白血病、急性髓性白血病、慢性淋巴细胞白血病、慢性髓性白血病、慢性粒单核细胞白血病、肝癌、非小细胞肺癌、小细胞肺癌、肺类癌肿瘤、皮肤淋巴瘤、恶性间皮瘤、多发性骨髓瘤、骨髓增生异常综合征、鼻腔和鼻窦癌、鼻咽癌、神经母细胞瘤、非霍奇金淋巴瘤、口腔和口咽癌、骨肉瘤、卵巢癌、胰腺癌、阴茎癌、垂体瘤、前列腺癌、视网膜母细胞瘤、横纹肌肉瘤、唾液腺癌、成人软组织肉瘤、基底和鳞状细胞皮肤癌、黑色素瘤、小肠癌、胃癌、睾丸癌、喉癌、胸腺癌、甲状腺癌、子宫肉瘤、阴道癌、外阴癌、华氏巨球蛋白血症、肾母细胞瘤(wilms tumor)和癌症治疗引起的继发性癌症。
[0806]
在一些方面,所述肿瘤是实体肿瘤。“实体肿瘤”包括但不限于肉瘤、黑色素瘤、癌或其他实体肿瘤癌症。“肉瘤”是指由类似胚胎结缔组织的物质组成并且通常由嵌入纤维状或均质物质中的紧密堆积的细胞构成的肿瘤。肉瘤包括但不限于软骨肉瘤、纤维肉瘤、淋巴肉瘤、黑色素肉瘤、粘液肉瘤、骨肉瘤、abemethy肉瘤(abemethy's sarcoma)、脂肉瘤、脂肪肉瘤、肺泡状软部分肉瘤、成釉细胞肉瘤、葡萄状肉瘤、绿色肉瘤、绒毛膜癌、胚胎肉瘤、肾母细胞瘤肉瘤、子宫内膜肉瘤、间质肉瘤、尤文肉瘤、筋膜肉瘤、成纤维细胞肉瘤、巨细胞肉瘤、粒细胞肉瘤、霍奇金肉瘤、特发性多发性色素沉着出血性肉瘤、b细胞免疫母细胞肉瘤、淋巴瘤、t细胞免疫母细胞肉瘤、延森氏肉瘤(jensen's sarcoma)、卡波西肉瘤、kupffer细胞肉瘤、血管肉瘤、白血病性肉瘤、恶性间充质肉瘤、骨旁肉瘤、网状细胞肉瘤、劳斯肉瘤(rous sarcoma)、浆液囊性肉瘤、滑膜肉瘤或毛细血管扩张型肉瘤。
[0807]
术语“黑色素瘤”是指由皮肤和其他器官的黑色素细胞系统产生的肿瘤。黑色素瘤包括例如肢端皮损性黑色素瘤、无黑色素性黑色素瘤、良性幼年黑色素瘤、克劳德曼黑色素瘤(cloudman's melanoma)、s91黑色素瘤、哈-帕黑色素瘤(harding-passey melanoma)、幼年黑色素瘤、恶性雀斑样黑色素瘤(lentigo maligna melanoma)、恶性黑素瘤、转移性黑素瘤、结节性黑素瘤、甲下黑素瘤或浅表性扩散黑色素瘤。
[0808]
术语“癌”是指由倾向于浸润周围组织并引起转移的上皮细胞组成的恶性新生长物。示例性癌包括例如腺泡癌、腺泡状癌、腺囊性癌、腺样囊性癌、腺癌、肾上腺皮质癌、肺泡癌、肺泡细胞癌、基底细胞癌(basal cell carcinoma)、基底细胞癌(carcinoma basocellulare)、基底细胞样癌、基底鳞状细胞癌、支气管肺泡癌、细支气管癌、支气管癌、脑状癌、胆管细胞癌、绒毛膜癌、胶状癌、粉刺癌、子宫体癌、筛状癌、铠甲状癌、皮肤癌、柱状癌、柱状细胞癌、导管癌、硬癌(carcinoma durum)、胚胎性癌、脑状癌、表皮样癌、腺样上皮细胞癌、外植癌、溃疡性癌、纤维癌、胶样癌(gelatiniform carcinoma)、胶状癌(gelatinous carcinoma)、巨细胞癌(giant cell carcinoma)、巨细胞癌(carcinoma gigantocellulare)、腺癌、粒层细胞癌、基底细胞癌(hair-matrix carcinoma)、血样癌、肝细胞癌、许特尔细胞癌(hurthle cell carcinoma)、玻质状癌、肾上腺样癌(hypemephroid carcinoma)、幼稚型胚胎性癌、原位癌、表皮内癌、上皮内癌、克罗姆佩柯赫尔氏肿瘤(krompecher's carcinoma)、库尔契茨基氏细胞癌(kulchitzky-cell carcinoma)、大细胞癌、扁豆状癌(lenticular carcinoma)、扁豆状癌(carcinoma lenticulare)、脂瘤样癌、淋巴上皮癌、髓样癌、髓质癌、黑色素癌、软癌、粘液素癌、粘液癌(carcinoma muciparum)、粘液细胞癌、粘液表皮样癌、粘液癌(carcinoma mucosum)、粘液癌(mucous carcinoma)、粘液瘤样癌、鼻咽癌、燕麦状细胞癌、骨化性癌、骨质癌、乳头状癌、门静脉周癌、原位癌(preinvasive carcinoma)、刺细胞癌、髓样癌、肾脏的肾细胞癌、储备细胞癌、肉瘤样癌、施耐德氏癌(schneiderian carcinoma)、硬癌(scirrhous carcinoma)、阴囊癌、印戒细胞癌、单纯癌、小细胞癌、马铃薯状癌、球形细胞癌、梭形细胞癌、髓样癌(carcinoma spongiosum)、鳞状癌、鳞状细胞癌、绳捆癌、毛细管扩张癌(carcinoma telangiectaticum)、毛细管扩张癌(carcinoma telangiectodes)、移行细胞癌、块状癌、结节性皮癌、疣状癌或绒毛状癌。
[0809]
可以根据本文公开的方法治疗的额外癌症包括例如白血病、霍奇金病、非霍奇金淋巴瘤、多发性骨髓瘤、神经母细胞瘤、乳腺癌、卵巢癌、肺癌、横纹肌肉瘤、原发性血小板增多症、原发性巨球蛋白血症、小细胞肺肿瘤、原发性脑瘤、胃癌、结肠癌、恶性胰岛瘤、恶性类癌、尿膀胱癌、癌前皮肤病灶、睾丸癌、淋巴瘤、甲状腺癌、甲状腺乳头状癌、神经母细胞瘤、神经内分泌癌、食管癌、泌尿生殖系道癌、恶性高钙血症、宫颈癌、子宫内膜癌、肾上腺皮质癌、前列腺癌、m
ü
llerian癌症(m
ü
llerian cancer)、卵巢癌、腹膜癌、输卵管癌或子宫乳头状浆液性癌。
[0810]
iii.试剂盒和制品
[0811]
本公开还提供了一种试剂盒,其包括(i)能够特异性检测编码来自表1的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2的基因生物标记物的rna的多个寡核苷酸探针。还提供了一种制品,其包括(i)能够特异性检测编码来自表1的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2的基因生物标记物的rna的多个寡核苷酸探针,其中所述制品包括微阵列。
[0812]
此类试剂盒和制品可以包括容器,每个容器具有一种或多种用于所述方法的各种试剂(例如,呈浓缩形式),包括例如一种或多种寡核苷酸(例如,能够与对应于本文公开的生物标记物基因的mrna杂交的寡核苷酸),或抗体(即,能够检测本文公开的生物标记物基因的蛋白质表达产物的抗体)。
[0813]
可以提供已经附着到固体支撑物的一种或多种寡核苷酸或抗体(例如捕获抗体)。可以提供已经缀合到可检测标记的一种或多种寡核苷酸或抗体。
[0814]
试剂盒还可以提供试剂、缓冲液和/或仪器以支持本文提供的方法的实践。
[0815]
在一些方面,试剂盒包括一个或多个核酸探针(例如,包含天然存在的和/或化学修饰的核苷酸单元的寡核苷酸),其能够例如在高度严格条件下与本文公开的生物标记物基因的基因序列的子序列杂交。在一些方面,能够例如在高度严格条件下与本文公开的生物标记物基因的基因序列的子序列杂交的一个或多个核酸探针(例如,包含天然存在的和/或化学修饰的核苷酸单元的寡核苷酸)附着到微阵列,例如微阵列芯片。在一些方面,微阵列是例如affymetrix、agilent、applied微阵列、arrayjet或illumina微阵列。在一些方面,阵列是dna微阵列。在一些方面,微阵列是cdna微阵列、rna微阵列、寡核苷酸微阵列、蛋白质微阵列、肽微阵列、组织微阵列或表型微阵列。
[0816]
根据本公开提供的试剂盒还可以包括描述本文公开的方法或其实际应用以对患者的癌症样品进行分类的小册子或说明书。试剂盒中所包括的说明书可以粘贴于包装材料上或可以作为包装说明书包括在内。虽然说明书通常是书写或印刷的材料,但它们不限于此类。设想能够存储此类说明并将其传达给终端用户的任何媒介。此类媒介包括但不限于电子存储媒介(例如,磁盘、磁带、磁片盒、芯片)、光学媒介(例如,cd rom)等。如本文所用,术语“说明书”可以包括提供说明书的互联网网站的地址。
[0817]
在一些方面,试剂盒是htg molecular edge-seq测序试剂盒。在其他方面,试剂盒是illumina测序试剂盒,例如用于novaseq、nextseq或hiseq 2500平台。
[0818]
iv.伴随诊断系统
[0819]
本文公开的方法可以作为伴随诊断提供,例如通过网络服务器可用,以告知临床医生或患者潜在的治疗选择。本文公开的方法可以包括采集或以其他方式获得生物样品并进行分析方法(例如,应用基于群体的分类器,诸如本文公开的基于标志1和标志2的分类器;或非基于群体的分类器,诸如基于本文公开的ann的分类模型),以将来自患者肿瘤的样品单独或与其他生物标记物组合分类为tme类别,并且基于tme类别分配(例如,特定基质表型的存在或不存在,即受试者对于基质表型或其组合是生物标记物阳性的和/或生物标记物阴性的)提供用于向患者施用的合适治疗(例如,本文公开的tme类别特定疗法或其组合)。
[0820]
由于所涉及的计算的复杂性,例如标志分数的计算、输入数据的预处理以应用ann模型、输入数据的预处理以训练ann、后处理ann的输出、训练ann或其任何组合,本文所述的方法的至少一些方面都可以使用计算机来实现。在一些方面,计算机系统包括通过总线电耦合的硬件元件,包括处理器、输入装置、输出装置、存储装置、计算机可读存储介质读取器、通信系统、处理加速(例如,dsp或专用处理器)和存储器。计算机可读存储介质读取器可以进一步耦合到计算机可读存储介质,所述组合综合表示远程、本地、固定和/或可移动存储装置加用于临时和/或更永久地包含计算机可读信息的存储介质、存储器等,其可以包括存储装置、存储器和/或任何其他此类可访问的系统资源。
[0821]
可以利用单个架构来实现一个或多个服务器,其可以根据当前希望的协议、协议变体、扩展等来进一步配置。然而,对于本领域的技术人员显而易见的是,各个方面可以根据更具体的应用要求良好地利用。也可以利用定制的硬件,并且/或者可以在硬件、软件或
两者中实现特定元件。此外,虽然可以采用与其他计算装置诸如网络输入/输出装置(未示出)的连接,但是应理解,也可以利用与其他计算装置的一个或多个有线、无线、调制解调器和/或其他连接。
[0822]
在一方面,所述系统还包括一个或多个用于向一个或多个处理器提供输入数据的装置。所述系统还包括用于存储排序数据元素的数据集的存储器。在另一方面,用于提供输入数据的装置包括用于检测数据元素的特征的检测器,例如像荧光板读取器、质谱仪或基因芯片读取器。
[0823]
所述系统可以另外包括数据库管理系统。用户请求或查询可以以数据库管理系统理解的适当语言格式化,所述数据库管理系统处理查询以从训练集的数据库中提取相关信息。所述系统可以连接到网络服务器和一个或多个客户端连接到的网络。如本领域已知的,网络可以是局域网(lan)或广域网(wan)。优选地,服务器包括运行计算机程序产品(例如,软件)以访问用于处理用户请求的数据库数据所必需的硬件。所述系统可以与输入装置通信以向系统提供关于数据元素的数据(例如,表达值)。在一方面,输入装置可以包括基因表达谱系统,其包括例如质谱仪、基因芯片或阵列读取器等。
[0824]
本文所述的一些方面可以被实现为包括计算机程序产品。计算机程序产品可以包括计算机可读介质,其具有包含在介质中的计算机可读程序代码,以用于使应用程序在具有数据库的计算机上执行。如本文所用,“计算机程序产品”是指呈自然或编程语言语句形式的有组织的指令集,其包含在任何性质(例如,书面、电子、磁性、光学或其他)物理介质上,并且可以与计算机或其他自动化数据处理系统一起使用。此类编程语言语句,在由计算机或数据处理系统执行时,使计算机或数据处理系统根据语句的特定内容进行动作。
[0825]
计算机程序产品包括但不限于:源代码和目标代码中的程序和/或嵌入在计算机可读介质中的测试或数据文库。此外,可以以多种形式提供使计算机系统或数据处理设备装置能够以预选方式起作用的计算机程序产品,其包括但不限于原始源代码、汇编代码、目标代码、机器语言、前述的加密或压缩版本以及任何和所有等价物。在一方面,提供了一种计算机程序产品,以实施本文公开的治疗、诊断、预后或监测方法,例如以基于根据本文公开的分类器,例如基于群体的分类器(例如,基于如本文公开的标志1和标志2)或非基于群体的分类器(例如,基于如本文公开的ann的分类模型)对来自患者的肿瘤样品或肿瘤微环境样品进行的分类来确定是否施用某种疗法。
[0826]
计算机程序产品包括体现可由计算装置或系统的处理器执行的程序代码的计算机可读介质,所述程序代码包括:
[0827]
(a)检索归因于来自受试者的生物样品的数据的代码,其中所述数据包括对应于生物样品中的生物标记物基因(例如,来自表1用于导出标志1的基因套组和来自表2用于导出标志2的基因套组,或来自表1和表2、或来自图28a-g中公开的基因集中的任一个、或来自用于训练ann的表5的基因套组)的表达水平数据(或以其他方式衍生自表达水平值的数据)。这些值也可以与对应的值组合,例如,患者当前的治疗方案或缺乏治疗方案;和
[0828]
(b)执行分类方法的代码,所述分类方法指示例如基于患者癌症的tme分类,例如基于群体的分类器(例如,基于如本文公开的标志1和标志2)或非基于群体的分类器(例如,基于如本文公开的ann的分类模型)是否向有需要的患者施用治疗剂。
[0829]
虽然各个方面已被描述为方法或装置,但是应理解,可以通过与计算机耦合的代
码(例如,驻留在计算机上或可由计算机访问的代码)来实现各个方面。例如,可以利用软件和数据库来实现以上讨论的许多方法。因此,除了通过硬件实现的方面以外,还应注意,这些方面可以通过使用包括计算机可用介质的制品来实现,所述计算机可用介质具有包含在其中的计算机可读程序代码,这使得能够实现在本说明书中公开的功能。因此,希望各方面在其程序代码方式中也被认为受到本专利的保护。
[0830]
此外,一些方面可以是存储在几乎任何类型的计算机可读存储器中的代码,包括但不限于ram、rom、磁介质、光学介质或磁光介质。甚至更通常,一些方面可以以软件或以硬件或其任何组合实现,包括但不限于在通用处理器上运行的软件、微代码、pla或asic。
[0831]
还设想一些方面可以作为体现在载波中的计算机信号以及通过传输介质传播的信号(例如,电信号和光信号)来实现。因此,以上讨论的各种类型的信息可以以结构,诸如数据结构格式化,并且作为电信号通过传输介质传输或存储在计算机可读介质上。
[0832]
v.额外的技术和测试
[0833]
可以例如与靶序列表达的测量或与本文公开的方法组合采用本领域已知的用于诊断和/或建议、选择、指定、推荐或以其他方式确定怀疑患有癌症的患者或患者类别的治疗过程的因素。因此,本文公开的方法可以包括额外的技术,诸如细胞学、组织学、超声分析、mri结果、ct扫描结果和psa水平的测量。
[0834]
用于分类疾病状态和/或指定治疗方式的经认证的测试也可以用于诊断、预测和/或监测受试者中癌症的状态或结果。经认证的测试可以包括用于表征一个或多个感兴趣的靶序列的表达水平的方式,以及来自认可使用所述测试来对生物样品的疾病状态进行分类的政府监管机构的认证。
[0835]
在一些方面,经认证的测试可以包括用于扩增反应的试剂,其用于检测和/或定量待在测试中表征的靶序列的表达。可以在有或没有在先前靶扩增的情况下使用探针核酸的阵列,用于测量靶标序列表达。
[0836]
所述测试可以提交给有权认证所述测试用于区分疾病状态和/或结果的机构。测试中使用的靶序列的表达水平的检测结果以及与疾病状态和/或结果的相关性可以提交给所述机构。可以获得授权测试的诊断和/或预测用途的认证。
[0837]
还提供了包括本文公开的基因集中的任一个的多个归一化表达水平的表达水平组合。在一些方面,基因集中的基因选自表1。在一些方面,基因集中的基因选自表2。在一些方面,基因集中的基因选自表1和表2(或图28a-g中公开的基因套组(基因集)中的任一个)。在一些方面,基因集选自表3或表4中公开的基因集,或图28a、28b、28c、28d、28e、28f或28g中公开的基因集中的任一个。可以通过进行本文所述的方法从个体患者或从一组患者获得表达水平来提供此类组合。表达水平可以通过本领域已知的任何方法归一化;可以在各个方面中使用的示例性归一化方法包括稳健多芯片平均(rma)、探针对数强度误差估计(plier)、基于非线性拟合(nlfit)分位数和非线性归一化及其组合。还可以对表情数据进行背景校正;可用于背景校正的示例性技术包括使用中值抛光探针建模和草图归一化进行归一化的强度模式。
[0838]
在一些方面,建立组合,使得组合中的基因组合相对于已知方法表现出改进的灵敏度和特异性。在考虑将一组基因包含在组合中时,表达测量中的小标准偏差与更大的特异性相关。其他变化的测量,诸如相关系数也可以用于这种能力。本公开还涵盖以上方法,
其中表达水平以至少约45%的特异性、至少约50%的特异性、至少约55%、至少约60%的特异性、至少约65%的特异性、至少约70%的特异性、至少约75%的特异性、至少约80%的特异性、至少约85%的特异性、至少约90%的特异性或至少约95%的特异性确定受试者中的癌症的状态或结果。
[0839]
在一些方面,本文公开的方法用于诊断、监测和/或预测癌症的状态或结果的准确度是至少约45%、至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约75%、至少约80%、至少约85%、至少约90%或至少约95%。
[0840]
分类器或生物标记物的准确度可以通过95%置信区间(ci)来确定。通常,如果95%ci不与1重叠,则认为分类器或生物标记物具有良好的准确度。在一些方面,分类器或生物标记物的95%ci是至少约1.08、至少约1.10、至少约1.12、至少约1.14、至少约1.15、至少约1.16、至少约1.17、至少约1.18、至少约1.19、至少约1.20、至少约1.21、至少约1.22、至少约1.23、至少约1.24、至少约1.25、至少约1.26、至少约1.27、至少约1.28、至少约1.29、至少约1.30、至少约1.31、至少约1.32、至少约1.33、至少约1.34、或至少约1.35或更大。分类器或生物标记物的95%ci可以是至少约1.14、至少约1.15、至少约1.16、至少约1.20、至少约1.21、至少约1.26或至少约1.28。分类器或生物标记物的95%ci可以是小于约1.75、小于约1.74、小于约1.73、小于约1.72、小于约1.71、小于约1.70、小于约1.69、小于约1.68、小于约1.67、小于约1.66、小于约1.65、小于约1.64、小于约1.63、小于约1.62、小于约1.61、小于约1.60、小于约1.59、小于约1.58、小于约1.57、小于约1.56、小于约1.55、小于约1.54、小于约1.53、小于约1.52、小于约1.51、小于约1.50或更小。分类器或生物标记物的95%ci可以是小于约1.61、小于约1.60、小于约1.59、小于约1.58、小于约1.56、1.55或1.53。分类器或生物标记物的95%ci可以在约1.10至约1.70之间、约1.12至约1.68之间、约1.14至约1.62之间、约1.15至约1.61之间、约1.15至约1.59之间、约1.16至约1.160之间、约1.19至约1.55之间、约1.20至约1.54之间、约1.21至约1.53之间、约1.26至约1.63之间、约1.27至约1.61之间或约1.28至约1.60之间。
[0841]
在一些方面,生物标记物或分类器的准确度取决于95%ci范围的差值(例如,95%ci区间的高值和低值的差异)。通常,与具有95%ci区间范围的较小差值的生物标记物或分类器相比,具有95%ci区间范围的较大差值的生物标记物或分类器具有更大的可变性并且被认为准确度更小。在一些方面,如果95%ci范围的差值小于约0.60、小于约0.55、小于约0.50、小于约0.49、小于约0.48、小于约0.47、小于约0.46、小于约0.45、小于约0.44、小于约0.43、小于约0.42、小于约0.41、小于约0.40、小于约0.39、小于约0.38、小于约0.37、小于约0.36、小于约0.35、小于约0.34、小于约0.33、小于约0.32、小于约0.31、小于约0.30、小于约0.29、小于约0.28、小于约0.27、小于约0.26、小于约0.25或更小,则生物标记物或分类器被认为更准确。生物标记物或分类器的95%ci范围的差值可以是小于约0.48、小于约0.45、小于约0.44、小于约0.42、小于约0.40、小于约0.37、小于约0.35、小于约0.33或小于约0.32。在一些方面,生物标记物或分类器的95%ci范围的差值在约0.25至约0.50之间、在约0.27至约0.47之间或在约0.30至约0.45之间。
[0842]
在一些方面,本文公开的方法的灵敏度是至少约45%。在一些方面,灵敏度是至少约50%。在一些方面,灵敏度是至少约55%。在一些方面,灵敏度是至少约60%。在一些方面,灵敏度是至少约65%。在一些方面,灵敏度是至少约70%。在一些方面,灵敏度是至少约
75%。在一些方面,灵敏度是至少约80%。在一些方面,灵敏度是至少约85%。在一些方面,灵敏度是至少约90%。在一些方面,灵敏度是至少约95%。
[0843]
在一些方面,本文公开的分类器或生物标记物是临床上重要的。在一些方面,分类器或生物标记物的临床重要性通过auc值确定。为了是临床上重要的,auc值是至少约0.5、至少约0.55、至少约0.6、至少约0.65、至少约0.7、至少约0.75、至少约0.8、至少约0.85、至少约0.9或至少约0.95。分类器或生物标记物的临床重要性可以通过准确度百分比确定。例如,如果分类器或生物标记物的准确度是至少约50%、至少约55%、至少约60%、至少约65%、至少约70%、至少约72%、至少约75%、至少约77%、至少约80%、至少约82%、至少约84%、至少约86%、至少约88%、至少约90%、至少约92%、至少约94%、至少约96%或至少约98%,则分类器或生物标记物被确定为临床上重要的。
[0844]
在其他方面,分类器或生物标记物的临床重要性通过中值倍数差值(mdf)值确定。为了是临床上重要的,mdf值是至少约0.8、至少约0.9、至少约1.0、至少约1.1、至少约1.2、至少约1.3、至少约1.4、至少约1.5、至少约1.6、至少约1.7、至少约1.9或至少约2.0。在一些方面,mdf值大于或等于1.1。在一些方面,mdf值大于或等于1.2。可替代地或另外,分类器或生物标记物的临床重要性通过t检验p值确定。在一些方面,为了是临床上重要的,t检验p值小于约0.070、小于约0.065、小于约0.060、小于约0.055、小于约0.050、小于约0.045、小于约0.040、小于约0.035、小于约0.030、小于约0.025、小于约0.020、小于约0.015、小于约0.010、小于约0.005、小于约0.004或小于约0.003。t检验p值可以是小于约0.050。可替代地,t检验p值是小于约0.010。
[0845]
在一些方面,分类器或生物标记物的临床重要性通过临床结果确定。例如,不同的临床结果可以具有不同的auc值、mdf值、t检验p值和准确度值的最小或最大阈值,其将确定分类器或生物标记物是否是临床上重要的。在另一个实例中,如果t检验p值小于约0.08、小于约0.07、小于约0.06、小于约0.05、小于约0.04、小于约0.03、小于约0.02、小于约0.01、小于约0.005、小于约0.004、小于约0.003、小于约0.002或小于约0.001,则分类器或生物标记物被认为是临床上重要的。
[0846]
在一些方面,分类器或生物标记物的性能是基于优势比。如果优势比是至少约1.30、至少约1.31、至少约1.32、至少约1.33、至少约1.34、至少约1.35、至少约1.36、至少约1.37、至少约1.38、至少约1.39、至少约1.40、至少约1.41、至少约1.42、至少约1.43、至少约1.44、至少约1.45、至少约1.46、至少约1.47、至少约1.48、至少约1.49、至少约1.50、至少约1.52、至少约1.55、至少约1.57、至少约1.60、至少约1.62、至少约1.65、至少约1.67、至少约1.70或更高,则分类器或生物标记物可以被认为具有良好的性能。在一些方面,分类器或生物标记物的优势比是至少约1.33。
[0847]
分类器和/或生物标记物的临床重要性可以是基于单变量分析优势比p值(uvaorpval)。分类器和/或生物标记物的单变量分析优势比p值(uvaorpval)可以在约0与约0.4之间。分类器和/或生物标记物的单变量分析优势比p值(uvaorpval)可以在约0与约0.3之间。分类器和/或生物标记物的单变量分析优势比p值(uvaorpval))可以在约0与约0.2之间。分类器和/或生物标记物的单变量分析优势比p值(uvaorpval))可以是小于或等于0.25、小于或等于约0.22、小于或等于约0.21、小于或等于约0.20、小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于
约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。
[0848]
分类器和/或生物标记物的单变量分析优势比p值(uvaorpval)可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的单变量分析优势比p值(uvaorpval)可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0849]
分类器和/或生物标记物的临床重要性可以是基于多变量分析优势比p值(mvaorpval)。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以在约0与约1之间。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以在约0与约0.9之间。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以在约0与约0.8之间。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval)可以是小于或等于约0.90、小于或等于约0.88、小于或等于约0.86、小于或等于约0.84、小于或等于约0.82或小于或等于约0.80。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以是小于或等于约0.78、小于或等于约0.76、小于或等于0.74、小于或等于约0.72、小于或等于约0.70、小于或等于约0.68、小于或等于约0.66、小于或等于约0.64、小于或等于约0.62、小于或等于约0.60、小于或等于约0.58、小于或等于约0.56、小于或等于约0.54、小于或等于约0.52或小于或等于约0.50。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval)可以是小于或等于约0.48、小于或等于约0.46、小于或等于约0.44、小于或等于约0.42、小于或等于约0.40、小于或等于约0.38、小于或等于约0.36、小于或等于约0.34、小于或等于约0.32、小于或等于约0.30、小于或等于约0.28、小于或等于约0.26、小于或等于约0.25、小于或等于约0.22、小于或等于约0.21、小于或等于约0.20、小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的多变量分析优势比p值(mvaorpval))可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0850]
分类器和/或生物标记物的临床重要性可以是基于kaplan meier p值(km p值)。分类器和/或生物标记物的kaplan meier p值(km p值)可以在约0与约0.8之间。分类器和/或生物标记物的kaplan meier p值(km p值)可以在约0与约0.7之间。分类器和/或生物标记物的kaplan meier p值(km p值)可以是小于或等于约0.80、小于或等于约0.78、小于或等于约0.76、小于或等于0.74、小于或等于约0.72、小于或等于约0.70、小于或等于约0.68、小于或等于约0.66、小于或等于约0.64、小于或等于约0.62、小于或等于约0.60、小于或等于约0.58、小于或等于约0.56、小于或等于约0.54、小于或等于约0.52或小于或等于约0.50。分类器和/或生物标记物的kaplan meier p值(km p值)可以是小于或等于约0.48、小
于或等于约0.46、小于或等于约0.44、小于或等于约0.42、小于或等于约0.40、小于或等于约0.38、小于或等于约0.36、小于或等于约0.34、小于或等于约0.32、小于或等于约0.30、小于或等于约0.28、小于或等于约0.26、小于或等于约0.25、小于或等于约0.22、小于或等于约0.21、小于或等于约0.20、小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。分类器和/或生物标记物的kaplan meier p值(km p值)可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的kaplan meier p值(km p值)可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0851]
分类器和/或生物标记物的临床重要性可以是基于存活auc值(survauc)。分类器和/或生物标记物的存活auc值(survauc)可以在约0-1之间。分类器和/或生物标记物的存活auc值(survauc)可以在约0至约0.9之间。分类器和/或生物标记物的存活auc值(survauc)可以是小于或等于约1、小于或等于约0.98、小于或等于约0.96、小于或等于约0.94、小于或等于约0.92、小于或等于约0.90、小于或等于约0.88、小于或等于约0.86、小于或等于约0.84、小于或等于约0.82或小于或等于约0.80。分类器和/或生物标记物的存活auc值(survauc)可以是小于或等于约0.80、小于或等于约0.78、小于或等于约0.76、小于或等于0.74、小于或等于约0.72、小于或等于约0.70、小于或等于约0.68、小于或等于约0.66、小于或等于约0.64、小于或等于约0.62、小于或等于约0.60、小于或等于约0.58、小于或等于约0.56、小于或等于约0.54、小于或等于约0.52或小于或等于约0.50。分类器和/或生物标记物的存活auc值(survauc)可以是小于或等于约0.48、小于或等于约0.46、小于或等于约0.44、小于或等于约0.42、小于或等于约0.40、小于或等于约0.38、小于或等于约0.36、小于或等于约0.34、小于或等于约0.32、小于或等于约0.30、小于或等于约0.28、小于或等于约0.26、小于或等于约0.25、小于或等于约0.22、小于或等于约0.21、小于或等于约0.20、小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。分类器和/或生物标记物的存活auc值(survauc)可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的存活auc值(survauc)可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001
[0852]
分类器和/或生物标记物的临床重要性可以是基于单变量分析风险比p值(uvahrpval)。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以在约0至约0.4之间。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以在约0至约0.3之间。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以是小于或等于约0.40、小于或等于约0.38、小于或等于约0.36、小于或等于约0.34或小于或等于约0.32。
分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以是小于或等于约0.30、小于或等于约0.29、小于或等于约0.28、小于或等于约0.27、小于或等于约0.26、小于或等于约0.25、小于或等于约0.24、小于或等于约0.23、小于或等于约0.22、小于或等于约0.21或小于或等于约0.20。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以是小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的单变量分析风险比p值(uvahrpval)可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0853]
分类器和/或生物标记物的临床重要性可以是基于多变量分析风险比p值(mvahrpval)mva hrpval。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以在约0至约1之间。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以在约0至约0.9之间。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以是小于或等于约1、小于或等于约0.98、小于或等于约0.96、小于或等于约0.94、小于或等于约0.92、小于或等于约0.90、小于或等于约0.88、小于或等于约0.86、小于或等于约0.84、小于或等于约0.82或小于或等于约0.80。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以是小于或等于约0.80、小于或等于约0.78、小于或等于约0.76、小于或等于0.74、小于或等于约0.72、小于或等于约0.70、小于或等于约0.68、小于或等于约0.66、小于或等于约0.64、小于或等于约0.62、小于或等于约0.60、小于或等于约0.58、小于或等于约0.56、小于或等于约0.54、小于或等于约0.52或小于或等于约0.50。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以是小于或等于约0.48、小于或等于约0.46、小于或等于约0.44、小于或等于约0.42、小于或等于约0.40、小于或等于约0.38、小于或等于约0.36、小于或等于约0.34、小于或等于约0.32、小于或等于约0.30、小于或等于约0.28、小于或等于约0.26、小于或等于约0.25、小于或等于约0.22、小于或等于约0.21、小于或等于约0.20、小于或等于约0.19、小于或等于约0.18、小于或等于约0.17、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12或小于或等于约0.11。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)mva hrpval可以是小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0854]
分类器和/或生物标记物的临床重要性可以是基于多变量分析风险比p值
(mvahrpval)。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)可以在约0至约0.60之间。分类器和/或生物标记物的重要性可以是基于多变量分析风险比p值(mvahrpval)。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)可以在约0至约0.50之间。分类器和/或生物标记物的重要性可以是基于多变量分析风险比p值(mvahrpval)。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)可以是小于或等于约0.50、小于或等于约0.47、小于或等于约0.45、小于或等于约0.43、小于或等于约0.40、小于或等于约0.38、小于或等于约0.35、小于或等于约0.33、小于或等于约0.30、小于或等于约0.28、小于或等于约0.25、小于或等于约0.22、小于或等于约0.20、小于或等于约0.18、小于或等于约0.16、小于或等于约0.15、小于或等于约0.14、小于或等于约0.13、小于或等于约0.12、小于或等于约0.11或小于或等于约0.10。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)可以是小于或等于约0.10、小于或等于约0.09、小于或等于约0.08、小于或等于约0.07、小于或等于约0.06、小于或等于约0.05、小于或等于约0.04、小于或等于约0.03、小于或等于约0.02或小于或等于约0.01。分类器和/或生物标记物的多变量分析风险比p值(mvahrpval)可以是小于或等于约0.01、小于或等于约0.009、小于或等于约0.008、小于或等于约0.007、小于或等于约0.006、小于或等于约0.005、小于或等于约0.004、小于或等于约0.003、小于或等于约0.002或小于或等于约0.001。
[0855]
本文公开的分类器和/或生物标记物在提供来自受试者的样品的临床相关分析方面可以优于当前分类器或临床变量。在一些方面,与当前分类器或临床变量相比,所述分类器或生物标记物可以更准确地预测临床结果或状态。例如,分类器或生物标记物可以更准确地预测转移性疾病。可替代地,分类器或生物标记物可以更准确地预测无疾病证据。在一些方面,分类器或生物标记物可以更准确地由无疾病预测死亡。本文公开的分类器或生物标记物的性能可以是基于auc值、优势比、95%ci、95%ci范围的差值、p值或其任何组合。
[0856]
本文公开的分类器和/或生物标记物的性能可以通过auc值来确定,并且性能的改善可以通过本文公开的分类器或生物标记物的auc值与当前分类器或临床变量的auc值的差值来确定。在一些方面,当本文公开的分类器和/或生物标记物的auc值比当前分类器或临床变量的auc值大至少约0.05、至少约0.06、至少约0.07、至少约0.08、至少约0.09、至少约0.10、至少约0.11、至少约0.12、至少约0.13、至少约0.14、至少约0.15、至少约0.16、至少约0.17、至少约0.18、至少约0.19、至少约0.20、至少约0.022、至少约0.25、至少约0.27、至少约0.30、至少约0.32、至少约0.35、至少约0.37、至少约0.40、至少约0.42、至少约0.45、至少约0.47、或至少约0.50或更大时,本文公开的分类器和/或生物标记物优于当前分类器或临床变量。在一些方面,本文公开的分类器和/或生物标记物的auc值比当前分类器或临床变量的auc值大至少约0.10。在一些方面,本文公开的分类器和/或生物标记物的auc值比当前分类器或临床变量的auc值大至少约0.13。在一些方面,本文公开的分类器和/或生物标记物的auc值比当前分类器或临床变量的auc值大至少约0.18。
[0857]
本文公开的分类器和/或生物标记物的性能可以通过优势比来确定,并且性能的改善可以通过将本文公开的分类器或生物标记物的优势比与当前分类器或临床变量的优势比进行比较来确定。两个或更多个分类器、生物标记物和/或临床变量的性能的比较通常可以是基于第一分类器、生物标记物或临床变量的(1-优势比)的绝对值与第二分类器、生物标记物或临床变量的(1-优势比)的绝对值的比较。通常,与具有较小的(1-优势比)的绝
对值的分类器、生物标记物或临床变量相比,具有较大的(1-优势比)的绝对值的分类器、生物标记物或临床变量可以被认为具有更好的性能。
[0858]
在一些方面,分类器、生物标记物或临床变量的性能是基于优势比和95%置信区间(ci)的比较。例如,第一分类器、生物标记物或临床变量可以具有比第二分类器、生物标记物或临床变量更大的(1-优势比)的绝对值,然而,第一分类器、生物标记物或临床变量的95%ci可以与1重叠(例如,准确度差),而第二分类器、生物标记物或临床变量的95%ci不与1重叠。在这种情况下,第二分类器、生物标记物或临床变量被认为优于第一分类器、生物标记物或临床变量,因为第一分类器、生物标记物或临床变量的准确度小于第二分类器、生物标记物或临床变量的准确度。在另一个实例中,基于优势比的比较,第一分类器、生物标记物或临床变量可以优于第二分类器、生物标记物或临床变量;然而,第一分类器、生物标记物或临床变量的95%ci的差值是第二分类器、生物标记物或临床变量的95%ci的至少约2倍。在这种情况下,第二分类器、生物标记物或临床变量被认为优于第一分类器。
[0859]
在一些方面,本文公开的分类器或生物标记物比当前分类器或临床变量更准确。如果本文公开的分类器或生物标记物的95%ci的范围不跨越1或与1重叠并且当前分类器或临床变量的95%ci的范围跨越1或与1重叠,则本文公开的分类器或生物标记物比当前分类器或临床变量更准确。
[0860]
在一些方面,本文公开的分类器或生物标记物比当前分类器或临床变量更准确。当本文公开的分类器或生物标记物的95%ci范围的差值是当前分类器或临床变量的95%ci范围的差值的约0.70、约0.60、约0.50、约0.40、约0.30、约0.20、约0.15、约0.14、约0.13、约0.12、约0.10、约0.09、约0.08、约0.07、约0.06、约0.05、约0.04、约0.03或约0.02倍小时,本文公开的分类器或生物标记物比当前分类器或临床变量更准确。当本文公开的分类器或生物标记物的95%ci范围的差值是当前分类器或临床变量的95%ci范围的差值的约0.20至约0.04倍小时,本文公开的分类器或生物标记物比当前分类器或临床变量更准确。
[0861]
vi.实施方式
[0862]
本公开提供了用于确定有需要的受试者中的癌症的肿瘤微环境(tme)的群体方法。在一些方面,所述群体方法包括确定组合生物标记物,其包括(a)标志1分数;和(b)标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定。
[0863]
还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用ia类别tme疗法,其中在所述施用之前,所述受试者的肿瘤被鉴定为具有特定tme。此tme可以例如被定义为组合生物标记物,其包括(a)负标志1分数;和(b)正标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定。
[0864]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括(a)在施用之前,鉴定表现出包括以下的组合生物标记物的受试者:(a)负标志1分数;和(b)正标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第
二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定;和(b)向所述受试者施用ia类别tme疗法。
[0865]
还提供了一种用于鉴定罹患适合用ia类别tme疗法治疗的癌症的人受试者的方法,所述方法包括(i)通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定标志1分数;和(ii)通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定标志2分数,其中在施用之前,包括(a)负标志1分数和(b)正标志2分数的组合生物标记物的存在指示可以施用ia类别tme疗法来治疗所述癌症。
[0866]
在一些方面,所述ia类别tme疗法包括检查点调节剂疗法。在一些方面,所述检查点调节剂疗法包括施用刺激性免疫检查点分子的激活剂。在一些方面,所述刺激性免疫检查点分子的激活剂是针对gitr、ox-40、icos、4-1bb或其组合的抗体分子。在一些方面,所述检查点调节剂疗法包括施用rorγ激动剂。
[0867]
在一些方面,所述抑制性免疫检查点分子的抑制剂是针对单独pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体,或与以下组合:tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂、cd86激动剂或其组合。
[0868]
在一些方面,所述抑制性免疫检查点分子的抑制剂是针对单独pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体,或与以下组合:tim-3的调节剂(例如,激动剂或拮抗剂)、lag-3的调节剂(例如,激动剂或拮抗剂)、btla的调节剂(例如,激动剂或拮抗剂)、tigit的调节剂(例如,激动剂或拮抗剂)、vista的调节剂(例如,激动剂或拮抗剂)、tgf-β或其受体的调节剂(例如,激动剂或拮抗剂)、lair1的调节剂(例如,激动剂或拮抗剂)、cd160的调节剂(例如,激动剂或拮抗剂)、2b4的调节剂(例如,激动剂或拮抗剂)、gitr的调节剂(例如,激动剂或拮抗剂)、ox40的调节剂(例如,激动剂或拮抗剂)、4-1bb(cd137)的调节剂(例如,激动剂或拮抗剂)、cd2的调节剂(例如,激动剂或拮抗剂)、cd27的调节剂(例如,激动剂或拮抗剂)、cds的调节剂(例如,激动剂或拮抗剂)、icam-1的调节剂(例如,激动剂或拮抗剂)、lfa-1(cd11a/cd18)的调节剂(例如,激动剂或拮抗剂)、icos(cd278)的调节剂(例如,激动剂或拮抗剂)、cd30的调节剂(例如,激动剂或拮抗剂)、cd40的调节剂(例如,激动剂或拮抗剂)、baffr的调节剂(例如,激动剂或拮抗剂)、hvem的调节剂(例如,激动剂或拮抗剂)、cd7的调节剂(例如,激动剂或拮抗剂)、light的调节剂(例如,激动剂或拮抗剂)、nkg2c的调节剂(例如,激动剂或拮抗剂)、slamf7的调节剂(例如,激动剂或拮抗剂)、nkp80的调节剂(例如,激动剂或拮抗剂)、cd86的调节剂(例如,激动剂或拮抗剂)或其任何组合。
[0869]
在一些方面,所述抗pd-1抗体包括纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、tsr-042、信迪利单抗、替雷利珠单抗或其抗原结合部分。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单
072、ly3300054、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗交叉竞争与人pd-1的结合。
[0875]
在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。在一些方面,所述抗ctla-4抗体包括伊匹单抗(ipilimumab)或双特异性抗体xmab20717(抗pd-1/抗ctla-4)或其抗原结合部分。在一些方面,所述抗ctla-4与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)结合相同的ctla-4表位。在一些方面,所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188和tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;(iii)抗ctla-4抗体,其是伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4),或(iv)其组合。
[0876]
在一些方面,所述抗血管生成疗法包括施用选自由以下组成的组中的抗vegf抗体:伐利苏单抗、贝伐珠单抗、那赛昔珠单抗(抗dll4/抗vegf双特异性)及其组合。在一些方面,所述抗血管生成疗法包括施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2抗体。在一些方面,所述抗vegfr2抗体包括雷莫卢单抗(ramucirumab)。
[0877]
在一些方面,所述抗血管生成疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。在一些方面,所述抗免疫抑制疗法包括施用抗ps抗体、抗ps靶向抗体、结合β2-糖蛋白1的抗体、pi3kγ的抑制剂、腺苷通路抑制剂、ido的抑制剂、tim的抑制剂、lag3的抑制剂、tgf-β的抑制剂、cd47抑制剂或其组合。在一些方面,所述抗ps靶向抗体是巴维妥昔单抗或结合β2-糖蛋白1的抗体。在一些方面,所述pi3kγ抑制剂是ly3023414(samotolisib)或ipi-549。
[0878]
在一些方面,所述腺苷通路抑制剂是ab-928。在一些方面,所述tgfβ抑制剂是ly2157299(galunisertib),或者所述tgfβr1抑制剂是ly3200882。在一些方面,所述cd47抑制剂是莫洛利单抗(magrolimab,5f9)。在一些方面,所述cd47抑制剂靶向sirpα。在一些方面,所述免疫抑制疗法包括施用tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂、cd86激动剂或其组合。
[0879]
在一些方面,所述抗免疫抑制疗法包括施用tim-3的调节剂(例如,激动剂或拮抗剂)、lag-3的调节剂(例如,激动剂或拮抗剂)、btla的调节剂(例如,激动剂或拮抗剂)、tigit的调节剂(例如,激动剂或拮抗剂)、vista的调节剂(例如,激动剂或拮抗剂)、tgf-β或其受体的调节剂(例如,激动剂或拮抗剂)、lair1的调节剂(例如,激动剂或拮抗剂)、cd160的调节剂(例如,激动剂或拮抗剂)、2b4的调节剂(例如,激动剂或拮抗剂)、gitr的调节剂(例如,激动剂或拮抗剂)、ox40的调节剂(例如,激动剂或拮抗剂)、4-1bb(cd137)的调节剂(例如,激动剂或拮抗剂)、cd2的调节剂(例如,激动剂或拮抗剂)、cd27的调节剂(例如,激动剂或拮抗剂)、cds的调节剂(例如,激动剂或拮抗剂)、icam-1的调节剂(例如,激动剂或拮抗
剂)、lfa-1(cd11a/cd18)的调节剂(例如,激动剂或拮抗剂)、icos(cd278)的调节剂(例如,激动剂或拮抗剂)、cd30的调节剂(例如,激动剂或拮抗剂)、cd40的调节剂(例如,激动剂或拮抗剂)、baffr的调节剂(例如,激动剂或拮抗剂)、hvem的调节剂(例如,激动剂或拮抗剂)、cd7的调节剂(例如,激动剂或拮抗剂)、light的调节剂(例如,激动剂或拮抗剂)、nkg2c的调节剂(例如,激动剂或拮抗剂)、slamf7的调节剂(例如,激动剂或拮抗剂)、nkp80的调节剂(例如,激动剂或拮抗剂)、cd86的调节剂(例如,激动剂或拮抗剂)或其任何组合。
[0880]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用id类别tme疗法,其中在所述施用之前,所述受试者被鉴定为表现出包括以下的组合生物标记物:(a)负标志1分数;和(b)负标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定。
[0881]
还提供了一种用于治疗罹患癌症的人受试者的方法,其包括(a)在施用之前,鉴定表现出包括以下的组合生物标记物的受试者:(a)负标志1分数;和(b)负标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定;和(b)向所述受试者施用id类别tme疗法。
[0882]
还提供了一种用于鉴定罹患适合用id类别tme疗法治疗的癌症的人受试者的方法,所述方法包括(i)通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定标志1分数;和(ii)通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定标志2分数,其中在施用之前,包括(a)负标志1分数和(b)负标志2分数的组合生物标记物的存在指示可以施用id类别tme疗法来治疗所述癌症。
[0883]
在一些方面,所述id类别tme疗法包括在施用引发免疫应答的疗法的同时或之后施用检查点调节剂疗法。在一些方面,所述引发免疫应答的疗法是疫苗、car-t或新表位疫苗。在一些方面,所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。在一些方面,所述抑制性免疫检查点分子的抑制剂是针对pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体。
[0884]
在一些方面,所述抗pd-1抗体包括纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042或其抗原结合部分。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042交叉竞争与人pd-1的结合。在一些方面,所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042结合相同的表位。在一些方面,所述抗pd-l1抗体包括阿维单抗、阿替利珠单抗、cx-072、ly3300054、德瓦鲁单抗或其抗原结合部分。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗交叉竞争与人pd-l1的结合。在一些方面,所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。
[0885]
在一些方面,所述抗ctla-4抗体包括伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)或其抗原结合部分。在一些方面,所述抗ctla-4与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)交叉竞争与人ctla-4的结合。在一些方面,所述抗ctla-4抗体与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)结合相同的ctla-4表位。在一些方面,所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗和tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;(iv)抗ctla-4抗体,其是伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4),或(iii)其组合。
[0886]
本公开提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用a类别tme疗法,其中在所述施用之前,所述受试者被鉴定为表现出包括以下的组合生物标记物:(a)正标志1分数;和(b)负标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定。
[0887]
还提供了一种用于治疗罹患癌症的人受试者的方法,其包括(a)在施用之前,鉴定表现出包括以下的组合生物标记物的受试者:(a)正标志1分数;和(b)负标志2分数,在施用之前,其中(i)标志1分数通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定;和(b)向所述受试者施用a类别tme疗法。
[0888]
还提供了一种用于鉴定罹患适合用a类别tme疗法治疗的癌症的人受试者的方法,所述方法包括(i)通过测量从所述受试者获得的第一样品中选自表3(或选自图28a-28g)的基因套组的表达水平来确定标志1分数;和(ii)通过测量从所述受试者获得的第二样品中选自表4(或选自图28a-28g)的基因套组的表达水平来确定标志2分数,其中在施用之前,包括(a)正标志1分数和(b)负标志2分数的组合生物标记物的存在指示可以施用a类别tme疗法来治疗所述癌症。
[0889]
在一些方面,所述a类别tme疗法包括vegf靶向疗法和其他抗血管生成剂、血管生成素1(ang1)的抑制剂、血管生成素2(ang2)的抑制剂、dll4的抑制剂、抗vegf和抗dll4的双特异性抑制剂、tki抑制剂、抗fgf抗体、抗fgfr1抗体、抗fgfr2抗体、抑制fgfr1的小分子、抑制fgfr2的小分子、抗plgf抗体、针对plgf受体的小分子、针对plgf受体的抗体、抗vegfb抗体、抗vegfc抗体、抗vegfd抗体、针对vegf/plgf捕获分子的抗体诸如阿柏西普或ziv-阿柏西普、抗dll4抗体或抗notch疗法,诸如γ-分泌酶的抑制剂。
[0890]
在一些方面,所述tki抑制剂选自由以下组成的组:卡博替尼、凡德他尼、替沃扎尼、阿西替尼、乐伐替尼、索拉非尼、瑞戈非尼、舒尼替尼、呋喹替尼、帕唑帕尼及其任何组合。在一些方面,tki抑制剂是呋喹替尼。在一些方面,所述vegf靶向疗法包括施用抗vegf抗体或其抗原结合部分。
[0891]
在一些方面,所述抗vegf抗体包括伐利苏单抗、贝伐珠单抗或其抗原结合部分。在一些方面,所述抗vegf抗体与伐利苏单抗或贝伐珠单抗交叉竞争与人vegf a的结合。在一
些方面,所述抗vegf抗体与伐利苏单抗或贝伐珠单抗结合相同的表位。在一些方面,所述vegf靶向疗法包括施用抗vegfr抗体。在一些方面,所述抗vegfr抗体是抗vegfr2抗体。在一些方面,所述抗vegfr2抗体包括雷莫卢单抗或其抗原结合部分。
[0892]
在一些方面,所述双特异性抗vegf/抗dll4抗体包括那赛昔珠单抗或其抗原结合部分。在一些方面,所述双特异性抗vegf/抗dll4抗体与那赛昔珠单抗交叉竞争与人vegf和/或dll4的结合。在一些方面,所述双特异性抗vegf/抗dll4抗体与那赛昔珠单抗结合相同的vegf和/或dll4表位。
[0893]
在一些方面,所述a类别tme疗法包括施用血管生成素/tie2靶向疗法。在一些方面,所述血管生成素/tie2靶向疗法包括施用内皮糖蛋白和/或血管生成素。在一些方面,所述a类别tme疗法包括施用dll4靶向疗法。在一些方面,所述dll4靶向疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。在本文公开的方法的一些方面,所述方法还包括(a)施用化学疗法;(b)进行手术;(c)施用放射疗法;或(d)其任何组合。
[0894]
在一些方面,选自表4的基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60或61个选自表2的基因或1至124个选自图28a-28g的基因套组。在一些方面,所述基因套组是选自表4或图28a-28g的基因套组。在一些方面,选自表3的基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个选自表1的基因或1至124个选自图28a-28g的基因套组。在一些方面,所述基因套组是选自表3或图28a-28g的基因套组。
[0895]
在一些方面,所述第一样品和所述第二样品是相同的样品。在一些方面,所述第一样品和所述第二样品是不同的样品。在一些方面,所述第一样品和/或所述第二样品包括瘤内组织。在一些方面,所述表达水平是表达蛋白质水平。在一些方面,所述表达水平是转录rna表达水平。在一些方面,所述rna表达水平使用测序或测量rna的任何技术来确定。在一些方面,所述测序是下一代测序(ngs)。在一些方面,所述ngs选自由以下组成的组:rna-seq、edgeseq、pcr、nanostring或其组合。在一些方面,所述rna表达水平使用荧光来确定。在一些方面,所述rna表达水平使用affymetrix微阵列或agilent微阵列来确定。在一些方面,rna表达水平经受分位数归一化。在一些方面,所述分位数归一化包括将输入rna水平值分箱成分位数。在一些方面,所述输入rna水平被分箱成100个分位数。在一些方面,所述分位数归一化包括将所述rna表达水平分位数转换为正态输出分布函数。
[0896]
在一些方面,标志分数的计算包括(i)测量来自受试者的测试样品中基因套组中的每个基因的表达水平;(ii)对于每个基因,从步骤(i)的表达水平中减去从参考样品中此基因的表达水平获得的平均表达值;(iii)对于每个基因,将步骤(ii)中获得的值除以从参考样品的表达水平获得的每个基因的标准偏差;和(iv)将步骤(iii)中获得的所有值相加,并且将所得的数除以基因套组中基因数的平方根;其中如果(iv)中获得的值大于零,则标志分数为正标志分数,并且其中如果(iv)中获得的值小于零,则标志分数为负标志分数。
[0897]
在一些方面,参考样品包括参考表达水平的集合。在一些方面,参考表达值是标准化参考值。在一些方面,参考表达值从样品群体获得。在一些方面,参考表达水平来源于公
开可用的数据库或相互归一化的数据库组合。在一些方面,参考样品包括从不同群体获得的组织样品。在一些方面,参考样品包括在不同时间点采集的样品。在一些方面,不同时间点是较早的时间点。
[0898]
在一些方面,所述癌症是肿瘤。在一些方面,所述肿瘤是癌。在一些方面,所述肿瘤选自由以下组成的组:胃癌、结直肠癌、肝癌(肝细胞癌,hcc)、卵巢癌、乳腺癌、nsclc、膀胱癌、肺癌、胰腺癌、头颈癌、淋巴瘤、子宫癌、肾癌或肾癌(renal/kidney cancer)、胆管癌、前列腺癌、睾丸癌、尿道癌、阴茎癌、胸癌、直肠癌、脑癌(胶质瘤和胶质母细胞瘤)、子宫颈腮腺癌(cervicalparotid cancer)、食管癌、胃食管癌、喉癌、甲状腺癌、腺癌、神经母细胞瘤、黑色素瘤和默克尔细胞癌(merkel cell carcinoma)。在一些方面,所述癌症是复发性的。在一些方面,所述癌症是难治性的。在一些方面,在至少一种包括施用至少一种抗癌剂的先前疗法后,所述癌症是难治性的。在一些方面,所述癌症是转移性的。
[0899]
在一些方面,所述施用有效地治疗所述癌症。在一些方面,所述施用减少癌症负荷。在一些方面,与所述施用之前的癌症负荷相比,癌症负荷减少至少约10%、至少约20%、至少约30%、至少约40%或约50%。在一些方面,在所述初始施用之后,所述受试者表现出至少约1个月、至少约2个月、至少约3个月、至少约4个月、至少约5个月、至少约6个月、至少约7个月、至少约8个月、至少约9个月、至少约10个月、至少约11个月、至少约一年、至少约18个月、至少约两年、至少约三年、至少约四年或至少约五年的无进展存活。
[0900]
在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的稳定疾病。在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的部分应答。
[0901]
在一些方面,在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的完全应答。
[0902]
在一些方面,与未表现出组合生物标记物的受试者的无进展存活概率相比,所述施用使无进展存活概率提高至少约10%、至少约20%、至少约30%、至少约40%、至少约50%、至少约60%、至少约70%、至少约80%、至少约90%、至少约100%、至少约110%、至少约120%、至少约130%、至少约140%或至少约150%。
[0903]
在一些方面,与未表现出组合生物标记物的受试者的总体存活概率相比,所述施用使总体存活概率提高至少约25%、至少约50%、至少约75%、至少约100%、至少约125%、至少约150%、至少约175%、至少约200%、至少约225%、至少约250%、至少约275%、至少约300%、至少约325%、至少约350%或至少约375%。
[0904]
本公开还提供了一种试剂盒,其包括(i)能够特异性检测编码来自表1(或来自图28a-28g)的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2(或来自图28a-28g)的基因生物标记物的rna的多个寡核苷酸探针。还提供了一种制品,其包括(i)能够特异性检测编码来自表1(或来自图28a-28g)的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2(或来自图28a-28g)的基因生物标记
物的rna的多个寡核苷酸探针,其中所述制品包括微阵列。
[0905]
还提供了一种用于确定有需要的受试者中的肿瘤的肿瘤微环境的基因套组,其至少包括来自表1(或来自图28a-28g)的生物标记物基因和来自表2(或来自图28a-28g)的生物标记物基因,其中所述肿瘤微环境用于(i)鉴定适合抗癌疗法的受试者;(ii)确定正在经历抗癌疗法的受试者的预后;(iii)启动、暂停或修改抗癌疗法的施用;或(iv)其组合。
[0906]
本公开提供了一种用于鉴定罹患适合用抗癌疗法治疗的癌症的人受试者的组合生物标记物,其中所述组合生物标记物包括在从所述受试者获得的样品中测量的标志1分数和标志2分数,其中(i)标志1分数通过测量从所述受试者获得的第一样品中表3(或图28a-28g)的基因套组中的基因的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中表4(或图28a-28g)的基因套组中的基因的表达水平来确定;并且其中(a)如果标志1分数为负并且标志2分数为正,则所述疗法是ia类别tme疗法;(b)如果标志1分数为正并且标志2分数为正,则所述疗法是is类别tme疗法;(c)如果标志1分数为负并且标志2分数为负,则所述疗法是id类别tme疗法;或者(d)如果标志1分数为正并且标志2分数为负,则所述疗法是a类别tme疗法。
[0907]
还提供了一种用于治疗有需要的人受试者中的癌症的抗癌疗法,其中所述受试者被鉴定为表现出包括标志1分数和标志2分数的组合生物标记物,其中(i)标志1分数通过测量从所述受试者获得的第一样品中表3(或图28a-28g)的基因套组中的基因的表达水平来确定;并且(ii)标志2分数通过测量从所述受试者获得的第二样品中表4(或图28a-28g)的基因套组中的基因的表达水平来确定,并且其中(a)如果标志1分数为负并且标志2分数为正,则所述疗法是ia类别tme疗法;(b)如果标志1分数为正并且标志2分数为正,则所述疗法是is类别tme疗法;(c)如果标志1分数为负并且标志2分数为负,则所述疗法是id类别tme疗法;或者(d)如果标志1分数为正并且标志2分数为负,则所述疗法是a类别tme疗法。
[0908]
e1.一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)的方法,其包括确定组合生物标记物,所述组合生物标记物包括:
[0909]
(a)标志1分数;和
[0910]
(b)标志2分数,
[0911]
其中
[0912]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平来确定;并且
[0913]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平来确定。
[0914]
e2.一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用ia类别tme疗法,其中在所述施用之前,所述受试者被鉴定为表现出组合生物标记物,所述组合生物标记物包括:
[0915]
(a)负标志1分数;和
[0916]
(b)正标志2分数,
[0917]
其中
[0918]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平来确定;并且
[0919]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平来确定。
[0920]
e3.一种用于治疗罹患癌症的人受试者的方法,其包括:
[0921]
(a)在施用之前鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[0922]
(a)负标志1分数;和
[0923]
(b)正标志2分数,
[0924]
其中
[0925]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平来确定;并且
[0926]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平来确定;
[0927]
以及
[0928]
(b)向所述受试者施用ia类别tme疗法。
[0929]
e4.一种用于鉴定罹患适合用ia类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[0930]
(i)通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平确定标志1分数;以及
[0931]
(ii)通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平确定标志2分数,
[0932]
其中在施用之前包括:
[0933]
(a)负标志1分数;和
[0934]
(b)正标志2分数的组合生物标记物的存在,
[0935]
指示可以施用ia类别tme疗法来治疗所述癌症。
[0936]
e5.如实施方式e2至e4中任一项所述的方法,其中所述ia类别tme疗法包括检查点调节剂疗法。
[0937]
e6.如实施方式e2至e5中任一项所述的方法,其中所述检查点调节剂疗法包括施用刺激性免疫检查点分子的激活剂。
[0938]
e7.如实施方式e6所述的方法,其中所述刺激性免疫检查点分子的激活剂是针对gitr、ox-40、icos、4-1bb或其组合的抗体分子。
[0939]
e8.如实施方式e5所述的方法,其中所述检查点调节剂疗法包括施用rorγ激动剂。
[0940]
e9.如实施方式e5所述的方法,其中所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。
[0941]
e10.如实施方式e9所述的方法,其中所述抑制性免疫检查点分子的抑制剂是针对单独pd-1(例如,信迪利单抗、替雷利珠单抗、派姆单抗或其抗原结合部分)、pd-l1、pd-l2、ctla-4或其组合的抗体,或与以下组合:tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)
28g)的基因套组的表达水平来确定;
[0962]
以及
[0963]
(b)向所述受试者施用is类别tme疗法。
[0964]
e20.一种用于鉴定罹患适合用is类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[0965]
(i)通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平确定标志1分数;以及
[0966]
(ii)通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平确定标志2分数,
[0967]
其中在施用之前包括:
[0968]
(a)正标志1分数;和
[0969]
(b)正标志2分数的组合生物标记物的存在,
[0970]
指示可以施用is类别tme疗法来治疗所述癌症。
[0971]
e21.如实施方式e18至e20所述的方法,其中所述is类别tme疗法包括施用(1)检查点调节剂疗法和抗免疫抑制疗法,和/或(2)抗血管生成疗法。
[0972]
e22.如实施方式e21所述的方法,其中所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。
[0973]
e23.如实施方式e22所述的方法,其中所述抑制性免疫检查点分子的抑制剂是针对pd-1、pd-l1、pd-l2、ctla-4或其组合的抗体。
[0974]
e24.如实施方式e23所述的方法,其中所述抗pd-1抗体包括纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、tsr-042、信迪利单抗、替雷利珠单抗或其抗原结合部分。
[0975]
e25.如实施方式e23所述的方法,其中所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042交叉竞争与人pd-1的结合。
[0976]
e26.如实施方式e23所述的方法,其中所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042结合相同的表位。
[0977]
e27.如实施方式e23所述的方法,其中所述抗pd-l1抗体包括阿维单抗、阿替利珠单抗、cx-072、ly3300054、德瓦鲁单抗或其抗原结合部分。
[0978]
e28.如实施方式e23所述的方法,其中所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗交叉竞争与人pd-1的结合。
[0979]
e29.如实施方式e23所述的方法,其中所述抗pd-l1抗体与阿维单抗、阿替利珠单抗、cx-072、ly3300054或德瓦鲁单抗结合相同的表位。
[0980]
e30.如实施方式e23所述的方法,其中所述抗ctla-4抗体包括伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)或其抗原结合部分。
[0981]
e31.如实施方式e23所述的方法,其中所述抗ctla-4与伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4)交叉竞争与人ctla-4的结合。
[0982]
e32.如实施方式e23所述的方法,其中所述抗ctla-4抗体与伊匹单抗或双特异性
抗体xmab20717(抗pd-1/抗ctla-4)结合相同的ctla-4表位。
[0983]
e33.如实施方式e21所述的方法,其中所述检查点调节剂疗法包括施用(i)选自由以下组成的组中的抗pd-1抗体:纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗和tsr-042;(ii)选自由以下组成的组中的抗pd-l1抗体:阿维单抗、阿替利珠单抗、cx-072、ly3300054和德瓦鲁单抗;(iii)抗ctla-4抗体,其是伊匹单抗或双特异性抗体xmab20717(抗pd-1/抗ctla-4),或(iv)其组合。
[0984]
e34.如实施方式e21至e33所述的方法,其中所述抗血管生成疗法包括施用选自由以下组成的组中的抗vegf抗体:伐利苏单抗、贝伐珠单抗、那赛昔珠单抗(抗dll4/抗vegf双特异性)及其组合。
[0985]
e35.如实施方式e21至e34所述的方法,其中所述抗血管生成疗法包括施用抗vegfr抗体。
[0986]
e36.如实施方式e35所述的方法,其中所述抗vegfr抗体是抗vegfr2抗体。
[0987]
e37.如实施方式e36所述的方法,其中所述抗vegfr2抗体是雷莫卢单抗。
[0988]
e38.如实施方式e21至e37所述的方法,其中所述抗血管生成疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。
[0989]
e39.如实施方式e21至e38所述的方法,其中所述抗免疫抑制疗法包括施用抗ps抗体、抗ps靶向抗体、结合β2-糖蛋白1的抗体、pi3kγ的抑制剂、腺苷通路抑制剂、ido的抑制剂、tim的抑制剂、lag3的抑制剂、tgf-β的抑制剂、cd47抑制剂或其组合。
[0990]
e40.如实施方式e39所述的方法,其中所述抗ps靶向抗体是巴维妥昔单抗或结合β2-糖蛋白1的抗体。
[0991]
e41.如实施方式e39所述的方法,其中所述pi3kγ抑制剂是ly3023414(samotolisib)或ipi-549。
[0992]
e42.如实施方式e39所述的方法,其中所述腺苷通路抑制剂是ab-928。
[0993]
e43.如实施方式e39所述的方法,其中所述tgfβ抑制剂是ly2157299(galunisertib),或者所述tgfβr1抑制剂是ly3200882。
[0994]
e44.如实施方式e39所述的方法,其中所述cd47抑制剂是莫洛利单抗(5f9)。
[0995]
e45.如实施方式e39所述的方法,其中所述cd47抑制剂靶向sirp。
[0996]
e46.如实施方式e21至e45所述的方法,其中所述免疫抑制疗法包括施用tim-3的抑制剂、lag-3的抑制剂、btla的抑制剂、tigit的抑制剂、vista的抑制剂、tgf-β或其受体的抑制剂、lair1的抑制剂、cd160的抑制剂、2b4的抑制剂、gitr的抑制剂、ox40的抑制剂、4-1bb(cd137)的抑制剂、cd2的抑制剂、cd27的抑制剂、cds的抑制剂、icam-1的抑制剂、lfa-1(cd11a/cd18)的抑制剂、icos(cd278)的抑制剂、cd30的抑制剂、cd40的抑制剂、baffr的抑制剂、hvem的抑制剂、cd7的抑制剂、light的抑制剂、nkg2c的抑制剂、slamf7的抑制剂、nkp80的抑制剂、cd86激动剂或其组合。
[0997]
e47.一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用id类别tme疗法,其中在所述施用之前,所述受试者被鉴定为表现出组合生物标记物,所述组合生物标记物包括:
[0998]
(a)负标志1分数;和
[0999]
(b)负标志2分数,
[1000]
其中
[1001]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平来确定;并且
[1002]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平来确定。
[1003]
e48.一种用于治疗罹患癌症的人受试者的方法,其包括:
[1004]
(a)在施用之前鉴定表现出组合生物标记物的受试者,所述组合生物标记物包括:
[1005]
(a)负标志1分数;和
[1006]
(b)负标志2分数,
[1007]
其中
[1008]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平来确定;并且
[1009]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平来确定;
[1010]
以及
[1011]
(b)向所述受试者施用id类别tme疗法。
[1012]
e49.一种用于鉴定罹患适合用id类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[1013]
(i)通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平确定标志1分数;以及
[1014]
(ii)通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平确定标志2分数,
[1015]
其中在施用之前包括:
[1016]
(a)负标志1分数;和
[1017]
(b)负标志2分数的组合生物标记物的存在,
[1018]
指示可以施用id类别tme疗法来治疗所述癌症。
[1019]
e50.如实施方式e47至e49中任一项所述的方法,其中所述id类别tme疗法包括在施用引发免疫应答的疗法的同时或之后施用检查点调节剂疗法。
[1020]
e51.如实施方式e50所述的方法,其中所述引发免疫应答的疗法是疫苗、car-t或新表位疫苗。
[1021]
e52.如实施方式e50所述的方法,其中所述检查点调节剂疗法包括施用抑制性免疫检查点分子的抑制剂。
[1022]
e53.如实施方式e52所述的方法,其中所述抑制性免疫检查点分子的抑制剂是针对pd-1、pd-l1、pd-l2、ctla-4或其组合的抗体。
[1023]
e54.如实施方式e53所述的方法,其中所述抗pd-1抗体包括纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗、tsr-042或其抗原结合部分。
[1024]
e55.如实施方式e53所述的方法,其中所述抗pd-1抗体与纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042交叉竞争与人
28g)的基因套组的表达水平来确定;
[1046]
以及
[1047]
(b)向所述受试者施用a类别tme疗法。
[1048]
e66.一种用于鉴定罹患适合用a类别tme疗法治疗的癌症的人受试者的方法,所述方法包括:
[1049]
(i)通过测量从所述受试者获得的第一样品中选自表3(或图28a-28g)的基因套组的表达水平确定标志1分数;以及
[1050]
(ii)通过测量从所述受试者获得的第二样品中选自表4(或图28a-28g)的基因套组的表达水平确定标志2分数,
[1051]
其中在施用之前包括:
[1052]
(a)正标志1分数;和
[1053]
(b)负标志2分数的组合生物标记物的存在,
[1054]
指示可以施用a类别tme疗法来治疗所述癌症。
[1055]
e67.如实施方式e64至e66所述的方法,其中所述a类别tme疗法包括vegf靶向疗法和其他抗血管生成剂、血管生成素1(ang1)的抑制剂、血管生成素2(ang2)的抑制剂、dll4的抑制剂、抗vegf和抗dll4的双特异性抑制剂、tki抑制剂、抗fgf抗体、抗fgfr1抗体、抗fgfr2抗体、抑制fgfr1的小分子、抑制fgfr2的小分子、抗plgf抗体、针对plgf受体的小分子、针对plgf受体的抗体、抗vegfb抗体、抗vegfc抗体、抗vegfd抗体、针对vegf/plgf捕获分子的抗体诸如阿柏西普或ziv-阿柏西普、抗dll4抗体或抗notch疗法,诸如γ-分泌酶的抑制剂。
[1056]
e68.如实施方式e67所述的方法,其中所述tki抑制剂选自由以下组成的组:卡博替尼、凡德他尼、替沃扎尼、阿西替尼、乐伐替尼、索拉非尼、瑞戈非尼、舒尼替尼、呋喹替尼、帕唑帕尼及其任何组合。
[1057]
e69.如实施方式e68所述的方法,其中所述tki抑制剂是呋喹替尼。
[1058]
e70.如实施方式e67所述的方法,其中所述vegf靶向疗法包括施用抗vegf抗体或其抗原结合部分。
[1059]
e71.如实施方式e70所述的方法,其中所述抗vegf抗体包括伐利苏单抗、贝伐珠单抗或其抗原结合部分。
[1060]
e72.如实施方式e70所述的方法,其中所述抗vegf抗体与伐利苏单抗或贝伐珠单抗交叉竞争与人vegf a的结合。
[1061]
e73.如实施方式e70所述的方法,其中所述抗vegf抗体与伐利苏单抗或贝伐珠单抗结合相同的表位。
[1062]
e74.如实施方式e67所述的方法,其中所述vegf靶向疗法包括施用抗vegfr抗体。
[1063]
e75.如实施方式e74所述的方法,其中所述抗vegfr抗体是抗vegfr2抗体。
[1064]
e76.如实施方式e75所述的方法,其中所述抗vegfr2抗体包括雷莫卢单抗或其抗原结合部分。
[1065]
e77.如实施方式e64至e76中任一项所述的方法,其中所述a类别tme疗法包括施用血管生成素/tie2靶向疗法。
[1066]
e78.如实施方式e77所述的方法,其中所述血管生成素/tie2靶向疗法包括施用内皮糖蛋白和/或血管生成素。
[1067]
e79.如实施方式e64至e78中任一项所述的方法,其中所述a类别tme疗法包括施用dll4靶向疗法。
[1068]
e80.如实施方式e79所述的方法,其中所述dll4靶向疗法包括施用那赛昔珠单抗、abl101(nov1501)或abt165。
[1069]
e81.如实施方式e1至e80中任一项所述的方法,其包括:
[1070]
(a)施用化学疗法;
[1071]
(b)进行手术;
[1072]
(c)施用放射疗法;或
[1073]
(d)其任何组合。
[1074]
e82.如实施方式e1至e81中任一项所述的方法,其中所述选自表4的基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60或61个选自表2的基因,或1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123或124个选自图28a-28g的基因。
[1075]
e83.如实施方式e1至e82中任一项所述的方法,其中所述基因套组是选自表4或图28a-28g的基因套组。
[1076]
e84.如实施方式es1至e83中任一项所述的方法,其中所述选自表3的基因套组包括1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62或63个选自表1的基因,或1、2、3、4、5、6、7、8、9、10、11、12、13、14、15、16、17、18、19、20、21、22、23、24、25、26、27、28、29、30、31、32、33、34、35、36、37、38、39、40、41、42、43、44、45、46、47、48、49、50、51、52、53、54、55、56、57、58、59、60、61、62、63、64、65、66、67、68、69、70、71、72、73、74、75、76、77、78、79、80、81、82、83、84、85、86、87、88、89、90、91、92、93、94、95、96、97、98、99、100、101、102、103、104、105、106、107、108、109、110、111、112、113、114、115、116、117、118、119、120、121、122、123或124个选自图28a-28g的基因。
[1077]
e85.如实施方式e1至e84中任一项所述的方法,其中所述基因套组是选自表3或图28a-28g的基因套组。
[1078]
e86.如实施方式e1至e85中任一项所述的方法,其中所述第一样品和所述第二样品是相同的样品。
[1079]
e87.如实施方式e1至e85中任一项所述的方法,其中所述第一样品和所述第二样品是不同的样品。
[1080]
e88.如实施方式e1至e87中任一项所述的方法,其中所述第一样品和/或所述第二样品包括瘤内组织。
[1081]
e89.如实施方式e1至e88中任一项所述的方法,其中所述表达水平是表达蛋白质水平。
[1082]
e90.如实施方式e1至e88中任一项所述的方法,其中所述表达水平是转录rna表达水平。
[1083]
e91.如实施方式e1至e90中任一项所述的方法,其中所述rna表达水平使用测序或测量rna的任何技术来确定。
[1084]
e92.如实施方式e91所述的方法,其中所述测序是下一代测序(ngs)。
[1085]
e93.如实施方式e92所述的方法,其中所述ngs选自由以下组成的组:rna-seq、edgeseq、pcr、nanostring、wes或其组合。
[1086]
e94.如实施方式e90所述的方法,其中所述rna表达水平使用荧光来确定。
[1087]
e95.如实施方式e90所述的方法,其中所述rna表达水平使用affymetrix微阵列或agilent微阵列来确定。
[1088]
e96.如实施方式e90至e95所述的方法,其中所述rna表达水平经受分位数归一化。
[1089]
e97.如实施方式e96所述的方法,其中所述分位数归一化包括将输入rna水平值分箱成分位数。
[1090]
e98.如实施方式e97所述的方法,其中所述输入rna水平分箱成100个分位数。
[1091]
e99.如实施方式e96至e98所述的方法,其中所述分位数归一化包括将所述rna表达水平分位数转换为正态输出分布函数。
[1092]
e100.如实施方式e1至e99中任一项所述的方法,其中所述标志分数的计算包括
[1093]
(i)测量来自所述受试者的测试样品中的所述基因套组中的每个基因的表达水平;
[1094]
(ii)对于每个基因,从步骤(i)的所述表达水平中减去从参考样品中的此基因的表达水平获得的平均表达值;
[1095]
(iii)对于每个基因,将步骤(ii)中获得的值除以从所述参考样品的表达水平获得的每个基因的标准偏差;以及
[1096]
(iv)将步骤(iii)中获得的所有值相加并且将所得的数除以所述基因套组中的基因数的平方根;
[1097]
其中如果(iv)中获得的值大于零,则所述标志分数是正标志分数,并且其中如果(iv)中获得的值小于零,则所述标志分数是负标志分数。
[1098]
e101.如实施方式e100所述的方法,其中所述参考样品包括参考表达水平的集合。
[1099]
e102.如实施方式e101所述的方法,其中所述参考表达值是标准化参考值。
[1100]
e103.如实施方式e101所述的方法,其中所述参考表达值从样品群体获得。
[1101]
e104.如实施方式e101所述的方法,其中所述参考表达水平来源于公开可用的数据库或相互归一化的数据库组合。
[1102]
e105.如实施方式e100所述的方法,其中所述参考样品包括从不同群体获得的组织样品。
[1103]
e106.如实施方式e100至e105中任一项所述的方法,其中所述参考样品包括在不同时间点采集的样品。
[1104]
e107.如实施方式e106所述的方法,其中所述不同时间点是较早的时间点。
[1105]
e108.如实施方式e1至e107中任一项所述的方法,其中所述癌症是肿瘤。
[1106]
e109.如实施方式e108所述的方法,其中所述肿瘤是癌。
[1107]
e110.如实施方式e108所述的方法,其中所述肿瘤选自由以下组成的组:胃癌、结直肠癌、肝癌(肝细胞癌,hcc)、卵巢癌、乳腺癌、nsclc、膀胱癌、肺癌、胰腺癌、头颈癌、淋巴瘤、子宫癌、肾癌或肾癌、胆管癌、前列腺癌、睾丸癌、尿道癌、阴茎癌、胸癌、直肠癌、脑癌(胶质瘤和胶质母细胞瘤)、子宫颈腮腺癌、食管癌、胃食管癌、喉癌、甲状腺癌、腺癌、神经母细胞瘤、黑色素瘤和默克尔细胞癌。
[1108]
e111.如实施方式e1至e110中任一项所述的方法,其中所述癌症是复发性的。
[1109]
e112.如实施方式e1至e110中任一项所述的方法,其中所述癌症是难治性的。
[1110]
e113.如实施方式e112所述的方法,其中在至少一种包括施用至少一种抗癌剂的先前疗法后,所述癌症是难治性的。
[1111]
e114.如实施方式e1至e113中任一项所述的方法,其中所述癌症是转移性的。
[1112]
e115.如实施方式e2至e114中任一项所述的方法,其中所述施用有效地治疗癌症。
[1113]
e116.如实施方式e2至e115中任一项所述的方法,其中所述施用减少癌症负荷。
[1114]
e117.如实施方式e116所述的方法,其中与所述施用之前的癌症负荷相比,癌症负荷减少至少约10%、至少约20%、至少约30%、至少约40%或约50%。
[1115]
e118.如实施方式e2至e117中任一项所述的方法,其中在所述初始施用之后,所述受试者表现出至少约1个月、至少约2个月、至少约3个月、至少约4个月、至少约5个月、至少约6个月、至少约7个月、至少约8个月、至少约9个月、至少约10个月、至少约11个月、至少约一年、至少约18个月、至少约两年、至少约三年、至少约四年或至少约五年的无进展存活。
[1116]
e119.如实施方式e2至e118中任一项所述的方法,其中在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的稳定疾病。
[1117]
e120.如实施方式e2至e119中任一项所述的方法,其中在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的部分应答。
[1118]
e121.如实施方式e2至e120中任一项所述的方法,其中在所述初始施用之后,所述受试者表现出约1个月、约2个月、约3个月、约4个月、约5个月、约6个月、约7个月、约8个月、约9个月、约10个月、约11个月、约一年、约18个月、约两年、约三年、约四年或约五年的完全应答。
[1119]
e122.如实施方式e2至e121中任一项所述的方法,其中与未表现出所述组合生物标记物的受试者的无进展存活概率相比,所述施用使无进展存活概率提高至少约10%、至少约20%、至少约30%、至少约40%、至少约50%、至少约60%、至少约70%、至少约80%、至少约90%、至少约100%、至少约110%、至少约120%、至少约130%、至少约140%或至少约150%。
[1120]
e123.如实施方式e2至e122中任一项所述的方法,其中与未表现出所述组合生物标记物的受试者的总体存活概率相比,所述施用使总体存活概率提高至少约25%、至少约
50%、至少约75%、至少约100%、至少约125%、至少约150%、至少约175%、至少约200%、至少约225%、至少约250%、至少约275%、至少约300%、至少约325%、至少约350%或至少约375%。
[1121]
e124.一种试剂盒,其包括(i)能够特异性检测编码来自表1的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2的基因生物标记物的rna的多个寡核苷酸探针。
[1122]
e125.一种制品,其包括(i)能够特异性检测编码来自表1(或图28a-28g)的基因生物标记物的rna的多个寡核苷酸探针,和(ii)能够特异性检测编码来自表2(或图28a-28g)的基因生物标记物的rna的多个寡核苷酸探针,其中所述制品包括微阵列。
[1123]
e126.一种用于确定有需要的受试者中的肿瘤的肿瘤微环境的基因套组,其至少包括来自表1(或图28a-28g)的生物标记物基因和来自表2(或图28a-28g)的生物标记物基因,其中所述肿瘤微环境用于
[1124]
(i)鉴定适合抗癌疗法的受试者;
[1125]
(ii)确定正在经历抗癌疗法的受试者的预后;
[1126]
(iii)启动、暂停或修改抗癌疗法的施用;或
[1127]
(iv)其组合。
[1128]
e127.一种用于鉴定罹患适合用抗癌疗法治疗的癌症的人受试者的组合生物标记物,其中所述组合生物标记物包括在从所述受试者获得的样品中测量的标志1分数和标志2分数,其中
[1129]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中表3(或图28a-28g)的基因套组中的基因的表达水平来确定;并且
[1130]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中表4(或图28a-28g)的基因套组中的基因的表达水平来确定,
[1131]
并且其中
[1132]
如果所述标志1分数为负并且所述标志2分数为正,则所述疗法是ia类别tme疗法;
[1133]
如果所述标志1分数为正并且所述标志2分数为正,则所述疗法是is类别tme疗法;
[1134]
如果所述标志1分数为负并且所述标志2分数为负,则所述疗法是id类别tme疗法;
[1135]
如果所述标志1分数为正并且所述标志2分数为负,则所述疗法是a类别tme疗法。
[1136]
e128.一种用于治疗有需要的人受试者中的癌症的抗癌疗法,其中所述受试者被鉴定为表现出包括标志1分数和标志2分数的组合生物标记物,其中
[1137]
(i)所述标志1分数通过测量从所述受试者获得的第一样品中表3(或图28a-28g)的基因套组中的基因的表达水平来确定;并且
[1138]
(ii)所述标志2分数通过测量从所述受试者获得的第二样品中表4(或图28a-28g)的基因套组中的基因的表达水平来确定,
[1139]
并且其中
[1140]
如果所述标志1分数为负并且所述标志2分数为正,则所述疗法是ia类别tme疗法;
[1141]
如果所述标志1分数为正并且所述标志2分数为正,则所述疗法是is类别tme疗法;
[1142]
如果所述标志1分数为负并且所述标志2分数为负,则所述疗法是id类别tme疗法;
[1143]
如果所述标志1分数为正并且所述标志2分数为负,则所述疗法是a类别tme疗法。
[1144]
实施例
[1145]
实施例1
[1146]
肿瘤微环境(tme)分类:基于群体的分类器
[1147]
本公开描述了创建能够基于基因表达将肿瘤样品分层(或分类)为四种类别的基于群体的z分数分类器(基于群体的分类器)的方法。如本文所用,四种类别也可以被称为肿瘤微环境(tme)、基质类型、基质亚型、或表型或其变体。本文还描述了用于由原始微阵列(rna)和rna测序数据生成表达值的分析管道。
[1148]
对于数据预处理,存在各种用于测量基因表达的技术,其中每种平台技术都需要对原始数据进行特定的预处理。基于群体的分类器支持affymetrix dna微阵列、高通量下一代rna测序,并且在一些方面可以扩展到其他技术。
[1149]
对于微阵列数据,affymetrix芯片程序测量存储在cel文件中的每个细胞(每个含有一个独特的探针)的强度像素值。使用affy r包处理cel文件。使用以下参数应用expresso函数:rma(稳健多芯片平均)背景校正方法、分位数归一化、非探针特定校正和中值抛光汇总(j.w.tukey,exploratory data analysis,addison-wesley,1977)。对通过expresso函数返回的表达值进行log2转换。最后,将表达式分位数转换为正态输出分布,将输入值分箱为100个分位数(图1)。
[1150]
通过清理读取、将它们与参考基因组比对并定量基因表达来处理illumina rna-seq测序读取。因此,分析步骤包括三个关键步骤:修剪(bbduk)、映射(star)和表达定量(featurecounts)。参考人类基因组是ensembl,92版本,参考常见掺入标准进行扩展(ercc和sirv)。作为额外的质量控制步骤,将一百万个读取的子样品(seqtk工具)映射到所选物种的rrna和珠蛋白序列以确定样品中这些种类的读取的总体比例。结果报告在multiqc报告的汇总表中。
[1151]
使用基于云的genialis expressions软件生成原始和归一化(tpm、fpkm)表达值,并且与重现它们所需的所有技术细节一起报告。在使用基于z分数的模型对样品进行分层之前,将tpm归一化表达式分位数转换为正态输出分布,将输入值分箱为100个分位数(图1)。
[1152]
对于其他平台技术,例如edgeseq(htg molecular diagnostics,inc.),分位数归一化应用于跨平台分析,将输入值分箱为100个分位数并应用正态输出分布函数。任何方法的准确度随着群体分布达到正态分布而增加。
[1153]
样品的分类。本公开的基于群体的分类器(或基于群体的方法)假定基因表达水平的以零为中心的正态分布(μ=0)。
[1154]
在整个患者群体中,根据每个基因的表达水平计算此基因的平均值和标准偏差。对于个体患者,针对每个基因,取患者的标准化表达水平,减去群体平均值,然后除以标准偏差。这是z分数。在一些方面,没有对于对自由度的校正。
[1155]
对于个体患者,将标志内的所有z分数相加,并且然后除以基因数的平方根。结果是根据等式1的激活分数zs:
[1156][1157]
其中z是指z分数,s是指样品(患者),g是指基因,并且g是指标志基因集。|g|指示
基因集g的大小。当激活分数大于零时,即zs》=0,则称此标志为正,并且因此zs《0意味着标志为负。z
s,g
是描述远离群体的平均值的量级和方向的向量,并且是无单位的;激活分数zs也是无单位的。
[1158]
通过将激活分数与表13相关联来进行预后或预测。换言之,基于患者z分数的符号和所使用的阈值(正或负zs),通过应用表13中的规则(基于加和标志1和标志2z分数的符号的患者分类规则),将患者分类为四种基质亚型中的一种。还参见图10。
[1159]
表13.基于标志1和标志2基因的激活分数的四种类别的基质亚型的预后或预测生物学。
[1160]
标志1标志2基质亚型的类别-+ia(免疫活性型)++is(免疫抑制型)
‑‑
id(免疫沙漠型)+-a(血管生成型)
[1161]
通过表1中的一个或多个(例如,至少1、2、3、4、5、10、15、20、25、30、40、50、60、61、62或63个)基因的激活分数zs来确定第一生物标志,标志1。
[1162]
在一些方面,在本公开中也可以被称为生物标记物的基因包括以下中的一种或多种:abcc9、afap1l2、bace1、bgn、bmp5、col4a2、col8a1、col8a2、cpxm2、cxcl12、ebf1、ecm2、ednra、eln、epha3、fbln5、gnas、gnb4、gucy1a3、hey2、hspb2、il1b、itga9、itpr1、jam2、jam3、kcnj8、lamb2、lhfp、ltbp4、meox1、mgp、mmp12、mmp13、naalad2、nfatc1、nov、olfml2a、pcdh17、pde5a、pdgfrb、peg3、plscr2、plxdc2、rgs4、rgs5、rnf144a、rras、runx1t1、cav2、selp、serpine2、sgip1、smarca1、spon1、stab2、steap4、tbx2、tek、tgfb2、tmem204、ttc28和utrn。
[1163]
通过表2中的一个或多个(例如,至少1、2、3、4、5、10、15、20、25、30、40、50、60或61个)基因的激活分数zs的总和来确定第二生物标志,标志2。
[1164]
在一些方面,所述基因包括以下中的一种或多种:agr2、c11orf9、dusp4、eif5a、etv5、gad1、iqgap3、mst1、mt2a、mta2、pla2g4a、reg4、srsf6、strn3、trim7、usf1、zic2、c10orf54、ccl3、ccl4、cd19、cd274、cd3e、cd4、cd8b、ctla4、cxcl10、ifna2、ifnb1、ifng、lag3、pdcd1、pdcd1lg2、tgfb1、tigit、tnfrsf18、tnfrsf4、tnfsf18、tlr9、havcr2、cd79a、cxcl11、cxcl9、gzmb、ido1、igll5、adamts4、capg、ccl2、ctsb、folr2、hfe、hmox1、hp、igfbp3、mest、plau、rac2、rnh1、serpine1和timp1。
[1165]
实施例2
[1166]
将分类器应用于公共数据集
[1167]
实施例1中所述的分类器用于根据如本文所述的基于群体的方法或分类器分析三个公开可用的数据集。如本文所述对数据集进行归一化(图1)。在图1中,直方图的顶行显示标志1和2基因的log2表达分布,并且显示数据集具有不同的范围和分布。acrg和singapore中的rna表达水平通过微阵列(affymetrix)进行分析,而tcga数据中的rna表达水平来源于rna测序。
[1168]
在图1的图的中间行中,计算群体中值和z分数。如所预期,分布全部以0为中心,但由于平台差异(微阵列和rna-seq),分布的整体形状不同。图1的图的底行显示分位数归一
化之后的表达(z分数)值。作为归一化的结果,可以相对于全部三个数据集的中位数进行分类。
[1169]
本公开的基于群体的方法用于将亚洲癌症研究组(acrg)数据集的298个患者分类为四种基质亚型。acrg是一个非营利性制药行业联盟,其提供受亚洲最常诊断出的癌症(肝癌、胃癌和肺癌)影响的患者的经过验证且综合性的基因组数据集。acrg数据集中的rna表达数据作为affymetrix微阵列数据提供。胃癌数据集中有300个患者,其中两个患者的结果数据(总体存活率)不可用。因此,本公开中的一些表涉及298个患者,而其他表或图表可以涉及300个患者。患者仅接受化学疗法,并且总体存活率由所述联盟进行验证。
[1170]
来自癌症基因组图谱(tcga)计划的胃癌数据(可在www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga获得)用于本公开的基于群体的方法,用于将388个患者分为四种基质亚型。tcga中的rna表达数据作为rna-seq提供,并且结果数据作为388个患者的总体存活率提供,然而并非所有的共变量数据都可用,因此本文中的某些表和数字是指患者的较小子集。
[1171]
发明人使用的singapore胃癌数据集或singapore群组来自gastric cancer project’08,如发现于www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=gse15459。在affymetrix基因芯片人基因组u133加2.0阵列上分析了200个原发性胃肿瘤,其中使用192个(liu等人,(2013)gastroenterology)。结果数据在以下中报告为总体存活率:lei z,tan ib,das k,deng n等人identification of molecular subtypes of gastric cancer with different responses to pi3-kinase inhibitors and 5-fluorouracil.gastroenterology 2013年9月;145(3):554-65。
[1172]
将阈值设置为平均值或零的基于群体的方法用于对三个数据集中的每一个进行分类。表14示出了在分类之后三个群组中的每一个中患者的四种基质亚型的分布。
[1173]
表14.本公开的四种类型的基质亚型在三个可公开获得的胃癌数据集(acrg、tcga和singapore)中的流行率。
[1174]
基质亚型acrgtcgasingaporea15.2%19.5%24.4%ia26.5%20.7%27.5%id34.8%32.6%23.1%is23.5%27.2%25.1%
[1175]
将如acrg定义的肿瘤亚型与四种基质亚型进行比较。数据集中的acrg肿瘤亚型与本公开的基质亚型没有强关联性。acrg数据被描述为具有4种肿瘤亚型:msi-微卫星不稳定;mss-微卫星稳定/emt-上皮-间质转化(发生在伤口愈合和癌症转移开始期间);tp53-是(肿瘤)蛋白p53的正常表型;并且tp53+是(肿瘤)蛋白p53的异常表型(表15)。
[1176]
表15.acrg数据集中的肿瘤亚型(n=300)与四种基质亚型没有强关联性。
[1177][1178]
tcga描述了四种胃癌亚型。将tcga胃癌亚型c1、c2、c3和c4(n=232)与如根据本公开分类的间质亚型进行比较,并且分析揭示胃癌亚型和基质亚型之间没有强关联性(表16)。
[1179]
表16.tcga胃癌亚型c1、c2、c3和c4(n=232)与基质亚型进行比较揭示没有强关联性。
[1180][1181]
singapore胃癌数据集报告了四种不同的癌症亚型:间质型、代谢型、增殖型和不稳定型。表17显示用本公开的基于群体的方法(阈值为平均值或零)分类的192个患者的基质亚型之间缺乏相关性。
[1182]
表17.singapore数据集胃癌亚型(间质型、代谢型、增殖型和不稳定型)与基质亚型没有强关联性。
[1183][1184]
对于报告了年龄的共变量的三个数据集的所有患者,探索了分类患者的年龄与四种基质亚型的关系(表18)。当所有三个数据集的患者用本公开的基于群体的方法(阈值为平均值或零)分类时,年龄与四种基质亚型之间没有明显的关联。
[1185]
表18.在三个可公开获得的胃癌数据集中,年龄的共变量与本公开的基质亚型没有关联。tcga数据群组的388个受试者中仅252个报告了年龄,而所有300个acrg和192个singapore患者报告了年龄。
[1186]
[1187][1188]
对于报告了性别的共变量的三个数据集的所有患者,探索了分类患者的性别与四种基质亚型的关系(表19)。当所有三个数据集的患者用本公开的基于群体的方法(阈值为平均值或零)分类时,性别与四种基质亚型之间没有明显的关联。
[1189]
表19.在三个可公开获得的胃癌数据集中,性别的共变量与本公开的基质亚型没有关联。tcga数据群组的388个受试者中仅254个报告了性别。
[1190][1191]
对于报告了癌症期的共变量的三个数据集的所有患者,探索了分类患者的癌症期与四种基质亚型的关系(表20)。当所有三个数据集的患者用本文公开的基于群体的方法(阈值为平均值或零)分类时,癌症期与四种基质亚型之间没有明显的关联。
[1192]
表20.在三个可公开获得的胃癌数据集中,癌症期的共变量与本公开的基质亚型不相关。acrg中300受试者中的298个报告了疾病期;388个tcga数据受试者中的375个报告了时期;192个singapore受试者报告了时期。
[1193][1194]
对于报告了lauren肿瘤分类的共变量的acrg的所有患者,探索了分类患者的lauren肿瘤分类与四种基质亚型的关系(表21)。胃肿瘤的lauren肿瘤分类是本领域已知的;存在三种类型:弥漫型、肠型和混合型。当acrg患者用本公开的基于群体的方法(阈值为平均值或零)分类时,lauren肿瘤分类与四种基质亚型之间没有明显的关联。
[1195]
表21.acrg胃癌数据集(n=300)的本公开的基质亚型(基于群体)与lauren肿瘤分
类的比较。
[1196][1197]
根据本公开的基于群体的方法(阈值设定为平均值或零,除非另有指示),单个地基于三个数据集生成存活曲线(在本领域中被称为kaplan-meier曲线)并组合。
[1198]
图2,kaplan-meier图描绘了分类acrg群组的存活曲线,绘制为y轴上的存活概率相对于x轴上的时间(以月为单位)。基质亚型id与ia以及id与a之间的存活结果在统计学上不同,但id与is之间没有不同;还参见表22。对存活率最有利的基质亚型是ia或免疫活性型,这与患有免疫炎性肿瘤的胃癌患者具有最好的预后的观察一致。a和is组代表最差的存活风险。
[1199]
在ia患者中,免疫细胞对于癌症的新抗原负荷产生应答。is或免疫抑制型患者对于癌症不产生免疫应答。id或免疫沙漠型患者没有大量转录本公开的表1和表2中所列的基质基因。患者似乎没有产生免疫应答,但他们也没有血管生成增殖。a或血管生成型患者可能具有快速增殖的肿瘤脉管系统。
[1200]
表22.对应于使用基于群体的方法(阈值设定为平均值或零)分类的acrg数据集的图2的存活风险曲线的数据。
[1201][1202]
表22揭示id与ia以及id与a之间的存活结果在统计学上不同,但id与is之间没有不同。在此存活分析中,风险比(hr)是对应于两个水平的解释变量所描述的条件的风险率的比率。在此实施例中,id与ia之间的hr为0.519,表明id基质亚型的死亡风险增加。
[1203]
图3,kaplan-meier图描绘了分类tcga群组的存活曲线,绘制为y轴上的存活概率相对于x轴上的时间(以月为单位)。在tcga数据集中,若干种基质亚型之间的存活结果在统
计学上没有不同,如在acrg数据集中所见(表23;也与图2和表22进行比较)。然而,当组合所有三个数据集(singapore数据集在下文描述)时,四种类别的基质亚型的存活结果,数据变得统计学上显著(参见表25)。
[1204]
表23.对应于使用基于群体的方法分类的tcga数据集的图3的存活风险曲线的数据。
[1205][1206]
图4,kaplan-meier图描绘了分类singapore群组的存活曲线,绘制为y轴上的存活概率相对于x轴上的时间(以月为单位)。在singapore数据集中,若干种基质亚型之间的存活结果在统计学上没有不同,如在acrg数据集中所见(表24;也与图2和表22进行比较)。然而,当组合所有三个数据集时,数据变得统计学上显著(参见表25)。
[1207]
表24.对应于使用基于群体的方法分类的singapore数据集的图3的存活风险曲线的数据。
[1208][1209]
用零或平均值的阈值分类的三个组合数据集的kaplan-meier图可以见于图5。相对于x轴上时间(以月为单位),在y轴上绘制存活概率。统计结果报告在表25中。将所有acrg、tcga和singapore数据集组合时,每个类别的患者数量如下:id类别,n=286或32.5%;ia类别,n=199或22.6%;a类别,n=182或20.7%;is类别,n=213或24.2%。基质亚型id与ia之间的存活结果在统计学上不同,但id与a或id与is之间没有不同;还参见表25。这些数据表明这些亚型描述的不同基质生物学与癌症结果有不同的相关性。
[1210]
表25.对应于使用基于群体的方法分类的图5的组合存活风险曲线的数据。
[1211][1212]
进行基因本体分析。图6a示出了acrg数据中作为四种基质亚型的函数的来自treg标志(angelova等人(2015)genome biol.16:64)的表达水平的值的中值和范围的盒式图。图6b示出了acrg数据中作为四种基质亚型的函数的炎性应答标志(如通过go(基因本体,go_ref:0000022定义)的表达水平的值的中值和范围的盒式图。
[1213]
进行两个标志,标志1和标志2的进一步的基因本体分析。对于acrg群组,每个患者的标志1通路激活分数绘制在x轴上,并且内皮细胞标志激活绘制在y轴上。趋势线代表线性回归。内皮细胞标志从bhasin等人,bmc genomics 11:342,2010获得。正斜率指示acrg群组中患者的标志1基因与内皮细胞标志之间的正相关性(图7a)。每个患者的标志2通路激活分数绘制在x轴上,并且指定通路的通路激活分数绘制在y轴上。趋势线代表线性回归。正斜率指示acrg群组中患者的标志2通路激活分数与图的标题中指示的通路之间的正相关性。从趋势线的斜率可以看出,参与巨噬细胞通路的基因与标志2基因的相关性最小,而参与炎性应答通路(如通过go(基因本体,go_ref:0000022)定义)以及treg和t细胞通路(angelova等人)的基因是正相关的(图7b)。用tcga数据集(图8a和图8b)和singapore数据集(图9a和图9b)进行类似的分析。
[1214]
采用各种阈值对acrg数据集的患者进行分层或分类(表26)。可以看出,对每个个体z分数(无单位)应用+或
–
0.4(例如)的阈值将导致患者的zs或激活分数的变化,并且因此导致分配到四种基质亚型中的每一种的患者数量的变化。在一些方面,不同的阈值以及标志1和2中的每一个的不同阈值适用于本公开的方法。
[1215]
表26.在acrg群组的分类期间改变标志1(
″1″
)和标志2(
″2″
)的阈值。
[1216] 阈值=02》=+0.41》=-0.42》=0.4,1》=0.41》=-0.4ia24.8%21.1%29.2%22.8%22.5%id30.2%33.9%25.8%37.2%28.5%a18.8%21.8%15.8%18.5%20.5%is26.2%23.2%29.2%21.5%28.5%
[1217]
实施例3
[1218]
治疗前胃肿瘤微环境rna标志与对于检查点抑制剂疗法的临床应答相关
[1219]
总结:回顾性数据分析指示,当患者用靶向疗法(诸如检查点抑制剂)治疗时,胃癌肿瘤微环境表型与临床应答相关。所述分析包括45个胃癌肿瘤样品。数据指示相对于免疫抑制型(is)、免疫沙漠型(id)和血管生成型(a)表型,免疫活性型(ia)表型对于检查点抑制
剂有独特的应答。
[1220]
背景信息、方法和结果:根据本公开的基于群体的方法对接受派姆单抗的45个胃癌患者的回顾性分类进行分类。通过配对末端rna-seq测量rna表达水平并在分类之前标准化。根据recist标准报告数据,例如完全应答者(cr)、部分应答者(pr)和sd/pd(疾病稳定/进展性疾病(参见表27)。总体应答率(orr)在此定义为cr+pr患者的数量除以患者总数。所有患者的orr为27%(12/45)。类别应答率在此定义为此基质亚型类别中cr+pr患者的数量除以此类别中患者的数量。当对患者进行回顾性分析并置于ia类别中时,应答率为80%;并且在is类别中,应答率为18%。回顾性地落入id类别中的患者的应答率为12%,并且a类别患者的应答率为0%。
[1221]
表27.针对胃癌接受派姆单抗的患者的治疗前分类(平均值阈值),n=45。
[1222][1223]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1224]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1225]
(b)肿瘤是来自胃癌的肿瘤;并且
[1226]
(c)tme类别特定疗法包括施用派姆单抗。
[1227]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1228]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1229]
(b)肿瘤是来自胃癌的肿瘤;并且
[1230]
(c)tme类别特定疗法包括施用派姆单抗。
[1231]
实施例4
[1232]
与对于抗血管生成疗法的临床应答相关的治疗前胃肿瘤微环境rna标志
[1233]
总结:回顾性数据分析指示,当患者用靶向疗法(诸如血管生成抑制剂)治疗时,胃癌基质表型与临床应答相关。所述分析包括49个胃癌肿瘤样品。数据指示相对于免疫活性型(ia)和免疫沙漠型(id)表型,血管生成型(a)和免疫抑制型(is)表型对于抗血管生成疗法有独特的应答。
[1234]
背景信息、方法和结果:由雷莫卢单抗、vegf抑制剂和紫杉醇组成的药物组合是pdl-1阴性胃癌患者中二线治疗的常用方案。为了测试当患者用雷莫卢单抗和紫杉醇治疗时基质表型是否与临床结果相关,分析了来自49个胃癌患者的治疗前存档组织中的rna基因标志并且根据本公开的基于群体的方法进行分类。针对临床结果数据测试了每种基质表型之间的相关性。在将患者分层为四种表型中的一种的情况下,与历史数据相比,效应量和临床显著性发生了变化(wilke等人2014)。根据recist标准报告数据,例如完全应答者(cr)、部分应答者(pr)和sd/pd(疾病稳定/进展性疾病(参见表28)。通过配对末端rna-seq测量rna表达水平并在分类之前标准化。总体应答率(orr)在此定义为cr+pr患者的数量除以患者总数。对于本实施例中的49个患者,所有患者的orr为39%(19/49)。类别应答率在此定义为此基质亚型类别中cr+pr患者的数量除以此类别中患者的数量。当对患者进行回顾性分析并置于is类别中时,类别应答率为56%;并且在a类别中,类别应答率为37%。回顾性地落入ia类别中的患者的类别应答率为33%,并且id类别患者的类别应答率为25%。总体而言,在此相对较小的患者样品集中,a和is肿瘤微环境表型与使用抗血管生成疗法的改善的临床结果特异性相关。
[1235]
表28.针对胃癌接受雷莫卢单抗和紫杉醇的患者的治疗前分类(平均值阈值),n=49。
[1236][1237]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1238]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下
组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1239]
(b)肿瘤是来自胃癌的肿瘤;并且
[1240]
(c)tme类别特定疗法包括施用vegf抑制剂,例如雷莫卢单抗和紫杉醇。
[1241]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1242]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1243]
(b)肿瘤是来自胃癌的肿瘤;并且
[1244]
(c)tme类别特定疗法包括施用vegf抑制剂,例如雷莫卢单抗和紫杉醇。
[1245]
实施例5
[1246]
治疗前胃肿瘤微环境rna标志与对于化学疗法的临床应答相关。
[1247]
总结:回顾性数据分析指示,当患者用化学疗法治疗时,胃癌肿瘤微环境表型与临床应答相关。所述分析包括50个胃癌肿瘤样品。数据指示相对于免疫活性型(ia)和免疫沙漠型(id)表型,血管生成型(a)和免疫抑制型(is)表型对于化学疗法有更少的应答。
[1248]
背景信息、方法和结果:folfox是一种常用的化学疗法组合方案,其由氟尿嘧啶、亚叶酸和奥沙利铂组成。在未经治疗的晚期胃癌患者中,folfox的总体应答率(orr)报告为34.8%(al-batran等人j clin oncol.2008年3月20日;26(9):1435-42)。中位进展时间(pfs)和总体存活期(os)分别为5.8个月和10.7个月。为了测试当患者用化学疗法治疗时基质表型是否与临床结果相关,分析了来自50个胃癌患者(44个原发性肿瘤样品,6个转移性肿瘤样品)的治疗前存档组织中的rna表达。针对临床结果数据测试了每种基质表型之间的相关性。在a和is患者中,与分类为ia和id表型的患者相比,使用folfox赋予的益处较少:在ia和id患者中,中位pfs和os分别延长至大约7.8个月和14.7个月。总体而言,在此相对较小的患者样品集中,a和is肿瘤微环境表型与改善的临床结果特异性相关,这表明表型对于化学疗法益处具有预测性。
[1249]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1250]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1251]
(b)肿瘤是来自胃癌的肿瘤;并且
[1252]
(c)tme类别特定疗法包括施用化学疗法,例如folfox。
[1253]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1254]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1255]
(b)肿瘤是来自胃癌的肿瘤;并且
[1256]
(c)tme类别特定疗法包括施用化学疗法,例如folfox。
[1257]
实施例6
[1258]
结直肠癌肿瘤微环境rna标志与对于抗血管生成疗法的临床应答相关。
[1259]
总结:回顾性数据分析指示,当患者用靶向疗法(包括血管生成抑制剂)治疗时,结直肠癌肿瘤微环境表型与临床应答相关。所述分析包括分析642个结直肠癌肿瘤样品。数据指示相对于免疫活性型(ia)和免疫沙漠型(id)表型,血管生成型(a)和免疫抑制型(is)表型对于抗血管生成疗法有独特的应答。
[1260]
背景信息、方法和结果:贝伐珠单抗与化学疗法的组合增加晚期结直肠癌患者的pfs和os(snyder等人rev recent clin trials.2018;13(2):139-149)。先前未经治疗的转移性结直肠癌患者的总体应答率(rr)报告为左侧肿瘤中的80%和右侧肿瘤中的83%。左侧肿瘤和右侧肿瘤两者中中位进展时间(pfs)和总体存活期(os)分别为13个月和37个月。为了测试当患者用血管生成抑制剂治疗时肿瘤微环境表型是否与临床结果相关,分析了来自从642个胃癌患者(321个左侧,321个右侧)采集的存档组织的肿瘤rna基因标志。针对临床结果数据测试了每种肿瘤表型之间的相关性。在将肿瘤分层为四种表型中的一种的情况下,与历史数据相比,效应量和显著性发生了变化。在a和is患者中,与分类为ia和id表型的患者相比,使用贝伐珠单抗赋予些许益处:在a和is患者中,预测中位pfs和os分别偏移至15个月和39个月。ia和id患者的无进展存活和os数据与历史值一致。总体而言,a和is肿瘤微环境表型与使用血管生成抑制剂的改善的临床结果特异性相关,并且对于pfs具有预测性作用。
[1261]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1262]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1263]
(b)肿瘤是来自结直肠癌的肿瘤;并且
[1264]
(c)tme类别特定疗法包括施用贝伐珠单抗与化学疗法的组合。
[1265]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者
施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1266]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1267]
(b)肿瘤是来自结直肠癌的肿瘤;并且
[1268]
(c)tme类别特定疗法包括施用贝伐珠单抗与化学疗法的组合。
[1269]
实施例7
[1270]
巴维妥昔单抗ii期临床试验
[1271]
此实施例涉及使用巴维妥昔单抗增强免疫治疗剂在人体中的活性,并且特别涉及在根据本公开表征患者的基质亚型的情况下,使用巴维妥昔单抗与抗pd-1或抗pd-l1抗体的组合治疗癌症患者。
[1272]
巴维妥昔单抗与派姆单抗在以下患者中的开放标签ii期试验:(i)在使用任何检查点抑制剂治疗之后实现确认的疾病控制(cr、pr或sd)之后复发的患者;或(ii)在晚期胃癌或胃食管癌中未接受抗pd-1或抗pd-l1疗法的患者。试验在全球大约19个中心进行,包括美国和亚洲。试验的目标是(i)观察与使用抗pd-1或抗pd-l1单一疗法的历史结果相比,所述组合是否安全并为组合治疗提供临床上有意义的改善,以及(ii)观察是否存在一个生物标记物亚组,其中在ruo(仅限研究使用)情景中,对组合疗法的应答比其他生物标记物亚组有意义。
[1273]
测试产品、剂量和施用如下:提供作为无菌不含防腐剂的具有10mm醋酸盐、ph 5.0的溶液的巴维妥昔单抗和注射用水。根据临床方案,每周以至少3mg/kg体重的静脉内(iv)输注方式施用巴维妥昔单抗。q3w施用200mg固定剂量的派姆单抗。
[1274]
根据全rna测序技术公司制定的方案,使用来自最近活检的福尔马林固定组织生成rna序列。
[1275]
基质亚型为ia或is(如通过基于群体的方法分析)或生物标记物阳性(如通过ann方法分析)的患者从巴维妥昔单抗和派姆单抗(一种代表性的检查点抑制剂)的组合治疗中受益。
[1276]
表29列出了将具有适当阈值、截止值或参数的ann方法应用于38个患者的数据的结果,所述患者的rna测序数据,以及orr、dcr和最佳客观应答(cr、pr、sd和pd)可获得。
[1277]
表29.生物标记物数据可获得的38个用巴维妥昔单抗和派姆单抗组合疗法治疗胃癌/胃食管癌的患者的生物标记物阳性和阴性。使用ann方法确定生物标记物阳性(即,存在生物标记物)或阴性(即,不存在生物标记物)。
[1278][1279]1确认的应答&下一次扫描待定情况下的未确认
[1280]
疾病控制率(dcr)被定义为在抗癌剂的临床试验中对治疗干预实现完全应答(cr)、部分应答(pr)或稳定疾病(sd)的患有晚期或转移性癌症的患者的百分比。pd是进展性疾病。
[1281]
将oncg100试验中38个存在生物标记物数据(即,如通过ann方法分类的rna表达数据)的患者的回顾性分析与nlr(嗜中性粒细胞-白细胞比率)数据组合。性能数据在表30中给出。
[1282]
表30.具有生物标记物数据且nlr《4的22个患者的性能值。
[1283][1284]
准确度(acc):正确预测数/总预测数
[1285]
roc auc:接受者操作特征曲线下面积;模型能够区分类别的程度
[1286]
灵敏度:真生物标记物应答者/总实际应答者
[1287]
特异性:真生物标记物非应答者/总实际非应答者
[1288]
阳性预测值(ppv):真生物标记物应答者/总预测生物标记物应答者
[1289]
阴性预测值(npv):真生物标记物非应答者/总预测生物标记物非应答者
[1290]
在正在经历巴维妥昔单抗和派姆单抗的组合疗法的80个胃癌/胃食管癌患者的组中,生物标记物阳性率为大约30%。
[1291]
表31示出了23个具有生物标记物数据的患者的基于群体的z分数基质表型分类和最佳客观应答。
[1292]
表31. 23个具有生物标记物数据的患者的基于群体的z分数分类和最佳客观应答。
[1293][1294][1295]
表32示出了全部44个患者的试验的中期结果。在9个患者中观察客观应答,所有登记患者的总体应答率(orr)为20%。并非所有的患者都具有确认的应答。
[1296]
表32.胃癌/胃食管癌研究中的巴维妥昔单抗和派姆单抗组合疗法(未确认的结果;n=44,mss,pd-l1阳性和阴性患者)。
[1297] 所有患者(n=44)orr[cr+pr]9/44(20%)dcr[cr+pr+sd]17/44(39%)cr2/44(5%)pr7/44(16%)sd8/44(18%)pd27/44(61%)
[1298]
此外,其他非基于rna标志的生物标记物用于评定患者的基线免疫状态。这些包括微卫星不稳定性(msi-h)、错配修复缺陷(例如,通过ihc确定)、ebv(埃-巴二氏病毒)或hpv(人乳头瘤病毒)阳性(存在或不存在)、基线β2gp1(β2-糖蛋白1)表达水平、ifnγ表达水平和pd-1或pd-l1表达水平,使用组合阳性分数(cps)。cps是pd-l1染色细胞(例如,肿瘤细胞、淋巴细胞、巨噬细胞)的数量除以活肿瘤细胞的总数,再乘以100。
[1299]
本领域已知msi-h(即,微卫星不稳定性高)和/或具有阳性ebv信号和/或pd-l1表达水平高的患者对于抗pd-1或抗pd-l1单一疗法具有更好的应答。在此临床试验中,预期mss(微卫星稳定,与msi-h相反)、ebv阴性或pd-l1低的患者将从巴维妥昔单抗中受益,使得患者能够更好地对派姆单抗产生应答。在mss(微卫星稳定性)的患者子集分析中,对于28个mss患者,orr为21.0(n=6);16个患者具有未知的mss状态。百分之二十(20%)的cps《1的患者对治疗产生应答;两个为完全应答者(cr)的患者具有小于1的cps分数。
[1300]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1301]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1302]
(b)肿瘤是来自晚期胃癌或胃食管癌的肿瘤;并且
[1303]
(c)tme类别特定疗法包括施用巴维妥昔单抗和抗pd-1免疫疗法抗体(例如,纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042)或抗pd-l1免疫疗法抗体。
[1304]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1305]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1306]
(b)肿瘤是来自晚期胃癌或胃食管癌的肿瘤;并且
[1307]
(c)tme类别特定疗法包括施用巴维妥昔单抗和抗pd-1免疫疗法抗体(例如,纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042)或抗pd-l1免疫疗法抗体。
[1308]
实施例8
[1309]
巴维妥昔单抗iii期临床试验
[1310]
本实施例描述了使用本公开的方法作为患者选择工具,即iuo(仅限研究人员使用),巴维妥昔单抗和抗pd-1免疫疗法抗体在胃癌中的iii期关键试验。
[1311]
所述试验与先前实施例中所述的临床试验类似地进行,但是在30个试验中心中以300个晚期腺癌胃癌或胃食管癌患者进行。对胃癌患者进行活检,测量标志1和标志2基因的rna表达水平,并使用适当的阈值与基于群体的参考进行比较。is患者对于巴维妥昔单抗和检查点抑制剂产生最佳应答,并且与方案统计部分定义的组合治疗相比有临床上有意义的改善。ia患者也产生应答,但是id和a患者没有资格参加试验。
[1312]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1313]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1314]
(b)肿瘤是来自胃癌的肿瘤;并且
[1315]
(c)tme类别特定疗法包括施用巴维妥昔单抗和抗pd-1免疫疗法抗体(例如,纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042)。
[1316]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习
分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1317]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1318]
(b)肿瘤是来自胃癌的肿瘤;并且
[1319]
(c)tme类别特定疗法包括施用巴维妥昔单抗和抗pd-1免疫疗法抗体(例如,纳武单抗、派姆单抗、西米普利单抗、pdr001、cbt-501、cx-188、信迪利单抗、替雷利珠单抗或tsr-042)。
[1320]
实施例9
[1321]
抗vegf疗法i/ii期试验
[1322]
本实施例涉及基于根据本公开的患者的基质亚型,使用具有一种与vegf缔合以增强活性的组分的抗血管生成抗体(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体)和/或双特异性抗体(例如,抗vegf/抗dll4双特异性那赛昔珠单抗)作为单一剂或与标准护理诸如化学疗法组合。
[1323]
本实施例描述了在患有以下疾病的患者中单独或与标准护理组合的抗vegf疗法的开放标签i/ii期试验;使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线)。试验在全球大约10个中心进行,包括美国、欧洲和亚洲。试验的目标是观察单一疗法抗vegf治疗或组合治疗是否安全并且与历史结果相比是否有临床上有意义的改善。将生物标记物阳性亚组(a和is)中的潜在预测结果包括到vegf治疗或与vegf的组合治疗中在ruo(仅限究使用)情景中是临床上有意义的。
[1324]
测试产品、剂量和施用模式如下:根据临床方案作为静脉内(iv)输注施用。
[1325]
根据全rna测序技术公司诸如htg molecular diagnostics(tucson,arizona,usa)或almac(craigavon,northern ireland,uk)制定的方案,使用来自最近活检的福尔马林固定组织生成rna序列。基质亚型为a或is的患者从抗vegf治疗或抗vegf组合治疗中受益。
[1326]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1327]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1328]
(b)肿瘤是来自以下的肿瘤:使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线);并且
[1329]
(c)tme类别特定疗法包括在患有(b)的癌症的患者中施用具有一种与vegf缔合以
增强活性的组分的抗血管生成抗体(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体)和/或双特异性抗体(例如,抗vegf/抗dll4双特异性那赛昔珠单抗)作为单一剂或与标准护理诸如化学疗法组合。
[1330]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1331]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1332]
(b)肿瘤是来自以下的肿瘤:使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线);并且
[1333]
(c)tme类别特定疗法包括在患有(b)的癌症的患者中施用具有一种与vegf缔合以增强活性的组分的抗血管生成抗体(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体)和/或双特异性抗体(例如,抗vegf/抗dll4双特异性那赛昔珠单抗)作为单一剂或与标准护理诸如化学疗法组合。
[1334]
实施例10
[1335]
抗vegf疗法iii期试验
[1336]
本实施例描述了使用本公开的方法作为分层工具,即iuo(仅限研究人员使用),用抗vegf疗法(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体,和/或双特异性抗体,例如,抗vegf/抗dll4双特异性那赛昔珠单抗)作为单一剂或与标准护理组合进行的先前实施例的适应症中的一种在患有以下疾病的患者中的iii期关键试验:使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线)。
[1337]
对患有以上癌症的患者进行活检,测量标志1和标志2基因的rna表达水平并用ann模型(在基于群体的参考上训练)分析,并且使用适当的阈值与基于群体的参考进行比较。a或is的患者,即生物标记物阳性的患者对于抗vegf治疗或组合抗vegf疗法产生最佳应答,并且与方案中预定义的统计计划相比相对于组合治疗有临床上有意义的改善。id或ia患者没有资格参加研究。
[1338]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1339]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、
121、166、169、179、185、232、200、216、241、250、263和278;
[1340]
(b)肿瘤是来自以下的肿瘤:使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线);并且
[1341]
(c)tme类别特定疗法包括在患有(b)的癌症的患者中施用单独或与标准护理组合的抗vegf疗法(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体,和/或双特异性抗体,例如,抗vegf/抗dll4双特异性那赛昔珠单抗)。
[1342]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1343]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1344]
(b)肿瘤是来自以下的肿瘤:使针对晚期疾病批准的所有线的治疗(例如,4线)失败的晚期铂类耐药卵巢癌,在至少两种先前的标准化学疗法方案(例如,3线)之后的难治性结肠或直肠腺癌,或术后晚期腺癌胃癌或胃食管癌(例如,1线);并且
[1345]
(c)tme类别特定疗法包括在患有(b)的癌症的患者中施用单独或与标准护理组合的抗vegf疗法(例如,特异性针对vegf的单克隆抗体或抗dll4单克隆抗体,和/或双特异性抗体,例如,抗vegf/抗dll4双特异性那赛昔珠单抗)。
[1346]
实施例11
[1347]
非群体机器学习分类器
[1348]
提供了三种类型的基于机器学习的非基于群体的分类器的机制。根据本公开的非群体分类器涵盖例如逻辑回归、随机森林和人工神经网络(例如,下文呈现的多层感知器)。提供了拟合模型(分类器)、映射函数和参数。
[1349]
逻辑回归
[1350]
逻辑回归对某个事件(例如,患者表达某种表型)的概率进行建模。这可以扩展到对若干类事件,例如,表型的四种不同表现方式进行建模。
[1351]
逻辑回归使用以下逻辑函数预测目标类别(例如,tme类别)的概率:
[1352][1353]
逻辑函数(图11)可以解释为采用对数几率和输出概率。当泛化到多个特征时,我们可以将t表示为以下:
[1354]
t=β0+β1x1+β2x2+...+βmxm[1355]
并且一般逻辑函数p可以写成:
[1356][1357]
模型拟合:模型学习参数β,对于所述参数,预测器(逻辑函数)针对训练数据集x
(例如,对应于本文公开的基因套组的一组rrna表达水平以及例如由本文公开的基于群体的分类器的应用产生的分配的tme分类)产生最小误差。拟合模型表示为一组参数β和逻辑函数。直观地,逻辑回归搜索在其参数中做出最少假设的模型。逻辑回归也受益于正则化,否则它可能会过拟合。逻辑回归可以泛化到多个结果(例如,当目标变量有多个,例如四个不同的值时)。多项逻辑回归是一种将逻辑回归泛化到多类问题的分类方法。
[1358]
在此,针对每个类别(例如,tme类别)学习一组参数β(例如,基因套组中的mrna表达水平)。根据预测,每个类别(例如,一个tme类别)都被分配概率,并且样品(例如,基因套组中的一组mrna表达水平)被分类到具有最高概率的tme类别。在acrg数据集上拟合的最终逻辑回归模型的参数在下表中定义。
[1359]
表33:最终逻辑回归模型的参数。
[1360][1361]
*来自98个基因的集的示例性基因
[1362]
随机森林
[1363]
随机森林(breiman l,2001)是一种训练成百上千个决策树的集成方法。单个树是简单的预测器(类似流程图的结构),其中每个内部节点代表对特征(基因)的测试,每个分支表示测试的结果(表达高于或低于给定阈值),并且每个叶具有类别标签(表型)。随机森
林模型在树的数量上增长而不受过度拟合的影响。更复杂的分类器(具有更多树的更大森林)变得更准确的这种观察与复杂性的增长几乎总是导致过度拟合的其他技术形成对比。这使得随机森林成为通用分类器,其可以应用于小型数据集,也可以应用于大型数据集。参见图12a和图12b。
[1364]
模型拟合:通过首先从训练集中抽取具有替换的随机样品,并且然后在随机抽取的样品上拟合分类树来拟合单个树。所述模型由一组树表示,每个树都有一组学习规则和针对特征的决策阈值。在acrg数据集上拟合的最终随机森林模型的参数在图13中定义。
[1365]
人工神经网络
[1366]
多层感知器(mlp)是一类前馈型人工神经网络。mlp由至少三层节点组成:输入层、隐藏层和输出层。除输入节点外,每个节点都是使用非线性激活函数的神经元。mlp利用被称为反向传播的监督学习技术进行训练。mlp可以区分不是线性可分的数据。
[1367]
训练集:使用acrg基因表达数据集作为训练集。acrg训练集包括298个可用样品中的235个样品,因为63个样品被鉴定为接近类别标签的决策边界;这些样品影响了模型的稳健性,并且因此未包括在训练集中。还包括98个连续变量(98个基因的套组,其包括标志1和标志2表,即表1和表2中呈现的基因子集),并且对应于四个目标类别(a、ia、is和id肿瘤微环境)。可以使用其他训练集,例如表5中公开的那些。如图14所示,每个样品包括基因套组中每个基因的值(例如,mrna水平)及其分类为特定类别(例如,使用基于本文公开的两个标志的群体方法分配)。
[1368]
神经层架构:所使用的ann是多层感知器(mlp),其包括一个输入层、输出层和一个隐藏层,如图15中以简化形式所示。输入层中的每个神经元连接到隐藏层中的两个神经元,并且隐藏层中的每个神经元连接到输出层中的每个神经元。可以使用其他架构来实践本发明,例如图16中所示的任何架构。
[1369]
训练:训练过程的目标是鉴定每个输入的权重wi和隐藏层中的偏差b,使得神经网络最小化训练集上的预测误差。参见图17。如图17所示,基因套组中的每个基因(x1..xn)被用作隐藏层中每个神经元的输入,并且通过训练过程鉴定隐藏层的偏差b值。来自每个神经元的输出是每个基因表达水平(xi)、权重(wi)和偏差(b)的函数,如图17所示。
[1370]
可以在隐藏层中应用多个激活函数,如图18所示。使用范围为-1至1的双曲正切激活函数(tanh)来生成如本文所述的ann分类器
[1371]
y(vi)=tanh(vi)
[1372]
其中yi是第i个节点(神经元)的输出,并且vi是输入连接的加权和。
[1373]
如上所述,人工神经网络分类器包括输入层中的基因表达值(对应于98个基因的套组),隐藏层中编码两个基质标志之间的关系的两个神经元,以及预测四种基质表型的概率的四个输出。参见图19。通过应用包括softmax函数的逻辑回归分类器支持将输出层值多类别分类为四种表型类别(ia、id、a和is)。softmax将十进制概率分配到加起来必须为1.0的每个类别。此额外的限制有助于训练更快地收敛。softmax就在输出层之前的神经网络层实施并且具有与输出层相同数量的节点。
[1374]
作为额外的改进,根据所使用的特定数据集,对softmax函数的结果应用各种截止值(例如,参见以下实施例中讨论的应用于派姆单抗神经网输出的截止值。
[1375]
对人工神经网络分类器的检查揭示了训练算法确实已经学习了权重(在表34中列
出),所述权重表示引入基于z分数算法的群体模型(即,本公开的基于群体的分类器,其用于生成训练数据集)中的标志1和标志2标志的基于符号的规则。
[1376]
所述规则是从训练数据中自动推断出来的。除隐藏层包括两个神经元外,所述算法没有给出关于标志1和2的任何假设。对于每个隐藏神经元,来自标志1和标志2的基因至少在一定程度上通过正基因权重或负基因权重做出贡献,然而一个隐藏神经元更受一个标志支配,并且反之亦然(图29a和图29b)。
[1377]
表34:输出层上的人工神经网络权重。
[1378][1379][1380]
在acrg数据集上拟合的最终人工神经网络模型的参数列表在表35中示出。
[1381]
表35:最终人工神经网络模型的参数。
[1382][1383]
*来自98个基因的集的示例性基因
[1384]
实施例12
[1385]
将ann方法应用于派姆单抗单一疗法
[1386]
图20示出了在用派姆单抗单一疗法治疗胃癌之后,只有tme is和ia类别患者显示完全应答,并且tme ia类别中完全应答的数量远高于is中的数量。此外,部分应答者的数量也远高于ia类别中的数量。
[1387]
图21示出了可以用包括基因表达数据的数据集训练ann分类器,所述基因表达数据包括来自患有特定癌症(胃癌)并用特定疗法(派姆单抗)治疗的患者的基因表达数据。分类器的输出被分为tme a、is、id、ia类别,但是完全应答者(cr)和部分应答者(pr)聚类在一个输出值接近1的神经元处。因此,可以在softmax函数中实施一个新阈值,其可以有效地鉴定is和ia tme类别内更有可能是派姆单抗单一疗法的完全或部分应答者的患者。如果选择包括is和ia类别患者(选项a;深色区域),则选择中将包括许多非应答者。然而,如果选择仅
包括ia类别患者(选项b;深色区域),则整个群体可能仅由完全应答者和部分应答者构成。
[1388]
选项1,即优化阈值但同时采用is和ia组,将生物标记物阳性总体应答率(orr)的优化从80%适度降低至70%orr(10/14)。此选项使生物标记物阴性结果最小化并最大化从8/12至10/12的总应答者的捕获。
[1389]
选项2,即优化阈值但同时仅采用ia子组,将生物标记物阳性orr的优化从80%提高至100%orr(8/8)。然而,最小化生物标记物阴性结果或最大化总应答者的捕获没有变化。
[1390]
为了找到应答的边界,对概率分数进行额外的优化。与0.50的概率分数相比,这导致生物标记物阳性(ia)组中的应答者最大化,从而允许更准确地预测对派姆单抗产生应答的患者,同时这也使应答者组中生物标记物阴性患者的数量最小化。
[1391]
在0.5概率分数下,性能为80%ppv(阳性预测值)和94%特异性。在0.87概率分数下,性能上升至100%ppv和100%特异性,而不损害灵敏度和npv(阴性预测值)。灵敏度是指真生物标记物应答者的数量除以实际应答者的数量;特异性是指真生物标记物非应答者的数量除以实际非应答者的数量;ppv是指真生物标记物应答者的数量除以预测生物标记物应答者的总数(生物标记物阳性评级的表现如何);并且npv
–
是指真生物标记物非应答者的数量除以预测生物标记物非应答者的总数(生物标记物阴性评级的表现如何)。
[1392]
表36示出了在对73个胃癌患者进行二线治疗(77%派姆单抗,23%nivolizumab)之后,ann生物标记物(ia)的特异性为83%。
[1393]
表36.与pd-1的行业黄金标准生物标记物相比的ann概率分数优化以及73个患者的msi-高状态(77%派姆单抗,23%nivolizumab)。
[1394][1395]
准确度(acc):正确预测数/总预测数。
[1396]
roc auc:接受者操作特征曲线下面积;模型能够区分类别的程度。
[1397]
灵敏度:真生物标记物应答者/总实际应答者。
[1398]
特异性:真生物标记物非应答者/总实际非应答者。
[1399]
阳性预测值(ppv):真生物标记物应答者/总预测生物标记物应答者。
[1400]
阴性预测值(npv):真生物标记物非应答者/总预测生物标记物非应答者
[1401]
表37.用派姆单抗或nivolizumab治疗的二线胃癌中生物标记物的比较(n=73)。
[1402][1403][1404]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1405]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1406]
(b)肿瘤是来自胃癌的肿瘤;并且
[1407]
(c)tme类别特定疗法包括施用派姆单抗单一疗法。
[1408]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习
分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1409]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1410]
(b)肿瘤是来自胃癌的肿瘤;并且
[1411]
(c)tme类别特定疗法包括施用派姆单抗单一疗法。
[1412]
实施例13
[1413]
将ann方法应用于雷莫卢单抗和紫杉醇
[1414]
针对实施例4的雷莫卢单抗加紫杉醇数据的ann模型性能。雷莫卢单抗靶向血管生成,因此预期a和is tme中的应答者。因此,将结果合并到a/is(两者均为血管生成应答性tme)中,以比较灵敏度和特异性相对于ia/id tme。
[1415]
表38:使用ann模型来分类对于血管生成疗法的应答者。
[1416][1417][1418]
此方法可以类似地应用于其他类型的癌症和其他疗法,例如,选择哪些个体将成为使用这种特定疗法进行治疗的候选者。
[1419]
在没有任何患者选择的情况下,应答者与非应答者的总体比率(19/48)为39.6%(表39)。使用ann方法,并且为了找到应答的边界,进行额外的优化。这导致生物标记物阳性组中的应答者最大化,从而允许更准确地预测对雷莫卢单抗和紫杉醇的组合疗法产生应答的患者,同时这也使应答者组中生物标记物阴性患者的数量最小化。在优化之后,与39.6%的未选择百分比相比,73.7%的应答者是生物标记物阳性的。与27.7%的生物标记物阴性率相比,生物标记物阳性患者具有约2.5倍的应答率:73.7%。生物标记物阳性组的中位存
活期为19个月,而生物标记物阴性组为16.5个月。
[1420]
表39
[1421]
生物标记物+/-的tme表型应答者(pr)(sd/pd) n=19(n=29)生物标记物阳性n=3014(73.7%)16(55.5)生物标记物阴性n=185(27.8%)13(44.8)
[1422]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1423]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1424]
(b)tme类别特定疗法包括施用雷莫卢单抗和紫杉醇。
[1425]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1426]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1427]
(b)tme类别特定疗法包括施用雷莫卢单抗和紫杉醇。
[1428]
实施例14
[1429]
那赛昔珠单抗1a期试验
[1430]
患有实体瘤的患者的1a期剂量递增试验的回顾性数据分析。患者必须具有组织学确认的转移性或不可切除的恶性肿瘤,对于所述恶性肿瘤没有剩余的标准治愈性疗法,也没有证明存活益处的疗法,或者他们必须没有资格接受这种疗法。对患有上述癌症的患者采集活检。通过免疫组织化学在ffpe肿瘤样本中测量探索性预测生物标记物,诸如dll4和vegf,在研究开始时存档或新鲜核心针活检(尽可能优选2个ffpe核心)。从存档的ffpe肿瘤样本回顾性地测量标志1和标志2基因的rna表达水平,并且使用肿瘤类型的适当阈值应用基于群体的方法(z分数)和非基于群体的ann算法两者,因为每个肿瘤类型具有特定阈值。排除没有结果标签的患者,在1a期试验数据的总生物标记物子集中留下39个患者。在此全员剂量递增试验中,38%的患者实现sd(稳定疾病)或更好(recist 1.1标准)。在生物标记物阳性子集中,48%的患者实现sd或更好。
[1431]
值得注意地,在妇科癌症(n=18)中,具有sd或更好的所有患者都落入生物标记物阳性组,其中58%的生物标记物阳性(n=12)和0%的生物标记物阴性(n=6)受益。模型性能在表40中列出;并且缩写和定义如下;acc是准确度;auc roc是接受操作者特征曲线下面
积;灵敏度是真生物标记物应答者的数量除以实际应答者的数量;特异性是真生物标记物非应答者的数量除以实际非应答者的数量;ppv是阳性预测值,即真生物标记物应答者的数量除以预测生物标记物应答者的总数;npv是阴性预测值,即真生物标记物非应答者的数量除以预测生物标记物非应答者的总数。
[1432]
表40.所有患者(n=39)中和妇科癌症(n=18)中的z分数和ann模型性能。
[1433][1434][1435]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中
所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1436]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1437]
(b)肿瘤是来自妇科癌症的肿瘤;并且
[1438]
(c)tme类别特定疗法包括施用那赛昔珠单抗。
[1439]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1440]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1441]
(b)肿瘤是来自妇科癌症的肿瘤;并且
[1442]
(c)tme类别特定疗法包括施用那赛昔珠单抗。
[1443]
实施例15
[1444]
那赛昔珠单抗1b期试验
[1445]
在回顾性分析中应用ann方法的本实施例描述了那赛昔珠单抗加紫杉醇的1b期剂量递增和扩展研究。此试验招募了44个铂类耐药卵巢癌(proc)患者,所述患者的》2个先前疗法失败/或之前接受过贝伐珠单抗。截至2019年第1季度末的最后一次中期数据分析,未确认应答率为43%,并且确认应答率为36%。
[1446]
获得了在试验中具有确认应答或进展性疾病(recist标准)的患有proc、子宫癌或输卵管癌的意向治疗群体中44个患者的应答数据。参见表41。
[1447]
表41.那赛昔珠单抗1b生殖癌症意向治疗群体应答率和疾病控制率
[1448][1449]
从患者活检中测量标志1和标志2基因的rna表达水平。在登记时采集活检或使用
存档的活检。使用生殖癌症的适当阈值应用基于群体(z分数)和非基于群体的ann算法。排除没有结果标签的患者,在1b数据集的总生物标记物子集中留下23个患者。
[1450]
在应用ann模型之后呈阳性的那些患者被认为是生物标记物阳性的。具有已知生物标记物状态的患者的orr和dcr在表42和表43中给出。
[1451]
表42.那赛昔珠单抗1b试验:具有rna表达数据和卵巢癌、子宫癌和输卵管癌的确认应答数据的23个患者的生物标记物状态。
[1452][1453][1454]
表43.那赛昔珠单抗1b试验生殖癌症群组中23个具有生物标记物数据和确认应答的患者的基于群体的z分数分类和最佳客观应答。
[1455]
tmen#cr#pr#sd#pdia6/231221is9/230531a2/230200id6/230132
[1456]
确认应答意指根据协议,在第一次成像扫描之后拍摄的第二次成像扫描确认应答。根据定义,进展性疾病(pd)不是确认应答;pd患者被包括在计算orr和dcr的分母中。生物标记物阳性患者的无进展存活(pfs)益处为9.2个月,而生物标记物阴性患者为3.5个月(p=0.0037)。kaplan-meier存活曲线在图22中提供。
[1457]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1458]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1459]
(b)肿瘤是选自卵巢癌、子宫癌和输卵管癌的生殖肿瘤;并且
[1460]
(c)tme类别特定疗法包括施用那赛昔珠单抗和紫杉醇。
[1461]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1462]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1463]
(b)肿瘤是选自卵巢癌、子宫癌和输卵管癌的生殖肿瘤;并且
[1464]
(c)tme类别特定疗法包括施用那赛昔珠单抗和紫杉醇。
[1465]
实施例16
[1466]
肿瘤不可知模型
[1467]
图26示出了将ann模型应用于1200个患者样品序列的结果,使用rna外显子组测序技术对400个患者的样品进行测序,三种不同肿瘤类型中的每一种
–
结直肠癌、胃癌和卵巢癌。跨可能的基质表型的结果的一致性揭示了本公开的ann模型对肿瘤类型是不可知的。
[1468]
针对患者数据(n=704)使用基于z分数群体的方法和ann模型以回顾性分类来自身体中至少17个不同起源的肿瘤的基质表型(表44)。结果数据都不与分类相关联,但是四种表型的分布与1,099个样品的分析中分类的四种表型的分布相似,所述样品代表卵巢癌(n=392)、结直肠癌(n=370)和胃癌(n=337)的样品,通过rna外显子组技术测序,如图27示。
[1469]
表44.来自至少17个不同起源的704个患者的基质表型。
[1470]
生物标记物调用n/总患者样品百分比ia(z分数)102/70414.5%ia(ann)120/70417.1%is_(z分数)246/70434.9%is_(ann)234/70433.2%a(z分数)108/70415.3%a_(ann)104/70414.7%id(z分数)247/70435.1%id(ann)245/70434.8%
[1471]
实施例17
[1472]
潜在空间
[1473]
将通过将ann模型应用于实施例7和12的数据产生的概率函数的投影绘制在由疾病分数符号表示的潜在空间中(完全应答,cr;部分应答,pr;稳定疾病,sd;进展性疾病,pd)。图23示出了潜在空间可视化,其提供亚型调用的概率并且可以用于告知医师生物标记物置信度以帮助治疗决策。图24示出了二级逻辑回归模型的潜在空间可视化,所述二级逻辑回归模型在潜在空间上训练以便基于患者结果标签学习生物标记物阳性相对于生物标
记物阴性决策边界。
[1474]
图25示出了用实施例12的患者数据训练的潜在空间可视化(逻辑回归),其中受试者具有大于3个月的无进展存活(pfs)。所有患者的疾病分数用作符号来标记概率分数。在图26中,在潜在空间上训练二级逻辑回归模型,以便基于实施例7的oncg100数据的患者结果标签来学习生物标记物阳性相对于生物标记物阴性的决策边界,并且绘制其具有生物标记物的患者的疾病分数。
[1475]
图中的曲线轮廓由于模型中特征之间的相互作用项而出现。在潜在空间图中,特征是标志1分数(例如,基因激活与内皮细胞标志激活相关的标志)和标志2分数(例如,激活与炎性和免疫细胞标志激活相关的标志)。在这种情况下,术语相互作用是指一个特征对预测的影响取决于另一个特征的值的情况,即当两个特征的影响不相加时。例如,在模型中添加或减去特征意味着没有相互作用;然而,在模型中对特征进行相乘、相除或配对意味着相互作用。
[1476]
在预测二元患者应答的图中,轮廓是平行的,因为基础逻辑回归不模拟特征之间的相互作用。相互作用项不存在是逻辑回归的基本特性之一,这使得它不太容易过度拟合并在小型数据集上获得良好的性能。因此,如果模型中没有相互作用项,则轮廓始终是平行的。
[1477]
另一方面,预测表型(四种类别,对应于四种tme)的图具有曲线轮廓。尽管每个单个表型类别的基础模型(神经元)等同于逻辑回归,但四个逻辑回归发生了四个表型类别概率的重新归一化,所以四个表型类别概率之和等于1。这使用softmax函数来实现,所述函数是标志1分数与标志2分数之间发生相互作用的函数。因此,此模型产生曲线轮廓。
[1478]
实施例18
[1479]
将ann方法应用于癌症中的检查点抑制剂单一疗法
[1480]
在使用抗pd-1或pd-1疗法(诸如替雷利珠单抗、信迪利单抗、派姆单抗或纳武单抗)的任何实体瘤的临床试验中,基于患者的tme的rna表达分析选择患者进行治疗。对患者进行实体瘤活检,将其处理为例如福尔马林固定石蜡包埋的块,并且将从所述块上切下的最近的载玻片转移到服务提供商以通过测序(例如,使用rna-seq、rna外显子组或微阵列测序)进行rna表达确定。根据本发明的算法对rna表达数据进行归一化和分析。
[1481]
试验中的治疗资格是基于大于60%的生物标记物阳性概率(或ia+is概率》60%),或基于逻辑回归算法,所述逻辑回归算法例如在实施例7的数据上训练并且基于大于例如5个月的无进展存活率(pfs》5)应用于潜在空间,使得pfs》5子集中的患者有资格接受治疗。
[1482]
由根据研究装置豁免(ide)使用的此临床试验测定为临床医生提供以下输出中的一个或多个:二元是/否答案、每个tme类别的概率、在具有概率轮廓和历史结果数据的潜在空间图上绘制的患者的概率、或覆盖有基于pfs》5的概率逻辑回归的患者的概率绘制的潜在空间图。
[1483]
基于先前的生物标记物分析(例如,pd-l1cps》1),此临床试验招募了未接受检查点抑制剂或没有资格接受现有检查点抑制剂的患者。在此试验中,超过20%的患者对基于pr或cr评定(recist标准)的治疗产生应答。
[1484]
本公开提供了一种用于确定有需要的受试者中的癌症的肿瘤微环境(tme)(并且任选地选择tme类别特定疗法的受试者)的方法,其包括对从来自所述受试者的肿瘤组织样
品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann),其中所述机器学习分类器将所述受试者鉴定为表现出或不表现出选自由以下组成的组中的tme:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1485]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;
[1486]
(b)肿瘤是实体瘤;并且
[1487]
(c)tme类别特定疗法包括施用抗pd-1或pd-1疗法,诸如替雷利珠单抗、信迪利单抗、派姆单抗或纳武单抗
[1488]
本公开还提供了一种用于治疗罹患癌症的人受试者的方法,其包括向所述受试者施用tme类别特定疗法,其中在所述施用之前,所述受试者被鉴定为表现出或不表现出通过对从由所述受试者获得的肿瘤组织样品的基因套组获得的多个rna表达水平应用机器学习分类器(例如,本文公开的ann)确定的tme,其中所述tme选自由以下组成的组:is(免疫抑制型)、a(血管生成型)、ia(免疫活性型)、id(免疫沙漠型)及其组合,其中
[1489]
(a)基因套组是(i)包括表1和表2的基因或其组合的基因套组,或(ii)选自由以下组成的组中的基因集:图28a-28g的基因集23、30、46、51、60、82、108、116、139、158、73、91、121、166、169、179、185、232、200、216、241、250、263和278;并且
[1490]
(b)肿瘤是实体瘤;并且
[1491]
(c)tme类别特定疗法包括施用抗pd-1或pd-1疗法,诸如替雷利珠单抗、信迪利单抗、派姆单抗或纳武单抗。
[1492]
***
[1493]
应理解,具体实施方式部分而非发明内容以及摘要部分旨在用于解释实施方式。发明内容和摘要部分可以阐述一个或多个但并非如诸位发明人所设想的本发明的全部示例性实施方式,并且因此不旨在以任何方式限制本发明和所附实施方式。
[1494]
上文已经借助于说明指定功能的实现及其关系的功能性构建块对本发明进行了描述。为了便于描述,本文任意地限定了这些功能性构建块的边界。可以限定替代性边界,只要指定功能及其关系得以适当地进行。
[1495]
具体实施方式的以上描述将充分地揭示本发明的总体性质,使得在不脱离本发明的一般概念的情况下,其他人可以无需过多的实验通过应用本领域的技术内的知识,容易地针对此类具体实施方式的各种应用进行修改和/或改编。因此,基于本文提出的传授内容和指导,此类改编和修改旨在处于所公开的实施方式的等同方案的含义和范围内。应理解,本文的措辞或术语是出于描述而非限制的目的,使得本说明书的术语或措辞应由技术人员根据传授内容和指导进行解释。
[1496]
本发明的广度和范围不应受任何上述示例性实施方式限制,而应仅根据以下实施方式和其等效物定义。
[1497]
在本公开全文中可以引用的所有引用的参考文献(包括文献参考文献、专利、专利申请和网站)的内容出于任何目的特此明确地通过引用以其整体并入,其中引用的参考文献也是如此。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1