使用无细胞DNA片段尺寸以确定拷贝数变异的制作方法
使用无细胞dna片段尺寸以确定拷贝数变异
1.相关申请的交叉引用
2.本申请是中国专利申请201680084307.1号的分案申请,并要求于2016年2月3日提交的题为:using cell
‑
free dna fragment size to determine copy number variations的美国临时专利申请no.62/290,891,以及2016年12月16日提交的题为:using cell
‑
free dna fragment size to determine copy number variations的美国专利申请no.15/382,508的权益,其出于所有目的通过引用整体并入本文。
背景技术:
3.人类医学研究的关键努力之一是发现产生不良健康后果的遗传异常。在许多情况下,在基因组的以异常拷贝存在的部分中已鉴定出特定基因和/或关键诊断标记物。例如,在产前诊断中,整个染色体的额外或缺失拷贝是经常发生的遗传病变。在癌症中,整个染色体或染色体区段的拷贝的缺失或倍增,以及基因组的特定区域的更高水平的扩增是常见的事件。
4.关于拷贝数变异(cnv)的大部分信息都是通过允许识别结构异常的细胞遗传学解析所提供的。用于遗传筛选和生物剂量测定的常规方法利用侵入性方法,例如羊膜穿刺术、脐带穿刺术或绒毛膜绒毛取样(cvs),以获得用于分析核型的细胞。认识到需要不需要细胞培养的更快速的测试方法,荧光原位杂交(fish)、定量荧光pcr(qf
‑
pcr)和阵列
‑
比较基因组杂交(阵列
‑
cgh)已被开发为分子
‑
细胞遗传学方法以用于分析拷贝数变异。
5.人类医学研究的关键努力之一是发现产生不良健康后果的遗传异常。在许多情况下,在基因组的以异常拷贝存在的部分中已鉴定出特定基因和/或关键诊断标记物。例如,在产前诊断中,整个染色体的额外或缺失拷贝是经常发生的遗传病变。在癌症中,整个染色体或染色体区段的拷贝的缺失或倍增,以及基因组的特定区域的更高水平的扩增是常见的事件。
6.关于拷贝数变异(cnv)的大部分信息都是通过允许识别结构异常的细胞遗传学解析所提供的。用于遗传筛选和生物剂量测定的常规方法利用侵入性方法,例如羊膜穿刺术、脐带穿刺术或绒毛膜绒毛取样(cvs),以获得用于分析核型的细胞。认识到需要不需要细胞培养的更快速的测试方法,荧光原位杂交(fish)、定量荧光pcr(qf
‑
pcr)和阵列
‑
比较基因组杂交(阵列
‑
cgh)已被开发为分子
‑
细胞遗传学方法以用于分析拷贝数变异。
7.允许在相对短的时间内对整个基因组进行测序的技术的出现,以及循环的无细胞dna(cfdna)的发现提供了将来自一条染色体的遗传物质与另一条染色体的遗传物质进行比较的机会而没有与侵入性采样方法相关的风险,这提供了一种工具来诊断目标遗传序列的各种拷贝数变异。
8.非侵入性产前诊断中现有方法的局限性,包括源于有限水平的cfdna的灵敏度不足,以及源于基因组信息固有性质的技术的测序偏差,这些是对提供任何或所有特异性、灵敏度和适用性以在各种临床环境中可靠地诊断拷贝数变化的非侵入性方法的持续需求的基础。已经显示胎儿cfdna片段的平均长度短于孕妇血浆中的母体cfdna片段。母体和胎儿
cfdna之间的这种差异在本文的实施方式中被利用以确定cnv和/或胎儿分数。本文公开的实施方案满足了一些上述需求。一些实施方案可以用无pcr的文库制备物与成对的末端dna测序结合实施。一些实施方案为非侵入性产前诊断和各种疾病的诊断提供高分析灵敏度和特异性。
9.发明概述
10.在一些实施方案中,提供了用于确定任何胎儿非整倍性的拷贝数变异(cnv)和已知或疑似与多种医学病况相关的cnv的方法。可以根据本方法确定的cnv包括1
‑
22号染色体、x染色体和y染色体中的任一个或多个的三体性和单体性、其它染色体多体性以及所述染色体中的任一个或多个的区段的缺失和/或重复。在一些实施方案中,该方法涉及在测试样品中鉴定目标核酸序列如临床相关序列的cnv。该方法评估了特定目标序列的拷贝数变异。
11.在一些实施方案中,该方法在计算机系统中实施,该计算机系统包括一个或多个处理器和系统存储器以评估包含一个或多个基因组的核酸的测试样品中的目标核酸序列的拷贝数。
12.本公开的一方面涉及一种用于确定包括源自两个或更多个基因组的无细胞核酸片段的测试样品中的目标核酸序列的拷贝数变异(cnv)的方法。该方法包括:(a)接收通过对测试样品中的无细胞核酸片段进行测序所获得的序列读取;(b)将无细胞核酸片段的序列读取或含有序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中参考基因组被分为多个箱;(c)确定测试样品中存在的至少一些无细胞核酸片段的片段尺寸;(d)通过以下,对于每个箱,计算参考基因组箱的序列标签覆盖率:(i)确定与箱比对的序列标签的数量,和(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与箱比对的序列标签的数量归一化;(e)使用目标序列中的箱覆盖率和目标序列的参考区域中的箱覆盖率来确定目标序列的t
‑
统计量;以及(f)使用从t
‑
统计量和关于无细胞核酸片段尺寸的信息所计算的似然比,确定目标序列中的拷贝数变异。
13.在一些实施方式中,该方法包括执行(d)和(e)两次,对第一尺寸域中的片段执行一次并对第二尺寸域中的片段再执行一次。在一些实施方式中,第一尺寸域包括样品中基本上所有尺寸的无细胞核酸片段,并且第二尺寸域仅包括小于限定尺寸的无细胞核酸片段。在一些实施方式中,第二尺寸域仅包括小于约150bp的无细胞核酸片段。在一些实施方式中,由使用第一尺寸范围内的片段的序列标签的目标序列的第一t
‑
统计量,以及使用第二尺寸范围内的片段的序列标签的目标序列的第二t
‑
统计量,计算似然比。
14.在一些实施方式中,将似然比计算为测试样品是非整倍体样品的第一似然性除以测试样品是整倍体样品的第二似然性。
15.在一些实施方式中,除了t
‑
统计量和关于无细胞核酸片段尺寸的信息之外,从胎儿分数的一个或多个值计算似然比。
16.在一些实施方式中,胎儿分数的一个或多个值包括使用关于无细胞核酸片段尺寸的信息所计算的胎儿分数值。在一些实施方式中,通过以下,计算胎儿分数值:获得片段尺寸的频率分布;并且将频率分布应用于使胎儿分数与片段尺寸频率相关联的模型,以获得胎儿分数值。在一些实施方式中,将胎儿分数与片段尺寸频率相关联的模型包括具有对于多个片段尺寸的多个项和系数的一般线性模型。
17.在一些实施方式中,胎儿分数的一个或多个值包括使用参考基因组的箱的覆盖率信息所计算的胎儿分数值。在一些实施方式中,通过以下,计算胎儿分数值:将多个箱的覆盖率值应用于使胎儿分数与箱的覆盖率相关联的模型,以获得胎儿分数值。在一些实施方式中,使胎儿分数与箱覆盖率相关联的模型包括具有对于多个箱的多个项和系数的一般线性模型。在一些实施方式中,多个箱在训练样品中具有胎儿分数和覆盖率之间的高度相关性。
18.在一些实施方式中,胎儿分数的一个或多个值包括使用在读取中发现的多个8
‑
聚体的频率所计算的胎儿分数值。在一些实施方式中,通过以下,计算胎儿分数值:将多个8
‑
聚体的频率应用于使胎儿分数与8
‑
聚体频率相关联的模型,以获得胎儿分数值。在一些实施方式中,使胎儿分数与8
‑
聚体频率相关联的模型包括具有对于多个8
‑
聚体的多个项和系数的一般线性模型。在一些实施方式中,多个8
‑
聚体具有胎儿分数和8
‑
聚体频率之间的高度相关性。
19.在一些实施方式中,胎儿分数的一个或多个值包括使用性染色体箱的覆盖率信息所计算的胎儿分数值。
20.在一些实施方式中,似然比是从胎儿分数、短片段的t
‑
统计量和所有片段的t统计量所计算的,其中短片段是在小于标准尺寸的第一尺寸范围内的无细胞核酸片段,以及所有片段是包括短片段和长于标准尺寸的片段的无细胞核酸片段。在一些实施方式中,如下计算似然比:
[0021][0022]
其中p1表示数据来自代表3拷贝或1拷贝模型的多元正态分布的似然性,p0表示数据来自代表2拷贝模型的多元正态分布的似然性,t
短
、t
所有
是由短片段和所有片段产生的染色体覆盖率所计算的t评分,以及q(ff
总
)是胎儿分数的密度分布。
[0023]
在一些实施方式中,除了t
‑
统计量和关于无细胞核酸片段尺寸的信息之外,从胎儿分数的一个或多个值计算似然比。
[0024]
在一些实施方式中,计算x单体、x三体、13三体、18三体或21三体的似然比。
[0025]
在一些实施方式中,将序列标签的数量归一化包括:针对样品的gc含量归一化、针对训练组的变异的全局波谱(global wave profile)归一化,和/或针对从主组分分析获得的一个或多个组分归一化。
[0026]
在一些实施方式中,目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0027]
在一些实施方式中,参考区域是所有稳定染色体、不包含目标序列的稳定染色体、至少目标序列之外的染色体,和/或选自稳定染色体的染色体亚组。在一些实施方式中,参考区域包括已被确定为一组训练样品提供最佳信号检测能力的稳定染色体。
[0028]
在一些实施方式中,该方法还包括:对于每个箱,计算箱的尺寸参数的值:(i)由箱中无细胞核酸片段的尺寸确定尺寸参数的值,和(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将尺寸参数的值归一化。该方法还包括,使用目标序列中的箱的尺寸参数的值和目标序列的参考区域中的箱的尺寸参数的值来确定目标序列的基于尺寸的t
‑
统计量。
在一些实施方式中,由t
‑
统计量和基于尺寸的t
‑
统计量计算(f)的似然比。在一些实施方式中,由基于尺寸的t
‑
统计量和胎儿分数计算(f)的似然比。
[0029]
在一些实施方式中,该方法还包括,将似然比与调用标准(call criterion)进行比较以确定目标序列中的拷贝数变异。在一些实施方式中,将似然比在与调用标准进行比较之前转换为log似然比。在一些实施方式中,通过将不同标准应用于训练样品的训练组并选择提供限定的灵敏度和限定的选择性的标准来获得调用标准。
[0030]
在一些实施方式中,该方法还包括,获得多个似然比并将多个似然比应用于决策树以确定样品的倍性情况。
[0031]
在一些实施方式中,该方法还包括,获得多个似然比和目标序列的一个或多个覆盖率值,并将多个似然比和目标序列的一个或多个覆盖率值应用于决策树以确定样品的倍性情况。
[0032]
本公开的另一方面涉及一种用于确定包括源自两个或更多个基因组的无细胞核酸片段的测试样品中的目标核酸序列的拷贝数变异(cnv)的方法。该方法包括:(a)接收通过对测试样品中的无细胞核酸片段进行测序所获得的序列读取;(b)将无细胞核酸片段的序列读取或含有序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中参考基因组被分为多个箱;(c)通过以下,对于每个箱,计算参考基因组箱的序列标签的覆盖率:(i)确定与箱比对的序列标签的数量,和(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与箱比对的序列标签的数量归一化。该方法还包括:(d)使用目标序列中的箱覆盖率和目标序列的参考区域中的箱覆盖率来确定目标序列的t
‑
统计量;(e)估计测试样品中的无细胞核酸片段的一个或多个胎儿分数值;以及(f)使用t
‑
统计量和一个或多个胎儿分数值,确定目标序列中的拷贝数变异。
[0033]
在一些实施方式中,(f)包括从t
‑
统计量和一个或多个胎儿分数值计算似然比。在一些实施方式中,计算x单体、x三体、13三体、18三体或21三体的似然比。
[0034]
在一些实施方式中,将序列标签的数量归一化包括:针对样品的gc含量归一化、针对训练组的变异的全局波谱归一化、和/或针对从主组分分析获得的一个或多个组分归一化。
[0035]
在一些实施方式中,目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0036]
本公开的另一方面涉及一种用于确定包括源自两个或更多个基因组的无细胞核酸片段的测试样品中的目标核酸序列的拷贝数变异(cnv)的方法。该方法包括:(a)接收通过对测试样品中的无细胞核酸片段进行测序所获得的序列读取;(b)将无细胞核酸片段的序列读取或含有序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中参考基因组被分为多个箱;(c)确定测试样品中存在的无细胞核酸片段的片段尺寸;(d)使用具有在第一尺寸域内的尺寸的无细胞核酸片段的序列标签,计算参数基因组的箱的序列标签的覆盖率;(e)使用具有在第二尺寸域内的尺寸的无细胞核酸片段的序列标签,计算参数基因组的箱的序列标签的覆盖率,其中第二尺寸域不同于第一尺寸域;(f)使用(c)中确定的片段尺寸计算参考基因组的箱的尺寸特征;以及(g)使用(d)和(e)中计算的覆盖率和(f)中计算的尺寸特征确定目标序列中的拷贝数变异。
[0037]
在一些实施方式中,第一尺寸域包括样品中基本上所有尺寸的无细胞核酸片段,
并且第二尺寸域仅包括小于限定尺寸的无细胞核酸片段。在一些实施方式中,第二尺寸域仅包括小于约150bp的无细胞核酸片段。
[0038]
在一些实施方式中,目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0039]
在一些实施方式中,(g)包括使用在(d)和/或(e)中计算的目标序列中的箱覆盖率来计算目标序列的t
‑
统计量。在一些实施方式中,其中计算目标序列的t
‑
统计量包括使用目标序列中的箱覆盖率和目标序列的参考区域中的箱覆盖率。
[0040]
在一些实施方式中,(g)包括使用在(f)中计算的目标序列中的箱的尺寸特征来计算目标序列的t
‑
统计量。在一些实施方式中,计算目标序列的t
‑
统计量包括使用目标序列中的箱的尺寸特征和目标序列的参考区域中的箱的尺寸特征。
[0041]
在一些实施方式中,箱的尺寸特征包括尺寸小于限定值的片段与箱中总片段的比率。
[0042]
在一些实施方式中,(g)包括从t
‑
统计量计算似然比。
[0043]
在一些实施方式中,(g)包括由使用(d)中所计算的覆盖率的目标序列的第一t
‑
统计量,以及使用(e)中所计算的覆盖率的目标序列的第二t
‑
统计量来计算似然比。
[0044]
在一些实施方式中,(g)包括由使用(d)中所计算的覆盖率的目标序列的第一t
‑
统计量、使用(e)中所计算的覆盖率的目标序列的第二t
‑
统计量以及使用(f)中所计算的尺寸特征的目标序列的第三t
‑
统计量来计算似然比。
[0045]
在一些实施方式中,除了至少第一和第二t
‑
统计量之外,从胎儿分数的一个或多个值计算似然比。在一些实施方式中,该方法还包括使用关于无细胞核酸片段的尺寸的信息来计算胎儿分数的一个或多个值。
[0046]
在一些实施方式中,该方法还包括使用参考基因组的箱覆盖率信息来计算胎儿分数的一个或多个值。在一些实施方式中,胎儿分数的一个或多个值包括使用性染色体的箱覆盖率信息来计算的胎儿分数值。在一些实施方式中,计算x单体、x三体、13三体、18三体或21三体的似然比。
[0047]
在一些实施方式中,(d)和/或(e)包括:(i)确定与箱比对的序列标签的数量,和(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与箱比对的序列标签的数量归一化。在一些实施方式中,将序列标签的数量归一化包括:针对样品的gc含量归一化、针对训练组的变异的全局波谱归一化、和/或针对从主组分分析获得的一个或多个组分归一化。
[0048]
在一些实施方式中,(f)包括,对于每个箱,计算箱的尺寸参数的值:(i)由箱中无细胞核酸片段的尺寸来确定尺寸参数的值,以及(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将尺寸参数的值归一化。
[0049]
本发明的另一方面涉及一种用于评估测试样品中目标核酸序列的拷贝数的系统,该系统包括:用于接收来自测试样品的核酸片段并提供测试样品的核酸序列信息的测序仪;处理器;和一个或多个计算机可读存储介质,其上存储有用于在所述处理器上执行的指令。该指令包括针对以下的指令:(a)接收通过对测试样品中的无细胞核酸片段进行测序所获得的序列读取;(b)将无细胞核酸片段的序列读取或含有序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中参考基因组被分为多个箱;(c)确定测试样品中存在的至少一些无细胞核酸片段的片段尺寸;以及(d)通过以下,对于
每个箱,计算参考基因组箱的序列标签的覆盖率:(i)确定与箱比对的序列标签的数量,和(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与箱比对的序列标签的数量归一化。该方法还包括:(e)使用目标序列中的箱覆盖率和目标序列的参考区域中的箱覆盖率来确定目标序列的t
‑
统计量;以及(f)使用从t
‑
统计量和关于无细胞核酸片段尺寸的信息计算的似然比,确定目标序列中的拷贝数变异。
[0050]
在一些实施方式中,该系统被配置为执行上述方法中任一种。
[0051]
本公开的另一方面涉及一种计算机程序产品,其包括一个或多个计算机可读的非暂时性存储介质,其上存储有计算机可执行指令,所述指令当由计算机系统的一个或多个处理器执行时,使计算机系统实施上述方法中任一种。
[0052]
作为非限制性实例,本申请提供了以下实施方案:
[0053]
1.用于确定测试样品中的目标核酸序列的拷贝数变异(cnv)的方法,所述测试样品包含源自两个或更多个基因组的无细胞核酸片段,所述方法包括:
[0054]
(a)接收通过对所述测试样品中的所述无细胞核酸片段进行测序所获得的序列读取;
[0055]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中所述参考基因组被分为多个箱;
[0056]
(c)确定所述测试样品中存在的至少一些无细胞核酸片段的片段尺寸;
[0057]
(d)对于每个箱,通过以下计算所述参考基因组箱的序列标签的覆盖率:
[0058]
(i)确定与所述箱比对的序列标签的数量,和
[0059]
(ii)基于由于拷贝数变异以外的因素导致的箱间变异,将与所述箱比对的序列标签的数量归一化;
[0060]
(e)使用所述目标序列中的箱覆盖率和所述目标序列的参考区域中的箱覆盖率来确定所述目标序列的t
‑
统计量;以及
[0061]
(f)使用从所述t
‑
统计量计算的似然比和关于所述无细胞核酸片段尺寸的信息,确定所述目标序列中的拷贝数变异。
[0062]
2.如实施方案1所述的方法,其包括进行(d)和(e)两次,一次用于第一尺寸域中的片段以及另一次用于第二尺寸域中的片段。
[0063]
3.如实施方案2所述的方法,其中所述第一尺寸域包括所述样品中基本上所有尺寸的无细胞核酸片段,并且所述第二尺寸域仅包括小于限定尺寸的无细胞核酸片段。
[0064]
4.如实施方案2所述的方法,其中所述第二尺寸域仅包括小于约150bp的无细胞核酸片段。
[0065]
5.如实施方案2所述的方法,其中由使用第一尺寸范围内的片段的序列标签的目标序列的第一t
‑
统计量,以及使用第二尺寸范围内的片段的序列标签的目标序列的第二t
‑
统计量来计算所述似然比。
[0066]
6.如实施方案1所述的方法,其中将所述似然比计算为所述测试样品是非整倍体样品的第一似然性除以所述测试样品是整倍体样品的第二似然性。
[0067]
7.如实施方案1所述的方法,其中除了所述t
‑
统计量和关于无细胞核酸片段尺寸的信息之外,从胎儿分数的一个或多个值计算所述似然比。
[0068]
8.如实施方案7所述的方法,其中所述胎儿分数的一个或多个值包括使用所述关于无细胞核酸片段尺寸的信息所计算的胎儿分数的值。
[0069]
9.如实施方案8所述的方法,其中通过以下计算所述胎儿分数的值:
[0070]
获得所述片段尺寸的频率分布;和
[0071]
将所述频率分布应用于使胎儿分数与片段尺寸频率相关联的模型,以获得所述胎儿分数值。
[0072]
10.如实施方案8所述的方法,其中所述使胎儿分数与片段尺寸频率相关联的模型包括具有对于多个片段尺寸的多个项和系数的一般线性模型。
[0073]
11.如实施方案7所述的方法,其中所述胎儿分数的一个或多个值包括使用所述参考基因组的箱的覆盖率信息所计算的胎儿分数的值。
[0074]
12.如实施方案11所述的方法,其中通过以下来计算所述胎儿分数的值:将多个箱的覆盖率值应用于使胎儿分数与箱覆盖率相关联的模型,以获得胎儿分数值。
[0075]
13.如实施方案12所述的方法,其中所述使胎儿分数与箱覆盖率相关联的模型包括具有对于多个箱的多个项和系数的一般线性模型。
[0076]
14.如实施方案13所述的方法,其中所述多个箱在训练样品中具有胎儿分数和覆盖率之间的高度相关性。
[0077]
15.如实施方案7所述的方法,其中所述胎儿分数的一个或多个值包括使用在所述读取中发现的多个8
‑
聚体的频率所计算的胎儿分数的值。
[0078]
16.如实施方案15所述的方法,其中通过以下计算所述胎儿分数的值:将多个8
‑
聚体的频率应用于使胎儿分数与8
‑
聚体频率相关联的模型,以获得所述胎儿分数值。
[0079]
17.如实施方案16所述的方法,其中所述使胎儿分数与8
‑
聚体频率相关联的模型包括具有对于多个8
‑
聚体的多个项和系数的一般线性模型。
[0080]
18.如实施方案17所述的方法,其中所述多个8
‑
聚体具有胎儿分数和8
‑
聚体频率之间的高度相关性。
[0081]
19.如实施方案7所述的方法,其中所述胎儿分数的一个或多个值包括使用性染色体箱的覆盖率信息所计算的胎儿分数的值。
[0082]
20.如实施方案7所述的方法,其中所述似然比是从胎儿分数、短片段的t
‑
统计量和所有片段的t统计量所计算的,其中所述短片段是在小于标准尺寸的第一尺寸范围内的无细胞核酸片段,并且所述所有片段是包括短片段和长于标准尺寸的片段的无细胞核酸片段。
[0083]
21.如实施方案20所述的方法,其中由以下计算所述似然比:
[0084][0085]
其中p1表示数据来自代表3拷贝或1拷贝模型的多元正态分布的似然性,p0表示数据来自代表2拷贝模型的多元正态分布的似然性,t
短
、t
所有
是由短片段和所有片段产生的染色体覆盖率所计算的t评分,以及q(ff
总
)是胎儿分数的密度分布。
[0086]
22.如实施方案1所述的方法,其中除了所述t
‑
统计量和关于无细胞核酸片段尺寸的信息之外,从胎儿分数的一个或多个值计算所述似然比。
[0087]
23.如实施方案1所述的方法,其中计算x单体、x三体、13三体、18三体或21三体的似然比。
[0088]
24.如实施方案1所述的方法,其中将序列标签的数量归一化包括:针对所述样品的gc含量归一化,针对训练组的变异的全局波谱归一化,和/或针对从主组分分析获得的一个或多个组分归一化。
[0089]
25.如实施方案1所述的方法,其中所述目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0090]
26.如实施方案1所述的方法,其中所述参考区域选自:所有稳定染色体、不包含所述目标序列的稳定染色体、至少所述目标序列之外的至少一条染色体,以及选自所述稳定染色体的染色体亚组。
[0091]
27.如实施方案26所述的方法,其中所述参考区域包括已被确定能为一组训练样品提供最佳信号检测能力的稳定染色体。
[0092]
28.如实施方案1所述的方法,其还包括:
[0093]
对于每个箱,通过以下计算箱的尺寸参数的值:
[0094]
(i)由所述箱中无细胞核酸片段的尺寸确定所述尺寸参数的值,和
[0095]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将所述尺寸参数的值归一化;和
[0096]
使用所述目标序列中的箱的尺寸参数的值和所述目标序列的参考区域中的箱的尺寸参数的值,来确定所述目标序列的基于尺寸的t
‑
统计量。
[0097]
29.如实施方案28所述的方法,其中由所述t
‑
统计量和所述基于尺寸的t
‑
统计量计算(f)的似然比。
[0098]
30.如实施方案28所述的方法,其中由所述基于尺寸的t
‑
统计量和胎儿分数计算(f)的似然比。
[0099]
31.如实施方案1所述的方法,其还包括将所述似然比与调用标准进行比较,以确定所述目标序列中的拷贝数变异。
[0100]
32.如实施方案31所述的方法,其中在与所述调用标准进行比较之前,将所述似然比转换为log似然比。
[0101]
33.如实施方案31所述的方法,其中通过将不同标准应用于训练样品的训练组并选择提供限定的灵敏度和限定的选择性的标准来获得所述调用标准。
[0102]
34.如实施方案1所述的方法,其还包括获得多个似然比,并将所述多个似然比应用于决策树以确定所述样品的倍性情况。
[0103]
35.如实施方案1所述的方法,其还包括获得多个似然比和目标序列的一个或多个覆盖率值,并将多个似然比和所述目标序列的一个或多个覆盖率值应用于决策树以确定所述样品的倍性情况。
[0104]
36.用于评估测试样品中目标核酸序列的拷贝数的系统,所述系统包括:
[0105]
测序仪,其用于接收来自所述测试样品的核酸片段并提供所述测试样品的核酸序列信息;
[0106]
处理器;和
[0107]
一个或多个计算机可读存储介质,其上存储有用于在所述处理器上执行的以下指
令:
[0108]
(a)接收通过对所述测试样品中的无细胞核酸片段进行测序而获得的序列读取;
[0109]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含所述目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中所述参考基因组被分为多个箱;
[0110]
(c)确定所述测试样品中存在的至少一些无细胞核酸片段的片段尺寸;
[0111]
(d)对于每个箱,通过以下计算所述参考基因组的箱的序列标签的覆盖率:
[0112]
(i)确定与所述箱比对的序列标签的数量,和
[0113]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与所述箱比对的序列标签的数量归一化;
[0114]
(e)使用所述目标序列中的箱覆盖率和所述目标序列的参考区域中的箱覆盖率来确定所述目标序列的t
‑
统计量;和
[0115]
(f)使用由所述t
‑
统计量和关于无细胞核酸片段尺寸的信息所计算的似然比,确定所述目标序列中的拷贝数变异。
[0116]
37.用于确定包含源自两个或更多个基因组的无细胞核酸片段的测试样品中的目标核酸序列的拷贝数变异(cnv)的方法,所述方法包括:
[0117]
(a)接收通过对所述测试样品中的所述无细胞核酸片段进行测序而获得的序列读取;
[0118]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中参考基因组被分为多个箱;
[0119]
(c)对于每个箱,通过以下计算所述参考基因组箱的序列标签的覆盖率:
[0120]
(i)确定与所述箱比对的序列标签的数量,和
[0121]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与所述箱比对的序列标签的数量归一化;
[0122]
(d)使用所述目标序列中的箱覆盖率和所述目标序列的参考区域中的箱覆盖率来确定所述目标序列的t
‑
统计量;
[0123]
(e)估计所述测试样品中的无细胞核酸片段的一个或多个胎儿分数值;和
[0124]
(f)使用所述t
‑
统计量和所述一个或多个胎儿分数值,确定所述目标序列中的拷贝数变异。
[0125]
38.如实施方案37所述的方法,其中(f)包括从所述t
‑
统计量和所述一个或多个胎儿分数值计算似然比。
[0126]
39.如实施方案38所述的方法,其中计算x单体、x三体、13三体、18三体或21三体的似然比。
[0127]
40.如实施方案37所述的方法,其中将序列标签的数量归一化包括:针对所述样品的gc含量归一化,针对训练组的变异的全局波谱归一化,和/或针对从主组分分析获得的一个或多个组分归一化。
[0128]
41.如实施方案37所述的方法,其中所述目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0129]
42.用于评估测试样品中目标核酸序列的拷贝数的系统,所述系统包括:
[0130]
测序仪,其用于接收来自所述测试样品的核酸片段并提供所述测试样品的核酸序列信息;
[0131]
处理器;和
[0132]
一个或多个计算机可读存储介质,其上存储有用于在所述处理器上执行的以下指令:
[0133]
(a)接收通过对所述测试样品中的无细胞核酸片段进行测序而获得的序列读取;
[0134]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含所述目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中所述参考基因组被分为多个箱;
[0135]
(c)对于每个箱,通过以下计算所述参考基因组的箱的序列标签的覆盖率:
[0136]
(i)确定与所述箱比对的序列标签的数量,和
[0137]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与所述箱比对的所述序列标签的数量归一化;
[0138]
(d)使用所述目标序列中的箱覆盖率和所述目标序列的参考区域中的箱覆盖率来确定所述目标序列的t
‑
统计量;
[0139]
(e)估计所述测试样品中的无细胞核酸片段的胎儿分数;和
[0140]
(f)使用所述t
‑
统计量以及关于胎儿分数和无细胞核酸片段尺寸的信息,确定所述目标序列中的拷贝数变异。
[0141]
43.用于确定测试样品中的目标核酸序列的拷贝数变异(cnv)的方法,所述测试样品包括来源于两个或多个基因组的无细胞核酸片段,所述方法包括:
[0142]
(a)接收通过对所述测试样品中的无细胞核酸片段进行测序而获得的序列读取;
[0143]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中所述参考基因组被分为多个箱;
[0144]
(c)确定所述测试样品中存在的无细胞核酸片段的片段尺寸;
[0145]
(d)使用具有在第一尺寸域内的尺寸的无细胞核酸片段的序列标签,计算所述参数基因组的箱的序列标签覆盖率;
[0146]
(e)使用具有在第二尺寸域内的尺寸的无细胞核酸片段的序列标签,计算所述参数基因组的箱的序列标签覆盖率,其中所述第二尺寸域不同于所述第一尺寸域;
[0147]
(f)使用(c)中确定的片段尺寸,计算所述参考基因组的箱的尺寸特征;和
[0148]
(g)使用(d)和(e)中计算的覆盖率和(f)中计算的尺寸特征,确定所述目标序列中的拷贝数变异。
[0149]
44.如实施方案43所述的方法,其中所述第一尺寸域包括所述样品中基本上所有尺寸的无细胞核酸片段,并且所述第二尺寸域仅包括小于限定尺寸的无细胞核酸片段。
[0150]
45.如实施方案44所述的方法,其中所述第二尺寸域仅包括小于约150bp的无细胞核酸片段。
[0151]
46.如实施方案43所述的方法,其中所述目标序列是选自以下的人染色体:13号染色体、18号染色体、21号染色体、x染色体和y染色体。
[0152]
47.如实施方案43所述的方法,其中(g)包括使用(d)和/或(e)中计算的目标序列
中的箱覆盖率来计算所述目标序列的t
‑
统计量。
[0153]
48.如实施方案47所述的方法,其中计算所述目标序列的t
‑
统计量包括使用所述目标序列中的箱覆盖率和所述目标序列的参考区域中的箱覆盖率。
[0154]
49.如实施方案43所述的方法,其中(g)包括使用(f)中计算的目标序列中的箱的尺寸特征来计算所述目标序列的t
‑
统计量。
[0155]
50.如实施方案49所述的方法,其中计算所述目标序列的t
‑
统计量包括使用所述目标序列中的箱的尺寸特征和所述目标序列的参考区域中的箱的尺寸特征。
[0156]
51.如实施方案43所述的方法,其中箱的尺寸特征包括所述箱中的小于限定值的尺寸的片段与总片段的比率。
[0157]
52.如实施方案43所述的方法,其中(g)包括由所述t
‑
统计量来计算似然比。
[0158]
53.如实施方案43所述的方法,其中(g)包括由使用(d)中计算的覆盖率的来自所述目标序列的第一t
‑
统计量以及使用(e)中计算的覆盖率的来自所述目标序列的第二t
‑
统计量,计算似然比。
[0159]
54.如实施方案43所述的方法,其中(g)包括由使用(d)中计算的覆盖率的来自所述目标序列的第一t
‑
统计量、使用(e)中计算的覆盖率的来自所述目标序列的第二t
‑
统计量以及使用(f)中计算的尺寸特征的来自所述目标序列的第三t
‑
统计量,计算似然比。
[0160]
55.如实施方案53或54所述的方法,其中除了至少第一t
‑
统计量和第二t
‑
统计量之外,由胎儿分数的一个或多个值计算所述似然比。
[0161]
56.如实施方案55所述的方法,其还包括使用关于无细胞核酸片段尺寸的信息来计算所述胎儿分数的一个或多个值。
[0162]
57.如实施方案55所述的方法,其还包括使用所述参考基因组的箱覆盖率信息来计算所述胎儿分数的一个或多个值。
[0163]
58.如实施方案55所述的方法,其中所述胎儿分数的一个或多个值包括使用性染色体的箱覆盖率信息计算的胎儿分数的值。
[0164]
59.如实施方案55所述的方法,其中计算x单体、x三体、13三体、18三体或21三体的似然比。
[0165]
60.如实施方案43所述的方法,其中(d)和/或(e)包括:
[0166]
(i)确定与所述箱比对的序列标签的数量,和
[0167]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将与所述箱比对的序列标签的数量归一化。
[0168]
61.如实施方案60所述的方法,其中将序列标签的数量归一化包括:针对所述样品的gc含量归一化,针对训练组的变异的全局波谱归一化,和/或针对从主组分分析获得的一个或多个组分归一化。
[0169]
62.如权利要求43所述的方法,其中(f)包括对于每个箱,通过以下计算箱尺寸参数的值:
[0170]
(i)由所述箱中无细胞核酸片段的尺寸来确定所述尺寸参数的值,和
[0171]
(ii)基于由于拷贝数变异以外的因素所导致的箱间变异,将所述尺寸参数的值归一化。
[0172]
63.用于评估测试样品中目标核酸序列的拷贝数的系统,所述系统包括:
[0173]
测序仪,其用于接收来自所述测试样品的核酸片段并提供所述测试样品的核酸序列信息;
[0174]
处理器;和
[0175]
一个或多个计算机可读存储介质,其上存储有用于在所述处理器上执行的以下指令:
[0176]
(a)接收通过对所述测试样品中的无细胞核酸片段进行测序而获得的序列读取;
[0177]
(b)将所述无细胞核酸片段的序列读取或含有所述序列读取的片段与包含所述目标序列的参考基因组的箱进行比对,从而提供测试序列标签,其中所述参考基因组被分为多个箱;
[0178]
(c)确定所述测试样品中存在的无细胞核酸片段的片段尺寸;
[0179]
(d)使用具有在第一尺寸域内的尺寸的无细胞核酸片段的序列标签,计算所述参考基因组箱的序列标签覆盖率;
[0180]
(e)使用具有在第二尺寸域内的尺寸的无细胞核酸片段的序列标签,计算所述参考基因组箱的序列标签覆盖率,其中所述第二尺寸域不同于所述第一尺寸域;
[0181]
(f)使用(c)中确定的片段尺寸,计算所述参考基因组的箱的尺寸特征;和
[0182]
(g)使用(d)和(e)中计算的覆盖率以及(f)中计算的尺寸特征,确定所述目标序列中的拷贝数变异。
[0183]
尽管本文的实施例涉及人类并且语言主要针对人类的关注点,但本文描述的概念适用于来自任何植物或动物的基因组。根据以下描述和所附权利要求,本公开的这些和其它目的和特征将变得更加明显,或者可以通过如下文所述的本公开的实践来了解。
[0184]
通过参考并入
[0185]
本文提及的所有专利、专利申请和其它出版物,包括在这些参考文献中公开的所有序列,通过引用明确地并入本文,其程度如同每个单独的出版物、专利或专利申请被明确地和单独地指出要通过引用并入一样。引用的所有文献在相关部分中通过引用整体并入本文,用于本文引用其的上下文中指出的目的。然而,任何文献的引用不应被解释为承认它是关于本公开的现有技术。
[0186]
附图简述
[0187]
图1是用于确定包含核酸混合物的测试样品中存在或缺失拷贝数变异的方法100的流程图。
[0188]
图2a主题性地说明了配对的末端测序如何可以用于确定片段尺寸和序列覆盖率。
[0189]
图2b显示了使用基于尺寸的覆盖率来确定测试样品中目标核酸序列的拷贝数变异的方法的流程图。
[0190]
图2c描绘了用于确定用于评估拷贝数的目标核酸序列的片段尺寸参数的方法的流程图。
[0191]
图2d显示了工作流程的两个重叠通路的流程图。
[0192]
图2e显示了用于评估拷贝数的三途径方法(three
‑
pass process)的流程图。
[0193]
图2f显示了将t
‑
统计量应用于拷贝数分析以提高分析精确度的实施方式。
[0194]
图2g显示了根据本公开的一些实施方式的用于从覆盖率信息确定胎儿分数的示例方法。
[0195]
图2h显示了根据一些实施方式的用于根据尺寸分布信息确定胎儿分数的方法。
[0196]
图2i显示了根据本公开的一些实施方式的用于从8
‑
聚体频率信息确定胎儿分数的示例方法。
[0197]
图2j显示了用于处理序列读取信息的工作流程,其可用于获得胎儿分数估值。
[0198]
图3a显示了用于降低来自测试样品的序列数据中的噪声的方法的实例的流程图。
[0199]
图3b
‑
3k呈现了在图3a中所述的方法的各个阶段获得的数据的分析。
[0200]
图4a显示了用于创建用于降低序列数据中的噪声的序列掩码的方法的流程图。
[0201]
图4b显示了mapq评分与归一化覆盖量(normalized coverage quantities)的cv具有强的单一相关性。
[0202]
图5是用于处理测试样品并最终进行诊断的分散系统的框图。
[0203]
图6示意性地说明了处理测试样品的不同操作可以如何被分组以由系统的不同元件处理。
[0204]
图7a和图7b显示根据实施例1a(图7a)中描述的缩略方案和实施例1b中描述的方案(图7b)制备的cfdna测序文库的电泳图。
[0205]
图8显示了与标准实验室工作流程相比,新版nipt的整体工作流程和时间线。
[0206]
图9显示了作为输入提取的cfdna的函数的测序文库产量(yield),其表明与文库浓度至输入浓度的强线性相关性,具有高转化效率。
[0207]
图10显示了如从具有男性胎儿的妊娠的324个样品测量的cfdna片段尺寸分布。
[0208]
图11显示了来自定位的配对末端读取的总计数与来自小于150bp的配对末端读取的计数相比的相对胎儿分数。
[0209]
图12显示了用于检测21三体样品的以下各项的组合t
‑
统计量非整倍性评分:(a)所有片段的计数;(b)仅短片段(<150bp)计数;(c)短片段的分数(在80与150bp之间计数/计数<250bp);(d)来自(b)和(c)的组合t
‑
统计量;和(e)使用illumina redwood city clia实验室方法用平均16m计数/样品获得的相同样品的结果。
[0210]
图13显示了从所选的箱估计的胎儿分数相对于用x染色体的归一化染色体值(ref)测量的胎儿分数。组1(set 1)用于校准胎儿分数值,并且独立组2用于测试相关性。
[0211]
发明详述
[0212]
定义
[0213]
除非另有说明,否则本文公开的方法和系统的实践涉及常用于分子生物学、微生物学、蛋白质纯化、蛋白质工程、蛋白质和dna测序以及重组dna领域的常规技术和装置,这些技术和装置在本领域的技术范围内。此类技术和装置是本领域技术人员已知的并且在许多文本和参考文献中有所描述(参见如,sambrook等人,
″
molecular cloning:a laboratory manual,
″
第三版(cold spring harbor),[2001]);和ausubel等人,
ꢀ″
current protocols in molecular biology
″
[1987])。
[0214]
数字范围包括限定范围的数字。在本说明书全文中给出的每个最大数值限制意在包括每个较低的数值限制,如同这些较低的数值限制在本文中明确写出一样。在本说明书全文中给出的每个最小数值限制将包括每个较高的数值限制,如同此类较高数值限制在本文中明确写出一样。在本说明书全文中给出的每个数值范围将包括落入此类更宽的数值范围内的每个较窄的数值范围,如同这些较窄的数值范围都在本文中明确写出一样。
[0215]
本文提供的标题不旨在限制本公开。
[0216]
除非本文另有定义,否则本文使用的所有技术和科学术语具有与本领域普通技术人员通常理解的含义相同的含义。包括本文包括的术语的各种科学词典是本领域技术人员公知的并且是可获得的。尽管与本文描述的那些方法和材料类似或等同的任何方法和材料可用于实践或测试本文公开的实施方案,但描述了一些方法和材料。
[0217]
通过参考整个说明书,可以更全面地描述紧接的下面定义的术语。应理解,本公开不限于所描述的特定方法、方案和试剂,因为这些可以根据本领域技术人员使用的上下文而变化。如本文所用,除非上下文另有明确说明,否则单数术语
″
一个
″
、
″
一种
″
和
″
该
″
包括复数指代。
[0218]
除非另有说明,分别地,核酸以5'至3'方向从左至右书写,并且氨基酸序列以氨基至羧基方向从左至右书写。
[0219]
本文使用的术语
″
参数
″
表示物理特征,其值或其它特征对诸如拷贝数变异的相关条件具有影响。在一些情况下,术语参数用于参考影响数学关系或模型的输出的变量,所述变量可以是自变量(即,模型的输入)或基于一个或多个自变量的中间变量。根据模型的范围,一个模型的输出可能成为另一个模型的输入,从而成为其它模型的参数。
[0220]
术语
″
片段尺寸参数
″
是指涉及片段(如核酸片段;例如从体液中获得的cfdna片段)或片段集合的尺寸或长度的参数。如本文所用,当1)对于片段尺寸或尺寸范围,参数有利地被加权,如当与该尺寸或尺寸范围的片段相关联时,计数加权比其它尺寸或范围的计数更大;或者2)从对于该片段尺寸或尺寸范围有利地加权的值获得参数,如当与该尺寸或尺寸范围的片段相关联时,从计数获得比率加权更大时,参数
″
偏向片段尺寸或尺寸范围
″
。当基因组产生相对于来自另一基因组或相同基因组的其它部分的核酸片段富含或具有更高浓度的尺寸或尺寸范围的核酸片段时,片段尺寸或尺寸范围可以是基因组或其部分的特征。
[0221]
术语
″
加权
″
是指使用被认为是
″
权
″
的一个或多个值或函数来修改数量诸如参数或变量。在某些实施方案中,参数或变量乘以权。在其它实施方案中,参数或变量以指数方式进行修改。在一些实施方案中,函数可以是线性或非线性函数。适用的非线性函数的实例包括但不限于海维塞德阶跃函数(heaviside step functions)、箱车函数(box
‑
car functions)、阶梯函数或s型函数。对原始参数或变量加权可以系统地增加或减少经加权的变量的值。在某些实施方案中,加权可以导致正值、非负值或负值。
[0222]
术语
″
拷贝数变异
″
在本文中是指相较于参考样品中存在的核酸序列的拷贝数,测试样品中存在的核酸序列的拷贝数的变异。在某些实施方案中,核酸序列为1kb或更大。在一些情况下,核酸序列是整个染色体或其重要部分。
″
拷贝数变体
″
是指其中通过将测试样品中的目标核酸序列与预期水平的目标核酸序列进行比较而发现拷贝数差异的核酸序列。例如,将测试样品中的目标核酸序列的水平与合格样品中存在的目标核酸序列的水平进行比较。拷贝数变体/变异包括缺失(包括微缺失)、插入(包括微插入)、复制、增殖和易位。cnv包括染色体非整倍性和部分非整倍性。
[0223]
术语
″
非整倍性
″
在本文中是指由整个染色体或染色体的一部分的丢失或获得引起的遗传物质的不平衡。
[0224]
术语
″
染色体非整倍性
″
和
″
完全染色体非整倍性
″
在本文中是指由整个染色体的
丢失或获得引起的遗传物质的不平衡,并且包括种系非整倍性和镶嵌非整倍性。
[0225]
术语
″
部分非整倍性
″
和
″
部分染色体非整倍性
″
在本文中是指由染色体的一部分丢失或获得引起的遗传物质的不平衡,如部分单体和部分三体,并涵盖由易位、缺失和插入导致的不平衡。
[0226]
术语
″
多个
″
是指不止一个要素。例如,术语在本文中用于指代使用本文公开的方法足以鉴定测试样品和合格样品中拷贝数变异的显著差异的核酸分子或序列标签的数量。在每个实施方案中,为每个测试样品获得约20至40bp的至少约3
×
106个序列标签。在一些实施方案中,每个测试样品提供至少约5
×
106、8
×
106、10
×
106、15
×
106、20
×
106、30
×
106、40
×
106或50
×
106个序列标签的数据,每个序列标签包含约20至40bp。
[0227]
术语
″
配对末端读取
″
是指来自配对末端测序的读取,该配对末端测序从核酸片段的每一端获得一个读取。配对末端测序可以涉及将多核苷酸链片段化为称为插入物的短序列。对于相对短的多核苷酸如无细胞dna分子,片段化是任选的或不必要的。
[0228]
术语
″
多核苷酸
″
、
″
核酸
″
和
″
核酸分子
″
可互换使用,并且是指核苷酸(即对于rna是核糖核苷酸和对于dna是脱氧核糖核苷酸)的共价连接序列,其中一个核苷酸的戊糖的3'位置通过磷酸二酯基团连接到下一个核苷酸的戊糖的5'位置。核苷酸包括任何形式的核酸的序列,包括但不限于rna和dna分子,诸如cfdna分子。术语
″
多核苷酸
″
包括但不限于单链和双链多核苷酸。
[0229]
术语
″
测试样品
″
在本文中是指通常来源于生物流体、细胞、组织、器官或生物体的包含核酸或核酸混合物的样品,所述核酸混合物包含至少一条待筛选拷贝数变异的核酸序列。在某些实施方案中,该样品包含至少一条核酸序列,其拷贝数疑似已经发生变异。此类样品包括但不限于痰/口腔液、羊水、血液、血液级分或细针活检样品(如,手术活检、细针活检等)、尿液、腹膜液、胸膜液等。虽然样品通常取自人类受试者(例如,患者),但测定可用于任何哺乳动物(包括但不限于狗、猫、马、山羊、绵羊、牛、猪等)的样品中拷贝数变异(cnv)。样品可以从生物来源获得便直接使用或在改变样品特性的预处理后使用。例如,这种预处理可包括从血液制备血浆、稀释粘性流体等。预处理方法还可包括但不限于过滤、沉淀、稀释、蒸馏、混合、离心、冷冻、冻干、浓缩、扩增、核酸片段化、使干扰组分失活、试剂的添加、裂解等。如果对样品采用这种预处理方法,则这种预处理方法通常使得一个或多个目标核酸有时以与未处理的测试样品(如,即,未经过任何一种或多种这种预处理方法的样品)中的浓度成比例的浓度保留在测试样品中。这种
″
经处理的
″
或
″
经加工的
″
样品仍被认为是关于本文所述方法的生物
″
测试
″
样品。
[0230]
术语
″
合格样品
″
或
″
未受影响的样品
″
在本文中是指包含将与测试样品中的核酸进行比较的以已知的拷贝数存在的核酸的混合物的样品,并且它对于目标核酸序列是正常的、即非整倍体的样品。在一些实施方案中,合格样品用作训练组的未受影响的训练样品,以推导出序列掩码或序列谱(sequence profiles)。在某些实施方案中,合格样品用于识别所考虑的染色体的一个或多个归一化染色体或区段。例如,合格样品可用于识别21号染色体的归一化染色体。在这种情况下,合格样品是并非21三体样品的样品。另一个实例涉及仅使用女性作为x染色体的合格样品。合格样品也可以用于其它目的,诸如确定用于识别受影响样品的阈值、鉴定用于限定参考序列上的掩码区域的阈值、确定基因组的不同区域的预期覆盖量等。
[0231]
术语
″
训练组
″
在本文中是指一组训练样品,其可以包括受影响的和/或未受影响的样品并且用于开发用于分析测试样品的模型。在一些实施方案中,训练组包括未受影响的样品。在这些实施方案中,使用不受目标拷贝数变异影响的样品的训练组来建立用于确定cnv的阈值。训练组中未受影响的样品可用作鉴定归一化序列、如归一化染色体的合格样品,并且未受影响的样品的染色体剂量用于设定目标序列(如,染色体)中的每一个的阈值。在一些实施方案中,训练组包括受影响的样品。训练组中受影响的样品可用于验证受影响的测试样品是否可以很容易地与未受影响的样品区分开来。
[0232]
训练组也是目标群体中的统计样品,所述统计样品不应与生物样品混淆。统计样品通常包括多个个体,所述个体的数据用于确定对群体可推广的一个或多个目标定量值。统计样品是目标群体中的个体的亚组。个体可以是人、动物、组织、细胞、其它生物样品(即,统计样品可以包括多个生物样品),以及提供用于统计分析的数据点的其它个体实体。
[0233]
通常,训练组与验证组一起使用。术语
″
验证组
″
用于指代统计样品中的个体组,所述个体的数据用于验证或评估使用训练组确定的目标定量值。在一些实施方案中,例如,训练组提供用于计算参考序列的掩码的数据,而验证组提供评估掩码的有效性或效能的数据。
[0234]
″
拷贝数的评估
″
在本文中用于参考与序列拷贝数相关的遗传序列的状态的统计评估。例如,在一些实施方案中,评估包括确定遗传序列的存在或缺失。在一些实施方案中,评估包括确定遗传序列的部分或完全非整倍性。在其它实施方案中,评估包括基于遗传序列的拷贝数在两个或更多个样品之间进行区分。在一些实施方案中,评估包括基于遗传序列的拷贝数的统计分析,如,归一化和比较。
[0235]
术语
″
合格核酸
″
可与
″
合格序列
″
互换使用,其是与目标序列或核酸的量进行比较的序列。合格序列是优选以已知表现存在于生物样品中的序列,即合格序列的量是已知的。通常,合格序列是
″
合格样品
″
中存在的序列。
″
合格目标序列
″
是合格序列,其数量在合格样品中是已知的并且是与对照受试者和患有医学病况的个体之间的目标序列的差异相关联的序列。
[0236]
术语
″
目标序列
″
或
″
目标核酸序列
″
在本文中是指与健康和患病个体之间的序列表示的差异相关的核酸序列。目标序列可以是染色体上的序列,其在疾病或遗传病况中被错误表现,即过度表现或表现不足。目标序列可以是染色体的一部分,即染色体区段,或整个染色体。例如,目标序列可以是在非整倍性病况中过度表现的染色体,或在癌症中表现不足的编码肿瘤抑制子的基因。目标序列包括在总群体或受试者细胞的亚群中过度表现或表现不足的序列。
″
合格目标序列
″
是合格样品中的目标序列。
″
测试目标序列
″
是测试样品中的目标序列。
[0237]
术语
″
归一化序列
″
在本文中是指用于将定位到与所述归一化序列相关联的目标序列的序列标签的数量归一化的序列。在一些实施方案中,归一化序列包括稳定染色体。
″
稳定染色体
″
是不太可能是非整倍体的染色体。在涉及人染色体的一些情况下,稳定染色体是除x染色体、y染色体、13号染色体、18号染色体和21号染色体之外的任何染色体。在一些实施方案中,归一化序列显示在样品和测序运行中定位到它的序列标签的数量的变异性,该变异性近似于用作归一化参数的目标序列的变异性。归一化序列可以将受影响的样品与一种或多种未受影响的样品区分开来。在一些实施方式中,当与其它潜在的归一化序列诸
如其它染色体相比时,归一化序列最佳或有效地区分受影响的样品与一种或多种未受影响的样品。在一些实施方案中,归一化序列的变异性计算为样品和测序运行中的目标序列的染色体剂量的变异性。在一些实施方案中,在一组未受影响的样品中鉴定归一化序列。
[0238]
″
归一化染色体
″
、
″
归一化标准染色体
″
或
″
归一化染色体序列
″
是
″
归一化序列
″
的实例。
″
归一化染色体序列
″
可由单个染色体或一组染色体组成。在一些实施方案中,归一化序列包括两个或更多个稳定染色体。在某些实施方案中,稳定染色体是x染色体、y染色体、13号染色体、18号染色体和21号染色体以外的常染色体。
″
归一化区段
″
是
″
归一化序列
″
的另一个实例。
″
归一化区段序列
″
可以由染色体的单个区段组成,或者它可以由相同或不同染色体的两个或更多个区段组成。在某些实施方案中,归一化序列旨在针对变异性诸如方法相关的、染色体间(运行内(intra
‑
run))和测序间(运行间(inter
‑
run))变异性进行归一化。
[0239]
术语
″
可区分性
″
在本文中是指归一化染色体的特征,其使人们能够将一种或多种未受影响的即正常的样品与一种或多种受影响的即非整倍体样品区分开。展示最佳
″
可区分性
″
的归一化染色体是染色体或染色体组,它在一组合格样品中的目标染色体的染色体剂量分布与在一种或多种受影响的样品中相应的染色体中相同的目标染色体的染色体剂量之间提供最大的统计差异。
[0240]
术语
″
变异性
″
在本文中是指归一化染色体的另一个特征,其使人们能够将一种或多种未受影响的即正常的样品与一种或多种受影响的即非整倍体样品区分开。在一组合格的样品中测量的归一化染色体的变异性是指定位至它的序列标签数量的变异性,其近似于定位至目标染色体的序列标签数量的变异性(对于目标染色体其用作归一化参数)。
[0241]
术语
″
序列标签密度
″
在本文中是指定位至参考基因组序列的序列读取的数量,如21号染色体的序列标签密度是由测序方法产生的定位至参考基因组的21号染色体的序列读取的数量。
[0242]
术语
″
序列标签密度比
″
在本文中是指定位至参考基因组的染色体(如,21号染色体)的序列标签的数量与参考基因组染色体的长度之比。
[0243]
术语
″
序列剂量
″
在本文中是指与序列标签的数量相关的参数或针对目标序列和序列标签数量鉴定的另一参数或针对归一化序列鉴定的其它参数。在一些情况下,序列剂量是指目标序列的序列标签覆盖率或其它参数与归一化序列的序列标签密度或其它参数的比率。在一些情况下,序列剂量是指将目标序列的序列标签密度与归一化序列的序列标签密度相关联的参数。
″
测试序列剂量
″
是将目标序列(如21号染色体)的序列标签密度或其它参数与测试样品中测定的归一化序列(如9号染色体)的序列标签密度或其它参数相关联的参数。类似地,
″
合格序列剂量
″
是将目标序列的序列标签密度或其它参数与合格样品中确定的归一化序列的序列标签密度或其它参数相关联的参数。
[0244]
术语
″
覆盖率
″
是指定位至定义序列的序列标签的丰度。覆盖率可以通过序列标签密度(或序列标签计数)、序列标签密度比、归一化的覆盖量、调整的覆盖值等来定量地表示。
[0245]
术语
″
覆盖量
″
是指原始覆盖率的修改,并且通常表示基因组区域诸如箱中序列标签(有时称为计数)的相对数量。覆盖量可通过归一化、调整和/或校正基因组区域的原始覆盖率或计数来获得。例如,可以通过将定位至该区域的序列标签计数除以定位至整个基因
组的总数序列标签来获得区域的经归一化的覆盖量。经归一化的覆盖量允许对跨越不同样品的箱的覆盖率进行比较,其可具有不同的测量深度。它与序列剂量的不同之处在于,后者通常通过除以定位至整个基因组的亚组的标签计数来获得。该亚组是一个或多个归一化区段或染色体。覆盖量,无论是否归一化,可以针对基因组上的区域至区域的全局谱变异、g
‑
c部分变异、稳定染色体中的异常值等进行校正。
[0246]
术语
″
下一代测序(ngs)
″
在本文中是指允许克隆扩增分子和单个核酸分子的大规模平行测序的测序方法。ngs的非限制性实例包括使用可逆染料终止子的边合成边测序以及边连接边测序。
[0247]
术语
″
参数
″
在本文中是指表征系统性质的数值。通常,参数在数值上表征定量数据集和/或定量数据集之间的数值关系。例如,定位至染色体的序列标签数量与标签定位其上的染色体的长度之间的比率(或比率的函数)是参数。
[0248]
术语
″
阈值
″
和
″
合格阈值
″
在本文中是指用作表征样品诸如含有来自疑似患有医学病况的生物体的核酸的测试样品的截断的任何数量。可以将阈值与参数值进行比较,以确定产生此类参数值的样品是否表明该生物体具有医学病况。在某些实施方案中,使用合格数据集计算合格阈值,并且该合格阈值用作生物体中的拷贝数变异(如非整倍性)的诊断限值。如果通过从本文公开的方法获得的结果超过阈值,则可以将受试者诊断为具有拷贝数变异,如21三体。通过分析针对样品的训练组计算的经归一化的值(如染色体剂量、ncv或nsv)来鉴定本文所述的方法的适当阈值。可以使用训练组中的合格(即,未受影响的)样品来鉴定阈值,该训练组包括合格的(即,未受影响的)样品和受影响的样品。已知具有染色体非整倍性的训练组中的样品(即受影响的样品)可用于确认所选择的阈值可用于区分测试组中受影响的样品和未受影响的样品(参见本文的实施例)。阈值的选择取决于用户希望进行分类的置信水平。在一些实施方案中,用于鉴定适当的阈值的训练组包括至少10个、至少20个、至少30个、至少40个、至少50个、至少60个、至少70个、至少80个、至少90个、至少100个、至少200个、至少300个、至少400个、至少500个、至少600个、至少700个、至少800个、至少900个、至少1000个、至少2000个、至少3000个、至少4000个或更多个合格样品。使用更大的合格样品组来改善阈值的诊断效用可能是有利的。
[0249]
术语
″
箱
″
是指序列的区段或基因组的区段。在一些实施方案中,箱在基因组或染色体内彼此邻接。每个箱可以限定参考基因组中的核苷酸的序列。根据特定应用和序列标签密度所要求的分析,箱的尺寸可以是1kb、100kb、1mb等。除了它们在参考序列内的位置外,箱可具有其它特征,诸如样品覆盖率和序列结构特征,诸如g
‑
c分数。
[0250]
术语
″
掩蔽阈值
″
在本文中用于指代与基于序列箱中的序列标签的数量的值进行比较的量,其中掩蔽具有超过掩蔽阈值的值的箱。在一些实施方案中,掩蔽阈值可以是百分等级、绝对计数、定位质量评分或其它适合的值。在一些实施方案中,掩蔽阈值可以被定义为跨多个未受影响的样品的变异系数的百分等级。在其它实施方案中,掩蔽阈值可以被定义为定位质量分数,如mapq评分,其涉及将序列读取与参考基因组比对的可靠性。注意,掩蔽阈值不同于拷贝数变异(cnv)阈值,后者是表征含有来自疑似患有与cnv相关的医学病况的生物体的核酸的样品的截断。在一些实施方案中,相对于本文其它地方描述的归一化的染色体值(ncv)或归一化的片段值(nsv)来定义cnv阈值。
[0251]
术语“经归一化的值”在本文中是指一个数值,该数值将针对目标序列(如染色体
或染色体区段)所鉴定的序列标签的数量与针对归一化序列(如归一化染色体或归一化染色体区段)所鉴定的序列标签的数量相关联。例如,
″
经归一化的值
″
可以是如本文其它地方所述的染色体剂量,或者它可以是ncv,或者它可以是如本文其它地方所述的nsv。
[0252]
术语
″
读取
″
是指从核酸样品的一部分获得的序列。通常,尽管不是必须的,读取表示样品中连续碱基对的短序列。读取可以由样品部分的碱基对序列(以a、t、c或g)象征性地表示。它可以存储在存储设备中并且适当地处理以确定它是否与参考序列匹配或满足其它标准。可以直接从测序装置获得读取,或者间接地从关于样品的存储序列信息获得读取。在一些情况下,读取是足够长度(如,至少约25bp)的dna序列,其可以用于鉴定更大的序列或区域,如可以将其比对并特异性地指定至染色体或基因组区域或基因。
[0253]
术语
″
基因组读取
″
用于指代个体的整个基因组中任何区段的读取。
[0254]
术语
″
序列标签
″
在本文中可与术语
″
定位的序列标签
″
互换使用以指代序列读取,其通过比对被特定地分配,即定位到较大的序列(如参考基因组)。定位的序列标签被唯一地定位至参考基因组,即它们被分配到参考基因组的单个位置。除非另有说明,定位至参考序列上相同序列的标签被计数一次。标签可以作为数据结构或其它数据集合物提供。在某些实施方案中,标签含有读取序列和该读取的相关信息,诸如基因组中的序列的位置,如染色体上的位置。在某些实施方案中,该位置被指定用于正链取向。可以定义标签以允许与参考基因组比对中有限量的错配。在一些实施方案中,可以定位至参考基因组上的多于一个位置的标签,即不唯一地定位的标签,可以不包括在分析中。
[0255]
术语
″
非冗余序列标签
″
是指未定位至相同位点的序列标签,在一些实施方案中为了确定归一化的染色体值(ncv)而考虑在内。有时将多个序列读取与参考基因组上的相同位置比对,产生冗余或重复的序列标签。在一个实施方案中,为了确定ncv,将定位至相同位置的复制序列标签省略或计数作为一个
″
非冗余序列标签
″
。在一些实施方案中,对与非排除的位点比对的非冗余序列标签进行计数以产生用于确定ncv的
″
非排除的位点计数
″
(nes计数)。
[0256]
术语
″
位点
″
是指参考基因组上的唯一位置(即染色体id、染色体位置和取向)。在一些实施方案中,位点可以为序列上的残基、序列标签或区段提供位置。
[0257]
″
排除的位点
″
是在参考基因组的区域中发现的位点,为了对序列标签计数而被排除。在一些实施方案中,排除的位点存在于染色体的含有重复序列的区域中,如着丝粒和端粒中,以及多于一个染色体共有的染色体的区域中,如存在于y染色体上的也存在于x染色体上的区域。
[0258]
″
非排除的位点
″
(nes)是为了对序列标签计数而未被排除的位点。
[0259]
″
非排除的位点计数
″
(nes计数)是定位至参考基因组上的nes的序列标签的数量。在一些实施方案中,nes计数是定位至nes的非冗余序列标签的数量。在一些实施方案中,覆盖率和相关参数,诸如归一化的覆盖量、去除全局谱的覆盖量和染色体剂量是基于nes计数的。在一个实例中,染色体剂量计算为目标染色体的nes计数与归一化染色体的计数的比率。
[0260]
经归一化的染色体值(ncv)将测试样品的覆盖率与一组训练/合格样品的覆盖率相关联。在一些实施方案中,ncv基于染色体剂量。在一些实施方案中,ncv涉及测试样品中目标染色体的染色体剂量与一组合格样品中相应的染色体剂量的平均值之间的差异,并且
可以计算为:
[0261][0262]
其中和分别是一组合格样品中第j染色体剂量的估计平均值和标准偏差,并且x
ij
是观察到的测试样品i的第j染色体比(剂量)。
[0263]
在一些实施方案中,ncv可以通过将测试样品中的目标染色体的染色体剂量与相同流动池上测序的多路复用样品中的相应染色体剂量的中值相关联来
″
在执行中(on the fly)
″
如下进行计算:
[0264][0265]
其中m
j
是同一流动池上测序的一组多路复用样品中第j染色体剂量的估计中值;是一个或多个流动池上测序的一组或多组多路复用样品中的第j染色体剂量的标准偏差,并且x
ij
是观察到的测试样品i的第j染色体剂量。在该实施方案中,测试样品i是在测定m
j
的相同流动池上测序的多路复用样品之一。
[0266]
例如,对于测试样品a中的目标21号染色体,作为在一个流动池上的64个多路复用样品之一进行测序,测试样品a中的21号染色体的ncv被计算为样品a中21号染色体的剂量减去在64个多路复用样品中测定的21号染色体的剂量的中值,除以在流动池1或另外的流动池上64个多路复用样品确定的21号染色体的剂量的标准偏差。
[0267]
如本文所用,术语
″
比对的(aligned)
″
、
″
比对(alignment)
″
或
″
比对(aligning)
″
是指将读取或标签与参考序列进行比较从而确定参考序列是否含有读取序列的过程。如果参考序列含有读取,则读取可以定位至参考序列,或者在某些实施方案中,定位至参考序列中的特定位置。在一些情况下,比对简单地告知读取是否是特定参考序列的成员(即,读取是否存在于参考序列中)。例如,读取与人13号染色体的参考序列的比对将判断读取是否存在于13号染色体的参考序列中。提供此信息的工具可称为集合成员测试器。在一些情况下,比对另外地表示读取或标签定位至的参考序列中的位置。例如,如果参考序列是整个人基因组序列,则比对可以表示读取存在于13号染色体上,并且还可以指示读取位于13号染色体的特定链和/或位点上。
[0268]
比对的读取或标签是被鉴定为其核酸分子顺序与来自参考基因组的已知序列顺序匹配的一条或多条序列。尽管可以手动完成比对,但通常通过计算机算法实现,因为在实现本文公开的方法的合理时间段内不可能比对读取。来自比对序列的算法的一个实例是作为illumina genomics analysis流水线的一部分分布的核苷酸数据有效局部比对(efficient local alignment of nucleotide data,eland)计算机程序。或者,布隆过滤器或类似的集合成员测试器可用于将读取与参考基因组比对。参见于2011年10月27日提交的美国专利申请no.61/552,374,其通过引用整体并入本文。序列读取在比对中的匹配可以是100%序列匹配或小于100%(非完美匹配)。
[0269]
本文中所用的术语
″
定位
″
是指通过比对将序列读取特定地分配给更大的序列(如参考基因组)。
[0270]
如本文所用,术语
″
参考基因组
″
或
″
参考序列
″
是指无论是部分的还是完整的任何
特定的已知基因组序列,所述任何生物或病毒的基因组序列可用于从受试者中引用所鉴定的序列。例如,用于人类受试者的参考基因组以及许多其它生物体可在国家生物技术中心信息(national center for biotechnology information)中找到,网址为ncbi.nlm.nih.gov。
″
基因组
″
是指核酸序列中表达的生物体或病毒的完整遗传信息。
[0271]
在某些实施方案中,参考序列明显大于与其比对的读取。例如,它可以是大至少约100倍、或大至少约1000倍,或大至少约10,000倍,或大至少约105倍,或大至少约106倍,或大至少约107倍。
[0272]
在一个实例中,参考序列是全长人基因组的参考序列。此类序列可以被称为基因组参考序列。在另一个实例中,参考序列限于特定的人染色体诸如13号染色体。在一些实施方案中,参考y染色体是来自人基因组hgl9型的y染色体序列。此类序列可以被称为染色体参考序列。参考序列的其它实例包括其它物种的基因组,以及任何物种的染色体、亚染色体区域(诸如链)等。
[0273]
在某些实施方案中,参考序列是来源于多个个体的共有序列或其它组合。然而,在某些申请中,参考序列可以取自特定的个体。
[0274]
术语
″
临床上相关的序列
″
在本文中是指已知或疑似与遗传或疾病状况相关或涉及遗传或疾病状况的核酸序列。确定临床上相关的序列的缺失或存在可用于确定诊断医学病况或确认医学病况的诊断,或提供疾病发展的预后。
[0275]
当在核酸或核酸混合物的上下文中使用时,术语
″
衍生的
″
在本文中是指从其来源的来源获得一种或多种核酸的手段。例如,在一个实施方案中,来源于两个不同的基因组的核酸的混合物意味着核酸(如cfdna)是由细胞通过天然存在的过程诸如坏死或凋亡而自然释放的。在另一个实施方案中,来源于两个不同的基因组的核酸的混合物意味着核酸是从来自受试者的两种不同类型的细胞中提取的。
[0276]
当在获得特定定量值的上下文中使用时,术语
″
基于
″
在本文中是指使用另一个量作为输入来计算特定定量值作为输出。
[0277]
术语
″
患者样品
″
在本文中是指从患者(即医疗看护、护理或治疗的接收者)获得的生物样品。患者样品可以是本文描述的任何样品。在某些实施方案中,患者样品是通过非侵入性程序,如外周血液样品或粪便样品获得的。本文描述的方法不限于人。因此,考虑了各种兽医应用,在此类情况下,患者样品可以是来自非人哺乳动物(如猫科动物、猪科动物、马科动物、牛科动物等)的样品。
[0278]
术语
″
混合样品
″
在本文中是指含有来源于不同的基因组的核酸的混合物的样品。
[0279]
术语
″
母体样品
″
在本文中是指从妊娠受试者、如女性获得的生物样品。
[0280]
术语
″
生物体液
″
在本文中是指取自生物来源的液体,并且包括例如血液、血清、血浆、痰液、灌洗液、脑脊液、尿液、精液、汗液、泪液、唾液等。如本文所用,术语
″
血液
″
、
″
血浆
″
和
″
血清
″
明确涵盖其分级或其加工部分。类似地,如果从活组织检查、拭子、涂片等取样,
″
样品
″
明确地涵盖来源于活组织检查、拭子、涂片等的加工级分或部分。
[0281]
术语
″
母体核酸
″
和
″
胎儿核酸
″
在本文中分别是指妊娠女性受试者的核酸和由妊娠女性携带的胎儿的核酸。
[0282]
如本文所用,术语
″
对应于
″
有时是指核酸序列,如基因或染色体,其存在于不同受试者的基因组中并且在所有基因组中不一定具有相同的序列,但用于提供目标序列(如基
因或染色体)的身份而不是遗传信息。
[0283]
如本文所用,术语
″
胎儿分数
″
是指含有胎儿和母体核酸的样品中存在的胎儿核酸的分数。胎儿分数通常用于表征母亲血液中的cfdna。
[0284]
如本文所用,术语
″
染色体
″
是指活细胞的携带遗传性的基因载体,其来源于包含dna和蛋白组分(尤其是组蛋白)的染色质链。本文采用传统的国际认可的单独人基因组染色体编号系统。
[0285]
如本文所用,术语
″
多核苷酸长度
″
是指参考基因组的序列或区域中核苷酸的绝对数量。术语
″
染色体长度
″
是指参考基因组的序列或区域中核苷酸的绝对数量。术语
″
染色体长度是指以碱基对给出的染色体的已知长度,如提供于万维网上的|genome|.|ucsc|.|edu/cgi
‑
bin/hgtracks?hgsid=167155613&chrominfopage=找到的人染色体的ncbi36/hgi8组装中。
[0286]
术语
″
受试者
″
在本文中是指人受试者以及非人受试者,诸如哺乳动物、无脊椎动物、脊椎动物、真菌、酵母、细菌和病毒。尽管本文的实例涉及人并且文字主要涉及人类关注的,但本文公开的概念适用于来自任何植物或动物的基因组,并且可用于兽医学、动物科学、研究实验室等领域。
[0287]
术语
″
病况
″
在本文中是指
″
医学状况
″
,作为广义术语,包括所有疾病和病症,但可包括可能影响个人健康、从医疗援助中受益或有对医学治疗的影响的损伤和正常健康情况,诸如妊娠。
[0288]
术语
″
完整
″
用于指染色体非整倍性时,在本文中是指整个染色体的获得或丢失。
[0289]
术语
″
部分
″
当用于指染色体非整倍性时,在本文中是指染色体的一部分即区段的获得或丢失。
[0290]
术语
″
镶嵌性
″
在本文中是指表示在一个从单个受精卵发育的个体中存在两个具有不同核型的细胞群。镶嵌现象可能是由于只传播到成体细胞的一个亚组的发育过程中的突变造成的。
[0291]
术语
″
非镶嵌性
″
在本文中是指由一个核型的细胞组成的生物体,如人胎儿。
[0292]
如本文所用的术语
″
灵敏度
″
是指当存在目标病况时测试结果为阳性的概率。它可以计算为真阳性的数量除以真阳性和假阴性的总和。
[0293]
如本文所用的术语
″
特异性
″
是指当不存在目标病况时测试结果将是阴性的概率。它可以计算为真阴性的数量除以真阴性和假阳性的总和。
[0294]
术语
″
富集
″
在本文中是指扩增包含在母体样品的一部分中的多态性靶核酸并将扩增产物与除去该部分的母体样品的其余部分合并的过程。例如,母体样品的其余部分可以是原始的母体样品。
[0295]
术语
″
原始的母体样品
″
在本文中是指从用作来源的妊娠受试者(如女性)获得的非富集生物样品,从其中移除一部分以扩增多态性靶核酸。
″
原始样品
″
可以是从妊娠受试者获得的任何样品,以及其处理的级分,例如,从母体血浆样品中提取的经纯化的cfdna样品。
[0296]
如本文所用的术语
″
引物
″
是指经分离的寡核苷酸,当置于诱导延伸产物合成的条件下时,它能作为合成的起始点(如,该条件包括核苷酸、诱导剂诸如dna聚合酶以及适合的温度和ph)。引物优选是用于最大扩增效率的单链,但可以可替代地是双链的。如果是双链
的,则在用于制备延伸产物之前,首先处理引物以分离其链。优选地,引物是寡脱氧核糖核苷酸。引物必须足够长,以在诱导剂存在下引发延伸产物的合成。引物的确切长度将取决于许多因素,包括温度、引物来源、方法的使用以及用于引物设计的参数。
[0297]
引言和背景
[0298]
人基因组中的cnv显著影响人类多样性和对疾病的易感性(redon等人,nature23:444
‑
454[2006],shaikh等人genome res 19:1682
‑
1690[2009])。此类疾病包括但不限于癌症、感染性和自身免疫性疾病、神经系统疾病、代谢和/或心血管疾病等。
[0299]
已知cnv通过不同的机制促成遗传疾病,导致在大多数情况下基因剂量的不平衡或基因破坏。除了其与遗传病症的直接相关性之外,已知cnv介导可能有害的表型变化。最近,一些研究报道,与正常对照相比,复杂病症诸如自闭症、adhd和精神分裂症中的罕见或从头cnv的负担增加,突出了罕见或独特cnv的潜在致病性(sebat等人,316:445
‑
449[2007];walsh等人,science 320:539
‑
543[2008])。cnv起源于基因组重排,主要是由于缺失、重复、插入和不平衡的易位事件。
[0300]
已经表明,胎儿来源的cfdna片段平均比母体来源的那些片段短。基于ngs数据的nipt(非侵入性产前检测)已成功实施。目前的方法涉及使用短读取(25bp
‑
36bp)对母体样品进行测序,与基因组比对,相较于与正常二倍体基因组相关的预期归一化覆盖率,计算和归一化亚染色体覆盖率,并最终评估靶染色体(13/18/21/x/y)的过表达。因此,传统的nipt测定和分析依赖于计数或覆盖率来评估胎儿非整倍性的可能性。
[0301]
由于母体血浆样品代表母体和胎儿cfdna的混合物,任何给定的nipt方法的成功取决于其检测低胎儿分子样品中的拷贝数变化的灵敏度。对于基于计数的方法,它们的灵敏度由以下确定:(a)测量深度,以及(b)数据归一化以减少技术差异的能力。本公开通过从配对末端读取推导出片段尺寸信息并在分析流水线中使用该信息来提供nipt和其它应用的分析方法。提高的分析灵敏度提供了以降低的覆盖率(如,减少的测量深度)应用nipt方法的能力,这使得该技术能够用于进行平均风险妊娠的较低成本测试。
[0302]
本文公开了用于在测试样品中确定不同的目标序列的拷贝数和拷贝数变异(cnv)的方法、装置和系统,所述测试样品包含来源于两个或更多个不同基因组的核酸的混合物,并且已知或疑似与一条或多条目标序列的数量不同。由本文公开的方法和装置确定的拷贝数变异包括整个染色体的获得或丢失,涉及非常大的染色体区段的微观可见的变化,以及dna区段的大量亚微观拷贝数变异,尺寸范围从单个核苷酸至数千碱基(kb)至数兆碱基(mb)。
[0303]
在一些实施方案中,提供了使用含有母体和胎儿无细胞dna的母体样品来确定胎儿的拷贝数变异(cnv)的方法。一些实施方式使用cfdna的片段长度(或片段尺寸)来提高母体血浆中cfdna的胎儿非整倍性检测的灵敏度和特异性。一些实施方案是用无pcr文库制备结合配对末端dna测序实现的。在一些实施方案中,利用片段尺寸和覆盖率来增强胎儿非整倍性检测。在一些实施方案中,该方法涉及将较短片段的独立计数与基因组上箱中较短片段的相对分数组合。
[0304]
本文公开的一些实施方案提供了通过去除样品内gc
‑
含量偏差来提高序列数据分析的灵敏度和/或特异性的方法。在一些实施方案中,去除样品内gc偏差是基于针对未受影响的训练样品中常见的系统变异校正的序列数据。
[0305]
公开的一些实施方案提供从无细胞核酸片段获得具有高信噪比的参数,用于确定与拷贝数和cnv相关的各种遗传条件的方法,其相对于常规方法具有提高的灵敏度、选择性和/或效率。参数包括但不限于覆盖率、片段尺寸加权覆盖率、限定范围内的片段的分数或比率、片段的甲基化水平、从覆盖率获得的t
‑
统计量、从覆盖率信息获得的胎儿分数估值等。已经发现所描述的方法在改善具有来自所考虑的基因组(例如,胎儿的基因组)的相对低分数的dna的样品中的信号方面特别有效。此类样品的实例是来自怀有异卵双胞胎、三胞胎等的个体的母体血液样品,其中该方法评估其中一个胎儿的基因组中的拷贝数变异。
[0306]
在一些实施方案中,可以使用非常低的cfdna输入(不需要pcr扩增)以简单文库制备来实现高分析灵敏度和特异性。无pcr方法简化了工作流程,提高了周转时间并消除了pcr方法固有的偏差。在一些实施方案中,从母体血浆检测胎儿非整倍性可以比常规方法更稳定和有效,需要更少的独特cfdna片段。结合起来,在显著较低的cfdna片段数量下,以非常快的周转时间实现了提高的分析灵敏度和特异性。这可能使nipt以显著更低的成本进行,以便于在一般产科人群中应用。
[0307]
在各种实施方式中,利用所公开的方法可能实现无pcr文库制备。一些实施方式消除了pcr方法的固有偏差,降低了测量复杂度,减少了所需的测量深度(低了2.5倍),提供了更快的周转时间(如在一天内周转),使得能够进行过程中的胎儿分数(ff)测量,便于使用片段尺寸信息区分母体和胎儿/胎盘cfdna。
[0308]
评估cnv
[0309]
用于确定cnv的方法
[0310]
使用由本文公开的方法提供的序列覆盖率值、片段尺寸参数和/或甲基化水平,可以确定与序列、染色体或染色体区段的拷贝数和cnv相关的各种遗传病况,相对于使用通过常规方法获得的序列覆盖率值,其具有提高的灵敏度、选择性和/或效率。例如,在一些实施方案中,掩蔽的参考序列用于确定包含胎儿和母体核酸分子的母体测试样品中存在或缺失任何两种或更多种不同的完整胎儿染色体非整倍性。下面提供的示例性方法将读取与参考序列(包括参考基因组)进行比对。比对可以在未掩蔽或掩蔽的参考序列上进行,从而产生定位至参考序列的序列标签。在一些实施方案中,只考虑落在参考序列的未掩蔽区段上的序列标签以确定拷贝数变异。
[0311]
在一些实施方案中,评估cnv的核酸样品涉及通过三种类型的调用之一来表征染色体或区段非整倍性的状态:
″
正常的
″
或
″
未受影响的
″
,
″
受影响的
″
和
″
无调用的
″
。用于调用正常和受影响的阈值是典型的设置。在样品中测量与非整倍性或其它拷贝数变异相关的参数,并将测量值与阈值进行比较。对于重复型非整倍体,如果染色体或区段剂量(或其它测量值序列含量)高于为受影响的样品设定的限定阈值,则进行受影响的调用。对于此类非整倍体,如果染色体或区段剂量低于为正常样品设定的阈值,则产生正常的调用。相比之下,对于缺失型非整倍性,如果染色体或区段剂量低于受影响的样品的限定阈值,则产生受影响的调用,并且如果染色体或区段剂量高于为正常的样品设定的阈值,则进行正常的调用。例如,在三体存在的情况下,
″
正常的
″
调用由参数值确定,如低于用户定义的可靠性阈值的测试染色体剂量,并且
″
受影响的
″
调用由参数确定,如高于用户定义的可靠性阈值的测试染色体剂量。
″
无调用
″
结果由参数确定,如位于用于产生
″
正常的
″
或
″
受影响的
″
调用的阈值之间的测试染色体剂量。术语
″
无调用的
″
与
″
未分类的
″
可互换使用。
[0312]
可用于确定cnv的参数包括但不限于覆盖率、片段尺寸偏差/加权的覆盖率、片段在限定的尺寸范围内的分数或比率、以及片段的甲基化水平。如本文所讨论,从与参考基因组的区域比对的读取的计数获得覆盖率,并且任选地归一化以产生序列标签计数。在一些实施方案中,序列标签计数可以通过片段尺寸来加权。
[0313]
在一些实施方案中,片段尺寸参数偏向于基因组之一的片段尺寸特征。片段尺寸参数是涉及片段尺寸的参数。在下列情况下,参数偏向片段尺寸:1)参数对于片段尺寸有利地加权,如,对于该尺寸加权的计数比对其它尺寸更重;或者2)参数是从对于片段尺寸有利地加权的值获得的,如从对于尺寸加权更重的计数获得的比率。当基因组相对于另一基因组或相同基因组的另一部分具有富集或更高浓度的该尺寸的核酸时,尺寸是基因组的特征。
[0314]
在一些实施方案中,用于确定母体测试样品中存在或缺失任何完整胎儿染色体非整倍性的方法包括:(a)获得母体测试样品中胎儿和母体核酸的序列信息;(b)使用上述序列信息和方法鉴定选自1
‑
22号染色体、x染色体和y染色体的每个目标染色体的序列标签数量、序列覆盖量、片段尺寸参数或另一参数,以鉴定一条或多条归一化染色体序列的序列标签数量或另一参数;(c)使用为每个目标染色体鉴定的序列标签数量或其它参数,以及为每个归一化染色体鉴定的序列标签数量或其它参数,来计算每个目标染色体的单个染色体剂量;以及(d)将每种染色剂剂量与阈值进行比较,从而确定母体测试样品中存在或缺失任何完整的胎儿染色体非整倍性。
[0315]
在一些实施方案中,上述步骤(a)可包括对测试样品的核酸分子的至少一部分进行测序,以获得测试样品的胎儿和母体核酸分子的所述序列信息。在一些实施方案中,步骤(c)包括计算每个目标染色体的单个染色体剂量,作为对于每个目标染色体所鉴定的序列标签数量或其它参数和对一条或多条归一化染色体序列所鉴定的序列标签数量或其它参数的比率。在一些其它实施方案中,染色体剂量基于来源于序列标签数量或另一参数的经处理的序列覆盖量。在一些实施方案中,仅使用唯一的非冗余的序列标签来计算处理的序列覆盖量或另一参数。在一些实施方案中,经处理的序列覆盖量是序列标签密度比,它是由序列长度归一化的序列标签密度数量。在一些实施方案中,经处理的序列覆盖量或其它参数是归一化的序列标签或另一归一化的参数,其是目标序列的序列标签数量或其它参数除以所有或大部分的基因组的序列标签数量或其它参数。在一些实施方案中,根据目标序列的全局谱来调整经处理的序列覆盖量或其它参数诸如片段尺寸参数。在一些实施方案中,根据所测试的样品的gc含量和序列覆盖率之间的样品间相关性来调整经处理的序列覆盖量或其它参数。在一些实施方案中,经处理的序列覆盖量或其它参数由这些方法组合产生,所述方法在本文其它地方进一步描述。
[0316]
在一些实施方案中,染色体剂量被计算为每个目标染色体与归一化染色体序列的经处理的序列覆盖率或其它参数的比率。
[0317]
在上述实施方案中的任一个中,完全染色体非整倍性选自完全染色体三体性、完全染色体单体性和完全染色体多体性。完全染色体非整倍性选自1
‑
22号染色体、x染色体和y染色体中的任一种的完全非整倍体。例如,所述不同的完全胎儿染色体非整倍性选自三体2、三体8、三体9、三体20、21三体、13三体、三体16、18三体、三体22、47,xxx、47,xyy和x单体。
[0318]
在上述实施方案中的任一个中,对来自不同母体受试者的测试样品重复步骤(a)
‑
(d),并且该方法包括确定每个测试样品中任两种或更多种不同的完全胎儿染色体非整倍性的存在或缺失。
[0319]
在上述实施方案中的任一个中,该方法还可以包括如下计算归一化的染色体值(ncv),其中ncv将染色体剂量与一组合格样品中相应的染色体剂量的平均值相关联:
[0320][0321]
其中和分别是一组合格样品中第j染色体剂量的估计平均值和标准偏差,并且x
ij
是观察到的测试样品i的第j染色体剂量。
[0322]
ncv可以通过将测试样品中的目标染色体的染色体剂量与相同流动池上测序的多路复用样品中的相应染色体剂量的中值相关联来
″
在执行中
″
如下进行计算:
[0323][0324]
其中m
j
是同一流动池上测序的一组多路复用样品中第j染色体剂量的估计中值;是一个或多个流动池上测序的一组或多组多路复用样品中的第j染色体剂量的标准偏差,并且x
i
是观察到的测试样品i的第j染色体剂量。在该实施方案中,测试样品i是在测定m
j
的相同流动池上测序的多路复用样品之一。
[0325]
在一些实施方案中,提供了用于确定包含胎儿和母体核酸的母体测试样品中存在或缺失不同部分胎儿染色体非整倍性的方法。该方法涉及类似于如上所述的用于检测完全非整倍性的方法的程序。然而,不是分析完整的染色体,而是分析染色体的区段。参见美国专利申请公开no.2013/0029852,其通过引用并入。
[0326]
图1显示了用于根据一些实施方案确定拷贝数变异的存在的方法。图1所示的方法100使用基于序列标签数量(即,序列标签计数)的序列标签覆盖率来确定cnv。然而,类似于上面用于计算ncv的描述,可以使用其它变量或参数,诸如尺寸、尺寸比和甲基化水平来代替覆盖率。在一些实施方式中,两个或更多个变量组合以确定cnv。此外,覆盖率和其它参数可以基于从其获得标签的片段的尺寸来加权。为了便于读取,在图1所示的方法100中仅涉及覆盖率,但应注意其它参数,诸如尺寸、尺寸比和甲基化水平、按尺寸加权的计数等,可以用来代替覆盖率。
[0327]
在操作130和135中,确定合格序列标签覆盖率(或另一参数的值)和测试序列标签覆盖率(或另一参数的值)。本公开提供了确定相对于常见方法提供提高的灵敏度和选择性的覆盖量的方法。操作130和135由星号标记并由粗线框突出显示,以指示这些操作有助于改进现有技术。在一些实施方案中,将序列标签覆盖量归一化、调整、修整和以其它方式处理,以提高分析的灵敏度和选择性。这些方法在本文其它地方进一步描述。
[0328]
从全局角度来看,该方法利用合格训练样品的归一化序列来确定测试样品的cnv。在一些实施方案中,合格训练样品不受影响且具有正常的拷贝数。归一化序列提供一种机制来归一化测量运行内和运行间的变异性。将归一化序列用来自一组合格样品的序列信息进行鉴定,所述样品获自已知包含对任何一条目标序列(如染色体或其区段)具有正常拷贝数的细胞的受试者。在图1中描绘的方法的实施方案的步骤110、120、130、145和146中概述了归一化序列的确定。在一些实施方案中,归一化序列用于计算测试序列的序列剂量。参见
步骤150。在一些实施方案中,归一化序列也用于计算针对其比较测试序列的序列剂量的阈值。参见步骤150。从归一化序列和测试序列获得的序列信息用于确定测试样品中染色体非整倍性的有统计学意义的鉴定(步骤160)。
[0329]
转到用于根据一些实施方案确定拷贝数变异的存在的方法的细节,图1提供了用于确定生物样品中的目标序列(如,染色体或其区域)的cnv的实施方案的流程图100。在一些实施方案中,生物样品是从受试者获得的并且包含由不同基因组贡献的核酸的混合物。不同的基因组可以由两个个体贡献给样品,如不同的基因组由胎儿和携带胎儿的母亲贡献。此外,不同的基因组可以由三个或更多个体贡献给样品,如不同的基因组由两个或更多个胎儿和携带胎儿的母亲贡献。可替代地,基因组由来自同一受试者的非整倍体癌细胞和正常整倍体细胞贡献给样品,如来自癌症患者的血浆样品。
[0330]
除了分析患者的测试样品之外,为每个可能的目标染色体选择一个或多个归一化染色体或者一个或多个归一化染色体区段。鉴定归一化染色体或区段与患者样品的正常测试不同时,这可能在临床环境中发生。换言之,在测试患者样品之前鉴定归一化染色体或区段。归一化染色体或区段与目标染色体或区段之间的关联性被存储以供测试期间使用。如下面所解释,此类关联性通常在跨越多个样品的测试的时间段内保持。以下讨论涉及针对各个目标染色体或区段选择归一化染色体或染色体区段的实施方案。
[0331]
获得一组合格样品以鉴定合格的归一化序列,并提供方差值用于确定测试样品中cnv的有统计意义的鉴定。在步骤110中,从已知包括具有任一个目标序列的正常拷贝数的细胞的多个受试者中获得多个生物合格样品。在一个实施方案中,合格样品是从怀有胎儿的母亲获得的,该胎儿已经使用细胞遗传学方法确认具有正常拷贝数的染色体。生物合格样品可以是生物流体,如血浆或如下所述的任何适合的样品。在一些实施方案中,合格样品含有核酸分子(如cfdna分子)的混合物。在一些实施方案中,合格样品是母体血浆样品,其含有胎儿和母体cfdna分子的混合物。使用任何已知的测序方法,通过对至少一部分核酸,如胎儿和母体核酸进行测序来获得归一化染色体和/或其区段的序列信息。优选地,本文其它地方描述的下一代测序(ngs)方法中的任何一种用于将胎儿和母体核酸序列测序为单个或克隆扩增的分子。在某些实施方案中,在测序之前和期间,如下所公开,处理合格样品。它们可以使用如本文公开的装置、系统和试剂盒处理。
[0332]
在步骤120中,对合格样品中包含的所有合格核酸中的每一个的至少一部分进行测序,以产生数百万次序列读取,如36bp读取,将其与参考基因组(如hgl8)比对。在一些实施方案中,序列读取包含约20bp、约25bp、约30bp、约35bp、约40bp、约45bp、约50bp、约55bp、约60bp、约65bp、约70bp、约75bp、约80bp、约85bp、约90bp、约95bp、约l00 bp、约110bp、约120bp、约130、约140bp、约150bp、约200bp、约250bp、约300bp、约350bp、约400bp、约450bp或约500bp。预期技术进步将使得单末端读取大于500bp,在生成配对末端读取时使得能够实现大于约1000bp的读取。在一个实施方案中,定位的序列读取包含36bp。在另一个实施方案中,定位的序列读取包含25bp。
[0333]
将序列读取与参考基因组比对,并且唯一定位至参考基因组的读取被称为序列标签。落入掩蔽的参考序列的掩蔽区段上的序列标签不计入cnv的分析。
[0334]
在一个实施方案中,从唯一定位至参考基因组的读取获得包含20至40bp读取的至少约3
×
106个合格序列标签、至少约5
×
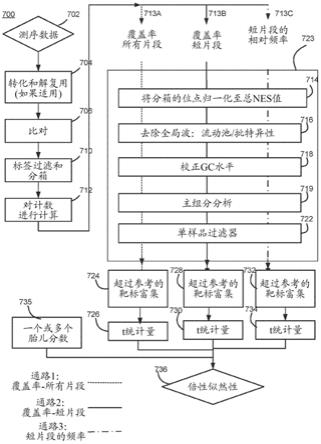
106个合格序列标签、至少约8
×
106个合格序列标
签、至少约10
×
106个合格序列标签、至少约15
×
106个合格序列标签、至少约20
×
106个合格序列标签、至少约30
×
106个合格序列标签、至少约40
×
106个合格序列标签或至少约50
×
106个合格序列标签。
[0335]
在步骤130中,对从合格样品中的核酸测序获得的所有标签进行计数,以获得合格的序列标签覆盖率。类似地,在操作135中,对从测量样品获得的所有标签进行计数以获得测试序列标签覆盖率。本公开提供了确定提供相对于常规方法提高的灵敏度和选择性的覆盖量的方法。操作130和135由星号标记并由粗线框突出显示,以指示这些操作有助于改进现有技术。在一些实施方案中,将序列标签覆盖量归一化、调整、修整和以其它方式处理,以提高分析的灵敏度和选择性。这些方法在本文其它地方进一步描述。
[0336]
由于所有合格序列标签都在每个合格的样品中被定位和计数,因此确定了合格样品中的目标序列(如临床相关序列)的序列标签覆盖率,也确定了随后从其鉴定出归一化序列的另外序列的序列标签覆盖率。
[0337]
在一些实施方案中,目标序列是与完全染色体非整倍性(如21号染色体)相关的染色体,并且合格的归一化序列是完整染色体,所述完整染色体与染色体非整倍性无关并且其序列标签覆盖率的变异近似于目标序列(即染色体)如21号染色体的变异。所选的一个或多个染色体可以是最近似目标序列的序列标签覆盖率的变异的一个或一组。1
‑
22号染色体、x染色体和y染色体中的任一种或多种可以是目标序列,并且一种或多种染色体可以被鉴定为合格样品中的任一种1
‑
22号染色体、x染色体和y染色体中的每一种的归一化序列。归一化染色体可以是单独的染色体,或者它可以是如本文其它地方所述的一组染色体。
[0338]
在另一个实施方案中,目标序列是与部分非整倍性(如染色体缺失或插入,或不平衡的染色体易位)相关的染色体的区段,并且归一化序列是染色体区段(或区段组),所述染色体区段(或区段组)与部分非整倍性无关并且其序列标签覆盖率的变异近似于与部分非整倍性相关的染色体区段的变异。所选的一个或多个归一化染色体区段可以是最近似目标序列的序列标签覆盖率中的变异的一个或多个。任一种或多种染色体1
‑
22、x和y中的任一个或多个区段可以是目标序列。
[0339]
在其它实施方案中,目标序列是与部分非整倍性相关的染色体的区段,并且归一化序列是一个或多个整个染色体。在其它实施方案中,目标序列是与非整倍性相关的整个染色体,并且归一化序列是与非整倍性无关的一个或多个染色体区段。
[0340]
无论单条序列还是一组序列在合格的样品中被鉴定为任何一条或多条目标序列的归一化序列,可以选择合格的归一化序列以具有序列标签覆盖率的变异或最佳或有效地近似于如在合格样品中确定的目标序列的片段尺寸参数。例如,合格的归一化序列是当用于归一化目标序列时产生跨合格的样品的最小变异性的序列,即归一化序列的变异性最接近合格样品中确定的目标序列的变异性。换言之,合格的将归一化序列是经选择以产生跨合格样品的序列剂量(对于目标序列)的最小变异的序列。因此,该过程选择一条当用作归一化染色体时预期产生目标序列的连续运行(run
‑
to
‑
run)染色体剂量的最小变异性的序列。
[0341]
对于任一条或多条目标序列,在合格样品中鉴定的归一化序列仍然是用于确定数日、数周、数月和可能数年内测试样品中存在或缺失非整倍性的所选择的归一化序列,前提是需要生成测序文库所需的程序并且对样品进行测序随着时间的推移基本上没有改变。如
上所述,选择用于确定非整倍性存在的归一化序列用于(可能还有其它原因)在序列标签数量或片段尺寸参数值的变异性,将其在样品(如,不同样品)和测序运行(如,在同一天和/或不同天发生的测序运行)之间定位其上,所述变异性最近似其用作归一化参数的目标序列的变异性。这些过程的显著改变将影响定位至所有序列的标签的数量,这继而将确定哪一个或一组序列将具有在同一天或不同天在相同的和/或不同的测序运行中的跨样品的变异性,所述变异性最接近于目标序列的变异性,这将要求重新确定归一化序列组。程序的显著改变包括用于制备测序文库的实验室方案的变化,其包括与为多重测序而不是单重测序制备样品相关的变化以及测序平台的变化,其包括用于测序的化学变化。
[0342]
在一个实施方案中,选择用于归一化特定目标序列的归一化序列是最好地将一个或多个合格样品与一个或多个受影响的样品区分开的序列,这意味着归一化序列是具有最大可区分性的序列,即归一化序列的可区分性是使得它为受影响的测试样品中的目标序列提供最佳分化,以容易地区分开受影响的测试样品与其它未受影响的样品。在其它实施方案中,归一化序列是具有最小变异性和最大可区分性的组合。
[0343]
如下所述并且如实施例所示,可以将可区分性水平测定为合格样品群中的序列剂量(如染色体剂量或区段剂量)与一个或多个测试样品中的一个或多个染色体剂量之间的统计差异。例如,可区分性可以用数字表示为t
‑
检验值,其表示合格样品群中的染色体剂量与一个或多个测试样品中的一个或多个染色体剂量之间的统计差异。类似地,可区分性可以基于区段剂量而不是染色体剂量。可替代地,可区分性可以用数字表示为归一化的染色体值(ncv),只要ncv的分布正常,它就是染色体剂量的z
‑
评分。类似地,在染色体区段是目标序列的情况下,区段剂量的可区分性可以用数字表示为归一化的区段值(nsv),只要ncv的分布正常,它就是染色体区段剂量的z
‑
评分。在确定z
‑
评分时,可以使用一组合格样品中染色体或区段剂量的平均值和标准偏差。可替代地,可以使用包含合格样品和受影响样品的训练组中染色体或区段剂量的平均值和标准偏差。在其它实施方案中,归一化序列是具有最小变异性和最大可区分性或小变异性和大可区分性的最佳组合的序列。
[0344]
该方法鉴定下述序列,其本身具有相似的特征并且在样品和测序运行之间易于发生类似的变化,并且可用于确定测试样品中的序列剂量。
[0345]
序列剂量的确定
[0346]
在一些实施方案中,如图1所示的步骤146中所述,在所有合格样品中确定一个或多个目标染色体或区段的染色体或区段剂量,并且归一化染色体或区段序列是在步骤145中鉴定的。在计算序列剂量之前提供一些归一化序列。然后根据如下进一步描述的各种标准来鉴定一条或多条归一化序列,参见步骤145。在一些实施方案中,如经鉴定的归一化序列导致跨所有合格样品的目标序列的序列剂量的最小变异性。
[0347]
在步骤146中,基于计算的合格标签密度,将目标序列的合格序列剂量,即染色体剂量或区段剂量确定为目标序列的序列标签覆盖率与另外序列的合格序列标签覆盖率的比率,随后在步骤145中从其鉴定归一化序列。随后使用经鉴定的归一化序列来确定测试样品中的序列剂量。
[0348]
在一个实施方案中,合格样品中的序列剂量是染色体剂量,其计算为合格样品中的目标染色体的序列标签数量或片段尺寸参数与归一化染色体序列的序列标签数量的比率。归一化染色体序列可以是单个染色体、一组染色体、一个染色体的区段或一组来自不同
染色体的区段。因此,目标染色体的染色体剂量在合格的样品中确定为目标染色体的标签数量与以下序列的标签数量的比率:(i)由单个染色体组成的归一化染色体序列,(ii)由两个或更多个染色体组成的归一化染色体序列,(iii)由染色体的单个区段组成的归一化区段序列,(iv)由来自一个染色体的两个或更多个区段组成的归一化区段序列,或(v)由两个或更多个染色体的两个或更多个区段组成的归一化区段序列。根据(i)
‑
(v)确定目标21号染色体的染色剂剂量的实例如下:目标染色体(如21号染色体)的染色体剂量被确定为21号染色体的序列标签覆盖率与以下序列标签覆盖率之一的比率:(i)所有剩余的染色体中的每一个,即1
‑
20号染色体、22号染色体、x染色体和y染色体;(ii)两个或更多个剩余染色体的所有可能组合;(iii)另一个染色体如9号染色体的区段;(iv)另一个染色体的两个区段,如,9号染色体的两个区段;(v)两个不同染色体的两个区段,如9号染色体的区段和14号染色体的区段。
[0349]
在另一个实施方案中,合格样品中的序列剂量是与染色体剂量相反的区段剂量,所述区段剂量被计算为合格样品中的目标区段(即并非整个染色体)的序列标签数量与归一化区段序列的序列标签数量的比率。归一化区段序列可以是上面讨论的归一化染色体或区段序列中的任一种。
[0350]
归一化序列的鉴定
[0351]
在步骤145中,为目标序列鉴定归一化序列。在一些实施方案中,如,归一化序列是基于计算的序列剂量的序列,如导致跨所有合格的训练样品的目标序列的序列剂量的最小变异性。该方法鉴定序列,所述序列本身具有相似的特征并且在样品和测试运行之间易于发生类似的变异并且可用于确定测试样品中的序列剂量。
[0352]
用于一条或多条目标序列的归一化序列可以在一组合格样品中进行鉴定,并且随后使用在合格样品中鉴定的序列来计算每个测试样品中的一条或多条目标序列的序列剂量(步骤150)以确定每种测试样品中存在或缺失非整倍性。当使用不同的测序平台时和/或当待测序的核酸的纯化和/或测序文库的制备中存在差异时,对于目标染色体或区段鉴定的归一化序列可能不同。使用根据本文描述的方法的归一化序列提供了染色体或其区段的拷贝数的变异的特异性和灵敏性度量,而与所使用的样品制备和/ 或测量平台无关。
[0353]
在一些实施方案中,鉴别多于一个归一化序列,即可以针对一条目标序列确定不同的归一化序列,并且可以针对一条目标序列确定多个序列剂量。例如,当使用14号染色体的序列标签覆盖率时,目标21号染色体的染色体剂量的变化,如变异系数(cv=标准偏差/平均值)是最小的。然后,可以鉴定两条、三条、四条、五条、六条、七条、八条或更多条归一化序列以用于确定测试样品中目标序列的序列剂量。例如,任何一种测试样品中的21号染色体的第二剂量可以使用7号染色体、9号染色体、11号染色体或12号染色体作为归一化染色体序列来确定,因为这些染色体的cv均接近于14号染色体的cv。
[0354]
在一些实施方案中,当选择单个染色体作为目标染色体的归一化染色体序列时,归一化染色体序列将是这样的染色体,其导致具有跨测试的所有样品(如,合格样品)的最小变异性的目标染色体的染色体剂量。在一些情况下,最佳的归一化染色体可能没有最小的变异,但可能具有最佳区分一种或多种测试样品与合格样品的合格剂量分布,即最佳归一化染色体可能没有最低的变异,但可能具有最大的可区分性。
[0355]
在一些实施方案中,归一化序列包括一条或多条稳定常染色体序列或其区段。在
一些实施方案中,稳定常染色体包括除目标染色体之外的所有常染色体。在一些实施方案中,稳定常染色体包括除了x、y、13、18和21之外的所有常染色体。在一些实施方案中,稳定常染色体包括除了从样品确定的偏离正常二倍体状态的那些之外的所有常染色体,其可用于确定具有相对于正常二倍体基因组的异常拷贝数的癌症基因组。
[0356]
测试样品中的非整倍性的测定
[0357]
基于合格样品中的归一化序列的鉴定,确定测试样品中的目标序列的序列剂量,所述测试样品包含来源于一条或多条目标序列不同的基因组的核酸的混合物。
[0358]
在步骤115中,从疑似或已知携带目标序列的临床相关cnv的受试者获得测试样品。测试样品可以是生物流体,如,血浆或如下所述的任何适合的样品。如所解释,可以使用非侵入性程序诸如简单抽血来获得样品。在一些实施方案中,测试样品含有核酸分子、如cfdna分子的混合物。在一些实施方案中,测试样品是母体血浆样品,其含有胎儿和母体cfdna分子的混合物。
[0359]
在步骤125中,测试样品中测试核酸的至少一部分如针对合格样品所描述进行测序,以产生数百万次序列读取,如36bp读取。在某些实施方案中,2
×
36bp配对末端读取用于配对末端测序。如在步骤120中那样,从测量样品中的核酸的测序产生的读取被唯一地定位或比对至参考基因组以产生标签。如步骤120所述,至少约3
×
106个合格序列标签、至少约5
×
106个合格序列标签、至少约8
×
106个合格序列标签、至少约10
×
106个合格序列标签、至少约15
×
106个合格序列标签、至少约20
×
106个合格序列标签、至少约40
×
106个合格序列标签、或至少约50
×
106个合格序列标签,包括20至40bp的读取,是从唯一地定位至参考基因组的读取获得的。在某些实施方案中,由测序装置产生的读取以电子格式提供。如下所讨论,使用计算装置完成比对。将各个读取与参考基因组进行比较以鉴定其中读取与参考基因组唯一对应的位点,所述参考基因组通常是很大的(数百万个碱基对)。在一些实施方案中,比对程序允许读取和参考基因组之间的有限错配。在一些情况下,允许读取中的1、2或3个碱基对在参考基因组中与相应的碱基对错配,但仍然进行定位。
[0360]
在步骤135中,使用如下所述的计算装置对从测试样品中的核酸的测序获得的所有或大多数标签进行计数以确定测试序列标签覆盖率。在一些实施方案中,将每个读取与参考基因组的特定区域(在大多数情况下为染色体或区段)比对,并且通过将位点信息附加到读取来将读取转换为标签。随着该过程展开,计算装置可以保持定位至参考基因组的每个区域(在大多数情况下为染色体或区段)的标签/读取的数量的连续计数。为每个目标染色体或区段和每个相应的归一化染色体或区段存储计数。
[0361]
在某些实施方案中,参考基因组具有一个或多个经排除的区域,这些区域是真正生物基因组的一部分但不包括在参考基因组中。不对潜在地与这些经排除的区域比对的读取进行计数。经排除的区域的实例包括长重复序列的区域、有x染色体和y染色体之间的相似性的区域等。使用通过上述掩蔽技术获得的经掩蔽的参考序列,仅考虑参考序列的未掩蔽区段上的标签用于cnv分析。
[0362]
在一些实施方案中,该方法确定当多个读取与参考基因组或序列上的相同位点比对时是否对标签多次计数。可能存在两个标签具有相同序列并且因此与参考序列上的相同位点比对的情况。用于对标签计数的方法在某些情况下可以从计数中排除源自相同测序样品的相同标签。如果在给定样品中不成比例的标签数量相同,则表明程序中存在强偏差或
其它缺陷。因此,根据某些实施方案,计数方法不对与来自先前计数的样品的标签相同的来自给定样品的标签计数。
[0363]
可以设置各种标准以选择何时忽略来自单个样品的相同标签。在某些实施方案中,经计数的标签的定义百分比必须是唯一的。如果比该阈值更大的标签不是唯一的,则忽略它们。例如,如果定义的百分比要求至少50%是唯一的,则不对相同的标签计数直至唯一标签的百分比超过样品的50%。在其它实施方案中,唯一标签的阈值数量为至少约60%。在其它实施方案中,唯一标签的阈值百分比是至少约75%、或至少约90%、或至少约95%、或至少约98%、或至少约99%。对于21号染色体,阈值可以设定为90%。如果30m标签与21号染色体比对,那么它们中的至少27m必须是唯一的。如果3m计数的标签不是唯一的并且3000万和第一标签不是唯一的,则不对其计数。可以使用适当的统计分析来选择用于确定何时不对另外相同标签进行计数的特定阈值或其它标准的选项。影响该阈值或其它标准的一个因素是经测序的样品与可以与标签比对的的基因组尺寸的相对量。其它因素包括读取的尺寸和类似的考虑因素。
[0364]
在一个实施方案中,将定位至目标序列的测试序列标签的数量归一化至它们被定位其上的目标序列的已知长度,以提供测试序列标签密度比。如针对合格样品所描述的,不需要归一化为目标序列的已知长度,并且可以将其包括为减少数字中的数字位数以简化其用于人类理解的步骤。由于所有经定位的测试序列标签都在测试样品中进行计数,因此确定测试样品中的目标序列(如,临床
‑
相关序列)的序列标签覆盖率,也测定对应于合格样品中鉴定的至少一条归一化序列的另外序列的序列标签覆盖率。
[0365]
在步骤150中,基于合格样品中的至少一条归一化序列的同一性,确定测试样品中的目标序列的测试序列剂量。在某些实施方案中,使用如本文所述的目标序列和相应的归一化序列的序列标签覆盖率通过计算确定测试序列剂量。负责该任务的计算装置将以电子方式访问目标序列与其相关的归一化序列之间的关联性,其可以存储在数据库、表格、图表中,或者作为代码包含在程序指令中。
[0366]
如本文其它地方所述,至少一条归一化序列可以是单个序列或一组序列。测试样品中目标序列的序列剂量是对测试样品中目标序列确定的序列标签覆盖率与测试样品中确定的至少一条归一化序列的序列标签覆盖率的比率,其中测试样品中的归一化序列对应于用于特定目标序列的在合格样品中鉴定的归一化序列。例如,如果在合格样品中鉴定的用于21号染色体的归一化序列被确定为染色体,如14号染色体,那么21号染色体(目标序列)的测试序列剂量被确定为各自在测试样品中确定的21号染色体的序列标签覆盖率和14号染色体的序列标签覆盖率的比率。类似地,确定13号染色体、18号染色体、x染色体、y染色体和与染色体非整倍性相关的其它染色体的染色体剂量。用于目标染色体的归一化序列可以是染色体中的一个或一组,或染色体区段中的一个或一组。如前所述,目标序列可以是染色体的一部分,如染色体区段。因此,染色体区段的剂量可以确定为为测试样品中的区段确定的序列标签覆盖率与测试样品中的归一化染色体区段的序列标签覆盖率的比率,其中测试样品中的归一化区段对应于特定目标区段的在合格样品中鉴定的归一化区段(单个或一组区段)。染色体区段的尺寸范围可以是数千碱基(kb)至数兆碱基(mb)(如,约1kb至10kb、或约10kb至100kb、或约100kb至1mb)。
[0367]
在步骤155中,阈值是从为在多个合格样品中确定的合格序列剂量和确定的已知
作为目标序列的非整倍体的样品的序列剂量建立的标准偏差值推导的。请注意,此操作通常与患者测试样品的分析不同时执行。它可以与从合格样品中选择归一化序列同时执行。准确的分类取决于不同类别的概率分布之间的差异,即非整倍性的类型。在一些实例中,阈值选自每种类型的非整倍性(如21三体)的经验分布。如实施例所述为对13三体、18三体、21三体和x单体非整倍性进行分类而建立的可能阈值,所述实施例描述了通过对从包含胎儿和母体核酸的混合物的母体样品中提取的cfdna进行测序来确定染色体非整倍性的方法的用途。确定用于区分受染色体的非整倍性影响的样品的阈值与用于不同的非整倍性的阈值可以相同或可以不同。如实施例中所示,每个目标染色体的阈值由跨样品和测定运行的目标染色体剂量的变异性确定。用于任何目标染色体的染色体剂量的变异性越小,跨所有未受影响的样品的目标染色体的剂量中的扩展越窄,其用于设定用于确定不同的非整倍性的阈值。
[0368]
返回与对患者测试样品进行分类相关联的过程,在步骤160中,通过将目标序列的测试序列剂量与从合格序列剂量建立的至少一个阈值进行比较,在测试样品中确定目标序列的拷贝数变异。该操作可以通过用于测量序列标签覆盖率和/或计算区段剂量的相同计算装置来执行。
[0369]
在步骤160中,将测试目标序列的计算剂量与设定为根据用户定义的
″
可靠性阈值
″
选择的阈值的计算剂量相比较,以将样品分类为
″
正常的
″
和
″
受影响的
″
或者
″
无调用的
″
。
″
无调用的
″
样品是不能对其进行可靠的确定性诊断的样品。每类受影响的样品(如,21三体、部分21三体、x单体)都有自己的阈值,一个用于调用正常(未受影响)的样品并且另一个用于调用受影响的样品(尽管在一些情况下两个阈值重合)。如本文其它地方所述,在一些情况下,如果测试样品中的核酸的胎儿分数足够高,则可以将无调用的转换为调用的(受影响的或正常的)。可以通过在该过程流程的其它操作中采用的计算装置来报告测试序列的分类。在一些情况下,分类以电子格式报告,并且可以向目标人员显示、发电子邮件、发短信等。
[0370]
在一些实施方案中,cnv的确定包括计算ncv或nsv,所述ncv或nsv将染色体或区段剂量与如上所述的一组合格样品中的相应的染色体或区段剂量的平均值相关联。然后可以通过将ncv/nsv与预定的拷贝数评估阈值进行比较来确定cnv。
[0371]
可以选择拷贝数评估阈值来优化假阳性率和假阴性率。拷贝数评估阈值越高,出现假阳性的可能性越小。类似地,阈值越低,出现假阴性的可能性越小。因此,在第一理想阈值(在第一理想阈值之上仅对真阳性进行分类)和第二理想阈值(在第二理想阈值之下仅对真阴性进行分类)之间存在权衡。
[0372]
主要根据如在一组未受影响的样品中确定的特定目标染色体的染色体剂量的变异性来设定阈值。变异性取决于许多因素,包括样品中存在的胎儿cdna的分数。变异性(cv)由跨未受影响的样品群的染色体剂量的平均值或中值和标准偏差确定。因此,用于对非整倍性进行分类的一个或多个阈值根据以下使用ncv:
[0373][0374]
(其中和分别是一组合格样品中的第j染色体剂量的估计平均值和标准偏差,并且x
ij
是观察到的测试样品i的第j染色体剂量。)
[0375]
其相关的胎儿分数:
[0376][0377]
因此,对于目标染色体的每个ncv,可以根据跨未受影响的样品群的目标染色体的染色体比的平均值和标准偏差,从cv计算出与给定的ncv值相关的预期胎儿分数。
[0378]
随后,基于胎儿分数和ncv值之间的关系,可以选择决策边界,在该决策边界之上,基于正态分布分位数确定样品是阳性的(受影响的)。如上所述,在一些实施方案中,设定阈值以在真阳性率和假阴性率的检测结果之间进行最佳权衡。即,选择阈值以使真阳性和真阴性的总和最大化,或最小化假阳性和假阴性的总和。
[0379]
某些实施方案提供了一种在包含胎儿和母体核酸分子的生物样品中提供胎儿染色体非整倍性的产前诊断的方法。诊断是通过以下进行:基于从来源于生物测试样品(如母体血浆样品)的胎儿和母体核酸分子的混合物的至少一部分获得序列信息,从测序数据计算用于一个或多个目标染色体的归一化染色体剂量和/或用于一个或多个目标区段的归一化区段剂量,并分别确定测试样品中的目标染色体的染色体剂量和/或区段剂量之间的统计学显著性差异,以及在多个合格(正常)样品中建立的阈值,并基于统计差异提供产前诊断。如该方法的步骤160中所述,进行正常或受影响的诊断。如果没有把握作出正常或受影响的诊断,则提供
″
无调用
″
。
[0380]
在一些实施方案中,可以选择两个阈值。选择第一阈值以使假阳性率最小化,高于该值时样品将被分类为
″
受影响的
″
,并且选择第二阈值以使假阴性率最小化,低于该值时样品将被分类为
″
未受影响的
″
。ncv高于第二阈值但低于第一阈值的样品可被分类为
″
非整倍性疑似
″
或
″
无调用
″
样品,其中非整倍性的存在或缺失可通过独立手段确认。第一和第二阈值之间的区域可以被称为
″
无调用
″
区域。
[0381]
在一些实施方案中,表1中显示了疑似和无调用阈值。可以看出,ncv的阈值在不同的染色体之间变化。在一些实施方案中,阈值根据如上所解释的样品的ff而变化。在一些实施方案中,这里应用的阈值技术有助于提高灵敏度和选择性。
[0382]
表1.疑似的和受影响的ncv阈值包围无调用范围
[0383] 疑似的受影响的chr 133.54.0chr 183.54.5chr 213.54.0chr x(xo,xxx)4.04.0chr y(xx对比xy)6.06.0
[0384]
片段尺寸和序列覆盖率分析
[0385]
如上所述,片段尺寸参数以及覆盖率可用于评估cnv。无细胞核酸片段(如cfdna片段)的片段尺寸可以通过配对末端测序、电泳(如基于微芯片的毛细管电泳)和本领域已知的其它方法获得。图2a主要显示了可以如何使用配对末端测序来确定片段尺寸和序列覆盖率。
[0386]
图2a的上半部分显示了胎儿无细胞dna片段和母体无细胞dna片段的图,所述片段提供用于配对末端测序方法的模板。通常,长核酸序列在配对末端测序方法中被片段化为
待读取的更短的序列。此类片段还被称为插入物。片段化对于无细胞dna是不必要的,因为它们已经存在于片段中,大多数短于300个碱基对。已经表明母体血浆中的胎儿无细胞dna片段比母体无细胞dna片段长。如图2a的顶部所示,胎儿来源的无细胞dna具有约167个碱基对的平均长度,而母体来源的无细胞dna具有约175个碱基对的平均长度。在某些平台上的配对末端测序中,例如,如下文进一步描述的合成平台的illumina测序,将接头序列、索引序列和/或引物序列连接到片段的两端(图2a中未示出)。片段是一个方向上的第一读取,从片段的一端提供读取1。然后第二读取从片段的相对端开始,提供rea 2序列。读取1和读取2之间的对应性可以通过它们在流动池中的坐标来鉴定。然后将读取1和读取2定位至参考序列作为彼此靠近的一对标签,如图2a的下半部分所示。在一些实施方案中,如果读取足够长,则两个读取可以在插入物的中间部分重叠。在该对与参考序列比对之后,两个读取之间的相对距离和片段的长度可以从两个读取的位置确定。因为配对末端读取提供两倍于相同读取长度的单末端读取的碱基对,它们有助于提高比对质量,尤其是对于具有许多重复或非唯一序列的序列。在许多实施方案中,将参考序列分为箱,诸如100k碱基对箱。在将配对末端读取与参考序列比对之后,可以确定与箱比对的读取的数量。也可以确定箱的数量以及插入物(如,cfdna片段)的长度。在一些实施方案中,如果插入物横跨两个箱,则插入物的一半可归因于每个箱。
[0387]
图2b显示了提供方法220以用于使用基于尺寸的覆盖率来确定测试样品中的目标核酸序列(包括源自两个或更多个基因组的无细胞核酸片段)的拷贝数变异的实施方案。如本文所公开的,当1)参数对片段尺寸或尺寸范围有利地加权,如当与该尺寸或尺寸范围的片段相关联时,比其它尺寸或范围的片段加权更大的计数时;或者当2)参数是从对于该片段尺寸或尺寸范围有利地加权的值获得的,如当与该尺寸或尺寸范围的片段相关联时,从加权更大的计数获得的比率时,参数
″
偏向片段尺寸或尺寸范围
″
。当基因组产生相对于来自另一基因组或相同基因组的其它部分的核酸片段富含或具有更高浓度的尺寸或尺寸范围的核酸片段时,片段尺寸或尺寸范围可以是基因组或其部分的特征。
[0388]
方法220通过接收通过对测试样品中的无细胞核酸片段进行测序获得的序列读取开始。参见框222。测试样品中的两个或更多个基因组可以是妊娠母亲的基因组和由妊娠母亲携带的胎儿的基因组。在其它应用中,测试样品包括来自肿瘤细胞和未受影响的细胞的无细胞dna。在一些实施方案中,由于由尺寸偏差的覆盖率提供的高信噪比,进行无细胞核酸片段的测序而无需使用pcr扩增核酸片段。方法200还涉及将无细胞核酸片段的序列读取与包含目标序列的参考基因组比对,并将其分成多个箱。成功的比对产生测试序列标签,其包含序列及其在参考序列上的位置。参见框224。然后,方法220通过确定测试样品中存在的无细胞核酸片段的尺寸来进行。应用配对末端测序的一些实施方案提供与序列标签相关的插入物的长度。参见框226。术语
″
尺寸
″
和
″
长度
″
在就核酸序列或片段而言时可互换使用。在这里所示的实施方案中,方法220还涉及基于从其获得标签的无细胞核酸片段的尺寸来对测试序列标签进行加权。参见框228。如本文所用,
″
加权
″
是指使用一个或多个变量或函数修改数量。一个或多个变量或函数被认为是
″
权重
″
。在许多实施方案中,变量乘以权。在其它实施方案中,变量可以指数或其它方式进行修改。在一些实施方案中,通过将覆盖率偏向从测试样品中的一个基因组的特征性尺寸或尺寸范围的无细胞核酸片段获得的测试序列标签来进行对测试序列标签的加权。如本文所公开的,当基因组相对于另一基因组或相
同基因组的另一部分具有富集的或更高浓度的核酸时,尺寸是基因组的特征。
[0389]
在一些实施方案中,加权函数可以是线性或非线性函数。适用的非线性函数的实例包括但不限于海维塞德阶跃函数、箱车函数、阶梯函数或s型函数。在一些实施方案中,使用海维塞德函数或箱车函数,使得在特定的尺寸范围内的标签乘以权重1,并且在该范围之外的标签乘以权重0。在一个实施方案中,在80和150个碱基对之间的片段给予权重1,而在该范围之外的片段给予权重0。在这些实例中,加权是谨慎的,根据所有值的参数是否落在特定范围之内或之外,为零或一。可替代地,权重被计算为片段尺寸或相关参数值的其它方面的连续函数。
[0390]
在一个实施方案中,一个尺寸范围内的片段的权重是正的,而另一个范围内的那些片段的权重是负的。当两个基因组之间的差值方向具有相反的符号时,这可以用于帮助增强信号。例如,读取计数对于80
‑
150个碱基对的插入物具有1的权重,并且对于160
‑
200个碱基对的插入物具有
‑
1的权重。
[0391]
可以给予计数以及其它参数以权重。例如,加权也可以应用于使用片段尺寸的分数或比率参数。例如,该比率可以给在某些子范围内的片段比片段和其它尺寸箱更大的加权。
[0392]
然后基于加权的测试序列标签计算箱的覆盖率。参见框230。此类覆盖率被认为是有尺寸偏差的。如上所解释,当参数对于片段尺寸或尺寸范围有利地加权时,值偏向该片段尺寸或尺寸范围。方法200还涉及从计算的覆盖率中鉴定目标序列中的拷贝数变异。参见框232。在一些实施方案中,如下文结合图2c、3a
‑
3k和4进一步解释的,可以调整或校正覆盖率以去除数据中的噪声,从而增加信噪比。在一些应用中,基于在方法220中获得的加权标签的覆盖率在确定拷贝数变异方面提供了比未加权的覆盖率更高的灵敏度和/或更高的选择性。在一些应用中,下面提供的示例性工作流程可以进一步提高cnv分析的灵敏度和选择性。
[0393]
用于分析片段尺寸和/或序列覆盖率的工作流程实例
[0394]
公开的一些实施方案提供了确定具有低噪声和/或高信号的序列覆盖量从而提供用于确定与拷贝数和cnv相关的各种遗传病况的数据的方法,该方法具有相对于通过常规方法获得的序列覆盖量提高的灵敏度、选择性和/或效率。在某些实施方案中,来自测试样品的序列被处理以获得序列覆盖量。
[0395]
该方法利用可从其它来源获得的某些信息。在一些实施方式中,所有这些信息都是从已知不受影响的(如,不是非整倍体)样品的训练组获得的。在其它实施方案中,部分或全部信息是从其它测试样品中获得的,其当在同一过程中分析多个样品时,可以
″
在执行中
″
提供。
[0396]
在某些实施方案中,采用序列掩码来减少数据噪声。在一些实施方案中,目标序列及其归一化序列被掩蔽。在一些实施方案中,当考虑不同的目标染色体或区段时,可以采用不同的掩码。例如,当13号染色体是目标染色体时可以使用一个掩码(或一组掩码),并且当21号染色体是目标染色体时可以使用不同的掩码(或一组掩码)。在某些实施方案中,掩码是以箱的分辨率定义的。因此,在一个实例中,掩码分辨率是100kb。在一些实施方案中,可以将不同的掩码应用于y染色体。如在2013年6月17日提交的美国临时专利申请no.61/836,057[代理人案号artep008p]中所述,y染色体的经掩蔽的排除区域可以以比其它目标染色
体更高的解析率(l kb)提供。掩码以鉴别经排除的基因组区域的文件的形式提供。
[0397]
在某些实施方案中,该方法利用经归一化的覆盖率的预期值来去除目标序列谱中的箱间变异,所述变异不提供测试样品的cnv的信息。该方法根据跨整个基因组中的每个箱或者至少参考基因组中的稳定染色体的箱的经归一化的覆盖率的预期值来调整经归一化的覆盖量(用于下面的操作317)。通过该方法也可以提高除覆盖率以外的参数。预期值可以从未受影响的样品的训练组确定。作为实例,预期值可以是跨训练组样品的中值。样品的预期覆盖率值可以被确定为与箱比对的唯一非冗余标签的数量除以在参考基因组的稳定染色体中的与所有箱比对的唯一非冗余标签的总数。
[0398]
图2c描绘了用于确定目标序列的片段尺寸参数的方法200的流程图,所述参数用于在框214中评估测试样品中的目标序列的拷贝数。该方法去除了在未受影响的训练样品之间共有的系统变异,所述变异增加了用于cnv评估的分析中的噪声。它还去除了测试样品特有的gc偏差,从而提高了数据分析中的信噪比。值得注意的是,无论覆盖率是否对尺寸有偏置,方法200也可以应用于覆盖率。类似地,图2d、3和4中的方法同样适用于覆盖率、片段尺寸加权的覆盖率、片段尺寸、在限定的尺寸范围内的片段的分数或比率、片段的甲基化水平等。
[0399]
如框202所示,方法200通过提供测试样品的序列读取开始。在一些实施方案中,序列读取通过对从孕妇血液获得的dna区段(包括母亲和胎儿的cfdna)进行测序获得。该方法继续使序列读取与包括目标序列的参考基因组比对,从而提供测试序列标签。框204。在一些实施方案中,排除了与多于一个位点比对的读取。在一些实施方案中,与同一位点的多个读取比对被排除或减少到单个读取计数。在一些实施方案中,也排除了与经排除的位点比对的读取。因此,在一些实施方案中,仅对与非排除的位点比对的唯一比对的非冗余标签进行计数,以提供用于确定每个箱的覆盖率或其它参数的非排除的位点计数(nes计数)。
[0400]
方法200提供测试样品中存在的无细胞核酸片段的尺寸。在一些实施方案中,使用配对末端测序,可以从插入物的末端处的一对读取的位置获得插入物尺寸/长度。其它技术可用于确定片段尺寸。参见框205。然后,在参考基因组的箱(包括目标序列中的箱)中,方法200确定偏向基因组之一的特征性片段尺寸的片段尺寸参数的值。术语
″
片段尺寸参数
″
是指涉及核酸片段的片段或片段集合的尺寸或长度的参数;如,从体液中获得的cfdna片段。如果本文所用,当1)参数对片段尺寸或尺寸范围有利地加权,如当与该尺寸或尺寸范围的片段相关联时,比其它尺寸或范围加权更大的计数时;或者当2)参数是从对于该片段尺寸或尺寸范围有利地加权的值获得的,如当与该尺寸或尺寸范围的片段相关联时,从加权更大的计数获得的比率时,参数
″
偏向片段尺寸或尺寸范围
″
。当基因组产生相对于来自另一基因组或相同基因组的其它部分的核酸片段富含或具有更高浓度的尺寸或尺寸范围的核酸片段时,片段尺寸或尺寸范围可以是基因组或其部分的特征。
[0401]
在一些实施方案中,片段尺寸参数是尺寸加权的计数。在一些实施方案中,将片段在一个范围内加权1,并且在该范围之外加权0。在其它实施方案中,片段尺寸参数是尺寸范围内的片段的分数或比率参见框206。在一些实施方案中,每个箱的片段尺寸参数(或覆盖率,如上所述)的值除以同一样品中的归一化序列的参数值,从而提供归一化参数。
[0402]
然后,方法200提供目标序列的全局谱。全局谱包括从未受影响的训练样品的训练组获得的每个箱中的预期参数值。框208。方法200通过根据预期参数值调整测试序列标签
的归一化参数值来去除训练样品中共有变异,以获得目标序列的参数的全局谱校正值。框210。在一些实施方案中,从框208中提供的训练组获得的参数的预期值是跨训练样品的中值。在一些实施方案中,操作2010通过从参数的经归一化的值中减去参数的预期值来调整参数的归一化值。在其它实施方案中,操作210将参数的经归一化的值除以每个箱的参数的预期值,以产生参数的全局谱校正值。
[0403]
除了全局谱校正之外或代替全局谱校正,方法200通过调整参数值来去除测试样品特有的gc偏差。如框212所示,该方法基于gc含量水平与测试样品中存在的全局谱校正的覆盖率之间的关系调整全局谱校正的参数值,从而获得片段尺寸参数的样品
‑
gc校正值。在调整了未受影响的训练样品中共有的系统变异和受试者内部gc偏差后,该方法提供了针对全局谱和/或gc变量校正的片段尺寸值,该值用于以提高的灵敏度和特异性评估样品的cnv。在一些实施方式中,可以使用主组分分析方法调整片段尺寸值,以去除与目标序列的拷贝数变异无关的方差组分,如参考图2f的框719进一步描述。在一些实施方式中,可以通过去除样品内的异常值箱来确定(curate)片段尺寸值,如参考图3a的框321所描述。
[0404]
使用多个参数进行的拷贝数确定的多程方法
[0405]
如上所强调,本文公开的方法适用于使用多个参数确定cnv,所述参数包括但不限于覆盖率、片段尺寸加权的覆盖率、片段尺寸、在限定的尺寸范围内的片段的分数或比率、片段的甲基化水平等。这些参数中的每一个可以被分别处理以单独地有助于最终拷贝数变异确定。
[0406]
在一些实施方案中,类似的方法可以应用于尺寸加权的覆盖率分析和片段尺寸分析,两者都是片段尺寸参数。图2d显示了工作流程600的两个重叠通路的流程图,通路1用于尺寸加权的覆盖率以及通路2用于片段尺寸分析。在此处未示出的另一个实施方案中,甲基化水平可以在一次另外的通路中进行处理。两个通路可以包括可比较的操作以获得经调整的覆盖率信息,cnv的确定基于所述覆盖率信息。
[0407]
该方法的初始单通路部分通过接收测序数据开始,参见框602,并继续通过计算进行如上所述的计数,参见框612。在此之后,所描绘的方法分成两个通路,如上所述。返回到方法的初始部分,工作流程将测序数据转换为序列读取。当测序数据来源于多路复用测序时,序列读取也被解复用以鉴别数据源。参见框604。然后将序列读取与参考序列比对,其中比对的序列读取作为序列标签提供。参见框606。然后,将序列标签进行过滤以获得非排除的位点(nes),其是明确定位的非重复的序列标签。序列标签被组织成特定序列长度的箱,诸如1kb、100kb或1mb。参见框610。在涉及综合征特异性区域分析的一些实施方案中,箱为100kb。在一些实施方案中,可以使用从多个未受影响的样品获得的序列掩码来以如图3a、框313中所述的方式掩蔽表现出高变异性的箱。然后对nes中的标签进行计数以提供经归一化并调整用于cnv分析的覆盖率。参见框612。
[0408]
在所描绘的实施方案中,操作604、606、610和612被执行一次并且大部分剩余的操作被执行两次,一次用于尺寸加权的覆盖率分析(过程1)并且一次用于片段尺寸分析(过程2)。在其它实施方案中,显示为在两次过程中执行的一次或多次操作仅执行一次,并且结果在两个过程中共享。这种共享操作的实例包括操作614、616和618。
[0409]
在所描绘的实施方案中,所获得的nes的覆盖率(尺寸加权的计数)或片段尺寸参数(尺寸分数或比率)通过如将箱的nes值除以基因组或一组归一化染色体的总nes而被归
一化。在一些实施方案中,只有覆盖率被归一化,而片段尺寸参数不需要归一化,因为它不以与覆盖率一样的方式受测序深度的影响。参见框614。然后,在一些实施方案中,去除包括未受影响的样品的训练组共有的方差,所述方差与目标cnv 无关。在所描绘的实施方案中,共有方差表示为以与上述全局波谱类似的方式从未受影响的样品获得的全局波谱。在一些实施方案中,如图6所示,用于获得全局波谱的未受影响的样品包括来自相同流动池或加工批次的样品。参见框616。下文进一步解释流动池特异性全局波的计算。在所描绘的实施方案中,在去除全局波谱之后,在样品特异性的基础上校正gc水平的覆盖率。参见框616。用于gc校正的一些算法在下文中与图3a、框319相关联的文本中进一步详细描述。
[0410]
在所描绘的实施方案中,在用于加权的覆盖率分析的通路1和用于片段尺寸分析的通路2中,针对单个样品特异性的噪声,可以进一步过滤数据,如具有极大地不同于其它箱的覆盖率的异常值箱的数据可能从分析中去除,所述差异不能归因于目标拷贝数变异。参见框622。该样品内过滤操作可对应于图3a中的框321。
[0411]
在一些实施方案中,经过单次样品过滤后,相对于参考,通路1的加权覆盖率值和通路2的片段尺寸参数在靶信号中得到富集。参见框624和628。然后,染色体的覆盖率和片段尺寸参数分别用于计算如上所述的染色体剂量和归一化的染色体值(ncv)。然后可以将ncv与标准进行比较以确定指示cnv概率的评分。参见框626和630。然后可以组合来自两个通路的评分以提供复合的最终评分,其确定是否应该调用非整倍性。在一些实施方案中,626和630的评分是t
‑
检验统计量或z值。在一些实施方案中,最终的评分是卡方值。在其它实施方案中,最终评分是两个t值或z评分的均方根。组合来自两条通路的两个评分的其它手段可用于提高cnv检测中的总体灵敏度和选择性。或者,可以通过逻辑运算(如和(and)运算或者或(or)运算)来组合来自两个通路的两个评分。例如,当优选高灵敏度以确保低假阴性时,当来自通路1或通路2的评分满足调用标准时,可以进行cnv调用。另一方面,如果需要高选择性以确保低假阳性,则只有当来自通路1或通路2的评分满足调用标准时,才能进行cnv调用。
[0412]
值得注意的是,使用上述此类逻辑操作在灵敏度和选择性之间存在权衡。在一些实施方案中,应用两步测序方法来克服权衡,如下文进一步描述。简言之,将样品的初始评分与设计用于增加灵敏度的相对低的第一阈值进行比较,并且如果样品评分高于第一阈值,则其经历比第一轮更深的第二轮测序。然后在类似于上述的工作流程中重新处理和分析此类样品。然后将得到的评分与经设计以提高灵敏度的相对高的第二阈值进行比较。在一些实施方案中,样品经历了在高于第一阈值的那些中相对低的第二轮测序评分,从而减少了需要重新测序的样品的数量。
[0413]
在一些实施方案中,可以采用使用第三参数的第三通路。该第三通路的一个实例是甲基化。甲基化可以通过测量来自样品的核酸的甲基化直接测定,或间接测定为与无细胞核酸的片段尺寸相关的参数。
[0414]
在一些实施方案中,该第三参数是第二覆盖率或基于计数的参数,其中计数基于在基于第一计数的参数中使用的主片段尺寸之外的片段尺寸。当使用80至150个碱基对之间的片段来产生计数或覆盖率参数时,它们从测序中排除了约70%的读取。在这些被排除的读取仍然具有一些潜在有用的信号的程度上,它们可以用于第三参数,所述第三参数包括被排除的读取或基于尺寸的分数中的读取,其在用于第一参数中的基于尺寸的分数之外
或与其重叠。在这方面,从排除的片段取得的读取和相关的覆盖率值可以被给予较低的权重。换句话说,使用这些读取计算的拷贝数变异参数在产生最终拷贝数变异调用时可能不太重要。或者,如上所述,当两个基因组在两个尺寸范围内具有相反特征时,第一参数中的尺寸范围之外的标签可以采取正值。
[0415]
在各种实施方式中,方法200、220和600中的覆盖率偏向来自在片段尺寸光谱的更短端处的片段的标签。在一些实施方案中,覆盖率偏向于尺寸小于指定值的片段的标签。在一些实施方案中,覆盖率偏向来自比指定值更短的尺寸的片段的标签,并且该范围的上端为约150个碱基对或更少。
[0416]
在方法200、220和600的各种实施方式中,通过对无细胞核酸片段进行测序而不是首先使用pcr来扩增无细胞核酸片段的核酸来获得序列读取。在某些实施方案中,通过对无细胞核酸片段进行测序至每个样品的深度不大于约6m片段获得测序读取。在一些实施方案中,测量深度不大于每样品约1m片段。在一些实施方案中,测序读取是通过多路复用测量获得的,并且多路复用的样品的数量是至少约24。
[0417]
在方法200、220和600的各种实施方式中,测试样品包括来自个体的血浆。在一些实施方案中,该方法还包括从测试样品中获得无细胞核酸。在一些实施方案中,该方法还包括对源自两个或多个基因组的无细胞核酸片段进行测序。
[0418]
在方法200、220和600的各种实施方式中,两个或更多个基因组包括来自母亲和胎儿的基因组。在一些实施方式中,目标序列中的拷贝数变异包含胎儿的基因组中的非整倍性。
[0419]
在方法200、220和600的各种实施方式中,两个或更多个基因组包含来自癌症和体细胞的基因组。在一些实施方式中,该方法包括使用癌症基因组中的拷贝数变异来诊断癌症、监测癌症进展和/或确定癌症的治疗。在一些实施方式中,拷贝数变异导致遗传异常。
[0420]
在方法200、220和600的各种实施方式中,覆盖率偏向来自在片段尺寸谱的较长端处的片段的标签。在一些实施方式中,覆盖率偏向来自于尺寸大于指定值的片段的标签。在一些实施方式中,覆盖率偏向来自在片段尺寸范围内的片段的标签,并且其中该范围的下端是约150个碱基对或更多。
[0421]
在方法200、220和600的各种实施方式中,该方法还涉及:在参考基因组的箱(包括目标序列)中确定所述箱中无细胞核酸片段的甲基化水平,并使用除了经计算的覆盖率或片段尺寸参数值之外或代替经计算的覆盖率或片段尺寸参数值的甲基化水平来鉴定拷贝数变异。在一些实施方式中,使用甲基化水平来鉴定拷贝数变异涉及为目标序列的箱提供总体甲基化谱。总体甲基化谱包括至少目标序列箱中的预期的甲基化水平。在一些实施方式中,预期的甲基化水平是从包含核酸的未受影响的训练样品的训练组中的无细胞核酸片段的长度获得的,所述核酸被测序并且以与测试样品的核酸片段基本相同的方式比对,所述预期的甲基化水平展现出箱至箱变异。在一些实施方式中,该方法涉及使用至少目标序列的箱中的预期甲基化水平来调整甲基化水平的值,从而获得目标序列的甲基化水平的全局谱校正值,该方法进一步涉及使用全局谱校正的覆盖率和全局谱校正的甲基化水平来鉴定拷贝数变异。在一些实施方式中,使用全局谱校正的覆盖率和全局谱校正的甲基化水平鉴定拷贝数变异还包括:调整基于gc含量水平的全局谱校正的覆盖率和全局谱校正的甲基化水平,从而获得gc校正的覆盖率和gc校正的目标序列的甲基化水平值;并使用gc校正的
覆盖率和gc校正的甲基化水平鉴定拷贝数变异。
[0422]
在方法200、220和600的各种实施方式中,片段尺寸参数包括分数或比率,其包括具有比阈值更短或更长的片段尺寸的测试样品中的无细胞核酸片段的一部分。在一些实施方式中,片段尺寸参数包括分数,其包括:(i)在包括110个碱基对的第一尺寸范围内的测试样品中的多个片段,以及(ii)在包括第一尺寸范围和第一尺寸范围之外的尺寸的第二尺寸范围内的测试样品中的多个片段。
[0423]
使用三通路方法、似然比、t统计量和/或胎儿分数进行的拷贝数确定
[0424]
图2e显示了用于评估拷贝数的三通路方法的流程图。它包括工作流程700的三个重叠通路,其包括通路1(或713a)分析与所有尺寸的片段相关的读取的覆盖率,通路2(或713b)分析与更短的片段相关的读取的覆盖率,以及通路3(或713c)分析更短的读取相对于所有读取的相对频率。
[0425]
方法700在其整体组织中类似于方法600。由框702、704、706、710、712指示的操作可以以与由框602、604、606和610以及612指示的操作相同或相似的方式执行。在获得读取计数之后,在通路713a中使用来自所有尺寸的片段的读取来确定覆盖率。在通路713b中使用来自短片段的读取来确定覆盖率。在通路713c中确定来自短片段的读取相对于所有读取的频率。在本文的其它地方,相对频率还被称为尺寸比率或尺寸分数。其是片段尺寸特征的一个实例。在一些实施方式中,短片段是比约150个碱基对短的片段。在各种实施方式中,短片段可以为约50
‑
150、80
‑
150或110
‑
150个碱基对的尺寸。在一些实施方式中,第三通路或通路713c是任选的。
[0426]
三个通路713a、713b和713c的数据都经历归一化操作714、716、718、719和722,以去除与目标序列的拷贝数无关的方差。将这些归一化操作在框723中加框。操作714涉及通过将分析的数量除以参考序列的数量的总值来对目标序列的分析的数量进行归一化。该归一化步骤使用从测试样品获得的值。类似地,操作718和722使用从测试样品获得的值来归一化经分析的数量。操作716和719使用从未受影响的样品的训练组获得的值。
[0427]
操作716去除从未受影响的样品的训练组获得的总体波的方差,其使用如参考框616描述的相同或类似的方法。操作718使用如参考框618所述的相同或相似的方式方法去除个体特异性gc方差的方差。
[0428]
操作719使用主组分分析(pca)方法去除进一步的方差。通过pca方法去除的方差是由于与目标序列的拷贝数无关的因素导致的。每个箱中的经分析的数量(覆盖率、片段尺寸比等)为pca提供因变量,并且未受影响的训练组的样品供给这些因变量的值。训练组的样品都包括具有与目标序列相同的拷贝数的样品,如体细胞染色体的两个拷贝、x染色体的一个拷贝(当男性样品用作未受影响的样品时),或者x染色体的两个拷贝(当女性样品用作未受影响的样品时)。因此,样品的方差不是由非整倍性或拷贝数的其它差异引起的。训练组的pca产生与目标序列的拷贝数无关的主要组分。然后可以使用主要组分来去除与目标序列的拷贝数无关的测试样品中的方差。
[0429]
在某些实施方案中,使用从在目标序列之外的区域中的未受影响的样品数据估计的系数,从测试样品数据中去除一个或多个主要组分的方差。在一些实施方式中,该区域代表所有稳定染色体。例如,对在训练正常样品的归一化的箱覆盖率数据执行pca,从而提供对应于可以捕获数据中的大多数方差的维度的主要组分。如此捕获的方差与目标序列中的
拷贝数变异无关。在从训练普通样品中获得主要组分之后,将它们应用于测试数据。跨目标序列之外的区域的箱生成具有测试样品作为响应变量并且主要组分作为因变量的线性回归模型。得到的回归系数用于通过减去由估计的回归系数限定的主要组分的线性组合来归一化目标区域的箱覆盖率。这样可以从目标序列中去除与cnv无关的方差。参见框719。残差数据用于下游分析。另外,操作722使用参考框622描述的方法来移除异常值数据点。
[0430]
在框723中进行归一化操作之后,所有箱的覆盖率值已经
″
归一化
″
以去除除了非整倍性或其它拷贝数变异以外的变异源。在某种意义上,出于拷贝数变异检测的目的,相对于其它箱,目标序列的箱被富集或改变。参见框724,其不是操作但表示所得的覆盖率值。大框723中的归一化操作可以增加信号和/或降低分析数量噪声。类似地,对箱的短片段的覆盖率值进行了归一化,以去除除了非整倍性或其它拷贝数变异之外的变异源,如框728所示,并且箱的短片段的相对频率(或尺寸比)已被类似地归一化以去除除了非整倍性或其它拷贝数变异之外的变异源,如框732所示。如框724一样,框728和732不是操作而是代表在加工大框723之后的覆盖率和相对频率值。应当理解,可以修改、重新排列或去除框723中的操作。例如,在一些实施方案中,不执行pca操作719。在其它实施方案中,不执行gc操作718的校正。在其它实施方案中,改变操作的顺序;如,在校正gc操作718之前,执行pca操作719,
[0431]
在框724中所示的归一化和方差去除之后的所有片段的覆盖率用于获得在框726中的t
‑
统计量。类似地,在框728中所示的归一化和方差去除之后的短片段的覆盖率用于获得在框730中的t
‑
统计量,并且在框732中所示的归一化和方差去除之后的短片段的相对频率用于获得在框734中的t
‑
统计量。
[0432]
图2f说明了为什么将t
‑
统计量应用于拷贝数分析可有助于提高分析的精确度。图2f在每个图中显示了目标序列和参考序列的归一化箱覆盖率的频率分布,其中目标序列分布重叠并模糊参考序列分布。在顶部图中,显示了具有更高覆盖率的样品的箱覆盖率,具有超过600万次读取;在底部图中,显示了具有更低覆盖率的样品的箱覆盖率,具有少于2百万次读取。横轴表示相对于参考序列的平均覆盖率归一化的覆盖率。纵轴表示与具有平均覆盖率值的箱的数量相关的相对概率密度。因此,图2f是一类直方图。目标序列的分布显示在前面,并且参考序列的分布显示在后面。目标序列的分布平均值低于参考序列的分布平均值,表明样品中的拷贝数降低。目标序列和参考序列之间的平均差异对于顶部图中的高覆盖率样品和底部图中的低覆盖率样品是类似的。因此,在一个实施方式中,可以使用平均值的差异来鉴定目标序列中的拷贝数变异。注意,高覆盖率样品的分布具有小于低覆盖率样品的那些分布的方差。仅使用均值来区分这两个分布并不能捕获两个分布之间的差异,并使用均值和方差。t
‑
统计量可以反映分布的均值和方差。
[0433]
在一些实施方式中,操作726如下计算t
‑
统计量:
[0434][0435]
其中x1是目标序列的箱覆盖率,x2是参考区域/序列的箱覆盖率,s1是目标序列的覆盖率的标准偏差,s2是参考区域的覆盖率的标准偏差,n1是目标序列的箱数量;并且n2是参考区域的箱的数量。
[0436]
在一些实施方式中,参考区域包括所有稳定染色体(如,除了最可能具有非整倍性的染色体那些染色体)。在一些实施方式中,参考区域包括目标序列之外的至少一个染色体。在一些模仿中,参考区域包括不包含目标序列的稳定染色体。在其它实施方式中,参考区域包括一组染色体(如,选自稳定染色体的染色体亚组),其已被确定为一组训练样品提供最佳信号检测能力。在一些实施方案中,信号检测能力基于参考区域区分具有拷贝数变异的箱与不具有拷贝数变异的箱的能力。在一些实施方案中,参考区域以类似于用于确定
″
归一化序列
″
或
″
归一化染色体
″
的方式鉴定,如标题为
″
归一化序列的鉴定(identification of normalizing sequences)
″
的章节所述。
[0437]
返回图2e,可以将一个或多个胎儿分数估值(框735)与框726、730和734中的任何t统计量组合以获得倍性情况的似然估值。参见框736。在一些实施方式中,框740的一个或多个胎儿分数通过图2g中的方法800、图2h中的方法900或图2i的方法1000中的任一个获得。该方法可以使用工作流程(如图2j中的工作流程1100)平行实现。
[0438]
图2g显示了根据本公开的一些实施方式的用于从覆盖率信息确定胎儿分数的示例性方法800。方法800通过从训练组获得训练样品的覆盖率信息(如,序列剂量值)开始。参见框802。训练组的每个样品都是从已知携带男性胎儿的孕妇获得的。即,该样品含有男性胎儿的cfdna。在一些实施方式中,操作802可以获得以与本文所述的序列剂量不同的方式归一化的序列覆盖率,或者它可以获得其它覆盖率值。
[0439]
然后,方法800涉及计算训练样品的胎儿分数。在一些实施方式中,胎儿分数可以从序列剂量值计算:
[0440][0441]
其中rx
j
是男性样品的序列剂量,中值(rx
i
)是女性样品的序列剂量的中值。在其它实施方式中,可以使用平均或其它集中趋势量度。在一些实施方式中,ff可以通过其它方法获得,诸如x染色体和y染色体的相对频率。参见框804。
[0442]
方法800还涉及将参考序列分成子序列的多个箱。在一些实施方式中,参考序列是完整的基因组。在一些实施方式中,箱是100kb箱。在一些实施方式中,将基因组分成约25,000个箱。然后该方法获得箱的覆盖率。参见框806。在一些实施方式中,在框806中使用的覆盖率是在经历了图2j的框1123中所示的归一化操作之后获得的。在其它实施方式中,可以使用来自不同尺寸范围的覆盖率。
[0443]
每个箱与训练组中的样品的覆盖率相关联。因此,对于每个箱,可以在样品的覆盖率和样品的胎儿分数之间获得相关性。方法800涉及获得所有箱的胎儿分数和覆盖率之间的相关性。参见框808。然后,该方法选择具有高于阈值的相关值的箱。参见框810。在一些实施方式中,选择具有6000最高相关值的箱。目的是鉴定在训练样品中显示覆盖率和胎儿分数之间高度相关性的箱。然后箱可用于预测测试样品中的胎儿分数。虽然训练样品是男性样品,但胎儿分数与覆盖率之间的相关性可以推广到男性和女性的测试样品。
[0444]
使用具有高相关性值的所选箱,该方法获得将胎儿分数与覆盖率相关联的线性模型。参见框812。每个选定的箱为线性模型提供自变量。因此,所获得的线性模型还包括每个箱的参数或权重。调整箱的权重以使模型拟合数据。在获得线性模型之后,方法800涉及将测试样品的覆盖率数据应用于模型以确定测试样品的胎儿分数。参见框814。测试样品的所
应用的覆盖率数据用于在胎儿分数和覆盖率之间具有高相关性的箱。
[0445]
图2j显示了用于加工序列读取信息的工作流程1100,其可用于获得胎儿分数估值。工作流程1100与图2d中的工作流程600共有类似的处理步骤。框1102、1104、1106、1110、1112、1123、1114、1116、1118和1122分别对应于框602、604、606、610、612、623、614、616、618和622。在一些实施方式中,123框中的一个或多个归一化操作是任选的。通路1提供了覆盖率信息,其可以在图2g中所示的方法800的框806中使用。然后,方法800可以在图2j中产生胎儿分数估值1150。
[0446]
在一些实施方式中,可以组合多个胎儿分数估值(如图2j中的1150和1152)以提供复合胎儿分数估值(如,1154)。可以使用各种方法来获得胎儿分数估值。例如,胎儿分数可以从覆盖率信息获得。参见图2j的框1150和图2g的方法800。在一些实施方式中,胎儿分数也可以从片段的尺寸分布估计。参见图2j的框1152和图2h的方法900。在一些实施方式中,胎儿分数也可以从8聚体频率分布估计。参见图2j的框1152和图2i的方法1000。
[0447]
在包含男性胎儿的cfdna的测试样品中,也可以从y染色体和/或x染色体的覆盖率估计胎儿分数。在一些实施方式中,通过使用选自以下的信息获得推定男性胎儿的胎儿分数(参见,如框1155)的复合估值:从箱的覆盖率信息获得的胎儿分数,从片段尺寸信息获得的胎儿分数,从y染色体的覆盖率获得的胎儿分数,从x染色体获得的胎儿分数,以及它们的任何组合。在一些实施方式中,通过使用y染色体的覆盖率获得胎儿的推定性别。可以以各种方式组合两个或更多个胎儿分数(如,1150和1152)以提供胎儿分数的复合估值(如,1155)。例如,可以在一个实施方式中使用均值或加权均值方法,其中加权可以基于胎儿分数估值的统计置信度。
[0448]
在一些实施方式中,通过使用选自以下的信息获得推定男性胎儿的胎儿分数的复合估值:从箱的覆盖率信息获得的胎儿分数,从片段尺寸信息获得的胎儿分数,以及它们的任何组合。
[0449]
图2h显示了根据一些实施方式从尺寸分布信息确定胎儿分数的方法。方法900通过从训练组获得男性训练样品的覆盖率信息(如,序列剂量值)开始。参见框902。然后,方法900涉及使用上面参考框804描述的方法计算训练样品的胎儿分数。参见框904。
[0450]
方法900继续将尺寸范围划分为多个箱,以提供基于片段尺寸的箱,并确定基于片段尺寸的箱的读取的频率。参见框906。在一些实施方式中,获得基于片段尺寸的箱的频率而没有对框1123中所示的因子进行归一化。参见图2j的途径1124。在一些实施方式中,在任选地进行图2j的框1123中所示的归一化操作之后,获得基于片段尺寸的箱的频率。在一些实施方式中,将尺寸范围分为40个箱。在一些实施方式中,低端的箱包括尺寸小于约55个碱基对的片段。在一些实施方式中,低端的箱包括在约50
‑
55个碱基对范围的尺寸的片段,其排除了短于50bp的读取的信息。在一些实施方式中,高端的箱包括尺寸大于约245个碱基对的片段。在一些实施方式中,高端的箱包括在约245
‑
250个碱基对范围的尺寸的片段,其排除了长于250bp的读取的信息。
[0451]
方法900通过使用训练样品的数据获得将基于片段尺寸的箱的胎儿分数与读取频率相关的线性模型。参见框908。所获得的线性模型包括用于基于尺寸的箱的读取的频率的自变量。模型还包括每个基于尺寸的箱的参数或权重。调整箱的权重以将模型拟合至数据。在获得线性模型之后,方法900涉及将测试样品的读取频率数据应用于模型以确定测试样
品的胎儿分数。参见框910。
[0452]
在一些实施方式中,可以使用8聚体频率来计算胎儿分数。图2i显示了根据本公开的一些实施方式从8聚体频率信息确定胎儿分数的示例性方法1000。方法1000通过从训练组获得男性训练样品的覆盖率信息(如,序列剂量值)开始。参见框1002。方法1000然后涉及使用针对框804描述的任何方法计算训练样品的胎儿分数。参见框1004。
[0453]
方法1000还涉及从每个训练样品的读取获得8聚体的频率(如,8个位置处的4个核苷酸的所有可能诱变)。参见框1006。在一些实施方式中,获得高达65,536个或接近那么多的8聚体和它们的频率。在一些实施方式中,获得8聚体的频率而没有对框1123中所示的因素进行归一化。参见图2j的途径1124。在一些实施方式中,在任选地进行图2j的框1123中所示的归一化操作之后,获得8聚体频率。
[0454]
每个8聚体与训练组中的样品的频率相关联。因此,对于每个8聚体,可以在样品的8聚体频率和样品的胎儿分数之间获得相关性。方法1000涉及获得所有8聚体的胎儿分数和8聚体频率之间的相关性。参见框1008。然后,该方法选择具有高于阈值的相关性值的8聚体。参见框1010。目的是鉴定8聚体,其证明训练样品中的8聚体频率和胎儿分数之间的高相关性。然后箱可用于预测测试样品中的胎儿分数。虽然训练样品是男性样品,但胎儿分数与8聚体频率之间的相关性可以推广到男性和女性测试样品。
[0455]
使用具有高相关性值的所选8聚体,该方法获得使胎儿分数与8聚体频率相关联的线性模型。参见框1012。每个选定的箱为线性模型提供自变量。因此,所获得的线性模型还包括每个箱的参数或权重。在获得线性模型之后,方法1000涉及将测试样品的8聚体频率数据应用于模型以确定测试样品的胎儿分数。参见框1014。
[0456]
返回图2e,在一些实施方式中,方法700涉及在操作736中使用基于由操作726提供的所有片段的覆盖率的t
‑
统计量、由操作726提供的胎儿分数估值和基于由操作730提供的短片段的覆盖率的t
‑
统计量,获得最终倍性似然性。这些实施方式使用多元正态模型组合来自通路1和通路2的结果。在用于评估cnv的一些实施方式中,倍性似然性是非整倍性似然性,其是具有非整倍体假设(如,三体或单体)的模型的似然性减去具有整倍体假设的模型的似然性,其中该模型使用基于所有片段的覆盖率的t
‑
统计量、胎儿分数估值以及基于短片段的覆盖率的t
‑
统计量作为输入并提供似然性作为输出。
[0457]
在一些实施方式中,倍性似然性表示为似然比。在一些实施方式中,将似然比建模为:
[0458][0459]
其中p1表示数据来自代表3拷贝或1拷贝模型的多元正态分布的似然性,p0表示数据来自代表2拷贝模型的多元正态分布的似然性,t
短
、t
所有
是从由短片段和所有片段产生的染色体覆盖率计算的t评分,而q(ff
总
)是考虑与胎儿分数估计相关的误差的的胎儿分数密度分布(从训练数据估计)。该模型组合了由短片段产生的覆盖率和由所有片段产生的覆盖率,这有助于提高受影响和未受影响的样品的覆盖率评分之间的分离。在所描绘的实施方案中,该模型也利用胎儿分数,从而进一步提高了区分受影响和未受影响的样品的能力。这里,如上所述使用基于所有片段的覆盖率的t
‑
统计量(726)、基于短片段的覆盖率的t
‑
统计
量(730)以及由方法800(或框726)、900或1000提供的胎儿分数估值来计算似然比。在一些实施方式中,该似然比用于分析13号染色体、18号染色体和21号染色体。
[0460]
一些实施方式,通过操作736获得的倍性似然性仅使用基于通路3的操作734提供的短片段的相对频率以及由操作726、方法800、900或1000提供的胎儿分数估值而获得的t
‑
统计量。似然比可以根据以下等式计算:
[0461][0462]
其中p1表示数据来自代表3拷贝或1拷贝模型的多元正态分布的似然性,p0表示数据来自代表2拷贝模型的多元正态分布的似然性,t
短_freq
是从由短片段的相对频率计算的t评分,而q(ff
总
)是考虑与胎儿分数估计相关的误差的胎儿分数密度分布(根据训练数据估计)。此处,使用基于短片段(734)的相对频率的t
‑
统计量以及由如上所述的方法800(或框726)、900或1000提供的胎儿分数估值来计算似然比。在一些实施方式中,该似然比用于分析x染色体。
[0463]
在一些实施方式中,使用基于所有片段的覆盖率的t
‑
统计量(726)、基于短片段的覆盖率的t
‑
统计量(730)以及短片段的相对频率(734)计算似然比。此外,如上所述获得的胎儿分数可以与t
‑
统计量组合以计算似然比。通过组合来自三个通路713a、713b和713c中的任一个的信息,可以提高倍性评估的辨别能力。参见,如实施例2和图12。在一些实施方式中,可以使用不同的组合来获得染色体的似然比,如来自所有三个通路的t统计量、来自第一通路和第二通路的t统计量、胎儿分数和三个t
‑
统计量、胎儿分数和一个t统计量等。然后,可以基于模型性能选择最佳组合。
[0464]
在用于评估常染色体的一些实施方式中,经建模的似然比表示相对于已经从二倍体样品获得的经建模的数据的似然比的已经从三体或单体样品获得的经建模的数据的似然比。在一些实施方案中,此类似然比可用于确定常染色体的三体或单体。
[0465]
在用于评估性染色体的一些实施方式中,评估x单体的似然比和x三体的似然比。此外,也评估了x染色体和y染色体的染色体覆盖率测量(如,cnv或覆盖率z评分)。在一些实施方式中,使用决策树来确定性染色体的拷贝数以评估这四个值。在一些实施方式中,决策树允许确定xx、xy、x、xxy、xxx或xyy的倍性情况。
[0466]
在一些实施方式中,似然比被转换成log似然比,并且用于调用非整倍性或拷贝数变异的标准或阈值可以凭经验设定以获得特定的灵敏度和选择性。例如,当应用于训练组时,可以基于模型的灵敏度和选择性来设定用于调用13三体或18三体的log似然比为1.5。此外,例如,在某些应用中,可以将21号染色体的三体的调用标准值设定为3。
[0467]
确定序列覆盖率的示例性方法的细节
[0468]
图3a显示了用于降低来自测试样品的序列数据中的噪声的方法301的实例。图3b
‑
3j给出了该方法的各个阶段的数据分析。这提供了可以在图2d中描绘的多通路方法中使用的方法流程的一个实例。
[0469]
图3a所示的方法301使用基于序列标签数量的序列标签覆盖率来评估拷贝数。然而,类似于参考图1的上面关于用于确定cnv的方法100的描述,可以使用其它变量或参数,诸如尺寸、尺寸比和甲基化水平来代替方法400的覆盖率。在一些实施方式中,两个或更多
个变量可分别经历相同的方法以得出指示cnv概率的两个评分,如上面参考图2d所示。然后可以组合两个评分以确定cnv。此外,覆盖率和其它参数可以基于从中得到标签的片段的尺寸来加权。为了便于读取,在方法300中仅涉及覆盖率,但是应当注意,可以使用其它参数,诸如尺寸、尺寸比和甲基化水平、按尺寸加权的计数等来替代覆盖率。
[0470]
如图3a所示,所描绘的方法开始于从一个或多个样品中提取cfdna。参见框303。适合的提取方法和装置在本文其它地方描述。在一些实施方案中,2013年3月15日提交的美国专利申请no.61/801,126(通过引用整体并入本文)中描述的方法提取cfdna。在一些实施方式中,该装置一起处理来自多个样品的cfdna以提供多路复用文库和序列数据。参见图3a中的框305和307。在一个实施方案中,该装置平行处理来自八个或更多个测试样品的cfdna。如本文其它地方所述,测序系统可以处理经提取的cfdna以产生编码的(如,条形编码的)cfdna片段的文库。测序仪对cfdna文库进行测序以产生非常大量的序列读取。每样品编码允许多路复用样品中的读取的去多路复用(demultiplexing)。八个或更多个样品中的每一个可具有数十万或数百万次读取。该方法可以在图3a中的另外操作之前过滤读取。在一些实施方案中,读取过滤是由测序仪中实施的软件程序实现的质量过滤过程,以过滤掉错误和低质量的读取。例如,illumina的测序控制软件(scs)与序列和变异同感评估软件(illumina's sequencing control software(scs)and consensus assessment of sequence and variation software)程序通过将由测序反应产生的原始图像数据转换为强度评分、碱基调用、质量评分的比对和另外的格式来过滤掉错误和低质量的读取,从而为下游分析提供生物相关的信息。
[0471]
在测序仪或其它装置产生样品的读取之后,该系统的元件将读取与参考基因组经计算地比对。参见框309。比对在本文其它地方描述。比对产生标签,所述标签含有带有注释的位置信息的读取序列,该信息指定参考基因组上的唯一位置。在某些实施方式中,该系统进行第一遍比对而不考虑重复读取(具有相同序列的两个或更多个读取)并且随后去除重复读取或将重复的读取计数为单个读取产生非重复的序列标签。在其它实施方式中,该系统不去除重复的读取。在一些实施方案中,该方法考虑去除了与基因组上的多个位置比对的读取,以产生唯一比对的标签。在一些实施方案中,将定位至非排除的位点(nes)的唯一比对的非冗余序列标签考虑在内,以产生非排除的位点计数(nes计数),其提供估计覆盖率的数据。
[0472]
如其它地方所解释,排除的位点是在参考基因组的区域中存在的位点,其为了对序列标签进行计数而被排除在外。在一些实施方案中,排除的位点存在于含有重复序列的染色体区域(如着丝粒和端粒)中,以及对于多于一种染色体是共有的染色体区域(如y染色体上存在的且也存在于x染色体上的区域)中。非排除的位点(nes)是为了对序列标签进行计数的目的在参考基因组中不排除的位点。
[0473]
接下来,该系统将比对的标签分成参考基因组上的箱。参见框311。箱沿参考基因组的长度间隔开。在一些实施方案中,整个参考基因组被分成连续的箱,其可以具有限定的相等尺寸(如,100kb)。或者,箱可能基于每个样品,可以具有动态确定的长度。测序深度影响最佳箱选择。动态尺寸的箱可以具有由文库尺寸确定的尺寸。例如,可以将箱尺寸确定为平均容纳1000个标签所需的序列长度。
[0474]
每个箱都有来自正在考虑的样品的一定数量的标签。反映比对序列的
″
覆盖率
″
的
这一数量的标签用作过滤和另外的清理样品数据的起点,以可靠地确定样品中的拷贝数变异。图3a显示了框313至321中的清理操作。
[0475]
在图3a所描绘的实施方案中,该方法将掩码应用于参考基因组的箱。参见框313。在以下一些或所有方法操作中,该系统可以排除经掩蔽的箱中的覆盖率。在许多情况下,来自经掩蔽的箱的覆盖率值不被认为是图3a中的任何剩余操作。
[0476]
在各种实施方式中,应用一个或多个掩码以去除被发现展现出样品至样品的高变异性的基因组区域的箱。为目标染色体(如chr13、18和21)和其它染色体提供此类掩码。如在其它地方所解释,目标染色体是所考虑可能具有拷贝数变异或其它畸变的染色体。
[0477]
在一些实施方式中,使用以下方法从合格样品的训练组中鉴别掩码。最初,根据图3a中的操作315至319对每个训练组样品进行处理和过滤。然后记录每个箱的归一化和校正的覆盖量,并且计算每个箱的统计量,诸如标准偏差、绝对中值偏差和/或变异系数。可以针对每个目标染色体评估各种过滤器组合。过滤器组合为目标染色体的箱提供一个过滤器,并且为所有其它染色体的箱提供不同的过滤器。
[0478]
在一些实施方式中,在获得掩码(例如,通过选择如上所述的目标染色体的截断)之后,重新考虑归一化染色体(或一组染色体)的选择。在应用序列掩码之后,可以如本文其它地方所述进行选择一个或多个归一化染色体的方法。例如,将染色体的所有可能组合评估为归一化染色体并根据它们区分受影响和未受影响的样品的能力进行排序。该方法可能(或可能不)找到不同的最佳归一化染色体或染色体组。在其它实施方案中,归一化染色体是导致跨所有合格样品的目标序列的序列剂量的最小变异性的那些。如果鉴定出不同的归一化染色体或染色体组,则该方法任选地执行上述箱的鉴定以进行过滤。可能一个或多个新的归一化染色体产生不同的截断。
[0479]
在某些实施方案中,对y染色体应用不同的掩码。适合的y染色体掩码的实例描述于2013年6月17日提交的美国临时专利申请no.61/836,057[代理人案号artep008p]中,其出于所有目的通过引用并入本文。
[0480]
在该系统对箱进行经计算地掩蔽之后,它对未被掩码排除的箱中的覆盖率值进行经计算地归一化。参见框315。在某些实施方案中,该系统针对参考基因组或其一部分中的大部分或全部覆盖率(如,参考基因组的稳定染色体中的覆盖率),对每个箱中的测试样品覆盖率值(如,nes计数/箱)进行归一化。在一些情况下,该系统通过将所考虑的箱的计数除以与参考基因组中的所有稳定染色体比对的所有未排除的位点的总数,对测试样品覆盖率值(每箱)进行归一化。在一些实施方案中,系统通过执行线性回归来对测试样品覆盖率值(每箱)进行归一化。例如,系统首先计算稳定染色体中的箱亚组的覆盖率为y
a
=截距+斜率*gwp
a
,其中y
a
是箱a的覆盖率,gwp
a
是同一箱的全局谱。然后该系统将归一化的覆盖率z
b
计算为:z
b
=y
b
/(截距+斜率*gwp
b
)
‑
1。
[0481]
如上所解释,稳定染色体是不太可能是非整倍体的染色体。在某些实施方案中,稳定染色体均为13号染色体、18号染色体和21号染色体以外的常染色体。在一些实施方案中,稳定染色体为除了确定偏离正常的二倍体基因组的染色体之外的所有常染色体。
[0482]
箱的转换计数值或覆盖率被称为
″
归一化覆盖量
″
以供进一步处理。使用每个样品唯一的信息进行归一化。通常,没有使用来自训练组的信息。归一化允许来自具有不同文库尺寸(并且因此不同的读取数量和标签数量)的样品的覆盖量在平等的基础上被处理。随后
的一些方法操作使用来源于训练样品的覆盖量,其可以是从比所考虑的测试样品所用的文库更大或更小的文库进行测序。在没有基于与整个参考基因组(或至少稳定染色体)比对的读取的数量进行归一化的情况下,在一些实施方式中使用来源于训练组的参数的处理可能不是可靠的或可推广的。
[0483]
图3b显示了许多样品的跨21号染色体、13号染色体和18号染色体的覆盖率。以彼此不同的方式处理一些样品。结果,可以在任何给定的基因组位置看到广泛的样品
‑
至
‑
样品变异。归一化去除了一些样品
‑
至
‑
样品变异。图3c的左图描绘了跨整个基因组的归一化覆盖量。
[0484]
在图3a的实施方案中,该系统从操作315中产生的归一化覆盖量中去除或减少
″
全局谱
″
。参见框317。该操作去除了由基因组结构、文库生成方法和测序方法产生的归一化覆盖量中的系统偏差。此外,该操作经设计以校正与任何给定样品中的预期谱的任何系统线性偏差。
[0485]
在一些实施方式中,全局谱去除涉及将每个箱的归一化覆盖量除以每个箱的相应的预期值。在其它实施方案中,全局谱去除涉及从每个箱的归一化覆盖量中减去每个箱的预期值。预期值可以从未受影响的样品(或x染色体的未受影响的女性样品)的训练组获得。未受影响的样品是来自已知对于目标染色体不具有非整倍性的个体的样品。在一些实施方式中,全局谱去除涉及从每个箱的归一化覆盖量中减去每个箱的预期值(从训练组获得)。在一些实施方案中,该方法使用如使用训练组确定的每个箱的归一化覆盖量的中值。换言之,中值是预期值。
[0486]
在一些实施方案中,全局谱去除是使用针对样品覆盖率对全局谱的依赖性的线性校正实现的。如所指出的,全局谱是如从训练组确定的每个箱的预期值(例如每个箱的中值)。这些实施方案可采用通过将测试样品的归一化覆盖量针对对每个箱获得的总体中值谱拟合而获得的稳定线性模型。在一些实施方案中,线性模型是通过将样品的观察到的归一化覆盖量针对总体中值(或其它期望值)谱回归而获得的。
[0487]
线性模型基于以下假设:样品覆盖量与全局谱值具有线性关系,所述线性关系对于稳定染色体/区域和目标序列两者应当都适用。参见图3d。在这种情况下,对全局谱的预期覆盖量进行的样品归一化覆盖量的回归将产生具有斜率和截距的线。在某些实施方案中,此类线的斜率和截距用于从箱的全局谱值计算
″
预测的
″
覆盖量。在一些实施方式中,全局谱校正涉及通过箱的预测覆盖量来对每个箱的归一化覆盖量进行建模。在一些实施方式中,通过以下调整测试序列标签的覆盖率:(i)获得测试序列标签的覆盖率相对于一个或多个稳定染色体或区域中的多个箱中的预期覆盖率之间的数学关系,以及(ii)将数学关系应用于目标序列中的箱。在一些实施方式中,使用来自未受影响的训练样品的预期覆盖率值和稳定染色体或基因组的其它稳定区中的测试样品的覆盖率值之间的线性关系来校正测试样品中的覆盖率的变异。调整导致全局谱校正的覆盖率。在一些情况下,调整涉及获得稳定染色体或区域中的箱亚组的测试样品的覆盖率,如下:
[0488]
y
a
=截距+斜率*gwp
a
[0489]
其中y
a
是一个或多个稳定染色体或区域中的测试样品的箱a的覆盖率,并且gwp
a
是未受影响的训练样品的箱的全局谱。然后该方法将目标序列或区域的全局谱校正的覆盖率zb计算为:
[0490]
z
b
=y
b
/(截距+斜率*gwp
b
)
–1[0491]
其中y
b
是所观察到的目标序列中的测试样品的箱b的覆盖率(其可以驻留在稳定的染色体或区域之外),并且gwp
b
是未受影响的训练样品的箱的全局谱。分母(截距+斜率*gwp
b
)是箱b的覆盖率,其基于从基因组的稳定区域估计的关系,其预测在未受影响的测试样品中观察到。在具有拷贝数变异的目标序列的情况下,箱b的观察到的覆盖率以及因此全局谱校正的覆盖率值将显著偏离未受影响的样品的覆盖率。例如,在受影响的染色体上的箱的三体样品的情况下,校正的覆盖率z
b
将与胎儿分数成比例。这一方法通过计算稳定染色体的截距和斜率来在样品内归一化,然后评估目标基因组区域如何偏离同一样品内稳定染色体保持的关系(如通过斜率和截距所述)。
[0492]
斜率和截距是从如图3d中所示的线获得的。全局谱去除的一个实例如图3c所描绘。左图显示了跨许多样品的归一化覆盖量的高的箱间变异。右图显示了在如上所述的全局谱去除之后相同的归一化覆盖量。
[0493]
在系统去除或减少在框317上的全局谱变异后,其校正了样品内gc(鸟嘌呤
‑
胞嘧啶)含量变异。参见框319。每个箱都有自己的gc分数贡献。通过将箱中的g和c核苷酸的数量除以箱中的核苷酸的总数(如,100,000)来确定分数。一些箱的gc分数比其它的箱更高。如图3e和3f所示,不同的样品表现出不同的gc偏差。这些差异及其校正将在下面进一步解释。图3e
‑
g显示了全局谱校正的、归一化的覆盖量(每箱)作为gc分数(每箱)的函数。令人惊讶的是,不同的样品表现出不同的gc依赖性。一些样品显示单调减少的依赖性(如图3e中),而其它样品表现出逗号形状的依赖性(如图3f和3g中)。因为这些谱对于每个样品可以是唯一的,所以在该步骤中描述的校正是针对每个样品单独且唯一地进行的。
[0494]
在一个实施方案中,该系统基于gc分数经计算地排列箱,如图3e
‑
g所示。然后,它使用来自具有相似gc含量的其它箱的信息来校正箱的全局谱校正的、归一化的覆盖量。这一校正适用于每个未掩蔽的箱。
[0495]
在一些方法中,以下列方式校正每个箱的gc含量。该系统经计算地选择具有类似于所考虑的箱的那些gc分数的gc分数的箱,然后根据所选箱中的信息确定校正参数。在一些实施方案中,使用任意定义的相似性截断来选择具有相似gc分数的那些箱。在一个实例中,选择所有箱的2%。这些箱是与所考虑的箱最相似的具有gc 含量箱的2%。例如,选择具有略多gc含量的箱的1%和具有略少gc含量的1%。
[0496]
使用所选择的箱,该系统经计算地确定校正参数。在一个实例中,校正参数是所选箱中的归一化覆盖量(在全局谱去除之后)的代表值。此类代表值的实例包括所选箱中的归一化覆盖量的中值或平均值。该系统将所考虑的箱的经计算的校正参数应用于所考虑的箱的归一化覆盖量(在全局谱去除之后)。在一些实施方式中,从所考虑的箱的归一化覆盖量中减去代表值(如,中值)。在一些实施方案中,仅使用稳定常染色体的覆盖量(除13号染色体、18和21之外的所有常染色体)选择归一化覆盖量的中值(或其它代表值)。
[0497]
在使用如100kb箱的一个实例中,每个箱将具有唯一的gc分数值,并且该箱基于它们的gc分数含量被分成组。例如,该箱被分成50组,其中组边界对应于%gc分布的(0、2、4、6和100)分位数。计算来自定位至相同gc组(在样品中)的稳定常染色体的每组箱的中值归一化覆盖量,然后从归一化覆盖量中减去中值(对于在同一gc组中的跨整个基因组的所有箱)。这将从任何给定样品内的稳定染色体估计的gc校正应用于同一样品中可能受影响的
染色体。例如,将gc含量在0.338660和0.344720之间的稳定染色体上的所有箱分组在一起,计算该组的中值并从该gc范围内的箱的归一化覆盖率中减去,所述箱可以是在基因组的任何地方找到(不包括13号染色体、18号染色体、21号染色体和x染色体)。在某些实施方案中,y染色体被排除在gc校正方法之外。
[0498]
图3g显示了使用中值归一化覆盖量作为刚刚描述的校正参数的gc校正的应用。左图显示了未校正的覆盖量相对于gc分数谱。如所示,该谱具有非线性形状。右图显示了经校正的覆盖量。图3h显示了在gc分数校正之前(左图)和gc分数校正之后(右图)的许多样品的归一化覆盖率。图3i显示了在gc分数校正之前(红色)和gc分数校正之后(绿色)的许多测试样品的归一化覆盖率的变异系数(cv),其中gc校正导致归一化的覆盖率中的实质上更小的变异。
[0499]
上述方法是gc校正的相对简单的实施方式。用于校正gc偏差的替代方法采用样条(spline)或其它非线性拟合技术,其可以应用于连续gc空间并且不涉及通过gc含量对覆盖量进行分箱。适合的技术的实例包括持续的勒斯(loess)校正和平滑样条校正。拟合函数可以来源于逐个箱归一化覆盖量相对于所考虑的样品的gc含量。通过将考虑到箱的gc含量应用于拟合函数来计算每个箱的校正。例如,归一化覆盖量可以通过减去在所考虑的箱的gc含量中的样条的预期覆盖率值来调整。或者,可以通过根据样条拟合划分预期的覆盖率值来实现调整。
[0500]
在操作319中校正gc依赖性之后,该系统经计算地去除所考虑的样品中的异常值箱
‑
参见框321。这一操作可以被称为单个样品过滤或修整。图3j显示了即使在gc校正之后,覆盖率在小区域内仍具有样品特异性变异。参见例如12号染色体上的位置1.1e8处的覆盖率,其中出现与预期值的意外高偏差。这种偏差可能是由于材料基因组中的小拷贝数变异引起的。或者,这可能是由于测序中与拷贝数变异无关的技术原因导致的。通常,这一操作仅适用于稳定染色体。
[0501]
作为一个实例,系统经计算地过滤具有gc校正的归一化覆盖量的任何箱,其与跨染色体中的所有箱(具有考虑用于过滤的箱)的经gc校正的归一化覆盖量的中值相差超过3个中值绝对偏差。在一个实例中,截断被定义为经调整与标准偏差一致的3个中值绝对偏差,因此实际上截断是与中值的1.4826*绝对偏差。在某些实施方案中,该操作应用于样品中的所有染色体,包括稳定染色体和疑似有非整倍性的染色体。
[0502]
在某些实施方式中,执行可以表征为质量控制的另外操作。参见框323。在一些实施方案中,质量控制度量涉及检测任何潜在的分母染色体,即
″
归一化染色体
″
或
″
稳定染色体
″
是否是非整倍体或另外地不适合于确定测试样品是否在目标序列中具有拷贝数变异。当该方法确定稳定染色体是不适当的时,该方法可以忽略测试样品并且不做识别。或者,这一qc度量的失败可能触发使用一组替代的归一化染色体进行调用。在一个实例中,质量控制方法将稳定染色体的实际归一化覆盖率值与稳定常染色体的期望值进行比较。期望值可以通过以下来获得:将多元正态模型拟合到未受影响的训练样品的归一化谱,根据数据的似然性性或贝叶斯标准(bayesian criteria)选择最佳模型结构(如,使用赤池(akaike)信息标准或可能的贝叶斯信息标准选择模型)并修正用于qc的最佳模型。稳定染色体的正态模型可以通过例如使用聚类技术来获得,该聚类技术鉴定具有正常样品中的染色体覆盖率的平均值和标准偏差的概率函数。当然,可以使用其它模型形式。该方法评估了在被给予固
定模型参数的任何输入测试样品中观察到的归一化覆盖率的似然性。它可以通过用该模型对每个输入测试样品进行评分来获得似然性,并从而鉴定了相对于正常样品组的异常值。测试样品的似然性与训练样品的似然性的偏差可能表明归一化染色体的异常或可能导致不正确的样品分类的样品处理/测定伪影(artifact)。这一qc度量可用于减少与这些样本伪影中的任一个相关联的分类中的误差。图3k,右图,在x轴上显示染色体数量,并且y轴显示基于与如上所述获得的qc模型的比较的归一化染色体覆盖率。该图显示了对于2号染色体具有过大覆盖率的一个样品和对于20号染色体具有过大覆盖率的其它样品。这些样品将使用此处描述的qc度量消除或转向使用一组替代的归一化染色。图3k的左图显示了ncv相对于染色体的似然性。
[0503]
图3a中描绘的序列可用于基因组中所有染色体的所有箱。在某些实施方案中,将不同的方法应用于y染色体。为计算染色体或区段剂量、ncv和/或nsv,使用来自剂量、ncv和/或nsv中所用的染色体或区段中的箱的经校正的归一化覆盖量(如图3a中所确定)。参见框325。在某些实施方案中,平均归一化覆盖量是从目标染色体、归一化染色体、目标区段中的所有箱计算的,和/或归一化区段用于计算如本文其它地方所述的序列剂量、ncv和/或nsv。
[0504]
在某些实施方案中,将y染色体差异处理。可以通过掩蔽y染色体特有的一组箱来将其过滤。在一些实施方案中,y染色体过滤器根据美国临时专利申请no.61/836,057中的方法确定,该美国临时专利申请先前通过引用并入。在一些实施方案中,过滤器掩码箱小于其它染色体的过滤器中的那些。例如,y染色体掩码可以在1kb水平下进行过滤,而其它染色体掩码可以在100kb水平下进行过滤。然而,y染色体可以在与其它染色体相同的箱尺寸(如,100kb)下归一化。
[0505]
在某些实施方案中,将经过滤的y染色体如上文在图3a的操作315中所述进行归一化。然而,另外,不进一步校正y染色体。因此,不使y染色体箱进行全局谱去除。类似地,不使y染色体箱进行gc校正或之后执行的其它过滤步骤。这是因为当处理样品时,该方法不知道样品是男性还是女性。女性样品应该没有与y参考染色体比对的读取。
[0506]
创建序列掩码
[0507]
本文公开的一些实施方案采用使用序列掩码在目标序列上滤除(或掩蔽)非判别式序列读取的策略,这导致在用于cnv评估的覆盖率值中的相对于通过常规方法计算的值更高的信号和更低的噪声。可以通过各种技术来鉴定此类掩码。在一个实施方案中,使用图4a
‑
4b中所示的技术鉴定掩码,如下面进一步详细说明。
[0508]
在一些实施方式中,使用已知具有目标序列的正常拷贝数的代表性样品的训练组来鉴定掩码。可以使用一项这样的技术来鉴定掩码,所述技术首先归一化训练组样品,然后校正跨序列范围(例如,谱)的系统变异,然后如下所述校正它们的gc变异性。归一化和校正是对来自训练组的样品而不是测试样品进行的。将掩码鉴定一次,然后应用于许多测试样品。
[0509]
图4a显示了用于创建这样的序列掩码的方法400的流程图,其出于拷贝数评估的考虑可以应用于一个或多个测试样品以去除目标序列上的箱。图4中所示的方法400使用基于序列标签数量的序列标签覆盖率以获得序列掩码。然而,类似于参考图1的上面关于用于确定cnv的方法100的描述,除了方法400的覆盖率之外或代替方法400的覆盖率,可使用其
它变量或参数,诸如尺寸、尺寸比和甲基化水平。在一些实施方式中,可产生两个或更多个参数的一个掩码。此外,覆盖率和其它参数可以基于从中得到标签的片段的尺寸来加权。为了便于读取,在方法400中仅涉及覆盖率,但是应当注意,可以使用其它参数,诸如尺寸、尺寸比和甲基化水平、按尺寸加权的计数等来替代覆盖率。
[0510]
方法400通过提供包含来自多个未受影响的训练样品的序列读取的训练组开始。框402。然后,该方法将训练组的序列读取与包含目标序列的参考基因组比对,从而为训练样品提供训练序列标签。框404。在一些实施方案中,仅将定位至非排除的位点的唯一比对的非冗余标签用于进一步分析。该方法涉及将参考基因组分成多个箱,并且针对每个未受影响的训练样品确定在每个训练样品的每个箱中的训练序列标签的覆盖率。框406。该方法还针对每个箱确定跨所有训练样品的训练序列标签的预期覆盖率。框408。在一些实施方案中,每个箱的预期覆盖率是跨训练样品的中值或平均值。预期的覆盖率构成全局谱。然后该方法通过去除全局谱中的变异来调整每个训练样品的每个箱中的训练序列标签的覆盖率,从而获得每个训练样品的箱中的训练序列标签的全局谱校正的覆盖率。然后该方法创建序列掩码,其包括跨参考基因组的未掩蔽和经掩蔽的箱。每个经掩蔽的箱具有超过掩蔽阈值的分布特性。为跨训练样品的箱中的训练序列标签的经调整的覆盖率提供分布特征。在一些实施方式中,掩蔽阈值可以涉及在跨训练样品的箱内的归一化覆盖率中的观察到的变异。可以基于各个度量的经验分布,鉴定具有跨样品的高的变异系数或归一化覆盖率的中值绝对偏差的箱。在一些可选的实施方式中,掩蔽阈值可以涉及在跨训练样品的箱内的归一化覆盖率中的观察到的变异。可以基于各个度量的经验分布,掩蔽具有跨样品的高的变异系数或归一化覆盖率的中值绝对偏差的箱。
[0511]
在一些实施方式中,为目标染色体和所有其它染色体定义了用于鉴定经掩蔽的箱的单独截断,即掩蔽阈值。此外,可以分别为每个目标染色体定义单独的掩蔽阈值,并且对于所有未受影响的染色体的组定义单个的掩蔽阈值。作为一个实例,为13号染色体定义基于某个掩蔽阈值的掩码,并且使用另一个掩蔽阈值来定义其它染色体的掩码。未受影响的染色体也可以根据染色体定义其掩蔽阈值。
[0512]
可以评估每个目标染色体的各种掩蔽阈值组合。掩蔽阈值组合为目标染色体的箱提供一个掩码,并且为所有其它染色体的箱提供不同的掩码。
[0513]
在一种方法中,变异系数(cv)或样品分布截断的度量的值范围被定义为箱cv值的经验分布的百分位数(如,95、96、97、98、99),并且这些截断适用于除目标染色体之外的所有常染色体。此外,cv的百分位数截断的范围被定义为经验cv分布,并且这些截断被应用于目标染色体(如,chr 21)。在一些实施方案中,目标染色体是x染色体和13号染色体、18和21。当然,可以考虑其它方法;例如,可以对每个染色体进行单独的优化。总之,待平行优化的范围(如,所考虑的目标染色体的一个范围和所有其它染色体的另一个范围)定义了cv截断组合的网格。参见图4b。跨两个截断评估该系统对训练组的性能(一个用于归一化染色体(或除目标染色体之外的常染色体)和一个用于目标染色体),并且最佳表现组合被选择用于最终配置。对于每种目标染色体,这种组合可以是不同的。在某些实施方案中,性能是对验证组而不是训练组评估的,即交叉验证用于评估性能。
[0514]
在一些实施方案中,经优化用于确定截断范围的性能是染色体剂量的变异系数(基于归一化染色体的暂定选择)。该方法选择使用当前选择的归一化染色体(或多条染色
体)使目标染色体的染色体剂量(如,比率)的cv最小化的截断的组合。在一种方法中,该方法如下测试网格中每个截断组合的性能:(1)应用截断组合来定义所有染色体的掩码并应用那些掩码来过滤训练组的标签;(2)通过将图3a的方法应用于过滤标签,计算跨未受影响的样品的训练组的归一化覆盖率;(3)通过如对所考虑的染色体的箱归一化覆盖率求和,确定每个染色体的代表性归一化覆盖率;(4)使用当前归一化染色体计算染色体剂量,以及(5)确定染色体剂量的cv。该方法可以通过将它们应用于与训练组的原始部分分离的一组测试样品来评估所选过滤器的性能。也就是说,方法将原来的训练组分成训练亚组和测试亚组。如上所述,训练亚组用于定义掩码截断。
[0515]
在可选的实施方案中,代替基于覆盖率的cv定义掩码,可以通过来自跨箱内的训练样品的比对结果的定位质量评分的分布来定义掩码。定位质量评分反映了读取定位至参考基因组的唯一性。换句话说,定位质量评分量化了读取未比对的概率。低定位质量评分与低唯一性(未比对的高概率)相关联。唯一性考虑了读取序列中的一个或多个错误(由测序仪生成)。定位质量评分的详述呈现于li h,ruan j,durbin r.(2008)mapping short dna sequencing reads and calling variants using mapping quality scores.genome research 18:1851
‑
8中,其通过引用整体并入本文。在一些实施方式中,本文的定位质量评分被称为mapq评分。图4b显示了mapq评分与经处理的覆盖率的cv具有强烈的单调相关性。例如,cv高于0.4的箱几乎完全聚类在图4b中的图的左侧,其具有低于约4的mapq评分。因此,具有小mapq的掩蔽箱可以产生与由具有高cv的掩蔽箱定义的掩码非常相似的掩码。
[0516]
样品和样品处理
[0517]
样品
[0518]
用于确定cnv(如染色体非整倍性、部分非整倍性等)的样品可包括取自其中一条或多条目标序列的拷贝数变异待确定的任何细胞、组织或器官的样品。理想地,样品含有存在于细胞中的核酸和/或为
″
无细胞
″
的核酸(如cfdna)。
[0519]
在一些实施方案中,有利的是获得无细胞核酸,如无细胞dna(cfdna)。无细胞核酸,包括无细胞dna,可以通过本领域已知的各种方法从包括但不限于血浆、血清和尿液的生物样品中获得(参见,如,fan等人,proc natl acad sci 105:16266
‑
16271[2008];koide等人,prenatal diagnosis 25:604
‑
607[2005];chen等人,nature med.2:1033
‑
1035[1996];lo等人,lancet 350:485
‑
487[1997];botezatu等人,clin chem.46:1078
‑
1084,2000;和su等人,j mol.diagn.6:101
‑
107[2004])。为了从样品中的细胞中分离无细胞dna,可以使用各种方法包括但不限于分级分离、离心(如,密度梯度离心)、dna特异性沉淀或高通量细胞分选和/或其它分离方法。可获得用于手动和自动分离cfdna的可商购获得的试剂盒(roche diagnostics,indianapolis,in,qiagen,valencia,ca,macherey
‑
nagel,duren,de)。包含cfdna的生物样品已经用于测定中以通过可以检测染色体非整倍性和/或各种多态性的测序测定来确定染色体异常(如,21三体)的存在或缺失。
[0520]
在各种实施方案中,样品中存在的cfdna可以在使用前(如,在制备测序文库之前)特异性地富集或非特异性地富集。样品dna的非特异性富集是指在制备cfdna测序文库之前可用于提高样品dna水平的样品基因组dna片段的全基因组扩增。非特异性富集可以是包含多于一个基因组的样品中存在的两个基因组之一的选择性富集。例如,非特异性富集可以选择母体样品中的胎儿基因组,其可以通过增加样品中胎儿与母体dna的相对比例的已知
方法获得。可替代地,非特异性富集可以是样品中存在的两种基因组的非选择性扩增。例如,非特异性扩增可以是包含来自胎儿和母体基因组的dna的混合物的样品中的胎儿和母体dna的。全基因组扩增的方法是本领域已知的。简并寡核苷酸引发的pcr(dop)、引物延伸pcr技术(pep)和多重置换扩增(mda)是全基因组扩增方法的实例。在一些实施方案中,针对包含来自不同基因组的cfdna的混合物的样品,未富集混合物中存在的基因组的cfdna。在其它实施方案中,针对包含来自不同基因组的cfdna的混合物的样品,非特异性富集样品中存在的任何一种基因组。
[0521]
例如,如上所述,包含通常应用本文所述方法的一种或多种核酸的样品包括生物样品(
″
测试样品
″
)。在一些实施方案中,通过许多众所周知的方法中的任一种纯化或分离待筛选一种或多种cnv的核酸。
[0522]
因此,在某些实施方案中,该样品包括经纯化或经分离的多核苷酸或由经纯化或经分离的多核苷酸组成,或者它可包括样品,诸如组织样品、生物流体样品、细胞样品等。适合的生物流体样品包括但不限于血液、血浆、血清、汗液、眼泪、痰液、尿液、痰液、耳流液(ear flow)、淋巴液、唾液、脑脊液、ravages、骨髓悬液、阴道流液、经宫颈灌洗液、脑液、腹水、乳汁、呼吸道、肠道和泌尿生殖道的分泌物、羊水、乳汁和白细胞去除法(leukophoresis)样品。在一些实施方案中,该样品是易于通过非侵入性程序获得的样品,如血液、血浆、血清、汗液、泪液、痰液、尿液、痰液、耳流液、唾液或粪便。在某些实施方案中,该样品是外周血样品,或外周血样品的血浆和/或血清级分。在其它实施方案中,生物样品是拭子或涂片、活检样本或细胞培养物。在另一个实施方案中,该样品是两种或更多种生物样品的混合物,如,生物样品可包括两种或更多种生物流体样品、组织样品和细胞培养物样品。如本文所用,术语
″
血液
″
、
″
血浆
″
和
″
血清
″
明确涵盖其级分或加工部分。类似地,当从活检组织、拭子、涂片等取样时,
″
样品
″
明确涵盖来源于活检组织、拭子、涂片等的加工级分或部分。
[0523]
在某些实施方案中,样品可以从来源获得,包括但不限于来自不同个体的样品、来自相同或不同个体的不同发育阶段的样品、来自不同患病个体(如,患有癌症或疑似患有遗传病症的个体)的样品、正常个体、在个体疾病的不同阶段获得的样品、从经受不同疾病治疗的个体获得的样品、来自经受不同环境因素的个体的样品、来自对病理具有易感性的个体的样品、与暴露于传染病因子(如,hiv)的个体等。
[0524]
在一个示例性但非限制性的实施方案中,样品是从妊娠女性(例如孕妇)获得的母体样品。在这种情况下,可以使用本文所述的方法分析样品,以提供胎儿中潜在的染色体异常的产前诊断。母体样品可以是组织样品、生物流体样品或细胞样品。作为非限制性实例,生物流体包括血液、血浆、血清、汗液、泪液、痰液、尿液、痰液、耳流液、淋巴液、唾液、脑脊液、ravages、骨髓悬液、阴道流液、经宫颈灌洗液、脑液、腹水、乳汁、呼吸道、肠道和泌尿生殖道的分泌物,以及白细胞去除法样品。
[0525]
在另一个说明性但非限制性的实施方案中,母体样品是两种或更多种生物样品的混合物,如,生物样品可包括两种或更多种生物流体样品、组织样品和细胞培养物样品。在一些实施方案中,该样品是易于通过非侵入性程序获得的样品,如血液、血浆、血清、汗液、泪液、痰液、尿液、乳汁、痰液、耳流液、唾液和粪便。在一些实施方案中,生物样品是外周血样品,和/或其血浆和血清级分。在其它实施方案中,生物样品是拭子或涂片、活检样本或细
胞培养物样品。如上文所公开,术语
″
血液
″
、
″
血浆
″
和
″
血清
″
明确涵盖其级分或加工部分。类似地,当从活检组织、拭子、涂片等取样时,
″
样品
″
明确涵盖来源于活检组织、拭子、涂片等的加工级分或部分。
[0526]
在某些实施方案中,样品也可以从体外培养的组织、细胞或其它含多核苷酸的来源获得。经培养的样品可以取自各来源,包括但不限于维持在不同培养基和条件(如,ph、压力或温度)中的培养物,维持不同长度的时间段的培养物(如,组织或细胞),用不同因子或试剂(如药物候选物或调节剂)处理的培养物(如组织或细胞),或不同类型组织和/或细胞的培养物。
[0527]
从生物来源分离核酸的方法是众所周知的,并且根据来源的性质而不同。本领域的技术人员可根据本文所述方法的需要从来源中容易地分离一种或多种核酸。在一些情况下,对核酸样品中的核酸分子进行片段化可能是有利的。片段化可以是随机的,或者它可以是特异性的,如例如,使用限制性内切核酸酶消化实现。用于随机片段化的方法是本领域熟知的,并且包括例如有限的dna酶消化、碱处理和物理剪切。在一个实施方案中,样品核酸是作为cfdna获得的,其未经过片段化。
[0528]
测序文库制备
[0529]
在一个实施方案中,本文描述的方法可以利用下一代测量技术(ngs),其允许多个样品作为基因组分子(即,单重(singleplex)测序)或作为包含索引的基因组分子的合并样品(如,多重(multiplex)测序)在单个测序运行中进行单独地测序。这些方法可以产生高达数亿次的dna序列读取。在各种实施方案中,基因组核酸和/或索引的基因组核酸的序列可以使用例如本文所述的下一代测序技术(ngs)来确定。在各种实施方案中,可以使用如本文所述的一个或多个处理器来执行使用ngs获得的大量序列数据的分析。
[0530]
在各种实施方案中,此类测量技术的用途不涉及测量文库的制备。
[0531]
然而,在某些实施方案中,本文考虑的测量方法涉及测序文库的制备。在一个说明性方法中,测序文库制备涉及产生准备进行测序的接头修饰的(adapter
‑
modified)dna片段(如多核苷酸)的随机集合。多核苷酸的测序文库可以通过逆转录酶的作用从dna或rna制备,包括dna或cdna的等效物、类似物,例如dna或cdna,其是互补的或由rna模板产生的拷贝dna。多核苷酸可以起源于双链形式(如,dsdna诸如基因组dna片段、cdna、pcr扩增产物等),或者在某些实施方案中,多核苷酸可能起源于单链形式(如,ssdna、rna等)并已转化为dsdna形式。以实例说明,在某些实施方案中,可以将单链mrna分子复制成适合用于制备测序文库的双链cdna。一级多核苷酸分子的精确序列通常不是文库制备方法的材料,并且可能是已知的或未知的。在一个实施方案中,多核苷酸分子是dna分子。更具体地,在某些实施方案中,多核苷酸分子代表生物体的整个遗传互补物或生物体的基本上整个遗传互补物,并且是基因组dna分子(如,细胞dna、无细胞dna(cfdna)等),其通常包括内含子序列和外显子序列(编码序列),以及非编码调控序列诸如启动子和增强子序列。在某些实施方案中,一级多核苷酸分子包含人基因组dna分子,即存在于妊娠受试者的外周血中的cfdna分子。
[0532]
通过使用包含特定范围的片段尺寸的多核苷酸来促进一些ngs的测序平台的测序文库的制备。此类文库的制备通常涉及大的多核苷酸(如细胞基因组dna)的片段化,以获得所需尺寸范围的多核苷酸。
[0533]
片段化可以通过本领域技术人员已知的许多方法中的任一种来实现。例如,片段
化可以通过机械手段实现,包括但不限于雾化、超声处理和水剪切。然而,机械片段化通常在c
‑
o、p
‑
o和c
‑
c键处裂解dna主链,导致平末端和3'
‑
和5'
‑
突出端与破坏的c
‑
o、p
‑
o和c
‑
c键的异质混合物(参见,如,alnemri和liwack,j biol.chem 265:17323
‑
17333[1990];richards和boyer,j mol biol 11:327
‑
240[1965]),其可能需要修复,因为它们可能缺少用于随后的酶促反应(如制备用于测序的dna所需的测序接头的连接)所必要的5'
‑
磷酸根。
[0534]
相反,cfdna通常以小于约300个碱基对的片段存在,并随后片段化通常不是使用cfdna样品生成测序文库所必需的。
[0535]
通常,无论多核苷酸被强制片段化(如,在体外片段化),还是以片段天然存在,它们都被转化为具有5'
‑
磷酸根和3'
‑
羟基的平末端dna。标准方案,如用于使用例如如本文别处所述的illumina平台进行测序的方案,指导用户末端修复样品dna,以在da加尾之前纯化末端修复的产品,并在文库制备的接头连接步骤之前纯化da加尾产品。
[0536]
本文所述的序列文库制备方法的各种实施方案消除了执行标准方案通常要求的一个或多个步骤以获得可由ngs测序的经修饰的dna产物的需要。缩略方法(abb方法),1步法和2步法是用于制备测量文库的方法的实例,其可以在2012年7月20日提交的专利申请13/555,037中找到,该申请通过引用整体并入。
[0537]
用于跟踪和验证样品完整性的标记核酸
[0538]
在各种实施方案中,样品完整性验证和样品跟踪可以通过在处理之前对样品基因组核酸,如cfdna和附带的标记核酸的混合物(已被引入到样品中)进行测序来完成。
[0539]
可以将标记核酸与测试样品(如生物源样品)组合并经受这样的方法,该方法包括例如以下的一个或多个步骤:对生物源样品进行分级分离,如获得来自全血样品的基本上无细胞的血浆级分,从分级分离(如,血浆)或未分级分离的生物源样品(如组织样品)纯化核酸,并测序。在一些实施方案中,测序包括制备测序文库。选择对于源样品是独特的与源样品组合的标记分子的序列或序列组合。在一些实施方案中,样品中的独特标记分子都具有相同的序列。在其它实施方案中,样品中的独特标记分子是多条序列,如两条、三条、四条、五条、六条、七条、八条、九条、十条、十五条、二十条或更多条不同序列的组合。
[0540]
在一个实施方案中,可以使用具有相同序列的多个标记核酸分子来验证样品的完整性。或者,样品的身份可以使用多个标记核酸分子来验证,所述分子具有至少2条、至少3条、至少4条、至少5条、至少6条、至少7条、至少8条、至少9条、至少10条、至少11条、至少12条、至少13条、至少14条、至少15条、至少16条、至少17m条、至少18条、至少19条、至少20条、至少25条、至少30条、至少35条、至少40条、至少50条或更多条不同的序列。验证多个生物样品(即两种或更多种生物样品)的完整性,要求两种或更多种样品中的每一个用标记核酸标记,所述标记核酸具有对于待标记的多个测试样品中的每一个都是唯一的序列。例如,第一样品可以用具有序列a的标记核酸标记,并且第二样品可以用具有序列b的标记核酸标记。或者,第一样品可以用都具有序列a的标记核酸分子标记并且第二样品可以用序列b和c的混合物标记,其中序列a、b和c是具有不同序列的标记分子。
[0541]
一种或多种标记核酸可以在样品制备的任何阶段添加到样品,该样品制备在文库制备(如果要制备文库)和测序之前发生。在一个实施方案中,标记分子可以与未处理的源样品组合。例如,标记核酸可以在用于收集血液样品的收集管中提供。或者,标记核酸可以在抽血后添加到血液样品。在一个实施方案中,将标记核酸添加到用于收集生物流体样品
的容器中,如将一种或多种标记核酸添加到用于收集血液样品的血液收集管。在另一个实施方案中,将一种或多种标记核酸添加到生物流体样品的级分。例如,将标记核酸添加到血液样品的血浆和/或血清级分,如母体血浆样品。在又一个实施方案中,将标记分子加入到纯化样品,如从生物样品中纯化的核酸样品。例如,将标记核酸添加到经纯化的母体和胎儿cfdna的样品。类似地,可以在处理样本之前将标记核酸添加到活检样本中。在一些实施方案中,标记核酸可以与递送标记分子至生物样品细胞中的载体组合。细胞
‑
递送载体包括ph敏感性脂质体和阳离子脂质体。
[0542]
在某些实施方案中,标记分子具有反基因组(antigenomic)序列,所述反基因组序列是生物源样品的基因组中不存在的序列。在一个示例性实施方案中,用于验证人生物源样品完整性的标记分子具有人基因组中不存在的序列。在一个替代的实施方案中,标记分子具有源样品和任一种或多种其它已知基因组中不存在的序列。例如,用于验证人生物源样品完整性的标记分子具有人基因组和小鼠基因组中不存在的序列。该替代方案允许验证包含两个或更多个基因组的测试样品的完整性。例如,从受病原体(如细菌)感染的受试者获得的人无细胞dna样品的完整性可以使用具有在人基因组和感染细菌的基因组中都不存在的序列的标记分子来验证。许多病原体(如细菌、病毒、酵母、真菌、原生动物等)的基因组的序列可在万维网ncbi.nlm.nih.gov/genomes公开获得。在另一个实施方案中,标记分子是具有任何已知基因组中不存在的序列的核酸。标记分子的序列可以通过算法随机生成。
[0543]
在各种实施方案中,标记分子可以是天然存在的脱氧核糖核酸(dna)、核糖核酸或人工核酸类似物(核酸模拟物),包括肽核酸(pna)、吗啉代核酸、锁定核酸、乙二醇核酸及苏阿糖核酸,所述人工核酸类似物(核酸模拟物)通过改变分子的主链或不具有磷酸二酯主链的dna模拟物来与天然存在的dna或rna相区分。脱氧核糖核酸可以来自天然存在的基因组,或者可以通过使用酶或通过固相化学合成在实验室中生成。化学方法也可用于产生自然界中未发现的dna模拟物。dna的衍生物是可用的,其中磷酸二酯键联被替换但其中脱氧核糖被保留,包括但不限于具有由硫代甲醛或羧酰胺键联形成的主链的dna模拟物,其已显示为良好的结构dna模拟物。其它dna模拟物包括吗啉代衍生物和肽核酸(pna),其含有基于n
‑
(2
‑
氨基乙基)甘氨酸的假肽主链(ann rev biophys biomol struct 24:167
‑
183[1995])。pna是一种非常好的dna(或核糖核酸[rna])结构模拟物,并且pna寡聚体能够与watson
‑
crick互补dna和rna(或pna)寡聚体形成非常稳定的双链体结构,并且它们可以也通过螺旋入侵与双链体dna中的靶标结合(mol biotechnol 26:233
‑
248[2004]。可用作标记分子的dna类似物的另一种良好结构模拟物/类似物是硫代磷酸酯dna,其中非桥接氧中的一个被硫替代。这种修饰减少了内切
‑
和外切
‑
核酸酶2的作用,包括5'至3'和3'至5'dna pol 1外切核酸酶、核酸酶s1和p1、rna酶、血清核酸酶和蛇毒磷酸二酯酶。
[0544]
标记分子的长度可以与样品核酸的长度不同或模糊(indistinct),即标记分子的长度可以与样品基因组分子的长度相似,或者它可以大于或小于样品基因组分子的长度。标记分子的长度通过构成标记分子的核苷酸或核苷酸类似物碱基的数量来测量。可以使用本领域已知的分离方法将具有与样品基因组分子长度不同的长度的标记分子与源核酸区分开。例如,标记和样品核酸分子的长度差异可以通过电泳(如毛细管电泳)分离确定。尺寸分化可以有利于定量和评估标记和样品核酸的质量。优选地,标记核酸比基因组核酸短,并且具有足够的长度以排除它们被定位至样品的基因组。例如,因为需要30个碱基的人序列
将其唯一地定位至人基因组。因此,在某些实施方案中,用于人样品的测序生物测定中的标记分子的长度应为至少30bp。
[0545]
标记分子长度的选择主要由用于验证源样品完整性的测序技术决定。也可以考虑经测序的样品基因组核酸的长度。例如,一些测序技术采用多核苷酸的克隆扩增,这可能要求待克隆扩增的基因组多核苷酸具有最小长度。例如,使用illumina gaii序列分析仪进行的测序包括通过最小长度为110bp的多核苷酸的桥式pcr(也称为簇扩增)进行的体外克隆扩增,所述多核苷酸上连接有接头以提供可经克隆扩增并测序的至少200bp且小于600bp的核酸。在一些实施方案中,接头连接的标记分子的长度为约200bp至约600bp、约250bp至550bp、约300bp至500bp或约350至450。在其它实施方案中,接头连接的标记分子的长度为约200bp。例如,当对存在于母体样品中的胎儿cfdna进行测序时,标记分子的长度可以选择为与胎儿cfdna分子的长度相似。因此,在一个实施方案中,用于包括母体样品中的cfdna的大规模平行测序以确定胎儿染色体非整倍性的存在或缺失的测定中的标记分子的长度可以为约150bp、约160bp、170bp、约180bp、约190bp或约200bp;优选地,标记分子为约170pp。其它测序方法,如,solid测序,polony测序和454测序使用乳液pcr来克隆扩增用于测序的dna分子,并且每种技术决定待扩增的分子的最小和最大长度。作为经克隆扩增的核酸被测序的标记分子的长度可以高达约600bp。在一些实施方案中,待测序的标记分子的长度可以大于600bp。
[0546]
单分子测序技术,不采用分子克隆扩增并且能够在非常宽的模板长度范围内测量核酸,在大多数情况下不要求待测序的分子具有任何特定长度。然而,每单位质量的序列的产率取决于3'末端羟基基团的数量,并因此具有相对短的测序模板比具有长模板更有效。如果以长度超过1000nt的核酸开始,通常建议将核酸剪切至平均长度100至200nt,以便可以从相同质量的核酸产生更多的序列信息。因此,标记分子的长度的范围可以从数十个碱基到数千个碱基。用于单分子测序的标记分子的长度可高达约25bp、高达约50bp、高达约75bp、高达约100bp、高达约200bp、高达约300bp、高达约400bp、高达约500bp、高达约600bp、高达约700bp、高达约800bp、高达约900bp、高达约1000bp或更长。
[0547]
为标记分子选择的长度也由正在测序的基因组核酸的长度决定。例如,cfdna在人血流中作为细胞基因组dna的基因组片段循环。在孕妇血浆中发现的胎儿cfdna分子一般比母体cfdna分子短(chan等人,clin chem 50:8892[2004])。循环胎儿dna的尺寸分级分离证实了循环胎儿dna的平均长度片段<30bp,而母体dna估计在约0.5和1kb之间(li等人,clin chem,50:1002
‑
1011[2004])。这些发现与fan等人的发现一致,他们使用ngs确定胎儿cfdna很少>340bp(fan等人,clin chem56:1279
‑
1286[2010])。用标准二氧化硅基法从尿液中分离的dna由两部分组成,即来自脱落细胞的高分子量dna和经肾dna(tr
‑
dna)的低分子量(150
‑
250个碱基对)部分(botezatu等人,clin chem.46:1078
‑
1084,2000;和su等人,j mol.diagn.6:101
‑
107,2004)。应用新开发的从体液中分离无细胞核酸到分离经肾核酸的技术,揭示了尿液中存在远短于150个碱基对的dna和rna片段(美国专利申请公开号20080139801)。在其中cfdna是经测序的基因组核酸的实施方案中,所选择的标记分子可以达到接近cfdna的长度。例如,作为单个核酸分子或作为经克隆扩增的核酸进行测序的母体cfdna样品中使用的标记分子的长度可以在约100bp至600之间。在其它实施方案中,样品基因组核酸是更大分子的片段。例如,经测序的样品基因组核酸是片段化的细胞dna。在实施
方案中,当片段化的细胞dna经测序时,标记分子的长度可以达到dna片段的长度。在一些实施方案中,标记分子的长度至少是将序列读取唯一地定位至适当的参考基因组所需的最小长度。在其它实施方案中,标记分子的长度是排除标记分子被定位至样品参考基因组所需的最小长度。
[0548]
此外,标记分子可用于验证样品,所述样品不通过核酸测序进行测定并且可通过除测序之外的常用生物技术如实时pcr来验证。
[0549]
样品对照(例如,用于测序和/或分析的方法中阳性对照)。
[0550]
在各种实施方案中,如如上所述引入至样品中的标记序列可以起阳性对照的作用以验证测序以及随后的加工和分析的精确度和功效。
[0551]
因此,提供了用于为样品中的dna进行测序而提供方法中阳性对照(ipc)的组合物和方法。在某些实施方案中,提供了用于对包含基因组混合物的的样品中的cfdna进行测序的阳性对照。ipc可以用于关联从不同的样品组(如在不同的测序运行中在不同时间测序的样品)获得的序列信息中的基线偏移。因此,例如,ipc可以将针对母体测试样品获得的序列信息与从在不同时间测序的一组合格样品获得的序列信息相关联。
[0552]
类似地,在区段分析的情况下,ipc可以将从受试者获得的一个或多个特定区段的序列信息与从在不同时间测序的一组合格样品(类似序列)获得的序列相关联。在某些实施方案中,ipc可以将从受试者获得的特定癌症相关基因座的序列信息与从一组合格样品(例如,已知的扩增/缺失等)获得的序列信息相关联。
[0553]
此外,ipc可用作标记以通过测序方法跟踪一种或多种样品。ipc还可提供定性的阳性序列剂量值,如ncv,用于目标染色体的一个或多个非整倍性(如,21三体、13三体、18三体),以提供正确的解释,并确保数据的可靠性和精确度。在某些实施方案中,可以创建ipc以包含来自男性和女性基因组的核酸,以提供母体样品中x染色体和y染色体的剂量,以确定胎儿是否是男性。
[0554]
方法中对照的类型和数量取决于所需测试的类型或性质。例如,对于需要对包含基因组混合物的样品dna进行测序以确定是否存在染色体非整倍性的测试,方法中对照可以包括从已知包含正在测试的相同染色体非整倍性的样品获得的dna。在一些实施方案中,ipc包含来自已知包含目标染色体的非整倍性的样品的dna。例如,用于确定母体样品中存在或缺失胎儿三体(如21三体)的测试的ipc包含从具有21三体的个体获得的dna。在一些实施方案中,ipc包含从两个或多个具有不同非整倍性的个体获得的dna的混合物。例如,对于确定存在或缺失13三体、18三体、21三体和x单体的测试,ipc包含从各自携带胎儿的孕妇获得的dna样品的组合,其中三体性中的一个被测试。除了完全染色体非整倍性之外,可以创建ipc以提供用于确定部分非整倍性的存在或缺失的测试的阳性对照。
[0555]
用作检测单一非整倍性的对照的ipc可以使用从两名受试者获得的细胞基因组dna的混合物创建,其中一名受试者是非整倍体基因组的贡献者。例如,可以通过将携带三体染色体的男性或女性受试者的基因组dna与已知不携带三体染色体的女性受试者的基因组dna组合,来创建作为用于确定胎儿三体(如,21三体)的测试的对照的ipc。基因组dna可从两名受试者的细胞中提取,并剪切以提供约100
‑
400bp、约150
‑
350bp或约200
‑
300bp的片段,以模拟母体样品中的循环cfdna片段。选择来自携带非整倍性(如21三体)的受试者的片段化dna的比例来模拟在母体样品中发现的循环胎儿cfdna的比例,以提供包含片段化dna
的混合物的ipc,所述dna的混合物包含来自携带非整倍性的受试者的dna的约5%、约10%、约15%、约20%、约25%、约30%。ipc可以包含来自各自携带不同的非整倍性的不同受试者的dna。例如,ipc可以包含约80%的未受影响的女性dna,并且剩余的20%可以是来自各自携带21号染色体三体、13号染色体三体和18号染色体三体的三名不同受试者的dna。制备片段化dna的混合物用于测序。片段化dna的混合物的加工可以包括制备测序文库,其可以使用任何大规模平行方法以单重或多重方式进行测序。基因组ipc的储存溶液可以储存并用于多种诊断性测试中。
[0556]
或者,可以使用从已知携带具有已知染色体非整倍性的胎儿的母亲获得的cfdna来创建ipc。例如,cfdna可以从携带有21三体的胎儿的孕妇获得。将cfdna从母体样品中提取,并克隆到细菌载体中并在细菌中生长以提供ipc的持续来源。可以使用限制酶从细菌载体中提取dna。或者,经克隆的cfdna可以通过如pcr扩增。可以处理ipc dna以用于在与待被分析染色体非整倍性的存在或缺失的测试样品的cfdna相同的运行中进行测序。
[0557]
尽管上面关于三体性描述了ipc的创建,但是应当理解,可以创建ipc以反映其它部分非整倍性,包括例如各种区段扩增和/或缺失。因此,例如,当已知各种癌症与特定的扩增相关联(如,与20q13相关的乳腺癌)时,可以创建掺入那些已知的扩增的ipc。
[0558]
测序方法
[0559]
如上所指示,对所制备的样品(如,测序文库)进行测序,作为用于鉴定一种或多种拷贝数变异的方法的一部分。可以使用许多测序技术中的任一种。
[0560]
一些测序技术可商购获得,诸如来自affymetrix inc.(sunnyvale,ca)的边杂交边测序平台,和来自454life sciences(bradford,ct)、illumina/solexa(hayward,ca)和helicos biosciences(cambridge,ma)的边合成边测序平台,及来自applied biosy stems(foster city,ca)的边连接边平台,如下所述。除了使用helicos biosciences的边合成边测序进行的单分子测序之外,其它单分子测序技术包括但不限于pacific biosciences的smrt
tm
技术,ion torrent
tm
技术,以及由例如oxford nanopore technologies开发的纳米孔测序。
[0561]
虽然自动化桑格(sanger)方法被认为是
″
第一代
″
技术,但包括自动化桑格测序在内的桑格测序也可以用于本文所述的方法中。另外的适合的测序方法包括但不限于核酸成像技术,如原子力显微术(afm)或透射电子显微术(tem)。以下更详细地描述说明性测序技术。
[0562]
在一个说明性但非限制性的实施方案中,本文描述的方法包括使用illumina边合成边测序和基于可逆终止子的测序化学获得测试样品中的核酸(如母体样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息(例如,如bentley等人,nature 6:53
‑
59[2009]中所述)。模板dna可以是基因组dna,如细胞dna或cfdna。在一些实施方案中,来自经分离的细胞的基因组dna用作模板,并且将其片段化为数百个碱基对的长度。在其它实施方案中,cfdna用作模板,并且片段化不是所需的,因为cfdna作为短片段存在。例如胎儿cfdna在血流中以长约170个碱基对(bp)的片段循环(fan等人,clin chem 56:1279
‑
1286[2010]),并且在测序之前不需要dna片段化。illumina的测序技术依赖于片段化的基因组dna与光学透明平面的附接,所述平面上结合了寡核苷酸锚。模板dna被末端修复以产生5'
‑
磷酸化的平末端,并且klenow片段的聚合酶活性用于将单个a碱基添加到平的磷
酸化的dna片段的3'末端。该添加制备dna片段用于连接至寡核苷酸接头,其在3'末端具有单个t碱基的突出以提高连接效率。接头寡核苷酸与流动池锚定寡核苷酸互补(在重复扩增的分析中不要与锚/锚定的读取混淆)。在有限的稀释条件下,将接头修饰的单链模板dna添加到流动池中并通过杂交固定到锚寡聚物。将附接的dna片段延伸并桥接扩增以产生具有数亿个簇的超高密度测序流动池,每个簇含有相同模板的约1,000个拷贝。在一个实施方案中,使用pcr扩增随机片段化的基因组dna,之后使其进行簇扩增。或者,使用无扩增(如,无pcr)基因组文库制备,并仅使用簇扩增来富集随机片段化的基因组dna(kozarewa等人,nature methods 6:291
‑
295[2009])。
[0563]
将模板使用采用可逆终止子与可移除的荧光染料的稳定四色dna边合成边测序技术测序。使用激光激发和全内反射光学器件实现高灵敏度荧光检测。将约数十至几百个碱基对的短序列读取与参考基因组比对,并使用专门开发的数据分析管道软件鉴定短序列读取与参考基因组的唯一定位。完成第一读取后,模板可以原位再生,以便从片段的另一端进行第二读取。因此,可以使用dna片段的单末端或配对末端测序。
[0564]
本公开的各种实施方案可以使用边合成边测序,其允许配对末端测量。在一些实施方案中,illumina的合成平台的测序涉及使片段聚类。聚类是一种其中每个片段分子被等温扩增的方法。在一些实施方案中,作为这里描述的实例,该片段具有两个不同的接头附接至该片段的两端,所述接头允许片段与流动池泳道表面上的两种不同的寡核苷酸杂交。该片段还包括或附接至该片段两端的两条索引序列,所述索引序列提供鉴定多路复用测序中的不同样本的标签。在一些测序平台中,待测序的片段还被称为插入物。
[0565]
在一些实施方式中,用于在illumina平台中聚类的流动池是具有泳道的玻璃载玻片。每个泳道都是涂覆有两种类型的寡核苷酸坪的玻璃通道。杂交由表面上两种类型的寡核苷酸的第一种来实现。该寡核苷酸与片段一端的第一接头互补。聚合酶产生杂交的片段的补体链。使双链分子变性,并洗去原始模板链。剩余的链,与许多其它剩余的链平行,通过桥接应用克隆扩增。
[0566]
在桥接扩增中,链折叠,并且链的第二末端上的第二接头区域与流动池表面上的第二类的寡核苷酸杂交。聚合酶产生互补链,从而形成双链桥接分子。使这个双链分子变性,导致两个单链分子通过两种不同的寡核苷酸与流动池连接。然后该方法反复重复,并且对数百万个簇同时进行该方法,导致所有片段的克隆扩增。在桥接扩增后,反义链被裂解并洗掉,仅留下正义链。3'端被封闭以防止不需要的引发。
[0567]
在聚类之后,测序开始于延伸第一测序引物以产生第一读取。通过每个循环,荧光标记的核苷酸竞争添加至增长的链中。基于模板的序列仅掺入一个。在每个核苷酸添加后,簇被光源激发,并发射特征荧光信号。循环次数决定了读取的长度。发射波长和信号强度决定了碱基识别。对于给定的簇,同时读取所有相同的链。以大规模并行方式对数以亿计的簇进行测序。在第一读取完成时,洗掉读取产物。
[0568]
在涉及两个索引引物的方案的下一步中,将索引1引物引入并杂交至模板上的索引1区域。索引区域提供片段的鉴定,这对于在多重测序方法中对样品进行解复用是有用的。类似于第一读取生成索引1读取。在完成索引1读取之后,洗掉读取产物并且将链的3'端去保护。然后模板链折叠并结合至流动池上的第二寡核苷酸。以与索引1相同的方式读取索引2序列。然后,在步骤完成时洗掉索引2读取产物。
[0569]
在读取两个索引之后,读取2通过使用聚合酶启动以扩展第二流动池寡核苷酸,形成双链桥。使该双链dna变性,并且封闭3'端。将原始正义链裂解开并洗掉,留下反义链。读取2以引段2测序引物的引入开始。与读取1一样,重复测序步骤直到达到所需的长度。将读取2产物洗掉。该整个方法产生了代表所有片段的数百万个读取。基于样品制备期间引入的独特索引分离来自汇集的样品文库的序列。对于每个样品,类似的碱基识别段的读取局部聚类。正向和反向读取配对,从而创建连续的序列。这些连续序列与参考基因组比对以进行变体鉴定。
[0570]
上述边合成边测序实例涉及配对末端读取,其用于所公开方法的许多实施方案中。配对末端测序涉及来自片段两端的2个读取。当一对读取被定位至参考序列时,可以确定两个读取之间的碱基对距离,然后可以使用该距离来确定从其获得读取的片段的长度。在一些情况下,跨越两个箱的片段将使其成对末端读取与一个箱比对,而另一个与相邻箱比对。随着箱变长或读取变短,这种情况变得越来越少。可以使用各种方法来解释这些片段的箱成员资格。例如,在确定箱的片段尺寸频率时可以省略它们;它们可以计入两个相邻的箱;它们可以被指定给涵盖两个箱的大量碱基对的箱;或者它们可以被指定给两个箱,其权重与每个箱中的碱基对的部分相关。
[0571]
配对末端读取可以使用不同长度的插入物(即,待测序的不同的片段尺寸)。作为本公开中的默认含义,配对末端读取用于指代从各种插入物长度获得的读取。在一些情况下,为了区分短插入物配对末端读取与长插入物配对末端读取,后者还被称为配偶配对读取。在涉及配偶配对读取的一个实施方案中,两个生物素接合接头首先附接至相对长插入物的两端(如,数kb)。然后生物素接合接头连接插入物的两端以形成环化分子。然后可以通过进一步片段化环化分子来获得涵盖生物素接合接头的子片段。然后,可以通过与上述短插入物配对末端测序相同的程序来对包括原始片段的两端的子片段以相反的序列顺序进行测序。使用illumina平台的配偶配对测序的进一步细节在以下url的在线出版物中示出,其通过引用整体并入:resl.lilluminal.lcom/documents/products/technotes/
[0572]
technote_nextera_matepair_data_processing。关于配对末端测序的另外信息可以见于美国专利no.7601499和美国专利公布no.2012/0,053,063,其关于配对末端测序方法和装置的材料通过引用并入。
[0573]
在对dna片段进行测序之后,将预定长度(如100bp)的序列读取定位或比对至已知的参考基因组。参考序列上的经定位或比对的读取及其相应的位置被称为标签。在一个实施方案中,参考基因组序列是ncbi36/hgl8序列,其可在万维网上以genome.ucsc.edu/cgi
‑
bin/hggateway?org=human&db=hg18&hgsid=166260105)获得。或者,参考基因组序列是grch37/hgl9,其可以在万维网上以genome.ucsc.edu/cgi
‑
bin/hggateway获得。其它公开的序列信息源包括genbank、dbest、dbsts、embl(欧洲分子生物学实验室(the european molecular biology laboratory))和ddbj(日本的dna数据库(the dna databank of japan))。许多计算机算法可用于比对序列,包括但不限于blast(altschul等人,1990)、blitz(mpsrch)(sturrock&collins,1993)、fasta(person&lipman,1988)、bowtie(langmead等人,genome biology10:r25.1
‑
r25.10[2009])或eland(illumina,inc.,san diego,ca,usa)。在一个实施方案中,对血浆cfdna分子的克隆扩增拷贝的一端进行测序并且通过illumina基因组分析仪的生物信息学比对分析进行加工,其使用核苷酸数据库的高
效大规模比对(eland)软件。
[0574]
在一个说明性但非限制性的实施方案中,本文描述的方法包括使用helicos真单分子测序(helicos true single molecule sequencing,tsms)技术的单分子测序技术获得测试样品中核酸(如母体样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息(如harris t.d.等人,science 320:106
‑
109[2008]中所述)。在tsms技术中,将dna样品裂解成约100至200个核苷酸的链,并将聚腺苷酸序列添加至每条dna链的3'端。将每条链通过添加荧光标记的腺苷核苷酸来标记。然后将dna链杂交至流动池,所述流动池含有固定至流动池表面的数百万个寡
‑
t捕获位点。在某些实施方案中,模板可以处于约1亿个模板/cm2的密度。然后将流动池加载到仪器中,如heliscope
tm
测序仪,并且激光照射流动池的表面,揭示了每个模板的位置。ccd相机可以将模板的位置定位在流动池表面上。然后将模板荧光标记裂解并洗掉。测序反应通过引入dna聚合酶和荧光标记的核苷酸开始。寡
‑
t核酸用作引物。聚合酶以模板指导的方式将经标记的核苷酸掺入引物中。去除聚合酶和未掺入的核苷酸。通过对流动池表面成像来识别已经指导掺入荧光标记的核苷酸的模板。成像后,裂解步骤去除荧光标记,并用其它荧光标记的核苷酸重复方法,直到达到所需的读取长度。用每个核苷酸添加步骤收集序列信息。通过单分子测序技术进行的全基因组测序在测序文库的制备中排除或通常避免基于pcr的扩增,并且该方法允许直接测量样品,而不是测量该样品的拷贝。
[0575]
在另一个说明性但非限制性的实施方案中,本文描述的方法包括使用454测序(roche)获得测试样品中的核酸(如母体测试样品中的cfdna、筛选癌症的受试者中的cfdna或细胞dna等)的序列信息(例如,如margulies,m.等人nature 437:376
‑
380[2005]中所述)。454测序通常涉及两个步骤。在第一步中,将dna剪切成约300
‑
800个碱基对的片段,并且该片段是平末端的。然后将寡核苷酸接头连接到该片段的末端。接头用作片段的扩增和测序的引物。可以如使用含有5'
‑
生物素标签的接头b将片段附接至dna捕获珠粒,如链霉亲和素
‑
涂覆的珠粒。附接至珠粒的片段在油
‑
水乳液的液滴内进行pcr扩增。结果是每个珠粒上克隆扩增的dna片段的多个拷贝。在第二步中,珠粒被捕获在孔中(如,皮升尺寸的孔)。对每个dna片段并行进行焦磷酸测序。添加一个或多个核苷酸产生光信号,其由ccd照相机记录在测序仪器中。信号强度与掺入的核苷酸数成比例。焦磷酸测序利用焦磷酸盐(ppi),其在核苷酸添加后释放。在腺苷5'磷酰硫酸盐存在下,ppi被atp硫酸化酶转化为atp。荧光素酶使用atp将荧光素转化为氧化荧光素,并且这种反应产生的光被测量和分析。
[0576]
在另一个说明性但非限制性的实施方案中,本文描述的方法包括使用solid
tm
边连接边测序技术(applied biosystems)获得测试样品中核酸(如母体测试样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息。在solid
tm
边连接边测序中,将基因组dna剪切成片段,并将接头附接至片段的5'和3'末端以产生片段文库。可替代地,内部接头可以通过以下步骤来引入:将接头连接至片段的5'和3'端,环化该片段、消化经环化的片段以产生内部接头,并附接接头至所得到的片段的5'和3'端以生成配偶配对文库。接下来,在含有珠粒、引物、模板和pcr组分的微反应器中制备克隆珠粒群。pcr后,使模板变性并富集珠粒以分离珠粒与经延伸的模板。所选珠粒上的模板经过允许键合至载玻片的3'修饰。该序列可以通过部分随机寡核苷酸与中心确定的碱基(或碱基对)的依序杂交和连接来确定,其由特定荧光团鉴定。记录颜色后,将连接的寡核苷酸裂解并去除,然后重复该方
法。
[0577]
在另一个说明性但非限制性的实施方案中,本文描述的方法包括使用pacific biosciences的单分子实时(smrt
tm
)测序技术获得测试样品中核酸(如母体测试样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息。在smrt测序中,染料标记的核苷酸的连续掺入在dna合成期间成像。单个dna聚合酶分子附接至单个零模式波长检测器(zmw检测器)的底部表面,其获得序列信息同时磷酸连接的核苷酸被掺入生长的引物链中。zmw检测器包括限制结构,该限制结构使得能够观察到针对荧光核苷酸本底的dna聚合酶对单个核苷酸的掺入,所述荧光核苷酸在zmw外快速扩散(如,以微秒计)。将核苷酸掺入生长链中通常需要几毫秒。在此期间,荧光标记被激发并产生荧光信号,并且荧光标签被裂解掉。测量染料的相应荧光表明掺入了哪种碱基。重复该方法以提供序列。
[0578]
在另一个说明性但非限制性的实施方案中,本文描述的方法包括使用纳米孔测序获得测试样品中的核酸(如母体测试样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息(如如soni gv和meller a.clin chem 53:1996
‑
2001[2007]中所述)。纳米孔测序dna分析技术由许多公司开发,包括例如oxford nanopore technologies(oxford,united kingdom)、sequenom、nabsys等。纳米孔测序是一种单分子测序技术,从而在单个dna分子通过纳米孔时对其直接测序。纳米孔是小孔,通常直径为1纳米级。将纳米孔浸入导电流体中并在其上施加电势(电压)导致由于离子通过纳米孔的传导而产生的轻微电流。流动的电流量对纳米孔的尺寸和形状敏感。当dna分子穿过纳米孔时,dna分子上的每个核苷酸都会阻塞纳米孔到不同程度,从而以不同程度改变通过纳米孔的电流大小。因此,当dna分子通过纳米孔时,电流的这种变化提供了dna序列的读取。
[0579]
在另一个说明性但非限制性的实施方案中,本文描述的方法包括使用化学敏感场效应晶体管(chemfet)阵列获得测试样品中的核酸(如母体测试样品中的cfdna、针对癌症进行筛选的受试者中的cfdna或细胞dna等)的序列信息(例如,如美国专利申请公布no.2009/0026082中所述)。在该技术的一个实例中,可以将dna分子置于反应室中,并且模板分子可以与结合至聚合酶的测序引物杂交。在测序引物的3'端将一个或多个三磷酸盐掺入新的核酸链可以被chemfet鉴定为电流变化。阵列可以具有多个chemfet传感器。在另一个实例中,可以将单个核酸附接至珠粒,并且该核酸可以在珠粒上扩增,并且可以将各个珠粒转移至chemfet阵列上的各个反应室,其中每个室具有chemfet传感器,并且可以对该核酸进行测序。
[0580]
在另一个实施方案中,本方法包括使用透射电子显微术(tem)获得测试样品中的核酸(如母体测试样品中的cfdna)的序列信息。该方法称为单个分子放置快速纳米转移(eviprnt),包括利用选择性地用重原子标记物标记的高分子量(150kb或更高)dna的单原子分辨率透射电子显微镜成像,并将这些分子以具有一致的碱基
‑
至
‑
碱基间距的超密集(3nm链
‑
至
‑
链)平行阵列的方式排列在超薄膜上。电子显微镜用于对膜上的分子成像以确定重原子标记物的位置并从dna中提取碱基序列信息。该方法在pct专利公开wo 2009/046445中进一步描述。该方法允许在小于十分钟内对全人基因组进行测序。
[0581]
在另一个实施方案中,dna测序技术是ion torrent单分子测序,它在半导体芯片上将半导体技术与简单的测序化学组合成一对,以直接将化学编码的信息(a、c、g、t)转换为数字信息(0、1)。实际上,当通过聚合酶将核苷酸掺入dna链时,作为副产物释放出氢离
子。ion torrent使用高密度的微加工孔阵列以大规模并行方式执行这种生化方法。每个孔都持有不同的dna分子。在孔下方是离子敏感层,并且在其下方是离子传感器。当将核苷酸(例如c)添加到dna模板然后掺入dna链中时,将释放氢离子。来自此离子的电荷将改变溶液的ph,这可以通过ion torrent的离子传感器检测到。测序仪
‑
基本上是世界上最小的固态ph计
‑
识别碱基,直接从化学信息到数字信息。然后,离子个人基因组成机器(pgm
tm
)测序仪依次用一个核苷酸接着另一个对芯片进行充满。如果充满芯片的下一个核苷酸不匹配,不会记录电压变化并且也不会识别碱基。如果dna链上有两个相同的碱基,则电压将加倍,并且芯片将记录经识别的两个相同的碱基。直接检测允许以秒记录核苷酸掺入。
[0582]
在另一个实施方案中,本方法包括使用边杂交边测序获得测试样品中的核酸(如母体测试样品中的cfdna)的序列信息。边杂交边测序包括使多条多核苷酸序列与多个多核苷酸探针接触,其中多个多核苷酸探针中的每一个可任选地束缚至基底。基底可以是包含已知核苷酸序列的阵列的平坦表面。阵列的杂交模式可用于确定样品中存在的多核苷酸序列。在其它实施方案中,每个探针都被束缚至珠粒,如磁珠等。可以确定与珠粒的杂交并将其用于鉴定样品中的多条多核苷酸序列。
[0583]
在本文所述的方法的一些实施方案中,经定位的序列标签包括序列约20bp、约25bp、约30bp、约35bp、约40bp、约45bp、约50bp、约55bp、约60bp、约65bp、约70bp、约75bp、约80bp、约85bp、约90bp、约95bp、约l00 bp、约110bp、约120bp、约130、约140bp、约150bp、约200bp、约250bp、约300bp、约350bp、约400bp、约450bp或约500bp的读取。预期技术进步将使大于500bp的单末端读取能够在生成配对末端读取时实现大于约1000bp的读取。在一个实施方案中,经定位的序列标签包含36bp的序列读取。通过比较标签的序列与参考序列来实现序列标签的定位,以确定经测序的核酸(如cfdna)分子的染色体来源,并且不需要特定的遗传序列信息。可以允许小程度的错配(每个序列标签0
‑
2个错配)来解释参考基因组和混合样品中的基因组之间可能存在的微小多态性。
[0584]
通常获得每个样品的多个序列标签。在一些实施方案中,包含20至40bp(如36bp)的读取的至少约3
×
106个序列标签、至少约5
×
106个序列标签、至少约8
×
106个序列标签、至少约10
×
106个序列标签、至少约15
×
106个序列标签、至少约20
×
106个序列标签、至少约30
×
106个序列标签、至少约40
×
106个序列标签或至少约50
×
106个序列标签通过将读取定位至每样品的参考基因组获得。在一个实施方案中,将所有序列读取都定位至参考基因组的所有区域。在一个实施方案中,对已经定位至参考基因组的所有区域(如所有染色体)的标签进行计数,并且确定混合dna样品中的cnv,即目标序列的过度表现或表现不足,如染色体或其部分。该方法不需要在两个基因组之间进行分类。
[0585]
正确确定样品中存在或缺失cnv(如,非整倍性)所需的精确度是基于测序运行中样品之间定位至参考基因组的序列标签数量的变异(染色体间变异性)以及在不同的测序运行中定位至参考基因组的序列标签的数量的变异(测序间变异性)进行预测的。例如,对于定位至富含gc或贫gc的参考序列的标签,变异可能特别明显。其它变异可以由使用不同的核酸提取和纯化方案、测序文库的制备以及不同测序平台的使用导致。本方法基于归一化序列(归一化染色体序列或归一化区段序列)的知识,使用序列剂量(染色体剂量或区段剂量),以内在地解释来自染色体间(运行内)和测序间(运行间)的应计变异性和平台
‑
依赖性变异性。染色体剂量基于归一化染色体序列的知识,其可以由单一染色体或者由选自1
‑
22号染色体、x染色体和y染色体的两种或更多种染色体组成。或者,归一化染色体序列可由单个染色体区段,或由一个染色体或两个或更多个染色体的两个或更多个区段组成。区段剂量基于归一化区段序列的知识,其可以由任一种染色体的单个区段,或由1
‑
22号染色体、x染色体和y染色体中的任两个或更多个的两个或更多个区段组成。
[0586]
cnv和产前诊断
[0587]
在母体血液中循环的无细胞胎儿dna和rna可用于越来越多的遗传病况的早期非侵入性产前诊断(nipd),两者用于妊娠管理和辅助生殖决策。已知在血流中循环的无细胞dna的存在超过50年。最近,在妊娠期间在母体血流中发现了少量循环胎儿dna的存在(lo等人,lancet 350:485
‑
487[1997])。认为源于垂死的胎盘细胞,无细胞胎儿dna(cfdna)已被证明由通常长度小于200bp的短片段组成(chan等人,clin chem 50:88
‑
92[2004]),其早在妊娠4周就可以辨别出来(illanes等人,early human dev 83:563
‑
566[2007]),并且已知在递送数小时内从母体血液循环中清除(lo等人,am j hum genet 64:218
‑
224[1999])。除了cfdna之外,无细胞胎儿rna(cfrna)的片段也可以在母体血流中辨别,源自胎儿或胎盘中转录的基因。来自母体血液样品的这些胎儿遗传元件的提取和随后分析为nipd提供了新的机会。
[0588]
本方法是一种不依赖于多态性的方法,其用于nipd中并且不需要将胎儿cfdna与母体cfdna区分开以能够确定胎儿非整倍性。在一些实施方案中,非整倍性是完全染色体三体或单体,或部分三体或单体。部分非整倍性是由染色体的一部分的丢失或增加引起的,并且涵盖由不平衡的易位、不平衡的倒置、缺失和插入引起的染色体不平衡。到目前为止,与生命相容的最常见的已知非整倍性是21三体,即唐氏综合征(down syndrome,ds),其由21号染色体的部分或全部的存在引起。极少地,ds可能由遗传或偶发的缺陷引起,从而21号染色体的全部或部分的额外拷贝变得附着至另一种染色体(通常是14号染色体)以形成单个的异常染色体。ds与智力障碍、严重的学习困难和因长期健康问题(诸如心脏病)引起的死亡率过高相关。具有已知临床意义的其它非整倍性包括爱德华综合征(edward syndrome)(18三体)和帕托综合征(patau syndrome)(13三体),它们在生命的最初几个月内经常是致命的。与性染色体数量相关的异常也是已知的,并包括女性出生时的x单体如特纳综合征(turner syndrome)(xo)和三重x综合征(xxx)以及男性出生时的克兰费尔特综合征(kleinefelter syndrome)(xxy)和xyy综合征,其都与各种表型(包括不育和智力技能降低)有关。x单体[45,x]是早期妊娠丧失的常见原因,占自然流产的约7%。基于45,x(也称为特纳综合征)的活产频率为1
‑
2/10,000,估计不到1%的45,x受孕将存活到足月。约30%的特纳综合征患者镶嵌有45,x细胞系与46,xx细胞系或含有重排的x染色体的细胞系(hook和warburton 1983)。考虑到高胚胎致死率,活产婴儿的表型相对温和,并且假设可能所有患有特纳综合征的活产女性携带含有两种性染色体的细胞系。x单体在女性中可以以45,x或45,x/46xx存在,并且在男性可以以45,x/46xy存在。人的常染色体单体通常被认为与生命不相容;然而,有相当多的细胞遗传学报告描述了活产儿童中一个21号染色体的完整单体(vosranova i等人,molecular cytogen.1:13[2008];joosten等人,prenatal diagn.17:271
‑
5[1997]。本文描述的方法可用于在产前诊断这些和其它染色体异常。
[0589]
根据一些实施方案,本文公开的方法可以确定1
‑
22号染色体、x染色体和y染色体中的任一种的染色体三体的存在或缺失。可以根据本方法检测的染色体三体的实例包括但
不限于21三体(t21;唐氏综合征(down syndrome))、18三体(t18;爱德华氏综合征(edward's syndrome))、三体16(t16)、三体20(t20)、三体22(t22;猫眼综合征)、三体15(t15;普瑞德威利综合征(prader willi syndrome))、13三体(t13;帕陶综合征(patau syndrome)),三体8(t8;warkany综合征)、三体9和xxy(克兰费尔特综合征)、xyy或xxx三体。以非镶嵌状态存在的其它常染色体的完整三体是致命的,但是当以镶嵌状态存在时可以与生命相容。应当理解,根据本文提供的教导,可以在胎儿cfdna中确定各种完整的三体(无论是以镶嵌或非镶嵌状态存在)和部分三体。
[0590]
可以通过本方法确定的部分三体的非限制性实例包括但不限于,部分三体1q32
‑
44、三体9p、三体4镶嵌性、三体17p、部分三体4q26
‑
qter、部分2p三体、部分三体1q和/或部分三体6p/单体6q。
[0591]
本文公开的方法还可用于确定x染色体单体、21号染色体单体和部分单体,诸如单体13、单体15、单体16、单体21和单体22,它们已知参与妊娠流产。通常参与完全非整倍性的染色体的部分单体也可通过本文所述的方法确定。可以根据本方法确定的缺失综合征的非限制性实例包括由染色体的部分缺失引起的综合征。可以根据本文描述的方法确定的部分缺失的实例包括但不限于1号染色体、4号染色体、5号染色体、7号染色体、11号染色体、18号染色体、15号染色体、13号染色体、17号染色体、22号染色体和10号染色体的部分缺失,其描述于下文中。
[0592]
1q21.1缺失综合征或1q21.1(复发性)微缺失是1号染色体的罕见畸变。在缺失综合征旁边,还有1q21.1重复综合征。虽然在特定部位上缺失综合征丢失了一部分dna,但在重复综合征的同一部位上有两个或三个拷贝的相似dna部分。文献是指缺失和重复两者作为1q21.1拷贝
‑
数目变异(cnv)。1q21.1缺失可与tar综合征(血小板减少症伴桡骨缺乏)相关。
[0593]
沃夫
‑
贺许宏氏综合征(wolf
‑
hirschhorn syndrome,whs)(omin#194190)是一种与染色体4p16.3的半合子缺失有关的连续基因缺失综合征。沃夫
‑
贺许宏氏综合征是一种先天性畸形综合征,其特征是出生前和出生后生长不足、不同程度的发育障碍、特征性颅面特征(鼻子'希腊战士头盔'外观,高额头,突出的眉间,眼距过远,高拱眉,眼睛突出,内眦赘皮,短人中,嘴角向下的鲜明嘴和小颌畸形),以及癫痫症。
[0594]
5号染色体的部分缺失,也称为5p
‑
或5p负,并命名为猫叫综合征(cris du chat syndrome)(omin#123450),是由5号染色体的短臂(p臂)缺失(5p15.3
‑
p15.2)引起的。患有这种病况的婴儿经常会听到像猫一样高亢的哭声。该病症的特征是智力残疾和发育迟缓、小头尺寸(小头畸形)、低出生体重以及婴儿期肌肉张力弱(张力减退)、独特的面部特征和可能的心脏缺陷。
[0595]
威廉
‑
博伊伦综合征(williams
‑
beuren syndrome),也被称为染色体7q11.23缺失综合征(omin 194050),是一种连续基因缺失综合征,导致由染色体7q11.23上含有大约28个基因的1.5
‑
1.8mb的半合子缺失引起的多系统病症。
[0596]
雅各布森综合征(jacobsen syndrome),也被称为11q缺失病症,是一种罕见的先天性病症,由11号染色体的包含带11q24.1的末端区域的缺失引起。它可以导致智力障碍、独特的面部外观和各种身体问题,包括心脏缺陷和出血性病症。
[0597]
18号染色体的部分单体,称为单体18p,是一种罕见的染色体病症,其中18号染色
体的全部或部分短臂(p)被缺失(单体的)。该病症的特征通常为身材矮小、不同程度的精神发育迟滞、言语延迟、颅骨和面部(颅面)区畸形和/或另外的身体异常。相关的颅面缺陷在范围和严重程度上可能因病例而异。
[0598]
由15号染色体的结构或拷贝数变化引起的病况包括安格尔曼综合征(angelman syndrome)和普瑞德
‑
威利综合征(prader
‑
willi syndrome),其涉及15号染色体的相同部分(15q11
‑
q13区域)中基因活性的丧失。应当理解,在载体亲本中几种易位和微缺失可以是无症状的,但是可以在后代中引起主要的遗传疾病。例如,携带15q11
‑
q13微缺失的健康母亲可以生下患有安格尔曼综合征的孩子,这是一种严重的神经退行性病症。因此,本文所述的方法、装置和系统可用于鉴定胎儿中的这种部分缺失和其它缺失。
[0599]
部分单体13q是一种罕见的染色体病症,其当13号染色体的长臂(q)的一块丢失(单体的)时产生。出生时有部分单体13q的婴儿可能表现出低出生体重、头部和面部(颅面部)畸形、骨骼异常(特别是手和脚)以及其它身体异常。精神发育迟滞是这种病况的特征。出生时患有这种病症的个体中,婴儿期的死亡率很高。部分单体13q的几乎所有情况都是随机出现的,没有明显的原因(零星的)。
[0600]
史密斯
‑
马吉利综合征(smith
‑
magenis syndrome)(sms
‑
omim#182290)是由17号染色体的一个拷贝上的缺失或遗传物质丢失引起的。这种众所周知的综合征与发育迟缓、精神发育迟滞、先天性异常诸如心脏和肾脏缺陷,以及神经行为异常诸如严重的睡眠障碍和自我伤害行为有关。史密斯
‑
马吉利综合征(sms)在大多数情况下(90%)由染色体17p11.2中的3.7
‑
mb中间缺失引起。
[0601]
22q11.2缺失综合征,也被称为迪乔治综合征(digeorge syndrome),是一种由一小块的22号染色体缺失引起的综合征。缺失(22q11.2)发生在染色体对之一的长臂上的染色体的中间附近。这种综合征的特征即使在同一家庭的成员中差异很大,并影响身体的许多部分。特征性体征和症状可能包括出生缺陷诸如先天性心脏病,最常见的与闭合性神经肌肉问题(腭咽闭合不全)相关的腭缺陷,学习障碍,面部特征的轻微差异和复发性感染有关。染色体区域22q11.2中的微缺失与精神分裂症的风险增加20至30倍相关。
[0602]
10号染色体短臂上的缺失与迪乔治综合征样表型有关。染色体10p的部分单体是罕见的,但已经在显示迪乔治综合征特征的一部分患者中观察到。
[0603]
在一个实施方案中,本文描述的方法、装置和系统用于确定部分单体,包括但不限于1号染色体、4号染色体、5号染色体、7号染色体、11号染色体、18号染色体、15号染色体、13号染色体、17号染色体、22号染色体和10号染色体的部分单体,如部分单体1q21.11、部分单体4p16.3、部分单体5p15.3
‑
p15.2、部分单体7q11.23、部分单体11q24.1、部分单体18p、染色体15的部分单体(15q11
‑
q13)、部分单体13q、部分单体17p11.2、22号染色体的部分单体(22q11.2)和部分单体10p也可以使用该方法确定。
[0604]
可以根据本文描述的方法确定的其它部分单体包括不平衡易位t(8;11)(p23.2;p15.5);11q23微缺失;17p11.2缺失;22q13.3缺失;xp22.3微缺失;10p14缺失;20p微缺失,[del(22)(q11.2q11.23)]、7q11.23和7q36缺失;1p36缺失;2p微缺失;神经纤维瘤病1型(17q11.2微缺失),yq缺失;4p16.3微缺失;1p36.2微缺失;11ql4缺失;19q13.2微缺失;鲁宾斯坦
‑
泰比(rubinstein
‑
taybi)(16p13.3微缺失);7p21微缺失;米勒
‑
狄克综合征(miller
‑
dieker syndrome)(17p13.3);和2q37微缺失。部分缺失可以是染色体的一部分的小缺失,
或者它们可以是其中可以发生单个基因缺失的染色体的微缺失。
[0605]
已经鉴定了由染色体臂的部分重复引起的几种重复综合征(参见omin[在线人孟德尔遗传,在网址ncbi.nlm.nih.gov/omim查看])。在一个实施方案中,本方法可用于确定1
‑
22号染色体、x染色体和y染色体中的任一个的区段的重复和/或倍增的存在或不存在。可根据本方法确定的重复综合征的非限制性实例包括8号染色体、15号染色体、12号染色体和17号染色体的部分的重复,其在下面描述。
[0606]
8p23.1重复综合征是一种由人8号染色体区域重复引起的罕见遗传性病症。这种重复综合征的估计患病率为64,000例出生中的1例,并且是8p23.1缺失综合征的倒数。8p23.1重复与可变表型相关,包括言语延迟、发育迟缓、轻度畸形、具有突出的前额和弓形眉毛以及先天性心脏病(chd)中的一种或多种。
[0607]
染色体15q重复综合征(dup15q)是一种由染色体15q11
‑
13.1的复制引起的临床可鉴定的综合征。具有dup15q的婴儿通常有肌张力减退(肌张力差)、生长迟缓;他们可能出生时就具有唇裂和/或腭裂或心脏、肾脏或其它器官的畸形;他们表现出一定程度的认知延迟/残疾(精神发育迟滞)、言语和语言延迟以及感觉处理障碍。
[0608]
帕里斯特
‑
吉利恩综合征(pallister killian syndrome)是额外的#12染色体材料的结果。通常存在细胞的混合物(镶嵌性),有些具有额外的#12材料并且有些是正常的(46种染色体,没有额外的#12材料)。患有这种综合征的婴儿存在许多问题,包括严重的精神发育迟滞、肌肉张力差、
″
粗糙
″
的面部特征和突出的前额。他们往往有非常薄的上唇、下唇较厚并且鼻子较短。其它健康问题包括癫痫发作、喂养不良、关节僵硬、成年期白内障,听力丧失和心脏缺陷。患有帕里斯特
‑
吉利恩的人寿命缩短。
[0609]
患有指定为dup(17)(p11.2p11.2)或dup17p的遗传病况的个体在17号染色体的短臂上携带额外的遗传信息(称为复制)。染色体17p11.2的重复是波托茨基
‑
鲁普斯基综合征(potocki
‑
lupski syndrome,ptls)的基础,这是一种新发现的遗传病况,在医学文献中仅报道了数十例病例。具有这种重复的患者通常具有低肌肉张力、不良喂养和在婴儿期期间不能茁壮成长,并且还表现出运动和言语阶段的延迟发育。许多患有ptls的个体都难以进行清晰发音和语言处理。此外,患者可能具有与自闭症或自闭症谱系障碍患者相似的行为特征。患有ptls的个体可能有心脏缺陷和睡眠呼吸暂停。已知染色体17p12中包含基因pmp22的大区域的重复会引起沙尔科
‑
玛丽
‑
图思(charcot
‑
marie tooth disease)。
[0610]
cnv与死胎有关。然而,由于常规细胞遗传学的固有局限性,cnv对死胎的贡献被认为不足(harris等人,prenatal diagn 31:932
‑
944[2011])。如实施例中所示和本文其它地方所述,本方法能够确定染色体区段的部分非整倍性(如,缺失和倍增)的存在,并且可用于鉴定和确定存在或不存在与死产有关的cnv。
[0611]
临床病症的cnv的测定
[0612]
除了出生缺陷的早期确定之外,本文描述的方法可以应用于确定基因组内遗传序列的表现中的任何异常。基因组内遗传序列的表现的许多异常与各种病理有关。此类病理包括但不限于癌症、传染病和自身免疫性疾病、神经系统疾病、代谢和/或心血管疾病等。
[0613]
因此,在各种实施方案中,预期在诊断和/或监测和或治疗此类病理中使用本文所述的方法。例如,可应用该方法以确定疾病的存在或缺失,监测疾病的进展和/或治疗方案的功效,以及确定病原体如病毒的核酸的存在或缺失;确定与移植物抗宿主病(gvhd)相关
的染色体异常,并确定个体在法医分析中的贡献。
[0614]
癌症中的cnv
[0615]
已经表明,来自癌症患者的血浆和血清dna含有可测量的量的肿瘤dna,可以被回收并用作肿瘤dna的替代来源,并且肿瘤的特征在于非整倍性,或者基因序列或甚至整个染色体的不适当数量。因此,在来自个体的样品中确定给定序列(即目标序列)的量的差异可以用于医学病况的预后或诊断。在一些实施方案中,本方法可用于确定疑似或已知患有癌症的患者中染色体非整倍性的存在或不存在。
[0616]
本文的一些实施方式提供了基于循环cfdna样品,使用样品的浅测序和配对末端方法并且使用可从配对末端读取获得的片段尺寸信息来鉴定在正常细胞的本底中存在来自癌细胞的差异性甲基化的凋亡dna,来检测癌症、跟踪治疗性响应和最小残留疾病的方法。已显示,在一些癌症中,肿瘤来源的cfdna比非肿瘤来源的cfdna短。因此,本文所述的基于尺寸的方法可用于确定包括与这些癌症相关的非整倍性在内的cnv,使得能够(a)检测筛选或诊断性设置中存在的肿瘤;(b)监测对疗法的响应;(c)监测最小残留疾病。
[0617]
在某些实施方案中,非整倍性是受试者的基因组的特征,并且导致癌症易感性通常增加。在某些实施方案中,非整倍性是特定细胞(如肿瘤细胞、原肿瘤赘生性细胞等)的特征,其是瘤形成或具有增加的瘤形成易感性。如下所述,特定的非整倍性与特定癌症或对特定癌症的易感性有关。在一些实施方案中,可以使用非常浅的配对末端测序方法以成本有效的方式检测/监测癌症存在。
[0618]
因此,本文所述的方法的各种实施方案提供了对来自受试者的测试样品中的目标序列(如一条或多条临床相关序列的)的拷贝数变异的确定,其中拷贝数中的某些变异提供了癌症的存在和/或易感性的指标。在某些实施方案中,该样品包含来源于两种或更多种类型的细胞的核酸的混合物。在一个实施方案中,核酸的混合物来源于源于患有医学病况如癌症的受试者的正常和癌细胞。
[0619]
癌症的发展通常伴随着由被称为染色体不稳定性(cin)的方法引起的整个染色体数目的改变(即完全染色体非整倍性)和/或染色体区段数的改变(即部分非整倍性)(thoma等人,swiss med weekly 2011:141:wl3170)。据信许多实体瘤,诸如乳腺癌,通过几种遗传畸变的积累从起始进展到转移。[sato等人,cancer res.,50:7184
‑
7189[1990];jongsma等人,j clin pathol:mol path 55:305
‑
309[2002])]。此类遗传畸变,因为它们积累,可以赋予增殖优势、遗传不稳定性和伴随的快速进化耐药性的能力,以及增强的血管形成、蛋白水解和转移。遗传畸变可能影响隐性
″
肿瘤抑制基因
″
或主要作用致癌基因。据信导致杂合性缺失(loh)的缺失和重组通过揭示突变的肿瘤抑制子等位基因而在肿瘤进展中起主要作用。
[0620]
cfdna已在被诊断患有恶性肿瘤的患者的循环中被发现,包括但不限于肺癌(pathak等人clin chem 52:1833
‑
1842[2006])、前列腺癌(schwartzenbach等人clin cancer res 15:1032
‑
8[2009])和乳腺癌(schwartzenbach等人,可在breast
‑
cancer
‑
research.com/content/11/5/r71[2009]在线获得)。可以在癌症患者的循环cfdna中确定的与癌症相关的基因组不稳定性的鉴定是潜在的诊断性和预后工具。在一个实施方案中,本文描述的方法用于确定样品中一条或多条目标序列的cnv,如包含来源于疑似或已知患有癌症(如癌、肉瘤、淋巴瘤、白血病、生殖细胞肿瘤和胚细胞瘤)的受试者的核酸混合物的
样品。在一个实施方案中,该样品是从外周血来源(加工)的血浆样品,其可包含来源于正常细胞和癌细胞的cfdna的混合物。在另一个实施方案中,确定cnv是否存在所需的生物样品来源于如果存在癌症则包含来自其它生物组织的癌细胞和非癌细胞的混合物的细胞,所述其它生物组织包括但不限于生物流体诸如血清、汗液、眼泪、痰液、尿液、痰液、耳流液、淋巴液、唾液、脑脊液、ravages、骨髓悬液、阴道流液、宫腔灌洗液、脑液、腹水、乳汁、呼吸道、肠道和泌尿生殖道的分泌物及白细胞去除法样品,或在组织活检、拭子或涂片中。在其它实施方案中,生物样品是粪便(排泄物)样品。
[0621]
本文描述的方法不限于cfdna的分析。应该认识到,可以对细胞dna样品进行类似的分析。
[0622]
在各种实施方案中,一条或多条目标序列包含一条或多条已知或疑似在癌症的发展和/或进展中起作用的核酸序列。目标序列的实例包括染色体的核酸序列,如完全染色体和/或区段,其如下所述在癌细胞中被扩增或缺失。
[0623]
cnv总数和癌症风险。
[0624]
常见的癌症snp以及类似的常见癌症cnv可能各自仅赋予疾病风险的轻微增加。然而,它们可能共同导致癌症风险显著增加。在这方面,值得注意的是,大dna区段的种系增加和丢失已被报道为使个体易患神经母细胞瘤、前列腺癌和结直肠癌、乳腺癌和brca1相关卵巢癌的因素(参见,如,krepischi等人breast cancer res.,14:r24[2012];diskin等人nature 2009,459:987
‑
991;liu等人cancer res 2009,69:2176
‑
2179;lucito等人cancer biol ther 2007,6:1592
‑
1599;thean等人genes chromosomes cancer 2010,49:99
‑
106;venkatachalam等人int j cancer 2011,129:1635
‑
1642;和yoshihara等人genes chromosomes cancer 2011,50:167
‑
177)。值得注意的是,经常在健康群体中发现的cnv(常见的cnv)被认为在癌症病因学中起作用(参见,如,shlien和malkin(2009)genome medicine,1(6):62)。在一项测试了常见cnv与恶性肿瘤相关的假设的研究(shlien等人proc natl acad sci usa 2008,105:11264
‑
11269)中,创建了每个已知cnv的图谱,其基因座与真正的癌症相关基因的基因座一致(如由higgins等人nucleic acids res 2007,35:d721
‑
726分类)。这些被称为
″
癌症cnv
″
。在初始分析(shlien等人proc natl acad sci usa 2008,105:11264
‑
11269)中,使用具有5.8kb的平均探针间距离的affymetrix 500k阵列组评估770个健康基因组。由于cnv通常被认为在基因区域被耗竭(redon等人(2006)nature 2006,444:444
‑
454),令人惊讶的是在大型参考群体中,在一个以上的人中发现49种直接被cnv涵盖或重叠的癌症基因。在前十种基因中,可以在四个或更多个人中发现癌症cnv。
[0625]
因此认为cnv频率可用作癌症风险的度量(参见,如,美国专利公布no:2010/0261183 a1)。cnv频率可以简单地通过生物体的组成型基因组确定,或者它可以代表来源于一个或多个肿瘤(赘生性细胞)的部分(如果存在的话)。
[0626]
在某些实施方案中,测试样品(如,包含构成性(种系)核酸的样品)或核酸混合物(如,来源于赘生性细胞的一种或多种种系核酸)中的许多cnv使用本文所述的用于拷贝数变异的方法确定。在测试样品中鉴定例如与参考值比较增加的数目的cnv,指示受试者中癌症的风险或易感性。应当理解,参考值可以随给定的群体而变化。还应当理解,cnv频率增加的绝对值将根据用于确定cnv频率和其它参数的方法的分辨率而变化。通常,确定cnv频率增加至少约为参考值的1.2倍,以指示癌症的风险(参见,如,美国专利公布no:2010/
0261183 a1),例如cnv频率增加至少或约为参考值的1.5倍或更大,诸如参考值的2
‑
4倍是癌症风险增加的指标(如,与正常健康参考群体相比)。
[0627]
还认为确定与参考值相比哺乳动物的基因组中的结构变异指示癌症的风险。在该上下文中,在一个实施方案中,术语
″
结构变异
″
可被定义为哺乳动物中的cnv频率乘以哺乳动物中的平均cnv尺寸(以bp计)。因此,由于cnv频率增加和/或由于大量基因组核酸缺失或重复的发生而导致高结构变异评分。因此,在某些实施方案中,使用本文所述的方法确定测试样品(如包含构成性(种系)核酸的样品)中的许多cnv以确定拷贝数变异的尺寸和数量。在某些实施方案中,基因组dna内的总结构变异评分大于约1兆碱基、或大于约1.1兆碱基、或大于约1.2兆碱基、或大于约1.3兆碱基、或大于约1.4兆碱基、或大于约1.5兆碱基、或大于约1.8兆碱基、或大于约2 兆碱基的dna指示癌症的风险。
[0628]
据信这些方法可以任何癌症的风险的量度,包括但不限于急性和慢性白血病,淋巴瘤,间充质或上皮组织、脑、乳房、肝、胃的许多实体瘤,结肠癌,b细胞淋巴瘤,肺癌,支气管癌,结直肠癌,前列腺癌,乳腺癌,胰腺癌,胃癌,卵巢癌,膀胱癌,脑或中枢神经系统癌,周围神经系统癌症,食道癌,子宫颈癌,黑素瘤,子宫或子宫内膜癌,口腔癌或咽癌,肝癌,肾癌,胆道癌,小肠癌或阑尾癌,唾液腺癌,甲状腺癌,肾上腺癌,骨肉瘤,软骨肉瘤,脂肪肉瘤,睾丸癌及恶性纤维组织细胞瘤和其它癌症。
[0629]
完整染色体非整倍性。
[0630]
如上所述,癌症中存在高频率的非整倍性。在检查癌症中的体细胞拷贝数改变(scna)流行的某些研究中,已发现典型癌细胞的基因组的四分之一受到非整倍性的全臂scna或全染色体scna的影响(参见,如beroukhim等人nature 463:899
‑
905[2010])。在数种癌症类型中反复观察到全染色体改变。例如,在10
‑
20%的急性髓性白血病(aml)以及一些实体瘤(包括尤因氏肉瘤(ewing's sarcoma)和硬纤维瘤)病例中可以看到8号染色体的获得(参见,如barnard等人leukemia 10:5
‑
12[1996];maurici等人cancer genet.cytogenet.100:106
‑
110[1998];qi等人cancer genet.cytogenet.92:147
‑
149[1996];barnard,d.r.等人blood 100:427
‑
434[2002];等。表2中显示了人类癌症中染色体获得和丧失的说明性但非限制性的列表。
[0631]
表2.人类癌症中说明性、特异性、复发性染色体获得和丧失(参见,如gordon等人(2012)nature rev.genetics.13:189
‑
203)。
[0632]
chromosomes cancer 48:366
‑
380(2009);beroukhim等人nature.463(7283):899
‑
905[2010])。另外,观察到臂水平拷贝数变异的频率随着染色体臂的长度而减小。根据这一趋势进行调整后,大多数染色体组展现出在多种癌症谱系中优先获得或丧失的强有力证据,但很少见到这两种情况(参见,如beroukhim等人nature.463(7283):899
‑
905[2010])。
[0637]
因此,在一个实施方案中,本文所述的方法用于确定样品中的臂水平cnv(包括一个染色体臂或基本上一个染色体臂的cnv)。cnv可以在包含构成性(种系)核酸的测试样品中的cnv中测定,并且臂水平cnv可以在那些构成性核酸中鉴定。在某些实施方案中,在包含核酸混合物(如,来源于正常细胞的核酸和来源于肿瘤细胞的核酸)的样品中鉴定(如果存在)臂水平cnv。在某些实施方案中,该样品来源于疑似或已知患有癌症(如癌、肉瘤、淋巴瘤、白血病、生殖细胞肿瘤、胚细胞瘤等)的受试者。在一个实施方案中,该样品是从外周血来源(加工)的血浆样品,其可包含来源于正常细胞和癌细胞的cfdna的混合物。在另一个实施方案中,用于确定cnv是否存在的生物样品来源于细胞,如果存在癌症,则包含来自其它生物组织的癌细胞和非癌细胞的混合物,所述生物组织包括但不限于生物流体诸如血清、汗液、眼泪、痰液、尿液、痰液、耳流液、淋巴液、唾液、脑脊液、ravages、骨髓悬液、阴道流液、宫腔灌洗液、脑液、腹水、乳汁、呼吸道、肠道和泌尿生殖道的分泌物及白细胞去除法样品,或在组织活检、拭子或涂片中。在其它实施方案中,生物样品是粪便(排泄物)样品。
[0638]
在各种实施方案中,被鉴定为指示癌症存在或癌症风险增加的cnv包括但不限于表3中列出的臂水平cnv。如表3中所示,某些cnv包含大量的臂水平获得,其表明存在癌症或某些癌症的风险增加。因此,例如,1q的获得指示急性成淋巴细胞性白血病(all)、乳腺癌、gist、hcc、肺nsc、成神经管细胞瘤、黑素瘤、mpd、卵巢癌和/或前列腺癌。3q的获得指示食管鳞状细胞癌、肺sc和/或mpd存在或增加的风险。7q的获得指示结直肠癌、神经胶质瘤、hcc、肺nsc、成神经管细胞瘤、黑素瘤、前列腺癌和/或肾癌的存在或增加的风险。7p的获得指示乳腺癌、结直肠癌、食管腺癌、神经胶质瘤、hcc、肺nsc、成神经管细胞瘤、黑素瘤和/或肾癌的存在或增加的风险。20q的获得指示乳腺癌、结直肠癌、去分化脂肪肉瘤、食管腺癌、食管鳞状细胞癌、胶质瘤癌、hcc、肺nsc、黑素瘤、卵巢癌和/或肾癌的存在或增加的风险,等。
[0639]
类似于表3中所示,包含显著的臂水平丧失的某些cnv指示某些癌症的存在和/ 或增加的风险。因此,例如,1p的丧失指示胃肠道基质瘤的存在或增加的风险。4q的丧失指示结直肠癌、食管腺癌、肺sc、黑素瘤、卵巢癌和/或肾癌的存在或增加的风险,17p的丧失指示乳腺癌、结直肠癌、食管腺癌、hcc、肺nsc、肺sc和/或卵巢癌存在或增加的风险,等。
[0640]
表3.16种癌症亚型(乳腺癌、结直肠癌、去分化的脂肪肉瘤、食管腺癌、食管鳞状细胞癌、gist(胃肠道基质瘤)、神经胶质瘤、hcc(肝细胞癌)、肺nsc、肺sc、成神经管细胞瘤、黑素瘤、mpd(骨髓增殖性疾病)、卵巢癌、前列腺癌、急性成淋巴细胞性白血病(all)和肾癌)中的每一种的显著性臂水平染色体区段拷贝数改变(参见,如beroukhim等人nature(2010)463(7283):899
‑
905)。
[0641]
[0642]
[0643][0644]
臂水平拷贝数变异之间关联的实例旨在说明而非限制。其它臂水平拷贝数变异和它们的癌症关联性是本领域技术人员已知的。
[0645]
较小的,如局部(focal),拷贝数变异。
[0646]
如上所指示,在某些实施方案中,本文所述的方法可用于确定染色体扩增的存在或缺失。在一些实施方案中,染色体扩增是一个或多个完整染色体的获得。在其它实施方案中,染色体扩增是染色体的一个或多个区段的获得。在其它实施方案中,染色体扩增是两个或更多个染色体的两个或更多个区段的获得。在某些实施方案中,染色体扩增可以涉及一种或多种致癌基因的获得。
[0647]
与人实体瘤相关的显性作用基因通常通过过表达或改变的表达发挥其作用。基因扩增是导致基因表达上调的常见机制。来自细胞遗传学研究的证据表明,超过50%的人乳腺癌发生了显著的扩增。最值得注意的是,位于17号染色体(17(17q21
‑
q22))上的原癌基因人表皮生长因子受体2(her2)的扩增导致细胞表面上her2受体的过表达,导致乳腺癌和其它恶性肿瘤中过多和失调的信号传导(park等人,clinical breast cancer 8:392
‑
401[2008])。已发现多种致癌基因在其它人类恶性肿瘤中被扩增。人肿瘤中的细胞致癌基因扩增的实例包括以下的扩增:早幼粒细胞白血病细胞系hl60和小细胞肺癌细胞系中的c
‑
myc,原代神经母细胞瘤(iii期和iv期)、神经母细胞瘤细胞系、前列腺癌细胞系和原发性肿瘤以及小细胞肺癌细胞系和肿瘤中的n
‑
myc,小细胞肺癌细胞系和肿瘤中的l
‑
myc,急性髓性白血病和结肠癌细胞系中的c
‑
myb,表皮样癌细胞和原发性神经胶质瘤中的c
‑
erbb,肺、结肠、膀胱和直肠的原发癌中的ck
‑
ras
‑
2,乳腺癌细胞系中的n
‑
ras(varmus h.,ann rev genetics 18:553
‑
612(1984)[引用于watson等人,molecular biology of the gene(第4
版;benjamin/cummings publishing co.1987)中]。
[0648]
致癌基因的重复是许多类型癌症的常见原因,如p70
‑
s6激酶1扩增和乳腺癌的情况。在此类情况下,遗传重复发生在体细胞中,并且仅影响癌细胞本身的基因组,而不影响整个生物体,更不用说任何随后的后代。在人癌症中扩增的致癌基因的其它实例包括乳腺癌中的myc、erbb2(efgr)、ccndl(细胞周期蛋白dl)、fgfr1和fgfr2,子宫颈癌中的myc和erbb2,结直肠癌中的hras、kras和myb,食道癌中的myc、ccndl和mdm2,胃癌中的ccne、kras和met,成胶质细胞瘤中的erbb 1和cdk4,头颈癌中的ccndl、erbb1和myc,肝细胞癌中的ccndl,神经母细胞瘤中的mycb,卵巢癌中的myc、erbb2和akt2,肉瘤中的mdm2和cdk4,及小细胞肺癌中的myc。在一个实施方案中,本方法可用于确定与癌症相关的致癌基因的扩增的存在或不存在。在一些实施方案中,经扩增的致癌基因与乳腺癌、子宫颈癌、结直肠癌、食道癌、胃癌、成胶质细胞瘤、头颈癌、肝细胞癌、神经母细胞瘤、卵巢癌、肉瘤和小细胞肺癌有关。
[0649]
在一个实施方案中,本方法可用于确定染色体缺失的存在或缺失。在一些实施方案中,染色体缺失是一种或多种全染色体的丢失。在其它实施方案中,染色体缺失是染色体的一个或多个区段的丢失。在其它实施方案中,染色体缺失是两个或更多个染色体的两个或更多个区段的丢失。染色体缺失可能涉及一种或多种肿瘤抑制子基因的丢失。
[0650]
涉及肿瘤抑制基因的染色体缺失被认为在实体瘤的发展和进展中起重要作用。前列腺癌肿瘤抑制基因(rb
‑
1)位于13号染色体q14中,是最广泛表征的肿瘤抑制基因。rb
‑
1基因产物,105kda核磷蛋白,明显地在细胞周期调控中起重要作用(howe等人,proc natl acad sci(usa)87:5883
‑
5887[1990])。rb蛋白的表达改变或丢失是通过点突变或染色体缺失由两个基因等位基因的失活引起的。已经发现rb
‑
i基因改变不仅存在于前列腺癌中,而且存在于其它恶性肿瘤诸如骨肉瘤、小细胞肺癌(rygaard等人,cancer res 50:5312
‑
5317[1990)])和乳腺癌中。限制性片段长度多态性(rflp)研究表明,这种肿瘤类型在13q处经常失去杂合性,这表明rb
‑
1基因等位基因中的一个因总染色体缺失而丢失(bowcock等人,am j hum genet,46:12[1990])。1号染色体异常,包括重复、缺失和涉及6号染色体和其它伴侣染色体的不平衡易位,表明1号染色体的区域,特别是1q21
‑
1q32和1p11
‑
13,可能具有与骨髓增殖性赘生物的慢性和晚期致病性相关的致癌基因或肿瘤抑制基因(caramazza等人,eur j hematol 84:191
‑
200[2010])。骨髓增殖性赘生物也与5号染色体的缺失有关。5号染色体的完全丢失或中间缺失是骨髓增生异常综合征(mds)中最常见的核型异常。孤立的del(5q)/5q
‑
mds患者比具有另外的核型缺陷的患者具有更好的预后,这些患者倾向于发展骨髓增殖性赘生物(mpn)和急性髓性白血病。不平衡的5号染色体缺失的频率已经导致5q具有一个或多个肿瘤抑制子基因的想法,这些基因在造血干/祖细胞(hsc/hpc)的生长控制中具有基本作用。以5q31和5q32为中心的常见缺失区域(cdr)的细胞遗传学定位鉴定了候选肿瘤抑制子基因,包括核糖体亚基rps14、转录因子egrl/krox20和细胞骨架重构蛋白、α
‑
连环蛋白(eisenmann等人,oncogene 28:3429
‑
3441[2009])。新鲜肿瘤和肿瘤细胞系的细胞遗传学和等位基因分型研究表明,染色体3p上几个不同区域的等位基因丢失,包括3p25、3p21
‑
22、3p21.3、3p12
‑
13和3p14,是最早和最频繁的基因组异常,其参与广泛的肺、乳房、肾脏、头颈部、卵巢、子宫颈、结肠、胰腺、食管、膀胱和其它器官的主要上皮癌。几种肿瘤抑制基因已被定位至染色体3p区域,并且认为中间缺失或启动子超甲基化在癌症发展过程中
3p或整个3号染色体的丢失之前(angeloni d.,briefings functional genomics 6:19
‑
39[2007])。
[0651]
患有唐氏综合征(ds)的新生儿和儿童经常出现先天性短暂性白血病,并且患急性髓性白血病和急性成淋巴细胞性白血病的风险增加。含有约300个基因的21号染色体可能参与白血病、淋巴瘤和实体瘤中的许多结构畸变,如易位、缺失和扩增。此外,已经鉴定出位于21号染色体上的基因在肿瘤发生中起重要作用。体细胞数畸变以及结构21号染色体畸变与白血病相关,并且位于21q的特定基因(包括runx1、tmprss2和tff)在肿瘤发生中起作用(fonatsch c gene chromosomes cancer49:497
‑
508[2010])。
[0652]
鉴于前述内容,在各种实施方案中,本文描述的方法可用于确定已知包含一种或多种原癌基因或肿瘤抑制基因和/或已知与癌症或癌症风险增加相关的区段cnv。在某些实施方案中,可以在包含构成性(种系)核酸的测试样品中测定cnv,并且可以在那些构成性核酸中鉴定区段。在某些实施方案中,在包含核酸(如,来源于正常细胞的核酸和来源于赘生性细胞的核酸)的混合物的样品中鉴定区段cnv(如果存在)。在某些实施方案中,该样品来源于疑似或已知患有癌症(如癌、肉瘤、淋巴瘤、白血病、生殖细胞肿瘤、胚细胞瘤等)的受试者。在一个实施方案中,该样品是从外周血来源(加工)的血浆样品,其可包含来源于正常细胞和癌细胞的cfdna的混合物。在另一个实施方案中,用于确定cnv是否存在的生物样品来源于细胞,如果癌症存在,则其包含来自其它生物组织的癌细胞和非癌细胞的混合物,所述生物组织包括但不限于生物流体诸如血清、汗液、眼泪、痰液、尿液、痰液、耳流液、淋巴液、唾液、脑脊液、ravages、骨髓悬液、阴道流液、宫腔灌洗液、脑液、腹水、乳汁、呼吸道、肠道和泌尿生殖道的分泌物及白细胞去除法样品,或在组织活检、拭子或涂片中。在其它实施方案中,生物样品是粪便(排泄物)样品。
[0653]
用于确定癌症存在和/或癌症风险增加的cnv可包括扩增或缺失。
[0654]
在各种实施方案中,被鉴定为指示癌症存在或癌症风险增加的cnv包括表4中所示的一种或多种扩增。
[0655]
表4.特征在于与癌症相关的扩增的说明性但非限制性的染色体区段。列出的癌症类型是beroukhim等人nature 18:463:899
‑
905中鉴定的那些。
[0656]
[0657]
[0658]
[0659][0660]
在某些实施方案中,与上文(本文)所述的扩增组合,或单独地,被鉴定为指示癌症存在或癌症风险增加的cnv包括表5所示的一种或多种缺失。
[0661]
表5.特征在于与癌症相关的缺失的说明性但非限制性的染色体区段。列出的癌症
类型是beroukhim等人nature 18:463:899
‑
905中鉴定的那些。
[0662]
[0663]
[0664]
[0665]
[0666][0667]
被鉴定为各种癌症特征的非整倍性(如表4和5中鉴定的非整倍性)可含有已知与癌症病因有关的基因(如肿瘤抑制基因、致癌基因等)。这些非整倍性也可以被探测,以鉴定相关但以前未知的基因。
[0668]
例如,beroukhim等人,同上,使用grail(gene relationships among implicated l0c120)评估了拷贝数改变中的潜在致癌基因,该算法搜索基因组区域之间的功能性关系。grail根据引用所述基因的所有论文的已发表摘要之间的文本相似性,基于一些靶基因将在共同途径中起作用的概念,对基因组区域集合中的每个基因就其与其它区域中基因的'相关性'进行评分。这些方法允许鉴定/表征先前与所讨论的特定癌症无关的基因。表6说明
已知在鉴定的扩增区段和预测基因内的靶基因,并且表7说明已知在已鉴定的缺失区段和预测基因内的靶基因。
[0669]
表6.已知或预测存在于以各种癌症中的扩增为特征的区域中的说明性但非限制性的染色体区段和基因(参见,如beroukhim等人,同上)。
[0670]
[0671]
[0672]
[0673][0674]
表7.已知或预测存在于以各种癌症中的扩增为特征的区域中的示例性但非限制性的染色体区段和基因(参见,如beroukhim等人,同上)。
[0675]
[0676]
[0677]
[0678]
[0679][0680]
在某些实施方案中,考虑使用本文鉴定的方法鉴定包含表6中鉴定的扩增区域或基因的区段的cnv,和/或使用本文鉴定的方法鉴定包含表7中鉴定的缺失区域或基因的区段的cnv。
[0681]
在一个实施方案中,本文描述的方法提供了评估基因扩增和肿瘤进化程度之间的关联性的手段。扩增和/或缺失与癌症分期或分级之间的相关性可能具有预后重要性,因为这种信息可能有助于定义基于遗传的肿瘤分级,从而更好地预测未来病程,其中更晚期的肿瘤具有最差的预后。此外,关于早期扩增和/或缺失事件的信息可用于将这些事件与后续疾病进展的预测因子相关联。
[0682]
如通过该方法鉴定的基因扩增和缺失可以与其它已知的参数相关联,诸如肿瘤分级、组织学、brd/urd标记指数、激素状态、淋巴结受累、肿瘤尺寸、存活持续时间和其它可从流行病学和生物统计学研究获得的肿瘤性质。例如,待通过该方法测试的肿瘤dna可包括非典型增生、原位导管癌、i
‑
iii期癌症和转移性淋巴结,以便允许鉴定扩增和缺失与分期之
间的关联性。所建立的关联性可能使有效的治疗性干预成为可能。例如,始终扩增的区域可能含有经过表达的基因,其产物可能能够受到治疗性影响(例如,生长因子受体酪氨酸激酶、p285
her2
)。
[0683]
在某些实施方案中,本文所述的方法可用于通过确定来自原发性癌症的核酸序列的拷贝数变异与转移至其它位点的细胞的那些拷贝数变异来鉴定与耐药性相关的扩增和/或缺失事件。如果基因扩增和/或缺失是允许耐药性快速发展的核型不稳定性表现,则预期来自化学抗性患者的原发性肿瘤中的扩增和/或缺失比化学敏感性患者中的肿瘤中的更多。例如,如果特定基因的扩增导致耐药性的发展,那么围绕那些基因的区域将预期在来自化学抗性患者的胸膜积液的肿瘤细胞中始终扩增,但在原发性肿瘤中则不然。发现基因扩增和/或缺失与耐药性发展之间的关联性可以允许鉴定将会或不会从辅助疗法中受益的患者。
[0684]
以与确定在母体样品中的完整和/或部分胎儿染色体非整倍性的存在或缺失的方式类似的方式,可以使用本文所述的方法、装置和系统来确定在包含核酸如dna或cfdna的任何患者样品(包括不是母体样品的患者样品)中存在或缺失完整和/或部分染色体非整倍性。患者样品可以是如本文其它地方所述的任何生物样品类型。优选地,通过非侵入性程序获得样品。例如,该样品可以是血液样品,或其血清和血浆级分。或者,该样品可以是尿液样品或粪便样品。在其它实施方案中,样品是组织活检样品。在所有情况下,该样品包含核酸如cfdna或基因组dna,其经纯化并且使用先前描述的任何ngs测序方法测序。
[0685]
可以根据本方法确定与形成相关的完全和部分染色体非整倍性和癌症的进展。
[0686]
在某些实施方案中,当使用本文所述的方法来确定癌症的存在和/或增加的风险时,可以针对确定cnv的一个或多个染色体进行数据的归一化。在某些实施方案中,可以针对确定cnv的一个或多个染色体臂进行数据的归一化。在某些实施方案中,可以针对确定cnv的一个或多个特定区段进行数据的归一化。
[0687]
除了cnv在癌症中的作用之外,cnv还与越来越多的常见复杂疾病相关,包括人类免疫缺陷病毒(hiv)、自身免疫性疾病和一系列神经精神病症。
[0688]
传染性和自身免疫性疾病中的cnv
[0689]
迄今为止,许多研究报道了参与炎症和免疫响应和hiv、克罗恩氏病(crohn’s disease)和其它自身免疫性病症的基因中的cnv之间的关联性(fanciulli等人,clin genet 77:201
‑
213[2010])。例如,ccl3l1中的cnv参与hiv/aids易感性(ccl3l1,17q11.2缺失)、类风湿性关节炎(ccl3l1,17q11.2缺失)和川崎病(kawasaki disease) (ccl3l1,17q11.2重复)有关;hbd
‑
2中的cnv已报道对慢性克罗恩氏病(hdb
‑
2,8p23.1缺失)和银屑病(hdb
‑
2,8p23.1缺失)易感;fcgr3b中的cnv显示对全身性红斑狼疮中的肾小球性肾炎(fcgr3b,1q23缺失,1q23重复),抗嗜中性粒细胞胞浆抗体(anca)
‑
相关的血管炎(fcgr3b,1q23缺失)易感并且增加发展类风湿性关节炎的风险。至少有两种已经显示与不同基因座处的cnv相关的炎症或自身免疫性疾病。例如,克罗恩氏病与hdb
‑
2处的低拷贝数相关,但也与编码p47免疫相关gtp酶家族成员的igrm基因上游的常见缺失多态性相关。除了与fcgr3b拷贝数的关联性之外,sle易感性据报道在补体组分c4的拷贝数较低的受试者之间显著增加。
[0690]
在许多独立研究中已经报道了在gstm1(gstm1,1q23缺失)和gstt1(gstt1,
22q11.2缺失)基因座的基因组缺失的与特应性哮喘的风险增加之间的关联性。在一些实施方案中,本文描述的方法可用于确定与炎症和/或自身免疫性疾病相关的cnv的存在或缺失。例如,该方法可用于确定疑似患有hiv、哮喘或克罗恩氏病的患者中的cnv的存在。与此类疾病相关的cnv的实例包括但不限于17q11.2、8p23.1、1q23和22q11.2处的缺失,以及17q11.2和1q23处的复制。在一些实施方案中,本方法可用于确定cnv在基因中的存在,所述基因包括但不限于ccl3l1、hbd
‑
2、fcgr3b、gstm、gstt1、c4和irgm。
[0691]
神经系统的cnv疾病
[0692]
在自闭症、精神分裂症和癫痫以及一些神经退行性疾病情况(诸如帕金森氏病、肌萎缩侧索硬化症(als)和常染色体显性阿尔茨海默氏病)中报道了从头和遗传的cnv与几种常见的神经疾病和精神病学疾病之间的关联性(fanciulli等人,clin genet77:201
‑
213[2010])。在患有自闭症和自闭症谱系障碍(asd)的患者中观察到细胞遗传学异常,其在15q11
‑
q13处具有重复。根据自闭症基因组项目联盟(autism genome project consortium),154个cnv(包括几种复发性cnv),在染色体15q11
‑
q13上或新基因组位置处,包括染色体2p16、1q21和与史密斯
‑
马吉利综合征相关的区域中与asd重叠的17p12。染色体16p11.2上的复发性微缺失或微重复突出了以下观察结果:在诸如shank3(22q13.3缺失)、轴突蛋白1(nrxn1,2p16.3缺失)和neuroglin(nlgn4,xp22.33缺失)的基因的基因座处检测到从头cnv,其已知调控突触分化和调控谷氨酸能神经递质释放。精神分裂症也与多种从头cnv相关。与精神分裂症相关的微缺失和微重复含有属于神经发育和谷氨酸能途径的基因的过表达,这表明影响这些基因的多个cnv可能直接导致精神分裂症的发病,如erbb4,2q34缺失,slcja3,5p13.3缺失;rapegf4,2q31.1缺失;cit,12.24缺失;和具有从头cnv的多个基因。cnv还与其它神经系统病症相关,包括癫痫(chrna 7,15q13.3缺失)、帕金森氏病(snca4q22重复)和als(smn1,5q12.2.
‑
q13.3缺失;和smn2缺失)。在一些实施方案中,本文描述的方法可用于确定与神经系统疾病相关的cnv的存在或缺失。例如,该方法可用于确定疑似患有孤独症、精神分裂症、癫痫、神经退行性疾病诸如帕金森氏病、肌萎缩侧索硬化症(als)或常染色体显性阿尔茨海默氏病的患者中cnv的存在。该方法可用于确定与神经系统疾病(包括但不限于自闭症谱系障碍(asd)、精神分裂症和癫痫中的任一种)相关的基因的cnv,以及与神经退行性病症(诸如帕金森氏病)相关的基因的cnv。与此类疾病相关的cnv的实例包括但不限于15q11
‑
q13、2p16、1q21、17p12、16p11.2和4q22处的重复,以及22q13.3、2p16.3、xp22.33、2q34、5p13.3、2q31.1、12.24、15q13.3和5q12.2处的缺失。在一些实施方案中,该方法可用于确定基因中cnv的存在,所述基因包括但不限于shank3、nlgn4、nrxn1、erbb4、slc1a3、rapgef4、cit、chrna7、snca、smnl和mn2。
[0693]
cnv和代谢性或心血管疾病
[0694]
在许多研究中已经报道了代谢性状和心血管性状(诸如家族性高胆固醇血症(fh)、动脉粥样硬化和冠状动脉疾病)之间的关联性,并且在多项研究中报道了cnv(fanciulli等人,clin genet 77:201
‑
213[2010])。例如,在一些不携带其它ldlr突变的fh患者中,在ldlr基因中观察到种系重排,主要是缺失(ldlr,19p13.2缺失/重复)。另一个实例是编码载脂蛋白(a)(apo(a))的lpa基因,其血浆浓度与冠状动脉疾病、心肌梗塞(mi)和中风的风险相关。含有脂蛋白lp(a)的apo(a)的血浆浓度在个体之间变化超过1000倍,并且90%的这种变异性在lpa基因座处经遗传测定,其中血浆浓度和lp(a)同种型尺寸与'
kringle 4'重复序列的高度可变数量(范围5
‑
50)成比例。这些数据表明在至少两个基因中的cnv可能与心血管风险相关。本文描述的方法可用于大型研究中以特异性搜索cnv与心血管病症的相关性。在一些实施方案中,本方法可用于确定与代谢疾病或心血管疾病相关的cnv的存在或缺失。例如,本方法可用于确定疑似患有家族性高胆固醇血症的患者中cnv的存在。本文描述的方法可用于确定与代谢疾病或心血管疾病如高胆固醇血症相关的基因的cnv。与此类疾病相关的cnv的实例包括但不限于ldlr基因的19p13.2缺失/重复,以及lpa基因的倍增。
[0695]
用于确定cnv的装置和系统
[0696]
通常使用各种计算机执行的算法和程序来执行测序数据的分析和从中导出的诊断。因此,某些实施方案采用涉及存储在一个或多个计算机系统或其它处理系统中或者通过一个或多个计算机系统或其它处理系统传输的数据的方法。本文公开的实施方案还涉及用于执行这些操作的装置。该装置可以为所需目的而专门构造,或者它可以是通过计算机程序和/或存储在计算机中的数据结构选择性地激活或重新配置的通用计算机(或一组计算机)。在一些实施方案中,一组处理器协同地(例如,经由网络或云计算)和/或并行地执行所述分析操作中的一些或全部。用于执行本文描述的方法的处理器或处理器组可以是各种类型的,包括微控制器和微处理器,诸如可编程设备(如,cpld和fpga)和非可编程设备,诸如门阵列asic或通用微处理器。
[0697]
此外,某些实施方案涉及有形和/或非暂时性计算机可读介质或计算机程序产品,其包括用于执行各种计算机
‑
实现的操作的程序指令和/或数据(包括数据结构)。计算机可读介质的实例包括但不限于,半导体存储设备,磁性介质诸如磁盘驱动器、磁带,光学介质诸如cd,磁光介质以及专门配置用于存储和执行程序指令的硬件设备,诸如只读存储设备(rom)和随机访问存储器(ram)。计算机可读介质可以由终端用户直接控制,或者介质可以由终端用户间接控制。直接控制的介质的实例包括位于用户设施处的介质和/或不与其它实体共享的介质。间接控制的介质的实例包括用户经由外部网络和/或经由提供共享资源的服务(诸如
″
云
″
)间接访问的介质。程序指令的实例包括诸如由编译器产生的机器代码,以及包含可由计算机使用解释器执行的更高级代码的文件。
[0698]
在某些实施方案中,所公开的方法和装置中采用的数据或信息以电子格式提供。这样的数据或信息可以包括来源于核酸样品的读取和标签,与参考序列的特定区域比对(如,与染色体或染色体区段比对)的此类标签的计数或密度,参考序列(包括仅提供或主要提供多态性的参考序列),染色体和区段剂量,识别诸如非整倍性调用、归一化染色体和区段值、染色体对或区段对及相应的归一化染色体或区段、咨询建议、诊断等。如本文所用,以电子格式提供的数据或其它信息可用于存储在机器上并在机器之间传输。通常,电子格式的数据以数字方式提供,并且可以作为位和/或字节存储在各种数据结构、列表、数据库等中。数据可以以电子、光学方式等体现。
[0699]
一个实施方案提供了用于在测试样品中产生指示存在或不存在非整倍性(如胎儿非整倍性)或癌症的输出的计算机程序产品。计算机产品可以含有用于执行用于确定染色体异常的任一种或多种上述方法的指令。如所解释的,计算机产品可以包括非暂时性和/或有形计算机可读介质,其上记录有计算机可执行或可编译逻辑(如,指令)用于使处理器能够确定染色体剂量,并且在一些情况下,确定存在还是缺失胎儿非整倍性。在一个实例中,
计算机产品包括计算机可读介质,其上记录有计算机可执行或可编译逻辑(如,指令)用于使处理器能够诊断胎儿非整倍性,其包括:用于从来自母体生物样品的至少一部分的核酸分子接收测序数据的接收程序,其中所述测序数据包括计算的染色体和/或区段剂量;用于从所述接收数据分析胎儿非整倍性的计算机辅助逻辑;和输出程序,用于产生指示所述胎儿非整倍性的存在、不存在或种类的输出。
[0700]
可以将来自所考虑的样品的序列信息定位至染色体参考序列,以鉴定任一个或多个目标染色体中的每一个的序列标签数目,并鉴定用于所述任一种或多种目标染色体中的每一个的归一化区段序列的多个序列标签。在某些实施方案中,将参考序列存储在数据库中,诸如关系数据库或对象数据库,例如。
[0701]
应当理解,在大多数情况下,对于未受协助的人来说,执行本文公开的方法的计算操作是不实际的,或者甚至是不可能的。例如,在无计算装置帮助的情况下,将来自样品的单个30bp的读取定位至任何一个人染色体可能需要多年的努力。当然,问题是复杂的,因为可靠的非整倍性识别通常需要将数千(如,至少约10,000)或甚至数百万的读取定位至一个或多个染色体。
[0702]
可以使用用于评估测试样品中遗传目标序列的拷贝数的系统来执行本文公开的方法。该系统包括:(a)测序仪,其用于接收来自测试样品的核酸,提供来自样品的核酸序列信息;(b)处理器;以及(c)一个或多个计算机可读存储介质,其上存储有用于在所述处理器上执行的指令,以执行鉴定任何cnv(如染色体或部分非整倍性)的方法。在一些实施方案中,该方法由计算机可读介质指示,其上存储有计算机可读指令,用于执行鉴定任何cnv(如染色体或部分非整倍性)的方法。因此,一个实施方案提供了计算机程序产品,其包括一个或多个计算机可读的非暂时性存储介质,其上存储有计算机可执行指令,所述指令当由计算机系统的一个或多个处理器执行时,使计算机系统实现一种用于评估包含胎儿和母体无细胞核酸的测试样品中目标序列的拷贝数的方法。该方法包括:(a)接收通过对测试样品中的无细胞核酸片段进行测序获得的序列读取;(b)将无细胞核酸片段的序列读取与包含目标序列的参考基因组比对,从而提供测试序列标签,其中参考基因组被分成多个箱;(c)确定测试样品中存在的无细胞核酸片段的尺寸;(d)基于从其获得标签的无细胞核酸片段的尺寸对测试序列标签进行加权;(e)基于(d)的加权标签,计算箱的覆盖率;以及(f)从计算的覆盖率中鉴定目标序列中的拷贝数变异。在一些实施方式中,加权测试序列标签涉及将覆盖率偏向从测试样品中的一个基因组的特征性尺寸或尺寸范围的无细胞核酸片段获得的测试序列标签。在一些实施方式中,对测试序列标签进行加权涉及将值1赋予给从尺寸或尺寸范围的无细胞核酸片段获得的标签,并将值0指定给其它标签。在一些实施方式中,该方法还涉及在包含目标序列的参考基因组的箱中确定片段尺寸参数值,其包括具有比阈值更短或更长的片段尺寸的测试样品中的无细胞核酸片段的量。这里,鉴定目标序列中的拷贝数变异涉及使用片段尺寸参数值以及在(e)中计算的覆盖率。在一些实施方式中,该系统经配置使用上述各种方法和过程来评估测试样品中的拷贝数。
[0703]
在一些实施方案中,指令可进一步包括在提供母体测试样品的人类受试者的患者医疗记录中自动记录与该方法相关的信息,诸如染色体剂量和存在或缺失胎儿染色体非整倍性。患者医疗记录可以由例如实验室、医生办公室、医院、健康维护组织、保险公司或个人医疗记录网站维护。此外,基于处理器实现的分析的结果,该方法可以进一步涉及对从其取
得母体测试样品的人类受试者开处方、开始和/或改变治疗。这可以涉及对取自受试者的另外样品进行一次或多次另外的测试或分析。
[0704]
所公开的方法也可以使用计算机处理系统来执行,该计算机处理系统适于或者被配置为执行用于鉴定任何cnv(如染色体或部分非整倍性)的方法。一个实施方案提供了一种计算机处理系统,其适于或者被配置为执行如本文所述的方法。在一个实施方案中,该装置包括适于或者被配置为对样品中的至少一部分核酸分子进行测序的测序装置,以获得本文其它各处所述的序列信息类型。该装置还可包括用于加工样品的组件。此类组件在本文其它地方描述。
[0705]
序列或其它数据,可以直接或间接地输入计算机或存储在计算机可读介质上。在一个实施方案中,计算机系统直接耦合至测序装置,该测序装置从样品读取和/或分析核酸序列。来自此类工具的序列或其它信息经由计算机系统中的界面提供。或者,由该系统处理的序列由序列存储源诸如数据库或其它存储库提供。一旦可用于该处理装置,存储设备或大容量存储设备至少暂时地缓冲或存储核酸序列。此外,存储设备可以存储各个染色体或基因组等的标签计数。存储器还可以存储用于分析呈现序列或定位的数据的各种例程和/或程序。此类程序/例程可以包括用于执行统计分析等的程序。
[0706]
在一个实例中,用户将样品提供到测序装置中。由连接到计算机的测序装置收集和/或分析数据。计算机上的软件允许数据收集和/或分析。可以将数据存储、显示(通过监视器或其它类似设备)和/或发送到另一个位置。计算机可以连接到互联网,互联网用于将数据传输到远程用户(如医生、科学家或分析员)使用的手持设备。应当理解,可以在传输之前存储和/或分析数据。在一些实施方案中,收集原始数据并将其发送到将分析和/或存储数据的远程用户或装置。传输可以经由互联网进行,但也可以经由卫星或其它连接进行。或者,数据可以存储在计算机可读介质上,并且该介质可以被运送到终端用户(如,经由邮件)。远程用户可以位于相同或不同的地理位置,包括但不限于建筑物、城市、州、国家或大陆。
[0707]
在一些实施方案中,该方法还包括收集关于多条多核苷酸序列(如,读取、标签和/或参考染色体序列)的数据,并将数据发送到计算机或其它计算系统。例如,计算机可以连接到实验室设备,如样品收集装置、核苷酸扩增装置、核苷酸测序装置或杂交装置。然后,计算机可以收集由实验室设备收集的可用数据。数据可以在任何步骤存储在计算机上,如在发送之前、发送期间或与发送一起或发送之后实时收集。可以将数据存储在可以从计算机中提取的计算机可读介质上。可以如经由本地网络或诸如因特网的广域网将收集或存储的数据从计算机传输到远程位置。在远程位置,可以对传输的数据执行各种操作,如下所述。
[0708]
在本文公开的系统、装置和方法中可以存储、传输、分析、和/或操纵的电子格式数据类型如下:
[0709]
通过对测试样品中的核酸进行测序来获得的读取
[0710]
通过将读取与参考基因组或其它参考序列进行比对来获得的标签
[0711]
参考基因组或序列
[0712]
序列标签密度
‑
参考基因组或其它参考序列的两个或更多个区域(通常是染色体或染色体区段)中的每一个的标签的计数或数量
[0713]
对特定目标染色体或染色体区段的归一化染色体或染色体区段的标识
[0714]
从目标染色体或区段和相应的归一化染色体或区段获得的染色体或染色体区段(或其它区域)的剂量
[0715]
用于调用染色体剂量作为受影响、不受影响或无调用的阈值
[0716]
染色体剂量的实际调用
[0717]
诊断(与调用相关的临床病况)
[0718]
来源于调用和/或诊断的进一步测试的建议
[0719]
来源于调用和/或诊断的治疗和/或监测方案
[0720]
可以使用不同的装置在一个或多个位置处获得、存储、传输、分析和/或操纵这些各种类型的数据。处理选项涉及广泛的范围。在范围的一端,所有或大部分信息在处理测试样品的位置处被存储并使用,如医生办公室或其它临床环境。在另一个极端情况下,在一个位置获得样品,将它处理并且任选地在不同的位置处进行测序,将读取进行比对并且在一个或多个不同的位置处进行调用,并且在另一个位置(其可以是获得样品的位置)准备诊断、建议和/或计划。
[0721]
在某些实施方案中,利用测序装置生成读取,然后将其传输到远程站点,在远程站点处理它们以产生非整倍性识别。在这个远程位置,作为一个实例,将读取与参考序列比对以产生标签,这些标签被计数并分配给目标染色体或区段。同样在远程位置,使用相关的归一化染色体或区段将计数转换为剂量。此外,在远程位置,剂量用于产生非整倍性识别。
[0722]
可以在不同位置使用的加工操作包括:
[0723]
样品收集
[0724]
测序之前的样品处理
[0725]
测序
[0726]
分析序列数据并推导非整倍性调用
[0727]
诊断
[0728]
报告诊断和/或调用给患者或健康护理提供者
[0729]
制定用于进一步处理、测试和/或监测的计划
[0730]
执行计划
[0731]
咨询
[0732]
这些操作中的任一个或多个可以如本文其它地方所述自动化。通常,对序列数据进行的测序和分析以及导出非整倍性调用将在计算上执行。另一个操作可以手动或自动执行。
[0733]
可以进行样品收集的位置的实例包括卫生从业者办公室、诊所、患者家(其中提供样品收集工具或试剂盒)和移动健康护理车辆。可以在测序之前执行样品处理的位置的实例包括健康从业者办公室、诊所、患者家(其中提供样品加工装置或试剂盒)、移动健康护理车辆和非整倍性分析提供者设施。可以为测试发生的位置提供专用网络连接,用于以电子格式传输序列数据(通常是读取)。此类连接可以是有线的或无线的,并且具有并且可以被配置为将数据发送到可以在传输到处理站点之前处理和/或聚合数据的站点。数据聚合器可由健康组织诸如健康维护组织(health maintenance organizations,hmo)维护。
[0734]
分析和/或推导操作可以在任何前述位置处执行,或者可以在专用于计算和/或分析核酸序列数据服务的另一远程站点处执行。此类位置包括例如,集群诸如通用服务器农
场、非整倍性分析服务业的设施等。在一些实施方案中,用于执行分析的计算装置是租赁的或租用的。计算资源可以是因特网可访问的处理器集合的一部分,诸如俗称为云的处理资源。在一些情况下,计算由并行或大规模并行的处理器组执行,这些处理器彼此关连或不关连。可以使用分布式处理诸如集群计算、网格计算等来完成处理。在此类实施方案中,计算资源的集群或网格共同形成超级虚拟计算机,该超级虚拟计算机由多个处理器或计算机(一起作用以执行本文所述的分析和/或推导)组成。这些技术以及更多常规的超级计算机可用于处理如本文所述的序列数据。每种都是依赖于处理器或计算机的并行计算形式。在网格计算的情况下,这些处理器(通常是整个计算机)通过常规网络方案诸如以太网通过网络(专用、公共或因特网)连接。相比之下,超级计算机有许多通过本地高速计算机总线连接的处理器。
[0735]
在某些实施方案中,诊断(如,胎儿患有唐氏综合征或患者患有特定类型的癌症)在与分析操作相同的位置产生。在其它实施方案中,它在不同的位置进行。在一些实例中,报告诊断是在取得样品的位置处执行的,尽管不一定是这种情况。可以生成或报告诊断和/或进行计划开发的位置的实例包括有线或无线连接到网络的健康从业者办公室、诊所、可通过计算机访问的因特网站点、以及诸如手机、平板电脑、智能电话等的手持设备。进行咨询的位置的实例包括健康从业者办公室、诊所、可通过计算机访问的互联网站点、手持设备等。
[0736]
在一些实施方案中,在第一位置执行样品收集、样品处理和测序操作,并且在第二位置执行分析和推导操作。然而,在一些情况下,样品收集在一个位置(如,医疗从业者办公室或诊所)收集,并且样品加工和测序在不同的位置处执行,该位置任选地是进行分析和推导的相同位置。
[0737]
在某些实施方案中,以上列出的操作的序列可以由启动样品收集、样品处理和/或测序的用户或实体触发。在一个或多个这些操作开始执行之后,其它操作可以自然地跟随。例如,测序操作可以使读取段自动收集并发送到处理装置,然后该处理装置通常自动并且可能无需进一步的用户干预地进行非整倍性操作的序列分析和推导。在一些实施方式中,然后将该处理操作的结果自动递送(可能重新格式化为诊断)到处理向健康专业人员和/或患者报告信息的系统组件或实体。如所解释的,此类信息也可以被自动处理以产生治疗、测试和/或监测计划,可能连同咨询信息。因此,启动早期操作可以触发端至端序列,其中向健康专业人员、患者或其它相关方提供诊断、计划、咨询和/或对于作用于身体状况有用的其它信息。即使整个系统的部分是物理分离的并且可能远离如样品和序列装置的位置,这也可以实现。
[0738]
图5显示了用于从测量样品产生调用或诊断的分散系统的一种实施方式。样品收集位置01用于从患者诸如妊娠女性或推定的癌症患者获得测试样品。然后将样品提供给处理和测量位置03,其中可以如上所述处理测试样品并且进行测序。位置03包括用于处理样品的装置以及用于对经处理的样品进行测序的装置。如本文其它地方所述,测序的结果是读取的集合,其通常以电子格式提供并且提供给诸如因特网的网络,其由图5中的参考号05指示。
[0739]
将序列数据提供给远程位置07,在远程位置07执行分析和调用生成。该位置可以包括一个或多个强大的计算设备,诸如计算机或处理器。在位置07处的计算资源完成其分
析并从所接收的序列信息生成调用之后,调用被中继回网络05。在一些实施方式中,不仅在位置07处生成调用而且也生成相关诊断。然后,调用和或诊断通过网络传输并返回到样品收集位置01,如图5所示。如所解释,这仅仅是关于如何在各个位置之间划分与生成调用或诊断相关联的各种操作的许多变化之一。一种常见变体涉及在单个位置提供样品收集和处理以及测序。另一种变化涉及在与分析和调用生成相同的位置提供处理和测序。
[0740]
图6详细说明了在不同位置处执行各种操作的选项。在图6中描绘的最精细意义上,以下每个操作在单独的位置执行:样品收集、样品处理、测序、读取比对、调用、诊断和报告和/或计划开发。
[0741]
在聚集这些操作中的一些的实施方案中,在一个位置执行样品处理和测序,并且在单独的位置执行读取比对、调用和诊断。参见由参考字符a标识的图6的部分。在图6中由字符b标识的另一个实施方式中,样品收集、样品处理和测序都在相同位置处执行。在这种实施方式中,读取比对和调用在第二位置中执行。最后,诊断和报告和/或计划开发在第三个位置中进行。在图6中由字符c描绘的实施方式中,样品收集在第一位置处执行,样品处理、测序、读取比对、调用和诊断都在第二位置处一起执行,并且报告和/或计划开发在第三位置处进行。最后,在图6中d标记的实施方式中,样品收集在第一位置处执行,样品处理、测序、读取比对和调用都在第二位置处执行,并且诊断和报告和/或计划管理在第三位置处进行。
[0742]
一个实施方案提供了用于在包含胎儿和母体核酸的母体测试样品中确定存在或缺失任一种或多种不同的完整胎儿染色体非整倍性的系统,该系统包括用于接收核酸样品并提供来自样品的胎儿和母体核酸序列信息的测序仪;处理器;以及包括在所述处理器上执行的指令的机器可读存储介质,所述指令包括:
[0743]
(a)用于获得样品中所述胎儿和母体核酸的序列信息的代码;
[0744]
(b)用于使用所述序列信息来计算地鉴定选自1
‑
22号染色体、x染色体和y染色体的一个或多个目标染色体中的每一个的来自胎儿和母体核酸的序列标签数量,并鉴定所述任一个或多个目标染色体中的每一个的至少一条归一化染色体序列或归一化染色体区段序列的序列标签数量的代码;
[0745]
(c)用于使用对所述任一个或多个目标染色体中的每一个所鉴定的所述序列标签数量和对每条归一化染色体序列或归一化染色体区段序列所鉴定的所述序列标签数量以计算任一个或多个目标染色体中的每一个的单个染色体剂量的代码;和
[0746]
(d)用于比较任一个或多个目标染色体中的每一个的单个染色体剂量的每一个与一个或多个目标染色体中的每一个的相应阈值,并从而确定样品中存在或缺失任一个或多个完整的不同胎儿染色体非整倍性的代码。
[0747]
在一些实施方案中,用于计算任一个或多个目标染色体的每一个的单个染色剂剂量的代码包括用于计算所选择的一个目标染色体的染色体剂量为对所选目标染色体鉴定的序列标签数量与对所选目标染色体的相应至少一条归一化染色体序列或归一化染色体区段序列所鉴定的序列标签数量的比率的代码。
[0748]
在一些实施方案中,该系统还包括用于重复计算任一个或多个目标染色体的任一个或多个区段的任何剩余染色体区段中的每一个的染色体剂量的代码。
[0749]
在一些实施方案中,选自1
‑
22号染色体、x染色体和y染色体的一个或多个目标染
色体包括选自1
‑
22号染色体、x染色体和y染色体的至少二十个染色体,并且其中指令包括用于确定存在或缺失至少二十个不同的完整胎儿染色体非整倍性的指令。
[0750]
在一些实施方案中,至少一条归一化染色体序列是选自1
‑
22号染色体、x染色体和y染色体的一组染色体。在其它实施方案中,至少一条归一化染色体序列是选自1
‑
22号染色体、x染色体和y染色体的单一染色体。
[0751]
另一实施方案提供了一种用于确定包含胎儿和母体核酸的母体测试样品中存在或缺失任一种或多种不同部分胎儿染色体非整倍性的系统,该系统包括:用于接收核酸样品的测序仪并提供来自样品的胎儿和母体核酸序列信息;处理器;和包括在所述处理器上执行的指令的机器可读存储介质,所述指令包括:
[0752]
(a)用于获得所述样品中所述胎儿和母体核酸的序列信息的代码;
[0753]
(b)用于使用所述序列信息来计算地鉴定选自1
‑
22号染色体、x染色体和y染色体的任一个或多个目标染色体的任一个或多个区段的来自胎儿和母体核酸的序列标签数量以鉴定所述任一个或多个目标染色体的任一个或多个区段中的每一个的至少一条归一化区段序列的序列标签数量的代码;
[0754]
(c)用于使用对所述任一个或多个目标染色体的任一个或多个区段中的每一个所鉴定的所述序列标签数量和对所述归一化区段序列所鉴定的所述序列标签数量以计算所述任一个或多个目标染色体的任一个或多个区段中的每一个的单个染色体区段剂量的代码;和
[0755]
(d)用于比较所述任一个或多个目标染色体的任一个或多个区段中的每一个的单个染色体区段剂量与所述任一个或多个目标染色体的任一个或多个染色体区段中的每一个的相应阈值,并从而确定所述样品中存在或缺失一个或多个不同的部分胎儿染色体非整倍性的代码。
[0756]
在一些实施方案中,用于计算单个染色体区段剂量的代码包括用于将所选染色体区段中的一个的染色体区段剂量计算为对所选染色体区段鉴定的序列标签数量与对所选染色体区段的相应归一化区段序列所鉴定的序列标签数量的比率的代码。
[0757]
在一些实施方案中,该系统还包括用于重复计算任一个或多个目标染色体的任一个或多个区段的任何剩余染色体区段中的每一个的染色体区段剂量的代码。
[0758]
在一些实施方案中,该系统还包括(i)用于对来自不同母体受试者的测试样品重复(a)
‑
(d)的代码,以及(ii)用于确定在所述样品的每一个中存在或缺失任一个或多个不同的部分胎儿染色体非整倍性代码。
[0759]
在本文提供的任何系统的其它实施方案中,该代码还包括用于自动记录在提供母体测试样品的人受试者的患者医疗记录中存在或缺失如在(d)中所确定的胎儿染色体非整倍性的代码,其中使用处理器进行记录。
[0760]
在本文提供的任何系统的一些实施方案中,测序仪经配置用于执行下一代测序(ngs)。在一些实施方案中,测序仪经配置用于使用具有可逆染料终止子的边合成边测序来执行大规模并行测序。在其它实施方案中,测序仪经配置用于执行边连接边测序。在其它实施方案中,测序仪经配置用于执行单分子测序。
实施例
[0761]
实施例1
[0762]
初始和经富集的测序文库的制备和测序
[0763]
a.制备测序文库
‑
缩略方案(abb)
[0764]
所有测序文库,即初始和经富集的文库,是由从母体血浆中提取的大约2ng的纯化cfdna制备的。文库制备使用nebnext
tm
dna样品制备dna试剂组1(nebnext
tm
dna sample prep dna reagent set 1,部件号e6000l;new england biolabs,ipswich,ma)的试剂进行,用于如下的因为无细胞血浆dna在自然界中是片段化的,所以没有对血浆dna样品通过雾化或超声处理进行进一步的片段化。根据末端修复模块,通过在20℃,在1.5ml微量离心管中孵育cfdna与5μl 10x磷酸化缓冲液、2μl脱氧核苷酸溶液混合物(10mm,各dntp)、1μl1:5稀释的dna聚合酶i、1μl t4 dna聚合酶及1μl t4多核苷酸激酶(在nebnext
tm dna样品制备dna试剂组1中提供)持续15分钟,来将包含在40μl中的约2ng纯化cfdna片段的突出端转化为经磷酸化的平末端。然后通过将反应混合物在75℃下孵育5分钟来热灭活酶。将混合物冷却至4℃,并使用10μl含有klenow片段(3'至5'外切
‑
)的da加尾主混合物(nebnext
tm
dna样品制备dna试剂组1)完成平末端dna的da加尾,并在37℃孵育15分钟。随后,通过将反应混合物在75℃孵育5分钟使klenow片段热灭活。在klenow片段灭活之后,1μl 1:5稀释的illumina基因组接头寡核苷酸混合物(部件号1000521;illumina inc.,hayward,ca)用于通过将反应混合物在25℃孵育15分钟,使用nebnext
tm
dna样品制备dna试剂组1中提供的4μl t4 dna连接酶,来连接illumina接头(非
‑
索引y
‑
接头)至da
‑
加尾的dna。将混合物冷却至4℃,并且使用agencourt ampure xp pcr纯化系统(部件号a63881;beckman coulter genomics,danvers,ma)中提供的磁珠,将接头连接的cfdna从未连接的接头、接头二聚体和其它试剂中纯化。使用高保真主混合物(25μl;finnzymes,woburn,ma)和与接头互补的illumina的pcr引物(各0.5μμ)(部件号1000537和1000537),进行18个循环的pcr以选择性富集接头连接的cfdna(25μl)。根据制造商的说明书,使用illumina基因组pcr引物(部件号100537和1000538)和nebnext
tm
dna样品制备dna试剂组1中提供的phusion hf pcr主混合物,使接头
‑
连接的dna进行pcr(98℃持续30秒;18个循环的98℃持续10秒,65℃持续30秒和72℃持续30;最终延伸在72℃下,持续5分钟,并在4℃保持)。根据可从www.beckmangenomics.com/products/ampurexpprotocol_000387v001.pdf获得的制造商的说明书,使用agencourt ampure xp pcr纯化系统(agencourt bioscience corporation,beverly,ma)将经扩增的产物纯化。将经纯化的扩增产物在40μl qiagen eb缓冲液中洗脱,并使用用于2100生物分析仪的agilent dna1000试剂盒(agilent technologies inc.,santa clara,ca)分析经扩增的文库的浓度和尺寸分布。
[0765]
b.制备测序文库—全长方案
[0766]
本文所述的全长方案基本上是由illumina提供的标准方案,并且仅与illumina方案在经扩增的文库的纯化方面不同。illumina方案指导使用凝胶电泳将经扩增的文库纯化,而本文所述的方案使用磁珠进行相同的纯化步骤。基本上根据制造商的说明书,使用nebnext
tm
dna样品制备dna试剂组1(部件号e6000l;new england biolabs,ipswich,ma),使用从母体血浆中提取的约2ng纯化cfdna制备初始测序文库用于除了使用agencourt磁珠和试剂代替纯化柱进行的接头连接产物的最终纯化之外的所有步骤,均根
据伴随用于使用gail进行测序的基因组dna文库的样品制备的nebnext
tm
试剂的方案进行。nebnext
tm
方案基本上按照可从grcf.jhml.edu/hts/protocols/11257047_chip_sample_prep.pdf获得的由illumina提供的方案进行。
[0767]
根据末端修复模块(end repair module),通过在20℃,在200μl微量离心管中孵育40μ1cfdna与5μl 10x磷酸化缓冲液、2μl脱氧核苷酸溶液混合物(10mm,各dntp)、1μl的1:5稀释的dna聚合酶i、1μl的t4 dna聚合酶及1μl的t4多核苷酸激酶(在nebnext
tm
dna样品制备dna试剂组1中提供)持续30分钟,来将包含在40μl中的约2ng纯化cfdna片段的突出端转化为经磷酸化的平末端。将样品冷却至4℃,并如下使用qiaquick pcr纯化试剂盒(qiagen inc.,valencia,ca)中提供的qiaquick柱进行纯化。将50μl反应物转移至1.5ml微量离心管中,并加入250μl的qiagen缓冲液pb。将得到的300μl转移至qiaquick柱,将其在微量离心机中以13,000rpm离心1分钟。用750μl qiagen缓冲液pe洗涤柱,并再次离心。通过以13,000rpm再离心5分钟,除去残留的乙醇。通过离心将dna在39μl qiagen缓冲液eb中洗脱。根据制造商的da
‑
加尾模块(da
‑
tailing module),使用16μl含有klenow片段(3'至5'外切
‑
)的da
‑
加尾主混合物(nebnext
tm
dna样品制备dna试剂组1)并在37℃孵育30分钟,实现34μl平末端dna的da加尾。将样品冷却至4℃,并如下使用minelute pcr纯化试剂盒(minelute pcr purification kit,qiagen inc.,valencia,ca)中提供的柱进行纯化。将50μl反应物转移至1.5ml微量离心管,并加入250μl qiagen缓冲液pb。将300μl转移至minelute柱,将其在微量离心机中以13,000rpm离心1分钟。将柱用750μl qiagen缓冲液pe洗涤,并再次离心。通过以13,000rpm再离心5分钟,除去残留的乙醇。通过离心将dna在15μl qiagen缓冲液eb中洗脱。根据快速连接模块(quick ligation module),将10微升dna洗脱液与1μl 1:5稀释的illumina基因组接头寡核苷酸混合物(部件号1000521)、15μl 2x快速连接反应缓冲液和4μlquickt4 dna连接酶在25℃一起孵育15分钟。将样品冷却至4℃,并如下使用minelute柱纯化。将150微升qiagen缓冲液pe加入到30μl反应物,并将整个体积转移至minelute柱,将其在微量离心机中以13,000rpm离心1分钟。将柱用750μlqiagen缓冲液pe洗涤,并再次离心。通过以13,000rpm再离心5分钟,除去残留的乙醇。通过离心将dna在28μl qiagen缓冲液eb中洗脱。根据制造商的说明书,使用illumina基因组pcr引物(部件号100537和1000538)和nebnext
tm
dna制备dna试剂组1中提供的phusion hf pcr主混合物(phusion hf pcr master mix),使23微升接头连接的dna洗脱液进行18个pcr循环(98℃持续30秒;18个循环的98℃持续10秒,65℃持续30秒,和72℃持续30;最终延伸72℃持续5分钟并在4℃下维持)。根据可从www.beckmangenomics.com/products/ampurexpprotocol_000387v001.pdf获得的制造商的说明书,使用agencourt ampure xp pcr纯化系统(agencourt bioscience corporation,beverly,ma)将扩增产物纯化。agencourt ampure xp pcr纯化系统可去除未掺入的dntp、引物、引物二聚体、盐和其它污染物,并回收大于100bp的扩增子。在40μl qiagen eb缓冲液中从agencourt珠粒洗脱经纯化的扩增产物,并使用用于2100生物分析仪的agilent dna1000试剂盒(agilent technologies inc.,santa clara,ca)分析文库的尺寸分布。
[0768]
c. 分析根据缩略(a)和全长(b)方案制备的测序文库
[0769]
由生物分析仪(bioanalyzer)产生的电泳图示于图7a和7b中。图7a显示了使用(a)
中描述的全长方案从由血浆样品m24228纯化的cfdna制备的文库dna的电泳图,并且图7b显示了使用(b)中描述的全长方案从由血浆样品m24228纯化的cfdna制备的文库dna的电泳图。在两个图中,峰1和4分别代表15bp的下标记(lower marker)和1,500上标记(upper marker);峰值以上的数字表示文库片段的迁移时间;并且水平线表示积分(integration)的设定阈值。图7a中的电泳图显示187bp的片段的次要峰和263bp的片段的主峰,而图7b中的电泳图仅显示265bp处的一个峰。峰面积的积分导致图7a中187bp峰的dna的计算浓度为0.40ng/μl,图7a中263bp峰的dna的浓度为7.34ng/μl,并且图7b中265bp峰的dna的浓度为14.72ng/μl。已知连接到cfdna的illumina接头是92bp,其当从265bp中减去时,表明cfdna的峰尺寸是173bp。187bp处的次要峰可能代表端
‑
至
‑
端连接的两个引物的片段。当使用缩略方案时,直链双引物片段从最终的文库产物中消除。缩略方案还消除了小于187bp的其它较小片段。在该实施例中,经纯化的接头连接的cfdna的浓度是使用全长方案产生的接头连接的cfdna的浓度的两倍。已经注意到,接头连接的cfdna片段的浓度总是大于使用全长方案获得的浓度(数据未显示)。
[0770]
因此,使用缩略方案制备测序文库的一个优势是获得的文库始终只包含262
‑
267bp范围内的一个主峰,而使用全长方案制备的文库的质量如除了代表cfdna之外的峰数目和迁移率所反映的那样变化。非cfdna产物将占据流动池上的空间并降低簇扩增和随后的测序反应成像的质量,这是非整倍性状态的总体分配的基础。缩略方案显示不影响文库的测序。
[0771]
使用缩略方案制备测序文库的另一个优势是平末端、d
‑
a加尾和接头连接的三个酶促步骤需要不到一个小时来完成,以支持快速非整倍体诊断性服务的验证和实施。
[0772]
另一个优势是平末端、d
‑
a加尾和接头连接的三个酶促步骤在相同的反应管中进行,从而避免了可能导致材料丢失的多个样品转移,并且更重要的是可能的样品混淆和样品污染。
[0773]
实施例2
[0774]
使用片段尺寸进行的非侵入性产前检测
[0775]
引言
[0776]
自2011年底和2012年初商业引入以来,母体血浆中无细胞dna(cfdna)的非侵入性产前检测(nipt)已迅速成为筛选有高胎儿非整倍性风险的孕妇的首选方法。该方法主要基于对孕妇血浆中的cfdna进行分离和测序,并对与参考人基因组的特定区域比对的cfdna片段的数量进行计数(参考文献:fan等人,lo等人)。这些dna测序和分子计数方法允许高精度地确定跨基因组的每个染色体的相对拷贝数。在多个临床研究中可重复地实现对21三体、18和13的检测的高灵敏度和特异性(参考文献,引用gil/nicolaides元分析)。
[0777]
最近,另外的临床研究表明,这种方法可以扩展到一般的产科人群。高风险人群和平均风险人群之间的胎儿分数没有可检测的差异(参考文献)。临床研究结果表明,使用通过cfdna测序进行的分子计数的nipt在两个群体中表现相同。已经证实了正预测值(ppv)相对于标准血清筛选的统计学显著提高(参考文献)。与血清生化和颈部半透明度测量相比,较低的假阳性测试结果显著降低了对侵入性诊断性程序的需求(参见来自abuhamad's group的larion等人的参考文献)。
[0778]
鉴于在一般产科群体中良好的nipt表现,工作流程的简单性和成本现已成为在一
般产科群体中实施全染色体非整倍性检测的cfdna测序的主要考虑因素(参考文献:ispd辩论1,brisbane)。大多数nipt实验室方法在文库制备和单末端测序之后利用聚合酶链式反应(pcr)扩增步骤,其需要1000
‑
2000万个独特的cfdna片段以实现合理的灵敏度以检测非整倍性。基于pcr的工作流程的复杂性和更深的测序要求限制了nipt测定的潜力,并导致成本增加。
[0779]
这里证明了使用非常低的cfdna输入而不需要pcr扩增的简单文库制备可以实现高分析灵敏度和特异性。无pcr方法简化了工作流程,提高了周转时间并消除了pcr方法固有的偏差。无扩增工作流程可以与配对末端测序结合,以允许确定每个样品中的每个标签的片段长度和总胎儿分数。由于胎儿cfdna片段比母体片段短[参考文献quake 2010,也应该引用lo's science clin translation文章],从母体血浆检测胎儿非整倍性可以更加稳定和高效,需要更少的独特cfdna片段。相结合地,在显著更低的cfdna片段数量下,以非常快的周转时间实现了提高的分析灵敏度和特异性。这可能使nipt以显著更低的成本进行,以便于在一般产科群体中应用。
[0780]
方法
[0781]
将外周血样品吸入bct管(streck,omaha,ne,usa)中并运送到redwood city的illumina clia实验室进行商业nipt测试。签署的患者同意书允许第二血浆等分试样脱鉴定(de
‑
identified)并用于临床研究,但从纽约州发送的患者样品除外。选择用于该项工作的血浆样品以包括具有一系列cfdna浓度和胎儿分数的未受影响的和非整倍体胎儿。
[0782]
文库加工的简化
[0783]
使用经过微小修改以容纳更大的裂解物输入的nucleospin 96孔血液纯化试剂盒(macherey
‑
nagel,diiren,germany),从900μl的母体血浆中提取cfdna。使经分离的cfdna直接进入测序文库方法中,没有cfdna输入的任何归一化。用truseq无pcr dna文库试剂盒(truseq pcr free dna library kit,illumina,san diego,ca,usa)制备测序文库,其具有双重指标以用于条形码化cfdna片段以用于样品鉴定。对文库方案的以下修改用于改善文库制备与输入cfdna的低浓度的相容性。模板输入体积增加,而末端修复、a
‑
加尾和连接主混合物和接头浓度降低。另外,在末端修复后,引入热灭杀步骤以使酶失活,去除后端修复spri(供应商)珠粒纯化步骤,并且在后连接spri珠粒纯化步骤期间的洗脱使用ht1缓冲液(illumina)。
[0784]
使用经配置为具有96个通道头和8个1ml移液通道的单个star(hamilton,reno,nv,usa)液体处理器,一次批量处理96个血浆样品。液体处理器通过dna提取、测序文库制备和定量处理每种个体血液样品。用accuclear(biotium,hayward,ca,usa)定量个体样品文库,并用归一化输入制备48个样品的汇集物,得到用于测序的最终浓度为32pm。
[0785]
配对末端测序
[0786]
利用2x 36bp配对末端测序,加上另外16个循环的用于对样品条形码进行测序,用illumina nextseq 500仪器进行dna测序。共有364个样品在8个独立的测序批次中运行。
[0787]
使用bcl2fastq(illumina)对配对的dna序列进行解复用,并将其使用bowtie2对准器算法[参考文献landmead]定位至参考人基因组(hg19)。成对的读取必须匹配待计数的正义链和反义链。超过为10的定位质量评分(具有全局唯一的第一读取)(ruan等人)的所有
计数的定位对被分配给大小为100kb的非重叠连续固定宽度基因组箱。大约2%的基因组在独立一组nipt样品中显示出高度可变覆盖率,并被排除在进一步分析之外。
[0788]
使用可从经测序的cfdna片段两端的每一个的定位位置获得的基因组位置信息和片段尺寸,推导出每个100kb窗的两个变量:(a)长度低于150个碱基对的短片段的总计数,和(b)在低于250个碱基对的所有片段组内的80和150个碱基对之间的片段的分数。将片段的尺寸限制为小于150个碱基对,富集了源自胎盘的片段,所述胎盘是胎儿dna的代用品。短片段的分数表征了血浆混合物中的相对胎儿cfdna量。与对于该染色体是二体的整倍体胎儿相比,预期来自三体胎儿的cfdna具有更高分数的定位至三体染色体的短读取。
[0789]
利用图2d所示的方法,将短片段的计数和分数独立地归一化,以去除归因于基因组鸟嘌呤胞嘧啶(gc)含量的系统性测定偏差和样品特异性变异。通过去除偏离全染色体中值超过3个稳定的标准偏差度量的箱来修整归一化值。最后,对于两个变量中的每一个,将与靶染色体相关的修整的归一化值与归一化参考染色体上的那些进行比较,以构建t
‑
统计量。
[0790]
来自每个配对末端测序运行的数据遵循分析的四个步骤:1)读取转换,2)100kb分辨率下的特征分箱,3)在100kb分辨率下的每个特征(计数和分数)的归一化,以及4)组合特征和评分以用于非整倍性检测。在步骤1中,将样品数据从各个条形码解复用,与基因组比对,并对序列质量进行过滤。步骤2,对每个箱,确定长度低于150个碱基对的短片段的总计数,和低于250个碱基对的所有片段组内的80至150个碱基对之间的片段的分数。在步骤3中去除测定偏差和样品特异性变异。最后,确定对参照物的富集并使用t
‑
检验对每个计数和分数进行评分,并将其组合用于非整倍性检测的最终评分。
[0791]
检测胎儿全染色体非整倍性
[0792]
我们测试了计数和分数数据是否可以组合起来以增强检测胎儿21三体的能力。将来自携带具有核型确认为21三体的胎儿的孕妇的16个血液样品和来自未受影响的妊娠的294个样品随机分布于加工批次中,得到9个用于测序的流动池。分别检查每个算法步骤以确定每个步骤和步骤组合检测非整倍性的能力。将组合情况下的胎儿非整倍性检测的最终评分定义为两个单独t
‑
统计量的平方和的平方根,并且应用单个阈值来产生
″
非整倍性检出
″
相对于
″
非整倍性未检出
″
的调用。
[0793]
胎儿分数的计算
[0794]
对于每种样品,使用基因组100kb箱的亚组内的尺寸[111,136bp]的片段总数与尺寸[165,175bp]的片段总数之比来估计胎儿分数。使用来自携带已知男性胎儿的妇女的样品,确定了与来源于x染色体的拷贝数的胎儿分数具有最高相关性的前10%基因组箱[参考文献rava]。与胎儿分数来源于相关的基因组箱的最高10%与x染色体[ref rava]的拷贝数相关。使用包括箱选择和回归模型参数估计两者的留一交叉验证[ref]分析来计算基于片段尺寸的胎儿分数估值与已知男性胎儿中的来源于x染色体的那些之间的相关性。然后使用来自片段尺寸比的线性回归模型推导出估计的胎儿分数。
[0795]
结果
[0796]
文库加工的简化
[0797]
图8显示了与标准实验室工作流程相比,该新版nipt的整体工作流程和时间表。用于血液分离、cfdna提取、文库构建、定量和汇集的整个96
‑
样品制备工作流程能够在单个
hamilton star上在不到6小时的总制备时间内处理样品。这是与使用clia实验室中所用的基于pcr的方法的9小时和两个hamilton star相比。每个样品提取的cfdna的量平均为60pg/μl,并且测量文库输出的产率与cfdna输入线性相关(r2=0.94),如图9所示。平均回收率大于70%(添加范围),表明在spri珠粒纯化后高效回收cfdna。每个测试运行使用多路复用的归一化量的48个样品,并且花费大约14小时来完成。唯一定位成对读取的中值为xxx m,其中95%的样品高于yyy。
[0798]
配对末端测序
[0799]
nextseq 500上每48
‑
样品批次的总测序时间少于14小时。这与hiseq 2500上的实验室方法为40小时(1个流通池,96个样品)或50个小时(2个流通池,192个样品)相比较。cfdna片段两端的定位基因组位置提供了cfdna片段尺寸信息。图10显示了从具有男性胎儿的妊娠的324个样品测量的cfdna片段尺寸分布。定位至已知为整倍体的常染色体且主要代表母体染色体的片段的尺寸由薄曲线表示。插入物的平均尺寸为175bp,其中xx%的片段测量在100bp和200bp之间。粗曲线表示仅由代表胎儿cfdna片段的y染色体产生的片段尺寸。来自y染色体特定序列的尺寸分布较小,平均167bp,在较短的片段尺寸下具有10个碱基的周期性。
[0800]
由于cfdna的较短片段富含胎儿dna,因此由于优先选择胎儿读取,预期仅使用较短片段的选择性分析会增加相对的胎儿表征。图11显示了与来自小于150bp的配对末端读取的计数相比的来自定位的配对末端读取的总计数的相对胎儿分数。总之,中值胎儿分数与总数相比增加了2倍,尽管方差有所增加。发现150bp的尺寸截断提供了计数的最佳权衡,胎儿表征与计数的方差相比增加。
[0801]
检测胎儿全染色体非整倍性
[0802]
对每个可用的度量、总计数、小于150bp的计数、富含胎儿cfdna的计数分数(在80和150bp之间的计数/<250bp的计数)和较短的片段计数与分数的组合进行了测试以区分21三体样品与21号染色体中的那些整倍体的能力。图12显示了这些指标中的每一个的结果。总计数具有xx计数的中值,而小于150bp的计数具有yy计数的中值。然而,可如图12(a)和12(b)所示,较小的计数显示21三体和整倍体之间的更好分离,主要是因为该度量对于胎儿cfdna而言是富集的。单独的分数几乎与区分非整倍性的总计数一样有效(图12(c)),但当与短片段计数组合使用时(图12(d))仅提供相对于仅短片段计数的改善的区分。这表明该分数提供了增强了21三体的检测的独立信息。当与使用用pcr扩增进行并且中值为16m计数/样品的文库制备的目前clia实验室工作流程相比时,无pcr的配对末端测序工作流程显示相等的性能,具有显著更少的计数/样品(如,6m计数/样品或更少)和更简单、更短的样品制备工作流程。
[0803]
胎儿分数的计算
[0804]
使用来自具有男性胎儿的妊娠的x染色体结果,可以利用归一化染色体值来确定计数的胎儿分数(clinchem参考文献),并比较不同的cfdna片段尺寸。来源于x染色体的胎儿分数用于校准一组140个样品的比率,并使用留一交叉验证来估计性能。图13显示了交叉验证的胎儿分数预测的结果,并证明了两个数据集之间的相关性,表明一旦测量了校准集,胎儿分数估值可以从任何样品中获得,包括来自携带女性胎儿的妇女的样品。
[0805]
讨论
[0806]
已经证明,使用无pcr文库制备结合配对末端dna测序,可以实现母体血液中cfdna的胎儿非整倍性检测的高分析灵敏度和特异性。该方法简化了工作流程,提高了周转时间(图8),并且应该消除pcr方法固有的一些偏差。配对末端测序允许确定片段长度尺寸和胎儿分数,与目前实施的商业方法相比,其可以进一步用于增强在显着更低的标签计数下的非整倍性的检测。无pcr配对末端实施方式的表现似乎类似于使用高达三倍的标签数量的单末端测序方法。
[0807]
文库加工的简化
[0808]
无pcr工作流程具有若干个用于临床实验室的优势。由于文库制备的高产率和线性行为,可以直接从各个样品文库浓度制作用于测序的样品的归一化汇集物。由此消除了文库制备方法的pcr扩增中固有的偏差。此外,不需要在pcr活动前和后分离单独的液体处理器;这减少了实验室的资金负担。这个简化的工作流程允许在临床实验室的单个轮班内准备各批次的样品,然后进行测序并分析过夜。总之,减少资本支出,减少
″
实际操作
″
时间和快速周转允许潜在地显著减少nipt的成本和整体稳定性。
[0809]
配对末端测序
[0810]
在nextseq 500系统上使用配对末端测序对于对cfdna片段进行计数有数个优势。首先,使用双索引条形码,可以以高水平多路复用样品,从而允许具有高统计置信度的运行
‑
至
‑
运行变异的归一化和校正。此外,由于每次运行多路复用多样化48个样品,并且用于聚类的流动池所需的量是有限的,每样品的输入要求显著降低,允许使用无pcr文库工作流程。凭借其典型的cfdna产率(约为5ng/样品),研究人员即使没有pcr扩增也能够获得每样品2
‑
3次测序。这与需要从多个血液管输入大量血液以产生足够的cfdna用于非整倍性测定(ref)的其它方法形成对比。最后,配对末端测序允许确定胎儿cfdna的cfdna片段尺寸和分析富集。
[0811]
胎儿全染色体非整倍性的检测
[0812]
我们的结果表明,低于150bp的cfdna片段的计数能够比总计数更好地区分非整倍性和整倍体染色体。这一观察结果与fan等人的结果形成对比,后者认为由于可用计数的数量减少,使用较短的片段(fan等人)可以减少计数统计量的精确度。如yu等人所暗示的,短片段的分数也为21三体检测提供了一些区分,尽管动态范围小于计数。然而,结合计数和分数度量导致三体样品与整倍体的最佳分离,并暗示这两个度量是染色体表征的互补测量。其它生物度量,如甲基化,也可能提供可以增强非整倍性检测的信噪比的正交信息。
[0813]
胎儿分数的计算
[0814]
此处呈现的方法还允许估计每样品中的胎儿分数,而无需产生额外的实验室工作。在每个流动池上有许多样品,其中大约一半是雄性,通过用从男性样品中确定的胎儿分数校准来自片段尺寸信息的胎儿分数测量值,可以获得所有样品的精确胎儿分数估值。在商业环境中,研究人员的临床经验表明,即使没有特定的胎儿分数测量(ref),使用大量单末端标签的标准计数方法也导致非常低的假阴性率。鉴于此处观察到的检测的类似限制,预期有等效的测试性能。
[0815]
结论
[0816]
已经证明,使用无pcr文库制备结合配对末端dna测序,可以实现母体血液中cfdna的胎儿非整倍性检测的高分析灵敏度和特异性。这种简化的工作流程具有非常快的周转时
间,可能允许以显著更低的成本执行nipt以用于一般产科群体。此外,配对末端测序技术有测量其它生物现象以及提供其它临床应用的潜能。例如,来自基因组或cpg岛的甲基化特定区域的尺寸信息可以提供另一个正交度量,用于增强跨基因组的拷贝数变体的检测。
[0817]
在不脱离本公开的精神或本质特征的情况下,本公开可以以其它特定形式实施。所描述的实施方案在所有方面都应被认为仅是说明性的而非限制性的。因此,本公开的范围由所附权利要求而不是前面的描述指示。在权利要求的等效性的含义和范围内的所有变化都应包含在其范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1