脑卒中多基因遗传风险综合评分的构建方法与装置及应用
本发明是关于一种脑卒中多基因遗传风险综合评分(metaprs)的构建方法与装置及其应用。
背景技术:
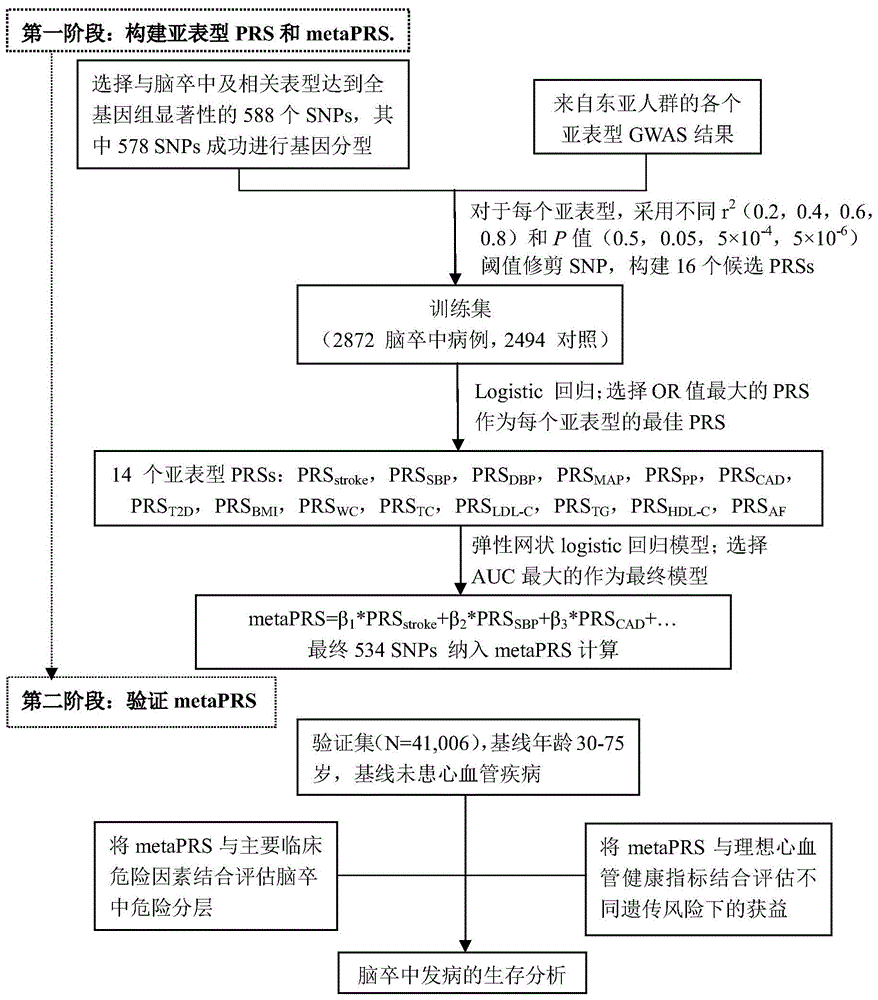
:脑卒中死亡是全球主要的健康威胁之一。据估计,全球25岁以上成人脑卒中终生风险约为25%,其中东亚人群风险最高,达到39%。在中国,脑卒中是居民死亡的主要原因,2017年脑卒中死亡人数达207万。因此,尽早识别高危人群,针对主要危险因素(例如高血压,糖尿病,血脂异常等)进行健康生活方式管理和药物干预对于中国乃至全球的脑卒中一级预防具有重要意义。脑卒中是由遗传和环境因素共同引起的复杂疾病。全基因组关联研究(genome-wideassociationstudy,gwas)已确定至少42个与脑卒中相关的遗传易感基因,以及上百个与脑卒中相关表型有关的遗传基因,包括血压、2型糖尿病(type2diabetes,t2d)、血脂水平、体质指数(bodymassindex,bmi)、房颤(atrialfibrillation,af)等。整合这些遗传变异构建脑卒中多基因遗传风险评分(polygenicriskscore,prs)将有助于开展心血管疾病早期风险预测进而指导一级预防。然而,现有脑卒中多基因遗传风险评分的评估准确性有待提高。此外,几乎所有现有遗传评分都是基于欧洲人群构建(stroke2014;45:394-402,stroke2014;45:403-412,stroke2014;45:2856-2862,bmj2018;363:k4168,jamacardiology2018;3:693-702,natcommun2019;10:5819),而脑卒中的流行病学特征因国家而异,与西方人群相比,东亚人群特别是中国人群的脑卒中发病率和出血性脑卒中事件比例要高得多。现有的基于欧洲人群构建的脑卒中多基因遗传风险评分并不适用于东亚人群。因此,构建东亚人群脑卒中prs并在前瞻性队列人群中严格评估其遗传风险预测价值至关重要。技术实现要素:本发明的一个目的在于提供一种脑卒中多基因遗传风险评分的构建方法。本发明的另一目的在于提供一种构建脑卒中多基因遗传风险评分的装置。一方面,本发明提供了一种脑卒中多基因遗传风险评分的构建方法,其是一种脑卒中多基因遗传风险综合评分的构建方法,该方法包括步骤:(1)筛选与脑卒中相关或与脑卒中相关表型相关(达到全基因组显著关联)的单核苷酸多态性位点(snp)的集合;(2)基于步骤(1)中的单核苷酸多态性位点进行基因分型;(3)从全基因组关联研究结果中分别提取所测snp对应于多个亚表型的危险等位基因、效应值及p值,构建多个候选亚表型prs并筛选最佳亚表型prs;其中,所述多个亚表型包括:脑卒中,冠心病,2型糖尿病,房颤,收缩压,舒张压,平均动脉压,脉压,体质指数,腰围,总胆固醇,低密度脂蛋白胆固醇,甘油三酯和高密度脂蛋白胆固醇;(4)确定各个亚表型prs的权重;(5)将亚表型prs的权重转化为snp水平的权重;(6)构建脑卒中多基因遗传风险综合评分metaprs。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,脑卒中相关表型包括血压(收缩压,舒张压,平均动脉压,脉压)、2型糖尿病、血脂(总胆固醇,低密度脂蛋白胆固醇,甘油三酯和高密度脂蛋白胆固醇)、肥胖(体质指数,腰围)、房颤和冠心病。即,本发明的脑卒中多基因遗传风险评分的构建方法中,构建的多个候选亚表型prs包括:脑卒中、冠心病、2型糖尿病、房颤、收缩压、舒张压、平均动脉压、脉压、体质指数、腰围、总胆固醇、低密度脂蛋白胆固醇、甘油三酯和高密度脂蛋白胆固醇的亚表型prs。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,所述脑卒中多基因遗传风险评分是用于评估东亚人群脑卒中发病风险,所述单核苷酸多态性位点的集合中纳入:所有人群的与脑卒中或冠心病相关的单核苷酸多态性位点,以及东亚人群的血压、2型糖尿病、血脂、肥胖、房颤相关的单核苷酸多态性位点。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,进行基因分型的队列人群为东亚人群。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,使用多重聚合酶链反应靶向扩增子测序技术进行基因分型。中位测序深度为979×。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,基因分型过程中,可排除基因型检出率低于95%的snp,得到检测合格的snp集合。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,是从大规模东亚人群全基因组关联研究结果中分别提取所测snp对应于多个亚表型的危险等位基因、效应值及p值。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,构建各个亚表型prs的过程包括:根据提取的p值大小分出多组snp,对于每组snp,基于队列人群数据,使用plink软件clumping命令按照不同的连锁不平衡r2修剪,得到多组snp组合;利用基因型数据,将个体snp风险等位基因数(0、1或2)根据其对应的效应值进行加权并求和构建多个纳入不同组合snp的候选prs,采用logistic回归模型评估这些候选prs与脑卒中的关联,比值比(oddsratio,or)最大(prs每增加一个标准差)的评分被选作最佳亚表型prs。根据提取的p值大小分出多组snp,对于每组snp,基于队列人群数据,使用plink软件clumping命令按照不同的连锁不平衡r2修剪,得到多组snp组合;利用基因型数据,将个体snp风险等位基因数(0、1或2)根据其对应的效应值进行加权并求和构建多个纳入不同组合snp的候选prs,采用logistic回归模型评估这些候选prs与脑卒中的关联,比值比(oddsratio,or)最大(prs每增加一个标准差)的评分被选作最佳亚表型prs。根据本发明的更具体实施方案,上述构建各个亚表型prs的过程中,可以根据提取的p值大小分出n组snp,n大于等于2。例如,可按照p值0.5、0.05、5×10-3、5×10-4、5×10-5、5×10-6从中选出3组、4组、5组或6组。根据本发明的更具体实施方案,上述构建各个亚表型prs的过程中,不同的连锁不平衡r2例如可以是选自0.2、0.4、0.6、0.8等。根据本发明的更具体实施方案,上述构建各个亚表型prs的过程中,当根据提取的p值大小分出n组snp,不同的连锁不平衡r2为0.2、0.4、0.6和0.8时,可得到4n组snp组合,即可构建4n个纳入不同组合snp的候选prs。例如,当根据提取的p值大小分出4组snp,不同的连锁不平衡r2为0.2、0.4、0.6和0.8时,可构建16个纳入不同组合snp的候选prs。本发明中,可进一步通过pearson相关分析计算各个亚表型prs两两之间的相关系数r和p值。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,可从所有队列人群按照预定比例选出部分人群作为训练集(其余部分人群可作为验证集)。所述构建亚表型prs、确定各个亚表型prs的权重的过程在训练集中进行。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,确定各个亚表型prs的权重的过程包括:将各个亚表型prs转化为均值为0、标准差为1的标准化评分;利用训练集,将标化后的各个亚表型prs及要调整的协变量(年龄、性别)共同放入弹性网状logistic回归模型,选择auc最高的模型作为最终模型,从中获得每个prs的系数(β1…β14)作为权重。在本发明的一些具体实施方案中,弹性网状logistic回归模型可校正各个亚表型prs之间的相关性,本发明利用该模型评估了14个亚表型prs与脑卒中的关联,对比分析了弹性网状logistic回归估计的or值与单变量logistic回归估计的or值。进一步地,本发明通过整合14种亚表型prs,将亚表型prs的权重转化为snp水平的权重,构建脑卒中metaprs并进行验证。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,将亚表型prs的权重转化为snp水平的权重的过程按照以下模型进行:其中,σ1,…,σ14是训练集中每个亚表型prs的标准差,αj1,…,αj14是第i个snp对应于每个亚表型的效应值,如果第k个评分中未包含某个snp,则该snp的效应值大小αjk设为0。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,构建的脑卒中多基因遗传风险综合评分metaprs为:metaprs=∑βsnp_i×ni其中,βsnp_i是指第i个snp的效应值,ni指个体所携带第i个snp的效应等位基因数目。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险综合评分的构建方法,还可进一步包括评价所构建的metaprs对脑卒中风险预测和分层的作用的过程。根据本发明的具体实施方案,本发明的脑卒中多基因遗传风险评分的构建方法中,优选地,以队列人群所有个体metaprs的20%和80%百分位数为切点,划分个体脑卒中遗传发病风险为低、中、高危人群。另一方面,本发明还提供了一种用于构建脑卒中多基因遗传风险综合评分的装置,该装置包括:基因分型模块,用于进行基因分型;亚表型prs构建模块,用于从全基因组关联研究结果中分别提取所测snp对应于多个亚表型的危险等位基因、效应值及p值,并构建候选亚表型prs、筛选最佳亚表型prs;模型训练模块,用于在训练集中确定各个亚表型prs的权重;metaprs构建模块,用于将亚表型prs的权重转化为snp水平的权重并构建脑卒中多基因遗传风险综合评分(metaprs)。根据本发明的具体实施方案,本发明的构建脑卒中多基因遗传风险综合评分的装置中,还可选择性地包括snp筛选模块,用于筛选与脑卒中相关或与脑卒中相关表型相关的单核苷酸多态性位点(snp)的集合。根据本发明的具体实施方案,本发明的构建脑卒中多基因遗传风险综合评分的装置中,基因分型模块还可用于在基因分型后排除基因型检出率低于95%的snp。根据本发明的具体实施方案,本发明的构建脑卒中多基因遗传风险综合评分的装置中,选择性地,所述metaprs构建模块可进一步用于评价所构建的metaprs对脑卒中风险预测和分层的作用。另一方面,本发明还提供了一种计算机设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,其中,所述处理器执行所述计算机程序时实现利用本发明所述方法构建的脑卒中多基因遗传风险综合评分评估个体脑卒中发病风险。在本发明的具体实施方案中,本发明依托于脑卒中及相关表型的大型东亚人群gwas结果数据开发了一个包含多个遗传变异的多基因遗传风险综合评分,并在41,006名研究对象的大型前瞻性队列中,评价其对脑卒中风险分层的效果。研究发现,遗传风险高的个体(遗传风险上20%)发生脑卒中的风险比遗传风险低的个体(遗传风险下20%)高约2倍(hr:1.99,95%ci:1.66-2.38),两组的脑卒中终生风险分别为25.2%(95%ci:22.5%-27.7%)和13.6%(95%ci:11.6%-15.5%)。并且本发明的脑卒中发病风险评估对于出血性和缺血性脑卒中均适用。本研究证实多基因遗传风险综合评分可以实现脑卒中风险精细化分层,本发明对于构建脑卒中多基因遗传风险综合评分以及脑卒中一级预防方面具有重要应用前景。附图说明图1显示本发明的脑卒中多基因遗传风险评分的构建研究设计和工作流程。图2显示成功基因分型的578snps的测序深度。箱式图代表578snps测序深度的4分位分布。矩形中线代表测序深度中位数(979×),上边和下边代表75%(1376×)和25%分位数(738×)。图3显示训练集中采用东亚和欧美gwas效应值比较脑卒中prs与脑卒中的关联。采用logistic回归模型计算比值比(ors)和95%可信区间(cis),调整年龄和性别。分别使用日本生物样本库和megastroke联盟欧洲人群脑卒中gwas数据的效应值作为snps权重计算评分。设定不同的p值阈值(5×10-6,5×10-4,0.05,0.5)分别构建4个包含不同snps组合的prs(连锁不平衡r2<0.6)。图4显示训练集中候选多基因风险评分(每增加一个标准差)与脑卒中的关联。采用logistic回归计算比值比(or)和95%可信区间(ci),调整年龄和性别。对于每个表型,基于汇总数据采用不同的连锁不平衡r2(0.2、0.4、0.6、0.8)和显著性阈值(p值=0.5、0.05、5×10-4、5×10-6)构建16个候选prs。图5显示训练集中各个亚表型prss之间的相关性。采用pearson相关分析计算每对prs的相关系数和p值。*p<0.05,**p<10-3,***p<10-10。图6显示训练集中亚表型多基因风险评分(每增加一个标准差)与脑卒中的关联。分别采用logistic回归和弹性网状logistic回归计算比值比(or)和95%可信区间(ci),调整年龄和性别。图7显示前瞻性队列中metaprs和亚表型prs与脑卒中发病的关联。采用队列分层、年龄为时间尺度的cox比例风险回归模型计算风险比(hr)和95%可信区间(ci),调整性别。图8显示metaprs五分位与脑卒中发病的关联。采用队列分层、年龄为时间尺度的cox比例风险回归模型计算风险比(hr)和95%可信区间(ci),调整性别。图9显示不同遗传风险分层下脑卒中的相对风险和终生风险。采用队列分层、年龄为时间尺度的cox比例风险回归模型计算风险比(hr)和80岁前脑卒中累积发病曲线,调整性别。图10显示不同遗传风险分层下缺血性脑卒中和出血性脑卒中的相对风险和终生风险。采用队列分层、年龄为时间尺度的cox比例风险回归模型计算风险比(hr)和80岁前缺血性脑卒中和出血性脑卒中累积发病曲线,调整性别。具体实施方式为了对本发明的技术特征、目的和有益效果有更加清楚的理解,现结合具体实施例及对本发明的技术方案进行以下详细说明,应理解这些实例仅用于说明本发明而不用于限制本发明的范围。实施例中,各原始试剂材料均可商购获得,未注明具体条件的实验方法为所属领域熟知的常规方法和常规条件,或按照仪器制造商所建议的条件。实施例1研究设计流程与研究人群研究设计流程参见图1所示。本研究利用病例对照设计的训练集来构建metaprs,并在一个大型前瞻性队列“中国动脉粥样硬化性心血管疾病风险预测项目(predictionforatheroscleroticcardiovasculardiseaseriskinchina,china-par)”中验证和评价其应用于脑卒中风险预测的临床价值。训练集包括2872例脑卒中病例(2548例缺血性和324例出血性脑卒中)和2494例对照(表1)。所有病例均有脑卒中的有效诊断,并且由神经科医师根据计算机断层扫描(ct)和/或磁共振成像(mri)的医疗记录确诊。对照组为参加社区心血管危险因素调查的个体,并通过病史,临床检查和标准调查问卷确定未发生过脑卒中。表1.训练集的人群特征特征对照(n=2494)脑卒中病例(n=2872)参与研究时的年龄,years66.1(10.3)-发病年龄,years-66.6(9.8)男性,n(%)934(37.4)1,617(56.3)当前吸烟者,n(%)554(22.2)622(21.8)收缩压,mmhg132.4(15.9)149.7(23.7)舒张压,mmhg82.9(8.5)87.9(25.9)总胆固醇,mg/dl188.1(36.8)182.3(64.5)高血压,n(%)1,176(47.2)2,242(78.9)糖尿病,n(%)285(11.4)578(20.3)血脂异常,n(%)895(35.9)1,330(48.5)连续型变量以均值(标准差)表示,分类变量以数目(百分比)表示。验证人群来自china-par项目的三个队列:中国心血管流行病学多中心协作研究1998(chinamulti-centercollaborativestudyofcardiovascularepidemiology1998,chinamuca1998),中国心血管健康多中心合作研究(internationalcollaborativestudyofcardiovasculardiseaseinasia,interasia)和中国代谢综合征的社区干预研究暨中国家庭健康研究(communityinterventionofmetabolicsyndromeinchina&chinesefamilyhealthstudy,cimic)。这些队列的建立和随访已在现有技术文章中进行了详细介绍(circulation2016;134:1430-1440)。简而言之,chinamuca1998,interasia和cimic分别建立于1998、2000-2001和2007-2008年。在2012-2015年期间,采用统一的问卷和方案对这三个队列进行了最新的随访。在43,881例有血液样本和随访信息的参与者中,本发明进一步排除了561例基因型缺失率高(>5.0%)或平均测序深度低(<30×),1352例基线年龄<30岁或>75岁,962例基线患有心血管疾病(脑卒中和心肌梗塞)的参与者,最后共有41,006例参与者纳入分析。这些研究均已获得中国医学科学院阜外医院伦理审查委员会的批准。在收集数据之前,每位参与者均签署了书面知情同意。基线主要传统危险因素收集在基线调查中,对每位参与者进行标准问卷调查、体格检查和实验室检测。由经过专业培训并考核合格的调查员根据统一制定的调查方案收集了一系列生活方式危险因素和心血管代谢指标。基线脑卒中传统危险因素主要包括高血压、血脂异常、糖尿病、肥胖(bmi≥28kg/m2)和脑卒中家族史。高血压定义为收缩压(systolicbloodpressure,sbp)≥140mmhg和/或舒张压(diastolicbloodpressure,dbp)≥90mmhg和/或过去两周内使用降压药。血脂异常定义为总胆固醇(totalcholesterol,tc)≥240mg/dl和/或高密度脂蛋白胆固醇(high-densitylipoproteincholesterol,hdl-c)<40mg/dl和/或甘油三酯(triglycerides,tg)≥200mg/dl和/或低密度脂蛋白胆固醇(low-densitylipoproteincholesterol,ldl-c)≥160mg/dl和/或使用降脂药。糖尿病定义为空腹血糖≥126mg/dl和/或使用胰岛素或口服降糖药。脑卒中家族史定义为任何一级亲属(父亲,母亲或兄弟姐妹)有脑卒中病史。脑卒中事件随访三个队列采用相同的研究方案进行随访,通过约访和入户调查的方式获取研究对象的脑卒中发病死亡信息,并进一步获得病历和死亡证明以供核实。所有医疗和死亡记录均由中国医学科学院阜外医院终点评价委员会的两位专家独立审查。如果两位专家意见不一致,则与委员会内其他专家共同讨论以达成最终诊断。死亡原因按照icd-10(国际疾病分类,第10版)进行编码。脑卒中定义为随访期间确诊的首次致死或非致死性脑卒中事件(i60-i69)。脑卒中亚型分为缺血性脑卒中(i63),出血性脑卒中(i60-i62)和未定亚型脑卒中(i64-i69)。单核苷酸多态性位点的选择和基因分型本发明基于既往全基因组关联研究选择了588个与脑卒中或脑卒中相关表型达到全基因组显著关联的单核苷酸多态性(singlenucleotidepolymorphism,snp)位点,包括脑卒中(n=42)和一系列脑卒中的主要危险因素包括血压(n=46)、2型糖尿病(n=89)、血脂(n=126)、肥胖(n=79)和房颤(n=16)(表2和表3)。本发明中也纳入了与冠状动脉疾病(coronaryarterydisease,cad)相关的snp(n=199)。东亚和欧洲人群报道的与脑卒中和冠心病相关的所有snp均被纳入,其他表型主要纳入东亚人群报道的snp。表2.本研究所选snps的数目traitsno.ofsnpsstroke(as,is,hs)42bp(sbp,dbp,pp,map,hypertension)46cad199t2d89obesity(bmi,wc,whr)79lipids(tc,ldl-c,tg,hdl-c)126af16total588**由于不同表型间易感snp的重叠总数目相加不等于588(等于597)。snp,单核苷酸多态性;as,所有脑卒中;is,缺血性脑卒中;hs,出血性脑卒中;bp,血压;sbp,收缩压;dbp,舒张压;pp,脉压;map,平均动脉压;cad,冠状动脉疾病;t2d,2型糖尿病;bmi,体质指数;wc,腰围;whr,腰臀比;tc,总胆固醇;ldl-c,低密度脂蛋白胆固醇;tg,甘油三酯;hdl-c,高密度脂蛋白胆固醇;af,房颤。使用多重聚合酶链反应靶向扩增子测序技术对所有训练集参与者进行基因分型。使用illuminahiseqxten测序仪扩增目标区域,以进行高通量测序。排除10个基因型检出率低于95%的snp之后,保留578个常染色体snp供后续分析,平均基因型检出率为99.9%,中位测序深度为979×(图2)。为评估基因分型的可重复性,对1648份重复样本进行检测,基因分型一致率>99.4%。metaprs的构建(1)从gwas结果数据提取snp效应值,计算各个亚表型prs从表3参考文献提供的网址中获取14个亚表型(脑卒中,冠心病,2型糖尿病,房颤,收缩压,舒张压,平均动脉压,脉压,体质指数,腰围,总胆固醇,低密度脂蛋白胆固醇,甘油三酯和高密度脂蛋白胆固醇)的全基因组关联研究结果,从中分别提取所测snp对应于14个亚表型的危险等位基因、效应值及p值。表3.用于多基因风险评分计算的汇总数据来源prs,多基因风险评分;gwas,全基因组关联研究;ewas,全外显子关联研究;is,缺血性脑卒中;bp,血压;sbp,收缩压;dbp,舒张压;pp,脉压;map,平均动脉压;cad,冠状动脉疾病;t2d,2型糖尿病;bmi,体质指数;wc,腰围;whr,腰臀比;tc,总胆固醇;ldl-c,低密度脂蛋白胆固醇;tg,甘油三酯;hdl-c,高密度脂蛋白胆固醇;af,房颤。以亚表型stroke为例,从文章(large-scalegenome-wideassociationstudyinajapanesepopulationidentifiesnovelsusceptibilitylociacrossdifferentdiseases.naturegenetics2020;52:669-679)提供的网站http://jenger.riken.jp/en/result下载脑卒中(17,671病例,192,383对照)gwas结果,从中提取出所测snp的危险等位基因、效应值及p值。根据提取的p值,按照0.5、0.05、5×10-4、5×10-6筛选出4组snp,对于每组snp,基于队列人群数据,使用plink软件(version1.9)clumping命令按照不同的连锁不平衡r2(0.2、0.4、0.6、0.8)修剪,最终得到16组snp组合。利用训练集基因型数据,将个体snp风险等位基因数(0、1或2)根据其对应的效应值进行加权并求和构建16个纳入不同组合snp的候选prs,采用logistic回归模型评估这些候选prs与脑卒中的关联,比值比(oddsratio,or)最大(prs每增加一个标准差)的评分被选作最佳脑卒中prs。重复以上过程,分别得到其他13个亚表型prs。其中,最佳脑卒中亚表型(stroke)prs利用的snp位点及效应值见表4。(2)在训练集中计算各个亚表型prs的权重将14个亚表型prs转化为均值为0,标准差为1的评分。利用训练集,将标化后的14个亚表型prs及要调整的协变量(年龄、性别)共同放入弹性网状logistic回归模型(cv.glmnet函数,r包“glmnet”),该模型采用10倍交叉验证的方法评估一系列不同惩罚项(设置alpha=0、0.1、0.2、0.3、0.4、0.5、0.6、0.7、0.8、0.9或1.0)的模型,将模型参数type.measure设置为“auc”,模型自动筛选auc(areaunderreceiving-operatorcharacteristiccurve,接收者操作特征曲线下面积)最高的模型作为最终模型,从中获得每个prs的系数(β1…β14)作为权重。(3)将亚表型prs的权重转化为snp水平的权重利用以上公式将prs水平的权重转换为snp水平的权重,其中σ1,…,σ14是训练集中每个亚表型prs的标准差,αj1,…,αj14是第i个snp对应于每个亚表型的效应值,如果第k个评分中未包含某个snp,则该snp的效应值大小αjk设为0。(4)计算metaprs利用公式:metaprs=∑βsnp_i×ni计算个体的metaprs,其中βsnp_i是指第i个snp的效应值(即第4步得到的snp水平的权重),ni指个体所携带第i个snp的效应等位基因数目。经过统计处理步骤,最终共有534个snp纳入metaprs的计算,表4中提供了所有符合条件snp的信息和权重。(5)metaprs切点划分以队列人群所有个体metaprs的20%和80%百分位数为切点,划分个体脑卒中遗传风险为低、中、高危人群。表4.本发明所确定snps的信息和权重统计分析研究对象基线特征中的连续性变量以均值(标准差)表示,分类变量以频数(百分比)表示。根据metaprs水平将研究对象分为低(metaprs的最低五分位数),中(metaprs的第2-4五分位数)和高(metaprs的最高五分位数)遗传风险组。采用性别调整、年龄为时间尺度的分层cox比例风险回归模型计算遗传风险评分脑卒中发病的风险比(hazardratio,hr)和95%置信区间(confidenceinterval,cis)。采用“survfit.coxph”(r包“survival”)绘制校正性别的累积发病率曲线,从而评估不同遗传风险分层下研究对象80岁时的脑卒中终生风险。所有分析均采用r软件版本3.6.0(rfoundationforstatisticalcomputing,vienna,austria)或sas统计软件9.4版(sasinstituteinc,cary,nc)进行。研究人群遗传风险分组表5显示了队列人群中41,006例研究对象的基线特征。总人群的平均年龄为51.9(10.6)岁,男性占43.1%。遗传风险高(metaprs上20%)的参与者具有较高的心血管代谢危险因素(高血压,糖尿病,血脂异常)。经过367,750人年的随访(平均随访9.0年),有1227例参与者在80岁之前发生脑卒中,包括769例缺血性脑卒中,355例出血性脑卒中,21例缺血性脑卒中并发出血性脑卒中,以及124例未定亚型脑卒中。表5.前瞻性队列的基线信息连续型变量以均值(标准差)表示,分类变量以数目(百分比)表示。多基因遗传风险评分构建以及对脑卒中的预测本发明首先依据日本生物样本库脑卒中gwas结果p值设定4个阈值(5×10-6,5×10-4,0.05,0.5)筛选4组不同snps组合,然后在训练集采用欧洲人群的gwas结果数据作为snp效应值计算脑卒中prs,并进一步评估它们与脑卒中的关联强度。如图3所示,与使用日本生物样本库脑卒中gwas效应值相比,当使用来自欧洲人群的效应值时,4个纳入不同snp组合的prs(每增加一个sd)与脑卒中关联的or(95%ci)值均显著下降。因此,本研究采用东亚人群的gwas效应值构建各个亚表型prs,训练集中每个候选亚表型prs与脑卒中的关联强度见图4,选择or值最大的一个评分作为最终的亚表型prs。通过pearson相关分析计算各个亚表型prs两两之间的相关系数r和p值,如图5所示,各个亚表型prs之间存在不同程度的相关性,其中舒张压和平均动脉压(r=0.90)、收缩压和平均动脉压(r=0.86)、收缩压和舒张压(r=0.77)以及总胆固醇和ldl-c(r=0.85)prs之间的相关性较强。进一步利用弹性网状logistic回归模型评估14个亚表型prs与脑卒中的关联,该模型可校正各个亚表型prs之间的相关性,弹性网状logistic回归估计的or值与单变量logistic回归估计的or值对比见图6(图6中ldl-c和hdl-c权重为0)。最后,通过整合14种亚表型prs构建脑卒中metaprs并在队列人群中进行验证。本发明的脑卒中多基因遗传风险评分的构建过程中,最佳脑卒中亚表型(stroke)prs确定了一组与东亚人群相关的脑卒中风险相关基因,其包括表4所示的280个stroke相关单核苷酸多态性位点,检测这些stroke相关单核苷酸多态性位点,通过∑βi×ni获得发病风险的遗传风险评分,能良好地评估东亚人群的脑卒中发病风险。其中各stroke相关各snp的效应值可以统一采用表4中亚表型prs栏内的snp的效应值,也可以统一采用表4中metaprs栏内的snp的效应值。遗传风险评分越高,个体脑卒中发病的风险越高。本发明的评估脑卒中发病风险方案,可以在检测表4所示的280个stroke相关snp基础上,进一步选择性地检测表4所示的159个cad相关snp、4个sbp相关snp、1个wc相关snp、55个t2d相关snp、22个tc相关snp、9个pp相关snp、4个af相关snp中的一组或多组snp,通过∑βi×ni获得发病风险的遗传风险评分,可以更好地评估东亚人群的脑卒中发病风险。当本发明的评估脑卒中发病风险方案包括检测cad、sbp、wc、t2d、tc、pp、af相关snp中的一组或多组时,这些snp的效应值可以统一采用表3中亚表型prs栏内的snp的效应值,优选统一采用表3中metaprs栏内的snp的效应值。遗传风险评分越高,个体脑卒中发病的风险越高。在验证人群中,包含表4所示534个snp的metaprs与脑卒中的关联强度高于其他任何亚表型prs,metaprs每增加一个标准差,总脑卒中、缺血性脑卒中和出血性脑卒中的hr(95%ci)分别为1.28(1.21-1.36),1.29(1.20-1.39)和1.30(1.17-1.45)(图7)。进一步调整包括脑卒中家族史在内的临床危险因素(表6),metaprs与脑卒中发病的hr值仅有稍许降低,表明本发明的metaprs可独立于传统临床危险因素之外用于评估脑卒中发病风险。表6.metaprs(每增加一个标准差)与脑卒中发病的关联,调整或不调整临床危险因素采用队列分层、年龄为时间尺度的cox比例风险回归模型计算风险比(hr)和95%可信区间(ci),调整性别,调整或不调整临床危险因素。本发明中,根据总人群metaprs遗传风险评分进行了metaprs遗传风险分层(表7)。metaprs遗传风险评分<-0.140可判断为个体脑卒中发病低遗传风险(metaprs0-20%),metaprs遗传风险评分>0.305可判断为个体脑卒中发病高遗传风险(metaprs80-100%)。表7metaprs遗传风险分层速查表将人群按照metaprs的5等分分组后,各组人群的脑卒中风险显示出明显的梯度(趋势p值<0.001)(图8)。与低遗传风险者(metaprs下20%)相比,高遗传风险者(metaprs上20%)发生脑卒中的风险约高2倍(hr:1.99,95%ci:1.66-2.38,p=1.11×10-13),遗传风险高的个体脑卒中的终生风险(80岁发生脑卒中风险)也比遗传风险低的个体高近2倍(分别为25.2%,95%ci:22.5%-27.7%和13.6%,95%ci:11.6%-15.5%)(图9)。上述遗传风险评分对缺血性和出血性脑卒中的预测效果类似(图10)。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1