使用机器学习对核酸序列进行分类的方法与流程
1.本发明涉及生物技术领域,具体涉及使用机器学习对核酸序列进行分类的方法。
背景技术:
2.目前对核酸序列进行分类多采用序列比对的方法。首先获取带有明确标签的核酸序列构建数据库,然后将待检测的核酸序列与数据库中的核酸序列进行逐一比对,找出与其相似性最高的序列,最后此相似性最高的序列所属标签将赋予待检测序列作为它的类别。
3.序列比对方法虽然使用十分广泛,但是其局限性也十分明显。由于目前数据库均是基于已知分类序列进行构建,通过序列比对方法对新出现的核酸序列则不能正确地进行分类;同时在基因序列测定过程中,受技术和资金所限,获得的核酸序列可能只是其完整基因组序列的一部分,有时长度甚至短于1000bp,而序列比对方法对于短序列的区分度更低;随着数据库中参考序列数目的不断增加,检测目标序列的运算量也会不断加大,甚至在普通计算机上难以完成。
技术实现要素:
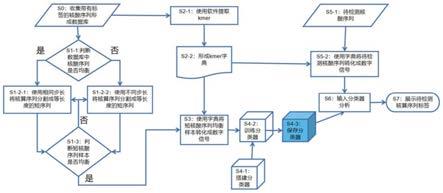
4.为了解决上述问题,本发明提供一种使用机器学习对核酸序列进行分类的方法,所述方法包括以下步骤:s0:收集带有准确分类标签的核酸序列构建核酸序列数据库;s1:将核酸序列数据库中所有核酸序列分割成等长度的短序列,从而形成短核酸序列均衡样本,该短序列的长度为4bp~核酸序列数据库中最短序列的长度,短核酸序列均衡样本适合于机器学习,短核酸序列均衡样本是指最多分类标签数目的短核酸序列数量n
most
与最少分类标签数目的短核酸序列数量n
least
的比值不超过5时的短核酸序列样本,优选的所述比值不超过2;s2:根据所述核酸序列数据库中的核酸序列,提取核酸kmer,核酸kmer长度为4≤kmer长度≤所述短序列的长度,形成核酸序列的kmer字典;s3:使用s2中的kmer字典将s1中的短核酸序列均衡样本转化成数字信号;s4:使用s3中的数字信号训练一个能对带有分类标签的核酸序列进行多分类的神经网络分类器,该神经网络分类器用于识别和判断待检测核酸序列在核酸数据库中的分类;s5:使用s2中的kmer字典将待检测核酸序列转化成数字信号;s6:将s5中的数字信号输入到s4中训练好的神经网络分类器中,对待检测核酸序列进行分类。
5.在一种实施方式中,本发明方法还包括步骤s7:展示待检测核酸序列的分类结果。
6.在一种实施方式中,s7中展示部分为每条序列的id和该id序列可能的分类标签;并显示该分类标签的可靠性分值,分值越高则表明这条id序列为此分类标签可靠性越高。
7.在一种实施方式中,所述s1包括以下步骤:
8.s1
‑
1:判断核酸序列数据库中所有核酸序列是否均衡;
9.s1
‑2‑
1:如果均衡,则使用相同分割步长将核酸序列数据库中所有核酸序列分割成等长度的短序列,所述相同分割步长是分割时后移长度,该后移长度不大于所述短序列
的长度,分割核酸序列数据库中所有核酸序列获得短序列样本;
10.s1
‑2‑
2:如果不均衡,则对核酸序列数据库中每类标签核酸序列使用各自分割步长将每类标签核酸序列分割成等长度的短序列,每类标签核酸序列分割步长为分割各类标签核酸序列时后移长度,该后移长度不大于所述短序列的长度,分割核酸序列数据库中所有核酸序列获得短序列样本;
11.s1
‑
3:判断s1
‑2‑
1或者s1
‑2‑
2获得短核酸序列的样本是否为均衡并适合于机器学习的样本,如果是,则停止s1
‑2‑
1或者s1
‑2‑
2,如果不是,则重复s1
‑2‑
1或者s1
‑2‑
2,直到获得均衡并适合于机器学习的短核酸序列的样本。
12.在一种实施方式中,所述s1
‑2‑
2中,包括以下步骤:(1)获得核酸序列数据库中带有准确分类标签的核酸序列中每类核酸序列数目与特定值的比率,特定值的选择范围在核酸序列数据库中核酸序列中最多标签数目的核酸序列数量n
most
与最少标签数目的核酸序列数量n
least
之间的任意值,比率值=特定值
÷
每类核酸数目,比率值小于1的则人为将比率值设定为1;和(2)根据每类核酸的比率值求得此类核酸的分割步长,分割步长=短序列长度
÷
比率值。
13.在一种实施方式中,s2包括:s2
‑
1:使用软件提取核酸序列数据库中所有的kmer组成集合;和,s2
‑
2:构建kmer字典,将s2
‑
1中的所有kmer按字母顺序从小到大排序,并且排序好后的kmer中的第一位置插入<unk>表示字典中不存在的kmer。
14.在一种实施方式中,所述字典的查询方式为kmer:kmer所在字典中的位置。
15.在一种实施方式中,s2
‑
1中软件是jellyfish软件。
16.在一种实施方式中,所述s4包括以下步骤:s4
‑
1:搭建一个含有输入层、embedding层、最大池化层、平均池化层、中间合并层、全连接层一、全连接层二、输出层的模型;s4
‑
2:把s3得到的数字信号输入到s4
‑
1搭建的模型中进行训练,直到该神经网络成为对输入核酸序列进行多分类的神经网络分类器,分类标签为s0核酸数据库中的分类标签;s4
‑
3:把s4
‑
2中训练好的模型保存,用于后续对待检测序列进行分析。
17.在一种实施方式中,使用s3中50%
‑
90%数字信号作为训练集,s3中10%
‑
50%数字信号作为测试集,将训练集输入s4
‑
2中搭建的神经网络,并对该网络进行反复训练,并利用测试集不断测试,直到该网络的分类表现达到成为对输入核酸序列进行多分类的神经网络分类器的要求,其分类标签为核酸数据库中核酸序列的分类标签。
18.在本发明方法中,在步骤s1中,根据数据库中各类别核酸的数目设定相应的步长对各类别的核酸进行分割,从而获得短序列均衡样本,使其适合于机器学习并得到可靠的神经网络分类器,短序列的长度可根据后续待检测样本的长度灵活选择。
19.在s2步骤中,使用kmer提取软件直接提取数据库中所有的kmer组成字典,可以减少后续步骤中与字典对应的embedding数组的运算量,减少模型的存储空间,增加模型的可移植性。
20.在s4步骤中,训练一个核酸序列分类器并保存,可以快速将待检测核酸序列与数据库中的核酸序列标签进行对应分类。
21.在s7步骤中,展示部分为每条序列的id和该id序列多种可能的分类标签;并显示可能分类标签的可靠性分值,分值越高则表明这条id序列为此分类标签可靠性越高,为研究该id序列与多类标签之间的关系提供参考。
22.通过本发明的方法,能够准确快速地将待检测核酸序列与数据库中的核酸序列标签进行对应分类,并展示每个标签的可靠性分值,为后续研究提供参考,增加了模型的使用价值。
23.当数据库中核酸序列不均衡时,本发明方法对核酸序列数据库中每类标签核酸序列使用各自分割步长将每类标签核酸序列分割成等长度的短序列组成短核酸序列均衡样本,避免因数据库核酸序列不平衡导致数据库不可用或需要付出额外努力增加核酸样本的现象,增加了机器学习对核酸序列进行分类的适用范围。
24.由于某一长度的kmer的全部数目为4
k
/2+1(k值为kmer的长度),因此当k值足够大时,kmer的全部数目会变得非常大,本地计算机资源难以处理。例如当k=20,kmer的数目约为5497亿,而与之对应的embedding数组储存的数据则会更加庞大。本发明方法使用kmer提取软件直接提取数据库中所有的kmer组成字典,可以减少后续步骤中与字典对应的embedding数组的运算量,减少模型的存储空间,增加模型的可移植性。
附图说明
25.为了更清楚地说明本申请实施例中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本申请中记载的一些实施例,对于本领域普通技术人员来说,在不付出创造性劳动的前提下,还可以根据这些附图获得其它的附图。
26.图1是本发明方法的流程图;
27.图2是本发明实施例的核酸序列数据库中各亚型所对应的数目示意图,共184个亚型,根据亚型的数目从大到小排序,截取前26种亚型名称及其数目;
28.图3是通过本发明实施例核酸数据库中各亚型的数目求得每种亚型相较于某一特定值的比率结果图,共184种亚型的比率值,根据比率值从大到小排序,截取前20种亚型的名称及比率值;
29.图4是将本发明实施例核酸数据库中184种亚型的基因组全长dna序列分割后,根据短序列的亚型数目排序结果示意图,截取前21种亚型名称及所对应的短序列数目;
30.图5是本发明实施例使用jellyfish软件提取本发明核酸数据库中的所有20mer形成的txt文件的示意图,截取前13种20mer和数目,
‘
>49’表示下面20mer(
‘
aaaaaaaaaaaaaaaaaaaa’)序列在数据库中的数目;
31.图6是本发明将20mer集合按字母顺序从小到大排列形成20mer字典示意图,不存在的20mer序列标记为<unk>,从本本发明核酸数据库中提取出的字典数目共计6942463个不同的20mer序列,截取前25种20mer;
32.图7显示本发明实施例前20个1000bp dna序列的数字信号示意图,转化后每个序列的信号长度为981,9284表示为20mer字典中第9824位20mer表示的序列;
33.图8是本发明实施例模型构建流程图;
34.图9是本发明实施例的模型训练过程示意图;
35.图10是本发明实施例的待检测核酸序列样本fasta文件部分展示示意图,
‘
>1’表示序列的名称,下面为其核酸序列,此图展示了两条待检测序列的名称(
‘
>1’和
‘
>14’)和核酸序列;
36.图11是本发明实施例的待检测核酸序列输入到模型后的分类结果示意图,id为fasta文件中每条序列的名称,top
‑
1至top
‑
5为这条id序列前5种可能的亚型,括号中的值为此种亚型的可靠性分值,分值越高则表明这条id序列为此亚型的可靠性越高。
具体实施方式
37.为了使本领域技术人员更好地理解本申请中的技术方案,下面将结合实施例对本发明作进一步说明,显然,所描述的实施例仅仅是本申请一部分实施例,而不是全部的实施例。基于本申请中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其它实施例,都应当属于本申请保护的范围。下面结合附图及实施例对本发明作进一步描述。
38.如图1所示,本发明的目的是利用机器学习对核酸序列进行分类,本实施例以hiv
‑
1型病毒为目标,具体方法如下:
39.s0:收集带有准确分类标签的核酸序列构建核酸序列数据库:收集带有准确分类标签的hiv基因组dna数据构建基础数据库,在本实施例中分类标签是hiv各种亚型标签。
40.本数据库来源于hiv sequence database(https://www.hiv.lanl.gov/components/sequence/hiv/search/search.html)中的12825条完整的hiv
‑
1型dna序列(截止到2020年9月6日),然后将其中每种亚型含有的dna序列数目多于2的基因组dna序列构建核酸序列数据库,共计184种亚型。
41.s1:将核酸序列数据库中所有核酸序列分割成等长度的短序列,从而形成短核酸序列均衡样本,该短序列的长度为4bp~核酸序列数据库中最短序列的长度,短核酸序列均衡样本适合于机器学习,短核酸序列均衡样本是指最多分类标签数目的短核酸序列数量n
most
与最少分类标签数目的短核酸序列数量n
least
的比值不超过5时的短核酸序列样本,优选地所述比值不超过2;
42.s1
‑
1:判断核酸序列数据库中所有核酸序列是否均衡。在本发明实施例中,由于带有hiv
‑
1_b亚型标签的序列有5703条,带有hiv
‑
1_ahju亚型标签的序列有2条(未展示),所以数据是不均衡的,如图2所示。
43.s1
‑2‑
2:对核酸序列数据库中每类标签核酸序列使用各自分割步长将每类标签核酸序列分割成等长度的短序列,每类标签核酸序列分割步长为分割各类标签核酸序列时后移长度,该后移长度不大于所述短序列的长度,分割核酸序列数据库中所有核酸序列获得短序列样本。
44.(1)获得核酸序列数据库中带有准确分类标签的核酸序列中每类核酸序列数目与特定值的比率,特定值的选择范围在核酸序列数据库中核酸序列中最多标签数目的核酸序列数量n
most
与最少标签数目的核酸序列数量n
least
之间的任意值,比率值=特定值
÷
每类核酸数目,比率值小于1的则人为将比率值设定为1;
45.(2)根据每类核酸的比率值求得此类核酸的分割步长,分割步长=短序列长度
÷
比率值。
46.在本实施例中短序列长度为1000bp,不同步长的计算方法如下:
47.(1)通过核酸序列数据库中每种亚型的数目求得每种亚型与特定值的比率。特定值的选择范围在数据库中含有核酸序列最多的标签数目(n
most
)与含有核酸序列最少的标
签数目(n
least
)之间(n
least
≤特定值≤n
most
),本实施例中选择hiv
‑
1_a1亚型在数据库中的数目作为特定值,为497,如图1所示。然后求得各亚型数目与497的比率形成txt格式文件,比率值=497
÷
亚型数目,比率值小于1的则人为将比率值设定为1。例如hiv
‑
1_01bc亚型在数据库中的数目为50,则比率ratio=497/50=9.94,如图3所示。
48.(2)根据每类核酸的比率值求得此类核酸的分割步长,分割步长=短序列长度
÷
比率值。例如,hiv
‑
1_b亚型的步长为step=1000bp
÷
1.0=1000bp,hiv
‑
1_01bc亚型的步长为step=1000bp
÷
9.94=100.603≈100bp。
49.根据每种亚型的步长将核酸序列数据库中对应亚型的全长基因组dna序列分割成1000bp短dna序列。分割核酸序列数据库后可得到hiv
‑
1_c亚型含1000bp短dna序列最多,数目为5066条;hiv
‑
1_ahju亚型含1000bp短dna序列最少,数目为2954条(在图中未显示),如图4所示。
50.s1
‑
3:判断s1
‑2‑
1或者s1
‑2‑
2获得短核酸序列的样本是否为均衡并适合于机器学习的样本,如果是,则停止s1
‑2‑
1或者s1
‑2‑
2,如果不是,则重复s1
‑2‑
1或者s1
‑2‑
2,直到获得均衡并适合于机器学习的短核酸序列的样本。在本实施例中,由于n
most
÷
n
least
≤2,所以分割后的样本是均衡的。
51.s2
‑
1:使用软件提取核酸序列数据库中所有的kmer组成集合。在本实施例中,使用jellyfish软件提取数据库种的所有20mer。kmer的取值范围为:4≤kmer长度≤短序列长度,在本实施例中短序列长度为1000bp,kmer选择20mer,如图5所示。
52.s2
‑
2:构建kmer字典,将s2
‑
1中的所有kmer按字母顺序从小到大排序,并且排序好后的kmer中的第一位置插入<unk>表示字典中不存在的kmer。
53.在本实施例中,构建20mer字典。将s2
‑
1中的所有20mer形成集合并按字母顺序从小到大排序,考虑到在待检测序列中的20mer可能不存在s2
‑
1中的20mer集合中,因此在排序好后的20mer中的第一位置插入<unk>表示字典中不存在的20mer。字典的查询方式为20mer:20mer所在字典中的位置。如图6所示。
54.s3:使用s2中的kmer字典将s1中的短核酸序列均衡样本转化成数字信号。在本实施例中,使用20mer字典将s1
‑
3中的均衡样本转化成数字信号,根据样本序列的kmer在kmer字典中的位置将样本转化成数字信号,如图7所示。
55.s4:使用s3中的数字信号训练一个能对带有分类标签的核酸序列进行多分类的神经网络分类器,该神经网络分类器用于识别和判断待检测核酸序列在核酸数据库中核酸分类。
56.s4
‑
1:搭建一个含有输入层、embedding层、最大池化层、平均化层、中间合并层、全连接层一、全连接层二、输出层的模型。在s4
‑
1中,输入层维度为1
×
981,维度计算公式为:短dna序列长度
‑
kmer+1,在此实施例中得到的信号长度为1000
‑
20+1=981;embedding层维度为6942463
×
100,其中6942463为构建字典中kmer的种类;最大池化层为获取embs(981
×
100)中每一列的最大值,得到一维数组max_pool(1
×
100);平均池化层为获取embs中每一列的平均值,得到一维数组avg_pool(1
×
100);中间合并层为将max_pool与avg_pool两者通过numpy模块中的concatenate函数进行拼接,得到一维数组input(1
×
200);全连接层一含有150个神经元;全连接层二含有150个神经元;输出层含有184个神经元,最后采用softmax激活函数把输出层每个神经元的值转化成概率值。如图8所示。
57.s4
‑
2:取s3数据集中80%的数据作为训练集,剩余20%的数据作为测试集。在选取时,需要保证训练集和测试集中各亚型的比例近似相同。将训练集送入s4
‑
1中搭建的神经网络中,对网络进行反复训练,并利用测试集不断测试。直到该网络的分类表现达到要求,如图9所示。
58.s4
‑
3:将上一步训练好的模型分类器保存到磁盘中。对后续待检测核酸序列进行分析。
59.s5:使用s2中的kmer字典将待检测序列转化成数字信号。
60.s5
‑
1:获得待检测hiv核酸序列65条,长度1000bp左右,如图10所示。
61.s5
‑
2:使用s2
‑
2中的20mer字典将s5
‑
1中的65条hiv核酸序列转化成数字信号。
62.s6:将s5中的数字信号输入到s4中训练好的神经网络分类器中,对待检测核酸序列进行分类。
63.s7:展示待检测核酸序列的分类结果。展示结果如图11所示,id为fasta文件中每条序列的id,top
‑
1至top
‑
5为这条id序列前5种可能的亚型。括号中的值为此种亚型的可靠性分值,分值越高则表明这条id序列为此亚型的可靠性越高。
64.本实施例使用本地计算机进行分析,本地计算机配置为:处理器intel(r)core(tm)i5
‑
9400 cpu@2.90ghz,内存8.00gb。神经网络训练好后,9秒内可对6万条核酸序列进行分类,分类结果kappa系数=0.982。并且由于在输出阶段可以看出检测核酸序列前5种可能的亚型,因此可以根据其概率值的分配评估其是否由多种亚型重组而成,若前两种亚型的概率值均在0.4以上则可判定此序列是由前两种亚型重组而成。
65.应该理解到披露的本发明不仅仅限于描述的特定的方法、方案和物质,因为这些均可变化。还应理解这里所用的术语仅仅是为了描述特定的实施方式方案的目的,而不是意欲限制本发明的范围,本发明的范围仅受限于所附的权利要求。
66.本领域的技术人员还将认识到,或者能够确认使用不超过常规实验,在本文中所述的本发明的具体的实施方案的许多等价物。这些等价物也包含在所附的权利要求中。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1