Bap1作为肿瘤治疗靶点在制备预防或治疗肿瘤药物中的应用
本发明属于肿瘤药物
技术领域:
,具体涉及bap1作为肿瘤治疗靶点在制备预防或治疗肿瘤药物中的应用。
背景技术:
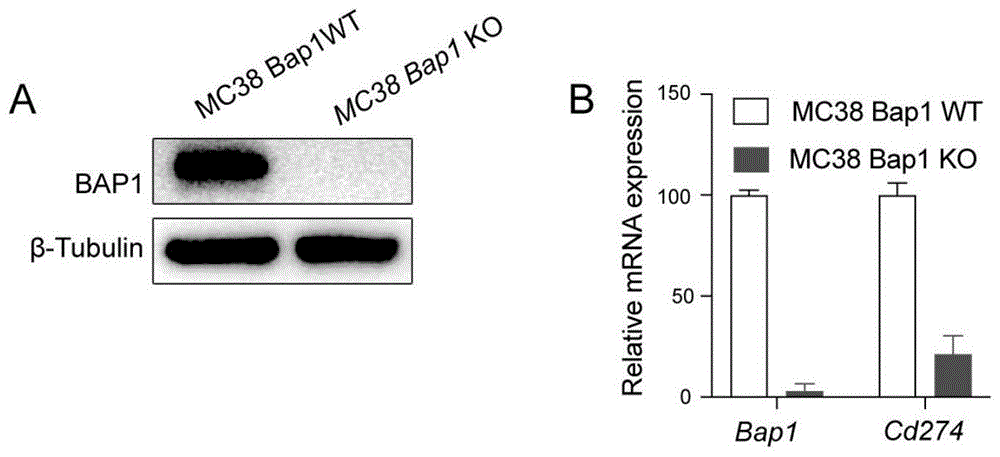
:brca1相关蛋白1(brca1associatedprotein1),即bap1,在人基因组中命名为bap1,为泛素羟基末端水解酶家族中的一员,其基本功能是去除底物蛋白质的泛素化修饰。bap1蛋白参与多种细胞功能的调控,如细胞增殖、细胞分化、程序性细胞死亡、dna损失修复和染色质修饰等。bap1基因突变在葡萄膜黑色素瘤、恶性间皮瘤和肾细胞癌中频繁发生,大部分早期研究认为bap1是一个抑癌基因。但是,近年来也有研究报道bap1可以通过其去泛素化的功能来促进乳腺癌的发生发展。技术实现要素:有鉴于此,本发明的目的在于提供一种bap1的新应用,即bap1作为肿瘤治疗靶点在制备预防或治疗肿瘤药物中的应用。本发明提供了抑制bap1表达或活性的试剂在制备具有以下(1)~(4)中至少一种功能的产品中的应用;(1)预防或治疗肿瘤;(2)抑制肿瘤细胞生长;(3)提高对肿瘤细胞的杀伤力;(4)延长肿瘤机体存活时间;(5)增加肿瘤细胞表面抗原的递呈。优选的,所述提高对肿瘤细胞的杀伤力包括细胞因子或细胞因子组合对肿瘤细胞的杀伤力和免疫细胞对肿瘤细胞的杀伤力。优选的,所述细胞因子包括fasl、tnfα、ifnγ或ifnβ;所述细胞因子组合包括fasl、tnfα、ifnγ和ifnβ中的两种或多种;所述免疫细胞包括t细胞;所述t细胞包括cd8t细胞。优选的,所述抑制肿瘤细胞生长包括促进肿瘤细胞的凋亡、抑制肿瘤细胞成瘤和肿瘤细胞被免疫系统清除。本发明还提供了缺失bap1的肿瘤细胞在制备肿瘤免疫治疗的药物中的应用。优选的,所述缺失bap1的肿瘤细胞依赖于cd8t细胞介导的抗肿瘤免疫反应。优选的,所述抑制bap1表达或活性的试剂包括抑制bap1基因的表达、抑制bap1蛋白的表达或丧失bap1蛋白活性的试剂。优选的,所述抑制bap1基因的表达是采用基因编辑的方式造成bap1基因发生碱基插入突变、碱基缺失突变、非同义的碱基突变或整段基因丢失。优选的,所述抑制bap1基因的表达的试剂包括干扰bap1基因的rna分子。优选的,所述抑制bap1蛋白的表达或丧失bap1蛋白活性的试剂包括bap1蛋白抗体或bap1蛋白小分子抑制剂。优选的,所述肿瘤包所述肿瘤包括葡萄膜黑色素瘤、睾丸生殖细胞癌、肺鳞癌、甲状腺癌、肾透明细胞癌、皮肤黑色素瘤、浸润性乳腺癌、脑低级别胶质瘤、前列腺癌、结肠癌、肾癌和肺癌中的一种或几种。本发明提供了抑制bap1表达或活性的试剂在制备具有以下(1)~(4)中至少一种功能的产品中的应用;(1)预防或治疗肿瘤;(2)抑制肿瘤细胞生长;(3)提高对肿瘤细胞的杀伤力;(4)延长肿瘤机体存活时间。本发明以bap1为肿瘤治疗靶点,通过敲除或抑制bap1基因或蛋白的功能,能够增强机体的抗肿瘤免疫功能,显著增强多种细胞因子以及t细胞对该肿瘤细胞的杀伤能力,抑制肿瘤细胞的生长或被清除,促进肿瘤细胞凋亡,延长肿瘤机体的存活时间,因此通过缺失bap1功能能显著增强肿瘤免疫治疗的效果,因此以bap1蛋白作为肿瘤治疗的靶点,拥有广阔的应用前景,具有极高的医学价值。本发明提供缺失bap1的肿瘤细胞在制备肿瘤免疫治疗的药物中的应用。实验证明,肿瘤细胞中的bap1缺失,能够使得小鼠对该肿瘤细胞产生长久的免疫记忆能力,使同种bap1缺失的肿瘤细胞快速被小鼠机体清除,实现了肿瘤免疫治疗目的。同时,本发明提供的应用,对多种肿瘤有治疗效果。实验证明,对黑色素瘤、结肠癌、乳腺癌、肾癌和肺癌等多种肿瘤细胞敲除bap1基因,均能够达到对应肿瘤的免疫治疗效果。附图说明图1为经基因编辑的mc38肿瘤细胞中bap1蛋白不能正常表达的检测结果;图2是敲除ct26肿瘤细胞中bap1蛋白后,注射该肿瘤细胞于正常balb/c小鼠的皮下,该肿瘤细胞的生长受到抑制直致消失的生长曲线图及小鼠的生存曲线图;图3为bap1缺失和野生型4t1肿瘤细胞在正常balb/c小鼠皮下注射后,肿瘤的生长曲线图和小鼠的生存曲线图;图4为bap1缺失和野生型emt6肿瘤细胞在正常balb/c小鼠皮下注射后,肿瘤的生长曲线图和小鼠的生存曲线图;图5为bap1缺失和野生型mc38肿瘤细胞在正常c57bl/6小鼠皮下注射后,肿瘤细胞的生长曲线图及小鼠的生存曲线图;图6为bap1缺失和野生型b16.f10肿瘤细胞在正常c57bl/6小鼠皮下注射后,肿瘤生长曲线图和小鼠的生存曲线图;图7为缺失bap1mc38肿瘤细胞可引起系统性的免疫记忆,其中a为野生型c57bl/6小鼠清除bap1缺失mc38细胞54天后,仍能清除野生型mc38细胞;b为c57bl/6小鼠对肿瘤细胞的记忆性杀伤依赖其cd8+t细胞;图8细胞因子tnfα、ifnγ或ifnβ处理bap1缺失的mc38肿瘤细胞后凋亡率统计结果;图9为敲除肿瘤细胞中bap1蛋白后,注射该肿瘤细胞于多种免疫缺陷小鼠皮下,肿瘤的生长曲线图;图10为通过敲除肿瘤细胞中的b2m、tap基因和fas基因验证cd8t淋巴细胞杀伤肿瘤依赖mhci介导的抗原提呈作用;图11为bap1蛋白缺失的肿瘤和bap1野生型肿瘤中免疫细胞浸润对比图,每克肿瘤样品中免疫细胞数目在bap1蛋白缺失的肿瘤显著增多;图12为肿瘤细胞中的bap1蛋白缺失致使c57bl/6小鼠肿瘤微环境中产生更多肿瘤特异性杀伤的cd8t细胞;图13为缺失bap1肿瘤细胞更易被t细胞杀伤;其中图13a为car-t细胞体内杀伤实验结果;图13b为ot-it细胞体内杀伤实验结果;图14为以tcga数据库人肿瘤数据为样本,分析患者中cd8+t细胞和活化cdc等免疫细胞的免疫浸润评分结果。具体实施方式本发明提供了抑制bap1表达或活性的试剂在制备具有以下(1)~(4)中至少一种功能的产品中的应用;(1)预防或治疗肿瘤;(2)抑制肿瘤细胞生长;(3)提高对肿瘤细胞的杀伤力;(4)延长肿瘤机体存活时间;(5)增加肿瘤细胞表面抗原的递呈。在本发明中,所述肿瘤包括葡萄膜黑色素瘤、睾丸生殖细胞癌、肺鳞癌、甲状腺癌、肾透明细胞癌、皮肤黑色素瘤、浸润性乳腺癌、脑低级别胶质瘤、前列腺癌、结肠癌、肾癌和肺癌中的一种或几种。在本发明中,所述抑制bap1表达或活性的试剂优选包括抑制bap1基因的表达、抑制bap1蛋白的表达或丧失bap1蛋白活性的试剂。所述抑制bap1蛋白的表达或丧失bap1蛋白活性的试剂优选包括bap1蛋白抗体或bap1蛋白小分子抑制剂。所述抑制bap1基因的表达优选是采用基因编辑的方式造成bap1基因(seqidno:1)发生碱基插入突变、碱基缺失突变、非同义的碱基突变或整段基因丢失。本发明通过向肿瘤细胞递送cas9和向导rna(sgrna),靶向编辑肿瘤细胞中bap1基因,使得肿瘤细胞bap1蛋白不能正常表达。所述抑制bap1基因的表达的试剂优选包括干扰bap1基因的rna分子,更优选为sgrna。所述sgrna优选包括两套grna,其中bap1_grnai包括bap1_grna_1f(tggcagccgctggacaata,seqidno:2)和bap1_grna_1r(tattgtccagcggctgcca,seqidno:3);bap1_grnaii包括bap1_grna_2f(ccttcattcggctcagcgt,seqidno:4)和bap1_grna_2r(acgctgagccgaatgaagg,seqidno:5)。采用两套grna分别对多种肿瘤细胞进行基因编辑,利用bap1_grna_i编辑对mc38细胞bap1基因编辑结果mc38bap1ko1如表1所示。表1mc38细胞bap1基因编辑后mc38bap1ko1序列注:---:删除序列,加粗:插入序列;-bp:删除片段大小,+bp:插入片段大小;起始突变位点:距离bap1基因的核苷酸序列5'端。下同。利用bap1_grna_i编辑对mc38细胞bap1基因编辑结果mc38bap1ko2如表2所示。表2mc38细胞bap1基因编辑后mc38bap1ko2序列利用bap1_grna_i编辑对ct26细胞bap1基因编辑结果ct26bap1ko2如表3所示。表3ct26细胞bap1基因编辑后ct26bap1ko1序列利用bap1_grna_i编辑对ct26细胞bap1基因编辑结果ct26bap1ko2如表4所示。表4ct26细胞bap1基因编辑后ct26bap1ko2序列利用bap1_grna_ii对mc38细胞bap1基因编辑结果mc38bap1ko3如表5所示。表5mc38细胞bap1基因编辑后mc38bap1ko3序列利用bap1_grna_ii对b16.f10细胞bap1基因编辑结果b16.f10bap1ko1如表6所示。表6b16f10细胞bap1基因编辑后b16f10bap1ko1序列利用bap1_grna_ii对b16.f10细胞bap1基因编辑结果b16.f10bap1ko2如表7所示。表7b16.f10细胞bap1基因编辑后b16.f10bap1ko2序列利用bap1_grna_ii对4t1细胞bap1基因编辑结果b16.f10bap1ko1如表8所示。表84t1细胞bap1基因编辑后4t1bap1ko1序列利用bap1_grna_ii对4t1细胞bap1基因编辑结果b16.f10bap1ko2如表9所示。表94t1细胞bap1基因编辑后4t1bap1ko2序列利用bap1_grna_ii对4t1细胞bap1基因编辑结果b16.f10bap1ko3如表10所示。表104t1细胞bap1基因编辑后4t1bap1ko3序列利用bap1_grna_ii对emt6细胞bap1基因编辑结果emt6bap1ko1如表10所示。表10emt6细胞bap1基因编辑后emt6bap1ko1序列利用bap1_grna_ii对emt6细胞bap1基因编辑结果emt6bap1ko2如表11所示。表11emt6细胞bap1基因编辑后emt6bap1ko2序列利用bap1_grna_ii对emt6细胞bap1基因编辑结果emt6bap1ko3如表12所示。表12emt6细胞bap1基因编辑后emt6bap1ko3序列所述的bap1_grna_i编辑的细胞利用sanger测序法所需的引物序列为上游引物:5'-acctcccacccctattatttt-3'(seqidno:39),下游引物:5'-gcagctagggaggaagataac-3'(seqidno:40);bap1_grna_ii编辑的细胞利用sanger测序法所需的引物序列为上游引物:5'-gactgggtaagacaaaggaa-3'(seqidno:41),下游引物:5'-gagcaacacaacagaaaagg-3'(seqidno:42)。在本发明中,所述预防或治疗肿瘤表现为将所述试剂抑制bap1基因或蛋白表达或丧失活性后,机体内细胞因子和免疫细胞对肿瘤细胞杀伤力强,抑制肿瘤细胞生长,促进其凋亡,具有去除肿瘤的能力,并对机体形成长久的保护作用,从而达到抗肿瘤的目的。在本发明中,所述提高对肿瘤细胞的杀伤力优选包括细胞因子或细胞因子组合对肿瘤细胞的杀伤力和免疫细胞对肿瘤细胞的杀伤力。所述细胞因子优选包括fasl、tnfα、ifnγ或ifnβ;所述细胞因子组合优选包括fasl、tnfα、ifnγ和ifnβ中的两种或多种,更优选为ifnγ+ifnβ组合。实验表明,缺失bap1肿瘤细胞凋亡水平上升,说明细胞因子fasl、tnfα、ifnγ,ifnβ,ifnγ+ifnβ对缺失bap1肿瘤细胞与对照肿瘤细胞相比具有更高的杀伤力,bap1的缺失增强了肿瘤对细胞因子的敏感性。肿瘤细胞中的bap1蛋白缺失能够使得小鼠肿瘤微环境中产生更多特异性杀伤该肿瘤的免疫细胞。肿瘤细胞bap1蛋白的缺失能够显著提高小鼠体内肿瘤的免疫细胞的浸润。所述免疫细胞优选包括t细胞。所述t细胞包括cd8t细胞。所述缺失bap1的肿瘤细胞依赖于cd8t细胞介导的抗肿瘤免疫反应。实验表明,所述bap1缺失的肿瘤细胞对cd8t细胞杀伤更敏感。缺失bap1肿瘤细胞的清除依赖于fas-fasl杀伤途径激活cd8t细胞在体内清除肿瘤细胞的杀伤途径,而不依赖于穿孔素-颗粒酶的杀伤途径。在本发明中,所述抑制肿瘤细胞生长优选包括促进肿瘤细胞的凋亡、抑制肿瘤细胞成瘤和肿瘤细胞被免疫系统清除。实验表明,bap1缺失的肿瘤细胞生长被体内的免疫系统显著抑制直至清除,延长了小鼠存活时间。同时肿瘤细胞中的bap1蛋白缺失,促进肿瘤细胞表型的改变,下调pd-l1基因的表达,提示bap1蛋白缺失影响肿瘤免疫刹车分子pd-l1的表达。bap1缺失肿瘤细胞生长被体内的免疫系统显著抑制直至清除,延长了小鼠存活时间。本发明还提供了缺失bap1的肿瘤细胞在制备肿瘤免疫治疗的药物中的应用。优选的肿瘤细胞中的bap1蛋白缺失,能够使得小鼠对该肿瘤细胞产生长久的免疫记忆能力。实验表明,所述的肿瘤细细胞被体内免疫系统清除后能够帮助机体抵抗同种肿瘤的形成,而不同细胞系来源的肿瘤细胞生长则不受影响。在本发明中,所述抑制bap1表达或活性的试剂在预防或治疗肿瘤中的应用。缺失bap1的肿瘤细胞在肿瘤免疫治疗中应用。所述试剂的种类与上述通过基因编辑技术编辑所述的bap1蛋白的基因使得肿瘤细胞内不再表达bap1蛋白的试剂;或通过抗体结合bap1蛋白使得肿瘤细胞内的bap1蛋白失去活性的抗体;或通过小分子抑制bap1蛋白使肿瘤细胞内的bap1蛋白失去活性的小分子抑制剂相同,在此不做赘述。所述缺失bap1的肿瘤细胞的制备方法优选采用基因编辑方法获得。所述基因编辑采用的试剂为上述sgrna。本发明实验结果明,bap1蛋白在多种肿瘤细胞中的缺失,均能够显著抑制或清除这些肿瘤细胞在具有正常免疫系统的小鼠体内的生长,表明其在抗肿瘤免疫治疗中具有广阔的应用前景。下面结合实施例对本发明提供的bap1作为肿瘤治疗靶点在制备预防或治疗肿瘤药物中的应用进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。实施例1一.mc38-cas9细胞的制备通过无内毒素质粒大抽试剂盒,提取编号为68343的质粒,该载体上带有cas9基因和抗blasiticidin基因。通过慢病毒感染将载体68343转到mc38细胞中,用含有blasiticidin的培养基进行培养,杀死未转68343的mc38细胞,连续杀毒约两周的时间便可得到mc38-cas9细胞。二.bap1sgrna的制备采用常规方法设计bap1_grnai基因编辑用引物:①bap1_grna_1f:cacctggcagccgctggacaata(seqidno:3);②bap1_grna_1r:aaactattgtccagcggctgcca(seqidno:4);其中下划线碱基是酶切位点;采用常规方法设计bap1_grnaii基因编辑用引物:③bap1_grna_2f:ccttcattcggctcagcgt(seqidno:4)④bap1_grna_2r:acgctgagccgaatgaagg(seqidno:5)。1.合成引物①bap1-grna-1f②bap1-grna-1r按生工说明书操作1)开盖前先离心,条件为4000rpm,60s;2)慢慢打开管盖,按照说明加入适量的dd|h20,将其稀释成100μm浓度;3)盖上盖子后,充分震荡混匀,瞬时离心,4000rpm,1min。2.单链rna的磷酸化与退火(单链变双链)表13反应体系的配制bap1-grna-1foligo1(100μm)1μlbap1-grna-1roligo2(100μm)1μlt4dnaligasebuffer(10×)(neb)1μlddh2o6.5μlt4pnk(neb)0.5μl总计10μl1)37℃水浴30min;2)微波开水至沸腾(高火约25min),将1)中样品放到95℃开水中,使其自然缓慢降至室温;3)取2)中液体3μl+597μlddh2o(1:200稀释),混匀后离心。3.质粒载体的克隆表14克隆体系的配制67974线性化载体(28.4ng/μlbpil酶切产物)2μl退火双链(3)中1:200稀释的)1μlt4dnaligasebuffer(10×)(neb)1μlddh2o6μlt4dnaligase1μl总计11μl1)涡旋混匀后,离心;2)25℃水浴30min,室温放置。3)转化(1)取感受态细胞;(2)冰上3min待其融化成冰水混合物,将3ul的连接产物加入20μl的感受态,放冰上静置30min;(3)42℃热激30s,冰上放置2min;(4)将其加入500μllb,放到摇菌锥形瓶中摇1h(37℃220rpm);(5)取100~150μl的菌加入含有amp抗性的培养板中,摇匀培养;(6)将板子放到37℃培养箱中,过夜培养;(7)第二天早上挑单克隆,到含amp抗体的la培养基中培养;(8)4~5h后,送生工测序;(9)利用seqman对每隔克隆细胞进行分析,挑选sgrna成功连接在67974载体上的克隆。三、将上述步骤得到的sgrna进行质粒提取、慢病毒包装、慢病毒感染cas9表达的细胞系,利用流式分选bfp阳性的单克隆细胞(67974载体上含有bfp蓝色荧光蛋白),待细胞扩大培养后,利用sanger法将其送生工测序,分析结果。具体方法如下:1.病毒包装:将hek293ft细胞接种到10厘米细胞培养皿中,当细胞密度达到70%~80%进行慢病毒包装;在3mlopti-mem中加入5.4μg重组质粒、5.4μgpspax2、1.2μgpmd2.g和12μlplus(11514015,invitrogen),室温孵育5分钟;在该混合物中加入36μllipofectamineltx(15338100,invitrogen),轻轻混匀,室温孵育30分钟;将hek293ft的培养基换成5mlopti-mem后,在培养皿中加入孵育好的复合物,细胞培养箱中孵育4~8小时后弃掉上清,加入10ml完全培养基;48~72小时后,用10ml注射器吸取培养皿中的上清,用0.45μm的过滤器(slhv033rb,millipore)过滤到离心管中,过滤后的病毒液至于-80℃保存。2.病毒感染:取1×106个细胞于离心管中,加入适量的病毒液(moi<0.7)重悬细胞,培养箱中孵育30分钟;在6孔板中加入3ml培养基,在培养箱中预热,将孵育结束的细胞病毒混合液加入培养基中;48小时后进行细胞传代即可。3.单细胞分选用重组质粒进行病毒包装和病毒感染cas9稳定表达的细胞系,方法参考上述,病毒感染5~7天后进行单细胞分选。为了提高单细胞的存活率,可以在96孔板每孔加入100μl的0.1%的明胶,室温孵育1h,每孔加入100μldpbs洗3次(非必须步骤);在96孔板加入100μl含2%ps的培养基,用封口膜封上口,4℃或者冰上分选备用。病毒感染5~7天的细胞在t25培养瓶中培养,加1ml1×dpbs洗一次,加500μl0.25%trypsin消化1min,加1ml培养基dmem培养基终止消化,350g,3min离心,弃上清,facs缓冲液重悬,用200目滤网过滤到无菌流式管中,冰上保存;因为重组质粒表达蓝色荧光蛋白(bfp)(addgene#67974)、利用分选型流式细胞仪,选择bfp阳性,上机4℃分选,分选结束后在96孔板中补加100μl的dmem培养基,放进培养箱培养。对于比较脆弱的细胞可以每隔3~4天换液,10天左右能够在96孔板中观察到单克隆细胞。4.sanger法测序利用oligo引物设计软件,在sgrna靶向的基因序列前后设计引物,需要注意的是,引物的位置最好距离靶点100bp以上,因为测序会导致靠近引物的序列结果不准确。设计的引物使pcr产物的长度在300~800bp以内。引物用ddh2o溶解成10nm备用,-20℃保存。从96孔板中取适量单克隆细胞,加入200μlpcr管或96孔pcr板子中,做好样品标记,加入20μldna裂解(加入1%的蛋白酶k溶液,试剂盒中提供)(macherey-nagel货号73200.500),放入pcr仪,25℃裂解3min,95℃终止裂解5min。pcr反应体系见表15。表15pcr反应体系其中bap1_grna_i基因编辑的引物序列为上游引物:5'-acctcccacccctattatttt-3'(seqidno:39),下游引物:5'-gcagctagggaggaagataac-3'(seqidno:40);bap1_grna_ii编辑的引物序列为上游引物:5'-gactgggtaagacaaaggaa-3'(seqidno:41),下游引物:5'-gagcaacacaacagaaaagg-3'(seqidno:42)。pcr反应条件见表16。表16pcr反应条件pcr产物在1.2%琼脂糖凝胶电泳,胶回收目标大小的dna片段。dna片段克隆到pmd19-t载体中(6013,takara),反应体系见表17,形成含dna片段的pmd19-t的载体。表17克隆体系的配制dna片段2.25μlpmd19-t0.25μlsolutioni2.5μl16℃30min4℃hold冰上融化100μl的感受态细胞(cd501,全式金)到冰水混合物状态,约2min左右,取16μl感受态细胞加入3μl连接了待测dna片段的pmd19-t的载体。感受态反应条件见表18。表18感受态反应条件在la固体培养板中加入10~20颗平板涂布玻璃珠(生工货号b529319-0025),将100μl或全部的菌加入6厘米培养皿中,震荡涂布摇匀,37℃恒温箱,倒置过夜培养(注:摇匀后la固体培养基表面最好呈现干燥状态);在1.5ml离心管中加入0.5~1mlla液体培养基,用10μl枪头沾取单个克隆加入1.5ml离心管中,37℃,200rpm扩大培养,选pmd19-t载体上的通用引物送公司测序。利用bap1_grna_i编辑分别对mc38细胞、ct26细胞的bap1进行基因编辑,得到敲除bap1的mc38细胞和ct26细胞,敲除后的细胞bap1基因序列变化具体见表1~表4。利用bap1_grna_ii编辑分别对mc38细胞、b16.f10细胞、4t1细胞、emt6细胞的bap1进行基因编辑,得到敲除bap1的mc38细胞、b16.f10细胞、4t1细胞、emt6细胞,敲除后的细胞bap1基因序列变化具体见表5~表13。实施例2western-blot检测1.取上述制备的bap1缺失的mc38肿瘤细胞,贴壁细胞弃上清,用1×pbs洗一遍,加胰酶消化成单细胞,加完全培养基终止消化,转移到离心管,350g,3min离心,弃上清。加1×pbs重悬清洗并计数,取1×106个细胞,转移到1.5ml离心管,350g,3min离心弃上清,置于冰上。2.裂解细胞:ripa裂解液配制:从-20℃冰箱取出分装的ripa(1ml/管)(碧云天,货号p0013b;thermoscientific,货号89900),室温融化,1mlripa加入10μl蛋白酶抑制剂(雅酶,货号crf101),现配现用;5×loadingbuffer配制:从4℃取出配制的5×sds-page蛋白上样缓冲液(500μl/管)按照体积比5%加入20μl2-巯基乙醇(gibco货号21985023),配置好室温可以保存1个月,长期保存放入4℃冰箱;步骤1中1×106个细胞加80μl含蛋白酶抑制剂的ripa移液枪混匀,冰上裂解10min震荡5s,重复2次,共裂解30min,加入20μl含有2-巯基乙醇的5×loadingbuffer,振荡混匀,100℃金属浴加热5min,样本-20℃保存。3.制胶:ddh2o清洗玻璃板,对齐放置于制胶架上,倒置控干,同时从4℃冰箱拿出page凝胶快速制备试剂盒(7.5%)(雅酶,货号rg11升级),从-20℃拿出改良型过硫酸铵溶液室温融化,快融化时可以震荡10s加速融化并混匀;分离胶为4mla试剂加4mlb试剂,加改良型过硫酸铵80μl,震荡5s混匀,加入玻璃板中,加入2ml乙醇压平,20min左右倒出乙醇并等待乙醇挥发;浓缩胶为2mla试剂加2mlb试剂,加改良型过硫酸铵40μl,震荡5s混匀,加入玻璃板中,插入梳子,30min左右凝固后加入电泳槽,电泳槽可以同时跑两块胶,若只跑一块胶,另一面用塑料板固定夹紧,在电泳槽中加入1×sds-page电泳缓冲液,并确保两块胶中间的液面高于外面。4.加样:加样顺序为蛋白marker(雅酶,货号wj101),对照组样本,实验组样本,1.5mm梳子最多上14个样品,用10μl枪头上样,每个孔加10μl。加样注意不要将细胞碎片加入孔中,胶板中间补满1×sds-page电泳缓冲液,80v恒压跑浓缩胶,跑到浓缩胶下面换成120v恒压跑分离胶,跑到溴酚蓝到胶板边缘,停止。5.转膜:100ml10×转膜液加700mlddh2o加200ml甲醇配制成1×转膜液。在盒子中加入50ml左右的1×转膜液,在另一个盒子中加入50ml甲醇。将浓缩胶割掉,将溴酚蓝割掉,在胶左上角切角标记,量胶的长宽后,将胶浸泡在1×转膜液中,剪切厚滤纸两张和胶同长宽,放在1×转膜液中,剪切pvdf膜和胶同长宽,放在甲醇中浸泡活化5min,在转膜架正极在下(透明),负极在上(黑色)放置,透明面上依次对齐放上塑料棉,滤纸,pvdf膜,胶(切角位于右上方),滤纸,塑料棉,注意过程中保持胶膜湿润,需要倒入1×转膜液保持湿润但是不能有气泡,夹紧后放入转膜槽中,空白位置放入冰盒,在冰浴中,330v,转膜1.5h。pvdf膜贴胶的一面朝上(正面),在膜左上角切角标记,按照目标条带大小裁膜。6.封闭:pvdf膜正面朝上用5%(tbst为溶剂)脱脂奶粉(全称difcotmskimmilk,公司bd,货号232100)摇床室温封闭2h。7.抗体染色:5%(tbst)脱脂奶粉,1:2000配制bap1抗体(cst,货号13271s),1:2000配制β-tublin抗体(cst,货号86298s)。4℃摇床过夜。8.洗掉非特异性结合一抗:用tbst洗3次,每次10分钟。9.孵育二抗:5%(tbst)脱脂奶粉,抗兔二抗(cst货号7074s),抗鼠二抗(cst货号7076s),抗山羊二抗(abcam货号ab6741)按照1:2000比例加入二抗抗体,室温摇床2h。10洗掉非特异性结合二抗:tbst洗3次,每次10分钟。11成像:在膜上加200μlecl蛋白印迹底物(thermoscientific货号21232),使用bio-rad化学发光成像系统(型号chemidocimagingsystem)成像。结果见图1。由结果可知,bap1缺失和对照的mc38肿瘤细胞中,bap1缺失的肿瘤中没有bap1蛋白质的表达。bap1缺失和对照的mc38肿瘤细胞中,bap1缺失的mc38肿瘤细胞不能正常表达mrna,在基因编辑位点之后的mrna表达水平显著降低。实施例3将上述制备的缺失bap1的ct26细胞(ct26bap1ko)与野生型ct26细胞(ct26bap1wt)分别皮下注射至balb/c小鼠,注射一百万细胞后,观察bap1缺失组和对照组的ct26肿瘤生长情况。同时将上述制备的缺失bap1的4t1细胞、缺失bap1的emt6及其野生型细胞注射小鼠皮下,来研究bap1缺失是否能够肿瘤细胞系生长受到抑制。结果见图2~图4。结果表明,缺失bap1的ct26肿瘤细胞、缺失bap1的emt6和缺失bap1的4t1细胞生长被显著抑制,且延长了该组小鼠的存活时间。实施例4将上述制备的敲除bap1的mc38细胞(mc38bap1ko)与野生型mc38细胞(mc38bap1wt)分别皮下注射至c57bl6/j小鼠皮下注射一百万细胞后,观察bap1缺失组和对照组的肿瘤生长情况,来研究bap1缺失是否能够肿瘤细胞系生长受到抑制。结果见图5。结果表明,bap1缺失的mc38肿瘤细胞生长被显著抑制,且延长了该组小鼠的存活时间。实施例5将上述制备的敲除bap1的b16.f10细胞(b16.f10bap1ko)与野生型b16.f10细胞(b16.f10bap1wt)分别皮下注射至c57bl6/j小鼠注射一百万细胞后,观察bap1缺失组和对照组的肿瘤生长情况,来研究bap1缺失是否能够肿瘤细胞系生长受到抑制。结果见图6。结果表明,bap1缺失的b16.f10肿瘤细胞生长被显著抑制,且延长了该组小鼠的存活时间。说明缺失bap1的肿瘤细胞生长被体内的免疫系统显著抑制或清除,延长了小鼠存活时间。实施例6当小鼠中bap1komc38肿瘤细胞被机体彻底清除以后,再次向小鼠皮下分别注射野生型mc38细胞和b16.f10细胞,发现mc38细胞生长受到抑制并被清除,而b16.f10细胞可成瘤且不能被清除,说明小鼠体内产生了免疫记忆并且只识别相同类型的肿瘤抗原;此外,利用anti-cd8抗体(yts169.4,bioxcell)剔除已获得免疫记忆的小鼠体内cd8+t细胞后,再次注射相同的野生型肿瘤细胞,其生长不受抑制(见图7)。这些结果表明缺失bap1mc38肿瘤细胞可引起系统性的免疫记忆。实施例7将上述制备得到的bap1缺失mc38肿瘤细胞与野生型mc38肿瘤细胞分别进行细胞培养,培养期间,分别采用20ng/mltnfα、100ng/mlifnγ,1000u/mlifnβ,1000u/mlifnβ+100ng/mlifnγ刺激,刺激72小时后,通过annexinv-fitc/pi凋亡检测试剂盒对细胞进行流式染色,结果显示检测各组中bap1缺失及野生型mc38肿瘤细胞凋亡水平。结果见图8。与野生型mc38肿瘤细胞相比,bap1缺失mc38肿瘤细胞在ifnγ、ifnβ和ifnβ+ifnγ刺激72小时后,bap1缺失的mc38肿瘤细胞凋亡水平上升;而bap1缺失mc38肿瘤细胞在tnfα刺激72小时后,bap1缺失的mc38肿瘤细胞凋亡水平上升。实施例8将敲除bap1的mc38肿瘤细胞皮下注射该肿瘤细胞于免疫缺陷小鼠(例如:缺乏成熟t、b细胞的rag2-/-小鼠,tcra-/-小鼠,batf3-/-小鼠)或用腹腔注射抗体的方式清除小鼠体内的cd4+t细胞和cd19+b和cd8+t细胞,绘制由于t细胞和dc细胞的缺失该肿瘤细胞无法被免疫系统完全清除的生长曲线图。结果见图9。bap1缺失的肿瘤细胞能够被小鼠清除的主要原因是树突状细胞介导的cd8+t淋巴细胞对肝癌细胞的特异性杀伤效应。实施例9通过敲除肿瘤细胞中的b2m、tap基因和fas基因验证cd8t淋巴细胞杀伤肿瘤的途径。按照实施例1中记载的基因敲除方法,对mc38肿瘤细胞中b2m(b2msgrna1caccgtatactcacgccacccac,seqidno:43)和bap1进行双基因敲除,筛选验证得到双基因敲除肿瘤细胞(记为b2mbap1dko),对mc38肿瘤细胞中tap(tapsgrna1caccctaggactaggggtccgcg,seqidno:44)和bap1进行双基因敲除,筛选验证得到双基因敲除肿瘤细胞(记为tapbap1dko),对mc38肿瘤细胞中fas基因(fassgrna1caccctccgagagtttaaagctg,seqidno:45)和bap1基因进行双基因敲除,筛选验证得到双基因敲除肿瘤细胞(记为fasbap1dko)。分别将上述制备的三个双基因敲除的肿瘤细胞皮下注射至c57bl6/j小鼠,注射一百万细胞后,观察并统计三组双基因缺失的肿瘤生长情况。结果见图10。将bap1和tap1和bap1和b2m基因敲除的肿瘤细胞系皮下注射到c57bl/6小鼠,发现肿瘤的生长得到恢复,说明mhci介导的抗原递呈作用对机体产生免疫反应非常重要。将bap1fasdko肿瘤细胞注射到c57bl/6体内,结果显示缺失bap1肿瘤细胞的清除依赖于fas-fasl杀伤途径而不依赖于穿孔素-颗粒酶的杀伤途径。实施例10流式细胞术分析肿瘤微环境的免疫细胞c57bl/6小鼠皮下mc38肿瘤细胞生长6~14天,分离肿瘤。用肿瘤解离试剂盒(miltenyibiotec货号130-096-730),将肿瘤解离成单细胞,用红细胞裂解液(gibco货号a10492-01)去除红细胞。用去死细胞试剂盒(miltenyibiotec货号130-090-101)去掉肿瘤细胞中的死细胞,用cd45+细胞富集试剂盒(miltenyibiotec货号130-110-618)富集肿瘤细胞中的cd45+的细胞,进行抗体染色。用注射器研磨肿瘤淋巴结,70目滤网过滤细胞,facs缓冲液冲洗滤网和研磨的培养皿,ack裂解红细胞后,进行抗体染色。bap1蛋白缺失的肿瘤和野生型bap1蛋白表达的肿瘤内部浸润的免疫细胞类型对比图见图11。由图11所示,与野生型肿瘤细胞相比,bap1缺失的肿瘤中的cd45阳性细胞的比例显著增高,同时cd4阳性的t细胞、cd8阳性的t细胞以及γδt细胞的比例均有显著的增高,且cd8阳性的t细胞的增高最大。这表明bap1蛋白的缺失增强了肿瘤微环境中的免疫细胞浸润,尤其是cd8阳性的t细胞增高最为明显。实施例11肿瘤细胞中的bap1蛋白缺失,能够使得c57bl/6小鼠肿瘤微环境中产生更多特异性杀伤该肿瘤的cd8+t细胞将上述制备的缺失bap1mc38肿瘤细胞(mc38bap1ko)和野生型mc38肿瘤细胞(mc38bap1wt)分别表达ova基因,分别得到表达ova蛋白的缺失bap1mc38肿瘤细胞(bap1ko-mc38-ova)和野生型mc38肿瘤细胞(bap1wt-mc38-ova)。将表达ova的肿瘤细胞分别移植到c57bl/6小鼠皮下和batf3-/-的小鼠皮下,利用ovatetramer-siinfekl分析bap1的缺失是否促进小鼠产生anti-ova特异性cd8+t细胞。结果见图12。结果显示在batf3-/-体内识别siinfekl抗原特异性的cd8+t细胞的比例很低,而在c57bl/6体内,在缺失bap1肿瘤细胞中,结合肿瘤表位抗原siinfekl的cd8+t细胞比例明显增多,这表明bap1的缺失增加了肿瘤细胞表面抗原的递呈。实施例12分别将表达cd19的野生型肿瘤细胞(mc38-wt(cd19))和表达cd19的bap1缺失肿瘤细胞(mc38-ko(cd19))注射至rag2-/-小鼠皮下,在肿瘤注射后第三天,尾静脉注射激活的car-t细胞,统计肿瘤体积变化情况。结果表明,mc38-ko(cd19)组较mc38-wt(cd19)组肿瘤生长速度明显降低(图13a)。同时将表达ova的肿瘤细胞注射至rag2-/-小鼠皮下,分离ot-i小鼠的cd8+t细胞,在肿瘤注射后第三天,尾静脉注射激活的ot-1t细胞,统计肿瘤体积变化情况。结果表明,mc38-ko(ova)肿瘤组较bap1wt(ova)组肿瘤生长速度明显降低(图13b),说明缺失bap1肿瘤细胞更易被cd8+t细胞杀伤。实施例13tcga数据集中33种癌症的mrna表达谱可从网站cbioportal(http://www.cbioportal.org/)获得。所有分析和绘图均使用r软件完成。基因gzmb和ifng的平均mrna表达水平用于测量t细胞的细胞毒性。使用计算方法xcell(https://github.com/dviraran/xcell)计算了患者中cd8+t细胞和活化cdc等免疫细胞的免疫浸润评分,应用wilcoxon检验比较两组之间的免疫评分分布。结果见图14,其中图14中各癌症的中文名称及英文全称见表19。表19各癌症中文及英文名称对应表注:-log10(p值)>2的癌症种类为图14中灰点部分,-log10(p值)<2的癌症种类为图14中黑点部分。对tcga数据库人肿瘤样本进行分析发现,在葡萄膜黑色素瘤(uvm)、睾丸生殖细胞癌(tgct)和肺鳞癌(lusc)等多种肿瘤中bap1的表达水平与t细胞毒性呈负相关(图14中a)。在lusc和和brca中,bap1的表达水平与t细胞毒性、cd8+t细胞浸润、活化cdc的浸润呈负相关。这表明bap1的功能在多种癌症中都与t细胞毒性、cd8+t细胞浸润、活化cdc的浸润相关。以上所述仅是本发明的优选实施方式,应当指出,对于本
技术领域:
的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。序列表<110>同济大学附属第一妇婴保健院<120>bap1作为肿瘤治疗靶点在制备预防或治疗肿瘤药物中的应用<160>42<170>siposequencelisting1.0<210>1<211>8530<212>dna<213>智人(homosapiens)<400>1gcctccgcccccgccgttcggccggcggaagaccagcccccgggttaggctcgtcgtgtg60atgatgacgttgtacgtccggcgcgggcgagtgacgcggcgcggcccgttgtctgtgcgg120ggctgagaagccctaagggcggtgggcgctcccggttgggggcggcgcgggtttagcagg180gcctggacatggcgctgagaggccgctccgcgggaagatgaataagggctggctggagct240ggagagtgacccaggtacggaggggcctcggcgggccgggggctggggaggccggatggg300ccccggacgcacctgcatgacaaatccctcttcttgtcgtcccacccaggcctcttcacc360ctcctggtggaagatttcggtaagaaccccgttacccacgaccttcggtccctggctggt420gtggatgtgggggttggtccttgagttctcgcagcagagcctgggactgccctgccgacc480tctcctttcctgatcgtaggtgtcaaaggggtgcaagtggaggagatctatgaccttcag540agtaaatgccaggggtgagtggctgggacgcccccttacctccggagggccgagaaaggg600caggggaatagtaccccattcaagggaggcagtactagaaaacctgagctatataaaata660tcacataagaaagtgtcactctttgtgggtgccctcttggacttttcttttcttctcagt720ctttcctcagtgtttctccttagaaagctgtagccctccttttaaccctaagcactcgtg780gagtacagttgagctgcctttctggttttgcagccggaaccctccgttgcatagtgttgc840accttttttgtaagtgtacagtccattaattacatcaccacatgtgctgtgcctttaatc900accacacaacatttgtcctgaccattagtggcgctctcctctagtcccaatgcttgggag960gcagaggcaggcagatctctgagtttgagtccaacctacgctacttagtgagaccatgtg1020tcaacaaaactgaacacagttgtcctgtattgattggttagtgtgagaaggcctttttta1080ggtacctaaactcagagtcttgttttggaaatctatagaatagaggcatagagaacctaa1140ctgcttccaagggaaggaactggtttgggagttcacgcactatactcattttactaggac1200tggaggccgaggtccaccagtgagttggtacttaattcttcatcaacgtttttgtgcctt1260ttattgtgtccctaccttgctccatcctgggccagcagttccagatttccttcttctctt1320gcctagacctcatagaccgagtggggtttgagagcttcaggctcatacttgactgcctgt1380gataggcttctggggtttgaggtctcagagccatgttggatacagtttggttaagagtga1440gggactcagtgcaagagcatttcagtgcaaaggattagggagagaaagaaggggatgctg1500attgtcatctgcctcttgcttctctaggcctgtgtatggatttatcttcctgttcaaatg1560gatcgaagagcgcaggtcccgccgcaaggtttctacgttggtggatgacacgtctgtgat1620tgatgatgatattgtgaataacatgttctttgctcaccaggtctgctgagttctgcgctt1680gtttcgggccaggatgctatttttgtctttgtttgtttctggtattgtgtggaaggtggc1740agtgggggagcaggaagaagcgctagactggaaccaagagaaactgttgcagaaaccctg1800tggcagacagacctaactctttttcattagagggaatacctctgtagttggtggtgtttt1860agtacattctccatgtccctagataggaggtattttagttggttcatgttaaatgaataa1920agactgggtaagacaaaggaaactaaaggatgttgacatctttctttttagtcttcattc1980tcaataagtgtgggcatggggatgtctggggtaaagagtatatgaggtatatgagtgatt2040gtttttgatttatagctgattcccaactcttgtgccactcacgccttgctaagcgtgctt2100ctgaactgcagcaatgtggatctggggcccacgctgagccgaatgaaggatttcaccaaa2160ggcttcagtcctgaggtacgctgcagtaccttagatcatgcttacagatggctgggcaca2220tctgatcctcagagctattggtaatgccactgcatggtatctggtattttttggtcattt2280ctttttcaaatgtttactaacatctatttgactcataagtaatgtggaaggtagttgatt2340ttaggctttgctgctaaatgagatgatagaaggaaaagacttaagtgatctgggtcagac2400cttttctgttgtgttgctccccagtagtcactgatggctgtgcacggccaatatggccca2460tctctgtaggaaatcctaagcaaggtctcatttgccaccactgccctttcagaacggtca2520tctgtgctgcttgggattcctggaatgcctctaagcctgctttatggaactcttccccta2580ttcagaggctcagctgagaatctgtatttatgtagtcttcttttatttttagagcaaagg2640atatgcaattggcaatgcccctgaactggccaaggcacataatagccatgccaggtatgt2700gagagctatggttgctgatgtggggtcggggtggtggaggatgtctctgattttgtcctt2760ttccaggccagaaccacgtcaccttcctgagaaacagaatggcctcagtgcagtgcggac2820catggaggcgtttcattttgtcagctatgtgcctatcacagggaggctctttgaattgga2880tggattgaaagtctacccaattgatcatggtaggccttctgaggaaggggcctgttgggt2940ttttcatgcttccttctgattttctgggtctcagggactttagatccttgagatgtagga3000gcgtgagtggataccagggacatctaactcttgccatggtttcatcttatactgtttatg3060tgtctagcttgtagcttctcaaccacacgaatctgttaaccctggcctaccccatctcag3120ggtttcccacctgctgattcctcatcttggtcttccctagggccctggggagaggacgag3180gagtggacagataaagctcgaagggtcatcatggagcgaatcggtcttgctactgcaggg3240taagaacccttggttgtgtccttctggagctatgtcccctttggggggaaagagagaagc3300gtaatgatctgtagattgtaaggccccatggtatctggcaagatcaggcttggggtttgg3360gatttaaattttaggttgcctgtggcctgctctgggctgggctggaggagggacaagcct3420tgtgtgtgtgtgtgtgtgcggggggcgtgttaagaagcacaggtgtgctggcttacttcc3480tgacaagcctctatcagagctcatgggaaggtgtagctgggtttcagatgtgtgtctggc3540tgctcgctgcctattggagtggagcctatacttagagctacagatgtgactgtgagagga3600gctctggcaagacacttgcctcagtcttaacagcaggtcaggtctggggtgggaactcct3660ctcatggttgccactgtgattgcagggagccctatcatgacattcgcttcaacctgatgg3720cagtggtgcctgaccgcaggattaagtatgagaccaggctacatgtactgaaggtgaacc3780gacaaacagtcctggaggccctgcagcaggtaagttgcctggctttgccaacccagtgct3840ccctgtctctcctacattttatttagtatctgtctctaattcccaaatcagtgtcttgta3900gattgaggctttaactaaccatttctttcctttgcttcctttgtaagggtggtagaactt3960aacttctaaaacgatgttgtggcttggcctttttattttccaataaataaataaataaat4020aaataagcaggcaggcagggtcaaggcccttagctatctatggtggaaacctggctctat4080ccttctctcctaagcttgagttttgttttaagctgattagagtaacccagccggagctga4140ttcagacccacaaatctcaagagtcacagctgcctgaggagagcaagccagccagcagca4200agtccccccttggactggaggcaggcagaaccccagtggcctctgagtgcactcagacag4260gtactagagggacttgggcatcagccatccatcgtagcttgacataggaagcacaagcac4320ttcaagaaggtacttggtacaggggctctagcaggaggcctgaaatcccagtacctgaag4380ctttcccattaaaacagggagagtagagtgagattttcctaaaggatacgtgaaaaggac4440tatcatgaccctagacccacacctctctgtattgggtggtccatcccatcccatacctcc4500cacccctattattttatctataggctgccttcttgttgtgtcttgttttgttttttgttt4560ttttttttttttttcattgtgtgacttagagcttacctgcctatgctgtttgctgtcttc4620actgggggaaacaactgtcacaggattatggactgtagcttttcttaaggacacagctga4680atgtcctatttatactttgccttgtctcagatggtgcagaggaggtggctggttcgtgcc4740cacaaactacaacccatagtcctcccagcaaatgtaagctggtggtgaagcctccaggga4800gcagcctcaatggggttcccccaaaccctgcccctattgtccagcggctgccagccttcc4860tagacaatcacaattatgccaaatcccctatgcaggtaagatggaagtaccctcaactga4920gaggccacaatgttggttcccagtgttattctttgactctctgattggttcttgatgacc4980aggctcctctagtactcataggccccaacagagcgcatggccttaaggtggctaagagag5040ctgcttctctgacttttaagtccagttgcctgccacacagttgggtatactccttggcac5100atgggaaagtttctcctggtaactcaaaagtatgtggcaaggcattctgtttcttttaga5160agcacttcgcacagctaagagcctagttatcttcctccctagctgcgataccatgcctgg5220cttgggtgagctctctcattaccgctcttgcctgccccaggaggaggaagacctggcggc5280aggagtgggtcgcagccgcgttcccgtccgagcgccccagcagtactctgaagacgagga5340tgactacgaggatgaggacgaggacgtgcagaacaccaaccctgccatcaggtcagtcct5400gactcggagacggtgtcaaactctgggtacctcagatcctgttctcaatattccaccctg5460cgtctgagagaacagtagacagaatgcaggccctggaggaggctggcttgaggggctaac5520ccataagtttagggggaggtaaacatcttaccatgtatgtgtcagtacattttgttgagg5580tgatctttcagcccgacctcaaagaattagggtaggagaagaggttggagagatgatttg5640ctgagtaggatggcacttttgttggaggatagagcactagaaggttagaagtgagatagc5700tagagtagaggaggccacctgcctatactctggcacttaggaagctcttgctaagagatt5760gtttagaccaagtgtaagagcttccatttgggcttgggaagaagatactatatttgagcc5820attctggacactgctgggtgtggtcacctggcccattccctgtttttacctcttattgga5880ccccacagatacaagcggaaggggacagggaagccaggatcgttgagcaattcttcagat5940gggcagctgtcagtgctgcagcccaacaccatcaatgtcttaactgagaagcttcaagag6000tctcagaaagacctttcagttcctctgtccatcaagactagcagtggggctgggagtcca6060gctgtggctgtgcccacacactcgcaaccttcacccacccctagcaatgagagcacggac6120acagcctctgagattggcagtgctttcaactcacccttgcgctcgcccatccgctcggcc6180aacccaacacggccctctagccctgtcacctctcacatctccaaggtgctttttggagaa6240gatgacagcctacttcgtgttgactgcatacgctacaaccgtgctgtccgtgacctgggt6300cctgtcattagcacgggcctgctgcaccttgctgaagatggtgtactgagtcccctggca6360ctcacaggtgggccttggactccctcactggtcacttggtgtacccaaggaggaggaggg6420aggtggcccggtgactacagagtatcatgtactctgatgcacccttttattcctctctag6480agggtgggaagggttcctcaccttctaccagatcaagccaaggcagccaggggtccagtg6540gcctagaggagaaggaggtggtggaagtcacagagagcagagacaagcctgggctgaaca6600ggtccagtgagcccttgagtggagagaagtactcacccaaggtgagtgtctttcagcctt6660ctcctagcaggggaaggacctgggcccctactcccaggttttgggtacttgattggaatt6720ttggagtgaaagttggtggctaaagccagggagctctttgggattggatggcattggtag6780ccccctggactgtggctgttcacccttgtttctagttgcttcttagtaatggagctttgg6840tgccagtcaggcccctaaggtatacaatgttgggttgtggcaggagctgctagcactgct6900aaagtgtgtagaagccgagattgcaaactatgaggcctgtctcaaggaggaggtggagaa6960aaggaagaagttcaaggtgggtgacccctctactgcccaaggacagctgctctgagaaga7020tgggctgggatgccctcatgtgccttctttttcacagattgatgaccagcgaaggaccca7080caactatgacgagtttatctgtaccttcatatccatgctggctcaggaaggtgaggaggt7140gcctactatgtcttaattgtcctaccctcttgagggtcatgtacctcaaactcctccccc7200tccctcccccaagacttcacctttggccgatgagactcaagctcagggagggctgtgaga7260gcctgggagacagccacctactaaggagtttcctgctttacacaggaatgctggccaacc7320tagtggaacagaacatctcagtgcgccggcgccaaggggtcagcattgggcggcttcaca7380agcagcggaagcctgaccggcggaaacgctctcgcccctacaaggccaaacgccagtgag7440aacctctggccctgactctgcagcctctcttgccgcaaggccctcatcagggcccttcac7500tgtccctcttttccaagtattactgagtagttcaacgagagcccaggtcctgggagtggg7560agtgacatgtattctgcactgtgctctggggtctggcagggacaaggcagctcctcaatg7620ctcaggaagcagcagctggaactggagggcatccaccatggcctccacagccatctgagg7680agcagcaggacctggcctttctgcctgggcagcaggatgtacattctacctattggagat7740gtttaggttttgggcttttgtccatcttgccactgtattaacatggcagcttcctgactc7800tgctgtctctcccagcagctgtcattctgctgggccaggtctcttagaatacatcacagt7860cccagctgagaaacacaccaccactagcctgtcctcgttgggaagaagcttggcctacac7920ttcattcctctgggagagcaccaaactcagggacctggctgctgagctggaatgggcatg7980gggtatcctaataggatgtacagcctatcagggtcccacggctatagtggagacaagtgt8040ttgaactgtcccagtggcatgcagactgtcctgttagtgagtcagagcaaggctctgggg8100ctctttgccttcagtgttgtggccctggccatgggtctgccttaggctcctaatctctgt8160ctctggggcccaggggagcctcgacctagcccctcagtattaccatgtcttctcttagga8220atatcagaggcagggctgcttgaatggagctggtactattcaattctcccgactgtgcca8280gcttccttgtcctcttgcaaggtactcagggtagggacagggttgagaggagttagaact8340tctgtatccccatagggcttggtagtaagtacagcactgcttcaaaatccagcccccaaa8400agccctgtaccctctgctagatacaggcgatccaagcaggccccttatctgtacatagtg8460actgtaggggacccctctagtttctgtaaatattgaatcagtgagtgaataaaaatgtgc8520taataaatga8530<210>2<211>19<212>dna<213>人工序列(artificialsequence)<400>2tggcagccgctggacaata19<210>3<211>19<212>dna<213>人工序列(artificialsequence)<400>3tattgtccagcggctgcca19<210>4<211>19<212>dna<213>人工序列(artificialsequence)<400>4ccttcattcggctcagcgt19<210>5<211>19<212>dna<213>人工序列(artificialsequence)<400>5acgctgagccgaatgaagg19<210>6<211>45<212>dna<213>人工序列(artificialsequence)<400>6gggttcccccaaaccctgcccctattgtccagcggctgccagcct45<210>7<211>37<212>dna<213>人工序列(artificialsequence)<400>7gggttcccccaaaccctgccccagcggctgccagcct37<210>8<211>46<212>dna<213>人工序列(artificialsequence)<400>8gggttcccccaaaccctgcccctatttgtccagcggctgccagcct46<210>9<211>48<212>dna<213>人工序列(artificialsequence)<400>9tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>10<211>36<212>dna<213>人工序列(artificialsequence)<400>10tgaactgcagcaatgtggatctgggccgaatgaagg36<210>11<211>37<212>dna<213>人工序列(artificialsequence)<400>11tgaactgcagcaatgtggatctggggccgaatgaagg37<210>12<211>46<212>dna<213>人工序列(artificialsequence)<400>12gggttcccccaaaccctgcccctattgtccagcggctgccagcctt46<210>13<211>26<212>dna<213>人工序列(artificialsequence)<400>13gggttcccccaaaccctgccagcctt26<210>14<211>47<212>dna<213>人工序列(artificialsequence)<400>14gggttcccccaaaccctgcccctatttgtccagcggctgccagcctt47<210>15<211>46<212>dna<213>人工序列(artificialsequence)<400>15gggttcccccaaaccctgcccctattgtccagcggctgccagcctt46<210>16<211>26<212>dna<213>人工序列(artificialsequence)<400>16gggttcccccaaaccctgccagcctt26<210>17<211>48<212>dna<213>人工序列(artificialsequence)<400>17tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>18<211>49<212>dna<213>人工序列(artificialsequence)<400>18tgaactgcagcaatgtggatctggggcccacgactgagccgaatgaagg49<210>19<211>37<212>dna<213>人工序列(artificialsequence)<400>19tgaactgcagcaatgtggatctggggccgaatgaagg37<210>20<211>48<212>dna<213>人工序列(artificialsequence)<400>20tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>21<211>30<212>dna<213>人工序列(artificialsequence)<400>21tgaactgcagcaatgtgagccgaatgaagg30<210>22<211>94<212>dna<213>人工序列(artificialsequence)<400>22tttgatttatagctgattcccaactcttgtgccactcacgccttgctaagcgtgcttctg60aactgcagcaatgtggatctggggcccacgctga94<210>23<211>90<212>dna<213>人工序列(artificialsequence)<400>23tttgatttatagctgattcccaactcttgtgccactcacgccttgctaagcgtgcttctg60aactgcagcaatgtggatctggggcccaca90<210>24<211>48<212>dna<213>人工序列(artificialsequence)<400>24tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>25<211>37<212>dna<213>人工序列(artificialsequence)<400>25tgaactgcagcaatgtggatctggggccgaatgaagg37<210>26<211>48<212>dna<213>人工序列(artificialsequence)<400>26tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>27<211>43<212>dna<213>人工序列(artificialsequence)<400>27tgaactgcagcaatgtggatctggggcccacgccgaatgaagg43<210>28<211>37<212>dna<213>人工序列(artificialsequence)<400>28tgaactgcagcaatgtggatctggggccgaatgaagg37<210>29<211>113<212>dna<213>人工序列(artificialsequence)<400>29tgattgtttttgatttatagctgattcccaactcttgtgccactcacgccttgctaagcg60tgcttctgaactgcagcaatgtggatctggggcccacgctgagccgaatgaag113<210>30<211>90<212>dna<213>人工序列(artificialsequence)<400>30tgattgtttttgatttatagctgattcccaactcttgtgccactcacgccttgctaagcg60tgcttctgaactgcagcaatgtggatctgg90<210>31<211>10<212>dna<213>人工序列(artificialsequence)<400>31tcgaatgaag10<210>32<211>48<212>dna<213>人工序列(artificialsequence)<400>32tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>33<211>37<212>dna<213>人工序列(artificialsequence)<400>33tgaactgcagcaatgtggatctggggcccaatgaagg37<210>34<211>48<212>dna<213>人工序列(artificialsequence)<400>34tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>35<211>40<212>dna<213>人工序列(artificialsequence)<400>35tgaactgcagcaatgtggatctggggcccacgaatgaagg40<210>36<211>37<212>dna<213>人工序列(artificialsequence)<400>36tgaactgcagcaatgtggatctggggccgaatgaagg37<210>37<211>48<212>dna<213>人工序列(artificialsequence)<400>37tgaactgcagcaatgtggatctggggcccacgctgagccgaatgaagg48<210>38<211>40<212>dna<213>人工序列(artificialsequence)<400>38tgaactgcagcaatgtggatctggggccccagaatgaagg40<210>39<211>21<212>dna<213>人工序列(artificialsequence)<400>39acctcccacccctattatttt21<210>40<211>21<212>dna<213>人工序列(artificialsequence)<400>40gcagctagggaggaagataac21<210>41<211>20<212>dna<213>人工序列(artificialsequence)<400>41gactgggtaagacaaaggaa20<210>42<211>20<212>dna<213>人工序列(artificialsequence)<400>42gagcaacacaacagaaaagg20当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1