一种差异性甲基化区域筛选方法及其装置与流程
1.本发明涉及生物医学领域,具体涉及一种差异性甲基化区域筛选方法及其装置。
背景技术:
2.循环肿瘤dna(circulating tumor dna;ctdna)是一种携带了肿瘤特异性遗传和表观遗传变异的生物标志物,由于无创以及低成本的优势,ctdna标志物正在广泛应用于肿瘤诊断和预后预测,其中包括甲基化、拷贝数变异、体细胞突变、单核苷酸变异等。对比于其它ctdna生物标志物,dna甲基化作为肿瘤标志物的优势在于ctdna的甲基化模式与它们来源的细胞或组织一致,同时在肿瘤基因组中,dna甲基化具有高度的一致性,因此检测患者血浆中肿瘤特异性dna的甲基化是一种可行的血液检测方法。
3.对于早期肿瘤来说,由于ctdna含量很低,信号微弱,因此,在肿瘤早筛领域中,提高筛查结果的灵敏性是评价模型性能的重要指标,传统采用靶向wgbs策略与甲基化芯片平台的早筛技术所使用的甲基化标记物(即差异性甲基化区域)数量较少,只能捕获部分基因组区间的信息,并且其只关注了单个cpg位点的差异化特征,未考虑到cpg之间所具有的连锁效应,因此传统的甲基化标志物筛选方法影响了早筛模型应用时的灵敏度(亦称敏感性)。
技术实现要素:
4.根据第一方面,在一实施例中,提供一种差异性甲基化区域筛选方法,包括:
5.cpg簇的提取步骤,包括从参考基因组中提取cpg簇;
6.cpg簇的筛选步骤,包括根据提取的cpg簇,对比对到参考基因组的肿瘤组织样本测序数据、相应的对照样本测序数据、健康样本的cfdna测序数据、患病样本的cfdna测序数据进行过滤;
7.肿瘤组织中特异的差异性甲基化区域筛选步骤,包括以过滤后的cpg簇为单位,在所述肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析,获得高差异性甲基化区域、低差异性甲基化区域;
8.肿瘤患者cfdna中特异的差异性甲基化区域筛选步骤,包括以所述健康样本的cfdna测序数据和所述患病样本的cfdna测序数据作为背景数据集,对高差异性甲基化区域、低差异性甲基化区域进行过滤,获得过滤后的高差异性甲基化区域、低差异性甲基化区域。
9.根据第二方面,在一实施例中,提供一种差异性甲基化区域筛选装置,包括:
10.cpg簇的提取模块,用于从参考基因组中提取cpg簇;
11.cpg簇的筛选模块,用于根据提取的cpg簇,对比对到参考基因组的肿瘤组织样本测序数据、相应的对照样本测序数据、健康样本的cfdna测序数据、患病样本的cfdna测序数据进行过滤;
12.肿瘤组织中特异的差异性甲基化区域筛选模块,用于以过滤后的cpg簇为单位,在
肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析,获得高差异性甲基化区域、低差异性甲基化区域;
13.肿瘤患者cfdna中特异的差异性甲基化区域筛选模块,用于以健康样本的cfdna测序数据和患病样本的cfdna测序数据作为背景数据集,对高差异性甲基化区域、低差异性甲基化区域进行过滤,获得过滤后的高差异性甲基化区域、低差异性甲基化区域。
14.根据第三方面,在一实施例中,提供一种预测癌症的装置,包括第二方面所述装置,以及预测模块,所述预测模块用于根据待测样本cfdna测序数据,以及所述过滤后的高差异性甲基化区域、低差异性甲基化区域,预测待测样本所属生物体为患病个体或健康个体。
15.根据第四方面,在一实施例中,提供一种装置,包括:
16.存储器,用于存储程序;
17.处理器,用于通过执行所述存储器存储的程序以实现如第一方面所述的方法。
18.根据第五方面,在一实施例中,提供一种计算机可读存储介质,所述介质上存储有程序,所述程序能够被处理器执行以实现如第一方面所述的方法。
19.依据上述实施例的差异性甲基化区域筛选方法及其装置,本发明基于cpg位点距离与甲基化信号连锁性的高度相关性,动态地将基因组划分为具有连锁关系的cpg簇,结合数据库中的肿瘤群体数据和健康个体数据,筛选获得肿瘤cfdna中特异的差异性甲基化区域,有效提高甲基化标记物(即差异性甲基化区域)筛选的灵敏性与特异性。
附图说明
20.图1为一种实施例中全基因组范围内cpg簇窗口的划分流程图。
21.图2为一种实施例中全基因组范围内cpg簇窗口的筛选及质控流程图。
22.图3为一种实施例的dmr类型统计结果图。
具体实施方式
23.下面通过具体实施方式结合附图对本发明作进一步详细说明。其中不同实施方式中类似元件采用了相关联的类似的元件标号。在以下的实施方式中,很多细节描述是为了使得本技术能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他元件、材料、方法所替代。在某些情况下,本技术相关的一些操作并没有在说明书中显示或者描述,这是为了避免本技术的核心部分被过多的描述所淹没,而对于本领域技术人员而言,详细描述这些相关操作并不是必要的,他们根据说明书中的描述以及本领域的一般技术知识即可完整了解相关操作。
24.另外,说明书中所描述的特点、操作或者特征可以以任意适当的方式结合形成各种实施方式。同时,方法描述中的各步骤或者动作也可以按照本领域技术人员所能显而易见的方式进行顺序调换或调整。因此,说明书和附图中的各种顺序只是为了清楚描述某一个实施例,并不意味着是必须的顺序,除非另有说明其中某个顺序是必须遵循的。
25.本文中为部件所编序号本身,例如“第一”、“第二”等,仅用于区分所描述的对象,不具有任何顺序或技术含义。而本技术所说“连接”、“联接”,如无特别说明,均包括直接和间接连接(联接)。
26.根据第一方面,在一实施例中,提供一种甲基化标记物筛选方法,包括:
27.cpg簇的提取步骤,包括从参考基因组中提取cpg簇;
28.cpg簇的筛选步骤,包括根据提取的cpg簇,对比对到参考基因组的肿瘤组织样本测序数据、相应的对照样本测序数据、健康样本的cfdna测序数据、患病样本的cfdna测序数据进行过滤;
29.肿瘤组织中特异的差异性甲基化区域筛选步骤,包括以过滤后的cpg簇为单位,在所述肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析,获得高差异性甲基化区域、低差异性甲基化区域;
30.肿瘤患者cfdna中特异的差异性甲基化区域筛选步骤,包括以所述健康样本的cfdna测序数据和所述患病样本的cfdna测序数据作为背景数据集,对高差异性甲基化区域、低差异性甲基化区域进行过滤,获得过滤后的高差异性甲基化区域、低差异性甲基化区域。健康样本相当于基线,将患病样本与健康样本对比,找出高差异、低差异的区域。
31.需要说明的是,该方法筛选得到的是过滤后的高差异性甲基化区域、低差异性甲基化区域,属于中间结果,不是最终的疾病诊断结果,因此,该方法不属于疾病的诊断方法,更不属于疾病的治疗方法。
32.在一实施例中,cpg簇的提取步骤中,包括对每个cpg位点上下游延伸预设长度的区域,合并,获得合并的cpg簇。
33.在一实施例中,预设长度为100~125bp,优选为100bp,该长度范围内的cpg位点的甲基化信号相关性较强,因此可固定设置为100bp。
34.在一实施例中,cpg簇的提取步骤中,获得合并的cpg簇后,筛选并保留至少包含m个cpg位点的窗口,获得cpg簇。
35.在一实施例中,m=3。
36.在一实施例中,cpg簇的提取步骤中,保留至少包含m个cpg位点的窗口,获得cpg簇后,还包括筛选并保留与测序芯片位点存在重叠的区域。
37.在一实施例中,筛选并保留与测序芯片位点存在重叠的区域时,保留至少包含1个测序芯片信号的cpg簇。
38.在一实施例中,所述测序芯片包括但不限于hm450k芯片、hm850k芯片中的至少一种。
39.在一实施例中,cpg簇的提取步骤中,还包括按预设的阈值对cpg簇进行第一次划分,获得>阈值的cpg簇以及≤阈值的cpg簇;对于>阈值的cpg簇,按照预设宽度和预设步长进行第二次划分,获得第二次划分后的cpg簇;将≤阈值的cpg簇与第二次划分后的cpg簇汇总,获得汇总后的cpg簇。
40.在一实施例中,所述阈值可以为1kb。
41.在一实施例中,所述预设宽度可以为1kb。
42.在一实施例中,所述预设步长可以为500bp。
43.在一实施例中,cpg簇的筛选步骤中,按如下条件中的至少一种进行过滤:
44.1)过滤在≥第一预设比例的患病样本中无法检测到的探针;
45.2)过滤其所包含的探针信号中≥第二预设比例无法检测的cpg簇;
46.3)过滤其所包含的探针信号中≥第三预设比例无法检测的患病样本。
47.在一实施例中,cpg簇的筛选步骤中,还包括对过滤后的cpg簇进行质控。
48.在一实施例中,对过滤后的cpg簇进行质控时,具体是根据黑名单对所述过滤后的cpg簇进行再次过滤,获得再次过滤后的cpg簇。
49.在一实施例中,所述黑名单包括存在多比对现象以及对应snp热点区域的cpg位点。
50.在一实施例中,依次按照条件1)、2)、3)进行过滤。
51.在一实施例中,第一预设比例可以为5%。
52.在一实施例中,第二预设比例可以为50%。
53.在一实施例中,第三预设比例可以为20%。
54.在一实施例中,肿瘤组织中特异的差异性甲基化区域筛选步骤中,高差异性甲基化区域是指肿瘤组织样本中的甲基化水平高于对照组织样本甲基化水平的差异性甲基化区域,低差异性甲基化区域是指肿瘤组织样本中的甲基化水平低于对照组织样本甲基化水平的差异性甲基化区域。例如,如果肿瘤组织样本中该dmr的平均甲基化率为0.8,对照样本中为0.5,则为肿瘤hyper-dmr(高差异性甲基化区域);反之,如果肿瘤组织样本中该dmr的平均甲基化率为0.5,对照样本中为0.8,则为肿瘤hypo-dmr(低差异性甲基化区域)。
55.在一实施例中,肿瘤患者cfdna中特异的差异性甲基化区域筛选步骤中,以中位值作为每一个cpg簇的特征值,对高差异性甲基化区域、低差异性甲基化区域进行过滤。中位值能更好反映数据的特征。
56.在一实施例中,以过滤后的cpg簇为单位,在肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析时,使用的统计检验方法包括但不限于moderated-t test、fisher test、wilcox rank test中的至少一种。
57.在一实施例中,在肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析时,设置两种阈值作为筛选标准,一种阈值为p-value,另一种阈值为δmethylation level(甲基化水平差值)。
58.在一实施例中,肿瘤患者cfdna中特异的差异性甲基化区域筛选步骤中,获得过滤后的高差异性甲基化区域、低差异性甲基化区域后,还包括根据差异性甲基化区域窗口内相邻cpg的聚集密度进行筛选,获得筛选后的高差异性甲基化区域、低差异性甲基化区域。
59.在一实施例中,肿瘤患者cfdna中特异的差异性甲基化区域筛选步骤中,根据差异性甲基化区域窗口内相邻cpg的聚集密度进行筛选时,使得每个差异性甲基化区域窗口内至少存在1条可捕获到≥3个cpg位点的序列。
60.在一实施例中,cpg簇的提取步骤中,在全基因组范围内,从参考基因组中提取cpg簇。
61.在一实施例中,所述患病样本为体液样本。
62.在一实施例中,所述体液样本包括但不限于血液、血浆、尿液、唾液等等中的至少一种。
63.在一实施例中,所述患病样本cfdna测序数据为甲基化测序数据。
64.在一实施例中,所述相应的对照样本是指与肿瘤组织来源于同一生物体的样本。
65.在一实施例中,所述生物体包括人,具体可以是灵长目人科人属智人种。
66.在一实施例中,所述相应的对照样本可以包括癌旁组织样本。
67.在一实施例中,所述肿瘤包括实体瘤。实体瘤即有形瘤,可通过临床检查如x线摄片、ct扫描、b超,或触诊扪及到的有形肿块称实体瘤。x线、ct扫描、b超及触诊无法看到或扪及到的肿瘤,如血液病中的白血病,则属于非实体瘤。
68.在一实施例中,所述肿瘤包括但不限于肝癌、乳腺癌、卵巢癌、子宫癌、宫颈癌、脑瘤、甲状腺癌、食管癌、肺癌、胃癌、胰腺癌、肾癌、结直肠癌、膀胱癌、淋巴癌、黑色素瘤、前列腺癌、睾丸癌、阴茎癌等等。此处仅仅为示例性列举,适用于本发明的癌症种类不受限制。
69.根据第二方面,在一实施例中,提供一种差异性甲基化区域筛选装置,包括:
70.cpg簇的提取模块,用于从参考基因组中提取cpg簇;
71.cpg簇的筛选模块,用于根据提取的cpg簇,对比对到参考基因组的肿瘤组织样本测序数据、相应的对照样本测序数据、健康样本的cfdna测序数据、患病样本的cfdna测序数据进行过滤;
72.肿瘤组织中特异的差异性甲基化区域筛选模块,用于以过滤后的cpg簇为单位,在肿瘤组织样本测序数据与相应的对照样本测序数据中进行差异性甲基化分析,获得高差异性甲基化区域、低差异性甲基化区域;
73.肿瘤患者cfdna中特异的差异性甲基化区域筛选模块,用于以健康样本的cfdna测序数据和患病样本的cfdna测序数据作为背景数据集,对高差异性甲基化区域、低差异性甲基化区域进行过滤,获得过滤后的高差异性甲基化区域、低差异性甲基化区域。
74.根据第三方面,在一实施例中,提供一种预测癌症的装置,包括第二方面所述装置,以及预测模块,所述预测模块用于根据待测样本cfdna测序数据,以及所述过滤后的高差异性甲基化区域、低差异性甲基化区域,预测待测样本所属生物体为患病个体或健康个体。
75.所述预测模块的预测方法如下:如果待测样本中的异常差异性甲基化区域>所述阈值,则预测所述待测样本所属生物体为患病个体;如果待测样本中的异常差异性甲基化区域≤所述阈值,则预测所述待测样本所属生物体为健康个体。
76.在一实施例中,所述cfdna测序数据包括甲基化测序数据。
77.在一实施例中,所述cfdna测序数据为全基因组测序数据或区域捕获测序数据。
78.在一实施例中,所述待测样本为体液样本。
79.在一实施例中,所述体液样本包括血液、血浆、尿液、唾液中的至少一种。
80.根据第四方面,在一实施例中,提供一种装置,包括:
81.存储器,用于存储程序;
82.处理器,用于通过执行所述存储器存储的程序以实现如第一方面所述的方法。
83.在一实施例中,所述处理器还用于通过执行所述存储器存储的程序以实现预测步骤,所述预测步骤包括根据待测样本cfdna测序数据,以及所述过滤后的高差异性甲基化区域、低差异性甲基化区域,预测待测样本所属生物体为患病个体或健康个体。
84.根据第五方面,在一实施例中,提供一种计算机可读存储介质,所述介质上存储有程序,所述程序能够被处理器执行以实现如第一方面所述的方法。
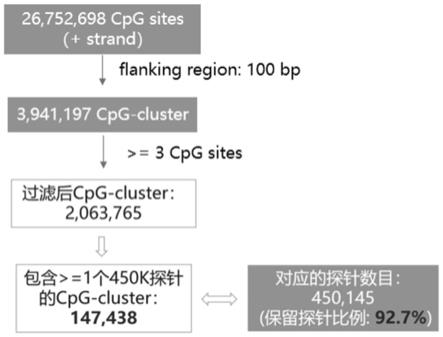
85.在一实施例中,所述程序还能够被处理器执行以实现预测步骤,所述预测步骤包括根据待测样本cfdna测序数据,以及所述过滤后的高差异性甲基化区域、低差异性甲基化区域,预测待测样本所属生物体为患病个体或健康个体。
86.在一实施例中,本发明的方法可应用于液体活检行业,包括基于全基因组甲基化信号的肿瘤早筛领域与生物信息领域。
87.在一实施例中,本发明以人类参考基因组(例如hg19基因组)中所包含的所有cpg位点为背景,基于cpg位点距离与甲基化信号连锁性的高度相关性,动态地将基因组划分为具有连锁关系的cpg簇。以cpg簇为单位,结合tcga与geo数据库中的肿瘤群体数据与健康个体数据,筛选获得一组肿瘤cfdna(circulating free dna;cfdna)中特异的差异性甲基化区域(differentially methylated region,dmr)。
88.在一实施例中,基因组聚类算法中所使用到的参数(包括cpg位点的数目以及flanking region的长度)都是可变的,从而实现动态地将基因组划分为具有连锁关系的cpg簇。
89.实施例1:全基因组范围cpg簇的产生
90.操作流程如图1、图2所示,图1为全基因组范围内cpg簇窗口的划分流程图,图2为全基因组范围内cpg簇窗口的筛选及质控流程图。
91.步骤1:全基因组范围内cpg簇窗口的划分
92.如图1所示,针对全基因组中26,752,698个cpg位点(此处的26,752,698个cpg位点是hg19基因组的固定位置),对每个位点上下扩增100bp区域,通过相互合并后获得3,941,197个cpg簇。为了保证有目标区域有充足的可用cpg信号以及在后期应用中降低测序错误率所带来的信号干扰,只保留至少包含3个cpg位点的窗口,获得2,063,765个cpg簇。
93.考虑到公共数据库中的数据为450k芯片信号,因此,基于illumina的hm450k芯片的设计信息,在这一步中保留了只与hm450k芯片位点存在重叠的区域,保留至少包含1个450k芯片信号的cpg簇,在这一步之后共获得147,438个cpg簇。
94.步骤2:全基因组范围内cpg簇窗口的筛选
95.如图2所示,针对147,438个cpg簇进行窗口特征分析,存在部分》1kb的超宽区域,考虑到后期应用,对这部分窗口进行了二次划分。窗口大小设置为1kb,步长设置为500bp。通过将二次划分后的cpg簇与原有69,264个≤1kb的cpg簇进行汇总,获得312,973个原始cpg簇,这些窗口对应了759,300个cpg位点。
96.步骤3:全基因组范围内中cpg簇窗口的质控
97.由于illumina的hm450k芯片在设计中存在一些缺陷,例如部分cpg位点可能存在多比对现象以及对应snp热点区域,因此,在下一步中,建立了这两种特征相关的黑名单探针集合,用于过滤上一步获得的cpg簇中存在问题的探针。过滤后,共获得277,418个cpg簇,这些区域将作为后续dmr流程筛选的起点。
98.x染色体、y染色体以及1至22号染色体上均有黑名单区间,本实施例共有38941个黑名单区间。部分黑名单区间如表1所示。
99.表1
100.染色体起点终点染色体起点终点11426189491426190001311489167211489172321144267871144268381422674498226745493448007494480080015829253338292538441546098061546098571625692812569332
5180662641180662692172141623021416281672294826722948771815000434150004857745087557450880619547468945474694588888525488885305204701381447013865913999787313999792421463967234639677410429708424297089322429087454290879611123899065123899116x125299352125299403124911368749113738y85530098553060
101.实施例2:肿瘤cfdna特异dmr的筛选
102.步骤1:cpg簇数据的质控
103.在实际数据应用中,由于所用的数据来自不同的公共数据集合,可能存在质量参差不一的问题。因此,针对这些数据,本实施例设计了一套质控方法,针对所有在后续dmr筛选环节中使用到的肿瘤群体数据以及对照数据进行过滤。过滤原则与优先级顺序如下所示:
104.(1)过滤在≥5%的患病样本中所无法检测到的探针。
105.(2)过滤其所包含的探针信号中≥50%无法检测的cpg簇。例如,对于某个包含10个探针信号的cpg簇,如果该簇中≥5个以上的探针信号无法检测到,则该cpg簇没有通过筛选,弃之。
106.(3)过滤其所包含的探针信号中≥20%无法检测的患病样本。
107.步骤2:肿瘤组织特异dmr的筛选
108.考虑到产品应用的场景,由于需要获得肿瘤cfdna中具有足够特异性与灵敏度的dmr,并基于该特征区间来捕获所需要的连锁甲基化信号。在第一轮筛选中,以过滤后的cpg簇为单位,首先在肿瘤组织与对应的癌旁组织(即对照组织样本)中进行差异性甲基化分析,设置3种统计检验方法,分别为moderated-t test、fisher test、wilcox rank test,并设置两种阈值作为筛选标准。阈值1为p-value,阈值2为δmethylation level。对于高甲基化dmr(hy per dmr定义:肿瘤组织样本中的甲基化水平高于对照组织样本),设置δmethylation level为0.2的阈值,低甲基化dmr(hypo dmr定义:肿瘤组织样本中的甲基化水平低于对照组织样本)设置为0.1的阈值,在p-value为0.01的条件下,本实施例共筛选获得肿瘤组织特异的26,315组dmr区域,包含8,687组hyper dmr与17,628组hypo dmr。
109.步骤3:肿瘤cfdna特异dmr的筛选
110.在第二轮筛选中,为了使得这些信号在cfdna样本中具有较高的特异性,以健康样本的cfdna测序数据以及肝硬化等肝疾病患者样本的数据作为背景数据集(健康样本cfdna测序数据来自全基因组甲基化测序数据,肝硬化患者cfdna样本的测序数据来自geo数据库,为芯片数据),以中位值作为每一个cpg簇的特征值进性dmr的过滤,筛选获得16,269组dmr区域,包含5,992组hyper dmr与10,277组hypo dmr。在此基础上,为了使得dm r区域具有高密度的cpg信号,本实施例根据dmr窗口内相邻cpg的聚集密度进行了筛选,保证1个dmr窗口内至少存在1条可以捕获到3个cpg位点的序列,最终共获得肿瘤cfd na特异的15,144组dmr区域,包含5,964组hyper dmr与9,180组hypo dmr。
111.实施例3
112.本实施例的实验数据来自自测的52例肝细胞癌(hepatocellular carcinoma,hcc)与33例健康个体的全基因组甲基化测序样本(此处的85例样本测序数据均来自受试者的血浆样本)。依据实施例2中的高甲基化dmr(hyper dmr)与低甲基化dmr(hypo dmr),检测肝癌患者与健康人。利用公开软件cancerdetector进行目标dmr的肿瘤纯度(tumor fracti on;tf)计算(参考文献:li w,li q,kang s,et al.cancerdetector:ultrasensitive and non-invasive cancer detection at the resolution of individual reads using cell-free dna methylati on sequencing data.nucleic acids res 2018;46:e89.),以33例健康人样本中的tf上限值作为预测待测样本所属受试者是否为肝癌患者、健康人的阈值界限。如果dmr中所包含的甲基化信号与健康人数据库中的背景信号差异高于20%,则将该dmr视为异常dmr。如果待测样本中的异常dmr的比例>阈值,则预测为肝癌患者,如果待测样本中的异常dmr的比例≤阈值,则预测为健康人。
113.结果表现如图3与表2所示,可见,该方法具有高特异性、高敏感性的优势。
114.表2
115.dmr类型敏感性(n=52)特异性(n=33)all(n=15144)84.0%100.0%hyper(n=5964)74.0%100.0%hypo(n=9180)94.2%100.0%
116.敏感性是指在判断有病(阳性)的人群中,检测出阳性的概率。
117.特异性是指在判断无病(阴性)的人群中,检测出阴性的概率。
118.从图3可见,all dmr、hyper dmr、hypo dmr的auc(area under curve,roc曲线下与坐标轴围成的面积)分别高达98.1%、96.6%、98.1%,非常接近于100%。
119.本领域技术人员可以理解,上述实施方式中各种方法的全部或部分功能可以通过硬件的方式实现,也可以通过计算机程序的方式实现。当上述实施方式中全部或部分功能通过计算机程序的方式实现时,该程序可以存储于一计算机可读存储介质中,存储介质可以包括:只读存储器、随机存储器、磁盘、光盘、硬盘等,通过计算机执行该程序以实现上述功能。例如,将程序存储在设备的存储器中,当通过处理器执行存储器中程序,即可实现上述全部或部分功能。另外,当上述实施方式中全部或部分功能通过计算机程序的方式实现时,该程序也可以存储在服务器、另一计算机、磁盘、光盘、闪存盘或移动硬盘等存储介质中,通过下载或复制保存到本地设备的存储器中,或对本地设备的系统进行版本更新,当通过处理器执行存储器中的程序时,即可实现上述实施方式中全部或部分功能。
120.以上应用了具体个例对本发明进行阐述,只是用于帮助理解本发明,并不用以限制本发明。对于本发明所属技术领域的技术人员,依据本发明的思想,还可以做出若干简单推演、变形或替换。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1