直接RNA测序的多组学分析方法、系统、设备及存储介质与流程
直接rna测序的多组学分析方法、系统、设备及存储介质
技术领域
1.本技术涉及生物信息处理技术领域,具体而言,涉及一种直接rna测序的多组学分析方法、系统、设备及存储介质。
背景技术:
2.目前,第三代测序技术中,纳米孔测序技术能够直接对rna分子测序,又称为直接rna测序技术;由于直接rna测序技术测序得到的测序结果包含rna分子多个维度信息,但是目前没有一种能够挖掘该直接rna测序结果的方法,因此实现对直接rna测序结果的多维度信息获取具有重要的现实意义。
3.现有技术中,对于第三代测序技术的rna分子数据分析过程中,主要存在以下几种技术:一是结合二代测序技术进行全长转录本定量分析,该分析一般采用第二代测序技术所得rna序列结合三代测序技术测得rna序列进行定量分析,缺点是在定量分析中还需要对样本进行一次第二代测序,对样品量要求多,价格昂贵;二是全长转录本鉴定分析,该分析采用定性方式只鉴定第三代测序结果中rna分子的全长结构序列,缺点是不能够有效利用rna直接测序的其他维度数据;三是rna分子的甲基化修饰鉴定,该分析采用建立模型对rna分子甲基化位点进行预测,缺点是预测准确性有待提高,且不能判定甲基化位点位于原始rna分子的那个位子。国内外对直接rna测序技术的分析方案较少,都只分析了rna分子的一到两个维度信息,且第三代测序技术价格昂贵,并没有充分利用直接rna测序结果。
技术实现要素:
4.本技术实施例的目的在于提供一种直接rna测序的多组学分析方法、系统、设备及存储介质,可以实现提高测序精度的技术效果。
5.第一方面,本技术实施例提供了一种直接rna测序的多组学分析方法,包括:
6.获取直接rna测序的测序数据;
7.将所述测序数据与参考基因组比对,获得测序比对数据;
8.根据所述测序比对数据进行全长转录本鉴定,获得全长转录本序列数据;
9.基于所述全长转录本序列数据对所述测序数据进行转录本定量处理,获得转录本定量数据;
10.根据甲基化修饰预测模型处理所述测序数据,获得甲基化修饰数据;
11.根据新生mrna预测模型处理所述全长转录本序列数据,获得新生mrna数据;
12.根据所述全长转录本序列数据、所述转录本定量数据、所述甲基化修饰数据和所述新生mrna数据进行关联性分析,获得直接rna测序多维度信息。
13.在上述实现过程中,该直接rna测序的多组学分析方法通过对直接rna测序的测序数据及测序比对数据进行处理,依次获得全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据,能够对直接rna测序的rna序列进行多维度数据分析,一个测序数据分析4个维度信息,从而实现多组学的数据整合;因此,该直接rna测序的多组学分析方法
通过一次实验即可同时获得多套数据方案,减少了多次生物学实验带来的实验误差和批次效应,实现提高测序精度的技术效果。
14.进一步地,所述根据所述测序比对数据进行全长转录本鉴定,获得全长转录本序列数据的步骤,包括:
15.对所述测序比对数据进行矫正,获得校正后的测序比对数据;
16.对所述校正后的测序比对数据进行聚类处理,获得所述全长转录本序列数据。
17.进一步地,所述基于所述全长转录本序列数据对所述测序数据进行转录本定量处理,获得转录本定量数据的步骤,包括:
18.将所述全长转录本序列数据作为定量转录本的参考序列,基于所述参考序列将所述测序数据进行转录本定量,获得所述转录本定量数据。
19.进一步地,所述根据甲基化修饰预测模型处理所述测序数据,获得甲基化修饰数据的步骤,包括:
20.根据所述测序数据,获取原始读长;
21.对所述原始读长的电信号进行重新计算,获取甲基化修饰引起的电信号变化信息;
22.根据原始读长和所述电信号变化信息进行甲基化修饰鉴定,获得甲基化修饰位点数据;
23.过滤所述甲基化修饰位点数据,获得所述甲基化修饰数据。
24.进一步地,所述根据新生mrna预测模型处理所述全长转录本序列数据,获得新生mrna数据的步骤,包括:
25.对加入5eu孵育的rna直接测序读长和未加入5eu孵育的rna直接测序读长序列进行处理,得到多个特征信息;
26.建立随机森林模型;
27.根据所述随机森林模型对所述多个特征信息进行训练,获得训练模型;
28.将所述测序数据输入所述训练模型,获得所述新生mrna数据。
29.进一步地,所述直接rna测序多维度信息包括第一关联信息、第二关联信息和第三关联信息,所述根据所述全长转录本序列数据、所述转录本定量数据、所述甲基化修饰数据和所述新生mrna数据进行关联性分析,获得直接rna测序多维度信息的步骤,包括:
30.将所述全长转录本序列数据与所述转录本定量数据进行关联分析,获得所述第一关联信息;
31.将所述转录本定量数据和所述甲基化修饰数据进行关联性分析,获得所述第二关联信息;
32.将所述甲基化修饰数据和所述新生mrna数据进行关联分析,获得所述第三关联信息。
33.第二方面,本技术实施例提供了一种直接rna测序的多组学分析系统,包括:
34.获取模块,用于获取直接rna测序的测序数据;
35.测序比对模块,用于将所述测序数据与参考基因组比对,获得测序比对数据;
36.全长转录本模块,用于根据所述测序比对数据进行全长转录本鉴定,获得全长转录本序列数据;
37.定量模块,用于基于所述全长转录本序列数据对所述测序数据进行转录本定量处理,获得转录本定量数据;
38.甲基化修饰模块,用于根据甲基化修饰预测模型处理所述测序数据,获得甲基化修饰数据;
39.新生mrna模块,用于根据新生mrna预测模型处理所述全长转录本序列数据,获得新生mrna数据;
40.关联分析模块,用于根据所述全长转录本序列数据、所述转录本定量数据、所述甲基化修饰数据和所述新生mrna数据进行关联性分析,获得直接rna测序多维度信息。
41.进一步地,所述测序比对模块包括:
42.矫正单元,用于对所述测序比对数据进行矫正,获得校正后的测序比对数据;
43.测序比对单元,用于对所述校正后的测序比对数据进行聚类处理,获得所述全长转录本序列数据。
44.进一步地,定量模块还用于将所述全长转录本序列数据作为定量转录本的参考序列,基于所述参考序列将所述测序数据进行转录本定量,获得所述转录本定量数据。
45.进一步地,甲基化修饰模块包括:
46.原始读长单元,用于根据所述测序数据,获取原始读长;
47.电信号变化单元,用于对所述原始读长的电信号进行重新计算,获取甲基化修饰引起的电信号变化信息;
48.修饰位点单元,用于根据原始读长和所述电信号变化信息进行甲基化修饰鉴定,获得甲基化修饰位点数据;
49.甲基化修饰单元,用于过滤所述甲基化修饰位点数据,获得所述甲基化修饰数据。
50.进一步地,新生mrna模块包括:
51.特征信息单元,用于对加入5eu孵育的rna直接测序读长和未加入5eu孵育的rna直接测序读长序列进行处理,得到多个特征信息;
52.模型建立单元,用于建立随机森林模型;
53.训练单元,用于根据所述随机森林模型对所述多个特征信息进行训练,获得训练模型;
54.新生mrna单元,用于将所述测序数据输入所述训练模型,获得所述新生mrna数据。
55.进一步地,关联分析模块包括:
56.第一关联单元,用于将所述全长转录本序列数据与所述转录本定量数据进行关联分析,获得所述第一关联信息;
57.第二关联单元,用于将所述转录本定量数据和所述甲基化修饰数据进行关联性分析,获得所述第二关联信息;
58.第三关联单元,用于将所述甲基化修饰数据和所述新生mrna数据进行关联分析,获得所述第三关联信息。
59.第三方面,本技术实施例提供的一种电子设备,包括:存储器、处理器以及存储在所述存储器中并可在所述处理器上运行的计算机程序,所述处理器执行所述计算机程序时实现如第一方面任一项所述的方法的步骤。
60.第四方面,本技术实施例提供的一种存储介质,所述存储介质上存储有指令,当所
述指令在计算机上运行时,使得所述计算机执行如第一方面任一项所述的方法。
61.第五方面,本技术实施例提供的一种计算机程序产品,所述计算机程序产品在计算机上运行时,使得计算机执行如第一方面任一项所述的方法。
62.本技术公开的其他特征和优点将在随后的说明书中阐述,或者,部分特征和优点可以从说明书推知或毫无疑义地确定,或者通过实施本技术公开的上述技术即可得知。
63.为使本技术的上述目的、特征和优点能更明显易懂,下文特举较佳实施例,并配合所附附图,作详细说明如下。
附图说明
64.为了更清楚地说明本技术实施例的技术方案,下面将对本技术实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本技术的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
65.图1为本技术实施例提供的直接rna测序的多组学分析方法的流程示意图;
66.图2为本技术实施例提供的获得全长转录本序列数据的流程示意图;
67.图3为本技术实施例提供的获得甲基化修饰数据的流程示意图;
68.图4为本技术实施例提供的获得新生mrna数据的流程示意图;
69.图5为本技术实施例提供的获得直接rna测序多维度信息的流程示意图;
70.图6为本技术实施例提供的基因表达isoform图;
71.图7为本技术实施例提供的rna表达箱线图;
72.图8为本技术实施例提供的差异rna热图;
73.图9为本技术实施例提供的差异rna火山图;
74.图10为本技术实施例提供的m6apeak在rna结构上的分布pie图;
75.图11为本技术实施例提供的m6apeak在rna结构上的分布metageneplot图;
76.图12为本技术实施例提供的m6a位点差异热图;
77.图13为本技术实施例提供的新生mrna表达箱线图;
78.图14为本技术实施例提供的新生mrna差异热图;
79.图15为本技术实施例提供的基因水平多功能分析图;
80.图16为本技术实施例提供的差异基因分布diu饼图;
81.图17为本技术实施例提供的diu分析火山图;
82.图18为本技术实施例提供的m6a与isodorm联合分析四象限图;
83.图19为本技术实施例提供的直接rna测序的多组学分析系统的结构示意图;
84.图20为本技术实施例提供的测序比对模块的结构示意图;
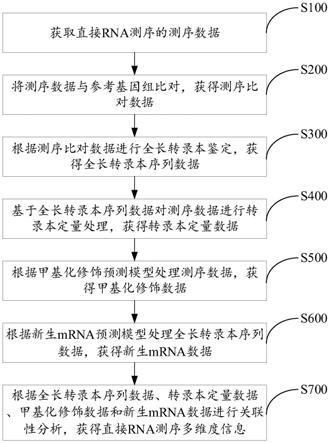
85.图21为本技术实施例提供的甲基化修饰模块的结构示意图;
86.图22为本技术实施例提供的新生mrna模块的结构示意图;
87.图23为本技术实施例提供的关联分析模块的结构示意图;
88.图24为本技术实施例提供的一种设备的结构框图。
具体实施方式
89.下面将结合本技术实施例中的附图,对本技术实施例中的技术方案进行描述。
90.应注意到:相似的标号和字母在下面的附图中表示类似项,因此,一旦某一项在一个附图中被定义,则在随后的附图中不需要对其进行进一步定义和解释。同时,在本技术的描述中,术语“第一”、“第二”等仅用于区分描述,而不能理解为指示或暗示相对重要性。
91.本技术实施例提供了一种直接rna测序的多组学分析方法、系统、设备及存储介质,可以应用于rna测序分析中;该直接rna测序的多组学分析方法通过对直接rna测序的测序数据及测序比对数据进行处理,依次获得全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据,能够对直接rna测序的rna序列进行多维度数据分析,一个测序数据分析4个维度信息,从而实现多组学的数据整合;因此,该直接rna测序的多组学分析方法通过一次实验即可同时获得多套数据方案,减少了多次生物学实验带来的实验误差和批次效应,实现提高测序精度的技术效果。
92.请参见图1,图1为本技术实施例提供的直接rna测序的多组学分析方法的流程示意图,该直接rna测序的多组学分析方法包括如下步骤:
93.s100:获取直接rna测序的测序数据。
94.示例性地,rna(ribonucleic acid,核糖核酸),存在于生物细胞以及部分病毒、类病毒中的遗传信息载体。rna由核糖核苷酸经磷酸二酯键缩合而成长链状分子。一个核糖核苷酸分子由磷酸,核糖和碱基构成。rna的碱基主要有4种,即a(腺嘌呤)、g(鸟嘌呤)、c(胞嘧啶)、u(尿嘧啶),其中,u(尿嘧啶)取代了dna(deoxyribonucleic acid,脱氧核糖核酸)中的t。核糖核酸在体内的作用主要是引导蛋白质的合成。
95.在一些实施方式中,本技术实施例通过将生物组织样本或细胞样本经过5eu孵育,提取rna,经过三代测序仪得到rna直接测序的结果,即直接rna测序的测序数据。
96.s200:将测序数据与参考基因组比对,获得测序比对数据。
97.示例性地,基因组(genome),是有关特定生物全部染色体的遗传物质的总和,dna碱基对总数来表示。对单倍体而言,基因组表示这种生物的总dna;对于二倍体的高等生物,其配子的dna总和即为一组基因组。二倍体有两份同源的基因组,真核生物细胞中有几个染色体组,故有几个基因组。细菌的基因组是唯一的染色体。
98.在一些实施方式中,使用比对工具minimap2(li heng)将直接rna测序的测序数据与参考基因组比对,输出比对bam文件结果,即测序比对数据。
99.s300:根据测序比对数据进行全长转录本鉴定,获得全长转录本序列数据。
100.示例性地,转录本是由一条基因通过转录形成的一种或多种可供编码蛋白质的成熟的mrna。
101.在一些实施方式中,全长转录本鉴定,根据比对bam文件结果,可以进行全长转录本鉴定和发现新全长转录本,例如,使用flair(van baren,m.j)软件将比对bam文件结果进行全长转录本鉴定,从而获得全长转录本序列数据。
102.s400:基于全长转录本序列数据对测序数据进行转录本定量处理,获得转录本定量数据。
103.在一些实施方式中,转录本定量时,可以将全长转录本序列数据作为定量转录本的参考序列,通过salmon(rob patro)定量软件,将每个样本进行直接rna测序,获得测序结
果,进而得到转录本精确定量,即转录本定量数据。
104.s500:根据甲基化修饰预测模型处理测序数据,获得甲基化修饰数据。
105.示例性地,甲基化是指从活性甲基化合物(如s-腺苷基甲硫氨酸)上将甲基催化转移到其他化合物的过程。可形成各种甲基化合物,或是对某些蛋白质或核酸等进行化学修饰形成甲基化产物。在生物系统内,甲基化是经酶催化的,这种甲基化涉及重金属修饰、基因表达的调控、蛋白质功能的调节以及核糖核酸(rna)加工。在真核生物中,5’端的cap以及3’端的ploy a修饰在转录调控中起到了十分重要的作用,而mrna的内部修饰则用于维持mrna的稳定性。mrna最常见的内部修饰包括n6-腺苷酸甲基化(m6a)、n1-腺苷酸甲基化(m1a)、胞嘧啶羟基化(m5c)等。
106.示例性地,当rna分子腺苷酸上第六位氮原子处发生甲基化时,就是我们所说的m6a;应理解,本技术实施例以m6a甲基化修饰鉴定作为一般化示例。
107.在一些实施方式中,m6a甲基化修饰鉴定,将直接rna测序的测序数据,通过软件tombo(stoiber,m.h)和mines(gene w.yeo)预测rna分子中的甲基化修饰信息。
108.s600:根据新生mrna预测模型处理全长转录本序列数据,获得新生mrna数据。
109.在一些实施方式中,新生mrna鉴定,将全长转录本序列数据,通过深度学习建立的预测模型鉴定新生mrna。
110.s700:根据全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据进行关联性分析,获得直接rna测序多维度信息。
111.在一些实施方式中,多组学融合分析,将步骤s300,步骤s400,步骤s500和步骤s600所获得的rna分子多维度信息(全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据)进行关联性分析,获得直接rna测序多维度信息。
112.在一些实施场景中,将生物组织样本或细胞样本经过5eu孵育,提取rna,经过三代测序仪得到rna直接测序的结果。本技术针对直接rna测序数据包含rna分子多维度信息,设计了基于三代测序技术——直接rna测序的多组学分析方法,该方法克服了现有技术只能分析直接rna测序结果的1种或2种维度信息、不能完全挖掘直接rna测序结果的多维度信息的缺陷,同时降低了样本进行多次实验带来的实验误差和批次效应。本技术通过一次测序分析出多套结果,并实现了数据之间有机结合,一次性实现了多组学分析内容。因此,该直接rna测序的多组学分析方法通过一次实验即可同时获得多套数据方案,减少了多次生物学实验带来的实验误差和批次效应,实现提高测序精度的技术效果。
113.请参见图2,图2为本技术实施例提供的获得全长转录本序列数据的流程示意图,包括如下步骤:
114.s301:对测序比对数据进行矫正,获得校正后的测序比对数据;
115.s302:对校正后的测序比对数据进行聚类处理,获得全长转录本序列数据。
116.在一些实施方式中,进行全长转录本鉴定,根据测序比对数据,可以进行全长转录本鉴定和发现新全长转录本,例如,使用flair(van baren,m.j)软件将比对bam文件进行全长转录本鉴定:
117.第一步:对测序比对数据中的测序错误碱基进行矫正,得到校正后的测序比对数据。
118.第二步:对校正后的测序比对数据进行聚类,得到转录本同工型序列,即鉴定出全
长转录本序列fasta文件(全长转录本序列数据)。
119.在一些实施方式中,s400:基于全长转录本序列数据对测序数据进行转录本定量处理,获得转录本定量数据的步骤中,包括:
120.将全长转录本序列数据作为定量转录本的参考序列,基于参考序列将测序数据进行转录本定量,获得转录本定量数据。
121.示例性地,转录本定量,将全长转录本序列数据作为定量转录本的参考序列,通过salmon(rob patro)定量软件,将每个样本取直接rna测序的测序数据,得到转录本精确定量:
122.第一步:将全长转录本序列数据作为定量转录本的参考序列;
123.第二步:将直接rna测序的测序数据通过salmon软件进行转录本定量,得到每条全长转录本的读长(reads)计数,进而得到转录本精确定量,即转录本定量数据。
124.示例性地,由于受目前测序水平的限制,基因组测序时需要先将基因组打断成dna片段,然后再建库测序。reads(读长)指的是测序仪单次测序所得到的碱基序列,也就是一连串的atcgggta之类的,它不是基因组中的组成。不同的测序仪器,reads长度不一样。对整个基因组进行测序,就会产生成百上千万的reads。
125.请参见图3,图3为本技术实施例提供的获得甲基化修饰数据的流程示意图,包括如下步骤:
126.s501:根据测序数据,获取原始读长;
127.s502:对原始读长的电信号进行重新计算,获取甲基化修饰引起的电信号变化信息;
128.s503:根据原始读长和电信号变化信息进行甲基化修饰鉴定,获得甲基化修饰位点数据;
129.s504:过滤甲基化修饰位点数据,获得甲基化修饰数据。
130.示例性地,以m6a甲基化修饰鉴定为例进行说明,将直接rna测序的测序数据,通过软件tombo(stoiber,m.h)和mines(gene w.yeo)预测rna分子中的甲基化修饰信息:
131.第一步:通过tombo(stoiber,m.h)软件,将直接rna测序data的原始reads写入原始数据对应的存储数据电流信号的fast5文件;
132.第二步:通过tombo(stoiber,m.h)软件,对reads的电信号进行重新计算,找出m6a修饰引起的电信号变化,并将该变化重新写入fast5文件;
133.第三步:通过tombo(stoiber,m.h)软件,对第一步和第二步处理的fast5文件,进行m6a修饰鉴定,并输出鉴定m6a修饰的wig文件(甲基化修饰位点数据);
134.第四步:通过mines(gene w.yeo)软件对上步所得wig文件进行过滤得到过滤后的m6a修饰位点文件,即rna分子中的甲基化修饰信息(甲基化修饰数据)。
135.请参见图4,图4为本技术实施例提供的获得新生mrna数据的流程示意图,包括如下步骤:
136.s601:对加入5eu孵育的rna直接测序读长和未加入5eu孵育的rna直接测序读长序列进行处理,得到多个特征信息;
137.s602:根据随机森林模型对多个特征信息进行训练,获得训练模型;
138.s603:将测序数据输入训练模型,获得新生mrna数据。
139.示例性地,新生mrna鉴定时,根据全长转录本序列数据和通过深度学习建立的预测模型,可以鉴定新生mrna,获得新生mrna数据:
140.第一步:首先进行前期模型训练数据处理,将加入5eu孵育的rna直接测序reads(实验组)和未加入5eu孵育的rna直接测序reads(对照组)进行处理,得到9个特征信息(每条reads的特征为:u碱基错配率,u碱基错配测序质量平均值,u测为a碱基错配率,u测为a碱基错配测序质量平均值,u测为c碱基错配率,u测为c碱基错配测序质量平均值,u测为g碱基错配率,u测为g碱基错配测序质量平均值,碱基删除率);
141.第二步:建立随机森林模型,将上述得到的9个特征信息进行训练,用于预测新生mrna,得到训练模型;
142.第三步:将直接rna测序的测序数据输入至训练模型,进行新生mrna预测,获得新生mrna的reads序列,即新生mrna数据。
143.请参见图5,图5为本技术实施例提供的获得直接rna测序多维度信息的流程示意图,包括如下步骤:
144.s701:将全长转录本序列数据与转录本定量数据进行关联分析,获得第一关联信息;
145.s702:将转录本定量数据和甲基化修饰数据进行关联性分析,获得第二关联信息;
146.s703:将甲基化修饰数据和新生mrna数据进行关联分析,获得第三关联信息。
147.示例性地,进行多组学融合分析时,s300-s600各个步骤所获得的rna分子多维度信息(全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据)进行关联性分析,获得直接rna测序多维度信息:
148.第一步:将全长转录本序列数据与转录本定量数据进行关联分析,通过将转录本定量,得到isoform矩阵,对isoform关联性,可以查看差异isoform在基因上的表达;
149.第二步:将转录本定量数据和甲基化修饰数据进行关联性分析,可通过isoform矩阵和甲基化修饰数据进行关联,可以找到m6a修饰影响的isoform;
150.第三步:将甲基化修饰数据和新生mrna数据进行关联分析,通过甲基化修饰数据和新生mrna数据进行关联,可以找到新生mrna变化的原因。
151.示例性地,isoform指同一个基因的不同版本的蛋白。
152.在一些实施方式中,本技术实现了直接rna测序数据4个维度多组学分析方法(如图1至图5所示),打破了直接rna测序数据只能分析1-2个维度数据方法。其中转录本定量建立了原始数据过滤标准,m6a甲基化修饰鉴定构建了深度学习预测模型,auc达到88%高于现有技术的预测准确率,新生mrna鉴定同时构建了深度学习模型,auc达到90%高于现有技术预测标准。此外,本技术实现了一次实验同时获得多套数据方案,减少了多次生物学实验带来的实验误差和批次效应,多组学融合分析模块提供了4个维度数据的交叉融合,带来生物学研究新方向。
153.在一些实施方式中,根据图1至图5所述的方法实施例进行实际应用,获得的结果示例如下:
154.(1)维度1全长转录本结果:
[0155][0156]
表1-fastq数据统计表
[0157]
如表1所示,sample name为样品名;readnum为序列条数;base(m)为总碱基数;n50为n50长度;meanlength为reads平均长度;maxlength为最长reads长度;meanqscore为平均质量值。
[0158]
请参见图6,图6为本技术实施例提供的基因表达isoform图,图6中黑色块代表转录本的外显子区域;未有enst号的转录本是新鉴定的转录本。
[0159]
(2)维度2全长转录本结果:
[0160][0161]
表2-比对率统计表
[0162]
如表2所示,sample为样品名;allread为总测序reads数;mapread为比对上参考序列的reads数;map为比对率。
[0163]
请参见图7,图7为本技术实施例提供的rna表达箱线图,横坐标是样本名称,纵坐标是基因计数值。
[0164]
请参见图8,图8为本技术实施例提供的差异rna热图,横坐标是样本名称,纵坐标是差异转录本的表达量,颜色越深表达量越高。
[0165]
请参见图9,图9为本技术实施例提供的差异rna火山图,横坐标是两种条件下转录本表达量的倍数差异,纵坐标是显著性fdr值,其中fdr《0.05为显著性差异结果,图中深色点为显著性差异结果。
[0166]
(3)rna甲基化修饰模块结果:
[0167]
请参见图10,图10为本技术实施例提供的m6apeak在rna结构上的分布pie图;如图10所示,该饼图分别是:3’utr为基因3端区域,5’utr为基因5端区域,cds为基因编码区,ncrna为非编码rna的甲基化修饰位置的统计图。
[0168]
请参见图11,图11为本技术实施例提供的m6apeak在rna结构上的分布metageneplot图,图11中的横坐标是基因的区域位置,纵坐标是甲基化修饰的比例。
[0169]
请参见图12,图12为本技术实施例提供的m6a位点差异热图,图12中的横坐标是样本名称,纵坐标是差异转录本的表达量,颜色越深表达量越高。
[0170]
(4)新生mrna结果:
[0171]
请参见图13,图13为本技术实施例提供的新生mrna表达箱线图,图13中的横坐标是样本名称,纵坐标是基因计数值。
[0172]
请参见图14,图14为本技术实施例提供的新生mrna差异热图,图14中的横坐标是样本名称,纵坐标是差异转录本的表达量,颜色越深表达量越高。
[0173]
(5)基于(1)至(4)的联合分析模块结果:
[0174]
请参见图15,图15为本技术实施例提供的基因水平多功能分析图,图15中的横坐标是基因占比,纵坐标是基因区域统计指标个数统计值。
[0175]
请参见图16,图16为本技术实施例提供的差异基因分布diu饼图,其中diu指显著性差异基因,major isoform switching代表转录本具有结构性变化。
[0176]
请参见图17,图17为本技术实施例提供的diu分析火山图,其中横坐标是两种条件下转录本表达量的倍数差异,纵坐标是显著性fdr值,其中fdr《0.05为显著性差异结果,图中深色点为显著性差异结果。
[0177]
请参见图18,图18为本技术实施例提供的m6a与isodorm联合分析四象限图,其中横坐标是两种条件下转录本表达量的倍数差异,纵坐标是两种条件下甲基化修饰表达量的倍数差异。
[0178]
请参见图19,图19为本技术实施例提供的直接rna测序的多组学分析系统的结构示意图,该直接rna测序的多组学分析系统包括:
[0179]
获取模块100,用于获取直接rna测序的测序数据;
[0180]
测序比对模块200,用于将测序数据与参考基因组比对,获得测序比对数据;
[0181]
全长转录本模块300,用于根据测序比对数据进行全长转录本鉴定,获得全长转录本序列数据;
[0182]
定量模块400,用于基于全长转录本序列数据对测序数据进行转录本定量处理,获得转录本定量数据;
[0183]
甲基化修饰模块500,用于根据甲基化修饰预测模型处理测序数据,获得甲基化修饰数据;
[0184]
新生mrna模块600,用于根据新生mrna预测模型处理全长转录本序列数据,获得新生mrna数据;
[0185]
关联分析模块700,用于根据全长转录本序列数据、转录本定量数据、甲基化修饰数据和新生mrna数据进行关联性分析,获得直接rna测序多维度信息。
[0186]
请参见图20,图20为本技术实施例提供的测序比对模块的结构示意图。
[0187]
示例性地,测序比对模块200包括:
[0188]
矫正单元301,用于对测序比对数据进行矫正,获得校正后的测序比对数据;
[0189]
测序比对单元302,用于对校正后的测序比对数据进行聚类处理,获得全长转录本序列数据。
[0190]
示例性地,定量模块400还用于将全长转录本序列数据作为定量转录本的参考序列,基于参考序列将测序数据进行转录本定量,获得转录本定量数据。
[0191]
请参见图21,图21为本技术实施例提供的甲基化修饰模块的结构示意图。
[0192]
示例性地,甲基化修饰500模块包括:
[0193]
原始读长单元501,用于根据测序数据,获取原始读长;
[0194]
电信号变化单元502,用于对原始读长的电信号进行重新计算,获取甲基化修饰引起的电信号变化信息;
[0195]
修饰位点单元503,用于根据原始读长和电信号变化信息进行甲基化修饰鉴定,获得甲基化修饰位点数据;
[0196]
甲基化修饰单元504,用于过滤甲基化修饰位点数据,获得甲基化修饰数据。
[0197]
请参见图22,图22为本技术实施例提供的新生mrna模块的结构示意图。
[0198]
示例性地,新生mrna模块600包括:
[0199]
特征信息单元601,用于对加入5eu孵育的rna直接测序读长和未加入5eu孵育的rna直接测序读长序列进行处理,得到多个特征信息;
[0200]
模型建立单元602,用于建立随机森林模型;
[0201]
训练单元603,用于根据随机森林模型对多个特征信息进行训练,获得训练模型;
[0202]
新生mrna单元604,用于将测序数据输入训练模型,获得新生mrna数据。
[0203]
请参见图23,图23为本技术实施例提供的关联分析模块的结构示意图。
[0204]
示例性地,关联分析模块700包括:
[0205]
第一关联单元701,用于将全长转录本序列数据与转录本定量数据进行关联分析,获得第一关联信息;
[0206]
第二关联单元702,用于将转录本定量数据和甲基化修饰数据进行关联性分析,获得第二关联信息;
[0207]
第三关联单元703,用于将甲基化修饰数据和新生mrna数据进行关联分析,获得第三关联信息。
[0208]
应理解,图19至图23所示的直接rna测序的多组学分析系统与图1至图5所示的方法实施例相互对应,为避免重复,此处不再赘述。
[0209]
本技术还提供一种电子设备,请参见图24,图24为本技术实施例提供的一种设备的结构框图。电子设备可以包括处理器510、通信接口520、存储器530和至少一个通信总线540。其中,通信总线540用于实现这些组件直接的连接通信。其中,本技术实施例中电子设备的通信接口520用于与其他节点设备进行信令或数据的通信。处理器510可以是一种集成电路芯片,具有信号的处理能力。
[0210]
上述的处理器510可以是通用处理器,包括中央处理器(cpu,central processing unit)、网络处理器(np,network processor)等;还可以是数字信号处理器(dsp)、专用集成电路(asic)、现成可编程门阵列(fpga)或者其他可编程逻辑器件、分立门或者晶体管逻辑器件、分立硬件组件。可以实现或者执行本技术实施例中的公开的各方法、步骤及逻辑框图。通用处理器可以是微处理器或者该处理器510也可以是任何常规的处理器等。
[0211]
存储器530可以是,但不限于,随机存取存储器(ram,random access memory),只读存储器(rom,read only memory),可编程只读存储器(prom,programmable read-only memory),可擦除只读存储器(eprom,erasable programmable read-only memory),电可擦除只读存储器(eeprom,electric erasable programmable read-only memory)等。存储器
530中存储有计算机可读取指令,当所述计算机可读取指令由所述处理器510执行时,电子设备可以执行上述图1至图5方法实施例涉及的各个步骤。
[0212]
可选地,电子设备还可以包括存储控制器、输入输出单元。
[0213]
所述存储器530、存储控制器、处理器510、外设接口、输入输出单元各元件相互之间直接或间接地电性连接,以实现数据的传输或交互。例如,这些元件相互之间可通过一条或多条通信总线540实现电性连接。所述处理器510用于执行存储器530中存储的可执行模块,例如电子设备包括的软件功能模块或计算机程序。
[0214]
输入输出单元用于提供给用户创建任务以及为该任务创建启动可选时段或预设执行时间以实现用户与服务器的交互。所述输入输出单元可以是,但不限于,鼠标和键盘等。
[0215]
可以理解,图24所示的结构仅为示意,所述电子设备还可包括比图24中所示更多或者更少的组件,或者具有与图24所示不同的配置。图24中所示的各组件可以采用硬件、软件或其组合实现。
[0216]
本技术实施例还提供一种存储介质,所述存储介质上存储有指令,当所述指令在计算机上运行时,所述计算机程序被处理器执行时实现方法实施例所述的方法,为避免重复,此处不再赘述。
[0217]
本技术还提供一种计算机程序产品,所述计算机程序产品在计算机上运行时,使得计算机执行方法实施例所述的方法。
[0218]
在本技术所提供的几个实施例中,应该理解到,所揭露的装置和方法,也可以通过其它的方式实现。以上所描述的装置实施例仅仅是示意性的,例如,附图中的流程图和框图显示了根据本技术的多个实施例的装置、方法和计算机程序产品的可能实现的体系架构、功能和操作。在这点上,流程图或框图中的每个方框可以代表一个模块、程序段或代码的一部分,所述模块、程序段或代码的一部分包含一个或多个用于实现规定的逻辑功能的可执行指令。也应当注意,在有些作为替换的实现方式中,方框中所标注的功能也可以以不同于附图中所标注的顺序发生。例如,两个连续的方框实际上可以基本并行地执行,它们有时也可以按相反的顺序执行,这依所涉及的功能而定。也要注意的是,框图和/或流程图中的每个方框、以及框图和/或流程图中的方框的组合,可以用执行规定的功能或动作的专用的基于硬件的系统来实现,或者可以用专用硬件与计算机指令的组合来实现。
[0219]
另外,在本技术各个实施例中的各功能模块可以集成在一起形成一个独立的部分,也可以是各个模块单独存在,也可以两个或两个以上模块集成形成一个独立的部分。
[0220]
所述功能如果以软件功能模块的形式实现并作为独立的产品销售或使用时,可以存储在一个计算机可读取存储介质中。基于这样的理解,本技术的技术方案本质上或者说对现有技术做出贡献的部分或者该技术方案的部分可以以软件产品的形式体现出来,该计算机软件产品存储在一个存储介质中,包括若干指令用以使得一台计算机设备(可以是个人计算机,服务器,或者网络设备等)执行本技术各个实施例所述方法的全部或部分步骤。而前述的存储介质包括:u盘、移动硬盘、只读存储器(rom,read-only memory)、随机存取存储器(ram,random access memory)、磁碟或者光盘等各种可以存储程序代码的介质。
[0221]
以上所述仅为本技术的实施例而已,并不用于限制本技术的保护范围,对于本领域的技术人员来说,本技术可以有各种更改和变化。凡在本技术的精神和原则之内,所作的
任何修改、等同替换、改进等,均应包含在本技术的保护范围之内。应注意到:相似的标号和字母在下面的附图中表示类似项,因此,一旦某一项在一个附图中被定义,则在随后的附图中不需要对其进行进一步定义和解释。
[0222]
以上所述,仅为本技术的具体实施方式,但本技术的保护范围并不局限于此,任何熟悉本技术领域的技术人员在本技术揭露的技术范围内,可轻易想到变化或替换,都应涵盖在本技术的保护范围之内。因此,本技术的保护范围应所述以权利要求的保护范围为准。
[0223]
需要说明的是,在本文中,诸如第一和第二等之类的关系术语仅仅用来将一个实体或者操作与另一个实体或操作区分开来,而不一定要求或者暗示这些实体或操作之间存在任何这种实际的关系或者顺序。而且,术语“包括”、“包含”或者其任何其他变体意在涵盖非排他性的包含,从而使得包括一系列要素的过程、方法、物品或者设备不仅包括那些要素,而且还包括没有明确列出的其他要素,或者是还包括为这种过程、方法、物品或者设备所固有的要素。在没有更多限制的情况下,由语句“包括一个
……”
限定的要素,并不排除在包括所述要素的过程、方法、物品或者设备中还存在另外的相同要素。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1