一种碳末端酰胺化多肽结构的动力学预测方法和装置
1.本发明涉及一种碳末端酰胺化多肽结构的动力学预测方法和装置,尤其涉及一种基于全原子分子动力学与增强采样的碳末端酰胺化多肽结构的动力学预测方法和装置,属于生物信息学、蛋白质结构和分子动力学领域。
背景技术:
2.多肽是一种由α-氨基酸脱水缩合而形成的化合物,通常含有10~50个氨基酸。多肽是生物体内重要的活性物质,例如酶、激素、神经间质、细胞生长和生殖因子等,具有调节细胞的生理功能、激活生物体内的相关酶系、传递神经系统信息、运输物质等功能。多肽的结构决定其功能,因此确定多肽的结构对理解其生物学活性具有重要意义,对多肽类产品的开发也具有实用价值。
3.多肽药物,因其适应症广、安全性高且疗效显著,目前已广泛应用于糖尿病、骨质疏松、癌症等疾病的预防、诊断和治疗。基因工程重组技术和化学合成技术是多肽药物制备过程中的两种常用技术。基因工程重组技术通过生物表达和提取过程,能获得与天然多肽完全一致的空间结构和生物学活性。而化学合成多肽,通常按多肽的氨基酸序列顺序从碳末端向氮末端进行合成,因其合成过程中多肽的碳末端会携带游离的羧基,因此将其碳末端酰胺化。当基因工程重组多肽和化学合成多肽具有相同的氨基酸序列时,人们往往不会关注末端结构的微小差异。随着循证医学的逐渐发展和重视,对于药物在治疗过程中的安全性和有效性,更加关联至分子本身的空间构象的影响。末端酰胺化和天然羧基的不同,很可能导致多肽分子理化性质、空间结构、免疫原性等不同。由于多肽的序列短、柔性较大,现有的结构解析技术难以在相同的生理仿真环境中测定末端酰胺化和天然多肽的结构,这不利于揭示末端酰胺化与天然多肽的结构异同。鉴于此,设计一种能快速且准确度高的构建多肽结构的方法具有十分重要的意义。然而,化学合成的多肽与天然多肽的氨基酸序列完全相同,基于生物信息学的结构预测技术难以进行特异性结构建模。采用常规的分子动力学进行从头算的折叠模拟,难以充分地进行构象采样,且计算量大。
4.本发明致力于解决上述技术问题,提出了一种从多肽的天然结构出发,使用全原子分子动力学与增强采样相结合的动力学计算来预测碳末端酰胺化多肽结构的方法和装置。
技术实现要素:
5.本发明的目的是针对目前还没有基于多肽的天然结构特异性地预测碳末端酰胺化多肽结构的技术现状,提出了一种碳末端酰胺化多肽结构的动力学预测方法和装置。
6.本发明所述方法基于多肽的天然结构,构建碳末端酰胺化多肽的初始构象,在溶剂环境中对碳末端酰胺化多肽进行原子水平的增强构象采样计算,在采样空间上重建其自由能,找寻其自由能极小,在构象空间中提取自由能极小的结构,从而获得碳末端酰胺化多肽结构。
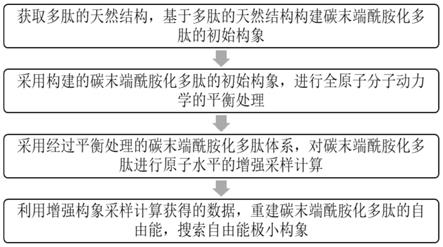
7.为此,本发明一方面提供了一种碳末端酰胺化多肽结构的动力学预测方法,包括以下步骤:
8.1、获取多肽的天然结构,基于多肽的天然结构构建碳末端酰胺化多肽的初始构象;
9.2、采用步骤1构建的碳末端酰胺化多肽的初始构象,进行全原子分子动力学的平衡处理;
10.3、采用步骤2经过平衡处理的碳末端酰胺化多肽体系,对碳末端酰胺化多肽进行原子水平的增强采样计算;
11.4、利用步骤3的增强采样计算获得的数据,重建碳末端酰胺化多肽的自由能,搜索自由能极小构象。
12.为达到上述目的,本发明采取如下技术方案。
13.所述一种碳末端酰胺化多肽结构的动力学预测方法,包括以下步骤:
14.步骤1:获取多肽的天然结构,构建碳末端酰胺化多肽的初始构象;
15.其中,多肽的天然结构从蛋白质数据银行(pdb)下载;
16.其中,碳末端酰胺化多肽的初始构象是基于多肽的天然结构构建,具体包含如下子步骤:
17.步骤1.1对多肽的天然结构进行预处理,具体为:
18.删除多肽的天然结构中的水分子、离子等无用的成分,检查多肽的天然结构是否存在残基和原子的缺失,补全缺失的残基和原子,如果多肽的天然结构仅包含重原子则添加氢原子;
19.步骤1.2采用步骤1.1经过预处理的多肽的天然结构,构建碳末端酰胺化多肽的初始构象,具体为:
20.采用工具软件pdbviewer显示步骤1.1经过预处理的多肽的天然结构,找到碳末端氨基酸残基的羧基(-cooh),将羧基中的羟基(-oh)删除,编辑氨基(-nh2)构象并使氮原子与碳原子成键,即为碳末端酰胺化多肽的初始构象。
21.步骤2:采用步骤1构建的碳末端酰胺化多肽的初始构象,进行全原子分子动力学的平衡处理,具体为:
22.采用步骤1.2构建的碳末端酰胺化多肽的初始构象,选择全原子力场和水模型,生成拓扑文件,定义溶剂盒子的形状和大小,添加水分子和抗衡离子,先对溶剂化的碳末端酰胺化多肽体系进行能量极小化,然后采用恒温器对溶剂化的碳末端酰胺化多肽体系进行nvt平衡,最后采用恒压器对溶剂化的碳末端酰胺化多肽分子体系进行npt平衡;
23.其中,全原子分子动力学计算的一种优选方案是采用gromacs软件;
24.步骤3:采用步骤2经过npt平衡的溶剂化的碳末端酰胺化多肽体系,对碳末端酰胺化多肽进行原子水平的增强采样计算;
25.步骤3具体包含如下子步骤:
26.步骤3.1建立碳末端酰胺化多肽的全局集体坐标:天然原子关联分数和螺旋含量;
27.其中,天然原子关联分数和螺旋含量是基于步骤2的经过npt平衡的碳末端酰胺化多肽结构建立;
28.其中,天然原子关联分数表述为如下公式(1):
[0029][0030]
其中,i和j是碳末端酰胺化多肽中重原子的序号,r
ij
是增强构象采样计算过程中t时刻碳末端酰胺化多肽结构中重原子i和j的距离,是经过npt平衡的碳末端酰胺化多肽结构中重原子i和j之间的实际距离,参数β的优选值为50,参数λ的优选值为1.8;
[0031]
其中,天然原子关联分数的计算只考虑碳末端酰胺化多肽中的非相邻残基;
[0032]
其中,螺旋含量表述为如下公式(2)和(3):
[0033][0034][0035]
其中,n(rmsd)是切换函数,其值在0~1范围;ω
α
是在碳末端酰胺化多肽中依次选取的6残基组;是6残基组ω
α
的重原子坐标;{r0}是选取的6残基理想螺旋组的重原子坐标;rmsd是与{r0}之间的均方根误差;
[0036]
步骤3.2在二维构象空间中,对碳末端酰胺化多肽进行全原子分子动力学和增强采样相结合的动力学计算,保存动力学计算获得的能量、轨迹和全局集体坐标;
[0037]
其中,二维构象空间由步骤3.1的天然原子关联分数qn和螺旋含量sh构成;
[0038]
其中,本步骤的动力学计算是在步骤2的全原子分子动力学的平衡处理的基础上进行;
[0039]
其中,本步骤的增强采样由历史依赖偏置势来驱动;
[0040]
其中,历史依赖偏置势由高斯函数的求和来构建,表述为如下公式(4):
[0041][0042]
其中,c(x)为步骤3.1的全局集体坐标,x是碳末端酰胺化多肽中重原子的坐标,t是动力学计算的时间,ω为高斯函数的高度,δσ为高斯函数的宽度,τg为高斯函数的沉积率;
[0043]
其中,全原子分子动力学和增强采样相结合的动力学计算的一种优选方案是采用gromacs与plumed软件;
[0044]
其中,全原子分子动力学和增强采样相结合的动力学计算时,ω的值一种优选方案为0.1~1kj/mol,δσ的值一种优选方案为步骤3.1建立的天然原子关联分数qn和螺旋含量sh变化范围的1%,τg的一种优选方案为每隔500~1000的全原子分子动力学时间步沉积一次高斯函数;
[0045]
步骤3.3监测天然原子关联分数qn和螺旋含量sh随时间的演化,适时停止步骤3.2的动力学计算;
[0046]
其中,为了使步骤3.2的动力学计算能充分地进行构象采样,一种优选方案是当天然原子关联分数qn和螺旋含量sh低于步骤1的碳末端酰胺化多肽的初始构象的30%时停止动力学计算;
[0047]
步骤4:利用步骤3的增强构象采样计算获得的数据,重建碳末端酰胺化多肽的自
由能,搜索自由能极小构象,具体为:
[0048]
利用步骤3的增强构象采样计算获得的能量数据,以天然原子关联分数和螺旋含量为坐标轴重建碳末端酰胺化多肽的自由能曲面,在自由能曲面上搜索自由能极小,确定自由能极小对应的天然原子关联分数和螺旋含量的值,进而在步骤3获得的轨迹中找到并提取自由能极小对应的结构,即为预测的碳末端酰胺化多肽结构;
[0049]
至此,从步骤1到步骤4,完成了一种碳末端酰胺化多肽结构的动力学预测方法。
[0050]
本发明再一方面提供了一种碳末端酰胺化多肽结构的动力学预测装置,包括以下模块:
[0051]
1、获取和构建模块,用于获取多肽的天然结构,基于多肽的天然结构构建碳末端酰胺化多肽的初始构象;
[0052]
2、平衡处理模块,用于采用获取和构建模块构建的碳末端酰胺化多肽的初始构象,进行全原子分子动力学的平衡处理;
[0053]
3、计算模块,用于采用平衡处理模块经过平衡处理的碳末端酰胺化多肽体系,对碳末端酰胺化多肽进行原子水平的增强采样计算;
[0054]
4、重建模块,用于采用计算模块获得的增强采样计算数据,重建碳末端酰胺化多肽的自由能,搜索自由能极小构象。
[0055]
在本发明所述的装置中,获取和构建模块还包括:
[0056]
预处理模块,用于在获得多肽的天然结构之后,构建碳末端酰胺化多肽的初始构象之前,对获得的多肽的天然结构进行预处理。
[0057]
在本发明所述的装置中,多肽的天然结构从蛋白质数据银行(pdb)下载。
[0058]
在本发明所述的装置中,预处理包括删除多肽的天然结构中的水分子、离子等无用的成分,检查多肽的天然结构是否存在残基和原子的缺失,补全缺失的残基和原子,如果多肽的天然结构仅包含重原子则添加氢原子。
[0059]
在本发明所述的装置中,构建碳末端酰胺化多肽的初始构象包括采用工具软件pdbviewer显示经过预处理的多肽的天然结构,找到碳末端氨基酸残基的羧基(-cooh),将羧基中的羟基(-oh)删除,编辑氨基(-nh2)构象并使氮原子与碳原子成键。
[0060]
在本发明所述的装置中,平衡处理模块进一步用于:
[0061]
采用获取和构建模块构建的碳末端酰胺化多肽的初始构象,选择全原子力场和水模型,生成拓扑文件,定义溶剂盒子的形状和大小,添加水分子和抗衡离子,先对溶剂化的碳末端酰胺化多肽体系进行能量极小化,然后采用恒温器对溶剂化的碳末端酰胺化多肽体系进行nvt平衡,最后采用恒压器对溶剂化的碳末端酰胺化多肽分子体系进行npt平衡。
[0062]
在本发明所述的装置中,平衡处理模块的全原子分子动力学平衡处理采用gromacs软件进行。
[0063]
在本发明所述的装置中,计算模块包括:
[0064]
a、建立全局集体坐标模块,用于基于平衡处理模块获得的经过npt平衡的碳末端酰胺化多肽结构,建立碳末端酰胺化多肽的全局集体坐标:即天然原子关联分数qn和螺旋含量sh;
[0065]
b、动力学计算模块,用于在二维构象空间中,对碳末端酰胺化多肽进行全原子分子动力学和增强采样相结合的动力学计算,保存动力学计算获得的能量、轨迹和全局集体
坐标;
[0066]
c、动力学计算停止模块,用于监测天然原子关联分数qn和螺旋含量sh随时间的演化,适时停止动力学计算。
[0067]
在本发明所述的装置中,天然原子关联分数表述为如下公式(1)
[0068][0069]
其中,i和j是碳末端酰胺化多肽中重原子的序号,r
ij
是增强构象采样计算过程中t时刻碳末端酰胺化多肽结构中重原子i和j的距离,是经过npt平衡的碳末端酰胺化多肽结构中重原子i和j之间的实际距离,参数β的优选值为50,参数λ的优选值为1.8;
[0070]
其中,天然原子关联分数的计算只考虑碳末端酰胺化多肽中的非相邻残基;
[0071]
其中,螺旋含量表述为如下公式(2)和(3):
[0072][0073][0074]
其中,n(rmsd)是切换函数,其值在0~1范围;ω
α
是在碳末端酰胺化多肽中依次选取的6残基组;是6残基组ω
α
的重原子坐标;{r0}是选取的6残基理想螺旋组的重原子坐标;rmsd是与{r0}之间的均方根误差。
[0075]
在本发明所述的装置中,二维构象空间由天然原子关联分数qn和螺旋含量sh构成。
[0076]
在本发明所述的装置中,动力学计算是在平衡处理模块的全原子分子动力学的平衡处理的基础上进行;
[0077]
其中增强采样由历史依赖偏置势来驱动,历史依赖偏置势由高斯函数的求和来构建,表述为如下公式(4):
[0078][0079]
其中,c(x)为步骤3.1的全局集体坐标,x是碳末端酰胺化多肽中重原子的坐标,t是动力学计算的时间,ω为高斯函数的高度,δσ为高斯函数的宽度,τg为高斯函数的沉积率。
[0080]
在本发明所述的装置中,全原子分子动力学和增强采样相结合的动力学计算采用gromacs与plumed软件进行。
[0081]
在本发明所述的装置中,全原子分子动力学和增强采样相结合的动力学计算时,ω的值为0.1~1kj/mol,δσ的值为天然原子关联分数qn和螺旋含量sh变化范围的1%,τg为每隔500~1000的全原子分子动力学时间步沉积一次高斯函数。
[0082]
在本发明所述的装置中,当天然原子关联分数qn和螺旋含量sh达到获取和构建模块的碳末端酰胺化多肽的初始构象的30%时停止动力学计算。
[0083]
在本发明所述的装置中,重建模块进一步用于:
[0084]
利用增强构象采样计算获得的能量数据,以天然原子关联分数和螺旋含量为坐标轴重建碳末端酰胺化多肽的自由能曲面,在自由能曲面上搜索自由能极小,确定自由能极
小对应的天然原子关联分数和螺旋含量的值,进而在获得的轨迹中找到并提取自由能极小对应的结构,即为预测的碳末端酰胺化多肽结构。
[0085]
有益效果
[0086]
本发明的一种碳末端酰胺化多肽结构的动力学预测方法,与现有的蛋白质及多肽结构建模方法相比,具有如下有益效果:
[0087]
1、本发明的方法是通过理论计算来预测碳末端酰胺化多肽结构,与nmr实验解析多肽结构相比,能节省大量的时间、费用;
[0088]
2、本发明的方法是基于天然多肽的结构来预测碳末端酰胺化多肽的结构,与传统的结构建模技术相比,不仅能够提高碳末端酰胺化多肽结构建模的可靠性,还可能体现碳末端酰胺化多肽结构的特异性;
[0089]
3、本发明的方法是在生理仿真环境中进行全原子分子动力学与增强采样相结合的计算获得碳末端酰胺化多肽结构,不仅能够节省计算量,而且预测的结构更接近生理环境中的构象;
[0090]
4、本发明预测的碳末端酰胺化多肽结构可以直接与相应的天然多肽结构进行比较,揭示碳末端酰胺化对多肽结构的影响,展现基因工程重组技术获得的多肽在空间结构、生物活性等方面的优越性;
[0091]
5、本发明的方法可广泛应用于生物医药领域中多肽类药物分子的结构建模,能够节约大量的人力和研发成本。
附图说明
[0092]
图1为本发明预测方法的整体流程图;
[0093]
图2为本发明预测方法的优选流程图;
[0094]
图3为本发明预测装置的整体结构图;
[0095]
图4为本发明预测装置的优选结构图;
[0096]
图5为艾塞那肽的天然结构图;
[0097]
图6为碳末端氨基酸残基的酰胺化前后的结构通式图;
[0098]
图7为碳末端酰胺化艾塞那肽的自由能等高线图;
[0099]
图8为预测的碳末端酰胺化艾塞那肽的结构图。
具体实施方式
[0100]
为了更好的说明本方法的目的和优点,结合附图及具体实施例对本发明具体实施内容做进一步详细说明。
[0101]
实施例1
[0102]
本实施例详细阐述了本发明所述的预测方法在具体实施时针对艾塞那肽的碳末端酰胺化后结构的动力学预测。
[0103]
艾塞那肽的碳末端酰胺化后结构的动力学预测,具体实施过程如下:
[0104]
步骤1:基于艾塞那肽的天然结构构建碳末端酰胺化艾塞那肽的初始构象;
[0105]
其中,艾塞那肽的天然结构从蛋白质数据银行下载,pdb代码为1jrj,保存为1jrj.pdb文件,该天然结构由nmr实验解析,1jrj.pdb文件有36个model;
[0106]
其中,艾塞那肽的天然结构如图5所示,由图中可以看出,1-6残基片段是柔性的loop结构,7-28残基片段形成一个长的α-helix结构,21-38残基片段形成一个疏水的色氨酸笼;
[0107]
其中,基于艾塞那肽的天然结构构建碳末端酰胺化艾塞那肽的初始构象,包含如下子步骤:
[0108]
步骤1.1对艾塞那肽的天然结构进行预处理,具体为:
[0109]
查看艾塞那肽的天然结构1jrj.pdb文件,没有水分子、离子等成分;采用pdbviewer软件检查1jrj.pdb文件,没有残基和原子的缺失;1jrj.pdb含有氢原子,不用再添加氢原子;删除1jrj.pdb文件中的注释,选取model 1的坐标数据,保存为1jrj0.pdb文件;
[0110]
步骤1.2构建碳末端酰胺化艾塞那肽的初始构象,具体为:
[0111]
采用工具软件pdbviewer打开经过预处理的艾塞那肽的天然结构(1jrj0.pdb),找到第39位丝氨酸中的羧基(-cooh),将该羧基中的羟基(-oh)删除,编辑氨基(-nh2)构象并使氮原子与碳原子成键,即形成酰胺基(-conh2),保存该结构为1jrj1.pdb文件,即为碳末端酰胺化艾塞那肽的初始构象;
[0112]
其中,图6显示了碳末端氨基酸残基的酰胺化前后的结构通式;
[0113]
步骤2:对碳末端酰胺化艾塞那肽的初始构象进行全原子分子动力学的平衡处理,具体为:
[0114]
采用步骤1.2构建的碳末端酰胺化艾塞那肽的初始构象(1jrj1.pdb),使用gromacs软件进行全原子分子动力学平衡处理;全原子力场采用charmm36,水模型采用tip3p;溶剂盒子采用立方体盒子,抗衡离子采用na
+
和cl-;先对溶剂化的碳末端酰胺化艾塞那肽体系进行能量极小化;然后,对此体系进行nvt平衡,恒温器使用修正的berendsen恒温器,温度设为310k;最后,对此体系进行npt平衡,恒压器的使用parrinello-rahman耦合器,压强设为1bar;
[0115]
步骤3:采用步骤2经过npt平衡的溶剂化的碳末端酰胺化艾塞那肽体系,对碳末端酰胺化艾塞那肽进行原子水平的增强采样计算;
[0116]
步骤3具体包含如下子步骤:
[0117]
步骤3.1构建碳末端酰胺化艾塞那肽的天然原子关联分数和螺旋含量;
[0118]
其中,天然原子关联分数qn采用发明内容步骤3.1所述公式(1)构建;
[0119]
其中,i和j是碳末端酰胺化艾塞那肽中c、o、n等重原子的序号,i和j值为1~575的整数;r
ij
是增强构象采样计算过程中,t时刻的碳末端酰胺化艾塞那肽的结构中重原子i和j的距离;是碳末端酰胺化艾塞那肽中重原子i和j的初始距离,它们通过提取步骤2中经过npt平衡的碳末端酰胺化艾塞那肽结构,进而计算重原子i和j的距离获得;β取值为50;λ取值为1.8;
[0120]
其中,天然原子关联分数仅考虑碳末端酰胺化艾塞那肽的非相邻氨基酸残基中重原子之间的关联;
[0121]
其中,螺旋含量sh采用发明内容步骤3.1所述公式(2)和(3)构建;
[0122]
其中,螺旋含量sh的构建,具体为:
[0123]
根据步骤1.2的碳末端酰胺化艾塞那肽的初始构象(1jrj1.pdb),氨基酸7-28片段
为α-helix,依次选择(7,8,
…
,12)、(8,9,
…
,13)、
…
、(23,24,
…
,28)为6氨基酸残基组ω
α
;从碳末端酰胺化艾塞那肽中提取这些ω
α
的重原子坐标与6残基理想螺旋组件的重原子坐标{r0}进行比对,计算与{r0}的均方根误差rmsd;将rmsd值代入公式(2)和(3)得到螺旋含量sh;
[0124]
步骤3.2对碳末端酰胺化艾塞那肽执行原子水平的增强采样动力学计算,具体为:
[0125]
在天然原子关联分数和螺旋含量形成二维构象空间中,对碳末端酰胺化艾塞那肽进行全原子分子动力学和增强采样相结合的动力学计算,保存计算获得的能量、轨迹、天然原子关联分数和螺旋含量;
[0126]
其中,二维构象空间由步骤3.1构建的天然原子关联分数和螺旋含量构成;
[0127]
其中,本步骤的动力学计算是在步骤2经过npt平衡的溶剂化的碳末端酰胺化艾塞那肽体系的基础上进行;
[0128]
其中,本步骤的增强采样由历史依赖偏置势来驱动,历史依赖偏置势的定义为发明内容步骤3.2所述公式(4);
[0129]
其中,全原子分子动力学计算使用gromacs软件,增强采样计算使用plumed软件;
[0130]
其中,全原子分子动力学和增强采样相结合的动力学计算中,高斯函数的高度ω设为0.1kj/mol;高斯函数的宽度δσ,对于天然原子关联分数设为0.01,对于螺旋含量设为0.25;每隔500时间步的全原子分子动力学计算添加一次高斯函数,即高斯函数的沉积率τg设为500;
[0131]
其中,每500个全原子分子动力学时间步保存一次能量、轨迹、天然原子关联分数和螺旋含量;
[0132]
步骤3.3监测天然原子关联分数和螺旋含量随时间的演化,适时停止增强采样动力学计算;
[0133]
其中,监测到天然原子关联分数和螺旋含量随时间的演化特征为:天然原子关联分数和螺旋含量的初始值分别为0.99和20.9,天然原子关联分数和螺旋含量随时间逐渐减少;
[0134]
其中,在计算时间为250ns时,天然原子关联分数和螺旋含量分别减少到0.3和6.2,停止增强采样动力学计算;
[0135]
步骤4:重建碳末端酰胺化艾塞那肽的自由能,搜索自由能极小构象,具体为:
[0136]
利用步骤3的增强采样计算获得的能量数据,以天然原子关联分数和螺旋含量为坐标轴重建碳末端酰胺化艾塞那肽的自由能曲面,在自由能曲面上搜索自由能极小,确定自由能极小对应的天然原子关联分数和螺旋含量的值,进而在步骤3获得的轨迹中找到并提取自由能极小对应的结构,即为预测的碳末端酰胺化艾塞那肽结构;
[0137]
其中,重建的碳末端酰胺化艾塞那肽的自由能曲面等高线图见图7,图中的横坐标为天然原子关联分数qn,qn的范围是0.3~1.0,纵坐标为螺旋含量sh,sh的范围是4~22,图中的曲线为自由能等高线,同一条等高线有相同的自由能值,相邻两条等高线的自由能间隔为1.5kj/mol,图中显示,该自由能曲面存在3个主要的自由能极小f1、f2和f3;
[0138]
其中,自由能极小f1、f2和f3对应的天然原子关联分数和螺旋含量如下表1所示:
[0139]
表1自由能极小的自由能、天然原子关联分数和螺旋含量
[0140]
自由能极小点自由能值(kj/mol)qnshf1-14.10.9721.2f2-25.70.5413.3f3-11.10.356.4
[0141]
从表中可以看出,自由能极小f1的qn等于0.97,sh等于21.2,与碳末端酰胺化艾塞那肽的初始构象的qn和sh值基本相同,说明f1对应的结构类似于初始构象;自由能极小f2的自由能为-25.7kj/mol,比f1和f3的自由能值小10kj/mol以上,表明自由能全局极小f2的构象最稳定;
[0142]
其中,预测的碳末端酰胺化艾塞那肽的自由能全局极小结构如图8所示,它是根据自由能极小f2的qn和sh值,从轨迹中找到并提取的结构;从图中可以看出,碳末端酰胺化艾塞那肽的9-24残基片段形成一个α-helix,1-8和25-39残基片段形成了柔性loop,碳末端的25-39残基片段弯曲并靠近这个α-helix;
[0143]
其中,预测的碳末端酰胺化艾塞那肽结构可以与艾塞那肽的天然结构进行比较;艾塞那肽的天然结构的特点如本实施例步骤1.1所述,其碳端的色氨酸笼有利于绑定glp-1受体的细胞外结构域,中部的长α-helix结构有利于嵌入glp-1受体的活性部位,氮端的柔软loop有利于结合glp-1受体的跨膜结构域并激活glp-1受体;与之相比,碳末端酰胺化艾塞那肽的结构缺乏疏水的色氨酸笼,其中部的α-helix明显比艾塞那肽的天然结构的α-helix短,这种结构不利于它通过“双结构域模式”结合并激活glp-1受体;第39位丝氨酸的酰胺化对碳末端酰胺化艾塞那肽的空间结构、生物活性会产生不利影响;而且,由于基因工程重组技术能够获得与天然多肽完全一致的空间结构和生物学活性,本实施例的结果可以提示通过基因工程重组技术获取的艾塞那肽比化学合成的碳末端酰胺化艾塞那肽在空间结构、生物活性等方面具有优越性。
[0144]
以上所述为本发明的较佳实施例而已,本发明不应该局限于该实施例和附图所公开的内容。凡是不脱离本发明所公开的精神下所公开的精神下完成的等效或修改,都落入本发明保护的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1