一种基因多序列比对方法、设备和系统
1.本发明属于生物信息学技术领域,具体涉及一种基因多序列比对方法、设备和系统。
背景技术:
2.基因的多序列比对是生物信息学的基本组成和重要基础。序列比对的基本思想是,基于生物学中序列决定结构,结构决定功能的普遍规律,将核酸序列和蛋白质一级结构上的序列都看成由基本字符组成的字符串,检测序列之间的相似性,发现生物序列中的功能、结构和进化的信息。
3.例如,裂腹鱼类cytb基因多序列对比是一种重要的基因多序列比对研究体系。细胞色素b基因(cytb)是真核生物线粒体基因组中的一个编码基因,因其单拷贝、进化速率适中、多态性位点丰富等优点,是常用于研究生物遗传进化及多样性的靶基因。裂腹鱼类是青藏高原及周边地区特有的一个鲤科裂腹鱼亚科的鱼类物种类群,是我国青藏高原地区重要的渔业资源和生态关键物种。在物种的起源和进化上,裂腹鱼类的物种都有较近的亲缘关系,形态上较为相似,在cytb基因的序列结构上既有不同物种特异的多态位点,也有裂腹鱼类共有的保守基序。因此,cytb基因是区分不同裂腹鱼物种,研究其系统进化和物种分类常用的靶基因。在研究方法上,最重要和最基础的是cytb基因的测序和序列比对分析。首先利用pcr技术,根据裂腹鱼类cytb基因的通用引物(l14724和h15915)体外扩增获得包含cytb基因开放阅读框(orf)全长的基因序列,接着,利用焦磷酸测序技术,对扩增产物的脱氧核糖核苷酸组成顺序进行测定,获得由不同碱基构成的序列信息,然后,利用基因序列软件对不同样本的cytb基因序列进行比对分析,从而获得遗传变异信息,用于物种鉴定、分类和系统进化等研究。
4.现有的基因序列比对方法和软件已经商业化,具体包括clustalx、mega、muscle、mafft等在线软件或pc安装软件。其中mega软件集成了clustalx、muscle等多种比对方法,是最常用的序列比对分析软件,有较强的可视化效果。
5.虽然这些软件已经较为成熟,但是它们在使用时需要输入的基因序列数据必须是标准化的格式。然而,在实际工作中,测序得到的基因序列通常是不符合这些软件的输入要求的。例如,测序得到的基因序列可能出现如下情况:是正义链序列或反义链序列,可能包含引物、接头和终止密码子之后的“垃圾”序列,可能包括不在对比范围内的冗余序列,存在5’和3’端残缺,包含测序错误的位点等。
6.因此,现有的测序数据在利用软件进行比对前,还需要研究者根据自身对相关物种的研究经验,对序列进行编辑、校正。这大大增加研究者的工作量,不利于基因多序列比对工作的高效进行。
技术实现要素:
7.针对现有技术的缺陷,本发明提供一种基因多序列比对方法、设备和系统,目的在
于提供一种包含对基因序列进行自动编辑、校正步骤的基因多序列比对方法,减少相关研究工作中研究者的工作量,提高基因多序列比对工作的效率。
8.一种基因多序列比对方法,包括如下步骤:
9.步骤1,设置模板信息,所述模板信息包括待分析基因的模板序列和保守基序的信息;
10.步骤2,根据步骤1的模板信息作为参照,对待分析序列进行校正和多序列全局比对,形成数据集。
11.优选的,步骤1中,所述模板信息是通过待分析基因的正义链简并序列获得的。
12.优选的,步骤2中,进行校正和多序列全局比对的具体步骤如下:
13.步骤2.1,根据步骤1的模板信息作为参照,识别待分析序列中的反义链序列,将所述反义链序列处理为正义链序列;
14.步骤2.2,根据步骤1的模板信息作为参照,识别经过步骤2.1处理后的待分析序列中的引物、接头和终止密码子之后的测序载体序列,并将所述测序载体序列剪切舍弃;
15.步骤2.3,对经过步骤2.2处理后的待分析序列进行多序列全局对比,形成比对后的数据集;
16.步骤2.4,根据步骤1的模板信息作为参照,校对步骤2.3得到的数据集,填补5
’‑
和3
’‑
端残缺的序列,删除非比对区序列;
17.步骤2.5,对经过步骤2.4处理后的数据集进行多序列全局对比,识别包含测序错误位点的序列样本,删除包含测序错误位点的序列样本或对包含测序错误位点的序列样本进行调整。
18.优选的,步骤2.5中,识别包含测序错误位点的序列样本的方法为:对经过步骤2.4处理后的数据集中的序列样本的每个位点基因型或缺失型的出现频率进行统计,若某个位点的某种基因型或缺失型只出现一次,则具有该基因型或缺失型的该位点为测序错误位点。
19.优选的,步骤2中,采用matff、clustalx、mega或muscle进行多序列全局比对。
20.优选的,所述待分析基因的模板序列为其间并序列,所述间并序列如seq id no.1所述;
21.所述保守基序的数量为三个,序列分别为aaaattgctaa、attgcccg和gtaattac;
22.所述保守基序的位置为从5’开始的第34位至第44位、第292位至第299位、第433位至第440位。
23.优选的,所述待分析序列和数据集的文件格式为fasta格式。
24.本发明还提供一种计算机设备,用于基因多序列比对,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现上述基因多序列比对方法。
25.本发明还提供一种基因多序列比对的系统,包括:
26.上述计算机设备;
27.服务器,用于存储和传输所述待分析序列的原始数据。
28.本发明还提供一种计算机可读存储介质,其上存储有用于实现上述基因多序列比对方法的计算机程序。
29.本技术提供了一种根据待分析基因的模板信息对测序数据进行自动校正和多序列全局比对的方法。特别的,在优选方案中,本发明提供了裂腹鱼类cytb基因的模板信息,能够准确地对裂腹鱼类的测序数据进行自动校正和多序列全局对比。本发明的方法将现有技术中需要人工完成的校正过程通过程序自动实现,大大减少了多序列比对研究工作中研究者的工作量,能够显著提高相关工作的效率。因此,本发明具有很好的应用前景。
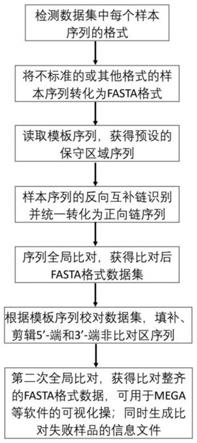
30.显然,根据本发明的上述内容,按照本领域的普通技术知识和惯用手段,在不脱离本发明上述基本技术思想前提下,还可以做出其它多种形式的修改、替换或变更。
31.以下通过实施例形式的具体实施方式,对本发明的上述内容再作进一步的详细说明。但不应将此理解为本发明上述主题的范围仅限于以下的实例。凡基于本发明上述内容所实现的技术均属于本发明的范围。
附图说明
32.图1为本发明实施例1的流程示意图。
具体实施方式
33.需要特别说明的是,实施例中未具体说明的数据采集、传输、储存和处理等步骤的算法,以及未具体说明的硬件结构、电路连接等均可通过现有技术已公开的内容实现。
34.实施例1
35.本实施例提供一种基因多序列比对的系统,其包括服务器和计算机设备。服务器用于存储和传输所述待分析序列的原始数据,计算机设备用于基因多序列比对,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现基因多序列比对方法。
36.利用上述系统进行裂腹鱼类cytb基因多序列比对的方法如图1所示,具体步骤如下:
37.步骤1:对测序仪器产生的裂腹鱼类cytb基因序列的格式进行逐条检测,所有样本的基因序列文件必须在同一个文件夹(路径)下。如果程序检测到的序列格式是标准的fasta格式,则将序列信息提取出来,合并入一个在该步骤运行时产生的文本文件中,该文件名称定义为allseq.fas(再次出现简称为allseq.fas文件);如果程序检测到的序列格式不是标准的fasta格式,则首先将序列格式转化成标准的fasta格式,然后合并入allseq.fas文件。
38.步骤2:程序自动读取模板序列,并在模板序列中提取预先设置好位置和长度的保守基序(motif)。模板序列是根据多个裂腹鱼物种的cytb基因序列获得的正义链简并序列,程序中预设了根据前期的研究积累分析获得的模板序列,并提供了用于识别序列一致性motif的位置和长度信息(表1)。同时模板序列、motif的位置和长度参数在需要时也可以手动修改和设定。motif的位置和长度参数直接影响下游分析是否能够正常运行,以及分析结果的准确性。
39.本实施例中,所述待分析基因的模板序列为其间并序列,所述间并序列如seq id no.1所述。seq id no.1序列如下:
[0040]5’‑
atggcaagcctacgaaaaactcaccccctaattaaaattgctaa cagtgcactagttgacctgc
cagcaccatccaacatctcagcatgatgaaactttggctctcttctaggrctatgcctagccactcaaatcctaaccg gcctattcctagccatacactatacctcagaygtttcaaccgcattctcatcagtagtccatatttgccgggacgtaaattacggctgactaatccgcaacgtrcacgccaacggagcatcrttcttctttatctgyatttatatacatattgcccgaggcctatactacggatcttacctctacaaagaaacctgaaatattggtgtrgtccttctrcttcttgttatratgacggccttcgtaggvtatgtcctgccatgrggtcaaatrtctttttgaggtgcyacagtaattacmaatctcctrtccgctgtrccataygtaggtgaygttctagtccaatggatttgaggcggattctcagtagataaygcaacactaacrcgattcttcgcatttcactttctatttccatttgtaattgctgctataaccatcttrcacctcctrtttttacatgaaactggrtcaaayaacccgattggsctcaactcagacgcagataaaatccccttccacccatactttacatataaagayttrcttggcttcgtaattatactttttttacttatgcttttagcactattttctccgaayctgctrggagacccagaaaacttcacccccgccaaccccctagtcacaccaccacacattaarccagartgatatttcctgttcgcctatgccatyctmcgrtcyatcccaaacaarcttggtggtgtacttgcwctactattttctattctrgthttaatagttgtgcctctscttcacacctccaagctacgaggactaacattccgcccaatcacccaattcttattctgaactctrgtggcagacatrattatcytracatgaattggcggcataccagtagaacacccatttattattattggacaagtcgcatccgccctdtactttgcactgtttctcrtttttataccactagcagggtgrgtagaaaataaagcactggaattagcc-3’。
[0041]
motif的信息如下表所示。
[0042]
表1保守基序
[0043][0044]
步骤3:程序自动对样本序列进行编辑。供的模板序列作为标准参照,逐条检测allseq.fas文件中的样本序列,若样本序列为正义链序列,则不做处理;若样本序列为反义互补序列,则自动对齐进行反向互补处理,转化成正义链序列。接着,程序自动逐条识别allseq.fas文件中样本序列的引物、接头和终止密码子之后的“垃圾”序列,并将这些序列剪切舍弃。通过步骤3处理后的数据,自动生成fasta格式文件allseqedited.fas。allseqedited.fas文件的第一条序列是模板序列,接着是分析样本序列。分析样本序列的顺序与allseq.fas文件中的样本顺序相同。
[0045]
步骤4:程序自动调用matff软件对allseqedited.fas进行多序列全局比对。如果matff软件已经设置在环境变量中,则无需再进行路径配置,否则matff软件的绝对路径需要提前设置在配置文件中。程序在执行matff比对之前,对系统线程数(虚拟核)进行检测,获得线程数,以最大线程数的2/3整数作为线程数参数,例如,如果检测到的最大线程数是56,则线程数参数为37。接着程序自动执行matff软件,对allseqedited.fas文件进行多序列全局比对,并生成比对后的fasta格式文件allseqeditedaligned.fas。
[0046]
除了本实施例选择的matff软件,也可采用其他能够完成多序列比对的软件。
[0047]
步骤5:程序自动校正序列,补齐5’和3’端残缺的序列,删除冗余非比对序列。此步骤生成fasta格式的allseqproofed.fas
[0048]
步骤6:程序自动调用matff软件对allseqproofed.fas进行多序列全局比对。比对完成后,将输出结果转化成有序的位点矩阵,逐个位点检测每个样本每个位点的碱基型或
缺失型在allseqproofed.fas文件所有样本中的频率,若某个样本某个位点的碱基型或缺失型在所有样本中仅出现一次,则该样本该位点信息被定义为疑似测序错误位点记录到日志文件中,同时以该位点最高频率的碱基型作为该样本该位点建议校正碱基型记录到日志文件中。最终,该步骤生成fasta格式文件allseqproofedaligned.fas和日志文件。
[0049]
日志文件中列出了疑似测序错误位点和建议调整信息,该结果仅供参考,可根据实际情况调整allseqproofedaligned.fas文件中相应样本的相应位点。allseqproofedaligned.fas文件是fasta格式,可以在mega等软件中进行可视化展示,可以在dnasp等软件中直接进行遗传进化分析,也可转化成其他格式文件用于进化树构建。
[0050]
以100条裂腹鱼类cytb基因测序数据为测试样本,利用本方法对原始测序序列进行矫正、分析,并以人工矫正分析结果为标准进行比较。本专利方法对100条裂腹鱼类cytb基因序列的处理、分析过程耗时为41s,而人工处理、分析过程耗时5.3h,本方法耗时显著减小。为了验证本方法的准确性,对每个测试样本的人工矫正序列和本方法得到的矫正序列进行逐一比对,结果序列完全一致,因此验证得到本方法结果的准确性为100%。
[0051]
通过上述实施例可以看到,本发明提供了一种具有自动校正功能的基因多序列比对方法,本发明的方法相比于现有技术省略了人工校正待分析序列的步骤,大大减少了相关领域研究者的工作量,显著提高了多序列比对工作的效率。因此,本发明具有很好的应用前景。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1