一种结合参考库先验知识的宏基因组学分箱方法及系统与流程
1.本技术涉及宏基因组学和数据科学技术领域,特别是涉及结合参考库先验知识的宏基因组学分箱方法及系统。
背景技术:
2.本部分的陈述仅仅是提到了与本技术相关的背景技术,并不必然构成现有技术。
3.宏基因组学直接从自然环境样本中研究微生物的遗传物质,提供了一种研究真实微生物世界的有效方法,避免了实验室培养产生的偏差。宏基因组学分箱是将基因序列进行分类来区分不同的微生物物种或亚种,其分箱结果将直接影响宏基因组学研究的准确度,因而宏基因组学分箱已成为宏基因组学研究中的一个关键问题。
4.目前,宏基因组学分箱研究主要分为重叠群(contig)分箱和长读段(long read)分箱两大类。其中,重叠群是由短读段(short read)通过末端的重叠序列相互连接形成的长基因片段;而长读段则是伴随第三代测序技术(tgs)产生的长基因序列。此两类皆因序列更长、包含更多基因特征而比短读段更适合分箱。
5.就分箱方法而言,已有的宏基因组学分箱方法大致分为基于参考库(reference-based)的分箱和参考库无关(reference-free)的分箱两大类。其中,基于参考库的分箱通过将目标序列数据集与已知物种参考库(reference database)进行比对来实现对目标序列数据集的分类,这种分箱方法对于已知物种基因序列可以达到很高的分箱精度,但无法处理未知物种;参考库无关的分箱则不依赖参考库,而是基于基因序列的区分性特征,采用特征工程和聚类方法来实现分箱。此类分箱方法可对未知物种进行分类,但其分箱精度通常较低,尤其是当区分特征差异较小或者物种数量较大时。
6.近年来,随着新物种的不断发现和登记,参考库得到很大补充和完善,能够对目标序列数据集的物种信息提供便捷的评估,尽管此评估结果还不够准确,但却是非常有价值的先验知识,充分利用这些先验信息能够极大提升分箱的精度。目前已有学者组合两种分箱方法实现宏基因组分箱,其分箱过程分为两个相对独立的阶段,首先采用特征库无关的分箱方法对进行初次分箱,然后对分箱质量不足的序列采用基于特征库的方法进行重新分箱;其本质是将基于特征库分箱作为特征库无关分箱的一种补充策略,并未将两种分箱方法真正融合。当区分特征差异较小或者物种数量较大时,这种方法的分箱效果将依赖基于参考库的重新分箱,因此会影响对未知物种序列的识别。
技术实现要素:
7.为了解决现有技术的不足,本技术提供了一种结合参考库先验知识的宏基因组学分箱方法及系统,充分利用已有参考库所提供的先验知识,将此先验知识纳入特征无关的宏基因组学分箱。与现有技术相比,此方法从实质上实现了此两类分箱方法的融合,解决了现有宏基因组分箱不能处理未知物种序列或分箱精度不足等问题,既能对未知物种基因序列进行分箱,也比参考库无关的分箱方法具有更优的分箱性能。
8.为实现上述目的,本发明的一个或多个实施例提供了如下技术方案:
9.一种结合参考库先验知识的宏基因组学分箱方法,所述参考库存储已知物种的标准基因序列,包括以下步骤:
10.获取目标序列数据集,对其中的每个序列样本进行特征提取,得到其初始区分特征向量,经特征变换后,得到该序列的分箱特征向量;
11.将目标序列数据集与参考库进行对比,得到所述目标序列数据集包含的物种数量估算值和各序列样本属于不同物种的置信度;根据所述物种数量估算值生成分箱数量可行区间,在此区间内,每个可行分箱数量均大于等于物种数量估算值;根据所述各序列样本属于不同物种的置信度,得到先验聚类中心集,将所述分箱数量可行区间和先验聚类中心集作为先验知识;
12.对所述分箱数量可行区间中的每个分箱数量,确定相应聚类中心集,所述聚类中心集包括先验聚类中心集和扩增聚类中心集;对于每个可行分箱数量,基于相应聚类中心集,对分箱特征向量集进行聚类分析,选取最优聚类结果,获取所述最优聚类结果中各簇对应的序列样本集,即为分箱结果。
13.进一步地,所述特征变换用于获取所述初始区分特征向量的低维表示,即分箱特征向量。
14.进一步地,所述特征变换采用以下任一方法:
15.(1)深度学习模型vae;(2)降维模型umap;(3)先采用深度学习模型vae,再对得到的隐向量进一步采用降维模型umap。
16.进一步地,根据所述各序列样本属于不同物种的置信度,得到先验聚类中心集包括:
17.对于其中的每类物种,根据各序列样本属于该类物种的置信度,选择此物种的多个标杆序列样本,并基于分箱特征向量集获取这些标杆样本的分箱特征向量,这些分箱特征向量的中心即该类物种的先验聚类中心。
18.进一步地,所述扩增聚类中心采用在分箱特征向量集中随机方式选取,且保证每两个扩增聚类中心之间、以及扩增聚类中心与各先验聚类中心之间的距离均不小于设定阈值。
19.进一步地,根据轮廓系数或ch指数,对所有可行分箱数量对应的聚类结果进行对比,选择最优聚类结果。
20.进一步地,获取所述最优聚类结果中各簇对应的序列样本集包括:
21.假设x为目标序列数据集,对任意序列样本x∈x,记为x对应的分箱特征向量,若最优聚类结果为则计算bj={x|x∈x且j=1,2,
…
,k
*
,得到由各簇所对应的序列样本集即为分箱结果。
22.一个或多个实施例提供了一种结合参考库先验知识的宏基因组学分箱系统,所述参考库存储已知物种的标准基因序列,包括:
23.特征提取模块,用于获取目标序列数据集,对其中的每个序列样本进行特征提取,得到其初始区分特征向量,经特征变换后,得到该序列的分箱特征向量;
24.先验知识获取模块,用于将目标序列数据集与参考库进行对比,得到所述目标序
列数据集包含的物种数量估算值和各序列样本属于不同物种的置信度;根据所述物种数量估算值生成分箱数量可行区间,在此区间内,每个可行分箱数量均大于等于物种数量估算值;根据所述各序列样本属于不同物种的置信度,得到先验聚类中心集,将所述分箱数量可行区间和先验聚类中心集作为先验知识;
25.分箱模块,用于对所述分箱数量可行区间中的每个可行分箱数量,确定相应聚类中心集,所述聚类中心集包括先验聚类中心集和扩增聚类中心集;对于每个可行分箱数量,基于相应聚类中心集,对分箱特征向量集进行聚类分析,选取最优聚类结果,获取所述最优聚类结果中各簇对应的序列样本集,即为分箱结果。
26.一个或多个实施例提供了一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现所述结合参考库先验知识的宏基因组学分箱方法。
27.一个或多个实施例提供了一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现所述结合参考库先验知识的宏基因组学分箱方法。
28.以上一个或多个技术方案存在以下有益效果:
29.本技术首先基于参考库估算物种数量并选取这些物种的置信序列样本,将这些置信序列样本的分箱特征向量的中心作为先验聚类中心,然后以先验聚类中心为基础,扩展未知物种聚类中心,然后基于先验聚类中心和扩展的聚类中心,对序列分箱特征集进行聚类,最后依据最优聚类结果实现对目标序列数据集的分箱。本技术充分利用已有参考库所提供的先验知识,将此先验知识纳入特征无关的宏基因组学分箱,从实质上实现了此两类分箱方法的融合。
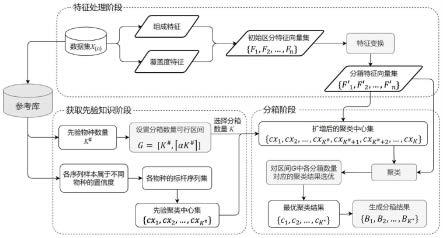
30.相对于基于参考库的分箱,本技术实现了对未知物种序列的分箱;相对于参考库无关的分箱,由于采用来自参考库的物种信息作为先验知识,具有比现有参考库无关分箱方法更优的分箱性能。
附图说明
31.构成本技术的一部分的说明书附图用来提供对本技术的进一步理解,本技术的示意性实施例及其说明用于解释本技术,并不构成对本技术的不当限定。
32.图1为本技术一个或多个实施例中结合参考库先验知识的宏基因组学分箱方法主要步骤流程示意图。
具体实施方式
33.应该指出,以下详细说明都是示例性的,旨在对本发明提供进一步的说明。除非另有指明,本文使用的所有技术和科学术语具有与本发明所属技术领域的普通技术人员通常理解的相同含义。
34.需要注意的是,这里所使用的术语仅是为了描述具体实施方式,而非意图限制根据本发明的示例性实施方式。如在这里所使用的,除非上下文另外明确指出,否则单数形式也意图包括复数形式,此外,还应当理解的是,当在本说明书中使用术语“包含”和/或“包括”时,其指明存在特征、步骤、操作、器件、组件和/或它们的组合。
35.在不冲突的情况下,本发明中的实施例及实施例中的特征可以相互组合。
36.实施例一
37.本实施例提供了一种结合参考库先验知识的宏基因组学分箱方法,包括特征处理阶段、获取先验知识和分箱三个阶段。包括以下步骤:
38.步骤1:获取目标序列数据集,对其中的每个序列样本进行特征提取,得到其初始区分特征向量,经特征变换后,得到该序列的分箱特征向量。
39.所述步骤1即对应特征处理阶段,具体包括:
40.步骤1-1:获取目标序列数据集,设目标序列数据集为x={x1,x2,...,xn},n表示序列样本的数量。
41.步骤1-2:对所述目标序列数据集中的所有序列样本进行特征提取,得到初始区分特征集。具体地,对任一序列样本xi∈x,计算其初始区分特征,得到该序列样本的初始区分特征向量fi=(f
i,1
,f
i,1
,
…
,f
i,m
),m为特征维数;由此得到目标序列数据集x所对应的初始区分特征集{f1,f2,
…
,fn}。对于长读段和重叠群,此初始区分性特征主要包括组成(composition)和覆盖度(coverage)信息。
42.步骤1-3:对初始区分特征集进行特征变换,得到分箱特征向量集。具体地,对初始区分特征集{f1,f2,
…
,fn},采用机器学习或深度学习模型进行特征变换,得到分箱特征向量集{f'1,f'2,
…
,f'n},其中,f'i=(f'
i,1
,f'
i,2
,
…
,f'
i,s
)为序列样本xi的分箱特征向量,i=1,2,
…
,n,s为分箱特征的维数。
43.所述特征变换的目的是获取所述初始区分特征向量的低维表示,即分箱特征向量,可通过深度学习模型vae或降维模型umap实现。作为一种优选实现方式,先采用深度学习模型vae,再对得到的隐向量进一步采用降维模型umap。采用深度学习模型vae能够在实现降维的同时,最大限度的保留初始区分特征集中的显著性信息,再通过降维模型umap得到最终低维表示,保证了分箱特征向量集中不同物种分箱特征向量之间的差异性,同时有利于提高后续聚类效率。
44.以长读段数据集为例,设有长读段数据集为x={x1,x2,...,xn},n为长读段样本数量。对任一长读段样本xi∈x,计算其3核苷酸组成和k-mer覆盖度作为初始区分特征,得到初始区分特征集{f1,f2,
…
,fn};然后对此初始区分特征集采用深度学习模型vae和降维模型umap进行变换,得到分箱特征向量集{f'1,f'2,
…
,f'n},其中,f'i=(f'
i,1
,f'
i,2
,
…
,f'
i,s
),i=1,2,
…
,n,s为分箱特征向量的维数。本实施例中,s=2。
45.步骤2:将目标序列数据集与参考库进行对比,得到所述目标序列数据集包含的物种数量估算值和各序列样本属于不同物种的置信度,根据所述物种数量估算值生成分箱数量可行区间,在此区间内,每个可行分箱数量大于等于物种数量估算值,根据所述各序列样本属于不同物种的置信度得到先验聚类中心集,将所述分箱数量可行区间和先验聚类中心集作为先验知识。
46.所述步骤2即对应获取先验知识阶段,具体包括:
47.步骤2-1:将目标序列数据集x与参考库进行比对,得到数据集x包含的物种数量估算值k
#
,及各序列样本属于不同物种的置信度。
48.步骤2-2:设置分箱数量可行区间其中α为区间宽度因子,取值为大于1的经验值,本实施例中,α=1.5。
49.步骤2-3:对于每类物种,根据各序列样本属于该类物种的置信度,选择此物种的多个标杆样本,并基于分箱特征向量集获取这些标杆样本的分箱特征向量,这些分箱特征向量的中心即为该类物种对应的先验聚类中心。本实施例中,采用多个分箱特征向量的均值作为所述多个分箱特征向量的中心。
50.具体地,根据步骤2-1所得各序列样本属于不同物种的置信度,对物种v,v=1,2,
…
,k
#
,选定置信度最大的t个序列样本作为该物种标杆样本;然后,在{f'1,f'2,
…
,f'n}中找到这些标杆样本的分箱特征向量,计算这些分箱特征向量的中心作为物种v的置信聚类中心cxv,据此得到所有物种的先验聚类中心集
51.以长读段数据集为例,首先将目标序列数据集x与参考库进行比对,得到数据集x包含的物种数量的估算值k
#
,及各序列样本属于不同物种的置信度,然后,对任一物种v,v=1,2,
…
,k
#
,选定置信度最大的50个长读段样本作为标杆样本;然后,在{f'1,f'2,
…
,f'n}中找到这50个标杆样本的分箱特征向量,计算这些分箱特征向量的中心作为物种v的置信聚类中心cxv,据此得到所有物种的先验聚类中心集
52.所述步骤3对应分箱阶段。
53.步骤3:对所述分箱数量可行区间中的每个可行分箱数量,确定相应聚类中心集,所述聚类中心集包括先验聚类中心集和扩增聚类中心。对于每个可行分箱数量,基于相应聚类中心集,对分箱特征向量集进行聚类分析,选取最优聚类结果,获取所述最优聚类结果中各簇对应的序列样本集,即为分箱结果。
54.所述步骤3具体包括:
55.步骤3-1:对每个可行分箱数量k∈g,选取该分箱数量所对应的聚类中心集其中,为来自参考库的先验聚类中心,为扩增聚类中心。扩增聚类中心的选取采用在分箱特征向量集中随机选取且保证每两个扩增聚类中心之间、以及扩增聚类中心与各先验聚类中心分箱特征向量之间的距离均不小于阈值d。其中,所述分箱特征向量之间的距离的计算可采用欧氏距离、余弦相似度等,在此不做限定。
56.步骤3-2:选择基于中心的聚类模型,如k-means模型,以uk为聚类中心集,对分箱特征向量集{f'1,f'2,
…
,f'n}进行聚类,得到每个可行分箱数量k所对应的聚类结果{c1,c2,
…
,ck}。
57.步骤3-3:对中所有分箱数量对应的聚类结果进行对比,选择最优聚类结果,记为其中,k
*
为最优分箱数量。
58.其中,聚类结果的评估可采用轮廓系数或ch指数等指标,基于多种指标对比聚类结果,综合评估得到最优聚类结果。
59.步骤3-4:对任意序列样本x∈x,设为x对应的分箱特征向量,则按最优聚类结果计算bj={x|x∈x且},j=1,2,
…
,k
*
,得到由各簇所对应的序列样本集即为分箱结果。
60.实施例二
61.基于实施例一中所述方法,本实施例提供了一种结合参考库先验知识的宏基因组学分箱系统,所述参考库存储已知物种的标准基因序列,包括:
62.特征提取模块,用于获取目标序列数据集,对其中的每个序列样本进行特征提取,得到其初始区分特征向量,经特征变换后,得到该序列的分箱特征向量;
63.先验知识获取模块,用于将目标序列数据集与参考库进行对比,得到所述目标序列数据集包含的物种数量估算值和各序列样本属于不同物种的置信度;根据所述物种数量估算值生成分箱数量可行区间,在此区间内,每个可行分箱数量均大于等于物种数量估算值;根据所述各序列样本属于不同物种的置信度,得到先验聚类中心集,将所述分箱数量可行区间和先验聚类中心集作为先验知识;
64.分箱模块,用于对所述分箱数量可行区间中的每个可行分箱数量,确定相应聚类中心集,所述聚类中心集包括先验聚类中心集和扩增聚类中心集;对于每个可行分箱数量,基于相应聚类中心集,对分箱特征向量集进行聚类分析,选取最优聚类结果,获取所述最优聚类结果中各簇对应的序列样本集,即为分箱结果。
65.实施例三
66.本实施例的目的是提供一种电子设备。
67.一种电子设备,包括存储器、处理器及存储在存储器上并可在处理器上运行的计算机程序,所述处理器执行所述程序时实现实施例一中所述的方法。
68.实施例四
69.本实施例的目的是提供一种计算机可读存储介质。
70.一种计算机可读存储介质,其上存储有计算机程序,该程序被处理器执行时实现实施例一中所述的方法。
71.以上实施例二至四与方法实施例一相对应,具体实施方式可参见实施例一的相关说明部分。术语“计算机可读存储介质”应该理解为包括一个或多个指令集的单个介质或多个介质;还应当被理解为包括任何介质,所述任何介质能够存储、编码或承载用于由处理器执行的指令集并使处理器执行本发明中的任一方法。
72.以上一个或多个实施例充分利用已有参考库所提供的先验知识,将其纳入宏基因组学分箱。与现有技术相比,此方法解决了现有宏基因组分箱不能处理未知物种序列或分箱精度不足等问题,既能对未知物种基因序列进行分箱,也比参考库无关的分箱方法具有更优的分箱性能。
73.本领域技术人员应该明白,上述本发明的各模块或各步骤可以用通用的计算机装置来实现,可选地,它们可以用计算装置可执行的程序代码来实现,从而,可以将它们存储在存储装置中由计算装置来执行,或者将它们分别制作成各个集成电路模块,或者将它们中的多个模块或步骤制作成单个集成电路模块来实现。本发明不限制于任何特定的硬件和软件的结合。
74.上述虽然结合附图对本发明的具体实施方式进行了描述,但并非对本发明保护范围的限制,所属领域技术人员应该明白,在本发明的技术方案的基础上,本领域技术人员不需要付出创造性劳动即可做出的各种修改或变形仍在本发明的保护范围以内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1