一种一次性检测多种肉源性成分的检测引物、方法及试剂盒与流程
本发明属于生物技术领域,具体涉及一种能够一次性检测多种肉源性成分的检测引物、检测方法及检测试剂盒。
背景技术:
:
对于肉源性成分的鉴定,普遍使用的是实时荧光定量PCR方法。该方法有较高的灵敏度,适合加工食品微量肉源成分检测,且能相对定量,因此该方法被广泛使用。但该方法存在一定的缺点,如有些物种的特异性探针和引物难以设计,以及一套探针、引物对应某特定的单一的物种成分。为解决这一问题,也有人使用通用引物和克隆测序方法,对多种肉源性成分进行检测。但这种方法需筛选阳性克隆,工作量大、成本高、还存在漏检的情况。而关于混合样品的检出方法,如多重PCR、限制性片段多态法(RFLP)、和单链构象多态(SSCP)、随机扩增多态片段法(RAPD)都属于传统的检测方法,前三种方法不能检测样品中未知的肉源性成分,而RAPD-PCR法则存在重复性差和假阳性、假阴性等问题。
如今,二代测序技术为物种鉴定的研究提供了一种新型的方法,并已经广泛应用于环境微生物的宏基因组的研究。因其高通量的优势可对混合样品中多种的、未知的物种成分进行鉴定。但由于二代测序有读长限制,对于100bp以上的序列,测序准确率会降低,而且混合样品不能直接用标准的DNA条形码(>500bp)进行物种鉴定,否则多重复片段的拼接会大大增加错误率。
技术实现要素:
:
本发明的目的是提供一种快速、灵敏、重复性高地检测多种肉源性成分的检测引物、检测方法及检测试剂盒,本发明设计一对全新的通用检测引物,以生鲜的肌肉组织为材料提取DNA,对提取DNA的方法进行了优化,结合二代测序技术,一次性检测出多种肉源性成分,并且还能够检出未知的肉源性成分,达到快速、灵敏、省时省力且准确度高的效果,从而实现了本发明的目的。
本发明的一次性检测多种肉源性成分的检测引物,其特征在于,所述的检测引物如下所示:
正向引物F:GGTCAACAAATCATAAAGATATTGG
反向引物R:ACTATAAAGAAGATTATTACAAAGGC
本发明的一次性检测多种肉源性成分的检测试剂盒,包括PCR反应液、Ex-Taq DNA聚合酶、裂解缓冲液、TE缓冲液、苯酚、和检测引物,其特征在于,所述的检测引物如下所示:
正向引物F:GGTCAACAAATCATAAAGATATTGG
反向引物R:ACTATAAAGAAGATTATTACAAAGGC
所述的裂解缓冲液为10mmol/L pH8.0的Tris-HCl,0.1mol/L pH8.0的EDTA和质量体积比(m/V)为0.5%的SDS。
所述的TE缓冲液为100mmol/L pH8.0的Tris-HCl和10mmol/L pH8.0的EDTA。
所述的苯酚为经0.1mol/L pH8.0的Tris-HCl平衡到pH值为8.0。
本发明的一次性检测多种肉源性成分的检测方法,用于检测肉类食品或者其他的混和的动物样品的物种鉴定,其特征在于,包括以下步骤:
(1)、采用蛋白酶K-苯酚法提取肉类样品的基因组DNA,具体为:将肉类样品研磨成肉糜,加入裂解缓冲液,每200μL的裂解缓冲液对应加20μL浓度为20mg/mL的蛋白酶K,水浴,再加入等体积的经0.1mol/L pH8.0的Tris-HCl平衡的苯酚,其pH值平衡到8.0,离心分离两相,转移水相再用苯酚抽提两次,收集水相再次用苯酚抽提第三次,转移水相加入醋酸铵及乙醇,离心收集DNA沉淀,70%酒精洗涤离心,加入TE缓冲液溶解产物,得到样品的基因组DNA提取液;
(2)、使用上述检测引物、PCR反应液及Ex-Taq DNA聚合酶对基因组DNA进行PCR扩增,得到目的扩增片段;
(3)、采用二代测序方法对上述目的扩增片段进行测序以明确样品中含有哪些肉源性成分。
所述的PCR扩增,优选,PCR扩增反应体系为:每50μL的反应体系中含有10×PCR Buffer5μL、正向引物(20nmol/μL)1μL、反向引物(20nmol/μL)1μL、dNTP(2.5mM)2μL、Ex-Taq(5U/μL)1μL、模板DNA(50ng/μL)5μL,并最终用双蒸水定容到50μL。
所述的PCR扩增,优选,PCR扩增反应程序为:94℃变性3min,98℃、10s;48℃、30s;68℃、1min;30个循环,68℃延伸7min。
所述的二代测序,优选,先测定各样品PCR产物的目的DNA片段的浓度,对于检测合格的样品构建文库,再用合格的文库进行cluster制备和高通量测序;测序采用Illumina HiSeq4000平台和PE150bp的测序策略,以及Q20(>85%)策略对测序的准确性进行控制;测序后对原始数据进行过滤处理,过滤低质量的reads,剩余高质量的序列(有效序列)用于后期分析;由原始序列数和有效序列数得出序列的有效利用率,以判断测序质量和DNA提取的效果,再做后续的序列聚类和注释;把97%相似度的有效序列聚成同一个操作分类单元(Operational taxonomic unit,OTU),然后通过OTU与数据库比对进行序列的物种注释;注释后,省去与库中参考序列低于98%相似度的序列,以保证序列物种注释的准确性。
本发明由原始序列数和有效序列数得出序列的有效利用率为96.59%,反映了测序质量和DNA提取的效果较好。OTU序列注释后,有98.18%的有效序列能准确鉴定到对应的物种,能与参考序列达到高于98%的相似度。
本发明相对于现有技术方法具有如下的优点及效果:无需每种肉源性成分对应一套特异性引物和探针;能一次性检出多种肉源性成分;省去纯化、连接、转化等繁琐的操作步骤;准确地检出未知的肉源性成分,且重复性高。其检测结果与传统的克隆测序方法相比,更全面,更高灵敏度,具有更广的适用性,可应用于多种肉源性成分的鉴定、未知肉源性成分的溯源和微量肉源性成分的检出。
附图说明:
图1是样品肉源性成分的克隆测序结果;
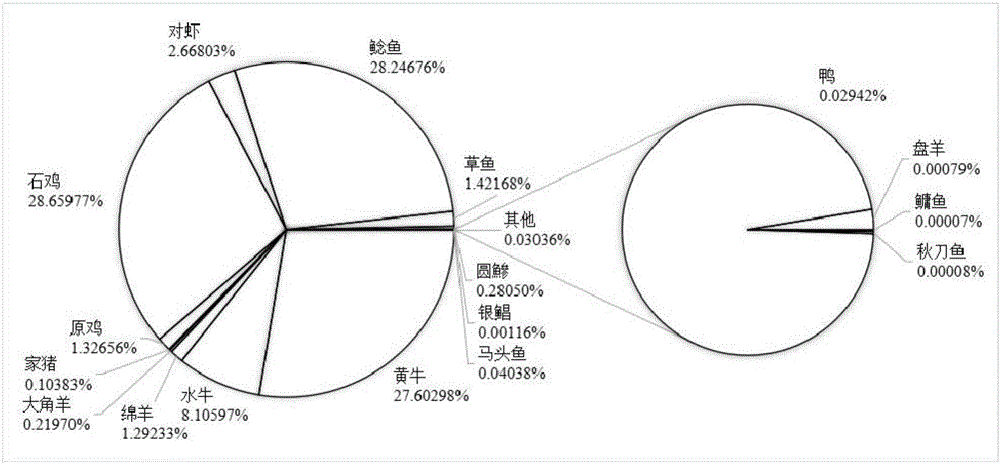
图2是样品肉源性成分的二代测序结果,各个物种的百分比源于三个平行样品中各个物种百分比的均值。
具体实施方式:
以下实施例是对本发明的进一步说明,而不是对本发明的限制。
实施例1:
一、通用检测引物的设计
从NCBI数据库中获取45个远缘物种序列,涵盖了哺乳动物(n=14)、鱼类(n=19)、鸟类(n=8)和甲壳纲类(n=4)。在标准DNA条形码COI的基础上,经软件分析,选出较理想的区域片段miniCOI,长度为136bp,既能满足一般的二代测序平台的读长限制,又具有适度的变异性使不同的物种得以区分,以及良好的保守性使同种个体聚类。
引物与模板序列的保守性:经多序列比对后,得出引物的保守碱基(阴影标记),与标准DNA条形码COI的通用引物相比,新设计的引物与模板间有更好的保守性,体现了该引物具有较好的通用性。
MiniCOI序列的引物(本发明的通用检测引物):
F(5’-3’) R(5’-3’)
GGAAAATATATT--------AGGTTAG
标准COI序列的引物:
F(5’-3’) R(5’-3’)
GGAAAATATATT--------ATAGAAATC
二、检测
1、基因组DNA提取
用传统的试剂盒提取全基因组DNA时,以过柱方式获取膜上DNA会产生约30%的损失,会对混合样品中微量DNA成分的检测产生一定的影响。而优化后提取方法不需滤过吸附柱,而选用蛋白酶K-苯酚法提取,以尽量减少DNA的损失。
从中山市某市场购买了一些常见的生鲜肉,包括鱼(5种)、鸡(2种)、猪(1种)、牛(2种)、羊(2种)和虾(1种)。肉类样品在实验处理前放置-20℃储存。选定多种不同的肉作为混合样品的组成成分。混合前,选取肉的内部肌肉组织,用无菌的剪刀剪取0.02g。为了防止不同肉之间的交叉污染,样品处理时每把剪刀都经高温高压和照紫外处理,而且在同一次混合操作中,一种肉对应一把剪刀。每种肉在重量上等比例混合,形成一个混合样品,再进行样品基因组DNA的提取,具体步骤如下:
A.把混合肉类样品置于1.5mL的离心管中,用无菌研磨棒把混合肉样品研磨成糜状;
B.加入200μL的裂解缓冲液,混匀后再加20μL的浓度为20mg/mL的蛋白酶K,56℃的水浴3个小时,每隔15min旋转黏滞的溶液,充分裂解后,用移液枪不断吹打混匀,按体积等比例分成三份,形成三个平行样品;
C.待溶液冷却至室温,加入与步骤B所获得的溶液相等体积的pH值8.0的苯酚(经0.1mol/L Tris-HCl,pH8.0平衡),将离心管置于旋转器上缓慢颠转10min以温和地混合两相,溶液呈乳浊状则证明两相充分混合;
D.室温12000rpm离心15min以分离两相;
E.用宽口的移液管(出口直径0.3cm)将滞留的水相转移至另一离心管;
F.用苯酚再抽提两次,收集水相;
G.第三次用苯酚抽提后,将水相转至另一离心管中,加入0.2倍体积10mol/L醋酸铵及2倍体积乙醇,旋转离心管至溶液彻底混匀为止,室温12000rpm离心5min,收集DNA沉淀;
H.用70%酒精50μL洗涤DNA沉淀2次,室温12000rpm离心5min,小心移除液体,管口敞开,室温晾干直至可见的痕量乙醇挥发殆尽;
I.加入50μL的TE缓冲液(pH8.0)溶解沉淀,获得样品基因组DNA提取液。
2、PCR扩增反应
对上述各个基因组DNA提取样品进行目的DNA序列的扩增获取。
PCR扩增反应体系为:每50μL的反应体系中含有10×PCR Buffer 5μL、正向引物(20nmol/μL)1μL、反向引物(20nmol/μL)1μL、dNTP(2.5mM)2μL、Ex-Taq(5U/μL)1μL、模板DNA(50ng/μL)5μL,并最终用双蒸水定容到50μL。PCR扩增反应程序为:94℃变性3min,98℃、10s;48℃、30s;68℃、1min;30个循环,68℃延伸7min。获得目的扩增序列。
3、不同测序方法的数据产生、处理与分析
克隆测序
为了比较一代测序与二代测序应用于多种肉源性成分检测上的差异,如结果的准确性、检测方法的灵敏度和工作效率等,所以在同样的扩增条件下,取3个平行的扩增产物继续进行传统的挑克隆方法。产物经纯化后把目的片段连接到pGM-T载体(购自天根生化科技(北京)有限公司),再把载体转化至DH5α大肠杆菌中。菌液涂平板后,于37℃过夜培养。蓝白斑测试检验阳性克隆。每个样品选取130个阳性克隆菌,于37℃摇菌过夜培养。菌液经过稀释,提取质粒后进行一代测序。
测序后产生的序列经过寻找引物,选取两端引物以内的序列部分与NCBI数据库比对,再把与参考序列的相似度达到98%的序列进行物种注释。三个样品中共有374条序列(占总序列的95.89%)能测序成功并注释。
二代测序
测定各个PCR产物的目的DNA片段的浓度,对于检测合格的样品构建文库,用合格的文库进行cluster制备和高通量测序。测序采用Illumina HiSeq4000平台和PE150bp的测序策略,以及Q20(>85%)策略对测序的准确性进行控制。
测序后对原始数据进行过滤处理,过滤低质量的reads,剩余高质量的序列(有效序列)用于后期分析。由原始序列数和有效序列数得出序列的有效利用率,以判断测序质量和DNA提取的效果,再做后续的序列聚类和注释。把97%相似度的有效序列聚成同一个操作分类单元(Operational taxonomic unit,OTU),然后通过OTU与数据库比对进行序列的物种注释。注释后,省去与库中参考序列低于98%相似度的序列,以保证序列物种注释的准确性。
由原始序列数和有效序列数得出序列的有效利用率为96.59%,反映了测序质量和DNA提取的效果较好。OTU序列注释后,有98.18%的有效序列能准确鉴定到对应的物种,能与参考序列达到高于98%的相似度。
4、不同测序方法的结果比对
克隆测序
如图1所示,在挑克隆的丰度为混合的物种数的10倍的情况下,能检测出10种肉源性成分,其余的3个物种中有一个物种在PCR扩增中呈现阴性结果,其他两个物种均能被单独的扩增和克隆,而最终没有被检测出来。该方法虽然能检测出大部分的成分,但有一种常见的鸡肉成分未被检出。经过实时荧光定量PCR对样品中鸡源性成分的检测,结果呈阳性,由此证明了该方法的缺点,即人为筛选或丰度不足而致的假阴性。
二代测序
如图2所示,二代测序方法能把混合样品中的13种肉源性成分全都被检出,所扩增出的目的片段序列分别如SEQ ID1-13所示,其中一种肉源性成分可能由于PCR产物含量低而在琼脂糖凝胶电泳中呈阴性,却在该方法中呈阳性。除了这13种已知成分,还有4种未知的肉源性成分也一并检出,包括鸭、鳙鱼、秋刀鱼、盘羊,所扩增出的目的片段序列分别如SEQ ID14-17所示。这四种成分是在预知以外的,很大可能源于采购样品时,某肉类样品曾与其他肉类样品有接触而被粘连或附着,即使微量的成分也能被检出。
经结果比对,使用本发明的通用检测引物进行扩增并结合二代测序方法具有更高的灵敏度、省时省力、而且保持一定的准确度。不仅能避免传统克隆方法中假阴性结果的缺点,还能检出未知的肉源性成分。因此,本发明具有更高的适应性和应用性,可应用于多种肉源性成分的鉴定、未知肉源性成分的溯源和微量肉源性成分的检出。
序列表
<110>中山出入境检验检疫局检验检疫技术中心
<120>一种一次性检测多种肉源性成分的检测引物、方法及试剂盒
<160>18
<210>1
<211>136
<212>DNA
<213>鲶鱼(Pangasianodon hypophthalmus)
<400>1
CACCCTCTAC CTAGTATTTG GTGCCTGAGC AGGAATAGTT GGTACGGCCC TCAGCCTCCT 60
AATTCGGGCA GAGCTAGCCC AACCCGGCGC CCTTCTAGGC GACGACCAAA TTTATAATGT 120
TATTGTCACT GCCCAT 136
<210>2
<211>136
<212>DNA
<213>马头鱼(Branchiostegus argentatus)
<400>2
CACCCTTTAT TTAGTATTTG GTGCTTGAGC CGGTATAGTA GGCACAGCCT TAAGCTTGCT 60
CATTCGAGCA GAACTTAGCC AACCAGGCGC CCTCCTCGGG GATGACCAGA TTTATAATGT 120
TATTGTTACA GCACAT 136
<210>3
<211>136
<212>DNA
<213>圆鰺(Decapterus maruadsi)
<400>3
CACCCTTTAT CTAGTATTTG GTGCTTGAGC TGGAATAGTA GGAACTGCTT TAAGCCTACT 60
TATTCGGGCA GAATTAAGCC AACCTGGCGC CCTTCTAGGG GATGACCAAA TTTACAACGT 120
AATTGTTACG GCCCAC 136
<210>4
<211>136
<212>DNA
<213>草鱼(Ctenopharyngodon idella)
<400>4
TACCCTCTAT CTTGTATTTG GKKSCTGAGC CGGAATAGTG GGAACCGCTC TAAGCCTTCT 60
CATTCGAGCC GAACTAAGCC AACCCGGATC ACTTCTGGGC GATGATCAAA TTTATAATGT 120
TATTGTCACT GCCCAT 136
<210>5
<211>136
<212>DNA
<213>对虾(Litopenaeus vannamei)
<400>5
AACATTATAC TTTATCTTCG GGGCTTGAGC TGGAATAGTA GGTACCGCTC TTAGACTTAT 60
TATCCGAGCT GAATTAGGTC AACCTGGGAG CCTCATTGGG GATGATCAAA TTTATAACGT 120
AGTTGTCACA GCTCAC 136
<210>6
<211>136
<212>DNA
<213>原鸡(Gallus gallus)
<400>6
CACTCTTTAC CTAATTTTCG GCACATGGGC GGGCATAGCC GGCACAGCAC TTAGCCTTCT 60
AATTCGCGCA GAACTAGGAC AGCCCGGAAC TCTCTTAGGA GACGATCAAA TTACAAATGT 120
AATCGTCACA GCCCAT 136
<210>7
<211>136
<212>DNA
<213>石鸡(Alectoris chukar)
<400>7
CACCCTTTAT CTAATTTTCG GCACATGAGC AGGTATAGCC GGCACAGCAC TTAGCCTGCT 60
TATTCGCGCA GAACTAGGAC AACCGGGCAC CCTCTTAGGA GATGACCAAA TCTATAATGT 120
AATCGTTACA GCCCAT 136
<210>8
<211>136
<212>DNA
<213>绵羊(Ovis aries)
<400>8
CACCCTTTAC CTTCTATTTG GTGCCTGAGC TGGTATAGTA GGAACCGCCT TAAGCCTACT 60
AATTCGCGCC GAACTAGGCC AACCCGGAAC TCTACTCGGA GATGACCAAA TCTACAACGT 120
AATTGTAACC GCACAT 136
<210>9
<211>136
<212>DNA
<213>大角羊(Ovis canadensis)
<400>9
TACCCTATAC TTACTATTTG GTGCCTCAGC CAACATAGTA GGAACAGCCC TAAGCCTGCT 60
GATTCGTGCT GAACTAGGTC AATGTGGAAC CTTGCTCAGA GATGACCAGA TTCATAATGT 120
AATTGTAACT GCACAC 136
<210>10
<211>136
<212>DNA
<213>黄牛(Bos taurus)
<400>10
TACCCTTTAT CTACTATTTG GTGCTTGGGC CGGTATAGTA GGAACAGCTC TAAGCCTTCT 60
AATTCGCGCT GAATTAGGCC AACCCGGAAC TCTGCTCGGA GACGACCAAA TCTACAACGT 120
AGTTGTAACC GCACAC 136
<210>11
<211>136
<212>DNA
<213>水牛(Bubalus bubalis)
<400>11
CACCCTGTAC TTGCTATTTG GTGCCTGAGC CGGCATAGTA GGGACAGCCC TAAGCCTGCT 60
GATTCGCGCT GAATTAGGTC AACCTGGGAC CCTACTCGGA GATGACCAAA TCTACAACGT 120
AGTTGTAACC GCACAC 136
<210>12
<211>136
<212>DNA
<213>家猪(Sus scrofa)
<400>12
CACCCTGTAC CTACTATTTG GTGCCTGAGC AGGAATAGTG GGCACTGCCT TGAGCCTACT 60
AATTCGCGCT GAACTAGGTC AGCCCGGAAC CCTACTTGGC GATGATCAAA TCTATAATGT 120
AATTGTTACA GCTCAT 136
<210>13
<211>136
<212>DNA
<213>银鲳(Pampus chinensis)
<400>13
CACCCTTTAT CTAGTATTTG GTGCATGAGC TGGTATAGTA GGCACAGCCC TAAGCTTACT 60
TATTCGAGCT GAATTAAACC AACCAGGTGC CCTCCTTGGG GATGACCAAA TTTATAATGT 120
AATCGTTACA GCACAT 136
<210>14
<211>136
<212>DNA
<213>鸭(Anser canagica)
<400>14
CACTCTTTAC CTAATTTTCG GCACATGGGC GGGCATAGCC GGCACAGCAC TTAGCCTTCT 60
AATCTGCGCA GAACTAGGAC AGCCCGGAAC TCTCTTAGGA GACGACCAAA TCTATAATGT 120
AATCGTTACA GCCCAC 136
<210>15
<211>136
<212>DNA
<213>鳙鱼(Hypophthalmichthys nobilis)
<400>15
TACCCTTTAT CTTGTATTTG GTGCCTGAGC CGGAATAGTG GGAACCGCCC TAAGCCTTCT 60
CATTCGAGCC GAACTAAGCC AACCCGGATC ACTTCTGGGC GATGACCAAA TTTATAACGT 120
TATTGTTACT GCCCAT 136
<210>16
<211>136
<212>DNA
<213>秋刀鱼(Cololabis saira)
<400>16
CACCCTTTAT CTAGTATTTG GTGCTTGAGC TGGAATAGTA GGCACTGCCC TAAGCCTTCT 60
TATTCGAGCA GAACTAAGTC AACCGGGCTC TCTTCTAGGA GATGATCAAA TTTATAATGT 120
CATCGTTACA GCCCAC 136
<210>17
<211>136
<212>DNA
<213>盘羊(Ovis ammon darwini)
<400>17
CACCCTTTAC CTCCTATTTG GTGCCTGAGC TGGCATAGTA GGAACCGCCT TAAGCCTACT 60
AATTCGCGCC GAACTAGGCC AACCCGGAAC TCTACTTGGA GATGATCAAA TCTACAACGT 120
AATTGTAACC GCACAC 136
- 还没有人留言评论。精彩留言会获得点赞!