一种目标区域多重PCR与快速文库构建的方法与流程
本发明属于遗传学与精准医学领域,涉及到一种靶向特异区段超高重PCR与快速文库构建的新方法。
背景技术:
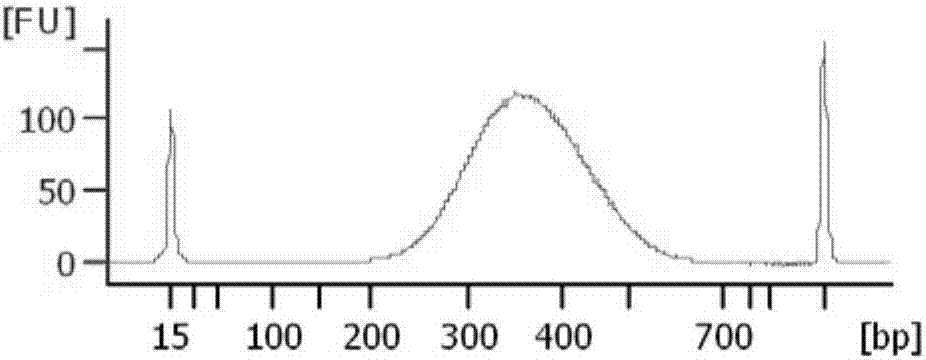
:多重PCR技术在遗传学与精准医学领域最大的价值在于它能提高诊断的敏感性。敏感性可以从两方面分析:检测敏感性和临床敏感性。检测敏感性强调的是检测结果:如果标本中含有被检测物,检测成功的几率是多少。如果是PCR反应,就是能检测到的最低拷贝数。临床敏感性是从临床角度看问题:实验结果给病人出诊断的成功率有多高。如果一个病仅有一个病因,检测敏感性和临床敏感性无差别。如果疾病的诊断需要同时分析多病因,仅对一个指标进行检测,检测敏感性再高其临床敏感性也要大打折扣。qPCR的检测敏感性是无可争议的,可是因为无法做多重,所以临床敏感性不够好。而多重PCR临床敏感性要高很多。精准医学时代,靶向特异区段重测序的需求日益增加,样本量常达上千,只用传统的Sanger法或目标区域杂交捕获测序价格昂贵。因此基于多重或超高重PCR的扩增子测序适用性强,可以针对连续区段,外显子靶标以其他指定位点,设计特异引物,一次获得所有位点,有着重要的临床意义和广泛的应用前景,但是仍然存在很多限制性因素,其中多重PCR扩增效率的一致性一直是最大的难题,扩增子越多、不均一性越高等。本领域需要解决多重PCR扩增效率的一致性。技术实现要素:本发明旨在通过多重PCR快速构建DNA文库并解决其中多重PCR扩增效率的一致性问题。具体地:本发明提供一种通过多重PCR扩增DNA并构建文库的方法,包括:(1)获取样本DNA(2)使用添加了锚定接头且经过修饰的特异性引物对的集合,对步骤(1)得到的样本DNA进行第一轮PCR,所述特殊接头为且经过修饰的特异性引物对具有如下结构:5’修饰的锚定接头序列+针对待扩增区域的特异性序列(3)不添加新的引物,将步骤(2)得到的产物在新的反应体系中进行第二轮PCR,使得全部引物被用于扩增,(4)使用特殊设计的文库构建引物对,以所述特异性引物对上的特殊接头为引物锚定区域,进行扩增的同时在包含目标基因/区段的扩增子两端加上文库接头,所述文库构建引物对包含的引物具有如下结构(5’到3’)5’修饰的文库接头+锚定接头序列(5)构建文库。在一个实施方案中,所述特异性引物对的集合包括50-2000个引物对。在一个实施方案中,所述待扩增区域的特异性序列的长度为5-20kb个碱基。在一个实施方案中,步骤(2)所述的5’修饰是NH2-C6修饰。在一个实施方案中,步骤(2)中正向引物的锚定接头序列为:5’-NH2-C6-GCTCTTCCGATCTMM(SEQIDNO:1),M为A或C;步骤(2)中反向引物的锚定接头序列为:5’-NH2-C6-GCTCTTCCGATCTKK(SEQIDNO:2),M为G或T。在一个实施方案中,步骤(4)所述的5’修饰是NH2-C6修饰。在一个实施方案中,其中所述文库构建引物的引物之一还包括索引(index)序列,所述索引序列为4-10个任意碱基,作为区分文库的标签。在一个优选实施方案中,步骤(4)中正向引物的文库接头为:5’NH2-C6-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGAC(SEQIDNO:3)反向引物的文库接头为:5’NH2-C6-CAAGCAGAAGACGGCATACGAGATNNNNNNGTGACTGGAGTTCAGACGTGT(SEQIDNO:4),其中N为任意碱基。在一个优选实施方案中,步骤(4)中正向引物(即文库构建正向引物)为:5’NH2-C6-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCTMM(SEQIDNO:5)反向引物为(即文库构建反向引物):5’NH2-C6-CAAGCAGAAGACGGCATACGAGATNNNNNNGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTKK(SEQIDNO:6)其中M为A或C,K为G或T,NNNNNN为索引序列,N为任意碱基。在一个示例中,第一轮PCR中添加了锚定接头且经过修饰的特异性正向引物为:5’NH2-C6-GCTCTTCCGATCTMMx......x(x......x代表特异性区域);添加了锚定接头且经过修饰的特异性反向引物为:5’NH2-C6-GCTCTTCCGATCTKKx......x(x......x代表特异性区域);第二轮PCR不添加引物。第三轮PCR所用的文库构建正向引物为:5’NH2-C6-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCTMM;文库构建反向引物为:5’NH2-C6-CAAGCAGAAGACGGCATACGAGATNNNNNNGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTKK(NNNNNN为索引序列,N为任意碱基);通过上述三轮PCR所获得的文库结构为:5’-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCT[MMx......xMM]AGATCGGAAGAGCACACGTCTGAACTCCAGTCACNNNNNNATCTCGTATGCCGTCTTCTGCTTG-3’。有益效果:本发明能在2-3小时内,由2.5-50ng经提取的DNA快速构建文库。具体地,以添加了不同特殊接头且经过修饰的多个特异性正反向引物对,对DNA上的目标靶点进行第一轮扩增。为了保证各个目标区域被均等的扩增出来,我们将第一轮PCR产物投入到新的反应体系中,但不加入新的引物,以图引物被完全消耗从而保证各个目标区域扩增的均一性。在第三轮PCR中,以特异性正反向引物对上的特殊接头为引物锚定区域,进行扩增的同时在包含目标基因/区段的扩增子两端加上Illumina文库接头,以获得符合下游分析要求的高质量文库,并且在建库过程中引物端自动加上样本区分barcode,无需二次建库。下机数据各扩增子均一性高,1MReads前提下,99.5%扩增子上的热点达到1200X覆盖;特异性高,100%的靶序列均能被有效扩增。本发明文库接头的添加具有方向性,可提高数据利用率和文库质量。附图说明图1是用Agilent2100Bioanalyzer检测制备DNA文库中的片段长度分布范围的结果。L:DNALadder;S:使用20ngcfDNA构建的多重PCR产物文库,磁珠进行选择回收后结果。图2是用Agilent2100Bioanalyzer检测制备DNA文库中的片段长度分布范围的结果。图3是采用Samtools软件分析测序结果得到的位点测序覆盖深度数据,将各位点的深度做Log运算,然后将位点名称和深度的Log值反应在柱状图中,得到位点覆盖均一性数据(模板为CellFreeDNA,cfDNA),其中横坐标为位点所在的基因名称和Cosmic数据库查询编号,纵坐标为测序覆盖深度数据的Log值,显示热点最大碱基覆盖度:93470X,最小覆盖度:256X。具体实施方式为更进一步阐述本发明所采取的技术手段及其效果,以下通过具体实施例来进一步说明本发明的技术方案,但本发明并非局限在实施例范围内。实施例1多重PCR扩增步骤一:获取样本的DNA采用全血为样本,选择合适的市售常规cfDNA提取试剂盒提取cfDNA,依据说明书进行提取。步骤二:第一轮PCR富集目标片段根据下表配制PCR反应缓冲体系注意:X和Y根据引物混合物储存液浓度(通常为100uM)和引物对数量进行调整本实施例所用正向引物混合物和反向引物混合物共包括针对人肺癌突变热点(例如EGFR基因p.E746_A750delELREA、p.V769_D770insASV、p.T790M、p.C797S、p.L858R)设计的92对特异性引物,所述特异性引物包括5’修饰的锚定接头序列+针对待扩增区域的特异性序列,所述5’修饰的锚定接头序列为5’-NH2-C6-GCTCTTCCGATCTMM(对于正向引物)或5’-NH2-C6-GCTCTTCCGATCTKK(对于反向引物),此例中X为1.09,Y为0.688。PCR条件如下:步骤三:第二轮PCR消耗多余引物根据下表配制PCR反应缓冲体系:PCR条件如下:此步骤完成扩增后,取下离心,将产物200倍稀释,即1uL产物溶于199uLNucleaseFreeWater,混匀后取出20uL开展后续实验。步骤四:第三轮PCR添加文库接头根据下表配制PCR反应缓冲体系:其中Illumina_MM通用引物为:5’NH2-C6-AATGATACGGCGACCACCGAGATCTACACTCTTTCCCTACACGACGCTCTTCCGATCTMM(SEQIDNO:5)Illumina_KK索引引物为:5’NH2-C6-CAAGCAGAAGACGGCATACGAGATNNNNNNGTGACTGGAGTTCAGACGTGTGCTCTTCCGATCTKK(SEQIDNO:6),其中M为A或C;K为G或T。PCR条件如下:实施例2文库构建和检测步骤一:纯化1)将扩增产物转移至离心管中,取0.8×(40uL)AmpureXP磁珠或CMpure磁珠,与扩增产物混匀后置于磁力架上静置10min;2)待磁珠全部吸附到管壁上(约5min),弃上清后用新配制的80%乙醇清洗磁珠两次,弃上清;3)室温静置5min,待磁珠干燥后(请注意不要过分干燥致使磁珠开裂,以免影响回收效率),根据下游需要以17.5uLTEbuffer、EBbuffer或去核酸水重悬磁珠;4)室温静置5min后将离心管置于磁力架上,吸取15uL上清,此上清为多重PCR文库。步骤二:文库质量检测1.文库浓度为了得到高质量的测序结果,需要对文库进行精确定量,首先推荐使用RealtimePCR方法对DNA文库进行绝对定量。此外,还可使用荧光染料法,如Qubit法或荧光染料Picogreen法,此处请勿使用基于吸光度测量的定量方法。最终可使用以下近似公式换算DNA文库的摩尔浓度。2.文库长度分布制备好的DNA文库可用琼脂糖凝胶电泳或Agilent2100Bioanalyzer检测DNA文库中的片段长度分布范围。结果见图1。3.测序上机(数据量为1Mreads)将文库以Illumina公司Next550测序仪进行测序。测序结果如图3和下表1-2所示。表1以人类cfDNA为模板(梯度投入),针对肺癌突变热点设计92对特异性引物,扩增测序后数据情况(此表格由Samtools软件输出):表2热点变异检出灵敏性数据(固定比例阳性样本掺合):突变名称掺合比例检出的突变频率EGFR基因L858R2.92%2.29%EGFR基因T790M1%1.52%EGFR基因L861Q1.78%2.01%本发明经测序后,下机数据显示:位点覆盖深度高,各个扩增子均一性好;固定掺合比例的突变核酸掺入的测试显示:与掺合比例相比没有数量级的差异,说明检测灵敏性好。申请人声明,本发明通过上述实施例来说明本发明的详细方法,但本发明并不局限于上述详细方法,即不意味着本发明必须依赖上述详细方法才能实施。所属
技术领域:
的技术人员应该明了,对本发明的任何改进,对本发明产品各原料的等效替换及辅助成分的添加、具体方式的选择等,均落在本发明的保护范围和公开范围之内。序列表<110>上海亿康医学检验所有限公司<120>一种目标区域多重PCR与快速文库构建的方法<141>2017-12-15<160>6<170>SIPOSequenceListing1.0<210>1<211>15<212>DNA<213>人工序列()<220><221>misc_feature<222>(1)<223>5‘端被NH2-C6基团封闭<400>1gctcttccgatctmm15<210>2<211>15<212>DNA<213>人工序列()<220><221>misc_feature<222>(1)<223>5‘端被NH2-C6基团封闭<220><221>misc_feature当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1