与乳腺癌相关的染色体相互作用的检测的制作方法
本发明涉及检测染色体相互作用。
背景技术:
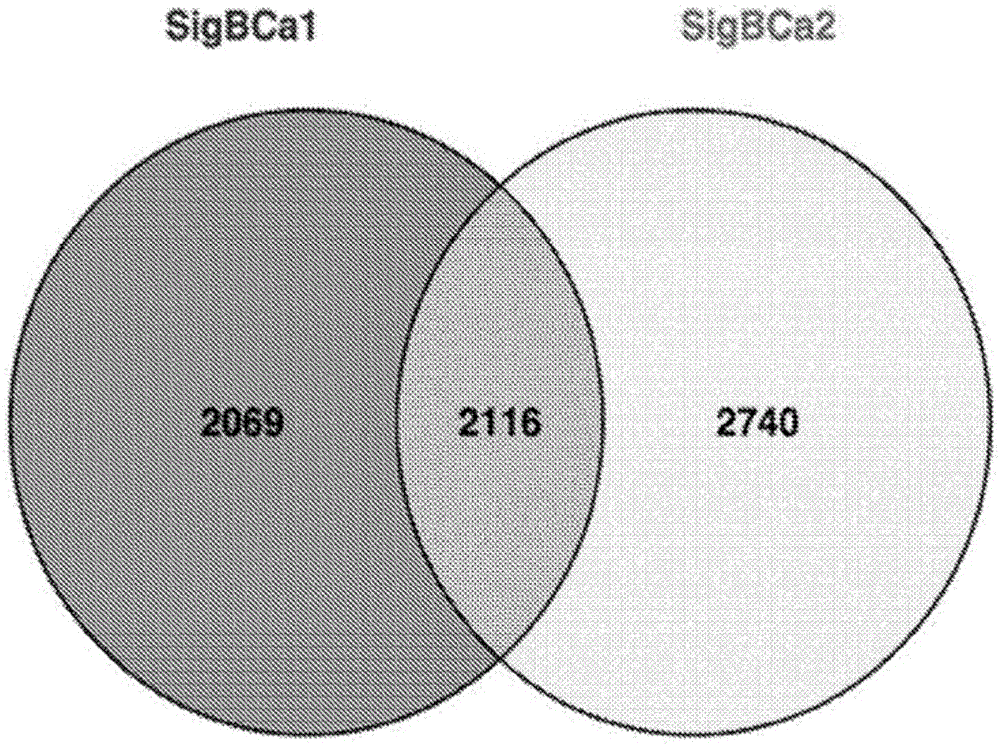
:癌症是由细胞生长和分裂中的调控丧失引起的。当细胞dna中的突变发生而细胞无法修复该突变时发生这种情况,该突变可以是遗传的(种系)或发育的(后天的)。存在两种类型的癌症:良性的和恶性的,当细胞分裂中的调控丧失发生但肿瘤未扩散到身体的其他部位时,良性癌症发生。恶性(或转移性)癌症是更严重的并且当癌细胞经由血流或淋巴系统迁移到身体的其他部位时发生。乳腺癌是对于从乳腺开始的癌症的名称,并且是全世界第二大常见癌症。2012年估计发生了1410万新的癌症病例。目前使用乳房x线照片的癌症筛查是检查任何乳腺异常的黄金标准,并且如果检测到肿块,则进行活组织检查。侵袭性乳腺癌的组织学分级用于将具有侵袭性乳腺癌的患者分成具有不同的预后的三组:良好、中等和差。技术实现要素:由于与病理或治疗关联的调控表观遗传控制设置,在基因座处的特异性染色体构象标签/标志/特点(ccs)存在或不存在。ccs具有温和的解离速率,并且当代表特定的表型或病理时,它们将仅随着生理信号转换而改变为新的表型,或这是外部干预的结果。此外,这些事件的测量是二元的,并且因此该读数与不同水平的dna甲基化、组蛋白修饰和大多数非编码rna的连续读数形成鲜明对比。迄今为止用于大多数分子生物标志物的连续读数对数据分析提出了挑战,因为特定生物标志物的变化的幅度在患者与患者之间变化很大,当它们被用于对患者群组分层时,其引起分类统计数据的问题。这些分类统计数据更适合使用不存在该幅度的生物标志物,并且仅提供表型差异的“是或否”二元评分——预示episwitchtm生物标志物是潜在诊断、预后和预测性生物标志物的优秀资源。本发明人已经使用允许识别群体中亚组的方法识别了染色体相互作用与乳腺癌相关的基因组区域。因此,本发明提供了用于检测染色体状态的工艺(方法),该染色体状态代表群体中的亚组,包括确定在基因组的限定的疾病相关的区域内存在或不存在染色体相互作用,其中所述疾病是乳腺癌。通过确定哪种染色体相互作用与对应于群体的乳腺癌亚组的染色体状态相关的方法,染色体相互作用可任选地已经被识别或是可识别的(或可导出的),该方法包括将来自具有不同染色体状态的亚组的第一核酸集与第二指标核酸集接触,并且允许互补序列杂交,其中第一和第二核酸集中的核酸代表(表现为)连接产物,包括来自已经在染色体相互作用中聚集在一起的两个染色体区域的序列,并且其中第一和第二核酸集之间的杂交模式允许确定哪种染色体相互作用对乳腺癌亚组是特异性的。附图说明图1.来自bca1和bca2阵列的显著性探针的比较。探针调整p值<0.05。图2.此维恩图(venndiagram)示出了患者集分为标志物减少集(118,集1)和模型验证集(50,集2)。另一患者集指的是从位点2集群(shipment)122中排除的对照。图3.glmnet模型的交叉验证图用于选择λ(逻辑模型的惩罚值)和系数(在最小误差处)。y轴是均方误差。x轴是log(λ)。图4.对于集群122的异常值质量控制。使用来自最终brca模型的8个标志物对来自位点2的对照样品的因子分析(主成分分析)图。显示为三角形(位点2_b2)的患者来自集群122(30名患者),并且这些被排除在总分析之外,而显示为圆形(位点_2)的患者是来自集群113(25名患者)的位点2对照,其被使用。该图显示,在来自相同位置的这些对照内,存在大的变化成分,其将在与brca相比较的背景下意味着在生物学上相似的患者分开。这是值得关注的,因为这种变化将在最终模型中竞争brca与对照之间的差异;因此,位点2批次2集群122样品被移除。y轴为dim216.79%。x轴为dim118.11%。图5示出了使用来自最终brca模型的8个标志物对分析中使用的所有对照样品(69个对照)的因子分析(主成分分析)图。患者中存在轻微的地域变化,但这小于位点2对照(集群122)的异常值组的变化。圆形是位点1。三角形是位点2。深色正方形是位点3。交叉形是位点4。交叉方框是位点5。y轴为dim211.57%。y轴为dim115.47%。图6示出了使用来自最终brca模型的8个标志物对分析中使用的所有对照样品(69个对照)的因子分析(主成分分析)图,加上位点2的30个排除的异常值组(集群122显示为交叉圆形)。数据的分散主要是由于位点2集群122中的问题。深色菱形是位点1。深色圆形是位点2。深色正方形是位点3。深色三角形是位点4。交叉形是位点5。浅色三角形是位点6。交叉圆形是位点2_b2。y轴为dim211.14%。x轴为dim112.36%。图7示出了关于标志物atm_11_108118137_108126372_108155279_108156687_rf的结果。第一个图示出了用atm引物54和56扩增,472bp分析二。第二个图示出了标准曲线。使用fam。发现效率为91.7%,r2为0.996,斜率为-3.539,y轴截距(y-int)为39.706。图8示出了关于atm_11_108118137_108126372_108155279_108156687_rf的扩增线。第一个图示出了用atm引物54和56扩增,472bp分析二,行c。第二个图示出了用atm引物54和56扩增,472bp分析二,行d。图9示出了关于标志物cdc6_17_38421089_38423079_38451196_38457050_ff的结果。第一个图示出了pcr2cdc6ff的扩增。第二个图示出了标准曲线。使用fam。发现效率为90.7%,r2为0.990,斜率为-3.568,y轴截距为40.652。图10示出了关于标志物foxc1_6_1577253_1581989_1604206_1605973_fr的结果。第一个图示出了atm208bpfoxc1的扩增。第二个图示出了标准曲线。使用fam。发现效率为101.6%,r2为0.992,斜率为-3.284,y轴截距为37.746。图11示出了关于标志物foxc1_6_1577253_1581989_1604206_1605973_fr的扩增线。第一个图示出了用atm208bp的扩增,行c。第二个图示出了用atm208bp的扩增,行d。图12示出了关于标志物map3k1_5_56102259_56110500_56140227_56144076_ff的结果。第一个图示出了扩增pcr9map3k1细胞c1-c6(rfu相对于循环)。第二个图示出了关于c1至c6孔的熔融峰。y轴示出-d(rfu)/dt。x轴示出了摄氏温度。图13示出了关于标志物map3k1_5_56102259_56110500_56140227_56144076_ff的结果。第一个图示出了map3k1495bp的扩增。第二个图示出了标准曲线。使用fam。发现效率为91.9%,r2为0.999,斜率为-3.533,y轴截距为40.940。图14示出了关于标志物me3_11_86300063_86304401_86420537_86426200_fr的结果。第一个图示出了扩增me3pcr12,a7-a12(rfu相对于循环)。第二个图示出了熔融峰。y轴示出了-d(rfu)/dt。x轴示出了摄氏温度。图15示出了关于标志物me3_11_86300063_86304401_86420537_86426200_fr的结果。第一个图示出了me3291bp的扩增。第二个图示出了标准曲线。使用fam。发现效率为96.8%,r2为0.998,斜率为-3.400,y轴截距为39.596。图16示出了关于标志物melk_9_36577630_36579243_36637050_36643005_rf的结果。第一个图示出了melk207bp的扩增。第二个图示出了标准曲线。使用fam。发现效率为91.3%,r2为0.995,斜率为-3.550,y轴截距为42.000。图17示出了关于标志物msh3_5_80021913_80025030_80153948_80159012_rf的结果。第一个图示出了msh3207bp的扩增。第二个图示出了标准曲线。使用fam。发现效率为97.1%,r2为0.990,斜率为-3.394,y轴截距为41.876。图18示出了关于标志物nf1_17_29477103_29483764_29651799_29657368_ff的结果。第一个图示出了nf1401bp的扩增。第二个图示出了标准曲线。使用fam。发现效率为99.0%,r2为0.987,斜率为-3.347,y轴截距为40.192。图19示出了关于标志物srd5a1_5_6634973_6639025_6667775_6669711_rf的结果。仅示出了标准曲线,没有患者数据。第一个图示出了srda51的扩增。第二个图示出了标准曲线。使用fam。发现效率为95.5%,r2为0.997,斜率为-3.434,y轴截距为39.761。图20示出了关于标志物tspyl5_8_98276431_98282736_98316421_98318720_ff的结果。仅显示标准曲线,没有患者数据。第一个图示出了tspyl5的扩增。第二个图示出了标准曲线。使用fam。发现效率为94.2%,r2为0.998,斜率为-3.469,y轴截距为41.344。图21示出了对应于实施例2的标志物集2的数据。环检测数据示出了标志物是否与癌症疾病样品或对照样品相关。图22示出了对应于实施例3的标志物集3的数据。环检测数据示出了标志物是否与恶性疾病样品或对照样品相关。示出的统计数据用于巢式pcr工作。对于示出了扩增曲线的所有图,y轴是rfu,x轴是循环;并且对于行c中的孔,患者样品的扩增线用x标记,在行d中,患者扩增曲线用三角形(δ)指示。对于示出了标准曲线的所有图,y轴为cq,x轴为log起始量;并且圆形是标准,并且交叉形是未知。具体实施方式本发明的工艺本发明的工艺(方法)包括用于检测与乳腺癌相关的染色体相互作用的分型系统。这种分型可以使用本文提到的episwitchtm系统实行,其基于已经在染色体相互作用中聚集在一起的染色体的交联区域,使染色体dna经历断裂,并且然后连接存在于交联实体中的核酸,以获得具有来自形成染色体相互作用的两个区域的连接核酸。该连接核酸的检测允许确定特定染色体相互作用的存在或不存在。可以使用上述方法识别染色体相互作用,在该方法中使用第一和第二核酸的群体。这些核酸也可以使用episwitchtm技术产生。与本发明相关的表观遗传相互作用如本文所用,术语“表观遗传”和“染色体”相互作用通常是指染色体远端区域之间的相互作用,所述相互作用是动态的,并且取决于染色体区域的状态改变、形成或破坏。在本发明的特定工艺中,通过首先产生包括来自作为相互作用的部分的染色体的两个区域二者的序列的连接核酸来检测染色体相互作用。在这些工艺中,可以通过任何合适的手段交联该区域。在优选的实施方式中,相互作用使用甲醛交联,但也可以通过任何醛或d-生物素基-e-氨基己酸-n-羟基琥珀酰亚胺酯或地高辛-3-o-甲基羰基-e-氨基己酸-n-羟基琥珀酰亚胺酯交联。多聚甲醛可以交联dna链,间隔4埃。染色体相互作用可以反映染色体区域的状态,例如,如果染色体区域被转录或抑制以响应生理条件的变化。特异于如本文限定的亚组的染色体相互作用已经被发现是稳定的,因此提供了测量两个亚组之间差异的可靠手段。另外,特异于特性(诸如疾病状态)的染色体相互作用将通常在生物过程的早期发生,例如与其他表观遗传标志物诸如甲基化或对组蛋白蛋白质结合的改变相比较。因此,本发明的工艺能够检测生物过程的早期阶段。这允许早期干预(例如治疗),因此可能更有效。此外,同一亚组内的个体之间的相关染色体相互作用几乎没有变化。检测染色体相互作用是高度信息性的,每个基因具有最高达50种不同的可能相互作用,并且因此本发明的工艺可以提出500,000种不同的相互作用。优选的标志物集本文公开了特异性标志物,其中任何一种都可用于本发明。可以使用其他标志物集,例如以本文公开的组合或其中一个。标志物集1、2和3是优选的。这些可以通过任何合适的方法分型,例如本文公开的基于pcr或探针的方法,包括qpcr方法。标志物在本文中通过位置或通过探针和/或引物序列来限定。表观遗传相互作用的位置和起因表观遗传染色体相互作用可以重叠并且包括显示出编码相关的基因或未描述的基因的染色体区域,但同样可能在基因间区域中。还应当注意,本发明人已发现所有区域中的表观遗传相互作用在确定染色体基因座的状态方面同样重要。这些相互作用不一定在位于基因座的特定基因的编码区域中,并且可能在基因间区域中。在本发明中检测到的染色体相互作用可以由下述引起:由基础dna序列的改变、由环境因子、dna甲基化、非编码反义rna转录、非诱变致癌物、组蛋白修饰、染色质重塑和特异性局部dna相互作用。导致染色体相互作用的改变可由本身不直接影响基因产物或基因表达的模式的基础核酸序列的改变引起。这种改变可以是,例如基因内和/或外的snp,基因间dna、微小rna和非编码rna的基因融合和/或缺失。例如,已知大约20%的snp在非编码区域中,并且因此所描述的工艺在非编码情况下也是信息性的。在一种实施方式中,聚集在一起以形成相互作用的染色体的区域在同一染色体上分隔开的小于5kb、3kb、1kb、500个碱基对或200个碱基对。检测到的染色体相互作用优选位于表9中提到的任何基因内。检测到的染色体相互作用可以位于标志物集1、2或3提到的任何基因内。然而,它也可以是基因的上游或下游,例如来自基因或来自编码序列的最高达50,000、最高达30,000、最高达20,000、最高达10,000或最高达5000个碱基的上游,或者来自基因或来自编码序列的最高达50,000、最高达30,000、最高达20,000、最高达10,000或最高达5000个碱基的下游。亚组、诊断和个性化治疗本发明的目的是允许检测与乳腺癌亚组相关的染色体相互作用。因此,该工艺可以用于或可以不用于诊断乳腺癌。本发明的工艺可用于诊断恶性乳腺癌,并且优选地,在这样的实施方式中使用来自标志物集3的标志物。如本文所用,“亚组”优选是指群体亚组(群体中的亚组),更优选是特定动物,诸如特定的真核生物或哺乳动物(例如人类、非人类、非人类灵长类动物、或啮齿动物,例如小鼠或大鼠)的群体中的亚群。最优选地,“亚组”是指人类群体中的亚组。本发明包括检测和治疗群体中的特定亚组。本发明人已经发现,给定群体中的子集(例如,两个或至少两个子集)之间的染色体相互作用不同。识别这些差异将允许医师将他们的患者分类为如该工艺中所述的群体的一个子集的一部分。因此,本发明为医师提供了基于患者的表观遗传染色体相互作用为患者个性化用药的方法。产生连接核酸本发明的某些实施方式利用连接核酸,特别是连接dna。这些包括来自在染色体相互作用中聚集在一起的两个区域二者的序列,并且因此提供关于相互作用的信息。本文描述的episwitchtm方法使用这种连接核酸的产生来检测染色体相互作用。因此,本发明的工艺可包括通过以下步骤(包括包含这些步骤的方法)产生连接核酸(例如dna)的步骤:(i)交联存在于染色体基因座处的表观遗传染色体相互作用,优选在体外;(ii)任选地从所述染色体基因座中分离交联的dna;(iii)对所述交联的dna进行切割,例如通过用至少切割其一次的酶(特别是在所述染色体基因座内切割至少一次的酶)的限制性消化;(iv)连接所述交联的断裂的dna末端(特别是形成dna环);以及(v)任选地识别所述连接dna和/或所述dna环的存在,特别是使用诸如pcr(聚合酶链式反应)的技术,以识别特异性染色体相互作用的存在。可以进行这些步骤以检测本文提及的任何实施方式的染色体相互作用,诸如用于确定个体是否是乳腺癌亚组的一部分。还可以进行这些步骤以产生本文提到的第一和/或第二核酸集。pcr(聚合酶链式反应)可以用于检测或识别连接核酸,例如产生的pcr产物的大小可以指示存在的特异性染色体相互作用,并且因此可以用于识别基因座的状态。在优选的实施方式中,在pcr反应中使用如表10中所示的至少1、2、3、4、5、6、7或8个引物或引物对。在其他优选的实施方式中,在pcr反应中使用与标志物集2或3相关或如所示用于标志物集2或3的至少1、2、3、4、5、6、7或8个引物或引物对。技术人员将了解许多限制性酶,这些限制性酶可用于切割感兴趣的染色体基因座内的dna。显而易见的是,所用的特定酶将取决于所研究的基因座和位于基因座的dna的序列。如本发明所述,可用于切割dna的限制酶的非限制性实例是taqi。实施方式,诸如episwitchtm技术episwitchtm技术还涉及使用微阵列episwitchtm标志物数据检测特异于表型的表观遗传染色体构象标签。以本文所述方式利用连接核酸的实施方式,诸如episwitchtm具有若干优点。它们具有低水平的随机噪声,例如因为来自本发明的第一核酸组的核酸序列与第二核酸集杂交或杂交失败。这提供了二元结果,允许以相对简单的方式测量表观遗传水平的复杂机制。episwitchtm技术还具有快速处理时间和低成本。在一种实施方式中,处理时间为3小时至6小时。样品和样品处理本发明的工艺将通常在样品上进行。样品将通常含有来自个体的dna。样品将通常包含细胞。在一种实施方式中,通过微创性手段获得样品,并且可以例如是血液样品。可以提取dna并且用标准的限制性酶切割。这可以预先确定保留哪些染色体构象,并且将使用episwitchtm平台检测。由于组织和血液之间的染色体相互作用的同步,包括水平转移,血液样品可用于检测组织中的染色体相互作用,诸如与疾病相关的组织。对于某些病症,诸如癌症,由于突变的遗传噪声可以影响相关组织中的染色体相互作用“信号”,并且因此使用血液是有利的。本发明的核酸的性质本发明涉及某些核酸,诸如本文所述的如在本发明的工艺中使用或产生的连接核酸。它们可与本文提及的第一和第二核酸相同或具有本文提及的第一和第二核酸的任何性质。本发明的核酸通常包括两个部分,每个部分包括来自在染色体相互作用中聚集在一起染色体的两个区域之一的序列。通常,每个部分的长度为至少8、10、15、20、30或40个核苷酸,例如长度为10至40个核苷酸。优选的核酸包括来自任何表中提到的任何基因的序列。通常优选的核酸包括表9中提到的特异性探针序列;或这些序列的片段和/或同源物。通常优选的核酸还包括与标志物集2或3相关和/或提及的用于标志物集2或3的特异性探针序列;或这些序列的片段和/或同源物。优选地,核酸是dna。应当理解,在提供特异性序列的情况下,本发明可以使用如特定实施方式中所需的互补序列。表10中所示的引物也可用于如本文所述的本发明中。在一种实施方式中,使用的引物包括以下任何一种:表10中所示的序列、或表10中所示任何序列的片段和/或同源物。与标志物集2或3相关和/或示出的用于标志物集2或3的引物也可用于如本文所述的本发明中。在一种实施方式中,使用的引物包括以下任何一种:所示用于标志物集2或3的序列;或所示用于标志物集2或3的任何序列的片段和/或同源物。第二核酸集——“指标”序列第二核酸集序列具有作为指标序列集的功能,并且本质上是适合于识别亚组特异性序列的核酸序列集。它们可以代表“背景”染色体相互作用,并且可能以某些方式被选择或未被选择。它们一般是所有可能的染色体相互作用的子集。第二核酸集可以通过任何合适的方法获得。它们可以从计算上获得,或者它们可以基于个体中的染色体相互作用。它们通常代表比第一核酸集更大的群体组。在一种特定的实施方式中,第二核酸集代表特异性基因集中的所有可能的表观遗传染色体相互作用。在另一特定实施方式中,第二核酸集代表本文所述群体中存在的所有可能的表观遗传染色体相互作用的大部分。在一种特定实施方式中,第二核酸集代表位于至少20、50、100或500个基因中(例如在20至100或50至500个基因中)的表观遗传染色体相互作用的至少50%或至少80%。第二核酸集通常代表至少100种可能的表观遗传染色体相互作用,表观遗传染色体相互作用修饰、调控或以任何方式介导群体中的疾病状态/表型。第二核酸集可以代表影响物种中疾病状态的染色体相互作用,例如该基因编码与任何疾病状态、疾病易感性或疾病表型相关的细胞因子、激酶或调节物的基因中的染色体相互作用。第二核酸集通常包括代表与乳腺癌亚组相关和不相关的表观遗传相互作用。在一种特定的实施方式中,第二核酸集至少部分地源自群体中天然存在的序列,并且通常通过电脑模拟方法获得。与存在于天然存在的核酸中的核酸的相应部分相比,所述核酸可以还包括单个或多个突变。突变包括一个或多个核苷酸碱基对的缺失、替换和/或添加。在一种特定的实施方式中,第二核酸集可以包括表现为同源物和/或直系同源物的序列,该序列与存在于天然存在的物种中的核酸的相应部分具有至少70%的序列同一性。在另一特定实施方式中,提供了与存在于天然存在的物种中的核酸的相应部分至少80%的序列同一性或至少90%的序列同一性。第二核酸集的性质在一种特定实施方式中,在第二核酸集中存在至少100种不同的核酸序列,优选至少1000、2000或5000种不同的核酸序列,具有至多100,000、1,000,000或10,000,000种不同的核酸序列。典型的数量为100至1,000,000,诸如1,000至100,000种不同的核酸序列。全部或至少90%或至少50%或这些将对应于不同的染色体相互作用。在一种特定实施方式中,第二核酸集代表位于至少20个不同基因座或基因中的染色体相互作用,优选至少40个不同的基因座或基因,并且更优选至少100、至少500、至少1000或至少5000个不同的基因座或基因,诸如100到10,000个不同的基因座或基因。第二核酸集的长度适合于它们根据沃森与克里克(watsoncrick)碱基配对与第一核酸集特异性杂交,以允许识别对亚组特异性的染色体相互作用。通常,第二核酸集将包括序列中的两个部分,该两个部分对应于在染色体相互作用中聚集在一起的两个染色体区域。第二核酸集通常包括长度为至少10个,优选20个,并且仍优选30个碱基(核苷酸)的核酸序列。在另一实施方式中,核酸序列的长度可以是至多500个,优选至多100个,并且仍优选至多50个碱基对。在优选的实施方式中,第二核酸集包括17至25个碱基对之间的核酸序列。在一种实施方式中,第二核酸集序列的至少100、80%或50%具有如上所述的长度。优选地,不同的核酸不具有任何重叠序列,例如至少100%、90%、80%或50%的核酸在至少5个连续核苷酸上不具有相同的序列。鉴于第二核酸集充当“指标”,那么相同的第二核酸集可以与代表不同特性的亚组的不同的第一核酸集一起使用,即第二核酸集可以代表“通用”的核酸集合,其可用于识别与不同特性相关的染色体相互作用。第一核酸集第一核酸集通常来自患有乳腺癌的个体。第一核酸可具有本文提及的第二核酸集的任何特性和性质。第一核酸集通常源自已经历如本文所述的处理和加工(特别是episwitchtm交联和断裂步骤)的个体的样品。通常,第一核酸集代表存在于取自个体的样品中的全部或至少80%或50%染色体相互作用。通常,与由第二核酸集代表的染色体相互作用相比,第一核酸集代表了跨第二核酸集代表的基因座或基因的染色体相互作用的更小的群体,即第二核酸集代表了限定的基因座或基因的集中的相互作用的背景或指标集的。核酸库本文提及的任何类型的核酸群体可以以包括至少200、至少500、至少1000、至少5000或至少10000种不同的该类型的核酸(诸如“第一”或“第二”核酸)的库的形式存在。这样的库可以是结合到阵列的形式。杂交本发明需要用于允许来自第一核酸集和第二核酸集的完全或部分互补的核酸序列杂交的手段。在一种实施方式中,在单一测定中,即在单一杂交步骤中,使所有第一核酸集与所有第二核酸集接触。然而,可以使用任何合适的测定。标记的核酸和杂交模式可以标记本文提到的核酸,优选使用独立的标记,诸如荧光团(荧光分子)或放射性标记,其有助于检测成功的杂交。某些标记可以在紫外线下检测。杂交模式,例如在本文所述的阵列上,代表两个亚组之间的表观遗传染色体相互作用中的差异,并且因此提供了比较表观遗传染色体相互作用并且确定哪些表观遗传染色体相互作用特异于本发明的群体中的亚组的工艺。术语“杂交模式”广泛地涵盖第一和第二核酸集之间存在和不存在杂交,即来自第一集的哪些特异性核酸与来自第二集的哪些特异性核酸杂交,并且因此它不限于任何特定的测定或技术,或需要具有在其上可以检测“模式”表面或阵列。选择具有特定特性的亚组本发明提供了包括检测染色体相互作用的存在或不存在的工艺,通常为5至20或5至500个这样的相互作用,优选20至300或50至100个相互作用,以便确定个体中与乳腺癌相关的特性的存在或不存在。优选地,染色体相互作用是位于本文提及的任何基因中的那些。在一种实施方式中,分型的染色体相互作用是由表9中的核酸代表的那些。表9中标题为“环检测”的列示出了哪个亚组(乳腺癌或对照)被每个探针检测到。可以看出,作为测试的一部分,本发明的工艺可以检测乳腺癌亚组和/或对照亚组(非乳腺癌)。被测试的个体本文提及了被测试的个体源自的物种的实例。此外,在本发明的工艺中被测试的个体可以已经以某种方式被选择。例如,个体可以是雌性。优选的基因区域、基因座、基因和染色体相互作用对于本发明的所有方面,优选的基因区域、基因座、基因和染色体相互作用在表9中提及。通常在本发明的工艺中,从表9中列出的相关基因的至少1、2、3、4、5、6、7或8个中检测染色体相互作用。优选地,检测由表9中探针序列所代表的相关特异性染色体相互作用的至少1、2、3、4、5、6、7或8个的存在或不存在。疾病相关的区域可以是本文提及的任何基因的上游或下游,例如50kb上游或20kb下游,例如来自编码序列。对于本发明的所有方面,优选的基因区域、基因座、基因和染色体相互作用在其他表中提及。通常在本发明的工艺中,从表中列出的相关基因的至少1、2、3、4、5、6、7或8个中检测染色体相互作用,例如对于标志物集2或3。优选地,检测了由表中探针序列所代表的相关特异性染色体相互作用的至少1、2、3、4、5、6、7或8个的存在或不存在。疾病相关的区域可以是本文提及的任何基因的上游或下游,例如50kb上游或20kb下游,例如来自编码序列。在一种实施方式中,基因座(包括检测到染色体相互作用的基因和/或位置)可包括ctcf结合位点。这是能够结合转录抑制子ctcf的任何序列。该序列可以包括或有下述组成:可以在基因座处以1、2或3个拷贝存在的序列ccctc。ctcf结合位点序列可包括序列ccgcgnggnggcag(以iupac命名法)。ctcf结合位点可以在染色体相互作用的至少100、500、1000或4000个碱基内或在表9示出的任何染色体区域内。ctcf结合位点可以在染色体相互作用的至少100、500、1000或4000个碱基内或在任何表中示出的任何染色体区域内,例如用于标志物集2或3的。在一种实施方式中,检测到的染色体相互作用存在于表9示出的任何基因区域。在该工艺中检测到连接核酸的情况下,则可以检测表9中的任何探针序列中示出的序列。在另一实施方式中,检测到的染色体相互作用存在于其他表中示出的任何基因区域,例如用于标志物集2或3的。在该工艺中检测到连接核酸的情况下,则可以检测表中任何探针序列中示出的序列,例如对于标志物集2或3。因此,通常可以检测来自探针的两个区域(即来自染色体相互作用的两个位点)的序列。在优选的实施方式中,在该工艺中使用的探针包括或有下述组成:与任何表中示出的探针相同的或互补的序列。在一些实施方式中,使用包括与表中示出的任何探针序列同源的序列的探针。本文提供的表表9示出了代表与乳腺癌相关的染色体相互作用的探针(episwitchtm标志物)数据和基因数据。其他探针和基因数据在其他表中示出,例如用于标志物集2或3的。探针序列示出了可用于检测下述连接产物的序列:该连接产物产生自已经在染色体相互作用中聚集在一起的两个基因区域的位点二者,即探针将包括与连接产物中的序列互补的序列。前两个起始-终止位置集合示出了探针位置,并且后两个起始-终止位置集合示出了相关的4kb区域。探针数据表中提供了以下信息:——hyperg_stats:基于超几何富集参数在基因座中发现显著性episwitchtm标志物数目的概率的p值——探针总计数:在基因座处测试的episwitchtm构象总数目——探针显著性(sig)计数:在基因座处被发现具有统计学显著性的episwitchtm构象数目——fdrhyperg:多重检验(假发现率)校正的超几何p值——显著性(sig)百分比:显著性episwitchtm标志物相对于在基因座处测试的标志物数目的百分比——logfc:表观遗传率(fc)的以2为底的对数——aveexpr:所有阵列和通道上的探针的平均log2表达——t:调节t-统计量——p值:原始p值——adj.p值:调整的p值或q值——b—b统计量(对数概率(lods)或b)是该基因差异表达几率的对数。——fc—非对数倍数变化——fc_1—以零为中心的非对数倍数变化。——ls—二元值,这与fc_1值有关。fc_1值低于-1.1,它设置为-1,并且如果fc_1值高于1.1,它设置为1。在这些值之间,值为0表9示出了已发现相关染色体相互作用存在的基因。其他表示出了类似的数据。基因座表中的p值与hyperg_stats相同(基于超几何富集参数在基因座中发现显著性episwitchtm标志物数目的概率的p值)。探针设计为距离taq1位点30bp。在pcr的情况下,pcr引物还被设计为检测连接产物,但它们离taq1位点的位置改变。探针位置:起始1——在片段1上的taqi位点的30个上游碱基终止1——片段1上的taqi限制性位点起始2——片段2上的taqi限制性位点终止2——在片段2上的taqi位点的30个下游碱基4kb序列位置:起始1——在片段1上的taqi位点的4000个上游碱基终止1——片段1上的taqi限制性位点起始2——片段2上的taqi限制性位点终止2——在片段2上的taqi位点的4000个下游碱基表10和其他表示出了每个靠前的pcr标志物:glmnet——用于拟合整个lasso或弹性网络规则的程序。λ设为0.5(弹性网络)用于样品制备和染色体相互作用检测的优选实施方式本文描述了制备样品和检测染色体构象的方法。可以使用这些方法的优化(非常规)版本,例如,如本节所述。通常,样品将包括至少2×105个细胞。样品可包括最多达5x105个细胞。在一种实施方式中,样品将包括2×105至5.5×105个细胞。本文描述了存在于染色体基因座处的表观遗传染色体相互作用的交联。这可以在细胞裂解发生之前进行。细胞裂解可以进行3至7分钟,诸如4至6或约5分钟。在一些实施方式中,细胞裂解进行至少5分钟并且少于10分钟。本文描述了用限制性酶消化dna。通常,dna限制在约55℃至约70℃,诸如约65℃,进行约10至30分钟的时间,诸如约20分钟。优选使用产生具有平均片段大小最高达4000个碱基对的连接dna的片段的常见的切割限制酶。任选地,限制酶使得连接dna的片段具有约200至300个碱基对的平均片段大小,诸如约256个碱基对。在一种实施方式中,典型的片段大小为从200个碱基对至4,000个碱基对,诸如400至2,000或500至1,000个碱基对。在episwitch方法的一种实施方式中,dna沉淀步骤不在dna限制性消化步骤和dna连接步骤之间进行。本文描述了dna连接。通常,dna连接进行5至30分钟,诸如约10分钟。样品中的蛋白质可以被酶促消化,例如使用蛋白酶,任选的蛋白酶k。可以酶促消化蛋白质约30分钟至1小时的时间,例如约45分钟。在一种实施方式中,在消化蛋白质后,例如蛋白酶k消化,没有交联反转或酚dna提取步骤。在一种实施方式中,pcr检测能够检测连接核酸的单拷贝,优选对于连接核酸存在/不存在具有二元读数。本发明的工艺和用途可以以不同方式描述本发明的工艺。它可以描述为制备连接核酸的方法,包括(i)在染色体相互作用中已经聚集在一起的染色体区域的体外交联;(ii)对所述交联dna进行切割或限制性消化断裂;和(iii)连接所述交联的断裂的dna末端以形成连接核酸,其中连接核酸的检测可用于确定基因座处的染色体状态,并且其中优选地:——基因座可以是表9中提到的任何基因座、区域或基因,——和/或其中染色体相互作用可以是本文提及的任何染色体相互作用或对应于表9中公开的任何探针的染色体相互作用,和/或——其中连接产物可具有或包括(i)与表9中公开的任何探针序列相同的或同源的序列;或(ii)与(ii)互补的序列。本发明的工艺可以描述为用于检测代表群体中不同亚组的染色体状态的工艺,包括确定在基因组的限定的表观遗传活性(疾病相关的)区域内存在或不存在染色体相互作用,其中优选:——该亚组由乳腺癌的存在或不存在限定,和/或——染色体状态可以在表9中提到的任何基因座、区域或基因处;和/或——染色体相互作用可以是表9中提到的那些中的任何一种,或者对应于该表中公开的任何探针的那些。本发明包括检测表9提到的任何基因座、基因或区域处的染色体相互作用。本发明包括使用本文提及的核酸和探针以检测染色体相互作用,例如使用至少1、2、4、6或8种这样的核酸或探针来检测位于至少1、2、4、6或8个不同的基因座或基因中的染色体相互作用。本发明包括使用表10中列出的任何引物或引物对或使用如本文所述的这些引物的变体(包含引物序列或包含引物序列的片段和/或同源物的序列)来检测染色体相互作用。在特定实施方式中:——基因座可以是任何表中提到的任何基因座、区域或基因,例如对于标志物集2或3的,——和/或其中染色体相互作用可以是本文提到的任何染色体相互作用或对应于任何表中公开的任何探针的染色体相互作用,例如对于标志物集2或3的,和/或——其中连接产物可具有或包括(i)与任何表中公开的任何探针序列相同的或同源的序列,例如对于标志物集2或3;或(ii)与(ii)互补的序列。本发明的工艺可以描述为用于检测代表群体中不同亚组的染色体状态的工艺,包括确定在基因组的限定的表观遗传活性(疾病相关的)区域内存在或不存在染色体相互作用,其中优选:——该亚组由乳腺癌的存在或不存在限定,和/或——染色体状态可以位于任何表中提到的任何基因座、区域或基因处,例如对于标志物集2或3的;和/或——染色体相互作用可以是任何表中提到的那些中的任何一种,例如对于标志物集2或3的;或对应于该表中公开的任何探针的那些。本发明包括检测位于任何表中提到的任何基因座、基因或区域处的染色体相互作用,例如对于标志物集2或3的。本发明包括使用本文提及的核酸和探针以检测染色体相互作用,例如使用至少1、2、4、6或8种这样的核酸或探针来检测位于至少1、2、4、6或8个不同的基因座或基因中的染色体相互作用。本发明包括使用任何表中列出的任何引物或引物对,例如用于标志物集2或3的或使用如本文所述的这些引物的变体(包括引物序列的序列或包括引物序列的片段的序列和/或包括引物序列的同源物的序列)来检测染色体相互作用。本发明的方法识别新治疗的用途染色体相互作用的知识可用于识别用于病症的新治疗。本发明提供了本文限定的染色体相互作用的方法和用途,以识别或设计用于乳腺癌的新治疗剂。同源物本文提及多核苷酸/核酸(例如dna)序列的同源物。这样的同源物通常具有至少70%的同源性,优选至少80%,至少85%,至少90%,至少95%,至少97%,至少98%或至少99%的同源性,例如跨至少10、15、20、30、100或更多个连续核苷酸的区域,或跨来自参与染色体相互作用中的染色体的区域的核酸的一部分。可以基于核苷酸同一性(有时被称为“严格同源性”)计算同源性。因此,在特定实施方式中,在本文中通过参考百分比序列同一性来提及多核苷酸/核酸(例如dna)序列的同源物。通常,这样的同源物具有至少70%的序列同一性,优选至少80%,至少85%,至少90%,至少95%,至少97%,至少98%或至少99%的序列同一性,例如跨至少10、15、20、30、100或更多个连续核苷酸的区域,或跨越来自参与染色体相互作用中的染色体的区域的核酸的一部分。例如,uwgcg包提供可用于计算同源性和/或%序列同一性的bestfit程序(例如在其默认设置下使用)(devereux等(1984)nucleicacidsresearch12,p387-395)。pileup和blast算法可用于计算同源性和/或%序列同一性和/或排列序列(诸如识别等同的或相应的序列(通常在其默认设置下)),例如如altschuls.f.(1993)jmoievol36:290-300;altschul,s,f等(1990)jmoibiol215:403-10中所述。用于进行blast分析的软件可通过国家生物技术信息中心公开获得。该算法涉及首先通过识别查询序列中长度为w的短字来识别高评分序列对(hsp),其在与在数据库序列中的相同长度的字对准时匹配或满足某些正值阈值评分t。t被称为邻域字得分阈值(altschul等,同上)。这些初始邻域字命中(hits)作为种子,用于启动搜索以找到包含它们的hsp。字命中沿每个序列在两个方向上延伸直到可以增加累积对准评分。在以下情况下,在每个方向上的字命中的延伸停止:累积对准评分从其最大实现值减少数量x;由于一个或多个负评分残基对准的累积,累积评分为零或低于零;或达到任一序列的末端。blast算法参数w5t和x确定对准的灵敏度和速度。blast程序使用11的字长(w)作为默认值,blosum62评分矩阵(参见henikoff和henikoff(1992)proc.natl.acad.sci.usa89:10915-10919)对准(b)为50,期望值(e)为10,m=5,n=4,以及两条链的比较。blast算法进行两个序列之间的相似性的统计分析;参见,例如,karlin和altschul(1993)proc.natl.acad.sci.usa90:5873-5787。由blast算法提供的一种相似性测量是最小和概率(p(n)),其提供了指示两个多核苷酸序列之间偶然发生匹配的概率。例如,如果在第一序列与第二序列的比较中的最小和概率小于约1,优选地小于约0.1,更优选地小于约0.01,并且最优选地小于约0.001,则序列被认为与另一序列相似。同源序列通常有1、2、3、4或更多个碱基,诸如少于10、15或20个碱基(其可以是核苷酸的替换、缺失或插入)不同。这些变化可以跨越上述与计算同源性和/或%序列同一性有关的任何区域中测量。阵列第二核酸集可以与阵列结合,并且在一种实施方式中,存在与阵列结合的至少15,000、45,000、100,000或250,000个不同第二核酸,其优选代表至少300、900、2000或5000个基因座。在一种实施方式中,第二核酸的不同群体中的一个或多个或全部与阵列的多于一个不同区域结合,在实际中,在阵列上重复以允许错误检测。该阵列可以基于agilentsureprintg3定制cgh微阵列平台。检测第一核酸与阵列的结合可以通过双色系统进行。治疗剂本文提及治疗剂。本发明提供了这样的药剂以用于预防或治疗某些个体,例如通过本发明的工艺识别的那些个体中的乳腺癌。这可以包括向有需要的个体给药治疗有效量的药剂。本发明提供了该药剂在制备用于预防或治疗某些个体的乳腺癌的药物中的用途。优选的治疗剂是用于破坏癌细胞生长的细胞毒性药物。有许多不同的化疗药物常用于治疗乳腺癌。这些药物包括环磷酰胺、表柔比星、氟尿嘧啶(5fu)、氨甲蝶呤、丝裂霉素、米托蒽醌、阿霉素、多烯紫杉醇(泰素帝)和吉西他滨(健择)。通常患者服用约三种化疗药物在一起的组合。治疗剂可以降低触发癌细胞生长的激素水平。用于激素疗法的各种药物包括阿纳托唑(瑞宁得)、依西美坦(阿诺新)、来曲唑(弗隆)和它莫西芬。治疗剂可以是生物疗法,诸如中断癌细胞之间相互作用的药物,并且从而停止细胞分裂和生长。常用的用于生物疗法的药物包括赫塞汀(曲妥珠单抗)、拉帕替尼(泰嘉锭)、帕妥珠单抗(贺疾妥)和依维莫司(飞尼妥)。药剂的制剂将取决于药剂的性质。该药剂将以含有药剂和药学上可接受的载体或稀释剂的药物组合物的形式提供。合适的载体和稀释剂包括等渗盐水溶液,例如磷酸盐缓冲盐水。典型的口服剂型组合物包括片剂、胶囊、液体溶液和液体混悬剂。该药剂可以配制用于肠胃外、静脉内、肌肉内、皮下、透皮或口服给药。药剂的剂量可根据各种参数确定,尤其是根据所用物质;待治疗个体的年龄,重量和病症;给药途径;和所需的方案。医师将能够确定任何特定药剂的所需给药途径和剂型。然而,合适的剂量可以是从0.1至100mg/kg体重,诸如1至40mg/kg体重,例如,每天服用从1至3次。本文提及的物质的形式本文提及的任何物质,诸如核酸或治疗剂,可以是纯化的或分离的形式。它们的形式可能不同于自然界中发现的形式,例如它们可以与它们在自然界中不存在的其他物质组合存在。核酸(包括本文限定的序列的部分)可以具有不同于自然界中发现的序列的序列,例如关于同源性部分所述的序列中具有至少1、2、3、4或更多个核苷酸改变。核酸可以在5'或3'末端具有异源序列。核酸可以在化学上不同于自然界中发现的核酸,例如它们可以以某些方式修饰,但优选仍然能够沃森克里克(watson-crick)碱基配对。适当时,核酸将以双链或单链形式提供。本发明以单或双链形式提供本文提及的所有特异性核酸序列,并且因此包括所公开的任何序列的互补链。本发明还提供了用于实施本发明的包括检测与乳腺癌相关的染色体相互作用或诊断乳腺癌的任何工艺的试剂盒。这种试剂盒可包括能够检测相关染色体相互作用的特异性结合剂,诸如能够检测由本发明的工艺产生的连接核酸的试剂。试剂盒中存在的优选试剂包括探针(能够与连接核酸杂交)或引物对,例如如本文所述的,能够在pcr反应中扩增连接核酸。本发明还提供了能够检测相关染色体相互作用的装置。该装置优选包含能够检测染色体相互作用的任何特异性结合剂、探针或引物对,诸如本文所述的任何这样的试剂、探针或引物对。检测方法在一种实施方式中,使用在pcr反应期间在活化后可检测的探针进行与染色体相互作用相关的连接序列的定量检测,其中所述连接序列包括来自在表观遗传染色体相互作用中聚集在一起的两个染色体区域的序列,其中所述方法包括在pcr反应期间使连接序列与探针接触,并且检测探针的活化程度,并且其中所述探针结合连接位点。该方法通常允许使用双标记荧光水解探针以符合miqe的方式检测特定相互作用。该探针一般被标记有可检测的标记,该标记具有无活性和活性状态,使得该标记仅在活化时检测到。活化程度将与pcr反应中存在的模板(连接产物)的程度有关。可以在全部或一些pcr期间进行检测,例如至少50%或80%的pcr循环。探针可以包括共价附连到寡核苷酸一个末端的荧光团和附连到核苷酸另一末端的猝灭基团,使得荧光团的荧光被猝灭基团猝灭。在一种实施方式中,荧光团被附连到寡核苷酸的5'末端,并且猝灭基团共价附连到寡核苷酸的3'末端。可用于本发明的方法的荧光团包括fam、tet、joe、yakimayellow、hex、花菁(cyanine)3、atto550、tamra、rox、德克萨斯红(texasred)、花菁3.5、lc610、lc640、atto647n、花菁5、花菁5.5和atto680。可与适当的荧光团一起使用的猝灭基团包括tam、bhq1、dab、eclip、bhq2和bbq650,任选地其中所述荧光团选自hex、德克萨斯红(texasred)和fam。荧光团和猝灭基团的优选组合包括fam与bhq1以及德克萨斯红与bhq2。探针在qpcr测定中的用途本发明的水解探针通常是用浓度匹配的阴性对照优化的温度梯度。优选地,优化单步pcr反应。更优选地,计算标准曲线。使用结合在连接序列的接合处的特异性探针的优点是可以在不使用巢式pcr方法的情况下实现对连接序列的特异性。本文描述的方法允许精确并且精密地量化低拷贝数目的靶标。在温度梯度优化之前,可以纯化靶标连接序列,例如凝胶纯化。靶标连接序列可以测序。优选地,使用约10ng,或5至15ng,或10至20ng,或10至50ng,或10至200ng模板dna进行pcr反应。设计正向和反向引物使得一个引物结合由连接dna序列代表的染色体区域之一的序列,并且另一引物结合由连接dna序列代表的另一染色体区域,例如,通过与序列的互补。连接dna靶标的选择本发明包括选择用于如本文所限定的pcr方法中的引物和探针,包括基于引物结合和扩增连接序列的能力选择引物,并且基于探针序列将结合的靶标序列的性质选择探针序列,特别是靶标序列的曲率。通常设计/选择探针以结合跨越限制性位点的并列限制性片段的连接序列。在本发明的一种实施方式中,计算与特定染色体相互作用相关的可能的连接序列的预测曲率,例如使用本文引用的特异性算法。曲率可以表示为每螺旋转弯的度数,例如,每螺旋转弯10.5°。在连接序列具有每螺旋转弯至少5°的曲率倾向峰值分数,通常每螺旋转弯至少10°、15°或20°,例如每螺旋转弯5°至20°的情况下,选择该连接序列用于靶向。优选地,针对至少20、50、100、200或400个碱基计算每螺旋转弯的曲率倾向分数,诸如针对连接位点的20至400个上游和/或下游碱基。因此,在一种实施方式中,连接产物中的靶标序列具有任何这些水平的曲率。还可以基于最低热力学结构自由能来选择靶标序列。特定实施方式在特定实施方式中,igfbp3中的染色体相互作用未被分型/检测。在某些实施方式中,本文提及的任何基因中的染色体相互作用未被分型/检测。在一种实施方式中,任何以下基因未被分型/检测:bcas1、znf217、tshz2、sumo1p1、mir4756、bcas3、tbx2、c17orf82、tbx4、bca54、linc00651、ube2v1、tmem189、cebpb、loc284751、ptpni、mir645、fam65cpard68、adnp、linc00494、prex1、arfgef2、cse1l、pde4dip、sec22b、notch2nlnbp10、hfe2、txnip、polr3gl、ankrd34a、lix1l、rbm8a、gnrhr2、pex11b、itga10、ankrd35、pias3、nudti7、polr3c、rnf115、cd160、pdzk1、gpr89a、znf334.ocstamp、slc13a3、tp53rk、slc2a10、eya2、mir3616、zmynd8、l0c100131496、dlg1、mir4797、dlg1-as1、bdh1、loc220729、kiaa0226、mir922、fyttdl、lrch3、iqcg、rpl35a、lmln、ankrd18dp、ddx59、camsap2、gpr25、c1orf106、kif21b、cacna15、ascls、tmem9、igfn1、pkp1、tnn2、lad1、tnni1、phlda3、ncoa1、ptrhd1、cenpo、adcy3、dnajc27、dnajc27-as1、efr3b.pomc、dnmt3a、mir1301、dtnb、spon2、loc100130872、ctbp1、ctbp1-as1、maea、uvssa、cripak、fam53a、slbp、tmem129、tacc3、fgfr3、letm1、whsc1、scarna22、whsc2、mir943、c4orf48、nat8l、poln、haus3、mxd4、mir4800、zfyve28、loc402160、rnf4、loc1oo506190、c9orf50、ntmt1、asb6、prrx2、ptges、tor1b、tor1a、c9orf78、usp20、fnbp1、gpr107、nc51、ass1。在一种实施方式中,仅染色体内相互作用被分型/检测,并且染色体外相互作用(在不同染色体之间)未被分型/检测。出版物本文提及的所有出版物的内容通过引用并入本说明书中,并且可用于进一步限定与本发明相关的特征。特异性实施方式episwitchtm平台技术检测基因座处正常和异常情况之间调控变化的表观遗传调控标签。episwitchtm平台识别并且监测与人类染色体的调控高级结构(也称为染色体构象标签)相关的基因调控的基本表观遗传水平。染色体标签是基因失调的连续反应中的不同的主要步骤。它们是具有一组针对生物标志物平台的独特优势的高级生物标志物,该生物标志物平台利用晚期表观遗传和基因表达生物标志物,诸如dna甲基化和rna谱。episwitchtm阵列测定定制的episwitchtm阵列筛查平台具有15k、45k、100k和250k独特的染色体构象的4种密度,每个嵌合片段在阵列上重复4次,形成有效密度分别为60k、180k、400k和1百万。定制设计的episwitchtm阵列15kepiswitchtm阵列可以筛查整个基因组,包括使用episwitchtm生物标志物发现技术查询的约300个基因座。episwitchtm阵列以agilentsureprintg3定制cgh微阵列平台为基础;该技术提供4种密度,60k、180k、400k和1百万探针。每个阵列的密度降低到15k、45k、100k和250k,因为每个episwitchtm探针都被表现为一式四份,因此允许重现性的统计评估。每个遗传基因座查询的潜在episwitchtm标志物的平均数目为50;因此,可以研究的基因座的数目是300、900、2000和5000。episwitchtm定制阵列流程episwitchtm阵列是在一组样品情况下的双色系统,在episwitchtm库生成后,用cy5中标记,并且待比较/分析的另一样品(对照)用cy3中标记。使用agilentsurescan扫描仪扫描阵列,并且使用agilent特征提取软件提取所得特征。然后使用r中的episwitchtm阵列处理脚本以处理数据。使用r:limma*中的bioconductor中的标准双色程序包处理阵列。使用limma*中normalizewithinarrays函数进行阵列的归一化,并且这是对芯片上agilent阳性对照和episwitchtm阳性对照进行的。基于agilentflag调用过滤数据,移除agilent对照探针并且对技术复制探针进行平均,以便使用limma*对它们分析。基于它们的被比较的2个场景之间的差异对探针建模,并且然后通过使用假发现率校正。具有变异系数(cv)<=30%的探针被用于进一步筛查,其<=-1.1或=>1.1并且通过p<=0.1fdrp值。为了进一步减少探针组,使用r中的factorminer程序包进行多因子分析。*注意:limma是用于评估微阵列实验中的差异表达的线性模型和经验贝叶斯(bayes)法。limma是用于分析来自微阵列或rna-seq的基因表达数据的r程序包。最初基于调整的p值、fc和cv<30%(任意截止点)参数选择探针池用于最终拣选。仅基于前两个参数(adj.p值;fc)绘制进一步的分析和最终列表。本文提及的基因tspyl5——tspy类5srd5a1——类固醇5阿尔法-还原酶1map3k1——丝裂原活化蛋白激酶激酶激酶1vav3——vav鸟嘌呤核苷酸交换因子3atm——atm丝氨酸/苏氨酸激酶slc16a10——溶质载体家族16成员10me3——苹果酸酶3通过以下非限制性实施例说明本发明。实施例1统计流程使用r中的episwitchtm分析包处理episwitchtm筛查阵列,以便选择高值episwitchtm标志物,以转换到episwitchtmpcr平台上。步骤1基于它们校正的p值(假发现率,fdr)选择探针,校正的p值是修正的线性回归模型的结果。选择低于p值<=0.1的探针,并且然后通过它们的表观遗传率(er)进一步降低,探针er必须<=-1.1或=>1.1,以便被选择用于进一步分析。最后的过滤是变异系数(cv),探针必须低于<=0.3。步骤2基于它们的er选择来自统计列表的前40个标志物,以选择作为用于pcr转换的标志物。具有最高负er负载的前20个标志物和具有最高正er负载的前20个标志物形成列表。步骤3来自步骤1的所得标志物,统计学上显著性探针形成使用超几何富集(he)富集分析的基础。该分析能够使来自显著性探针列表的标志物减少,并且与来自步骤2的标志物一起形成转换到episwitchtmpcr平台上的探针列表。通过he处理统计探针以确定哪些遗传位置具有统计学上显著性探针的富集,指示哪些遗传位置是表观遗传差异的中心。选择基于校正的p值的最显着富集的基因座,用于探针列表生成。选择低于0.3或0.2的p值的遗传位置。使用来自步骤2的标志物定位到这些遗传位置的统计探针形成了用于episwitchtmpcr转换的高值标志物。阵列设计和处理阵列设计1.遗传基因座使用sii软件(目前为v3.2)处理:a.在这些特异性遗传基因座上抽取出基因组序列(上游50kb的基因序列和下游20kb的基因序列)b.限定该区域内的序列涉及ccs的概率c.使用特异性re切割该序列d.确定哪些限制性片段可能以某种取向相互作用e.排列不同cc在一起相互作用的可能性。2.确定阵列大小,并且从而确定可用探针位置的数目(x)3.抽取出x/4相互作用。4.对于每个相互作用,限定了至来自部分1的限制性位点30bp的序列以及至部分2的限制性位点的30bp的序列。检查这些区域是否重复,如果是,排除并且在列表中采取下一个相互作用。加入两个30bp以限定探针。5.创建x/4探针加限定的对照探针的列表,并且复制4次以创建在阵列上待创建的列表6.将探针列表上传到agilentsure设计网站,用于定制cgh阵列。7.使用探针组以设计agilent定制cgh阵列。阵列处理1.使用episwitchtm标准操作程序(sop)处理样品用于模板生产。2.阵列处理实验室乙醇沉淀清除。3.依据agilentsuretag完整dna标记试剂盒处理样品——基于agilent寡核苷酸阵列的cgh,用于基因组dna分析酶标记,用于血液、细胞或组织。4.使用agilentc扫描仪使用agilent特征提取软件扫描。乳腺癌概述年龄特异性发病率表明,亚洲和西方群体中侵袭性乳腺癌的年龄效应更为相似。事实上,近几代亚洲乳腺癌率甚至超过了美国历史上的高发率,凸显了亚洲群体中对有效的预防和治疗策略的迫切需要。然而,一项大规模的25年研究的结果显示乳房x光检查并未降低乳腺癌相关的死亡率。在肿瘤变得肉眼可见之前,乳腺癌的早期检测将意味着医疗干预可以在癌症更可治疗时的阶段开始。episwitchtm技术概述episwitchtm平台为与异常和响应性基因表达相关的主要疾病提供了高度有效的筛查、早期检测、伴随诊断、监测和预后分析手段。这种方式的主要优点是其是非侵入性的、快速的,并且依赖于高度稳定的dna类靶标作为染色体标签的部分,而不是依赖于不稳定的蛋白质/rna分子。episwitchtm生物标志物标签在复杂疾病表型的分层中表现出高稳健性、灵敏性和特异性。该技术利用表观遗传学的科学中的最新突破,监测并且评估作为一类高度信息性的表观遗传生物标志物的染色体构象标签(chromosomeconformationsignature)。目前在学术环境中开展的研究方法对于细胞材料的生化处理需要3到7天,以便检测ccs。这些程序具有有限的灵敏度和重现性;并且此外,这些程序在设计阶段没有由episwitchtm分析包提供的有针对性的见解的益处。episwitchtm分析包episwitchtm平台技术检测作为主要表观遗传调控框架的部分的人类染色体的高级结构中的变化。并置染色体中的远距离位点形成特异性类型的生物标志物——调控染色体构象标签。该工艺中最大的挑战之一是识别染色体中基因/基因座中形成高级结构部分的潜在位点。这通过使用识别给定序列内的潜在位点的专有模式识别软件来执行。episwitchtm分析包软件,包括机器学习算法,识别dna中可能形成ccs的高级结构的模式。电脑模拟的episwitchtm阵列标志物识别通过episwitchtm阵列对来自测试群组的临床样品直接评估跨越基因组中的ccs位点,以识别所有相关的分层引导生物标志物。episwitchtm阵列平台由于其高通量容量以及其快速筛查大量基因座的能力而用于标志物识别。使用的阵列是agilent定制cgh阵列,其允许通过电脑模拟软件识别的标志物被查询。episwitchtmpcr然后通过episwitchtmpcr或dna测序仪(即roche454,纳米孔minion等)验证由episwitchtm阵列识别的潜在标志物。在统计学上显着并且显示出最佳重现性的排名居前的pcr标志物被选择用于进一步减少到最终的episwitchtm标签组中,并且在独立的样品群组上验证。episwitchtmpcr可由经过培训的技术人员按照建立的标准化操作程序方案执行。所有方案和试剂的制造均在iso13485和9001认证下进行,以确保工作质量和转移方案的能力。episwitchtmpcr和episwitchtm阵列生物标志物平台兼容全血和细胞系两者的分析。测试足够灵敏,以使用小体积的血液检测极低拷贝数上的异常。综述本发明人已使用表观遗传染色体相互作用作为用于识别生物标志物的基础,以用作乳腺癌诊断中的伴随诊断方法。episwitchtm生物标志物发现平台由本发明人发展以检测表观遗传调控标签变化,诸如那些驱动乳腺癌中涉及的表型变化。episwitchtm生物标志物发现平台识别限定将环境线索整合到表观遗传和转录机制中的初始调控过程的ccs。因此,ccs是基因调控连续反应中的主要步骤。由episwitchtm生物标志物发现平台分离的ccs具有若干记载的良好的优点:严格的生化和生理稳定性;他们的二元性质和读数;以及它们在真核生物的基因调控连续反应中的主要地位。episwitchtm阵列筛查平台应用于本发明,并且其结果转换到episwitchtmpcr平台上,以满足以下目标:1.识别episwitchtm标志物,episwitchtm标志物将乳腺癌患者与健康个体区分开来;2.识别episwitchtm标志物,episwitchtm标志物可以被发展成提供灵敏度、特异性或阳性预测值(ppv)的标准的测试,与目前现有的临床实践相关。在该乳腺癌生物标志物发现项目中,使用了8x60k阵列,这允许以一式四份用于研究多达56,964个潜在的染色体构象。使用来自一系列背景的八个ii/iii期乳腺癌患者样品,相对于八个合并的健康对照患者样品,单独测试,产生了两个阵列。每个阵列包含56,964个episwitchtm探针。为每个样品制备episwitchtm模板。第一个阵列是在亚洲乳腺癌样品上进行的。第二个阵列使用欧洲和亚洲样品。亚洲和欧洲乳腺癌在er+和er-状态之间可能不同。在多个群体中发现了对类似癌症的重叠探针。然后测试每个探针的数据的统计质量,并且然后如其后所述分析。血液样品质量控制结果研究中使用的样品来自马来西亚。适用于episwitchtm测定的血液样品的生化质量直接受样品氧化和蛋白质变性程度的影响,以血红蛋白为例。这两个参数是在样品处理之前评估血液质量的标准手段。简而言之,当氧合的血红蛋白(氧合血红蛋白)被氧化时,形成高铁血红蛋白,并且如果球蛋白结构域变性,则高铁血红蛋白被转化为半血色质(hemichrome)。通过winterbourn(1990),oxidativereactionsofhemoglobin.methodsenzymol.1990;186:265-72所描述的质量控制方法,使用光谱变化来计算每个级分的丰度,这基于各血红蛋白级分的消光系数。根据该文献,作为用于每个样品质量控制的部分,将血液在pbs中稀释并且在分光光度计(epochmicroplate(biotek))上在560、577和630nm处进行分析。根据标准计算监测三个血红蛋白级分中的每一个的微摩尔浓度:μm氧合血红蛋白=119*a577-39*a630-89*a560,μm高铁血红蛋白=28*a577+307*a630-55*a560,μm半血色质=-133*a577-114*a630+233*a560。表现出氧合血红蛋白:高铁血红蛋白比率≥0.75的样品通过质量控制,并且被认为适合于episwitchtm处理。11个样品,由于它们的氧化和变性的生化状态,未通过血红蛋白qc(样品brcama132、brcama136、brcama137、brcama147、brcama164、brcama165、brcama166、brcama167、brcama168、brcama169和brcama170)并且被排除。表1.样品的oxy/met-hb比率阈值及其在统计方法中的使用。所有处理样品的表观遗传分析包括对于异常值的第二质量控制。集群122(位点2批次2对照)显示出与所有其他位点和集群根本不同的分布和质量。根据异常值控制的标准实践,来自位点2批次2(集群122)的30个样品被排除在测试的发展之外。episwitchtm阵列结果·两个数据集都产生了许多显著性探针;·阵列1,bca14185,在乳腺癌与健康对照的分析中识别的显著性episwitchtm标志物;·阵列2,bca24856,在乳腺癌与健康对照的分析中识别的显著性episwitchtm标志物;·然而,在2个研究之间存在两个分析二者之间一致的2116个显著性探针的重叠(参见图1)所有数据是最初采集的并且移出所有饱和探针。然后对它们归一化以均匀化通道之间的数据。然后将每个数据集的所有四个重复组合在一起,并且确定变异系数。使用归一化的相关值缩减该2116个探针以对阵列上变化最大的基因排序。富集分析被用于发现高于其随机机会的差异表达最大的基因。总共有138个标志物来自组合的bca1和bca2阵列,其表现出差异的上调或下调表达。采用前80个episwitchtm标志物(参见附录i),包括来自阵列1的41个标志物和来自阵列2的39个标志物,用于通过episwitchtmpcr测定验证以在乳腺癌和健康对照之间分层。episwitchtmpcr平台和标志物验证使用集成dna技术(idt)软件(和primer3web版本4.0.0软件,如果需要)根据微阵列上识别的标志物设计引物。对每个引物组进行引物测试。在合并的样品子集上测试每个集以确保合适的引物可以研究潜在的相互作用。如果引物测试成功,那么将对引物组进行筛查。使用168个样品。这些样品被分为2组:将118个患者样品(68个brca和50个对照)用于标志物减少和模型发展,并且将剩余的50个样品(31个brca和19个对照)用作独立群组以验证发展自最初的118个患者组的最终模型。来自位点2集群122(限定为批次2)的30个对照样品,因为它们被证明是质量控制程序中的异常值,未在最终患者组中使用。引物筛查该测试用于消除非特异性引物,并且以确定引物是否能够检测3c构象环。所有提取的血液样品均以1:2-1:64稀释。初步结果以二元形式生成;即“1”——是,带在正确的大小位置存在,或“0”——否,带在正确的大小位置不存在。通过episwitchtmpcr的所有读数均在阳性和阴性对照两者的检测准确度>95%的存在下且情况下进行。筛查151个引物组成功通过了引物测试阶段,并且在8个brca和8个对照血液样品上测试。在第一筛查中,样品与阵列上使用的样品匹配。表2.阵列1上使用的和在进一步的pcr验证中的样品。筛查2然后显示差异化的引物组用另外12个brca和12个对照血液样品进行筛查。使用1:2至1:64稀释系列来识别测定灵敏度的范围。将来自筛查1和2的结果合并在一起,以给出所有20个使用的样品的完整表示。测试另外24个brca和24个对照和最后的剩余样品。筛查3然后使用最具信息性的三个稀释度筛查最后的20个brca和20个对照样品,覆盖每个引物组测定的灵敏检测范围。总计13个标志物用于最后的20个样品筛查中。将来自筛查3的结果与90个brca和90个对照样品合并在一起,以给出用于brca和对照两者的100个样品的完整表示。然后测试这些在区分brca患者和对照样品中的有效性。生成卡方检验(fisher精确)以给出最终标志物。表3.最终标志物和引物组。13个引物组合与3个稀释因子,39个种标志物的标志物减少为了将最终选择的13个位置和39个标志物减少到工作分类模型,使用了具有r统计语言的glmnet程序包。glmnet执行惩罚(弹性网络惩罚)回归建模,允许岭回归或lasso回归(其忽略标志物的共线性)。对患者组1进行了使用lasso回归的多变量逻辑回归分析。[见图3]基因标志物glmnetsrd5a1_5prmr.177.179_20.233358596nf1_17prmr.261.263_40.145129097tspyl5_8prmr.129.131_20.04597074me3_11prmr.173.175_40.019318541vav3_1prmr.185.187_8-0.008248717atm_11prmr.53.55_32-0.029412806map3k1_5prmr.161.163_8-0.045528058slc16a10_6prmr.113.115_4-0.0174300311表4.使用来自组1的118个患者将具有其3个稀释度的13-标志物集减少到8个标志物集;标志物的glmnet系数如上表中所示。前4个标志物是具有brca表型趋势的标志物,并且蓝色的底部四个标志物是具有对照表型趋势的标志物。逻辑回归分析使用怀卡托智能分析环境(waikatotoenvironmentforknowledgeanalysis)(weka)软件版本3.6.12进行逻辑回归分析。使用该分析,针对患者组1(118个患者,68个brca和50个对照)建立了灵敏度和特异性的分类函数,8个标志物通过glmnet分析识别。表5.以上示出了对于118个患者,8个标志物模型的模型测试统计数据。分类基于80%的训练(94个已知样品)和20%的测试(24个盲样品)分析。该模型的auc为0.832。模型验证然后在患者组2(31个brca和19个对照)上测试8个标志物逻辑模型,这些患者不用于减少标志物并且是独立的数据集。表6.上表示出了对于独立的50个患者组上的8-标志物模型的模型测试统计数据。分类基于80%的训练(40个已知样品)和20%的测试(10个盲样品)分析。该模型的auc为0.98。主成分分析(pca)是用于简化复杂数据集的探索性多变量统计技术。给定对n个变量的m个观察,pca的目标是通过找到r个新变量来减少数据矩阵的维数,其中r小于n。作为主成分,这些r个新变量一起占据原始n个变量中尽可能多的方差,同时保持相互不相关且正交。每个主成分都是原始变量的线性组合,并且因此通常可以将含义归于成分所代表的内容。主成分分析已用于大范围的生物医学问题,包括分析微阵列数据以寻找异常基因以及分析其他类型的表达数据。表7.使用在分析中使用的所有168个样品显示对于8-标志物brca模型验证的标签的因子分析数据(主成分分析):项目的发展(118个患者)和验证(50个患者)步骤。结论质量控制程序识别并且排除了集群122(位点2控制),其作为谱和质量与来自其他位点和集群的所有其他样品根本不同的样品。通过episwitchtm方法的结果的染色体构象分析和逻辑回归,已发展出8种生物标志物的标签,该标签对118个乳腺癌患者和健康对照的样品分层,交叉验证结果为85.7%敏感性、80%特异性、85.7%ppv和80%npv。对50个样品的独立群组验证显示出生物标志物的83.3%灵敏度、100%特异性、100%ppv和80%npv。附录1:主要标志物列表阵列1表8.经由episwitchtm技术识别的80个标志物,其将乳腺癌与对照样品分层。实施例2oxfordbiodynamics(obd)是医疗保健服务公司,在异常基因表达和表观遗传学领域提供新颖的专利平台技术。获得专利的episwitchtm平台技术检测表观遗传调控标签变化。episwitchtm生物标志物发现平台识别限定将环境线索整合到表观遗传和转录机制中的初始调控过程的染色体构象标签(ccs)。因此,ccs是基因调控连续反应中的主要步骤。由episwitchtm生物标志物发现平台分离的ccs具有若干优点:→严格的生化和生理稳定性;→他们的二元性质和读数;→它们在真核生物的基因调控连续反应中的主要地位。由于与病理或治疗相关的调控表观遗传控制设置,在基因座处的特异性构象标签存在或不存在。ccs具有温和的解离速率,并且当代表特定的表型或病理时,它们将仅随着生理信号转换或由于外部干预而改变为新的表型。此外,这些事件的测量是二元的,并且因此该读数与不同水平的dna甲基化、组蛋白修饰和大多数非编码rna的连续读数形成鲜明对比。迄今为止对于所使用的大多数分子生物标志物的连续读数对数据分析提出了挑战,因为对于特定生物标志物的变化的幅度在患者与患者之间变化很大,导致用于对患者分层的分类统计数据的问题。这些分类统计数据和推理方法更适合使用不存在该幅度的生物标志物,并且仅提供表型差异的“是或否”二元评分,预示episwitchtmccs生物标志物是用于潜在诊断、预后和预测性生物标志物的优秀资源。obd一直在其所有发展的应用中都观察到了的高度传播的episwitchtm标志物,对主要和次要受影响的组织具有高度一致性,并且具有强大的验证结果。episwitchtm生物标志物标签在复杂疾病表型的分层中表现出高稳健性和高灵敏度以及特异性。obd技术利用表观遗传学的科学中的最新突破,并且提供独特并且唯一的工业质量iso认证平台,用于作为一类高度信息性的表观遗传生物标志物的染色体构象标签的发现、监测和评估。episwitchtm技术为与异常和响应性基因表达相关的主要疾病提供了高度有效的筛查、早期检测、伴随诊断、监测和预后分析的手段。obd方法的主要优点是其是非侵入性的、快速的,并且依赖于基于高度稳定dna的靶标作为部分染色体标签,而不是不稳定的蛋白质/rna分子。技术概述ccs形成了稳定的表观遗传控制的调控框架,并且获得跨越整个细胞基因组的遗传信息。在结果将其本身表现为明显异常之前,ccs的变化良好地反映了调控和基因表达的模式的早期变化。ccs的简单研究方式是它们是拓扑排列,其中dna的不同远距离调控部分紧密接近以影响彼此的功能。这些连接不是随机进行的;它们被高度调控,并且被公认为具有显著性生物标志物分层能力的高水平调控机制。在应用表观遗传学的快速发展领域,ccs提供了与其他生物标志物平台相比显着优势。作为新的生物标志物实体,ccs的发现、监测和验证需要在其质量、稳定性、灵敏度、重现性、成本和运营周转时间上的性能方面行业可接受的技术。通过episwitchtm阵列在来自测试群组的临床样品上直接评估可能形成跨基因组的ccs的高级结构的dna,用于识别所有相关的分层引导生物标志物。在episwitchtm阵列筛查之后,统计学上显着的分层生物标志物池通常超过300个引导。然后将许多引导转换到episwitchtmcr中。分层生物标志物的最小标签(<15)经过标准验证,并且一旦确认,验证的标签包含作为具有特异性病理的患者中的表观遗传调控的条件生物标志物的二元ccs(存在或不存在)。obd技术利用表观遗传学的科学中的最新突破,并且提供独特并且唯一的工业质量iso认证平台,用于染色体构象标签的发现、监测和评估。episwitchtm测定临床样品的专有生化处理提供将表观遗传ccs生物标志物快速并且有效(<4hrs)转换为基于序列的分析物,分析物然后通过episwitchtm阵列(agilentcgh阵列平台的修改版本)、episwitchtmpcr或dna测序仪即roche454、纳米孔minion等读取episwitchtm阵列分析episwitchtm阵列平台由于其高通量以及快速筛查大量基因座的能力而用于标志物识别。该项目中使用的阵列是agilent定制-cgh阵列,其允许obd查询通过电脑模拟软件识别的标志物。该项目将使用阵列进行,其具有使用15kepiswitchtm阵列的来自组1(第i、ii、iii和iv期)的样品,但为了增加分析范围,样品与不同的种族合作使用以增加来源于阵列的数据的广度。因此,替代地,我们使用两个8x60k阵列,其允许一式四份地研究多达56,964个潜在的染色体构象,因此在该项目中使用了60k阵列。这可用于在四个重复中查看最多达14,000个探针中的染色体构象标签。使用来自一系列背景的8个ii/iii期乳腺癌患者样品产生两个阵列,相对于8个合并的健康对照患者样品进行单独测试。为每个样品制备episwitchtm模板。第一阵列是在通过obd获取的亚洲乳腺癌样品上进行的。第二阵列使用波兰群组和独立的亚洲样品群组。亚洲和欧洲乳腺癌在er+和er-状态之间以及其他亚型和表观遗传谱的流行性可能不同。在多个群体中发现了对类似癌症的重叠探针。分析的主要成果是:·两个数据集都产生了许多显著性探针;·阵列1,brca14185,在乳腺癌与健康对照的分析中识别的显著性episwitchtm标志物;·阵列2,brca24856,在乳腺癌与健康对照的分析中识别的显著性episwitchtm标志物;·在2个研究之间存在两个分析二者之间一致的2116个显著性探针的重叠。图1示出了来自brca1(表11)和brca2(表12,包括波兰群组)阵列的显著性探针的比较。探针调整p值<0.05。所有数据是最初采集的并且移出所有饱和探针。发生归一化以均匀化通道之间的数据。然后将每个数据集的所有四个重复组合在一起,并且确定变异系数。使用归一化的相关值缩减该2116个探针以对阵列上变化最大的基因排序。富集分析被用于发现高于其随机机会的差异表达最大的基因。因此,总共有138个标志物来自组合的brca1和brca2阵列,其表现出差异的上调或下调表达。阵列中使用的样品在年龄、阵列、年龄范围33-68岁,阵列232至65岁中尽可能接近地匹配。样品id患者id种族年龄类型阶段病理erprherbrcama050039位点1印度46idct3,n1,0iiia++-brcama051040位点1马来西亚47idct3,n2,0iiian/an/an/abrcama060049位点1中国68idc4c,n2,0iiibn/an/an/abrcama061050位点1印度59idc4a,0,0iiibn/an/an/abrcama062051位点1马来西亚33idct3,0,0iibn/an/an/abrcama064053位点1马来西亚50idc4c,n1,0iiib(-)(-)(-)brcama089023位点2印度66idcct4,n+,0iiib(-)(-)+brcama041003位点4印度48ilct2,0,0iii++(-)表11,brca阵列1上使用的样品。表12,brca阵列2上使用的样品。筛查1,episwitchtm标志物验证episwitchtmpcr测定是分子生物学测试,其可由经过培训的技术人员按照标准化操作程序方案执行。所有方案和试剂制造均根据iso13485和9001规范进行,以确保工作质量和转移方案的能力。使用集成dna技术(idt)软件(和primer3web版本4.0.0软件,如果需要)根据识别自微阵列的标志物设计引物。使用mmp1引物在单份提取的样品上进行样品质量控制。所有样品均显示对mmp1的阳性结果,允许样品继续进行episwitchtmpcr。所有提取的血液样品均以1:2-1:64稀释,并且进行巢式pcr。初步结果以二元形式生成,即“1”——是,带在正确的大小处存在,或“0”——否,带在正确的大小处不存在。在统计分析之后,采用前80个episwitchtm标志物,包括来自阵列1的41个标志物和来自阵列2的39个标志物,用于通过episwitchtmpcr测定进行验证以在乳腺癌样品之间进行分层。在对8个brca和8个对照样品的第一轮筛查后,标志物减少到51个,第二轮使用另外36个brca和36个对照样品,标志物被减少到13个能够在brca和对照患者之间分层的良好标志物(表13)。表13,用于通过pcr在格利维策(gliwice)样品上评估的良好标志物筛查2,对格利维策(gliwice)样品的episwitchtmpcr验证使用13个良好标志物对50个格利维策(gliwice)样品和22个对照样品进行筛查,使用1:2至1:64稀释系列。有关二元数据结果,参见附录中的表18。在进行筛查后,通过使用卡方检验(fisher精确)测试二元结果在区分brca和对照样品中的有效性,以得到最终标志物。然后使用glmnet和贝叶斯逻辑建模统计数据评估13个标志物的结果。然后10个标志物(表4)突显出具有良好的评分。表14示出了良好glmnet评分的标志物额外的统计分析进一步减少了标志物;使用具有66%的训练集和34%的测试集的分类随机树,其中使用24个样品。正确分类的实例为19(79.1667%),错误分类的实例为5(20.8333%),并且这给出卡帕(kappa)统计量为0.5,并且平均绝对误差为0.2322。平均绝对误差为0.4656,具有的相对绝对误差为55.2934%,均方误差为108.4286%。分类的详细准确度tp率fp率精确率召回率f-测量roc面积分类0.75010.750.8570.925brca10.250.44410.6150.925对照0.7920.0420.9070.7920.8170.925混淆矩阵ab155a=brca04b=对照使用glmnet产生最终的8个标志物表15最终的8个标志物样品的独立分类最后阶段是使用逻辑建模和5倍交叉验证对25个样品的独立群组测试标志物分层。真阳性率0.836num真阳性8.36假阳性率0.09num_假阳性0.44真阴性率0.91num真阴性4.08假阴性率0.164num假阴性1.64ir精确率0.953505ir召回率0.836f测量0.887459roc下面积0.9026这表明在独立群组验证中,该分类法基于能够具有83.6%的灵敏度和91.0%的特异性的选定的标志物,roc值为0.903。这意味着二元分类法的性能是高标准的,最高的roc为1,并且最低为0.5。结论该研究的目的是确定患有乳腺癌或易患乳腺癌的女性的全血中的表观遗传变化,然后使用生物标志物用于诊断分层。发展了60kepiswitchtm阵列,以研究与乳腺癌患者对照患者的诊断相关的56964个潜在的染色体相互作用。产生了两个阵列,第一个具有亚洲brca和对照患者样品,第二个具有亚洲和波兰brca样品和对照两者,这允许我们看到两个阵列之间是否存在任何类似的标志物。这最终将允许在不同族群之间找到更大深度的标志物。在分析阵列后,发现4185和4856的探针具有2116个显著性探针的重叠。进行了探针的校正归一化,并且发现了138个潜在标志物,潜在标志物可用于确定乳腺癌与对照患者的诊断。进行进一步的统计学减少以产生进行pcr筛查的前80个标志物。在几轮筛查后,13个标志物示出了其在筛查能力中的稳健性,每个具有的p值>0.3。然后这13个标志物用于筛查来自纪念癌症中心(memorialcancercenter)和肿瘤研究所,格利维策分部(instituteofoncology,gliwicebranch)(iog)的50个brca样品加上22个对照患者样品。在系列稀释后进行巢式pcr筛查,然后分析二元读数以确定哪些标志物能够在brca和对照之间进行区分,最终缩减到8个标志物,请参见表16。表16示出了产生的最终标志物分析的最后阶段是确定8个标志物是否可用于乳腺癌患者与对照患者的诊断。使用25个样品的独立子集来运行逻辑建模以确定标志物是否可以正确地预测样品。在25个样品中,标志物示出了83.6%的灵敏度和91.0%的特异性,具有的roc值为0.903。通过episwitchtm筛查发现的标志物也示出了在癌症诊断中的有趣特征。共济失调毛细管扩张突变激酶(atm)在dna损伤应答中起关键作用,功能丧失可能导致癌症发展;它们还与持续肿瘤生长中的信号传导通路相关联。atm促进her2(人类表皮生长因子受体2)在阳性乳腺癌细胞系中的致肿瘤性。atm参与具有hsp90(热休克蛋白)和her2的三聚化合物,并且已在几种肿瘤中被识别。乳腺癌的显著性风险与chek2、palb2和tp53相关联,加上atm中突变的中度风险。基因slc16a10,溶质载体家族16(芳香族氨基酸转运体),成员10,参与了选择性转运芳香氨基酸的系统t,已知其在肾、肝和肠中强烈表达。其相关途径包括葡萄糖和其他糖、胆汁盐和有机酸、金属离子和胺化合物的转运、蛋白质消化和吸收。与该基因相关的go注释包括转运体活性。单向转运体tat1(slc16a10)需要平衡特异性膜上的aaa浓度。vav3是在前列腺癌肿瘤发生中起着显著作用的致癌基因,它也在乳腺癌中表达和上调。vav蛋白是用于rho家族的gtp酶的鸟嘌呤核苷酸交换因子。它们参与细胞信号传导和肿瘤发生。vav3增强细胞生长和增殖。乳腺和前列腺癌是激素非依赖性肿瘤,其生长由它们各自的激素受体介导。vav3在乳腺癌的发展中是在表观遗传上调控的。msh3、muts同系物3与几种不同类型的癌症相关,诸如结肠直肠癌、乳腺癌、前列腺癌、膀胱癌、甲状腺癌、卵巢癌和食道癌。错配修复通路涉及细胞周期调控、细胞凋亡和dna损伤。在人类中有7个错配修复基因,已经报道了msh3基因的180个snp。msh3蛋白表达的缺失与结肠直肠癌相关,并且多态性rs26279g与乳腺癌风险相关。foxc1、叉头框(forkheadbox)c1是转录因子,其参与胚胎发育期间中胚层、脑和眼的发育,它可能是基底样乳腺癌的关键诊断标志物。升高的foxc1水平预示癌症,诸如肺和肝细胞癌的低生存率。foxc1蛋白仅在基底细胞中表达。foxc1已被识别为经由gli2转录因子的直接相互作用的hedgehog信号传导的smoothhead(smo)非依赖性激活物。这些结果示出了非常稳健和特异性的标志物组,在特异性遗传基因座的3d染色质结构水平上作为表观遗传失调监测,这可以帮助将乳腺癌患者样品与对照样品进行具有高可靠性水平的分层。附录ii表17,来自mariasklodowska-curie纪念癌症中心(memorialcancercenter)和肿瘤研究所、格利维策分部(instituteofoncology,gliwicebranch)(iog)的bca样品表18使用前13个标志物的二元分析结果。较暗的结果示出了p值>0.3。较亮的结果示出了最佳稀释度。实施例3本实施例中描述的工作涉及13个嵌套标志物,其通过聚合酶链式反应(pcr)和qpcr分型(请参见下表19)。发展这些标志物以区分患有乳腺癌的患者与非恶性个体。qpcr发展的概述是:——巢式pcr引物——单步sybrpcr(温度梯度优化)——凝胶纯化——荧光计测量、测序、同源性和基因组定位检查——水解探针优化——患者样品的标准曲线(stdcurve)测试。该工作旨在识别表观遗传变化,其可用于使用小体积的血液样品区分乳腺癌患者和非恶性材料。来自亚洲群组的血液样品,其包括来自原始测试评估工作的血液样品,将用于根据miqe(定量实时pcr实验公布的最小信息)指南验证qpcr探针测定。发展了每个标志物qpcr探针和个体检测测定,并且在代表性样品池(4x4)上跨温度梯度测试,以满足以下用于质量检测的符合miqe的标准:1.特异性:通过测序验证预测的pcr扩增子。2.线性标准曲线(r2>0.98)。3.效率(e),e>90%。4.基因组非特异性交叉反应对照与所有测定一起使用。要求是使用水解探针发展至少70%的嵌套标志物用于检测,其中测定的性能满足上述四个标准。episwitchtmqpcr测定发展数据通过巢式pcr确认了ccs生物标志物。所有发展pcr均使用qiagility进行。使用单步温度梯度pcr和基于sybr的检测,在与阴性对照物匹配的浓度下筛查每孔10ng的3c样品模板。10种相互作用被识别并且测序。将测序数据提交给ensembl,并且使用ensemblblat和needleman-wunsch算法确认了每个预测的3c相互作用的基因组定位。设计水解探针用于每个确认的相互作用的接合区域并且通过温度梯度优化。所有样品对于稳定的独立3c相互作用(mmp1)均为阳性。用n=8个患者样品(4=乳腺癌,4=非恶性),标准曲线和浓度匹配的阴性对照测试所有测定。来自发展过程的引物数据包括原始测序电泳图,并且以清晰易于检查的形式呈现每个qpcr测定。测定按字母顺序排列。在探针温度梯度优化期间,将106个拷贝的标准品用作阳性对照。用1-106个拷贝之间的曲线测试患者样品。对于报告中描述的每种测定,记录患者筛查期间标准曲线分析中的任何变化。episwitchtmqpcr测定总结atm_11_108118137_108126372_108155279_108156687_rfi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为472bp。iii.在样品pcr产物的直接测序后的ensemblblat(图1)。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.996。v.一个扩增子加倍产生两个=100%有效。测定效率=91.7%(>90%miqe指南)。vi.该测定示出了患者(n=8)子集(c01-c12=乳腺癌,d01至d12=非恶性)之间的显着的拷贝数差异(表20)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。样品pcr产物直接测序后的ensemblblat。对该pcr产物测序并且定位到染色体11q22.3。两个3c片段在taqi(tcga)处连接。序列迹线上方是ensemblblat定位数据(红色为序列同源性)。表19cdc6_17_38421089_38423079_38451196_38457050_ffi.3c模板以单步扩增。ii.实验室芯片图像。注释:带仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为428bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是101-106个拷贝的线性曲线。r2=0.99。v.一个扩增子加倍产生两个=100%有效。效率90.7%(>90%miqe指南)。vi.该测定示出了患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的显着的拷贝数差异(表2)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。表20foxc1_6_1577253_1581989_1604206_1605973_fr.i.3c模板以单步扩增。ii.实验室芯片图像。注释:带仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为208bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是101-106个拷贝的线性曲线。r2=0.992。v.一个扩增子加倍产生两个=100%有效。该测定的效率为101.6%,(>90%miqe指南)。vi.该测定示出了患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的显着的拷贝数差异(表21)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。样品pcr产物直接测序后的ensemblblat。foxc1孔b7208bp单步扩增(内引物)的实验室芯片图像。对该pcr产物测序并且定位到染色体6p。表21map3k1_5_56102259_56110500_56140227_56144076_ffi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为495bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.999。v.一个扩增子加倍产生两个=100%有效。测定效率=91.9%(>90%miqe指南)。vi.患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的拷贝数差异(表22)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。样品pcr产物直接测序后的ensemblblat。对该pcr产物测序并且定位到染色体5q11.2。表22me3_11_86300063_86304401_86420537_86426200_fri.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为291bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.998。v.一个扩增子加倍产生两个=100%有效。测定效率=96.8%(>90%miqe指南)。vi.患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的测定差异(表5)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。表23melk_9_36577630_36579243_36637050_36643005_rfi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为265bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.995。v.一个扩增子加倍产生两个=100%有效。测定效率=91.3%(>90%miqe指南)。vi.患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的测定差异(表24)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。样品pcr产物直接测序后的ensemblblat。对该pcr产物测序并且定位到染色体9p13.2。表24msh3_5_80021913_80025030_80153948_80159012_rfi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为207bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.99。v.一个扩增子加倍产生两个=100%有效。测定效率=97.1%(>90%miqe指南)。vi.患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的测定差异(表25)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。样品pcr产物直接测序后的ensemblblat。对该pcr产物测序并且定位到染色体5q14.1。表25nf1_17_29477103_29483764_29651799_29657368_ffi.3c模板以单步扩增。ii.实验室芯片图像。注释:预期大小的扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为401bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.987。v.一个扩增子加倍产生两个=100%有效。测定效率=99%(>90%miqe指南)。vi.该测定显示患者(n=8)子集(c01-c12=乳腺癌,d01-d12=非恶性)之间的差异(表26)。sq=起始量,在20ng模板中拷贝。nan=0拷贝。表26srd5a1_5_6634973_6639025_6667775_6669711_rfi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为219bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.997。v.一个扩增子加倍产生两个=100%有效。测定效率=95.5%(>90%miqe指南)。tspyl5_8_98276431_98282736_98316421_98318720_ffi.3c模板以单步扩增。ii.实验室芯片图像。注释:扩增子仅在多聚甲醛固定的样品中可见(10ng模板)。单步pcr产物具有预期的大小为507bp。iii.样品pcr产物直接测序后的ensemblblat。注释:高质量测序(正向和反向引物)与预测的3c相互作用具有100%的同源性。iv.定量pcr测定标准的性能。标准曲线是102-106个拷贝的线性曲线。r2=0.998。v.一个扩增子加倍产生两个=100%有效。测定效率=94.2%(>90%miqe指南)。结论1.对于两个引物组二者,3c标志物atm、foxc1和tspyl1产生了单步产物。2.atm拷贝数在乳腺癌中增加(n=4,表1)。行c(恶性晚期阶段疾病乳腺癌)中的样品与行d(非恶性早期阶段)不同,其中p值为0.009037772。3.cdc6_ff拷贝数在乳腺癌中减少(n=4,表2)。4.foxc1_fr拷贝数在乳腺癌中减少。行c与行d不同,其中p值为0.004112668。>atm_11_108118137_108126372_108155279_108156687_rf下划线=正向,双下划线=反向,虚线下划线=taqi.>cdc6_17_38421089_38423079_38451196_38457050_ff>foxc1_6_1577253_1581989_1604206_1605973_fr>map3k1_5_56102259_56110500_56140227_56144076_ff>me3_11_86300063_86304401_86420537_86426200_frmelk_9_36577630_36579243_36637050_36643005_rfmsh3_5_80021913_80025030_80153948_80159012_rf>nf1_17_29477103_29483764_29651799_29657368_ff>srd5a1_5_6634973_6639025_6667775_6669711_rf>tspyl5_8_98276431_98282736_98316421_98318720_ff>cdc6_17_38421089_38423079_38467677_38474960_fr>slc16a10_6_111441989_111447305_111492951_111498421_fr>vav3_1_108148303_108158073_108220200_108227533_rf双标记水解探针用于检测用5'-fam/bhq1-3'标记的测序的相互作用。探针是温度梯度优化的,并且设计为跨3c片段的接合点,使3c产物的检测完全特异性。qpcr标准曲线(106拷贝-1拷贝)由报告图中使用的测序的产物产生。mmp1拷贝测试作为内部对照用于3c库产生。使用的引物组和探针显示在下面的参考序列中。taqi位点突出显示。探针跨越两个片段的接合点,并且在66.4℃的退火温度下是特异性的。ggggagtggatgggataaggtggaatgttgggtgaactaaaaggcctttaaggcccctctgaaatccagcatcgaagagggaaactgcatcacagttgatggaagtctgttggcctcttaacaaagctaatgcttgcccttctggcttagcttacataagaaccacaaggaatctttgttgaattgtttctttcagatcatcgggacaactctccttttgatggacctggaggaaatcttgctcatgcttttcaaccaggcccammp1-42f5'-ggggagtggatgggataaggtg-3’mpp1f5'-tgggcctggttgaaaagcat-3’mmp1f1b2探针5'-fam-atccagcatcgaagagggaaactgcatca-bhq1-3’用于水解qpcr的正向和反向引物和探针序列在前面的表中描述了。对于内部对照标志物mmp1的3c库拷贝数测试。mmp-1的3c相互作用用作episwitchtm库的内部对照。双标记5'fam-bhq1-3'标记的水解探针用于检测测序的相互作用。以20ng筛查样品并且记录拷贝数。如上定量了264bp产物,在用3c靶标筛查之前,所有样品在实验室芯片(labchip)上运行。靶标表示为对每个实验的mmp1比率。用标准曲线筛查qpcr并且估计3c片段拷贝数。将qpcr模板调节至20ng的3c库dna,并且与浓度匹配的阴性对照一起使用,该阴性对照包括源自正常血液的3c库。另外的阴性对照包括没有甲醛固定的患者材料,消化和连接的库材料,以及正常基因组dna。3c相互作用mmp-1用作episwitchtm库合成的内部对照。我们已经使用了具有匹配的猝灭基团的hex、德克萨斯红(texasred)和fam。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1