用于大熊猫的STR基因座集及用途的制作方法
本发明涉及遗传学测定领域,具体涉及一种用于大熊猫的str基因座集及用途,尤其涉及一种str基因座集、用来扩增所述str基因座集的引物组,试剂盒、基因分型的方法、基因分型的系统、用于大熊猫个体识别和亲缘鉴定的方法和设备。
背景技术:
目前用于鉴别大熊猫个体及性别的技术有咬节识别法、足迹识别法、粪便dna识别法等。其中,咬节识别法为已知大熊猫99%的食物都是竹子,但由于大熊猫咀嚼不细、消化率低,导致粪便中会残留大量长度及形状均保存较完好的竹茎,这些竹茎片段被称之为咬节。一般通过提取1-3团完整的大熊猫粪便,打散后测量其中100个咬节的长度并取其平均值,便可以判断大熊猫的个体差异。足迹识别法:足迹类似于人的指纹,同样隐含着个体特异的信息。通过对圈养已知大熊猫的足迹进行收集分析,研究人员根据大熊猫野外地表介质复杂、地形复杂等特点,利用稳健的交互验证判别分析和聚类分析法,研发出针对大熊猫的个体识别算法及模型。通过利用已知圈养大熊猫个体在圈养及野外条件下的足迹进行模型验证,个体识别准确率可以达到90%。粪便dna识别法:通过提取大熊猫粪便外黏膜中的dna来鉴别个体。一般先将大熊猫的新鲜粪便用无水乙醇浸泡,粪便外层黏膜中的细胞就会被分解,以便提取dna。
然而用于大熊猫的个体识别和亲缘鉴定的方法还有待进一步改进。
技术实现要素:
本发明旨在至少在一定程度上解决相关技术中的技术问题之一。为此,本发明的一个目的在于提出一种str基因座集、用来扩增所述str基因座集的引物组,试剂盒、基因分型的方法、基因分型的系统、用于大熊猫个体识别和亲缘鉴定的方法和设备。
本发明的发明人在研究过程中发现,针对大熊猫个体识别的已有方法,存在各种弊端。其中咬节识别法的准确率较低,个体识别准确率最多仅能达到90%。足迹识别法需要同时收集6个以上的足迹方能保证预测结果的准确性。粪便dna识别法对于粪便的新鲜度要求很高,需要非常新鲜的粪便才能有效提取dna,同时相比于血液样本而言,从粪便中提取dna的难度更大。
因此本发明提供了一种痕量样本条件下准确率更高,速度更快,操作更简便的新型个体识别方法,能够突破现有大熊猫个体识别方法的局限性,而且该方法在用于大熊猫的个体识别过程中,在个体出生时就已经完全表现,并且终生不变,不受性别、年龄、疾病、环境等因素影响,准确率高且不受样本数量的限制。
为此,在本发明的第一方面,本发明提出了一种str基因座集,所述str基因座集选自以下基因座中的至少一种:pt01、pt02、pt03、pt04、pt05、pt06、pt07、pt08、pt09、pt10、pt11、pt12、pt13、pt14、pt15、pt16、pt17、pt18、pt19、pt20。微卫星(microsatellite),又称短串联重复序列(shorttandemrepeats,str),是由2-6个碱基作为核心单位串联重复而成的dna序列,因其在基因组中分布广泛、具有高度的遗传多态性、检测方法简单而被广泛应用于法医学领域,尤其在个体识别和亲权鉴定中发挥了重要的作用。根据本发明的实施例,本发明提供了str基因座集,其可以包括pt01、pt02、pt03、pt04、pt05、pt06、pt07、pt08、pt09、pt10、pt11、pt12、pt13、pt14、pt15、pt16、pt17、pt18、pt19、pt20基因座中的一种、两种或多种。例如,所述str基因座集可以仅包括pt01,也可以包括pt02和pt03,或者也可以包括pt09、pt10和pt11等。利用本发明提供的str基因座集,可以应用于大熊猫的精准个体识别和精细化管理,能够对大熊猫进行有效的个体识别,提高大熊猫的个体识别能力,且痕量样本即可以实现,不受性别、年龄、疾病、环境等因素影响,准确率高且不受样本数量的限制。
在本发明的第二方面,本发明提供了一种引物组,所述引物组适于特异性扩增根据本发明第一方面所述的str基因座集中的至少之一的核酸序列。
根据本发明的实施例,以上引物组可以进一步附加如下技术特征:
在本发明的一些实施中,所述引物组根据本发明第一方面所述的基因座集中的至少之一的核心序列两端250bp以内的保守序列设计而成。利用每一个基因座的核心序列两端250bp以内的保守序列设计引物组,实现对于每一个基因座的扩增,从而可以保证扩增的准确性。其中核心序列指的是每一个基因座所对应的重复单元所形成的短串联重复结构。
根据本发明的一些实施例,所述核酸序列位于基因组中。
根据本发明的一些实施例,所述引物组选自下列至少一对:seqidno:1至seqidno:44所示。在进行多重pcr反应的时候,引物序列的选择应该非常小心。因为引物的不恰当选择可能会产生不期望的结果,例如扩增缺乏、在预期的靶基因座之外的一个或多个位点扩增、形成引物二聚体、不同的基因座的引物间发生非期望的相互作用等等。本发明提供的引物对可以同时实现基因座的共扩增,能够同时实现各基因座的多重pcr扩增。需要说明的是,seqidno:1~seqidno:44中含有22对引物,分别对应pt01~pt22。
根据本发明的一些实施例,所述引物组中的至少一个引物上连接有可检测标记,所述可检测标记包括荧光标记。当不同的引物组上连接有不同的可检测标记时,通过检测相应的标记,来反映不同的str基因座的情况。
根据本发明的一些实施例,所述引物组中包含有多种不同的可检测标记。当所述引物组中包含不止是一对引物时,即通过一个扩增体系,实现对于两个以上的str位点的同时扩增,可以根据需要,将不同的引物标记上不同的荧光标记,然后根据荧光的不同,得到带有不同的荧光的dna扩增产物;对于带有同种荧光的dna扩增产物,可以结合dna扩增产物的片段大小,区分得到具体对应是哪个str基因座。例如,利用不同的荧光染料标记引物,使得扩增出来的产物可以分别检测为蓝色,绿色,黄色或者红色等,可以实现每一个str基因座的快速检测。具体可以为采用fam标记为蓝色,hex标记为绿色,tmra标记为黄色,rox标记为红色。
根据本发明的第三方面,本发明提供了一种试剂盒。根据本发明的实施例,所述试剂盒包括用于扩增str基因座集的试剂,所述str基因座集包括根据本发明第一方面所述的str基因座集。本发明提供的试剂盒能够实现str基因座集的扩增,应用于大熊猫的精准个体识别和精细化管理,能够对大熊猫进行有效的个体识别,提高大熊猫的个体识别能力。
根据本发明的实施例,以上试剂盒可以进一步附加如下技术特征:
根据本发明的一些实施例,所述试剂盒进一步包括根据本发明第二方面所述的引物组。利用所述引物组可以实现str基因座集的有效扩增,而且可以实现str基因座集的共扩增,从而可以实现快速检测。
根据本发明的一些实施例,所述试剂盒进一步包括等位基因分型标准物,所述等位基因分型标准物包括根据本发明第一方面所述的每一个基因座的纯合子dna。将每一个基因座的纯合子dna作为等位基因分型标准物,用来实现对于未知样本的str基因座的分型。在本文中,所用到的术语“等位基因分型标准物(allelicladder)”指的是一组长度与各str基因座所对应等位基因形成的dna。试剂盒中可以含有不同的等位基因分型标准物,以便应用所述试剂盒区别不同的str基因座。
在本发明的第四方面,本发明提出了一种检测基因座集的试剂在制备试剂盒中的用途,所述试剂盒用于大熊猫个体识别和亲缘鉴定。根据本发明的实施例,所述基因座集为根据本发明第一方面所述的基因座集。
在本发明的第五方面,本发明提出了一种基因分型的方法,包括:对待测样品内的基因座集中的至少一个基因座进行扩增,得到经扩增的等位基因,所述基因座集为根据本发明第一方面所述的str基因座集;对所述经扩增的等位基因进行分析,以确定在所述待测样品内的基因组座集中的至少一个基因座上存在的等位基因。
根据本发明的实施例,以上基因分型的方法可以进一步附加如下技术特征:
根据本发明的一些实施例,利用多重扩增反应对待测样品内的基因座集中的至少两个基因座进行共扩增,得到经扩增的等位基因。
根据本发明的一些实施例,对所述待测样品内的dna上的基因座集中的至少一个基因座进行扩增。
根据本发明的一些实施例,所述待测样品来自于大熊猫。
根据本发明的一些实施例,所述待测样品来自于血液、精液、阴道细胞、毛发、唾液、尿、骨、口腔样品、含有胎盘细胞的羊膜水、含有胎儿细胞的羊膜水中的一种或多种。
根据本发明的一些实施例,在进行所述分析之前,还包括对所述经扩增的等位基因进行分离。
根据本发明的一些实施例,通过毛细管凝胶电泳对所述经扩增的等位基因进行分离。
根据本发明的一些实施例,使用引物组特异性扩增含有本发明第一方面所述str基因座集中的至少之一的核酸序列,所述引物组为根据本发明第二方面所述的引物组。
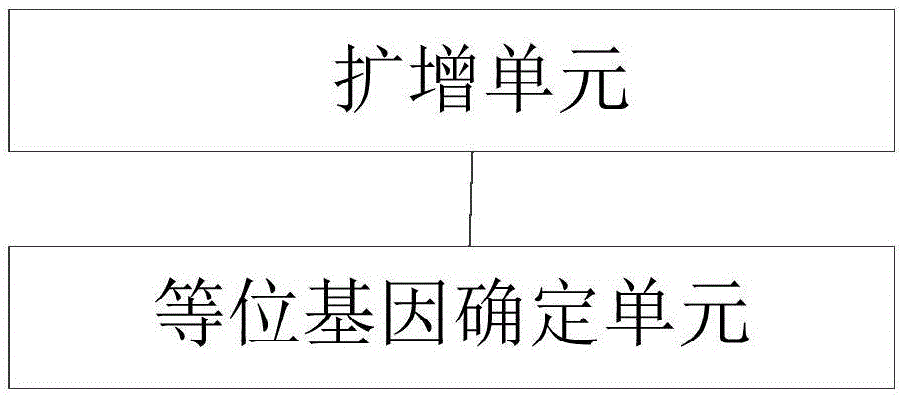
在本发明的第六方面,本发明提出了一种基因分型的系统,所述系统包括:扩增单元,所述扩增单元用于对待测样品内的基因座集中的至少一个基因座进行扩增,得到经扩增的等位基因,所述基因座集为本发明第一方面所述的str基因座集;等位基因确定单元,所述等位基因确定单元与所述扩增单元相连,所述等位基因确定单元用于对所述经扩增的等位基因进行分析,以确定在所述待测样品内的基因组座集中的至少一个基因座上存在的等位基因。
根据本发明的实施例,以上基因分型的系统,可以进一步附加如下技术特征:
根据本发明的一些实施例,所述扩增单元利用多重扩增反应对所述待测样品内的基因座集中的至少两个基因座进行共扩增,得到经扩增的等位基因。
根据本发明的一些实施例,所述扩增单元用于对所述待测样品内的dna上的基因座集中的至少一个基因座进行扩增。
根据本发明的一些实施例,所述待测样品来自于大熊猫。
根据本发明的一些实施例,所述待测样品来自于血液、精液、阴道细胞、毛发、唾液、尿、骨、口腔样品、含有胎盘细胞的羊膜水、含有胎儿细胞的羊膜水中的一种或多种。
根据本发明的一些实施例,所述系统还包括分离单元,所述分离单元分别与所述扩增单元和所述等位基因确定单元相连,所述分离单元用来对所述经扩增的等位基因进行分离。
根据本发明的一些实施例,所述分离单元通过毛细管凝胶电泳对所述经扩增的等位基因进行分离。
根据本发明的一些实施例,所述扩增单元使用引物组特异性扩增含有本发明第一方面所述str基因座集中的至少之一的核酸序列,所述引物组为根据本发明第二方面所述的引物组。
在本发明的第七方面,本发明提出了一种str基因座集在大熊猫个体识别和亲缘鉴定领域中的用途。根据本发明的实施例,所述str基因座集包括本发明第一方面所述的str基因座集。
在本发明的第八方面,本发明提出了一种用于大熊猫个体识别和亲缘鉴定的方法,包括:获取大熊猫待测个体的dna;基于str基因座集,获得所述dna的基因分型结果,所述str基因座集包括本发明第一方面所述的str基因座集;根据所述基因分型结果,对所述大熊猫待测个体进行个体识别和亲缘鉴定。
根据本发明的一些实施例,所述基因分型结果根据本发明第五方面任一实施例所述的方法获得。
在本发明的第九方面,本发明提出了一种用于大熊猫个体识别和亲缘鉴定的设备,包括:dna获取系统,所述dna获取系统用来获取大熊猫待测个体的dna;基因分型系统,所述基因分型系统与所述dna获取系统相连,所述基因分型系统基于srt基因座集,获得所述dna的基因分型结果,所述str基因座集包括本发明第一方面所述的str基因座集;分析系统,所述分析系统与所述基因分型系统相连,所述分析系统根据所述基因分型结果,对所述大熊猫待测个体进行个体识别和亲缘鉴定。
根据本发明的实施例,所述基因分型系统为根据本发明第六方面任一实施例所述的系统。
本发明所取得的有益效果为:本发明提供的str基因座集中的每一个基因座的基因型频率均符合hardy-weinberg平衡(p>0.05)。独立性检验证实22个基因座之间均无明显关联,处于连锁平衡状态。利用上述22个str位点对未知大熊猫进行个体识别,计算得到杂合度(he)在0.6238-0.7846之间,个体识别能力(pd)为0.7898-0.9206,多态信息含量为0.5874-0.7580,随机匹配概率均在0.0794-0.2102之间,累积随机匹配概率(cpm)为1.82e-18;累积个体识别能力(cpd)达0.9999999999999999981804870639,远超出行业标准,利用本发明提供的str基因座集可以实现大熊猫的个体识别和亲缘鉴定。
附图说明
图1是根据本发明的一个实施例提供的基因分型系统的示意图。
图2是根据本发明的一个实施例提供的基因分型系统的示意图。
图3是根据本发明的一个实施例提供的用于大熊猫个体识别和亲缘鉴定的设备。
图4是根据本发明的一个实施例提供的信息分析流程示意图。
具体实施方式
下面详细描述本发明的实施例,所述实施例的示例在附图中示出,其中自始至终相同或类似的标号表示相同或类似的元件或具有相同或类似功能的元件。下面通过参考附图描述的实施例是示例性的,旨在用于解释本发明,而不能理解为对本发明的限制。
为了实现对于大熊猫个体的科学和精确识别,本发明提出了一种对未知大熊猫生物检材进行个体识别的方法,并提供了一套完整的信息分析流程,获得了一套包含22个str基因座在内的遗传标记系统,应用于大熊猫精准个体识别和精细化管理。
为此,根据本发明的一个方面,本发明提出了一种str基因座集,所述str基因座集选自以下基因座中的至少一种:pt01、pt02、pt03、pt04、pt05、pt06、pt07、pt08、pt09、pt10、pt11、pt12、pt13、pt14、pt15、pt16、pt17、pt18、pt19、pt20、pt21、pt22。
利用包含本发明22个str基因座中的一种、两种或者多种组成的str基因座集,能够对大熊猫进行有效的个体识别。本发明中的str基因座集也可以称为str遗传标记系统。根据本发明的一些实施例,所述基因座的每一个的随机匹配概率小于30%,杂合度在0.6以上。每一个基因座的随机匹配概率小于30%,杂合度在0.6以上,各基因座具有很高的多态性,可以有效地对个体进行识别和鉴定。其中在本文中,随机匹配概率(probabilityofmatching,pm)即为在同一群体中,随机抽取两名个体,基因型一致的概率。杂合度(heterozygosity)是指某群体某一遗传标记所有基因型中杂合子所占到的比例。即为杂合子数与个体总数的比值,杂合度用来反映群体内遗传变异的水平。
下面对本发明中出现的某些术语进行解释和说明,以便能够更好的理解本发明。需要说明的是,这些解释和说明,仅用来帮助对于本发明的理解,而不应看做是对于本发明的限制。
在本文中,正如本领域通常的理解,“dna”指的是以其各种形式存在的脱氧核糖核酸,诸如基因组dna、cdna、分离的核酸分子、载体dna以及染色体dna。“核酸”是指任何形式的dna或rna(核糖核酸)。
在本文中,基因座指的是基因在染色体上所占的位置,在分子水平上,基因座是指有遗传效应的dna序列。一个基因座可以是一个基因,一个基因的一部分,或具有某种调控作用的dna序列。染色体中,编码在相同基因座上的dna被称为等位基因。
在本文中,所用到的术语“str基因座”或“多个str基因座”或者“str基因座集”中的str基因座是指由在某一个染色体处或者给定的靶核酸处,由两个或多个核苷酸重复形成的核苷酸序列。其中“多个str基因座”或者“str基因座集”包括两个以上的str基因座组成的集合。在本文中,所用到的术语“str等位基因”或“等位基因”指的是个体基因组中发现的str基因座的存在形式。针对某一个str基因座,其可以是杂合的,意味着两个等位基因(分别来自于两个生物学亲本)的长度不同且碱基对组成不同,或者可以是纯合的,意味着两个等位基因长度相同(碱基对组成通常相同但并不总是相同)。某些情况下,对于给定的str基因座,个体可能具有三个或更多个等位基因。在某些情况下,由于一个或多个突变,给定str基因座的个体的等位基因可能不同于它的父母。
在对待测个体或者待测样本体内的基因座进行分析时,可以从待测个体或者待测样本的组织样品中制备得到dna,所述组织样品例如可以是血液、精液、阴道细胞、毛发、唾液、尿、骨、口腔样品、含有胎盘细胞或胎儿细胞的羊膜水、绒膜绒毛、和/或任意这些的混合物或其他组织的一种或更多种。
在本文中,“引物组”包含至少一对引物,所述引物适于特异性扩增所述str基因座集中的至少一个str基因座的核酸序列。因此,在某些情况下,引物组也可以表述为引物对。引物组中可以仅包含一对引物,用来特异性扩增一个str基因座;也可以包含两对以上的引物,可以用来特异性扩增两个以上的str基因座。例如,可以仅包含seqidno:1和seqidno:2,仅用来特异性扩增pt01基因座。也可以同时包含seqidno:3和seqidno:4以及seqidno:5和seqidno:6,仅用来特异性扩增pt02基因座和pt03基因座。
本发明还提供了一种试剂盒。所述试剂盒可以用来对待测样本或者待测个体的str基因座进行扩增或者检测。在本发明的一些具体实施方式中,所述试剂盒包括容器,所述容器中具有用来扩增一个或者更多个基因座的特异性引物对。所述试剂盒还可任选地包含使用说明书。所述试剂盒还可以包含其它任选的试剂盒成分,例如包括以下中的一种或者两种或者多种:用于扩增的足量的酶、促进扩增的缓冲液、促进酶活性的盐溶液、在扩增期间用于链延伸的核苷酸(dntp)、用于制备用于电泳的扩增材料的上样溶液、作为模板对照的基因组dna、保证材料如预期的模式在分离介质中迁移的大小标记物、以及教导用户以及减少使用中的误差的方案和手册。本发明的试剂盒包括任何其他形式的例如用于手工应用的测试试剂盒或使用自动化检测仪或分析仪的测试试剂盒等,这些均包含在本发明所述的试剂盒的范围之内。其中,盐和缓冲液例如,可以包括氯化镁以及tris-hcl和kcl。缓冲液中可以含有添加剂,如表面活性剂、二甲基亚砜(dmso)、甘油、牛血清白蛋白(bsa)和聚乙二醇(peg),以及本领域技术人员熟知的其他添加剂。核苷酸通常是脱氧核糖核苷三磷酸,例如脱氧腺苷三磷酸(datp)、脱氧胞苷三磷酸(dctp)、脱氧鸟苷三磷酸(dgtp)和脱氧胸苷三磷酸(dttp),也以适当的量加至反应室用于靶核酸扩增。
利用本发明提供的str基因座集可以实现亲子鉴定。在进行亲子鉴定时,可将待测样本的str基因座的扩增结果或dna概况与取自假定匹配源的已知样品的str基因座的扩增结果或dna概况进行比较,或者也可以与衍生自该假定匹配源的样品的str基因座的扩增结果或dna概况进行比较。
利用本发明提供的str基因座,还可以用来丰富大熊猫的基因库。
根据本发明的又一方面,本发明提供了一种基因分型系统,如图1所示,所述基因分型系统包括:扩增单元和等位基因确定单元,所述等位基因确定单元与所述扩增单元相连,所述扩增单元用于对待测样品内的基因座集中的至少一个基因座进行扩增,得到经扩增的等位基因,所述基因座集选自以下基因座中的至少一种:pt01、pt02、pt03、pt04、pt05、pt06、pt07、pt08、pt09、pt10、pt11、pt12、pt13、pt14、pt15、pt16、pt17、pt18、pt19、pt20、pt21、pt22。所述等位基因确定单元用于对所述经扩增的等位基因进行分析,以确定在所述待测样品内的基因组座集中的至少一个基因座上存在的等位基因。
根据本发明的实施例,所述基因分型系统还可以包括分离单元,如图2所示,所述分离单元分别与所述扩增单元和所述等位基因确定单元相连,所述分离单元用来对所述经扩增的等位基因进行分离。例如,利用所述分离单元通过毛细管凝胶电泳对所述经扩增的等位基因进行分离。
在本发明的又一方面,本发明提供了一种用于大熊猫个体识别和亲缘鉴定的设备,如图3所示,所述设备包括dna获取系统,基因分型系统和分析系统,所述基因分型系统与所述dna获取系统相连,所述分析系统与所述基因分型系统相连。所述dna获取系统用来获取大熊猫待测个体的dna;所述基因分型系统基于srt基因座集,获得所述dna的基因分型结果,所述str基因座集选自以下基因座中的至少一种:pt01、pt02、pt03、pt04、pt05、pt06、pt07、pt08、pt09、pt10、pt11、pt12、pt13、pt14、pt15、pt16、pt17、pt18、pt19、pt20;所述分析系统根据所述基因分型结果,对所述大熊猫待测个体进行个体识别和亲缘鉴定。
下面将结合实施例对本发明的方案进行解释。本领域技术人员将会理解,下面的实施例仅用于说明本发明,而不应视为限定本发明的范围。实施例中未注明具体技术或条件的,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购获得的常规产品。
实施例1
在本实施例中,本发明提供了一组对大熊猫进行个体识别的str位点,共计有22个位点。这22个位点是通过对20只大熊猫的全基因组测序数据中分析得到的,并在另外27只无关大熊猫样本中得到了验证。基于上述位点可以对未知大熊猫个体来源的痕量血液、血痕、唾液等生物检材进行个体识别,以确定检材的个体来源,应用于大熊猫个体识别。
本实施例具体分析流程如下,如图4所示:
(1)群体数据获取:
从ncbi-sra数据库中获取47只野生大熊猫全基因组重测序数据(sra053353,illuminanthiseq2000,pe100),47只大熊猫个体之间均无生物学关联,其中20个样本用于筛选str位点(训练集),其余27个样本作为验证集。
(2)参考基因组获取:
从ucsc数据库(http://genome.ucsc.edu/)中获取最新版本大熊猫参考基因组文件:ailmel1,约2.2g。
(3)原始数据格式转换:
使用bzip2将原始数据.bz2格式转换为.fq格式,再使用gzip将.fa压缩为.fq.gz格式。
(4)数据质控:
使用fastqc(v0.11.5)对原始数据进行质控,测序质量基本合格,可以用于后续分析。
(5)构建参考基因组索引:
根据lobstr脚本文件lobstr_index.py和getstrinfo.py为大熊猫参考基因组构建索引文件。
(6)序列比对:
使用lobstr将reads比对到上述参考基因组上,并使用samtools对生成的bam文件进行排序和索引。
(7)str分型:
使用allelotype程序对训练样本进行str分型,得到vcf文件,共计1,138,361个候选str位点,包括每个str所在位置、起始碱基、核心序列、分型结果等信息。
(8)str位点筛选:
为了从上述1,138,361个候选位点中筛选出最优str位点,以便应用于大熊猫个体识别,然后根据如下标准,对位点进行筛选:
a)重复单元长度在3-5之间,
b)个体区分度阈值设置为:1,
c)随机匹配率阈值设置为:0.0000000000001,
d)位点出现率≥90%(即至少在18个样本中均存在的位点),
e)等位基因数目≥3,
f)杂合度期望值≥60%,
g)随机匹配概率≤30%,
基于上述标准,最终得到22个符合条件的高辨性str位点,22个位点的基本信息详见表1。
表1用于大熊猫个体识别的22个str位点基本信息
其中本发明中的基因座名称是本发明为区分不同基因座,所给出的编号,每个基因座的详细信息如表1所示。其中,表1中给出的坐标即为scaffold编号。其中scaffold指的是将测序得到的若干片段进行重叠拼接后形成contigs(重叠群),然后确定contigs之间的排列关系,这些contigs按照顺序排列形成scaffold。由于目前大熊猫的基因组序列还未定位到具体的染色体上,所以利用ucsc数据库(http://genome.ucsc.edu/)中大熊猫参考基因组文件:ailmel1(约2.2g),根据scaffold编号和起始碱基位置即可获得相应的str位点信息。
(9)参数统计:
利用genepop程序进行hardy-weinberg平衡检验和连锁不平衡(1inkagedisequilibrium,ld)检验。其中hardy-weinberg定律是指在一个无限大的、不发生突变、迁移和选择并随机交配的群体中,基因频率和基因型频率在世代传递中保持不变,处于平衡状态。22个基因座的个体识别能力(discriminationpower,pd)、随机匹配概率(probablityofmatching,pm)、多态性信息含量(polymorphisminformationcontent,pic)、杂合度(expeetedhcterozygosity,he)均由modified-powerstates软件计算得到。采用乘积原理计算22个str基因座的累积随机匹配概率(cpm)和累积个体识别能力(cpd)。22个位点的相关法医学参数见表2。
表2.22个str位点的相关法医学参数
其中,pic用来评价一个多态性基因座使用价值的一个量化概念,即在某一个群体内科利用该基因座进行遗传分析的概率。pd(powerofdiscrimination)也简写为dp,是指在同一群体中随机抽取两名个体,其基因型不同的概率,即利用某一遗传标记可以识别无关个体的能力。随机匹配概率(probabilityofmatching,pm)即为在同一群体随机抽取两名个体,基因型一致的概率。杂合度(he)用来反映群体内遗传变异的水平。通常在行业内,一般如下法医学参数达到下列阈值要求的str位点即可作为法医学鉴定依据,包括:he>=0.6,pd>=0.6,pic>=0.5,cpd>=0.99999。从表2可以看出,本发明的22个str基因座中的每一个基因座的杂合度(he)在0.6238-0.7846之间,个体识别能力(pd)为0.7898-0.9206,多态信息含量为0.5874-0.7580,随机匹配概率均在0.0794-0.2102之间,累积随机匹配概率(cpm)为1.82e-18;累积个体识别能力(cpd)达0.9999999999999999981804870639,远超出行业标准,利用本发明提供的str基因座集可以实现大熊猫的个体识别和亲缘鉴定。
10)str位点验证:
利用27个无关大熊猫样本对这22个基因座进行验证,使用allelotype对27个验证样本进行str分型,得到各自的vcf文件,并通过python脚本从vcf文件中获取每个验证样本在上述所选取的22个str位点处的分型数据,绘制热图,统计所有验证集中两两样本之间的差异位点数目,结果显示在27只验证样本中有27种等位基因的组合方式,100%能够将验证样本区分开,这说明没有任何两个样本在这22个位点的基因型完全一致,该套str遗传标记系统可用于对大熊猫进行个体识别。
实施例2
实施例2提供了针对实施例1中的每一个str基因座,设计了引物。
使用在线引物设计软件sgd,在上述22个str位点核心序列两端250bp以内的保守序列中,分别设计用于pcr反应的正向引物和反向引物,见表3。
表322个str位点的引物
经过试验验证,利用以上引物,能够实现每一个str基因座的扩增。更重要的是,将这些引物混合形成混合物,能够在一个扩增体系中同时实现对于所有str基因座的扩增。每条引物退火温度均在60℃左右,彼此之间不会产生引物二聚体,也不会发生非特异性扩增,也没有其他的相互作用或者交叉反应。
实施例3
实施例3提供了一种利用本发明提供的str基因座集对大熊猫进行个体识别的方法。包括如下步骤:
(1)样本采集:使用采样拭子采样头擦拭大熊猫两侧口腔壁各15-20次,将采样拭子放入保存管,折断拭子杆部,留下拭子头于保存管中,拧紧保存管盖,从而获得大熊猫口腔细胞样本。
(2)dna提取:根据天根dna提取试剂盒(货号:dp318-03)说明书的操作步骤,对步骤(1)中采集的样本提取基因组dna,并将得到的dna片段用琼脂糖凝胶电泳和紫外分光光度计检测浓度与纯度,od260/od280比值应为1.7-1.9符合要求。从而获得高纯度的全基因组dna样本。
(3)引物合成:采用固相亚磷酰胺三酯法合成实施例2中提供的22对引物序列。
(4)pcr扩增:配制pcr反应体系,依次按照高温变性、低温退火(复性)及适温延伸等反应进行pcr复合扩增,从而得到pcr扩增产物。
(5)毛细管电泳:采用abi3730xl全自动dna测序仪对pcr产物进行毛细管电泳检测,包括自动灌胶、上样、电泳分离和检测。
(6)数据分析:采用专业法医鉴定软件genemapper(v4.0)对分型结果进行判读,最后统计本发明中每个snp位点的分型数据。
(7)出具报告:根据22个基因座的分型结果为每只大熊猫出具”dna档案”报告,用于个体识别,从而建立大熊猫的dna档案。
按照如上方法,利用实施例2提供的引物序列,对多个待测大熊猫样本的22个基因座扩增,并进行分型测定。结果,对于被测试的各大熊猫样本来说,每个样本的分型结果图谱清晰完整,而且没有任何两个样本的分型数据完全一致,表明基于本发明的22个基因座,完全可以实现对于大熊猫的基因分型。以所得到的分型结果作为每只大熊猫的dna档案报告,用作大熊猫个体的识别,以此来建立大熊猫的dna档案库。
在本发明的描述中,需要理解的是,术语“中心”、“纵向”、“横向”、“长度”、“宽度”、“厚度”、“上”、“下”、“前”、“后”、“左”、“右”、“竖直”、“水平”、“顶”、“底”“内”、“外”、“顺时针”、“逆时针”、“轴向”、“径向”、“周向”等指示的方位或位置关系为基于附图所示的方位或位置关系,仅是为了便于描述本发明和简化描述,而不是指示或暗示所指的装置或元件必须具有特定的方位、以特定的方位构造和操作,因此不能理解为对本发明的限制。
此外,术语“第一”、“第二”仅用于描述目的,而不能理解为指示或暗示相对重要性或者隐含指明所指示的技术特征的数量。由此,限定有“第一”、“第二”的特征可以明示或者隐含地包括至少一个该特征。在本发明的描述中,“多个”的含义是至少两个,例如两个,三个等,除非另有明确具体的限定。
在本发明中,除非另有明确的规定和限定,术语“安装”、“相连”、“连接”、“固定”等术语应做广义理解,例如,可以是固定连接,也可以是可拆卸连接,或成一体;可以是机械连接,也可以是电连接或彼此可通讯;可以是直接相连,也可以通过中间媒介间接相连,可以是两个元件内部的连通或两个元件的相互作用关系,除非另有明确的限定。对于本领域的普通技术人员而言,可以根据具体情况理解上述术语在本发明中的具体含义。
在本发明中,除非另有明确的规定和限定,第一特征在第二特征“上”或“下”可以是第一和第二特征直接接触,或第一和第二特征通过中间媒介间接接触。而且,第一特征在第二特征“之上”、“上方”和“上面”可是第一特征在第二特征正上方或斜上方,或仅仅表示第一特征水平高度高于第二特征。第一特征在第二特征“之下”、“下方”和“下面”可以是第一特征在第二特征正下方或斜下方,或仅仅表示第一特征水平高度小于第二特征。
在本说明书的描述中,参考术语“一个实施例”、“一些实施例”、“示例”、“具体示例”、或“一些示例”等的描述意指结合该实施例或示例描述的具体特征、结构、材料或者特点包含于本发明的至少一个实施例或示例中。在本说明书中,对上述术语的示意性表述不必针对的是相同的实施例或示例。而且,描述的具体特征、结构、材料或者特点可以在任一个或多个实施例或示例中以合适的方式结合。此外,在不相互矛盾的情况下,本领域的技术人员可以将本说明书中描述的不同实施例或示例以及不同实施例或示例的特征进行结合和组合。
尽管上面已经示出和描述了本发明的实施例,可以理解的是,上述实施例是示例性的,不能理解为对本发明的限制,本领域的普通技术人员在本发明的范围内可以对上述实施例进行变化、修改、替换和变型。
sequencelisting
<110>深圳市华大司法技术协同创新研究院,深圳华大法医科技有限公司
<120>用于大熊猫的str基因座集及用途
<130>pidc3181693
<160>44
<170>patentinversion3.5
<210>1
<211>20
<212>dna
<213>人工序列
<400>1
aacacccacttacctctcaa20
<210>2
<211>21
<212>dna
<213>人工序列
<400>2
ctgggaaggaataaaagtggc21
<210>3
<211>21
<212>dna
<213>人工序列
<400>3
ggacacacaaacttttggttg21
<210>4
<211>21
<212>dna
<213>人工序列
<400>4
gggaactttaaagataccttg21
<210>5
<211>20
<212>dna
<213>人工序列
<400>5
atctatccatcacccagtga20
<210>6
<211>21
<212>dna
<213>人工序列
<400>6
tgtctggtatggtagccactt21
<210>7
<211>21
<212>dna
<213>人工序列
<400>7
tgctttcctagcttttaactt21
<210>8
<211>21
<212>dna
<213>人工序列
<400>8
tctttcctggcttcttagcct21
<210>9
<211>19
<212>dna
<213>人工序列
<400>9
tcatccatttcgttgcaag19
<210>10
<211>20
<212>dna
<213>人工序列
<400>10
gccaacaggtatacactgaa20
<210>11
<211>20
<212>dna
<213>人工序列
<400>11
aaaaaagagagagggagcca20
<210>12
<211>20
<212>dna
<213>人工序列
<400>12
actttcttctctgcctatcc20
<210>13
<211>21
<212>dna
<213>人工序列
<400>13
gaccaagggtaagagtctgaa21
<210>14
<211>18
<212>dna
<213>人工序列
<400>14
aaaggcctcatggggcac18
<210>15
<211>21
<212>dna
<213>人工序列
<400>15
ttacaaatggtgacagggtgc21
<210>16
<211>18
<212>dna
<213>人工序列
<400>16
atccccaagaaagaaata18
<210>17
<211>21
<212>dna
<213>人工序列
<400>17
tcaataaaatatacctgtcgg21
<210>18
<211>20
<212>dna
<213>人工序列
<400>18
tggaatgtagaccaactctg20
<210>19
<211>21
<212>dna
<213>人工序列
<400>19
gggctttttcccaacaaaaac21
<210>20
<211>20
<212>dna
<213>人工序列
<400>20
ttcaaattctggctgctgct20
<210>21
<211>21
<212>dna
<213>人工序列
<400>21
ggatacagggtctttaaaaag21
<210>22
<211>21
<212>dna
<213>人工序列
<400>22
tgccacaacaaagtagcatgg21
<210>23
<211>21
<212>dna
<213>人工序列
<400>23
ggctaaaataacccagttatc21
<210>24
<211>20
<212>dna
<213>人工序列
<400>24
accttttgtgttgcacagga20
<210>25
<211>20
<212>dna
<213>人工序列
<400>25
gttaataatgggaaggctga20
<210>26
<211>20
<212>dna
<213>人工序列
<400>26
ttgtctgggttccacagatt20
<210>27
<211>21
<212>dna
<213>人工序列
<400>27
acaatacacacccacacacca21
<210>28
<211>20
<212>dna
<213>人工序列
<400>28
ctggtcaaaatggcctcaaa20
<210>29
<211>20
<212>dna
<213>人工序列
<400>29
agttaccccaatccccaaaa20
<210>30
<211>20
<212>dna
<213>人工序列
<400>30
acttggtaatgcgcttcagg20
<210>31
<211>20
<212>dna
<213>人工序列
<400>31
agaatactggcctgcatgat20
<210>32
<211>20
<212>dna
<213>人工序列
<400>32
ctcctgaatttattggatcg20
<210>33
<211>21
<212>dna
<213>人工序列
<400>33
tgaaactggaatacagacaga21
<210>34
<211>21
<212>dna
<213>人工序列
<400>34
gacacaagaatgtgtatcaag21
<210>35
<211>21
<212>dna
<213>人工序列
<400>35
ctttgttagagcctttcctac21
<210>36
<211>20
<212>dna
<213>人工序列
<400>36
gtcgtctccattggctttta20
<210>37
<211>20
<212>dna
<213>人工序列
<400>37
tgacagctaagggaacagaa20
<210>38
<211>21
<212>dna
<213>人工序列
<400>38
cacagttgtatcataagctga21
<210>39
<211>21
<212>dna
<213>人工序列
<400>39
ctatctttgggcatctgagtt21
<210>40
<211>21
<212>dna
<213>人工序列
<400>40
attcctatggctgggtcacct21
<210>41
<211>21
<212>dna
<213>人工序列
<400>41
agatcatgaggtaaaaatctg21
<210>42
<211>21
<212>dna
<213>人工序列
<400>42
ctttgtcaagtcctagttcca21
<210>43
<211>20
<212>dna
<213>人工序列
<400>43
ggatgtgtgagaaactacca20
<210>44
<211>20
<212>dna
<213>人工序列
<400>44
gattatcctaggtgatccca20
- 还没有人留言评论。精彩留言会获得点赞!