第三代测序用标签序列、接头序列、试剂盒和第三代测序建库方法与流程
本发明涉及第三代测序技术领域,具体涉及一种第三代测序用标签序列、接头序列、试剂盒和第三代测序建库方法。
背景技术:
第三代测序(如pacbio平台测序)基于边合成边测序的原理,以smrt(单分子实时荧光测序技术)芯片为载体进行测序反应。测序时将基因组dna打断成许多小片段,制成液滴后将其分散到不同的zmw(zero-modewaveguides,零模波导)纳米孔中。当zmw纳米孔底部聚合反应发生时,被不同荧光标记的核苷酸会在小孔的荧光探测区域中被聚合酶滞留,根据荧光的种类和荧光持续时间就可以判定模板dna碱基组成的种类。
pacbio平台上每个smrt芯片有100万个zmw测序孔,平均可以产出5-15g数据,但是对于基因组较小的物种,所需要的数据量较少(数据需求小于1g),往往需要把每个样本加上不同的分子标签(也称“标签序列”),混合测序,最后通过标签序列拆分每个样本的序列。
第二代测序平台,每张芯片可以产生多达1t的数据,但是单个样本一般不需要这么多数据,所以为了充分了利用第二代平台的测序数据,科学家发明设计了dna标签序列(barcode),将已知的标签序列连接在dna文库的两端,然后将带有不同标签序列的样本混合到一起测序,将测序的数据进行标签序列拆分到对应样本。目前由于第二代测序在测序过程中只能读取识别atgc四种碱基,所以科学家随机使用atgc四种碱基设计标签序列,不同测序平台标签序列碱基数不同。例如bgiseq500平台,一般在文库的两端各加入10个碱基的标签序列(atcg四种碱基随机分布)。
第三代测序在设计标签序列时,参考了第二代测序平台标签序列设计方法,使用atgc四种碱基设计标签序列(例如,长度为16个碱基的标签)连接到文库的两端,然后根据标签序列对测序数据进行拆分。
如图1所示,第三代测序的接头为环状接头,16个碱基的标签序列(barcode)位于插入片段(insert)与接头之间。当文库的插入片段较短时,文库可以反复读多次,标签序列也会反复被识别,根据文库两端的标签序列即可拆分出子文库的对应信息。当文库较长时,聚合酶可能读不到标签序列,此时大部分数据无法拆分到各个子文库,造成数据浪费。第三代测序文库插入片段长度大致在5-8kb,没有充分的发挥第三代测序读长的优势(目前读长15-20kb),同时由于标签序列设计方面的问题,拆分率大致在60-70%左右,造成30-40%的数据浪费,无形中增加了测序成本,限制了第三代测序平台的发展。
技术实现要素:
本发明提供一种提高第三代测序数据拆分率的标签序列、接头序列、试剂盒和第三代测序建库方法。
根据第一方面,一种实施例中提供一种第三代测序用标签序列,该标签序列由若干连续的碱基组成,上述碱基中至少部分碱基是甲基化的碱基。
作为优选的技术方案,上述碱基中至少一种类型的碱基全部是甲基化的碱基;优选地,上述碱基中只有一种类型的碱基全部是甲基化的碱基;更优选地,上述碱基中腺嘌呤碱基全部是6-甲基腺嘌呤(6ma);或者,上述碱基中胞嘧啶碱基全部是4-甲基胞嘧啶(4mc)或5-甲基胞嘧啶(5mc)。
作为优选的技术方案,上述标签序列由6至20个碱基组成;优选地,上述标签序列由16个碱基组成。
根据第二方面,一种实施例中提供一种第三代测序用接头序列,该接头序列包括标签序列和与上述标签序列连接的其他序列,上述标签序列由若干连续的碱基组成,上述碱基中至少部分碱基是甲基化的碱基。
作为优选的技术方案,上述标签序列的碱基中至少一种类型的碱基全部是甲基化的碱基;优选地,上述标签序列的碱基中只有一种类型的碱基全部是甲基化的碱基;更优选地,上述标签序列的碱基中腺嘌呤碱基全部是6-甲基腺嘌呤(6ma);或者,上述标签序列的碱基中胞嘧啶碱基全部是4-甲基胞嘧啶(4mc)或5-甲基胞嘧啶(5mc)。
作为优选的技术方案,上述标签序列由6至20个碱基组成;优选地,上述标签序列由16个碱基组成。
根据第三方面,一种实施例中提供一种第三代测序用试剂盒,该试剂盒包括第二方面的接头序列;任选地,还包括建库用试剂组分。
根据第四方面,一种实施例中提供第一方面的标签序列或第二方面的接头序列在构建第三代测序文库中的用途。
根据第五方面,一种实施例中提供一种第三代测序文库构建方法,该方法包括使用第二方面的接头序列与待连接的核酸片段进行连接形成带有上述接头序列的测序文库。
作为优选的技术方案,上述方法还包括:在连接上述接头序列之前,对上述待连接的核酸片段进行末端修复或末端修复并加a碱基反应,形成适合与上述接头序列连接的核酸片段;在连接上述接头序列之后,使用消化酶消化未连接的核酸片段和未连接的接头序列。
根据第六方面,一种实施例中提供一种第三代测序方法,该方法包括:
第三代测序文库构建,其包括使用第二方面的接头序列与待连接的核酸片段进行连接形成带有上述接头序列的测序文库;和
对上述测序文库进行第三代上机测序。
作为优选的技术方案,上述第三代上机测序是pacbio平台测序。
本发明使用甲基化的标签序列能够对常规拆分方法无法拆分的第三代测序数据进行拆分,大大提高了第三代测序数据的拆分率,本发明能够使第三代测序数据的整体拆分率达到85%左右。
附图说明
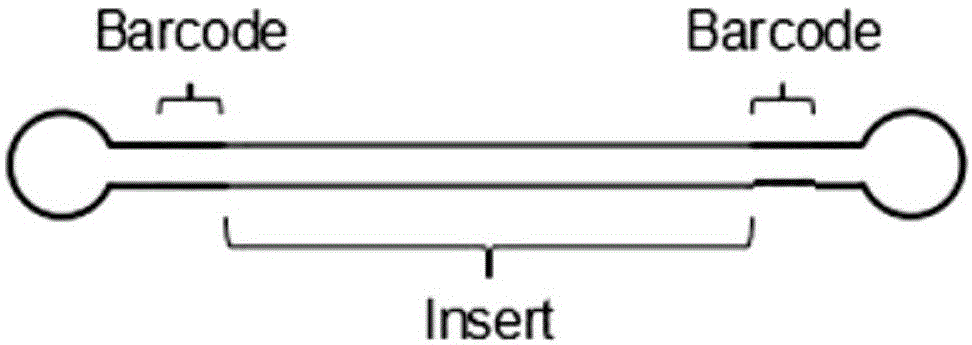
图1为本发明中第三代测序文库结构示意图,两端连接有环状接头,接头中的标签序列(barcode)位于插入片段(insert)与环状接头之间。
图2为本发明实施例中使用含有6-甲基腺嘌呤(6ma)的标签序列进行第三代测序的碱基读取原理示意图,横坐标表示时间(time),纵坐标表示荧光强度(fluorescenceintensity);图中示出当遇到6ma甲基化修饰碱基时(上图),光强持续时间较长、光强较弱;当碱基上无修饰时(下图),光强相对较强、持续时间较短。
具体实施方式
下面通过具体实施方式结合附图对本发明作进一步详细说明。在以下的实施方式中,很多细节描述是为了使得本发明能被更好的理解。然而,本领域技术人员可以毫不费力的认识到,其中部分特征在不同情况下是可以省略的,或者可以由其他元件、材料、方法所替代。
另外,说明书中所描述的特点、操作或者特征可以以任意适当的方式结合形成各种实施方式。同时,方法描述中的各步骤或者动作也可以按照本领域技术人员所能显而易见的方式进行顺序调换或调整。因此,说明书和附图中的各种顺序只是为了清楚描述某一个实施例,并不意味着是必须的顺序,除非另有说明其中某个顺序是必须遵循的。
本发明涉及第三代单分子测序技术领域,目的在于通过设计一种甲基化的接头,使得有效样本的整体拆分率提升至85%以上。
由于第三代测序为单分子实时荧光测序技术,在测序过程中,可以检测聚合酶荧光强度和持续时间直接读取到甲基化修饰。在建库测序过程中,不需要进行任何出库,是一种直接获取甲基化修饰的方法。
具体而言,第三代单分子测序技术,在获取超长读长的同时,可利用测序过程聚合酶反应的动力学变化直接检测碱基修饰。其原理在于,聚合酶每合成一个碱基,都有一个时间段,而当模板碱基带有修饰时,就像行车过程中遇到路障聚合酶会慢下来,使带有修饰的碱基两个相邻的脉冲峰之间的距离和参考序列的距离之间的比值结果即ipd值(interpulseduration,脉冲间隔持续时间)大于1,由此就可以推断这个位置有修饰。如图2所示,上图中模板上将与t碱基配对的a碱基是6-甲基腺嘌呤(6ma),聚合酶遇到6ma碱基会慢下来,即图中显示a和t两个相邻的脉冲峰之间的距离变长;下图中模板上将与t碱基配对的a碱基是未修饰的碱基,因此两个相邻的脉冲峰之间的距离正常。因此,根据相邻的脉冲峰之间的距离和参考序列的距离之间的比值可以判断位点是否为甲基化修饰位点。
需要说明的是,基于本发明的原理,即碱基上的甲基化修饰如同行车过程中遇到路障,会使聚合酶慢下来的原理,任何碱基的任何甲基化修饰均可用于本发明中实现相同的发明目的,达到相同的技术效果。因此,本发明中碱基的甲基化修饰不限于6-甲基腺嘌呤(6ma),例如,还可以是4-甲基胞嘧啶(4mc)和5-甲基胞嘧啶(5mc)等甲基化修饰。同时,atgc四种碱基中任何一种或多种碱基的甲基化修饰均可用于本发明。
因此,本发明的一种实施例中提供一种第三代测序用标签序列,该标签序列由若干连续的碱基组成,这些碱基中至少部分碱基是甲基化的碱基。
本发明中,所谓“标签序列”即能够区分不同样本来源的分子标签,对于第三代单分子测序技术而言,可以是指能够区分每个单分子样本来源的分子标签。
需要说明的是,本发明对甲基化的碱基的数量和种类没有限制,可以是所有碱基均甲基化,也可以是仅有部分碱基甲基化;可以是所有种类的碱基(例如atgc四种碱基)均甲基化,也可以是只有一种、二种或三种碱基甲基化。作为优选的技术方案,碱基中至少一种(即一种、二种或三种)类型的碱基全部是甲基化的碱基,例如,a碱基全部是甲基化的碱基、或t碱基全部是甲基化的碱基、或g碱基全部是甲基化的碱基、或c碱基全部是甲基化的碱基、或at碱基全部是甲基化的碱基、或gc碱基全部是甲基化的碱基、或atg碱基全部是甲基化的碱基等。在本发明的优选实施例中,上述碱基中只有一种类型的碱基全部是甲基化的碱基,更优选,碱基中腺嘌呤碱基(a)全部是6-甲基腺嘌呤(6ma);或者,碱基中胞嘧啶碱基全部是4-甲基胞嘧啶(4mc)或5-甲基胞嘧啶(5mc)。
本发明中,标签序列的长度没有特别限定,通常标签序列由6至20个碱基组成。在本发明的优选实施例中,标签序列由16个碱基组成。
本发明的一种实施例中提供一种第三代测序用接头序列,该接头序列包括标签序列和与标签序列连接的其他序列,标签序列由若干连续的碱基组成,碱基中至少部分碱基是甲基化的碱基。
需要说明的是,上述关于标签序列的所有描述均适用于接头序列中的标签序列部分。另外,与标签序列连接的“其他序列”可以是任何序列,优选测序平台接头序列,更优选pacbio测序平台接头序列,这些接头序列属于本领域公知的序列。“其他序列”与标签序列的连接和位置关系没有特别限定,标签序列可以位于其他序列两端,形成反向互补结构,即两段反向互补的标签序列分别连接在它们中间的其他序列两端,并且这两段反向互补的标签序列中最少一段是上述描述的甲基化修饰情况,优选两段反向互补的标签序列均是上述描述的甲基化修饰情况。在其他实施例中,标签序列可以位于其他序列内部,即标签序列将其他序列间隔成两段或多段,并且标签序列可以形成反向互补,也可以不形成反向互补。
本发明的一种实施例中提供一种第三代测序用试剂盒,该试剂盒包括本发明的接头序列。
除了本发明的接头序列,本发明的试剂盒还可以包括建库用试剂组分,例如用于打断基因组dna的酶,如tn5转座酶等;用于修复打断的dna的酶,如t4多聚核苷酸激酶等;用于连接接头序列和待连接的核酸片段的连接酶,如t4dna连接酶等;用于消化未连接的核酸片段和未连接的接头序列的消化酶等。
本发明的一种实施例中提供本发明的标签序列或本发明的接头序列在构建第三代测序文库中的用途,尤其是本发明的标签序列在制备第三代测序文库的接头序列中的用途。
本发明的一种实施例中提供一种第三代测序文库构建方法,该方法包括使用本发明的接头序列与待连接的核酸片段进行连接形成带有接头序列的测序文库。待连接的核酸片段,一般是指dna片段,本发明优选是长片段,例如15-20kb或5-8kb等长度的片段,这样的dna片段可以是基因组打断后的片段,也可以是mrna经反转后的片段等。
对于打断的dna片段,在连接接头序列之前,可以对其进行末端修复或末端修复并加a碱基反应,形成适合与接头序列连接的核酸片段。同时,为了避免未连接的核酸片段和未连接的接头序列对后续测序的影响,在连接接头序列之后,可以使用消化酶消化未连接的核酸片段和未连接的接头序列。
本发明的一种实施例中提供一种第三代测序方法,该方法包括:第三代测序文库构建,其包括使用本发明的接头序列与待连接的核酸片段进行连接形成带有接头序列的测序文库;以及对测序文库进行第三代上机测序,例如pacbio平台测序。然后对测序数据根据样本来源进行数据拆分。
现有的第三代测序接头上标签序列没有甲基化修饰,按照常规方法进行拆分,拆分率只能达到65%左右。常规拆分方法一般是,分析整张芯片的下机数据,根据两端标签序列(一般是16bp)对应子样本文库信息,如果只读到一端的标签序列信息,系统会直接将其视为未匹配的测序读长(reads)。但是在测序过程中,经常出现测序聚合酶活性不够读取整个插入片段的长度,只能读取到一端的标签序列信息,这种情况下的测序数据会被归类为无效信息,从而导致样本的拆分率大致只有65%左右。
本发明的方法,第三代测序接头上标签序列经过甲基化修饰,测序后首先按照常规方法拆分数据,大致能够拆分出65%数据,另外还有35%左右的数据会被归类到“无效信息”。然后,根据本发明的甲基化修饰的标签序列,筛选出只读到一端标签序列的测序读长,根据上面的甲基化位点再次拆分数据,使得原本浪费的“无效信息”数据达到二次利用的效果,经过二次拆分能够使得整体拆分率达到85%左右。具体而言,对于标签序列上使用6ma的情况,当dna上面存在6ma,荧光的颜色、持续时间,会与常规未经修饰的碱基存在差异。在设计合成标签序列时,将所有(也可以是部分)a碱基加上了6ma修饰,每一种标签序列上面a碱基的位置和数目是不同的,相当于每个标签序列都有自己独特的6ma信息,根据6ma的数目和位置信息,可以将原本未能正确拆分的信息重新利用。类似地,对于任何甲基化碱基,每一种标签序列上面甲基化碱基的位置和数目不同,相当于每个标签序列都有自己独特的甲基化碱基信息。
本发明能够应用于目前的第三代测序平台(pacbio平台)的多种产品,包括denovo动植物,denovo微生物,16s全长测序,全长转录组,pcr产物等产品。本发明能够使第三代测序平台测序上机更加灵活,能够降低建库测序的成本,市场前景较为广阔。
以下通过比较例和实施例详细说明本发明的技术方案,应当理解,比较例和实施例仅是示例性的,不能理解为对本发明保护范围的限制。
比较例1
使用的商业建库试剂盒:100-991-900#smrtbelltmtemplateprepkit1.0reagentquantitiessupport10librarypreparations.smrtbell.模板制备试剂盒(500bp-20kb)品牌@pacificbiosciences/c/规格&10个反应/盒((pacificbiosciences,lot:0101995217))。纯化磁珠:诺唯赞vahtstmdnacleanbeads(lot:n411)。
实验步骤如下:
(1)合成pacbio平台官方公布的带标签序列的接头,如下表1所示,其中下划线部分为标签序列,其他部分为测序平台接头序列部分。
表1带标签序列的接头
将表1中的带标签序列的接头在如下表2所示的退火体系和退火反应条件中进行退火反应。
表2退火体系和退火反应条件
(2)采用covarisg-tube打断基因组dna,0.45倍pacbio磁珠纯化样本,然后检测浓度和片段分布。
(3)采用酶7消化打断后的dna。
(4)采用损伤修复酶修复dna上面的缺口,然后0.45倍pacbio磁珠纯化样本。
(5)采用t4dna连接酶、10xtempbuffer、atplow和退火的带标签序列的接头进行连接。
(6)接头连接完成之后,高温让t4dna连接酶失活,然后加入消化酶3和消化酶7,用0.45pacbio磁珠纯化。
(7)对分选纯化后的产物进行浓度和片段大小质检,然后按照规则混合。
(8)连接测序引物和测序聚合酶后,进行pacbio平台测序。
测序后进行数据拆分,结果如下表3所示:
表3
表3数据显示:4个细菌样本,按照常规pacbio公布的流程操作,未拆分出来的无效数据占比为32.7%,拆分率为67.3%。
实施例1
使用的商业建库试剂盒:100-991-900#smrtbelltmtemplateprepkit1.0reagentquantitiessupport10librarypreparations.smrtbell.模板制备试剂盒(500bp-20kb)品牌@pacificbiosciences/c/规格&10个反应/盒((pacificbiosciences,lot:0101995217))。纯化磁珠:诺唯赞vahtstmdnacleanbeads(lot:n411)。
实验步骤如下:
(1)合成带甲基化修饰标签序列的接头,接头序列如表1所示,与比较例不同点在于,标签序列(下划线部分)中的a碱基是6ma修饰碱基;并按照表2所示的退火体系和退火反应条件完成接头的退火。
(2)采用covarisg-tube打断基因组dna,0.6倍诺唯赞磁珠纯化样本,然后检测浓度和片段分布。
(3)采用酶7消化打断后的dna。
(4)采用损伤修复酶修复dna上面的缺口,然后0.6倍诺唯赞磁珠纯化dna。
(5)采用t4dna连接酶、10xtempbuffer、atplow和退火的带标签序列的接头进行连接。
(6)接头连接完成之后,高温让t4dna连接酶失活,然后加入消化酶3和消化酶7,用0.6倍的诺唯赞磁珠纯化样本后,进行片段分选及其纯化。
(7)对分选纯化后的产物进行浓度和片段大小质检,然后按照规则混合。
(8)连接测序引物和测序聚合酶后,进行pacbio平台测序。
测序后进行数据拆分,结果如下表4所示:
表4
表4数据显示:4个细菌样本,使用加6ma甲基化修饰的标签序列,未能拆分出的数据为12%,所以有效数据拆分率为88%。
以上应用了具体个例对本发明进行阐述,只是用于帮助理解本发明,并不用以限制本发明。对于本发明所属技术领域的技术人员,依据本发明的思想,还可以做出若干简单推演、变形或替换。
sequencelisting
<110>武汉华大医学检验所有限公司
<120>第三代测序用标签序列、接头序列、试剂盒和第三代测序建库方法
<130>18i26828
<160>16
<170>patentinversion3.3
<210>1
<211>77
<212>dna
<213>人工序列
<400>1
cacatatcagagtgcgatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tcgcactctgatatgtg77
<210>2
<211>77
<212>dna
<213>人工序列
<400>2
acacacagactgtgagatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tctcacagtctgtgtgt77
<210>3
<211>77
<212>dna
<213>人工序列
<400>3
cacgcacacacgcgcgatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tcgcgcgtgtgtgcgtg77
<210>4
<211>77
<212>dna
<213>人工序列
<400>4
acagtcgagcgctgcgatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tcgcagcgctcgactgt77
<210>5
<211>77
<212>dna
<213>人工序列
<400>5
acacacgcgagacagaatctctctcttttcctcctcctccgttgttgttgttgagagaga60
ttctgtctcgcgtgtgt77
<210>6
<211>77
<212>dna
<213>人工序列
<400>6
acgcgctatctcagagatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tctctgagatagcgcgt77
<210>7
<211>77
<212>dna
<213>人工序列
<400>7
acactagatcgcgtgtatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tacacgcgatctagtgt77
<210>8
<211>77
<212>dna
<213>人工序列
<400>8
ctcactacgcgcgcgtatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tacgcgcgcgtagtgag77
<210>9
<211>77
<212>dna
<213>人工序列
<400>9
cgcatgacacgtgtgtatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tacacacgtgtcatgcg77
<210>10
<211>77
<212>dna
<213>人工序列
<400>10
catagagagatagtatatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tatactatctctctatg77
<210>11
<211>77
<212>dna
<213>人工序列
<400>11
cacacgcgcgctatatatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tatatagcgcgcgtgtg77
<210>12
<211>77
<212>dna
<213>人工序列
<400>12
tcacgtgctcactgtgatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tcacagtgagcacgtga77
<210>13
<211>77
<212>dna
<213>人工序列
<400>13
acacactctatcagatatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tatctgatagagtgtgt77
<210>14
<211>77
<212>dna
<213>人工序列
<400>14
cacgacacgacgatgtatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tacatcgtcgtgtcgtg77
<210>15
<211>77
<212>dna
<213>人工序列
<400>15
ctatacatagtgatgtatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tacatcactatgtatag77
<210>16
<211>77
<212>dna
<213>人工序列
<400>16
cactcacgtgtgatatatctctctcttttcctcctcctccgttgttgttgttgagagaga60
tatatcacacgtgatgt77
- 还没有人留言评论。精彩留言会获得点赞!